Изобретение относится к ветеринарной микробиологии, в частности к методам определения видовой принадлежности мяса с помощью полимеразной цепной реакции.
Известно использование ПЦР в реальном времени для определения ДНК следующих животных - лошади, коровы, свиньи, осла, курицы, индейки, кошки, собаки и кролика (https://stylab.ru/netcat_files/userfiles/Files/Articles/Meat/Meat_1_04_2013.pdf.).
Наиболее близким по технической сущности является способ идентификации видовой принадлежности тканей животного в продовольственном сырье, кормах и пищевых продуктах (патент РФ №№2694713, кл. C12Q 1/68, 2019 г.), включающий выделение ДНК из ткани животного сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: JOE/Yellow для специфического сигнала для животного и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, положительного контрольного образца в виде смеси, содержащую фрагменты геномов животного и бактериофага Т4 с нуклеотидной последовательностью:
 - прямой праймер
- прямой праймер
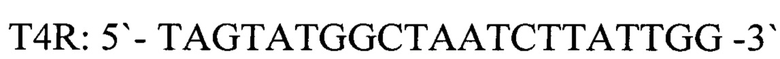 - обратный праймер
- обратный праймер
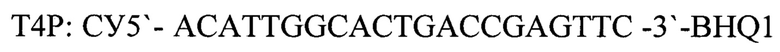 - зонд, взятых в соотношении 1:1 и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей:, интерпретацию результатов проводят на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный.
- зонд, взятых в соотношении 1:1 и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей:, интерпретацию результатов проводят на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный.
Недостатком известного технического решения является недостаточная точность из-за использования суспензии бактериофага, которая требует предварительную обработку, включая центрифугирование, концентрирование и перевод в определенный буферный раствор, что влечет за собой значительную трудоемкость и финансовые затраты.
Техническим результатом является повышение точности идентификации видовой принадлежности, упрощение процесса подготовки внутреннего контрольного образца и уменьшение его стоимости.
Технический результат достигается тем, что в способе идентификации ДНК ткани медведей (Ursus) в сухих кормах и мясных полуфабрикатах, включающем выделение ДНК из ткани животного сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, положительного контрольного образца в виде смеси, содержащую фрагменты геномов животного и бактериофага Т4 с нуклеотидной последовательностью:
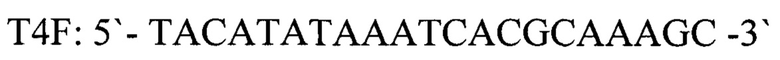 - прямой праймер
- прямой праймер
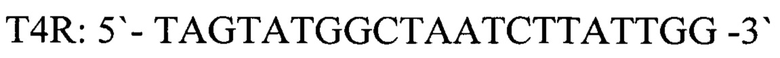 - обратный праймер
- обратный праймер
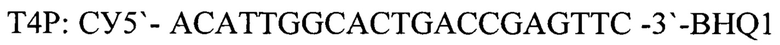 - зонд, взятых в соотношении 1:1 и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный, согласно изобретению выделяют ДНК из ткани медведя (Ursus) и для внутреннего контрольного образца используют фаголизат бактериофага Т4, а для положительного контрольного образца - фрагменты геномов нативного бактериофага Т4 и ткани медведя (Ursus) со следующей нуклеотидной последовательностью:
- зонд, взятых в соотношении 1:1 и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный, согласно изобретению выделяют ДНК из ткани медведя (Ursus) и для внутреннего контрольного образца используют фаголизат бактериофага Т4, а для положительного контрольного образца - фрагменты геномов нативного бактериофага Т4 и ткани медведя (Ursus) со следующей нуклеотидной последовательностью:
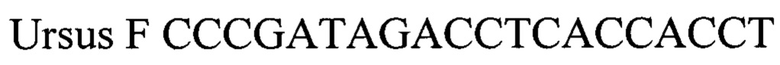 прямой праймер
прямой праймер
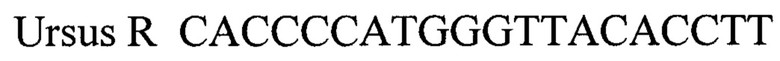 обратный праймер
обратный праймер
 зонд.
зонд.
Новизна заявляемого способа состоит в идентификации видовой принадлежности тканей медведя с помощью полимеразной цепной реакции (ПЦР) с флуоресцентной детекцией в режиме реального времени, что в свою очередь позволяет с высокой точностью определить наличие их ингредиентов в продовольственном сырье, кормах и пищевых продуктах.
Признаки, отличающие заявляемое техническое решение от прототипа, направлены на достижение технического результата и не выявлены при изучении данной и смежной областей науки и техники и, следовательно, соответствуют критерию «изобретательский уровень».
Заявляемый способ рекомендовано использовать в специализированных ветеринарных, санитарно-эпидемиологических, животноводческих, сельскохозяйственных предприятиях, что соответствует критерию «промышленная применимость».
Способ идентификации ДНК ткани медведей (Ursus) в сухих кормах и мясных полуфабрикатах осуществляется следующим образом.
Для исследования сухих кормов и мясных полуфабрикатов на содержание ДНК ткани медведя проводят полимеразную цепную реакцию с флуоресцентной детекцией с применением термоциклера типа Rotor-Gene Q при соответствующих температурно-временных режимах амплификации и измеряют накопление флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей: JOE/Yellow для специфического сигнала для ДНК ткани медведя (Ursus) и Cy5/Red - для внутреннего контрольного образца. Интерпретацию результатов проводят на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный.
Для повышения точности идентификации ткани медведя для внутреннего контрольного образца используют фаголизат бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, если концентрация фаговых частиц отклоняется в большую или меньшую сторону, то наблюдаются повторности сомнительных образцов. Для положительного контрольного образца используют смесь, содержащую фрагменты геномов ДНК ткани медведей (Ursus) и нативного бактериофага Т4 взятых в соотношении 1:1 со следующими нуклеотидными последовательностями:
 прямой праймер
прямой праймер
 обратный праймер
обратный праймер
 зонд.
зонд.
 - прямой праймер
- прямой праймер
 - обратный праймер
- обратный праймер
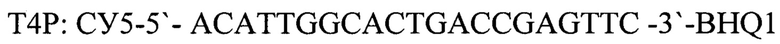 - зонд.
- зонд.
Использование для разных видов контроля различные формы материала бактериофага Т4: фаголизата и фрагмента генома со специфическими к нему праймерами и зондом обусловлено тем, что это позволяет контролировать корректное прохождение реакции в каждой пробирки, а также контролируется этап выделения ДНК из образцов. Кроме того, использование фаголизата бактериофага Т4, представляющего собой суспензию бактериофага, полученную после лизиса зараженных фагом клеток ткани, повышает чувствительность и упрощает процесс идентификации ткани медведей в продуктах. Использование нативного бактериофага, т.е. неповрежденного при исследовании, улучшает синтез ДНК, что также улучшает качество процесса идентификации.
При конструировании праймеров и зонда основными требованиями были: степень гомологии (комплементарность) с выбранным участком гена; отсутствие самокоплементарных участков внутри олигонуклеотидов и комплементарности друг другу, чтобы не допускать возникновения устойчивых вторичных структур (димеров); близость значений температуры отжига праймеров.
Конструирование специфических праймеров и зонда осуществляли с помощью компьютерных программ на основании анализа нуклеотидных последовательностей референтных штаммов и изолятов, опубликованных на ресурсе GenBank и подбора условий для проведения ПЦР в реальном времени с применением разработанных праймеров и зонда, несущего флуорофор и тушитель, и комплементарного части амплифицируемого со специфическими праймерами фрагмента.
Праймеры, специфичные для ДНК медведей (Ursus) были отобраны на основе гена 12S РНК (Ursus arctos 12S ribosomal RNA gene, partial sequence; and tRNA-Val gene, complete sequence; mitochondrial, код доступа AY012153.1 complete genome, участок между 500 и 700). Праймеры были спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность. Для детекции продуктов амплификации был подобран оли-гонуклеотидные флуоресцентно-меченный зонд Ursus Р (комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров Ursus F и Ursus R) Зонд был помечен красителем FAM. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР. Ни одна из выбранных последовательностей не обнаружена в геномах каких либо животных (не включая представителей рода Ursus) и любых видов растений, которые потенциально могут быть использованы при производстве кормов и пищевых продуктов.
В качестве внутреннего контроля использовался бактериофаг Т4, имеющий геномную ДНК порядка 170 тысяч пар нуклеотидов (Enterobacteria phage Т4Т, complete genome GenBank: HM137666.1). В результате анализа был выбран участок между 400 и 600 нуклеотидами, содержащий уникальные нуклеотидные последовательности, рассчитаны первичные структуры олигонуклеотидных праймеров, фланкирующих выбранный участок генома. Праймеры были спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность.
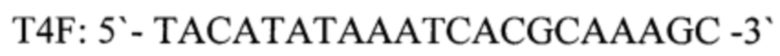 - прямой праймер
- прямой праймер
 - обратный праймер
- обратный праймер
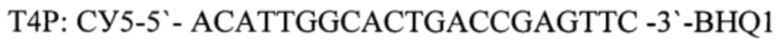 - зонд.
- зонд.
Для детекции продуктов амплификации подобран олигонуклеотидный флуоресцентно-меченный зонд Т4Р, комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров T4F и T4R. Зонд был помечен красителем Су5. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР.
Пример конкретного осуществления способа идентификации
Для подтверждения эффективности способа были использованы сухие корма в виде рыбной и мясной муки; сырые и термически обработанные мясные продукты, т.е. мясные полуфабрикаты.
От пробы плотной консистенции отбирают на исследование общую пробу весом 10-50 г. Гранулированную или консервированную продукцию перед исследованием (10-20 г) растирают в ступке до гомогенного состояния.
Лабораторные пробы (20 - 40 мг) отбирают на исследование в одноразовые микропробирки вместимостью 1,5 мл в двух повторах. Отобранные лабораторные пробы направляют на выделения ДНК.
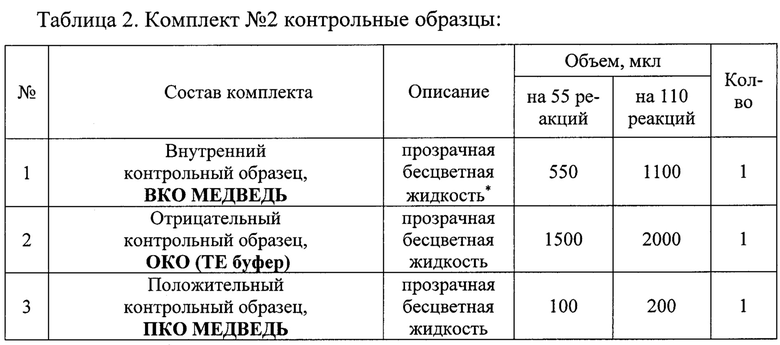
Исследование проводят с помощью набора реагентов «ПЦР-МЕДВЕДЬ-ФАКТОР». Набор состоит из комплекта реагентов для проведения мультиплексной ПНР (комплект №1) и комплекта контрольных образцов (комплект №2). Набор выпускается в двух вариантах: 1) Для анализа 55 образцов (включая контрольные образцы)
2) Для анализа 110 образцов (включая контрольные образцы).
Наборы используют в соответствии с инструкцией по применению набора реагентов «ПЦР-МЕДВЕДЬ-ФАКТОР» для определения ДНК ткани медведей (Ursus) методом полимеразной цепной реакции (ПЦР) с флуоресцентной детекцией в РВ ТУ 21.10.60-163-51062356-2018, для диагностики in vitro, http://www.vetfaktor.ru/.
Состав набора приведен в Таблицах 1 и 2.

Исследования состоит из трех этапов:
- экстракция нуклеиновая кислота (НК);
- проведение реакции ПЦР РВ;
- учет результатов анализа.
Для экстракции (выделение) НК из исследуемых проб отбирают необходимое количество одноразовых пробирок объемом 1,5 мл, включая отрицательный контроль выделения. Во все пробирки с исследуемыми образцами, включая пробирку для отрицательного контрольного образца (ОКО), вносят по 10 мкл внутреннего контрольного образца (ВКО) для ткани медведей, в качестве которого, используют фаголизат бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл.
Следующий этап это подготовка образцов к проведению ПЦР.
Общий объем реакционной смеси - 25 мкл, объем ДНК-пробы - 10 мкл.
Успешное прохождение реакции контролируют использованием положительного контрольного образца (ПКО) МЕДВЕДЬ, ВКО МЕДВЕДЬ и ДНК буфера. В качестве ПКО используют смесь содержащую фрагменты геномов ткани медведей и нативного бактериофага Т4 взятых в соотношении 1:1, со следующими нуклеотидными последовательностями:
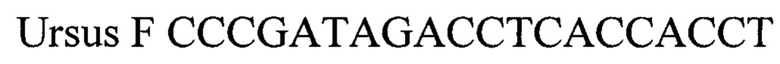 прямой праймер
прямой праймер
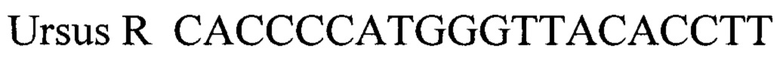 обратный праймер
обратный праймер
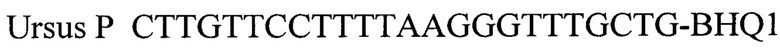 зонд.
зонд.
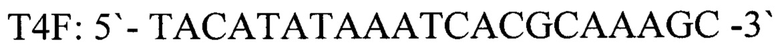 - прямой праймер
- прямой праймер
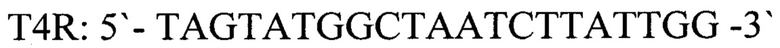 - обратный праймер
- обратный праймер
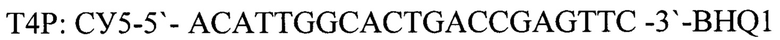 - зонд.
- зонд.
В отдельной пробирке смешивают компоненты набора из расчета на каждую реакцию:
5 мкл ПЦР СМЕСЬ МЕДВЕДЬ;
10 мкл ПЦР БУФЕР МЕДВЕДЬ;
0,5 мкл TAQ POLYMERASE
Перемешивают смесь на вортексе и сбросывают капли кратковременным центрифугированием.
Отбирают необходимое количество пробирок для амплификации ДНК исследуемых и контрольных проб. Вносят по 15 мкл приготовленной реакционной смеси.
Помещают подготовленные для проведения ПЦР пробирки в ячейки амплификатора и используют программное обеспечение прибора. Далее проводят ПЦР РВ с флуоресцентной детекцией.
Параметры температурно-временного режима амплификации на приборе «Rotor-Gene Q» представлены в таблице 3.

Интерпретация результатов анализа.
Полученные данные - кривые накопления флуоресцентного сигнала анализируются с помощью программного обеспечения используемого прибора для проведения ПЦР в соответствии с инструкцией производителя к прибору.
Учет результатов ПЦР РВ проводится по наличию или отсутствию пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию или отсутствию значения порогового цикла «Ct» для исследуемого образца).
Результат считается достоверным в случае корректного прохождения положительных и отрицательных контролей амплификации и экстракции ДНК в соответствии с таблицей 4.

Появление любого значения Ct в таблице 4 результатов для отрицательного контроля этапа экстракции ВК- на канале JOE/Yellow и для отрицательного контроля этапа ПЦР К- на любом из каналов свидетельствует о наличии контаминации реактивов или образцов. В этом случае результаты анализа для всех проб считаются недействительными. Требуется повторить анализ всех проб, а также предпринять меры по выявлению и ликвидации источника контаминации.
Образцы, для которых значение Ct по каналу Cy5/Red отсутствует или превышает 35 цикл (и при этом не получен положительный результат на канале JOE/Yellow) требуют повторного проведения исследования с этапа экстракции ДНК. Задержка в значениях пороговых циклов для исследуемых образцов указывает на присутствие ингибиторов в пробе(ах) или на ошибки при экстракции ДНК или при постановке реакции ПЦР РВ.
В образце обнаружена ДНК ткани медведей (Ursus), если наблюдается экспоненциальный рост сигнала на канале JOE/Yellow, при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4).
Если для исследуемого образца по каналам JOE/Yellow значение Ct определяется позднее 37 цикла при корректном прохождении положительных и отрицательных контролей, образец исследуется повторно с этапа экстракция ДНК. Если при повторной постановке Ct более 37 результат считается отрицательным.
Образец считается отрицательным ДНК (Ursus) не обнаружена), если не определяется значение Ct (не наблюдается рост специфического сигнала) на канале JOE/Yellow при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4), а значение Ct по каналу Cy5/Red менее 35.
Для исследуемых образцов (сухой корм и мясные полуфабрикаты) предел точности содержания тканей медведей представлен в таблице 5.

Для доказательства эффективности использования ПЦР с флуоресцентной детекцией в режиме реального времени проводился сравнительный анализ чувствительности заявляемого с прототипом, в котором использовался метод ПЦР с использованием внутреннего контроля в виде суспензии бактериофага, а в заявляемом - использовался фаголизат бактериофага и геном нативного бактериофага. Оказалось чувствительность ПЦР при обнаружении примеси ткани медведей в кормах и в мясных фаршах примерно в 1,5 раза выше. Трудоемкость и стоимость процесса определения ДНК ткани медведей в кормах и фаршах снизилась на 3-5%.
--->
Перечень последовательностей
<110> Федеральное государственное бюджетное образовательное учреждение высшего образования «Кубанский государственный аграрный университет имени И.Т. Трубилина».
<120> Способ идентификации ДНК ткани медведя (Ursus) в сухих кормах и мясных полуфабрикатах
<140> 2019133133
< 160> 6
< 210> 1
< 211> 20
< 212> ДНК
< 213> Ursus
< 400> 1
cccgatagacctcaccacct 20
< 210> 2
< 211> 20
< 212> ДНК
< 213> Ursus
< 400> 2
caccccatgggttacacctt 20
< 210> 3
< 211> 24
< 212> ДНК
< 213> Ursus
< 400> 3
cttgttccttttaagggtttgctg 24
< 210> 4
< 211> 21
< 212> ДНК
< 213> Бактериофаг Т4
< 400> 4
tacatataaatcacgcaaagc 21
< 210> 5
< 211> 21
< 212> ДНК
< 213> Бактериофаг Т4
< 400> 5
tagtatggctaatcttattgg 21
< 210> 6
< 211> 21
< 212> ДНК
< 213> Бактериофаг Т4
< 400> 6
acattggcactgaccgagttc 21
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Тест-система для идентификации ДНК ткани медведя (Ursus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726429C1 |
| Способ идентификации ДНК ткани кошки домашней (Felis silvestris catus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2728662C1 |
| Способ идентификации видовой принадлежности тканей крыс и мышей в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2742952C1 |
| Способ идентификации ДНК ткани ежа обыкновенного (Erinaceus europaeus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726433C1 |
| Способ определения ДНК ткани дятла (Picidae) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2714287C1 |
| Способ идентификации ДНК ткани перепелки обыкновенной (Coturnix coturnix) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2734035C1 |
| Тест-система для идентификации ДНК ткани ежа обыкновенного (Erinaceus europaeus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2725215C1 |
| Тест-система для идентификации ДНК тканей крыс и мышей в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2725539C1 |
| Тест-система для определения ДНК ткани дятла (Picidae) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2725216C1 |
| Тест-система для идентификации ДНК ткани кошки домашней (Felis silvestris catus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2728639C1 |
Изобретение относится к ветеринарной микробиологии, в частности к методам определения видовой принадлежности мяса с помощью полимеразной цепной реакции. Для повышения точности идентификации видовой принадлежности, упрощения процесса подготовки и уменьшения его стоимости, в способе идентификации ДНК ткани медведей (Ursus) в сухих кормах и мясных полуфабрикатах, включающем выделение ДНК из ткани животного сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 копий нуклеотидных последовательностей на 1 мкл, положительного контрольного образца в виде смеси, содержащей фрагменты геномов животного и бактериофага Т4 с нуклеотидной последовательностью:
 - прямой праймер
- прямой праймер
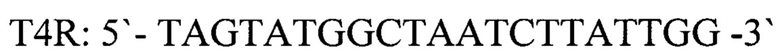 - обратный праймер
- обратный праймер
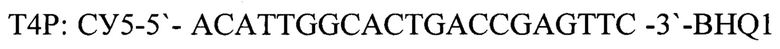 - зонд, взятых в соотношении 1:1, и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный, согласно изобретению выделяют ДНК из ткани медведя (Ursus) и для внутреннего контрольного образца используют фаголизат бактериофага Т4, а для положительного контрольного образца - фрагменты геномов нативного бактериофага Т4 и ткани медведя (Ursus) со следующей нуклеотидной последовательностью:
- зонд, взятых в соотношении 1:1, и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный, согласно изобретению выделяют ДНК из ткани медведя (Ursus) и для внутреннего контрольного образца используют фаголизат бактериофага Т4, а для положительного контрольного образца - фрагменты геномов нативного бактериофага Т4 и ткани медведя (Ursus) со следующей нуклеотидной последовательностью:
 - прямой праймер,
- прямой праймер,
 - обратный праймер,
- обратный праймер,
 - зонд. 5 табл.
- зонд. 5 табл.
Способ идентификации ДНК ткани медведей (Ursus) в сухих кормах и мясных полуфабрикатах, включающий выделение ДНК из ткани животного сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, положительного контрольного образца в виде смеси, содержащей фрагменты геномов животного и бактериофага Т4 с нуклеотидной последовательностью:
T4F: 5'-TACATATAAATCACGCAAAGC-3' - прямой праймер,
T4R: 5'-TAGTATGGCTAATCTTATTGG-3' - обратный праймер,
Т4Р: СУ5-5'-ACATTGGCACTGACCGAGTTC-3'-BHQ1 - зонд, взятых в соотношении 1:1, и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный, отличающийся тем, что выделяют ДНК из ткани медведя (Ursus) и для внутреннего контрольного образца используют фаголизат бактериофага Т4, а для положительного контрольного образца - фрагменты геномов нативного бактериофага Т4 и ткани медведя (Ursus) со следующей нуклеотидной последовательностью:
Ursus F CCCGATAGACCTCACCACCT - прямой праймер,
Ursus R CACCCCATGGGTTACACCTT - обратный праймер,
Ursus Р CTTGTTCCTTTTAAGGGTTTGCTG-BHQ1 - зонд.
| Способ идентификации видовой принадлежности баранины и говядины в продовольственном сырье, кормах и пищевых продуктах | 2018 |
|
RU2694713C1 |
| Колосник для воспринятия золы и шлака в механических топках | 1925 |
|
SU2560A1 |
| ANIS DTN-AMAR, et al, Universal direct PCR amplification system: a time- and cost-effective tool for high-throughput applications, Biotech, 2017, p | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
Авторы
Даты
2020-07-14—Публикация
2019-10-16—Подача