Изобретение относится к области медицины, биотехнологии, молекулярной биологии и может быть использовано для одновременной детекции возбудителя сапа Burkholderia mallei и возбудителя мелиоидоза Burkholderia pseudomallei методом полимеразной цепной реакции в режиме реального времени, как в практическом здравоохранении, так и для научных исследований в области микробиологии, эпидемиологии и патогенеза сапа и мелиоидоза.
Два близкородственных вида рода Burkholderia - В. mallei и В. pseudomallei - вызывают тяжелые, потенциально смертельные заболевания у людей. В. mallei является возбудителем сапа - заболевания, которое встречается по всему миру. Болезнь в основном поражает непарнокопытных, но люди могут заразиться воздушно-капельным путем или при контакте с больными животными или инфицированным материалом через поврежденную кожу или слизистые оболочки. Время от времени сообщается о случаях заболеваний у людей, также есть несколько сообщений о заражении людей в лабораторных условиях [Srinivasan A., Kraus С.N., Deshazer D., Becker P.М., Dick J.D., Spacek L., Bartlett J.G., Byrne W.R., Thomas D.L. (2001). Glanders in a military research microbiologist. N. Engl. J. Med. 345, 256-25810]. Клинические проявления заболевания зависят от способа заражения. При сапе наблюдаются лихорадка, пневмония, абсцессы в печени, селезенке, на коже и мышцах, бактериемия. При отсутствии лечения смерть наступает через 7-10 дней в 95% случаев и в 50% случаев при лечении антибиотиками [Spickler A.R. Glanders. In: Dvorak G., Spickler A.R., Roth J.A., editors. Handbook For Zoonotic Diseases of Companion Animals. 1 ed. Ames: Center for Food Security and Public Health; 2008. p.133±5].
Кроме того, микроорганизмы являются потенциальным агентом биотерроризма: для инфицирования требуются низкие дозы, заболеваемость и смертность высокие, возбудитель устойчив ко многим антибиотикам, их можно распространить в виде аэрозоля [http://www.selectagents.gov/resources/List_of_Select_Agents_and_Toxins_2012-12-4.pdf].
В. pseudomallei является возбудителем мелиоидоза - заболевания человека и животных, похожего на сап. Мелиоидоз распространен во всех тропических и субтропических областях, особенно в Южной и Юго-Восточной Азии и северной части Австралии. Случаи заболеваний, привезенных туристами, зарегистрированы за пределами эндемичных районов [Cuadros J., Gil Н., Miguel J.D., Marabe G., Gomez-Herruz T.A., Lobo В., Marcos R., Anda P: Case report: melioidosis imported from west Africa to Europe. AmJTrop Med Hyg 2011, 85(2):282-284]. Существует угроза заноса возбудителя на территорию России из эндемичных стран из-за увеличения транспортных связей, грузо- и пассажироперевозок, расширения туристических поездок российских граждан в эндемичные регионы, привлечения иностранной рабочей силы и незаконной миграции населения.
Бактерии обычно обитают во влажных почвах и стоячих водах, например, на рисовых полях в эндемичных регионах. В группе риска находятся рабочие рисовых полей, коренное население южной и восточной Азии, туристы и люди, страдающие от иммуносупрессивных заболеваний, в т.ч. сахарного диабета, цирроза печени, талассемии, почечной недостаточности. Заражение происходит воздушно-капельным путем, при порезах или приеме загрязненной воды. Общая смертность составляет 40% в северном Таиланде, 39% в Сингапуре и 19% в Австралии. Число случаев мелиоидоза, зарегистрированных за пределами Восточной Азии и северной Австралии, растет.
Для диагностики сапа и мелиоидоза применяются микробиологические методы, иммунологические тесты на основе моноклональных антител и молекулярные методы для идентификации нуклеотидных последовательностей возбудителя. Существующие бактериологические и иммунологические методы идентификации В. mallei недостаточно эффективны для экспресс-диагностики. Для выявления и идентификации возбудителя сапа в последнее время все более активно используют молекулярно-генетические методы, большинство из них основано на амплификации нуклеиновых кислот (традиционная ПЦР, ПЦР в режиме реального времени и др.). ПЦР в режиме реального времени (ПЦР-РВ) - полимеразная цепная реакция, при которой детекция ПЦР-продукта происходит в реальном времени по мере его накопления. В основе метода лежит процесс экспоненциального увеличения количества ампликона при повторении циклов амплификации специфичных фрагментов, включающих температурную денатурацию ДНК, отжиг праймеров и последующую достройку полинуклеотидных цепей с этих праймеров ДНК-полимеразой. Наличие положительного сигнала оценивается по накоплению флуоресценции в режиме реального времени. Преимущества данной методики - возможность анализировать динамику накопления ампликонов, высокая специфичность детекции, отсутствие постамплификационных этапов (не нужно проводить электрофорез), нет риска контаминации продуктами амплификации, возможность количественной оценки ДНК, использование флуоресцентных зондов.
Сложность при разработке молекулярно-биологических тестов заключается в том, что генетически В. mallei и В. pseudomallei очень близки - более 99% их последовательностей гомологичны, поэтому дифференцировать их друг от друга с помощью молекулярно-генетических методов затруднительно.
Имеются сведения о разработанных ПЦР тест-системах, в том числе мультиплексных, для детекции и дифференциации В. mallei и В. pseudomallei [Janse I., Hamidjaja R.A., Hendriks A.C., van Rotterdam B.J. Multiplex qPCR for reliable detection and differentiation of Burkholderia mallei and Burkholderia pseudomallei. BMC Infect Dis. 2013;13:86]. Для дифференциации обоих микроорганизмов в качестве мишеней были использованы сигнатурные последовательности mau (локус ВМА2524.1) и psu (локус BPSS1387), которые являлись уникальными для В. mallei и В. pseudomallei, соответственно. Однако, позже было установлено, что праймеры и зонд, комплементарные последовательности mau, позволяли обнаруживать ДНК В. pseudomallei не всех штаммов. А олигонуклеотиды к гену mau выявляли ДНК некоторых штаммов возбудителя мелиоидоза.
Известно о применении ПЦР-РВ с праймерами к последовательностям гена fliP, который кодирует белок флагеллин Р, что позволило выявить В. mallei в клинических образцах. Предложенные авторами праймеры были специфичными и не направляли синтез с ДНК 27 штаммов В. pseudomallei, 29 штаммов других буркхолдерий и гетерологичных микроорганизмов [Tomaso Н., Scholz Н.С, Al Dahouk S., Eickhoff М, Treu Т.М., Wernery R., Wernery U., Neubauer H. Development of a 5'-Nuclease Real-Time PCR Assay Targeting fliP for the Rapid Identification of Burkholderia mallei in Clinical Samples. Clin Chem. 2006; 52(2):307-10].
Проведенный поиск по патентным базам и научно-техническим источникам информации показал, что наиболее близким аналогом являются специфичные праймеры на основе фрагментов гена fliP. (Патент RU №2551208 «Набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации В. mallei и дифференциации его от В. pseudomallei».) Однако, данное изобретение не позволяет одновременно выявлять ДНК обоих патогенов.
Поэтому, существует необходимость создания диагностикума в формате мультиплексной ПЦР-РВ для одновременной детекции ДНК возбудителей сапа и мелиоидоза.
Техническим результатом предлагаемого изобретения является разработка высокоспецифичных олигонуклеотидных праймеров для быстрого, специфичного и чувствительного способа одновременной детекции ДНК возбудителей сапа и мелиоидоза.
Технический результат достигается, тем, что предложен набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для выявления ДНК возбудителей сапа и мелиоидоза методом ПЦР в режиме реального времени, содержащий олигонуклеотиды SEQ ID №3; SEQ ID №4, SEQ ID №7; комплементарные к гену-мишени IS407A/fliP бактерий вида В. mallei, кодируемому нуклеотидной последовательностью SEQ ID №: 1 и олигонуклеотиды SEQ ID №5; SEQ ID №6, SEQ ID №8; комплементарные к гену-мишени Orf12 области кластера генов TTS1 бактерий вида В. pseudomallei кодируемому нуклеотидной последовательностью SEQ ID №: 2
Технический результат также достигается, тем, что предложен способ выявления ДНК возбудителей сапа и мелиоидоза методом ПЦР с детекцией продукта в режиме реального времени, с использованием праймеров
SEQ ID №:3 (прямой праймер BmF IS407A-fliP В. mallei),
SEQ ID №:4 (обратный праймер BmR IS407A-fliP В. mallei),
SEQ ID №:6 (прямой праймер BpF orf12 кластера генов TTS1B. pseudomallei),
SEQ ID №:7 (обратный праймер BpR orj12 кластера генов TTS1.S. pseudomallei)
и флуоресцентно-меченых зондов:
SEQ ID №:5 Bm_FAM (IS407A-fliP B. mallei Bm_FAM с флуоресцентным красителем FAM и тушителем флуоресценции RTQ1),
SEQ ID №:8 Вр_Су5 (orf12 кластера генов TTS1 В. pseudomallei Вр_Су5 с флуоресцентным красителем Су5 и тушителем флуоресценции BHQ2), которые направляют синтез и накопление в одной пробирке фрагментов гена-мишени IS407A/fliP бактерий вида В. mallei фрагмента гена Orf12 области кластера генов TTS1 бактерий вида В. pseudomallei в следующих условиях: предварительный прогрев смеси 95°С - 10 мин, затем 40 циклов денатурации при 94°С - 15 сек и амплификации-элонгации при 60°С -1 мин, детекция продукта амплификации с ДНК В. mallei осуществляется при регистрации флуоресцентного сигнала от метки FAM, детекция продукта амплификации с ДНК В. pseudomallei при регистрации флуоресцентного сигнала от метки Су5.
Технический результат также достигается благодаря использованию в качестве целевых мишеней нуклеотидной последовательности IS407A-fliP, характерной только для В. mallei, и нуклеотидной последовательности гена orf12 кластера генов TTS1 (секреторной системы типа III), которая считается надежной мишенью для видоспецифической идентификации В. pseudomallei.
Для выявления ДНК В. mallei и В. pseudomallei проводят реакцию с использованием одновременно двух пар олигонуклеотидных праймеров, гомологичных фрагментам генов SEQ ID №: 1 В. mallei и SEQ ID №: 2 В. pseudomallei, а также двух соответствующих олигонуклеотидных TaqMan зондов с флуоресцентными красителями, отличающихся спектром поглощения и эмиссии, что позволяет выявлять сигналы от различных мишеней.
Обоснование выбора олигонуклеотидных праймеров и ДНК-мишени.
В геноме обоих микроорганизмов присутствует инсерционный элемент IS407A. Было обнаружено, что при встраивании этого элемента в последовательность гена флагеллина Р (fliP) В. mallei теряет подвижность. Кроме того, усеченный ген fliP имеет противоположную ориентацию. Эта генетическая особенность и неподвижность клетки характерна для всех видов В. mallei. У В. pseudomallei вставки IS407A внутри гена fliP нет, кроме того, последовательности fliP у В. pseudomallei и других видов буркхолдерий, таких как В. cepacia, отличаются. Область IS407A/fliP является уникальной для вида В. mallei. Различия этих генетических структур В. mallei и В. pseudomallei были использованы для расчета праймеров с целью идентификации двух микроорганизмов. Нуклеотидная последовательность гена orf12 кластера генов TTS1 является уникальной для В. pseudomallei.
Последовательности SEQ ID №1 и SEQ ID №2 (гены-мишени) для подбора праймеров с целью детекции ДНК возбудителей сапа и мелиоидоза методом ПЦР-РВ были получены из базы GenBank NCBI (National Center for Biotechnology Information, США).
На основании последовательностей выбранных фрагментов генов IS407A/fliP В. mallei и orf12 кластера генов TTS1 В. pseudomallei с помощью программы GenScript Online PCR Primers Designs Tool (https://www.genscript.com/tools/pcr-primers-designer) подобраны олигонуклеотидные праймеры, обеспечивающие амплификацию фрагментов указанных генов-мишеней размером 201 п. н., 194 п. н., соответственно, а также гибридизационные зонды формата TaqMan. Выбор праймеров осуществляли с учетом возможности их использования в мультиплексной ПЦР с регистрацией результатов в режиме «реального времени».
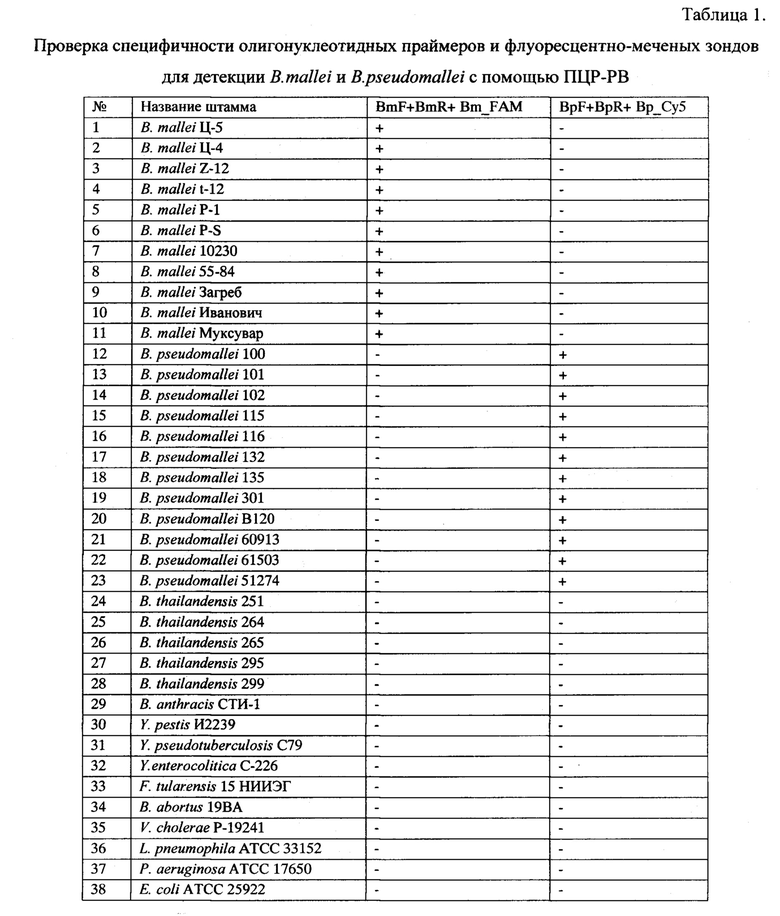
Апробация праймеров была осуществлена на 12 штаммах Burkholderia mallei: В. mallei 10230, В. mallei 55-84, В. mallei Загреб, В. mallei Иванович, В. mallei Muksuwar, В. mallei P-l, В. mallei P-S, В. mallei Ц-5, В. mallei Z-12, В. mallei Ц-4, В. mallei t-12, В. mallei В120; 11 штаммах Burkholderia pseudomallei: В. pseudomallei 100, В. pseudomallei 61503, В. pseudomallei 101, В. pseudomallei 51274, В. pseudomallei 115, В. pseudomallei 116, В. pseudomallei 60913, В. pseudomallei 301, В. pseudomallei 102, В. pseudomallei 135, В. pseudomallei 132 из Государственной коллекции патогенных микроорганизмов и клеточных культур «ГКПМ-Оболенск» (табл. 1).
В экспериментах по определению чувствительности использовали штаммы В. mallei Ц-5 и В. pseudomallei 100, используя для выделения ДНК обеззараженные суспензии микроорганизмов в концентрациях от 1×106 м.к./мл до 1×101 м.к./мл.
Изобретение иллюстрируется следующими графическими материалами (табл. 2, фиг. 1-5) и примерами (1-3).
Таблица 1. Проверка специфичности олигонуклеотидных праймеров и флуоресцентно-меченых зондов для детекции В. mallei и В. pseudomallei с помощью ПЦР-РВ.
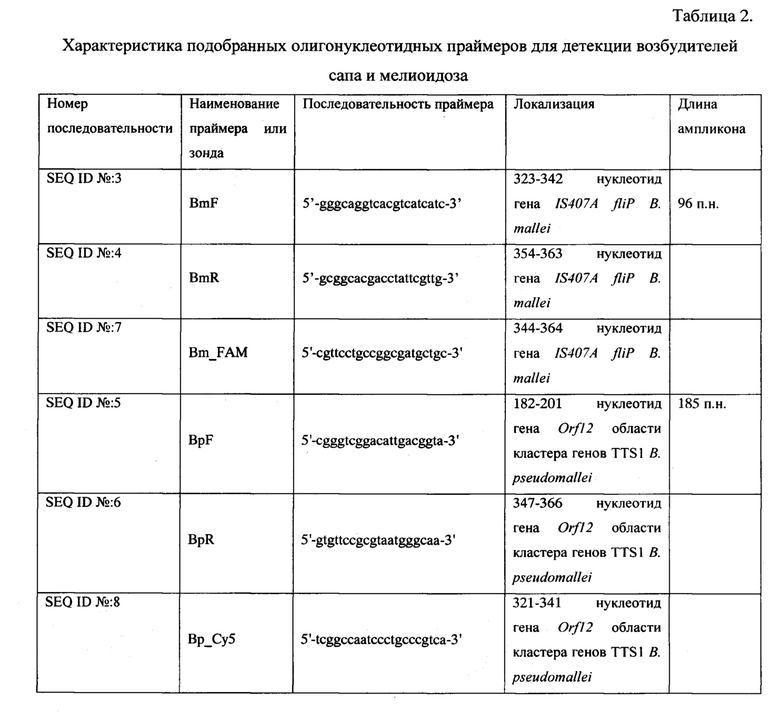
Таблица 2. Характеристика подобранных олигонуклеотидных праймеров для детекции возбудителей сапа и мелиоидоза.
Фиг. 1. Последовательность SEQ ID №1 - нуклеотидная последовательность участка генома IS407A/fliP штамма В. mallei АТСС 23344. Серыми прямоугольниками выделены участки отжига праймеров и зонда и комплементарные им последовательности.
Фиг 2. Последовательность SEQ ID №2 - нуклеотидная последовательность гена orf12 кластера генов TTS1 В. pseudomallei. Серыми прямоугольниками выделены участки отжига праймеров и зонда и комплементарные им последовательности.
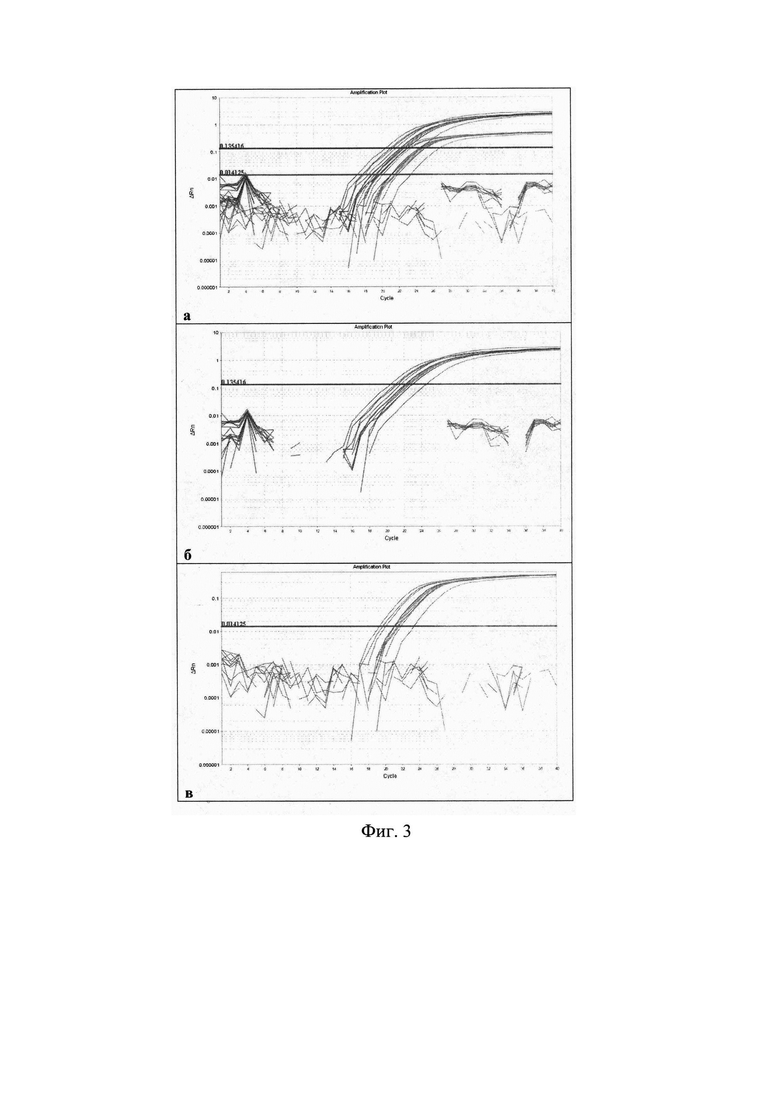
Фиг 3. Определение специфичности олигонуклеотидов SEQ ID №3; SEQ ID №4, SEQ ID №7 для детекции В. mallei и олигонуклеотидов SEQ ID №5; SEQ ID №6, SEQ ID №8 для обнаружения В. pseudomallei с помощью ПЦР-РВ в мультиплексном формате.
а - графики амплификации с ДНК В. mallei с детекцией продукта по каналу FAM (красного цвета) и графики амплификации с ДНК В. pseudomallei с детекцией продукта по каналу Су5 (синего цвета).
б - графики амплификации с ДНК В. mallei с детекцией продукта по каналу FAM (красного цвета)
в - графики амплификации с ДНК В. pseudomallei с детекцией продукта по каналу Су5 (синего цвета).
Фиг 4. Результаты определения чувствительности набора праймеров SEQ ID №3; SEQ ID №4 и TaqMan-зонда SEQ ID №7.
Использована ДНК из l×106 м.к./мл; ДНК из 1×105 м.к./мл; 3 - ДНК из 1×104 м.к./мл; 4 - ДНК из l×103 м.к./мл; 5 - ДНК из l×102 м.к./мл; 6 - ДНК из l×101 м.к./мл штамма В. mallei Ц-5.
Фиг 5. Результаты определения чувствительности набора праймеров SEQ ID №5; SEQ ID №6 и TaqMan-зонда SEQ ID №8.
Использована ДНК из l06 м.к./мл; ДНК из l×105 м.к./мл; 3 - ДНК из l×104 м.к./мл; 4 - ДНК из l×103 м.к./мл; 5 - ДНК из l×102 м.к./мл; 6 - ДНК из l×101 м.к./мл штамма В. pseudomallei 100.
Примеры конкретного выполнения.
Пример 1. Методика конструирования олигонуклеотидных праймеров для детекции ДНК возбудителей сапа и мелиоидоза методом ПЦР с детекцией продукта в режиме реального времени.
На основе анализа in silico нуклеотидных последовательностей возбудителя сапа, присутствующих в базе данных Genbank, для конструирования прямого и обратного праймеров и TaqMan-зонда был выбран участок генома IS407A/fliP В. mallei размером 545 п. н. К участку гена SEQ ID №1 (фиг. 1) были подобраны праймеры SEQ ID №3, SEQ ID №4 и SEQ ID №7. Расчетная длина предполагаемого ампликона, фланкируемого праймерами SEQ ID №3 и SEQ ID №4, составила 96 п. н. (табл. 2). На Фиг. 1 показаны участки отжига праймеров и TaqMan-зонда, а также комплементарные им последовательности, которые выделены прямоугольниками серого цвета.
Для конструирования прямого и обратного праймеров и TaqMan-зонда для выявления ДНК В. pseudomallei в качестве мишени использовали ген Orf12 области кластера генов TTS1 В. pseudomallei размером 456 п. н (GenBank: AF074878.2). К участку гена SEQ ID №2 (фиг. 2) были подобраны праймеры SEQ ID №5, SEQ ID №6 и SEQ ID №8. Расчетная длина предполагаемого ампликона, фланкируемого праймерами SEQ ID №5 и SEQ ID №6, составила 185 п. н. (табл. 2). На Фиг. 2 показаны участки отжига праймеров и TaqMan-зонда, а также комплементарные им последовательности, которые выделены прямоугольниками серого цвета.
При выборе олигонуклеотидных праймеров использовали программу для расчета праймеров он-лайн GenScript Online PCR Primers Designs Tool (https://www.genscript.com/tools/pcr-primers-designer). При подборе праймеров руководствовались требованиями к олигонуклеотидам, используемым в ПЦР. Анализ формирования вторичных структур (димеров, шпилек) выбранными праймерами проводили с помощью компьютерной программы mfold (http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form). Структуры всех олигонуклеотидов сравнивались с помощью информационного ресурса NCBI BLAST (http://blast.ncbi.nlm.nih.gov/Blast.cgi) с последовательностями ДНК, размещенными в базе данных GenBank. При проведении этого анализа олигонуклеотиды с существенной гомологией с ДНК каких-либо других организмов исключали. Для определения специфичности праймеров моделировали ПЦР in silico в отношении секвенированных геномов микроорганизмов с помощью In silico PCR amplification (http://insilico.ehu.es/PCR/). Показана теоретическая пригодность праймеров для успешной инициации реакции амплификации и гибридизации.
Пример 2. Амплификация специфических фрагментов ДНК с помощью разработанных праймеров для детекции возбудителей сапа и мелиоидоза методом мультиплексной ПЦР-РВ.
В работе использовали программируемый термоциклер (амплификатор) для проведения ПЦР с детекцией результата в режиме реального времени 7500 Real Time PCR System (Applied Biosystems).
Реакционная смесь объемом 25 мкл содержала 1х ПЦР-буфер-Б для Taq ДНК-полимеразы (Синтол, Россия), 1 ед. Taq ДНК-полимераз с ингибирующими активность фермента антителами (Синтол, Россия), 2,5 мМ MgCl2 (Синтол, Россия), 0,25 мМ каждого дНТФ (Синтол, Россия), 4 праймера (Синтол, Россия): 1,0 мкМ SEQ ID №3 (BmF), 1,0 мкМ SEQ ID №4 (BmR), 1,0 мкМ SEQ ID №5 (BpF), 1,0 мкМ SEQ ID №6 (BpR), 2 TaqMan зонда (Синтол, Россия): 0,5 мкМ SEQ ID №7 (Bm_FAM), 0,5 мкМ SEQ ID №8 (Bp_Cy5), 5 мкл матрицы. Реакцию проводили по следующей программее: предварительный прогрев смеси 95°С - 10 мин, затем 40 циклов денатурации при 94°С - 15 сек и амплификации-элонгации при 60°С - 1 мин, детекцию продукта амплификации с ДНК В. mallei осуществляли при регистрации флуоресцентного сигнала от метки FAM, детекцию продукта амплификации с ДНК В. pseudomallei - при регистрации флуоресцентного сигнала от метки Су5.
Детекцию специфических фрагментов ДНК патогенов осуществляли с использованием флуоресцентных зондов, которые представляют собой олигонуклеотиды с флуоресцентной меткой на 5'-конце и гасителем флуоресценции на 3'-конце (TaqMan-зонд). TaqMan зонд комплементарен участку амплифицируемой области. Гаситель поглощает испускаемое флуоресцентной меткой излучение. В присутствии матрицы зонд гибридизуется с участком ДНК. Во время стадии элонгации полимераза синтезирует комплементарную цепь ДНК и, дойдя до участка, гибридизованного с пробой, начинает расщеплять пробу за счет 5'-экзонуклеазной активности. В результате флуоресцентная метка отделяется от гасителя, и ее свечение измеряет устройство, связанное с термоциклером. Сигнал накапливается в реальном времени с каждым циклом ПЦР. Интенсивность флуоресценции на этапе отжига в каждом цикле амплификации прямо пропорциональна концентрации ампликона.
Результат амплификации считали положительными при наличии флуоресцентного сигнала, пересекающего пороговую линию. Фиг. 3 отображает графики амплификации с ДНК штаммов возбудителей сапа и мелиоидоза с набором праймеров и флуоресцентно-меченых зондов, подобранных к последовательностям SEQ ID №1 и SEQ ID №2.
Пример 3. Определение специфичности и чувствительности разработанных олигонуклеотидных праймеров и TaqMan-зондов в мультиплексной реакции к последовательностям SEQ ID №1 для идентификации ДНК возбудителя сапа и SEQ ID №2 для идентификации ДНК возбудителя мелиоидоза.
Специфичность разработанных праймеров оценена на коллекции из 38 штаммов, из которых 12 штаммов В. mallei, 11 штаммов В. pseudomallei и 15 штаммов гетерологичных микроорганизмов (5 штаммов В. thailandensis, и по 1 штамму В. anthracis, F. tularensis, Y. pestis, Y. pseudotuberculosis, Y. enterocolitica, V. cholerae, B. abortus, L. pneumophila, P. aeruginosa, E.coli). Оценка специфичности показала, что праймеры SEQ ID №3 и SEQ ID №4 направляли синтез только с ДНК В. mallei, а праймеры SEQ ID №5 и SEQ ID №6 - только с ДНК В. pseudomallei, продукты амплификации с ДНК гетерологичных штаммов отсутствовали. Таблица 1 и Фиг. 3 отображает результат оценки специфичности соответствующих олигонуклеотидных праймеров и зондов при одновременной детекции обоих возбудителей.
Чувствительность реакции амплификации с разработанными специфичными олигонуклеотидами SEQ ID №3, SEQ ID №4 и SEQ ID №7 оценивали при исследовании проб ДНК, выделенных из бактериальных взвесей клеток В. mallei Ц-5.
Чувствительность реакции амплификации с разработанными специфичными олигонуклеотидами SEQ ID №5, SEQ ID №6 и SEQ ID №8 оценивали при исследовании проб ДНК, выделенных из бактериальных взвесей клеток В. pseudomallei 100.
Культуры патогенных буркхолдерий и близкородственных микроорганизмов из ампул высевали в бульон Хоттингера с 5% содержанием глицерина по ГОСТ 6259-75, рН 7,0±0,2 и инкубировали при температуре (37±1)°С в течение 18-24 ч. Затем культуры пересевали на агар Хоттингера с 5% содержанием глицерина и инкубировали в течение 24-48 ч при (37±1)°С.
Для культивирования гетерологичных микроорганизмов использовали общепринятые питательные среды для соответствующих видов («FT-агар» - для F. tularensis; бульон Хоттингера и агар Хоттингера - для В. anthracis, Y. pestis, Y. pseudotuberculosis и E.coli; бруцеллагар - для бруцелл; легионелбакагар - для легионелл). Инкубацию посевов культур проводили при 37°С и при 28°С - для Y. pestis.
Для дальнейшего использования в ПЦР готовили взвеси каждого штамма в концентрации, соответствующей 10 ME по стандартному образцу мутности бактерийных взвесей (комплект БАК) СОП №1-98 ООО «ОРМЕТ», Екатеринбург, эквивалентной 1⋅109 м.к./мл, которые затем разводили 0,9% раствором натрия хлорида от 1⋅106 м.к./мл до концентрации 1×10 м.к./мл для В.pseudomallei и В.mallei.
Из каждой культуры готовили отдельно суспензии в 2 мл 0,9% раствора натрия хлористого по отраслевому стандартному образцу мутности 10 единиц ГИСК им. Л.А. Тарасевича, что соответствует 1⋅108 м.к./мл для В. anthracis. 1⋅109 м.к./мл для Y. pestis, Y. pseudotuberculosis, Brucella, Legionella и E.coli, 5⋅1010 м.к./мл для F. tularensis. Суспензии с гетерологичными микроорганизмами разводили до концентрации 1⋅106 м.к./мл.
Учитывая, что возбудители сапа и мелиоидоза относятся к агентам II группы патогенности, пробоподготовку и обеззараживание материала для проведения ПЦР осуществляли согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Для обеззараживания в пробирки с тестируемыми микробными взвесями добавляли мертиолят натрия до конечной концентрации 0,01% (1:10000) и прогревали на водяной бане при температуре (56±1)°С в течение 30 мин. Затем в отдельные микроцентрифужные пробирки объемом 1,5 мл переносили по 100 мкл обработанных мертиолятом натрия проб. Выделение ДНК из проб микроорганизмов проводили путем нуклеосорбции и лизиса клеток гуанидинтиоцианатом с помощью набора «ДНК-сорб-В» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора) в соответствии с инструкцией по применению.
Чувствительность праймеров оценивали с помощью проб ДНК, выделенных из десятикратных разведений штаммов В. mallei Ц-5 и В. pseudomallei 100. Анализ результатов показал, что чувствительность разработанных праймеров составила l×102 м.к./мл для обоих возбудителей. Фиг. 4 и Фиг. 5 отображают результат определения чувствительности олигонуклеотидных праймеров.
Таким образом, разработанные праймеры и флуоресцентно-меченые зонды могут быть использованы для одновременного обнаружения ДНК возбудителей сапа и мелиоидоза и позволяют в короткий срок с высокой чувствительностью и специфичностью детектировать возбудителей сапа и мелиоидоза в пробах чистых культур. В результате проведенной оценки установлено, что выбранные праймеры и зонды для детекции возбудителей сапа и мелиоидоза методом ПЦР-РВ в мультиплексном формате обладают 100% специфичностью и чувствительностью - l×102 м.к./мл.
--->
Перечень последовательностей
Burkholderia mallei ATCC 23344
CDS complement(1..264)
/product="IS407A, transposase OrfA"
CDS complement(330..545)
/note="This gene is disrupted by a
65 kb insertion that is flanked by IS407
elements.identified by similarity to SP:P33133"
/product="flagellar biosynthetic protein FliP,
interruption-N"
1 tcagggcttt cccttgacga caaccttcaa cgcttccata tcgagcattg cttcggccag
61 cagtttcttc agtcgggcat tctccacctc gaggcccttg agccggcggg cttccgagac
121 ttccatgccg ccgaacttcg cgcgccaggt gtagaacgac gcgtcactga acccatgctt
181 cctgcacagt tccttgaccg gcataccggc ctcggcttcc ttcagaaacc cgatgatttg
241 ctgttccgta aagcgcttct tcatgttcgt cttcttctcc gaaaacgaac tttactagac
301 tccggctggc cctgtttgta gggggcaggt cacgtcatca tcagcagcat cgccggcagg
361 aacgacagca tcgtgagcag cagcatcgtc tgcacgctca acgaataggt cgtgccgccg
421 ttcgggcccg gcgccgagtt gaacgcaggc agcccggccg cctgcgcgca cgcgagcgcg
481 ggcgcgaggc cgatcaggat cgcgggcagc cagcgcgccg cgccgcgcag gcaagcggtt
541 ttcat
GenBank: AF074878.2
Burkholderia pseudomallei putative type III secretion system-associated gene cluster, complete sequence
organism= Burkholderia pseudomallei
gene="Orf12"
1 atgagcagcg agcgctatat ccaattgatt caggatctgt gcgcaaccgt cggactcgcc
61 aacgtcgata acgtgctcga agctcgcacg attgaagtgg aaggcttcga cgtgcgcctc
121 gattgcttcg acagcgatgc ggaggcgatg tacgccaatt tccatttcgg cacggtaacg
181 gcgggtcgga cattgacggt attcaggttg atgctggagg cgaacctgct cgtctatgcg
241 caggatcagg ctcagctcgg catcgatacc aacacgggcg gcgtcatact gataatccgg
301 ttgccgttta gtctcggcat tgacgggcag ggattggccg atctgcttgc ccattacgcg
361 gaacacggcc gctactggcg gcagagcatt atcgaatcga gcgacgaaat gttcgaaggc
421 atcgcttcgg gcgagtattt ctggctacga gcctga
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ Burkholderia mallei И ДИФФЕРЕНЦИАЦИИ ЕГО ОТ Burkholderia pseudomallei | 2014 |
|
RU2551208C1 |
| ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЕ ОЛИГОНУКЛЕОТИДНЫЕ ЗОНДЫ ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЕЙ САПА И МЕЛИОИДОЗА B. pseudomallei И B. mallei | 2010 |
|
RU2435861C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ РНК ВОЗБУДИТЕЛЕЙ САПА BURKHOLDERIA MALLEI И МЕЛИОИДОЗА BURKHOLDERIA PSEUDOMALLEI НА ОСНОВЕ ТРАНСКРИПЦИОННОЙ АМПЛИФИКАЦИИ (NASBA) В РЕЖИМЕ "РЕАЛЬНОГО ВРЕМЕНИ" | 2018 |
|
RU2699180C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ Burkholderia pseudomallei | 2014 |
|
RU2556810C1 |
| ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЙ ОЛИГОНУКЛЕОТИДНЫЙ ЗОНД ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЕЙ САПА И МЕЛИОИДОЗА B.pseudomallei И B.mallei | 2010 |
|
RU2435860C1 |
| ОЛИГОНУКЛЕОТИДНЫЙ ЗОНД ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЕЙ САПА И МЕЛИОИДОЗА B. pseudomallei И B. mallei | 2010 |
|
RU2439159C1 |
| ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ BmVAT5-Ch2s/BmVAT5-Ch2as ДЛЯ ОБНАРУЖЕНИЯ ФРАГМЕНТА ДИФФЕРЕНЦИАЦИИ ВМАА0107 ШТАММОВ ВОЗБУДИТЕЛЯ САПА | 2012 |
|
RU2474620C1 |
| Набор олигонуклеотидных праймеров для выявления вариантных штаммов Burkholderia thailandensis, содержащих кластер генов биосинтеза капсульного полисахарида, высоко гомологичный Burkholderia pseudomallei | 2019 |
|
RU2728356C1 |
| НАБОР ФЛУОРЕСЦЕНТНО-МЕЧЕНЫХ ОЛИГОНУКЛЕОТИДНЫХ ЗОНДОВ ДЛЯ ТИПИРОВАНИЯ ШТАММОВ Burkholderia mallei МЕТОДОМ АМПЛИФИКАЦИИ ДИФФЕРЕНЦИРУЮЩИХ ФРАГМЕНТОВ ДНК | 2014 |
|
RU2551227C1 |
| ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ ДЛЯ ГЕНОТИПИРОВАНИЯ B. mallei МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2012 |
|
RU2474618C1 |




Изобретение относится к областям биотехнологии. Предложен набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для ПЦР-РВ, который позволяет одновременно детектировать специфические для бактерий вида В. mallei фрагменты ДНК целевого гена, имеющего нуклеотидную последовательность SEQ ID №:1, и специфические для бактерий вида В. pseudomallei фрагменты ДНК целевого гена, имеющего нуклеотидную последовательность SEQ ID №:2. Для амплификации специфических фрагментов ДНК В. mallei ПЦР-РВ набор специфических олигонуклеотидов включает: прямой праймер - «BmF», имеющий нуклеотидную последовательность SEQ ID №:3, обратный праймер - «BmR», имеющий нуклеотидную последовательность SEQ ID№:4, флуоресцентно-меченый зонд - «Bm_FAM», имеющий нуклеотидную последовательность SEQ ID№:7. Для амплификации специфических фрагментов ДНК В. pseudomallei ПЦР-РВ набор олигонуклеотидов включает: прямой праймер - «BpF», имеющий нуклеотидную последовательность SEQ ID №:5, обратный праймер - «BpR», имеющий нуклеотидную последовательность SEQ ID №:6, флуоресцентно-меченый зонд - «Вр_Су5», имеющий нуклеотидную последовательность SEQ Ш№:8. Для выявления ДНК В. mallei и В. pseudomallei проводят реакцию с использованием одновременно двух пар олигонуклеотидных праймеров, гомологичных фрагментам генов SEQ ID №: 1 В. mallei и SEQ ID №: 2 В. pseudomallei, а также двух соответствующих олигонуклеотидных TaqMan зондов с флуоресцентными красителями, отличающихся спектром поглощения и эмиссии, что позволяет выявлять сигналы от различных мишеней. Заявленное изобретение позволяет в течение 2 часов с высокой специфичностью одновременно детектировать наличие возбудителя сапа - В. mallei и возбудителя мелиоидоза - В. pseudomallei - в пробах чистых культур в концентрации 100 м.к./мл. 2 н.п. ф-лы, 5 ил., 2 табл., 3 пр.
1. Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для выявления ДНК возбудителей сапа и мелиоидоза методом ПЦР с детекцией продукта в режиме реального времени, содержащий олигонуклеотиды SEQ ID №3; SEQ ID №4, SEQ ID №7, комплементарные к гену-мишени IS407A/fliP бактерий вида В. mallei, кодируемому нуклеотидной последовательностью SEQ ID №: 1, и олигонуклеотиды SEQ ID №5; SEQ ID №6, SEQ ID №8, комплементарные к гену-мишени Orf12 области кластера генов TTS1 бактерий вида В. Pseudomallei, кодируемому нуклеотидной последовательностью SEQ ID №:2.
2. Способ выявления ДНК возбудителей сапа и мелиоидоза методом ПЦР с детекцией продукта в режиме реального времени, отличающийся тем, что используют праймеры из набора по п. 1, которые направляют синтез и накопление в одной пробирке фрагментов гена-мишени IS407A/fliP бактерий вида В. mallei фрагмента гена Orf12 области кластера генов TTS1 бактерий вида В. pseudomallei при условии предварительного прогрева смеси 95°С - 10 мин, затем 40 циклов денатурации при 94°С - 15 сек и амплификации-элонгации при 60°С - 1 мин, детекция продукта амплификации с ДНК В. mallei осуществляется при регистрации флуоресцентного сигнала от метки FAM, детекция продукта амплификации с ДНК В. pseudomallei при регистрации флуоресцентного сигнала от метки Су5.
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ Burkholderia mallei И ДИФФЕРЕНЦИАЦИИ ЕГО ОТ Burkholderia pseudomallei | 2014 |
|
RU2551208C1 |
| TOMASO Н., et al, Development of a 5¢-Nuclease Real-Time PCR Assay Targeting fliP for the Rapid Identification of Burkholderia mallei in Clinical Samples | |||
| Clin Chem | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| JANSE I., Multiplex qPCR for reliable detection and differentiation of Burkholderia mallei and Burkholderia pseudomallei | |||
| BMC | |||
Авторы
Даты
2020-12-11—Публикация
2019-12-11—Подача