Изобретение относится к области генной инженерии и биотехнологии, а именно к числу средств - направляющих РНК, которые могут быть использованы в системе CRISPR-Cas12 в составе рибонуклеопротеиновых комплексов для выявления (обнаружения, детекции) ДНК вируса гепатита В.
Изобретение позволяет in vitro выявлять единичные копии ДНК вируса гепатита В.
Направляющие РНК, описанные в настоящей заявке, могут быть использованы для детекции ДНК вируса гепатита В после проведения специфической амплификации фрагментов генома вируса гепатита В. Амплификация при этом может быть проведена различными способами, среди которых полимеразная цепная реакция (PCR); петлевая изотермическая амплификация (LAMP); геликаза-зависимая амплификация (HDA); рекомбиназа-опосредованная амплификация (RPA); амплификация со смещением цепи (SDA); амплификация, основанная на последовательности нуклеиновых кислот (NASBA); опосредованная транскрипцией амплификация (ТМА); амплификация, опосредованная никирующим ферментом (NEAR); круговая амплификация (RCA) и многие другие виды амплификации.
Направляющие РНК, описанные в настоящей заявке, могут быть использованы для разработки высокочувствительных и высокотехнологичных диагностических систем нового поколения на основе CRISPR технологий для совершенствования методов диагностики инфекционных заболеваний.
Для решения эпидемиологических задач по расшифровке вспышек инфекционных болезней, выявления и идентификации возбудителя, а также детекции специфических бактериальных и вирусных генов необходимы разработка и внедрение в практику работы надзорных и мониторинговых служб современных технологий молекулярной эпидемиологии. Одной из таких технологий является использование элементов генетического редактирования системы CRISPR-Cas. Данная технология развивается достаточно эффективно в отношении создания средств лечения некоторых болезней, несмотря на ряд трудностей, связанных с возникновением непредвиденных мутаций. При углубленных исследованиях в области применения CRISPR-Cas системы, было выяснено, что она может быть использована для тонких диагностических процедур при выявлении возбудителя/ей инфекции у человека, а также их генотипирования.
В 2018 г. было показано, что один из ферментов CRISPR системы - Cas12 после распознавания своей целевой ДНК-мишени начинает неспецифически гидролизовать одноцепочечную ДНК. Такое свойство Cas12 можно использовать в качестве индикатора присутствия определенной мишени, например, генома вируса или бактерии. Исследователи использовали это открытие для создания технологической платформы обнаружения нуклеиновых кислот, известной как DETECTR (DNA Endonuclease Targeted CRISPR Trans Reporter - ДНК-нацеленная эндонуклеаза CRISPR трансрепортер). Предложенная платформа объединяет нуклеазу Cas12a, ее направляющую РНК, специфичную к нуклеиновой кислоте, и флуоресцентную репортерную молекулу. Впервые технология DETECTR была использована для выявления и генотипирования вируса папилломы человека. DETECTR в течение 1 ч позволила дифференцировать HPV16 и HPV18 в неочищенных экстрактах ДНК из культивируемых клеток человека и клинических образцов. При этом DETECTR корректно (сопоставимо с результатами ПЦР-анализа) идентифицировала HPV16 в 25 и HPV18 - в 23 из 25 клинических образцов [J.S. Chen, Ε. Ma, L.B. Harrington et al., CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity, Science 360(6387) (2018) 436-9, doi: 10.1126/science.aar6245].
He менее важным приложением системы CRISPR-Cas является идентификация патогенов и детекция специфических бактериальных генов с помощью платформы SHERLOCK (specific high-sensitivity enzymatic reporter unlocking - ферментативная специфическая высокочувствительная разблокировка репортера). Предложенная платформа объединяет нуклеазу Cas13a, ее направляющую РНК, специфичную к нуклеиновой кислоте, и флуоресцентную репортерную молекулу. Комплекс Cas13a связывает и расщепляет предварительно амплифицированную нуклеиновую кислоту-мишень с высокой специфичностью. С помощью SHERLOCK удалось корректно дифференцировать близкородственные штаммы вируса Зика и Денге [J.S. Gootenberg, О.О. Abudayyeh, J.W. Lee et al., Nucleic acid detection with CRISPR-Cas 13a/C2c2, Science 356(6336) (2017) 438-42, doi: 10.1126/science.aam9321].
SHERLOCK была усовершенствована (SHERLOCKv2) и позволила выявлять в одном анализе до 4 мишеней. Мультиплексирование было достигнуто путем объединения нескольких нуклеаз Cas13 и нуклеазы Cas12 со специфичными флуоресцентными репортерными комплексами, которые обеспечивали детекцию сигнала на разных длинах волн. Количественное обнаружение было достигнуто путем оптимизации концентраций олигонуклеотидов, используемых на этапе предварительной амплификации, чтобы входной сигнал и интенсивность сигнала тесно коррелировали в широком диапазоне концентраций образца. Повышенная чувствительность была достигнута путем добавления Csm6 для увеличения интенсивности расщепления флуоресцентного репортера. Стоит отметить, что SHERLOCKv2 является портативным анализом за счет того, что обнаружение показаний флуоресценции в предложенной технологии заменено на визуальную детекцию на тест-полосках [J.S. Gootenberg, О.О. Abudayyeh, M.J. Kellner et al., Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6, Science 360(6387) (2018) 439-44, doi: 10.1126/science.aaqO 179].
На основе SHERLOCK была разработана технология HUDSON (heating unextracted diagnostic samples to obliterate nucleases - нагревание неэкстрагированных диагностических образцов для уничтожения нуклеаз), которая устраняет необходимость в экстракции нуклеиновых кислот и позволяет обнаруживать патогены непосредственно в биологических образцах пациента (образец крови, сыворотки или плазмы крови, клеток крови, слюны, мокроты, лимфоидных тканей, тканей кроветворных органов и других биологических материалов). В HUDSON нагревание и химическое восстановление инактивируют нуклеазы, присутствующие в высоких концентрациях в биологических образцах пациента, после чего вирусные частицы лизируются, высвобождая нуклеиновые кислоты в раствор. HUDSON позволяет в течение 2 ч с высокой чувствительностью обнаружить вирус Денге в образцах цельной крови, сыворотки и слюны пациентов. Кроме того, HUDSON позволяет дифференцировать четыре серотипами вируса Денге и выявлять 6 наиболее распространенных мутаций обратной транскриптазы ВИЧ [С. Myhrvold, С.А. Freije, J.S. Gootenberg et al., Field-deployable viral diagnostics using CRISPR-Cas13, Science 360(6387) (2018) 444-8, doi: 10.1126/science.aas8836].
В связи с этим крайне актуальной является задача разработки новых эффективных методик выявления нуклеиновых кислот возбудителей инфекционных заболеваний, основанных на генетических технологиях, таких как CRISPR-Cas.
В ходе изучения уровня техники были найдены научные статьи, описывающие разработку технологий для выявления вируса гепатита В in vitro, основанных на применении белков систем направленного редактирования генома.
В 2020 году Zhou R. и соавторы описали способ выявления ДНК вируса гепатита В с помощью белка Cas12a и технологии плазмонной петлевой изотермической амплификации (LAMP) с ортогональным цветовым выводом. Чувствительность описанного метода составляла 140 копий ДНК вируса гепатита В на реакцию [R. Zhou, Υ. Li, Т. Dong, Y. Tang, F. Li, A sequence-specific plasmonic loop-mediated isothermal amplification assay with orthogonal color readouts enabled by CRISPR Cas12a, Chemical communications (Cambridge, England) 56(24) (2020) 3536-3538, https://doi.org/10.1039/d0cc00397b].
Cas12a также был использован для выявления ДНК вируса гепатита В в методе, основанном на применении петлевой изотермической амплификации (LAMP) для предварительной амплификации. Визуализация результатов анализа возможна как с помощью флуоресцентного индикатора, так и с помощью тест-полосок. Чувствительность описанной платформы составляла 25 копий ДНК вируса гепатита В на реакцию [R. Ding, J. Long, Μ. Yuan, Χ. Zheng, Υ. Shen, Υ. Jin, Η. Yang, Η. Li, S. Chen, G. Duan, CRISPR/Cas12-Based Ultra-Sensitive and Specific Point-of-Care Detection of HBV, International journal of molecular sciences 22(9) (2021) 4842, https://doi.org/10.3390/ijms220948421.
Cas12a также был использован для выявления ДНК вируса гепатита В в методе, основанном на применении петлевой изотермической амплификации (LAMP) для предварительной амплификации. Визуализация результатов анализа возможна как с помощью флуоресцентного индикатора, так и с помощью тест-полосок. Чувствительность описанной платформы составляла 25 копий ДНК вируса гепатита В на реакцию [R. Ding, J. Long, Μ. Yuan, Χ. Zheng, Υ. Shen, Υ. Jin, Η. Yang, Η. Li, S. Chen, G. Duan, CRISPR/Cas12-Based Ultra-Sensitive and Specific Point-of-Care Detection of HBV, International journal of molecular sciences 22(9) (2021) 4842, https://doi.org/10.3390/ijms22094842].
X. Chen и соавторы в 2021 году для выявления ДНК вируса гепатита В использовали многократную амплификацию с перекрестным смещением (MCDA) для быстрой предварительной амплификации, с последующим детектированием последовательностей-мишеней с помощью Cas12b. Чувствительность описанной платформы составляла 10 копий ДНК вируса гепатита В на реакцию [X. Chen, Y. Tan, S. Wang, X. Wu, R. Liu, X. Yang, Y. Wang, J. Tai, S. Li, A CRISPR-Cas 12b-Based Platform for Ultrasensitive, Rapid, and Highly Specific Detection of Hepatitis В Virus Genotypes В and С in Clinical Application, Frontiers in bioengineering and biotechnology 9 (2021) 743322. https://doi.org/10.3389/fbioe.2021.7433221.
Кроме того, в ходе изучения уровня техники были найдены патенты на изобретения, описывающие разработку и получение терапевтических направляющих РНК для удаления ДНК вируса гепатита В из клетки-хозяина, инфицированной HBV, с помощью технологии CRISPR-Cas (RU 2694396, RU 2652899, WO 2017213898, WO 2017070284, CN 110656109, EP 3365447, MX 2016007324). Из перечисленных выше материалов ни один нельзя отнести к ближайшим аналогам настоящего изобретения в виду того, что направляющие РНК, описанные в настоящей заявке, предназначены для разработки высокочувствительных и высокотехнологичных диагностических (но не терапевтических) систем нового поколения на основе CRISPR технологий для совершенствования методов диагностики инфекционных заболеваний.
Исходя из вышеизложенного, возникает техническая проблема, заключающаяся в необходимости разработки и получения направляющих РНК для выявления единичных копий (менее 3 копий ДНК на реакцию) ДНК вируса гепатита В in vitro с помощью Cas12.
Предложенная технология перспективна для разнообразных применений, включая количественное определение ДНК/РНК, быструю мультиплексную детекцию экспрессии, другие виды чувствительной детекции, например, выявление загрязнения образцов нуклеиновыми кислотами. Технология, основанная на CRISPR-Cas, является многофункциональной, устойчивой к ошибкам технологией детекции ДНК, пригодной для быстрой постановки диагнозов, включая инфекционные заболевания, генотипирование инфекционных агентов и выявление генов антибиотикоустойчивости бактериальных патогенов.
Применение предложенной технологии делает возможным создание диагностических систем нового поколения, которые будут обладать следующими свойствами:
высокая чувствительность;
возможность проведения диагностики у постели больного;
возможность проведения диагностики в полевых условиях без применения специализированного высокотехнологичного оборудования;
скорость и простота анализа;
сниженная стоимость анализа;
отсутствие необходимости оснащения диагностической лаборатории дорогостоящим оборудованием;
отсутствие необходимости проведения выделения нуклеиновых кислот возбудителя.
Изобретение относится к новым средствам - направляющим РНК, которые могут быть использованы в системе CRISPR-Cas12 для ультрачувствительного выявления, идентификации, обнаружения или детекции ДНК вируса гепатита В в биологических образцах.
Технической задачей предложенного изобретения является разработка новых средств направляющих РНК, которые могут быть использованы в системе CRISPR-Cas 12 с белками Cas12, например, LbCpf1 из Lachnospiraceae, для ультрачувствительного выявления ДНК вируса гепатита В.
При осуществлении настоящего изобретения, согласно приведенной в формуле изобретения совокупности существенных признаков, достигается неожиданный технический результат - возможность ультрачувствительного выявления ДНК вируса гепатита В до единичных (1,74) копий ДНК в одной реакции. Изобретение обеспечивает повышение эффективности выявления ДНК вируса гепатита В до 5 раз.
Технический результат достигается за счет:
разработки молекул направляющих РНК, которые могут быть использованы в системах CRISPR-Cas12 для ультра чувствительного выявления ДНК вируса гепатита В, где указанные направляющие РНК выбраны из последовательностей SEQ ID NO: 1-6, способны связываться с целевыми высоко консервативными участками ДНК вируса гепатита В, содержат РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae, с обеспечением выявления единичных копий ДНК вируса гепатита В.
применения РНК-направляемой ДНК-эндонуклеазы LbCpf1 из Lachnospiraceae, полученной согласно способу, разработанному авторами ранее (Патент РФ №2707542, дата приоритета 28.03.2019), для создания рибонуклеопротеиновых комплексов (РПК) системы CRISPR-Cas, пригодных для детекции ДНК вируса гепатита В в ультра низких концентрациях (единичные копии).
разработки набора специфических олигонуклеотидов, выбранных из SEQ ID NO: 7-18, для предварительной амплификации фрагментов генома вируса гепатита В.
оптимизации условий проведения предварительной амплификации фрагментов генома вируса гепатита В.
определения условий проведения ультрачувствительной детекции ДНК вируса гепатита В и установления последовательности стадий метода.
Предложена молекула направляющей РНК системы CRISPR-Cas, которая способна связываться с целевыми высококонсервативными участками генома вируса гепатита В. Нуклеотидная последовательность, раскрытой в настоящей заявке, направляющей РНК выбрана из последовательностей SEQ ID NO: 1-6.
Молекула направляющей РНК содержит в своем составе РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae.
Молекула направляющей РНК обеспечивает выявление единичных копий ДНК вируса гепатита В.
Направляющие РНК, согласно настоящему изобретению, соответствуют высоко консервативным фрагментам генома вируса гепатита В. Наиболее предпочтительны направляющие РНК, распознающиеся РНК-направляемыми ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae, характеризующиеся, имеющие или содержащие нуклеотидную последовательность, выбранную из:
SEQIDNO: 1;
SEQ ID NO: 2;
SEQ ID NO: 3;
SEQ ID NO: 4;
SEQ ID NO: 5;
SEQ ID NO: 6;
или идентичной любой из них по меньшей мере на 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%,
или комплементарной любой из них,
или гибридизующейся с любой из них в строгих условиях.
Согласно предложенному изобретению, получают рибонуклеопротеиновые комплексы (РПК), состоящие из по меньшей мере одной направляющей РНК и РНК-направляемой ДНК-эндонуклеазы системы CRISPR-Cas (LbCpf1 из Lachnospiraceae), пригодные для использования для выявления ДНК вируса гепатита В в ультранизких концентрациях (единичные копии).
Рибонуклеопротеиновый комплекс системы CRISPR-Cas для выявления ДНК вируса гепатита В, сформированный из LbCpf1 из Lachnospiraceae, и по меньшей мере одной направляющей РНК.
Предложенный рибонуклеопротеиновый комплекс системы CRISPR-Cas пригоден для выявления единичных копий ДНК вируса гепатита В.
Препараты РПК представляют собой растворы, содержащие направляющую РНК, выбранную из SEQ ID NO: 1-6, объединенную с белком системы CRISPR-Cas (LbCpf1 из Lachnospiraceae) или лиофильно высушенные РПК.
Полученные направляющие РНК могут быть использованы в составе набора для обнаружения ДНК вируса гепатита В, с инструкцией по применению.
Набор для обнаружения ДНК вируса гепатита В, может содержать рибонуклеопротеиновый комплекс и инструкцию по применению.
При этом по меньшей мере одна направляющая РНК в составе набора может находиться в комплексе с белком системы CRISPR-Cas (LbCpf1 из Lachnospiraceae) в одном контейнере или отдельно в разных контейнерах.
Набор может дополнительно включать компоненты для проведения предварительной амплификации высококонсервативных фрагментов генома вируса гепатита В, в том числе один или несколько специфических олигонуклеотидов, выбранных из SEQ ID NO: 7-18.
Специфические олигонуклеотиды для проведения предварительной амплификации фрагмента генома вируса гепатита В, согласно настоящему изобретению, соответствуют высоко консервативным участкам генома вируса гепатита В. Наиболее предпочтительны олигонуклеотиды, характеризующиеся, имеющие или содержащие нуклеотидную последовательность, выбранную из:
SEQ ID NO: 7;
SEQ ID NO: 8;
SEQ ID NO: 9;
SEQ ID NO: 10;
SEQ ID NO: 11;
SEQ ID NO: 12;
SEQ ID NO: 13;
SEQ ID NO: 14;
SEQ ID NO: 15;
SEQ ID NO: 16;
SEQ ID NO: 17;
SEQ ID NO: 18;
или идентичной любой из них по меньшей мере на 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%,
или комплементарной любой из них,
или гибридизующейся с любой из них в строгих условиях.
Предложенная технология позволяет определить единичные копии ДНК вируса гепатита В, в биологических образцах пациента, выбранных из жидкости и/или ткани, предположительно содержащих вирус гепатита В. Биологическим образцом может быть образец крови, сыворотки или плазмы крови, спинномозговой жидкости, клеток крови, слюны, мокроты, лимфоидных тканей, тканей кроветворных органов и других биологических материалов от пациента, которые могут быть использованы для анализа на наличие ДНК вируса гепатита В.
Краткое описание чертежей
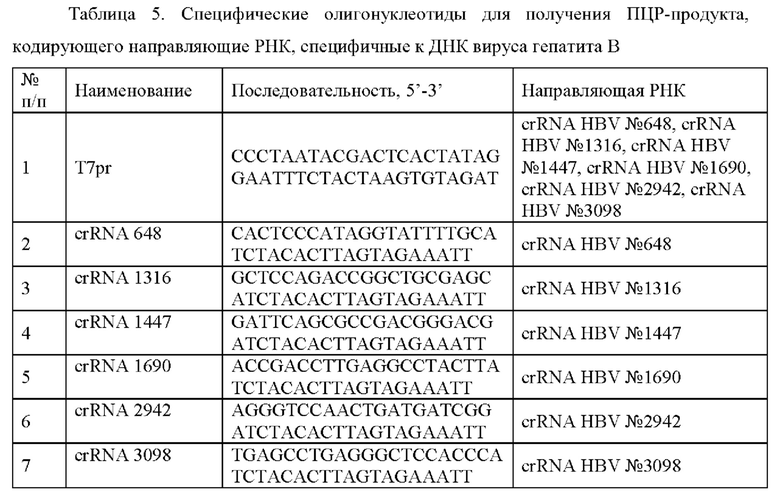
Фиг. 1. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 169 п.о.) после предварительной амплификации с использованием олигонуклеотидов For HBsAg и Rev HBsAg при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20000 фг модельной матрицы pGEM-T-HBV-HBsAg;
2 - Продукт, полученный в ходе амплификации 2000 фг модельной матрицы pGEM-T-HBV-HBsAg;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-HBsAg;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-HBsAg;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-HBsAg;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-HBsAg;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-HBsAg;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-HBsAg;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-HBsAg;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
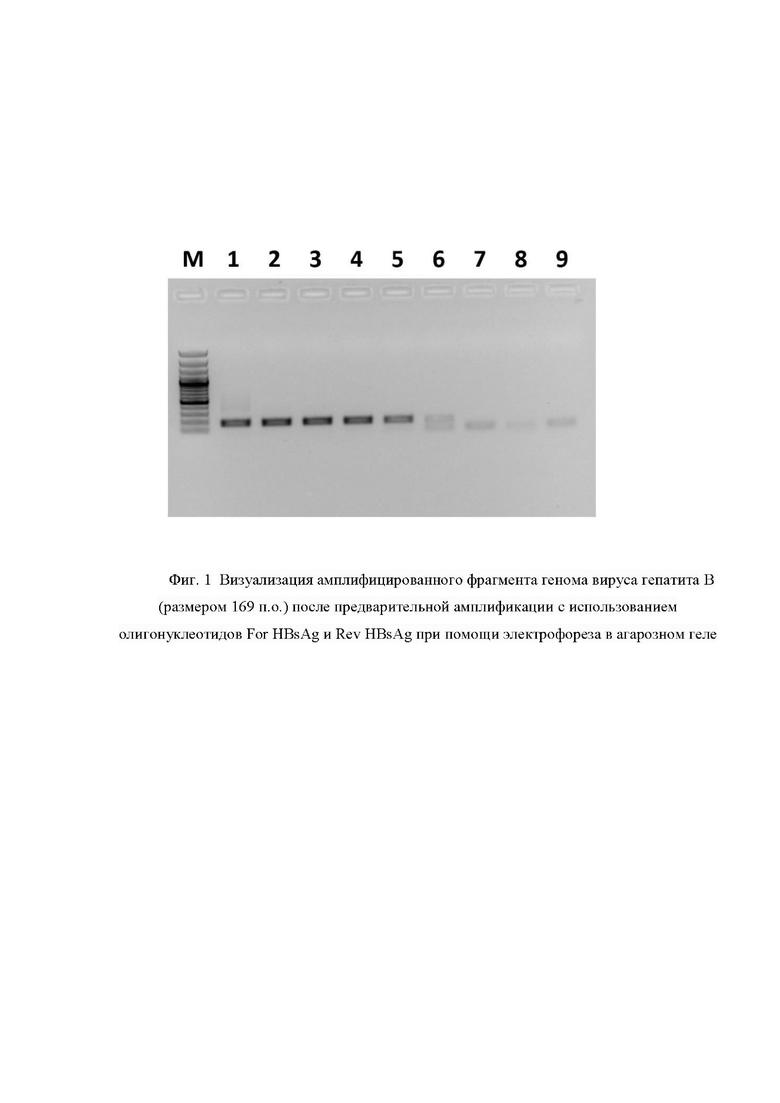
Фиг. 2. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 304 п.о.) после предварительной амплификации с использованием олигонуклеотидов For HBV1286 и Rev HBV 1523 при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20 ООО фг модельной матрицы pGEM-T-HBV-1286-1523;
2 - Продукт, полученный в ходе амплификации 2 ООО фг модельной матрицы pGEM-T-HBV-1286-1523;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-1286-1523;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-1286-1523;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-1286-1523;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-1286-1523;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-1286-1523;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-1286-1523;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-1286-1523;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
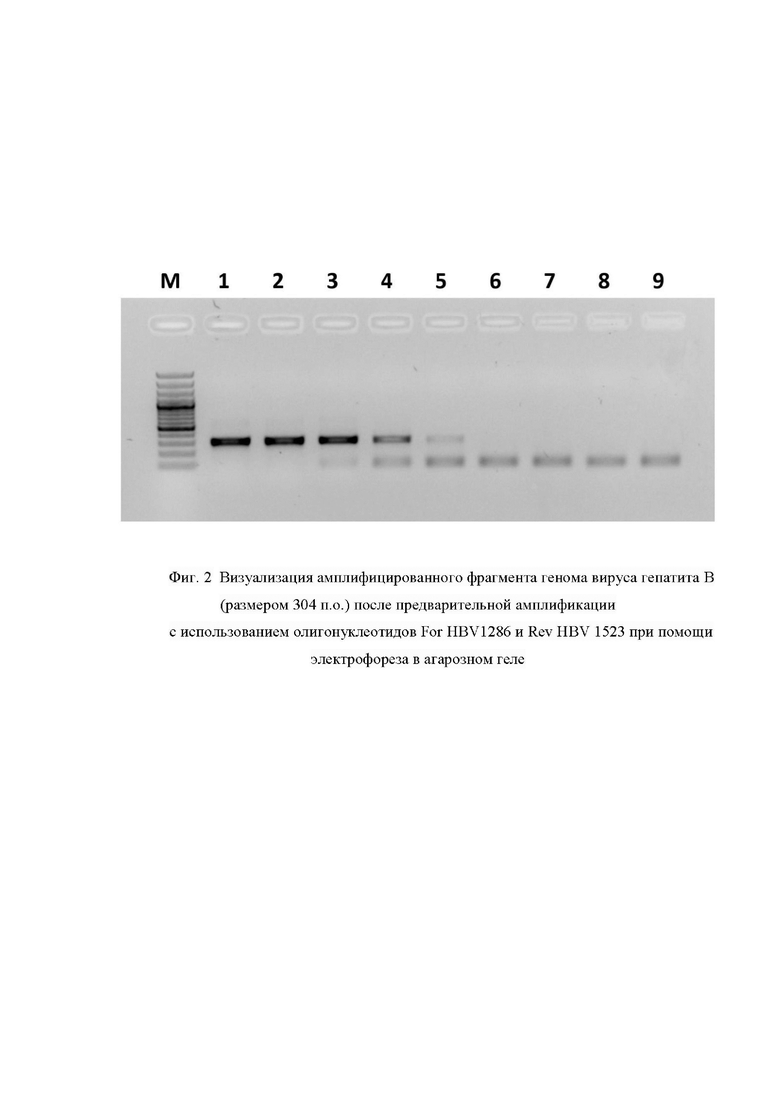
Фиг. 3. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 330 п.о.) после предварительной амплификации с использованием олигонуклеотидов For HBV 1286 и Rev HBV 1550 при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20000 фг модельной матрицы pGEM-T-HBV-1286-15 50;
2 - Продукт, полученный в ходе амплификации 2000 фг модельной матрицы pGEM-T-HBV-1286-1550;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-1286-1550;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-1286-1550;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-1286-1550;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-1286-1550;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-1286-1550;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-1286-1550;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-1286-1550;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
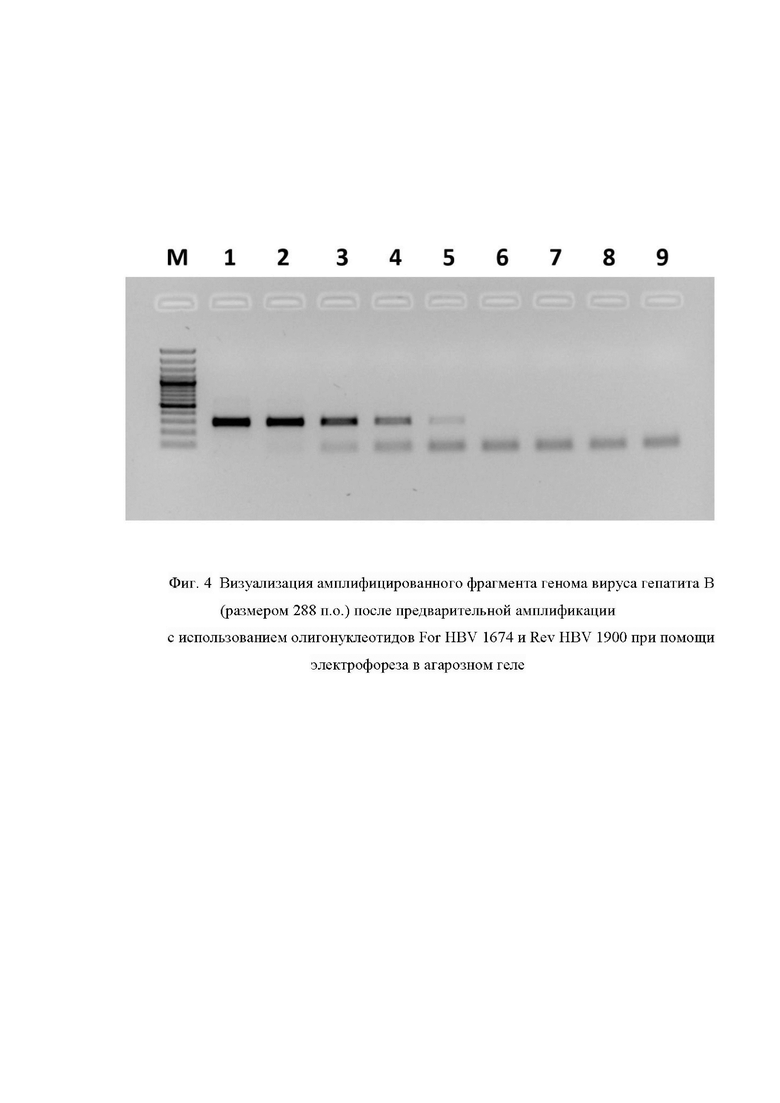
Фиг. 4. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 288 п.о.) после предварительной амплификации с использованием олигонуклеотидов For HBV 1674 и Rev HBV 1900 при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20000 фг модельной матрицы pGEM-T-HBV-1674-1900;
2 - Продукт, полученный в ходе амплификации 2000 фг модельной матрицы pGEM-T-HBV-1674-1900;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-1674-1900;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-1674-1900;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-1674-1900;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-1674-1900;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-1674-1900;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-1674-1900;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-1674-1900;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
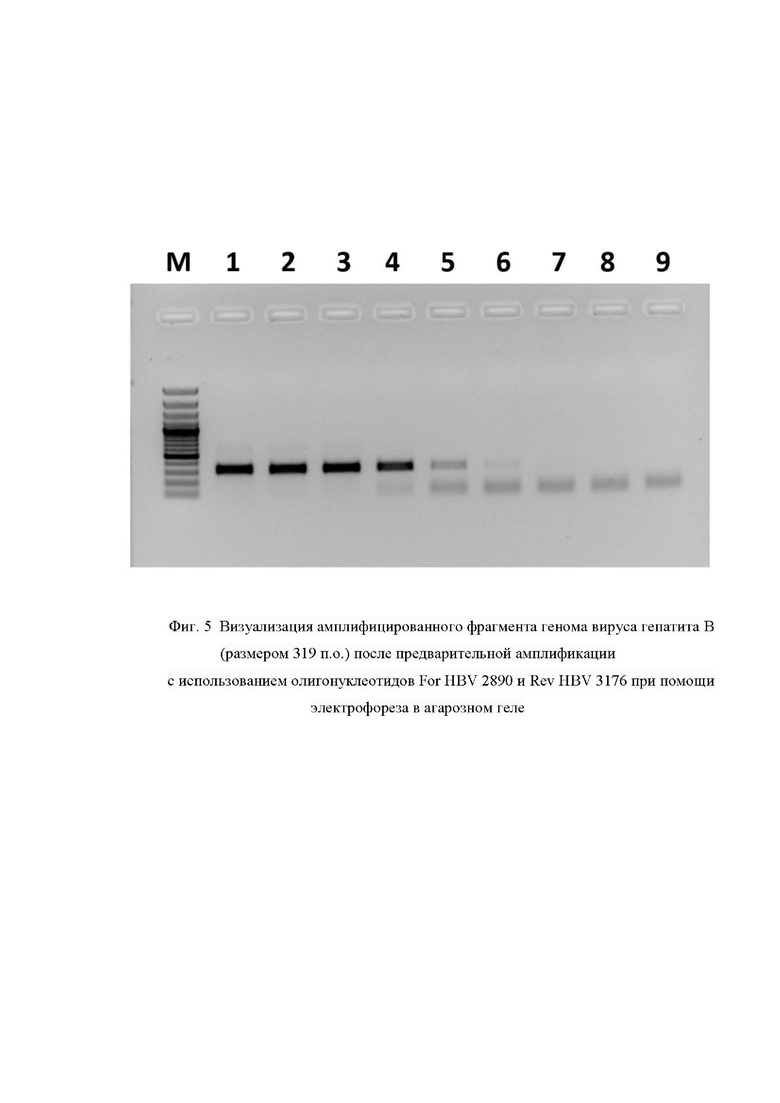
Фиг. 5. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 319 п.о.) после предварительной амплификации с использованием олигонуклеотидов For HBV 2890 и Rev HBV 3176 при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20000 фг модельной матрицы pGEM-T-HBV-2890-3176;
2 - Продукт, полученный в ходе амплификации 2000 фг модельной матрицы pGEM-T-HBV-2890-3176;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-2890-3176;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-2890-3176;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-2890-3176;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-2890-3176;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-2890-3176;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-2890-3176;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-2890-3176;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
Фиг. 6. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных фрагментов модельной матрицы pGEM-T-HBV-HBsAg, обработанных рибонуклеопортеиновыми комплексами, содержащими белок LbCpf1 и направляющие РНК crRNA HBV №648.
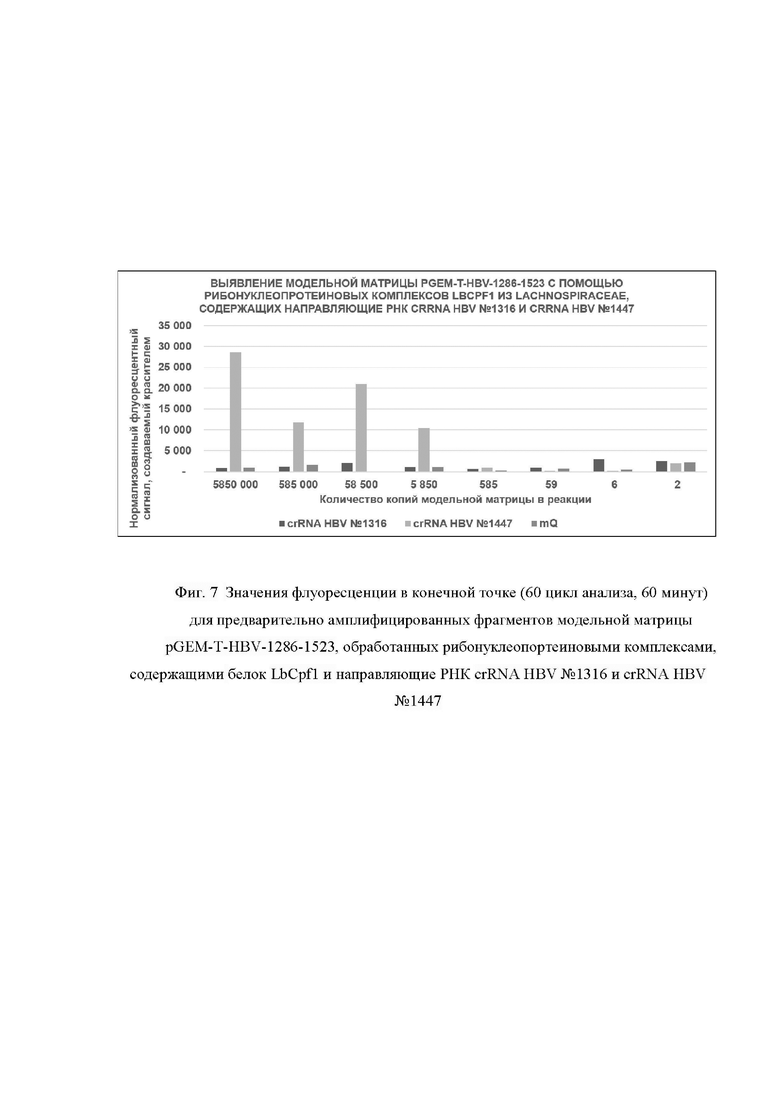
Фиг. 7. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных фрагментов модельной матрицы pGEM-T-HBV-1286-1523, обработанных рибонуклеопортеиновыми комплексами, содержащими белок LbCpf1 и направляющие РНК crRNA HBV №1316 и crRNA HBV №1447.
Фиг. 8. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных фрагментов модельной матрицы pGEM-T-HBV-1286-1550, обработанных рибонуклеопортеиновыми комплексами, содержащими белок LbCpf1 и направляющие РНК crRNA HBV №1316 и crRNA HBV №1447.
Фиг. 9. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных фрагментов модельной матрицы pGEM-T-HBV-1674-1900, обработанных рибонуклеопортеиновыми комплексами, содержащими белок LbCpf1 и направляющие РНК crRNA HBV №1690.
Фиг. 10. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных фрагментов модельной матрицы pGEM-T-HBV-2890-3176, обработанных рибонуклеопортеиновыми комплексами, содержащими белок LbCpf1 и направляющие РНК crRNA HBV №2942 и crRNA HBV №3098.
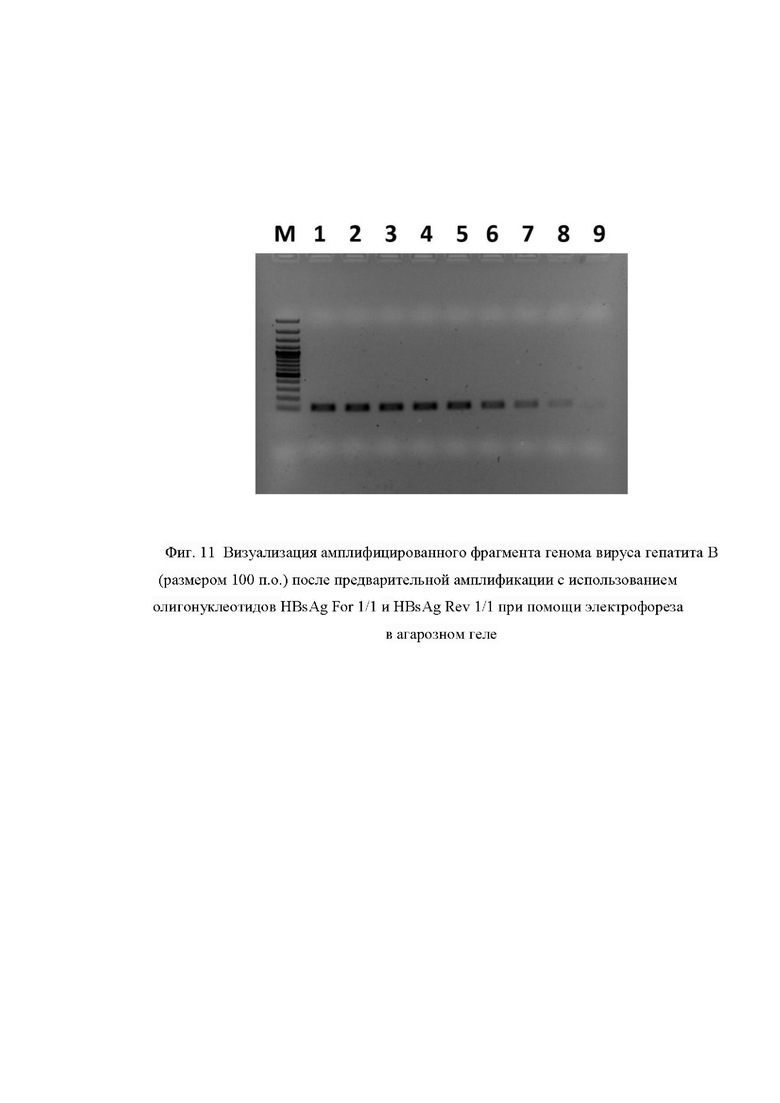
Фиг. 11. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 100 п.о.) после предварительной амплификации с использованием олигонуклеотидов HBsAg For 1/1 и HBsAg Rev 1/1 при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20000 фг модельной матрицы pGEM-T-HBV-HBsAg;
2 - Продукт, полученный в ходе амплификации 2000 фг модельной матрицы pGEM-T-HBV-HBsAg;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-HBsAg;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-HBsAg;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-HBsAg;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-HBsAg;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-HBsAg;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-HBsAg;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-HBsAg;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
Фиг. 12. Визуализация амплифицированного фрагмента генома вируса гепатита В (размером 126 п.о.) после предварительной амплификации с использованием олигонуклеотидов HBsAg For 1/2 и HBsAg Rev 1/1 при помощи электрофореза в агарозном геле, где цифрами 1-9 обозначены:
1 - Продукт, полученный в ходе амплификации 20000 фг модельной матрицы pGEM-T-HBV-HBsAg;
2 - Продукт, полученный в ходе амплификации 2000 фг модельной матрицы pGEM-T-HBV-HBsAg;
3 - Продукт, полученный в ходе амплификации 200 фг модельной матрицы pGEM-T-HBV-HBsAg;
4 - Продукт, полученный в ходе амплификации 20 фг модельной матрицы pGEM-T-HBV-HBsAg;
5 - Продукт, полученный в ходе амплификации 2 фг модельной матрицы pGEM-T-HBV-HBsAg;
6 - Продукт, полученный в ходе амплификации 0,2 фг модельной матрицы pGEM-T-HBV-HBsAg;
7 - Продукт, полученный в ходе амплификации 0,02 фг модельной матрицы pGEM-T-HBV-HBsAg;
8 - Продукт, полученный в ходе амплификации 0,002 фг модельной матрицы pGEM-T-HBV-HBsAg;
9 - отрицательный контроль, не содержащий модельной матрицы pGEM-T-HBV-HBsAg;
Μ - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bp Plus, Thermo Fisher Scientific, США).
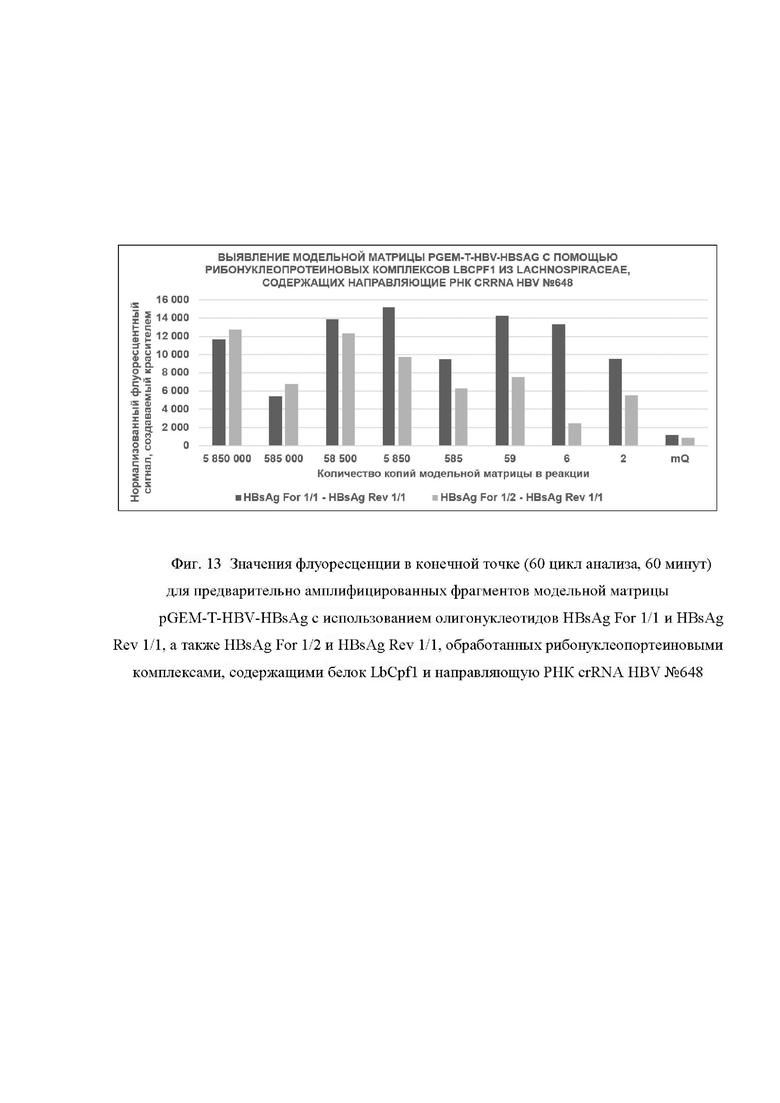
Фиг. 13. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных фрагментов модельной матрицы pGEM-T-HBV-HBsAg с использованием олигонуклеотидов HBsAg For 1/1 и HBsAg Rev 1/1, а также HBsAg For 1/2 и HBsAg Rev 1/1, обработанных рибонуклеопортеиновыми комплексами, содержащими белок LbCpf1 и направляющую РНК crRNA HBV №648.
Примеры осуществления изобретения
ПРИМЕР 1: АНАЛИЗ НУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ПОЛНЫХ ГЕНОМОВ ВИРУСА ГЕПАТИТА В И ДИЗАЙН СПЕЦИФИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ПРЕДВАРИТЕЛЬНОЙ АМПЛИФИКАЦИИ ФРАГМЕНТОВ ГЕНОМОВ
Для поиска высококонсервативных участков 50 нуклеотидных последовательностей полных геномов вируса гепатита В, принадлежащих генотипу D, который является наиболее распространенным генотипом в Российской Федерации [Ю.В. Останкова, А.В. Семенов, Е.Б. Зуева, А.А. Тотолян, Первые случаи выявления вируса гепатита В субгенотипа D4 у больных хроническим, острым и скрытым вирусным гепатитом В в Российской Федерации, Молекулярная генетика, микробиология и вирусология 38 (4) (2020) 180-187; R. Ozaras, I. Inane Balkan, Μ. Yemisen, F. Tabak, Epidemiology of HBV subgenotypes D, Clin Res Hepatol Gastroenterol 39 (1) (2015) 28-37], были выровнены в программе MEGA7.0.26 [S. Kumar, G. Stecher, K. Tamura, MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets, Mol. Biol. Evol. 33 (2016) 1870-1874]. Полногеномные последовательности были выровнены с использованием алгоритма ClustalW «Align DNA». Для попарных и множественных выравниваний были установлены следующие параметры: «Gap Opening Penalty» - 15; «Gap Extension Penalty» - 6,66. Для сравнения последовательностей нуклеиновых кислот по умолчанию использовалась оценочная матрица ШВ (параметр «Transition Weight» равен 0,5). Отрицательная матрица не использовалась для выравнивания, а параметр «Delay Di-vergent Cutoff» был установлен на уровне 30%.
В результате анализа полученных выравниваний для разработки наборов специфических олигонуклеотидов были выбраны области генома вируса гепатита В, кодирующие поверхностный антиген гепатита В, ДНК-полимеразу, Х-белок и прекоровый/коровый белок вируса гепатита В.
Для предварительной амплификации целевых высококонсервативных последовательностей генома вируса гепатита В были подобраны 9 специфических олигонуклеотидов (Таблица 1), с помощью которых нарабатываются 5 фрагментов (Таблица 2).
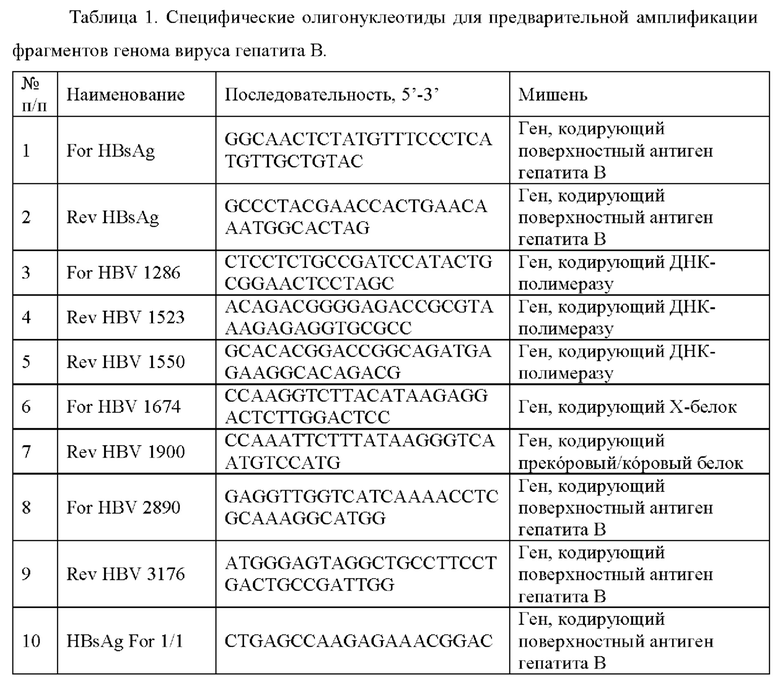

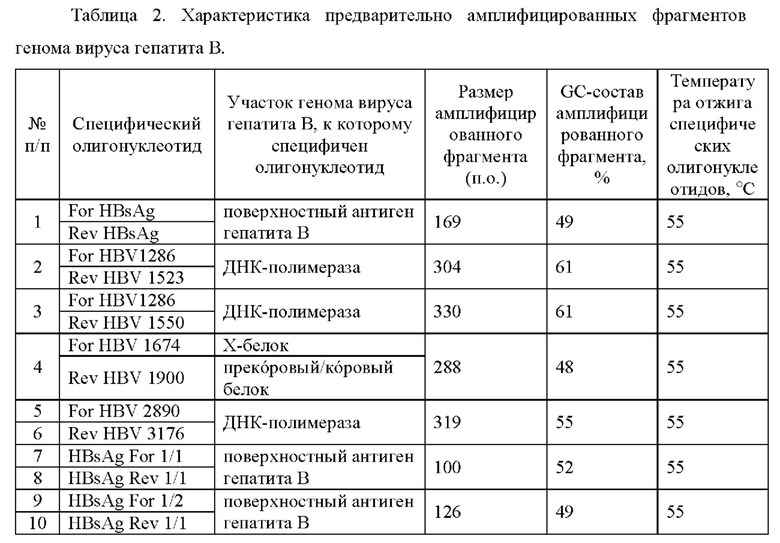
ПРИМЕР 2: ПОДБОР ПОСЛЕДОВАТЕЛЬНОСТЕЙ-МИШЕНЕЙ В ГЕНОМЕ ВИРУСА ГЕПАТИТА В ДЛЯ СОЗДАНИЯ НАПРАВЛЯЮЩИХ РНК
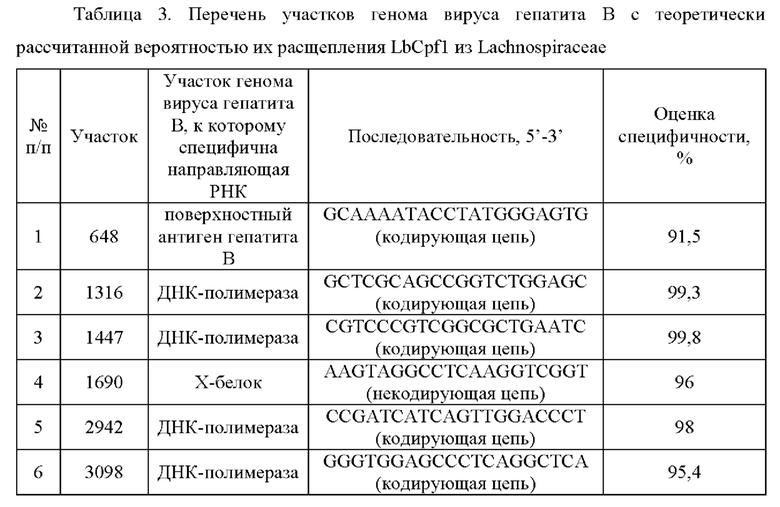
Для подбора последовательностей-мишеней в геноме вируса гепатита В для создания направляющих РНК были использованы современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Benchling (https://www.benchling.com/molecular-biology/). Был составлен перечень участков в геноме вируса гепатита В с теоретически рассчитанной вероятностью их расщепления в высоко консервативных участках (Таблица 3). Направляющие РНК, специфически узнающие фрагменты в геноме вируса гепатита В, представлены уникальными последовательностями SEQ ID NO: 1-6.
ПРИМЕР 3: КОНСТРУИРОВАНИЕ МОДЕЛЬНЫХ МАТРИЦ (КОНТРОЛЬНЫХ ОБРАЗЦОВ)
В качестве модельной матрицы фрагмента генома вируса гепатита В были использованы плазмидные ДНК pGEM-T-HBV-HBsAg, pGEM-T-HBV-1286-1523, pGEM-T-HBV-1286-1550, pGEM-T-HBV-1674-1900 и pGEM-T-HBV-2890-3176, содержащие в своем составе фрагменты генов, кодирующих поверхностный антиген гепатита В (фрагмент 169 п.о., полученный с использованием олигонуклеотидов For HBsAg и Rev HBsAg), ДНК-полимеразу (фрагмент 304 п.о., полученный с использованием олигонуклеотидов For HBV 1286 и Rev HBV 1523; фрагмент 330 п.о., полученный с использованием олигонуклеотидов For HBV 1286 и Rev HBV 1550; а также фрагмент 319 п.о., полученный с использованием олигонуклеотидов For HBV 2890 и Rev HBV 3176), а также Х-белок и прекоровый/коровый белок (фрагмент 288 п.о., полученный с использованием олигонуклеотидов For HBV 1674 и Rev HBV 1900).
Для конструирования модельных матриц была проведена амплификация соответствующих фрагментов с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов (ГенТерра, Россия), перечисленных в Таблице 1. Полученные фрагменты были визуализированы с помощью электрофореза в 1% агарозном геле, после чего были очищены с использованием коммерчески доступного набора ISOLATE II PCR and Gel Kit (BioLine, США) согласно инструкции производителя. Очищенные ПЦР-продукты были лигированы в вектор pGEM®-T (Promega6 США). Полученные лигазные смеси были трансформированы в химически компетентные клетки XLIO-Gold (TetrD(mcrA)183 D(mcrCB-hsdSMR-mrr)173 endA1 supE44 thi-1 recA1 gyrA96 relA1 lac Hte [F' proAB lacIqZDM15 Tn10 (Tetr) Amy Camr]). Клоны, содержащие вставку длиной требуемого размера, идентифицировали при помощи ПЦР с праймерами M13For и M13Rev (ГенТерра, Россия). Плазмидную ДНК выделяли и верифицировали корректность встроенной последовательности методом капиллярного секвенирования. Полученные плазмидные ДНК pGEM-T-HBV-HBsAg, pGEM-T-HBV-1286-1523, pGEM-T-HBV-1286-1550, pGEM-T-HBV-1674-1900 и pGEM-T-HBV-2890-3176 использовали в качестве модельных матриц для выявления ДНК вируса гепатита В.
ПРИМЕР 4: ПОДГОТОВКА МАТЕРИАЛА ДЛЯ ОБНАРУЖЕНИЯ ДНК ВИРУСА ГЕПАТИТА В МЕТОДОМ ПРЕДВАРИТЕЛЬНОЙ АМПЛИФИКАЦИИ
Подготовку материала для обнаружения ДНК вируса гепатита В проводили методом предварительной амплификации. В качестве модельных матриц фрагментов генома вируса гепатита В биологического образца использовали плазмидные ДНК, описанные в Примере 3.
Предварительную амплификацию фрагмента генома вируса гепатита В проводили методом ПЦР с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов (ГенТерра, Россия), перечисленных в Таблице 1. Размер амплифицированных фрагментов перечислен в таблице 2. Температурный профиль амплификации приведен ниже:
1. денатурация: 95°С в течение 3 минут;
2. 35 циклов амплификации: 95°С - 15 сек, 55°С - 45 сек, 72°С - 30 сек;
3. финальная элонгация: 72°С в течение 5 минут.
В ходе подготовки материала для обнаружения ДНК вируса гепатита В методом предварительной амплификации проводили титрование модельных матриц путем приготовления серийных разведений (Таблица 4).
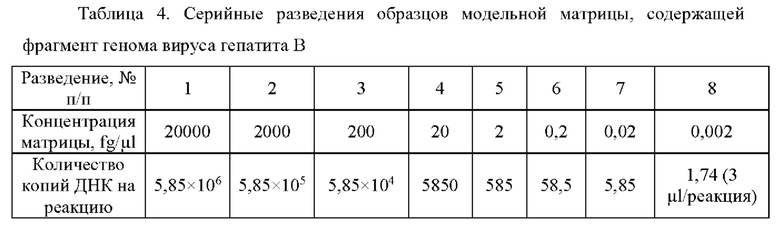
Для оценки эффективности предварительной амплификации полученные продукты, соответствующие фрагментам генома вируса гепатита В, визуализировали при помощи электрофореза в агарозном геле (фиг. 1-5).
Подготовленный описанным способом материал без предварительной очистки использовали для экспериментов по выявлению ДНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов LbCpf1 из Lachnospiraceae, содержащих направляющие РНК с уникальными последовательностями SEQ ID NO: 1-6.
ПРИМЕР 5: ПОЛУЧЕНИЕ НАПРАВЛЯЮЩИХ РНК И СОЗДАНИЕ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ ДЛЯ ОБНАРУЖЕНИЯ ДНК ВИРУСА ГЕПАТИТА В
Для получения направляющих РНК для обнаружения ДНК вируса гепатита В был разработан набор специфических олигонуклеотидов, приведенных в Таблице 5. Получение направляющих РНК проводят в несколько этапов:
1. получение ПЦР-продукта с использованием набора специфических олигонуклеотидов (Таблица 5), кодирующего направляющую РНК, способную связываться с целевым фрагментом генома вируса гепатита В, содержащую РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae;
2. очистка ПЦР-продукта, кодирующего направляющую РНК, специфичную к фрагменту генома вируса гепатита В;
3. синтез направляющей РНК, специфичной к фрагменту генома вируса гепатита В;
4. очистка направляющей РНК, специфичной к фрагменту генома вируса гепатита В.
ПЦР-продукт, кодирующий направляющую РНК crRNA HBV №648, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и crRNA 648 (ГенТерра, Россия). Размер амплифицированного фрагмента, кодирующего crRNA HBV №648, составляет 62 пары нуклеотидов.
ПЦР-продукт, кодирующий направляющую РНК crRNA HBV №1316, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и crRNA 1316 (ГенТерра, Россия). Размер амплифицированного фрагмента, кодирующего crRNA HBV №1316, составляет 62 пары нуклеотидов.
ПЦР-продукт, кодирующий направляющую РНК crRNA HBV №1447, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и crRNA 1447 (ГенТерра, Россия). Размер амплифицированного фрагмента, кодирующего crRNA HBV №1447, составляет 62 пары нуклеотидов.
ПЦР-продукт, кодирующий направляющую РНК crRNA HBV №1690, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и crRNA 1690 (ГенТерра, Россия). Размер амплифицированного фрагмента, кодирующего crRNA HBV №1690, составляет 62 пары нуклеотидов.
ПЦР-продукт, кодирующий направляющую РНК crRNA HBV №2942, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и crRNA 2942 (ГенТерра, Россия). Размер амплифицированного фрагмента, кодирующего crRNA HBV №2942, составляет 62 пары нуклеотидов.
ПЦР-продукт, кодирующий направляющую РНК crRNA HBV №3098, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и crRNA 3098 (ГенТерра, Россия). Размер амплифицированного фрагмента, кодирующего crRNA HBV №3098, составляет 62 пары нуклеотидов.
Температурный профиль амплификации для получения ПЦР-продуктов, кодирующих направляющие РНК, содержащие РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae:
1. денатурация: 95°С в течение 3 минут;
2. 35 циклов амплификации: 95°С- 15 сек, 55°С - 45 сек, 72°С - 30 сек;
3. финальная элонгация: 72°С в течение 5 минут.
ПЦР-продукты, кодирующие направляющие РНК, специфичные к фрагменту генома вируса гепатита В, визуализировали при помощи электрофореза в агарозном геле.
Очистку ПЦР-продуктов, кодирующих направляющие РНК, специфичные к фрагменту генома вируса гепатита В, проводили с использованием коммерчески доступного набора ISOLATE II PCR and Gel Kit (BioLine, США) согласно инструкции производителя. Очищенные ПЦР-продукты, кодирующие направляющие РНК, специфичные к фрагменту генома вируса гепатита В, использовали в качестве матрицы для синтеза направляющих РНК.
Синтез направляющих РНК, специфичных к фрагменту генома вируса гепатита В, осуществляли методом in vitro транскрипции с использованием коммерчески доступных наборов реагентов (HiScribe™ Т7 High Yield RNA Synthesis Kit, NEB, США) согласно инструкции производителя. Продукты реакции in vitro транскрипции переосаждали из реакционной смеси добавлением хлорида натрия до конечной концентрации 400 тМ и равного объема изопропилового спирта. Такие модификации протокола производителя, внесенные авторами, позволяют увеличить выход продукта реакции и получить желаемую концентрацию финального препарата направляющей РНК.
Создание готовых рибонуклеопротеиновых комплексов, содержащих белок семейства CRISPR-Cas LbCpf1 из Lachnospiraceae, и соответствующую направляющую РНК авторы проводили по стандартному протоколу с некоторыми модификациями [С. Anders, Μ. Jinek, In vitro enzymology of Cas9, Methods Enzymol. 546 (2014) 1-20, https://doi.org/10.1016/B978-0-12-801185-0.00001-5].
Непосредственно перед объединением с Cas-белком препарат направляющей РНК (в количестве 250 нг) прогревали при 90°С в течение 5 минут и позволяли медленно остыть до комнатной температуры (инкубация при комнатной температуре не менее 10 минут). Такое прогревание необходимо для формирования корректной конформации шпильки, содержащейся в направляющей РНК. Многие производители коммерческих препаратов Cas-белков пропускают данный этап при подготовке рибонуклеопротеинового комплекса.
Для формирования готового рибонуклеопротеинового комплекса 250 нг Cas-белка LbCpf1 из Lachnospiraceae и подготовленную соответствующую направляющую РНК смешивали и инкубировали 15 минут при комнатной температуре. Полученный таким способом рибонуклеопротеиновый комплекс готов для выявления фрагментов генома вируса гепатита В.
ПРИМЕР 6: ОБНАРУЖЕНИЕ ДНК ВИРУСА ГЕПАТИТА В С ПОМОЩЬЮ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ CRISPR-CAS НА ПРИМЕРЕ МОДЕЛЬНЫХ МАТРИЦ
Предварительно амплифицированный материал, полученный способом, описанным в Примере 4, использовали в качестве матрицы для обнаружения РНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, полученных способом, описанным в Примере 5.
Для обнаружения ДНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, содержащих LbCpf1 из Lachnospiraceae, готовили реакционную смесь, содержащую следующие компоненты:
10 × буфер (100 mM TrisHCl рН 8,0, 1 Μ NaCl);
50 mM MgCl2 (конечная концентрация в реакционной смеси 10 mM);
250 нг рибонуклеопротеинового комплекса (LbCpf1 из Lachnospiraceae и направляющие РНК crRNA HBV №648 или crRNA HBV №1316 или crRNA HBV №1447 или crRNA HBV №1690 или crRNA HBV №2942 или crRNA HBV №3098);
10 pmol флуоресцентный зонд (FAM-TTA-TT-BHQ1);
Мишень (предварительно амплифицированный фрагмент генома вируса гепатита В);
вода mQ.
Реакционные смеси, содержащие все необходимые компоненты, помещали в амплификатор QuantStudio 5 (Thermo Fisher Scientific, США) и задавали следующие параметры реакции:
60 циклов:
37°С - 35 сек,
37°С - 25 сек, съемка флуоресценции.
В первую очередь были проведены эксперименты по обнаружению ДНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, сформированных на основе LbCpf1 из Lachnospiraceae, с использованием в качестве мишени модельных матриц pGEM-T-HBV-HBsAg, pGEM-T-HBV-1286-1523, pGEM-T-HBV-1286-1550, pGEM-T-HBV-1674-1900 и pGEM-T-HBV-2890-3176. На Фиг. 6-10 приведены типичные результаты анализа на примерах значений флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных мишеней, обработанных рибонуклеопротеиновыми комплексами CRISPR-Cas, содержащими соответствующие направляющие РНК. Наилучший результат - выявление 6 копий модельной матрицы в реакционной смеси был показан для направляющих РНК crRNA HBV №648 (фиг. 6), crRNA HBV №1690 (фиг. 9) и crRNA HBV №3098 (фиг. 10). Направляющая РНК crRNA HBV №1447 была способна обнаружить от 585 до 5850 копий модельных матриц pGEM-T-HBV-1286-1523 или pGEM-T-HBV-1286-1550 в реакционной смеси (фиг. 7, 8). Было показано, что рибонуклеопротеиновые комплексы CRISPR-Cas, содержащие в своем составе направляющие РНК crRNA HBV №1316 и crRNA HBV №2942 не обладают способностью выявлять ДНК вируса гепатита В (фиг. 7, 8, 10).
ПРИМЕР 7: ОБНАРУЖЕНИЕ ЕДИНИЧНЫХ КОПИЙ ДНК ВИРУСА ГЕПАТИТА В С ПОМОЩЬЮ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ CRISPR-CAS
Для обнаружения единичных копий ДНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов CRISPR-Cas было принято решение оптимизировать процесс предварительной амплификации. Для этого были разработаны дополнительные олигонуклеотиды (Таблица 1) для амплификации фрагмента гена, кодирующего поверхностный антиген вируса гепатита В, входящего в состав модельной матрицы pGEM-T-HBV-HBsAg.
Для предварительной амплификации с новыми олигонуклеотидами модельную матрицу pGEM-T-HBV-HBsAg титровали путем приготовления серийных разведений (Таблица 4) как описано в Примере 4.
ПЦР-продукты, кодирующие фрагменты гена, кодирующего поверхностный антиген вируса гепатита В, входящего в состав модельной матрицы pGEM-T-HBV-HBsAg, получали в реакции амплификации с использованием ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) как описано в Примере 4.
Температурный профиль амплификации для получения ПЦР-продуктов:
1. денатурация: 95°С в течение 3 минут;
2. 30, 35 или 40 циклов амплификации: 95°С - 15 сек, 55°С - 45 сек, 72°С - 30 сек;
3. финальная элонгация: 72°С в течение 5 минут.
Для оценки эффективности предварительной амплификации полученные продукты, соответствующие фрагментам генома вируса гепатита В, визуализировали при помощи электрофореза в агарозном геле (фиг. 11, 12).
Полученный таким способом материал использовали в качестве матрицы для обнаружения единичных копий ДНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов CRISPR-Cas, полученных способом, описанным в Примере 5.
Обнаружение единичных копий провирусной ДНК вируса гепатита В с помощью рибонуклеопротеиновых комплексов CRISPR-Cas проводили способом, описанным в Примере 6.
В ходе проведенного анализа было показано, что рибонуклеопротеиновые комплексы CRISPR-Cas, сформированные на основе LbCpf1 из Lachnospiraceae и направляющей РНК crRNA HBV №648 обладают способностью выявлять единичные копии ДНК вируса гепатита В (2 копии/реакция). Применение оптимизированных олигонуклеотидов HBsAg For 1/1, HBsAg For 1/2 и HBsAg Rev 1/1 позволило обнаруживать до 2 копий ДНК, внесенных в реакцию предварительной амплификации (фиг. 13). При этом в среднем уже на 20 цикле (20 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) вдвое; к 31 циклу анализа - более чем в 5 раз, а к 43 циклу - более чем в 10 раз.
Таким образом, разработанные направляющие РНК позволяют ультрачувствительно выявлять единичные копии ДНК вируса гепатита В в образце после предварительной амплификации в составе рибонуклеопротеиновых комплексов CRISPR-Cas.
--->
Перечень последовательностей
<110> ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
<120>
<160> NUMBER OF SEQ ID NO: NOS: 18
<210> SEQ ID NO: NO 1
<211> 40
<212> RNA
<213> artificial
<400> SEQUENCE 1 (crRNA HBV №648):
aau uuc uac uaa gug uag aug caa aau acc uau ggg agu g 40
<210> SEQ ID NO: NO 2
<211> 40
<212> RNA
<213> artificial
<400> SEQUENCE 2 (crRNA HBV №1316):
aau uuc uac uaa gug uag aug cuc gca gcc ggu cug gag c 40
<210> SEQ ID NO: NO 3
<211> 40
<212> RNA
<213> artificial
<400> SEQUENCE 3 (crRNA HBV №1447):
aau uuc uac uaa gug uag auc guc ccg ucg gcg cug aau c 40
<210> SEQ ID NO: NO 4
<211> 40
<212> RNA
<213> artificial
<400> SEQUENCE 4 (crRNA HBV №1690):
aau uuc uac uaa gug uag aua agu agg ccu caa ggu cgg u 40
<210> SEQ ID NO: NO 5
<211> 40
<212> RNA
<213> artificial
<400> SEQUENCE 5 (crRNA HBV №2942):
aau uuc uac uaa gug uag auc cga uca uca guu gga ccc u 40
<210> SEQ ID NO: NO 6
<211> 40
<212> RNA
<213> artificial
<400> SEQUENCE 6 (crRNA HBV №3098):
aau uuc uac uaa gug uag aug ggu gga gcc cuc agg cuc a 40
<210> SEQ ID NO: NO 7
<211> 32
<212> DNA
<213> artificial
<400> SEQUENCE 7 (For HBsAg):
ggc aac tct atg ttt ccc tca tgt tgc tgt ac 32
<210> SEQ ID NO: NO 8
<211> 31
<212> DNA
<213> artificial
<400> SEQUENCE 8 (Rev HBsAg):
gcc cta cga acc act gaa caa atg gca cta g 31
<210> SEQ ID NO: NO 9
<211> 34
<212> DNA
<213> artificial
<400> SEQUENCE 9 (For HBV 1286):
ctc ctc tgc cga tcc ata ctg cgg aac tcc tag c 34
<210> SEQ ID NO: NO 10
<211> 34
<212> DNA
<213> artificial
<400> SEQUENCE 10 (Rev HBV 1523):
aca gac ggg gag acc gcg taa aga gag gtg cgc c 34
<210> SEQ ID NO: NO 11
<211> 33
<212> DNA
<213> artificial
<400> SEQUENCE 11 (Rev HBV 1550):
gca cac gga ccg gca gat gag aag gca cag acg 33
<210> SEQ ID NO: NO 12
<211> 33
<212> DNA
<213> artificial
<400> SEQUENCE 12 (For HBV 1674):
cca agg tct tac ata aga gga ctc ttg gac tcc 33
<210> SEQ ID NO: NO 13
<211> 30
<212> DNA
<213> artificial
<400> SEQUENCE 13 (Rev HBV 1900):
cca aat tct tta taa ggg tca atg tcc atg 30
<210> SEQ ID NO: NO 14
<211> 33
<212> DNA
<213> artificial
<400> SEQUENCE 14 (For HBV 2890):
gag gtt ggt cat caa aac ctc gca aag gca tgg 33
<210> SEQ ID NO: NO 15
<211> 34
<212> DNA
<213> artificial
<400> SEQUENCE 15 (Rev HBV 3176):
atg gga gta ggc tgc ctt cct gac tgc cga ttg g 34
<210> SEQ ID NO: NO 16
<211> 20
<212> DNA
<213> artificial
<400> SEQUENCE 16 (HBsAg For 1/1):
ctg agc caa gag aaa cgg ac 20
<210> SEQ ID NO: NO 17
<211> 19
<212> DNA
<213> artificial
<400> SEQUENCE 17 (HBsAg For 1/2):
acc act gaa caa atg gca c 19
<210> SEQ ID NO: NO 18
<211> 22
<212> DNA
<213> artificial
<400> SEQUENCE 18 (HBsAg Rev 1/1):
aac cta cgg atg gaa att gca c 22
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ обнаружения ДНК вируса гепатита B в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2021 |
|
RU2782951C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas12 и препарат для выявления ДНК вируса гепатита B в ультранизких концентрациях | 2021 |
|
RU2792891C1 |
| Система CRISPR-Cas12 для выявления РНК вируса гепатита С генотипов 1b и 3a в ультранизких концентрациях | 2022 |
|
RU2800421C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления РНК вируса гепатита С генотипов 1b и 3a в ультранизких концентрациях | 2022 |
|
RU2799430C1 |
| Способ обнаружения РНК вируса гепатита С генотипов 1b и 3a в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2022 |
|
RU2800420C1 |
| Способ обнаружения РНК вируса SARS-CoV-2 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2021 |
|
RU2764021C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR/Cas12 и препарат для выявления РНК вируса SARS-CoV-2 в ультранизких концентрациях | 2021 |
|
RU2764015C1 |
| Система CRISPR-Cas12 для выявления гена exoU, кодирующего экзотоксин системы секреции третьего типа, Pseudomonas aeruginosa, в ультранизких концентрациях | 2021 |
|
RU2791879C1 |
| Способ получения препарата рибонуклеопротеинового комплекса CRISPR-Cas и препарат для выявления гена exoU, кодирующего экзотоксин системы секреции третьего типа, Pseudomonas aeruginosa | 2021 |
|
RU2782739C1 |
| Способ обнаружения гена exoU, кодирующего экзотоксин системы секреции третьего типа, Pseudomonas aeruginosa в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2021 |
|
RU2791880C1 |




Изобретение относится к биотехнологии, а именно к молекуле направляющей РНК системы CRISPR-Cas, рибонуклеопротеиновому комплексу системы CRISPR-Cas, содержащему вышеуказанную молекулу, а также к наборам, содержащим рибонуклеопротеиновый комплекс системы CRISPR-Cas и специфические олигонуклеотиды для предварительной амплификации высококонсервативного участка генома вируса гепатита В. Изобретение позволяет эффективно выявлять ДНК вируса гепатита В после проведения специфической амплификации фрагмента генома этого вируса. Изобретение обеспечивает выявление единичных копий ДНК вируса гепатита В. 4 н. и 3 з.п. ф-лы, 13 ил., 5 табл., 7 пр.
1. Молекула направляющей РНК системы CRISPR-Cas, которая способна связываться с целевыми высококонсервативными участками генома вируса гепатита B, где нуклеотидная последовательность направляющей РНК выбрана из последовательностей SEQ ID NO: 1-6.
2. Молекула направляющей РНК по п.1, отличающаяся тем, что содержит в своем составе РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой LbCpf1 из Lachnospiraceae.
3. Молекула направляющей РНК по пп. 1, 2, отличающаяся тем, что обеспечивает выявление единичных копий ДНК вируса гепатита B.
4. Рибонуклеопротеиновый комплекс системы CRISPR-Cas для выявления ДНК вируса гепатита B, сформированный из РНК-направляемой ДНК-эндонуклеазы LbCpf1 из Lachnospiraceae, и по меньшей мере одной направляющей РНК из пп. 1-3.
5. Рибонуклеопротеиновый комплекс системы CRISPR-Cas по п. 4, пригодный для выявления единичных копий ДНК вируса гепатита B.
6. Набор для обнаружения ДНК вируса гепатита B, содержащий
(i) (a) молекулу направляющей РНК системы CRISPR-Cas по любому из пп. 1-3 и направляемую ДНК-эндонуклеазу LbCpf1 из Lachnospiraceae;
или
(i) (b) рибонуклеопротеиновый комплекс по любому из пп. 4 и 5;
и
(ii) инструкцию по применению.
7. Набор для обнаружения ДНК вируса гепатита B, содержащий
(i) (a) молекулу направляющей РНК системы CRISPR-Cas по любому из пп. 1-3 и направляемую ДНК-эндонуклеазу LbCpf1 из Lachnospiraceae;
или
(i) (b) рибонуклеопротеиновый комплекс по любому из пп. 4 и 5;
и
(ii) один или несколько специфических олигонуклеотидов, выбранных из SEQ ID NO: 7-18,
и
(iii) инструкцию по применению.
| XU CHEN et al., A CRISPR-Cas 12b-Based Platform for Ultrasensitive, Rapid, and Highly Specific Detection of Hepatitis В Virus Genotypes В and С in Clinical Application, Front Bioeng Biotechnol, 2021, 9:743322 | |||
| RONGHUA DING et al., CRISPR/Cas12-Based Ultra-Sensitive and Specific Point-of-Care Detection of HBV, Int | |||
| J | |||
| Mol | |||
| Sci., 2021, 22, 4842 |
Авторы
Даты
2022-11-01—Публикация
2021-12-27—Подача