1. Область изобретения
Настоящее раскрытие относится к новому варианту регулятора А метаболизма ацетата, к полинуклеотиду, кодирующему данный вариант, к микроорганизму для продуцирования L-аминокислот с разветвленной цепью, к микроорганизму, включающему данный вариант или полинуклеотид, и к способу продуцирования L-аминокислот с разветвленной цепью с использованием данного микроорганизма.
2. Описание связанной области
L-аминокислоты представляют собой основные структурные элементы белков и используются в качестве важного сырья для фармацевтического сырья, пищевых добавок, кормов для животных, питательных веществ, инсектицидов, бактерицидов и т.д. В частности, аминокислота с разветвленной цепью (ВСАА) представляет собой общий термин для L-валина, L-лейцина и L-изолейцина, которые представляют собой незаменимые аминокислоты. Известно, что аминокислоты с разветвленной цепью имеют антиоксидантные эффекты и эффект прямого стимулирования синтеза белка в мышечных клетках.
Тем временем, продукция аминокислот с разветвленной цепью с использованием микроорганизмов, главным образом, проводится посредством микроорганизмов рода Escherichia или микроорганизмов рода Corynebacterium, и известно, что аминокислоты с разветвленной цепью биосинтезируются с использованием 2-кетоизокапроата в качестве предшественника из пировиноградной кислоты посредством нескольких стадий (Корейский патент №100220018, Корейский патент №100438146). Однако проблемой с продукцией L-аминокислот с разветвленной цепью с использованием микроорганизмов является то, что массовое промышленное производство не является легким.
С данным предшествующим уровнем техники авторы настоящего изобретения обнаружили то, что способность к продукции L-аминокислот с разветвленной цепью может примечательно увеличиваться при введении варианта микроорганизма с усиленной активностью регулятора А метаболизма ацетата (далее именуемого RamA, J Bacteriol. 2006 Apr; 188 (7): 2554-67, Applied Microbiology and Biotechnology (2018) 102: 5901-5910) с целью улучшения способности к продукции L-аминокислот с разветвленной цепью посредством микроорганизмов.
Авторы настоящего изобретения разработали новый вариант регулятора А метаболизма ацетата, который улучшает продукцию L-аминокислот с разветвленной цепью, полинуклеотид, кодирующий данный вариант, микроорганизм для продуцирования L-аминокислот с разветвленной цепью, микроорганизм, включающий данный вариант или полинуклеотид, и способ продуцирования L-аминокислот с разветвленной цепью с использованием данного микроорганизма, посредством этого осуществляя настоящее раскрытие.
КРАТКОЕ ИЗЛОЖЕНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Целью настоящего раскрытия является предложение варианта регулятора А метаболизма ацетата, у которого аминокислота, соответствующая положению 56 в аминокислотной последовательности SEQ ID NO: 3, заменена на другую аминокислоту.
Другой целью настоящего раскрытия является предложение полинуклеотида, кодирующего вариант по настоящему раскрытию.
Еще одной другой целью настоящего раскрытия является предложение микроорганизма для продуцирования L-аминокислот с разветвленной цепью, включающего вариант по настоящему раскрытию или полинуклеотид, кодирующий данный вариант.
Еще одной другой целью настоящего раскрытия является предложение способа продуцирования L-аминокислот с разветвленной цепью, включающего стадию культивирования данного микроорганизма в среде.
Еще одной другой целью настоящего раскрытия является предложение композиции для продуцирования L-аминокислот с разветвленной цепью, включающей вариант по настоящему раскрытию, полинуклеотид, кодирующий данный вариант, вектор, включающий данный полинуклеотид, или микроорганизм, включающий полинуклеотид по настоящему раскрытию; среды, в которой культивируется данный микроорганизм; или комбинации двух или более чем двух их них.
Еще одной другой целью настоящего раскрытия является предложение применения варианта регулятора А метаболизма ацетата, у которого аминокислота, соответствующая положению 56 в аминокислотной последовательности SEQ ID NO: 3, заменена на другую аминокислоту, в продукции L-аминокислот с разветвленной цепью.
ПОДРОБНОЕ ОПИСАНИЕ ПРЕДПОЧТИТЕЛЬНЫХ ВОПЛОЩЕНИЙ
Настоящее раскрытие будет подробно описано следующим образом. Тем временем, каждое описание и воплощение, раскрытое в данном раскрытии, также может применяться к другим описаниям и воплощениям. То есть, все комбинации разных элементов, раскрытых в данном раскрытии, попадают в пределы объема настоящего раскрытия. Кроме того, объем настоящего раскрытия не ограничивается конкретным описанием, описанным ниже. Кроме того, целый ряд статей и патентных документов приводятся в виде ссылок и цитируются во всем данном описании изобретения. Раскрытия процитированных статей и патентных документов включаются сюда посредством ссылки во всей их полноте для дальнейшего прояснения уровня и объема объекта, к которому относится настоящее раскрытие.
Согласно одному аспекту настоящего раскрытия предложен вариант регулятора А метаболизма ацетата, у которого аминокислота, соответствующая положению 56 в аминокислотной последовательности SEQ ID NO: 3, заменена на другую аминокислоту.
Варианты по настоящему раскрытию могут представлять собой варианты, имеющие усиленную активность, посредством замены аминокислоты в конкретном сайте в аминокислотной последовательности существующего регулятора А метаболизма ацетата, но не ограничивающиеся ими.
В одном воплощении вариант регулятора А метаболизма ацетата может представлять собой вариант регулятора А метаболизма ацетата, включающий одну или более чем одну, или две или более чем две аминокислотные замены в аминокислотной последовательности SEQ ID NO: 3, но не ограничивается ими.
В частности, в отношении варианта по настоящему раскрытию аминокислота(ты), соответствующая(щие) положению 56 и/или положению 52 в SEQ ID NO: 3, может(гут) быть заменена(ны) другой(ими) аминокислотой(ами), но не ограничиваясь ими. Более конкретно, в отношении данного варианта, аминокислоты в любом одном или более чем одном положении, или в обоих положениях, или в соответствующих им положениях могут быть заменены другой аминокислотой, но не ограничиваясь ими.
Термин «другая аминокислота» не ограничивается, при условии, что она является отличной от аминокислоты перед заменой. Например, при описании как «осуществление замены аминокислоты, соответствующей положению 56 в SEQ ID NO: 3, другой аминокислотой», это означает замену фенилаланином, глицином, аланином, аргинином, аспартатом, цистеином, глутаминовой кислотой (глутаматом), аспарагином, глутамином, гистидином, пролином, серином, тирозином, изолейцином, лизином, триптофаном, валином, метионином или треонином, исключая лейцин, и при описании как «осуществление замены аминокислоты, соответствующей положению 52 в SEQ ID NO: 3, другой аминокислотой», это означает замену аспарагином, глицином, аргинином, аспартатом, цистеином, глутаминовой кислотой, глутамином, гистидином, пролином, серином, тирозином, изолейцином, лейцином, лизином, фенилаланином, триптофаном, валином, метионином или треонином, исключая аланин, но не ограничиваясь ими.
Тем временем специалисты в данной области могут идентифицировать аминокислоты, соответствующие положениям 56 и 52 SEQ ID NO: 3 по настоящему раскрытию, в любой аминокислотной последовательности посредством выравнивания последовательности, известного в данной области, и, хотя и определяется в настоящем раскрытии иначе, очевидно, что описание «аминокислота в конкретном положении в конкретной последовательности номер» подразумевает включение «аминокислоты в положении, соответствующем ему» в произвольной аминокислотной последовательности. Следовательно, аминокислотная последовательность, в которой любая одна или более чем одна аминокислота, выбранная из группы, состоящей из аминокислот, соответствующих положениям 56 и 52 SEQ ID NO: 3, заменяется другой аминокислотой, также включается в объем настоящего раскрытия.
Например, когда одна или более чем одна, или две или более чем две аминокислоты среди аминокислот, соответствующих положениям 56 и 52 SEQ ID NO: 3, заменены другой аминокислотой, возможно предложение варианта, имеющего более высокую активность, чем аминокислотная последовательность без замен (немодифицированная).
В частности, вариант по настоящему раскрытию может представлять собой вариант, в котором аминокислоты, соответствующие положениям 56 и 52 SEQ ID NO: 3, заменяются другой аминокислотой, но не ограничивается им.
Для конкретного примера, вариант по настоящему раскрытию может представлять собой вариант, в котором лейцин, который представляет собой аминокислоту, соответствующую положениям 56 SEQ ID NO: 3, заменяется аланином, и аланин, который представляет собой аминокислоту, соответствующую положениям 52 SEQ ID NO: 3, заменяется валином, но не ограничивается им.
Для более конкретного примера вариант по настоящему раскрытию может иметь аминокислотную последовательность, представленную SEQ ID NO: 1 или SEQ ID NO: 5, или может по существу состоять из данной аминокислотной последовательности.
Кроме того, вариант по настоящему раскрытию может включать замену аминокислоты(лот), соответствующей(щих) положениям 56 и/или 52 с N-конца SEQ ID NO: 3, на другие аминокислоты в аминокислотной последовательности, имеющей по меньшей мере 70%-ную, 75%-ную, 80%-ную, 85%-ную, 90%-ную, 95%-ную, 96%-ную, 97%-ную, 98%-ную, 99%-ную, 99,5%-ную, 99,7%-ную или 99,9%-ную или большую гомологию или идентичность с аминокислотной последовательностью, представленной SEQ ID NO: 1 или SEQ ID NO: 5. Также очевидно то, что вариант, имеющий аминокислотную последовательность, имеющую делецию, модификацию, замену, консервативную замену или присоединение некоторых аминокислот, также попадает в пределы объема настоящего раскрытия, при условии, что данная аминокислотная последовательность имеет такую гомологию или идентичность и демонстрирует эффективность, соответствующую эффективности варианта по настоящему раскрытию.
Его примеры включают примеры, имеющие добавление или делецию последовательности, которые не изменяют функцию варианта по настоящему раскрытию, на N-конце, С-конце аминокислотной последовательности и/или внутри аминокислотной последовательности, встречающуюся в природе мутацию, молчащую мутацию или консервативную замену.
Термин «консервативная замена» в том виде, как он здесь используется, означает замену одной аминокислоты другой аминокислотой, имеющей аналогичные структурные и/или химические свойства. Такая аминокислотная замена обычно может происходить на основе сходства в полярности, заряде, растворимости, гидрофобности, гидрофильности и/или амфипатической природе остатков. Например, положительно заряженные (основные) аминокислоты включают аргинин, лизин и гистидин; отрицательно заряженные (кислотные) аминокислоты включают глутаминовую кислоту и аспартат; ароматические аминокислоты включают фенилаланин, триптофан и тирозин, гидрофобные аминокислоты включают аланин, валин, изолейцин, лейцин, метионин, фенилаланин, тирозин и триптофан. Кроме того, аминокислоты могут подразделяться на аминокислоты, имеющие электрически заряженную боковую цепь, и аминокислоты, имеющие незаряженную боковую цепь. Аминокислоты, имеющие электрически заряженную боковую цепь, включают аспарагиновую кислоту, глутаминовую кислоту, лизин, аргинин, гистидин, и аминокислоты, имеющие незаряженную боковую цепь, также могут подразделяться на неполярные аминокислоты и полярные аминокислоты. Неполярные аминокислоты могут включать глицин, аланин, валин, лейцин, изолейцин, метионин, фенилаланин, триптофан и пролин, и полярные аминокислоты могут включать серин, треонин, цистеин, тирозин, аспарагин и глутамин. Обычно консервативная замена может едва влиять или не влияет на активность продуцированных полипептидов. Обычно консервативная замена может едва влиять или не влияет на активность белков или полипептидов.
Кроме того, данный вариант может включать делеции или добавления аминокислот, которые имеют минимальный эффект на свойства и вторичную структуру данного полипептида. Например, данный полипептид может быть конъюгирован с N-концевой сигнальной (или лидерной) последовательностью белка, которая котрансляционно или посттрансляционно участвует в транслокации белка. Кроме того, данный полипептид может быть конъюгирован с другими последовательностями или линкерами таким образом, чтобы быть идентифицированным, очищенным или синтезированным.
Термин «вариант» в том виде, как он здесь используется, относится к полипептиду, который имеет отличную аминокислотную последовательность от последовательности данного варианта перед тем, как она изменяется посредством консервативной замены и/или модификации одной или более чем одной аминокислоты, но сохраняет функции или свойства. Такой вариант обычно можно идентифицировать посредством модифицирования одной или более чем одной аминокислоты аминокислотной последовательности полипептида и оценки свойств модифицированного полипептида. Другими словами, способность данного варианта может увеличиваться, оставаться неизменной или снижаться по сравнению со способностью полипептида перед изменением. Кроме того, некоторые варианты могут включать варианты, в которых одна или более чем одна часть, такая как N-концевая лидерная последовательность или трансмембранный домен, была удалена. Другие варианты могут включать варианты, в которых от зрелого белка была удалена часть N- и/или С-конца. Термин «вариант» можно использовать взаимозаменяемо с такими терминами, как модификация, модифицированный полипептид, модифицированный белок, мутант, мутеин и дивергент, и он не ограничивается ими, при условии, что он представляет собой термин, используемый со значением изменения. Относительно целей настоящего раскрытия, данный вариант может представлять собой полипептид, включающий аминокислотную последовательность, представленную SEQ ID NO: 1, в которой лейцин, который представляет собой аминокислоту, соответствующую положению 56 SEQ ID NO: 3, заменен аланином, или полипептид, включающий аминокислотную последовательность, представленную SEQ ID NO: 5, в которой лейцин, который представляет собой аминокислоту, соответствующую положению 56 SEQ ID NO: 3, заменен аланином, и аланин, который представляет собой аминокислоту, соответствующую положению 52 SEQ ID NO: 3, заменен валином.
Кроме того, данный вариант может включать делеции или добавления аминокислот, которые имеют минимальный эффект на свойства и вторичную структуру данного полипептида. Например, с N-концом данного варианта может быть конъюгирована сигнальная (или лидерная) последовательность, которая котрансляционно или постгрансляционно участвует в транслокации белка. Данный вариант может быть конъюгирован с другими последовательностями или линкерами таким образом, чтобы быть идентифицированным, очищенным или синтезированным.
Термин «гомология» или «идентичность» в том виде, как он здесь используется, означает степень сходства между двумя данными аминокислотными последовательностями или последовательностями оснований и может выражаться в виде процентной доли. Термины «гомология» и «идентичность» часто могут использоваться взаимозаменяемо.
Гомологию или идентичность последовательности консервативного полинуклеотид а или полипептида определяют стандартными алгоритмами выравнивания, и можно совместно использовать штраф за пробел, устанавливаемый программой по умолчанию. По существу, гомологичные или идентичные последовательности обычно способны к гибридизации с полной последовательностью или с частью последовательности при умеренно или сильно жестких условиях. Очевидно, что гибридизация также включает гибридизацию с полинуклеотидом, включающим кодон общего типа или кодон с учетом вырожденности кодонов.
Имеют ли или нет любые две последовательности полинуклеотид а или полипептида гомологию, сходство или идентичность, можно определять с использованием известных компьютерных алгоритмов, таких как программа «FASTA», например, с использованием параметров по умолчанию, как в Pearson et at (1988) Proc. Natl. Acad. Sci. USA 85: 2444. В качестве альтернативы, гомологию, сходство или идентичность можно определять с использованием алгоритма Needleman-Wunsch (Needleman and Wunsch, 1970, J. Mol. Biol. 48:443-453), как осуществляется в программе Needleman пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al. (2000), Trends Genet. 16:276-277) (версия 5.0.0 или более поздняя) (включая программный пакет GCG (Devereux, J., et al., Nucleic Acids Research 12:387 (1984)), BLASTP, BLASTN, FASTA (Atschul, S.F. et al., JMOLEC BIOL 215:403 (1990); Guide to Huge Computers, Martin J. Bishop, ed., Academic Press, San Diego, 1994 и CARILLO et al. (1988) SIAM J Applied Math 48:1073). Например, для определения гомологии, сходства или идентичности можно использовать BLAST Национального центра биотехнологической информации или ClustalW.
Гомологию, сходство или идентичность полинуклеотидов или полипептидов можно определять сравнением информации по последовательности с использованием, например, компьютерной программы GAP, такой как в Needleman et al. (1970), JMolBiol. 48:443, как анонсировано, например, в Smith and Waterman, Adv. Appl. Math (1981) 2:482. В заключение, результат программы GAP может быть определен как значение, полученное делением числа аналогично выровненных символов (т.е. нуклеотидов или аминокислот) на общее число символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать (1) матрицу двоичных сравнений (включающую значения 1 для идентичности и 0 для неидентичности) и матрицу взвешенных сравнений Gribskov et al., (1986) Nucl. Acids Res. 14:6745 (или матрицу замен EDNAFULL (EMBOSS версии NCBI NUC4.4)), как раскрыто в Schwartz and Dayhoff, eds., Atlas Of Protein Sequence And Structure, National Biomedical Research Foundation, pp. 353-358 (1979); (2) штраф 3,0 для каждого пропуска и дополнительный штраф 0,10 для каждого символа в каждом пропуске (или штраф 10 за открытие пропуска, штраф 0,5 за удлинение пропуска); и (3) отсутствие штрафа за концевые пропуски.
Для одного примера настоящего раскрытия вариант по настоящему раскрытию может иметь активность регулятора А метаболизма ацетата. Кроме того, вариант по настоящему раскрытию может иметь активность для увеличения способности к продуцированию L-аминокислоты с разветвленной цепью по сравнению с полипептидом дикого типа, имеющим активность регулятора А метаболизма ацетата.
Термин «регулятор А метаболизма ацетата» в том виде, как он здесь используется, представляет собой белок-мишень по настоящему раскрытию и относится к регуляторному белку, относящемуся к метаболизму ацетата, и может кодироваться геном ramA.
В настоящем раскрытии экспрессия регулятора А метаболизма ацетата может усиливаться, приводя к увеличению продуктивности в отношении L-аминокислот с разветвленной цепью.
Термин «соответствующий» в том виде, как он здесь используется, относится к аминокислотным остаткам в положениях, перечисленных в данном полипептиде, или к аминокислотным остаткам, которые являются аналогичными, идентичными или гомологичными остаткам, перечисленным данном полипептиде. Осуществление идентификации аминокислоты в соответствующем положении может быть определяющим для конкретной аминокислоты в последовательности, которая относится к специфической последовательности. Термин «соответствующая область» в том виде, как он здесь используется, обычно относится к аналогичному или соответствующему положению в родственном белке или эталонном белке.
Например, произвольную аминокислотную последовательность выравнивают с SEQ ID NO: 3, и на основе этого каждый аминокислотный остаток данной аминокислотной последовательности может быть пронумерован по отношению к числовому положению аминокислотного остатка, соответствующего аминокислотному остатку SEQ ID NO: 3. Например, алгоритм выравнивания последовательности, как описано в настоящем раскрытии, может определять положение аминокислоты или положение, в котором происходит модификация, такая как замена, вставка или делеция, посредством сравнения с положением в запрашиваемой последовательности (также именуемой «эталонная последовательность»).
Для таких выравниваний, например, можно использовать алгоритм Needleman-Wunsch (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443-453), программу Needleman пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000, Trends Genet. 16:276-277) и т.д., но не ограничиваясь ими, и можно подходящим образом использовать программу выравнивания последовательности, алгоритм попарного сравнения последовательностей и т.д., которые известны в данной области.
Согласно другому аспекту настоящего раскрытия предложен полинуклеотид, кодирующий вариант по настоящему раскрытию.
Термин «полинуклеотид» в том виде, как он здесь используется, представляет собой нить ДНК или РНК, имеющую определенную длину, или более конкретно - полимер нуклеотидов, в котором мономеры нуклеотидов соединяются в длинную цепь ковалентными связями, и, более конкретно, означает фрагмент полинуклеотид а, кодирующий указанный вариант.
Полинуклеотид, кодирующий вариант по настоящему раскрытию, может включать нуклеотидную последовательность, кодирующую аминокислотную последовательность, представленную SEQ ID NO: 1 или SEQ ID NO: 5. В качестве примера настоящего раскрытия полинуклеотид по настоящему раскрытию может иметь или включать последовательность SEQ ID NO: 2 или SEQ ID NO: 6. Кроме того, полинуклеотид по настоящему раскрытию может состоять или по существу состоит из последовательности SEQ ID NO: 2 или SEQ ID NO: 6.
В полинуклеотиде по настоящему раскрытию могут быть сделаны разные модификации в кодирующей области, при условии, что аминокислотная последовательность варианта по настоящему раскрытию не изменяется при рассмотрении вырожденности кодонов или предпочтительных кодонов в организмах, которые предназначены для экспрессии варианта по настоящему раскрытию. В частности, полинуклеотид по настоящему раскрытию может иметь или включает последовательность оснований, имеющую 70%-ную или большую, 75%-ную или большую, 80%-ную или большую, 85%-ную или большую, 90%-ную или большую, 95%-ную или большую, 96%-ную или большую, 97%-ную или большую, 98%-ную или большую и меньше, чем 100%-ную гомологию или идентичность с последовательностью SEQ ID NO: 2 или SEQ ID NO: 6, или может состоять или по существу состоит из последовательности оснований, имеющей 70%-ную или большую, 75%-ную или большую, 80%-ную или большую, 85%-ную или большую, 90%-ную или большую, 95%-ную или большую, 96%-ную или большую, 97%-ную или большую, 98%-ную или большую и меньше, чем 100%-ную гомологию или идентичность с последовательностью SEQ ID NO: 2 или SEQ ID NO: 6, но не ограничиваясь ими. Здесь в последовательности, имеющей гомологию или идентичность, кодон, кодирующий аминокислоту, соответствующую положению 56 SEQ ID NO: 1 или SEQ ID NO: 5, может представлять собой один из кодонов, кодирующих аланин, и кодон, кодирующий аминокислоту, соответствующую положению 52 SEQ ID NO: 5, может представлять собой один из кодонов, кодирующих валин.
Кроме того, полинуклеотид по настоящему раскрытию может включать зонд, который может быть получен из известной последовательности гена, например, последовательности без ограничения, при условии, что она представляет собой последовательность, которая способна гибридизоваться с комплементарной последовательностью, со всей или с частью полинуклеотидной последовательности по настоящему раскрытию при жестких условиях. «Жесткие условия» означают условия, которые обеспечивают специфичную гибридизацию между полинуклеотидами. Данные условия конкретно описываются в документах (см. J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York, 9.50 9.51, 11.7 11.8). Их примеры включают условия, в которых полинуклеотиды, имеющие более высокую гомологию или идентичость, т.е. полинуклеотиды, имеющие 70%-ную или большую, 75%-ную или большую, 80%-ную или большую, 85%-ную или большую, 90%-ную или большую, 95%-ную или большую, 96%-ную или большую, 97%-ную или большую, 98%-ную или большую, или 99%-ную или большую гомологию или идентичность, гибридизуются друг с другом, тогда как полинуклеотиды, имеющие меньшую гомологию или идентичность, не гибридизуются друг с другом, или условия, при которых промывка осуществляется один раз, в частности, от двух до трех раз при концентрации соли и температуре, эквивалентных 60°С, 1× SSC (раствор хлорида и цитрата натрия), 0,1% SDS (додецилсульфат натрия), в частности, при 60°С, 0,1× SSC, 0,1% SDS и, более конкретно, при 68°С, 0,1× SSC, 0,1% SDS, которые представляют собой условия промывки для обычной саузерн-гибридизации.
Для гибридизации требуется, чтобы две нуклеиновые кислоты имели комплементарные последовательности, хотя допускаются и несоответствия между основаниями, в зависимости от жесткости гибридизации. Термин «комплементарный» используется для описания связи между нуклеотидными основаниями, способными к гибридизации друг с другом. Например, в отношении ДНК, аденин является комплементарным тимину, и цитозин является комплементарным гуанину. Следовательно, полинуклеотид по настоящему раскрытию также может включать по существу аналогичные последовательности нуклеиновой кислоты, а также фрагменты выделенной нуклеиновой кислоты, которые являются комплементарными всей последовательности.
В частности, полинуклеотид, имеющий гомологию или идентичность с полинуклеотидом по настоящему раскрытию, может быть выявлен с использованием условий гибридизации, включающих стадию гибридизации при значении Tm 55°С и вышеописанных условиях. Кроме того, значение Tm может составлять 60°С, 63°С или 65°С, но не ограничивается ими, и может подходящим образом корректироваться специалистами в данной области согласно цели.
Подходящая жесткость для гибридизации данного полинуклеотида зависит от длины и степени комплементарности данного полинуклеотида, и переменные хорошо известны в данной области (например, J. Sambrook et al., выше).
Согласно еще одному другому аспекту настоящего раскрытия предложен вектор, включающий полинуклеотид по настоящему раскрытию. Данный вектор может представлять собой экспрессионный вектор для осуществления экспрессии полинуклеотида у микроорганизма, но не ограничивается им.
Термин «вектор» в том виде, как он здесь используется, может включать ДНК-конструкцию, включающую последовательность полинуклеотида, кодирующую интересующий полипептид, который связан функциональным образом с подходящей регуляторной областью экспрессии (или последовательностью контроля экспрессии) таким образом, что интересующий полипептид может экспрессироваться в подходящем хозяине. Регуляторная область экспрессии может включать промотор, способный инициировать транскрипцию, любую последовательность оператора для осуществления контроля транскрипции, последовательность, кодирующую подходящий сайт связывания мРНК с рибосомой, и последовательность, контролирующую терминацию транскрипции и трансляции. Данный вектор можно трансформировать в подходящий микроорганизм, и затем он может реплицироваться или функционировать независимо от генома хозяина, или может сам интегрироваться в геном.
Вектор, используемый в настоящем раскрытии, конкретно не ограничен, но можно использовать любой вектор, известный в данной области. Примеры обычно используемых векторов включают природные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, в качестве фагового вектора или космидного вектора можно использовать pWE15, М13, MBL3, MBL4, IXII, ASHII, APII, t10, t11, Charon4A, Charon21A и т.д., и в качестве плазмидного вектора можно использовать систему pDZ, систему pBR, систему pUC, систему pBluescript II, систему pGEM, систему pTZ, систему pCL, систему рЕТ и т.д. В частности, можно использовать вектор pDZ, pDC, pDCM2, pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118, pCC1BAC, и т.д.
Например, полинуклеотид, кодирующий интересующий полипептид, можно вставлять в хромосому посредством вектора для внутриклеточной хромосомной вставки. Вставку указанного полинуклеотида в хромосому можно осуществлять любым способом, известным в данной области, например, гомологичной рекомбинацией, но не ограничиваясь ей. Указанный вектор может дополнительно включать селективный маркер для подтверждения вставки в хромосому. Указанный селективный маркер служит для отбора клеток, трансформированных векторами, то есть, для подтверждения вставки интересующей молекулы нуклеиновой кислоты, и можно использовать маркеры, которые придают селектируемые фенотипы, такие как устойчивость к лекарственным средствам, ауксотрофия, устойчивость к цитотоксическим агентам или экспрессия поверхностных полипептидов. В среде при обработке селективным агентом выживают или демонстрируют другие фенотипические признаки только клетки, экспрессирующие селективный маркер, и, таким образом, могут быть отобраны трансформированные клетки.
Термин «трансформация» в том виде, как он используется в данном документе, означает то, что вектор, включающий полинуклеотид, кодирующий целевой полипептид, вводится в микроорганизм таким образом, что полипептид, кодируемый данным полинуклеотидом, может экспрессироваться в данном микроорганизме. Трансформированный полинуклеотид может локализоваться, независимо от положения, либо вставленным в хромосому микроорганизма, либо локализованным вне хромосомы, при условии, что он может экспрессироваться в данном микроорганизме. Указанный полинуклеотид включает ДНК и/или РНК, кодирующую интересующий полипептид. Указанный полинуклеотид может быть введен в любой форме, при условии, что он может быть введен в микроорганизм и может экспрессироваться. Например, указанный полинуклеотид может быть введен в микроорганизм в виде экспрессионной кассеты, которая представляет собой генетическую конструкцию, содержащую все элементы, требующиеся для автономной экспрессии. Указанная экспрессионная кассета обычно может включать промотор, связанный функциональным образом с полинуклеотидом, сигнал терминации транскрипции, сайт связывания рибосомы и сигнал терминации трансляции. Указанная экспрессионная кассета может находиться в виде экспрессионного вектора, способного к автономной репликации. Кроме того, указанный полинуклеотид может быть введен в микроорганизм сам по себе и связан функциональным образом с последовательностью, требующейся для экспрессии в микроорганизме, но не ограничиваясь ей.
Кроме того, термин «связанный функциональным образом» означает то, что последовательность полинуклеотида функционально связана с последовательностью промотора, которая инициирует и опосредует транскрипцию полинуклеотида, кодирующего интересующий вариант по настоящему раскрытию.
Согласно еще одному другому аспекту настоящего раскрытия предложен микроорганизм для продуцирования L-аминокислот с разветвленной цепью, включающий вариант по настоящему раскрытию или полинуклеотид по настоящему раскрытию.
В частности, указанный микроорганизм может представлять собой микроорганизм рода Corynebacterium, и более конкретно Corynebacterium glutamicum, но не ограничивается ей.
Микроорганизм по настоящему раскрытию может включать модифицированный полипептид по настоящему раскрытию, полинуклеотид, кодирующий данный полипептид, или вектор, включающий полинуклеотид по настоящему раскрытию.
Термин «микроорганизм (или штамм)» в том виде, как он здесь используется, включает все микроорганизмы дикого типа или естественно или искусственно генетически модифицированные микроорганизмы, и он может представлять собой микроорганизм, в котором ослаблен или усилен специфический механизм из-за вставки чужеродного гена или усиления активности, или инактивации эндогенного гена, и он может представлять собой микроорганизм, включающий генетическую модификацию для продукции интересующего полипептида, белка или продукта.
Штамм по настоящему раскрытию может представлять собой штамм, включающий любой один или более чем один вариант по настоящему раскрытию, полинуклеотид по настоящему раскрытию и вектор, включающий полинуклеотид по настоящему раскрытию; штамм, модифицированный для экспрессии варианта по настоящему раскрытию или полинуклеотида по настоящему раскрытию; штамм (например, рекомбинантный штамм), экспрессирующий вариант по настоящему раскрытию или полинуклеотид по настоящему раскрытию; или штамм (например, рекомбинантный штамм), имеющий активность варианта по настоящему раскрытию, но не ограничивается ими.
Штамм по настоящему раскрытию может представлять собой штамм, имеющий способность к продуцированию L-аминокислот с разветвленной цепью.
Штамм по настоящему раскрытию может представлять собой микроорганизм, имеющий в природе регулятор А метаболизма ацетата или способность к продуцированию L-аминокислот с разветвленной цепью, или микроорганизм, у которого вариант по настоящему раскрытию или полинуклеотид, кодирующий данный вариант (или вектор, включающий данный полинуклеотид), вводится в родительский штамм, который не имеет регулятора А метаболизма ацетата или способности к продуцированию L-аминокислот с разветвленной цепью, и/или у которого способность к продуцированию L-аминокислот с разветвленной цепью придается родительскому штамму, но не ограничивается ими.
Например, штамм по настоящему раскрытию представляет собой клетку или микроорганизм, который трансформирован полинуклеотидом по настоящему раскрытию или вектором, включающим полинуклеотид, кодирующий вариант по настоящему раскрытию, для экспрессии варианта по настоящему раскрытию. В отношении целей настоящего раскрытия штамм по настоящему раскрытию может включать любой микроорганизм, который включает вариант по настоящему раскрытию и способен к продукции L-аминокислот с разветвленной цепью. Например, штамм по настоящему раскрытию может представлять собой рекомбинантный штамм, в котором полинуклеотид, кодирующий вариант по настоящему раскрытию, вводят в природный микроорганизм дикого типа или микроорганизм, продуцирующий L-аминокислоты с разветвленной цепью, для того, чтобы экспрессировать вариант регулятора А метаболизма ацетата и иметь повышенную способность к продуцированию L-аминокислоты с разветвленной цепью. Рекомбинантный штамм, имеющий повышенную способность к продуцированию L-аминокислоты с разветвленной цепью, может представлять собой микроорганизм, имеющий повышенную способность к продуцированию L-аминокислоты с разветвленной цепью по сравнению с природным микроорганизмом дикого типа или микроорганизмом, в котором регулятор А метаболизма ацетата не модифицирован (т.е. микроорганизмом, экспрессирующим регулятор А метаболизма ацетата дикого типа (SEQ ID NO: 3), или микроорганизмом, который не экспрессирует указанный модифицированный белок (SEQ ID NO: 1 или SEQ ID NO: 5), но не ограничивается ими. Например, штамм, имеющий повышенную способность к продуцированию L-аминокислоты с разветвленной цепью по настоящему раскрытию может представлять собой микроорганизм, имеющий повышенную способность к продуцированию L-аминокислоты с разветвленной цепью по сравнению с микроорганизмом, включающим полипептид SEQ ID NO: 3 или полинуклеотид, кодирующий данный полипептид, но не ограничивается им.
Например, рекомбинантный штамм, имеющий повышенную способность к продуцированию, может иметь повышенную способность к продуцированию L-аминокислот с разветвленной цепью примерно на 1% или более, в частности, примерно на 1% или более, примерно на 2,5% или более, примерно на 5% или более, примерно на 6% или более, примерно на 7% или более, примерно на 8% или более, примерно на 9% или более, примерно на 10% или более, примерно на 10,5% или более, примерно на 11% или более, примерно на 11,5% или более, примерно на 12% или более, примерно на 12,5% или более, примерно на 13% или более, примерно на 13,5% или более, примерно на 14% или более, примерно на 14,1% или более, примерно на 14,2% или более, примерно на 14,3% или более, примерно на 14,4% или более, примерно на 14,5% или более, или примерно на 14,6% или более (верхняя граница конкретно не ограничивается, но может составлять, например, примерно на 200% или менее, примерно на 150% или менее, примерно на 100% или менее, примерно на 50% или менее, примерно на 45% или менее, примерно на 40% или менее, примерно на 35% или менее, примерно на 30% или менее, примерно на 25% или менее, примерно на 20% или менее, или примерно на 15% или менее) по сравнению со способностью к продуцированию L-аминокислоты с разветвленной цепью родительского штамма перед изменением или с немодифицированным микроорганизмом, но повышенное количество не ограничивается им, при условии, что способность к продуцированию имеет увеличенное количество значения + по сравнению со способностью к продуцированию родительского штамма перед изменением или с немодифицированным микроорганизмом. В другом примере рекомбинантный штамм, имеющий повышенную способность к продуцированию, может иметь повышенную способность к продуцированию L-аминокислоты с разветвленной цепью примерно в 1,1 раза или более, примерно в 1,12 раза или более, примерно в 1,13 раза или более, или в 1,14 раза или более (верхняя граница конкретно не ограничивается, но может быть, например, примерно в 10 раз или менее, примерно в 5 раз или менее, примерно в 3 раза или менее, примерно в 2 раза или менее, примерно в 1,5 раза или менее, или примерно в 1,2 раза или менее) по сравнению со способностью к продуцированию L-аминокислоты с разветвленной цепью родительского штамма перед изменением или с немодифицированным микроорганизмом, но не ограничивается ими. Термин «примерно» относится к интервалу, который включает все из плюс/минус 0,5, плюс/минус 0,4, плюс/минус 0,3, плюс/минус 0,2, плюс/минус 0,1 и т.д. и включает все из значений, которые являются эквивалентными или аналогичными значениям после термина «примерно», но данный интервал не ограничивается ими.
Термин «немодифицированный микроорганизм» в том виде, как он здесь используется, не исключает штаммы, включающие мутацию, которая может встречаться у микроорганизмов в природе, и может представлять собой штамм дикого типа или сам природный штамм, или может представлять собой штамм перед изменением признака посредством генетического изменения из-за природных или искусственных факторов. Например, немодифицированный микроорганизм может представлять собой штамм, в который не вводится или еще не был введен вариант регулятора А метаболизма ацетата, описанный в настоящем описании изобретения. Термин «немодифицированный микроорганизм» может использоваться взаимозаменяемо с терминами «штамм перед модификацией», «микроорганизм перед модификацией», «неизмененный штамм», «немодифицированный штамм», «неизмененный микроорганизм» или «эталонный микроорганизм».
В другом примере настоящего раскрытия микроорганизм по настоящему раскрытию может представлять собой Corynebacterium glutamicum, Corynebacterium crudilactis, Corynebacterium deserti, Corynebacterium efficiens, Corynebacterium callunae, Corynebacterium stationis, Corynebacterium singulare, Corynebacterium halotolerans, Corynebacterium striatum, Corynebacterium ammoniagenes, Corynebacterium pollutisoli, Corynebacterium imitans, Corynebacterium testudinoris или Corynebacterium flavescens.
Термин «усиление» активности полипептида в том виде, как он здесь используется, означает то, что активность полипептида увеличивается по сравнению с эндогенной активностью. Термин «усиление» можно использовать взаимозаменяемо с такими терминами, как активация, повышающая регуляция, сверхэкспрессия и увеличение и т.д. Здесь активация, усиление, повышающая регуляция, сверхэкспрессия и увеличение могут включать и демонстрирование активности, которой исходно не обладал, и демонстрирование улучшенной активности по сравнению с эндогенной активностью или активностью перед модификацией. Термин «эндогенная активность» означает активность специфического полипептида, которой исходно обладал родительский штамм перед изменением признака или немодифицированный микроорганизм, когда признак меняется генетическим изменением из-за природных или искусственных факторов. Это можно использовать взаимозаменяемо с термином «активность перед модификацией». Тот факт, что активность полипептида «усиливается», «подвергается повышающей регуляции», «сверхэкспрессируется» или «увеличивается» по сравнению с эндогенной активностью, означает то, что активность полипептида улучшается по сравнению с активностью и/или концентрацией (уровнем экспрессии) специфического полипептида, которым исходно обладал родительский штамм перед изменением признака или немодифицированный микроорганизм.
Усиление может достигаться через введение чужеродного полипептида или увеличение эндогенной активности и/или концентрации (уровня экспрессии) данного полипептида. Усиление активности полипептида может быть подтверждено посредством увеличения уровня активности и уровня экспрессии соответствующего полипептида или количества продукта, продуцированного из соответствующего полипептида.
Для усиления активности полипептида можно применять разные способы, хорошо известные в данной области, и данный способ не ограничивается, при условии, что активность интересующего полипептида может усиливаться по сравнению с активностью микроорганизма перед модификацией. В частности, можно использовать генную инженерию и/или белковую инженерию, хорошо известные специалистам в данной области, которые являются традиционными способами молекулярной биологии, но данный способ не ограничивается ими (например, Sitnicka et al. Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol. 2. 1-16; Sambrook et al. Molecular Cloning. 2012 и т.д.).
В частности, усиление активности полипептида по настоящему раскрытию может представлять собой:
1) увеличение числа внутриклеточных копий полинуклеотида, кодирующего полипептид;
2) замену регуляторной области экспрессии гена на хромосоме, кодирующей полипептид, последовательностью, демонстрирующей сильную активность;
3) модификацию инициирующего кодона транскрипта гена, кодирующего полипептид, или последовательности оснований, кодирующей 5'-UTR область;
4) модификацию аминокислотной последовательности полипептида для усиления активности данного полипептида;
5) модификацию последовательности полинуклеотида, кодирующей полипептид, для усиления активности данного полипептида (например, модификацию последовательности полинуклеотида гена полипептида для кодирования полипептида, который был модифицирован для усиления активности данного полипептида);
6) введение чужеродного полипептида, демонстрирующего активность данного полипептида, или чужеродного полинуклеотида, кодирующего данный полипептид;
7) оптимизацию кодонов полинуклеотида, кодирующего полипептид;
8) анализ третичной структуры полипептида для отбора и модификации или химической модификации экспонированного сайта; или
9) комбинацию двух или более чем двух, выбранных из (1)-(8), но конкретно не ограничиваясь ими.
Более конкретно:
1) Увеличение числа внутриклеточных копий полинуклеотида, кодирующего полипептид, может быть достигнуто посредством введения в микроорганизм вектора, который может реплицироваться и функционировать независимо от хозяина, и с которым полинуклеотид, кодирующий соответствующий полипептид, связан функциональным образом. В качестве альтернативы, увеличение может быть достигнуто введением одной копии или двух или более чем двух копий полинуклеотида, кодирующего соответствующий полипептид, в хромосому микроорганизма. Введение в хромосому может осуществляться введением вектора, способного вставлять полинуклеотид в хромосому микроорганизма, в микроорганизм, но не ограничивается им. Данный вектор является таким, как описано выше.
2) Замена регуляторной области экспрессии гена (или последовательности контроля экспрессии) на хромосоме, кодирующей полипептид, последовательностью, демонстрирующей сильную активность, может представлять собой, например, появление изменения последовательности из-за делеции, вставки, неконсервативной или консервативной замены или их комбинации, или замену последовательности, демонстрирующей более сильную активность, таким образом, что активность регуляторной области экспрессии дополнительно усиливается. Регуляторная область экспрессии может включать, но конкретно не ограничивается промотором, последовательностью оператора, последовательностью, кодирующей сайт связывания рибосомы, последовательностью, контролирующей терминацию транскрипции и трансляции и т.д. Например, замена может представлять собой замену исходного промотора сильным промотором, но не ограничивается ей.
Примеры известных сильных промоторов включают промоторы CJ1-CJ7 (патент США US 7662943 В2), промотор lac, промотор trp, промотор trc, промотор tac, промотор PR фага лямбда, промотор PL, промотор tet, промотор gapA, промотор SPL7, промотор SPL13(sm3) (патент США US 10584338 В2), промотор O2 (патент США US 10273491 В2), промотор tkt, промотор уссА и т.д., но не ограничиваются ими.
3) Модификация инициирующего кодона транскрипта гена, кодирующуего полипептид, или нуклеотидной последовательности, кодирующей область 5'-UTR, может представлять собой, например, замену последовательностью оснований, кодирующей другой инициирующий кодон, имеющий более высокую скорость экспрессии полипептида по сравнению с эндогенным инициирующим кодоном, но не ограничивается ей.
4) и 5) Модификация аминокислотной последовательности или последовательности полинуклеотида может представлять собой появление изменения в последовательности из-за делеции, вставки, неконсервативной или консервативной замены аминокислотной последовательности полипептида или последовательности полинуклеотида, кодирующей данный полипептид, или их комбинации, или замены аминокислотной последовательностью или последовательностью полинуклеотида, модифицированной для демонстрации более сильной активности, или аминокислотной последовательностью или последовательностью полинуклеотида, модифицированной для того, чтобы быть более активной, таким образом, что активность данного полипептида усиливается, но не ограничивается ими. Данная замена может конкретно осуществляться посредством вставки полинуклеотида в хромосому гомологичной рекомбинацией, но не ограничивается ей. Используемый здесь вектор может дополнительно содержать селективный маркер для подтверждения вставки в хромосому. Данный селективный маркер является таким, как описано выше.
6) Введение чужеродного полинуклеотида, демонстрирующего активность полипептида, может представлять собой введение в микроорганизм чужеродного полинуклеотида, кодирующего полипептид, демонстрирующий идентичную или аналогичную данному полипептиду активность. Нет ограничения по его происхождению или последовательности, при условии, что данный чужеродный полинуклеотид демонстрирует идентичную или аналогичную данному полипептиду активность. Используемый в введении способ может осуществляться посредством подходящего выбора способа трансформации, известного специалистам в данной области. Поскольку введенный полинуклеотид экспрессируется в клетке-хозяине, может продуцироваться полипептид и может увеличиваться его активность.
7) Оптимизация кодонов полинуклеотида, кодирующего данный полипептид, может представлять собой оптимизацию кодонов эндогенного полинуклеотида таким образом, чтобы увеличивать транскрипцию или трансляцию в микроорганизме, или оптимизацию кодонов чужеродного полинуклеотида таким образом, чтобы осуществлять оптимизированную транскрипцию и трансляцию в микроорганизме.
8) Анализ третичной структуры полипептида для выбора и для модификации или химическая модификация экспонированного сайта могут осуществляться, например, для определения шаблонного белка-кандидата согласно степени сходства последовательности посредством сравнения информации по последовательности полипептида, подлежащего анализу, с хранилищем информации по последовательностям известных белков базы данных для подтверждения структуры на основе этого и для модификации или химической модификации экспонированного сайта, подлежащего модификации или химической модификации.
Такое усиление активности полипептида может представлять собой увеличение активности или концентрации, или уровня экспрессии соответствующего полипептида на основе активности или концентрации полипептида, экспрессируемого в микробном штамме дикого типа или в микробном штамме перед модификацией, или увеличение количества продукта, продуцируемого из соответствующего полипептида, но не ограничивается ими.
В микроорганизме по настоящему раскрытию частичная или полная модификация полинуклеотида может индуцироваться (а) гомологичной рекомбинацией с использованием вектора для вставки в хромосому в микроорганизме или геномным редактированием с использованием генетически модифицированной нуклеазы (например, CRISPR-Cas9), и/или (б) обработкой светом, таким как ультрафиолетовые лучи и излучение, и/или химическими агентами, но не ограничиваясь ими. Способ осуществления модификации части или всего гена может включать способ с использованием технологии рекомбинантной ДНК. Например, посредством введения в микроорганизм нуклеотидной последовательности или вектора, включающего нуклеотидную последовательность, гомологичную интересующему гену, для вызова гомологичной рекомбинации можно удалить часть гена или весь данный ген. Нуклеотидная последовательность или вектор, подлежащий введению, может включать доминантный селективный маркер, но не ограничивается им.
В микроорганизме по настоящему раскрытию вариант, полинуклеотид, L-аминокислота с разветвленной цепью и т.д. являются такими, как описано в других аспектах.
Согласно еще одному другому аспекту настоящего раскрытия предложен способ продуцирования L-аминокислот с разветвленной цепью, включающий стадию культивирования микроорганизма в среде.
В частности, способ продуцирования L-аминокислот с разветвленной цепью по настоящему раскрытию может включать стадию культивирования штамма Corynebacterium glutamicum, включающего вариант по настоящему раскрытию или полинуклеотид по настоящему раскрытию, или вектор по настоящему раскрытию, в среде, но не ограничивается ими.
Термин «культивирование» в том виде, как он здесь используется, означает выращивание микроорганизма по настоящему раскрытию в условиях среды, контролируемых подходящим образом. Способ культивирования по настоящему раскрытию можно осуществлять в соответствии с подходящей средой и условиями культивирования, известными в данной области. Такой способ культивирования может быть легко скорректирован и использован специалистами в данной области согласно выбранному штамму. В частности культивирование может быть периодического типа, непрерывного типа и/или типа с подпиткой, но не ограничивается ими.
Термин «среда» в том виде, как он здесь используется, означает смешанное вещество, содержащее питательные вещества, требующиеся для культивирования микроорганизма по настоящему раскрытию в качестве главного компонента, и данная среда поставляет питательные вещества, факторы роста и т.д., включая воду, которые являются незаменимыми для выживания и развития. В частности, в качестве среды и других условий культивирования, используемых для культивирования микроорганизма по настоящему раскрытию, можно использовать любые без конкретного ограничения, при условии, что она представляет собой среду, используемую для обычного культивирования микроорганизмов. Микроорганизм по настоящему раскрытию можно культивировать в обычной среде, содержащей правильные источники углерода, источники азота, источники фосфора, неорганические соединения, аминокислоты и/или витамины, и т.д., при одновременном осуществлении контроля температуры, рН и т.д. при аэробных условиях.
Например, культуральную среду для штамма рода Corynebacterium можно найти в документе ("Manual of Methods for General Bacteriology" by the American Society for Bacteriology (Washington, D.C., USA, 1981)).
В настоящем раскрытии источники углерода включают углеводы, такие как глюкоза, сахароза, лактоза, фруктоза, сахароза, мальтоза и т.д.; сахароспирты, такие как маннит, сорбит и т.д.; органические кислоты, такие как пировиноградная кислота, молочная кислота, лимонная кислота и т.д.; аминокислоты, такие как глутаминовая кислота, метионин и лизин и т.д. Можно использовать природные органические питательные вещества, такие как гидролизат крахмала, мелассу, сырую мелассу, рисовые отруби, маниок, остаток сахарного тростника и жидкий кукурузный экстракт. В частности, можно использовать углеводы, такие как глюкоза и стерилизованные предобработанные мелассы (а именно: мелассы, превращенные в восстанавливающий сахар), и можно использовать подходящие количества других источников углерода разными способами без ограничения. Данные источники углерода можно использовать одиночно или в комбинации двух или более чем двух из них, но не ограничиваясь ими.
В качестве источников азота можно использовать неорганические источники азота, такие как аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония, нитрат аммония и т.д.; и органические источники азота, такие как аминокислоты, такие как глутаминовая кислота, метионин, глутамин и т.д., пептон, NZ-амин, мясной экстракт, дрожжевой экстракт, солодовый экстракт, жидкий кукурузный экстракт, гидролизат казеина, рыба или продукты ее разложения и обезжиренный соевый жмых или продукты его разложения. Данные источники азота можно использовать одиночно или в комбинации двух или более чем двух из них, но не ограничиваясь ими.
Источники фосфора могут включать монокалия фосфат, дикалия фосфат или натрийсодержащие соли, соответствующие им. В качестве неорганических соединений можно использовать хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и т.д. Кроме того, можно включать аминокислоты, витамины и/или подходящие предшественники и т.д. Данные компоненты или предшественники можно добавлять в среду порционно или непрерывно, но не ограничиваясь ими.
Кроме того, во время культивирования микроорганизма по настоящему раскрытию рН среды можно корректировать добавлением в данную среду правильным способом таких соединений, как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота, серная кислота и т.д. Кроме того, во время культивирования пенообразование можно подавлять посредством применения пеногасителя, такого как сложный полигликолевый эфир жирной кислоты. Кроме того, в среду можно инъецировать кислород или кислородсодержащий газ для того, чтобы поддерживать аэробное состояние данной среды, или газ можно не инъецировать, или можно инъецировать газообразный азот, водород или диоксид углерода для того, чтобы поддерживать анаэробное и микроаэробное состояния, но не ограничиваясь ими.
При культивировании по настоящему раскрытию можно поддерживать температуру культивирования от 20°С до 45°С, в частности, от 25°С до 40°С, и данный штамм можно культивировать в течение примерно от 10 до 160 часов, но не ограничиваясь ими.
L-аминокислоты с разветвленной цепью, продуцируемые посредством культивирования по настоящему раскрытию, могут секретироваться в среду или могут оставаться в клетках.
Способ продуцирования L-аминокислот с разветвленной цепью по настоящему раскрытию может дополнительно включать стадии получения микроорганизма по настоящему раскрытию, приготовления среды для культивирования данного штамма или их комбинацию (в любом порядке), например, перед стадией культивирования.
Способ продуцирования L-аминокислот с разветвленной цепью по настоящему раскрытию может дополнительно включать стадию выделения L-аминокислот с разветвленной цепью из среды в соответствии с культивированием (среда, подвергнувшаяся воздействию культуры) или из микроорганизма по настоящему раскрытию. После стадии культивирования может быть дополнительно включена стадия выделения.
Выделение может служить для сбора интересующих L-аминокислот с разветвленной цепью посредством подходящего способа, известного в данной области, согласно способу культивирования микроорганизма по настоящему раскрытию, например, способу периодического, непрерывного культивирования или культивирования с подпиткой. Например, можно использовать центрифугирование, фильтрование, обработку осадителем кристаллизованного белка (высаливание), экстракцию, обработку ультразвуком, ультрафильтрацию, диализ, разные виды хроматографии, такие как хроматография на молекулярных ситах (гель-фильтрация), адсорбционная хроматография, ионообменная хроматография, аффинная хроматография и т.д., ВЭЖХ (высокоэффективная жидкостная хроматография) или их комбинацию. Интересующие L-аминокислоты с разветвленной цепью можно выделять из среды или микроорганизма посредством подходящего способа, известного в данной области.
Способ продуцирования L-аминокислот с разветвленной цепью по настоящему раскрытию может дополнительно включать стадию очистки. Очистку можно осуществлять посредством подходящего способа, известного в данной области. В одном примере, когда способ продуцирования L-аминокислот с разветвленной цепью по настоящему раскрытию включает и стадию выделения, и стадию очистки, данные стадию выделения и стадию очистки можно осуществлять непрерывно или прерывисто, независимо от порядка, или можно осуществлять одновременно, или посредством объединения в одну стадию, но не ограничиваясь ими.
В способе по настоящему раскрытию вариант, полинуклеотид, вектор, микроорганизм и т.д. являются такими, как описано в других аспектах.
Согласно еще одному другому аспекту настоящего раскрытия предложена композиции для продуцирования L-аминокислот с разветвленной цепью, включающая вариант по настоящему раскрытию, полинуклеотид, кодирующий данный вариант, вектор, включающий данный полинуклеотид, микроорганизм, включающий полинуклеотид по настоящему раскрытию; его культуральная среда; или комбинация двух или более чем двух из них.
Композиция по настоящему раскрытию может дополнительно включать произвольные подходящие эксципиенты, которые обычно используют в композициях для продуцирования аминокислот. Такие эксципиенты могут представлять собой, например, консервант, увлажнитель, диспергирующий агент, суспендирующий агент, буферизующий агент, стабилизатор или изотоничный агент и т.д., но не ограничиваются ими.
В композиции по настоящему раскрытию вариант, полинуклеотид, вектор, штамм, среда, L-аминокислоты с разветвленной цепью и т.д. являются такими, как описано в других аспектах.
В еще одном другом аспекте настоящего раскрытия предложено применение варианта регулятора А метаболизма ацетата, в котором аминокислота, соответствующая положению 56 в аминокислотной последовательности SEQ ID NO: 3, заменяется другой аминокислотой, в продукции L-аминокислот с разветвленной цепью.
При применении по настоящему раскрытию вариант, L-аминокислоты с разветвленной цепью и т.д. являются такими, как описано в других аспектах.
Ниже настоящее раскрытие будет более подробно описано посредством типичных воплощений. Однако следующие типичные воплощения являются лишь предпочтительными воплощениями для иллюстрации настоящего раскрытия и, таким образом, не предназначены для ограничения объема настоящего раскрытия ими. Тем не менее технические вопросы, не описанные в настоящем описании изобретения, могут быть в достаточной степени поняты и легко воплощены специалистами в технической области настоящего раскрытия или в аналогичных технических областях.
Пример 1. Отбор мутантных штаммов с повышенной способностью к продуцированию валина посредством искусственного мутагенеза
Пример 1-1. Искусственный мутагенез посредством УФ (ультрафиолетового) излучения
Для того, чтобы отобрать мутантные штаммы с повышенной способностью к продуцированию валина, Corynebacterium glutamicum KCCM11201P (корейский патент №101117022), который представляет собой валин-продуцирующий штамм, распределяли на питательной среде, содержащей агар, и культивировали при 30°С в течение 36 часов. Затем сотни колоний, полученных таким образом, облучали УФ при комнатной температуре для индукции случайных мутаций в геноме данного штамма.
Пример 1-2. Оценка титра ферментации штамма с индуцированными мутациями и скрининг указанного штамма
Для того чтобы отобрать мутантные штаммы с повышенной способностью к продуцированию L-валина по сравнению с Corynebacterium glutamicum KCCM11201P, который использовали в качестве родительского штамма, проводили анализ титра ферментации на штаммах, в которых были индуцированы случайные мутации. После субкультивирования каждой колонии в питательной среде каждый штамм инокулировали в 250 мл колбе с угловыми перегородками, содержащей 25 мл продукционной среды, и культивировали при 30°С в течение 72 часов со встряхиванием при 200 об/мин. Композиции питательной среды и продукционной среды являются следующими.
Питательная среда (рН 7,2)
10 г глюкозы, 5 г мясного экстракта, 10 г полипептона, 2,5 г хлорида натрия, 5 г дрожжевого экстракта, 20 г агара, 2 г мочевины (на основе 1 л дистиллированной воды).
Продукционная среда (рН 7,0)
100 г глюкозы, 40 г сульфата аммония, 2,5 г соевого белка, 5 г твердых веществ кукурузного экстракта, 3 г мочевины, 1 г двухосновного фосфата калия, 0,5 г магния сульфата гептагидрата, 100 мкг биотина, 1 мг тиамина-HCl, 2 мг кальция пантотената, 3 мг никотинамида, 30 г карбоната кальция (на основе 1 л дистиллированной воды).
После завершения культивирования количества продукции L-валина измеряли с использованием ВЭЖХ (высокоэффективная жидкостная хроматография), и концентрации L-валина, проанализированные таким образом, показаны в Таблице 1 ниже.

Как показано в Таблице 1, был выбран штамм С13, который демонстрировал наивысшее увеличение продукции валина, по сравнению с контрольным штаммом КССМ11201Р.
Пример 2. Подтверждение мутации посредством секвенирования генов
Ключевые гены штамма С13 с повышенной способностью к продуцированию валина секвенировали и сравнивали с генами штамма KCCM11201P и штаммом Corynebacterium glutamicum АТСС14067 дикого типа. В результате подтвердили то, что штамм С13 включал мутации нуклеотидной последовательности в конкретных положениях в области открытой рамки считывания (ORF) гена ramA. В частности, подтвердили то, что штамм С13 имел вариант регулятора А метаболизма ацетата (ramA), у которого были введены две мутации в последовательность оснований, расположенную в 166-167 п.н. ниже кодона инициации гена ramA, приводя к изменению существующего CTG до GCG, и замена лейцина аланином, который представляет собой аминокислоту в положении 56 с N-конца.
В результате осуществления анализа мутантной области подтвердили то, что подвергался влиянию домен связывания эффектора белка регулятора А метаболизма ацетата, и ожидалось, что активность данного белка усиливалась бы.
Пример 3. Получение штамма KCCM11201P с введенной мутацией ramA и проверка способности к продуцированию валина
Пример 3-1. Получение штамма с введенной мутацией ramA из штамма Corynebacterium glutamicum KCCM11201P и оценка способности к продуцированию L-валина
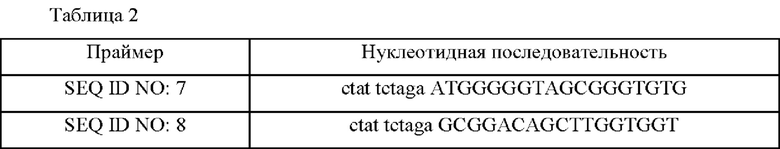
Для того чтобы вставить мутанта ramA(L56A), представленного SEQ ID NO: 1, в Corynebacterium glutamicum KCCM11201P, конструировали вектор, содержащий целевую мутацию. Описывая подробно, геномную ДНК штамма С13 экстрагировали с использованием мининабора для выделения общей ДНК G-spin (Intron, кат. №17045) согласно протоколу, предложенному в данном наборе, и проводили ПЦР с использованием геномной ДНК в качестве матрицы. ПЦР проводили при условиях денатурации при 94°С в течение 5 минут; с последующими 25 циклами денатурации при 94°С в течение 30 секунд, отжигом при 55°С в течение 30 секунд и полимеризацией при 72°С в течение 150 секунд; полимеризацией при 72°С в течение 7 минут, и получали ПЦР-продукт из 1000 п.н. (далее именуемый «фрагмент 1 с введенной мутацией») с использованием SEQ ID NO: 7 и SEQ ID NO: 8.
Фрагмент 1 с введенной мутацией, полученный выше, обрабатывали рестрикционным ферментом XbaI (New England Biolabs, Beverly, MA) и затем лигировали с вектором pDZ, обработанным таким же рестрикционным ферментом (корейский патент №100924065 и международная патентная публикация №2008033001) с использованием лигазы Т4 (New England Biolabs, Beverly, MA). Полученный ген трансформировали в Е. coli DH5α, которую отбирали в среде LB (Лурия-Бертани), содержащей канамицин, и получали ДНК с использованием набора для очистки плазмидной ДНК DNA-spin (iNtRON) с получением вектора pDZ-ramA, включающего фрагмент 1 с введенной мутацией (L56A).
Затем для того, чтобы далее ввести в Corynebacterium glutamicum KCCM11201P мутацию ramA(A52V), при которой аланин, который представляет собой аминокислоту в положении 52 гена ramA, связанного с известной способностью к продуцированию лизина, мутирован до валина (Metabolic Engineering, Volume 48, 2018, страницы 1-12, ISSN 1096-7176, https://doi.org/10.1016/j.ymben.2018.05.004.), получали вектор, включающий целевую мутацию. Описывая подробно, геномную ДНК штамма С13 выделяли с использованием мини-набора для выделения общей ДНК G-spin (Intron, кат. №17045) согласно протоколу, предложенному в данном наборе, и проводили ПЦР с использованием геномной ДНК в качестве матрицы. ПЦР проводили при условиях денатурации при 94°С в течение 5 минут; с последующими 25 циклами денатурации при 94°С в течение 30 секунд, отжигом при 55°С в течение 30 секунд и полимеризацией при 72°С в течение 150 секунд; полимеризацией при 72°С в течение 7 минут, и получали ПЦР-продукт из 502 п.н. (далее именуемый «фрагмент 2 с введенной мутацией») с использованием SEQ ID NO: 9 и SEQ ID NO: 10, и получали ПЦР-продукт из 500 п.н. (далее именуемый «фрагмент 3 с введенной мутацией») с использованием SEQ ID NO: 11 и SEQ ID NO: 12.
Фрагменты 2 и 3 с введенной мутацией, полученные выше, лигировали с вектором pDZ, обработанным XbaI (New England Biolabs, Beverly, MA) (корейский патент №100924065 и международная патентная публикация №2008033001) с использованием набора для клонирования infusion (Takara Bio Inc., Otsu, Japan) и затем трансформировали в Е. coli DH5α. Полученный ген трансформировали в Е. coli DH5α, затем осуществляли отбор в среде LB, содержащей канамицин, и получали ДНК с использованием набора для очистки плазмидной ДНК DNA-spin (iNtRON) с получением вектора pDZ-ramA(A52V+L56A), включающего фрагменты 2 и 3 с введенной мутацией.

Затем pDZ-ramA(L56A) трансформировали в Corynebacterium glutamicum KCCM11201P посредством гомологичной рекомбинации на хромосоме (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Штамм, в котором данный вектор был встроен в хромосому посредством рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Затем трансформанта Corynebacterium glutamicum, у которого была завершена вторичная рекомбинация, подвергали ПЦР с использованием SEQ ID NO: 7 и SEQ ID NO: 8 с идентификацией штамма, имеющего замену лейцина на аланин в положении 56 SEQ ID NO: 3 в ORF гена ramA на хромосоме. Данный рекомбинантный штамм называли Corynebacterium glutamicum KCCM11201P::ramA(L56A).
pDZ-ramA(A52V+L56A) трансформировали в Corynebacterium glutamicum KCCM11201P посредством гомологичной рекомбинации на хромосоме (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Штамм, в котором данный вектор был встроен в хромосому посредством рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Затем трансформанта Corynebacterium glutamicum, у которого была завершена вторичная рекомбинация, подвергали ПЦР с использованием SEQ ID NO: 9 и SEQ ID NO: 12 с идентификацией штамма, имеющего замену аланина на валин в положении 52 и замену лейцина на аланин в положении 56 SEQ ID NO: 3 в ORF гена ramA на хромосоме. Данный рекомбинантный штамм называли Corynebacterium glutamicum KCCM11201P::ramA(A52V+L56A).
Проводили анализ в колбе для сравнения способности к продуцированию валина между штаммами, продуцирующими валин: Corynebacterium glutamicum KCCM11201P, KCCM11201P::ramA(L56A) и KCCM11201P::ramA(A52V+L56A). Каждый штамм субкультивировали в питательной среде и затем инокулировали в 250 мл колбу с угловыми перегородками, содержащую 25 мл продукционной среды, и культивировали при 30°С в течение 72 часов со встряхиванием при 200 об/мин соответственно.
Питательная среда (рН 7,2)
10 г глюкозы, 5 г мясного экстракта, 10 г полипептона, 2,5 г хлорида натрия, 5 г дрожжевого экстракта, 20 г агара, 2 г мочевины (на основе 1 л дистиллированной воды). Продукционная среда (рН 7,0)
100 г глюкозы, 40 г сульфата аммония, 2,5 г соевого белка, 5 г твердых веществ кукурузного экстракта, 3 г мочевины, 1 г двухосновного фосфата калия, 0,5 г сульфата магния гептагидрата, 100 мкг биотина, 1 мг тиамин-HCl, 2 мг пантотената кальция, 3 мг никотинамида, 30 г карбоната кальция (на основе 1 л дистиллированной воды).
После завершения культивирования измеряли способность к продуцированию L-валина с использованием ВЭЖХ (высокоэффективная жидкостная хроматография). Концентрации L-валина, проанализированного таким образом, показаны в Таблице 4 ниже.
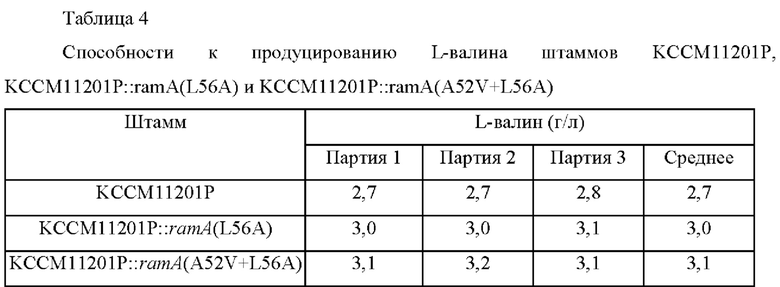
Как показано в Таблице 4, подтвердили то, что штаммы KCCM11201P::ramA(L56A) и KCCM11201P::ramA(A52V+L56A), в которые был введен каждый вариант, демонстрировали 11%-ное и 14,6%-ное увеличение способности к продуцированию L-валина, соответственно, по сравнению со штаммом дикого типа KCCM11201P, который представляет собой родительский штамм.
Пример 3-2: получение штамма с введением мутации RamA из штамма Corynebacterium glutamicum CJ7V и оценка способности к продуцированию L-валина
Для того, чтобы проверить эффект увеличения способности к продуцированию L-валина у штаммов, принадлежащих к другому продуцирующему L-валин Corynebacterium glutamicum, в Corynebacterium glutamicum ATCC14067 дикого типа вводили вид мутации (ilvN(A42V); Biotechnology and Bioprocess Engineering, June 2014, Volume 19, Issue 3, pp.456-467) с получением штамма, имеющего улучшенную способность к продуцированию L-валина.
Описывая подробно, геномную ДНК штамма Corynebacterium glutamicum ATCC 14067 дикого типа выделяли с использованием мининабора для выделения общей ДНК G-spin (Intron, кат. №17045) согласно протоколу, предложенному в данном наборе. Проводили ПЦР с использованием геномной ДНК в качестве матрицы. Для конструирования вектора для введения мутации A42V в ген ilvN получали фрагменты гена (А, В) с использованием пары праймеров SEQ ID NO: 13 и SEQ ID NO: 14, и пары праймеров SEQ ID NO: 15 и SEQ ID NO: 16 соответственно. ПЦР проводили при условиях денатурации при 94°С в течение 5 минут; с последующими 25 циклами денатурации при 94°С в течение 30 секунд, отжигом при 55°С в течение 30 секунд и полимеризацией при 72°С в течение 60 секунд; полимеризацией при 72°С в течение 7 минут.
В результате получали оба фрагмента А и В в виде полинуклеотидов из 537 п.н. Проводили ПЦР с перекрывающимися праймерами с использованием данных двух фрагментов в качестве матрицы и SEQ ID NO: 13, и SEQ ID NO: 16 с получением ПЦР-продукта из 1044 п. н. (далее именуется «фрагмент 2 с введенной мутацией»).
Фрагмент 2 с введенной мутацией, полученный выше, обрабатывали рестрикционным ферментом XbaI (New England Biolabs, Beverly, MA) и затем лигировали с вектором pDZ, обработанным таким же рестрикционным ферментом, с использованием лигазы Т4 (New England Biolabs, Beverly, MA). Полученный ген трансформировали в Е. coli DH5α, которую отбирали в среде LB, содержащей канамицин, и получали ДНК с использованием набора для очистки плазмидной ДНК DNA-spin (iNtRON). Вектор для введения мутации A42V гена ilvN называли pDZ-ilvN(A42V).

Затем pDZ-ilvN(A42V) трансформировали в Corynebacterium glutamicum ATCC 14067 дикого типа посредством гомологичной рекомбинации на хромосоме (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Штамм, в котором данный вектор был встроен в хромосому посредством рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Затем трансформанта Corynebacterium glutamicum, у которого была завершена вторичная рекомбинация, подвергали ПЦР с использованием SEQ ID NO: 13 и SEQ ID NO: 16 для амплификации данного фрагмента гена, и затем штамм с введенной мутацией идентифицировали посредством генетического анализа последовательности. Данный рекомбинантный штамм называли Corynebacterium glutamicum CJ7V.
Затем Corynebacterium glutamicum CJ7V трансформировали каждым вектором таким же способом, как и в Примере 3-1, с получением штаммов, которые называли Corynebacterium glutamicum CJ7V::ramA(L56A), CJ7V::ramA(A52V+L56A) соответственно. Для сравнения способности к продуцированию L-валина между полученными штаммами данные штаммы культивировали таким же способом, как и в Примере 3-1, и анализировали их концентрации L-валина. Концентрации L-валина, проанализированные таким образом, показаны в Таблице 6 ниже.
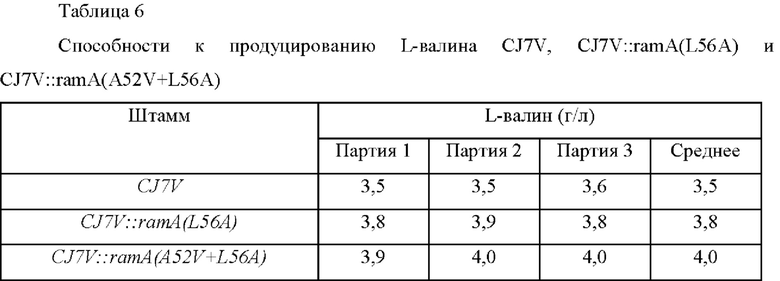
Как показано в Таблице 6, подтвердили то, что штаммы CJ7V::ramA(L56A) и CJ7V::ramA(A52V+L56A), в которые был введен каждый вариант, демонстрировали 8,5%-ное и 12,3%-ное увеличение способности к продуцированию L-валина, соответственно, по сравнению со штаммом CJ7V.
Пример 4. Получение штамма с введенной мутацией ramA из штамма Corynebacterium glutamicum KCCM11248P, имеющего способность к продуцированию L-изолейцина, и оценка способности к продуцированию L-изолейцина
Для того, чтобы проверить эффект увеличения способности к продуцированию L-изолейцина у штамма Corynebacterium glutamicum, имеющего способность к продуцированию L-изолейцина, получали штамм, в котором мутация ramA была введена в продуцирующий L-изолейцин штамм Corynebacterium glutamicum KCCM11248P (корейский патент №101335789).
Описывая подробно, вектор pDZ-ramA(L56A), полученный в Примере 3-1, трансформировали в Corynebacterium glutamicum KCCM11248P посредством гомологичной рекомбинации на хромосоме (van der Rest et al., Appl Microbiol Biotechnol 52:541 545, 1999). Штамм, в котором данный вектор был встроен в хромосому посредством рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Затем трансформанта Corynebacterium glutamicum, у которого была завершена вторичная рекомбинация, подвергали ПЦР с использованием SEQ ID NO: 7 и SEQ ID NO: 8 для идентификации штамма, имеющего замену лейцина на аланин в положении 56 SEQ ID NO: 3 в ORF (открытая рамка считывания) гена ramA на хромосоме. Данный рекомбинантный штамм называли Corynebacterium glutamicum KCCM11248P::ramA(L56A).
Вектор pDZ-ramA(A52V+L56A), полученный в Примере 3-1, трансформировали в Corynebacterium glutamicum KCCM11248P посредством гомологичной рекомбинации на хромосоме (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Штамм, в котором вектор был встроен в хромосому посредством рекомбинации гомологичной последовательности, отбирали в среде, содержащей 25 мг/л канамицина. Затем трансформанта Corynebacterium glutamicum, у которого была завершена вторичная рекомбинация, подвергали ПЦР с использованием SEQ ID NO: 9 и SEQ ID NO: 12 для идентификации штамма, имеющего замену аланина на валин в положении 52 и замену лейцина на аланин в положении 56 SEQ ID NO: 3 в ORF гена ramA на хромосоме. Данный рекомбинантный штамм называли Corynebacterium glutamicum KCCM11248P::ramA(A52V+L56A).
Проводили анализ в колбе для сравнения способности к продуцированию изолейцина между штаммами, продуцирующими изолейцин: Corynebacterium glutamicum KCCM11248P, KCCM11248P::ramA(L56A) и KCCM11248P::ramA(A52V+L56A). Каждый штамм инокулировали в 250 мл колбу с угловыми перегородками, содержащую 25 мл продукционной среды изолейцина, и культивировали при 32°С в течение 60 часов со встряхиванием при 200 об/мин соответственно.
Продукционная среда (рН 7,2)
100 г глюкозы, 2 г дрожжевого экстракта, 16 г сульфата аммония, 1 г одноосновного фосфата калия, 1 г сульфата магния гептагидрата, 10 мг сульфата железа гептагидрата, 10 мг сульфата марганца моногидрата, 200 мкг биотина (на основе 1 л дистиллированной воды).
После завершения культивирования измеряли способность к продуцированию L-изолейцина с использованием ВЭЖХ. Концентрации L-изолейцина, проанализированного таким образом, показаны в Таблице 7 ниже.
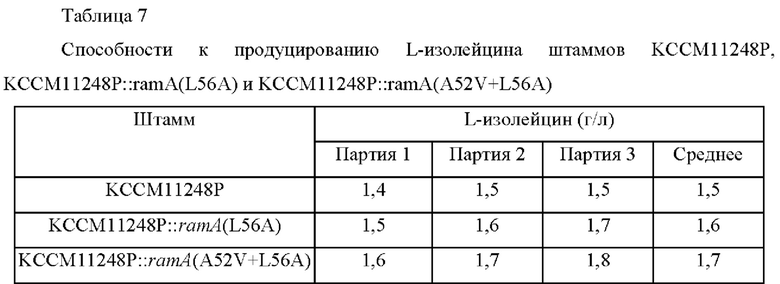
Как показано в Таблице 7, подтвердили то, что штаммы KCCM11248P::ramA(L56A) и KCCM11248P::ramA(A52V+L56A), в которые был введен каждый вариант, демонстрировали 6,8%-ное и 13,6%-ное увеличение способности к продуцированию L-изолейцина, соответственно, по сравнению с родительским штаммом KCCM11248P.
На основе приведенного выше описания специалистам в данной области будет понятно то, что настоящее раскрытие может осуществляться в разной конкретной форме без изменения его технической сущности или существенных характеристик. В данном отношении следует понимать то, что приведенное выше воплощение не является ограничивающим, но во всех аспектах иллюстративным. Объем данного раскрытия определяется приложенной формулой изобретения, а не предшествующим ей описанием, и, следовательно, подразумевается, что все изменения и модификации, которые попадают в пределы данной формулы изобретения, или эквиваленты таких пределов, следовательно, охватываются данной формулой изобретения.
Эффект изобретения
При культивировании микроорганизма, включающего вариант регулятора А метаболизма ацетата по настоящему раскрытию возможно продуцировать с высоким выходом L-аминокислоты с разветвленной цепью по сравнению с существующим микроорганизмом, имеющим немодифицированный полипептид.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="P23871RU-st26-RU.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2024-04-18">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/010784</ApplicationNumberText>
<FilingDate>2022-07-22</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>P23871RU</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0114069</ApplicationNumberText>
<FilingDate>2021-08-27</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">СИДЖЕЙ ЧЕИЛДЖЕДАНГ КОРПОРЕЙШН
</ApplicantName>
<ApplicantNameLatin>CJ CHEILJEDANG CORPORATION</ApplicantNameLatin>
<InventionTitle languageCode="ru">Новый вариант регулятора А
метаболизма ацетата и способ продуцирования L-аминокислоты с
разветвленной цепью с его применением</InventionTitle>
<SequenceTotalQuantity>16</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>281</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..281</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>regulators of acetate metabolism A
variant (L56A)</INSDQualifier_value>
<NonEnglishQualifier_value>вариант регулятора А метаболизма
ацетата (L56A)</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..281</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VDTQRIKDDEDAIRSALTSLKTATGIPVTMFATVLQDNRLQITQWVGLR
TPALQNAVIEPGVGVGGRVVATRRPVGVSDYTRANVISHEKDSAIQDEGLHSIVAVPVIVHREIRGVLYV
GVHSAVRLGDTVIEEVTMTARTLEQNLAINSALRRNGVPDGRGSLKASRVMNGAEWEQVRSTHSKLRMLA
NRVTDEDLRRDLEELCDQMVTPVRIKQTTKLSARELDVLACVALGHTNVEAAEEMGIGAETVKSYLRSVM
RKLGAHTRYEAVNAARRIGALP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>846</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..846</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>regulators of acetate metabolism A
variant (L56A)</INSDQualifier_value>
<NonEnglishQualifier_value>вариант регулятора А метаболизма
ацетата (L56A)</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..846</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggatacccagcggattaaagatgacgaagatgctattcgttcggcgc
tgacatcgctgaaaaccgcaacaggcatcccagtcaccatgttcgccactgtgttgcaggacaatcgcct
gcaaattactcagtgggttgggttgcgtaccccggctctgcagaatgcggtcattgaaccaggtgtgggc
gttggtggacgcgtcgtcgcaacccgtcgtccggttggtgtgagtgattacaccagggcaaatgtcattt
cacatgagaaggattccgcgattcaggatgagggccttcattccattgtcgcagttcccgtgatcgtgca
ccgcgaaatccgtggcgttttgtatgttggcgttcactctgcggtgcgtctcggcgacactgttattgaa
gaagtcaccatgactgcgcgcacgttggaacaaaacctggcgatcaactccgcgcttcgccgcaatggcg
ttcctgatggtcgcggttccctcaaagctagccgcgtgatgaatggggcggagtgggagcaggttcgttc
cactcattccaagctgcgcatgctggcaaatcgtgtgaccgatgaggatctgcgccgcgatttggaagag
ctttgcgatcagatggtcaccccagtccgcatcaagcagaccaccaagctgtccgcgcgtgagttggacg
tgctggcttgtgtcgcgctcggtcacaccaacgtcgaagctgctgaagagatgggcatcggcgcggaaac
cgtcaagagctacctgcgctcggtcatgcgcaagctcggcgcccacacgcgctacgaggcagtcaacgca
gcacgccggatcggcgcactgccttaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>281</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..281</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>regulators of acetate metabolism
A</INSDQualifier_value>
<NonEnglishQualifier_value>регулятор А метаболизма
ацетата</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..281</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
<NonEnglishQualifier_value>неопределено</NonEnglishQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VDTQRIKDDEDAIRSALTSLKTATGIPVTMFATVLQDNRLQITQWVGLR
TPALQNLVIEPGVGVGGRVVATRRPVGVSDYTRANVISHEKDSAIQDEGLHSIVAVPVIVHREIRGVLYV
GVHSAVRLGDTVIEEVTMTARTLEQNLAINSALRRNGVPDGRGSLKASRVMNGAEWEQVRSTHSKLRMLA
NRVTDEDLRRDLEELCDQMVTPVRIKQTTKLSARELDVLACVALGHTNVEAAEEMGIGAETVKSYLRSVM
RKLGAHTRYEAVNAARRIGALP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>846</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..846</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>regulators of acetate metabolism
A</INSDQualifier_value>
<NonEnglishQualifier_value>регулятор А метаболизма
ацетата</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..846</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
<NonEnglishQualifier_value>неопределено</NonEnglishQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggatacccagcggattaaagatgacgaagatgctattcgttcggcgc
tgacatcgctgaaaaccgcaacaggcatcccagtcaccatgttcgccactgtgttgcaggacaatcgcct
gcaaattactcagtgggttgggttgcgtaccccggctctgcagaatctggtcattgaaccaggtgtgggc
gttggtggacgcgtcgtcgcaacccgtcgtccggttggtgtgagtgattacaccagggcaaatgtcattt
cacatgagaaggattccgcgattcaggatgagggccttcattccattgtcgcagttcccgtgatcgtgca
ccgcgaaatccgtggcgttttgtatgttggcgttcactctgcggtgcgtctcggcgacactgttattgaa
gaagtcaccatgactgcgcgcacgttggaacaaaacctggcgatcaactccgcgcttcgccgcaatggcg
ttcctgatggtcgcggttccctcaaagctagccgcgtgatgaatggggcggagtgggagcaggttcgttc
cactcattccaagctgcgcatgctggcaaatcgtgtgaccgatgaggatctgcgccgcgatttggaagag
ctttgcgatcagatggtcaccccagtccgcatcaagcagaccaccaagctgtccgcgcgtgagttggacg
tgctggcttgtgtcgcgctcggtcacaccaacgtcgaagctgctgaagagatgggcatcggcgcggaaac
cgtcaagagctacctgcgctcggtcatgcgcaagctcggcgcccacacgcgctacgaggcagtcaacgca
gcacgccggatcggcgcactgccttaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>281</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..281</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>regulators of acetate metabolism A
variant (L56A+A52V)</INSDQualifier_value>
<NonEnglishQualifier_value>вариант регулятора А метаболизма
ацетата (L56A+A52V)</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..281</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VDTQRIKDDEDAIRSALTSLKTATGIPVTMFATVLQDNRLQITQWVGLR
TPVLQNAVIEPGVGVGGRVVATRRPVGVSDYTRANVISHEKDSAIQDEGLHSIVAVPVIVHREIRGVLYV
GVHSAVRLGDTVIEEVTMTARTLEQNLAINSALRRNGVPDGRGSLKASRVMNGAEWEQVRSTHSKLRMLA
NRVTDEDLRRDLEELCDQMVTPVRIKQTTKLSARELDVLACVALGHTNVEAAEEMGIGAETVKSYLRSVM
RKLGAHTRYEAVNAARRIGALP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>846</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..846</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>regulators of acetate metabolism A
variant (L56A+A52V)</INSDQualifier_value>
<NonEnglishQualifier_value>вариант регулятора А метаболизма
ацетата (L56A+A52V)</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..846</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggatacccagcggattaaagatgacgaagatgctattcgttcggcgc
tgacatcgctgaaaaccgcaacaggcatcccagtcaccatgttcgccactgtgttgcaggacaatcgcct
gcaaattactcagtgggttgggttgcgtaccccggttctgcagaatgcggtcattgaaccaggtgtgggc
gttggtggacgcgtcgtcgcaacccgtcgtccggttggtgtgagtgattacaccagggcaaatgtcattt
cacatgagaaggattccgcgattcaggatgagggccttcattccattgtcgcagttcccgtgatcgtgca
ccgcgaaatccgtggcgttttgtatgttggcgttcactctgcggtgcgtctcggcgacactgttattgaa
gaagtcaccatgactgcgcgcacgttggaacaaaacctggcgatcaactccgcgcttcgccgcaatggcg
ttcctgatggtcgcggttccctcaaagctagccgcgtgatgaatggggcggagtgggagcaggttcgttc
cactcattccaagctgcgcatgctggcaaatcgtgtgaccgatgaggatctgcgccgcgatttggaagag
ctttgcgatcagatggtcaccccagtccgcatcaagcagaccaccaagctgtccgcgcgtgagttggacg
tgctggcttgtgtcgcgctcggtcacaccaacgtcgaagctgctgaagagatgggcatcggcgcggaaac
cgtcaagagctacctgcgctcggtcatgcgcaagctcggcgcccacacgcgctacgaggcagtcaacgca
gcacgccggatcggcgcactgccttaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctattctagaatgggggtagcgggtgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctattctagagcggacagcttggtggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggggatcctctagactaataccgcaaatgg</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaccagattctgcagaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttctgcagaatctggtca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggtcgactctagaggtggtctgcttgatg</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctagaggcagaccctattctatgaagg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtgtttcggtctttacagacacgagggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer odhA_down_F</INSDQualifier_value>
<NonEnglishQualifier_value>праймер
odhA_down_F</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtccctcgtgtctgtaaagaccgaaacact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>primer</INSDQualifier_value>
<NonEnglishQualifier_value>праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatttctagacgtgggagtgtcactcgcttgg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии и представляет собой вариант регулятора А метаболизма ацетата, обладающий активностью регулятора А метаболизма ацетата, в котором аминокислота, соответствующая положению 56 в аминокислотной последовательности SEQ ID NO: 3, заменена другой аминокислотой или в котором аминокислота, соответствующая положению 56, и аминокислота, соответствующая положению 52 в аминокислотной последовательности SEQ ID NO: 3, заменены другой аминокислотой. Изобретение относится также к способу продуцирования L-аминокислот с разветвленной цепью, включающему стадию культивирования микроорганизма, содержащего указанный вариант регулятора А метаболизма ацетата. Изобретение позволяет получать L-аминокислоты с разветвленной цепью с высокой степенью эффективности. 5 н. и 8 з.п. ф-лы, 7 табл., 3 пр.
1. Вариант регулятора А метаболизма ацетата, обладающий активностью регулятора А метаболизма ацетата, в котором аминокислота, соответствующая положению 56 в аминокислотной последовательности SEQ ID NO: 3, заменена другой аминокислотой.
2. Вариант регулятора А метаболизма ацетата, обладающий активностью регулятора А метаболизма ацетата, в котором аминокислота, соответствующая положению 56, и аминокислота, соответствующая положению 52 в аминокислотной последовательности SEQ ID NO: 3, заменены другой аминокислотой.
3. Вариант по п. 1, в котором аминокислота, соответствующая положению 56 SEQ ID NO: 3, заменена на аланин.
4. Вариант по п. 2, в котором аминокислота, соответствующая положению 56, и аминокислота, соответствующая положению 52 SEQ ID NO: 3, заменены на аланин и валин соответственно.
5. Вариант по п. 1, состоящий из аминокислотной последовательности, представленной SEQ ID NO: 1.
6. Вариант по п. 2, состоящий из аминокислотной последовательности, представленной SEQ ID NO: 5.
7. Полинуклеотид, кодирующий вариант по любому из пп. 1-6.
8. Микроорганизм для продуцирования L-аминокислот с разветвленной цепью, содержащий вариант по любому из пп. 1-6 или полинуклеотид, кодирующий указанный вариант.
9. Микроорганизм для продуцирования L-аминокислот с разветвленной цепью по п. 8, имеющий повышенную способность к продуцированию L-аминокислот с разветвленной цепью по сравнению с микроорганизмом, включающим полипептид SEQ ID NO: 3 или кодирующий его полинуклеотид.
10. Микроорганизм для продуцирования L-аминокислот с разветвленной цепью по п. 8, представляющий собой микроорганизм рода Corynebacterium.
11. Микроорганизм для продуцирования L-аминокислот с разветвленной цепью по п. 10, где указанный микроорганизм рода Corynebacterium представляет собой Corynebacterium glutamicum.
12. Способ продуцирования L-аминокислот с разветвленной цепью, включающий стадию культивирования микроорганизма по п. 8 в среде.
13. Способ по п. 12, дополнительно включающий стадию выделения целевого вещества из среды.
| US 7332310 B2, 19.02.2008 | |||
| KR 101117022 B1, 16.03.2012 | |||
| KR 101335789 B1, 02.12.2013 | |||
| Микроорганизм рода Corynebacterium, обладающий L-изолейцин-продуцирующей способностью, и способ получения L-изолейцина с использованием этого микроорганизма | 2016 |
|
RU2698394C1 |
Авторы
Даты
2025-04-10—Публикация
2022-07-22—Подача