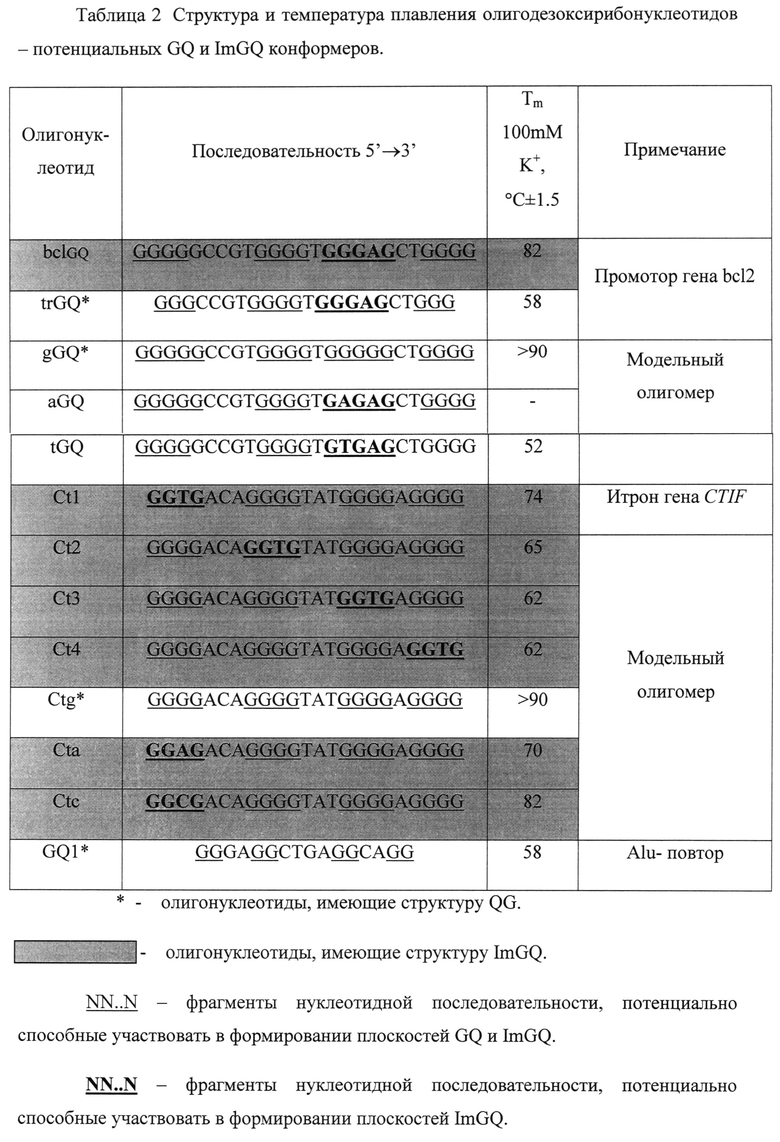
Область техники, к которой относится изобретение
Предлагаемое изобретение относится к области геномики и структурной биологии и может быть использовано для анализа геномных текстов, исследования структурно-функциональных свойств и механизмов функционирования ДНК и РНК. Прогнозирование существования новых неканонических третичных структур полинуклеотидов, а именно несовершенных G-квадруплексов (ImGQ), потенциально стабильных в физиологических условиях, позволит также найти точные молекулярные мишени и биомаркеры патологических состояний человека, разработать новые лекарства направленного действия. Предметом настоящего патента являются алгоритм поиска ImGQ, потенциально стабильных в физиологических условиях (1), и способ получения олигонуклеотидов, способных формировать ImGQ (2).
Уровень технического состояния
В последние годы показано, что в составе нативной ДНК кроме превалирующей B-формы существуют другие неканонические пространственные структуры (конформеры, ncDNA). Такие сайты рассматривают как структурно-опосредованные регуляторы работы геномов, например, регуляторы транскрипции генов [1]. Среди известных ncDNA можно назвать G-квадруплексы (GQ) и I-мотивы [2, 3], триплексы и фрагменты параллельных дуплексов [4-6]. Наиболее изучены из ncDNA G-квадруплексы.
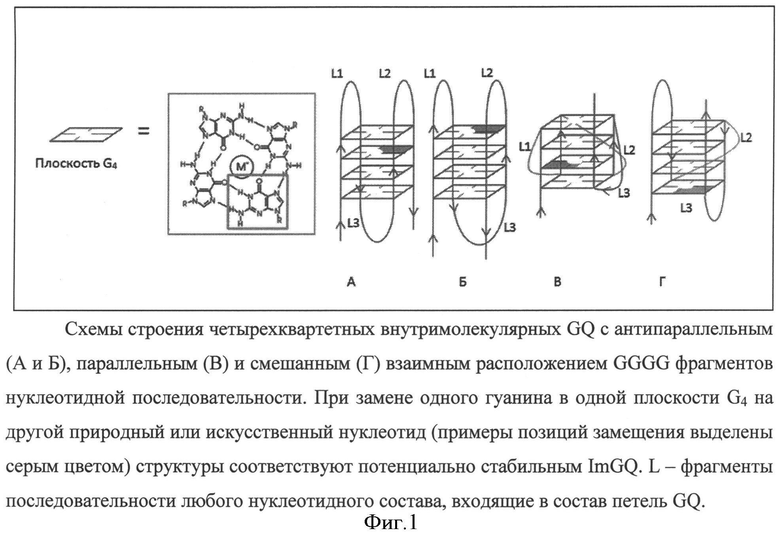
Внутримолекулярный G-квадруплекс (англ. G-quadruplex, а также G-tetrads, GQ) - олигонуклеотид или фрагмент полинуклеиновой кислоты, способный образовать четырехцепочечную спираль, стабилизированную взаимодействием четырех гуаниновых оснований. Каждый G-квартет скреплен в сумме восемью водородными связями и образует плоскую структуру (Фигура 1).
GQ найдены in vivo в промоторах генов человека c-kit, c-myc, hTERT и др. [7-12]. Последовательности нуклеотидов, способные формировать QG, широко представлены в нетранслируемой 5'-области многих генов и могут принимать непосредственное участие в регуляции их экспрессии [13]. К образованию GQ склонны также многие микро- и минисателлитные повторы генома человека, например, теломерный повтор (TTAGGG)n или сателлит (GGGTCT)n. Показано, что мутации в области GQ лежат в основе многих заболеваний [14]. В настоящее время GQ-сайты полинуклеотидов рассматриваются как перспективные терапевтические мишени и в онкологии [15].
Известно, что для образования GQ необходимо, чтобы фрагмент полинуклеотида соответствовал определенным требованиям. Последовательность, потенциально способная к формированию GQ, должна соответствовать следующей формуле (GxLyi)Gx, где x=2, 3, 4… - количество G-квартетов GQ; y=1, 2, 3… - количество нуклеотидов в i-той петле, соединяющей G4 плоскости.
Для поиска PQS (Potential Quadruplex Sequence) во фрагментах полинуклеотидов и геномных текстах был разработан алгоритм [5, 16] (ближайший прототип) и созданы доступные онлайн программы, например, QGRS (http://bioinformatics.ramapo.edu/QGRS/analyze.php) и quadfinder (http://miracle.igib.res.in/quadfinder/).
Для подтверждения существования каждого найденного GQ необходимо получить соответствующий олигонуклеотид и определить его конформацию инструментальными и физико-химическими методами.
В настоящей заявке мы впервые доказываем, что G-квадруплексы не описывают все конформационные состояния полинуклеотидов, стабилизированных стекинг взаимодействием четырех гуаниновых оснований. Несовершенный G-квадруплекс (англ. ImG-quadruplex, а также ImG-tetrads, ImGQ) - олигонуклеотид или фрагмент полинуклеиновой кислоты, сходной с GQ последовательностью, но несущий, в отличие от GQ, одну замену квартет-образующего гуанина на другой нуклеотид или его аналог (Фигура 1А-Г), также может иметь стабильную конформацию в естественных условиях.
Ни один из известных алгоритмов анализа первичных последовательностей ДНК по признаку образования потенциальных неканонических конформаций не учитывает правила формирования несовершенных квадруплексов, а поисковые программы, созданные на их основе, не обеспечивают выявление сайтов, способных формировать ImGQ.
Раскрытие изобретения и технический результат
Техническим результатом настоящего изобретения является создание нового инструмента биоинформационного анализа, позволяющего выявлять в первичных последовательностях полинуклеотидов сайты, способные формировать потенциально стабильную в физиологических условиях новую конформацию - несовершенные G-квадруплексы (ImGQ). Для его достижения необходимо было разработать способ получения ImGQ-олигонуклеотидов природных и модельных последовательностей и на их примерах доказать существование ImGQ и подтвердить их высокую стабильность (см. данные в таблице 2).
Технический результат достигается также тем, что на основании исследования свойств олигонуклеотидов и анализа литературных данных выработаны правила формирования ImGQ (алгоритм).
Предлагаемый алгоритм поиска ImGQ предусматривает описание последовательностей потенциальных несовершенных квадруплексов набором формул, которые могут быть представлены в виде таблицы (Таблица 1) форматом k×4, где n (число строк) соответствует числу квартетов, образованных четырьмя последовательными фрагментами сайта и не может быть меньше 4 (k≥4). Сумма элементов таблицы описывает все мононесовершенные ImGQ, способные принять конформацию ImGQ в физиологических и близких к физиологическим условиях (например, в 50-100 mM K+Cl, 10 mМ TrisHCl, pH 7-8).
Важно отметить, что в данном подходе описанные ранее GQ (совершенные GQ) становятся частным случаем ImGQ, реализующимся при N=G.
Для обоснования предложенного алгоритма, направленного на поиск стабильных конформеров, необходимо пояснить ряд ключевых позиций.
1. Рассматривается возможность формирования только внутримолекулярных GQ.
В условиях in vivo концентрация полинуклеотидов относительно невелика, кроме того молекулы ДНК и РНК входят в состав сложных супрамолекулярных комплексов с различными биополимерами и низкомолекулярными соединениями, что в большинстве случаев препятствует образованию межмолекулярных GQ.
2. ImGQ, стабильные в физиологических условиях, способны формироваться в случае четырех и более квартетных GQ, несущих единичные замены в G4 плоскостях.
Известно, что нуклеотидные замены в G4 плоскостях и даже изменение природы химической связи между гуанинами приводят к дестабилизации двух и трехквартетных GQ [16-19]. Пример 1 иллюстрирует влияние на стабильность четырехквартетных GQ - gGQ (sequence 1) и Ctg (sequence 2) (Таблица 2) - одно- и двух-нуклеотидных замен G на другие нуклеотиды в квадруплекс-образующих блоках GGGG. Из приведенных в Таблице 2 данных видно, что единичные замены приводят к незначительному снижению температуры плавления GQ (до 82°C, для bclGQ (sequence 3) и до 62-82°C для олигонуклеотидов серии Ct). Отметим, что стабильность структур этих ImGQ остается высокой и значительно превышает физиологические нормы.
Замена двух гуанинов в G4-плоскости приводит к снижению стабильности третичной структуры олигонукелотида и размыванию спектров КД (Таблица 1. Фигура 2А, олигомеры aGQ (G/A, G/A) (sequence 4) и tGQ (G/A,G/T) (sequence 5)). Так, например, температура плавления tGQ (sequence 5) не превышает 52°C, следовательно, он не может быть достоверно отнесен к стабильным в физиологических условиях GQ, а олигомер aGQ (sequence 4) образует межмолекулярные структуры.
О формировании четырехквартетного высокостабильного несовершенного квадруплекса свидетельствует и тот факт, что термостабильность ImQG bclGQ (G/A) (sequence 3) - природной последовательности из промоторной области гена bcl2 - значительно (на 27°C) превышает стабильность трехквартетного гомолога trGQ (sequence 6), образование которого предсказывало использование прежнего алгоритма поиска QG (например, по программе quadfinder, основанной на известном алгоритме [16]).
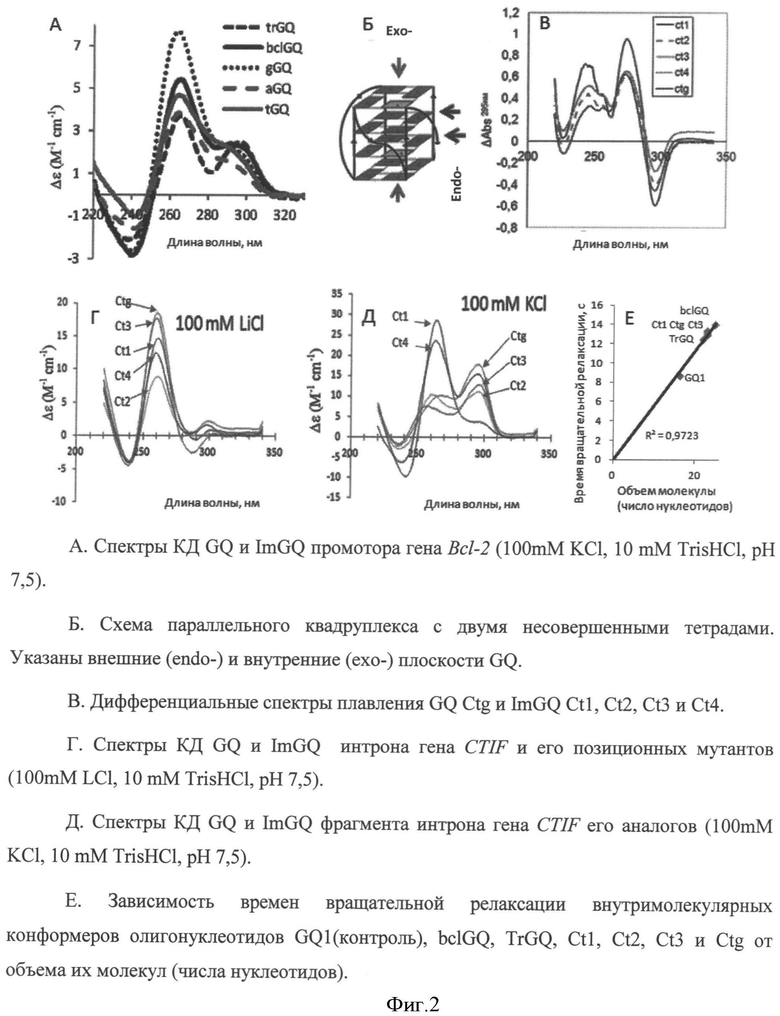
Характер КД-спектров ImGQ (Фигура 2А и Д) в присутствии ионов калия, стабилизирующих GQ, и дифференциальных кривых плавления ImGQ (Фигура 2В) типичен для GQ. Причем, ImGQ так же, как и совершенные квадруплексы (в данном примере Ctg (sequence 2)) теряют стабильность конформации в присутствии ионов лития (Фигура 2Г).
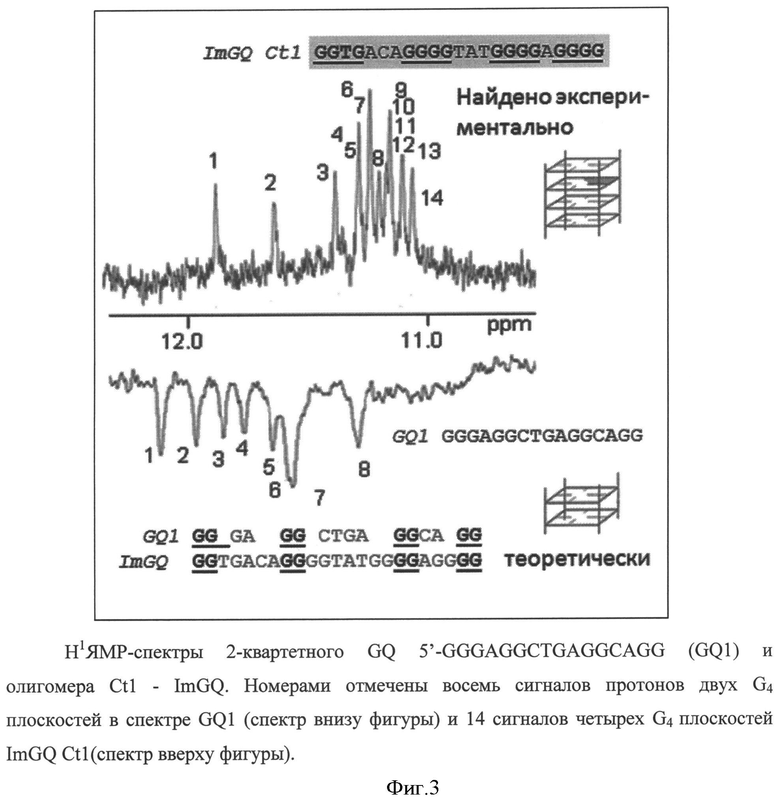
Надежным доказательством формирования ImGQ служит сравнение спектров ПМР (в области 12 ppm) природного ImQG Ct1 (sequence 7) и спектра олигонуклеотида GQ1 (sequence 8). Обе последовательности в соответствии с известным алгоритмом поиска PQS способны к образованию 2-тетрадных GQ, поскольку максимальная длина четырех G-блоков в их составе равна двум. Это означает, что в области Хугстиновских взаимодействий (область 12 ppm) должны наблюдаться 8 сигналов, что и соответствует спектру GQ1 (sequence 8) (Фигура 3, нижняя часть). Однако, в спектре олигомера Ct1 (sequence 7) (Фигура 3, верхний спектр) обнаруживается не 8, а 14 сигналов протонов, задействованных в образовании плоскостей ImGQ структуры. Этот факт хорошо описывает четырехтетрадная схема структурообразующего ImGQ (Фигура 3).
Совокупность приведенных аргументов позволяет сделать вывод о том, что новый алгоритм поиска высокостабильных ImGQ должен относиться к четырех- и более квартетным структурам ImGQ и верен для n≥4.
Длина петель ImQG по аналогии с GQ рассматривается в пределах от 1 до 7 нуклеотидов [20]. В состав петель может входить любой нуклеотид.
Интервал 1-7 не является обязательным и может быть расширен. Однако, ограничение длины петель до 7 звеньев увеличивает вероятность выявления наиболее стабильных в физиологических условиях GQ [20, 21], эта закономерность может быть отнесена и к ImGQ.
3. Алгоритм предусматривает проведение поиска потенциальных ImGQ, способных существовать одновременно, т.е., например, блок GGGTGG при поиске четырехквартетных ImGQ учитывается как один, а не как три фрагмента разных GQ, полученных в результате альтернативного фолдинга.
С учетом приведенных выше правил была разработана компьютерная программа для поиска ImGQ в геномных нуклеотидных данных (Таблица 1), названная ImGQfinder (дата подачи заявки на госрегистрацию: 06.09.2012). С ее помощью можно анализировать большие объемы данных, получать список последовательностей ImGQ с указанием положения в геноме, статистику вхождений дефектов (N) и другие результаты. Программа написана на С#. Выходной формат может быть изменен.
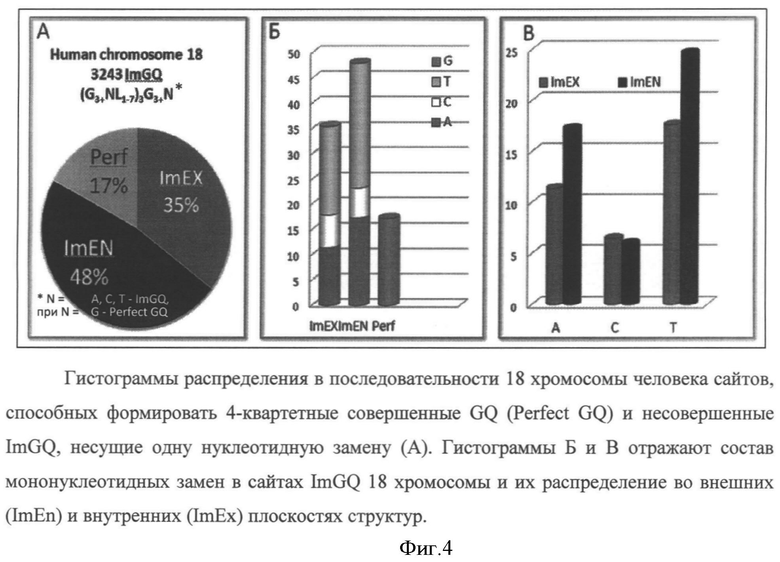
В результате анализа первичной нуклеотидной последовательностей прямой и обратной цепей 18 хромосомы человека (http://www.ncbi.nlm.nih.gov/nuccore/NC_000018.9) с помощью описываемой программы ImGQfinder были выявлены 3243 четырехтетрадные (для n=4) ImGQ. Причем, только 17% из них является перфектными GQ (N=G) и могли быть выявлены с помощью известных ранее алгоритмов (Пример 2. Фигура 4А). Таким образом, анализ последовательности только одной хромосомы позволил выявить более 2,5 тысяч новых потенциально высокостабильных неканонических структур. Анализ найденных последовательностей ImGQ показал, что в природе замены могут встречаться как во внутренних (ImEn, 48%), так и во внешних (ImEx, 35%) плоскостях структуры. Причем замещающий G нуклеотид N наиболее часто представлен Т и А и редко (менее 13%) С (Фигура 4А-В).
Для подтверждения формирования неканонических структур ImGQ и исследования их термостабильности в физиологических условиях были синтезированы олигонуклеотиды bclGQ (sequence 3) и Ct1 (sequence 7). Последовательности олигомеров соответствуют природным сайтам, найденным с помощью программы ImGQfinder, промоторной области гена bcl2 (одного из ключевых генов онкогенеза) и интрона гена CTIF (компонент СВР80/СВР20 комплекса инициации трансляции [22]). Результаты исследований подтвердили, что рассматриваемые олигомеры действительно формируют высокостабильные (Tплавления >70°C), стерически-затрудненные неканонические структуры ImGQ (Примеры 1.1-1.5. Фигуры 2 и 3).
На основе найденных правил формирования ImGQ были сконструированы и получены олигомеры, моделирующие различное положение и природу мононуклеотидных замен в составе искусственных ImGQ (Таблица 2). Исследования показали, что во всех случаях наблюдалось формирование стабильных ImGQ (Пример 1, Таблица 2, Фигуры 2-3), что может служить надежным подтверждением адекватности разработанных правил формирования ImGQ и программы ImGQfinder.
Способ поиска и прогнозирование формирования новых неканонических третичных структур полинуклеотидов, а именно несовершенных G-квадруплексов, потенциально стабильных в физиологических условиях, позволит найти новые молекулярные мишени и биомаркеры патологических состояний человека, разработать на этой основе новые лекарства направленного действия.
Описание чертежей
На Фигуре 1 приведены схемы строения четырехквартетных внутримолекулярных GQ с антипараллельным (А и Б), параллельным (В) и смешанным (Г) взаимным расположением GGGG фрагментов нуклеотидной последовательности. При замене одного гуанина в одной плоскости G4 на другой природный или искусственный нуклеотид (примеры позиций замещения выделены серым цветом) структуры соответствуют потенциально стабильным ImGQ. L1, L2, L3 - фрагменты последовательности любого нуклеотидного состава, входящие в состав петель GQ.
На Фигуре 2 представлены спектры КД GQ и ImGQ двух природных фрагментов генома человека - промотора гена Bcl-2 (А) и интрона гена CTIF (Д) - и их мутантов (100 mM KCl, 10 mМ TrisHCl, pH 7,5). Схема параллельного квадруплекса с двумя несовершенными тетрадами представлена на Фигуре 2Б. Указаны внешние (endo-) и внутренние (ехо-) G4 плоскости квадруплексов. На Фигуре 2В приведены дифференциальные спектры плавления GQ Ctg (sequence 2) и ImGQ Ct1 (sequence 7), Ct2, Ct3 и Ct4 (sequences 9-11), а на Фигуре 2Г представлены спектры КД GQ и ImGQ интрона гена CTIF и его позиционных мутантов, полученные в присутствии 100 mM иона лития, препятствующего образованию GQ структуры. На Фигуре 2Е представлена зависимость времен вращательной корреляции интеркалятора (бромистый этидий) в комплексах с GQ1 (sequence 8) (контроль), bclGQ (sequence 3), TrGQ (sequence 6), Ct1 (sequence 7), Ct2 (sequence 9), Ct3 (sequence 10) и Ctg (sequence 2) от объема их молекул (числа нуклеотидов).
На Фигуре 3 приведено сравнение H1 ЯМР-спектров 2-квартетного квадруплекса GQ1 (sequence 2) и олигомера Ct1 (sequence 7) - ImGQ. Номерами отмечены восемь сигналов протонов двух G4 плоскостей в спектре GQ1 (sequence 2) (спектр внизу фигуры) и 14 сигналов четырех G4 плоскостей ImGQ Ct1 (sequence 7) (спектр вверху фигуры), который в соответствии с известными ранее подходами должен был также содержать только 8 сигналов протонов в области 12 ppm.
На Фигуре 4 приведены результаты анализа последовательности 18 хромосомы человека с помощью нового алгоритма поиска сайтов, способных формировать новые неканонические структуры - ImGQ. Представлены гистограммы распределения в последовательности 18 хромосомы человека сайтов, способных формировать 4-квартетные совершенные GQ (Perfect GQ) и несовершенные ImGQ, несущие одну нуклеотидную замену (А). Гистограммы Б и В отражают состав мононуклеотидных замен в сайтах ImGQ 18 хромосомы и их распределение во внешних (ImEn) и внутренних (ImEx) плоскостях структур.
Описание примеров реализации изобретения
Пример 1 описывает получение олигодезоксирибонуклеотидов природных и модельных последовательностей, приведенных в Таблице 2, (Пример 1.1) и исследование их термодинамических и спектральных свойств (Примеры 1.2-1.4).
В Примерах 1.2-1.3 показана высокая стабильность природных несовершенных GQ из промоторной области гена bcl2 и интрона гена CTIF при физиологических условиях. Представлены доказательства существования олигонуклеотида с последовательностью bcl2 ImGQ (sequence 1) в растворе именно в виде дефектного 4-квартетного, а не совершенного 3-квартетного GQ, найденного по стандартному алгоритму [16] с помощью программы quadfinder. Изучена возможность образования дважды дефектных квадруплексов - производных ImGQ bcl2 (sequences 2-5) (Таблица 2).
Аналогичные данные получены для искусственных неприродных последовательностей Ct2, Ct3, Ct4 (sequences 9-11), Cta (sequence 12), Ctc (sequence 13) и показано, что они формируют ImGQ, а олигомер Ctg (sequence 2) - совершенный GQ, что полностью соответствует нашим расчетам.
Характерное для GQ влияние ионов калия (стабилизация) и лития (дестабилизация) наблюдалась и для ImGQ. КД-спектры олигонуклетидов Ct2, Ct3 и Ct4 (sequences 9-11) - позиционных изомеров природного Ct1 и совершенного GQ Ctg, полученного заменой T в третьей позиции Ct1 на G, соответствуют спектрам GQ с разными вкладами параллельных и антипараллельных структур в присутствии ионов K+ (Фигура 2Д). Присутствие Li+ подавляет образование неканонических структур, что наглядно отражают схожие спектры олигомеров на Фигуре 2Г.
Пример 1.4. описывает эксперименты, подтверждающие образование внутримолекулярных структур. Мономолекулярность квадруплексов следует из данных, приведенных на Фигуре 2Е, отражающих пропорциональность изменения времен вращательной релаксации комплексов олигомеров с бромистым этидием объему молекулы, т.е. числу нуклеотидных звеньев в составе олигомеров. Кроме того, независимость показателей температуры плавления ImGQ от их концентрации также свидетельствует об образовании внутримолекулярных конформаций.
Пример 1.5. описывает получение H1 ЯМР-спектров 2-квартетного GQ 5'-GGGAGGCTGAGGCAGG (GQ1) и олигомера Ct1. Исходя из известных ранее представлений олигомер Ct1 также должен представлять 2-квартетный GQ, поскольку максимальная длина четырех G-блоков в его составе не превышает двух. Однако, в отличие от спектра GQ1, представленного в области 12 ppm 8 сигналами, соответствующими двум G4-плоскостям, в спектре Ct1 наблюдается 14 сигналов (область 12 ppm). Этот факт подтверждает предположение о том, что олигонуклеотид Ct1 формирует ImGQ-конформацию, состоящую из трех G4 и одной G3T (см. схемы на Фигуре 1А, В).
Таким образом, образование несовершенных 4-квартетных ImGQ доказано совокупностью ряда экспериментальных данных: В том числе формирование ImGQ следует из существенного различия температур плавления и КД-спектров олигонуклеотидов, последовательности которых соответствуют полной и укороченной bcl2 PQS (Тпл. bclGQ ~82°C >> Тпл. trGQ ~58°C, trGQ = техквартетный GQ, укороченная последовательность), приведенных термодинамических и спектральных характеристик (Таблица 2, Фигура 2А-Е и Фигура 3).
Таблица 2. Температуры плавления bcl2 ImGQ и его производных в Tris-HCl буфере (pH 7.5).
В Примере 2 представлены результаты анализа первичных последовательностей прямой и обратной цепей 18 хромосомы человека (общая длина составляет ~140 миллионов нуклеотидов) ImGQ с помощью найденного алгоритма и программы ImGQfinder и их сравнение с результатами поиска GQ в среде известной программы quadfinder.
Пример 2.1 описывает параметры поиска
Пример 2.2 описывает обработку полученный данных и сопоставление встречаемости G/A, G/T и G/C замен в несовершенных квадруплексах. Кроме того в примере 2.2 проводится сравнение количества совершенных и монодефектных несовершенных 4-квартетных GQ в 18 хромосоме (Фигура 4А-В).
Показано, что наиболее часто встречаются замены G/T (47% от общего количества ImGQ). Замены G/A также представлены достаточно широко (23% от общего количества GQ). Замены G/C представлены в меньшей степени (13%). Замены во внутренних (центральных) квартетах преобладают (48% от общего количества замен).
На долю совершенных GQ приходится лишь 17% от общего числа потенциально стабильных квадруплексов. Таким образом, основная часть возможных 4-квартетных неканонических структур (более ¾) была выявлена в результате применения нового алгоритма. Полученные данные стали основой для исследования механизмов функционирования геномов и являются важными новыми молекулярными мишенями для разработки лекарств направленного действия.
Ниже приведены примеры реализации изобретения.
Примеры реализации изобретения
Пример 1. Получение олигодезоксирибонуклеотидов природных и модельных последовательностей (Табл.2) и исследование их термодинамических и спектральных свойств.
Пример 1.1. Синтез олигодезоксирибонуклеотидов
Твердофазным амидофосфитным методом на автоматическом синтезаторе ДНК АСМ 800 ("Биоссет", Россия) были получены олигонуклеотиды, последовательности которых приведены в Таблице 2.
В синтезе использовали стандартные коммерческие нуклеозид-амидофосфиты и нуклеозид-полимеры (все реагенты - Glen Research, США). Удаление синтезированного олигомера с носителя и деблокирование осуществляли аммонолизом (28% аммиак, 5 часов, 50°C). 5'-O-Диметокситритильные (DMTr-) производные олигонуклеотидов выделяли ВЭЖХ (Agilent Chemstation) в градиенте ацетонитрила в 0,1 М ацетате аммония. Колонка Teknokroma (Испания), 1×25 см, 45°C, 1,5 мл/мин. Удаление DMTr-группы проводили 80% водной раствором уксусной кислоты (20 мин). Структуру и чистоту олигонуклеотидов подтверждали данными MALDI-TOF-масс-спектров. Масс-спектры получали в режиме регистрации положительных ионов на масс-спектрометре Bruker Microflex (Германия). Олигонуклеотиды обессоливали на ZipTipC18 (Millipore, USA) перед MALDI-TOF-MS анализом.
Пример 1.2. Определение термодинамических параметров образования конформеров ДНК по профилям термической диссоциации. Спектры кругового дихроизма олигонуклеотидов.
Профили термической диссоциации квадруплексов регистрировали на спектрофотометре Jasco V-550 (США), оборудованном термостатирующей приставкой, при λ=295 нм в диапазоне температур 20-90°C. Плавление при данной длине волны считали доказательством наличия квадруплексной структуры. Термодинамические параметры образования квадруплексов, в частности температуру плавления, рассчитывали методом нелинейной регрессии в программе KaleidaGraph v. 4.0 (Synergy, Великобритания), основываясь на модели двух состояний (1).
КД спектры олигонуклеотидов регистрировали на спектрополяриметре Jasco 715 (США) с термостатируемой ячейкой при температуре 20°C в диапазоне длин волн 220-330 нм.
Непосредственно перед регистрацией КД спектров и кривых плавления растворы олигонуклеотидов в 20 мМ Tris-HCl буфере (pH 7.5) с различными концентрациями KCl и LiCl нагревали до 90°C, выдерживали при данной температуре 3 мин и быстро охлаждали до 0°C.
Пример 1.3. Определение состава стабильных стерических конформаций олигонуклеотидов - потенциальных ImGQ по временам вращательной релаксации комплексов этидий бромид (EtBr) : квадруплекс.
Времена вращательной релаксации (ρ) комплексов рассчитывали по уравнению Перрен-Вебера [23]: 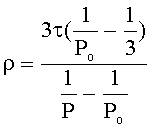
Поляризацию флуоресценции EtBr в комплексе с олигонуклеотидом определяли по соотношению интенсивностей вертикальной и горизонтальной компонент флуоресценции при возбуждении вертикально поляризованным светом. Интенсивность флуоресценции (λ=610 нм) измеряли на спектрофлуориметре Cary Eclipse (Varian, США) при возбуждении светом с длиной волны 540 нм при 20°C. Поляризацию флуоресценции рассчитывали по формуле [24]:
P=(I║-I┴)/(I║+I┴)
Время жизни флуоресценции комплексов EtBr : квадруплекс определяли с помощью спектрофлуориметра Easy Life V (Optical Building Blocks, США) в импульсном режиме.
Пример 1.4. H1 ЯМР-спектры олигонуклеотидов получали (0,1 мМ раствор олигонуклеотида в воде, H2O/D2O, 9/1, 100 mM KCl, 20 mM TrisHCl, pH 7,5). Спектры получали на приборе Bruker AMX400 (Германия), при температуре 19°C с подавлением сигнала воды. Для интерпретации спектров использовали программу MestReNova version 7.0.3 (Mestrelab Research SL, Испания).
Пример 2. Описание алгоритма поиска ImGQ и его применение для анализа первичных нуклеотидных последовательностей и конструирования искусственных фрагментов ДНК - потенциальных ImGQ.
Пример 2.1. Описание алгоритма поиска сайтов потенциальных ImGQ в составе полинуклеотидов.
Формулы последовательностей фрагментов полинуклеотидов (ImGQ), потенциально способных принять конформацию несовершенных квадруплексов приведены в Таблице 1.
При составлении алгоритма учитываются только внутримолекулярные ImGQ, способные существовать одновременно. Например, фрагмент GGGTGG фиксируется только один раз, без учета возможности реализации альтернативных конформеров.
Для выборки стабильных в физиологических условиях последовательностей несовершенных GQ были введены следующие граничные условия:
- рассматриваются только моно-несовершенные ImGQ, т.е. во всех плоскостях GQ возможна только одна замена гуанина на другой нуклеотид;
- n≥4 (n - число квартетов, образованных четырьмя последовательными G-богатыми фрагментами последовательности);
- x=1÷7 (x - длина петель в составе ImGQ);
При N=G, формула описывает совершенный GQ.
На основании данного алгоритма написана программа поиска стабильных ImGQ в первичных нуклеотидных последовательностях - ImGQfinder (Заявление на Госрегистрацию от 06.09.2012)
Пример 2.2. Поиск GQ и потенциально стабильных ImGQ в первичной последовательности 18 хромосомы человека проводили с помощью программы ImGQFinder (Заявление на Государственную регистрацию программы для ЭВМ от 06.09.2012).
Анализировали последовательность 18 хромосомы человека из базы данных NCBI (http://www.ncbi.nlm.nih.gov/nuccore/NC_000018.9) Поиск ImGQ проводили отдельно по прямой и обратной цепям. Учитывали совершенные и монодефектные 4-квартетные GQ. Каждый поливариантный GQ (PQS, способную образовывать несколько GQ различной структуры) учитывали лишь единожды. Этого достигали сопоставлением координат найденных GQ. При пересечении координат дефектных и совершенных GQ дефектный квадруплекс отбрасывали, фиксировали совершенный как термодинамически более вероятный.
Сравнение представленности совершенных и несовершенных GQ в 18 хромосоме. Определение частот встречаемости различных типов дефектов в несовершенных GQ.
Рассчитывали отдельно количество совершенных GQ и GQ с заменами G/A, G/C и G/T. Результаты поиска по прямой и обратной цепям суммировали. Количество GQ с внешними дефектами рассчитывали как сумму замен в первом и четвертом квартетах, с внутренними дефектами - как сумму замен во втором и третьем квартетах.
Литература
1. Raiber E.A., Kranaster R., Lam E., Nikan M. Balasubramanian S. A non-canonical DNA structure is a binding motif for the transcription factor SP1 in vitro // Nucleic Acids Res. - 2011.
2. Kouzine F. Levens D. Supercoil-driven DNA structures regulate genetic transactions // Front Biosci. - 2007; 12 4409-4423.
3. Brown R.V. Hurley L.H. DNA acting like RNA // Biochem Soc Trans. - 2011; 39 (2): 635-640.
4. Nelson L.D., Bender С., Mannsperger H., Buergy D., Kambakamba P., Mudduluru G., Korf U., Hughes D., Van Dyke M.W. Allgayer H. Triplex DNA-binding proteins are associated with clinical outcomes revealed by proteomic measurements in patients with colorectal cancer // Mol Cancer. - 2012; 11 (1): 38.
5. Cer R.Z., Bruce K.H., Donohue D.E., Temiz N.A., Mudunuri U.S., Yi M., Volfovsky N., Bacolla A., Luke B.T., Collins J.R. Stephens R.M. Searching for non-B DNA-forming motifs using nBMST (non-B DNA motif search tool) // Curr Protoc Hum Genet. - 2012; Chapter 18 Unit 18 17 11-22.
6. Shchyolkina A.K., Borisova O.F., Livshits M.A., Pozmogova G.E., Chernov B.K., Klement R. Jovin T.M. Parallel-stranded DNA with mixed AT/GC composition: role of trans G.C base pairs in sequence dependent helical stability // Biochemistry. - 2000; 39 (33): 10034-10044.
7. Hsu S.T., Varnai P., Bugaut A., Reszka A.P., Neidle S. Balasubramanian S. A G-rich sequence within the c-kit oncogene promoter forms a parallel G-quadruplex having asymmetric G-tetrad dynamics // J Am Chem Soc. - 2009; 131 (37): 13399-13409.
8. Shahid R., Bugaut A. Balasubramanian S. The BCL-2 5' untranslated region contains an RNA G-quadruplex-forming motif that modulates protein expression // Biochemistry. - 2011; 49 (38): 8300-8306.
9. Tolmachov O.E. Self-entanglement of long linear DNA vectors using transient non-B-DNA attachment points: A new concept for improvement of non-viral therapeutic gene delivery // Med Hypotheses. - 2012.
10. Fisette J.F., Montagna D.R., Mihailescu M.R. Wolfe M.S. A G-Rich element forms a G-quadruplex and regulates BACE1 mRNA alternative splicing // J Neurochem. - 2012.
11. Cheng L.C., Pai T.W. Li L.A. Regulation of human CYP11B1 and CYP11B2 promoters by transposable elements and conserved cis elements // Steroids. - 2012; 77 (1-2): 100-109.
12. Zhou W., Brand N.J. Ying L. G-quadruplexes-novel mediators of gene function // J Cardiovasc Transl Res. - 2011; 4 (3): 256-270.
13. Huppert J.L. Balasubramanian S. G-quadruplexes in promoters throughout the human genome // Nucleic Acids Res. - 2007; 35 (2): 406-413.
14. Wu Y. Brosh R.M., Jr. G-quadruplex nucleic acids and human disease // FEBS J. - 2010; 277 (17): 3470-3488.
15. Balasubramanian S., Hurley L.H. Neidle S. Targeting G-quadruplexes in gene promoters: a novel anticancer strategy? // Nat Rev Drug Discov. - 2011; 10 (4): 261-275.
16. Huppert J.L. Balasubramanian S. Prevalence of quadruplexes in the human genome // Nucleic Acids Res. - 2005; 33 (9): 2908-2916.
17. Gonzalez V., Guo K., Hurley L. Sun D. Identification and characterization of nucleolin as a c-myc G-quadruplex-binding protein // J Biol Chem. - 2009; 284 (35): 23622-23635.
18. Dai J., Dexheimer T.S., Chen D., Carver M., Ambrus A., Jones R.A. Yang D. An intramolecular G-quadruplex structure with mixed parallel/antiparallel G-strands formed in the human BCL-2 promoter region in solution // J Am Chem Soc. - 2006; 128 (4): 1096-1098.
19. Zaitseva M., Kaluzhny D., Shchyolkina A., Borisova O., Smirnov I. Pozmogova G. Conformation and thermostability of oligonucleotide d(GGTTGGTGTGGTTGG) containing thiophosphoryl internucleotide bonds at different positions // Biophysical Chemistry. - 2010; 146 (1): 1-6.
20. Hazel P., Huppert J., Balasubramanian S. Neidle S. Loop-length-dependent folding of G-quadruplexes // J Am Chem Soc. - 2004; 126 (50): 16405-16415.
21. Guedin A., De Cian A., Gros J., Lacroix L. Mergny J.L. Sequence effects in single-base loops for quadruplexes // Biochimie. - 2008; 90 (5): 686-696.
22. Kim K.M., Cho H., Choi K., Kim J., Kim B.W., Ко Y.G., Jang S.K. Kim Y.K. A new MIF4G domain-containing protein, CTIF, directs nuclear cap-binding protein CBP80/20-dependent translation // Genes Dev. - 2009; 23 (17): 2033-2045.
23. Marky L.A. Breslauer K.J. Calculating thermodynamic data for transitions of any molecularity from equilibrium melting curves // Biopolymers. - 1987; 26 (9): 1601-1620.
24. Weber G. Anderson S.R. The effects of energy transfer and rotational diffusion upon the fluorescence polarization of macromolecules // Biochemistry. - 1969; 8 (1): 361-371.
| название | год | авторы | номер документа |
|---|---|---|---|
| КОМБИНАТОРНЫЙ СПОСОБ ПОЛУЧЕНИЯ ДНК-АПТАМЕРНЫХ ИНГИБИТОРОВ ТРОМБИНА И АПТАМЕРНЫЕ ОЛИГОНУКЛЕОТИДЫ (ВАРИАНТЫ) | 2013 |
|
RU2520094C1 |
| ДНК-АПТАМЕРЫ, ИНГИБИРУЮЩИЕ ТРОМБИН, И СПОСОБ СТАБИЛИЗАЦИИ ИХ СТРУКТУРЫ | 2009 |
|
RU2429293C1 |
| Лекарственная форма ДНК-аптамера | 2019 |
|
RU2730000C1 |
| СПОСОБ СОЗДАНИЯ КВАДРУПЛЕКСА И СПОСОБ ОБНАРУЖЕНИЯ НУКЛЕИНОВОЙ КИСЛОТЫ | 2002 |
|
RU2304168C2 |
| АПТАМЕРЫ ДЛЯ ПРИМЕНЕНИЯ ПРИ ИНГИБИРОВАНИИ И/ИЛИ ПОДАВЛЕНИИ АКТИВАЦИИ TLR9 | 2017 |
|
RU2789087C2 |
| ЛЕЧЕНИЕ ПРОЛИФЕРАТИВНЫХ ЗАБОЛЕВАНИЙ С ИСПОЛЬЗОВАНИЕМ АНТИСМЫСЛОВОГО ОЛИГОМЕРА IAP И ХИМИОТЕРАПЕВТИЧЕСКОГО ПРЕПАРАТА | 2004 |
|
RU2376018C2 |
| ИММУНОСТИМУЛИРУЮЩИЕ ОЛИГОНУКЛЕОТИДЫ | 2018 |
|
RU2795701C2 |
| ДНК-аптамеры к праймазе и ДНК-полимеразе PrimPol человека | 2020 |
|
RU2770691C2 |
| АНТИКОАГУЛЯНТ ПРЯМОГО ДЕЙСТВИЯ НА ОСНОВЕ МОДУЛЬНОГО БИВАЛЕНТНОГО ДНК АПТАМЕРА, СЕЛЕКТИВНОГО К ТРОМБИНУ | 2016 |
|
RU2631829C1 |
| Способ определения пространственной структуры биомолекул | 2020 |
|
RU2751797C1 |



Изобретение относится к области биотехнологии, конкретно к способу получения неприродных искусственных олигонуклеотидов, потенциально способных образовывать стабильные в физиологических и близких к физиологическим условиях неканонические структуры - несовершенные G-квадруплексы (ImGQ), включающие одну нуклеотидную замену в G4 плоскости в G-квадруплексах (GQ). Указанный способ включает использование алгоритма описания нуклеотидных последовательностей в виде определенного набора формул для дальнейшего синтеза выбранных олигонуклеотидов. Изобретение позволяет с помощью биоинформационного анализа получать неприродные искусственные олигонуклеотиды, потенциально способные формировать новую конформацию - несовершенные G-квадруплексы. 4 ил., 2 табл., 2 пр.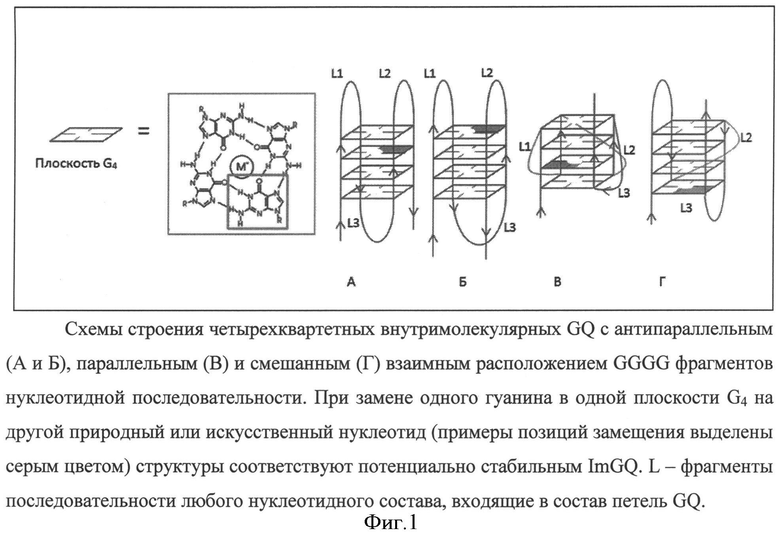
Способ получения неприродных искусственных олигонуклеотидов, потенциально способных образовывать стабильные в физиологических и близких к физиологическим условиях неканонические структуры - несовершенные G-квадруплексы (ImGQ), включающие одну нуклеотидную замену в G4 плоскости в G-квадруплексах (GQ), включающий использование алгоритма описания нуклеотидных последовательностей в виде набора нижеследующих структурных формул

(для k≥4; x=1÷7), где k - число квартетов, образованных четырьмя последовательными G - богатыми фрагментами нуклеотидной последовательности; L - любой нуклеотид в петле ImGQ; x - количество нуклеотидов в петле; N - любой не-G нуклеотид в плоскости ImGQ; для дальнейшего синтеза неприродных искусственных олигонуклеотидов, потенциально способных образовывать ImGQ и характеризующихся выбранными последовательностями.
| HUPPERT J.L | |||
| et al, Prevalence of quadruplexes in the human genome, Nucleic Acids Res., 2005, v.33, n.9, p.2908-2916 | |||
| KIKIN O | |||
| et al, QGRS Mapper: a web-based server for predicting G-quadruplexes in nucleotide sequences, Nucleic Acids Res | |||
| (Web Server issue), 2006, v.34, p.W676-W682 | |||
| HERSHMAN S.G | |||
| et al | |||
| Genomic distribution and functional analyses |
Авторы
Даты
2014-03-20—Публикация
2012-10-04—Подача