ОБЛАСТЬ ТЕХНИКИ
Новые гены, композиции и способы повышения устойчивости сельскохозяйственной культуры к фитофторозу пасленовых.
УРОВЕНЬ ТЕХНИКИ
Картофель (Solatium tuberosum L.) является четвертой по важности сельскохозяйственной культурой и наиболее важной незерновой сельскохозяйственной культурой в мире. При выращивании картофеля основным природным фактором, ограничивающим урожайность, является фитофтороз пасленовых, вызываемый патогеном Phytophthora infestans (Mont.) группы оомицетов, обнаруженным деБари. В отсутствие контроля это разрушительное заболевание может привести к полной потере урожая сельскохозяйственной культуры (Świeżyński и Zimnoch-Guzowska 2001). Обработка фунгицидом на сегодняшний день является наиболее распространенным способом контролирования фитофтороза пасленовых. Тем не менее, высокая стоимость применения фунгицида является проблемой, поскольку применение фунгицида может отрицательно сказываться на здоровьи и экологической безопасности, применение химических препаратов начинают ограничивать. Кроме того, данный патоген быстро эволюционирует, и некоторые из новых его вариантов нечувствительны к распространенным фунгицидам (Day и Shattock 1997; Goodwin и др. 1996). Следовательно, придание генетической устойчивости культивируемому картофелю рассматривают как важный способ достижения длительной устойчивости к фитофторозу пасленовых.
Описано два основных типа устойчивости к фитофторозу картофеля (Umaerus и Umaerus 1994). Первый, общий вид устойчивости часто основан на мажорных локусах количественных признаков (QTL) и нескольких минорных QTL и обеспечивает частичную устойчивость. Второй, специфичный вид устойчивости основан на мажорных доминантных генах устойчивости (R-генов). В ранних программах по разведению в первой половине прошлого века было идентифицировано 11 R-генов (R1-R11), полученных из S. demissum. Девять R-генов, R3 (в настоящее время разделены на R3a и R3b) и R5-R11 локализованы на хромосоме 11 (Bradshaw и др. 2006; EI-Kharbotly 1994, 1996; Huang и др. 2004; Huang 2005). Другие R-гены, происходящие из S. demissum, были картированы в различных положениях, включая R1 на хромосоме 5 (EI-Kharbotly и др. 1994; Leonards-Schippers и др. 1992) и R2 на хромосоме 4 (Li и др., 1998). Все R-гены из S. demissum, внедренные в культивируемый картофель, были преодолены патогеном, поскольку новые штаммы быстро эволюционировали и приобретали вирулентность в отношении ранее устойчивых хозяев (Umaerus и Umaerus 1994). Соответственно, частичная устойчивость, обеспечиваемая QTL, считалась более длительной, чем устойчивость, придаваемая отдельными R-генами (Turkensteen 1993). Тем не менее, частичная устойчивость в значительной степени коррелировала с типом созревания, что усложняло сохранение устойчивости при разведении (Wastie 1991). Также положения QTL в геноме часто соответствовали участку кластеров R-генов (Gebhart и Valkonen 2001; Grube и др. 2000).
Таким образом, недавние усилия, направленные на идентификацию устойчивости к фитофторозу пасленовых, были сосредоточены на мажорных R-генах, обуславливающих широкий спектр устойчивости, полученных из различных диких видов Solarium. Сообщалось, что помимо S. demissum другие дикие виды Solarium, такие как S. acaule, S. chacoense, S. berthaultii, S. brevidens, S. bulbocastanum, S. microdontum, S. sparsipilum, S. spegazzinii, S. stoloniferum, S. sucrense, S. toralapanum, S. vernei и S. verrucosum, являются новыми источниками устойчивости к фитофторозу пасленовых (обзор подготовлен Jansky 2000; Hawkes 1990). На сегодняшний день три R-гена, RB/Rpi-blb1, Rpi-blb2 и Rpi-blb3 из S. bulbocastanum были картированы на хромосомах 8, 6 и 4, соответственно (Nasess и др. 2000; Park и др. 2005а; van der Vossen и др. 2003, 2005). Другой R-ген, Rpi-abpt, вероятно, из S. bulbocastanum, был локализован на хромосоме 4 (Park и др. 2005b). Rpi1 из S. pinnatisectum на хромосоме 7 (Kuhl и др. 2001), Rpi-mcq1 из S. mochiquense (Smilde и др. 2005) и Rpi-phu1 из S. phureja на хромосоме 9 (Śliwka и др. 2006), о чем также сообщалось.
Как следует из обзора уровня техники в этой области, существует значительная потребность в новых генах, композициях и способах придания устойчивости к фитофторозу пасленовых. Описанное в настоящем документе изобретение удовлетворяет эту потребность путем скрининга диких видов Solanum и путем клонирования, а также введения и экспрессирования новых генов устойчивости Rpi в картофеле.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Авторы настоящего изобретения выделили, идентифицировали и описали несколько различных R-генов фитофтороза пасленовых, полученных из диких видов картофеля S. okadae, а также из S. mochiquense и S. neorossii.
Согласно настоящему изобретению предложены новые последовательности генов, композиции и способы повышения устойчивости сельскохозяйственных культур, в частности, но не ограничиваясь, картофеля, к фитофторозу пасленовых, вызванному патогенным оомицетом Phytophthora infestans.
КРАТКОЕ ОПИСАНИЕ ФИГУР
Фиг.1 Происхождение генотипов, используемых для создания ВАС-библиотек. (а): К39 и (b): K182.
Фиг.2 Анализ размера вставки отобранных случайным образом ВАС-клонов из двух ВАС-библиотек, К39 (а) и K182 (b), с помощью гель-электрофореза в пульсирующем поле. ВАС-клоны расщепляли рестриктазой NotI. Получали полосы, соответствующие 7.5 т.п.н., из вектора клонирования plndigoBAC-5. Значения молекулярных масс по маркерам фага лямбда (Sigma Chemical) показаны на фигурах справа.
Фиг.3 Положения генов устойчивости к фитофторозу пасленовых Rpi-oka1, Rpi-oka2 и Rpi-oka3 из S. okadae u Rpi-nrs1 из S. neorossii.
Фиг.4 Изображения SCAR-маркеров, использовавшихся для скрининга двух ВАС-библиотек. TG551, расщепленный рестриктазой TaqI (a), связан с Rpi-oka1, TG551, расщепленным рестриктазой MwoI (b), и TG35 (с) связаны с Rpi-oka1, и U296361 (d) и TG591 (е) связаны с Rpi-mcq1. Указаны маркер размера 1 т.п.н. и исходные генотипы (А624, А613, А618 и А988), а остальные представляют собой ВАС-пулы, положительные по некоторым маркерам, идентифицированным с помощью ПЦР с последующим расщеплением рестриктазой. Устойчивая аллель помечена '<<' справа на каждой фигуре.
Фиг.5 Контиг ВАС-клонов, идентифицированный из ВАС-библиотеки К39 и покрывающий участок генома, содержащий Rpi-oka1 и Rpi-oka2.
Фиг.6 Картирование генов Rpi, полученных из Solarium okadae и S. neorossiic высоким разрежением и на малом участке.
Фиг.7 Выравнивание выявленных последовательностей белков Rpi-oka1, Rpi-oka2 и Tm-22. Показана полная последовательность аминокислот Rpi-oka1, точками отмечены идентичные остатки в двух других белках. В тех случаях, где остатки из Rpi-oka2 и Tm-22 отличаются от Rpi-oka1, показаны остатки в этих белках. Два различия в аминокислотах между Rpi-oka1 и Rpi-oka2 указаны жирным шрифтом. Предсказанные спиральные домены подчеркнуты, первый и четвертый гидрофобные остатки каждого повтора группы из семи остатков подчеркнуты двойной чертой. Консервативные мотивы в домене NB-ARC отмечены курсивными строчными буквами. Предполагаемые лейцин-богатые повторы (LRR) показаны над последовательностью.
Фиг.8 Выравнивание последовательностей белков Rpi-oka1, Rpi-nrs1 и Tm-22. Домены СС, NB-ARC и LRR выделены красным, зеленым и оранжевым, соответственно. Консервативные мотивы в домене NB-ARC подчеркнуты и выделены курсивом.
Фиг.9 Карты генетических связей на хромосоме IX локусов Rpi-oka1 (а) и Rpi-nrs1 (b), картированные в популяциях 7698 и 7663, соответственно. Числа слева показывают расстояния между генами в сантиморганах (сМ). Относительные положения картированных локусов обозначены горизонтальными линиями. Буква п обозначает размер каждой популяции.
Фиг.10 Расположение на карте гена устойчивости к фитофторозу пасленовых Rpi-mcq1 из S. mochiquense.
Фиг.11 Контиг ВАС-клонов, идентифицированный в ВАС-библиотеке К182 и покрывающий участок генома, содержащий Rpi-mcq1.
ПОДРОБНОЕ ПИСАНИЕ ПРЕДПОЧТИТЕЛЬНЫХ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
Следующие последовательности приложены к данной заявке на Фиг.12:
Приведенные выше последовательности представляют собой последовательности, удлиненные по сравнению с описанными в GB 714241.7, по которой заявлен приоритет настоящей заявки. В частности, они были удлинены, как указано далее:
SEQ ID 1a - удлинена на 99 дополнительных оснований в начале
SEQ ID 1b - удлинена на 141 дополнительное основание в начале
SEQ ID 3 - удлинена на те же 141 дополнительное основание в начале
SEQ ID 4a - удлинена на 33 дополнительных аминокислоты в начале
SEQ ID 4b - удлинена на 47 дополнительных аминокислот в начале
SEQ ID 6 - удлинена на те же 47 дополнительных аминокислот в начале.
Тем не менее, более не отказываются от приятзаний на более раннее изобретение. Таким образом, в тех случаях, когда какой-либо аспект или вариант реализации настоящего изобретения описан в отношении удлиненных последовательностей, описанных выше, следует понимать, что с соответствующими изменениями то же самое применимо к описанной ранее более короткой последовательности. Таким образом, все аспекты или варианты реализации настоящего изобретения по отдельности и в совокупности также будут с соответствующими изменениями охватывают:
SEQ ID 1а - нуклеотиды 100-2676
SEQ ID 1b - нуклеотиды 142-2718
SEQ ID 3 - нуклеотиды 142-2718
SEQ ID 4a - аминокислоты 34-891
SEQ ID 4b - аминокислоты 48-905
SEQ ID 6 - аминокислоты 48-905.
Следующие последовательности представляют собой кассеты экспрессии, включающие некоторые из описанных выше последовательностей:
В SEQ ID 1с (Rpi-oka1) - промотор Rpi-oka1 включен в участок, содержащий основания 1-709, включая нетранслируемую 5'-область (НТО), состоящую из оснований 627-709. Открытая рамка считывания (OPC) Rpi-oka1 присутствует на участке, содержащем основания 710-3382, а терминатор располагается от основания 3383 и далее. Эту последовательность клонировали в pSLJ21152 и затем использовали для трансформирования S. tuberosum и S. lycopersicum, чтобы придать устойчивость к Р. infestans.
В SEQ ID 2b (Rpi-mcq1.1) - промотор Rpi-mcq1.1 включен в участок, содержащий основания 1-2262, открытая рамка считывания (OPC) Rpi-mcq1.1 присутствует на участке, содержащем основания 2263-4848, а терминатор располагается от основания 4849 и далее. Эту последовательность клонировали в pSLJ21153 и затем применяли для трансформирования S. tuberosum и S. lycopersicum, чтобы придать устойчивость к Р. infestans.
В SEQ ID 2d (Rpi-mcq1.2) - промотор Rpi-mcq1.2 включен в участок, содержащий основания 1-1999, открытая рамка считывания (OPC) Rpi-mcq1.2 присутствует на участке, содержащем основания 2000-4567, и терминатор располагается от основания 4568 и далее. Эту последовательность клонировали в pSLJ21148 и затем применяли для трансформирования S. tuberosum и S. lycopersicum, чтобы придать устойчивость к Р. infestans.
На Фигуре 8 показано, что последовательности Rpi-oka1 и Rpi-nrs1 очень близки.
Как описано ниже, полагают, что последовательности Rpi-oka2 фактически идентичны Rpi-nrs1, но для полноты описания приведены обе.
Тем не менее, последовательности Rpi-oka3, описанные ниже в данной заявке, идентичны Rpi-oka2, и, соответсвенно, не описаны в явной форме.
Наконец, различные кандидатные гены Rpi были идентифицированы у S. mochiquense и они оба приведены в указанных последовательностях. Полагают, что они оба являются функциональными R-генами с различными специфичностями узнавания.
Таким образом, в первом аспекте настоящего изобретения описаны изолированные молекулы нуклеиновых кислот, кодирующие функциональный ген Rpi, который, возможно, может быть выбран из S. okadae, S. mochiquense и S. neorossii.
В частности, варианты реализации настоящего изобретения обеспечивают изолированный ген устойчивости Rpi, имеющий последовательность, обозначенную в списке последовательностей SEQ. ID. 1a, 1b, 2а, 2b или 3.
Молекулы нуклеиновых кислот согласно настоящему изобретению могут быть изолированными и/или очищенными от их природного окружения, в по существу чистой или гомогенной форме, или свободными или по существу свободными от других нуклеиновых кислот видов, из которых они получены. В данной заявке в объем термина "изолированные" входят все указанные возможности.
Молекулы нуклеиновых кислот могут быть полностью или частично синтетическими. В частности, они могут быть рекомбинантными в том смысле, что последовательности нуклеиновых кислот, которые не встречаются вместе в природе (не прилегают друг к другу), были лигированы или объединены другим искусственным способом.
В качестве альтернативы они могли быть синтезированы непосредственно, например, с применением автоматизированного устройства для синтеза.
Предпочтительные нуклеиновые кислоты состоят по существу из обсуждаемого гена, возможно в векторе экспрессии, описанном более подробно ниже.
Нуклеиновая кислота согласно настоящему изобретению может включать кДНК, РНК, геномную ДНК и модифицированные нуклеиновые кислоты или аналоги нуклеиновых кислот. Во всех случаях, когда описана последовательность ДНК, например, со ссылкой на фигуру, если в контексте не указано другое, эквивалентная РНК с заменой Т на U везде, где он встречается, входит в объем настоящего изобретения. Во всех случаях, когда нуклеиновая кислота согласно настоящему изобретению описана в данной заявке, комплементарная нуклеиновая кислота будет также входить в объем настоящего изобретения. Комплементарной к данной нуклеиновой кислоте (последовательности) называют нуклеиновую кислоту(последовательность) такой же длины, что и данная нуклеиновая кислота (последовательность), но на 100% комплементарную ей.
Если описаны геномные последовательности нуклеиновых кислот согласно настоящему изобретению, нуклеиновые кислоты, включающие любой один или более (например, 2) интронов или экзонов из любой из этих последовательностей, также включены.
Ген устойчивости в контексте настоящего изобретения представляет собой ген, который контролирует устойчивость к фитофторозу пасленовых, вызванную Р. infestans. Такой ген может кодировать полипептид, способный узнавать и активировать защитный ответ у растения, в ответ на введение указанного патогена, или раздражителем, или его продуктом гена Avr.
Нуклеиновые кислоты согласно первому аспекту можно успешно использовать, например, для картофеля.
Нуклеиновая кислота согласно настоящему изобретению может кодировать одну из последовательностей аминокислот, описанных выше (4а, 4b, 5a, 5b, 6), например может быть вырожденным эквивалентом соответствующих последовательностей нуклеотидов.
В дополнительном аспекте настоящего изобретения описаны нуклеиновые кислоты, которые представляют собой варианты последовательностей согласно первому аспекту.
Вариант молекулы нуклеиновой кислоты гомологичен или идентичен части кодирующей последовательности или целой кодирующей последовательности, описанной выше. В целом, варианты могут кодировать полипептиды или их можно применять для выделения или амплификации нуклеиновых кислот, которые кодируют полипептиды, способные опосредовать ответ на Р. infestans, и/или которые будут специфично связываться с антителом, которое образуется в ответ на полипептиды, описанные выше (4a, 4b, 5a, 5b, 6).
Вариантами согласно настоящему изобретению могут быть искуссвенные нуклеиновые кислоты (т.е. содержащие последовательности, которые имеют неприродное происхождение), которые специалист сможет получить, руководствуясь описанием настоящего изобретения. В качестве альтернативы они могут быть новыми, встречающимися в природе, нуклеиновыми кислотами, которые были выделены или могут быть выделены с применением последовательностей согласно настоящему изобретению, например, из S. mochiquense, S. okadae и S. neorossii.
Таким образом, вариант может быть отличительной частью или фрагментом (полученным любым способом), соответствующим части приведенной последовательности. Указанные фрагменты могут кодировать определенные функциональные части полипептида, например LRR-участки, или сайты терминации.
В равной мере фрагменты могут найти применение при зондировании или амплификации приведенных последовательностей, или близких им последовательностей. Подходящие длины фрагментов и условия для таких процессов обсуждаются более подробно ниже.
Также включены нуклеиновые кислоты, которые были удлинены с 3'- или 5'-конца.
Варианты последовательности, которые встречаются в природе, могут включать аллели или другие гомологи (которые могут включать полиморфизмы или мутации по одному или более основаниям).
Искусственные варианты (производные) могут получить специалисты в данной области, например, с помощью сайт-направленного или неспецифического мутагенеза, или путем непосредственного синтеза. Предпочтительно, вариант нуклеиновой кислоты получают либо непосредственно либо опосредованно (например, с помощью одного или более этапов амплификации или репликации) из исходной нуклеиновой кислоты, включающей часть или всю последовательность согласно первому аспекту. Предпочтительно, она кодирует ген устойчивости к Р. infestans.
В объем термина "вариант" нуклеиновой кислоты в данном описании входят все указанные возможности. Когда термин используется применительно к полипептидам или белкам, он указывает на кодируемый вариантом нуклеиновой кислоты продукт экспрессии.
Некоторые из аспектов настоящего изобретения, относящиеся к указанным вариантам, далее будут описаны более подробно.
Вычисленные идентичности нуклеотидов были такими, как указано далее:
Расчет идентичности аминокислот были такими, как указано далее:
Указанные выше множественные сравнения выполняли, применяя AlignX (Vector NTI Suite Invitrogen) с ядром на основе матрицы CLUSTAL.
В более общем смысле, гомологию (т.е. близость или идентичность) можно определить, сравнивая последовательности с помощью программ BestFit и GAP из пакета программного обеспечения GCG, Wisconsin Package 10.0 от Genetics Computer Group, Мэдисон, Висконсин. CLUSTAL также представляет собой матрицу, используемую в BestFit. Параметры предпочтительно устанавливают на значения по умолчания, как указано далее: штраф за открытие гэпа (пробела): 9; штраф за продление гэпа: 2. Гомологию можно определить на уровне последовательности нуклеотидов и/или кодируемой последовательности аминокислот. Предпочтительно, последовательность нуклеиновой кислоты и/или аминокислот имеет по меньшей мере приблизительно 50%, или 60%, или 70%, или 80% гомологии, наиболее предпочтительно по меньшей мере приблизительно 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% гомологии с последовательностью SEQ. ID. 1a, 1b, 2a, 2b или 3, или 4а, 4b, 5a, 5b, или 6 в соответствующих случаях.
В частности, настоящее изобретение обеспечивает изолированный ген устойчивости Rpi, имеющий последовательность, которая на по меньшей мере приблизительно 80% гомологична последовательности нуклеиновой кислоты, обозначенной в списке последовательностей SEQ. ID. 1a, 1b, 2a, 2b или 3.
Настоящее изобретение дополнительно обеспечивает изолированный белок, включающий последовательность аминокислот, которая на по меньшей мере 80% гомологична последовательности аминокислот, обозначенной в списке последовательностей SEQ. ID. 4а, 4b, 5a, 5b, или 6. Таким образом, вариант полипептида в соответствии с настоящим изобретением может включать в пределах последовательностей, представленных в данной заявке, одно изменение аминокислоты или 2, 3, 4, 5, б, 7, 8, или 9 изменений, приблизительно 10, 15, 20, 30, 40 или 50 изменений, или больше, чем приблизительно 50, 60, 70, 80, 90, 100, 200, 400 изменений. Вдобавок к одному или более изменениям в представленной последовательности аминокислот вариант полипептида может включать дополнительные аминокислоты на С-конце и/или N-конце.
Таким образом, в дополнительном аспекте настоящего изобретения описан способ получения производного нуклеиновой кислоты, включающий этап модифицирования кодирующей последовательности нуклеиновой кислоты согласно настоящему изобретению, например SEQ. ID. 1a, 1b, 2a, 2b или 3.
Для получения производного в последовательность можно внести изменения с помощью одного или более присоединений, вставок, делеций или замен одного или более нуклеотидов в нуклеиновой кислоте, что приводит к присоединению, вставке, делеций или замене одной или более аминокислот в кодируемом полипептиде. Изменения могут быть желательны для ряда причин, включая включение или удаление следующих свойств: последовательности сайтов для эндонуклеаз рестрикции; использование кодона; другие сайты, необходимые для посттрансляционной модификации; сайты расщепления в кодируемом полипептиде; мотивы в кодируемом полипептиде (например, сайты связывания). Лидерные или другие нацеливающие последовательности могут быть удалены из экспрессирующегося белка, чтобы определить его положение после экспрессии. Все это может способствовать эффективному клонированию и экспрессированию активного полипептида в рекомбинантной форме (описано ниже).
Другой желательной мутацией может быть неспецифический или сайт-направленный мутагенез, позволяющий измененить активность (например, специфичности) или стабильность кодируемого полипептида. Изменения могут представлять собой консервативное изменение, т.е. замену одного гидрофобного остатка, такого как изолейцин, валин, лейцин или метионин, на другой, или замену одного полярного остатка на другой, как, например, аргинина на лизин, глутаминовой кислоты на аспарагиновую кислоту, или глутамина на аспарагин. Специалистам в данной области хорошо известно, изменение первичной структуры полипептида путем консервативной замены не может значительно изменить активность этого пептида, так как боковая цепь аминокислоты, выстраиваемой в последовательность, сможет образовывать связи и контакты, аналогичные тем, которые образовывала боковая цепь аминокислоты, которая была замещена. Это в равной степени справедливо в отношении участка, имеющего критическое значение для определения конформации пептида.
Также включены варианты, включающие неконсервативные замены. Что хорошо известно специалистам в данной области, замены на участках пептида, которые не важны для определения его конформации, не могут сильно влиять на его активность, так как они сильно не изменяют трехмерную структуру пептида.
На участках, которые важны для определения конформации пептидов или их активности, такие изменения могут придать полипептиду полезные свойства. В действительности, изменения, такие как изменения, описанные выше, могут придать несколько полезных свойств пептиду, например измененную стабильность или специфичность. Например, манипуляции с LRR-участками полипептидов, кодируемых нуклеиновыми кислотами согласно настоящему изобретению, могут позволить получить новую избирательность устойчивости, например, по отношению к культурам Р. infestans.
LRR-участки также можно привить на другие NBS-участки (например, из других генов устойчивости). Таким образом, способы получения новой избирательности могут включать смешивание или введение последовательностей из родственных генов устойчивости в последовательности Rpi, описанные в данной заявке. В альтернативной стратегии модификации последовательностей Rpi используют ПЦР, описанную ниже (Но и др., 1989, Gene 77, 51-59) или перетасовка ДНК (Crameri и др., 1998, Nature 391).
Подробный анализ некоторых ОРС согласно настоящему изобретению приведен в Примерах ниже, включая обозначение вариантов, включающих замены, и обозначение интересующих участков.
В дополнительном аспекте настоящего изобретения предусмотрен способ идентификации и/или клонирования варианта нуклеиновой кислоты из растения, в указанном способе используют отличную последовательность нуклеотидов Rpi (например, которая представлена в последовательности SEQ. ID. 1А, 1В, 2А, 2В или 3, или комплементарную ей, или вырожденные праймеры, основанные на ней).
Олигонуклеотид для применения в реакциях зондирования или амплификации включает или состоит из приблизительно 30 или менее нуклеотидов в длину (например 18, 21 или 24). Как правило, специфичные праймеры состоят из не менее чем 14 нуклеотидов в длину. Для оптимальной специфичности и экономической эффективности могут быть предпочтительными праймеры, состоящие из 16-24 нуклеотидов в длину. Специалисты в данной области имеют достаточный опыт в разработке праймеров для применения в таких процессах, как ПЦР. В случае необходимости, зондирование можно осуществить с целыми рестрикционными фрагментами гена, описанного в данной заявке, которые могут состоять из сотен или даже тысяч нуклеотидов в длину.
Предпочтительно зонд/праймер отличается в том смысле, что он присутствует во всех или некоторых из последовательностей Rpi, описанных в данной заявке, но не в последовательности гена устойчивости из известного уровня техники.
Например, результаты относительно функциональных аллелей, представленные в настоящем описании (см., например, Фиг.10 или Фиг. 11), позволяют идентифицировать функциональные аллели Rpi, как указано далее.
В дополнительном варианте реализации вариант в соответствии с настоящим изобретением также возможно получен способом, который включает:
(а) обеспечение препарата нуклеиновой кислоты, например, из растительных клеток,
(b) обеспечение молекулы нуклеиновой кислоты, которая представляет собой зонд, описанный выше,
(c) приведение во взаимодействие нуклеиновой кислоты в указанном препарате с указанной молекулой нуклеиновой кислоты в условиях, подходящих для гибридизации указанной молекулы нуклеиновой кислоты с любым указанным геном или гомологом в указанном препарате, и идентификацию указанного гена или гомолога, если таковой присутствует, по его гибридизации с указанной молекулой нуклеиновой кислоты.
При зондировании можно применять стандартную методику саузерн-блоттинга. Например, ДНК можно экстрагировать из клеток и расщепить различными рестриктазами. Фрагменты рестрикции затем можно разделить с помощью электрофореза в агарозном геле, с последующей денатурацией и переносом на нитроцеллюлозный фильтр или нейлон. Меченый зонд можно гибридизировать с фрагментами ДНК на фильтре и определить связывание. ДНК для зондирования возможно получен из препаратов РНК из клеток.
Тестируемая нуклеиновая кислота может быть получена из клетки в виде геномной ДНК, кДНК или РНК, или смеси любых из перечисленных молекул, предпочтительно в виде библиотеки в подходящем векторе. При использовании геномной ДНК можно применять зонд для идентифицирования нетранскрибируемых участков указанного гена (например, промоторов и т.д.), как описано далее в данной заявке. Зондирование, возможно, можно осуществить с использованием так называемых чипов нуклеиновых кислот (для обзора см. Marshall & Hodgson (1998) Nature Biotechnology 16: 27-31).
Предварительные эксперименты можно осуществить путем гибридизации в условиях низкой жесткости. Для зондирования предпочтительными условиями являются достаточно строгие условия для получения простого паттерна с небольшим количеством гибридизаций, идентифицированных как положительные, которые можно исследовать далее.
Например, скрининг первоначально можно вести в условиях, которые включают температуру, равную приблизительно 37°С или менее, концентрацию формамида, равную менее чем приблизительно 50%, и от средней до низкой концентрации соли (например, стандартный раствор хлорида и цитрата натрия ("SSC")=0.15 М хлорид натрия; 0.15 М натрия цитрат; pH 7).
В качестве альтернативы условия могут включать температуру, равную приблизительно 50°С или менее, и высокую концентрацию соли (например, "SSPE" 0.180 мМ хлорид натрия; 9 мМ вторичный кислый фосфат натрия; 9 мМ первичный кислый фосфат натрия; 1 мМ натрия EDTA; pH 7.4). Предпочтительно, скрининг проводят при приблизительно 37°С, концентрации формамида, равной приблизительно 20%, и концентрации соли, равной приблизительно 5×SSC, или температуре, равной приблизительно 50°С, и концентрации соли, равной приблизительно 2×SSPE. Эти условия позволяют идентифицировать последовательности, которые имеют существенную степень гомологии (схожести, идентичности) с последовательностью зонда, не требуя идеальной гомологии для идентификации стабильного гибрида.
Подходящие условия включают, например, для обнаружения последовательностей, которые приблизительно на 80-90% идентичны, гибридизацию в течение ночи при 42°С в 0.25 М Na2HPO4, pH 7.2, 6.5% ДСН, 10% сульфате декстрана и окончательную промывку при 55°С в 0.1× SSC, 0.1% ДСН. Для обнаружения последовательностей, которые более чем приблизительно на 90% идентичны, подходящие условия включают гибридизацию в течение ночи при 65°С в 0.25 М Na2HPO4, pH 7.2, 6.5% ДСН, 10% сульфате декстрана и окончательную промывку при 60°С в 0.1× SSC, 0.1% ДСН.
В данной области хорошо известно, что следует постепенно повышать жесткость условий гибридизации до тех пор, пока не останется лишь несколько положительных клонов. Когда получают большое число гибридизующихся фрагментов при низкой фоновой гибридизации, считают, что подходящие условия достигнуты.
Используя эти условия, можно проводить поиск в библиотеках нуклеиновых кислот, например в библиотеках кДНК, типичных для экспрессированых последовательностей. Специалисты в данной области смогут легко применить подходящие условия желательной жесткости для избирательной гибридизации, принимая во внимание такие факторы, как длина олигонуклеотида и состав оснований, температура и т.п. Одна общая формула для расчета жесткости условий, необходимых для достижения гибридизации между молекулами нуклеиновых кислот с определенной гомологией последовательностей, представляет собой (Sambrook и др., 1989):
Tm = 81.5°C + 16.6Log [Na+] + 0.41 (% G+C) - 0.63 (% формамида) - 600/#п.о. в дуплексе.
В качестве иллюстрации приведенной выше формулы: если использовать [Na+] = [0.368] и 50-% формамид, при содержании GC, равном 42%, и среднем размере зонда, равном 200 оснований, Тт будет равна 57°С. Tm дуплекса ДНК уменьшается на 1-1.5°С с каждый снижением гомологии на 1%. Таким образом, мишени с более чем приблизительно 75% идентичностью последовательностей можно будет наблюдать при температуре гибридизации, равной 42°С. Такая последовательность будет считаться по существу гомологичной последовательности нуклеиновых кислот согласно настоящему изобретению.
Связывание зонда с целевой нуклеиновой кислотой (например, ДНК) можно измерить, используя любую из множества методик, которые доступны специалистам в данной области. Например, зонды можно снабдить радиоактивной, флуоресцентной или ферментативной меткой. Другие способы, в которых не используется мечение зонда, включают амплификацию с использованием ПЦР (см. ниже) или расщепление РНКазой. После идентификации успешной гибридизации осуществляют выделение гибридизированной нуклеиновой кислоты, которое может включать один или более этапов ПЦР или амплификации вектора в подходящем хозяине.
Таким образом, один вариант реализации данного аспекта настоящего изобретения представляет собой нуклеиновую кислоту, которая включает или состоит по существу из последовательности нуклеотидов, комплементарных последовательности нуклеотидов, гибридизующихся с любой кодирующей последовательностью, приведенной в данной заявке. В качестве альтернативы нуклеиновая кислота согласно данному аспекту может представлять собой нуклеиновую кислоту, гибридизующуюся с последовательностью нуклеотидов, комплементарной любой кодирующей последовательности, приведенной в данной заявке. Разумеется, ДНК, как правило, двунитевая, и методики блоттинга, такие как гибридизация по Саузерну, часто осуществляют после разделения нитей, вне зависимости от того, которая из нитей гибридизируется. Предпочтительно гибридизируемая нуклеиновая кислота или комплементарная ей кодирует продукт, способный влиять на свойство устойчивости растения, в частности на реакцию устойчивости, связанную с Rpi.
В дополнительном варианте реализации гибридизацию молекулы нуклеиновой кислоты с вариантом можно определить или идентифицировать опосредованно, например, применяя реакцию амплификации нуклеиновых кислот, в частности, полимеразную цепную реакцию (ПЦР) (см. "PCR protocols; A Guide to Methods and Applications", ред. Innis и др., Academic Press, Нью-Йорк, (1990)).
Способы, описанные выше, также можно применять для определения присутствия одной из последовательностей нуклеотидов согласно настоящему изобретению в рамках генетического контекста отдельного растения. Это может оказаться полезным для программ разведения растений, например, для непосредственного выбора растений, содержащих аллели, которые отвечают за желательные признаки в этих видах растения либо в родительских растениях, либо в потомстве (например, гибриды, F1, F2 и т.д.).
В данной заявке, если по контексту не требуется иное, термин "нуклеиновая кислота Ppi" включает любую из нуклеиновых кислот согласно настоящему изобретению, описанных выше, включая функциональные варианты.
В одном аспекте настоящего изобретения нуклеиновая кислота Rpi, описанная выше, представлена в виде рекомбинантного и предпочтительно реплицируемого вектора. "Вектор" включает, среди прочего, любую плазмиду, космиду, фаг или бинарный вектор Agrobacterium в дву- или однонитевой линейной или кольцевой форме, который обладает или не обладает способностью самостоеятельно передаваться или активироваться и который обладает способностью трансформировать прокариотического или эукариотического хозяина либо путем встраивания в геном клетки, либо путем внехромосомного присутствия (например, автономно реплицирующаяся плазмида с сайтом начала репликации). В частности, включены челночные векторы, под которыми подразумевается ДНК-носитель, способный, благодаря своей природе или конструкции, реплицироваться в двух различных организмах-хозяевах, которые могут быть выбраны из актиномицетов, и родственных видов, бактерий и эукариот (например, высшего растения, дрожжей или клеток грибка).
Вектор, включающий нуклеиновую кислоту согласно настоящему изобретению, не обязательно включает промотор или другую регуляторную последовательность, в частности, если вектор используется для введения нуклеиновой кислоты в клетки для рекомбинации в геном.
Предпочтительно, нуклеиновая кислота в векторе находится под контролем и функционально связана с подходящим промотором или другими регуляторными элементами, что обеспечивает транскрипцию в клетке-хозяине, такой как микробная, например, бактериальная или растительная клетка. Вектор может представлять собой бифункциональный экспрессионный вектор, который функционирует во множестве хозяев. В случае геномной ДНК нуклеиновая кислота может содержать собственный промотор или другие регуляторные элементы, а в случае кДНК она может находиться под контролем подходящего промотора или других регуляторных элементов для экспрессии в клетке-хозяине.
Под "промотором" подразумевают последовательность нуклеотидов, с которых может инициироваться транскрипция функционально связанной ДНК, находящейся по ходу транскрипции (т.е. в 3'-направлении на смысловой нити двунитевой ДНК). "Функционально связанный" обозначает фрагмент, присоединенный в виде части той же молекулы нуклеиновой кислоты, соответствующим образом расположенный и ориентированный для инициирования транскрипции с указанного промотора. ДНК, функционально связанная с промотором, "находится под регуляцией инициирования транскрипции" указанного промотора.
Таким образом, данный аспект настоящего изобретения предусматривает генную конструкцию, предпочтительно реплицируемый вектор, включающий промотор, функционально связанный с последовательностью нуклеотидов согласно настоящему изобретению, такой как SEQ. ID. 1а, 1b, 2а, 2b или 3, или вариантом указанной последовательности.
В целом, специалисты в данной области легко смогут сконструировать векторы и разработать протоколы для экспрессирования рекомбинантного гена. Подходящие векторы можно выбрать или сконструировать таким образом, чтобы они содержали подходящие регуляторные последовательности, включая последовательности промотора, фрагменты терминатора, последовательности полиаденилирования, последовательности энхансера, маркерные гены и другие последовательности в случае необходимости. Для дополнительных подробностей см., например, Molecular Cloning: a Laboratory Manual: 2oe издание, Sambrook и др., 1989, Cold Spring Harbor Laboratory Press (или более поздние издания этого труда).
Множество известных методик и протоколов для манипулирования нуклеиновой кислотой, например, для получения конструкций нуклеиновых кислот, мутагенеза (см. выше обсуждение в отношении вариантов), секвенирования, введения ДНК в клетки и экспрессирования гена, и анализа белков подробно описаны в Current Protocols in Molecular Biology, второе издание, Ausubel и др. ред., John Wiley & Sons, 1992. Содержание публикаций Sambrook и др. и Ausubel и др. включены в данную заявку посредством ссылки.
В одном варианте реализации данного аспекта настоящего изобретения предусмотрена генная конструкция, предпочтительно реплицируемый вектор, включающий индуцибельный промотор, функционально связанный с последовательностью нуклеотидов согласно настоящему изобретению. Термин "индуцибельный", применительно к промотору, хорошо понятен специалистам в данной области. По существу, экспрессия под контролем индуцибельного промотора "включатся" или повышается в ответ на внешний стимул. Природа стимула может быть различной для разных промоторов. Некоторые индуцибельные промоторы вызывают небольшие или недетектируемые уровни экспрессии (или отсутствие экспрессии) в отсутствии подходящего стимула. Другие индуцибельные промоторы вызывают детектируемую конститутивную экспрессию в отсутствии стимула. Каким бы ни был уровень экспрессии в отсутствии стимула, экспрессия с любого индуцибельного промотора повышается в присутствии подходящего стимула.
Особенно выжными в данном контексте являются конструкции нуклеиновых кислот, которые выполняют функцию векторов для растений. Конкретные процедуры и векторы, ранее используемые с большим успехом в растениях, описаны у Guerineau и Mullineaux (1993) (Plant transformation and expression vectors. B: Plant Molecular Biology Labfax (ред. Croy RRD) Oxford, BIOS Scientific Publishers, стр.121-148).
Подходящие промоторы, которые работают в растениях, включают 35S вируса мозаики цветной капусты (CaMV 35S). Другие примеры описаны на стр. 120 у Lindsey & Jones (1989) "Plant Biotechnology in Agriculture" публ. OU Press, Milton Keynes, Великобритания. Промотор можно выбрать таким образом, чтобы он включал один или более мотивов или элементов последовательности, придающих связанный с развитием и/или тканеспецифичный регуляторный контроль экспрессии. Индуцибельные промоторы растений включают индуцируемый этанолом промотор, описанный у Caddick и др. (1998) Nature Biotechnology 16: 177-180.
Исключительно в качестве примера в SEQ. ID. 1с, 2с и 2d представлены последовательности нуклеотидов, включающие oka1, mcq1.1 и mcq1.2, и последовательности промотора и терминатора, которые можно применять в конструкциях, используемых для трансформирования как картофеля, так и томата.
Может оказаться желательным использование сильного конститутивного промотора. При желании, в конструкцию могут быть включены генетические маркеры селекции, такие как маркеры, придающие селектируемые фенотипы, как, например, устойчивость к антибиотикам или гербицидам (например, к канамицину, гигромицину, фосфинотрицину, хлорсульфурону, метотрексату, гентамицину, спектиномицину, имидазолинонам и глифосату).
Настоящее изобретение также обеспечивает способы, включающие введение такой конструкции в клетку-хозяина, в частности в растительную клетку.
В дополнительном аспекте настоящего изобретения описана клетка-хозяин, содержащая гетерологичную конструкцию согласно настоящему изобретению, в частности, клетка растения или микроорганизма. Термин "гетерологичный" широко используется в данном аспекте для указания на то, что обсуждаемый ген/последовательность нуклеотидов (ген Rpi) был внедрен в указанные клетки растения или его предшественника с помощью генной инженерии, т.е. путем вмешательства человека. Гетерологичный ген может заместить эндогенный эквивалентный ген, т.е. такой ген, который в норме выполняет такую же или близкую функцию, или встроенная последовательность может быть дополнительной по отношению к эндогенному гену или другой последовательности.
Нуклеиновая кислота, гетерологичная по отношению к растительной клетке, может представлять собой нуклеиновую кислоту, не встречающуюся в природе в клетках такого типа, сорта или вида. Таким образом, гетерологичная нуклеиновая кислота может включать кодирующую последовательность или может быть получена из конкретного типа клеток растения, или видов, или сортов растения, помещенную в клетку растения другого типа или вида, или сорта растения. Дополнительной возможностью является помещение последовательности нуклеиновой кислоты в клетку, в которой она или ее гомолог присутствует в природе, но при этом указанная последовательность нуклеиновой кислоты связана и/или прилегает к нуклеиновой кислоте, которая не встречается в природе в клетке, или клетки этого типа или вида, или сорта растения, например, она функционально связана с одной или более регуляторными последовательностями, такими как последовательность промотора, для контролирования экспрессии.
Клетку-хозяина (например, растительную клетку) предпочтительно трансформируют некоторой конструкцией, при этом говорят, что конструкцию вводят в клетку, что приводит к изменению одного или более свойств (особенностей, признаков) данной клетки и, следовательно, фенотипа, например, в отношении Р. infestans.
Нуклеиновую кислоту можно трансформировать в клетки растения с применением любой подходящую методики, такой как применение нейтрализованного Ti-плазмидного вектора, который несет Agrobacterium, используя его природную способность к переносу генов (ЕР-А-270355, ЕР-А-0116718, NAR 12(22) 8711-87215 1984), бомбардировка частицами или микрочастицами (US 5100792, ЕР-А-444882, ЕР-А-434616), микроинъекция (WO 92/09696, WO 94/00583, ЕР 331083, ЕР 175966, Green и др. (1987) Plant Tissue and Cell Culture, Academic Press), электропорация (ЕР 290395, WO 8706614 Gelvin Debeyser), другие виды непосредственного поглощения ДНК (DE 4005152, WO 9012096, US 4684611), опосредованное липосомами поглощение ДНК (например, Freeman и др. Plant Cell Physiol. 29: 1353 (1984)), или метод встряхивания (например, Kindle, PNAS U.S.A. 87: 1228 (1990d). Физические способы трансформирования клеток растения рассмотрены у Oard, 1991, Biotech. Adv.9: 1-11.
Исключительно в качестве примера, ниже в данной описании в Примере 6 приведены стратегии трансформирования для картофеля, томата и табака приведены далее в данной заявке. Другие стратегии, особенно подходящие для ролда Solanum, хорошо известны специалистам в данной области (см., например, Mansure и Magioli, Acta Botanica Brasilica, 2005 (том 19) (№1) 139-148). Выбор конкретной методики трансформации будет определяться ее эффективностью для трансформирования определенных видов растений, а также опытом и предпочтением человека, применяющего на практике настоящее изобретение с помощью конкретной выбранной методики. Для специалиста будет очевидно, что выбор конкретной системы трансформации для введения нуклеиновой кислоты в клетки растения не является определяющим или ограничивающим настоящее изобретение, так же как и выбор методики регенерации растения. Таким образом, дополнительный аспект настоящего изобретения обеспечивает способ трасформации клетки растения, включающий введение конструкции, описанной выше, в клетку растения и создание условий, обеспечивающих или допускающих рекомбинацию между вектором и геномом клетки растения, для введения нуклеиновой кислоты согласно настоящему изобретению в геном клетки.
В объем настоящего изобретения дополнительно входит клетка-хозяин, трансформированная нуклеиновой кислотой или вектором согласно настоящему изобретению, в частности клетка растения или микроорганизма. В клетке трансгенного растения (т.е. трансгенного по обсуждаемой нуклеиновой кислоте) указанный трансген может находиться на внегеномном векторе или может быть внедрен, предпочтительно стабильно, в геном. Может присутствовать более чем одна гетерологичная последовательность нуклеотидов на гаплоидный геном.
В целом, после трасформации растение можно регенерировать, например, из одной клетки, каллусной ткани или листовых дисков, что является стандартной методикой в данной области. Почти любое растение можно полностью регенерировать из клеток, тканей и органов указанного растения. Доступные методики описаны в Vasil и др., Cell Culture and Somatic Cell Genetics of Plants, тома I, II и III, Laboratory Procedures and Their Applications, Academic Press, 1984, и Weissbach и Weissbach, Methods for Plant Molecular Biology, Academic Press, 1989.
Также предусмотрены растения, которые включают клетку растения согласно настоящему изобретению.
Вдобавок к регенерированному растению настоящее изобретение включает все следующее: клон такого растения, потомство и потомков, полученные путем самоопыления или скрещивания (например, потомков F1 и F2), и любую часть любого из перечисленных вариантов. Настоящее изобретение также предусматривает части таких растений, например любую часть, которую можно использовать для репродукции или размножения, полового или неполового, включая побеги, семена и т.п., или которая может быть сама по себе представлять продукт, например клубень.
Настоящее изобретение дополнительно обеспечивает способ влияния или воздействия на степень устойчивости растения к патогену, в частности к Phytophthora infestans, а конкретнее к любой из культур, обсуждаемых в данной заявке, причем указанный способ включает этап обеспечения условий, вызывающих или допускающих экспрессию гетерологичной последовательности нуклеиновых кислот, обсуждаемых выше, в клетках указанного растения.
Этому этапу может предшествовать более ранний этап введения нуклеиновой кислоты в клетку растения или ее предшественник.
Предпочтительными растениями для трасформации являются растения семейства Solanaceae, более предпочтительно рода Solanum. Возможно, растение может представлять собой S. tuberosum или S. lycopersicum.
Указанные способы также могут включать манипуляции другими генами, например, которые могут задействованы в передаче сигнала устойчивости или в формировании ответа устойчивости.
Таким образом, предусмотрен способ влияния или воздействия на степень устойчивости растения к Р. infestans, причем указанный способ включает этап сохдания условий, вызывающих или допускающих экспрессию гетерологичной нуклеиновой кислоты, описанной выше, в клетке растения.
В предпочтительных способах, в указанное растение вводят более чем один ген Rpi. В других стратегиях предусмотрено множество растений, каждое из которых включает отличный эндогенный или гетерологичный ген Rpi (при этом по меньшей мере одно из указанных растений включает гетерологичный ген Rpi согласно настоящему изобретению, т.е. полученный с помощью технических способов, описанных выше). Множество растений можно посадить вместе на одном участке, чтобы максимизировать степень или продолжительность устойчивости сельскохозяйственной культуры к Р. infestans. В качестве альтернативы множество растений можно сажать последовательно на этом участке (например, с чередованием), чтобы достичь такого же эффекта.
Предшествующее описание, в целом, было посвящено применению нуклеиновых кислот согласно настоящему изобретению для получения функциональных полипептидов Rpi в растении, с обеспечением повышения его устойчивости к патогену. Исключительно для полноты описания следует отметить, что информацию, приведенную в данной заявке, также можно использовать для снижения активности или уровней таких полипептидов в клетках, в которых желательно это сделать (например, в экспериментальной модели). Нуклеиновые кислоты и основанные на них методики для осуществления отрицательной регуляции (например, комплементарные последовательности) являются частью настоящего изобретения.
Выше отмечено, что в объем настоящего изобретения также входит продукт экспрессии любой из последовательностей нуклеиновых кислот Rpi (особенно функционального Rpi), описанных выше, а также способы получения продукта экспрессии путем экспрессии кодирующей нуклеиновой кислоты при подходящих для этого условиях, которая может происходить в подходящих клетках-хозяевах.
Предпочтительный полипептид включает последовательность аминокислот, представленную в SEQ. ID. 4a, 4b, 5a, 5b, или 6. Тем не менее, полипептид согласно настоящему изобретению может представлять собой вариант (аллель, фрагмент, производное, мутант или гомолог и т.д.) указанных полипептидов.
Также в объем настоящего изобретения входят полипептиды, которые хотя и явно родственны функциональным полипептидам Rpi (например, они проявляют иммунологическую кросс-реактивность с указанным полипептидом или включают характерные мотивы в последовательности, общие с указанным полипептидом), но не имеют функции Rpi.
После экспрессии рекомбинантный продукт можно, в случае необходимости, выделить из системы экспрессии. Как правило, тем не менее, полипептиды согласно настоящему изобретению используют in vivo (в частности, in planta).
Очищенные белки Rpi или варианты белков согласно настоящему изобретению, которые получали путем рекомбинанантной экспрессии кодирующей нуклеиновой кислоты, можно применять для получения антител, используя методики, которые являются стандартными в данной области. Способы получения антител включают иммунизацию млекопитающего (например, мыши, крысы, кролика, лошади, козы, овцы или обезьяны) белком или его фрагментом. Антитела могут быть получены из иммунизированых животных, с использованием любой из множества методик, известных в данной области, и можно осуществить скрининг, предпочтительно по связыванию антитела с целевым антигеном. Например, можно использовать методики вестерн-блоттинга или иммунопреципитации (Armitage и др., 1992, Nature 357: 80-82). Антитела могут быть поликлональными или моноклональными. В качестве альтернативы или дополнения к иммунизированию млекопитающего антитела с подходящей специфичностью связывания могут быть получены из полученной рекомбинантным методом библиотеки экспрессированных вариабельных доменов иммуноглобулинов, например, применяя бактериофаг лямбда или нитевидный бактериофаг, которые выставляют на поверхностях функциональные домены связывания иммуноглобулинов; например, см. WO 92/01047.
Антитела, полученные в ответ на введение полипептида или пептида, можно применять для идентификации и/или выделения гомологичных полипептидов, а затем кодирующих генов. Таким образом, согласно настоящему изобретению предложен способ идентификации или выделения полипептида с функцией Rpi (в соответствии с вариантами реализации, описанными в данной заявке), включающий скрининг кандидатных пептидов или полипептидов с помощью полипептида, включающего антигенсвязывающий домен антитела (например, целое антитело или его фрагмент), который способен связывать пептид, полипептид Rpi или его фрагмент или вариант, или предпочтительно имеет специфичность связывания с таким пептидом или полипептидом, который имеет последовательность аминокислот, определенную в данной заявке.
Специфично связывающие агенты, такие как антитела и полипептиды, включая антигенсвязывающие домены антител, которые связывают и предпочтительно специфичны к полипептидам с последовательностями SEQ. ID. 4а, 4b, 5а, 5b, или 6 или мутантным вариантам, вариантам или производным указанных последовательностей, а также их применение и способы, в которых они используются, представляют собой дополнительные варианты воплощения настоящего изобретения.
Приведенное выше описание было, в целом, посвящено транслированным и кодирующим частям генов Rpi. Также в настоящее изобретение включены нетранскрибируемые части (НТО) указанных генов.
Таким образом, дополнительный аспект настоящего изобретения представляет собой изолированную молекулу нуклеиновой кислоты, кодирующую промотор, или другую НТО (3' или 5'), гена Rpi, описанного в данной заявке.
Выше было отмечено, что в последовательностях SEQ. ID. 1с, 2с и 2d представлены последовательности нуклеотидов oka1, mcq1.1 и mcq1.2 и включены последовательности промотора и терминатора, которые можно применять в конструкциях, используемых для трасформации как картофеля, так и томата.
Таким образом, видно, что авторы настоящего изобретения выделили, идентифицировали и описали несколько различных R-генов фитофтороза пасленовых, полученных из картофеля из диких видов S. okadae, а также из S. mochiquense и S. neorossii. Соответственно, согласно настоящему изобретению предложены новые последовательности генов, композиции и способы повышения устойчивости у сельскохозяйственных культур, в частности, но не ограничиваясь, у картофеля, к фитофторозу пасленовых, вызванному оомицетным патогеном Phytophthora infestans.
Для того чтобы клонировать указано R-гены фитофтороза пасленовых, была разработана новая комбинация различных методик. В Примерах ниже более подробно описано, что авторы изобретения создали две ВАС-библиотеки из геномной ДНК двух видов. В настоящем описании описано конструирование и анализ двух ВАС-библиотек. Кроме того, были идентифицированы и описаны ВАС-клоны, связанные с R-генами фитофтороза пасленовых, применяя основанные на ПЦР маркеры, разработанные во время предварительных экспериментов по картированию. Этот процесс облегчил тонкое картирование R-генов, поиск целевых генов в хромосоме, физическое картирование и, наконец, клонирование генов.
Конструирование библиотек с большими вставками геномной ДНК представляет собой один из существенных этапов стратегий клонирования генов, основанных на картировании. Некоторые способы были разработаны для конструирования библиотек, включая искусственную дрожжевую хромосому (YAC), искусственную хромосому фага Р-1 (РАС), клон, основанный на плазмиде (РВС), подходящую для трасформации искусственную хромосому растения (ТАС), бактериальную искусственную хромосому (ВАС) и бинарную бактериальную синтетическую хромосому (BIBAC) (описаны у Feng и др. 2006). В течение последних нескольких лет были созданы ВАС-библиотеки на основе различных видов растений, включая основные сельскохозяйственные культуры: рис, пшеницу и картофель (Тао и др. 2002; Nilmalgoda и др. 2003; Chen и др. 2004), и другие виды, таких как персик, чеснок, банан, сахарная свекла, соевый боб, арахис и подсолнечник (Georgi и др. 2002; Lee и др. 2003; Vilarinhos и др. 2003; McGrath и др. 2004; Wu и др. 2004; Yuksel и др. 2005; Bouzidi и др. 2006; Feng и др. 2006).
Мы работали над выделением генов из диких видов S. okadae и S. mochiquense, придающих устойчивость к фитофторозу пасленовых у картофеля, применяя основанные на картировании подходы к клонированию генов. О гене, полученном из S. mochiquense, уже сообщали ранее, однако до настоящего момента он не был выделен (Smilde и др. 2005). Также недавно были идентифицированы гены, полученные из S. okadae (Foster и др. неопубликованные результаты). Шагом по направлению к основанному на картировании клонированию этих R-генов явилось создание авторами настоящего изобретения двух ВАС-библиотек из К39, включающих Rpi-oka1 и Rpi-oka2, и из К182, включающего Rpi-mcq1. Для создания высококачественных ВАС-библиотек решающей является оптимизация условий частичного гидролиза и тщательный выбор по размеру частично расщепленных фрагментов ДНК. Меньшие фрагменты часто позволяют получить клоны с меньшими вставками с более высокой эффективностью трасформации, но большие фрагменты часто обеспечивают более высокий процент клонов, в которых отсутствуют вставки, и более низкую эффективность трасформации (Feng и др. 2006). В данном исследовании для ВАС-библиотек были отобраны фрагменты размером 100-200 т.п.н.
Размер гаплоидного генома видов Solanum лежит в диапазоне от 800 Мб до 1,200 Мб в зависимости от видов. Согласно Arumuganathan и Earie (1991), размер гаплоидного генома S. berthaultii составляет 840 Мб, а у S. tuberosum составляет 800-930 Mb. В настоящем исследовании в общей сложности 105,216 и 100,992 ВАС-клонов со средними размерами вставок 103.5 т.п.н. и 85.5 т.п.н. получали для библиотек К39 и К182, соответственно. Исходя из того, что размер гаплоидного генома картофеля составляет 1,000 Мб, мы оценили, что эти библиотеки содержат приблизительно 10.9 и 8.6 геномных эквивалентов для К39 и К182 библиотек, соответственно. Хотя отбирали фрагменты ДНК в диапазоне 100-200 т.п.н., средние размеры вставок для обеих библиотек были меньше, чем ожидалось. Это несоответствие наблюдалось и другими исследователями (Danesh и др. 1998; Meksem и др. 2000; Yuksel и Paterson 2005) и могло быть вызвано присутствием меньших фрагментов, которые не были полностью удалены на этапах отбора по размеру, что предположил Frijters и др. (1997). Учитывая покрытие генома, ожидали, что все участки генома должны быть хорошо представлены. Авторы проверили это, применяя основанные на ПЦР маркеры, про которые известно, что они связаны с R-генами Rpi-oka1, Rpi-oka2 и Rpi-mcq1.
Для того чтобы свести к минимуму количество требующихся реакций ПЦР, мы использовали стратегию объединения в пул для скрининга библиотек. Ранее отличные стратегии объединения в пул использовались для скрининга ВАС-библиотек (Klein и др. 2000; Ozdemir и др. 2004; Bouzidi и др. 2006). В нашем исследовании мы использовали стратегию объединения пула с планшета вместе со стратегией объединения пула колонок и рядов планшета. Объединение в пул осуществляли для каждого 384-луночного планшета и получали плазмидную ДНК из каждого пула. С учетом геномных эквивалентов каждой библиотеки теоретически мы ожидали, что 11 и 9 пулов будут положительными по конкретному маркеру и что половина этих пулов после расщепления рестриктазами для идентифицирования устойчивых аллель-специфичных маркеров будет содержать ВАС-клоны из гаплотипа, соответствующего каждому гену.
Положительные по основанным на ПЦР маркерам ВАС-клоны в библиотеках К39 и К182 соответствовали или были несколько лучше, чем предполагаемые геномные эквиваленты, и были идентифицированы в средних количествах 15 и 12.5, соответственно. Оба этих количества были несколько большими, чем ожидаемые количества на основании оценки геномных эквивалентов. Это могло быть вызвано переоценкой размера гаплоидного генома картофеля или недооценкой средних размеров вставок полученных ВАС-клонов. С другой стороны, ВАС-библиотеки, которые мы сконструировали, могут отличаться вследствие пере- или недопредставления сайтов для HindIII в пределах интересующего участка. Для того чтобы достигнуть лучшего представления, другие исследователи при конструировании ВАС-библиотек использовали две или три различные рестриктазы, богатые либо АЛ, либо G/C (Chang и др. 2001; Тао и др. 2002; Chen и др. 2004).
На основании результатов скрининга ВАС с помощью ПЦР-маркеров, связанных с генами Rpi, мы секвенировали ВАС-концы восьми отдельных ВАС-клонов для каждой библиотеки. Из них один идентифицированный из каждой из библиотек К39 и К182 был аналогичен Tm-22, R-гену вируса мозаики томата на хромосоме 9 томата (Lanfermeijer и др. 2003). Дополнительно две другие ВАС-концевые последовательности из библиотеки К182 были близки нескольким различным белкам устойчивости. Эти результаты, объединенные с генетическими картами связей Rpi-oka1, Rpi-oka2 и Rpi-mcq1, созданными в дополнительных исследованиях авторов настоящего изобретения (Foster и др. неопубликовано; Zhu и др. неопубликовано), указывают на то, что мы идентифицировали ВАС-клоны, которые покрыли участок генома, содержащий указанные гены.
ВАС-библиотеки с большими вставками представляют собой важный инструмент для поиска в хромосоме (т.н. «прогулке»), конструирования ВАС-контига и физического картирования участков, содержащих R-гены. Хотя точное физическое расположение R-генов не было определено, как показано в результатах скрининга ВАС с помощью подхода на основе ПЦР и ВАС-концевых последовательностей отобранных ВАС-клонов, конструирование ВАС-библиотек, покрывающих в 10.9 и в 8.6 раз гаплоидный геном картофеля, из S. okadae и S. mochiquense облегчило клонирование генов Rpi и будет представлять ценность для дополнительных исследований генома картофеля, которые требуют основанные на картировании этапы клонирования.
После описания настоящего изобретения в общих чертах, включая способы получения и применения композиций, полезных для придания устойчивости к фитофторозу пасленовых, ниже приведены примеры, дополняющие и обеспечивающие воплощение настоящего изобретения, включая лучший вариант воплощения и его эквиваленты. Тем не менее, для специалиста в данной области очевидно, что настоящее изобретение, которое иллюстрируют эти примеры, не ограничено подробностями примеров, приведенных в данной заявке. Для понимания объема настоящего изобретения, следует обратиться к формуле изобретения, приложенной к настоящему описанию.
ПРИМЕРЫ
ПРИМЕР 1. КОНСТРУИРОВАНИЕ ВАС-БИБЛИОТЕК ИЗ ДИКИХ ВИДОВ КАРТОФЕЛЯ SOLANUM OKADAE И SOLANUM MOCHIQUENSE И ИДЕНТИФИКАЦИЯ КЛОНОВ ОКОЛО ЛОКУСОВ УСТОЙЧИВОСТИ К ФИТОФТОРОЗУ ПАСЛЕНОВЫХ
а. Материалы растений
Растения, используемые для создания двух ВАС-библиотек, показаны на Фиг.1. Растение К39 S. okadae получены из трансгетерозиготы, несущей как Rpi-oka1, происходящий из родительского А618, так и Rpi-oka2 из А624 (Foster и др. неопубликованные результаты). Растение К182 S. mochiquense гетерозиготно по Rpi-mcq1 (ранее называемому Rpi-moc1; Smilde и др. 2005), и его получали из популяции ВС1.
b. Получение высокомолекулярной вставки ДНК
Способ, используемый для получения высокомолекулярной (ВМ) ДНК, был в некоторой степени модифицирован Liu и Whittier (1994) и Chalhoub и др. (2004). Растительный материал выращивали in vitro на среде Мурасиге-Скуга (MS) без сахарозы, ткани молодых листьев собирали и хранили при -80°С. Двадцать грамм замороженной ткани листьев использовали для получения сгустков ДНК, содержащих ВМ (высокомолекулярную) ДНК. Сгустки ДНК получали в 0.7% агарозе inCert (Biozym, Ольдендорф, Германия), промывали лизирующим буферным раствором (1% лаурилсаркозин натрия, 0.2 мг/мл протеиназы К и 3.8 мг/мл диэтилдитиокарбамата натрия, растворенными в 0.5 М EDTA, pH 8.5) и хранили при 4°С в 0.5 М EDTA до того момента, когда они требуются, без снижения качества ДНК, согласно предположению Osoegawa и др. (1998). Сгустки после хранения пропитывали буфером ТЕ, мелко рубили и частично расщепляли 5 единицами рестриктазы HindIII в течение 1 часа, на основании результатов предшествующих экспериментов по оптимизаци, которые показали, что эти условия позволяли получить ДНК с размером в диапазоне 50-300 т.п.н.
Осуществляли трехкратный отбор по размеру, чтобы улучшить размер и однородность вставок, как описано у Chalhoub и др. (2004). Первый отбор по размеру выполняли на 1% агарозе Seakem LE (Biozym, Ольдендорф, Германия), используя пульс-электрофорез в контролируемом гомогенном электрическом поле (CHEF) (Bio-rad, Геркулес, США) при 1-40 секундах, 120°, 16 часах и 200 В в 0.25х буфере ТВЕ, за тем непосредственно следовал второй отбор по размерам в том же геле при 4-5 секундах, 120°, 6 часах и 180 В в том же буфере. Участки геля, содержащие частично расщепленную ДНК размером между 100 и 200 т.п.н., вырезали и разделяли пополам. Для третьего отбора по размеру вырезанные кусочки геля отдельно прогоняли на 1 % агарозе с низкой температурой плавления Sea Plaque GTG (Biozym, Ольдендорф, Германия) при 3-4.5 секундах, 120°, 14 часах и 180 В. Отобранные по размеру фрагменты ДНК вырезали из геля и хранили при 4°С в 0.5 М EDTA (pH 8). ДНК извлекали в 40 мкл 1х буфера ТАЕ путем электроэлюции, применяя систему электроэлюции BioRad (Bio-rad, Геркулес, США).
с. Конструирование ВАС-библиотеки
Лигирование и трансформирование выполняли согласно методам, описанным у Allouis и др. (2003) и Chalhoub и др. (2004), с небольшой модификацией. Тотальную элюированную ДНК из отобранных по размеру фрагментов ДНК лигировали в 100 мкл реакционной смеси с 10 нг вектора plndigoBAC-5 (Epicentre Biotechnologies, Мэдисон, США) и 800 ед. ДНК-лигазы Т4 (New England Biolabs, Ипсуич, США). Лигазную смесь диализировали против 0.5 × буфера ТЕ в течение 3 часов, применяя мембрану Millipore (Millipore, Биллерика, США). Три микролитра диализированной смеси для лигирования смешивали с 20 мкл электрокомпетентных клеток ElectroMax DH10B (Invitrogen, Пейсли, Великобритания), инкубировали в течение 1 минуты на льду и подвергали электропорации при 180 В, 200 Ом и 25 мкФ. Трансформированные клетки вносили в 1 мл среды SOC (Invitrogen, Пейсли, Великобритания), инкубировали при 37°С в течение 1 часа, высевали на избирательную среду LB с 17 мкг/мл хлорамфеникола, 125 мкг/мл IPTG (изопропилтио-β-D-галактозида) и 100 мкг/мл X-Gal (5-бром-4-хлор-3-индолил-β-D-галактозида) и растили при 37°С в течение ночи. Белые колонии переносили в 384-луночные микротитрационные планшеты (Genetix Ltd., Дорсет, Великобритания), содержащие питательную среду для замораживания (1% триптон, 0.5% дрожжевой экстракт, 0.5% NaCl, 0.63% K2HPO4, 0.045% цитрат натрия, 0.009% MgSO4, 0.09% (NH4)2SO4, 0.18% KH2PO4, 4.4% глицерин и 17 мкг/мл хлорамфеникола, pH 7.2), применяя прибор Q-Pix (Genetix Ltd., Дорсет, Великобритания), инкубировали при 37°С в течение ночи и хранили при -80°С.
d. Определение размера вставки в ВАС
Чтобы определить размер вставки в ВАС-клонах, произвольно отобранные ВАС-клоны культивировали в 3 мл LB, содержащей 17 мкг/мл хлорамфеникола, при 37°С в течение ночи. ВАС-ДНК изолировали, применяя в некоторой степени модифицированный способ относительно предложенного в наборе Qiagen plasmid midi (Qiagen Ltd, Кроули, Великобритания) и расщепляли рестриктазой Nott в течение 3 часов для высвобождения вставки ДНК из вектора. Расщепленную ДНК разделяли на 1% агарозном геле, применяя гель-электрофорез в CHEF (Bio-rad, Геркулес, США) при 5-15 секундах, 120°, 16 часах и 200 В в 0.5× буфере ТВЕ.
е. Скрининг ВАС-библиотеки и описание ВАС-клонов
ВАС-клоны, которые хранили в отдельном 384-луночном планшете, объединяли в пулы и получали плазмидную ДНК из каждого пула. По объединенной в пул ДНК осуществляли скрининг с помощью восьми ПЦР-маркеров (Таблица 1а), про которые известно, что они связаны с идентифицированными генами Rpi. После того как положительные пулы были идентифицированы с помощью определенных маркерных праймеров, делали реплику исходного 384-луночного планшета с библиотекой на твердой среде LB при помощи инструмента для получения реплик высокой плотности и осуществляли скрининг рядов и колонок клонов с помощью ПЦР, применяя те же праймеры для выбора единичных положительных клонов. Секвенировали ВАС-конец отобранных положительных клонов, применяя реагенты для циклического секвенирования Big Dye v.3.1 (Applied Biosystems, Фостер-Сити, США). Реакции секвенирования вели на ABI 3730 в лаборатории John Innes Centre Genome Laboratory (Норидж, Великобритания).
Дополнительно, объединенную в пул плазмидную ДНК из ВАС-пулов библиотеки К39 подвергали спот-блоттингу на мембране Hybond-N+ и зондировали путем гибридизации с 32Р-меченым маркером okaNBS-Hae в качестве зонда. 384-луночные планшеты с ВАС, соответствующие пулам, идентифицированным с помощью этого зонда, затем наносили двойными пятнами на мембрану Hybond-N+ и гибридизировали с тем же зондом, чтобы идентифицировать отдельные ВАС-клоны из пулов. ВАС-ДНК изолировали из идентифицированных ВАС-клонов и применяли метод "отпечатков пальцев" SNaPshot для создания контигов из ВАС, содержащих последовательности, гомологичные зонду. Отобранные ВАС-клоны, которые были положительными при ПЦР с выбранными маркерными праймерами (TG551 и TG35), также включали в анализ SNaPshot.
f. Результаты. Конструирование и описание ВАС-библиотеки
С целью выделения R-генов фитофтороза картофеля создали две ВАС-библиотеки из двух растений - К39 и К182 (Фиг. 1). Результаты ауткросса с восприимчивым генотипом S. okadae указывают на то, что К39 трансгетерозиготно по Rpi-oka1 и Rpi-oka2. Анализ фенотипа и генотипов растений, происходящих от К182, указывает на то, что К182 гетерозиготно по Rpi-mcq1.
Две ВАС-библиотеки были созданы из частично расщепленной HindIII ДНК картофеля. Библиотеки из К39 и К182 состояли из 105,216 и 100,992 клонов, которых хранили в 274×384- и 263×384-луночных микротитрационных планшетах, соответственно. Средние размеры вставок оценивали на основании анализа геля после электрофоретического разделения в пульсирующем поле расщепленной NotI ДНК из 38 и 40 произвольно отобранных клонов из библиотек К39 и К182, соответственно. Паттерны расщепленных NotI ДНК клонов из указанных двух библиотек представлены на Фиг.2. Оцененные размеры вставок лежали в диапазоне от 60 до 165 т.п.н., при этом средний размер составлял 103.5 т.п.н., для библиотеки К39 и от 50 до 130 т.п.н., при этом средний размер составлял 85.5 т.п.н. для библиотеки К182. Размер гаплоидного генома картофеля по нашей оценке составил приблизительно 1,000 Мб, следовательно, предположили, что эквивалентов генома всего 10.9× и 8.6× для библиотек К39 и К182, соответственно.
ПРИМЕР 2. ИДЕНТИФИКАЦИЯ, КАРТИРОВАНИЕ И КЛОНИРОВАНИЕ ГЕНОВ Rpi ИЗ Solanum okadae и S. neorossii
а. Условия роста растений
Семена от 12 Solanum okadae и 4 S. neorossii (Таблица 1b) получали из Центра генетических ресурсов в Вагенингене, Нидерланды (CGN). Поверхность семян стерилизовали в 70% этаноле в течение 1 минуты, дезинфицировали 1.5% гипохлоритом в течение 5 минут, ополаскивали 3 раза в стерильной дистиллированной воде и помещали на твердую среду MS (среду Мурасиге-Скуга) (2% агароза), содержащую 3% сахарозы, для проращивания. Пророщенную рассаду переносили в парниковое помещение и регулярно обрабатывали фунгицидами и пестицидами, чтобы не допустить развития трипсов, афид, паутинного клеща, настоящей мучнистой росы и альтернариоза (Alternaria solani).
b. Штаммы Phytophthora infestans, заражение и оценка в патологическом тесте
Изолят Р. infestans 98.170.3 (штамм 1.3.4.10.11; Smilde и др. 2005) был предоставлен Dr David Shaw из Университета Бангора, Великобритания. Культура 90128 (штамм 1.3.4.7.8.9.10.11), IPO-complex (штамм 1.2.3.4.6.7.10.11), IPO-0 (спектр вирулентности неизвестен) и ЕС1 (штамм 3.4.7.11) были предоставлены Dr Edwin van der Vossen из Plant Research International, Вагенинген, Нидерланды. Изолят SuperBlight был предоставлен Dr Paul Birch, SCRI, Данди, Великобритания, и представляет собой изолят, на сегодняшний день вирулентный для большого количества выращиваемых в промышленных масштабах культурных сортов картофеля в Великобритании и Европе. Изоляты МР324, МР717, МР778, МР674, МР622, МР618 и МР650 получали из IHAR, Польша.
Изоляты поддерживали на 18°С на агаре Rye В. Свежий спорангий получали два раза в неделю путем субкультивирования на свежих чашках. Периодически подтверждали способность изолятов инфицировать материал хозяина на отжеленных листьях подходящего, восприимчивого растения. Зрелый, свежий спорангий собирали после 10 дней роста на среде Rye В, при этом наполняли чашку стерильной деионизированной водой и оставляли собранную суспензию спор на 20 минут в свежей чашке Петри. После этого большая часть спорангия налипала на пластиковые поверхности чашки. Воду из исходной суспензии заменяли на свежую холодную воду, спорангий ресуспендировали и инкубировали при 4°С в течение от 1 до 4 часов, чтобы индуцировать высвобождение зооспоры.
Тест с отделенным листом использовали для скрининга на устойчивость к Р. infestans (модифицированный у (Vleeshouwers и др. 1999)). Отсоединяли по два листа от каждого растения, вставляли в небольшую часть влажной флористической губки и помещали в 9 см чашку Петри. Листья заражали каплями по 10 мкл суспензии зооспор (от 20,000 до 50,000 зооспор мл-1) и инокулят осторожно распределяли по нижней поверхности листа с помощью художественной кисти. Чашки Петри заворачивали в полимерную пленку и инкубировали в течение от 7 до 12 дней в контролируемых условиях окружающей среды (18°С; цикл 18 ч света/6 ч темноты) перед оценкой фенотипов. Растения с листьями, на которых были видны повреждения в процессе спорообразования, оценивали как восприимчивые; растения с листьями, на которых отсутствовали видимые симптомы или некроз и отсутствовало спорообразование, оценивали как устойчивые. Если у двух листьев была различная реакция, фенотип растения рассматривали как промежуточный (слабая устойчивость). Для подтверждения этих промежуточных фенотипов осуществляли по меньшей мере три независимых заражения. Для ярковыраженных фенотипов (либо оба листа устойчивы либо оба восприимчивы) два независимых раунда заражений считали достаточными.
с. Выделение ДНК
ДНК выделяли из материала растений, применяя либо набор DNeasy 96 Plant (Qiagen) либо протокол из (Park и др. 2005). Вкратце, приблизительно 50 мг материала листьев собирали в 250 мкл лизирующего ядра буфера (200 мМ Tris-HCl pH 7.5, 50 мМ EDTA, 2 М NaCl, 2% СТАВ), к которому добавляли 200 мкл буфера для экстрагирования ДНК (100 мМ Tris-HCl pH 7.5, 350 мМ сорбит, 20 мМ бисульфит натрия). Материал листьев затем имельчали, применяя размалывающий прибор Retsch MM300 с двумя 3 мм стальными шарикоподшипниками для каждого образца, и инкубировали при 65°С в течение 1 часа. Добавляли двести пятьдесят микролитров ледяного хлороформа, образцы размешивали и центрифугировали на 3500 об/мин в течение 10 минут. Супернатант переносили в чистую пробирку и ДНК преципитировали путем добавления равного объема изопропанола, а затем центрифугировали на 3500 об/мин в течение 60 мин. Осажденную ДНК сушили на воздухе и ресуспендировали в 100 мкл ТЕ.
d. Анализ ПДАФ и SSR и основанное на ПЦР картирование
Анализ полиморфизма длин амплифицированных фрагментов (ПДАФ) выполняли по существу как описано у (Thomas и др. 1995) и (Vos и др. 1995) на расщепленной PstI/MseI матрице ДНК, используя этап предварительной амплификации с праймерами PstI+0 и MseI+1 и этап избирательной амплификации, применяя праймеры PstI+2 и MseI+3. Продукты реакции ПДАФ денатурировали и разделяли с помощью электрофореза на геле с 4.5% акриламидом/7.5 М мочевиной/0.5 × ТВЕ (45 мМ Tris-борат, 1 мМ EDTA), который вели на 100 Вт в течение 2.5 ч. После электрофореза гели переносили на 3 мм бумагу Whatman, сушили без фиксации и экспонировали на рентгеновскую пленку (Х-ОМАТ AR, Kodak) в течение 1-7 дней.
Информативные полоски ПДАФ вырезали из геля и регидратировали в ТЕ (10 мМ Tris-HCI pH 8.0, 0.1 мМ EDTA). Кусочки геля затем переносили в свежий ТЕ, измельчали и обломки удалял путем центрифугирования при 14000 g в течение 1 мин. Для клонирования фрагменты ПДАФ сначала снова амплифицировали с помощью ПЦР, используя 2 мкл супернатанта и те же условия цикла и праймеры, что и для исходной амплификации. Полученные продукты клонировали в pGEM-T Easy (Promega, Мэдисон, Висконсин), следуя инструкциям производителя, и секвенировали, применяя набор ABI PRISM Big Dye (v.3.1) Terminator Cycle Sequencing Ready Reaction (PE Applied Biosystems), следуя инструкциям производителя.
Реакции микросателлитной (SSR) ПЦР вели в объеме 25 мкл, содержащем 20 мМ Tris-HCI (pH 8.4), 50 мМ KCl, 2.5 мМ MgCl2, 0.4 мМ каждого из dCTP, dTTP и dGTP, 0.012 мМ немеченого dATP, 370 кБк [□-33P)]dATP (Amersham Biosciences), 0.4 мкМ каждого праймера, 1 ед. ДНК-полимеразы Taq (Invitrogen, Карлсбад, Калифорния) и 100 нг матричной ДНК. Температурные условия циклов включали первоначальный этап денатурации при 94°С в течение 4 мин, затем этап отжига праймеров (либо при 50°С либо при 55°С в зависимости от используемой пары праймеров; см. Таблицу 2) в течение 2 мин и этап удлинения при 72°С в течение 90 с. Последующие циклы проводили, как указано далее: 29 циклов по 94°С в течение 1 мин, температура отжига праймеров в течение 2 мин, 72°С в течение 90 с, а затем заключительный этап удлинения при 72°С в течение 5 мин. Продукты амплификации денатурировали путем добавления равного количества стоп-реагента (95% формамид, содержащий бромфеноловый синий и ксиленцианол) и грели при 98°С в течение 10 мин. От двух до пяти микролитров реакции прогоняли на 6% денатурирующих полиакриламидных гелях, содержащих 6 М мочевину, при 100 Вт в течение 2-4 часов. Гели сушили и экспонировали на рентгеновскую пленку, как и для реакций ПДАФ.
Обычные реакции ПЦР вели в объеме реакции 15 мкл, содержащем 20 мМ Tris-HCl (pH 8.4), 50 мМ KCl, 1.5 мМ MgCl2, 200 мкМ каждого dNTP, 0.4 мкМ каждого праймера, 0.5 ед. полимеразы Taq (Invitrogen) и 10-100 нг матричной ДНК. Температурные условия циклов обычно включали первоначальный этап денатурации при 94°С в течение 2 мин, а затем 35 циклов по 94°С в течение 15 с, температура отжига праймеров (Таблица 2) в течение 30 с, 72°С в течение 1 мин на т.п.н. амплифицируемого продукта, а затем заключительный этап удлинения при 72°С в течение 10 мин. Для секвенирования праймеры и dNTP удаляли из продуктов ПЦР путем инкубирования с 1.2 ед. экзонуклеазы I и ×1.2 ед. SAP (устойчивой к кислотам протеазы) при 37°С в течение 30 мин, с последующим инкубированием при 80°С в течение 20 мин, чтобы денатурировать ферменты. Секвенирование осуществляли, применяя набор ABI PRISM Big Dye (v. 3.1) Terminator Cycle Sequencing Ready Reaction (PE Applied Biosystems), следуя инструкциям производителя. Последовательности исследовали на наличие однонуклеотидных полиморфизмов (ОНП) между устойчивыми и восприимчивыми гаплотипами, которые можно было бы использовать для разработки маркеров CAPS (рестрикционный полиморфизм амплифицированных последовательностей) для картирования в сегрегирующих популяциях.
е. Результаты. Изменение устойчивости к Р. infestans у образцов CGN
Скрининг 12 выборок S. okadae с использованием изолята 98.170.3 Р. infestans в тестах на отсоединенных листьях выявил фенотипическое изменение устойчивости в шести из них (Таблица 1b). Все оставшиеся шесть образцов были восприимчивы к этому конкретному изоляту, несмотря на результаты, полученные CGN, указывающие на то, что по меньшей мере три из этих образцов были умеренно или очень устойчивы к Р. infestans. Устойчивость была очевидна при полном отсутствии спорообразования на ткани листьев, тогда как экстенсивный рост мицелия был очевиден на листьях восприимчивых растений, начиная с 4 дня после заражения (Фиг.1). Восприимчивые листья часто становились полностью черными через семь дней после заражения.
Разработка популяций S. okadae и S. neorossii для картирования
Устойчивые растения из пяти выборок S. okadae скрещивали с восприимчивыми растениями либо из тех же либо из других выборок (Таблица 3). В каждом из скрещиваний устойчивость к Р. infestans разделялась 1:1 в полученном потомстве, указывая на присутствие потенциально пяти гетерозиготных генов Rpi в устойчивых родителях. Осуществляли межвидовое скрещивание между восприимчивым растением S. okadae и устойчивым растением S. neorossii. Анализ устойчивости в полученном потомстве указывал на присутствие доминантного гена Rpi, который сегрегировал в отношении 1:1.
Картирование генов Rpi в S. okadae
Rpi-oka1
Всего использовали 72 комбинации праймеров для ПДАФ для скрининга устойчивых и восприимчивых пулов Soka014, чтобы получить фрагменты, связанные с Rpi-oka1. С помощью комбинации праймеров PstI+CT/Msel+AGA получали фрагмент 97 п.о., который амплифицировался только у устойчивого родителя и устойчивых пулов популяции Soka014. Последовательность этого фрагмента была близка к экспрессирующейся маркерной последовательности (EST) SGN-U214221 из объединенной библиотеки EST Lycopersicon (http://www.sgn.cornell.edu). Праймеры для ПЦР разрабатывали по последовательности SGN-U214221 и использовали для амплификации бэнда размером приблизительно 2 т.п.н. из Solarium lycopersicum (ранее Lycopersicon esculentum, Le) и S. pennellii (ранее L pennellii, Lp), который проявил полиморфизм между двумя указанными видами при расщеплении рестриктазой AluI. Анализ полиморфизма в интрогрессивных линиях Le/Lp (Eshed и Zamir 1994) позволил определить расположение этого маркера в IL 9.2, что указывает на то, что этот маркер мог быть на любом плече хромосомы IX. Этот полиморфизм был не в IL 9.1, который значительно перекрывается с IL 9.2, позволяя предположить, что указанный маркер расположен близко к центромере на любом плече хромосомы. Применяя отличные праймеры для ПЦР (SokaM2.9LF5 и SokaM2.9LR5) и расщепление рестриктазой Ddel, этот маркер (SokaM2.9L) был картирован в популяции Soka014 на расстоянии, равном приблизительно 6 см (3 рекомбинанта из 48 сегрегантов) от Rpi-oka1 (Фиг. 3).
Дополнительные маркеры были разработаны путем конструирования праймеров для ПЦР для известной маркерной последовательности RFLP хромосомы IX в базе данных SGN секвенирования продуктов ПЦР, амплифицированных по обеим устойчивой и восприимчивой родительским ДНК, и идентификации ОНП, которые можно было бы использовать для разработки маркеров CAPS (Таблица 2). Таким образом, Rpi-oka1 был картирован на участке 6.0 см хромосомы IX, ограниченном маркерами C2_At4g02680 и TG186. Было обнаружено, что маркеры TG551 и TG35 косегрегировали с Rpi-oka1 (Фиг.3).
Rpi-oka3
Осуществляли скрининг устойчивых и восприимчивых пулов в популяции Soka040, используя всего 48 комбинаций праймеров для ПДАФ. Комбинация праймеров PsfI+AT/Msel+GCT позволяла получить связанный фрагмент длиной 108 п.о. Для этого фрагмента не обнаружили значительной схожести последовательности, и хотя можно было амплифицировать фрагмент из Le и Lp, применяя праймеры для ПЦР, разработанные для этой последовательности, не было обнаружено полиморфизмов, которые можно было бы использовать для картирования маркера в интрогрессивных линиях. Тем не менее, присутствовал полиморфизм Ddel между родителями популяции Soka040, который позволил картировать преобразованный ПЦР-маркер (М6.44) приблизительно в 23 см от Rpi-oka3 (Фиг.3).
Комбинация праймеров для ПДАФ PsfI+AA/Msel+GTC позволяла получить связанный фрагмент из приблизительно 260 п.о. Праймеры, разработанные для этой последовательности, позволяли амплифицировать фрагмент из Le и Lp, в котором обнаруживался полиморфизм при переваривании рестриктазой либо HaeIII, либо Sau3AI. Этот полиморфизм присутствовал в интрогрессивной линии IL 9-2. Не было обнаружено полиморфизмов среди родителей популяции Soka040 и поэтому полученный маркер не мог быть картирован по отношению к Rpi-oka3.
Для подтверждения расположения Rpi-oka3 на хромосоме IX исследовали четыре маркера SSR (Stm0010, Stm 0017, Stm 1051 и Stm 3012; (Milbourne и др. 1998)) на связание с Rpi-oka3. Stm0017 не амплифицировался успешно с восприимчивого родителя и поэтому не мог быть использован для картирования. Stm0010, Stm1051 и Stm3012, которые картировались на коротком плече хромосомы IX (Milbourne и др. 1998), все проявили полиморфизм между устойчивыми и восприимчивыми родителями и пулами и поэтому обеспечили дополнительное доказательство того, что указанный ген расположен на хромосоме IX (Фиг.3).
Rpi-oka2
В общей сложности 72 комбинации праймеров ПДАФ использовали для скрининга устойчивых и восприимчивых пулов Soka013, чтобы получить фрагменты, связанные с Rpi-oka1. Комбинация праймеров для ПДАФ PsfI+AA/Msel+CAT позволяла получить связанный фрагмент, который был преобразован в основанный на ПЦР аллель-специфичный маркер размером приблизительно 200 п.о. (Soka13M5.17). Этот маркер был картирован на расстоянии в приблизительно 6.5 см от Rpi-oka2 (Фиг.3).
Дополнительно, на карту связей Rpi-oka2 поместили 3 дополнительных маркера ПДАФ (Р12М44_103, Р13М42_228 и Р17М33_472) (Фигура 3).
Так как было показано, что Rpi-oka1 и Rpi-oka3 сильно связаны с маркерами TG551 и TG35 хромосомы IX, эти маркеры также исследовали на связание с Rpi-oka1. В популяции Soka013, TG551 и TG35 были картированы в 0.7 сМ к центромере от Rpi-oka2 на хромосоме IX (Фигура 3), фланкированные маркером Т1421.
Картирование генов Rpi в S. neorossii Rpi-nrsi
В общей сложности 11 маркеров ПДАФ были помещены на карту связей Rpi-nrsi (Фигура 3). Были предприняты попытки преобразовать эти маркеры в SCAR-маркеры, чтобы исследовать полиморфизмы, которые можно было бы использовать для помещения этих маркеров на интрогрессивные линии Le/Lp (Eshed и Zamir 1994). Тем не менее, ни один из этих маркеров не был информативным, и поэтому расположение Rpi-nrsi на хромосоме невозможно было подтвердить, используя эти маркеры. Маркер TG551 действительно проявил сильное связывание с Rpi-nrsi и поэтому мы пришли к заключению, что Rpi-nrsi также расположен на хромосоме IX, вероятно, в том же локусе, что и Rpi-oka1-3.
Использование NBS-маркера, сильно связанного с Rpi-oka1, 2, 3 и Rpi-nrsi, для картирования
NBS-маркер NBS3B (см. ПРИМЕР 3) был преобразован в основанный на ПЦР SCAR-маркер, который можно амплифицировать, используя праймеры okaNBSHae-F и okaNBSHae-R (Таблица 2). Эти праймеры позволяли амплифицировать фрагмент размером 555 п.о. (маркер okaNBSHae) из устойчивых растений, содержащих Rpi-oka1, Rpi-oka2 и Rpi-nrs1. Было показано, что в каждой популяции этот маркер косегрегировал с геном устойчивости (Фиг.3). Для Rpi-oka3 продукт ПЦР был амплифицирован как с устойчивых, так и с восприимчивых растений. Тем не менее, маркер был преобразован в CAPS-маркер путем расщепления рестриктазой MaeIII. Было показано, что этот CAPS-маркер косегрегировал с Rpi-oka3 в популяции Soka040 (Фиг. 3).
Скрининг ВАС-библиотеки и конструирование контига
Мы использовали девять ПЦР-маркеров, связанных с Rpi-oka1, Rpi-oka2 и Rpi-mcq1, для скрининга ВАС-библиотек. Как показано в Таблице 1а, TG551, TG35 и TG186 связаны с Rpi-oka1. TG551 также связан с Rpi-oka2. Маркер okaNBSHae связан как с Rpi-oka1, так и с Rpi-oka2. Хотя TG551 связан с обоими генами из S. okadae, аллели этого маркера из гаплотипов Rpi-oka1 и Rpi-oka2 можно различить путем рестрикционного расщепления, как указано в Таблице 1а. Все из U282757, U296361, TG591 и U279465 связаны с Rpi-mcq1. Количество ВАС-пулов, для которых было показано, что они положительны по этим маркерам, варьировало от 11 до 17 для библиотеки К39 и от 9 до 14 для библиотеки К182 (Таблица 1а).
В библиотеке К39 маркер TG186 был амплифицирован в 17 пулах. Было невозможно определить, из какого гаплотипа (Rpi-oka1 или Rpi-oka2) этот маркер был амплифицирован. TG186, несмотря на то, что он был CAPS-маркером, был исключающе связан с Rpi-oka1, и аллель, присутствующий в гаплотипе Rpi-oka1, неотличим от такового в гаплотипе Rpi-oka2. Маркер okaNBSHae был амплифицирован в 18 пулах. Так как этот маркер не полиморфный между гаплотипами Rpi-oka1 и Rpi-oka2, было невозможно определить гаплотип этих ВАС на основании этого маркера. TG551 амплифицировали с помощью ПЦР в 16 пулах, и расщепление рестриктазами позволило выявить шесть пулов, положительных по гаплотипу Rpi-oka1, и семь пулов, положительных по гаплотипу Rpi-oka2. TG35 амплифицировали с помощью ПЦР в 11 пулах, восемь из которых, как было показано, имели гаплотип Rpi-oka1 при расщеплении рестриктазами, позволяя предположить, что оставшиеся три имели гаплотип Rpi-oka2. Изображения гелей, на которых показаны основанные на ПЦР маркеры и их паттерны рестрикции (в соответствующих случаях), представлены на Фиг.4а-4с.
В результате гибридизации зонда okaNBSHae с объединенными в пул ВАС-ДНК было идентифицировано всего 67 пулов, содержащих ВАС-клоны с гомологичными последовательностями. В результате скрининга с высокой плотностью мембран с двойными пятнами, содержащими каждый отдельный ВАС-клон из идентифицированных пулов, всего было идентифицировано 85 ВАС-клонов, из которых выделяли ДНК и использовали ее в методе "отпечатков пальцев" ВАС SNaPshot, вместе с дополнительными 10 отобранными клонами, которые были положительны по маркерам TG551 и/или TG35. Из полученных контигов один контиг содержал ВАС-клоны, идентифицированные с помощью а) основанного на ПЦР скрининга с применением связанных маркеров TG551, TG35 и косегрегирующегося маркера okaNBSHae и b) гибридизации с применением маркера okaNBSHae в качестве зонда. Этот ВАС-контиг представлен на Фиг.5.
Пулы из библиотеки К39, которые были положительны по любому из маркеров TG551, TG35, или okaNBSHae, которые сильно связаны или, в случае okaNBSHae, косегрегируют с Rpi-oka1 или Rpi-oka2, выбирали произвольно. Исходные 384-луночные планшеты для каждой из ВАС-библиотек реплицировали на твердую среду LB. Колонии из каждого планшета соскабливали по рядам и колонкам и осуществляли скрининг на присутствие соответствующего маркера. Из 384-луночнх планшетов отбирали отдельные клоны.
После отбора отдельных клонов ВАС-ДНК выделяли и ВАС-концы секвенировали. Поиск гомологии в BLAST (http://www.ncbi.nlm.nih.gov/BLASTT) показал, что два клона из библиотеки К39 (К39_272М11 и К39_256М23) имели ВАС-концевые последовательности, которые были значительно похожи друг на друга и на R-ген вируса мозаики томата Tm-22 (Lanfermeijer и др. 2003).
Праймеры для PCR разрабатывали для каждой из концевых последовательностей ВАС и использовали для амплификации продуктов по родительским генотипам Rpi-oka1 и Rpi-oka2. Продукты ПЦР секвенировали и анализировали на присутствие ОНП, что позволило бы применение продуктов ПЦР в качестве маркеров в соответствующих популяциях. Успешно преобразованные маркеры наносили на генетическую карту с более высоким разрешением для Rpi-oka1 (1213 растений) и Rpi-oka2 (1706 растений). Положение этих маркеров по отношению к Rpi-oka1 и Rpi-oka2 представлено на Фиг.6.
Картирование с высоким разрешением и клокирование Rpi-oka1
Осуществляли скрининг в популяции Soka014 Rpi-oka1, состоящей из 1214 растений, на рекомбинанты между маркерами SokaM2.9L и TG186. Всего было идентифицировано 169 рекомбинантов, покрывающих генетический интервал в 14 сМ. Предварительный скрининг этих рекомбинантов на фенотип заболевания показал, что локус устойчивости был расположен к югу от маркера TG35. Следовательно, подмножество из 53 рекомбинантов между маркерами TG35 и TG186 было отобрано из большего подмножества рекомбинантов. Среди этих рекомбинантов проводили скрининг на устойчивость или восприимчивость к Р. infestans и с маркерами, разработанными для концевых последовательностей ВАС. Результаты показали, что Rpi-oka1 был расположен в пределах генетического интервала длиной 0.33 сМ, ограниченного маркерами TG35 и 185L21R (ВАС-концевой маркер) (Фиг.5, 6). Исходя из физической карты, созданной с помощью анализа ПЦР и методом "отпечатков пальцев" ВАС-клонов из библиотеки К39 (Фиг.5), предположили, что Rpi-oka1 присутствует на физическом участке, покрытом двумя ВАС-клонами К39_148Р20 и К39_26619. Эти два ВАС-клона были секвенированы, и была идентифицирована одна кандидатная открытая рамка считывания (ОРС) для Rpi-oka1.
Картирование с высоким разрешением и клонирование Rpi-oka2
Для создания карты с высоким разрешением мы использовали два фланкирующих основанных на ПЦР маркера (TG551 и Т1421; Фигура 1а), чтобы провести скрининг расширенной популяции для отбора рекомбинантов вокруг локуса устойчивости. Было отобрано 46 рекомбинантов из расширенной популяции 1706 генотипов, представляя интервал в 2.9 сМ между двумя указанными маркерами ПЦР. Эти рекомбинанты фенотипировали по устойчивости к фитофторозу пасленовых и генотипировали, применяя ВАС-концевые маркеры из ВАС-контига. Результаты показали, что Rpi-oka2 был расположен в пределах генетического интервала в 0.12 сМ и ограничен ВАС-концевыми маркерами 266I9F и 185L21R (Фиг.5, 6). Исходя из физической карты, созданной с помощью анализа ПЦР и методом "отпечатков пальцев" ВАС-клонов из библиотеки К39, предположили, что Rpi-oka2 расположен на том же физическом участке, на котором был идентифицирован Rpi-oka1. Тем не менее, в отличие от ВАС-клона К39_272М11 (у которого один конец содержал частичный гомолог Tm22), клоны, которые покрывали тот же физический участок, что и Rpi-oka1, не были идентифицированы в библиотеке (Фиг.5). В качестве альтернативного подхода праймеры, которые были разработаны для амплификации полной ОРС Rpi-oka1, использовали в реакции ПЦР с ДНК из родительского растения популяции Rpi-oka2. Полученный продукт ПЦР клонировали в pGEM-T Easy и 4 клона секвенировали, чтобы получить консенсусную последовательность для кандидата Rpi-oka2.
Картирование с высоким разрешением Rpi-nrs1
Для создания карты с высоким разрешением мы использовали три основанных на ПЦР фланкирующих маркера (SheoM2.9a, TG551 и ТР25; Фигура 3) для скрининга расширенной популяции и отбора рекомбинантов вблизи локуса устойчивости Rpi-nrsi. TP25 был преобразован из ПДАФ-маркера, P13M34_370[R]. Первоначально было отобрано 323 рекомбинанта из расширенной популяции 1402 генотипов, в результате чего получали интервал генетического расстояния в 23 см между SneoM2.9a и TP25. В то же время были разработаны более близкие к Rpi-nrsi маркеры ПЦР (U317500 и U270442). Последовательно было отобрано 40 рекомбинантов и эти рекомбинанты фенотипировали на устойчивость к фитофторозу пасленовых. RGA-маркер, обозначенный okaNBSHae, был картирован в том же генетическом положении, что и Rpi-nrs1, и несколько основанных на концевых последовательностях ВАС ПЦР-маркеров было разработано из Rpi-oka1/2 контига из ВАС-библиотеки К39, что позволило построить мелкомасштабную генетическую карту вблизи Rpi-nrs1 (Фигура 6). Дополнительно, рекомбинантов тестировали на устойчивость, используя три различных изолята, и их фенотипы по-разному сегрегировали, указывая на то, что в популяции присутствовало несколько генов. Ожидали, что второй ген, обозначенный Rpi-nrs1b, будет расположен к югу от Rpi-nrs1a (Фигура 6).
Анализ Rpi-oka1, Rpi-oka2 и Rpi-oka3
OPC Rpi-oka1 имеет длину 2673 п.о. и транслируется в белковую последовательность, состоящую из 891 аминокислоты, с рассчитанной молекулярной массой, равной 102 кДа, и pI 8.05. OPC Rpi-oka2 включает 2715 п.о. и транслируется в белковую последовательность, состоящую из 905 аминокислот, с рассчитанной молекулярной массой 103.6 кДа и pI 8.16. Последовательность продукта ПЦР, амплифицированного по материалу, содержащему Rpi-oka3, была идентична таковой для Rpi-oka2. Белок Rpi-okal имел все черты, характерные для CC-NB-LRR-класса устойчивых белков (класса белков с биспиральным участком связывания нуклеотидов - лейцин-богатым повтором). В пределах первых 215 аминокислот N-концевой части белка находилось 4 участка, каждый из которых включал 3 предсказанных повторяющихся мотива группы из семи остатков, типичных для биспиральных доменов (Фиг.7). Все домены NB-ARC (van der Biezen и Jones 1998) присутствовали в последовательности аминокислот на участке 216-505. За доменом NB-ARC следовал участок, включающий ряд из 15 неправильных LRR мотивов, которые можно было выровнять согласно консенсусной последовательности LxxLxxLxxLxLxxC/N/Sx(x)LxxLPxx (где L мог быть L, I, М, В, Y или F и х представлял собой любую аминокислоту) (McHale и др. 2006).
Последовательность Rpi-oka2 отличается от Rpi-oka1 на вставку из 42 нуклеотидов в 5'-конце гена (Фиг.7). Дополнительные 14 аминокислот, присутствующие в соответствующем участке Rpi-oka2, не нарушают какой-либо из предсказанных биспиральных доменов. Также существует 3 однонуклеотидных полиморфизма (ОНП) между Rpi-oka1 и Rpi-oka2; А1501Т, Т1767С и G2117A (Фиг.7). Эти отличия в нуклеотидах приводят к отличию в двух аминокислотах между Rpi-oka1 и Rpi-oka2 (Фиг.7). Отличие в положении 501 находится в концевом участке домена NB-ARC, прямо перед участком LRR, и приводит к замене аспарагина в Rpi-oka1 на тирозин в Rpi-oka2. Эта замена аминокислоты не влияет на любой из описанных доменов NB-ARC. В положении 706, в пределах 9ого LRR, аргинин в Rpi-oka1 заменяется на лизин в Rpi-oka2; оба этих остатка представляют собой положительно заряженные полярные аминокислоты и, следовательно, эта замена может рассматриваться как тождественная.
Rpi-oka1 и Rpi-oka2 идентичны на 80.9% и 79.7%, соответственно, с Tm-22 на уровне нуклеиновых кислот. На уровне аминокислот это соответствует 72.1% и 71.1% идентичности, соответственно. Как и ожидали, учитывая его роль в специфичности узнавания, процент схожести был наиболее низким в домене LRR, где Rpi-oka1/2 и Tm-22 имели лишь 57.5% схожести. В противоположность схожесть последовательностей среди биспиральных и NB-ARC доменов Rpi-oka1/2 и Tm-22 составляла 81.8% и 79.7%; в пределах консервативных доменов участка NB-ARC, Tm-22 и Rpi-oka1 отличались лишь по 1 аминокислоте.
Праймеры oka1long-F и oka1long-R (Таблица 2) использовали для амплификации гомологичных Rpi-oka1 последовательностей из родительского материала, содержащего Rpi-oka3. Полученные продукты ПЦР клонировали в pGEM-T и секвенировали. Полученные последовательности были идентичны Rpi-oka2.
Было невозможно амплифицировать полноразмерные паралоги Rpi-oka1 из восприимчивого родительского А613 S. okadae. Это наблюдение вместе с тем фактом, что маркер okaNBSHae можно было амплифицировать только из устойчивых генотипов, позволяет предположить, что восприимчивый фенотип вызван скорее отсутствием Rpi-oka1, чем нефункциональной его копией.
Rpi-oka1 также присутствует в устойчивых генотипах S. neorossii и является ортологом Rpi-phu1.
Картирование гена Rpi в сегрегировавшей популяции, полученной из устойчивого растения S. neorossii с номером доступа CGN1800, также выявило сильное связание между идентифицированным геном (Rpi-nrs1) и маркером TG551, что указывает на то, что этот ген был расположен в том же участке, что и Rpi-oka1. Аналогично сообщали, что Rpi-phu1 из S. phureja также картируется на этом участке (Sliwka и др. 2006). Маркер okaNBSHae-F/R Rpi-oka1 косегрегировал с устойчивостью в популяции 149 растений S. tuberosum после расщепления по Rpi-phu1. Полноразмерные паралоги Rpi-oka1 амплифицировали по ДНК 3 устойчивых генотипов, содержащих Rpi-phu1. Получили один продукт и секвенированием показали, что он идентичен Rpi-oka1. Аналогично, амплификация материала устойчивого S. neorossii показала, что Rpi-oka2 присутствовал в этом материале, и присутствие этого гена коррелировало с устойчивостью у 40 предварительно отобранных рекомбинантов. Было показано, что материал устойчивых растений, содержащий Rpi-nrs1 или Rpi-phu1, также устойчив к каждому из изолятов Р. infestans, использованных в данном исследовании, за исключением ЕС1. Таким образом, мы пришли к заключению, что Rpi-oka1=Rpi-phu1 и Rpi-oka2=Rpi-oka3= Rpi-nrs1.
bКоличество растений, проявивших устойчивые (У) или восприимчивые (В) фенотипы
ПРИМЕР 3. КАРТИРОВАНИЕ И КЛОНИРОВАНИЕ Rpi-oka1 И RPI-nrs1 С ПРИМЕНЕНИЕМ ПОДХОДА ПОИСКА КАНДИДАТНОГО ГЕНА/АЛЛЕЛИ
На сегодняшний день клонирование R-генов обычно осуществляют с помощью стратегии позиционного клонирования. После того как функциональный ген клонируют из специфичного R-локуса, можно попытаться клонировать функциональные аллели из того же или отличного вида для того, чтобы определить частоту аллеля и аллельные варианты в данном локусе. В данном примере мы демонстрируем, что анализ профиля NBS (Linden и др., 2004) при объединении с общим анализом сегрегантов (ОАС) (Michelmore и др., 1991) представляет собой эффективный инструмент для получения маркеров кандидатных генов, который может предсказать положение исследуемого R-локуса и при этом обозначить начальную позицию для клонирования гена с помощью стратегии поиска функциональной аллели.
Материал растений
Образцы Solarium okadae и Solanum neorossii запрашивали у Centre for Genetic Resources (CGN) в Вагенингене, Нидерланды. После скрининга Phytophthora infestans устойчивые генотипы конкретных образцов использовали для создания популяций для вне- или внутривидового картирования. Популяцию 7698 для картирования Rpi-oka1 создали путем скрещивания ОКА7014-9 (устойчивого растения F1, полученного при скрещивании ОКА367-1 и ОКА366-8, оба из которых получены из образца CGN18108) с восприимчивым растением NRS735-2 (CGN18280). Все генотипы S. okadae были получены из образца CGN18108. Популяцию 7663 для картирования Rpi-nrs1 создали путем скрещивания устойчивого растения NRS365-1 (CGN18000) с NRS735-2.
Тесты на заболевание
Тесты с отделенными листьями (ТОЛ) проводили на видах Solanum, как описано у Vleeshouwers и др. (1999). Листья заражали, капая 10 мкл инокулята (5×104 зооспор/мл) на нижню сторону и инкубируя при 15°С в течение 6 дней в камере искусственного климата со световым периодом 16 ч день/8 ч ночь. На 6 день после заражения листья, на которых выявили спорообразование, оценивали как восприимчивые, тогда как листья, на которых не проявилось симптомов или некротических повреждений, оценивали как устойчивые.
Разработка маркера
Маркеры подходящих положений на хромосоме отбирали из базы данных Solanaceae Genomics Network (SGN), а затем разрабатывали на их основе полиморфные маркеры для каждой из соответствующих популяций для картирования. Дополнительные маркеры для кандидатных генов разрабатывали путем анализа профиля NBS, описанного у van der Linden и др. (2004). Матрицы получали при расщеплении рестриктазами геномной ДНК, применяя рестриктазы MseI, HaeIII, AluI, RsaI или TaqI. Адаптеры лигировали с рестрикционными фрагментами. ПЦР-фрагменты получали с помощью радиоактивно-меченых праймеров (nbs1, nbs2, nbs3, nbs5a6 или nbs9), разработанных на основе консервативных доменов NBS-домена (мотивы Р-петля, Киназа-2 и GLPL (Calenge, 2005; Syed, 2006)).
ПЦР-амплификация кандидатных R-генов
Для ПЦР длинных фрагментов с Taq-полимеразой или полимеразой Pfu Turbo готовили 50 мкл реакционной смеси, содержащей 50 нг геномной ДНК, 1 мкл прямого праймера (10 мкМ), 1 мкл обратного праймера (10 мкМ), 0.8 мкл dNTP (5 мМ каждого), 5 мкл 10Х буфера, 5 единиц Taq-полимеразы (Perkin Elmer) или 1 мкл pfu Turbo (Invitrogen). Использовали следующую программу ПЦР: 94°С в течение 3 мин, 94°С в течение 30 сек, 55°С в течение 30 сек, 72°С в течение 4 мин, 72°С в течение 5 мин на протяжении 29 циклов.
«Прогулка по геному»
Маркерные последовательности удлинняли путем клонирования фланкирующих фрагментов ДНК с помощью набора ClonTech Genome Walker, следуя инструкциям производителя и применяя адаптер с тупыми концами, включающий комплементарные последовательности:
5-GTAATACGACTCACTATAGGGCACGCGTGGTCGACGGCCCGGGCTGGA-3 и 5'-PO4TCCAGCCC и специфичные праймеры к адаптеру АР1 (5'-TAATACGACTCACTATAGGGC) и АР2 (5'-ACTATAGGGCACGCGTGGT). Выполняли одновременную реакцию рестрикции-лигирования, а затем два раунда ПЦР. Готовили 50 мкл смеси для реакции рестрикции-лигирования (RL), содержащей 250 нг геномной ДНК, 5 единиц фермента, режущего по тупым концам (Bsh1236I, AluI, DpnI, HaeIII, RsaI, HincII, DraI, ScaI, HpaI или SspI), 1 мкл адаптера Genome Walker (25 мкМ), 10 мМ АТР, 10 мкл 5× RL буфера, 1 единица ДНК-лигазы Т4 (Invitrogen, 1 ед/мкл). Расщепляемую смесь инкубировали при 37°С в течение 3 часов. Образцы разбавляли в 50 раз перед проведением ПЦР. Для первого раунда ПЦР, готовили 20 мкл реакционной смеси, содержащей 5 мкл разбавленной RL-ДНК, 0.6 мкл специфичного прямого праймера 1 (10 мкМ), 0.6 мкл АР1 (10 мкМ), 0.8 мкл dNTP (5 мМ каждого), 2 мкл 10× буфера (Perkin Elmer), 5 единиц Taq-полимеразы (Perkin Elmer). Первый раунд ПЦР вели, используя следующую программу циклов: 30 сек при 94°С в качестве этапа денатурации, 30 сек при 56°С в качестве этапа отжига и 60 сек при 72°С в качестве этапа удлинения. Выполняли 35 циклов. Второй раунд ПЦР, в котором использовали такие же условия, что и в первом, вели, применяя специфичный праймер 2 и АР2 и 5 мкл разбавленного в 50 раз продукта первого раунда ПЦР. 5 мкл второго продукта ПЦР наносили на гель (1% агароза) и наибольшие ампликоны клонировали в вектор pGEM®-T Easy Vector от Promega и секвенировали.
Саtеwау®-клонирование кандидатных R-генов в бинарный экспрессионный вектор
Методику клонирования Gateway® применяли, следуя инструкциям производителя, чтобы эффективно клонировать кандидатные гены вместе с подходящими последовательностями промотора и терминатора в бинарный вектор pKGW-MGW Gateway®. В плазмиде pKGW кассета Gateway® была заменена на множественную кассету Gateway®, амплифицированную по pDESTr4r3, что позволило получить pKGW-MGW. В данном исследовании мы использовали промотор и терминатор Rpi-blb3 (Lokossou и др., в подготовке), которые были клонированы в векторы pDONR Gateway® pDONRP4P1R и pDONRP2RP3, соответственно, с получением pENTR-Blb3P и pENTR-Blb3T. ПЦР-ампликоны, полученные с помощью полимеразы Pfu Turbo, клонировали в pDONR221 с получением клонов pENTR-RGH, а затем клонировали вместе с фрагментами промотора и терминатора Rpi-blb3 в pKGW - MGW, применяя набор для клонирования с множественной Gateway® (Invitrogen). Клоны pENTR были получены путем проведения реакции BP-Reaction II в течение ночи (http://www.untergasser.com/lab/protocols/bp gateway reaction ii v1 0.shtml).
Компетентные клетки DH5α (Invitrogen) трансформировали посредством теплового шока 5 мкл смеси BP-Reaction II. Клетки отбирали на среде LB, содержащей 50 мг/мл канамицина. Колонии проверяли на наличие соответствующих вставок путем осуществления ПЦР колонии. ДНК подходящих клонов pENTR экстрагировали из Е. coli и использовали для клонирования реакции LR с множественной Gateway®, чтобы получить окончательные клоны для бинарной экспрессии (http://www.untergasser.com/lab/protocols/lr_multiple_gateway_reaction_v1_0.sh tml). Компетентные клетки DH5a (Invitrogen) трансформировали посредством теплового шока 5 мкл реакционной смеси LR. Клетки отбирали на среде LB, содержащей 100 мг/мл спектиномицина. Колонии проверяли с помощью ПЦР на присутствие правильных вставок. Положительные колонии выращивали в течение ночи в среде LB, дополненной 100 мг/мл спектиномицина, чтобы экстрагировать окончательный экспрессионный вектор. Окончательный экспрессионный вектор переносили в штамм COR308 Agrobacterium tumefaciens посредством электропорации. Колонии отбирали на среде LB, дополненной 100 мг/мл спектиномицина и 12.5 мг/мл тетрациклина, в течение ночи при 30°С.
Секвенирование
Клонированные фрагменты или продукты ПЦР, которые получали либо с помощью Taq-полимеразы (Perkin Elmer) либо с помощью полимеразы Pfu Turbo (Invitrogen), секвенировали, как указано далее: 10 мкл реакционных смесей для секвенирования получали, используя 5 мкл продукта ПЦР или 5 нг плазмиды, 3 мкл буфера, 1 мкл DETT (Amersham) и 1 мкл прямого или обратного праймера. Используемая программа ПЦР включала 25 циклов по 94°С в течение 20 сек, 50°С в течение 15 сек, 60°С в течение 1 мин. Последовательности получали на секвенаторах ABI 3730XL.
РЕЗУЛЬТАТЫ
Генетическая основа и спектр устойчивости к фитофторозу пасленовых у образцов S. okadae и S. neorossii.
Чтобы определить генетическую основу устойчивости к фитофторозу пасленовых у S. okadae и S. neorossii, осуществляли скрининг 14 и 5 образцов, соответственно, с помощью тестов с отделенными листьями (ТОЛ) со сложным изолятом IPO-C P. infestans. Устойчивые генотипы, отобранные среди ока, номер доступа CGN18108, и nrs, номер доступа CGN18000, использовали для получения популяций S. okadae и S. neorossii для картирований 7698 [oka7014-9 (oka367-1 × oka366-8) × nrs735-2) и 7663 (nrs365-1 x nrs735-2), соответственно. После тестов ТОЛ с 50 потомками F1 растений из популяции 7698 30 оценили как устойчивые и 22 как восприимчивые, что позволило предположить присутствие одного доминантного R-гена, который мы назвали Rpi-oka1. В результате скрининга из 60 потомков F1 растений из популяции 7663, 24 оценили как устойчивые и 36 как восприимчивые, что позволило предположить, что nrs365-1 также содержал один доминантный R-ген, который мы назвали Rpi-nrs1.
Спектр устойчивости обоих генов проанализировали, подвергая их действию некоторых изолятов с различным уровнем сложности и агрессивности (Таблица 4). Результаты показали, что Rpi-oka1 и Rpi-nrs1 имели одинаковую специфичность. Штамм ЕС1 был единственным способным преодолеть оба R-гена.
Картирование Rpi-oka1 и Rpi-nrs1 на хромосоме IX
Чтобы определить, находится ли ген Rpi-oka1, сегрегирующий в популяции 7698, на хромосоме IX, мы попробовали разработать и картировать специфичные для хромосомы IX маркеры TG35, TG551, TG186, СТ183 и Т1421 в первых 50 потомках F1 растений из популяции 7698. Лишь TG35 и TG186 были полиморфны между родительскими генотипами и были действительно связаны с Rpi-oka1 (Фиг. 9). С целью разработать дополнительные маркеры для Rpi-oka1, а также маркеры для Rpi-nrs1 мы провели общий анализ сегрегантов (ОАС) в сочетании с анализом профиля NBS в обеих популяциях для картирования. Это позволило идентифицировать всего 9 специфичных маркеров для Rpi-oka1 в 7698 и 8 для Rpi-nrs1 в 7663. Наконец, лишь два устойчивых специфичных фрагмента: один - полученный с помощью комбинации праймер-фермент NBS2/RsaI и другой - полученный с помощью NBS3/HaeIII, косегрегировало с устойчивостью в первых популяциях 7698 и 7663 из 50 и 60 потомков F1 растений, соответственно. Эти фрагменты были, соответственно, клонированы и секвенированы. После анализа с помощью BLAST обе последовательности оказались значительно схожими с геном Tm-22 на хромосоме IX томата (Lanfermeijer и др., 2003; 2005). Клонированные фрагменты NBS2/RsaI и NBS3/HaeIII имели размер 350 и 115 п.о. и разделяли 88.3% и 80.3% идентичности последовательностей ДНК с Tm-22. Эти открытия позволили предположить, что Rpi-nrs1 мог быть расположен на том же участке на хромосоме IX, что и Rpi-oka1. С целью проверить это, были разработаны специфичные праймеры для каждого фрагмента, и они использовались для разработки SCAR-маркеров в обеих популяциях для картирования Rpi-oka1 и Rpi-nrs1. Таким образом, были разработаны два маркера, полученные в результате анализа профиля NBS, NBS3A и NBS3B для популяций 7663 и 7698, соответственно. Впоследствии определяли положения этих маркеров относительно Rpi-nrs1 и специфичных маркеров хромосомы IX TG35 и TG551 при Rpi-nrs1 картировании 7698. Косегрегация TG35, TG551 и NBS3B с Rpi-nrs1 в первой популяции F1 из 60 растений подтвердила, что Rpi-nrs1 был действительно расположен на хромосоме IX, в том же участке, что и Rpi-oka1 (Фиг.9).
Для того чтобы разработать фланкирующие маркеры для рекомбинантного анализа, маркеры, связанные с TG35 или TG551, отбирали из SGN и осуществляли скрининг в обеих популяциях. Несмотря на низкую частоту встречаемости полиморфизма, основанные на EST маркеры U276927 и U270442 I были разработаны и картированы в популяциях 7698 и 7663, соответственно (Таблица 4 и Фигура 9). U276927 был картирован в 2 сМ к северу от Rpi-oka1, тогда как U270442 I был картирован в 3.5 сМ к югу от Rpi-nrs1. Впоследствии рекомбинантный анализ выполняли на 500 потомках популяции 7698 и 1005 потомках популяции 7663, применяя фланкирующие маркеры U276927 / TG186 и NBS3B / U270442, соответственно. Это позволило осуществить картирование Rpi-oka1 и Rpi-nrs1 с генетическимий интервалами 4 сМ и 3.6 сМ, соответственно (Фигура 9).
Основанный на Tm-22 поиск аллели в S. okadae и S. neorossi
Авторы настоящего изобретения использовали стратегию поиска аллели, основанную на гомологии, чтобы клонировать Rpi-oka1 и Rpi-nrs1.
Первым этапом была разработка вырожденных праймеров путем введения предполагаемых инициирующих и стоп-кодонов от кандидатных гомологов гена Tm-2 (Tm2GH) в локусы Rpi-oka1 и Rpi-nrs1. На основании выравнивания всех доступных полученных из картофеля и томата Tm-22-подобных последовательностей в общедоступных базах данных последовательностей мы разработали праймеры ATG-Tm2-F и TGA-Tm2-R (Таблица 5). Тем не менее, не было получено ампликонов ожидаемого размера, когда этот набор праймеров тестировали на родительских генотипах обеих картируемых популяций. Так как последовательность праймера ATG-Tm2-F присутствовала в полученной в результате анализа профиля NBS косегрегирующей маркерной последовательности, были разработаны три новых обратных праймера (REV-A, -В и -С), расположенные в 100 п.о. против хода транскрипции от изначального сайта праймера TGA-Tm2-R, на участке, который консервативен во всех выровненных Tm-22-подобных последовательностях. При комбинировании либо с ATG-Tm2-F либо с NBS3B-F отдельный ампликон, размером приблизительно 2.5 т.п.н., специфично амплифицировался лишь с устойчивых родительских генотипов, т.е. ока7014-9 и nrs365-1. Эти фрагменты клонировали в вектор pGEM®-T Easy и приблизительно 96 отдельных клонов из каждого генотипа секвенировали, применяя стратегию праймер-опосредсредованной прогулки. Все полученные последовательности имели 75-80% схожести с Tm-22. Всего 5 различных классов можно было различить среди полученных из ока7014-9 последовательностей, тогда как последовательности nrs365-1 распадались лишь на 3 различных класса. Эти различные классы впоследствии назвали NBS3B-подобные или NBS3B-неподобные на основании степени гомологии с последовательностью NBS3B (Таблица 6).
С целью найти недостающую С-концевую часть амплифицированного 7m2GH мы осуществили 3'-прогулку по геному, применяя праймеры GSP1-1, GSP1-2 и GSP2 (Таблица 5), которые были разработаны расположенными приблизительно в 100 п.о. против хода транскрипции от праймеров REV-A, -В и -С, для того чтобы получить перекрывание 100 п.о. между клонированными NBS3B-подобными последовательностями и клонами, полученными с помощью прогулки по геному. Было получено три ампликона по ~200 п.о. из oka7014-9 и один ~1 т.п.н. из nrs365-1. После клонирования, секвенирования и выравнивания с клонированным 7m2GH все четыре клона, похоже, соответствовали клону 7m2GH-nrs8bis, так как перекрывающиеся 100 п.о. точно совпадали. Чтобы иметь возможность впоследствии амплифицировать полноразмерный 7m2GH из локусов Rpi-oka1 и Rpi-nrs1, мы разработали новый обратный праймер (TAA-8bis-R) (Таблица 5) на основании выравнивания полноразмерной последовательности 7m2GH-nrs8bis с последовательностью Tm22 из томата (Фигура 2). Так как исходный стоп-кодон TGA не присутствовал в последовательности 7m2GH-nrs8bis, мы включили следующий стоп-кодон (ТАА) в пределах рамки считывания, который был расположен 12 п.о. по ходу транскрипции.
Полноразмерную амплификацию 7m2GH из oka7014-9 и nrs365-1 впоследствии осуществляли с помощью высокоточной полимеразы Pfu Turbo, применяя праймеры ATG-Tm2-F и TAA-8bis-R. Ампликоны размером ~2.6 т.п.н. клонировали в вектор pGEM®-T Easy и секвенировали. Из ОКА7014-9 получили три различных типа клонов, один из которых нес ОРС ожидаемого размера (7m2GH-oka1bis). Все клоны, полученные из NRS365-1, были идентичны друг другу и также включали ожидаемую ОРС. Клон Tm2GH-nrs1.9 выбрали вместе с 7m2GH-oka1bis для дополнительного генетического анализа.
Перед проведением анализа комплементарности Tm2GH-oka1bis и Tm2GH-nrs1.9, было необходимо удостовериться в том, что выбранные Tm2GH в действительности картированы в локусах Rpi-oka1 и Rpi-nrs1. При тестировании в качестве SCAR-маркеров в изначальных популяциях для картирования оба маркера косегрегировали с устойчивостью. При амплификации ATG-Tm2-F и TAA-8bis-R в наборе рекомбинантов, которые определяли локусы Rpi-oka1 и Rpi-nrs1, ампликоны ожидаемого размера действительно получали лишь из устойчивых к фитофторозу пасленовых рекомбинантов, подтверждая, что оба Tm2GA действительно были хорошими кандидатами в Rpi-oka1 и Rpi-nrs1. Тем не менее, были устойчивые рекомбинанты 2 в популяции для картирования Rpi-oka1 и 1 в популяции для картирования Rpi-nrs1, которые не образовали ожидаемый продукт ПЦР, позволяя предположить, что оба локуса могли в действительности включать тандем из двух функциональных R-генов.
Анализ ОРС Rpi-oka1 и Rpi-nrs1
Структура генов Rpi-oka1 и Rpi-nrs1
5'-концевую структуру Rpi-oka1 и Rpi-nrs1 определили путем сравнения последовательностей ампликонов с фрагментами кДНК, полученными путем быстрой амплификации 5'-концов кДНК (RACE). Идентифицированные с помощью RACE специфичные 5'-фрагменты кДНК Rpi-oka1 и Rpi-nrs1 включали 5'-нетранслируемые участки размером 83 нуклеотида (нт) у Rpi-oka1 и 5 нт у Rpi-nrs1. Оба гена не включали интроны. Открытые рамки считывания Rpi-oka1 и Rpi-nrs1 кодировали предсказанные пептиды по 891 и 905 аминокислот, соответственно. Вдобавок к вставке в 14 аминокислот в N-концевом участке Rpi-nrs1 лишь две другие аминокислоты отличались между Rpi-oka1 и Rpi-nrs1. В положениях 548 и 753, Rpi-oka1 включает остатки аспарагина и аргинина, тогда как соответствующими остатками в Rpi-nrs1 были тирозин и лизин, соответственно (Фигура 8). Тем не менее, замещенные остатки имели такие же свойства. Аспарагин и тирозин принадлежат к группе гидрофобных остатков, тогда как аргинин и лизин являются положительно заряженными остатками. Белковые последовательности обоих генов несли консервативные мотивы класса CC-NBS-LRR R-белков (Фигура 8). Биспиральный (СС) домен расположен в N-концевой части белков между аминокислотами 1 и 183 для Rpi-oka1 и между 1 и 198 для Rpi-nrs1. В первых 183 или 198 остатках можно узнать 4 пары предполагаемых мотивов, состоящих из семи гидрофобных остатков, в последовательностях Rpi-oka1 и Rpi-nrs1, соответственно. Домен NB-ARC (сайт связывания нуклеотидов, апоптоз, R-продукты гена, CED-4) можно узнать в участке аминокислот между остатками 183 или 198 и 472 или 486, соответственно (Р-петля, Киназа-2, GLPL) (Van der Bierzen и Jones 1998). С-концевая половина Rpi-oka1 и Rpi-nrs1 включает ряд 15 мотивов LRR неправильного размера, которые можно выровнять согласно следующей консенсусной последовательности LxxLxxLxxLxLxxC/N/Sx(x)LxxLPxx (где х представляет собой любую аминокислоту) (McHale и др. 2006). Анализ PROSITE (Hofmann и др. 1999) позволил идентифицировать 4 сайта N-гликозилирования, 7 сайтов фосфорилирования казеинкиназой II, 10 сайтов фосфорилирования протеинкиназой С, 6 сайтов N-миристоилирования и 1 сайт фосфорилирования Camp- и Cgmp-зависимой протеинкиназой.
На уровне белка Rpi-oka1 и Rpi-nrs1 имеют 75% идентичных аминокислот с белковой последовательностью Tm-22. Примечательно, что наиболее низкий уровень гомологии был обнаружен в LRR домене, где Tm-22 имеет лишь 62% идентичности с Rpi-oka1 и Rpi-nrs1. В противоположность биспиральный и NB-ARC домены Rpi-oka1 и Rpi-nrs1 имеют 87% идентичности последовательности аминокислот с теми же участками Tm-22.
Характеристика изолятов Phytophthora infestans, используемых для определения специфичности Rpi-oka1 и Rpi-nrs1
ния
ния
ния

ПРИМЕР 4А. ВРЕМЕННАЯ КОМПЛЕМЕНТАЦИЯ В NICOTIANA BENTHAMIANA
В зависимости от разрешения генетической карты при соответствующих исследованиях и размера семейства кандидатных генов подход к поиску аллели может дать множество кандидатных генов, которые необходимо функционально проанализировать. На сегодняшний день функциональные анализы гомологов кандидатных R-генов (RGH) обычно требуют стабильной трасформации восприимчивого генотипа с целью комплементации. Это трудоемкий и неэффективный подход, так как он требует по меньшей мере нескольких месяцев для получения трансгенного растения, которое можно функционально проанализировать. В настоящем исследовании мы использовали открытие, что Nicotiana benthamiana восприимчив к Р. infestans, несмотря на предыдущие сообщения (ссылка Kamoun и др., 1998), чтобы разработать тест на временную комплементацию Agrobacterium (ATCA) для R-генов, которые придают устойчивость к Р. infestans.
Тесты на временную трансформацию Agrobacterium (ATTA)
Тесты на временную трансформацию Agrobacterium (ATTA) выполняли на Nicotiana benthamiana, после чего проводили тесты с отделенными листьями, применяя подходящие изоляты Р. infestans. Растения в возрасте четырех недель пропитывали раствором Agrobacterium tumefaciens штамма COR308 (Hamilton и др., 1996), несущего предполагаемые кандидатные R-гены. За два дня до пропитывания A. tumefaciens растили в течение ночи при 30°С в среде LB с тетрациклином (12.5 мг/мл) и спектиномицином или канамицином (100 мг/мл и 50 мг/мл, соответственно). После 16 ч роста измеряли OD-i и 50 мл среды YEB заражали × мкл культуры в LB и растили в течение ночи при 30°С для того, чтобы достичь OD2, равной 0.8 на следующий день [x=z/OD1, где z=80000 (2 степень (дельта t/2)]. На следующий день 45 мл культуры в YEB центрифугировали в течение 8 мин при 4000 об/мин. Осадок ресуспендировали в мл ММА, содержащей 1 мл/л ацетосирингона. Y=22×OD2 позволял стандартизировать различные культуры на уровне OD3 равной 2.0. Каждый ресуспендированный осадок инкубировали в течение часа при комнатной температуре. Затем нижнюю сторону листа пропитывали культурой ММА при OD4, равной 0.1, применяя 2 мл шприц. Через два дня после пропитывания выполняли тест ТОЛ, как описано выше. Фенотипы по инфицированию (устойчивые или восприимчивые) оценивали через 4-7 дней после заражения.
Тесты с отделенными листьями осуществляли, как описано у Vleeshouwers и др. (1999), используя два изолята Р. infestans, IPO-комплекс, который не вирулентен при наличии Rpi-oka1, 2 или Rpi-nrs1 и изолят ЕС1, который вирулентен при наличии всех трех генов. Листья заражали, капая по 10 мкл инокулята Phytophthora infestans (5×104 зооспор/мл) на нижнюю сторону листьев, и инкубировали при 15°С в течение 6 дней в камере искусственного климата со световым периодом 16 ч день/8 ч ночь. Через 6 дней после заражения листья, на которых выявили спорообразование, оценивали как восприимчивые, тогда как листья, на которых не выявили симптомов или некротических повреждений, оценивали как устойчивые. Проводили три независимых теста на временную комплементацию в трех повторностях с обоими изолятами. Для каждой повторности листья с номерами 4, 5 и 6, если считать от нижней части растения, пропитывали и впоследствии провоцировали Р. infestans. Через пять дней после заражения IPO-С, 60-70% листьев, временно экспрессирующих кандидатные гены Rpi-oka1 или Rpi-nrs1, проявили типичный гиперчувствительный ответ, что происходило и у положительных контрольных растений, временно экспрессирующих функциональный ген Rpi-sto1 (Vleeshouwers и др., 2008), хотя в последнем случае эффективность комплементации была значительно более высокой (80-90% провоцированных листьев проявили гиперчувствительный ответ). В противоположность листья, экспрессирующие abptGH-а, нефункциональный паралог Rpi-abpt (Lokossou и др., в подготовке) были полностью восприимчивыми. В случае ЕС1 все пропитанные листья были восприимчивы, за исключением пропитанных Rpi-sto1, который придает устойчивость к ЕС1. Эти результаты совпадали со спектром устойчивости Rpi-oka1 и Rpi-nrs1 и, следовательно, позволили предположить, что кандидатные гены представляли собой Rpi-oka1 и Rpi-nrs1.
ПРИМЕР 4В
Анализ комплементации путем стабильной трансформации cv. Desiree
Для подтверждения результатов, полученных с помощью тестов на временную комплементацию в N. benthamiana, бинарные конструкции Gateway, несущие Tm2GH-oka1b и Tm2GH-oka1.9, переносили в восприимчивый картофель культурного сорта Desiree путем опосредованной Agrobacterium трансформации. Для контроля мы также трансформировали культурный сорт Desiree конструкцией pSLJ21152, бинарной конструкцией, несущей фрагмент 4.3 т.п.н., влючающий предполагаемые последовательности промотора, ОРС и терминатора Rpi-oka1 (см. ПРИМЕР 6). Первичные трансформанты, несущие интересующие трансгены, тестировали на устойчивость к Р. infestans в тестах с отделенными листьями. Неожиданно, лишь генетическая конструкция, несущая фрагмент 4.3 т.п.н. Rpi-oka1, оказалась способной дополнять восприимчивый фенотип; 8 из 9 первичных трансформантов оказались устойчивыми. Все 22 содержащие Tm2GH-oka1b и 17 7m2GH-oka1.9 первичные трансформанты были восприимчивы к Р. infestans.
Выравнивание последовательностей Tm2GH-oka1b и T/772GH-oka1.9 с фрагментом 4.4 т.п.н. Rpi-oka1 вявило присутствие дополнительного инициирующего кодона ATG в пределах рамки считывания в 99 нт против хода транскрипции от инициирующего кодона, который первоначально использовали в качестве основы для экспериментов по поиску аллелей. Это открытие вместе с отрицательными результатами комплементации, полученными для Tm2GH-oka1b и Tm2GH-oka1.9, и положительным результатом комплементации для фрагмента 4.3 т.п.н. Rpi-oka1, позволили предположить, что расположенный ближе к 5'-концу инициирующий кодон представляет собой фактический инициирующий кодон для ОРС функциональных Rpi-oka1 и Rpi-nrs1.
Тесты на временную комплементацию с применением 5'-удлиненных продуктов для поиска аллелей
Для обнаружения предполагаемых полноразмерных генов Rpi-oka1 и Rpi-nrs1 в oka7014-9 nrs365-1, соответственно, геномную ДНК обоих генотипов подвергали ПЦР длинных фрагментов с применением праймеров ATG2-Tm2F и TAA-8bR (Таблица 2). Ампликоны ожидаемого размера клонировали в вектор pGEM®-T Easy и секвенировали. Клоны, полученные из oka7014-9, были все одинаковы и идентичны с соответствующей последовательностью в pSLJ21152 (см. ПРИМЕР 6). Клоны, полученные из nrs365-1, также были все идентичны, но включали вставку из 42 нт в 5'-удлиненном участке по сравнению с таковыми, полученными из oka7014-9. Обе последовательности впоследствии вставляли в бинарный экспрессионный вектор Gateway® между регуляторными элементами гена Rpi-blb3 (Lokossou и др., в подготовке) и проводили анализ временной комплементации в N. benthamiana вместе с исходными конструкциями Tm2GH-oka1b и Tm2GH-oka1.9 и pSLJ21152. Оба полноразмерных гена и 4.3 т.п.н. ген Rpi-oka1 проявили сравнимые уровни устойчивости с таковыми для положительного контроля Rpi-sto1 (80-90% провоцированных листьев проявили гиперчувствительный ответ), тогда как более короткие генные конструкции снова проявили значительно более низкие уровни устойчивости (60-70% проявили гиперчувствительный ответ), что указывает на то, что полноразмерные ампликоны, полученные из oka7014-9 и nrs365-1, представляют собой Rpi-oka1 и Rpi-nrs1, соответственно.
ПРИМЕР 5. ИДЕНТИФИКАЦИЯ, КАРТИРОВАНИЕ И КЛОНИРОВАНИЕ ГЕНОВ Rpi ИЗ S. mochicluense
Картирование генов Rpi в S. mochiauense
Rpi-mcq1
Rpi-mcq1 был ранее картирован в основном на нижней части длинного плеча хромосомы IX (Smilde и др., 2005), хотя не были описаны его тонкое картирование или характеристика. Были разработаны фланкирующие маркеры, которые покрывают расстояние в 20 сМ, вдобавок к маркеру (TG328), который косегрегировал с Rpi-mcq1 в популяции из 68 растений. Для тонкого картирования Rpi-mcq1 использовали всего 72 комбинации праймеров для ПДАФ, чтобы найти более сильно сцеппленные маркеры. Был идентифицирован один полиморфный бэнд Р13М32_472 для картирования южной стороны гена. Было разработано 5 дополнительных CAPS-маркеров (Таблица 7) из высвобожденной последовательности ВАС-клонов томата С09НВа0165Р17 и другие известные RFLP-маркеры хромосомы IX из базы данных SGN. Таким образом, Rpi-mcq1 был картирован на участке 11.6 сМ, фланкированном маркерами Т0156 и S1d11, и косегрегировал с CAPS-маркерами TG328, U286446, U296361, и TG591 (Фиг.10).
Картирование с высоким разрешением и клонирование Rpi-mcql Скрининг ВАС-библиотеки
В библиотеке К182 маркер U279456 был амплифицирован в девяти пулах (Таблица 1а). Этот маркер аллель-специфичный и связан с восприимчивым гаплотипом в К182. Другие три маркера (U282757, U296361 и TG591) представляют собой CAPS-маркеры, и поэтому расщепление рестриктазами использовали для определения гаплотипа идентифицированных ВАС. Анализ этих трех маркеров показал, что от трех до семи ВАС-пулов содержали ВАС-клоны с маркерными аллелями из устойчивого гаплотипа (Таблица 1а, Фиг.4d-4e).
Картирование с высоким разрешением и клонирование Rpi-mcq1
Произвольно выбирали восемь ВАС-пулов из библиотеки К182, которые были положительны по U282757, U296361, TG591 или U279465, маркерам, которые сильно связаны с Rpi-mcq1. Исходные 384-луночные планшеты для каждой из ВАС-библиотек реплицировали на твердую среду LB. Колонии из каждого планшета соскабливали по рядам и колонкам и осуществляли скрининг на присутствие соответствующего маркера. Отбирали отдельные клоны из 384-луночных планшетов. Одна из ВАС-концевых последовательностей из клонов, отобранных из библиотеки К182, была значительно похожа на ген Tm-22. Дополнительно, две из ВАС-концевых последовательностей из библиотеки К182 были похожи на несколько различных белков устойчивости, идентифицированных в Solanum tuberosum, Malus baccata, Populus balsamifera, Populus trichocarpa, Medicago truncatula и Lens culinaris.
Использовали расширенную популяцию, состоящую из 2502 растений, чтобы идентифицировать рекомбинанты между фланкирующими маркерами Т0156 и S1d11. Всего было обнаружено 163 рекомбинанта и их использовали для анализа косегрегирующих маркеров TG328, U286446, U296361, и TG591. В результате Rpi-mcq1 был картирован на участке длиной 0.32 сМ на хромосоме IX, фланкированном маркерами U286446 и 9C23R (маркер, разработанный по ВАС-концевой последовательности), и косегрегирующем с маркерами U272857 и TG591 (Фиг.10). На основании порядка CAPS-маркеров соответствующие ВАС-клоны были упорядочены и использовались для создания контига, который покрывал участок от 9C23R до TG328 (Фиг.11). Участок, на котором предположительно находился Rpi-mcq1, был покрыт двумя перекрывающимися ВАС-клонами, 9С23 и 43В09, которые были идентифицированы из устойчивых гаплотипов. Эти два ВАС-клона секвенировали и субклонировали в бинарный космидный вектор pCLD04541 путем неполного гидролиза рестриктазой Sau3AI. Анализ последовательности ВАС показал, что она содержит 2 полные ОРС и 2 неполные ОРС, которые похожи на ген устойчивости к ToMV Tm22. Предположили, что 2 полные ОРС были кандидатами на Rpi-mcq1, и космидные клоны, содержащие эти две ОРС (Rpi-mcq1.1 и Rpi-mcq1.2), были идентифицированы и введены в восприимчивый сорт картофеля Desiree, культурный сорт томата Moneymaker и N. benthamiana путем опосредованной Agrobacterium трансформации.
Анализ OPC Rpi-mcg1
Мы секвенировали два кандидатных ВАС-клона 9С23 и 43В09. ВАС-клон К182_43В09 имел длину 103,863 п.о. и был полностью секвенирован. ВАС-клон К182_9С23 был секвенирован до 3 контигов, К182_9С23_2699 (62,389 п.о.), К182_9С23_2732 (22,072 п.о.), и К182_9С23_2737 (10,119 п.о.). После выравнивания последовательностей ВАС было обнаружено, что К182_43В09 содержит весь К182_9С23_2737 и приблизительно половину К182_9С23_2732, что указывает на наличие перекрывающихся участков между двумя ВАС-клонами.
Было обнаружено, что К182_9С23_2699 содержит 3 OPC и К182_9С23_2732 и К182_9С23_2737 каждый содержат 1 OPC длиннее 300 п.о. OPC из К182_9С23_2732 и К182_9С23_2737 были идентичны первой и второй OPC из К182_43В09. Первая OPC К182_9С23_2699 кодировала предполагаемую NAD-зависимую эпимеразу (тот же ген, что и CAPS-маркер U272857). Вторая кодировала целый белок гена устойчивости растения CC-NBS-LRR типа, который был значительно похож на ген устойчивости к ToMV Tm22 и, таким образом, представлял собой кандидатный ген for Rpi-mcq1 (Rpi-mcq1.1). Третья OPC кодировала нецелый Tm22-подобный белок, который содержал часть NBS-мотива и полный LRR-мотив. Также было 5 дополнительных OPC, предсказанных по последовательности К182_43В09. Предположили, что первая и третья OPC кодируют РНК-зависимые ДНК-полимеразы и белки ретротранспозонов, пятая OPC кодировала тот же ген, что и CAPS-маркер U296361. Их не рассматривали в качестве Rpi-mcq1 кандидатов, так как такие белки не связаны с функцией гена устойчивости растения. Вторая и четвертая OPC были похожи на ген устойчивости Tm22. Вторая OPC кодировала полный ген, в котором были обнаружены все домены СС, NB и LRR; его рассматривали как второй кандидат в Rpi-mcq1 (Rpi-mcq1.2). Четвертая OPC предположительно кодировала укороченный белок, так как в нем присутствовал ранний стоп-кодон после аминокислоты 110.
Два кандидатных гена Rpi-mcq1 субклонировали в бинарный космидный вектор pCLD04541. Rpi-mcq1.1 имел длину 2,589 п.о. и предположительно кодировал белок с CC-NB-LRR, состоящий из 862 аминокислот, с рассчитанной молекулярной массой 98.2 кДа и pI 7.75. Биспиральный (СС) домен был расположен в N-концевой части белка между аминокислотами 1 и 173. В первых 173 остатках можно было узнать 12 предполагаемых мотивов групп из семи гидрофобных остатков. Домен NB-ARC, который содержал все описанные мотивы, присутствовал в последовательности аминокислот с 173 по 478 (van der Biezen & Jones, 1998). Домен LRR присутствовал в последовательности аминокислот на участке с 479 по 862, который включал ряд из 15 мотивов LRR неправильного размера, которые можно было выровнять согласно следующей консенсусной последовательности LxxLxxLxxLxLxxC/N/Sx(x)LxxLPxx (где х представляет собой любую аминокислоту) (McHale и др., 2006). Мотив 5 NB-ARC (положения аминокислот 481-485) перекрывался с началом домена LRR.
Rpi-mcq1.2 имел длину 2,571 п.о. и предположительно кодировал белок СС-NBS-LRR, состоящий из 856 аминокислот, с рассчитанной молекулярной массой 98.0 кДа и pI 7.81. Биспиральный (СС) домен был расположен в М-концевой части белка между аминокислотами 1 и 170. Домен NB-ARC присутствовал в последовательности аминокислот с 171 по 476 и содержал все описанные мотивы (van der Biezen & Jones, 1998). Домен LRR присутствовал в последовательности аминокислот с 477 по 856, и включал ряд из 15 мотивов LRR неправильного размера, которые можно было выровнять согласно следующей консенсусной последовательности LxxLxxLxxLxLxxC/N/Sx(x)LxxLPxx (где х представляет собой любую аминокислоту) (McHale и др., 2006). Мотив 5 NB-ARC перекрывался с началом домена LRR.
Rpi-mcq1.1 и Rpi-mcq1.2 были приблизительно на 77% и 75% идентичны, соответственно, белку Tm22 на уровне аминокислот, и на 81% идентичны друг другу.
ПРИМЕР 6. ВВЕДЕНИЕ НОВЫХ ГЕНОВ УСТОЙЧИВОСТИ В ГЕНОТИПЫ КАРТОФЕЛЯ И ТОМАТА, ВОСПРИИМЧИВЫЕ К PHYOPHTHORA INFESTANSб И В N. BENTHAMIANA
Бинарный вектор с геном Rpi-oka1 под контролем его собственного промотора и терминатора (исходный ген)
Фрагмент 4.3 т.п.н. (SEQ. ID. No. 1с), несущий промотор, открытую рамку считывания (ОРС) и терминатор Rpi-oka1, амплифицировали с помощью ПЦР, используя праймеры oka1longF (5'-AGTTATACACCCTACATTCTACTCG-3') и oka1longR (5'-CTTTGAAAAGAGGCTTCATACTCCC-3'), из ВАС-клона К39_26619. Этот фрагмент клонировали в pGEM-T Easy (Promega) и секвенировали для подтверждения того, что во время ПЦР не было введено ошибок. Полученную плазмиду расщепляли рестриктазой EcoRI и фрагмент, содержащий исходный ген, клонировали в сайт EcoRI в pBin19. Полученную плазмиду назвали pSLJ21152. Плазмиду pSLJ21152 внедряли в штамм AGL1 Agrobacterium tumefaciens.
Бинарный вектор с генами Rpi-mcq1.1 и Rpi-mcq1.2 под контролем их собственных промоторов и терминаторов (исходные гены)
ВАС-клоны К182_9С23 и К182_43В09 субклонировали путем неполного гидролиза рестриктазой Sau3AI в сайт BamHI бинарного космидного вектора pCLD04541. Рекомбинантный космидный вектор упаковывали, применяя GigaPack Gold (Stratagene), и внедряли в штамм DH5[альфа]Е.coli. Осуществляли скрининг полученных клонов с помощью ПЦР, используя праймеры TG591-F и TG591-R, чтобы идентифицировать клоны, содержащие кандидаты в Rpi-mcq1, и положительных клонов отбирали для секвенирования концов. Клоны, несущие два кандидата Rpi-mcq1, идентифицировали исходя из целых ВАС-последовательностей ВАС-клонов К182_9С23 и К182_43В09. Клон pSLJ21153 включал целую последовательность Rpi-mcq1.1, включая последовательности промотора и терминатора (SEQ. ID. No 2с). Этот клон также содержал дополнительный гомолог гена устойчивости, в котором отсутствовал биспиральный домен и часть домена NBS и, следовательно, предположили, что он не функциональный. Для подтверждения этого, дополнительный клон, обозначенный D5, который содержал указанный укороченный ген при отсутствии Rpi-mcq1.1, также был идентифицирован. Клон pSLJ21148 включал целую последовательность Rpi-mcq1.2, включая последовательности промотора и терминатора (SEQ. ID. No 2d). Клоны, несущие полноразмерные кандидатные гены Rpi-mcq1 и укороченный ген, внедряли в штаммы AGL1 и LBA4404 Agrobacterium tumefaciens. Чтобы удостовериться в том, что не произошло перестроек плазмид, плазмиду выделяли из полученных трансконъюгантов, трансформировали обратно в штамм DH10-[бета]E. coli, расщепляли рестриктазами и сравнивали с рестрикциями исходных стоков плазмид.
Трансформирование картофеля
Создали культуру(ы) Agrobacterium tumefaciens в условиях селекции подходящим антибиотиком и растили в течение 24 часов при встряхивании на 28°С. Собирали срезы междоузлия стеблей (без узлов) растений картофеля культурного сорта Desiree возрастом 4-6 недель, которые растили в асептической культуре на среде MS (2% сахароза). Междоузлия разрезали на 1 см срезы и помещали в 20 мл питательной среды LSR. 100 мкл ночной культуры Agrobacterium tumefaciens добавляли на срезы стебля и инкубировали в течение 20 минут при 40 об/мин в темноте при 24°С. Срезы стебля удаляли из суспензии Agrobacterium tumefaciens, насухо промокали и инкубировали в условиях слабого освещения при 18С в течение 3 дней на твердой среде LSR1 (на чашку высевали около 15-20 кусочков ткани). Совместно культивируемые срезы стебля затем переносили в среду LSR1 с антибиотиками для селекции в количестве около 10 кусочков ткани на чашку. Кусочки стебля субкультивировали на свежей LSR1 среде каждые 7-10 дней в течение приблизительно 3-6 недель или до момента появления первых небольших каллюсов. После того как каллюсы достаточно развились, срезы стебля переносили на среду LSR2 с антибиотиками для селекции. Срезы стебля субкультивировали каждые 7-10 дней до тех пор, пока не начали появляться всходы. Всходы появлялись в течение 2 месяцев с момента начала трансформации. Всходы удаляли с помощью острого скальпеля и высаживали на твердую среду MS2R с антибиотиками для селекции. Трансгенные растения, несущие подходящие гены устойчивости к антибиотику или гербициду, начинали образовывать корни, как правило, в течение 2 недель, и их отсаживали из культуры ткани в стерильные торфяные блоки перед пересадкой в теплицу.
Среда
Среда MS для саженцев картофеля
1Х среда Мурасиге-Скуга
2% сахарозы
0.6% агарозы
100 мг/л гидролизата казеиновой кислоты
pH 5.7
Питательная среда LSR
1Х среда Мурасиге-Скуга
3% сахарозы
pH 5.7
Среда LSR1
1Х среда Мурасиге-Скуга
3% сахарозы
2.0 мг/л зеатинрибозида
0.2 мг/л α-нафтилуксусной кислоты (NAA)
0.02 мг/л GA3
0.6% агарозы
pH 5.7
Среда LSR2
1Х среда Мурасиге-Скуга
3% сахарозы
2.0 мг/л зеатинрибозида
0.02 мг/л NAA
0.02 мг/л GA3
0.6% агарозы
pH 5.7
MS2R
1Х среда Мурасиге-Скуга
2% сахарозы
100 мг/л миоинозитола
2.0 мг/л глицина
0.2% Gelrite
Трансформирование томата
Поверхность семян томата стерилизовали в течение 2 минут в 70% этаноле, чтобы очистить семя от желатиновой оболочки, а затем один раз ополаскивали стерильной водой. Затем семена стерилизовали в 10% растворе бытового отбеливателя (например, Domestos/Vortex) в течение 3 часов при встряхивании и промывали 4 раза в стерильной воде. Семена помещали в кадки (20-30 семян/кадку), содержащие среду для проращивания, и инкубировали при 25°С в помещение для культивирования (световой период 16 часов, дополняемый освещением Gro-Lux или лампами накаливания). Рассаду выращивали в течение 7-10 дней и использовали на стадии, когда семядоли достаточно молодые, продолжают расти и не наблюдается явного образования листьев. Десять миллилитров минимальной среды А, содержащей подходящие антибиотики, заражали штаммом LBA4404 A. tumefaciens и растили при встряхивании на 28С. Один миллилитр тонкой суспензионной культуры табака помещали на чашки, содержащие среду для суспензионной культуры, отвержденную 0.6% агарозой, или среду MS, дополненную 0.5 мг/л 2,4-D и 0.6% агарозой. Клетки распределяли, чтобы получить ровный слой, и чашки помещали незапечатанными и составленными в стопку в помещение для культивирования при 25°С при слабом свете до наступления следующего дня. Кусок фильтровальной бумаги Whatman no.1 помещали на поверхность чашек с фидером, соблюдая осторожность, чтобы исключить образование пузырьков воздуха и удостовериться в том, что бумага полностью смочена. Для трасформации использовали семядоли, так как гипокотиль давал большое количество тетраплоидов. В чашке Петри отрезали кончики от семядолей, а затем осуществляли еще два поперечных разреза с получением двух кусочков ткани приблизительно по 0.5 см в длину. Кусочки ткани переносили в чистую чашку Петри с водой, чтобы предотвратить какое-либо повреждение во время дополнительного разрезания. После того как было собрано достаточно кусочков ткани, их высушивали промоканием на стерильной фильтровальной бумаге и помещали приблизительно 30-40 штук на чашку с фидером, нижней поверхностью кверху (в перевернутом положении). Чашки Петри помещали незапечатанными и сложенными в стопку на 25°С при низкой интенсивности света в течение 8 часов. Культуру Agrobacterium откручивали и ресуспендировали в среде MS, содержащей 3% сахарозу, до OD600, равной 0.4-0.5. Бактериальную суспензию переносили в чашку Петри и кусочки ткани из одной чашки с фидером погружали в суспензию. Их затем удаляли и сушили промоканием на стерильной фильтровальной бумаге перед их возвращением в исходную чашку с фидером, снова заботясь о том, чтобы не повредить ткань. Нет конкретного требуемого периода нахождения кусочков ткани в среде с бактериями, достаточно лишь удостовериться в полном погружении кусочков в жидкость. Чашки возвращали в те же условия, которые использовали в фазе предварительного инкубирования (25°С при низкой интенсивности света) и кокультивировали в течение 40 часов. Кусочки ткани с фидерных слоев помещали (по 12 кусочков ткани на чашку Петри) на чашки для регенерации томата, содержащие аугментин или карбенициллин при концентрации 500 мкг/мл и канамицин при концентрации 100 мкг/мл, чтобы провести селекцию по маркеру трасформации Т-ДНК. Семядоли помещали нужной стороной кверху, так чтобы они закручивались внутрь в среде, обеспечивая хороший контакт между краями разреза листа и питательными веществами и антибиотиками в среде. Агар-гель использовали в качестве сгущащего агента, так как он позволял получить мягкую среду, в которую можно аккуратно протолкнуть кусочки. Чашки оставляли незапечатанными и возвращали в прежние условия культивирования (25°С при низкой интенсивности света). Кусочки ткани переносили в свежую среду каждые 2-3 недели. После того как регенерировавший материал становился слишком большим для чашек Петри, его помещали в большие стеклянные банки с завинчивающейся крышкой. Всходы срезали с кусочков ткани и помещали в среду для корнеобразования с аугментином при концентрации 200 мкг/мл и канамицином при концентрации 50 мкг/мл. Для переноса в почву максимально возможное количество среды удаляли путем аккуратной промывки корней в проточной воде. Растения аккуратно переносили в гидратированные, автоклавированные горшки Jiffy (торфоперегнойные горшки) и держали их закрытыми, чтобы поддерживать высокую влажность при нахождении в комнате для роста. Влажность постепенно снижалась. После того как стало видно, что корни прорастают сквозь горшки Jiffy, растения переносили в теплицу.
При 1000х не все витамины переходят в раствор. Хранить при 4°С и втряхивать перед применением.
Для чашек:
Приготовить описанную выше среду в 500 мл и автоклавировать.
Отдельно автоклавировать 15 г бактоагара в 490 мл H2O
Добавить MgSO4 и глюкозу и объединить.
Трансформирование Nicotiana benthamiana
Растения N. benthamiana растили до тех пор, пока они не стали размером 10-20 см в высоту, но перед этим они начали цвести. Культуры Agrobacterium tumefaciens начали вести в условиях селекции подходящим антибиотиком и растили в течение 24 часов при встряхивании на 28°С. На следующий день культуры A. tumefaciens откручивали и ресуспендировали в среде Мурасиге-Скуга, содержащей 3% сахарозу. Молодые листья N. benthamiana (вплоть до 10 см в диаметре) собирали и поверхность стерилизовали в 1% свежем гипохлорите натрия, содержащем несколько капель Tween 20, действующего в качестве поверхностно-активного вещества, в течение 20 минут. Листья затем хорошо промывали стерильной водой, разрезали на 1-2 см квадраты с помощью острого скальпеля и погружали в суспензию Agrobacterium tumefaciens. Удостоверившись в том, что листья были полностью смочены, их затем быстро высушивали промоканием и помещали на среду для совместного культивирования на 3 дня. После этого кокультивируемые кусочки листьев переносили на селективную среду с подходящими антибиотиками приблизительно по 10 кусочков ткани на чашку. Кусочки ткани субкультивировали на свежей среде каждые 7-10 дней в течение приблизительно 1-2 месяцев до момента появления первых всходов. Всходы удаляли с помощью острого скальпеля и высаживали в среду для корнеобразования с антибиотиками для селекции. Трансгенные растения, несущие подходящие гены устойчивости к антибиотику или гербициду, начинали образовывать корни, как правило, в течение 2 недель, и их можно было отсаживать из культуры ткани в стерильные торфяные блоки перед пересадкой в теплицу.
Питательная среда MS
1Х среда Мурасиге-Скуга
3% сахарозы
pH 5.7
Среда для совместного культивирования
1Х основная смесь солей Мурасиге-Скуга
1Х витамин В5 Gamborg
3% сахарозы
0.59 г/л MES
1.0 мг/л ВАР
0.1 мг/л NAA
0.6% агарозы
pH 5.7
Селективная среда
1Х основная смесь солей Мурасиге-Скуга
1Х витамин В5 Gamborg
3% сахарозы
0.59 г/л MES
1.0 мг/л ВАР
0.1 мг/л NAA
0.4% агар-гель
pH 5.7
Среда для образования корней
1/2 концентрации среды Мурасиге-Скуга
0,5% сахарозы
0.25% Gelrite
pH 5.8
Анализ комплементации [Rpi-oka1).
В общей сложности 37 растений S. tuberosum культурного сорта Desiree, способные расти на канамицине, были отобраны как предполагаемые трансформанты Rpi-oka1. После переноса в теплицу листья вырезали и использовали в тесте с отделенными листьями с изолятами Р. infestans 90128 и Superblight, чтобы определить, придавал ли трансген устойчивость к фитофторозу. Из 37 трансформантов, 31 действительно оказались устойчивыми и не проявили каких-либо признаков фитофторозной инфекции. Некоторые растения проявили признаки сверхвосприимчивости, локализованные в месте заражения. Оставшиеся 6 растений были восприимчивы к обоим изолятам, так же как и контроль (нетрансформированные Desiree). Фенотип трансгенных растений точно коррелировал с амплификацией ОРС Rpi-oka1 методом ПЦР для всех растений, из которых амплифицировался Rpi-oka1, подтвердили их устойчивость. Трансген Rpi-oka1 также придавал устойчивость к ряду изолятов Р. infestans, что подробно описано в Таблице 6.1. Все тестированные трансгенные растения были восприимчивы к изоляту ЕС1, показывая, что в трансгенных растениях сохранялась специфичность Rpi-oka1 и что фенотип устойчивости возникал не вследствие конститутивной активации трансгеном сигнальных путей защиты. Для трансгенных растений томата культурного сорта Moneymaker, несущих Rpi-oka1, также было показано, что они устойчивы к изоляту 90128Р. infestans. Всего 21 растение культурного сорта Moneymaker S. lycopersicum, способное расти на канамицине, отбирали как предполагаемые трансформанты Rpi-oka1. После переноса в теплицу листья вырезали и использовали в тесте с отделенными листьями с изолятом 90128 Р. infestans, чтобы определить придавал ли трансген устойчивость к фитофторозу. Из 21 трансформанта 13 действительно оказались устойчивыми и не проявили каких-либо признаков фитофторозной инфекции. Некоторые растения проявили признаки сверхвосприимчивости, локализованные в месте заражения. Оставшиеся 8 растений оказались восприимчивыми к изолятам 90128, таке как и контроль (нетрансформированный Moneymaker). Фенотип трансгенных растений, как правило, коррелировал с амплификацией ОРС Rpi-oka1 с помощью ПЦР, для большинства растений, из которых амплифицировали Rpi-oka1, подтвердили их устойчивость. Тем не менее, два растения, которые содержали Rpi-oka1, что определили с помощью ПЦР, все еще были восприимчивыми, что указывает на то, что трансген либо был подавлен либо был вставлен в транскрипционно неактивный участок реципиентного генома томата. Все растения, из которых невозможно было амплифицировать Rpi-oka1, были восприимчивы к Р. infestans.
Спектр изолятов Р. Infestans. устойчивость к которым придает Rpi-oka1
Отсоединенные листья трансгенного картофеля культурного сорта Desiree, несущего Rpi-oka1, заражали рядом изолятов Р. infestans (Таблица 6.1), чтобы определить диапазон изолятов, к которым придает устойчивость Rpi-oka1. Из 11 тестированных изолятов только изолят ЕС1 из Эквадора смог преодолеть Rpi-oka1 и вызвать заболевание на зараженных растениях.
Анализ комплементации (Rpi-mcq1.1 и Rpi-mcq1.2)
Всего 22 и 20 предполагаемых трансгенных линий S. tuberosum культурного сорта Desiree получали после трасформации pSLJ21153 (Rpi-mcq1.1) и pSLJ21148 (Rpi-mcq1.2), соответственно. После переноса в теплицу проводили тесты с отделенными листьями, применяя изоляты 90128, ЕС1, Hica и IPO-complex P. infestans. Для конструкции pSLJ21153, для 12 трансгенных линий было показано, что они устойчивы к изолятам 90128 и ЕС1, но восприимчивы к Hica и IPO-complex. Для конструкции pSLJ21148, 10 трансгенный линий проявили повышенную устойчивость к Hica при заражении при низкой концентрации (1×104 зооспор мл-1), но были восприимчивы к 90128, ЕС1 и IPO-complex. Rpi-mcq1.2, присутствующий в этой конструкции, также придавал частичную устойчивость к изолятам Superblight и МР618 (Таблица 6). Для линий трансгенного картофеля, трансформированных космидой D5, которая содержала укороченный гомолог гена устойчивости, также присутствующий в pSLJ21153, было показано, что они восприимчивы ко всем тестируемым изолятам Р. infestans.
Двумя конструкциями (pSLJ21153 и pSLJ21148), несущими Rpi-mcq1.1 и Rpi-mcq1.2, соответственно, трансформировали культурный сорт томата Moneymaker. Было идентифицировано две линии, положительные по Rpi-mcq1.1, и 11 линий, положительных по Rpi-mcq1.2, с помощью ПЦР с применением специфичных для этих генов праймеров. В двух трансгенных линиях Rpi-mcq1.1 придавал устойчивость к 90128 и ЕС1. Трансгенные линии Moneymaker, несущие Rpi-mcq1.2 или укороченный гомолог R-гена, который также присутствовал в конструкции pSLJ21153 с Rpi-mcq1.1 (космида D5), были восприимчивы к 90128 и ЕС1.
Спектр изолятов Р. Infestans, устойчивость к которым придают Rpi-mca1.1 и Rpi-mcq1.2
Тестировали более широкий диапазон изолятов Р. infestans, чтобы определить их вирулентность/невирулентность на Rpi-mcq1.1 и Rpi-mcq1.2 (Таблица 6.1). Из 12 тестируемых изолятов Rpi-mcq1.1 придавал устойчивость к 6 изолятам, и Rpi-mcq1.2 придавал частичную устойчивость к 3 изолятам (Таблица 6.1).
ПРИМЕР 7. СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ИЗБЕЖАНИЯ РАЗВИТИЯ УСТОЙЧИВОСТИ К НОВЫМ ГЕНАМ
Гены устойчивости в диких популяциях обычно высокополиморфны (Jones 2001, DangI и Jones 2001, Bergelson и др., 2001), и эта гетерогенность, вероятно, имеет критическое значение для их эффективности, поскольку они подвержены частотозависимому отбору (если какой-либо из R-генов доминирует, отбор усиливается в направлении штаммов патогена, которые могут преодолеть его). В сельском хозяйстве монокультуры являются стандартом, который облегчает развитие эпидемии любого патогена, который способен к росту и быстрому воспроизведени на конкретном сорте сельскохозяйственной культуры. Авторы настоящего изобретения предположили, что если идентифицировать достаточно R-генов и применять их в смесях на генетическом фоне, который иначе однороден по агрономическим и потребительским качествам, тогда можно достичь длительной устойчивости сельскохозяйственных культур (Pink и Puddephat 1999, Jones 2001). Чтобы эта стратегия сработала и чтобы решить проблемы, связанные с преодолением связанных с R-генами устойчивости и эволюции, необходимо выделить так много генов Rpi, после того как возможно.
Указанная задача была решена согласно настоящему изобретению путем выделения множества генов Rpi из диких родственников картофеля, включения этих генов по отдельности в один сорт, объединения (смешивания) полученных линий и посадки. Эта стратегия позволяет обойти проблему, состоящую в том, что сортовые монокультуры становятся полностью восприимчивыми к любому штамму фитофтороза, который может преодолеть конкретный ген Rpi в этом сорте, и этот штамм затем доминирует в популяции паразитов. Применяя 3 гена Rpi в смеси, любой штамм фитофтороза, который преодолеет один из этих генов Rpi, сможет расти лишь на 33% растений в поле. Альтернативная стратегия согласно настоящему изобретению включает применение того же сорта, но несущего каждый год различный ген Rpi. При этом сценарии штаммы патогена, которые успешны в один год, на следующий год оказываются неуспешными или менее успешными, что приводит к снижению потерь. В частности, настоящее изобретение предусматривает:
1. Замедление эпидемий
Если штамм Р. infestans, который вирулентен на одном из R-генов, попадет в сельскохозяйственную культуру, распространение этого штамма будет ограничено растениями, которые несут этот R-ген, другие растения, несущие отличные R-гены, останутся устойчивыми, что приведет к ограничению распространения патогена и суммарных потерь. Для патогена значительно сложнее развить вирулентность против множества R-генов, и поэтому развитие штаммов, способных вызывать заболевание растения с более чем одним R-геном, будет минимизировано. Поскольку растения с различными R-генами будут смешаны, будет обеспечено пространственное разделение между растениями с одним и тем же R-геном, что ограничит скорость распространения в поле.
2. Вирулентность на растении с более чем 1 геном Rpi приводит к снижению способности в приспособлении по сравнению с таковой у вирулентного только на растении с 1 геном
Продукты R-генов растений узнают молекулы, продуцируемые конкретными штаммами патогенов растения. Согласно принятой на сегодняшний день гипотезе, патоген приобретает вирулентность путем потери гена, который кодирует эффекторную молекулу, которая, как правило, требуется патогену для преодоления защитных ответов хозяина и обычно распознается R-геном растения. Таким образом, приобретение вирулентности для растения с более чем одним R-геном включает потерю патогеном более одной эффекторной молекулы. Такой штамм патогена будет менее эффективно конкурировать со штаммами, которые не теряли эффекторов или потеряли один эффектор, при росте на хозяевах, которые содержат различные комплементы R-генов.
3. Несовместимые штаммы инициируют системную приобретенную устойчивость
В дополнение к обусловленной Р-генами реакции устойчивости растения имеют способность запускать неспецифичную защитную реакцию, известную как системная приобретенная устойчивость (SAR). После узнавания патогена растение может инициировать защитные реакции, которые действуют против широкого диапазона патогенов и не ограничены штаммом патогена, который узнается R-геном. Таким образом, узнавание конкретного штамма патогена введеным R-геном также будет способствоать защите против других штаммов, которые не обязательно должны узнаваться какими-либо введенными R-генами.
4. Будет слабый отбор по вирулентности на редком гене Rpi
Ген Rpi, который нечасто присутствует в популяции, будет оказывать меньшее селекционное давление на патоген, чтобы преодолеть эту устойчивость. Такую стратегию можно использовать, чтобы защитить важные R-гены и удостовериться в том, что их долговечность максимизирована, путем их рационального размещения, при котором популяция патогена не встречается с большим количеством сельскохозяйственных культур с конкретным R-геном. Такому разумному размещению будет способствовать знание приблизительной частоты соответствующего аллеля вирулентности в популяции патогена.
СПИСОК ЛИТРЕРАТУРЫ
Allouis S, Moore G, Bellec A, Sharp R, Faivre Rampant P, Mortimer K, Pateyron S, Foote TN, Griffiths S, Caboche M, Chalhoub В (2003) Construction and characterisation of a hexaploid wheat (Triticum aestivum L.) ВАС library from the reference germplasm 'Chinese Spring'. Cereal Res Commun 31:331-338
Arumuganathan K, Earle ED (1991) Nuclear DNA content of some important plant species. Plant Mol Biol Rep 9:208-218
Ballvora A, Ercolano MR, WeiB J, Meksem K, Bormann CA, Oberhagemann P, Salamini F, Gebhardt С (2002) The R1 gene for potato resistance to late blight (Phytophthora infestans) belongs to the leucine zipper/NBS/LRR class of plant resistance genes. Plant J 30:361-371
Black W, Mastenbroek C, Mills WR, Peterson LC (1953) A proposal for an international nomenclature of races of Phytophthora infestans and of genes controlling immunity in Solanum demissum derivatives. Euphytica 2:173-240
Bouzidi MF, Franchel J, Tao Q, Stormo K, MrazA, Nicolas P, Mouzeyar S (2006) A sunflower ВАС library suitable for PCR screening and physical mapping of targeted genomic regions. Theor AppI Genet 113:81-89
Bradshaw JE, Bryan GJ, Lees AK, McLean K, Solomon-Blackburn RM (2006) Mapping the R10 and R11 genes for resistance to late blight (Phytophthora infestans) present in the potato (Solanum tuberosum) R-gene differentials of Black. Theor Appl Genet 112:744-751
Chalhoub В, Belcram H, Caboche M (2004) Efficient cloning of plant genomes into bacterial artificial chromosome (ВАС) libraries with larger and more uniform insert size. Plant Biotechnol J 2:181-188
Chang Y-L, Tao Q, Scheuring C, Ding K, Meksem K, Zhang H-B (2001) An integrated map of Arabidopsis thaliana for functional analysis of its genome sequence. Genetics 159:1231-1242
Chen Q, Sun S, Ye Q, McCuine S, Huff E, Zhang HH (2004) Construction of two ВАС libraries from the wild Mexican diploid potato Solarium pinnatisectum, and the identification of clones near the late blight and Colorado potato beetle resistance loci. Theor Appl Genet 108:1002-1009
Costanzo S, Simko I, Christ BJ, Haynes KG (2005) QTL analysis of late blight resistance in a diploid potato family of Solanum phureja × S. stenotomum. Theor Appl Genet 111:609-617
Danesh D, Penuela S, Mudge J, Denny RL, Nordstrom H, Martinez JP, Young ND (1998) A bacterial artificial chromosome library for soybean and identification of clones near a major cyst nematode resistance gene. Theor Appl Genet 96:196-202
De Wit, P.J.G.M. (1997) Pathogen avirulence and plant resistance: a key role for recognition. Trends In Plant Science. 2, 452-458.
EI-Kharbotly A, Leonards-Schippers C, Huigen DJ, Jacobsen E, Pereira A, StiekemaWJ, Salamini F, Gebhardt С (1994) Segregation analysis and RFLP mapping of the R1 and R3 alleles conferring race-specific resistance to Phytophthora infestans in progeny of dihaploid potato parents. Mol Gen Genet 242:749-754
EI-Kharbotly A, Palomino-Sanchez C, Salamini F, Jacobsen E, Gebhardt С (1996) R6 and R7 alleles of potato conferring race-specific resistance to Phytophthora infestans (Mont.) de Bary identified genetic loci clustering with the R3 locus on chromosome XI. Theor Appl Genet 92:880-884
Eshed Y, Zamir D (1994) A genomic library of Lycopersicon pennellii in Lycopersicon esculentum: a tool for fine mapping of genes. Euphytica 79:175-179
Ewing ЕЕ, Simko I, Smart CD, Bonierbale MW, Mizubuti ESG, May GD, Fry WE (2000) Genetic mapping from field of qualitative and quantitative resistance to Phytophthora infestans in a population derived from Solanum tuberosum and Solanum berthaultii. Mol Breeding 6:25-36
Feng J, Vick BA, Lee MK, Zhang HB, Jan CC (2006) Construction of ВАС and BIBAC libraries from sunflower and identification of linkage group-specific clones by overgo hybridization. Theor Appl Genet 113:23-32
Flor, H.H. (1971) Current status of the gene-for-gene concept. Annual Review of Phytopathology 78, 275-298
Frijters ACJ, Zhang Z, van Damme M, Wang GL, Ronald PC, Michelmore RW (1997) Construction of a bacterial artificial chromosome library containing large EcoRI and HindIII genomic fragments of lettuce. Theor Appl Genet 94:390-399
Gebhardt, C. and Valkonen, J.P. (2001) Organization of genes controlling disease resistance in the potato genome. Anna Rev Phytopathol 39, 79-102
Georgi LL, Wang Y, Yvergniaux D, Ormsbee T, Inigo M, Reighard G, Abbott AG (2002) Construction of а ВАС library and its application to the identification of simple sequence repeats in peach [Prunus persica (L.) Batsch]. Theor Appl Genet 105:1151-1158
Ghislain M, Trognitz B, Herrera MaDeIR, Solis J, Casallo G, Vasquez C, Hurtade O, Castillo R, Portal L, Orrillo M (2001) Genetic loci associated with field resistance to late blight in offspring of Solarium phureja and S. tubersosum grown under short-day conditions. Theor AppI Genet 103:433-442
Grube, R.C., Radwanski, E.R. and Jahn, M. (2000) Comparative genetics of disease resistance within Solanaceae. Genetics 155, 873-887
Helgeson JP, Pohlman JD, Austin S, Haberlach GT, Wielgus SM, Ronis D, Zambolim L, Tooley P, McGrath JM, James RV, Stevenson WR (1998) Somatic hybrids between Solarium bulbocastanum and potato: a new source of resistance to late blight. Theor AppI Genet 96:738-742
Hofmann, K., Bucher, P., Falquet, L. and Bairoch, A. (1999) The PROSITE database, its status in 1999. Nucl. Acids Res. 27, 215-219.
Huang S, Vleeshouwers VGAA, Werij JS, Hutten RCB, van Eck HJ, Visser RGF, Jacobsen E (2004) The R3 resistance to Phytophthora infestans in potato is conferred by two closely linked R genes with distinct specificities. Mol Plant Microb Interact 17:428-435
Huang S, van derVossen EAG, Kuang H, VIeeshouwers VGAA, Zhang N, Borm TJA, van Eck HJ, Baker B, Jacobsen E, Visser RGF (2005) Comparative genomics enabled the isolation of the R3a late blight resistance gene in potato. Plant J 42:251-261
Huang, S. (2005) Discovery and characterization of the major late blight resistance complex in potato: genomic structure, functional diversity, and implications. PhD thesis.
Jansky S (2000) Breeding for disease resistance in potato. Plant Breeding Rev 19:69-155
Jiang J, Gill BS, Wang GL, Ronald PC, Ward DC (1995) Metaphase and interphase fluorescence in situ hybridization mapping of the rice genome with bacterial artificial chromosomes. Proc Natl Acad Sci USA 92:4487-4491
Kamoun S, van West P, Vleeshouwers VGAA, de Groot KE, Govers F (1998) Resistance of Nicotiana benthamiana to Phytophthora infestans is mediated by the recognition of the elicitor protein 1NF1. Plant Cell 10:1413-1425
Klein PE, Klein RR, Cartinhour SW, Ulanch PE, Dong J, Obert JA, Morishige DT, Schlueter SD, Childs KL, Ale M, Mullet JE (2000) A high-throughput AFLP-based method for constructing integrated genetic and physical maps: progress toward a sorghum genome map. Genome Res 10:789-807
Kuhl JC, Hanneman Jr RE, Havey MJ (2001) Characterization and mapping of Rpi1, a late-blight resistance locus from diploid (1EBN) Mexican Solanum pinnatisectum. Mol Genet Genomics 265:977-985
Lanfermeijer FC, Dijkhuis J, Sturre MJG, de Haan P, Hille J (2003) Cloning and characterization of the durable tomato mosaic virus resistance gene Tm-22 from Lycopersicon esculentum. Plant Mol Biol 52:1037-1049
Lanfermeijer, F.C., Warmink, J. and Hille, J. (2005) The products of the broken Tm-2 and the durable Tm-2(2) resistance genes from tomato differ in four amino acids. J Exp Bot 56, 2925-2933
Lee H-R, Eom E-M, Lim Y-P, Bang J-W, Lee D-H (2003) Construction of a garlic ВАС libaray and chromosomal assignment of ВАС clones using the FISH technique. Genome 46:514-520
Leonards-Schippers C, Gieffers W, Salamini F, Gebhardt С (1992) The R1 gene conferring race-specific resistance to Phytophthora infestans in potato is located on potato chromosome V. Mol Gen Genet 233:278-283
Linden, C.C.G.v.d., Wouters, D.D.C.A.E., Mihalka, V.V., Kochieva, E.E.Z., Smulders, M.M.J.M. and Vosman, B.B. (2004) Efficient targeting of plant disease resistance loci using NBS profiling. Theoretical and applied genetics 109, 384-393.
Liu YG and Whittier RF (1994) Rapid preparation of megabase plant DNA from nuclei in agarose plugs and microbeads. Nucleic Acids Res 22:2168-2169
Li X, van Eck HJ, Rouppe van derVoort JNAM, Huigen DJ, Stam P, Jacobsen E (1998) Autotetraploids and genetic mapping using common AFLP markers: the R2 allele conferring resistance to Phytophthora infestans mapped on potato chromosome 4. TheorAppI Genet 96:1121-1128
Malcolmson JF, Black W (1966) New R genes in Solanum demissum Lindl. and their complementary races of Phytophthora infestans (Mont.) de Bary. Euphytica 15:199-203
Mastenbroek С (1953) Experiments on the inheritance of blight immunity in potatoes derived from Solatium demissum Lindl. Euphytica 2:197-206
McGrath JM, Shaw RS, de los Reyes BG, Weiland JJ (2004) Construction of a sugar beet ВАС library from a hybrid with diverse traits. Plant Mol Biol Reporter 22:23-28
McHale, L, Tan, X., Koehl, P. and Michelmore, R.W. (2006) Plant NBS-LRR proteins: adaptable guards. Genome Biol 7, 212
Meksem К, Zobrist К, Ruben E, Hyten D, Quanzhou T, Zhang H-B, Lightfoot D (2000) Two large-insert soybean genomic libraries constructed in a binary vector: applications in chromosome walking and genome wide physical mapping. Theor AppI Genet 101:747-755
Michelmore, R.W., Paran, I. and Kesseli, R.V. (1991) Identification of markers linked to disease-resistance genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions by using segregating populations. Proc Natl Acad Sci USA 88, 9828-9832.
Milbourne D, Meyer RC, Collins AJ, Ramsay LD, Gebhardt C, Waugh R (1998) Isolation, characterisation and mapping of simple sequence repeat loci in potato. Molecular and General Genetics 259:233-245
Naess SK, Bradeen JM, Wielgus SM, Haberlach GT, McGrath JM, Helgeson JP (2000) Resistance to late blight in Solanum bulbocastanum is mapped to chromosome 8. Theor Appl Genet 101:697-704
Nilmalgoda SD, Cloutier S, Walichnowski AZ (2003) Construction and characterization of a bacterial artificial chromosome (ВАС) library of hexaploid wheat (Triticum aestivum L.) and validation of genome coverage using locus-specific primers. Genome 46:870-878
Osoegawa, K, Woon PY, Zhao B, Frengen E, Tateno M, Catanese JJ, de Jong PJ (1998) An improved approach for construction of bacterial artificial chromosome libraries. Genomics 52:1-8
Ozdemir N, Horn R, Friedt W (2004) Construction and characterization of а ВАС library for sunflower (Helianthus annuus L.). Euphytica 138:177-183
Pan, Q., Liu, Y.-S., Budai-Hadrian, O., Sela, M., Carmel-Goren, L., Zamir, D. and Fluhr, R. (2001) Comparative Genetics of Nucleotide Binding Site-Leucine Rich Repeat Resistance Gene Homologues in the Genomes of Two Dicotyledons: Tomato and Arabidopsis. Genetics 159, 1867 с-.
Park T-H, VIeeshouwers VGAA, Hutten RCB, van Eck HJ, van der Vossen E, Jacobsen E, Visser RGF (2005a) High-resolution mapping and analysis of the resistance locus Rpi-abpt against Phytophthora infestans in potato. Mol Breeding 16:33-43
Park T-H, Gros A, Sikkema A, VIeeshouwers VGAA, Muskens M, Allefs S, Jacobsen E, Visser RGF, van der Vossen EAG (2005b) The late blight resistance locus Rpi-blb3 from Solarium bulbocastanum belongs to a major late blight R gene cluster on chromosome 4 of potato. Mol Plant Microb Interact 18:722-729
Park TH, VIeeshouwers VG, Huigen DJ, van der Vossen EA, van Eck HJ, Visser RG (2005с) Characterization and high-resolution mapping of a late blight resistance locus similar to R2 in potato. Theor AppI Genet 111:591-597
Rauscher, G.M., Smart, C.D., Simko, I., Bonierbale, M., Mayton, H., Greenland, A. and Fry, W.E. (2006) Characterization and mapping of RPi-ber, a novel potato late blight resistance gene from Solanum berthaultii. Theoretical and applied genetics 112, 674-687
Sandbrink JM, Colon LT, Wolters PJCC, Stiekema WJ (2000) Two related genotypes of Solanum microdontum carry different segregating alleles for field resistance to Phytophthora infestans. Molecular Breeding 6:215-225
Śliwka J, Jakuczum H, Lebecka R, Marczewski W, Gebhardt C, Zimnoch-Guzowska E (2006) The novel, major locus Rpi-phu1 for late blight resistance maps to potato chromosome IX and is not correlated with long vegetation period. Theor Appl Genet 113:685-695
Smilde WD, Brigneti G, Jagger L, Perkins S, Jones JD (2005) Solanum mochiquense chromosome IX carries a novel late blight resistance gene Rpi-moc1. Theor Appl Genet 110:252-258Song J, Bradeen JM, Naess SK, Raasch JA, Wielgus SM, Haberlach GT, Liu J, Kuang H, Austin-Phillips S, Buell CR, Helgeson JP, Jiang J (2003) Gene RB cloned from Solanum bulbocastanum confers broad spectrum resistance to potato late blight. Proc NatlAcad Sci USA 100:9128-9133
Tao Q, Wang A, Zhang MB (2002) One large-insert plant-transformation-competent BIBAC library and three ВАС libraries of Japonica rice for genome research in rice and other grasses. TheorAppI Genet 105:1058-1066
Thomas CM, Vos P, Zabeau M, Jones DA, Norcott KA, Chadwick BP, Jones JD (1995) Identification of amplified restriction fragment polymorphism (AFLP) markers tightly linked to the tomato Cf-9 gene for resistance to Cladosporium fulvum. Plant J 8:785-794
van der Vossen E, Sikkema A, Hekkert BL, Gros J, Stevens P, Muskens M, Wouters D, Pereira A, Stiekema W, Allefs S (2003) An ancient R gene from the wild potato species Solarium bulbocastanum confers broad-spectrum resistance to Phytophthora infestans in cultivated potato and tomato. Plant J 36:867-882
van der Vossen EAG, Gros J, Sikkema A, Muskens M, Wouters D, Wolters P, Pereira A, Allefs S (2005) The Rpi-blb2 gene from Solarium bulbocastanum is an Mi-1 gene homolog conferring broad-spectrum late blight resistance in potato. Plant J 44:208-222
Vilarinhos AD, Piffanelli P, Lagoda P, Thibivilliers S, Sabau X, Carreel F, Hont AD (2003) Construction and characterization of a bacterial artificial chromosome library of banana (Musa acuminate Colla). Theor Appl Genet 106:1102-1106
Vleeshouwers VGAA, van Dooijeweert W, Keizer LCP, Sijpkes L, Covers F, Colon LT (1999) A laboratory assay for Phytophthora infestans resistance in various Solanum species reflects the field situation. European Journal of Plant Pathology 105:241-250
Vos P, Hogers R, Bleeker M, Reijans M, Vandelee T, Homes M, Frijters A, Pot J, Peleman J, Kuiper M, Zabeau M (1995) AFLP: A new technique for DNA fingerprinting. Nucleic Acids Research 23:4407-4414
Wang GL, Holsten ТЕ, Song WY, Wang HP, Ronald PC (1995) Construction of a rice bacterial artificial chromosome library and identification of clones linked to the Xa-21 disease resistance locus. Plant J 7:525-533
Wastie RL (1991) Breeding for resistance. In: Ingram DS, William PH (eds) Advances in plant pathology Vol. 7. Academic press, London, pp 193-224
Woo S-S, Jiang J, Gill BS, Paterson AH, Wing RA (1994) Construction and charaterization of a bacterial artificial chromosome library of Sorghum bicolor. Nucleic Acids Res 22:4922-4931
Wu CC, Nimmakayala P, Santos FA, Springman R, Scheuring C, Meksem K, Lightfoot DA, Zhang H-B (2004) Construction and characterization of a soybean bacterial artificial chromosome library and use of multiple complementary libraries for genome physical mapping. TheorAppI Genet 109:1041-1050YQksel B, Paterson AH (2005) Construction and characterization of a peanut HindIII ВАС library. Theor Appl Genet 111:630-639.
| название | год | авторы | номер документа |
|---|---|---|---|
| ГЕН, ПРИДАЮЩИЙ УСТОЙЧИВОСТЬ К PHYTOPHTHORA INFESTANS (ФИТОФТОРОЗУ) В СЕМЕЙСТВЕ SOLANACEAE | 2003 |
|
RU2361920C2 |
| КЛОНИРОВАНИЕ И ИСПОЛЬЗОВАНИЕ ФУНКЦИОНАЛЬНОГО R-ГЕНА ИЗ SOLANUM CHACOENSE | 2010 |
|
RU2586509C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817376C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2814154C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2824558C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817378C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817379C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817383C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817374C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2800828C1 |
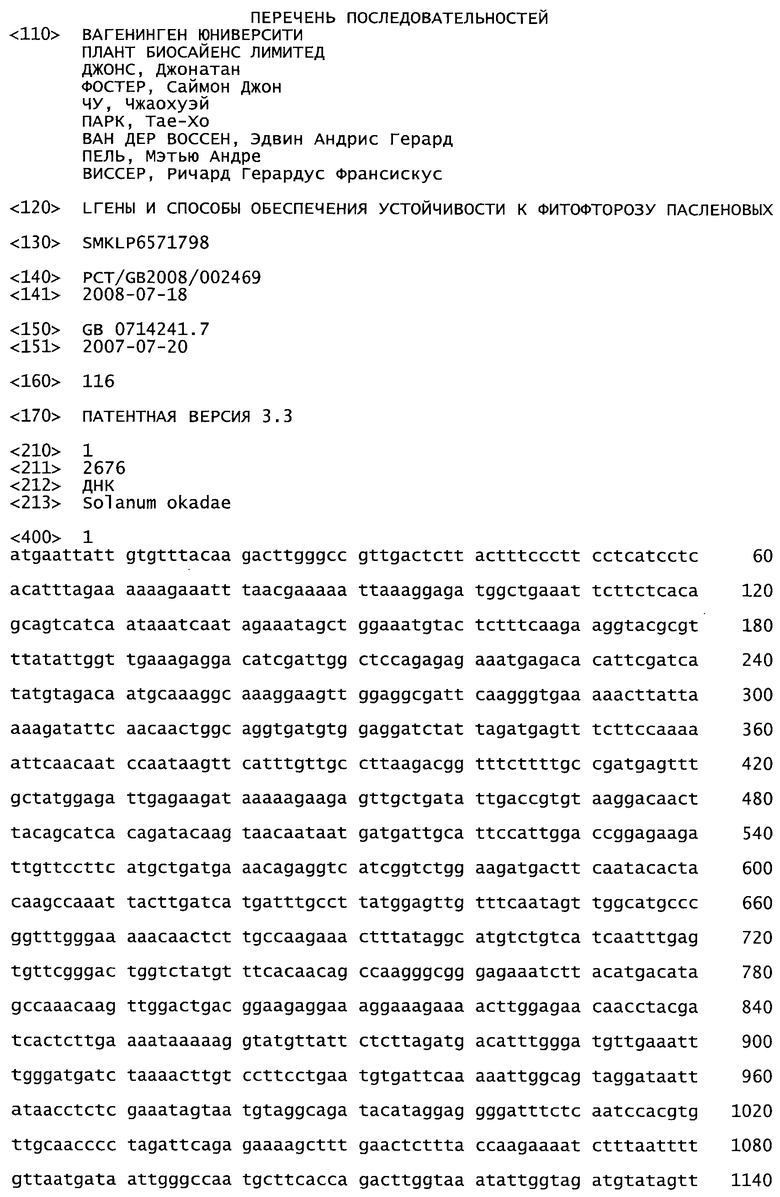




























































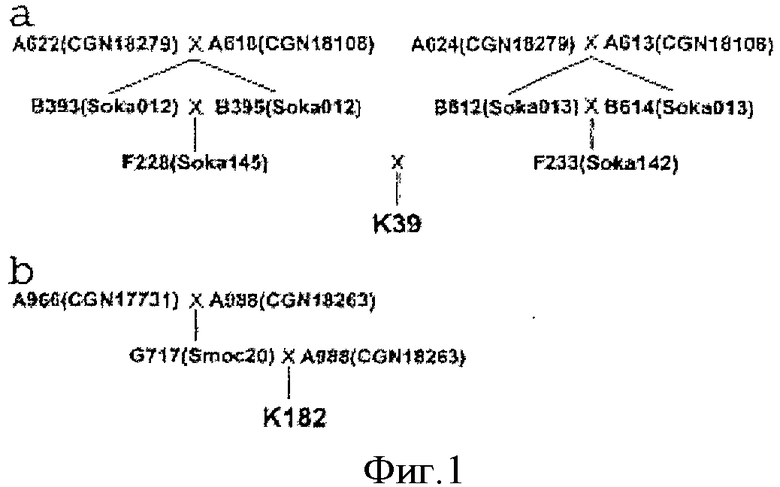
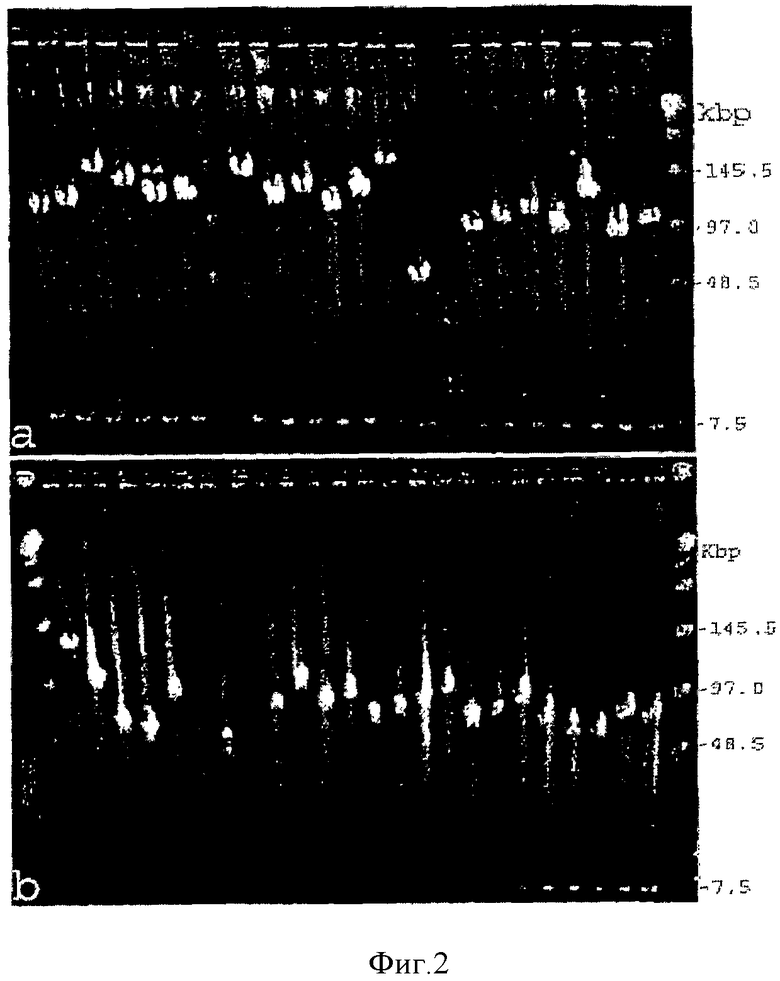


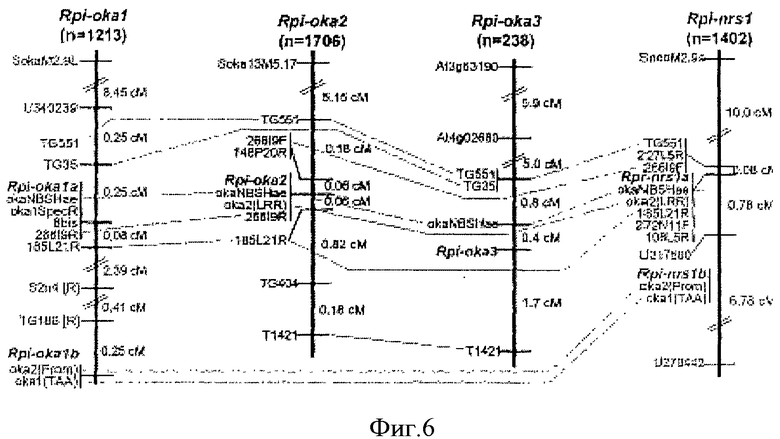


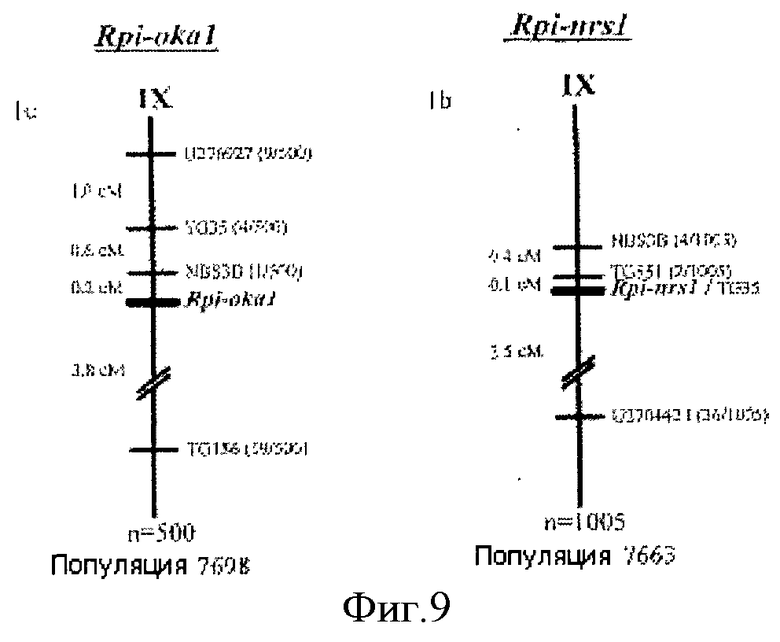
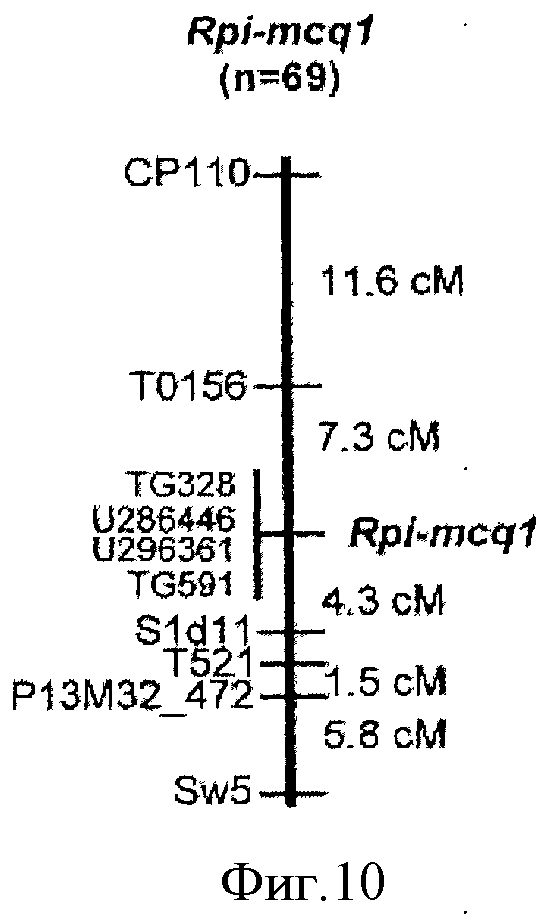










Изобретение относится к области биохимии, в частности к генам устойчивости Rpi для повышения устойчивости к фитофторозу пасленовых у растений семейства пасленовых, а также к белкам, способным опосредовать ответ против Р.infestans. Раскрыты трансгенные растения семейства пасленовых, обладающие повышенной устойчивостью к фитофторозу пасленовых, содержащие вышеуказанные гены, а также способ получения таких растений. Описаны векторы для экспрессии белка, содержащие вышеуказанные гены. Также раскрыты способы обеспечения длительной устойчивости к заболеванию у картофеля с использованием вышеуказанных генов. Использование изобретения позволяет получить растения семейства пасленовых, обладающие повышенной устойчивостью к фитофторозу пасленовых по сравнению с растением семейства пасленовых дикого типа. 9 н. и 6 з.п. ф-лы, 12 ил., 6 табл., 7 пр.
1. Изолированный ген устойчивости Rpi для повышения устойчивости к фитофторозу пасленовых у растения семейства пасленовых, имеющий последовательность, которая по меньшей мере на 85% гомологична последовательности нуклеиновой кислоты, обозначенной в списке последовательностей SEQ. ID. 1 или SEQ. ID. 7.
2. Изолированный ген устойчивости Rpi по п.1, имеющий последовательность, которая по меньшей мере на 90% гомологична последовательности нуклеиновой кислоты, обозначенной в списке последовательностей SEQ. ID. 1.
3. Изолированный ген устойчивости Rpi по п.2, который включает последовательность нуклеиновой кислоты, обозначенную в списке последовательностей SEQ. ID. 7.
4. Изолированный ген устойчивости Rpi по п.3, который включает последовательность нуклеиновой кислоты, обозначенную в списке последовательностей SEQ. ID. 8.
5. Изолированный белок, который имеет последовательность аминокислот, которая на по меньшей мере 80% гомологична последовательности аминокислот, обозначенной в списке последовательностей SEQ. ID. 4 или SEQ. ID. 12, или последовательности Rpi-nsr1, обозначенной в списке последовательностей SEQ ID. 16, и который способен опосредовать ответ против Р.infestans.
6. Изолированный белок по п.5, имеющий последовательность, которая по меньшей мере на 90% гомологична последовательности аминокислот, обозначенной в списке последовательностей SEQ. ID. 4.
7. Изолированный белок по п.6, имеющий последовательность, которая включает последовательность аминокислот, обозначенную в списке последовательностей SEQ. ID. 4, или последовательность Rpi-nsr1, обозначенную в списке последовательностей SEQ. ID. 16.
8. Изолированный ген, кодирующий белок по любому из пп. с 5 по 7.
9. Трансгенное растение семейства пасленовых, обладающее повышенной устойчивостью к фитофторозу пасленовых, несущее ген согласно любому из пп. с 1 по 4 или 8, который был введен в указанное растение.
10. Растение по п.9, которое представляет собой картофель.
11. Способ получения трансгенного растения семейства пасленовых, обладающего повышенной устойчивостью к фитофторозу пасленовых, который включает введение в клетку указанного растения или в часть указанного растения гена согласно любому из пп. с 1 по 4 или 8 и получение целого растения из указанной клетки или из указанной части указанного растения.
12. Трансгенное растение семейства пасленовых, обладающее повышенной устойчивостью к фитофторозу пасленовых, полученное способом по п. 11.
13. Вектор для экспрессии белка, способного опосредовать ответ против Р.infestans, включающий ген согласно любому из пп. с 1 по 4 или 8, и подходящие регуляторные последовательности, функционально связанные с указанным геном с обеспечением его экспрессии при помещении в подходящую систему in vitro или in vivo.
14. Способ обеспечения длительной устойчивости к заболеванию у картофеля, который включает выделение множества генов Rpi из диких родственников картофеля, введение указанных генов по отдельности в промышленную линию или сорт картофеля, смешивание и посадку полученной смеси линий, полученных таким образом, причем указанные гены представляют собой гены по любому из пп. 1-4 и 8.
15. Способ обеспечения длительной устойчивости к заболеванию у картофеля, который включает выделение множества генов Rpi из диких родственников картофеля, введение указанных генов отдельно в промышленную линию или сорт картофеля и посадку каждый год сорта, несущего другой ген Rpi, причем указанные гены представляют собой гены по любому из пп. 1-4 и 8.
| J | |||
| SONG, Gene RB cloned from Solanum bulbocastanum confers | |||
| broad spectrum resistance to potato late blight, PNAS, 2003, v | |||
| Облицовка комнатных печей | 1918 |
|
SU100A1 |
| Устройство для электрической сигнализации | 1918 |
|
SU16A1 |
| Устройство для подачи в мешалку смешиваемых сыпучих материалов | 1928 |
|
SU9128A1 |
| S | |||
| HUANG, Comparative genomics enabled the isolation of the R3a late blight resistance gene in potato, The Plant Journal, 2005, v | |||
| Устройство для усиления микрофонного тока с применением самоиндукции | 1920 |
|
SU42A1 |
| Способ модулирования для радиотелефона | 1921 |
|
SU251A1 |
| ГЕН, ПРИДАЮЩИЙ УСТОЙЧИВОСТЬ К PHYTOPHTHORA INFESTANS (ФИТОФТОРОЗУ) В СЕМЕЙСТВЕ SOLANACEAE | 2003 |
|
RU2361920C2 |
| БИОЛОГИЧЕСКИЙ | |||
Авторы
Даты
2014-04-10—Публикация
2008-07-18—Подача