Область техники, к которой относится изобретение
Изобретение относится к гену устойчивости, выделенному из S.chacoense. Кроме того, изобретение относится к использованию указанного гена устойчивости, например, для клонирования функциональных гомологов, и к использованию указанного(ых) гена(ов) устойчивости в способе повышения или придания, по меньшей мере, частичной устойчивости растениям к заражению оомицетами. Конкретнее, изобретение относится к гену устойчивости, который способен повышать или придавать, по меньшей мере, частичную устойчивость к Phytophtora sp.(например, Phytophtora infestans) посредством методов генной инженерии или маркер-опосредованных методов селекции.
Уровень техники
Фитофтороз, вызываемый оомицетом Phytophtora infestans, является одним из наиболее серьезных заболеваний в мировом производстве картофеля. Он ответственен за Ирландский картофельный голод в середине 19-го века, приведший к гибели одного миллиона людей. Хотя много усилий в исследованиях приложено в борьбе с этим болезнетворным организмом, химическая борьба с Р.infestans все еще является основной стратегией ухода за культурой, но безопасность окружающей среды становится более важной, и патоген иногда способен вырабатывать устойчивость к обработке фунгицидами. Поэтому внедрение устойчивости в современные сорта картофеля является наиболее желательной стратегией борьбы с заболеванием.
В прошлом веке Solanum demissum, который представляет собой гексаплоидный мексиканский вид, широко использовался при выведении картофеля, устойчивого к фитофторозу. Сначала был описан ряд генов 11R, полученных из S. demissum. Из них R1, R2, R3a/b, R6 и R7 локализованы на генетических картах картофеля (Solanum tuberosum). Однако указанные R-гены придают специфическую устойчивость к патогенным вариантам, и гены, которые были интрогрессированы в сорта картофеля, главным образом, R1, R2, R3, R4 и R10, быстро преодолевались патогеном. Следовательно, требуются новые источники для устойчивости, и в настоящее время имеются сообщения о нескольких других диких видах Solanum как потенциальных источниках устойчивости, многие из которых генетически охарактеризованы (таблица 6).
Последние усилия по идентификации устойчивости к фитофторозу сосредоточены на основных R-генах, придающих устойчивость широкого спектра, полученных из различных диких видов Solanum. Сообщается о других, кроме S.demissum, диких видах Solanum как новых источниках для устойчивости к фитофторозу, таких как S.acaule, S.chacoense, S.berthaultii, S.brevidens, S.bulbocastanum, S.microdontum, S.sparsipilum, S.spegazzinii, S.,stolonifemm, S.sucrense, S.toralapanum, S.vemei и S.verrucosum (обзор в (Jansky, 2000)).
S.chacoense представляет собой самонесовместимый диплоидный вид из Южной Америки и, как полагают, является источником для устойчивости к фитофторозу. Последняя таксономическая перестановка в отделе Petota выявила его родство с видами, подобными S.berthaultii и S.tarijense. Некоторые образцы S.chacoense (CHC543-1), S. berthaultii (BER481-3, BER94-2031) и S.tarijense (TAR852-5) испытаны в анализе отделенного листа (DLA) со многими изолятами (таблица 5) и в повторных полевых испытаниях с изолятом IPO-C. Во всех испытаниях CHC543-1, BER94-2031, BER481-3 и TAR852-5 оставались непораженными, что подчеркивает релевантность экспрессированных R-генов для улучшения устойчивости.
Молекулярное клонирование генов, ответственных за устойчивость, и последующее введение генов в сорта картофеля составляют третий способ, который обходит многие из проблем, с которыми сталкиваются в двух предшествующих стратегиях.
На сегодняшний день клонированы многие R-гены фитофтороза, подобные аллельным генам RB и Rpi-blb1 на хромосоме 8 и Rpi-blb2 на хромосоме 6 (таблица 6). Недавно также выделен ген устойчивости Rpi-blb3 (WO 2008/091153). Хотя первые результаты, полученные с RB и Rpi-blb1, -2 и -3 являются многообещающими, все еще остается потребность в других R-генах.
Сущность изобретения
Настоящее изобретение относится к способу придания, по меньшей мере, частичной устойчивости или повышения устойчивости растения против заражения оомицетом, включающему предоставление растению или его части нуклеиновой кислоты, кодирующей аминокислотную последовательность Rpi-che1 с фигуры 4, или ее функциональный фрагмент или функциональный гомолог, предпочтительно, при этом указанное растение представляет собой растение из семейства Solanaceae, предпочтительнее, Solanum tuberosum. Предпочтительно, указанный оомицет включает Phytophtora, предпочтительнее, Phytophtora infestans. В конкретном воплощении вышеуказанный функциональный гомолог выбирают из группы аминокислотных последовательностей, состоящей из 493-7_G12, 543-5_С2, 849-1_М8_М18_М20, 487-1_I4_I6_I8, 94-2031_L4_L7_18, 561-2_K4_K14_K22, 324-2_J1_J3_J8, 852-5_E14_E23, 852-5_Е28, 493-9_Н5_Н30, 493-7_G14_G22, 561-2_K6_K30_K31 и 493-7_G21. В других конкретных воплощениях нуклеотидная последовательность, определенная выше, включает нуклеотидную последовательность, отображенную на фигуре 7, или нуклеотидную последовательность, кодирующую аминокислотные последовательности 493-7_G12, 543-5_С2, 849-1_М8_М18_М20, 487-1_I4_I6_I8, 94-2031_L4_L7_18, 561-2_К4_К14_К22, 324-2_J1_J3_J8, 852-5_Е14_Е23, 852-5_Е28, 493-9_Н5_Н30, 493-7_G14_G22, 561-2_К6_К30_К31 и 493-7_G21, отображенные на фиг.13.
Изобретение также включает способ выведения тетраплоидного растения, устойчивого к оомицету, предпочтительно, Phytophtora, включающий
a) повышение уровня плоидности гамет диплоидного растения, которое уже содержит нуклеотидную последовательность, определенную выше;
b) использование указанных гамет в скрещивании с гаметами тетраплоидного растения; и
c) отбор потомства указанного скрещивания по наличию указанной нуклеотидной последовательности. Предпочтительно, в указанном способе диплоидное растение на стадии а) представляет собой растение из рода S.chacoense, S.berthaultii, S.sucrense или S.tarijense.
Изобретение также относится к способу отбора растения или растительного материала или его потомства по его подверженности или устойчивости к заражению оомицетом, причем указанный способ включает стадии испытания, по меньшей мере, части указанного растения или растительного материала или его потомства на присутствие или отсутствие нуклеиновой кислоты, определенной выше. Конкретно в таком способе испытание включает обнаружение наличия одного или нескольких маркеров из таблиц 2 и 8, и его выполняют с праймером или зондом, которые специфически связываются с указанной нуклеиновой кислотой.
Следовательно, изобретение также относится к маркеру для маркер-опосредованной селекции для выведения растений, устойчивых против оомицетов, при этом указанный маркер выбирают из маркеров, представленных в таблицах 2 и 8.
В другом воплощении изобретение также относится к изолированной или рекомбинантной нуклеотидной последовательности, включающей нуклеотидную последовательность, кодирующую аминокислотную последовательность Rpi-che1 с фигуры 4, или ее функциональный фрагмент, или нуклеотидную последовательность, кодирующую аминокислотную последовательность из 493-7_G12, 543-5_C2, 849-1_M8_M18_M20, 487-1J4J6J8, 94-2031_L4_L7_18, 561-2_K4_K14_K22, 324-2_J1_J3_J8, 852-5_Е14_Е23, 852-5_Е28, 493-9_Н5_Н30, 493-7_G14_G22, 5б1-2_К6_К30_К31 и 493-7_G21 или ее функциональный фрагмент. Предпочтительно указанный фрагмент включает, по меньшей мере, домен LRR аминокислотной последовательности. Также предпочтительным является воплощение, где изолированная или рекомбинантная нуклеотидная последовательность по п.10 включает нуклеотидную последовательность, отображенную на фигуре 7 или на фигуре 13.
Изобретение также относится к трансгенной или тетраплоидной клетке, включающей нуклеиновую кислоту по изобретению.
Также частью изобретения является вектор, включающий нуклеотидную последовательность по изобретению. Предпочтительно указанный вектор также включает промотор и/или терминатор, с которым ген естественно ассоциируется, предпочтительнее, укороченный промотор, имеющий менее 1000 нуклеотидов вверх от последовательности гена.
Изобретение также относится к трансгенной или тетраплоидной клетке-хозяину, включающей нуклеиновую кислоту по изобретению или вектор по изобретению, предпочтительно, при этом такой клеткой-хозяином является клетка Agrobacterium или растительная клетка.
Изобретение также относится к трансгенной или тетраплоидной растительной клетке, включающей нуклеиновую кислоту по изобретению или вектор по изобретению, предпочтительно, при этом такой растительной клеткой является клетка из Solanaceae, предпочтительнее, Solanum tuberosum, предпочтительнее, тетраплоидного Solanum tuberosum. В другом предпочтительном воплощении изобретение относится к трансгенному или тетраплоидному растению, включающему такую клетку, и также части растения, полученной от такого растения, при этом такой частью предпочтительно является клубень.
В настоящее изобретение также включается белок, кодированный изолированной или рекомбинантной нуклеиновой кислотой по изобретению, или его функциональный фрагмент, при этом указанный белок имеет аминокислотную последовательность Rpi-chc1, отображенную на фигуре 4.
Изобретение также относится к антителу, которое (специфически) связывается с белком по п.20.
Пояснения к фигурам
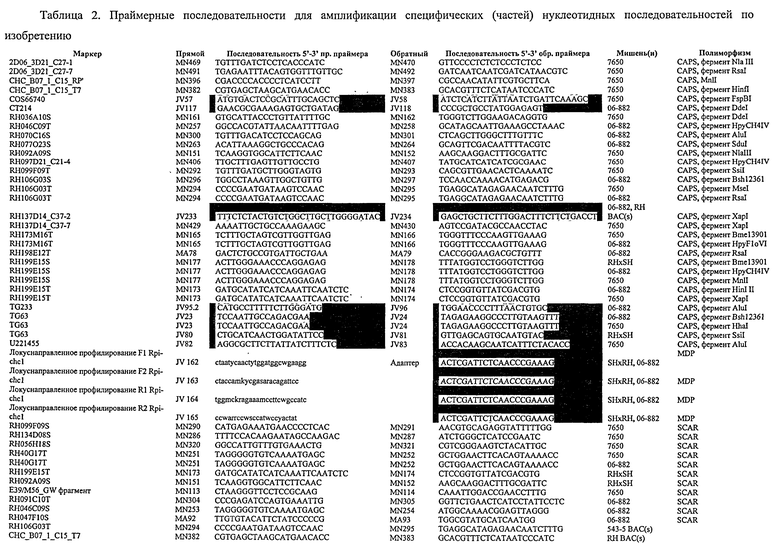
Фигура 1. Генетические и физические карты локусов Rpi-chc1 (А) и Rpi-ber (В) (популяции 7650 и 06-882, соответственно). Указаны относительные позиции маркеров, число рекомбинантов, идентифицированных между маркерами, перекрывающиеся клоны ВАС, которые охватывают R-локусы, и относительные позиции RGA на физических картах СНС543-5 и RH89-039-16.
Фигура 2. Аннотация последовательности Chr10 ВАС.
Секвенированы и аннотированы два перекрывания по типу черепичной кровли (Tiling path), состоящих из 3 и 4 перекрывающихся ВАС из RH89-039-16 (RH106G038, RH137D014, RH009D021 и RH122B15, RH77023, RH04G12, RH199E15) и двух перекрывающихся ВАС из СНС543-5. Позиции маркеров и концевых последовательностей ВАС у перекрывающихся ВАС указаны остриями стрелок. Позиции контигов последовательностей показаны горизонтальными стрелками. Позиции генов, предсказанные алгоритмом FGENESH, указаны заштрихованными прямоугольниками. Гомология последовательностей белков, обнаруженная поиском BlastP в базе данных NR, показана вертикальными стрелками. RGA нумеруются соответственно - A: RH106G03, В: RH137D14, С: RH97D21, D: RH122B15, Е: RH77023, F: CHC B1 (В07-1-05), G: CHC B2 (2-D06_3-D21).
Фигура 3. Временная комплементация чувствительности к Phytophtora в листьях Nicotiana benthamiana. Через 2 дня после агроинфильтрации листья заражают инокуляцией суспензии зооспор изолята 90128 Р.infestances (авирулентного на СНС543-5) в анализе отделенного листа. Типичные фенотипы заболевания развиваются через 6 дней после инокуляции контрольных растений, которые агрофильтрованы pBINplus без вставки. Полную устойчивость наблюдают у контрольных растений, агрофильтрованных pBINplus:Rpi-blb1. Агроинфильтрация pBINplus:CHCB2-3 - одним из трех RGA из интервала картирования Rpi-chc1, также придает полную устойчивость к заражению Р.infestances, в то время как листья, инфильтрованные pBINplus:CHCB2-l и pBINplus:CHCB2-2, остаются чувствительными.
Фигура 4. Выравнивание аминокислотной последовательности RGA из S. chacoense (CHC B1-1, CHC B1-2, CHC B2-1, CHC B2-2 и CHC B2-3 = Rpi-chc1) и из родственных последовательностей, происходящих из образцов S.tuberosum RH89-039-16 (77023 с5794, 77023 с5795, 77023 с671, 77023 с7063, 77023 с7064, 122 В15С88, 122 В15С247, 137D14c131, и 137D14c132).
Белок ABF81421 с неизвестной функцией кодирован геном из Populus trichocarpa.
Фигура 5. Организация белкового домена Rpi-chc1
N-Концевой СС-домен включает аминокислоты 1-231. Для аминокислот, показанных затемненными, с использованием алгоритма «спираль» с размером окна 14 прогнозирована укладка в спиральную структуру. Центральный домен NB-ARC включает аминокислоты 232-557. Домены в затемненных участках демонстрирует сходство с описанными ранее доменами Kinase 1a, Kinase 2, kinase 3а, GLPL, RNBS-D и MHD, соответственно. С-Концевой LRR-домен состоит из 20 несовершенных повторов, богатых лейцином. Консервативные гидрофобные аминокислоты (А, V, L и F) в данном случае отмечены затемнением. Консенсус показан внизу.
Фигура 6. Позиции на генетической карте родственных последовательностей Rpi-chc1 и генов устойчивости к фитофторозу на хромосоме 10.
Карты UHD хромосом SH и RH показаны слева (van Os et al., 2006). 06-882 и 7677, полученные в данном исследовании, показаны в середине. Позиции Rpi-ber (Rauscher et al., 2006), Rpi-ber1 и Rpi-ber2 (Park et al., 2008) показаны справа. Красные линии показывают расположение генов устойчивости к фитофторозу.
Фигура 7. Нуклеотидная последовательность клона СНС В2-3 (7907 п.о.), содержащая Rpi-chc1-кодирующую и регуляторную последовательности. Кодирующий Rpi-chc1 участок в 4550 п.о. выделен затемнением (3358-7266). Вышерасположенные 3357 нуклеотидов (1-3357) и нижерасположенные 641 нуклеотид (7267-7907) содержат регуляторные последовательности.
Фигура 8. Функциональная комплементация чувствительности к Phytophtora infestsns (Pi) в трансгенных растениях сорта Дезире. Сорт Дезире, трансформированный кандидатами в гены Rpi-chc1 (RGC-1, -2 и -3), заражают изолятом Pi 90128 в анализе отделенного листа. Учитывают картину через 6 дней после инокуляции. Устойчивость наблюдают только в трансгенах, содержащих RGC-3.
Фигура 9. Скрининг набора РЕХ с использованием совместной инфильтрации. Клоны РЕХ инфильтруют в листья N. benthamiana одни или инфильтруют совместно с Rpi-chc1. Оценивают картину через одну неделю после инфильтрации. Лист А-РЕХ1=RD31, РЕХ2=RD36. Лист В-РЕХ1=RD12-1, РЕХ2=RD12-2. Лист С-РЕХ1=INF1, РЕХ2=pGR106. На каждом листе пятно на листе внизу слева инфильтровано R3a+avr3a. Пятна внизу справа инфильтрованы Rpi-chc1. Лист А не показывает идентификации соответствующего эффектора. В показывает некроз в случае взаимодействия Rpi-chc1 и RD12. С показывает аутонекроз в случае INF1.
Фигура 10. Регуляторные элементы, управляющие экспрессией Rpi-chc1.
ORF Rpi-chc1 клонируют в промежутке между одной из четырех промоторных/терминаторных последовательностей - ее собственным промотором в 3 т.п.о. и терминатором в 0,5 т.п.о. (p-chc1-long), ее собственным промотором в 0,9 т.п.о. и терминатором в 0,5 т.п.о. (p-chc1-short), двойным промотором 35S в pMDC32 или комбинацией промотор/терминатор Rpi-blb3 (Lokosson et al., 2009). Совместную агорофильтрацию с PEX-RD12 выполняют при пяти серийных разведениях (OD600=2,0, 1,0, 0,5, 0,2, 0,1) как указано. R3a, смешанный с Avr3a, используют в качестве положительного контроля (+), и Rpi-chc1 используют в качестве отрицательного контроля (-). Изображения получают через 6 дней после инокуляции.
Фигура 11. Отбор пар Rpi-chc1-специфических праймеров, используемых для скринига зародышевой плазмы.
А. Отбор пар Rpi-chc1-специфических праймеров. Комбинации праймеров а: 581+582, b: 585+587, с: 585+589, d: 586+587, e: 586+589, f: 588+589 относятся к таблице 8. Используемые матрицы: 1-chc543-5 (растение-донор для Rpi-chc1), 2-chc544-5 (чувствительный родитель популяции картирования), 3-RH89-39-16 (чувствительное растение, донор гомологичных последовательностей Rpi-chc1), 4-СНС ВАС-1 (клон ВАС, содержащий три неактивных RGA), 5-СНС ВАС-2 (клон ВАС, содержащий Rpi-chc1), 6-MQ.B. 225 генотипов из таксономических групп 10-12 до 10-17, перечисленных в таблице 7, скринированы с комбинацией праймеров D. Светлые стрелки показывают фрагменты ожидаемого размера в 6 генотипах.
Фигура 12. Филогенетический анализ гомологов Rpi-chc1.
Зеленый цвет-последовательности, выделенные ПЦР гомологов Rpi-chc1 (пример 2).
Черный цвет-гомологи Rpi-chc1, идентифицированные во время клонирования на основе картирования (пример 1).
Фигура 13. Нуклеотидные последовательности 21 упомянутого гомолога Rpi-chc1.
Фигура 14. Выравнивание по Clustal W последовательностей белков, кодированных гомологами Rpi-chc1 с фигуры 11, и последовательностей гомологов Rpi-chc1, описанных в примере 1.
Подробное описание
Используемый в данном описании термин «растение или его часть» обозначает любое целое или нецелое растение, отдельные клетки и клетки тканей, такие как растительные клетки, которые являются интактными в растениях, скопления клеток и тканевые культуры, из которых могут быть регенерированы растения картофеля. Примеры частей растения включают, но не ограничиваются указанным, отдельные клетки и ткани из пыльцы, семяпочек, листьев, зародышей, корней, кончиков корней, пыльников, цветков, плодов, боковых побегов, клубней, в том числе, клубней картофеля для потребления или «семенных клубней» для выращивания или кленового размножения, и семян; а также пыльцу, семяпочки, листья, зародыши, корни, кончики корней, пыльники, цветки, плоды, стебли, побеги, отпрыски, корневые побеги, семена, протопласты, каллюс и т.п.
Используемый в данном описании термин «популяция» обозначает генетически неоднородное собрание растений, имеющих общее генетическое происхождение.
Используемый в данном описании термин «сорт» определяется соглашением UPOV и относится к любой группировке растений в пределах одного ботанического таксона наименьшего известного ранга, которая может (а) определяться проявлением признаков, которые обусловлены данным генотипом или комбинацией генотипов, (b) отличаться от любой другой группировки растений за счет проявления, по меньшей мере, одного из указанных признаков, и (с) рассматриваться как единица в отношении ее способности не изменяться при размножении.
Термин «культивар» (для культивируемого сорта), используемый в данном описании, определяется как сорт, который обычно не обнаруживается в природе, но который выращен людьми, т.е. имеет биологический статус иной, чем «дикий» статус, где «дикий» статус показывает исходное некультивированное или природное состояние растения или образца. Термин «культивар» конкретно относится к растению картофеля, имеющему уровень плоидности, который соответствует тетраплоидному. Термин «культивар» также включает, но не ограничивается указанным, полунатуральный, полудикий, сорный традиционный культивар, ландрас, селекционный материал, исследовательский материал, селекционную линию, синтетическую популяцию, гибрид, семенную/основную популяцию, инбредную линию (родителя гибридного культивара), расщепляющуюся популяцию, мутантный/генетический материал и прогрессивный/улучшенный культивар.
Используемый в данном описании термин «скрещивание» обозначает оплодотворение женских растений (или гамет) мужскими растениями (или гаметами). Термин «гамета» относится к гаплоидной или диплоидной половой клетке (яйцеклетке или спермии), вырабатываемой в растениях путем мейоза или путем первой или второй реституции или двойной реституции из гаметофита, и вовлеченной в половое размножение, во время которого две гаметы противоположного пола сливаются с образованием диплоидной или полиплоидной зиготы. Термин обычно включает отсылку к пыльце (включающей сперматогенную клетку) и семяпочке (включающей яйцеклетку). Поэтому термин «скрещивание» обычно относится к оплодотворению семяпочки одной особи пыльцой другой особи, в то время как термин «самоопыление» относится к оплодотворению семяпочек особи пыльцой генетически той же особи.
Термин «обратное скрещивание», используемый в данном описании, обозначает процесс, в котором растение, полученное от скрещивания между двумя родительскими линиями, скрещивают с одной из его родительских линий, при этом родительскую линию, используемую при обратном скрещивании, называют рекуррентным родителем. Повторное обратное скрещивание приводит к геному, становящемуся все более и более похожим на рекуррентного родителя, насколько это может быть достигнуто для данного уровня гомо- или гетерозиготности указанного родителя.
Используемый в данном описании термин «самоопыление» определяется как относящийся к самооплодотворению, при этом особь опыляется или оплодотворяется своей собственной пыльцой.
Термин «маркер», используемый в данном описании, обозначает любой индикатор, который используется в способах для обнаружения различий в характеристиках геномных последовательностей. Примерами таких индикаторов являются маркеры полиморфизма длин рестрикционных фрагментов (RFLP), маркеры полиморфизма длин амплифицированных фрагментов (AFLP), однонуклеотидного полиморфизма (SNP), инсерционных мутаций, микросателлитные маркеры (SSR), амплифицированные участки с охарактеризованной последовательностью (SCAR), маркеры рестрикционного полиморфизма амплифицированной последовательности (CAPS) или изозимные маркеры или комбинации маркеров, описанных в данном описании, которые определяют специфическую генетическую и хромосомную локацию.
Используемый в данном описании термин «локус» определяется как генетическая или физическая позиция, которую данный ген занимает на хромосоме растения.
Термин «аллель(и)», используемый в данном описании, обозначает любую одну или несколько альтернативных форм гена, и все аллели соотносятся с присутствием или отсутствием определенного фенотипического признака или свойства у растения. В диплоидной клетке или организме два аллеля данного гена занимают соответствующие локусы на паре гомологичных хромосом. В некоторых случаях вместо «аллеля» это точнее можно назвать «гаплотипами» (т.е. аллелем хромосомного сегмента), однако в таких случаях термин «аллель» следует понимать как включающий термин «гаплотип».
Термин «гетерозиготный», используемый в данном описании, только в отношении диплоидов обозначает генетическое состояние, существующее в том случае, когда в соответствующих локусах на гомологичных хромосомах находятся различные аллели.
Используемый в данном описании только в отношении диплоидов термин «гомозиготный» определяется как генетическое состояние, существующее в том случае, когда в соответствующих локусах на гомологичных хромосомах находятся идентичные аллели.
Используемый в данном описании только в отношении тетраплоидов термин «нуллиплекс», «симплекс», «дуплекс», «триплекс» и «квадруплекс» определяется как генетическое состояние, существующее в том случае, когда в соответствующем локусе на соответствующих гомологичных хромосомах специфический аллель присутствует 0, 1, 2, 3 или 4 раза, соответственно. На тетраплоидном уровне фенотипический признак, ассоциированный с рецессивным аллелем, наблюдают только тогда, когда аллель присутствует в состоянии квадруплекса, в то время как фенотипический признак, ассоциированный с доминантным аллелем, наблюдают уже тогда, когда аллель присутствует в состоянии симплекса или более высоком состоянии.
Термины «гаплоид», «диплоид» и «тетраплоид», используемые в данном описании, определяются как наличие соответственно одной, двух и четырех пар каждой хромосомы в каждой клетке (исключая половые клетки).
Термин «гаплотип», используемый в данном описании, обозначает комбинацию аллелей в нескольких локусах, которые передаются вместе в составе одной хромосомы. Включаются гаплотипы, относящиеся всего к двум локусам, и гаплотипы, относящиеся ко всей хромосоме, в зависимости от числа событий рекомбинации, которые происходят между данным набором локусов.
Используемый в данном описании термин «делать вывод» или «заключение» в отношении оценки присутствия устойчивости к грибам связанной с экспрессией гена Rpi-chc1, обозначает формулирование вывода о присутствии указанного гена в растении или его части с использованием способа анализа встречаемости отдельного нуклеотида или сочетания нуклеотидов указанного гена в образце нуклеиновой кислоты растения или его части. Как раскрывается в данном описании, встречаемость нуклеотидов можно идентифицировать непосредственно путем проверки качественных различий или количественных различий в уровнях экспрессии молекул нуклеиновой кислоты или косвенно путем проверки (уровня экспрессии) белка Rpi-chc1.
Термин «праймер», используемый в данном описании, относится к олигонуклеотиду, который способен к отжигу с мишенью амплификации, давая возможность ДНК-полимеразе присоединяться, причем посредством этого служит в качестве точки инициации синтеза ДНК, когда помещен в условия, в которых вызывается синтез продукта удлинения праймера, который комплементарен цепочке нуклеиновой кислоты, т.е. в присутствии нуклеотидов и агента полимеризации, такого как ДНК-полимераза, и при подходящих температуре и рН. Праймер (амплификации) предпочтительно является одноцепочечным для максимальной эффективности амплификации. Предпочтительно прайме представляет собой олигодезоксирибонуклеотид. Праймер должен быть достаточно длинным для инициирования синтеза продуктов удлинения в присутствии агента полимеризации. Точная длина праймеров будет зависеть от многих факторов, включая температуру и источник праймера. Термин «пара двунаправленных праймеров», используемый в данном описании, относится к одному прямому и одному обратному праймеру, как это обычно используется в технике амплификации ДНК, такой как ПЦР амплификация.
Используемый в данном описании термин «зонд» обозначает последовательность одноцепочечного нуклеотида, которая будет узнавать и образовывать через водородную связь дуплекс с комплементарной последовательностью в аналите нуклеотидной последовательности-мишени или ее к ДНК-производного.
Термины «жесткость» или «жесткие условия гибридизации» относятся к условиям гибридизации, которые влияют на устойчивость гибридов, например, температуре, концентрации соли, рН, концентрации формамида и т.п. Такие условия эмпирически оптимизированы для максимизации специфического связывания и минимизации неспецифического связывания праймера или зонда с его нуклеотидной последовательностью-мишенью. Используемые термины включают обращение к условиям, в которых зонд или праймер будут гибридизировать с последовательностью-мишенью в большей степени, чем с другими последовательностями, что поддается обнаружению (например, по меньшей мере, в 2 раза больше, чем фон). Жесткие условия зависят от последовательности и будут различаться в различных обстоятельствах. Более длинные последовательности гибридизируют специфически при более высоких температурах. Как правило, жесткие условия выбирают такими, что температура примерно на 5°С ниже, чем температура плавления (Тпл (Тm)) специфической последовательности при определенных ионной силе и рН. Тпл является температурой (при определенных ионной силе и рН), при которой 50% комплементарной последовательности-мишени гибридизирует с полностью соответствующим зондом или праймером.
Типично жесткие условия будут такими, когда концентрация соли меньше примерно 1,0 М иона Na+, типично, концентрация иона Na+ (или других солей) примерно 0,01-1,0 М, при рН 7,0-8,3 и температура составляет, по меньшей мере, примерно 30°С для коротких зондов или праймеров (например, в 10-50 нуклеотидов) и, по меньшей мере, примерно 60°С для длинных зондов или праймеров (например, свыше 50 нуклеотидов). Жестких условий также можно достичь добавлением дестабилизирующих агентов, таких как формамид. Примеры условий низкой жесткости или «условий пониженной жесткости» включают гибридизацию с буферным раствором с 30% формамида, 1 М NaCl, 1% SDS при 37°С и промывку в 2х SSC при 40°С. Примеры жестких условий включают гибридизацию с 50% формамида, 1 М NaCl, 1% SDS при 37°С и промывку в 0,1х SSC при 60°С. Процедуры гибридизации хорошо известны в технике и описаны, например, в Ausubel F.M., Brent R., Kingston R.E., Moore D.D., Seidman J.G., Smith J.A., Struhl K., eds. (1998), Current protocols in molecular biology. V.B.Chanda, series ed. New York: John Wiley & Sons.
В настоящем изобретении описывается клонирование гена Rpi-chc1. Rpi-chc1 был картирован в новом локусе R-гена на хромосоме 10 с использованием картирования популяций S. chacoense. Маркеры, прочно соединенные с Rpi-chc1, используют для получения физической карты локуса R. Три аналога R-гена (RGA), присутствующие на одном из двух клонов ВАС, которые охватывают локус Rpi-chc1, намечают для анализа комплементации, из которых один, как оказывается, является функциональным геном Rpi-chc1. Вне кластеров R-гена, описанных в данном изобретении, Rpi-chc1 имеет наибольшую идентичность аминокислотных последовательностей (40%). с белком, кодированным геном из тополя (Populas trichocarpa) с неизвестной функцией, обозначенным ABF81421. Более низкий процент гомологии (<30%) обнаруживают с R-белками, идентифицированными ранее в Solanaceae (таблица 3).
В первом воплощении изобретение относится к изолированной или рекомбинантной нуклеотидной последовательности, включающей нуклеотидную последовательность, кодирующую аминокислотную последовательность Rpi-chc1 (=СНС_В2-3), представленную на фигуре 4, или ее функциональный фрагмент или функциональный гомолог, т.е. функциональный фрагмент или функциональный гомолог аминокислотной последовательности, показанной на фигуре 4.
Термин «нуклеиновая кислота» обозначает молекулу одно- или двухцепочечной ДНК или РНК.
Также включены последовательности, комплементарные описанным здесь нуклеотидным последовательностям.
Термин «его функциональный фрагмент» типично используют для обозначения фрагмента белка Rpi-chc1, который способен придавать, по меньшей мере, частичную устойчивость или возрастающую устойчивость растения семейства Solanaceae против заражения оомицетами. Такой фрагмент представляет собой, например, укороченный вариант белка Rpi-chc1, представленного на фигуре 4. Укороченный вариант/фрагмент белка Rpi-chc1 представляет собой фрагмент, который включает меньше 1302 аминокислот и, предпочтительно, включает часть домена LRR (т.е. часть домена из богатых лейцином повторов, который простирается примерно от аминокислоты 557 до аминокислоты 1302 Rpi-chc1) и/или N-концевые части белка Rpi-chc1.
Термин «функциональный гомолог» типично используют для обозначения последовательности белка, которая высокогомологична или имеет высокую идентичность с описанным здесь белком Rpi-chc1, который способен придавать, по меньшей мере, частичную устойчивость или возрастающую устойчивость растения семейства Solanaceae против заражения оомицетами. Включаются искусственные изменения или замены аминокислотных остатков, которые, по меньшей мере, частично сохраняют действие белка Rpi-chc1. Например, некоторые аминокислотные остатки могут быть традиционно заменены на другие остатки сравнимой природы, например, основной остаток на другой основной остаток, кислый остаток на другой кислый остаток, гидрофобный остаток на другой гидрофобный остаток и т.д. Примерами гидрофобных аминокислот являются валин, лейцин и изолейцин. Фенилаланин, тирозин и триптофан являются примерами аминокислот с ароматической боковой цепью, и цистеин, а также метионин являются примерами аминокислот с серосодержащими боковыми цепями. Серин и треонин содержат алифатические гидроксильные группы и рассматриваются как гидрофильные. Аспарагиновая кислота и глутаминовая кислота являются примерами аминокислот с кислой боковой цепью. Коротко, термин «его функциональный гомолог» включает варианты белка Rpi-chc1, в которых аминокислоты встроены, заменены или делегированы, и которые, по меньшей мере, частично, сохраняют действие белка Rpi-chc1 (т.е. по меньшей мере, частичное придание или возрастание устойчивости растения семейства Solanaceae против заражения оомицетами). Предпочтительными вариантами являются варианты, которые содержат только традиционные замены, описанные выше. Высокая идентичность, упомянутая выше, означает идентичность, по меньшей мере, на 80, 85 или 90%. Даже более предпочтительными являются аминокислоты, которые имеют идентичность 91, 92, 93, 94 или 95%. Наиболее предпочтительными являются аминокислоты, которые имеют идентичность с аминокислотной последовательностью Rpi-chc1 96, 97, 98 или 99%. Гомологичные белки, например, представляют собой последовательности, выровненные с CHC_B2-3 на фиг.5 и с ORF Rpi-chc1 на фиг.14.
Функциональная гомологичная нуклеотидная последовательность представляет собой нуклеотидную последовательность, которая кодирует функциональный гомологичный белок, описанный выше.
Процент гомологии и/или идентичности можно определить, например, с использованием компьютерных программ, таких как BLAST, ClustalW или ClustalX.
Многие нуклеотидные последовательности кодируют белок, который на 100% идентичен белку Rpi-chc1, представленному на фигуре 4. Это так, поскольку нуклеотиды в нуклеотидном триплете могут изменяться без изменения соответствующей аминокислоты (неоднозначность в нуклеотидных триплетах). Таким образом, нуклеотидная последовательность, кодирующая такой белок, может быть изменена не повлияв на аминокислотную последовательность белка. Однако в предпочтительном воплощении изобретение относится к изолированной или рекомбинантной нуклеотидной последовательности, отображенной на фигуре 7. В предпочтительном воплощении изобретение относится к изолированной, синтетической или рекомбинантной нуклеиновой кислоте, которая представляет собой кодирующую последовательность (CDS) белка Rpi-chc1, т.е. нуклеотиды 3358-7266 на фигуре 7 (затемненные), или его функционального гомолога. Нуклеотидные последовательности гомологов представлены на фиг.13, и соответствующие аминокислотные последовательности приводятся в выравнивании на фиг.14.
Фрагменты, а также гомологи описанного в данном описании гена и белка Rpi-chc1 можно проверить, например, на их функциональность с использованием анализов временной трансформации Agrobacterium tumefaciens (агрофильтрация) и/или с использованием анализа отделенного листа.
В экспериментальной части, например, описывается функциональный скрининг для испытания кандидатов в гены с использованием агроинфильтрации, в соответствии с чем 4-недельные растения Nicotiana benthamiana дикого типа инфильтруют штаммами Agrobacterium, содержащими кандидаты в гомологи Rpi-chc1. Затем в анализе отделенного листа инфильтрованные листья через одни сутки после инфильтрации заражают штаммом Р. infestans, который является вирулентным для N. benthamiana, например, IPO-C или 90128. Такая система в равной степени подходит для испытания фрагментов кандидатов в гомологи Rpi-chc1. Специалист в данной области техники таким образом может легко определить, можно или нет гомолог или фрагмент Rpi-chc1 рассматривать как функциональный гомолог или фрагмент.
Временная экспрессия гена, которая достигается через агрофильтрацию, является быстрым, гибким и воспроизводимым подходом к экспрессии полезных белков на высоком уровне. В растениях рекомбинантные штаммы Agrobacterium tumefaciens можно использовать для временной экспрессии генов, которые встраивают в участок Т-ДНК бактериальной Ti-плазмиды. Бактериальную культуру инфильтруют в листья, и после переноса Т-ДНК в клетках растения происходит эктопическая экспрессия интересующего гена. Однако применимость системы ограничена, поскольку эктопическая экспрессия РНК прекращается через 2-3 дня. Показано, что основной причиной такой потери эффективности является посттраскрипционный сайленсинг гена (PTGS). Система, основанная на коэкспрессии кодированного вирусом супрессора сайленсинга гена белка р19 вируса кустистой карликовости томата (TBSV), предотвращает начало PTGS в инфильтрованных тканях и создает возможность для высокого уровня временной экспрессии. Экспрессия ряда белков в присутствии р19 усиливается в 50 раз или больше, так что очистки белка можно достичь из такого небольшого количества материала инфильтрованного листа как 100 мг. Хотя ясно, что использование р19 имеет преимущества, агроинфильтрацию без р19 также можно использовать для испытания функциональности предполагаемых фрагментов и функциональных гомологов.
С другой стороны, каждый кандидат в генную конструкцию (например, являющуюся фрагментом или гомологом) является мишенью для трансформации в восприимчивый культивар картофеля, например Дезире. Первичные трансформанты заражают в анализах отделенного листа с использованием, например, изолятов IPO-O, IPO-C или 90128. Трансформанты, которые устойчивы к таким изолятам, содержат, например, функциональные фрагменты или гомологи Rpi-chc1.
В еще одном воплощении изобретение относится к вектору, включающему нуклеиновую кислоту, предлагаемую в данном описании, т.е. нуклеиновую кислоту, способную придавать, по меньшей мере, частичную устойчивость или повышенную устойчивость растения семейства Solanaceae против заражения оомицетами. Более определенно, изобретение относится к вектору, включающему изолированную, синтетическую или рекомбинантную нуклеотидную последовательность, включающую нуклеотидную последовательность, кодирующую аминокислотную последовательность Rpi-chc1 на фигуре 4 или ее функциональный фрагмент или функциональный гомолог. Изобретение также относится к вектору, включающему нуклеотидную последовательность, отображенную на фигуре 7.
Примерами подходящего вектора являются pBeloBACII, pBINplus, pKGW-MG или любой коммерчески доступный клонирующий вектор.
Как будет отмечено ниже, существует несколько путей, которыми нуклеиновую кислоту по изобретению можно перенести в растение. Один подходящий способ переноса опосредуется Agrobacterium, когда нуклеиновая кислота, которую переносят, представляет собой часть бинарного вектора, и поэтому предпочтительно, чтобы вышеописанный вектор представлял собой бинарный вектор. Другим подходящим способом является скрещивание растения, которое содержит ген, кодирующий Rpi-chc1, с растением, которое не содержит ген, и идентификация потомства гибрида, которое наследует ген Rpi-chc1.
Изобретение также относится к клетке-хозяину, включающей нуклеиновую кислоту, описанную в данном описании, или вектор, описанный в данном описании. Примерами предпочтительной клетки-хозяина являются клетка Е.coli, подходящая для клонов ВАС (например, DH10B), или клетка(хозяин) Agrobacterium. В другом воплощении указанная клетка-хозяин является растительной клеткой. Предпочтительной растительной клеткой является клетка, полученная от члена семейства Solanaceae, и даже более предпочтительная указанная клетка является клеткой от Solanum tuberosum, Solanum lycopersicum, прежде известного как Lycopersicon esculentum, перца и баклажана. Из каждой такой клетки можно получить трансгенное или генетически модифицированное растение (например, растение картофеля или томата) методами, известными специалистам (например, по протоколам регенерации).
Изобретение также относится к листу, клубню, плоду или семени или части или потомству генетически модифицированного растения, описанного в данном описании.
В еще одном воплощении изобретение относится к белку, кодированному описанной здесь изолированной или рекомбинантной нуклеиновой кислотой, или его функциональному фрагменту или функциональному гомологу. В предпочтительном воплощении изобретение относится к белку, кодированному нуклеотидной последовательностью, отображенной на фигуре 7. В еще одном предпочтительном воплощении изобретение относится к белку, включающему аминокислотную последовательность на фигуре 4 или ее функциональный фрагмент или функциональный гомолог. Также предпочтительными являются функциональные (активные) белки, отображенные на фигуре 14, конкретнее, белки, обозначенные как 493-7_G12, 543-5_C2, 849-1_М8_М18_М20, 487-1_I4_I6_I8, 94-2031_L4_L7_18, 561-2_K4_K14_K22, 324-2_J1_J3_J8, 852-5_E14_E23, 852-5_E28, 493-9_H5_H30, 493-7_G14_G22, 561-2_К6_К30_К31 и 493-7_G21.
Описанный здесь белок Rpi-chc1 содержит 1302 аминокислоты, и домены LRR Rpi-chc1 состоят из 29 несовершенных повторов (фигура 5). Представляет интерес, что Rpi-chc1 имеет наибольшую гомологию (75-90%) с другими RGA из кластера генов Rpi-chc1 из S. chacoense и с генами из синтетических кластеров на хромосоме 10 из S. tuberosum (таблица 3). Меньшая (40%), но существенная степень гомологии обнаружена с белком, кодированным геном из тополя (образец номер ABF81421, таблица 3) с неизвестной функцией. Различные домены Rpi-chcl показывают различные степени гомологии с соответствующими доменами белка тополя, кодированного ABF81421. Домен NBS является наиболее консервативным (примерно 48% идентичность), за ним домен СС (примерно 34% идентичность). Домен LRR является наименее консервативным (примерно 21% идентичность). Общая гомология менее 33% найдена с белком из огурца FOM2, который придает устойчивость к патогенному грибу Fusarium oxysporum, Rpi-blb1 из S. bulbocastanum, R3a из S. demissum и RPS1 из сои (Glycine max), которые придают устойчивость к Phytophthora sp. Такая гомология последовательностей показывает, что Rpi-chc1 является членом древнего семейства R-генов, которые ранее не характеризовались в Solanaceae.
Как уже описано, функциональный фрагмент или функциональный гомолог Rpi-chc1 представляет собой фрагмент или гомолог, который способен придавать, по меньшей мере, частичную устойчивость или повышенную устойчивость растения семейства Solanaceae против заражения оомицетами.
Способы испытания функциональности функционального фрагмента или функционального гомолога Rpi-chc1 упомянуты выше.
На основе описанных здесь нуклеотидных последовательностей изобретение также предоставляет зонды и праймеры (т.е. олигонуклеотидные последовательности, комплементарные к одной из (комплементарных) цепочек ДНК, как описано в данном описании). Зонды, например, применимы в Саузерн- или нозерн-анализе, и праймеры применимы, например, в анализе методом ПЦР. Праймеры на основе описанных здесь нуклеотидных последовательностей вполне применимы как помощники растениеводов, работающих в области классической селекции и/или селекции путем генетической модификации нуклеотидного содержания растения (предпочтительно, указанное растение представляет собой Solanum tuberosum, Solanum lycopersicum, ранее известный как Lycopersicon esculentum, перец или баклажан), при селекции растения, которое способно экспрессировать, например, Rpi-chc1 или его функциональный фрагмент или гомолог.
Следовательно, в другом воплощении изобретение относится к связывающей молекуле, способной связываться с нуклеиновой кислотой, кодирующей Rpi-chc1 или его функциональный фрагмент или гомолог, описанные здесь, или его комплементарной нуклеиновой кислотой. В предпочтительном воплощении указанная связывающая молекула представляет собой праймер или зонд. Как указывалось, такая связывающая молекула очень полезна для растениеводов, и поэтому изобретение также относится к способу отбора растения или растительного материала или его потомства по его чувствительности или устойчивости к заражению оомицетами. Предпочтительно испытываемую нуклеиновую кислоту растения выделяют из указанного растения, и полученную изолированную нуклеиновую кислоту приводят в контакт с одной или несколькими (предпочтительно, различными) связывающими молекулами. Можно использовать, например, анализ методом ПЦР для испытания растений на присутствие или отсутствие Rpi-chc1 в геноме растения. Такой способ может быть особенно предпочтительным в протоколах трансформации без маркеров, как описано в WO 03/010319.
Описанный здесь белок Rpi-chc1 также можно использовать для выявления антител способами, известными специалистам. Таким образом, изобретение также относится к антителу, которое (специфически) связывается с белком, кодированным описанной здесь изолированной или рекомбинантной нуклеиновой кислотой (например, нуклеотидной последовательностью на фигуре 7), или антителу, которое (специфически) связывается с белком, отображенным на фигуре 4, или его функциональным фрагментом или функциональным гомологом. Такое антитело применимо, например, в методах анализов белков, таких как Вестерн-блоттинг или ELISA, и поэтому может использоваться при селекции растений, которые успешно экспрессируют ген Rpi-chc1.
На основе описанных здесь нуклеотидных последовательностей изобретение также предоставляет способы придания или увеличения устойчивости растения против заражения оомицетами. Следовательно, изобретение также относится к способу придания растению, по меньшей мере, частичной устойчивости или повышенной устойчивости против заражения оомицетами, включающему предоставление растению или его части
- изолированной или рекомбинантной нуклеотидной последовательности, включающей нуклеотидную последовательность, кодирующую аминокислотную последовательность Rpi-chc1 на фигуре 4 или ее функциональный фрагмент или функциональный гомолог, или
- изолированной или рекомбинантной нуклеотидной последовательности, отображенной на фигуре 7, или
- вектора, включающего описанные здесь нуклеотидные последовательности, или
- клетки-хозяина, описанной в данном описании.
Такой способ придания растению, по меньшей мере, частичной устойчивости или повышенной устойчивости против заражения оомицетами может основываться на классической селекции, начиная с родительского растения, которое уже содержит ген Rpi-chc1 или его функциональный гомолог, или он включает перенос ДНК в растение, т.е. включает способ трансформации растительной клетки, включающий предоставление указанной растительной клетке нуклеиновой кислоты, описанной здесь, или вектора, описанного здесь, или клетки-хозяина, описанной здесь.
Существует несколько способов, которыми рекомбинантную нуклеиновую кислоту можно перенести в растительную клетку, например, трансформация, опосредуемая Agrobacterium. Однако кроме заражения Agrobacterium существуют другие способы эффективной доставки ДНК в реципиентные растительные клетки, когда изобретение желают осуществить на практике. Полагают, что подходящие способы доставки ДНК в растительные клетки включают фактически любой способ, которым ДНК можно ввести в клетку, такой как прямая доставка ДНК, такая как ПЭГ-опосредованная трансформация протопластов, поглощение ДНК, опосредуемое потерей влаги/инигибированием (Potrykus et al., Mol. Gen. Genet, 199: 183-188, 1985), электропорация (пат. США №5384253), перемешивание с волокнами карбида кремния (Kaeppler et al., 1990; пат. США №5302523; и пат. США №5464765) и акселерация частиц, покрытых ДНК (пат. США №5550318; пат. США №5538877 и пат. США №5538880). С применением таких методов, как указанные, можно устойчиво трансформировать клетки из фактически растения любого вида, и такие клетки могут развиваться в трансгенные растения.
В случае использования трансформации, опосредуемой Agrobacterium, предпочтительно использовать, по существу, вирулентную Agrobacterium, такую как А. tumefaciens, как, например, штамм А281 или штамм, полученный из него, или другой вирулентный штамм, доступный в технике. Такие штаммы Agrobacterium содержат участок ДНК, происходящий от вирулентного участка Ti-плазмиды pTiBo542, которая координирует процессинг Т-ДНК и ее перенос в растительные клетки. Трансформация растений на основе Agrobacterium хорошо известна в технике (как, например, описанная в, например, Komari Т. et al.: Plant Transformation Technology: Agrobacterium-Mediated Transformation, in Handbook of Plant Biotechnology, Eds. Christou P. and Klee H., John Wiley & Sons, Ltd, Chichester, UK 2004, pp.233-262). Предпочтительно используют протокол трансформации без маркеров, такой как описанный в WO 03/010319.
С другой стороны, нуклеиновую кислоту гена Rpi-chc1 или его функционального гомолога можно ввести в растение скрещиванием. Схема такого скрещивания начинается с отбора подходящего родительского растения. Это может быть, например, исходный сорт Solanum chacoense (такой как образец СНС543-5), исходный сорт S.tarijense (такой как образец TAR852), исходный сорт S. berthaultii (такой как образец BER481-2 или BER94-2031) или растение, которое получило нужную нуклеиновую кислоту методами генной инженерии, описанными выше.
Любой подходящий способ, известный в технике для скрещивания выбранных растений, может быть применен в способе по изобретению. Это включает способы как in vivo, так и in vitro. Специалист в данной области техники должен иметь в виду, что методы in vivo, такие как слияние протопластов или спасение зародыша, можно применять, когда они считаются подходящими.
Выбранные растения, которые используют для целей скрещивания в способах по изобретению, могут иметь любой тип плоидности. Например, выбранные растения могут быть гаплоидными, диплоидными или тетраплоидными. Однако скрещивание диплоидных растений, таких как S.chacoense, S.tarijense и S.berthaultii, будет давать только диплоидное потомство. Скрещивание диплоидного растения с тетраплоидным растением будет приводить к триплоидному потомству, которое является стерильным.
Таким образом, когда отбирают растения, которые являются диплоидными, их плоидность следует повысить до тетраплоидного уровня перед тем, как их можно скрещивать с другим тетраплоидным растением в способах по изобретению. Способы повышения плоидности растения известны в технике и могут быть легко применимы специалистом в данной области техники. Например, плоидность диплоидного растения для целей скрещивания можно повысить путем использования гамет 2N указанного диплоидного растения. Плоидность также можно повысить путем ингибирования сегрегации хромосом во время мейоза, например, обработкой диплоидного растения колхицином. Применяя такие методы к диплоидному растению, получают зародыши или гаметы, которые включают удвоенное обычное число хромосом. Затем такие зародыши или гаметы можно использовать для целей скрещивания. В случае картофеля предпочтительно устойчивое тетраплоидное растение, так как известно, что тетраплоидные растения имеют более высокий выход клубней.
Так как оказывается, что характеристика устойчивости является доминантным признаком, достаточно, если присутствует только один аллель с функциональным геном.
Предпочтительно выбранные растения скрещивают друг с другом с использованием классических методов скрещивания in vivo, которые включают одну или несколько стадий скрещивания, в том числе, самоопыление. Применяя такие стадии классического скрещивания, в потомстве можно объединить характеристики обоих родителей. Например, растение, которое дает высокий урожай, можно скрестить с растением, которое содержит большое количество определенных питательных веществ. Такое скрещивание может дать потомство, включающее обе характеристики, т.е. растения, которые не только содержат большие количества питательных веществ, но также дают высокие урожаи.
Когда применяют обратное скрещивание, потомство F1 скрещивают с одним из его высокоурожайных родителей Р для гарантии того, что характеристики потомства F2 будут походить на свойства высокоурожайного родителя. Например, выбранное диплоидное растение картофеля с устойчивостью к оомицетам делают тетраплоидным, используя колхицин, и затем скрещивают с выбранным тетраплоидным высокоурожайным культиваром картофеля с целью получения в итоге высокоурожайного тетраплоидного потомства с устойчивостью к оомицетам. Также можно применить самоопыление. Выбранные растения или потомство затем скрещивают с ними самими для получения инбредных сортов для разведения. Например, выбранные образцы из вышеуказанного потомства F1 скрещивают с ними самими для получения потомства F2, из которого можно отобрать образцы, которые имеют повышенный уровень устойчивости.
После переноса нуклеиновой кислоты в растение или растительную клетку следует определить, какие растения или растительные клетки снабжены указанной нуклеиновой кислотой. При селекции и скрещивании в способе по изобретению, по меньшей мере, на одной стадии селекции в помощь селекции используют маркер. В технике известно, что маркеры, указывающие на определенный признак или состояние, можно обнаружить in vivo и in vitro на различных биологических уровнях. Например, маркеры можно обнаружить на пептидном уровне или генном уровне. На генном уровне маркер можно детектировать на уровне РНК или уровне ДНК. Предпочтительно, в настоящем изобретении присутствие такого маркера детектируют на уровне ДНК с использованием вышеописанных праймеров и/или зондов. С другой стороны, надлежащую экспрессию белка Rpi-chc1 или его функционального гомолога можно оценить в частях растения путем осуществления иммуноанализа с антителами, которые специфически связывают белок. После праймеров и зондов по изобретению также можно использовать специфические маркеры, которые должны обнаруживаться по соседству с кодирующей последовательностью. Такие маркеры указаны ниже в экспериментальной части и включают маркеры, указанные в таблице 2. Маркеры получены из сопровождающих последовательностей ВАС.
В случае трансгенных подходов селекцию трансформированного растения можно выполнять с использованием селектируемого маркера или репортерного гена. Среди селектируемых" маркеров или селекционных генов, которые наиболее широко используются при трансформации растений, находятся гены бактериальной неомицин-фосфотрансферазы (гены nptI, nptII и nptIII), придающие устойчивость к селективному агенту канамицину, как предлагается в ЕР 131623, и бактериальный ген aphIV, предложенный в ЕР 186425, придающий устойчивость к гигромицину. В ЕР 275957 раскрывается использование гена ацетилтрансферазы из Streptomyces viridochromogenes, который придает устойчивость к гербициду фосфинотрицину. Гены растений, придающие относительную устойчивость к гербициду глифозату, предлагаются в ЕР 218571. Подходящими примерами репортерных генов являются бета-глюкуронидаза (GUS), бета-галактозидаза, люцифераза и зеленый флуоресцентный белок (GFP).

В предпочтительном воплощении изобретение относится к способу придания растению, по меньшей мере, частичной устойчивости или повышенной устойчивости против заражения оомицитом, включающему предоставление растению
- изолированной или рекомбинантной нуклеотидной последовательности, включающей нуклеотидную последовательность, кодирующую аминокислотную последовательность Rpi-chc1 на фигуре 4 или ее функциональный фрагмент или функциональный гомолог,
изолированной или рекомбинантной нуклеотидной последовательности, отображенной на фигуре 7, или изолированной или рекомбинантной нуклеотидной последовательности, кодирующей белок, выбранный из группы из 493-7_G12, 543-5_C2, 849-1_М8_М18_М20, 487-1_I4_I6_I8, 94-2031_L4_L7_18, 561-2_K4_K14_K22, 324-2_J1_J3_J8, 852-5_Е14_Е23, 852-5_Е28, 493-9_Н5_Н30, 493-7_G14_G22, 561-2_К6_К30_К31 и 493-7_G21, отображенной на фигуре 13,
- вектора, включающего описанные здесь нуклеотидные последовательности, или
- клетки-хозяина, описанной здесь,
при этом указанный оомицит включает Phytophthora, предпочтительно, Phytophthora infestans, и/или при этом указанное растение включает растение из семейства Solanaceae, предпочтительно, растение картофеля или томата, предпочтительнее, тетраплоидное растение картофеля.
Изобретение также относится к растению, которое можно получить с использованием способа придания растению, по меньшей мере, частичной устойчивости или повышенной устойчивости против заражения оомицитом, описанного выше. Предпочтительное растение представляет собой растение из семейства Solanaceae, и даже предпочтительнее, указанное растение представляет собой Solanum tuberosum или Solanum lycopersicum, ранее известный как Lycopersicon esculentum, Solanum melononga, виды Capsicum, такие как С.annuum, С.baccatum, С.chinense, С.frutescens и С.pubescens. Изобретение также относится к растению, которое снабжено нуклеиновой кислотой, кодирующей белок Rpi-chc1 или его функциональный фрагмент или функциональный гомолог.
Изобретение также относится к части растения или потомству растения по изобретению, включающим нуклеиновую кислоту, кодирующую аминокислотную последовательность Rpi-chc1 на фигуре 4 или ее функциональный фрагмент или функциональный гомолог.
В предпочтительном воплощении описанную в данном описании нуклеиновую кислоту переносят в сорт Solanum иной, чем Solanum chacoense, т.е. описанную в данном описании нуклеиновую кислоту предпочтительно предоставляют предшественнику не-chacoense, предпочтительно, S.lycopersicon или S.tuberosum. Из последних наиболее предпочтительным является тетраплоидный сорт, и предпочтительнее, сорт, представляющий коммерческий интерес, такой как Bintje, Desiree или Premiere, Spunta, Nicola, Favorit, Russet Burbank, Aveka или Lady Rosetta.
Также возможно придание устойчивости по изобретению растению, которое уже частично резистентно к заражению оомицетом, при этом указанному растению предоставляется нуклеиновая кислота, кодирующая другой ген устойчивости, такой как Rpi-blb1, -2, -3, Rpi-vnt1 или Rpi-meq1.
Изобретение также относится к применению изолированной или рекомбинантной нуклеотидной последовательности, включающей нуклеотидную последовательность, кодирующую аминокислотную последовательность Rpi-chc1 на фигуре 4 или ее функциональный фрагмент или функциональный гомолог, или к применению изолированной или рекомбинантной нуклеотидной последовательности, отображенной на фигуре 7, или к применению вектора, включающего любую из указанных нуклеотидных последовательностей, или к применению клетки-хозяина, включающей любую из указанных нуклеотидных последовательностей или указанный вектор, для придания растению, по меньшей мере, частичной устойчивости против заражения оомицетом. В предпочтительном воплощении указанный оомицет включает Phytophthora, и даже предпочтительнее, Phytophthora infestans. В еще одном предпочтительном воплощении указанное растение включает Solanum tuberosum или Solanum lycopersicum, ранее известный как Lycopersicon esculentum.
В еще одном воплощении изобретение относится к способу получения белка Rpi-chc1 или его функционального фрагмента или функционального гомолога, включающему функциональную связь нуклеиновой кислоты, описанной здесь, с регуляторной последовательностью и создание возможности экспрессии указанной нуклеиновой кислоты в клетке-хозяине. Примерами регуляторной последовательности являются промоторная и/или терминаторная последовательность. Кроме того, как станет ясно из примера 2, предпочтительно, чтобы последовательность Rpi-chc1 экспрессировалась под контролем своего собственного промотора и терминатора. Поэтому изобретение также относится к промоторной и/или терминаторной последовательностям Rpi-chc1 (фигура 7). Фигура 7 показывает нуклеотидную последовательность клона СНС В2-3 (7907 п.о.), содержащего ген Rpi-chc1 и регуляторные последовательности. Кодирующий Rpi-chc1 участок в 4550 п.о. выделен затемнением (нукл. 3358-7266). Вышерасположенные 3357 нукл. (1-3357) и нижерасположенные 641 нуклеотид ниже (нукл. 7267-7907) содержат регуляторные последовательности, которые обеспечивают правильную экспрессию гена. Специалист вполне способен клонировать (частично) указанные регуляторные последовательности и испытать их эффективность в транскрипции. Также обнаружено, что получают даже еще лучшую экспрессию с укороченным промотором, т.е. промотором, содержащим менее 1000, предпочтительно, не более 900 пар оснований выше последовательности гена.
Изобретение будет поясняться подробнее приведенными далее примерами, не являющимися ограничительными.
Экспериментальная часть
Пример 1. Развитие популяций
Последняя таксономическая перегруппировка отдела Petota Solanum показывает недостаток структуры вида в указанном отделе (Jacobs et al., 2008). Для того чтобы идентифицировать признаки устойчивости к фитофторозу, из таксономической группы 10-14 (Jacobs et al., 2008) отбирают несколько образцов и проверяют их уровни устойчивости к Phytophthora infestans в полевых испытаниях. Отбирают пять образцов с высокими уровнями устойчивости, которые определены ранее как S. tarijense (TAR), S. berthaultii (BER) и S. chacoense (CHC) (TAR852-5, BER94-2031-01, BER481-3, BER493-7, CHC543-5). Для того чтобы изучить генетическую основу их устойчивости, получают гибриды с использованием устойчивых BER493-7, CHC543-5 и BER94-2031-01 в качестве родителей. Полученные популяции F1 испытывают не расщепление устойчивости к Р. infestans в анализе отделенного листа (таблица 1).
Таблица 1. Анализ популяций
Анализы отделенного листа выполняют на потомках указанных гибридов. Соотношение расщепления растений определяют с фенотипами у(стойчивый), ч(увствительный) или с(омнительный).
В популяциях 7650 и 06-882 обнаруживают явное расщепление 1:1 - характеристика для расщепления единственного доминантного гена устойчивости. В популяции 7767 также обнаружено расщепление 1:1, однако также обнаружена группа из 10 растений с промежуточными (с) уровнями устойчивости.
Позиции картирования Rpi-chc1 и Rpi-ber
Из литературы известно, что ген устойчивости к фитофторозу из S.berthaultii (Rpi-ber) тесно связан с TG63 на длинном плече хромосомы 10 (Rauscher et al., 2006) - участком, к которому также картируется Ph-2 QTL томата из S.pimpenellifolium (Moreau et al., 1998). Поэтому авторы разработали маркеры CAPS в TG63 в трех популяциях. С использованием полиморфизма, описанного в таблице 2, обнаружено, что устойчивость в 06-882 и 7650 тесно связана с TG63, так как найдены один и два рекомбинанта, соответственно. Также устойчивость в 7677 связана с TG63, хотя наблюдают более высокую частоту рекомбинаций (15 рекомбинаций). Следует вывод, что указанная область на хромосоме 10 является весьма важной для устойчивости к фитофторозу. Поэтому авторы намеревались использовать хорошо охарактеризованную физическую карту RH89-039-16 для того, чтобы генерировать ссылочную карту локуса TG63. С использованием полиморфизма, описанного в таблице 2, TG63 картируют к RH10B41. В такой позиции картирования закрепляют контиг 6701. Концевые последовательности ВАС в указанном контиге используют для генерации маркеров, подходящих для картирования в популяции 7650. Обнаруживают, что RH199E15S (таблица 2) сорасщепляется с устойчивостью в 7650 и 06-882, что указывает, что 6710 из RH89-039-16 находится в локусе, синтеническом с локусом Rpi-chc1 и Rpi-ber.
Кроме закрепления TG63 генетически, его также размещают на физической карте RH89-039-16 с помощью скрининга библиотеки RH ВАС методом ПЦР. Обнаруживают положительный контиг 2203. Что примечательно, контиг 2203 закрепляют на RH10B38 с использованием двух независимых маркеров (Jan de Boer, PGSC). Разрабатывают маркеры CAPS на основе концевых последовательностей ВАС в контиге 2203 и картируют в популяциях 06-882 и 7650. Также указанные маркеры тесно связаны с устойчивостью, что показывает, что такой контиг также находится в локусе, синтеничном с локусом Rpi-chc1 и Rpi-ber.
С использованием концевых последовательностей ВАС идентифицируют три дополнительных контига RH ВАС, фланкирующие контиги 2203 и 6701 (фигура 1А). Для того чтобы получить достаточную информацию о последовательностях для точного картирования, составляют и секвенируют два перекрывания по типу черепичной кровли, состоящие из 3 и 4 перекрывающихся ВАС (106G038, 137D014, 009D021 и 122В15, 77023, 04G12, 199Е15). Аннотация последовательности RH ВАС (фигура 2) показывает присутствие двух RGA в первом перекрывании (который картирован к RH10B38) и 7 RGA во втором перекрывании (который картирован к RH10B41, 42), показанных остриями стрелок на фигуре 1А. Некоторые маркеры, полученные из них и других последовательностей хромосомы 10, картированы в популяции S.chacoense 7650 (фигура 1В) и популяции S.berthaultii 06-882 (фигура 1А). Размер таких популяций возрастает до 2357 и 2532, соответственно. Рекомбинанты в релевантных участках генома скринируют для использования маркеров RH099F09T и RH092A09S в популяции 7650 и маркеров RH91C10T и RH199 Е15S в популяции 06-882. Маркеры, которые получают из одних и тех же RH ВАС (RH137D14), 137D14-C37-7 и 137D14-C37-2 находятся в RH89-039-16 только на расстоянии 15 т.п.о. и совместно расщепляются в популяции 7650 (два рекомбинанта) и в популяции 06-882 (нет рекомбинантов), соответственно. Это решительно предполагает, что Rpi-chc1 и Rpi-ber находятся в синтеничных кластерах генов, и что может существовать аллельное соотношение между генами.
Клонирование Rpi-chc1
Для того чтобы клонировать Rpi-chc1, конструируют две библиотеки ВАС с использованием ДНК, полученной из устойчивого клона СНС543-5. Первую библиотеку конструируют в векторе pCC1BAC ВАС, и она содержит приблизительно 22000 клонов со средним размером вставки ~70 т.п.о., что соответствует 1 геномному эквиваленту. Вторую библиотеку конструируют в векторе pIndigoBAC-5 ВАС, и она содержит приблизительно 110000 клонов со средним размером вставки ~45 т.п.о., что соответствует 3 геномным эквивалентам. Первую библиотеку скринируют с маркером RH106G03T (таблица 2, фигура 1В), который совместно расщепляется с устойчивостью в популяции 7650 с только тремя событиями рекомбинации. Таким путем идентифицируют клоны СНС В1. Оба конца ВАС СНС В1 (В07_1_С15) картируют, и конец RP (маркер B07_1_C15_RP'), который показывает только одно событие рекомбинации с геном устойчивости Rpi-chc1, используют для скрининга второй библиотеки ВАС, и идентифицируют СНС В2 (2-D06_3-D21) (фигура 1В). СНС В2 получают как содержащий маркер RH137D14 С37-7. Два события рекомбинации находят с RH137D14 С37-7 на другом участке гена устойчивости Rpi-chc1. Поэтому делают вывод, что локус Rpi-chc1 ограничивается до интервала 0,2 сМ (рекомбинанты 5/2357), который физически перекрывается двумя частично перекрывающимися клонами ВАС СНС В1 и СНС В2 (фигура 1В).
Путем секвенирования таких двух ВАС находят, что СНС В1 содержит два RGA, и СНС В2 содержит три RGA, которые называют СНС В 1-1, СНС В 1-2, СНС В2-1, СНС В2-2 и СНС В2-3, соответственно (фигура 2). Последние три RGA находятся в интервале картирования, ограниченным B07_1_C15_RP' и RH137D14 С37-7. Поэтому три гена субклонируют в pBINplus под контролем их нативных регуляторных элементов методом ПЦР протяженных участков с использованием высокоточной полимеразы Phusion®. Полученные субклоны полностью секвенируют и находят, что они идентичны последовательностям матрицы их ВАС.
Комплементационный анализ осуществляют в Nicotiana benthamiana с использованием анализа временной (инфильтрации) Agrobacterium tumefaciens (агроинфильтрация), когда 4-недельные растения N. benthamiana дикого типа инфильтруют штаммом Agrobacterium AGL1+virG, содержащим pBINplus:CHC B2-1, pBINplus:CHC B2-2 и pBINplus:CHC В2-3, соответственно. В качестве контроля используют pBINplus без вставки и pBINplus:Rpi-blb1. Через два дня инфильтрованные листья заражают штаммом Р.infestans 90128 в анализах отделенного листа (DLA). Листья, нфильтрованные pBINplus:CHC В2-3 и pBINplus:Rpi-blb, показывают устойчивость к заражению, в то время как pBINplus:CHC B2-1, pBINplus:CHC B2-2 и pBINplus без вставки заселяются Phytophtora, как явствует из споруляции повреждений (фигура 3). Данный эксперимент ясно показывает, что СНС В2-3 является активным геном устойчивости против Р.infestans. Так как ни один другой ген из присутствующих в интервале генетического картирования Rpi-chc1 не показывает активность, можно сделать вывод, что СНС В2-3 представляет собой ген Rpi-chc1.
Гомология и структура Rpi-chc1
Представляет интерес, что Rpi-chc1 имеет наибольшую гомологию (75-98%) с другими RGA из кластера генов из S.chacoense и с генами из синтенических кластеров на хромосоме 10 из клона S.tuberosum RH89-039-16 (таблица 3, фигура 4). Обнаруживают меньшую (40%), но существенную степень гомологии с белком, кодированным геном с неизвестной функцией из тополя (образец номер ABF81421, таблица 3, фигура 4). Различные домены белка Rpi-chc1 имеют гомологию различной степени с соответствующими доменами белка тополя, кодированными ABF81421. Домен NBS является наиболее консервативным (идентичность 48%), затем следует домен СС (идентичность 34%). Домен LRR является наименее консервативным (идентичность 21%). Найдена общая гомология менее 33% с белком FOM2 из огурца (Joobeur et al., 2004), который придает устойчивость к патогенному грибу Fusarium oxysporum, Rpi-blb1 из S. bulbocastanum (Song et al., 2003; van der Vossen et al., 2003), R3a из S. demissum (Huang et al., 2005) и RPS1-k из сои (Glycine max)(Gao et al., 2005), который придает устойчивость к Phytophthora sp.
Rpi-chcl включает ORF из 3909 нуклеотидов (нук), которая кодирует белок в 1302 аминокислоты, включающий все последовательности, характеризующие R-белки CC-NB-LRR (фигура 6). На N-конце можно выделить 5 тяжей аминокислот по их способности к укладке в спиральную структуру. Центральный домен NB-ABC содержит тяжи аминокислот, которые проявляют сходство с субдоменами Kinase 1a, Kinase 2, Kinase 3а, GLPL, RNBS-D и MHD (Bendahmane et al., 2002; van der Biezen and Jones, 1998). В отличие от других белков NB-LRR белок Rpi-chc1 характеризуется отсутствием очевидного субдомена RNBS-A и присутствием двойного субдомена MHD. С-Концевой домен содержит 29 несовершенных повторов, богатых лейцином (LRR). Оба LRR 3 и 4 содержат характерный признак LDL, который обычно присутствует в LRR3. Как MHD, так и LRR3 вовлекаются в регуляцию активности и предполагаемые внутримолекулярные взаимодействия (Bendahmane et al., 2002; Tameling et al., 2006). Дупликация обоих таких субдоменов может предполагать обычный регуляторный механизм.
Локусы генома гомологичные Rpi-chc1; локус направленное профилирование
Для того чтобы идентифицировать позиции в геноме, которые содержат нуклеотидные последовательности, родственные Rpi-chc1, разработан новый метод, который основан на профилировании NBS (Brugmans et al., 2008; van der Linden et al., 2004), который будет называться «локуснаправленное профилирование». Вместо используемых ранее праймеров, которые нацелены на домены, которые обычно присутствуют во всех R-генах, теперь используются праймеры, которые консервативны в последовательностях семейства Rpi-chc1 (таблица 2). В таком случае ожидается, что целью являются только гены, родственные Rpi-chc1. Геномную ДНК от родителей и потомства различных популяций (SH×RH, 06-882) гидролизуют с RsaI, HaeIII, AluI или MseII. К продуктам гидролиза лигируют адаптер, и с использованием адапторного праймера в комбинации со специфическим праймером семейства Rpi-chc1 реакцией ПЦР создают несколько фрагментов с различной молекулярной массой. Полосы полиморфов в двух популяциях детектируют с использованием полиакриламидного геля Licor. Полосы полиморфов оценивают в 40 растениях потомства популяций SH×RH, и впоследствии картины расщепления маркеров приводят в соответствие с картой UHD (van Os et al., 2006). К длинному плечу хромосомы 10, где располагается ген Rpi-chc1, картируются 73% маркеров. Также анализ последовательностей изолированных фрагментов маркеров показывает значительную гомологию с семейством гена Rpi-chc1 (таблица 4b). В целом такие данные показывают, что «локус направленное профилирование» является успешным подходом к получению маркеров в специфической области генома. На хромосоме 10 с высокой частотой маркируются три различных локуса (таблица 4а). Представляет интерес, что первые два локуса совпадают с позициями картирования контигов 2203 и 6701 на карте RH10B38-39 и RH10B41-42, соответственно. Третья группа маркеров картируется к RH10B54. Представляет интерес, что ген Rpi-ber (Park et al., 2008) находится в том же интервале маркеров, что и кластер RH10B54. Для того чтобы проверить является ли ген Rpi-ber потенциально гомологом Rpi-chc1, в популяции 06-882 получают 58 маркеров локус направленного профилирования Rpi-chc1. От устойчивого родителя получают 34 маркера из указанных. Из них 28 связывают с устойчивостью (9 в фазе сцепления, 19 в фазе отталкивания). Полностью связывают с устойчивостью в первых 1771 особи популяции 2 маркера фазы сцепления и 7 маркеров фазы отталкивания. Это решительно приводит к мысли, что Rpi-ber является гомологом Rpi-chc1. Среди 28 связанных маркеров в локус направленном профилировании Rpi-chc1 можно различить четыре группы картин рекомбинации, и каждая группа отмечается названием характерного маркера на фигуре 1А. Три группы маркеров соответствуют кластеру RH10B38-39, одна группа маркеров соответствует кластеру RH10B41-42. Такой результат подтверждает результат авторов, полученный с популяцией SH×RH, что семейство родственных Rpi-chc1 последовательностей на хромосоме 10 располагается в, по меньшей мере, двух тесно связанных кластерах.
oryza.pro
В другой популяции (7677), происходящей от S.berthaultii, образец 493-7, обнаружено, что маркер профиля NBS, полученный с описанным ранее праймером NBS5a6, в этой популяции тесно связан с устойчивостью к Phytophthora. Он картирован к теломерному сайту, относящемуся к TG403 на длинном плече хромосомы 10 (фигура 6). Анализ последовательности данного фрагмента показывает высокую гомологию с членами семейства Rpi-chc1. Все вместе такие результаты показывают, что на хромосоме 10 присутствуют, по меньшей мере, четыре генетически различных кластера. Это схоже с ситуацией на длинном плече хромосомы 9, где идентифицированы три различных родственных Тт2-2 кластера (Foster et al., 2009; Pel et al., 2009).
Растительный материал и изоляты Phytophthora infestans
В данном исследовании используют четыре устойчивых к фитофторозу клона - TAR852-5 (происходящий от CGN22729), BER94-2031-01 (происходящий от PI473331), BER481-3 (происходящий от CGN18190), BER493-7 (происходящий от CGN17823), СНС543-5 (происходящий от BGRC63055). СНС543-5 скрещивают с СНС544-5, и получают популяцию 7650. BER94-2031-01 скрещивают с чувствительным клоном G254, и получают популяцию 06-882. BER493-7 скрещивают с RH89-039-16, и получают популяцию 7677. Используют для трансформации культивар картофеля Дезире. Растения Nicotiana benthamiana дикого типа используют для анализов временной комплементации.
Характеристики и происхождение изолятов Р.infestans, используемых в данном исследовании, указаны в таблице 5.
Конструирование библиотек ВАС
В качестве источника для конструирования библиотек ВАС используют клон СНС543-5. Получение высокомолекулярной ДНК и конструирование библиотек ВАС осуществляют так, как описано Rouppe van der Voort et al., 1999. Для первой библиотеки используют в качестве основы pCC1BAC. Для второй библиотеки используют pIndigoB AC-5, оба из Epicenter. Получают приблизительно 22000 клонов со средним размером вставки ~70 т.п.о., соответствующей 1 геномному эквиваленту, для библиотеки 1, и для библиотеки 2 получают приблизительно 110000 клонов со средним размером вставки ~45 т.п.о., соответствующей 3 геномным эквивалентам. Клоны ВАС хранят в виде бактериальных пулов, содержащих приблизительно 700-1000 белых колоний. Их получают, снимая колонии с агаровых пластинок с последующим ресуспендированием в среде LB, содержащей 18% глицерина и 12,5 мкг·мл-1 хлорамфеникола, с использованием стерильной стеклянной лопатки. Такие так называемые суперпулы хранят при -80°С. Осуществляют скрининг маркеров библиотек ВАС, сначала изолируя плазмидную ДНК из каждого пула с использованием стандартного протокола щелочного лизиса, и осуществляют ПЦР для идентификации положительных пулов. Бактерии, соответствующие положительным пулам, разбавляют и высевают на агаровую пластинку LB, содержащую хлорамфеникол (12,5 мкг·мл-1). Отдельные белые колонии пикируют в 384-луночные титрационные микропланшеты, и затем идентифицируют отдельные положительные клоны скринигом маркеров, как описано Rouppe van der Voort et al., 1999. Названия клонов ВАС, выделенных из суперклонов, содержат префикс СНС, за которым следует номер (В1 и В2), соответствующий порядку, в котором их идентифицируют. Субклонирование кандидатов в гены ПЦР протяженных участков
Кандидатов в RGA субклонируют из клона ВАС СНС В2 следующим образом. Создают праймеры приблизительно на 3 т.п.о. выше прогнозируемого иницирующего кодона и приблизительно на 700 п.о. ниже прогнозируемого терминирующего кодона. (СНС B2-1F=MN459: tgaccctgcaggGGACCCCTTAACAAGTGATGTG,
СНС B2-1R=MN462: tgacggcgcgccAAAAAGTCCCGCTTTGATACC,
СНС B2-2F- MN483: tgaccctgcaggCCCCTTAACAAGTGATGTGATG,
СНС B2-2R=MN484: tgacggcgcgccTCAGGTTCCCTTACAAGATTCC,
СНС B2-3F=MN479: tgaccctgcaggACGCATCAGGAAGAGAGGAG,
СНС B2-3R=MN480: tgacggcgcgccGCGGTTCCTCTGTGAAACAC)
Секвенирование ДНК и компьютерный анализ
Секвенирование клонов ВАС в библиотеках в 2 т.п.о. и 6 т.п.о. осуществляют с использованием метода дробовика, и выполняется Macrogen (Южная Корея). Реакции секвенирования выполняют с использованием метода с красителем-терминатором. Контиги последовательностей собраны Macrogen. Закрытие пробела осуществляют с использованием блуждающей затравки у «дробленых» клонов или непосредственно у ВАС.
Последовательности контигов анализируют с использованием сетевого приложения FGENESH (Softberry) для того, чтобы предсказать структуру генов. RGA и RGA из публично доступных баз данных выравнивают для анализа гомологии и дистанционного анализа с использованием пакета программ DNA star (Lasergene). Консервативные домены идентифицируют с использованием сетевого приложения SMART (EMBL).
Анализ устойчивости
Анализы отделенного листа используют для определения устойчивости фенотипов первичных трансформантов и листьев N. benthamiana. Для фенотипирования популяции СНС используют изолят 90128. Для фенотипирования популяции ber используют изолят IPO-C. Спектры устойчивости резистентных родителей определяют с использованием изолятов, описанных в таблице 5. Получение инокулята и инокуляцию выполняют так, как описано в литературе (Vieeshouwers et al., 1999). Через шесть дней после инокуляции определяют фенотипы растений. Листья, не показывающие симптомы или локализованный некроз в месте инокуляции, оценивают как устойчивые, а листья с четкими повреждениями от спорообразования как чувствительные.
Временная комплементация в N. benthamiana
Анализы временной трансформации Agrobacterium (агроинфильтрацию) осуществляют на N. benthamiana. Рекомбинантные культуры A.tumefaciens AGL1 + выращивают в среде LB (10 г бактериологического пептона, 10 г Nad и 5 г дрожжевого экстракта в 1 л воды MQ) с добавлением 5 мг/л тетрациклина и 50 г/л канамицина для конструкций pBINplus. Через один или два дня рассчитанное количество культуры (соответствующей OD 0,5 при 600 нм) переносят в среду YEB (5 г мясного экстракта, 5 г бактериологического пептона, 5 г сахарозы, 1 г дрожжевого экстракта, 2 мл 1 М раствора MgSO4 в 1 л воды MQ) с добавлением канамицина для всех штаммов. Через 1 день ночные клетки центрифугируют при 3500 об/мин и ресуспендируют в среде ММА (20 г сахарозы, 5 г солей MS и 1,95 г MES) с добавлением 1 мл 200 мМ раствора ацетосирингона до конечного OD 0,2 и инфильтруют в 4-недельные растения 3-мл шприцом. Затем через два дня инфильтрованные листья заражают штаммом Р.infestans 90128 в анализе отделенного листа (DLA). Гиперчувствительную реакцию (HR) или спорообразование Р.infestans оценивают через 5-7 дней после инокуляции.
Пример 2. Rpi-chc1 является функциональным геном устойчивости против Phytophtora infestans
Методы
Растительный материал и изоляты Phytophtora infestans
В данном исследовании используют 225 растений Solanum, их названия, используемые в данном исследовании, и номера образцов приводятся в таблице 7. Девять устойчивых к фитофторозу растений используют для выделения функциональных гомологов Rpi-chc1 (tar852-5, ber94-2031-01, которые происходят от PI473331, ber481-3, ber493-5, -7, -9, chc543-5, ber324-2, ber487-l, ber561-2, и scr849-l). CHC543-5 скрещивают с СНС544-5, и получают популяцию 7650. BER94-2031-01 скрещивают с чувствительным клоном G254, и получают популяцию 06-882. BER493-7 скрещивают с RH89-039-16, и получают популяцию 7677. Культивар картофеля Дезире используют для трансформации. Растения Nicotiana benthamiana дикого типа используют для анализов временной комплементации.
Характеристики и источник изолятов Р. infestans, используемых в данном исследовании, указаны в таблице 5.
Клонирование генов кандидатов
Гомологи Rpi-chc1 амплифицируют ПЦР с использованием высокоточной термоустойчивой ДНК-полимеразы для ПЦР протяженных участков Phusion® согласно инструкциям изготовителя (New England Biolabs). Создают праймеры, перекрывающие инициирующий и терминирующий ко доны Rpi-chc1 и содержащие удлинения AttB1 и AttB2 (MN595 и MN597, таблица 8). Продукты ПЦР рекомбинируют в pDONR221 с использованием клоназы® ВР согласно инструкциям изготовителя (InVitroGen). Секвенирование ДНК выполняют в Baseclear (Нидерланды) с использованием стандартного и изготовленного праймеров (MN622-MN650, таблица 8). Последовательности анализируют и выравнивают для анализа гомологии и филогении с использованием пакета программ DNA star (Lasergene).
Промоторно-терминаторные конструкции
Для того чтобы получить клоны, содержащие промотор и терминатор Rpi-chc1 для конструирования экспрессирующих конструкций, опосредуемых применением технологии «triple point gateway», создают специфические праймеры (MN598, MN599, MN601, MN670; таблица 8), соответствующие промоторной и терминаторной последовательностям Rpi-chc1, к которым добавляют сайты рекомбинации AttB4, AttB1 и AttB2, АttВ3, соответственно. Продукты ПЦР получают с использованием высокоточной термоустойчивой ДНК-полимеразы для ПЦР протяженных участков Phusion® согласно инструкциям изготовителя. Продукты ПЦР рекомбинируют с использованием клоназы® ВР. Появление ошибок ПЦР исключают путем анализа последовательностей полученных клонов с использованием праймеров MN651 и 652, указанных в таблице 8. Реакции «triple point gateway» выполняют с помощью клоназы LR с использованием таких конструкций и последовательностей ORF в pDONR221.
Анализ устойчивости
Анализы отделенного листа используют для определения устойчивости фенотипов первичных трансформантов и листьев N. benthamiana. Для фенотипирования СНС используют трансгенный изолят 90128. Для фенотипирования гомологов Rpi-chc1 в N.benthamiana используют изолят IPO-C. Получение инокулята и инокуляцию выполняют так, как описано Vieeshouwers et al., 1999. Через шесть дней после инокуляции определяют фенотипы растений. Листья, не показывающие симптомы или локализованный некроз в месте инокуляции, оценивают как устойчивые, а листья с четкими повреждениями от спорообразования как чувствительные.
Временная комплементация в N. benthamiana
Анализы временной трансформации Agrobacterium (агроинфильтрацию) осуществляют на N.benthamiana. Рекомбинантные культуры A. tumefaciens COR308 выращивают в среде LB (10 г бактериологического пептона, 10 г Nad и 5 г дрожжевого экстракта в 1 л воды MQ) с добавлением 5 мг/л тетрациклина и 50 г/л канамицина для конструкций pBINplus. Через один или два дня рассчитанное количество культуры (соответствующей OD 0,5 при 600 нм) переносят в среду YEB (5 г мясного экстракта. 5 г бактериологического пептона, 5 г сахарозы, 1 г дрожжевого экстракта, 2 мл 1 М раствора MgSO4 в 1 л воды MQ) с добавлением канамицина для всех штаммов. Через 1 день ночные клетки центрифугируют при 3500 об/мин и ресуспендируют в среде ММА (20 г сахарозы, 5 г солей MS и 1,95 г MES) с добавлением 1 мл 200 мМ раствора ацетосирингона до конечного OD 0,2 и инфильтруют в 4-недельные растения 3-мл шприцом. Затем через два дня инфильтрованные листья заражают штаммом Р.infestans 90128 в анализах отделенного листа (DLA). Гиперчувствительную реакцию (HR) или спорообразование Р.infestans оценивают через 5-7 дней после инокуляции.
Совместная инфильтрация
В плазмиде PVX в Agrobacterium tumefaciens COR308 присутствует набор из 90 эффекторов (набор РЕХ). Бинарные плазмиды содержат эффектор из Pi, клонированного внутри генома PVX. После агрофильтрации будут экспрессироваться как эффектор, так и PVX. В ходе эксперимента PVX не может распространяться системно, и представляет интерес только локальная экспрессия эффектора. После узнавания кодированного эффектора R-геном HR можно наблюдать 3-5 точек на дюйм. Симптомы PVX видны через 6 дней, и их обычно сначала наблюдают в неинфильтрованных листьях.
В качестве позитивного контроля используют R3a и Аur3а-KI, комбинацию R-ген -Avr-ген, которая, как известно, дает сильную реакцию (Armstrong et al., 2005). Скрининг с кандидатом в Rpi-chc1 показывает некротические пятна с двумя потенциальными эффекторными генами RD12-1 и RD12-2 (фигура 8).
В предыдущем примере описано основанное на картировании клонирование гена Rpi-chc1 из Solanum chacoense, номер образца 543-5. Rpi-chc1 является основателем ранее неописанного семейства R-генов класса CC-NB-LRR и локализуется на хромосоме 10 вблизи маркера TG63. Ген присутствует в кластере генов с пятью гомологами. Генетический анализ показывает, что только три из указанных гомологов (СНС В2-1, СНС В2-2 и СНС В2-3) могут потенциально кодировать Rpi-chc1. Анализ временной комплементации в N.benthamiana предполагает, что СНС В2-3 является активной копией.
В данном эксперименте показывают путем устойчивой трансформации чувствительного cv. Дезире, что действительно СНС В2-3 может соответствовать чувствительности к Phytophtora infestans (фигура 8). Такой результат поддерживает предыдущее предположение о том, что СНС В2-3 представляет собой Rpi-chc1. Также такой результат показывает, что Rpi-chc1 может быть функциональным в широком спектре видов Solanum, таких как S. chacoense и N. benthamiana, а также в S. tuberosum.
Rpi-chc1 специфически узнает эффекторный белок RXLR
Для того чтобы представить спектр активности Rpi-chc1, исследуют, какой компонент Pi распознается. До тех пор, пока все компоненты Pi распознаются хозяином, R-белки являются эффекторами класса RXLR. Изолят Pi T30-4 является а-вирулентным на растениях, экспрессирующих Rpi-chc1, и поэтому в этом изоляте должен экспрессироваться родственный компонент. Недавно секвенирован геном T30-4, и оказывается, его геном кодирует сотни эффекторов RXLR (Hans et al., 2009). В растительный экспрессирующий вектор pGR106 клонированы шестьдесят пять эффекторов RXLR, включающих все известные Avr (Avr1, Avr2, Avr3a, Avr4, Avr-blb1, Avr-blb2), и также пять эффекторов не-RXLR (Inf1, PiNIP), и названы набором РЕХ (Vieeshouwers et al., 2008). Набор РЕХ скринируют совместной агрофильтрацией с Rpi-chc1 в N. benthamiana. Таким путем как выбранный эффектор, так и ген Rpi-chc1 экспрессируются в одних и тех же клетках. В случае, когда эффектор узнается Rpi-chc1, он будет вызывать гиперчувствительную реакцию (HR), и результатом будет некротическое повреждение в инфильтрованном участке листа. Такое явление хорошо описано для совместной инфильтрации R3a и Avr3a (Armstrong et al., 2005), что включено в эксперименты в данном описании как положительный контроль (фигура 9). Участки листа, которые агроинфильтрованы одним Rpi-chc1, остаются зелеными, что показывает, что сам Rpi-chc1 не вызывает гибели клеток. Также совместная инфильтрация с описанными ранее Avr (Avr1, Avr2, Avr3a, Avr4, Avr-blb1, Avr-blb2) не вызывает HR, что показывает, что Rpi-chc1 узнает новый компонент Pi, и что он имеет уникальный путь индукции устойчивости. С другой стороны, некоторые эффекторы из набора РЕХ вызывают независимую от Rpi-chc1 гиперчувствительную реакцию (фигура 9, лист С). Однако также имеются два клона в наборе РЕХ, только которые показывают зависимую от Rpi-chc1 гибель клеток (фигура 9, лист В). Оба клона (RD12-1 и RD12-2) являются высоко гомологичными друг с другом и фактически кодируют идентичные белки. RD31, который кодирует белок с 60% идентичностью с RD12, не узнается (фигура 9, лист А), что показывает, что узнавание Rpi-chc1 является довольно специфическим. Для того чтобы проверить специфичность узнавания со стороны R-гена, RD12 совместно инфильтруют с Rpi-blb1, Rpi-blb3 и R3a. Также совместной инфильтрацией проверяют паралоги Rpi-chc1 СНС В2-1 и СНС В2-2 (см. пример 1), которые показывают 78% и 83% идентичность, соответственно, с Rpi-chc1 по аминокислотному уровню. Ни один из таких R-генов или паралогов R-генов не вызывает гиперчувствительной реакции после совместной инфильтрации с RD12 (данные не приводятся). Такие результаты ясно показывают, что Rpi-chc1 может специфически узнавать компонент Pi RD12. RD12 (=PITG_16245) имеет несколько паралогов в геноме Pi (PITGJ6418, PITGJ6427, PITGJ6233, PITG_16240, PITGJ20934, PITG_20936, PITGJ20336, и PITG_23230), последовательности которых приводятся ниже.

Нельзя исключить, что такие паралоги также узнаются Rpi-chc1 при взаимодействии с Pi. Нельзя исключить, что могут узнаваться дополнительные неродственные Pi компоненты, так как описана двойственная специфичность R-генов (Jones and Dang1, 2006).
Требование к промотору для экспрессии Rpi-chc1
Для того чтобы определить, какие регуляторные последовательности наиболее подходят для управления экспрессией открытой рамки считывания Rpi-chc1, используют стратегию, описанную ранее (Lokossou et al., 2009), по которой кандидатов в OFR клонируют между нужными промоторами и терминаторами с использованием стратегии triple point gatway. OFR Rpi-chc1 клонируют между ее собственными промотором в 3 т.п.о. и терминатором в 0,5 т.п.о. (p-chc1-long), которые также присутствуют в анализах начальной комплементации, как представлено на фигуре 8. Кроме того, OFR Rpi-chc1 клонируют между тремя альтернативными комбинациями промотор/терминатор:
укороченный вариант (0,8 т.п.о.) ее собственного промотора и ее собственного терминатора в 0,6 т.п.о. (p-chc1-short); двойной промотор 35S в pMDC32 и комбинация промотор/терминатор Rpi-blb3 (Lokossou et al., 2009). Для того чтобы проверить, какая комбинация промотора и терминатора является оптимальной, четыре конструкции Rpi-chc1 трансформируют в AGL-1+virG, культуры смешивают, 1:1, с A. tumefaciens COR308, содержащим PEX-RD12. Серийные разведения в среде ММА инфильтрируют в листья N. benthamiana (фигура 10). Конструкция p-chc1-long вызывает HR в смесях с RD12 с OD600 2,0 и 1,0. Конструкция p-chc1-short также экспрессирует HR в концентрации, меньшей в два раза (OD600=0,5). Примечательно, что конструкции промотор/терминатор 35S и Rpi-blb3 не подходят для функциональной экспрессии гена Rpi-chc1. Такие результаты показывают, что промотор Rpi-chc1 функционально отличается от других испытываемых промоторов. Кроме того, считается, что последовательности в обратном направлении (<-900 п.о.) в промоторе Rpi-chc1 содержат элементы ингибирования экспрессии.
Скрининг зародышевой плазмы на последовательности, схожие с Rpi-chc1
Для того чтобы дополнительно подтвердить предположение, что Rpi-chc1 может быть активным в широком интервале видов Solanum и также исследовать дивергенцию аллельной последовательности Rpi-chc1 и активность в зародышевой плазме, проводят скрининг 225 генотипов (таблица 7) из авторской коллекции зародышевой плазмы на присутствие родственных последовательностей Rpi-chc1 с использованием выравнивания последовательностей активного Rpi-chc1 и нескольких родственных последовательностей, идентифицированных в первоначальной заявке, полученных от RH89-039-16 и от неактивных паралогов в chc543-5. Пары праймеров (таблица 8) создают таким образом, что предположительно только активная копия амплифицируется ПЦР. Как видно на фигуре 11А, комбинации праймеров D и Е являются высокоспецифичными, так как продукты ПЦР наблюдают только в реакционных смесях, которые содержат матрицу Rpi-chc1, и амплификацию не обнаруживают из матриц, которые содержат близкородственные последовательности. Комбинации праймеров D и Е используют для скрининга рекомбинантов при точном картировании популяции S.chacoence (n=2400) и S.berthaultii (n=2600; Rpi-ber; инвентарный №PI265858; 94-2031 *G254), в которых устойчивость к Pi расщепляется. В любой популяции не обнаруживают рекомбинантов между маркером и устойчивостью (данные не приводятся). Это показывает, что оба маркера являются высокоспецифичными. Также это показывает, что ген Rpi-ber родственен Rpi-chc1, и что молекулярные маркеры, полученные из Rpi-chc1, можно использовать для мечения таких генов устойчивости.
Генотип chc543-5, из которого выделен Rpi-chc1, размещается в таксономической группе 10-14 (Jacobs et al., 2008). Для того чтобы осуществить скрининг для других гомологичных последовательностей Rpi-chc1, отбирают 225 генотипов из авторской коллекции зародышевой плазмы (таблица 7), размещенных в таксономических группах с 10-12 по 10-17. Целостность ДНК подтверждают с использованием ПЦР Ef1-α (данные не приводятся), и затем используют комбинацию праймера D для скрининга родственных последовательностей Rpi-chc1. В таком скрининге обнаружено, что шесть генотипов являются положительными (фигура 11В). Первым из всех обнаружен chc543-5, что подтверждает надежность скрининга. Кроме того, обнаружено пять других генотипов, среди которых растения S. berthaultii 324-2, 481-3 и 561-2, что подтверждает предшествующее предположение о том, что Rpi-chc1 и Rpi-ber действительно являются родственными. Также метят два других вида S. tarijense (852-5) и S. sucrense (849-1).
Выявление гомологов Rpi-chc1
Для того чтобы охарактеризовать функциональное и последовательное сохранение или расхождение Rpi-chc1, клонируют открытые рамки считывания из растений, которые являются положительными при скрининге зародышевой плазмы, и кроме того, из растений, известных как содержащие гены устойчивости на хромосоме 10 (описанные на фигуре 6). Создают праймеры, перекрывающие инициирующий и терминирующий кодоны Rpi-chc1, и добавляют удлинения tat1 и AttB2 для клонирования ВР в pDONR221. Реакции ПЦР с использованием исправляющей ошибки полимеразы Phusion® приводят к специфическим продуктам для всех выбранных генотипов. Такие ПЦР-фрагменты клонируют, и в случае каждого генотипа отбирают и секвенируют по концам шесть колоний. Некоторые генотипы продуцируют только один тип последовательности, и для таких генотипов делают вывод, что амплифицируется только один ген-мишень. Для генотипов с двумя или большим числом типов последовательностей еще 16 колоний секвенируют по концам и группируют. Из каждой группы последовательностей три клона секвенируют полностью с использованием полученных из Rpi-chc1 внутренних праймеров. Это приводит к идентификации 21 новой последовательности, схожей с Rpi-chc1 (фигура 13). Кодированные последовательности белков выравнивают с использованием clustal-W вместе с ранее идентифицированными гомологами Rpi-chc1 (фигура 14). Это приводит к филогенетическому древу, представленному на фигуре 12. Из chc543-5 выделяют два типа последовательностей. Первый тип идентичен Rpi-chc1. Второй тип последовательностей локализуется на другой ветви (ветвь 1 на фигуре 12) с несколькими последовательностями, причем все происходят от растений S. berthaultii, что показывает, что такой подход является удачным при идентификации гомологов Rpi-chc1. Четыре генотипа 849-1, RH89-39, 487-1 и 94-2031-1 дают только один тип последовательности. Первые три размещаются на одной и той же ветви 2 (ветвь 2 на фигуре 12). Последовательности RH89-39 RHJD3, D4 и D7 идентичны друг с другом и показывают два ошибочных спаривания нуклеотидов с RH137D14 с 13-2 -последовательностью, которая генерируется во время конструирования физической карты RH в начальной заявке. Обе последовательности размещаются на ветви 2, которая также содержит последовательности S. sucrense 849-1JMS, M18 и М20, и также последовательности S. berthaultii 487-1, 14, 16 и 18. Кроме того, на ветви 2 присутствует 852-5_Е3S. tarijense. Поскольку RH89-39 чувствителен к заражению Pi, предполагается, что указанные последовательности представляют собой неактивные гомологи. Две другие последовательности, выделенные из S. tarijense 852-5, размещаются на ветви 3, которая также содержит ген Rpi-chc1. Более того, на этой ветви обнаружены три последовательности из растений S. berthaultii 94-2031-1, 561-2 и 324-2, которые показывают только минимальное отклонение последовательности и кодируют идентичные аминокислотные последовательности. Ветвь 4 содержит последовательности только из растений S. berthaultii. Ветвь 5 содержит только последовательности, которые идентифицированы ранее, так же как в случае оставшейся группы, названной группой 6. Ветви с 1 по 4 имеют N-концевое приращение кодированного белка в 45 а.к. по сравнению с белками на ветви 5 и группе 6. Последовательности на ветвях 2, 3 и 5 картированы к кластеру R-генов в пределах 0,1 сМ к TG63. Последовательности на ветвях 1 и 4 генетически не картированы. Сравнение с недавно доступными последовательностями генома S.phureja показывает, что последовательности на ветвях с 1 по 5 имеют самые близкие гомологи в кластере TG63. Сравнение с геномом томата показывает, что здесь также существует кластер Rpi-chc1 вблизи TG63. Как показано ранее, в таком генетическом местоположении картирован ген устойчивости к Pi Ph-2. Некоторые растения томата, которые были секвенированы, не содержат ген устойчивости Ph-2, но может присутствовать потенциально неактивный аллель (фигура 13). Последовательности группы 6 имеют самую близкую гомологию с кластером родственных R-генов вблизи TG403 на хромосоме 10 - участком, где также картирована устойчивость к Pi (см. фиг.6), что показывает, что гомологичные последовательности Rpi-chc1 из указанного кластера потенциально кодируют устойчивость к Pi.
Функциональный анализ гомологов Rpi-chc1
Теперь, когда идентифицирован 21 новый гомолог Rpi-chc1, и авторы показали разнообразие последовательностей, возникает вопрос, сохраняется или варьируется функциональность среди таких последовательностей. Все идентифицированные последовательности, которые представляют собой ORF, субклонируют с использованием рекомбинации triple point gateway под контролем промотора Rpi-chc1-short и терминатора Rpi-chc1 в бинарном векторе pDEST236. На основании результатов, показанных на фигуре 10, это считается наилучшим комплексом для управления экспрессией выявленных гомологов Rpi-chc1. Затем конструкции трансформируют в штамм A. tumefaciens COR308 для анализов временной комплементации в N.benthamiana. С другой стороны, для совместной экспрессии с близким Pi эффектором RD12 гомологи Rpi-chc1 трансформируют в штамм A. tumefaciens AGL1+virG. Оба эксперимента комплементарны, так как анализ временной комплементации может показать, может ли Rpi-chc1 индуцировать устойчивость, а совместная инфильтрация может показать специфичность узнавания гена. Все эксперименты повторяют, по меньшей мере, дважды, и результаты суммируют в таблице 9. Можно наблюдать несколько комбинаций реактивности с RD12 и устойчивости к IPO-C. Можно различить две четкие группы. Первая группа не реагирует с RD12 и чувствительна к IPO-C (группа 1; таблица 9). Такие последовательности являются неактивными гомологами и в основном располагаются на филогенетической ветви 1 (фигура 12). Вторая группа (группа 2; таблица 9) представляет собой функциональные гомологи Rpi-chc1, так как они активно вызывают устойчивость против Pi, и они узнают один и тот же компонент Pi (RD12). Последовательности этой группы также четко отличаются от других последовательностей, так как все они располагаются на ветви 3 (фигура 12). Клон S. tarijense 852-5 Е28 индуцирует HR в отсутствие RD12, и в этом смысле является уникальным по картине своей активности и составляет группу активности 3. Так как он не вызывает устойчивость, он, вероятно, представляет собой неактивный аллель. Другой аллель из того же растения (клон Е14) не узнает RD12, но вызывает сильную устойчивость. Поэтому группа активности 4 отличается от группы 2, поскольку она наиболее вероятно узнает другой компонент из Pi. Группа активности 5 достаточно схожа с группой 4; различие состоит только в том, что устойчивость к заболеванию не настолько сильная. Это предполагает, что группа 5 также узнает другие компоненты из Pi и будет иметь спектр различной устойчивости. Последняя группа (группа 6) отличается, поскольку RD12 узнается только слабо, и устойчивость также слабая. В сумме такие данные показывают, что наиболее близкие гомологи Rpi-chc1 имеют консервативный механизм устойчивости, в то время как наименее родственные последовательности имеют более разнообразной механизм устойчивости. Все вместе такие данные показывают, что многие члены семейства R-генов, которые различаются по степени подобия, являются функциональными при предоставлении устойчивости против Pi.
7.8.10
7.8.11
Таблица 9. Функциональный анализ недавно идентифицированных гомологов Rpi-chc1. В колонке «Реактивность к RD12» R обозначает «реагирующий», N обозначает «не реагирующий», *обозначает «самоактивирующийся», r обозначает слабую реакцию. В колонке «Устойчивость к IPO-C» R обозначает сильную устойчивость, r обозначает слабую устойчивость, S обозначает чувствительность.
Ссылки
Armstrong, M.R., Whisson, S.C., Pritchard, L., Bos, J.I.B., Venter, E., Avrova, A.O., Rehmany, A.P., BAhme, U., Brooks, K., Cherevach, I., Hamlin, N., White, В., Fraser, A., Lord, A., Quail, M.A., Churcher, C., Hall, N., Berriman, M., Huang, S., Kamolin, S., Beynon, J.L. and Birch, P.R.J. (2005) An ancestral oomycete locus contains late blight avirulence gene Avr3a, encoding a protein that is recognized in the host cytoplasm. Proceedings of the National Academy of Sciences of the United States of America, 102, 7766-7771.
Ballvora, A., Ercolano, M.R., Weiss, J., Meksem, K., Bormann, C.A., Oberhagemann, P., Salamini, F. and Gebhardt, C. (2002) The R1 gene for potato resistance to late blight {Phytophthora infestans) belongs to the leucine zipper/NBS/LRR class of plant resistance genes. Plant J., 30, 361-371.
Bendahmane, A., Farnham, G., Moffett, P. and Baulcombe, D.C. (2002) Constitutive gain-of-function mutants in a nucleotide binding site-leucine rich repeat protein encoded at the Rx locus of potato. Plant J., 32, 195-204.
Bradshaw, J.E., Bryan, G.J., Lees, A.K., McLean, K. and Solomon-Blackburn, R.M.
(2006) Mapping the RIO and R11 genes for resistance to late blight {Phytophthora infestans) present in the potato (Solanum tuberosum) R-gene differentials of Black
Theor. Appl. Genet., 112, 744-751.
Brugmans, В., Wouters, D., van Os, H., Hutten, R.C.B., van der Linden, G., Visser, R.G.F., van Eck, H.J. and van der Vossen, E.A.G. (2008) Genetic mapping and transcription analyses of resistance gene loci in potato using NBS profiling. Theor Appl Genet, 117, 1379-1388.
Foster, S.J., Park, Т.Н., Pel, M., Brigneti, G., Sliwka, J., Jagger, L., van der Vossen, E.A.G. and Jones, J.D. (2009) Rpi-vnt1.1, a Tm-2(2) Homolog from Solanum venturii, Confers Resistance to Potato Late Blight. Mol. Plant Microbe Interact., 22, 589-600.
Gao, H., Narayanan, N.N., Ellison, L. and Bhattacharyya, M.K. (2005) Two Classes of Highly Similar Coiled Coil-Nucleotide Binding-Leucine Rich Repeat Genes Isolated from the Rps1-k Locus Encode Phytophthora Resistance in Soybean. Mol. Plant Microbe Interact., 18, 1035-1045.
Haas, B.J., Kamoun, S., Zody, M.C., Jiang, R.H., Handsaker, R.E., Cano, L.M., Grabherr, M., Kodira, C.D., Raffaele, S., Torto-Alalibo, Т., Bozkurt, Т.О., Ah-Fong, A.M., Alvarado, L., Anderson, V.L., Armstrong, M.R., Avrova, A., Baxter, L., Beynon, J., Boevink, P.C., Bollmann, S.R., Bos, J.I., Bulone, V., Cai, G., Cakir, C., Carrington, J.C., Chawner, M., Conti, L., Costanzo, S., Ewan, R., Fahlgren, N., Fischbach, M.A., Fugelstad, J., Gilroy, E.M., Gnerre, S., Green, P.J., Grenville-Briggs, L.J., Griffith, J., Grunwald, N.J., Horn, K., Horner, N.R., Hu, C.H., Huitema, E., Jeong, D.H., Jones, A.M., Jones, J.D., Jones, R.W., Karlsson, E.K., Kunjeti, S.G., Lamour, K., Liu, Z., Ma, L., Maclean, D., Chibucos, M.C., McDonald, H., McWalters, J., Meijer, H.J., Morgan, W., Morris, P.F., Munro, C.A., O'Neill, K., Ospina-Giraldo, M., Pinzon, A., Pritchard, L., Ramsahoye, В., Ren, Q., Restrepo, S., Roy, S., Sadanandom, A., Savidor, A., Schornack, S., Schwartz, D.C., Schumann, U.D., Schwessinger, В., Seyer, L., Sharpe, Т., Silvar, C., Song, J., Studhoime, D.J., Sykes, S., Thines, M., van de Vondervoort, P.J., Phuntumart, V., Wawra, S., Weide, R., Win, J., Young, C., Zhou, S., Fry, W., Meyers, B.C., van West, P., Ristaino, J., Govers, F., Birch, P.R., Whisson, S.C., Judelson, H.S. and Nusbaum, C. (2009) Genome sequence and analysis of the Irish potato famine pathogen Phytophthora infestans. Nature, 461,393-398.
Huang, S., van der Vossen, E.A.G., Kuang, H., Vieeshouwers, V.G.A.A., Zhang, N., Borm, T.J., van Eck, H.J., Baker, В., Jacobsen, E. and Visser, R.G.F. (2005) Comparative genomics enabled the isolation of the R3a late blight resistance gene in potato. Plant J., 42, 251-261.
Jacobs, M.M., van den Berg, R.G., Vieeshouwers, V.G., Visser, M., Mank, R., Sengers, M., Hoekstra, R. and Vosman, B. (2008) AFLP analysis reveals a lack of phylogenetic structure within Solanum section Petota. BMC evolutionary biology, 8,145.
Jansky, S. (2000) Breeding for disease resistance in potato.
Plant Breeding Rev., 19, 69-155.
Jones, J.D. and Dangl, J.L. (2006) The plant immune system. Nature, 444,323-329.
Joobeur, Т., King, J.J., Nolin, S.J., Thomas, C.E. and Dean, R.A. (2004) The Fusarium wilt resistance locus Fom-2 of melon contains a single resistance gene with complex features. Plant J„ 39,283-297.
Kuhl, J.C., Hanneman, R.E., Jr. and Havey, M.J. (2001) Characterization and mapping of Rpil, a late-blight resistance locus from diploid (1EBN) Mexican Solarium pinnatisectum. Mol. Genet. Genomics, 265, 977-985.
Lokossou, A.A., Park, Т.Н., van Arkel, G., Arens, M., Ruyter-Spira, C., Morales, J., Whisson, S.C., Birch, P.R., Visser, R.G., Jacobsen, E. and van der Vossen, E.A. (2009) Exploiting knowledge of R/Avr genes to rapidly clone a new LZ-NBS-LRR family of late blight resistance genes from potato linkage group IV. Mol. Plant Microbe Interact., 22, 630-641.
Moreau, P., Thoquet, P., Olivier, J., Laterrot, H. and Grimsley, N. (1998) Genetic Mapping of Ph-2, a Single Locus Controlling Partial Resistance to Phytophthora infestans in Tomato. Mol. Plant Microbe Interact., 11,259-269.
Park, Т.Н., Foster, S.J., Brigneti, G. and Jones, J.D.G. (2008) Two distinct potato late blight resistance genes from Solarium berthaultii are located on chromosome 10. Euphytica.
Park, Т.Н., Gros, J., Sikkema, A., Vieeshouwers, V.G.A.A., Muskens, M., Allefs, S., Jacobsen, E., Visser, R.G.F. and van der Vossen, E.A.G. (2005a) The late blight resistance locus Rpi-bib3 from Solanum bulbocastanum belongs to a major late blight R gene cluster on chromosome 4 of potato. Mol. Plant Microbe Interact., 18, 722-729.
Park, Т.Н., Vieeshouwers, V.G.A.A., Huigen, D.J., van der Vossen, E.A.G., van Eck, H.J. and Visser, R.G.F. (2005b) Characterization and high-resolution mapping of a late blight resistance locus similar to R2 in potato. TAG. Theoretical and applied genetics. 111, 591-597.
Pel, M.A., Foster, S.J., Park, Т.Н., Rietman, H., van Arkel, G., Jones, J.D., Van Eck, H.J., Jacobsen, E., Visser, R.G.F. and Van der Vossen, E.A.G. (2009) Mapping and Cloning of Late Blight Resistance Genes from Solanum venturii Using an Interspecific Candidate Gene Approach. Mol. Plant Microbe Interact., 22, 601-615.
Rauscher, G.M., Smart, C.D., Simko, L, Bonierbale, M., Mayton, H., Greenland, A. and Fry, W.E. (2006) Characterization and mapping of RPi-ber, a novel potato late blight resistance gene from Solanum berthaultii. Theor. Appl. Genet., 112, 674-687.
Rouppe van der Voort, J.N.A.M., Kanyuka, K., van der Vossen, E.A.G., Bendahmane, A., Mooijman, P., Klein-Lankhorst, R.M., Stiekema, W.J., Baulcombe, D.C. and Bakker, J. (1999) Tight physical linkage of the nematode resistance gene Gpa2 and the virus resistance gene Rx on a single segment introgressed from the wild species Solanum tuberosum subsp. andigena CPC 1673 into cultivated potato., Mol. Plant Microbe Interact., 12, 197-206.
Song, J., Bradeen, J.M., Naess, S.K., Raasch, J.A., Wielgus, S.M., Haberlach, G.T., Liu, J., Kuang, H., Austin-Phillips, S., Buell, C.R., Helgeson, J.P. and Jiang, J. (2003) Gene RB cloned from Solanum bulbocastanum confers broad spectrum resistance to potato late blight. Proc. Natl. Acad. Sci. USA, 100, 9128-9133.
Tameling, W.I., Vossen, J.H., Albrecht, M., Lengauer, Т., Berden, J.A., Haring, M.A., Cornelissen, B.J. and Takken, F.L. (2006) Mutations in the NB-ARC domain of 1-2 that impair ATP hydrolysis cause autoactivation. Plant Physiol., 140, 1233-1245.
Tan, M.Y., Hutten, R.C., Celis, C., Park, Т.Н., Niks, R.E., Visser, R.G.F. and van Eck, H.J. (2008) The R(Pi-mcd1) locus from Solanum microdontum involved in resistance to Phytophthora infestans, causing a delay in infection, maps on potato chromosome 4 in a cluster of NBS-LRR genes. Mol. Plant Microbe Interact., 21, 909-918.
van der Biezen, E.A. and Jones, J.D. (1998) The NB-ARC domain: a novel signalling motif shared by plant resistance gene products and regulators of cell death in animals. Curr. Biol., 8,R226-227.
van der Linden, C.G., Wouters, D.C., Mihalka, V., Kochieva, E.Z., Smulders, M.J.
and Vosman, B. (2004) Efficient targeting of plant disease resistance loci using NBS profiling. Theor. Appl. Genet., 109, 384-393.
van der Vossen, E.A.G., Sikkema, A., Hekkert, B.L., Gros, J., Stevens, P., Muskens, M., Wouters, D., Pereira, A., Stiekema, W.J. and Allefs, S. (2003) An ancient R gene from the wild potato species Solanum bulbocastanum confers broad-spectrum resistance to Phytophthora infestans in cultivated potato and tomato. Plant J., 36, 867-882.
van Os, H., Andrzejewski, S., Bakker, E., Barrena, I., Bryan, G.J., Caromel, В., Ghareeb, В., Isidore, E., de Jong, W., van Koert, P., Lefebvre, V., Milbourne, D., Ritter, E., van der Voort, J.N.A.M., Rousselle-Bourgeois, F., van VIiet, J., Waugh, R., Visser, R.G.F., Bakker, J. and van Eck, H.J. (2006) Construction of a 10,000-marker ultradense genetic recombination map of potato: providing a framework for accelerated gene isolation and a genomewide physical map. Genetics, 173, 1075-1087.
Vieeshouwers, V.G.A.A., Rietman, H., Krenek, P., Champouret, N., Young, C., Oh, S.K., Wang, M., Bouwmeester, K., Vosman, В., Visser, R.G.F., Jacobsen, E., Govers, F., Kamoun, S. and Van der Vossen, E.A.G. (2008) Effector genomics accelerates discovery and functional profiling of potato disease resistance and phytophthora infestans avirulence genes. PLoS ONE, 3, e2875.
Vieeshouwers, V.G.A.A., van Dooijweert, W., Paul Keizer, L.C., Sijpkes, L., Govers, F. and Colon, L.T. (1999) A laboratory assay for Phytophthora infestans resistance in various Solanum species reflects the field situation. Eur. J. Plant Patbol., 105,241-250.
Wang, M., Allefs, S., van den Berg, R.G., Vieeshouwers, V.G.A.A., van der Vossen, E.A.G. and Vosman, B. (2008) Allele mining in Solanum: conserved homologues of Rpi-blb1 are identified in Solanum stoloniferum. Theor. Appl. Genet., 116, 933-943.
| название | год | авторы | номер документа |
|---|---|---|---|
| ГЕНЫ И СПОСОБЫ ОБЕСПЕЧЕНИЯ УСТОЙЧИВОСТИ К ФИТОФТОРОЗУ ПАСЛЕНОВЫХ | 2008 |
|
RU2511423C2 |
| ГЕН, ПРИДАЮЩИЙ УСТОЙЧИВОСТЬ К PHYTOPHTHORA INFESTANS (ФИТОФТОРОЗУ) В СЕМЕЙСТВЕ SOLANACEAE | 2003 |
|
RU2361920C2 |
| БОЛЕЗНЕУСТОЙЧИВЫЕ РАСТЕНИЯ | 2006 |
|
RU2393227C2 |
| АУТОАКТИВИРУЮЩИЙСЯ БЕЛОК УСТОЙЧИВОСТИ | 2006 |
|
RU2375453C2 |
| УСТОЙЧИВЫЕ К ГРИБКАМ РАСТЕНИЯ И ИХ ИСПОЛЬЗОВАНИЕ | 2004 |
|
RU2377248C2 |
| РОЗА, УСТОЙЧИВАЯ К МУЧНИСТОЙ РОСЕ | 2018 |
|
RU2769284C2 |
| НУКЛЕИНОВЫЕ КИСЛОТЫ И СПОСОБЫ ПОЛУЧЕНИЯ СЕМЯН, ИМЕЮЩИХ В ЗАРОДЫШЕ ПОЛНЫЙ ДИПЛОИДНЫЙ ГЕНОМ, КОМПЛЕМЕНТАРНЫЙ МАТЕРИНСКОМУ | 2006 |
|
RU2438297C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817378C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817374C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2800828C1 |
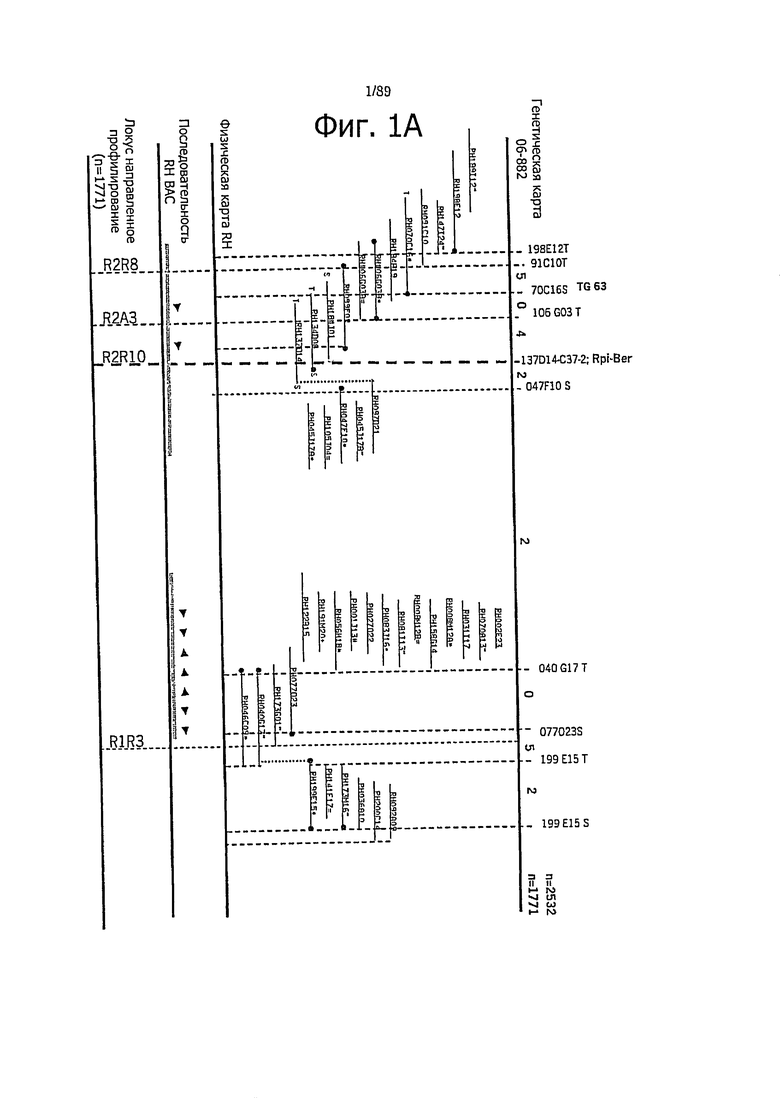
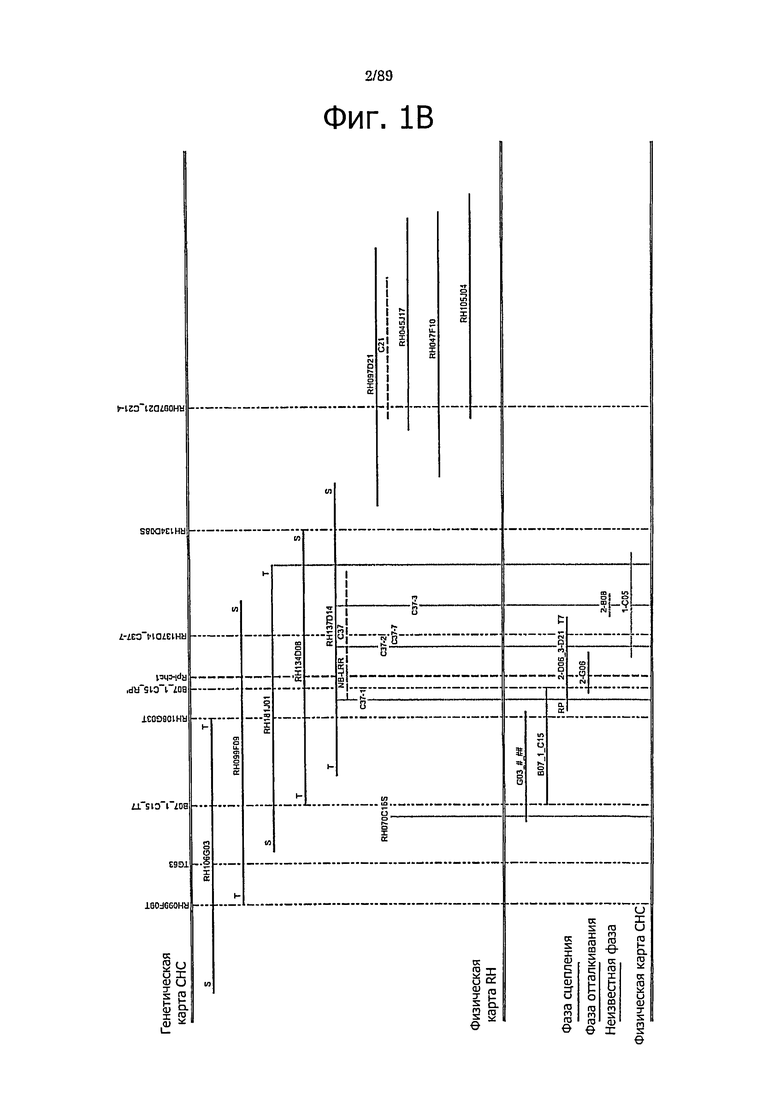

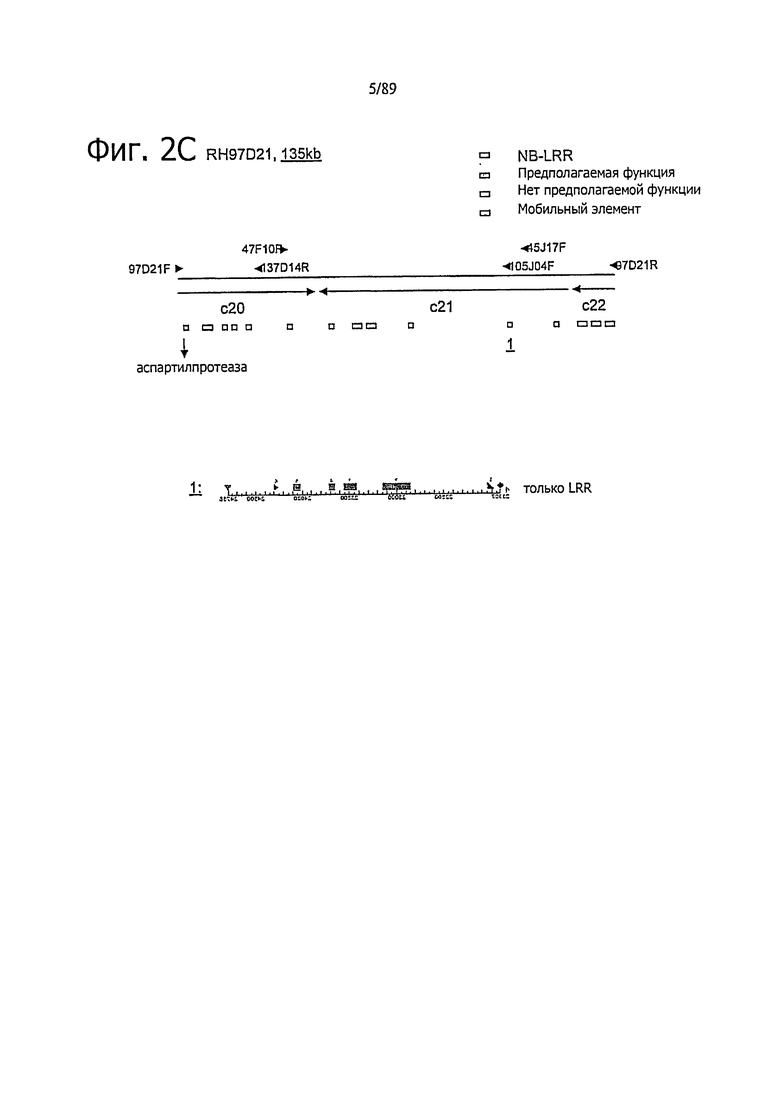

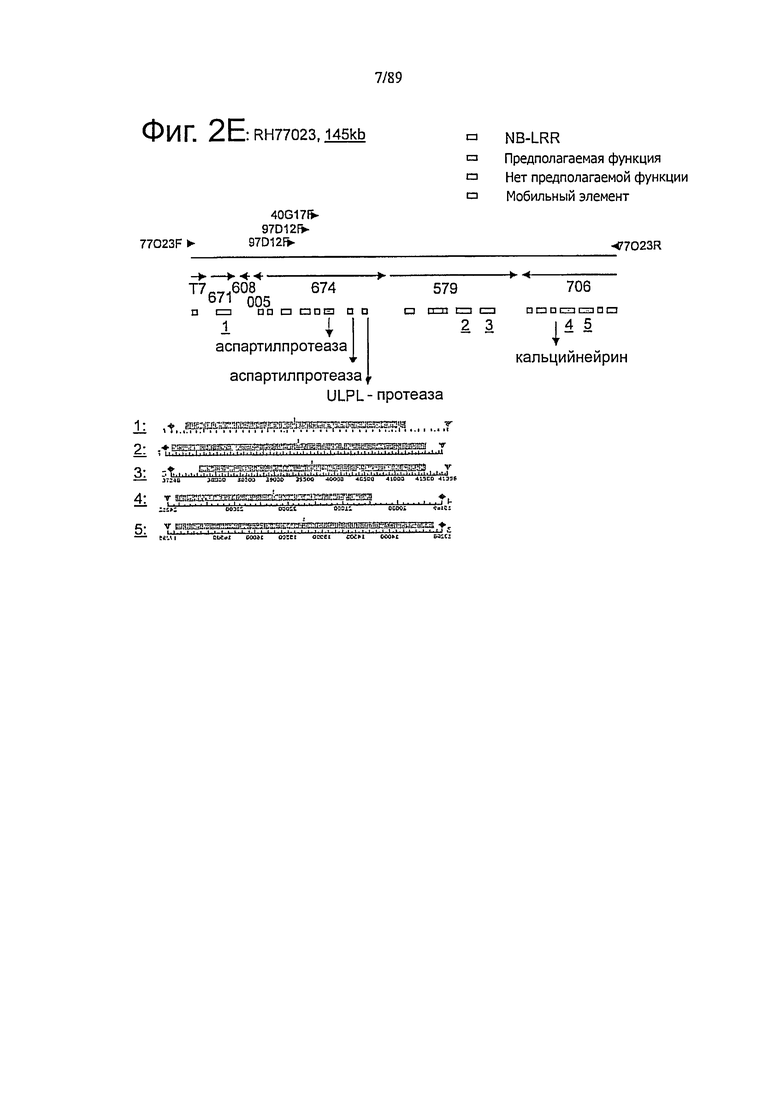


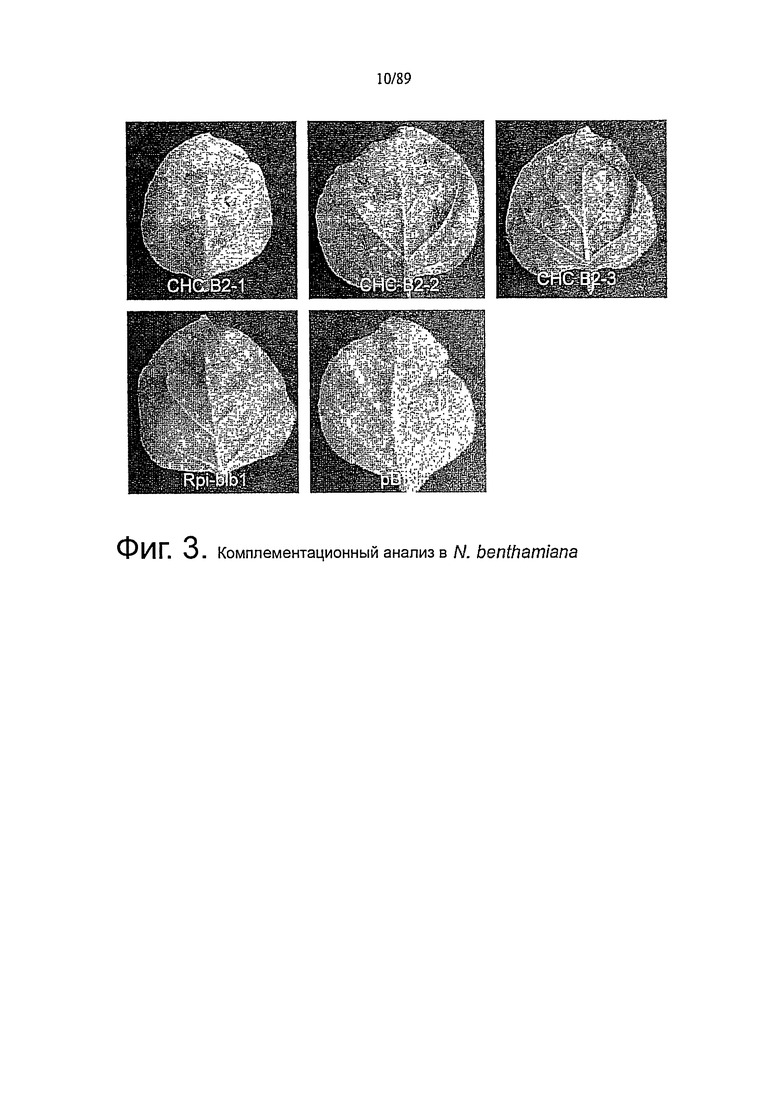
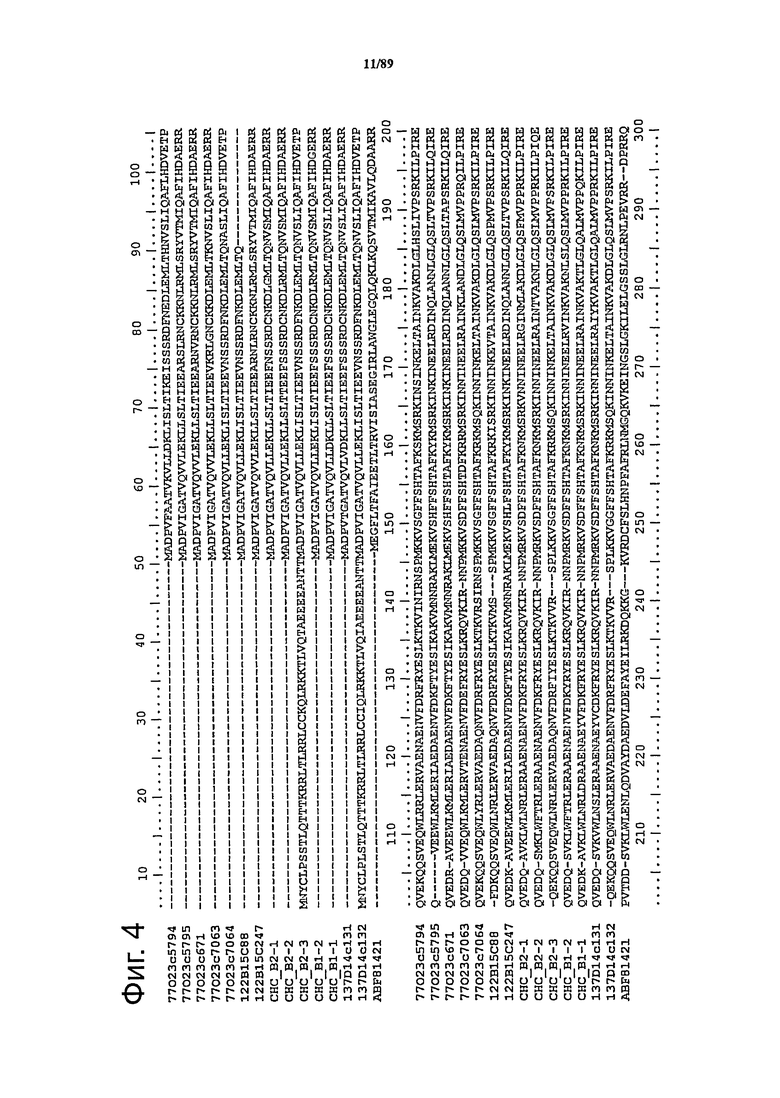










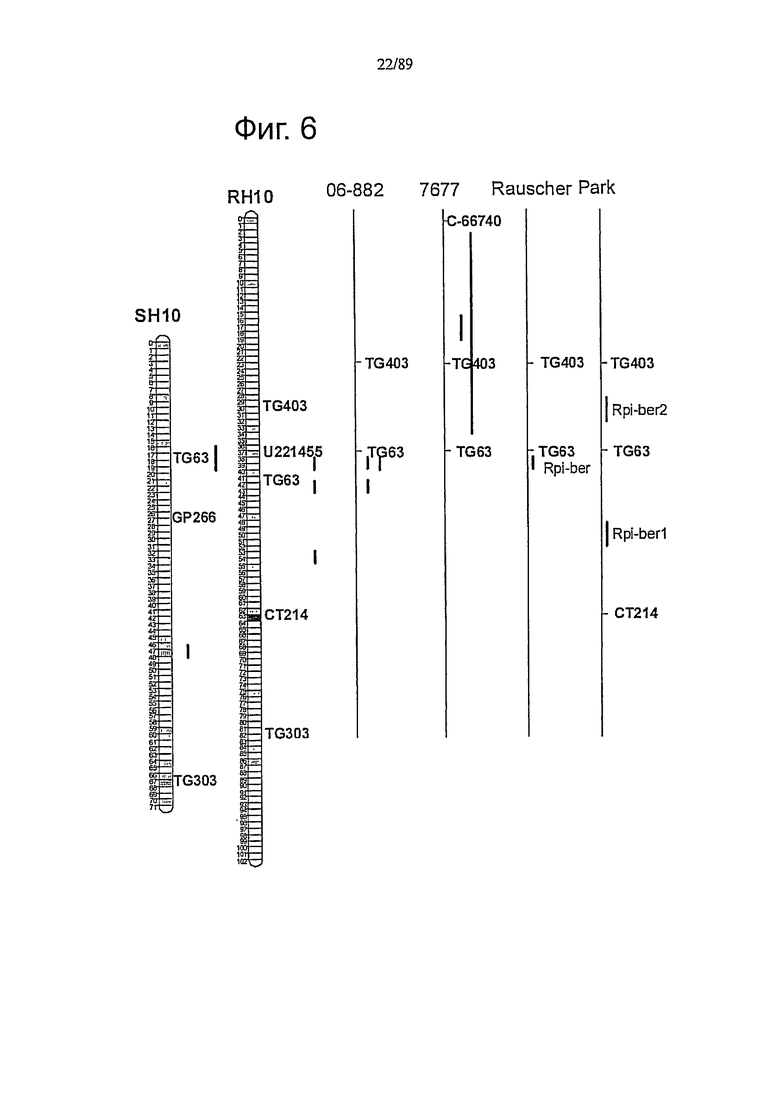


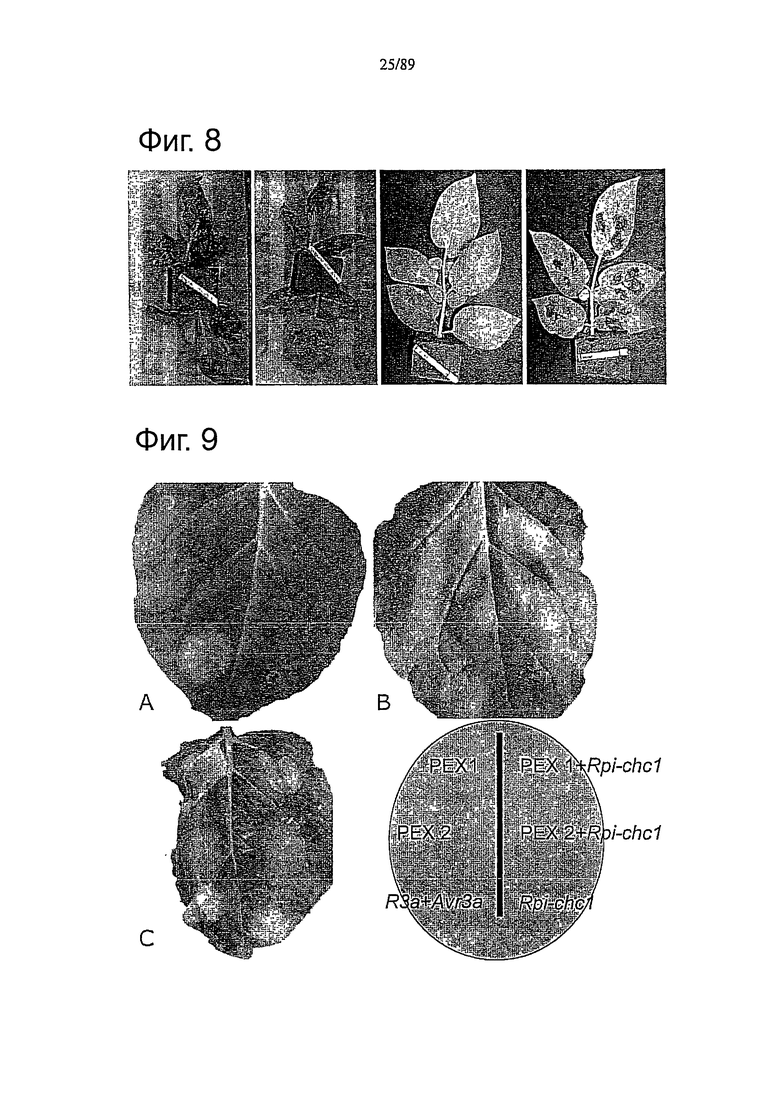
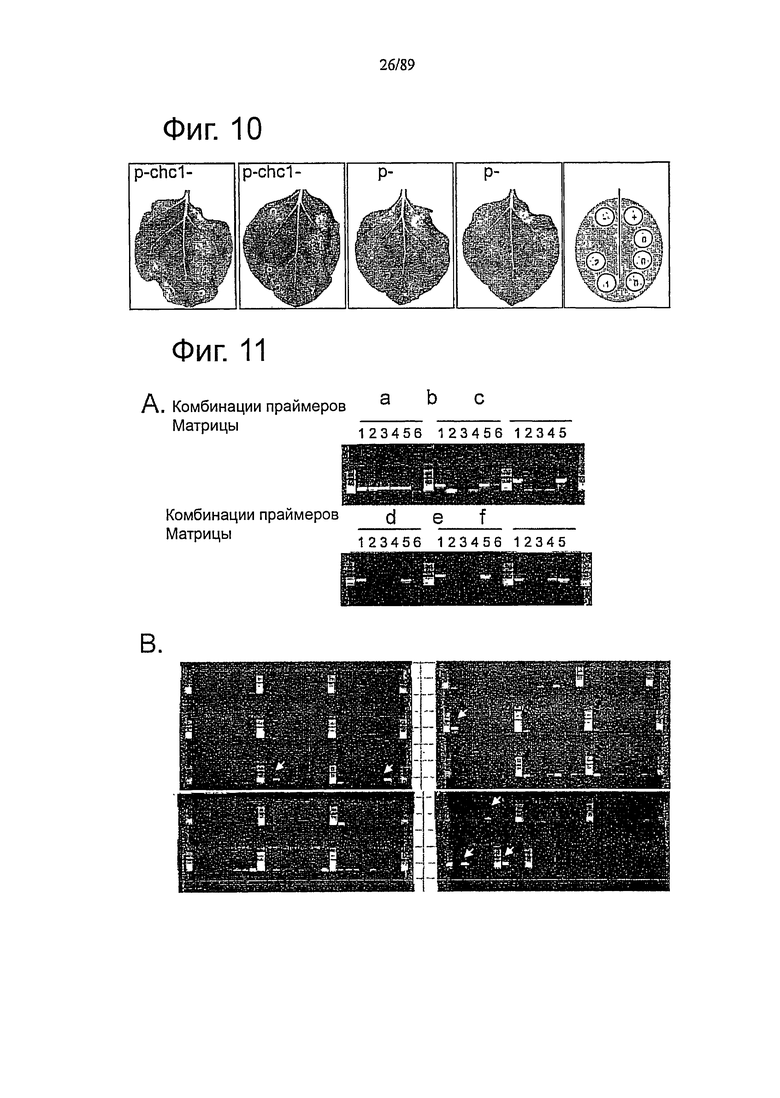
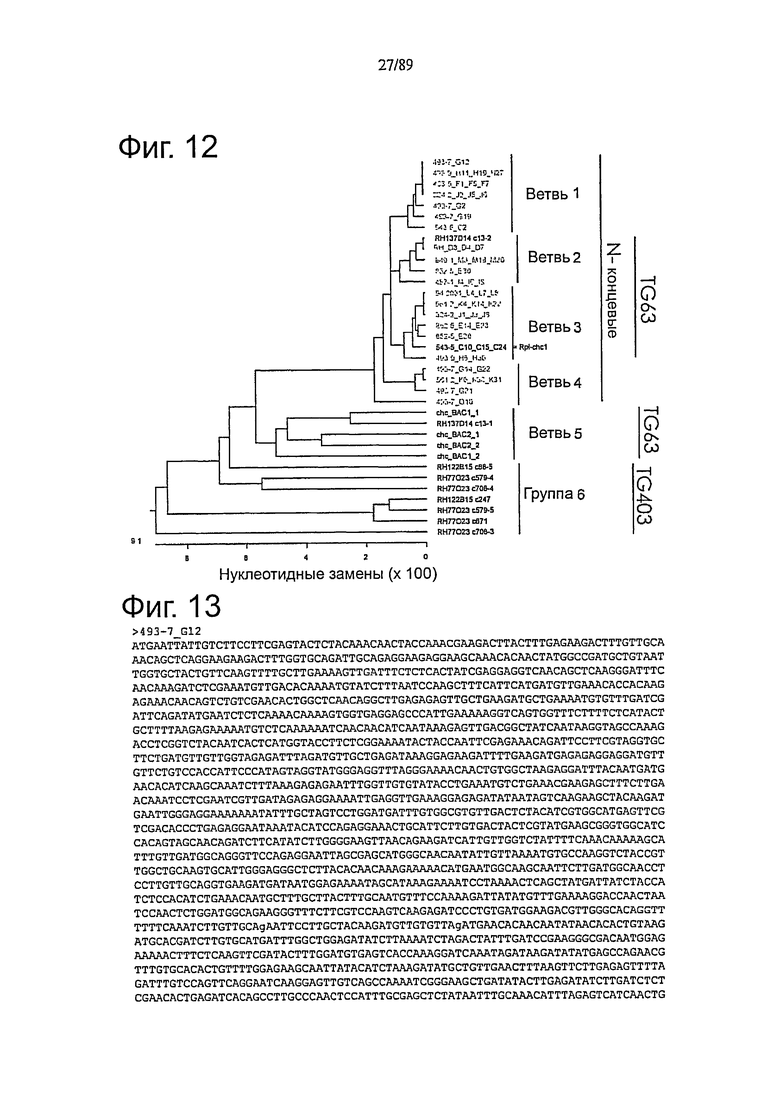




















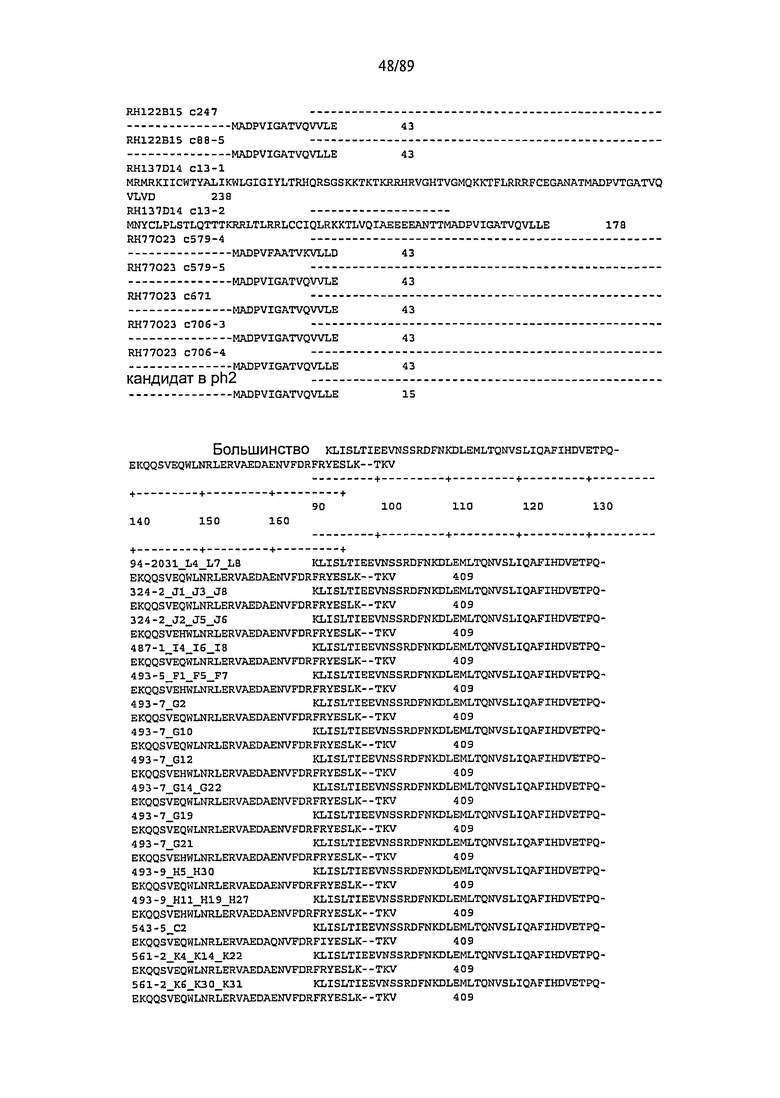









































Изобретение относится к области биохимии, в частности к способу придания, по меньшей мере, частичной устойчивости или повышения устойчивости растения против заражения Phytophthora infestans, к нуклеиновой кислоте, способной обеспечить устойчивость к Phytophthora infestans, а также к экспрессионной конструкции, клетке и вектору, содержащим вышеуказанную нуклеиновую кислоту. Также раскрыт способ получения тетраплоидного растения, представляющего собой растение из семейства Solanaceae, устойчивого к Phytophthora infestans с использованием вышеуказанной нуклеиновой кислоты. Изобретение также относится к растению, устойчивому к Phytophthora infestans и его части. Также раскрыт белок, способный усиливать устойчивость к Phytophthora infestans. Изобретение позволяет эффективно придавать или повышать устойчивость растения против заражения Phytophthora infestans. 11 н. и 6 з.п. ф-лы, 14 ил., 9 табл., 2 пр.
1. Способ придания, по меньшей мере, частичной устойчивости или повышения устойчивости растения против заражения Phytophthora infestans, включающий предоставление растению или его части нуклеиновой кислоты, кодирующей аминокислотную последовательность Rpi-chc1, представленную SEQ ID NO: 110, или ее функциональный фрагмент, где функциональный фрагмент содержит домен LRR и/или N-концевые части белка Rpi-chc1, или функциональный гомолог, который имеет по меньшей мере 80% идентичности с SEQ ID NO: 110, при этом указанное растение представляет собой растение из семейства Solanaceae, предпочтительнее Solanum tuberosum.
2. Способ по п. 1, при этом функциональный гомолог выбирают из группы аминокислотных последовательностей, состоящей из SEQ ID NO: 194, SEQ ID NO: 198, SEQ ID NO: 203, SEQ ID NO: 205, SEQ ID NO: 207, SEQ ID NO: 209, SEQ ID NO: 211, SEQ ID NO: 212, SEQ ID NO: 216, SEQ ID NO: 219, SEQ ID NO: 221, SEQ ID NO: 223.
3. Способ по пп. 1-2, при этом нуклеотидная последовательность, определенная в п. 1, включает нуклеотидную последовательность SEQ ID NO: 183, SEQ ID NO: 185, SEQ ID NO: 187, SEQ ID NO: 189, SEQ ID NO: 191, SEQ ID NO: 193, SEQ ID NO: 195, SEQ ID NO: 197, SEQ ID NO: 199, SEQ ID NO: 202, SEQ ID NO: 204, SEQ ID NO: 206, SEQ ID NO: 208, SEQ ID NO: 210, SEQ ID NO: 212, SEQ ID NO: 214, SEQ ID NO: 216, SEQ ID NO: 218, SEQ ID NO: 220, SEQ ID NO: 222, SEQ ID NO: 224, или нуклеотидную последовательность, кодирующую аминокислотные последовательности SEQ ID NO: 194, SEQ ID NO: SEQ ID NO: 198, SEQ ID NO: 203, SEQ ID NO: 205, SEQ ID NO: 207, SEQ ID NO: 209, SEQ ID NO: 211, SEQ ID NO: 212, SEQ ID NO: 216, SEQ ID NO: 219, SEQ ID NO: 221, SEQ ID NO: 223.
4. Изолированная или рекомбинантная нуклеиновая кислота, способная обеспечить устойчивость к Phytophthora infestans, характеризующаяся нуклеотидной последовательностью, кодирующей аминокислотную последовательность Rpi-chc1, представленную SEQ ID NO: 110, или ее функциональный фрагмент, где функциональный фрагмент содержит домен LRR и/или N-концевые части белка Rpi-chc1, или нуклеотидную последовательность, кодирующую аминокислотную последовательность, выбранную из SEQ ID NO: 194, SEQ ID NO: 198, SEQ ID NO: 203, SEQ ID NO: 205, SEQ ID NO: 207, SEQ ID NO: 209, SEQ ID NO: 211, SEQ ID NO: 212, SEQ ID NO: 216, SEQ ID NO: 219, SEQ ID NO: 221, SEQ ID NO: 223 или ее функциональный фрагмент,
где функциональный фрагмент содержит домен LRR и/или N-концевые части белка Rpi-chc1.
5. Изолированная или рекомбинантная нуклеиновая кислота по п. 4, при этом указанный фрагмент включает, по меньшей мере, домен LRR аминокислотной последовательности.
6. Изолированная или рекомбинантная нуклеиновая кислота по п. 4, включающая нуклеотидную последовательность SEQ ID NO: 125 или SEQ ID NO: 183, SEQ ID NO: 185, SEQ ID NO: 187, SEQ ID NO: 189, SEQ ID NO: 191, SEQ ID NO: 193, SEQ ID NO: 195, SEQ ID NO: 197, SEQ ID NO: 199, SEQ ID NO: 202, SEQ ID NO: 204, SEQ ID NO: 206, SEQ ID NO: 208, SEQ ID NO: 210, SEQ ID NO: 212, SEQ ID NO: 214, SEQ ID NO: 216, SEQ ID NO: 218, SEQ ID NO: 220, SEQ ID NO: 222, SEQ ID NO: 224.
7. Экспрессионная конструкция, содержащая нуклеиновую кислоту по любому из пп. 4-6.
8. Способ получения тетраплоидного растения, представляющего собой растение из семейства Solanaceae, устойчивого к Phytophthora infestans, включающий:
a) удвоение уровня плоидности гамет диплоидного растения, которое трансформировано нуклеотидной последовательностью, определенную в любом из пп. 4-6;
b) использование указанных гамет в скрещивании с гаметами тетраплоидного растения;
c) отбор потомства указанного скрещивания по наличию указанной нуклеотидной последовательности, и
d) получение устойчивого растения.
9. Способ по п. 8, при этом диплоидное растение на стадии а) представляет собой растение из рода S. chacoense, S. berthaultii, S. sucrense или S. tarijense.
10. Трансгенная или тетраплоидная клетка для экспрессии Rpi-chc1, включающая нуклеиновую кислоту по любому из пп. 4, 5 или 6.
11. Экспрессирующий вектор, включающий нуклеиновую кислоту по любому из пп. 4-6.
12. Вектор по п. 11, также включающий промотор и/или терминатор, с которым ген естественно ассоциируется, предпочтительнее укороченный промотор, имеющий менее 1000 нуклеотидов вверх от последовательности гена.
13. Трансгенная или тетраплоидная клетка-хозяин, для экспрессии Rpi-chc1, включающая нуклеиновую кислоту по любому из пп. 4-6 или вектор по п. 11 или 12, предпочтительно клетка Agrobacterium или растительная клетка.
14. Трансгенная или тетраплоидная растительная клетка, для получения растения или для экспрессии Rpi-chc1, включающая нуклеиновую кислоту по любому из пп. 4-6 или вектор по п. 11 или 12, при этом указанная растительная клетка представляет собой клетку из Solanaceae, предпочтительнее Solarium tuberosum, предпочтительнее тетраплоидного Solarium tuberosum.
15. Трансгенное или тетраплоидное растение, устойчивое к Phytophthora infestans, представляющее собой растение из семейства Solanaceae, включающее клетку по п. 14.
16. Часть растения по п. 15, для регенерации растения или для употребления или для культивирования, при этом указанная часть растения содержит нуклеиновую кислоту по пп. 4-6 и предпочтительно при этом указанная часть представляет собой клубень.
17. Белок, способный усиливать устойчивость к Phytophthora infestans, кодированный изолированной или рекомбинантной нуклеиновой кислотой по любому из пп. 4-6, или его функциональный фрагмент, где функциональный фрагмент содержит домен LRR и/или N-концевые части белка Rpi-chc1, предпочтительно при этом указанный белок имеет аминокислотную последовательность Rpi-chc1, представленную SEQ ID NO: 110.
Авторы
Даты
2016-06-10—Публикация
2010-09-20—Подача