Область техники, к которой относится изобретение
Изобретение относится к области биотехнологии и генной инженерии растений, а именно к способу получения двудольного растения при помощи технологии редактирования генома растений CRISPR/Cas9. путем целенаправленного воздействия на нуклеотидную последовательность кодирующей области гена EDR1 картофеля системами редактирования генома CRISPR/Cas9, в которых используются молекулы РНК-проводника или сконструированный на их основе экспрессионный ДНК-вектор.
Предшествующий уровень техники
Картофель (Solarium tuberosum L.) является одной из ведущих сельскохозяйственных культур. Актуальными задачами картофелеводства являются повышение урожайности и переход к возделыванию новых сортов с комплексом технологических свойств, востребованных промышленностью.
Фитофтороз картофеля, вызываемый фитопатогенным оомицетом Phytophthora infestans, является наиболее вредоносным и экономическим значимым заболеванием картофеля [1, 2, 3]. В настоящий момент в мире активно ведется селекционная работа по созданию сортов картофеля, устойчивых к фитофторозу, с использованием доминантных генов вертикальной устойчивости (так называемых R-генов) [4]. Недостатком такого подхода является относительно низкая долговечность и расоспецифичность устойчивости, придаваемой такими генами. Быстрая эволюция P. infestans приводит к изменению структуры микробных эффекторов, узнаваемых растительными рецепторами - продуктами R-генов. Вследствие этого, рецепторы картофеля теряют способность эффективно связывать эффекторы P. infestans и вызывать каскад защитных реакций, запускаемый в растении при их узнавании.
Альтернативой устойчивости, придаваемой доминантными R-генами, является рецессивная устойчивость, придаваемая определенными аллелями так называемых «генов восприимчивости», или S-генов. Этот вид устойчивости в ряде случаев характеризуется повышенной долговечностью и эффективностью [5]. В защите растения от биотрофных и гемибиотрофных патогенов, к которым относится Phytophthora infestans, ключевую роль играет защитный путь, регулируемый гормоном салициловой кислотой (SA). Путь ответа на SA вовлекает в себя большое количество генов, среди которых есть как положительные, так и отрицательные регуляторы защитного ответа. Одним из таких генов является ген EDR1. Было обнаружено, что растения арабидопсиса с инактивированным геном EDR1 являются более устойчивыми к гемибиотрофной бактерии Pseudomnas syringae pv. tomato и биотрофному грибу Erysiphe dehoracearum [6]. При этом, инактивация этого гена не приводит к конститутивной активации SA-опосредованного защитного ответа в растениях, который выражается в различных неблагоприятных фенотипических проявлениях - карликовости, хлорозе, повреждениях на листьях. Белковый продукт гена EDR1 является ферментом киназой, регулирующей каскад МАР-киназ, в частности МРК3, МКК4, МКК5, МРК6, что выражается в подавлении опосредованного салициловой кислотой защитного ответа.
Традиционные методы селекции позволяют получать сорта картофеля, характеризующиеся высокой устойчивостью к болезням и стрессовым факторам. Однако создание сорта с желаемыми признаками является очень длительным процессом, а полезные гены могут быть получены только от совместимых генотипов. Возможность контролировать устойчивость растения картофеля к биотрофным и гемибиотрофным патогенам путем точечного изменения нуклеотидной последовательности гена EDR1 позволит упростить процесс получения новых высокоустойчивых сортов, соответствующих современным требованиям. Однако получение растений, имеющих мутации в кодирующей области гена EDR1 на неизменном генетическом фоне, представляет собой техническую проблему, для решения которой необходимо расширение арсенала средств, доступных селекционерам. В качестве решения этой проблемы настоящее изобретение предлагает способ получения растения картофеля с измененной при помощи технологии редактирования генома растений CRISPR/Cas9 последовательностью кодирующей области гена EDR1.
Сущность изобретения
Задачей изобретения является разработка способа получения растения картофеля с тетрааллельными мутациями в кодирующей области гена EDR1 для дальнейшего использования этого растения в селекционном процессе.
Для решения данной задачи предлагается способ, согласно которому получают конструкцию pKSE401-sgRNA2-2-l, несущую последовательности, которые кодируют нуклеазу Cas9 и молекулу РНК-проводника (sgRNA), переносят эту конструкцию в клетки картофеля с помощью бактерии Agrobacterium tumefaciens для внесения мутаций в кодирующую область гена EDR1 и проводят отбор растений-регенерантов с отредактированным геномом.
В другом аспекте настоящее изобретение также относится к растению картофеля, полученному этим способом.
Использование РНК-проводника, в составе экспрессионного вектора pKSE401 (Addgene Plasmid #62202) в описываемом примере, позволило эффективно внести мутации в целевую последовательность кодирующей области гена EDR1 растения картофеля, не затрагивая другие участки генома, и в дальнейшем закрепить полученные мутации в последующем поколении растений.
Краткое описание чертежей
Фигура 1. Выравнивание нуклеотидных последовательности аллелей дикого типа гена EDR1 картофеля сорта Вектор.
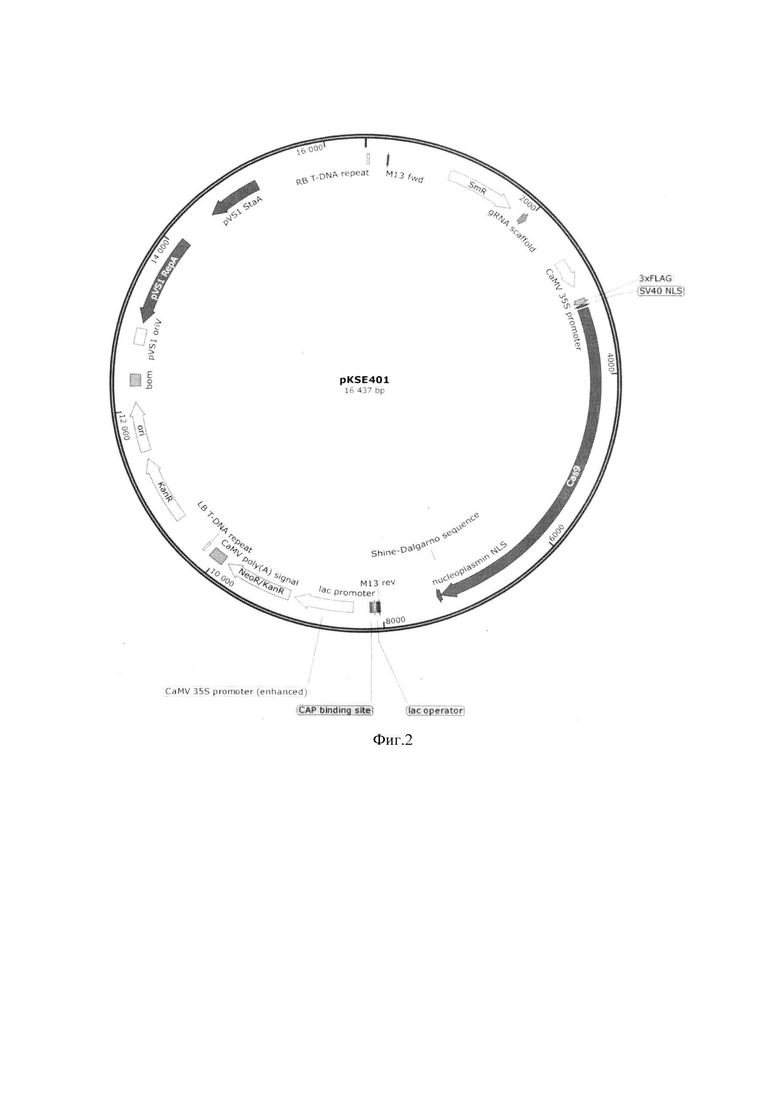
Фигура 2. Карта вектора pKSE401. KanR - ген устойчивости к антибиотику канамицину для селекции в бактериях, ori- ориджин репликации, RB T-DNA repeat и LB T-DNA repeat -левая и правая границы Т-ДНК соответственно, Cas9 - нуклеаза, gRNA scaffold - скаффолд гидовой РНК, NeoR/KanR - ген устойчивости к антибиотику канамицину в растениях.
Фигура 3. Выравнивание нуклеотидных последовательности аллелей гена EDR1 у подвергнутого геномному редактированию растения 3-2-4/10-1/46 картофеля сорта Вектор. В результате таргетного NGS-секвенирования первого экзона гена EDR1 у сорта картофеля Вектор было выявлено три варианта последовательностей.
Фигура 4. События геномного редактирования целевой нуклеотидной последовательности участка гена EDR1 у растения 3-2-4/10-1/46. Показана нуклеотидная последовательность молекулы РНК-проводника (sgRNA2) и нуклеотидные последовательности аллелей, образующихся в результате геномного редактирования.
Пример 1
Получение трансгенного растения культурного картофеля Solanum tuberosum, содержащего тетрааллельные мутации в кодирующей области гена EDR1, путем геномного редактирования с помощью технологии CRISPR/Cas9.
Выбор гена-мишени для геномного редактирования
В качестве гена-мишени для редактирования был выбран ген EDR1, влияющий на проявление хозяйственно значимого признака у картофеля - устойчивости к фитофторозу.
Секвенирование гена EDR1 у растений картофеля сорта Вектор и дизайн молекулы РНК-проводника (sgRNA2)
Культурный картофель является тетраплоидом, что делает секвенирование его генов по методу Сэнгера весьма трудоемким. Гораздо более удобной, хотя и дорогостоящей альтернативой является таргетное NGS-секвенирование, позволяющее получить много прочтений для одного фрагмента и достоверно выявить все аллельное разнообразие.
С точки зрения инактивации гена наиболее перспективным является внесение мутаций в его начало так, чтобы сбой рамки считывания происходил как можно раньше. Поэтому нами были подобраны праймеры для секвенирования фрагмента первого экзона гена EDR1 картофеля. Далее, с использованием образца тотальной ДНК амплифицировали участок первого экзона гена EDR1 с помощью этой пары праймеров:
StEDR-ex1-NGS-F
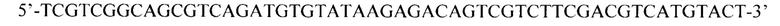
StEDR-ex1-NGS-R
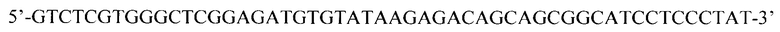
Полученные ПЦР-продукты размером 395 п. н. выделяли из агарозного геля с помощью набора GeneJET Gel Extraction Kit (Thermo Fisher Scientific) и использовали для приготовления библиотек по протоколу 16S Metagenomic Sequencing Library Preparation (Preparing 16S Ribosomal RNA Gene Amplicons for the Illumina MiSeq System). Далее библиотеки секвенировали на приборе Illumina MiSeq.
Для каждого секвенируемого образца длина прочтения, качество и количество ридов в полученных fastq-файлах оценивались с помощью программы FastQC 0.11.9. Первичная обработка ридов осуществлялась с помощью программы Trimmomatic-0.39. После тримминга fastq файлы конвертировались в fasta файлы, которые анализировались в программе Unipro UGENE 33.0. Количество прочтений на образец варьировало от 3798 до 12567.
В результате таргетного NGS-секвенирования первого экзона гена EDR1 у сорта картофеля Вектор было выявлено четыре аллеля данного гена (Фигура 1). Аллели 1, 2 и 3 идентичны между собой имеют нуклеотидную последовательность SEQ ID NO: 1, аллель 4 отличается от них заменами по положениям 103, 156, 242 и 294 и имеет нуклеотидную последовательность SEQ ID NO: 2. Аллели 1-3 по данным положениям можно обозначить, как GATT; аллель 4 - как AGCG.
Анализ доступного генома картофеля (сорт Solyntus, BioProject: PRJNA631911) показал, что ген EDR1 содержится в геноме картофеля только в одном локусе, что косвенно подтверждается тем, что при секвенировании было обнаружено только четыре аллеля.
Для дизайна sgRNA1 использовали пакет приложений «CRISPR RGEN Tools» (http://www.rgenome.net/) [7]. При дизайне sgRNA2 руководствовались следующими критериями:
Во-первых, наличие РАМ последовательности в 5' области относительно последовательности протоспейсера. Для используемой в настоящем изобретении spCas9 РАМ последовательность NGG, где N - любой нуклеотид.
Во-вторых, минимизация возможного нецелевого редактирования - «off-target» эффекта.
В-третьих, отсутствие в вариабельной части sgRNA2 вторичных структур, наличие которых может снижать эффективность редактирования.
Таким образом, была подобрана последовательность РНК-проводника SEQ ID NO: 3 для инактивации гена EDR1 картофеля сорта Вектор на консервативные участки первого экзона этого гена.
Создание генетических конструкций для инактивации гена StEDR1
Конструкции для инактивации гена EDR1 получали на основе вектора pKSE401 [8] (Фигура 2).
Последовательность SEQ ID NO: 3 подобранной нами молекулы sgRNA2 клонировали в вектор pKSE401.
Клонирование осуществляли по технологии Golden Gate. Для каждого из двух вариантов конструкций был установлен клон, секвенирование которого продемонстрировало отсутствие мутаций в последовательностях кассет экспрессии sgRNA2.
Готовым генетическим вектором трансформировали и агробактерии штамма AGLO. Далее эти бактерии использовали для получения трансгенных растений картофеля сорта Вектор.
Агробактериалъная трансформация растений картофеля
Предварительный подбор условий позволил эффективно провести агробактериальную трансформацию растений картофеля сорта Вектор. 100 листовых эксплантов ко-культивировали с агробактерией и далее культивировали на среде Р-2. На 45-й день культивирования (с момента ко-культивации) у 60-70% эксплантов отметили формирования каллуса по краям эксплантов при использовании каждой генетической конструкции. Через 120 дней культивирования наблюдали формирование морфогенного каллуса, регенерирующего побеги, у 52-60% эксплантов при использовании каждой генетической конструкции.
В результате генетической трансформации отобрали растение 3-2-4/10-1/46, демонстрирующее устойчивость к селективному гербициду.
Наличие инсерционных/делеционных мутаций в целевой кодирующей области гена EDR1 подтвердили путем таргетного высокопроизводительного секвенирования. Для этого соответствующий фрагмент гена EDR1 амплифицировали с помощью вышеуказанных праймеров StEDR-ex1-NGS-F и StEDR-ex1-NGS-R.
Полученные ПЦР-продукты очищали набором GeneJET PCR Purification Kit (Thermo) и использовали для приготовления библиотек по протоколу 16S Metagenomic Sequencing Library Preparation (Preparing 16S Ribosomal RNA Gene Amplicons for the Illumina MiSeq System). Далее библиотеки секвенировали на приборе Illumina MiSeq, как указано выше.
Генотиприрование нуклеотидной последовательности гена EDR1 отобранного растения 3-2-4/10-1/46 показало делецию 2 нуклеотидов в аллеле 1 (SEQ ID NO: 4 в перечне последовательностей), делецию 3 нуклеотидов в аллелях 2 и 3 (SEQ ID NO: 5 в перечне последовательностей) и делецию 5 нуклеотидов в аллеле 4 (SEQ ID NO: 6 в перечне последовательностей) в сравнении с не модифицированной последовательностью EDR1 (Фигура 3). Согласно генотипированию, растение 3-2-4/10-1/46 является гетерозиготным тетрааллельным. Эти мутации возникли в результате функциональной активности РНК-проводника sgRNA2 SEQ ID NO: 3, поскольку изменения обнаружены в области ожидаемого сайта расщепления молекулы ДНК комплексом Cas9-PHK-проводник (Фигура 4). Таким образом, получено растение картофеля, несущее тетра-аллели кодирующей области гена EDR1 с нуклеотидными последовательностями SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6 в перечне последовательностей, из которых все являются мутантными.
Финансирование работ по созданию настоящего изобретения проводилось из средств Соглашения №075-15-2019-1667 от «31» октября 2019 г. о предоставлении из федерального бюджета грантов в форме субсидий в соответствии с пунктом 4 статьи 78.1 Бюджетного кодекса Российской Федерации на осуществление государственной поддержки создания и развития центра геномных исследований мирового уровня «Курчатовский геномный центр» в рамках реализации федерального проекта «Развитие научной и научно-производственной кооперации» национального проекта «Наука».
Список литературы
1. Akino S., Takemoto D., Hosaka K. Phytophthora infestans: a review of past and current studies on potato late blight // Journal of general plant pathology. - 2014. - T. 80. - №. 1. - C. 24-37.
2. Irshad G., Naz M. F. A. F. Important fungal diseases of potato and their management-a brief review // Mycopath. - 2014. - Т. 11. - №. 1.
3. Bain R.A., Moller K. Factors influencing potato tuber infection by Phytophthora infestans //Schepers, H. und E. Bouma. - 1999. - C. 210-227.
4. Aguilera-Galvez C. et al. Two different R gene loci co-evolved with Avr2 of Phytophthora infestans and confer distinct resistance specificities in potato // Studies in mycology. - 2018. - T. 89. - C. 105-115.
5. Pavan S. et al. Loss of susceptibility as a novel breeding strategy for durable and broad-spectrum resistance // Molecular Breeding. - 2010. - T. 25. - №. 1. - C. 1-12.
6. Frye C. A., Innes R. W. An Arabidopsis mutant with enhanced resistance to powdery mildew // The Plant Cell. - 1998. - T. 10. -№. 6. - C. 947-956.
7. Hwang G. H., Kim J. S., Bae S. Web-based CRISPR toolkits: Cas-OFFinder, Cas-Designer, and Cas-Analyzer // CRISPR Guide RNA Design. - Humana, New York, NY, 2021. - C. 23-33.
8. Xing H. L. et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants // BMC plant biology. -2014. - T. 14. - №. 1. - C. 1-12.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Перечень
последовательностей 46.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2022-08-23">
<ApplicantFileReference>46</ApplicantFileReference>
<ApplicantName languageCode="ru">ФГБНУ ВНИИСБ</ApplicantName>
<ApplicantNameLatin>All-Russia Research Institute of Agricultural
Biotechnology</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ
КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1
ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ
CRISPR/CAS9</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>328</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..328</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Solanum tuberosum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcttcgacgtcatgtactaccgatcatcggaattccagttcggttc
ttcaatcaccgtcgagtcctcccacgatttcaaatgcgtcaacggcgaccactgctgtttcggtgagtgc
gggtggtggaggagggaatatttcgacgatcaatcgacagcaggactattatacttcggaggaagagtat
caggtgcaacttgccctagcgttgagtgtgtcatcctcgcaatctcaagctctgtctgatgttaattcaa
gcaacggtcagatattagggagagggcgaagcgctgttgacttggcacgtgatagggaggatgccgctg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>328</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..328</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Solanum tuberosum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcttcgacgtcatgtactaccgatcatcggaattccagttcggttc
ttcaatcaccgtcgagtcctcccacgatttcaaatgcgtcaacggcgaccactactgtttcggtgagtgc
gggtggtggaggagggaatatttcgacgatcaatcggcagcaggactattatacttcggaggaagagtat
caggtgcaacttgccctagcgttgagtgtgtcatcctcgcaatctcaagctccgtctgatgttaattcaa
gcaacggtcagatattagggagagggcgaagcgcggttgacttggcacgtgatagggaggatgccgctg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caagcaacggtcagatatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>326</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..326</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Solanum tuberosum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcttcgacgtcatgtactaccgatcatcggaattccagttcggttc
ttcaatcaccgtcgagtcctcccacgatttcaaatgcgtcaacggcgaccactgctgtttcggtgagtgc
gggtggtggaggagggaatatttcgacgatcaatcgacagcaggactattatacttcggaggaagagtat
caggtgcaacttgccctagcgttgagtgtgtcatcctcgcaatctcaagctctgtctgatgttaattcaa
gcaacggtcatattagggagagggcgaagcgctgttgacttggcacgtgatagggaggatgccgctg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>325</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..325</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Solanum tuberosum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcttcgacgtcatgtactaccgatcatcggaattccagttcggttc
ttcaatcaccgtcgagtcctcccacgatttcaaatgcgtcaacggcgaccactgctgtttcggtgagtgc
gggtggtggaggagggaatatttcgacgatcaatcgacagcaggactattatacttcggaggaagagtat
caggtgcaacttgccctagcgttgagtgtgtcatcctcgcaatctcaagctctgtctgatgttaattcaa
gcaacggtcaattagggagagggcgaagcgctgttgacttggcacgtgatagggaggatgccgctg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>323</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..323</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Solanum tuberosum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcttcgacgtcatgtactaccgatcatcggaattccagttcggttc
ttcaatcaccgtcgagtcctcccacgatttcaaatgcgtcaacggcgaccactactgtttcggtgagtgc
gggtggtggaggagggaatatttcgacgatcaatcggcagcaggactattatacttcggaggaagagtat
caggtgcaacttgccctagcgttgagtgtgtcatcctcgcaatctcaagctccgtctgatgttaattcaa
gcaacggtattagggagagggcgaagcgcggttgacttggcacgtgatagggaggatgccgctg</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817384C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817376C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817374C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817379C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТЕТРААЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817383C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С ТРИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2817378C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2824558C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ КАРТОФЕЛЯ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В КОДИРУЮЩЕЙ ОБЛАСТИ ГЕНА EDR1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2022 |
|
RU2814154C1 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С БИАЛЛЕЛЬНЫМИ МУТАЦИЯМИ В ПРОТОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772575C2 |
| СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЯ ПШЕНИЦЫ С НУКЛЕОТИДНОЙ ДЕЛЕЦИЕЙ В ПРОМОТОРНОЙ ОБЛАСТИ ГЕНА VRN-A1 ПРИ ПОМОЩИ ТЕХНОЛОГИИ РЕДАКТИРОВАНИЯ ГЕНОМА РАСТЕНИЙ CRISPR/CAS9 | 2020 |
|
RU2772577C2 |



Изобретение относится к области биотехнологии, а именно к способу получения растения картофеля с тетрааллельными мутациями в кодирующей области гена EDR1 при помощи технологии редактирования генома растений CRISPR/Cas9. Способ предусматривает получение конструкции pKSE401-sgRNA2-2-1, содержащей нуклеотидную последовательность РНК-проводника, перенесение этой конструкции в клетки картофеля с помощью бактерии Agrobacterium tumefaciens для внесения мутаций в кодирующую область гена EDR1 и отбор растений-регенерантов с тетрааллельными мутациями в кодирующей области гена EDR1. Изобретение эффективно для получения растения картофеля. 2 н.п. ф-лы, 4 ил., 1 пр.
1. Способ получения растения картофеля с тетрааллельными мутациями в кодирующей области гена EDR1, включающий в себя следующие этапы:
получение конструкции на основе вектора pKSE401, содержащей нуклеотидную последовательность РНК-проводника SEQ ID NO: 3,
перенесение этой конструкции в клетки картофеля с помощью бактерии Agrobacterium tumefaciens для внесения мутаций в кодирующую область гена EDR1 и
отбор растений-регенерантов с тетрааллельными мутациями в кодирующей области гена EDR1, где растения имеют аллели кодирующей области гена EDR1, имеющие нуклеотидные последовательности SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6.
2. Растение картофеля с тетрааллельными мутациями в кодирующей области гена EDR1 для дальнейшего применения в селекционном процессе, полученное способом по п. 1, где растение имеет отличающиеся аллели кодирующей области гена EDR1, имеющие нуклеотидные последовательности SEQ ID NO: 4, SEQ ID NO: 5 и SEQ ID NO: 6.
| WO2013016477 A1, 31.01.2013 | |||
| CN109609358 А1, 12.04.2019 | |||
| СПОСОБ СЕЛЕКЦИИ СПЕРМАТОЗОИДОВ ДЛЯ ЭКСТРАКОРПОРАЛЬНОГО ОПЛОДОТВОРЕНИЯ | 2013 |
|
RU2535359C1 |
| BALDINI D | |||
| et al | |||
| Sperm Selection for ICSI: Do We Have a Winner? Cells, 2021, vol.10 | |||
| LEUNG E.T | |||
| et al | |||
| Simulating nature in sperm selection for assisted reproduction, Nature reviews urology, 2022, vol.19. | |||
Авторы
Даты
2023-07-28—Публикация
2022-09-07—Подача