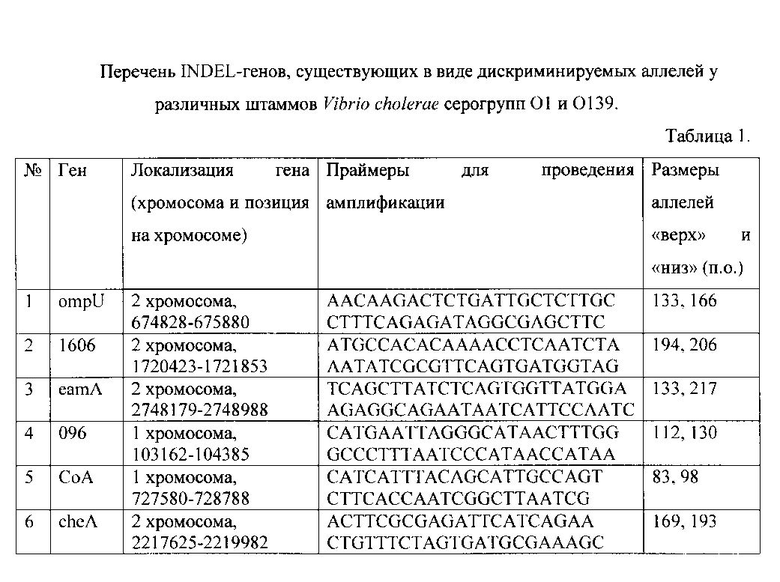
Изобретение относится к области медицинской микробиологии, а именно к лабораторной диагностике, и может быть использовано при молекулярно-генетическом внутривидовом типировании штаммов Vibrio cholerae O1 и O139 серогрупп.
Среди множества лабораторных тестов, позволяющих определить происхождение штаммов возбудителя холеры, только методики молекулярного типирования позволяют определить уникальные генетические характеристики штаммов холерных вибрионов, что позволяет не только характеризовать циркулирующие штаммы возбудителя, но и устанавливать причину заболевания и идентифицировать источники заноса на основании полного совпадения генетических маркеров.
Наибольшей информативностью обладает метод анализа вариабельных тандемных повторов - VNTR-анализ [1, 2], позволяющий определять уникальную аллельную формулу для каждой культуры и тем самым четко дифференцировать штаммы самых различных возбудителей.
Сущность VNTR-анализа заключается в том, что проводят амплификацию ДНК исследуемого штамма с набором специфических праймеров к индивидуальному VNRT-локусу. Полученные специфические фрагменты исследуют различными методами с целью точного определения молекулярной массы амплифицированного фрагмента ДНК. В зависимости от установленной массы каждого фрагмента вычисляют число тандемных повторов для исследуемого локуса. Подобное исследование проводят для нескольких локусов. Сочетание чисел, характеризующих число повторов каждого из изученных локусов, составляет уникальную аллельную VNTR-формулу для анализируемого штамма холерного вибриона.
Однако данный способ имеет ряд существенных недостатков. Индивидуальные амплифицированные VNTR-фрагменты обладают большой молекулярной массой, и соседние аллели отличаются весьма незначительно: обычно всего шесть нуклеотидов, что делает весьма затруднительным определение их истинного веса с точностью до нуклеотида. Для точного определения размера аллелей необходимы сложные и трудоемкие методики определения молекулярной массы амплифицированного фрагмента.
В одном случае это может быть электрофорез в геле полиакриламда или агарозы с набором маркеров известного молекулярного веса [3]. Известно, что электрофоретическая подвижность молекул нуклеиновых кислот обратно пропорциональна логарифму молекулярной массы. Поэтому, точно измерив после электрофореза в геле длину пробега исследуемого фрагмента и стандартных фрагментов известной массы, можно точно определить молекулярную массу ДНК [4].
Известны исследования, которые показали наличие в генах различных живых организмов особых стабильно наследуемых генетических характеристик, заключающихся во вставках-делециях (insertions/deletions) коротких фрагментов ДНК, при этом такие гены обозначают как INDEL-гены [5]. Изучение полиморфизма INDEL-генов в настоящее время широко используется при молекулярном типировании [6].
За прототип выбран способ VNTR-анализа [7], который заключается в выделении ДНК из исследуемой культуры путем теплового лизиса, постановки ПЦР со специфическими праймерами, меченными флюоресцентными метками к VNTR-локусам, и учете полученных результатов, последний проводят путем электрофореза продуктов амплификации в секвенаторе ДНК ABI Prism 310 в присутствии флюоресцентно меченных маркеров MapMaker LOW (lot 030600; BioVentures, Inc).
Однако данный способ имеет ряд недостатков. Прежде всего для его осуществления необходим набор праймеров, меченных флюоресцентными метками. Это резко усложняет и удорожает процедуру синтеза праймеров. Для проведения исследования необходим сложный и дорогостоящий импортный ДНК секвенатор, а само проведение анализа требует использования дорогостоящих расходных материалов, которые поставляются производителем.
Кроме того, проведение реакции требует использования импортных дорогостоящих флюоресцентно меченных маркеров. Эти обстоятельства приводит к крайне высокой стоимости анализа и полной зависимости исследователя от поставок производителем расходных материалов из-за рубежа.
Технической задачей предлагаемого изобретения является разработка нового невысокой себестоимости способа, позволяющего определить идентичность штаммов возбудителя холеры O1 и O139 серогрупп, выделенных в различных регионах, областях и странах путем поиска в геноме различных штаммов холерных вибрионов генов, имеющих у различных штаммов четко дискриминируемые INDEL-аллели.
Поставленная задача достигается тем, что в известном способе молекулярно-генетического внутривидового типирования V. cholerae O1 и O139 серогрупп, включающем выделение ДНК из исследуемого штамма V. cholerae серогруппы O1 или O139, постановку ПЦР со специфическими праймерами и учет реакции путем электрофореза, амплификацию исследуемой ДНК проводят сконструированными специфическими праймерами к каждому из шести INDEL-генов, имеющих по две дискриминируемых аллели, а именно:
с праймерами к гену ompU AACAAGACTCTGATTGCTCTTGC,
CTTTCAGAGATAGGCGAGCTTC,
с длиной амплифицированного фрагмента 166 п. о. или 133 п.о.;
с праймерами к гену 1606 ATGCCACACAAAACCTCAATCTA,
AATATCGCGTTCAGTGATGGTAG,
с длиной амплифицированного фрагмента 206 п.о. или 194 п.о.;
с праймерами к гену eamA TCAGCTTATCTCAGTGGTTATGGA,
AGAGGCAGAATAATCATTCCAATC,
с длиной амплифицированного фрагмента 217 п.о. или 133 п.о.;
с праймерами к гену 096 CATGAATTAGGGCATAACTTTGG,
GCCCTTTAATCCCATAACCATAA,
с длиной амплифицированного фрагмента 130 п.о. или 112 п.о.;
с праймерами к гену СоА CATCATTTACAGCATTGCCAGT,
CTTCACCAATCGGCTTAATCG,
с длиной амплифицированного фрагмента 98 п.о. или 83 п.о.;
с праймерами к гену cheA ACTTCGCGAGATTCATCAGAA,
CTGTTTCTAGTGATGCGAAAGC,
с длиной амплифицированного фрагмента 193 п.о. или 169 п.о.,
при этом учет результатов типирования проводят как визуальную идентификацию после электрофореза, причем для каждого штамма устанавливают уникальный INDEL-генотип по двум или большему числу INDEL-генов, а путем сравнения выявленных INDEL-генотипов устанавливают общее или различное происхождение исследуемых штаммов V. cholerae O1 и O139 серогрупп.
Способ осуществляется следующим образом.
Перед постановкой способа молекулярно-генетического типирования выделяют ДНК исследуемой культуры. Для этого массу бактерий, сформировавших колонию или газон, стандартной бактериологической петлей (диаметром 2 мм) вносят в микропробирку Эппендорф объемом 1,5 мл, содержащую 200 мкл дистиллированной воды, после чего выделяют ДНК, как принято (Методические указания МУ 1.3.1791-9 «Организация работы при исследовании методом ПЦР материала, инфицированного микроорганизмами 1 и 2 группы патогенности. - М., 2003-38 с.).
Следующим этапом способа является проведение ПЦР.
В процессе компьютерного анализа нуклеотидных последовательностей Vibrio cholerae авторы идентифицировали ряд INDEL-генов, отличающихся по длине у штаммов холерного вибриона различного происхождения (см. таблицу 1). К варьирующим участкам указанных генов ompU, hutB, 1606, eamA, his, CoA были сконструированы специфические праймеры.
Анализ коллекции штаммов холерного вибриона Ростовского противочумного института различного происхождения и степени вирулентности подтвердил существование полиморфизма длин указанных генов. Обнаружено существование всего двух четко дискриминируемых по электрофоретической подвижности аллелей (см. таблицу 1).
Далее проводят амплификацию выделенной ДНК с сконструированными специфическими праймерами к генам ompU, 1606, eamA, 096, СоА и cheA.
Условия проведения реакции амлификации.
Амплификацию проводят по следующей схеме: денатурация при 95°C - 3 мин (1 цикл); затем 35 циклов: денатурация при 95°C - 20 с, отжиг при 63°C - 20 с, синтез при 72°C - 20 с; синтез при 72°C - 7 мин (1 цикл).
Инкубационную смесь объемом 25 мкл готовят из расчета: 1,5 мМ Mg-буфер, 0,2 мМ смеси дНТФ, 1,0 мкМ смеси праймеров (по 0,5 мкМ каждого праймера) 25 нг ДНК-матрицы, 0,25 ед. ДНК-полимеразы, оставшийся объем - вода. В качестве матрицы используют геномную ДНК (объемом 5 мкл), полученную из разных штаммов вибрионов.
Учет результатов амплификации проводят с помощью электрофореза в 2% агарозном или 10% полиакриламидном геле. Полученный типичный результат амплификации по шести локусам представлен на рисунке.
На рисунке показан эталонный маркер локусов отражающий результат электрофореза в 8% геле полиакриламида продуктов амплификации ДНК различных штаммов V. cholerae со специфическими праймерами к INDEL-генам. 1A - распределение аллелей по гену che, лунки 1, 3 аллель 193 п.о. обозначена как «верх»; 2, 4 аллель 169 п.о. обозначена как «низ». 1B - распределение аллелей по гену 1606 (1, 2 верхняя аллель 206 п.о., 3, 4 - нижняя аллель 194 п.о. 1C - распределение аллелей по гену 096 (1, 2 «верх» 130 п. о., 3, 4 - «низ» 112 п.о.), 1D - распределение аллелей по гену СоА (1, 3 аллель «низ» 83 п.о., 2 - аллель «верх» 98 п.о.). 1Е - распределение аллелей по гену ompU (1 - «низ» 133 п.о., 2 и 3 «верх» 166 п.о.). и 1F - распределение аллелей по гену eamA (1, 3 «верх» 217 п.о., 2 - «низ» 133 п.о.).
Следовательно, на основании электрофореза продуктов амплификации в геле полиакриламида заявителем получен эталонный маркер локусов, сравнивая с которым проводят визуальную идентификацию аллелей INDEL-генов как «низ», или «верх» у изучаемых штаммов V. cholerae O1 и O139 серогрупп.
Пример 1, подтверждающий внутривидовую дифференциацию штаммов Vibrio cholerae серовара O1, выделенных из объектов внешней среды, при анализе INDEL-генов
Предварительно выделяют ДНК исследуемых штаммов (МУ 1.3.1791-9) и далее проводят ПЦР с видоспецифическими олигонуклеотидными праймерами к генам ompU и cheA и проводят амплификацию по вышеуказанной схеме. Результат электрофоретического разделения фрагментов ДНК в 10% геле полиакриламида представлен в таблице 2.
Из таблицы 2 видно, что, сравнивая с эталонным маркером 4 штаммов, выделенных в 2013 году, по двум локусам ompU и cheA, обнаружено три различных INDEL-генотипа. Так, штаммы 65 и 28 имеют идентичный генотип "ompU верх и cheA верх»; штамм 27 «ompU низ и cheA» верх, а штамм 43 «ompU верх и cheA низ». Выявленные различия в INDEL-генотипах позволяют заключить, что штаммы 65 и 28 имеют общее происхождение, а штаммы 27 и 43 отличаются по своему происхождению.
Вывод. Для молекулярного типирования штаммов холерного вибриона, выделенных в РФ в 2013 году из объектов внешней среды, достаточно проведения амплификации всего по двум локусам ompU и cheA, которые визуально идентифицируют для каждого штамма.
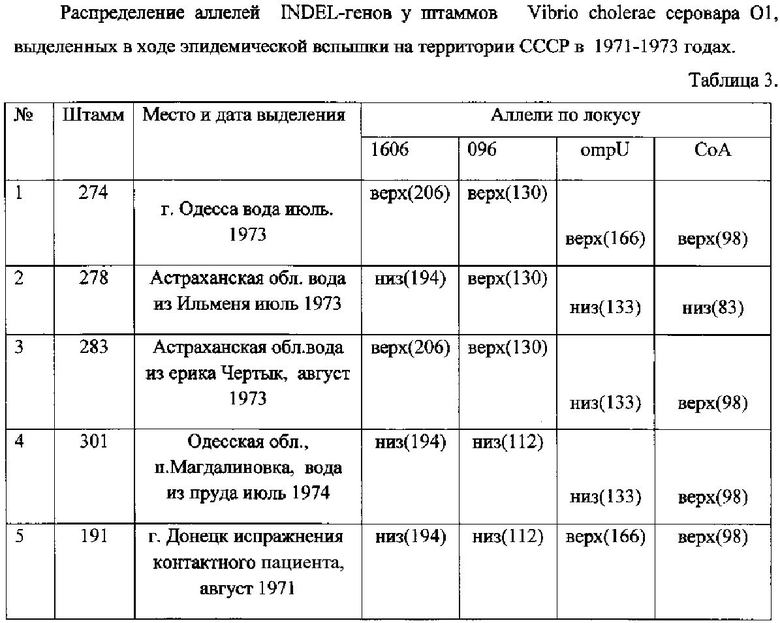
Пример 2, подтверждающий внутривидовую дифференциацию штаммов Vibrio cholerae O1, выделенных в ходе эпидемических проявлений холеры при анализе INDEL-генов
Исследуют штаммы холерных вибрионов, выделенных в ходе эпидемической вспышки на территории СССР в 1973-1974 годах. Предварительно выделяют ДНК исследуемых штаммов, как описано выше, и далее проводят ПЦР по вышеуказанной схеме с видоспецифическими олигонуклеотидными праймерами к генам 1606, 096, ompU и СоА амплификацию. Результат электрофоретического разделения фрагментов ДНК в 10% геле полиакриламида представлен в таблице 3.
Сравнительный анализ с эталонным маркером пяти штаммов, выделенных в ходе эпидемической вспышки в 1973-1974 годах, по четырем локусам 1606,096, ompU и СоА показал, что каждый из изученных штаммов обладает уникальным INDEL-генотипом, что свидетельствует о различном происхождении каждого изученного штамма. На основании полученных результатов можно заключить, что в 1971-1974 годах на территории СССР имела место циркуляция штаммов холерного вибриона, имевших происхождение из разных источников.
Таким образом, для молекулярно-генетического типирования штаммов холерного вибриона, выделенных на территории СССР в ходе вспышки холеры в 1971-1973 годах, достаточно изучения всего четырех INDEL-генов.
Пример 3, подтверждающий видовую дифференциацию штаммов Vibrio cholerae серовара O139, выделенных от человека и из объектов внешней среды, при анализе INDEL-генов
Предварительно выделяют ДНК исследуемых штаммов (МУ 1.3.1791-9) и далее проводят ПЦР с видоспецифическими олигонуклеотидными праймерами к генам eamA и cheA и проводят амплификацию по вышеуказанной схеме. Результат электрофоретического разделения фрагментов ДНК в 10% геле полиакриламида представлен в таблице 4.
Из данных таблицы 4 делаем заключение, что при сравнительном анализе INDEL-генов eamA cheA у четырех Vibrio cholerae серовара O139, выделенных от человека и из объектов внешней среды, обнаружено, что токсигенные штаммы, выделенные от человека, по распределению аллелей INDEL-генов отличаются от нетоксигенных штаммов, выделенных из объектов окружающей среды. Так, нетоксигенные штаммы серовара O139 имеют генотип «eamА верх и cheA верх», а токсигенные «eamА низ и cheA низ». Следовательно, штаммы Vibrio cholerae серовара O139, выделенные от человека, имеют общее происхождение и отличаются от штаммы Vibrio cholerae серовара O139, выделенных из объектов внешней среды. Таким образом, на основании анализа всего двух INDEL-генов eamA и cheA можно проводить типирование штаммов Vibrio cholerae серовара O139.
Использование предлагаемого метода INDEL-типирования позволяет на основании изучения INDEL-генов ompU, 1606, eamA, 096, СоА и cheA выявлять уникальные генетические маркеры штаммов Vibrio cholerae сероваров O1 и O139.
Новое тестирование и невысокая себестоимость дает возможность применения предлагаемого способа в практике работы специализированных учреждений, занимающихся мониторингом холеры.
Кроме того, на основании полученной информации об INDEL-генотипе можно создавать базы данных о штаммах холерного вибриона, циркулирующих в различных регионах мира, сравнивая данные с эталонным маркером осуществлять молекулярное типирование.
Источники информации
1. Belkum van, A., S. Sherer, W. van Leeuwen Variable number of tandem repeats in clinical strains of Haemophilus influenzae // Infect. Immun. - 1997. - N. 65. - P. 5017-5027.
2. Jackson, P.J., E.A. Walthers, A.S. Kalif, K.L. Richmond et al. Characterization of the variable number of tandem repeats in vvrA from different Bacillus anthracis isolates. // Appl. Environ. Microbiol.. - 1997. - Vol. 63, N 4. - P. 1400-1405.
3. Водопьянов C.O., Гончаров E.K., Дуванова O.B., Мишанькин М.Б., Сучков И.Ю., Олейников И.П., Водопьянов А.С., Шишияну М.В., Щербакова Е.В., Корниенко И.В., Мишанькин Б.Н..Вариабельный тандемный повтор VcA Vibrio cholerae. // Молекулярная биология, 2002, том 36, №6, С. 1074-1079.
4. Методы генетической инженерии. Молекулярное клонирование: Пер с англ. / Маниатис Т., Фрич Э., Сэмбрук Дж. - М.: Мир, 1984. - 480 с., ил. (157-185.)
5. Larsson P., Svensson K., Karlsson L., Guala D., Granberg M., Forsman M., and Johansson A. Canonical insertion-deletion markers for rapid DNA typing of Francisella tularensis.// Emerging Infectious Diseases 2007. Vol. 13, No. 11, P. 1725-1732.
6. Natsoulis G., Zhang N., Welch K., Bell J., and Hanlee P.J. Identification of insertion deletion mutations from deep targeted resequencing.// J Data Mining Genomics Proteomics.; 4(3). doi: 10.4172/2153-0602.1000132.
7. Danin-Poleg, Y., Cohen, H. Gancz, Y.Y. Broza, H. Goldshmidt, E. Malul, L. Valinsky, L. Lerner, M. Broza, Y. Kashi Vibrio cholerae strain typing and phylogeny study based on simple sequence repeats // J. Clin. Microbiol. - 2007. - Vol. 45. - P. 736-746.

Изобретение относится к области биохимии. Заявлен способ молекулярно-генетического внутривидового типирования Vibrio cholerae O1 и O139 серогрупп. Способ включает выделение ДНК из исследуемого штамма V. cholerae серогруппы O1 или O139, постановку ПЦР со специфическими праймерами и учет реакции путем электрофореза. Амплификацию исследуемой ДНК проводят сконструированными специфическими праймерами к каждому из шести INDEL-генов, а именно к генам ompU, 1606, eamA, 096, CoA и cheA, каждый из которых имеет по две дискриминируемых аллели. Учет результатов типирования проводят как визуальную идентификацию после электрофореза, причем для каждого исследуемого штамма устанавливают уникальный INDEL-генотип по двум или большему числу INDEL-генов. Путем сравнения выявленных INDEL-генотипов устанавливают общее или различное происхождение исследуемых штаммов V.cholerae O1 и O139 серогрупп. Изобретение позволяет выявлять уникальные генетические маркеры штаммов V.cholerae серогрупп O1 и O139, что может быть использовано в практике работы специализированных учреждений, занимающихся мониторингом за холерой. 1 ил., 4 табл., 3 пр.
Способ молекулярно-генетического внутривидового типирования V. cholerae O1 и O139 серогрупп, включающий выделение ДНК из исследуемого штамма V. cholerae серогруппы O1 или O139, постановку ПЦР со специфическими праймерами и учет реакции путем электрофореза, отличающийся тем, что амплификацию исследуемой ДНК проводят сконструированными специфическими праймерами к каждому из шести INDEL-генов, имеющих по две дискриминируемых аллели, а именно:
с праймерами к гену ompU AACAAGACTCTGATTGCTCTTGC,
CTTTCAGAGATAGGCGAGCTTC,
с длиной амплифицированного фрагмента 166 п.о. или 133 п.о.;
с праймерами к гену 1606 ATGCCACACAAAACCTCAATCTA,
AATATCGCGTTCAGTGATGGTAG,
с длиной амплифицированного фрагмента 206 п.о. или 194 п.о.;
с праймерами к гену eamA TCAGCTTATCTCAGTGGTTATGGA,
AGAGGCAGAATAATCATTCCAATC,
с длиной амплифицированного фрагмента 217 п.о. или 133 п.о.;
с праймерами к гену 096 CATGAATTAGGGCATAACTTTGG,
GCCCTTTAATCCCATAACCATAA,
с длиной амплифицированного фрагмента 130 п.о. или 112 п.о.;
с праймерами к гену СоА CATCATTTACAGCATTGCCAGT,
CTTCACCAATCGGCTTAATCG,
с длиной амплифицированного фрагмента 98 п.о. или 83 п.о.;
с праймерами к гену cheA ACTTCGCGAGATTCATCAGAA,
CTGTTTCTAGTGATGCGAAAGC,
с длиной амплифицированного фрагмента 193 п.о. или 169 п.о.,
при этом учет результатов типирования проводят как визуальную идентификацию после электрофореза, причем для каждого штамма устанавливают уникальный INDEL-генотип по двум или большему числу INDEL-генов, а путем сравнения выявленных INDEL-генотипов устанавливают общее или различное происхождение исследуемых штаммов V. cholerae O1 и O139 серогрупп.
| YEAL DANIN-POLEG et al., Vibrio cholerae strain typing and phylogeny study based on simple sequence repeats, Journal of Clinical Microbiology, 2007, vol.45 no.3, pp.736-746 | |||
| I | |||
| G | |||
| RIVERA et al., Enterobacterial Repetitive Intergenic Consensus Sequences and the PCR To Generate Fingerprints of Genomic DNAs from Vibrio cholerae O1, O139, and Non-O1 |
Авторы
Даты
2016-02-10—Публикация
2014-05-06—Подача