Предлагаемое изобретение относится к области медицинской микробиологии, а именно к лабораторной диагностике, и может быть использовано при молекулярно-генетическом внутривидовом типировании штаммов Klebsiella pneumoniae, циркулирующих в объектах на различных территориях, с целью их дифференциации.
В настоящее время K. pneumoniae является частым и очень распространенным обитателем кишечника млекопитающих. Ее можно легко выделить с поверхностей слизистых оболочек человека, загрязненных фекалиями рук, в сточных водах и с поверхностей в отделениях, [2] что может указывать на существование нескольких изолированных популяций возбудителя. Клебсиеллы могут выдерживать пересыхание в естественных условиях, при этом сохраняя свою жизнеспособность, а также формировать биопленки на поверхностях медицинского оборудования. Данные свойства способствуют развитию нозокомиальных инфекций [3,4].
K. pneumoniae легко колонизирует слизистые оболочки человека, включая желудочно-кишечный тракт и верхние отделы дыхательных путей, при этом себя никак не проявляя. После колонизации слизистых она легко диссиминирует в различные органы и ткани, вызывая пневмонию и другие тяжелые инфекции человека (абсцесс почек и печени, остеомиелит, тромбоз кавернозного синуса, абсцесс яремной луковицы, менингит, абсцесс головного мозга, спонтанный бактериальный перитонит и абсцессы мягких тканей шеи и рук) [1].
Известные молекулярные методы типирования изолятов K. pneumoniae, используемые для эпидемиологического расследования, включают случайно амплифицированную полиморфную ДНК [5], гель-электрофорез в импульсном поле [6] и полиморфизм длин амплифицированных фрагментов [7, 8]. Однако эти методы в основном используются для исследования вспышек на локальном уровне. Это обусловлено низкой воспроизводимостью при постановке в различных лабораториях. Риботипирование является более воспроизводимым методом, особенно в автоматизированном формате [9], однако имеет практические и теоретические ограничения и требует наличия сложного оборудования [10].
Известен способ молекулярного типирования Klebsiella pneumoniae путем анализа сиквенсов ряда генов домашнего хозяйства (MLST) [11]. Данный способ высокоинформативен, однако требует точного секвенирования набора изучаемых генов, что обуславливает необходимость наличия дорогостоящего оборудования и реагентов, часто импортных. Поэтому данный прием недоступен для многих лабораторияй.
За прототип выбран способ генетической дифференциации штаммов Yersinia pseudotuberculosis путем молекулярно-генетического типирования) [12], характеризующийся вставками-делециями (insertions/deletions) коротких фрагментов ДНК, которые обозначают как INDEL-маркеры.
Использование ПЦР со специфическими праймерами, фланкирующими INDEL-локус, позволяет получать воспроизводимые результаты и проводить дифференцировку между «вставкой» и «делецией» оценивая размер синтезированного ампликона.
Простота и высокая воспроизводимость метода обусловила его применение для типирования холерных вибрионов [13], иерсиний, бруцелл [14], но не применялась для исследований Klebsiella pneumoniae.
Технической задачей предполагаемого изобретения является необходимость разработки нового способа, позволяющего достоверно, быстро и с невысокой стоимостью при минимальном использовании дорогостоящих импортных приборов и реагентов осуществлять типирование штаммов Klebsiella pneumoniae выделенных в различных регионах от человека и из объектов внешней среды.
Поставленная задача достигается тем, что в способе молекулярно-генетического типирования штаммов Klebsiella pneumoniae с использованием INDEL-маркеров, включающем выделение ДНК из исследуемого штамма, постановку ПЦР со специфическими праймерами и учет реакции после электрофореза, отличие заключается в том, что с ДНК исследуемого штамма амплификацию проводят с набором 11 праймеров к INDEL-маркерам Klebsiella pneumoniae, имеющих два альтернативных аллеля:
с праймерами к гену MsyB
Forward: ATCGGTCATGGCCTGGTG
Reverse: TTTTCACCAATTTCCGCGGT
с длиной фрагмента амплификации 152 п.о. или 176п.о.
с праймерами к гену АrаС
Forward: GAGCCTTGAACAACTGCCC
Reverse: GCAGATCCATCGCCAGGC
с длиной фрагмента амплификации 107п.о. или 125 п. о
с праймерами к гену TraD
Forward: GACTACGGCGACCTTAACGA
Reverse: GCGTCAGACAATGCATCCAG
с длиной фрагмента амплификации 86п.о. или 101п.о.
с праймерами к гену pol-deac
Forward: ACAACTCGACGCCAAACTAC
Reverse: CACGCCATGGAACGGATTAC
с длиной фрагмента амплификации 77п.о. или 86п.о.
с праймерами к гену Galactose
Forward: GGTTTTCAGCTGGTGGAAGG
Reverse: CGCAATATCCGCTTCCACG
с длиной фрагмента амплификации 96п.о. или 102п.о.
с праймерами к гену ОmрС
Forward: CGTAACGTCCTGGACCGAC
Reverse: GGACTGCAGGAAGTTGTCAG
с длиной фрагмента амплификации 73 п. о. или 79 п. о.
с праймерами к гену 8455
Forward: GAACGCTTAACCGCCATCC
Reverse: CTCACCGCTTTCACGCTG
с длиной фрагмента амплификации 98 п. о. или 122 п. о.
с праймерами к гену 23015
Forward: AAGGTGAGGTCGATCTGGC
Reverse: CGCAATATCCGCTTCCACG
с длиной фрагмента амплификации 80 п. о. или 86 п. о.
с праймерами к гену 23215
Forward: AAGGTGAGGTCGATCTGGC
Reverse: CACGCCATGGAACGGATTAC
с длиной фрагмента амплификации 77 п. о. или 86 п. о.
с праймерами к гену 12905
Forwa rd: GGAGCCTGATTTCTTGCCG
Reverse: AGAGTACGCTGAAGTCACGC
с длиной фрагмента амплификации 89 п. о. или 97 п. о.
с праймерами к гену 13875
Forward: TGGCAACCATGATGAGTACCT
Reverse: CTTTCTGTACCTGCGTCGTG
с длиной фрагмента амплификации 90 п. о. или 99 п. о.
при этом учет результатов амплификации проводят с помощью электрофореза в 10% полиакраламидном геле с последующим детектированием размера каждого INDEL-локуса, а затем по полученным размерам аллелей выстраивают с помощью алгоритма UPGMA дендограмму, по которой осуществляют типирование штаммов Klebsiella pneumoniae, определяя общее или различное происхождение исследуемых штаммов и возможных путей их распространения
Кроме того ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 EдTaq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, 0,5 мкл 50Х SYBRGreenI, 1,0 мкМ каждого из праймеров, 5 мкл ДНК - ДНК матрицы, оставшийся объем - вода.
При этом амплификацию проводят с соблюдением следующих этапов и режимов:
1) 95°С этап денатурации - 3 мин (1 цикл),
2) 95°С этап денатурации - 20 сек (35 циклов),
3) отжиг при 60°С - 20 с,
4) синтез при 72°С - 20 с,
5) синтез при 72°С - 1 мин (1 цикл)
Обоснование выбора праймеров.
Важнейшим этапом при разработке ПЦР, обеспечивающим получение корректного результата, является правильный подбор мишеней для посадки праймеров. Основной проблемой при подборе праймеров для выявления INDEL-локусов Klebsiella pneumoniae явилась их унификация, все реакции должны успешно проходить при одинаковых условиях амплификации и формировать только два специфических ампликона для каждого локуса в интервале 78-180 нуклеотидных пар, что позволяет упростить проведение учета предлагаемого способа. Были проанализированы полные геномы 35 штаммов Klebsiella pneumoniae находящиеся в общедоступных базах данных. Все праймеры были сконструированы авторами с помощью комплекса программного обеспечения, разработанного во ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора (г. Ростов-на-Дону).
Объектом защиты настоящего изобретения является набор 11 праймеров к INDEL-локусам Klebsiella pneumoniae (см. таблицу 1), с последующим проведением ПЦР реакции и учета результатов путем построения дендрограммы для молекулярного типирования Klebsiellap neumoniae

Способ осуществляется следующим образом.
Перед постановкой ПЦР проводят предварительную подготовку материала (выделение ДНК). Массу бактерий, сформировавших газон на плотной питательной среде, стерильной палочкой перемещают в пробирку, содержащую 3-5 мл физиологического раствора. Доводят плотность суспензии до 109 клеток на мл среды по оптической плотности после чего выделяют ДНК для постановки ПЦР [7] с помощью любого коммерческого набора для выделения ДНК или прогревания в течении 30 минут при 99°С.
С полученной таким образом ДНК проводят реакцию амплификации с набором 11 праймеров к INDEL-локусам Klebsiellapneumoniae.
Условия проведения реакции ПЦР:
Готовят 11 пробирок для реакционных смесей, маркируют согласно анализируемому INDEL-локусу. Инкубационная смесь объемом 25 мкл содержит: 2,5 мкл буфера, 1 EдTaq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, (все компоненты производства ЗАО Евроген, Москва), 1,0 мкМ каждого из И пар праймеров, 5 мкл ДНК - ДНК матрицы, оставшийся объем - вода.
После внесения всех компонентов амплификацию проводят в амплификаторе «Терцик» (производства «ДНК-Технология Москва) по следующей схеме: денатурация при 95°С - 3 мин (1 цикл);
затем 35 циклов: денатурация при 95°С - 20 с, отжиг при 60°С - 20 с, синтез при 72°С -20 с; синтез при 72°С - 1 мин (1 цикл).
Учет результатов амплификации проводят с помощью электрофореза в 2% агарозном или 10% полиакриламидном геле. На основании электрофореза продуктов амплификации в геле полиакриламида легко детектируется размер каждого INDEL-локуса (Таблица 1).
Далее полученные размеры аллелей INDEL-локусов используют для построения итоговой дендрограммы. По расположению штаммов на дендрограмме судят о INDEL-генотипах и их взаимосвязи.
Детектированные размеры каждого INDEL-локуса согласно данным Таблицы 1 используют для построения дендрограммы по алгоритму UPGMA (методом парных групп со средним арифметическим и иерархической кластеризации) [15].
Сущность изобретения поясняется следующими примерами.
Пример 1.
Анализировали 21 штамм Klebsiella pneumoniae из коллекции живых культур ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора.
Шесть штаммов были выделены из воды поверхностного водоема Ростовской области (1W, 2W, 3W, 4W, 5W, 6W) и 15 штаммов (72138, 71505, 71514, 71704, 71320, 71374, 72244, 72375, 71381, 70622, 70966, 43592, 44712, 44716, 454730 выделены в 2021 году из различных лечебных учреждений Ростовской области.
Перед постановкой ПЦР проводят выделение ДНК путем прогревания суспензии до 109 клеток в течении 30 минут при 99°С. С полученной таким образом ДНК проводят реакцию амплификации с набором 11 праймеров к INDEL-локусам Klebsiella pneumoniae. Далее с помощью электрофореза в 10% полиакриламидном геле для изучаемого штамма детектировали размер каждого из 11 анализируемых INDEL-локусов согласно данным Таблицы 1.
Итоговая дендрограмма на основании анализа 21 штамма Klebsiella pneumoniae представлена на Фиг. 1.
Фиг. 1. отражает INDEL-генотипы 21 штамма Klebsiella pneumoniae. Шесть «водных» штаммов из воды поверхностного водоема Ростовской области (обозначены как 1W-6W) и 15 «клинических» штаммов из различных лечебных учреждений Ростовской области (каждый штамм имеет пятизначный номер). Штаммы с идентичным INDEL-генотипом на графике отмечены вертикальными линиями. Например, штаммы 5W b 4W объеденены вертикальной линией и имеют идентичный INDEL-генотип. Такой же результат отмечают на штаммах: 44712 и 71704; 70622 и 72244; 71374 и 71514.
В ходе анализа 21 штаммов Klebsiella pneumoniae выявлено 14 INDEL-генотипов (Фиг. 1). При этом 6 «водных» штаммов сформировали 4 INDEL-генотипа, а 15 «клинических» - 10 INDEL-генотипов. Для удобства восприятия локализация 6 «водных» INDEL-генотипов отмечена литерой W. Характер распределения INDEL-генотипов 21 штамма Klebsiella pneumoniae на дендрограмме свидетельствует о раздельном происхождении «водных» и «клинических» штаммов, циркулирующих на территории Ростовской области. Таким образом, предложенный способ позволяет четко определять происхождение выделенных культур Klebsiella pneumoniae и таким образом выявлять штаммы «клинического» происхождения.
Пример 2.
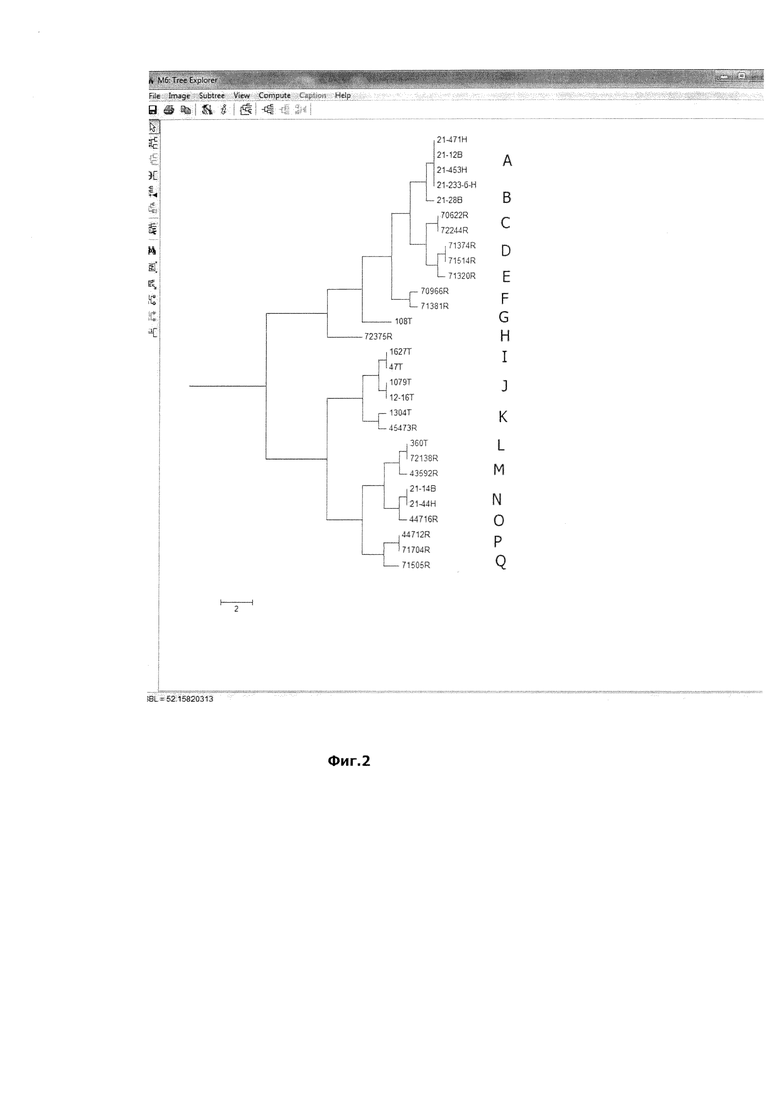
Анализировали 29 штаммов Klebsiella pneumoniae из коллекции живых культур ФКУЗ Ростовский-на-Дону противочумный институт Роспотребнадзора выделенных в 2021 году различных лечебных учреждений Ростовской области-15 культур 72138R, 71505R, 71514R, 71704R, 71320R, 71374R, 72244R, 72375R, 71381R, 70622R, 70966R, 43592R, 44712R, 44716R, 45473R - (обозначены в конце пятизначного номера литерой «R»), Тюмени 7 культур -108Т,47Т, 12-16Т, 1079Т, 360Т, 1304Т, 1627Т (литера «Т»), Хабаровска 4 культуры 21-233(б)Н, 21-453Н, 21-44Н, 21-471Н (литера «Н») и Благовещенска 3 культуры 21-28В, 21-12В, 21-14В (литера «В»).
Условия постановки реакции и учета результатов, как в Примере 1.
Полученный результат представлен на Фигуре 2. В результате анализа 29 «клинических» штаммов выявлено их распределение в 17 INDEL-генотипов, обозначенных на Фиг. 2 для удобства восприятия литерами A-Q. Полученный результат свидетельствовал о высокой дискриминирующей способности предложенного метода типирования штаммов Klebsiella pneumoniae с использованием INDEL-маркеров.
Установлена четкая географическая привязанность INDEL-генотипа. Так, генотипы C, D, Е, F, Н, М, P, Q сформировали только штаммы, изолированные из лечебных учреждений Ростовской области. В Тюмени циркулировали штаммы INDEL-генотипов G, I, J. Часть генотипов сформировали штаммы циркулирующие в нескольких регионах одновременно. Например, генотипы «А» и «N» включали штаммы из Хабаровска и Благовещенска, генотипы«и» и «К» содержали штаммы из Тюмени и Ростова.
Таким образом, предлагаемый метод может быть использован для определения происхождения «клинических» штаммов и установки возможных путей их распространения на основании анализа картины распределения INDEL-генотипов Klebsiella pneumoniae.
Источники информации
1. Choby J.Е., Howard-Anderson J., Weiss D.S. HypervirulentKlebsiellapneumoniae-clinical and molecular perspectives // Journal of internal medicine. - 2020. - T. 287. - №. 3. - C. 283-300.
2. Чеботарь И.В. и др. Почему Klebsiellapneumoniae становится лидирующим оппортунистическим патогеном // Клиническая микробиология и антимикробная химиотерапия. - 2020. - Т. 22. - №. 1. - С. 4-19.
3. Wang G. et al. The characteristic of virulence, biofilm and antibiotic resistance of Klebsiellapneumoniae // International Journal of Environmental Research and Public Health. - 2020. - T. 17. - №. 17. - C. 6278.
4. Paczosa M. K., Mecsas J. Klebsiella pneumoniae: going on the offense with a strong defense //Microbiology and Molecular Biology Reviews. - 2016. - T. 80. - №. 3. -C. 629-661.
5. Brisse, S., and J. Verhoef.2001. Phylogenetic diversity of Klebsiellapneumoniae and Klebsiellaoxytoca clinical isolates revealed by randomly amplified polymorphic DNA, gyrA and parC genes sequencing and automated ribotyping. Int. J. Syst. Evol. Microbiol. 51: 915-924.
6. Arlet, G., M. Rouveau, I. Casin, P.J. Bouvet, P.H. Lagrange, and A. Philippon.1994. Molecular epidemiology of Klebsiellapneumoniae strains that produce SHV-4 beta-lactamase and which were isolated in 14 French hospitals. J. Clin. Microbiol. 32: 2553-2558.
7. Jonas, D., B.  F. D. Daschner, J. Verhoef, and S. Brisse.2004. Discrimination of Klebsiellapneumoniae and Klebsiellaoxytoca phylogenetic groups and other Klebsiella species by use of AFLP. Res. Microbiol.155:17-23.
F. D. Daschner, J. Verhoef, and S. Brisse.2004. Discrimination of Klebsiellapneumoniae and Klebsiellaoxytoca phylogenetic groups and other Klebsiella species by use of AFLP. Res. Microbiol.155:17-23.
8. Van Westreenen, M., A. Paauw, A.C. Fluit, S. Brisse, W. Van Dijk, and J. Verhoef.2003. Occurrence and spread of SHV extended-spectrum beta-lactamase-producing Klebsiellapneumoniae isolates in Curacao. J. Antimicrob. Chemother. 52: 530-532.
9. Brisse, S., V. Fussing, B. Ridwan, J. Verhoef, and R. J. Willems.2002. Automated ribotyping of vancomycin-resistant Enterococcus faecium isolates. J. Clin. Microbiol. 40: 1977-1984.
10. Grimont, P.A.D., and F. Grimont.2001. rRNA gene restriction pattern determination (ribotyping) and computer interpretation, p. 107-133. In L. Dijkshoorn, K.J. Towner, and M.J. Struelens (ed.), New approaches for the generation and analysis of microbial typing data. Elsevier, Amsterdam, TheNetherlands.
11. Diancourt L. et al. Multilocus sequence typing of Klebsiellapneumoniae nosocomial isolates // Journal of clinical microbiology. - 2005. - T. 43. - №. 8. - C. 4178-4182.
12. «Способ генетической дифференциации штаммов Yersinia pseudotuberculosis путем молекулярно-генетического типирования», пат. №2736649 кл. C12N 1/00, опубл.19.11.2020 г. Бюл. №32.
13. «Способ молекулярно-генетического внутривидового типирования токсигенных штаммов Vibrio cholerae 01 Eltor», пат. №2627193,кл. C12N 15/10, опубл. 03.08.2017 г. Бюл.№22.
14. «Способ генетического INDEL-типирования штаммов Brucella melitensis», пат. №2732425,кл. C12Q 1/68, опубл. 16.09.2020 г.
15. https: //kodomo.fbb.msu.ru/~nihilenia/term2/tree.htm/
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Alexey.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2022-10-14">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022121086/20</ApplicationNumberText>
<FilingDate>2022-08-02</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>none</ApplicantFileReference>
<ApplicantName languageCode="ru">ФКУЗ Ростовский-на-Дону
противочумный институт Роспотребнадзора</ApplicantName>
<ApplicantNameLatin>FGHI Rostov-on-Don Plague Control Research
Institute of the Rospotrebnadzor</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ молекулярно-генетического
типирования штаммов Klebsiella pneumoniae с использованием
INDEL-маркеров</InventionTitle>
<SequenceTotalQuantity>22</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcggtcatggcctggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttttcaccaatttccgcggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagccttgaacaactgccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcagatccatcgccaggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactacggcgaccttaacga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgtcagacaatgcatccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acaactcgacgccaaactac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgccatggaacggattac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggttttcagctggtggaagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcaatatccgcttccacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtaacgtcctggaccgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggactgcaggaagttgtcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaacgcttaaccgccatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcaccgctttcacgctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggtgaggtcgatctggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcaatatccgcttccacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acaactcgacgccaaactac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgccatggaacggattac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagcctgatttcttgccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagtacgctgaagtcacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcaaccatgatgagtacct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Klebsiella
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttctgtacctgcgtcgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ дифференциации штаммов Pseudomonas aeruginosa с помощью молекулярно-генетического типирования | 2023 |
|
RU2816184C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| Способ генетической дифференциации штаммов Yersinia pseudotuberculosis путем молекулярно-генетического типирования | 2019 |
|
RU2736649C1 |
| Способ оценки внутривидовой конкуренции токсигенных и нетоксигенных штаммов Vibrio cholerae О1 и О139 серогрупп с помощью ПЦР в режиме реального времени | 2023 |
|
RU2806564C1 |
| СПОСОБ ГЕНЕТИЧЕСКОГО INDEL-ТИПИРОВАНИЯ ШТАММОВ BRUCELLA MELITENSIS | 2019 |
|
RU2732425C1 |
| Способ идентификации SNP-генотипов возбудителя чумы средневекового биовара филогенетических ветвей 2.MED1 и 2.MED4 из очагов Северного и Северо-Западного Прикаспия методом секвенирования по Сэнгеру | 2024 |
|
RU2831366C1 |
| Набор праймеров для выявления возбудителей бактериальной пневмонии человека методом мультиплексной рекомбиназной полимеразной амплификации | 2023 |
|
RU2813995C1 |
| Способ выявления гена холодового шока csh1 у штаммов Vibrio cholerae O1 и неО1/неО139 методом петлевой изотермической амплификации (LAMP) | 2023 |
|
RU2819101C1 |
| Способ выявления рекомбинантных форм вируса гепатита C и мутаций лекарственной устойчивости вируса гепатита С к препаратам прямого противовирусного действия методом полимеразной цепной реакции с последующим секвенированием | 2023 |
|
RU2824565C1 |
| Способ дифференциации штаммов Helicobacter pylori путем молекулярно-генетического типирования | 2018 |
|
RU2688434C1 |

Изобретение относится к области медицинской микробиологии, а именно к лабораторной диагностике, и может быть использовано при молекулярно-генетическом внутривидовом типировании штаммов Klebsiella pneumoniae, циркулирующих в объектах на различных территориях, с целью их дифференциации. Способ молекулярно-генетического типирования с использованием INDEL-маркеров включает выделение ДНК из исследуемого штамма, постановку ПЦР со специфическими праймерами и учет реакции после электрофореза. Амплификацию ДНК исследуемого штамма проводят с набором 11 праймеров к INDEL-маркерам Klebsiella pneumoniae, имеющих два альтернативных аллеля, а именно: с праймерами к гену MsyB, к гену AraC, к гену TraD, к гену pol-deac, к гену Galactose, к гену OmpC, к гену 8455, к гену 23015, к гену 23215, к гену 12905 и к гену 13875. Учет результатов амплификации проводят с помощью электрофореза в 10% полиакриламидном геле с последующим детектированием размера каждого INDEL-локуса. По полученным размерам аллелей выстраивают с помощью алгоритма UPGMA дендограмму, по которой осуществляют типирование штаммов Klebsiella pneumoniae, определяя общее или различное происхождение исследуемых штаммов и возможных путей их распространения. Осуществление способа позволяет достоверно и быстро осуществлять типирование штаммов. 2 з.п. ф-лы, 2 ил, 1 табл., 2 пр.
1. Способ молекулярно-генетического типирования штаммов Klebsiella pneumoniae с использованием INDEL-маркеров, включающий выделение ДНК из исследуемого штамма, постановку ПЦР со специфическими праймерами и учет реакции после электрофореза, отличающийся тем, что с ДНК исследуемого штамма амплификацию проводят с набором 11 праймеров к INDEL-маркерам Klebsiella pneumoniae, имеющих два альтернативных аллеля:
с праймерами к гену MsyB
Forward: ATCGGTCATGGCCTGGTG
Reverse: TTTTCACCAATTTCCGCGGT
с длиной фрагмента амплификации 152 п.о. или 176 п.о.,
с праймерами к гену AraC
Forward: GAGCCTTGAACAACTGCCC
Reverse: GCAGATCCATCGCCAGGC
с длиной фрагмента амплификации 107 п.о. или 125 п.о.,
с праймерами к гену TraD
Forward: GACTACGGCGACCTTAACGA
Reverse: GCGTCAGACAATGCATCCAG
с длиной фрагмента амплификации 86 п.о. или 101 п.о.,
с праймерами к гену pol-deac
Forward: ACAACTCGACGCCAAACTAC
Reverse: CACGCCATGGAACGGATTAC
с длиной фрагмента амплификации 77 п.о. или 86 п.о.,
с праймерами к гену Galactose
Forward: GGTTTTCAGCTGGTGGAAGG
Reverse: CGCAATATCCGCTTCCACG
с длиной фрагмента амплификации 96 п.о. или 102 п.о.,
с праймерами к гену OmpC
Forward: CGTAACGTCCTGGACCGAC
Reverse: GGACTGCAGGAAGTTGTCAG
с длиной фрагмента амплификации 73 п.о. или 79 п.о.,
с праймерами к гену 8455
Forward: GAACGCTTAACCGCCATCC
Reverse: CTCACCGCTTTCACGCTG
с длиной фрагмента амплификации 98 п.о. или 122 п.о.,
с праймерами к гену 23015
Forward: AAGGTGAGGTCGATCTGGC
Reverse: CGCAATATCCGCTTCCACG
с длиной фрагмента амплификации 80 п.о. или 86 п.о.,
с праймерами к гену 23215
Forward: AAGGTGAGGTCGATCTGGC
Reverse: CACGCCATGGAACGGATTAC
с длиной фрагмента амплификации 77 п.о. или 86 п.о.,
с праймерами к гену 12905
Forward: GGAGCCTGATTTCTTGCCG
Reverse: AGAGTACGCTGAAGTCACGC
с длиной фрагмента амплификации 89 п.о. или 97 п.о.,
с праймерами к гену 13875
Forward: TGGCAACCATGATGAGTACCT
Reverse: CTTTCTGTACCTGCGTCGTG
с длиной фрагмента амплификации 90 п.о. или 99 п.о.,
при этом учет результатов амплификации проводят с помощью электрофореза в 10% полиакраламидном геле с последующим детектированием размера каждого INDEL-локуса, а затем по полученным размерам аллелей выстраивают с помощью алгоритма UPGMA дендограмму, по которой осуществляют типирование штаммов Klebsiella pneumoniae, определяя общее или различное происхождение исследуемых штаммов и возможных путей их распространения.
2. Способ по п. 1, отличающийся тем, что ПЦР проводят в объеме 25 мкл и реакционная смесь содержит: 2,5 мкл буфера, 1 EдTaq ДНК-полимеразы, 0,2 мкМ смеси дНТФ, 0,5 мкл 50Х SYBRGreenI, 1,0 мкМ каждого из праймеров, 5 мкл ДНК - ДНК матрицы, оставшийся объем - вода.
3. Способ по п. 1, отличающийся тем, что амплификацию проводят с соблюдением следующих этапов и режимов:
1) 95°С этап денатурации - 3 мин (1 цикл),
2) 95°С этап денатурации - 20 с (35 циклов),
3) отжиг при 60°С - 20 с,
4) синтез при 72°С - 20 с,
5) синтез при 72°С - 1 мин (1 цикл).
| DIANCOURT L., ET AL | |||
| Multilocus sequence typing of Klebsiella pneumoniae nosocomial isolates // Journal of clinical microbiology | |||
| T | |||
| Зубчатое колесо со сменным зубчатым ободом | 1922 |
|
SU43A1 |
| Шест для прыжков | 1924 |
|
SU4178A1 |
| Способ генетической дифференциации штаммов Yersinia pseudotuberculosis путем молекулярно-генетического типирования | 2019 |
|
RU2736649C1 |
| СПОСОБ ГЕНЕТИЧЕСКОГО INDEL-ТИПИРОВАНИЯ ШТАММОВ BRUCELLA MELITENSIS | 2019 |
|
RU2732425C1 |
| АЛЕКСЕЕВА А | |||
| Е., БРУСНИГИНА Н | |||
| Ф., СОЛНЦЕВ Л | |||
| А., ГОРДИНСКАЯ Н | |||
| А | |||
| Молекулярное типирование клинических изолятов Klebsiella | |||
Авторы
Даты
2023-05-23—Публикация
2022-08-02—Подача