Изобретение относится к области биотехнологии, молекулярной биологиии и может быть использовано для выявления ДНК возбудителей кокцидиоидомикоза Coccidioides immitis и Coccidioides posadasii в биологическом материале и объектах окружающей среды специалистами учреждений как практического здравоохранения, службы Роспотребнадзора, так и в научных исследованиях.
Необходимость исследований, направленных на выявление этих патогенных микромицетов, определяется отсутствием средств специфической профилактики кокцидиоидомикоза, а также выраженной резистентностью Coccidioides spp. ко многим химиотерапевтическим препаратам.
Трудности диагностики кокцидиоидомикоза обусловлены диморфным характером этих микроскопических грибов - способностью существования в мицелиальной (сапробиотической) форме во внешней среде и тканевой (паразитической) в организме человека и животных. Существующие методы идентификации возбудителей кокцидиоидомикоза оказываются недостаточно эффективными для их ускоренной диагностики. Традиционная схема лабораторного анализа с выделением культуры и использованием биопробных животных занимает до 30 и более суток. Общим для всех иммуносерологических методов, направленных на выявление антигенов грибов, является их недостаточная чувствительность составляющая 1×104-1×107 артроспор/мл.
В качестве альтернативного способа выявления данных патогенов наиболее перспективным является полимеразная цепная реакция (ПЦР), которую отличает универсальность, высокая чувствительность и относительная простота исполнения. ПЦР позволяет исследовать любой материал как биологический (мокроту, бронхоальвеолярный лаваж, плевральную жидкость, мазки из ротоглотки, кровь, микро- и макроаутоптаты), так и пробы из объектов окружающей среды (воду, почву, смывы), которые инфицированы возбудителями кокцидиоидомикоза, а также чистые культуры.
Известны способы идентификации возбудителя кокцидиоидомикоза, согласно которым ДНК-мишенями для амплификации служат нуклеотидные последовательности рибосомальных генов (18S рРНК, 5,8S рРНК, а также спейсерные области рибосомальных генов) [Greene D.R., Koenig G., Fisher M.C. et al. Soil isolation and molecular identification of Coccidioides immitis. Mycologia. 2000; 92:406-10; Lindsley M.D., Hurst S.F., Iqbal N.J. et al. Rapid identification of dimorphic and yeast-like fungal pathogens using specific DNA probes. J. Clin. Microbiol. 2001; 39:3505-11]. Однако, учитывая высокую консервативность этих регионов у различных видов грибов, существует вероятность получения ложноположительных результатов, в первую очередь при исследовании материала из объектов внешней среды. Это указывает на необходимость проведения секвенирования всех продуктов амплификации, если в ПЦР-тест-системе используются праймеры, разработанные на основе фрагментов рибосомальных генов возбудителей кокцидиоидомикоза [Millar B.C., Jim X., Walker M.J. … et al. False identification of Coccidioides immitis: do molecular methods always get it right? J. Clin. Microbiol. 2003; 41(12):5778-80]. Такой подход является достаточно трудоемким и приводит к значительному увеличению времени и стоимости анализа.
Наряду с рибосомальными генами, в качестве мишени амплификации использовались гены, кодирующие видоспецифические белки: 19 кДа специфичный белок (CS-Ag) [Pan. S., Cole G.T. Molecular and biochemical characterization of a Coccidioides immitis-specific antigen. Infect. Immun. 1995; 63(10):3994-4002], пролин-богатый иммунодоминантный белок клеточной стенки (Ag2/PRA) [Bialek R., Kern J., Herrmann Т. et al. PCR assays for identification of Coccidioides posadasii based on the nucleotide sequence of the antigen 2/Proline-Rich Antigen. J. Clin. Microbiol. 2004; 42:778-83], иммунодоминантный гликопротеин внешней стенки сферул данных микромицетов (SOWgp82) [Ткаченко Г.А., Гришина М.А., Антонов В.А. и др. Идентификация возбудителей кокцидиоидомикоза методом полимеразной цепной реакции. Мол. генет., микробиол. и вирусол. 2007; 4:25-31]. Однако детекция продуктов амплификации в этих случаях осуществляется с помощью электрофореза, что создает риск контаминации продуктами ПЦР и, соответственно, увеличивает риск возникновения ложноположительных результатов.
Использование в качестве специфической мишени фрагмента гена МВР-1 позволило разработать олигонуклеотидные праймеры, позволяющие выявлять С.immitis и С.posadasii методом ПЦР в биологическом материале и объектах окружающей среды [патент РФ №2346046, МПК C12N 15/30, C12Q 1/68, опубл. 10.02.2009]. Однако существенным недостатком этого набора олигонуклеотидов, так же как и вышеупомянутых аналогов, является электрофорезная детекция продуктов амплификации.
Детекция продуктов 1ТДР с помощью специальных флуоресцентных меток позволяет отказаться от стадии электрофореза, что не только приводит к снижению риска контаминиции, т.к. учет результатов реакции происходит без открывания пробирок, но и сокращает время проведения анализа. Этот подход позволяет проводить автоматическую интерпретацию полученных результатов и снимает проблему субъективной оценки электрофореграмм.
Существует множество флуоресцентных технологий, различающихся по способам генерации репортерной флуоресценции. Для обнаружения возбудителей кокцидиоидомикоза известна методика, основанная на использовании зондов с резонансным переносом энергии флуоресценции (LightCycler assay) [Binnicker M.J., Buckwalter S.P., Eisberner J.J. et al. Detection of Coccidioides species in clinical specimen by real-time PCR. J. Clin. Microbiol. 2007; 45:173-78]. Применение данного подхода сопряжено с необходимостью использования двух олигонуклеотидных зондов, меченных разными флуорофорами, что повышает стоимость проведения анализа и предъявляет высокие требования к выбору пары флуоресцентных красителей, между которыми возможен резонансный перенос энергии.
K.W. Sheff с соавт. для дифференциации С. immitis и С. posadasii предложена технология TaqMan-ПЦР в формате реального времени [Sheff K.W., York E.R., Driebe Е.М. et al. Development of a rapid, cost-effective TaqMan Real-Time PCR Assay for identification and differentiation of Coccidioides immitis and Coccidioides posadasii. Med. Mycol. 2010; 48(3):466-9]. Используемый в данной работе нефлуоресцентный гаситель MGB (Minor Groove Binder) повышает температуру плавления зонда и стабильность его взаимодействия с ампликоном. Однако подобная модификация зонда значительно увеличивает стоимость синтеза олигонуклеотида, а также на данный момент сложно осуществима на территории Российской Федерации.
Из флуоресцентных методов детекции известен гибридизационный олигонуклеотидный зонд PR-SOW для идентификации возбудителя кокцидиоидомикоза С. posadasii (патент РФ 2539108, МПК C12Q 1/68, опубл. 10.01.2015 г.). Однако с помощью этого зонда нельзя выявлять С. immitis.
Наиболее близким аналогом является работа S. Gago с соавт., в которой приведены праймеры и зонд, сконструированные на основе нуклеотидной последовательности внутреннего транскрибирующегося спейсерного региона ITS1 генов рРНК [Gago S., Buitrago M.J., Clemons K.V. Development and validation of a quantitative real-time PCR assay for the early diagnosis of coccidioidomycosis. Diagn Microbiol Infect Dis. 2014; 79(2):214-21]. Однако праймеры и зонд, рассмотренные в данной работе, были апробированы только при исследовании клинических образцов. Высокая степень гомологии возбудителей кокцидиоидомикоза с близкородственными сапрофитными грибами и консервативность рибосомальных генов грибов определяют возможность получения ложноположительных результатов при исследовании объектов окружающей среды с помощью этих олигонуклеотидов.
Недостатки описанных подходов устраняются в настоящем изобретении. Вышеупомянутые зонды отличаются по структуре от зонда, предлагаемого в заявляемом техническом решении. В отличие от S. Gago с соавт. разработанный гибридизационный зонд сконструирован таким образом, что взаимно-комплементарные концевые последовательности, образующие «шпильку» по принципу «молекулярного маяка» (molecular beacon), полностью комплементарны целевой последовательности амплификации, что существенно повышает специфичность гибридизации. Кроме того, предлагаемые олигонуклеотидные праймеры, входящие в состав набора, имеют оригинальные места посадки, а также больше по размеру, тем самым увеличивая специфичность разработанного набора олигонуклеотидов.
Целью настоящего изобретения является разработка набора олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации ДНК возбудителей кокцидиоидомикоза С. immitis и С. posadasii методом полимеразной цепной реакции с гибридизационно-флуоресцентным учетом результатов.
Цель достигается созданием набора олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации ДНК возбудителей кокцидиоидомикоза С. immitis и С. posadasii методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией результатов, содержащего пару олигонуклеотидов, обладающих активностью прямого и обратного праймеров, и зонд с комплементарными концевыми последовательностями по типу «молекулярного маяка», имеющих следующую структуру:

где FAM - карбоксифлуоресцеин, флуоресцентный краситель, длина волны поглощения которого составляет 492 нм, а длина волны флуоресценции - 520 нм. RTQ1 - гаситель флуоресценции с диапазоном гашения 470-570 нм.
Характеристика олигонуклеотидных праймеров, зонда и ДНК-мишени для гибридизации
Основываясь на данных, представленных в базе GenBank NCBI (National Center for Biotechnology Information, США), были подобраны праймеры, обозначенные CimMBPSs/CimMBP4as, комплементарные фрагменту гена МВР-1 (macrophage binding protein) и обеспечивающие амплификацию специфического фрагмента размером 373 п.н.
Для детекции продуктов 1ТДР в режиме реального времени был сконструирован зонд формата «молекулярный маяк», обозначенный Р/-МВР-1, на разных концах которого расположены флуорофор и гаситель флуоресценции. Подобная структура зонда обеспечивает максимальный эффект тушения и низкую фоновую флуоресценцию, поскольку молекулы флуорофора и гасителя сближены в пространстве.
Апробация разработанного набора олигонуклеотидов была осуществлена на наборе штаммов возбудителей кокцидиоидомикоза коллекционного центра ФКУЗ Волгоградский научно-исследовательский противочумный институт Роспотребназора. В качестве положительного контроля в экспериментах использовали референтные штаммы С. immitis 158 и С. posadasii 36-S. Выделение ДНК проводили из обеззараженных суспензий микромицетов в концентрациях от 1×109 до 1×10 артроспор/мл.
Примеры конкретного выполнения
Пример 1. Методика конструирования олигонуклеотидных праймеров и флуоресцентно-меченого гибридизационного зонда для идентификации ДНК возбудителей кокцидиоидомикоза методом ПЦР в режиме реального времени
На основе изучения in silico секвенированных нуклеотидных последовательностей возбудителей кокцидиоидомикоза, присутствующих в генетических базах данных (EMBL, Genbank, DDBJ), для конструирования прямого CimMBP3s и обратного CimMBP4as праймеров была выбрана последовательность гена С. immitis МВР-1 (macrophage binding protein), размер которой составляет 1534 п.н. (GenBank NCBI, AF326276). Наиболее перспективной для подбора праймеров при разработке ПНР была выявлена последовательность гена, фланкируемая с 453 по 825 нуклеотид. Расчетная длина специфического фрагмента составила 373 п.н. (табл. 1).
При использовании компьютерных программ была проанализирована структура выбранной пары праймеров CimMBP3s/CimMBP4as (образование димеров, шпилек и других вторичных структур) и показана их теоретическая пригодность для успешной инициации реакции амплификации.
Для флуоресцентной детекции продуктов ПЦР в режиме реального времени сконструирован зонд Pf-MBP-1 в формате «молекулярный маяк» размером 27 п.н., который полностью гибридизовался на участке гена МВР-1 с 601 по 627 нуклеотид, фланкируемого праймерами CimMBP3s/CimMBP4as.
Вторичную структуру и термодинамические параметры разработанного зонда оценивали в онлайн-режиме с использованием программы Mfold на интернет-сайте http://mfold.rna. albany. edu.
Специфичность разработанных олигонуклеотидов оценивали методом множественного сравнения последовательностей с помощью компьютерной программы BLAST на web-сервере (bttp://www.ncbi.nlm.nih.gov/BLAST/) Национального Центра Биотехнологической Информации (NCBI) для установления гомологии между сконструированными праймерами и зондом и нуклеотидными последовательностями близкородственных возбудителей особо опасных микозов и гетерологичных микроорганизмов. На момент проведения компьютерного анализа гомологии выявлено не было.
Пример 2. Амплификация специфических фрагментов ДНК с помощью разработанного набора олигонуклеотидов для идентификации ДНК возбудителей кокцидиоидомикоза С. immitisi и С. posadasii с детекцией результатов ПЦР в режиме реального времени.
Инактивацию образцов и выделение ДНК проводили согласно требованиям СП 1.3.3118-13 «Безопасность работы с микроорганизмами I-II групп патогенности (опасности)» и МУ 1.3. 2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности».
Обеззараживание исследуемых проб проиводили добавлением раствора мертиолята натрия до конечной концентрации 0,1% и последующим прогреванием в течение 40 мин при температуре температуре 56°С и инкубировании при комнатной температуре до 24 ч. Выделение ДНК из чистых культур микромицетов осуществляли с помощью метода гуанидинтиоцианат-фенольной экстракции с переосаждением ДНК изопропанолом [Sandhu G.S. et al., 1995].
Для проведения ПЦР в режиме реального времени с разработанными праймерами CimMBP3s/CimMBP4as и олигонуклеотидным зондом Pf-MBP-1 готовили смесь компонентов следующего состава (из расчета на одну пробу в 25 мкл смеси): 200 мкМ каждого из дезоксинуклеозидтрифосфатов, 2 мМ MgCl2, 5 мкл 5х ПЦР-буфер 0mM Mg2+ (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), по 0,48 мкМ специфических олигонуклеотидных праймеров, 0,24 мкМ флуоресцентно-меченого зонда, 2 ед. TaqF ДНК-полимеразы (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), 4,96 мкл Н2О для ПЦР и 5 мкл исследуемой ДНК.
В качестве отрицательного контрольного образца в реакционную смесь добавляли ТЕ-буфер.
Амплификацию в режиме реального времени проводили на приборе «Rotor-Gene 6000» («Corbett Research)), Австралия), в микроцентрифужных пробирках объемом 0,2 мл, с использованием «горячего старта» по следующей программе: предварительный прогрев 95°С - 15 мин, 45 циклов (95°С - 20 с, 55°С - 60 с), из них первые 5 циклов без флуоресцентной детекции, затем 40 циклов с флуоресцентной детекцией сигнала на этапе отжига праймеров.
Анализ продуктов ПЦР осуществляли в режиме реального времени на приборе «Rotor-Gene 6000» («Corbett Research», Австралия) по каналу Green. Регистрацию результатов проводили в табличной и графической форме с помощью программного обеспечения прибора. Результаты анализировали по наличию или отсутствию пересечения кривой флуоресценции с пороговой линией, что определяется значением порогового цикла «Ct» в соответствующей графе в таблице результатов. Результат считали положительным в случае, если кривая накопления флуоресценции для соответствующего образца имела характерную «сигмовидную» форму и пересекала пороговую линию. При этом значение Ct для исследуемых образцов было не более 33.
Пример 3. Определение чувствительности и специфичности реакции амплификации с помощью разработанного набора олигонуклеотидных праймеров CimMBP3s/CimMBP4as и флуоресцентно-меченого зонда Pf-МВР-1 для идентификации ДНК возбудителей кокцидиоидомикоза Coccidioides immitis и Coccidioides posadasii.
Исследования проводили на штаммах микромицетов из коллекционного центра ФКУЗ Волгоградский научно-исследовательский противочумный институт Роспотребнадзора. При подготовке проб для ПЦР исследуемые культуры грибов в мицелиальной фазе роста культивировали на агаре Сабуро при 28°С в течение 30 суток. Дрожжевые формы H. capsulatum и P. brasiliensis выращивали на FT-агаре с добавлением цельной крови и L-цистеина при 37°С в течение 10-14 суток, в зависимости от штамма. Из выросших культур готовили суспензии в 0,15 М растворе хлорида натрия, рН 7,1±1.
После обеззараживания мертиолятом натрия (1:1000) и фильтрования суспензий грибов проводили подсчет клеток исходных взвесей артроспор и дрожжевой фазы микромицетов в камере Горяева. Затем взвеси микромицетов разводили 0,15 М раствором хлорида натрия таким образом, чтобы концентрации для Coccidioides spp. составляли 1×103-1×105 артроспор/мл, а для гетерологичных микроорганизмов до 1×107 м.к./мл.
Выделение ДНК, постановку и учет результатов реакции ПЦР в режиме реального времени осуществляли, как описано в примере 2.
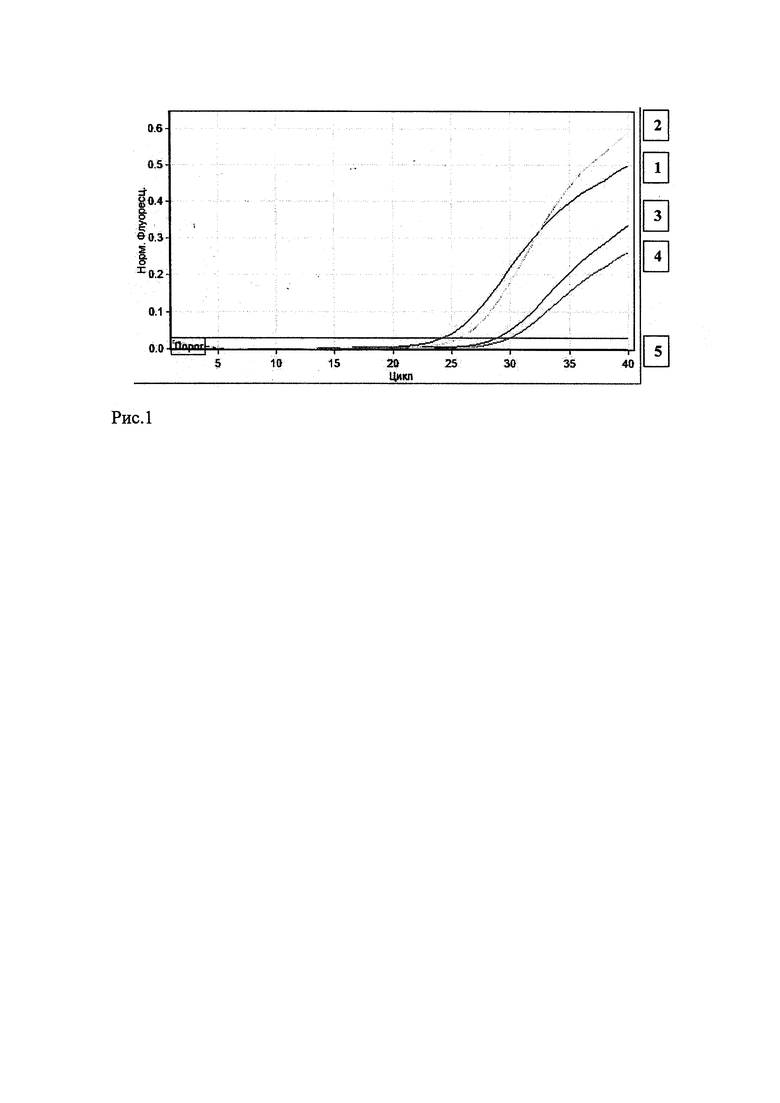
Чувствительность реакции амплификации с разработанным набором специфичных праймеров CimMBP3s/CimMBP4as и флуоресцентно-меченого зонда Pf-MBP-1 оценивалась при исследовании проб ДНК, выделенных из десятикратных разведении артроспор чистых культур возбудителей кокцидиоидомикоза. При тестировании коллекции грибных культур С. immitis и C. posadasii продукт амплификации синтезировался с ДНК всех штаммов возбудителей кокцидиоидомикоза с чувствительностью 1×104 клеток/мл (рис. 1).
Рисунок 1 отображает график нарастания флуоресцентных кривых, полученных при амплификации ДНК штаммов возбудителей кокцидиоидомикоза с праймерами CimMBP3s/CimMBP4as и зондом Pf-МВР-1 (изображены кривые 1 - С.posadasii 36-S в концентрации 105 м.к./мл; 2 - С. immitis 158 концентрации 105 м.к./мл; 3 - С. posadasii 36-S в концентрации 104 м.к./мл; 4 - С. immitis 158 в концентрации 104 м.к./мл; 5 - отрицательный контроль и кривые накопления флуоресценции проб С. immitis 158 и C. posadasii 36-S в концентрации меньше 10 м.к./мл, не пересекающие пороговую линию).
Специфичность разработанного набора праймеров
CimMBP3s/CimMBP4as и флуоресцентно-меченого зонда Pf-MBP-1 оценена на коллекции из 8 штаммов С. immitis, 14 штаммов C. posadasi и 15 штаммов гетерологичных микроорганизмов (3 штамма Н. capsulatum, 3 штамма H. duboisii, 3 штамма В. dermatitidis, 2 штамма P. brasiliensis, 1 штамм Cryptococcus neoformans, 1 штамм Aspergillus fumigates, 1 штамм Candida glabrata, 1 штамм Scopulariopsis brevicaulis). Оценка специфичности показала отсутствие перекрестных реакций с близкородственными гетерологичными штаммами и наличие специфической амплификации со всеми штаммами С. immitis и С. posadasi.
Таким образом, из вышеизложенного следует, что достигнута поставленная цель, а именно: разработан набор праймеров CimMBP3s/CimMBP4as и флуоресцентно-меченого зонда Pf-MBP-1 для идентификации ДНК возбудителей кокцидиоидомикоза Coccidioides immitis и Coccidioides posadasii. Подтверждена специфичность и определена аналитическая чувствительность сконструированного набора олигонуклеотидов для детекции данных микромицетов, которая составила 1×104 артроспор/мл, что позволяет использовать сконструированный набор олигонуклеотидов для детекции возбудителей кокцидиоидомикоза в короткий срок с высокой чувствительностью и специфичностью.

| название | год | авторы | номер документа |
|---|---|---|---|
| ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЙ ОЛИГОНУКЛЕОТИДНЫЙ ЗОНД PR-SOW ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЯ КОКЦИДИОИДОМИКОЗА Coccidioides posadasii | 2013 |
|
RU2539108C1 |
| ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЕЙ КОКЦИДИОИДОМИКОЗА Coccidioides immitis И Coccidioides posadasii | 2007 |
|
RU2346046C1 |
| ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ ДЛЯ ИДЕНТИФИКАЦИИ Coccidioides posadasii | 2007 |
|
RU2346045C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЯ БЛАСТОМИКОЗА BLASTOMYCES DERMATITIDIS | 2016 |
|
RU2639498C1 |
| ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЙ ОЛИГОНУКЛЕОТИДНЫЙ ЗОНД MS8 Flip-R ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЯ ГИСТОПЛАЗМОЗА Histoplasma capsulatum | 2013 |
|
RU2532845C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ ВИРУСА ЗАПАДНОГО НИЛА 1 ГЕНОТИПА | 2019 |
|
RU2715625C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ ВИРУСА ЗАПАДНОГО НИЛА 4 ГЕНОТИПА (WEST NILE VIRUS LINEAGE 4) | 2020 |
|
RU2737396C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ ВИРУСА ЗАПАДНОГО НИЛА 2 ГЕНОТИПА | 2019 |
|
RU2715617C1 |
| ФЛУОРЕСЦЕНТНО-МЕЧЕНЫЕ ОЛИГОНУКЛЕОТИДНЫЕ ЗОНДЫ ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЕЙ САПА И МЕЛИОИДОЗА B. pseudomallei И B. mallei | 2010 |
|
RU2435861C1 |
| ОЛИГОНУКЛЕОТИДНЫЙ ЗОНД ДЛЯ ИДЕНТИФИКАЦИИ ВОЗБУДИТЕЛЕЙ САПА И МЕЛИОИДОЗА B. pseudomallei И B. mallei | 2010 |
|
RU2439159C1 |
Изобретение относится к области биотехнологии, молекулярной биологиии. Представлен набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации ДНК возбудителей кокцидиоидомикоза С. immitis и С. posadasii методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией результатов, содержащего пару олигонуклеотидов, обладающих активностью прямого и обратного праймеров, и зонд с комплементарными концевыми последовательностями по типу «молекулярного маяка», имеющих следующую структуру:

Использование разработанного набора олигонуклеотидных праймеров CimMBP3s/CimMBP4as и флуоресцентно-меченого зонда Pf-MBP-1 позволяет в короткий срок с высокой чувствительностью и специфичностью проводить идентификацию ДНК возбудителей кокцидиоидомикоза С. immitis и С. posadasii в пробах чистых культур, биологическом материале и объектах окружающей среды. 1 ил., 1 табл., 3 пр.
Набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации ДНК возбудителей кокцидиоидомикоза immitis и Coccidioides posadasii методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией результатов, содержащий пару олигонуклеотидов, обладающих активностью прямого и обратного праймеров, и зонд, сконструированный по типу «молекулярного маяка», имеющих следующую структуру:

где FAM - карбоксифлуоресцеин, флуоресцентный краситель, длина волны поглощения которого составляет 490 нм, а длина волны флуоресценции - 520 нм; RTQ1 - гаситель флуоресценции с диапазоном гашения 470-570 нм.
Авторы
Даты
2016-04-27—Публикация
2015-04-09—Подача