Область техники, к которой относится изобретение
[0001] Настоящее изобретение относится к экспрессии представляющих интерес белков в растениях. В настоящем изобретении также предусматриваются способы и композиции для продуцирования представляющих интерес белков в растениях.
Уровень техники
[0002] Растения предоставляют большие возможности как системы для продуцирования рекомбинантных белков. Одним из способов продуцирования чужеродных белков в растениях является создание стабильной трансгенной линии в растениях. Однако этот способ занимает много времени и является трудоемким. Альтернативным в трансгенных растениях является применение экспрессионных векторов на основе вирусов растений. Векторы на основе вирусов растений делают возможной быструю высокоуровневую транзиторную (временную) экспрессию белков в растениях.
[0003] Один из способов осуществления высокоуровневой транзиторной экспрессии чужеродных белков в растениях включает применение векторов на основе РНК-вирусов растений, включая комовирусы, такие как вирус мозаики коровьего гороха Cowpea mosaic virus (CPMV; см., например, Международные заявки WO 2007/135480; WO 2009/087391; патент США US 2010/0287670, Sainsbury F. et al., 2008, Plant Physiology; 148: 121-1218; Sainsbury F. et al., 2008, Plant Biotechnology Journal; 6: 82-92; Sainsbury F. et al., 2009, Plant Biotechnology Journal; 7: 682-693; Sainsbury F. et al. 2009, Methods in Molecular Biology, Recombinant Proteins From Plants, vol. 483: 25-39).
[0004] Комовирусы представляют собой двухкомпонентные (бипартитные) РНК-содержащие вирусы. Сегменты генома РНК комовируса называются RNA-1 (РНК-1) и RNA-2 (РНК-2). RNA-1 кодирует VPg, белки репликазу и протеазу. Репликаза необходима вирусу для репликации вирусного генома. RNA-2 комовируса, вируса мозаики коровьего гороха (CPMV), кодирует полипротеин протяженностью 105 кДа или 95 кДа, процессируемый в 4 функциональных пептидах.
[0005] Область 5' CPMV RNA-2 содержит инициирующие (стартовые, старт-) кодоны (AUG) в положениях 115, 161, 512 и 524. Стартовые кодоны в положениях 161 и 512 находятся в одной и той же триплетной рамке считывания. Инициация в старт-кодоне в положении 161 приводит к синтезу полипротеина 105K, тогда как инициация в старт-кодоне в положении 512 направляет синтез 95K полипротеина. Инициация трансляции со стартового кодона в положении 512 в CPMV является более эффективной, чем инициация в положении 161, и приводит к продуцированию большего количества 95К полипротеина, чем 105K полипротеина. Стартовый кодон в положении 115 не является существенным для репликации вирусов (Wellink et al., 1993 Biochimie. 75(8):741-7).
[0006] Сохранение рамки считывания между сайтами инициации в положениях 161 и 512 в CPMV RNA-2 необходимо для эффективной репликации RNA-2 под действием RNA-1-кодируемой репликазы (Holness et al., 1989; Virology 172, 311-320; van Bokhoven et al. 1993, Virology 195, 377-386; Rohll et al., 1993 Virology 193, 672-679; Wellink et al., 1993, Biochimie. 75(8):741-7). Это требование влияет на протяженность последовательностей, которые можно вводить (вставлять) выше (в направлении 3'-5') стартового кодона 512 в репликативных формах CPMV RNA-2 экспрессионных векторов. Кроме того, применять полилинкеры следует осторожно, поскольку они могут сдвигать открытую рамку считывания кодонов (ORF) между этими сайтами инициации.
[0007] CPMV послужил основой для разработки векторных систем, пригодных для продуцирования гетерологичных полипептидов в растениях (Liu et al., 2005 Vaccine 23, 1788-1792; Sainsbury et al., 2007 Virus Expression Vectors (Hefferon, K. ed), pp. 339-555). Эти системы основаны на модификации RNA-2, но отличаются в зависимости от того, используются ли полноразмерные варианты или варианты с делециями. Репликацию модифицированной RNA-2 осуществляют совместной инокуляцией с RNA-1. Чужеродные белки связываются (сливаются) с С-концом полипротеинов, процессированных при использовании RNA-2. Высвобождение N-концевого полипептида опосредуется действием каталитической пептидной последовательности 2А вируса ящера (Gopinath et al., 2000, Virology 267: 159-173). Полученные молекулы RNA-2 способны распространяться как внутри растения, так и от растения к растению. Эта стратегия была применена для экспрессии ряда рекомбинантных белков, например, таких как коровый антиген гепатита С (HBcAg) и малые иммунные белки (SIP), в растениях коровьего гороха (Mechtcheriakova et al. J. Virol. Methods 131, 10-15; 2006; Monger et al., 2006, Plant Biotechnol. J. 4, 623-631; Alamillo et al., 2006, Biotechnol. J. 1, 1103-1111). Несмотря на успешный результат, использование полноразмерного вирусного вектора ограничивает по размеру встраиваемые последовательности, а перемещения между растениями вызывает озабоченность биоизоляцией (биобезопасностью) вируса.
[0008] Чтобы устранить проблемы, связанные с биобезопасностью и размером вставки, Canizares et al. (2006 Plant Biotechnol, J 4:183-193) заменили  часть кодирующей области RNA-2 на нужную последовательность, чтобы получить деактивированный вариант CPMV RNA-2 (deIRNA-2). Последовательность для экспрессии связывали с AUG в положении 512 RNA-2, непосредственно выше (upstream) 3' нетранслируемой области (UTR), чтобы создать молекулу, которая имитирует RNA-2. Такие конструкции были способны к репликации при введении их в растения в присутствии RNA-1 и супрессора сайленсинга, и обеспечивали (направляли) синтез гетерологичных белков на значительном уровне (Sainsbury et al., 2008 Plant Biotechnol J 6:82-92).
часть кодирующей области RNA-2 на нужную последовательность, чтобы получить деактивированный вариант CPMV RNA-2 (deIRNA-2). Последовательность для экспрессии связывали с AUG в положении 512 RNA-2, непосредственно выше (upstream) 3' нетранслируемой области (UTR), чтобы создать молекулу, которая имитирует RNA-2. Такие конструкции были способны к репликации при введении их в растения в присутствии RNA-1 и супрессора сайленсинга, и обеспечивали (направляли) синтез гетерологичных белков на значительном уровне (Sainsbury et al., 2008 Plant Biotechnol J 6:82-92).
[0009] Мутация стартового кодона в положении 161 в векторе CPMV RNA-2 (U162C; НТ) повышает уровень экспрессии белка, кодируемого последовательностью, встроенной после стартового кодона в положении 512. Такая система способствует продуцированию чужеродных белков на высоком уровне при отсутствии необходимости в вирусной репликации, и она была названа CPMV-HT системой (Международная заявка WO 2009/087391; Sainsbury and Lomonossoff, 2008, Plant Physiol. 148, 1212-1218). В экспрессионных плазмидах pEAQ (Sainsbury et al., 2009, Plant Biotechnology Journal, 7, pp 682-693; патент США US 2010/0287670) экспрессируемую последовательность позиционируют между 5'UTR и 3'UTR. 5'UTR в ряду pEAQ несет мутацию U162C (НТ).
Сущность изобретения
[0010] Настоящее изобретение относится к экспрессии представляющих интерес белков в растениях. В настоящем изобретении также предусматриваются способы и композиции для получения представляющих интерес белков в растениях.
[0011] Согласно настоящему описанию в изобретении предусматривается энхансер экспрессии, содержащий нуклеотидную последовательность CPMV 5'UTR, состоящую из X нуклеотидов (CMPVX), где Х=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1, или состоящий из нуклеотидной последовательности, которая примерно на 80%-100% сходна с последовательностью CMPVX, где Х=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1. Энхансер экспрессии может содержать нуклеотидную последовательность, выбранную из группы, включающей SEQ ID NO: 24, 27, 68, 69, 70 and 71.
[0012] В настоящем изобретении также предусматривается энхансер экспрессии по определению выше, который также содержит спейсерную последовательность (CPMVX+, где Х=160, 155, 150, 114 нуклеотидов последовательности SEQ ID NO: 1). Спейсерная последовательность может иметь протяженность от 0 примерно до 100 нуклеотидов или любое промежуточное значение между этими двумя величинами, может содержать одну или более последовательностей (Мэрилин) Козак для растений, множественный сайт клонирования, одну или более линкерных последовательностей, один или более рекомбинационных сайтов или их комбинацию. В настоящем изобретении также предусматривается энхансер экспрессии по определению выше, при этом последовательность Козак выбрана из группы последовательностей, показанных как SEQ ID NO: 5-17. Энхансер экспрессии по определению выше (CPMVX+, где Х=160, 155, 150 или 114 из SEQ ID NO: 1) может содержать нуклеотидную последовательность, выбранную из группы, включающей SEQ ID NO: 2, 72, 73, 74, 75, 76 и 77.
[0013] Также предусматривается растительная система экспрессии (экспрессионная система на основе растений), содержащая нуклеотидную последовательность, включающую регуляторную область, функционально связанную с энхансером экспрессии CPMVX, CPMVX+, по определению выше, при этом энхансер экспрессии функционально связан с представляющей интерес нуклеотидной последовательностью. Растительная экспрессионная система может также содержать 3'UTR комовируса. Растительная экспрессионная система может также содержать вторую нуклеотидную последовательность, кодирующую супрессор сайленсинга, например, HcPro или р19.
[0014] Представляющая интерес нуклеотидная последовательность растительной системы экспрессии по определению выше может кодировать вирусный белок или антитело. Например, вирусный белок может представлять собой гемагглютинин гриппа и может быть выбран из группы, включающей H1, Н2, Н3, Н4, Н5, Н6, Н7, Н8, Н9, Н10, H11, Н12, Н13, H14, Н15, Н16 и гемагглютинин вируса гриппа типа В. Нуклеотидная последовательность, кодирующая вирусный белок или антитело, может содержать нативную (собственную, родную) сигнальную пептидную последовательность или ненативный сигнальный пептид, например, ненативный сигнальный пептид может быть получен из протеин-дисульфид-изомеразы (PDI, ПДИ).
[0015] Согласно настоящему изобретению предусматривается способ продуцирования представляющего интерес белка в растении или в части растения, включающий введение в растение или в часть растения растительной системы экспрессии, содержащей CPMVX или CPMVX+, по определению выше, и инкубацию растения или части растения в условиях, которые допускают экспрессию нуклеотидной последовательности, кодирующей белок, представляющий интерес.
[0016] В настоящем изобретении также предусматривается растение или часть растения, транзиторно трансфецируемые с использованием растительной системы экспрессии, описанной выше.
[0017] Экспрессионные системы на основе растений по данному описанию приводят к повышению или усилению экспрессии нуклеотидной последовательности кодирующей гетерологичной открытой рамки считывания, которая функционально связана с энхансером экспрессии, либо CPMVX, либо CPMVX+ по данному описанию. Повышение экспрессии можно определять, сравнивая уровень экспрессии, достигнутый с помощью энхансеров на основе CPMVX или на основе CPMVX+, с уровнем экспрессии той же самой нуклеотидной последовательности кодирующей гетерологичной рамки считывания, функционально связанной с энхансерной последовательностью из уровня техники (CPMV НТ), содержащей неполноценный М белок (описанный в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в данное изобретение посредством отсылки). Пример последовательности CPMV НТ из уровня техники приводится в SEQ ID NO: 4.
[0018] Экспрессионные системы на основе растений по данному описанию могут также обладать рядом свойств, например, содержать "удобные" сайты для клонирования генов, могут легко заражать (инфицировать) растения экономичным способом, могут вызывать эффективное локальное или системное заражение инокулированных растений. Кроме того, заражение должно обеспечить хороший выход полезного белкового материала.
[0019] В данном разделе "Раскрытие изобретения" необязательно описаны все признаки изобретения.
Краткое описание чертежей
[0020] Эти и другие признаки изобретения станут более очевидными с учетом нижеприведенного описания, в котором дается ссылка на прилагаемые фигуры, где:
[0021] На ФИГУРЕ 1А дается общее схематическое изображение нескольких энхансерных последовательностей, CPMVX и CPMVX+ (содержащих CPMVX, и спейсерный фрагмент, который не является ограничивающим примером, содержит множественный сайт клонирования и последовательность Козак для растений) по настоящему описанию. Каждый из СРМСХ и CPMVX+ показан как функционально связанный с регуляторной областью последовательности растений по их 5-' и их 3'-концам, последовательно, представляющая интерес нуклеотидная последовательность (включая инициирующий кодон ATG и СТОП-кодон), 3'UTR и терминаторная последовательность. Примером конструкции CPMVX по настоящему описанию является CPMV160. Примером конструкции CPMVX+ по настоящему описанию является CPMV160+. На ФИГУРЕ 1В даны примеры нескольких вариантов конструкций, содержащих энхансерные последовательности по настоящему описанию (CPMV160, полная последовательность, представленная как SEQ ID NO: 1; CPMV155, полная последовательность, представленная как SEQ ID NO: 24; CPMV150, полная последовательность, представленная как SEQ ID NO: 27; и CPMV114, полная последовательность, представленная как SEQ ID NO: 68), функционально связанные с областью регуляторной последовательности (регуляторной областью) растения (в данных неограничивающих примерах 2X35S) по их 5'-концам и их 3'-концам, представляющая интерес (изучаемая) нуклеотидная последовательность, или "GOI", которая включает последовательность Козак для растений, прилегающую к инициирующему сайту (стартовому кодону) ATG (элементы, показанные в квадратных скобках, включены в качестве контекста, и они не являются частью энхансерных последовательностей CPMVX или CPMVX+). На ФИГУРЕ 1С даны примеры некоторых вариантов конструкций, содержащих энхансерные последовательности по данному описанию (CPMV160+, полная последовательность, представленная как SEQ ID NO: 2; CPMV155+, полная последовательность, представленная как SEQ ID NO: 72; CPMV150+, полная последовательность, представленная как SEQ ID NO: 73; и CPMV114+, полная последовательность, представленная как provided as SEQ ID NO: 74), функционально связанные с регуляторной последовательностью (регуляторной областью) растения (в данных неограничивающих примерах 2X35S) по их 5'-концам и их 3'-концам, и спейсерный фрагмент (в данных неограничивающих примерах содержащий множественный сайт клонирования и последовательность Козак для растений), представляющая интерес изучаемая последовательность, "GOI", содержащая инициирующий (стартовый) кодон ATG (элементы, показанные в квадратных скобках, включены в качестве контекста, и они не являются частью энхансерных последовательностей CPMVX или CPMVX+).
[0022] На ФИГУРЕ 2 показан титр относительной гемагглютинации (HMG) в неочищенных экстрактах белков, продуцированных в растениях, содержащих экспрессионные конструкции на основе CPMV-HT (из уровня техники) и экспрессионные конструкции на основе CPMV160+, функционально связанные с изучаемой представляющей интерес нуклеотидной последовательностью. Представлены данные по экспрессии НА при использовании H1 A/California/07/2009 с PDI сигнальным пептидом (PDI-H1 Cal; конструкция номер 484 5'UTR: CPMV НТ; и конструкция номер 1897, 5'UTR: CPMV160+; см. Пример 5), Н3 A/Victoria/361/2011 с PDI сигнальным пептидом (PDI-H3 Vic; конструкция номер 1391, 5'UTR: CPMV НТ; и конструкция номер 1800, 5'UTR: CMPV160+; см. Примеры 1 и 2, соответственно), Н5 вируса гриппа А/(Indonesia)/5/2005 с нативным сигнальным пептидом (WtSp-H5 Indo; конструкция номер 489, 5'UTR: CMPV НТ; и конструкция номер 1880, 5'UTR: CMPV160+; см. Пример 6), и B/Wisconsin/1/2010 с делегируемой протеолитической петлей и с нативным сигнальным пептидом (WtSp-B Wis-PrL; конструкция номер 1445, 5'UTR: CMPV НТ; и конструкция номер 1975, 5'UTR: CMPV 160+, см. Пример 13). PDI: сигнальный пептид протеин-дисульфид-изомеразы; PrL-: делетируемая протеолитическая петля.
[0023] На ФИГУРЕ 3 показан титр относительной гемагглютинации (HMG) в неочищенных экстрактах белков, продуцированных в растениях, содержащих экспрессионные конструкции на основе CPMV-HT (из уровня техники) и экспрессионные конструкции на основе CPMV160+. Представлены данные по экспрессии НА при использовании H1 A/California/07/2009 с PDI сигнальным пептидом (конструкция номер 484, 5'UTR: CMPV НТ; и конструкция номер 1897 5'UTR: CMPV160+, см. Пример 5), Н3 A/Victoria/361/2011 с PDI сигнальным пептидом (конструкция номер 1391, 5'UTR: CMPV НТ; и конструкция номер 1800 5'UTR: CMPV 160+, см. Примеры 1 и 2, соответственно), В Brisbane/60/08 с делетируемой протеолитической петлей и с PDI сигнальным пептидом (конструкция номер 1039, 5'UTR: CMPV НТ; и конструкция номер 1937, 5'UTR: CMPV 160+; см. Пример 9), В Brisbane/60/08+H1Tm, с делетируемой протеолитической петлей, трансмембранный домен и цитоплазматический концевой сегмент заменены на трансмембранный домен и цитоплазматический концевой сегмент H1 A/California/07/2009, и с PDI сигнальным пептидом (конструкция номер 1067, 5'UTR: CMPV НТ; и конструкция номер 1977, 5'UTR: CMPV160+; см. Пример 10), В Massachusetts/2/2012 2012 с делетируемой протеолитической петлей и с PDI сигнальным пептидом (конструкция номер 2072, 5'UTR: CMPV НТ; и конструкция номер 2050, 5'UTR: CMPV 160+; см. Пример 11), В Massachusetts/2/2012+H1Tm с делетируемой протеолитической петлей, трансмембранный домен и цитоплазматический концевой сегмент заменены на трансмембранный домен и цитоплазматический концевой сегмент H1 A/California/07/2009 и с PDI сигнальным пептидом (конструкция номер 2074, 5'UTR: CMPV НТ; и конструкция номер 2060, 5'UTR: CMPV160+; см. Пример 12), В Wisconsin/1/2010 с делетируемой протеолитической петлей и с нативным сигнальным пептидом (конструкция номер 1445, 5'UTR: CMPV НТ; и конструкция номер 1975, 5'UTR: CMPV160+; см. Пример 13), и В Wisconsin/1/2010+H1Tm с делетируемой протеолитической петлей, трансмембранный домен и цитоплазматический концевой сегмент заменены на трансмембранный домен и цитоплазматический концевой сегмент H1 A/California/07/2009 и с нативным сигнальным пептидом (конструкция номер 1454, 5'UTR: CMPV НТ; и конструкция номер 1893, 5'UTR: CMPV160+; см. Пример 14).
[0024] На Фигуре 4А даны примеры вариантов тестированных последовательностей Козак в растениях. Конструкции включают частичную последовательность CPMVX+, регуляторную область последовательности растения, спейсерный фрагмент и изучаемую представляющую интерес нуклеотидную последовательность (GOI). В данном неограничивающем примере конструкция включает регуляторную область 2X35S, CPMV160+, спейсерный фрагмент, содержащий множественный сайт клонирования, и последовательность Козак для растений (также указан 5'-конец представляющей интерес (исследуемой) нуклеотидной последовательности: "ATG…GOI"; где GOI представляет собой Н3 A/Victoria/361). Варианты последовательностей Козак для растений также показаны под последовательностью (см. также Фигуру 9). Каждый вариант последовательности Козак для растений конденсировали с 3' концом спейсерного фрагмента и с 5'-ATG сайтом исследуемой нуклеотидной последовательности (в этих неограничивающих примерах Н3 A/Victoria/361). Другие элементы конструкций оставались теми же самыми). На ФИГУРЕ 4В показаны НА титры исследуемой (представляющей интерес) последовательности, продуцируемой в растениях, содержащей экспрессионную конструкцию на основе CMPV160+ и вариантную последовательность Козак для растений.
[0025] На ФИГУРЕ 5 показана экспрессия антитела ритуксимаба (ритуксана) под контролем CPMV-HT (конструкции номер 5001 и 5002, см. Примеры 15 и 16) или CPMV160 (номера конструкций 2100 и 2109, см. Примеры 15 и 16) и либо с нативным сигнальным пептидом, либо с сигнальным пептидом протеин-дисульфид-изомеразы (PDI), замещающим нативный сигнальный пептид.
[0026] На ФИГУРЕ 6 показаны компоненты последовательностей, использованные для создания конструкции номер 1391 (A-2X35S CPMV-HT PDISP H3 Victoria NOS; см. пример 1). Конструкция номер 1391 включает последовательность CPMV-HT из уровня техники (последовательность CMPV 5'UTR с мутантным старт-кодоном в положении 161 связана (соединена) с последовательностью, кодирующей неполноценный М белок, и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/H3 Victoria)). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминаторный кодон (терминатор, стоп-кодон) нопалин-синтазы. На ФИГУРЕ 6А показана последовательность праймера IF-PDI.S1=3c (SEQ ID NO: 67). На ФИГУРЕ 6В показана последовательность праймера IF-H3V36111.s1-4r (SEQ ID NO: 17). А ФИГУРЕ 6С показана последовательность PDISP/H3 Victoria (SEQ ID NO: 18). На ФИГУРЕ 6D схематически показана конструкция 1191. На ФИГУРЕ 6Е показана конструкция 1191; с левой до правой границ Т-ДНК (подчеркнуто), 2X35S CPMV-HT NOS, с кассетой экспрессии (экспрессионной кассетой) ингибитора сайленсинга Пластоцианин-Р19-Пластоцианина (SEQ ID NO: 19). На ФИГУРЕ 6F показана кассета экспрессии номер 1391 от промотора 2X35S до терминатора NOS. Подчеркнута нуклеотидная последовательность PDISP/H3 Victoria; CPMV 5'UTR показана жирным шрифтом; последовательность, кодирующая неполноценный М-белок, показана курсивом (SEQ ID NO: 20). На ФИГУРЕ 6G показана аминокислотная последовательность PDISP/H3 Victoria (SEQ ID NO: 21). На ФИГУРЕ 6Н схематически представлена конструкция номер 1391 (референсная (эталонная) конструкция).
[0027] На ФИГУРЕ 7 представлены компоненты последовательности, использованные для получения конструкции номер 1800 (A-2X35S CPMV160+ PDISP H3 Victoria NOS; см. Пример 2). Конструкция номер 1800 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминаторный кодон нопалин-синтазы. На ФИГУРЕ 7А показана последовательность праймера IF**(SacII)-PDI.s1+4c (SEQ ID NO: 22). На ФИГУРЕ 7В показана последовательность праймера IF-H3V36111.s1-4r (SEQ ID NO: 23). Последовательность PDISP/H3 Victoria показана на Фигуре 6С (SEQ ID NO: 18). На ФИГУРЕ 7С схематически представлена конструкция 2171 (указаны сайты узнавания рестриктазами SacII и StuI, используемые для линеаризации плазмиды). На ФИГУРЕ 7D показана конструкция 2171 с левой до правой границ Т-ДНК (подчеркнуто), 2X35S/CPMV160+/NOS с кассетой экспрессии ингибитора сайленсинга Пластоцианин-Р19-Пластоцианина, трансмембранным сегментом и цитоплазматическим хвостом H1 California и CPMV3'UTR (SEQ ID NO: 25). На ФИГУРЕ 7Е показана кассета экспрессии номер 1800 от 2X35S промотора до NOS терминатора. Подчеркнута нуклеотидная последовательность PDISP/H3 Victoria; 5'UTR последовательность показана жирным шрифтом; последовательность Козак для растений подчеркнута двойной чертой; спейсерный фрагмент (множественный сайт клонирования) из 16 пар оснований позиционирован между последовательностью 5'UTR и последовательностью Козак для растений (SEQ ID NO: 26). Аминокислотная последовательность PDISP/H3 Victoria показана на Фигуре 6G (SEQ ID NO: 27). На ФИГУРЕ 7F схематически представлена конструкция номер 1800 (конструкция на основе CPMVX+, где Х=160).
[0028] На ФИГУРЕ 8 показаны компоненты последовательности, используемые для получения конструкции номер 1935 (2X35S/CPMV160/PDISP/H3 Victoria/NOS; см. Пример 3). Конструкция номер 1935 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, и не включает спейсерный фрагмент (множественный сайт клонирования) или последовательность Козак для растений (эта конструкция также не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160 (CPMVX, где Х=160). PDISP: сигнальный белок протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы. На ФИГУРЕ 8А показана последовательность праймера IF-CPMV(fl5'UTR)_SpPDI.c (SEQ ID NO: 28). На ФИГУРЕ 8В схематически представлена конструкция 1190. На ФИГУРЕ 8С нуклеотидная последовательность конструкции 1190 с левой до правой границ Т-ДНК (подчеркнуто), 2X35S/CPMV160/NOS с кассетой экспрессии ингибитора сайленсинга Пластоцианин-Р19-Пластоцианина и CPMV3'UTR (SEQ ID NO: 29). На ФИГУРЕ 8D показана кассета экспрессии 1935 от 2X35S промотора до NOS терминатора. Нуклеотидная последовательность PDISP/H3 Victoria подчеркнута, 5'UTR последовательность показана жирным шрифтом (SEQ ID NO: 30). Эта кассета не включает последовательность Козак для растений или спейсерный фрагмент (множественный сайт клонирования). На ФИГУРЕ 8Е схематически показана конструкция номер 1935 (конструкция на основе CPMVX, где Х=160).
[0029] На ФИГУРЕ 9 показаны последовательности, содержащие варианты в последовательности Козак для растений, используемые для разработки отбора конструкций на основе "CPMV160+" (конструкций от номера 1992 до номера 1999). Показаны изменения в последовательности между сайтом рестрикции SacII и ATG PDISP/H3 Victoria в системах экспрессии 2X35S/CPMV160+/NOS, включающие варианты (изменения) в последовательности Козак для растений (последовательности показаны как модификации соответствующей последовательности из конструкции 1800; см. Пример 4). Последовательность Козак для растений в варианте подчеркнута. PDISP: сигнальный пептид протеин-дисульфид-изомеразы. На ФИГУРЕ 9А нуклеотидная последовательность IF-HT1*(-Mprot)-PDI.c (SEQ ID NO: 31; используемая для создания конструкции номер 1992). На ФИГУРЕ 9В показана нуклеотидная последовательность IF-HT2*(-Mprot)-PDI.c (SEQ ID NO: 32; используемая для создания конструкции номер 1993). На ФИГУРЕ 9С показана нуклеотидная последовательность IF-HT3*(-Mprot)-PDI.c (SEQ ID NO: 33; используемая для создания конструкции номер 1994). На ФИГУРЕ 9D показана нуклеотидная последовательность IF-HT4*(-Mprot)-PDI.c (SEQ ID NO: 34; используемая для создания конструкции номер 1995). На ФИГУРЕ 9Е показана нуклеотидная последовательность IF-HT5*(-Mprot)-PDI.c (SEQ ID NO: 35; используемая для создания конструкции номер 1996). На ФИГУРЕ 9F показана нуклеотидная последовательность IF-HT6*(-Mprot)-PDI.c (SEQ ID NO: 36; используемая для создания конструкции номер 1997). На ФИГУРЕ 9G показана нуклеотидная последовательность IF-HT7*(-Mprot)-PDI.c (SEQ ID NO: 37; используемая для создания конструкции номер 1998). На ФИГУРЕ 9Н показана нуклеотидная последовательность IF-HT8*(-Mprot)-PDI.c (SEQ ID NO: 38; используемая для создания конструкции номер 1999). На ФИГУРЕ 9I схематически показана конструкция номер 1992, содержащая последовательность Козак для растений (Kozak1), с использованием SEQ ID NO: 31 (ФИГУРА 9А). Конструкции 1993-1999 содержат те же признаки, что и конструкция 1992, за исключением того, что каждая конструкция (1993-1999) содержит модифицированную последовательность Козак для растений (Kozak1), показанную на Фигурах с 9В до 9Н (SEQ ID NOs: с 32 до 38), соответственно.
[0030] На ФИГУРЕ 10 показаны компоненты последовательности, используемые для получения конструкций номер 484 и номер 1897 (2X35S/CPMV НТ PDISP/H1 California NOS и 2X35S/CPMV160+ PDISP/H1 California NOS, соответственно; см. Пример 5). Конструкция номер 484 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак для растений между последовательностью 5'UTR и исследуемой (представляющей интерес) нуклеотидной последовательностью (PDISP/H1 California). Конструкция номер 1897 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполный белок М) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы. На ФИГУРЕ 10А показана нуклеотидная последовательность PDISP/H1 California (SEQ ID NO: 39). На ФИГУРЕ 10В показана аминокислотная последовательность PDISP/H1 California (SEQ ID NO: 40). На ФИГУРЕ 10С схематически представлена конструкция номер 484 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 10D схематически представлена конструкция номер 1897 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0031] На ФИГУРЕ 11 показаны компоненты последовательности, применяемые для получения конструкций номер 489, номер 1880 и номер 1885 (2X35S/CPMV НТ Н5 Indonesia NOS; CPMV160+ Н5 Indonesia NOS, и CPMV160 Н5 Indonesia NOS, соответственно; см. Пример 6). Конструкция номер 489 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/H1 California). Конструкция номер 1880 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок), и является примером конструкции на основе CPMV 160+ (CPMVX+, где Х=160). Конструкция номер 1885 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, и не включает спейсерный фрагмент (множественный сайт клонирования) или последовательность Козак для растений (эта конструкция не включает также последовательность, кодирующую неполноценный М белок) и является примером конструкции на основе CPMV160 (CPMVX, где Х=160). NOS: терминатор нопалин-синтазы. На ФИГУРЕ 11А показана нуклеотидная последовательность нативного Н5 Indonesia (SEQ ID NO: 41). На ФИГУРЕ 11В показана аминокислотная последовательность нативного Н5 Indonesia (SEQ ID NO: 42). На ФИГУРЕ 11С схематически показана конструкция номер 489 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 11D схематически показана конструкция номер 1880 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160). На ФИГУРЕ 11Е схематически показана конструкция номер 1885 (2X35S/CPMV160, конструкция на основе CPMVX, где Х=160).
[0032] На ФИГУРЕ 12 показаны компоненты последовательности, используемые для создания конструкций номер 1240 и номер 2168 (2X35S/CPMV НТ PDISP/H7 Hangzhou NOS и 2X35S/CPMV160+ PDISP/H7 Hangzhou NOS, соответственно; см. Пример 7). Конструкция номер 1240 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связывается с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой последовательностью (PDISP/H7 Hangzhou). Конструкция номер 1897 включает CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы. На ФИГУРЕ 12А показана нуклеотидная последовательность PDISP/H7 Hangzhou (SEQ ID NO: 43). На ФИГУРЕ 12В показана аминокислотная последовательность PDISP/H7 Hangzhou (SEQ ID NO: 44). На ФИГУРЕ 12С схематически представлена конструкция номер 2140 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 12D схематически представлена конструкция номер 2168 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0033] На ФИГУРЕ 13 показаны компоненты последовательности, используемые для создания конструкций номер 2130 и номер 2188 (2X35S/CPMV НТ PDISP/H7 Hangzhou + H5 Indonesia ТМСТ NOS и 2X35S/CPMV160+ PDISP/H7 Hangzhou + H5 Indonesia ТМСТ NOS, соответственно; см. Пример 8). Конструкция номер 2130 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным старт-кодоном в положении 161 связывается с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/H7 Hangzhou + H5 Indonesia ТМСТ). Конструкция номер 1897 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы; ТМСТ: трансмембранный домен и цитоплазматический хвост (концевой сегмент). На ФИГУРЕ 13А показана нуклеотидная последовательность PDISP/H7 Hangzhou + H5 Indonesia ТМСТ (SEQ ID NO: 45). На ФИГУРЕ 13В показана аминокислотная последовательность PDISP/H7 Hangzhou + H5 Indonesia ТМСТ (SEQ ID NO: 46). На ФИГУРЕ 13С схематически представлена конструкция номер 2130 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 13D схематически представлена конструкция номер 2188 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0034] На ФИГУРЕ 14 показаны компоненты последовательности, используемые для создания конструкций номер 1039 и номер 1937 (2X35S/CPMV НТ PDISP/HA В Brisbane (PrL-) NOS и 2X35S/CPMV160+ PDISP/HA В Brisbane (PrL-) NOS, соответственно; см. Пример 9). Конструкция номер 1039 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связывается с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/HA В Brisbane (PrL-)). Конструкция номер 1937 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV 160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы; PrL-: делетируемая протеолитическая петля. На ФИГУРЕ 14А показана нуклеотидная последовательность PDISP/HA В Brisbane (PrL-) (SEQ ID NO: 47). На ФИГУРЕ 14B показана аминокислотная последовательность PDISP/HA В Brisbane (PrL-) (SEQ ID NO: 48). На ФИГУРЕ 14C схематически представлена конструкция номер 1039 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 14D схематически представлена конструкция номер 1937 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0035] На ФИГУРЕ 15 показаны компоненты последовательности, используемые для создания конструкций номер 1067 и номер 1977 (2X35S/CPMV НТ PDISP/HA В Brisbane (Prl-) + H1 California ТМСТ NOS и 2X35S/CPMV160+PDISP/HA В Brisbane (PrL-) + H1 California ТМСТ NOS, соответственно; см. Пример 10). Конструкция номер 1067 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным старт-кодоном в положении 161 связывается с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/HA В Brisbane (PrL-) + Н1 California ТМСТ). Конструкция номер 1977 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы; PrL-: делетируемая протеолитическая петля; ТМСТ: цитоплазматический хвост трансмембранного домена. На ФИГУРЕ 15А показана нуклеотидная последовательность PDISP/HA В Brisbane (PrL-) + Н1 California ТМСТ (SEQ ID NO: 49). На ФИГУРЕ 15В показана аминокислотная последовательность PDISP/HA В Brisbane (PrL-) + H1 California ТМСТ (SEQ ID NO: 50). На ФИГУРЕ 15C схематически представлена конструкция номер 1067 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 15D схематически представлена конструкция номер 1977 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0036] На ФИГУРЕ 16 показаны компоненты последовательности, используемые для создания конструкций номер 2072 и номер 2050 (2X35S/CPMV НТ PDISP/HA В Massachusetts (PrL-) NOS и 2X35S/CPMV160+ PDISP/HA В Massachusetts (PrL-) NOS, соответственно; см. Пример 11). Конструкция номер 2072 включает последовательность CPMV-HT из уровня техники (последовательность CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/HA В Massachusetts (PrL-)). Конструкция номер 2050 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы. NOS: терминатор нопалин-синтазы; PrL-: делетируемая протеолитическая петля. На ФИГУРЕ 16А показана нуклеотидная последовательность PDISP/HA В Massachusetts (PrL-) (SEQ ID NO: 51). На ФИГУРЕ 16В показана аминокислотная последовательность PDISP/HA В Massachusetts (PrL-) (SEQ ID NO: 52). На ФИГУРЕ 16C схематически представлена конструкция номер 2072 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 16D схематически представлена конструкция номер 2050 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0037] На ФИГУРЕ 17 показаны компоненты последовательности, используемые для создания конструкций номер 2074 и номер 2060 (2X35S/CPMV НТ PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ NOS и 2X35S/CPMV160+ PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ NOS, соответственно; см. Пример 12). Конструкция номер 2074 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 слита с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ). Конструкция номер 2060 включает последовательность, CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы; NOS: терминатор нопалин-синтазы; PrL-: делетируемая протеолитическая петля; ТМСТ: трансмембранный домен и цитоплазматический хвост. На ФИГУРЕ 17А показана нуклеотидная последовательность PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ (SEQ ID NO: 53). На ФИГУРЕ 17В показана аминокислотная последовательность PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ (SEQ ID NO: 54). На ФИГУРЕ 17C схематически представлена конструкция номер 2074 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 17D схематически представлена конструкция номер 2060 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0038] На ФИГУРЕ 18 показаны компоненты последовательности, используемые для создания конструкций номер 1445, номер 1820 и номер 1975 (2X35S/CPMV НТ НА В Wisconsin (PrL-) NOS, 2X35S/CPMV160+ НА В Wisconsin (PrL-) NOS и 2X35S/CPMV160 НА В Wisconsin (PrL-) NOS, соответственно; см. Пример 13). Конструкция номер 1445 включает последовательность CPMV-HT из уровня техники (последовательность CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующую неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и исследуемой нуклеотидной последовательностью (НА В Wisconsin (PrL-)). Конструкция номер 1820 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). Конструкция номер 1975 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, и не включает спейсерный фрагмент (множественный сайт клонирования) или последовательность Козак для растений (эта конструкция также не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе "CPMV160" (CPMVX). PrL-: делетируемая протеолитическая петля; NOS: терминатор нопалин-синтазы. На ФИГУРЕ 18А показана нуклеотидная последовательность НА В Wisconsin (PrL-) (SEQ ID NO: 55). На ФИГУРЕ 18В показана аминокислотная последовательность НА В Wisconsin (PrL-) (SEQ ID NO: 56). Ha ФИГУРЕ 18C схематически представлена конструкция номер 1445 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 18D схематически представлена конструкция номер 1820 (2X35S/CPMV160+; конструкция на основе CPMVX+). На ФИГУРЕ 18Е схематически представлена конструкция номер 1975 (2X35S/CPMV160; конструкция на основе CPMVX, где Х=160).
[0039] На ФИГУРЕ 19 показаны компоненты последовательности, используемые для создания конструкций номер 1454 и номер 1893 (2X35S/CPMV НТ НА В Wisconsin (PrL-) + Н1 California ТМСТ NOS и 2X35S/CPMV160+ НА В Wisconsin (PrL-) + H1 California ТМСТ NOS, соответственно; см. Пример 14). Конструкция номер 1454 включает последовательность CPMV-HT из уровня техники (последовательность CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и изучаемой нуклеотидной последовательностью (НА В Wisconsin (PrL-) + H1 California ТМСТ). Конструкция номер 1893 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). NOS: терминатор нопалин-синтазы; PrL-: делетируемая протеолитическая петля; ТМСТ: трансмембранный домен и цитоплазматический хвост. На ФИГУРЕ 19А показана нуклеотидная последовательность НА В Wisconsin (PrL-) + H1 California ТМСТ (SEQ ID NO: 57). Ha ФИГУРЕ 19В показана аминокислотная последовательность PDISP/HA В Wisconsin (PrL-) + H1 California ТМСТ (SEQ ID NO: 58). На ФИГУРЕ 19C схематически представлена конструкция номер 1454 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 19D схематически представлена конструкция номер 1893 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0040] На ФИГУРЕ 20 показаны компоненты последовательности, используемые для создания конструкций номер 5001 и номер 2100 (2X35S/CPMV НТ НС ритуксимаба (ритуксан) NOS и 2X35S/CPMV160+ НС ритуксимаба (Ритуксан) NOS, соответственно; см. Пример 15). Конструкция номер 5001 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и изучаемой нуклеотидной последовательностью (НС ритуксимаба (Ритуксан)). Конструкция номер 2100 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). НС: тяжелая цепь; NOS: терминатор нопалин-синтазы. На ФИГУРЕ 20А показана нуклеотидная последовательность НС ритуксимаба (Ритуксан; SEQ ID NO: 59). На ФИГУРЕ 20В показана аминокислотная последовательность НС ритуксимаба (Ритуксан; SEQ ID NO: 60). На ФИГУРЕ 20С схематически представлена конструкция номер 5001 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 20D схематически представлена конструкция номер 2100 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0041] На ФИГУРЕ 21 показаны компоненты последовательности, используемые для создания конструкций номер 5002 и номер 2109 (2X35S/CPMV НТ PDISP/HC ритуксимаба (ритуксан) NOS и 2X35S/CPMV160+ PDISP/HC ритуксимаб (Ритуксан) NOS, соответственно; см. Пример 16). Конструкция номер 5001 включает последовательность CPMV-HT из уровня техники (последовательность CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связывается с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и изучаемой нуклеотидной последовательностью (PDISP/HC Rituzan (Ритуксан?)). Конструкция номер 2100 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (этак конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы; НС: тяжелая цепь; NOS: терминаторный кодон нопалин-синтазы. На ФИГУРЕ 21А показана нуклеотидная последовательность PDISP/HC ритуксимаба (Ритуксан; SEQ ID NO: 61). На ФИГУРЕ 21В показана аминокислотная последовательность PSISP/HC ритуксимаба (Ритуксан; SEQ ID NO: 62). На ФИГУРЕ 21С схематически представлена конструкция номер 5002 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 21D схематически представлена конструкция номер 2109 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0042] На ФИГУРЕ 22 показаны компоненты последовательности, используемые для создания конструкций номер 5021 и номер 2120 (2X35S/CPMV НТ LC ритуксимаба (Ритуксан) NOS и 2X35S/CPMV160+ LC ритуксимаба (Ритуксан) NOS, соответственно; см. Пример 17). Конструкция номер 5021 включает последовательность CPMV-HT из уровня техники (CMPV 5'UTR с мутантным кодоном в положении 161 связана с последовательностью, кодирующей неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и изучаемой нуклеотидной последовательностью (LC ритуксимаба (Ритуксан)). Конструкция номер 2120 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). LC: легкая цепь; NOS: терминатор нопалин-синтазы. На ФИГУРЕ 22А показана нуклеотидная последовательность LC ритуксимаба (Ритуксан; SEQ ID NO: 63). На ФИГУРЕ 22В показана аминокислотная последовательность LC ритуксимаба (Ритуксан; SEQ ID NO: 64). На ФИГУРЕ 22С схематически представлена конструкция номер 5021 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 22D схематически представлена конструкция номер 2120 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
[0043] На ФИГУРЕ 23 показаны компоненты последовательности, используемые для создания конструкций номер 5022 и номер 2129 (2X35S/CPMV НТ PDISP/LC ритуксимаба (Ритуксан) NOS и 2X35S/CPMV160+ PDISP/LC ритуксимаба (Ритуксан) NOS, соответственно; см. Пример 18). Конструкция номер 5001 включает последовательность CPMV-HT (CMPV 5'UTR с мутантным стартовым кодоном в положении 161 связана с последовательностью, кодирующую неполноценный М белок) и не содержит гетерологичной последовательности Козак между 5'UTR и изучаемой нуклеотидной последовательностью (PDISP/LC ритуксимаба (Ритуксан)). Конструкция номер 2100 включает последовательность CPMV 5'UTR, содержащую 160 нуклеотидов, спейсерный фрагмент (множественный сайт клонирования) и последовательность Козак для растений (эта конструкция не содержит последовательности, кодирующей неполноценный М белок) и является примером конструкции на основе CPMV160+ (CPMVX+, где Х=160). PDISP: сигнальный пептид протеин-дисульфид-изомеразы; НС: тяжелая цепь; NOS: терминатор нопалин-синтазы. На ФИГУРЕ 23А показана нуклеотидная последовательность PDISP/LC ритуксимаба (Ритуксан; SEQ ID NO: 65). На ФИГУРЕ 23В показана аминокислотная последовательность PSISP/LC ритуксимаба (Ритуксан; SEQ ID NO: 66). На ФИГУРЕ 23С схематически представлена конструкция номер 5022 (2X35S/CPMV НТ; референсная конструкция). На ФИГУРЕ 23D схематически представлена конструкция номер 2129 (2X35S/CPMV160+; конструкция на основе CPMVX+, где Х=160).
Подробное описание изобретения
[0044] Настоящее изобретение относится к экспрессии представляющих интерес белков в растениях. В настоящем изобретении также предусматриваются способы и композиции для продуцирования представляющих интерес белков в растениях.
[0045] В приведенном далее описании широко используется ряд терминов, указанные ниже определения даются с целью облегчить понимание различных аспектов изобретения. Примеры, приведенные в описании, включая примеры терминов, даются лишь с целью иллюстрации и не претендуют на ограничение объема и смысла вариантов настоящего изобретения.
[0046] В данном контексте слова, определяющие существительное единственном числе, в сочетании с термином "содержащий" могут означать "один", но они также соответствуют значению "один или более", "по меньшей мере один" и "один или более одного". Термин "примерно", "около" относится к отклонению приблизительно +/-10% от данного значения. Термины "совокупность", "ряд" означают более одного, например, два или более, три или более, четыре или более и т.п.
[0047] В настоящем изобретении предусматривается энхансер экспрессии, содержащий CPMV 5' нетранслируемую область (UTR), "CPMVX", содержащую X нуклеотидов последовательности SEQ ID NO: 1, где X=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1 или последовательности, которая на 80%-100% сходна с последовательностью CPMVX, где Х=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1. Этот энхансер экспрессии обычно именуется CPMVX (См. Фигуру 1А).
[0048] Энхансерная последовательность CPMVX может быть также связана со спейсерной последовательностью, причем CMPVX содержит X нуклеотидов последовательности SEQ ID NO: 1, где Х=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1 или последовательности, которая на 80%-100% сходна с последовательностью CPMVX, где Х=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1, и спейсерная последовательность содержит от 1 до 100 нуклеотидов, связанных с 3'-концом CMPVX последовательности. Например, спейсерная последовательность может содержать примерно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 85, 90, 95 или 100 нуклеотидов или любое число нуклеотидов между этими значениями.
[0049] Если последовательность CMPVX содержит спейсерный фрагмент, тогда этот энхансер экспрессии можно обозначать как CPMVX+ (см. Фигуру 1А), где Х=160, 155, 150, 114 нуклеотидов последовательности SEQ ID NO: 1, его можно также называть как последовательность CMPVX, содержащую спейсерную последовательность, или его можно обозначать как CPMV160+; CPMV155+; CPMV150+; CPMV114+, когда Х-160, 155, 150 или 114, соответственно. Конструкции, содержащие CPMVX, которые не включают спейсерную последовательность, можно обозначать (называть) CPMVX+, где Х=160, 155, 150, 114 последовательности SEQ ID NO: 1 и где длина (протяженность) спейсерной последовательности составляет 0 нуклеотидов.
[0050] Спейсерная последовательность может быть модифицирована процессингом (процессированием), делецией или заменой нативной CMPV5'UTR последовательности, расположенной 3' к нуклеотиду 160. Модифицированную спейсерную последовательность можно удалить, заменить, процессировать или сделать более короткой по сравнению с исходной или немодифицированной (т.е. нативной) спейсерной последовательностью, ассоциированной с 5'UTR (описанной в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218). Спейсерная последовательность может содержать один или более сайтов рестрикции (полилинкер, множественный сайт клонирования, один или более сайтов клонирования), одну или более последовательностей Козак для растений, одну или более линкерных последовательностей, один или более сайтов рекомбинации или их комбинацию. Например, но это не следует рассматривать как ограничение, спейсерная последовательность может содержать, последовательно, множественный сайт клонирования нужной длины, связанный с последовательностью Козак для растений. Спейсерная последовательность не содержит нуклеотидной последовательности из нативной 5'UTR последовательности, которая позиционирована 3' к нуклеотиду 160 нативной последовательности CPMV 5'UTR, например, нуклеотиды с 161 до 512, как показано на Фигуре 1 в публикации Sainsbury F., and Lomonossoff G.P. (2008, Plant Physiol. 148: pp. 1212-1218; которая включена в настоящую заявку посредством отсылки), или нуклеотиды 161-509 последовательности SEQ ID NO: 4. То есть неполноценный М белок, присутствующий в последовательности CPMV НТ из уровня техники (Фигура 1 в публикации Sainsbury F., and Lomonossoff G.P., 2008) удален из 5'UTR по настоящему изобретению.
[0051] Энхансер экспрессии CPMVX, или CPMVX+, может быть функционально связан на 5'конце энхансерной последовательности с регуляторной областью, которая активна в растении, и функционально связан с представляющей интерес (изучаемой) нуклеотидной последовательностью на 3'конце энхансера экспрессии (Фигура 1А), для того, чтобы управлять экспрессией представляющей интерес последовательности в растении-хозяине.
[0052] Также предусматриваются экспрессионные системы для продуцирования одного или более представляющих интерес белков в растительных клетках с применением либо CMPVX, либо CPMVX+. Экспрессионные системы по данному описанию содержат кассету экспрессии, содержащую CPMVX, или последовательность, которая на 80% сходна с последовательностью CPMVX, и, необязательно, спейсерная последовательность связана с CMPVX (CPMVX+). Экспрессионная кассета, содержащая CMPVX или CMPVX+, может также содержать регуляторную область, активную в растении, которая функционально связана с 5'концом энхансера экспрессии. Представляющая интерес изучаемая нуклеотидная последовательность может быть функционально связана с 3'концом кассеты экспрессии с тем, чтобы после введения в растительную клетку в растении осуществлялась экспрессия представляющей интерес нуклеотидной последовательности.
[0053] В настоящем изобретении также предусматриваются растительные клетки, растительные ткани, цельные растения, инокулят (посевной материал, посевная культура), нуклеиновые кислоты, конструкции, содержащие представляющие интерес нуклеотидные последовательности, кассеты экспрессии или экспрессионные системы, содержащие CPMVX or CPMVX+ по настоящему описанию и способы осуществления экспрессии представляющего интерес белка в растениях.
[0054] Что касается Фигур 1А, 1В и 1С, то на них представлены неограничивающие примеры энхансера экспрессии, содержащего последовательность CPMV 5'UTR (CPMVX), включающую нуклеотиды из X нуклеотидов последовательности SEQ ID NO: 1, где Х=160, 155, 150 или 114 последовательности SEQ ID NO: 1. Энхансер экспрессии CMPVX может обозначаться как CPMV160; CPMV155; CPMV150; CPMV114, если Х=160, 155, 150 или 114, соответственно.
[0055] Представляющая интерес нуклеотидная последовательность может быть соединена, связана (функционально связана) с энхансерной последовательностью, содержащей регуляторную область растений, различными методами. Например, но без ограничения:
1) Представляющая интерес нуклеотидная последовательность, кодирующая представляющий интерес белок, может быть связана с 3'концом энхансера экспрессии непосредственно после 5'UTR последовательности, например, CPMVX, где Х=160, 155, 150, 114 нуклеотидов последовательности SEQ ID NO: 1. В данном примере представляющая интерес нуклеотидная последовательность связывается с 5'UTR без множественного сайта клонирования, и представляющая интерес нуклеотидная последовательность может включать на своем 5'конце последовательность Козак для растений непосредственно выше (upstream, 5') инициирующего (стартового) кодона ATG представляющей интерес нуклеотидной последовательности (см. Фигуру 1В). Если Х=160 (т.е. CPMV160), тогда представляющей интерес нуклеотидной последовательности, которая функционально связана с CPMV160, может быть не нужна последовательность Козак для растений, связанная с ее 5' концом, поскольку нуклеотиды 150-160, или 155-160, последовательности SEQ ID NO: 1 содержат последовательность, подобную последовательности Козак. Однако при необходимости последовательность Козак для растений может быть включена в конструкции, содержащие CPMV160 (см. Фигуру 1В: "+/-послед. Козак для растений"). Если Х=155, 150 или 114, тогда, для оптимальной экспрессии представляющей интерес нуклеотидной последовательности, рекомендуется включение последовательности Козак для растений, связанной с 5'концом представляющей интерес нуклеотидной последовательности, в конструкции, содержащие CPMV155, CPMV150 или CPMV114.
2) Представляющая интерес нуклеотидная последовательность может быть связана с CMPVX+ энхансером экспрессии (где Х=160, 155, 150, 114 нуклеотидов последовательности SEQ ID NO: 1), который содержит последовательность Козак для растений на своем 3' конце, таким образом, что представляющая интерес нуклеотидная последовательность расположена непосредственно после последовательности Козак для растений. В данном примере представляющая интерес нуклеотидная последовательность, которая связана с CPMVX+, не включает множественный сайт клонирования или последовательность Козак для растений (полученная конструкция будет аналогичной конструкции, представленной на Фигуре 1В).
3) Представляющая интерес нуклеотидная последовательность может быть связана с CPMVX+ энхансером экспрессии (где Х=160, 155, 150, 114 нуклеотидов последовательности SEQ ID NO: 1), содержащим множественный сайт клонирования (MCS) на своем 3'конце, с помощью множественного сайта клонирования. В данном примере представляющая интерес нуклеотидная последовательность может включать на своем 5' конце соответствующую последовательность, которая допускает связывание с множественным сайтом клонирования энхансера экспрессии, и последовательность Козак для растений непосредственно выше ("upstream") инициирующего (стартового) кодона ATG представляющей интерес нуклеотидной последовательности (см. Фигуру 1С).
[0056] В целом результатом любого из вышеприведенных методов является конструкция (или кассета экспрессии), содержащая регуляторную область растения, находящуюся в функциональной связи (функционально связанную) с последовательностью CPMV 5'UTR, содержащей нуклеотиды X, где Х=160, 155, 150, 114 последовательности SEQ ID NO: 1 (или энхансерной последовательностью, которая на 80% сходна с последовательностью CPMV 5'UTR), 3' конец последовательности CPMV 5'UTR связан с 5' концом последовательности Козак для растений, 3' конец последовательности Козак для растений связан с 5' концом и прилегает к 5' концу представляющей интерес нуклеотидной последовательности, содержащей инициирующую последовательность ATG. Конструкция может содержать, или может не содержать множественный сайт клонирования, локализованный между 5'UTR и последовательностью Козак для растений. Конструкция может также содержать последовательность 3' нетранслируемой области (UTR), например, 3'UTR комовируса или 3'UTR пластоцианина, и терминаторную последовательность, например, NOS терминаторную последовательность, функционально связанную с 3' концом представляющей интерес нуклеотидной последовательности (см. Фигуру 1А).
[0057] Также предусматривается растительная экспрессионная система, содержащая нуклеиновую кислоту, включающую регуляторную область, функционально связанную с одним или более энхансеров экспрессии по настоящему описанию (например, CPMVX), и представляющую интерес нуклеотидную последовательность. Также описывается нуклеиновая кислота, содержащая промоторную последовательность (регуляторную область), функционально связанную с энхансером экспрессии, содержащим CPMV 5'UTR и модифицированную или делетируемую спейсерную последовательность (например, CPMVX+) и представляющую интерес нуклеотидную последовательность. Нуклеиновая кислота может содержать также последовательность, кодирующую 3'UTR, например 3'UTR комовируса или 3'UTR пластоцианина, и терминаторную последовательность, например, NOS терминатор, так что представляющая интерес нуклеотидная последовательность вставлена выше ("upstream") 3'UTR.
[0058] Под выражением "функционально связанный" понимают, что специфические последовательности взаимодействуют либо непосредственно, либо опосредованно, чтобы осуществить целевую функцию, например, такую как опосредование или модуляция экспрессии нуклеотидной последовательности. Взаимодействие функционально связанных последовательностей может быть опосредовано, например, белками, которые взаимодействуют с функционально связанными последовательностями.
[0059] В данном контексте выражения "энхансер(-ы) экспрессии", "энхансерная(-ые) последовательность(-и)" или "энхансерный(-е) элемент(-ы)" включают последовательности, полученные из участков или обладающие сходством с последовательностями участков CPMV 5'UTR сегмента RNA-2 (РНК-2) генома. Энхансерная последовательность может усиливать экспрессию 3' ("downstream") гетерологичной открытой рамки считывания (ORF), с которой она связана.
[0060] Термин "5'UTR", или "5' нетранслируемая последовательность", или "5' лидерная последовательность" относится к нетранслируемым областям мРНК. Обычно 5'UTR начинается в сайте начала транскрипции и заканчивается непосредственно перед сайтом инициации трансляции или стартовым кодоном (обычно AUG в последовательности мРНК, ATG в последовательности ДНК) кодирующей области. Протяженность 5'UTR можно модифицировать с помощью мутации, например, замены, делеции или вставки (инсерции) 5'UTR. 5'UTR можно также модифицировать посредством мутации стартового кодона или сайта инициации трансляции природных нуклеиновых кислот (мРНК, ДНК), так что кодон более не функционирует как стартовый кодон, и трансляция может начинаться с другого сайта инициации.
[0061] 5'UTR из нуклеотидов 1-160 последовательности CPMV RNA-2 (SEQ ID NO: 1), начинается с сайта начала транскрипции до первого узнаваемого стартового кодона в рамке (в положении 161), который служит сайтом инициации для продуцирования более длинного из двух имеющих общие карбокси-концы (котерминальных) белков, кодируемых сегментом генома комовируса. Кроме того, 'третий' сайт инициации в положении 115 (или соответствующем ему положении) в CPMV RNA-2 геномной последовательности также может быть мутантным, делегированным или по-другому измененным. Было показано, что удаление AUG 115, помимо удаления AUG 161, усиливает экспрессию в сочетании с неполноценным М белком (Sainsbury and Lomonossoff, 2008, Plant Physiology; 148: 1212-1218; Международная заявка WO 2009/087391; которая включена в настоящее изобретение посредством отсылки).
[0062] Энхансер экспрессии может содержать CPMV 5' нетранслируемую область (UTR), включающую нуклеотиды из X последовательности SEQ ID NO: 1, где Х=160, 155, 150 или 114 последовательности SEQ ID NO: 1 (CPMVX), или последовательность, которая обладает 80%-ным сходством с последовательностью CPMVX (где Х=160, 155, 150 или 114 нуклеотидов последовательности SEQ ID NO: 1; см. Фигуры 1А и 1В) и проявляет свойство усиления экспрессии нуклеотидной последовательности кодирующей гетерологичной открытой рамки считывания, которая функционально связана с энхансером экспрессии, по сравнению с экспрессией той же самой нуклеотидной последовательности кодирующей гетерологичной открытой рамки считывания, которая функционально связана с энхансерной последовательностью CPMV НТ из уровня техники, содержащей неполноценный М белок (как описано в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в настоящее изобретение посредством отсылки).
[0063] Энхансерная последовательность CPMVX может быть также связана со спейсерной последовательностью, например, с множественным сайтом клонирования (MCS), или MCS, связанным с последовательностью Козак для растений, при этом CMPVX содержит нуклеотиды из X нуклеотидов последовательности SEQ ID NO: 1, где Х=160, 155, 150 или 114 последовательности SEQ ID NO: 1, или последовательность, которая на 80% сходна с последовательностью CPMVX (where Х=160, 155, 150, or 114 of SEQ ID NO: 1) и проявляет свойство усиления экспрессии нуклеотидной последовательности кодирующей гетерологичной открытой рамки считывания, функционально связанной с энхансером экспрессии, по сравнению с экспрессией той же самой нуклеотидной последовательности кодирующей гетерологичной открытой рамки считывания, которая функционально связана с энхансерной последовательностью CPMV НТ из уровня техники, содержащей неполноценный М белок (как описано в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в настоящее изобретение посредством отсыпки). Спейсерная последовательность содержит от 0 до 500 нуклеотидов, связанных с 3' концом CMPVX последовательности. Предпочтительно, спейсерная последовательность содержит множественный сайт клонирования (MCS), или MCS, связанный с последовательностью Козак для растений и не включает М белок. Если CMPVX последовательность содержит спейсерный фрагмент (без М белка), тогда этот энхансер экспрессии может именоваться (обозначаться) как "CPMVX+" (см. Фигуры 1А м 1С), как "CMPVX, содержащую спейсерную последовательность и последовательность Козак для растений ", или как "CMPVX, содержащую MCS наряду с последовательностью Козак для растений".
[0064] Энхансер экспрессии CPMVX, где Х=160, состоит из нуклеотидов 1-160 последовательности SEQ ID NO: 1:
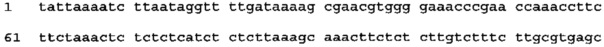
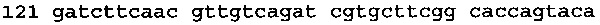 (SEQ ID NO: 1)
(SEQ ID NO: 1)
Если энхансер экспрессии состоит из нуклеотидов 1-160 последовательности SEQ ID NO: 1 (CPMV160), тогда представляющая интерес нуклеотидная последовательность, в присутствии или в отсутствие 5' последовательности Козак для растений, локализованной на 5' конце, прилегающей к инициирующей последовательности (ATG), может быть связана с 3' концом 5'UTR (после нуклеотида 160 последовательности SEQ ID NO: 1), так что целая конструкция напоминает конструкцию, показанную на Фигуре 1B (CPMV160). Конструкция, содержащая CPMV160, может также содержать регуляторную область, функционально связанную с 5' концом энхансера экспрессии, и последовательность, кодирующую 3'UTR, например, 3' нетранслируемую область (UTR) комовируса или 3'UTR пластоцианина, и терминаторную последовательность, например, NOS терминатор, связанный с 3' концом представляющей интерес последовательности. Без связи с какой-либо теорией можно сказать, что CPMV160 может не потребоваться добавление последовательности Козак для растений на 5' конце представляющей интерес последовательности, так как последовательность в положениях 150-155, 155-160 или 150-160 нуклеотиды последовательности SEQ ID NO: 1 могут действовать как активная (нативная) последовательность Козак в растении. Конструкция номер 1935 (см. Пример 3) и конструкция номер 1885 (см. Пример 6) являются примерами конструкций на основе CPMV160 (CPMVX, где Х=160).
[0065] Энхансер экспрессии может представлять собой CPMVX+, где Х=160. Неограничивающим примером такого энхансера является CPMV160+ (см. Фигуру 1С), содержащий последовательность SEQ ID NO: 2 (5'UTR: нуклеотид 1-160; множественный сайт клонирования, показан курсивом, нуклеотиды 161-176; последовательность Козак для растений показана жирным шрифтом заглавными буквами, нуклеотиды 177-181):

 (SEQ ID NO: 2)
(SEQ ID NO: 2)
[0066] Примеры конструкций, использующих последовательность SEQ ID NO: 2 в качестве энхансера экспрессии, включают конструкции 1800, 1897, 1880, 2168, 2188, 1937, 1977, 2050, 2060, 1975, 1893, 2100, 2109, 2120, 2129 (см. Пример 3 и 5-18, соответственно).
[0067] Как будет очевидно для специалиста в данной области техники, любой множественный сайт клонирования (MCS) или MCS различной длины (либо короче, либо длиннее) можно использовать вместо нуклеотидов 161-176 последовательности SEQ ID NO: 2. Далее, последовательность Козак для растений в SEQ ID NO: 2 (показана как нуклеотиды 177-181) может являться любой последовательностью Козак для растений, включая, но без ограничения, одну из последовательностей, выбранных из SEQ ID NO's: 5-17 (см. также Фигуру 4А; конструкция на Фигуре 4 включает SEQ ID NO: 2, с указанными вариантами последовательности Козак для растений, и содержит регуляторную область растения, связанную с 5' концом 5'UTR, и сайт инициации транскрипции, ATG, представляющей интерес нуклеотидной последовательности, локализованный 3' к последовательности Козак для растений).
[0068] Энхансер экспрессии, CPMVX, может включать нуклеотид "А" в положении 115 (115А), вследствие чего CMPVX, 115А, где Х=160, 155 или 150, содержит последовательность дикого типа CPMV RNA2 генома (см. Международную заявку WO 2009/087391, которая включена в настоящее изобретение посредством отсылки, о полной последовательности сегмента дикого типа CPMV RNA-2 генома). Примером энхансера экспрессии CPMVX, 115А является "CPMV160, 115А", по определению SEQ ID NO: 69 (нуклеотид "А" показан жирным шрифтом и подчеркнут):
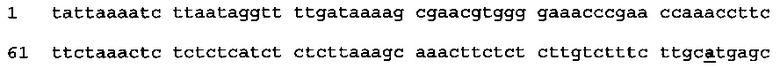
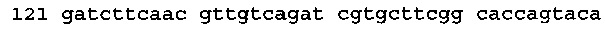 (SEQ ID NO: 69)
(SEQ ID NO: 69)
[0069] Энхансер экспрессии CPMVX+также может включать нуклеотидный остаток "А" в положении 115 (115А), так что CMPVX+, 115А, где Х=160, 155 или 150, содержит последовательность дикого типа CPMV RNA2 генома (Международная заявка WO 2009/087391, которая включена в настоящее изобретение посредством отсылки). Неограничивающим примером энхансера экспрессии CPMVX+, 115А является "CPMV160+, 115А", как определено в SEQ ID NO: 75 (нуклеотид "А" показан жирным шрифтом и подчеркнут):

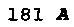 (SEQ ID NO: 75)
(SEQ ID NO: 75)
[0070] Как отмечалось выше для последовательности SEQ ID NO: 2, любой MCS, или MCS другой длины может применяться вместо MCS последовательности SEQ ID NO: 75, и последовательность Козак для растений может представлять собой любую последовательность Козак для растений.
[0071] Если энхансер экспрессии состоит из нуклеотидов 1-155 последовательности SEQ ID NO: 1 (CPMV 155):
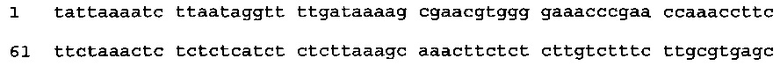
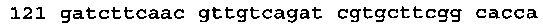 (SEQ ID NO: 24),
(SEQ ID NO: 24),
тогда представляющая интерес нуклеотидная последовательность с последовательностью Козак для растений, локализованной на 5' конце, прилегающем к инициирующей последовательности (ATG), может быть связана с 3' концом 5'UTR (после нуклеотида 155 последовательности SEQ ID NO: 1), так что общая конструкция напоминает конструкцию, показанную на Фигуре 1B (CPMV155). Конструкция, содержащая CPMV155, может также содержать регуляторную область, функционально связанную с 5' концом энхансера экспрессии, и последовательность, кодирующую 3'UTR, например, 3' нетранслируемую область (UTR) комовируса или 3' UTR пластоцианина, и терминаторную последовательность, например, NOS терминаторную последовательность, связанную с 3' концом представляющей интерес последовательности. В данном примере представляющая интерес нуклеотидная последовательность содержит последовательность Козак для растений на своем 5' конце, так как нативная последовательность Козак или участок этой последовательности (нуклеотиды 155-160 of SEQ ID NO: 1) удалены.
[0072] Энхансер экспрессии может представлять собой конструкцию CPMV 155+, содержащую SEQ ID NO: 72 (5'UTR: нуклеотиды 1-155; множественный сайт клонирования, показанный курсивом, 156-171; последовательность Козак для растений, показанная заглавными буквами и жирным шрифтом, нуклеотиды 172-176):
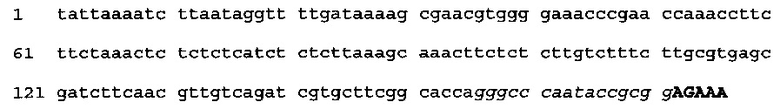
(SEQ ID NO: 72)
[0073] Как указывалось выше, для CPMV160+ (SEQ ID NO: 2), любой MCS, или MCS другой длины, может применяться вместо MCS последовательности SEQ ID NO: 72, и последовательность Козак для растений может представлять собой любую последовательность Козак для растений.
[0074] Энхансер экспрессии CPMV 155 может включать нуклеотид "А" в положении 115 (115А), так что "CMPV155, 115А" содержит последовательность дикого типа CPMV RNA2 генома (см. Международную заявку WO 2009/087391, которая включена в данный патент посредством отсылки), как определено в SEQ ID NO: 70 ("А" показан жирным шрифтом и подчеркнут):
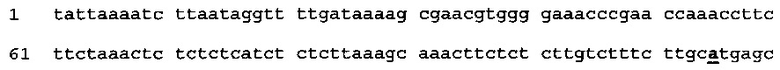
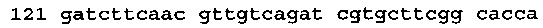 (SEQ ID NO: 70)
(SEQ ID NO: 70)
[0075] Энхансер экспрессии CPMV155+ может также включать нуклеотид "А" в положении 115 (115А), так что "CMPV155+, 115а" содержит последовательность дикого типа CPMV RNA2 генома (см. Международную заявку WO 2009/087391, которая включена в данный патент посредством отсылки), как определено в SEQ ID NO: 76 (нуклеотид "А" показан жирным шрифтом и подчеркнут):
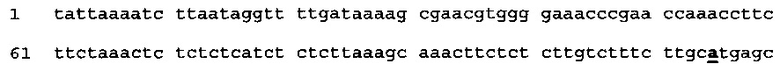
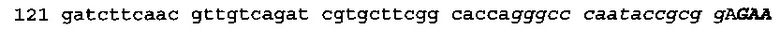
 (SEQ ID NO: 76)
(SEQ ID NO: 76)
[0076] Как указано выше для SEQ ID NO: 2, любой MCS, или MCS другой длины, может применяться вместо MCS последовательности SEQ ID NO: 76, и последовательность Козак для растений может представлять собой любую последовательность Козак для растений.
[0077] Если энхансер экспрессии состоит из нуклеотидов 1-150 последовательности SEQ ID NO: 1 (CPMV 150):
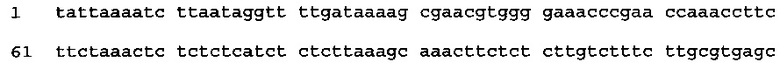
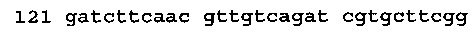 (SEQ ID NO: 27),
(SEQ ID NO: 27),
тогда представляющая интерес нуклеотидная последовательность с последовательностью Козак для растений, локализованной на 5' конце, прилегающем к инициирующей последовательности (ATG), может быть связана с 3' концом 5'UTR (после нуклеотида 150 последовательности SEQ ID NO: 1), так что общая конструкция напоминает конструкцию, показанную на Фигуре 1B (CPMV150). Конструкция, содержащая CPMV150, может также содержать регуляторную область, функционально связанную с 5' концом энхансера экспрессии, и последовательность, кодирующую 3'UTR, например, 3' нетранслируемую область (UTR) комовируса или 3' UTR пластоцианина, и терминаторную последовательность, например, NOS терминаторную последовательность, связанную с 3' концом представляющей интерес нуклеотидной последовательности. В данном примере представляющая интерес нуклеотидная последовательность содержит последовательность Козак для растений на своем 5' конце, так как нативная последовательность Козак (нуклеотиды 150-160 of SEQ ID NO: 1) удалена.
[0078] Энхансер экспрессии может представлять собой CPMV150+, содержащий последовательность SEQ ID NO: 73 (5'UTR: нуклеотид 1-150; множественный сайт клонирования, показан курсивом, нуклеотиды 156-166; последовательность Козак для растений, показанная заглавными буквами жирным шрифтом, нуклеотиды 167-171):
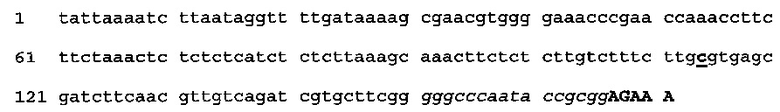
(SEQ ID NO: 73)
[0079] Как указывается выше для CPMV160+ (SEQ ID NO: 2), любой MCS, или MCS другой длины, может применяться вместо MCS последовательности SEQ ID NO: 73, и последовательность Козак для растений может представлять собой любую последовательность Козак для растений.
[0080] Энхансер экспрессии CPMV 150 может включать нуклеотид "А" в положении 115 (115А), так что "CMPV150, 115А" содержит последовательность дикого типа CPMV RNA2 генома (см. Международную заявку WO 2009/087391, которая включена в данный патент посредством отсылки), как определено в SEQ ID NO: 71 (нуклеотид "А" показан жирным шрифтом и подчеркнут):
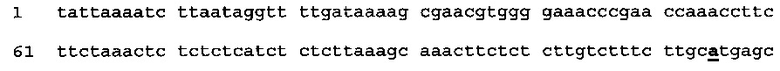
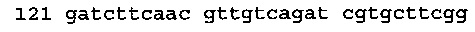 (SEQ ID NO: 71)
(SEQ ID NO: 71)
[0081] Энхансер экспрессии CPMV150+ может также включать нуклеотид "А" в положении 115 (115А), так что "CMPV150+, 115А" содержит последовательность дикого типа CPMV RNA2 генома (см. Международную заявку WO 2009/087391, которая включена в данный патент посредством отсылки), как определено в SEQ ID NO: 77 (нуклеотид "А" показан жирным шрифтом и подчеркнут):

 (SEQ ID NO: 77)
(SEQ ID NO: 77)
[0082] Как отмечается выше для SEQ ID NO: 2, любой MCS, или MCS другой длины, может применяться вместо MCS последовательности SEQ ID NO: 73, и последовательность Козак для растений может представлять собой любую последовательность Козак для растений.
[0083] Если энхансер экспрессии состоит из нуклеотидов 1-114 последовательности SEQ ID NO: 1:
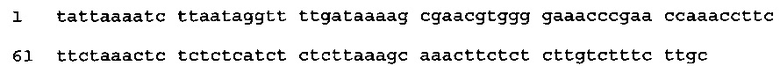
(SEQ ID NO: 68)
тогда представляющая интерес нуклеотидная последовательность с последовательностью Козак для растений, локализованной на 5' конце, прилегающем к инициирующей последовательности (ATG), может быть связана с 3' концом 5'UTR (после нуклеотида 114 последовательности SEQ ID NO: 1), так что общая конструкция напоминает конструкцию, показанную на Фигуре 1B (CPMV114). Конструкция, содержащая CPMV114, может также содержать регуляторную область, функционально связанную с 5' концом энхансера экспрессии, и последовательность, кодирующую 3'UTR, например, 3' нетранслируемую область (UTR) комовируса или 3'UTR пластоцианина, и терминаторную последовательность, например, NOS терминаторную последовательность, связанную с 3' концом представляющей интерес нуклеотидной последовательности. В данном примере представляющая интерес нуклеотидная последовательность содержит последовательность Козак для растений на своем 5' конце, так как последовательность, подобная последовательности Козак, находится 5' к нуклеотиду 114 последовательности SEQ ID NO: 1.
[0084] Энхансер экспрессии может представлять собой CPMV114+, содержащий последовательность SEQ ID NO: 74 (5'UTR: нуклеотид 1-114; множественный сайт клонирования, показанная курсивом, нуклеотиды 115-130; последовательность Козак для растений, показанная заглавными буквами жирным шрифтом, нуклеотиды 131-135):

(SEQ ID NO: 74)
[0085] Как указано выше для CPMV160+ (SEQ ID NO: 2), любой MCS, или MCS другой длины, может применяться вместо MCS последовательности SEQ ID NO: 73, и последовательность Козак для растений может представлять собой любую последовательность Козак для растений.
[0086] Энхансер экспрессии может также содержать нуклеотиды 1-160 последовательности SEQ ID NO: 1, связанные с последовательностью Козак для растений, локализованной ниже (3', 'downstream") положения 160 последовательности SEQ ID NO: 1. Последовательность Козак для растений может быть локализована таким образом, чтобы непосредственно прилегать к нуклеотиду 160 последовательности SEQ ID NO: 1, или энхансер экспрессии может содержать спейсерный фрагмент, включающий примерно от 0 примерно до 500 нуклеотидов, или любое количество нуклеотидов между этими значениями, непосредственно прилегающий к нуклеотиду 160 последовательности SEQ ID NO: 1 (CPMVX+), и последовательность Козак для растений, связанную с 3' концом спейсерного фрагмента. Спейсерный фрагмент может содержать множественный сайт клонирования (MCS), включающий примерно от 4 примерно до 100 нуклеотидов или любое количество нуклеотидов между этими значениями, и представляющая интерес нуклеотидная последовательность, содержащая последовательность Козак для растений и соответствующий сайт клонирования на своем 5' конце, может быть функционально связана с энхансером экспрессии CMPVX посредством MCS, или спейсерный фрагмент может содержать множественный сайт клонирования, включающий от примерно 4 до 100 нуклеотидов, связанных с последовательностью Козак для растений, а представляющая интерес нуклеотидная последовательность может быть связана с энхансером экспрессии непосредственно 3' (ниже, "downstream") последовательности Козак для растений. Предпочтительно, спейсерный фрагмент не содержит последовательности, кодирующей М белок.
[0087] Примером конструкции, который не следует рассматривать, как ограничивающий, содержащей, последовательно: регуляторную область растения, связанную с последовательностью CPMV 5'UTR, состоящей из нуклеотидов 1-160 SEQ ID NO: 1, которая связана со спейсерным фрагментом, является CPMV160+, показанная на Фигуре 1С (на Фигуре 1С, ATG, стартовый сайт (кодон) представляющей интерес нуклеотидной последовательности "GOI", также показан для наглядности). В этом примере спейсерный фрагмент связан с 3' концом CPMV 1-160 последовательности и содержит, последовательно, множественный сайт клонирования, связанный с последовательностью Козак для растений (в данном примере, который не следует считать ограничивающим, последовательность Козак для растений представляет собой: AGAAA). Спейсерный фрагмент не содержит какой-либо последовательности, кодирующей М белок. Если конструкция CPMV160+ связана с представляющей интерес нуклеотидной последовательностью (как показано на Фигуре 1С), тогда последовательность Козак для растений расположена 5' к представляющей интерес нуклеотидной последовательности и прилегает к ATG инициирующему кодону (сайту) представляющей интерес нуклеотидной последовательности. Как должно быть ясно специалисту в данной области техники, множественный сайт клонирования может содержать один или более подходящих сайтов рестрикции, а последовательность множественного сайта клонирования не ограничивается примером, показанным на Фигуре 1С. Кроме того, последовательность Козак для растений может представлять собой любую последовательность Козак для растений, а не ограничиваться последовательностью, представленной на Фигуре 1С. Конструкции номер 1800, 1897, 1880, 2168, 2188, 1937, 1977, 2050, 2060, 1975, 1893, 2100, 2109, 2120, 2129 (см. Примеры 3 и 5-18, соответственно) являются примерами конструкций на основе CPMV160+ (CPMVX+, где Х=160).
[0088] На Фигуре 1С также представлен пример энхансеров экспрессии CPMV155+, CPMV150+ и CPMV114+, каждый из которых содержит нуклеотиды 1-155, 1-150 или 1-114 последовательности SEQ ID NO: 1, соответственно, связанные со спейсерным фрагментом способом, аналогичным способу, описанному выше для CPMV160+. На Фигуре 1С также показан стартовый сайт (кодон) ATG представляющей интерес нуклеотидной последовательности (GOI) для каждой из конструкций CPMV155+, CPMV150+ и CPMV114+. В этих примерах спейсерный фрагмент, связанный с 3' концом CPMV энхансерной последовательности содержит, последовательно, множественный сайт клонирования, связанный с последовательностью Козак для растений. Спейсерный фрагмент не содержит какой-либо последовательности, кодирующей М белок. Как должно быть ясно специалисту в данной области техники, множественный сайт клонирования может содержать один или более подходящих сайтов рестрикции, а последовательность множественного сайта клонирования не ограничивается примером, показанным на Фигуре 1С. Кроме того, последовательность Козак для растений может представлять собой любую последовательность Козак для растений, а не ограничиваться последовательностью, представленной на Фигуре 1С (AGAAA).
[0089] Энхансер экспрессии может также представлять собой энхансер экспрессии CPMVX, где Х=160, 155, 150 или 114 последовательности SEQ ID NO: 1, в сочетании с множественным сайтом клонирования (полилинкер, сайт рестрикции; сайт клонирования), связанным с 3' концом последовательности 5'UTR и не содержащим последовательности Козак для растений (т.е. CPMVX+, где Х=160, 155, 150 или 114 последовательности SEQ ID NO: 1). В этих случаях нуклеотидная последовательность, кодирующая представляющий интерес белок (представляющая интерес нуклеотидная последовательность), которая предназначена для присоединения к энхансеру, содержит, последовательно от 5' конца к 3' концу представляющей интерес нуклеотидной последовательности, множественный сайт клонирования (комплементарный сайту спейсерного фрагмента; спейсерный фрагмент не содержит какой-либо последовательности, кодирующей М белок), связанный с последовательностью Козак для растений, расположенной 5' (выше, "upstream") и прилегающий к ATG сайту инициации (сайт инициации транскрипции) представляющей интерес нуклеотидной последовательности.
[0090] Энхансер экспрессии может содержать также одну или более "консенсусных последовательностей Козак", или "последовательностей Козак". Последовательности Козак играют главную роль в инициации трансляции. Скорость трансляции можно оптимизировать, если обеспечить, чтобы любые мРНК нестабильные последовательности были исключены из трансгенной конструкции и чтобы сайт инициации трансляции, или участок инициации, был совместим с консенсусной последовательностью Козак для растений (Gutierrrez, R.A. et al., 1999, Trends Plant Sci. 4, 429-438; Kawaguchi, R. and Bailey-Serres, J., 2002, Curr. Opin. Plant Biol. 5, 460-465). Наиболее высококонсервативным остатком в этом мотиве является пурин (который чаще всего представляет собой А) в положении на три нуклеотида выше (5', "upstream") кодона ATG, который служит сигналом начала трансляции (Kozak, М., 1987, J. Mol. Biol. 20:947-950, вводится в настоящее изобретение посредством отсылки). Консенсусные последовательности Козак для растений известны в уровне техники (см., например Rangan et al. Mol. Biotechnol., 2008, July 39(3), pp. 207-213). Как природные, так и синтетические последовательности Козак могут использоваться в энхансере экспрессии или могут связываться с представляющей интерес нуклеотидной последовательностью по настоящему изобретению.
[0091] Последовательность Козак для растений может являться любой известной последовательностью Козак для растений (см., например, L. Rangan et. al. Mol. Biotechnol., 2008, July 39(3), pp. 207-213), включая, но без ограничения, следующие консенсусные последовательности для растений:
саА(А/С)а (SEQ ID NO: 5; царство растений)
ааА(А/С)а (SEQ ID NO: 6; двудольные растения)
aa(A/G)(А/С)а (SEQ ID NO: 7; арабидопсис)
Последовательность Козак для растений может быть выбрана также из группы (см. Фигуру 4):
AGAAA (SEQ ID NO: 8)
AGACA (SEQ ID NO: 9)
AGGAA (SEQ ID NO: 10)
AAAAA (SEQ ID NO: 11)
AAACA (SEQ ID NO: 12)
AAGCA (SEQ ID NO: 13)
AAGAA (SEQ ID NO: 14)
AAAGAA (SEQ ID NO: 15)
AAAGAA (SEQ ID NO: 16)
(A/-)A(A/G)(A/G)(A/C)A. (SEQ ID NO: 3; Консенсусная последовательность)
[0092] Энхансер экспрессии может содержать также один или более элементов, называемых: "сайт(ы) рестрикции", или "сайт(ы) узнавания рестриктазами", "множественный сайт клонирования", "MCS", "сайт(ы) клонирования", "полилинкерная последовательность" или "полилинкер" с целью способствовать инсерции (вставке, включению) целевого нуклеотида в растительную экспрессионную систему. Сайты рестрикции представляют собой мотивы со специфической последовательностью, которые узнаются рестриктазами как общеизвестные в уровне техники. Энхансер экспрессии может содержать один или более сайтов рестрикции, или сайтов клонирования, которые расположены ниже (3', "downstream") последовательности 5'UTR. Один или более сайтов рестрикции, или сайтов клонирования, может быть также локализован выше (5') одной или более последовательностей Козак, и располагаться между 5'UTR и последовательностью Козак. Полилинкерная последовательность (множественный сайт клонирования) может содержать любую последовательность нуклеиновых кислот, которые пригодны для добавления и удаления нуклеотидных последовательностей, включая нуклеотидную последовательность, кодирующую представляющий интерес (целевой) белок, к 3' концу (и от 3' конца) последовательности 5'UTR. Полилинкерная последовательность может содержать от 4 до примерно 100 нуклеиновых кислот или любое количество между этими значениями.
[0093] Энхансер экспрессии может также содержать последовательность SEQ ID NO: 1, функционально связанную с регуляторной областью у растений и сайтом начала транскрипции (ATG), связанным с представляющей интерес нуклеотидной последовательностью (GOI), как показано на Фигуре 1B (CPMVX; где Х=160, 155, 150 или 114). CPMVX может также содержать любую последовательность Козак для растений, включая, но без ограничения, одну из последовательностей, выбранных из SEQ ID NO's: 5-17.
[0094] 5'UTR последовательность для применения в энхансере экспрессии по настоящему описанию (CPMVX or CPMVX+, where Х=160, 155, 150 или 144) может быть получена при использовании двухкомпонентного РНК-содержащего вируса, например, сегмента RNA-2-генома двухкомпонентного РНК-содержащего вируса, такого как комовирус, при условии, что эта последовательность на 100%, 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентична последовательности, представленной либо SEQ ID NO: 1, либо SEQ ID NO: 2. Например, идентичность между энхансерной последовательностью и последовательностями SEQ ID NO's: 1 и 2 может составлять от примерно 80% до примерно 100%, или любое значение между этими величинами, идентичность последовательностям SEQ ID NO's: 1 и 2 может составлять от примерно 90% до примерно 100%, или любое значение между этими величинами, идентичность последовательностям SEQ ID NO's: 1 и 2 может составлять от примерно 95% до примерно 100%, или любое значение между этими величинами, идентичность последовательностям SEQ ID NO's: 1 и 2 может составлять от примерно 98% до примерно 100%, или любое значение между этими величинами, при этом энхансер экспрессии, будучи функционально связан с регуляторной последовательностью растений и последовательностью Козак для растений по настоящему описанию, повышает уровень экспрессии представляющей интерес нуклеотидной последовательности, которая функционально связана с энхансером экспрессии, по сравнению с уровнем экспрессии представляющей интерес нуклеотидной последовательности, связанной с CMPV НТ (SEQ ID NO: 4; энхансерной последовательностью из уровня техники, содержащей неполноценный М белок, описанной в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в настоящее изобретение посредством отсылки), при использовании той же самой регуляторной области растений.
[0095] SEQ ID NO: 4 содержит CPMV НТ энхансер экспрессии, известный из уровня техники (см., например, Фигуру 1 в публикации Sainsbury and Lomonossoff 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в настоящее изобретение посредством отсылки). "CPMV НТ" включает 5'UTR последовательность из нуклеотидов 1-160 последовательности SEQ ID NO: 4 с модифицированными нуклеотидами в положениях 115 (gtg) и 162 (acg), и (последовательность, кодирующую) неполноценный М белок, и не содержит последовательности Козак для растений (5'UTR: нуклеотиды 1-160; (последовательность, кодирующая) неполноценный М белок, подчеркнута, нуклеотиды 161-509). SEQ ID NO: 4 включает также множественный сайт клонирования (изображен курсивом, нуклеотиды 510-528), который не присутствует в последовательности CPMV НТ из уровня техники:

[0096] Конструкции, содержащие CPMV НТ, применяются в данном изобретении в качестве референсных конструкций с тем, чтобы можно было сравнивать уровниэкспрессии представляющей интерес нуклеотидной последовательности, или продукта, кодируемого представляющей интерес нуклеотидной последовательностью, продуцируемых с использованием конструкции, содержащей CPMVX или CPMVX+. Конструкции 1391, 484, 489, 2140, 2130, 1039, 1067, 2072, 2074, 1445, 1454, 5001, 5002, 5021 и 5022 (см. Примеры 1 и 5-18, соответственно) содержат референсную конструкцию CPMV НТ.
[0097] Как показано на Фигурах 2-5, применение энхансеров экспрессии по данному изобретению привело к повышению экспрессии представляющей интерес нуклеотидной последовательности по сравнению с экспрессией той же самой представляющей интерес нуклеотидной последовательности с использованием тех же самых промоторных и 3'UTR и терминаторных последовательностей. Например, на Фигурах 2, 3 и 5, дается сравнение экспрессии белков, продуцируемых в растениях, с использованием экспрессионных конструкций, содержащих CPMV-HT (из уровня техники), и экспрессионных конструкций на основе CPMV160+, функционально связанных с ними (конструкциями):
H1 A/California/07/2009 ("PDI-H1 Cal", или "H1 A/California/07/2009"): конструкция на основе CPMV160+ номер 1897, конструкция на основе CPMV НТ номер 484 (см. Пример 5);
Н3 A/Victoria/361/2011 ("PDI-H3 Vic", or "Н3 A/Victoria/361/2011"): конструкция на основе CPMV160+ номер 1800; конструкция на основе CPMV НТ номер 1391 (см. Примеры 1 и 2, соответственно);
Н5 вируса гриппа A/Indonesia/5/2005 с нативным сигнальным пептидом (WtSp-H5 Indo): конструкция на основе CPMV160+ номер 1880; конструкция на основе CPMV НТ номер 489 (см. Пример 6);
B/Wisconsin/1/2010 с делетируемой протеолитической петлей и с нативным сигнальным пептидом ("WtSp-B Wis-PrL", или "B/Wisconsin/1/2010"): конструкция на основе CPMV160+ номер 1975; конструкция на основе CPMV НТ номер 1445 (см. Пример 13);
В Brisbane/60/08 с делетируемой сигнальной петлей и с PDI сигнальным пептидом ("В Brisbane/60/08"): конструкция на основе CPMV160+ номер 1937; конструкция на основе CMPV НТ номер 1039 (см. Пример 9);
В Brisbane/60/08+H1Tm, с делетируемой протеолитической петлей, связанной с трансмембранным доменом и цитоплазматическим хвостом и с PDI сигнальным пептидом ("В Brisbane/60/08+H1Tm"): конструкция на основе CPMV160+ номер 1977; конструкция на основе CMPV НТ номер 1067 (см. Пример 10),
В Massachusetts/2/2012 2012 с делетируемой протеолитической петлей и с PDI сигнальным пептидом ("В Massachusetts/2/2012 2012"): конструкция на основе CPMV160+ номер 2050; конструкция на основе CPMV НТ номер 2072 (см. Пример 11),
В Massachusetts/2/2012+H1Tm с делетируемой протеолитической петлей, связанной с трансмембранным доменом и цитоплазматическим хвостом и с PDI сигнальным пептидом ("В Massachusetts/2/2012+H1Tm"): конструкция на основе CPMV160+ номер 2060; конструкция на основе CPMV НТ номер 2074 (см. Пример 12),
В Wisconsin/1/2010+H1Tm с делетируемой протеолитической петлей, связанной с трансмембранным доменом и цитоплазматическим хвостом и с нативным сигнальным пептидом ("В Wisconsin/1/2010+H1Tm"): конструкция на основе CPMV160+ номер 1893; конструкция на основе CPMV НТ номер 1454 (см. Пример 14);
Ритуксимаб (Ритуксан) под контролем CPMV-HT с нативным или PDI сигнальным пептидом ("CPMV-HT/SP дикого типа" и "CPMV-HT/PDISP"; конструкции номер 5001 и 5002, соответственно, см. Примеры 15 и 16), или CPMV160+ ("CPMV160+/SP дикого типа" и "CPMV160+/PDISP"; конструкции номер 2100 и 2109, соответственно, см. Примеры 15 и 16).
[0098] В каждом случае уровень экспрессии (определенной как активность гемагглютинации или экспрессия ритуксимаба (ритуксана, Rituxan), если это происходит) увеличивается в конструкции на основе CMPV160+ по сравнению с экспрессией известной конструкции на основе CPMV. Кроме того, некоторые из нуклеотидных последовательностей, представляющих интерес, кодировали химерные или модифицированные белки, например, содержащие гетерологичные сигнальные пептиды (например, PDI), гетерологичные последовательности трансмембранной области и цитоплазматического хвоста (TDCT) и/или модифицированные последовательности, включающие делегируемую протеолитическую петлю (PrL-).
[0099] Повышение уровня экспрессии, наблюдаемое при использовании конструкций на основе CPMV160+, выявлено также, когда последовательность Козак для растений, применяемая в упомянутых конструкциях на основе CPMV160+, заменена другими последовательностями Козак для растений, например, одной из последовательностей Козак для растений, представленных SEQ ID NO: 8-16. Например, на Фигуре 4 дается сравнение уровня экспрессии белков, продуцируемых в растениях, включающих конструкции на основе CPMV160+, функционально связанные с нуклеотидной последовательностью, представляющей интерес (Н3 A/Victoria/361), при этом каждая из них соединена с различными последовательностями Козак для растений. В каждом случае экспрессия (определенная как титр гемагглютинации) конструкции на основе CMPV160+ характеризуется значительными уровнями экспрессии, которые превышают уровень экспрессии известной конструкции на основе CMPV НТ.
[00100] Термины "процент сходства" или "процент идентичности" (когда они используются для конкретной последовательности, применяются, например, так, как отражено в компьютерной программе Висконсинского университета (University of Wisconsin) GCG, или при ручном выравнивании и визуальном изучении (см., например, Current Protocols in Molecular Biology, Ausubel et al., eds. 1995 supplement). Методы выравнивания последовательностей для сравнения хорошо известны в предшествующем уровне техники. Оптимальное выравнивание последовательностей для сравнения может быть осуществлено, например, с применением алгоритма Смита-Ватермана (1981, Adv. Appl. Math. 2:482), алгоритма выравнивания Нидлмана-Вунша (Needleman & Wunsch, 1970, J. Mol. Biol. 48:443), метода сходства Пирсона и Липмана (Pearson & Lipman, (1988, Proc. Nat'l. Acad. Sci. USA 85:2444)), с помощью компьютерной реализации этих алгоритмов (например: GAP, BESTFIT, FASTA и TFASTA в программе Winconsin Genetics Software Package, Genetics Computer Group (GCG), 575 Science Dr., Madison, Wis.).
[00101] Примерами алгоритмов, подходящих для определения процента идентичности последовательностей и сходства последовательностей являются алгоритмы BLAST и BLAST 2.0, которые описаны в публикациях Altschul et al., (1977, Nuc. Acids Res. 25:3389-3402) и Altschul et al., (1990, J. Mol. Biol. 215:403-410), соответственно. BLAST и BLAST 2.0 используются вместе с параметрами, описанными в этих публикациях, для определения процента идентичности последовательностей в нуклеиновых кислотах и белках по изобретению. Например, в компьютерной программе BLASTN (для нуклеотидных последовательностей) в качестве параметров по умолчанию могут использоваться: длина слова (W), равная 11, ожидание (Е), равное 10, М=5, N=-4 и сравнение обеих нитей. Для аминокислотных последовательностей в программе BLASTP в качестве параметров по умолчанию могут быть использованы: длина слова, равная 3, ожидание (Е), равное 10, и выравнивания (В) с матрицей замен BLOSUM62 (см. Henikoff & Henikoff, 1989, Proc. Natl. Acad. Sci. USA 89:10915), равные 50, ожидание (E), равное 10, M=5, N=-4, и сравнение обеих нитей. Компьютерная программа для осуществления определений BLAST является общедоступной в Национальном центре биотехнологической информации (National Center for Biotechnology Information)) (см. стандартный адресный формат URL: ncbi.nlm.nih.gov/).
[00102] Представляющая интерес нуклеотидная последовательность, которая кодирует белок, требует наличия "сайта инициации трансляции" или "сайта инициации", или "инициирующего кодона", или "старт-кодона", расположенного 5' (выше, "upstream") экспрессируемого гена. Такие сайты инициации могут быть или частью энхансерной последовательности, или частью представляющий интерес нуклеотидной последовательности, кодирующей белок.
[00103] Термин "экспрессионная кассета" относится к нуклеотидной последовательности, включающей представляющую интерес нуклеиновую кислоту, под контролем соответствующего промотора или других регуляторных элементов и эффективно (функционально) связанную с соответствующим промотором или другими регуляторными элементами для транскрипции представляющей интерес нуклеиновой кислоты в клетке-хозяине.
[00104] Под "протеолитической петлей" или "сайтом расщепления"
подразумевают консенсусную последовательность протеолитического сайта, который принимает участие в расщеплении предшественника НА0. Термины "консенсус" или "консенсусная последовательность", используемые в данной заявке, означают последовательность (или аминокислотную или нуклеотидную последовательность), которая включает вариабельность родственных последовательностей, основанную на анализе выравнивания множества последовательностей, например, подтипов конкретной последовательности белка-предшественника НА0 вируса гриппа. Консенсусная последовательность сайта расщепления белка-предшественника НА0 гриппа может включать консенсусные аминокислотные последовательности гена гемагглютинина вируса гриппа А, включая, например, консенсусную H1, консенсусную Н3, консенсусную Н5 последовательности или аминокислотные последовательности гена гемагглютинина вируса гриппа типа В, например, но без ограничения, вирусов В Florida, В Malaysia, В Wisconsin и В Massachusetts. Неограничивающие примеры последовательностей участка протеолитической петли показаны на Фигурах 15 и 18В предварительной заявки США на патент №61/806,227 (поданной 28 марта 2013 г, которая включена в данную заявку посредством отсылки; см. также статью Bianchi et al., 2005, Journal of Virology, 79:7380-7388; включенную в данную заявку посредством отсылки).
[00105] Остатки в протеолитической петле или протеолитическом сайте расщепления могут быть мутантными, например, но без ограничения, в точках мутации, замены, инсерции (вставки) или делеции. Термин "аминокислотная мутация" или "модификация аминокислоты", используемый в данной заявке, охватывает аминокислотные замены, делеции, инсерции и модификации. Может быть осуществлена любая комбинация замены, делеции, инсерции и модификации, как описано в предварительной заявке США на патент №61/806,227 (поданной 28 марта 2013 г, которая включена в данную заявку посредством отсылки), чтобы получить конечную конструкцию при условии, что эта конечная конструкция обладает нужными характеристиками, например, пониженной степенью расщепления протеолитической петли или сайта расщепления под действием протеазы.
[00106] Как описано в данной заявке, предусмотрена также конструкция нуклеиновой кислоты (экспрессионная система), включающая последовательность энхансера экспрессии, функционально связанную с нуклеотидной последовательностью, представляющей интерес, которая кодирует белок, представляющий интерес. Предусмотрены также экспрессионные системы на основе растений, включающие энхансерную последовательность, описанную в данной заявке. Предусмотрена также экспрессионная система на основе растений, включающая регуляторную область (участок) растений, функционально связанную с энхансерной последовательностью, которая функционально связана с нуклеотидной последовательностью, представляющей интерес, при этом представляющая интерес нуклеотидная последовательность кодирует белок, представляющий интерес. Энхансерная последовательность может быть выбрана из любой из следующих последовательностей: SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77, или нуклеотидной последовательности, которая характеризуется 100%, 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичностью с последовательностью, представленной любой из SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77, при этом энхансер экспрессии, когда он функционально связан с регуляторной областью растения и последовательностью Козак для растений, как описано в данной заявке, повышает уровень экспрессии нуклеотидной последовательности, представляющей интерес, которая функционально связана с энхансером экспрессии, по сравнению с уровнем экспрессии представляющей интерес нуклеотидной последовательности, связанной с CMPV НТ (SEQ ID NO: 4; известной энхансерной последовательностью, включающей неполноценный белок М, как описано в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в настоящую заявку посредством отсылки) с использованием той же самой регуляторной области растений.
[00107] Энхансерная последовательность по данному изобретению может быть использована для экспрессии белка, представляющего интерес, в организме хозяина, например, растения. В этом случае белок, представляющий интерес, может также быть гетерологичным по отношению к рассматриваемому организму-хозяину и может быть введен в клетки растения с использованием методов трансформации, известных из предшествующего уровня техники. Гетерологичный ген в организме может заменить эндогенный эквивалентный ген, а именно ген, который обычно выполняет ту же самую или подобную функцию, или инсерционная последовательность может быть дополнительной к эндогенному гену или другой последовательности.
[00108] Энхансерная последовательность, функционально связанная с нуклеотидной последовательностью, представляющей интерес, может быть также функционально связана с промотором или регуляторной областью растения и некодирующим участком 3'UTR и терминаторными последовательностями. Энхансерная последовательность может быть, например, любой из последовательностей SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77 или нуклеотидной последовательностью, которая характеризуется 100%, 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичностью с последовательностью, представленной в любой из SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77. Таким образом, нуклеотидная последовательность, представляющая интерес, размещена между энхансерной последовательностью и терминаторной последовательностью (см. Фигуру 1А). Или же энхансер экспрессии или нуклеотидная последовательность, представляющая интерес, может включать последовательность Козак в составе растений.
[00109] Настоящее изобретение предусматривает также кассету экспрессии, включающую последовательно промотор или регуляторную область растения, функционально связанный(ую) с последовательностью энхансера экспрессии, как описано в данной заявке, которая является связанной с представляющей интерес нуклеотидной последовательностью, последовательностью 3'UTR и терминаторной последовательностью. Энхансерная последовательность может быть, например, любой из последовательностей SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77 или нуклеотидной последовательностью, которая характеризуется 100%, 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичностью с последовательностью, представленной в любой из SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77. Или же энхансер экспрессии или нуклеотидная последовательность, представляющая интерес, может включать последовательность Козак в составе растений.
[00110] Как известно специалисту в данной области техники, терминационная (терминаторная) последовательность может быть любой последовательностью, которая является активной в растении-хозяине, например, терминаторная последовательность может быть получена из сегмента генома RNA-2 двухкомпонентного РНК-вируса, например, генома комовируса, или же терминаторная последовательность может быть терминатором NOS.
[00111] Конструкции согласно настоящему изобретению могут также содержать 3'-нетранслируемый участок (UTR). 3'-нетранслируемый участок содержит сигнал полиаденилирования и другие регуляторные сигналы, способные осуществлять процессинг мРНК или экспрессию генов. Сигнал полиаденилирования обычно характеризуется осуществлением добавления участков полиадениловой кислоты к 3'-концу предшественника мРНК. Сигналы полиаденилирования обычно распознаются по наличию гомологии канонической формы 5'-ААТААА-3', хотя вариации не являются необычными. Неограничивающими примерами подходящих 3'-участков являются 3'-транскрибированные нетранслируемые области, содержащие сигнал полиаденилирования индуцирующих опухоль (Ti) плазмидных генов Agrobacterium, таких как гены нопалинсинтазы (Nos ген), и гены растений, такие как гены запасных белков соевых бобов, ген небольшой субъединицы рибулозы-1, ген 5-бифосфаткарбоксилазы (ssRUBISCO; см. патент США №4,962,028; который включен в данную заявку посредством отсылки), промотор, используемый для регуляции экспрессии пластоцианина (публикация Pwee and Gray 1993; которая включена в данную заявку посредством отсылки). Терминационная (терминаторная) последовательность может быть получена из участка 3'UTR гена альфа-пластоцианина.
[00112] Под "нуклеотидной последовательностью (или последовательностью нуклеиновой кислоты), представляющей интерес", или "областью кодирования, представляющей интерес", подразумевают любую нуклеотидную последовательность или участок кодирования (эти термины можно использовать как взаимозаменяемые), которые должны экспрессироваться в организме-хозяине, например, в растении, для получения белка, представляющего интерес. Такая нуклеотидная последовательность, представляющая интерес, может кодировать, но без ограничения, нативные или модифицированные белки, промышленно важный фермент или модифицированный промышленно важный фермент, белок из сельскохозяйственных отходов или модифицированный белок из сельскохозяйственных отходов, хелперный белок, белковую добавку, фармацевтически активный белок, биологически активную добавку, продукт с добавленной стоимостью или его фрагмент для пищи, пищевой продукт или и питательную, и пищевую добавку.
[00113] Белок, представляющий интерес, может содержать нативный или ненативный сигнальный пептид; ненативный сигнальный пептид может иметь растительное происхождение. Например, сигнальный пептид может представлять собой сигнальный пептид протеин-дисульфид-изомеразы (PDI). Нативный сигнальный пептид может соответствовать сигнальному пептиду экспрессируемого белка, представляющего интерес.
[00114] Нуклеотидная последовательность, представляющая интерес, или участок кодирования может также содержать нуклеотидную последовательность, которая кодирует фармацевтически активный белок, например, факторы роста, регуляторы роста, антитела, антигены и их фрагменты или их производные, используемые для иммунизации или вакцинации и т.п. Такие белки включают, но без ограничения, белок, который является патогеном для человека, вирусный белок, например, но без ограничения, VLP-образующие антигены (антигены, образующие вирусоподобные частицы), один или более белков из: респираторно-синцитиального вируса (RSV), ротавируса, вируса гриппа, вируса иммунодефицита человека (ВИЧ, HIV), вируса бешенства, вируса папилломы человека (HPV), энтеровируса 71 (EV71) или интерлейкинов, например, одного или более интердейкинов из ряда IL-1 - IL-24, IL-26 и IL-27, цитокинов, эритропоэтина (ЕРО), инсулина, G-CSF, GM-CSF, hPG-CSF, M-CSF или их комбинаций, интерферонов, например, интерферона-альфа, интерферона-бета, интерферона-гамма, факторов свертывания крови, например, фактора VIII, фактора IX или рецепторов tPA hGH, рецепторов, агонистов рецепторов, антител, например, но без ограничения, ритуксимаба (Rituxan), нейропептидов, инсулина, вакцин, факторов роста, например, но без ограничения, эпидермального фактора роста, фактора роста керацитов, фактора трансформации роста, регуляторов роста, антигенов, аутоантигенов, их фрагментов или их комбинаций.
[00115] Белок, представляющий интерес, может также представлять собой гемагглютинин вируса гриппа (НА; см. заявку WO 2009/009876, которая включена в данную заявку посредством отсылки). НА представляет собой гомотримерный мембранный гликопротеин 1 типа, обычно содержащий сигнальный пептид, домен НА1 и домен, включающий трансмембранный якорный сайт на С-конце и небольшой цитоплазматический хвост. Нуклеотидные последовательности, кодирующие НА, хорошо известны и доступны (см. например, BioDefense and Public Health Database (Influenza Research Database; Squires et al., 2008 Nucleic Acids Research 36:D497-D503) на URL: biohealthbase.org/GSearch/home.do?; или базы, поддерживаемые Национальным центром биотехнологической decorator=Influenza информации (см. URL: ncbi.nlm.nih.gov), обе эти публикации включены в данную заявку посредством отсылки).
[00116] Белок НА может быть белком вируса гриппа типа А, вируса гриппа типа В, или белком НА вируса гриппа подтипа А, выбранного из группы, состоящей из вариантов H1, Н2, Н3, Н4, Н5, Н6, Н7, Н8, Н9, Н10, Н11, Н12, Н13, Н14, Н15 и Н16. В соответствии с некоторыми аспектами настоящего изобретения НА может быть белком вируса гриппа типа А, выбранного из группы, состоящей из вариантов H1, Н2, Н3, Н5, Н6, Н7 и Н9. Фрагменты Has, перечисленные выше, можно рассматривать как белки, представляющие интерес. Кроме того, домены НА типа или подтипа, перечисленных выше, могут быть соединены с получением химерного HA's (см, например, заявку WO 2009/076778, которая включена в данную заявку посредством отсылки).
[00117] Примеры подтипов, содержащих белки НА, включают варианты A/New Caledonia/20/99 (H1N1), A/Indonesia/5/2006 (H5N1), А/цыпленка/New York/1995, А/серебристой чайки/DE/677/88 (H2N8), A/Texas/32/2003, А/дикой утки/MN/33/00, А/утки/Shanghai/1/2000, А/шилохвоста/ТХ/828189/02, А/индейки/Ontario/6118/68 (H8N4), А/широконоски/Iran/G54/03, А/цыпленка/Germany/1949 (H10N7), А/утки/England/56 (H11N6), A/утки/Alberta/60/76 (H12N5), А/чайки/Maryland/704/77 (H13N6), А/дикой утки/Gurjev/263/82, A/утки/Australia/341/83 (H15N8), А/черноголовой чайки/Sweden/5/99 (H16N3), B/Lee/40, C/Johannesburg/66, A/PuertoRico/8/34 (H1N1), A/Brisbane/59/2007 (H1N1), A/Solomon Islands 3/2006 (H1N1)/, A/Brisbane 10/2007 (H3N2), A/Wisconsin/67/2005 (H3N2), B/Malaysia/2506/2004, B/Florida/4/2006, A/Singapore/1/57 (H2N2), A/Anhui/1/2005 (H5N1), A/Vietnam/1194/2004 (H5N1), A/чирка/HongKong/W312/97 (H6N1), A/лошади/Prague/56 (H7N7), A/HongKong/1073/99 (H9N2)).
[00118] Белок НА может быть белком подтипа H1, Н2, Н3, Н5, Н6, Н7 или Н9. Например, белок H1 может быть белком штамма вирусов A/New Caledonia/20/99 (H1N1), A/PuertoRico//8/34 (H1N1), A/Brisbane/59/2007 (H1N1), A/Solomon Islands 3/2006 (H1N1), A/California/04/2009 (H1N1) или A/California/07/2009 (H1N1). Белок Н3 может быть белком штамма вирусов A/Brisbane 10/2007 (H3N2), A/Wisconsin/67/2005 (H3N2), A/Victoria/361/2011 (H3N2), A/Texas/50/2012 (H3N2), A/Hawaii/22/2012 (H3N2), A/New York/39/2012 (H3N2) или A/Perth/16/2009 (H3N2). Согласно еще одному аспекту данного изобретения белок Н2 может быть белком штамма вирусов A/Singapore/1/57 (H2N2). Белок Н5 может быть белком штамма вирусов A/Anhui/1/2005 (H5N1), A/Vietnam/1194/2004 (H5N1) или A/Indonesia/5/2005. Согласно одному из аспектов данного изобретения белок Н6 может быть белком штамма вирусов A/чирка/HongKong/W312/97 (H6N1). Белок Н7 может быть белком штамма вирусов A/лошади/Prague/56 (H7N7) или Н7 A/Hangzhou/1/2013, A/Anhui/1/2013 (H7N9) или A/Shanghai/2/2013 (H7N9). Согласно еще одному аспекту настоящего изобретения белок Н9 может быть белком штамма вирусов A/HongKong/1073/99 (H9N2). Согласно еще одному аспекту настоящего изобретения белок НА может быть белком вируса гриппа типа В, включая варианты B/Malaysia/2506/2004, B/Florida/4/2006, B/Brisbane/60/08, B/Massachusetts/2/2012-подобного вируса (линии Ямагата) или B/Wisconsin/1/2010 (линии Ямагата). Неограничивающие примеры аминокислотных последовательностей белков НА из подтипов H1, Н2, Н3, Н5, Н6, Н7, Н9 или В включают последовательности, описанные в заявках WO 2009/009876, WO 2009/076778, WO 2010/003225 (которые включены в данную заявку посредством отсылки). Белок НА вируса гриппа может быть белком варианта вируса Н5 Indonesia.
[00119] НА может также быть химерным НА, где нативный трансмембранный домен НА заменен гетерологичным трансмембранным доменом. Трансмембранный домена белков НА является высококонсервативным (см., например, Фигуру 1С в заявке WO 2010/148511; которая включена в данную заявку посредством отсылки). Гетерологичный трансмембранный домен может быть получен из любого трансмембранного домена НА, например, но без ограничения, трансмембранного домена вирусных источников H1 California, B/Florida/4/2006 (GenBank, номер доступа АСА33493.1), B/Malaysia/2506/2004 (GenBank, номер доступа ABU99194.1), H1/Bri (GenBank, номер доступа ADE28750.1), H1 A/Solomon Islands/3/2006 (GenBank, номер доступа ABU99109.1), H1/NC (GenBank, номер доступа AAP34324.1), H2 A/Singapore/1/1957 (GenBank, номер доступа AAA64366.1), Н3 А/Brisbane/10/2007 (GenBank, номер доступа ACI26318.1), Н3 A/Wisconsin/67/2005 (GenBank, номер доступа АВO37599.1), Н5 A/Anhui/1/2005 (GenBank, номер доступа ABD28180.1), Н5 A/Vietnam/1194/2004 (GenBank, номер доступа ACR48874.1), H5-Indo (GenBank, номер доступа ABW06108.1). Трансмембранный домен может быть также представлен следующей консенсусной аминокислотной последовательностью:
 (SEQ ID NO: 78)
(SEQ ID NO: 78)
[00120] НА может содержать нативный или ненативный сигнальный пептид; при этом ненативный пептид может иметь растительное происхождение. Нативный сигнальный пептид может соответствовать нативному сигнальному пептиду экспрессируемого гемагглютинина, или может соответствовать второму гемагглютинину. Кроме того, сигнальный пептид может происходить из структурного белка или гемагглютинина вируса, отличающегося от вируса гриппа. Неограничивающие примеры сигнальных пептидов, которые могут быть использованы, включают пептид протеин-дисульфид-изомеразы (из люцерны) (PDI SP; нуклеотиды 32-103, номер доступа в GenBank Z11499), или сигнальный пептид пататина (PatA SP; нуклеотиды 1738-1806, номер доступа в GenBank А08215). Нуклеотидная последовательность PatA SP под этим номером доступа имеет вид:
 (SEQ ID NO: 79)
(SEQ ID NO: 79)
Аминокислотная последовательность сигнального пептида пататина А имеет следующий вид:
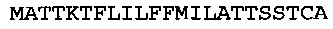 (SEQ ID NO: 80)
(SEQ ID NO: 80)
[00121] Настоящее изобретение предусматривает также молекулы нуклеиновой кислоты, содержащие последовательности, кодирующие белок НА. Молекулы нуклеиновой кислоты могут также содержать одну или более регуляторных областей, функционально связанных с последовательностью, кодирующей белок НА. Молекулы нуклеиновой кислоты могут включать последовательность, кодирующую белки вариантов H1, Н2, Н3, Н4, Н5, Н6, Н7, Н8, Н9, Н10, Н11, Н12, Н13, Н14, Н15, Н16 или НА вируса гриппа типа В. Например, белок НА, кодируемый молекулой нуклеиновой кислоты, может быть белком подтипов гемагглютинина вариантов H1, Н2, Н3, Н5, Н6, Н7, Н9 вируса гриппа типа В. Белок H1, кодируемый нуклеиновой кислотой, может быть белком вариантов из штаммов A/New Caledonia/20/99 (H1N1), А/Пуэрто-Рико/8/34 (H1N1), A/Brisbane/59/2007 (H1N1), A/Solomon Islands 3/2006 (H1N1), A/California/04/2009 (H1N1) или A/California/07/2009 (H1N1). Н3 белок, кодируемый молекулой нуклеиновой кислоты, может быть белком вариантов из штаммов: A/Brisbane 10/2007 (H3N2), A/Wisconsin/67/2005 (H3N2), A/Victoria/361/2011 (H3N2), A/Texas/50/2012 (H3N2), А/Гавайи/22/2012 (H3N2), A/New York/39/2012 (H3N2) или А/Перт/16/2009 (H3N2). H2 белок, кодируемый нуклеиновой кислотой, может быть белком вариантов из штаммов A/Singapore/1/57 (H2N2). Н5 белок, кодируемый молекулой нуклеиновой кислоты, может быть белком вариантов из штаммов A/Anhui/1/2005 (H5N1), A/Vietnam/1194/2004 (H5N1) или A/IIndonesia/5/2005. Н6 белок, кодируемый молекулой нуклеиновой кислоты, может быть белком вариантов из штаммов А/дикой утки/HongKong/W312/97 (H6N1). Н7 белок, кодируемый молекулой нуклеиновой кислоты, может быть белком вариантов из штаммов А/лошади/Prague/56 (H7N7) или Н7 A/Hangzhou/1/2013, A/Anhui/1/2013 (H7N9) или A/Shanghai/2/2013 (H7N9). Далее, Н9 белок, кодируемый молекулой нуклеиновой кислоты, может быть белком из штамма A/HongKong/1073/99 (H9N2). Белок НА, кодируемый молекулой нуклеиновой кислоты, может быть белком вируса гриппа типа В, включая варианты B/Malaysia/2506/2004, B/Florida/4/2006, B/Brisbane/60/08, B/Massachusetts/2/2012-подобного вируса (линии Ямагата) или B/Wisconsin/1/2010 (линии Ямагата). Неограничивающие примеры аминокислотных последовательностей белков НА из подтипов H1, Н2, Н3, Н5, Н6, Н7, Н9 или типа В включают последовательности, описанные в заявках WO 2009/009876, WO 2009/076778, WO 2010/003225 (которые включены в настоящую заявку посредством отсылки). Белок НА вируса гриппа может происходить из варианта Н5 Indonesia.
[00122] Таблица 1: Примеры конструкций, которые были получены, как описано в данной заявке:
Конструкции на основе CMPV-HT
(конструкции, содержащие SEQ ID NO: 4; уровень техники)
Конструкции на основе CPMV160+
(конструкции, содержащие SEQ ID NO: 2)

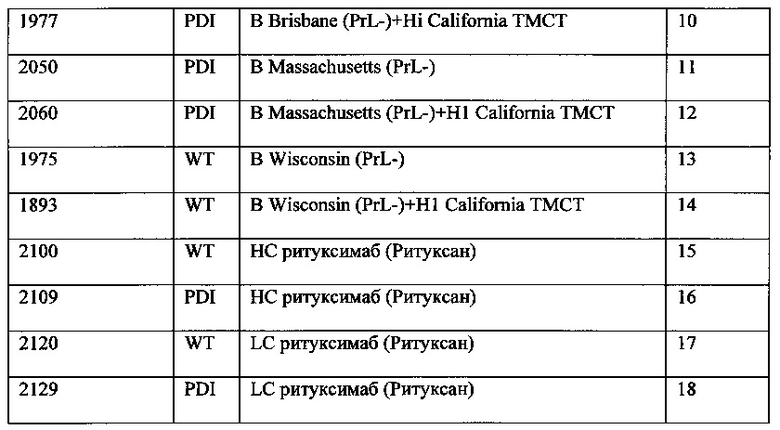
Конструкции на основе CPMV160
(конструкции, содержащие SEQ ID NO: 1)

[00123] Если нуклеотидная последовательность, представляющая интерес, кодирует продукт, который оказывает, прямо или косвенно, токсическое действие на растение, тогда такая токсичность может быть снижена путем селективной экспрессии нуклеотидной последовательности, представляющей интерес, в нужной ткани или на нужной стадии развития растения.
[00124] Участок кодирования, представляющий интерес, или нуклеотидная последовательность, представляющая интерес, может экспрессироваться в любом подходящем растении-хозяине, которое или трансформировано, или содержит нуклеотидные последовательности или молекулы нуклеиновой кислоты, или генетические конструкции, или векторы согласно данному изобретению. Примеры подходящих растений-хозяев включают, но без ограничения, Arabidopsis (резуховидку Таля), сельскохозяйственные культуры, включая, например, канолу (разновидность рапса), Brassica spp (капусту декоративную), маис, Nicotiana spp. (табак), например, Nicotiana benthamiana (табак Бенхама), люцерну, картофель, сладкий картофель (Ipomoea batatus), женьшень, горох, овес, рис, соевые бобы, пшеницу, ячмень, подсолнечник, хлопок, кукурузу, рожь (Secale cereale), сорго (Sorghum bicolor, Sorghum vulgare), сафлор (Carthamus tinctorius).
[00125] Термины "биомасса" и "растительный материал", используемые в настоящей заявке, относятся к любому материалу, происходящему из растения. Биомасса или растительный материал может включать целое растение или часть растения, включающую листья, корни, стебель, цветы, семена, она может также включать любую ткань растения, любые клетки растения или любую часть растения, ткани или клетки. Кроме того, биомасса или растительный материал могут включать внутриклеточные компоненты растений, внеклеточные компоненты растений, жидкие или твердые экстракты растений или их комбинацию. Кроме того, биомасса или растительный материал могут включать растения, клетки растений, ткань, жидкий экстракт или их комбинацию из листьев растений, стеблей, фруктов, корней или их комбинацию. Часть растения может содержать растительный материал или биомассу.
[00126] Под "регуляторной областью" ("регуляторным участком"), "регуляторным элементом" или "промотором" подразумевают часть нуклеиновой кислоты, обычно, но не всегда, выше (5', "upstream") участка гена, кодирующего белок, которая может состоять или из ДНК или РНК, или из ДНК и РНК. Когда регуляторная область является активной и находится в функциональной ассоциации или функционально связана с геном, представляющим интерес, это может привести к экспрессии гена, представляющего интерес. Регуляторный элемент может быть способен к опосредованию специфичности органа или к регуляции активации гена в процессе развития или к временной активации гена. "Регуляторная область" включает элементы промотора, коровые промоторные элементы, проявляющие базальную промоторную активность, элементы, которые являются индуцибельными в ответ на внешние раздражители, элементы, которые опосредуют активность промотора, такие как негативные регуляторные элементы или энхансеры транскриции. Термин "регуляторная область", используемый в настоящей заявке, включает также элементы, которые являются активными после транскрипции, например, регуляторные элементы, которые модулируют экспрессию генов, такие как энхансеры трансляции и транскрипции, репрессоры трансляции и транскрипции, 5' активирующие последовательности и детерминанты нестабильности мРНК. Некоторые из этих последних элементов могут быть расположены вблизи кодирующей области.
[00127] В контексте данной заявки термин "регуляторная область" или "регуляторный элемент" обычно относится к последовательности ДНК, обычно, но не всегда, выше (5') кодирующей последовательности структурного гена, который контролирует экспрессию кодирующей области путем обеспечения распознавания РНК полимеразы и/или других факторов, требующихся для транскрипции при создании конкретного сайта. Однако следует иметь в виду, что другие нуклеотидные последовательности, расположенные в интронах, или в 3' UTR последовательности, могут также вносить свой вклад в регуляцию экспрессии кодирующего участка, представляющего интерес. Примером регуляторного элемента, который обеспечивает узнавание для РНК-полимеразы (полимеразой) или других факторов транскрипции для обеспечения инициации в конкретном сайте, является промоторный элемент. Большинство, но не все, промоторные элементы у эукариот содержит ТАТА-бокс, консервативную нуклеотидную последовательность, состоящую из пар оснований аденозина и тимидина, обычно расположенных на участке приблизительно за 25 пар оснований до сайта инициации транскрипции. Промоторный элемент может содержать базальный промоторные элемент, который отвечает за инициацию транскрипции, а также другие регуляторные элементы (перечисленные выше), которые модифицируют экспрессию генов.
[00128] Существуют несколько типов регуляторных областей, включая те, которые являются эволюционно регулируемыми, индуцибельными или конститутивными. Регуляторная область, которая является эволюционно (в процессе развития) или контролирует дифференциальную экспрессию гена, активируется в некоторых органах или в тканях органа в специфический момент времени во время развития этого органа или ткани. Однако некоторые регуляторные области, регулируемые в процессе развития, предпочтительно могут быть активными в некоторых органах или тканях на специфических стадиях развития, они также могут быть активными в процессе развития или на базальном уровне в других органах и тканях в растении. Примеры тканеспецифических регуляторных областей, например, семяспецифической регуляторной области, включают промотор напина и промотор круциферина (Rask et al., 1998, J. Plant Physiol. 152: 595-599; Bilodeau et al., 1994, Plant Cell 14: 125-130). Пример специфического для листьев промотора включает промотор пластоцианина (см. патент США №7,125,978, который включен в данную заявку посредством отсылки).
[00129] Индуцибельная регуляторная область представляет собой участок, который способен прямо или косвенно активировать транскрипцию одной или более последовательностей или генов ДНК в ответ на индуцирующий фактор (индуктор). В отсутствие индуцирующего фактора последовательности или гены не будут транскрибироваться. Обычно белковый (протеиновый) фактор, который специфически связывает индуцибельную регуляторную область для активации транскрипции, может находиться в неактивной форме, которая затем прямо или косвенно превращается в активную форму под действием индуцирующего фактора. Однако белковый фактор может отсутствовать. Индуктор может представлять собой химический агент, такой как белок, метаболит, регулятор роста, гербицид или фенольное соединение или физиологический стресс, вызванный непосредственно воздействием тепла, холода, соли или токсичных элементов или непосредственно вызванный действием патогена или болезнетворного начала, такого как вирус. Растительная клетка, содержащая индуцибельную регуляторную область, может подвергаться воздействию индуктора при внешнем воздействии индуктора на клетку или растение, например, при распылении, увлажнении, нагревании или при применении других методов. Индуцибельные регуляторные элементы могут происходить или из генов растения, или из генов, отличных от генов растения (см., например, публикацию Gatz, С. and Lenk, I.R.P., 1998, Trends Plant Sci. 3, 352-358; которая включена в данную заявку посредством отсылки). Примеры потенциальных индуцибельных промоторов включают, но без ограничения, тетрациклин-индуцируемый промотор (публикация Gatz, С., 1997, Ann. Rev. Plant Physiol. Plant Mol. Biol. 48, 89-108; которая включена в данную заявку посредством отсылки), стероид-индуцируемый промотор (публикация Aoyama, Т. and Chua, N.H., 1997, Plant J. 2, 397-404; которая включена в данную заявку посредством отсылки) и этанол-индуцируемый промотор (публикации Salter, M.G., et al., 1998, Plant Journal 16, 127-132; Caddick, M.X., et al., 1998, Nature Biotech. 16, 177-180, которые включены в данную заявку посредством отсылки), цитокин-индуцируемые гены IB6 and CKI1 (публикации Brandstatter, I. and Kieber, J.J., 1998, Plant Cell 10, 1009-1019; Kakimoto, Т., 1996, Science 274, 982-985; которые включены в данную заявку посредством отсылки) и ауксин-индуцируемый элемент, DR5 (публикация Ulmasov, Т., et al., 1997, Plant Cell 9, 1963-1971; которая включена в данную заявку посредством отсылки).
[00130] Конститутивная регуляторная область управляет экспрессией гена в различных частях растения и непрерывно в процессе развития растения. Примеры известных конститутивных регуляторных элементов включают промоторы, связанные с транскриптом CaMV 35S (p35S; Odell et al., 1985, Nature, 313: 810-812), промоторы актина 1 риса (Zhang et al., 1991, Plant Cell, 3: 1155-1165), актина 2 (An et al., 1996, Plant J., 10: 107-121) или tms 2 (см. патент США №5,428,147, который включен в данную заявку посредством отсылки) и промоторы: гена триозофосфатизомеразы 1 (Xu et al., 1994, Plant Physiol. 106: 459-467), гена убиквитина 1 маиса (Cornejo et al., 1993, Plant Mol. Biol. 29: 637-646), генов убиквитина 1 и 6 Arabidopsis (арабидопсиса) (Holtorf et al., 1995, Plant Mol. Biol. 29: 637-646), гена 4A фактора инициации трансляции табака (Mandel et al., 1995 Plant Mol. Biol. 29: 995-1004), промотор вируса мозаики маниоки, pCAS, (Verdaguer et al., 1996); промотор малой субъединицы рибулозобифосфаткарбоксилазы, pRbcS: (Outchkourov et al., 2003), pUbi (для однодольных и двудольных растений).
[00131] Как описано в настоящей заявке, было обнаружено, что регуляторные области, содержащие энхансерные последовательности с проявленной эффективностью при экспрессии в листьях, являются эффективными в процессе транзиторной экспрессии. Не ограничиваясь какой-либо конкретной теорией, можно полагать, что связывание 5' (upstream) регуляторных элементов фотосинтетического гена с ядерным матриксом может опосредовать сильную экспрессию. Например, в положении -784 от начала трансляции пластоцианина гороха (см. патент США №7,125,978, который включен в данную заявку посредством отсылки) может быть использована сильная экспрессия репортерного гена.
[00132] Термин "конститутивный, используемый в данной заявке, не обязательно указывает, что нуклеотидная последовательность под контролем конститутивного регуляторной области экспрессируется на том же уровне во всех типах клеток, но эта последовательность экпрессируется в большом числе клеток разных типов, даже в тех случаях, когда имеются различные разновидности.
[00133] Экспрессионные конструкции, описанные выше, могут находиться в составе вектора. Вектор может включать граничные последовательности, которые обеспечивают передачу и интеграцию экспрессионной кассеты в геном организма или хозяина. Такая конструкция может представлять собой бинарный вектор, например, бинарный вектор трансформации на основе pPZP (см. Hajdukiewicz, et al., 1994). Другой пример такой конструкции включает pBin19 (см. Frisch, D.A., L.W. Harris-Haller, et al. 1995, Plant Molecular Biology 27: 405-409).
[00134] Если это желательно, на конструкции по изобретению можно воздействовать путем включения маркеров. Однако это может не потребоваться. Подходящие маркеры включают ферменты, которые обеспечивают стойкость к химическим агентам, таким как антибиотик, например, гентамицин, гигромицин, канамицин, или гербициды, такие как фосфинотрицин, глифосат, хлорсульфурон и т.п. Точно так же могут быть использованы ферменты, обеспечивающие получение соединения, идентифицируемого по изменению цвета, такой как GUS (бета-глюкуронидаза), или по люминесценции, такой как люцифераза или GFP.
[00135] Вектор может также включать энхансер экспрессии, описанный в данной заявке. Энхансер экспрессии может находиться в Т-ДНК (транспортной ДНК), которая содержит также супрессор сайленсинга генов и гена NPTII (неомицин фосфотрансферазы II). По ли линкер может также кодировать один или два набора 6 х гистидиновых остатков для обеспечения соединения N- или С-концевых меток в представляющем интерес белке для облегчения очистки белка.
[00136] Пост-трансляционный сайленсинг генов (PTGS) может участвовать в ограничении экспрессии трансгенов в растениях, и совместная экспрессия супрессора сайленсинга из вируса Y картофеля (HcPro) может быть использована для противодействия специфическому расщеплению "трансгенных" мРНК (см. публикацию Brigneti et al., 1998, EMBO J. 17, 6739-6746, которая включена в данную заявку посредством отсылки). Альтернативные супрессоры сайленсинга хорошо известны в предшествующем уровне техники и могут быть использованы, как описано в данной заявке (см. публикацию Chiba et al., 2006, Virology 346:7-14; которая включена в данную заявку посредством отсылки), например, но без ограничения, это могут быть TEV-p1/HC-Pro (ген p1 вируса гравировки табака/HC-Pro), BYV-p21, ген р19 вируса кустистой карликовости томата (TBSV р19; конструкция р19 описана в заявке WO 2010/0003225, которая включена в данную заявку посредством отсылки), капсидный белок вируса курчавости томата (TCV-CP), ген 2b вируса мозаики огурца; CMV-2b), ген р25 вируса X картофеля (PVX-p25), ген p11 вируса М картофеля (PVM-p11), ген p11 вируса S картофеля (PVS-p11), ген р16 вируса ожога черники (BScV-p16), ген р23 вируса тристецы цитрусовых (CTV-p23), ген р24 вируса-2 скручивания виноградных листьев (GLRaV-2 р24), ген р10 вируса А винограда (GVA-p10), ген р14 вируса В винограда (GVB-p14), ген р10 латентного вируса Heracleum (борщевика)) (HLV-p10) или ген р16 латентного вируса чеснока (GCLV-p16).
[00137] Следовательно, один или более супрессоров сайленсинга, например, но без ограничения, HcPro, TEV-p1/HC-Pro, BYV-p21, TBSV р19, TCV-CP, CMV-2b, PVX-p25, белок rgsCaM, белок В2 из FHV (вируса Дома Скопления), малый белок вирусной оболочки CPMV и белок вирусной оболочки из TCV (вируса морщинистости репы), PVM-p11, PVS-p11, BScV-p16, CTV-p23, GLRaV-2 p24, GBV-p14, HLV-p10, GCLV-p16 или GVA-p10 могут экспрессироваться вместе с элементом для амплификации экспрессионной кассеты на основе комовируса, элементом амплификации на основе геминивируса и нуклеотидной последовательностью, кодирующей представляющий интерес белок, для дальнейшего обеспечения высоких уровней продуцирования белков в растении.
[00138] Конструкции по изобретению могут быть введены в клетки растений с использованием Ti-плазмид (плазмид, индуцирующих образование опухолей), Ri-плазмид (плазмид, индуцирующих образование корней), вирусных векторов в растениях, непосредственной трансформации ДНК, микроинъекции, электропорации и т.д. Обзор таких методов можно найти, например, в публикациях Weissbach and Weissbach, Methods for Plant Molecular Biology, Academy Press, New York VIII, pp. 421-463 (1988); Geierson and Corey, Plant Molecular Biology, 2nd Ed. (1988); и Miki and Iyer, Fundamentals of Gene Transfer in Plants. In Plant Metabolism, 2d Ed. DT. Dennis, DH Turpin, DD Lefebrve, DB Layzell (eds), Addison Wesly, Langmans Ltd. London, pp. 561-579 (1997). Другие методы включают непосредственное поглощение ДНК, применение липосом, электропорацию, например, использование протопластов, микроинъекции, применение микрочастиц или "усов" и вакуум-инфильтрации. См., например, публикации Bilang, et al. (1991, Gene 100: 247-250), Scheid et al. (1991, Mol. Gen. Genet. 228: 104-112), Guerche et al. (1987, Plant Science 52: 111-116), Neuhause et al. (1987, Theor. Appl Genet. 75: 30-36), Klein et al, (2987, Nature 327: 70-73); Freeman et al. (1984, Plant Cell Physiol. 29: 1353), Howell et al. (1980, Science 208: 1265), Horsch et al. (1985, Science 221: 1229-1231), DeBlock et al., (1989, Plant Physiology 91: 694-701), Methods for Plant Molecular Biology (Weissbach and Weissbach, eds., Academic Press Inc., 1988), Methods in Plant Molecular Biology (Schuler and Zielinski, eds., Academic Press Inc., 1989), WO 92/09696, WO 94/00583, EP 331083, EP 175966, Liu and Lomonossoff (2002, J Virol Meth., 105:343-348), EP 290395; WO 8706614; патенты США №№4,945,050; 5,036,006; и 5,100,792, заявки США на патент №08/438,666, поданная 10 мая 1995 г, и №07/951,715, поданная 25 сентября 1992 г. (все из которых включены в данную заявку посредством отсылки).
[00139] Для осуществления экспрессии конструкций по изобретению могут быть использованы методы транзиторной экспрессии (см. публикации D'Aoust et al., 2009, Methods in molecular biology, Vol 483, pages 41-50; Liu and Lomonossoff, 2002, Journal of Virological Methods, 105:343-348; которые включены в данную заявку посредством отсылки). Альтернативно, может быть использован метод вакуумной транзиторной экспрессии, описанный в публикации Kapila et al., (1997, Plant Sci. 122, 101-108; которая включена в данную заявку посредством отсылки) или в заявках WO 00/063400, WO 00/037663 (которых включены в данную заявку посредством отсылки). Эти методы могут включать, например, но без ограничения, метод агроинокуляции или агроинфильтрации, метод инфильтрации при помощи шприца, однако, могут быть применены и другие методы транзиторной экспрессии, описанные выше. При осуществлении агроинокуляции или агроинфильтрации или инфильтрации при помощи шприца смесь Agrobacteria (агробактерий), содержащих нужную нуклеиновую кислоту, вводится во внутриклеточное пространство ткани, например, листьев, воздушную часть растения (включая стебель, листья и цветы), другую часть растения (стебель, корень, цветок) или в целое растение. После прохождения через эпидермис Agrobacteria инфицируют растение и переносят копии т-ДНК в клетки. т-ДНК транскрибируется в эписомальной форме и транслируется в мРНК, в результате в инфицированных клетках продуцируется целевой белок, однако, перемещение т-ДНК внутрь ядра является транзиторным.
[00140] Часть настоящего изобретения составляют трансгенные растения, растительные клетки или семена, содержащие генетическую конструкцию по изобретению, которая может быть использована как платформа, подходящая для транзиторной экспрессии белков, описанных в данной заявке. Методы регенерации целых растений из растительных клеток хорошо известны из уровня техники (см., например, Guerineau and Mullineaux (1993, Plant transformation and expression vectors. In: Plant Molecular Biology Labfax (Croy RRD ed) Oxford, BIOS Scientific Publishers, pp 121-148). В общем, трансформированные клетки растений инкубируются в подходящей среде, которая может содержать селективные агенты, такие как антибиотики, где используются выбранные маркеры для облегчения идентификации трансформированных растительных клеток. Сразу же после образования каллуса можно ускорить образование побегов путем применения соответствующих гормонов растений в соответствии с известными способами и перемещение побегов в корневую среду для регенерации растений. Затем растения могут быть использованы для получения повторяющихся поколений, или из семян, или с применением методики вегетативного размножения. Трансгенные растения могут быть также получены без использования тканевой культуры. Способы стабильной трансформации и регенерации этих организмов описаны в предшествующем уровне техники и известны специалисту в данной области. Доступные способы описаны в публикациях Vasil et al., (Cell Culture and Somatic Cell Genetics of Plants, Vol. I, Il and III, Laboratory Procedures and Their Applications, Academic Press, 1984) и Weissbach and Weissbach, (Methods for Plant Molecular Biology, Academic Press, 1989). Способ получения трансформированных и регенерированных растений не является критическим для настоящего изобретения.
[00141] Если нужно трансформировать или совместно трансформировать растения, часть растений или растительную клетку с применением двух или более конструкций нуклеиновых кислот, конструкция нуклеиновых кислот может быть введена в Agrobacterium в едином событии трансфекции, когда формируются пулы нуклеиновых кислот и бактериальные клетки трансфецируются, как описано. Или же конструкции могут вводиться последовательно. В этом случае первая конструкция помещается в Agrobacterium, как описано, при этом клетки растут в селективной среде (например, в присутствии антибиотика), когда могут расти лишь бактерии-одиночные трансформанты. После этой первой стадии селекции в Agrobacterium, как описано, вводится вторая конструкция нуклеиновой кислоты, при этом клетки растут в двойной селективной среде, когда могут расти только бактерии-двойные трансформанты. Затем бактерии-двойные трансформанты могут быть использованы для трансформации растения, части растения или растительной клетки, как описано в данной заявке, или могут быть подвергнуты дальнейшей трансформации для размещения третьей конструкции нуклеиновой кислоты.
[00142] Альтернативно, если растения, части растения или растительная клетка должны трансформироваться или совместно трансформироваться при помощи двух или более конструкций нуклеиновых кислот, конструкция нуклеиновой кислоты может быть введена в растение путем совместной инфильтрации смеси клеток Agrobacterium с растением, частью растения или растительной клеткой, причем каждая клетка Agrobacterium может содержать одну или более конструкций, которые должны быть введены в растение. Для того чтобы варьировать уровни относительной экспрессии представляющей интерес нуклеотидной последовательности в конструкции в растении, части растения или в растительной клетке, можно менять концентрации различных популяций Agrobacteria, содержащих целевые конструкции, во время проведения стадии инфильтрации.
[00143] Настоящее изобретение предусматривает также трансгенное растение, содержащее экспрессионную систему, описанную в данной заявке, при этом наблюдается повышенный уровень экспрессии представляющей интерес гетерологичной нуклеиновой кислоты в кассете по сравнению с уровнями экспрессии других аналогичных экспрессионных систем, которые лишены одного или более компонентов экспрессионной системы, описанной в данной заявке, например, CMPV НТ (SEQ ID NO: 4).
[00144] Настоящее изобретение включает также способ получения представляющего интерес белка, включающий стадии предоставления растения или части растения, которые экспрессируют экспрессионную систему, описанную в данной заявке, сбора по меньшей мере одной ткани, в которой был экспрессирован представляющий интерес белок, и необязательно выделения представляющего интерес белка из ткани.
[00145] Таким образом, согласно различным аспектам и без ограничения настоящее изобретение предусматривает:
- энхансер экспрессии, содержащий область 5'UTR комовируса, выбранную из последовательностей SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77, или нуклеотидной последовательности, которая характеризуется 100%, 99%, 98%, 97%, 96%, 95%, 90%, 85% или 80% идентичностью с последовательностью, представленной любой из SEQ ID NO's: 1, 2, 24, 27, 68, 69 и 70-77, при этом энхансер экспрессии, будучи функционально связан с регуляторной областью растения и последовательностью Козак для растений, согласно данной заявке, повышает уровень экспрессии представляющей интерес нуклеотидной последовательности, функционально связанной с энхансером экспрессии, по сравнению с уровнем экспрессии представляющей интерес нуклеотидной последовательности, связанной с CMPV НТ (SEQ ID NO: 4; известной энхансерной последовательности, включающей неполноценный белок М, как описано в публикации Sainsbury F., and Lomonossoff G.P., 2008, Plant Physiol. 148: pp. 1212-1218; которая включена в данную заявку посредством отсылки) с использованием той же самой регуляторной области растений.
- одну или более экспрессионных систем, содержащих энхансер экспрессии на основе комовируса, или экспрессионную кассету, описанную в данной заявке, промотор (регуляторную область), необязательно полилинкер, последовательность Козак, нуклеиновую кислоту, кодирующую белок, представляющий интерес, и терминатор.
- способы экспрессии представляющего интерес белка в организме-хозяине, таком как растение, с использованием одной или более экспрессионных систем или одного или более векторов по описанию в данной заявке.
- клетки-хозяева или организмы-хозяева, экспрессирующие представляющие интерес белки с использованием одной или более экспрессионных систем или одного или более векторов по изобретению, и способы получения хозяев и организмов.
[00146]
Примеры
[00147] Пример 1 - 2X35S/CPMV-HT/PDISP/H3 Victoria/NOS (конструкция номер 1391)
[00148] Последовательность, кодирующую белки вируса гриппа Н3 линии A/Victoria/361/2011, в которой нативный сигнальный пептид был заменен нативным сигнальным пептидом протеин-дисульфид-изомеразы (PDISP/H3 Victoria), клонировали в экспрессионную систему 2X35S-CPMV-HT-NOS (CMPV-HT) с использованием метода ПЦР. Фрагмент, содержащий кодирующую последовательность PDISP/H3 Victoria, амплифицировали, используя праймеры IF-PDI.S1+3с (Фигура 6А, SEQ ID NO: 67) и IF-H3V36111.s1-4r (Фигура 6В, SEQ ID NO: 17), используя в качестве матрицы последовательность PDISP/H3 Victoria (Фигура 6С, SEQ ID NO: 18). Продукт ПЦР клонировали в экспрессионную систему 2X35S/CPMV-HT/NOS, используя систему клонирования In-Fusion (Clontech, Mountain View, СА). Конструкцию номер 1191 (Фигура 6D) расщепляли рестриктазами SacII и StuI и для In-Fusion реакции сборки использовали линеаризованную плазмиду. Конструкция номер 1191 представляла собой плазмиду-акцептор, предназначенную для "In Fusion" клонирования представляющих интерес генов в экспрессионную кассету на основе CPMV-HT. Она также встраивает генетическую конструкцию для совместной экспрессии супрессора сайленсинга TBSV P19, под контролем промотора и терминатора гена пластоцианина люцерны. Скелетом является бинарная плазмида pCAMBIA, и последовательность от левой до правой границ тДНК представлена на Фигуре 6Е (SEQ ID NO: 19). Полученная конструкция получила номер 1391 (Фигура 6F, SEQ ID NO: 20). Аминокислотная последовательность зрелого вируса гриппа Н3 A/Victoria/361/2011, связанная с PDISP, представлена на Фигуре 6G (SEQ ID NO: 21). Конструкция плазмиды 1391 показана на Фигуре 6Н.
[00149] Пример 2 - 2X35S/CPMV160+/PDISP/H3 Victoria/NOS (конструкция номер 1800)
[00150] Последовательность, кодирующую белки вируса гриппа Н3 линии A/Victoria/361/2011, в которой нативный сигнальный пептид был заменен сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/H3 Victoria), клонировали в экспрессионную систему 2X35S/CPMV160+/NOS (CPMV160+) методом ПЦР. Фрагмент, содержащий кодирующую последовательность PDISP/H3 Victoria, амплифицировали, используя праймеры IF**(SacII)-PDI.s1+4c (Фигура 7А, SEQ ID NO: 22) и IF-H3V36111.s1-4r (Фигура 7В, SEQ ID NO: 23), применяя в качестве матрицы последовательность PDISP/H3 Victoria (Фигура 7С, SEQ ID NO: 24). Продукт ПЦР клонировали в экспрессионную систему 2X35S/CPMV160/NOS, используя систему клонирования In-Fusion (Clontech, Mountain View, СА). Конструкцию номер 2171 (Фигура 7D) расщепляли рестриктазами SacII и StuI и для реакции сборки In-Fusion использовали линеаризованную плазмиду. Конструкция номер 2171 представляла собой плазмиду-акцептор, предназначенную для "In Fusion" клонирования представляющих интерес генов в экспрессионную кассету на основе CPMV160+. Она также встраивает генетическую конструкцию для совместной экспрессии супрессора сайленсинга TBSV P19, под контролем промотора и терминатора гена пластоцианина люцерны. Скелетом является бинарная плазмида pCAMBIA, и последовательность от левой до правой границ тДНК представлена на Фигуре 7Е (SEQ ID NO: 25). Полученная конструкция получила номер 1800 (Фигура 7F, SEQ ID NO: 26). Аминокислотная последовательность зрелого вируса гриппа Н3 A/Victoria/361/2011, связанная с PDISP, представлена на Фигуре 7G (SEQ ID NO: 21). Конструкция плазмиды 1800 показана на Фигуре 7Н.
[00151] Пример 3 - 2X35S/CPMV160/PDISP/H3 Victoria/NOS (конструкция номер 1935)
[00152] Последовательность, кодирующую белки вируса гриппа Н3 линии A/Victoria/361/2011, в которой нативный сигнальный пептид был заменен сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/H3 Victoria), клонировали в экспрессионную систему 2X35S/CPMV160-NOS методом ПЦР. Фрагмент, содержащий кодирующую последовательность PDISP/H3 Victoria, амплифицировали, используя праймеры IF-CPMV(fl5'UTR)_SpPDI.c (Фигура 8А, SEQ ID NO: 28) и IF-H3V36111.s1-4r (Фигура 7В, SEQ ID NO: 23), применяя в качестве матрицы последовательность PDISP/H3 Victoria (Фигура 7С, SEQ ID NO: 24). Продукт ПЦР клонировали в экспрессионную систему 2X35S/CPMV160/NOS, используя систему клонирования In-Fusion (Clontech, Mountain View, СА). Конструкцию номер 1190 (Фигура 8В) расщепляли рестриктазами SacII и StuI и для реакции сборки In-Fusion использовали линеаризованную плазмиду. Конструкция номер 1190 представляла собой плазмиду-акцептор, предназначенную для "In Fusion" клонирования представляющих интерес генов в экспрессионную кассету на основе CPMV160. Она также встраивает генетическую конструкцию для совместной экспрессии супрессора сайленсинга TBSV Р19 под контролем промотора и терминатора гена пластоцианина люцерны. Скелетом является бинарная плазмида pCAMBIA, и последовательность от левой до правой границ тДНК представлена на Фигуре 8С (SEQ ID NO: 29). Полученная конструкция получила номер 1935 (Фигура 8D, SEQ ID NO: 30). Аминокислотная последовательность зрелого вируса гриппа Н3 A/Victoria/361/2011, соединенная с PDISP, представлена на Фигуре 7G (SEQ ID NO: 27). Конструкция плазмиды 1935 показана на Фигуре 8Е.
[00153] Пример 4 - Изменения в последовательности между сайтом рестрикции SacII и ATG PDISP/H3 Victoria в экспрессионной системе 2X35S/CPMV160+/NOS (конструкции номер 1992-1999)
[00154] Восемь конструкций, содержащих варианты последовательности между сайтом рестрикции SacII и ATG PDISP/H3 Victoria в экспрессионной системе 2X35S/CPMV160+/NOS, были получены с применением того же метода ПЦР, что и конструкция номер 1800 (см. Пример 2), и с использованием модифицированного прямого праймера, остальные компоненты были теми же самыми. Варианты НТ1*-НТ8* амплифицировали, используя праймеры, указанные на Фигурах 9А-9Н:
IF-HT1*(-Mprot)-PDI.c (Фигура 9А, SEQ ID NO: 31),
IF-HT2*(-Mprot)-PDI.c (Фигура 9В, SEQ ID NO: 32),
IF-HT3*(-Mprot)-PDI.c (Фигура 9C, SEQ ID NO: 33)
IF-HT4*(-Mprot)-PDI.c (Фигура 9D, SEQ ID NO: 34)
IF-HT5*(-Mprot)-PDI.c (Фигура 9E, SEQ ID NO: 35)
IF-HT6*(-Mprot)-PDI.c (Фигура 9F, SEQ ID NO: 36)
IF-HT7*(-Mprot)-PDI.c (Фигура 9G, SEQ ID NO: 37) и
IF-HT8*(-Mprot)-PDI.c (Фигура 9H, SEQ ID NO: 38),
для получения конструкций номер 1992-1999, соответственно. Конструкция плазмиды 1992 представлена на Фигуре 91. Аналогичные приемы были использованы при получении конструкций 1993-1999.
[00155] Пример 5 - 2X35S/CPMV НТ (конструкция номер 484) и 2X35S/CPMV160+ (конструкция номер 1897) для PDISP/H1 California
[00156] Кодирующую последовательность, соответствующую H1 вирусу гриппа A/California/7/2009, в которой нативный сигнальный пептид был заменен сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/H1 California) (Фигура 10А, SEQ ID NO: 39), клонировали в исходные CPMV-HT и CPMV160 с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и номер 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/H1 California. Аминокислотная последовательность зрелого вируса гриппа H1 линии A/California/7/2009, соединенная с PDISP, представлена на Фигуре 10В (SEQ ID NO: 40). Конструкции плазмид 484 и 1897 представлены на Фигурах 10С и 10D.
[00157] Пример 6 - 2X35S/CPMV НТ (конструкция номер 489). 2X35S/CPMV160+ (конструкция номер 1880) и 2X35S/CPMV160 (конструкция номер 1885) для Н5 Indonesia
[00158] Кодирующую последовательность, соответствующую нативному Н5 вирусу гриппа штамма A/Indonesia/5/2005 (Фигура 11А, SEQ ID NO: 41), клонировали в исходные CPMV-HT, CPMV160+ и CPMV160, используя метод ПЦР, как и в случае конструкций 1391 (см. Пример 1), 1800 (см. Пример 2) и 1935 (см. Пример 3), соответственно, но с модифицированными праймерами, специально сконструированными для Н5 Indonesia. Аминокислотная последовательность нативного белка вируса гриппа Н5 A/Indonesia/5/2005 представлена на Фигуре 11В (SEQ ID NO: 42). Конструкции плазмид 489, 1880 и 1885 представлены на Фигурах 11С-11Е.
[00159] Пример 7 - 2X35S/CPMV НТ (конструкция номер 2140) и 2X35S/CPMV160+ (конструкция номер 2168) для PDISP-H7 Hangzhou
[00160] Кодирующую последовательность, соответствующую вирусу гриппа Н7 линии A/Hangzhou/1/2013, в которой нативный сигнальный пептид был заменен сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/H7 Hangzhou) (Фигура 12А, SEQ ID NO: 43), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/H7 Hangzhou. Аминокислотная последовательность зрелого вируса гриппа Н7 линии A/Hangzhou/1/2013, соединенная с PDISP, представлена на Фигуре 12В (SEQ ID NO: 44). Конструкции плазмид 2140 и 2168 представлены на Фигурах 12С и 12D.
[00161] Пример 8 - 2X35S/CPMV НТ (конструкция номер 2130) и 2X35S/CPMV160+ (конструкция номер 2188) для PDISP/H7 Hangzhou + H5 Indonesia ТМСТ
[00162] Последовательность, кодирующую химерный гемагглютинин, соответствующую эктодомену белка вируса гриппа Н7 A/Hangzhou/1/2013, связанную с трансмембранным доменом и цитоплазматическим хвостом (ТМСТ) Н5 вируса гриппа A/Indonesia/5/2005 и с сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/H7 Hangzhou + H5 Indonesia ТМСТ) (Фигура 13А, SEQ ID NO: 45), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/H7 Hangzhou + H5 Indonesia ТМСТ. Аминокислотная последовательность зрелого вируса гриппа Н7 Hangzhou + H5 Indonesia ТМСТ, связанная с PDISP, представлена на Фигуре 13В (SEQ ID NO: 46). Конструкции плазмид 2130 и 2188 представлены на Фигурах 13С и 13D.
[00163] Пример 9 - 2X35S/CPMV НТ (конструкция номер 1039) и 2X35S/CPMV160+ (конструкция номер 1937) для PDISP/HA В Brisbane (PrL-)
[00164] Кодирующую последовательность, соответствующую НА вируса гриппа линии B/Brisbane/60/2008 с делетируемой протеолитической петлей (PrL-) (см. предварительную заявку США на патент №61/806,227, поданную 28 марта 2013 г, которая включена в данную заявку посредством отсылки, для дополнительной информации об участках делетируемой протеолитической петли в последовательностях НА), в которой нативный сигнальный пептид был заменен сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/HA В Brisbane (PrL-)) (Фигура 14А, SEQ ID NO: 47), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/HA В Brisbane (PrL-). Аминокислотная последовательность зрелого НА В Brisbane (PrL-), соединенная с PDISP, показана на Фигуре 14В (SEQ ID NO: 48). Конструкции плазмид 1039 и 1937 представлены на Фигурах 14С и 14D.
[00165] Пример 10 - 2X35S/CPMV НТ (конструкция номер 1067) и 2X35S/CPMV160+ (конструкция номер 1977) для PDISP/HA В Brisbane (PrL-) + H1 California ТМСТ
[00166] Последовательность, кодирующую химерный гемагглютинин, соответствующую эктодомену белка вируса гриппа НА линии B/Brisbane/60/08 с делетируемой протеолитической петлей (PrL-) (см. предварительную заявку США на патент №61/806,227, поданную 28 марта 2013 г, которая включена в данную заявку посредством отсылки, для дополнительной информации об участках делетируемой протеолитической петли в последовательностях НА), соединенную с трансмембранным доменом и цитоплазматическим хвостом (ТМСТ) H1 линии A/California/7/2009 и с сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/HA В Brisbane (PrL-) + H1 California ТМСТ) (Фигура 15А, SEQ ID NO: 49), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/HA В Brisbane (PrL-) + H1 California ТМСТ. Аминокислотная последовательность зрелого НА В Brisbane (PrL-), соединенная с PDISP, показана на Фигуре 15В (SEQ ID NO: 50). Конструкции плазмид 1067 и 1977 представлены на Фигурах 15С и 15D.
[00167] Пример 11 - 2X35S/CPMV НТ (конструкция номер 2072) и 2X35S/CPMV160+ (конструкция номер 2050) для PDISP/HA В Massachusetts (PrL-)
[00168] Кодирующую последовательность, соответствующую НА вируса гриппа линии B/Massachusetts/2/2012 с делетируемой протеолитической петлей (PrL-) (см. предварительную заявку США на патент №61/806,227, поданную 28 марта 2013 г., которая включена в данную заявку посредством отсылки, для дополнительной информации об участках делетируемой протеолитической петли в последовательностях НА), в которой нативный сигнальный пептид был заменен сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/HA В Massachusetts (PrL-)) (Фигура 16А, SEQ ID NO: 51), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/HA В Massachusetts (PrL-). Аминокислотная последовательность зрелого НА В Massachusetts (PrL-), соединенная с PDISP, показана на Фигуре 16В (SEQ ID NO: 52). Конструкции плазмид 2072 и 2050 представлены на Фигурах 16С и 16D.
[00169] Пример 12 - 2X35S/CPMV НТ (конструкция номер 2074) и 2X35S/CPMV160+ (конструкция номер 2060) для PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ
[00170] Последовательность, кодирующую химерный гемагглютинин, соответствующую эктодомену НА вируса гриппа линии B/Massachusetts/2/2012 с делетируемой протеолитической петлей (PrL-) (см. предварительную заявку США на патент №61/806,227, поданную 28 марта 2013 г, которая включена в данную заявку посредством отсылки, для дополнительной информации об участках делетируемой протеолитической петли в последовательностях НА), соединенную с трансмембранным доменом и цитоплазматическим хвостом (ТМСТ) H1 A/California/7/2009 и с сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ) (Фигура 17А, SEQ ID NO: 53), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/HA В Massachusetts (PrL-) + H1 California ТМСТ. Аминокислотная последовательность зрелого НА В Massachusetts (PrL-)-) + H1 California ТМСТ, соединенная с PDISP, показана на Фигуре 17В (SEQ ID NO: 54). Конструкции плазмид 2074 и 2060 представлены на Фигурах 17С и 17D.
[00171] Пример 13 - 2X35S/CPMV НТ (конструкция номер 1445), 2X35S/CPMV160+ (конструкция номер 1820) и CPMV 160 (конструкция номер 1975) для НА В Wisconsin (PrL-)
[00172] Кодирующую последовательность, соответствующую НА вируса гриппа B/Wisconsin/1/2010 с делетируемой протеолитической петлей (PrL-) (см. предварительную заявку США на патент №61/806,227, поданную 28 марта 2013 г, которая включена в данную заявку посредством отсылки, для дополнительной информации об участках делетируемой протеолитической петли в последовательностях НА), с его нативным сигнальным пептидом (НА В Wisconsin (PrL-)) (Фигура 18А, SEQ ID NO: 55) клонировали в исходные CPMV-HT, CPMV 160+ и CPMV 160 с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для НА В Wisconsin (PrL-). Аминокислотная последовательность зрелого НА В Wisconsin (PrL-) с его нативным сигнальным пептидом показана на Фигуре 18В (SEQ ID NO: 56). Конструкции плазмид 1445, 1820 и 1975 are представлены на Фигурах 18С, 18D и 18Е, соответственно.
[00173] Пример 14 - 2X35S/CPMV НТ (конструкция номер 1454) и 2X35S/CPMV160+ (конструкция номер 1893) для НА В Wisconsin (PrL-) + H1 California ТМСТ.
[00174] Последовательность, кодирующую химерный гемагглютинин, соответствующую эктодомену НА вируса гриппа B/Wisconsin/2/2012 с делетируемой протеолитической петлей (PrL-) (см. предварительную заявку США на патент №61/806,227, поданную 28 марта 2013 г, которая включена в данную заявку посредством отсылки, для дополнительной информации об участках делетируемой протеолитической петли в последовательностях НА), соединенную с трансмембранным доменом и цитоплазматическим хвостом (ТМСТ) H1 A/California/7/2009 с нативным сигнальным пептидом НА В Wisconsin (НА В Wisconsin (PrL-) + H1 California ТМСТ) (Фигура 19A, SEQ ID NO: 57), клонировали в исходные CPMV-HT, CPMV160+ и CPMV 160 с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для НА В Wisconsin (PrL-) + H1 California ТМСТ. Аминокислотная последовательность НА В Wisconsin (PrL-) + H1 California ТМСТ показана на Фигуре 19 В (SEQ ID NO: 58). Конструкции плазмид 1454 и 1893 представлены на Фигурах 19С и 19D, соответственно.
[00175] Пример 15 - 2X35S/CPMV НТ (конструкция номер 5001) и 2X35S/CPMV160+ (конструкция номер 2100) для НС ритуксимаба (Ритуксан)
[00176] Кодирующую последовательность, соответствующую тяжелой цепи моноклонального антитела IgG1 ритуксимаба (НС ритуксимаба (Ритуксан); Фигура 20А, SEQ ID NO: 59), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для НС ритуксимаба (Ритуксан). Аминокислотная последовательность НС ритуксимаба (Ритуксан) показана на Фигуре 20В (SEQ ID NO: 60). Конструкции плазмид 5001 и 2100 представлены на Фигурах 20С и 20D, соответственно.
[00177] Пример 16 - 2X35S/CPMV НТ (конструкция номер 5002) и 2X35S/CPMV160+ (конструкция номер 2109) для PDISP/HC ритуксимаба (Ритуксан)
[00178] Кодирующую последовательность, соответствующую тяжелой цепи моноклонального антитела IgG1 ритуксимаба, в которой нативный сигнальный пептид был заменен нативным сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/HC ритуксимаба (Ритуксан); Фигура 21A, SEQ ID NO: 61), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/HC ритуксимаба (Ритуксан). Аминокислотная последовательность зрелого НС ритуксимаба (Ритуксан), соединенная с PDISP, показана на Фигуре 21В (SEQ ID NO: 62). Конструкции плазмид 5002 и 2109 представлены на Фигурах 21С и 21D, соответственно.
[00179] Пример 17 - 2X35S/CPMV-HT (конструкция номер 5021) и 2X35S/CPMV160+ (конструкция номер 2120) для LC ритуксимаба (Ритуксан)
[00180] Кодирующую последовательность, соответствующую легкой цепи моноклонального антитела IgG1 ритуксимаба (LC ритуксимаба (Ритуксан); (Фигура 22А, SEQ ID NO: 63), клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для LC ритуксимаба (Ритуксан). Аминокислотная последовательность LC ритуксимаба (Ритуксан), соединенная с PDISP, показана на Фигуре 22В (SEQ ID NO: 64). Конструкции плазмид 5021 и 2120 представлены на Фигурах 22С и 22D, соответственно.
[00181] Пример 18 - 2X35S/CPMV-HT (конструкция номер 5022) и 2X35S/CPMV160+ (конструкция номер 2129) для PDISP/LC ритуксимаба (Ритуксан)
[00182] Кодирующую последовательность, соответствующую легкой цепи моноклонального антитела IgG1 ритуксимаба, в которой нативный сигнальный пептид был заменен нативным сигнальным пептидом протеин-дисульфид-изомеразы люцерны (PDISP/HC ритуксимаба (Ритуксан); Фигура 23А, SEQ ID NO: 65) клонировали в исходные CPMV-HT и CPMV160+ с применением того же метода ПЦР, что и для конструкций номер 1391 (см. Пример 1) и 1800 (см. Пример 2), соответственно, но с использованием модифицированных праймеров ПЦР, специально сконструированных для PDISP/LC ритуксимаба (Ритуксан). Аминокислотная последовательность зрелой LC ритуксимаба (Ритуксан), соединенная с PDISP, показана на Фигуре 23В (SEQ ID NO: 66). Конструкции плазмид 5022 и 2129 представлены на Фигурах 23С и 23D.
[00183] Пример 19 - трансфекция агробактерий
[00184] Штамм агробактерий AGL1 трансфецировали методом электропорации с конструкциями ДНК, используя методы, описанные D'Aoust et al., 2008 (Plant Biotechnology Journal 6:930-940). Трансфецированные Agrobacterium выращивали в среде YEB, дополненной 10 мМ 2-(N-морфолино)этансульфокислоты (MES), 20 мкМ ацетосирингона, 50 мкг/мл канамицина и 25 мкг/мл карбенициллина, с pH 5.6, до OD600 между 0.6 и 1.6. Суспензии агробактерий (Agrobacterium) перед использованием центрифугировали и ресуспендировали в инфильтрационной среде (10 мМ MgCl2 и 10 мМ MES рН 5.6).
Приготовление растительной биомассы, инокулюм (инокулят, посевной материал) и агроинфильтрация
[00185] Растения Nicotiana benthamiana (табак Бентхама) выращивали из семян в лотках (плоской посуде), заполненных продажным субстратом с белым мхом. Растения выращивали в теплице с фотопериодом (световым днем) 16/8 при температуре 25°С днем/20°С ночью. Через три недели после посева отдельные ростки выдергивали, пересаживали в горшки и оставляли расти в теплице еще в течение трех недель в тех же условиях.
[00186] Agrobacteria, трансфецированные с использованием каждой конструкции, выращивали в среде YEB, дополненной 10 мМ 2-(N-морфолино)этансульфокислоты (MES), 20 мкМ ацетосирингона, 50 мкг/мл канамицина и 25 мкг/мл карбенициллина, с рН 5.6, до OD600 между 0.6 и 1.6 Суспензии агробактерий (Agrobacterium) перед использованием центрифугировали, ресуспендировали в инфильтрационной среде (10 мМ MgCl2 и 10 мМ MES с pH 5.6) и хранили в течение ночи при 4°С. В день инфильтрации партии культур разводили в 2.5 объемах культур и оставляли нагреваться перед использованием. Целые растения N. benthamiana помещали кверху корнями в бактериальную суспензию в воздухонепроницаемом баке из нержавеющей стали под вакуумом 20-40 Торр (~2.67÷~5.33 кПа) на 2 мин. Затем растения возвращали в теплицу на 2-6 дней для инкубации до их сбора.
Сбор листьев и экстракция тотального белка
[00187] После инкубации собирали воздушную часть растений, замораживали при температуре -80°С и измельчали. Тотальные растворимые белки экстрагировали путем гомогенизации (Polytron) каждого образца замороженного и измельченного растительного материала в 3 объемах холодного 50 мМ Tris, pH 8.0, 0.15 М NaCl, 0.1% Triton Х-100 и 1 мМ фенилметансульфофторида. После гомогенизации суспензии центрифугировали при 10,000 g в течение 10 мин при 4°С и эти очищенные сырые экстракты (супернатант) хранили для анализа.
[00188] Пример 20 - Анализ белков и иммуноблоттинг
[00189] Содержание тотального белка в очищенных сырых экстрактах определяли методом (Мэрион) Бредфорд (Bio-Rad, Hercules, СА), используя бычий сывороточный альбумин в качестве референсного стандарта. Белки выделяли методом SDS-PAGE и осуществляли электрофоретический перенос (электроблоттинг) на поливинилиденфторидные (PVDF) мембраны (Roche Diagnostics Corporation, Indianapolis, IN) для иммунодетекции. Перед иммуноблоттингом мембраны обрабатывали 5% снятым (обезжиренным) молоком и 0.1% Tween-20 в физиологическом растворе с Tris-буфером (TBS-T) в течение 16-18 ч при 4°С.
[00190] Иммуноблоттинг проводили после первой инкубации с первичным антителом (в Таблице 4 показаны антитела и условия, использованные для детекции каждого НА) в 2 мкг/мл 2% обезжиренного молока с TBS-Tween 20 (0.1%). Вторичными антителами, использованными для детекции хемилюминесцентным методом, были антитела, указанные в Таблице 4, разведенные в 2% обезжиренном молоке с TBS-Tween 20 (0.1%). Иммунореактивные комплексы были обнаружены методом хемилюминесценции с использованием люминола в качестве субстрата (Roche Diagnostics Corporation).
[00191] 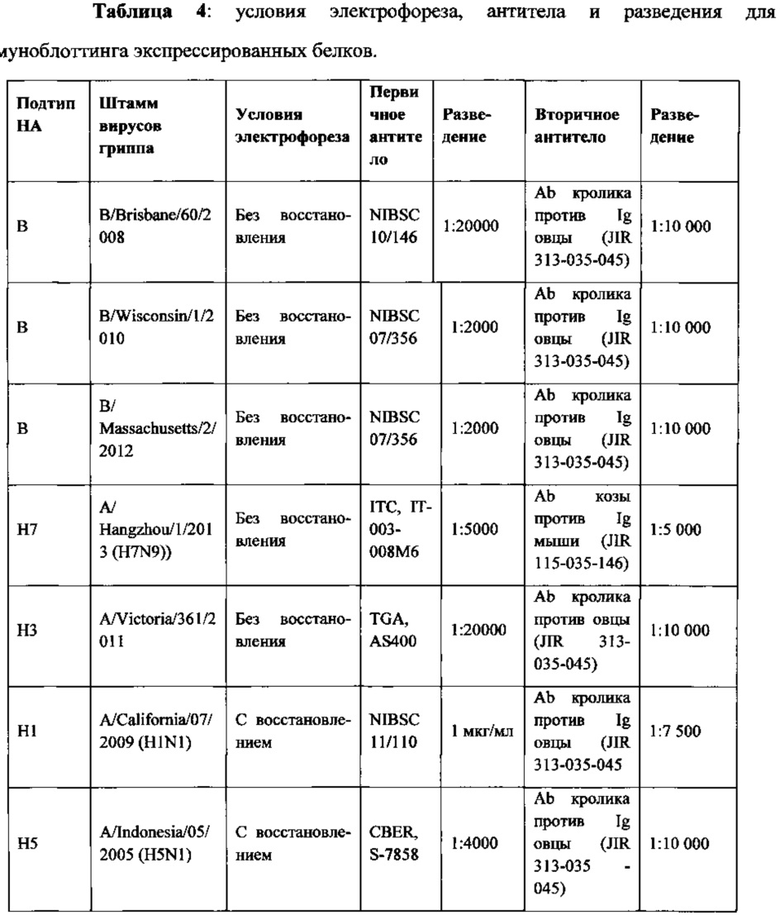
JIR: Jackson ImmunoResearch, West Grove, PA, USA;
CBER: Center for Biologies Evaluation and Research, Rockville, MD, USA.
Sino: Sino Biological inc., Beijing, China.
TGA: Therapeutic Goods Administration, Australia.
NIBSC: National Institute for Biological Standards and Control, United Kingdom
ITC: Immune Technology Corp., New York, NY, USA
[00192] Пример 21 - определение гемагглютинации
[00193] Определение гемагглютинации основано на использовании метода, описанного Nayak and Reichl (2004). Вкратце, серийные двойные разведения испытуемых образцов (100 мкл) получали в 96-луночном титрационном микропланшете с V-образным дном, содержащем 100 мкл PBS, в каждую лунку помещали 100 мкл разведенного образца. В каждую лунку добавляли 1000 мкл 0.25% суспензии красных кровяных телец (эритроцитов) индейки (Bio Link Inc., Syracuse, NY; для всех штаммов В, H1, Н5 и Н7) или 0.5% суспензии красных кровяных телец морской свинки (для Н3) и проводили инкубацию в течение 2 ч при комнатной температуре. Величину, обратную предельному разведению, свидетельствующему о полной гемагглютинации, регистрировали как активность НА.
[00194] Все приведенные публикации включены в данную заявку посредством отсылки.
[00195] Настоящее изобретение было описано на примере одного или более вариантов. Однако специалисту в данной области очевидно, что может быть сделан ряд изменений и модификаций без выхода за рамки данного изобретения, объем которого определяется формулой изобретения.
| название | год | авторы | номер документа |
|---|---|---|---|
| ПОЛУЧЕНИЕ ВИРУСОПОДОБНЫХ ЧАСТИЦ ВИРУСА ГРИППА В РАСТЕНИЯХ | 2014 |
|
RU2705555C2 |
| ПОЛУЧЕНИЕ ВИРУСОПОДОБНЫХ ЧАСТИЦ ВИРУСА ГРИППА В РАСТЕНИЯХ | 2014 |
|
RU2742607C1 |
| ПОВЫШЕНИЕ ВЫХОДА ВИРУСОПОДОБНЫХ ЧАСТИЦ В РАСТЕНИЯХ | 2012 |
|
RU2682752C2 |
| Получение вирусоподобной частицы вируса бешенства в растениях | 2012 |
|
RU2655433C2 |
| ПОЛУЧЕНИЕ ВИРУСОПОДОБНЫХ ЧАСТИЦ В РАСТЕНИЯХ | 2011 |
|
RU2655431C2 |
| ХИМЕРНЫЕ ВИРУСОПОДОБНЫЕ ЧАСТИЦЫ, СОДЕРЖАЩИЕ ГЕМАГГЛЮТИНИН, СХОДНЫЕ С ЧАСТИЦАМИ ВИРУСА ГРИППА | 2010 |
|
RU2569195C9 |
| МУТАНТЫ ГЕМАГГЛЮТИНИНА ВИРУСА ГРИППА | 2019 |
|
RU2800938C2 |
| СИНТЕТИЧЕСКИЕ 5 UTR (НЕТРАНСЛИРУЕМЫЕ ОБЛАСТИ), ЭКСПРЕССИОННЫЕ ВЕКТОРЫ И СПОСОБ ПОВЫШЕНИЯ ТРАНСГЕННОЙ ЭКСПРЕССИИ | 2008 |
|
RU2524431C2 |
| МОДИФИКАЦИЯ ПРОДУЦИРОВАНИЯ БЕЛКОВ В РАСТЕНИЯХ | 2015 |
|
RU2731295C2 |
| ПОЛУЧЕНИЕ ПИКОРНАВИРУСОПОДОБНЫХ ЧАСТИЦ В РАСТЕНИЯХ | 2013 |
|
RU2693431C2 |
































































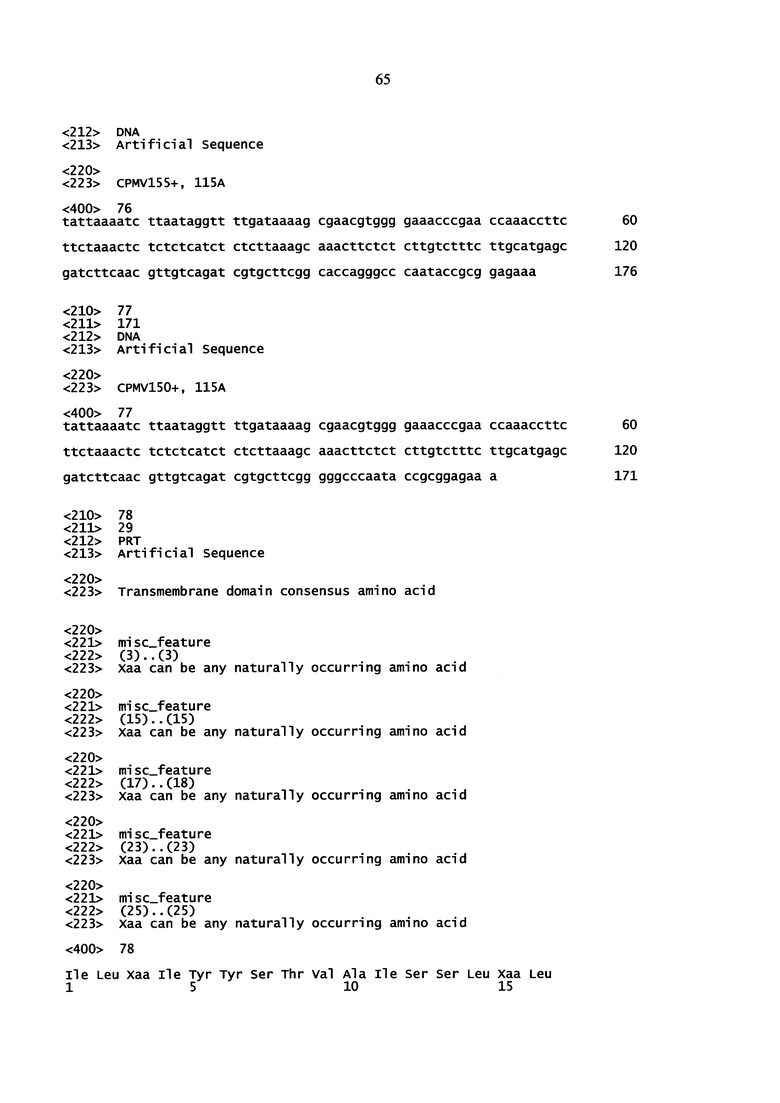

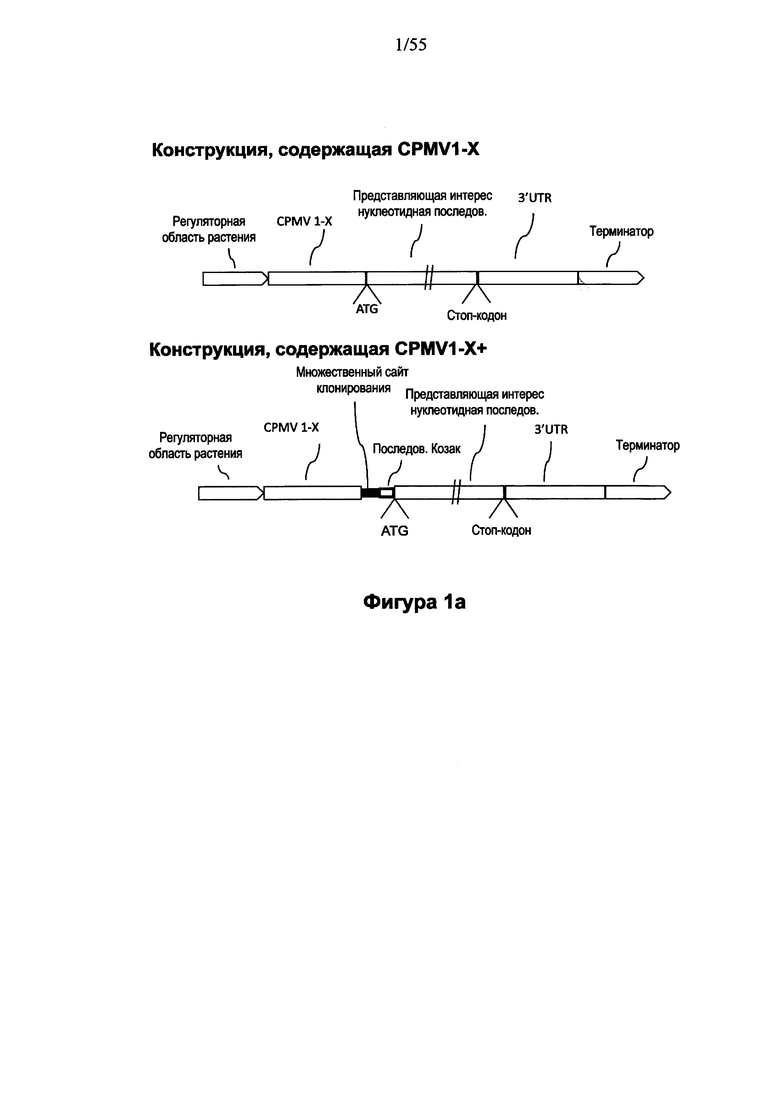


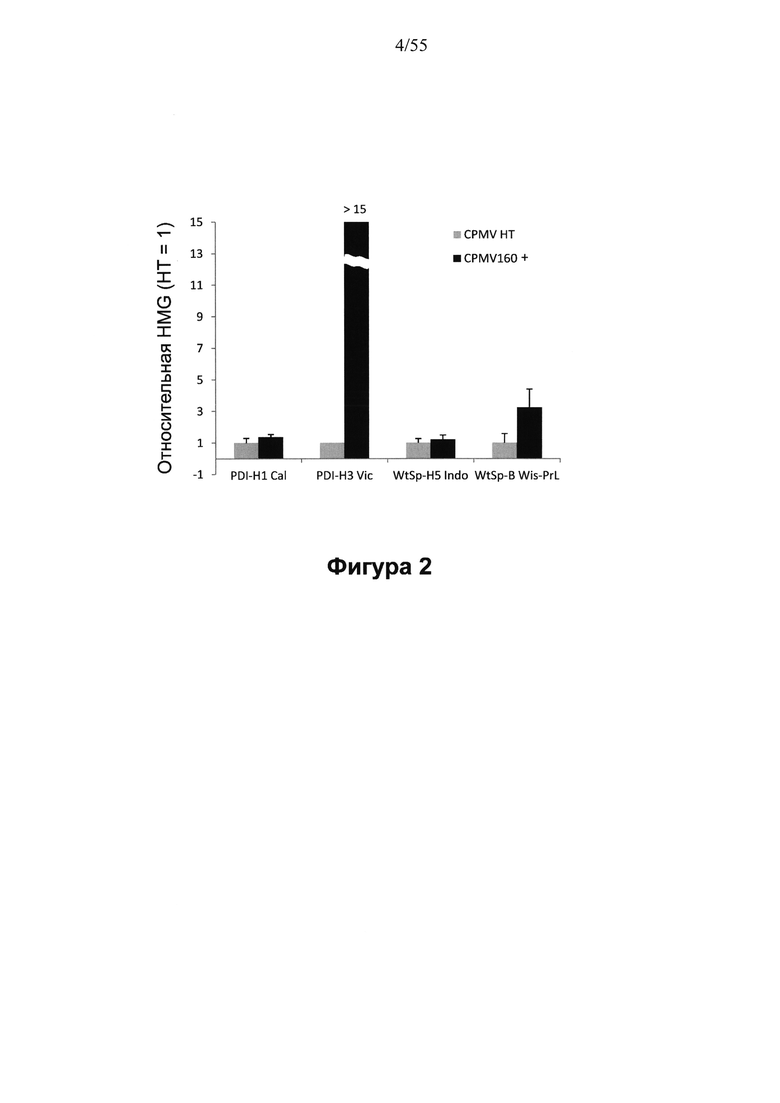
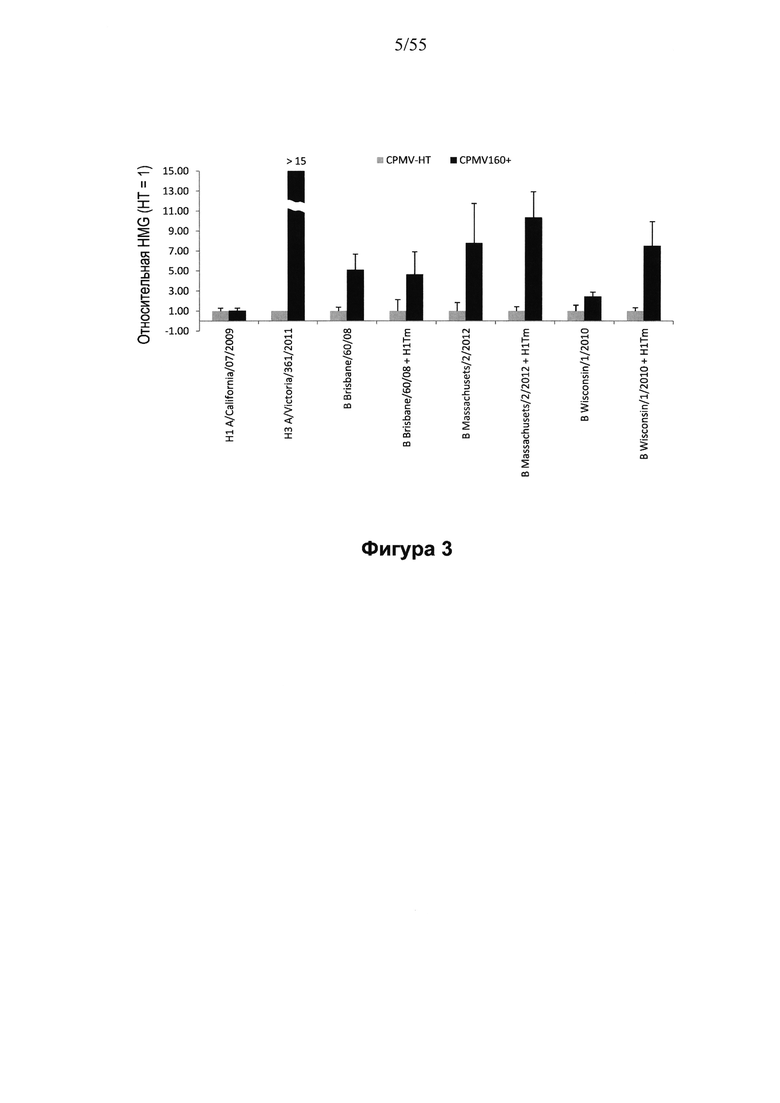
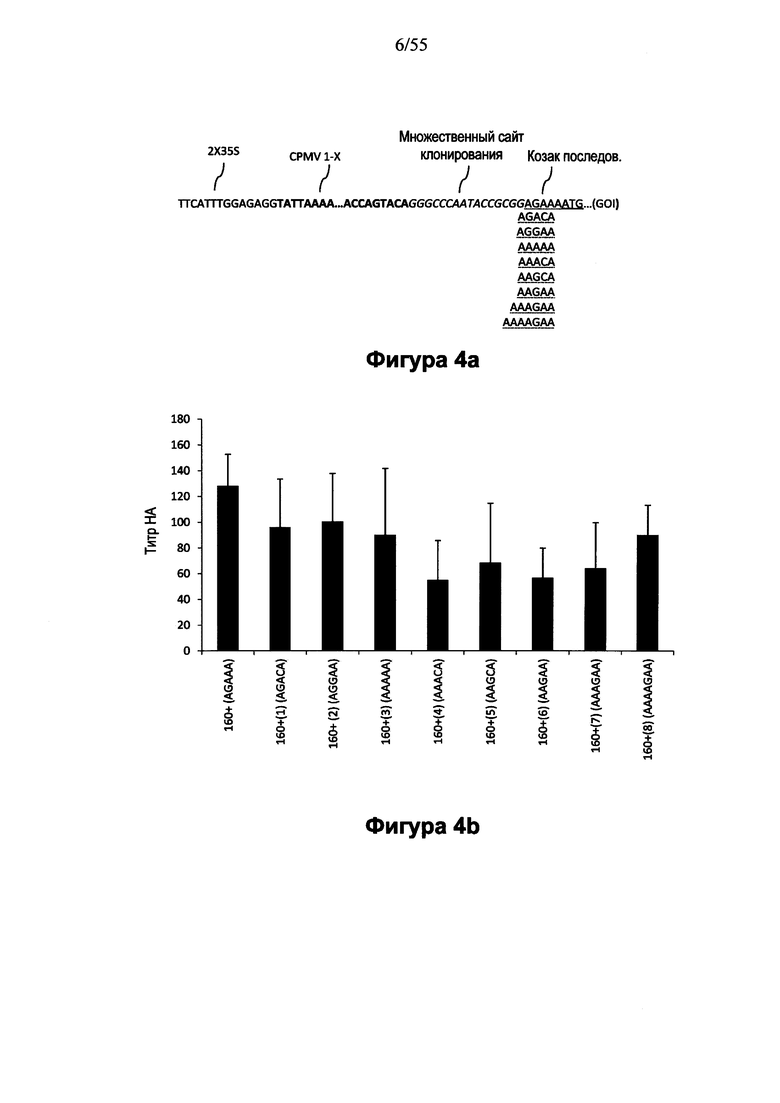
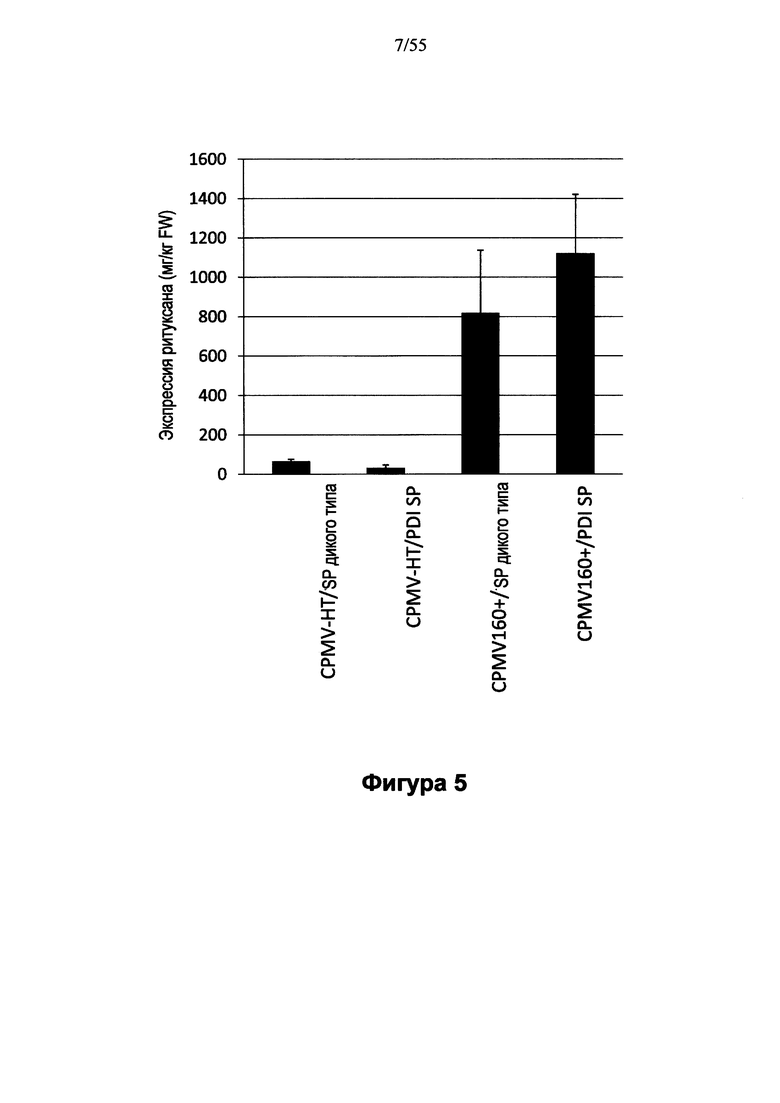














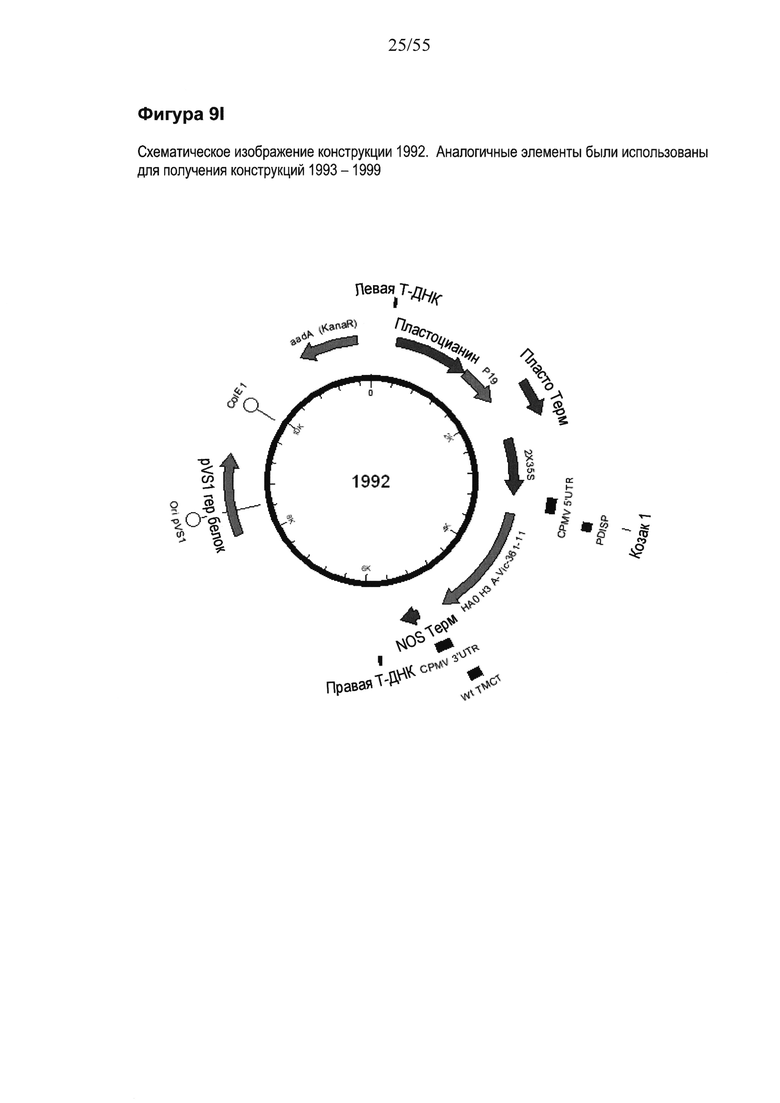



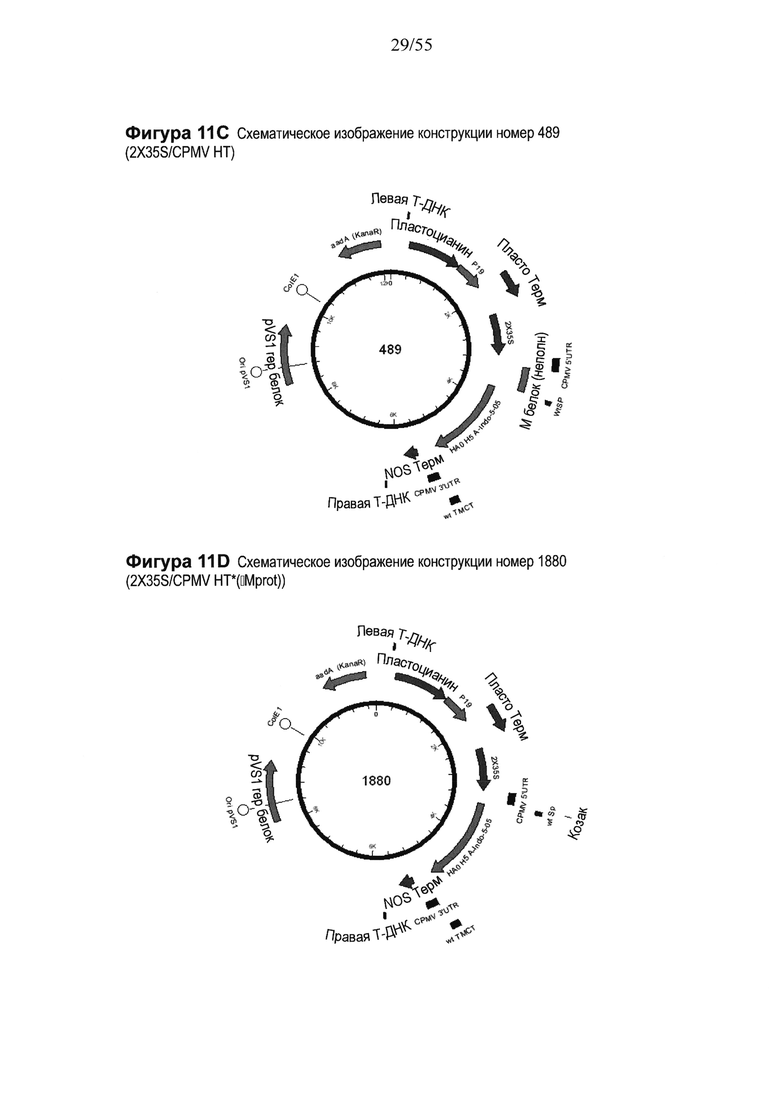
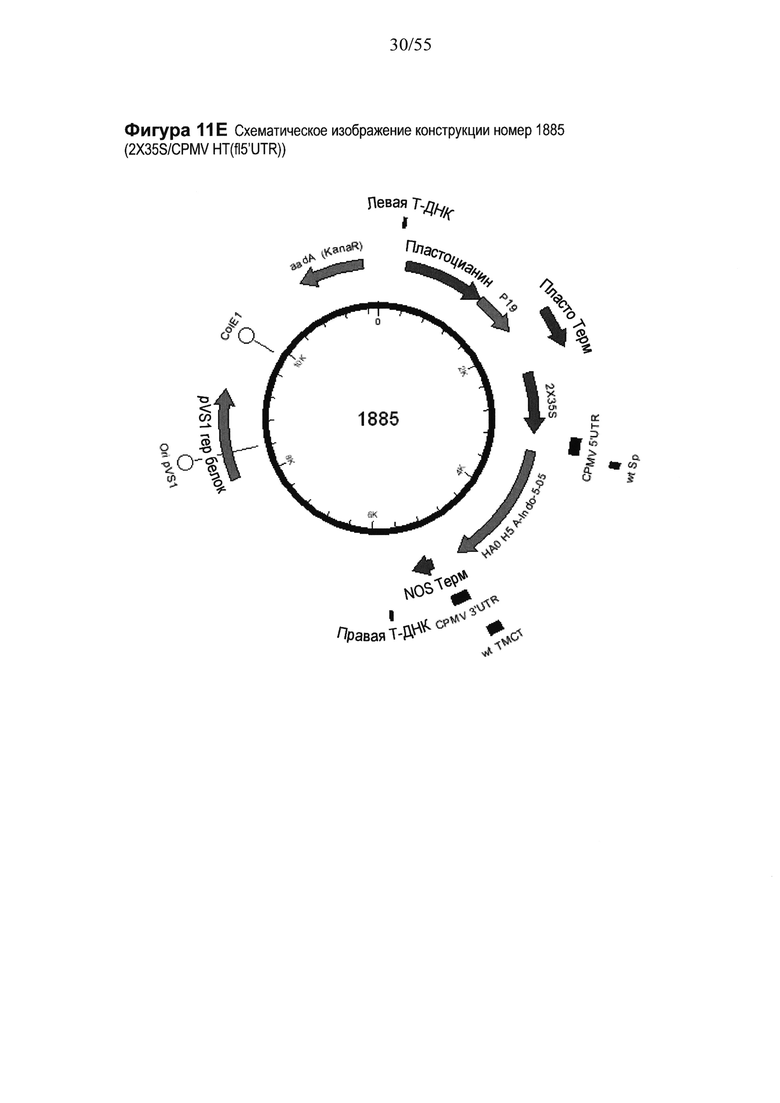

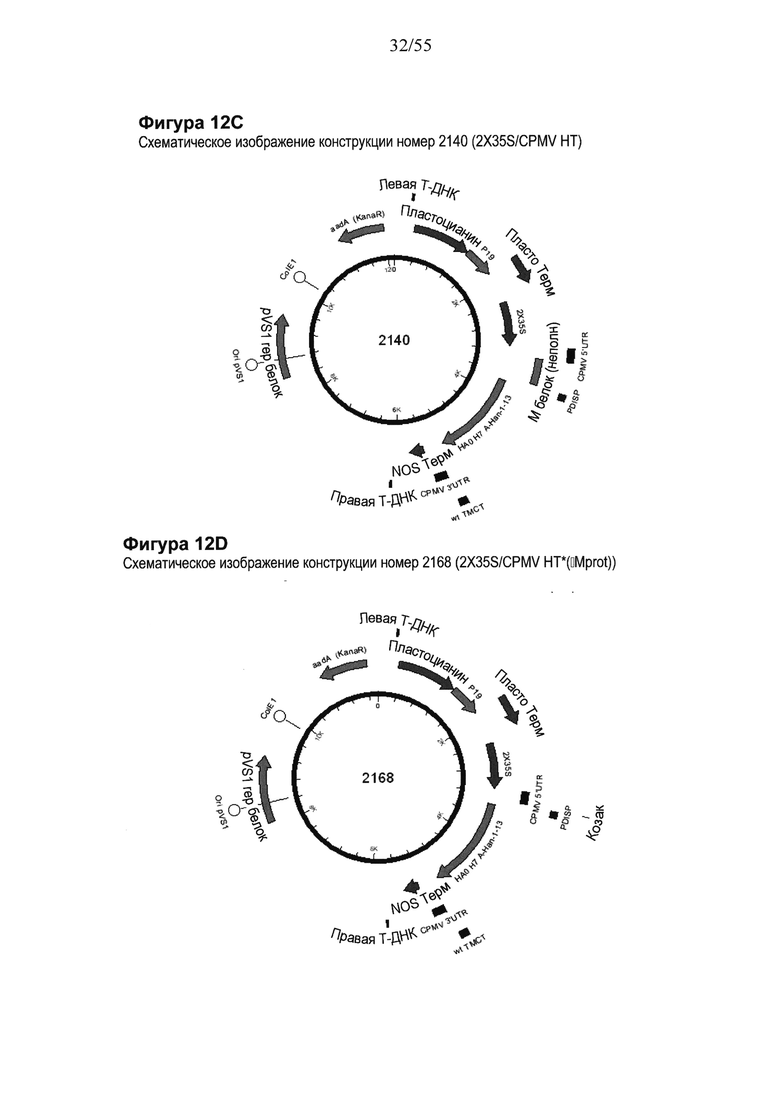













Изобретение относится к области биохимии, в частности к экспрессионной конструкции нуклеиновой кислоты, содержащей последовательность энхансера экспрессии, функционально связанную с представляющей интерес гетерологичной последовательностью, расположенной 3' к последовательности энхансера экспрессии, а также к растительной экспрессионной системе, содержащей вышеуказанную конструкцию. Также раскрыт способ продуцирования представляющего интерес белка в растении или в части указанного растения, предусматривающий использование вышеуказанной экспрессионной системы. Изобретение также относится к растению, а также к части растения, содержащим вышеуказанную растительную экспрессионную систему. Изобретение позволяет эффективно получать представляющий интерес белок в растении или в части растения. 5 н. и 19 з.п. ф-лы, 23 ил., 4 табл., 21 пр.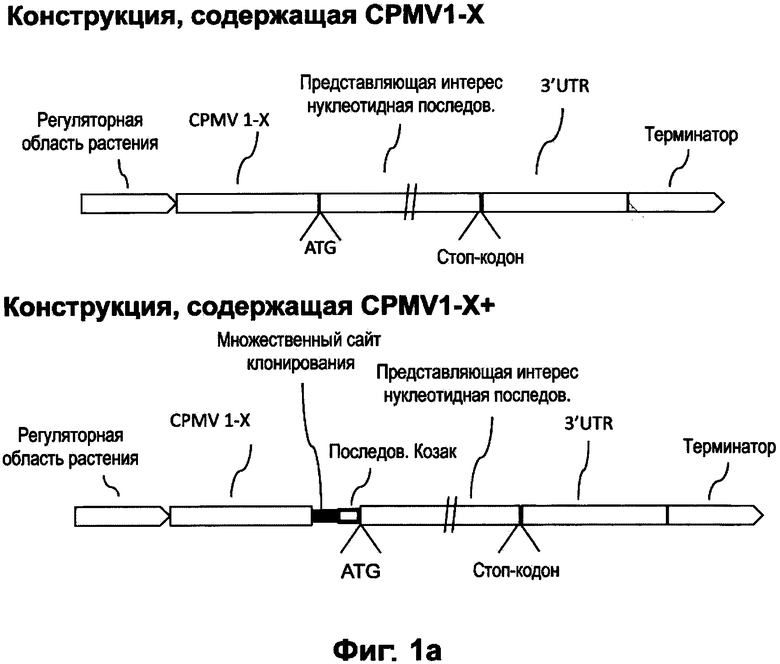
1. Экспрессионная конструкция нуклеиновой кислоты, содержащая последовательность энхансера экспрессии, функционально связанную с представляющей интерес гетерологичной последовательностью, расположенной 3' к последовательности энхансера экспрессии, причем последовательность энхансера экспрессии по существу состоит из 1-160 нуклеотидов последовательности SEQ ID NO: 1 или состоит по существу из 1-160 нуклеотидов последовательности SEQ ID NO: 1 с заменой G115A, и при этом ни последовательность энхансера экспрессии, ни представляющая интерес гетерологичная последовательность не содержат нуклеотиды 161-509 последовательности SEQ ID NO: 4.
2. Экспрессионная конструкция нуклеиновой кислоты по п. 1, в которой представляющая интерес гетерологичная последовательность выбрана из группы, состоящей из последовательности Козак для растений, множественного сайта клонирования, линкера, полилинкера, сайта рекомбинации, нуклеотидной последовательности, кодирующей белок, представляющий интерес, и их комбинации.
3. Экспрессионная конструкция нуклеиновой кислоты по п. 2, в которой представляющая интерес гетерологичная последовательность содержит последовательность Козак для растений.
4. Экспрессионная конструкция нуклеиновой кислоты по п. 3, в которой представляющая интерес гетерологичная последовательность содержит также множественный сайт клонирования.
5. Экспрессионная конструкция нуклеиновой кислоты по п. 3, в которой последовательность Козак выбрана из группы последовательностей, представленных в SEQ ID NO: 5-17.
6. Экспрессионная конструкция нуклеиновой кислоты по п. 2, содержащая нуклеотидную последовательность, представленную SEQ ID NO: 2.
7. Экспрессионная конструкция нуклеиновой кислоты по п. 1, в которой последовательность энхансера экспрессии состоит из SEQ ID NO: 69.
8. Экспрессионная конструкция нуклеиновой кислоты по п. 2, содержащая нуклеотидную последовательность SEQ ID NO: 75.
9. Растительная экспрессионная система, содержащая экспрессионную конструкцию нуклеиновой кислоты по п. 1, в которой последовательность энхансера экспрессии функционально связана с регуляторной областью.
10. Растительная экспрессионная система по п. 9, в которой экспрессионная конструкция нуклеиновой кислоты содержит также 3' UTR комовируса.
11. Растительная экспрессионная система по п. 9, содержащая также вторую нуклеотидную последовательность, при этом вторая нуклеотидная последовательность кодирует супрессор сайленсинга.
12. Растительная экспрессионная система по п. 11, в которой супрессор сайленсинга выбран из HcPro и р19.
13. Растительная экспрессионная система по п. 9, в которой регуляторная область выбрана из промотора пластоцианина, промотора CaMV 35S, промотора 2х CaMV35S, промотора CAS, промотора RbcS, промотора Ubi или промотора гена актина.
14. Растительная экспрессионная система по п. 9, в которой представляющая интерес гетерологичная последовательность содержит нуклеотидную последовательность, кодирующую вирусный белок или антитело.
15. Растительная экспрессионная система по п. 14, в которой вирусный белок представляет собой гемагглютинин гриппа, выбранный из группы, состоящей из H1, Н2, Н3, Н4, Н5, Н6, Н7, Н8, Н9, Н10, H11, Н12, Н13, Н14, Н15, Н16 гемагглютинина и гемагглютинина вируса гриппа типа В.
16. Растительная экспрессионная система по п. 14, в которой нуклеотидная последовательность, кодирующая вирусный белок или антитело, содержит последовательность нативного сигнального пептида или ненативного сигнального пептида.
17. Растительная экспрессионная система по п. 16, в которой ненативный сигнальный пептид получен из протеин-дисульфид-изомеразы (PDI).
18. Способ продуцирования представляющего интерес белка в растении или в части указанного растения, включающий введение в растение или в часть указанного растения растительной экспрессионной системы по п. 9, в которой представляющая интерес гетерологичная последовательность содержит нуклеотидную последовательность, кодирующую белок, представляющий интерес, и инкубацию растения или части указанного растения в условиях, которые допускают экспрессию нуклеотидной последовательности, кодирующей белок, представляющий интерес.
19. Растение для экспрессии белка, представляющего интерес, при этом растение содержит растительную экспрессионную систему по п. 9, в которой представляющая интерес гетерологичная последовательность содержит нуклеотидную последовательность, кодирующую белок, представляющий интерес.
20. Часть растения для экспрессии белка, представляющего интерес, при этом часть растения содержит растительную экспрессионную систему по п. 9, в которой представляющая интерес гетерологичная последовательность содержит нуклеотидную последовательность, кодирующую белок, представляющий интерес.
21. Экспрессионная конструкция нуклеиновой кислоты по п. 2, в которой представляющий интерес белок представляет собой гемагглютинин гриппа, выбранный из В НА, С, H1, Н2, Н3, Н4, Н5, Н6, Н7, Н8, Н9, Н10, H11, Н12, Н13, Н14, Н15 и Н16.
22. Экспрессионная конструкция нуклеиновой кислоты по п. 21, в которой НА является химерным НА, при этом нативный трансмембранный домен белка НА заменен гетерологичным трансмембранным доменом.
23. Экспрессионная конструкция нуклеиновой кислоты по п. 22, в которой гетерологичный трансмембранный домен получен из вируса H1 California.
24. Экспрессионная конструкция нуклеиновой кислоты по п. 2, в которой последовательность энхансера экспрессии слита с представляющей интерес гетерологичной последовательностью.
| SAINSBURY F | |||
| et al., pEAQ: versatile expression vectors for easy and quick transient expression of heterologous proteins in plants, Plant Biotechnol J., 2009, Vol.7, N.7, pp.682-693 | |||
| WO2013068593 A1, 16.05.2013 | |||
| WO2009076778 A1, 25.06.2009 | |||
| ИСКУССТВЕННЫЙ ПРОМОТОР ДЛЯ ЭКСПРЕССИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ ДНК В РАСТИТЕЛЬНЫХ КЛЕТКАХ | 2003 |
|
RU2326167C2 |
Авторы
Даты
2019-09-11—Публикация
2015-01-08—Подача