ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
[0001] По настоящей заявке испрашивается приоритет предварительной заявки США 61/875845, поданной 10 сентября 2013 года. Полное содержание указанной заявки включено в настоящую заявку в качестве ссылки.
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
[0002] Настоящее изобретение относится к композициям и способам идентификации растений канолы, обладающих резистентностью к болезни черная ножка, где в способах используют молекулярно-генетические маркеры для идентификации, отбора и/или конструирования растений, резистентных к черной ножке, и, в частности, для отбора или конструирования растений с геном резистентности к черной ножке Rlm4. Настоящее изобретение также относится к растениям канолы, которые являются резистентными к черной ножке, которые получают способами по настоящему изобретению.
УРОВЕНЬ ТЕХНИКИ
[0003] Черная ножка является серьезным заболеванием канолы, которое может привести к значительным потерям урожая у восприимчивых сортов в Канаде, Европе и Австралии. Черная ножка является наиболее распространенным названием, но эту болезнь также называют стеблевым раком или фомозом стеблей. Черную ножку вызывает гриб Leptosphaeria maculans. Высокоагрессивный штамм гриба черной ножки был впервые обнаружен в 1975 году в Саскачеване, но сейчас он встречается в большинстве областей выращивания канолы в западной части Канады.
[0004] Ежегодно проводятся полевые исследования болезни канолы в основных областях ее выращивания в западной части Канады. Исследования болезни, проведенные в последние годы, свидетельствуют о том, что черная ножка обычно встречается в 35-55 процентах исследованных культур. Средние значения заболеваемости (процент растений с симптомами черной ножки), как правило, составляют один процент от базального стеблевого рака и три процента от поражений, возникающих в других местах на стебле. Поражения, происходящие в других местах на стебле, имеют меньшее влияние на урожай и качество семян, чем базальный стеблевой рак. Самые высокие значения заболеваемости в основном наблюдаются в зерновых культурах, которые получили повреждения градом.
[0005] Как указывают исследования болезни, симптомы черной ножки не являются необычными для посевов канолы, даже если выращиваются резистентные к ней сорта. Тем не менее, для предотвращения отрицательного влияния черной ножки на урожай семян и их качество, важно знать симптомы черной ножки, цикл заболевания, а также способы контроля заболевания.
[0006] Черная ножка является наиболее серьезным заболеванием канолы в Австралии. Тяжесть заболевания черной ножки возросла в последние годы в связи с увеличением посевных площадей и интенсивности производства. Хотя и не повсеместно, но в некоторых сезонах потери урожая были зафиксированы на уровне 50 процентов и выше, и вплоть до 90 процентов потери урожая происходит в тех случаях, когда в определенных сортах L.maculans преодолевает основную генетическую резистентность к черной ножке.
[0007] В осенне-зимний период осадки вызывают выход спор из пожнивных остатков. В течение двух недель споры распространяются на семядоли канолы и на молодые листья, хорошо видно развитие повреждений грязно-белого цвета, в пределах поражения пикнидиевые плодовые тела (темные точки) высвобождают споры, распространяемые дождем. Инфекция черной ножкой может возникать на семядолях, листьях, стеблях и стручках. Растение чувствительно к инфекции черной ножкой от стадии рассады до стадий образования стручка. Поражения, возникающие на листьях, имеют грязно-белый цвет и неправильную округлую форму. На стеблях повреждения, вызываемые черной ножкой, могут быть весьма различны, но обычно располагаются у основания стебля или в местах прикрепления листа. После того, как поражение сформировалось, гриб растет внутри сосудистой системы растения в направлении кроны, где вызывает гниль кроны растения, что приводит к раку. Тяжелая форма рака будет разрывать связь корней и стебля, а менее тяжелая форма инфекции будет приводить к внутренней инфекции кроны, ограничивая ток воды и питательных веществ внутри растения. Поражения ствола могут доходить до нескольких дюймов в длину, и, как правило, имеют белый или серый цвет с темной каймой. Стволовые поражения могут также возникать как почернение по всему основанию. Тяжелая форма инфекции обычно приводит к сухой гнили или раку у основания стебля. Стебель становится окольцованным и, после того, как растения преждевременно созревают, урожай, скорее всего, погибнет. Семена могут быть сморщенными, а стручки легко разрушаются при сборе урожая, что приводит к потере семян.
[0008] В последние годы симптомы черной ножки были также обнаружены в корнях растений, эта корневая инфекция в тяжелых случаях, по-видимому, вызывает преждевременную гибель всего растения. Форма заболевания, называемая корневая гниль, вызывается одними и теми же штаммами черной ножки, которые вызывают стеблевой рак, а способы контроля нормальной черной ножки являются такими же, как для формы заболевания, называемой корневой гнилью.
[0009] С увеличением акров высевания канолы и более частым севооборотом, болезнь черная ножка снова стала важным заболеванием, снижающим урожайность и качество.
[0010] Резистентность сортов является лучшей защитой на сегодняшний день против болезни черная ножка. Развитие сортов, резистентных к черной ножке, привело к снижению экономических потерь, связанных с черной ножкой. В результате, многие фермеры выращивают резистентные сорта и, таким образом, могут уйти от более частого севооборота. Предварительные наблюдения, начиная с 2010 года, при исследовании болезни канолы показывают, что в Манитобе у сортов категории R (resistant) отмечена более высокая частота возникновения заболевания черная ножка и более тяжелое ее течение. У L.maculans возникли новые варианты, и теперь эти новые варианты способны инфицировать сорта канолы, раннее относящиеся к категории R.
[0011] Когда впервые были разработаны сорта, резистентные к черной ножке, исследователи классифицировали патоген на патогруппы "PG", на основании инфицирующей способности изолята в отношение 3 сортов - Westar, Glacier и Quinta. В связи с изменением популяции черной ножки классификации PG стало недостаточно для описания новейших вариантов. Это означает, что система PG не может идентифицировать видоизменяемость изолятов, в результате чего источники резистентности не обнаружены в «Quinta» и «Glacier». На основе молекулярных маркеров изоляты патогенов стало возможно классифицировать на группы PG гораздо быстрее, чем традиционными способами инокуляции растений (Dusabenyagasani, M., and Fernando, W. G. D. 2008).
[0012] При новом подходе, используя сорта или линии Brassica spp., несущие 14 специфических гена резистентности, новые изоляты L.maculans могут быть дифференцированы или охарактеризованы как отдельные расы на основе наблюдаемых реакций. В документе Rimmer (2007) сообщалось об этих 14 генах специфической резистентности, обозначенных как Rlm 1 - Rlm 10 и LepR1 - LepR4. Изучение 96 изолятов гриба черной ножки западной части Канады, используя десять генов резистентности, показало значительное различие в популяции патогена для большого числа этих генов (Kutcher et al. 2010).
[0013] При получении нового желательного генетического материала растения существует множество стадий. Программы селекции растений объединяют желательные признаки двух или более культурных сортов или различных универсальных источников в фонд селекции, где культурные сорта выращивают при помощи самоопыления и отбора желаемых фенотипов. Новые культурные сорта оценивают для определения того, какие из них имеют коммерческий потенциал. Селекция растений начинается с анализа и определения проблем и недостатков имеющейся на данный момент генетического материала, создания программных целей, а также определения конкретных целей селекции. Следующей стадией является отбор генетического материала, который обладает признаками, соответствующими программным целям. Эта цель состоит в объединении в одном сорте улучшенного сочетания желательных признаков родительского генетического материала. Эти важные признаки могут включать более высокую урожайность семян, резистентность к болезням и насекомым, лучшие показатели стеблей и корней, резистентность к засухе и жаре, а также улучшенное агрономическое качество.
[0014] Выбор способов селекции или отбора зависит от способа размножения растений, наследуемости улучшенного признака(ов) и типов культурных сортов, используемых в коммерческих целях (например, F1 гибридный культурный сорт, культурный сорт чистой линии и тому подобное). Для высоконаследуемых признаков выбор наилучших индивидуальных растений, оцененных в одном местоположении, может быть эффективным, при этом для низконаследуемых признаков отбор должен основываться на средних значениях, полученных из воспроизводимых оценок семейств родственных растений. Популярные методы отбора обычно включают селекцию на базе родословной, модифицированную селекцию на базе родословной, массовый отбор и рекуррентную селекцию.
[0015] Сложность наследования влияет на выбор способа селекции. Возвратное скрещивание используют для переноса желательных аллелей одного или нескольких генов высоконаследуемого признака в желаемый культурный сорт. Такой подход широко используется для селекции резистентных к болезням культурных сортов. Различные способы рекуррентной селекции используют для улучшения количественно-наследуемых признаков под контролем многочисленных генов. Использование рекуррентной селекции в самоопыляющихся культурах зависит от легкости опыления, частоты успешных гибридов от каждого опыления и количества гибридного потомства от каждого успешного скрещивания.
[0016] Каждая программа селекции должна включать периодическую объективную оценку эффективности способа селекции. Критерий оценки изменяется в зависимости от цели и задач, но должен включать положительный эффект отбора в течение года (на основании сравнений с соответствующим стандартом), общий объем улучшенных линий селекции, а также число успешных культурных сортов, производимых на единицу затрат (например, в год, на один затраченный доллар и тому подобное). Перспективные улучшенные линии селекции затем тщательно проверяют и сравнивают с соответствующими стандартами в характерной окружающей среде коммерческой целевой зоны(зон) в течение трех или более лет. Кандидаты на новые коммерческие культурные сорта выбирают из лучших линий; те линии, у которых сохраняются некоторые недостатки, могут использоваться в качестве родительских для создания новых популяций для дальнейшего отбора. Эти процессы, ведущие к конечной стадии маркетинга и поставкам, как правило, занимают от 8 до 12 лет с момента осуществления первого скрещивания. Таким образом, разработка новых культурных сортов является длительным процессом, который требует точного перспективного планирования, эффективного использования ресурсов, а также минимальных изменений в направлении.
[0017] Наиболее трудной задачей при селекции растений является выявление отдельных растений с наилучшей генетикой. Один из способов выявления растения с наилучшей генетикой состоит в наблюдении за продуктивностью растения по сравнению с другими экспериментальными растениями и по сравнению с повсеместно используемым стандартным культурным сортом. Если единственное наблюдение не дает требуемых результатов, то повторные наблюдения обеспечивают более точную оценку его генетической ценности. Эта задача очень сложна, потому что (для большинства признаков) истинная ценность генотипа маскируется другими смешивающимися признаками растений или факторами окружающей среды.
[0018] Целью селекции растений канолы является разработка новых, уникальных и улучшенных культурных сортов канолы и ее гибридов. Селекционер вначале отбирает и скрещивает две или более родительских линий с последующим повторным самоопылением и отбором, производя большое число новых генетических комбинаций. Селекционер может теоретически получить миллиарды различных генетических комбинаций путем скрещивания, самоопыления и мутагенеза. Такой селекционер не имеет прямого контроля над процессом на клеточном уровне. Таким образом, два селекционера никогда не смогут создать одну и ту же линию или даже очень схожие линии, имеющие одинаковые признаки канолы.
[0019] Каждый год растениевод-селекционер отбирает генетический материал, чтобы продвинуться к следующему поколению. Такой генетический материал выращивают под воздействием уникальных и различных географических, климатических и почвенных условий. Затем проводят дальнейший отбор в процессе вегетационного периода и в его конце. Созданные культурные сорта являются непредсказуемыми. Такая непредсказуемость обусловлена отбором, проводимым селекционером, который происходит в уникальных условиях окружающей среды и который не позволяет контролировать уровень ДНК (используя традиционные способы селекции), при наличии миллиона возможных различных генетических комбинаций, которые были получены. Селекционер, являющийся средним специалистом в данной области, не может спрогнозировать окончательные полученные линии, которые он разрабатывает, за исключением, возможно, в очень грубой и общей форме. Аналогично, тот же селекционер не может получить одинаковый культурный сорт дважды, используя одинаковые родительские линии и одинаковые способы отбора. Такая непредсказуемость приводит к расходованию большого количества ресурсов, финансов и к другим расходам, при создании новых улучшенных культурных сортов канолы.
[0020] Создание новых культурных сортов канолы требует создания и отбора сортов канолы, скрещивания этих сортов, а также отбора наилучших гибридных линий скрещивания. Гибридные семена получают путем ручного скрещивания выбранных родительских линий с мужской фертильностью, или используя систему мужской стерильности. Эти гибриды отбирают по определенному генному признаку (например, цвету стручка, цвету цветка, цвету опушенности стебля и резистентности к гербицидам), который указывает на то, что семя действительно является гибридом. Данные о родительских линиях, а также о фенотипе гибрида, влияют на решение селекционера о дальнейшем продолжении скрещивания этого конкретного гибрида.
[0021] Селекция по родословной широко используется для улучшения самоопыляющихся культур. При селекции по родословной две родительские линии, которые обладают подходящими дополнительными признаками, скрещивают с получением потомства F1. Популяцию F2 получают при самоопылении одного или нескольких растений из поколения потомства F1. Отбор лучших особей можно начать в популяции F2; затем, начиная с F3, отбирают лучшие особи в лучших семействах. Для повышения эффективности отбора по низконаследуемым признакам повторное тестирование семейств можно начинать с поколения F4. На заключительной стадии инбридинга (например, F6 или F7) лучшие линии или смеси линий со сходными фенотипами тестируют на возможное возникновение новых культурных сортов. Массовая и рекуррентная селекция могут быть использованы для улучшения популяций либо самоопыляющихся культур, либо перекрестно-опыляющихся культур. Генетически регулируемая популяции гетерозиготных особей может быть либо идентифицирована, либо создана скрещиванием нескольких разных родителей. Лучшие растения могут быть отобраны на основе индивидуальных преимуществ, выдающегося потомства или прекрасной скрещиваемости. Отобранные растения скрещивают с получением новой популяции, в которой могут быть продолжены дальнейшие циклы отбора.
[0022] Возвратное скрещивание используют для переноса генов низконаследуемого и высоконаследуемого признака в желательный гомозиготный культурный сорт или инбредную линию, которая является рекуррентной родительской линией. Источником признака для переноса является «родитель-донор». Полученное растение должно иметь свойства рекуррентной родительской линии (например, культурного сорта растения), и желательный признак, перенесенный от родителя-донора. После первоначального скрещивания отбирают особи с фенотипом родителя-донора, и их повторно скрещивают (возвратное скрещивание) с рекуррентной родительской линией. В результате ожидается, что растение будет иметь свойства рекуррентной родительской линии и желаемый признак, перенесенный от родителя-донора.
[0023] При селекции канолы «способ поколение из одного семени» относится к посадке сегрегирующейся популяции, после чего происходит сбор образца одного семени с каждого полученного растения, и использованию собранного образца одного семени для посадки следующего поколения. Когда популяция вырастет из поколения F2 до желаемого уровня инбридинга, растения, из которых выведены линии, будут каждый раз давать различные особи F2. С каждым поколением число растений в популяции снижается из-за неспособности некоторых семян к прорастанию или неспособности некоторых растений дать по крайней мере одно семя. В результате, не все растения F2, первоначально взятые в популяции, будут представлены потомством после завершения выращивания поколения.
[0024] При способе со множеством семян селекционеры канолы обычно собирают семена с каждого растения в популяции, и обмолачивают их вместе с формированием основной массы. Часть этой массы используют для посадки следующего поколения, а часть оставляют в резерве. Этот способ называется модифицированным способом поколения из одного семени. Способ со множеством семян используется для экономии труда, связанного со сбором. Сбор семян при помощи машин намного быстрее, чем сбор одного семени от каждого растения вручную, как при способе с одним семенем. Способ со множеством семян также позволяет посадить аналогичное число семян популяции для каждого поколения инбридинга. Собирают достаточное количество семян для компенсации числа растений, которые не прорастают или не дают семян.
[0025] Правильный контроль должен обнаруживать любые крупные ошибки и устанавливать уровень преимущества или улучшения нового культурного сорта по сравнению с существующими культурными сортами. Кроме наличия улучшенных характеристик должен быть спрос на новый культурный сорт, который согласуется с промышленными стандартами или для него должен быть создан новый рынок. При тестировании, предшествующем выпуску нового сорта культурного растения, следует принимать во внимание исследования и затраты на разработку, а также техническое преимущество конечного культурного сорта. Внедрение нового культурного сорта может повлечь за собой дополнительные затраты производителя семян, растениевода, технолога и потребителя, из-за специальных требований к рекламе и маркетингу, изменению семян и к практике коммерческого производства и использованию нового продукта. Для культурных сортов, предназначенных для получения семян, должна существовать возможность легкого и экономичного получения семян.
[0026] Целью растениевода-селекционера является отбор растений и обогащение популяции растений особями, которые обладают желаемыми признаками, например, резистентностью к болезням или резистентностью к черной ножке, что в конечном итоге приводит к повышению аграрной продуктивности. В соответствии с вышеизложенным, постоянной целью селекционеров канолы является создание стабильных высокоурожайных культурных сортов, которые являются агрономически значимыми. Существующие в настоящее время цели включают максимальное увеличение количества зерна, выращенного на используемой земле, и поставок продуктов питания как для животных, так и для людей. Для достижения этих целей селекционер канолы должен отбирать и создавать растения канолы, у которых имеются признаки, приводящие к улучшению культурных сортов, и он должен осуществлять это наиболее рентабельным способом. Молекулярные маркеры могут быть использованы в способе отбора с помощью маркеров (MAS) для содействия в идентификации и отборе особей или семейств особей, которые обладают унаследованными свойствами, которые сцеплены с маркерами.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[0027] Молекулярные маркеры, которые сцеплены с резистентностью к черной ножке, могут быть использованы для упрощения отбора при помощи маркеров по признаку резистентности к черной ножке у канолы. Отбор при помощи маркеров дает значительные преимущества по времени, финансовым затратам и трудоемкости по сравнению с фенотипированием резистентности к черной ножке. В настоящем документе описаны конкретные маркеры, идентифицированные в пределах областей резистентности к черной ножке или вблизи них в геноме канолы, которые являются полиморфными в родительских генотипах и которые сцеплены (например, тесно сцеплены) с фенотипом резистентности к черной ножке. Эти маркеры, обеспечивают превосходную практическую ценность при отборе при помощи маркеров растений канолы и культурных сортов, обладающих резистентностью к черной ножке.
[0028] В настоящем документе представлены способы идентификации первого растения канолы, которое проявляет резистентность к черной ножке, или генетического материала, содержащийся в таком растении канолы. Первое растение канолы или генетический материал, которые проявляют резистентность к черной ножке, могут в некоторых случаях представлять собой растение или генетический материал, которые обладают резистентностью к черной ножке (или улучшенной резистентностью к черной ножке), по сравнению с родительским растением или генетическим материалом первого растения или генетическим материалом. Первое растение канолы или генетический материал, которые проявляют устойчивость к черной ножке, могут в некоторых случаях представлять собой растение или генетический материал, обладающие улучшенной резистентностью к черной ножке, чем наблюдаемая в том или ином конкретном обычном растении или генетическом материале того же вида (например, канола) как первое растение или генетический материал. Некоторые варианты осуществления таких способов могут включать детекцию в первом растении канолы или генетическом материале по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке, где по меньшей мере один маркер выбран из группы, состоящей из: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705; и маркеров, связанных (например, тесно связанных ) с любым из DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705.
Также описаны способы получения растения канолы или генетического материала, обладающих резистентностью к черной ножке. Некоторые варианты осуществления таких способов могут включать интрогрессию по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке, из первого растения канолы или генетического материала во второе растение канолы или генетический материал с получением растения канолы или генетического материала, которые, вероятно, будут обладать резистентностью к черной ножке. В таких примерах по меньшей мере один маркер может быть выбран из группы, состоящей из: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705. Растение канолы или генетический материал, полученные вышеуказанными способами, также включены в конкретные варианты осуществления.
[0029] Также описаны способы получения растения канолы или генетический материал, обладающих резистентностью к черной ножке. Некоторые варианты осуществления таких способов могут включать интрогрессию по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке, из первого растения канолы или генетического материала во второе растение канолы или генетический материал с получением растения канолы или генетического материала, которые, вероятно, будут обладать резистентностью к черной ножке. В таких примерах по меньшей мере один маркер может быть выбран из группы, состоящей из: DBSNP09246, DBSNP01407, DBSNP05863 и DBSNP01261. Растение канолы или генетический материал, полученные вышеуказанными способами, также включены в конкретные варианты осуществления.
[0030] Некоторые варианты осуществления включают способы получения трансгенного растения канолы. Примеры таких способов могут включать введение одной или более экзогенных молекул нуклеиновой кислоты в мишеневое растение канолы или его потомство, где по меньшей мере одна из одной или нескольких экзогенных молекул нуклеиновой кислоты содержит геномную нуклеотидную последовательность канолы, которая сцеплена по меньшей мере с одним маркером, который сцеплен с резистентностью к черной ножке, или где по меньшей мере одна из одной или нескольких экзогенных молекул нуклеиновой кислоты содержит нуклеотидную последовательность, которая специфически гибридизуется с нуклеотидной последовательностью, которая сцеплена по меньшей мере с одним маркером, сцепленным с резистентностью к черной ножке. Маркер, который сцеплен с резистентностью к черной ножке, может быть выбран из группы, состоящей из: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590, и маркеров, сцепленных с любым из DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705. В некоторых примерах приведенных выше способов получения трансгенного растения канолы, полученное трансгенное растение канолы может обладать резистентностью к черной ножке.
[0031] Некоторые варианты осуществления включают системы и наборы для идентификации растения канолы, которое, вероятно, обладает резистентностью к черной ножке. Конкретные примеры таких систем и наборов могут включать набор зондов нуклеиновых кислот, каждый из которых содержит нуклеотидную последовательность, которая специфически гибридизуется с нуклеотидной последовательностью, которая сцеплена в каноле по меньшей мере с одним маркером, который сцеплен с резистентностью к черной ножке. Маркер, который сцеплен в каноле с резистентностью к черной ножке, может быть выбран из группы, состоящей из: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590, и маркеров, сцепленных с любым из DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590. Конкретные примеры систем и наборов для идентификации растения канолы, которое может обладать резистентностью к черной ножке, также могут включать детектор, конфигурация которого позволяет детектировать один или несколько выходных сигналов набора зондов нуклеиновых кислот, или их ампликона, таким образом, идентифицируя наличие или отсутствие по меньшей мере одного маркера, который сцеплен с резистентностью к черной ножке. Конкретные примеры включают инструкции, которые устанавливают зависимость наличия или отсутствия по меньшей мере одного маркера с вероятной резистентностью к черной ножке.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Описание некоторых вариантов осуществления
[0032] По ряду причин желательно вырастить канолу, обладающую резистентностью к черной ножке. Варианты осуществления настоящего изобретения включают, например, Композиции и способы идентификации растений канолы, обладающих резистентностью к черной ножке и/или генетического материала, несущей генотип, который является прогностическим и определяющим фенотип резистентности к черной ножке. Способы получения таких растений канолы и генетического материала включены в некоторые варианты осуществления. Такие способы могут включать, например, но ими не ограничиваясь, интрогрессию желаемых маркерных аллелей резистентности к черной ножке и/или способы генетической трансформации. Растения канолы и/или генетический материал, полученные с помощью таких способов, как указано выше, включены в конкретные варианты осуществления. Системы и наборы для отбора растений канолы, обладающих резистентностью к черной ножке, и/или генетического материала, несущей генотип, который является прогностическим и определяющим резистентность к черной ножке, также являются признаком некоторых вариантов осуществления.
[0033] Идентификация и отбор растений канолы, обладающих резистентностью к черной ножке, с помощью MAS, способны обеспечить эффективный и экологически чистый подход к созданию растений с резистентностью к желаемому заболеванию. Варианты осуществления настоящего изобретения относятся к ряду маркерных локусов канолы и к хромосомным интервалам QTL, которые демонстрируют статистически значимую совместную сегрегацию (и, следовательно, являются прогностическими и определяющими) с резистентностью к черной ножке. Детекция этих маркеров, или дополнительных локусов, сцепленных с этими маркерами, которые, следовательно, эквивалентны им, может использоваться в программе селекции канолы с помощью маркеров с получением растений, устойчивых к черной ножке, и генетического материала.
[0034] Некоторые варианты осуществления относятся к способам идентификации первого растения канолы или генетического материала (например, линия или сорт), которые проявляют устойчивость к черной ножке. В некоторых примерах по крайней мере один аллель одного или нескольких маркерных локусов (например, множество маркерных локусов), которые сцеплены (например, тесно сцеплены) с признаком резистентности к черной ножке, детектируют в первом растении канолы или генетическом материале. Маркер локуса может быть выбран из локусов, указанных на ФИГ.2, включая: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590 и другие маркеры, которые сцеплены по меньшей мере с одним из маркеров QTL, указанных выше. Более конкретно, маркерные локусы могут быть выбран из локусов, перечисленных на ФИГ.2, включая: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705, а также другие маркеры, которые сцеплены по меньшей мере с одним из маркеров QTL, указанных выше.
[0035] В некоторых примерах множество маркерных локусов могут быть выбраны или идентифицированы в одном и том же растении или генетическом материале. Все комбинации, например, DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590, и других маркеров, которые сцеплены по меньшей мере с одним из маркеров QTL, как указано выше, могут быть включены во множество маркерных локусов для отбора или идентификации в растении или генетическом материале.
[0036] В аспектах некоторых вариантов осуществления резистентность к черной ножке растения канолы может быть количественно оценена, используя любое подходящее средство или способ, известный в данной области.
II. Сокращения
[0037] AFLP полиморфизм длин амплифицированных фрагментов
[0038] ASH аллель специфическая гибридизация
[0039] CCD устройство для подсоединения заряда
[0040] EST маркер экспрессируемой последовательности
[0041] FAME метиловый эфир жирной кислоты
[0042] FID пламенно-ионизационный детектор
[0043] GC газовая хроматография
[0044] LCR лигазная цепная реакция
[0045] LG группа сцепления
[0046] LNA закрытая нуклеиновая кислота
[0047] LOD логарифм (по основанию 10) нечетных чисел
[0048] MAS отбор с помощью маркеров
[0049] NASBA амплификация на основе последовательностей нуклеиновых кислот
[0050] NIR ближняя часть инфракрасного диапазона (спектроскопия)
[0051] ЯМР ядерно-магнитный резонанс (спектроскопия)
[0052] ORF открытая рамка считывания
[0053] ПЦР полимеразная цепная реакция
[0054] PNA нуклеиновая кислота пептида
[0055] QTL локус количественных признаков
[0056] RAPD случайным образом амплифицированная полиморфная ДНК
[0057] RFLP полиморфизм длин рестрикционных фрагментов
[0058] ОТ-ПЦР ПЦР с обратной транскриптазой
[0059] SNP однонуклеотидный полиморфизм
[0060] SSCP однонитевой конформационный полиморфизм
[0061] SSR простая последовательность повтора
Термины
[0062] Как используется в настоящей заявке, включая формулу изобретения, термины в единственном числе и в единственных формах включают множественное число, если из контекста с очевидностью не следует обратное. Таким образом, например, ссылка на "растение" также относится к "растениям". Кроме того, в зависимости от контекста, использование термина "растение" также может относиться к генетически-идентичному или идентичному потомству этого растения. Аналогично, термин "нуклеиновая кислота" может относиться к множеству копий молекулы нуклеиновой кислоты. Аналогично, термин "зонд" может относиться ко множеству сходных или идентичных молекул зонда.
[0063] Числовые диапазоны включают числа, определяющие диапазон, и включают каждое целое и нецелое дробное число в пределах этого определенного диапазона. Если не указано иного, то все технические и научные термины, используемые в настоящем документе, имеют значения, которые обычно понятно специалисту в данной области.
[0064] Для упрощения рассмотрения различных вариантов осуществления, описанных в настоящем документе, приводится следующее разъяснение специфических терминов:
[0065] Выделенный: "Выделенный" биологический компонент (такой как нуклеиновая кислота или белок) по существу отделен, получен независимо или очищен от других биологических компонентов клетки организма, в которой в естественных условия находится этот компонент (т.е., от других хромосомных и экстра-хромосомных ДНК и РНК, а также белков), при этом осуществляя химическое или функциональное изменение компонента (например, нуклеиновая кислота может быть выделена из хромосомы путем разрыва химических связей, связывающих нуклеиновую кислоту с остальной частью ДНК в хромосоме). Молекулы нуклеиновых кислот и белков, которые были "выделены", включают молекулы нуклеиновых кислот и белков, очищенные стандартными способами очистки. Термин также охватывает нуклеиновые кислоты и белки, полученные рекомбинантной экспрессией в клетке-хозяине, а также химически синтезированные молекулы нуклеиновых кислот, белков и пептидов.
[0066] Картирование популяция: Используемый в настоящем документе термин "картирование популяции" может относиться к популяции растений (например, к популяции растений канолы), используемых для картирования генов. Картирование популяций обычно получают при контролируемых скрещиваниях родительских генотипов, которые могут быть представлены двумя инбредными линиями. Решение об отборе родительских линий, программе скрещивания для проведения картирования популяции, а также о типе используемых маркеров, зависит от картируемого гена, наличия маркеров и молекулярной карты. Родительские линии растений в пределах картируемой популяции должны иметь достаточную вариацию интересуемого(ых) признака(ов) как на уровне последовательности нуклеиновой кислоты, так и на уровне фенотипа. Вариации в родительской последовательности нуклеиновой кислоты используют для отслеживания событий рекомбинации в растениях картируемой популяции. Наличие информативных полиморфных маркеров зависит от количества вариаций последовательности нуклеиновой кислоты. Таким образом, информативные маркеры могут быть не идентифицированы при конкретных скрещиваниях родительских генотипов, хотя такие маркеры могут существовать.
[0067] "Генетическая карта" представляет собой описание генетической связи между локусами на одной или нескольких хромосомах (или группах сцепления) в пределах данного вида, как может быть определено анализом картирования популяции. В некоторых примерах генетическая карта может быть изображена схематично или в форме таблицы. Термин "генетическое картирование" может относиться к процессу определения связей сцепления локусов посредством использования генетических маркеров, картирования популяций, сегрегирующих в отношении маркеров, и стандартных генетических принципов частоты рекомбинации. "Генетическая карта о расположении" относится к расположению на генетической карте (по отношению к окружающим генетическим маркерам на той же самой группе сцепления или хромосоме), где специфический маркер может быть обнаружен в пределах данного вида. Напротив, "физическая карта генома" относится к абсолютным расстояниям (например, измеренных в парах оснований или выделенных и перекрывающихся смежных генетических фрагментах) между маркерами в пределах данного вида. Физическая карта генома не обязательно отражает реальную частоту рекомбинации, наблюдаемую при тестируемом скрещивании вида, между различными точками на физической карте.
[0068] Скрещивание: Как используется в настоящем описании термин "скрещивание" или "скрещенный" относится к слиянию гамет путем опыления с получением потомства (например, клеток, семян и растений). Этот термин включает как половое скрещивание (т.е., опыление одного растения другим), так и само-воспроизводство (т.е., самоопыление, например, с использованием пыльцы и яйцеклетки одного и того же растения).
[0069] Возвратное скрещивание: Способы возвратного скрещивания могут быть использованы для введения последовательности нуклеиновой кислоты в растения. Методика возвратного скрещивания широко используется на протяжении десятилетий для введения новых признаков в растения. Jensen, N., Ed. Plant Breeding Methodology, John Wiley & Sons, Inc., 1988. При обычном протоколе возвратного скрещивания представляющий интерес первоначальный сорт (рекуррентный родитель) скрещивают со вторым сортом (не рекуррентный родитель), который несет представляющий интерес ген, подлежащий переносу. Полученное в результате этого скрещивания потомство затем снова скрещивают с рекуррентным родителем, и процесс повторяют до тех пор, пока не будет получено растение, в котором по существу все желаемые морфологические и физиологические характеристики рекуррентного растения воссозданы в трансформированном растении в дополнение к перенесенному из нерекуррентного родителя гену.
[0070] Интрогрессия: Как используется в настоящем документе термин "интрогрессия" относится к переносу аллеля на генетическом локусе в фоновый генотип. В некоторых вариантах осуществления интрогрессия специфической аллельной формы в локусе может происходить путем передачи аллельной формы по меньшей мере одному потомству посредством полового скрещивания двух родителей одного и того же вида, где по крайней мере один из родителей имеет специфическую аллельную форму в своем геноме. Потомство, содержащее специфическую аллельную форму, может быть повторно возвратно скрещено с линией, имеющей желаемый фоновый генотип. Потомство от возвратного скрещивания может быть отобрано по специфической аллельной форме так, чтобы получить новый сорт, в котором специфическая аллельная форма была бы закреплена в фоновом генотипе. В некоторых вариантах осуществления интрогрессия специфической аллельной формы может происходить в результате рекомбинации двух донорных геномов (например, в слитом протопласте), где по крайней мере один из донорных геномов имеет специфическую аллельную форму в своем геноме. Интрогрессия может включать перенос специфической аллельной формы, которая может быть, например, но ими не ограничиваясь, выбранной аллельной формой маркерного аллеля; QTL; и/или трансгеном.
[0071] Генетический материал: Как используется в настоящем документе термин "генетический материал" относится к генетическому материалу определенного растения, группы растений (например, линии, сорта и семейства растения) или полученному из них, к клону, полученному из растения или из группы растений. Генетический материал может быть частью организма или клетки, или же может быть отделена (например, выделена) из организма или клетки. В целом, генетический материал относится к генетическому материалу со специфическим молекулярным набором, который является основой для наследственных качеств растения. Как используется в настоящем документе термин "генетический материал" относится к клеткам определенного растения; семени; ткани конкретного растения (например, ткани, из которой могут быть выращены новые растения); и частям определенного растения, не являющимся семенами (например, лист, стебель, пыльца и клетки).
[0072] Как используется в настоящем документе термин "идиоплазма" может быть использован для обозначения семени (или другого растительного материала), из которого можно размножить растение. "Банк генетического материала" может относиться к организованной коллекции различных семян или другого генетического материала (в котором каждый генотип однозначно идентифицирован), из которого может быть культивирован известный культурный сорт, и из которого может быть создан новый сорт. В вариантах осуществления генетический материал, используемый в способе или в растении, как описано в настоящем документе, является генетическим материалом из линии или сорта канолы. В конкретных примерах генетический материал представляет собой семя линии или сорта канолы. В конкретных примерах генетический материал представляет собой образец нуклеиновой кислоты линии или сорта канолы.
[0073] Ген: Как используется в настоящем документе термин "ген" (или "генетический элемент") может относиться к наследуемой геномной последовательности ДНК с функциональной значимостью. Термин "ген" также может быть использован для обозначения, например, но ими не ограничиваясь, кДНК и/или мРНК, кодируемых наследуемой геномной последовательностью ДНК.
[0074] Генотип: Как используется в настоящем описании термин "генотип" относится к генетической конституции особи (или группы особей) в одном или нескольких конкретных локусах. Генотип особи или группы особей определен и описан аллельными формами в одном или нескольких локусах, которые особь унаследовала от своих родителей. Термин генотип также может быть использован для обозначения генетической конституции особи в одном локусе, множественных локусах или во всех локусах ее генома. "Гаплотип" представляет собой генотип особи во множестве генетических локусов. В некоторых примерах генетические локусы, описываемые гаплотипом, могут быть физически и генетически сцеплены; т.е., локусы могут быть расположены на том же сегменте хромосомы.
[0075] Локус количественных признаков: Специфические хромосомные локусы (или интервалы) могут быть картированы в геноме организма, который коррелирует с конкретными количественными фенотипами. Такие локусы называют локусами количественных признаков, или QTL. Как используется в настоящем документе термин "локус количественных признаков" (QTL) может относиться к участкам ДНК, которые были идентифицированы, вероятно, как последовательности ДНК (например, гены, некодирующие последовательности и/или межгенные последовательности), которые лежат в основе количественного признака или фенотипа, который изменяется по степени, и могут быть отнесены к взаимодействию двух или более последовательностей ДНК (например, генов, некодирующих последовательностей и/или межгенных последовательностей) или продуктов их экспрессии и их окружения. Таким образом, термин "локус количественных признаков" включает полиморфные генетические локусы по меньшей мере с двумя аллелями, которые избирательно влияют на экспрессию фенотипического признака по меньшей мере в одном фоновом генотипе (например, по крайней мере в одной селекционной популяции или потомстве). На практике, QTL могут быть идентифицированы на молекулярном уровне для облегчения картирования областей генома, которые содержат последовательности, вовленченные в определение количественного признака, такого как резистентность к черной ножке.
[0076] Как используется в настоящем документе термин "интервал QTL" может относиться к участкам ДНК, которые сцеплены с генами, лежащими в основе признака QTL. Интервал QTL, как правило, но не обязательно, больше самого QTL. Интервал QTL может содержать участки ДНК, которые расположены в направлении 5' и/или 3' по отношению к QTL.
[0077] Было разработано множество экспериментальных моделей для идентификации и анализа QTL. См., например, Jansen (1996) Trends Plant Sci 1:89. Большая часть опубликованных работ по картированию QTL в сортах сельскохозяйственных культур основаны на использовании двухродительского скрещивания (Lynch and Walsh (1997) Genetics and Analysis of Quantitative Traits, Sinauer Associates, Sunderland). Как правило, эти модели включают скрещивание одной или нескольких родительских пар, которые могут быть, например, одной парой, полученной из двух инбредных линий, или несколькими связанными или не связанными между собой родительскими растениями разных инбредных линий, каждая из которых демонстрирует различные характеристики по сравнению с представляющим интерес фенотипическим признаком. Как правило, этот экспериментальный протокол включает получение от 100 до 300 сегрегирующих потомств от одного скрещивания двух расходящихся инбредных линий, которые, например, выбраны для максимального увеличения фенотипического различия и различия молекулярных маркеров между линиями. Родители и сегрегирующее потомство генотипируют на множественные маркерные локусы и оценивают от одного до нескольких количественных признаков (например, устойчивость к болезням). QTL затем идентифицируют как значимые статистические связи между генотипическими значениями и фенотипической изменчивостью в пределах сегрегирующего потомства. Значимость этого экспериментального протокола основана на применении инбредного скрещивания, так как получающиеся родители F1 все имеют одинаковую фазу сцепления (как если аллели объединены в родительском поколении). Таким образом, после самоопыления растений F1, все сегрегирующее потомство F2 является информативным, а неравновесное сцепление - максимально, фаза сцепления известна, присутствуют только две аллели QTL и (за исключением потомства возвратного скрещивания) частота каждого аллеля QTL равна 0,5.
[0078] Многочисленные статистические способы определения являются ли маркеры генетически сцеплены с QTL (или с другим маркером) известны специалистам в данной области и включают, например, но ими не ограничиваясь, модели стандартных линий, такие как ANOVA или регрессионное картирование (Haley and Knott (1992) Heredity 69:315); и способы максимальной вероятности, такие как алгоритмы максимального правдоподобия (например, Lander and Botstein (1989) Genetics 121:185-99; Jansen (1992) Theor. Appl. Genet. 85:252-60; Jansen (1993) Biometrics 49:227-31; Jansen (1994) "Mapping of quantitative trait loci by using genetic markers: an overview of biometrical models," In J. W. van Ooijen and J. Jansen (eds.), Biometrics in Plant breeding: applications of molecular markers, pp. 116-24, CPRO-DLO Metherlands; Jansen (1996) Genetics 142:305-11; и Jansen and Stam (1994) Genetics 136:1447-55).
[0079] Примеры статистических способов включают одноточечный маркерный анализа; интервальное картирование (Lander and Botstein (1989) Genetics 121:185); композиционное интервальное картирование; регрессионый анализ со штрафом; комплексный анализ по родословной; анализ MCMC; анализ MQM (Jansen (1994) Genetics 138:871); анализ HAPLO-IM+, анализ HAPLO-MQM и анализ HAPLO-MQM+; байесовский MCMC; гребень-регрессию; анализ идентичности происхождения; и регрессию Хаземана-Эльстона, каждый из которых может быть использован в контексте конкретных вариантов осуществления изобретения. Альтернативные статистические способы, применимые для комплексной селекции популяций, которые могут использоваться для идентификации и локализации QTL в конкретных примерах, описаны в патенте США 6399855 и международной патентной публикации РСТ WO0149104 A2. Все эти подходы требуют большого объема вычислений и, как правило, осуществляются с помощью компьютерной системы и специализированного программного обеспечения. Соответствующие статистические пакеты доступны из различных общедоступных и коммерческих источников, и они известны специалистам в данной области.
[0080] Маркер: Хотя специфические последовательности ДНК, которые кодируют белки, как правило, являются консервативными для вида, другие области ДНК (например, некодирующие ДНК и интроны) имеют тенденцию к развитию и накоплению полиморфизма, и, следовательно, могут изменяться между особями одного и того же вида. Геномная изменчивость может быть любого происхождения, например, изменчивость может быть обусловлена вставками, делециями, дупликациями ДНК, повторяющимися элементами ДНК, точечными мутациями, событиями рекомбинации, а также наличием и последовательностью транспозонов. Такие области могут содержать молекулярно-генетические маркеры, которые могут быть использованы. В целом, любой дифференцированно наследуемый полиморфный признак (в том числе полиморфизмы нуклеиновых кислот), который сегрегируется среди потомства, является возможным маркером.
[0081] Как используется в настоящем описании термины "маркер" и "молекулярный маркер" относятся к нуклеотидной последовательности или кодируемому им продукту (например, белку), используемым в качестве точки отсчета при идентификации сцепленного локуса. Таким образом, маркер может относиться к последовательности гена или к нуклеотидной последовательности, которые могут быть использованы для идентификации растений, имеющих конкретную аллель. Маркер может быть описан как вариант в данном геномном локусе. Генетический маркер может быть короткой последовательностью ДНК, такой как последовательность, окружающая изменение в одноосновной паре (однонуклеотидный полиморфизм или "SNP"), или длинной, например, микросателлитом/простой повторяющейся последовательностью ("SSR"). "Маркерный аллель" или "форма маркерного аллеля" относится к варианту маркера, который присутствует у конкретной особи. Термин "маркер", как используется в настоящем документе, может относиться к клонированному сегменту хромосомной ДНК, а также может относиться или, альтернативно, относится к молекуле ДНК, которая комплементарна клонированному сегменту хромосомной ДНК. Этот термин также относится к последовательностям нуклеиновых кислот, комплементарным последовательностям геномных маркеров, таким как нуклеотидные праймеры и зонды.
[0082] Маркер может быть описан, например, как специфический полиморфный генетический элемент в специфическом месте на генетической карте организма. Генетическая карта может быть графическим представлением генома (или части генома, например, единичной хромосомы), где расстояние между "ориентирами" на хромосоме измеряют по частоте рекомбинации между этими "ориентирами". Генетический "ориентир" быть любым из множества известных полиморфных маркеров, например, но ими не ограничиваясь: маркеры простой повторяющейся последовательности (SSR); маркеры полиморфизма длин рестрикционных фрагментов (RFLP); и маркеры однонуклеотидного полиморфизма (SNP). В качестве примера, маркеры SSR могут быть получены из геномных или экспрессированных нуклеиновых кислот (например, маркеры экспрессируемой последовательности (EST)).
[0083] Дополнительные маркеры включают, например, но ими не ограничиваются, EST; полиморфизмы длин амплифицированных фрагментов (AFLP) (Vos et al. (1995) Nucl. Acids Res. 23:4407; Becker et al. (1995) Mol. Gen. Genet. 249:65; Meksem et al. (1995) Mol. Gen. Genet. 249:74); случайным образом амплифицированную полиморфную ДНК (RAPD) и изоферментные маркеры. Маркеры изоферментов могут быть использованы в качестве генетических маркеров, например, для отслеживания маркеров изоферментов или других типов маркеров, которые сцеплены с конкретным первым маркером. Изоферменты представляют собой множественные формы ферментов, которые отличаются друг от друга аминокислотной последовательностью (и, следовательно, кодирующей их последовательностью нуклеиновых кислот). Некоторые ферменты представляют собой множественные ферменты, содержащие несколько различающиеся субъединицы. Другие изоферменты являются либо мультимерными, либо мономерными, но отщепляются от про-фермента в различных сайтах аминокислотной последовательности про-фермента. Изоферменты могут быть охарактеризованы и проанализированы либо на уровне белка, либо на уровне нуклеиновой кислоты. Таким образом, любой из способов, основанных на нуклеиновых кислотах, описанный в настоящем документе, может быть использован для анализа маркеров изоферментов в конкретных примерах.
[0084] "Генетические маркеры" включают аллели, которые являются полиморфными в популяции, где аллели могут быть детектированы и дифференцированы с помощью одного или нескольких аналитических способов (например, с помощью анализа RFLP, анализа AFLP, анализа маркеров изоферментов, анализа SNP и анализа SSR). Термин "генетический маркер" может также относиться к генетическому локусу (к "маркерному локусу"), который может быть использован в качестве точки отсчета при идентификации генетически сцепленного локуса (например, QTL). Такой маркер может также называться "маркером QTL".
[0085] Природа вышеуказанных физических "ориентиров" (и способы, используемые для их детекции) различна, но все эти маркеры физически различаются друг от друга (а также от множества аллелей любого конкретного маркера) по длине и/или последовательности полинуклеотидов. Многочисленные способы детекции молекулярных маркеров и идентификации маркерных аллелей хорошо известны. Для обнаружения такой изменчивости специалисту в данной области известен широкий диапазон протоколов, и эти протоколы зачастую характерны для типа полиморфизма, для обнаружения которого они разработаны. Такие протоколы включают, например, но ими не ограничиваются, ПЦР-амплификацию; детекцию одноцепочечного конформационного полиморфизма (SSCP), например, посредством электрофореза; и самоподдерживающуюся репликацию последовательностей (3SR) (см. Chan and Fox (1999) Reviews in Medical Microbiology 10:185-96).
[0086] Основная мотивация для разработки методик с использованием молекулярных маркеров для селекционеров растений заключалась в повышении эффективности селекции посредством MAS. Молекулярный маркерный аллель, который демонстрирует неравновесное сцепление с желаемым фенотипическим признаком (например, QTL) обеспечивает эффективный инструмент для отбора желаемого признака в популяции растений. Ключевым компонентом для реализации подхода MAS является создание полной (информационно богатой) генетической карты молекулярных маркеров в генетическом материале растения; детекция по меньшей мере одного QTL на основе статистических связей между маркером и фенотипической изменчивостью; определение набора конкретных эффективных для использования маркерных аллелей на основе результатов анализа QTL; а также использование и/или экстраполяция этой информации на существующий набор селекционного генетического материала для принятия решений об отборе на основе маркеров.
[0087] Генетическую изменчивость, например, как определено по картированию популяции, можно наблюдать среди различных популяций одного вида (например, канолы). Несмотря на изменчивость в генетической карте, которая может возникать среди популяций одного вида, генетическая карта и информация о маркерах, полученная из одной популяции, в целом остается эффективной для использования среди множества популяций для целей идентификации и/или отбора растений и/или генетического материала, включающих признаки, которые сцеплены с маркерами, и для негативного отбора растений и/или генетический материал, включающих нежелательные признаки.
[0088] В настоящем документе описано два типа маркеров, используемых в конкретных протоколах MAS, - это маркеры SSR и SNP. Маркеры SSR включают любой тип молекулярной гетерогенности, которая приводит к вариабельности длины последовательности нуклеиновой кислоты. Примерами маркеров SSR являются короткие (до нескольких сотен пар оснований) сегменты ДНК, которые состоят из нескольких тандемных коротких повторов (т.е., от двух до десяти) пар оснований в последовательности. В повторяющихся последовательностях, следовательно, может быть две, три, четыре, пять, шесть, семь, восемь, девять или десять пар оснований. Эти повторяющиеся последовательности дают высокополиморфные участки ДНК различной длины в виду плохой точности репликации (например, в результате проскальзывания полимеразы). По всей видимости, SSR случайным образом рассеяны по геному, и, как правило, фланкированы консервативными областями. Маркеры SSR также могут быть получены из генных последовательностей (в форме кДНК, частичной кДНК или EST), а также из негенетического материала.
[0089] Неоднородность маркеров SSR делает их подходящими для использования в качестве молекулярных генетических маркеров. Например, геномная изменчивость SSR является наследуемой, и мульти-аллельной, кодоминантной и воспроизводимо обнаруживаемой. Увеличение числа более совершенных способов детекции на основе амплификации (например, методик на основе ПЦР) представляет различные чувствительные способы детекции неоднородности нуклеотидной последовательности между образцами. Зонды (например, праймеры нуклеиновых кислот) могут быть сконструированы для гибридизации с консервированными областями, которые фланкируют SSR, и зонды могут быть использованы для амплификации вариабельной области SSR. Ампликоны разных размеров, полученные из области SS, имеют характерные и воспроизводимые размеры. Ампликоны SSR разного размера, наблюдаемые из двух гомологичных хромосом одной особи или разных особей в популяции растений, определяют маркерные аллели SSR. До тех пор, пока существует по крайней мере два маркерных аллеля SSR, которые дают продукты ПЦР различных размеров, SSR может быть использован в качестве маркера.
[0090] (Не)равновесное сцепление: Как используется в настоящем описании термин “равновесное сцепление” относится к ситуации, когда два маркера сегрегируют независимо; то есть, маркеры распределяются среди потомства случайным образом. Маркеры, которые демонстрируют равновесное сцепление считаются несцепленными (независимо от того находятся ли одни на одной хромосоме или нет). Как используется в настоящем документе термин “неравновесное сцепление” относится к ситуации, когда два маркера сегрегируют неслучайным образом; то есть, маркеры имеют частоту рекомбинации, равную менее 50% (и, таким образом, в соответствии с определением, они отделены друг от друга менее, чем на 50 сМ в одной группе сцепления). В некоторых примерах маркеры, которые демонстрируют неравновесное сцепление, считаются сцепленными.
[0091] Сцепленные, тесно сцепленные и чрезвычайно тесно сцепленные: как используется в настоящем документе связь между генами или маркерами может относиться к явлению, при котором гены или маркеры на хромосоме обнаруживают значительную вероятность быть переданными вместе особи в следующем поколении. Таким образом, связывание одного маркера с другим маркером или геном может быть измерено и/или выражено как частота рекомбинации. Чем ближе два гена или маркера друг к другу, тем ближе к "1" становится эта вероятность. Таким образом, термин "сцепленный" может относиться к одному или более генам или маркерам, которые передаются вместе с геном с вероятностью более 0,5 (что, как ожидается, при независимой сортировке происходит когда маркеры/гены расположены на разных хромосомах). Если присутствие гена способствует проявлению фенотипа у особи, то можно говорить о том, что маркеры, которые сцеплены с геном, сцеплены с фенотипом. Таким образом, термин "сцепленный" может относиться к взаимосвязи между маркером и геном, или между маркером и фенотипом.
[0092] Относительное генетическое расстояние (определяется как частота кроссинговера и измеряется в сантиморганах (сМ)), как правило, пропорционально физическому расстоянию (измеряемому в парах оснований), которое разделяет два сцепленных маркера или гена друг от друга на хромосоме. Один сантиморган определяется как расстояние между двумя генетическими маркерами, которые демонстрируют частоту рекомбинации, равную 1% (т.е., событие кроссинговера происходит между двумя маркерами один раз на каждые 100 клеточных делений). В целом, чем ближе один маркер к другому маркеру или гену (независимо от того измеряется ли расстояние между ними в терминах генетического расстояния или физического расстояния), тем более тесно они сцеплены. Поскольку хромосомное расстояние приблизительно пропорционально частоте событий рекомбинации между признаками, то существует приблизительное физическое расстояние, которое коррелирует с частотой рекомбинации. Например, в каноле 1 сМ коррелирует в среднем с приблизительно 400 кб.
[0093] Таким образом, термин "сцепленный" может относится в настоящем документе к одному или более генам или маркерам, которые физически расположены в пределах приблизительно 4,0 Мб друг от друга на одной и той же хромосоме канолы (т.е., приблизительно 10 сМ). Таким образом, два "сцепленных" гена или маркера могут быть разделены расстоянием в 4,1 Мб; приблизительно 4,0 Мб; приблизительно 3,0 Мб; приблизительно 2,5 Мб; 2,1 Мб; 2,00 Мб; приблизительно 1,95 Мб; приблизительно 1,90 Мб; приблизительно 1,85 Мб; приблизительно 1,80 Мб; приблизительно 1,75 Мб; приблизительно 1,70 Мб; приблизительно 1,65 Мб; приблизительно 1,60 Мб; приблизительно 1,55 Мб; приблизительно 1,50 Мб; приблизительно 1,45 Мб; приблизительно 1,40 Мб; приблизительно 1,35 Мб; приблизительно 1,30 Мб; приблизительно 1,25 Мб; приблизительно 1,20 Мб; приблизительно 1,15 Мб; приблизительно 1,10 Мб; приблизительно 1,05 Мб; приблизительно 1,00 Мб; приблизительно 0,95 Мб; приблизительно 0,90 Мб; приблизительно 0,85 Мб; приблизительно 0,80 Мб; приблизительно 0,75 Мб; приблизительно 0,70 Мб; приблизительно 0,65 Мб; приблизительно 0,60 Мб; приблизительно 0,55 Мб; приблизительно 0,50 Мб; приблизительно 0,45 Мб; приблизительно 0,40 Мб; приблизительно 0,35 Мб; приблизительно 0,30 Мб; приблизительно 0,25 Мб; приблизительно 0,20 Мб; приблизительно 0,15 Мб; приблизительно 0,10 Мб; приблизительно 0,05 Мб; приблизительно 0,025 Мб; и приблизительно 0,01 Мб.
[0094] Как используется в настоящем документе термин "тесно сцепленный" может относиться к одному или более генам или маркерам, которые расположены в пределах приблизительно 2,0 Мб друг от друга на одной хромосоме. Таким образом, два "тесно сцепленных" гена или маркера могут быть разделены расстоянием в 2,1 Мб; приблизительно 1,75 Мб; приблизительно 1,5 Мб; приблизительно 1,0 Мб; приблизительно 0,9 Мб; приблизительно 0,8 Мб; приблизительно 0,7 Мб; приблизительно 0,6 Мб; приблизительно 0,55 Мб; 0,5 Мб; приблизительно 0,45 Мб; приблизительно 0,4 Мб; приблизительно 0,35 Мб; приблизительно 0,3 Мб; приблизительно 0,25 Мб; приблизительно 0,2 Мб; приблизительно 0,15 Мб; приблизительно 0,1 Мб; и приблизительно 0,05 Мб.
[0095] Как используется в настоящем документе термин “чрезвычайно тесно сцепленные” может относится к одному или более генам или маркерам, которые расположены в пределах приблизительно 500 кб друг от друга на одной хромосоме. Таким образом, два "чрезвычайно тесно сцепленных" гена или маркера могут быть разделены расстоянием в 600 кб; приблизительно 450 кб; приблизительно 400 кб; приблизительно 350 кб; приблизительно 300 кб; приблизительно 250 кб; приблизительно 200 кб; приблизительно 175 кб; приблизительно 150 кб; приблизительно 125 кб; приблизительно 120 кб; приблизительно 115 кб; приблизительно 110 кб; приблизительно 105 кб; 100 кб; приблизительно 95 кб; приблизительно 90 кб; приблизительно 85 кб; приблизительно 80 кб; приблизительно 75 кб; приблизительно 70 кб; приблизительно 65 кб; приблизительно 60 кб; приблизительно 55 кб; приблизительно 50 кб; приблизительно 45 кб; приблизительно 40 кб; приблизительно 35 кб; приблизительно 30 кб; приблизительно 25 кб; приблизительно 20 кб; приблизительно 15 кб; приблизительно 10 кб; приблизительно 5 кб; и приблизительно 1 кб.
[0096] Чем ближе конкретный маркер к гену, который кодирует полипептид, который оказывает влияние на конкретный фенотип (независимо от того измеряется ли оно в терминах генетического или физического расстояния), тем более тесно сцепленный конкретный маркер с фенотипом. С учетом вышеизложенного, следует принимать во внимание, что маркеры, сцепленные с конкретным геном или фенотипом, включают такие маркеры, которые тесно сцеплены, и такие маркеры, которые чрезвычайно тесно сцеплены с геном или фенотипом. В некоторых вариантах осуществления, чем ближе конкретный маркер к гену, который кодирует полипептид, который оказывает влияние на фенотип резистентности к черной ножке (независимо от того измеряется ли оно в терминах генетического или физического расстояния), тем более тесно сцеплены этот конкретный маркер и фенотип резистентности к черной ножке. Таким образом, сцепленные, тесно сцепленные и чрезвычайно тесно сцепленные генетические маркеры и фенотип резистентности к черной ножке в каноле могут быть эффективно использованы в программах MAS для идентификации сортов канолы, обладающих улучшенной резистентностью к черной ножке (по сравнению с родительскими сортами и/или по меньшей мере с одним конкретным обычным сортом), для идентификации отдельных растений канолы, обладающих резистентностью к черной ножке (или улучшенной резистентностью к черной ножке), и для селекции этого признака в других сортах канолы для получения устойчивости к черной ножке или для ее улучшения.
[0097] В некоторых вариантах осуществления связь между молекулярным маркером и фенотипом может быть выражена как "вероятность" или "скорректированная вероятность." В этом контексте, значение вероятности является статистической вероятностью того, что конкретная комбинация фенотипа и наличия или отсутствия определенной формы маркерного аллеля является случайной. Таким образом, чем меньше значение вероятности, тем больше вероятность того, что фенотип и конкретная форма маркерного аллеля будут сегрегировать совместно. В некоторых примерах оценка вероятности может быть описана как "значимая" или "незначимая." В конкретных примерах значение вероятности случайной сортировки, равное 0,05 (р=0,05 (5% вероятность)), считается "значимым" указанием на совместную сегрегацию. Тем не менее, в других примерах значимая вероятность может быть любой вероятностью, равной меньше 50% (р=0,5). Например, значимая вероятность может быть меньше 0,25; меньше 0,20; меньше 0,15; или меньше 0,1.
[0098] В некоторых вариантах осуществления маркер, который сцеплен с резистентностью к черной ножке, может быть выбран из маркеров QTL группы сцепления N10 канолы, как проиллюстрировано на ФИГ.2. В некоторых вариантах осуществления, маркер, который сцеплен с резистентностью к черной ножке, может быть выбран из маркеров, которые расположены в пределах приблизительно 10 сМ от маркера QTL, как проиллюстрировано на ФИГ.2. Таким образом, маркер, который сцеплен н с фенотипом резистентности к черной ножке, может быть, например, в пределах 10 сМ; 9 сМ; 8 сМ; 7 сМ; 6 сМ; 5 сМ; 4 сМ; 3 сМ; 2 сМ; 1 сМ; 0,75 сМ; 0,5 сМ; 0,25 сМ; или менее, от маркера QTL, как проиллюстрировано на ФИГ.2.
[0099] Растениевод-селекционер может успешно использовать молекулярные маркеры для идентификации желаемых особей путем идентификации маркерных аллелей, которые демонстрируют статистически значимую вероятность совместной сегрегации с желаемым фенотипом (например, резистентностью к черной ножке), что проявляется как неравновесное сцепление. Идентифицируя молекулярный маркер или кластеры молекулярных маркеров, которые сегрегируют совместно с количественным признаком, селекционер, таким образом, идентифицирует QTL. Идентифицируя и отбирая маркерный аллель (или желаемые аллели множественных маркеров), который сцеплен с желаемым фенотипом, растениевод-селекционер способен быстро выбрать фенотип путем отбора подходящего молекулярного маркерного аллеля (т.е., MAS). Чем больше молекулярных маркеров помещено на генетическую карту, тем больше становится вероятная эффективность этой карты для проведения MAS.
[0100] Набор маркеров: Как используется в настоящем документе “набор” маркеров или зондов относится к специфической коллекции маркеров и зондов (или данных, полученных на их основе), которые могут быть использованы для идентификации особей, содержащих представляющий интерес признак. В некоторых вариантах осуществления набор маркеров, сцепленных с фенотипом резистентности к черной ножке, может быть использован для идентификации растений канолы, обладающих резистентностью к черной ножке. Данные, соответствующие набору маркеров или набору зондов (или данные, полученные при использовании таких маркеров или зондов), могут храниться на электронном носителе. В то время как каждый маркер в наборе маркеров может быть применим для идентификации признака, отдельные маркеры, выбранные из набора или поднаборов, включая некоторые, но не все, из маркеров, также могут быть эффективны для идентификации особей, содержащих представляющий интерес признак.
[0101] Аллель: Как используется в настоящем документе термин "аллель" относится к одной из двух или более различных нуклеотидных последовательностей, которые встречаются в специфическом локусе. Например, первый аллель может встречаться на одной хромосоме, в то время как второй аллель может встречаться на второй гомологичной хромосоме; например, как в случае различных хромосом гетерозиготной особи, или между различными гомозиготными или гетерозиготными особями в популяции. В некоторых вариантах осуществления конкретный аллель в конкретном локусе может быть сцеплен с агрономически желательным фенотипом (например, с резистентностью к черной ножке). В некоторых вариантах осуществления конкретный аллель в локусе может обеспечить идентификацию растений, которые не содержат агрономически желательного фенотипа (например, резистентности к черной ножке), в результате чего эти растения могли быть исключены из программы селекции или посадки. Маркерный аллель может сегрегировать вместе благоприятным фенотипом, тем самым давая преимущество для идентификации растений, имеющих этот фенотип.
[0102] “Аллельная форма хромосомного сегмента” может относится к хромосомному сегменту, который содержит нуклеотидную последовательность маркерного аллеля, который влияет на или сцеплен с конкретным фенотипом, на одном или нескольких генетических локусах, физически расположенных на хромосомном сегменте. "Частота аллелей" может относиться к частоте (выраженной как доля или процент), при которой аллель присутствует в локусе в пределах растения, в пределах линии или в пределах линий в популяции. Таким образом, для аллеля "А" диплоидная особь с генотипом "АА", "Аа" или "АА" имеет частоту аллелей 1,0, 0,5 или 0,0, соответственно. Частота аллелей в пределах линии может быть оценена путем усреднения частоты аллелей образца особей этой линии. Аналогично, частота аллелей в пределах линий в популяции может быть вычислена путем усреднения частот аллелей линий, которые составляют популяцию. Для популяции с конечным ограниченным числом особей или линий, частота аллелей может быть выражена как количество особей или линий (или любой другой определенной группы), содержащих эту аллель.
[0103] Маркерный аллель "положительно" коррелирует с признаком, если маркер сцеплен с этим признаком, и, если присутствие маркерного аллеля является показателем того, что желаемый признак или форма признака будет находится в растении, содержащем этот аллель. Маркерный аллель "отрицательно" коррелирует с признаком, если маркер сцеплен с этим признаком, и, если присутствие маркерного аллеля является показателем того, что желаемый признак или форма признака не будет находится в растении, содержащем этот аллель.
[0104] "Гомозиготная" особь имеет только одну аллельную форму в заданном локусе (например, диплоидное растение имеет копию такой же аллельной формы в конкретном локусе для каждой из двух гомологичных хромосом). Особь является “гетерозиготной”, если в локусе присутствует более одной аллельной формы (например, диплоидная особь имеет копию первой аллельной формулы и одну копию второй аллельной формы в этом локусе). Термин "гомогенность" относится к представителям группы, которые имеют один и тот же генотип (т.е., одну и ту же частоту аллелей) на одном или нескольких специфических представляющих интерес локусах. Напротив, термин “гетерогенность” относится к особям в пределах группы, которых отличаются генотипом на одном или более специфических представляющих интерес локусах.
[0105] Любая методика, которая может быть использована для описания нуклеотидной последовательности в локусе, может быть использована для идентификации маркерного аллеля. Способы обнаружения маркерного аллеля включают, например, но ими не ограничиваются, способы молекулярной идентификации (например, амплификацию и детекцию маркерного ампликона). Например, аллельная форма маркера SSR или маркера SNP может быть обнаружена с помощью методики на основе амплификации. В обычном способе детекции на основе амплификации маркерный локус или часть маркерного локуса амплифицируют (используя, например, ПЦР, LCR и транскрипцию с использованием нуклеиновой кислоты, выделенной из представляющего интерес растения канолы, в качестве матрицы амплификации), а полученный амплифицированный маркерный ампликон детектируют. В некоторых вариантах осуществления РНК растений может быть использована в качестве матрицы для реакции амплификации. В некоторых вариантах осуществления геномная ДНК растений может быть использована в качестве матрицы для реакции амплификации. В некоторых примерах маркер QTL является маркером SNP, и обнаруженный аллель является маркерным аллелем SNP, а метод детекции является аллель-специфической гибридизацией (ASH). В некоторых примерах маркер QTL представляет собой маркер SSR, а обнаруженный аллель является маркерным аллелем SSR.
[0106] Методика ASH основана на стабильном отжиге короткого одноцепочечного олигонуклеотидного зонда с полностью комплементарной одноцепочечной нуклеиновой кислотой-мишенью. Детекция может быть осуществлена путем детекции изотопной или неизотопной метки, присоединенной к зонду. Для каждого полиморфизма могут быть сконструированы два или более различных зонда ASH для того, чтобы иметь идентичные последовательности ДНК, исключая сайт полиморфизма. Каждый зонд может быть абсолютно гомологичным одной последовательности аллеля, так чтобы диапазон зондов может распознать все известные альтернативные последовательности аллеля. Если каждый зонд гибридизован с ДНК-мишенью при соответствующей конструкции зонда и условиях гибридизации, то одноосновное несоответствие между зондом и ДНК-мишенью будет препятствовать гибридизации. Таким образом, только один из альтернативных зондов гибридизуется с мишеневым образцом, который является гомозиготным по аллелю. Образцы, которые являются гетерозиготными или гетерогенными для двух аллелей, гибридизуются с обоими из двух альтернативных зондов.
[0107] Маркеры ASH могут использоваться в качестве доминирующих маркеров, где присутствие или отсутствие только одного аллеля определяется по гибридизации или отсутствию гибридизации только с одним зондом. По отсутствию гибридизации можно сделать вывод об альтернативном аллеле. В примерах, зонды ASH и молекулы-мишени могут быть молекулами РНК или ДНК; молекула-мишень может содержать нуклеотиды любой длины вне последовательности, которая комплементарна зонду; зонд может быть разработан для гибридизации с любой цепью ДНК-мишени; и размер зонда может изменяться, чтобы соответствовать требованиям различных условий гибридизации.
[0108] Амплифицированные вариабельные последовательности относятся к амплифицированным последовательностям генома растения, которые демонстрируют высокую степень изменчивости остатков нуклеиновой кислоты среди представителей одного и того же вида. Все организмы имеют вариабельные геномные последовательности, и каждый организм (за исключением клона) имеет различный набор вариабельных последовательностей. После идентификации наличие специфической вариабельной последовательности может быть использовано для прогнозирования фенотипических признаков. ДНК из растения может, в некоторых примерах, быть использована в качестве матрицы для амплификации с праймерами, которые фланкируют вариабельную последовательность ДНК. Вариабельная последовательность может быть амплифицирована и затем секвенирована.
[0109] Самоподдерживающаяся репликация последовательностей также может, и в качестве альтернативы, использоваться для идентификации генетических маркеров. Самоподдерживающаяся репликация последовательностей относится к способу амплификации нуклеиновых кислот с использованием последовательностей мишеневых нуклеиновых кислот, которые реплицируются экспоненциально in vitro по существу в изотермических условиях, используя три ферментативные активности, вовлеченные в репликацию ретровируса: активность обратной транскриптазы; РНКазы Н; и ДНК-зависимой РНК-полимеразы. Guatelli et al. (1990) Proc. Natl. Acad. Sci. USA 87:1874. Имитируя ретровирусную стратегию репликации РНК с помощью промежуточных продуктов кДНК, эта реакция накапливает копии кДНК и РНК исходной мишени.
[0110] Данные, представляющие обнаруженный маркерный(ые) аллель(и), могут быть переданы (например, в электронном виде; и посредством инфракрасной, беспроводной или оптической передачи) на компьютер или на машиночитаемый носитель для анализа или хранения.
[0111] Например, праймер амплификации или пара праймеров амплификации могут быть смешаны с геномной нуклеиновой кислотой, выделенной из первого растения канолы или с генетическим материалом, где праймер или пара праймеров комплементарны или частично комплементарны по меньшей мере части маркерного локуса, и праймер или пара праймеров способна инициировать полимеризацию ДНК с помощью ДНК-полимеразы, используя геномную нуклеиновую кислоту канолы в качестве матрицы. Праймер или пара праймеров (например, пара праймеров, приведенная в Таблица 3, удлиняется при реакции полимеризации ДНК с использованием ДНК-полимеразы и матрицы геномной нуклеиновой кислоты с получением по меньшей мере одного ампликона.
[0112] "Позиционное клонирование" относится к конкретной методике клонирования, при которой мишеневую нуклеиновую кислоту идентифицируют и выделяют по геномной близости к маркеру. Например, геномный клон нуклеиновой кислоты может включать все или часть еще двух хромосомных областей, которые проксимальны друг другу. Если маркер может быть использован для идентификации клона геномной нуклеиновой кислоты из геномной библиотеки, то стандартные методы, такие как субклонирование и/или секвенирование могут быть использованы для идентификации и/или выделения подпоследовательностей клона, которые расположены рядом с маркером.
[0113] Локус: Как используется в настоящем описании термин "локус" относится к положению на геноме, которое соответствует измеряемой характеристике (например, признаку) или полиморфизму. Локус SNP определяют с помощью зонда, который гибридизуется с ДНК, содержащейся в пределах локуса.
[0114] Селекция с помощью маркеров: Как используется в настоящем документе термин "селекция с помощью маркеров" может относиться к подходу для селекции, непосредственно используя MAS, по одному или нескольким признакам (например, по улучшенной резистентности к черной ножке). В современной практике растениеводы-селекционеры пытаются идентифицировать легко обнаруживаемые признаки, такие как цвет цветка, внешний вид семян или варианты изоферментов, которые сцеплены с агрономически желаемым признаком. Растениеводы-селекционеры затем следят за агрономическим признаком при селекции, популяционной селекции, отлеживая сегрегацию легко обнаруживаемого признака. Тем не менее, существует лишь несколько таких сцепленных связей, доступных для использования при селекции растений.
[0115] Селекция с помощью маркеров представляет экономически эффективный способ с минимальными затратами времени для улучшения сортов растений. Некоторые примеры использования селекции с помощью маркеров включает применение изоферментных маркеров. See, e.g., Tanksley and Orton, eds. (1983) Isozymes in Plant Breeding and Genetics, Amsterdam: Elsevier. Одним из примеров является изоферментный маркер, сцепленный с геном резистетности к нематодам, вредителям томатов. Резистентность, контролируемая геном, обозначенным как Mi, расположена на 6-ой хромосоме томатов и очень тесно сцепленным с Aps1, изоферментом кислой фосфатазы. Преимуществом применения изоферментного маркера Aps1 для опосредованной селекции гена Mi состоит в том, что сегрегация в популяции может быть однозначно определена стандартными электрофоретическими способами; изоферментый маркер может быть подсчитан в ткани саженца без необходимости в доведении растений до созревания; и совместное доминирование аллелей изоферментного маркера позволяет различать гомозиготы и гетерозиготы. См. Rick (1983) in Tanksley and Orton, выше.
[0116] Зонд: В некоторых вариантах осуществления присутствие маркера в растении может быть обнаружено посредством применения зонда нуклеиновой кислоты. Зонд может быть молекулой ДНК или молекулой РНК. РНК-зонды могут быть синтезированы способам, известными специалистам в данной области, например, используя матрицу молекулы ДНК. Зонд может содержать всю или часть нуклеотидной последовательности маркера и дополнительную прилегающую нуклеотидную последовательность генома растения. В данном случае такой зонд называется "непрерывный зонд." Дополнительная прилегающая нуклеотидная последовательность указывается как "вверху" или "внизу" от исходного маркера, в зависимости от того находится ли прилегающая нуклеотидная последовательность хромосомы растения в направлении 5 'или 3' от исходного маркера, в обычном понимании. Как понятно среднему специалисту в данной области, процесс получения дополнительной прилегающей нуклеотидной последовательности для включения в маркер может повторяться почти до бесконечности (ограничен лишь длиной хромосомы), тем самым идентифицируя дополнительные маркеры на хромосоме.
[0117] Последовательность олигонуклеотидного зонда может быть получена синтетически или путем клонирования. Подходящие векторы клонирования хорошо известны специалистам в данной области. Олигонуклеотидный зонд может быть меченными или не меченными. Существует большое разнообразие методик введения меток в молекулы нуклеиновых кислот, в том числе, например, но ими не ограничиваясь: введение радиоактивных меток ник-трансляцией; случайное праймирование; введение метки в концевую часть с помощью терминальной дезокситрансферазы; или тому подобное, где используемые нуклеотиды мечены, например, радиоактивным32P. Другие метки, которые могут быть использованы, включают, например, но ими не ограничиваются: флуорофоры (например, FAM и VIC); ферменты; субстраты ферментов; кофакторы ферментов; ингибиторы ферментов; и тому подобное. Альтернативно, использование метки, которая обеспечивает детектируемый сигнал, самостоятельно или в сочетании с другими реакционно-способными агентами, может быть заменено лигандами, с которыми связываются рецепторы, где рецепторы помечены (например, с помощью указанных выше меток) для получения детектриуемых сигналов, либо сами по себе, либо в сочетании с другими реагентами. См., например, Leary et al. (1983) Proc. Natl. Acad. Sci. USA 80: 4045-9.
[0118] Зонд может содержать нуклеотидную последовательность, которая не является прилегающей для исходного маркера; этот зонд называется в настоящем документе как "неприлегающий зонд." Последовательность неприлегающего зонда расположена достаточно близко к последовательности исходного маркера на геноме, так что неприлегающий зонд генетически сцеплен с тем же геном или признаком (например, с резистентностью к черной ножке). Например, в некоторых вариантах осуществления неприлегающий зонд расположен в пределах приблизительно 10 сМ; 9 сМ; 8 сМ; 7 сМ; 6 сМ; 5 cM; 4 cM; 3 cM; 2 cM; 1 cM; 0,75 cM; 0,5 cM; 0,25 cM; или менее, от маркера QTL, как проиллюстрировано на ФИГ.2.
[0119] Зонд может быть точной копией детекируемого маркера. Зонд также может быть молекулой нуклеиновой кислоты, содержащей или состоящей из нуклеотидной последовательности, которая по существу идентична клонированному сегменту хромосомной ДНК организма особи (например, канолы). Как используется в настоящем документе термин "по существу идентичные" может относиться к нуклеотидным последовательностям, которые идентичные более чем на 85%. Например, по существу идентичная нуклеотидная последовательности может быть на 85,5%; 86%; 87%; 88%; 89%; 90%; 91%; 92%; 93%; 94%; 95%; 96%; 97%; 98%; 99% или 99,5% идентична основной последовательности.
[0120] Зонд также может быть молекулой нуклеиновой кислоты, которая "специфически гибридизуется" или "специфически комплементарна" точной копии детектируемого маркера ("ДНК-мишень"). "Специфически гибридизуется" и "специфически комплементарна" являются терминами, которые указывают на достаточную степень комплементарности, при которой происходит стабильное и специфическое связывание между молекулой нуклеиновой кислоты и ДНК-мишенью. Молекула нуклеиновой кислоты не обязательно должна быть на 100% комплементарна своей мишеневой последовательности для специфической гибридизации. Молекула нуклеиновой кислоты специфически гибридизуется, если имеется достаточная степень комплементарности, чтобы исключить неспецифическое связывание нуклеиновой кислоты с немишеневыми последовательностями в условиях, когда желательно специфическое связывание, например, в жестких условиях гибридизации.
[0121] Условия гибридизации, дающие, в частности, условия жесткой степени могут изменяться в зависимости от природы выбранного способа гибридизации и композиции и длины гибридизующихся последовательностей нуклеиновых кислот. Как правило, температура гибридизации и ионная сила буфера гибридизации (особенно концентрация Na+ и/или Mg++) определяют жесткость гибридизации, хотя количество промывок также отказывает влияет на жесткость. Расчеты условий гибридизации, необходимых для достижения определенной степени жесткости, известны среднему специалисту в данной области, и описаны, например, в Sambrook et al. (ed.) Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1989, chapters 9 and 11; и Hames and Higgins (eds.) Nucleic Acid Hybridization, IRL Press, Oxford, 1985. Дополнительные подробные инструкции и руковдство по гибридизации нуклеиновых кислот можно найти, например, в Tijssen, “Overview of principles of hybridization and the strategy of nucleic acid probe assays,” in Laboratory Techniques in Biochemistry and Molecular Biology-Hybridization with Nucleic Acid Probes, Part I, Chapter 2, Elsevier, NY, 1993; и Ausubel et al., Eds., Current Protocols in Molecular Biology, Chapter 2, Greene Publishing and Wiley-Interscience, NY, 1995.
[0122] Как используется в настоящем документе термин "жесткие условия" включает условия, при которых гибридизация будет происходить только при наличии менее 50% несоответствия между молекулой гибридизации и ДНК-мишенью. "Жесткие условия" включают дополнительные конкретные уровни жесткости. Таким образом, как используется в настоящем документе условиями "умеренной жесткости" являются условия, при которых не гибридизуются молекулы с более 50% несоответствием последовательностей; условиями "высокой жесткости" являются условия, при которых не гибридизуются последовательности с более 20% несоответствием; и условиями "очень высокой жесткости" являются условия, при которых не гибридизуются последовательности с более чем 10% несоответствием.
[0123] Ниже приведены характерные не ограничивающие условия гибридизации.
[0124] Очень высокая жесткость (детекция последовательностей, которые имеют по меньшей мере 90% идентичность последовательностей): гибридизация в 5× буфере SSC при 65°С в течение 16 часов; промывка два раза в 2× буфере SSC при комнатной температуре в течение 15 минут в каждом случае; и промывка два раза в 0,5× буфере SSC при 65°С в течение 20 минут в каждом случае.
[0125] Высокая жесткость (детекция последовательностей, которые имеют по меньшей мере 80% идентичность последовательностей): гибридизация в 5×-6× буфере SSC при 65-70°С в течение 16-20 часов; промывка два раза в 2× буфере SSC при комнатной температуре в течение 5-20 минут в каждом случае; и промывка два раза в 1× буфере SSC при 55-70°С в течение 30 минут в каждом случае.
[0126] Умеренная жесткость (детекция последовательностей, которые имеют по меньшей мере 50% идентичность последовательностей): гибридизация в 6× буфере SSC при температуре от комнатной до 55°С в течение 16-20 часов; промывка по меньшей мере два раза в 2×-3× буфере SSC при температуре от комнатной до 55°С в течение 20-30 минут в каждом случае.
[0127] Что касается всех зондов, описанных выше, то зонд может содержать дополнительные последовательности нуклеиновых кислот, например, промоторы; транскрипционные сигналы; и/или последовательности векторов. Любой из зондов, описанных выше, может использоваться для определения дополнительных маркеров, которые сцеплены с геном, вовлеченным в резистентность к черной ножке у канол, и маркеры, идентифицированные таким образом, могут быть эквивалентны маркерам, приведенным в качестве примеров в настоящем описании, и, таким образом, находятся в рамках настоящего изобретения.
[0128] Идентичность последовательностей: Термин "идентичность последовательностей" или "идентичность", как используется в настоящем документе в контексте двух последовательностей нуклеиновых кислот или полипептидов, может относиться к остаткам двух последовательностей, которые являются одинаковыми при выравнивании на максимальное соответствие в определенном окне сравнения.
[0129] Как используется в настоящем документе термин "процент идентичности последовательностей" может относиться к значению, определенному при сравнении двух оптимально выровненных последовательностей (например, последовательностей нуклеиновых кислот) в окне сравнения, где часть последовательности в окне сравнения может содержать добавления или делеции (т.е., пропуски) по сравнению с основной последовательностью (которая не содержит добавлений или делеций) для оптимального выравнивания двух последовательностей. Процент рассчитывают путем определения числа положений, в которых находятся идентичные нуклеотиды или аминокислотные остатки в обеих последовательностях, с получением числа совпадающих положений, деля число совпадающих положений на общее число положений в окне сравнения и умножая результат на 100 с получением процента идентичности последовательностей.
[0130] Способы выравнивания последовательностей для сравнения хорошо известны в данной области. Различные программы и алгоритмы выравнивания описаны, например: Smith и Waterman (1981) Adv. Appl. Math. 2:482; Needleman and Wunsch (1970) J. Mol. Biol. 48:443; Pearson and Lipman (1988) Proc. Natl. Acad. Sci. U.S.A. 85:2444; Higgins and Sharp (1988) Gene 73:237-44; Higgins and Sharp (1989) CABIOS 5:151-3; Corpet et al. (1988) Nucleic Acids Res. 16:10881-90; Huang et al. (1992) Comp. Appl. Biosci. 8:155-65; Pearson et al. (1994) Methods Mol. Biol. 24:307-31; Tatiana et al. (1999) FEMS Microbiol. Lett. 174:247-50. Подробное рассмотрение методов выравнивания последовательностей и вычисления гомологии можно найти, например, в Altschul et al. (1990) J. Mol. Biol. 215:403-10.
[0131] Инструмент the National Center for Biotechnology Information (NCBI) Basic Local Alignment Search Tool (BLAST™; Altschul et al. (1990)) доступен из нескольких источников, включая the National Center for Biotechnology Information (Bethesda, MD), и в интернете, для использования применительно к нескольким программам анализа последовательностей. Описание того, как определить идентичность последовательностей, используя эту программу, можно найти в интернете в разделе "Help" для BLAST™. Для сравнения последовательностей нуклеиновых кислот, может быть использована функция "последовательности Blast 2" программы BLAST™ (BLASTN), используя по умолчанию матрицу BLOSUM62 с параметрами по умолчанию. При оценке этим способом последовательности нуклеиновых кислот с еще большей схожестью с основными последовательностями покажут увеличение процента идентичности.
[0132] Молекула нуклеиновой кислоты: Как используется в настоящем документе термин "молекула нуклеиновой кислоты" может относиться к полимерной форме нуклеотидов, которые могут включать как смысловые, так и антисмысловые цепи РНК, кДНК, геномной ДНК и синтетических форм, и смешанных полимеров указанных выше молекул. Нуклеотид может относиться к рибонуклеотиду, дезоксирибонуклеотиду или модифицированной форме любого типа нуклеотида. "Молекула нуклеиновой кислоты", как используется в настоящем документе является синонимом "нуклеиновой кислоте" и "полинуклеотиду". Термин включает одно- и двухцепочечные формы ДНК. Молекула нуклеиновой кислоты может включать как природные, так и модифицированные нуклеотиды, или оба этих типа, связанные друг с другом природными и/или неприродными нуклеотидными связями.
[0133] "Экзогенная" молекула представляет собой молекулу, которая не является природной для определенной системы (например, генетического материала, сорта, элитного сорта и/или растения) относительно нуклеотидной последовательности и/или расположению в геноме полинуклеотида, и по аминокислотной последовательности и/или клеточной локализации полипептида. В вариантах осуществления, экзогенные или гетерогенные полинуклеотиды или полипептиды могут быть молекулами, которые искусственно введены в биологическую систему (например, в растительную клетку, геном растения, конкретный вид растения или сорт и/или хромосому растения) и не являются природными для этой конкретной биологической системы. Таким образом, обозначение нуклеиновой кислоты как "экзогенная" может указывать на то, что нуклеиновая кислота происходит из источника, отличного от природного источника, либо может указывать на то, что нуклеиновая кислота имеет неприродную конфигурацию, генетическое расположение или расстановку элементов.
[0134] В отличие от этого, например, "природные" или "эндогенные" нуклеиновые кислоты представляет собой нуклеиновую кислоту (например, ген), которая не содержит элемента нуклеиновой кислоты, кроме тех, которые обычно присутствуют в хромосоме или другом генетическом материале, в котором обычно обнаруживается нуклеиновая кислота в природе. Эндогенный генный транскрипт кодируется нуклеотидной последовательностью в природном хромосомном локусе, и искусственно не вводится в клетку.
[0135] Термин "рекомбинантный" относится к веществу (например, к рекомбинантной нуклеиновой кислоте, рекомбинантному гену, рекомбинантному полинуклеотиду и/или рекомбинантному полипептиду), которое был изменено путем вмешательства человека. Например, расстановка частей или элементов рекомбинантной молекулы, может быть отлична от природной расстановки и/или первичная последовательность рекомбинантной молекулы может до некоторой степени быть измерена по сравнению с ее природной последовательностью. Вещество может быть изменено с получением рекомбинантного вещества в пределах природного окружения или состояния или выделена из них. Открытая рамка считывания нуклеиновой кислоты является рекомбинантной, если нуклеотидная последовательность открытой рамки считывания была удалены из природного окружения и клонирована в любой тип искусственной нуклеиновой кислоты (например, в вектор). Методики и реагенты для получения рекомбинантных молекул, особенно рекомбинантных нуклеиновых кислот, широко представлены и являются обычными в данной области. Термин "рекомбинантный" также может в настоящем описании относятся к клетке или организму, который содержит рекомбинантное вещество (например, к растению и/или клетке растения, которая содержит рекомбинантную нуклеиновую кислоту). В некоторых примерах рекомбинантный организм представляет собой трансгенный организм.
[0136] Как используется в настоящем документе термин "введен", если речь идет о транслокации гетерологичной или экзогенной нуклеиновой кислоты в клетку, относится к введению нуклеиновой кислоты в клетку, используя любую методику, доступную в данной области. Этот термин включает способы введения нуклеиновых введение кислот, включая, например, но ими не огранчиваясь, трансфекцию; трансформацию; и трансдукцию.
[0137] Как используется в настоящем документе термин "вектор" относится к полинуклеотиду или другим молекулам, которые способны переносить по меньшей мере один сегмент нуклеиновой кислоты в клетку. Вектор может необязательно включать компоненты/элементы, которые обеспечивают поддержание вектора и возможность осуществления его предполагаемого применения (например, последовательности, необходимые для репликации, гены, придающие резистентность к лекарственному средству или антибиотику, сайт множественного клонирования и/или функционально связанные промоторные/энхансерные элементы, которые делают возможным экспрессию клонированного гена). Векторы могут быть получены, например, из плазмид, бактериофагов или вирусов растений или животных. "Клонирующий вектор", "челночный вектор" или "субклонированный вектор" обычно содержит функционально-связанные элементы для упрощения стадий клонирования или субклонирования (например, сайт множественного клонирования, содержащий несколько сайтов рестрикции эндонуклеазы).
[0138] Термин "вектор экспрессии", как используется в настоящем документе относится к вектору, содержащему функционально-связанные полинуклеотидные последовательности, которые могут упрощать экспрессию кодирующей последовательности в конкретном организме-хозяине. Например, бактериальный вектор экспрессии может упрощать экспрессию кодирующей последовательности в бактерии. Растительный вектор экспрессии может упрощать экспрессию кодирующей последовательности в клетке растения. Полинуклеотидные последовательности, упрощающие экспрессию у прокариот, могут включать, например, но ими не ограничиваясь, промотор; оператор; и сайт связывания рибосом. Векторы экспрессии у эукариот (например, вектор экспрессии растений) содержат промоторы, энхансеры, сигналы терминации и полиаденилирования (и другие последовательности), которые обычно отличаются от последовательностей, которые используют в векторах экспрессии прокариот.
[0139] Однонуклеотидный полиморфизм: Как используется в настоящем документе термин "однонуклеотидный полиморфизм" (SNP) может относиться к варианту последовательности ДНК, возникающему, когда один нуклеотид в геноме (или в другой общей последовательности) отличается у представителей видов или между парами хромосом у индивидуума. В пределах популяции SNP могут быть отнесены к меньшей частоте аллелей, которая является максимально низкой частотой аллелей, которая наблюдается в конкретной популяции. Для однонуклеотидных полиморфизмов эта частота меньше двух частот аллелей. Различные популяции, как ожидается, обладают по меньшей мере несколько различными частотами аллелей. Конкретные популяции могут проявлять значительно отличающиеся частоты аллелей. В некоторых примерах, маркеры, сцепленные с резистентностью к SCN, являются маркерами SNP.
[0140] SNP могут находится в пределах кодирующих последовательностей генов, некодирующих областей генов или в межгенных областях между генами. SNP в пределах кодирующей последовательности не обязательно изменяют аминокислотную последовательность белка, который ей продуцируется, из-за вырожденности генетического кода. SNP, в котором обе формы дают одну и ту же полипептидную последовательность, называется "синонимичным" (иногда называется "сайленс" мутацией). Если продуцируется другая последовательность, то их называют "несинонимичными". Несинонимичное изменение могут быть либо миссенс, либо нонсенс, где миссенс изменение приводит к другой аминокислоте, а нонсенс изменение приводит к преждевременному стоп-кодону. SNP, которые находятся вне белок-кодирующих областей, все еще могут оказывать влияние на генный сплайсинг, на связывание транскрипционных факторов или на последовательности некодирующих РНК. SNP, как правило, являются биаллельными и, таким образом, их легко можно анализировать в растениях и животных. Sachidanandam (2001) Nature 409:928-33.
[0141] Растение: Как используется в настоящем документе термин "растение" может относиться ко всему растению, клетке или культуре ткани, полученной из растения и/или любой части любого вышеуказанного. Таким образом, термин "растение" включает, например, но ими не ограничивается, целые растения; компоненты и/или органы растений (например, листья, стебли и корни); растительную ткань; семена; и растительную клетку. Растительная клетка может быть, например, но ими не ограничиваться, клеткой в растении и/или клеткой растения, клеткой, выделенной из растения и клеткой, полученной путем культивирования клетки, выделенной из растения. Таким образом, термин "растение канолы" может относиться, например, но ими не ограничиваться, ко всему растению канолы; к множественным растениям канолы; растительной клетке(ам) канолы; к генетическому материалу растения канолы; культуре ткани канолы (например, из которой может быть регенерировано растение канолы); каллуса растения канолы; частей растения канолы (например, семени канолы, цветка канолы, семядолей канолы, листа канолы, стебля канолы, почки канолы, корня канолы и концевой части корня канолы); и клеток растения канолы, которые интактны в растениях канолы или в частях растений канолы.
[0142] "Трансгенное растение" представляет собой растение, содержащее по меньшей мере в одной из своих клеток экзогенный полинуклеотид. В примерах экзогенный полинуклеотид стабильно интегрирован в геном клетки, таким образом, что полинуклеотид может наследоваться в последующие поколения. В некоторых примерах, гетерологичный полинуклеотид может быть интегрирован в геном как часть рекомбинантной кассеты экспрессии. Термин "трансгенный", как используется в настоящем описании, обозначает любую клетку, клеточную линию, каллус, ткань, часть растения или растение, генотип которого был изменен присутствием экзогенной нуклеиновой кислоты. Таким образом, этот термин охватывает трансгенные организмы и клетки, которые были первоначально изменены и содержат экзогенный полинуклеотид, и эти организмы и клетки созданы путем скрещивания или бесполого размножения исходного трансгенного организма или клетки. Термин "трансгенный", как используется в настоящем документе, не включает геномные (хромосомные или экстра-хромосомные) изменения, введенные обычными способами селекции растений ( например, скрещиванием только нетрансгенных организмов) или природными событиями (например, случайным перекрестным опылением, нерекомбинантным вирусным инфицированием, нерекомбинантной бактериальной трансформацией, нерекомбинантной транспозицией и спонтанной мутацией).
[0143] "Линия” или “сорт” представляет собой группу отдельных растений, имеющих одинаковое происхождение. Растения одной линии в целом до некоторой степени являются инбредными, и, как правило, гомозиготными и гомогенными в большинстве генетических локусов. "Подлиния" может относиться к инбредному подмножеству потомков от общего предка, которые генетически отличаются от других аналогичных инбредных подмножеств того же родителя. В некоторых вариантах осуществления "подлиния" может быть получена в результате инбридинга семян отдельного растения канолы, выбранного из поколений от F3 до F5 до того момента, пока остаточные сегрегирующие локусы остаются гомозиготными в большинстве или во всех локусах.
[0144] Коммерческие сорта канолы, как правило, получают путем объединения самоопыляемого потомства ("партия") одного растения от F3 до F5 при контролируемом скрещивании 2-х генетически разных родителей. В то время как такое разнообразие, как правило, является однородном, самоопыляющееся разнообразие, полученное из выбранного растения, в конечном счете (например, в поколении F8) становится смесью гомозиготных растений, которые могут изменятся в генотипе в любом локусе, который был гетерозиготным в первоначально выбранном растении от F3 до F5. В вариантах осуществления, описанных в настоящем документе, подлинии на основе маркера, которые отличаются друг от друга по качественному маркерному полиморфизму на уровне ДНК в одном или более специфических локусах, получают путем генотипирования образца семян, полученных от индивидуального самоопыляющегося потомства, полученного из отобранных растений от F3 до F5. Такой образец семян может быть генотипирован как семя, или как ткань растения, выращенная из семени. В некоторых примерах, семена, имеющие общий генотип в одном или нескольких определенных маркерных локусах, объединяют в партию с получением подлинии, которая является генетически гомогенной в одном или более локусах, которые сцеплены с представляющим интерес признаком (например, с резистентностью к черной ножке).
[0145] "Наследственная линия" относится к родительской линии, которая является источником, или была использована в качестве источника, генетического материала, например, для создания элитных линий. "Наследственная популяция" относится к группе предков, которые внесли основную часть генетической изменчивости, которая была использована для создания элитной линии. "Потомки" являются потомками предков, и потомки могут быть отделены от своих предков многими поколениями селекции. Например, элитные линии являются потомками своих предков. Родословная может быть использована для описания отношений между потомком и каждым из его предков. Родословная может охватывать одно или несколько поколений, и, таким образом, может описывать отношения между потомком и его предками, удаленными на 1, 2, 3, 4 и тому подобное, поколений.
[0146] "Элитная линия" относится к агрономически превосходной линии, которая была выведена и отобрана (зачастую на протяжении многих циклов) в виду агрономически превосходных характеристик. Многочисленные элитные линии канолы доступны и известны специалистам в данной области. Элитная популяция представляет собой ассортимент элитных линий или особей из элитных линий, которые могут быть использованы как представители уровня техники с точки зрения агрономически превосходных генотипов данного вида сельско-хозяйственных культур (например, канолы). Аналогично, элитный генетический материал или элитная линия генетического материала является агрономически превосходным генетическим материалом. Элитный генетический материал может быть получен из растения с агрономически превосходными характеристиками, и может быть использована для создания растения с агрономически превосходными характеристиками, такого как канола уже существующей или недавно разработанной элитной линии.
[0147] В отличие от элитных линий, "экзотическая линия" (или "экзотический генетический материал") относится к линии или генетическому материалу, полученных из канолы, не принадлежащей к доступной элитной линии канолы или генетическому материалу. В контексте скрещивания двух растений канолы или генетического материала, экзотический генетический материал не связан тесно по происхождению с элитным генетическим материалом, с которым его скрещивают. Чаще всего, экзотический генетический материал отобран для введения нового генетического элемента (например, представляющей интерес аллельной формы) в программу селекции.
[0148] Признак или фенотип: Термины "признак" и "фенотип" используются в настоящем описании взаимозаменяемо для обозначения измеряемой или наблюдаемой наследственной характеристики. Фенотип может в некоторых примерах находится непосредственно под контролем одного гена или генетического локуса (т.е., одного признака гена). В других примерах, фенотип может быть результатом взаимодействия нескольких генов (комплексный признак). Таким образом, QTL может действовать через механизм одного гена или полигенный механизм. В некоторых примерах, признак или фенотип могут быть обозначены как "фенотипическое значение", что соответствует количественному значению, измеренному в отношении фенотипического признака.
[0149] Термин "молекулярная фенотип" может относиться к фенотипу, который детекцируют на уровне популяции (одной или более) молекул. В некоторых примерах, молекулярный фенотип может быть детектируемым только на молекулярном уровне. Детектируемые молекулы фенотипа могут быть нуклеиновыми кислотами (например, геномной ДНК или РНК); белками; и/или метаболитами. Например, молекулярный фенотип может быть профилем экспрессии одного или нескольких генных продуктов (например, на специфической стадии развития растений, или в ответ на состояние окружающей среды или стресс).
[0150] Резистентность к черной ножке: для целей настоящего описания признаком, представляющим особый интерес, является "резистентность к черной ножке.” Специалисту в данной области будет понятно, что резистентность к черной ножке в основном определяется наследственными генетическими факторами. Так, например, отбор конкретного сорта канолы для культивирования может быть основан по меньшей мере частично на резистентности к характерному заболеванию (например, к черной ножке) этого конкретного сорта при нормальных условиях выращивания в поле (например, в условиях без засухи, болезней и при адекватном количестве питательных веществ в почве). Например, растение канолы с чувствительностью к черной ножке может иметь оценку чувствительности, равную 0, 1, 3, 5, 7 или 9 (см. Таблицу 1), где оценка 9 указывает на высокую чувствительность, или ограниченную резистентность, к болезни черная ножка.
[0151] В некоторых вариантах осуществления "резистентность к черной ножке" определяется путем сравнения с характерной резистентностью к черной ножке у сорта дикого типа или родительского сорта. Таким образом, первое растение канолы, содержащее фенотип резистентности к черной ножке, может иметь "повышенные" или "увеличенные" уровни резистентности (или "сниженные" или "уменьшенные" уровни резистентности к черной ножке) по сравнению с канолой дикого типа, или по сравнению с родительским сортом канолы, из которого получено первое растение канолы. Повышенный или увеличенный являются относительными терминами, которые указывают, что растение обладает лучшей резистентностью к болезни черная ножка, или обладает ей в большей степени, чем аналогичное растение дикого типа. Сниженный и уменьшенный являются относительными терминами, которые указывают, что растение более восприимчиво к болезни черная ножка или подвержено ей в большей степени, чем аналогичное растение дикого типа.
[0152] Описание того, как оценивают степень резистентности к черной ножке.
Обнаружение маркеров резистентности в каноле
[0153] Способы детекции (выявления) растений канолы или генетического материала, которые несут определенные аллели маркерных локусов резистентности к черной ножке, являются признаками некоторых вариантов осуществления. В некоторых вариантах осуществления любая из множества методик детекции маркера, доступная из уровня техники, может быть использована для детекции маркерного аллеля, в зависимости от типа детектируемого маркера. Например, подходящие способы детекции маркеров могут включать амплификацию и идентификацию полученного амплифицированного маркера, например, но ими не ограничиваясь, с помощью ПЦР; LCR; и методами аплификации, основанными на транскрипции (например, ASH, детекция SSR, анализ RFLP и многие другие).
[0154] В целом, генетический маркер сцеплен с одним или несколькими свойствами нуклеиновых кислот для его детекции. Например, некоторые методики детекции генетических маркеров используют гибридизацию зонда нуклеиновой кислоты с нуклеиновой кислотой, соответствующей генетическому маркеру (например, амплифицированная нуклеиновая кислота, полученная с использованием геномных молекул ДНК канолы в качестве матрицы). Форматы гибридизация включают, например, но ими не ограничиваются, фазу раствора; твердую фазу; смешанную фазу; а также могут быть использованы анализы гибридизации in situ для детекции аллеля в конкретных вариантах осуществления. Подробное руководство по гибридизации нуклеиновых кислот можно найти, например, в Tijssen (1993) Laboratory Techniques in Biochemistry and Molecular Biology-Hybridization with Nucleic Acid Probes Elsevier, NY.
[0155] Маркеры, соответствующие генетическим полиморфизмам между представителями популяции, могут быть детектированы любым из многочисленных способов, включая, например, но ими не ограничиваясь, способы амплификации на основе нуклеиновых кислот; и нуклеотидное секвинирование области полиморфного маркера. Многие способы детекции (включая способы на основе амплификации и секвенирования) могут быть без труда адаптированы к высокоэффективному анализу в некоторых примерах, например, используя доступные высокоэффективные способы секвенирования, такие как секвенирование путем гибридизации.
[0156] Праймеры для амплификации в случае амплификации маркерных локусов типа SSR, включены в качестве конкретных примеров некоторых вариантов осуществления. В таблице 6 представлены специфические праймеры для амплификации конкретных маркеров, описанных в настоящем документе. Тем не менее, специалисту в данной области будет понятно, что вместо указанных праймеров могут быть использованы другие последовательности по обе стороны от указанных праймеров, если эти праймеры способны амплифицировать нуклеотидную последовательность, содержащую детектируемую аллель. Кроме того, точный зонд, используемый для детекции аллеля, может изменен. Например, любой зонд, способный идентифицировать область детектируемого маркерного ампликона, может быть заменен на зонды, указанные в настоящем описании в качестве примера. Кроме того, конфигурация праймеров амплификации и зондов для детекции, может также различаться. Таким образом, варианты осуществления изобретения не ограничены праймерами и зондами, конкретно указанными в настоящем описании. Хотя в настоящем описании приведено большое число примеров характерных праймеров (см. Таблицу 6), подходящие праймеры для использования по настоящему изобретению могут быть сконструированы, используя любой подходящий способ. Например, эквивалентные праймеры могут быть сконструированы, используя любую подходящую программу, например, но ими не ограничиваясь, LASERGENE®.
[0157] Молекулярные маркеры могут быть детектированы с помощью принятых способов, доступных в данной области, включая, например, но ими не ограничиваясь, ASH, или другие способы обнаружения SNP; детекцию AFLP; детекцию амплифицированных вариабельных последовательностей; детекцию RAPD; детекцию RFLP; детекцию самоподдерживающейся репликации последовательностей; детекцию SSR; детекцию SSCP; и детекцию маркеров изоферментов. Хотя приведенные в качестве примеров маркеров, показанные на Фиг. 1 и в Таблице 6, являются маркерами SSR, любой из вышеупомянутых типов маркеров может быть использован в конкретных вариантах осуществления для идентификации сегментов хромосом, включающих генетический элемент, который вносит вклад в фенотип резистентности к черной ножке у канолы.
[0158] Например, маркеры, которые содержат RFLP, могут быть детектированы, например, гибридизацией зонда (который, как правило, является субфрагментом или синтетическим олигонуклеотидом, соответствующим субфрагменту) детектируемой нуклеиновой кислоты с геномной ДНК, расщепленной ферментами рестрикции. Фермент рестрикции выбирают таким образом, чтобы получить ферменты рестрикции по крайней мере двух альтернативных (или полиморфных) длин у разных особей или популяций. Определение одного или несколько ферментов рестрикции, которые дают информативные фрагменты для каждого скрещивания, является простой процедурой, которая без труда может быть осуществлена специалистом в данной области после получения последовательности ДНК-мишени. После разделения по длине в соответствующей матрице (например, агарозном или полиакриламидном геле) и переноса на мембрану (например, нитроцеллюлозную или мембрану из нейлона) меченый зонд может быть гибридизован в условиях, которые приводят к равновесному связыванию зонда с мишенью, с последующим удалением избытка зонда путем промывки и обнаружением меченного зонда.
[0159] В некоторых вариантах осуществления стадию амплификации используют как часть способа детекции/генотипирования маркерного локуса. Тем не менее, стадия амплификации не во всех случаях является обязательным требованием для детекции маркера. Например, неамплифицированная геномная ДНК может быть детектирована путем простого проведения Саузерн-блоттинга на образце геномной ДНК. Отдельные зонды детекции также могут не использоваться в способах амплификации/детекции, например, но ими не ограничиваясь, при осуществлении реакции амплификации в реальном времени, при которой детектируют образование продукта путем модификации праймера для амплификации после введения в продукт; введении меченых нуклеотидов в ампликон; и путем мониторинга изменений свойств молекулярного вращения ампликонов по сравнению с неамплифицированными предшественниками (например, с помощью флуоресцентной поляризации).
[0160] ПЦР, ОТ-ПЦР, ПЦР в реальном времени и LCR, имеют, в частности, широкое применение в качестве способов амплификации и амплификации-детекции для амплификации и детекции нуклеиновых кислот (например, нуклеиновых кислот, содержащих маркерные локусы). Подробное описание, касающееся применения этих и других способов амплификации, можно найти в любом из множества стандартных текстов, включая Sambrook et al., Molecular Cloning- A Laboratory Manual (2000) 3rd Ed., Vol. 1-3, Cold Spring Harbor Laboratory, Cold Spring Harbor, NY; Current Protocols in Molecular Biology, (с добавлениями до 2002) F. M. Ausubel et al., eds., Current Protocols, совместное предприятие Greene Publishing Associates, Inc. и John Wiley & Sons, Inc.; и PCR Protocols A Guide to Methods and Applications (1990) Innis et al. eds) Academic Press Inc., San Diego, CA. Дополнительные сведения, касающиеся детекции нуклеиновых кислот в растениях, также могут быть найдены, например, в Plant Molecular Biology (1993) Croy (ed.) BIOS Scientific Publishers, Inc.
[0161] Дополнительные сведения, касающиеся методик, достаточных для специалистов, непосредственно в данной области, о конкретных способах амплификации и детекции in vitro, включая полимеразную цепную реакцию (ПЦР), лигазную цепную реакцию (LCR), амплификацию с Qβ-репликазой и другие методики, опосредованные РНК полимеразой (например,, NASBA), и их примеры, могут быть найдены, например, в: патенте США 4683202; Arnheim and Levinson (1991) J. NIH Res. 3:81-94; Kwoh et al. (1989) Proc. Natl. Acad. Sci. USA 86:1173; Guatelli et al. (1990), выше; Lomell et al. (1989) J. Clin. Chem. 35:1826; Landegren et al. (1988) Science 241:1077-80; Van Brunt (1990) Biotechnology 8:291-4; Wu and Wallace (1989) Gene 4:560; Barringer et al. (1990) Gene 89:117; и Sooknanan and Malek (1995) Biotechnology 13:563-4. Усовершенствованные способы амплификации крупных нуклеиновых кислот с помощью ПЦР, которые могут быть использованы в некоторых случаях применения позиционного клонирования, дополнительно описаны в статье Cheng et al. (1994) Nature 369: 684, и приведенных в ней ссылках, в которых получают ампликоны ПЦР размером до 40 к.б.
[0162] Большое число доступных текстов по биологии также подробно разбирают ПЦР и связанные с ней способы амплификации. Специалисту будет понятно, что по существу любая РНК может быть преобразована в двухцепочечную ДНК, которая может быть использована для расщепления рестриктазами, для ПЦР-амплификации и секвенирования с использованием обратной транскриптазы и полимеразы (например, ОТ-ПЦР).
[0163] В некоторых вариантах осуществления зонд нуклеиновой кислоты может быть использован для детекции нуклеиновой кислоты, которая содержит маркерный аллель нуклеотидной последовательности. Такие зонды могут быть использованы, например, при позиционном клонировании для выделения нуклеотидных последовательностей, которые сцеплены с последовательностью маркерного аллеля. Зонды нуклеиновых кислот, которые могут быть использованы в конкретных вариантах осуществления не ограничены каким-либо конкретным ограниченным размером. В некоторых вариантах осуществления зонд нуклеиновой кислоты может, например, но ими не ограничиваясь, иметь длину по меньшей мере 20 нуклеотидов; по меньшей мере 50 нуклеотидов; по меньшей мере 100 нуклеотидов; и по меньшей мере 200 нуклеотидов. Зонды нуклеиновых кислот к маркерному локусу могут быть клонированы и/или синтезированы.
[0164] Любая подходящая метка может быть использована вместе с зондом в конкретных примерах. Детектируемые метки, подходящие для применения вместе с зондами нуклеиновых кислот, включают любую композицию, детектируемую спектроскопическими, радиоизотопными, фотохимическими, биохимическими, иммунохимическими, электрическими, оптическими или химическими средствами. Таким образом, зонд для гибридизации может быть детектирован, используя, например, авторадиографию, флюорографию или другие подобные методики детекции, в зависимости от конкретной детектируемой метки. Метки, которые могут быть использованы, включают биотин (для окрашивания меченого стрептавидином конъюгата), магнитные бусы, флуоресцентные красители, радиоактивные метки, ферменты и колориметрические метки. Другие метки включают лиганды, которые связываются с антителами или со специфическими связывающими мишенями, меченными флуорофорами, хемилюминесцентными агентами и ферментами. Зонд также может содержать радиоактивно-меченные ПЦР -праймеры, которые используются для создания радиоактивно-меченного ампликона. Дополнительная информация о подходах введения меток при мечении нуклеиновых кислот, а также соответствующие подходы детекции можно найти, например, в Haugland (1996) Handbook of Fluorescent Probes and Research Chemicals, Sixth Edition, Molecular Probes, Inc., Eugene OR; and Haugland (2001) Handbook of Fluorescent Probes and Research Chemicals, Eigth Edition, Molecular Probes, Inc., Eugene, OR (доступно на CD ROM). В конкретных примерах детекцию с помощью ПЦР и количественное определение проводят с использованием олигонуклеотидных зондов с двойной флуорогенной меткой, например, зондов TaqMan® (Applied Biosystems).
[0165] В некоторых вариантах осуществления праймеры не метят, а маркерные ампликоны ПЦР могут быть визуализированы, например, после их разделения по размеру (например, после электрофореза в агарозном геле). В конкретных примерах окрашивание этидий бромидом ПЦР-ампликона, после их разделения по размеру, позволяет визуализировать различные по размеру ампликоны, соответствующие различным маркерным аллелям.
[0166] Праймеры для использования в вариантах осуществления изобретения не ограничены праймерами, способными давать ампликон любого конкретного размера. Например, праймеры, использованные для амплификации конкретных маркерных локусов и аллелей, не ограничены праймерами, амплифицирующими всю область соответствующего локуса. Праймеры могут давать ампликона любой подходящей длины, которая длиннее или короче, чем указанные при определении термина "аллель". Например, маркер амплификации может давать ампликон, который, например, но ими не ограничиваясь, может иметь длину по меньшей мере 20 нуклеотидов; по меньшей мере 50 нуклеотидов; по меньшей мере 100 нуклеотидов; и по меньшей мере 200 нуклеотидов.
[0167] Способы синтеза для получения олигонуклеотидов и используемых композиции, содержащих олигонуклеотиды (например, зонды, праймеры, молекулярные маяки, PNAS и LNA), как правило, хорошо известны специалистам в данной области. Например, олигонуклеотиды могут быть синтезированы химически в соответствии со способом твердофазного синтеза с использованием фосфорамидит триэфира, как описано, например, Beaucage and Caruthers (1981) Tetrahedron Letts. 22(20):1859-62. Такие способы могут использовать автоматизированный синтезатор, например, но ими не ограничиваясь, как описано Needham-VanDevanter et al. (1984) Nucleic Acids Res. 12:6159-68. Олигонуклеотиды (включая модифицированные олигонуклеотиды) также могут быть заказаны из различных коммерческих источников, в том числе, например, но ими не ограничиваясь, The Midland Certified Reagent Company; The Great American Gene Company; ExpressGen Inc.; и Operon Technologies Inc. Аналогично, РНК могут быть специально заказаны в любом из множества источников, включая, например, но ими не ограничиваясь, PeptidoGenic; HTI Bio-Products, Inc.; BMA Biomedicals Ltd (U.K.); and Bio.Synthesis, Inc.
[0168] В некоторых вариантах осуществления способ in silico может быть использован для обнаружения маркерного аллеля. Например, последовательность нуклеиновой кислоты, содержащая последовательность маркера, может храниться на компьютере. Желаемая последовательность маркерного локуса (или ее гомолог) может быть идентифицирована, используя подходящий алгоритм поиска нуклеиновой кислоты, как предложено, например, но ими не ограничиваясь, BLAST™, или еще более простыми текстовыми процессорами.
[0169] В некоторых вариантах осуществления маркерный аллель детектируют с использованием способа детекции на основе ПЦР, где размер или последовательность ампликона ПЦР, содержащая маркер, указывает на отсутствие или наличие конкретного маркерного аллеля. В некоторых примерах, ПЦР-праймеры гибридизуются с консервативными областями, фланкирующими область полиморфного маркера. ПЦР-праймеры, используемые таким образом для амплификации молекулярного маркера, иногда в данной области называют "маркерами ПЦР" или просто "маркерами".
[0170] Основной мотивацией для разработки молекулярных маркеров для сортов сельскохозяйственных культур является возможное повышение эффективности селекции растений посредством отбора с помощью маркеров (MAS). Генетические маркеры, которые сцеплены с представляющим интерес признаком или геном могут быть использованы для идентификации растений, которые содержат желаемый маркерный аллель в одном или нескольких локусах, который как ожидается, растения передадут наряду с представляющим интерес признаком или геном своему потомству. Генетические маркеры могут быть использованы для идентификации растений, которые содержат конкретный генотип в одном локусе или в нескольких несцепленных или сцепленных локусах (например, гаплотип). Аналогично, маркерные аллели, описанные в настоящем документе, могут быть введены с помощью и в желаемый фоновый генотип канолы, генетический материал, растение, линию, сорт и тому подобное, как часть всех программ селекции MAS, разработанных для улучшения урожая канолы.
[0171] В соответствии с некоторыми вариантами осуществления, маркеры, описанные в настоящем документе, представляют средства для идентификации растений канолы и генетического материала, которые обладают резистентностью к черной ножке (или повышенной резистентностью к черной ножке, или сниженной чувствительностью к черной ножке) путем идентификации растений и генетического материала, содержащих специфическую аллель в локусе, такую как DBSNP09246, DBSNP01407, DBSNP05863, DBSNP01261, и/или маркерный локус, сцепленный по меньшей мере с одним из указанных выше аллелей. Благодаря идентификации растения без маркерного аллеля, который сегрегирует вместе с резистентностью к черной ножке, могут быть идентифицированы растения и генетический материал, чувствительные к черной ножке (или растения со сниженной степенью резистентности к черной ножке), например, для исключения их из последующих скрещиваний и селекции.
[0172] В соответствии с вышеизложенным, варианты осуществления настоящего изобретения включают молекулярные маркеры, которые имеют значительную вероятность совместной сегрегации с QTL, что способствует фенотипу резистентности к черной ножке или придает его. Эти маркеры QTL находят применение в селекции с помощью маркеров по желательным признакам (по резистентности к черной ножке), а также для других вариантов применения. Варианты осуществления настоящего изобретения не ограничиваются каким-либо конкретным способом детекции или анализа этих маркеров.
Интрогрессия маркеров резистентности к черной ножке в линии канолы
[0173] Как было указано, выше, идентификация растений канолы или генетического материала, которые включают маркерный аллель или аллели, которые сцеплены с фенотипом с резистентностью к черной ножке, является основой для проведения отбора канолы с помощью маркеров. В некоторых вариантах осуществления отбирают по меньшей мере одно растение канолы, которое содержит по меньшей мере один маркерный аллель, который положительно коррелирует с резистентностью к черной ножке, при этом растения канолы, которые содержат маркерные аллели, которые имеют отрицательную корреляцию с резистентностью к черной ножке, могут быть не отобраны.
[0174] Желаемые маркерные аллели, которые положительно коррелируют с резистентностью к черной ножке, могут быть введены путем интрогрессии в канолу, имеющую конкретный (например, элитный или экзотический) фоновый генотип, таким образом, чтобы получить растение или генетический материал с интрогрессией резистентности к черной ножке. В некоторых вариантах осуществления множество маркеров резистентности к черной ножке может быть последовательно или одновременно отобрано и/или введено интрогрессией в канолу. Конкретные комбинации маркеров резистентности к черной ножке, которые могут быть отобраны для одного растения или генетического материала, не ограничены и могут включать комбинацию маркеров, таких как комбинации, представленные на Фиг.2, все маркеры, сцеплены с маркерами, перечисленными на Фиг.2, или любые маркеры, расположенные в пределах интервалов QTL, как определено в настоящем документе.
[0175] В вариантах осуществления способность идентифицировать маркерные аллели QTL, которые положительно коррелируют с резистентностью к черной ножке растения канолы, также обеспечивает способ отбора растений, которые имеют благоприятные маркерные локусы. Например, может быть отобрано любое растение, которое идентифицировано как содержащее желаемый маркерный аллель (например, маркерный аллель, который положительно коррелирует с резистентностью к черной ножке), а растения, которые не имеют этот аллель (или, которые содержат аллель, который отрицательно коррелирует с резистентностью к черной ножке) могут быть не отобраны. Таким образом, в конкретных вариантах осуществления, после идентификации маркерного аллеля в первом растении или генетическом материале, способ интрогрессии включает отбор первого растения канолы или генетического материала, или отбор потомства первого растения или генетического материала. В некоторых примерах, полученное при отборе растение канолы или генетический материал могут быть скрещены со вторым растением канолы или генетическим материалом (например, элитной канолой или экзотической канолой) таким образом, чтобы дать потомство, содержащее маркерный аллель и желаемые характеристики и/или аллели второго растения или генетического материала.
[0176] В некоторых вариантах осуществления способ интрогрессии QTL резистентности к черной ножке может предусматривать, например, наличие по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке (например, маркера, который сегрегирует вместе с резистентностью к черной ножке); определение маркерного аллеля в первом растении или генетическом материале, содержащих QTL резистентности к черной ножке; и интрогрессию маркерного аллеля во второе растение канолы или генетический материал, с получением, таким образом, растения канолы или генетического материала с интрогрессией. В конкретных вариантах осуществления второе растение канолы или генетический материал могут обладать улучшенной резистентностью к черной ножке по сравнению с первым растением канолы или генетическим материалом, в то время как растение канолы или генетический материал с интрогрессией будут обладать резистентность к черной ножке по сравнению со вторым растением или генетическим материалом. Как будет описано более подробно ниже, растение канолы или генетический материал с итрогрессией, полученные в этих и других вариантах осуществления, также включены в варианты осуществления по настоящему изобретению.
[0177] В некоторых вариантах осуществления, если растение канолы или генетический материал с интрогрессией получены любым из способов, приведенных в настоящем описании, то растение канолы или генетический материал с интрогрессией могут быть охарактеризованы по чувствительности или резистентности к черной ножке растения. Растение или генетический материал с интрогрессией могут содержать, например, но ими не ограничиваясь, уровень чувствительности к черной ножке, равный 0, 1, 3 или 5. В некоторых примерах, такое растение канолы или генетический материал с интрогрессией имеет уровень чувствительности к черной ножке, равный 0, 1, 3, 5 или 7.
[0178] Кроме отобранных маркерных аллелей (например, с помощью стандартных способов селекции), введенных интрогрессией в желаемые фоновые генотипы, так, что QTL резистентности к черной ножке была введена интрогрессией в фоновый генотип, в некоторых вариантах осуществления трансгенные подходы могут быть использованы для получения растений канолы и/или генетического материала с резистентностью к черной ножке. В некоторых вариантах осуществления экзогенная нуклеиновая кислота (например, ген или открытая рамка считывания), которая сцеплена по меньшей мере с одним маркером, как описано в настоящем документе, в каноле, может быть введена в мишеневые растение или генетический материал. Например, нуклеиновая кислота, кодирующая последовательность, сцепленную по меньшей мере с одним маркером, как описано в настоящем документе, может быть клонирована из геномной ДНК канолы (например, с помощью позиционного клонирования) и введена в мишеневые растение или генетический материал.
[0179] Таким образом, конкретные варианты осуществления включают способы получения растения канолы или генетического материала, обладающих фенотипом резистентности к черной ножке, где способ включает введение экзогенной нуклеиновой кислоты в мишеневое растение канолы или его потомство, где экзогенная нуклеиновая кислота по существу идентична нуклеотидной последовательности, которая сцеплена по меньшей мере с одним положительно коррелированным маркерным аллелем на одном или более маркерных локусах, которые сцеплены с резистентностью к черной ножке. В некоторых примерах, маркерный локус может быть выбран из: DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705; и маркера, который сцеплен (например, демонстрирует не более 10% частоты рекомбинации) по меньшей мере с одним из указанных выше. В некоторых вариантах осуществления для создания трансгенного растения может быть использовано множество сцепленных маркеров. Каждый из маркеров, описанных в настоящем документе, который используют в таком множестве, находится в компетенции специалиста.
[0180] Любой из разнообразных способов может быть использован для введения экзогенной нуклеиновой кислоты в растение канолы или генетический материал. В некоторых вариантах осуществления нуклеотидную последовательность выделяют с помощью позиционного клонирования и идентифицируют по сцеплению с маркерным аллелем, который положительно коррелирует с резистентностью к черной ножке. Например, нуклеотидная последовательность может соответствовать открытой рамке считывания (ORF), которая кодирует полипептид, который при экспрессии в растении канолы, приводит к резистентности к черной ножке растения канолы или способствует такой резистентности. Нуклеотидная последовательность может быть затем введена в экзогенную молекулу нуклеиновой кислоты. Точный состав экзогенной нуклеиновой кислоты может изменяться. Например, экзогенная нуклеиновая кислота может содержать вектор экспрессии для экспрессии нуклеотидной последовательности в растении, в которое введена экзогенная нуклеиновая кислота.
[0181] Маркеры, сцепленные с резистентностью к черной ножке, могут может быть введены интрогрессией (например, таким образом, вводя интрогрессией фенотип резистентности к черной ножке) в растение канолы или генетический материал, используя способ, включающий отбор при помощи маркера. В вариантах осуществления MAS осуществляют, используя полиморфные маркеры, которые идентифицированы как имеющие значительную вероятность совместной сегрегации с признаком резистентности к черной ножке. Такие маркеры (например, маркеры, представленные на ФИГ. 2), как предполагают, располагаются в пределах или вблизи гена или генов, которые способствуют проявлению фенотипа резистентности к черной ножке (по сравнению с растением, содержащим ген или гены дикого типа). Такие маркеры могут рассматриваться как индикаторы признака, и могут быть отнесены к маркерам QTL. В вариантах осуществления изобретения растение или генетический материал проверяют на наличие положительно коррелирующего аллеля по крайней мере в одном маркере QTL.
[0182] В вариантах осуществления анализ сцепления используют для определения того, какие полиморфные маркерные аллели демонстрирует статистическую вероятность совместной сегрегации с фенотипом резистентности к черной ножке. После идентификации такого маркерного аллеля с положительной корреляцией с фенотипом резистентности к черной ножке, маркер затем может быть использован для быстрого и точного скрининга линий растений на аллель резистентности к черной ножке без необходимости выращивания растений в жизненном цикле и ожидания фенотипических оценок. Кроме того, идентификация маркера обеспечивает генетический отбор на конкретный аллель резистентности к черной ножке, даже если молекулярная идентичность фактического QTL резистентности к черной ножке неизвестна. Небольшой образец ткани (например, первого листа растения) может быть взят у потомства растения канолы, полученного путем скрещивания, и скринирован вместе с соответствующим молекулярным маркером. Таким образом, можно быстро определить следует ли использовать потомство для дальнейшего разведения.
[0183] В некоторых вариантах осуществления, включающих MAS, полиморфный маркерный локус QTL может быть использован для отбора растения, которое содержит маркерный аллель (или аллели), который положительно коррелирует с резистентностью к черной ножке. Например, нуклеиновая кислота, соответствующая нуклеиновой кислоте маркерного аллеля может быть детектирована в биологическом образце, взятом у растения для отбора. Такая детекция может иметь форму гибридизации зонда нуклеиновой кислоты с маркерным аллелем или его ампликоном (например, используя аллель-специфическую гибридизацию, Саузерн-блоттинг, Нозерн-блоттинг, гибридизацию in situ и гибридизацию праймеров с последующей ПЦР-амплификацией области маркера). После проверки наличия (или отсутствия) конкретного маркерного аллеля в биологическом образце, растение отбирают и, в некоторых примерах, его можно использовать для получения потомства растений путем селекции.
[0184] Растениеводы-селекционеры хотят иметь комбинацию маркерных локусов резистентности к черной ножке и маркеров/генов других желательных признаков (например, высокого урожая) для разработки улучшенных сортов канолы. Скрининг большого числа образцов с помощью немолекулярных методов (например, оценка признака в растениях канолы), как правило, является дорогостоящим, требующим много времени и ненадежным. Использование полиморфных маркеров, описанных в настоящем документе, которые сцеплены с QTL резистентности к черной ножке, обеспечивает эффективный способ отбора желательных сортов в селекционных программах. Преимущества отбора при помощи маркеров по сравнению с полевыми оценками на резистентность к черной ножке заключаются, например, в том, что MAS может быть проведен в любое время года, независимо от сезона выращивания. Кроме того, как указано выше, воздействия окружающей среды в значительной степени не имеют значения для отбора при помощи маркеров.
[0185] Если в популяции происходит сегрегация множества маркерных локусов, сцепленных с одним или несколькими признаками (например, множества маркеров, сцепленных с резистентностью к черной ножке), то эффективность MAS по сравнению с фенотипическим скринингом становится еще больше, так как все маркерные локусы, могут быть оценены в лаборатории в единственном образце ДНК. В конкретных вариантах осуществления настоящего изобретения, DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590 маркеры, а также маркеры, сцепленные по меньшей мере с одним из указанных выше, могут быть проанализированы одновременно или последовательно в одном образце, или во множестве параллельных образцов.
[0186] Еще одним применением MAS в селекции растений является содействие восстановлению рекуррентного родительского генотипа путем селекции возвратным скрещиванием. Возвратное скрещивание обычно проводят с целью интрогрессии одного или нескольких маркеров или локусов QTL от донорного родителя (например, родителя, содержащего желаемый маркерный локус резистентности к черной ножке) в другой желательный фоновый генотип рекуррентного родителя (например, в другую высокоурожайную линию канолы). Чем больше проводят циклов возвратного скрещивания, тем больше генетический вклад рекуррентного родителя в полученный сорт с интрогрессией. В некоторых примерах, может быть проведено большое число циклов возвратного скрещивания, например, из-за того, что растения с резистентностью к черной ножке могут быть по каким-либо причинам нежелательны, например, из-за низкой урожайности, низкой плодовитости и тому подобное. Напротив, линии, которые являются результатом интенсивных программ селекции, могут иметь прекрасную урожайность, плодовитость и тому подобное, но единственно не иметь желательного признака, например, резистентности к черной ножке. При возвратном скрещивании при помощи маркеров из специфических маркеров донорного источника, которые могут или не могут основывать элитный фоновый генотип для элитного сорта, который будет служить в качестве реккурентной линии, специалист может отобрать в пределах потомства возвратного скрещивания в качестве донорного маркера, а затем использовать повторное возвратное скрещивание с рекуррентной линией для восстановления такого объема генома рекуррентной линии, который только возможен.
[0187] В соответствии с вышеизложенным, маркеры и способы, описанные в настоящей заявке, могут быть использованы для того, чтобы проводить отбор при помощи маркеров или селекцию сортов канолы с желаемым компонентом (набором) аллельных форм сегментов хромосом, сцепленных с агрономически превосходными характеристиками (например, резистентностью к черной ножке наряду с любыми другими доступными маркерами урожайности, устойчивости к болезням и тому подобное). Любой из описанных маркерных аллелей может быть введен в линию канолы с помощью интрогрессии (например, путем традиционной селекции, посредством трансформации или с помощью их обоих) с получением растения канолы с агрономически превосходными характеристиками. Если нуклеиновые кислоты растения являются положительными в отношении желаемого генетического маркерного аллеля, то растение может самоопыляться в некоторых вариантах осуществления с получением линией истинной селекции с тем же генотипом, или же может быть скрещено с растением, содержащим тот же маркерный аллель или другие желаемые маркеры и/или характеристики, с получением гибридного поколения половым скрещиванием.
[0188] Часто, способ по настоящему изобретению используют по крайней мере в отношении одного соответствующего растения канолы, такого как растение из линий предшественников или линий потомства в родословной растений канолы, являющихся объектом способа, таким образом, чтобы наследование желаемого аллеля резистентности к черной ножке аллель могло бы быть прослежено. Число поколений, отделяющих растения канолы, являющиеся объектом способа по настоящему изобретению, как правило, будет составлять от 1 до 20, обычно от 1 до 5, и, как правило, 1, 2 или 3 разделенных поколения, и довольно часто прямой потомок или родитель растения канолы будет являться объектом способа (то есть, одно разделенное поколение).
[0189] Генетическое разнообразие играет важную роль в селекционных программах. При ограниченном разнообразии, генетический эффект, достигаемый в программе разведения, в конечном счете достигнет плато, когда все благоприятные аллели будут закреплены в пределах элитной популяции. Таким образом, одной из целей селекции растений является включение разнообразия в элитный пул без потери генетического эффекта, который уже был достигнут, и с минимальными возможными инвестициями. MAS указывает какие геномные области и какие благоприятные аллели первоначальных предков были отобраны и сохраняются в течение долгого времени, что облегчает усилия по введению благоприятного изменения из источников экзотического генетического материала (родителей, которые не связаны с элитным генофондом) в надежде обнаружить благоприятные аллели, которые в настоящее время отсутствуют в элитном генофонде. Таким образом, в некоторых вариантах осуществления маркеры, описанные в настоящем документе, могут быть использованы для MAS при скрещивании с участием (элитная х экзотическая) линий канолы, обеспечивая в сегрегирующем потомстве MAS сохранение главных аллелей урожайности, наряду с маркерными аллелями резистентности к черной ножке, как описано в настоящем документе.
[0190] Молекулярные маркерные локусы и аллели, описанные в настоящем документе (например, DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787 и DBSNP01590, и маркеры, сцепленные по меньшей мере с одним из выше перечисленных) могут быть использованы в некоторых вариантах осуществления, как указано выше, для идентификации QTL резистентности к черной ножке, который затем может быть клонирован известными методиками. Такие клоны с повышенной резистентностью к черной ножке могут быть сначала идентифицированы по генетическому сцеплению с маркерами, как описано в настоящем документе. Например, при "позиционном клонировании генов" используют преимущества физической близости к маркеру резистентности к черной ножке для определения выделенного фрагмента хромосомы, содержащего ген QTL резистентности к черной ножке. Выделенный фрагмент хромосомы может быть получен такими известными способами, как, например, но ими не ограничиваясь, перевариванием хромосомной ДНК одним или несколькими ферментами рестрикции, амплификацией хромосомной области, используя ПЦР, а также любую подходящую альтернативную реакцию амплификации. Переваренный ферментами или амплифицированный фрагмент может быть затем лигирован в вектор, подходящий для репликации и/или экспрессии встроенного фрагмента. Маркеры, которые прилегают к ORF, сцепленной с фенотипическим признаком, могут быть специфически гибридизированы с клоном ДНК (например, с клоном из геномной библиотеки ДНК), таким образом идентифицируя клон, на котором расположен ORF (или фрагмент ORF). Если маркер отдален от гена QTL резистентности к черной ножке на большее расстояние, то фрагмент, содержащий ORF, может быть идентифицирован путем последовательных циклов скрининга и выделения клонов, которые вместе содержат непрерывную последовательность ДНК. Этот процесс обычно называют “прогулкой по хромосоме,” и он может использоваться для получения "контига” или "карты контига”.
[0191] Протоколы, достаточные в качестве руководства для специалиста по выделению клонов, связанных со сцепленными маркерами, можно найти, например, в Sambrook et al. (ed.) Molecular Cloning: A Laboratory Manual, 2nd ed., vol. 1-3, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1989; and Ausubel et al., Eds., Current Protocols in Molecular Biology, Chapter 2, Greene Publishing and Wiley-Interscience, NY, 1995.
Растения, содержащие маркеры резистентности к черной ножке
[0192] Некоторые варианты осуществления включают способы получения растения канолы, и дополнительно включают собственно эти растения канолы. В конкретных вариантах осуществления, такой способ может включать скрещивание первого родительского растения канолы, содержащего по меньшей мере один маркерный аллель, который положительно коррелирует с резистентностью к черной ножке, со вторым растением канолы в качестве маркера, сцепленного с резистентностью к черной ножке, как описано в настоящем документе, и выращивание женской особи растения канолы в условиях роста растений с получением потомства растения канолы. Такое потомство канолы может быть проанализировано на маркерные аллели, сцепленные с резистентностью к черной ножке, и желаемое потомство может быть отобрано. Такие растения потомства или их семена могут иметь различное применение, в том числе, например, но ими не ограничиваясь, они могут быть проданы на коммерческой основе для выращивания канолы; использованы в пищу; обработаны с получением желаемого продукта канолы (например, масла канолы или муки из канолы); и/или дополнительно использованы в последующих раундах селекции. Растения канолы в соответствии с некоторыми вариантами осуществления включают потомство растений, которое содержат по меньшей мере одну из аллельных маркерных форм, описанных в настоящем документе, таким образом, что потомство в дальнейшем способно наследовать маркерный аллель.
[0193] Некоторые варианты осуществления включают способы получения растений канолы, обладающих резистентностью к черной ножке. В конкретных вариантах осуществления такие способы могут включать получение такого растения с помощью обычной селекции растений или путем введения экзогенной ДНК (например, трансгена) в сорта канолы или растения канолы.
[0194] Таким образом, некоторые варианты осуществления включают клетки-хозяева и организмы, которые трансформированы нуклеиновыми кислотами, соответствующими QTL резистентности к черной ножке, идентифицированной с использованием по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке, как описано в настоящем документе. В некоторых примерах, такие нуклеиновые кислоты могут включать интервалы хромосом (например, геномные фрагменты), ORF и/или кДНК, которые кодируют продукты экспрессии, которые придают фенотип резистентности к черной ножке.
[0195] Клетки-хозяева могут быть получены методами генной инженерии (например, трансдукцией, трансфекцией, трансформацией и тому подобное) с помощью вектора (например, клонирующего вектора, челночного вектора или вектора экспрессии,), который содержит ORF, сцепленную с маркером резистентности к черной ножке. Векторы включают, например, но ими не ограничиваются, плазмиды; фагмиды; Agrobacterium; вирусы; "голые" полинуклеотиды (линейные или круговые); и конъюгированные полинуклеотиды. Многие векторы могут быть введены в бактерии, особенно с целью размножения и увеличения их количества.
[0196] Векторы могут быть введены в ткани растения, культивируемые клетки растений и в растительные протопласты любым из множества стандартных способов, известных в данной области, включая, например, но ими не ограничиваясь: электропорацию (From et al. (1985) Proc. Natl. Acad. Sci. USA 82:5824); инфекцию с помощью вирусных векторов, таких как вирус мозаики цветной капусты (CaMV) (см., например, патент США 4407956); баллистическую пенетрацию мелкими частицами, содержащими нуклеиновую кислоту (Klein et al. (1987) Nature 327:70); применение пыльцы в качестве вектора (международная патентная публикация PCT No. WO 85/01856); и применение Agrobacterium tumefaciens или A. rhizogenes, несущих плазмиду T-DNA, в которой клонированы фрагменты ДНК (Fraley et al. (1983) Proc. Natl. Acad. Sci. USA 80:4803). Любой подходящий способ, в том числе, но ими не ограничиваясь, конкретные способы, использованные в настоящем описании, который обеспечивает эффективное введение нуклеиновой кислоты в клетку или протопласт, может быть использован в определенных вариантах осуществления настоящего изобретения.
[0197] Сконструированные клетки-хозяева могут культивированы в обычные питательные среды или среды, модифицированные, например, для активации промоторов или селекции трансформантов. В некоторых вариантах осуществления растительные клетки-хозяева могут культивированы в трансгенные растения. Регенерация растений из культивированных протопластов описана, например, Evans et al. (1983) “Protoplast Isolation and Culture,” In Handbook of Plant Cell Cultures 1, MacMillan Publishing Co., NY, pp. 124-176; Davey (1983) “Recent Developments in the Culture and Regeneration of Plant Protoplasts,” In Protoplasts, Birkhauser, Basel, pp. 12-29; Dale (1983) “Protoplast Culture and Plant Regeneration of Cereals and Other Recalcitrant Crops,” In Protoplasts, выше, pp. 31-41; и Binding (1985) “Regeneration of Plants,” In Plant Protoplasts, CRC Press, Boca Raton, FL, pp. 21-73. Дополнительные источники, в которых представлено подробное описание, касающиеся культуры клеток растений и регенерации, включают Payne et al. (1992) Plant Cell and Tissue Culture in Liquid Systems, John Wiley & Sons, Inc., NY; Gamborg and Phillips (eds.) (1995) Plant Cell, Tissue and Organ Culture; Fundamental Methods, Springer Lab Manual, Springer-Verlag (Berlin Heidelberg NY); и R. R. D. Croy (Ed.) Plant Molecular Biology (1993) Bios Scientific Publishers, Oxford, UK (ISBN 0 12 198370 6).
[0198] Трансформированные растительные клетки, которые получены с использованием любого из указанных выше способов трансформации, могут быть культивированы с целью регенерации всего растения, которое обладает трансформированным генотипом и, таким образом, желаемым фенотипом. Такие методики регенерации обычно основаны на манипуляции с определенными фитогормонами в питательной среде для культуры ткани, как правило, используя биоцидные и/или гербицидные маркеры, которые введены в клетку вместе с желаемыми нуклеотидными последовательностями. Процессы регенерации и роста, используемые для производства всего растения, как правило, включают стадии селекции трансформированных клеток и побегов; укоренения побегов трансформанта; и рост всходов в почве.
[0199] Трансформация растений с помощью нуклеиновых кислот, которые обеспечивают устойчивость к черной ножке (например, которые включают маркеры, описанные в настоящем описании) могут быть использованы для трансформации видов, отличных от канолы. Например, предполагается, что продукты экспрессии QTL, которые способствуют или обеспечивают фенотип резистентности к черной ножке у канолы, могут также придавать резистентность к черной ножке при трансформировании и экспрессии в других агрономически важных видах растений и в растениях, важных с точки зрения садоводства. Такие виды включают двудольные растения, например, но ими не ограничиваясь, родов: Sinapis, Raphanus, Descurainia, Sisymbrium и Thlaspi, а также другие представители семейства Brassicaceae, включая масличный рапс (канола), брюкву (репа), семена репы масличной, турнепс, капусту, кресс-салат, редис, хрен, левкой, китайскую капусту, брокколи, и овощи, и семена сельскохозяйственных культур капусты. Обычные культурные растения, которые могут быть использованы в конкретных примерах, включают, например, но ими не ограничиваются: канолу, брокколи, брюссельскую капусту, капусту, цветную капусту, кормовую капусту и кольраби.
Системы детекции и/или маркеры, коррелирующие с резистентностью к черной ножке
[0200] Системы, включая автоматизированные системы, идентификации растений, которые содержат по меньшей мере один маркер, сцепленный с фенотипом резистентности к черной ножке в каноле, и/или для корреляции наличия специфических сцепленных маркерных аллелей с резистентностью к черной ножке, также включены в некоторые варианты осуществления. Характерные системы могут включать зонды, которые можно использовать для детекции аллелей в маркерном локусе, как описано в настоящем описании; детектор для детекции меток в зондах; соответствующие элементы для манипуляции с жидкостями и терморегуляторы, например, такие, которые смешивают зонды и матрицы и/или матрицы амплификации; и/или инструкции к системе, которые устанавливают связь между детекцией зондов и присутствием конкретного маркерного локуса или аллеля.
[0201] В конкретных вариантах осуществления изобретение относится к системе для идентификации растения канолы, в котором прогнозируется наличие резистентности к черной ножке. Такая система может включать, например, но ими не ограничиваясь: набор маркерных праймеров и/или зондов, сконфигурированных для детекции по меньшей мере одного аллеля по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке (например, DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787, DBSNP01590, и маркер, сцепленный по меньшей мере с одним из указанных выше); детектор, который сконфигурирован для возможности обнаружения одного или более выходных сигналов набора маркерных зондов или праймеров, или их ампликона, тем самым идентифицируя наличие или отсутствие аллеля; и инструкций к системе, которые устанавливают связь между наличием или отсутствием аллеля резистентности к черной ножке.
[0202] Система, которая осуществляет обнаружение маркера и/или устанавливает корреляцию, может включать детектор, который сконфигурирован для обнаружения одного или нескольких выходных сигналов набора маркерных зондов или праймеров, или их ампликона. Точная конфигурация детектора может зависеть от типа используемой метки для детекции маркерного аллеля. Конкретные примеры могут включать световые детекторы и/или детекторы радиоактивности. Например, детекция светового излучения или другого свойства меченого зонда может свидетельствовать о наличии или отсутствии маркерного аллеля, взаимодействующих с зондом (например, посредством специфической гибридизации). Детектор(ы) необязательно контролирует один или множество сигналов реакции амплификации. Например, детектор может контролировать оптические сигналы, которые соответствуют результатам анализа амплификации в "реальном времени".
[0203] Доступно широкое разнообразие устройств для детекции сигнала, включая, например, но ими не ограничиваясь, фотоумножители; спектрофотометры; CCD-матрицы; матрицы и сканеры матриц; сканирующие детекторы; фотоумножители и фотодиоды; станции с микроскопами; гальво-сканеры; и микрофлюидальные приборы для детекции амплификации нуклеиновых кислот. Кроме типа метки, используемой для детекции маркерного аллеля, точная конфигурация детектора может зависеть, частично, от измерительных приборов, которые наиболее удобно получить пользователю. В некоторых примерах могут быть использованы детекторы, которые обнаруживают флуоресценцию, фосфоресценцию, радиоактивность, рН, заряд, оптическую плотность, люминесценцию, температуру или магнетизм.
[0204] Конкретная форма инструкций, приложенных к системе в соответствии с некоторыми вариантами осуществления, может аналогичным образом изменяться в зависимости от компонентов системы. Например, инструкции могут быть представлены в виде системного программного обеспечения в одной или более интегрированных единицах системы, или они могут присутствовать на одном или нескольких компьютерах или машиночитаемых носителей, функционально связанных с детектором. В некоторых примерах инструкции к системе включают по меньшей мере одну справочную таблицу, которая включает установление связи между наличием или отсутствием конкретного маркерного аллеля в растении или генетическом материале, и наличие или отсутствие резистентности к черной ножке. Инструкции также могут включать указания по установке пользовательского интерфейса в системе; например, позволяющие пользователю просматривать результаты анализа образца и входные параметры системы.
[0205] Система может включать, в конкретных вариантах осуществления компоненты для хранения или передачи машиночитаемых данных, представляющие или характеризующие детектированные маркерные аллели, например, в автоматизированной (например, полностью автоматизированной) системе. Например, может быть предусмотрено, что машиночитаемый носитель включает кэш-память, оперативную память и запоминающее устройство и/или другие компоненты для электронного хранения данных (например, жесткие диски, флоппи-диски и накопители) для хранения компьютерного кода. Данные об аллелях, детектированные способом по настоящему изобретению, также могут передаваться в электронном, оптическом или магнитном виде в сигналы компьютерных данных, осуществляемых в передающую среду по сети, такой как интранет или интернет, или их комбинации. Система также, или альтернативно, может передавать данные с помощью беспроводной, инфракрасной или других доступных альтернативных вариантов передачи.
[0206] Во время работы система обычно содержит образец, который анализируют, такой как ткань растения или материал, выделенный из ткани, такой как геномная ДНК, амплифицированная геномная ДНК, кДНК, амплифицированная кДНК, РНК, амплифицированная РНК или тому подобное.
[0207] В некоторых вариантах осуществления система может состоять из отдельных элементов, или, в качестве альтернативы, может быть интегрирована в единый блок для удобного обнаружения маркерных аллелей, и, необязательно, дополнительно для установления связи маркер-фенотип. В конкретных вариантах осуществления система может также включать образец, например, но им не ограничиваясь, геномную ДНК; амплифицированную геномную ДНК; кДНК; амплифицированную кДНК; РНК; и амплифицированную РНК, из канолы или из выбранной растительной ткани канолы.
[0208] Автоматизированные системы, предусмотренные в некоторых вариантах осуществления, необязательно включают компоненты для манипуляции с образцом; например, роботизированные устройства. Например, автоматизированная система может включать роботизированную основу для контроля за жидкостью для передачи растворов (например, экстрактов растительных клеток) от источника к пункту назначения (например, от микротитровального планшета к матричному субстрату), который может быть функционально связан с цифровым компьютером (например, в интегрированной компьютерной системе). Устройство ввода для ввода данных в цифровой компьютер для контроля за высокоэффективным переносом жидкости с помощью роботизированной основы для контроля за жидкостью (и, при необходимости, для контроля за переносом этим основанием к твердому носителю) также может быть признаком автоматизированной системы. Многие автоматизированные роботизированные системы манипулирования с жидкостями являются коммерчески доступными. Например, множество автоматизированных систем, которых используют различные системы Zymate™, и, как правило, включают роботы и модули для манипуляции с жидкостями, доступны от Caliper Technologies Corp. (Hopkinton, MA). Аналогично, обычный робот ORCA®, который используют в различных лабораторных системах (например, для манипуляций с микротрировальным планшетом) также коммерчески доступны от, например, Beckman Coulter, Inc. (Fullerton, CA). В качестве альтернативы обычной робототехнике, в настоящее время широко доступны микрофлюидальные системы для осуществления манипуляций с жидкостями и детекции от Caliper Technologies and Agilent technologies (Palo Alto, CA).
[0209] В конкретных вариантах осуществления система для анализа молекулярных маркеров может включать, например, но ими не ограничиваясь, цифровой компьютер, содержащий высокоэффективное программное обеспечение для контроля за жидкостью; цифровой компьютер, содержащий программное обеспечение для анализа изображения для анализа данных от маркерных меток; цифровой компьютер, содержащий программное обеспечение для интерпретации данных; роботизированную основу для контроля за жидкостью для передачи растворов от источника к месту назначения; устройство ввода (например, компьютерная клавиатура) для ввода данных в систему (например, для контроля за высокоэффективным переносом жидкости роботизированной основой для контроля за жидкостью); и сканер изображений для оцифровки сигналов меток меченых зондов.
[0210] Оптические изображения (например, характеристики гибридизация), просматриваемые и/или записываемые с помощью камеры или другого устройства (например, фотодиод и устройство для хранения данных), могут быть дополнительно обработаны в любом из вариантов осуществления настоящего изобретения. Например, но ими не ограничиваясь, такие изображения могут быть обработаны с помощью цифрового преобразования изображения и/или хранения и анализа изображения на компьютере. Разнообразное коммерчески доступное периферийные оборудование и программное обеспечение доступно для оцифровки, хранения и анализа, оцифрованного видео или оцифрованного оптического изображения, например, используя различные компьютерные платформы и платформы для программирования.
[0211] Некоторые варианты осуществления также включают наборы, которые могут быть использованы, для идентификации растений, которые содержат по меньшей мере один маркер, сцепленный с резистентностью к черной ножке у канолы, и/или для установления связи между наличием специфического сцепленного маркерного аллеля и резистентностью к черной ножке. В некоторых примерах, такой набор может включать соответствующие праймеры или зонды для детекции по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке, и конкретных маркерных аллелей; и инструкции для использования праймеров или зондов для детекции по меньшей мере одного маркера и установления связи между маркерным аллелем и резистентностью к черной ножке. Набор может в некоторых примерах включать упаковочные материалы для упаковки зондов, праймеров и/или инструкций; и контроли (например, контрольные реакции амплификации, которые включают зонды, праймеры или матричные нуклеиновые кислоты для амплификации и маркеры молекулярного размера).
[0212] В некоторых вариантах осуществления набор или системы для идентификации растений, которые содержат по меньшей мере один маркер, сцепленный с резистентностью к черной ножке у канолы, и/или для установления связи между специфическим сцепленным маркерным аллелем и резистентностью к черной ножке может включать нуклеиновые кислоты, которые детектируют конкретные маркеры QTL, описанные в настоящем документе. Например, система или набор могут включать пару праймеров амплификации, способную инициировать полимеризацию ДНК с помощью ДНК-полимеразы на матрице нуклеиновой кислоты канолы с получением маркерного ампликона канолы, где маркерный ампликон соответствует маркеру канолы, выбранному из DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704, DBSNP05705 DBSNP28066, DBSNP27644, DBSNP28099, DBSNP33158, DBSNP14607, DBSNP04906, DBSNP07219, DBSNP08872, DBSNP08485, DBSNP00547, DBSNP08169, DBSNP00787, DBSNP01590, и маркера, сцепленного по меньшей мере с одним из указанных выше. Например, пара праймеров, которые специфичны для маркера, могут быть выбраны из пары праймеров, приведенных в Таблице 3, или их эквивалентов.
ПРИМЕРЫ
[0213] Следующие примеры приведены для иллюстрации, а не для ограничения, некоторых вариантов осуществления изобретения. Понятно, что примеры и варианты осуществления, описанные в настоящем документе, даны только в иллюстративных целях, и специалистам в данной области будет понятно, что различные реагенты, методики, системы и параметры могут быть изменены без отхода от сущности или объема изобретения.
Пример 1: Экстракция растительного материала и ДНК
[0214] Популяцию Nex845CL/NT152346//Nex845CL, состоящую из 731 потомков BC1F1, использовали для точного картирования Rlm4. Линия весенней канолы Nex845CL, Dow AgroSciences (DAS), с набором характеристик масла омега-9, является рекуррентным родителем. Линия весенней канолы NT152346 является растением-донором Rlm4.
[0215] ДНК экстрагировали, используя способ экстракции ДНК MagAttract (Qiagen, Valencia, CA) и Biocel 1800 (Agilent Technologies, Santa Clara, CA). ДНК количественно определяли, используя спектрофотометр NanoDrop 8000 (Thermo Scientific, Rockford, IL), в соответствии с инструкциями изготовителя.
Пример 2: Фенотипирование картированной популяции
Выращивание растения
[0216] Семена высевали в почву в 36-луночные поддоны со вставками в камерах роста. Одно растение инокулировали на 4 долях семядоли. Поддерживали нормальные условия для роста растений (16:8 фотопериод свет/темнота, 19°C в дневное время и 16°C в ночное время). Полив и внесение удобрений применяли в обычном режиме. Инокуляция
[0217] Инокулирование проводили на 10 день после посадки, когда семядоли были полностью раскрыты. Каждая доля семядоли была повреждена при помощи пинцета для тканей зубов. Десять микролитров (мкл) суспензии пикнидиоспор Leptosphaeria maculans (L. maculans) в H2O при концентрации 2×107 спор / мл, используя пипетку с повтором, капали на каждую долю. На две семядоли одного растения капали четыре капли. В случае, когда единичное растение использовали из сегрегирующей популяции, например, популяции F2 возвратного скрещивания, 4 рейтинга одного растения обрабатывали как повторы. Инокулированные семядоли поддерживали при комнатной температуре в течение 6-8 часов перед тем как возвращали обратно в камеры роста.
Баллы
[0218] Симптомы заболевания регистрировали на 11 и 12 день после инокуляции (д.п.и.). Симптомы начали появляться на 7 д.п.и. в виде ограниченной некротической ткани вокруг повреждения в случае фенотипов с резистентностью и в виде слабого побеления вокруг раны в случае фенотипов с чувствительностью. Оценку по шкале 0-9 баллов присваивали каждому сайту инокуляции в момент оценки в зависимости от размера поражения, количества темных краев вокруг раны, а также наличия тканевого коллапса и споруляции (таблица 1).
Оценка болезни и описание L. maculans.
Расчет индекса заболевания и классификация
[00219] Индекс заболевания (DI) рассчитывали на основании формулы:
[00220]
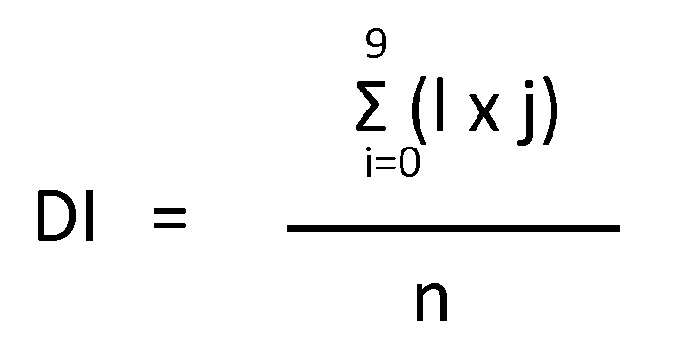
[0221] где n=общее число растений, i=баллы оценки и j=число растений/баллы. Растения с DI<3 классифицировали как резистентные; растения с DI=3-5 классифицировали как растения с промежуточной резистентностью; растения с DI>5 оценивали как чувствительные.
Пример 3: Система генотипирования на основе специфического ПЦР в отношении конкуррентного аллеля KBioscience (KASPar™)
[0222] Система генотипирования KASPar™ состоит из двух компонентов (1) специфического анализа SNP-(комбинации трех немеченных праймеров), и (2) универсальной реакционной смеси в 2х концентрации и содержащей фермент полимеразу Taq, пассивный эталонный краситель, ROX, 50 мМ MgCl2 и DMSO. Три праймера: аллель-специфический праймер 1 (A1), аллель-специфический праймер 2 (A2) и обычный (C1) или обратный праймер, были разработаны, используя алгоритм анализа диспетчера потока операций, Kraken (KBiosciences, Hoddesdon, Hertfordshire, UK).
[0223] Готовили аналитическую смесь 3 праймеров, состоящую из 12 микромолей (мкМ) каждого A1 и A2 и 30 мкМ C1. Универсальную реакционную смесь разбавляли до 1x и добавляли дополнительное количество MgCl2, так, чтобы конечная концентрация MgCl2 реакционной смеси при концентрации 1х равнялась 1,8 миллимолям (мМ). ДНК распределяют в 384-луночные планшеты для ПЦР при концентрации 1-5 нг/мкл на лунку и сушат в планшетах в печи при 65°C в течение 1 часа и 15 минут. Смесь для анализа и универсальную реакционную смесь объединяют в пропорции 1:54 и распределяют 4 мкл в планшеты ДНК, используя робот для манипуляций с жидкостью, так, чтобы конечное количество смеси для анализа в планшете было 0,07 мкл и конечное количество разбавленной реакционной смеси было 3,93 мкл. Аппараты GeneAmp PCR System 9700 (Applied Biosystems, Foster City, CA) использовали для термоцикла со следующими условиями: 94°С в течение 15 минут, 20 циклов 94°С в течение 10 секунд, 57°С в течение 5 секунд, 72°С в течение 10 секунд; 22 цикла 94°C в течение 10 секунд, 57°С в течение 20 секунд, 72°C в течение 40 секунд. После завершения термоцикла аллель-специфическую интенсивность флуоресценции считывали с использованием спектрофлуорометра PHERAStar® (BMG LabTech, Cary, NC) при комнатной температуре и данные загружали в систему Kraken для анализа.
[0224] KASPar™ использует флуорофоры FAM и VIC для разделения генотипов. Пассивный эталонный краситель ROX также используют для обеспечения нормализации вариаций сигнала, вызванных различиями в объеме жидкости в лунках. В Kraken данные FAM и VIC наносили на оси x- и y-, соответственно. Генотип может быть определен в соответствии с образцом кластеров (Фигура 1).
[0225] Маркеры SNP и консенсусную карту SNP с высокой плотностью, разработанная DAS, использовали, отбирая 66 маркеров SNP, которые были картированы на LG N7 на консенсусной карте, и которые были полиморфными среди картированных родителей, Nex845CL и NT152346. Тридцать семь из 66 выбранных маркеров SNP были успешно преобразованы в анализах KASPar™ и генотипированы на картированную популяцию BC1F1. В таблице 2 приведен список маркеров 37 SNP, которые были преобразованы в анализах KASPar™ и были использованы для точного картирования Rlm4. В таблице 3 приведены последовательности праймеров KASPar™ для восьми маркеров, которые наиболее тесно сцеплены с Rlm4.
[0226]
Последовательности 37 маркеров SNP, использованных для точного картирования Rlm4. ГР: Генетическое расстояние; Nex845CL: Rlm4 родитель с чувствительностью; NT152346: Rlm4 родитель с резистентностью.
[0227]
Последовательности праймеров для анализов KASPar™, разработанные для восьми маркеров SNP (DBSNP30220, DBSNP31606, DBSNP10503, DBSNP10504, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705) с 1,0 cM Rlm4.
Пример 4: Анализ сцепления и построение карты
[0228] MAPMAKER/EXP 3.0 (Lander et al. 1987) использовали для построения группы сцепления (LG) для подтверждения того, маркеры были картированы вместе с фенотипическим признаком на LG N7. Для MAPMAKER/EXP 3.0 требуется только один входной файл от каждой популяции, названный как факт локусного генотипа. В файле локусного генотипа популяции F2 аллели элитного родителя названы “A”, аллели донороного родителя названы “B”, а гетерозиготные аллели названы “H”. Отсутствующие данные были представлены как дефис (-) в файле локусного генотипа. Была построена карта генетического сцепления LG N7 и 37 маркеров SNP. Поскольку Rlm4 является качественным одиночным признаком, специфичным для расы, то фенотипические данные классифицировали как R (DI≤5) и S (DI>5), и картировали как маркер на карте сцепления LG7. Было идентифицировано восемь маркеров SNP, DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220, DBSNP01654, DBSNP01910, DBSNP05704 и DBSNP05705, в пределах 1,0 сМ Rlm4 резистентности к черной ножке (Фигура 2). Маркеры SNP DBSNP10503, DBSNP10504, DBSNP31606, DBSNP30220 и DBSNP01654 точно сегрегируют вместе с Rlm4 (Фигура 2). Восемь высокоэффективных маркеров SNP, идентифицированных и разработанных в DAS, значительно упрощая селекцию при помощи маркеров Rlm4 в программе селекции DAS.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Dow AgroSciences LLC
Tang, Shunxue
Zhao, Jianwei
<120> Молекулярные маркеры гена резистентности к черной ножке RLM4
в BRASSICA NAPUS и способы их применения
<130> 72075-US-NP
<160> 61
<210> 1
<211> 530
<212> ДНК
<213> Brassica napus
<400> 1
ctgaccccac cgaatgaaaa ccaggatcag ccgagtccag tttcggtttt acagcatcct 60
ttggaagagg aatatgtggg aaatccagaa tgttctgggt caaccaagcc atggaccagc 120
caaggaggaa aagaaatgtc tctgaaatgt agtcttatag acaaatcacc tccaatagga 180
tcaatcgctc gtgttttctc ttgggaagat gagtcataca cagacatrac taaacccgga 240
aatggaataa aggaagatga agactggtat tacttcatca aaacgctttt aaaaacatcc 300
ggtttcagcg gcagtgatcc gcttatgacc cggtggcact caccagatag ccctttggaa 360
ccatcattaa gagacaggtt tgccaacaag gaacccatca aacgcaggaa ccagcgatca 420
aaccgcaagc ttgtgtttga ctgcgtcaat gccatcataa cagagacaac atcaacagct 480
gcacgcactg gcttgacctc tggattcgac aatgtggaac atgtttggac 530
<210> 2
<211> 601
<212> ДНК
<213> Brassica napus
<400> 2
tttttttttt ttttttcccg aaaatagtaa tttccattaa ccaaaaagag gcaataccat 60
ccttaagatt ccaacaaact caataatttc tcaaaccttg ttactaactt actacattgc 120
aatattacac gataacgaag agaagaaggc attgtcagtt aactgaagaa gaagaggagc 180
tcaactcaac atrtacttag caaaagcttt ggatataatc gaaaagccca tgtactaatc 240
cttgagagtt tttcttgaga gaaaagtcta tcactttccc gtgttcatca tcagcgttat 300
gttgtctcct ttaagaagaa tccttccaag ttgttttctg gtgttcttct tgatgctcac 360
ttcttcagct tcatccaaca ctaggttcat gtattcgtca aacccagtga ttcttccttc 420
aatcctcaaa tctttctgct caaaaagcca aatctggatc ctagctttgc tttgaagaaa 480
cctaaaaatc aagttgatag gttgagtcat aatcctttga actttggtgc tcgccattgc 540
tcccgacgtt tcaattctct ctctctctct ctcaagcttc ggtggacaag aagacggacg 600
c 601
<210> 3
<211> 1698
<212> ДНК
<213> Brassica napus
<400> 3
agatagaggg agagaggttc gttttgcatc cctgagatcg gcggcggagt tataacgacg 60
gtggaatcat gcaaacgtag tcaccaacga aaccgtcttt cttcccggag acaaagaggc 120
agcgatgatt gtctctgttt cttcgttttt tctctccgat ctggcctctc tgatccggtg 180
aagaagaaga acaataagct ccccgccgat cgaatcttgc agctccttct tttctcagaa 240
gagatttaat ttcaatcatt tcgtcgtttg atccatcgat ttcatcgagt ttttggcatg 300
ctgtaaaaag agagatgtct tttaaaagca tccttcgtga tctgaaggaa gtgagggatg 360
gacttggagg gatctctaag cgaggctggt caaagtcttc ccacattgct cctgatcata 420
cartgacacc atcggagaac atcccgcaga gcccctgggc ttctttgccg cctgagttgc 480
tccatgagat catcaggagg gttgaagaga gcgagaccgc ttggcccgcc cgagcggctg 540
ttgtctcttg tgcttctgtc tgtaaatcgt ggagaggaat cacaatggag actgtgagag 600
tccctgagca gtgtgggaaa ctcactttcc ctatctcatt gaaacagccg ggtccgcgag 660
accttcctat tcaatgcttt attaaaagga acagagcaac agctacatat attctctact 720
atggcttgat gccttctgag atggagaatg acaagctatt gttagcagca agaagagtta 780
gaagagcgac atgcactgac tttgtaatct cactttctgc caaaaacttc tcaaggagaa 840
gcagcactta tgttggcaag ctaaggtctg gttttctagg aaccaagttc acaatatatg 900
acaaccaaac aggatcatcc actgcacaag cccaacctaa gcaagcatct cctaagttac 960
ctgctactag ctatacctca ggaagcataa cctacgagct caatgttctt cgcacaaggg 1020
ggcctagaag gatgcattgc gttatggact ctataccact ctcttctgtt atctctgagc 1080
catcagtagt taaagaagaa gtatcttcag gcgaaaccag ctcaacagac aaagagaata 1140
tctctccaag cttgttggat cagccgctgg ttctcaaaaa caagtccccg agatggcacg 1200
agcagttgca gtgctggtgc ctcaacttca aggggagagt gaccgttgct tcggtcaaga 1260
acttccagct tgtggcagac attgaccctt ctttagatgc gctgcctgaa gagcatgaga 1320
gagtgatctt acagtttggc aaaatcggca aggacatttt caccatggat tatcgttatc 1380
ctctctctgc ttttcaagct tttgctatat gcatcagcag ctttgacacc aaaccggctt 1440
gcgaaggtta aagttaactg ctgagtgaaa atccaattat ctctgcaaat catccatcat 1500
agaaggcctt tctgatcttc tctgagccat gaaaatgctt tttgtattat gctttaaaat 1560
tgcctatcct aattcactgt gacgaaaact tgatggggct cttcggtgga taagctttac 1620
tgaatgtaat gattgttgta tccaacgtaa tatattataa acagaaagct ttacatataa 1680
tctttcacaa ttaaactg 1698
<210> 4
<211> 676
<212> ДНК
<213> Brassica napus
<400> 4
cgattccgtg gatcgactct atgtagagga atttgcaata cttttaggga atcacttttc 60
ctccttttat ccacaggttt acgctgctat tgttaatatc attgagaagc cgtgggagcg 120
tgtatgcatt gatggaaagc cccatttaca tggtttcaag cttgggtcag agaaccatac 180
agtggaggct acagtacaaa agtctggtgc actaacctta acttctggtg ttgcaggact 240
agctttgctc aagacagccc aggtacacag crttttgttt ttctttttga attttctgtc 300
tgtggcgagt taaatgtcgt ttggctagtg tttcctgcga tgcatagagt tattggtttg 360
caaaatgaat ttttttttgg tgtttttctt cttttattgg cggtcttctc tcgtttagtc 420
ccttgccgag tctactctac aagaaaggga aacacaatgt agatcgttat acatgtcctt 480
gtttgtctat agctgttaga gatacaagtc tgctcctcta acttatgaag ttgagggaga 540
ggaaaagcaa agaagaaatg atatgaaata cgagattcga tgttagttta tcagtctgtt 600
ttagaaagat actgataact cattactcac aaaatgccac tttaatgaaa atgaacagtc 660
aggatttgag aagttt 676
<210> 5
<211> 681
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (403)..(403)
<223> n равно a, c, g или t
<400> 5
aagatcatgt cggatctgaa gacggtgagg gataagatca cgcaatcgat gttcagggaa 60
cacgcgctcg aggacgaaga agaagagggg aagaccgacg agtccggccg cgagaaggtt 120
tctccggcga agccgtggaa tctgaggaaa aggagggccg cgtgcaagga gcctgtttcg 180
gagaggatag ygaatccttc gccgccgagg gtgaaggaga gaggcggagt ggtggaagcc 240
gagacggcgg cgaaggagat gatgatgcgg cggggcaggt tctccgtgaa gctgtcgaag 300
aaggagatcg aggaggattt cgtggcggct cttgggcacc gaccaccgcg ccggccaaag 360
aagcggccga ggaccgttca gaagaagctc gacagcttgc atncctgggt tttatctgag 420
tgaagtgacg cttgatgcct ataaggtccc tgaagaaacc aagaatatac agcgatgatg 480
atgtaaaaga ttatgtaatt cagttgccgt gaagtttcaa agagttgtca atcttttgaa 540
attgggtcaa gtcttgaagc tcccttccct gtcctgttct gctaatttct cgttttgtta 600
atgatttttt ttcttcccaa cttgttctga ataaacggta caaaaagctt tggagaatgt 660
gtaggttccg gttgattaat a 681
<210> 6
<211> 755
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (15)..(15)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (167)..(167)
<223> n равно a, c, g или t
<400> 6
gtcgacgatt tcgtngcaaa acatccgtca gaaaaaatgg tacaggctac gatgcagtct 60
tatatggtag gggagattga gacaagtatt gagatcaamg cttccgccca gaagtactac 120
cacatgctca ccggaaaacc aaaagatctt ccggaagcca gtcctgnaca acctccagag 180
atgtgatgtg ctcgagggag agcctggaaa agttggccgt attctctcct ggaactatgt 240
aactaatgga cagccaaagg tgatgaagga gaggatcgag gcactggagc cggagaagaa 300
tctgatcgtg gctagggtta ttgggggaga tctaatgaaa gagttcaaga acttcttcct 360
cactattcag gcgaccccga aacaaagagg acccggaagt gttgttaagt gtcacctgaa 420
gtatgagagg attgacaaga aggtggctga cccagaggac atactcgtgt tgtttgtcaa 480
tgcatccaga gacatggaca aaatgctttc ttctgaattc tagaggattc ttcggaaatc 540
ttttcatcca tctatcgatc tatcatctat ccgtccgact atgtgtctat ctatctatat 600
attgtgtctg ggtgtttagt atatatttct caataatgtg atctctctat aactgaagag 660
atctaatata tgcaagtatg tcataatcat gcacgatgcc tagtgcgtac gtttgaagtt 720
gtaatttgta aagtgtcact tgtttcgttg ctatc 755
<210> 7
<211> 706
<212> ДНК
<213> Brassica napus
<400> 7
actatcagct acccaaatct aactctggac tgaacttgga ccagcacaac aactcaatcc 60
cgacaatgac cggctccatc ggtgcatgcg acgacaagaa caagactatc ttgccgcagc 120
aacaaccaag catgcctcgt gagcaagacc aatacatgcc aatcgcaaac gtgataagga 180
tcatgcgtaa aatcttaccg ccacacgcca aaatctctga cgacgcaaaa gaaacgattc 240
aagaatgcgt ctccgagtac atcagcttcg tgaccggtga agctaacgag cgttgccaac 300
gtgagcaacg taagacaata actgctgaag atatcctttg ggcaatgagc aaacttgggt 360
tygatgatta cgttggacca ctcaacgtgt tcattaaccg gtaccgtgag ttcgagaccg 420
atcgtgggtg ttcacttaga ggtgagtcat catttaaacc ggtctatgga ggaagtggta 480
tggggtttca cggcccacct ccaccgggtt cttatggtta tggtatgttg gatcagtcta 540
tggtcatggg tggtggtcgg tactaccata acggatcggg tccggatgga tcagtaggtg 600
gtggcggtgg atcttcctct tctatgaatg gaatgccggt ttatgaccag tatggtcagt 660
ataagtgaag atgaacattt ctttgatttt ctgcatgtct attcaa 706
<210> 8
<211> 1017
<212> ДНК
<213> Brassica napus
<400> 8
ggctttggag aagtttgtga aagaatcgag cattccgctt atcaccgtct ttgacaagga 60
tccaaacaac cacccttatg ttatcaaatt ctttgacagc cctaacacta aggcgatgtt 120
tttcattaac ttcaccggag aatcagctga gactcttaaa tcaaagtacc gtgaagttgc 180
tacatccaac aagggacagg gtcttagctt ccttctcggt gatgctgaga acagccaagg 240
cgcattccag tactttggac tcgaagagag ccaagttcct ctcatcatca tccaaactgc 300
tgacgacaag aagtacctga aaacaaatgt ggaggttgac cagattggtt catggatcaa 360
ggacttcaaa gatggaaaag tgtcccctca caaaaaatct caacctgtcc caaccgaaaa 420
caacgagcca gtgaaggttg ttgttggtga gagccttgac gacatggtct tcaactctgg 480
aaagaacgtg ttgcttgagt tctatgcacc atggtgtgga cactgccaaa agcttgttcc 540
aatyttggac gaagtggctg tgtcatacca aagcgatcca agtgttgtca tagctaagct 600
agatgcaact gcaaacgact tcccacgtga taccttcgat gtgaagggat tcccaaccat 660
ttacttcaga tcagcgagcg gaaacgttgt cctgtacgaa ggagacagga caaaggaaga 720
ctttataagc ttcattgaca agaacaagga cacagctgga gagcctaaga cggaggacaa 780
gacagctgag gccactaaag acgagctctg aaaaggagaa gcgtgttccg tctcttagct 840
gctagcctag tttggtagtt ttgaggaaaa tacacagaag accacacttc ttagcgaatg 900
tttctttgtc tattagtttt gtttttttat atgcagaatt ttctaaacaa taaccgaata 960
acgcctgcct cgtttcttgt tttttggtta attgttcgtt ttttgacctc tgtcctc 1017
<210> 9
<211> 931
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (342)..(342)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (846)..(846)
<223> n равно a, c, g или t
<400> 9
cgtcttctca gatttctcct ccgttctcct cgaaaaaagc tccaaacttc actcccacaa 60
accctactct tccgatgtcg atcttcgagt acaatggaag tgcggtcgta gctatggtgg 120
ggaagaactg cttcgcyatc gccagtgatc gaaggctcgg tgtgcagctt cagacaatcg 180
ccaccgattt ccagagaatc tccaagatcc acgatcgcgt cttcatcggc ctctctggtc 240
tcgccaccga tgtccaaaca ctgtaccagc gcttggtgtt tcgtcataag ctgtaccagc 300
ttcgtgaaga gagagacatg aagcctgaaa ctttcgctag tnctcgtctc tgccattctt 360
tacgagaaga gatttggtcc ttacttatgc caaccagtga ttgctggatt tgggagaaga 420
tgacaagcct ttcatctgca ccatggactc cattggcgcc aaggagttgg ctaaagattt 480
cgttgtatct ggaactgctt cagaatcact gtatggagcc tgcgaagcaa tgtacaagcc 540
tgatatggaa gcggaggagc tgtttgagac gatatcgcaa gcacttctct cgtcagttga 600
ccgtgattgt ctgagtggtt ggggaggcca tgtctacgtt gtaacaccga ctgagattaa 660
agaaaggatc ctaaagggaa ggatggatta atctaatcca tcaacatcat catctgcttc 720
tatccaagtt gttgtctttt ctctgttaac cggtttaaag tagaataccc atcacatccc 780
ggttaattca tagaaccatt ccttctctga gattgtggat gaagtttgat tttctcctct 840
ttggantatt ttattagagt taacgcctgt tctgtaaaaa aaaaaaaaaa aaaaaaaaaa 900
aaaaagcttg tcctcggccg cgaccacgct a 931
<210> 10
<211> 688
<212> ДНК
<213> Brassica napus
<400> 10
tctattttga tgtcttatcc aaatccaacc aatgagcatg cagatataaa ttgcaatact 60
acaactctat cttctatttg tgtctaccaa gtctttgttc cttgctttta cgcattcttg 120
gattagctat gacacattca ctctctcctc tcctaccggc tgctcacgcc ggtagcccct 180
cctctccatc accgaggtca caaccgcaga ctcctcccat catccttcaa gttccaccaa 240
ttaatcgcag ggatattgtg gtgggattag gcagtgcgct atggagttgg gacgccctga 300
acggtaagga cgaggcaatg gcggcagcaa gacggccgcc tccaccacca gcagttgaga 360
agaaagatcc gaatgtgagt ggagttcagg ctaaggtatt agcgagtaag aagcgtaaag 420
aggcaatgaa ggcctccatg gctaaactta gagagagagg caaatctgtt gttgatgaag 480
aaaaaccatc ttcatcttct ccttctgctm ctgttgttgt taaagatgag ccaactcctc 540
cttctgctgc tcccgttgtt gttgaagctg aacaaactcc ttcttctgct tctgatcaat 600
agtatccaag caaaatggtc taaacctttc tatttatctt ctctaccaaa ttttatccta 660
tcatgttaat tcacgctact gtacttta 688
<210> 11
<211> 1830
<212> ДНК
<213> Brassica napus
<400> 11
aagatacaat acagtctcac ctaaaaaccg tgaaggaggc aaaatccgaa tcaaaatcgt 60
ctttgcatca tccatggatc cttacagggt tcgtccttca agcgctcatg attcgccctt 120
cttcacgaca aactcgggtg ctcctgtctg gaacaacaac tcctctttga ctgctggaac 180
cagaggtccg attcttttgg aagactacca tctgcttgag aaactcgcca actttgacag 240
ggagcggatt cctgagaggg tggttcacgc cagaggtgcc agtgccaaag gtttctttga 300
agtcactcat gacatcacac accttacttg tgctgatttt ctccgaggac ctggcgtcca 360
gactcctgtc attgttcgtt tctctactgt gatccacgag cgtggcagcc ccgagactct 420
cagagatcct cgtggtttcg ctgtcaagtt ctacaccaga gaggggaact ttgatcttgt 480
cgggaacaac ttccctgtct tcttcatccg cgacgggatg aagttccctg acatggtcca 540
cgccctgaaa ccgaacccca aatcccacat tcaggagaat tggaggatac tggacttctt 600
ctcacaccac cctgagagtc ttcacatgtt ttcattcctc tttgacgatc tcggtatccc 660
acaggactac aggcacatgg atggctttgg tgtcaacacc tacatgctta tcaacaaagc 720
cgggaaagct cactacgtga agttccactg gaaaccctct tgtggggtta aatgcctcct 780
tgatgaggaa gctatcaagg tcggaggatc caatcacagc catgcaacca aggatctcta 840
tgactcaatc gctgctggga actatccaga gtggaatctc ttcgttcaag tgatggatcc 900
tgctcacgag gacaagtttg acttcgaccc tcttgatgtg acaaagatct ggcctgaaga 960
tatcttacct ctgcagcctg ttggacgctt ggtcttgaac aaaaacattc gacaacttct 1020
ttaatgagaa tgagcagatt gctttctgcc ctgctattgt ggttcctggc gtccattact 1080
cagacgataa gctactccag accaggatct tctcctacgc tgatagtcag agacaccgtc 1140
ttggaccaaa ctatctgcag ctgccggtca atgcccccaa atgtgctcac cataacaatc 1200
accatgaagg tttcatgaac ttcatgcaca gagatgagga ggtgaattac ttcccttcaa 1260
ggttgaaccc ggttcgccat gctgacaaat accctacaac tcctgttttc tgctctggaa 1320
atcgtgagaa gtgcatgatt gagaaggaga acaactttca gcagccaggg gaacgatacc 1380
gatcctggga cgcagacagg caagagcgtt tcgtgaagcg ttttgttgaa gcacttgcgg 1440
agcctcgcgt gacgcacgag atccgcagca tttggatatc ytactggact caggcagaca 1500
aatctctggg acagaaacta gcttcccgtc ttaacgtgag gccaaagtac tgaatggtat 1560
catctcaaaa gactaagtga acccaatatg tgctcttatt gcggtttggt ttctgtgtaa 1620
gctctttggt aatagttcat ttaaataaaa accgtgacaa tgaaatgttt aaagaagggt 1680
actactagtt catgttgtaa tctttctatt cacaatgtca acgaatattt tctaaggcaa 1740
ataattaaaa ttaaaaaaaa aaaaaaaggg cggccgctct agaggatcca agcttacgta 1800
cgcgtgcatg cgacgtcata gctcttctat 1830
<210> 12
<211> 1753
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (492)..(492)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (847)..(847)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (988)..(988)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (999)..(999)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1574)..(1574)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1674)..(1674)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1726)..(1726)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1738)..(1738)
<223> n равно a, c, g или t
<400> 12
ctggacgcct gtgggaccgg tccggaattt cgggtcgacg aagcgtccgc ccacgcgtcc 60
gcggacgcgt gggcggacgc gtgggcgcgt acaacgctcc gttttacacc acaaacggcg 120
gtgctccggt ctcgaacaac atctcctccc tcaccatcgg agaaagaggt ccggttcttc 180
ttgaggacta ccatctgatc gagaaggttg ctaacttcac cagggagagg attcctgaga 240
gagtggttca tgccagaggg atcagtgcta agggcttctt tgaggtcacc catgacatct 300
ccaacctcac ttgtgctgat ttcctcagag cccctggtgt tcagactccg gtcatcgtcc 360
gtttctccac tgtcgttcac gagcgtgcca gccctgaaac catgagagac atccgtggct 420
tcgccgtcaa gttttacacc agagagggga actttgatct cgttggaaac aacactccgg 480
tgttcttcat cncgcgacgg gattcagttc ccggatgttg tccacgccct gaagccgaac 540
ccgaaaacaa atatccagga gtactggagg atactggact acatgtccca cttaccagag 600
agtctcctca catggtgctg gatgtttgat gacgtcggta tcccacaaga ctacaggcac 660
atggaaggtt tcggcgtcca cacctacact ctagtctcca aatccggaaa ggttctcttc 720
gtgaagttcc actggaaacc aacttgtggg atcaagaatc tcactgatga agaggctaag 780
gtagttggag gagccaatca cagccacgcg actaaggatc ttcacgatgc tattgcgtct 840
ggtaacntac cctgagtgga agctttttat tcagacaatg gatcctgcgg atgaggataa 900
gtttgacttc gacccgcttg atgtgaccaa gatctggcct gaggatatct tgcctctgca 960
gcctgttggt cggttggttc tgaacagnga ctattgatna acttctttaa tgagactgag 1020
cagcttgctt tcaaccctgg tcttgtggtt cctgggatct actactcgga tgacaagctg 1080
cttcagtgta ggatctttgc gtatggtgac actcagagac atcgtcttgg accgaactat 1140
ctgcagcttc cggttaatgc tcccaaatgt gctcaccaca acaatcacca tgaagggttt 1200
atgaacttca tgcacagaga tgaggagatc aattactacc catcaaagtt tgatcctgtc 1260
cggtgcgcag agaaagttcc tatccctaac aaatcataca ctggaatcag aacaaagtgc 1320
atcatcaaga aggagaacaa cttcaagcag ccaggagaca gatacagatc atgggcacca 1380
gacaggcaag acaggtttgt gaagagatgg gttgagatac tgtcagagcc gcgtctgact 1440
catgagatcc gcagcatctg gatctcttac tggtctcagg ctgatcgatc actgggacag 1500
aaactagcaa gccgtctcaa cgtgaggcca agcatctagg gagaccartc tcttgtcatc 1560
ggaaaggtac aaantcatat gatactctcc atcatcactt aaaattcctc aagaagatta 1620
tgttgttgtt gttgctgttt cgtgtctgta taataaaaat gatgctttgt aatncctttt 1680
aaaccaaatg tttccttggt attttacttg tcaaactctg ggcttngtaa tcatctcntc 1740
agaaaaaaat cag 1753
<210> 13
<211> 1007
<212> ДНК
<213> Brassica napus
<400> 13
tattaaaaaa aactgcaatt gggaatatgt gtccctcttg tctcctccat tccattactt 60
caagagaaca catcgcgtgc cttccttcca tcaaccatgg cttactcagc tcccatatct 120
ccatccctat ccttcttcaa aaacccccaa ctcaccagac tctcgttccc tttctcccca 180
ctttactctc gccctagggt tcagaatcca agtctgctca cgaagaacaa gacgtggtcg 240
tcgtccgtgg tggtggcggc gacggcggcg gagaagcaga agaagagata ccctggagaa 300
tcgaaagggt ttgtggagga gatgaggttt gtggcgatga gacttcacac gaaggagcag 360
gccaaggaag gtgagaaaga gacaaaakct cccgaggaac gtcctgtcgc taaatgggaa 420
ccgactgttg aaggttactt gaggtttttg gtggatagca agttggttta cgacacgctt 480
gaagggatta ttcatcagtc cactttccca acttatgcgg aattcaagaa cactgggctg 540
gaaagggcag agaaattgga cactgatctg aagtggttca aagaacaagg ctacgagatt 600
ccagaaccaa ctgatactgg taaaaaatat tctcagtatt taaaggattt agcagacaag 660
gatcctccat cattcatttg tcacttctac aacatctact tcgcccacag cgctggtggt 720
cgaatgattg gaagaaaggt ggcggaaagg attctcgaca atagagaact cgagttttac 780
aaatgggacg gcgacctttc cggattgttg cagaacgtga gggaaaaact gaacaaagtt 840
gcagaggagt ggacgagaga agagaagaat cattgtttgg aagagaccga gaaatcgttc 900
aagtattctg gtgagatact tcgtctcata ttgtcctgat tcctctcttt atatacgcgc 960
ttgtggcaac taaaatccct gctcaagttt tttattcctg tattata 1007
<210> 14
<211> 1007
<212> ДНК
<213> Brassica napus
<400> 14
tattaaaaaa aactgcaatt gggaatatgt gtccctcttg tctcctccat tccattactt 60
caagagaaca catcgcgtgc cttccttcca tcaaccatgg cttactcagc tcccatatct 120
ccatccctat ccttcttcaa aaacccccaa ctcaccagac tctcgttccc tttctcccca 180
ctttactctc gccctagggt tcagaatcca agtctgctca cgaagaacaa gacgtggtcg 240
tcgtccgtgg tggtggcggc gacggcggcg gagaagcaga agaagagata ccctggagaa 300
tcgaaagggt ttgtggagga gatgaggttt gtggcgatga gacttcacac gaaggagcag 360
gccaaggaag gtgagaaaga gacaaaatct cccgaggaac gtcctgtcgc taaatgggaa 420
ccgactgttg aaggytactt gaggtttttg gtggatagca agttggttta cgacacgctt 480
gaagggatta ttcatcagtc cactttccca acttatgcgg aattcaagaa cactgggctg 540
gaaagggcag agaaattgga cactgatctg aagtggttca aagaacaagg ctacgagatt 600
ccagaaccaa ctgatactgg taaaaaatat tctcagtatt taaaggattt agcagacaag 660
gatcctccat cattcatttg tcacttctac aacatctact tcgcccacag cgctggtggt 720
cgaatgattg gaagaaaggt ggcggaaagg attctcgaca atagagaact cgagttttac 780
aaatgggacg gcgacctttc cggattgttg cagaacgtga gggaaaaact gaacaaagtt 840
gcagaggagt ggacgagaga agagaagaat cattgtttgg aagagaccga gaaatcgttc 900
aagtattctg gtgagatact tcgtctcata ttgtcctgat tcctctcttt atatacgcgc 960
ttgtggcaac taaaatccct gctcaagttt tttattcctg tattata 1007
<210> 15
<211> 637
<212> ДНК
<213> Brassica napus
<400> 15
ttcccgggtc gacggcagat ttaagctctt cgagagtcac gccatcttga tttatcttgc 60
ctcagcattc ccaagtgtag ctgatcattg gtaccccaat gatctttcca agagagccaa 120
gattcactct gtcttggatt ggcatcacac caatttacgc cctggtgcag ctggatatgt 180
aatgaatact gttctagctc catttcttgg ccgttctcta gattcraaag cagctgctga 240
agctgagaag atactaacca agtctctgtc cactctagag actttttggc ttaagggcaa 300
tgctaaattc ttgctgggaa gtagccaacc atccgtagct gatcttagcc ttgtatgcga 360
gattatgcaa ctccaggttt tggatgagaa agatcgtctt aggctgttta gtccttacaa 420
gaaagttgaa gaatggattg agaatacgag aaaggcgaca cagcctcact ttgatgtggt 480
tcataaaacc ctttacggag ccaaagacaa atttgacaag cagcggaaga tgtcgaacgc 540
gggtcttcaa tctaagatgt aagaaaccaa acttgtattg gtgacggttt ggccaaaacc 600
ggtttaacgg ttcactgtaa tacctcggta caacatt 637
<210> 16
<211> 779
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (14)..(15)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (22)..(24)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (34)..(34)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (43)..(43)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (54)..(54)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (93)..(93)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (103)..(103)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (120)..(120)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (128)..(128)
<223> n равно a, c, g или t
<400> 16
attatagctg gacnntacta tnnntttatg attngaaact tgnatgccag atancagaca 60
atttaagata aaggtaataa caataattca gcnagaaatc canaaaaaaa aagcagagtn 120
aaaacganag agtttacaac aataattgcc agatattatt ttttgctttg gtcttttgtg 180
ttcaatatta tatctgtaaa aactgttctt gaaaaacatc catggctgaa gcaggaagtc 240
caagaagcac attgatgctc ctcctctgtt ctccatgaga caaaaacaaa gtcacttctt 300
tctcaggcaa agctactgga ccggacaaaa ccggttctcc ccaaccgaag tctgtggtat 360
gaaaacccaa tctcgaccat gtagtgatca gaagagtgga agaaagagaa ggtcttgmtc 420
ttgttacctc aaagtaatca atggcagatc tcatgtatcc atcagttacc atcttaatgg 480
cttctcttac taatccaaca gcgtagccca aaggtttctc ggttagctct ccagcttcac 540
atattgagtt tgtgagaact attccattcc cgaaataccc tttgggaagt ggaggctcaa 600
acttggctct accatcaaag gcgaagagaa gctttgtttt ctgatcactc aacatcttca 660
gtgacttggt tcttgctctc cacacaaaag cagacaaagc ttcaaaagtt gtgcaggaga 720
gagattcact gttctctgtt gcttggagct taagtttctt gattttctct gggtcaaag 779
<210> 17
<211> 272
<212> ДНК
<213> Brassica napus
<400> 17
tttttccagg aatagcaaca aacattcctt agggtgaaaa tatttaaaac aaacatcaaa 60
atccgccgtt acctgtgggc aaggcctctg gaccaagcaa aaatagaggc tatgctgttg 120
gtgcttgttt crccaccttt ttggtgaact ctgtagtggc gtgttactgt gccatgagct 180
gcctctgctt caatggtctt tccatcagga caaacctaca attttccccc aaaatattaa 240
ttcaggaaga ttcatgggaa acatgttaac aa 272
<210> 18
<211> 315
<212> ДНК
<213> Brassica napus
<400> 18
tacaaacacc ttataacaga gaagccattt gactgtgcag ttagctcctt ctggactgct 60
ctaagtttct tgttgctctt tgcttatccc accccygaga acccaactcc aagctacgag 120
tatcacaccg ccttcactgg ggttgcacta ggaattgtga gcattcaaga attgttatat 180
agcgcagttt tgtaagtggt ttgttcagat agtgtcctta atcaatgttt ttgctcgtct 240
taggtgactg gagttcagca aacatacagc caattccacc acgaaggagc tccacgaatc 300
ttctcaccag agctt 315
<210> 19
<211> 274
<212> ДНК
<213> Brassica napus
<400> 19
ttagctcctt ctggactgct ctaagtttct tgttgctctt tgcttatccc accccygaga 60
acccaactcc aagctacgag tatcacaccg ccttcactgg ggttgcacta ggaattgtga 120
gcattcaaga gatgctatat agtgcagttt tgtaaatggt ttattcagag agtagcctta 180
atcaatggtt ttgaatgtct taggtgactg gcgttcagca aacatacagc cagttccacc 240
acgaaggggc tccacgaatc ttctcaccag agct 274
<210> 20
<211> 292
<212> ДНК
<213> Brassica napus
<400> 20
ctgaacggcg ggttctaaat cagcatcatc tccaaacgga ttaacttcac ggtcccatcc 60
tccacctctg ttgttccagc caccaccacc gccaccacct cctcgtccac cataaccttg 120
gccgggacga cctacatcgt tcctgtagcc accgcctcca ccaactccac ctccaggagc 180
ccatcgagaa ggytgaccac cataccctgg acgatcattc tgtggcgaag gagcaacagg 240
gtctggttgc ctgttcctta aatgaggagg aacataagca ggctttgccc tg 292
<210> 21
<211> 764
<212> ДНК
<213> Brassica napus
<400> 21
ttattattaa agtagtccaa acaaaagcat tcactcacgc ctacaacttc caaaaccatc 60
gtctttcttc aaaacaagat tacataggaa agagactgat aacatttcaa acactgataa 120
cacactctag ttggactgac gcagctctgg tcgaacaaaa tctgcatctt gaggcactga 180
gatatccacg gtaggcctga gctctgcgca gacctggata gcaccatgga ctggatcagc 240
gaagaagttg ctgatcaagt ctttgttgat tggtgcacca ttgtccacag ccaccagagc 300
ttgttgggtc aggttgtagt cgaaccttgg tgcccatttc ctcgatccca atcttgctgt 360
ggtcgagcag ttgtccacca tgaacgacac tcctctagcg tgcataaacg ggttcagaga 420
atcaacagac tcratcacac tctcgttgat gatctctgag taagagtgac ccttcttcct 480
caagatctca atctgagcca tcatgagggc aacgtaaact ccagcggtga agggatacaa 540
gggacccaag tcaccagctg gtctggactt cctgacgcgt tcaccgacct tccacattct 600
tgtctgatca atctttccca ttgggaatgc aggtaaaccc tccttatcgt agaagcgacg 660
accggctaag acaacactcc tgatttcgct gccagctgct acatcctcgt agcattcata 720
gaggatctcc catacaggat agaaggatgc actgtatgca gtct 764
<210> 22
<211> 252
<212> ДНК
<213> Brassica napus
<400> 22
aaaaaattta agatgattga caccgacatt taaaaaaaaa aaggactgat ggtatwgcct 60
tcttatccga agaacaaatg tacaagaaag attccttcac aatttgatac tttcacggct 120
caaaatatcc tcatgattct catcaataca cgcaacacac atttttgttt tgtggatcac 180
gttaaaacag atgattgaat caagcgttct tataggttac aagcaaatga gcaaaaggca 240
aaccagtttg aa 252
<210> 23
<211> 268
<212> ДНК
<213> Brassica napus
<400> 23
actgccgcct tccatttaca aagctgtgaa gacagagttt ctaaaatagt tctctgggtt 60
tccttcagct ccacgctatt caatcttaga cacttgtttt aacctcacaa gttatgaaga 120
cgtcagcata ccaactatca aaatgatctt ccaaggtagg gctgagcttg aagtggatgt 180
cactggtgtg ttctactttg tcaagccwga tgcatctcta gtctgcttgg ccttggcaag 240
tctttcatat gaaaacgaag tcggtatt 268
<210> 24
<211> 404
<212> ДНК
<213> Brassica napus
<400> 24
ctgcaggaag atacttgtca acttcctaat aagtatctcc agccacttat tcaccgaact 60
ctaagtatcg caaggtttat ctccaaattt tagctctgaa gttctctatt tgragttttc 120
attttgttat ctacttgtct ggaacaacta gagggaaata aactgagcaa ataaattttg 180
gttgggcagg gataaatctg atgatgatat aagtgttgcc agccccagtt atgataacta 240
tgtatattcc tctccccggc atgaggatct atctctaaat aatgttgcaa gtgcgcatgc 300
aatcaatggg aaatcaacgg attccacatc tatcgcaacc aatcaaccaa cagagattcc 360
agcaggaagc tgtgtcagta agggagagag gttagtcctt ttaa 404
<210> 25
<211> 404
<212> ДНК
<213> Brassica napus
<400> 25
ctgcaggaag atacttgtca acttcctaat aagtatctcc agccacttat tcaccgaact 60
ctaagtatcg caaggtttat ctccaaattt tagctctgaa gttctctatt tgaagttttc 120
attttgttat ctacttgtct ggaacaacta gagggaaata aactgarcaa ataaattttg 180
gttgggcagg gataaatctg atgatgatat aagtgttgcc agccccagtt atgataacta 240
tgtatattcc tctccccggc atgaggatct atctctaaat aatgttgcaa gtgcgcatgc 300
aatcaatggg aaatcaacgg attccacatc tatcgcaacc aatcaaccaa cagagattcc 360
agcaggaagc tgtgtcagta agggagagag gttagtcctt ttaa 404
<210> 26
<211> 1006
<212> ДНК
<213> Brassica napus
<400> 26
cccgggctgc aggattcggc acgagcaaga tcttctctaa tctctaccta tctctctctc 60
atctctctct ttcatggctg ttactggctt ctctctctga ttttctcgaa gttacacgat 120
gagtctcgtg gctagtcact cgccgcgttt aactctaacc ggcgacggcg tttccttacg 180
caattcgaga agaaacggtg aaaaatccaa acttttcttg ctaaatcgaa ggagatcagc 240
gcgtgcagct cttgttcaag ctaagcccag agaagacgga gcggtggcaa gctcttcccc 300
ctcctcaaaa cctccggtta tccaataccg acgagctgat ctcgcggatg atctccaagc 360
ggaggcacga gctttaagtc gagccgttgg tgcttctgtt tattctccag aactaatcgc 420
tagaaaacat ggctctcagc ctctcaaggc tttgcagaga agtctggaga tattgtcagc 480
tttaggtggc ttcgcgttca agttagggat tgatcagagg caagggaagc tagagcwgaa 540
catgaagaag agagctggtg agctcagaaa gattttcact cgtctggggc ccacttttgt 600
taagttgggt caaggtttat ccacccgacc cgacctctgt ccacctgatt acctcgaaga 660
acttgctgag cttcaggatg ctttgccaac cttccctgac gcagaagcct ttacttgcat 720
cgaaagagag ttagactcat ctctagaatc catcttctcc tctgtatccc ctaagccaat 780
agcagcagct accctcggcc aggtttacaa agctcacctc aggtactcag ggcaagttgt 840
cgctgtcaaa gtccaacgcc ccgggatcga agaagccatc ggtctcgact tctacctcat 900
cagaggagta gggaaactca taaacaaata cgcagacttc atcacccacg acgtcctcgc 960
cctcatcgcg agttcgcctg cagagtctac caggagctca actacg 1006
<210> 27
<211> 1130
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (1040)..(1040)
<223> n равно a, c, g или t
<400> 27
ccacgcgtcc gctcagtttg aaggaaaacc tggtcgatct ttcgatatag actttgagga 60
acgtctagaa accattagaa ggtcagctct tgagaagaaa aagacagaag tagttaacga 120
gtacggtcca atagactatg atgcaccagc accagtccag acagatcaga aaacaattgg 180
tcttggtacc aaggttggag ttggcatagc tgttgttgtc tttggtttag tttttgctct 240
tggagacttt ctccccacag gaagtgatag tccaacggag aagacctctg tggttaaaaa 300
caaaatttcr gaagaggaga aagccacgct tcagcaaagg ctcaaagagt ttgaaacaac 360
tctcgatggg gctcctaatg atcaaactgc tctagagggc gcagctgtga cactgactaa 420
tctaggagag tattcaggtg ctgctacgct tcttgagaaa ttggctaagg agagaccaac 480
tgatcctgat gtctttcgat tgcttggaga agtaaatttt gaacttaaga actatgaagg 540
cagtatcgct gcttacaaaa tttctgagaa ggtttctaat ggcattgatc ttgaagtcac 600
acgaggcctc atgaacgcgt atctagctgc taagaaacca gacgaggctg tcaaatttct 660
tctggatact cgcgaacgac tgaatacaag gaaaacaagc acagcagaca gtgtttccgc 720
tgaaactgaa ccagatctag acccaattca ggttgagttg cttcttggaa aagcttactc 780
agactgggga catatcagcg acgctatagc tgtttatgac cggcttatct atgaacatcc 840
tgaagacttc cgagggtact tggctaaggg aattttactg aaagaaaatg gaagcagagg 900
agatgcagag agaatgttca tccaggcacg gtttttcgca ccagacaaag ccaaggcctt 960
tgtggataga tattcgaagc tgtgatcagt agctggtttt gtatatatgt acgcacatgg 1020
gcttgtcttt ttttcaaccn taagtttata gccagagttg taacaagaga atattccaag 1080
gtcaataaga ttcatctgcg cagtttatac aaaaaaaaaa aaaaaaaagg 1130
<210> 28
<211> 890
<212> ДНК
<213> Brassica napus
<400> 28
tgcctcgtgg cgcatcattt cgtcgatcga gcagaaggaa gagagccgcg gcaacgacga 60
ccacgtcacg gcgatccgcg attacagatc taagatcgag acggagctct ctggaatctg 120
cgacgggatc cttaagctcc tcgattcgag gctcgtcccc gccgctgctt ccggcgattc 180
caaggtgttt tacctcaaga tgaaaggtga ttaccacagg tacttggctg agtttaagac 240
tggycaggag aggaaagacg ccgccgagaa cactctctcc gcttacaaag ccgctcagga 300
tattgcgaat gcagagctag caccaactca cccaatccgt ctaggtctgg cattgaactt 360
ctctgtgttc tactacgaga tccttaactc tcctgatcgt gcctgcagcc tcgccaaaca 420
ggcctttgat gacgcgatag ccgagttgga cactctaggt gaagagtcgt acaaagacag 480
caccttgatc atgcagctgc tccgtgacaa cctcactctc tggacatccg acatgcagga 540
tgatgctgcg gatgagatca aggaagcatc agctccaaaa ccaacagagg aacagcagta 600
acaatctttt gagtgaactg ctaagtttct agggggtttg aaactttgtt ttctctttcg 660
tccatatgtt tcctaggtcg tcgcttgccc attcgctttt attcattcat cataatttct 720
ggcaagtctg aaccatcgat catcttctat ctcagttgct tgattttctt ttgtttttcc 780
ttttttttta tgtgggttaa tgatttcagt gtctgccatc gaaccttgct ttgtgtgttt 840
ctcatcaatt tgaagatact tttgtcggtc tgaaaaaaaa aaaaaaaaaa 890
<210> 29
<211> 890
<212> ДНК
<213> Brassica napus
<400> 29
tgcctcgtgg cgcatcattt cgtcgatcga gcagaaggaa gagagccgcg gcaacgacga 60
ccacgtcacg gcgatccgcg attacagatc taagatcgag acggagctct ctggaatctg 120
cgacgggatc cttaagctcc tcgattcgag gctcgtcccc gccgctgctt ccggcgattc 180
caaggtgttt tacctcaaga tgaaaggtga ttaccacagg tacttggctg agtttaagac 240
tggccaggag aggaaagacg ccgccgagaa cactctctcc gcttacaaag ccgctcagga 300
tattgcgaat gcagagctag caccaactca cccaatccgt ctaggtctgg cattgaactt 360
ctctgtgttc tactacgaga tccttaactc tcctgatcgt gcctgcagcc tcgccaaaca 420
ggcctttgat gacgcgatag ccgagttgga cactctaggt gaagagtcgt acaaagacag 480
caccttgatc atgcagctkc tccgtgacaa cctcactctc tggacatccg acatgcagga 540
tgatgctgcg gatgagatca aggaagcatc agctccaaaa ccaacagagg aacagcagta 600
acaatctttt gagtgaactg ctaagtttct agggggtttg aaactttgtt ttctctttcg 660
tccatatgtt tcctaggtcg tcgcttgccc attcgctttt attcattcat cataatttct 720
ggcaagtctg aaccatcgat catcttctat ctcagttgct tgattttctt ttgtttttcc 780
ttttttttta tgtgggttaa tgatttcagt gtctgccatc gaaccttgct ttgtgtgttt 840
ctcatcaatt tgaagatact tttgtcggtc tgaaaaaaaa aaaaaaaaaa 890
<210> 30
<211> 875
<212> ДНК
<213> Brassica napus
<400> 30
aaattctagg gttttgcacc acacaatcgc cgaacgaatc caacaccatg ggacgaagac 60
cagcgagatg ctaccgtcag atcaagggga agccctaccc gaagtcccgc tactgccgcg 120
gtgtccccga ccccaagatc cgcatctacg acgtcggcat gaagaagaaa ggagtcgacg 180
agttcccttt ctgcgtccac ctcgtctcct gggagaagga gaacgtctcc agcgaagccc 240
tcgaggctgc tcgtatcgcc tgcaacaagt acatggtcaa atccgccgga aaagacgcct 300
tccatttgag gattagggtt caccctttcc atgtcctgag gatcaacaag atgctctcgt 360
gcgccggggc tgataggctc cagactggta tgagaggtgc gtttggtaag gccttgggga 420
cttgcgcgag ggttgcgatt gggcaggtgc ttttgtctgt gaggtgtaag gataaccatg 480
gggctcatgc tcaggaggcg cttaggaggg ctaagtttaa gttcccgggg cgtcagaaga 540
tcattgtcag caggaaatgg ggtttcacca agttcaaccg tgcggattac actaggttga 600
gacagtccaa gagggttgtt ccggatggtg tcaatgctaa gtttctgtcg aaccatggtc 660
cgttggctaa ccgtcagcct ggaagtgcct tcatatcagc caccagcgat taagaatgaa 720
gatgatttgt tgttgtcgaa ctgagaatyt agtttctgca gttttttgtt ttgtttttgt 780
tgttacagtt gaaaacatga ttcatcgtat gactacattt tttcttggac aaatgttttt 840
agtatcaata tctcagcctc tagtttatct acttt 875
<210> 31
<211> 916
<212> ДНК
<213> Brassica napus
<400> 31
tccaagcccc tccgaccaat ggcggcgaca atggcaacat cagccacatg catgtcctta 60
aacccatctc ctcctaagtc cccaaaccaa accaaaccca tctcctcatc aaaacccttc 120
atcaccctcc caacaccact aaaacccacc gtctctctcg ccgtcacaag caccgccctc 180
gccggcgccg tattctccac cctcagctac tccgaacccg ccttcgctgc ccaccagatc 240
gccgagctcg cggcagcggc cgcgggaggt agcgacaacc gaggactagc tcttctcctc 300
ccgatcgtcc cggcgatcgg atgggtgctc ttcaacattc tccagccagc tctcaaccag 360
atcaacaaga tgcgcgagag caaaggagtc gtcgcgggtc ttggcrtcgg cggtggtctc 420
gctgcgtcag ggcttttgac tatgcctccg gaggcctccg ccagtgtatt taaccagatg 480
gcggcggtgg cggaagcggc tgccaaaggt gaagacaaca ggggacagtt gttgttgttc 540
gtggttgcac cggctctact ttgggttctt tacaatatat tgcagcctgc tttgaaccaa 600
ctcaataaaa tgaggtctga gtaataatat attatcatcg ttgggattca aatgtaaaat 660
taatcctttg ttttatggac agttttttat gtttaaaatt ttggacatat cctttgttat 720
atatatctcg cctactatat actcgatgtc ttataagttt tcaaccgcca tcactagtcc 780
ttatgcgatt ttggtgtttc aaatccatct ataccagaac ctagtgttat gtcttggact 840
taccctaagg ttaaggatta aaacctccca tgttttgtag atgcttccac atattatttg 900
catatcaata acactc 916
<210> 32
<211> 182
<212> ДНК
<213> Brassica napus
<400> 32
ctgcaggtaa gcctatatat agctggackc atcatattat tagtggtcat tctataaagc 60
atgtgtgcgc tgctatggtc cctgtcttcc ttaccctcat gctcgctaaa agaagcgttc 120
aaaccgagag gtatacatat gaaatgatga cacaccctta ttctcggact ttgacgcttt 180
aa 182
<210> 33
<211> 1298
<212> ДНК
<213> Brassica napus
<400> 33
ccacgcgtcc gtggatctcc gtgttcctat ttgagatgtg tgatatagga gagatctgat 60
gaaacagaga catgtcttca ggtgaggggg cttctgtgta gtgagtctgg tcaccataag 120
cttcaaagaa acaccaatca ggtatatccg taatcccaac cattgatgca atgttgcata 180
caggattcct cgcagctgct gccacaaact tatccggcgc ctggccaatc aagtgtgtgg 240
tcagaaaccc accatgagaa ccacctagca cggttattct agacgggtct gcaagtccca 300
tctcaacagc atgatctacc gctgagagca catcgtttac gtcctgtgat ccaatttttc 360
caggtacaga ctgcagagca tcttccccaa atcccaacga acccctgtaa tttacaatca 420
gcagactgta tccaattgag gagagatatg ccagctgctt ggaaaagctg catggtgaaa 480
ctgtatgagg gcctccatgg aggacaacaa ctaaaggatc acatttccca ttctccttgg 540
tctccgaaga cgatacatat atagcttcaa aaggtctttt ggccccttcg gtgagacatt 600
cagaaacatt actggtccga gtatcccatc cctgcgtcgc cggatttgaa aactccaccg 660
tgaaagatgc agcacgacga ggtcatatgg caagtcatca gacacaagca ctgcagttac 720
atggccaaga tcgaaaccgg aatcttctgt agaaaccctt acaacgtaac gggtatttgy 780
aaccgaagct cttgtcctct cgctaacagc cgttacgcca ccatcagaga ccacgatggt 840
gtgttttatt tgtatatgaa gacaatagag agagcccata tgccaaagaa cttgtgggag 900
agggtgaagt tgcctagaaa ctacgagaag gctcttgaaa ccattgacaa gcacttgttg 960
tattggccta agttattaca gcacaagatc aagcaaagac tgaccaaaat gactcagatg 1020
cgtattcgta tgaggaaact tgctctcaag accagggaga agataatgac gacaccaagg 1080
agggatatca agagagaatt aagaagagag gagaaggctg ttaaagcagc agtcttggat 1140
aaggctatcg agaccgagtt gctagagcgt ttgaagaagg gtatttatgg tgacatatac 1200
aactacccgg agcttgagtg gaacatgttc ttgatgaaga aaagaaattg gcagaaggtg 1260
ttgaggagga ggaagaagag gaggagccag agattgaa 1298
<210> 34
<211> 267
<212> ДНК
<213> Brassica napus
<400> 34
ttaaactagc ttcttctttt gtagatattc tcaatcaaca gttgaaaatg tacaactccc 60
tctcacaagc ccagtctgat actaagatgg ttcagtcgct tgtggcartt tgggaggttc 120
atcctaactt tattagtttt attctttgaa gttattttgt tggtctcttc cctgacctaa 180
ccttgtcact gttttctcag gaggagtatg aaaggactgg cgtgtatgat gcacctatac 240
cccctgatcc tggcaaaccc cctgcag 267
<210> 35
<211> 237
<212> ДНК
<213> Brassica napus
<400> 35
ctgcaggcat catggcacgt aaccggaagg gtgcgggcac cacgcacgtt ttccttcatg 60
atgttgaccg catagtggag aagacgtamg ccaacgagtt cctatgcgag aagtacagag 120
tcaattcagc tggtaggctc tggcactttg agatacccaa cgctgctaac atgagcgacc 180
agcctgggga ccgattttgc taggttgaac cgatctttga ttcttcgtta cttttaa 237
<210> 36
<211> 2463
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (1590)..(1590)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1648)..(1648)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1650)..(1650)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1658)..(1658)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1660)..(1660)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1670)..(1670)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1675)..(1675)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1678)..(1678)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1689)..(1689)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (1698)..(1698)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (2174)..(2174)
<223> n равно a, c, g или t
<400> 36
cgggtcgacg atcccgtgcg caagctggga gaagtctgga ggaagccaaa aagttagttg 60
agaagagttt cggaaactat gtgagcagta acgtgatggt tgctgccaag gaggagcttg 120
ctgaaatcga caagaagatc gagattctga cgtcagagat aagcgatgag gcgatagata 180
agaagagcag gaagcttctg tcagcgaatc agtacaagga gatcactgca cttcagggag 240
rgctgcgtga agagaaacgc aagcgtactg aatttcgaaa aaggatggaa ctcgaaaggt 300
tctcagcatt aaaaccgctt ctaaaaggga tggaagacgg aaacttaccg tttatatgtt 360
tggacttcaa agattctgaa gggatgcagc agtcagtccc tgcggtttac ttggggcata 420
tcgattcatt taacggttca aagcttcaga agatgatgtc tttggatgag tcctttgcgt 480
tgaacgtaat cgaggatgaa ccggcggctg atgagcctat tgttaaacca tcttactatg 540
tggcccttgg ctctgataac tcatggtatc tcttcactga gaaatggata aggactgttt 600
acaggactgg cttccccaac actgctctag cgacaggaga tgctttgcct cgagaaatca 660
tgaaggctct tcttgacaaa gcagatatgc agtgggataa actatccgat tccgagctcg 720
gaagtatgtg gagaatggaa ggatctctag agacatggtc ctggagttta aacgtgcctg 780
tcctgagcag cctctccgag gaagacgagg tgttgcacat gtcccaagag tatgacaacg 840
ctgctgaaca atacaaggaa caaagaagca aagtgtcgcg gttgaagaag aggatatcac 900
gatcagcagg gttcagagaa tacaagaaaa ttctggagaa cgctaagctg acggttgaga 960
agatgaaacg gctcaaggcg agatcgagac gcctcataaa ccgtttagag cagatcgaac 1020
cgtctggttg gaaagacttc atgcgcataa gcaatgtgat tcacgagagc agagcgttgg 1080
acattaacac acacttgata tttcctctgg gtgaaacagc tgctgctatc agaggagaga 1140
acgagctctg gctcgcaatg gtgctccgga acaaagtcct ggtcgatctc aagcctcctc 1200
aactagctgg cgtatgtgcg agtttagtct ccgaaggcat taaagttcga ccgtggagag 1260
acaacaacta tatatacgag ccgtctgata cagtagttga tgtggttaac ttcttagagg 1320
aacaaagaag ctcgcttata aagcttcaag agaagcatga ggttgagatt tcttgttgtt 1380
tggatgttca gttctctggt atggttgaag cttgggcctc tgggctaagc tggaaagaga 1440
tgatgatgga atgtgcaatg gacgaaggtg atcttgctcg tttgttgcga cgcaccattg 1500
atctcttggc tcagattcct aagttgcctg atattgaccc caagctgcaa cgtagtgcag 1560
ctgctgcagc agatatcatg gaccgtccan ccaatcagcg agcttgccgg ttaaagaggc 1620
tccatgttag caatgtcttt agtcaacntn aaaaatancn attagaaccn tgagngtntg 1680
tacactatnt taaggatncc agaacaaatt tttcattcag gtgattaatg ttggaaagaa 1740
taggaccaaa caagagaact ttgagttaat gttacaacat acaaaagctt tatatacaca 1800
actgtggaag ctaatccctt ccagtattcc tctcttccga caaagctcaa tgctgagtat 1860
atcaaccaaa agaatgtctt taaaagcgtg agaagttgct ctgttcaaca gtggcaagtg 1920
tgagaaaggt cattggccgg tgagaagttt ttggtaactt tgtatcctcc tttgcctgat 1980
tgaagagcca ctggcaacaa cagggtcttg tctgctttgg tttgaaacct tcttcctctt 2040
aagacgatgc aaaacatcct ctgtgtcgaa tacataggag acctttgaag aactgcaggt 2100
tatcttacac tccaaaccgt tactaatctt aacttgatcc tcaccatttt tacgcttatt 2160
cctgtttaga tagnattcga ggattctcct tcacattagg ttgactggaa aggaatttat 2220
tgtggaggta agatgcaaaa gtttgatcca tggatatgga tgtcacatca ggcttatcat 2280
gtccgttgtt cagaagcatc atagataatt ttgtcggtgt cgacagttca cattcaatgc 2340
ggtatatatc ttcatcagga aggagaacac gagcattgtc ataataaaca gcatcaagat 2400
atttttcagg ctttcttgat ttctcatata agtatagctg gtgtagcttg ttgtccatct 2460
cat 2463
<210> 37
<211> 815
<212> ДНК
<213> Brassica napus
<220>
<221> misc_feature
<222> (153)..(153)
<223> n равно a, c, g или t
<220>
<221> misc_feature
<222> (241)..(241)
<223> n равно a, c, g или t
<400> 37
ccacgcgtcc gcggacgcgt gggcggacgc gtgggcgtag tttgtttccg cgtgctcgaa 60
gaggacgaga catataactt ctacatagag agagaaagag acagcaaaac ccaagcagtt 120
gaaacagaga aggacgagta aaacagaaca agngaaggaa gacagagatt ctggttctcc 180
aacgtttgag agagaacaca ccaaaacaat gtcgttccgt agcatagtac gcgatgtgag 240
nagacagtat aggcaacctg tctcgccgta gcttcgactt caagctgagc agccttcaca 300
aagaaggagg tggtaagtct cgcggttcgg ttcaagactg tcacgaggaa caacaacaac 360
aacaacagcc tttagcactg gtggttcaag aaaccccttg ggcgaatctg cctcccgagc 420
tgttgaggga tgtgatcaaa aggctggaag agagcgagag cgcgtggcct gcgaggaagc 480
acgtggtrgc ttgtgcttct gtttgcaggt cgtggagaga tatgtgcaaa gagattgttc 540
aaaggcctga ggtttctggg aagatcacgt ttcctgtttc gcttaaacag cctggaccaa 600
gagatgcaac aatgcaatgc tttatcaaga gggataagtc taacttgact taccatttgt 660
acctttgtct cagtcctgct ttgttggtcg agaatggaaa gtttcttctc tcggcgaaac 720
gcataagaag aaccacttac acagagtacg tcatctccat gcacgcagac accatttcaa 780
gatcaagcaa cacctacatt ggcaagatca ggtct 815
<210> 38
<211> 53
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 38
gaaggtgacc aagttcatgc tgacatttaa aaaaaaaaag gactgatggt ata 53
<210> 39
<211> 53
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 39
gaaggtcgga gtcaacggat tgacatttaa aaaaaaaaag gactgatggt att 53
<210> 40
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 40
gaatctttct tgtacatttg ttcttcggat 30
<210> 41
<211> 45
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 41
gaaggtgacc aagttcatgc tggtgtgttc tactttgtca agcca 45
<210> 42
<211> 45
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 42
gaaggtcgga gtcaacggat tggtgtgttc tactttgtca agcct 45
<210> 43
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 43
aaggccaagc agactagaga tgcat 25
<210> 44
<211> 51
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 44
gaaggtgacc aagttcatgc tcaaatttta gctctgaagt tctctatttg a 51
<210> 45
<211> 50
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 45
gaaggtcgga gtcaacggat taaattttag ctctgaagtt ctctatttgg 50
<210> 46
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 46
cctctagttg ttccagacaa gtagataa 28
<210> 47
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 47
gaaggtgacc aagttcatgc tatccctgcc caaccaaaat ttatttgt 48
<210> 48
<211> 46
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 48
gaaggtcgga gtcaacggat tccctgccca accaaaattt atttgc 46
<210> 49
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 49
cttgtctgga acaactagag ggaaataaa 29
<210> 50
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 50
gaaggtgacc aagttcatgc tagaggcaag ggaagctaga gca 43
<210> 51
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 51
gaaggtcgga gtcaacggat tagaggcaag ggaagctaga gct 43
<210> 52
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 52
gctcaccagc tctcttcttc atgtt 25
<210> 53
<211> 44
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 53
gaaggtgacc aagttcatgc tgaagcgtgg ctttctcctc ttct 44
<210> 54
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 54
gaaggtcgga gtcaacggat taagcgtggc tttctcctct tcc 43
<210> 55
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 55
caacggagaa gacctctgtg gttaa 25
<210> 56
<211> 46
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 56
gaaggtgacc aagttcatgc tgtacttggc tgagtttaag actggt 46
<210> 57
<211> 44
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 57
gaaggtcgga gtcaacggat tacttggctg agtttaagac tggc 44
<210> 58
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 58
gagtgttctc ggcggcgtct tt 22
<210> 59
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 59
gaaggtgacc aagttcatgc tgagagtgag gttgtcacgg aga 43
<210> 60
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 60
gaaggtcgga gtcaacggat tagagtgagg ttgtcacgga gc 42
<210> 61
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 61
gagtcgtaca aagacagcac cttgat 26
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ НИЗКОГО СОДЕРЖАНИЯ ПАЛЬМИТИНОВОЙ КИСЛОТЫ В ПОДСОЛНЕЧНИКЕ (HELIANTHUS ANNUS) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2013 |
|
RU2670517C2 |
| SNP-МАРКЕРЫ И ОТБОР В ОТНОШЕНИИ НИЗКОГО СОДЕРЖАНИЯ КЛЕТЧАТКИ В ПРЕДСТАВИТЕЛЯХ РОДА BRASSICA | 2019 |
|
RU2834673C2 |
| КАНОЛА HO/LL С УСТОЙЧИВОСТЬЮ К ЗАБОЛЕВАНИЮ КИЛОЙ КРЕСТОЦВЕТНЫХ | 2012 |
|
RU2618846C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| КАНОЛА HO/LL С УСТОЙЧИВОСТЬЮ К ЗАБОЛЕВАНИЮ КИЛОЙ КРЕСТОЦВЕТНЫХ | 2012 |
|
RU2711934C2 |
| ГЕНЕТИЧЕСКИЕ ЛОКУСЫ, СВЯЗАННЫЕ С УСТОЙЧИВОСТЬЮ К ПЫЛЬНОЙ ГОЛОВНЕ У МАИСА | 2009 |
|
RU2562864C2 |
| РАСТЕНИЕ, УСТОЙЧИВОЕ К НАСЕКОМЫМ | 2008 |
|
RU2522477C2 |
| РАСТЕНИЕ, УСТОЙЧИВОЕ К НАСЕКОМЫМ | 2014 |
|
RU2617955C2 |
| РАСТЕНИЕ АРБУЗА, ХАРАКТЕРИЗУЮЩЕЕСЯ ПРОДУКТИВНЫМ ЦВЕТЕНИЕМ | 2017 |
|
RU2792674C2 |


Изобретение относится к области биохимии, в частности к способу идентификации растения канолы, обладающего резистентностью к черной ножке. При этом способ включает детекцию в растении канолы по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке. Изобретение также относится к способу получения растения канолы, обладающего резистентностью к черной ножке. Указанный способ включает скрещивание первого растения канолы, содержащего по меньшей мере один маркерный аллель, который положительно коррелирован с резистентностью к черной ножке, со вторым растением канолы для получения популяции растений канолы и идентификацию растения канолы из популяции, обладающего резистентностью к черной ножке. Изобретение позволяет эффективно идентифицировать растение канолы, обладающее резистентностью к черной ножке. 2 н. и 10 з.п. ф-лы, 2 ил., 3 табл., 4 пр.
1. Способ идентификации растения канолы, обладающего резистентностью к черной ножке, где способ включает: детекцию в растении канолы по меньшей мере одного маркера, сцепленного с резистентностью к черной ножке, где по меньшей мере один маркер выбран из группы, состоящей из SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36, SEQ ID NO:37, и маркера, сцепленного по меньшей мере с одним из SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36 или SEQ ID NO:37.
2. Способ по п.1, где способ включает детекцию в растении канолы маркера, сцепленного по меньшей мере с одним из SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36 и SEQ ID NO:37.
3. Способ по п.2, где детектируемый маркер отражает частоту генетической рекомбинации, равную менее приблизительно 10%, по меньшей мере с одним из маркеров SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36 и SEQ ID NO:37.
4. Способ по п.1, где детектируемый маркер выбран из группы, состоящей из SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36 и SEQ ID NO:37.
5. Способ по п.1, где детекция включает детекцию по меньшей мере одной аллельной формы однонуклеотидного полиморфизма (SNP).
6. Способ по п.1, где детекция включает амплификацию маркера или части маркера с получением амплифицированного маркерного ампликона, и детекцию полученного амплифицированного маркерного амликона.
7. Способ по п.6, где амплификация включает:
смешивание праймера амплификации или пары праймеров амплификации с нуклеиновой кислотой, выделенной из растения канолы, где праймер или пара праймеров комплементарны или частично комплементарны по меньшей мере части маркера и способны инициировать полимеризацию ДНК с помощью ДНК-полимеразы, используя нуклеиновую кислоты канолы в качестве матрицы; и
удлинение праймера или пары праймеров в реакции полимеризации ДНК, включающей ДНК-полимеразу и матрицу нуклеиновой кислоты, с получением по меньшей мере одного ампликона.
8. Способ по п.6, где амплификация включает использование полимеразной цепной реакции (ПЦР) или лигазной цепной реакции (LCR), используя нуклеиновую кислоту, выделенную из растения канолы в качестве матрицы в ПЦР или LCR.
9. Способ получения растения канолы, которое обладает резистентностью к черной ножке, где способ включает: скрещивание первого растения канолы, содержащего по меньшей мере один маркерный аллель, который положительно коррелирован с резистентностью к черной ножке, со вторым растением канолы для получения популяции растений канолы, скрининг популяции на присутствие по меньшей мере одного маркерного аллеля, где по меньшей мере один маркер выбран из группы, состоящей из SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36, SEQ ID NO:37, и маркера, сцепленного по меньшей мере с одним из SEQ ID NO:24, SEQ ID NO:25, SEQ ID NO:23, SEQ ID NO:22, SEQ ID NO:26, SEQ ID NO:27, SEQ ID NO:28, SEQ ID NO:29, SEQ ID NO:17, SEQ ID NO:18, SEQ ID NO:19, SEQ ID NO:20, SEQ ID NO:21, SEQ ID NO:31, SEQ ID NO:30, SEQ ID NO:32, SEQ ID NO:34, SEQ ID NO:33, SEQ ID NO:35, SEQ ID NO:36 или SEQ ID NO:37, и
идентификацию растения канолы из популяции, которое обладает резистентностью к черной ножке, способом по п.1.
10. Способ по п.9, где второе растение канолы проявляет более низкий уровень резистентности к черной ножке по сравнению с первым растением канолы, и где полученное растение канолы проявляет повышенную резистентность к болезни черная ножка по сравнению со вторым растением.
11. Способ по п.9, где второе растение канолы является растением элитного сорта канолы или экзотического сорта канолы.
12. Способ по п.2, где детектированный маркер определяют с использованием картирования популяции Nex845CL/NT152344//Nex845CL.
| RAMAN R | |||
| ET AL: Molecular mapping of qualitative and quantitative loci for resistance to Leptosphaeria maculans; causing blackleg disease in canola (Brassica napus L.), THEOR APPL GENET., vol | |||
| Плуг с фрезерным барабаном для рыхления пласта | 1922 |
|
SU125A1 |
| Аппарат для передачи изображений неподвижных и движущихся предметов | 1923 |
|
SU405A1 |
| WO2009048847 A1, 16.04.2009 | |||
| YUNMING LONG et al., Identification of two blackleg resistance genes and fine mapping of one of these | |||
Авторы
Даты
2020-04-08—Публикация
2014-09-08—Подача