Область техники
Изобретение относится к области генной инженерии и биотехнологии, а именно к числу средств - направляющих РНК, которые могут быть использованы в системах CRISPR-Cas12 в составе рибонуклеопротеиновых комплексов для выявления, обнаружения, детекции провирусной ДНК вируса иммунодефицита человека (ВИЧ-1), интегрированной в геном человека.
Изобретение позволяет in vitro выявлять единичные копии молекул провирусной ДНК ВИЧ-1.
Направляющие РНК, описанные в настоящей заявке, могут быть использованы для детекции провирусной ДНК ВИЧ-1 после проведения специфической амплификации фрагментов ДНК ВИЧ-1. Амплификация при этом может быть проведена различными способами, среди которых полимеразная цепная реакция (PCR); петлевая изотермическая амплификация (LAMP); геликаза-зависимая амплификация (HDA); рекомбиназа-опосредованная амплификация (RPA); амплификация со смещением цепи (SDA); амплификация, основанная на последовательности нуклеиновых кислот (NASBA); опосредованная транскрипцией амплификация (TMA); амплификация, опосредованная никирующим ферментом (NEAR); круговая амплификация (RCA) и многие другие виды амплификации.
Направляющие РНК, описанные в настоящей заявке, могут быть использованы для разработки высокочувствительных и высокотехнологичных диагностических систем нового поколения на основе CRISPR технологий для борьбы с распространением социально-значимых инфекций, таких как ВИЧ, для диагностики ВИЧ-инфекции у детей, рожденных от ВИЧ-инфицированных матерей, для мониторинга инфекционной безопасности донорской крови, оценки вирусной нагрузки и лекарственной устойчивости вируса иммунодефицита человека.
Уровень техники
Для решения эпидемиологических задач по расшифровке вспышек инфекционных болезней, выявления и идентификации возбудителя необходимы разработка и внедрение в практику работы надзорных и мониторинговых служб современных технологий молекулярной эпидемиологии.
Одной из таких технологий является использование элементов генетического редактирования системы CRISPR/CAS. Данная технология развивается достаточно эффективно в отношении создания средств лечения некоторых болезней, несмотря на ряд трудностей, связанных с возникновением непредвиденных мутаций. При углубленных исследованиях в области применения CRISPR/CAS системы было выяснено, что она может быть использована для тонких диагностических процедур при выявлении возбудителя/ей инфекции у человека, а также их генотипирования.
В 2018 году было показано, что один из ферментов CRISPR системы - Cas12 после распознавания своей целевой ДНК-мишени начинает неспецифически гидролизовать одноцепочечную, а также двухцепочечную ДНК. Такое свойство Cas12 можно использовать в качестве индикатора присутствия определенной мишени, например, генома вируса или бактерии. Исследователи использовали это открытие для создания технологической платформы обнаружения нуклеиновых кислот, известной как DETECTR (DNA EndonucleaseTargeted CRISPR TransReporter - ДНК-нацеленная эндонуклеаза CRISPR транс репортер). Впервые DETECTR была использована для выявления и генотипирования вируса папилломы человека (HPV).
Предложенная платформа объединяет нуклеазу Cas12a, ее направляющую РНК, специфичную к нуклеиновой кислоте HPV, флуоресцентную репортерную молекулу. Технология DETECTR используется для обнаружения целевой ДНК-мишени после предварительной амплификации (ChenJS, MaE, HarringtonLB, DaCostaM, TianX, PalefskyJM, DoudnaJA. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 2018 Apr 27; 360(6387):436-439).
В связи с этим крайне актуальной является задача разработки новых эффективных методик выявления нуклеиновых кислот возбудителей социально-значимых инфекционных заболеваний, таких как ВИЧ, гепатиты В и С, туберкулез и др., основанных на генетических технологиях, таких как CRISPR/CAS.
В ходе изучения уровня техники не было найдено источников, описывающих разработку и получение направляющих РНК для выявления единичных копий провирусной ДНК ВИЧ-1 in vitro.
Исходя из этого, возникает техническая проблема, заключающаяся в необходимости разработки и получения направляющих РНК для выявления единичных копий провирусной ДНК ВИЧ-1 in vitro.
Раскрытие сущности
Предложенная технология перспективна для разнообразных применений, включая количественное определение ДНК/РНК, быструю мультиплексную детекцию экспрессии, другие виды чувствительной детекции, например, выявление загрязнения образцов нуклеиновыми кислотами. Технология, основанная на CRISPR/CAS, является многофункциональной, устойчивой к ошибкам технологией детекции ДНК, пригодной для быстрой постановки диагнозов, включая инфекционные заболевания, и генотипирования инфекционных агентов.
Применение предложенной технологии делает возможным создание диагностических систем нового поколения, которые будут обладать следующими свойствами:
• высокая чувствительность;
• возможность проведения диагностики у постели больного;
• возможность проведения диагностики в полевых условиях без применения специализированного высокотехнологичного оборудования;
• скорость и простота анализа;
• сниженная стоимость анализа;
• отсутствие необходимости оснащения диагностической лаборатории дорогостоящим оборудованием;
• отсутствие необходимости проведения выделения нуклеиновых кислот возбудителя.
Изобретение относится к новым средствам - направляющим РНК, которые могут быть использованы в системах CRISPR-Cas12 для ультрачувствительного выявления, идентификации, обнаружения или детекции провирусной ДНК вируса иммунодефицита человека (ВИЧ-1), интегрированной в геном человека, в биологических образцах.
Технической задачей настоящего изобретения является разработка новых средств - направляющих РНК, которые могут быть использованы в системах CRISPR-Cas12 с белками Cas12, такими как белки AsCpf1 из Acidaminococcus или LbCpf1 из Lachnospiraceae, для ультрачувствительного выявления провирусной ДНК ВИЧ-1, интегрированной в геном человека.
При осуществлении настоящего изобретения достигается неожиданный технический результат - возможность ультрачувствительного выявления провирусной ДНК ВИЧ-1 до единичных копий провирусной ДНК ВИЧ-1 в одной реакции.
Изобретение обеспечивает повышение эффективности выявления провирусной ДНК ВИЧ-1 с 100 до 10 копий/мл (ГЭ/мл).
Технический результат достигается за счет:
• разработки молекул направляющих РНК, которые могут быть использованы в системах CRISPR-Cas12, для ультра чувствительного выявления провирусной ДНК ВИЧ-1, интегрированной в геном человека, где указанные направляющие РНК выбраны из последовательностей SEQ ID NO: 1 и SEQ ID NO: 2, способны связываться с целевыми высоко консервативными участками генома ВИЧ-1, содержат РНК-шпильку, которая распознается РНК-направляемыми ДНК-эндонуклеазами AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, с обеспечением выявления единичных копий провирусной ДНК ВИЧ-1;
• применения РНК-направляемых ДНК-эндонуклеаз AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, полученных согласно способу, разработанному авторами ранее (Патент РФ № 2707542, опубл.27.11. 2019), для создания рибонуклеопротеиновых комплексов (РПК) системы CRISPR/CAS, пригодных для детекции провирусной ДНК ВИЧ-1 в ультранизких концентрациях (единичные копии);
• разработки набора специфических олигонуклеотидов, выбранных из SEQ ID NO: 3-6, для предварительной амплификации фрагментов генома ВИЧ-1;
• оптимизации условий проведения предварительной амплификации фрагментов генома ВИЧ-1;
• определения условий проведения ультрачувствительной детекции провирусной ДНК ВИЧ-1 и установления последовательности стадий метода.
Предложена молекула направляющей РНК системы CRISPR/CAS, которая способна связываться с целевыми высоко консервативными участками генома вируса иммунодефицита человека (ВИЧ-1). Нуклеотидная последовательность раскрытой в настоящей заявке направляющей РНК выбрана из последовательностей SEQ ID NO: 1 и SEQ ID NO: 2.
Молекула направляющей РНК содержит в своем составе РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой, выбранной из AsCpf1 из Acidaminococcus или LbCpf1 из Lachnospiraceae.
Молекула направляющей РНК обеспечивает выявления единичных копий провирусной ДНК ВИЧ-1.
Направляющие РНК согласно настоящему изобретению соответствуют высоко консервативным фрагментам генома ВИЧ-1. Наиболее предпочтительны направляющие РНК, распознающиеся РНК-направляемыми ДНК-эндонуклеазами AsCpf1 из Acidaminococcus или LbCpf1 из Lachnospiraceae, характеризующиеся, имеющие или содержащие нуклеотидную последовательность, выбранную из:
• SEQ ID NO: 1;
• SEQ ID NO: 2;
• или идентичной любой из них по меньшей мере на 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%,
• или комплементарной любой из них,
• или гибридизующейся с любой из них в строгих условиях.
Согласно предложенному изобретению получают рибонуклеопротеиновые комплексы (РПК), состоящие из по меньшей мере одной направляющей РНК и РНК-направляемой ДНК-нуклеазы системы CRISPR/CAS, выбранной из AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, пригодные для использования для выявления провирусной ДНК ВИЧ-1 в ультранизких концентрациях (единичные копии).
Рибонуклеопротеиновый комплекс системы CRISPR/CAS для выявления провирусной ДНК ВИЧ-1, сформированный из РНК-направляемой ДНК-эндонуклеазы, выбранной из AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, и по меньшей мере одной направляющей РНК.
Предложенный рибонуклеопротеиновый комплекс системы CRISPR/CAS пригоден для выявления единичных копий провирусной ДНК ВИЧ-1, интегрированной в геном человека.
Препараты РПК представляют собой растворы, содержащие направляющую РНК, выбранную из SEQ ID NO: 1 и SEQ ID NO: 2, объединенную с белком системы CRISPR/CAS (AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae) или лиофильно высушенные РПК.
Полученные направляющие РНК могут быть использованы в составе набора для обнаружения провирусной ДНК вируса иммунодефицита человека (ВИЧ-1), интегрированной в геном человека, с инструкцией по применению.
Набор для обнаружения провирусной ДНК ВИЧ-1, интегрированной в геном человека, может содержать рибонуклеопротеиновый комплекс и инструкцию по применению.
При этом, по меньшей мере одна направляющая РНК в составе набора может находиться в комплексе с белком системы CRISPR/CAS (AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae) в одном контейнере или отдельно в разных контейнерах.
Набор может дополнительно включать компоненты для проведения предварительной амплификации высоко консервативных фрагментов генома ВИЧ-1, в том числе один или несколько специфических олигонуклеотидов, выбранных из SEQ ID NO: 3-6.
Специфически олигонуклеотиды для проведения предварительной амплификации фрагментов генома ВИЧ-1 согласно настоящему изобретению соответствуют высоко консервативным участкам генома ВИЧ-1. Наиболее предпочтительны олигонуклеотиды, характеризующиеся, имеющие или содержащие нуклеотидную последовательность, выбранную из:
• SEQ ID NO: 3;
• SEQ ID NO: 4;
• SEQ ID NO: 5;
• SEQ ID NO: 6;
• или идентичной любой из них по меньшей мере на 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99%,
• или комплементарной любой из них,
или гибридизующейся с любой из них в строгих условиях.
Предложенная технология позволяет определить единичные копии провирусной ДНК вируса иммунодефицита человека (ВИЧ-1), интегрированной в геном человека, в биологических образцах пациента, выбранных из жидкости и/или ткани, предположительно содержащих провирусную ДНК ВИЧ-1. Биологическим образцом может быть образец крови, сыворотки или плазмы крови, клеток крови, слюны, мокроты, лимфоидных тканей, тканей кроветворных органов и других биологических материалов от пациента, которые могут быть использованы для анализа на наличие провирусной ДНК ВИЧ-1, интегрированной в геном человека.
Краткое описание чертежей
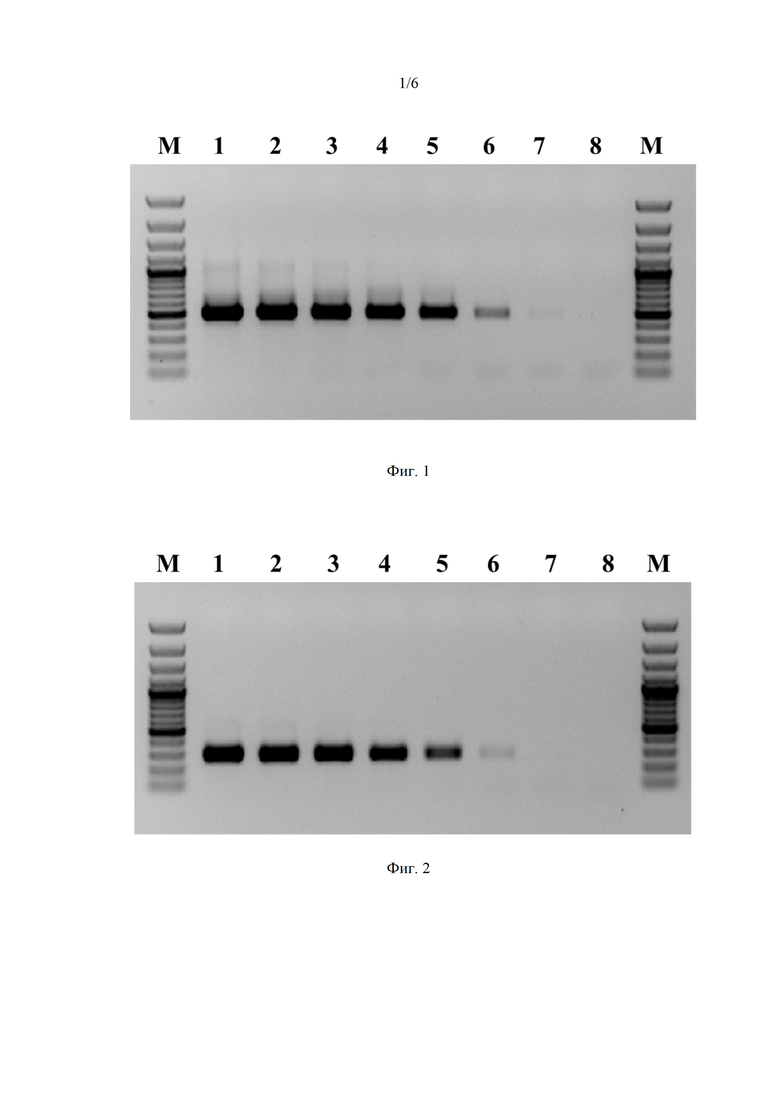
Фиг. 1. Визуализация фрагментов LTR после предварительной амплификации при помощи электрофореза в агарозном геле, где цифрами 1-8 обозначены:
1 - ПЦР-продукт LTR, полученный в ходе амплификации 1 нг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
2 - ПЦР-продукт LTR, полученный в ходе амплификации 0,1 нг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
3 - ПЦР-продукт LTR, полученный в ходе амплификации 10 пг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
4 - ПЦР-продукт LTR, полученный в ходе амплификации 1 пг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
5 - ПЦР-продукт LTR, полученный в ходе амплификации 0,1 пг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
6 - ПЦР-продукт LTR, полученный в ходе амплификации 10 фг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
7 - ПЦР-продукт LTR, полученный в ходе амплификации 1 фг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
8 - отрицательный контроль, не содержащий модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
М - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bpPlus, ThermoFisherScientific, США).
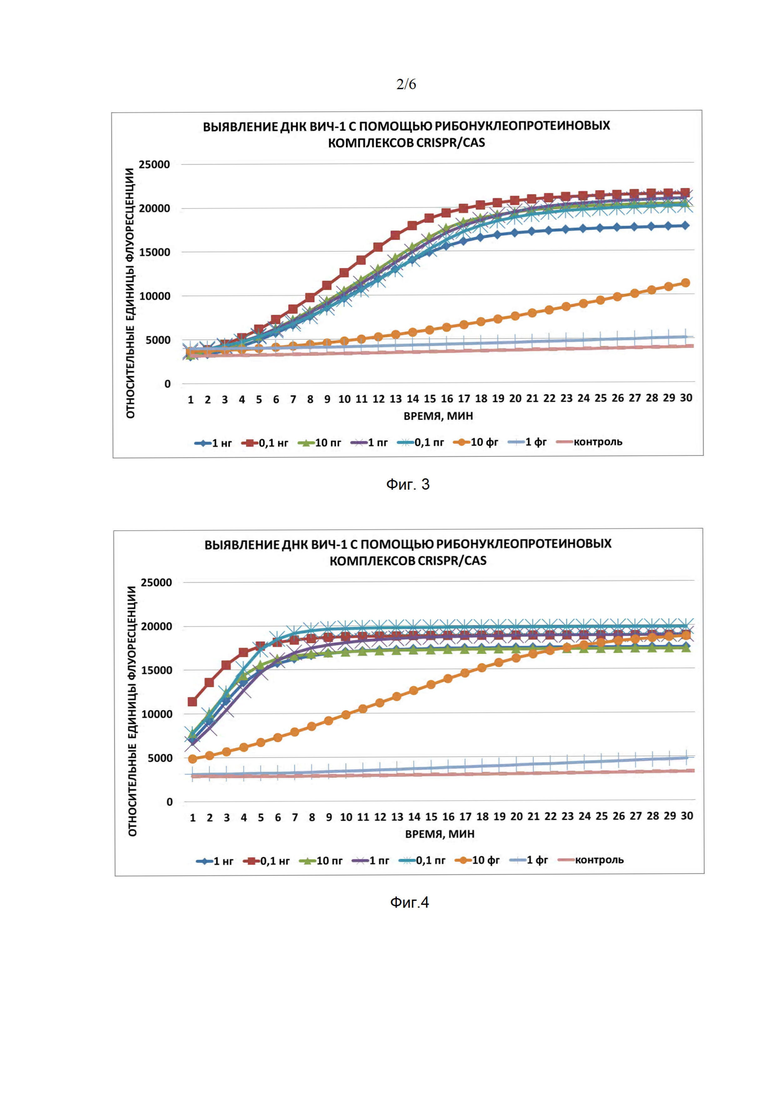
Фиг. 2. Визуализация фрагментов POL после предварительной амплификации при помощи электрофореза в агарозном геле, где цифрами 1-8 обозначены:
1 - ПЦР-продукт POL, полученный в ходе амплификации 1 нг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
2 - ПЦР-продукт POL, полученный в ходе амплификации 0,1 нг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
3 - ПЦР-продукт POL, полученный в ходе амплификации 10 пг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
4 - ПЦР-продукт POL, полученный в ходе амплификации 1 пг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
5 - ПЦР-продукт POL, полученный в ходе амплификации 0,1 пг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
6 - ПЦР-продукт POL, полученный в ходе амплификации 10 фг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
7 - ПЦР-продукт POL, полученный в ходе амплификации 1 фг модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
8 - отрицательный контроль, не содержащий модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3;
М - стандарты молекулярных масс: снизу вверх 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1200, 1500, 2000, 3000 пар нуклеотидов (GeneRuler 100 bpPlus, ThermoFisherScientific, США).
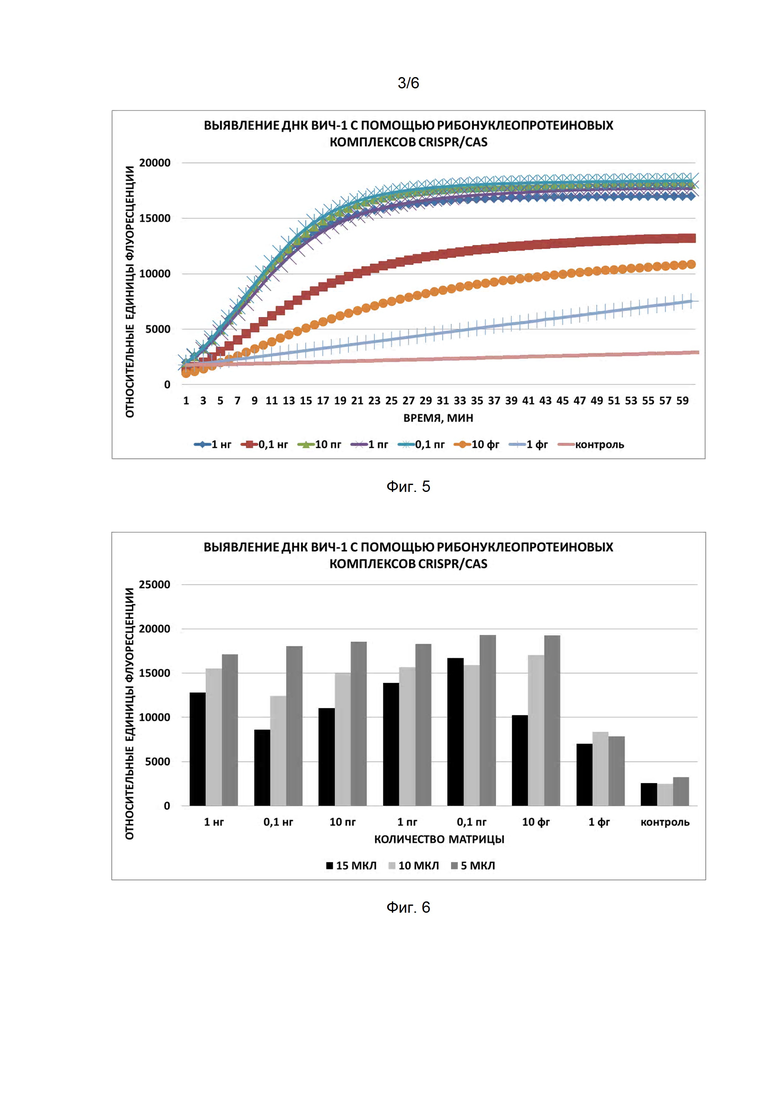
Фиг. 3. Профиль флуоресценции в реальном времени для предварительно амплифицированной мишени POL, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgPOL и белок AsCpf1.
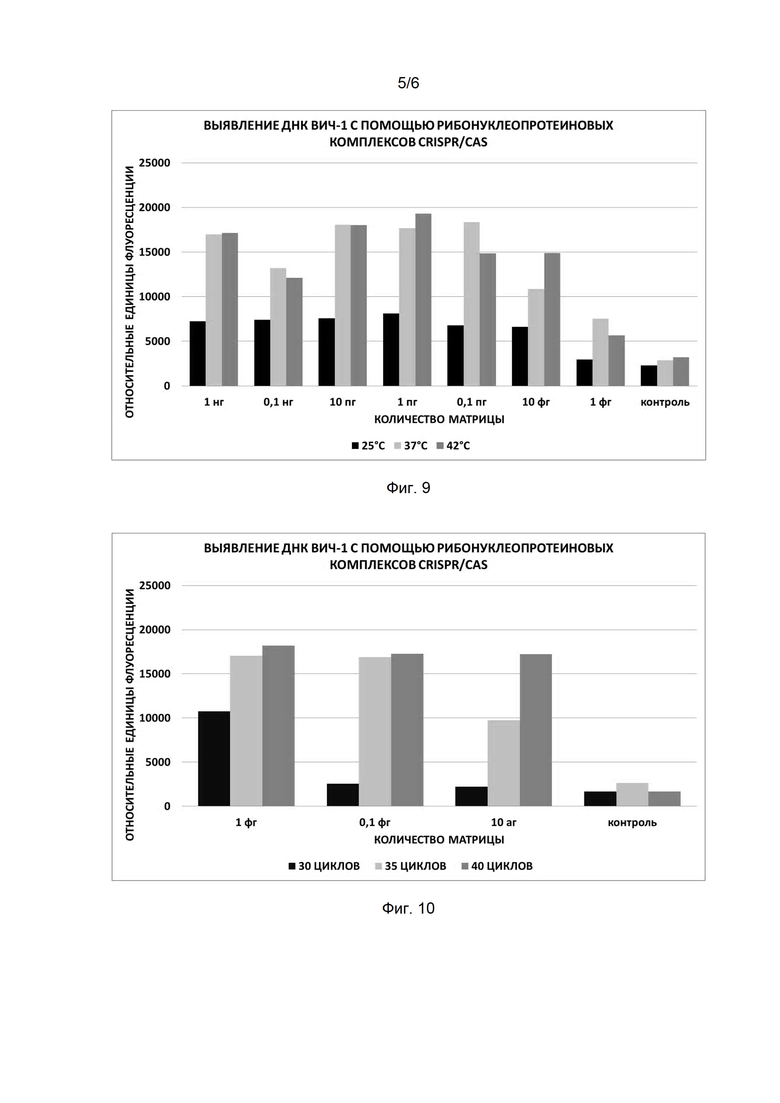
Фиг. 4. Профиль флуоресценции в реальном времени для предварительно амплифицированной мишени POL, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgPOL и белок LbCpf1.
Фиг. 5. Профиль флуоресценции в реальном времени для предварительно амплифицированной мишени LTR, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgLTR и белок LbCpf1. Предел обнаружения предварительно амплифицированной мишени LTR составляет 1 фг.
Фиг. 6. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированной мишени LTR, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgLTR и белок LbCpf1. Количество вносимой мишени не влияет существенно на значения флуоресценции в образце с минимальным количеством анализируемого материала (1 фг ДНК ВИЧ-1).
Фиг. 7. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированной мишени POL, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgPOL и белок LbCpf1. Количество вносимой мишени не влияет существенно на значения флуоресценции в образце с минимальным количеством анализируемого материала (1 фг ДНК ВИЧ-1).
Фиг. 8. Профиль флуоресценции в реальном времени для смеси предварительно амплифицированных мишеней LTR и POL, обработанных смесью рибонуклеопротеиновых комплексов, содержащих направляющие РНК sgLTR и sgPOL и белок LbCpf1.
Фиг. 9. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированной мишени LTR, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgLTR и белок LbCpf1. При 25°С и 42°С рибонуклеопротеиновый комплекс CRISPR/CAS способен обнаруживать до 10 фг ДНК ВИЧ-1, при 37°С - до 1 фг ДНК ВИЧ-1.
Фиг. 10. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированной мишени LTR, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgLTR и белок LbCpf1. Увеличение количества циклов предварительной амплификации до 35-40 позволяет обнаруживать до 2 аг ДНК ВИЧ-1(2×10-18 г, что соответствует 1,3 копиям ДНК, внесенным в реакцию предварительной амплификации).
Фиг. 11. Значения флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированной мишени POL, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgPOL и белок LbCpf1. Увеличение количества циклов предварительной амплификации до 35-40 позволяет обнаруживать до 2 аг ДНК ВИЧ-1 (2×10-18 г, что соответствует 1,3 копиям ДНК, внесенным в реакцию предварительной амплификации).
Список таблиц
Таблица 1. Специфические олигонуклеотиды для предварительной амплификации участков, соответствующих гену вирусной полимеразы (POL) и длинному концевому повтору (LTR).
Таблица 2. Серийные разведения модельной матрицы провирусной ДНК ВИЧ-1pNL4-3.
Таблица 3. Специфические олигонуклеотиды для получения ПЦР-продукта, кодирующего направляющую РНК (sgPOL и/или sgLTR).
Таблица 4. Дополнительные серийные разведения модельной матрицы провирусной ДНК ВИЧ-1pNL4-3.
Примеры осуществления изобретения
ПРИМЕР 1: ПОДБОР ПОСЛЕДОВАТЕЛЬНОСТЕЙ-МИШЕНЕЙ В ГЕНОМЕ ВИРУСА ИММУНОДЕФИЦИТА ЧЕЛОВЕКА (ВИЧ-1) ДЛЯ СОЗДАНИЯ НАПРАВЛЯЮЩИХ РНК
Для подбора последовательностей-мишеней в геноме вируса иммунодефицита человека (ВИЧ-1) для создания направляющих РНК были использованы современные алгоритмы insilico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая Benchling (https://www.benchling.com/molecular-biology/) и HIV databases (https://www.hiv.lanl.gov/content/sequence/HIV/mainpage.html). Был составлен перечень участков генома ВИЧ-1 с теоретически рассчитанной вероятностью их расщепления в высоко консервативных участках генома. Из составленного перечня выбраны участки, соответствующие гену вирусной полимеразы (POL) и длинному концевому повтору (LTR). Направляющая РНК, специфически узнающая высоко консервативный участок гена вирусной полимеразы (POL), представлена уникальной последовательностью SEQ ID NO: 1. Направляющая РНК, специфически узнающая высоко консервативный участок длинного концевого повтора (LTR), представлена уникальной последовательностью SEQ ID NO: 2.
ПРИМЕР 2: ПОДГОТОВКА МАТЕРИАЛА ДЛЯ ОБНАРУЖЕНИЯ ПРОВИРУСНОЙ ДНК ВИЧ-1 МЕТОДОМ ПРЕДВАРИТЕЛЬНОЙ АМПЛИФИКАЦИИ ФРАГМЕНТОВ ГЕНОМА ВИЧ-1
Подготовку материала для обнаружения провирусной ДНК ВИЧ-1 проводят методом предварительной амплификации. В качестве модельной матрицы провирусной ДНК ВИЧ-1 биологического образца используют плазмидную ДНК инфекционного молекулярного клона ВИЧ-1 pNL4-3. Физическая карта pNL4-3 доступна по адресу: https://aidsreagent.org/reagentdetail.cfm?t=molecular_clones&id=633.
Предварительную амплификацию участков, соответствующих гену вирусной полимеразы (POL) и длинному концевому повтору (LTR), проводят с использованием одного или нескольких специфических олигонуклеотидов с SEQ ID NO: 3-6, приведенных в Таблице 1.
Таблица 1. Специфические олигонуклеотиды для предварительной амплификации участков, соответствующих гену вирусной полимеразы (POL) и длинному концевому повтору (LTR).
ПЦР-продукт, кодирующий фрагмент гена вирусной полимеразы POL, получают в реакции амплификации с использованием ПЦР-смеси-2blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов Forpol и Revpol (Евроген, Россия). Размер амплифицированного фрагмента POL составляет 293 пары нуклеотидов.
ПЦР-продукт, кодирующий фрагмент длинного концевого повтора LTR, получают в реакции амплификации с использованием ПЦР-смеси-2blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов ForL и RevL (Евроген, Россия). Размер амплифицированного фрагмента LTR составляет 480 пар нуклеотидов.
Температурный профиль амплификации для получения ПЦР-продуктов, кодирующих фрагменты генома ВИЧ-1:
1. денатурация: 95°С в течение 3 минут;
2. 30 циклов амплификации:
95°С - 15 сек,
55°С - 45 сек,
72°С - 30 сек;
3. финальная элонгация: 72°С в течение 5 минут.
В ходе подготовки материала для обнаружения провирусной ДНК ВИЧ-1 методом предварительной амплификации проводят титрование модельной матрицы провирусной ДНК ВИЧ-1pNL4-3 путем приготовления серийных разведений (Таблица 2).
Таблица 2. Серийные разведения модельной матрицы провирусной ДНК ВИЧ-1 pNL4-3.
Для оценки эффективности предварительной амплификации полученные фрагменты генома ВИЧ-1 визуализируют при помощи электрофореза в агарозном геле (фиг. 1, фиг. 2).
Подготовленный описанным способом материал используют для экспериментов по выявлению провирусной ДНК вируса иммунодефицита человека (ВИЧ-1) с помощью рибонуклеопротеиновых комплексов AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, содержащих направляющие РНК sgPOL и/или sgLTR, без предварительной очистки.
ПРИМЕР 3: ПОЛУЧЕНИЕ НАПРАВЛЯЮЩИХ РНК И СОЗДАНИЕ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ ДЛЯ ОБНАРУЖЕНИЯ ПРОВИРУСНОЙ ДНК ВИРУСА ИММУНОДЕФИЦИТА ЧЕЛОВЕКА (ВИЧ-1)
Для получения направляющих РНК для обнаружения провирусной ДНК ВИЧ-1 разработан набор специфических олигонуклеотидов, приведенных в Таблице 3.
Таблица 3. Специфические олигонуклеотиды для получения ПЦР-продукта, кодирующего направляющую РНК (sgPOL и/или sgLTR).
Получение направляющих РНК проводят в несколько этапов:
1. получение ПЦР-продукта с использованием набора специфических олигонуклеотидов (Таблица 3), кодирующего направляющую РНК (sgPOL и/или sgLTR), способную связываться с целевым высоко консервативным участком генома ВИЧ-1 (POL или LTR, соответственно), содержащую РНК-шпильку, которая распознается РНК-направляемыми ДНК-эндонуклеазами AsCpf1 из Acidaminococcus, например, c SEQ ID NO: 7, или LbCpf1 из Lachnospiraceae, например, c SEQ ID NO: 8;
2. очистка ПЦР-продукта, кодирующего направляющую РНК (sgPOL и/или sgLTR);
3. синтез направляющей РНК (sgPOL и/или sgLTR);
4. очистка направляющей РНК (sgPOL и/или sgLTR).
ПЦР-продукт, кодирующий направляющую РНК sgLTR, получают в реакции амплификации с использованием ПЦР-смеси-2blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и tail L (Евроген, Россия). Размер амплифицированного фрагмента, кодирующего sgLTR, составляет 62 пары нуклеотидов.
ПЦР-продукт, кодирующий направляющую РНК sgPOL, получают в реакции амплификации с использованием ПЦР-смеси-2blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) и специфических олигонуклеотидов T7pr и tail pol (Евроген, Россия). Размер амплифицированного фрагмента, кодирующего sgPOL, составляет 62 пары нуклеотидов.
Температурный профиль амплификации для получения ПЦР-продуктов, кодирующих направляющие РНК:
1. денатурация: 95°С в течение 3 минут;
2. 35 циклов амплификации:
95°С - 15 сек,
55°С - 45 сек,
72°С - 30 сек;
3. финальная элонгация: 72°С в течение 5 минут.
Очистку ПЦР-продуктов, кодирующих направляющие РНК sgPOL и/или sgLTR, проводят с использованием коммерчески доступного набора ISOLATE II PCR and Gel Kit (BioLine, США) согласно инструкции производителя. Очищенные ПЦР-продукты, кодирующие направляющие РНК sgPOL и/или sgLTR, используют в качестве матрицы для синтеза направляющей РНК.
Синтез направляющих РНК sgPOL и/или sgLTR осуществляют методом in vitro транскрипции с использованием коммерчески доступных наборов реагентов (HiScribe™ T7 HighYield RNA SynthesisKit, NEB, США) согласно инструкции производителя. Продукты реакции in vitro транскрипции переосаждают из реакционной смеси добавлением хлорида натрия до конечной концентрации 400 mM и равного объема изопропилового спирта. Такие модификации протокола производителя, внесенные авторами, позволяют увеличить выход продукта реакции и получить желаемую концентрацию финального препарата направляющей РНК.
Создание готового рибонуклеопротеинового комплекса, содержащего белок семейства CRISPR/CAS AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, и направляющую РНК авторы проводят по стандартному протоколу с некоторыми модификациями (In vitro enzymology of Cas9 // Anders C, Jinek M.MethodsEnzymol. 2014;546:1-20. doi: 10.1016/B978-0-12-801185-0.00001-5).
Непосредственно перед объединением с CAS-белком препарат направляющей РНК (в количестве 250 нг) прогревают при 90°С в течение 5 минут и позволяют медленно остыть до комнатной температуры (инкубация при комнатной температуре не менее 10 минут). Такое прогревание необходимо для формирования корректной конформации шпильки, содержащейся в направляющей РНК. Многие производители коммерческих препаратов CAS-белков пропускают данный этап при подготовке рибонуклеопротеинового комплекса.
Для формирования готового рибонуклеопротеинового комплекса 250 нг CAS-белка AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae и подготовленную направляющую РНК смешивают и инкубируют 15 минут при комнатной температуре. Полученный таким способом рибонуклеопротеиновый комплекс готов для выявления провирусной ДНК вируса иммунодефицита человека (ВИЧ-1).
ПРИМЕР 4: ОБНАРУЖЕНИЕ ПРОВИРУСНОЙ ДНК ВИЧ-1 С ПОМОЩЬЮ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ CRISPR/CAS
Предварительно амплифицированный материал, полученный способом, описанным в Примере 2, используют в качестве матрицы для обнаружения провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS, полученных способом, описанным в Примере 3.
Для обнаружения провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS готовят реакционную смесь, содержащую следующие компоненты:
• 10× буфер (100 mM TrisHCl pH 8,0, 1 M NaCl);
• 50 mM MgCl2 (конечная концентрация в реакционной смеси 10 mM);
• 250 нг рибонуклеопротеинового комплекса (AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae и направляющие РНК sgPOL и/или sgLTR);
• 20 pmol флуоресцентный зонд (6FAM-TTATT-HQ1);
• Мишень (предварительно амплифицированный фрагмент ДНК ВИЧ-1 LTR и/или POL);
• вода mQ.
Реакционные смеси, содержащие все необходимые компоненты, помещают в амплификатор ДТП райм 5 (ДНК-Технология, Россия) и задают следующие параметры реакции:
30-60 циклов:
37°С - 35 сек,
37°С - 25 сек, съемка флуоресценции.
В первую очередь была предпринята попытка обнаружения провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS, сформированных на основе AsCpf1 из Acidaminococcus и LbCpf1 из Lachnospiraceae. Было показано, что оба типа рибонуклеопротеиновых комплексов CRISPR/CAS обладают способностью выявлять ДНК ВИЧ-1. Типичные результаты анализа приведены на примерах профилей флуоресценции в реальном времени для предварительно амплифицированной мишени POL, обработанной рибонуклеопротеиновыми комплексами, содержащими направляющую РНК sgPOL и белки AsCpf1 и LbCpf1, на Фиг. 3 и Фиг. 4, соответственно.
В ходе работ был оценен предел обнаружения провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS, сформированных на основе AsCpf1 из Acidaminococcus и LbCpf1 из Lachnospiraceae. Было показано, что рибонуклеопротеиновые комплексы CRISPR/CAS обладают способностью выявлять до 1 фг ДНК ВИЧ-1 (что соответствует 65 копиям ДНК, внесенным в реакцию предварительной амплификации). Уже на 30-ом цикле (30 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) вдвое. Типичные результаты анализа приведены на Фиг. 5 на примере профиля флуоресценции в реальном времени для предварительно амплифицированной мишени LTR, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgLTR и белок LbCpf1.
Была проведена оптимизация анализа. На первом этапе проведен эксперимент по изменению объема (количества) вносимой в анализ предварительно амплифицированной мишени. Было показано, что увеличение количества вносимой мишени не приводит к существенным изменениям значений флуоресценции в образце с минимальным количеством анализируемого материала (1 фг ДНК ВИЧ-1, что соответствует 65 копиям ДНК, внесенным в реакцию предварительной амплификации). Типичные результаты анализа приведены на примерах значений флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных мишеней LTR и POL, обработанных рибонуклеопротеиновыми комплексами, содержащими белок LbCpf1 и направляющие РНК sgLTR и sgPOL, на Фиг. 6 и Фиг. 7, соответственно.
На втором этапе был проведен эксперимент по комбинированию вносимых в анализ предварительно амплифицированных мишеней LTR и POL. Показано, что комбинирование вносимых в анализ мишеней не приводит к существенным изменениям значений флуоресценции в конечной точке в образце с минимальным количеством анализируемого материала (1 фг ДНК ВИЧ-1, что соответствует 65 копиям ДНК, внесенным в реакцию предварительной амплификации). Стоит отметить, что уже на 30-ом цикле (30 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) вдвое, а к 60 циклу анализа - более чем в 3 раза. Типичные результаты анализа приведены на Фиг. 8 на примере профиля флуоресценции в реальном времени для смеси предварительно амплифицированных мишеней LTR и POL, обработанных смесью рибонуклеопротеиновых комплексов, содержащих направляющие РНК sgLTR и sgPOL белок и LbCpf1.
На третьем этапе был проведен эксперимент по изменению температуры анализа. Было показано, что при 25°С и 42°С рибонуклеопротеиновые комплексы CRISPR/CAS способны обнаруживать до 10 фг ДНК ВИЧ-1 (что соответствует 650 копиям ДНК, внесенным в реакцию предварительной амплификации). Причем, на 60-ом цикле (60 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) в 4,6 раз, в случае проведения анализа при 42°С, и в 2,9 раз, в случае проведения анализа при 25°С. Оптимальной температурой для проведения анализа является 37°С, поскольку рибонуклеопротеиновые комплексы CRISPR/CAS способны обнаруживать до 1 фг ДНК ВИЧ-1 (что соответствует 65 копиям ДНК, внесенным в реакцию предварительной амплификации) и значение сигнала превышает значение «шума» более чем в 2,5 раза. Типичные результаты анализа приведены на Фиг. 9 на примере значений флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированной мишени LTR, обработанной рибонуклеопротеиновым комплексом, содержащим направляющую РНК sgLTR и белок LbCpf1.
ПРИМЕР 5: ОБНАРУЖЕНИЕ ЕДИНИЧНЫХ КОПИЙ ПРОВИРУСНОЙ ДНК ВИЧ-1 С ПОМОЩЬЮ РИБОНУКЛЕОПРОТЕИНОВЫХ КОМПЛЕКСОВ CRISPR/CAS
Для обнаружения единичных копий провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS было принято решение оптимизировать процесс предварительной амплификации за счет увеличения количества циклов.
Для предварительной амплификации титруют модельную матрицу провирусной ДНК ВИЧ-1 pNL4-3 путем приготовления дополнительных серийных разведений (Таблица 4).
Таблица 4. Дополнительные серийные разведения модельной матрицы провирусной ДНК ВИЧ-1pNL4-3.
ЦР-продукты, кодирующие фрагменты гена вирусной полимеразы POL и длинного концевого повтора LTR, получают в реакции амплификации с использованием ПЦР-смеси-2blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) как описано в Примере 2.
Температурный профиль амплификации для получения ПЦР-продуктов, кодирующих фрагменты генома ВИЧ-1:
1. денатурация: 95°С в течение 3 минут;
2. 30, 35 или 40 циклов амплификации:
95°С - 15 сек,
55°С - 45 сек,
72°С - 30 сек;
3. финальная элонгация: 72°С в течение 5 минут.
Полученный таким способом материал используют в качестве матрицы для обнаружения единичных копий провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS, полученных способом, описанным в Примере 3.
Обнаружение единичных копий провирусной ДНК ВИЧ-1 с помощью рибонуклеопротеиновых комплексов CRISPR/CAS проводят способом, описанным в Примере 4.
В ходе проведенного анализа было показано, что рибонуклеопротеиновые комплексы CRISPR/CAS обладают способностью выявлять единичные копии ДНК ВИЧ-1. Увеличение количества циклов предварительной амплификации до 35-40 позволяет обнаруживать до 2 аг ДНК ВИЧ-1(2×10-18 г, что соответствует 1,3 копиям ДНК, внесенным в реакцию предварительной амплификации). При этом уже на 10-15 цикле (10-15 минут) анализа значение сигнала превышало значение «шума» (неспецифической флуоресценции контрольного образца, не содержащего мишени) вдвое, а к 60 циклу анализа - более чем в 3-10 раз, при внесении в анализ материала, предварительно амплифицированного в течение 35 циклов. Типичные результаты анализа приведены на примерах значений флуоресценции в конечной точке (60 цикл анализа, 60 минут) для предварительно амплифицированных мишеней LTR и POL, обработанных рибонуклеопротеиновыми комплексами, содержащими направляющие РНК sgLTR и sgPOL и белок LbCpf1, на Фиг. 10 и Фиг. 11, соответственно.
Таким образом, разработанные направляющие РНК позволяют ультрачувствительно выявлять единичные копии провирусной ДНК ВИЧ-1 в образце после предварительной амплификации в составе рибонуклеопротеиновых комплексов CRISPR/CAS.
--->
Перечень последовательностей
<110> ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
<120>
<160> NUMBER OF SEQ ID NO: NOS: 6
<210> SEQ ID NO: NO 1
<211> 40
<212>RNA
<213>artificial
<400>SEQUENCE 1 (sgLTR):
acccacugcuuaagccucaaaucuacacuuaguagaaau u 40
<210> SEQ ID NO: NO 2
<211> 40
<212>RNA
<213>artificial
<400>SEQUENCE2 (sgPOL):
cugucucuguaauaaacccgaucuacacuuaguagaaauu 40
<210> SEQ ID NO: NO 3
<211> 31
<212>DNA
<213>artificial
<400>SEQUENCE3 (ForL):
tttggatggtgcttcaagctagtaccagtt g 31
<210> SEQ ID NO: NO 4
<211> 30
<212>DNA
<213>artificial
<400>SEQUENCE4 (RevL):
tggtctgagggatctctagttaccagagtc 30
<210> SEQ ID NO: NO 5
<211> 29
<212>DNA
<213>artificial
<400>SEQUENCE5 (For pol):
cacaattttaaaagaaaaggggggattgg 29
<210> SEQ ID NO: NO 6
<211> 29
<212>DNA
<213>artificial
<400>SEQUENCE6 (Rev pol):
caatcatcacctgccatctgttttccata 29
<210> SEQ ID NO: NO 7
<211> 1307
<212> protein
<213> artificial
<400> SEQUENCE 7 (AsCpf1):
MetHisHisHisHisHisHisHisGlySerProLysLysLysArgLysValMetThrGln
PheGluGlyPheThrAsnLeuTyrGlnValSerLysThrLeuArgPheGluLeuIlePro
GlnGlyLysThrLeuLysHisIleGlnGluGlnGlyPheIleGluGluAspLysAlaArg
AsnAspHisTyrLysGluLeuLysProIleIleAspArgIleTyrLysThrTyrAlaAsp
GlnCysLeuGlnLeuValGlnLeuAspTrpGluAsnLeuSerAlaAlaIleAspSerTyr
ArgLysGluLysThrGluGluThrArgAsnAlaLeuIleGluGluGlnAlaThrTyrArg
AsnAlaIleHisAspTyrPheIleGlyArgThrAspAsnLeuThrAspAlaIleAsnLys
ArgHisAlaGluIleTyrLysGlyLeuPheLysAlaGluLeuPheAsnGlyLysValLeu
LysGlnLeuGlyThrValThrThrThrGluHisGluAsnAlaLeuLeuArgSerPheAsp
LysPheThrThrTyrPheSerGlyPheTyrGluAsnArgLysAsnValPheSerAlaGlu
AspIleSerThrAlaIleProHisArgIleValGlnAspAsnPheProLysPheLysGlu
AsnCysHisIlePheThrArgLeuIleThrAlaValProSerLeuArgGluHisPheGlu
AsnValLysLysAlaIleGlyIlePheValSerThrSerIleGluGluValPheSerPhe
ProPheTyrAsnGlnLeuLeuThrGlnThrGlnIleAspLeuTyrAsnGlnLeuLeuGly
GlyIleSerArgGluAlaGlyThrGluLysIleLysGlyLeuAsnGluValLeuAsnLeu
AlaIleGlnLysAsnAspGluThrAlaHisIleIleAlaSerLeuProHisArgPheIle
ProLeuPheLysGlnIleLeuSerAspArgAsnThrLeuSerPheIleLeuGluGluPhe
LysSerAspGluGluValIleGlnSerPheCysLysTyrLysThrLeuLeuArgAsnGlu
AsnValLeuGluThrAlaGluAlaLeuPheAsnGluLeuAsnSerIleAspLeuThrHis
IlePheIleSerHisLysLysLeuGluThrIleSerSerAlaLeuCysAspHisTrpAsp
ThrLeuArgAsnAlaLeuTyrGluArgArgIleSerGluLeuThrGlyLysIleThrLys
SerAlaLysGluLysValGlnArgSerLeuLysHisGluAspIleAsnLeuGlnGluIle
IleSerAlaAlaGlyLysGluLeuSerGluAlaPheLysGlnLysThrSerGluIleLeu
SerHisAlaHisAlaAlaLeuAspGlnProLeuProThrThrLeuLysLysGlnGluGlu
LysGluIleLeuLysSerGlnLeuAspSerLeuLeuGlyLeuTyrHisLeuLeuAspTrp
PheAlaValAspGluSerAsnGluValAspProGluPheSerAlaArgLeuThrGlyIle
LysLeuGluMetGluProSerLeuSerPheTyrAsnLysAlaArgAsnTyrAlaThrLys
LysProTyrSerValGluLysPheLysLeuAsnPheGlnMetProThrLeuAlaSerGly
TrpAspValAsnLysGluLysAsnAsnGlyAlaIleLeuPheValLysAsnGlyLeuTyr
TyrLeuGlyIleMetProLysGlnLysGlyArgTyrLysAlaLeuSerPheGluProThr
GluLysThrSerGluGlyPheAspLysMetTyrTyrAspTyrPheProAspAlaAlaLys
MetIleProLysCysSerThrGlnLeuLysAlaValThrAlaHisPheGlnThrHisThr
ThrProIleLeuLeuSerAsnAsnPheIleGluProLeuGluIleThrLysGluIleTyr
AspLeuAsnAsnProGluLysGluProLysLysPheGlnThrAlaTyrAlaLysLysThr
GlyAspGlnLysGlyTyrArgGluAlaLeuCysLysTrpIleAspPheThrArgAspPhe
LeuSerLysTyrThrLysThrThrSerIleAspLeuSerSerLeuArgProSerSerGln
TyrLysAspLeuGlyGluTyrTyrAlaGluLeuAsnProLeuLeuTyrHisIleSerPhe
GlnArgIleAlaGluLysGluIleMetAspAlaValGluThrGlyLysLeuTyrLeuPhe
GlnIleTyrAsnLysAspPheAlaLysGlyHisHisGlyLysProAsnLeuHisThrLeu
TyrTrpThrGlyLeuPheSerProGluAsnLeuAlaLysThrSerIleLysLeuAsnGly
GlnAlaGluLeuPheTyrArgProLysSerArgMetLysArgMetAlaHisArgLeuGly
GluLysMetLeuAsnLysLysLeuLysAspGlnLysThrProIleProAspThrLeuTyr
GlnGluLeuTyrAspTyrValAsnHisArgLeuSerHisAspLeuSerAspGluAlaArg
AlaLeuLeuProAsnValIleThrLysGluValSerHisGluIleIleLysAspArgArg
PheThrSerAspLysPhePhePheHisValProIleThrLeuAsnTyrGlnAlaAlaAsn
SerProSerLysPheAsnGlnArgValAsnAlaTyrLeuLysGluHisProGluThrPro
IleIleGlyIleAspArgGlyGluArgAsnLeuIleTyrIleThrValIleAspSerThr
GlyLysIleLeuGluGlnArgSerLeuAsnThrIleGlnGlnPheAspTyrGlnLysLys
LeuAspAsnArgGluLysGluArgValAlaAlaArgGlnAlaTrpSerValValGlyThr
IleLysAspLeuLysGlnGlyTyrLeuSerGlnValIleHisGluIleValAspLeuMet
IleHisTyrGlnAlaValValValLeuGluAsnLeuAsnPheGlyPheLysSerLysArg
ThrGlyIleAlaGluLysAlaValTyrGlnGlnPheGluLysMetLeuIleAspLysLeu
AsnCysLeuValLeuLysAspTyrProAlaGluLysValGlyGlyValLeuAsnProTyr
GlnLeuThrAspGlnPheThrSerPheAlaLysMetGlyThrGlnSerGlyPheLeuPhe
TyrValProAlaProTyrThrSerLysIleAspProLeuThrGlyPheValAspProPhe
ValTrpLysThrIleLysAsnHisGluSerArgLysHisPheLeuGluGlyPheAspPhe
LeuHisTyrAspValLysThrGlyAspPheIleLeuHisPheLysMetAsnArgAsnLeu
SerPheGlnArgGlyLeuProGlyPheMetProAlaTrpAspIleValPheGluLysAsn
GluThrGlnPheAspAlaLysGlyThrProPheIleAlaGlyLysArgIleValProVal
IleGluAsnHisArgPheThrGlyArgTyrArgAspLeuTyrProAlaAsnGluLeuIle
AlaLeuLeuGluGluLysGlyIleValPheArgAspGlySerAsnIleLeuProLysLeu
LeuGluAsnAspAspSerHisAlaIleAspThrMetValAlaLeuIleArgSerValLeu
GlnMetArgAsnSerAsnAlaAlaThrGlyGluAspTyrIleAsnSerProValArgAsp
LeuAsnGlyValCysPheAspSerArgPheGlnAsnProGluTrpProMetAspAlaAsp
AlaAsnGlyAlaTyrHisIleAlaLeuLysGlyGlnLeuLeuLeuAsnHisLeuLysGlu
SerLysAspLeuLysLeuGlnAsnGlyIleSerAsnGlnAspTrpLeuAlaTyrIleGln
GluLeuArgAsnLysArgProAlaAlaThrLysLysAlaGlyGlnAlaLysLysLysLys 1340
<210> SEQ ID NO: NO 8
<211> 1231
<212> protein
<213> artificial
<400> SEQUENCE 8 (LbCpf1):
MetGlySerSerHisHisHisHisHisHisSerSerGlyLeuValProArgGlySerGly
ThrAlaThrMetSerLysLeuGluLysPheThrAsnCysTyrSerLeuSerLysThrLeu
ArgPheLysAlaIleProValGlyLysThrGlnGluAsnIleAspAsnLysArgLeuLeu
ValGluAspGluLysArgAlaGluAspTyrLysGlyValLysLysLeuLeuAspArgTyr
TyrLeuSerPheIleAsnAspValLeuHisSerIleLysLeuLysAsnLeuAsnAsnTyr
IleSerLeuPheArgLysLysThrArgThrGluLysGluAsnLysGluLeuGluAsnLeu
GluIleAsnLeuArgLysGluIleAlaLysAlaPheLysGlyAsnGluGlyTyrLysSer
LeuPheLysLysAspIleIleGluThrIleLeuProGluPheLeuAspAspLysAspGlu
IleAlaLeuValAsnSerPheAsnGlyPheThrThrAlaPheThrGlyPhePheAspAsn
ArgGluAsnMetPheSerGluGluAlaLysSerThrSerIleAlaPheArgCysIleAsn
GluAsnLeuThrArgTyrIleSerAsnMetAspIlePheGluLysValAspAlaIlePhe
AspLysHisGluValGlnGluIleLysGluLysIleLeuAsnSerAspTyrAspValGlu
AspPhePheGluGlyGluPhePheAsnPheValLeuThrGlnGluGlyIleAspValTyr
AsnAlaIleIleGlyGlyPheValThrGluSerGlyGluLysIleLysGlyLeuAsnGlu
TyrIleAsnLeuTyrAsnGlnLysThrLysGlnLysLeuProLysPheLysProLeuTyr
LysGlnValLeuSerAspArgGluSerLeuSerPheTyrGlyGluGlyTyrThrSerAsp
GluGluValLeuGluValPheArgAsnThrLeuAsnLysAsnSerGluIlePheSerSer
IleLysLysLeuGluLysLeuPheLysAsnPheAspGluTyrSerSerAlaGlyIlePhe
ValLysAsnGlyProAlaIleSerThrIleSerLysAspIlePheGlyGluTrpAsnVal
IleArgAspLysTrpAsnAlaGluTyrAspAspIleHisLeuLysLysLysAlaValVal
ThrGluLysTyrGluAspAspArgArgLysSerPheLysLysIleGlySerPheSerLeu
GluGlnLeuGlnGluTyrAlaAspAlaAspLeuSerValValGluLysLeuLysGluIle
IleIleGlnLysValAspGluIleTyrLysValTyrGlySerSerGluLysLeuPheAsp
AlaAspPheValLeuGluLysSerLeuLysLysAsnAspAlaValValAlaIleMetLys
AspLeuLeuAspSerValLysSerPheGluAsnTyrIleLysAlaPhePheGlyGluGly
LysGluThrAsnArgAspGluSerPheTyrGlyAspPheValLeuAlaTyrAspIleLeu
LeuLysValAspHisIleTyrAspAlaIleArgAsnTyrValThrGlnLysProTyrSer
LysAspLysPheLysLeuTyrPheGlnAsnProGlnPheMetGlyGlyTrpAspLysAsp
LysGluThrAspTyrArgAlaThrIleLeuArgTyrGlySerLysTyrTyrLeuAlaIle
MetAspLysLysTyrAlaLysCysLeuGlnLysIleAspLysAspAspValAsnGlyAsn
TyrGluLysIleAsnTyrLysLeuLeuProGlyProAsnLysMetLeuProLysValPhe
PheSerLysLysTrpMetAlaTyrTyrAsnProSerGluAspIleGlnLysIleTyrLys
AsnGlyThrPheLysLysGlyAspMetPheAsnLeuAsnAspCysHisLysLeuIleAsp
PhePheLysAspSerIleSerArgTyrProLysTrpSerAsnAlaTyrAspPheAsnPhe
SerGluThrGluLysTyrLysAspIleAlaGlyPheTyrArgGluValGluGluGlnGly
TyrLysValSerPheGluSerAlaSerLysLysGluValAspLysLeuValGluGluGly
LysLeuTyrMetPheGlnIleTyrAsnLysAspPheSerAspLysSerHisGlyThrPro
AsnLeuHisThrMetTyrPheLysLeuLeuPheAspGluAsnAsnHisGlyGlnIleArg
LeuSerGlyGlyAlaGluLeuPheMetArgArgAlaSerLeuLysLysGluGluLeuVal
ValHisProAlaAsnSerProIleAlaAsnLysAsnProAspAsnProLysLysThrThr
ThrLeuSerTyrAspValTyrLysAspLysArgPheSerGluAspGlnTyrGluLeuHis
IleProIleAlaIleAsnLysCysProLysAsnIlePheLysIleAsnThrGluValArg
ValLeuLeuLysHisAspAspAsnProTyrValIleGlyIleAspArgGlyGluArgAsn
LeuLeuTyrIleValValValAspGlyLysGlyAsnIleValGluGlnTyrSerLeuAsn
GluIleIleAsnAsnPheAsnGlyIleArgIleLysThrAspTyrHisSerLeuLeuAsp
LysLysGluLysGluArgPheGluAlaArgGlnAsnTrpThrSerIleGluAsnIleLys
GluLeuLysAlaGlyTyrIleSerGlnValValHisLysIleCysGluLeuValGluLys
TyrAspAlaValIleAlaLeuGluAspLeuAsnSerGlyPheLysAsnSerArgValLys
ValGluLysGlnValTyrGlnLysPheGluLysMetLeuIleAspLysLeuAsnTyrMet
ValAspLysLysSerAsnProCysAlaThrGlyGlyAlaLeuLysGlyTyrGlnIleThr
AsnLysPheGluSerPheLysSerMetSerThrGlnAsnGlyPheIlePheTyrIlePro
AlaTrpLeuThrSerLysIleAspProSerThrGlyPheValAsnLeuLeuLysThrLys
TyrThrSerIleAlaAspSerLysLysPheIleSerSerPheAspArgIleMetTyrVal
ProGluGluAspLeuPheGluPheAlaLeuAspTyrLysAsnPheSerArgThrAspAla
AspTyrIleLysLysTrpLysLeuTyrSerTyrGlyAsnArgIleArgIlePheArgAsn
ProLysLysAsnAsnValPheAspTrpGluGluValCysLeuThrSerAlaTyrLysGlu
LeuPheAsnLysTyrGlyIleAsnTyrGlnGlnGlyAspIleArgAlaLeuLeuCysGlu
GlnSerAspLysAlaPheTyrSerSerPheMetAlaLeuMetSerLeuMetLeuGlnMet
ArgAsnSerIleThrGlyArgThrAspValAspPheLeuIleSerProValLysAsnSer
AspGlyIlePheTyrAspSerArgAsnTyrGluAlaGlnGluAsnAlaIleLeuProLys
AsnAlaAspAlaAsnGlyAlaTyrAsnIleAlaArgLysValLeuTrpAlaIleGlyGln
PheLysLysAlaGluAspGluLysLeuAspLysValLysIleAlaIleSerAsnLysGlu
TrpLeuGluTyrAlaGlnThrSerValLysHisLysArgProAlaAlaThrLysLysAla
GlyGlnAlaLysLysLysLysGlySerGlyCys 1271
<---


Изобретение относится к области биотехнологии, а именно к направляющим РНК, рибонуклеопротеиновым комплексам системы CRISPR/CAS, содержащим направляющие РНК, и наборам, содержащим рибонуклеопротеиновые комплексы системы CRISPR/CAS и специфические олигонуклеотиды для предварительной амплификации высоко консервативных участков генома ВИЧ-1. Изобретение позволяет эффективно выявлять провирусную ДНК ВИЧ-1 после проведения специфической амплификации фрагментов ДНК ВИЧ-1. Изобретение обеспечивает выявление единичных копий провирусной ДНК вируса иммунодефицита человека (ВИЧ-1), интегрированной в геном человека. 4 н. и 3 з.п. ф-лы, 11 ил., 4 табл., 5 пр.
1. Молекула направляющей РНК системы CRISPR/CAS, которая способна связываться с целевыми высоко консервативными участками генома вируса иммунодефицита человека (ВИЧ-1), где нуклеотидная последовательность направляющей РНК выбрана из последовательностей SEQ ID NO: 1 и SEQ ID NO: 2.
2. Молекула направляющей РНК по п. 1, отличающаяся тем, что содержит в своем составе РНК-шпильку, которая распознается РНК-направляемой ДНК-эндонуклеазой, выбранной из AsCpf1 из Acidaminococcus или LbCpf1 из Lachnospiraceae.
3. Молекула направляющей РНК по п. 1 или 2, отличающаяся тем, что обеспечивает выявления единичных копий провирусной ДНК ВИЧ-1.
4. Рибонуклеопротеиновый комплекс системы CRISPR/CAS для выявления провирусной ДНК ВИЧ-1, сформированный из РНК-направляемой ДНК-эндонуклеазы, выбранной из AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae, и по меньшей мере одной направляющей РНК по пп. 1-3.
5. Рибонуклеопротеиновый комплекс системы CRISPR/CAS по п. 4, пригодный для выявления единичных копий провирусной ДНК ВИЧ-1, интегрированной в геном человека.
6. Набор для обнаружения провирусной ДНК ВИЧ-1, интегрированной в геном человека, содержащий
(i) (a) молекулу направляющей РНК системы CRISPR/CAS по любому из пп. 1-3 и по меньшей мере одну направляемую ДНК-эндонуклеазу, выбранную из AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae;
или
(i) (b) рибонуклеопротеиновый комплекс по любому из пп. 4-5;
и
(ii) инструкцию по применению.
7. Набор для обнаружения провирусной ДНК ВИЧ-1, интегрированной в геном человека, содержащий
(i) (a) молекулу направляющей РНК системы CRISPR/CAS по любому из пп. 1-3 и по меньшей мере одну направляемую ДНК-эндонуклеазу, выбранную из AsCpf1 из Acidaminococcus и/или LbCpf1 из Lachnospiraceae;
или
(i) (b) рибонуклеопротеиновый комплекс по любому из пп. 4-5;
и
(ii) один или несколько специфических олигонуклеотидов, выбранных из SEQ ID NO: 3-6,
и
(iii) инструкцию по применению.
| RU 2018101666 A, 19.07.2019 | |||
| CHEN J.S | |||
| et al | |||
| Способ гальванического снятия позолоты с серебряных изделий без заметного изменения их формы | 1923 |
|
SU12A1 |
| MA Y | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Chem., 2017, v.89, n.23, p.12896-12901 | |||
| US 20180179503 A1, 28.06.2018 | |||
| WO | |||
Авторы
Даты
2020-05-13—Публикация
2019-12-13—Подача