Область техники
Настоящее изобретение относится к области биотехнологии. Конкретнее, настоящее изобретение относится к применению высокотермостойкого Cas-белка и способу, и набору реактивов для обнаружения целевой молекулы нуклеиновой кислоты.
Уровень техники
Быстрое обнаружение молекул нуклеиновой кислоты является технологией, которая играет крайне важную роль в здравоохранении, контроле состояния окружающей среды, расследовании уголовных дел и других областях. Обнаружение молекул нуклеиновой кислоты может использоваться не только для выявления заражения людей, животных, растений и т.д. болезнетворными микроорганизмами, но также для выявления рисков генетических или онкологических заболеваний, для предоставления вспомогательных ссылочных материалов для индивидуальных лекарственных препаратов и для обнаружения загрязнения водоемов микроорганизмами. В настоящее время разработано много способов обнаружения нуклеиновой кислоты, например: ПЦР в режиме реального времени, метод гибридизации FISH (флуоресцентная гибридизация in situ) и т.д. Однако по-прежнему существует потребность в разработке быстрых, дешевых и чувствительных методов обнаружения нуклеиновой кислоты.
В последние годы хорошо зарекомендовал себя метод редактирования геномов на основе системы CRISPR (короткие палиндромные кластерные повторы). Эта задача, как правило, выполняется с помощью ДНК- (или РНК-) направленных эндонуклеаз, таких как Cas9. Специалисты могут направлять Cas9 и другие белки на любую последовательность-мишень нуклеиновой кислоты, содержащую мотив, смежный с протоспейсером (PAM) (например, последовательностью PAM Cas9 является NGG), просто путем конструирования направляющей РНК и вызвать двухцепочечный разрыв ДНК (или расщепление РНК).
Помимо редактирования геномов, CRISPR также может использоваться для регуляции действия гена, эпигенетического редактирования, скрининга функционального гена и геномной визуализации. В последнее время CRISPR, богатый возможностями набор инструментальных средств, начал появляться в области обнаружения нуклеиновой кислоты и привлек к себе внимание.
Первый метод обнаружения нуклеиновой кислоты на основе системы CRISPR основывался на белке dCas9 («мертвый» Cas9). dCas9 представляет собой мутацию Cas9 с двумя мутировавшими активными центрами каталитического расщепления. Таким образом, утрачивается двухцепочечная расщепляющая активность, но dCas9 сохраняет способность к наведению направляющей РНК и связыванию со специфичным участком ДНК. Чен (Chen) с соавторами использовали dCas9 для гибридизации с усиленным зеленым флуоресцентным белком (EGFP), который может наводиться специфичной направляющей РНК, а флуоресценцию на последовательности-мишени можно наблюдать через микроскоп. В 2017 г. Гук (Guk) с соавторами сообщил о системе ДНК-FISH на основе dCas9/sgRNA-SG I, которая может селективно обнаруживать метициллин-резистентный Staphylococcus aureus.
В 2013 г. Коллинз с командой создали способ обнаружения вируса Зика на листе бумаги. В этом способе используются процессы экстракции РНК, амплификации и реакции с детекцией на основе подхода Toehold. Согласно замыслу, если один штамм вируса отличается от другого штамма в последовательности сайта PAM (т.е. один является NGG, а другой отличается от него), штаммы, содержащие PAM, могут стать мишенью и могут быть расщеплены белком Cas9, таким образом влияя на процесс амплификации и детекции на основе подхода Toehold.
В последнее время суть другого способа обнаружения нуклеиновой кислоты на основе Cas9 заключается в изотермической амплификации, вызванной CRISPR/Cas9, которая также может использоваться для сайт-специфического обнаружения нуклеиновой кислоты. Этот способ использует расщепление CRISPR/Cas9, никазы и ДНК-полимеразы. Этот способ позволяет не только обнаруживать целевую концентрацию 0,82 моль, но также распознавать различие в одном основании в молекулах нуклеиновой кислоты. Распознавание целей в настоящем способе более универсально, чем в предыдущем способе.
Суть другого способа обнаружения нуклеиновой кислоты основана на эффекте «транс-расщепления» (или «обходного расщепления») некоторых белков Cas. В 2016 г. Чжан Фэн (Zhang Feng) с соавторами обнаружил, что белок Cas13a (ранее известный как C2c2) обладает обходной расщепляющей активностью. Другими словами, когда Cas13a связывается с последовательностью-мишенью РНК, он демонстрирует характеристики хаотичного разрезания других РНК, что используется для обнаружения специфической нуклеиновой кислоты, и этот способ называется SHERLOCK (Specific High Sensitivity Enzymatic Reporter UnLOCKing) (специфический высокочувствительный ферментативный репортер разблокировки).
Несмотря на то что есть множество способов обнаружения нуклеиновой кислоты на основе системы CRISPR, по-прежнему существует потребность в разработке более быстрых, простых и дешевых методов обнаружения нуклеиновой кислоты в этой области.
Сущность изобретения
Цель настоящего изобретения заключается в разработке более быстрых, простых и дешевых методов обнаружения нуклеиновой кислоты на основе системы CRISPR.
Первым аспектом настоящего изобретения является получение системы обнаружения целевых молекул нуклеиновой кислоты, которая включает:
(a) белок Cas12b, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
(b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и
(c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень.
В другом предпочтительном варианте осуществления система обнаружения включает качественное выявление или количественное выявление.
В другом предпочтительном варианте осуществления система обнаружения дополнительно содержит (d) буфер.
В другом предпочтительном варианте осуществления система обнаружения дополнительно содержит целевые молекулы нуклеиновой кислоты, подлежащие обнаружению.
В другом предпочтительном варианте осуществления система обнаружения дополнительно содержит:
(e1) полимеразу, которая используется для амплификации ДНК-мишени;
(e2) необязательную обратную транскриптазу, которая используется для обратной транскрипции;
(e3) dNTP, который используется для реакции амплификации и/или реакции обратной транскрипции.
В другом предпочтительном варианте осуществления система обнаружения дополнительно содержит реагенты, используемые для реакции петлевой изотермической амплификации (LAMP).
В другом предпочтительном варианте осуществления концентрация целевых молекул нуклеиновой кислоты, подлежащих обнаружению в системе обнаружения, составляет от 1×10-9 нМ до 1×103 нМ; предпочтительно от 1×10-8 нМ до 1×102 нМ.
В другом предпочтительном варианте осуществления концентрация целевых молекул нуклеиновой кислоты, подлежащих обнаружению в системе обнаружения, составляет от 1 до 100 копий/мкл или от 1 до 1×1015 копий/мкл, предпочтительно от 1 до 10 копий/мкл, более предпочтительно от 1 до 5 копий/мкл.
В другом предпочтительном варианте осуществления в системе обнаружения молярное отношение зонда на основе нуклеиновой кислоты к целевой молекуле нуклеиновой кислоты составляет от 103:1 до 1014:1, предпочтительно от 104:1 до 107:1.
В другом предпочтительном варианте осуществления длина направляющей РНК составляет от 16 до 25 нт, предпочтительно от 16 до 22 нт, более предпочтительно от 16 до 20 нт.
В другом предпочтительном варианте осуществления длина направляющей РНК составляет от 18 до 25 нт, предпочтительно от 18 до 22 нт, более предпочтительно от 18 до 20 нт.
В другом предпочтительном варианте осуществления, когда целевой молекулой нуклеиновой кислоты является одноцепочечная ДНК, сайт обнаружения целевой молекулы нуклеиновой кислоты находится в положении 9 или в положениях 10–16, предпочтительно в положениях 10–14, более предпочтительно в положениях 10–12 за последовательностью PAM направляющей РНК.
В другом предпочтительном варианте осуществления, когда сайт обнаружения находится в положении 9 за последовательностью PAM направляющей РНК, сайтом обнаружения является G.
В другом предпочтительном варианте осуществления сайт обнаружения находится в положениях 9–20, предпочтительно в положениях 10–18, более предпочтительно в положениях 10–16 за последовательностью PAM направляющей РНК.
В другом предпочтительном варианте осуществления сайт обнаружения целевой молекулы нуклеиновой кислоты находится в положениях 1–12, более предпочтительно в положении 1, 3 или 10 за последовательностью PAM направляющей РНК.
В другом предпочтительном варианте осуществления длина направляющей РНК составляет 15–30 нт, предпочтительно 15–18 нт.
В другом предпочтительном варианте осуществления ДНК-мишень содержит ДНК, образованную на основе обратной транскрипции РНК.
В другом предпочтительном варианте осуществления ДНК-мишень содержит кДНК.
В другом предпочтительном варианте осуществления ДНК-мишень выбирается из группы, состоящей из одноцепочечной ДНК, двухцепочечной ДНК и их комбинации.
В другом предпочтительном варианте осуществления зонд на основе нуклеиновой кислоты имеет флуоресцентную группу и группу гашения.
В другом предпочтительном варианте осуществления и флуоресцентная группа, и группа гашения независимо находятся на 5'-конце, 3'-конце и посередине зонда на основе нуклеиновой кислоты.
В другом предпочтительном варианте осуществления длина зонда на основе нуклеиновой кислоты составляет от 3 до 300 нт, предпочтительно от 5 до 100 нт, более предпочтительно от 6 до 50 нт, наиболее предпочтительно от 8 до 20 нт.
В другом предпочтительном варианте осуществления целевые молекулы нуклеиновой кислоты содержат целевые молекулы нуклеиновой кислоты, полученные из видов, выбранных из группы, состоящей из: растения, животного, насекомого, микроорганизма, вируса и их комбинации.
В другом предпочтительном варианте осуществления ДНК-мишень представляет собой синтетическую или естественно присутствующую ДНК.
В другом предпочтительном варианте осуществления ДНК-мишень содержит ДНК дикого типа или мутантную ДНК.
В другом предпочтительном варианте осуществления ДНК-мишень содержит ДНК, полученную обратной транскрипцией РНК или амплификацией, например кДНК и т.п.
В другом предпочтительном варианте осуществления белок Cas12b выбирается из группы, состоящей из: AacCas12b (Alicyclobacillus acidoterrestris), Aac2Cas12b (Alicyclobacillus acidiphilus), AkaCas12b (Alicyclobacillus kakegawensis), AmaCas12b (Alicyclobacillus macrosporangiidus), AheCas12b (Alicyclobacillus herbarius) и AcoCas12b (Alicyclobacillus contaminans).
В другом предпочтительном варианте осуществления зонд на основе нуклеиновой кислоты содержит одноцепочечную ДНК с детектируемой меткой.
В другом предпочтительном варианте осуществления одноцепочечная ДНК представляет собой одноцепочечную ДНК, меченную флуоресцентной меткой и биотином.
В другом предпочтительном варианте осуществления одноцепочечная ДНК представляет собой одноцепочечную ДНК, меченную флуоресцентной меткой.
В другом предпочтительном варианте осуществления одноцепочечная ДНК представляет собой флуоресцентный зонд, меченный флуоресцентной группой HEX на 5'-конце и группой гашения BHQ1 на 3'-конце.
Вторым аспектом настоящего изобретения является получение системы обнаружения SNPs (одиночных нуклеотидных полиморфизмов) или нуклеотидных мутаций (содержащих одноосновные или многоосновные мутации), причем система содержит первую систему обнаружения и вторую систему обнаружения;
в которой первая система обнаружения включает:
(a1) белок Cas12b, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
(b1) первую направляющую РНК, причем первая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и
(c1) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
а вторая система обнаружения включает:
(a2) белок Cas12b, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
(b2) вторую направляющую РНК, причем вторая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и
(c2) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень;
кроме того, первая направляющая РНК и вторая направляющая РНК нацелены на одну и ту же область последовательности нуклеиновой кислоты, содержащую SNP-сайт, а первая направляющая РНК нацелена на последовательность нуклеиновой кислоты дикого типа (или немутированной) SNP-сайта, а вторая направляющая РНК нацелена на последовательность мутированной нуклеиновой кислоты SNP-сайта.
В другом предпочтительном варианте осуществления система обнаружения также содержит систему холостой пробы.
В другом предпочтительном варианте осуществления система холостой пробы включает:
(a3) белок Cas12b, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
(c3) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК.
В другом предпочтительном варианте осуществления SNP-сайт находится в положении 9 или в положениях 10–16, предпочтительно в положениях 10–14, более предпочтительно в положениях 10–12 за последовательностью PAM направляющей РНК.
В другом предпочтительном варианте осуществления, когда SNP-сайт находится в положении 9 за последовательностью PAM направляющей РНК, а сайтом обнаружения является G.
В другом предпочтительном варианте осуществления SNP-сайт находится в положениях 9–20, предпочтительно в положениях 10–18, более предпочтительно в положениях 10–16 за последовательностью PAM направляющей РНК.
В другом предпочтительном варианте осуществления длина первой или второй направляющей РНК составляет от 16 до 25 нт, предпочтительно от 16 до 22 нт, более предпочтительно от 16 до 20 нт.
В другом предпочтительном варианте осуществления длина первой или второй направляющей РНК составляет от 18 до 25 нт, предпочтительно от 18 до 22 нт, более предпочтительно от 18 до 20 нт.
Третьим аспектом настоящего изобретения является получение набора для обнаружения целевых молекул нуклеиновой кислоты, включающего:
i) первый контейнер и белок Cas12b, находящийся в первом контейнере, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
ii) необязательный второй контейнер и вторую направляющую РНК, находящуюся во втором контейнере, причем вторая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты;
iii) третий контейнер и зонд на основе нуклеиновой кислоты, находящийся в третьем контейнере;
iv) необязательный четвертый контейнер и буфер, находящийся в четвертом контейнере;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень.
В другом предпочтительном варианте осуществления любые два, три или четыре (или все) из первого, второго, третьего и четвертого контейнеров могут быть одним и тем же контейнером или разными контейнерами.
В другом предпочтительном варианте осуществления зонд на основе нуклеиновой кислоты имеет флуоресцентную группу и группу гашения.
В другом предпочтительном варианте осуществления набор дополнительно содержит буфер.
В другом предпочтительном варианте осуществления набор дополнительно содержит:
v) пятый контейнер и полимеразу для амплификации ДНК-мишени, находящейся в пятом контейнере;
vi) необязательный шестой контейнер и обратную транскриптазу для обратной транскрипции, находящуюся в шестом контейнере;
vii) седьмой контейнер и dNTP для реакции амплификации и/или реакции обратной транскрипции, находящийся в седьмом контейнере.
В другом предпочтительном варианте осуществления система обнаружения дополнительно содержит реагенты, используемые для реакции петлевой изотермической амплификации (LAMP).
В другом предпочтительном варианте осуществления пятый контейнер, шестой контейнер и седьмой контейнер могут быть одним и тем же контейнером или разными контейнерами.
В другом предпочтительном варианте осуществления два, более двух или все с первого контейнера по седьмой контейнер могут быть одним и тем же контейнером или разными контейнерами.
Четвертым аспектом настоящего изобретения является разработка способа обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, который включает следующие этапы:
(i) получение системы обнаружения целевых молекул нуклеиновой кислоты по первому аспекту настоящего изобретения, и система обнаружения дополнительно содержит образец, подлежащий обнаружению; и
(ii) выяснение, расщеплен ли зонд на основе нуклеиновой кислоты в системе обнаружения белком Cas12b, причем расщепление представляет собой транс-расщепление обходной одноцепочечной ДНК;
при этом, если зонд на основе нуклеиновой кислоты расщепляется белком Cas12b, это указывает на то, что целевая молекула нуклеиновой кислоты присутствует в образце; а если зонд на основе нуклеиновой кислоты не расщепляется белком Cas12b, это указывает на указывает на отсутствие целевой молекулы нуклеиновой кислоты в образце.
В другом предпочтительном варианте осуществления белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b.
В другом предпочтительном варианте осуществления образец, подлежащий обнаружению, содержит неамплифицированный образец и амплифицированный (или амплифицированную нуклеиновую кислоту) образец.
В другом предпочтительном варианте осуществления образец, подлежащий обнаружению, представляет собой образец, получаемый путем амплификации.
В другом предпочтительном варианте осуществления способ амплификации нуклеиновой кислоты выбирается из группы, состоящей из таких: ПЦР-амплификация, петлевая изотермическая амплификация (LAMP), рекомбиназная полимеразная амплификация (RPA), лигазная цепная реакция, амплификация методом разветвленной ДНК, амплификация, основанная на последовательности нуклеиновых кислот (NASBA), амплификация с замещением цепей (SDA), транскрипционно-опосредованная амплификация, амплификация по типу катящегося кольца, геликаза-зависимая амплификация (HDA), изотермическая амплификация с одним праймером (SPIA), амплификация с использованием никирующих ферментов (NEAR), транскрипционно-опосредованная амплификация (TMA) и амплификация SMAP2.
В другом предпочтительном варианте осуществления ПЦР содержит высокотемпературную стадию ПЦР, ПЦР при нормальной температуре или низкотемпературную стадию ПЦР.
В другом предпочтительном варианте осуществления этот способ используется для выявления того, что присутствует на сайте-мишени — одиночный нуклеотидный полиморфизм (SNP), точечная мутация, делеция и/или вставка в нуклеиновые кислоты.
В другом предпочтительном варианте осуществления, когда PAM-последовательность отсутствует перед сайтом-мишенью или после него (в диапазоне от -20 нт до +20 нт, предпочтительно в диапазоне от -16 нт до +16 нт), амплификация нуклеиновой кислоты выполнялась с использованием праймеров, вводимых с помощью PAM.
В другом предпочтительном варианте осуществления праймер, вводимый с помощью PAM, имеет структурную формулу I от 5' до 3':
P1-P2-P3 (I)
где,
P1 — последовательность 5' сегментов на 5'-конце, которая комплементарна или не комплементарна по отношению к последовательности целевой молекулы нуклеиновой кислоты;
P2 — PAM-последовательность;
P3 — последовательность 3' сегментов на 3'-конце, которая комплементарна по отношению к последовательности целевой молекулы нуклеиновой кислоты.
В другом предпочтительном варианте осуществления PAM-праймер осуществляет специфическое связывание перед целевой молекулой нуклеиновой кислоты и после нее.
В другом предпочтительном варианте осуществления P1 имеет длину 0–20 нт.
В другом предпочтительном варианте осуществления P3 имеет длину от 5 до 20 нт.
В другом предпочтительном варианте осуществления PAM-праймер имеет длину от 16 до 50 нт, предпочтительно от 20 до 35 нт.
В другом предпочтительном варианте осуществления комплементация содержит полную комплементацию и частичную комплементацию.
В другом предпочтительном варианте осуществления, по меньшей мере, один праймер с РАМ-последовательностью используется при амплификации нуклеиновой кислоты.
В другом предпочтительном варианте осуществления, когда PAM-последовательность присутствует перед сайтом-мишенью или после него (в диапазоне от -20 нт до +20 нт, предпочтительно в диапазоне от -15 нт до +15 нт, более предпочтительно в диапазоне от -10 нт до +10 нт), могут использоваться праймеры с PAM-последовательностью или без нее, и продукт амплификации содержит PAM-последовательность.
В другом предпочтительном варианте осуществления обнаружение на этапе (ii) содержит флуоресцентный способ детектирования.
В другом предпочтительном варианте осуществления микропланшетный ридер или флуоресцентный спектрофотометр используется при флуоресцентном способе детектирования.
Пятым аспектом настоящего изобретения является получение способа обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, который содержит следующие этапы:
(i) получение системы обнаружения, которая содержит:
(a) белок Cas12b, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
(b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты;
(c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень;
(d) буфер;
(e1) полимеразу, которая используется для амплификации ДНК-мишени;
(e2) необязательную обратную транскриптазу, которая используется для обратной транскрипции;
(e3) dNTP, который используется для реакции амплификации и/или реакции обратной транскрипции; и
(f) образец для тестирования;
(ii) выполнение реакции обратной транскрипции и/или амплификации в системе обнаружения, получая таким образом обратно транскрибированную и/или амплифицированную систему обнаружения;
(iii) выяснение, расщеплен ли зонд на основе нуклеиновой кислоты в системе обнаружения, полученной на предыдущем этапе, белком Cas12b, причем расщепление представляет собой транс-расщепление обходной одноцепочечной ДНК;
при этом, если зонд на основе нуклеиновой кислоты расщепляется белком Cas12b, это указывает на то, что целевая молекула нуклеиновой кислоты присутствует в образце; а если зонд на основе нуклеиновой кислоты не расщепляется белком Cas12b, это указывает на указывает на отсутствие целевой молекулы нуклеиновой кислоты в образце.
В другом предпочтительном варианте осуществления на этапе (ii) температура системы обнаружения поддерживается на уровне 50–70°C, предпочтительно 50–65°C, более предпочтительно 55–65°C.
В другом предпочтительном варианте осуществления на этапе (iii) температура системы обнаружения поддерживается на уровне 25–70°C, предпочтительно 48–65°C.
В другом предпочтительном варианте осуществления способ амплификации нуклеиновой кислоты выбирается из группы, состоящей из таких: ПЦР-амплификация, петлевая изотермическая амплификация (LAMP), рекомбиназная полимеразная амплификация (RPA), лигазная цепная реакция, амплификация методом разветвленной ДНК, амплификация, основанная на последовательности нуклеиновых кислот (NASBA), амплификация с замещением цепей (SDA), транскрипционно-опосредованная амплификация, амплификация по типу катящегося кольца, геликаза-зависимая амплификация (HDA), изотермическая амплификация с одним праймером (SPIA), амплификация с использованием никирующих ферментов (NEAR), транскрипционно-опосредованная амплификация (TMA) и амплификация SMAP2.
В другом предпочтительном варианте осуществления амплификация нуклеиновой кислоты содержит LAMP-амплификацию.
В другом предпочтительном варианте осуществления в системе обнаружения концентрация нуклеиновой кислоты в образце, подлежащем тестированию, составляет от 1×10-11 нМ до 1×10-5 нМ, предпочтительно от 1×10-9 нМ до 1×10-6 нМ, более предпочтительно от 1×10-8 нМ до 1×10-7 нМ.
В другом предпочтительном варианте осуществления система обнаружения содержит качественное выявление или количественное выявление.
В другом предпочтительном варианте осуществления количественное выявление представляет собой абсолютное количественное выявление (например, количественное выявление в сочетании с технологией цифровой ПЦР).
В другом предпочтительном варианте осуществления общее время этапов (ii) и (iii) составляет ≤2 часов, предпочтительно ≤1,5 часа, более предпочтительно ≤1 часа (например, 30–60 минут).
В другом предпочтительном варианте осуществления целевая молекула нуклеиновой кислоты представляет собой последовательность метилированной нуклеиновой кислоты, или последовательность метилированной нуклеиновой кислоты представляет собой последовательность нуклеиновой кислоты, полученной после преобразования неметилированного С в урацил.
В другом предпочтительном варианте осуществления последовательность метилированной нуклеиновой кислоты обрабатывается бисульфитом для преобразования неметилированного С в урацил.
В другом предпочтительном варианте осуществления целевой молекулой нуклеиновой кислоты является молекула ДНК, полученная путем обратной транскрипции молекулы линейной или кольцевой РНК, или молекула ДНК, полученная путем ПЦР с обратной транскрипцией.
В другом предпочтительном варианте осуществления ПЦР с обратной транскрипцией содержит LAMP с обратной транскрипцией.
Шестым аспектом настоящего изобретения является применение белка Cas12b для подготовки реагента или набора для обнаружения целевых молекул нуклеиновой кислоты на основании обходной расщепляющей активности одноцепочечной ДНК, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b.
В другом предпочтительном варианте осуществления белок Cas12b выбирается из группы, состоящей из: AacCas12b (Alicyclobacillus acidoterrestris), Aac2Cas12b (Alicyclobacillus acidiphilus), AkaCas12b (Alicyclobacillus kakegawensis), AmaCas12b (Alicyclobacillus macrosporangiidus), AheCas12b (Alicyclobacillus herbarius) и AcoCas12b (Alicyclobacillus contaminans).
Седьмым аспектом настоящего изобретения является получение устройства обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, причем устройство содержит:
(a) модуль реакции амплификации — реакции обходного расщепления, который используется для выполнения реакций амплификации нуклеиновой кислоты и реакций обходного расщепления в цифровой реакционной системе, в которой реакция обходного расщепления опосредована белком Cas12b; и
(b) модуль обнаружения сигнала, который используется для выявления того, происходит ли реакция обходного расщепления, опосредованная белком Cas12b, в каждой цифровой реакционной системе.
В другом предпочтительном варианте осуществления реакция амплификации нуклеиновой кислоты и реакция обходного расщепления выполняются одновременно.
В другом предпочтительном варианте осуществления модуль реакции амплификации — реакции обходного расщепления дополнительно содержит устройство для контроля температуры, предназначенное для настройки температуры цифровой реакционной системы в устройстве при заранее заданной температуре.
В другом предпочтительном варианте осуществления заранее заданная температура составляет 50–70°C, предпочтительно 50–65°C, более предпочтительно 55–65°C.
В другом предпочтительном варианте осуществления заранее заданная температура составляет 25–70°C, предпочтительно 48–65°C.
В другом предпочтительном варианте осуществления заранее заданная температура практически такая же на протяжении всей реакции амплификации и реакции обходного расщепления.
В другом предпочтительном варианте осуществления диапазон колебания заранее заданной температуры составляет ±5°C, предпочтительно ±3°C, более предпочтительно ±1°C.
В другом предпочтительном варианте осуществления амплификация нуклеиновой кислоты представляет собой изотермическую амплификацию или амплификацию, в которой разница температур при денатурации-ренатурации-удлинении составляет ≤10°C (предпочтительно ≤5°C, более предпочтительно ≤3°C).
В другом предпочтительном варианте осуществления амплификация нуклеиновой кислоты содержит LAMP-амплификацию.
В другом предпочтительном варианте осуществления цифровая реакционная система содержит реакционную систему цифровой капельной ПЦР (ddPCR) или реакционную систему цифровой ПЦР на чипах (cdPCR).
В другом предпочтительном варианте осуществления цифровая реакционная система содержит несколько независимых устройств реакционной системы, и каждое устройство реакционной системы содержит:
(a) белок Cas12b, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
(b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты;
(c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень;
(d) буфер;
(e1) полимеразу, которая используется для амплификации ДНК-мишени;
(e2) необязательную обратную транскриптазу, которая используется для обратной транскрипции;
(e3) dNTP, который используется для реакции амплификации и/или реакции обратной транскрипции; и
(f) образец для тестирования;
при этом в связи с обработкой при разведении (т.е. цифровой обработкой) каждая независимая реакционная система содержит либо 1 копию нуклеиновой кислоты из образца, подлежащего тестированию, либо 0 копий нуклеиновой кислоты из образца, подлежащего тестированию.
В другом предпочтительном варианте осуществления каждое устройство реакционной системы одинаково, за исключением 1 или 0 копий нуклеиновой кислоты из образца, подлежащего тестированию.
В другом предпочтительном варианте осуществления устройство реакционной системы представляет собой каплю.
В другом предпочтительном варианте осуществления устройство реакционной системы представляет собой систему микрореакции, находящуюся в микропорах чипа.
В другом предпочтительном варианте осуществления устройство дополнительно содержит модуль загрузки образца и/или модуль управления.
В другом предпочтительном варианте осуществления модуль обнаружения сигнала содержит модуль визуализации.
В другом предпочтительном варианте осуществления модуль визуализации содержит устройство флуоресцентного детектирования.
В другом предпочтительном варианте осуществления устройство флуоресцентного детектирования облучает цифровую реакционную систему, чтобы возбудить реакционную систему для создания флуоресцентного сигнала и преобразует флуоресцентный сигнал в цифровой сигнал, который предпочтительно направляется в модуль управления.
В другом предпочтительном варианте осуществления модуль управления выполняет арифметический анализ на основе количества флуоресцентных сигналов (в предпочтительном варианте цифровых сигналов, преобразованных из флуоресцентных сигналов) и общего количества устройств реакционной системы, получая, таким образом, результат количественного выявления целевой нуклеиновой кислоты (например, концентрация или число копий).
В другом предпочтительном варианте осуществления устройство представляет собой цифровое ПЦР-устройство обнаружения.
Следует понимать, что в рамках настоящего изобретения различные вышеуказанные технические признаки настоящего изобретения и различные технические признаки, описанные далее (например, в вариантах осуществления), могут быть объединены друг с другом с целью создания нового или предпочтительного технического решения. Вследствие ограниченного объема не повторяется в настоящем документе.
Описание фигур
На фиг. 1 показана интенсивность флуоресценции, создаваемая транс-расщеплением зондов различной концентрацией Cas12b и одиночной направляющей РНК при использовании двухцепочечной ДНК и одноцепочечной ДНК в качестве ДНК-мишени соответственно.
На фиг. 2 показано влияние различных оснований в PAM-последовательности на интенсивность флуоресцентного детектирования транс-расщепления при использовании двухцепочечной ДНК и одноцепочечной ДНК в качестве ДНК-мишени соответственно.
На фиг. 3 показана скорость транс-расщепления белком Cas12b при использовании двухцепочечной ДНК или одноцепочечной ДНК в качестве мишени.
На фиг. 4 показана интенсивность флуоресценции транс-расщепления белком Cas12b при использовании двухцепочечной ДНК или одноцепочечной ДНК в качестве мишени в различных температурных режимах.
На фиг. 5 показана чувствительность обнаружения белком Cas12b (т.е. интенсивность флуоресценции транс-расщепления) при использовании двухцепочечной ДНК или одноцепочечной ДНК в качестве мишени в ходе градиентного разбавления последовательности-мишени.
На фиг. 6 показана чувствительность обнаружения белком Cas12b в сочетании с реакцией LAMP-амплификации (т.e. способ HOLMES v2.0) на мишень.
На фиг. 2 показано влияние одноосновных мутаций в положениях 1–12 последовательности-мишени одноцепочечной ДНК на интенсивность флуоресценции транс-расщепления белком Cas12b при использовании одиночных направляющих РНК с различной длиной направляющей последовательности.
На фиг. 8 показано влияние одноосновных мутаций в положениях 1–16 последовательности-мишени одноцепочечной ДНК на интенсивность флуоресценции транс-расщепления белком Cas12b при использовании одиночной направляющей РНК с направляющей последовательностью длиной 16 нт.
На фиг. 9 показано влияние одноосновных мутаций в любое из трех других оснований в положениях 1–16 последовательности-мишени одноцепочечной ДНК на интенсивность флуоресценции транс-расщепления белком Cas12b при использовании одиночной направляющей РНК с направляющей последовательностью длиной 16 нт.
На фиг. 10 показано влияние одноосновных мутаций в положениях 8–16 трех различных последовательностей-мишеней одноцепочечной ДНК (rs5082, rs1467558 и rs2952768) на интенсивность флуоресценции транс-расщепления белком Cas12b при использовании одиночной направляющей РНК с направляющей последовательностью длиной 16 нт.
На фиг. 11 показано влияние одноосновных мутаций в любом из положений 1–16 последовательности-мишени двухцепочечной ДНК на интенсивность флуоресценции транс-расщепления белком Cas12b при использовании одиночных направляющих РНК с различной длиной направляющей последовательности.
На фиг. 12 показана одноступенчатая реакция LAMP и обнаружение белком Cas12b при 50°C, 55°C, 60°C и 65°C.
На фиг. 13 показаны результаты тестов HOLMES v2.0 (LAMP в комбинации с белком Cas12b) для сайта gyrB E. coli с целью выявления присутствия E. coli и чувствительность обнаружения в ходе теста на градиентное разбавление. (После теста на покрытие число 0,005 OD E. coli составило около 7000)
На фиг. 14 показаны результаты тестов HOLMES v2.0 (LAMP в комбинации с белком Cas12b) для определяющего пол участка Y-хромосомы при использовании образцов слюны с целью установления пола (локус DNMT1-3 представляет собой генный локус на хромосоме, который использовался в качестве положительного контроля).
На фиг. 15 показано обнаружение SNP-сайта (rs5082) амплификацией асимметричной или симметричной ПЦР в комбинации с белком Cas12b. Видно, что, поскольку возле сайта rs5082 нет PAM-последовательности, асимметричная ПЦР, которая производит одноцепочечную ДНК, может более четко распознавать SNP-сайты.
На фиг. 16 показано обнаружение SNP-сайта (rs5082) путем LAMP-амплификации в комбинации с белком Cas12b, при этом были сконструированы праймеры и введена PAM-последовательность. Видно, что, поскольку возле сайта rs5082 нет PAM-последовательности, способ LAMP-амплификации, применяемый с PAM (с PAM-последовательностью в праймере), позволяет лучше распознавать SNP-сайты.
На фиг. 17 показано обнаружение вируса JEV (вирус японского энцефалита) РНК способом HOLMES v2.0 (LAMP в комбинации с белком Cas12b).
На фиг. 18 показано одноэтапное количественное определение следовой ДНК способом HOLMES v2.0 (LAMP в комбинации с белком Cas12b). С разбавлением матричной ДНК на различные концентрации одноэтапный способ LAMP-Cas12b использовался для выявления значения флуоресценции в режиме реального времени в флуорометре (реакция при 55°C).
На фиг. 19 показан примерный линейный тренд с использованием момента времени, когда значение флуоресценции достигает 600,000, как ось y (с использованием данных на фиг. 18), и с использованием абсолютного значения концентрации lg как ось х. Это уравнение можно использовать для количественного определения последовательности-мишени.
На фиг. 20 показано абсолютное количественное выявление образцов нуклеиновой кислоты с использованием цифрового метода HOLMES (LAMP в комбинации с белком Cas12b и чипом прозрачности).
На фиг. 21 показана схема обнаружения способом HOLMESv2 метилирования ДНК-мишени. После бисульфитной конверсии и ПЦР-амплификации основание C в неметилированном сайте CpG преобразуется в T. путем конструирования направляющей последовательности одиночной РНК, которая в точности совпадает с метилированным сайтом-мишенью, реакция HOLMESv2 (а именно, с добавлением белка Cas12b, одиночной РНК и зонда одноцепочечной ДНК, меченного флуоресцентной группой на одном конце и группой гашения на другом конце) использовалась для распознавания содержания метилированных сайтов в системе. Аналогичным образом также можно сконструировать направляющую последовательность одиночной РНК, которая в точности совпадает с неметилированным сайтом-мишенью, и использовать реакцию HOLMESv2 для распознавания содержания неметилированных сайтов в системе, а затем можно рассчитать соотношение метилированных сайтов.
На фиг. 22 показаны экспериментальные результаты обнаружения метилирования ДНК-мишени при использовании одиночных направляющих РНК с различными сайтами и различной длиной. Мишень-C означает результат тестирования при положении С (представляющем метилирование); тогда как мишень-T означает результат тестирования при положении T (представляющем неметилированное основание перед бисульфитной конверсией). Если последовательность одиночной направляющей РНК имеет высокое значение сигнала при нацеливании на мишень-C и низкое значение сигнала при нацеливании на мишень-T, последовательность одиночной направляющей РНК может использоваться для распознавания того, обладает ли сайт способностью к модификации метилирования.
На фиг. 23 показана схема обнаружения метилирования ДНК-мишени при использовании одиночной направляющей РНК при m3-C12-17. На левой картинке видно, что последовательность одиночной направляющей РНК полностью совпадает с последовательностью-мишенью; тогда как на правой картинке видно, что после бисульфитной конверсии и ПЦР-амплификации неметилированного сайта сайт М3 преобразуется в T, что приводит к несовпадению последовательности одиночной направляющей РНК с последовательностью-мишенью.
На фиг. 24 показаны результаты тестирования уровня метилирования сайта M3 CpG (COL1A2) в 4 клеточных линиях. Результаты 0%, 10%, 30% и 50% являются результатами тестирования для составления градуировочной кривой. 293T, SW480, NCI-N87 и MCF-7 являются фактическими тестовыми значениями в данном месте в 4 клеточных линиях. Слева направо показаны 3 независимых повторных эксперимента, и для каждого эксперимента были проведены 3 повторных теста.
На фиг. 25 показана градуировочная кривая уровня метилирования сайта M3 CpG (COL1A2). Диаграмма градуировочной кривой составлена на основании результата проверки градуировочной кривой на фиг. 5.
На фиг. 26 показаны результаты тестирования скорости метилирования сайта M3 CpG (COL1A2) в четырех клеточных линиях (293T, SW480, NCI-N87 и MCF-7). Как видно из фигуры, результат тестирования с помощью системы HOLMESv2 наиболее близок к результату секвенирования NGS (высокопроизводительное секвенирование).
На фиг. 27 показаны результаты обнаружения кольцевой РНК с использованием способа HOLMESv2. На верхней фигуре показана схема, изображающая обнаружение РНК с использованием способа HOLMESv2 и выбор сайта-мишени. На нижней левой фигуре показана схема, изображающая обнаружение целевого GAPDH с использованием способа HOLMESv2. На нижней правой фигуре показана схема, изображающая обнаружение целевого CDR1as с использованием способа HOLMESv2. Сайт-мишень означает сайт-мишень. NTC — это экспериментальная контрольная группа, в которой в качестве образца используется стерильная вода. Мишень — это экспериментальная группа, в которой в качестве образца используется образец РНК. Значения флуоресценции на нижней левой и нижней правой диаграммах представляют собой значения флуоресценции после вычитания экспериментальной фоновой флуоресценции. Обе группы реакции и NTC целевая группа были амплифицированы реакцией LAMP, и амплифицированные продукты использовались для теста на реакцию Cas12b; а фоновая флуоресценция представляла собой значение флуоресценции реакционной системы Cas12b, тестируемой добавлением непосредственно стерильной воды.
Подробное описание
В ходе широких и тщательных исследований авторы настоящего изобретения впервые разработали техническое решение по обнаружению целевой нуклеиновой кислоты путем изучения расщепляющих свойств ферментов Cas12b. Экспериментальные результаты показывают, что техническое решение по настоящему изобретению позволяет быстро, с высокой чувствительностью и точно обнаруживать нуклеиновую кислоту и что оно может применяться в различных областях, например, для быстрой идентификации пола с помощью образцов слюны, обнаружения заражения кишечной палочкой E. coli, быстрой идентификации генотипов SNP, обнаружения вируса РНК и определения концентрации следовой ДНК в образце. На этой основе было создано настоящее изобретение.
Термины
Термин «направляющая РНК», или «gRNA», или «sgRNA» (одиночная направляющая РНК) означает РНК, которая направляет белок Cas (например, белок Cas12b) для специфического связывания с последовательностью ДНК-мишени.
Термин «CRISPR» означает короткие палиндромные кластерные повторы, разделенные регулярными промежутками, которые представляют собой иммунные системы многих прокариотов.
Термин «белок Cas» означает CRISPR-ассоциированный белок, который представляет собой белок, связанный с системой CRISPR.
Термин «Cas12a» (ранее «Cpf1») означает крРНК-зависимую эндонуклеазу, которая представляет собой фермент типа V-A в классификации системы CRISPR.
Термины «Cas12b», «C2c1» используются взаимозаменяемо и означают sgRNA-зависимую эндонуклеазу, которая представляет собой фермент типа V-В в классификации системы CRISPR.
Термин «LAMP» означает способ петлевой изотермической амплификации и способ термостатической амплификации нуклеиновой кислоты, подходящий для генодиагностики.
Термин «РАМ» означает мотив, примыкающий к протоспейсеру, который необходим для расщепления белком Cas12b двухцепочечной ДНК, а PAM AacCas12b представляет собой последовательность TTN.
Термин «ПЦР» означает «полимеразная цепная реакция», способ, используемый для амплификации большого количества интересующих фрагментов ДНК.
HOLMES v2.0 означает одночасовую низкозатратную многоцелевую высокоэффективную систему версии 2.0, которая является способом обнаружения нуклеиновой кислоты на основе белка Cas12b.
Способ обнаружения
В настоящем изобретении предложен способ обнаружения целевых молекул нуклеиновой кислоты, который включает добавление направляющей РНК, белка Cas, зонда на основе нуклеиновой кислоты и буфера в реакционную систему, содержащую целевые молекулы нуклеиновой кислоты, а затем определение интенсивности флуоресценции.
В настоящем изобретении предложен способ быстрого обнаружения целевых молекул нуклеиновой кислоты с высокой специфичностью. После того как ДНК-мишень (одноцепочечная или двухцепочечная), одиночная направляющая РНК и белок Cas12b образуют тройной комплекс, этот комплекс расщепляет молекулы одноцепочечной ДНК в системе.
В настоящем способе на ДНК-мишень (последовательность ДНК, подлежащая обнаружению) нацелена сконструированная одиночная направляющая РНК; и одиночная направляющая РНК и белок Cas12b добавляются в систему обнаружения. Когда присутствует ДНК-мишень (одноцепочечная ДНК или двухцепочечная ДНК), белок Cas12b, одиночная направляющая РНК и ДНК-мишень образуют тройной комплекс, и этот комплекс расщепляет зонд на основе нуклеиновой кислоты, меченный флуоресцентным сигналом, используя свою обходную расщепляющую активность, излучая таким образом флуоресценцию.
Репрезентативный зонд на основе нуклеиновой кислоты представляет собой одноцепочечную ДНК с флуоресцентной группой и группой гашения, присоединенной к обоим концам, поэтому после расщепления зонда флуоресцентная группа может излучать свет.
В настоящем изобретении путем обнаружения флуоресценции можно узнать, содержится ли молекула ДНК-мишени в системе, подлежащей обнаружению.
В настоящем изобретении подходящим белком Cas является Cas12b, а предпочтительным белком Cas12b является: AacCas12b (Alicyclobacillus acidoterrestris), Aac2Cas12b (Alicyclobacillus acidiphilus), AkaCas12b (Alicyclobacillus kakegawensis), AmaCas12b (Alicyclobacillus macrosporangiidus), AheCas12b (Alicyclobacillus herbarius) или AcoCas12b (Alicyclobacillus contaminans).
Целевые молекулы нуклеиновой кислоты, подлежащие обнаружению в реакционной системе, содержащей целевые молекулы нуклеиновой кислоты, подлежащие обнаружению, могут быть неамплифицированными или быть полученными после амплификации и/или после амплификации с обратной транскрипцией.
Способ обнаружения по настоящему изобретению может выявлять молекулы нуклеиновой кислоты различных видов, например, молекулы нуклеиновой кислоты млекопитающих, растений, микроорганизмов и вирусов. Способ по настоящему изобретению, в частности, подходит для обнаружения болезнетворных микроорганизмов, генных мутаций или специфических ДНК- или РНК-мишеней.
Способ по настоящему изобретению позволяет быстро выяснять, содержится ли в образце специфическая последовательность ДНК. Кроме того, при сочетании с методом амплификации (например, LAMP, ПЦР, асимметричная ПЦР, RPA и т.д.) чувствительность способа обнаружения может быть значительно повышена.
В предпочтительном варианте обходное расщепление Cas12b можно скомбинировать с амплификацией нуклеиновой кислоты (например, изотермической амплификацией, LAMP-амплификацией) и другими методами (например, цифровой ПЦР).
В предпочтительном варианте осуществления настоящего изобретения, когда обнаружение на основе обходного расщепления Cas12b комбинируется с амплификацией нуклеиновой кислоты, чувствительность обнаружения может быть повышена до концентрации 10-8 нМ или ниже.
Кроме того, когда обнаружение на основе обходного расщепления Cas12b комбинируется с цифровой ПЦР, чувствительность обнаружения может быть повышена до 1 копии на реакционную систему (например, микрокапля), что отвечает практически всем потребностям чувствительности обнаружения.
Способ обнаружения нуклеиновой кислоты на основе белка Cas12b и HOLMES v2.0 (одночасовая низкозатратная многоцелевая высокоэффективная система версии 2.0)
Создание способов обнаружения нуклеиновой кислоты
С использованием характеристик белка Cas12b в настоящем изобретении разработан способ специфического обнаружения молекул нуклеиновой кислоты, который называется HOLMES (одночасовая низкозатратная многоцелевая высокоэффективная система версии 2.0). Как видно из названия, это быстрый (1 час), низкозатратный, многоцелевой, эффективный и простой способ тестирования.
Во всей реакционной системе его можно разделить на два больших этапа. Один этап — это амплификация матричной нуклеиновой кислоты, а другой — специфическое обнаружение нуклеиновой кислоты с помощью белка Cas12b. В альтернативном варианте два этапа могут быть объединены в один этап.
В настоящем изобретении, в предпочтительном варианте осуществления, для амплификации нуклеиновых кислот используется метод LAMP или асимметричная ПЦР. Но на практике любой метод амплификации можно объединить с обнаружением нуклеиновой кислоты второго этапа, например метод изотермической амплификации RPA или подобный метод.
Исходная нуклеиновая кислота не ограничивается двухцепочечной ДНК и может быть одноцепочечной ДНК или РНК, и, таким образом, этот способ применим к различным типам молекул нуклеиновой кислоты.
На этапе обнаружения нуклеиновой кислоты три компонента играют важную роль для эксперимента, а именно: белок Cas12b, одиночная направляющая РНК и зонд на основе нуклеиновой кислоты. Помимо AacCas12b, упомянутого в примерах, другие белки Cas12b одинаково подходят для настоящего способа. Кроме того, другие типы белков Cas (например, белок Cas12c) также входят в объем правовой охраны настоящего изобретения.
Что касается одиночной направляющей РНК в качестве гида, то после искусственной модификации и других модификаций она будет более устойчива в системе. При выборе зондов на основе нуклеиновой кислоты в настоящем изобретении выбрана короткая одноцепочечная ДНК, меченная группой HEX и BHQ1 (также меченная FAM и эклипсом), и любой другой метод с детектируемой меткой теоретически применим при условии, что зонд на основе нуклеиновой кислоты после расщепления может создавать обнаруживаемое различие. В качестве альтернативы зонд на основе нуклеиновой кислоты также может быть сконструирован для излучения флуоресценции после связи с соединением, чтобы определить, расщеплен зонд или нет.
С целью облегчения понимания далее описываются различные характеристики способа обнаружения нуклеиновой кислоты на основе белка Cas12b (включая предпочтительный способ HOLMES v2.0) по настоящему изобретению.
Идентификация транс-расщепляющей активности Cas12b: Хотя белки Cas12a и Cas12b совершенно отличаются по последовательности, экспериментальные результаты, полученные авторами настоящего изобретения, показывают, что белок Cas12b также обладает транс-расщепляющей активностью.
Изначально обходная ДНК была сконструирована как флуоресцентный зонд, состоящий из случайной последовательности длиной 12 нт, а флуоресцентная группа была мечена на 5’-конце, а группа гашения BHQ1 была мечена на 3’-конце (HEX-N12-BHQ1). Когда система содержит фрагмент ДНК-мишени (двухцепочечная ДНК или одноцепочечная ДНК), образуется тройной комплекс из ДНК-мишени, одиночной направляющей РНК и белка Cas12b. В этот момент зонд расщепляется, и флуоресценцию, излучаемую флуоресцентной группой HEX, можно обнаружить с помощью флуоресцентного детектора (свет возбуждения 535 нм, свет излучения 556 нм).
Как показано на фиг. 1, когда концентрация Cas12b и одиночной направляющей РНК составляла 250 нМ или 500 нМ, и двухцепочечная ДНК и одноцепочечная ДНК в качестве мишеней демонстрировали относительно высокую интенсивность флуоресценции. Когда концентрация Cas12b и одиночной направляющей РНК была снижена до 50 нМ и 100 нМ, одноцепочечная ДНК в качестве последовательности-мишени по-прежнему демонстрировала высокую интенсивность флуоресценции.
Характеристики PAM транс-расщепления Cas12b: Исследования показали, что во время цис-расщепления двухцепочечной ДНК белком Cas12b требуется последовательность, называемая PAM. Иными словами, только 5’-конец, за которым следует 5’-TTN-3’, может быть отрезан белком Cas12b. Чтобы узнать, требуется ли PAM для последовательности-мишени Cas12b во время транс-расщепления, авторы настоящего изобретения сконструировали различные последовательности (включая TTC, TAC, ATC, AAC, GGC и CCC) в месте расположения PAM, чтобы выяснить, требуется ли PAM во время транс-расщепления.
Как показано на фиг. 2, когда в качестве мишени используется двухцепочечная ДНК, транс-расщепление белком Cas12b более чувствительно к PAM-последовательности. Иными словами, повышенную интенсивность флуоресценции при транс-расщеплении можно получить, когда используется последовательность TTC (интенсивность флуоресценции, создаваемая после изменения последовательности на TAC слегка ниже, чем интенсивность флуоресценции при последовательности TTC). А когда в качестве мишени используется одноцепочечная ДНК, она нечувствительна к PAM-последовательности и всегда демонстрирует высокую интенсивность флуоресценции при транс-расщеплении.
Скорость транс-расщепления белком Cas12b: Когда одноцепочечная ДНК и двухцепочечная ДНК используются в качестве ДНК-мишени соответственно, измеряется скорость транс-расщепления белком Cas12b.
На фиг. 3 видно, что когда в качестве субстрата используется одноцепочечная ДНК, скорость транс-расщепления белком Cas12b очень высока, и она приближается к самому высокому значению всего через 6 минут. Когда в качестве субстрата используется двухцепочечная ДНК, скорость расщепления относительно низкая и происходит постепенное повышение. Это означает, что в некоторых случаях, когда требуется быстрое обнаружение (например, ≤10 минут), в качестве субстрата для обходного расщепления белком Cas12 должна использоваться одноцепочечная ДНК.
Применимый температурный диапазон для транс-расщепления белком Cas12b: Cas12b имеет очень широкий температурный диапазон для транс-расщепления, независимо от того, какая ДНК используется в качестве последовательности-мишени, одноцепочечная или двухцепочечная. Температура 45–65°C имеет хорошее значение отклика, особенно учитывая, что температурный диапазон для одноцепочечной ДНК-мишени шире, а именно 25–70°C (см. фиг. 4).
Чувствительность обнаружения Cas12b: Авторы настоящего изобретения проверяли чувствительность обнаружения Cas12b путем разбавления концентрации двухцепочечной ДНК-мишени или одноцепочечной ДНК-мишени. В предпочтительном варианте осуществления одноцепочечная ДНК-мишень (DNMT1-3(TTC PAM)-R) или двухцепочечная ДНК-мишень (полученная отжигом двух олигонуклеотидов DNMT1-3(TTC PAM)-F и DNMT1-3(TTC PAM)-R) разбавлялась до концентраций 100 нМ, 10 нМ, 1 нМ, ... до 10-4 нМ.
Результаты показывают, что когда концентрация ДНК составляет 1 нМ, целевая молекула по-прежнему может быть обнаружена с помощью Cas12b (фиг. 5).
Тест на чувствительность HOLMES v2.: В одном примере, когда Cas12b и реакция LAMP были объединены (т.e. HOLMES v2.0), чувствительность обнаружения могла достигать 10-8 нМ, что значительно повысило чувствительность обнаружения последовательности-мишени нуклеиновой кислоты (фиг. 6).
Тест SNP в сопоставлении с одноцепочечной ДНК-мишенью: Способ по настоящему изобретению очень подходит для обнаружения мутаций нуклеиновой кислоты, включая однонуклеотидные полиморфизмы, особенно однонуклеотидные полиморфизмы в одноцепочечных ДНК-мишенях.
Обнаружение SNP одноцепочечной ДНК-мишени показало, что, когда длина направляющей последовательности одиночной направляющей РНК составляла 18 нт или 20 нт, мутация основания положений 1–12 оказывала слабое воздействие на флуоресцентный сигнал, создаваемый транс-расщеплением. Когда длина направляющей последовательности одиночной направляющей РНК составляла 14 нт или 15 нт, значения флуоресценции, включая контроль дикого типа, были очень низкими. Когда длина направляющей последовательности одиночной направляющей РНК составляла 16 нт, после мутации положений 10–12, значение флуоресценции значительно снизилось по сравнению с контролем (фиг. 7).
В ходе дальнейшего исследования было установлено, что когда длина направляющей последовательности одиночной направляющей РНК составляла 16 нт, мутация основания положений 10–16 оказывала очевидное воздействие на значение флуоресценции, особенно учитывая, что значение флуоресценции было почти необнаруживаемым после мутации положений 10–14 (фиг. 8).
Кроме того, когда различные положения одноцепочечной ДНК-мишени мутировали в различные типы оснований, это не оказывало существенного воздействия на значение флуоресценции. При этом только мутация в G положения 9 значительно изменила значение флуоресценции (фиг. 9).
Кроме того, авторы настоящего изобретения также протестировали три другие последовательности-мишени. Хотя у различных последовательностей были различные изменения флуоресценции в результате мутации определенного сайта, положение, приводящее к значительному изменению, по-прежнему могло быть обнаружено для каждой последовательности среди положений 8–16, например, положений 8, 10, 11 и 12 в rs5082, положения 11 в rs1467558, положений 8–15 в rs2952768 (фиг. 10).
Тест SNP в сопоставлении с двухцепочечной ДНК-мишенью: Способ по настоящему изобретению подходит для обнаружения мутаций нуклеиновой кислоты, включая однонуклеотидные полиморфизмы в двухцепочечных ДНК-мишенях.
Обнаружение SNP двухцепочечной ДНК-мишени показало, что, флуоресцентный сигнал, создаваемый транс-расщеплением в связи с одноосновной мутацией, отличается от флуоресцентного сигнала одноцепочечной ДНК. Например, одиночная направляющая РНК, у которой длина направляющей последовательности составляет 18–20 нт, имеет различные степени чувствительности к мутациям оснований в положениях 10–16 (хотя флуоресцентный сигнал также уменьшается в различной степени после мутаций оснований в положениях 1 и 3) (фиг. 11). Как правило, эти одиночные направляющие РНК наиболее чувствительны к положениям 10 и 16.
Одноэтапное обнаружение при различных температурах: В настоящем изобретении также предложено одноэтапное обнаружение, которое объединяет амплификацию нуклеиновой кислоты и Cas12b.
Для проведения одноэтапного обнаружения предпочтительнее объединять LAMP-амплификацию с обнаружением белком Cas12b. В настоящем изобретении транс-расщепление белком Cas12b проверялось в различных температурных условиях (50°C, 55°C, 60°C и 65°C соответственно). Как показано на фиг. 12, одноэтапного обнаружения можно достичь при 50 и 55°C. Значение флуоресценции увеличивается быстрее особенно при 55°C.
Тестирование РНК-мишени: Способ по настоящему изобретению подходит не только для обнаружения ДНК-мишени, но и для обнаружения РНК-мишени. В настоящем изобретении РНК может быть обратно транскрибирована в форму ДНК, после чего можно обнаружить последовательность-мишень.
Предпочтительный способ заключается в добавлении обратной транскриптазы в реакционную систему для получения обратной транскрипции РНК. Затем, например, ДНК-полимераза Bst 2.0 используется для амплификации, а белок Cas12b используется для получения транс-расщепления. Другой способ заключается в непосредственном использовании ДНК-полимеразы Bst 3.0 (этот фермент может амплифицировать матричную РНК для получения ДНК), чтобы напрямую осуществить обратную транскрипцию и амплификацию РНК (Фиг. 17).
Количественное определение следовой ДНК-матрицы Способ по настоящему изобретению может использоваться для количественного определения.
В одном примере одноэтапный способ на основе LAMP-амплификации и Cas12b использовался при 55°C, выполняя, таким образом, количественное определение следовой ДНК. При разбавлении матриц на различные концентрации значение флуоресценции транс-расщепления определялось в режиме реального времени. После этого составлялась градуировочная кривая, а концентрацию матричной последовательности-мишени можно получить путем вычисления (фиг. 18 и 19).
Другой способ количественного определения заключается в объединении способа обнаружения на основе белка Cas12b по настоящему изобретению с методом цифровой ПЦР (см. «Цифровой способ HOLMES» ниже).
Конструирование праймеров и одиночной направляющей РНК
На основе идеи настоящего изобретения специалисты в этой области техники могут синтезировать соответствующие праймеры и/или одиночные направляющие РНК исходя из практических потребностей для обнаружения нуклеиновой кислоты на основе Cas12b по настоящему изобретению. В настоящем изобретении некоторые предпочтительные конструкции праймеров и одиночной направляющей РНК могут обозначать следующие предложения.
В отношении праймеров конструирование может обозначать следующее.
Ситуация 1. Если для предыдущей реакции амплификации выбран метод LAMP, можно использовать традиционные способы конструирования праймеров (http://primerexplorer.jp/elamp4.0.0/index.html). Рекомендуется добавлять праймеры LoopF и LoopB между участками праймеров F2/F3 и B2/B3, т.е. 6 пар праймеров.
Ситуация 2. Если для предыдущей реакции амплификации выбран метод ПЦР, необходимо приготовить несколько праймеров для испытания на наиболее подходящую комбинацию для получения одноцепочечной ДНК.
Ситуация 3. Для обнаружения SNP, если около сайта находится подходящий сайт PAM, то используется решение по Ситуации 1; если нет подходящего сайта РАМ, то сайт PAM необходимо ввести в праймер, чтобы продукт создал сайт PAM. Обычно SNP-сайт представляет собой 8–16 оснований на удалении от PAM, но специфическую последовательность-мишень необходимо протестировать.
Ситуация 4. Если затруднительно найти подходящие праймеры для LAMP-амплификации рядом с SNP-сайтом или затруднительно ввести подходящий PAM, для амплификации одноцепочечной ДНК можно использовать такие методы, как асимметричная ПЦР-амплификация.
В отношении конструирования одиночной направляющей РНК можно рассмотреть следующие ситуации.
Ситуация 1. Для обнаружения генов-мишеней обычно целесообразно выбрать комплементарно спаренную одиночную направляющую РНК 20 п.о. в случае последовательности-мишени с PAM-последовательностью.
Ситуация 2. Для обнаружения SNP, если на этапе амплификации используется двухцепочечная амплификация, например LAMP, в первую очередь необходимо проверить положение SNP-сайта. Одиночные направляющие РНК, длина направляющей последовательности которых составляет 18 нт, проверяются в первую очередь для положений 10–16.
Ситуация 3. Для обнаружения SNP, если на этапе амплификации используется одноцепочечная амплификация, например асимметричная ПЦР, в первую очередь необходимо проверить положение SNP-сайта. Одиночные направляющие РНК, длина направляющей последовательности которых составляет 16 нт, проверяются в первую очередь для положений 10–16, особенно для положений 10–12.
Цифровой способ HOLMES
В настоящем изобретении также предложен метод, который объединяет способ HOLMES по настоящему изобретению с цифровой ПЦР (dPCR), именуемый для краткости «цифровой способ HOLMES».
Цифровая ПЦР представляет собой метод абсолютного количественного выявления молекул нуклеиновой кислоты, который получил быстрое развитие за последние годы. В настоящее время в основном применяется два типа, цифровая капельная ПЦР (ddPCR) и цифровая ПЦР на чипах (cdPCR). Иначе говоря, перед традиционной ПЦР-амплификацией образец преобразуется в капли или добавляется к чипу, а реакционная система, содержащая молекулы нуклеиновой кислоты, разделяется на тысячи реакционных систем на уровне нанолитра. По сравнению с традиционной ПЦР, цифровая ПЦР дополнительно имеет этап предварительной обработки, а позднее этап флуоресцентного детектирования. В настоящее время обнаружение с помощью цифровой ПЦР по-прежнему требует использования традиционного устройства ПЦР для выполнения этапа амплификации, а условия ПЦР-реакции более жесткие и на ее проведение требуется больше времени (около 3-4 часов).
При использовании способа HOLMES в сочетании с существующим на сегодняшний день прибором для считывания флуоресценции для цифровой ПЦР на чипах (т. e. цифрового способа HOLMES) абсолютное количественное выявление может осуществляться только при постоянной температуре.
Кроме того, когда в цифровом способе HOLMES для амплификации используется метод LAMP или другие методы, амплификация может проводиться в широком температурном диапазоне (50–70°C или около 50–55°C), без потребности в точном температурном контроле, как в случае использования традиционных устройств ПЦР. Поскольку Cas12b также обладает обходной расщепляющей активностью в этом температурном диапазоне, он может не только эффективно интегрироваться, но также одновременно осуществлять амплификацию и обходное расщепление.
В настоящем изобретении также предложено устройство для цифрового обнаружения HOLMES, в частности устройство по седьмому аспекту настоящего изобретения.
Таким образом, цифровой способ HOLMES может использоваться для интегрирования предварительной обработки, амплификации и обнаружения в одном приборе, который может не только уменьшить человеческие ошибки при работе, но также ускорить процесс обнаружения и снизить затраты на обслуживание прибора.
Наборы
В настоящем изобретении также предложен набор, содержащий направляющую РНК, белок 12b и зонд на основе нуклеиновой кислоты. Кроме того, набор по настоящему изобретению может также содержать другие реагенты, такие как буферы, реагенты, необходимые для амплификации, реагенты, необходимые для обратной транскрипции, или их комбинация.
Как правило, набор по настоящему изобретению содержит:
i) первый контейнер и белок Cas12b, находящийся в первом контейнере, причем белок Cas12b представляет собой белок Cas12b или Cas, обладающий активностью, аналогичной обходной расщепляющей активности одноцепочечной ДНК белка Cas12b;
ii) необязательный второй контейнер и вторую направляющую РНК, находящуюся во втором контейнере, причем вторая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты;
iii) третий контейнер и зонд на основе нуклеиновой кислоты (предпочтительно флуоресцентный зонд), находящийся в третьем контейнере;
iv) необязательный четвертый контейнер и буфер, находящийся в четвертом контейнере;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень.
Кроме того, набор по настоящему изобретению может дополнительно содержать:
v) пятый контейнер и полимеразу для амплификации ДНК-мишени, находящейся в пятом контейнере;
vi) необязательный шестой контейнер и обратную транскриптазу для обратной транскрипции, находящуюся в шестом контейнере;
vii) седьмой контейнер и dNTP для реакции амплификации и/или реакции обратной транскрипции, находящийся в седьмом контейнере.
В настоящем изобретении один, несколько или все контейнеры могут быть одним и тем же контейнером или различными контейнерами.
Основные преимущества настоящего изобретения включают в себя:
Используя характеристики белка Cas12b, авторы настоящего изобретения разработали способ специфического обнаружения молекул нуклеиновой кислоты, который называется HOLMES (одночасовая низкозатратная многоцелевая высокоэффективная система версии 2.0). Он характеризуется как быстрый (1 час), низкозатратный, многоцелевой, эффективный и простой способ тестирования. Этот способ может использоваться в области быстрого обнаружения болезнетворных организмов, обнаружения SNP и т.п. По сравнению со способом HOLMES первого поколения на основе Cas12a, способ HOLMES второго поколения на основе высокотемпературного фермента Cas12b имеет больше преимуществ.
(1) Быстрота: Когда условия для проведения тестирования готовы, требуется всего около 1 часа с момента получения образца до момента получения результата.
(2) Низкая затратность: Проведение эксперимента не требует каких-либо специальных материалов или ферментов, а количество используемых материалов и реагентов невелико. Он может использоваться для тестирования и анализа следовых количеств.
(3) Эффективность: Способ по настоящему изобретению обладает чрезвычайно высокой чувствительностью и позволяет обнаруживать ДНК при концентрации 10 aM.
(4) Универсальность: Он позволяет обнаруживать различные образцы нуклеиновых кислот, включая образцы ДНК и образцы РНК.
(5) Простота: Не требует выполнения каких-либо специальных сложных этапов. Если набор готов и задана программа, требуется просто добавить образец или выполнить какой-либо аналогичный этап.
(6) Одноступенчатая реакция: реакция амплификации и реакция обнаружения могут проводиться одновременно, делая этот тест одноэтапным.
(7) Изотермичность: Поскольку и LAMP-амплификация и обнаружение белком Cas12b выполняются при постоянной температуре, то требования к прибору ниже.
(8) Широкий диапазон температуры реакции: Cas12b может использоваться для обнаружения при 45–65 °C, поэтому требования к прибору ниже.
(9) Два фермента могут осуществлять амплификацию и обнаружение ДНК или РНК: Поскольку ДНК-полимераза Bst 3.0 DNA обладает активностью ДНК-полимеразы с использованием ДНК или РНК в качестве матрицы, требуется только два фермента, Bst 3.0 и Cas12b, для осуществления амплификации и обнаружения ДНК или РНК, что снижает производственные затраты.
(10) Высокочувствительное обнаружение SNP: При надлежащем конструировании сайтов и одиночной направляющей РНК можно распознавать различия в одном основании с высокой чувствительностью.
(11) Возможно количественное выявление: Флуоресцентная детекция в реальном времени одноэтапным способом HOLMES или в сочетании с чипом и системой обнаружения прибора для цифровой ПЦР позволяет осуществлять количественное выявление образцов.
Ниже описание изобретения приводится вместе со специфическими вариантами осуществления. Следует понимать, что данные примеры не предназначены для ограничения объема изобретения. Экспериментальные способы в следующих примерах, в которых не указаны специфические условия, обычно выполняются в соответствии с традиционными условиями, например, такими, которые описаны в работе Сэмбрука (Sambrook) с соавторами «Молекулярное клонирование»: Лабораторное руководство (Нью-Йорк: Cold Spring Harbor Laboratory Press, 1989), либо в соответствии с условиями, рекомендуемыми производителем. Если не указано иное, проценты и части являются весовыми.
В этом применении, если не указано иное, система с транс-расщепляющей активностью белка Cas12b содержит: 250 нМ одиночной направляющей РНК и 250 нМ белка Cas12b, а условия протекания реакции: 48 °C в течение 30 минут.
Материалы
1. Ингибитор РНКаз был закуплен у компании TaKaRa. Высокоточная ДНК-полимераза KOD FX была закуплена у компании ToYoBo. Праймеры (олигонуклеотиды) были синтезированы компанией Shanghai Biotech. Полимераза T7 RNA была закуплена у компании Thermo. Набор для очистки и выделения РНК (RNA Clean & ConcentratorTM-5) был закуплен у компании Zymo Research. Система для очистки из геля и ПЦР Wizard® SV была закуплена у компании Promega. Среда (например, триптон, дрожжевой экстракт и т.д.) была закуплена у компании OXOID.
2. Формула среды: жидкая LB (1% триптона, 0,5% дрожжевого экстракта и 1% NaCl). При разработке рецептуры твердой LB необходимо добавить только 2% агара к жидкой LB.
Пример 1
Обнаружение двухцепочечной ДНК (дцДНК) или одноцепочечной ДНК (оцДНК) мишеней с помощью белка Cas12b
Одноцепочечная ДНК или двухцепочечная ДНК (мишень T1) была выбрана в качестве последовательности-мишени для проверки значений отклика белка Cas12b и одиночной направляющей РНК в различных концентрациях.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Сначала плазмида pUC18-sgRNA-T1 была сконструирована с pUC18 в качестве плазмидного остова. В этой плазмиде промотор T7 и матричная последовательность ДНК для транскрипции одиночной направляющей РНК были введены pUC18. (Примечание: Одиночная направляющая РНК, транскрибированная из данной матрицы, нацелена на последовательность, которая в этом исследовании называется T1.) В этом способе приготовления плазмида pUC18 использовалась в качестве матрицы, а pUC18-1-F и pUC18-1-R использовались в качестве праймеров, и был выполнен первый раунд ПЦР. Затем ДНК-лигаза T4 использовалась для соединения продуктов ПЦР, и продукт реакции трансформировался в DH10b. После секвенирования был получен правильный клон, который получил название pUC18-sgRNA-T1-pre. Затем плазмида pUC18-sgRNA-T1-pre использовалась в качестве матрицы, а pUC18-2-F и pUC18-2-R использовались в качестве праймеров, и был выполнен второй раунд ПЦР. Продукты ПЦР были соединены таким же образом и трансформированы. И наконец, была получена правильная секвенированная плазмида pUC18-sgRNA-T1.
Затем плазмида pUC18-sgRNA-T1 использовалась в качестве матрицы, а T7-crRNA-F и sgRNA-T1-R использовались в качестве праймеров, и ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована с помощью ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК (именуемая sgRNA-T1) была синтезирована с помощью набора для высокопродуктивной транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
И наконец, DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Приготовление ДНК-мишени
(1) Если ДНК-мишень была одноцепочечной, напрямую синтезировалась ДНК-мишень олигонуклеотидов (T1-R), которая содержит 20 п.о. последовательность-мишень (T1), распознаваемую одиночной направляющей РНК.
(2) Если ДНК-мишень была двухцепочечной, напрямую синтезировались два комплементарных олигонуклеотида (T1-F; T1-R), которые содержат 20 п.о. последовательность-мишень (T1), распознаваемую одиночной направляющей РНК. Два олигонуклеотида были отожжены, и, таким образом, была получена двухцепочечная ДНК-мишень. Конкретнее, спаренные олигонуклеотиды (2 мкМ) были смешаны в буфере 1×ПЦР (Transgen Biotech) общим объемом 20 мкл. После этого была выполнена процедура отжига: сначала была проведена денатурация при 95°C в течение 5 минут, затем охлаждение с 95 до 20°C со скоростью охлаждения 1°C в минуту, с использованием термоциклера.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 50, 100, 250 или 500 нМ), флуоресцентный зонд и зонд гашения (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48 °C в течение 30 минут. После этого она была инактивирована путем нагрева при 98 °C в течение 5 минут.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
Результаты показаны на фиг. 2. Когда концентрация Cas12b и одиночной направляющей РНК составляла 250 или 500 нМ, и двухцепочечная ДНК и одноцепочечная ДНК в качестве мишеней демонстрировали относительно высокую интенсивность флуоресценции. Когда концентрация Cas12b и одиночной направляющей РНК была снижена до 50 и 100 нМ, одноцепочечная ДНК в качестве последовательности-мишени по-прежнему демонстрировала высокую интенсивность флуоресценции.
Пример 2
Проверка чувствительности отклика с помощью способа HOLMES v2.0 (LAMP в сочетании с Cas12b)
Путем детектирования интенсивности возбужденной флуоресценции флуоресцентного зонда (HEX-N12-BHQ1) была определена концентрация ДНК-мишени, необходимая для обходной расщепляющей активности одноцепочечной ДНК белка Cas12b, иначе говоря, чувствительность обходной реакции расщепления одноцепочечной ДНК белком Cas12b.
1. Приготовление одиночной направляющей РНК
В первую очередь ранее сконструированная плазмида pUC18-sgRNA-T1 использовалась в качестве матрицы, и были сконструированы праймеры sgRNA-DNMT1-3-F и sgRNA-DNMT1-3-R. 20 оснований, нацеленных на ДНК-мишень T1, в одиночной направляющей РНК были заменены одиночной направляющей РНК, нацеленной на DNMT1-3 с помощью ПЦР, в результате чего была получена другая плазмида pUC18-sgRNA-DNMT1-3.
Затем плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, а T7-crRNA-F и ZLsgRNA-DNMT1-3-R использовались в качестве праймеров, и ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована с помощью ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован в водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК (sgRNA-DNMT1-3) была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37 °C.
И наконец, DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Приготовление ДНК-мишени
Для ДНК-мишени первой ситуацией было добавление белка Cas12b в реакционную систему напрямую, без амплификации. Описание способа приведено ниже:
(1) Если ДНК-мишень была одноцепочечной, олигонуклеотид длиной 50 п.о. (DNMT1-3) синтезировался напрямую, чтобы использоваться в качестве ДНК-мишени, которая содержит 20 п.о. последовательность-мишень (DNMT1-3), распознаваемую одиночной направляющей РНК.
(2) Если ДНК-мишень была двухцепочечной, напрямую синтезировались два комплементарных олигонуклеотида длиной 50 п. о. (DNMT1-3(TTC PAM)-F; DNMT1-3(TTC PAM)-R), которые содержат 20 п.о. последовательность-мишень (DNMT1-3), распознаваемую одиночной направляющей РНК. Два олигонуклеотида были отожжены, и, таким образом, была получена двухцепочечная ДНК-мишень. Конкретнее, спаренные олигонуклеотиды (2 мкМ) были отожжены в буфере 1×ПЦР (Transgen Biotech) общим объемом 20 мкл. После этого была выполнена процедура отжига: сначала была проведена денатурация при 95°C в течение 5 минут, затем охлаждение с 95 до 20°C со скоростью охлаждения 1°C в минуту, с использованием термоциклера.
(3) Одноцепочечная или двухцепочечная ДНК-мишень была разбавлена до 2 мкM, 0,2 μM, 0,02 мкM, 0,002 мкM и 0,0002 мкM для использования.
Вторая ситуация заключалась в том, что фрагмент, содержащий последовательность-мишень (DNMT1-3), вводился в плазмидный вектор и амплифицировался путем LAMP-реакции.
(1) Набор для клонирования pEasy-Blunt Zero Cloning Kit от компании Transgen использовался для введения фрагмента, содержащего последовательность-мишень (DNMT1-3), в клонирующий вектор pEasy-Blunt Zero Cloning Vector, и после проверки секвенирования был получен правильный клон.
(2) Реакция LAMP-амплификации
Вышеуказанная плазмида использовалась в качестве матрицы для выполнения LAMP-амплификации. Матрица добавлялась соответственно при 0,1 нМ, 0,1 нМ и концентрации с 10-кратным разведением до 10-11 нМ. Общий объем каждой реакционной системы составил 25 мкл. 1,6 мкМ LAMP-DNM-FIP и LAMP-DNM-BIP, 0,2 мкМ LAMP-DNM-F3 и LAMP-DNM-B3, и 0,4 мкМ LAMP-DNM-LoopF и LAMP-DNM-LoopB использовались в качестве праймеров. Для проведения LAMP-реакции использовался набор WarmStart® LAMP Kit (NEB). LAMP-реакция протекала при 65°C в течение 30 минут. После завершения LAMP-реакции продукт инактивировался при 85°C в течение 10 минут, а затем напрямую использовался в реакции Cas12b.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 250 нМ), 1 мкл ДНК-мишени или 1 мкл LAMP-продукта, флуоресцентный зонд (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 30 минут. После этого она была инактивирована путем нагрева при 98°C в течение 5 минут.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
На фиг. 5 видно, что Cas12b может обнаруживать одноцепочечную или двухцепочечную ДНК при концентрации 1 нМ. В сочетании с LAMP-амплификацией Cas12b (т.e. способ HOLMES v2.0) может обнаруживать ДНК при концентрации 10-8нМ (фиг. 6).
Пример 3 Обнаружение мишени одноосновной мутации с помощью белка Cas12b
Одноцепочечная ДНК или двухцепочечная ДНК использовалась в качестве последовательности-мишени для проверки изменения флуоресцентного сигнала транс-расщепления белком Cas12b при наличии одноосновной мутации в ДНК-мишени, выявляя таким образом одноосновные мутации.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, T7-crRNA-F использовался в качестве прямого праймера, а олигонуклеотиды, содержащие направляющие последовательности, комплементарные различным мишеням, использовались в качестве обратных праймеров. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а полноразмерная одиночная направляющая РНК (с длиной направляющей последовательности 20 нт) или усеченная одиночная направляющая РНК (с длиной направляющей последовательности менее 20 нт) была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37 °C в течение 30 минут. ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Приготовление ДНК-мишени
(1) Если ДНК-мишень была одноцепочечной, олигонуклеотид длиной 50 п.о. синтезировался напрямую, чтобы использоваться в качестве ДНК-мишени, которая содержит последовательность-мишень, распознаваемую одиночной направляющей РНК.
(2) Если ДНК-мишень была двухцепочечной, напрямую синтезировались два комплементарных олигонуклеотида длиной 50 п.о., которые содержат последовательность-мишень, распознаваемую одиночной направляющей РНК. Два олигонуклеотида были отожжены, и, таким образом, была получена двухцепочечная ДНК-мишень. Конкретнее, спаренные олигонуклеотиды (1 мкМ) были отожжены в буфере 1×ПЦР (Transgen Biotech) общим объемом 20 мкл. После этого была выполнена процедура отжига: сначала была проведена денатурация при 95°C в течение 5 минут, затем охлаждение с 95 до 20°C со скоростью охлаждения 1°C в минуту, с использованием термоциклера.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 250 нМ), ДНК-мишень (с окончательной концентрацией 50 нМ), флуоресцентный зонд и зонд гашения (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 30 минут. После этого она была инактивирована путем нагрева при 98 °C в течение 5 минут.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и затем проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
Прежде всего, как показано на фиг. 7, как в случае с последовательностью-мишенью DNMT1-3, так и в случае с одноцепочечной ДНК-мишенью, когда направляющая последовательность одиночной направляющей РНК составляла 20 нт, воздействие мутаций оснований в положениях 1–12 на флуоресцентный сигнал, создаваемый транс-расщеплением, было незначительным. Когда длина направляющей последовательности одиночной направляющей РНК составляла 14 или 15 нт, значения флуоресценции, включая контроль, были очень низкими. Когда длина направляющей последовательности одиночной направляющей РНК составляла 16 нт, после мутации положений 10–12 значение флуоресценции значительно снизилось.
В ходе дальнейшего исследования было установлено, что, как показано на фиг. 8, мутация основания положений 10–16 оказывала очевидное воздействие на значение флуоресценции, особенно учитывая, что значение флуоресценции было почти необнаруживаемым после мутации положений 10–14. Затем различные положения одноцепочечной ДНК-мишени мутировали в различные типы оснований, и это не оказывало существенного воздействия на значение флуоресценции. При этом только мутация в G положения 9 значительно изменила значение флуоресценции (фиг. 9).
Затем авторы настоящего изобретения также протестировали три другие последовательности-мишени. Хотя у различных последовательностей были различные изменения флуоресценции в результате мутации определенного сайта, положение, приводящее к значительному изменению, по-прежнему могло быть обнаружено для каждой последовательности среди положений 8–16, например, положений 8, 10, 11 и 12 в rs5082, положения 11 в rs1467558, положений 8–15 в rs2952768 (фиг. 10).
В случае с двухцепочечной ДНК-мишенью на фиг. 11 видно, что флуоресцентный сигнал, создаваемый транс-расщеплением, обусловленным воздействием одноосновной мутации, отличается от флуоресцентного сигнала одноцепочечной ДНК. При использовании последовательности-мишени DNMT1-3 в качестве примера одиночная направляющая РНК с длиной направляющей последовательности 18–20 была чувствительна к мутациям оснований в положениях 10–16. Кроме того, в данном примере несколько видов одиночной направляющей РНК наиболее чувствительны к положениям 10 и 16.
Одноступенчатая реакция LAMP и обнаружение белком Cas12b были осуществлены при 50°C, 55°C, 60°C и 65°C (фиг. 12).
Пример 4
Обнаружение кишечной палочки E. coli и других микроорганизмов в природных водах
Ген E. coli gyrB был выбран в качестве мишени обнаружения для косвенного обнаружения E. coli и других микроорганизмов в водоеме и определения предела обнаружения.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, а T7-crRNA-F использовался в качестве прямого праймера, а ZL-gyrB-crRNA2-R использовался в качестве обратного праймера. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37 °C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Реакция LAMP-амплификации
После культивирования E. coli MG1655 до OD600 1,0 часть бактериального раствора была подвергнута температурному воздействию в 100°C в течение 15 минут. После чего была проведена реакция LAMP-амплификации. В матрицу добавлялся бактериальный раствор различных концентраций, при этом разбавленные растворы имели оптическую плотность OD600 от 0, 5×10-3, 5×10-4 до 5×10-9 соответственно.
После теста на покрытие OD600 5×10-3 содержала около 7000 бактерий. Общий объем каждой реакционной системы составил 25 мкл. 1,6 мкМ LAMP-gyrB-FIP и LAMP-gyrB-BIP, 0,2 мкМ LAMP-gyrB-F3 и LAMP-gyrB-B3, и 0,4 мкМ LAMP-gyrB-LoopF и LAMP-gyrB-LoopB использовались в качестве праймеров. Для проведения LAMP-реакции использовался набор WarmStart® LAMP Kit (NEB). LAMP-реакция протекала при 65°C в течение 30 минут. После завершения LAMP-реакции продукт инактивировался при 85°C в течение 10 минут, а затем напрямую использовался в реакции Cas12b.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 250 нМ), 1 мкл LAMP-продукта, флуоресцентный зонд (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 30 минут. После этого она была инактивирована путем нагрева при 98°C в течение 5 минут.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
Как показано на фиг. 13, чувствительность обнаружения белком Cas12b в сочетании с LAMP-амплификацией (т. e. способ HOLMES v2.0) может достигать уровня обнаружения очень немногих E. coli (после теста на покрытие OD600=5×10-6 содержала около 7 E. coli).
Пример 5 Тест на определение пола по слюне
В этом примере специфический генный локус определяющего пол участка Y-хромосомы был выбран в качестве тестовой мишени, а пол определялся с помощью образца источника слюны способом HOLMES v2.0.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, а T7-crRNA-F использовался в качестве прямого праймера, а ZL-sry-crRNA3-R и ZLsgRNA-DNMT1-3-R использовались в качестве обратных праймеров. Две ДНК-матрицы, необходимые для транскрипции in vitro, были амплифицированы ПЦР соответственно. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Реакция LAMP-амплификации
Образцы мужской и женской слюны использовались в качестве матриц, и проводилась реакция LAMP-амплификации. Общий объем каждой реакционной системы составил 25 мкл. 1,6 мкМ LAMP-sry-FIP и LAMP-sry-BIP, 0,2 мкМ LAMP-sry-F3 и LAMP-sry-B3, и 0,4 мкМ LAMP-sry-LoopF и LAMP-sry-LoopB использовались в качестве праймеров. Для проведения LAMP-реакции использовался набор WarmStart® LAMP Kit (NEB). LAMP-реакция протекала при 65°C в течение 30 минут. После завершения LAMP-реакции продукт инактивировался при 85°C в течение 10 минут, а затем напрямую использовался в реакции Cas12b.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 250 нМ), 1 мкл LAMP-продукта, флуоресцентный зонд (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 30 минут. После этого она была инактивирована путем нагрева при 98°C в течение 5 минут.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
Как показано на фиг. 14, белок Cas12b в сочетании с LAMP-амплификацией (т.e. способ HOLMES v2.0) может эффективно определять пол человека с помощью образцов слюны.
Пример 6
Обнаружение SNP человека
В этом примере обнаруживался локус человека rs5082. ДНК-мишень была приготовлена асимметричной ПЦР или изотермической LAMP-амплификацией для определения типа SNP образцов, полученных из слюны.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, T7-crRNA-F использовался в качестве прямого праймера, а олигонуклеотиды, содержащие направляющие последовательности различной длины, комплементарные сайту rs5082, использовались в качестве обратных праймеров. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а усеченная одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C. В случае ДНК-мишени, создаваемой асимметричной ПЦР, усеченная одиночная направляющая РНК с направляющей последовательностью длиной 16 нт использовалась в реакции обнаружения белком Cas12b. В случае ДНК-мишени, создаваемой LAMP-амплификацией, усеченная одиночная направляющая РНК с направляющей последовательностью длиной 18 нт использовалась в реакции обнаружения белком Cas12b.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Реакция амплификации (асимметричная ПЦР или LAMP-амплификация)
Реакция асимметричной ПЦР-амплификации: Геном клетки человека HEK293T использовался в качестве матрицы, а праймеры ASP-праймер, ASP-rs5082-F, ASP-rs5082-R использовались для асимметричной ПЦР-амплификации (причем традиционная ПЦР не использует ASP-праймер), а фермент KOD FX использовался для реакции амплификации.
Реакция LAMP-амплификации: Геном клетки человека HEK293T использовался в качестве матрицы для проведения реакции LAMP-амплификации. Общий объем каждой реакционной системы составил 25 мкл. 1,6 мкМ LAMP-rs5082-FIP-10PAM (LAMP-rs5082-FIP в качестве контроля) и LAMP-rs5082-BIP, 0,2 мкМ LAMP-rs5082-F3 и LAMP-rs5082-B3, и 0,4 мкМ LAMP-rs5082-LoopF и LAMP-rs5082-LoopB использовались в качестве праймеров. Для проведения LAMP-реакции использовался набор WarmStart® LAMP Kit (NEB). LAMP-реакция протекала при 65°C в течение 30 минут. После завершения LAMP-реакции продукт инактивировался при 85°C в течение 10 минут, а затем напрямую использовался в реакции Cas12b. Поскольку к последовательности праймера LAMP-rs5082-FIP-10PAM была добавлена PAM-последовательность, амплифицированная ДНК имеет PAM-последовательность.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составила 500 нМ, а концентрация Cas12b при проведении теста с LAMP-амплификацией составила 250 нМ), 1 мкл ДНК-мишени или 1 мкл LAMP-продукта, флуоресцентный зонд (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 60 минут. После этого она была инактивирована путем нагрева при 98°C в течение 5 минут в приборе для ПЦР.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
Из фиг. 15 видно, что, поскольку возле сайта rs5082 нет PAM-последовательности, асимметричная ПЦР, которая производит одноцепочечную ДНК, может более четко распознавать SNP-сайты.
При сочетании с LAMP-амплификацией, поскольку возле сайта rs5082 не было PAM-последовательности, сайт обнаружения необходимо вводить с РАМ-последовательностью для лучшего распознавания сайтов SNP (фиг. 16).
Пример 7
Обнаружение вируса РНК
В этом примере был выбран вирус JEV (вирус японского энцефалита) РНК, а способ HOLMES v2.0 использовался для того, чтобы определить, заражен ли образец вирусом, и для тестирования были выбраны сайты E453 и NS170.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, T7-crRNA-F использовался в качестве прямого праймера, а олигонуклеотиды, содержащие направляющие последовательности, комплементарные соответствующим мишеням, использовались в качестве обратных праймеров. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80 °C для дальнейшего использования.
2. Реакция LAMP-амплификации
Клеточная жидкость, зараженная вирусом JEV, использовалась в качестве матрицы, и была проведена реакция LAMP-амплификации. Общий объем каждой реакционной системы составил 25 мкл. 1,6 мкМ LAMP-E453-FIP и LAMP-E453-BIP, 0.2 мкМ LAMP-E453-F3 и LAMP-E453-B3, и 0,4 мкМ LAMP-E453-LoopF и LAMP-E453-LoopB (или 1,6 мкМ LAMP-NS170-FIP и LAMP-NS170-BIP, 0.2 мкМ LAMP-NS170-F3 и LAMP-NS170-B3, и 0,4 мкМ LAMP-NS170-LoopF и LAMP-NS170-LoopB) использовались в качестве праймеров. Для LAMP-амплификации использовался набор WarmStart® LAMP Kit (NEB) или ДНК-полимераза Bst 3.0 (NEB). LAMP-реакция протекала при 65°C в течение 30 минут. После завершения LAMP-реакции продукт инактивировался при 85°C в течение 10 минут, а затем напрямую использовался в реакции Cas12b.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20 °C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 250 нМ), 1 мкл LAMP-продукта, флуоресцентный зонд (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 30 минут. После этого она была инактивирована путем нагрева при 98°C в течение 5 минут в приборе для ПЦР.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм).
Как показано на фиг. 17, белок Cas12b в сочетании с LAMP-амплификацией (т.e. способ HOLMES v2.0) может обнаруживать присутствие вируса JEV с высокой степенью чувствительности.
Пример 8
Относительное количественное определение следовой ДНК-матрицы (способ HOLMES в реальном времени)
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, T7-crRNA-F использовался в качестве прямого праймера, а олигонуклеотиды, содержащие направляющие последовательности, комплементарные мишеням, использовались в качестве обратных праймеров. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37 °C в течение 30 минут. Плазмидная ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Одноступенчатая реакция LAMP-амплификации-флуоресцентного детектирования (с использованием прибора для ПЦР в реальном времени)
Плазмиды DNMT1-3 различных концентраций использовались в качестве матриц для проведения реакции LAMP-амплификации. Общий объем каждой реакционной системы составил 20 мкл. 1,6 мкМ LAMP-DNM-FIP и LAMP-DNM-BIP, 0,2 мкМ LAMP-DNM-F3 и LAMP-DNM-B3, и 0,4 мкМ LAMP-DNM-LoopF и LAMP-DNM-LoopB использовались в качестве праймеров. Для проведения LAMP-реакции использовался набор WarmStart® LAMP Kit (NEB). Были добавлены смесь одиночной направляющей РНК и Cas12b (окончательная концентрация обоих компонентов составляла 500 нМ), флуоресцентный зонд (FAM-N12-Eclipse с окончательной концентрацией 500 нМ) и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания в флуорометре выполнялось флуоресцентное детектирование, и реакция протекала при 55°C в течение 120 минут.
3. Анализ результатов
На фиг. 18 показаны значения измерения флуоресценции ДНК-мишени в реальном времени при различных концентрациях.
Был получен примерный линейный тренд, с использованием момента времени, когда значение флуоресценции достигает 600,000 как ось y, и с использованием абсолютного значения концентрации lg как ось х.
Вышеуказанные результаты показывают, что способ по настоящему изобретению позволяет точно определять 10-1–10-6 нМ ДНК в течение 45 минут.
Пример 9
Абсолютное количественное определение следовой ДНК-матрицы (цифровой способ HOLMES)
В данном примере способ HOLMES был совмещен с цифровой ПЦР на чипах для абсолютного количественного определения при одной температуре реакции.
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, T7-crRNA-F использовался в качестве прямого праймера, а олигонуклеотиды, содержащие направляющие последовательности различной длины, комплементарные сайту rs5082, использовались в качестве обратных праймеров. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в этот продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37 °C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37 °C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Одноступенчатая реакция LAMP-амплификации-флуоресцентного детектирования (с использованием прибора JN Medsys для цифровой капельной ПЦР (ddPCR))
В качестве матрицы для абсолютного количественного определения использовался геном клетки человека 293T в различных концентрациях. Общий объем каждой реакционной системы составил 15 мкл. 1,6 мкМ LAMP-rs5082-FIP-10PAM и LAMP-rs5082-BIP, 0,2 мкМ LAMP-rs5082-F3 и LAMP-rs5082-B3, и 0,4 мкМ LAMP-rs5082-LoopF и LAMP-rs5082-LoopB использовались в качестве праймеров. Для проведения LAMP-реакции использовался набор WarmStart® LAMP Kit (NEB). Были добавлены смесь одиночной направляющей РНК и Cas12b (окончательная концентрация обоих компонентов составляла 500 нМ), флуоресцентный зонд (HEX-N12-BHQ1 и FAM-N12-Eclipse, оба с окончательной концентрацией 500 нМ), 0,375 мкл ингибитора РНКаз (40 ед/мкл) и 0,75 мкл раствора JN.
После смешивания с помощью предметного стекла образец был доставлен в чип (в пробирке для ПЦР) для разбиения системы. Затем чип был перенесен в прибор Sealing Enhancer для обработки.
Затем было добавлено 235 мкл блокирующего раствора, и реакция протекала в металлической ванне при температуре 55°C в течение 120 минут. Через 0, 60, 90 и 120 минут чип помещался в прибор View Jig для обнаружения флуоресценции.
3. Анализ результатов
Как показано на фиг. 20, через 120 минут реакции путем подсчета было обнаружено 51,75 копии в матрице 1 и 6,3 копии было обнаружено в матрице 2 с 10-кратным разведением. Ни одной копии не было обнаружено в контроле без матрицы.
Результаты показывают, что цифровой способ HOLMES по настоящему изобретению позволяет не только количественно определять, но также точно выявлять одну копию матрицы в системе микрообнаружения с исключительной чувствительностью, при этом значительно экономя время.
Пример 10
Обнаружение сайтов метилирования
В этом примере путем поискового запроса NCBI промоторная область (250 п.о.) гена коллагена α2(I) (COL1A2), содержащая 13 CpG-сайтов (M1-M13), была выбрана в качестве последовательности для обнаружения гена-мишени (учетный номер NCBI: AF004877.1). Ген коллагена α2(I) (COL1A2) кодирует коллаген и относится к онкогенезу.
В COL1A2(BSP)-C, все CpG-сайты были модифицированы метилированием. После бисульфитной конверсии (преобразование неметилированного С в урацил) и ПЦР-амплификации метилированное основание С в CpG по-прежнему оставалось С.
В COL1A2(BSP)-T ни один из CpG-сайтов не был модифицирован путем метилирования. После бисульфитной конверсии и ПЦР-амплификации основание C в CpG-сайте преобразуется в T.
Принцип способа обнаружения сайта метилирования по настоящему изобретению показан на фиг. 21.
Экспериментальный способ
1. Были извлечены геномы четырех линий раковых клеток (293T, SW480, NCI-N87 и MCF-7).
2. Геном был подвергнут воздействию бисульфитной конверсии. Была выбрана промоторная область гена COL1A2, содержащая сайт M3, и были сконструированы соответствующие BSP-праймеры (праймер COL1A2(BSP)-F/R). Геном клетки, подвергнутый воздействию бисульфитной конверсии, использовался в качестве матрицы для ПЦР-амплификации, а гель был восстановлен и очищен для получения продукта, который использовался в качестве ДНК-мишени для последующего обнаружения.
3. Приготовление целевой нуклеиновой кислоты для градуировочной кривой
(1) Праймеры (праймер COL1A2-F/R) использовались для амплификации фрагмента гена COL1A2 из SW480-gDNA, и он был собран в вектор pClone007S T (TSINGKE, TSV-007S). Затем вектор был трансформирован в DH10b. После культивирования в среде LB с подходящими антибиотиками при 37°C была извлечена и секвенирована плазмида, и, наконец, получена плазмида pClone007-COL1A2.
(2) Плазмида pClone007-COL1A2 была обработана метилтрансферазой CpG (M.SssI) (M0226V, NEB) так, чтобы остатки цитозина во всех последовательностях 5’-CG-3’ были метилированы. После этого плазмиды pClone007-COL1A2 и pClone007-COL1A2 обработанные метилтрансферазой CpG, были подвергнуты воздействию бисульфитной конверсии для преобразования неметилированного цитозина в урацил. Была сконструирована пара BSP-праймеров (праймер COL1A2(BSP)-F/R) для ПЦР-амплификации, и амплифицированные фрагменты были восстановлены и очищены гелем и собраны в вектор pClone007S T для конструирования плазмид pClone007-COL1A2(BSP)-C и pClone007-COL1A2(BSP) -T, получая таким образом фрагменты COL1A2(BSP)-C (мишень C) и COL1A2(BSP)-T (мишень T).
4. Обнаружение метилирования
Четыре линии раковых клеток gDNA (293T, SW480, NCI-N87 и MCF-7) были обработаны набором для прямого метилирования EZ DNA methylation-directTM (D5021, Zymo Research) для бисульфитной конверсии. После чего пара BSP-праймеров (праймер COL1A2(BSP)-F/R) использовалась для амплификации фрагментов гена COL1A2(BSP) (250bp) четырех линий раковых клеток и выполнены выделение из геля и очистка.
Способ бисульфитного секвенирования клонов
Четыре вышеуказанных фрагмента гена COL1A2 (BSP) были соединены с вектором pClone007S T, а затем трансформированы в DH10b. Более 10 позитивных моноклональных клонов были собраны на чашке и отправлена на анализ секвенирования. Инструмент для расчета скорости метилирования, QUantification tool for Methylation Analysis (QUMA) (инструмент количественного определения для анализа метилирования) использовался для расчета степени метилирования 13 CpG-сайтов в гене COL1A2.
Прямое бисульфитное секвенирование
Четыре вышеуказанных фрагмента гена COL1A2(BSP) были проанализированы способом секвенирования по Сенгеру, и была получена скорость метилирования путем расчета отношения пиков C/(C+T) 13 CpG-сайтов в гене COL1A2.
Способ высокопроизводительного бисульфитного секвенирования нового поколения (NGS)
Четыре вышеуказанных фрагмента гена COL1A2 (BSP) были подвергнуты высокопроизводительному бисульфитному секвенированию NGS, и был проведен анализ высокопроизводительного бисульфитного секвенирования NGS (проведен компанией Zhongke Purui Biotechnology Co., Ltd.) и степени метилирования 13 CpG-сайтов в гене COL1A2.
Обнаружение метилирования с помощью системы HOLMESv2
(1) Экстракция белка CRISPR-Cas12b
Кодон-оптимизированный полноразмерный ген AacCas12b был синтезирован компанией Shanghai Tolo Biotechnology Company Limited и клонирован в вектор pET28a. N-терминальный рекомбинантный белок Cas12b с HIS-меткой был экспрессирован и выработан в E. coli BL21 (DE3). Когда OD600 бактериального раствора достигла 0,6, было добавлено 0,25 мМ изопропилового тиогалактозида (IPTG), чтобы вызвать экспрессию, и бактериальный раствор культивировался при 16°C в течение 14–18 ч. После чего раствор был центрифугирован для сбора бактерий. Бактериальный раствор был суспендирован в буфере A (50 мМ Tris-HCl (pH 7,6), 150 мM NaCl, 20 мM имидазола и 1/500 толуолсульфонил фторида (об./об.)) и лизирован гомогенизатором высокого давления (Avestin), и центрифугирован при 15 000 об/мин в течение 30 мин. Ni-колонка (GE Healthcare) была предварительно выдержана в буфере A, а супернатант был перенесен в Ni-колонку. После этого был использован буфер B (50 мM Tris-HCl (pH 7,6), 200 мM NaCl и 30 мM имидазола), чтобы элюировать Ni-колонку, а буфер B (в котором концентрация имидазола была доведена до 300 мM), чтобы чтобы элюировать рекомбинантный белок. Элюированные фракции, содержащие белок AacCas12b, были собраны путем ультрафильтрации и сконцентрированы примерно до 5 мл. Затем он был загружен в колонку HiLoad 16/600 Superdex 200 pg (GE Healthcare), выдержанную в буфере C (40 мM Tris-HCl (pH 7,6), 200 мM NaCl, 2 мM DTT и 5% глицерина (об./об.))), и Элюированные фракции, содержащие белок AacCas12b, были собраны, сконцентрированы до 4 мг/мл с добавлением 50% глицерина и помещены на хранение при -20°C.
(2) Приготовление одиночной направляющей РНК
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, а T7-sgRNA-F использовался в качестве прямого праймера, и были сконструированы обратные праймеры, специфические для мишеней. ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в этот продукт ПЦР был добавлен DpnI (1 мкл DpnI на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C. И наконец, DNase I был добавлен в систему транскрипции (2 мкл DNase I на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК (Zymo Research). Полученная одиночная направляющая РНК получила название sgRNA-DNMT1-3. Количество продукта было определено с помощью NanoDrop 2000C, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
(3) Скрининг одиночной направляющей РНК
В случае с COL1A2 было сконструировано множество одиночных направляющих РНК, у которых направляющие последовательности различной длины совпадали с участками выше и ниже каждого CpG-сайта, а CpG-сайты совпадали с различными положениями гена. Одиночная направляющая РНК (с окончательной концентрацией 10 мкМ) была отожжена в приборе для ПЦР. Процедура отжига была следующей: 75°C в течение 5 минут; затем охлаждение со скоростью 1°C в 1 минуту до 20°C. После отжига одиночная направляющая РНК и белок AacCas12b (с окончательной концентрацией 5 мкМ) были смешаны в равных объемах и инкубированы при 30°C в течение 15–20 минут. После этого реакционная система 20 мкл Cas12b была готова (работа на льду):
Вода, свободная от РНКаз была добавлена к общему объему 20 мкл.
Реакция протекала в водяной бане при 48 C в течение 1 часа, и 20 мкл системы были перенесены в 96-луночный планшет. На микропланшетном ридере длина волны света возбуждения была задана на 535 нм, а длина волны света излучения была задана на 556 нм для измерения значения флуоресценции. Была отобрана наиболее подходящая одиночная направляющая РНК, используемая в группе реакций, в которой ДНК-мишень (COL1A2(BSP)-C/T) имела наибольшее различие со значением флуоресценции.
(4) Обнаружение метилирования на основе системы HOLMESv2
Фрагменты COL1A2(BSP)-C и COL1A2(BSP)-T были тщательно смешаны согласно соотношению COL1A2(BSP)-C как 0%, 10%, 30% и 50% соответственно, как ДНК-мишень градуировочной кривой обнаружения метилирования. Пробные фрагмнты COL1A2 (BSP) после обработки бисульфитом из четырех линий раковых клеток (293T, SW480, NCI-N87 и MCF-7) были смешаны с фрагментом COL1A2 (BSP)-T в эквимолярной концентрации соответственно, и затем смеси использовались в качестве ДНК-мишени для определения соотношения метилирования реальных образцов. Затем были определены соответственно градуировочная кривая метилирования М3-сайта промоторной области COL1A2 и соотношение метилирования М3-сайта в четырех линиях раковых клеток (293T, SW480, NCI-N87, MCF-7), используя sgRNA-COL1A2m3-C12-17 в системе HOLMESv2.
Одиночная направляющая РНК (с окончательной концентрацией 10 мкМ) была отожжена в приборе для ПЦР. Процедура отжига была следующей: 75 °C в течение 5 минут; затем охлаждение со скоростью 1 °C в 1 минуту до 20 °C. После отжига одиночная направляющая РНК и белок AacCas12b (с окончательной концентрацией 5 мкМ) были смешаны в равных объемах и инкубированы при 30 °C в течение 15–20 минут. После этого реакционная система 20 мкл Cas12b была готова (работа на льду):
Вода, свободная от РНКаз была добавлена к общему объему 20 мкл.
20 мкл системы были перенесены в 96-луночный планшет, который затем был помещен в прибор для ПЦР в реальном времени. Программа была выставлена на протекание реакции при 48 C в течение 1 часа, а значение флуоресценции измерялось каждые 2 минуты.
Градуировочная кривая метилирования на M3-сайте приняла значение флуоресценции в 30 минут и 10 минут как ординату Y, и приняла соотношение COL1A2(BSP)-C 0%, 10%, 30% и 50% как абсциссу X. Модель уравнения была следующей: Y=aX+b. Концентрация COL1A2 (BSP) в четырех линиях раковых клеток составила 1/2 ДНК-мишени на градуировочной кривой. Таким образом, модель расчета соотношения метилирования получила вид: Xнеизвестно=2(Yнеизвестнo-b)/a.
Экспериментальные результаты
1. Схема режима обнаружения
После того, как гены, содержащие метилированные CpG-сайты, были подвергнуты бисульфитной конверсии, все неметилированные CG-сайты в генах были преобразованы в UG, а все метилированные CG-сайты остались без изменений. Трансформированный ген использовался в качестве ДНК-матрицы для образования тройного комплекса с белком AacCas12b и одиночной направляющей РНК. В системе зонда одноцепочечной ДНК с флуоресцентной группой и группой гашения (т.е. в системе HOLMESv2) реакция была постоянной при 48 °C в течение определенного периода, и определялось значение флуоресценции. Если сайт обнаружения метилирован, зонд ДНК излучает свет, и значение флуоресценции высокое. Если сайт обнаружения не метилирован, зонд ДНК не излучает свет, и значение флуоресценции низкое или равно нулю.
2. Скрининг одиночной направляющей РНК
Как показано на фиг. 22, сравнивая значения флуоресценции каждой одиночной направляющей РНК и двух мишеней (мишень-C и мишень-T представляют фрагменты COL1A2 после обработки бисульфитом со степенью метилирования на всех CpG-сайтах 100 и 0% соответственно) в реакционной системе HOLMESv2-Cas12b, можно выбрать наиболее подходящую одиночную направляющую РНК. При этом, значения флуоресценции 9 sgRNAs, m3-C12-17, m4-C1-18, m4-C1-19, m4-C1-20, m5-C14-18, m6m7m8-C7-18, m6m7m8-19, m6m7m8-20 и m9m10-C1-18 имели очевидные различия в двух мишенях (мишень-C, мишень-T), все из которых могут использоваться для обнаружения метилирования. В данном исследовании выбрана одноточечная одиночная направляющая РНК (m3-C12-17) для последующего обнаружения метилирования. Одиночная РНК показана на фиг. 23.
3. Количественное определение метилирования на CpG-сайте M3 (COL1A2)
Фрагменты COL1A2 (BSP)-C и COL1A2 (BSP)-T были смешаны согласно соотношению содержания COL1A2 (BSP)-C 0%, 10%, 30%, 50% и sgRNA-COL1A2m3 -C12-17, используемому для определения значения флуоресценции реакции через 30 минут в системе HOLMESv. После анализа данных значение флуоресценции 0% использовалось в качестве нулевой точки для нормализации, и была построена количественная градуировочная кривая CpG-сайта M3 (COL1A2). Градуировочная кривая была построена соответственно для каждого теста. Авторы настоящего изобретения проводили каждый тест трижды с тремя повторами. Уравнения градуировочных кривых имели следующий вид: y = 758939x + 9320,9 (R2 = 0,9975); y = 671675x + 12415 (R2 = 0,9943); и y = 645621x + 8601,7 (R2 = 0,997) соответственно. Линейная корреляция трех градуировочных кривых была очень хорошей, при этом R2 достигало значения 0,99 или больше. Таким образом, значение флуоресценции данного генного локуса в реальных образцах можно определить с помощью системы HOLMESv2, чтобы рассчитать соответствующую степень метилирования (фиг. 25).
COL1A2 продемонстрировал различные уровни метилирования в различных формах рака. Для проверки применимости способа обнаружения метилирования с помощью системы HOLMESv2-Cas12b использовалась sgRNA-COL1A2m3-C12-17 для определения значений флуоресценции CpG-сайта M3 (COL1A2) в четырех видах раковых клеток (293T, SW480, NCI-N87 и MCF-7) через 30 минут реакции в системе HOLMESv2 (фиг. 24). При этом, концентрация гена-мишени, обнаруженная в раковых клетках, составила 1/2 концентрации градуировочной кривой. Их степени метилирования рассчитывались по вышеуказанным уравнениям градуировочной кривой соответственно: 63,3%, 81,3%, 54,8% и 77,1%.
Кроме того, одновременно использовался широко распространенный способ обнаружения метилирования для определения уровней метилирования гена COL1A2 в вышеуказанных четырех раковых клетках, и проводилось сравнение со способом по настоящему изобретению, чтобы проверить эффективность и применимость способа обнаружения метилирования с помощью системы HOLMESv2-Cas12b (фиг. 26).
Для количественного определения метилирования можно использовать способ высокопроизводительного бисульфитного секвенирования нового поколения (NGS). Скорости метилирования CpG-сайта M3 (COL1A2) в четырех раковых клетках (293T, SW480, NCI-N87 и MCF-7) составили: 65,26%, 81,06%, 48,27% и 67,3%, что соответствует результатам, полученным при обнаружении с помощью системы HOLMESv2-Cas12b.
В настоящее время способ прямого бисульфитного секвенирования и способ бисульфитного секвенирования клонов можно использовать только для полуколичественного определения метилирования. Результаты обнаружения метилирования на CpG-сайте M3 (COL1A2) в вышеуказанных четырех раковых клетках (293T, SW480, NCI-N87 и MCF-7) этими двумя способами составили 63,2%, 63,6%, 30,4%, 20% и 92,3% (12/13), 100% (15/15), 78,6 % (11/14), 71,4% (10/14). По сравнению с результатами, полученными способом высокопроизводительного бисульфитного секвенирования NGS разница была большой, при этом предыдущая была низкой, а последняя была высокой.
Вышеуказанные результаты показывают, что количественные результаты, полученные способом обнаружения с помощью системы HOLMESv2-Cas12b по настоящему изобретению, очень точные и их получение очень удобно и занимает мало времени.
Пример 11
Обнаружение альтернативно сплайсированной РНК и кольцевой РНК
Альтернативное сплайсирование пре-мРНК является ключевым звеном в посттранскрипционной регуляции высоких эукариот и играет важную роль в заболеваниях человека, например в прогрессировании опухоли. Некоторые пре-мРНК могут обрабатываться для образования кольцевой РНК.
В этом примере система HOLMESv2 использовалась для обнаружения сплайсирования мРНК со специфическим нацеливанием на сайт сплайсинга, чтобы обнаружить, в сочетании с RT-LAMP амплификацией, зрелую сплайсированную мРНК гена GAPDH (фиг. 27). Кроме того, аналогичная стратегия была принята при конструировании крРНК, и кольцевая РНК Cdr1as в HEK293T была успешно обнаружена в данном примере (фиг. 27).
1. Приготовление направляющей РНК (одиночной направляющей РНК)
Плазмида pUC18-sgRNA-DNMT1-3 использовалась в качестве матрицы, T7-sgRNA-F использовался в качестве прямого праймера, а олигонуклеотиды, содержащие направляющие последовательности, комплементарные соответствующим мишеням, использовались в качестве обратных праймеров (см. таблицу 2). ДНК-матрица, необходимая для транскрипции in vitro, была амплифицирована ПЦР. Затем в продукт ПЦР был добавлен DpnI (1 мкл DpnI (10 ед/мкл) на 50 мкл системы ПЦР), инкубирован на водяной бане при 37°C в течение 30 минут. Плазмидная ДНК-матрица была дигерирована, и для восстановления продукта ПЦР из геля использовался набор (Promega) с колонкой. Восстановленный продукт ПЦР использовался в качестве матрицы, а одиночная направляющая РНК была синтезирована с помощью набора для транскрипции T7 High Yield Transcription Kit (Thermo). Реакция протекала в ночное время (12–16 часов) при температуре 37°C.
Затем DNase I был добавлен в систему транскрипции (2 мкл DNase I (5 ед/мкл) на 50 мкл системы транскрипции), инкубирован на водяной бане при 37°C в течение 30 минут. ДНК-матрица была удалена, и для очистки РНК использовался набор для очистки и выделения РНК. Затем NanoDrop 2000C использовался для количественного определения, и продукт был помещен в холодильник на хранение при -80°C для дальнейшего использования.
2. Реакция LAMP-амплификации
Геном клетки 293T использовался для проведения реакции LAMP-амплификации. Общий объем каждой реакционной системы составил 25 мкл. 1,6 мкМ LAMP-FIP и LAMP-BIP, 0,2 мкМ LAMP-F3 и LAMP-B3, и 0,4 мкМ LAMP-LoopF и LAMP-LoopB использовались в качестве праймеров. Для LAMP-амплификации использовался набор WarmStart® LAMP Kit (NEB) или ДНК-полимераза Bst 3.0 (NEB). LAMP-реакция протекала при 65°C в течение 30 минут. После завершения LAMP-реакции продукт инактивировался при 85°C в течение 10 минут, а затем напрямую использовался в реакции Cas12b.
3. Реакция Cas12b
(1) Отжиг одиночной направляющей РНК: одиночная направляющая РНК была разбавлена до подходящей концентрации (5 мкМ) и отожжена в приборе для ПЦР. Процедура отжига: денатурация при 75°C в течение 5 минут, затем охлаждение с 75 до 20°C со скоростью охлаждения 1°C в минуту.
(2) Инкубация одиночной направляющей РНК с Cas12b: Отожженная одиночная направляющая РНК была инкубирована с Cas12b в эквимолярной концентрации и помещена при 30°C на 20–30 минут.
(3) Реакция Cas12b: В 20 мкл реакционную систему были добавлены смесь одиночной направляющей РНК и Cas12b, инкубированная на этапе (2) (окончательная концентрация обоих компонентов составляла 250 нМ), 1 мкл LAMP-продукта, флуоресцентный зонд (HEX-N12-BHQ1 с окончательной концентрацией 500 нМ) и 2 мкл буфера 3.1 10×NEB и 0,5 мкл ингибитора РНКаз (40 ед/мкл). После смешивания реакция протекала при 48°C в течение 30 минут. После этого она была инактивирована путем нагрева при 98°C в течение 5 минут в приборе для ПЦР.
4. Обнаружение обходной расщепляющей активности одноцепочечной ДНК белка Cas12b с использованием флуоресцентного микропланшетного ридера.
20 мкл инактивированного реакционного раствора было добавлено в 96-луночный планшет, и проведена детекция с помощью микропланшетного ридера (свет возбуждения 535 нм, свет излучения 556 нм). На фиг. 27 видно, что после LAMP-амплификации, Cas12b (т.е. способ HOLMES v2.0) может обнаруживать и определять, произошло ли ожидаемое сплайсирование РНК или же это кольцевая РНК.
Таблица 1. Олигонуклеотиды в качестве субстратов
Таблица 2. Последовательности-мишени, используемые для транскрипции одиночной направляющей РНК
Таблица 3. Олигонуклеотиды в качестве праймеров для амплификации
Все публикации, упомянутые в настоящем изобретении, включены в виде ссылок, как если бы каждый отдельный документ приводился в качестве примера в виде ссылки в настоящей заявке. Следует понимать, что после ознакомления с вышеуказанными идеями настоящего изобретения специалисты в этой области техники могут внести различные изменения и модификации. Эти эквивалентные формы также попадают в объем, определяемый прилагаемой формулой изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ШAНГХAИ ТОЛО БИОТЕХНОЛОДЖИ КОМПAНИ ЛИМИТЕД
<120> Применение высокотермостойкого Cas-белка, и способ и набор реактивов
для обнаружения целевой молекулы нуклеиновой кислоты
<130> 431117
<150> CN201810560284.8
<151> 2018-06-03
<160> 217
<170> PatentIn version 3.5
<210> 1
<211> 66
<212> ДНК
<213> искусственная последовательность
<400> 1
tttctgtttg ttatcgcaac tttctactga attcaagctt tactctagaa agaggagaaa 60
ggatcc 66
<210> 2
<211> 66
<212> ДНК
<213> искусственная последовательность
<400> 2
ggatcctttc tcctctttct agagtaaagc ttgaattcag tagaaagttg cgataacaaa 60
cagaaa 66
<210> 3
<211> 66
<212> ДНК
<213> искусственная последовательность
<400> 3
ggatcctttc tcctctttct agagtaaagc ttgaattcag tagaaagttg cgataacaaa 60
cagaaa 66
<210> 4
<211> 66
<212> ДНК
<213> искусственная последовательность
<400> 4
ggatcctttc tcctctttct agagtaaagc ttgaattcag tagaaagttg cgataacaaa 60
cagaaa 66
<210> 5
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 5
aatgttccct gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 6
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 6
gtcacgccac ttgacaggcg agtaacagac atggaccatc agggaacatt 50
<210> 7
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 7
aatgaaccct gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 8
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 8
gtcacgccac ttgacaggcg agtaacagac atggaccatc agggttcatt 50
<210> 9
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 9
aatgatccct gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 10
<211> 50
<212> DNA
<213> искусственная последовательность
<400> 10
gtcacgccac ttgacaggcg agtaacagac atggaccatc agggatcatt 50
<210> 11
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 11
aatgtaccct gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 12
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 12
gtcacgccac ttgacaggcg agtaacagac atggaccatc agggtacatt 50
<210> 13
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 13
gtcacgccac ttgacaggcg agtaacagac atggaccatc agggcccatt 50
<210> 14
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 14
gtcacgccac ttgacaggcg agtaacagac atggaccatc agggggcatt 50
<210> 15
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 15
aatgttcgct gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 16
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 16
gtcacgccac ttgacaggcg agtaacagac atggaccatc agcgaacatt 50
<210> 17
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 17
aatgttccgt gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 18
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 18
gtcacgccac ttgacaggcg agtaacagac atggaccatc acggaacatt 50
<210> 19
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 19
aatgttccca gatggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 20
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 20
gtcacgccac ttgacaggcg agtaacagac atggaccatc tgggaacatt 50
<210> 21
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 21
aatgttccct catggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 22
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 22
gtcacgccac ttgacaggcg agtaacagac atggaccatg agggaacatt 50
<210> 23
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 23
aatgttccct gttggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 24
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 24
gtcacgccac ttgacaggcg agtaacagac atggaccaac agggaacatt 50
<210> 25
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 25
aatgttccct gaaggtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 26
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 26
gtcacgccac ttgacaggcg agtaacagac atggaccttc agggaacatt 50
<210> 27
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 27
aatgttccct gatcgtccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 28
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 28
gtcacgccac ttgacaggcg agtaacagac atggacgatc agggaacatt 50
<210> 29
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 29
aatgttccct gatgctccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 30
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 30
gtcacgccac ttgacaggcg agtaacagac atggagcatc agggaacatt 50
<210> 31
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 31
aatgttccct gatggaccat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 32
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 32
gtcacgccac ttgacaggcg agtaacagac atggtccatc agggaacatt 50
<210> 33
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 33
aatgttccct gatggtgcat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 34
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 34
gtcacgccac ttgacaggcg agtaacagac atgcaccatc agggaacatt 50
<210> 35
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 35
aatgttccct gatggtcgat gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 36
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 36
gtcacgccac ttgacaggcg agtaacagac atcgaccatc agggaacatt 50
<210> 37
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 37
aatgttccct gatggtcctt gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 38
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 38
gtcacgccac ttgacaggcg agtaacagac aaggaccatc agggaacatt 50
<210> 39
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 39
aatgttccct gatggtccaa gtctgttact cgcctgtcaa gtggcgtgac 50
<210> 40
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 40
gtcacgccac ttgacaggcg agtaacagac ttggaccatc agggaacatt 50
<210> 41
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 41
aatgttccct gatggtccat ctctgttact cgcctgtcaa gtggcgtgac 50
<210> 42
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 42
gtcacgccac ttgacaggcg agtaacagag atggaccatc agggaacatt 50
<210> 43
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 43
aatgttccct gatggtccat gactgttact cgcctgtcaa gtggcgtgac 50
<210> 44
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 44
gtcacgccac ttgacaggcg agtaacagtc atggaccatc agggaacatt 50
<210> 45
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 45
aatgttccct gatggtccat gtgtgttact cgcctgtcaa gtggcgtgac 50
<210> 46
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 46
gtcacgccac ttgacaggcg agtaacacac atggaccatc agggaacatt 50
<210> 47
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 47
aatgttccct gatggtccat gtcagttact cgcctgtcaa gtggcgtgac 50
<210> 48
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 48
gtcacgccac ttgacaggcg agtaactgac atggaccatc agggaacatt 50
<210> 49
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 49
agacttagat ctgagccctc cctcttccca gcacaggcag gggtagaagc 50
<210> 50
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 50
agacttagat ctgagccctc cctcttccca gaacaggcag gggtagaagc 50
<210> 51
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 51
tgtgtttgga tttgcagtag gctgaagcgt tatactatga ctggagtcca 50
<210> 52
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 52
tgtgtttgga tttgcagtag gctgaagcgt tgtactatga ctggagtcca 50
<210> 53
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 53
aaaatagtgc tttttacttt tatctgaatg attgaaatgt ccttttccca 50
<210> 54
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 54
aaaatagtgc tttttacttt tatctgaatg actgaaatgt ccttttccca 50
<210> 55
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 55
gtcacgccac ttgacaggcg agtaacagac atggaccatc agtgaacatt 50
<210> 56
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 56
gtcacgccac ttgacaggcg agtaacagac atggaccatc agagaacatt 50
<210> 57
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 57
gtcacgccac ttgacaggcg agtaacagac atggaccatc aaggaacatt 50
<210> 58
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 58
gtcacgccac ttgacaggcg agtaacagac atggaccatc atggaacatt 50
<210> 59
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 59
gtcacgccac ttgacaggcg agtaacagac atggaccatc cgggaacatt 50
<210> 60
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 60
gtcacgccac ttgacaggcg agtaacagac atggaccatc ggggaacatt 50
<210> 61
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 61
gtcacgccac ttgacaggcg agtaacagac atggaccata agggaacatt 50
<210> 62
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 62
gtcacgccac ttgacaggcg agtaacagac atggaccatt agggaacatt 50
<210> 63
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 63
gtcacgccac ttgacaggcg agtaacagac atggaccacc agggaacatt 50
<210> 64
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 64
gtcacgccac ttgacaggcg agtaacagac atggaccagc agggaacatt 50
<210> 65
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 65
gtcacgccac ttgacaggcg agtaacagac atggaccctc agggaacatt 50
<210> 66
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 66
gtcacgccac ttgacaggcg agtaacagac atggaccgtc agggaacatt 50
<210> 67
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 67
gtcacgccac ttgacaggcg agtaacagac atggacaatc agggaacatt 50
<210> 68
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 68
gtcacgccac ttgacaggcg agtaacagac atggactatc agggaacatt 50
<210> 69
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 69
gtcacgccac ttgacaggcg agtaacagac atggaacatc agggaacatt 50
<210> 70
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 70
gtcacgccac ttgacaggcg agtaacagac atggatcatc agggaacatt 50
<210> 71
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 71
gtcacgccac ttgacaggcg agtaacagac atggcccatc agggaacatt 50
<210> 72
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 72
gtcacgccac ttgacaggcg agtaacagac atgggccatc agggaacatt 50
<210> 73
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 73
gtcacgccac ttgacaggcg agtaacagac atgaaccatc agggaacatt 50
<210> 74
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 74
gtcacgccac ttgacaggcg agtaacagac atgtaccatc agggaacatt 50
<210> 75
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 75
gtcacgccac ttgacaggcg agtaacagac atagaccatc agggaacatt 50
<210> 76
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 76
gtcacgccac ttgacaggcg agtaacagac attgaccatc agggaacatt 50
<210> 77
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 77
gtcacgccac ttgacaggcg agtaacagac acggaccatc agggaacatt 50
<210> 78
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 78
gtcacgccac ttgacaggcg agtaacagac agggaccatc agggaacatt 50
<210> 79
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 79
gtcacgccac ttgacaggcg agtaacagac ctggaccatc agggaacatt 50
<210> 80
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 80
gtcacgccac ttgacaggcg agtaacagac gtggaccatc agggaacatt 50
<210> 81
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 81
gtcacgccac ttgacaggcg agtaacagaa atggaccatc agggaacatt 50
<210> 82
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 82
gtcacgccac ttgacaggcg agtaacagat atggaccatc agggaacatt 50
<210> 83
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 83
gtcacgccac ttgacaggcg agtaacagcc atggaccatc agggaacatt 50
<210> 84
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 84
gtcacgccac ttgacaggcg agtaacaggc atggaccatc agggaacatt 50
<210> 85
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 85
gtcacgccac ttgacaggcg agtaacaaac atggaccatc agggaacatt 50
<210> 86
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 86
gtcacgccac ttgacaggcg agtaacatac atggaccatc agggaacatt 50
<210> 87
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 87
agacttagat ctgagccctc cctcttccca gcacacgcag gggtagaagc 50
<210> 88
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 88
agacttagat ctgagccctc cctcttccca gcactggcag gggtagaagc 50
<210> 89
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 89
agacttagat ctgagccctc cctcttccca gcagaggcag gggtagaagc 50
<210> 90
<211> 50
<212> ДНК
<213> artificial sequence
<400> 90
agacttagat ctgagccctc cctcttccca gctcaggcag gggtagaagc 50
<210> 91
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 91
agacttagat ctgagccctc cctcttccca ggacaggcag gggtagaagc 50
<210> 92
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 92
agacttagat ctgagccctc cctcttccca ccacaggcag gggtagaagc 50
<210> 93
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 93
agacttagat ctgagccctc cctcttccct gcacaggcag gggtagaagc 50
<210> 94
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 94
agacttagat ctgagccctc cctcttccga gcacaggcag gggtagaagc 50
<210> 95
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 95
agacttagat ctgagccctc cctcttcgca gcacaggcag gggtagaagc 50
<210> 96
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 96
tgtgtttgga tttgcagtag gctgaagcgt tatacaatga ctggagtcca 50
<210> 97
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 97
tgtgtttgga tttgcagtag gctgaagcgt tatagtatga ctggagtcca 50
<210> 98
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 98
tgtgtttgga tttgcagtag gctgaagcgt tattctatga ctggagtcca 50
<210> 99
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 99
tgtgtttgga tttgcagtag gctgaagcgt taaactatga ctggagtcca 50
<210> 100
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 100
tgtgtttgga tttgcagtag gctgaagcgt tttactatga ctggagtcca 50
<210> 101
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 101
tgtgtttgga tttgcagtag gctgaagcgt aatactatga ctggagtcca 50
<210> 102
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 102
tgtgtttgga tttgcagtag gctgaagcga tatactatga ctggagtcca 50
<210> 103
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 103
tgtgtttgga tttgcagtag gctgaagcct tatactatga ctggagtcca 50
<210> 104
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 104
tgtgtttgga tttgcagtag gctgaagggt tatactatga ctggagtcca 50
<210> 105
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 105
aaaatagtgc tttttacttt tatctgaatg attgatatgt ccttttccca 50
<210> 106
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 106
aaaatagtgc tttttacttt tatctgaatg attgtaatgt ccttttccca 50
<210> 107
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 107
aaaatagtgc tttttacttt tatctgaatg attcaaatgt ccttttccca 50
<210> 108
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 108
aaaatagtgc tttttacttt tatctgaatg atagaaatgt ccttttccca 50
<210> 109
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 109
aaaatagtgc tttttacttt tatctgaatg aatgaaatgt ccttttccca 50
<210> 110
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 110
aaaatagtgc tttttacttt tatctgaatg tttgaaatgt ccttttccca 50
<210> 111
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 111
aaaatagtgc tttttacttt tatctgaatc attgaaatgt ccttttccca 50
<210> 112
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 112
aaaatagtgc tttttacttt tatctgaaag attgaaatgt ccttttccca 50
<210> 113
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 113
aaaatagtgc tttttacttt tatctgattg attgaaatgt ccttttccca 50
<210> 114
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 114
ggatctcctg gcggaggtgg tggtagaagg tccaggagca ggggtagccg 50
<210> 115
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 115
ggatctcctg gcggaggtgg tggtagaagg tctaggagca ggggtagccg 50
<210> 116
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 116
agcacatgtt tgttgaatga gtcagtggat gacgtagatc agtatcttgt 50
<210> 117
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 117
agcacatgtt tgttgaatga gtcagtggat gatgtagatc agtatcttgt 50
<210> 118
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 118
gcttctttcc ctgcctgtgc tgggaagagg gagggctcag atctaagtct 50
<210> 119
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 119
agacttagat ctgagccctc cctcttccca gcacaggcag ggaaagaagc 50
<210> 120
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 120
gcttctttcc ctgcctgttc tgggaagagg gagggctcag atctaagtct 50
<210> 121
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 121
agacttagat ctgagccctc cctcttccca gaacaggcag ggaaagaagc 50
<210> 122
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 122
gcttttaccc ctgcctgtgc tgggaagagg gagggctcag atctaagtct 50
<210> 123
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 123
agacttagat ctgagccctc cctcttccca gcacaggcag gggtaaaagc 50
<210> 124
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 124
gcttttaccc ctgcctgttc tgggaagagg gagggctcag atctaagtct 50
<210> 125
<211> 50
<212> ДНК
<213> искусственная последовательность
<400> 125
agacttagat ctgagccctc cctcttccca gaacaggcag gggtaaaagc 50
<210> 126
<211> 12
<212> ДНК
<213> искусственная последовательность
<220>
<221> misc_feature
<222> (1)..(12)
<223> n is a, c, g, or t
<400> 126
nnnnnnnnnn nn 12
<210> 127
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 127
ctaccgtaat actaaaagtg ccacttctca gatttgagaa g 41
<210> 128
<211> 25
<212> ДНК
<213> искусственная последовательность
<400> 128
gaaattaata cgactcacta taggg 25
<210> 129
<211> 68
<212> ДНК
<213> искусственная последовательность
<400> 129
tcgcgcttgt cgcgcagacg aatgatctac aacagtagaa attccctata gtgagtcgta 60
ttaatttc 68
<210> 130
<211> 23
<212> ДНК
<213> искусственная последовательность
<400> 130
aacagacatg gaccatcagg gtg 23
<210> 131
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 131
catggaccat cagggtgcca c 21
<210> 132
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 132
acatggacca tcagggtgcc ac 22
<210> 133
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 133
gacatggacc atcagggtgc c 21
<210> 134
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 134
agacatggac catcagggtg c 21
<210> 135
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 135
cagacatgga ccatcagggt g 21
<210> 136
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 136
acagacatgg accatcaggg tg 22
<210> 137
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 137
ccagcacagg cagggggtgc cacttctcag atttgagaag 40
<210> 138
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 138
cgttatacta tgactggtgc cacttctcag atttgagaag 40
<210> 139
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 139
atgattgaaa tgtcctgtgc cacttctcag atttgagaag 40
<210> 140
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 140
ccagaacagg cagggggtgc cacttctcag atttgagaag 40
<210> 141
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 141
cgttgtacta tgactggtgc cacttctcag atttgagaag 40
<210> 142
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 142
atgactgaaa tgtcctgtgc cacttctcag atttgagaag 40
<210> 143
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 143
aggtccagga gcaggggtgc cacttctcag atttgagaag 40
<210> 144
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 144
aggtctagga gcaggggtgc cacttctcag atttgagaag 40
<210> 145
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 145
gatgacgtag atcagtgtgc cacttctcag atttgagaag 40
<210> 146
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 146
gatgatgtag atcagtgtgc cacttctcag atttgagaag 40
<210> 147
<211> 44
<212> ДНК
<213> искусственная последовательность
<400> 147
atatcttcta cttctccact gtgccacttc tcagatttga gaag 44
<210> 148
<211> 44
<212> ДНК
<213> искусственная последовательность
<400> 148
tctagagaat cccagaatgc gtgccacttc tcagatttga gaag 44
<210> 149
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 149
gcgttatact atgactgtgc cacttctcag atttgagaag 40
<210> 150
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 150
gcgttgtact atgactgtgc cacttctcag atttgagaag 40
<210> 151
<211> 42
<212> ДНК
<213> искусственная последовательность
<400> 151
cttcccagca caggcagggt gccacttctc agatttgaga ag 42
<210> 152
<211> 42
<212> ДНК
<213> искусственная последовательность
<400> 152
cttcccagaa caggcagggt gccacttctc agatttgaga ag 42
<210> 153
<211> 42
<212> ДНК
<213> искусственная последовательность
<400> 153
tcccagcaca ggcagggggt gccacttctc agatttgaga ag 42
<210> 154
<211> 42
<212> ДНК
<213> искусственная последовательность
<400> 154
tcccagaaca ggcagggggt gccacttctc agatttgaga ag 42
<210> 155
<211> 44
<212> ДНК
<213> искусственная последовательность
<400> 155
tgtgatccaa gacattcccc gtgccacttc tcagatttga gaag 44
<210> 156
<211> 44
<212> ДНК
<213> искусственная последовательность
<400> 156
ctcgtcagtg ctctcctctc gtgccacttc tcagatttga gaag 44
<210> 157
<211> 43
<212> ДНК
<213> искусственная последовательность
<400> 157
tcttcccagc acaggcaggg tgccacttct cagatttgag aag 43
<210> 158
<211> 43
<212> ДНК
<213> искусственная последовательность
<400> 158
tcttcccaga acaggcaggg tgccacttct cagatttgag aag 43
<210> 159
<211> 43
<212> ДНК
<213> искусственная последовательность
<400> 159
ttcccagcac aggcaggggg tgccacttct cagatttgag aag 43
<210> 160
<211> 43
<212> ДНК
<213> искусственная последовательность
<400> 160
ttcccagaac aggcaggggg tgccacttct cagatttgag aag 43
<210> 161
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 161
gtgaacgttc ccttagcact 20
<210> 162
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 162
gggagggcag aactagtcc 19
<210> 163
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 163
cgccacttga caggcgagta actgccactt attgggtcag c 41
<210> 164
<211> 39
<212> ДНК
<213> искусственная последовательность
<400> 164
gcgtgttccc cagagtgact tagcagcttc ctcctcctt 39
<210> 165
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 165
aggaaacatt aacgtactga tg 22
<210> 166
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 166
ttccttttat ttcccttcag c 21
<210> 167
<211> 18
<212> ДНК
<213> искусственная последовательность
<400> 167
cgacggcaaa gaagacca 18
<210> 168
<211> 18
<212> ДНК
<213> искусственная последовательность
<400> 168
agcctgccag gtgagtac 18
<210> 169
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 169
cgggtggatc ggcgttttgt tcactatgaa ggcggcatca 40
<210> 170
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 170
gtattggcgt cgaagtggcg ttcgctgcgg aatgttgttg 40
<210> 171
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 171
ttgttcagat attcaacgaa cg 22
<210> 172
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 172
gtggaacgat ggcttccagg 20
<210> 173
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 173
tctctgtgca tggcctgta 19
<210> 174
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 174
aacagtaaag gcaacgtcca 20
<210> 175
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 175
gcagctggga taccagtgga agtgcctcct ggaagaatgg 40
<210> 176
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 176
tctctagagc catcttgcgc ctgaagcgac ccatgaacgc 40
<210> 177
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 177
tgcttactga agccgaaaaa tg 22
<210> 178
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 178
tgatcgcgag accacacgat g 21
<210> 179
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 179
ggtttcggat gttacagcgt 20
<210> 180
<211> 35
<212> ДНК
<213> искусственная последовательность
<400> 180
caagcacccc acccgctcac ccacctcctc ctttg 35
<210> 181
<211> 43
<212> ДНК
<213> искусственная последовательность
<400> 181
ggtttcggat gttacagcgt gtgctggaag acttagatct gag 43
<210> 182
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 182
gctggaaagg tcaagggac 19
<210> 183
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 183
ggggtttgtt gcacagtcc 19
<210> 184
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 184
ggtagaagca aaggcaggag gtttgcccaa ggtcacacag 40
<210> 185
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 185
gaaagaagca aaggcaggag gtttgcccaa ggtcacacag 40
<210> 186
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 186
ctgggaagag ggagggctca gtgttgccac actttcactg g 41
<210> 187
<211> 18
<212> ДНК
<213> искусственная последовательность
<400> 187
gtgagcgggt ggggtgct 18
<210> 188
<211> 23
<212> ДНК
<213> искусственная последовательность
<400> 188
tctaagtctt ccagcacggg atc 23
<210> 189
<211> 18
<212> ДНК
<213> искусственная последовательность
<400> 189
tgacacagcc tgggactt 18
<210> 190
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 190
cacacctcct gtggctaag 19
<210> 191
<211> 40
<212> ДНК
<213> искусственная последовательность
<400> 191
gagtgttctg aaggcaccac cagctccatt ggaggggtct 40
<210> 192
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 192
acacaagggc taatgggtgc cgccaaagca attgatcggt c 41
<210> 193
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 193
ttggtgaacg gcttttccta tg 22
<210> 194
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 194
tgctctggat gggcgtcaac g 21
<210> 195
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 195
gagacaaagg aatgccctga 20
<210> 196
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 196
gccctctcaa gtttccatgt 20
<210> 197
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 197
cgggttgatg tgatgccaaa gcgagcacag agcttggaac a 41
<210> 198
<211> 42
<212> ДНК
<213> искусственная последовательность
<400> 198
ggagcgatca taggtacggc tggcgactct caatccagta cg 42
<210> 199
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 199
gaagtcttcg atttgcatg 19
<210> 200
<211> 22
<212> ДНК
<213> искусственная последовательность
<400> 200
acatgtggca gtccatagtg ac 22
<210> 201
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 201
agggatctcg ctcctggaag atcaccgtca aggctgagaa c 41
<210> 202
<211> 38
<212> ДНК
<213> искусственная последовательность
<400> 202
cgatgctggc gctgagtacg aatgagcccc agccttct 38
<210> 203
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 203
tccacccatg gcaaattcc 19
<210> 204
<211> 19
<212> ДНК
<213> искусственная последовательность
<400> 204
agggggcaga gatgatgac 19
<210> 205
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 205
tccattgatg acaagcttcc c 21
<210> 206
<211> 20
<212> ДНК
<213> искусственная последовательность
<400> 206
tcgtggagtc cactggcgtc 20
<210> 207
<211> 46
<212> ДНК
<213> искусственная последовательность
<400> 207
ccagatcttc caggaaaatc cacatctgta tttgatggaa gacctt 46
<210> 208
<211> 41
<212> ДНК
<213> искусственная последовательность
<400> 208
agaccagtaa ttgctggaag acttgtcttc caagaagctc c 41
<210> 209
<211> 21
<212> ДНК
<213> искусственная последовательность
<400> 209
agatttttct ggaagacatg g 21
<210> 210
<211> 18
<212> ДНК
<213> искусственная последовательность
<400> 210
atgtcttccg gacaatcc 18
<210> 211
<211> 23
<212> ДНК
<213> искусственная последовательность
<400> 211
tgctggaaga cttgatttac tgg 23
<210> 212
<211> 24
<212> ДНК
<213> искусственная последовательность
<400> 212
ggaggcaccc tagggccagg gaaa 24
<210> 213
<211> 25
<212> ДНК
<213> искусственная последовательность
<400> 213
gttactgcaa gcagcaacaa agtcc 25
<210> 214
<211> 24
<212> ДНК
<213> искусственная последовательность
<400> 214
ggaggtattt tagggttagg gaaa 24
<210> 215
<211> 25
<212> ДНК
<213> искусственная последовательность
<400> 215
attactacaa acaacaacaa aatcc 25
<210> 216
<211> 44
<212> ДНК
<213> искусственная последовательность
<400> 216
ggcgtcttca ccaccatgga gtgccacttc tcagatttga gaag 44
<210> 217
<211> 44
<212> ДНК
<213> искусственная последовательность
<400> 217
ctccaagtct tccagtaaat gtgccacttc tcagatttga gaag 44
<---
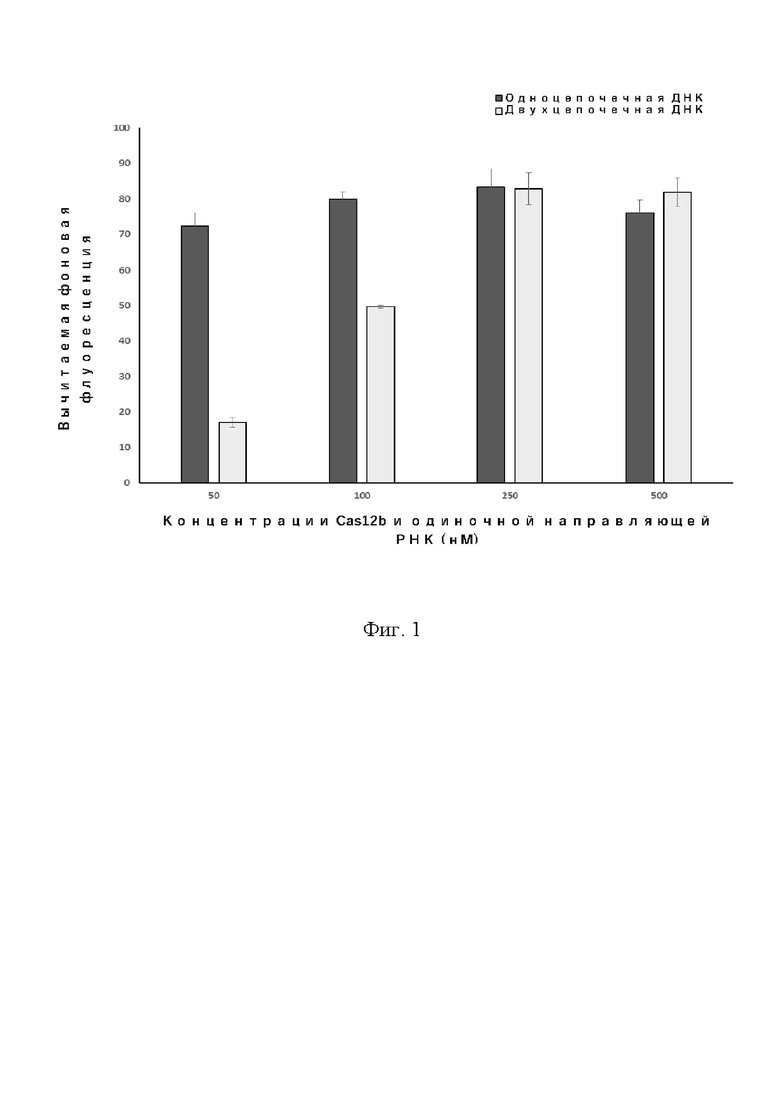
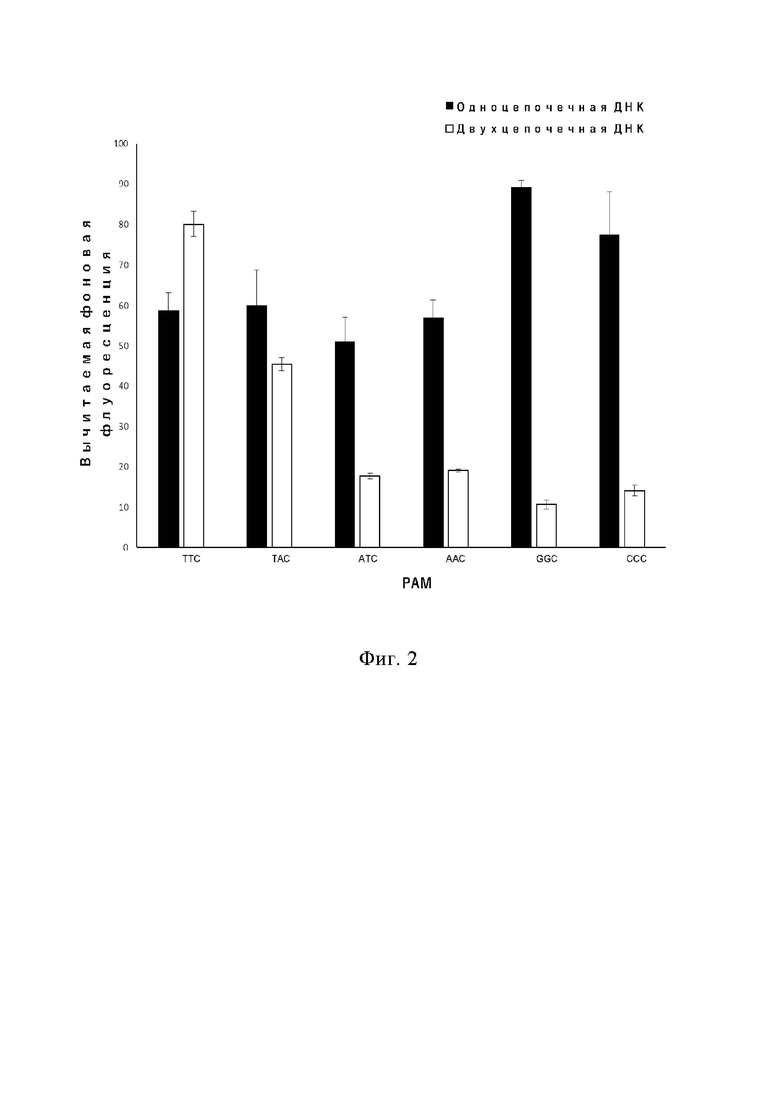
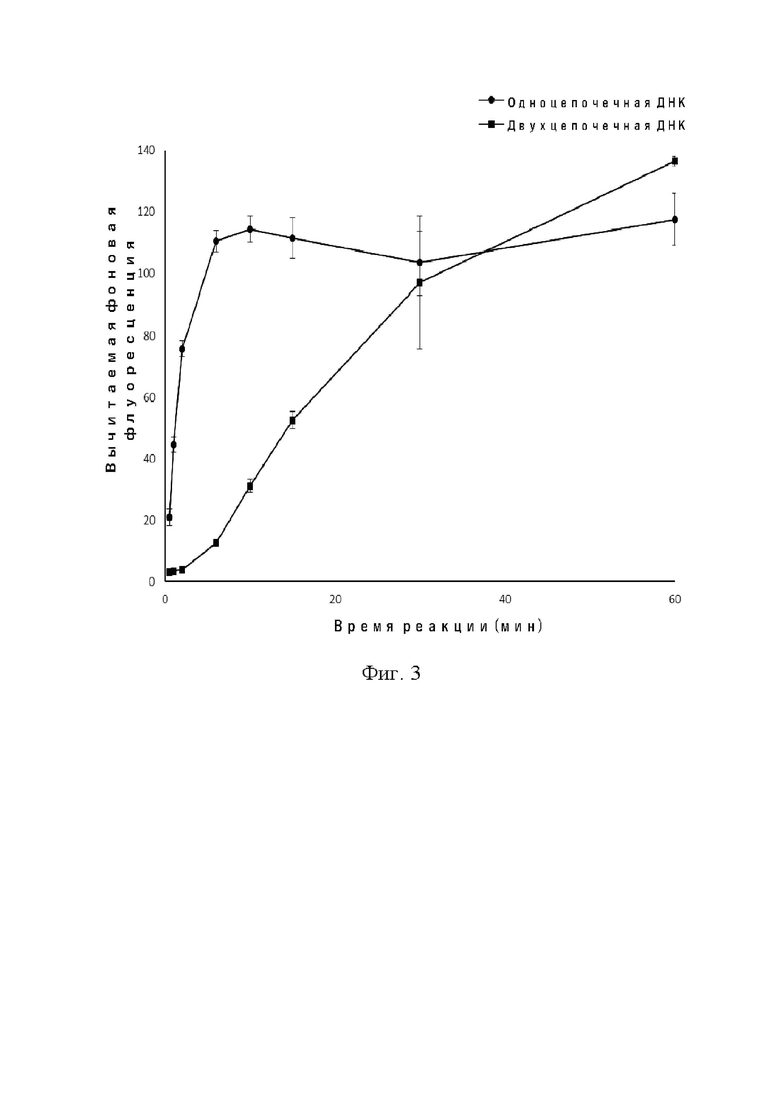
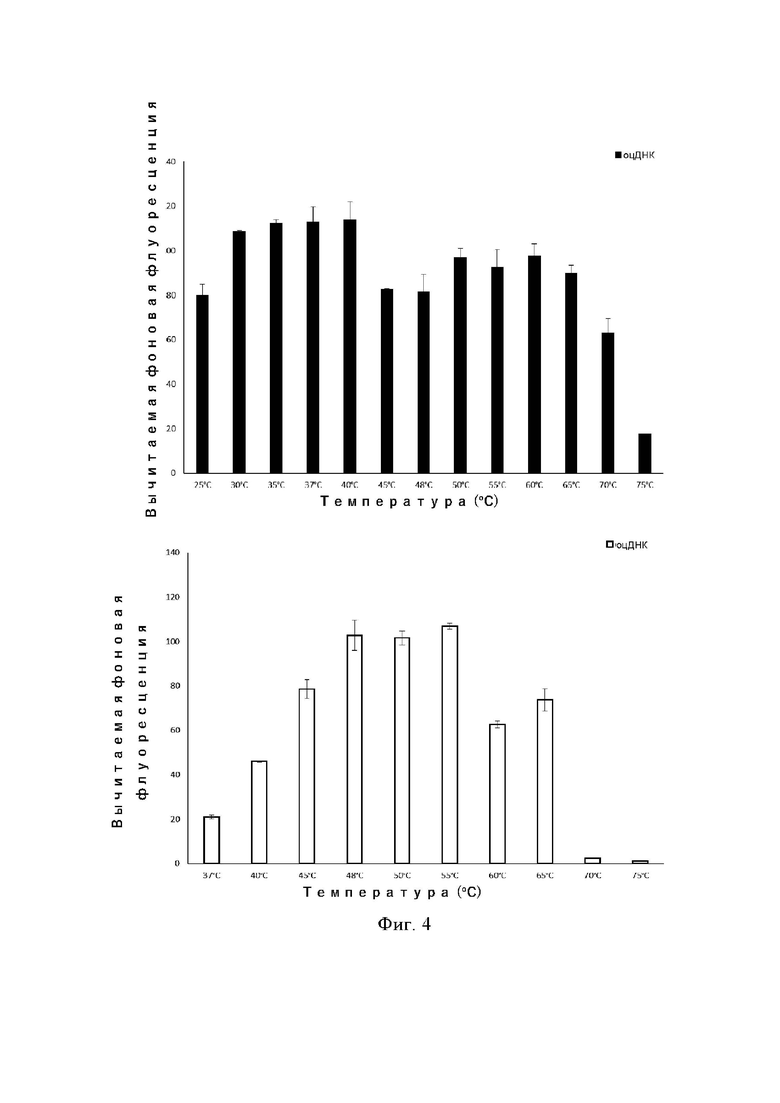
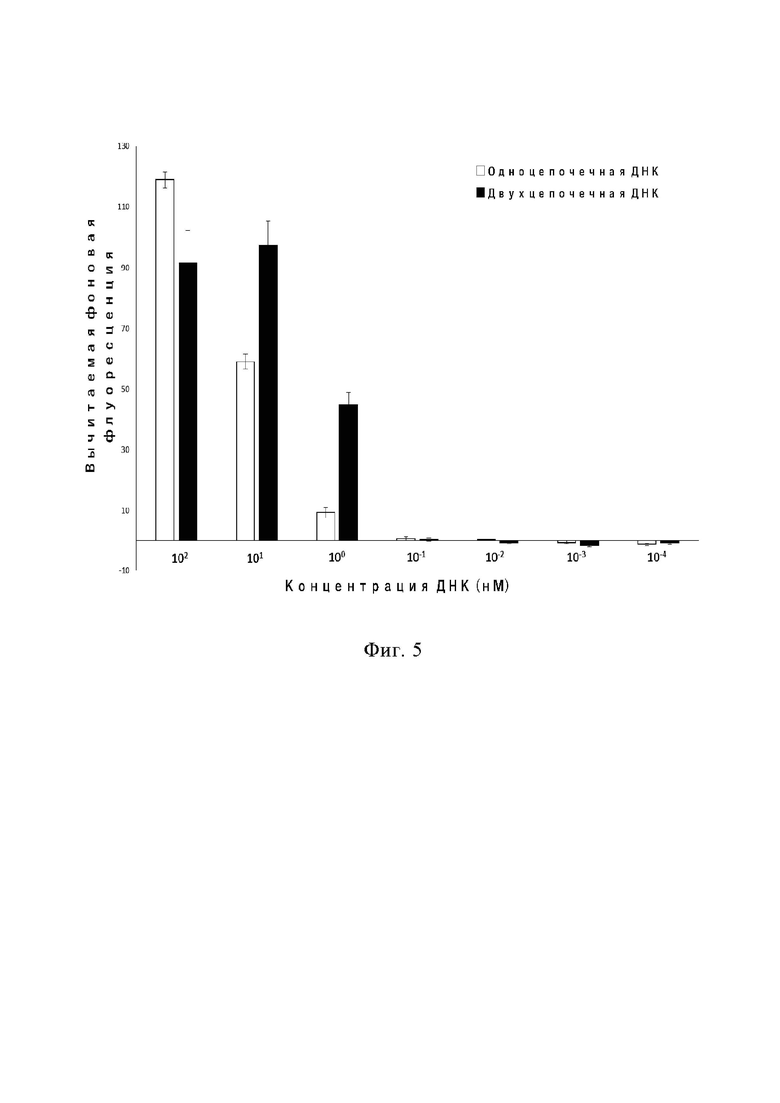
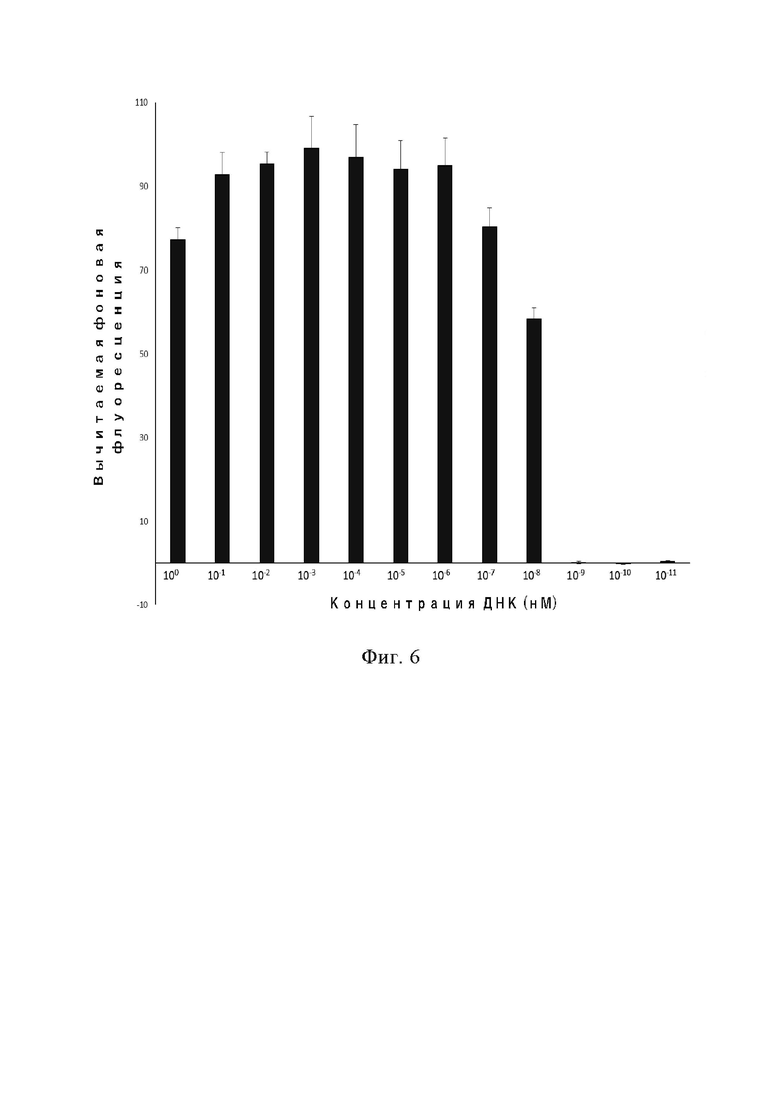
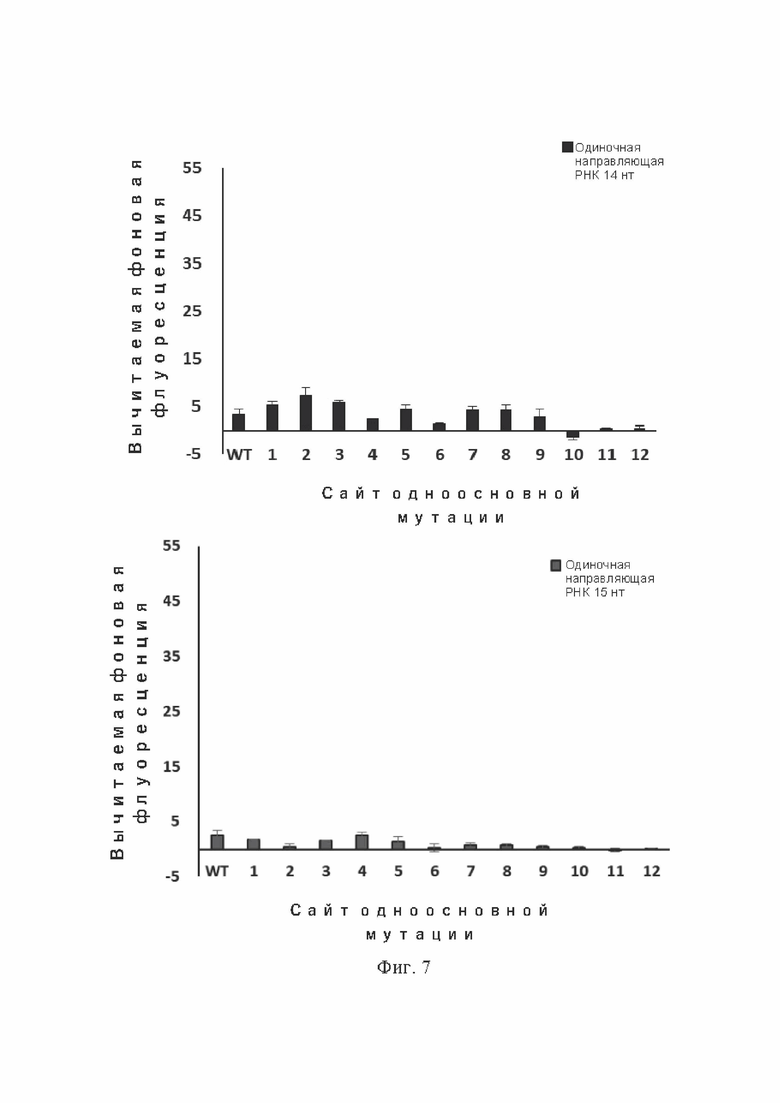
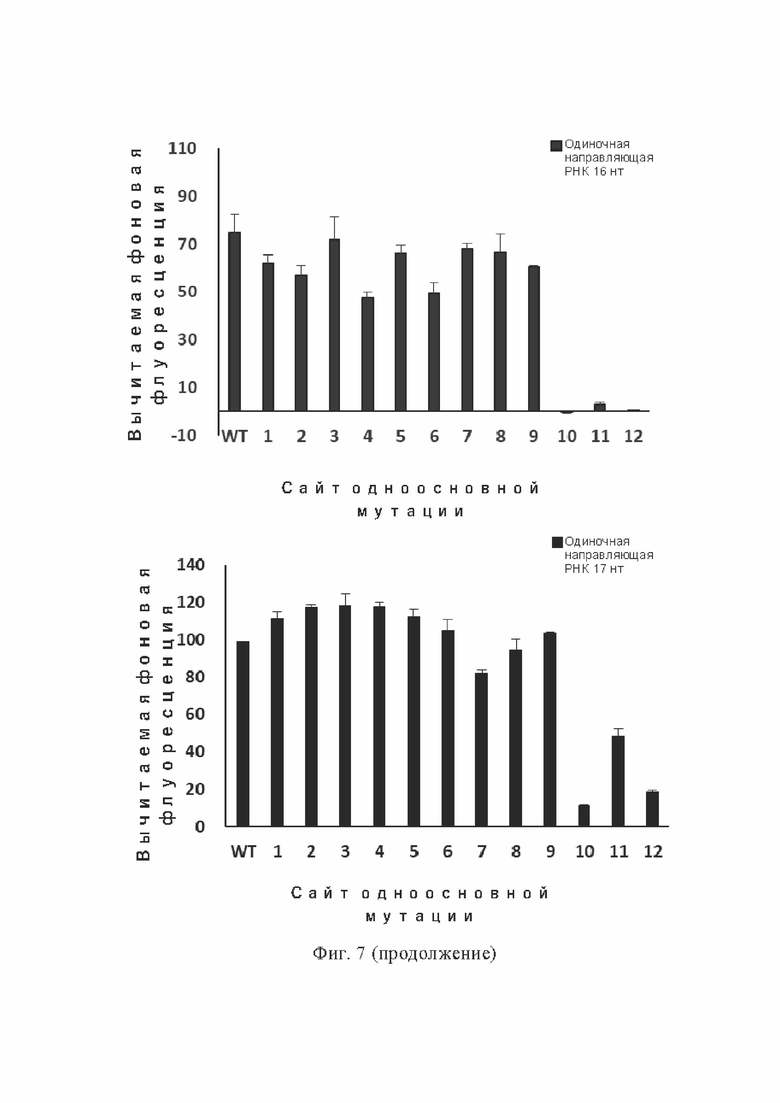
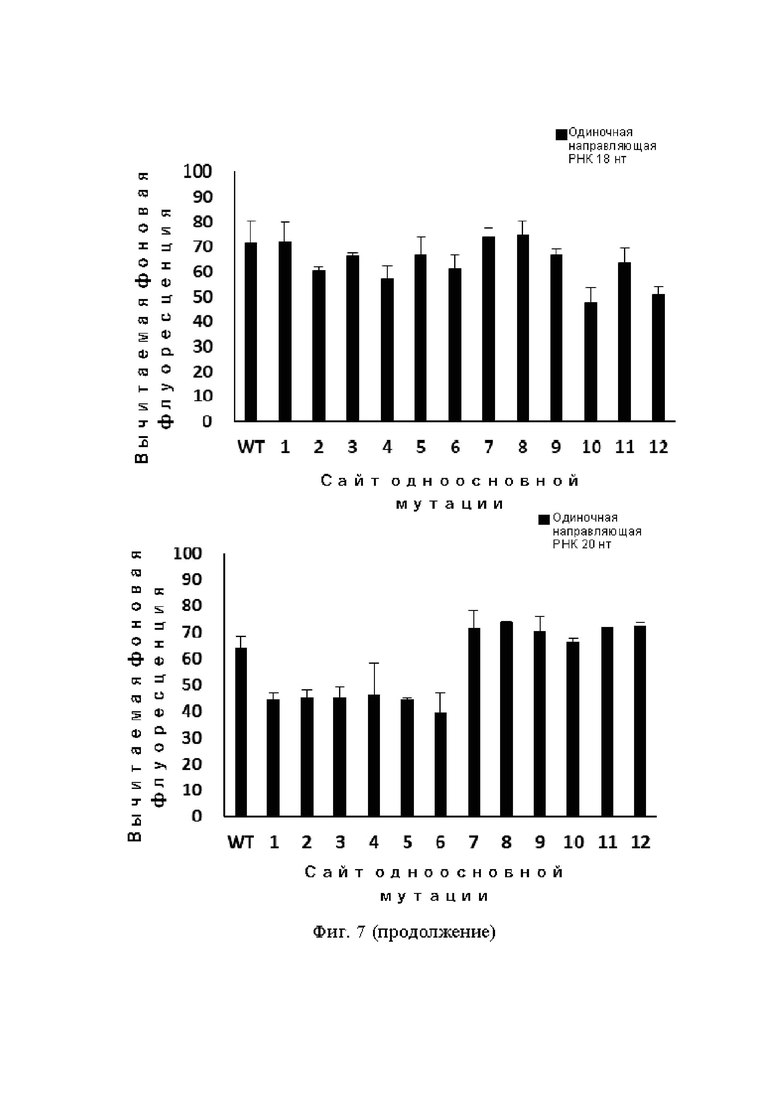
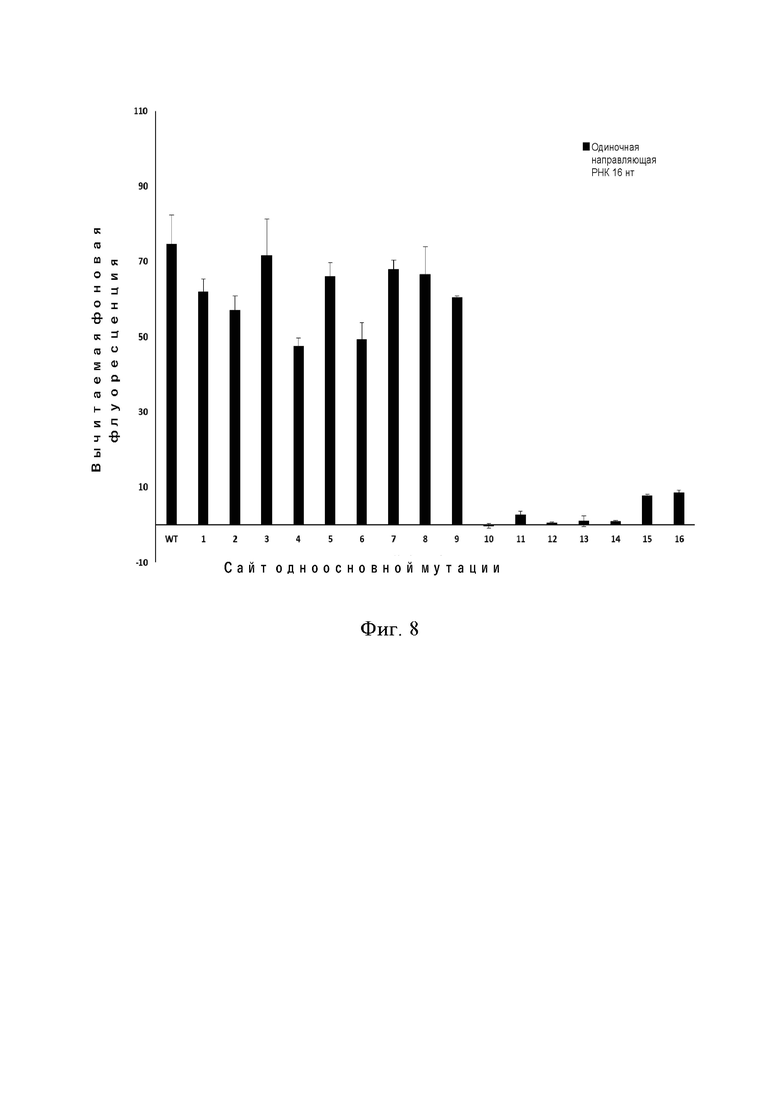
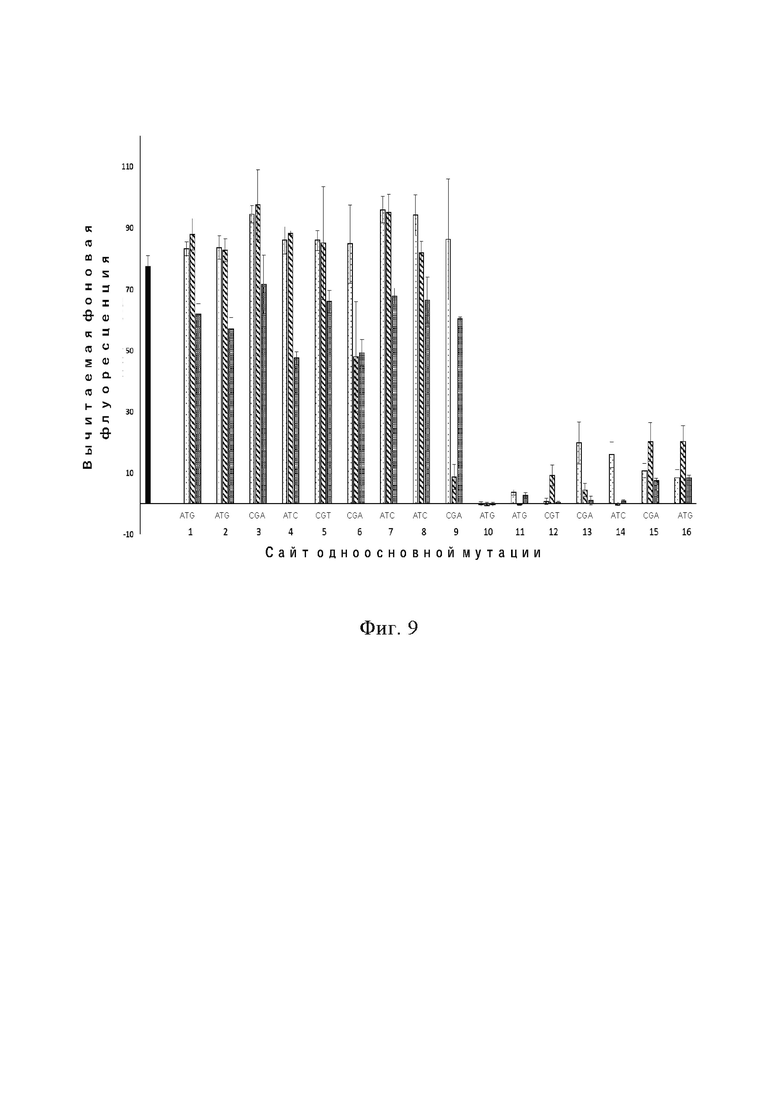
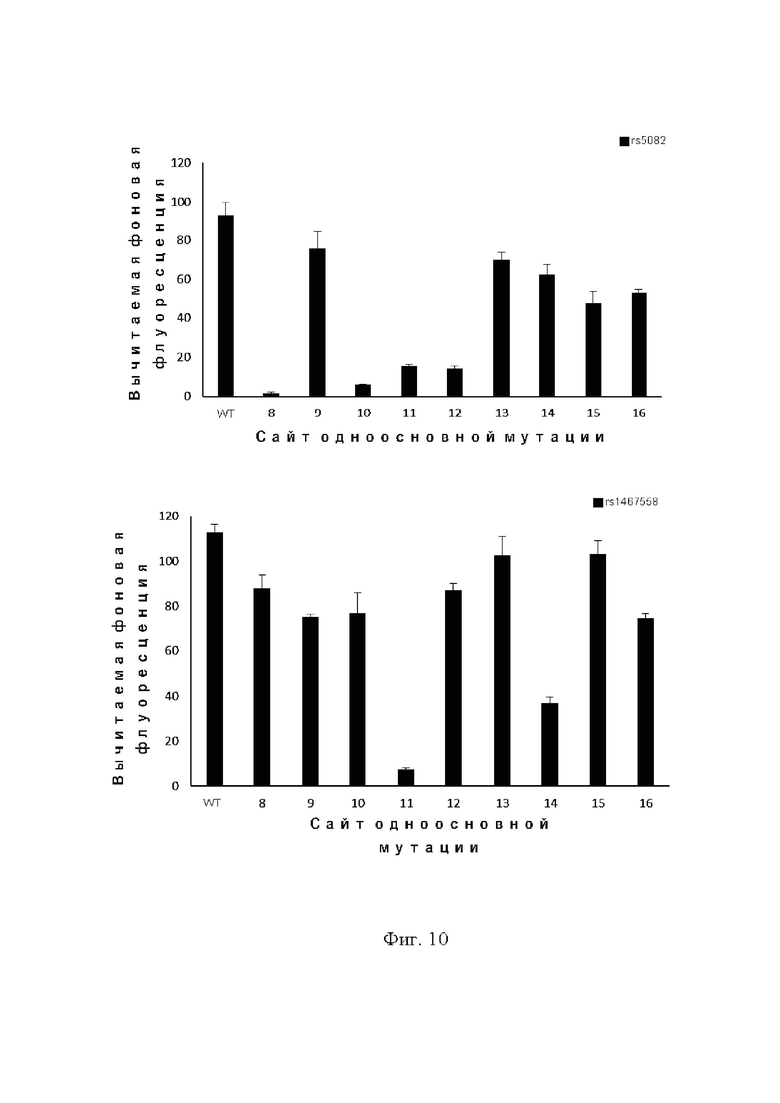
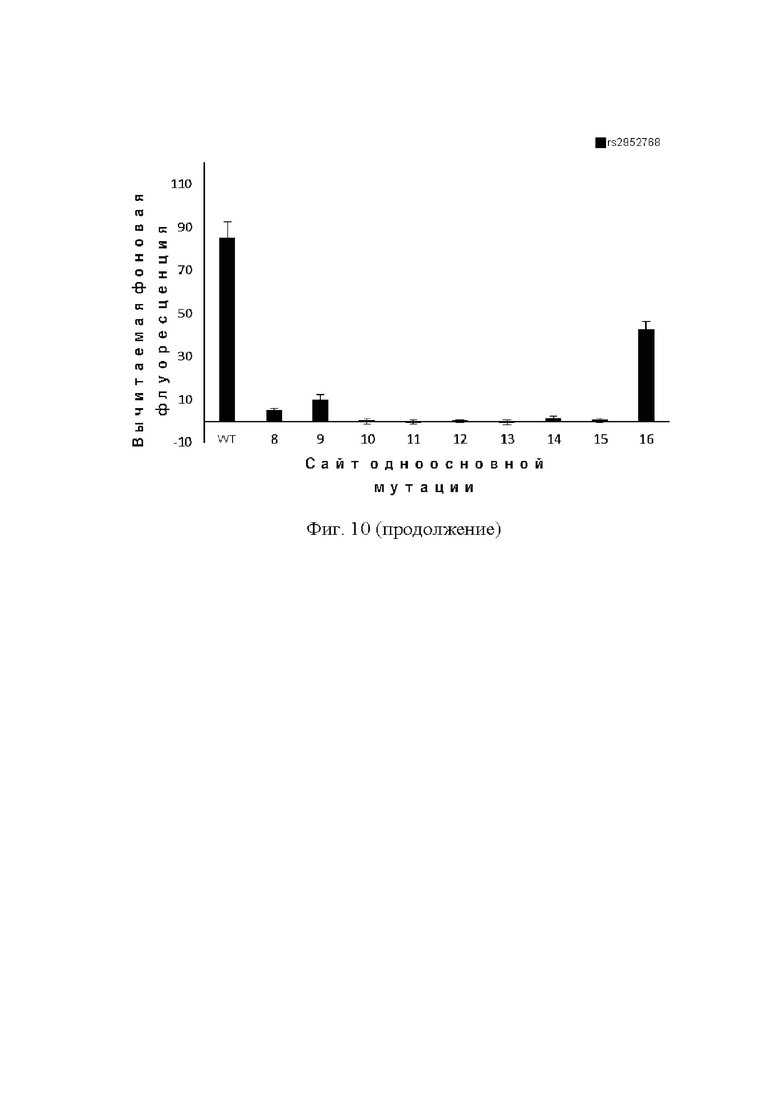
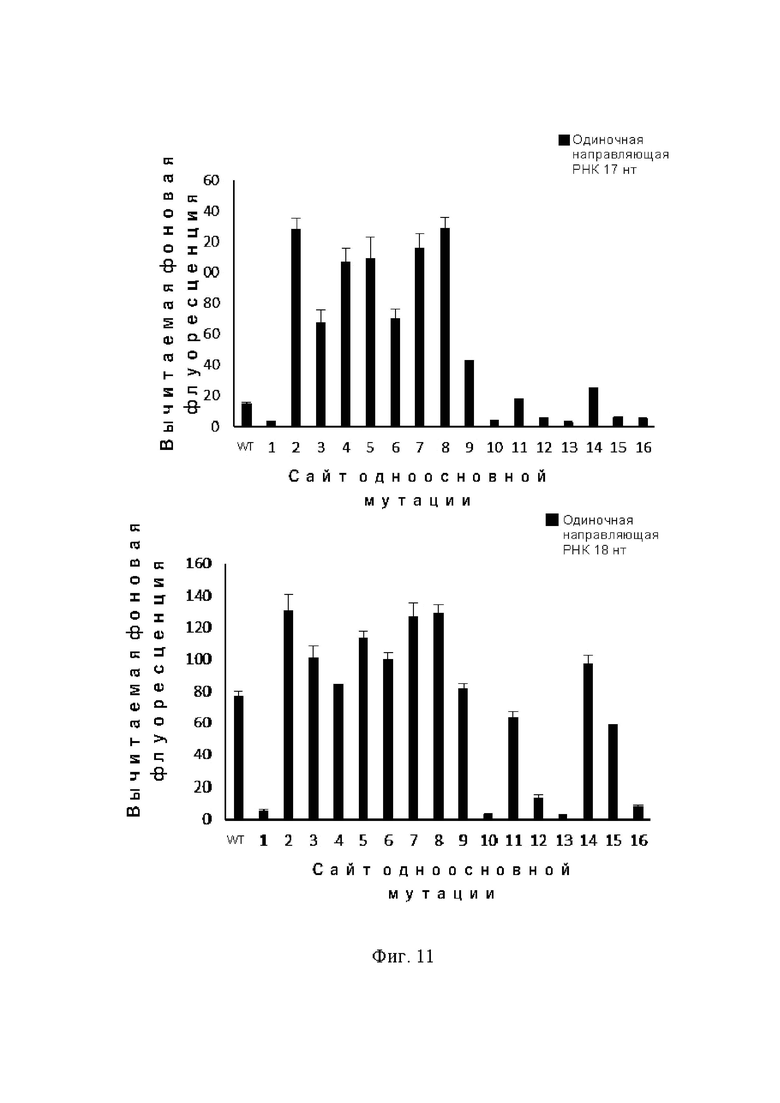
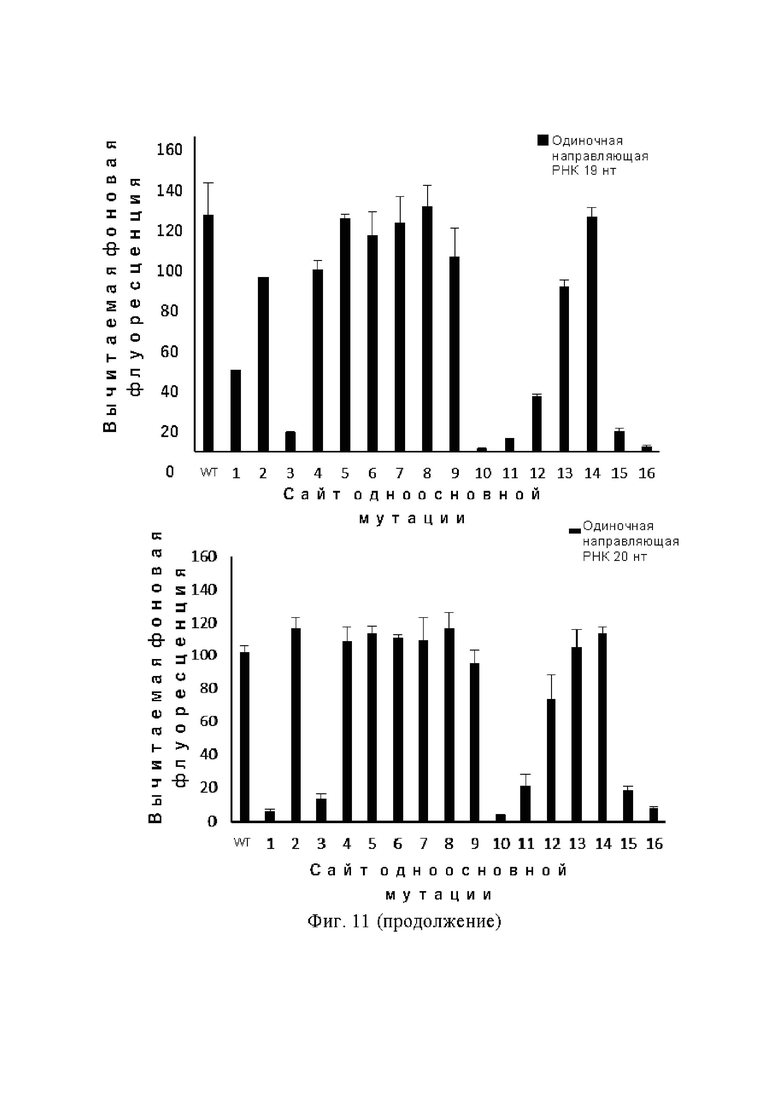
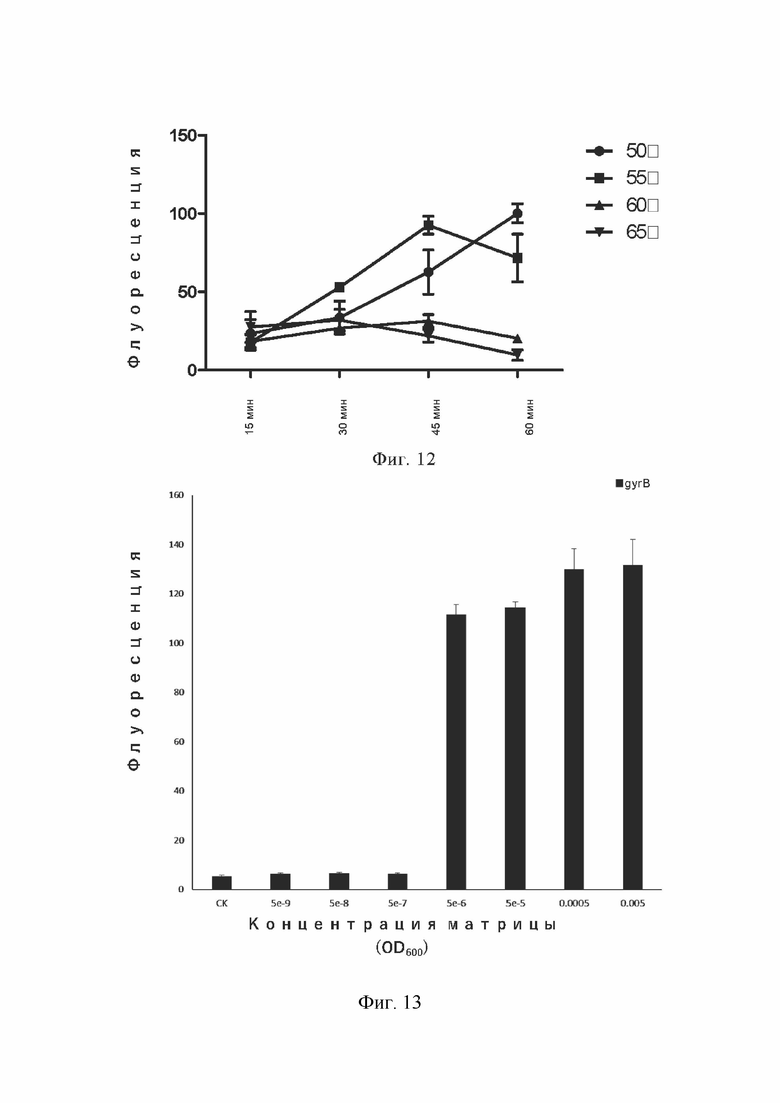
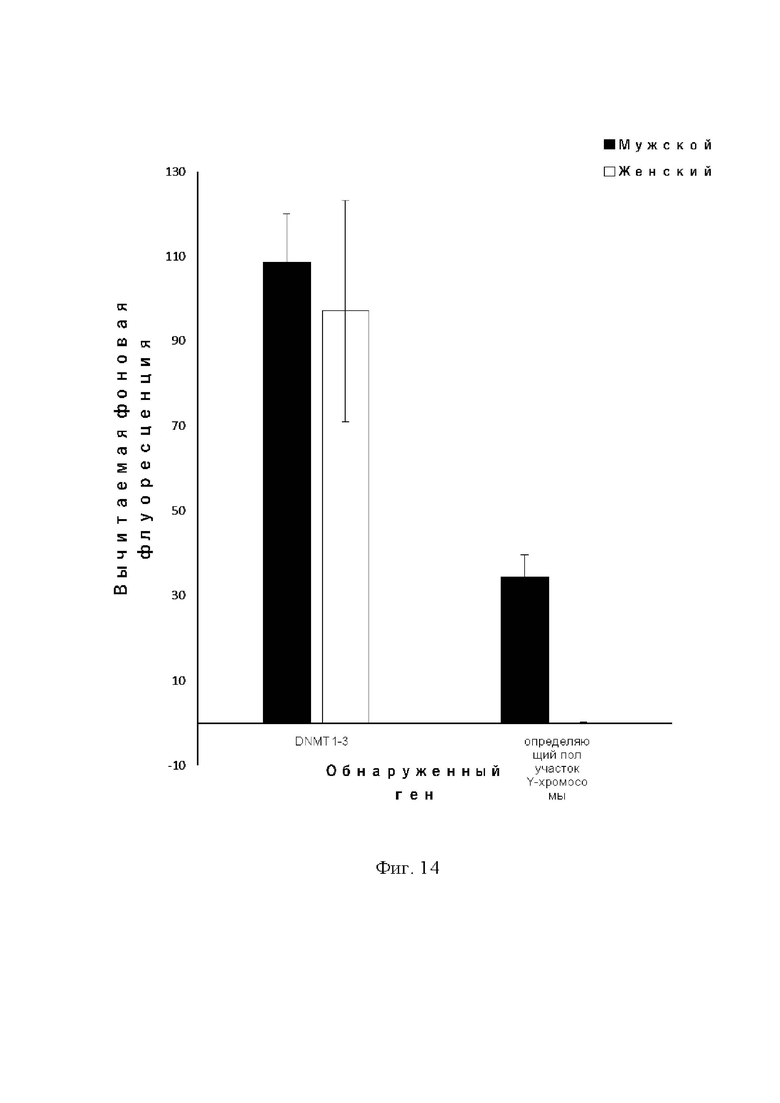
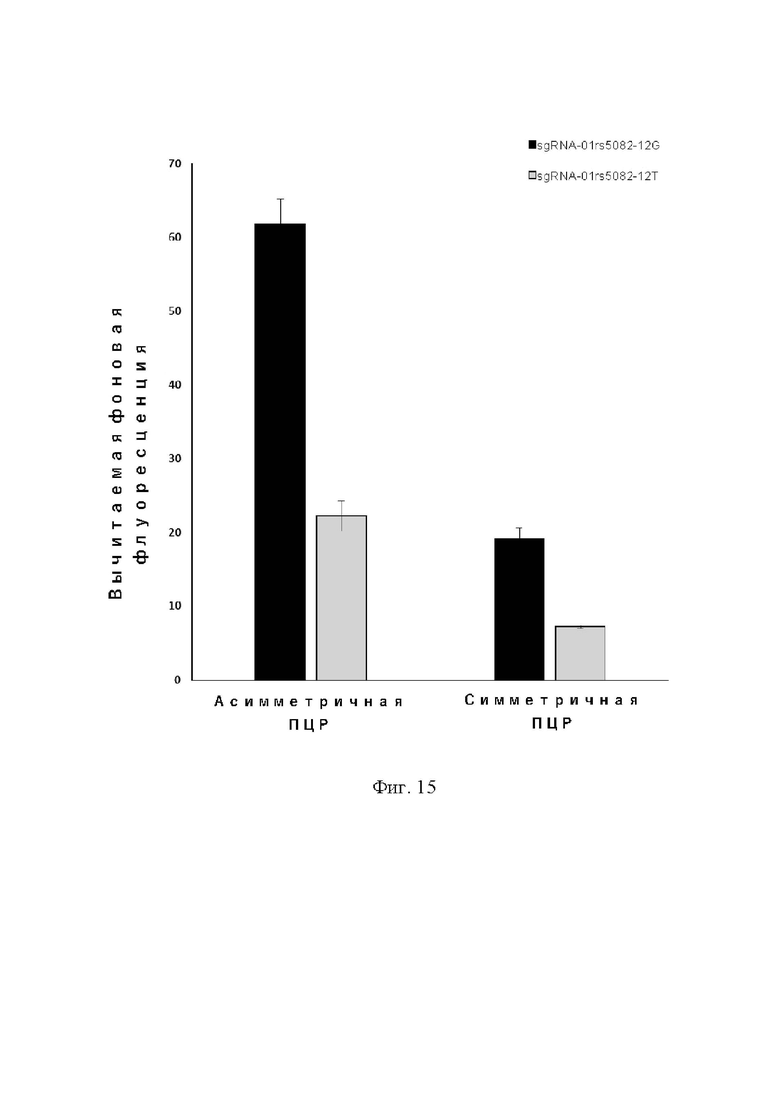
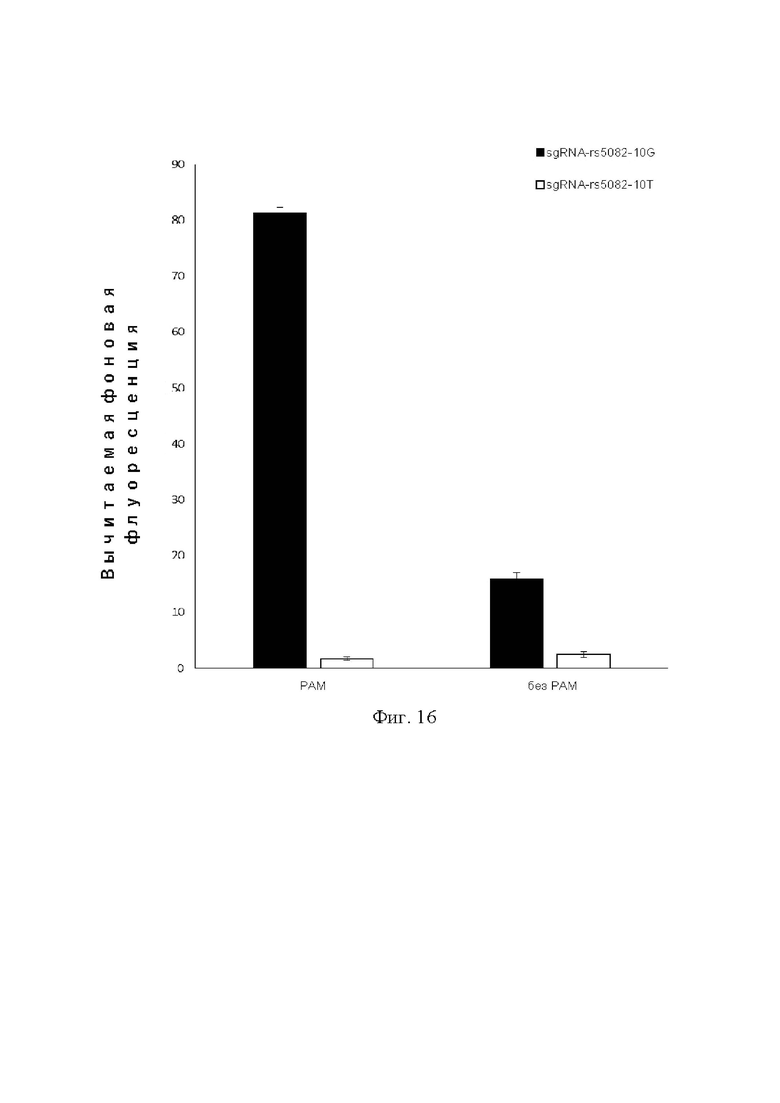
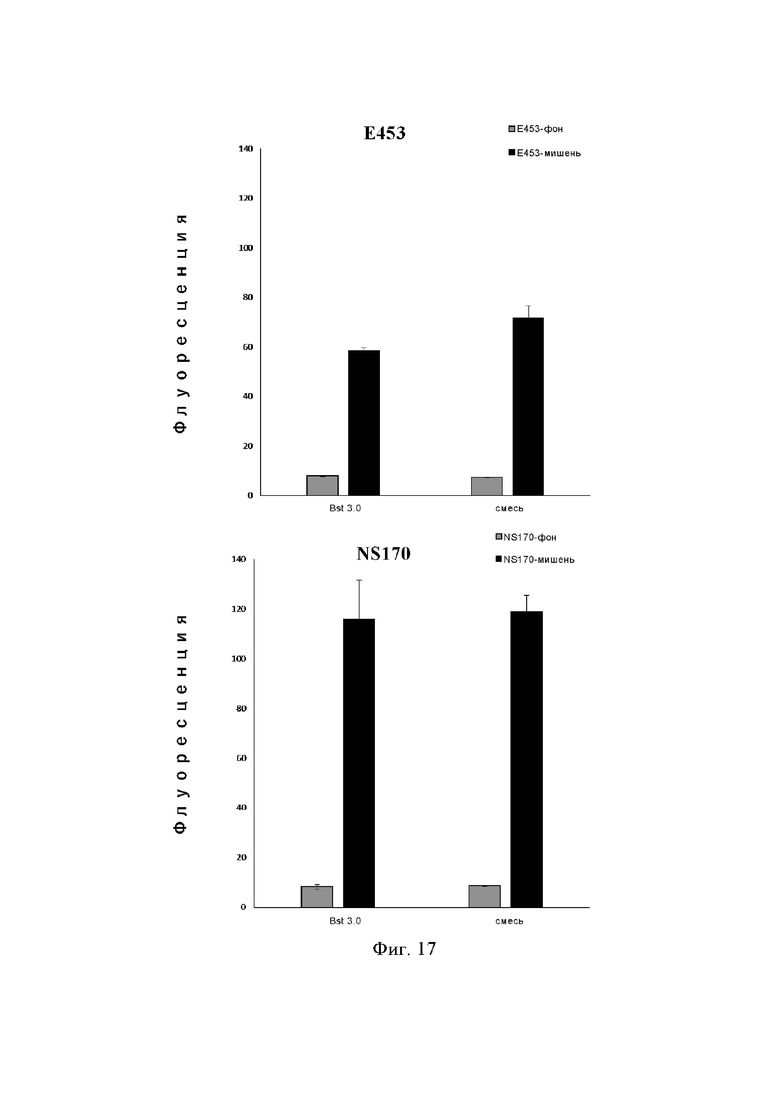
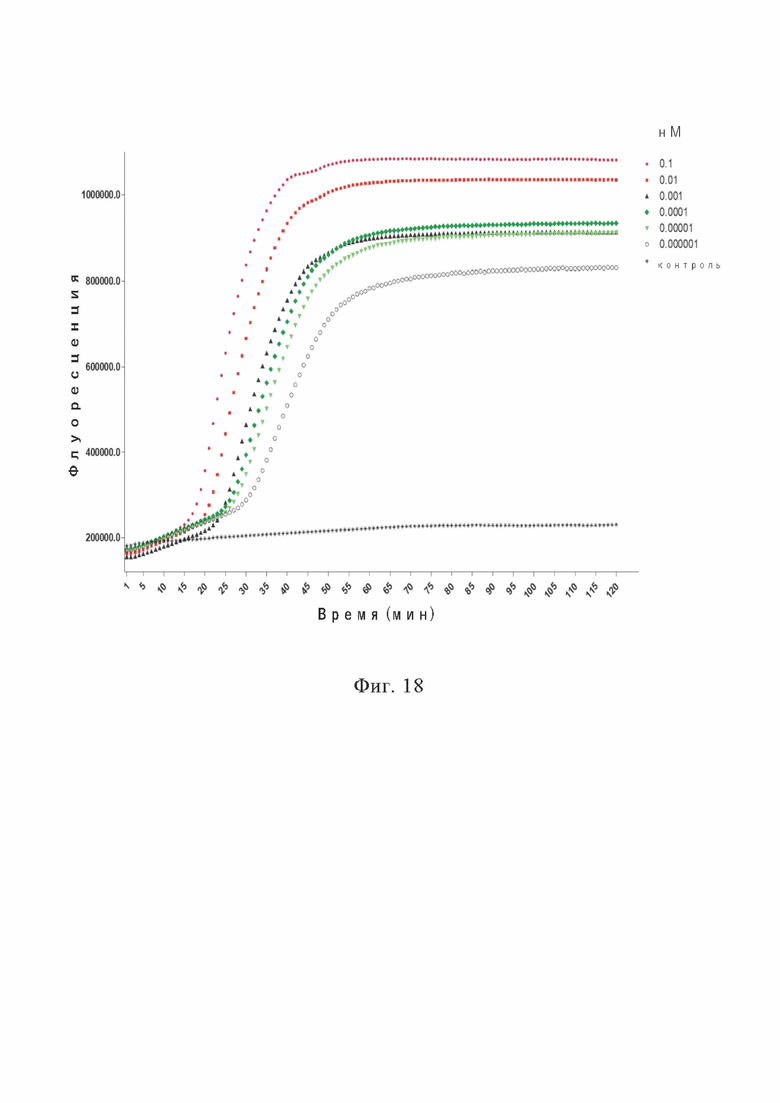
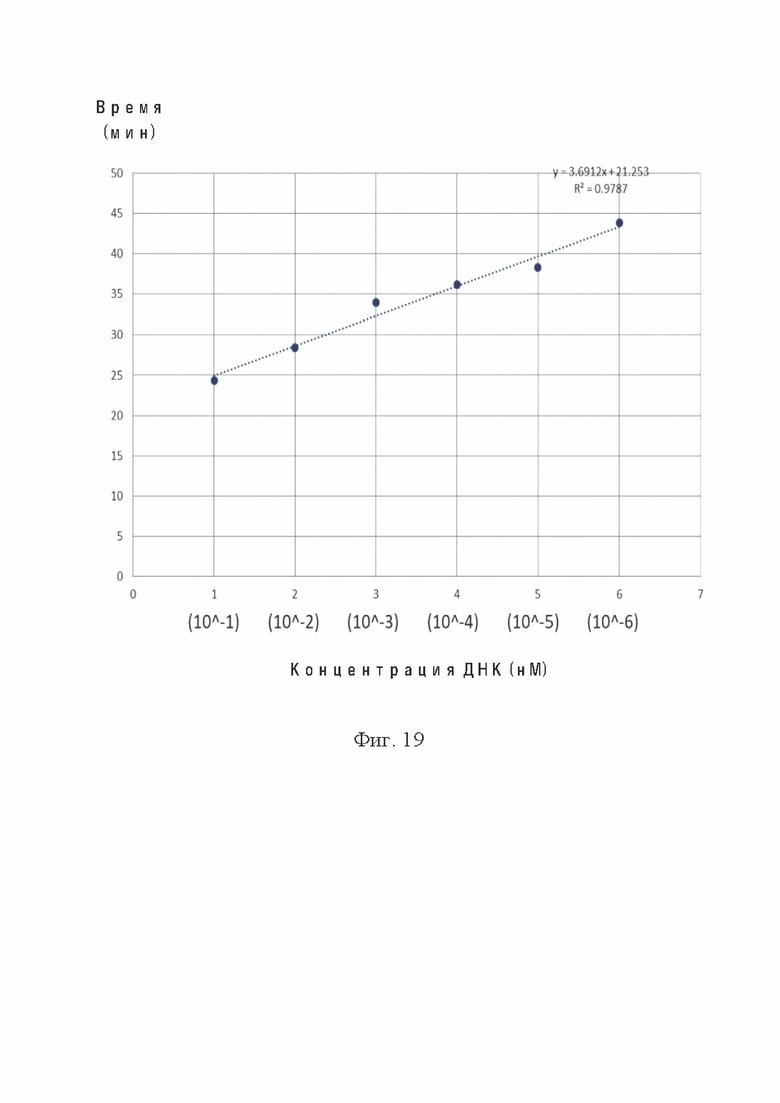
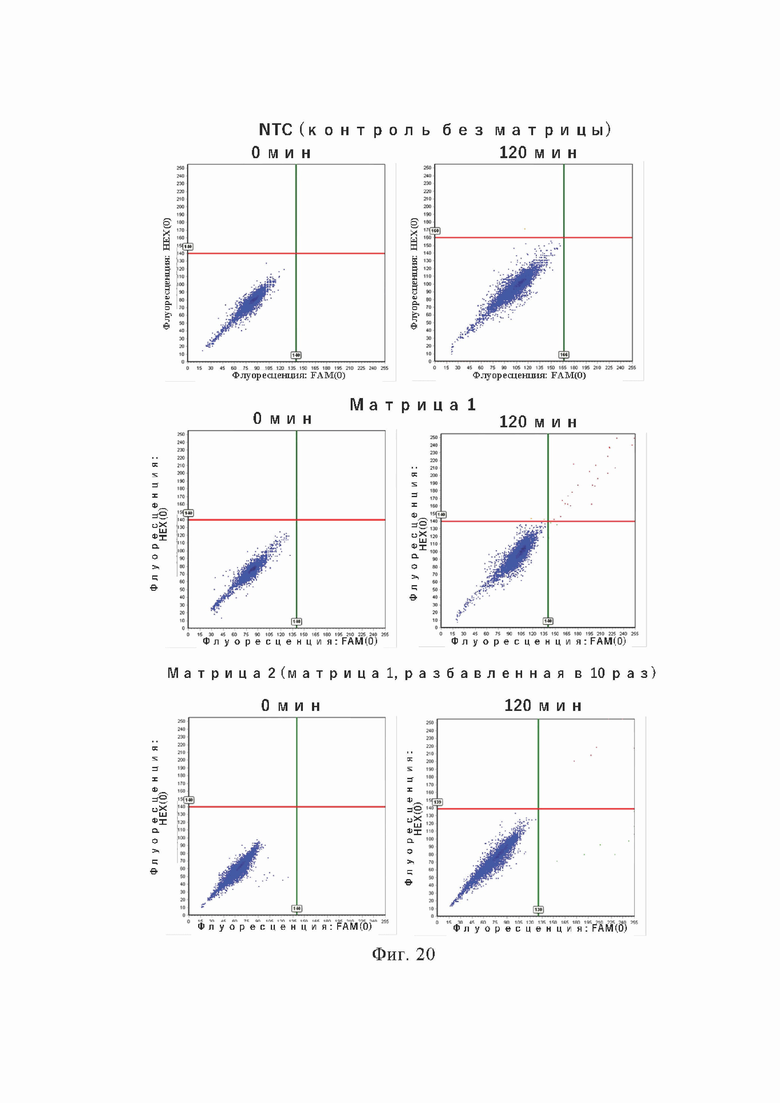
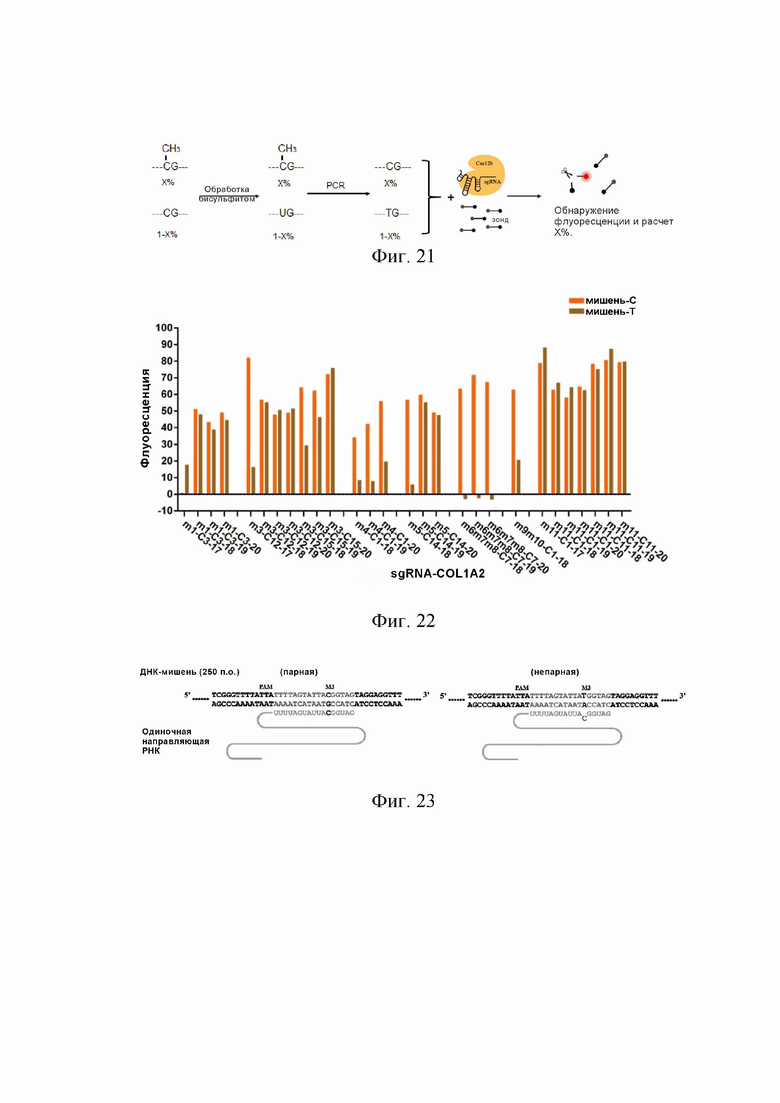
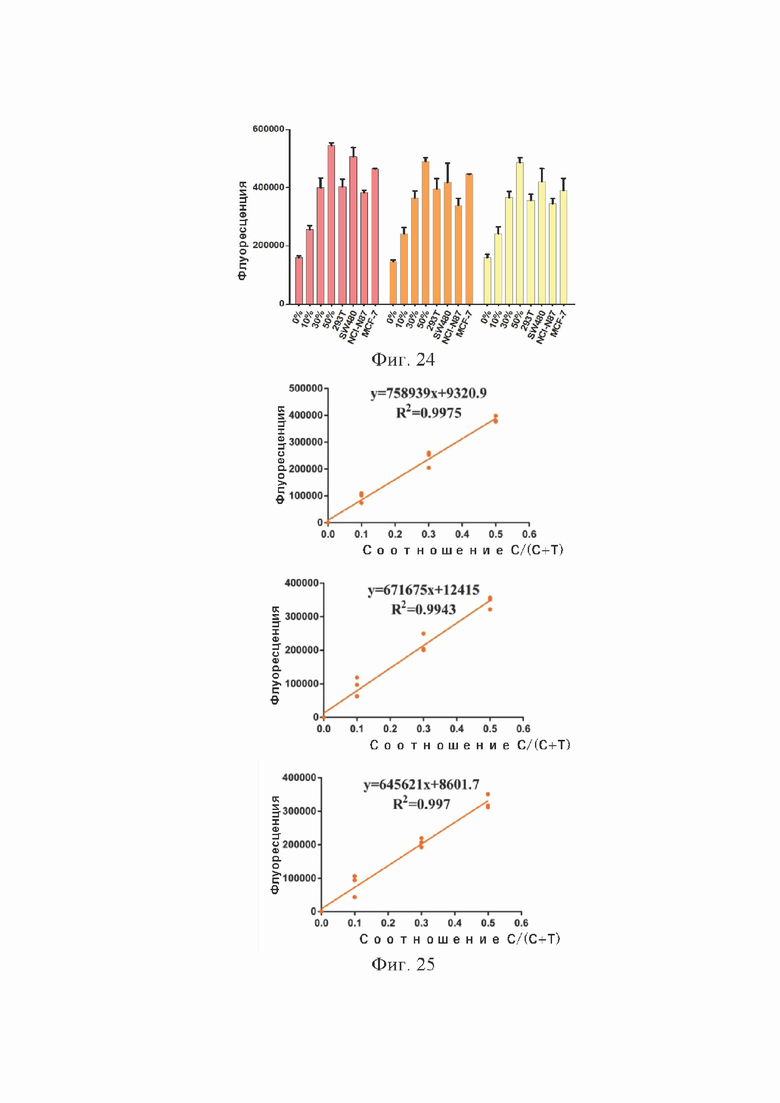
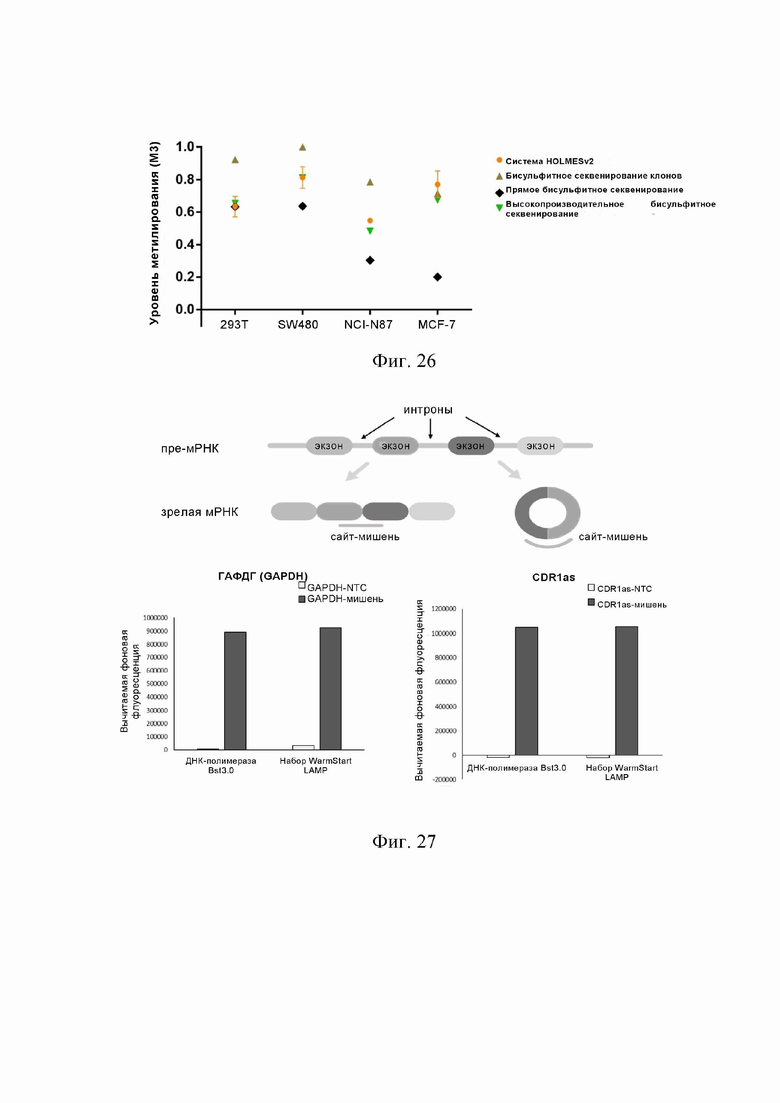
Изобретение относится к области биотехнологии, в частности к системе, включающей: (a) белок Cas12b; (b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и (c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК. Также раскрыто устройство, которое включает: (1) модуль реакции амплификации — реакции обходного расщепления, и (2) модуль обнаружения сигнала. Изобретение эффективно для обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, а также для обнаружения SNP (одиночных нуклеотидных полиморфизмов). 9 н. и 15 з.п. ф-лы, 27 ил., 3 табл., 11 пр.
1. Система обнаружения для обнаружения целевых молекул нуклеиновой кислоты, при этом система включает:
(a) белок Cas12b;
(b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и
(c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень.
2. Система обнаружения по п. 1, в которой система обнаружения включает реакцию амплификации нуклеиновой кислоты и реакцию обходного расщепления, при этом реакция амплификации нуклеиновой кислоты и реакция обходного расщепления выполняются одновременно, и система обнаружения представляет собой одноступенчатую реакционную систему.
3. Система обнаружения по п. 1, в которой белок Cas12b выбирается из группы, состоящей из: AacCas12b (Alicyclobacillus acidoterrestris), Aac2Cas12b (Alicyclobacillus acidiphilus), AkaCas12b (Alicyclobacillus kakegawensis), AmaCas12b (Alicyclobacillus macrosporangiidus), AheCas12b (Alicyclobacillus herbarius) и AcoCas12b (Alicyclobacillus contaminans).
4. Система обнаружения по п. 1, в которой, когда целевой молекулой нуклеиновой кислоты является одноцепочечная ДНК, сайт обнаружения целевой молекулы нуклеиновой кислоты находится в положении 9 или в положениях 10–16, предпочтительно в положениях 10–14 и более предпочтительно в положениях 10–12 за последовательностью PAM направляющей РНК.
5. Система обнаружения по п. 4, в которой, когда сайт обнаружения находится в положении 9 за последовательностью PAM направляющей РНК, сайтом обнаружения является G.
6. Система обнаружения по п. 4, в которой сайт обнаружения целевой молекулы нуклеиновой кислоты находится в положениях 1–12, предпочтительно в положении 1, 3 или 10 за последовательностью PAM направляющей РНК.
7. Система обнаружения SNP (одиночных нуклеотидных полиморфизмов), при этом данная система содержит первую систему обнаружения и вторую систему обнаружения;
в которой первая система обнаружения включает:
(a1) белок Cas12b;
(b1) первую направляющую РНК, причем первая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и
(c1) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
а вторая система обнаружения включает:
(a2) белок Cas12b;
(b2) вторую направляющую РНК, причем вторая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты; и
(c2) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень;
кроме того, первая направляющая РНК и вторая направляющая РНК нацелены на одну и ту же область последовательности нуклеиновой кислоты, содержащую SNP-сайт, а первая направляющая РНК нацелена на последовательность нуклеиновой кислоты дикого типа или немутированной SNP-сайта, а вторая направляющая РНК нацелена на последовательность мутированной нуклеиновой кислоты SNP-сайта.
8. Система обнаружения по п. 7, в которой система обнаружения включает реакцию амплификации нуклеиновой кислоты и реакцию обходного расщепления, при этом реакция амплификации нуклеиновой кислоты и реакция обходного расщепления выполняются одновременно, и система обнаружения представляет собой одноступенчатую реакционную систему.
9. Система обнаружения по п. 7, которая дополнительно содержит систему контроля холостой пробы, и система контроля холостой пробы включает:
(a3) белок Cas12b;
(c3) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК.
10. Набор для обнаружения целевых молекул нуклеиновой кислоты, который включает:
i) первый контейнер и белок Cas12b, находящийся в первом контейнере;
ii) второй контейнер и направляющую РНК, находящуюся во втором контейнере, причем вторая направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты;
iii) третий контейнер и зонд на основе нуклеиновой кислоты, находящийся в третьем контейнере;
iv) необязательно четвертый контейнер и буфер, находящийся в четвертом контейнере;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень.
11. Набор по п. 10, который дополнительно содержит:
v) пятый контейнер и полимеразу для амплификации ДНК-мишени, находящейся в пятом контейнере;
vi) необязательный шестой контейнер и обратную транскриптазу для обратной транскрипции, находящуюся в шестом контейнере;
vii) седьмой контейнер и dNTP для реакции амплификации и/или реакции обратной транскрипции, находящийся в седьмом контейнере.
12. Набор по п. 11, в котором полимераза представляет собой ДНК-полимеразу Bst 3.0.
13. Способ обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, который включает следующие этапы:
(i) предоставление системы обнаружения целевых молекул нуклеиновой кислоты по п. 1, и образец, подлежащий обнаружению; и добавление образца в систему обнаружения; и
(ii) выяснение, расщеплен ли зонд на основе нуклеиновой кислоты в системе обнаружения белком Cas12b, причем расщепление представляет собой транс-расщепление обходной одноцепочечной ДНК;
при этом, если зонд на основе нуклеиновой кислоты расщепляется белком Cas12b, это указывает на то, что целевая молекула нуклеиновой кислоты присутствует в образце; и если зонд на основе нуклеиновой кислоты не расщепляется белком Cas12b, это указывает на отсутствие целевой молекулы нуклеиновой кислоты в образце.
14. Способ обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, который включает следующие этапы:
(i) предоставление системы обнаружения и образца для тестирования, и добавление образца в систему обнаружения,
при этом система обнаружения включает:
(a) белок Cas12b;
(b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты;
(c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
причем целевой молекулой нуклеиновой кислоты является ДНК-мишень;
(d) буфер;
(e1) полимеразу, которая используется для амплификации ДНК-мишени;
(e2) необязательную обратную транскриптазу, которая используется для обратной транскрипции; и
(e3) dNTP, который используется для реакции амплификации и/или реакции обратной транскрипции;
(ii) выполнение реакции обратной транскрипции и/или амплификации в системе обнаружения, получая таким образом обратно транскрибированную и/или амплифицированную систему обнаружения;
(iii) выяснение, расщеплен ли зонд на основе нуклеиновой кислоты в системе обнаружения, полученной на предыдущем этапе, белком Cas12b, причем расщепление представляет собой транс-расщепление обходной одноцепочечной ДНК;
при этом, если зонд на основе нуклеиновой кислоты расщепляется белком Cas12b, это указывает на то, что целевая молекула нуклеиновой кислоты присутствует в образце; а если зонд на основе нуклеиновой кислоты не расщепляется белком Cas12b, это указывает на отсутствие целевой молекулы нуклеиновой кислоты в образце.
15. Способ по п.14, в которой система обнаружения включает реакцию амплификации нуклеиновой кислоты и реакцию обходного расщепления, при этом реакция амплификации нуклеиновой кислоты и реакция обходного расщепления выполняются одновременно, и система обнаружения представляет собой одноступенчатую реакционную систему.
16. Способ по п. 14, в котором обнаружение содержит: качественное выявление или количественное выявление;
в предпочтительном варианте количественное выявление представляет собой абсолютное количественное выявление; в более предпочтительном варианте количественное выявление представляет собой количественное выявление в сочетании с технологией цифровой ПЦР.
17. Способ по п. 14, в котором способ амплификации нуклеиновой кислоты выбирается из группы, состоящей из: ПЦР-амплификация, петлевая изотермическая амплификация (LAMP), рекомбиназная полимеразная амплификация (RPA), лигазная цепная реакция, амплификация методом разветвленной ДНК, амплификация, основанная на последовательности нуклеиновых кислот (NASBA), амплификация с замещением цепей (SDA), транскрипционно-опосредованная амплификация, амплификация по типу катящегося кольца, геликаза-зависимая амплификация (HDA), изотермическая амплификация с одним праймером (SPIA), амплификация с использованием никирующих ферментов (NEAR), транскрипционно-опосредованная амплификация (TMA) и амплификация SMAP2.
18. Способ по п. 14, в котором целевая молекула нуклеиновой кислоты представляет собой последовательность метилированной нуклеиновой кислоты, или последовательность метилированной нуклеиновой кислоты представляет собой последовательность нуклеиновой кислоты, полученной после преобразования неметилированного С в урацил.
19. Применение белка Cas12b для приготовления набора для обнаружения целевых молекул нуклеиновой кислоты на основании обходной расщепляющей активности одноцепочечной ДНК.
20. Применение белка Cas12b для приготовления реагента для обнаружения целевых молекул нуклеиновых кислот на основании обходной расщепляющей активности одноцепочечной ДНК.
21. Применение белка Cas12b в одноступенчатой реакционной системе для обнаружения целевых молекул нуклеиновых кислот на основании обходной расщепляющей активности одноцепочечной ДНК, при этом одноступенчатая реакционная система включает реакцию амплификации нуклеиновой кислоты и реакцию обходного расщепления, при этом реакция амплификации нуклеиновой кислоты и реакция обходного расщепления выполняются одновременно.
22. Устройство обнаружения присутствия в образце целевых молекул нуклеиновой кислоты, которое включает:
(1) модуль реакции амплификации — реакции обходного расщепления, который используется для выполнения реакций амплификации нуклеиновой кислоты и реакций обходного расщепления в цифровой реакционной системе, в которой реакция обходного расщепления опосредована белком Cas12b; и
(2) модуль обнаружения сигнала, который используется для выявления того, происходит ли реакция обходного расщепления, опосредованная белком Cas12b, в каждой цифровой реакционной системе, и модуль обнаружения сигнала включает модуль визуализации;
при этом реакция амплификации нуклеиновой кислоты и реакция обходного расщепления выполняются одновременно;
модуль реакции амплификации — реакции обходного расщепления содержит блок контроля температуры, для установки температуры цифровой реакционной системы в устройстве на заданную температуру; и заданная температура составляет 50-70°С, предпочтительно 50-65°С, более предпочтительно 55-65°С;
цифровая реакционная система содержит реакционную систему цифровой капельной ПЦР (ddPCR) или реакционную систему цифровой ПЦР на чипах (cdPCR);
при этом цифровая реакционная система содержит множество независимых устройств реакционной системы, и каждое устройство реакционной системы содержит:
(a) белок Cas12b;
(b) направляющую РНК, причем направляющая РНК направляет белок Cas12b для специфического связывания с целевыми молекулами нуклеиновой кислоты, причем целевой молекулой нуклеиновой кислоты является ДНК-мишень;
(c) зонд на основе нуклеиновой кислоты, который представляет собой одноцепочечную ДНК;
(d) буфер;
(e1) полимеразу, которая используется для амплификации ДНК-мишени;
(e2) необязательную обратную транскриптазу, которая используется для обратной транскрипции;
(e3) dNTP, который используется для реакции амплификации и/или реакции обратной транскрипции; и
(f) образец для тестирования;
при этом в связи с обработкой при разведении каждая независимая реакционная система содержит либо 1 копию нуклеиновой кислоты из образца, подлежащего тестированию, либо 0 копий нуклеиновой кислоты из образца, подлежащего тестированию; и
(3) модуль загрузки образца; и
(4) модуль управления.
23. Устройство по п. 22, в котором модуль визуализации содержит устройство флуоресцентного детектирования.
24. Устройство по п. 22, в котором модуль управления выполняет арифметический анализ на основе количества флуоресцентных сигналов и общего количества устройств реакционной системы, получая, таким образом, результат количественного выявления целевой нуклеиновой кислоты.
| SHI-YUAN LI et al., CRISPR-Cas12a-assisted nucleic acid detection, Cell Discovery, 2018 Apr 24;4:20 | |||
| CN107488710 A, 19.12.2017 | |||
| WO2016186946 A1, 24.11.2016 | |||
| JONATHAN S | |||
| GOOTENBERG et al., Nucleic acid detection with CRISPR-Cas13a/C2c2, Science, 2017, 356(6336), pp | |||
| Способ обогащения кислородных руд путем взбалтывания пены | 1911 |
|
SU438A1 |
| СМИРНОВ А.В | |||
| и др., Система CRISPR/Cas9 - универсальный инструмент | |||
Авторы
Даты
2023-07-28—Публикация
2019-06-03—Подача