Изобретение относится к разработке набора реагентов для выявления генетического материала (РНК) у обнаруженных относительно недавно представителей группы Jingmenvirus (JMV), таких как вирус Jingmen tick virus и Alongshan virus, в клинических образцах человека, культуральных вируссодержащих жидкостях и в членистоногих, с целью постановки диагноза, эпидемиологического расследования, а также для решения научно-исследовательских задач по мониторингу и изучению свойств новых для России вирусов, и может быть использовано в медицине, биотехнологии и эпидемиологии.
Род Flavivirus включает родственные, неклассифицированные сегментированные флавиподобные вирусы. Вирусы были отнесены к роду Flavivirus на основании того, что два сегмента у них имеют гомологию с флавивирусной РНК-зависимой РНК-полимеразой NS5 и геликазой-протеазой NS3. В группу JMV входят такие вирусы, как Jingmen tick virus (JMTV), Alongshan virus, Yanggou tick virus и другие. Представители группы JMV встречаются практически на всех континентах: Евразия [2,3,5,7,10-13], Африка [11,14], Южная Америка [11,14-17] и Северная Америка [18]. РНК этой группы вирусов обнаружены у разных видов насекомых (дрозофилы [19] и комаров [20]), клещей [2,3,5,7,10,11,13,17,20] и млекопитающих [3,11,14,15,21], в том числе у человека [10,12,20]. В первые ALSV был выявлен в Китае у пациента с лихорадкой неизвестного происхождения. Позже этот вирус был обнаружен у комаров [20], клещей [7,11,13,20] и млекопитающих [20,22]. Вирусы JMV были найдены в России. В 2020 г. Alongshan virus (ALSV) был найден в десяти пулах взрослых клещей Ixodes persulcatus, собранных в двух географически разнесенных регионах России, в Карелии и Челябинской области [la] Yanggou tick virus (YGTV) был впервые обнаружен у клещей Dermacentor nuttalli в Китае и оказался близок к JMTV [13], а в 2021 г. YGTV был найден у различных видов клещей в восьми регионах России [7]. Распространенность вируса колебалась от 0,5% до 8,0%.
Род Flavivirus относится к семейству Flaviviridae, представители которого небольшие оболочечные вирусы с одноцепочечной РНК (+), длиной около 10 т.п.н., которые кодируют одну открытую рамку считывания [1]. Флавиподобные вирусы с длиной генома размером более 16 т.п.н. [2] и сегментированным геномом [3] были включены в группу неклассифицированных вирусов, относящихся к роду Flavivirus [4]. Представители группы JMV, такие как клещевой вирус JMTV, ALSV, YGTV и другие имеют сегментированные ssRNA(+) геномы, два сегмента которых имеют гомологию с известной флавивирусной РНК-зависимой РНК- полимеразой NS5 (сегмент 1) и РНК-хеликазой-протеазой NS3 (сегмент 3) [3,5]. Остальные два сегмента характерны только для JMV и кодируют гликопротеины, белки VPla и VPlb (сегмент 2) [5-7] и другие вирусные белки - VP2 и VP3 (сегмент 4) [2,3,5].
Группе JMV в настоящее время уделяется повышенное внимание, поскольку, с одной стороны, изучение этой группы дает новую информацию о многообразии возможностей по организации генома РНК(+) вирусов и их эволюции, с другой стороны, существует неопределенность в отношении круга хозяев и вирулентности для людей и животных..
С целью контроля их распространения, определения круга хозяев и оценки эпидемического потенциала, чрезвычайно важным является разработка быстрых и эффективных средств диагностики этого опасного заболевания.
Данная заявка на изобретение посвящена разработке набора реагентов для выявления РНК вируса JMTV группы Jingmenvirus, в клинических образцах человека, культуральных вируссодержащих жидкостях и у членистоногих, методом ПЦР с гибридизационно-флуоресцентной детекцией.
Метод основан на амплификации кДНК в полимеразной цепной реакции (ПЦР) и гибридизационно-флуорецентной детекции продуктов амплификации в режиме реального времени в образцах биоматериала. Амплифицируемый участок кДНК, являясь маркерным, позволяет выявить вирусный агент в исследуемом образце. Для эффективного проведения ПЦР в режиме реального времени необходимы флуоресцентно-меченый ДНК-зонд и ДНК-затравки - праймеры (синтетические олигонуклеотиды) - строго специфичные к кДНК вирусного генома. Сложность выбора праймеров и зонда обусловлена требованием их строгой видоспецифичности. Праймеры должны быть комплементарны нуклеотидным последовательностям кДНК, ограничивая амлифицируемый участок справа и слева таким образом, чтобы синтез ДНК проходил строго в выбранном регионе. Флуоресцентно-меченый ДНК-зонд, в свою очередь, должен лежать внутри участка кДНК ограниченного праймерами. Правильный выбор праймеров позволяет осуществить в ПЦР экспоненциальное увеличение количества копий целевого участка кДНК. Правильный выбор сочетания пары праймеров и ДНК-зонда позволяет осуществлять детекцию накопления продуктов амплификации в режиме реального времени. В целом от правильности выбора олигодезоксирибонуклеотидных праймеров и ДНК-зонда зависит специфичность проводимой ПЦР, а значит и точность диагностики вирусного заболевания.
В литературе есть публикации, в которых предложены диагностические праймеры, обеспечивающие детекцию кДНК JMTV и ALSV.
Известен петлево-опосредованный набор (патент Китая CN 113604612, опубл. 05.11.2021 г., дата регистрации патента 01.08.2023 г.) [25] изотермических амплификационных праймеров для обнаружения вируса Алонгшан (ALSV), который характеризуется тем, что: набор праймеров состоит из 1 пары внешних праймера F3/B3 и 1 пары внутренних праймеров FIP/BIP. Последовательность праймеров следующая:
F3: TGATCTACATCCTCGCCGT
В3: CTGCTCCTGCTGTGAGTG
FIP: TGATTGCAGCTGCTGTCCCAGACCATGGGCAAGCCAAAC
BIP: TCATCAAGGAATTGCCCACGCATCTCCTCACCGGTTCTCAA.
Молярное отношение внешних праймеров к внутренним праймерам составляет 1:8.
Известен набор праймеров для обнаружения вируса Алонгшан для ПЦР (заявка на патент Китая №110273025, опубл. 05.11.2021 г.) [26], состоящий из праймера ALSV-F1, праймера ALSV-R1 и праймера ALSV- R2, нуклеотидная последовательность которых следующая:
Primer ALSV-F1: 5'-GTVTTYGTGCCRGGHCTGAC-3';
Primer ALSV-R1: 5'-GTGGABGGRGTTATBAYYCCYTTNG-3';
Primer ALSV-R2: 5'-TGGCABGTRTCRAAMACWGCRTC-3';
Wherein, V, Y, R, H, B, N, M, and W represent degenerate base codes, V=A/G/C; Y=C/T; R=A/G; Н=А/С/ T; В=G / С / T; N=A / T / C / G; M=A / C; W=A/T.
Описана система qPCR для обнаружения ALSV [23] на VP1a ген. Показано, что эта система может обнаруживать менее чем 104 копии ALSV в образце (инфицированная культура клеток РЕК). Система не выявляла другие распространенные клещевые инфекции родственных вирусов, например Yanggou tick virus (группа вирусов Jingmen), или других известных представителей рода Orthoflavivirus. Предложенная система qPCR не имела неспецифического сигнала для Ixodes ricinus, I. persulcatus, Dermacentor reticulatus, D. Marginatus, Haemaphysalis concinna и клещей H. japonica. Система qPCR не имела неспецифического сигнала на ДНК человека, а также сыворотки овец. В целом, описанная система qPCR может использоваться для обнаружения ALSV в образце, но не для обнаружения JMTV.
JMTV - это недавно обнаруженный в Китае клещевой вирус, который вызывает заболевание у людей. Позже оказалось, что вирус чрезвычайно широко распространен во всем мире и представляет значительную угрозу для населения. В WeiFang Medical University, в Китае, с помощью qRT-PCR были амплифицированы JMTV-специфичные гены и присутствие этого патогена в образцах было подтверждено секвенированием [24]. Из 35 собранных природных образцов клещей положительный результат qRT-PCR по JMTV был получен для А. Testudinarium и для Н. Longicornis. JMTV РНК была обнаружена у 11 клещей, в том числе у девяти клещей A. testudinarium [34,6%] и у двух клещей Н. Longicornis [22,2%]. Однако вирус не был выделен. Однако, дизайн праймеров (HaNS5 ген), используемых в вышеприведенным аналоге, не учитывал последовательности JMTV найденных в России в последнее время. Кроме того, не была проведена оценка чувствительности разработанной qPCR.
Вышеперечисленные диагностические наборы-аналоги либо не позволяют идентифицировать JMTV, либо не обладают достаточной чувствительностью.
Техническим результатом заявленного изобретения является создание такого набора олигонуклеотидных праймеров и флуоресцентно-меченого зонда, который обеспечивал бы высокую чувствительность детекции исключительно JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией.
Указанный технический результат достигается разработкой набора олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого ДНК-зонда для идентификации JMTV методом ПЦР с гибридизационнофлуоресцентной детекцией, содержащий одну пару олигонуклеотидов, обладающих активностью прямого и обратного праймеров в полимеразной цепной реакции, а также флуоресцентно-меченый ДНК-зонд, имеющие следующую структуру олигонуклеотидных последовательностей:
SEQ ID NO:1 - прямой праймер (F): 5’-GCTACGTCGGGCTCATGC-3’;
SEQ ID NO:2 - обратный праймер (R): 5’-CGTACGGTCCGAGTAGTCAG-3’
SEQ ID NO:3 - флуоресцентный зонд (Z): HEX-CGAGTGGAGGTTCTGGGTCGTC-BHQ2.
Таким образом, для qPCR на JMTV нами были подобраны специфические праймеры и рассчитан флуоресцентный зонд для области гена белка гликопротеина (glycoprotein) JMTV.
Учет и интерпретация результатов
Результат считать положительным, если по каналу HEX, кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. При этом значение Ct для данного образца должно быть менее 35. Амплитуда сигнала при этом не имеет значения.
Результат считать отрицательным, если значение Ct на канале HEX отсутствует. Если значение Ct больше 35, то необходимо повторить и считать положительным в случае повторения результата или при значении Ct меньше 35.
Задача по получению новых праймеров на известную последовательность геномной РНК JMTV является не очевидной. Результаты, полученные с использованием различных программ, отличаются друг от друга, и выбор оптимальной структуры праймеров становится нетривиальной задачей.
Следует отметить, что большинство пар праймеров, рассчитанных с использованием программ PeriPrimer и VectorNTI предназначены для наработки длинных ампликонов, что не совсем удачно для их применения в ПЦР с диагностической целью.
При разработке представленных в заявляемом наборе диагностических праймеров было синтезировано 4 пары праймеров с различной первичной структурой, с каждой из которых были проведены соответствующие исследования. Эмпирическим путем была определена наиболее подходящая пара праймеров, обладающая наибольшей аналитической чувствительностью и 100%-ной специфичностью к JMTV на панели заявленных образцов. Структура данных праймеров была рассчитанной с использованием приложения программного пакета Vector NTI 10 (Informax, США) и консенсусной последовательности множественного выравнивания по гену белка гликопротеина JMTV.
В работе, помимо дизайна структуры праймеров и олигонуклеотидного зонда, был получен положительный контроль, представляющий из себя рекомбинантную плазмиду, несущую синтетическую вставку кДНК, комплементарную участкам гена белка гликопротеина JMTV. С использованием положительного контроля эмпирическим путем была подобрана оптимальная температура отжига диагностических праймеров, которая отличается от теоретической, рассчитанной с использованием компьютерных программ. Также эмпирическим путем была установлена оптимальная концентрация пары праймеров/зонд для идентификации JMTV. Заявленные диагностические праймеры обладают 100%-ной специфичностью на панели положительных и отрицательных образцов. Поиск по международным базам данных нуклеиновых кислот показал, что выбранные праймеры и ДНК-зонд не имеют гомологии с человеческой ДНК и не взаимодействуют с геномной ДНК человека и мышей и других животных. Экспериментальным путем было показано, что выбранные праймеры и ДНК-зонд не взаимодействуют с человеческой ДНК и мышей.
На панели положительных образцов, содержащих генетический материал JMTV, произведена оценка чувствительности выбранной пары диагностических праймеров. С использованием положительного контроля определена аналитическая чувствительность заявляемой пары диагностических праймеров, которая составила не менее 50 геном- эквивалентов на реакцию (5×102 мл).
Таким образом, из приведенных выше доводов заявителя следует, что заявленный набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации генетического материала JMTV методом ПЦР в реальном времени соответствует критерию «изобретательский уровень».
Материалы и реактивы
В работе были использованы следующие материалы и реактивы: трис- (оксиметил)-аминометан, диметилсульфоксид (ДМСО), маркеры молекулярных масс белков, 3,3-диаминобензидин тетрагидрохлорид, натриевая соль MES (М3058), (“Sigma”, США); ПЭГ-20000 (“Merck”, США); твин-20, натрия азид, (“Serva”, США); набор для определения концентрации нуклеиновых кислот "Bio-Rad HS-dsDNA Assay Kit" (“BioRad”, США); акриламид, бисакриламид, натрия додецилсульфат, тетраметилэтилендиамин (ТЕМЭД), 2-меркаптоэтанол, бычий сывороточный альбумин (БСА) и др.
Вирусные штаммы
В работе были использованы вирусы и вируссодержащий материал из коллекции микроорганизмов ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора (Таблица 3).
Культуры клеток и среды
Клетки Vero получены из коллекции культур клеток ФБУН ГНЦ ВБ «Вектор». В работе использовали питательные среды: среда Игла МЕМ с двойным набором аминокислот и витаминов производства ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора.
Выделение вирусной РНК
Для экстракции нуклеиновых кислот были использованы тест-системы различных производителей:
С-004-(100)Набор «АмплиТест РИБО-преп» (ЦНИИ Эпидемиологии, Москва);
DU-10 Набор для выделения геномной ДНК из клеток, тканей и крови (БиоЛабМикс, Новосибирск);
Р-002/1 ПРОБА-НК, набор реагентов для выделения нуклеиновых кислот (ДНК-технология, Москва);
ВС032 Реагент ExtractRNA (Евроген, Москва), согласно инструкции по применению.
Оптимизацию ПЦР проводили с использованием амплификаторов Rotor Gene 6000 («Corbett Research», Австралия) и CFX96 Opus («BioRad», США). Электрофоретическую детекцию продуктов амплификации в агарозном геле проводили с использованием электрофоретических камер SE-20 (Хеликон, Россия), источников питания Эльф-8 (ЗАО НПФ ДНК- технология, Россия), системы видеодокументирования Molecular Imager Gel Doc XR System («BioRad», США) с прилагаемым ПО.
После проведения ПЦР полученные фрагменты ДНК разделяли электрофорезом в агарозном геле и очищали с использованием набора diaGene («Диа-М», Россия), клонировали в плазмиду pCR2.1. (“Invitrogen”, США). Определение нуклеотидных последовательностей проводилось на автоматическом секвенаторе ABI Genetic Analyzer 3500хl (ABI, США). Нуклеотидные последовательности анализировались с использованием базы данных NCBI Mega BLAST.
Нуклеотидные последовательности
Нуклеотидные последовательности были взяты из базы данных GenBank [http://www.ncbi.nlm.nih.gov/genbank/].
Статистическая обработка полученного материала проводилась с использованием пакетов программ Microsoft Excel 2010 for Windows и STATISTICA 64 for Windows (Stat Soft, USA). При статистической обработке данных для ненормально распределенных признаков использовали непараметрический критерий Манна-Уитни и коэффициент корреляции Спирмена. Сравнение частот встречаемости проводили по критерию χ2. Для показателей, чьи вариационные ряды приближались к нормальному, применяли дисперсионный анализ. При множественном сравнении средних использовали LSD-тест, а при сравнении 2-х средних - t-критерий Стьюдента.
Множественное выравнивание нуклеотидных последовательностей проводили при помощи приложения AlignX программного пакета Vector NTI Advance (Informax, США). Филогенетический анализ штаммов и изолятов вируса гриппа проводили при помощи программы Mega 7 [17]. Филогенетические деревья строили по методу объединения ближайших соседей на основе матриц генетических расстояний, рассчитанных по модели Юкса-Кантора. Индексы поддержки ветвей определяли перестановочным тестом (bootstrap) для 1 000 повторов.
В работе было использовано лицензионное программное обеспечение Microsoft Office 2010, Vector NTI Advance, Lasergene 7.
Изобретение иллюстрируется следующими графическими материалами.
На Фиг. 1. представлено множественное выравнивание нуклеотидных последовательностей JMTV и локализация на последовательности подобранной пары праймеров и флуоресцентно-меченого зонда.
На фиг. 2 представлена флюоресценция продуктов ПЦР в режиме реального времени для оценки чувствительности с 10-кратными разведениями плазмидной ДНК.
На фиг. 3 приведена флюоресценция продуктов ПЦР в режиме реального времени - анализ клинических образцов (мазки из зева и носа) инфицированных JMTV пациентов (образцы 1 и 2).
На фиг. 4 представлены данные флюоресценции продуктов ПЦР в режиме реального времени. Анализ образцов (клещи) инфицированных JMTV с использованием праймеров для выявления методом ПЦР JMTV.
На фиг. 5 изображен график, где представлен результат ПЦР (СТ-25,72), который свидетельствует об успешной экстракции РНК и построении кДНК на ее основе. На фиг.6 представлены последовательности SEQ ID: NO:1-3 олигонуклеотидов видоспецифические к JMTV: прямой (F) праймер 5'®3'; обратный (R) праймер 5'®3' и флуоресцентно-меченый ДНК-зонд (Z) 5'®3' соответственно.
Пример 1. Технология получения набора реагентов для выявления генетического материала РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией
После выделения РНК из исследуемого материала проводили реакцию обратной транскрипции с использованием набора реагентов «Реверта-L» (ЦНИИЭ, Россия) в соответствии с инструкцией по применению. ПЦР с кДНК, полученной после реакции обратной транскрипции, проводили с вирусспецифическими праймерами в реакционной смеси следующего состава (на 1 исследование):
Микропробирки помещали в амплификатор и проводили реакцию по программе (Таблица 1).
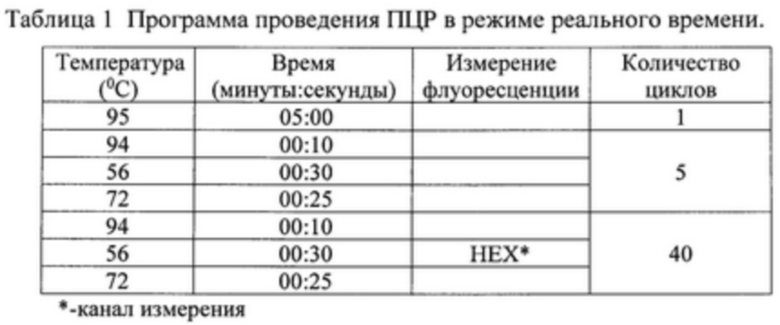
ПЦР в режиме реального времени и регистрацию результатов проводили в приборах CFX96 Opus («Bio-Rad», США) и “Rotor Gene 6000” («Corbet», Австралия) по каналу Orange (585 пт/ 610 пт).
Для выбора генотип-специфичных праймеров и ДНК-зондов предварительно был проведен анализ всех известных к настоящему моменту последовательностей геномов выбранных вирусов и представленных в базе данных GenBank. С использованием известных последовательностей были построены элайменты, соответствующие как полногеномным последовательностям, так и отдельным участкам генома вируса. На основе полученных данных для каждого из выбранных вирусов были найдены уникальные замены и отобраны пары праймеров, в максимальной степени, отвечающие необходимым требованиям.
Последовательности праймеров для проведения амплификации с последующей гибридизацией полученных ампликонов с зондами на поверхности микросфер определенного цвета представлены в Таблице 2.

Подбор и анализ свойств олигонуклеотидных праймеров и зондов проводился с использованием программного обеспечения Vector NTI Advance (InforMax).
Положительные контрольные образцы были получены методом молекулярной трансформации компетентных бактериальных клеток Escherichia coli бактериальными плазмидами pCR 2.1 (Invitrogen, США), включающими вставки ДНК, комплементарные участкам гена белка гликопротеина JMTV 200 п. н., с последующим выделением плазмидной ДНК из бактериальных клеток.
Условия проведения амплификации и реамплификации оптимизировались по концентрации праймеров и зондов в реакционной смеси и температуре отжига праймеров.
Подбор праймеров осуществлялся на кодирующую область геномной РНК (ген белка гликопротеина) JMTV. При этом праймеры подбирались с наименьшей гомологией к другим коронавирусам человека и животных, а также к геному человека. На фиг.1 представлено множественное выравнивание нуклеотидных последовательностей JMTV. Цветом помечены районы комплементарные диагностическим праймерам и зонду.
В качестве отрицательного контрольного образца в реакционную смесь вместо положительного контрольного образца добавляли TEx1 буфер.
ПЦР проводили на приборах Rotor Gene 6000 («Corbett Research», Австралия) или CFX96 Opus («BioRad», США).
Для определения аналитической чувствительности моновариантов систем «праймеры-зонд» для детекции вируса из концентрированных растворов положительных контрольных образцов были приготовлены последовательные 10-кратные разведения. Определение концентрации ДНК в разведениях при исследовании чувствительности праймеров осуществляли при помощи коммерческого набора «Quant-iT dsDNA, HS» («Invitrogen», США) и флуориметра QUBIT 2 («Invitrogen», США).
Для контроля повторяемости используют образец ПКО JMTV разведенный до концентрации 1,0×107 копий/мл (5 образцов). Процедура проводится одним оператором на одном наборе. Коэффициент вариации для повторяемости рассчитывается по формуле: CVp,%=Ct (стандотклон) / Ct (ср.знач.) ´ 100% для 5 проб ПКО JMTV и не должен превышать 3%.
Для контроля воспроизводимости используют образец ПКО JMTV разведенный до концентрации 1,0×107 копий/мл (10 образцов). Процедура проводится двумя разными операторами с двумя разными наборами одной серии в разные дни. Коэффициент вариации для воспроизводимости рассчитывается по формуле: CVp,%=Ct (стандотклон) / Ct (ср.знач.) ´ 100% для 10 проб ПКО JMTV и не должен превышать 6%.
Минимальное количество ДНК-матриц, детектируемое с применением наших праймеров и зондов после оптимизации условий проведения реакции, выраженное в ГЭ (геномных эквивалентах) в 24 мкл реакционной смеси, составило 50 ГЭ (Ct 39,5). На фиг. 2 представлена флюоресценция продуктов ПЦР в режиме реального времени. Оценка чувствительности с 10-кратными разведениями плазмидной ДНК. Прослеживается зависимость накопления флуоресцентного сигнала от концентрации матричной ДНК (кДНК JMTV).
Специфичность отжига праймеров и зондов проверяли постановкой ПЦР в режиме реального времени с использованием в качестве детектируемых образцов синтетической последовательности фрагмента генома JMTV (плазмида pCR-JMTV) и кДНК содержащей JMTV, построенной из суммарной РНК культуры клеток Vero инфицированной JMTV. На фиг. 3 приведена флюоресценция продуктов ПЦР в режиме реального времени. Анализ образцов (культуры клеток Vero) инфицированных JMTV (образцы 1 (Ct 20) и 2 (Ct 27); К+Ct 10).
Контроль специфичности осуществляли проведением ПЦР с флуоресцентной детекцией, где в качестве исследуемых образцов использовали пробы, содержащие генетический материал JMTV и пробы, содержащие генетический материал гетерологичных вирусов (вирус клещевого энцефалита, вирус Западного Нила, аденовирус 5 типа, вирус денге 2, вирус Повассан). Не специфических перекрестных реакций отмечено не было.
На фиг. 4 представлены данные по флюоресценции продуктов ПЦР в режиме реального времени образцов культуры клеток инфицированых JMTV с использованием специфических праймеров для JMTV с последующим подтверждением секвенированием по Сенгеру. Полученные данные свидетельствуют о специфичности выбранных нами праймеров для выявления JMTV.
Таким образом, разработан набор реагентов, предназначенный для выявления РНК JMTV методом, основанным на амплификации кДНК в полимеразной цепной реакции (ПЦР) и гибридизационно-флуорецентной детекции продуктов амплификации в режиме реального времени.
Пример 2. Исследование возможности использования реагентов для выделения РНК/ДНК из клинического материала для получения кДНК на матрице РНК и получения кДНК JMTV
Для определения возможности использования коммерчески доступных реагентов для выделения РНК/ДНК из полевого материала содержащего JMTV и для получения кДНК на матрице РНК, были использованы Комплект реагентов для выделения «РИБО-преп» (Per. №ФСР 2008/03147) и Комплект реагентов для получения кДНК на матрице РНК «РЕВЕРТА-L» (Per. №ФСР №ФСР 2008/03994) производства ФБУН ЦНИИ Эпидемиологии Роспотребнадзора. Данные наборы реагентов успешно используются для выявления возбудителей острых респираторных вирусных инфекций человека (ОРВИ) РНК респираторносинцитиального вируса (human Respiratory Syncytial virus - hRSv), метапневмовируса (human Metapneumovirus-hMpv), вирусов парагриппа 1, 2, 3 и 4 типов (human Parainfluenza virus-1-4-hPiv), коронавирусов (human Coronavirus - hCov), риновирусов (human Rhinoviru -hRv), ДНК аденовирусов групп В, С и E (human Adenovirus В,С,Е- hAdv) и бокавируса (human Bocavirus - hBov) в клиническом материале методом полимеразной цепной реакции (ПЦР) с гибридизационно-флуоресцентной детекцией.
Для определения возможности использования указанных комплектов реагентов для выделения РНК и получения кДНК JMTV использовали JMTV из лабораторной коллекции Отдела молекулярной вирусологии флавивирусов и вирусных гепатитов ФБУН ГНЦ ВБ «Вектор».
Экстракция РНК проводилась с использованием комплекта реагентов для выделения «РИБО-преп».
В пробирки с образцами в лизирующем растворе добавляли по 400 мкл раствора для преципитатции, перемешивали на вортексе и центрифугировли на микроцентрифуге в течение 5 мин при 15 тыс.об/мин. Аккуратно удаляли надосадочную жидкость, используя вакуумный отсасыватель и отдельный наконечник для каждой пробы.
Добавляли в пробы по 500 мкл раствора для отмывки 3, плотно закрывали крышки и осторожно промывали осадок, переворачивая пробирки 3-5 раз, после чего центрифугировли на микроцентрифуге в течение 2 мин при 13 тыс.об/мин.
Осторожно, не захватывая осадок, отбирали надосадочную жидкость, используя вакуумный отсасыватель и отдельный наконечник для каждой пробы, плотно закрывали крышки и осторожно промывали осадок, переворачивая пробирки 3-5 раз, после чего центрифугировли на микроцентрифуге в течение 2 мин при 13 тыс.об/мин.
После центрифугирования осторожно, не захватывая осадок, отбирали надосадочную жидкость, используя вакуумный отсасыватель и отдельный наконечник для каждой пробы.
Открытые пробирки помещали в твердотельный термостат при температуре 65°С на 5 мин. для подсушивания осадка.
Добавляли по 50 мкл РНК-буфера, перемешивании на вортексе и помещали в термостат при температуре 65°С на 5 мин плотно закрытыми, периодически встряхивая на вортексе.
Центрифугировали пробирки при 13 тыс.об/мин в течение 1 мин на микроцентрифуге. Надосадочную жидкость, содержащую РНК, использовали для проведения обратной транскрипции.
Для получения кДНК использовали комплект реагентов для получения кДНК на матрице РНК «РЕВЕРТА-L». В пробирку, содержащую 10 мкл РНК, добавляли 10 мкл смеси, приготовленной следующим образом: в пробирку с RT-mix вносили 5 мкл RT-G-mix-1 и 6 мкл ревертазы (MMulv), и перемешивали на вортексе. Инкубировали пробирки при 37°C 30 мин. Затем отдельным наконечником с аэрозольным барьером добавляли 20 мкл ДНК-буфера.
Надосадочную жидкость, содержащую кДНК использовали для постановки ПНР с ВКЭ специфическими праймерами и зондами. На фиг. 5 представлен положительный результат ПЦР (Ct 25,7), который свидетельствует об успешной экстракции РНК и построении кДНК на ее основе.
Таким образом, комплект реагентов для выделения «РИБО-преп» и комплект реагентов для получения кДНК на матрице РНК «РЕВЕРТА-L» могут быть использованы для выделения JMTV.
Пример 3. Проверка аналитической специфичности набора реагентов для выявления JMTV при тестировании на вирусе гриппа В, вирусах парагриппа 1-4, риновирусах, человеческих метапневмовирусах
Контроль аналитической специфичности набора реагентов для выявления JMTV осуществляли проведением ПЦР с флуоресцентной детекцией, где в качестве исследуемых образцов использовали инактивированные образцы культуральной жидкости, содержащие следующие штаммы из Государственной коллекции микроорганизмов ФБУН ГНЦ ВБ «Вектор»: коронавирус ближневосточного респираторного синдрома (БВРС-КоВ) штамм hCoV-EMC/2012); коронавирус SARS (штамм Frankfurt 1), вирус гриппа B/Chita/01/2010, вирус гриппа B/Chita/02/2010, вирус гриппа B/Chita/03/2010, вирус гриппа B/Chita/04/2010, РСВ штамм Long, аденовирус типа 5 штамм AD-GP (АВ_ AD-GP), а также охарактеризованные изоляты риновируса человека С (изолят RhV-NSC- 14/2012), риновируса человека А (изолят RhV-NSC-08/2011, изолят RhV- NSC-11/2012) и клинический материал, содержащий вирус парагриппа 1-4 и метапневмовирус человека (см. таблица 3).

Из представленных данных на фиг. 3 и табл. 3 следует, что набор реагентов для выявления JMTV не дает положительных результатов с образцами, содержащими гетерологичные вирусы: коронавирус ближневосточного респираторного синдрома (БВРС-КоВ); коронавирус SARS, вирус гриппа В, РСВ, метапневмовирус человека, аденовирус типа 5, риновируса человека, вирус парагриппа 1-4 и ДНК человека.
Пример 4. Результаты лабораторных испытаний набора реагентов для диагностики ин витро для выявления JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией
В связи с отсутствием полного аналога среди зарегистрированных на территории Российской Федерации медицинских изделий для диагностики ин витро - наборов реагентов для выявления РНК JMTV методом ПЦР, в качестве референтного метода использовали секвенирование по Сенгеру.
Подлинность используемых штаммов вирусов из Государственной коллекции микроорганизмов 1-4 групп патогенности ФБУН ГНЦ ВБ «Вектор» ранее подтверждена секвенированием.
Набор реагентов предназначен для выявления РНК JMTV методом, основанным на амплификации кДНК в полимеразной цепной реакции (ПЦР) и гибридизационно-флуорецентной детекции продуктов амплификации в режиме реального времени на приборах CFX96 (BioRad, США), Rotor-Gene 6000/3000 (Corbett Research, Австралия) или аналогичных приборах других производителей.
Диагностическая роль набора реагентов для выявления JMTV заключается в возможности его использования для ранней диагностики JMTV, индикации патогенных биологических агентов (ПБА); как ускоренного предварительного теста при выполнении культурального и биологического методов исследования и идентификации подозрительных культур; для эпидемиологического мониторинга.
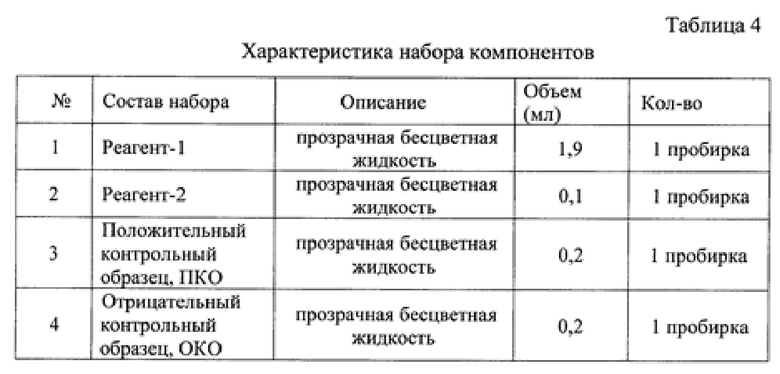
В состав набора входят следующие компоненты (таблица 4):
Примечание:
Реагент-1 расфасован по 1,9 мл в микропробирку вместимостью 2,0 мл, с завинчивающейся крышкой. Состав: 2х-кратный буфер для ОТ-ПЦР-РВ, олигонуклеотидные праймеры, флуоресцентный зонд, очищенная вода.
Реагент-2 расфасован по 100 мкл в микропробирку вместимостью 0,5 до 2,0 мл, с завинчивающейся крышкой. Состав: HS-Taq ДНК-полимераза в буферном растворе.
Метод основан на использовании ПЦР, представляющей собой повторяющиеся циклы амплификации специфической области кДНК- мишени в присутствии ДНК-полимеразы, дезоксирибонуклеозидтрифосфатов, буферного раствора и олигонуклеотидных праймеров, распознающих кДНК-мишень. Для амплификации фрагментов вирусной кДНК подобрана пара специфических олигонуклеотидных праймеров. Для детекции продукта амплификации применяется вирусспецифический флуоресцентно-меченый ДНК-зонд, и накопление флуоресцентного сигнала соответствует наличию в реакционной смеси специфических продуктов амплификации. Для проведения клинических испытаний были искусственно контаминированы генетическим материалом JMTV в концентрации 1×104 копий/мл.
Лабораторные испытания проведены с использованием 10 положительных проб (5 проб в 2-х повторах), содержащих генетический материал JMTV в концентрации не менее 1×104 копий/мл, и 10 отрицательных проб (5 проб в 2-х повторах), не содержащих генетический материал JMTV, в том числе:
- положительные образцы, содержащие генетический материал JMTV;
10 образцов мазков из ротоглотки, содержащих генетический материал JMTV;
10 образцов мазков из носоглотки, содержащих генетический материал JMTV;
5 образцов сыворотки крови, содержащих генетический материал JMTV;
- отрицательные образцы, не содержащие генетический материал JMTV;
4 образца ДНК человека;
Подлинность штаммов гетерологичных вирусов из Государственной коллекции микроорганизмов 1-4 групп патогенности ФБУН ГНЦ ВБ «Вектор» ранее подтверждена результатами секвенирования.
Референтный метод
В связи с отсутствием аналога среди зарегистрированных на территории Российской Федерации медицинских изделий для диагностики ин витро - наборов реагентов для выявления РНК JMTV методом ПЦР, в качестве референтного метода использовали секвенирование по Сенгеру.
Подлинность используемых штаммов вирусов из Государственной коллекции микроорганизмов 1-4 групп патогенности ФБУН ГНЦ ВБ «Вектор» ранее была подтверждена секвенированием.
Методика проведения исследований
Все образцы одновременно исследованы с использованием наборов реагентов для выявления JMTV, дата изготовления 20.10.2023.
Подготовка проб
Штаммы вирусов были получены из лабораторной коллекции ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора. Работы по наращиванию вирусов в клеточных культурах были проведены согласно стандартным операционным процедурам, разработанным в ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора.
Исследуемый биологический материал (культура клеток Vero) был контаминирован генетическим материалом JMTV в концентрации 1×104 копий/мл.
Обеззараживание проб
Обеззараживание проб проводили в соответствии с требованиями СП 1.3.3118-13 «Безопасность работы с микроорганизмами I-II групп патогенности (опасности)» и МУ 1.3.2569 -09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности».
Пробы, содержащие гетерологичные вирусы в объеме 1 мл переносили в микроцентрифужные пробирки вместимостью 1,5 мл, добавляли натрия мертиолят до концентрации 1:10000 (0,01%) и прогревали при (56±1)°C в течение 30 минут. Затем в отдельные микроцентрифужные пробирки вместимостью 1,5 мл переносили по 100 мкл обработанных мертиолятом натрия вирусосодержащих проб. Во все пробирки, добавляли лизирующий буфер на основе 6М гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения РНК, и инкубировали 15 минут при температуре (65±1)°C. После выполнения данного этапа материал считался обеззараженным.
Выделение РНК, получение кДНК
Все работы по выделению РНК проводились с соблюдением требованиий МУ 1.3. 2569 -09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Работа по выделению РНК осуществлялась из инактивированных проб за пределами «заразной» зоны в ПЦР боксе в защитном костюме IV типа, дополненном резиновыми перчатками, с соблюдением правил выноса предметов из «заразной» зоны (внешняя обработка контейнера).
Экстракция РНК проводилась с использованием зарегистрированного набора для выделения «Рибо-преп» (Per. №ФСР 2008/03147, производства ФБУН ЦНИИ Эпидемиологии Роспотребнадзора», Москва).
Для получения кДНК использовали зарегистрированный набор для получения кДНК на матрице РНК "PEBEPTA-L" (Per. №ФСР №ФСР 2008/03994, производства ФБУН ЦНИИ Эпидемиологии Роспотребнадзора», Москва).
В пробирку, содержащую 10 мкл РНК добавляли 10 мкл смеси, приготовленной следующим образом: в пробирку с RT-mix вносили 5 мкл RT-G-mix-1 и 6 мкл ревертазы (MMulv), и перемешивали на вортексе.
Инкубировали пробирки при 37°C 30 мин. Затем отдельным наконечником с аэрозольным барьером добавляли 20 мкл ДНК-буфера.
Надосадочную жидкость, содержащую кДНК, хранили при -20°С.
Проведение ПЦР
На одно определение расходуются:
18 мкл Реагент-1; 1 мкл Реагент-2.
На каждый анализ расходуются:
5 мкл ПОК; 5 мкл ОКО
Все реакционные компоненты добавляли отдельными наконечниками с аэрозольным барьером с помощью дозаторов переменного объема: 0,5-10, 2-20 и 20-200 мкл.
Последовательность проведения этапов анализа
Общий объем реакционной смеси 24 мкл, объем кДНК-пробы - 5 мкл.
Проведение амплификации:
Приготовили реакционную смесь для проведения ПЦР и внесли в пробирки.
В пробирке объемом 1,5 мл приготовили смесь компонентов следующего состава (из расчета на одну пробу):
перемешивали смесь пипетированием.
Внесли по 19 мкл смеси компонентов в заранее промаркированные пробирки.
Внесли в пробирки отдельным наконечником с аэрозольным барьером по 5 мкл препарата кДНК.
Поставили контрольные реакции амплификации:
отрицательный контроль - вместо препарата кДНК в пробирку внесли 5 мкл ОКО.
положительные контроли (вносили в последнюю очередь) - вместо препарата кДНК в пробирки внесли 5 мкл ПКО.
Общий (конечный) объем реакционной смеси в каждой пробирке должен составить 24 мкл.
Поставили пробирки в ячейки амплификатора и задали программу амплификации со следующими параметрами:
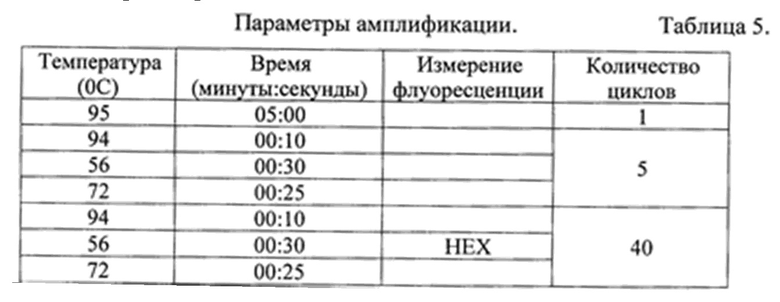
Далее запускали программу амплификации и вносили данные в таблицу исследуемых образцов.
Процедура измерения и оценки результатов анализа. ПЦР в режиме реального времени и регистрацию результатов проводили в приборе CFX 96 (BioRad, США) по каналу HEX.
Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию (или отсутствию) значения порогового цикла "Ct" в соответствующей графе в таблице результатов).
Результат подлежит учету в случае:
а) появления пересечения кривых флуоресценции с пороговой линией на канале HEX в пробах с положительными контрольными образцами (ПКО);
б) отсутствия положительного сигнала на канале HEX в пробе с отрицательными контрольными образцами (ОКО).
Результат считать положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. При этом значение Ct для данного образца должно быть менее 35. Амплитуда сигнала при этом не имеет значения.
Результат считать отрицательным, если значение Ct на канале HEX отсутствует.Если значение Ct больше 35, то ПЦР-РВ необходимо повторить и считать положительным в случае повторения результата или при значении Ct меньше 35.
Чувствительность. Набор реагентов для выявления JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией, при исследовании проб содержащих генетический материал JMTV в концентрации не менее 1×104 копий/мл, составляет 100%.
Внутрипостановочная, межпостановочная и межсерийная воспроизводимость для всех положительных образцов - 100%.
Специфичность. Набор реагентов для выявления РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией при исследовании проб содержащих генетический материал JMTV в концентрации не менее 1×104 копий/мл, составляет 100%.
Специфичность. Набор реагентов для выявления РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией при исследовании отрицательных образцов, содержащих НК гетерологичных микроорганизмов (вирус денге, аденовирус 5 типа, вирус гриппа типа А, вире гриппа типа В, вирус парагриппа 1 типа, респираторно-синцитиальный вирус), 4 отрицательных образца содержащих ДНК человека, образцов, для которых клинически подтвержден вирус парагриппа 1 типа, 4 клинических образца мазков из ротоглотки, для которых клинически подтвержден вирус гриппа типа А, 2 клинических образца мазков из ротоглотки, для которых клинически подтвержден вирус гриппа типа В, 2 клинических образца мазков из ротоглотки, для которых клинически подтвержден респираторносинцитиальный вирус отрицательный результат в ПЦР, диагностическая специфичность составила не менее 98%, с доверительной вероятностью 90%.
Статистическая обработка результатов проводилась в соответствии с «Методическими рекомендациями по порядку проведения экспертизы качества, эффективности и безопасности медицинских изделий», утв. ФГБУ «ЦМиКЭЭ» и ФГБУ «ВНИИМТ» 14.11.2013 г.
Статистическую достоверность полученных результатов испытаний оценивали в зависимости от числа параллельных опытов при доверительной вероятности 90%, используя формулу биноминального распределения Бернулли.
При лабораторных испытаниях набора реагентов для выявления JMTV на 10 положительных пробах (2 по 5 проб), содержащих генетический материал JMTV в концентрации не менее 1×104 копий/мл, был получен положительный результат в 100% случаев (10 проб из 10 заведомо положительных).
При лабораторных испытаниях набора реагентов для выявления JMTV на 36 пробах, не содержащих генетический материал JMTV (22 пробы, содержащих гетерологичные вирусы, по 10 проб вода и 4 пробы содержащих ДНК человека), отрицательный ответ получен в 200 пробах (100% случаев).
Таким образом, в ходе клинических испытаний с использованием 10 положительных проб, содержащих генетический материал JMTV и на отрицательных пробах, содержащих ДНК человека (4 проб) и НК гетерологичных вирусов (аденовирус 5 типа (1 проба), вирус денге (1 проба), вирус гриппа (4 пробы), вирус парагриппа (4 пробы), респираторносинцитиальный вирус (2 пробы), по одной пробе - ВКЭ, вирус Повассан, ВЗН), доказана диагностическая эффективность разработанного набора реагентов для выявления JMTV.
- при исследовании 10 положительных проб (2 по 5 проб, содержащих генетический материал JMTV положительный презультат получен в 10 случаях. Диагностическая чувствительность - не менее 95% с доверительной вероятностью 90%.
- при исследовании генетических проб содержащих ДНК человека (4 пробы) и НК гетерологических виросов (аденовирус 5 типа, вирус денге, вирус гриппа А и В, вирус парагриппа (4 пробы), респираторно-синцитиальный вирус (2 пробы)), с помощью набора реагентов для выявления JMTV, ложно положительных сигналов не обнаружено. Диагностическая специфичность - не менее 98% с доверительной вероятностью 90%.
Источники
1a. I.S. Kholodilov, A.G. Litov, A.S. Klimentov, О.A. Belova, А.Е. Polienko, N.A. Nikitin, А.М. Shchetinin, A.Y. Ivannikova, L. Bell-Sakyi, A.S. Yakovlev, S.V. Bugmyrin, L.A. Bespyatova, L.V. Gmyl, S.V. Luchinina, A.P. Gmyl, V.A. Gushchin, G.G. Karganova. Isolation and Characterisation of Alongshan Virus in Russia. Viruses. 2020 Mar 26;12(4):362. doi: 10.3390/V12040362
1. Simmonds, P.; Becher, P.; Bukh, J.; Gould, E.A.; Meyers, G.; Monath, T.; Muerhoff, S.; Pletnev, A.; Rico-Hesse, R.; Smith, D.B.; et al. ICTV virus taxonomy profile: Flaviviridae. J. Gen. Virol. 2017, 98, 2-3.
2. Shi, M.; Lin, X.-D.; Vasilakis, N.; Tian, J.-H.; Li, C.-X.; Chen, L.-L; Eastwood, G.; Diao, X.-N.; Chen, М.-H.; Chen, X.; et al. Divergent Viruses Discovered in Arthropods and Vertebrates Revise the Evolutionary History of the Flaviviridae and Related Viruses. J. Virol. 2016, 90, 659-669.
3. Qin, X.-C.; Shi, M.; Tian, J.-H.; Lin, X.-D.; Gao, D.-Y.; He, J.-R.; Wang, J.-B.; Li, C.-X.; Kang, Y.-J.; Yu, B.; et al. A tick-bome segmented RNA virus contains genome segments derived from unsegmented viral ancestors. Proc. Natl. Acad. Sci. USA2014, 111, 6744-6749.
4. International Committee on Taxonomy of Viruses Genus: Flavivirus. Available online: https://talk.ictvonline.org/ictv-reports/ictv_online_report/positive-sense-ma-viruses/w/flaviviridae/360/genus- flavivirus (accessed on 27 January 2021).
5. Dinner, E.; Hacio glu, S.; Kar, S.; Emanet, N.; Brinkmann, A.; Nitsche, A.; Ozkul, A.; Linton, Y.-M.; Ergiinay, K. Survey and Characterization of Jingmen Tick Virus Variants. Viruses 2019, 11, 1071.
6. Garry, C.E.; Garry, R.F. Proteomics Computational Analyses Suggest that the Envelope Glycoproteins of Segmented Jingmen Flavi-Like Viruses Are Class II Viral Fusion Proteins (_-Penetrenes) with Mucin-Like Domains. Viruses 2020, 12, 260.
7. Kholodilov, I.S.; Litov, A.G.; Klimentov, A.S.; Belova, O.A.; Polienko, A.E.; Nikitin, N.A.; Shchetinin, A.M.; Ivannikova, A.Y.; Bell-Sakyi, L.; Yakovlev, A.S.; et al. Isolation and characterisation of Alongshan virus in Russia. Viruses 2020, 12, 362.
8. Gordon, K.H.J.; Williams, M.R.; Hendry, D.A.; Hanzlik, T.N. Sequence of the genomic RNA of Nudaurelia virus (Tetraviridae) defines a novel virus genome organization. Virology 1999, 258, 42-53.
9. Yong, C.Y.; Yeap, S.K.; Omar, A.R.; Tan,W.S. Advances in the study of nodavirus. Peer J 2017, 2017, 1-31.
10. Jia, N.; Liu, H.B.; Ni, X.B.; Bell-Sakyi, L.; Zheng, Y.C.; Song, J.L.; Li, J.; Jiang, B.G.;Wang, Q.; Sun, Y.; et al. Emergence of human infection with Jingmen tick virus in China: A retrospective study. EBioMedicine 2019, 43, 317-324.
11. Temmam, S.; Bigot, T.; Chretien, D.; Gondard, M.; Perot, P.; Pommelet, V.; Dufou, E.; Petres, S.; Devillers, E.; Ноет, T.; et al. Insights into the Host Range, Genetic Diversity, and Geographical Distribution of Jingmenviruses. mSphere 2019, 4, 1-13.
12. Emmerich, P.; Jakupi, X.; von Possel, R.; Berisha, L.; Halili, B.; Gunther, S.; Cadar, D.; Ahmeti, S.; Schmidt-Chanasit, J. Viral metagenomics, genetic and evolutionary characteristics of Crimean-Congo hemorrhagic fever orthonairovirus in humans, Kosovo. Infect. Genet. Evol. 2018, 65, 6-11.
13. Kuivanen, S.; Levanov, L.; Kareinen, L.; Sironen, T.; Jaaskelainen, A.J.; Plyusnin, L; Zakham, F. Detection of novel tick-borne pathogen, Alongshan virus, in Ixodes ricinus ticks, south-eastern Finland. Eurosurveillance 2019, 24, 1900394.
14. Ladner, J.T.; Wiley, M.R.; Beitzel, B.; Auguste, A.J.; Dupuis, A.P.; Lindquist, M.E.; Sibley, S.D.; Kota, K.P.; Fetterer, D.; Eastwood, G.; et al. A Multicomponent Animal Virus Isolated from Mosquitoes. Cell Host Microbe 2016, 20, 357-367.
15. Maruyama, S.R.; Castro-Jorge, L.A.; Ribeiro, J.M.C.; Gardinassi, G.; Garcia, G.R.; Brandao, L.G.; Rodrigues, A.R.; Okada, M.I.; Abrao, E.P.; Ferreira, B.R.; et al. Characterisation of divergent flavivirus NS3 and NS5 protein sequences detected in Rhipicephalus microplus ticks from Brazil. Mem. Inst. Oswaldo Cruz 2014, 109, 38-50.
16. Villa, E.C.; Maruyama, S.R.; de Miranda-Santos, I.K.F.; Palacios, G.; Ladner, J.T. Complete Coding Genome Sequence for Mogiana Tick Virus, a Jingmenvirus Isolated from Ticks in Brazil. Genome Announc. 2017, 5, 17-18.
17. De Pascoal, J.O.; de Siqueira, S.M.; da Maia, R.C.; Szabo, M.P.J.; Yokosawa, J. Detection and molecular characterization of Mogiana tick virus (MGTV) in Rhipicephalus microplus collected from cattle in a savannah area, Uberlandia, Brazil. Ticks Tick. Borne. Dis. 2019, 10, 162-165.
18. Vandegrift, K.J.; Kumar, A.; Sharma, H.; Murthy, S.; Kramer, L.D.; Ostfeld, R.; Hudson, P.J.; Kapoor, A. Presence of segmented flavivirus infections in North America. Emerg. Infect. Dis. 2020, 26, 1810-1817.
19. Webster, C.L.; Waldron, F.M.; Robertson, S.; Crowson, D.; Ferrari, G.; Quintana, J.F.; Brouqui, J.M.; Bayne, E.H.; Longdon, B.; Buck, A.H.; et al. The discovery, distribution, and evolution of viruses associated with Drosophila melanogaster. PLoS Biol. 2015, 13, el002210.
20. Wang, Z.-D.;Wang, B.;Wei, F.; Han, S.-Z.; Zhang, L.; Yang, Z.-T.; Yan, Y.; Lv, X.-L.; Li, L.;Wang, S.-С.; et al. A New Segmented Virus Associated with Human Febrile Illness in China. N. Engl. J. Med. 2019, 380, 2116-2125.
21. De Souza, W.M.; Fumagalli, M.J.; de Carrasco, A.O.T.; Romeiro, M. F.; Modha, S.; Seki, M.C.; Gheller, J.M.; Daffre, S.; Nunes, M.R.T.; Murcia, P.R.; et al. Viral diversity of Rhipicephalus microplus parasitizing cattle in southern Brazil. Sci. Rep.2018, 8, 1-10.
22. Wang, Z.-D.; Wang, W.; Wang, N.N.; Qiu, K.; Zhang, X.; Tana, G.; Liu, Q. Prevalence of the emerging novel Alongshan virus infection in sheep and cattle in Inner Mongolia, northeastern China. Parasit. Vectors 2019, 12, 1-7.
23. Alexander G. Litov, Egor V. Okhezin, Ivan S. Kholodilov, Alexandra E. Polienko, Galina G. Karganova. Quantitative Polymerase Chain Reaction System for Alongshan Virus Detection. Methods Protoc. 2023, 6, 79. https://doi.org/10.3390/mps6050079
24. Yuli Zhang, Zhenfeng Li, Zheng Pang, Zhen Wu, Zhijuan Lin, Guoyu Niu. Identifcation of Jingmen tick virus (JMTV) in Amblyomma testudinarium from Fujian Province, southeastern China. Parasites & Vectors (2022) 15:339
25. Патент Китая CN 113604612, опубл. 05.11.2021 г., дата регистрации патента 01.08.2023 г. (аналог).
26. Заявка на патент Китая №110273025, опубл. 05.11.2021 г. (аналог).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Приложение_Набор
праймеров JMTV.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-11-01">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>123456</ApplicationNumberText>
<FilingDate>2023-10-29</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>12345</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2023-09-26</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Набор олигодезоксирибонуклеотидных
праймеров и флуоресцентно-меченого зонда для идентификации РНК JMTV
методом ПЦР с гибридизационно-флуоресцентной детекцией
</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctacgtcgggctcatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtacggtccgagtagtcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgagtggaggttctgggtcgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---




Изобретение относится к области биотехнологии. Описан набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого ДНК-зонда для идентификации РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией. Набор содержит одну пару олигонуклеотидов, обладающих активностью прямого (F) и обратного (R) праймеров в полимеразной цепной реакции, а также флуоресцентно-меченый ДНК-зонд (Z), имеющие следующие нуклеотидные последовательности SEQ ID NO: 1-3 соответственно. Техническим результатом заявленного изобретения является создание такого набора олигонуклеотидных праймеров и флуоресцентно-меченого зонда, который обеспечивал бы высокую чувствительность детекции исключительно JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией. 6 ил., 5 табл., 4 пр.
Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого ДНК-зонда для идентификации РНК JMTV методом ПЦР с гибридизационно-флуоресцентной детекцией, содержащий одну пару олигонуклеотидов, обладающих активностью прямого (F) и обратного (R) праймеров в полимеразной цепной реакции, а также флуоресцентно-меченый ДНК-зонд (Z), имеющие следующие нуклеотидные последовательности SEQ ID NO: 1-3 соответственно.
| US 20230203567 A1, 29.06.2023 | |||
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых ДНК-зондов для идентификации РНК энтеровирусов, ротовирусов, вирусов гепатита А и Е, аденовирусов, норовирусов и астровирусов из водной среды методом мультиплексной ПЦР | 2015 |
|
RU2610434C1 |
| Li L.F., Zhang M.Z., Zhu J.G., Cui X.M., Zhang C.F., Niu T.Y., Li J., Sun Y., Wei W., Liu H.B., Yuan T.T., Wei R., Wang Q., Xia L.Y., Zhao L., Lesley B.S., Jiang B.G., Jiang J.F., Frans J., Jia N., Cao W.C | |||
| Dermacentor silvarum, a Medically Important Tick, May Not Be a Competent Vector to | |||
Авторы
Даты
2024-05-07—Публикация
2023-12-04—Подача