Изобретение относится к набору праймеров и зондов для идентификации РНК вируса лихорадки Рифт-Валли методом изотермической ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени в клинических образцах от человека и животных и культуральных вируссодержащих жидкостях, с целью постановки диагноза, коррекции лечения, эпидемиологического расследования, а также для решения научно-исследовательских задач по мониторингу и изучению свойств вируса лихорадки Рифт-Валли и может быть использовано в медицине, биотехнологии и эпидемиологии.
Вирус лихорадки Рифт-Валли (ЛРВ) относится к роду флебовирусов. Это зоонозная арбовирусная природно-очаговая инфекционная болезнь, поражает людей, овец и крупный рогатый скот. ЛРВ была впервые выявлена в 1931 г. во время расследования эпидемии среди овец на ферме в Кении, в районе Рифтовой долины (Rift Valley). С тех пор к югу от Сахары регистрировались вспышки болезни ЛРВ. Так, в 1977 г. была зарегистрирована вспышка лихорадки Рифт-Валли в Египте, куда вирус был занесен инфицированным скотом перемещенным из районов расположенных вдоль ирригационных систем Нила. В результате торговли инфицированным скотом из стран Африканского Рога ЛРВ в сентябре 2000 г. был завезен в Саудовскую Аравию и Йемен.
Интересно, что возникновение эпидемических заболеваний передающихся комарами, таких как малярия, лихорадка денге и лихорадка долины Рифт, связывают с экстремальными погодными условиями, а точнее, с колебаниями температуры поверхностного слоя воды в экваториальной части Тихого океана. Данное явление называют Эль-Ниньо. Эль-Ниньо - фаза Южной осцилляции, в которой область нагретых приповерхностных вод смещается к востоку от Африканского континента. При этом ослабевают или вообще прекращаются пассаты, замедляется подъем глубинных вод океана к поверхности в восточной части Тихого океана, у берегов Перу. Противоположная фаза осцилляции называется Ла-Нинья. Установлено, что Эль-Ниньо коррелирует с циклами возникновения малярии в Индии, Венесуэле и Колумбии. Наблюдается связь со вспышками австралийского энцефалита (энцефалит долины Муррей), проявляющегося после сильных дождей и наводнений, вызванных Ла-Нинья на юго-востоке Австралии. Ярким примером является тяжелая вспышка лихорадки долины Рифт, произошедшая из-за Эль-Ниньо после экстремальных осадков в северовосточной части Кении, южной части Сомали и Танзании [https://www.allcountries.org/health/el_nino_and_its_health_impact.html].
За последнюю четверть века вирус лихорадки долины Рифт резко расширил ареал своего распространения и в настоящее время регистрируется не только в Африке, но и в Азии [Rift valley fever. Saudi Arabia: Yemen / Wkly Epidemiol. Rec. 2000. Vol. 76, N 40. P. 321.]. Кроме того, ЛРВ эволюционировал от малопатогенного до высокопатогенного для человека возбудителя, вызывая тяжелое геморрагическое заболевание и практически сравнявшись в этом отношении с некоторыми представителями группы особо опасных возбудителей [Маркин В.А., Марков В.А. Вирусные геморрагические лихорадки - эволюция эпидемического потенциала // ЖМЭИ. 2002. №1. С. 91-98.]. Так, летальность во время вспышки в ЮАР в 2010 г. составила 10%. В странах Восточного Рога (Кения, Сомали, Танзания) во время эпидемий 2006-2008 гг. летальность достигала 31,8-41% [Rift valley fever. URL: www.cdc.gov/ncidod/dvrd/spb/mnpages/ /dispages/rvf. htm (дата обращения: 10.07.2023)], а по некоторым наблюдениям - до 50% [Njenga M.K., Paweska J., Wanjala R. et al. Using a field quantitative real-time PCR test to rapidly identify highly viremic Rift valley fever cases // J. Clin. Microbiol. 2009. Vol. 47, N 4. P.1166-1171.]. Заболеваемость населения ЛДР в отдельных странах достигала от 5 до 25,5% [Faye О., Diallo М., Diop D. et al. Rift valley fever outbreak with East-Central African virus lineage in Mauritania, 2003 // Emerg. Infect. Dis. 2007. Vol. 13, N 7. P.1016-1023.]. 2016 г., в Республике Нигер Министерство здравоохранения сообщило о 105 предполагаемых случаях заболевания ЛРВ среди людей в регионе Тахуа, в том числе о 28 случаях смерти.
В связи с тем, что вспышки ЛРВ у животных предшествуют возникновению случаев заболевания людей, для обеспечения раннего оповещения ветеринарных служб и органов общественного здравоохранения, важное значение имеет создание эффективных диагностических систем раннего выявления новых случаев заболевания. Данная заявка на изобретение посвящена разработке набора реагентов для выявления РНК (кДНК) вируса ЛРВ методом изотермической ПНР с гибридизационно флуоресцентной детекцией. Амплифицируемый участок, являясь маркерным, позволяет выявить с помощью специфичных праймеров вирусную кДНК в исследуемом образце. Сложность выбора праймеров и зонда обусловлена требованием их строгой видоспецифичности. Праймеры должны быть комплементарны нуклеотидным последовательностям кДНК, ограничивая амлифицируемый участок справа и слева таким образом, чтобы синтез ДНК ДНК-полимеразой проходил строго в выбранном регионе. Правильный выбор праймеров позволяет осуществить экспоненциальное увеличение количества копий целевого участка кДНК. В целом от правильности выбора олигодезоксирибонуклеотидных праймеров зависит специфичность проводимой ПЦР, а значит и точность диагностики вирусного заболевания.
Аналогов, представляющих собой наборы праймеров/реагентов обеспечивающих детекцию РНК/кДНК вируса лихорадки Рифт-Валли в России не зарегистрировано. Однако, в литературе приводятся праймеры, используемые в лабораторных условиях в диагностических целях [Pepin М., Bouloy М., Bird В.Н. et al. Rift valley fever virus (Bunyaviridae: Phlebovirus): An update on pathogenesis, molecular epidemiology, vectors diagnostics and prevention // Vet. Res. 2010. Vol. 41, N 6. P. 61-101; Francis Wekesa, Mark Wamalwa, Richard Oduor, YatinderBinepa, Leonard Ateya, Noah Okumu, Angela M'kwenda, Christopher Masaba, Eugine Mukhaye. Development and Validation of Rapid Colorimetric Reverse Transcription Loop-Mediated Isothermal Amplification for Detection of Rift Valley Fever Virus Advances in Virology. Volume 2023, https://doi.org/10.1155/2023/1863980]. Так, был разработан одностадийный RT-LAMP тест с калориметрической детекцией, что позволило визуально обнаружить РНК В ЛРВ в течение 30 минут при 65°С. Диагностическая чувствительность и специфичность разработанного набора сравнивали с qRT-PCR ВЛРВ с использованием образцов тотальной РНК, извлеченных из 118 образцов крови. Колориметрический анализ RT-LAMP имел чувствительность 98,36% и специфичность 96,49%. Разработанная RT-LAMP оказалась в десять раз более чувствительной по сравнению с qRT-PCR анализа обычно используемого для подтверждения диагноза ЛРВ. Лабораторная диагностика ЛДР направлена на дифференциацию с эндемичными в этих же регионах инфекциями, имеющими достаточно схожий характер клинических проявлений - геморрагических лихорадок Эбола, Марбург, Ласса, денге, желтой лихорадки и рядом других особо опасных заболеваний.
Для выявления вируса лихорадки долины Рифт ранее была зарегистрирована Тест-система ФБУН ГНЦ ВБ "Вектор" (РЗН 2018/6931), на основе ПЦР в реальном времени (Ветеринария, 2007, N.6, стр. 53-57.), на которую получен патент РФ №2552795, C12Q 1/68, опубл. 07.05.2014 г. В качестве объекта изобретения заявлен набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации генетического материала ВЛДР в клинических и биологических образцах и объектах внешней среды методом ОТ-ПЦР в режиме реального времени. Подобраны олигонуклеотидные праймеры и флуоресцентно-меченый зонд, имеющие следующий нуклеотидный состав:
Праймер RVFV-2s: 5'-AGGAGCATCTGAAATAGG-3'
Праймер RVFV-2a: 5'-GGCTCAAATGATCAAMCC-3'
Зонд RVFV-2: 5'-FAM-TCATCATGGGAAACTCACGCA-BHQI-3'.
Данное изобретение позволяет эффективно идентифицировать вирус лихорадки долины Рифт. К недостаткам предлагаемой тест-системы можно отнести низкую чувствительность (100 копий/в реакции) и время реакции (1,5 часа).
Наиболее близким аналогом (прототипом) является патент Китая (CN105483293, МПК C12Q 1/70, опубл. 13.04.2016 г.) Fulminating-infectious-disease pathogen detecting primer pair and kit, Hao Rongzhang и др. Изобретение касается набора праймеров для обнаружения возбудителей опасных инфекционных заболеваний с применением RT-LAMP, вирусов Эбола, вирусов лихорадки Ласса, вирусов Марбурга, вирусов лихорадки Рифт-Валли, вирусов желтой лихорадки и вирусов лихорадки Чикунгунья. С помощью системы праймеров в RT-LAMP достигается хорошая чувствительность и специфичность. Набор праймеров предназначен для применения с микрофлюидным чипом. Время детектирования 1 час, заявленная чуствительность реакции в пределах 1 копии в реакции. Результат определяют по изменению цвета турбидиометрическим методом. Для окраски используют HNB. HNB - это гидроксинафтоловый синий, а гидроксинафтоловый синий - индикатор ионов кальция и магния. В ходе реакции ионы магния реагируют с dNTP с образованием пирофосфата магния, который поглощает ионы магния и цвет гидроксинафтолового синего меняется с фиолетового на синий.
Недостатком такой индикации RT-LAMP реакции является низкая чувствительность при малой концентрации образца в реакции. При концентрации 103-102 копий в реакции нет линейной зависимости между концентрацией определяемой РНК и временем реакции, высокий фон неспецифической флюоресценции. Именно поэтому, при концентрации 103-102 копий в реакции система с турбидиометрическим методом индикации RT-LAMP реакции дает ложно положительный результат.
Техническим результатом данного заявленного изобретения является создание отечественного набора реагентов для выявления РНК вируса лихорадки Рифт-Валли методом изотермической амплификации, который обеспечивал бы при высокой чувствительности высокую специфичность и детекцию только РНК вируса лихорадки Рифт-Валли в исследованных образцах.
Указанный технический результат достигается получением набора праймеров и зондов для выявления РНК вируса лихорадки Рифт-Валли методом RT-LAMP с гибридизационно-флуоресцентной детекцией в режиме реального времени, содержащем прямой внешний праймер F3 и обратный внешний праймер ВЗ, прямой внутренний праймер FIP и обратный внутренний праймер BIP к гену L РНК вируса лихорадки Рифт-Валли и гасящий зонд Fd DARQ для обнаружения амплификации. Также, для контроля этапа выделения нуклеиновых кислот, был разработан комплект праймеров, комплементарный нуклеотидной последовательности фага MS2 - прямой F1, обратный R1 праймеры и зонд Z1 с флуоресцентной меткой. Последовательности олигонуклеотидных праймеров и зондов представлены SEQ ID NO: 1-8 соответственно:
SEQ ID NO: 1

SEQ ID NO: 2

SEQ ID NO: 3
SEQ ID NO: 4
SEQ ID NO: 5

SEQ ID NO: 6
SEQ ID NO: 7

SEQ ID NO: 8
Полимеразная цепная реакция (ПЦР) - это амплификация специфических ДНК/кДНК последовательностей в реакции с циклическим изменением температуры реакционной смеси. Альтернативным подходом является амплификация специфического образца ДНК/кДНК при которой не требуется изменение температуры реакционной смеси, и поэтому часто ее называют изотермической амплификацией (LAMP - Loop-Mediated Isothermal Amplification). Проведение изотермической амплификации требует намного меньше времени (30 минут вместо полутора часов при классическом ПЦР), вместо дорогостоящего прибора - амплификатора, можно использовать небольшой и простой регистрирующий термостат (например LAMPIX 8, Россия), что делает методику особенно удобной для применения в полевых условиях.
Преимущества LAMP методики:
• Упрощенный протокол пробоподготовки;
• Амплификатор можно заменить на регистрирующий термостат;
• Изотермическая реакция с использованием Bst полимеразы мало чувствительна к ингибиторам ПНР (можно использовать упрощенную процедуру пробоподготовки);
• Достаточно высокая специфичность реакции, так как праймеры подбираются сразу к нескольким участкам внутри определенной последовательности;
• Детекцию результатов реакции можно проводить как с помощью оптического детектора, так и на глаз по цветной реакции с использованием цветной шкалы.
Из полученных экспериментальных результатов следует, что заявленный набор олигонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации генетического материала вируса лихорадки Рифт-Валли методом RT-LAMP в реальном времени соответствует критерию «изобретательский уровень».
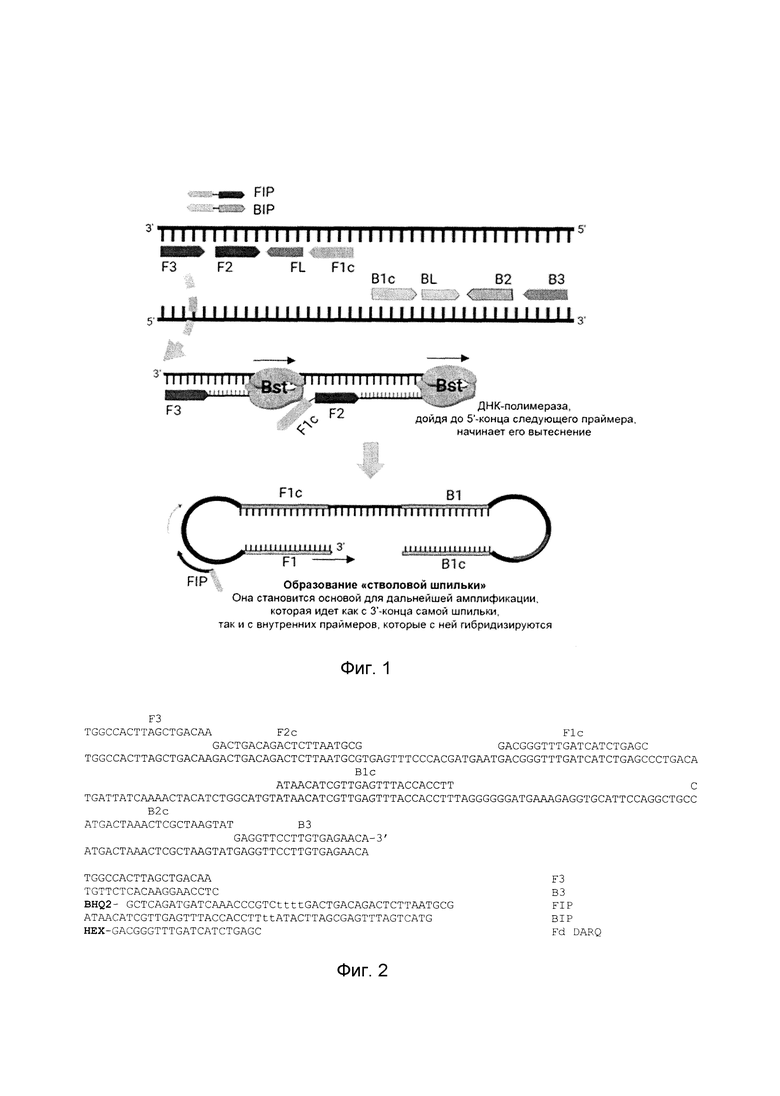
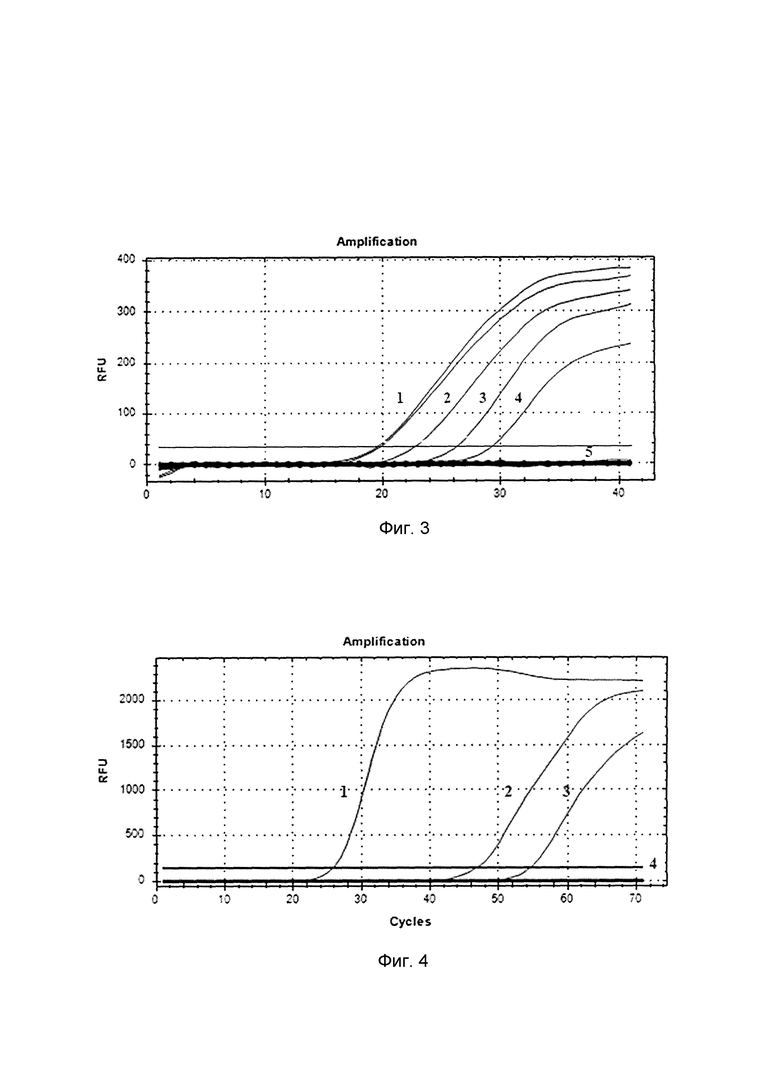
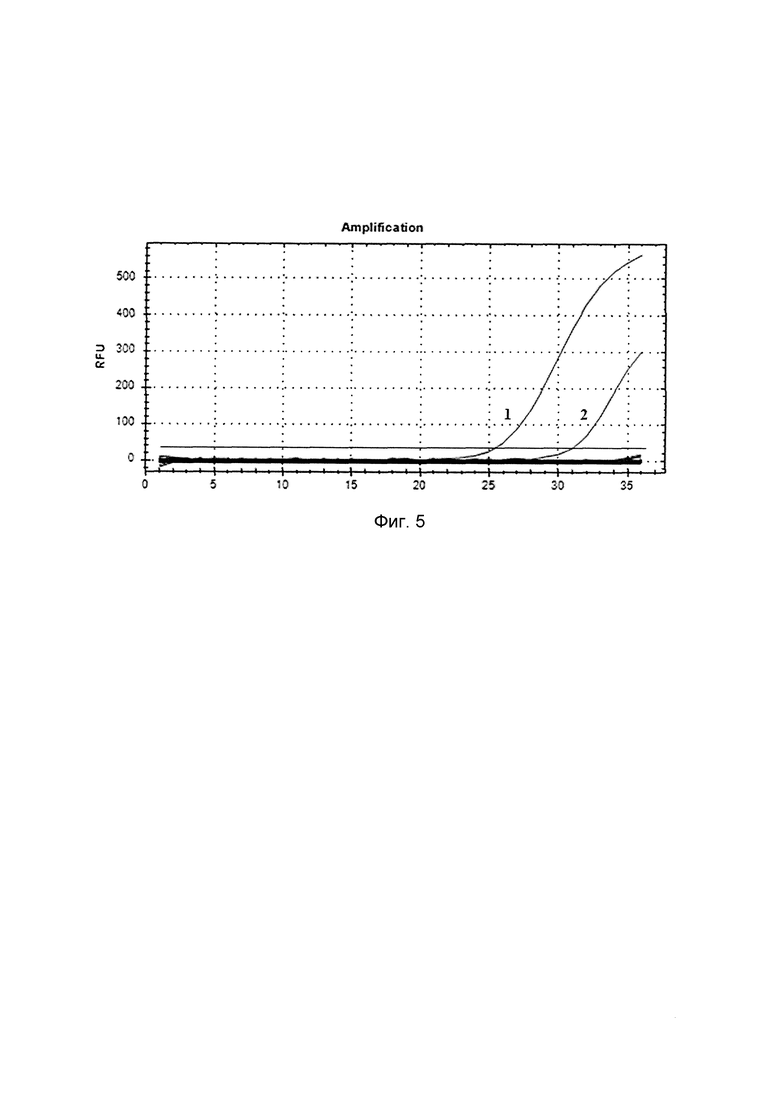
Изобретение иллюстрируется следующими графическими материалами. На фиг. 1 представлена схема прохождения LAMP с флюоресцентной детекцией (New England Biolabs). На фиг. 2 представлены дизайн праймеров для LAMP на мишень L ген (596-783 п. н.) - таргетный район РНК вируса лихорадки Рифт-Валли. На фиг. 3 представлена оценка чувствительности набора олигонуклеотидных праймеров и зонда в режиме реального времени по каналу HEX: в качестве образцов использовали 10-кратные разведения положительного контрольного образца (ПКО), представляющий собой рекомбинантную плазмиду pCR-VFRV содержащую встройку вирус-специфического фрагмента (L ген ВЛРВ, 596-783 п. н.). На фиг. 4 приведена оценка работы набора олигонуклеотидных праймеров и зонда в режиме реального времени по каналу FAM: образцами служили суспензии культур клеток Vero, инфицированные вирусом лихорадки Рифт-Валли (образцы 1-3), выделенные с добавлением внутреннего контрольного образца (ВКО) - очищенный раствор фага MS2. На фиг. 5 изображены результаты проверки аналитической специфичности набора праймеров в режиме реального времени по каналу FAM: использовали ПКО и РНК ВЛРВ Ниже представлены последовательности праймеров и зондов комплементарные нуклеотидной последовательности ВЛРВ (праймеры F3, В3, FIP, BIP и зонд Fd DARQ) и фага MS2 (праймеры F1, R1 и зонд Z1).
Метод LAMP был разработан для детекции таргентых нуклеиновых кислот без использования термоциклирования, то есть при постоянной температуре. LAMP основан на использовании ДНК-полимеразы со смещением цепи (например BST-полимераза и ее модификации) и специально разработанных наборов праймеров (4-6 праймеров), нацеленных на несколько областей целевого генома (4-8 областей), что позволяет накапливать большие количества амплифицированной ДНК/кДНК с высокой специфичностью.
Пример 1. Принцип работы LAMP с полученным набором олигонуклеотидных праймеров и зондов. На Фиг. 1 представлена общая схема проведения эксперимента при реализации петлевой изотермической амплификации.
Постановка реакции для выявления генетического материала ВЛРВ предполагает одновременное использование двух пар праймеров: внешних (прямой «forward» F3 и обратный «backward» В3) и внутренних («forward inner primer» - FIP и «backward inner primer» - BIP). Также для повышения чувствительности метода, улучшения кинетики протекающей реакции и регистрации флуоресцентного сигнала был подобран зонд (Fd DARQ).
Амплификация начинается с отжига всех специфических фрагментов ВЛРВ на кДНК-матрице с последующей их элонгацией. При этом используемая BST-полимераза с цепь-вытесняющей активностью при взаимодействии с 5'-концом последующего праймера начинает его вытеснение вместе с вновь синтезированной цепью. В результате освобождается одноцепочечная ДНК, на которой гибридизован праймер FIP, и за счет комплементарности свободного фрагмента F1c с участком F1 матрицы формируется петля. Образование петли с участием праймеров BIP и В3 происходит и на 3'-конце одноцепочечного фрагмента. В результате формируется гантелеобразная структура («стволовая шпилька»), которая становится основой для дальнейшей амплификации, идущей как с 3'-конца шпильки, так и с гибридизовавшихся с ней праймеров (рисунок). Продуктом LAMP является как сама «стволовая шпилька», так и конкатемеры амплифицируемой последовательности. Показано, что в результате реакции менее чем за час может быть сгенерировано до 109 копий целевого фрагмента ДНК, что обусловливает высокую чувствительность метода. Для идентификации и количественной оценки накопившихся ампликонов, в набор праймеров добавлен зонд, обладающий способность флуоресцентного свечения при связывании с комплементарным участком ДНК с детекцией по каналу HEX.
Амплификация ВКО (очищенного раствора фага MS2) происходит благодаря хеликазной и репликативной активности Bst-полимеразы, двух праймеров (F1 и R1), а детекция наличия ампликонов фрагмента нуклеотидной последовательности генома фага MS2 осуществляется благодаря наличию олигонуклеотидного зонда, обладающего способностью флуоресценции по каналу FAM.
LAMP реакции могут быть проведены при помощи термостата или водной бани, а детекция результатов, из-за большого количества копий, может быть проведена визуально по хромогенной реакции, без инструментального измерениями и тем самым с высокопропускной способностью.
С использованием положительных контрольных образцов (ПКО, ВКО, кДНК вируса лихорадки Рифт-Валли) нами была подобрана оптимальная температура отжига диагностических праймеров, была установлена аналитическая чувствительность праймеров/зонда для выявления РНК вируса лихорадки Рифт-Валли и была проведена оценка клинической и диагностической чувствительности.
Заявленные диагностические праймеры обладали 100%-ной специфичностью на панели отрицательных контрольных образцов и 100%-ной специфичностью на панели положительных контрольных образцов, состоящей из образцов, содержащих генетический материал вируса лихорадки Рифт-Валли. Экспериментальным путем было показано, что выбранные праймеры и флюоресцентно меченый зонд не имеют гомологии с человеческой ДНК и не взаимодействуют с геномной ДНК человека, летучих мышей и других животных. С использованием положительного контроля определена аналитическая чувствительность заявляемой пары диагностических праймеров, которая составила не менее 50 геном-эквивалентов на реакцию.
Пример 2. Материалы и методы.
В работе были использованы следующие реактивы: диметилсульфоксид (ДМСО), маркеры молекулярных масс нуклеиновых кислот 100+ bp DNA Ladder (Evrogen, Россия), 3,3-диаминобензидин тетрагидрохлорид, натриевая соль MES (М3058), ("Sigma", США); этилендиаминтетрауксусная кислота, твин-20, натрия азид, ("Serva", США); набор для определения концентрации нуклеиновых кислот "Bio-Rad dsDNA Assay Kit" ("BioRad", США).
Для получения ПКО использовали плазмидную ДНК pCR2.1-TOPO -3931 п. н. и клетки Е. coli штамма ТОРЮ («Thermo Fisher Scientific», США). Используемые приборы и оборудование:
LAMPIX8 (Россия) портативный амплификатор - используется для поддержания температурного режима изотермической ПЦР и обнаружения по флюоресценции (FAM) в реальном времени продуктов ПЦР;
Амплификатор OpusCFX96 (Bio-Rad): программируемый, диапазон температур до 99°С (±0,1); градиент температур до 20°С; термостатируемая крышка; термоблок 96×0,2 мл; цветная сенсорная панель; USB.
Microspin FV-2400 (BioSan, Латвия): вортекс-центрифуга 2400 об/мин. Ротор на 12×0,5 мл. и 12×0,2 мл.
Бокс микробиологической безопасности БМБ-II-"Ламинар-С по ТУ 9452-010-51495026-2011, в исполнении БМБ-II-"Ламинар-С.»-1.2. (Lamsystems, Россия).
Дозаторы механические (Eppendorf, Германия): объемом 1000-200 мкл, 200-20 мкл, 100-10 мкл, 10-0,1 мкл.
Культивирование вирусов.
Культивирование вируса проводили в стационарном режиме на монослое клеток 293 в культуральных флаконах объемом 1 л. Множественность заражения монослоя составляла 0,1 ЛД50 или 0,01 ЛД50 на клетку в зависимости от задачи исследования. Адсорбцию вируса проводили в течение 40 мин. при температуре 37°С. После этого монослой заливали питательной средой Игла MEM с двойным набором ингредиентов, в которую добавляли 0,06% глутамина и 0,05%, 0,4% или 2% человеческого сывороточного альбумина (в зависимости от условий эксперимента для получения вакцинного препарата) или 2% эмбриональную сыворотку КРС (для выделения анализа белков вируса) и антибиотиков (бензилпенициллина - 100 ед/мл, стрептомицина -100 мкг/мл). Объем питательной среды составлял 10-15% емкости культурального сосуда. Затем клетки инкубировали при температуре 37°С до появления ЦПД. КВЖ использовали для выделения РНК вируса.
Вирусные штаммы.
В работе были использованы вирусы и вируссодержащий материал из коллекции микроорганизмов ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора.
Культуры клеток и среды.
Клетки 293 получены из коллекции культур клеток ФБУН ГНЦ ВБ «Вектор». В работе использовали питательные среды: среда Игла MEM с двойным набором аминокислот и витаминов производства ФБУН ГНЦ ВБ «Вектор» Роспотребнадзора.
Дизайн праймеров.
Дизайн праймеров для изотермической амплификации ВЛРВ и фага MS2 осуществляли при помощи программы NEB LAMP Primer Design Tool (version 1.0.2), используя известные нуклеотидные последовательности геномов ВЛРВ и фага MS2, доступные в базе данных GenBank (http://www.ncbi.nlm.nih.gov/genbank). На фиг. 2 подробно представлены области к которым были подобраны специфичные к геному ВЛРВ праймеры. Множественное выравнивание нуклеотидных последовательностей проводили при помощи приложения AlignX программного пакета Vector NTI 10 (Informax, США). При конструировании олигонуклеотидных праймеров учитывались следующие требования: отсутствие протяженных участков повторяющихся нуклеотидов, отсутствие комплементарных последовательностей длиной более трех оснований внутри олигонуклеотидов, высокое содержание GC-оснований (не менее 50%). Анализ возможности образования олигонуклеотидами вторичных структур, понижающих эффективность ПЦР, проверяли также с помощью программы VectorNTI v.8 (InforMax, США). В работе было использовано лицензионное программное обеспечение Microsoft Office 2010, Vector NTI 11.5, Lasergene 7 и MEGA 7.
Для проверки работоспособности сконструированных праймеров были получены контрольные образцы:
• Для контроля чувствительности и специфичности - ПКО, представляющая собой рекомбинантную плазмидную ДНК pCR-VFRV, содержащая встройку вирус-специфического фрагмента (L ген ВЛРВ, 596-783 п.н.);
• Для контроля выделения нуклеиновых кислот - ВКО, представляющий собой очищенный раствор фага MS2.
Выделение вирусной РНК.
Для выделения РНК/ДНК было использовано решение от Nimagen (Голандия), с использованием полимерных материалов, а также набор «Рибо-ПРЕП» (ЦНИИ Эпидемиологии, Россия), согласно инструкции по применению.
Оптимизация LAMP.
Оптимизацию проводили с использованием амплификатора OpusCFX96 («BioRad», США) и LAMPIX 8 (Россия). Электрофоретическую детекцию продуктов амплификации в агарозном геле проводили с использованием электрофоретических камер SE-20 (Хеликон, Россия), источников питания PowerPac Basic (Bio-Rad), системы видео документирования Molecular Imager Gel Doc XR System («BioRad», США) с прилагаемым ПО.
Оценка специфичности продуктов ПЦР.
После проведения ПЦР с помощью праймеров F3 и В3 полученные фрагменты ДНК очищали с использованием набора QIAEX II Gel Extraction Kit («Qiagen», США) и определяли нуклеотидные последовательности на автоматическом секвенаторе ABI Genetic Analyzer 3500xl (ABI, США). Нуклеотидные последовательности анализировались с использованием базы данных NCBI MegaBLAST.
Пример 3. Экспериментальная постановка RT-LAMP-PCR.
Для идентификации РНК вируса лихорадки Рифт-Валли и фага MS2 (ВКО) формат теста был выбран как OneStep RT-LAMP-ПЦР - метод изотермической ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени и включал следующие этапы:
1. Экстракция РНК из образцов биологического материала.
Для выделения РНК/ДНК было использовано решение от Nimagen, с использованием полимерных материалов, что позволило в два раза ускорить процесс пробоподготовки, с 40 мин, до 20 мин. Реагент GenePurgeDirect® (Nimagen) состоит из полимерных материалов, которые обеспечивают быстрое извлечение нуклеиновых кислот из клеток в форме, которую можно использовать для ПЦР. Ингибиторы ПНР, высвобождающие во время лизиса и другие вещества, которые могут принять участие в реакции ПЦР, сегрегируются с полимерным материалом, и супернатант, содержащий нуклеиновые кислоты, может быть использован напрямую для ПЦР. Протокол выделения РНК занимает до 20 минут.
Для проведения лабораторных испытаний при выделении РНК/ДНК, были использованы зарегистрированные на территории Российской Федерации наборы реагентов «АмплиПрайм РИБО-преп». Реакцию обратной транскрипции и получение кДНК на матрице РНК и амплификацию целевых фрагментов кДНК при помощи специфичных к этим участкам пар праймеров проводили при помощи фермента Bst (Инова Плюс, Россия) в сочетании с обратной транскриптазой M-MuLV (Инова Плюс, Россия) в одной реакции.
2. Детекция результатов ПЦР.
Результаты непосредственно в ходе изотермической ПЦР регистрировали с использованием автоматических регистрирующих приборов: LAMPIX 8 (Россия) и OpusCFX96 (BioRad).
3. Получение стандартных положительных контрольных образцов (СОП), внутреннего контрольного образца (ВКО) и положительных контрольных образцов вируса лихорадки Рифт-Валли (ПКО).
ПКО был получены методом молекулярной трансформации компетентных бактериальных клеток Escherichia coli бактериальными плазмидами pCR2.1-TOPO («Thermo Fisher Scientific», США), включающими вставки ДНК, комплементарные участкам гена L вируса лихорадки Рифт-Валли, с последующим выделением плазмидной ДНК из бактериальных клеток. При этом праймеры подбирались с наименьшей гомологией к ДНК человека и животных, а также к другим вирусам.
Для формирования панели положительных и отрицательных контрольных образцов были использованы инфицированные изолятом вируса лихорадки Рифт-Валли и гетерологичных вирусных штаммов культуры клеток из коллекции микроорганизмов ФБУН ГНЦ ВБ Вектор Роспотребнадзора: вируса денге (DEN1), вируса Чикунгунья (RVFV), вируса желтой лихорадки (YEV), вируса Семлики Форест (SFV), вируса Японского энцефалита (JEV).
Контроль выделения вирусной РНК осуществляли по внутреннему контрольному образцу (ВКО). В качестве ВКО использовали фаг MS2, с добавлением специфических праймеров и зонда.
Постановка RT-LAMP-PCR. Реакцию RT-LAMP-PCR проводили в конечном объеме 15 мкл. Реагент 1 (5,5 мкл): 1,5 мкл PCR-mix ×10, 1.0 мкл 10 мМ dNTP, 0,7 мкл. MgSO4 100 mM, 1 мкл. F3/B3 (10 мкМ), 1 мкл. FIP/BIP (40 мкМ), 1 мкл. зонд (20 мкМ), в которую добавляют Реагент 2 (1,0 мкл): 0,5 мкл Ultra Fast Bst DNA полимераза (Инова Плюс), 4 единицы на реакцию и 0,5 мкл InovaScript обратная транскриптаза, 20 ед. на реакцию. В пробирку с 6,5 мкл реакционной смеси вносят от 1 до 8,5 мкл очищенной РНК и Н2О до конечного объема 15 мкл,.
В качестве положительного контрольного образца используют раствор (5 мкл) рекомбинантной плазмиды содержащей фрагмент кДНК вируса лихорадки Рифт-Валли (ВКО ВЛРВ).
В качестве отрицательного контрольного образца в реакционную смесь вместо положительного контрольного образца добавляли 5 мкл ТЕ-буфера (ОКО).
В качестве ВКО использовали раствор очищенного фага MS2 с добавлением вирус-специфических праймеров и зонда.
В качестве гетерологичных контрольных образцов использовали РНК вируса денге (DEN1), вируса Чикунгунья (RVFV), вируса желтой лихорадки (YEV), вируса Семлики Форест (SFV), вируса Японского энцефалита (JEV) и отрицательный контрольный образец (ОКО) - очищенную воду.
Визуализацию продуктов LAMP ВЛРВ проводили путем измерения интенсивности флюоресценции по каналу HEX, ВКО по каналу FAM. Полученные продукты LAMP-ПЦР дополнительно визуализировались путем электрофореза в 2% агарозном геле, окрашенном бромистым этидием.
Для определения аналитической чувствительности «праймеры-зонд», из концентрированного раствора положительного контрольного образца (ПКО) (107 копий/мл) были приготовлены последовательные 10-кратные разведения. Определение концентрации ДНК в разведениях, при исследовании чувствительности набора праймеров, осуществляли при помощи коммерческого набора «Quant-iT dsDNA, HS» («Invitrogen», США) и флуориметра QUBIT («Invitrogen», США). Для контроля повторяемости использовали образец СОП ПКО ВЛРВ разведенный до концентрации 1,0×107 копий/мл (5 образцов). Процедура проводится на одном наборе реагентов. Коэффициент вариации для повторяемости рассчитывается по формуле: CVp,%=Ct (станд отклон) / Ct (ср.знач.) × 100% для 5 проб ПКО ВЛРВ и не должен превышать 3%.
Для контроля воспроизводимости используют образец ПКО ВЛРВ с концентрацией 1,0×107 копий/мл (10 образцов). Процедура проводится с двумя разными наборами одной серии. Коэффициент вариации для воспроизводимости рассчитывается по формуле: CVp,%=Ct (станд отклон) / Ct (ср.знач.) × 100 % для 10 проб СОП ПКО ВЛРВ и не должен превышать 6%.
Минимальное количество ДНК-матриц, детектируемое с применением подобранных праймеров и зондов, после оптимизации условий проведения реакции, выраженное в ГЭ (геномных эквивалентах) в 24 мкл реакционной смеси, составило 50 ГЭ.
На фиг. 3 представлена флюоресценция продуктов LAMP ПЦР ВЛРВ в режиме реального времени. Оценка чувствительности с 10-кратными разведениями (кривые 1-4) положительного контрольного образца (кривая 1 (в двух повторах) ПКО, 107 копий/мл) - рекомбинантной плазмиды pCR-VFRV, содержащей встройку фрагмента L гена ВЛРВ (596-783 п.н.). Отрицательный контрольный образец (ОКО) - кривая 5.
На фиг. 4 представлена оценка работы набора олигонуклеотидных праймеров и зонда в режиме реального времени по каналу FAM: образцами служили суспензии культур клеток Vero, инфицированные вирусом лихорадки Рифт-Валли (образцы 1-3), выделенные с добавлением внутреннего контрольного образца (ВКО) - очищенный раствор фага MS2. В качестве отрицательного контроля использовали ОКО (образец 4). 1 цикл (по оси X) LAMP равен 40 секундам.
Специфичность праймеров и флюоресцентно меченых зондов проверяли постановкой LAMP в режиме реального времени с использованием в качестве детектируемых образцов культуры клеток Vero инфицированных вирусом лихорадки Рифт-Валли. Вирус лихорадки Рифт-Валли был получен из Государственной коллекции микроорганизмов ФБУН ГНЦ ВБ «Вектор».
Контроль специфичности осуществляли проведением LAMP с флуоресцентной детекцией, где в качестве исследуемых образцов использовали пробы, содержащие генетический материал ВЛРВ и пробы, содержащие генетический материал: РНК вируса денге (DEN1), вируса Чикунгунья (RVFV), вируса желтой лихорадки (YEV), вируса Семлики Форест (SFV) и вируса Японского энцефалита (JEV). Не специфических перекрестных реакций отмечено не было.
На фиг. 5 представлена флюоресценция продуктов LAMP в режиме реального времени. Оценка аналитической специфичности набора праймеров и реагентов для LAMP ВЛРВ. Кривые: 1 - ПКО; 2 - ВЛРВ. В качестве гетерологичных образцов в LAMP были использованы РНК вирусов денге (DEN1), Чикунгунья (RVFV), желтой лихорадки (YEV), Семлики Форест (SFV) и вируса Японского энцефалита (JEV). ОКО - вода.
Из представленных данных на фиг. 5 следует, что предлагаемые праймеры не дают положительных результатов с образцами, содержащими гетерологичные вирусы: вирусы денге (DEN1), Чикунгунья (RVFV), желтой лихорадки (YEV), Семлики Форест (SFV) и вируса Японского энцефалита (JEV) и ДНК человека.
Таким образом, был разработан набор праймеров, предназначенный для выявления ВЛРВ и фага MS2 (ВКО) методом LAMP в режиме реального времени.
Диагностическая роль набора праймеров заключается в возможности их использования для ранней диагностики ВЛРВ, как ускоренного предварительного теста при выполнении культурального и биологического методов исследования и идентификации подозрительных культур для эпидемиологического мониторинга.
Референтный метод (исходный, эталонный).
Для выявления РНК вируса лихорадки Рифт-Валли методом LAMP, в качестве референтного метода использовали Набор реагентов для выявления РНК вируса лихорадки долины Рифт методом ОТ-ПЦР с гибридизационно-флуоресцентной детекцией "Вектор-ОТ-ПЦРрв-ВЛДР" по ТУ 9398-056-05664012-2016, Регистрационное удостоверение на медицинское изделие РЗН 2018/6931.
Подлинность используемых штаммов ВЛРВ из Государственной коллекции микроорганизмов 1-4 групп патогенности ФБУН ГНЦ ВБ «Вектор» была подтверждена секвенированием.
Обеззараживание проб.
Обеззараживание проб проводили в соответствии с требованиями СП 1.3.3118-13 «Безопасность работы с микроорганизмами I-II групп патогенности (опасности)» и МУ 1.3.2569 -09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности».
Пробы, содержащие гетерологичные вирусы в объеме 1 мл переносили в микроцентрифужные пробирки вместимостью 1,5 мл, добавляли натрия мертиолят до концентрации 1:10000 (0,01%) и прогревали при (56±1)°С в течение 30 минут. Затем в отдельные микроцентрифужные пробирки вместимостью 1,5 мл переносили по 100 мкл обработанных мертиолятом натрия вирусосодержащих проб. Во все пробирки, добавляли лизирующий буфер на основе 6М гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения РНК (Литех, Россия, или Евроген, Россия), и инкубировали 15 минут при температуре (65±1)°С. После выполнения данного этапа материал считался обеззараженным.
Процедура измерения и оценки результатов анализа.
LAMP в режиме реального времени и регистрацию результатов проводили в приборе OpusCFX 96 (BioRad, США) по каналу Fam. Результаты интерпретируются на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию (или отсутствию) результатов).
Статистическая обработка результатов проводили в соответствии с «Методическими рекомендациями по порядку проведения экспертизы качества, эффективности и безопасности медицинских изделий», утв. ФГБУ «ГДМиКЭЭ» и ФГБУ «ВНИИМТ» 14.11.2013 г. Статистическую достоверность полученных результатов испытаний оценивали в зависимости от числа параллельных опытов при доверительной вероятности 90%, используя формулу биноминального распределения Бернулли.
Чувствительность изделия
Набор праймеров для выявления РНК вируса лихорадки Рифт-Валли методом LAMP с гибридизационно-флуоресцентной детекцией, при исследовании проб, содержащих генетический материал ВЛРВ в концентрации не менее 1×104 копий/мл, составляет 100%. Внутрипостановочная, межпостановочная и межсерийная воспроизводимость для всех положительных образцов - 100%.
Специфичность
Аналитическая специфичность.
Аналитическая специфичность оценивается по числу совпадений результатов с референс тест-системой. Аналитическая специфичность Набора праймеров для выявления РНК вируса лихорадки Рифт-Валли методом LAMP с гибридизационно-флуоресцентной детекцией, при исследовании 100 проб, содержащих генетический материал ВЛРВ в концентрации не менее 1×104 копий/мл, и отрицательные контрольные образцы составляет 100%.
Диагностическая специфичность
Диагностическая специфичность - число отрицательных реакций при тестировании материала образцов от больных контрольной группы (лица с подтвержденным отсутствием инфекции). Диагностическая специфичность с использованием заявляемых праймеров и зонда при исследовании 100 отрицательных образцов, содержащих нуклеиновую кислоту (НК) гетерологичных микроорганизмов (вирусы денге (DEN1), Чикунгунья (RVFV), желтой лихорадки (YEV), Семлики Форест (SFV) и вируса Японского энцефалита (JEV)), 20 отрицательных образцов, содержащих ДНК человека (сыворотка крови здоровых людей), 20 образцов мокроты и 20 образцов смывов из полости носоглотки, для которых клинически подтвержден вирус парагриппа 1 типа, 4 клинических образца мазков из ротоглотки, для которых клинически подтвержден вирус гриппа типа А, 2 клинических образца мазков из ротоглотки, для которых клинически подтвержден аденовирус 5 серотипа, 2 клинических образца мазков из ротоглотки, для которых клинически подтвержден респираторно-синцитиальный вирус. Отрицательный результат в LAMP получен для 100 образцов. Диагностическая специфичность - не менее 98% с доверительной вероятностью 90%.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Набор праймеров и
зондов вирус ЛРВ .xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-09-26">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2023-09-26</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>123456</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>123456789</ApplicationNumberText>
<FilingDate>2023-09-25</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Набор олигонуклеотидных праймеров
и зондов для идентификации РНК вируса лихорадки Рифт-Валли методом
изотермической ПЦР с гибридизационно-флуоресцентной детекцией в
режиме реального времени</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggccacttagctgacaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgttctcacaaggaacctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctcagatgatcaaacccgtcttttgactgacagactcttaatgcg</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>49</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataacatcgttgagtttaccaccttttatacttagcgagtttagtcatg
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgggtttgatcatctgagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcatggcttctaactttactcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attagtagatgccggagtttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtcgccccaagcaacttcgcta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |
| Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2834908C1 |
| Способ выявления вируса кори методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822430C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ РНК ВИРУСА ЛИХОРАДКИ ДОЛИНЫ РИФТ МЕТОДОМ ОТ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2014 |
|
RU2552795C1 |
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822161C1 |
| Способ выявления вируса клещевого энцефалита методом ОТ-ПЦР в реальном времени | 2019 |
|
RU2744187C1 |
| Способ выявления РНК модифицированного вакцинного полиовируса типа 2 (nOPV2) методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2795703C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
Изобретение относится к набору праймеров и зондов для идентификации РНК вируса лихорадки Рифт-Валли методом изотермической ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени в клинических образцах от человека и животных и культуральных вируссодержащих жидкостях с целью постановки диагноза, коррекции лечения, эпидемиологического расследования, а также для решения научно-исследовательских задач по мониторингу и изучению свойств вируса лихорадки Рифт-Валли и может быть использовано в медицине, биотехнологии и эпидемиологии. Техническим результатом изобретения является создание отечественного набора реагентов для выявления РНК вируса лихорадки Рифт-Валли методом изотермической амплификации, который обеспечивал бы при высокой чувствительности высокую специфичность и детекцию только РНК вируса лихорадки Рифт-Валли в исследованных образцах. Набор праймеров и зондов для выявления РНК вируса лихорадки Рифт-Валли методом RT-LAMP с гибридизационно-флуоресцентной детекцией в режиме реального времени содержит прямой внешний праймер F3 и обратный внешний праймер В3, прямой внутренний праймер FIP и обратный внутренний праймер BIP к гену L РНК вируса лихорадки Рифт-Валли и гасящий зонд Fd DARQ для обнаружения амплификации, а также прямой F1, обратный R1 праймеры и зонд Z1 с флуоресцентной меткой к фагу MS2, имеющие последовательности SEQ ID NO: 1-8 соответственно. 6 ил.
Набор олигонуклеотидных праймеров и зондов для выявления РНК вируса лихорадки Рифт-Валли методом RT-LAMP с гибридизационно-флуоресцентной детекцией в режиме реального времени, содержащий прямой внешний праймер F3 и обратный внешний праймер В3, прямой внутренний праймер FIP и обратный внутренний праймер BIP к гену L РНК вируса лихорадки Рифт-Валли и гасящий зонд Fd DARQ для обнаружения амплификации, а также прямой F1, обратный R1 праймеры и зонд Z1 с флуоресцентной меткой к фагу MS2, имеющие последовательности SEQ ID NO: 1-8 соответственно.
| CN105483293 А, 13.04.2016 | |||
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНО-МЕЧЕНОГО ЗОНДА ДЛЯ ИДЕНТИФИКАЦИИ РНК ВИРУСА ЛИХОРАДКИ ДОЛИНЫ РИФТ МЕТОДОМ ОТ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2014 |
|
RU2552795C1 |
| Francis Wekesa et al | |||
| Development and Validation of Rapid Colorimetric Reverse Transcription Loop-Mediated Isothermal Amplification for Detection of Rift Valley Fever Virus, Advances in Virology;Volume 2023, Article ID 1863980, 14 pages, 30 Jan | |||
Авторы
Даты
2024-02-12—Публикация
2023-10-18—Подача