Область техники
Изобретение относится к области медицины и молекулярной биологии. Заявлена тест-система для выявления в биоматериале ДНК возбудителей микобактериозов, входящих в Mycobacterium avium complex без дифференциации до вида (M. arosiense, M. avium, M. bouchedurhonense, M. chimaera, M. colombiense, M. indicus pranii, M. intracellulare, M. lepraemurium, M. mantenii, M. marseillense, M. paraintracellulare, M. timonense, M. vulneris, M. yongonense) методом ПЦР в реальном времени со специфично-подобранными праймерами и флуоресцентно-меченным зондом, используя в качестве мишени 16S-23S межгенный спейсер. Изобретение может быть использовано для создания медицинских изделий для диагностики in vitro, с целью выявления возбудителей микобактериозов службами клинико-лабораторной диагностики в лечебно-профилактических учреждениях, а также с целью осуществления эпидемиологического надзора населения за распространением возбудителей микобактериозов.
Уровень техники
Заболеваемость микобактериозом в мире неуклонно растёт. В России не ведется четкой статистики по данному заболеванию, тем не менее, по данным отдельных региональных исследований, распространенность микобактериозов составляет 1,5-4,3 на 100 000 населения (Белобородова Е. Н. и др. Клинико-эпидемиологическая характеристика микобактериоза в двух субъектах Российской Федерации //Инфекционные болезни. - 2020. - Т. 18. - №. 1. - С. 35-42.). Группами риска по развитию заболевания являются иммунокомпрометированные пациенты: лица с синдромом приобретенного иммунодефицита и другими иммунодефицитами; после трансплантации внутренних органов; пациенты с термическими травмами; больные пневмокониозами; больные хронической обструктивной болезнью легких; больные бронхиальной астмой; больные муковисцидозом; больные с бронхоэктазиями; больные саркоидозом; больные с легочными фиброзами различной природы; больные с онкологическими заболеваниями, в т.ч. гемобластозами; лица, длительно получающие глюкокортикостероидную или иммуносупрессивную терапию; лица пожилого возраста. Кроме этого, после перенесенного заболевания COVID-19 у многих пациентов формируется иммунодефицитное состояние и в легких формируются выраженные остаточные изменения в виде фиброза, что создает предпосылки к развитию микобактериозов, возбудитель которых широко распространен в природе (Yang F., Liu N., Hu J.Y. et al. Pulmonary rehabilitation guidelines in the principle of 4S for patients infected with 2019 novel coronavirus (2019-nCoV) (In Chinese)., Zhon-ghuaJie He He Hu Xi ZaZhi. 2020; 43 (3): 180-182.) Соответственно, с вопросами диагностики микобактериозов сталкиваются врачи самых разных специальностей. Учитывая прогнозируемый рост продолжительности жизни населения в РФ, старение популяции, увеличение доли иммунокомпрометированных пациентов и пандемию COVID-19, следует ожидать роста заболеваемости микобактериозом.
Значимым фактором роста заболеваемости является распространение ВИЧ-инфекции. Около 5-7% пациентов с диагнозом B20.0 (МКБ) страдают микобактериозом. Более чем у 90% таких пациентов выявляются виды, входящие в Mycobacterium avium complex (Савченко М. А. Клинические и эпидемиологические аспекты микобактериоза у пациентов с ВИЧ-инфекцией // ВИЧ-инфекция и иммуносупрессии. - 2019. - Т. 11. - №. 2. - С. 27-33.).
У больных СПИДом, на поздних стадиях иммунодепрессии при количестве CD4 + в крови менее 50 клеток/мкл, частота MAC-инфекции колеблется в пределах 20-40% и является наиболее частой причиной смертности (Nightingale SD1, Byrd LT, Southern PM, Jockusch JD, Cal SX, Wynne BA. Incidence of Mycobacterium avium-intracellulare complex bacteremia in human immunodeficiency virus-positive patients. J Infect Dis. 1992 Jun;165(6):1082-5).
MAC-инфекция часто маскируется под туберкулёз по своим симптомам, но вместе с тем лечение больных туберкулезом и микобактериозом отличается, т.к. виды Mycobacterium avium complex обладают природной резистентностью ко многим противотуберкулезным препаратам. Для правильного установления диагноза и назначения адекватного лечения необходимо проведение быстрого лабораторного исследования в выявлении микобактерий и, в частности, определении их принадлежности к Mycobacterium avium complex.
Диагностика MAC-инфекции осуществляется культивированием образцов от пациентов на плотных и жидких питательных средах и до настоящего времени этот метод остается «золотым стандартом» лабораторной диагностики микобактериальных инфекций.
Однако виды, входящие в Mycobacterium avium complex, относятся к медленнорастущим, соответственно, даже при использовании систем автоматического культивирования на жидких питательных средах (BACTEC MGIT и др.), время роста культуры может варьировать до 42-х дней. Кроме этого, после получения роста микобактерий, требуется дифференциация от Mycobacterium tuberculosis complex и идентификация Mycobacterium avium complex другими методами: биохимическими; времяпролетной масс-спектрометрии (MALDI-ToF MS); молекулярно-генетические (мультилокусное секвенирование, анализ полиморфизма длины рестрикционных фрагментов (ПДРФ), секвенирование гена 16S рРНК); высокопроизводительной жидкостной хроматографии миколовых кислот (HPLC), недостатком которых является длительность проведения исследований, т.к. требуется рост культуры микобактерий (до 2-3-х месяцев), трудоемкость и необходимость оснащения дорогостоящим оборудованием, высокая стоимость исследований. Молекулярные методы диагностики, основанные на методе полимеразной цепной реакции (ПЦР), позволяют сократить сроки диагностики микобактериальных инфекций до нескольких часов. Поэтому разработка и внедрение эффективных и быстрых новых методов лабораторной диагностики микобактериозов является чрезвычайно важной и актуальной.
Известные молекулярные методы и соответствующие тест-системы (комплекты реагентов) для диагностики микобактериозов основаны на определении специфичных фрагментов ДНК в геноме.
Коммерческий набор «GenoType Mycobacterium CM» (Hain Lifescience GmbH) основан на ПЦР с последующей гибридизацией на стрипах с иммобилизованными на них олигонуклеотидными зондами, и дифференцирует наиболее часто встречающиеся виды нетуберкулезных микобактерий, в том числе M. avium и M. intracellulare, входящие в M. avium complex. Тем не менее, этот способ является дорогим и трудоемким, для него невозможна автоматизация процесса, и он обладает низкой чувствительностью. Кроме этого, согласно официальной инструкции к набору, для исследования подходят только культуры микобактерий, что увеличивает сроки получения результатов до 2-3-х месяцев и ограничивает возможности использования данного метода.
В зарубежной литературе обнаруживается ряд патентов, касающихся выявления M. avium complex молекулярными методами. В них используются гибридизация с олигонуклеотидными пробами (US5153119, 1992; US5314801, 1994; US6136529, 2000), изотермическая амплификация по принципу вытеснения цепи (SDA) (US5681705, 1997; US5691143, 1997) и ПЦР (US5667994, 1997; US6465638, 2002).
Известен способ выявления Mycobacterium avium и Mycobacterium intracellulare с помощью ПЦР (US8084212, 2011), однако данным способом невозможно выявить другие виды, входящие в M. avium complex. Кроме этого, предложенная авторами в качестве мишени для M. avium последовательность IS1245 присутствуют не у всех подвидов данного вида (Johansen T. B. et al. Distribution of IS1311 and IS1245 in Mycobacterium avium subspecies revisited //Journal of Clinical Microbiology. - 2005. - Т. 43. - №. 5. - С. 2500-2502).
Из уровня техники также известен набор и способ выявления Mycobacterium avium (US2020181690), включающий праймеры 5′-AGTGGGCAACAATCCAAGAG-3′ и 5′-CCCGACACAACGAGGTTT-3, а в способе предложено использовать ПЦР с последующим построением кривых плавления. Однако для детекции в известном способе используют интеркалирующий краситель SYBR-Green I. Слабым местом интеркалирующих красителей является то, что они неспецифичны. Если во время ПЦР происходит амплификация не того участка ДНК, либо вовсе нескольких разных участков, все равно строится кривая амплификации, которая идентична кривой при амплификации целевых генов. Интеркалирующие красители генерируют флуоресцентный сигнал при связывании с любой двухцепочечной ДНК, независимо от ее нуклеотидной последовательности. То есть, необходимо проводить дополнительные анализы, которые подтверждают наличие специфичных продуктов амплификации. Подобные виды анализов требуют высокого уровня квалификации оператора.
Известен способ идентификации вида микобактерий комплекса MAIS (M. avium, M. intracellulare, M. scrofulaceum) и M. tuberculosis, включающий этапы выделения ДНК, рестрикции, разделения продуктов рестрикции в агарозном геле с последующим анализом профилей рестрикции изучаемых видов, при этом ДНК выделяют прогреванием микобактерий 12 мин при 95°C в ТЕ-буфере рН 8,0, полученные ампликоны обрабатывают рестриктазой Hin6I (объем ампликонов для рестрикции составляет 17 мкл и время рестрикции 1 ч 10 мин (RU2319748, 2008). Проведение анализа состоит из 3-х этапов:
1. Исследуемая последовательность ДНК изучаемого гена клонируется методом ПЦР.
2. Полученные ампликоны подвергаются воздействию рестриктаз, которые подбираются с помощью специальных программ, с учетом того, что фрагменты, на которые разрезаются полученные ампликоны, должны выявляться на агарозном геле.
3. Анализ полиморфизма профилей после разделения продуктов рестрикции в агарозном геле.
Однако в данном способе используется метод ПЦР-ПДРФ (полимеразной цепной реакции - полиморфизма длин рестрикционных фрагментов), который является более трудо- и времязатратным, менее чувствительным, а также контаминационно-опасным, чем ПЦР в реальном времени.
Известны олигонуклеотидные праймеры, комплементарные специфичной для Mycobacterium avium области mig-гена, использующиеся для выявления ДНК Mycobacterium avium, имеющие следующий нуклеотидный состав: 5'-CGT САА AAG CGA ACT GCA -3' и 5'-ТАА ТТС GTT GCC CGA СТС -3' и способ выявления ДНК Mycobacterium avium с помощью вышеуказанных олигонуклеотидных праймеров методом полимеразной цепной реакции (ПНР), включающей выделение ДНК, проведение амплификации ДНК на олигонуклеотидных праймерах, перенос продукта амплификации на гель с последующим детектированием результатов анализа на трансиллюминаторе, отличающийся тем, что праймеры имеют нуклеотидные последовательности: 5'-CGT САА AAG CGA ACT GCA -3'и 5'-ТАА ТТС GTT GCC CGA СТС -3' в случае положительной реакции синтезируется фрагмент, соответствующий размеру 157 п.н. (RU2554842, 2015). Для проведения электрофореза ДНК требуется специалист, обладающий специальным уровнем подготовки, т.к. после проведения реакции амплификации необходимо открыть пробирки (стрипы, планшеты) с ампликонами и поместить их в лунки агарозного или полиакриламидного геля вместе с красителем. Кроме того, процедура приготовления геля занимает в среднем 20 минут, а сам процесс электрофореза от 20 минут (в зависимости от длины ПЦР-продукта). Это увеличивает время проведения реакции минимум на 40 минут. Следовательно, использование электрофоретической детекции после проведения ПЦР удлиняет время- и трудозатраты, снижает чувствительность, является источником контаминации в лаборатории, обладает меньшей чувствительностью и не выявляет другие виды, входящие в M. avium complex.
Таким образом, существующие в настоящее время способы выявления M. avium complex включают в себя ПЦР с гибридизацией на стрипах, ПЦР-ПДРФ, ПЦР с электрофоретической детекцией. Все эти способы требуют открывания пробирок после этапа амплификации, что повышает риск внутрилабораторной контаминации и как следствие, появление ложноположительных результатов.
Техническая проблема, решаемая посредством заявляемого изобретения, заключается в необходимости преодоления недостатков, присущих аналогам и прототипу за счет создания тест-системы для выявления в биоматериале ДНК возбудителей микобактериозов, входящих в Mycobacterium avium complex без дифференциации до вида (M. arosiense, M. avium, M. bouchedurhonense, M. chimaera, M. colombiense, M. indicus pranii, M. intracellulare, M. lepraemurium, M. mantenii, M. marseillense, M. paraintracellulare, M. timonense, M. vulneris, M. yongonense) методом ПЦР в реальном времени со специфично-подобранными праймерами и флуоресцентно-меченным зондом, используя в качестве мишени 16S-23S межгенный спейсер.
Раскрытие изобретения
Техническим результатом предлагаемого изобретения является повышение качества лабораторной диагностики микобактериозов, т.е. разработка способа экспресс-выявления ДНК возбудителей микобактериозов (не более 121 мин.), характеризующийся уменьшением вероятности внутрилабораторной контаминации и, соответственно, вероятности получения ложноположительных результатов.
Указанный технический результат достигается комбинацией праймеров, специфичных к последовательности между генами 16S- и 23S-рРНК, и зонда для обнаружения видов Mycobacterium, где
(1) праймеры представляют собой олигонуклеотиды, содержащие нуклеотидную последовательность SEQ ID NO:1 - SEQ ID NO:4, и
(2) зонд с нуклеотидной последовательностью SEQ ID NO:5.
При этом 5'-конец зонда помечен репортерным флуоресцентным красителем ROX, а 3'-конец зонда помечен красителем-гасителем BHQ-2.
Технический результат также достигается набором реагентов для обнаружения видов Mycobacterium, характеризующийся тем, что включает вышеописанную комбинацию праймеров и зонда и реакционная смесь следующего состава: 5,1 мМ MgCl2; по 0,39 мМ каждого нуклеозидтрифосфата, выбранного из ряда - dATP, dGTP, dCTP и dTTP; 0,05 ед/мкл Taq-ДНК-полимеразы; 1х ПЦР-буфер. При этом используют ПЦР-буфер следующего состава: 0,1M Трис-HCl с pH = 8,6; 0,5М КCl; 1% Tween20.
Также технический результат достигается способом обнаружения видов Mycobacterium, заключающийся в проведении ПЦР в реальном времени с использованием вышеописанной комбинации праймеров и зонда или набора.
Краткое описание чертежей
Изобретение поясняется следующими чертежами.
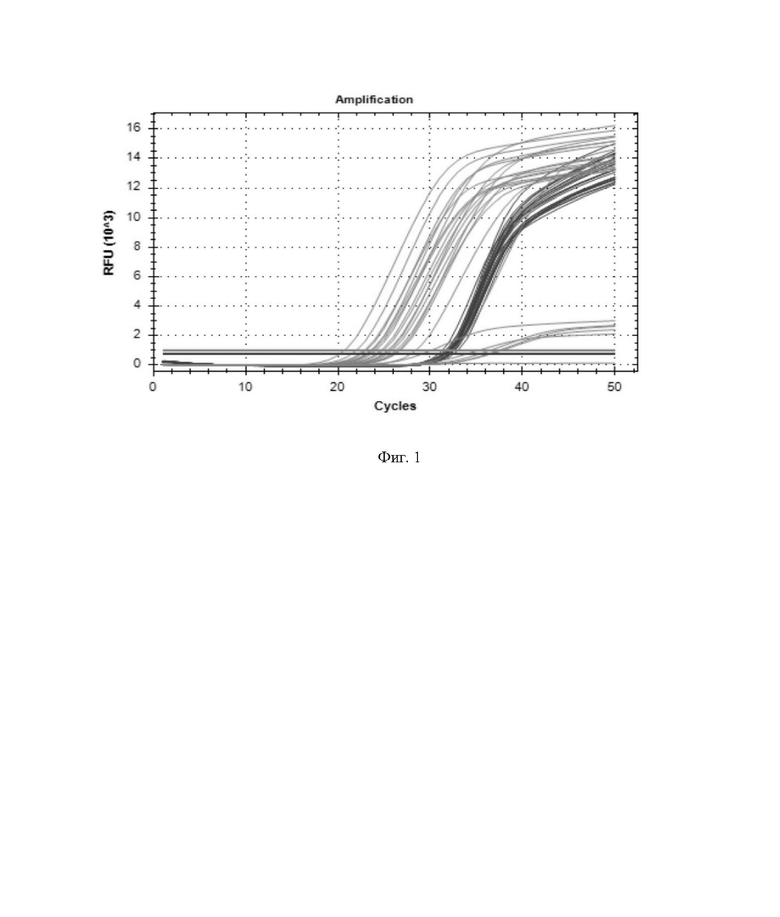
Фиг. 1. Кривые плавления ДНК исследуемых образцов, полученные методом ПЦР в реальном времени, с помощью разработанной тест-системы. На одном графике представлены оба канала, FAM(ВКО) - черные кривые и ROX(MAC) - серые кривые.
Осуществление изобретения
Ниже представлено более подробное описание заявляемого изобретения. Настоящее изобретение может подвергаться различным изменениям и модификациям, понятным специалисту на основе прочтения данного описания. Такие изменения не ограничивают объем притязаний.
В разработанной тест-системе используются 96 луночные планшеты, объёмом 0,2 мкл, с лиофильно высушенными реагентами для амплификации фрагментов ДНК, специфичных к M. avium complex (14 видов). При этом одна пробирка расходуется на один образец, что обеспечивает высокую процессивность анализа: так в амплификаторах на 96 лунок можно проанализировать 94 образца за одну постановку (ещё две точки включают в себя отрицательный и положительный контроли). Реакционная смесь содержит олигонуклеотиды для детекции внутреннего контрольного образца, добавляемого при выделении ДНК, что позволяет избежать ложноотрицательных результатов.
В состав реакционной смеси входят: 5,1 мМ MgCl2; по 0,39 мМ каждого нуклеозидтрифосфата (dATP, dGTP, dCTP и dTTP); 0,05 ед/мкл Taq-ДНК-полимеразы; 1х ПЦР-буфер (0,1M Трис-HCl с pH = 8,6; 0,5М КCl; 1% Tween20).
Кроме того, в состав смеси включены специфичные праймеры на последовательность между генами 16S- и 23S-рРНК (16S-23S межгенный спейсер) по 0,45 мкМ:
SEQ ID NO:1 5’-GTCGTCTGTAGTGGACGG (MAC_F11)
SEQ ID NO:2 5’-TCCACACGGACGGACCGA (MAC_R6)
SEQ ID NO:3 5’-TCCACACGGACTGACCG (MAC_R7)
SEQ ID NO:4 5’-TCCACACGGATCGACCGA (MAC_R8).
А также зонд, включающий последовательность SEQ ID NO:5 меченную флуорофором ROX 5’-(ROX)CAA+CAG+CAAA(T-BHQ2)GATTGC+CAGA+CA, знак «+» означает LNA-модификацию нуклеотида (Locked nucleic acids) в концентрации 0,3 мкМ.
SEQ ID NO:5 ROX 5’-(ROX)CAA+CAG+CAAA(T-BHQ2)GATTGC+CAGA+CA
5'-конец зонда помечен репортерным флуоресцентным красителем ROX, а 3'-конец зонда помечен красителем-гасителем BHQ-2
Подбор праймеров и зондов проводился с помощью программного обеспечения Unipro UGENE (Россия, Новосибирск). Предварительная оценка характеристик олигонуклеотидов, для дальнейшего подбора программы термоциклирования, проводилась с помощью встроенной в программное обеспечение функции «ПЦР in silico». Специфичность отжига праймеров и зондов оценивалась с помощью программного обеспечения BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). Синтез олигонуклеотидов осуществлен по стандартной технологии по заказу в компании АО «Вектор-бест» (Новосибирск, Россия).
При постановке ПЦР в реальном времени для определения MAC, обязательно ставится положительный отрицательный контроли (ПКО и К-). Положительный контрольный образец представляет собой ампликон размером 336 п.н., полученный методом амплификации ДНК культуры Mycobacterium avium с помощью праймеров MAC_F1seq (5’-TCGGTAATACCCGAAGC) и MAC_R1seq (5’-AGATGCTCGCGACCACTATC).
Предложенная нами тест-система для детекции M. avium complex использует в качестве мишени 16S-23S межгенный спейсер и позволяет выявлять виды M. Arosiense, M. Avium, M. Bouchedurhonense, M. Chimaera, M. Colombiense, M. Indicus pranii, M. Intracellulare, M. Lepraemurium, M. Mantenii, M. Marseillense, M. Paraintracellulare, M. Timonense, M. Vulneris, M. Yongonense без их дифференцировки.
Программа амплификации: 1 цикл 50°С - 2 мин, 95°C - 2 мин, далее 50 циклов 98°С - 10 сек и 60°С - 1 мин с детекцией сигнала флуоресценции по каналам FAM и ROX (общее время до получения результата 112 минут). Наличие сигнала на канале FAM свидетельствует об успешной экстракции ДНК. Тогда как, наличие сигнала на канале ROX, в пределах граничного значения (Ct <40), свидетельствует о наличии ДНК MAC. Для оценки адекватности прохождения ПЦР реакции обязательно ставятся положительный и отрицательные контроли (отрицательный контроль ПЦР реакции (К-) и отрицательный контроль опыта, прошедший через этап выделения (ОКО)).
После окончания реакции амплификации в родном ПО прибора для построения графиков плавления и получения значения Ct, выставляется значение линии Treshhold, равное среднему от максимального значения разгорания кривых плавления («плато»), деленное на 40.
Интерпретация результатов делится на 2 этапа:
1. Валидация образцов: оценивается форма полученных кривых плавления, она должна быть сигмоидной формы (если форма кривых не соответствует сигмоиде, такие образцы отправляются на повторную перестановку). Далее оценивается этап выделения образцов и выбраковка не валидных образцов по следующему алгоритму:
- Вычисляем среднее значение Ct по каналу «FAM» всех анализируемых образцов (включая ПКО и ОКО). Далее вычисляется разница между полученным пороговым значением (Ct) и средним значение Ct по каналу ВКО (FAM) - ΔCt. Если разница составила более 2-х циклов от среднего значения, такие образцы выбраковываются и подлежат повторной экстракции, с последующей постановкой тест-системой.
2. Интерпретация результатов по следующему алгоритму:
- Образец будет положительным (содержащим ДНК Mycobacterium avium complex) если Ct по каналу «ROX» меньше или равно 40.
- Образец будет отрицательным (не содержащим ДНК Mycobacterium avium complex) если для этого образца значение Ct по каналу «ROX» больше 40 или не определяется. Но если для такого образца значение ΔCt ВКО превышает 2, то результат не подлежит учёту как отрицательный. Необходимо провести повторный анализ данного образца, начиная с этапа выделения ДНК.
В случае если в К- наблюдается сигнал на одном из каналов или обоих сразу, не превышающий граничные значения, тогда регистрируется контаминация. Также если в отрицательном контроле опыта (ОКО) наблюдается сигнал на канале «ROX», не превышающий граничное значение 40, тогда констатируется контаминация. При контаминации все положительные результаты данной постановки ПЦР будут считаться недостоверными. Требуется повторить анализ положительных образцов. Образцы, анализ которых дал отрицательный результат, можно учитывать как отрицательные.
В том случае, если в образце положительного контроля реакции отсутствует сигнал или больше граничного (0) на одном или обоих каналах, постановка считается не валидной и требуется переставить все образцы, предварительно убедившись, что не были нарушены сроки годности реактивов.
Изобретение поясняется следующими примерами.
Пример 1.
По результатам культуральных исследований были отобраны и изучены 86 образцов культур микобактерий. Для данных культур вид микобактерий был определен с помощью MALDI-TOF Mass Spectrometry, а также секвенированием генов 16S рРНК, 16S-23S межгенного спейсера, hsp65, rpoB, IS1311. Совпадение результатов разработанной тест-системы и секвенирования отмечено в 84 из 86 (98%) случаев (Табл. 1). В одном образце (№4119) получен ложноположительный результат. Ещё в одном образце (№4837) получен ложноотрицательный результат, который может объясняться присутствием единичных копий ДНК M. avium. При дальнейшей проверке заявляемой тест-системы на M. avium с использованием многокопийной мишени IS1311 получены Ct = 38, что соответствует единичным копиям ДНК M. avium. По полученным результатам были определены диагностическая чувствительность и специфичность разработанной тест-системы, которые составили 98,18% [95ДИ 90,28-99,95] и 96,77% [95ДИ 83,3-99,92] соответственно.
*примечание: в некоторых образцах по результатам секвенирования выявлено несколько видов микобактерий.
Образцы с Ct>40 считаются отрицательными. На реакцию брали 5 мкл раствора ДНК образца.
Пример 2
Проведены испытания на 17 образцах гемокультур (культур микобактерий в крови), принадлежащих к M. avium, идентифицированных с помощью MALDI-TOF MS и секвенирования. Предложенная нами тест-система выявила 17 из 17 (100%) образцов. Результаты отображены в таблице 2.
Пример 3
Были изучены 253 клинических образца разных биоматериалов (мокрота, БАЛ, биоптаты, кал, моча, плевральная жидкость, асцитическая жидкость, раневое отделяемое) с помощью разработанной тест-системы. В дальнейшем, через 1,5-2 месяца из клинических образцов был получен рост культуры с помощью автоматизированной системы BACTEC MGIT 960 (BD, США), идентификация вида микобактерий в образцах проводилась с помощью MALDI-TOF Mass Spectrometry, а также секвенированием генов 16S рРНК, 16S-23S межгенного спейсера, hsp65, rpoB, IS1311. Диагностическая чувствительность и специфичность разработанной тест-системы в выявлении M. avium complex относительно культурального метода составили 75,42% [95ДИ 68,44-81,54] и 93,24 [95 ДИ 84,93-97,77]. Полученные результаты представлены в таблице 3.
Все испытания, описанные в 3-х примерах, проводились на амплификаторе в реальном времени CFX96, производства Bio-Rad (США); время, затраченное на выявление M. avium complex не превышает 121 мин. Пример полученных кривых отображен на фиг. 1.
Разработанная нами тест-система выявления M. avium complex демонстрирует высокую специфичность и чувствительность, что способствует повышению эффективности диагностики микобактериоза. Кроме того, детекция сигнала осуществляется без открывания пробирки (стрипов, планшета) после этапа амплификации, что значительно уменьшает вероятность внутрилабораторной контаминации. А также уменьшает время- и трудозатраты до получения конечного результата, т.к. детекция проводится в режиме реального времени.
Изобретение относится к области биотехнологии, а именно к тест-системе для выявления в биоматериале ДНК возбудителей микобактериозов, входящих в Mycobacterium avium complex без дифференциации до вида методом ПЦР в реальном времени. Предложена комбинация праймеров, специфичных к последовательности 16S-23S межгенного спейсера, и флуоресцентно-меченного зонда для обнаружения видов Mycobacterium. Также предложен набор реагентов, включающий указанную комбинацию и реакционную смесь для ПЦР. Также изобретение относится к способу обнаружения видов Mycobacterium. Изобретение может быть использовано для создания медицинских изделий для диагностики in vitro, с целью выявления возбудителей микобактериозов службами клинико-лабораторной диагностики в лечебно-профилактических учреждениях, а также с целью осуществления эпидемиологического надзора населения за распространением возбудителей микобактериозов. 3 н. и 2 з.п. ф-лы, 1 ил., 3 табл., 3 пр.
1. Комбинация праймеров, специфичных к последовательности между генами 16S- и 23S-рРНК, и зонда для обнаружения видов Mycobacterium, где
(1) праймеры представляют собой олигонуклеотиды, содержащие нуклеотидную последовательность SEQ ID NO:1 - SEQ ID NO:4, и
(2) зонд с нуклеотидной последовательностью SEQ ID NO:5.
2. Комбинация по п. 1, характеризующаяся тем, что 5'-конец зонда помечен репортерным флуоресцентным красителем ROX, а 3'-конец зонда помечен красителем-гасителем BHQ-2.
3. Набор реагентов для обнаружения видов Mycobacterium, характеризующийся тем, что включает комбинацию праймеров и зонда по п. 1 и реакционную смесь следующего состава: 5,1 мМ MgCl2; по 0,39 мМ каждого нуклеозидтрифосфата, выбранного из: dATP, dGTP, dCTP и dTTP; 0,05 ед/мкл Taq-ДНК-полимеразы; 1х ПЦР-буфер.
4. Набор по п. 3, характеризующийся тем, что используют ПЦР-буфер следующего состава: 0,1M Трис-HCl с pH = 8,6; 0,5М КCl; 1% Tween20.
5. Способ обнаружения видов Mycobacterium, характеризующийся тем, что проводят ПЦР в реальном времени с использованием комбинации праймеров и зонда по п. 1 или набора по п. 3.
| US 2020181690 A1, 11.06.2020 | |||
| US 5667994 A, 16.09.1997 | |||
| ОЛИГОНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ И СПОСОБ ВЫЯВЛЕНИЯ ДНК Mycobacterium avium МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2013 |
|
RU2554842C2 |
| ОДНОВРЕМЕННОЕ ОПРЕДЕЛЕНИЕ, ИДЕНТИФИКАЦИЯ И ДИФФЕРЕНЦИАЦИЯ ЭУБАКТЕРИАЛЬНЫХ ТАКСОНОВ С ПОМОЩЬЮ ГИБРИДИЗАЦИОННОГО АНАЛИЗА | 1995 |
|
RU2154106C2 |
| WO 2005061730 A1, 30.12.1998 | |||
| DEVALLOIS A | |||
| et al., Comparative evaluation of PCR and commercial DNA probes for detection and identification to species level of Mycobacterium avium and Mycobacterium intracellulare, JOURNAL | |||
Авторы
Даты
2024-05-28—Публикация
2023-09-14—Подача