Изобретение касается зондов, представляющих собой фрагменты нуклеиновых кислот из спейсерной области между генами 16S и 23S рибосомальной рибонуклеиновой кислоты (рРНК), которые предназначены для определения эубактериальных организмов в биологическом образце методом гибридизации, а также праймеров нуклеиновых кислот, предназначенных для амплификации указанной спейсерной области эубактериальных организмов в биологическом образце. Настоящее изобретение также касается нуклеотидных последовательностей новых спейсерных областей, на основании которых могут быть получены подобные зонды и праймеры.
С открытием полимеразной цепной реакции и некоторых других методик амплификации нуклеиновых кислот влияние технологии, использующей зонды ДНК для определения микроорганизмов в биологических образцах всех видов, усиливается. Будучи часто более специфичным и потенциально более чувствительным в случае использования адекватной системы амплификации и/или детекции, метод ДНК-зондов способен со временем вытеснить традиционные методики идентификации микроорганизмов.
Достоверность тестов, основанных на анализе нуклеиновых кислот, находится в существенной зависимости от чувствительности и специфичности используемых зондов и/или праймеров. Следовательно, краеугольным камнем подобного рода анализов является идентификация нуклеотидных последовательностей, являющихся уникальными для интересующей группы организмов.
Большинство описанных в литературе и/или коммерчески доступных тестов, основанных на использовании нуклеиновых кислот, нацелены на определение только одного отдельного организма в биологическом образце. В силу того, что большинство биологических образцов обычно могут содержать большое число разнообразных вызывающих заболевания микроорганизмов, требуется проведение большого числа отдельных анализов на каждый из них. Такого рода подход был трудоемким и требовал больших материальных и временных затрат. Вследствие этого число тестов, реально проводимых в большинстве обычных диагностических лабораторий для анализа отдельного образца, ограничено только несколькими, направленными на определение некоторых наиболее часто встречающихся организмов. Поэтому исключительно удобно было бы иметь в распоряжении систему, способную быстро, легко и одновременно проводить определение большого числа различных организмов. Чем большее число организмов может быть проверено в одном и том же анализе, тем более эффективной по стоимости будет такая процедура проверки.
Как было показано в ранее опубликованных работах, спейсерная область, расположенная между генами 16S рРНК и 23S рРНК, называемая также внутренним транскрибируемым спейсером (ITS), является выгодной мишенью для связывания зондов с целью определения патогенных микроорганизмов бактериального происхождения (международная заявка WO 91/16454 [44]; ЕР-А-0 395 292).
Одним из наиболее ценных преимуществ этой области является то, что нуклеотидные последовательности, уникальные для большого числа бактериальных таксонов, могут быть обнаружены в очень небольшой области бактериального генома. Эти свойства делают возможным создание "панелей зондов", позволяющих одновременно определить набор организмов, которые могут присутствовать в конкретном типе биологического образца. Кроме того, будучи фланкированными квази- универсальными консервативными нуклеотидными последовательностями, большей частью расположенными в 3'-концевой части гена 16S рРНК и 5'-концевой части гена 23S рРНК соответственно, практически все спейсеры могут быть одновременно амплифицированы с помощью ограниченного набора амплификационных праймеров. С другой стороны, на основании самих последовательностей спейсерных областей могут быть получены наборы специфических праймеров, позволяющих проводить видо- и группо- специфическую амплификации.
Спейсерная область 16S-23S рРНК представляет собой относительно короткий (примерно от 200 до 1000 пар нуклеотидов) участок ДНК, присутствующий в одной или множестве копий в геноме едва ли не всех эубактериальных организмов. В случае наличия многих копий в геноме одной бактерии эти копии могут быть или идентичными (как чаще всего бывает у некоторых представителей вида Neisseria), или могут отличаться одна от другой (как в случае Е. coli). Это отличие может быть ограничено несколькими нуклеотидами, но и делеции, и инсерции значительной протяженности могут иметь место.
К настоящему времени описаны и стали публичным достоянием спейсерные зонды лишь для ограниченного числа организмов, многие из которых приведены в международной заявке WO 91/16454. Как отмечено выше, осуществление возможности одновременного определения панели патогенов было бы очень выгодным, например панели патогенных микроорганизмов, которые могут присутствовать в одном и том же типе биологического образца, или панели патогенных микроорганизмов, которые могут вызывать болезненные симптомы одного и того же типа, трудно различимые клинически и/или биохимически, либо панели организмов, принадлежащих к одному и тому же таксону. Для того, чтобы сделать различные панели как можно более полными, необходимо иметь дополнительные зонды или наборы зондов к спейсерной области, позволяющие проводить идентификацию по меньшей мере следующих групп или видов бактерий:
- Mycobacterium species,
- Listeria species
- Chlamydia species
- Acinetobacter species
- Mycoplasma species
- Streptococcus spesies
- Staphylococcus species
- Salmonella species
- Brucella species
- Yersinia species
- Pseudomonas species
Эти дополнительные спейсерные зонды должны быть тщательно сконструированы так, чтобы их можно было использовать одновременно по меньшей мере с одним другим зондом в тех же условиях гибридизации и отмывки, что позволило бы проводить определение конкретной панели организмов.
Таким образом, задачей настоящего изобретения является подбор зондов или наборов зондов, имеющих своей мишенью спейсерную 16S-23S рРНК-область, и которые позволяют проводить определение и идентификацию по меньшей мере одного и предпочтительно более чем одного из упомянутых выше микроорганизмов. Зонды или наборы зондов подбираются таким образом, чтобы их можно было применять в сочетании по меньшей мере с одним другим зондом, преимущественно также из спейсерной 16S-23S рРНК-области, в тех же самых условиях гибридизации и отмывки для обеспечения возможности одновременного определения нескольких микроорганизмов в образце.
Задачей настоящего изобретения также является создание метода селекции указанных спейсерных зондов или наборов зондов.
Задачей настоящего изобретения также является создание быстрого и надежного метода гибридизации для определения и идентификации по меньшей мере одного микроорганизма в образце или одновременного определения и идентификации различных микроорганизмов в образце.
Более конкретной задачей настоящего изобретения является создание метода гибридизации, позволяющего одновременное определение и идентификацию набора микроорганизмов, присутствующих в образце конкретного типа.
Более конкретной задачей настоящего изобретения является создание зондов или наборов зондов, дающих возможность одновременного определения микроорганизмов в образце из дыхательного тракта.
Другой конкретной задачей настоящего изобретения является создание зондов или наборов зондов, дающих возможность одновременного определения микроорганизмов в образце из спинномозговой жидкости.
Еще одной конкретной задачей настоящего изобретения является создание зондов или наборов зондов, дающих возможность одновременного определения микроорганизмов в образце из мочеполового тракта.
Еще одной конкретной задачей настоящего изобретения является создание зондов или наборов зондов, дающих возможность одновременного определения микроорганизмов в образце, взятом из желудочно-кишечного тракта пациента.
Еще одной конкретной задачей настоящего изобретения является создание зондов или наборов зондов, дающих возможность одновременного определения микроорганизмов в пищевых образцах или в образцах, взятых для анализа окружающей среды.
Кроме того, задачей настоящего изобретения является создание способа определения и идентификации конкретного таксона в образце или набора таксонов, причем указанный таксон может представлять собой или полный род, или подгруппу внутри рода, вид или даже подвид внутри вида (подвиды, серовары, секвевары, биовары и т. п.).
Более конкретной задачей настоящего изобретения является создание зондов или наборов зондов для определения вида Mycobacterium и его подвидов, более конкретно - комплексных штаммов М. tuberculosis, штаммов Mycobacterium из MAIS-комплекса, М. avium и М. paratuberculosis, М. intracellulare и М. intracellulare-подобных штаммов, М. scrofulaceum, М. kansasii, М. chelonae, М. gordonae, М. ulcerans, М. genavense, М. xenopi, М. simiae, М. fortuitum, М. malmoense, М. celatum и М. haemophilum.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Mycoplasma, более конкретно М. pneumoniae и М. genitalium.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Pseudomonas, более конкретно P. aeruginosa.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Staphylococcus, более конкретно S. aureus и S. epidermidis.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Acinetobacter, более конкретно A. baumanii.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Listeria, более конкретно Listeria monocytogenes.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Brucella.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Salmonella.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Chlamydia, более конкретно С. trachomatis и С. psittaci.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Streptococcus.
Задачей настоящего изобретения также является создание зондов или наборов зондов для определения штаммов Yersinia enterolitica.
Задачей настоящего изобретения также является создание праймеров для специфической амплификации спейсерной 16S- 23S рРНК-области определенных организмов. Более конкретной задачей настоящего изобретения является создание праймеров для специфической амплификации спейсерной области штаммов Mycobacterium, Chlamydia, Listeria, Brucella и Yersinia enterolitica.
Задачей настоящего изобретения также является создание новых последовательностей спейсерных 16S-23S рРНК- областей, на основании которых могут быть получены полезные зонды или праймеры.
Задачей настоящего изобретения также является создание наборов для определения по меньшей мере одного организма в образце, в которых используются указанные выше зонды и/или праймеры.
Известно, что для нескольких вышеупомянутых организмов спейсерные последовательности уже опубликованы в литературе или имеются в доступных банках данных.
Однако следует внести ясность в понимание того, что спейсерные последовательности, приведенные в настоящем изобретении (фиг. 1-103), являются новыми и в случае, когда они относятся к тем же видам, для которых спейсерные последовательности были уже ранее описаны, они несколько отличаются от последних.
Кроме того принципиальная задача настоящего изобретения состоит в отборе на основании накопления данных по последовательностям спейсерных областей специфических зондов и наборов зондов, позволяющих проводить определение и идентификацию конкретных панелей организмов, принадлежащих общему таксону, или организмов, возможно присутствующих в образце того же типа.
Процедура отбора обычно состоит из теоретической и экспериментальной частей. Прежде всего различные спейсерные последовательности необходимо сравнить с соответствующими последовательностями "ближайших соседей" или спейсерными последовательностями других микроорганизмов, возможно присутствующих в данном образце. Для этого, конечно, требуется, как описано в примерах, определение нуклеотидной последовательности спейсерной области. Из такого сравнения могут быть определены области несовпадения, из которых готовят зонды с желаемыми гибридизационными свойствами, используя принципы, известные специалистам и более детально раскрытые ниже.
Во-вторых, созданные зонды необходимо проверить экспериментально и оценить их полезность в условиях специфической гибридизации и/или в сочетании с другими зондами. Экспериментальная проверка может быть осуществлена в соответствии с любым известным специалистам методом гибридизации, но преимущественным видом анализа для одновременного тестирования в одних и тех же условиях различных зондов является анализ методом обратной гибридизации. Специфическим форматом для обратной гибридизации различных одновременно применяемых в настоящем изобретении зондов является, как описано ниже, метод LiPA (Line Probe Assay).
В процессе экспериментального тестирования может быть выявлено неожиданное поведение в отношении гибридизации с нуклеиновой кислотой, являющейся мишенью, что может потребовать специфической адаптации зонда.
Кроме того специфичность и чувствительность зондов необходимо проверить на большой коллекции штаммов, как принадлежащих к определяемому таксону, так и к другим таксонам. Вследствие гетерогенности генома в спейсерной области или наличия у одного организма большого числа спейсерных областей с различными последовательностями, часто совершенно необходимым является секвенирование спейсерных областей дополнительных штаммов или секвенирование дополнительных спейсерных областей одного и того же штамма и изменение зондов в соответствии с новыми данными для достижения лучшей чувствительности и/или специфичности (см. , например, пример 3). В некоторых случаях бывает необходимо или предпочтительно использовать несколько зондов для одного и того же организма (см., например, примеры 2 и 7). Кроме этого при секвенировании спейсерной области некоторые организмы могут показать неожиданное родовое сходство (или отсутствие его), что может привести к пересмотру классификации штаммов в противоположность классическим таксономическим критериям (см., например, примеры 2 и 7).
В заключение можно отметить, что экспериментальная часть процедуры отбора зондов является необходимой и дополняет теоретическую часть. Создание зондов, особенно для фиксированных условий обратной гибридизации (одни и те же условия для каждого зонда), не происходит напрямую, и эффективность зондов должна быть скрупулезно оценена, перед тем как они могут быть использованы в формате обратной гибридизации. Следовательно, не всегда можно создать зонды на основании только теоретического знания нуклеотидной последовательности.
Для создания зондов с желаемыми характеристиками можно руководствоваться следующими полезными принципами.
В силу того, что степень и специфичность реакций гибридизации такого типа, как описаны здесь, зависят от ряда факторов, манипулирование одним или более из них будет определять точную чувствительность и специфичность конкретного зонда, либо полностью комплементарного к своей мишени, либо нет. Важность и влияние различных условий анализа, раскрываемые здесь ниже, известны специалистам.
Во-первых, стабильность гибрида нуклеиновых кислот типа [зонд:мишень] следует выбрать так, чтобы она была совместима с условиями анализа. Это может быть сделано путем исключения протяженных богатых А и Т последовательностей, за счет терминации гибридов парами оснований G-C и путем создания зондов с подходящей Тпл. Начальные и конечные участки зондов следует выбирать таким образом, чтобы длина и процентное содержание GC обуславливали бы Тпл примерно на 2-10oC выше, чем температура проведения заключительного анализа. Нуклеотидный состав зонда существенен, поскольку пары оснований G-C имеют большую температурную стабильность по сравнению с парами оснований А-Т ввиду наличия между основаниями дополнительной водородной связи. Следовательно, гибриды нуклеиновых кислот с более высоким содержанием G-C будут более стабильными при высоких температурах.
При конструировании зондов следует также принять во внимание такие условия, как ионная сила и температура инкубации, при которых зонды будут использоваться. Известно, что степень гибридизации увеличивается при увеличении ионной силы реакционной смеси, а температурная стабильность гибридов возрастает с увеличением ионной силы. С другой стороны, химические реагенты, такие как формамид, мочевина, ДМСО и спирты, разрушающие водородные связи, будут увеличивать строгость гибридизации. Дестабилизация водородных связей подобными реагентами может значительно понизить Тпл. В целом оптимальная гибридизация для синтетических олигонуклеотидных зондов длиной 10-50 нуклеотидных пар происходит при температуре приблизительно на 5oC ниже температуры плавления данного дуплекса. Инкубация при температурах ниже оптимума может вызывать ошибочное спаривание последовательностей оснований при гибридизации и приводить к уменьшению специфичности.
Желательно иметь зонды, которые гибридизуются только в условиях высокой строгости гибридизации. В подобных условиях будут образовываться гибриды нуклеиновых кислот, обладающих только высокой степенью комплементарности. Поэтому строгость условий анализа определяет степень комплементарности двух цепей нуклеиновых кислот, необходимую для образования гибрида. Строгость условий гибридизации выбирается с целью максимально увеличить разницу в стабильности между гибридами зонда с мишенью и со случайной нуклеиновой кислотой (не мишенью). В некоторых примерах настоящего изобретения, например когда необходимо различить два близких организма, бывает необходимо зафиксировать изменения в одной нуклеотидной паре. В таких случаях необходимы очень строгие условия гибридизации.
Во-вторых, зонды должны быть выбраны таким образом, чтобы максимально уменьшить число гибридов [зонд: не мишень]. Это может быть достигнуто минимизацией участков полной комплементарности с ДНК организмов, не являющихся мишенью, путем исключения богатых GC областей гомологии к последовательностям не-мишеней и таким выбором зондов, который обеспечивал бы как можно большее число дестабилизирующих ошибочных спариваний. Будет ли последовательность зонда пригодной для определения организма только специфического типа, во многом зависит от разницы в температурной стабильности гибридов [зонд: мишень] и [зонд: не мишень]. В создаваемых зондах разница значений этих Тпл должна быть как можно больше (например, по меньшей мере 2oC и предпочтительно 5oC).
Длина участка нуклеиновой кислоты, являющегося мишенью, и в соответствии с этим размер зонда могут также быть важными. В некоторых случаях могут существовать различные последовательности в рассматриваемой области, отличающиеся по расположению и длине, на основании которых могут быть созданы зонды с желаемыми гибридизационными свойствами. В других случаях одна последовательность может быть значительно лучше другой, отличающейся только на единственное основание. Несмотря на то, что для не полностью комплементарных нуклеиновых кислот возможна гибридизация, наиболее длинный участок с полностью комплементарной последовательностью оснований будет в норме изначально определять стабильность гибрида. Несмотря на то, что могут быть использованы олигонуклеотидные зонды различной длины и состава, в настоящем изобретении отдается предпочтение зондам длиной приблизительно от 10 до 50 оснований и с существенной гомологией к нуклеиновой кислоте - мишени.
В-третьих, выбор в качестве мишеней тех областей ДНК и РНК, о которых известно, что они образуют прочные внутренние структуры, ингибирующие гибридизацию, менее предпочтителен. Точно так же следует избегать применения зондов со значительной аутокомплементарностью. Как объяснялось выше, гибридизация представляет собой ассоциацию двух одиночных комплементарных цепей нуклеиновых кислот с образованием двойной цепи, связанной водородными связями. Очевидно, что если одна из двух цепей полностью или частично вовлечена в образование гибридной структуры, то она в меньшей степени способна принимать участие в образовании нового гибрида. Молекулы одного типа зонда с высокой степенью аутокомплементарности могут образовывать внутри- и межмолекулярные гибриды. Подобных структур следует избегать в процессе тщательной разработки зондов. Скорость и степень гибридизации могут быть существенно повышены за счет создания зондов, значительная часть представляющей интерес последовательности которых является одноцепочечной. Поиск подобного типа взаимодействия может быть осуществлен с помощью компьютерной программы. Тем не менее, в некоторых случаях взаимодействия подобного типа избежать не удается.
Зонды, описанные в настоящем изобретении, созданы для достижения максимального эффекта в одних и тех же условиях гибридизации, поэтому они могут быть использованы в наборах для одновременной гибридизации; это существенно повышает возможность их практического применения и приводит к значительной экономии времени и труда. Очевидно, что когда предпочтительны другие условия гибридизации, все зонды следует адаптировать к новым условиям соответственно либо путем добавления, либо удаления ряда концевых нуклеотидов. Следует понимать, что эти сопутствующие адаптации должны приводить к существенно одинаковому результату, а именно, чтобы соответствующие зонды продолжали специфически гибридизоваться с определенной мишенью. Подобные адаптации могут также быть необходимы, когда амплифицируемый материал будет представлять собой РНК, а не ДНК, как в случае системы NASBA.
Условия гибридизации могут быть с уверенностью проконтролированы по нескольким параметрам, таким как природа и концентрация компонентов среды и температура, при которой производится гибридизация и отмывка.
Верхнее значение температуры гибридизации и отмывки лимитируется строением зонда (его нуклеотидным составом, видом и длиной). Максимальная температура гибридизации или отмывки зондов, описанных в настоящем изобретении, колеблется от 40oC до 60oC, более предпочтительно - от 45oC до 55oC в специфических средах для гибридизации и отмывки, описанных в разделе Примеры. При более высоких температурах образование дуплексов (= гибридов) конкурирует с диссоциацией (или денатурацией) гибридов, образовавшихся между зондом и мишенью.
В предпочтительной гибридизационной среде по изобретению, содержащей 3 х SSC и 20% формамида, температура гибридизации может колебаться от 45oC до 55oC, составляя предпочтительно 50oC. Предпочтительная среда для отмывки содержит 3 х SSC и 20 % формамида, а предпочтительная температура отмывки такая же, как и для гибридизации, т.е. между 45oC и 55oC, и наиболее предпочтительной является температура 50oC.
Тем не менее при введении изменений как в зонды, так и в состав сред, значения температур, при которых зонды могут быть использованы с требуемой специфичностью, следует изменить в соответствии с известными соотношениями, такими, как описаны в работе: Hames В. и Higgins S. (eds.). Nucleic acid hybridization, A practical approach. IRL Press, Oxford, U.K., 1985.
Список отобранных зондов нуклеиновых кислот из 16S-23S спейсерной области рРНК, описанных в настоящем изобретении, приведен в Таблице la (SEQ ID N 1-64, 175-191, 193-201 и 210-212). Как описано в разделе Примеры, некоторые из этих зондов показывают большую чувствительность и/или специфичность, чем другие, и поэтому при определении интересующих организмов в биологическом образце предпочтение отдается лучшим с этой точки зрения зондам. Однако возможно, что для некоторых приложений (например, эпидемиология, типирование подштаммов и т.п.) очень информативным может оказаться набор зондов, включающий менее специфичные и/или менее чувствительные зонды (см. например, пример 7).
Ниже изложены следующие определения терминов и выражений, используемых в различных воплощениях настоящего изобретения.
Термин "спейсер" является аббревиатурой, относящейся к 16S-23S рРНК транскрибируемой спейсерной области.
Термин "зонд" относится к одноцепочечным олигонуклеотидам со специфической последовательностью оснований, в целях гибридизации существенно комплементарной к последовательности оснований мишени, которую необходимо определить.
Более узкий термин "спейсерный зонд" относится к определенному выше понятию зонда, имеющего последовательность оснований, в целях гибридизации существенно комплементарную к последовательности оснований мишени, расположенной в спейсерной области (областях) определяемого организма (или группы организмов).
Предпочтительными называются зонды, имеющие 70%, 80%, 90% или более 95% гомологии с определяемой последовательностью мишени. Данные последовательности-мишени могут представлять собой либо геномную ДНК или преРНК, либо их амплифицированные версии.
Предпочтительные размеры данных зондов составляют примерно от 5 до 50 нуклеотидов, более предпочтительны зонды длиной от 10 до 25 нуклеотидов. Используемые в настоящем изобретении нуклеотиды могут быть рибонуклеотидами, дезоксирибонуклеотидами и модифицированными нуклеотидами, такими как инозин, либо нуклеотидами, содержащими модифицированные группы, существенно не изменяющие их гибридизационные свойства. Более того, для специалистов очевидно, что любой из указанных ниже зондов может быть использован как в прямой форме, так и в форме комплементарной последовательности, либо в форме соответствующей РНК (где Т заменены на U).
Зонды по настоящему изобретению могут быть созданы клонированием рекомбинантных плазмид, содержащих вставки, включающие соответствующие нуклеотидные последовательности, при необходимости они могут быть вырезаны из клонированных плазмид с помощью соответствующих нуклеаз и извлечены, например, путем фракционирования в соответствии со значениями молекулярных масс. Зонды по настоящему изобретению также могут быть синтезированы химическим путем, например, обычным фосфотриэфирным способом.
Используемый здесь термин "комплементарные" нуклеиновые кислоты означает, что нуклеиновые кислоты данных последовательностей могут образовывать друг с другом совершенную двойную спираль за счет спаривания нуклеотидов.
Термин "гомологичный" используется в настоящей заявке как синоним термина "идентичный" и означает, что полинуклеиновая кислота, о которой говорится, что она обладает, например 80% гомологией, содержит 80% идентичных пар оснований в тех же позициях последовательности.
Термин "полинуклеиновая кислота" отвечает как двухцепочечной или одноцепочечной кДНК, так и геномной ДНК или РНК, содержащей по меньшей мере 10, 20, 30, 40 и 50 соединенных друг с другом нуклеотидов. Полинуклеиновая кислота длиной менее 100 нуклеотидов часто называется также олигонуклеотидом. Последовательности одноцепочечных полинуклеиновых кислот в настоящем изобретении всегда приведены, начиная от 5'-конца к 3'-концу.
Термин "ближайший сосед" означает таксон, о котором известно или о котором предполагается, что он наиболее близок к интересуемому организму в терминах гомологии ДНК и который необходимо отличить от интересуемого организма.
Выражение "желаемые гибридизационные свойства" означает, что зонд гибридизуется только с ДНК или РНК организмов, к которым он был разработан, но не с ДНК или РНК остальных организмов (ближайших соседей или организмов, которые могут присутствовать в том же образце). В частности, это означает, что интенсивность сигнала гибридизации по меньшей мере в два, три, четыре, пять, десять или более раз сильнее для ДНК- или РНК-мишени организма, для которого данный зонд был разработан, по сравнению с последовательностью, не являющейся мишенью.
Эти желаемые гибридизационные свойства соответствуют тому, что названо позже в тексте "специфической гибридизацией".
Выражение "таксон-специфическая гибридизация" или "таксон-специфический зонд" означает, что зонд гибридизуется только с ДНК или РНК таксона, для которого он был разработан, но не с ДНК и РНК других таксонов.
Термин таксон может относиться к полному роду или подгруппе внутри рода, виду или даже подтипу внутри вида (подвиды, серовары, секвевары, биовары и т.п.).
Термин "специфическая амплификация" или "специфические праймеры" означает тот факт, что указанные праймеры амплифицируют только спейсерную область тех организмов, для которых они были разработаны, но не других организмов.
Термин "чувствительность" означает число ошибочных отрицательных результатов: т. е., если 1 из определяемых 100 штаммов был пропущен, то говорят, что тест показывает чувствительность (100 - 1)/100 = 99%.
Термин "специфичность" означает число ошибочных положительных результатов: т.е., если на 100 определяемых штаммов 2 оказываются принадлежащими к организмам, для определения которых данный тест не предназначался, специфичность теста составляет (100 - 2)/100 = 98%.
Зонды, отобранные как "предпочтительные", показывают чувствительность и специфичность более 80%, предпочтительно более 90% и наиболее предпочтительно свыше 95%.
Термин "праймер" относится к одноцепочечной олигонуклеотидной последовательности ДНК, способной выступать в роли точки инициации для синтеза праймер-удлиненного продукта, комплементарного копируемой цепи нуклеиновой кислоты. Длина и последовательность нуклеотидов праймера должна быть такая, чтобы обеспечивать праймирование синтеза праймер-удлиненных продуктов. Предпочтительно праймер содержит примерно 5-50 нуклеотидов. Специфическая длина и последовательность праймера зависят от сложности необходимых ДНК или РНК мишеней, а также от условий использования праймера, таких как температура и ионная сила. Тот факт, что амплификационные праймеры могут не быть точной копией соответствующего участка последовательности, гарантируя подходящую амплификацию, полно подтвержден в литературе [29].
Используемый метод амплификации представляет собой полимеразную цепную реакцию (PCR; [45]), лигазную цепную реакцию (LCR; [30, 62, 2]), амплификацию, основанную на последовательности нуклеиновой кислоты (NASBA; [21, 11]), амплификационную систему, основанную на транскрипции (TAS; [28]), амплификацию замещением цепей (SDA; [14, 60]), амплификацию с помощью Qβ- репликазы [31, 33] или любой другой известный специалистам метод амплификации молекул нуклеиновых кислот.
Олигонуклеотиды, используемые в качестве праймеров или зондов, могут также включать аналоги нуклеотидов, такие как фосфоротиоаты [35], алкилфосфоротиоаты [37] или пептидные нуклеиновые кислоты [38, 39] либо могут содержать интеркалирующие агенты [1].
Как и большинство других изменений и модификаций, введенных в оригинальные последовательности ДНК, предусмотренные изобретением, эти изменения неизбежно повлекут за собой адаптации в отношении условий применения олигонуклеотидов для получения требуемой специфичности и чувствительности. Тем не менее в конечном счете результаты гибридизации будут существенно такими же, как и для немодифицированных олигонуклеотидов.
Введение подобных модификаций может быть выгодным для того, чтобы положительно повлиять на такие характеристики, как кинетика и обратимость гибридизации, биологическая стабильность молекул олигонуклеотидов и т.д.
Термин "твердая подложка" может относиться к любому материалу, к которому можно присоединить олигонуклеотидный зонд, обеспечивающему поддержание гибридизационных свойств зонда и низкий фоновый уровень гибридизации. Обычно в качестве твердого носителя используют планшет для микротитрования, мембрану (например, нейлоновую или нитроцеллюлозную) или микрошарики (гранулы). Перед нанесением на мембрану или перед фиксацией бывает удобно модифицировать зонд нуклеиновой кислоты с целью облегчения фиксации или улучшения эффективности гибридизации. Подобные модификации могут заключаться в присоединении гомополимерного хвоста, связанного с различными реакционноспособными группами, такими как алифатические группы, NH2-группы, SH-группы, карбоксильные группы, или связанного с биотином, гаптенами или белками.
Термин "меченый" относится к использованию меченых нуклеиновых кислот. Введение метки может быть осуществлено с использованием меченых нуклеотидов, включаемых во время полимеразной стадии амплификации, как показано в работах [45, 3], или меченых праймеров, или любым другим способом, известным специалистам. Метка может иметь изотопную природу (32P, 35S и т.д.) или не являться изотопом (биотин, дигоксигенин и т.д.).
"Образцом" может быть любой биологический материал, взятый как непосредственно у инфицированного человеческого существа (или животного), так и после культивирования (обогащения), или взятый из пищи или корма. Биологический материал может представлять собой, например, мокроты любого вида, бронхиальные смывы, кровь, кожную ткань, биоптаты, материал культуры лимфоцитов крови, колонии и т.д. Указанные образцы могут быть приготовлены или экстрагированы в соответствии с любой из известных специалистам методик.
Материалом "мишени" в данных образцах может быть как геномная ДНК или преРНК определяемого организма (организма-мишени), так и ее амплифицированная версия, как изложено выше. Более конкретно, последовательность нуклеотидов материала-мишени расположена в спейсерной области организма (ов)-мишени.
Определение и идентификация материала-мишени могут быть проведены с использованием одного из многочисленных электрофоретических, гибридизационных методов или методов секвенирования, описанных в литературе и общеизвестных специалистам в данной области. Однако очень удобной и полезной методикой одновременного определения нуклеиновых кислот, которые могут присутствовать в биологических образцах, является методика Линейного Зондового Анализа. Линейный Зондовый Анализ (LiPA) представляет собой обратный гибридизационный формат [46], использующий мембранные полоски, на которые могут быть нанесены в виде параллельных линий несколько олигонуклеотидных зондов (включая негативные и позитивные контрольные олигонуклеотиды).
Методика LiPA, как описано в [51] и в международной заявке WO 94/12670, обеспечивает проведение очень быстрого и благоприятного для пользователя гибридизационного теста. Результаты теста могут быть прочитаны в течение 4 ч после начала амплификации. После амплификации, во время которой в амплифицированный продукт обычно включается неизотопная метка, и щелочной денатурации амплифицированный продукт приводится в контакт с зондами на мембране, и гибридизация осуществляется в течение приблизительно от 1 до 1,5 ч. В результате образовавшиеся гибриды детектируются ферментативно, давая визуально наблюдаемый осадок пурпурно-коричневой окраски. Формат LiPA полностью совместим с коммерчески доступными сканирующими приборами, благодаря чему возможна автоматическая интерпретация результатов. Все эти преимущества делают возможным использование формата LiPA в обычных условиях.
Формат LiPA является не только полезным инструментом идентификации и определения патогенов на уровне вида, но также еще и на более высоких и низких уровнях. Например, конфигурация зондов на полосках LiPA может быть выбрана таким образом, что они смогут определять целый род (например, Neisseria, Listeria и т. д. ), или идентифицировать подгруппы внутри рода (например, подгруппы в Mycobacterium avium-intracellulare-scrofulaceum комплексе), или в некоторых случаях детектировать даже подтипы (подвиды, серовары, секвевары, биовары и т.п., что имеет отношение к клинике) внутри вида.
Следует подчеркнуть, что способность одновременного получения результатов гибридизации с рядом зондов является выдающимся преимуществом LiPA-технологии. Во многих случаях количество информации, которое может быть получено с использованием определенной комбинации зондов, значительно превосходит таковое, полученное с использованием анализов с единичным зондом. Поэтому выбор зондов на мембранной полоске является крайне важным ввиду того, что оптимальный набор зондов способен дать максимум возможной информации. Данное положение более конкретно рассмотрено далее в примерах.
Возможность комбинирования различных зондов на одной полоске позволяет преодолеть недостаток чувствительности, обусловленный гетерогенностью последовательности в области, являющейся мишенью, группы определяемых организмов. Из- за наличия такой гетерогенности могут потребоваться два или большее число зондов для позитивной идентификации всех организмов определенной группы. Данные зонды можно нанести на мембранные полоски в разные места, и результат интерпретируется как положительный, если по меньшей мере один из этих зондов окажется положительным. В альтернативном случае эти зонды можно нанести в виде смеси в одно и то же место, тем самым число линий на полоске уменьшится. Подобное уменьшение может быть удобным для того, чтобы сделать полоску более четкой или для расширения общего числа зондов на полоске. Другой альтернативный подход, с точки зрения его практической пользы, заключается в синтезе олигонуклеотидов, включающих последовательности двух (или более) различных зондов (= вырожденные зонды), которые далее могут быть подвергнуты процессингу и нанесены на полоску как одна олигонуклеотидная молекула. Данный подход будет значительно упрощать способы производства LiPA-полосок. Например, допустим, что зонды с нуклеотидными последовательностями А и В оба необходимы для определения всех штаммов таксона X. В случае последнего подхода можно синтезировать зонд, имеющий последовательность AB. Этот зонд будет объединять характеристики зондов А и В.
Благодаря достоинствам отмеченных выше свойств, система LiPA может рассматриваться как предпочтительный формат для гибридизационного метода, в котором необходимо одновременное определение нескольких организмов в образце. Кроме того, как описано в разделе Примеры, система LiPA является предпочтительным форматом метода отбора, заключающегося в экспериментальной оценке и отборе зондов, разработанных на основании теоретических данных.
Тем не менее должно быть ясно, что любой другой метод гибридизации, в котором различные зонды используются в одних и тех же условиях гибридизации и отмывки, может быть использован для описанных выше способов определения и/или отбора. Например, возможны иммобилизация нуклеиновой кислоты-мишени на твердой подложке и использование смеси различных зондов, помеченных различными метками, в результате чего получается различный детектируемый сигнал для каждого зонда, образовавшего гибрид с мишенью.
В качестве примера ниже приведена процедура, которой необходимо следовать для определения одного или более организмов в образце, используя формат LiPA.
- Во-первых, нуклеиновые кислоты определяемого в образце организма(ов) делают доступными для амплификации и/или гибридизации.
- Во-вторых, нуклеиновые кислоты, если они имеются, амплифицируют, как показано ниже, с помощью той или иной амплификационной системы. Обычно амплификация необходима для усиления последующего гибридизационного сигнала. Однако для некоторых образцов или некоторых организмов необходимости в амплификации нет. Это может быть также в случае использования для детекции образовавшихся гибридов высокочувствительных сигнал-усиливающих систем.
- В-третьих, по окончании стадии денатурации присутствующие в образце нуклеиновые кислоты или полученный амплификационный продукт приводят в контакт с полосками LiPA, на которых иммобилизованы один или более ДНК-зондов, позволяющих определять интересующие организмы, и проводят гибридизацию.
- В заключение, по окончании осуществления стадии отмывки гибриды детектируют с использованием удобной и совместимой детектирующей системы. Наличие или отсутствие в биологическом образце одного или нескольких организмов, на которые проводился анализ, может быть установлено на основании наблюдаемых гибридизационных сигналов или картин гибридизации.
Используемая амплификационная система может быть более или менее универсальной в зависимости от специфичности необходимой амплификации.
Применение универсальных праймеров с последовательностью, расположенной в консервативных фланкирующих участках (16S и 23S гены), спейсерной области рРНК позволяет амплифицировать спейсерную область большинства, если не всех, организмов эубактериального происхождения. Такой же результат может быть получен с использованием сочетания различных наборов праймеров с пониженной универсальностью (множественная амплификация, т.е. амплификационная процедура, в которой два или более набора праймеров используют одновременно в одной и той же реакционной смеси).
Для некоторых приложений пригодной может быть амплификация ДНК не всех присутствующих в образце организмов, а более специфическая, наперед заданных таксонов. Этого можно достичь благодаря использованию специфических праймеров, относящихся или к менее консервативным частям фланкирующих генов спейсерных областей (например, MYCP1-5 для амплификации спейсерной области микобактерий), или к самим спейсерным областям (например, LIS-P1-P7, BRU-P1-4, CHTR-P1-2 и YEC-P1-2 для специфической амплификации спейсерной области (ей) вида Lis-teria, вида Brucella, Chlamydia trachomatis и Yersinia enterocolitica, соответственно).
Таким образом настоящее изобретение предусматривает способ определения и идентификации по меньшей мере одного микроорганизма или одновременного определения различных микроорганизмов в образце, включающий стадии:
(i) при необходимости высвобождения, выделения и/или концентрирования полинуклеиновых кислот из микроорганизма(ов), определяемого в образце;
(ii) при необходимости амплификации 16S-23S рРНК спейсерной области или ее части из микроорганизма (ов), определяемого в образце, с помощью по меньшей мере одной подходящей пары праймеров;
(iii) гибридизации полинуклеиновых кислот, полученных на стадиях (i) или (ii), с набором зондов, включающим по меньшей мере два зонда, в одинаковых условиях гибридизации и отмывки, при этом указанные зонды выбраны из последовательностей, приведенных в Таблице 1а, или их эквивалентов, и/или из таксон-специфических зондов, принадлежащих любой из спейсерных последовательностей, представленных на фиг.1-103, при этом указанный таксон - специфический зонд выбран таковым, что он способен гибридизоваться в тех же самых условиях гибридизации и отмывки, что и по меньшей мере один из зондов Таблицы 1а;
(iv) детектирования гибридов, образованных на стадии (iii);
(v) идентификации присутствующего в образце микроорганизма (ов) на основании разностных гибридизационных сигналов, полученных на стадии (iv).
Все упоминаемые в Таблице 1а зонды отобраны таким образом, что проявляют при температуре гибридизации и отмывки, равной 50oC в предпочтительной среде для гибридизации и отмывки, представляющей собой 3XSSC и 20% формамид, требуемые гибридизационные свойства.
Термин "эквиваленты" зонда, называемые также "вариантами" или "гомологами" или "явными производными", относится к зондам, отличающимся по последовательности от зондов Таблицы 1а либо добавлением или удалением одного или нескольких нуклеотидов соответственно с любого из его концов, либо заменой одного или более нуклеотидов внутри указанной последовательности, либо и тем, и другим вместе, в результате чего указанные эквиваленты по-прежнему гибридизуются с той же РНК- и ДНК-мишенью, как и соответствующие немодифицированные зонды. Упоминаемые выше эквиваленты сохраняют по меньшей мере 75% гомологии, предпочтительно 80%, наиболее предпочтительно более 85% гомологии с последовательностью соответствующих немодифицированных зондов. Следует отметить, что при использовании эквивалента зонда может потребоваться изменение условий гибридизации для получения той же специфичности, что и у соответствующего немодифицированного зонда. Как следствие этого, ввиду того, что целью настоящего изобретения является применение набора зондов, работающих в одних и тех же условиях гибридизации и отмывки, будет необходимо изменить соответствующим образом последовательность других зондов, входящих в тот же самый набор, что и первоначальный немодифицированный зонд. Подобные модификации можно осуществить в соответствии с известными специалистам принципами, такими например, какие описаны в работе Hames В. и Higgins S. (Eds): Nucleic acid hybridization. Practical approach. IRL Press, Oxford, UK, 1985.
Изобретение также предусматривает способ отбора таксон- специфических зондов, принадлежащих спейсерной области (ям) указанных таксонов, причем упомянутые зонды выбираются так, что они проявляют свои требуемые гибридизационные свойства в унифицированных условиях гибридизации и отмывки.
Термин "унифицированные" условия означает, что эти условия, давая возможность определять различные таксоны, являются одними и теми же для различных зондов.
Предпочтительно, когда настоящее изобретение предусматривает описанный выше способ, в котором одновременно определяются по меньшей мере два микроорганизма.
В предпочтительном воплощении набор зондов, описанных на стадии (iii), включает по меньшей мере два зонда, выбираемые из нуклеотидных последовательностей Таблицы 1а, или их эквиваленты.
В другом воплощении набор зондов, описанных на стадии (iii), включает по меньшей мере один зонд, выбираемый из нуклеотидных последовательностей Таблицы 1а, или его эквиваленты, и по меньшей мере один таксон-специфический зонд, принадлежащий к любой из спейсерных последовательностей, представленных на фиг. 1- 103.
Еще в одном воплощении набор зондов, описанных на стадии (iii), включает по меньшей мере два таксон-специфических зонда, принадлежащих к любой из спейсерных последовательностей, представленных на фиг. 1-103.
Настоящее изобретение также предусматривает описанный выше способ, в котором зонды, указанные на стадии (iii), используются в сочетании по меньшей мере с еще одним зондом, предпочтительно также принадлежащим к 16S-23S рРНК спейсерной области, что дает возможность одновременного определения различных патогенных бактерий, возможно присутствующих в одном образце.
Наличие в биологических образцах организмов, вызывающих заболевания, может в значительной мере зависеть от происхождения образца. Наиболее общими патогенными бактериями, которые могут быть обнаружены в образцах мокроты, или образцах, взятых из дыхательного тракта, являются:
- Moraxella catarrhalis
- Streptococcus pneumoniae
- Haemophilus influenzae
- Pseudomonas aeruginosa
- Mycoplasma pneumoniae
- Acinetobacter species
- Mycobacterium species
- Staphylococcus aureus
- Legionella pneumophila.
LiPA- полоски, содержащие спейсерные зонды, позволяющие определять большинство, если не все эти организмы, будут чрезвычайно полезны по причинам, объясненным выше.
Очевидно, что это также применимо и к другим биологическим образцам, таким как: спинномозговая жидкость, пробы из мочеполового и желудочно-кишечного трактов, кровь, моча, продукты питания, почва и т.д. Например желательно, чтобы предпочтительная панель для анализа спинномозговой жидкости содержала бы сочетание зондов, дающих возможность определения и дифференциации следующих организмов:
- Neisseria meningitidis
- Streptococcus pneumoniae
- Streptococcus agalactiae
- Listeria monocytogenes
- Mycobacter iumtuberculosis.
Для некоторых из вышеупомянутых организмов спейсерные зонды уже разработаны и представлены в предыдущей заявке на изобретение (WO 91/16454). Чтобы иметь возможность определять большинство патогенных микроорганизмов в образце в одном испытании, зонды по настоящему изобретению можно применять в сочетании по меньшей мере с одним из зондов из WO 91/16454 или их очевидными производными, оговоренными в WO 91/16454. Для ясности эти зонды (1) приведены в конце текста.
Настоящее изобретение таким образом предусматривает описанный выше способ, в котором указанный образец представляет собой образец, взятый из дыхательного тракта, и в котором набор зондов, определенных на стадии (iii), включает по меньшей мере один зонд, выбранный из спейсерных зондов (2), приведенных в конце текста, и более предпочтительно из спейсерных зондов (3), приведенных в конце текста,
или эквивалентов указанных зондов,
и/или в котором набор зондов включает по меньшей мере один таксон-специфический зонд, принадлежащий последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, причем нуклеотидная последовательность указанной спейсерной области выбирается из любой из последовательностей, представленных как SEQ ID NO 76-106, 157-174, 124, 125, 111-115, 139-144 или 126-130, и при этом указанные зонды или эквиваленты возможно используются в сочетании с любым зондом, с помощью которого можно определить по меньшей мере один из следующих организмов: Haemophilus influenzae. Streptococcus pneumoniae, Moraxella catarrhalis или Bordetella pertussis.
Упомянутые выше зонды по изобретению разработаны для определения видов Mycobacterium (SEQ ID NO 1-33 и 175-191), Pseudomonas aeruginosa (SEQ ID NO 34-38), видов Mycoplasma (SEQ ID NO 49-52), Staphylococcus aureus (SEQ ID NO 53-56) и Acinetobacter baumanii (SEQ ID NO 57 и 58).
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть, семь, восемь или более вышеупомянутых зондов применяются одновременно.
Изобретение также касается описанного выше способа, в котором указанный образец представляет собой образец, взятый из спинномозговой жидкости, и в котором набор зондов, описанных на стадии (iii), включает по меньшей мере один зонд, выбранный из спейсерных зондов (4), приведенных в конце текста, и предпочтительно из спейсерных зондов (5), приведенных в конце текста,
или эквивалентов вышеупомянутых зондов,
и/или в котором набор зондов включает по меньшей мере один таксон-специфический зонд, принадлежащий последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, причем нуклеотидная последовательность указанной спейсерной области выбирается из любой из последовательностей, представленных как SEQ ID NO 116, 118-121 или 213-215, и при этом указанные зонды или эквиваленты возможно используются в сочетании с любым зондом, с помощью которого можно определить по меньшей мере один из следующих организмов: Neisseria meningitidis, Haemophilus influenzae или Streptococcus pneumoniae.
Упомянутые выше зонды по изобретению разработаны для определения видов Mycobacterium, в особенности Mycobacterium tuberculosis (SEQ ID NO 1-5), и видов Listeria, в особенности Listeria monocytogenes (SEQ ID NO 39-42).
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть, семь, восемь или более вышеупомянутых зондов применяются одновременно.
Изобретение также касается описанного выше способа, в котором вышеупомянутый образец представляет собой образец, взятый из мочеполового тракта, и в котором набор зондов, описанных на стадии (iii), включает по меньшей мере один зонд, выбранный из спейсерных зондов (6), приведенных в конце текста,
или эквивалентов вышеупомянутых зондов,
и/или в котором набор зондов включает по меньшей мере один таксон-специфический зонд, принадлежащий последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, причем нуклеотидная последовательность указанной спейсерной области выбирается из любой из последовательностей, представленных как SEQ ID NO 122, 123, 197, 124 или 125, при этом указанные зонды или эквиваленты возможно используются в сочетании с любым зондом, с помощью которого можно определить по меньшей мере один из следующих организмов: Neisseria gonorrhoeae, Haemophilus ducreyi или Streptococcus agalactiae.
Упомянутые выше зонды по изобретению разработаны для определения видов Chlamydia (SEQ ID NO 45-48 и 201) и видов Mycoplasma (SEQ ID NO 51 и 52).
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть или семь вышеупомянутых зондов применяются одновременно.
Изобретение также касается описанного выше способа, в котором вышеупомянутый образец представляет собой образец, взятый из пищи, и в котором набор зондов, определенных на стадии (iii), включает по меньшей мере один зонд, выбранный из спейсерных зондов (7), представленных в конце текста, и предпочтительно из спейсерных зондов (8), представленных в конце описания,
или эквивалентов указанных зондов,
и/или в котором набор зондов включает по меньшей мере один таксон-специфический зонд, принадлежащий последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, причем нуклеотидная последовательность указанной спейсерной области выбирается из любой из последовательностей, представленных как SEQ ID NO 116, 118-121, 213-215, 139-144, 131, 132, 154, 133-138, 195 или 196, при этом указанные зонды или эквиваленты возможно используются в сочетании с любым зондом, с помощью которого можно определить штаммы вида Campylobacter.
Упомянутые выше зонды по изобретению разработаны для определения видов Listeria (SEQ ID NO 39-44), видов Staphylococcus (SEQ ID NO 53-56), видов Brucella (SEQ ID NO 59, 60, 193 и 194), видов Salmonella (SEQ ID NO 61-64), Yersinia enterocolitica (SEQ ID NO 198-200).
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть, семь, восемь или более вышеупомянутых зондов применяются одновременно.
Изобретение также касается описанного выше способа, в котором вышеупомянутый образец представляет собой образец, взятый из желудочно-кишечного тракта пациента, и в котором набор зондов, определенных на стадии (iii), включает по меньшей мере один зонд, выбранный из спейсерных зондов (9), представленных в конце текста, и предпочтительно из спейсерных зондов (10), представленных в конце текста, или эквивалентов вышеупомянутых зондов,
и/или в котором набор зондов включает по меньшей мере один таксон-специфический зонд, принадлежащий последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, причем нуклеотидная последовательность указанной спейсерной области выбирается из любой из последовательностей, представленных как SEQ ID NO 133-138 или 195-196, при этом указанные зонды или эквиваленты возможно используются в сочетании с любым зондом, с помощью которого можно определить вид Campylobacter.
Упомянутые выше зонды по изобретению разработаны для определения видов Salmonella (SEQ ID NO 61-64) и Yersinia enterocolitica (SEQ ID NO 198-200).
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть или семь вышеупомянутых зондов применяются одновременно.
Изобретение также касается применения выбранных зондов или их эквивалентов для определения специфических бактериальных таксонов, причем указанные таксоны представляют собой либо полный род, либо подгруппу внутри рода, вид, либо даже подтип внутри вида.
Таким образом изобретение предусматривает описанный выше способ для определения и идентификации в образце одного или более штаммов вида и подвида Mycobacterium, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (11), представленных в конце текста, и более предпочтительно по меньшей мере с одним зондом из более узкой группы спейсерных зондов (12), представленных в конце текста, или с эквивалентами вышеупомянутых зондов,
и/или с любым зондом из SEQ ID NO 76-110 или 157-174, обеспечивающим специфическую гибридизацию указанного зонда с видом Mycobacterium.
Последовательности, представленные в SEQ ID NO 76-110 или 157-174, являются новыми.
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть, семь, восемь или более вышеупомянутых зондов применяются одновременно.
Как описано выше, предпочтительным более узким набором зондов являются те зонды, которые показывают чувствительность и специфичность более 80%, предпочтительно более 90%, наиболее предпочтительно - более 95% в специфических условиях гибридизации, описанных в разделе Примеры.
В одном из специфических воплощений изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более комплексных штаммов Mycobacterium tuberculosis, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MTB-ICG-1: GGGTGCATGACAACAAAGTTGGCCA (SEQ ID NO 3)
MTB-ICG-2: GACTTGTTCCAGGTGTTGTCCCAC (SEQ ID NO 4)
MTB-ICG-3: CGGCTAGCGGTGGCGTGTTCT (SEQ ID NO 5)
или с эквивалентами указанных зондов, и/или с любым зондом из SEQ ID NO 76, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с комплексом М. tuberculosis. Комплекс М. tuberculosis включает штаммы М.tuberculosis, М.bovis, М. bovis BCG, М. africanum и М.microti.
Последовательность, представленная в SEQ ID NO 76, является новой.
Предпочтительно, когда по меньшей мере два или три из вышеупомянутых зондов применяются одновременно.
В другом специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации одного или более штаммов Mycobacterium из MAIS-комплекса, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (13), представленных в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 77-100 или 108-110, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами MAIS-комплекса. По определению, принятому в настоящем изобретении, MAIS-комплекс включает все штаммы М. avium, М. intracellulare и М. scrofulaceum и все штаммы, близкородственные вышеупомянутым штаммам и неявно принадлежащие к другому определяемому виду Mycobacterium. Последняя группа штаммов определена в настоящем изобретении как "MIC-штаммы" (комплекс М. intracellulare).
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть, семь, восемь или более вышеупомянутых зондов применяются одновременно.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов М. avium и М. paratuberculosis, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MAV-ICG-1: TCGGTCCGTCCGTGTGGAGTC (SEQ ID NO 10)
MAV-ICG-22: GTGGCCGGCGTTCATCGAAA (SEQ ID NO 11)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 77 и 78, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. avium и М. paratuberculosis.
Последовательности, представленные в SEQ ID NO 77 и 78, являются новыми.
Предпочтительно, когда в данном воплощении оба зонда применяются в сочетании.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium intracellulare и MIC-штаммов, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (14), представленных в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами М. intracellulare и MIC-штаммами.
Последовательности, представленные в SEQ ID NO 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99, являются новыми.
Предпочтительно, когда по меньшей мере два, три, четыре, пять, шесть, семь, восемь или более вышеупомянутых зондов применяются одновременно.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium intracellulare, в котором стадия (iii) включает гибридизацию по меньшей мере со следующим зондом:
MIN-ICG-1: GCATAGTCCTTAGGGCTGATGCGTT (SEQ ID NO 12)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 89, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами М. intracellulare.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium scrofulaceum, в котором стадия (iii) включает гибридизацию со следующим зондом:
MSC-ICG-1: TCGGCTCGTTCTGAGTGGTGTC (SEQ ID NO 24)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 100, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. scrofulaceum.
Последовательность, представленная в SEQ ID NO 100, является новой.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium kansasii, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (15), представленных в конце текста, и более предпочтительно c зондами (16), представленными в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 101, 167, 168 или 169, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. kansasii.
Последовательности, представленные в SEQ ID NO 101, 167, 168 и 169, являются новыми.
Предпочтительно, когда по меньшей мере два, три или четыре вышеупомянутых зондов применяются одновременно.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium chelonae, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MCH-ICG-1: GGTGTGGACTTTGACTTCTGAATAG (SEQ ID NO 29)
MCH-ICG-2: CGGCAAAACGTCGGACTGTCA (SEQ ID NO 30)
MCH-ICG-3: GGTGTGGTCCTTGACTTATGGATAG (SEQ ID NO 210)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 102, 103 или 174, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. chelonae. В соответствии с другим предпочтительным воплощением эти три зонда применяются в сочетании.
Последовательности, представленные в SEQ ID NO 102, 103 и 174, являются новыми.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium gordonae, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MGO-ICG-1: AACACCCTCGGGTGCTGTCC (SEQ ID N0 31)
MGO-ICG-2: GTATGCGTТGTCGTTCGCGGC (SEQ ID N0 32)
MGO-ICG-5: CGTGAGGGGTCATCGTCTGTAG (SEQ ID N0 33)
и наиболее предпочтительно с:
MGO-ICG-5: CGTGAGGGGTCATCGTCTGTAG (SEQ ID NO 33)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 104, 105 или 106, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. gordonae.
Последовательности, представленные в SEQ ID NO 104 и 106, являются новыми.
Предпочтительно, когда по меньшей мере два или три вышеупомянутых зонда применяются одновременно.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium ulcerans или Mycobacterium marinum, в котором стадия (iii) включает гибридизацию со следующим зондом:
MUL-ICG-1: GGTTTCGGGATGTTGTCCCACC (SEQ ID NO 175)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 157, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. ulcerans и М. marinum.
Последовательность, представленная в SEQ ID NO 157, является новой.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium qenavense, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MGV-ICG-1: CGACTGAGGTCGACGTGGTGT (SEQ ID NO 176)
MGV-ICG-2: GGTGTTTGAGCATTGAATAGTGGTTGC (SEQ ID NO 177)
MGV-ICG-3: TCGGGCCGCGTGTTCGTCAAA (SEQ ID NO 211)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 158, 159, 160, 161 или 162, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. genavense.
Последовательности, представленные в SEQ ID NO 158 и 162, являются новыми.
Как описано в разделе Примеры, М. genavense включает штаммы М. genavense sensu strictu и группу близкородственных штаммов, названных М. simiae-подобными. Первая группа штаммов может быть специфически определена с помощью зонда MGV-ICG-1, в то время как последняя группа специфически гибридизуется с зондом MGV-ICG-3. С помощью зонда MGV-ICG-2 определяются обе группы.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium xenopi, в котором стадия (iii) включает гибридизацию со следующим зондом:
MXE-ICG-1: GTTGGGCAGCAGGCAGTAACC (SEQ ID NO 178)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 163, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. xenopi.
Последовательность, представленная в SEQ ID NO 163, является новой.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium simiae, в котором стадия (ili) включает гибридизацию со следующим зондом:
MSI-ICG-1: CCGGCAACGGTTACGTGTTC (SEQ ID NO 179)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 164 или 165, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М.simiae.
Последовательности, представленные в SEQ ID NO 164 или 165, являются новыми.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium fortuitum, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MFO-ICG-1: TCGTTGGATGGCCTCGCACCT (SEQ ID NO 180)
MFO-ICG-2: ACTTGGCGTGGGATGCGGGAA (SEQ ID NO 181)
или с эквивалентами указанных зондов или с любым зондом из SEQ ID NO 166, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. fortuitum.
Последовательность, представленная в SEQ ID NO 166, является новой.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium celatum, в ко тором стадия (iii) включает гибридизацию со следующим зондом:
MCE-ICG-1: TGAGGGAGCCCGTGCCTGTA (SEQ ID NO 190)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 170, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. celatum.
Последовательность, представленная в SEQ ID NO 170, является новой.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium haemophilum, в котором стадия (iii) включает гибридизацию со следующим зондом:
MHP-ICG-l: CATGTTGGGCTTGATCGGGTGC (SEQ ID NO 191)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 171, 172 или 173, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. haemophilum.
Последовательности, представленные в SEQ ID NO 171 и 173, являются новыми.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium malmoense, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MML-ICG-1: CGGATCGATTGAGTGCTTGTCCC (SEQ ID NO 188)
MML-ICG-2: TCTAAATGAACGCACTGCCGATGG (SEQ ID NO 189)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 107, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с М. malmoense.
Последовательность, представленная в SEQ ID NO 107, является новой.
В еще одном специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycobacterium, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MYC-ICG-1: ACTGGATAGTGGTTGCGAGCATCTA (SEQ ID NO 1)
MYC-ICG-22: CTTCTGAATAGTGGTTGCGAGCATCT (SEQ ID NO 2)
или с эквивалентами указанных зондов.
В соответствии с предпочтительным воплощением применяется комбинация обоих зондов.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycoplasma, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (17), представленных в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 124 или 125, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с видом Mycoplasma.
Предпочтительно, когда по меньшей мере два, три или четыре указанных зонда используются одновременно.
Более конкретно изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycoplasma pneumoniae, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
MPN-ICG-1: ATCGGTGGTAAATTAAACCCAAATCCCTGT (SEQ ID NO 49)
MPN-ICG-2: CAGTTCTGAAAGAACATTTCCGCTTCTTTC (SEQ ID NO 50)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 125, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с видом Mycoplasma pneumoniae. В соответствии с предпочтительным воплощением оба этих зонда используются в сочетании.
Последовательность, представленная в SEQ ID NO 125, является новой.
В другом конкретном воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Mycoplasma genitalium, в котором стадия (iii) включает гибридизацию со следующим зондом:
MGE-ICG-1: CACCCATTAATTTTTTCGGTGTTAAAACCC (SEQ ID NO 51)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 124, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с видом Mycoplasma genitalium.
Последовательность, представленная в SEQ ID NO 124, является новой.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Pseudomonas, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (18), представленных в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 111, 112, 113, 114 или 115, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами Pseudomonas.
Последовательности, представленные в SEQ ID NO 111 и 115, являются новыми.
Предпочтительно, когда по меньшей мере два, три или четыре вышеупомянутых зонда используются одновременно.
Более конкретно изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Pseudomonas aeruginosa, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (19), представленных в конце текста, и наиболее предпочтительно по меньшей мере с одним из зондов:
PA-ICG- 1: TGGTGTGCTGCGTGATCCGAT (SEQ ID NO 34)
PA-ICG-4:TGAATGTTCGT(G/A)(G/A)ATGAACATTGATTTCTGGTC (SEQ ID NO 37)
PA-I CG-5: CTCTTTCACTGGTGATCATTCAAGTCAAG (SEQ ID NO 38)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 111, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с Pseudomonas aeruginosa.
Последовательность, представленная в SEQ ID NO 111, является новой.
Предпочтительно, когда по меньшей мере два, три, четыре или пять вышеупомянутых зондов используются одновременно.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более видов Staphylococcus, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (20), представленных в конце текста,
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 139, 140, 141, 142, 143 или 144, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с видом Staphylococcus.
Последовательности, представленные в SEQ ID NO 139 и 144, являются новыми.
Предпочтительно, когда по меньшей мере два, три или четыре вышеупомянутых зонда используются одновременно.
Более конкретно изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Staphylococcus aureus, в котором стадия (iii) включает гибридизацию по меньшей мере с одним, а предпочтительно с обоими из следующих зондов:
STAU-ICG-3: AACGAAGCCGTATGTGAGCATTTGAC (SEQ ID NO 55)
STAU-ICG-4: GAACGTAACTTCATGTTAACGTTTGACTTAT (SEQ ID NO 56)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 139, 140, 141, 142 или 143, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с Staphylococcus aureus. В соответствии с предпочтительным воплощением оба этих зонда используются в сочетании.
В другом специфическом воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Staphylococcus epidermidis, в котором стадия (iii) включает гибридизацию с любым зондом, принадлежащим последовательности SEQ ID NO 144, такой длины, которая позволяет специфически гибридизоваться с Staphylococcus epidermidis.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более видов Acinetobacter, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
ACI-ICG-1: GCTTAAGTGCACAGTGCTCTAAACTGA (SEQ ID NO 57)
ACI-ICG-2: CACGGTAATTAGTGTGATCTGACGAAG (SEQ ID NO 58)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 126, 127, 128, 129 или 130, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с видом Acinetobacter. В соответствии с предпочтительным воплощением оба этих зонда используются в сочетании.
Последовательности, представленные в SEQ ID NO 126 и 130, являются новыми.
Более конкретно изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более видов Acinetobacter baumanii, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
ACI-ICG-1: GCTTAAGTGCACAGTGCTCTAAACTGA (SEQ ID NO 57)
ACI-ICG-2: CACGGTAATTAGTGTGATCTGACGAAG (SEQ ID NO 58)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 126, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с Acinetobacter baumanii. В соответствии с предпочтительным воплощением оба этих зонда используются в сочетании.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Listeria, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (21), представленных в конце текста, и наиболее предпочтительно по меньшей мере с одним из следующих зондов:
LIS-ICG-1: CAAGTAACCGAGAATCATCTGAAAGTGAATC (SEQ ID NO 39)
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC (SEQ ID NO 42)
LISP-ICG-1: CGTTTTCATAAGCGATCGCACGTT (SEQ ID NO 212)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 116, 118, 119, 120, 121, 213, 214 или 215, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с видом Listeria.
Как описано в разделе Примеры, вид Listeria включает штаммы Listeria sensu strictu и группу близкородственных организмов, названных "Listeria- подобными организмами". Последняя группа штаммов может быть специфически распознана с помощью зонда LISP-ICG-1.
Последовательности, представленные в SEQ ID NO 116, 118- 121 и 213-215, являются новыми.
Предпочтительно, когда по меньшей мере два, три, четыре, пять или шесть вышеупомянутых зондов используются одновременно.
Более конкретно изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Listeria monocytogenes, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
LMO-ICG-1: AAACAACCTTTACTTCGTAGAAGTAAATTGGTTAAG (SEQ ID NO 40)
LMO-ICG-2: TGAGAGGTTAGTACTTCTCAGTATGTTTGTTC (SEQ ID NO 41)
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC (SEQ ID NO 42)
и наиболее предпочтительно со следующим зондом:
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC (SEQ ID NO 42)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 120, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с Listeria monocytogenes.
Предпочтительно, когда по меньшей мере два или три вышеупомянутых зонда используются одновременно.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Brucella, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
BRU-ICG-1: CGTGCCGCCTTCGTTTCTCTTT (SEQ ID NO 59)
BRU-ICG-2: TTCGCTTCGGGGTGGATCTGTG (SEQ ID NO 60)
BRU-ICG-3: GCGTAGTAGCGTTTGCGTCGG (SEQ ID NO 193)
BRU-ICG-4: CGCAAGAAGCTTGCTCAAGCC (SEQ ID NO 194)
и наиболее предпочтительно по меньшей мере с одним из следующих зондов:
BRU-ICG-2: TTCGCTTCGGGGTGGATCTGTG (SEQ ID NO 60)
BRU-ICG-3: GCGTAGTAGCGTTTGCGTCGG (SEQ ID NO 193)
BRU-ICG-4: CGCAAGAAGCTTGCTCAAGCC (SEQ ID NO 194)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 131, 132 или 154, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами Brucella.
Последовательности, представленные в SEQ ID NO 131, 132 или 154, являются новыми.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Salmonella, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
SALM-ICG-1: CAAAACTGACTTACGAGTCACGTTTGAG (SEQ ID NO 61)
SALM-ICG-2: GATGTATGCTTCGTTATTCCACGCC (SEQ ID NO 62)
STY-ICG-1: GGTCAAACCTCCAGGGACGCC (SEQ ID NQ 63)
SED-ICG-1: GCGGTAATGTGTGAAAGCGTTGCC (SEQ ID NO 64)
и наиболее предпочтительно со следующим зондом:
SALM-ICG-1: CAAAACTGACTTACGAGTCACGTTTGAG (SEQ ID NO 61)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 133, 134, 135, 136, 137 или 138, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами Salmonella.
Последовательности, представленные в SEQ ID NO 133-138, являются новыми.
Предпочтительно, когда по меньшей мере два, три или четыре вышеупомянутых зонда используются одновременно.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Chlamydia, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (22), представленных в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 122, 123 или 197, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами Chlamydia.
Предпочтительно, когда по меньшей мере два, три, четыре или пять вышеупомянутых зондов используются одновременно.
Более конкретно изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Chlamydia trachomatis, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из зондов (23), представленных в конце текста, или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 123 или 197, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с Chlamydia trachomatis.
Последовательности, представленные в SEQ ID NO 123 и 197, являются новыми.
Предпочтительно, когда по меньшей мере два, три или четыре вышеупомянутых зонда используются одновременно.
В другом конкретном воплощении изобретение предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Chlamydia psittaci, в котором стадия (iii) включает гибридизацию по меньшей мере со следующим зондом:
CHPS-ICG-1: GGATAACTGTCTTAGGACGGTTTGAC (SEQ ID NO 48)
или с эквивалентами указанного зонда,
и/или с любым зондом из SEQ ID NO 122, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами Chlamydia psittaci.
Последовательность, представленная в SEQ ID NO 122, является новой.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Streptococcus, в котором стадия (iii) включает гибридизацию с любым из зондов, принадлежащих последовательностям SEQ ID NO 145, 146, 147, 148, 149, 150, 151, 152 или 153, обеспечивающим специфическую гибридизацию вышеупомянутого зонда со штаммами Streptococcus или с эквивалентами вышеупомянутых зондов.
Последовательности, представленные в SEQ ID NO 145, 146, 147, 148, 149, 150, 151, 152 или 153 являются новыми.
Изобретение также предусматривает описанный выше способ определения и идентификации в образце одного или более штаммов Yersinia enterocolitica, в котором стадия (iii) включает гибридизацию по меньшей мере с одним из следующих зондов:
YEC-ICG-1: GGAAAAGGTACTGCACGTGACTG (SEQ ID NO 198)
YEC-ICG-2: GACAGCTGAAACTTATCCCTCCG (SEQ ID NO 199)
YEC-ICG-3: GCTACCTGTTGATGTAATGAGTCAC (SEQ ID NO 200)
или с эквивалентами указанных зондов,
и/или с любым зондом из SEQ ID NO 195 или 196, обеспечивающим специфическую гибридизацию вышеупомянутого зонда с Yersinia enterocolitica.
Последовательности, представленные в SEQ ID NO 195 и 196, являются новыми.
В некоторых случаях бывает выгодно амплифицировать генетический материал не всех организмов, присутствующих в образце, а только тех более специфичных таксонов, которые считаются относящимися к делу. Для этих случаев настоящее изобретение предусматривает праймеры, позволяющие проводить специфическую амплификацию спейсерной области только этих заранее определенных таксонов.
Данное изобретение таким образом предусматривает описанный выше способ специфического определения и идентификации в образце Chlamydia trachomatis, в котором стадия (ii) включает амплификацию спейсерной области 16S- 23S рРНК или ее части с использованием по меньшей мере одного из следующих праймеров:
CHTR-P1: AAGGTTTCTGACTAGGTTGGGC (SEQ ID NO 69)
CHTR-P2: GGTGAAGTGCTTGCATGGATCT (SEQ ID NO 70)
или эквивалентов данных праймеров, причем вышеупомянутые эквиваленты, отличающиеся от указанных выше праймеров заменой в нуклеотидной последовательности одного или более нуклеотидов, сохраняют способность специфически амплифицировать спейсерную область Chlamydia trachomatis или ее часть.
Преимущественно используются оба праймера.
Изобретение также предусматривает описанный выше способ специфического определения и идентификации в образце вида Listeria, в котором стадия (ii) включает амплификацию спейсерной области 16S-23S рРНК или ее части с использованием по меньшей мере одного из праймеров (1), представленных в конце текста, или эквивалентов данных праймеров, причем вышеупомянутые эквиваленты, отличающиеся от указанных выше праймеров заменой в нуклеотидной последовательности одного или более нуклеотидов, сохраняют способность специфически амплифицировать спейсерную область вида Listeria или ее часть.
Изобретение также предусматривает описанный выше способ специфического определения и идентификации в образце вида Mycobacterium, в котором стадия (ii) включает амплификацию спейсерной области 16S-23S рРНК или ее части с использованием по меньшей мере одного из праймеров (2), представленных в конце текста, или эквивалентов данных праймеров, причем вышеупомянутые эквиваленты, отличающиеся от указанных выше праймеров заменой в нуклеотидной последовательности или более нуклеотидов, сохраняют способность специфически амплифицировать спейсерную область вида Mycobacterium или ее часть.
Изобретение также предусматривает описанный выше способ специфического определения и идентификации в образце вида Brucella, в котором стадия (ii) включает амплификацию спейсерной области 16S-23S pPHK или ее части с использованием по меньшей мере одного из праймеров (3), представленных в конце текста, или эквивалентов данных праймеров, причем вышеупомянутые эквиваленты, отличающиеся от указанных выше праймеров заменой в нуклеотидной последовательности одного или более нуклеотидов, сохраняют способность специфически амплифицировать спейсерную область вида Brucella или ее часть.
Изобретение также предусматривает описанный выше способ специфического определения и идентификации в образце вида Yersinia enterocolitica, в котором стадия (ii) включает амплификацию спейсерной области 16S-23S рРНК или ее части с использованием по меньшей мере одного из следующих праймеров:
YEC-P1: CCTAATGATATTGATTCGCG (SEQ ID NO 208)
YEC-P2: ATGACAGGTTAATCCTTACCCC (SEQ ID NO 209)
или эквивалентов данных праймеров, причем вышеупомянутые эквиваленты, отличающиеся от указанных выше праймеров заменой в нуклеотидной последовательности одного или более нуклеотидов, сохраняют способность специфически амплифицировать спейсерную область вида Yersinia enterocolitica или ее часть.
Настоящее изобретение также предусматривает составы, включающие по меньшей мере один из определенных выше зондов и/или праймеров.
Вышеупомянутые составы могут включать любой носитель, подложку, метку или растворитель, известные в данной области, более конкретно некоторые из меток и подложек подробно описаны в разделе Определения.
Более конкретно данное изобретение касается отдельных зондов и праймеров, определенных выше, в частности любого из зондов, специфицированных в таблице 1а, и любого из праймеров, специфицированных в таблице 1б.
В соответствии с другим воплощением настоящее изобретение касается также новых последовательностей спейсерных областей, определенных выше и показанных на фиг. 1-103 (SEQ ID NO 76-154, SEQ ID NO 157-174, SEQ ID NO 195 -197 и SEQ ID NO 213- 215).
В другом воплощении изобретение предусматривает метод обратной гибридизации, включающий любой из определенных выше зондов, в котором вышеупомянутые зонды иммобилизуются в известных позициях на твердой подложке, более предпочтительно на полоске мембраны.
В еще одном воплощении изобретение предусматривает набор для определения и идентификации по меньшей мере одного микроорганизма или одновременного определения и идентификации различных микроорганизмов в образце, включающий следующие составляющие:
(i) при необходимости, по меньшей мере одну подходящую пару праймеров, позволяющую проводить амплификацию внутрицистронной спейсерной области 16S-23S рРНК или ее части;
(ii) по меньшей мере один из определенных выше зондов;
(iii) буфер или компоненты, необходимые для приготовления буфера, позволяющего проводить реакцию гибридизации между вышеупомянутыми зондами и присутствующими в образце полинуклеиновыми кислотами или продуктами их амплификации;
(iv) раствор или компоненты, необходимые для приготовления раствора, позволяющего проводить отмывку образовавшихся гибридов в соответствующих условиях отмывки;
(v) при необходимости, средство для детекции гибридов, образовавшихся в предшествующей реакции гибридизации.
Подписи к чертежам
Фиг. 1 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium tuberculosis H37RV АТСС 27294 (SEQ ID NO 76).
Фиг. 2 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium avium ATCC 151.769 (ITG 4991) (SEQ ID NO 77).
Фиг. 3 показывает последовательность ДНК спейсерной области 16S-23S рРНК штаммов Mycobacterium paratuberculosis 316F и 2Е (SEQ ID NO 78).
Фиг. 4 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 5513 (SEQ ID NO 79).
Фиг. 5 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8695 (SEQ ID NO 80).
Фиг. 6 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8708 (SEQ ID NO 81).
Фиг. 7 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8715 (SEQ ID NO 82).
Фиг. 8 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8054 (SEQ ID NO 83).
Фиг. 9 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8737 (SEQ ID NO 84).
Фиг. 10 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8743 (SEQ ID NO 85).
Фиг. 11 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8745 (SEQ ID NO 86).
Фиг. 12 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8748 (SEQ ID NO 87).
Фиг. 13 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8752 (SEQ ID NO 88).
Фиг. 14 показывает последовательность ДНК спейсерной области 16S-23S _ рРНК _ штамма Mycobacterium intracellulare serovar 12 ITG 5915 (SEQ ID NO 89).
Фиг. 15 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium lufu ITG 4755 (SEQ ID NO 90).
Фиг. 16 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 5922 (SEQ ID NO 91).
Фиг. 17 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 1329 (SEQ ID NO 92).
Фиг. 18 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 1812 (SEQ ID NO 93).
Фиг. 19 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 5280 (SEQ ID NO 94).
Фиг. 20 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 5620 (SEQ ID NO 95).
Фиг.21 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 5765 (SEQ ID NO 96).
Фиг. 22 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 7395 (SEQ ID NO 97).
Фиг. 23 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8738 (SEQ ID NO 98).
Фиг. 24 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 926 (SEQ ID NO 99).
Фиг. 25 показывает последовательность ДНК спейсерной области 16S-23S рРНК _штамма Mycobacterium scrofulaceum ITG 4988 (SEQ ID NO 100).
Фиг. 26 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium kansasii АТСС 22478 (= ITG 4987) (SEQ ID NO 101).
Фиг. 27 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium chelonae abcessus ITG 4975 (SEQ ID NO 102).
Фиг. 28 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium chelonae chelonae ITG 9855 (SEQ ID NO 103).
Фиг. 29 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium gordonae ITG 7703 (SEQ ID NO 104).
Фиг. 30 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium gordonae ITG 7836 (SEQ ID NO 105).
Фиг. 31 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium gordonae ITG 8059 (SEQ ID NO 106).
Фиг. 32 показывает последовательность ДНК спейсерной области 16S-23S рРНК штаммов Mycobacterium malmoense ITG 4842 и ITG 4832 (SEQ ID NO 107).
Фиг. 33 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium 8757 (SEQ ID NO 108).
Фиг. 34 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8723 (SEQ ID NO 109).
Фиг. 35 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium ITG 8724 (SEQ ID NO 110).
Фиг. 36 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Pseudomonas aeruginosa UZG 5669 (SEQ ID NO 111).
Фиг. 37 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Pseudomonas pseudoalcaligenes LMG 1225 (SEQ ID NO 112).
Фиг. 38 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Pseudomonas stutzeri LMG 2333 (SEQ ID NO 113).
Фиг. 39 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Pseudomonas alcaligenes LMG 1224 (SEQ ID NO 114).
Фиг. 40 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Pseudomonas putida LMG 2232 (SEQ ID NO 115).
Фиг. 41 показывает последовательность ДНК малой спейсерной области 16S-23S рРНК штамма Listeria ivanovii CIP 7842 (SEQ ID NO 116).
Фиг. 42 показывает последовательность ДНК малой спейсерной области 16S-23S рРНК _штамма Listeria monocytogenes (SEQ ID NO 117).
Фиг. 43 показывает последовательность ДНК малой спейсерной области 16S-23S рРНК штамма Listeria seeligeri серовар 4А nr. 4268 (SEQ ID NO 118).
Фиг. 44 показывает часть последовательности ДНК большой спейсерной области 16S-23S рРНК............................. штамма Listeria ivanovii CIP 7842 (SEQ ID NO 119).
Фиг. 45 показывает последовательность ДНК большой спейсерной области 16S-23S рРНК штамма Listeria monocytogenes 1НЕ серовар 4В (SEQ ID NO 120).
Фиг. 46 показывает последовательность ДНК большой спейсерной области 16S-23S рРНК штамма Listeria seeligeri серовар 4А nr. 4268 (SEQ ID NO 121).
Фиг. 47 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Chlamydia psittaci 6ВС (SEQ ID NO 122).
Фиг. 48 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Chlamydia trachomatis (SEQ ID NO 123).
Фиг. 49 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycoplasma genitalium (U. Gobel) (SEQ ID NO 124).
Фиг. 50 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycoplasma pneumoniae АТСС 29432 (SEQ ID NO 125).
Фиг. 51 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Acinetobacter baumanii LMG 1041 (SEQ ID NO 126).
Фиг. 52 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Acinetobacter calcoaceticus LMG 1046 (SEQ ID NO 127).
Фиг. 53 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Acinetobacter haemolyticus LMG 996 (SEQ ID NO 128).
Фиг. 54 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Acinetobacter johnsonii LMG 999 (SEQ ID NO 129).
Фиг. 55 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Acinetobacter junii LMG 998 (SEQ ID NO 130).
Фиг. 56 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Brucella melitensis NIDO Биовар 1 (SEQ ID NO 131).
Фиг. 57 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Brucella suis NIDO Биовар 1 (SEQ ID NO 132).
Фиг. 58 показывает последовательность ДНК одной из спейсерных областей 16S-23S рРНК штамма Salmonella dublin (SEQ ID NO 133).
Фиг. 59 показывает последовательность ДНК одной из спейсерных областей 16S-23S рРНК штамма Salmonella dublin (SEQ ID NO 134).
Фиг. 60 показывает последовательность ДНК одной из спейсерных областей 16S-23S рРНК штамма Salmonella enteritidis (SEQ ID NO 135).
Фиг. 61 показывает последовательность ДНК одной из спейсерных областей 16S-23S рРНК штамма Salmonella enteritidis (SEQ ID NO 136).
Фиг. 62 показывает последовательность ДНК одной из спейсерных областей 16S-23S рРНК штамма Salmonella typhimurium (SEQ ID NO 137).
Фиг. 63 показывает последовательность ДНК одной из спейсерных областей 16S-23S рРНК штамма Salmonella typhimurium (SEQ ID NO 138).
Фиг. 64 показывает последовательность ДНК одной из спейсерных областей 16S-23S _рРНК _штамма Staphylococcus aureus UZG 5728 (SEQ ID NO 139).
Фиг. 65 показывает последовательность ДНК одной из спейсерных областей 16S-23S _рРНК _штамма Staphylococcus aureus UZG 6289 (SEQ ID NO 140).
Фиг. 66 показывает последовательность ДНК одной из спейсерных областей 16S-23S _рРНК _штамма Staphylococcus aureus UZG 6289 (SEQ ID NO 141).
Фиг. 67 показывает последовательность ДНК одной из спейсерных областей 16S-23S _рРНК _штамма Staphylococcus aureus UZG 6289 (SEQ ID NO 142).
Фиг. 68 показывает последовательность ДНК одной из спейсерных областей 16S-23S _рРНК _штамма Staphylococcus aureus UZG 6289 (SEQ ID NO 143).
Фиг. 69 показывает последовательность ДНК одной из спейсерных областей _ _ 16S-23S __рРНК_____штамма Staphylococcus epidermidis UZG CNS41 (SEQ ID NO 144).
Фиг. 70 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK _штaммa Streptococcus mitis UZG 2465 (SEQ ID NO 145).
Фиг. 71 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK _штaммa Streptococcus pyogenes UZG _3671 (SEQ ID NO 146).
Фиг. 72 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK __штамма Streptococcus sanguis UZG 1042 (SEQ ID NO 147).
Фиг. 73 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK _штaммa Streptococcus saprophyticus UZG CNS46 (SEQ ID NO 148).
Фиг. 74 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK _штамма Streptococcus species UZG 536 (84) (SEQ ID NO 149).
Фиг. 75 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK _штамма Streptococcus species UZG 4341 (SEQ ID NO 150).
Фиг. 76 показывает последовательность ДНК спейсерной области 16S-23S _ рРНК _штамма Streptococcus species UZG 457 (44В) (SEQ ID NO 151).
Фиг. 77 показывает последовательность ДНК спейсерной области 16S- 23S _ pPHK _штамма Streptococcus species UZG 97А (SEQ ID NO 152).
Фиг. 78 показывает последовательность ДНК спейсерной области 16S-23S _ pPHK _штамма Streptococcus species UZG 483 (76) (SEQ ID NO 153).
Фиг. 79 показывает последовательность ДНК спейсерной области 16S- 23S _ pPHK _штамма Brucella abortus NIDO Tulya биовар_3 (SEQ ID NO 154).
Фиг. 80 показывает последовательность ДНК спейсерной области 16S-23S рРНК штаммов Mycobacterium ulcerans ITG 1837 и Mycobacterium marinum ITG 7732 (SEQ ID NO 157).
Фиг. 81 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium genavense ITG 8777 (SEQ ID NO 158).
Фиг. 82 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium genavense ITG 92-742 (SEQ ID NO 159).
Фиг. 83 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium genavense ITG 9500 (SEQ ID NO 160).
Фиг. 84 показывает последовательность ДНК спейсерной области 16S-23S рРНК Mycobacterium simiae-подобного штамма ITG 7379 (SEQ ID NO 161).
Фиг. 85 показывает последовательность ДНК спейсерной области 16S-23S рРНК Mycobacterium simiae-подобного штамма ITG 9745 (SEQ ID NO 162).
Фиг. 86 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium xenopi ITG 4986 (SEQ ID NO 163).
Фиг. 87 показывает последовательность ДНК спейсерной области 16S- 23S рРНК штамма Mycobacterium simiae А_ ITG 4485 (SEQ ID NO 164).
Фиг. 88 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium simiae В ITG 4484 (SEQ ID NO 165).
Фиг. 89 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium fortuitum ITG 4304 (SEQ ID NO 166).
Фиг. 90 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium kansasii ITG 6328 (SEQ ID NO 167).
Фиг. 91 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium kansasii ITG 8698 (SEQ ID NO 168).
Фиг. 92 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium kansasii ITG 8973 (SEQ ID NO 169).
Фиг. 93 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium celatum ITG 94-123 (SEQ ID NO 170).
Фиг. 94 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium haemophilum ITG 776 (SEQ ID NO 171).
Фиг. 95 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium haemophilum ITG 778 (SEQ ID NO 172).
Фиг. 96 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Mycobacterium haemophilum ITG 3071 (SEQ ID NO 173).
Фиг. 97 показывает последовательность ДНК спейсерной области 16S-23S рРНК штаммов Mycobacterium chelonae ITG 94-330 и ITG 94-379 (SEQ ID NO 174).
Фиг. 98 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Yersinia enterocolitica P95 (SEQ ID NO 195).
Фиг. 99 показывает последовательность ДНК спейсерной области 16S-23S рРНК штамма Yersinia enterocolitica P95 (SEQ ID NO 196).
Фиг. 100 показывает последовательность ДНК спейсерной области 16S- 23S рРНК штамма Chlamydia trachomatis SSDZ 94 M 1961 (SEQ ID NO 197).
Фиг. 101 показывает последовательность ДНК спейсерной области 16S-23S рРНК Listeria-подобного изолята MB 405 (SEQ ID NO 213).
Фиг. 102 показывает последовательность ДНК спейсерной области 16S-23S рРНК Listeria-подобного изолята MB 405 (SEQ ID NO 214).
Фиг. 103 показывает последовательность ДНК спейсерной области 16S-23S рРНК Listeria-подобного изолята MB 405 (SEQ ID NO 215).
Подписи к таблицам
Таблица 1а: Список всех новых зондов, происходящих из спейсерной области 16S-23S рРНК.
Таблица 1б: Список возможных праймеров для таксон-специфической амплификации спейсерной области или ее части.
Таблица 2: Результаты гибридизации для Pseudomonas.
Таблица 3: Картина гибридизации различных зондов, полученная для микобактериальных (под) видов.
Таблица 4: Микобактериальные штаммы, протестированные в LiPA.
Таблица 5: Результаты гибридизации для Listeria (Зонды LMO1, 2, LSE1, LIV1, LIS1).
Таблица 6: Результаты гибридизации для Listeria (Зонды LMО3, LIS1).
Таблица 7: Результаты гибридизации для Chlamydia.
Таблица 8: Новые микобактериальные зонды и результаты гибридизации.
Таблица 9: Результаты гибридизации для Brucella.
Таблица 10: Результаты гибридизации для Staphylococcus.
Пример 1: Pseudomonas aeruginosa
Pseudomonas aeruginosa является важным, обычно в смысле серьезных скрытых заболеваний, патогеном человека. Кроме этого он представляет собой причину нозокоминальных инфекций, для которых характерна устойчивость к антимикробным агентам. Эта грамотрицательная, неферментирующая палочка может являться причиной различных клинических проявлений, подобных раневым инфекциям, бактериемии, инфекциям дыхательного и мочевого тракта, а также главной причиной заболеваемости и смертности пациентов от муковисцидоза.
В настоящее время виды Pseudomonas различают на основании ростовых характеристик и отдельных биохимических свойств, подразумевающих необходимость затрат времени от 24 до 72 ч для корректной идентификации патогена.
Полученные к настоящему времени моноклональные и поликлональные антитела значительно улучшили идентификацию видов Pseudomonas. Однако недавно было показано, что возможно прямое обнаружение данного типа микроорганизмов в клинических образцах с помощью очень чувствительного и специфичного метода, использующего зонды ДНК с предварительной или без предварительной амплификации ДНК-мишени.
ДНК-зонды, применяемые для изучения Pseudomonas aeruginosa, описаны и в основном используются для эпидемиологического типирования [41, 48, 36]. Однако ни один из этих зондов не принадлежит 16S-23S спейсерной области.
Спейсерная область между генами 16S-23S рРНК и часть гена 23S рРНК амплифицировали с помощью имеющихся праймеров (верхний праймер: TGGGGTGAAGTCGTAACAAGGTA, SEQ ID NO 155; нижний праймер: CCTTTCCCTCACGGTACTGGT, SEQ ID NO 156), используя полимеразную цепную реакцию для следующих видов:
- Pseudomonas aeruginosa 5669
- Pseudomonas alcaligenes LMG 1224T
- Pseudomonas fluorescens LMG 5167
- Pseudomonas putida LMG 2232
- Pseudomonas stutzeri LMG 2333T
- Pseudomonas pseudoalcaligenes LMG 1225T
Для облегчения клонирования полученных ампликонов в нижний праймер был введен сайт узнавания NotI. После очистки и обработки фрагмента с помощью NotI ампликон клонировали в обработанный EcoRV/NotI pBluescript SK+ плазмидный вектор.
Секвенирование спейсерной области 16S-23S рРНК выполняли с помощью дидезокситерминирующего рост цепи метода, либо используя двухцепочечную плазмидную ДНК, объединенную с праймерами, расположенными в плазмидном векторе, либо определяя последовательность непосредственно очищенных PCR- продуктов, объединенных с внешними PCR-праймерами.
На фиг. 36-40 представлены нуклеотидные последовательности спейсерных областей 16S-23S рРНК различных описанных выше видов Pseudomonas. Для P. fluorescens была получена только часть нуклеотидной последовательности.
На основании нуклеотидной последовательности спейсерной области штамма P. aeruginosa 5669 были выбраны и химически синтезированы пять олигонуклеотидных зондов следующих последовательностей:
РА1 = PA-ICG 1: TGGTGTGCTGCGTGATCCGATA
РА2 = PA-ICG 2: TGAATGTTCGTGGATGAACATTGATT
РА3 = PA-ICG 3: CACTGGTGATCATTCAAGTCAAG
Тестирование специфичности и чувствительности олигонуклеотидных зондов проводили с использованием метода обратной гибридизации. Геномную ДНК различных тестируемых бактерий амплифицировали, применяя биотинилированные праймеры (те же праймеры, что и для процедуры клонирования, см. выше). Полученный ампликон, перекрывающий 16S- 23S рРНК спейсерную область, денатурировали и гибридизовали с нанесенными в виде линий на мембранные полоски различными олигонуклеотидными зондами (LiPA). Гибридизацию проводили в смеси 3xSSC (1xSSC = 0,15 М NaCl, 0,015 М цитрат натрия, pH 7,0) и 20% формамида (FA) при температуре 50oC в течение 1 часа. Отмывку проводили в той же самой смеси при той же температуре в течение 15 мин.
Гибриды детектировали, используя конъюгат стрептовидина с щелочной фосфатазой, и визуализацию зондов осуществляли посредством реакции осаждения, применяя NBT (нитротетразолий синий) и BCIP (бромхлориндолилфосфат).
Результаты гибридизации с зондами РА1, РА2 и РА3, приведенные в таблице 4, показывают, что зонды РА1 и РА3 обладают 100% специфичностью к Pseudomonas aeruginosa и гибридизуются с генетическим материалом всех протестированых штаммов. Гибридизационный сигнал, получаемый с зондом РА3 при 50oC, не был оптимальным, поэтому данный олигонуклеотидный зонд был улучшен посредством добавления нескольких дополнительных нуклеотидов. Этот вновь созданный зонд получил номер РА5.
РА5: = PA-ICG 5: CTCTTTCACTGGTGATCATTCAAGTCAAG
Эксперименты по гибридизации с зондом РА5 подтвердили его 100% специфичность и 100% чувствительность к P. aeruginosa.
Олигонуклеотидный зонд РА2 гибридизовался только с 5 из 17 протестированных штаммов P. aeruginosa. Прямое секвенирование спейсерных областей 16S-23S рРНК тех штаммов, которые не гибридизовались с данным зондом, показало наличие некоторой гетерогенности между различными штаммами. Наблюдали несоответствие оснований в двух точках по отношению к первоначально разработанному зонду РА2. Для преодоления последствий гетерогенности между различными штаммами в области связывания зонда РА2, был разработан новый зонд РА4. В этом зонде заменены позиции, в которых не было спаривания оснований, и добавлено несколько дополнительных нуклеотидов для улучшения гибридизационного сигнала при 50oC.
PA4 = PA-ICG 4: TGAATGTTCGT(G/A)(G/A)ATGAACATTGATTTCTGGTC
Как показали результаты гибридизации, данный измененный зонд демонстрировал 100% специфичность и 100% чувствительность.
Пример 2: Mycobacterium
Большое число видов микобактерий может вызывать серьезные инфекционные заболевания человека. Известными примерами являются Mycobacterium tuberculosis и Mycobacterium leprae. В последнее время другие виды, такие как M.avium, М. intracellulare и М. kansasii, все более часто встречаются как патогены человека, особенно у людей с ослабленным иммунитетом.
Вследствие этого лабораторная диагностика микобактериальных инфекций не должна ограничиваться только диагностикой комплекса М. tuberculosis, а в идеале должна включать диагностику большинства других клинически важных видов микобактерий.
Идентификация и дифференциация патогенных микобактерий на видовом уровне с помощью обычных лабораторных методик в целом является трудной и требующей больших затрат времени процедурой.
Для преодоления этих проблем были введены в обращение методики, основанные на анализе ДНК. Описанные методики такого вида простираются от простого использования ДНК- зондов до автоматического секвенирования. О некоторых подходах недавно сообщалось в литературе [26, 17, 55, 47, 58, 54, 4].
Однако все эти методы имеют свои собственные недостатки и большинство из них остается рассчитанным на работу с культурами клеток. Более того, и это наиболее важно, ни одна из этих методик не позволяет проводить одновременное определение различных клинически значимых видов микобактерий в единичном тесте. Кроме того дифференциация отдельных групп внутри комплекса Mycobacterium avium-intracellulare является проблематичной и часто едва ли возможной.
Для преодоления вышеупомянутых недостатков был разработан LiPA-тест, позволяющий проводить одновременное и надежное определение и дифференциацию ряда видов и групп Mycobacterium.
Все наборы зондов, используемых для достижения этих целей, принадлежат спейсерной области 16S-23S рРНК. Применяемые способы аналогичны упомянутым в Примере 1.
Спейсерную область 16S-23S рРНК и часть фланкирующих 16S и 23S рРНК генов амплифицировали при помощи PCR с праймерами для рода Mycobacterium. По меньшей мере один из следующих праймеров, принадлежащих 16S-гену, использовали в качестве верхнего праймера:
MYC-P1: TCCCTTGTGGCCTGTGTG (SEQ ID NO 65)
MYC-P5: CCTGGGTTTGACATGCACAG (SEQ ID NO 192)
По меньшей мере один из следующих праймеров, принадлежащих 23S -гену, использовали при амплификации в качестве нижнего праймера:
MYC-P2: TCCTTCATCGGCTCTCGA (SEQ ID NO 66)
MYC-P3: GATGCCAAGGCATCCACC (SEQ ID NO 67)
MYC-P4: CCTCCCACGTCCTTCATCG (SEQ ID NO 68)
Все упомянутые выше праймеры амплифицировали спейсерную область всех протестированных штаммов Mycobacterium за исключением праймера MYC-P2, который не был функционален в отношении М. chelonae. Для повышения чувствительности определения иногда проводили nested PCR, используя Р5 и Р4 в качестве внешних праймеров и Р1 и Р3 в качестве внутренних.
Для создания и отбора зондов и комбинаций зондов, удовлетворяющих нашей задаче, было проведено секвенирование спейсерной области 16S-23S рРНК ряда штаммов микобактерий. Полученные нуклеотидные последовательности сравнивали между собой и с последовательностями, уже известными из литературы ([17, 18, 27, 52, EP-A-0395292, 57]) или из доступных банков данных. Соответствующие последовательности представлены на фиг. 1-35 (ОТ SEQ ID NO 76 ДО SEQ ID NO 110).
Все зонды, полученные на основании этих данных, были выверены таким образом, чтобы желаемый характер гибридизации достигался в унифицированных условиях гибридизации и отмывки (т.е., 3xSSC, 20% деионизованного формамида, 50oC). Набор выверенных зондов, используемых для гибридизации с целью дифференциации штаммов микобактерий, представлен в таблице 1а, SEQ ID NO 1-33. Отметьте, пожалуйста, что номенклатура зондов, используемая в данном примере, представляет собой сокращенную версию используемой в таблице 1а номенклатуры, а именно: буквы "ICG" везде опущены. Согласно полученной картине гибридизации протестированные штаммы могли быть приписаны к одному из следующих видов или видовых групп: комплексу М. tuberculosis, М. avium, М. intracellulare или комплексу М. intracellulare, М. kansasii, М. chelonae и М. gordonae. Протестированные штаммы, принадлежащие каждой группе, суммированы в таблице 4. Все штаммы были получены из Института Тропической Медицины, Антверпен, Бельгия. Результаты гибридизации различных зондов, полученные для каждой группы, приведены в таблице 3 и более детально обсуждены ниже.
Комплекс М. tuberculosis
Комплекс М. tuberculosis содержит все штаммы, принадлежащие к М. tuberculosis, М. bovis, М. africanum и М. microti. Зонды Mtbl, Mtb2 и Mtb3 гибридизуются с ДНК всех исследуемых штаммов М. tuberculosis. Ни один из других протестированных штаммов не гибридизовался с указанными зондами в использованных условиях. Вдобавок штаммы комплекса М. tuberculosis, так же как в случае со всеми другими протестированными штаммами микобактерий, гибридизуются либо с myc1, либо с myc22-зондом, либо с ними обоими. Последние два зонда были разработаны как общие зонды на Mycobacterium, используемые поодиночке или в сочетании друг с другом.
M.avium/M. paratuberculosis
Все изученные штаммы М. avium и М. paratuberculosis показывают одинаковую картину гибридизации с набором зондов. Для этого типа организмов позитивные гибридизационные сигналы получены с зондами myc1/myc22, mail, mil111, mav1, mah1 и mav22. Последние два зонда гибридизуются исключительно со штаммами М. avium и М. paratuberculosis и могут поэтому быть использованы в качестве видоспецифических зондов. Так как последовательности 16S-23S спейсерных областей изолятов М. avium и изолятов М. paratuberculosis являются идентичными или почти идентичными, эти два таксона не могут быть различимы между собой. Этот обнаруженный факт подтверждает данные секвенирования 16S рРНК, указывающие на то, что М. avium и М. paratuberculosis фактически следует считать принадлежащими к одному геновиду [43], М. avium ssp. avium и М. avium ssp. paratuberculosis.
М.intracellulare и комплекс М.intracellulare (MIC)
Генотипически МТС-штаммы представляют собой близкородственные организмы, которые в соответствии с данными последовательности 16S-23S спейсерной области принадлежат к отдельному кластеру, отличающемуся от остальных видов Mycobacterium. М. avium и М. scrofulaceum являются их ближайшими родственниками. Почти все протестированные штаммы, которые обычно относят к штаммам комплекса М. avium (MAC) (бывший MAIS-комплекс), могут быть обнаружены в группе MIC. Таким образом MIC-группа, определенная в настоящем изобретении, включает штаммы МАС-типа, описанные в [17], за исключением MAC-G, который, как оказалось, является М. scrofulaceum. Штаммы М. intracellulare sensu stricto (М.intracellulare s.s.) также являются частью данного кластера.
Ввиду того, что эта MIC-группа содержит довольно большую группу штаммов с подгруппами среди них, проявляя различные гибридизационные свойства с набором зондов, был рассмотрен вопрос о дальнейшем подразделении внутри MIC-типов.
Тип MICl включает М. intracellulare s.s. вместе с некоторыми другими МАС-штаммами. Все без исключения изоляты МIС1-типа гибридизуются со следующими зондами: myc1/myc22, mai1 и mac1. Следующие зонды могут быть использованы для дальнейших подразделений внутри MIC1-группы: mil11, min1, min2 до 2222, mil22 и mhef1. Штаммы М. intracellulare sensu stricto (тип MIC 1.1. а) можно отличить от других подтипов этой группы посредством использования зонда min1, который позитивен только для этой группы штаммов. Все штаммы из штаммов типа MIC 1.1.а позитивны, когда тестируются с помощью зонда на М.intracellulare диагностической системы Ген-Зонд для быстрого определения MAC.
Тип MIC 1.1. b и тип MIC 1.2 включают штаммы, являющиеся близкородственными с М.intracellulare. Они могут быть дифференцированы с помощью зондов mil11 и mil22 (см. таблицу 3). Дальнейшее подразделение внутри этих групп не предпринималось, хотя оно могло быть достигнуто с использованием зондов: min2, min22, min222 и min2222. Дальнейшее подразделение могло быть ценным по эпидемиологическим причинам.
Только два из протестированных штаммов нашей коллекции классифицируются как штаммы MIC 2-типа. Один из них представляет собой штамм "Mycobacterium lufu" (ITG 4755). Специфическая картина гибридизации с зондами, даваемая этими штаммами, характеризуется позитивным гибридизационным сигналом со следующими зондами: myc1/myc22, mai1, mil22, mah1 и mal1. Вариабельные результаты гибридизации получены для зондов min2222, mac1 и mhefl. Остальные зонды дают негативные результаты. Вполне вероятно, что, когда будет идентифицировано большее число штаммов этого типа, MIC 2 может в конечном счете оказаться гетерогенной группой. Зонды, дающие вариабельные результаты, могут помочь в будущей дифференциации, если это потребуется.
Тип MIC 3 объединяет довольно большое число MAC-штаммов, являющихся отдаленно родственными по отношению к штаммам М. intracellulare s.s. и к большинству других МАС-штаммов. Этот кластер следует считать генотипически отличным от М. avium и М. intracellulare. Все подтипы МIС 3 гибридизуются с зондами mic1/mic22, mai1, mil22 и mco1. Позитивный сигнал с последним зондом (mco1) характерен для штаммов MIC 3. Вариабельные результаты гибридизации получены со следующими зондами: mac1, mhef1 и mah1. MIC 3 можно подразделить далее на четыре подтипа с использованием трех зондов: mth11, mth2 и mef11. Зонд mth2 специфичен для типа MIC 3.1, который включает группу близкородственных МАС-штаммов, выделенных у людей с ослабленным иммунитетом. Большинство штаммов MIC 3 локализовано в MIC 3.1- подтипе. В конечном счете этой группе штаммов может быть приписан видовой статус, как и в случае других групп МАС-штаммов, до сих пор не имеющих названия. В подтипах MIC 3.4, MIC 3.3 и MIC 3.2 при тестировании нашей коллекции штаммов были обнаружены только два, один и один штамм, соответственно.
Тип MIC 4 представляет собой коллекцию "MAIS"-штаммов (включающих М. malmoense), отдаленно родственных М. intracellulare. Единственным зондом из вышеописанного набора, который гибридизуется с MIC 4 отдельно от общих зондов mic1/mic22, является зонд mai1. Данный зонд показывает широкую специфичность, гибридизуясь также с М. avium, М.intracellulare и другими MIC-штаммами, и М.scrofulaceum.
М.scrofulaceum
Все протестированные штаммы М. scrofulaceum показывают одинаковую гибридизационную картину с набором зондов. Позитивный сигнал с зондом msc1 является уникальным для штаммов М. scrofulaceum. Из других зондов позитивный сигнал на этот вид явно дают myc1/myc22 и также mai1.
М. kansasii
Зонды mka3 и mka4 специфичны для М. kansasii, то есть при использовании в гибридизации амплифицированной ДНК из М. kansasii на полоске LiPA получается отчетливый позитивный сигнал, пока со всеми другими тестируемыми организмами сигнал отсутствует. Несмотря на то, что нуклеотидная последовательность зондов mka1 и mka2 не является абсолютно комплементарной последовательности мишени (3 и 1 неспаренных нуклеотида, соответственно), полезность этих зондов также доказана в условиях их применения (50oC, 3xSSC, 20% формамида) ввиду их способности гибридизоваться исключительно с ДНК М. kansasii и ни с какой другой протестированной микобактериальной ДНК. Это показывает, что зонды не обязательно должны быть совершенно комплементарны последовательности мишени, чтобы быть полезными, и что изменения в последовательности и длине до некоторой степени являются допустимыми.
М. chelonae
Вид М. chelonae включает штаммы М. chelonae ssp. chelonae и М. chelonae ssp. abscessus. Секвенировали спейсерную область для одного штамма из каждого подтипа и между ними наблюдали небольшие отличия (SEQ ID NO 103 и SEQ ID NO 102). Зонды mch1 и mch2 гибридизуются с обоими штаммами. Все остальные зонды, за исключением myc1/myc22, негативны по отношению к этим двум штаммам.
В процессе тестирования зондов mch1 и mch2 с двумя дополнительными штаммами М. chelonae, не упоминаемыми в таблице 4, то есть со штаммами М. chelonae 94-379 и М. chelonae 94-330, полученными из Института Тропической Медицины (Антверпен, Бельгия), оказалось, что они не гибридизуются с зондом mch1. Данное наблюдение было подтверждено секвенированием спейсерной области этих двух штаммов (SEQ ID NO 184). Кластерный анализ спейсерной области с другими микобактериями показал, что штаммы М. chelonae могут быть подразделены на две группы. Для специфического определения этой второй группы штаммов, к которой принадлежат штаммы 94-379 и 94-330, был разработан третий зонд mch3.
Данный результат является иллюстрацией того, как использование ДНК- зондов, относящихся к спейсерной области 16S-23S рРНК, может быть полезным в дифференциации различных групп штаммов, принадлежащих одному виду по результатам классических методов идентификации, и что данные зонды возможно могут быть использованы для определения и описания новых видов среди микобактерий. В данном случае с помощью mch2 можно определять все штаммы М. chelonae, в то время как с помощью mch1 и mch3 можно проводить дифференциацию между различными подгруппами.
М. gordonae
Все протестированные штаммы М. gordonae гибридизуются зондом mgo5. Позитивные гибридизационные сигналы также получены с зондами mic1/mic22, и кроме того некоторые штаммы М. gordonae гибридизуются с зондами mgo1 и mgo2.
Другие виды микобактерий
Штаммы, принадлежащие к другим, нежели вышеупомянутые, видам микобактерий, гибридизуются только с общими зондами myc1/myc22. Это указывает на то, что данные штаммы вероятнее всего принадлежат роду Mycobacterium, но не принадлежат ни к одному из видов и групп, которые могут быть специфически идентифицированы с помощью одного или более из остальных описанных зондов.
В заключение мы может утверждать, что использование определенных сочетаний зондов по изобретению может обеспечить различный уровень проведения ДНК-зондовых тестов.
Когда все зонды применяют в одном и том же LiPA-тесте, может быть достигнута как дифференциация на видовом уровне, так и субтипирование конкретных групп микобактерий. Однако совокупность зондов на одной полоске ограничена теми зондами, которые являются видоспецифичными; в этом случае идентификацию проводят на видовом уровне. Дальнейшее уменьшение числа зондов на полоске может привести к специфическому определению только одного или лишь нескольких видов. Очевидно могут быть разработаны LiPA-полоски, с помощью которых можно попытаться проводить только субтипирование штаммов, например штаммов, принадлежащих комплексу М. intracellulare (MIC). В зависимости от конкретных нужд лабораторий, проводящих диагностику и/или типирование микобактерий, все эти различные приложения могут представлять ценность. Однако очевидно, что за счет использования сочетания зондов в LiPA-формате количество получаемой для идентификации присутствующих в клиническом образце организмов информации значительно больше по сравнению с ДНК-зондовыми тестами, использующими только единичный зонд. Для некоторых групп или, по меньшей мере, для дальнейшего подразделения некоторых групп невозможна разработка единичного зонда, гибридизующегося уникально с данной (под) группой. В этом случае необходимую информацию в состоянии обеспечить только анализ гибридизационных картин зондов. Для этих приложений LiPA представляет собой наиболее полезный формат.
Пример 3: Listeria
Вид Listeria представляет собой группу грамположительных палочек, широко распространенных в природе. Среди этой группы, по- видимому, только штамм L. monocytogenes является патогенным для человека и животных. L. monocytogenes является причиной листериоза, вызывающего менингит, выкидыши, энцефалит и септицемию. Индивидуумы с ослабленной иммунной системой, новорожденные и беременные женщины представляют собой группу повышенного риска в отношении этих болезней, распространяющихся через пищу. Большинство случаев заболевания вызывается употреблением пищи животного происхождения, в особенности мягких сыров. Поэтому пища не должна содержать L. monocytogenes. В некоторых странах система мер безопасности требует отсутствия всех видов Listeria в пищевых продуктах.
Классический способ идентификации L. monocytogenes в продуктах сыроварения включает обогащающее культивирование в течение 48 ч с последующим рассевом на селективной агаровой среде для образования колоний (48 ч) и полным набором биохимических и морфологических анализов [15]. Данная процедура может быть значительно упрощена за счет использования генетических зондов.
Несколько ДНК-зондов для идентификации L. monocytogenes уже описаны в литературе. Некоторые из них принадлежат генам, отвечающим за патогенность организма, например гену листериолизина О [12] или гену инвазия- ассоциированного белка (iap) [6].
Коммерчески доступная система идентификации, основанная на 16S рРНК зонде, была введена GenProbe [22, 40].
Специфические зонды применяют для проведения подтверждающих анализов, проводимых на колониях, полученных после обогащения и рассеивания на чашки с селективной агаровой средой.
Недавно в нескольких публикациях появились сообщения об использовании полимеразной цепной реакции для амплификации области, являющейся мишенью для ДНК-зондов, сокращающей время и не влияющей на специфичность и чувствительность анализа. Описаны различные наборы праймеров, специфически амплифицирующие ДНК L. monocytogenes. Данные наборы содержат праймеры, принадлежащие гену листериолизина О [20] и iap-гену [25].
Для разработки родоспецифичного зонда на Listeria и зонда, специфичного на Listeria monocytogenes, мы использовали в качестве мишени спейсерную область 16S-23S рРНК генов.
Спейсерную область амплифицировали посредством полимеразной цепной реакции, используя имеющиеся праймеры, принадлежащие 3'-концу гена 16S рРНК и 5'- концу гена 23S рРНК (нуклеотидные последовательности приведены в примере 1), с последующим клонированием в подходящий плазмидный вектор, следуя аналогичным методикам из примера 3.
Были получены два ампликона, различающиеся по длине (800 п.н. и 1100 п. н.). Оба PCR-фрагмента отклонировали для следующих видов Listeria:
- Listeria monocytogenes, серовар 4b, IHE (Институт Гигиены и Эпидемиологии, Бельгия).
- Listeria ivanovii, _CIP 72.48 (Национальная коллекция культур микроорганизмов института Пастера, Франция).
- Listeria seeligeri, серовар 4a, номер 42.68 (Институт Бактериологии, Sudd, Versuchs- und Forschungsanstalt fur Milchwirtschaft Weihenstephan, Германия).
Нуклеотидную последовательность спейсерной области между генами 16S и 23S рРНК установили, используя клонированный материал из PCR-фрагмента в 800 п. н. , и данная процедура была проделана для трех описанных видов Listeria. На фиг. 41-43 приведены полученные нуклеотидные последовательности различных коротких спейсерных областей. Последовательность такой короткой спейсерной области для L. monocytogenes кроме того была взята из EMBL банка данных (LMRGSPCR).
На основании этой информации по последовательностям были отобраны и химически синтезированы следующие олигонуклеотиды для видоспецифичного определения данных микроорганизмов:
LMO-ICG-1: AAACAACCTTTACTTCGTAGAAGTAAATTGGTTAAG
LMO -ICG-2: TGAGAGGTTAGTACTTCTCAGTATGTTTGTTC
LSE-ICG-1: AGTTAGCATAAGTAGTGTAACTATTTATGACACAAG
LIV-ICG-1: GTTAGCATAAATAGGTAACTATTTATGACACAAGTAAC
Кроме того был разработан родоспецифичный зонд на Listeria:
LIS-ICG-1: CAAGTAACCGAGAATCATCTGAAAGTGAATC
Олигонуклеотидные зонды иммобилизовали на мембранной полоске и после гибридизации с биотинилированными PCR-фрагментами гибриды визуализировали посредством реакции преципитации. Результаты гибридизации для различных видов Listeria суммированы в табл. 5.
Полученные результаты показывают, что зонд LIS1 пригоден для определения всех описанных видов Listeria, но также и что видоспецифичные зонды дают перекрестную гибридизацию. Следовательно, из этой короткой спейсерной области невозможно подобрать зонды с достаточной специфичностью.
К тому же из фрагмента в 1100 п.н. для Listeria monocytogenes был выделен спейсер между генами 16S и 23S рРНК. Нуклеотидная последовательность, полученная для этого вида, приведена на фиг. 45. Также получена информация о последовательности для L. seeligeri (см. фиг. 46), а для L. ivanovii установлена часть последовательности большого спейсера (см. фиг. 44).
На основании сравнения с последовательностью L.seeligeri был выбран следующий зонд для специфического определения L.monocytogenes:
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC
Предварительные результаты гибридизации (данные не приводятся) показали отсутствие какой-либо перекрестной гибридизации с другими видами Listeria при использовании данного LMO3-зoндa на Listeria monocytogenes и то, что все использованные штаммы Listeria гибридизовались с общим зондом LIS1.
Олигонуклеотидные зонды - LIS1 для определения всех видов Listeria и LMO3 для специфического определения L.monocytogenes - иммобилизовали на мембранной полоске и гибридизовали с мечеными ампликонами, содержащими спейсерную область 16S-23S рРНК из различных организмов. Результаты гибридизации показаны в таблице 6.
Для зондов LMO3 и LIS1 была получена отличная специфичность и чувствительность на родовом и видовом уровнях, соответственно.
Данные два зонда могут быть использованы для определения видов Listeria и Listeria monocytogenes непосредственно в образцах пищевых продуктов или после обогащения образцов в жидкой питательной среде. В обоих случаях могут возникнуть проблемы при амплификации с набором имеющихся праймеров, обусловленные высоким фоновым уровнем, даваемым флорой данных образцов.
Чтобы обойти эту проблему, мы разработали несколько наборов праймеров, принадлежащих спейсерной области 16S-23S рРНК видов Listeria.
Праймеры LIS-P1 и LIS-P2 являются верхними праймерами, в то время как LIS-P3 и LIS-P4 нижними. С помощью данных наборов праймеров для видов Listeria амплифицируется как меньшая спейсерная область 16S-23S рРНК, так и большая (за исключением L. grayi и L. murrayi). При необходимости данные праймеры могут быть использованы в nested PCR-анализе, в котором LIS-P1/LIS-P4 являются внешними праймерами, a LIS-P2/LIS-P3 - внутренними праймерами.
Для специфического определения Listeria monocytogenes был разработан зонд LMO-ICG-3, принадлежащий большой спейсерной области 16S-23S рРНК. Для специфической амплификации только этой большой спейсерной области с целью улучшенного определения данного патогена непосредственно в образцах был отобран набор праймеров из той части последовательности большой спейсерной области 16S-23S рРНК, которая отсутствует в меньшем рРНК спейсере. Для этой цели праймеры LIS-P5 и LIS-P6 использовали в качестве верхних праймеров, a LIS-P7 - в качестве нижнего.
LIS-P1: ACCTGTGAGTTTTCGTTCTTCTC (71)
LIS-P2: CTATTTGTTCAGTTTTGAGAGGTT (72)
LIS-P3: ATTTTCCGTATCAGCGATGATAC (73)
LIS-P4: ACGAAGTAAAGGTTGTTTTTCT (74)
LIS-P5: GAGAGGTTACTCTCTTTTATGTCAG (75)
LIS-P6: CTTTTATGTCAGATAAAGTATGCAA (202)
LIS-P7: CGTAAAAGGGTATGATTATTTG (203)
В процессе усовершенствования праймеров для видов Listeria был выделен организм из сыра, схожий с Listeria по результатам классических методов определения. Данный изолят (МБ 405) демонстрировал следующие свойства (схожие с видами Listeria): грамположителен, растет на Oxford и триптическом соевом агаре, обладает каталазной активностью. Единственное отличие от видов Listeria заключалось в подвижности, которое было негативным.
Используя для амплификации спейсерной области 16S-23S рРНК данного изолята MB 405 имеющиеся праймеры, описанные в примере 1, получили такой же образец ампликона, как и для видов Listeria. Гибридизация ампликона показала отсутствие сигнала с любым из зондов на вид Listeria.
Секвенирование 16S рРНК изолята MB 405 и последующее сравнение с видами Listeria и близкими им видами показало, что данный организм более близок к видам Listeria, чем к какому-либо другому описанному в литературе виду. Таксономическое изучение позволит установить, принадлежит или нет данный изолят к роду Listeria. Этот изолят и впоследствии выделенные организмы такого же типа названы в данной заявке Listeria-подобными организмами.
Изолят MB 405, по-видимому, содержит по меньшей мере три различных спейсерных области 16S-23S рРНК, которые были клонированы и секвенированы. В результате сравнения последовательностей с видами Listeria, была выбрана последовательность зонда для специфического определения Listeria-подобных штаммов:
LISP-ICG-1: CGTTTTCATAAGCGATCGCACGTT
Реакции обратной гибридизации данного зонда со спейсерными областями 16S-23S рРНК видов Listeria показали отсутствие перекрестной гибридизации.
Пример 4: Chlamydia trachomatis
Chlamydia trachomatis представляет собой небольшую облигатную внутриклеточную грамположительную бактерию, имеющую 15 сероваров (A-K, Ba, L1, L2 и L3), различающихся главным белком внешней мембраны (MOMP), и содержащую скрытую плазмиду, необходимую для внутриклеточного роста. Серовары A-K и Ba составляют трахома-биовар, в то время как серовары L1, L2 и L3- LGV-биовар.
Серовары А, В, Ba и С обычно ассоциированы с трахомой, основной причиной широко распространенной излечимой слепоты. Серовары D-K обнаружены в основном для инфекций, передающихся половым путем, и являются главной причиной воспалений шейки матки и почечных лоханок у женщин и уретрита и эпидидимита у мужчин. Серовары L1, L2 и L3 вовлечены в венерический лимфогранулематоз, редкое заболевание, передающееся половым путем.
Измерительным методом в лабораторной диагностике считается метод клеточных культур, хотя жизнеспособность образцов трудно поддерживать во время транспортировки и лабораторные методики требуют технического оборудования и затрат времени. Вследствие этого разработан ряд более быстрых тестовых наборов, таких как наборы для анализа с помощью фермент-связанных иммуносорбентов и с помощью прямой фиксации флюоресцентными антителами. Однако ни один из этих видов иммуноанализов не обладает высоким уровнем чувствительности и специфичности.
Коммерчески доступным является набор для проведения анализа с помощью неизотопных ДНК-зондов (Gen-Probe РАСЕ; [61]), с помощью которого можно определить наличие рРНК хламидий. Недавно для определения инфекций, вызываемых Chlamydia, был использован метод полимеразной цепной реакции (PCR). В качестве мишени использовали либо скрытую плазмиду [32], либо ген omp1, кодирующий главный белок внешней мембраны [53]. В сравнении с другими методиками PCR обладает наибольшей чувствительностью и специфичностью [42]. Ни один из этих анализов не использует ДНК- зонды, принадлежащие спейсерной области генов 16S-23S рРНК.
Для штаммов Chlamydia trachomatis L2 и Chlamydia psittaci 6BC часть цистрона рибосомальной РНК, содержащую 16S-23S спейсерную область, амплифицировали, используя имеющиеся праймеры (см. пример 1), с последующим клонированием в плазмидный вектор. Нуклеотидную последовательность спейсерной области 16S-23S рРНК определяли с помощью дидезокситерминирующего рост цепи метода.
Нуклеотидная последовательность спейсерной области обоих видов Chlamydia показана на фиг. 47, 48.
На основании полученной информации по последовательностям были химически синтезированы следующие олигонуклеотидные зонды:
CHTR-ICG-1: GGAAGAAGCCTGAGAAGGTTTCTGAC
CHTR-ICG-2: GCATTTATATGTAAGAGCAAGCATTCTATTTCA
CHTR-ICG-3: GAGTAGCGTGGTGAGGACGAGA
CHPS-ICG-1: GGATAACTGTCTTAGGACGGTTTGAC
Олигонуклеотидные зонды иммобилизовали в виде линий на мембранной полоске и далее использовали в методе обратной гибридизации с биотинилированными PCR-продуктами, содержащими в качестве мишени спейсерную область 16S-23S рРНК.
Гибридизацию проводили в растворе 3хSSC и 20% формамиде (FA) при температуре 50oC.
Результаты гибридизации с различными пробами приведены в таблице 7.
Как показано в табл. 7, при температуре гибридизации 50oС зонды CHTR1, CHTR2 и CHTR3 специфичны к Chlamydia trachomatis, а зонд CHPS1 специфичен к Chlamydia psittaci.
Несколько клинических изолятов, полученных из SSDZ (Делф, Нидерланды) и идентифицированных с помощью общеизвестных методик как Chlamydia trachomatis, исследовали методом обратной гибридизации с различными олигонуклеотидными зондами. Все зонды на вид Chlamydia trachomatis показывали позитивный сигнал гибридизации, и ни один из изолятов не взаимодействовал с зондом на Chlamydia psittaci. CHTR2-зонд значительно слабее взаимодействовал с некоторыми клиническими изолятами, нежели CHTR1 или CHTR3. Спейсерная область одного из этих изолятов (94 М 1961) была отсеквенирована (SEQ ID NO 197), и в данной последовательности было обнаружено расхождение только в одной паре оснований по сравнению со спейсерной последовательностью штамма L2. На основании этой новой спейсерной последовательности был получен дополнительный зонд (CHTR4):
CHTR-ICG-4: GAGTAGCGCGGTGAGGACGAGA (SEQ ID NO 201)
Этот зонд дает более сильный гибридизационный сигнал с некоторыми клиническими изолятами из Chlamydia trachomatis, чем CHTR2. Он может быть использован как самостоятельно, так и в сочетании с зондом CHTR2 (например, оба зонда применимы в одной LiPA-линии).
В целях разработки особо чувствительных методов анализа для определения Chlamydia trachomatis непосредственно в клинических образцах на основании спейсерной области 16S-23S рРНК был получен набор праймеров к данному виду, а именно: CHTR-P1 (верхний праймер) и CHTR-P2 (нижний праймер), специфически амплифицирующий спейсерную область вида Chlamydia.
CHTR-P1: AAGGTTTCTGACTAGGTTGGGC 69
CHTR-P2: GGTGAAGTGCTTGCATGGATCT 70
Пример 6: Mycoplasma pneumoniae и Mycoplasma genitalium
Микоплазмы представляют собой группу самых малых прокариотов, про которые известно, что они способны расти в свободной от клеток среде, не имеют клеточной стенки и обладают весьма малыми геномами с низким содержанием (G+C)-оснований. Более чем 100 различных видов было выделено у человека, животных, растений и насекомых.
У людей микоплазмы распознаются либо как патогенные организмы, либо как комменсалисты. Наиболее известным патогеном является Mycoplasma pneumoniae, причина основной атипичной пневмонии, особенно у детей и молодых совершеннолетних пациентов. Диагностика М. pneumoniae основывается на прямом выделении с помощью культуральных методов или на определении в сыворотке крови пациента специфических антител к М. pneumoniae.
Другой патоген, выделенный впервые из уретральных проб пациентов с негонококковым уретритом, был охарактеризован как Mycoplasma genitalium. Данная микоплазма имеет несколько общих свойств с М. pneumoniae. Оба вида являются патогенными и оба обладают способностью к адгезии к эритроцитам, клеткам различных тканей, стеклянным и пластиковым поверхностям. К тому же М. genitalium и М. pneumoniae имеют общие антигены, давая обширные перекрестные реакции в серологических тестах. Тот факт, что М. genitalium смогли обнаружить в пробах из дыхательного тракта пациентов, больных пневмонией, и выделить из смеси с М. pneumoniae, поставили под вопрос возможную патогенность М. genital ium.
Так как культивирование обоих штаммов подразумевает определенные затраты времени, а серологии не достает специфичности, для идентификации этих микоплазм, разработаны более быстрые и более специфичные методы анализа. Было описано использование гибридизационных анализов с ДНК-зондами для данных видов, однако несмотря на хорошую специфичность, эти тесты не позволяли определить низкие уровни М. pneumoniae или М. genitalium. Совсем недавно разработаны методики ДНК-гибридизации с использованием полимеразной цепной реакции. Докладывалось об использовании гена P1-адгезина [7] и гена 16S рРНК [59] в PCR- анализах, специфичных к М. pneumoniae. Были описаны специфичные PCR-анализы на М. genitalium с использованием последовательностей генов адгезина и 16S рРНК.
Были определены спейсерные последовательности клинических изолятов М. pneumoniae и М. genitalium (получены от U.Gobel, Фрайбургский университет, Германия). Они показаны на фиг. 49 и 50. Отмечаются некоторые отличия в последовательностях по сравнению с другими штаммами тех же видов, депонированных в EMBL-банке данных (MPMAC и MGG37, соответственно). На основании этой информации были получены четыре зонда: один общий зонд на Mycoplasma, два специфичных к М. pneumoniae и один специфичный к М. genitalium зонды:
Mycoplasma-ICG: CAAAACTGAAAACGACAATCTTTCTAGTTCC
MPN-ICG-1: ATCGGTGGTAAATTAAACCCAAATCCCTGT
MPN-ICG-2: CAGTTCTGAAAGAACATTTCCGCTTCTTTC
MGE-ICG-1: CACCCATTAATTTTTTCGGTGTTAAAACCC
Зонды наносили на LiPA-полоски и гибридизовали в стандартных условиях (3xSSC, 20% формамид при 50oC) с целью амплификации спейсерного материала из четырех штаммов М. pneumoniae, одного штамма М. genitalium и двадцати двух штаммов вида не-Mycoplasma. Общий зонд гибридизовался только с пятью исследуемыми штаммами вида Mycoplasma, в то время как специфичные зонды гибридизовались только со штаммами тех видов, для которых они были созданы.
Пример 7: Другие виды микобактерий
Благодаря постоянному усовершенствованию лабораторных методик, быстро растет информация по систематике и значению для клиники так называемых "потенциально патогенных микобактерий в окружающей среде".
С появлением по-новому распознаваемых заболеваний обнаружились и получили главное значение дополнительные синдромы, связанные с различными микобактериями.
С целью расширения возможностей LiPA-теста в сторону одновременного определения различных видов микобактерий, как описано в примере 2, был создан новый набор ДНК-зондов для специфической идентификации следующих видов: Mycobacterium ulcerans, Mycobacterium genavense, Mycobacterium xenopi, Mycobacterium simiae, Mycobacterium fortuitum, Mycobacterium malmoense, Mycobacterium celatum и Mycobacterium haemophilum.
Эти зонды были получены на основании последовательности спейсерной области 16S-23S рРНК. Для вышеупомянутых видов данную информацию получали прямым секвенированием PCR-продуктов или после клонирования PCR-амплифицированной спейсерной области. Обнаруженные последовательности представлены на фиг. 80-97 и на фиг. 38 для М. malmoense.
Последовательности спейсерной области вышеупомянутых видов микобактерий сравнивали с подобными, уже описанными в примере 2 или в опубликованных доступных источниках. На основании данных о расхождении в этих областях были сконструированы видоспецифичные ДНК-зонды. Зонды отбирали и создавали таким образом, чтобы желаемое гибридизационное поведение (т.е. видоспецифическая гибридизация) проявлялось в тех же самых условиях, что и для других микобактериальных зондов, упомянутых в примере 2, то есть 3xSSC, 20% формамид, 50oC. Это позволяет одновременно определять по меньшей мере два и, возможно, все микобактериальные виды, описанные в настоящем изобретении.
На основании последовательности спейсерной области видов М. ulcerans, М. genavense, М. xenopi, М. simiae, М. fortuitum, М. malmoense, М. celatum и М. haemophilum были созданы, соответственно, следующие олигонуклеотидные зонды:
MUL-ICG-1: GGTTTCGGGATGTTGTCCCACC
MGV-I CG-1: CGACTGAGGTCGACGTGGTGT
MGV-ICG-2: GGTGTTTGAGCATTGAATAGTGGTTGC
MXE- ICG-1: GTTGGGCAGCAGGCAGTAACC
MSI-ICG-1: GCCGGCAACGGTTACGTGTTC
MFO- ICG-1: TCGTTGGATGGCCTCGCACCT
MFO-ICG-2: ACTTGGCGTGGGATGCGGGAA
MML- ICG-1: CGGATCGATTGAGTGCTTGTCCC
MML-ICG-2: TCTAAATGAACGCACTGCCGATGG
MCE-ICG-1: TGAGGGAGCCCGTGCCTGTA
МНР-I CG-1: CATGTTGGGCTTGATCGGGTGC
Зонды иммобилизовали на LiPA-полоске и гибридизовали с амплифицированным биотинилированным материалом из набора репрезентативных микобактериальных видов так же, как описано в примере 2. Амплификацию спейсерной области проводили, как описано в примере 2, с помощью PCR и набора праймеров. В таблице 8 приведены наименования различных штаммов, использованных для тестирования специфичности, и полученные результаты гибридизации. Штаммы были получены из коллекции Института Тропической Медицины, Антверпен, Бельгия.
Протестированные зонды (MSI-ICG-1, MXE-ICG-1, MFO-ICG-1, MFO-ICG-2, MML-ICG-1, MML-ICG-2, MCE-ICG-1 и MHP-ICG-1) специфически определяли М. simiae, М. xenopi, М. fortuitum, М. malmoense, М. celatum и М. haemophilum, соответственно, и не давали перекрестной гибридизации с другими протестированными микобактериальными штаммами. Таким образом, данные зонды позволяют проводить специфическое определение тех микобактериальных видов, которые оставались недоступными для определения с помощью набора ДНК-зондов, описанного в примере 2. Штамм М. malmoense был классифицирован в примере 2 как штамм "MIC 4"-типа, в то время, как остальные вышеупомянутые виды гибридизовались только с общими для рода Mycobacterium зондами MYC1/MYC22 и были поэтому классифицированы в примере 2 как "другие микобактериальные виды".
Все протестированные изоляты М. genavense реагировали с MGV-ICG-1 и MGV-ICG-2, но не с MSI-ICG-1, разработанным для определения М.simiae, близкородственного виду М. genavense. Группа "промежуточных" организмов, располагающихся между М. simiae и М. genavense, была получена из Института Тропической Медицины, Антверпен, где они были классифицированы как "М. simiae-подобные" (штаммы 4358, 4824, 4833, 4844, 4849, 4857, 4859, 7375, 7379, 7730, 9745, 94-1228). Данные штаммы реагировали только с зондом MGV-ICG-2, но не с зондом MSI-ICG-1, предназначенным для специфического определения штаммов М. simiae sensu stricto. Секвенирование спейсерной области 16S-23S рРНК двух из этих "М. simiае-подобных" изолятов (штаммы 7379 и 9745) (см. SEQ ID NO 161 и 162) подтвердило, что они являются более родственными М. genavense, нежели М.simiae. Для специфического определения этой группы организмов, возможно принадлежащих новому виду, был разработан новый зонд - MGV- ICG-3.
MGV-ICG-3: TCGGGCCGCGTGTTCGTCAAA
Сказанное выше еще раз иллюстрирует, что применение ДНК-зондов, принадлежащих 16S-23S спейсерной области, может оказаться полезным для дифференциации различных групп штаммов, которые к тому же не могут быть классифицированы с помощью классических таксономических критериев. Использование этих ДНК-зондов вполне вероятно может привести к описанию новых (под) видов среди микобактерий. В этом случае зонд MGV-1 будет реагировать только со штаммами M. genavense sensu stricto, зонд MGV-3 будет реагировать только с промежуточными "М. simiae-подобными" штаммами, а зонд MGV-2 будет определять оба вида штаммов.
Зонд MUL-ICG-1 реагировал со всеми протестированными штаммами М. ulcerans, но также показывал перекрестную гибридизацию со штаммом М. marinum ITG 7732. Секвенирование спейсерной области этого штамма М. marinum действительно показало идентичность данной последовательности штамма М. ulcerans 1837 (см. фиг. 80). Дальнейшая дифференциация между М. marinum и М. ulcerans может быть осуществлена с использованием зонда, принадлежащего гену 16S-рРНК М. ulcerans, часть которого коамплифицирована вместе со спейсерной областью, когда для амплификации применяли праймеры MYC P1-P5. Видоспецифическим 165-рРНК зондом на М. ulcerans, который может работать в тех же самых условиях гибридизации, как и спейсерные зонды, предназначенные для дифференциации видов микобактерий, может быть например:
TGGCCGGTGCAAAGGGCTG (SEQ ID NO 216)
Предыдущий параграф показал, что хотя предпочтительно использовать зонды, принадлежащие спейсерной области, также возможно и иногда необходимо комбинировать спейсерные зонды с зондами, принадлежащими другим генетическим последовательностям, например 16S-pPHK гену. Здесь эти дополнительные зонды опять отобраны таким образом, что они показывают желаемые гибридизационные свойства в тех же самых условиях гибридизации и отмывки, что и спейсерные зонды.
Для вида М. kansasii с использованием описанных в примере 2 зондов MKA-ICG-1, 2, 3 и 4 были протестированны дополнительные к указанным в примере 2 штаммы. Ввиду того, что ни один из этих зондов не оказался полностью удовлетворительным, для определения М. kansasii были разработаны дополнительные зонды. С этой целью были секвенированы спейсерные области нескольких дополнительных штаммов М. kansasii ITG 6328, 8698 и 8973 (см. фиг.90-92). Эти штаммы были также получены из Института Тропической Медицины в Антверпене, Бельгия. По-видимому, штаммы М.kansasii составляют очень гетерогенную группу со значительными различиями в спейсерной последовательности между различными штаммами. Были разработаны дополнительные зонды МКА -ICG-5, 6, 7, 8, 9 и 10, все гибридизующиеся в тех же самых условиях, что были ранее описаны, то есть 3хSSC, 20% деионизованный формамид, 50oC. Зонды были протестированы с помощью коллекции тестовых штаммов, полученной из Института Тропической Медицины, Антверпен, Бельгия, и результаты показаны в таблице 8.
Ни один из зондов на М. kansasii не гибридизуется с протестированными видами, отличными от М. kansasii. Однако ввиду гетерогенного характера данных видов ни один из зондов на М. kansasii не гибридизуется со всеми штаммами М. kansasii. Различные зонды на М. kansasii узнают различные штаммы М. kansasii. Данное различие в гибридизации может иметь значение для клиники. С другой стороны, если желательно определить все штаммы М. kansasii, может быть рассмотрено сочетание различных зондов на М. kansasii.
Пример 8: Brucella
Бруцеллез является широко распространенным и экономически важным зоонозом, который также затрагивает людей.
Для идентификации видов Brucella используются главным образом бактериологические и иммунологические методики определения. Данные тесты требуют больших затрат времени и часто дают ошибочные положительные результаты. Разрабатываемые быстрые и надежные методы идентификации обычно основаны на методах амплификации и гибридизации ДНК.
Описано специфическое определение видов Brucella на основании амплификации гена белка внешней мембраны с молекулярной массой 43 кДа [16] или части гена 16S-pPHK [23].
С целью разработки специфических ДНК-зондов и праймеров для определения видов Brucella мы проанализировали спейсерную область 16S-23S генов рРНК. Используя имеющиеся праймеры (последовательности приведены в примере 1), спейсерную область амплифицировали с последующим клонированием в Bluescript SK+ вектор, следуя методике из примера 1.
Полученный ампликон длиной примерно в 1400 н.п. был клонирован для следующих видов Brucella:
- Brucella abortus NIDO Tulya биовар 3 (SEQ ID NO 154)
- Brucella melitensis NIDO биовар 1 (SEQ ID NO 131)
- Brucella suis NIDO биовар 1 (SEQ ID NO 132)
Обработка генно-инженерных конструкций Hind III с последующим субклонированием полученных фрагментов (n=3) облегчило секвенирование спейсерной области трех описанных видов Brucella. Нуклеотидные последовательности спейсерных областей вышеупомянутых штаммов Brucella melitensis, Brucella suis и Brucella abortus представлены на фиг. 56, 57 и 79, соответственно. Ввиду большой гомологии последовательностей данных спейсерных областей различных видов Brucella, на основании информации о данных последовательностях не удалось установить последовательностей видоспецифичных ДНК-зондов, а были разработаны только родоспецифичные зонды.
Для этой цели были химически синтезированы следующие зонды.
BRU-ICG-1: CGTGCCGCCTTCGTTTCTCTTT
BRU-ICG-2: TTCGCTTCGGGGTGGATCTGTG
BRU-IСG-3: GCGTAGTAGCGTTTGCGTCGG
BRU-ICG-4: CGCAAGAAGCTTGCTCAAGCC
Олигонуклеотидные зонды иммобилизовали на мембранной полоске и после обратной гибридизации с биотинилированными PCR-фрагментами гибриды визуализировали посредством реакции преципитации. Результаты гибридизации иммобилизованных зондов с различными видами Brucella и родственными организмами представлены в таблице 9.
Полученные результаты показывают, что зонды BRU-ICG-2, BRU-ICG- 3 и BRU-ICG-4 являются специфичными к видам Brucella и могут быть использованы в методе обратной гибридизации для определения этих патогенов. Зонд BRU-ICG-1 перекрестно гибридизуется со штаммами Ochrobactrum antropi и Rhizobium loti, которые представляют из себя два таксономически близкородственных организма, но присутствие которых не ожидается в тех образцах, которые используются для определения Brucella.
В целях специфической амплификации спейсерной области штаммов Brucella, так же как описано в предыдущих примерах (например, 3 и 4), специфические праймеры и для Brucella были выбраны на основании спейсерной области 16S-23S рРНК.
BRU-P1 и BRU-P2 применяли в качестве верхних праймеров, в то время как BRU-P3 и BRU-P4 использовали в качестве нижних. В nested PCR-анализе набором внешних праймеров является сочетание BRU-P1/BRU-P4, а внутренних - BRU-P2/BRU-P3
BRU-P1: TCGAGAATTGGAAAGAGGTC 204
BRU-P2: AAGAGGTCGGATTTATCCG 205
BRU-P3: TTCGACTGCAAATGCTCG 206
BRU-P4: TCTTAAAGCCGCATTATGC 207
Пример 9: Staphylococcus aureus
Staphylococcus aureus представляет собой вид стафилококка, наиболее часто связанного с инфекциями человека и животных. Штаммы Staphylococcus aureus идентифицированы как важные этиологические агенты как общих, так и нозокоминальных инфекций. В последнее время нозокоминальная инфекция, вызываемая метициллин-устойчивым штаммом S. aureus (MRSA), все шире распространяется во многих странах. Штаммы, принадлежащие этому виду, также являются агентами, вызывающими порчу и отравление пищи.
В литературе описано использование методик, основанных на применении ДНК-зондов и/или PCR для быстрого и специфического различения штаммов S. aureus от других стафилококковых штаммов. Примерами генов-мишеней, использованных для разработки этих, опирающихся на ДНК анализов, являются ген 16SрРНК [13, 19], mecA-ген [56, 50] и nuc-ген [5, 9].
В качестве мишени для разработки специфических ДНК-зондов мы выбрали спейсерную область генов 16S-23S рРНК. Амплификация с использованием имеющихся праймеров, принадлежащих генам 16S и 23S- рРНК (последовательности см. в примере 1), показала, что полученная картина не одинакова для всех протестированных штаммов S. aureus. Множество вариантов наблюдали как в количестве полученных фрагментов, так и в их размере.
Одна спейсерная область штамма UZG 5728 и четыре спейсерных области (различающиеся по длине) штамма UZG 6289 были отклонированы в Bluescript SK+вектор с последующим секвенированием. Нуклеотидные последовательности представлены на фиг. 64-68 (SEQ ID NO 139-143). Для разработки специфических ДНК-зондов провели сравнение нуклеотидных последовательностей этих различных спейсерных областей между собой и со спейсерной областью штамма Staphylococcus epidermidis UZG CNS41 (SEQ ID NO 144).
Были химически синтезированы следующие зонды:
STAU-ICG-1: TACCAAGCAAAACCGAGTGAATAAAGAGTT
STAU-ICG-2: CAGAAGATGCGGAATAACGTGAC
STAU-ICG-3: AACGAAGCCGTATGTGAGCATTTGAC
STAU-ICG-4: GAACGTAACTTCATGTTAACGTTTGACTTAT
Олигонуклеотиды иммобилизовали на мембранной полоске и после обратной гибридизации с биотинилированными PCR-фрагментами гибриды визуализировали с помощью колориметрической реакции преципитации.
Результаты гибридизации иммобилизованных зондов с различными видами Staphylococcus и нестафилококковыми организмами представлены в таблице 10.
Полученные результаты гибридизации показывают, что только зонды STAU-ICG-3 и STAU-ICG-4 специфичны для вида Staphylococcus aureus. Зонд STAU-ICG-1 реагирует со всеми протестированными видами Staphylococcus, а зонд STAU-ICG-2 перекрестно гибридизуется со штаммом S. lugdinensis. Ни зонд STAU- ICG-3, ни зонд STAU-ICG-4 не определяют все протестированные штаммы S. aureus, но когда в LiPA-анализе оба зонда используются одновременно, все протестированные штаммы S.aureus гибридизуются либо с одним из них, либо с обоими.
Литература
1. Asseline U, Deiarue M, Lancelot G, Touime F, Thuong N (1984) Nucleic acid-binding molecules with high affinity and base sequence specificity: intercalating agents covalently. linked to oligodeoxynucleotides. Proc. Natl. Acad. Sci. USA 81(ll): 3297-301.
2. Barany F (1991). Genetic disease detection and DNA amplification using cloned thermostable ligase. Proc Nad Acad Sci USA 88: 189-193.
3. Bej A, Mahbubani M, Miller R, Di Cesare J, Haff L, Atlas R (1990) Mutiplex PCR amplification and immobilized capture probes for detection of bacterial pathogens and indicators in water. Mol Cell Probes 4:353-365.
4. Boddinghaus B, Rogall T, Flohr T, Blocker H, Bottger E (1990). Detection and identification of Mycobacteria by amplification of rRNA. Journal of Clinical Microbiology, 28: 1751- 1759.
5. Brakstad, О.G., К. Aasbakk, and J.A. Maeland. 1992. Detection of Staphylococcus aureus by polymerase chain reaction amplification of the nuc gene. J. Clin. Microbiol. 30:1654- 1660.
6. Bubert A, Kohler S, Goebel W (1992). The homologous and heterologous regions within the iap gene allow genus- and species- specific identification of Listeria spp. by polymerase chain reaction. Applied and Environmental Microbiology, 58: 2625-2632.
7. Buck G, O'Hara L, Summersgill J (1992). Rapid, sensitive detection of Mycoplasma pneumoniae in simulated clinical specimens by DNA amplification. Journal of Clinical Microbiology, 30:3280- 3283.
8. Bukh Purcell R, Miller R (1993). At least 12 genotypes...PNAS 90, 8234-8238.
9. Chesneau, O., J. Allignet and N. El Solh. 1993. Thermonuclease gene as a target nucleotide sequence for specific recognition of Staphylococcus aureus. Mol. Cell. Probes. 2:301-310.
10. Chomczynski P, Sacchi N (1987) Single step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Anal Biochem 162:156-159.
11. Compton J (1991). Nucleic acid sequence-based amplification. Nature, 350: 91-92.
12. Datia A, Moore M, Wentz B, Lane J (1993). Identification and enumeration of Lisieria monocytogenes by nonradioactive DNA probe colony hybridization. Applid and Environmental Microbiology, 59: 144-149.
13. De Buyser, M. , A. Morvan, S. Aubert, F. Dilasser and N. El Solh. 1992. Evaluation of a ribosomal RNA gene probe for the identification of species and subspecies within the genus Staphylococcus. J. Gen. Microbiol. 118:889-899.
14. Duck P (1990). Probe amplifier system based on chimeric cycling oligonucleotides. Biotechniques 9,142-147.
15. Farber J, Peterkin P (1991). Listeria monocytogenes, a food-borne pathogen. Microbiological Reviews, 55:476-511.
16. Fekete, A., J.A. Bantle, S.M. Balling and M.R. Sanborn. 1990. Preliminary development of a diagnostic test for Brucella using polymerase chain reaction. J. Appl. Bacteriol. 69:216- 227.
17. Frothingham R, Wilson К (1993). Sequence-based differentiation of strains in the Mycobacterium avium complex. Journal of Bacteriology, 175.
18. Frothingham R, Wilson K. (1994). Molecular phylogeny of the Mycobacterium avium complex demonstrates clinically meaningful divisions. J Infect Diseases, 169: 305-312.
19. Geha, D.J., J.R. Uhl, C.A. Gustaferro, and D.H. Persing. 1994. Multiplex PCR for identification of methicillin-resistant staphylococci in the clinical laboratory. J. Clin. Microbiol. 32:1768-1772.
20. Golsteyn Thomas E, King R, Burchak J, Cannon V (1991). Sensitive and specific detection of Listeria monocytogenes in milk and ground beef with the polymerase chain reaction. Applied and Environmental Microbiology, 57: 2576-2580.
21. Guatelli J, Whitfield K, Kwoh D, Barringer K, Richman D, Gengeras T (1990) Isothermal, in vitro amplification of nucleic acids by a multienzyme reaction modeled after retroviral replication. Proc Natl Acad Sci USA 87: 1874-1878.
22. Herman L, De Ridder H (1993). Evaluation of a DNA-probe assay for the identification of Listeria monocytogenes. Milchwissenschaft, 48: 126-128.
23. Herman, L. and H. De Ridder. 1992. Identification of Brucella spp. by using the polymerase chain reaction. Appl. Env. Microbiol. 58:2099-2101.
24. Jacobs K, Rudersdorf R, Neill S, Dougherty J, Brown E. Fritsch E (1988) The thermal stability of oligonucleotide duplexes is sequence independent in tetraalkylammonium salt solutions: application to identifying recombinant DNA clones. Nucl Acids Res 16:4637-4650.
25. Jaton K, Sahli R, Bille J (1992). Development of polymerase chain reaction assays for detection of Listeria monocytogenes in clinical cerebrospinal fluid samples. Journal of Clinical Microbiology, 30: 1931-1936.
26. Jonas V, Aldan M, Curry J, Kamisango K, Knott C, Lankford R, Wolfe J, Moore D (1993). Detection and identification of Mycobacterium tuberculosis directly from sputum sediments by amplification of RNA. Journal of Clinical Microbiology, 31: 2410- 2416.
27. Kempsell K et al. (1992). The nucleotide sequence of the promotor, 16S rRNA and spacer region of the ribosomal RNA operon of Mycobacterium tuberculosis and comparison with M. leprae precursor rRNA. Journal of Gen Microbiol, 138: 1717-1727.
28. Kwoh D, Davis G, Whitfield K, Chappelle H, Dimichele L, Gingeras T (1989). Transcription-based amplification system and detection of amplified human immunodeficiency virus type 1 with a bead-based sandwich hybridization format. Proc Natl Acad Sci USA, 86: 1173-1177.
29. Kwok S, Kellogg D, McKinney N, Spasic D, Goda L, Levenson C, Sinisky J, (1990). Effects of primer-template mismatches on the polymerase chain reaction: Human immunodeficiency views type 1 model studies. Nucl. Acids Res., 18: 999.
30. Landgren U, Kaiser R, Sanders J, Hood L (1988). A ligase-mediated gene detection technique. Science 241:1077-1080.
31. Lizardi P. Guerra C, Lomeli H, Tussie-Luna I, Kramer F (1988) Exponential amplification of recombinant RNA hybridization probes. Bio/Technology 6:1197-1202.
32. Loeffelholz M, Lewinski C, Silver S, Purohit A, Herman S, Buonagurio D, Dragon E (1992). Detection of Chiamydia trachomatis in endocervical specimens by polymerase chain reaction. Journal of Clinical Microbiology, 30: 2847-2851.
33. Lomeli H, Tyagi S, Printchard C, Lisardi P, Kramer F (1989) Quantitative assays based on the use of replicatable hybridization probes. Clin Chem 35: 1826-1831.
34. Maniatis T, Fritsch E, Sambrook J (1982) Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY.
35. Matsukura M, Shinozuka K, Zon G, Mitsuya H, Reitz M, Cohen J, Broder S (1987) Phosphorothioate analogs of oligodeoxynucleotides: inhibitors of replication and cytopathic effects of human immunodeficiency virus. Proc. Nati. Acad. Sci. USA 84(21):7706-10.
36. Mcintosh I, Govan J, Brock D (1992). Detection of Pseudomonas aeruginosa in sputum from cystic fibrosis patients by the polymerase chain reaction. Molecular and Cellular Probes, 6: 299-304.
37. Miller P, Yano J, Yano E, Carroll C, Jayaram K, Ts'o P (1979) Nonionic nucleic acid analogues. Synthesis and characterization of dideoxyribonucleoside methylphosphonates. Biochemistry 18(23):5134-43.
38. Nielsen P, Egholm M, Berg R, Buchardt 0 (1991) Sequence- selective recognition of DNA by strand displacement with a thymine-substituted polyamide. Science 254(5037): 1497-500.
39. Nieisen P, Egholm M, Berg R, Buchardt O (1993) Sequence specific inhibition of DNA restriction enzyme cleavage by PNA. Nucleic-Acids-Res. 21(2): 197-200.
40. Ninet В, Bannerman E, Bille J (1992). Assessment of the accuprobe Lisieria monocytogenes culture identification reagent kit for rapid colony confirmation and its application in various enrichment broths. Applied and Environmental Microbiology, 58: 4055-4059.
41. Ogle J, Janda J, Woods D, Vasil M (1987). Characterization and use of a DNA probe as an epidemiological marker for Pseudomonas aeruginosa. The Journal of Infectious Diseases. 155: 119.
42. Ossewaarde J, Pieffe M, Rozenberg-Arska M, Ossenkoppele P, Nawrocki R, Van Loon A (1992). Development and clinical evaluation of a polymerase chain reaction test for detection of Chlamydia trachomatis. Journal of Clinical Microbiology, 30: 2122- 2128.
43. Rogall T, Wolters J, Flohr T, Bottger E (1990). Towards a phytogeny and definition of species at the molecular level within the genus Mycobactericum. Int. J. Syst. Bacteriol 40: 323-330.
44. Rossau R, Michielsen A, Jannes G, Duhamel M, Kersten K, Van Heuverswyn H. DNA probes for Bordetella species and a colorimetric reverse hybridization assay for the detection of Bordetella pertussis. Mol. Cell. Probes 6: 281-289, 1992.
45. Saiki R, Gelfand D, Stoffel S, Scharf S, Higuchi R, Horn G, Mullis K, Erlich H (1988). Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 239:487-491.
46. Saiki R, Walsh P, Levenson C, Erlich H (1989) Genetic analysis of amplified DNA with immobilized sequence-specific oligonucleotide probes (1989) Proc Nati Acad Sci USA 86:6230-6234.
47. Saito H, Tomioka H, Sato K, Hiromichi T, Tsukamura M, Kuze F, Asano К (1989). Identification and partial characterization of Mycobacterium avium and Mycobacterium imracellulare by using DNA probes. Journal of Clinical Microbiology, 27: 994-997.
48. Samadpour M, Moseley S, Lory S (1988). Biotinylated DNA probes for exotoxin A and pilin genes in the differentiation of Pseudomonas aeruginosa strains. Journal of Clinical Microbiology, 26:2319-2323.
49. Sano T, Smith C, Cantor С (1992) Immuno-PCR: very sensitive antigen detection by means of specific antibody-DNA conjugates. Science 258:120-122.
50. Shimaoka, M. , M. Yoh. A. Segawa, Y. Takarada, K. Yamamoto and T. Honda. 1994. Development of enzyme-labeled oligonucleotide probe for detection of mecA. gene in methicillin-resistant Staphylococcus aureus. J. Clin. Microbiol. 32:1866-1869.
51. Stuyver L, Rossau R, Wyseur A, Duhamel M, Vanderborght B, Van Heuverswyn H, Maeriens G (1993) Typing of hepatitis C virus (HCV) isolates and characterization of new (sub)types using a Line Probe Assay. J Gen Virology, 74: 1093-1102.
52. Suzuki Y et al. (1988). Complete nucleotide sequence of the 16S rRNA gene of Mycobacterium bovis BCG. J Bacteriol, 170: 2886-2889.
53. Taylor-Robinson D, Gilroy C, Thomas B, Keat A (1992). Detection of Chlamydia nachomatis DNA in joints of reactive arthritis patients by polymerase chain reaction. Lancet 340:81-82.
54. Telenti A, Marchesi F, Balz M, Bally F, Bottger E, Bodmer T (1993). Rapid identification of Mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. Journal of Clinical Microbiology, 31: 175-178.
55. Tomioka H, Saito H, Sato К, Tasaka H, Dawson J (1993). Identification of Mycobacterum avium complex strains belonging to serovars 21-28 by three commercial DNA probe tests. Tubercle and Lung Disease, 74: 91-95.
56. Ubukata, К., S. Nakagami, A. Nitta, A. Yamane, S. Kawakami, M. Suguria and M. Konno. 1992. Rapid detection of the mec A gene in methicillin-resistant staphylococci by enzymatic detection ofpolymerase chain reaction products. J. Clin. Microbiol. 30:1728-1733.
57. Van der Giessen, J et al. (1994). Comparison of the 23S rRNA genes and the spacer region between the 16S and 23S rRNA genes of the closely related M. avium and M. paratuberculosis and the fast-growing M. phlei. Microbiology, 140: 1103-1108.
58. Vaneechoutte M, De Beenhouwer H, Claeys G, Verschraegen G, De Rouck A, Paepe N, Elaichouni A, Portaels F (1993). Identification of Mycobacienum species by using amplified ribosomal DNA restriction analysis. Journal of Clinical Microbiology, 31: 2061-2065.
59. Van Kuppeveld F, Van Der Logt J, Angulo A, Van Zoest M, Quint W, Niesters H, Galama J, Melchers W (1992). Genus- and species-specific identification of mycoplasmas by 16S rRNA amplification. Applied and Environmental Microbiology, 58: 2606- 2615.
60. Walker G, Little M, Nadeau J, Shank D (1992). Isothermal in vitro amplification of DNA by a restriction enzyme/DNA polymerase system. Proc Natl Acad Sci USA 89:392-396.
61. Woods G, Young A, Scott J, Blair T, Johnson A (1990). Evaluation of a nonisotopic probe for detection of Chlamydia trachomatis in endocervical specimens. Journal of Clinical Microbiology, 28:370-372.
63. Wu D, Wallace В (1989). The ligation amplification reaction (LAR) - amplification of specific DNA sequences using sequential rounds of template-dependent ligation. Genomics 4:560-569.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ идентификации патогенных бактерий в пищевых субстратах с использованием высокопроизводительного секвенирования | 2018 |
|
RU2712527C2 |
| СПОСОБ ВЫЯВЛЕНИЯ УСТОЙЧИВОСТИ МИКОБАКТЕРИЙ К РИФАМПИЦИНУ С ВОЗМОЖНОЙ ИХ ИДЕНТИФИКАЦИЕЙ, СПОСОБ ГИБРИДИЗАЦИИ, ЗОНД И КОМПОЗИЦИЯ ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ, НАБОР ПРАЙМЕРОВ (ВАРИАНТЫ) | 1995 |
|
RU2204608C2 |
| СИСТЕМА ДЕТЕКЦИИ НАИБОЛЕЕ ЗНАЧИМЫХ ПРОКАРИОТИЧЕСКИХ ПРЕДСТАВИТЕЛЕЙ МИКРОБИОТЫ КИШЕЧНИКА ЧЕЛОВЕКА НА ОСНОВЕ ПЦР ПАНЕЛИ | 2017 |
|
RU2680268C1 |
| ОЛИГОНУКЛЕОТИДНЫЙ БИОЧИП ДЛЯ ИДЕНТИФИКАЦИИ ГЕНЕТИЧЕСКИХ ДЕТЕРМИНАНТ РЕЗИСТЕНТНОСТИ Neisseria gonorrhoeae К АНТИМИКРОБНЫМ ПРЕПАРАТАМ, НАБОР ОЛИГОНУКЛЕОТИДОВ, ИСПОЛЬЗУЕМЫХ ДЛЯ ИММОБИЛИЗАЦИИ НА БИОЧИПЕ | 2016 |
|
RU2636457C2 |
| СПОСОБ ИДЕНТИФИКАЦИИ И ДИФФЕРЕНЦИАЦИИ ПРОКАРИОТИЧЕСКИХ ОРГАНИЗМОВ | 2011 |
|
RU2486251C2 |
| Способ определения генетических детерминант резистентности возбудителя туберкулеза к бедаквилину и линезолиду | 2017 |
|
RU2677293C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ МИКОБАКТЕРИЙ С ПОМОЩЬЮ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2010 |
|
RU2455364C2 |
| СПОСОБ КОЛИЧЕСТВЕННОГО АНАЛИЗА КОЛИЧЕСТВА КЛЕТОК ПРЕДСТАВЛЯЮЩЕЙ ИНТЕРЕС БАКТЕРИИ В ЖИВОМ СОСТОЯНИИ С ПРИМЕНЕНИЕМ рРНК В КАЧЕСТВЕ МИШЕНИ | 2006 |
|
RU2420595C2 |
| ТЕСТ-СИСТЕМА ДЛЯ ЭКСПРЕСС-ВЫЯВЛЕНИЯ И ИДЕНТИФИКАЦИИ MYCOBACTERIUM AVIUM COMPLEX | 2023 |
|
RU2819877C1 |
| ЗОНД ПЕПТИДНО-НУКЛЕИНОВОЙ КИСЛОТЫ, НАБОР И СПОСОБ ОБНАРУЖЕНИЯ И/ИЛИ КОЛИЧЕСТВЕННОГО ОПРЕДЕЛЕНИЯ Salmonella spp. И ИХ ПРИМЕНЕНИЯ | 2011 |
|
RU2567011C2 |


































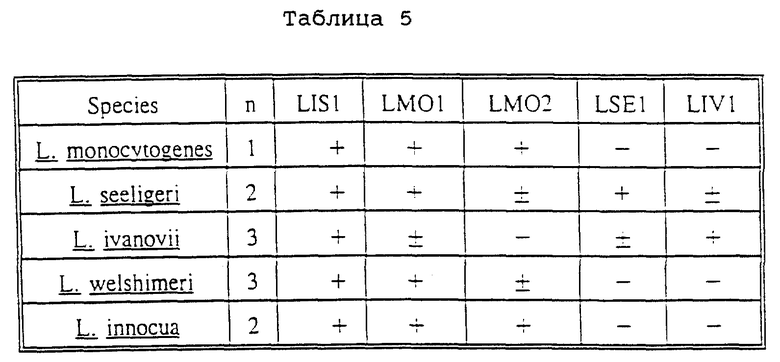


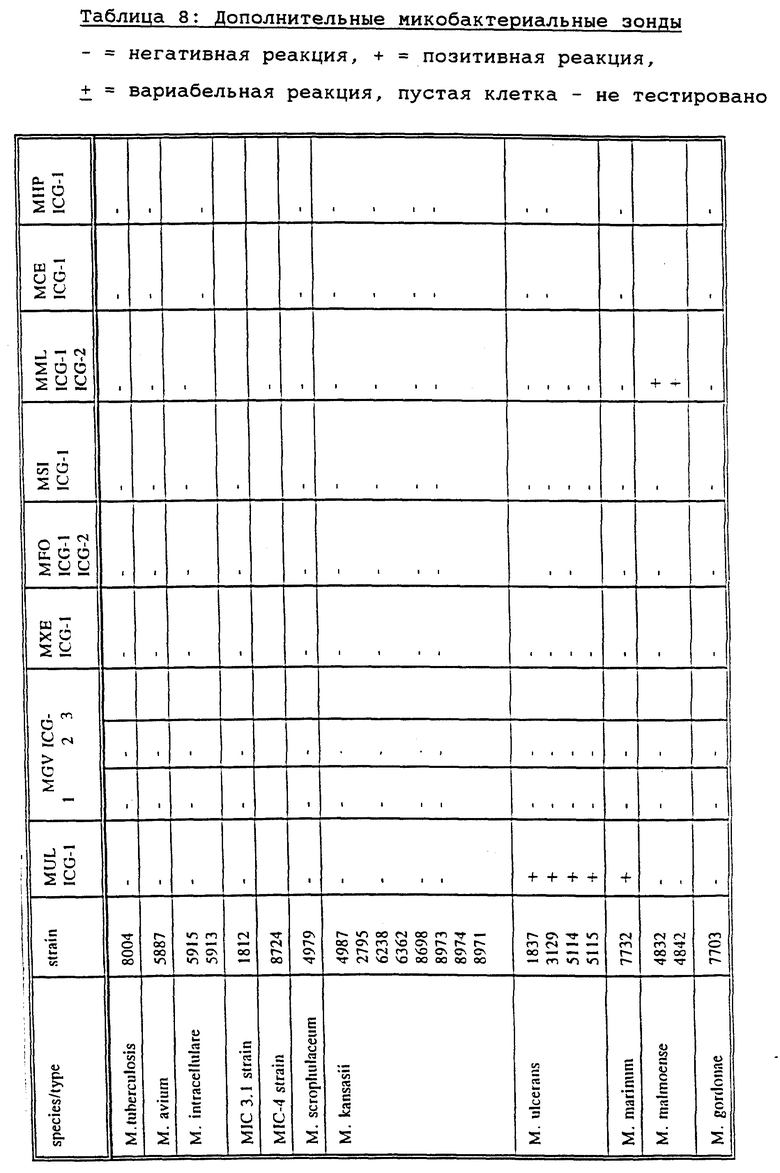

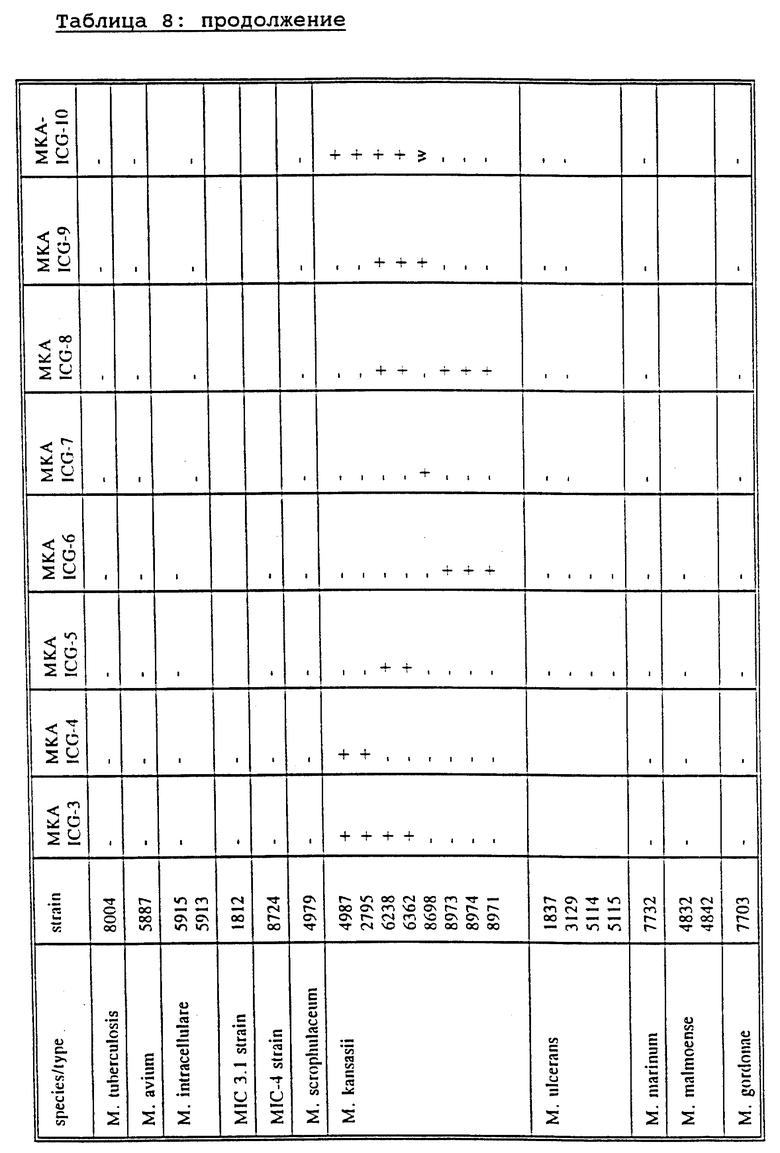
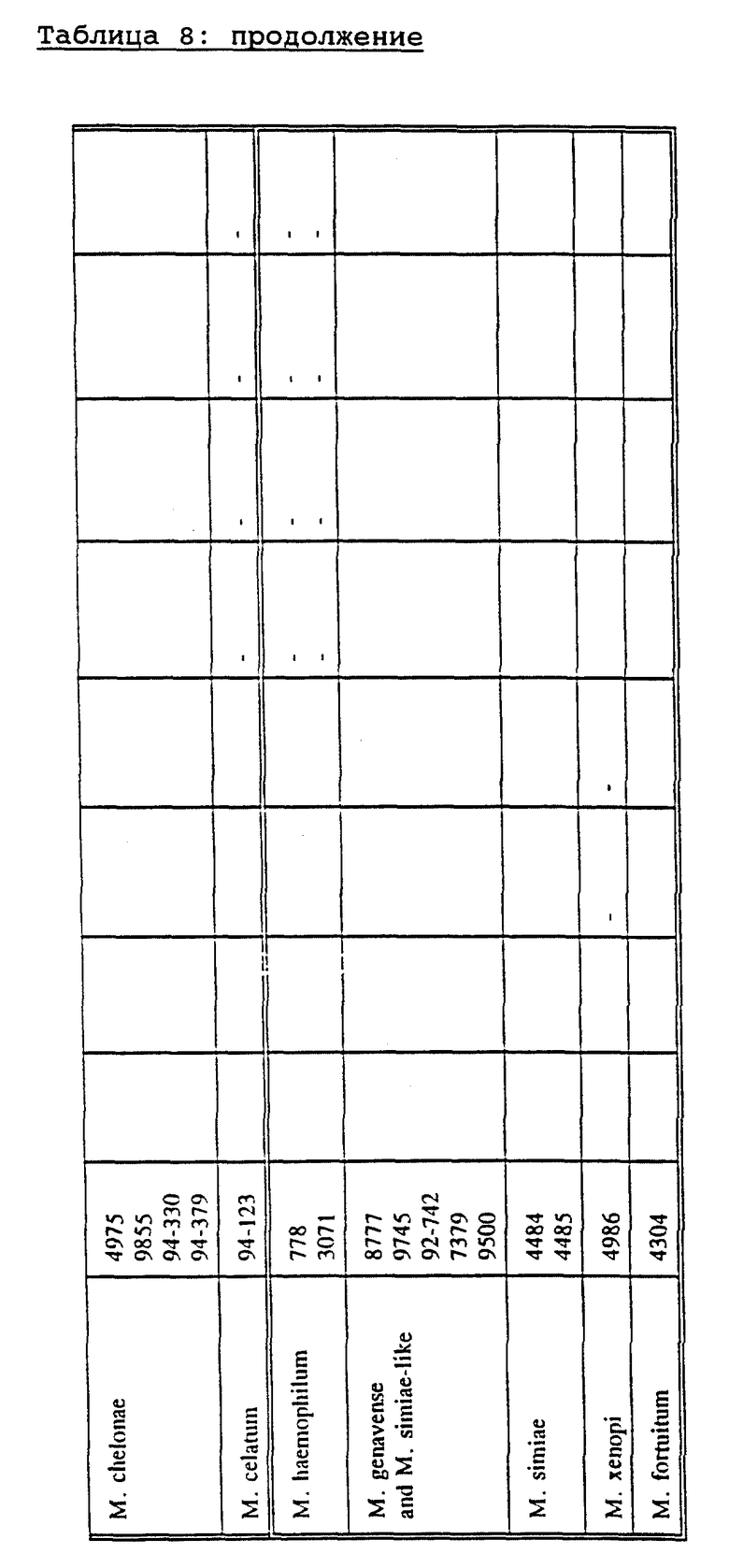









































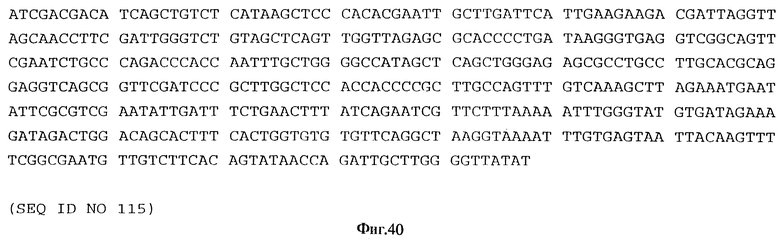































































Изобретение относится к медицине и касается способа определения и идентификации по меньшей мере одного микроорганизма или одновременного определения различных микроорганизмов в образце. Сущность изобретения состоит в (i) высвобождении, выделении и/или концентрировании полинуклеиновых кислот, присутствующих в образце; (ii) при необходимости амплификации 16S-23S pРНК спейсерной области или ее части с помощью по меньшей мере одной подходящей пары праймеров; (iii) гибридизации полинуклеиновых кислот, полученных на стадиях (i) или (ii), по меньшей мере с одним и предпочтительно более чем с одним спейсерным зондом, приведенным в табл.1а, или его эквивалентом в одинаковых условиях гибридизации и отмывки, и/или с таксон-специфическим зондом, принадлежащим любой из спейсерных последовательностей, представленных на фиг. 1-103, в тех же самых условиях гибридизации и отмывки; (iv) детектирования гибридов, образованных на стадии (iii) с каждым из зондов, примененных в соответствующих условиях гибридизации и отмывки; (v) идентификации присутствующего в образце микроорганизма(ов) на основании разностных гибридизационных сигналов, полученных на стадии (iv). Изобретение также включает зонд, состав, праймер и набор для определения и идентификации микроорганизмов. Способ повышает специфичность и чувствительность определения и идентификации микроорганизмов. 6 с. и 48 з.п. ф-лы, 103 ил., 11 табл.
и более предпочтительно из следующих спейсерных зондов:
или эквивалентов указанных зондов, и/или указанный набор зондов включает по меньшей мере один таксон-специфический зонд, полученный из последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, при этом последовательность указанной спейсерной области выбирают из любой из последовательностей, представленных согласно SEQ ID NO 76 - 106, 157 - 174, 124, 125, 111 - 115, 139 - 144 или 126 - 130, приведенным на фиг. 1 - 31, 80 - 97, 49, 50, 36 - 40, 64 - 69 и 51 - 55, и при этом указанные зонды или эквиваленты возможно используют в сочетании с любым зондом, с помощью которого можно определить по меньшей мере один из следующих организмов: Haemophilus influenzae, Streptococcus pneumoniae, Moraxella catarrhalis или Bordetella pertussis.
и предпочтительно из следующих спейсерных зондов:
или эквивалентов указанных зондов, и/или указанный набор зондов включает по меньшей мере один таксон-специфический зонд, полученный из последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, при этом последовательность указанной спейсерной области выбирают из любой из последовательностей, представленных согласно SEQ ID NO 116, 118 - 121 или 213 - 215, приведенным на фиг. 41, 43 - 46 и 101 - 103, и при этом указанные зонды или эквиваленты возможно используют в сочетании с любым зондом, с помощью которого можно определить по меньшей мере один из следующих организмов: Neisseria meningitidis, Haemophilus influenzae или Streptococcus pneumoniae.
или эквивалентов указанных зондов, и/или указанный набор зондов включает по меньшей мере один таксон-специфический зонд, полученный из последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, при этом последовательность указанной спейсерной области выбирают из любой из последовательностей, представленных согласно SEQ ID NO 122, 123, 197, 124 или 125, приведенным на фиг. 47, 48, 100, 49 и 50, при этом указанные зонды или эквиваленты возможно используют в сочетании с любым зондом, с помощью которого можно определить по меньшей мере один из следующих организмов: Neisseria gonorrhoeae, Haemophilus ducreyi или Streptococcus agalactiae.
и предпочтительно из следующих спейсерных зондов:
или эквивалентов указанных зондов, и/или указанный набор зондов включает по меньшей мере один таксон-специфический зонд, полученный из последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, при этом последовательность указанной спейсерной области выбирают из любой из последовательностей, представленных согласно SEQ ID NO 116, 118 - 121, 213 - 215, 139 - 144, 131, 132, 154, 133 - 138, 195 или 196, приведенным на фиг. 41, 43 - 46, 101 - 103, 64 - 69, 56, 57, 79, 58 - 63, при этом указанные зонды или эквиваленты возможно используют в сочетании с любым зондом, с помощью которого можно определить штаммы вида Campylobacter.
и предпочтительно из следующих спейсерных зондов:
или эквивалентов указанных зондов, и/или указанный набор зондов включает по меньшей мере один таксон-специфический зонд, полученный из последовательности спейсерной области одного из определяемых в указанном образце микроорганизмов, при этом последовательность указанной спейсерной области выбирают из любой из последовательностей, представленных согласно SEQ ID NO 133 - 138 или 195 - 196, приведенным на фиг. 58 - 63 и 98 - 99, при этом указанные зонды или эквиваленты возможно используют в сочетании с любым зондом, с помощью которого можно определить штаммы вида Campylobacter.
и более предпочтительно с по меньшей мере одним зондом из следующей более узкой группы спейсерных зондов:
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 76 - 110 или 157 - 174, приведенных на фиг. 1 - 35 и 80 - 97, обеспечивающим специфическую гибридизацию указанного зонда с видом Mycobacterium.
и/или с эквивалентами вышеупомянутых зондов, и/или с любым зондом, полученным из SEQ ID NO 76, приведенной на фиг. 1, с обеспечивающим специфическую гибридизацию указанного зонда с комплексом M. tuberculosis.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 77 - 100 или 108 - 110, приведенных на фиг. 2 - 25 и 33 - 35, обеспечивающим специфическую гибридизацию указанного зонда со штаммами MAIS-комплекса.
MAV-ICG-1: TCGGTCCGTCCGTGTGGAGTC (SEQ ID NO 10)
MAV-ICG-22: GTGGCCGGCGTTCATCGAAA (SEQ ID NO 11)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 77 и 78, приведенных на фиг. 2 и 3, обеспечивающим специфическую гибридизацию указанного зонда с M. avium и M. paratuberculosis.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98 или 99, приведенных на фиг. 4 - 24, обеспечивающим специфическую гибридизацию указанного зонда со штаммами M. intracellulare и MIC-штаммами.
MIN-ICG-1: GCATAGTCCTTAGGGCTGATGCGTT (SEQ ID NO 12)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 89, приведенной на фиг. 14, обеспечивающим специфическую гибридизацию указанного зонда с M. intracellulare.
MSC-ICG-1: TCGGCTCGTTCTGAGTGGTGTC (SEQ ID NO 24)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 100, приведенной на фиг. 25, обеспечивающим специфическую гибридизацию указанного зонда со штаммами M. scrofulaceum.
и более предпочтительно с:
или с эквивалентами указанного зондов, и/или с любым зондом, полученным из SEQ ID NO 101, 167, 168 или 169, приведенных на фиг. 26, 90 - 92, обеспечивающим специфическую гибридизацию указанного зонда со штаммами M. kansasii.
MCH-ICG-1: GGTGTGGACTTTGACTTCTGAATAG (SEQ ID NO 29)
MCH-ICG-2: CGGCAAAACGTCGGACTGTCA (SEQ ID NO 30)
MCH-ICG-3: GGTGTGGTCCTTGACTTATGGATAG (SEQ ID NO 210)
или с эквивалентами вышеупомянутых зондов, и/или с любым зондом, полученным из SEQ ID NO 102, 103 или 174, приведенных на фиг. 27 - 28 и 97, обеспечивающим специфическую гибридизацию указанного зонда со штаммами M. chelonae.
MGO-ICG-1: AACACCCTCGGGTGCTGTCC (SEQ ID NO 31)
MGO-ICG-2: GTATGCGTTGTCGTTCGCGGC (SEQ ID NO 32)
MGO-ICG-5: CGTGAGGGGTCATCGTCTGTAG (SEQ ID NO 33)
и наиболее предпочтительно с:
MGO-ICG-5: CGTGAGGGGTCATCGTCTGTAG (SEQ ID NO 33)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 104, 105 или 106, приведенных на фиг. 29 - 31, обеспечивающим специфическую гибридизацию указанного зонда с M. gordonae.
MUL-ICG-1: GGTTTCGGGATGTTGTCCCACC (SEQ ID NO 175)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 157, приведенной на фиг. 80, обеспечивающим специфическую гибридизацию указанного зонда с M. ulcerans или M. marinum.
MGV-ICG-1: CGACTGAGGTCGACGTGGTGT (SEQ ID NO 176)
MGV-ICG-2: GGTGTTTGAGCATTGAATAGTGGTTGC (SEQ ID NO 177)
MGV-ICG-3: TCGGGCCGCGTGTTCGTCAAA (SEQ ID NO 211)
или с эквивалентами вышеупомянутых зондов, и/или с любым зондом, полученным из SEQ ID NO 158, 159, 160, 161 или 162, приведенных на фиг. 81 - 85, обеспечивающим специфическую гибридизацию указанного зонда с M. genavense.
MXE-ICG-1: GTTGGGCAGCAGGCAGTAACC (SEQ ID NO 178)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 163, приведенной на фиг. 86, обеспечивающим специфическую гибридизацию указанного зонда с M. xenopi.
MSI-ICG-1: CCGGCAACGGTTACGTGTTC (SEQ ID NO 179)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 164 или 165, приведенных на фиг. 87 - 88, обеспечивающим специфическую гибридизацию указанного зонда с M. simiae.
MFO-ICG-1: TCGTTGGATGGCCTCGCACCT (SEQ ID NO 180)
MFO-ICG-2: ACTTGGCGTGGGATGCGGGAA (SEQ ID NO 181)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 166, приведенной на фиг. 89, обеспечивающим специфическую гибридизацию указанного зонда с M. fortuitum.
MCE-ICG-1: TGAGGGAGCCCGTGCCTGTA (SEQ ID NO 190)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 170, приведенной на фиг. 93, обеспечивающим специфическую гибридизацию указанного зонда с M. celatum.
MHP-ICG-1: CATGTTGGGCTTGATCGGGTGC (SEQ ID NO 191)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 171, 172 или 173, приведенных на фиг. 94 - 96, обеспечивающим специфическую гибридизацию указанного зонда с M. haemophilum.
MYC-ICG-1: ACTGGATAGTGGTTGCGAGCATCTA (SEQ ID NO 1)
MYC-ICG-22: CTTCTGAATAGTGGTTGCGAGCATCT (SEQ ID NO 2)
или с эквивалентами указанных зондов.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 124 или 125, приведенных на фиг. 49 и 50, обеспечивающим специфическую гибридизацию указанного зонда с видом Mycoplasma.
MPN-ICG-1: ATCGGTGGTAAATTAAACCCAAATCCCTGT (SEQ ID NO 49)
MPN-ICG-2: CAGTTCTGAAAGAACATTTCCGCTTCTTTC (SEQ ID NO 50)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 125, приведенной на фиг. 50, обеспечивающим специфическую гибридизацию указанного зонда с видом Mycoplasma pneumoniae.
MGE-ICG-1: CACCCATTAATTTTTTCGGTGTTAAAACCC (SEQ ID NO 51)
или с эквивалентами зонда, и/или с любым зондом, полученным из SEQ ID NO 124, приведенной на фиг. 49, обеспечивающим гибридизацию указанного зонда с видом Mycoplasma genitalium.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 111, 112, 113, 114 или 115, приведенных на фиг. 36-40, обеспечивающим специфическую гибридизацию указанного зонда со штаммами Pseudomonas.
и наиболее предпочтительно по меньшей мере с одним из следующих зондов:
PA-ICG-1: TGGTGTGCTGCGTGATCCGAT (SEQ ID NO 34)
PA-ICG-4: TGAATGTTCGT(G/A) V(G/A)ATGAACATTGATTTCTGGTC (SEQ ID NO 37)
PA-UCG-5: CTCTTTCACTGGTGATCATTCAAGTCAAG (SEQ ID NO 38)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 111, приведенной на фиг. 36, обеспечивающим специфическую гибридизацию указанного зонда с Pseudomonas aeruginosa.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 139, 140, 141, 142, 143 или 144, приведенных на фиг. 64-69, обеспечивающим специфическую гибридизацию указанного зонда с видом Staphylococcus.
STAU-ICG-3: AAGGAAGCCGTATGTGAGCATTTGAC (SEQ ID NO 55)
STAU-ICG-4: GAACGTAACTTCATGTTAACGTTTGACTTAT (SEQ ID NO 56)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 139, 140, 141, 142 или 143, приведенных на фиг. 64-68, обеспечивающим специфическую гибридизацию указанного зонда с Staphylococcus aureus.
ACI-ICG-1: GCTTAAGTGCACAGTGCTCTAAACTGA (SEQ ID NO 57)
ACI-ICG-2: CACGGTAATTAGTGTGATCTGACGAAG (SEQ ID NO 58)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 126, 127, 128, 129 или 130, приведенных на фиг. 51-55, обеспечивающим специфическую гибридизацию указанного зонда с видом Acinetobacter.
ACI-ICG-1: GCTTAAGTGCACAGTGCTCTAAACTGA (SEQ ID NO 57)
ACI-ICG-2: CACGGTAATTAGTGTGATCTGACGAAG (SEQ ID NO 58)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 126, приведенной на фиг. 51, обеспечивающим специфическую гибридизацию указанного зонда с Acinetobacter baumanii.
и наиболее предпочтительно, по меньшей мере, с одним из следующих зондов:
LIS-ICG-1: CAAGTAACCGAGAATCATCTGAAAGTGAATC (SEQ ID NO 39)
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC (SEQ ID NO 42)
LISP-ICG-1: CGTTTTCATAAGCGATCGCAAGTT (SEQ ID NO 212)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 116, 118, 119, 120, 121, 213, 214 или 215, приведенных на фиг. 41, 43-46, 101-103, обеспечивающим специфическую гибридизацию указанного зонда с видом Listeria.
LMO-ICG-1: AAACAACCTTTACTTCGTAGAAGTAAATTGGTTAAG (SEQ ID NO 40)
LMO-ICG-2: TGAGAGGTTAGTACTTCTCAGTATGTTTGTTC (SEQ ID NO 41)
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC (SEQ ID NO 42)
и наиболее предпочтительно со следующим зондом:
LMO-ICG-3: AGGCACTATGCTTGAAGCATCGC (SEQ ID NO 42)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 118 или 120, приведенных на фиг. 43 и 45, обеспечивающим специфическую гибридизацию указанного зонда с Listeria monocytogenes.
и наиболее предпочтительно со следующим зондом:
BRU-ICG-2: TTCGCTTCGGGGTGGATCTGTG (SEQ ID NO 60)
BRU-ICG-3: GCGTAGTAGCGTTTGCGTCGG (SEQ ID NO 193)
BRU-ICG-4: CGCAAGAAGCTTGCTCAAGCC (SEQ ID NO 194)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 131, 132 или 154, приведенных на фиг. 56-57 и 79, обеспечивающим специфическую гибридизацию указанного зонда со штаммами Brucella.
и наиболее предпочтительно со следующим зондом:
SALM-ICG-1: CAAAACTGACTTACGAGTCACGTTTGAG (SEQ ID NO 61)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 133, 134, 135, 136, 137 или 138, приведенных на фиг. 58-63, обеспечивающим специфическую гибридизацию указанного зонда со штаммами Salmonella.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 122, 123 или 197, приведенных на фиг. 47-48 и 100, обеспечивающим специфическую гибридизацию указанного зонда со штаммами Chlamydia.
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 123 или 197, приведенных на фиг. 48 и 100, обеспечивающим специфическую гибридизацию указанного зонда с Chlamydia trachomatis.
CHPS-ICG-1: GGATAACTGTCTTAGGACGGTTTGAC (SEQ ID NO 48)
или с эквивалентами указанного зонда, и/или с любым зондом, полученным из SEQ ID NO 122, приведенной на фиг. 47, обеспечивающим специфическую гибридизацию указанного зонда со штаммами Chlamydia psittaci.
CHTR-P1: AAGGTTTCTGACTAGGTTGGGC (SEQ ID NO 69)
CHTR-P2: GGTGAAGTGCTTGCATGGATCT (SEQ ID NO 70)
или эквивалентов данных праймеров, при этом указанные эквиваленты, отличающиеся по последовательности от упомянутых выше праймеров заменой одного или более нуклеотидов, по-прежнему сохраняют способность специфически амплифицировать спейсерную область Chlamydia trachomatis или ее часть.
или эквивалентов данных праймеров, при этом указанные эквиваленты, отличающиеся по последовательности от упомянутых выше праймеров заменой одного или более нуклеотидов, по-прежнему сохраняют способность специфически амплифицировать спейсерную область вида Listeria или ее часть.
или эквивалентов данных праймеров, при этом указанные эквиваленты, отличающиеся по последовательности от упомянутых выше праймеров заменой одного или более нуклеотидов, по-прежнему сохраняют способность специфически амплифицировать спейсерную область вида Mycobacterium или ее часть.
YEC-ICG-1: GGAAAAGGTACTGCACGTGACTG (SEQ ID NO 198)
YEC-ICG-2: GACAGCTGAAACTTATCCCTCCG (SEQ ID NO 199)
YEC-ICG-3: GCTACCTGTTGATGTAATGAGTCAC (SEQ ID NO 200)
или с эквивалентами указанных зондов, и/или с любым зондом, полученным из SEQ ID NO 195 или 196, приведенных на фиг. 98 - 99, обеспечивающим специфическую гибридизацию указанного зонда со штаммами Yersinia enterocolitica.
или эквивалентов данных праймеров, при этом указанные эквиваленты, отличающиеся по последовательности от упомянутых выше праймеров заменой одного или более нуклеотидов, по-прежнему сохраняют способность специфически амплифицировать спейсерную область вида Brucella или ее часть.
YEC-P1: CCTAATGATATTGATTCGCG (SEQ ID NO 208)
YEC-P2: ATGACAGGTTAATCCTTACCCC (SEQ ID NO 209)
или эквивалентов данных праймеров, при этом указанные эквиваленты, отличающиеся по последовательности от упомянутых выше праймеров заменой одного или более нуклеотидов, по-прежнему сохраняют способность специфически амплифицировать спейсерную область вида Yersinia enterocolitica или ее часть.
Приоритет по пунктам и признакам:
по пп.8 - 13, 16, 24 - 34, 36, 38, 41 - 43 от 24.06.94;
по пп.17, 19 - 23, 46 - 48, от 07.04.95;
по пп.1 - 7, 14, 15, 18, 35, 37, 39, 40, 44, 45, 49 - 54 в табл. 1а и 1б и на фиг. 1 - 103;
SEQ ID NO 1 - 154 от 24.06.94;
SEQ ID NO 157 - 209 от 07.04.95;
SEQ ID NO 210 - 215 от 23.06.95 (по дате подачи международной заявки).
| Автоматический огнетушитель | 0 |
|
SU92A1 |
| ЧУГУН | 0 |
|
SU398677A1 |
| Автоматический огнетушитель | 0 |
|
SU92A1 |
| HAMES B/ Higgins S., Nucleic acid hybridization, Practical approach, IRL | |||
| Press, Oxford7 ИК | |||
| Приспособление для установки двигателя в топках с получающими возвратно-поступательное перемещение колосниками | 1917 |
|
SU1985A1 |
| PAO C.C | |||
| et al., "The detection of Micobacterial DNA Seguences in incultured clinical specimens cloned mycobacterium tuberculosis DNA as probes", TUBERCULE, 1988, V.69, PP.27 - 36. | |||
Авторы
Даты
2000-08-10—Публикация
1995-06-23—Подача