Изобретение относится к области биотехнологии, а именно к индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex с помощью ПЦР-РВ. Метод полимеразной цепной реакции (ПЦР) обладает высокой специфичностью - выявляются уникальные фрагменты ДНК B. cereus, кодирующие выработку токсинов. Экстремальная чувствительность реакции, обеспечивает выявление клеток возбудителя даже при условии малой обсеменённости субстрата клетками B. cereus или при условии наличия сопутствующей микрофлоры, это позволяет использовать ПЦР в тех случаях, когда другие методы экспресс диагностики не дают положительного результата. Проведение ПЦР в формате «реального времени» (ПЦР-РВ) позволяет ускорить получение результата, что имеет большое значение в проведении массового микробиологического мониторинга [1].
Группа Bacillus cereus объединяет несколько близкородственных видов грамположительных палочковидных, спорообразующих факультативно аэробных бактерий, широко распространенных в природе [2, 4, 10]. Основными заболеваниями, с которым связывают B. cereus являются пищевые отравления. Данные по распространенности в мире пищевых токсикоинфекций, вызванных B. cereus на данный момент крайне неполны, но, по разным оценкам в разных регионах B. cereus ответственен за 1,5-15% от общего числа таких заболеваний [11]. Болезнь обычно проявляется болями в животе, водянистой диареей, а иногда также тошнотой и рвотой. Время инкубации колеблется в пределах 8-16 часов, но в редких случаях наблюдается более длительное время инкубации. Продолжительность заболевания обычно составляет 12-24 часа, но сообщалось о случаях, продолжающихся несколько дней [7]. Данную клиническую картину легко спутать с другими заболеванием алиментарного происхождения, вызываемым различными спорообразующим бактериями.
Этиологическими агентами пищевых токсикоинфекций B. cereus являются рвотный или диарейные токсины. Рвотный токсин цереулид представляет собой циклический додекадепсипептид кодируемой кластером генов цереулидсинтетазы (ces) размером 24 т.п.н. который расположен на мегаплазмиде pBCE4810 или pCER270 [4, 10]. Механизм, посредством которого цереулид вызывает рвоту у людей, точно не определён. Небольшой размер цереулида крайне усложняет его обнаружение, при проведении эпидрасследований.
Этиологическими агентами диарейных заболеваний пищевого происхождения, вызванных B. cereus в настоящее время считаются три цитотоксина: гемолизин BL (Hbl), негемолитический энтеротоксин ((Nhe) и цитотоксин К (CytK). Hbl и Nhe - это трехкомпонентные токсины. Hbl состоит из трех белков L2, L1 и B, кодируемых генами hblC, hblD и hblA, соответственно, которые котранскрибируются входя в состав одного оперона [6, 8].
Nhe состоит из белков NheA, NheB и NheC, кодируемых опероном nheABC [5].
CytK - это мономерный β-порин, один из двух основных β-поринов B. cereus [9].
Энтеротоксин FM (EntFM) - еще один фактор вирулентности B. cereus. Он не имеет существенного сходства с другими энтеротоксинами, и сам по себе EntFM не является цитотоксичным. Тем не менее, токсин участвует в обеспечении подвижности бактериальных клеток, формировании биопленок и адгезии к эпителиальным клеткам, которые важны для прогрессирования инфекционного процесса.
В настоящее время отечественных наборов для индикации и идентификации токсинов возбудителя пищевых токсико-инфекций, вызванных бактериями группы B. cereus complex методом ПЦР нет. Доступен набор реактивов для обнаружения ДНК Bacillus Cereus P608H. Производитель: TIANLONG (Китай). Набор предназначен для выявления ДНК условно-патогенных бактерий классов Bacilli, вызывающих нозокомиальные и внебольничные инфекции, полученных из биологического материала человека и бактериальных культур из этого биоматериала (мокрота, моча, мазки/соскобы из дыхательных путей, желудочно-кишечного и урогенитального тракта, фекалии, аспираты, экссудаты) методом ПЦР в режиме реального времени.
Недостатком данного набора является то, что исследователь может идентифицировать микроорганизм, но не может выявить гены, кодирующие токсины и оценить патогенный потенциал выделенной культуры.
Другой доступный набор SureFast® Bacillus cereus group PLUS представляет собой набор для ПЦР в реальном времени для прямого качественного обнаружения специфических последовательностей ДНК группы Bacillus cereus (Bacillus anthracis, Bacillus cereus, Bacillus cytoxis, Bacillus mycoides, Bacillus pseudomycoides, Bacillus thuringiensis и Bacillus weihenstephanensis). Производитель: R-Biopharm AG (Германия).
Данный набор способен дифференцировать между собой различных представителей группы Bacillus cereus complex, но не способен определить токсический профиль каждого представителя.
Иные зарубежные аналоги коммерчески недоступны для приобретения на территории РФ.
Известны классические микробиологические методы отбора проб, культивирование, микросопирование и диагностирования B. cereus complex, но они не охарактеризовывают патологический потенциал штаммов, а способствуют только определению таксономии возбудителя.
Индикация факторов патогенности возможна при использовании MALDI-TOF MS (matrix-assisted laser desorption ionization mass spectrometry), матрично-активированной лазерной деcорбционно/ионизационной времяпролетной масс-спектрометрии, для обнаружения цереулида, CytK и NheA в мазках, взятых из культивированных чистых культур B. cereus. Также существуют набор для тестирования обратной пассивной латексной аглютинации BCET-RPLA на фрагмент L2 Hbl производитель (Thermo Scientific). Тест Duopath Cereus на энтеротоксины NheB и фрагмент L2 Hbl производитель (Merck). Описан метод идентификации токсинов NheA и Hbl при использовании ELISA (enzyme linked immunoadsorbent assay) - метода определения с помощью иммуносорбентов, связанных с ферментами, который в настоящий момент в России также коммерчески недоступен. Альтернативный способ идентификации генов, кодирующих токсины - это секвенирование всего генома штамма возбудителя пищевой токсикоинфекции. Также для оценки токсического потенциала штаммов чистых культур B. cereus возможно использование перевиваемых клеточных линий. Например, клеточная линия Hep2 подходит для проверки штаммов на наличие церулида, а клеточные линии Vero и Сасо-2 обычно используются для проверки штаммов на энтеротоксический потенциал [3, 4].
Все описанные методы диагностики являются дорогостоящими и требуют специализированное оборудование и высококвалифицированных специалистов для проведения исследований. Таким образом, существует явная потребность в разработке способа экспресс-идентификации.
Технической задачей настоящего технического решения является разработка набора для быстрой идентификации ДНК генов кодирующих тоскины, синтезируемые бактериями группы B. cereus complex.
Техническим результатом заявленного технического решения является расширение арсенала технических средств, что позволяет без использования дополнительных методов выявлять ампликоны B. cereus complex в исследуемых образцах.
Указанный технический результат достигается тем, что набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации ДНК B. cereus complex методом гибридизационно-флуоресцентно полимеразной цепной реакции включает олигодезоксирибонуклеотиды, обладающие специфической активностью прямого и обратного праймеров, и флуоресцентно-меченый зонд, имеющие следующую структуру:
Используемые для расчета праймеров и зонда последовательности геномов были получены из международной базы данных GenBank. Для построения элайнментов, анализа и расчетов праймеров и зондов, изучения их специфичности использовали семейство компьютерных программ Vector NTI 9.0.0 (Informax, США) и онлайн-интернет-ресурсы базы данных Национального центра биотехнологической информации США (Blast NCBI).
Оценка специфичности праймеров проводилась на рабочей коллекции из 40 штаммов B. cereus депонированных в Государственную коллекцию патогенных микроорганизмов и клеточных культур «ГКПМ - Оболенск». Наличие генов кодирующих синтез токсинов в исследуемых штаммах B. cereus определяли по скринингу данных полногеномного секвенирования используя программу UGEN 50.0 (Унипро, Россия). Полученные для 40 штаммов результаты были верифицированы полногеномным секвенированием и эффективность определения наличия токсинов предлагаемым/разрабатываемым прототипом тест-системы составила 100%. Ложноположительных и ложноотрицательных результатов выявлено не было. Положительные контрольные образцы были получены in vitro методом ПЦР. Использовали метод наработки фрагмента локуса, кодирующего ген токсинообразования с ДНК матрицы штамма продуцента токсина B. cereus.
Условия проведения амплификации оптимизировали по концентрации ионов магния (Mg2+), концентрации праймеров и зондов в реакционной смеси, температуре отжига праймеров. С использованием полученной плазмидной конструкции были подобраны оптимальные концентрации ионов Mg2+ (2.5 мМ MgCl2), прямого и обратного праймеров (300 нМ), флуоресцентного ДНК-зонда (100 нМ). Важно отметить, что все этапы оптимизации проведения ПЦР были осуществлены с использованием коммерческих реагентов и приборов для амплификации, являясь коммерчески доступными, они предназначены для массового применения в диагностических лабораториях России. Это позволяет широко применять данное изобретение в лабораторной практике.
Для определения аналитической чувствительности метода были приготовлены последовательные 10-кратные разведения наработанной ДНК. Концентрацию ДНК определяли при помощи замера на Спектрофотометр NanoDrop One, 190-850 нм, от 1 мкл, (Thermo Scientific, США).
Аналитическая чувствительность разработанного метода с использованием праймеров составила 102 геномных эквивалентов (ГЭ).
Предлагаемое техническое решение даёт возможности разделения постановки реакции для индикации рвотного токсина и цитотоксина К отдельно от других диарейных токсинов. Это позволяет сократить стоимость проведения одного анализа.
Техническое решение обусловлено использованием идентичных каналов обнаружения FAM для индикации гемолизина BL и цереулида было вызвано невозможностью амплификаторов проводить четкую идетфикацию световых спектров при использовании более 4 флюорофоров. При использовании в реакционной смеси праймеров содержащих более 4 зондов происходит засвечивание при считывании светового канала и в результате наложении графиков кривых. Что в свою очередь приводит к образованию ложноположительных результатов. Поэтому мы предлагаем использовать одномоментную постановку сочетания из комбинации реакционных смесей 1 и 2 (Табл. 1).
Таблица 1. Состав реакционных смесей для ПЦР-анализа в формате двух независимых мультиплексных реакций
Таким образом, на основании вышеизложенного получаем комплект, включающий:
При этом ПЦР смесь 1 включает праймеры и флуоресцентно-меченые зонды для идентификации: цитотоксина К, цереулида и внутреннего положительного контроля. ПЦР смесь 2 включает праймеры и флуоресцентно-меченые зонды для идентификации: гемолизина BL, негемолитического энтеротоксина А, энтеротоксина FM и внутреннего положительного контроля. Внутренние положительные контроли смесей идентичны и включают любые обычно используемые для постановки ПЦР-РВ положительные контроли.
В основном исполнении изобретение имеет следующий вид:
Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для детекциии цереулида и диарейных энтеротоксинов (цитотоксина К, гемолизина BL, негемолитического энтеротоксина А, энтеротоксина FM) Bacillus cereus complex с помощью ПЦР-РВ, включающий:
а) набор праймеров для детекции цитотоксина К, состоящий из последовательностей SEQ ID No.1 (F), SEQ ID No.2 (R) и SEQ ID No.3 (Z);
b) набор праймеров для детекции негемолитического энтеротоксина А, состоящий из последовательностей SEQ ID No.4 (F), SEQ ID No.5 (R) и SEQ ID No.6 (Z);
с) набор праймеров для детекции гемолизин BL, состоящий из последовательностей SEQ ID No.7 (F), SEQ ID No.8 (R) и SEQ ID No.9 (Z);
d) набор праймеров для детекции энтеротоксина FM, состоящий из последовательностей SEQ ID No.10 (F), SEQ ID No.11 (R) и SEQ ID No.12 (Z);
e) набор праймеров для детекции цереулида, состоящий из последовательностей SEQ ID No.13 (F), SEQ ID No.14 (R) и SEQ ID No.15 (Z);
f) набор праймеров для внутреннего контроля ампликации, обычно применяемых для постановки ПЦР;
где F - прямой праймер, R - обратный праймер и Z - флуоресцентно-меченый ДНК-зонд,
где ДНК-зонды, состоящие из последовательностей SEQ ID No.3, SEQ ID No.6, SEQ ID No.9, SEQ ID No.12 и SEQ ID No.15 детектируются по каналам ROX, ROX, FAM, RG6 и FAM, соответственно, а положительный и отрицательный контроли, обычно используемые для постановки ПЦР-РВ, детектируются по каналу Cy5 и всем каналам (ROX, FAM, RG6), соответственно.
В одном из исполнений изобретения, оптимальная температура отжига праймеров составляет 57°С.
В одном из исполнений изобретения, в качестве внутреннего положительного контроля реакции используется плазмида E.coli pUC19, детектируемая по каналу Cy5.
В одном из исполнений изобретения, вместо детекции по каналу RG6 осуществляется детекция по каналу HEX.
Изобретение иллюстрируется следующими графическими материалами.
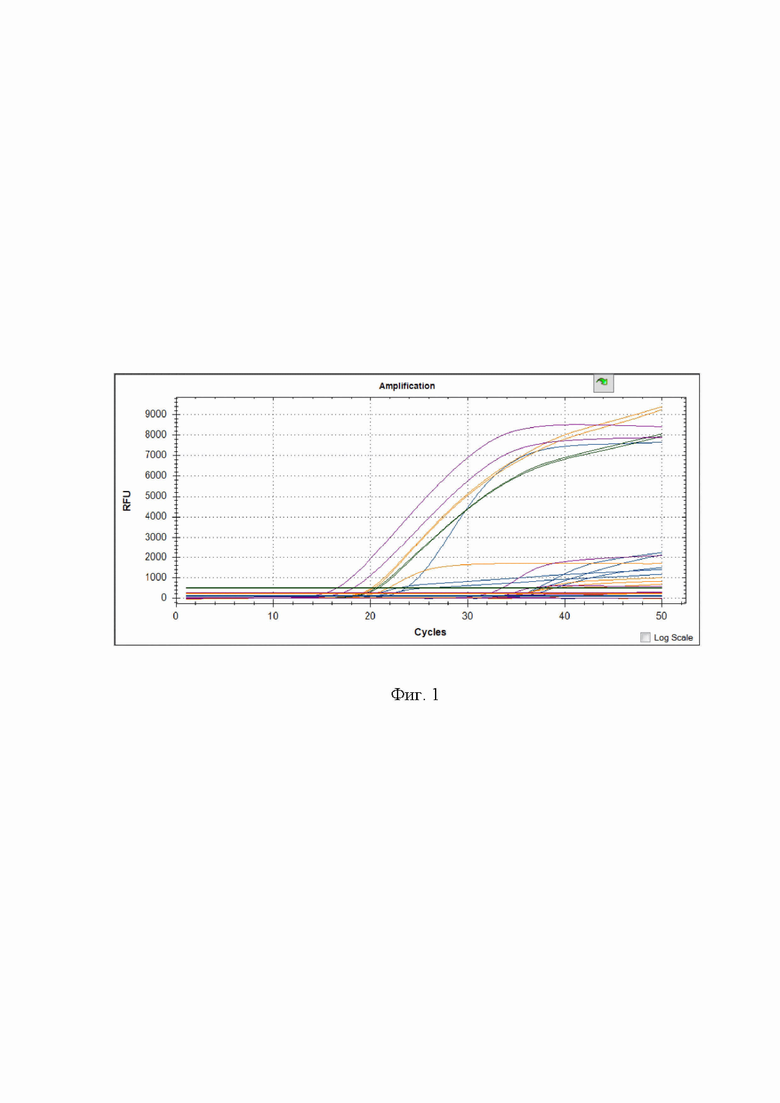
Фиг. 1. Общий вид итогового протокола при условии просмотра всех флюорофорных каналов и всех исследуемых образцов.
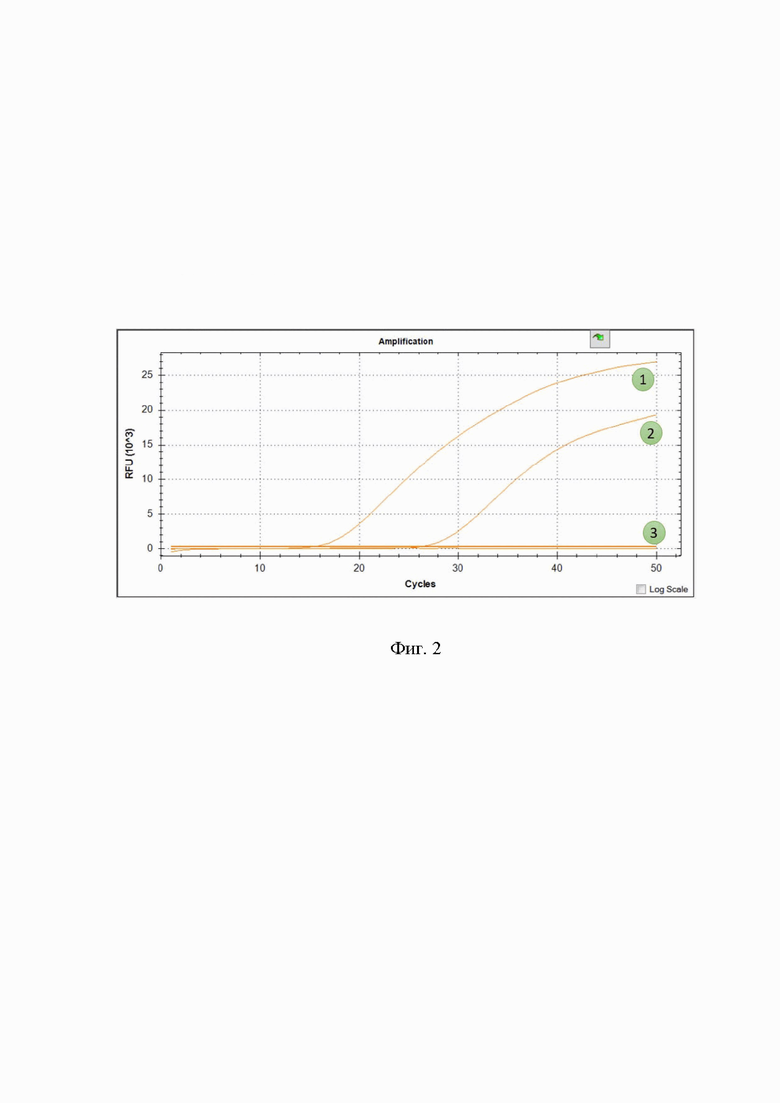
Фиг. 2. Идентификация цитотоксина К: 1 - положительный контроль; 2 - тестируемая ДНК; 3 - отрицательный контроль. Канал считывания флюорофора ROX.
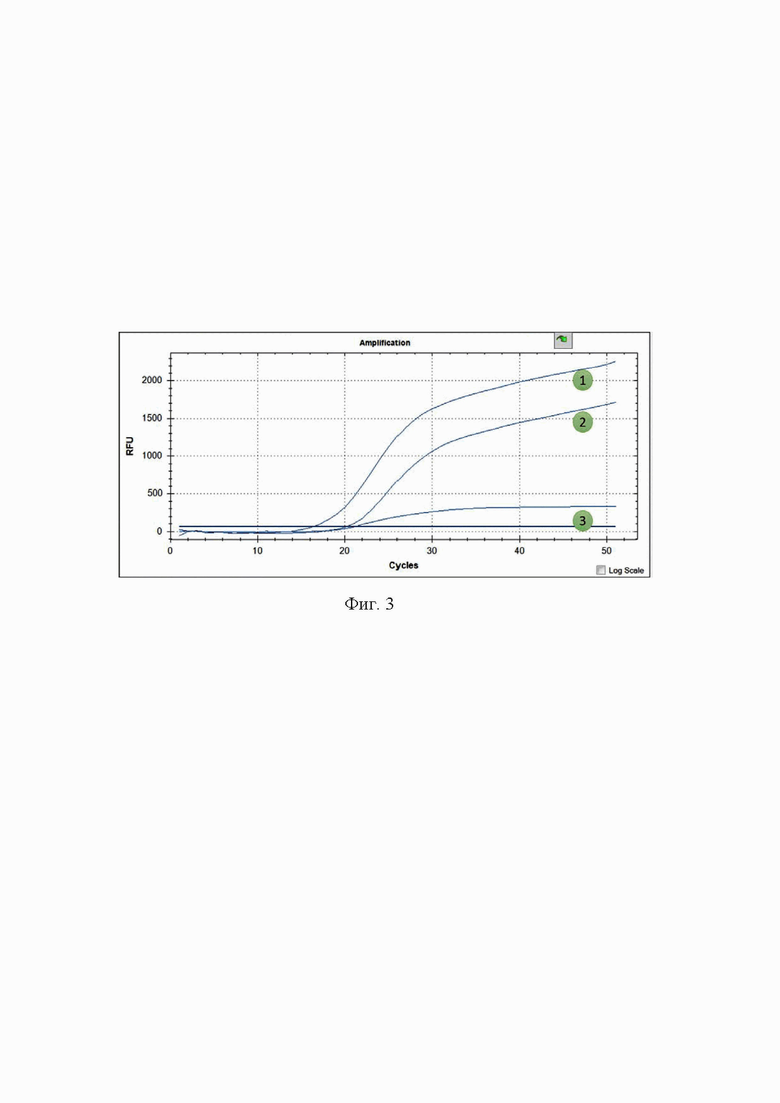
Фиг. 3. Идентификация цереулида: 1 - положительный контроль; 2 - тестируемая ДНК; 3 - отрицательный контроль. Канал считывания флюорофора FAM.
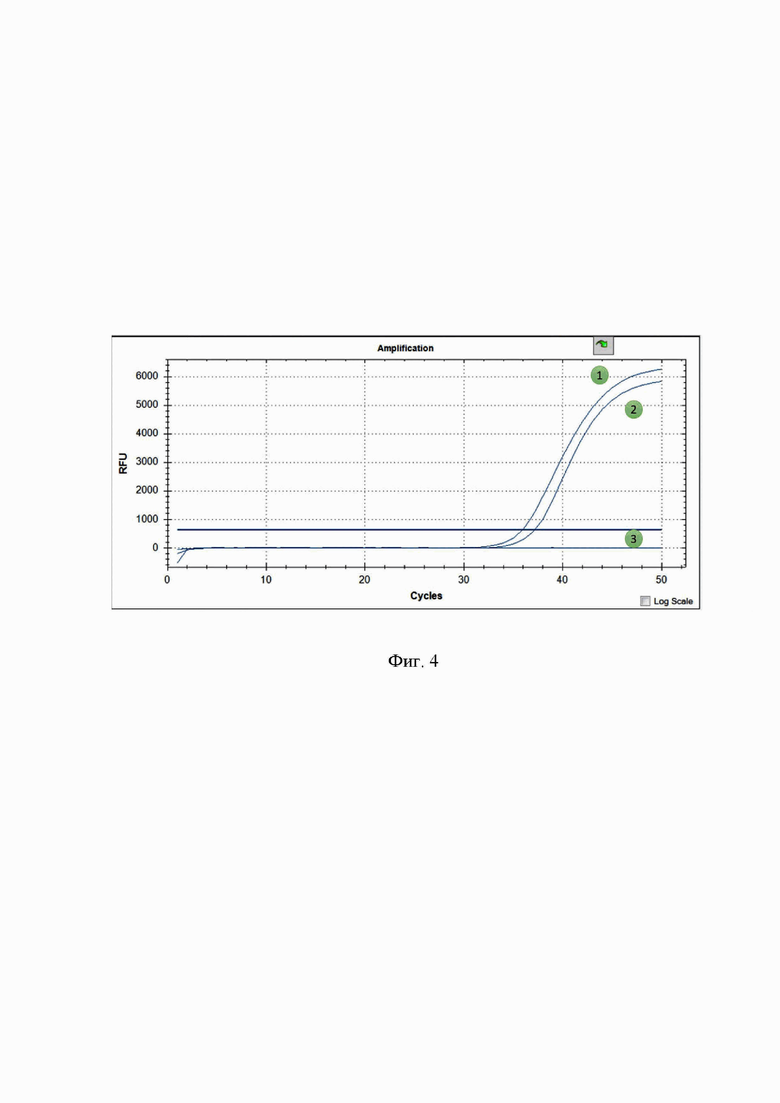
Фиг. 4. Идентификация гемолизина BL: 1 - положительный контроль; 2 - тестируемая ДНК; 3 - отрицательный контроль. Канал считывания флюорофора FAM.
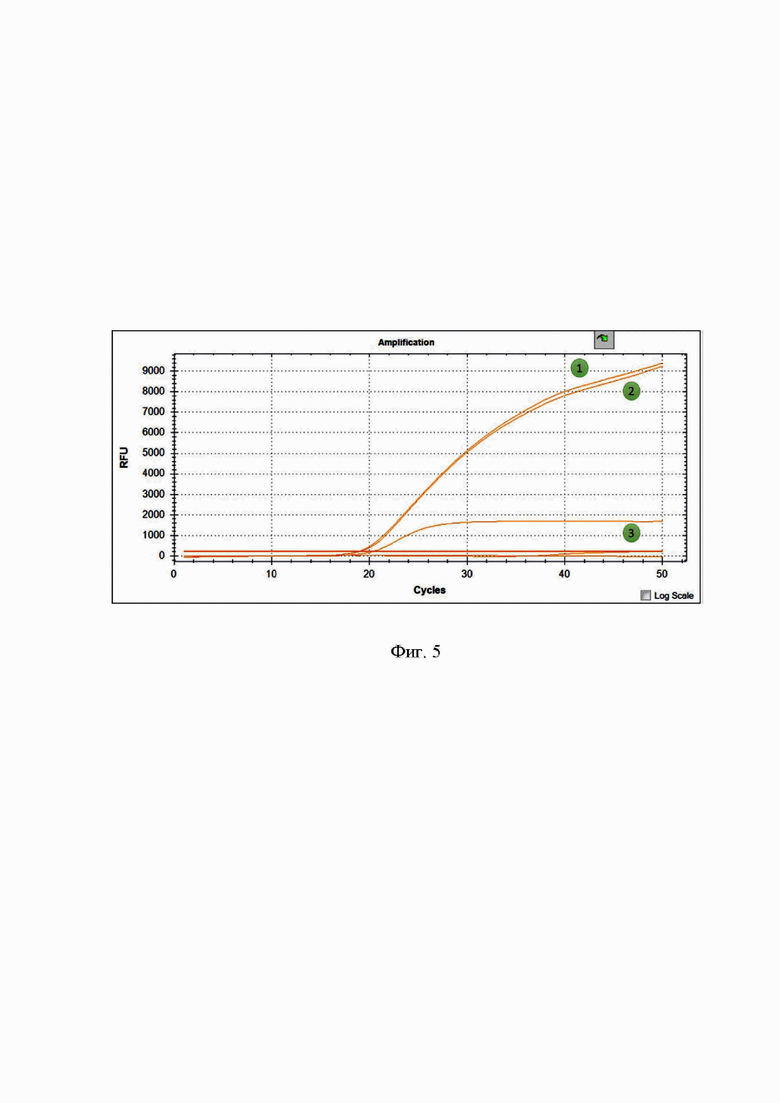
Фиг. 5. Идентификация негемолитического энтеротоксина А: 1 - положительный контроль; 2 - тестируемая ДНК; 3 - отрицательный контроль. Канал считывания флюорофора ROX.
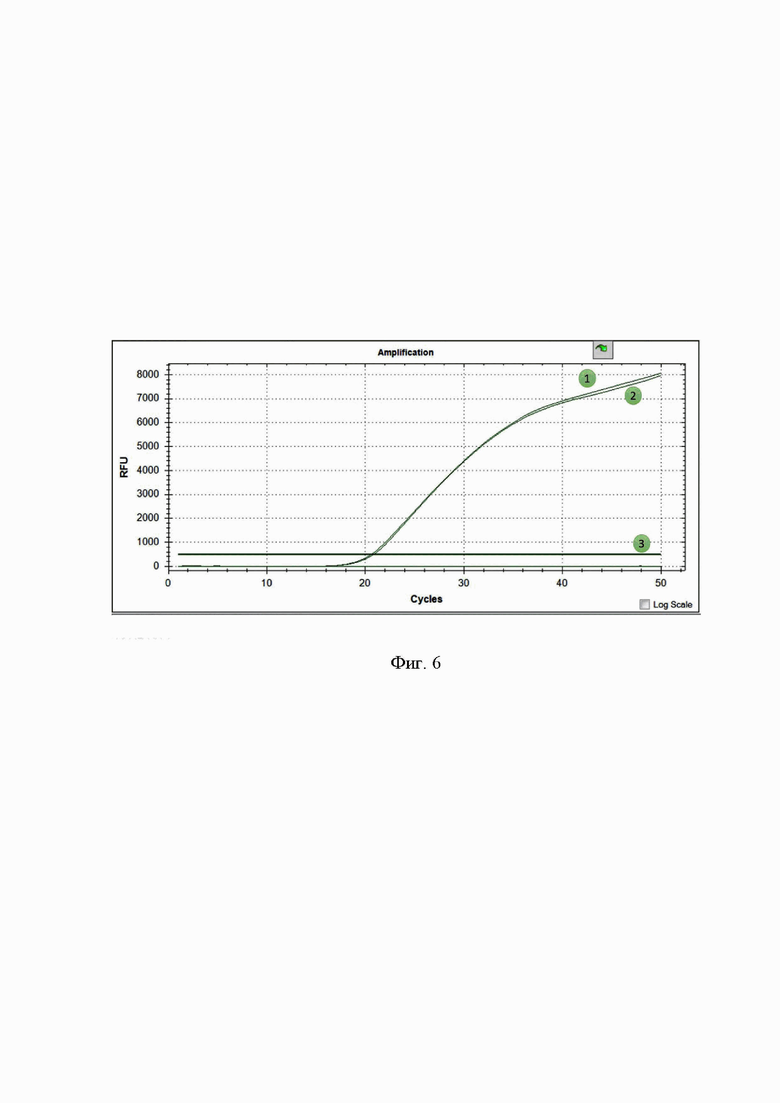
Фиг. 6. Идентификация энтеротоксина FM: 1 - положительный контроль; 2 - тестируемая ДНК; 3 - отрицательный контроль. Канал считывания флюорофора RG6, данные калибровки амплификатора попадают под световой диапазон HEX.
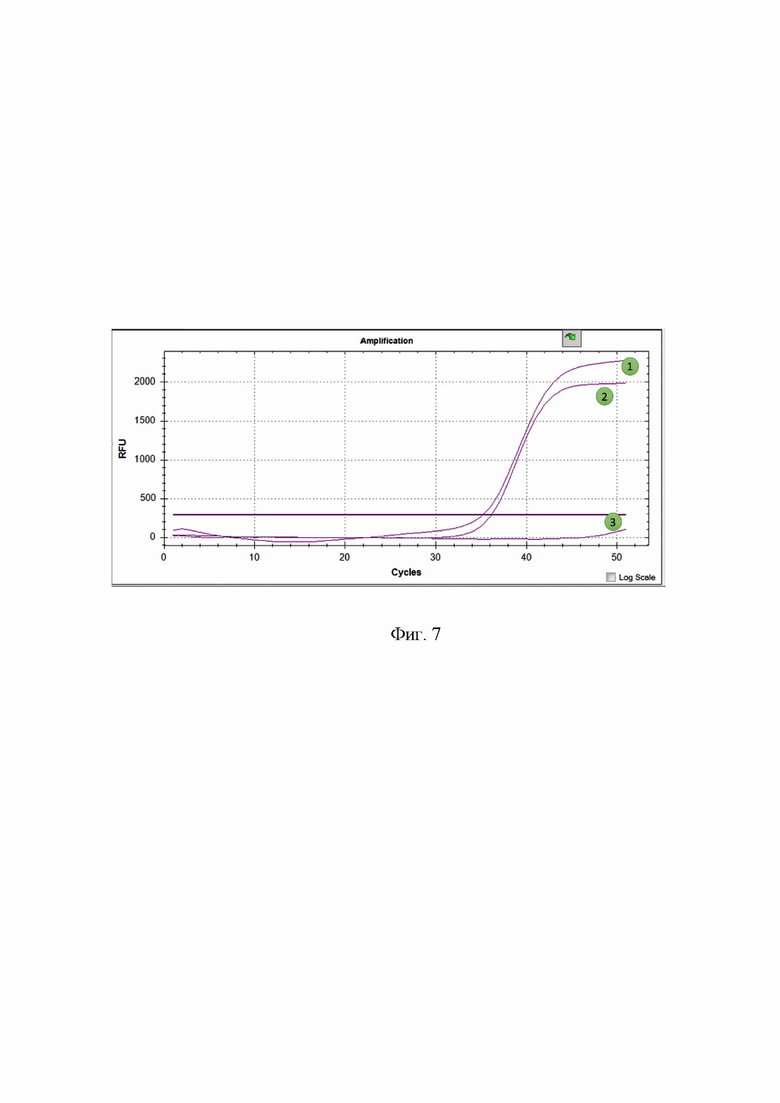
Фиг. 7. Контроль работы внутреннего положительного контроля тест-системы: 1 - положительный контроль (ПЦР смесь 1); 2 - положительный контроль (ПЦР смесь 2); 3 - отрицательный контроль. Канал считывания флюорофора Cy5.
ПРИМЕР
С целью раскрытия сущности заявленного технического решения для осуществления его специалистом в данной области техники приводится нижеследующий пример осуществления заявленного технического решения.
Для индикации цереулида и диарейных энтеротоксинов Bacillus cereus complex с помощью ПЦР-РВ были подготовлены штаммы B. cereus из рабочей коллекции как это принято в микробиологической практике и осуществлена экстракция ДНК с помощью коммерческих тес-наборов для экстракции ДНК из исследуемого материала. Затем была подготовлена реакционная смесь 1 и реакционная смесь 2 для постановки ПЦР-РВ. ПЦР смесь 1 включает праймеры и флуоресцентно-меченые зонды для идентификации: цитотоксин К, цереулида и внутреннего положительного контроля. ПЦР смесь 2 включает праймеры и флуоресцентно-меченые зонды для идентификации: гемолизина BL, негемолитического энтеротоксина А, энтеротоксина FM и внутреннего положительного контроля. Внутренние положительные контроли смесей идентичны и в данном случае представляют нуклеотидные последовательност 5'-3': tgtactgagagtgcaccatatgcg (PUC-F), 5'-3': aatttcacacaggaaacagctatgacc (PUC-R) и 5'-3': gatgtgcaaggcgattaagt (PUC-Z). Вместе с тем, в качестве положительных контролей могут использоваться любые контроли, обычно используемые на практике для постановки ПЦР-РВ, поэтому используемые в настоящем примере контроли не следует использовать как основание для ограничения объема исспрашиваемых прав по настоящей заявке.
В лунки микропланшета были внесены ПЦР смеси 1 и 2 по 10 мкл каждая. Затем были добавлены ДНК-полимераза, смесь дезоксинуклеозидтрифосфатов и ПЦР-буфер (Mg2+) по 5 мкл, а также деионизированная вода по 9 мкл. В качестве указанных компонентов могут использоваться коммерческие наборы. Затем в соответствующие лунки были добавлены контроли (в том числе соответствующие токсины для проверки правильности работы праймеров, которые не входят в набор и не используются в постановке) по 1 мкл и исследуемые образцы (ДНК, полученная на этапе экстракции) по 1 мкл. Таким образом, объем реакционного раствора составляет 25 мкл, а одна постановка включает:
ПК
О
Т
ОКО
ПК
О
Т
ОКО
где ПК - положительный контроль, ОКО - отрицательный контроль, ПЦР1 - ПЦР смесь 1 (включая ДНК-полимеразу, смесь дезоксинуклеозидтрифосфатов, ПЦР-буфер (Mg2+) и деионизированную воду), ПЦР2 - ПЦР смесь 2 (включая ДНК-полимеразу, смесь дезоксинуклеозидтрифосфатов, ПЦР-буфер (Mg2+) и деионизированную воду), О - образец, Т - смесь токсинов.
Для проведения амплификации использовали амплификатор CFX96, однако возможно использование любого амплификатора, обычно используемого на практике для постановки ПЦР-РВ, поэтому используемый в настоящем примере амплификатор не следует использовать как основание для ограничения объема исспрашиваемых прав по настоящей заявке.
Амплификацию проводили по следующей программе термоциклирования:
* считывание свечения
Полученные результаты (Фиг. 1) интерпретировали по каналам детекции FAM, HEX, ROX и Cy5.
ПЦР смесь 1:
цитотоксин К идентифицируется на основании характерного свечения, выраженного экспоненциальным графиком (Фиг. 2) по каналу ROX.
целеулид идентифицируется на основании характерного свечения, выраженного экспоненциальным графиком (Фиг. 3) по каналу FAM.
ПЦР смесь 2:
гемолизин BL идентифицируется на основании характерного свечения, выраженного экспоненциальным графиком (Фиг. 4) по каналу FAM.
негемолитический энтеротоксин А идентифицируется на основании характерного свечения, выраженного экспоненциальным графиком (Фиг.5) по каналу ROX.
энтеротоксин FM идентифицируется на основании характерного свечения, выраженного экспоненциальным графиком (Фиг. 6) по каналу HEX.
положительные контроли идентифицируются на основании характерного свечения, выраженного экспоненциальным графиком (Фиг. 7) по каналу Cy5. Отрицательный контроль идентифицируется на основании отсутствия свечения по любому из каналов (Фиг. 2 - Фиг. 7).
Список используемых источников литературы:
1. Ребриков, Д.В. ПЦР «в реальном времени» / Д.В. Ребриков, Г.А. Саматов, Д.Ю. Трофимов; под ред. д.б.н. Д.В. Ребрикова; предисл. Л.А. Остермана и акад. РАН и РАСХН Е.Д. Свердлова; 2-е изд., испр. и доп. - М.: БИНОМ. Лаборатория знаний, - 2009. - C. 233.
2. Rasko, D.A. Genomics of the Bacillus cereus group of organisms / D.A. Rasko, M.R. Altherr, C.S. Han, J. Ravel // FEMS Microbiol Rev. - 2005. - Vol. 29, N. 2. - P. 303-329.
3. Ehling-Schulz, M. Identification and partial characterization of the nonribosomal peptide synthetase gene responsible for cereulide production in emetic Bacillus cereus / M. Ehling-Schulz, N. Vukov, A. Schulz, R. Shaheen, M. Andersson, E. Märtlbauer, S. Scherer // Appl Environ Microbiol. - 2005. - Vol. 71, - Р. 105-113.
Ehling-Schulz, M. The Bacillus cereus Group: Bacillus species with pathogenic potential / M. Ehling-Schulz, D. Lereclus, T.M. Koehler // Microbiol Spect. - 2019. - Vol. 7, N3. - P. e. 1128.
4. Granum, P.E. The sequence of the non-haemolytic enterotoxin operon from Bacillus cereus / P.E. Granum, K. O'Sullivan, T. Lund // FEMS Microbiol Lett. - 1999. - Vol. 177 - Р. 225-229.
5. Heinrichs, J.H. Molecular cloning and characterization of the hblA gene encoding the B component of hemolysin BL from Bacillus cereus / J.H. Heinrichs, D.J. Beecher, J.D. MacMillan, B.A. Zilinskas // J Bacteriol. - 1993. - Vol. 175. - Р. 6760-6766.
6. Kramer, J.M. Bacillus cereus and other Bacillus species / J.M. Kramer, R.J. Gilbert // Foodborne Bacterial Pathogens. - 1989. - Р. 21-70.
7. Lindbäck, T. Insertional inactivation of hblC encoding the L2 component of Bacillus cereus ATCC 14579 haemolysin BL strongly reduces enterotoxigenic activity, but not the haemolytic activity against human erythrocytes / T. Lindbäck, O.A. Økstad, A.L. Rishovd, A.B. Kolstø // Microbiology - 1999. - Vol. 145. - Р. 3139-3146.
8. Lund, T.D. A new cytotoxin from Bacillus cereus that may cause necrotic enteritis / T.D. Lund, M.L. Buyser, P.E. Granum // Mol Microbiol. - 2000. - Vol. 38. - Р. 254-261.
9. Rasko, D.A. Complete sequence analysis of novel plasmids from emetic and periodontal Bacillus cereus isolates reveals a common evolutionary history among the B. cereus-group plasmids, including Bacillus anthracis pXO1 / D.A. Rasko, M.J. Rosovitz, O.A. Økstad, D.E. Fouts, L. Jiang, R.Z. Cer, A.B. KolstØ, S.R. Gill, J. Ravel // J Bacteriol. - 2007. - Vol. 189. - Р. 52-64.
10. Stenfors Arnesen, L.P. From soil to gut: Bacillus cereus and its food poisoning toxins / L.P. Stenfors Arnesen, A. Fagerlund, P.E. // Granum. FEMS Microbiol Rev. - 2008. - Vol. 32, N4. - Р. 579-606.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Real-time PCR test
system for the detection of cereulide and diarrheal enterotoxins
Bacillus cereus.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-09-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-05-08</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Federal Budget Institution of
Science «STATE RESEARCH CENTER FOR APPLIED MICROBIOLOGY AND
BIOTECHNOLOGY»</ApplicantName>
<ApplicantNameLatin>Federal Budget Institution of Science STATE
RESEARCH CENTER FOR APPLIED MICROBIOLOGY AND
BIOTECHNOLOGY</ApplicantNameLatin>
<InventorName languageCode="ru">Khlopova Kseniya</InventorName>
<InventorNameLatin>Khlopova Kseniya</InventorNameLatin>
<InventionTitle languageCode="ru">A set of oligodeoxyribonucleotide
primers and fluorescently labeled probes for the detection of
Bacillus cereus diarrheal and vomiting toxin DNA</InventionTitle>
<SequenceTotalQuantity>15</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctcgcacatatctatatgaatctgatgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agttccaaatgtgtagcctggacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtttctctcctggtatgatcgctgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagggttattggttacagcagtatctacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caattagcttcggattatattcatcaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttacgctaaggaggggcaaacggaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcctcttcattacgaaaattaggtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcatagccatttcttgaagtacttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctaatcaaacaagatatgaaggaatggtca</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaactggtggttcttatgttgttaacact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttacaaacttaacgaagtctgcacttacgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agctacatacaacgctgtaatcggtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctctggtaaatggctaatcgga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcatagtatgggtgctttggcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus cereus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgatatcgcggtgcttctcttcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области биотехнологии, в частности к набору олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для детекциии генов, кодирующих цереулид и диарейные энтеротоксины B. cereus complex, причем указанные энтеротоксины представляют собой цитотоксин К, гемолизин BL, негемолитический энтеротоксин А и энтеротоксин FM. Указанный набор содержит праймеры с последовательностями SEQ ID No. 1, 2, 4, 5, 7, 8, 10, 11, 13 и 14, а также флуоресцентно-меченые ДНК-зонды с последовательностями SEQ ID No. 3, 6, 9, 12 и 15. Настоящее изобретение обеспечивает выявление ампликонов в исследуемых образцах без использования дополнительных методов. 2 з.п. ф-лы, 7 ил., 1 табл., 1 пр.
1. Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченых зондов для детекциии генов, кодирующих цереулид и диарейные энтеротоксины, представляющие собой цитотоксин К, гемолизин BL, негемолитический энтеротоксин А и энтеротоксин FM Bacillus cereus complex с помощью метода ПЦР-РВ, включающий:
а) набор праймеров и зонда для детекции генов, кодирующих цитотоксин К, состоящий из прямого праймера с последовательностью SEQ ID No.1, обратного праймера с последовательностью SEQ ID No.2 и флуоресцентно-меченого ДНК-зонда с последовательностью SEQ ID No.3;
b) набор праймеров и зонда для детекции генов, кодирующих негемолитический энтеротоксин А, состоящий из прямого праймера с последовательностью SEQ ID No.4, обратного праймера с последовательностью SEQ ID No.5 и флуоресцентно-меченого ДНК-зонда с последовательностью SEQ ID No.6;
с) набор праймеров и зонда для детекции генов, кодирующих гемолизин BL, состоящий из прямого праймера с последовательностью SEQ ID No.7, обратного праймера с последовательностью SEQ ID No.8 и флуоресцентно-меченого ДНК-зонда с последовательностью SEQ ID No.9;
d) набор праймеров и зонда для детекции генов, кодирующих энтеротоксин FM, состоящий из прямого праймера с последовательностью SEQ ID No.10, обратного праймера с последовательностью SEQ ID No.11 и флуоресцентно-меченого ДНК-зонда с последовательностью SEQ ID No.12;
e) набор праймеров и зонда для детекции генов, кодирующих цереулид, состоящий из прямого праймера с последовательностью SEQ ID No.13, обратного праймера с последовательностью SEQ ID No.14 и флуоресцентно-меченого ДНК-зонда с последовательностью SEQ ID No.15;
f) набор праймеров для внутреннего контроля амплификации, применяемых для постановки ПЦР;
где ДНК-зонды, состоящие из последовательностей SEQ ID No.3, SEQ ID No.6, SEQ ID No.9, SEQ ID No.12 и SEQ ID No.15, детектируются по каналам ROX, ROX, FAM, RG6 и FAM соответственно, а положительный и отрицательный контроли, используемые для постановки ПЦР-РВ, детектируются по каналу Cy5 и по каналам ROX, FAM и RG6 соответственно.
2. Набор по п.1, в котором в качестве внутреннего положительного контроля реакции используется плазмида E.coli pUC19, детектируемая по каналу Cy5.
3. Набор по п.1, где вместо детекции по каналу R6G осуществляется детекция по каналу HEX.
| US 20130059756 A1, 07.03.2013 | |||
| DIETRICH, R | |||
| et al | |||
| The Food Poisoning Toxins of Bacillus cereus, Toxins, 2021, 13 (2), 98, pp | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| RAMARAO, N | |||
| et al | |||
| Advanced Methods for Detection of Bacillus cereus and Its Pathogenic Factors, Sensors, 2020, 20, no | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| РЯБИНИН И.А | |||
| Редкие возбудители кишечных и токсикоинфекций (краткий обзор), | |||
Авторы
Даты
2025-05-05—Публикация
2024-07-25—Подача