Изобретение относится к области биотехнологии и молекулярной биологии, а именно к способам молекулярно-генетической идентификации микроорганизмов. Изобретение может использоваться в фитосанитарном контроле, фитопатологии и микологии для специфического определения и обнаружения в растительных тканях септориоза (в англоязычной литературе - septoria leaf spot complex).
Предпосылки создания изобретения.
Болезни сельскохозяйственных растений, вызываемые грибами, наносят существенный ущерб урожаю и приводят к значительным экономическим потерям [1, 2].
В настоящее время септориоз является одним из наиболее вредоносных и экономически значимых заболеваний пшеницы в мире, особенно в странах с умеренным климатом. Потери от болезни в эпифитотийные годы могут достигать 20-40%. В связи с неблагоприятной обстановкой в стране по данному виду заболевания требуется быстрая диагностика фитопатогенов для своевременного реагирования и принятия необходимых мер и создания устойчивых генотипов [3].
Видовой состав возбудителей септориозных пятнистостей пшеницы на территории Российской Федерации представлен, главным образом, видами: Zymoseptoria tritici, Parastagonospora nodorum [4, 5, 6].
Z. tritici развивается на листьях, листовых влагалищах, реже на стеблях пшеницы. Заболевание характеризуется появлением на листьях продолговатой линейной пятнистости, располагающейся между жилками листа. Позднее на верхней и нижней стороне пораженного листа обнаруживаются хорошо заметные черные почковидные пикниды возбудителя, располагающиеся рядами вдоль жилок [7] (Пыжикова и др., 1989). P. nodorum является возбудителем септориоза колосковой чешуи и поражает пшеницу, тритикале, рожь.
Разработанные в настоящее время коммерческие тест-системы «Септориоз пшеницы (Septoria nodorum)» и «Септориоз пшеницы (Septoria tritici)» ООО «АгроДиагностика» для Real-time ПЦР (полимеразно-цепьевой реакции) позволяют выявить патогены с использованием дорогостоящего оборудования. Анализ публикаций [3, 9, 10] (Кожабергенов и др., 2020, Fraaije et al., 2001, Бейшова и др. 2015) свидетельствует об отсутствии праймеров на основе высокоспецифичного гена COXIII для фитопатогенов Z. tritici и P. nodorum.
По совокупности признаков наиболее близким по технической и технологической сущности к предлагаемому изобретению и принимается за прототип способ «Определение грибковых патогенов с использованием реакций полимеризации цепи» по патенту RU 2161196 C2 [8], недостатки которого те же - отсутствие праймеров на основе высокоспецифичного гена COXIII для фитопатогенов Z. tritici и P. Nodorum и высокая стоимость анализа.
Задачей изобретения является: разработка способа идентификации фитопатогенных грибов, позволяющего точно и быстро, с меньшими затратами идентифицировать фитопатогенный комплекс септориоза, включающий виды Z. tritici и P. nodorum в растительных тканях, с использованием метода ПЦР; достижение следующих технических результатов при использовании данного способа:
- прямое распознавание патогена и высокая специфичность анализа (идентификация Z. tritici и P. nodorum независимо от возможного присутствия в исследуемых пробах других видов грибов);
- высокая скорость проведения анализа (получение результатов анализа в течение 1 рабочего дня);
- определение фитопатогенного комплекса септориоза пшеницы на любых стадиях заболевания растений, в том числе самых ранних, еще при отсутствии внешних признаков поражения;
- решение других задач (мониторинг септориоза в посевах, подтверждение или дополнение результатов фитопатологической экспертизы, контроль заражения посадочного материала).
Решает поставленные задачи и достижение заявляемого технического результата изобретение - Способ идентификации фитопатогенных грибов Zymoseptoria tritic и Parastagonospora nodorum методом ПЦР.
Способ идентификации фитопатогенных грибов Zymoseptoria tritici и Parastagonospora nodorum методом ПЦР, отличающийся тем, что основан на использовании праймеров: COXIII-Nodorum-F: 5'-GTATTTACTTTATTATGTGCCCTC-3', COXIII-Nodorum-R: 5'-AATGGGTTAAATATATTATACTTAACG-3' для определения вида Parastagonospora nodorum и COXIII-Tritici-F: 5'-GCCTTAATACTTTTAATGTCAAGTATG-3', COXIII-Tritici-R: 5'-CGGTAATTCAAAAGCGTTAATTGC-3' для определения вида Zymoseptoria tritici, предусматривающих выделение ДНК из образцов биологического материала, проведение с выделенной ДНК полимеразной цепной реакции ПЦР и детекцию продуктов амплификации путем сравнения по размеру с маркерными ДНК-фрагментами маркера молекулярных масс, при котором наличие в пробе ПЦР-продуктов размером 194 или 255 пар нуклеотидов при использовании праймеров COXIII-Nodorum-F и COXIII-Nodorum-R или COXIII-Tritici-F и COXIII-Tritici-R соответственно, свидетельствует о присутствии в исследуемом образце фитопатогенов Parastagonospora nodorum или Zymoseptoria tritici соответственно.
Изобретение (способ) заключается в следующем.
Технический результат по идентификации фитопатогенных грибов достигается тем, что для проведения ПЦР подбираются олигонуклеотидные праймеры, специфичные к области ДНК, характерной для каждого из видов грибов, входящих в фитопатогенный комплекс септориоза пшеницы.
В соответствии с поставленной задачей проведен поиск генетических маркеров септориозного комплекса пшеницы: выбран ген COXIII, кодирующий фермент цитохром-с-оксидазу, представляющую собой часть белкового комплекса электрон-транспортной цепи. Биоинформатический анализ нуклеотидных последовательностей указанных маркеров проведен по базе данных GenBank (https://www.ncbi.nlm.nih.gov/nuccore). Проанализировано по 2 митохондриальных генома Z. tritici и P. nodorum. Проведено множественное выравнивание выбранных последовательностей внутри групп и между характерными представителями групп, выявлены полиморфизмы, отличающих ДНК видов Z. tritici и P. nodorum от других видов указанных родов, а также от других микромицетов.
Множественное выравнивание выбранных последовательностей проводили с помощью программы MEGA X, работающей на основе алгоритма ClustalW.
Дизайн праймеров, определение идентичности и специфичности олигонуклеотидных последовательностей проведены с помощью сервиса Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn).
Термодинамические характеристики, специфичность, отсутствие само- и взаимокомплементарностей, повторов определены в приложении Oligo Analysis. По выбранным последовательностям олигонуклеотиды были синтезированы фосфоамидитным методом.
Технический результат достигается тем, что проводится полимеразная цепная реакция (ПЦР), для которой используются олигонуклеотиды - праймеры, специфичные к области ДНК фрагмента COXIII, имеющей отличия в последовательности, характерные для каждого из видов Z. tritici и P. nodorum.
Способ основан на выделении ДНК из образцов биологического материала, проведении с выделенной ДНК полимеразной цепной реакции с использованием пар праймеров COXIII-Nodorum-F и COXIII-Nodorum-R для определения микроорганизма P. nodorum и COXIII-Tritici-F и COXIII-Tritici-R для определения микроорганизма Z. tritici. Праймеры специфичны для области генов COXIII митохондриальной ДНК и ограничивают участок ДНК размером 194 пар нуклеотидов у P. nodorum и участок ДНК размером 255 пар нуклеотидов у вида Z. tritici.
Анализ продуктов ПЦР выполняется с помощью электрофореза в 6% нативном полиакриламидном геле (ПААГ). Визуализация продуктов электрофоретического разделения и регистрацию результатов проводят на установке для гель-документации при ультрафиолетовой подсветке после предварительного окрашивания ДНК бромистым этидием.
Наличие на электрофореграмме ПЦР-продуктов размером 194 п.н. свидетельствует о присутствии в исследуемом образце ДНК P. nodorum, наличие ПЦР-продуктов размером 255 п.н свидетельствует о присутствии исследуемом образце ДНК Z. tritici.
При этом используемые праймеры имеют следующие нуклеотидные последовательности:
COXIII-Nodorum-F: 5'- GTATTTACTTTATTATGTGCCCTC -3',
COXIII-Nodorum-R: 5'- AATGGGTTAAATATATTATACTTAACG -3',
COXIII-Tritici-F: 5'-GCCTTAATACTTTTAATGTCAAGTATG -3',
COXIII-Tritici-R: 5'-CGGTAATTCAAAAGCGTTAATTGC -3',
ПЦР проводится в следующем режиме: 1 цикл - 94°С/5 мин; 35 циклов, включающих 94°С/30 сек, 42°С/30 сек, 72°С/90 сек; 1 цикл - 72°С/5 мин. Дополнительная элонгация 72°С/10 мин.
Выделение и подготовка ДНК для ПЦР-анализа микромицетов данным способом проводится любым коммерческим набором для выделения ДНК из растений и грибов. Образцы ДНК, пригодные для быстрого ПЦР-анализа могут быть получены путем механического лизиса, либо с помощью лизирующего буфера на основе цетилтриэтиламмония бромида либо гуанидинтиоционата и сорбента «диатомовая земля».
Разработанный способ позволяет проводить раннюю диагностику септориоза при отсутствии видимых морфологических признаков наличия фитопатогена напрямую с предположительно инфицированных листьев.
Пример 1.
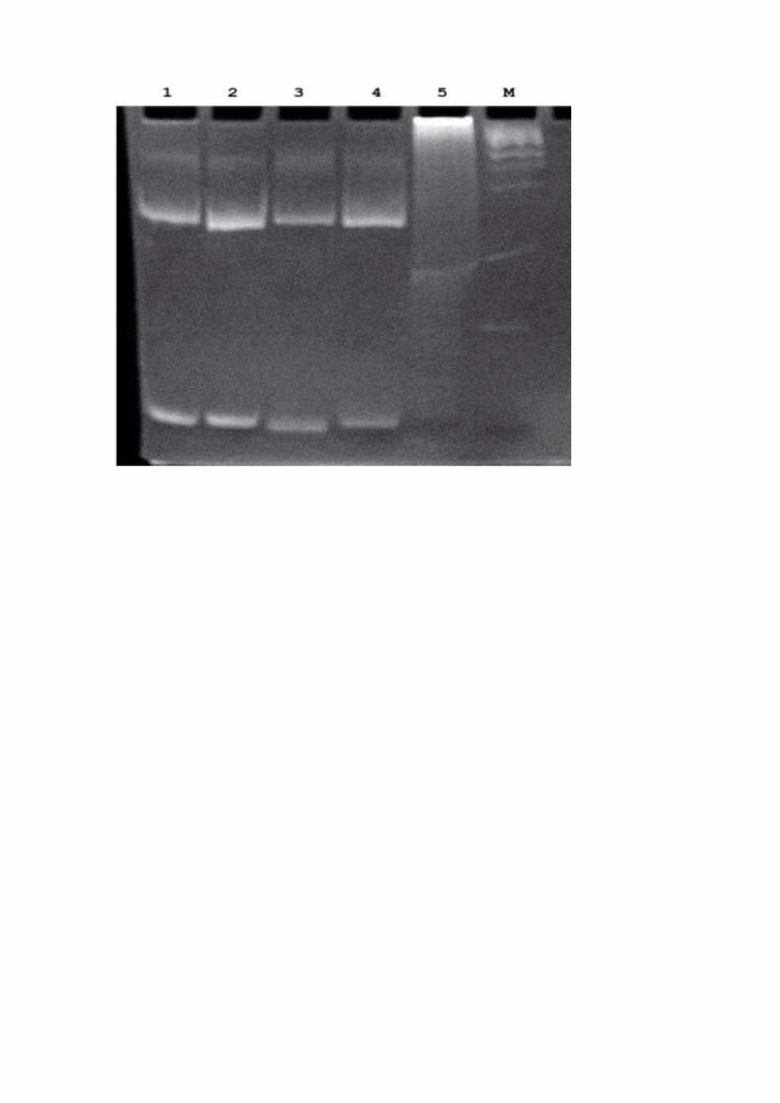
Апробация данного способа была проведена на микробиологических культурах Z. tritici, депонированных из коллекции Всероссийского научно-исследовательского института фитопатологии (ФГБНУ ВНИИФ) (штаммы НН 2-2, НН 4-4, НН 4-5, НН 4-7, выделенных в Нижегородской области), и представлена на фигуре 1.
Пример 2.
Апробация данного способа была проведена на микробиологических культурах Z. tritici, депонированных из коллекции Всероссийского научно-исследовательского института фитопатологии (ФГБНУ ВНИИФ) (штаммы НН 4-4, НН 4-5, выделенных в Нижегородской области), а также микробиологических культурах P. nodorum из коллекции ФАНЦ Северо-Востока имени Н.В. Рудницкого (штаммы 4w3511 и 4w3513, выделенные на территории Кировской области).
Мицелий фитопатогенных грибов отбирают с поверхности чашки Петри с агаризованной картофеле-глюкозной среды одноразовым шпателем в количестве 250 мг, переносят в пластиковую микропробирку объемом 2 мл, содержащую буфер ТЕ(10 мМ Трис, 1 мМ ЭДТА, рН 8,0) и микросферы для гомогенизации клеток, гомогенизируют механически ручным способом стерильным наконечником, центрифугируют при 14000g в течении 3 минут, надосадочную жидкость прогревают при переносят в чистую микропробирку, нагревают 10 мин при 95°С для дезактивирования возможных примесей нуклеаз и используют для ПЦР-амплификации.
Амплификацию ДНК проводят следующим образом:
Для постановки ПЦР используют, в микропробирки объемом 200 мкл, в которые вносят компоненты ПЦР-реакции из расчета: ДНК (50 нг) 2 мкл, 1 мкл 10-кратного буфера с Mg, 0,5 мкл смеси dNTP по 4 ммоль каждого нуклеотида, 10 мкл праймера в концентрации 10 пикомоль/мкл, 0,75 мкл taq-полимеразы (5ед. активности в мкл), воды до 10 мкл, 30 мкл минерального масла. Амплификацию проводили на программируемом термостате (амплификаторе) «Терцик» МС2 (ЗАО «НПФ ДНК-Технология», Москва, РФ) в следующем режиме: 1 цикл - 94°С/5 мин; 35 циклов, включающих 94°С/30 сек, 42°С/30 сек, 72°С/90 сек; 1 цикл - 72°С/7 мин.
Полученные продукты электрофоретического разделения в 6% нативном полиакриламидном геле, соответствующие ожидаемым длинам 194 п.н. для P. nodorum и 255 п.н. для Z. tritici, были элюированы из геля и отправлены на автоматическое секвенирование по Сэнгеру в ООО «Синтол».
Каждый полученный фрагмент ДНК был поочередно проверен прямым и обратным праймером. Таким образом получено 8 последовательностей продуктов ПЦР, которые проанализированы с помощью сервиса Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn).
- НН 4-4 COXIII-Tritici-F
AGGTTTAAATATGGGAGTATCCTTATTTATTATATCTGAAGCTTTATTTTTCTTAGCTATATTCTGAGCCTATTTTCATAGTGCTCTTTCACCTACTGTAGAATTAGGTGCACAATGACCTCCTATGGGTATAGAAGCAATTAACGTTTTTGAATTACCGA
Результат BLAST: Select seq MH374028.1 Zymoseptoria tritici isolate IPO323 mitochondrion, complete genome процент совпадения 99, 38%.
- НН 4-4 COXIII-Tritici-R
TTGTGCCCTAATTCTACGTAGGTGAAAGAGCACTATGAAAATAGGCTCAGAATATAGCTAAGAAAAATAAAGCTTCAGATATAATAAATAAGGATACTCCCATATTTAAACCTTTCTGTACAGCCAGAGTATGGTTACCTATGTAAGTACCTTCAGATATTACATCTCTAAATCAGAAAAACATACTTGACATTAAAGGGTATAAGACGCATAA
Результат BLAST: Select seq MH374028.1 Zymoseptoria tritici isolate IPO323 mitochondrion, complete genome процент совпадения 98.99%.
- НН 4-5 COXIII-Tritici-F
ATCTGAAGGTACTTACATAGGTAACCATACTCTGGCTGTACAGAAAGGTTTAAATATGGGAGTATCCTTATTTATTATATCTGAAGCTTTATTTTTCTTAGCTATATTCTGAGCCTATTTTCATAGTGCTCTTTCACCTACTGTAGAATTAGGTGCACAATGACCTCCTATGGGTATAGAAGCAATTAACGCTTTTGAATTACCGA
Результат BLAST: Select seq MH374028.1 Zymoseptoria tritici isolate IPO323 mitochondrion, complete genome процент совпадения 100%.
- НН 4-5 COXIII-Tritici-R
AGGTGAAAGAGCACTATGAAAATAGGCTCAGAATATAGCTAAGAAAAATAAAGCTTCAGATATAATAAATAAGGATACTCCCATATTTAAACCTTTCTGTACAGCCAGAGTATGGTTACCTATGTAAGTACCTTCAGATATTACATCTCTAAATCAGAAAAACATACTTGACATTAAAAGTATTAAGGCA
Результат BLAST: Select seq MH374028.1 Zymoseptoria tritici isolate IPO323 mitochondrion, complete genome процент совпадения 100%.
- 4W3511 COXIII-Nodorum-R:
CGTCCTTTGGTTTCCTGCTGATTAAGCATAGCTTACAAGATTTTTACGATATAAGTACTTATAAACACTGAGCTATTCCAGCATATAGTTAAATTTAAAGAGGGCACATAATAAAGTAAATACA
Результат BLAST: Phaeosphaeria nodorum SN15 mitochondrion, complete genome процент совпадения 99,19%.
- 4W3513 COXIII-Nodorum-F:
ATAAGTACTTAATCGTAAAAATCTTGTAAGCTATGCTTAATCAGCAGGAAACCAAAGGACGGATAAATATCCTAGTAGCATCCTCAGAGACTACACGCAAATAATTAATACGTTAAGTATAATATATTTAACCCATACA
Результат BLAST: Phaeosphaeria nodorum SN15 mitochondrion, complete genome процент совпадения 100%.
- 4W3513 COXIII-Nodorum-R:
CTAGGATATTTATCCGTCCTTTGGTTTCCAGCTGATTAAGCATAGCTTACAAGATTTTTACGATTAAGTACTTATAAACACTGAGCTATTCCAGCATATAGTTAAATTTAAAGAGGGCACATATAAAGTAAATACAATGGT
Результат BLAST: Phaeosphaeria nodorum SN15 mitochondrion, complete genome - процент совпадения составляет 98,55%.
Следовательно, подтверждена высокая специфичность праймеров на обнаружение возбудителей септориоза P. nodorum и Z. tritici.
Также достоинством разработанной нами тест-системы является более низкая стоимость, по сравнению с коммерческим аналогом. При необходимости использования ПЦР в режиме реального времени возможно разработать флуоресцирующие зонды.
Таким образом, новое изобретение «Способ идентификации фитопатогенных грибов Zymoseptoria tritici и Parastagonospora nodorum методом ПЦР» позволяет эффективно, с наименьшими затратами идентифицировать фитопатогенный комплекс септориоза видов Z. tritici и P. Nodorum, а также получить следующие технические результаты при использовании данного способа: прямое распознавание патогена и высокую специфичность анализа (идентификация Z. tritici и P. nodorum независимо от возможного присутствия в исследуемых пробах других видов грибов), высокую скорость (в течение 1 дня) проведения анализа, определение фитопатогенного комплекса септориоза пшеницы на любых стадиях заболевания растений, в том числе самых ранних, и др.
Литература
1. Eyal Z. The Septoria tritici and Stagonospora nodorum blotch diseases of wheat [Text] / Z. Eyal // Eur. J. Plant Pathol. - 1999. - V. 105, №7. - Р. 629-641.
2. Санина А.А., Пахолкова Е.В. Видовая структура популяций возбудителей септориоза пшеницы в различных регионах России [Текст] / А.А. Санина, Е.В. Пахолкова // Сборник трудов 1-го съезда микологов России. - Москва, 2002. Разд. 7. - 221 с.
3. Кожабергенов, Н.С., Тайлакова, Э. Т., Исабек, А.У., Бурашев, Е.Д., Султанкулова, К.Т., & Рсалиев, А.С. Разработка дуплексной тест-системы ПЦР для дифференциальной диагностики сеториоза пшеницы // Вестник КазНУ. Серия экологическая. - 2020. - Т. 63. - №. 2. - С. 63-70.
4. Санина А.А., Анциферова Л.В. Видовой состав грибов рода Septoria Sacc. на пшенице в европейской части СССР // Микология и фитопатология. - 1991. - Т. 25. - №.3. - С. 250-252.
5. Судникова В.П., Артемова С.В., Зеленева Ю.В. Патогенные свойства изолятов Septoria tritici Rob et Desm // Фитосанитарное оздоровление экосистем. - 2005. - С. 561-562.
6. Зеленева Ю.В. Методические подходы к выявлению источников устойчивости пшеницы к возбудителю Septoria tritici // Вестник российских университетов. Математика. - 2008. - Т. 13. - №. 5. - С. 333-337.
7. Пыжикова Г.В. Септориозы зерновых культур. М.: ВННИИТЭИСХ, 1984. 64с.
8. Патент RU 2161196 C2. Определение грибковых патогенов с использованием реакций полимеризации цепи. Патентообладатель: Новартис АГ. Заявка: 96122886/13, 1995.04.19. Опубликовано: 2000.12.27
9. Fraaije, B.A., Lovell, D.J., Coelho, J.M., Baldwin, S., & Hollomon, D. W. PCR-based assays to assess wheat varietal resistance to blotch (Septoria tritici and Stagonospora nodorum) and rust (Puccinia striiformis and Puccinia recondita) diseases // European Journal of Plant Pathology. - 2001. - Т. 107. - №. 9. - С. 905-917.
10. Бейшова И.С., Чужебаева Г.Д., Ковальчук А. М., Ульянов В. А. Разработка тест-системы на основе полимеразной цепной реакции в реальном времени для диагностики септориоза злаковых культур //Наука и современность. - 2015. - №. 40. - С. 7-14.
11. Фиг. 1 Нативный гель-электрофорез в 7 % полиакриламидном геле ПЦР продуктов амплификации COXIII-Tritici-F/COXIII-Tritici-R образцов коллеционных культур Z. tritici. 1 - HH 2-2, 2 - HH 4-4, 3 - HH 4-5, 4 - HH 4-7, 5 - отрицательный контроль, М - маркер Сибэнзим 100 b.p.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC"-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-02-16" softwareVersion="2.3.0"
softwareName="WIPO Sequence" fileName="СПОСОБ ПЦР – ИДЕНТИФИКАЦИИ
ФИТОПАТОГЕННЫХ ГРИБОВ ZYMOSEPTORIA TRITICI, PARASTAGONOSPORA NODORUM.xml"
dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023130393/20(067587)</ApplicationNumberText>
<FilingDate>2023-11-22</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Попыванов Дмитрий
Владимирович</ApplicantName>
<ApplicantNameLatin>Popyvanov D.V.</ApplicantNameLatin>
<InventorName languageCode="ru">Бессолицына Екатерина
Андреевна</InventorName>
<InventorNameLatin>Bessolitsina E.A.</InventorNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ПЦР - ИДЕНТИФИКАЦИИ ФИТОПАТОГЕННЫХ
ГРИБОВ ZYMOSEPTORIA TRITICI, PARASTAGONOSPORA NODORUM</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>PARASTAGONOSPORA NODORUM </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtatttactttattatgtgccctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>PARASTAGONOSPORA NODORUM</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatgggttaaatatattatacttaacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>ZYMOSEPTORIA TRITICI</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccttaatacttttaatgtcaagtatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>ZYMOSEPTORIA TRITICI</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggtaattcaaaagcgttaattgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ОПРЕДЕЛЕНИЕ ГРИБКОВЫХ ПАТОГЕНОВ С ИСПОЛЬЗОВАНИЕМ РЕАКЦИЙ ПОЛИМЕРИЗАЦИИ ЦЕПИ | 1995 |
|
RU2161196C2 |
| Тест-система для идентификации ДНК ткани ежа обыкновенного (Erinaceus europaeus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2725215C1 |
| Способ определения зараженности семян пшеницы септориозом | 2016 |
|
RU2646250C2 |
| Тест-система для идентификации ДНК тканей крыс и мышей в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2725539C1 |
| Способ идентификации видовой принадлежности тканей крыс и мышей в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2742952C1 |
| Способ идентификации ДНК ткани ежа обыкновенного (Erinaceus europaeus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726433C1 |
| Способ идентификации видовой принадлежности баранины и говядины в продовольственном сырье, кормах и пищевых продуктах | 2018 |
|
RU2694713C1 |
| Способ идентификации ДНК ткани сайры тихоокеанской (Cololabis saira) в пробе | 2023 |
|
RU2838063C2 |
| Тест-система для идентификации видовой принадлежности баранины и говядины в продовольственном сырье, кормах и пищевых продуктах | 2018 |
|
RU2702858C1 |
| Тест-система для выявления ДНК ткани домашнего осла (Equus asinus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726555C1 |
Изобретение относится к области биотехнологии. Описан способ идентификации фитопатогенных грибов Zymoseptoria tritici и Parastagonospora nodorum методом ПЦР. Он основан на использовании разработанных праймеров для определения вида Parastagonospora nodorum и для определения вида Zymoseptoria tritici. Технический результат заключается в разработке способа идентификации фитопатогенных грибов, позволяющего точно и быстро идентифицировать фитопатогенный комплекс септориоза, включающий виды Z. tritici и P. Nodoru в растительных тканях, с использованием метода ПЦР. 1 ил., 2 пр.
Способ идентификации фитопатогенных грибов Zymoseptoria tritici и Parastagonospora nodorum методом ПЦР, отличающийся тем, что основан на использовании праймеров: COXIII-Nodorum-F: 5'-GTATTTACTTTATTATGTGCCCTC-3', COXIII-Nodorum-R: 5'-AATGGGTTAAATATATTATACTTAACG-3' для определения вида Parastagonospora nodorum и COXIII-Tritici-F: 5'-GCCTTAATACTTTTAATGTCAAGTATG-3', COXIII-Tritici-R: 5'-CGGTAATTCAAAAGCGTTAATTGC-3' для определения вида Zymoseptoria tritici, предусматривающих выделение ДНК из образцов биологического материала, проведение с выделенной ДНК полимеразной цепной реакции ПЦР и детекцию продуктов амплификации путем сравнения по размеру с маркерными ДНК-фрагментами маркера молекулярных масс, при котором наличие в пробе ПЦР-продуктов размером 194 или 255 пар нуклеотидов при использовании праймеров COXIII-Nodorum-F и COXIII-Nodorum-R или COXIII-Tritici-F и COXIII-Tritici-R соответственно свидетельствует о присутствии в исследуемом образце фитопатогенов Parastagonospora nodorum или Zymoseptoria tritici соответственно.
| Автоглиссер | 1931 |
|
SU34994A1 |
| WO 2018098218 A1, 31.05.2018 | |||
| ОПРЕДЕЛЕНИЕ ГРИБКОВЫХ ПАТОГЕНОВ С ИСПОЛЬЗОВАНИЕМ РЕАКЦИЙ ПОЛИМЕРИЗАЦИИ ЦЕПИ | 1995 |
|
RU2161196C2 |
Авторы
Даты
2024-08-01—Публикация
2023-11-22—Подача