Область техники, к которой относится изобретение
Изобретение относится к применению видоспецифичных праймеров в опытах с использованием полимеразно-цепьевой реакции для обнаружения патогенных грибов. Применение таких праймеров позволяет обнаруживать определенные изоляты патогенных грибов и осуществлять наблюдение за развитием болезни в популяциях растений.
Предпосылки создания изобретения
Болезни растений ежегодно являются причиной существенных потерь урожая, нанося экономический ущерб фермерам и, кроме того, приводя во многих частях света к недостатку продовольственных ресурсов для местного населения. Широкое применение фунгицидов дало существенную защиту от поражения фитопатогенами. Однако несмотря на то, что затраты на фунгициды в мире составляют 1 миллиард долларов, мировые потери урожая по данным 1981 года (James, 1981; Seed Sci. & Technol. 9:679-685) составляли приблизительно 10%.
Серьезность обусловленного болезнью разрушительного процесса зависит от агрессивности патогена и реакции на него хозяина. Одной из целей большинства программ селекции растений является увеличение устойчивости к болезни растений-хозяев. Обычно различные расы патогенов по-разному взаимодействуют с различными сортами одних и тех же видов культурных растений, а многие механизмы, обусловливающие устойчивость хозяина, способствуют его защите только от определенных рас патогенов. Кроме того, некоторые расы патогенов вызывают ранние признаки симптомов болезни, но оказывают незначительное влияние на урожай. По данным Jones и Clifford (1983; Cereal Diseases, John Wiley) следует ожидать появления вирулентных форм патогена в популяции патогена в ответ на интродукцию устойчивости в сорта культурных растений-хозяев и поэтому представляется необходимым осуществлять наблюдение за популяциями патогенов. Кроме того, известно несколько зафиксированных случаев развития у штаммов грибов устойчивости к определенным фунгицидам. Еще в 1981 г. Fletcher и Wolfe (1981; Proc. 1981 Brit. Crop Prot. Conf.) установили, что 24% популяций настоящей мучнистой росы на яровом ячмене и 53% на озимом ячмене проявили значительную вариабельность в чувствительности к фунгициду триадименолу и что распределение этих популяций между сортами отличалось от наиболее чувствительного сорта, приводя к более частой встречаемости типов с пониженной чувствительностью. Подобные различия чувствительности грибов к фунгицидам зафиксированы для мучнистой росы пшеницы (также к триадименолу), Botrytis (к беномилу), Pyrenophora (к ртутьорганическим фунгицидам), Pseudocercosporella (к фунгицидам МВС-типа) и Mycosphaerella fijiensis к триазолам, как говорится у указанных выше авторов (Jones и Clifford Cereal Diseases, John Wiley, 1983).
Зерновые культуры выращивают во всем мире, и они составляют основную часть мировых продовольственных ресурсов. Хотя многие патогены приводят к потере урожая, вызывающие некроз патогены Septoria и Pseudocercosporella являются наиболее важными в основных зонах выращивания зерновых культур в Европе и Северной Америке (Jones и Clifford Cereal Diseases, John Wiley, 1983). В частности различные симптомы болезни, вызываемые различными изолятами и видами этих грибов, приводят к тому, что трудно точно предсказать потенциальные потери вследствие заболевания. Следовательно, для специалистов-практиков в области защиты растений представляет большую ценность разработка усовершенствованных методов диагностики для быстрого и точного выявления конкретных патогенов.
Четыре вида р. Septoria паразитируют на мелкозернистых видах зерновых. Septoria tritici является возбудителем окаймленной пятнистости и обладает вирулентностью для пшеницы, но также поражает тритикале и рожь. Обычно этот патоген вызывает некроз листьев. Septoria nodorum является возбудителем септориоза колосковой чешуи и поражает пшеницу, тритикале, рожь и ячмень и, хотя болезнь в основном ограничена колосковыми чешуями, она также распространяется и на листовые пластинки, и влагалище листа. Septoria avenae паразитирует на овсе, пшенице и тритикале, a Septoria passerinii - только на ячмене. Болезни, вызываемые возбудителями р. Septoria, приводят к ущербу в экономически значимых масштабах во всех областях выращивания пшеницы. Различные болезни, вызываемые возбудителями р. Septoria, часто конкурируют на полях, занятых культурой, и на отдельных растениях, причем симптомы болезней могут быть названы в целом "септориозным комплексом". Обычно наиболее часто встречаются такие виды, как S. tritici и S. nodorum. Согласно данным Wiese (1977; Compendium of Wheat Diseases, Amer. Phytopath. Soc. стр. 42-45) в настоящее время вследствие поражения септориозным комплексом ежегодно теряется около 2% мирового урожая пшеницы, а эти потери урожая в основном обусловлены недостаточным наливом зерна. Фунгицидные обработки могут спасти до 20% урожая в случаях серьезной зараженности возбудителями р. Septoria, но часто на начальной стадии заражения трудно распознать различные виды Septoria, что затрудняет решение вопроса о том, следует или нет применять фунгициды, поскольку различные сорта культурных растений проявляют неодинаковую степень устойчивости к различным видам Septoria.
Церкоспореллез злаковых трав вызывается грибом Pseudocercosporella herpotrichoides и ограничен базальной частью стебля злака. Пшеница, рожь, овес и другие злаки чувствительны к церкоспореллезу, который встречается в прохладном влажном климате и преобладает в Европе, Северной и Южной Америке, Африке и Австралии. Пшеница является наиболее чувствительным к этому возбудителю видом зерновых, однако выявлены изоляты, которые также являются вирулентными и для других злаков. Например, из ржи также был выделен R-штамм гриба, который медленнее растет на пшенице, чем W-штамм, который был выделен из пшеницы. Хотя церкоспореллез может приводить к гибели побегов или целых растений, наиболее часто он приводит к полеганию и/или приводит к уменьшению размера и количества зерен. Потери урожая от церкоспореллеза являются даже более существенными по сравнению с таковыми, вызываемыми Septoria tritici и Septoria nodorum. Обычные способы борьбы с церкоспореллезом включают обработку регуляторами роста растений для укрепления междоузлий и обработку фунгицидами. Однако различная чувствительность сортов культурных растений к различным штаммам гриба затрудняет прогнозирование ожидаемой эффективности фунгицидных обработок.
Пятнистость листьев бананов типа сигатока встречается в двух формах, каждая из которых вызывается различными грибами. Возбудителем важной в экономическом отношении черной сигатоки является Mycosphaerella fijiensis, тогда как возбудителем менее важной в экономическом отношении желтой сигатоки является Mycosphaerella musicola (Johanson и Jeger, 1993; Mycol. Res. 97: 670-674). Черная сигатока представляет собой главную проблему при выращивании бананов, приводя к серьезным потерям урожая от 30% и выше. Вследствие наличия у Mycosphaerella fijiensis устойчивости к фунгицидам следует ограничить применение фунгицидов для предотвращения дальнейшего развития устойчивости. Следовательно, разработка средств диагностики должна иметь важное значение для выявления таких обстоятельств, когда можно применять фунгициды без излишнего риска дальнейшего развития устойчивости к ним.
Таким образом, существует реальная необходимость разработки способа, позволяющего идентифицировать конкретные расы патогенных грибов на ранней стадии процесса заражения. С помощью идентификации конкретной расы патогена до того, как симптомы болезни станут очевидными в отношении урожая на корню, агроном может оценить возможное влияние дальнейшего развития патогена на сорт культурного растения, на котором он был обнаружен, и может выбрать соответствующий фунгицид, если такая обработка представляется необходимой.
Краткое изложение сущности изобретения
Настоящее изобретение относится к способам идентификации различных патотипов фитопатогенных грибов. Изобретение относится к последовательностям ДНК, которые позволяют выявить изменчивость различных патотипов грибов. Такие последовательности ДНК пригодны для способа по изобретению, поскольку они могут применяться для получения праймеров, пригодных для диагностических методов, основанных на полимеразно-цепьевой реакции (ПЦР). Эти праймеры генерируют уникальные фрагменты в ПЦР-реакциях, в которых ДНК- матрицу получают от определенных патотипов грибов и поэтому ее можно использовать для идентификации присутствия или отсутствия конкретных патотипов в материале растения-хозяина до начала проявления симптомов болезни.
Настоящее изобретение дает возможность оценить потенциальный ущерб для конкретного сорта культурного растения, связанный с данным патогенным штаммом, и обоснованно применять различный доступный ассортимент фунгицидов. Кроме того, изобретение может применяться для получения подробной информации о развитии и распространении конкретных рас патогенов в широких географических ареалах. В изобретении предлагается способ обнаружения, прежде всего пригодный для болезней с продолжительным латентным периодом, таких как болезни пшеницы, вызываемые Septoria nodorum или Septoria tritici, и болезни банана, вызываемые Mycosphaerella fijiensis.
Также разработаны наборы, пригодные для практического осуществления изобретения. Данные наборы прежде всего пригодны для идентификации патогенов p.p. Septoria, Pseudocercosporella, Fusarium и Mycosphaerella.
Краткое описание чертежей
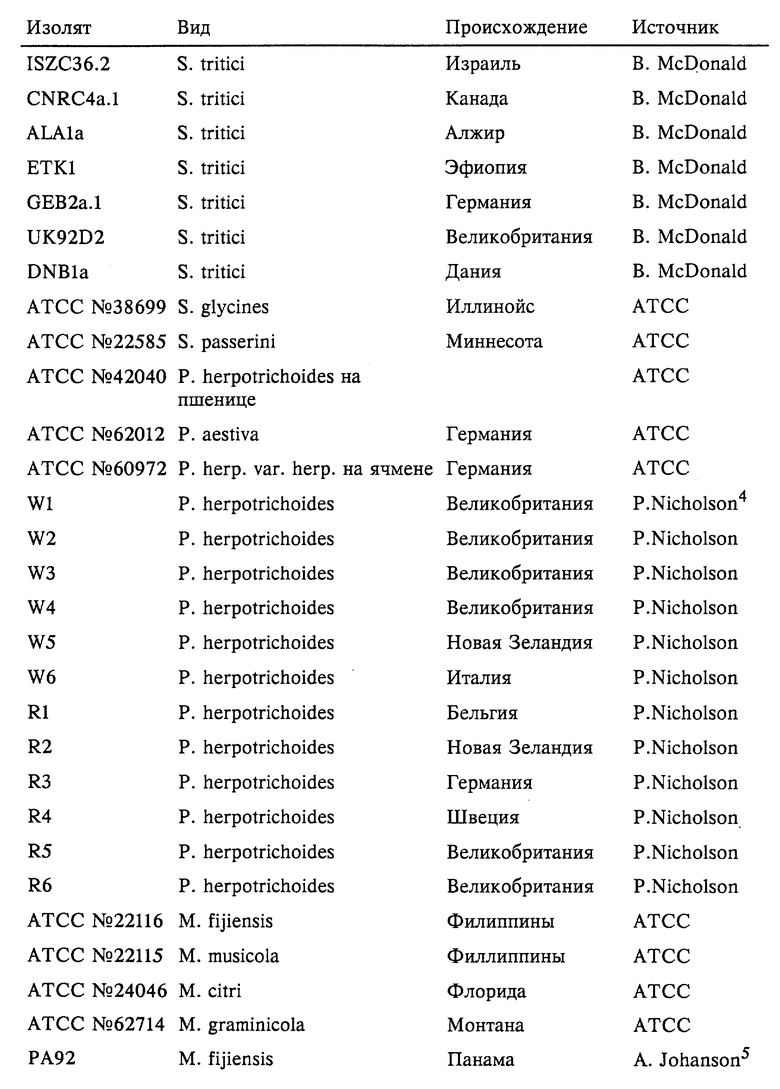
На фиг. 1 представлено сопоставление последовательностей внутренних транскрибирующихся спейсеров из Septoria tritici, Septoria nodorum, W-штамма Pseudocercosporella herpotrichoides (два варианта), R-штамма Pseudocercosporella herpotrichoides, Mycosphaerella fijiensis и Mycosphaerella musicola.
На фиг. 2 представлено сопоставление последовательностей внутренних транскрибирующихся спейсеров из Septoria nodorum и Septoria avenae f.sp. triticea.
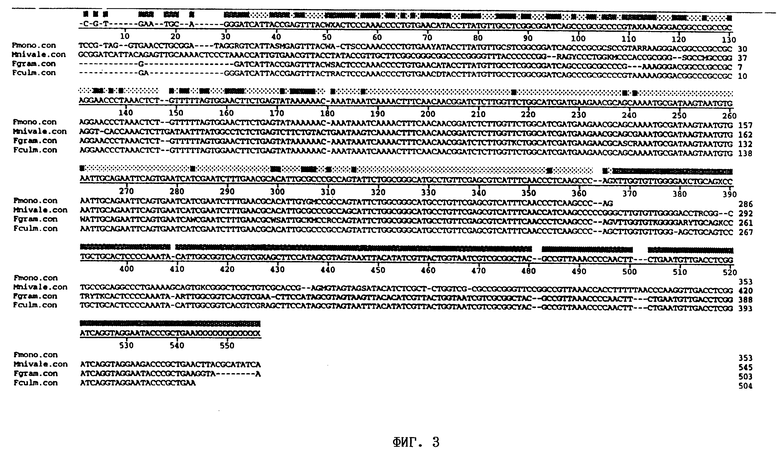
На фиг. 3 представлено сопоставление последовательностей внутренних транскрибирующихся спейсеров из Fusarium graminearum, Fusarium culmorum, Fusarium moniliforme и Microdochium nivale.
Подробное описание изобретения
Настоящее изобретение относится к уникальным последовательностям ДНК, которые пригодны для идентификации различных патотипов фитопатогенных грибов. В частности, последовательности ДНК могут применяться в качестве праймеров в анализах, основанных на использовании ПЦР, для идентификации патотипов грибов. Последовательности ДНК по изобретению содержат области внутреннего транскрибирующегося спейсера (ITS) гена рибосомной РНК конкретных патогенных грибов, а также праймеры, получаемые из этих областей, которые дают возможность идентифицировать конкретный патоген. Эти последовательности ITS-ДНК из различных патотипов видов или родов патогенов, которые могут быть разными для различных представителей видов или родов, могут применяться для идентификации этих определенных представителей.
Специалисты в области биомедицины в течение ряда лет использовали с определенным успехом методы, основанные на ПЦР, для обнаружения патогенов в зараженных тканях животных. Однако только в настоящее время этот метод был применен для выявления фитопатогенов. Например, с использованием ПЦР-последовательностей, специфичных для митохондриального генома патогена, было обнаружено присутствие Gaumannomyces graminis в зараженной пшенице (Schlesser и др., 1991; Applied and Environ. Microbiol. 57:553-556) и маркеры случайным образом амплифицированной полиморфной ДНК (т.е. САПД) оказались пригодными для распознавания многочисленных рас Gremmeniella abietina, возбудителя рака склеродерриса у хвойных.
Рибосомные гены пригодны для применения в качестве молекулярных зондов-мишеней вследствие наличия у них большого количества копий. Несмотря на высокую степень консервативности у зрелых последовательностей рРНК, нетранскрибирующиеся и транскрибирующиеся спейсерные последовательности обычно мало консервативны и, следовательно, пригодны в качестве последовательностей-мишеней для обнаружения недавней эволюционной дивергенции. Гены рРНК грибов организованы в единицы, каждая из которых кодирует три зрелых субъединицы 18S, 5,8S и 28S соответственно. Эти субъединицы разделены двумя внутренними транскрибирующимися спейсерами ITS1 и ITS2, длиной приблизительно 300 пар оснований (White и др., 1990; в PCR Protocols; ред. Innes и др., стр. 315-322). Кроме того, транскрибируемые единицы разделены нетранскрибирующимися спейсерными последовательностями (NTS). ITS- и NTS-последовательности прежде всего пригодны для обнаружения конкретных патотипов различных патогенных грибов.
Последовательности ДНК по изобретению имеют происхождение из области внутреннего транскрибирующегося спейсера (ITS) гена рибосомной РНК различных патогенных грибов. Последовательности ITS-ДНК из различных патотипов видов или родов патогенов могут быть разными для различных представителей видов или родов. Если определить ITS-последовательности патогена, то эти последовательности могут быть сопоставлены с другими ITS-последовательностями. Таким образом, из ITS-последовательностей могут быть получены праймеры. Это означает, что на основании данных об областях внутри ITS-областей могут быть сконструированы праймеры, которые имеют наиболее различающиеся последовательности среди патотипов грибов. Эти последовательности и праймеры, основанные на этих последовательностях, могут применяться для идентификации конкретных представителей патогенов.
Конкретные представляющие интерес последовательности ДНК включают последовательности ITS-ДНК грибов р. Septoria, в частности Septoria nodorum и Septoria tritici; p. Mycosphaerella, в частности Mycosphaerella fijiensis и Mycosphaerella musicola; p. Pseudocercosporella, в частности Pseudocercosporella herpotrichoides, прежде всего W-штамма и R-штамма Pseudocercosporella herpotrichoides; p. Fusarium, в частности F. graminearum, F. culmorum, F. moniliforme и Microdochium nivale. Такие ITS-ДНК-последовательности, а также праймеры, представляющие интерес, приведены в последовательностях SEQ ID NO: 1-47 и в SEQ ID NO: 50-86. Эти последовательности находят применение в основанной на использовании ПЦР идентификации представляющих интерес патотипов.
Способы использования последовательностей праймеров по изобретению в ПЦР-анализах хорошо известны в данной области техники. См., например, патенты США 4683195 и 4683202, а также у Schlesser и др., (1991) Applied and Environ. Microbiol. 57: 553-556. CM. также у Nazar и др. (1991; Physiol. and Molec. Plant Pathol. 39:1-11), которые применяли ПЦР-амплификацию для выявления различий в ITS-областях у Verticillium alboatrum и Verticillium dahliae и, следовательно, для разделения этих двух видов; и у Johanson и Jeger (1993; Mycol. Res. 97: 670-674), которые применяли аналогичные методы для разделения патогенов банана Mycosphaerella fijiensis и Mycosphaerella musicola.
Последовательности ITS-ДНК по изобретению могут быть выделены из патогенных грибов и клонированы способами, известными в данной области техники. В целом способы выделения ДНК из изолятов грибов известны. См., например, Raeder & Broda (1985) Letters in Applied Microbiology 2:17-20; Lee и др., (1990) Fungal Genetics Newsletter 35: 23-24; и Lee и Taylor (1990) в PCR Protocols: A Guide to Methods and Applications, ред. Innes и др.; стр. 282-287.
В другом варианте представляющие интерес ITS-области могут быть определены с помощью ПЦР-амплификации. Праймеры для амплификации полной ITS-области конструировали аналогично описанному у White и др., (1990; в PCR Protocols, ред. Innes и др.; стр. 315-322) и амплифицированные ITS-последовательности субклонировали в клонирующем векторе pCRII. Субклонированная последовательность включала левосторонний ITS (ITS1), правосторонний ITS (ITS2), а также расположенную в центре субъединицу 5,8S гена рРНК. Это было осуществлено для Septoria nodorum и Septoria tritici, многочисленных изолятов Pseudocercosporella и Mycosphaerella fijiensis, Mycosphaerella musicola, Septoria avenae triticea, F. graminearum, F. culmorum, F. moniliforme и Microdochium nivale.
Определяли ITS-последовательности и сравнивали последовательности в пределах каждой группы патогена для выявления месторасположения дивергенций, которые могут быть приемлемы при тестировании с помощью ПЦР для разделения различных видов и/или штаммов. Последовательности ITS-областей, которые были определены, представлены в виде последовательностей SEQ ID NO: 1-6, SEQ ID NO: 47 и SEQ ID NO: 82-86, а также показаны на фиг. 1, 2 и 3. На основе идентификации дивергенций синтезировали многочисленные праймеры и тестировали их с использованием ПЦР-амплификации. Матрицы, применяемые для тестирования с использованием ПЦР-амплификации, сначала представляли собой очищенную ДНК патогена, а затем ДНК, выделенную из зараженной ткани растения-хозяина. Таким образом, оказалось возможным идентифицировать пары праймеров, которые являлись диагностическими, т.е. которые позволяли идентифицировать один конкретный вид или штамм патогена, а не другой вид или штамм этого же патогена. Предпочтительные комбинации праймеров дают возможность разделить различные виды или штаммы в зараженной ткани растения-хозяина, т.е. в ткани хозяина, которая ранее была заражена конкретными видами или штаммами патогена.
Изобретение включает многочисленные комбинации праймеров для различных видов Septoria, Mycosphaerella и Fusarium и различных штаммов Pseudocercosporella, которые удовлетворяют этому критерию. Праймеры по изобретению конструируют на основе различий в последовательности между ITS-областями грибов. Различие между последовательностями минимум на одну пару оснований позволяет сконструировать дискриминирующий праймер. Для амплификации видоспецифичных ПЦР-фрагментов праймеры, сконструированные для конкретной ITS-области ДНК гриба, могут применяться в сочетании с праймером, созданным для консервативной области последовательности внутри кодирующей области рибосомной ДНК. Обычно для достижения хорошей чувствительности праймеры должны иметь теоретическую температуру плавления между приблизительно 60oC и 70oC и должны быть лишены значительных вторичных структур и перекрытий 3'-концов в комбинациях праймеров. Праймеры обычно имеют по крайней мере от приблизительно 5 до приблизительно 10 нуклеотидов.
Пригодность клонированных ITS-последовательностей для отбора праймеров для целей диагностики определяется в основном их быстрой эволюционной дивергенцией. Например, было обнаружено, что изоляты W-типа и R-типа патогена Pseudocercosporella herpotrichoides имеют дивергентные ITS-последовательности, из которых были получены диагностические праймеры. Однако быстрая дивергенция в пределах ITS-последовательности основана на том наблюдении, что были идентифицированы два различных варианта последовательности W-типа. Идентичность последовательностей в пределах W-типа составляла 99,4%, в то время как между W- и R-типами она составляла 98,6%, что позволяет предположить более близкую эволюционную взаимосвязь между двумя W-вариантами по сравнению с таковой, обнаруженной между W- и R-типами. Такая более близкая взаимосвязь также видна из сходной патогенности для хозяина двух изолятов с дивергентными ITS-последовательностями.
Помимо получения праймеров из последовательностей, имеющих происхождение из ITS, для ПЦР-диагностики изолятов грибов, изобретение также включает идентификацию праймеров из библиотеки праймеров САПД, которые при использовании в ПЦР дают возможность различать Septoria nodorum и Septoria tritici. Отобранные праймеры являются коммерчески доступными и поставляются Орегон Technologies Incorporated (Alameda, Калифорния). В результате скрининга геномной ДНК Septoria выделены два праймера, которые позволяют обнаруживать только S. tritici, и три праймера, которые позволяют обнаруживать только S. nodorum.
Настоящее изобретение дает возможность легко получать "наборы", содержащие элементы, необходимые для осуществления процесса. Такой набор может содержать носитель, разделенный на отсеки для размещения в нем в единой упаковке одной или нескольких единиц хранения, таких как пробирки или пузырьки. Одна из указанных единиц хранения может содержать немеченые или распознаваемые с помощью метки праймеры ДНК. Меченые праймеры ДНК могут находиться при необходимости как в лиофилизированной форме, так и в соответствующем буфере. Одна или несколько единиц хранения могут содержать один или несколько ферментов или реагентов для применения в ПЦР-реакциях. Эти ферменты могут присутствовать по отдельности или в смесях, в лиофилизированной форме или в соответствующих буферах.
И, наконец, набор может содержать все дополнительные элементы, необходимые для осуществления способа по изобретению, такие как буферы, реагенты для экстракции, ферменты, пипетки, планшеты, нуклеиновые кислоты, нуклеозидтрифосфаты, фильтровальную бумагу, материалы для получения геля, материалы для переноса, материалы для авторадиографии и т.п.
Приведенные ниже примеры иллюстрируют, не ограничивая объем изобретения, типовые экспериментальные протоколы, которые могут применяться для выделения ITS- последовательностей, выбора соответствующих последовательностей праймеров, тестирования праймеров на их избирательную и диагностическую эффективность, и применение таких праймеров для обнаружения болезней и изолята гриба. Указанные примеры приведены с целью иллюстрации и не ограничивают объем изобретения.
ПРИМЕРЫ
Пример 1: Изоляты грибов и экстракция геномной ДНК
Жизнеспособные изоляты грибов S. nodorum, S. tritici, S. passerini, S. glycines, Pseudocercosporella herpotrichoides, Pseudocercosporella aestiva, Mycosphaerella citri, Mycosphaerella graminicola, Mycosphaerella fijiensis и Mycosphaerella musicola получали из Американской коллекции типовых культур. Изоляты Fusarium culmorum и Fusarium graminearum были получены от доктора Paul Nelson из Пенсильванского государственного университета. Изолят Microdochium nivale (синоним Fusarium nivale) был получен от фирмы Ciba (Базель), а изолят Fusarium moniliforme был получен от доктора Loral Castor. Грибы выращивали в 150 мл бульона на основе картофельной декстрозы, инокулированного мицелярными фрагментами из культур, выращенных на КДА (агар с картофельной декстрозой). Культуры инкубировали на орбитальном шейкере при 28oC в течение 7-11 дней. Мицелии пеллетировали путем центрифугирования, затем погружали в жидкий азот и полную геномную ДНК экстрагировали в соответствии с протоколом Lee и Taylor (1990; в PCR Protocols: A Guide to Methods and Applications; ред. Innes и др., стр. 282-287).
Доктором Bruce McDonald из Техасского университета сельского хозяйства и шахт была предоставлена геномная ДНК из десяти изолятов S. nodorum и девяти изолятов S.tritici. Доктор Chris Caten из Бирмингемского университета предоставил очищенную ДНК шести изолятов гриба Septoria nodorum. Очищенные геномные ДНК из 12 изолятов Pseudocercosporella herpotrichoides были получены от доктора Paul Nicholson центра Джона Инна, Норвик, Великобритания. Шесть из этих изолятов были W-типа, а другие шесть изолятов были R-типа. Типы этих изолятов определяли на основе изучения патогенности и полиморфизма длин рестрикционных фрагментов (ПДРФ). Andrea Johanson из Института природных ресурсов предоставила геномную ДНК шести изолятов М (см. Табл. 1), musicola, шести изолятов М. fijiensis и одного изолята Mycosphaerella musae. Очищенная геномная ДНК Septoria avenae f.sp. triticea АТСС N 26380 была предоставлена доктором Peter Ueng из Министерства сельского хозяйства США (USDA), Белтсвил, Мэриленд.
Пример 2: Выделение областей внутреннего транскрибирующегося спейсера (ITS)
Фрагменты области внутреннего транскрибирующегося спейсера длиной приблизительно 550 пар оснований амплифицировали с помощью ПЦР из 25 нг геномной ДНК, выделенной из S.nodorum (АТСС N24425), S. tritici (АТСС N26517), R1-, R2-, W2- и W-изолятов Pseudocercosporella herpotrichoides, M. fijiensis (АТСС N22116) и M. musicola (АТСС N22115) с использованием 50 пмолей праймеров ITS1 (5'-TCCGTAGGTGAACCTGCGG-3'; SEQ ID NO: 38) и ITS4 (5'-TCCTCCGCTTATTGATATGC-3'; SEQ ID NO: 41). ПЦР осуществляли по методике примера 4, за исключением того, что реакции проводили в 100 мкл и отжиг осуществляли при 50oC. ITS- фрагменты очищали с помощью осаждения изопропанолом, как описано у Maniatis и др. (1982; Molecular Cloning; ред. Maniatis и др.; стр. 461-462). ДНК ресуспендировали в 50 мкл дН2О и клонировали с использованием набора для клонирования ТА Cloning Kit (часть N К2000-01) фирмы Invitrogen Corporation (Сан Диего, Калифорния), используя клонирующий вектор pCRII. Последовательности ДНК из ITS-областей определяли с помощью дидезокси-метода, используя автоматический секвенатор модели 373А Applied Biosystems (Foster City, Калифорния) с праймерами ITSI (см. вышеуказанную последовательность), ITS2 (5'-GCTGCGTTCTTCATCGATGC-3'; SEQ ID NO: 39), ITS4 (см. вышеуказанную последовательность) и с праймером М13 универсальный-20 (5'-GTAAAACGACGGCCAGT-3'; SEQ ID NO: 48) и с ревертивным праймером (5'-AACAGCTATGACCATG-3'; SEQ ID NO: 49). ITS-праймеры ITS1 (SEQ ID NO: 38), ITS2 (SEQ ID NO: 39), ITS3 (SEQ ID NO: 40) и ITS4 (SEQ ID NO: 41), применявшиеся для клонирования ITS-областей, подробно описаны у White и др. (1990; в PCR Protocols; ред. Innes и др., стр. 315-322).
Кроме того, области внутреннего транскрибирующегося спейсера амплифицировали с помощью ПЦР из 25 нг геномной ДНК, выделенной из S. avenae f. sp. triticea, M. nivale, F. moniliforme (N4551), изолятов F. graminearum R-8417, R-8546 и R-8422 и изолятов F. culmorum R-5126, R-5106 и R-5146. ПЦР-продукты очищали с помощью набора для очистки ДНК фирмы Promega Wizard (Madison, Висконсин). Последовательности ДНК из ITS-областей определяли аналогично описанному выше, используя праймеры ITS1 (SEQ ID NO: 38), ITS2 (SEQ ID NO: 39), ITS3 (SEQ ID NO: 40) и ITS4 (SEQ ID NO: 41). Реакции секвенирования объединяли для трех изолятов F. culmorum и F. graminearum для получения согласующей последовательности для F. culmorum и F. graminearum.
Пример 3: Экстракция ДНК из листьев пшеницы и банана
Из листьев пшеницы ДНК экстрагировали с использованием модифицированной версии протокола быстрой экстракции ДНК фирмы MicroProbe Corporation (Garden Grove, Калифорния), применяя набор для экстракции нуклеиновой кислоты IsoQuick (каталожный МХТ-020-100). Нормальные выходы составляли 5-10 мкг полной ДНК из 0,2 г листовой ткани. В каждом ПЦР-анализе использовали приблизительно 100 нг полной ДНК.
Модифицированная методика быстрой экстракции ДНК
Сначала перед применением набора все содержимое реагента 2A (20-кратная концентрация красителя) добавляли к реагенту 2 (матрица для экстракции).
(1) Приблизительно 0,2 г образца листьев добавляли в пробирку Эппендорфа объемом 1,5 мл, содержащую 50 мкл образца буфера А и 50 мкл 25 лизисного раствора N1. Образец листа растирали с помощью пестика Конте.
(2) Реагент 2 (матрица для экстракции) интенсивно встряхивали. К образцу лизата добавляли 350 мкл реагента 2.
(3) К образцу добавляли 200 мкл реагента 3 (буфер для экстракции). Образец интенсивно перемешивали в течение 20 с.
(4) Центрифугировали на микроцентрифуге при 12000xg в течение 5 мин.
(5) Водную фазу (верхний слой) переносили в новую микроцентрифужную пробирку. Этот объем обычно составлял около 200 мкл.
(6) К водной фазе образца добавляли 0,1-кратный объем водной фазы реагента 4 (ацетат натрия).
(7) К водной фазе образца добавляли такой же объем изопропанола и затем интенсивно перемешивали.
(8) Центрифугировали на микроцентрифуге при 12000xg в течение 10 мин.
(9) Супернатант отбрасывали, не нарушая дебрис нуклеиновой кислоты. К дебрису добавляли 0,5 мл 70%-ного при -20oC этанола. Пробирку интенсивно перемешивали до полного смешения.
(10) Центрифугировали на микроцентрифуге при 12000xg в течение 5 мин.
(11) Супернатант отбрасывали и дебрису давали высохнуть.
(12) Дебрис нуклеиновой кислоты растворяли в 50 мкл реагента 5 (безводная РНКаза).
Пример 4: Амплификация на основе полимеразно-цепьевой реакции
Полимеразно-цепьевые реакции осуществляли с использованием набора для амплификации генов типа GeneAmp фирмы Perkin-Elmer/Cetus (Norwalk, СТ; часть N N808-0009), применяя буфер, в состав которого входят 50 мМ KCl, 2,5 мМ MgCl2, 10 мМ Трис-HCI, pH 8,3, содержащий по 100 мкМ каждого из ТТФ, дАТФ, дЦТФ и дГТФ, 50 пМ праймера, 2,5 единицы Taq-полимеразы и 25 нг геномной ДНК в окончательном объеме 50 мкл. Реакции проводили в 30 циклов по 15 с при 94oC, 15 с при 50oC, 60oC или 70oC и 45 с при 72oC в термоциклизаторе типа Perkin- Elmer/Cetus модель 9600. Продукты анализировали с использованием гель-электрофореза, загружая 20 мкл образца ПЦР-продукта в 1,1-1,2%-ный агарозный гель.
Пример 5: Синтез и очистка олигонуклеотидов
Олигонуклеотиды (праймеры) синтезировали на синтезаторе ДНК типа Applied Biosystems 380A, используя B-цианотилфосфорамидитную химию.
Пример 6: Отбор видоспецифичных праймеров
Сопоставляли ITS-последовательности из S.nodorum, S. tritici, R- и W-штаммов P.herpotrichoides, M.fijiensis и M. musicola (фиг. 1). Сопоставляли ITS-последовательности из S.nodorum, S.avenae (фиг. 2). Также проводили сопоставление ITS- последовательностей из F. graminearum, F. culmorum, F. moniliforme и M. nivale (фиг. 3). Наборы праймеров синтезировали в соответствии с примером 5 на основе анализа сопоставленных последовательностей. Конструировали праймеры для областей, имеющих наибольшие различия в последовательностях для видов грибов, представленных на фиг. 1 и 2. В соответствии с фиг. 3 конструировали праймеры для областей с наибольшей гомологичностью в пределах ITS для грибов р. Fusarium. Кроме того, для тестирования в сочетании с праймерами, специфичными для ITS-области, синтезировали описанные у White и др., 1990; в PCR Protocols; ред. Innes и др., стр. 315-322, рибосомные ген-специфичные праймеры ITS1 (SEQ ID NO: 38), ITS2 (SEQ ID NO: 39), ITS3 (SEQ ID NO: 40) и ITS4 (SEQ ID NO: 41) (см. табл. 2).
Пример 7: Отбор праймеров для случайным образом амплифицированной полиморфной ДНК (САПД)
Две библиотеки САПД-праймеров (наборы В и Е), состоящие из двадцати олигонуклеотидов каждая, были получены от Ореrоn Technologies Incorporated (Alameda, Калифорния). Праймеры тестировали на их способность различать очищенные геномные ДНК S. nodorum, S. tritici, М.fijiensis и М. musicola. Условия проведения ПЦР были практически такими же, как и в примере 4, за исключением того, что количество циклов ПЦР было увеличено до 35, температура отжига составляла 30oC и было использовано только по 5 пмолей каждого праймера. Были выявлены пять САПД-праймеров, которые позволяли различать очищенные геномные ДНК S. nodorum, S. tritici, М. fijiensis и М. musicola. Праймеры ОРВ-12 и ОРЕ-6 производили отдельный фрагмент при амплификации с геномной ДНК S. tritici. Праймеры ОРЕ-12, ОРВ-19 и ОРЕ-15 производили отдельные фрагменты при амплификации с геномной ДНК S.nodorum. Под воздействием праймеров ОРВ-12 и ОРЕ-6 не получали никаких амплифицированных продуктов геномных ДНК из S. nodorum, М. fijiensis и М. musicola. Праймеры ОРЕ-12, ОРВ-19 и ОРЕ-15 не амплифицировали никакие фрагменты геномной ДНК S.tritici, М fijiensis или М. musicola (См.табл. 3).
Пример 8: Определение специфичности праймера для очищенной геномной ДНК гриба
Чтобы амплифицировать отдельный видоспецифичный фрагмент, проводили ПЦР в соответствии с примером 4, используя различные сочетания праймеров.
Продукты видоспецифичной ПЦР-амплификации получали от праймеров, сконструированных из ITS-области между 18S и 25S субъединицами рибосомной ДНК для каждого представляющего интерес штамма гриба (см. табл. 4).
Пример 9: Определение специфичности праймера для ткани растения, зараженной грибом
В соответствии с протоколом, описанным в примере 3, выделяли полную геномную ДНК из здоровых листьев пшеницы, из листьев пшеницы, зараженных S. nodorum, листьев пшеницы, зараженных S. tritici, и листьев пшеницы, зараженных как S. nodorum, так и S. tritici. ПЦР проводили по методике примера 4, тестируя комбинации праймеров, перечисленные в примере 8, в отношении ДНК из листьев пшеницы.
Специфичные для S. tritici праймеры JB446 (SEQ ID NO: 12) и ITS1 (SEQ ID NO: 38)(JB410) амплифицировали фрагмент длиной 345 пар оснований из очищенной ДНК S. tritici, из зараженной S.tritici листовой ткани пшеницы и из зараженного и S.tritici, и S. nodorum образца листовой ткани пшеницы. Набор праймеров не амплифицировал диагностический фрагмент ни из здоровой листовой ткани пшеницы, ни из ткани пшеницы, зараженной S.nodorum. Аналогично этому специфичные для S. tritici праймеры JB445 (SEQ ID NO: 11) и ITS4 (SEQ ID NO: 41)(JB415) амплифицировали фрагмент длиной 407 пар оснований из таких же тканей, что и комбинация праймеров JB446 (SEQ ID NO: 12) и ITS1 (SEQ ID NO: 38)(JB410), и также оказались пригодными для диагностики.
Аналогичные результаты в отношении диагностики были получены для специфичных для S. nodorum праймеров JB433 (SEQ ID NO: 7) и JB434 (SEQ ID NO: 8). Праймеры амплифицировали фрагмент длиной 448 пар оснований из зараженной S. nodorum ткани пшеницы, из образца листовой ткани пшеницы, зараженной и S. nodorum, и S. tritici, а также из очищенной геномной ДНК S. nodorum. Комбинация праймеров JB433 (SEQ ID NO: 7) и JB434 (SEQ ID NO: 8) не амплифицировала никакие фрагменты из здоровой ткани пшеницы, из ткани пшеницы, зараженной S. tritici, или из очищенной геномной ДНК S. tritici. Специфичные для S. nodorum праймеры JB527 (SEQ ID NO: 10) и JB525 (SEQ ID NO: 9) амплифицировали фрагмент длиной 458 пар оснований из таких же геномных ДНК и тканей пшеницы, что и комбинация праймеров JB433 (SEQ ID NO: 7) и JB434 (SEQ ID NO: 8).
Комбинации праймеров P.herpotrichoides, перечисленные в примере 8, тестировали с помощью ПЦР в отношении экстрактов из стеблей пшеницы, полученных аналогично описанному в примере 12. ПЦР проводили по методике примера 4 со следующими изменениями: 35 циклов проводили при 94oC в течение 15 с и при 70oC в течение 45 с, с использованием 1,5-2,5 мМ MgCl2 и по 200 мкМ каждого дНТФ. Для каждой ПЦР использовали по 1 мкл экстракта пшеницы.
Комбинация праймеров JB537 (SEQ ID NO: 15) и JB541 (SEQ ID NO: 19) амплифицировала фрагмент длиной 413 пар оснований из экстракта пшеницы, зараженного W-типом патотипа P.herpotrichoides. Никаких продуктов амплификации не получали при амплификации ни с экстрактом здоровой пшеницы, ни с экстрактом пшеницы, зараженной R-типом патотипа Р.herpotrichoides.
Комбинация праймеров JB539 (SEQ ID NO: 17) и JB544 (SEQ ID NO: 22) амплифицировала фрагмент длиной 487 пар оснований, а комбинация праймеров JB540 (SEQ ID NO: 18) и JB542 (SEQ ID NO: 20) амплифицировала фрагмент длиной 413 пар оснований из пшеницы, зараженной R-типом, но не из здоровой пшеницы или пшеницы, зараженной W-типом.
Полную геномную ДНК также выделяли из здоровых листьев банана и листьев банана, зараженных М. fijiensis, используя протокол, описанный в примере 3. ПЦР проводили по методике примера 4, тестируя комбинации праймеров М. fijiensis, перечисленные в примере 8, в отношении ДНК из листьев банана.
Специфичные для М.fijiensis праймеры JB549 (SEQ ID NO: 29) и ITS1 (SEQ ID NO: 38) (JB410) амплифицировали фрагмент длиной 489 пар оснований из очищенной ДНК М. fijiensis и из зараженной М. fijiensis листовой ткани банана. Набор праймеров не амплифицировал диагностический фрагмент из здоровой листовой ткани банана. Специфичные для М. fijiensis комбинации праймеров JB443 (SEQ ID NO: 26)/ITS4 (SEQ ID NO: 41)(JB415) и ITS1 (SEQ ID NO: 38)(JB410)/JB444 (SEQ ID NO: 30) амплифицировали фрагмент длиной 418 пар оснований и фрагмент длиной 482 пары оснований соответственно из такой же геномной ДНК и листовой ткани банана, что и комбинация праймеров JB549 (SEQ ID NO: 29) и ITS1 (SEQ ID NO: 38) (JB410).
Пример 10: Определение перекрестной реактивности видоспецифичных праймеров с другими видами и изолятами
Очищенные геномные ДНК грибов получали аналогично описанному в примере 1, а ПЦР проводили по методике примера 4, применяя видоспецифичные праймеры. Тестировали ДНК из других видов и изолятов грибов на способность видоспецифичных праймеров давать с ними перекрестную реакцию.
Специфичные для S.tritici праймеры JB446 (SEQ ID NO: 12) и ITS1 (SEQ ID NO:38)(JB410) амплифицировали фрагмент длиной 345 пар оснований из всех изолятов S.tritici, перечисленных в примере 1. Не было обнаружено перекрестной реактивности с очищенной геномной ДНК S. nodorum, S. glycines или S. passermi. Ни один из этих видов грибов не давал амплифицированный продукт со специфичными для S.tritici праймерами.
Фрагмент длиной 448 пар оснований амплифицировали из всех изолятов S. nodorum, перечисленных в примере 1, используя специфичные для S. nodorum праймеры JB433 (SEQ ID NO: 7) и JB434 (SEQ ID NO: 8). Аналогично этому специфичные для S. nodorum праймеры JB527 (SEQ ID NO: 10) и JB525 (SEQ ID NO: 9) амплифицировали фрагмент длиной 458 пар оснований из всех изолятов S.nodorum, перечисленных в примере 1. S. tritici, S. glycines и S. passerini не давали никаких продуктов амплификации при исследовании как с наборами специфичных для S. nodorum праймеров JB433 (SEQ ID NO: 7) и JB434 (SEQ ID NO: 8), так и с JB527 (SEQ ID NO: 10) и JB525 (SEQ ID NO: 9).
ПЦР в отношении ДНК других видов и изолятов грибов, перечисленных в примере 1, осуществляли с использованием условий, описанных в примере 9, и специфичных для P. herpotrichoides комбинации праймеров, перечисленных в примере 8.
Комбинации праймеров JB537 (SEQ ID NO: 15) и JB541 (SEQ ID NO: 19) давали фрагмент длиной 413 пар оснований из изолятов W-типа Р. herpotrichoides только тогда, когда их изучали в отношении изолятов Р. herpotrichoides и следующих патогенов зерновых культур: P. aestiva, С. cereale, P. sorokiniana, S. tritici и S. nodorum. Комбинации праймеров JB539 (SEQ ID NO: 17) и JB544 (SEQ ID NO: 22) давали фрагмент длиной 487 пар оснований из изолятов R-типа P. herpotrichoides только тогда, когда их изучали в отношении одних и тех же ДНК. Комбинации праймеров JB540 (SEQ ID NO: 18) и JB542 (SEQ ID NO: 20) давали фрагмент длиной 413 пар оснований из изолятов R-типа P.herpotrichoides только тогда, когда их изучали в отношении одних и тех же ДНК.
Пример 11: Источники зараженной Pseudocercosporella herpotrichoides пшеницы
Стебли пшеницы, зараженной церкоспореллезом, получали на стадии 1с программы фунгицидного скрининга фунгицидов фирмы Ciba (Базель). Растения пшеницы 8-дневного возраста заражали P. herpotrichoides путем опрыскивания оснований стеблей пшеницы суспензией конидий (5•105 конидий/мл) в 0,2%-ном Tween ® 20. После инокуляции растения покрывали пластиком и инкубировали в течение 1 дня при 20oC и относительной влажности 95-100%. Растения переносили в камеру для выращивания, где их инкубировали в течение 4 недель при 12oC и относительной влажности 60%. После этой инкубации растения переносили в теплицу и инкубировали при 18oC и относительной влажности 60%. Образцы растений пшеницы, инфицированных штаммом 311 W-типа P. herpotrichoides, отбирали на 8-9 неделе после заражения, тогда как растения, зараженные штаммом 308 патогена R-типа, собирали на 9-10 неделе после заражения.
Пример 12: Экстракция ДНК из стеблей пшеницы для определения Р. herpotrichoides
ДНК экстрагировали из стеблей пшеницы, используя протокол, описанный у Klimyuk и др. (The Plant Journal 3(3):493-494), с некоторыми модификациями. Кусочек стебля пшеницы длиной 2 см, отрезанный на расстоянии 0,5 см от основания стебля, помещали в 160 мкл 0,25 М NaOH и растирали с помощью пестика Конте до полной мацерации. Образец кипятили в течение 30 с. К образцу добавляли 160 мкл 0,25 М HCl и 80 мкл 0,5 М трис-Cl-буфера, pH 8,0/0,25% (объем/объем) Nonider ® P-40. Образцы дополнительно кипятили в течение 2 минут, затем помещали в баню с ледяной водой. Для ПЦР-анализа использовали 1 мкл экстракта.
Пример 13: Включение диагностических анализов в формат количественного колориметрического анализа
Колометрический анализ проводили по методике, описанной у Nikiforov и др. (PCR Methods and Applications 3:285-291), со следующими изменениями:
1) 30 мкл ПЦР-продукта R-типа и смесь 3 М NaCl/20 мМ ЭДТК добавляли в лунку с праймером захвата. В реакции гибридизации использовали 50 мкл ПЦР-продукта W-типа и смесь 3 М NaCl/20 мМ ЭДТК.
2) Обработку экзонуклеазой и реакцию гибридизации проводили в условиях инкубации при 37oC.
3) Применяли разведение 1:1000 моноклонального антитела против биотина пероксидазы из хрена (ПХ).
4) После 2 мин инкубации с О-фенилендиаминдигидрохлоридом (ОФД) в качестве субстрата в каждую лунку добавляли 50 мкл 3H HCl. 96-луночные планшеты считывали при 492 нм и соотносили со считыванием при 570 нм, используя обычный планшет-ридер, применяемый для твердофазного иммуноферментного анализа (ELISA).
Перечисленные в табл. 5 праймеры синтезировали по методике примера 5 для тестирования в качестве праймеров захвата при колориметрическом анализе.
Диагностические для S. nodorum праймеры JB527 (SEQ ID NO: 10) и JB525 (SEQ ID NO: 9) объединяли в формате для количественного колориметрического анализа. Праймер JB527 (SEQ ID NO: 10) синтезировали с помощью Midland Certified Reagent Complany (Midland, Техас) таким образом, чтобы он содержал биотиновую метку и чтобы 5'-конец имел четыре внутринуклеотидные тиофосфатные связи. ПЦР-амплификация, осуществленная по методике примера 4 с использованием модифицированных праймеров JB527 (SEQ ID NO: 10) и JB525 (SEQ ID NO: 9) из здоровой пшеницы и из пшеницы с низкой, средней и высокой степенью зараженности S. nodorum, не давала совсем или давала низкие, средние и высокие значения A492 соответственно при изучении колориметрическим методом с использованием праймера ITS2 (SEQ ID NO: 39) в качестве праймера захвата ПЦР-продукта.
5'-Праймеры JB539 (SEQ ID NO: 17) и JB540 (SEQ ID NO: 18), специфичные для R-типа P. herpotrichoides, и 5'-праймер JB537 (SEQ ID NO: 15), специфичный для W-типа P. herpotrichoides, также модифицировали таким образом, чтобы они содержали биотиновую метку и четыре внутринуклеотидные тиофосфатные связи. Вариант колориметрической оценки ПЦР-анализа для R-типа P.herpotrichoides разрабатывали с использованием модифицированного праймера JB540 (SEQ ID NO: 18), праймера JB542 (SEQ ID NO: 20) и праймера захвата JB539'15. Продукты, полученные в результате амплификации пшеницы, зараженной R-типом, и в результате амплификации геномной ДНК R-типа при использовании модифицированного праймера JB540 (SEQ ID NO: 18) и праймера JB542 (SEQ ID NO: 20), давали положительные колориметрические значения при исследовании колориметрическим методом. Положительные колориметрические значения также получали при колориметрических анализах ПЦР-продуктов, образовавшихся в результате амплификации с использованием модифицированного праймера JB537 (SEQ ID NO: 15) и специфичного для W-типа праймера JB541 (SEQ ID NO: 19) с пшеницей, зараженной W-типом, и геномной ДНК W-типа, когда в качестве праймера захвата использовали JB538'15. Кроме того, интенсивность колориметрического сигнала соответствовала интенсивности фрагмента ПЦР-продукта, проявляющейся на агарозном геле.
Ранее различные виды Septoria можно было идентифицировать путем исследования с помощью микроскопа, а идентификация различных штаммов Pseudocercosporella была возможна только с помощью патологических тестов. Аналогично этому четкая идентификация Mycosphaerella musicola и Mycosphaerella fijiensis была затруднена и даже выделение зрелых перитециев не всегда позволяло провести точную идентификацию (Pons, 1990; в Sigatoka Leaf Spot Deceases of Banana, ред. R.A.Fullerton и R.H.Stover, International Network for the Improvement of Banana and Plantain, Франция). В настоящее время для идентификации S. tritici, S. nodorum, Pseudocercosporella herpotrichoides и других патогенов обычно применяют иммунодиагностические наборы, основанные на использовании метода ELISA, однако этот метод не обладает необходимой точностью, имеет ограничения по разрешающей способности и не дает возможности отличить различные изоляты по настоящему изобретению. Следовательно, разработка теста на основе ДНК для быстрой идентификации различных штаммов указанных грибов дает реальные преимущества не только для специалистов в области систематики грибов, но также и для борьбы с болезнями и для применения в полевых условиях конкретного фунгицида.
Хотя настоящее изобретение описано со ссылкой на конкретные примеры его выполнения, очевидно, что возможны различные изменения, модификации и другие примеры выполнения, и, следовательно, все такие изменения, модификации и примеры выполнения следует рассматривать как подпадающие под объем настоящего изобретения.
Депонирование
Депонирование следующих культур было осуществлено 28 марта 1994 г. в Коллекции культур для целей процедуры патентования (NRRL) службы сельскохозяйственных исследований, Северный региональный исследовательский центр, 1815 North University Street, Peoria, Иллинойс 61604, США:
1. HB101 DH5d (pCRW2-1;SEQ ID NO: 3) Рег.номер NRRL B-21231.
2. HB101 DH5d (pCRW5-1;SEQ ID NO: 47) Рег.номер NRRL B-21232.
3. E.coli DH5d (pCRSTRIT1;SEQ ID NO: 1) Рег.номер NRRL B-21233.
4. E.coli DH5d (pCRR1-21;SEQ ID NO: 4) Рег.номер NRRL B-21234.
5. E.coli DH5d (pCRSNOD31;SEQ ID NO: 2) Рег.номер NRRL B-21235.
| название | год | авторы | номер документа |
|---|---|---|---|
| ДИАГНОСТИЧЕСКИЕ СПОСОБЫ ОБНАРУЖЕНИЯ МУТАЦИИ ЦИХРОМА B В ГРИБАХ | 2000 |
|
RU2244750C2 |
| СПОСОБ ИДЕНТИФИКАЦИИ ФИТОПАТОГЕННЫХ ГРИБОВ ZYMOSEPTORIA TRITICI И PARASTAGONOSPORA NODORUM МЕТОДОМ ПЦР | 2023 |
|
RU2824044C1 |
| ФУНГИЦИДНОЕ СРЕДСТВО ДЛЯ ЗАЩИТЫ РАСТЕНИЙ | 1994 |
|
RU2121792C1 |
| КОНСТРУКЦИЯ ДНК, КОНСТРУКЦИЯ РЕКОМБИНАНТНОЙ ДНК И СПОСОБ ПОЛУЧЕНИЯ РАСТЕНИЙ | 1994 |
|
RU2136757C1 |
| ЭНДОФИТНЫЕ ГРИБЫ EPICHLOE/NEOTYPHODIUM SPP. ДЛЯ ИСКУССТВЕННОГО ЗАРАЖЕНИЯ SECALE SPP. И ИХ ПРИМЕНЕНИЕ | 2014 |
|
RU2683379C2 |
| СПОСОБ КОНТРОЛЯ QoI РЕЗИСТЕНТНЫХ ПАТОГЕННЫХ ГРИБОВ | 2009 |
|
RU2527029C2 |
| СПОСОБ ОПРЕДЕЛЕНИЯ УСТОЙЧИВОСТИ ГРИБНОГО ФИТОПАТОГЕНА, АЛЛЕЛЬСПЕЦИФИЧЕСКИЙ ПРАЙМЕР И НАБОР | 2002 |
|
RU2296162C2 |
| ШТАММ MYCOSPHAERELLA SP., КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ БОРЬБЫ С ФУЗАРИОЗОМ | 2012 |
|
RU2705286C2 |
| ШТАММ MICROBACTERIUM, КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ БОРЬБЫ С ФУЗАРИОЗОМ | 2012 |
|
RU2634386C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ БОРЬБЫ С ФУЗАРИОЗОМ | 2019 |
|
RU2736382C1 |
















































Изобретение относится к фитопатологии, в частности к способу определения грибковых патогенов с использованием специфических праймеров в реакции полимеризации. Способ обнаружения фитопатогенного гриба включает стадии: выделение ДНК из листа растения, зараженного патогеном, амплификацию части интронной транскрибирующейся области патогена с использованием выделенной ДНК в качестве матрицы с помощью полимеразной цепной реакции со специфическим праймером, визуализацию амплифицированной части интронной транскрибирующейся области. Определена структура нескольких молекул ДНК, кодирующей интронную транскрибирующуюся последовательность, используемую для конструирования олигонуклеотидных праймеров для обнаружения фитопатогенного гриба. Сконструированы праймеры для определения фитопатогенных грибов. Разработан количественный эколориметрический анализ обнаружения фитопатогенного гриба. Способ позволяет обнаружить такие фитопатогенные грибы, как microdochium nivate, Septoria nodorum, Septoria tritici, Pseudocercosporella herpotrichoides, Mycospharella fijiensis, Mycosphaerella musicola, Fusarium culmorum, Fusarium graminearum, Fusarium moniliform. 6 с. и 12 з.п. ф-лы, 5 табл., 3 ил.
| WO 9114001, 19.09.1991 | |||
| Physiological and molecular plant pathology, 1992, vol | |||
| Механический грохот | 1922 |
|
SU41A1 |
| Вагонетка для движения по одной колее в обоих направлениях | 1920 |
|
SU179A1 |
| Phytopathology, 1994, vol | |||
| Способ приготовления сернистого красителя защитного цвета | 1921 |
|
SU84A1 |
Авторы
Даты
2000-12-27—Публикация
1995-04-19—Подача