Изобретение относится к области биотехнологии, в частности к получению (биосинтезу) антимикробного рекомбинантного пептида Hst1-E2A в клетках бактерий за счет введения в эти клетки плазмиды, несущей ген, кодирующий рекомбинантный пептид Hst1-E2A. Рекомбинантный пептид Hst1-E2A, обладающий антимикробным действием к широкому спектру как грамотрицательных, так и грамположительных бактерий.
Устойчивость бактериальных клеток к антибиотикам является очень серьезной проблемой. При этом разработка новых антибиотиков снижается. Связано это с тем, что разработка классических антимикробных препаратов очень затратна, а потенциальная прибыль может не покрыть затраты из-за быстрого развития устойчивости к ним [Plackett, В. Why Big Pharma Has Abandoned Antibiotics. Nature 2020, 586, S50-S52]. С этой точки зрения необходимо искать такие соединения, к которым бактерии не смогут сформировать устойчивость или формируют ее очень медленно (десятки лет). В последние годы большой интерес вызывают так называемые антимикробные пептиды, которые продуцируются всеми живыми видами. Нередко их называют натуральными антибиотиками, при этом к антимикробным пептидам значительно реже развивается резистентность, чем к обычным антибиотикам [Saint, J.K.; Henderson, K.D.; Chrom, C.L.; Abiuso, L.E.; Renn, L.M.; Caputo, G.A. Effects of Hydrophobic Amino Acid Substitutions on Antimicrobial Peptide Behavior. Probiotics Antimicrob. Proteins 2018, 10, 408-41].
Помимо антибактериального действия многие антимикробные пептиды обладают противовирусным, противогрибковым, противопаразитарным и противоопухолевым действием [Huan, Y.; Kong, Q.; Мои, Н.; Yi, Н. Antimicrobial Peptides: Classification, Design, Application and Research Progress in Multiple Fields. Front. Microbiol. 2020,0, 2559]. Антимикробные пептиды представляют собой природные пептиды длиной до 100 аминокислот и обладают большим структурным разнообразием [Wenyi, L.; Frances, S.; Neil, M.S.; John, D.W. Chemically modified and conjugated antimicrobial peptides against superbugs. Chem. Soc. Rev. 2021,50, 4932-4973]. Большинство механизмов действия антимикробных пептидов основано на электростатическом связывании положительно заряженного пептида с отрицательно заряженными компонентами внешней мембраны бактериальной клетки, что в итоге приводит к дестабилизации бактериальной мембраны и к гибели бактерии. Поэтому одним из наиболее важных свойств антимикробных пептидов, необходимых для проявления их антибактериальных свойств, является способность к образованию амфипатической альфа-спирали [Токе О. Antimicrobial peptides: New candidates in the fight against bacterial infections // Biopolym. - Pept. Sci. Sect. 2005. Vol. 80, №6. P. 717-735].
Особый интерес исследователей привлекают антимикробные пептиды, которые могут участвовать не только в иммунном ответе, но и в заживлении ран [Niyonsaba F. et al. Multifunctional antimicrobial proteins and peptides: natural activators of immune systems // Curr. Pharm. Des. Curr Pharm Des, 2009. Vol. 15, №21. P. 2393-2413]. Одним из интереснейших представителей эндогенных антимикробных пептидов человека являются гистатины. Гистатиновые пептиды представляют собой богатое гистидином семейство из 12 пептидов (гистатин 1-12), возникающее из двух генов HTN1 и HTN36. Гистатины были впервые описаны как противогрибковые агенты в слюне и слезах [Kalmodia, S., Son, KN., Cao, D. et al. Presence of Histatin-1 in Human Tears and Association with Aqueous Deficient Dry Eye Diagnosis: A Preliminary Study. Sci Rep 9, 10304 (2019). https://doi.org/10.1038/s41598-019-46623-9], но с тех пор было обнаружено, что они обладают противовирусной, антибактериальной, ранозаживляющей и даже противовоспалительной активностью. Показана эффективность гистатинов для восстановления костной ткани [Torres Р, Flores V, Silva Р, Gonzalez L. et al. The salivary peptide histatin-1 enhances bone repair in vivo. Biochemical and Biophysical Research Communications // V 676, 8 October 2023, P 207-212, https://doi.org/10.1016/j.bbrc.2023.07.047] и ускоренной эпитализации раневых поверхностей [Lei X, Cheng L, Lin H, Pang M, Yao Z, Chen C, Forouzanfar T, Bikker FJ, Wu G and Cheng В (2020) Human Salivary Histatin-1 Is More Efficacious in Promoting Acute Skin Wound Healing Than Acellular Dermal Matrix Paste. Front. Bioeng. Biotechnol. 8:999. doi: 10.3389/fbioe.2020.00999].
Так в публикациях Formulations for histatin therapeutics (US 20170224771, US 20170232064, US 20170239330, US 20170239331, WO 2016060916, WO 2016060917, WO 2016060921) описывается использование гистатинов в качестве заживляющего и противомикробного агента в гелях, мазях, кремах, тканевых клеях, пластырях, а также в различных инфузионных средствах, при этом в данная серия патентов распространяется и на химерные молекулы, сочетающие в себе последовательности как гистатина 1, так и гистатина 2 и различных цитокинов, но не распространяется на последовательности гистатинов, содержащих нефункциональные группы (остатки гидролизных пептидов). Также в данной группе патентов не указано, каким образом будут получены данные активные пептиды на основе гистатинов, также не указаны последовательности генов, кодирующих данные пептиды.
Использование гистатинов, ровно, как и использование иных антимикробных пептидов связано со сложностью получения их в промышленных масштабах.
Для получения достаточного количества антимикробных пептидов можно выделить три основных подхода: прямая изоляция из природных источников, химический синтез или рекомбинантная экспрессия [Parachin, N.S.; Mulder, К.С.; Viana, А.А.В; Dias, S.C.; Franco, O.L. Expression systems for heterologous production of antimicrobial peptides. Peptides 2012, 38, 446-56]. Поскольку большинство организмов содержат очень низкие концентрации антимикробных пептидов, изоляция пептидов непосредственно из природных источников в большинстве случаев является трудоемким и дорогостоящим процессом [Li, С; Haug, Т.; Styrvold, О.В.; Jorgensen, Т.О.; Stensvag, К. Strongylocins, novel antimicrobial peptides from the green sea urchin, Strongylocentrotus droebachiensis. Dev Comp Immunol. 2008, 32, 1430-1440]. Химический синтез пептидов позволяет получать как природные, так и синтетические антимикробные пептиды, однако крупномасштабный синтез пептидов является дорогостоящим [Fink, J.; Merrifield, R.B.; Boman, A.; Boman, H.G. The chemical synthesis of cecropin D and an analog with enhanced antibacterial activity. J Biol Chem. 1989, 264, 6260-6267]. Экспрессия в клетках считается наиболее эффективным методом с точки зрения времени и производственных затрат [Nijnik, A.; Hancock, R. Host defence peptides: antimicrobial and immunomodulatory activity and potential applications for tackling antibiotic-resistant infections. Emerg Health Threats J. 2009, 2, el]. Можно экспрессировать антимикробные пептиды без модификации, но существует угроза гибели клетки-продуцента под воздействием антимикробных пептидов. Можно экспрессировать антимикробные пептиды, слитые с белком-носителем, или экспрессировать антимикробные пептиды в виде мультимеров, в этом случае полученный продукт необходимо будет обработать специфическими протеазами [Esposito, D.; Chatterjee, D.K. Enhancement of soluble protein expression through the use of fusion tags. Curr Opin Biotechnol. 2006, 17, 353-358].
В патенте US 8,865,467 продукты генов Oct4, Klf4, Sox2 и с-Мус разделены последовательностями 2А-самовырезающегося пептида (2А self-cleaving peptide), при этом патент позволяет использовать все из четырех известных 2А-пептидов (F2A, Е2А, Т2А и Р2А). Однако здесь экспрессируются не антимикробные пептиды, а иные продукты летивируса, при этом разделение экспрессионных продуктов происходит в клетке-продуценте.
В работе Expression of the Antimicrobial Peptide SE-33-A2P, a Modified Analog of Cathelicidin, and an Analysis of Its Properties (https://doi.org/10.3390/antibiotics13020190), которая принята за ближайший аналог, показан способ экспрессии аналога кателицидина, когда экспрессионный продукт представлял собой несколько повторяющихся последовательностей пептида SE-33, разделенных последовательностями 2А-самовырезающегося пептида (2А self-cleaving peptide). В данной работе есть ряд отличий от настоящего изобретения: во-первых, речь там идет не о гистатинах, а об аналоге кателицидина; во-вторых, к качестве самовырезающегося пептида представлен пептид Т2А, а не пептид Е2А; в-третьих, самогидролиз продукта, описанного в статье, происходит в клетке, поэтому авторы подбирали условия для оптимальной экспрессии, а также стабилизировали мембрану бактериальных клеток, для того чтобы избежать токсического действия пептида SE-33-A2P; в-четвертых, очистка продукта проводилась двумя хроматографиями на mono Q.
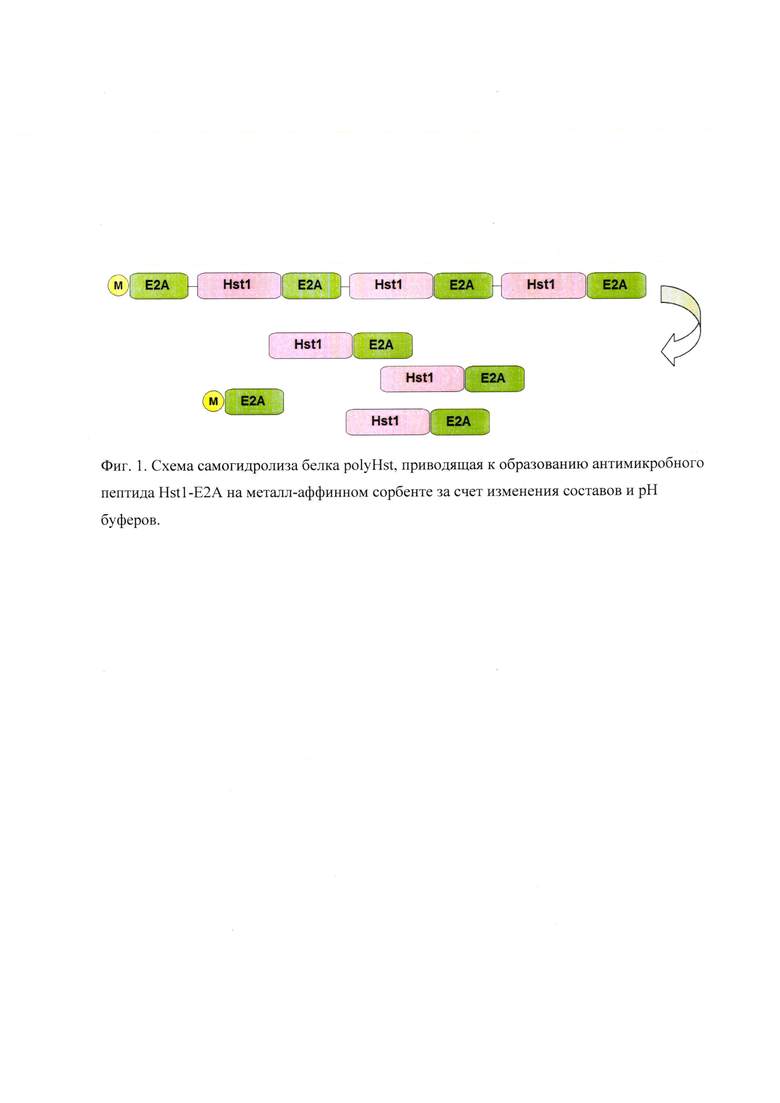
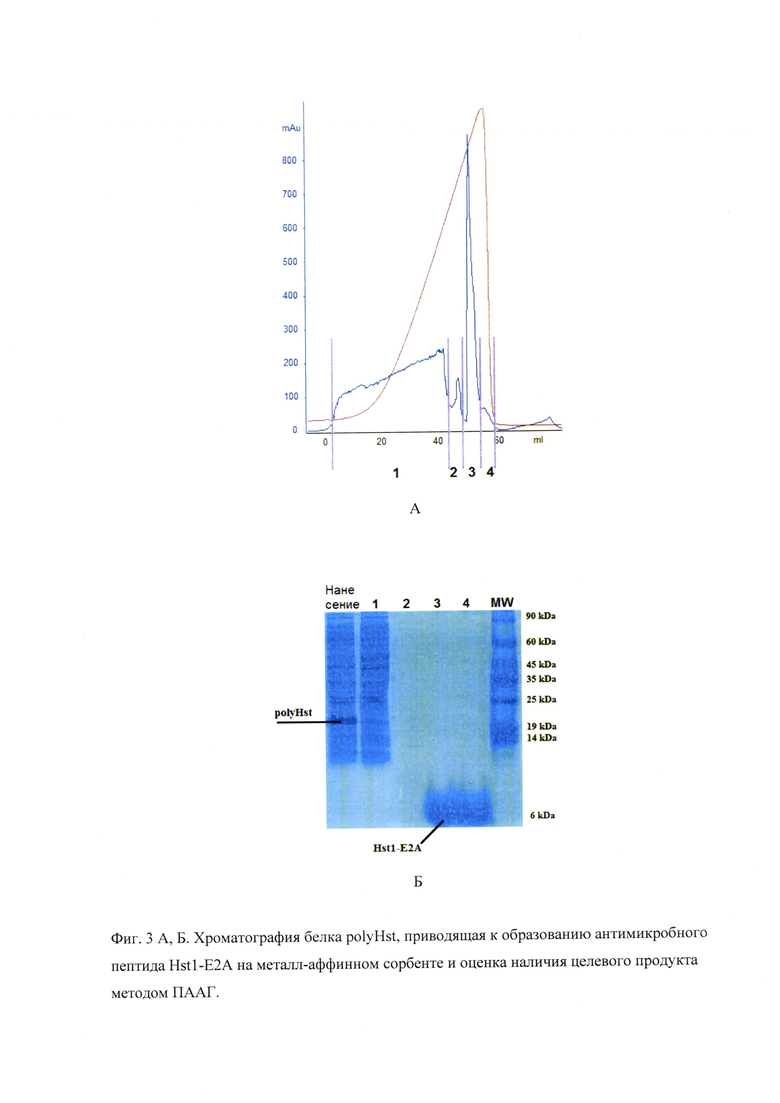
Заявителями разработан способ получения антимикробного пептида Hst1-E2A с SEQ ID NO: 1 методом рекомбинантной технологии. Для этого рекомбинантный белок polyHst с SEQ ID NO: 2, полученный в результате экспрессии бактериальной клеткой трансформированной плазмидой ppolyHst с SEQ ID NO: 3, после лизиса клеток сорбируется на металл-аффинную колонку, где после промывки и смены буфера и рН распадается на пептид Hst1-E2A, который и элюируется с колонки буфером, содержащим имидозол.
Техническим результатом, достигаемым при реализации данного изобретения, является получение выхода до 15 мг с 1 литра культуральной среды антимикробного пептида Hst1-E2A, белок-предшественник которого экспрессируется в клетках прокариот. Кроме того, технический результат заключается в упрощении способа в сравнении с ближайшим аналогом, так как очистка атимикробного пептида производится на одной колонке в рамках одной хроматографии. Также технический результат заключается в том, что клетки Е. coli стабильно экспрессируют белок polyHst после встраивания в них плазмиды по изобретению. Указанные технические результаты позволяют снизить себестоимость данного пептида по сравнению с химическим методом синтеза (получение гистатинов рекомбинантным способом не известно). Неожиданным техническим результатом стало то, что пептид Hst1-E2A проявляет 100% эффективной концентрации (ЕС 100) и 50% эффективной концентрации (ЕС50), при концентрациях 100-200 мкг/мл и 50 мкг/мл соответственно.
Антимикробный пептид Hst1-E2A имеет выраженную антимикробную активность в отношении граммположительных и граммотрицательных бактерий. Антимикробный пептид Hst1-E2A можно использовать в качестве антимикробного агента в составе лекарственных средств и медицинских изделий, прежде всего при заболеваниях, осложненных бактериальной инфекцией, устойчивой к антибиотикам, а также в косметологии.
Краткое описание фигур:
Фиг. 1. Схема самогидролиза белка polyHst по SEQ ID: 2, приводящая к образованию антимикробного пептида Hst1-E2A по SEQ ID: 1 на металл-аффинном сорбенте за счет изменения составов и рН буферов.
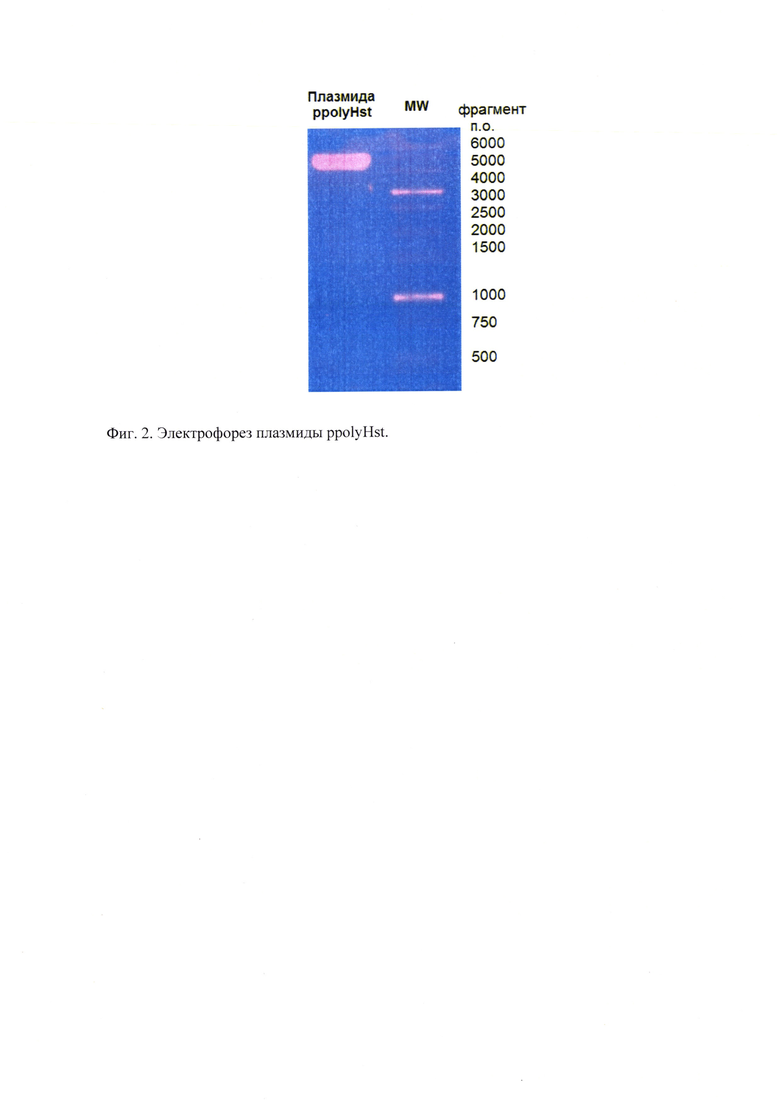
Фиг. 2. Электрофорез плазмиды ppolyHst по SEQ ID: 3.
Фиг. 3 А и Б. Хроматография белка polyHst по SEQ ID: 3, приводящая к образованию антимикробного пептида Hst1-E2A по SEQ ID: 1 на металл-аффинном сорбенте и оценка наличия целевого продукта методом ПААГ.
Фиг. 4. Аналитическая HPLC хроматографически очищенного пептида Hst1-E2A по SEQ ID: 1.
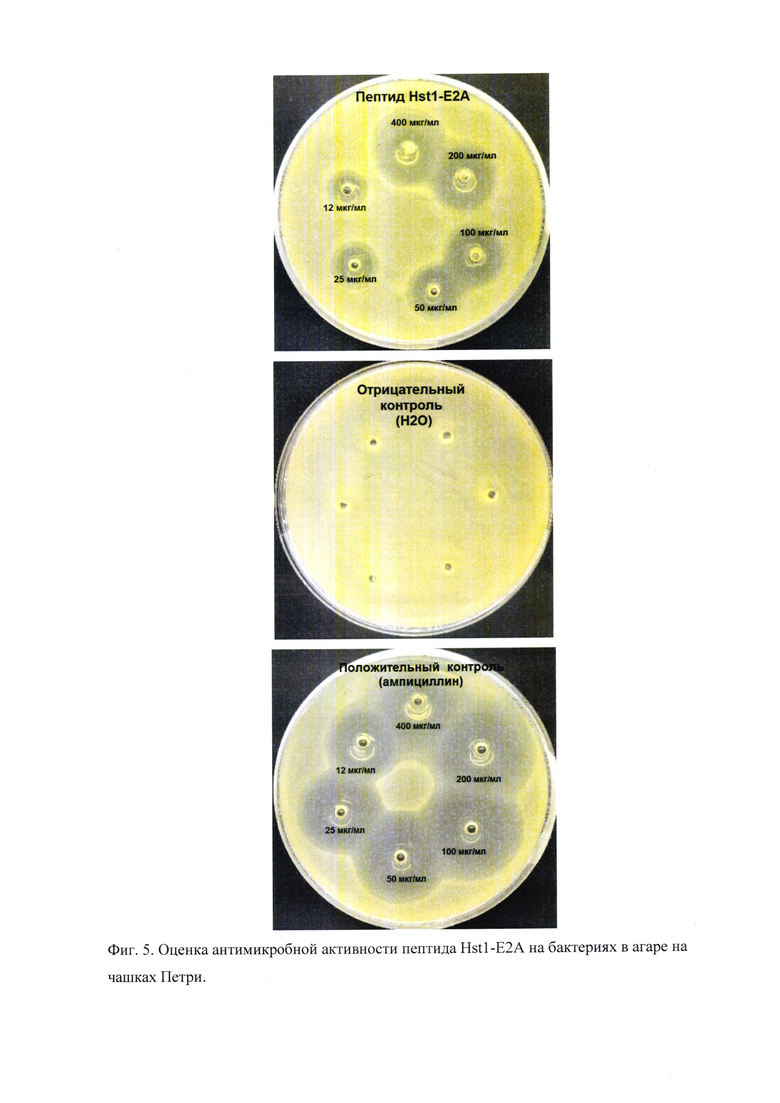
Фиг. 5. Оценка антимикробной активности пептида Hst1-E2A по SEQ ID: 1 на бактериях в агаре на чашках Петри.
ПРИМЕРЫ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ:
Пример 1. Получение плазмиды ppolyHst
Плазмиду рЕТ30(а) обрабатывали рестриктазами XbaI и XhoI. Проводили процедуру дефосфорилирования.
Химически синтезированную последовательность, кодирующую белок polyHst SEQ ID NO: 2, с триплетами, оптимизированными для экспрессии в бактериальных клетках, также обрабатывали рестриктазами XbaI и XhoI и десфорилировали.
Полученные фрагменты лигировали. Полученная плазмида, содержащая ген, кодирующий белок polyHst и имеющая 5674 оснований является плазмидой ppolyHst, имеющей SEQ ID NO: 3.
Пример 2. Трансформация плазмидой ppolyHst клеток Е. coli серии DE3 - получение клеток, стабильно экспрессирующих белок polyHst
Компетентные клетки одного из штаммов BL21(DE3) извлекали из кельвинатора (-80С°) и переносили в морозильную камеру, где инкубировали 8 часов при температуре -20С°. Затем пробирку с компетентными клеткам помещали на лед, где инкубировали в течение 2 часов. Далее в 100 мкл суспензии компетентных клеток вносили 100 нг плазмиды ppolyHst с SEQ ID NO: 3, аккуратно пипетировали и инкубировали на льду в течение 30 минут. Далее смесь помещали в термостат и инкубировали 45 секунд при 42С°, после чего смесь вновь помещали на лед на 30 минут. Далее в смесь добавляли 500 мкл буфера SOB и инкубировали 30 минут при 37С°. Затем смесь помещали в шейкер-инкубатор и выращивали клетки в течение 30 мину при 37С° и перемешивании при скорости 180 об/мин. Через 30 минут смесь разбавляли в 2 раза буфером LB, содержащим канамицин в концентрации 50 мкг/мл. Далее смесь помещали в шейкер-инкубатор и выращивали клетки в течение 3 часов при 37С° и перемешивании при скорости 180 об/мин. Через 3 часа культуру рассевали штрихом с использованием микробиологической петли на 2% агар в LB с 25 мкг/мл канамицином в чашки Петри.
Чашки Петри помещали в термостат на 37С° на 8 часов. Из выросших колоний выбирали несколько клонов. Выбранные клоны засевали в пробирки с LB объемом 2 мл с канамицином 25 мкг/мл. Выращивали на шейкере-инкубаторе при 180 об/мин при температуре 37С° в течение 4 часов до оптической плотности OD585=0,5OE. Весь объем культуры клеток пересевали в колбу с 10 мл LB с канамицином 25 мкг/мл и выращивали в течение двух часов. Через два часа весь объем пересевали в 100 мл LB с канамицином и вновь растили в тех же условиях в течение 2 часов до оптической плотности OD585=0,5OE. Далее вносили индуктор IPTG в концентрации 400 мкг/мл и добавляли гранулы PBS (две на 100 мл культуральной среды) для поддержания рН культуральной среды в районе 7,5. Через два часа культивирование прекращали. Уровень экспрессии белка polyHst с SEQ ID NO: 2 определяли электрофорезом в ПААГ. В пересчете на 1 литр культуральной среды он составил 20 мг.
Приме 3. Хроматография на металл-аффинном сорбенте, получение пептида Hst1-E2A
В культуральную среду засыпали мочевину из расчета 60 г на 100 мл культуральной среды. Вводили Tris-HCl рН8.0 до концентрации 50 мМ. Далее смесь перемешивали и центрифугировали при 20000 g в течение 30 минут.
Надосадочную жидкость наносили на хроматографическую колонку с Ni-Sepharose, уравновешенную 8 М мочевиной, 50 мМ NaCl и 50 мМ Tris-HCl рН 8.0. Колонку промывали раствором, содержащим 8 М мочевину, 50 мМ NaCl, 50 мМ Tris-HCl рН 8.0 в градиенте имидозола от 0 до 30 мМ. Объем промывки составлял 2 объема колонки.
Далее колонку переводили в раствор, содержащий 8 М мочевину, 50 мМ NaCl, 50 мМ Tris-HCl рН 6.0 в градиенте имидозола от 30 до 70 мМ. Объем промывки составлял 2 объема колонки.
Затем колонку переводили в раствор, содержащий 50 мМ NaCl, 50 мМ Tris-HCl рН 6.0, 70 мМ имидозола. Объем промывки составлял 1 объем колонки.
Элюировали пептид Hst1-E2A с SEQ ID NO: 1 в растворе, содержащем 50 мМ NaCl, 50 мМ Tris-HCl рН 6.0, в градиенте имидозола от 70 до 120 мМ. Объем элюции составил 2 объема колонки.
Чистоту пептида Hst1-E2A с SEQ ID NO: 1 определяли электрофорезом в ПААГ и на HPLC.
Пример 4. Изучение биологической активности (антимикробного действия) пептида Hst1-Е2А.
Антимикробные свойства пептида Hst1-E2A по SEQ ID NO: 1 были исследованы на грамотрицательных бактериях 6 штаммов E.coli: NB, BL21, BL21(DE3), BL21(DE3)pLysS, Origami(DE3), Origami(DE3)pLysS, в том числе и обладающих антибиотикоустойчивостью, и на грамположительных бактериях: Lactobacillus plantarum и Lactobacillus fermentum.
Готовили 2% агар LB в автоклаве. Далее агар остужали до 50С°. Затем в агар вносили по 1*10^9 бактериальных клеток на 20 мл агара. После этого агар заливали по 20 мл в чашки Петри диаметром 90 мм. После застывания агара специальным пробойником в агаре формировали лунки. В лунки вносили раствор антимикробного пептида Hst1-E2A с SEQ ID NO: 1. В лунки вносили раствор с концентрациями пептида Hst1-E2A с SEQ ID NO: 1: 400 мкг/мл; 200 мкг/мл; 100 мкг/мл; 50 мкг/мл; 25 мкг/мл и 12 мкг/мл.
Через сутки оценивали диаметр зоны отсутствия роста в агаре вокруг каждой из лунок.
Результаты расчета 100% эффективной концентрации (ЕС 100) и 50% эффективной концентрации (ЕС50) приведены в таблице.
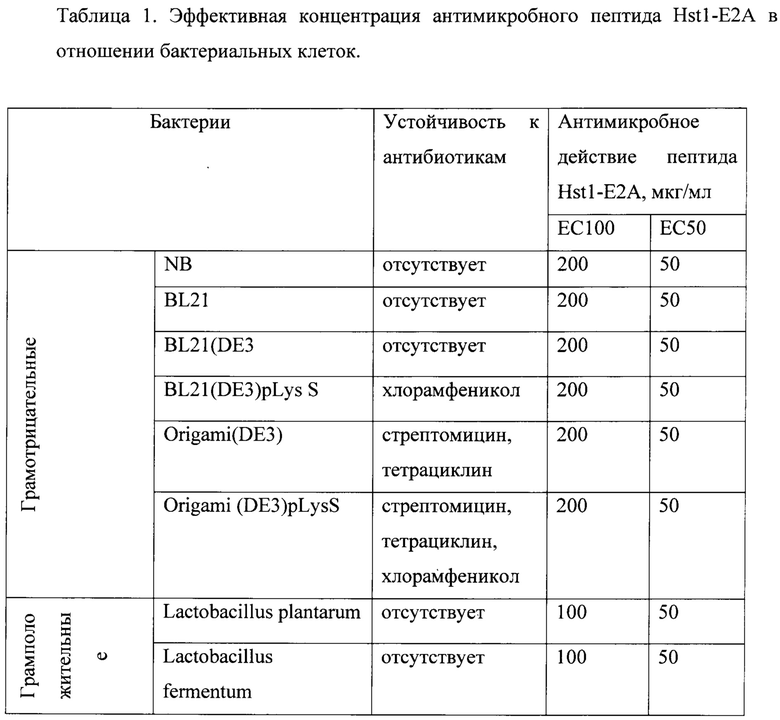
Следует отметить, что в уровне техники не выявлены сведения об эффективной концентрации пептида Hst1. Наши исследования показали, что непосредственно пептид Hst1, в сравнении с рекомбинантным пептидом Hst1-E2A, требует использования существенно более высоких концентраций, так 100% эффективной концентрацией для указанных выше штаммов является концентрация примерно на 10-15% выше, чем для заявляемого пептида. Тоже касается и 50% эффективности.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Плазмида, способ,
пептид.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-09-14">
<ApplicantFileReference>14092024</ApplicantFileReference>
<ApplicantName languageCode="ru">ООО "ЭВУАЗЬЕН
БИО"</ApplicantName>
<ApplicantNameLatin>OOO "EVUAZ'EN
BIO"</ApplicantNameLatin>
<InventorName languageCode="ru">Велесова Татьяна
Анатольевна</InventorName>
<InventorNameLatin>Velesova Tat'yana
Anatol'evna</InventorNameLatin>
<InventionTitle languageCode="ru">ПЛАЗМИДА, СПОСОБ РЕКОМБИНАНТНОЙ
ЭКСПРЕССИИ АНТИМИКРОБНОГО ПЕПТИДА, РЕКОМБИНАНТНЫЙ АНТИМИКРОБНЫЙ
ПЕПТИД</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>58</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..58</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DSHEKRHHGYRRKFHEKHHSHREFPFYGDYGSNYLYDNQCTNYALLKLA
GDVESNPGP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>195</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..195</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MQCTNYALLKLAGDVESNPGPDSHEKRHHGYRRKFHEKHHSHREFPFYG
DYGSNYLYDNQCTNYALLKLAGDVESNPGPDSHEKRHHGYRRKFHEKHHSHREFPFYGDYGSNYLYDNQC
TNYALLKLAGDVESNPGPDSHEKRHHGYRRKFHEKHHSHREFPFYGDYGSNYLYDNQCTNYALLKLAGDV
ESNPGP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>5674</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5674</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcgaatgggacgcgccctgtagcggcgcattaagcgcggcgggtgtg
gtggttacgcgcagcgtgaccgctacacttgccagcgccctagcgcccgctcctttcgctttcttccctt
cctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccgatt
tagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccc
tgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttccaaactg
gaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccgatttcggcctattg
gttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttacaatttca
ggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgt
atccgctcatgaattaattcttagaaaaactcatcgagcatcaaatgaaactgcaatttattcatatcag
gattatcaataccatatttttgaaaaagccgtttctgtaatgaaggagaaaactcaccgaggcagttcca
taggatggcaagatcctggtatcggtctgcgattccgactcgtccaacatcaatacaacctattaatttc
ccctcgtcaaaaataaggttatcaagtgagaaatcaccatgagtgacgactgaatccggtgagaatggca
aaagtttatgcatttctttccagacttgttcaacaggccagccattacgctcgtcatcaaaatcactcgc
atcaaccaaaccgttattcattcgtgattgcgcctgagcgagacgaaatacgcgatcgctgttaaaagga
caattacaaacaggaatcgaatgcaaccggcgcaggaacactgccagcgcatcaacaatattttcacctg
aatcaggatattcttctaatacctggaatgctgttttcccggggatcgcagtggtgagtaaccatgcatc
atcaggagtacggataaaatgcttgatggtcggaagaggcataaattccgtcagccagtttagtctgacc
atctcatctgtaacatcattggcaacgctacctttgccatgtttcagaaacaactctggcgcatcgggct
tcccatacaatcgatagattgtcgcacctgattgcccgacattatcgcgagcccatttatacccatataa
atcagcatccatgttggaatttaatcgcggcctagagcaagacgtttcccgttgaatatggctcataaca
ccccttgtattactgtttatgtaagcagacagttttattgttcatgaccaaaatcccttaacgtgagttt
tcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcg
taatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctacc
aactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtgtagccg
tagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccag
tggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggc
gcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactg
agatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccgg
taagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttatag
tcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagccta
tggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttct
ttcctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccg
cagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcctgatgcggtattttctc
cttacgcatctgtgcggtatttcacaccgcatatatggtgcactctcagtacaatctgctctgatgccgc
atagttaagccagtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacccgccaa
cacccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgaccgtctc
cgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagctgcggtaaagctca
tcagcgtggtcgtgaagcgattcacagatgtctgcctgttcatccgcgtccagctcgttgagtttctcca
gaagcgttaatgtctggcttctgataaagcgggccatgttaagggcggttttttcctgtttggtcactga
tgcctccgtgtaagggggatttctgttcatgggggtaatgataccgatgaaacgagagaggatgctcacg
atacgggttactgatgatgaacatgcccggttactggaacgttgtgagggtaaacaactggcggtatgga
tgcggcgggaccagagaaaaatcactcagggtcaatgccagcgcttcgttaatacagatgtaggtgttcc
acagggtagccagcagcatcctgcgatgcagatccggaacataatggtgcagggcgctgacttccgcgtt
tccagactttacgaaacacggaaaccgaagaccattcatgttgttgctcaggtcgcagacgttttgcagc
agcagtcgcttcacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccgccagcc
tagccgggtcctcaacgacaggagcacgatcatgcgcacccgtggggccgccatgccggcgataatggcc
tgcttctcgccgaaacgtttggtggcgggaccagtgacgaaggcttgagcgagggcgtgcaagattccga
ataccgcaagcgacaggccgatcatcgtcgcgctccagcgaaagcggtcctcgccgaaaatgacccagag
cgctgccggcacctgtcctacgagttgcatgataaagaagacagtcataagtgcggcgacgatagtcatg
ccccgcgcccaccggaaggagctgactgggttgaaggctctcaagggcatcggtcgagatcccggtgcct
aatgagtgagctaacttacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtg
ccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgccagggtggtttt
tcttttcaccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgcagcaag
cggtccacgctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatataacatg
agctgtcttcggtatcgtcgtatcccactaccgagatgtccgcaccaacgcgcagcccggactcggtaat
ggcgcgcattgcgcccagcgccatctgatcgttggcaaccagcatcgcagtgggaacgatgccctcattc
agcatttgcatggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcggctgaa
tttgattgcgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaatgggcc
cgctaacagcgcgatttgctggtgacccaatgcgaccagatgctccacgcccagtcgcgtaccgtcttca
tgggagaaaataatactgttgatgggtgtctggtcagagacatcaagaaataacgccggaacattagtgc
aggcagcttccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgacgcgttg
cgcgagaagattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacaccaccacg
ctggcacccagttgatcggcgcgagatttaatcgccgcgacaatttgcgacggcgcgtgcagggccagac
tggaggtggcaacgccaatcagcaacgactgtttgcccgccagttgttgtgccacgcggttgggaatgta
attcagctccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctggttcacc
acgcgggaaacggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtttcacat
tcaccaccctgaattgactctcttccgggcgctatcatgccataccgcgaaaggttttgcgccattcgat
ggtgtccgggatctcgacgctctcccttatgcgactcctgcattaggaagcagcccagtagtaggttgag
gccgttgagcaccgccgccgcaaggaatggtgcatgcaaggagatggcgcccaacagtcccccggccacg
gggcctgccaccatacccacgccgaaacaagcgctcatgagcccgaagtggcgagcccgatcttccccat
cggtgatgtcggcgatataggcgccagcaaccgcacctgtggcgccggtgatgccggccacgatgcgtcc
ggcgtagaggatcgagatcgatctcgatcccgcgaaattaatacgactcactataggggaattgtgagcg
gataacaattcccctctagaatggattcacatgaaaaaaggcaccacggataccgtagaaaattccacga
aaagcaccacagccatcgtgagttcccgttttacggtgactatggctcgaactatctgtacgacaatatg
gattcccacgagaagcgccatcatggctatcgacgtaagttccacgagaagcatcactcccatcgggaat
tcccgttttatggcgactacggcagcaattacctgtatgataacatggatagccacgagaaacgtcatca
cggttaccggcgtaaatttcatgaaaaacatcattctcaccgcgaatttccgttttatggtgattacggt
agcaactacctgtatgacaatatggactctcatgaaaagcgccatcatggttaccgccgcaagtttcacg
agaagcaccacagccaccgagagttcccattctatggtgattacggcagcaactatttgtacgacaacta
acctcgagcaccaccaccaccaccactgagatccggctgctaacaaagcccgaaaggaagctgagttggc
tgctgccaccgctgagcaataactagcataaccccttggggcctctaaacgggtcttgaggggttttttg
ctgaaaggaggaactatatccggat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ПЕПТИД НИКОМИЦИН ИЗ МОРСКОГО КОЛЬЧАТОГО ЧЕРВЯ NICOMACHE MINOR, ОБЛАДАЮЩИЙ АНТИМИКРОБНЫМ И ПРОТИВООПУХОЛЕВЫМ ДЕЙСТВИЕМ. | 2019 |
|
RU2721273C1 |
| БЕТА-ШПИЛЕЧНЫЙ ПОЛИПЕПТИД, ОБЛАДАЮЩИЙ АНТИМИКРОБНОЙ АКТИВНОСТЬЮ | 2015 |
|
RU2624020C2 |
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pЕ-Trx-Lc-def, ШТАММ Escherichia coli ДЛЯ ЭКСПРЕССИИ АНТИМИКРОБНОГО ПЕПТИДА ДЕФЕНСИНА ЧЕЧЕВИЦЫ Lens culinaris И СПОСОБ ПОЛУЧЕНИЯ УКАЗАННОГО ПЕПТИДА | 2010 |
|
RU2456345C1 |
| ПЕПТИД, ОБЛАДАЮЩИЙ ПРОТИВООПУХОЛЕВОЙ АКТИВНОСТЬЮ | 2017 |
|
RU2658781C1 |
| Пептид, проявляющий антибактериальные и противоопухолевые свойства | 2019 |
|
RU2702661C1 |
| Способ получения рекомбинантного антимикробного пептида UBI18-35, рекомбинантная плазмидная ДНК pET31b-2хUBI18-35 и штамм-продуцент Escherichia coli BL21 Rosetta DE3 pLysS/ pET31b-2хUBI18-35 антимикробного пептида UBI18-35 | 2018 |
|
RU2698037C1 |
| ПЛАЗМИДНЫЙ ВЕКТОР pE-Trx-Aur, ШТАММ ESCHERICHIA COLI ДЛЯ ЭКСПРЕССИИ АНТИМИКРОБНОГО ПЕПТИДА АУРЕЛИНА И СПОСОБ ПОЛУЧЕНИЯ УКАЗАННОГО ПЕПТИДА | 2009 |
|
RU2412999C1 |
| Бета-шпилечный пептид, обладающий антимикробной активностью в отношении бактерий с множественной лекарственной устойчивостью | 2023 |
|
RU2812977C1 |
| ПЛАЗМИДНЫЙ ВЕКТОР pET-His8-TrxL-Acip1, ШТАММ БАКТЕРИИ Escherichia coli BL21(DE3)/pET-His8-TrxL-Acip1 ДЛЯ ЭКСПРЕССИИ АНТИМИКРОБНОГО ПЕПТИДА АЦИПЕНСИНА-1 И СПОСОБ ПОЛУЧЕНИЯ УКАЗАННОГО ПЕПТИДА | 2015 |
|
RU2580031C2 |
| Плазмидный вектор pET-mChBac75Na, штамм бактерии Eschrichia coli BL21(DE3/ pET-mChBac75Na для экспрессии антимикробного пептида минибактенецина ChBac7.5 Nα и способ получения указанного пептида | 2016 |
|
RU2618850C2 |
Изобретение относится к области биотехнологии, в частности к получению (биосинтезу) антимикробного пептида Hst1-E2A в клетках бактерий за счет введения в эти клетки векторов (генетических конструкций), несущих ген, кодирующий пептид Hst1-E2A, обладающий антимикробным действием к широкому спектру как грамотрицательных, так и грамположительных бактерий. Описан способ получения антимикробного пептида Hst1-E2A по SEQ ID: 1 методом рекомбинантной технологии. Для этого рекомбинантный белок polyHst по SEQ ID: 2, полученный в результате экспрессии бактериальной клеткой, трансформированной плазмидой ppolyHst по SEQ ID: 3, после лизиса клеток сорбируется на металл-аффинную колонку, где после промывки и смены буфера и рН распадается на пептид Hst1-E2A, который и элюируется с колонки буфером, содержащим имидозол. Техническим результатом, достигаемым при реализации данного изобретения, является получение выхода антимикробного пептида Hst1-E2A не менее чем 15 мг с одного литра культуральной среды, белок-предшественник которого экспрессируется в клетках прокариот, что снижает себестоимость данного пептида по сравнению с химическим методом синтеза. Очистка атимикробного пептида производится на одной колонке в рамках одной хроматографии, что дополнительно снижает себестоимость продукта. Антимикробный пептид Hst1-E2A можно использовать в качестве антимикробного агента в составе лекарственных средств и медицинских изделий, прежде всего при заболеваниях, осложненных бактериальной инфекцией, устойчивой к антибиотикам, а также в косметологии. 4 н.п. ф-лы, 5 ил., 1 табл., 4 пр.
1. Плазмида ppolyHst для получения антимикробного пептида, имеющая SEQ ID NO: 3, кодирующая белок polyHst SEQ ID NO: 2, в состав которого входит пептид Hst1-E2A, имеющий SEQ ID NO: 1.
2. Антимикробный пептид Hst1-E2A, имеющий SEQ ID NO: 1.
3. Белок polyHst для получения пептида по п. 2, имеющий SEQ ID NO: 2, содержащий пептид Hst1-E2A, имеющий SEQ ID NO: 1.
4. Способ получения пептида Hst1-E2A по п. 2, включающий: встраивание плазмиды по п. 1 в клетку-хозяин; культивирование клетки-хозяина для экспрессии белка polyHst по п. 3; лизис клеток; хроматографию на металл-аффинном сорбенте, включающую сорбирование на металл-аффинную колонку, промывку, смену буфера и рН, в результате чего белок polyHst по п. 3 распадается с выделением пептида Hst1-E2A по п. 2; элюирование пептида по п. 2.
| US 20170239331 A1, 24.08.2017 | |||
| US 8865467 B2, 21.10.2014 | |||
| Способ получения рекомбинантного антимикробного пептида UBI18-35, рекомбинантная плазмидная ДНК pET31b-2хUBI18-35 и штамм-продуцент Escherichia coli BL21 Rosetta DE3 pLysS/ pET31b-2хUBI18-35 антимикробного пептида UBI18-35 | 2018 |
|
RU2698037C1 |
Авторы
Даты
2024-12-16—Публикация
2024-09-23—Подача