Область техники, к которой относится изобретение
Изобретение относится к области генетики, молекулярной биологии и медицины и касается способа определения чувствительности злокачественных опухолей к таргетной терапии посредством анализа соматических мутаций в гене PI3K с помощью технологии LNA-блокирующей мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом).
Уровень техники
Известно большое количество способов определения соматических мутаций, в которых используются различные инструменты для анализа уникальной нуклеотидной последовательности ДНК человека. Условно среди них можно выделить шесть групп:
1) Ферментативные подходы:
полиморфизм длин амплифицированных фрагментов (AFLP);
полиморфизм длин рестрикционных фрагментов (RFLP);
расщепление клевазой (CFLP);
расщепление резольвазой (EMD);
анализ, основанный на лигазной реакции (LDR, LCR);
ПЦР, обогащенная мутантной последовательностью;
PNA-блокирующая ПЦР в реальном времени (PNA-clamp PCR);
LNA-блокирующая ПЦР в реальном времени (LNA-clamp PCR);
ПЦР с прямой терминацией синтеза (DT-PCR);
аллель-специфическая ПЦР (AS-PCR, PCR-SSP);
SMart-амплификация;
PNA-LNA-блокирующая ПЦР в реальном времени (PNA- LNA-clamp PCR);
Секвенирование.
2) Химические методы: химическое расщепление гетеродупЛексов; химическое лигирование.
3) Методы, основанные на различной электрофоретической подвижности полиморфных участков ДНК:
анализ конформации одноцепочечных фрагментов (SSCP);
гетеродуплексный анализ (HA);
разделение продуктов амплификации посредством капиллярного электрофореза.
4) Детекция на твердой фазе: гибридизация на олигонуклеотидных матрицах; оптико-волоконный ДНК-гибридизационный анализ; элонгация иммобилизованных праймеров (минисеквенирование); пиросеквенирование.
5) Хроматографические методы: высокоэффективная жидкостная хроматография (HPLC).
6) Физические методы:
масс-спектрометрия;
резонансное тушение флуоресценции (FRET);
люминесценция, зависящая от локального окружения.
Наиболее распространенными из представленных методов являются:
1. Секвенирование амплифицированных фрагментов ДНК по Сенгеру
PIK3CA Activating Mutation in Colorectal Carcinoma: Associations with Molecular Features and Survival / C. Rosty, J.P. Young, M.D. Walsh et al. // PLoS One. - 2013. - Vol.8. - e65479. PIK3CA Gene Mutations in Pediatric and Adult Glioblastoma Multiforme / G.L. Gallia, V. Rand, I-M. Siu et al. // Mol Cancer Res. - 2006. - Vol.4. - P. 709-714.
2. Глубокое секвенирование
Novel Somatic Mutations to PI3K Pathway Genes in Metastatic Melanoma / A.Y. Shull, A. Latham-Schwark, P. Ramasamy et al. // PLoS One. - 2012. - Vol.7. - e43369.
3. Анализ конформации одноцепочечных фрагментов ДНК (SSCP)
PIK3CA exon 20 mutations are associated with poor prognosis in breast cancer patients / F.R. Mangone, I.G. Bobrovnitchaia, S. Salaorni et al. //Clinics (Sao Paulo). - 2012. - Vol.67. - P. 1285-1290.
Analysis of PIK3CA and B-RAF gene mutations in human astrocytomas: association with activation of ERK and AKT / Ε.A. El-Habr, P. Tsiorva, M. Theodorou et al. // Clin Neuropathol. - 2010. - Vol.29. - P. 239-245.
4. Аллель-специфичная ПЦР с визуализацией продуктов амплификации в агарозном геле
Mutation profiling identifies numerous rare drug targets and distinct mutation patterns in different clinical subtypes of breast cancers / L. Santarpia, Y. Qi, K. Stemke-Hale et al. // Breast Cancer Res Treat. - 2012. - Vol. 134. - P. 333-343.
5. Аллель-специфичная ПЦР в реальном времени
A real-time PCR assay for the detection of PIK3CA mutations in formalin-fixed paraffin embedded tissue (FFPET) specimens of breast cancer (ВС) / P. Schlag, C. Hoeppner, A. Bristol et al. // Cancer Res. - 2013. - Vol. 73. - Suppl. 1. - Abstr. 4217.
PIK3CA gene mutation associated with poor prognosis of lung adenocarcinoma / L. Zhang, L. Shi, X. Zhao et al. // Onco Targets Ther. - 2013. - Vol. 6. P. 497-502.
Mutations affecting BRAF, EGFR, PIK3CA, and KRAS are not associated with sporadic vestibular schwannomas / M. de Vries, I.B. Bruijn, A.M. Cleton-Jansen et. al. // Virchows Arch. - 2013. - Vol.462. - 211-217.
Rapid KRAS, EGFR, BRAF and PIK3CA mutation analysis of fine needle aspirates from non-small-cell lung cancer using allele-specific qPCR / R. van Eijk, J. Licht, M. Schrumpf et al. //PLoS One. - 2011. - Vol.6. - e17791.
6. Гибридизация с биочипами
Mutant PIK3CA accelerates HER2-driven transgenic mammary tumors and induces resistance to combinations of anti-HER2 therapies / A.B. Hanker, A.D. Pfefferle, J.M. Balko et al. // Proc Natl Acad Sci USA. - 2013. - Vol.110. - P. 14372-14377.
Lack of estrogen receptor-α is associated with epithelial-mesenchymal transition and PI3K alterations in endometrial carcinoma / E. Wik, M.B. Ræder, C. Krakstad et al. // Clin Cancer Res. - 2013. - Vol. 19. - P. 1094-1105.
A combined oncogenic pathway signature of BRAF, KRAS and PI3KCA mutation improves colorectal cancer classification and cetuximab treatment prediction / S. Tian, I. Simon, V. Moreno et al. // Gut. - 2013. - Vol. 62. - P. 540-549.
7. PNA-блокирующая ПЦР
Frequency of KRAS, BRAF, and PIK3CA mutations in advanced colorectal cancers: Comparison of peptide nucleic acid-mediated PCR clamping and direct sequencing in formalin-fixed, paraffin-embedded tissue / M.J. Kwon, S.E. Lee, S.Y. Kang, Y.L. Choi // Pathol Res Pract. - 2011. - Vol. 207. - P. 62-68.
8. LNA- блокирующая ПЦР с последующим секвенированием
Novel method for PIK3CA mutation analysis: locked nucleic acid - PCR sequencing / D. Ang, R. O′Gara, A. Schilling et al. // J Mol Diagn. - 2013. - Vol.15. - 312-318.
9. ПЦР, обогащенная мутантной последовательностью
Combined analysis of KRAS and PIK3CA mutations, MET and PTEN expression in primary tumors and corresponding metastases in colorectal cancer / A. Voutsina, M. Tzardi, A. Kalikaki et al. // Mod Pathol. - 2013. - Vol. 26. - P. 302-313.
10. Анализ кривых плавления высокого разрешения
High-Resolution Melting Analysis for Rapid Detection of KRAS, BRAF, and PIK3CA Gene Mutations in Colorectal Cancer / L. Simi, N. Pratesi, M. Vignoli et al. // Am J Clin Pathol. - 2008. - Vol.130. - P. 247-253.
Метод 1 является достаточно информативным, так как предоставляет полную информацию о последовательности ДНК в исследуемом локусе и позволяет определять мутации de novo. Однако у него есть несколько существенных недостатков: он не обладает достаточной чувствительностью для использования в клинической практике при анализе соматических мутаций (для данного метода образец должен содержать не менее 10% мутантной ДНК), характеризуется низкой производительностью, и необходимостью в дорогостоящем оборудовании и реактивах. Кроме того, для дальнейшей расшифровки хроматограмм необходима высокая квалификация персонала, что препятствует использованию данного метода в широкомасштабных исследованиях.
Метод 2, в отличии от секвенирования по Сенгеру, позволяет выявлять мутации, даже если мутантная ДНК составляет около 1% от общего пула ДНК. Этот метод, также как и секвенирование по Сенгеру, позволяет обнаруживать мутации de novo, поэтому его использование в клинико-диагностической лаборатории может быть очень полезно, однако из-за очень высокой стоимости оборудования и расходным материалов для анализа, его использование не получило широкое распространение в клинической диагностике. Кроме того, для работы на анализаторе требуется очень высокая квалификация персонала.
Метод 3 дает лишь косвенную информацию о нуклеотидной последовательности анализируемого участка гена, сложен в исполнении и трактовке результатов, не подлежит автоматизации и не позволяет делать однозначные выводы о генотипе исследуемого образца.
Метод 4 достаточно распространен, но требует постановки независимых реакций по количеству однонуклеотидных замен для выявления только одного генотипа. Кроме того, для оценки результатов требуется постановка гель-электрофореза, что резко снижает его ценность для широкомасштабного генотипирования.
Метод 5 широко распространен, на его основе разработано большое количество диагностических тест-систем, он обладает достаточно высокой аналитической чувствительностью. К недостаткам данного метода можно отнести необходимость постановки параллельных реакций при анализе большого количества соматических мутаций, необходимость дорогостоящего оборудования и достаточно высокой квалификации персонала.
Метод 6 наиболее перспективен для проведения широкомасштабного генотипирования, однако, на данный момент отсутствуют дешевые тест-системы, которые позволяли бы за один анализ определять большинство диагностически важных мутаций в исследуемом гене.
Методы 7-8 требуют постановки нескольких параллельных реакций по количеству анализируемых локусов для каждого пациента. Метод 7 позволяет определить только факт наличия/отсутствия мутации в образце, но не дает информации о конкретной генетической последовательности анализируемых локусов. В случае, если в одной пробирке анализируется присутствие только одного типа мутации, то данный подход весьма оправдан, а при одновременном анализе в одной пробирке сразу нескольких типов мутаций этот метод уже недостаточно информативен, поскольку позволяет обнаруживать лишь факт наличия/отсутствия мутаций. В таком случае более оптимально использование метода 8.
Метод 9 обладает достаточно высокой чувствительностью, но требует большого количества манипуляций. Кроме того, он также требует постановки нескольких параллельных реакций для каждого пациента.
Метод 10 позволяет с высокой точностью и достоверностью диагностировать мутации в гене PI3K, однако оборудование для него достаточно дорого.
Таким образом, в настоящий момент существует острая потребность в разработке способа одновременной диагностики соматических мутаций в гене ΡΙ3Κ, который бы выгодно отличался от известных решений простотой проведения анализа и низкой себестоимостью.
В настоящее время существует несколько патентов на методы анализа мутаций в гене PI3K, ассоциированных с чувствительностью опухоли к таргетной терапии. В американском патенте US 2011319477 приведены последовательности праймеров для амплификации и секвенирования экзонов ΡΙ3Κ. Данный метод характеризуется высокой надежностью и позволяет проводить поиск неизвестных мутаций, но он плохо применим для клинической диагностики так как является трудоемким и дорогостоящим. В британском патенте GB 2453173 описана методика поиска мутаций с помощью праймеров Scorpions с визуализацией ПЦР в реальном времени. Данный способ обладает рядом существенных преимуществ, таких как высокая аналитическая чувствительность и простота проведения анализа, однако не позволяет анализировать мутации E545K и Q546K.
В патентный заявке WO 2011031861 приведен способ детекции мутаций, основанный на методе ОТ-ПЦР и дальнейшим анализом результатов с помощью электрофореза в агарозном геле. Данный метод относительно дешев и не требует высокой квалификации персонала, но не удобен для рутинной клинической диагностики из-за использования электрофореза.
В американском патенте US 2011038862 предлагается анализировать мутации в гене PI3K с помощью SSCP анализа. Данный метод является устаревшим, сложным, неточным и не подлежит автоматизации.
Патентная заявка WO 2011087928 содержит описание методики определения мутаций в 12, 13, 61 кодонах гена KRAS и 542, 545 кодонах гена PI3K с помощью аллель-специфичной ПЦР в реальном времени. Данный метод в настоящее время является наиболее распространенным в лабораторной диагностике. К его главным достоинствам относятся высокая аналитическая чувствительность и быстрота анализа, однако для использования данного метода требуется дорогостоящее оборудование и достаточно высокая квалификация персонала.
В патентной заявке WO 2011131151 и американском патенте US 2011312523 описана методика обнаружения мутаций в 9 и 20 экзонах гена PI3K с помощью жидкостных чипов, данный метод обладает высокой специфичностью и чувствительностью, однако, в настоящее время отсутствуют доступные недорогие тест-системы, основанные на данном методе.
Поскольку все имеющиеся в настоящее время методы определения соматических мутаций в гене PI3K обладают теми или иными недостатками, существует реальная потребность в создании простого, недорогого и специфичного метода для выявления соматических мутаций в гене PI3K, ассоциированных с чувствительностью опухоли к таргетной терапии, с целью дальнейшего внедрения в любую стандартную клиническую лабораторию.
Такой способ обеспечивается настоящим изобретением.
Раскрытие изобретения
Сущность изобретения заключается в обеспечении способа определения соматических мутаций в гене PI3K, влияющих на чувствительность злокачественных опухолей к таргетной терапии. Способ основан на детекции точковых соматических мутаций E542K, E545K, Q546K, H1047L и H1047R.
Потенциальные эффекты влияния соматических мутаций в гене PI3K на эффективность лечения таргетными ингибиторами представлены в Табл. 1.
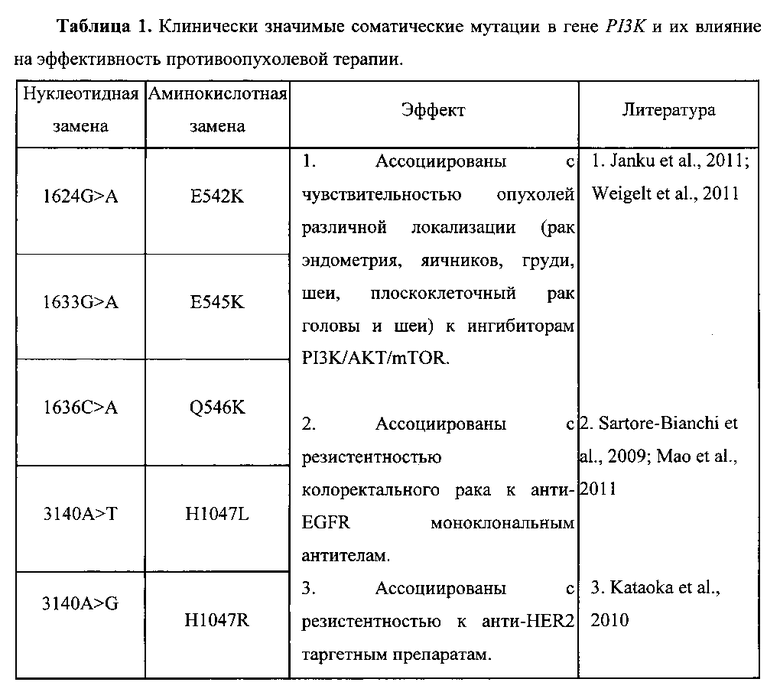
Список литературы к Таблице 1
1. PIK3CA Mutations in Patients with Advanced Cancers Treated with PI3K/AKT/mTOR Axis Inhibitors / F. Janku, A.M. Tsimberidou, I. Garrido-Laguna et al. // Mol Cancer Ther. - 2011. - Vol. 10. - P. 558-565.
2. PIK3CA mutation, but not PTEN loss of function, determines the sensitivity of breast cancer cells to mTOR inhibitory drugs / B. Weigelt, P.H. Warne, J. Downward // Oncogene. - 2011. - Vol. 30. - P. 3222-3233.
3. PIK3CA mutations in colorectal cancer are associated with clinical resistance to EGFR-targeted monoclonal antibodies / A. Sartore-Bianchi, M. Martini, F. Molinari et al. // Cancer Res. - 2009. - Vol.69. - P. 1851-1857.
4. PIK3CA exon 20 mutations as a potential biomarker for resistance to anti-EGFR monoclonal antibodies in KRAS wild-type metastatic colorectal cancer: a systematic review and meta-analysis / C. MaO, Z.Y. Yang, X.F. Hu et al. // Ann Oncol. - 2011. - doi: 10.1093/annonc/mdr464.
5. Association between gain-of-function mutations in PIK3CA and resistance to HER2-targeted agents in HER2-amplified breast cancer cell lines / Y. Kataoka, T. Mukohara, H. Shimada et al. // Ann Oncol. - 2010. - Vol. 21. - P. 255-262.
Основными аспектами данного изобретения являются LNA-блокирующая мультиплексная «гнездовая» ПЦР и биочип, содержащий набор иммобилизованных дифференцирующих олигонуклеотидов.
Первым важным аспектом изобретения являются праймеры для амплификации ДНК-локусов исследуемого гена PI3K, набор которых используется для получения изучаемых флуоресцентно меченых фрагментов ДНК в требуемом количестве. Праймеры обеспечивают избирательную амплификацию только локусов гена PI3K, избегая амплификации его псевдогена. Последовательности праймеров приведены в Табл. 2 и Перечне последовательностей (SEQ ID NO: 1-6).
Вторым важным аспектом изобретения являются LNA-олигонуклеотиды, обеспечивающие преимущественную амплификацию ДНК-локусов, содержащих соматические мутации. Преимущественная амплификация мутантных ДНК-локусов происходит в результате способности LNA-олигонуклеотидов узнавать и специфически связываться с фрагментами ДНК «дикого типа», препятствуя их амплификации. Последовательности LNA-олигонуклеотидов приведены в Табл. 3 и Перечне последовательностей (SEQ ID NO: 7-9).
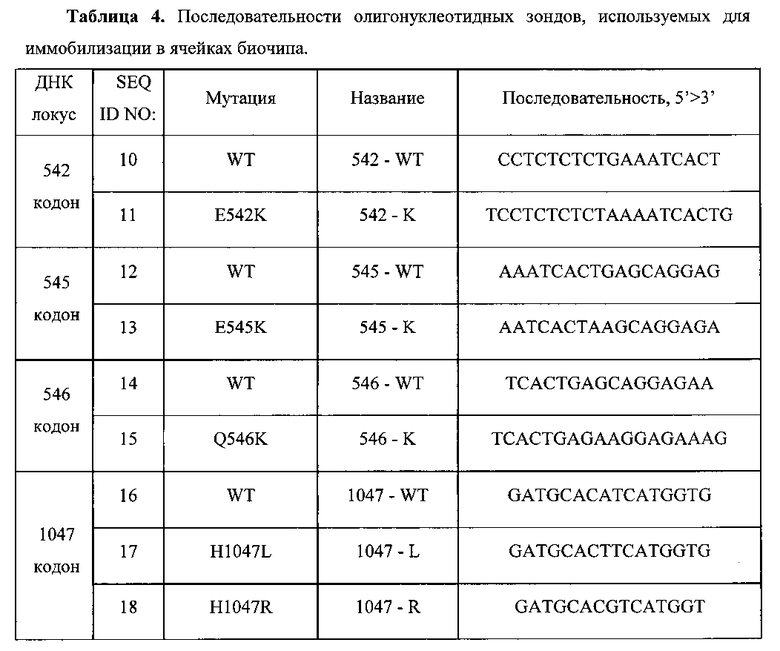
Третьим важным аспектом изобретения является биочип, содержащий набор иммобилизованных дифференцирующих олигонуклеотидов, последовательности которых приведены в Табл. 4 и Перечне последовательностей (SEQ ID NO: 10-18). Дифференцирующие олигонуклеотиды иммобилизуются в ячейках гидогелевого микрочипа, как описано в патенте [Мирзабеков А.Д., Рубина А.Ю., Паньков С. В., Перов А.Н., Чупеева В.В. (2003) Композиция для иммобилизации биологических макромолекул в гидрогелях, способ приготовления композиции, биочип, способ проведения ПЦР на биочипе. Патент RU 2206575 C2] в концентрации 200-400 мкМ. Схема расположения ячеек биочипа для анализа соматических мутаций в гене РБК приведена на Фиг. 1.
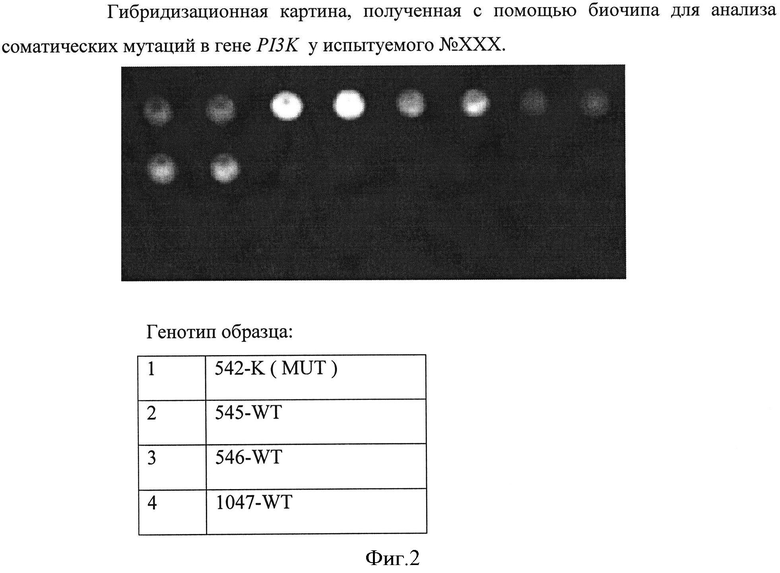
Набор праймеров и LNA-олигонуклеотидов используется на стадии предварительной амплификации ДНК-локусов посредством проведения двух параллельных мультиплексных LNΑ-блокирующих «гнездовых» ПЦР для подготовки ДНК-мишени к гибридизации на биочипе. На первом этапе, благодаря использованию LNA-блокирующей мультиплексной ПЦР проходит преимущественная амплификация ДНК-локусов, содержащих соматические мутации в гене PI3K (если образец содержит мутации). На втором этапе с амплифицированных фрагментов преимущественно нарабатывается одноцепочечная ДНК с одновременным введением флуоресцентной метки. Далее проводится гибридизация полученных флуоресцентно меченых одноцепочечных фрагментов ДНК с иммобилизованными в ячейках геля олигонуклеотидами, расположенными на пластиковой подложке. После проведения гибридизации и отмывки биочипа проводится анализ полученной флуоресцентной картины, на основании которого делается вывод о генотипе в исследуемом образце. Пример гибридизационной картины приведен на Фиг. 2.
Осуществление изобретения
Задача настоящего изобретения состоит в создании способа экспресс-анализа, позволяющего выявлять соматические мутаций E542K, E545K, Q546K, H1047L и H1047R гена ΡΙ3Κ.
Для обеспечения оптимального состава и структуры ПЦР-праймеров, они не должны образовывать между собой высокоэнергетических внутренних структур (шпильки и дуплексы) и должны обеспечивать специфичную амплификацию необходимого количества продукта и иметь примерно одинаковую температуру отжига. Для обеспечения сбалансированной эффективной амплификации исследуемых образцов также должны быть оптимизированы такие параметры как концентрация MgCl2 и праймеров в ПЦР-смеси, соотношение прямых и обратных праймеров, количество циклов амплификации на обоих этапах, время денатурации, отжига праймеров и элонгации на каждом этапе. В результате проведенной работы подобраны праймеры 1 и 2 этапа, которые позволяют осуществлять эффективную наработку выбранных фрагментов гена PI3K.

Для обеспечения оптимального состава и структуры LNA-олигонуклеотидов, используемых в протоколе LNA-блокирующей мультиплексной ПЦР, необходимы такие LNA-олигонуклеотиды, которые не образуют между собой и с праймерами высокоэнергетических внутренних структур (шпильки и дуплексы), обеспечивают специфичное связывание с целевой последовательностью и при этом подобраны таким образом, чтобы при условиях ПЦР они образовывали с ДНК-мишенью стабильные совершенные дуплексы и не образовывали несовершенные дуплексы. В результате проведенной работы подобраны LNA-олигонуклеотидов, которые позволяют осуществлять преимущественную наработку выбранных фрагментов гена PI3K, несущих соматические мутации.

Олигонуклеотиды для иммобилизации на биочипе для предсказания эффективности лечения пациентов с помощью таргетной терапии подбираются таким образом, чтобы идентифицировать все выбранные для анализа соматические мутации в гене PI3K.
Олигонуклеотиды должны соответствовать следующим критериям:
1) дискриминирующий зонд должен обладать высокой специфичностью к выбранному для анализа мутантному локусу, который представляет собой участок гена, амплифицированный с помощью ПЦР, с включенной флуоресцентной меткой;
2) вариабельный нуклеотид должен находиться в серединной области зонда, поскольку такое положение позволяет добиться большей дискриминации между совершенными и несовершенными дуплексами;
3) выбранные олигонуклеотиды не должны содержать высокостабильных вторичных структур, наличие которых может приводить к снижению эффективности гибридизации.
На биочипе для определения соматических мутаций в гене PI3K иммобилизованы 9 высокоспецифичных дифференцирующих олигонуклеотидных зондов (Табл. 4 и Перечень последовательностей (SEQ ID NO: 10-18)), структура которых обеспечивает связывание только с полностью комплементарными ДНК-мишенями, что, в свою очередь, обуславливает флуоресцентный сигнал в соответствующих им ячейках биочипа и дает наиболее четкие картины распределения гибридизационных сигналов. Неспецифическое связывание сведено к минимуму, что практически исключает ложноположительное «срабатывание» ячеек.
Так как при амплификации интересующих нас ДНК-локусов, используется LNA-блокирующая ПЦР, ячейки, содержащие зонды с последовательностью «дикого типа», может иметь низкий уровень флуоресцентного сигнала.
Приведем последовательность анализа с использованием данного метода. Наработка ПЦР-продуктов проводится в двух параллельных гнездовых реакциях. В первой пробирке происходит избирательная наработка фрагмента 9 экзона гена PI3K для анализа 542 кодона, во второй - фрагмента девятого экзона для анализ 545 и 546 кодонов и фрагмента 20 экзона для анализа 1047 кодона гена PI3K. В качестве матрицы используют опухолевую ДНК. LNA-олигонуклеотиды используются только на первом этапе ПЦР.
ПЦР может быть проведена с использованием любого вида термостабильной полимеразы (Taq-полимераза, Tth-полимераза, Tfl-полимераза, Pfu-полимераза, Vent-полимераза, DeepVent-полимераза и т.п.коммерчески доступные ферменты, выделенные из термофилов), работающей в соответствующем буфере. Для построения новой цепи в буфер добавляется смесь дНТФ (дАТФ, дГТФ, дЦТФ, дТТФ) в принятых концентрациях, при этом вместо дТТФ может быть использован дУТФ. Для проведения ПЦР могут быть использованы готовые коммерчески доступные наборы, содержащие все необходимые компоненты за исключением праймеров.
На первом этапе проходит амплификация ДНК-локусов гена PI3K, с преимущественной наработкой мутантных фрагментов (если образец содержит мутацию). Продукт первого этапа ПЦР используют в качестве матрицы на втором этапе, который проводят в реакционной смеси того же состава, но не добавляют LNA-олигонуклеотиды и добавляют избыток одного из праймеров для обеспечения избытка флуоресцентно меченого одноцепочечного ПЦР-продукта. Это позволяет получать одноцепочечный ампликон, способный к гибридизации с аллель-специфичными ДНК-зондами на биочипе. Флуоресцентное мечение проводят на втором этапе ПЦР одновременно с амплификацией одноцепочечных фрагментов ДНК посредством введения флуоресцентной метки. В качестве флуоресцентной метки используется дезоксинуклеотидтрифосфат, а именно Cy5-дУТФ, встраивающийся de novo в синтезируемую ДНК-цепь. В качестве флуоресцентной метки также может быть использован любой флуорохром без ограничения (например, FITC, Texas red, Cy-3 и т.д.), а также биотин.
Праймеры, LNA-олигонуклеотиды и олигонуклеотидные зонды синтезируют с использованием различных химических подходов, таких как фосфодиэфирный метод, гидрофосфорильный метод и т.д., при этом наиболее распространенным в настоящее время является фосфоамидитный метод синтеза. Синтез праймеров осуществляют, используя автоматические ДНК/РНК синтезаторы, например производства фирмы «Applied Biosystems» (США).
При изготовлении биочипа могут быть использованы олигонуклеотиды, несущие по 5′- или 3′-концу активную группу, обеспечивающую иммобилизацию. Модификация олигонуклеотида для введения активной группы может быть осуществлена как в автоматическом режиме при синтезе с использованием широкого спектра коммерчески доступных модификаторов, так и постсинтетически в ручном режиме. Например, при синтезе олигонуклеотидных зондов с помощью 3′-Amino-Modifier С7 CPG 500 («Glen Research», США) на 3′-конец олигонуклеотидов вводится спейсер со свободной аминогруппой, используемый для последующей иммобилизации олигонуклеотида на биочипе.
Для проведения мультиплексной LNA-блокирующей «гнездовой» ПЦР используют праймеры SEQ ID NO: 1-6 и LNA-олигонуклеотиды SEQ ID NO: 7-9, приведенные в Перечне последовательностей в Табл. 2 и в Табл. 3.
В первой реакции I этапа ПЦР используют праймеры (SEQ ID NO: 1, 2) и LNA-олигонуклеотид (SEQ ID NO: 7).
Во второй реакции I этапа ПЦР используют праймеры (SEQ ID NO: 1, 2, 4, 5) и LNA-олигонуклеотиды (SEQ ID NO: 8, 9).
В первой реакции II этапа ПЦР используют праймеры (SEQ ID NO: 1, 3).
Во второй реакции II этапа ПЦР используют праймеры (SEQ ID NO: 1, 3, 4, 6).
Далее проводится гибридизация флуоресцентно меченых фрагментов ДНК, полученных после проведения второго этапа ПЦР, с иммобилизованными в ячейках геля олигонуклеотидами, которые представляют собой участки гена PI3K и являются комплементарными последовательности «дикого типа» или последовательности, содержащей мутации.
Перед постановкой гибридизации ампликон денатурируют путем прогрева готовой гибридизационной смеси при 95°C в течение 5 минут с последующим быстрым охлаждением на льду в течение 2 минут. Гибридизация может быть проведена в любом известном специалисту в данной области гибридизационном буфере, например в гуанидиновом или SSPE-буфере. Типичное время гибридизации составляет 12-14 ч. при 37°C. Анализ генотипов и аллелей в исследуемом образце проводится с учетом расположения олигонуклеотидных зондов на биочипе, схема которого позволяет определить какие соматические мутации присутствуют в том или ином образце.
Анализируемые фрагменты ДНК образует совершенные гибридизационные дуплексы только с полностью комплементарными им олигонуклеотидами. Если последовательность анализируемой ДНК полностью комплементарна последовательности зонда, то образуется стабильный совершенный дуплекс (детектируется сигнал флуоресценции). В случае, если искомого фрагмента нет или в нем находится некомплементарное основание, то стабильного дуплекса не образуется (сигнал флуоресценции отсутствует). Дискриминацию совершенных и несовершенных дуплексов проводят после отмывки биочипа, сравнивая интенсивности сигналов флуоресценции соответствующих ячеек биочипа. Отмывка может быть проведена в любом известном в данной области техники буфере с добавлением соли (SSC, SSPE и т.п.) или в деионизованной воде, но за более короткое время [Sambrook J.F. & D. W. Russell (2001) «Molecular cloning: a laboratory manual» Cold Spring Harbor Laboratory Press].
После проведения гибридизации и отмывки биочипа проводится визуализация результатов гибридизации с помощью возбуждения флуоресценции меченого ПЦР-продукта второго этапа ПЦР, на основании которой делается вывод о генотипе в исследуемом образце. Регистрация гибридизационной картины может быть произведена с помощью любой детектирующей системы, распознающей флуоресцентный сигнал (флуоресцентный микроскоп с ПЗС-камерой, лазерный сканер, портативный анализатор биочипов и т.п. коммерчески доступные флуоресцентные анализаторы, например, портативный анализатор биочипов, снабженный ПЗС-камерой и специальным программным обеспечением, производства ООО «БИОЧИП-ИМБ» (Россия).
Биочипы могут быть изготовлены посредством последовательного нанесения на поверхность стеклянной подложки матрицы из ячеек акриламидного геля, активации ячеек и ковалентной иммобилизации в ячейках модифицированных олигонуклеотидов, несущих активные группы [Kolchinsky A, Mirzabekov A. Analysis of SNPs and other genomic variations using gel-based chips. Hum Mutat. 2002 Apr; 19 (4): 343-60. Review]. В качестве подложки помимо стекла может быть использован другой материал, в том числе металл, гибкие мембраны и пластик [Паньков С.В., Крейндлин Э.Я., Сомова О.Г., Моисеева О.В., Барский В.Е., Заседателев А.С. (2007) Применение немодифицированных полимерных материалов для изготовления подложки биочипов, биочип на их основе и способ его изготовления, способ иммобилизации гидрогелей на немодифицированных полимерных материалах. Патент RU 2309959]. Бочипы также могут быть изготовлены любыми другими известными специалисту в данной области способами [Seliger H, Hinz M, Happ E. Arrays of immobilized oligonucleotides - contributions to nucleic acids technology. Curr Pharm Biotechnol. 2003 Dec; 4 (6): 379-95].
Для изготовления биочипа в настоящем изобретении используется набор олигонуклеотидов (SEQ ID NO: 10-18), приведенных в Перечне последовательностей, а также в Табл. 4. В качестве контроля прохождения гибридизации в настоящем изобретении используют образец контрольной ДНК. Расположение конкретных олигонуклеотидных зондов на биочипе может варьироваться и определяется только удобством интерпретации результатов гибридизации.
Далее приводятся примеры, которые показывают применение способа анализа соматических мутаций в гене PI3K, ассоциированных с чувствительностью опухоли к таргетной терапии. Следует понимать, что приводимые примеры служат исключительно для иллюстрации и не предназначены для ограничения объема притязаний, выраженных в формуле изобретения. На основании настоящего описания специалист в данной области сможет легко предложить свои варианты и модификации осуществления изобретения, не отходя от общей концепции настоящего изобретения и без привлечения собственной изобретательской деятельности, так что должно быть понятно, что такие варианты и модификации также будут входить в объем притязаний настоящего изобретения.
Пример 1. Амплификация фрагмента гена PI3K, несущего мутацию Е542К методом «гнездовой» LNA-блокирующей ПЦР с целью получения флуоресцентно меченого мутантного ДНК-локуса гена в необходимом количестве
Из операционного материала, фиксированного в парафиновых блоках, образцы опухолевых тканей получали с помощью ручной микродиссекции под гистологическим контролем. Геномную ДНК выделяли с помощью набора QIAamp DNA FFPE Tissue Kit (Qiagen, Германия).
Для наработки локуса гена PI3K, несущего мутацию Е542К, использовали ПЦР-праймеры SEQ ID NO: 1-3. ПЦР проводили на приборе Dyad («Bio-Rad», США). ПЦР-смесь первого этапа общим объемом 25 мкл включала в себя: 1×ПЦР-буфер (67 мМ Трис-HCl, pH 8,6, 166 мМ (NH4)2SO4, 0,01% Тритон X-100), 1,5 мМ MgCl2, 0,2 мМ каждого из дНТФ («Силекс», Россия), 2,5 U Taq-полимеразы («Силекс», Россия), по 0,2 мкМ праймеров (SEQ ID NO: 1-2), 0,15 мкМ LNA-олигонуклеотида (SEQ ID NO: 7) и 25 нг ДНК. Амплификацию проводили по следующей схеме: денатурация при 94°C (3 мин 30 с), далее 35 циклов: 94°C (30 с), 60°C (20 с), 72°C (10 с), затем элонгация при 72°C в течение 3 мин. Смесь второго этапа ПЦР отличалась составом и концентрацией праймеров. Она содержала 0,2 мкМ прямого праймера (SEQ ID NO: 1), 2 мкМ обратного праймера (SEQ ID NO: 3) и не содержала LNA-олигонуклеотида. Для флуоресцентного мечения ПЦР-продукта второго раунда смесь содержала 0,2 нМ флуоресцентно меченого дУТФ-Су5, который встраивался в цепь в процессе амплификации. В качестве матрицы в смесь добавляли 2 мкл продукта I этапа ПЦР и проводили амплификацию по схеме: денатурация при 94°C (3 мин 30 с), далее 35 циклов:94°C (30 с), 60°C (20 с), 72°C (10 с), затем элонгация при 72°C в течение 3 мин.
Пример 2. Олигонуклеотидный биочип для соматических мутаций в гене PI3K
Олигонуклеотиды для иммобилизации на микрочипе синтезируют на автоматическом синтезаторе 394 DNA/RNA Synthesizer («Applied Biosystems», США) с использованием стандартной фосфоамидитной процедуры. 5′- или 3′-конец олигонуклеотидов содержит спейсер со свободной аминогруппой, который вводят в состав олигонуклеотида при синтезе путем использования 3′-Amino-Modifier С7 CPG 500 («Glen Research)), США). Присоединение флуоресцентной метки к олигонуклеотидам по свободной аминогруппе осуществляют в соответствии с рекомендациями производителя. Олигонуклеотиды, меченые цианиновым красителем Cy-3, очищают методом ВЭЖХ от олигонуклеотидов, не включивших флуоресцентный краситель, на колонке «Hypersil ODS» 5 мкм, 4,6×250 мм» («Sypelco Int», США).
Биочип изготовляют методом сополимеризации олигонуклеотида в акриламидном геле, как описано ранее [Паньков С.В., Крейндлин Э.Я., Сомова ОТ., Моисеева О.В., Барский В.Е., Заседателев А.С (2007) Применение немодифицированных полимерных материалов для изготовления подложки биочипов, биочип на их основе и способ его изготовления, способ иммобилизации гидрогелей на немодифицированных полимерных материалах. Патент RU 2309959], [Мирзабеков А.Д., Рубина А.Ю., Паньков СВ., Чернов Б.К. (2001) Способ иммобилизации олигонуклеотидов, содержащих непредельные группы, в полимерных гидрогелях при формировании микрочипа. Патент RU 2175972]. После изготовления биочипы проходят контроль качества нанесения, который включает в себя визуальный контроль физических размеров гелевых ячеек при помощи светового микроскопа в отраженном свете и контроль концентрации иммобилизованных олигонуклеотидов с помощью красителя Су-3 (ООО «БИОЧИП-ИМБ», Россия). Биочип содержит 9 иммобилизованных олигонуклеотидных зондов (SEQ ID NO: 10-18), список которых представлен также в Табл. 4. Ячейки наносят согласно схеме на Фиг. 1.
Пример 3. Гибридизация меченого продукта на биочипе
Реакционную смесь, полученную после проведения II этапа ПЦР, описанного в Примере 1, используют для гибридизации на биочипе. Гибридизационная смесь общим объемом 40 мкл содержала 10 мкл формамида (″Serva″, США), 10 мкл 20×SSPE (″Promega″, США) и 20 мкл амплификата. Готовую гибридизационную смесь перед гибридизацией денатурируют при 95°C в течение 5 мин, охлаждают во льду 2 мин и наносят на биочип в гибридизационную камеру объемом 40 мкл (ООО «БИОЧИП-ИМБ», Россия). Гибридизацию проводят в течение 12-14 ч при температуре 37°C. После завершения инкубации гибридизационную камеру удаляют вместе с непрореагировавшей гибридизационной смесью и проводят отмывку в 1×SSPE буфере («Promega», США) при комнатной температуре в течение 15 мин.
Пример 4. Регистрация и интерпретация результатов гибридизации
Регистрацию гибридизационной картины производят с помощью портативного анализатора биочипов, снабженного ПЗС-камерой, производимого ООО «БИОЧИП-ИМБ». Описание алгоритма автоматического анализа изображения с помощью программы Image Ware™ выходит за рамки настоящего изобретения.
Определим генотип по гибридизационной картине, представленной на Фиг. 2.
В олигонуклеотидах, соответствующих 542 кодону, флуоресцентный сигнал, наблюдается у олигонуклеотида 542-K, соответствующего соматической мутации, следовательно, анализируемый образец содержит мутацию E542K. Для локусов, соответствующих 545, 546 и 1047 кодонам, флуоресцентный сигнал наблюдается только у иммобилизованных олигонуклеотидов, соответствующих последовательности «дикого типа». Следовательно, данный образец содержит только мутацию E542K гена PI3K.
Изобретение относится к области биохимии, в частности к способу выявления соматических мутаций в гене PI3K, ответственных за чувствительность опухоли к таргетной терапии, с помощью технологии ДНК-биочипов. Определяют наличие соматических мутаций E542K, E545K, Q546K, H1047L и H1047R в гене PI3K с помощью LNA-блокирующей мультиплексной «гнездовой» ПЦР, последующей гибридизацией меченого ПЦР-продукта на биочипе, регистрацией и интерпретацией результатов. Изобретение позволяет быстро и эффективно выявить соматические мутации в гене PI3K, ассоциированные с чувствительностью опухоли к таргетной терапии. 2 з.п. ф-лы, 2 ил., 4 табл., 4 пр.
1. Способ выявления соматических мутаций E542K, E545K, Q546K, H1047L и H1047R в гене PI3K, предусматривающий следующие стадии:
(а) - амплификация фрагментов гена PI3K с помощью LNA-блокирующей мультиплексной «гнездовой» ПЦР, в которой в качестве матрицы для амплификации используется образец опухолевой ДНК:
в первой реакции I этапа ПЦР используются праймеры (SEQ ID NO: 1, 2) и LNA-олигонуклеотид (SEQ ID NO: 7);
во второй реакции I этапа ПЦР используются праймеры (SEQ ID NO: 1, 2, 4, 5) и LNA-олигонуклеотиды (SEQ ID NO: 8, 9);
в первой реакции II этапа ПЦР используются праймеры (SEQ ID NO: 1, 3);
во второй реакции II этапа ПЦР используются праймеры (SEQ ID NO: 1, 3, 4, 6); в результате образуется флуоресцентно меченый ПЦР-продукт;
(б) - обеспечение биочипа для идентификации соматических мутаций в гене PI3K, содержащего набор иммобилизованных олигонуклеотидов с последовательностями, представленными в SEQ ID NO: 10-18;
(в) - гибридизация меченого ПЦР-продукта на биочипе;
(г) - регистрация и интерпретация результатов гибридизации.
2. Способ по п. 1, в котором регистрацию результатов гибридизации проводят с помощью устройства, способного регистрировать и записывать сигналы флуоресценции.
3. Способ по п. 1, в котором для идентификации соматических мутаций в гене PI3K, амплифицированных с помощью набора праймеров и LNA-олигонуклеотидов, охарактеризованных в п.1, используется биочип, представляющий собой подложку с набором иммобилизованных на ней олигонуклеотидов с последовательностями, представленными в SEQ ID NO: 10-18.
| US 20110312523 A1, 22.12.2011 | |||
| ANG D | |||
| et al., Novel Method for PIK3CA Mutation Analysis: Locked Nucleic Acide-PCR Sequencing, The Journal of Molecular Diagnostics, May 2013, Vol | |||
| Прибор для нагревания перетягиваемых бандажей подвижного состава | 1917 |
|
SU15A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| СПОСОБ ПОЛУЧЕНИЯ КОНСЕРВОВ "МОРСКОЙ ОКУНЬ ОБЖАРЕННЫЙ В ТОМАТНОМ СОУСЕ" | 2011 |
|
RU2453173C1 |
| WO 2011087928 A1, 21.07.2011 | |||
| US 20130078631 A1, 28.03.2013 | |||
| RU 2010116772 A, 10.11.2011 | |||
| CATASUS L | |||
| et al., PIK3CA | |||
Авторы
Даты
2015-04-27—Публикация
2013-10-21—Подача