Область техники, к которой относится изобретение
Изобретение относится к молекулярной биологии, клинической генетике и фармакогеномике и обеспечивает способ диагностики генетической предрасположенности к онкологическим заболеваниям и индивидуальной чувствительности к фармацевтическим препаратам непосредственно в клиническом образце с использованием дифференцирующего олигонуклеотидного биологического микрочипа (биочипа).
Уровень техники
Для анализа генетического полиморфизма в клинической практике применяют чаще всего метод, сочетающий полимеразную цепную реакцию (ПЦР) и полиморфизм длин рестрикционных фрагментов (ПДРФ).
Существует огромное количество методов анализа генетического полиморфизма. Условно можно выделить шесть групп:
1) Ферментативные подходы:
полиморфизм длин рестрикционных фрагментов (RFLP);
полиморфизм длин аплифицированных фрагментов (AFLP);
расщепление эндонуклеазой Cleavase I (CFLP);
расщепление резольвазой (EMD);
анализы, основанные на лигазной реакции (LDR, LCR, Padlock);
инвазивное расщепление олигонуклеотидов (Invader);
случайная амплификация полиморфной ДНК (RAPD, AP-PCR);
ПЦР с прямой терминацией синтеза (DT-PCR);
аллель-специфический ПЦР (AS-PCR).
2) Химические методы;
химическое расщепление гетеродуплексов;
химическое лигирование.
3) Методы, основанные на различной электрофоретической подвижности полиморфных участков ДНК:
анализ конформации одноцепочечных фрагментов (SSCP);
гетеродуплексный анализ;
секвенирование ДНК.
4) Детекция на твердой фазе:
гибридизация на олигонуклеотидных матрицах;
оптиковолоконный ДНК-гибридизационный анализ;
элонгация иммобилизованных праймеров (минисеквенирование);
пиросиквенс.
5) Хроматографические методы:
денатурирующая ВЭЖХ.
6) Физические методы:
масс-спектрометрия;
резонансное тушение флуоресценции (FRET);
люминесценция, зависящая от локального окружения.
Однако наиболее распространенными из них являются:
1. Полиморфизм длин рестрикционных фрагментов (RFLP).
Joseph Т, Kusumakumary P, Chacko P, Abraham A, Radhakrishna Pillai M. Genetic polymorphism of CYP1A1, CYP2D6, GSTM1 and GSTT1 and susceptibility to acute lymphoblastic leukaemia in Indian children. Pediatr Blood Cancer. 2004 Oct; 43(5): 539-41.
Полиморфизм длин рестрикционных фрагментов заключается в ПЦР-амплификации интересующего фрагмента и его последующем расщеплении соответствующей рестриктазой. Благодаря простоте и надежности метод получил широкое распространение и до сих пор популярен, хотя и имеет некоторые ограничения - во-первых, он позволяет детектировать только SNPs, расположенные в сайтах рестрикции, во-вторых, годится лишь для детекции уже известных мутаций и, в-третьих, не может выявлять более одного-двух SNP одновременно. Кроме того, анализ является трудоемкой процедурой и занимает в среднем от 5 до 24 часов.
2. Аллель-специфическая ПЦР (AS-PCR).
Murata M, Shiraishi Т, Fukutome К, Watanabe M, Nagao M, Kubota Y, Ito H, Kawamura J, Yatani R. Cytochrome P4501A1 and glutathione S-transferase M1 genotypes as risk factors for prostate cancer in Japan. Jpn J Clin Oncol. 1998 Nov; 28(11): 657-60.
Аллельные варианты различаются за счет того, что 3'-концевой нуклеотид одного из праймеров в процессе ПЦР гибридизуется непосредственно с позицией SNP (т.е. если этот праймер комплементарен последовательности ДНК, то происходит наработка продукта). Метод достаточно сложен, т.к. в большой степени зависит от условий реакции и правильно выбранной последовательности праймера. Велико количество ложноположительных результатов. Кроме того, невозможно данным методом анализировать более 3-5 мутаций в одном анализе. Несмотря на все приведенные недостатки, при грамотно подобранных условиях, анализ SNP занимает всего 3-4 часа.
3. Анализ конформации одноцепочечных фрагментов (SSCP).
Broly F, Marez D, Sabbagh N, Legrand M, Millecamps S, Lo Guidice JM, Boone P, Meyer UA. An efficient strategy for detection of known and new mutations of the CYP2D6 gene using single strand conformation polymorphism analysis. Pharmacogenetics. 1995 Dec; 5(6): 373-84.
SSCP анализ включает денатурацию ПЦР-продуктов и разделение их в неденатурирующем геле. Электрофоретическая подвижность одноцепочечных фрагментов зависит от их нуклеотидной последовательности, определяющей характер вторичных и третичных структур, и меняется даже при отличии в один нуклеотид. Простота метода, умеренная стоимость и возможность использования флуоресцентной метки и капиллярного электрофореза позволяет легко автоматизировать процесс и анализировать одновременно большое количество локусов. Однако данным методом можно анализировать далеко не все SNP, особенно группы полиморфных замен, расположенные на близком расстоянии друг от друга. Кроме того, метод обладает невысокой воспроизводимостью результатов, особенно, при обнаружении de novo мутаций.
4. Macc-спектрометрия.
Nakai К, Habano W, Nakai К, Fukushima N, Fujita T, Gurwitz D. Ethnic differences of coronary artery disease-associated SNPs in two Israeli healthy populations using MALDI-TOF mass spectrometry. Life Sci. 2004 Jul 9; 75(8): 1003-10.
Метод заключается в том, что ионизированные молекулы ДНК (чаще всего специально подготовленные продукты ПЦР) отрываются от подложки методами MALDI (matrix-assisted laser desorption/ionisation) и ESI (electrospray ionisation). Они разгоняются в электрическом поле и направляются через вакуумную камеру к детектору. Время движения регистрируется. Время обратно пропорционально скорости молекулы, которая, в свою очередь, прямо пропорциональна отношению массы летящей молекулы к ее заряду. Эти параметры (отношение массы к заряду) уникальны для каждой молекулы и определяются исключительно ее нуклеотидной последовательностью, поэтому чувствительность метода чрезвычайно высока. Метод позволяет работать с нанолитрами образца. При всей привлекательности метода он является достаточно трудоемкой и дорогой процедурой (в основном из-за высокой стоимости оборудования), требует высокой квалификации сотрудников и не может быть применен далеко за пределами крупных медицинских центров мегаполисов.
5. Резонансный перенос энергии флуоресценции.
Schaeffeler E, Schwab M, Eichelbaum M, Zanger UM. CYP2D6 genotyping strategy based on gene copy number determination by TaqMan real-time PCR. Hum Mutat. 2003 Dec; 22(6): 476-85.
Данный метод позволяет следить за состоянием молекулы зонда в пробирке оптическими методами непосредственно в ходе реакции. Принцип явления заключается в том, что для детекции используют не способный удлиняться зонд, несущий флуорофор и тушитель и комплементарный средней части амплифицируемого фрагмента. Когда Taq полимераза разрушает его в ходе ПЦР реакции за счет экзонуклеазной активности, сопряженной с синтезом ДНК, флуоресцентная группа переходит в раствор и отдаляется от тушителя. Используя два аллель-специфических зонда с различными флуоресцентными метками, можно надежно различать гомо- и гетерозиготы. Основной привлекательностью метода является работа с закрытыми системами, исключающими контаминацию, и высокая скорость анализа (около 2-х часов). Минусы: высокая стоимость и невозможность анализа более 5 локусов у одного человека одновременно.
6. Мультиплексная ПЦР с последующим анализом на олигонуклеотидном микрочипе.
Wen SY, Wang H, Sun OJ, Wang SQ. Rapid detection of the known SNPs of CYP2C9 using oligonucleotide microarray. World J Gastroenterol. 2003 Jun; 9(6): 1342-6.
Данный метод предполагает предварительную мультиплексную амплификацию исследуемых фрагментов ДНК с использованием флуоресцентно меченных праймеров. В результате добавления избытка меченого праймера ПЦР достигается образование большого количества преимущественно меченого одноцепочечного продукта. Этот продукт добавляют на биочип, где происходит его гибридизация с иммобилизованными в геле олигонуклеотидными зондами. Если последовательность анализируемой ДНК полностью комплементарна последовательности зонда, то образуется стабильный дуплекс (детектируют сигнал флуоресценции), если искомого фрагмента нет, или же в нем находится некомплементарное основание, то стабильного дуплекса не образуется (сигнала флуоресценции нет). Простота метода, низкая стоимость и возможность использования флуоресцентной метки позволяет легко автоматизировать процесс. Однако данным методом можно анализировать далеко не все SNP (только точечные замены). Кроме того, метод не позволяет анализировать множество SNP одновременно.
7. Минисеквенирование на олигонуклеотидном микрочипе.
http://www.jurilab.com/default.asp?toc=66 DrugMEtTM microarray-based pharmacogenetic test processes 16 samples on one slide and includes 27 variations (SNPs) in 8 genes that code for major drug metabolising enzymes (DME).
При подготовке проб для этого метода используют предварительную амплификацию исследуемого фрагмента ДНК, содержащего SNP, и последующую гибридизацию его с зондом на биочипе. Последовательность зонда должна быть комплементарна последовательности исследуемой ДНК, причем последнее основание на 3'-конце зонда должно быть комплементарно основанию в ДНК, после которого следует вариабельный нуклеотид. Принцип детекции заключается в следующем: после гибридизации зонда с образцом в реакцию добавляют меченые дидезоксинуклеотиды и ДНК-полимеразу. Возможно присоединение только одного единственного нуклеотида к 3'-концу иммобилизованного зонда, поскольку дидезоксинуклеотиды не содержат на 3'-конце гидроксильной группы, с которой соединяется 5'-фосфатный остаток следующего нуклеотида. После отмывки чипа флуоресценция его ячеек определяется именно этим меченым дидезоксинуклеотидом. Метод позволяет с высокой степенью достоверности выявлять генотипы и анализировать более 20 мутаций одновременно, однако он достаточно дорог в настоящее время и не годится для анализа повторов и некартированных делеций и дупликаций.
Ближайшим аналогом настоящего изобретения является способ, описанный в (6). К существенным недостаткам способа можно отнести то, что не был разработан определенный подход, позволяющий "наращивать" число анализируемых генов.
Таким образом, все имеющиеся в настоящее время способы обладают теми или иными недостатками, и существует потребность в создании простого, недорогого, специфичного метода, позволяющего выявлять функционально-значимые полиморфные локусы, а также "наращивать" число анализируемых генов с целью дальнейшего внедрения в любую стандартную клиническую лабораторию.
Такой способ обеспечивается настоящим изобретением.
Раскрытие изобретения
Сущность изобретения заключается в обеспечении способа экспресс-анализа генетического полиморфизма для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям. Способ основан на проведении оригинального варианта мультиплексной полимеразной цепной реакции (ПЦР) в две стадии с последующей гибридизацией флуоресцентно меченного фрагмента ДНК на биологическом микрочипе, содержащем оригинальный набор дифференцирующих олигонуклеотидов, а также процедуры регистрации и интерпретации результатов.
Комбинированное использование блочного подхода при размещении и группировании генов, применение двухэтапной мультиплексной ПЦР и метода гибридизации на биочипах выгодно отличается от способов из уровня техники возможностью определения многих полиморфных вариантов с наименьшими затратами и наиболее специфичным способом за короткое время. Способ экономичен, не требует дорогостоящего оборудования и может быть введен в повсеместную клиническую практику. Данные, полученные с помощью способа настоящего изобретения, могут быть использованы, например, для определения генетической предрасположенности к раку легкого, раку молочной железы, раку мочевого пузыря, раку кожи, раку толстой и прямой кишки, аденоме прямой кишки, апластической анемии, лейкозам (у детей), алкогольному циррозу, бронхиальной астме, бронхиту, псориазу, мигрени. Кроме того, результаты исследования могут быть использованы для коррекции доз таких лекарственных препаратов, как, например: бета-адреноблокаторы, антидепрессанты, антипсихотропные, антиаритмические, антигипертензивные препараты, нейролептики, производные морфина, нейротрансмиттеры (допамины), анальгетики, опиаты (CYP2D6 обусловленные); антикоагулянты (варфарин), антигипертензивные препараты (лозартан), сахароснижающие препараты (глипизид), противосудорожные препараты (фенитоин, диазепам), антидепрессанты (амитриптилин, кломипрамин, имипрамин), ингибиторы протонных помп (омепразол), нестероидные противовоспалительные препараты (диклофенак, ибупрофен, пироксикам), толбутамин (CYP2C9 обусловленные); противосудорожные препараты, ингибиторы протонных помп (омепразол, лансопразол, пантопразол, эзомепразол и рабепразол), используемые при лечении кислотно-зависимых заболеваний, прогуанил и барбитураты (CYP2C19 обусловленные); бусульфан (GSTM1 и GSTT1 обусловленные); 2-аминофлюорен, р-анизид, изониазид, прокаинамид, амонафид (NAT2 обусловленные).
Важной особенностью изобретения является то, что последовательности праймеров 1-го этапа подобраны как в интронах, так и в экзонах для того, чтобы избежать амплификации других возможных гомологичных последовательностей в геноме человека. При этом флуоресцентное мечение фрагмента гена осуществляют одновременно с амплификацией при проведении второго этапа ПЦР.
Следующей особенностью изобретения является то, что идентификацию амплифицированных последовательностей осуществляют путем гибридизации полученного флуоресцентно меченного продукта амплификации с набором оригинальных олигонуклеотидных зондов, иммобилизованных на биочипе.
Одним из аспектов настоящего изобретения является способ экспресс-анализа генетического полиморфизма для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям, включающий верификацию полиморфных вариантов последовательности в клиническом образце ДНК, предусматривающий следующие стадии:
(а) - мультиплексная «гнездовая» (nested) двухэтапная полимеразная цепная реакция (ПЦР) с использованием набора праймеров, являющегося одним из отдельных аспектов данного изобретения, для амплификации фрагментов ДНК и мечения ПЦР-продукта на втором этапе ПЦР;
(б) - обеспечение биочипа, являющегося одним из отдельных аспектов данного изобретения, содержащего набор иммобилизованных олигонуклеотидов, являющийся одним из отдельных аспектов данного изобретения, для проведения гибридизации;
(в) - гибридизация амплифицированного меченого продукта на биочипе;
(г) - регистрация и интерпретация результатов гибридизации.
В одном из воплощений настоящего изобретения в качестве клинического образца используют образцы ДНК, выделенные из лимфоцитов периферической крови, клеток буккального эпителия и других материалов, полученных от обследуемого индивидуума.
В другом воплощении изобретения на втором этапе ПЦР на стадии (а) для флуоресцентного мечения фрагментов генов используют флуоресцентно меченные по 5'-концу праймеры.
В другом воплощении изобретения для обеспечения биочипа настоящего изобретения, содержащего набор иммобилизованных олигонуклеотидов настоящего изобретения, на стадии (б) используют метод фотоиндуцируемой сополимеризации в акриламидном геле.
В одном из воплощений изобретения используют условия гибридизации на стадии (в), обеспечивающие высокую степень дифференциации между совершенными и несовершенными гибридизационными дуплексами.
В еще одном из воплощений регистрацию результатов гибридизации на стадии (г) проводят с помощью портативного анализатора биочипов, снабженного ПЗС-камерой.
В другом воплощении изобретения регистрацию результатов гибридизации на стадии (г) проводят с помощью флуоресцентного микроскопа, снабженного ПЗС-камерой.
В другом воплощении изобретения интерпретацию результатов на стадии (г) проводят визуально.
В еще одном воплощении изобретения интерпретацию результатов на стадии (г) проводят с использованием программы автоматического анализа изображения.
Наконец, в еще одном воплощении интерпретированные результаты используют для целей медицинской диагностики, определения предрасположенности к различным заболеваниям и коррекции доз лекарственных препаратов.
Следующим аспектом изобретения является набор праймеров для амплификации полиморфных фрагментов генов, используемый в способе экспресс-анализа генетического полиморфизма настоящего изобретения. Последовательности праймеров приведены в Перечне последовательностей (SEQ ID NO:1-44).
Еще одним аспектом изобретения является набор олигонуклеотидов, иммобилизованных на биочипе, используемый в способе экспресс-анализа генетического полиморфизма настоящего изобретения для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям. Последовательности олигонуклеотидов приведены в Перечне последовательностей (SEQ ID NO:45-72).
Наконец, еще одним аспектом изобретения является биочип, представляющий собой подложку с набором иммобилизованных на ней олигонуклеотидов настоящего изобретения и используемый в способе экспресс-анализа генетического полиморфизма настоящего изобретения.
Перечень чертежей
Фигура 1. Схема биологического микрочипа. Ячейки темно-серого цвета соответствуют олигонуклеотидам, комплементарным последовательностям "дикого типа", черного цвета - последовательностям "мутантного типа". Ячейки светло-серого цвета соответствуют олигонуклеотидам, комплементарным последовательностям гена CYP1A1 с одной мутацией, а белого цвета - с двумя мутациями. Каждый олигонуклеотид на биочипе продублирован и имеет следующее диагностическое значение: 1 - Гаплотип 4887CYP1A1*C+4889CYP1A1*A; 2 - Гаплотип 4887CYP1A1*C+4889CYP1A1*G; 3 - Гаплотип 4887CYP1A1*A+4889CYP1A1*C; 4 - Гаплотип 4887CYP1A1*A+4889CYP1A1*G; 5 - Аллель 6235CYP1A1*T; 6 - Аллель 6235CYP1A1*C; 1 - Аллель 1934CYP2D6*G; 8 - Аллель 1934CYP2D6*A; 9 - Аллель 2637CYP2D6*A; 10 - Аллель 2637CYP2D6delA; 11 - Наличие гена GSTT1; 12 - Отсутствие гена GSTT1; 13 - Наличие гена GSTM1; 14 - Отсутствие гена GSTM1, 15 - Аллель 481NAT2*T; 16 - Аллель 481NAT2*C; 17 - Аллель 590NAT2*A; 18 - Аллель 590NAT2*G; 19 - Аллель 857NAT2*A; 20 - Аллель 857NAT2*G; 21 - Аллель 667MTHFR*C, 22 - Аллель 667MTHFR*T; 23 - Аллель 430CYP2C9*C; 24 - Аллель 430CYP2C9*T; 25 - Аллель 1075CYP2C9*A; 26 - Аллель 1075CYP2C9*C; 27 - Аллель 681CYP2C19*G; 28 - Аллель 681CYP2C19*A.
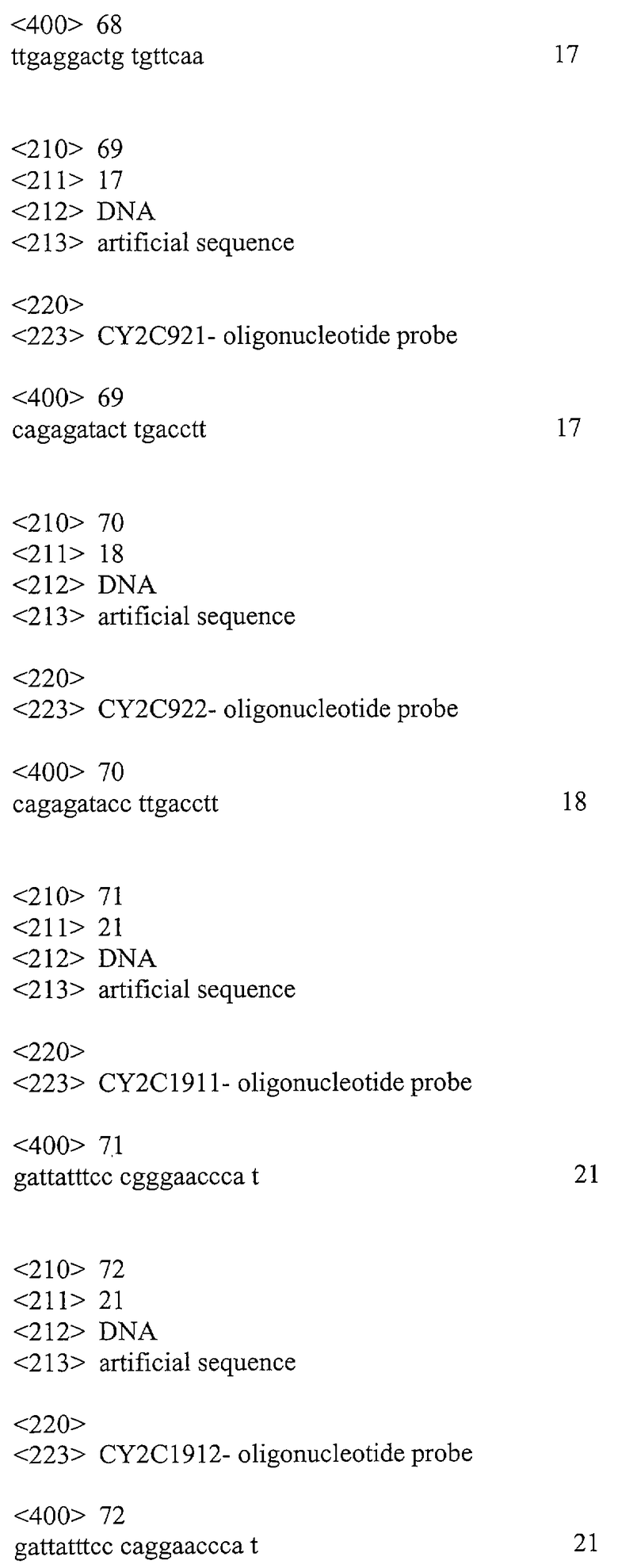
Фигура 2. Блок биочипа для выявления полиморфизма в генах CYP1A1 и CYP2D6. А - Схема и принцип расположения олигонуклеотидных зондов (указаны только вариабельные позиции). В двух верхних и двух нижних строках содержится идентичная информация (для продублированных на биочипе зондов). Б - Пример гибридизационной картины для образца с генотипом CYP1A1*2A/*1, CYP2D6*1/*1. Стрелками указан обнаруженный "мутантный" вариант. В - Диаграмма сравнения относительных сигналов флуоресценции. Значения сигналов в ячейках биочипа, содержащих С в позиции 4887 и А в позиции 4889 (первый столбец, две верхние строки), более чем в 2 раза превышают значения сигналов в ячейках, содержащих А в обеих позициях или С в позиции 4887 и G в позиции 4889 (верхние ячейки второго столбца и нижние ячейки первого столбца соответственно), а также в ячейках, содержащих двойную замену в позициях 4887 и 4889 (нижние ячейки второго столбца). Значения сигналов в верхних и нижних ячейках в третьем столбце (полиморфизм Т6235С) находятся на одном уровне, что свидетельствует о гетерозиготном состоянии этого локуса и наличии в искомой ДНК также аллеля *2А гена CYP1A1. Значения сигналов в верхних ячейках как четвертого, так и пятого столбца более чем в 2 раза превышают значения таковых в нижних ячейках, что свидетельствует об отсутствии в образце ДНК аллелей *4 и *3 гена CYP2D6.
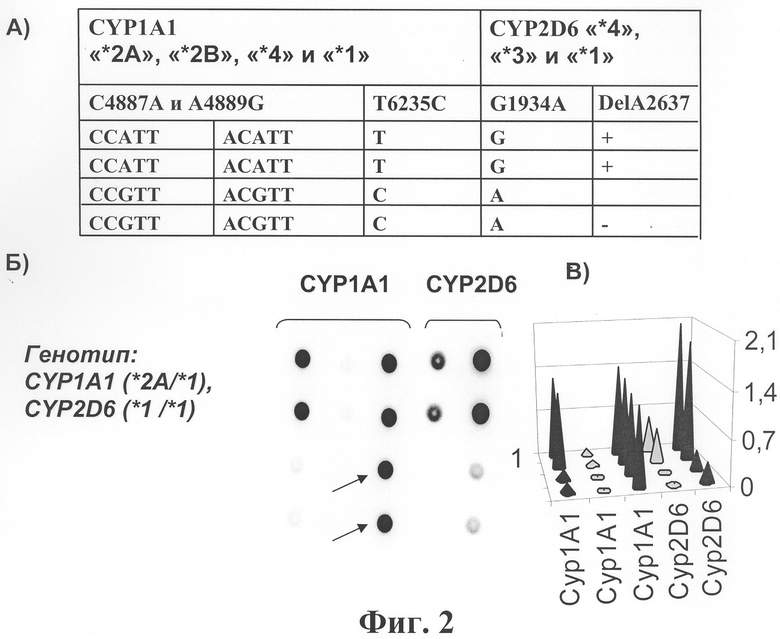
Фигура 3. Блок биочипа для выявления полиморфизма в генах GSTT1 и GSTM1. А-В названия подпунктов как на Фиг.2. На диаграмме (В) видно, что есть сигналы в верхних ячейках данного блока, и они превышают более чем в 2 раза сигналы в нижних ячейках, что свидетельствует о присутствии в образце хотя бы одной копии как гена GSTT1, так и гена GSTM1.
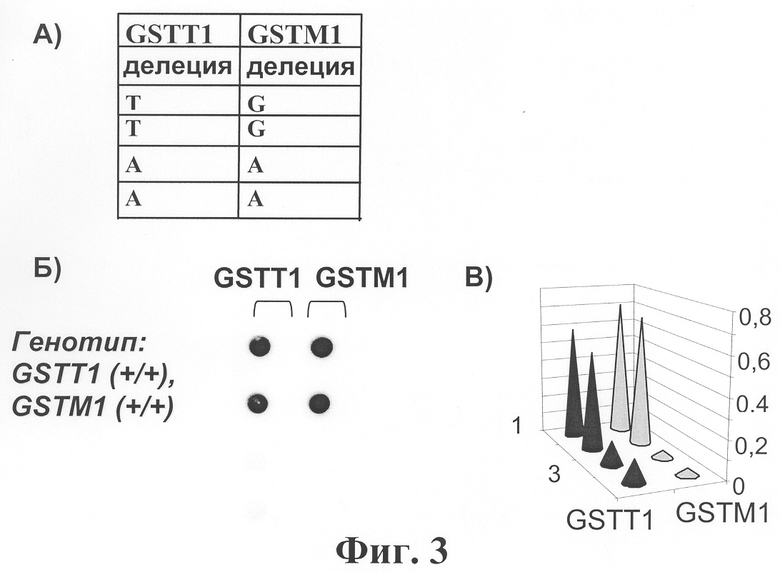
Фигура 4. Блок биочипа для выявления полиморфизма в генах NAT2 и MTHFR. А-В названия подпунктов как на Фиг.2. Сравнение значений сигналов ячеек проводили так же, как при выявлении мутаций в гене CYP2D6 блока №1.
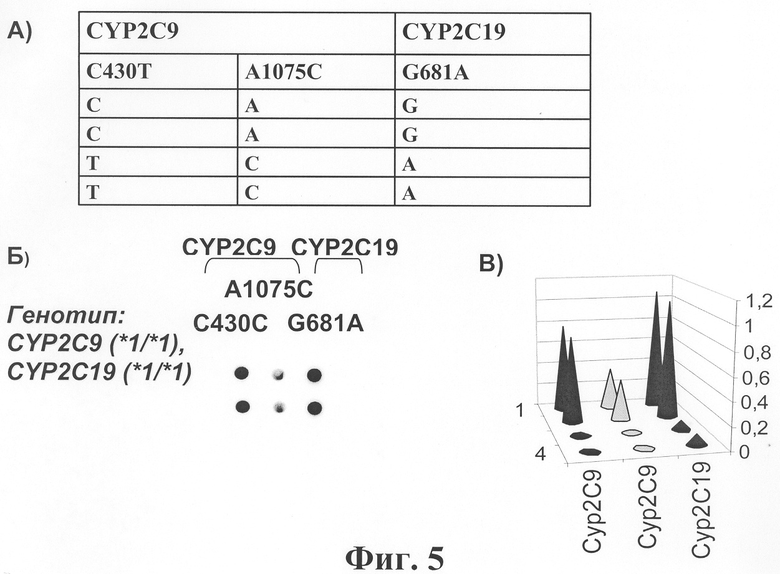
Фигура 5. Блок биочипа для выявления полиморфизма в генах CYP2C9 и CYP2C19. А-В названия подпунктов как на Фиг.2. Сравнение значений сигналов ячеек проводили так же, как при выявлении мутаций в гене CYP2D6 блока №1.
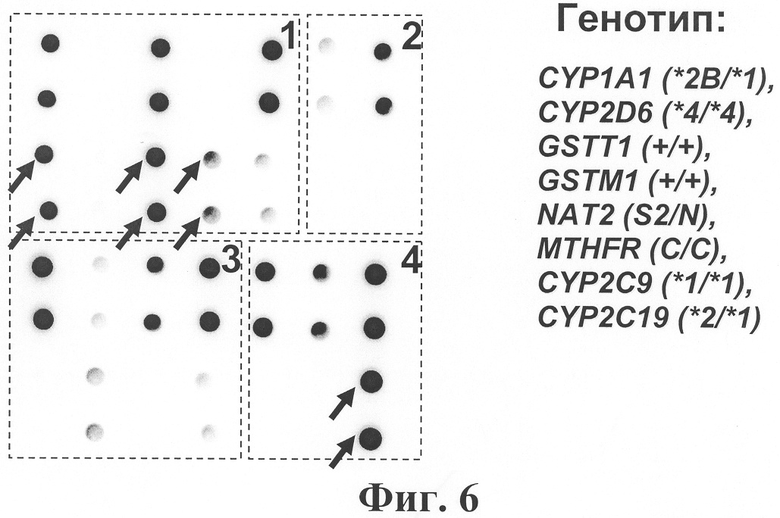
Фигура 6. Гибридизационная картина, полученная на биочипе для анализа полиморфизма по генам CYP1A1, CYP2D6, GSTM1, GSTT1, NAT2, MTHFR, CYP2C9 и CYP2C19. Стрелками указаны обнаруженные "мутантные" варианты.
Осуществление изобретения
Задача настоящего изобретения состоит в обеспечении способа экспресс-анализа генетического полиморфизма для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям в образцах ДНК, полученных от пациентов, с использованием олигонуклеотидного биочипа.
Образец ДНК используют как матрицу в мультиплексной «гнездовой» двухэтапной ПЦР. На втором этапе ПЦР получают флуоресцентно меченный амплифицированный фрагмент. Меченый ПЦР-продукт используют для дальнейшей гибридизации на биочипе, содержащем иммобилизованные олигонуклеотиды, представляющие собой участки последовательностей генов, комплементарных как последовательности "дикого типа", так и вариабельной последовательности. Анализируемый фрагмент ДНК образует совершенные гибридизационные дуплексы только с соответствующими полностью комплементарными ему олигонуклеотидами. Со всеми остальными олигонуклеотидами анализируемый фрагмент ДНК будет образовывать несовершенные дуплексы, стабильность которых существенно ниже стабильности совершенных дуплексов. Дискриминацию совершенных и несовершенных дуплексов проводят после отмывки биочипа, сравнивая интенсивности флуоресценции соответствующих ячеек биочипа. Интенсивность сигнала в случае образования совершенного дуплекса выше, чем в случае несовершенного. Используя схему расположения олигонуклеотидов на биочипе, определяют, какие варианты присутствуют в том или ином образце.
В предпочтительном воплощении способа настоящего изобретения, обеспечивающем удобство клинического применения, наличие альтернативных генетических вариантов определяют, используя образцы ДНК, выделенные из лимфоцитов периферической крови, полученных от пациента.
Полимеразная цепная реакция может быть проведена с использованием любого вида термостабильной полимеразы (например, Taq-полимеразы, фрагмента Стоффеля, Tth-полимеразы, Tfl-полимеразы, Pfu-полимеразы, Vent-полимеразы, DeepVent-полимеразы и т.п., которые коммерчески доступны от большого числа производителей), работающей в соответствующем буфере. Для построения новой цепи в буфер добавляется смесь дНТФ (дАТФ, дГТФ, дЦТФ, дТТФ) в принятых концентрациях, но вместо дТТФ может быть использован дУТФ. Для проведения ПЦР могут быть использованы готовые коммерчески доступные наборы, содержащие все необходимые компоненты за исключением праймеров.
В качестве праймеров для проведения мультиплексной «гнездовой» двухстадийной полимеразной цепной реакции используют праймеры SEQ ID NO:1-44, приведенные в Перечне последовательностей, а также в таблице 1. При этом проводят на первом этапе раздельную амплификацию с использованием праймеров с последовательностью SEQ ID NO:1-8, праймеров с последовательностью SEQ ID NO:9-12 и праймеров с последовательностью SEQ ID NO:13-18. На втором этапе раздельно проводят амплификацию с использованием праймеров с последовательностью SEQ ID NO:19-26, праймеров с последовательностью SEQ ID NO:27-30, праймеров с последовательностью SEQ ID NO:31-38 и праймеров с последовательностью SEQ ID NO:39-44. Существуют разнообразные химические подходы к синтезу олигонуклеотидных праймеров, например фосфодиэфирный метод, гидрофосфорильный метод и т.д., но наибольшее распространение в настоящее время имеет фосфорамидитный метод. Синтез праймеров осуществляют, используя автоматические ДНК/РНК синтезаторы, например (без ограничения) производства фирмы Applied Biosystems (США).
Последовательности праймеров, участвующих в мультиплексной ПЦР.
В одном из воплощений мечение ПЦР-продукта на втором этапе ПЦР проводят с помощью праймеров, несущих флуоресцентную метку на 5'-конце.
В качестве флуоресцентной метки может быть использован любой флуорохром (например, без ограничения, FITC, Texas red, Су-3, Су-5 и т.д.), а также биотин.
На второй стадии мультиплексной «гнездовой» двухэтапной ПЦР праймеры, входящие в состав одной пары, могут использоваться в эквимолярных количествах. Специалисту понятно, что образующийся при этом продукт амплификации будет по существу двухцепочечным, и для его эффективной гибридизации с олигонуклеотидами, иммобилизованными на биочипе, проводят предварительную денатурацию. Обычно денатурацию проводят непосредственно перед проведением гибридизации путем прогрева готовой гибридизационной смеси при 95°С в течение 5 мин с последующим быстрым охлаждением во льду. В предпочтительном воплощении изобретения вторую стадию мультиплексной «гнездовой» двухэтапной ПЦР проводят в асимметричном формате, т.е. флуоресцентно меченный праймер используют в молярном избытке по отношению к немеченому праймеру, например, при молярном соотношении около 10:1, соответственно. Специалисту понятно, что образующийся при этом продукт амплификации будет представлен преимущественно одноцепочечной флуоресцентно меченной ДНК. С учетом возможности образования вторичных структур для эффективной гибридизации такого продукта с олигонуклеотидами, иммобилизованными на биочипе, при необходимости также проводится его предварительная денатурация.
При подборе условий гибридизации создают условия, обеспечивающие наибольшую интенсивность флуоресценции совершенных дуплексов, специфичность взаимодействия и максимальную разницу в стабильности между совершенными и несовершенными дуплексами. Гибридизация может быть проведена в любом известном специалисту в данной области гибридизационном буфере, при необходимости содержащем реагенты, понижающие температуру плавления образуемых дуплексов и обеспечивающие специфичность взаимодействия между олигонуклеотидным зондом и ДНК-мишенью в диапазоне температур около 37°С. Это обеспечивает дополнительное удобство в использовании способа настоящего изобретения в случае клинической лаборатории, в комплект стандартного оборудования которой обычно входит термостат, установленный на температуру, например, 37°С. К таким реагентам относятся формамид, гуанидин гидрохлорид, гуанидин изотиоцианат и др. Гибридизация может проводиться и в отсутствии таких реагентов, но при более высоких температурах, соответствующих температурам плавления олигонуклеотидных зондов в используемом буфере. При подборе условий гибридизации и отмывки основное внимание должно быть обращено на специфичность прохождения гибридизации. Отмывка может быть проведена в любом известном в данной области техники буфере с добавлением соли (например, SSC, SSPE и т.п.) или в деионизованной воде, но за более короткое время (J.F.Sambrook and D.W.Russell, 2001, "Molecular cloning: a laboratory manual" Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York, v.l, p.6.50-6.62 (Protocol 10)).
Регистрация гибридизационной картины может быть произведена с помощью любой детектирующей системы, распознающей флуоресцентный сигнал (например, флуоресцентный микроскоп с ПЗС-камерой, лазерный сканер или портативный анализатор биочипов, доступные коммерчески от большого числа производителей, например портативный анализатор биочипов, производимый серийно ООО «Биочип-ИМБ», и снабженной регистрирующим устройством (например, ПЗС-камерой, видеокамерой, фотокамерой)). Анализ изображения может быть произведен как визуально, так и в автоматическом режиме с использованием специальной программы.
При изготовлении микрочипа могут быть использованы олигонуклеотиды, несущие по 5'- или 3'-концу активную группу, обеспечивающую иммобилизацию, существуют разнообразные химические подходы к синтезу олигонуклеотидов, например фосфодиэфирный метод, гидрофосфорильный метод и т.д., но наибольшее распространение в настоящее время имеет фосфорамидитный метод. Синтез олигонуклеотидов осуществляют, используя автоматические ДНК/РНК синтезаторы, например производства фирмы Applied Biosystems (США). Возможно использование широкого спектра групп для модификации иммобилизуемых олигонуклеотидов, таких как аминогруппа, тиол, гидразид, акриламидная группа, бензальдегид, тиофосфат и т.п. (Seliger H., Hinz М., Нарр Е. "Arrays of immobilized oligonucleotides - contributions to nucleic acids technology" Current Pharmaceutical Biotechnology, 2003, 4, 379-395). Модификация олигонуклеотида, имеющая целью введение активной группы, пригодной для последующей иммобилизации олигонуклеотида на биочипе, может быть осуществлена как в автоматическом режиме при синтезе с использованием широкого спектра модификаторов, которые коммерчески доступны, например, от Glen Research (USA), так и постсинтетически в ручном режиме.
Биочипы могут быть изготовлены путем создания на поверхности стеклянной подложки матрицы из ячеек полиакриламидного геля, активации ячеек и ковалентной иммобилизации в ячейках модифицированных олигонуклеотидов, несущих активные группы (Mirzabekov А. & Kolchinsky A. MAGIChip: Properties and applications in genomic studies. (2002) In Genomic Technologies: Present and Future. Eds. Galas D.J., S.J.McCormack. Caister Academic Press, pp.163-196). Также биочипы могут быть изготовлены методом химически индуцируемой или фотоиндуцируемой сополимеризации олигонуклеотида в акриламидном геле, как описано ранее (Vasiliskov V., Timofeev E., Surzhikov S., Drobyshev A., Shick V. & Mirzabekov A. 1999. Fabrication of microarray of gel-immobilized compounds on a chip by copolymerization. BioTechnique, 27, 592-606; Мирзабеков А.Д., Рубина А.Ю., Паньков С. В., Чернов Б.К. Патент на изобретение N 2175972 «Способ иммобилизации олигонуклеотидов, содержащих непредельные группы, в полимерных гидрогелях при формировании микрочипа». Приоритет от 28.12.1999). Также биочипы могут быть изготовлены любыми другими известными специалисту в данной области способами (Seliger H., Hinz М., Нарр Е. "Arrays of immobilized oligonucleotides - contributions to nucleic acids technology" Current Pharmaceutical Biotechnology, 2003, 4, 379-395), а в качестве подложки, помимо стекла, может быть использован другой материал (пластик, металл, гибкие мембраны и т.д.). Также для изготовления микрочипов могут быть использованы активированные подложки, коммерчески доступные от различных производителей (см., например, Ramakrishnan R, Dorris D, Lublinsky A, Nguyen A, Domanus M, Prokhorova A, Gieser L, Touma E, Lockner R, Tata M, Zhu X, Patterson M, Shippy R, Sendera TJ, Mazumder A. 2002. An assessment of Motorola CodeLink microarray performance for gene expression profiling applications. Nucleic Acids Res. 30: e30).
Для изготовления биочипа настоящего изобретения используют набор олигонуклеотидов SEQ ID NO:45-72, приведенных в Перечне последовательностей, а также таблице 2. В качестве контроля прохождения гибридизации в настоящем изобретении используют образец контрольной ДНК. Расположение конкретных олигонуклеотидных зондов на биочипе может варьировать и определяется только удобством для интерпретации результатов гибридизации.
Список олигонуклеотидов, иммобилизованных на микрочипе.
Ранняя диагностика и своевременная профилактика рака являются одними из самых актуальных проблем современной медицины. В структуре смертности на долю онкозаболеваний приходится около 17-20% всех случаев, и эта доля продолжает увеличиваться. Наиболее распространенными видами рака являются рак молочной железы у женщин (встречается у 5-10% всех женщин), рак легкого (встречается чаще у мужчин, от него умирают 3-4% населения) и рак прямой кишки (4-15% онкологических больных). Создание и внедрение в клиническую практику тестов на основе биочипов для определения генетической предрасположенности к развитию раков молочной железы, легкого и прямой кишки позволит разработать и внедрить комплекс профилактических мер, направленных на предотвращение этих заболеваний, а также повысить эффективность ранней, досимптоматической диагностики у лиц, выделенных в группы повышенного риска, скорректировать фармакотерапию заболеваний.
Биочип настоящего изобретения позволяет определять генетическую предрасположенность не только к раку легкого, раку молочной железы, раку мочевого пузыря, но и раку кожи, раку толстой и прямой кишки, аденоме прямой кишки, апластической анемии, лейкозам (у детей), алкогольному циррозу, бронхиальной астме, бронхиту, псориазу, мигрени.
Кроме того, результаты исследования на биочипе могут быть использованы для коррекции доз таких лекарственных препаратов, как, например: бета-адреноблокаторы, антидепрессанты, антипсихотропные, антиаритмические, нейролептики, антигипертензивные препараты, производные морфина, нейротрансмитеры (допамины), анальгетики, опиаты (CYP2D6 обусловленные); антикоагулянты (варфарин), антигипертензивные препараты (лозартан), сахароснижающие препараты (глипизид), противосудорожные препараты (фенитоин, диазепам), антидепрессанты (амитриптилин, кломипрамин, имипрамин), ингибиторы протонных помп (омепразол), нестероидные противовоспалительные препараты (диклофенак, ибупрофен, пироксикам), толбутамин (CYP2C9 обусловленные); противосудорожные препараты, ингибиторы протонных помп (омепразол, лансопразол, пантопразол, эзомепразол и рабепразол), используемые при лечении кислотно-зависимых заболеваний, прогуанил и барбитураты (CYP2C19 обусловленные); бусульфан (GSTM1 и GSTT1 обусловленные); 2-аминофлюорен, р-анизид, изониазид, прокаинамид, амонафид (NAT2 обусловленные).
Далее изобретение иллюстрируется примерами, которые демонстрируют применение способа экспресс-анализа генетического полиморфизма для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям. Следует, однако, понимать, что приводимые примеры служат исключительно для иллюстрации и не предназначены для ограничения объема притязаний, который определяется формулой изобретения.
Пример 1. Амплификация фрагментов генов методом двухэтапной «гнездовой» мультиплексной ПЦР с целью наработки флуоресцентно меченного фрагмента гибридного гена, специфичного для каждого полиморфного варианта.
1. Для проведения первого этапа готовят ПЦР смеси в трех пробирках объемом 25 мкл. Состав ПЦР-смеси: 67 мМ Трис-HCl, рН 8.6, 166 мМ (NH4)2SO4, 0,01% Тритон Х-100, 1,5 мМ MgCl2, 0,2 мМ каждого из дНТФ (Силекс, Россия), 1,5 единицы фермента Taq-полимераза фирмы «Силекс» (Россия), смесь праймеров первого раунда по 10 пмоль каждого: для пробирки №1 - SEQ ID NO:1-8, для пробирки №3 - SEQ ID NO: 9-12 и для пробирки №4 - SEQ ID NO:13-18. В каждую пробирку вносят 1 мкл образца ДНК. Всю смесь нагревают при 94°С в течение 5 мин, затем проводят 35 циклов амплификации по следующей схеме: 94°С - 30 с, 60°С - 30 с, 72°С - 1 мин. Завершающая инкубация: 72°С - 5 мин.
2. Для проведения второго этапа готовят ПЦР смеси в четырех пробирках объемом 25 мкл. На втором этапе проводят асимметричную ПЦР с праймерами второго этапа: для пробирки №1 - SEQ ID NO:19-26, для пробирки №2 - SEQ ID NO:27-30, для пробирки №3 - SEQ ID NO:31-38, для пробирки №4 - SEQ ID NO:39-44. В пробирку №2 вносят 1 мкл образца ДНК, в пробирки №1, 3 и 4 - по 1 мкл ПЦР-продукта первого этапа, соответственно. Для каждого фрагмента в реакции участвуют праймеры, меченные флуоресцентным красителем Су-5, по 50 пмоль каждого (SEQ ID NO:20, 22, 24, 26, 28, 30, 32, 34, 36, 38, 40, 42, 44) и немеченые праймеры по 5 пмоль каждого (SEQ ID NO:19, 21, 23, 25, 27, 29, 31, 33, 35, 37, 39, 41, 43), подобранные так, чтобы в каждой паре «верхний и нижний праймеры» один был меченым, а другой немеченым. Последовательности праймеров первого и второго этапа приведены также в таблице 1. Всю смесь нагревают при 94°С в течение 5 мин, затем проводят 35 циклов амплификации по следующей схеме: 94°С - 30 с, 62°С - 30 с, 72°С - 1 мин. Завершающая инкубация: 72°С - 5 мин. В результате реакции для каждого фрагмента нарабатывают преимущественно цепь одного знака (смысловая или антисмысловая), содержащую на 5'-конце флуоресцентную метку. ПЦР-анализ на наличие полиморфных вариантов проводят как несколько параллельных мультиплексных реакций.
Пример 2. Олигонуклеотидный биочип для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям.
3. Олигонуклеотиды для иммобилизации на микрочипе синтезируют на автоматическом синтезаторе 394 DNA/RNA Synthesizer (Applied Biosystems, США) с использованием стандартной фосфоамидитной процедуры. 5'- или 3'-конец олигонуклеотидов содержит спейсер со свободной аминогруппой, который вводят в состав олигонуклеотида при синтезе путем использования 3'-Amino-Modifier C7 CPG 500 (Glen Research, USA). Присоединение флуоресцентной метки к олигонуклеотидам по свободной аминогруппе осуществляют в соответствии с рекомендациями производителя. Олигонуклеотиды, меченные цианиновым красителем Су-5, очищают ВЭЖХ от олигонуклеотидов, не включивших флуоресцентный краситель, на колонке «Hypersil ODS 5 мкм, 4,6×250 мм» («Thermo Hypersil», США).
4. Биочип изготовляют методом сополимеризации олигонуклеотида в акриламидном геле, как описано ранее (Патент на изобретение N 2175972 «Способ иммобилизации олигонуклеотидов, содержащих непредельные группы, в полимерных гидрогелях при формировании микрочипа» Мирзабеков А.Д., Рубина А.Ю., Паньков С.В., Чернов Б.К. Приоритет от 28.12.1999). Биочип содержит 28 типов иммобилизованных олигонуклеотидов (SEQ ID NO:45-72), список которых представлен также в Таблице 2. На Фиг.1 представлена схема биочипа для определения фармакогенетического статуса человека и выявления генетической предрасположенности к онкологическим заболеваниям. Олигонуклеотидные пробы в составе биочипа сгруппированы в блоки. Блок №1 предназначен для выявления полиморфных вариантов генов CYP1A1 и CYP2D6 (фиг.2). В гене CYP1A1 с помощью биочипа можно анализировать полиморфные участки С4887А, A4889G и Т6235С, что позволяет выявлять две мутантные аллели этого гена: *2А (гаплотип 6235CYP1A1*C) и *4 (4887CYP1APA), а также мутантный гаплотип (аллель*2В) (4889CYP1A1*G+6235CYP1A1*C) и гаплотип дикого типа (аллель*1) (4887CYP1A1*C+4889CYP1A1*A+6235CYP1A1*T). В гене CYP2D6 можно идентифицировать полиморфные варианты G1934A и delA2637. Их определение позволяет выявлять две мутантные аллели этого гена: *4 (1934CYP2D6*A), *3 (2637CYP2D6de1A) и аллель дикого типа (*1) (1934CYP2D6*G+2637CYP2D6*A). Анализ результатов гибридизации на микрочипе и определение генотипа проводится следующим образом. При выявлении флуоресцентного сигнала во всех ячейках двух верхних строк (кроме ячеек второго столбца) считают, что образец содержит только аллели дикого типа генов CYP1A1 и CYP2D6. Аллели *2А гена CYP1A1 соответствует сигнал с двух нижних ячеек третьего столбца, аллели *4 - сигнал с двух верхних ячеек второго столбца, а аллель *2В определяют при наличии сигнала в двух нижних ячейках первого и третьего столбцов. Аллели *4 и *3 гена CYP2D6 определяют по появлению сигнала в двух нижних ячейках четвертого и пятого столбцов соответственно. На фиг.2 показан пример гибридизационной картины для образца, гетерозиготного по мутации Т6235С в гене CYP1A1 (*2А/*1) и гомозиготного по аллели дикого типа гена CYP2D6 (*1/*1). Блок №2 предназначен для обнаружения полиморфных вариантов генов GSTT1 и GSTM1 (фиг.3). С помощью чипа можно анализировать протяженные внутригенные делеции, что позволяет выявлять нуль-аллели этих генов (делеции в гомозиготе), а также находить образцы, в которых есть хотя бы одна копия гена GSTT1 или гена GSTM1 (делеции в гетерозиготе и гомозиготы дикого типа). Специфичность гибридизации ПЦР продукта с пробой подтверждают, используя в качестве пробы для сравнения олигонуклеотид с искусственно введенной заменой одного основания в центре пробы (фиг.3). В качестве внешнего контроля (прохождение ПЦР) используют сравнение с положительными контрольными образцами. На фиг.3 показаны гибридизационные картины образца, содержащего хотя бы одну копию как гена GSTT1, так и гена GSTM1 (в случае делеции в любом из генов сигнала нет как в верхних, так и в нижних ячейках). Блок №3 предназначен для выявления полиморфизма по генам NAT2 (С481Т, G590A и G857A) и MTHFR (C677T) (фиг.4). Обнаружение полиморфных участков С481Т, G590A и G857A позволяет выявлять четыре аллеля гена NAT2: S1 (481NAT2*G), S2 (590NAT2*G), S3 (857NAT2*G) и аллель дикого типа - N (481NAT2*T+590NAT2*A+857NAT2*A). В гене MTHFR анализируют полиморфный участок С667Т, что позволяет определять два аллели этого гена: аллель дикого типа 667MTHFR*C и мутантная аллель 667MTHFR*T. Сравнение значений сигналов ячеек проводят так же, как при выявлении мутаций в гене CYP2D6 блока №1. На фиг.4 приведена гибридизационная картина образца, гетерозиготного по мутации С481Т в гене NAT2 (S1/N) и C677T в гене MTHFR (С/Т). Блок №4 предназначен для обнаружения полиморфизма по генам CYP2C9 и CYP2C19 (фиг.5). Использование микрочипов позволяет анализировать полиморфные участки С430Т и А1075С в гене CYP2C9 и выявлять две мутантные аллели этого гена: *2 (430CYP2C9*T) и *3 (1075CYP2C9*C), а также аллель дикого типа, или *1 (430CYP2C9*C+1075CYP2C9*A). В гене CYP2C19 можно анализировать мутацию G681A, что позволяет определять аллель дикого типа, или *1 (681CYP2C19*G) и мутантная аллель *2 (681CYP2C19*A). Сравнение значений сигналов ячеек проводят так же, как при выявлении мутаций в гене CYP2D6 блока №1. На фиг.5 приведена гибридизационная картина образца "дикого типа", у которого не найдено соответствующих мутаций в генах CYP2C9 и CYP2C19. Разработанный биочип позволяет анализировать 14 мутаций в восьми различных генах. На фиг.6 приведен пример гибридизационной картины, включающей все блоки биочипа для образца со следующим генотипом: CYP1A1 (*2В/*1), CYP2D6 (*4/*4), GSTM1 (+/+), GSTT1 (+/+), NAT2 (S2/N), MTHFR (С/С), CYP2C9 (*1/*1), CYP2C19 (*2/*1).
Пример 3. Гибридизация меченого продукта на биочипе
5. Содержимое всех пробирок, полученных на стадии 2 Примера 1, используют для гибридизации на биочипе в буфере следующего состава: 25% формамид (Gibco BRL), 5×SSPE. Готовую гибридизационную смесь перед гибридизацией денатурируют при 95°С в течение 5 мин, охлаждают во льду и наносят на биочип под гибридизационную камеру объемом 20 мкл (Sigma, USA). Гибридизацию проводят в течение ночи (16-18 ч) при температуре 37°С. Отмывку проводят в буфере 1×SSPE при комнатной температуре в течение 10 мин.
Пример 4. Регистрация и интерпретация результатов гибридизации.
6. Регистрацию гибридизационной картины производят с помощью портативного анализатора биочипов, снабженного ПЗС-камерой, производимого ООО «Биочип-ИМБ». При визуальном анализе изображения интерпретацию результатов проводят как в примере 2. Описание алгоритма автоматического анализа изображения с помощью программы ImageWare™ выходит за рамки настоящего изобретения.
На основании настоящего описания специалист в данной области сможет легко предложить свои варианты и модификации осуществления изобретения, не отходя от изложенной выше общей концепции и без привлечения собственной изобретательской деятельности, так что должно быть понятно, что такие варианты и модификации также будут входить в объем притязаний настоящего изобретения, который определяется приводимой ниже формулой изобретения.
















Изобретение относится к области молекулярной биологии, клинической генетике и фармакогеномике и может быть использовано в медицине. Анализ генетического полиморфизма проводят путем амплификации полиморфных фрагментов генов посредством двухэтапной полимеразной цепной реакции и гибридизации полученных продуктов на биочипе с последующей регистрацией и интерпретацией результатов гибридизации. Для амплификации полиморфных фрагментов генов используют набор специфичных праймеров. В качестве биочипа на стадии гибридизации используют подложку с набором иммобилизованных на ней специфичных олигонуклеотидов. Применение изобретения обеспечивает выявление функционально-значимых полиморфных локусов. 4 н. и 8 з.п. ф-лы, 2 табл., 6 ил.
(а) - амплификацию полиморфных фрагментов генов «гнездовой» двухэтапной полимеразной цепной реакцией (ПЦР), где в качестве матрицы для амплификации используют клинический образец ДНК, амплификацию на первом этапе проводят с помощью праймеров с последовательностями, представленными в SEQ ID NO:1-18, а на втором - с помощью праймеров с последовательностями, представленными в SEQ ID NO:19-44, причем на втором этапе получают меченный ПЦР-продукт;
(б) - обеспечение биочипа, содержащего набор иммобилизованных олигонуклеотидов с последовательностями, представленными в SEQ ID NO:45-72;
(в) - гибридизацию меченного амплифицированного продукта на биочипе;
(г) - регистрацию и интерпретацию результатов гибридизации.
| US 6183963, 06.02.2001 | |||
| WO 03014387, 20.02.2003 | |||
| WO 03018837, 06.03.2003 | |||
| Способ получения производных циклического амина или их кислотно-аддитивных солей | 1988 |
|
SU1561823A3 |
| СПОСОБ ФИЛЬТРОВАНИЯ ЖИДКОСТЕЙ И ФИЛЬТР ДЛЯ ОЧИСТКИ ЖИДКОСТИ | 1988 |
|
RU2072882C1 |
| Газонаполненный электровакуумный прибор | 1959 |
|
SU129262A1 |
| JP 2004525448, 19.08.2004 | |||
| US 2002022225, 21.02.2002. | |||
Авторы
Даты
2007-07-27—Публикация
2005-10-11—Подача