Область техники, к которой относится изобретение
Изобретение относится к области генетики, молекулярной биологии и медицины и касается метода диагностики, определения прогноза и тактики ведения пациентов с глиальными опухолями головного мозга (глиомы) и опухолями хрящевой ткани (хондроидные опухоли) посредством анализа клинически значимых соматических мутаций в генах IDH1 и IDH2 с помощью комбинации LNA-блокирующей ПЦР и последующей гибридизации с олигонуклеотидным биологическим микрочипом (биочипом).
Уровень техники
Изоцитратдегидрогеназы 1 и 2 (IDH1 и IDH2) являются важными ферментами клеточного дыхания в цикле трикарбоновых кислот.Соматические мутации в генах IDH1 и IDH2 являются ранним событием в онкогенезе и распространены в таких опухолях, как, диффузные глиомы, хондросаркома, острый миелоидный лейкоз [Liu, X.; Ling, Z-Q. Role of isocitrate dehydrogenase 1/2 (IDH 1/2) gene mutations in human tumors. Histol Histopathol. 2015 Oct;30(10):l 155-60. doi: 10.14670/HH-11-643; Watanabe T, Nobusawa S, Kleihues P, Ohgaki H. IDH1 mutations are early events in the development of astrocytomas and oligodendrogliomas. Am J Pathol. 2009 Apr;174(4):l 149-53. doi: 10.2353/ajpath.2009.080958]. Повторяющиеся точечные мутации возникают в аминокислотных остатках аргинина гена IDH1 (аргинин 132 (R132)) либо гена IDH2 (аргинин 172 (R172) или аргинин 140 (R140)). Мутации генов ID1/2 относятся к активирующим мутациям и приводят к увеличению конверсии а-кетоглутората (α-KG) в D-2-гидроксиглутарат (D-2HG). D-2HG является онкогенным метаболитом, способным увеличивать пролиферацию клеток и метастазирование, что может быть связано с нарушением процессов метилирования ДНК и активацией рецептора фактора роста эндотелия сосудов VEGFR [Xu W, Yang Н, Liu Y, Yang Y, Wang P, Kim SH, Ito S, Yang C, Wang P, Xiao MT, Liu LX, Jiang WQ, Liu J, Zhang JY, Wang B, Frye S, Zhang Y, Xu YH, Lei QY, Guan KL, Zhao SM, Xiong Y. Oncometabolite 2-hydroxyglutarate is a competitive inhibitor of a-ketoglutarate-dependent dioxygenases. Cancer Cell. 2011 Jan 18;19(l):17-30. doi: 10.1016/j.ccr.2010.12.014; Carbonneau M, M Gagne L, Lalonde ME, Germain MA, Motorina A, Guiot MC, Secco B, Vincent EE, Tumber A, Hulea L, Bergeman J, Oppeimann U, Jones RG, Laplante M, Topisirovic I, Petrecca K, Huot ME, Mallette FA. The oncometabolite 2-hydroxyglutarate activates the mTOR signalling pathway. Nat Commun. 2016 Sep 14;7:12700. doi: 10.1038/ncommsl2700].
Мутации в генах IDH1/2 являются важным диагностическим маркером в опухолях хондроидного ряда [Amary MF, Bacsi К, Maggiani F, Damato S, Halai D, Berisha F, Pollock R, O'Donnell P, Grigoriadis A, Diss T, Eskandarpour M, Presneau N, Hogendoorn PC, Futreal A, Tirabosco R, Flanagan AM. IDH1 and IDH2 mutations are frequent events in central chondrosarcoma and central and periosteal chondromas but not in other mesenchymal tumours. J Pathol. 2011 Jul;224(3):334-43. doi: 10.1002/path.2913]. Также мутации IDH1/2 играют важную роль в развитии глиальных опухолей и служат важным диагностическим и прогностическим маркером диффузных глиом 2-4 степени злокачественности [Murugan АК, Alzahrani AS. Isocitrate Dehydrogenase IDH1 and IDH2 Mutations in Human Cancer: Prognostic Implications for Gliomas. Br J Biomed Sci. 2022 Jan 31;79:10208. doi: 10.3389/bjbs.2021.10208]. В настоящее время показана эффективность терапии ингибиторами IDH1 при остром миелоидном лейкозе и холангиокарциноме [Uson Junior PLS, Borad MJ. Clinical Utility of Ivosidenib in the Treatment of IDH1-Mutant Cholangiocarcinoma: Evidence To Date. Cancer Manag Res. 2023 Sep 18;15:1025-1031. doi: 10.2147/CMAR.S326060].
Известно большое количество методов анализа уникальной нуклеотидной последовательности ДНК человека с целью выявления соматических мутаций. Для детекции мутаций в генах ID1/2 наиболее часто используют следующие методы.
(1) Секвенирование по Сенгеру [Agarwal S, Sharma MC, Jha P, Pathak P, Suri V, Sarkar C, Chosdol K, Suri A, Kale SS, Mahapatra AK, Jha P. Comparative study of IDH1 mutations in gliomas by immunohistochemistry and DNA sequencing. Neuro Oncol. 2013 Jun;15(6):718-26. doi: 10.1093/neuonc/not015]. Метод (1) является высоко информативным и предоставляет полную информацию о последовательности ДНК в исследуемом локусе, позволяет определять мутации de novo. Однако он отличается низкой производительностью и недостаточно чувствителен для определения соматических мутаций в образцах опухоли, так как обладает низкой чувствительностью (образец должен содержать более 25-30% мутантной ДНК).
(2) Пиросеквенирование [Arita Н, Narita Y, Matsushita Y, Fukushima S, Yoshida A, Takami H, Miyakita Y, Ohno M, Shibui S, Ichimura K. Development of a robust and sensitive pyrosequencing assay for the detection of IDH1/2 mutations in gliomas. Brain Tumor Pathol. 2015 Jan;32(l):22-30. doi: 10.1007/s10014-014-0186-0].
Метод (2) является более чувствительным в сравнении с секвенированием по Сэнгеру (5% мутантной ДНК), однако он требует специализированного дорогостоящего оборудования и квалифицированного персонала.
(3) ПНР с последующим анализом кривых плавления высокого разрешения [Patel КР, Barkoh ВА, Chen Z, Ma D, Reddy N, Medeiros LJ, Luthra R. Diagnostic testing for IDH1 and IDH2 variants in acute myeloid leukemia an algorithmic approach using high-resolution melting curve analysis. J Mol Diagn. 2011 Nov;13(6):678-86. doi: 10.1016/j.jmoldx.2011.06.004].
(4) ПЦР с TaqMan-зондом и последующим анализом кривых плавления высокого разрешения [Табаков Д.В., Катаргин А.Н., Строганова A.M., Сендерович А.И., Насхлеташвили Д.Р., Киселева Н.П. Мутации изоцитратдегидрогеназ 1 и 2 и метилирование гена MGMT в глиомах. Успехи молекулярной онкологии. 2017;4(1), 53-59. doi: 10.17650/2313-805Х-2017-4-1-53-59].
Методы (3) и (4) являются достаточно чувствительными, способны определять около 5% мутантной ДНК, однако дают косвенную информацию о наличии мутации в образце, для идентификации мутации требуется дополнительный анализ (секвенирование по Сэнгеру).
(5) Аллель-специфическая ПЦР [Husain A, Mishra S, Siddiqui МН, Husain N. Detection of IDH1 Mutation in cfDNA and Tissue of Adult Diffuse Glioma with Allele-Specific qPCR. Asian Рас J Cancer Prev. 2023 Mar l;24(3):961-968. doi: 10.31557/APJCP.2023.24.3.961].
(6) Различные комбинированные подходы на основе аллель-специфической ПЦР, в том числе, с использованием LNA-нуклеотидов [Као HW, Kuo MC, Huang YJ, Chang Н, Ни SF, Huang CF, Hung YS, Lin TL, Ou CW, Lien MY, Wu JH, Chen CC, Shih LY. Measurable Residual Disease Monitoring by Locked Nucleic Acid Quantitative Real-Time PCR Assay for IDH1/2 Mutation in Adult AML. Cancers (Basel). 2022 Dec 15;14(24):6205. doi: 10.3390/cancersl4246205].
(7) Блокирование амплификации ДНК дикого типа в комбинации с методом ARMS (амплификация рефрактерной мутационной системы) [Catteau A, Girardi Н, Monville F, Poggionovo С, Carpentier S, Frayssinet V, Voss J, Jenkins R, Boisselier В, Mokhtari К, Sanson М, Peyro-Saint-Paul Н, Giannini С.A new sensitive PCR assay for one-step detection of 12 IDH1/2 mutations in glioma. Acta Neuropathol Commun. 2014 Jun 2;2:58. doi: 10.1186/2051-5960-2-58]. Методы (5) - (7) обладают высокой чувствительностью, дешевизной и простотой выполнения, однако требу.п постановки независимых параллельных ПЦР-реакций, соответствующих количеству анализируемых однонуклеотидных замен, что делает их трудоемкими при определении большого количества нуклеотидных замен.
(8) Массовое параллельное секвенирование [Sporikova Z, Slavkovsky R, Tuckova L, Kalita O, Megova Houdova M, Ehrmann J, Hajduch M, Hrabalek L, Vaverka M. IDH1/2 Mutations in Patients With Diffuse Gliomas: A Single Centre Retrospective Massively Parallel Sequencing Analysis. Appl Immunohistochem Mol Morphol. 2022 Mar 1;30(3):178-183.doi: 10.1097/РА1.0000000000000997].
Метод (8) является наиболее перспективным для одновременного анализа большого количество генетических локусов, также обладает высокой аналитической чувствительностью. Однако, его использование в рутинной лабораторной диагностике остается ограниченным из-за высокой стоимости оборудования и расходных материалов, а также необходимости привлечения высококвалифицированного персонала.
(9) Цифровая капельная ПЦР [Grassi S, Guerrini F, Ciabatti E, Puccetti R, Salehzadeh S, Metelli MR, Di Vita A, Domenichini C, Caracciolo F, Orciuolo E, Pelosini M, Mazzantini E, Rossi P, Mazziotta F, Petrini M, Galimberti S. Digital Droplet PCR is a Specific and Sensitive Tool for Detecting IDH2 Mutations in Acute Myeloid LeuKemia Patients. Cancers (Basel). 2020 Jun 30;12(7):1738. doi: 10.3390/cancers12071738].
Метод (9) обладает очень высокой чувствительностью (0,1% мутантной ДНК) и активно используется для анализа соматических мутаций в циркулирующей опухолевой ДНК. Для его применения требуется дорогостоящее оборудование и реактивы, а также высококвалифицированный персонал.
В настоящее время имеется ряд патентов, в которых раскрываются методы анализа мутаций в генах IDH1 и IDH2, имеющих диагностическое и прогностическое значение при различных видах злокачественных новообразований.
В патенте WO 2021206666 A1 "A Method for Intraoperatively or Postoperatively Detecting IDH1 and IDH2 gene Mutations in Glioma Tumors and Primers Used in This Method" (Турция, дата публикации 2021-10-14) исследуемые фрагменты генов IDH1 и IDH2 амплифицируются с помощью метода ARMS-ПЦР. Используют специально подобранные обратные праймеры, модифицированные на 3'-конце, для высокоспецифичной амплификации только мутантных последовательностей ДНК. Метод имеет высокую чувствительность, прост в дизайне и может быть реализован в течение 2 часов в одной пробирке. Однако, в этом случае определяется только присутствие мутантной ДНК в образце, но нет возможности определить тип мутации. Разработанная система позволяет выявлять ограниченное количество мутаций (3 мутации в гене IDH1 и 2 мутации в гене IDH2), дальнейшее увеличение числа анализируемых мишеней может приводить к снижению специфичности анализа.
В патенте WO 2021091803 А1 "IDH Mutation Detection Kit and Method ThereOf" (Тайвань, дата публикации 2021-05-14) описана комбинация ПЦР со специфичными для каждой мутации праймерами и последующей гибридизацией в растворе с детектирующими зондами, закрепленными на магнитных шариках. Магнитные шарики содержат также баркод для идентификации зонда, связавшегося с ДНК-мишенью. Данный метод позволяет идентифицировать большое количество мутаций в генах IDH1 и IDH2, но является многоэтапным и достаточно громоздким. Необходимо приготовление баркодированных магнитных частиц для связывания с зондами. Для мечения ДНК-мишени используется биотин, который далее связывается с коньюгатом стрептавидин-фикоэритрин, для регистрации флуоресцентного сигнала и считывания баркода необходимо использовать специализированное оборудование.
Метод, описанный в патенте WO 2016032947 A1 "Methods for Rapid and Sensitive Detection of Hotspot Mutations" (США, дата публикации 2016-03-03), использует количественную ПЦР с праймерами, содержащими на 3'-конце LNA-нуклеотиды, что повышает специфичность взаимодействия праймера и ДНК-мишени. Описана детекция мутаций в кодоне R132 гена IDH1 (R132H) и в кодоне R172 гена IDH2 (R172K) в комбинации с мутациями в гене TERT, что имеет важное значение в диагностике глиом и определении степени злокачественности опухоли. Метод описывает одновременный анализ ограниченного числа мутаций. Аналогичный подход представлен в китайском патенте CN 106795563 В "Method for the Rapid and Sensitive Detection of Hot Spot Mutations" (Китай, дата публикации 2021-08-06).
В другом китайском патенте CN 102732633 В "Detection Primer for Human IDH (Isocitrate Dehydrogenase) Gene Mutation and Reagent Kit" (Китай, дата публикации 2014-02-26) представлен набор реагентов для проведения ПЦР в реальном времени с последующим анализом кривых плавления высокого разрешения (High Resolution Melting, HRM) для дискриминации между «диким типом» и мутацией в кодонах R132 гена IDH1 и R172 гена IDH2. Использование анализа кривых плавления высокого разрешения не позволяет определять тип нуклеотидной замены, для ижентификации мутаций необходимо использовать дополнительные методы (секвенирование по Сэнгеру).
Таким образом, создание новых, более эффективных и доступных методов анализа мутаций в генах IDH1 и IDH2 остается актуальной задачей.
Раскрытие сущности изобретения
Технический результат заключается в возможности проводить детекцию 5 соматических мутаций R132H (c.395G>A), R132S (с.394С>А), R132G (c.394С>G), R132L (c.395G>T), R132C (с.394С>Т) в гене IDH1; и 2 мутаций R172K (c.515G>A), R172S(c.516G>C), в гене IDH2, в том числе, при низкой представленности мутантного аллеля (1-2%). Мутации в генах IDH1 и IDH2 являются важными диагностическими и прогностическими маркерами для целого ряда злокачественных новообразований (диффузные глиомы, хондроидные опухоли, острый миелоидный лейкоз).
Основными признаками данного изобретения являются наборы праймеров и LNA-зондов для проведения LNA-блокирующей «гнездной» ПЦР и набор иммобилизованных дифференцирующих олигонуклеотидов для иммобилизации на биочипе.
Первым важным признаком изобретения является набор праймеров для амплификации целевых локусов генов IDH1 и IDH2, который используется для получения изучаемых флуоресцентно меченых фрагментов ДНК в требуемом количестве. Последовательности праймеров приведены в Перечне последовательностей (Таблица 1, SEQ ID NO: 1 - SEQ ID NO: 8).
Вторым важным признаком изобретения является набор LNA-олигонуклеотидов, обеспечивающих преимущественную амплификацию ДНК-локусов, которые содержат соматические мутации. LNA-олигонуклеотиды обладают способностью специфически связываться с фрагментами ДНК «дикого типа» и препятствовать их амплификации. Последовательности LNA-олигонуклеотидов приведены в Перечне последовательностей (Таблица 2, SEQ ID NO: 9 - SEQ ID NO: 10).
Третьим важным признаком изобретения является биологический микрочип (биочип), содержащий набор иммобилизованных дифференцирующих олигонуклеотидов, последовательности которых приведены в Перечне последовательностей (Таблица 3, SEQ ID NO: 11 - SEQ ID NO: 19). Дифференцирующие олигонуклеотиды в концентрации 200-400 мкМ иммобилизуются в гидрогелевых ячейках биочипа, как описано в патенте RU 2206575 «Композиция для иммобилизации биологических макромолекул в гидрогелях, способ приготовления композиции, биочип, способ проведения ПЦР на биочипе» (Россия, дата публикации 2003-06-20). Схема расположения ячеек биочипа для анализа соматических мутаций в генах IDH1 и IDH2 приведена на Фигуре 1.
Одним из важных аспектов изобретения является использование наборов праймеров и LNA-олигонуклеотидов, представленных в Таблице 1 и Таблице 2, соответственно, которые необходимы для проведения двух параллельных LNA-блокирующих «гнездных» ПЦР при наработке ДНК-мишеней для гибридизации на биочипе. На первом этапе, благодаря использованию LNA- блокирующей ПЦР проходит преимущественная амплификация ДНК-локусов, содержащих соматические мутации (при их наличии в образце). На втором этапе с двухцепочечных ампликонов нарабатывается одноцепочечная ДНК, в которую в ходе ПЦР включается флуоресцентная метка.
Еще одним важным аспектом изобретения является гибридизация полученных флуоресцентно меченых одноцепочечных фрагментов ДНК с иммобилизованными в гидрогелевых ячейках биочипа олигонуклеотидами.
Следующим аспектом изобретения является отмывки биочипа и анализ полученной флуоресцентной картины, на основании которого делается вывод о генотипе в исследуемом образце.
Краткое описание фигур и таблиц
Фигура 1. Схема биочипа, используемая для анализа соматических мутаций в генах IDH1 и IDH2. В угловых позициях нанесены ячейки (М), содержащие флуоресцентный краситель Су5, светящийся перманентно, для ориентировки и контроля интенсивности свечения.
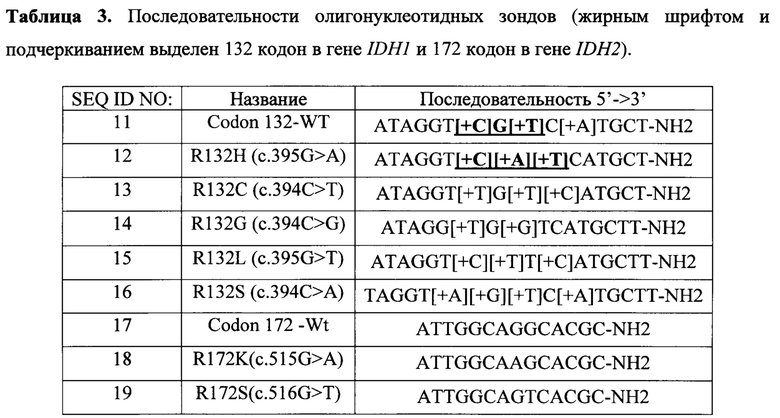
Фигура 2. Гибридизационные картины, полученные с помощью биочипа для анализа соматических мутаций в генах IDH1 и IDH2 в образцах хондроидных опухолей №1 (А) и №2 (Б). (А) - Образец №1 содержит мутацию R132G в гене IDH1, (Б) - Образец №2 содержит мутацию R172K в гене IDH2.
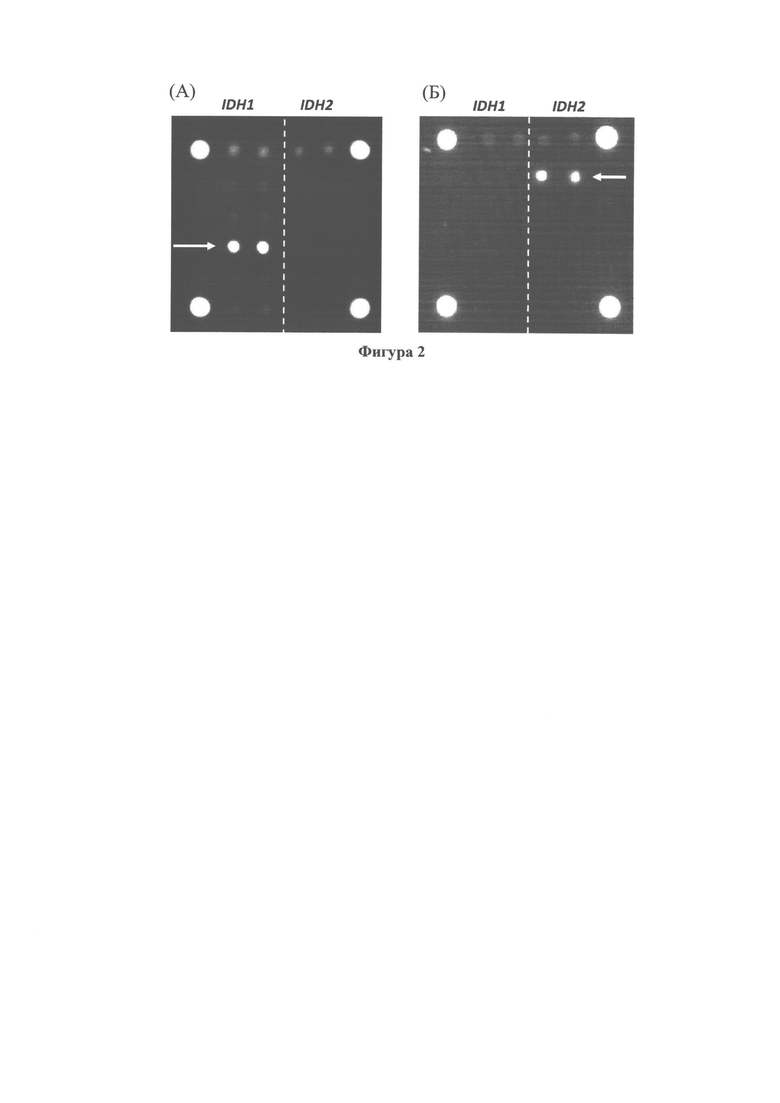
Фигура 3. Детекция мутации R132C в гене IDH1 в образце №3 при низком содержании мутантного алелля (не более 2%). Вверху - гибридизационные картины, внизу - нормированные значения сигналов флуоресценции. (А) - 0 μМ LNA-олигонуклеотида в реакции ПЦР, в отсутствие блокирования амплификации ДНК «дикого типа» мутация не выявляется; (Б) - при добавлении 0,1 μМ LNA-олигонуклеотида в образце выявляется мутация R132C в гене IDH1.
Таблица 1. Последовательности праймеров I и II этапа для проведения «гнездной» ПЦР.
Таблица 2. Последовательности LNA- олигонуклеотидов для блокирования амплификации ДНК дикого типа (LNA=[+А], [+Т], [+С], [+G], Р - фосфатная группа). На 3'- конце LNA-олигонуклеотидов находится фосфатная группа (Р).
Таблица 3. Последовательности олигонуклеотидных зондов (жирным шрифтом и подчеркиванием выделен 132 кодон в гене IDH1 и 172 кодон в гене IDH2).
Осуществление изобретения
Данное изобретение представляет собой анализ соматических мутаций в генах 1DH1 и IDH2 в образцах опухоли для осуществления которого необходимо разработать набор специфических синтетических олигонуклеотидов.
Для проведения амплификации фрагментов генов IDH1 и IDH2 были сконструированы праймеры 1 и 2 этапа для двух параллельных реакций «гнездной» ПЦР, проходящих при одинаковой температуре. Были оптимизированы условия реакции (концентрация ионов Mg++, праймеров, количество циклов амплификации на обоих этапах, время элонгации, денатурации и отжига праймеров на каждом этапе). Подобранные праймеры 1 и 2 этапа позволяют проводить эффективную наработку выбранных фрагментов генов IDH1 и IDH2 (Перечень последовательностей, Таблица 1).
Для обеспечения эффективного блокирования амплификации ДНК «дикого типа» были сконструированы LNA-олигонуклеотиды, которые не образуют между собой и с праймерами высокоэнергетических вторичных структур (шпильки и димеры), в то же время обеспечивают специфичное и высокоэнергетическое связывание с целевой последовательностью ДНК «дикого типа», что препятствует ее амплификации. Подобранные LNA-олигонуклеотиды позволяют осуществлять преимущественную наработку выбранных фрагментов генов IDH1 и IDH2, несущих соматические мутации (Перечень последовательностей, Таблица 2).
Олигонуклеотиды для иммобилизации на биочипе подбираются таким образом, чтобы идентифицировать все выбранные для анализа соматические мутации в генах IDH1 и IDH2. На биочипе для определения соматических мутаций в генах IDH1 и IDH2 иммобилизируют 9 высокоспецифичных дифференцирующих олигонуклеотидных зондов (Перечень последовательностей, Таблица 3), структура которых обеспечивает специфичное связывание с полностью комплементарными ДНК-мишенями, что обуславливает накопление флуоресцентного сигнала в соответствующих ячейках биочипа. Для повышения специфичности связывания зонда и мишени в состав иммобилизованного олигонуклеотидного зонда также вводят LNA-нуклеотиды, что позволяет свести к минимуму образование ложноположительных сигналов в ячейках биочипа.
Так как амплификация последовательностей ДНК «дикого типа» блокируется, ячейки, содержащие зонды с последовательностью «дикого типа», могут иметь низкий уровень флуоресцентного сигнала.
Приведем последовательность анализа с использованием данных наборов синтетических олигонуклеотидов. Наработка ПЦР-продуктов проводится в двух параллельных реакциях в два этапа. В первой пробирке происходит избирательная амплификация фрагмента гена IDH1, во второй - фрагмента гена IDH2. В качестве матрицы используют ДНК, выделенную их глиальных клеток опухолей головного мозга (глиомы) или клеток опухоли хрящевой ткани (хондроидные опухоли). LNA-олигонуклеотиды добавляют в реакционную смесь только на первом этапе ПЦР.
ПЦР может быть проведена с использованием любого вида термостабильной полимеразы, не имеющей 5'>3'экзонуклеазной активности (например, Hot Start Taq ДНК полимераза, ООО «СибЭнзим», Россия). Для построения новой цепи в буфер добавляется смесь дНТФ (дАТФ, дГТФ, дЦТФ, дТТФ) в необходимых концентрациях, при этом вместо дТТФ может быть использован дУТФ. Для проведения ПЦР также могут быть использованы готовые коммерчески доступные наборы, содержащие все необходимые компоненты за исключением праймеров.
На первом этапе проходит амплификация локусов генов IDH1 и IDH2, с преимущественной наработкой мутантной последовательности (если образец содержит мутацию). Продукт первого этапа ПЦР используют в качестве матрицы на втором этапе асимметричной ПЦР, используя реакционную смесь того же состава, но без добавления LNA- олигонуклеотидов. Также на втором этапе ПЦР, одновременно с амплификацией одноцепочечных фрагментов, проводят флуоресцентное мечение посредством введения в ДНК флуоресцентной метки. В качестве флуоресцентной метки используется дезоксинуклеотидтрифосфат с ковалентно присоединенным флуорохромом, а именно Су5-дУТФ, который встраивается в синтезируемую ДНК-цепь. В качестве флуоресцентной метки также может быть использован любой флуорохром без ограничения (например, FITC, Texas red, Су-3, Су-7), а также биотин. Это позволяет получать одноцепочечный ампликон, способный к гибридизации с аллель-специфичными ДНК-зондами на биочипе.
Праймеры, LNA-олигонуклеотиды и олигонуклеотидные зонды для иммобилизации синтезируют с использованием различных химических подходов (фосфодиэфирный метод, гидрофосфорильный метод и т.д.), при этом наиболее распространенным в настоящее время является фосфоамидитный метод синтеза. Синтез праймеров осуществляют, используя автоматические ДНК/РНК синтезаторы, например производства фирмы «Applied Biosystems» (США).
При изготовлении биочипа могут быть использованы олигонуклеотиды, несущие на 5'- или 3'-конце активную группу, обеспечивающую иммобилизацию. Модификация олигонуклеотида для введения активной группы может быть осуществлена как в автоматическом режиме при синтезе с использованием широкого спектра коммерчески доступных модификаторов, так и постсинтетически в ручном режиме. Например, при синтезе олигонуклеотидных зондов с помощью 3'-Amino-Modifier С7 CPG 500 («Glen Research», США) на З'-конец олигонуклеотидов вводится спейсер со свободной аминогруппой, используемый для последующей иммобилизации олигонуклеотида на биочипе.
Для проведения мультиплексной LNA-блокирующей «гнездной» ПЦР используют праймеры SEQ ID NO: 1 - SEQ ID NO: 8 и LNA-олигонуклеотиды SEQ ID NO: 9 и SEQ ID NO: 10, приведенные в Перечнях последовательностей.
В первой реакции I этапа ПЦР используют праймеры, представленные последовательностями SEQ ID NO: 1 и SEQ ID NO: 2, и LNA-олигонуклеотид с последовательностью SEQ ID NO: 9.
Во второй реакции I этапа ПЦР используют праймеры, представленные последовательностями SEQ ID NO: 5 и SEQ ID NO: 6, и LNA-олигонуклеотид с последовательностью SEQ ID NO: 10.
В первой реакции II этапа ПЦР используют праймеры с последовательностями SEQ ID NO: 3 и SEQ ID NO: 4.
Во второй реакции II этапа ПЦР используют праймеры с последовательностями SEQ ID NO: 7 и SEQ ID NO: 8.
Далее проводится гибридизация флуоресцентно меченых фрагментов ДНК, полученных после проведения второго этапа ПЦР, с иммобилизованными в гидрогелевых ячейках олигонуклеотидами, которые представляют собой короткие фрагменты генов IDH1 и IDH2 длиной 14-16 и.о. и являются комплементарными последовательности «дикого типа» или последовательности, содержащей мутации.
Гибридизация ПЦР-продукта с олигонуклеотидными зондами на биочипе может быть проведена в любом гибридизационном буфере, например, в SSPE-буфере с формамидом или буфере с гуанидином. Гибридизацию проводят 8-14 ч при 37°С. Отмывка биочипа после проведения гибридизации может быть проведена в любом известном в данной области техники буфере (SSC, SSPE и т.п.) или в дистиллированной воде.
Если последовательность анализируемой ДНК полностью комплементарна последовательности иммобилизованного зонда, то образуется стабильный совершенный дуплекс (яркий сигнал флуоресценции). В случае если хотя бы одно основание не комплементарно, то стабильного дуплекса не образуется и сигнал флуоресценции в ячейке отсутствует.Дискриминацию совершенных и несовершенных дуплексов проводят после отмывки биочипа, сравнивая интенсивности сигналов флуоресценции соответствующих ячеек биочипа.
Регистрация гибридизационной картины может быть произведена с помощью любой детектирующей системы, распознающей флуоресцентный сигнал (флуоресцентный микроскоп с ПЗС-камерой, лазерный сканер, портативный анализатор биочипов и т.п.коммерчески доступные флуоресцентные анализаторы, например, портативный анализатор биочипов, снабженный ПЗС-камерой и специальным программным обеспечением, производства ООО «БИОЧИП-ИМБ» (Россия). Анализ генотипа исследуемого образца проводится с учетом расположения олигонуклеотидных зондов на биочипе, схема которого позволяет определить, какие соматические мутации присутствуют в том или ином образце.
Биочипы могут быть изготовлены посредством последовательного нанесения на поверхность стеклянной подложки матрицы из ячеек полиакриламидного геля, активации ячеек и ковалентной иммобилизации в ячейках модифицированных олигонуклеотидов, несущих активные группы [Kolchinsky A, Mirzabekov A. Analysis of SNPs and other genomic variations using gel-based chips. Hum Mutat. 2002 Apr;19(4):343-60. doi: 10.1002/humu. 10077]. В качестве подложки, помимо стекла, может быть использован любой другой материал, в том числе металл, гибкие мембраны и пластик [Патент RU 2309959 «Применение немодифицированных полимерных материалов для изготовления подложки биочипов, биочип на их основе и способ его изготовления, способ иммобилизации гидрогелей на немодифицированных полимерных материалах», дата публикации 2007-11-10]. Биочипы также могут быть изготовлены любыми другими известными специалисту в данной области способами [Seliger Н, Hinz М, Нарр Е. Arrays of immobilized oligonucleotides--contributions to nucleic acids technology. Curr Pharm Biotechnol. 2003 Dec;4(6):379-95. doi: 10.2174/1389201033377454].
Для изготовления биочипа в настоящем изобретении используется набор олигонуклеотидных зондов с последовательностями SEQ ID NO: 11 - SEQ ID NO: 19, приведенными в Перечне последовательностей. Расположение иммобилизованных олигонуклеотидов на биочипе может варьировать и определяется только удобством интерпретации результатов гибридизации.
Далее приводятся примеры, которые показывают применение способа анализа соматических мутаций в генах IDH1 и IDH2. Приведенные примеры следует рассматривать в качестве иллюстрации, а не ограничения. Квалифицированный специалист сможет оценить, что варианты осуществления данного изобретения не ограничены примерами, приведенными в данном документе.
Пример 1. Амплификация фрагментов генов IDH1 и IDH2 методом «гнездной» LNA-блокирующей ПЦР с целью получения флуоресцентно меченого ПЦР-продукта в необходимом количестве.
Образцы опухолевых тканей получают с помощью микротома из операционного материала, фиксированного в парафиновых блоках. Геномную ДНК выделяют с помощью набора QIAamp DNA FFPE Tissue Kit (Qiagen, Hilden, Германия) в соответствии с протоколом производителя.
Амплификацию локусов генов IDH1 и IDH2 проводят в двух параллельных реакциях. Первая реакция содержит праймеры и LNA-олигонуклеотиды - SEQ ID NO:
1, SEQ ID NO: 2, SEQ ID NO: 9; вторая реакция - SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 10. ПЦР проводят на приборе T100 («Bio-Rad», США). ПЦР - смесь I этапа общим объемом 25 мкл включает в себя: 1 x ПЦР-буфер (67 мМ Трис- HCl, рН 8.6, 166 мМ (NH4)2S04, 0,01% Тритон Х-100), 2.5 мМ MgCl2, 0.2 мМ каждого из дНТФ («Силекс», Россия), по 0.2 мкМ праймеров, 0.1 мкМ LNA-олигонуклеотидов и 10-20 нг ДНК (оптимальная концентрация LNA-олигонуклеотида зависит от концентрации геномной ДНК в образце). Амплификацию проводят по следующей схеме: денатурация при 94°С (3 мин 30 с), далее 35 циклов: 94°С (30 с), 62°С (30 с), 72°С (30 с), затем элонгация при 72°С в течение 3 мин. Смесь II этапа ПЦР отличается составом и концентрацией праймеров. Она содержит по 0.2 мкМ прямых праймеров (первая реакция: SEQ ID NO: 3; вторая реакция: SEQ ID NO: 7), и по 2 мкМ обратных праймеров (первая реакция: SEQ ID NO: 4; вторая реакция: SEQ ID NO: 8) и не содержит LNA-олигонуклеотидов. Для флуоресцентного мечения ПЦР-продукта добавляют 0.2 нМ флуоресцентно меченого Су5-дУТФ, который встраивается в цепь в процессе амплификации. В качестве матрицы в смесь добавляют 2 мкл продукта I этапа ПЦР и проводят амплификацию по схеме: денатурация при 94°С (3 мин 30 с), далее 35 циклов: 94°С (30 с), 62°С (30 с), 72°С (30 с), затем элонгация при 72°С в течение 3 мин.
Пример 2. Олигонуклеотидный биочип для анализа соматических мутаций в генах IDH1 и IDH2.
Олигонуклеотидные зонды для иммобилизации на биологическом микрочипе синтезируют на автоматическом синтезаторе 394 DNA/RNA Synthesizer («Applied Biosystems», США) с использованием стандартной фосфоамидитной процедуры: на 3'-конец олигонуклеотидов вводят спейсер со свободной аминогруппой, используя метод 3'-Amino-Modifier С7 CPG 500 («Glen Research)), США).
Биочип изготавливают методом сополимеризации олигонуклеотида с компонентами акриламидного геля (патент RU 2309959 «Применение немодифицированных полимерных материалов для изготовления подложки биочипов, биочип на их основе и способ его изготовления, способ иммобилизации гидрогелей на немодифицированных полимерных материалах», дата публикации 2007-11-10 и RU 2175972 «Способ иммобилизации олигонуклеотидов, содержащих непредельные группы, в полимерных гидрогелях при формировании микрочипа», дата публикации 2001-11-20). Биочип содержит 9 иммобилизованных олигонуклеотидных зондов (SEQ ID NO: 11 - SEQ ID NO: 19). Ячейки наносят на пластиковую подложку биочипа согласно схеме, представленной на Фигуре 1.
Пример 3. Гибридизация меченого продукта на биочипе.
Реакционную смесь, полученную после проведения II этапа ПЦР, описанного в Примере 1, используют для гибридизации на биочипе. Гибридизационная смесь общим объемом 40 мкл содержит 10 мкл формамида ("Serva", США), 10 мкл 20xSSPE ("Promega", США), 10 мкл амплификата из первой реакции, 10 мкл амплификата из второй реакции. Полученную смесь наносят на биочип и оставляют на 12-14 ч при температуре 37°С. После проведения гибридизации биочип отмывают в 1× SSPE в течение 10 минут при комнатной температуре.
Пример 4. Регистрация и интерпретация результатов гибридизации.
Регистрацию гибридизационной картины проводят с помощью портативного анализатора биочипов, снабженного ПЗС-камерой (ООО «Биочип-ИМБ»). Описание алгоритма автоматического анализа изображения с помощью программы ImageWare™ выходит за рамки настоящего изобретения.
Определение генотипа проводят по гибридизационным картинам, представленным на Фигуре 2. На Фигуре 2(A), в ячейках биочипа, соответствующих 132 кодону гена IDH1, наблюдается интенсивный флуоресцентный сигнал в ячейках, соответствующих соматической мутации R132G (c.394C>G). Ячейки биочипа, соответствующие «дикому типу» (WT) данного локуса, обладают более низким уровнем флуоресценции. На Фигуре 2(Б), в ячейках биочипа, соответствующих 172 кодону гена IDH2, наблюдается интенсивный флуоресцентный сигнал в ячейках, соответствующих соматической мутации R172K (c.515G>A). Ячейки биочипа, соответствующие «дикому типу» данного локуса, обладают более низким уровнем флуоресценции.
Пример 5. Детекция соматической мутации в гене IDH1 при низкой представленности мутантного аллеля.
Геномную ДНК из образца опухоли с мутацией R132C в гене IDH1 (частота мутантного аллеля составляла 50%) смешивают с геномной опухолевой ДНК «дикого типа» и получают образцы ДНК, содержащие 10%, 5%, 2%, 1% мутантной ДНК. Далее проводят амплификацию образцов ДНК, как описано в Примере 1, с добавлением и без добавления LNA-олигонуклеотидов. Полученные ПЦР-продукты гибридизируют на биочипе (как описано в Примере 2 и Примере3), проводят регистрацию и интерпретацию результатов (аналогично Примеру 4). Гибридизационные картины для образца, содержащего 2% мутантной ДНК, полученные без добавления (А) и с добавлением (Б) LNA-олигонуклеотидов, представлены на Фигуре 3.
Таким образом, основными преимуществами настоящего изобретения являются простота проведения анализа, экономичность, высокая чувствительность и точность, а также возможность одновременного анализа всех клинически значимых мутаций в генах IDH1/2. В настоящий момент представлен анализ 5 мутаций в гене IDH1 и 2 мутаций в гене IDH2, однако этот список может быть расширен без существенного повышения сложности анализа и стоимости, а также увеличения трудозатрат.


Информация о перечнях последовательностей: Версия DTD: V1_3
Название файла: Способ высокочувствительного анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом (биочипом).xml
Название программного обеспечения: WIPO Sequence
Версия программного обеспечения: 2.3.0
Дата производства: 2024-05-22 Общая информация:
Текущая заявка / Ведомство ИС: RU
Текущая заявка / Номер дела заявителя: 20.05.2024
Имя заявителя: Федеральное государственное бюджетное учреждение науки Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук (ИМБ РАН)
Имя заявителя / Язык: ru
Имя заявителя / Имя латиницей: Engelhardt Institute of Molecular Biology, Russian Academy of Sciences
Название изобретения: Способ высокочувствительного анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом (биочипом) (ru)
Общее количество последовательностей: 19
Последовательности:
Номер последовательности (ID): 1
Длина: 22
Тип молекулы: DNA
Характеристики Местоположение/Квалификаторы:






--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ
высокочувствительного анализа соматических мутаций в генах IDH1 и
IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с
биологическим микрочипом (биочипом).xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-05-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>20.05.2024</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт молекулярной биологии им. В.А.
Энгельгардта Российской академии наук (ИМБ РАН)</ApplicantName>
<ApplicantNameLatin>Engelhardt Institute of Molecular Biology,
Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ высокочувствительного
анализа соматических мутаций в генах IDH1 и IDH2 с использованием
LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом
(биочипом)</InventionTitle>
<SequenceTotalQuantity>19</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH1-F1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccattatctgcaaaaatatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q6">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH1-R1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacatacaagttggaaatttctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH1-F2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaatatcccccggcttgtga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q12">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH1-R2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcaaaatcacattattgccaacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH2-F1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggactgtcttccgggag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q18">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH2-R1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacaccactgccatcttttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH2-F2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaacatcccacgcctagtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q24">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH2-R2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agaggatggctaggcgagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH1-LNA-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q28">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequence consists of locked nucleic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catgacgacct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>IDH2-LNA-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q32">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequence consists of locked nucleic
acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcctgccaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>Codon 132-WT</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q38">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataggtcgtcatgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q42">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R132H (c.395G>A)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>7..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q44">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataggtcatcatgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q47">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R132C (c.394C>T)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q48">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>9..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q49">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q50">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataggttgtcatgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q53">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R132G (c.394C>G)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q54">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q55">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q56">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataggtggtcatgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q59">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R132L (c.395G>T)</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R132L (c.395G>T)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>7..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q61">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q62">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q63">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataggtcttcatgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q65">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q66">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R132S (c.394C>A)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>6..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q67">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q68">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>locked nucleic acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q69">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taggtagtcatgctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q71">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q72">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>Codon 172 -Wt</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q73">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attggcaggcacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q75">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q76">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R172K(c.515G>A)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q77">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attggcaagcacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q79">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q80">
<INSDQualifier_name>standard_name</INSDQualifier_name>
<INSDQualifier_value>R172S(c.516G>T)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_difference</INSDFeature_key>
<INSDFeature_location>>14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q81">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>The 3' end of the sequence contains
an amino group</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attggcagtcacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ получения мышиной модели для изучения миодистрофии Дюшенна и вариантов ее терапии | 2023 |
|
RU2815936C1 |
| Способ сполиготипирования микобактерий туберкулезного комплекса с использованием ДНК-амплификации в иммобилизованной фазе и биологический микрочип для его осуществления | 2023 |
|
RU2807998C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |
| АНТИСМЫСЛОВЫЕ ОЛИГОНУКЛЕОТИДЫ, НАЦЕЛЕННЫЕ НА ГЕН CAV3.1, И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2839721C2 |
| Набор праймеров для выявления возбудителей бактериальной пневмонии человека методом мультиплексной рекомбиназной полимеразной амплификации | 2023 |
|
RU2813995C1 |
| СПОСОБ ИДЕНТИФИКАЦИИ ОДНОНУКЛЕОТИДНЫХ ПОЛИМОРФИЗМОВ В МНОГОКОПИЙНОМ ЛОКУСЕ ГЕНОМА МЕТОДОМ ПЕТЛЕВОЙ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ С ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2022 |
|
RU2809184C1 |
| СПОСОБ ГЕНОТИПИРОВАНИЯ ИЗОЛЯТОВ NEISSERIA GONORRHOEAE НА БИОЛОГИЧЕСКОМ МИКРОЧИПЕ | 2023 |
|
RU2816767C1 |
| Наборы олигонуклеотидов для выявления ДНК бактерии Helicobacter pylori в клиническом материале методом полимеразной цепной реакции в режиме реального времени | 2024 |
|
RU2839157C1 |
| Микроорганизм, имеющий ДНК-связывающий регулятор транскрипции семейства LacI с ослабленной активностью, и способ получения L-глутаминовой кислоты с его использованием | 2022 |
|
RU2833333C2 |
| Тест-система для определения статуса генов IDH ½ в тканях глиальных опухолей | 2023 |
|
RU2823028C1 |

Изобретение относится к области молекулярной биологии. Описан способ анализа соматических мутаций в генах IDH1 и IDH2 с использованием LNA-блокирующей ПЦР и гибридизации с биологическим микрочипом, включающий применение олигонуклеотидов с последовательностями SEQ ID NO: 1 - SEQ ID NO: 19. Также описан набор указанных синтетических олигонуклеотидов. Технический результат заключается в возможности проводить детекцию 5 соматических мутаций R132H (c.395G>A), R132S (с.394С>А), R132G (с.394С>G), R132L (c.395G>T), R132C (с.394С>Т) в гене IDH1; и 2 мутаций R172K (c.515G>A), R172S(c.516G>C) в гене IDH2, в том числе при низкой представленности мутантного аллеля (1-2%). 2 н.п. ф-лы, 3 ил., 3 табл., 5 пр.
1. Способ анализа соматических мутаций в генах IDH1 и IDH2, включающий следующие стадии:
(а) - амплификацию фрагментов генов IDH1 и IDH2 с помощью LNA-блокирующей «гнездной» ПЦР, в которой в качестве матрицы для амплификации используется образец опухолевой ДНК, при этом в первой реакции I этапа ПЦР используются праймеры, представленные последовательностями SEQ ID NO: 1 и SEQ ID NO: 2 и LNA-олигонуклеотидом с последовательностью SEQ ID NO: 9, а во второй реакции I этапа ПЦР используются праймеры, представленные последовательностями SEQ ID NO: 5 и SEQ ID NO: 6 и LNA-олигонуклеотидом с последовательностью SEQ ID NO: 10; в первой реакции II этапа ПЦР используются праймеры, представленные последовательностями SEQ ID NO: 3 и SEQ ID NO: 4; во второй реакции II этапа ПЦР используются праймеры, представленные последовательностями SEQ ID NO: 7 и SEQ ID NO: 8, в результате образуется флуоресцентно-меченый ПЦР-продукт;
(б) - обеспечение биочипа для идентификации соматических мутаций в генах IDH1 и IDH2, в гидрогелевых ячейках которого содержится набор иммобилизованных олигонуклеотидов, представленных последовательностями SEQ ID NO: 11 - SEQ ID NO: 19;
(в) - гибридизацию меченого ПЦР-продукта в гидрогелевых ячейках биочипа;
(г) - регистрацию и интерпретацию результатов гибридизации.
2. Набор синтетических олигонуклеотидов, представленный последовательностями SEQ ID NO: 1 - SEQ ID NO: 19, которые используются для выявления и идентификации соматических мутаций в генах IDH1 и IDH2 в способе по п. 1.
| KR 1020120127679 A, 23.11.2012 | |||
| СПОСОБ ДИАГНОСТИКИ И МОНИТОРИРОВАНИЯ ТЕЧЕНИЯ ЦЕРЕБРАЛЬНЫХ ГЛИОМ | 2017 |
|
RU2656182C1 |
| Rossetto M | |||
| et al | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Revue neurologique, 2011, Т.167(10), С | |||
| Приспособление для укладки спичечных или т.п. коробок рядами | 1925 |
|
SU699A1 |
Авторы
Даты
2025-04-29—Публикация
2024-06-04—Подача