Область техники, к которой относится изобретение
Настоящее изобретение относится в целом к генетическому контролю нападения видов насекомых-вредителей, в частности, предотвращению и/или борьбе с нападением вредителей на растения. Более конкретно, настоящее изобретение относится к подавлению экспрессии генов-мишеней у видов насекомых-вредителей с помощью интерферирующих молекул рибонуклеиновой кислоты (РНК). Также предусматриваются трансгенные растения, которые (i) экспрессируют или способны экспрессировать интерферирующие РНК по настоящему изобретению и (ii) устойчивы к нападению видов насекомых-вредителей.
Предпосылки создания изобретения
Существует множество видов насекомых-вредителей, которые могут поражать или нападать на большое множество местообитаний и организмов-хозяев. Насекомые-вредители включают разнообразные видов из отрядов насекомых Hemiptera (клопы), Coleoptera (жуки), Siphonaptera (блохи), Dichyoptera (тараканы и богомолы), Lepidoptera (моли и бабочки), Orthoptera (например, кузнечики) и Diptera (настоящие мухи). Нападение вредителей может приводить к значительному ущербу. Насекомые-вредители, которые нападают на виды растений, вызывают особые проблемы в сельском хозяйстве, поскольку они могут приводить к серьезному повреждению сельскохозяйственных культур и значительно снижать урожайность растений. Большое разнообразие различных типов растений, включая товарные культуры, такие как рис, хлопчатник, соя, картофель и кукуруза, чувствительны к нападению вредителей.
Традиционно, нападение насекомых-вредителей предотвращают или контролируют посредством применения химических пестицидов. Однако данные химические вещества не всегда пригодны для применения в обработке сельскохозяйственных культур, так как они могут являться токсичными для других видов и могут наносить значительный ущерб окружающей среде. За последние десятилетия исследователи разработали более экологически безопасные способы контроля нападения вредителей. Например, применялись микроорганизмы, такие как бактерии Bacillus thuringiensis, которые в естественных условиях экспрессируют белки, токсичные для насекомых-вредителей. Ученые также выделили гены, кодирующие эти инсектицидные белки, и использовали их для создания трансгенных культур, устойчивых к насекомым-вредителям, например, растения кукурузы и хлопчатника, генетически сконструированные для продуцирования белков семейства Cry.
Хотя бактериальные токсины являлись весьма успешными в борьбе с определенными типами вредителей, они не эффективны против всех видов вредителей. Исследователи, следовательно, искали другие, более целенаправленные подходы к борьбе с вредителями и, в частности, к РНК-интерференции или 'сайленсингу генов' в качестве средств для борьбы с вредителями на генетическом уровне.
РНК-интерференция или 'РНКi' является процессом, посредством которого экспрессия генов в отношении клетки или всего организма подавляется последовательность - специфическим образом. РНКi является в настоящее время общепризнанным методом в данной области для ингибирования или подавления экспрессии генов у самых разнообразных организмов, включая организмы-вредители, такие как грибы, нематоды и насекомые. Кроме того, предыдущие исследования показали, что подавление экспрессии генов-мишеней у видов насекомых-вредителей можно использовать в качестве средств для борьбы с нападением вредителей.
В WO 2007/074405 описаны способы ингибирования экспрессии генов-мишеней у беспозвоночных вредителей, включая колорадского жука. Кроме того, В WO 2009/091864 описаны композиции и способы подавления генов-мишеней у видов насекомых-вредителей, включая вредителей из рода Lygus.
Хотя применение РНКi для подавления экспрессии генов у видов вредителей в данной области известно, успех данного метода в применении в качестве меры борьбы с вредителями зависит от выбора наиболее подходящих генов-мишеней, а именно тех, у которых потеря функции приводит к значительному нарушению жизненно важного биологического процесса и/или гибели организма. Настоящее изобретение, таким образом, направлено на подавление конкретных генов-мишеней у насекомых-вредителей в качестве средства достижения более эффективного предотвращения и/или борьбы с нападением насекомых-вредителей, в частности на растения.
Краткое описание изобретения
Авторы настоящего изобретения пытались идентифицировать улучшенные средства для предотвращения и/или борьбы с нападением насекомых-вредителей с применением генетических подходов. В частности, они исследовали применение РНКi для подавления генов таким образом, чтобы снизить способность насекомых-вредителей выживать, расти, проходить через различные стадии жизненного цикла насекомых (например, через метаморфоз из куколки в имаго), колонизировать специфические местообитания и/или нападать на организмы-хозяева, и таким образом ограничить ущерб, наносимый вредителем.
Следовательно, в соответствии с одним аспектом настоящего изобретения предоставлено трансгенное растение или репродуктивный материал, или материал для размножения трансгенного растения, или культивируемая клетка трансгенного растения, которая экспрессирует или способна экспрессировать по меньшей мере одну интерферирующую рибонуклеиновую кислоту (РНК или двухцепочечную РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсируюший элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, состоящую любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, такой, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(v) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, где два ортологических гена являются подобными в последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или
(vi) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
В конкретном аспекте настоящего изобретения молекулы интерферирующей РНК, экспрессируемые растениями по настоящему изобретению, содержат по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsРНК содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени.
В одном варианте осуществления настоящее изобретение относится к трансгенному растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которая экспрессирует или способна экспрессировать молекулу интерферирующей РНК, которая содержит по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент молекулы интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsРНК содержит последовательность по меньшей мере из 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98%, 99% или 100% комплементарной последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени из комплекса тропонин/миофиламент.
В одном варианте осуществления ген-мишень кодирует белок wings up А (тропонин I) насекомых (например, у насекомых ортолог белка CG7178 Dm), при этом указанный ген-мишень представлен SEQ ID NO:1, 2, 174, 404, 175, 180, 181, 188 и 189. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:79, 349, 405, 352 или 356.
В одном варианте осуществления ген-мишень кодирует белок upheld (например, у насекомых ортолог белка CG7107 Dm), при этом указанный ген-мишень представлен SEQ ID NO:121, 130, 142, 143, 176, 177, 182 и 183. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:330, 350 или 353.
В одном варианте осуществления ген-мишень кодирует белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm) или белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), при этом указанный ген-мишень представлен SEQ ID NO:123 и 132. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:332.
В одном варианте осуществления ген-мишень кодирует тяжелую цепь миозина (например, у насекомых ортолог белка CG17927 Dm), при этом указанный ген-мишень представлен SEQ ID NO:122, 131, 144, 145, 178 и 179. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:331 или 351.
В одном варианте осуществления ген-мишень кодирует цитоплазматический белок на основе легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), при этом указанный ген-мишень представлен SEQ ID NO:124 и 133. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:333.
В одном варианте осуществления ген-мишень кодирует белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), при этом указанный ген-мишень представлен SEQ ID NO:125 и 134. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью с SEQ ID NO:334.
В одном варианте осуществления ген-мишень кодирует белок zipper (например, у насекомых ортолог белка CG15792 Dm), при этом указанный ген-мишень представлен SEQ ID NO:126 и 135. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью с SEQ ID NO:335.
В одном варианте осуществления ген-мишень кодирует тропонин С (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm), при этом указанный ген-мишень представлен SEQ ID NO:127 и 136, или 128 и 137, или 184 и 185. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:336, 337 и 354.
В соответствии с другим аспектом осуществления настоящее изобретение относится к трансгенному растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которая экспрессирует или способна экспрессировать молекулу интерферирующей РНК, которая содержит по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент молекулы интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsPHК содержит последовательность из по меньшей мере 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98%, 99% или 100% комплементарной последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени, который кодирует рибосомный белок насекомых.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), при этом указанный ген-мишень представлен SEQ ID NO:11, 12 и 141. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или обеими из SEQ ID NO:84 или 328.
В одном варианте осуществления ген-мишень кодирует рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), при этом указанный ген-мишень представлен SEQ ID NO:3 и 4. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:80.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), при этом указанный ген-мишень представлен SEQ ID NO:7 и 8. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:82.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), представленный SEQ ID NO:9 и 10. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:83.
В одном варианте осуществления ген-мишень кодирует рибосомный белок 318 (например, у насекомых ортолог белка CG8900 Dm), при этом указанный ген-мишень представлен SEQ ID NO:13 и 14. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:85.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), при этом указанный ген-мишень представлен SEQ ID NO:5 и 6. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:81.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), при этом указанный ген-мишень представлен SEQ ID NO:15 и 16, 204 и 205. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или обеими из SEQ ID NO:86 и 359.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), при этом указанный ген-мишень представлен SEQ ID NO:17 и 18. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:87.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), при этом указанный ген-мишень представлен SEQ ID NO:19 и 20. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:88.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), при этом указанный ген-мишень представлен SEQ ID NO:21 и 22. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:89.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), при этом указанный ген-мишень представлен SEQ ID NO:158 и 159. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:343.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), при этом указанный ген-мишень представлен SEQ ID NO:165, 166 и 167. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:347 и 348.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), при этом указанный ген-мишень представлен SEQ ID NO:156 и 157. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:342.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), при этом указанный ген-мишень представлен SEQ ID NO:160 и 161. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:344.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm), при этом указанный ген-мишень представлен SEQ ID NO:154 и 155. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:341.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), при этом указанный ген-мишень представлен SEQ ID NO:163 и 164. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:345.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), при этом указанный ген-мишень представлен SEQ ID NO:152 и 153. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:340.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S15Aa (например, у насекомых ортолог белка CG2033 Dm), при этом указанный ген-мишень представлен SEQ ID NO:150 и 151. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:339.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm), при этом указанный ген-мишень представлен SEQ ID NO:200 и 201. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:357.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm), при этом указанный ген-мишень представлен SEQ ID NO:386. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:390.
В одном варианте осуществления ген-мишень кодирует белок субъединицы II митохондриальной цитохром с-оксидазы (например, у насекомых ортолог белка CG34069 Dm), при этом указанный ген-мишень представлен SEQ ID NO:25 и 26. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:91.
В одном варианте осуществления ген-мишень кодирует γ-цепь АТФ-синтазы (например, у насекомых ортолог белка CG7610 Dm), при этом указанный ген-мишень представлен SEQ ID NO:129 и 138. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:338.
В одном варианте осуществления ген-мишень кодирует убиквитин-5Е (например, у насекомых ортолог белка CG32744 Dm), при этом указанный ген-мишень представлен SEQ ID NO:186 и 187, 202 и 203. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или обеими из SEQ ID NO:355 и 358.
В одном варианте осуществления ген-мишень кодирует протеасомную субъединицу бета-типа (например, у насекомых ортолог белка CG17331 Dm), при этом указанный ген-мишень представлен SEQ ID NO:387. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:391.
В одном варианте осуществления ген-мишень кодирует белок, который является у насекомого ортологом белка CG13704 Dm, при этом указанный ген-мишень представлен SEQ ID NO:388. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:392.
В одном варианте осуществления ген-мишень кодирует белок Rpn12 (например, у насекомых ортолог белка CG4157 Dm), при этом указанный ген-мишень представлен SEQ ID NO:389. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:393.
В соответствии с дополнительным аспектом настоящего изобретения предусматривается выделенный полинуклеотид, выбранный из группы, состоящей из:
(i) полинуклеотида, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, которая представлена любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, или
(ii) полинуклеотида, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, которая представлена любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, или
(iii) полинуклеотида, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, которая представлена в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) полинуклеотида, который содержит фрагмент по меньшей мере из 21 предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, который представлен в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и где указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью, такой, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(v) полинуклеотида, который содержит фрагмент по меньшей мере из 21 предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, который представлен в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и где указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью, при которой указанный фрагмент оптимально выравнивается и сравнивается с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(vi) полинуклеотида, кодирующего аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, и
где указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
Аминокислотные последовательности, кодируемые генами-мишенями по настоящему изобретению, представлены SEQ ID NO:79, 349, 405, 352, 356, 80, 326, 81, 327, 82, 83, 328, 84, 329, 85, 86, 359, 87-91, 330, 350, 353, 331, 351, 332-336, 337, 354, 338-344, 346, 345, 347, 348, 357, 355, 358, 390-393.
В конкретном аспекте по настоящему изобретению выделенный полинуклеотид является частью молекулы интерферирующей РНК, как правило, частью сайленсирующего элемента, содержащего по меньшей мере одну двухцепочечную область, содержащую смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsPHК содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени. Более конкретно, выделенный полинуклеотид клонирован в ДНК-конструкцию в смысловой и антисмысловой ориентации так, что при транскрипции смыслового и антисмыслового полинуклеотида формируется молекула dsPHК, которая функционирует при поглощении вредителем с ингибированием или подавлением экспрессии гена-мишени в указанном вредителе.
В определенных аспектах настоящее изобретение относится к выделенным полинуклеотидам, которые клонированы в ДНК-конструкцию в смысловой и антисмысловой ориентации так, что при транскрипции смыслового и антисмыслового полинуклеотида формируется молекула dsPHК, которая функционирует при поглощении насекомым с ингибированием или подавлением экспрессии гена-мишени в пределах комплекса тропонин/миофиламент.
В одном варианте осуществления ген-мишень кодирует белок wings up А (тропонин I) насекомых (например, у насекомых ортолог белка CG7178 Dm), при этом указанный ген-мишень представлен SEQ ID NO:1, 2, 174, 404, 175, 180, 181, 188 и 189. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:79, 349, 405, 352 или 356.
В одном варианте осуществления ген-мишень кодирует белок upheld (например, у насекомых ортолог белка CG7107 Dm), при этом указанный ген-мишень представлен SEQ ID NO:121, 130, 142, 143, 176, 177, 182 и 183. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:330, 350 или 353.
В одном варианте осуществления ген-мишень кодирует белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm), или белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), при этом указанный ген-мишень представлен SEQ ID NO:123 и 132. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:332.
В одном варианте осуществления ген-мишень кодирует тяжелую цепь миозина (например, у насекомых ортолог белка CG17927 Dm), при этом указанный ген-мишень представлен SEQ ID NO:122, 131, 144, 145, 178 и 179. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:331 или 351.
В одном варианте осуществления ген-мишень кодирует цитоплазматический белок на основе легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), при этом указанный ген-мишень представлен SEQ ID NO:124 и 133. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:333.
В одном варианте осуществления ген-мишень кодирует белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), при этом указанный ген-мишень представлен SEQ ID NO:125 и 134. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью с SEQ ID NO:334.
В одном варианте осуществления ген-мишень кодирует белок zipper (например, у насекомых ортолог белка CG15792 Dm), при этом указанный ген-мишень представлен SEQ ID NO:126 и 135. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью с SEQ ID NO:335.
В одном варианте осуществления ген-мишень кодирует тропонин С (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm), при этом указанный ген-мишень представлен SEQ ID NO:127 и 136, или 128 и 137, или 184 и 185. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или несколькими из SEQ ID NO:336, 337 и 354.
В соответствии с другими вариантами осуществления настоящее изобретение относится к выделенному полинуклеотиду, который клонирован в ДНК-конструкцию в смысловой и антисмысловой ориентации так, что при транскрипции смыслового и антисмыслового полинуклеотида формируется молекула dsPHК, которая функционирует при поглощении насекомым с ингибированием или подавлением экспрессии гена-мишени, который кодирует рибосомный белок насекомых.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), при этом указанный ген-мишень представлен SEQ ID NO:11, 12 и 141. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или обеими из SEQ ID NO:84 или 328.
В одном варианте осуществления ген-мишень кодирует рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), при этом указанный ген-мишень представлен SEQ ID NO:3 и 4. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:80.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), при этом указанный ген-мишень представлен SEQ ID NO:7 и 8. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:82.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), представленный SEQ ID NO:9 и 10. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:83.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S18 (например, у насекомых ортолог белка CG8900 Dm), при этом указанный ген-мишень представлен SEQ ID NO:13 и 14. В предпочтительном варианте осуществления ортолог унасекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:85.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), при этом указанный ген-мишень представлен SEQ ID NO:5 и 6. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:81.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), при этом указанный ген-мишень представлен SEQ ID NO:15 и 16, 204 и 205. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с одной или обеими из SEQ ID NO:86 и 359.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), при этом указанный ген-мишень представлен SEQ ID NO:17 и -18. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:87.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), при этом указанный ген-мишень представлен SEQ ID NO:19 и 20. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:88.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), при этом указанный ген-мишень представлен SEQ ID NO:21 и 22. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:89.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), при этом указанный ген-мишень представлен SEQ ID NO:158 и 159. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:343.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), при этом указанный ген-мишень представлен SEQ ID NO:165, 166 и 167. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:347 и 348.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), при этом указанный ген-мишень представлен SEQ ID NO:156 и 157. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:342.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), при этом указанный ген-мишень представлен SEQ ID NO:160 и 161. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:344.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm) у при этом указанный ген-мишень представлен SEQ ID NO:154 и 155. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:341.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), при этом указанный ген-мишень представлен SEQ ID NO:163 и 164. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:345.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), при этом указанный ген-мишень представлен SEQ ID NO:152 и 153. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:340.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S15Aa (например, у насекомых ортолог белка CG2033 Dm), при этом указанный ген-мишень представлен SEQ ID NO:150 и 151. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:339.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm), при этом указанный ген-мишень представлен SEQ ID NO:200 и 201. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:357.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm), при этом указанный ген-мишень представлен SEQ ID NO:386. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичностью аминокислотной последовательности с SEQ ID NO:390.
Предпочтительно, способы по настоящему изобретению находят практическое применение в предотвращении и/или борьбе с нападением насекомых-вредителей, в частности, борьбе с нападением вредителей сельскохозяйственных культур, таких как, но без ограничений, хлопчатник, картофель, рис, клубника, люцерна, сои, томат, канола, подсолнечник, сорго, просо, кукуруза, баклажан, перец и табак. В дополнение, интерферирующую РНК по настоящему изобретению можно вводить в растения, подлежащие защите, с помощью обычных методов генной инженерии.
Следовательно, в соответствии с другим аспектом настоящего изобретения предусматривается способ создания трансгенного растения, устойчивого к нападению любого вида вредителя, включающий:
(а) трансформацию растительной клетки с помощью ДНК-конструкции, содержащей полинуклеотидную последовательность, кодирующую интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного вида насекомого-вредителя, где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере, на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 или комплементарной ей последовательности, или
(v) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что, при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или
(vi) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая, при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389;
(b) регенерацию растения из трансформированной растительной клетки; и
(c) выращивание трансформированного растения в условиях, пригодных для экспрессии интерферирующей РНК из рекомбинантной ДНК-конструкции, при этом указанное растение, таким образом, является устойчивым к указанному вредителю по сравнению с нетрансформированным растением.
В дополнительном аспекте в данном описании предусматривается способ предотвращения и/или борьбы с нападением насекомых-вредителей в поле культурных растений, где указанный способ включает экспрессию в указанных растениях эффективного количества интерферирующей рибонуклеиновой кислоты (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного вида насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 1, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является, по меньшей мере на 75% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
Во всех аспектах настоящего изобретения, в предпочтительных вариантах осуществления ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233.
В предпочтительном варианте осуществления ген-мишень кодирует белок насекомых, выбранный из комплекса тропонин/миофиламент, выбранный из группы, содержащей белок тропонин I (например, у насекомых ортолог белка CG7178 Dm), белок upheld (например, у насекомых ортолог белка CG7107 Dm), белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm), белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), тяжелую цепь миозина (например, у насекомых ортолог белка CG17927 Dm), цитоплазматический белок на основе легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), белок zipper (например, у насекомых ортолог белка CG15792 Dm) и белок тропонин С (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm).
Во всех аспектах настоящего изобретения в предпочтительных вариантах осуществления ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной•последовательности, кодируемой любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273.
В предпочтительном варианте осуществления ген-мишень кодирует рибосомный белок насекомых, выбранный из группы, содержащей рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), рибосомный белок S18 (например, у насекомых ортолог белка CG8900 Dm), рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), и рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm), рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), рибосомный белок S15Aa (например, у насекомых ортолог белка CG2033 Dm), рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm) и рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm).
Во всех аспектах настоящего изобретения в предпочтительных вариантах осуществления ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401.
В предпочтительных вариантах осуществления ген-мишень может кодировать белок тропонин I насекомого (например, у насекомых ортолог белка CG7178 Dm). Белок тропонин I насекомого может иметь аминокислотную последовательность, которая по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентична аминокислотной последовательности, представленной в любой из SEQ ID NO:79, 349, 405, 352 или 356 (при оптимальном выравнивании указанных кодируемых белков).
Краткое описание таблиц и фигур
Таблица 1. Lygus hesperus новые мишени, идентифицированные из первого анализа.
Таблица 1B. Lygus hesperus новые мишени в пути Lh594.
Таблица 1C. Lygus hesperus новые мишени, идентифицированные из второго цикла анализа.
Таблица 2. Полинуклеотидные последовательности генов-мишеней, идентифицированные у Lygus hesperus.
Таблица 3. Аминокислотные последовательности генов-мишеней, идентифицированные у Lygus hesperus.
Таблица 4. dsPHК (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие Lygus hesperus генам-мишеням и праймерам для продуцирования dsPHК.
Таблица 5. Ранжирование мишеней Lygus hesperus в соответствии с кривыми дозовой зависимости (DRC) и по сравнению с исходными мишенями Lh423 и Lh105.
Таблица 6. Lygus hesperus мишени из второго цикла анализа-ранжирования в соответствии с DRC и по сравнению с исходными мишенями Lh423 и Lh594.
Таблица 7. Обзор тестирования трансгенного картофеля, несущего Lygus hesperus шпильки.
Таблица 8. Последовательность ампликонов для гена-мишени и два конститутивных гена для qRT-ПЦР.
Таблица 9. Полинуклеотидные последовательности генов-мишеней, идентифицированные у колорадского жука (СРВ).
Таблица 10. Аминокислотные последовательности генов-мишеней, идентифицированные у СРВ.
Таблица 11. dsPHК (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие СРВ генам-мишеням и праймерам для продуцирования dsPHК.
Таблица 12. Полинуклеотидные последовательности генов-мишеней, идентифицированные у коричневого дельфацида (ВРН).
Таблица 13. Аминокислотные последовательности генов-мишеней, идентифицированные у ВРН.
Таблица 14. dsPHК (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие ВРН генам-мишеням и праймерам для продуцирования dsPHК.
Таблица 15. Праймеры, используемые для амплификации сДНК тлей, на основе геномной последовательности гороховой тли.
Таблица 16. Полинуклеотидные последовательности генов-мишеней, идентифицированные у тлей.
Таблица 17. Аминокислотные последовательности генов-мишеней, идентифицированные у тлей.
Таблица 18. dsPHК (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие генам-мишеням тлей и праймерам для продуцирования dsPHК.
Таблица 19. Вырожденные праймеры, используемые для амплификации СРВ Ld594 сДНК.
Таблица 20. Вырожденные праймеры, используемые для амплификации ВРН сДНК.
Таблица 21. Leptinotarsa decemlineata новые мишени из данного теста.
Таблица 22. Nilaparvata lugens новые идентифицированные мишени.
Таблица 23. Acyrthosiphon pisum новые идентифицированные мишени.
Фиг. 1. Полоски Lh001_009 второго подтверждающего анализа. Темные полосы: смертность в день 3-6, светлые полосы: смертность в день 6-8. Кандидатные клоны названы с применением "Lygxxx" скрининговых кодов и "Lhxxx" номенклатурных кодов мишеней.
Фиг. 2. Полоски Lh010_020 второго подтверждающего анализа. Темные полосы: смертность в день 3-6, светлые полосы: смертность в день 6-8. Кандидатные клоны названы с применением "Lygxxx" скрининговых кодов и "Lhxxx" номенклатурных кодов мишеней.
Фиг. 3. Анализ смертности Lygus новых мишеней из полосок Lh001-Lh009, выраженный в % смертности за 10-дневный период. Контроли обозначены пунктирными линиями. Положительный контроль: Lh423 dsPHК (RpL19). Отрицательные контроли: GFP dsPHК и только корм (контроль).
Фиг. 4. Анализ смертности Lygus новых мишеней из полосок Lh010-Lh020, выраженный в % смертности за 10-дневный период. Контроли обозначены пунктирными линиями. Положительный контроль: Lh423 (RpL19). Отрицательные контроли: GFP и только корм (контроль).
Фиг. 5. Схематическое изображение растительного вектора экспрессии, несущего Lygus hesperus hpPHК кассету. RB: правая граница; LB: левая граница; P35S: промотор 35S вируса мозаики цветной капусты; T35S: терминатор 35S вируса мозаики цветной капусты; TNOS: терминатор нопалинсинтазы; GFP: репортерный ген зеленого флуоресцентного белка; NPT II: кодирующая последовательность гена неомицинфосфотрансферазы II; KmR: ген устойчивости к канамицину; pBR322 ori: pBR322 точка начала репликации; pBR322 bom: pBR322 мобилизация; pVS1 rep: pVS1 репликон; pVS1 sta: pVS1 элемент стабильности.
Фиг. 6. Картофель-Lygus in planta постановка анализа. Белые стрелки указывают повреждения насекомыми.
Фиг. 7-11. Lygus hesperus новые мишени - кривые дозовой зависимости при концентрациях очищенной синтетической dsPHК в диапазоне от 0,4 до 0,025 мкг/мкл (на фигуре единица "мкг/мклʺ не отображена). GFP dsPHК и воду milliQ применяли как отрицательные контроли. dsPHК мишеней получали с использованием праймеров, как описано в разделе примера 1.1.
Фиг. 12. Lh594 кривая дозовой зависимости, при концентрациях dsPHК в диапазоне от 0,05 до 0,001 мкг/мкл. GFP dsPHК и воду milliQ применяли как отрицательные контроли.
Фиг. 13. А dsPHК активность в Lygus hesperus биопробе в отсутствие tPHК. Lh594 (5 мкг/мкл); положительный контроль: Lh423 (5 мкг/мкл); отрицательные контроли: GFP dsPHК (5 мкг/мкл) и вода milliQ; В Идентификация Lh594 предела активности с использованием уменьшающейся концентрации dsPHК (от 5 мкг до 0,25 мкг). Отрицательные контроли: GFP dsPHК (5 мкг/мкл) и вода milliQ.
Фиг. 14. Полоски Lh010-Lh020 второго подтверждающего анализа мишеней второго теста. Темные полосы: смертность в день 4-8, светлые полосы: смертность в день 4-6. Кандидатные клоны названы с применением "Lygxxx" скрининговых кодов и "Lhxxx" номенклатурных кодов мишеней.
Фиг. 15. Результаты анализа для Lygus мишеней пути тропонина, тестированных при 0,5 мкг/мкл фиксировано.
Фиг. 16. А-В Lygus hesperus новые мишени из пути тропонина - кривые дозовой зависимости при концентрациях очищенной синтетической dsPHК в диапазоне от 0,4 до 0,025 мкг/мкл (на фигуре единица "мкг/мкл" не всегда отображена). GFP dsPHК и воду milliQ применяли как отрицательные контроли.
Фиг. 17. A-D Lygus hesperus новые мишени из мишеней второго теста - кривые дозовой зависимости при концентрациях очищенной синтетической dsPHК в диапазоне от 0,5 до 0,05 мкг/мкл. GFP dsPHК и воду milliQ применяли как отрицательные контроли.
Фиг. 18 А-В. Тестирование и отбор GUS трансгенных объектов. Восемь независимых объектов трансгенной линии GUS шпильки (Р001) тестировали в Lygus hesperus индивидуальном анализе в вегетационных сосудах и сравнивали с WT проростками. Все проростки подвергли одинаковой обработке. Однодневных Lygus hesperus нимф добавляли в каждый вегетационный сосуд, и выживаемость проверяли в течение 9 дней.
Фиг. 19. Тестирование Lh423 трансгенных объектов: 28 независимых трансгенных объектов (линия Р006) тестировали в Lygus hesperus индивидуальном анализе в вегетационных сосудах. Lh423 трансгенные проростки сравнивали с WT проростками и с GUS трансгенными объектами (линия Р001). Единичных однодневных Lygus hesperus нимф добавляли в каждый вегетационный сосуд, и выживаемость проверяли в течение 9 дней.
Фиг. 20. Тестирование Lh423 трансгенных объектов: показаны 6 независимых трансгенных объектов (Р006), ведущих к >60% выживаемости. Lh423 трансгенные проростки сравнивали с WT проростками и с GUS трансгенными линиями (Р001). Единичных однодневных Lygus hesperus нимф добавляли в каждый вегетационный сосуд, и выживаемость проверяли в течение 9 дней.
Фиг. 21. Тестирование Lh594 трансгенных объектов: 25 независимых трансгенных объектов (Р007) тестировали в Lygus hesperus индивидуальном анализе в вегетационных сосудах. Lh594 трансгенные проростки сравнивали с WT проростками и с GUS трансгенными объектами (Р001). Единичных однодневных Lygus hesperus нимф добавляли в каждый вегетационный сосуд, и выживаемость проверяли в течение 11 дней.
Фиг. 22. Тестирование Lh594 трансгенных объектов: показаны 6 независимых трансгенных объектов (Р007), ведущих к 60% выживаемости. Lh594 трансгенные проростки сравнивали с WT проростками и с GUS трансгенными линиями (Р001). Единичных однодневных Lygus hesperus нимф добавляли в каждый вегетационный сосуд, и выживаемость проверяли в течение 11 дней.
Фиг. 23. Относительное значение уровней Lh423 mPHК у насекомых после питания в течение 5 дней на трансгенных растениях, содержащих GUS шпильку или Lh423 шпильку. Образцы анализировали с помощью праймеров, амплифицирующих Lh423. Данные нормализовали, применяя GeNorm, с 2 конститутивными генами, Lh425 и Lh427.
Фиг. 24. Анализ выживаемости личинок СРВ, обработанных с помощью 1 мкг dsPHК Ld594, Ld619 и Ld620. Положительные контроли включали 1 мкг dsPHК исходных мишеней Ld513 и Ld049. Отрицательные контроли включали воду milliQ и FP.
Фиг. 25. Эффекты Ld594, Ld619 и Ld620 dsPHК на окукливание СРВ 4-ой возрастной стадии личинок, по сравнению с необработанным контролем (UTC). Жукам скармливали 1 мкг dsPHК, распределенной на листовых дисках картофеля, затем предоставляли возможность питаться на необработанных листьях картофеля (А) в течение 4 дней перед помещением на вермикулит. Для оценки эффекта dsPHК мертвых насекомых извлекали из вермикулита (из-за сильных эффектов, индуцированных с помощью Ld594 dsPHК, ни одну куколку невозможно восстановить из вермикулита и, следовательно, никакое изображение не доступно для данной dsРНК-мишени) (В).
Фиг. 26. Эффект СРВ Ld594, 619 и 620 dsPHК на выживаемость и приспособляемость имаго СРВ. Оценки проводили в дни 4, 6, 7, 8, 11 и 13. Контроль MQ: вода milliQ.
Фиг. 27. Активность dsPHК из N1594 пути у коричневого дельфацида. DsPHК тестировали при 0,5 мкг/мкл в присутствии 0,1% CHAPSO. Положительный контроль: N1537 dsPHК (0,5 мкг/мкл), отрицательные контроли: GFP dsPHК (0,5 мкг/мкл) и только корм.
Фиг. 28. Активность dsPHК из Ар594, Ар423, Ар537 и Ар560 на A. pisum. DsPHК тестировали при 0,5 мкг/мкл в присутствии 5 мкг/мкл tPHК. Отрицательный контроль: GFP dsPHК (0,5 мкг/мкл).
Фиг. 29. Проценты смертности личинок L. decemlineata на искусственном корме, обработанном с помощью dsPHК. Ld583, Ld584, Ld586 и Ld588 представляют клоны-мишени. Положительный контроль: Ld513; отрицательный контроль: FP.
Подробное описание настоящего изобретения
Авторы настоящего изобретения обнаружили, что подавление экспрессии конкретных генов-мишеней у видов насекомых-вредителей с помощью PHКi можно применять для эффективного предотвращения и/или борьбы с нападением указанного насекомого-вредителя. Использование PHКi для подавления экспрессии генов-мишеней у видов насекомых-вредителей применяется в данном описании для создания растений, устойчивых к нападению насекомых-вредителей.
Следовательно, в первом аспекте, настоящее изобретение предоставляет трансгенные растения, устойчивые к нападению видов насекомых-вредителей. В частности, в данном описании предоставлены трансгенные растения, которые экспрессируют или способны экспрессировать по меньшей мере одну интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видами насекомых-вредителей с подавлением экспрессии гена-мишени, как описано в других местах в данном описании, у указанного вредителя. Интерферирующей РНК может являться любая из тех, которые раскрыты в данном описании ниже. Предпочтительно, интерферирующая РНК содержит или состоит по меньшей мере из одного сайленсирующего элемента, при этом указанный сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых (смысловая цепь) содержит последовательность нуклеотидов» которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени. Подавление гена-мишени вредителя можно применять для нарушения жизненно важного биологического процесса или функции у вредителя, где 'жизненно важный' относится к тому факту, что процесс или функция являются необходимыми для инициирования или поддержания нападения вредителя.
Применяемый в данном описании термин 'растение' может содержать любой репродуктивный материал или материал для размножения растения. Ссылка на растение также может включать клетки растений, протопласты растений, культуры тканей растений, растительные каллусы, маточные корневища растений и растительные клетки, которые являются интактными в растениях или частях растений, такие как зародыши, пыльца, семяпочки, семена, листья, цветы, ветки, фрукты, ядра, колосья, початки, шелуха, стебли, корни, кончики корней и тому подобное. Потомство, варианты и мутанты любого из трансгенных растений, описанных в данном описании, находятся в рамках настоящего изобретения. Также включены семена, полученные от любого из указанных трансгенных растений.
Применяемый в данном описании термин "борьба" с нападением вредителей относится к любому воздействию на вредителя, которое служит для ограничения и/или уменьшения количества организмов вредителей и/или повреждения, вызываемого вредителем. Предпочтительные гены-мишени являются, следовательно, жизненно важными генами, которые контролируют или регулируют одну или более жизненно важные биологические функции у насекомых-вредителей, например, деление клеток, размножение, энергетический обмен, пищеварение, неврологическая функция и тому подобное. Подавление данных жизненно важных генов с помощью PHКi методов может вести к гибели насекомого или иным образом значительно замедлять рост и развитие, или нарушать способность вредителей колонизировать местообитание или нападать на организмы-хозяева.
Авторы настоящего изобретения в настоящее время идентифицировали лучшие гены-мишени видов насекомых-вредителей, принадлежащих к Lygus, Leptinotarsa, Nilaparvata и Acyrthosiphum родам, чьи мишени предусматриваются для применения в отдельности или в комбинации в качестве эффективных средств для PHКi - опосредованной борьбы с нападением насекомых на агрономически важные культуры. Ортологи этих недавно идентифицированных генов-мишеней можно использовать у других видов насекомых для борьбы с нападением насекомых на соответствующие значимые культуры.
Более конкретно, авторы настоящего изобретения описывают в данном описании, что гены, кодирующие белки комплекса тропонин/миофиламент, образуют отличные гены-мишени для подавления с помощью методики РНК-ингибирования. Один из данных генов-мишеней кодировал белок тропонин I (wings up А) насекомых, который является ортологом белка CG7178 дрозофилы. Данный белок участвует в мышечных сокращениях и принадлежит к физиологическому пути, который еще не полностью исследован для борьбы с (насекомыми) вредителями посредством РНК-ингибирования. Кроме того, поскольку данный белковый комплекс является специфичным для животных, не известен ни один гомолог или ортолог генов растений, что снижает риск нетипичных фенотипов растений при экспрессии dsPHК-мишени в растениях. В дополнение, у дрозофилы тропонин I описывается как гаплонедостаточный ген, проявляющий мутантный фенотип в гетерозиготном состоянии. Такие гены особенно восприимчивы к снижению уровней экспрессии mРНК, и как таковые могут считаться идеальными мишенями PHКi.
Дополнительные вызывающие интерес гены-мишени в данном комплексе тропонин/миофиламент перечислены ниже, и дополнительно исследуются для борьбы с помощью PHКi с Lygus hesperus и другими видам насекомых-вредителей:
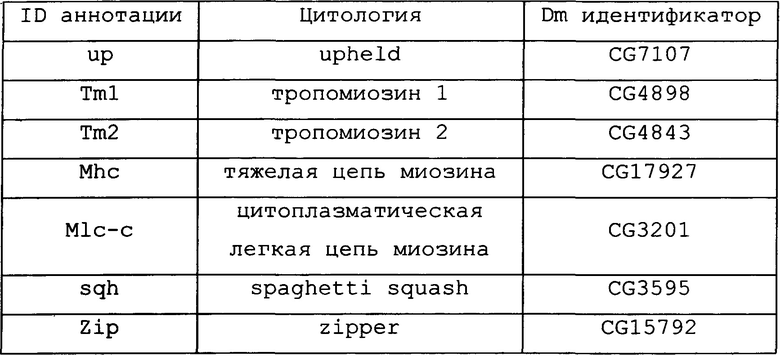
В одном аспекте настоящее изобретение предусматривает трансгенное растение у репродуктивный материал или материал для размножения, полученный из него, или культивируемую клетку растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени в указанном насекомом-вредителе.
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или имеющих нуклеотидную последовательность, так, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, и так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233.
В предпочтительном варианте осуществления ген-мишень кодирует белок насекомых, выбранный из комплекса тропонин/миофиламент, выбранный из группы, включающей тропонин I (например, у насекомых ортолог белка CG7178 Dm), белок upheld (например, у насекомых ортолог белка CG7107 Dm), белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm), белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), тяжелая цепь миозина (например, у насекомых ортолог белка CG17927 Dm), цитоплазматический белок на основе легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), белок zipper (например, у насекомых ортолог белка CG15792 Dm), тропонин С (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm).
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273.
В предпочтительном варианте осуществления ген-мишень кодирует рибосомный белок насекомых, выбранный из группы, содержащей рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), рибосомный белок S18 (например, у насекомых ортолог белка CG8900 Dm), рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), и рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm), рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), рибосомный белок S15Аа (например, у насекомых ортолог белка CG2033 Dm), рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm), рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm).
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей, мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401.
В предпочтительных вариантах осуществления ген-мишень может кодировать белок тропонин I насекомого (например, у насекомого ортолог белка CG7178 Dm). Белок тропонин I насекомого может иметь аминокислотную последовательность, которая по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентична аминокислотной последовательности, представленной в любой из SEQ ID NO:79, 349, 405, 352 или 356 (при оптимальном выравнивании указанных кодируемых белков).
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:141, 11 12 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность такой, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:141, 11, 12 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:141, 11, 12 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:141, 11, 12 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:141, 11, 12 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:141, 11, 12 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:141, 11, 12 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:141, 11, 12 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:141, 11, 12 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 7,5%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:141, 11, 12, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:141, 11 12.
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:17, 18 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность такой, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:17, 18 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:17, 18 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:17, 18 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:17, 18 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:17, 18 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:17, 18 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:17, 18 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:17, 18 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:17, 18, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:17, 18.
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:19, 20 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:19, 20 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:19, 20 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:19, 20 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:19, 20 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:19, 20 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:19, 20 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:19, 20 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:19, 20 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:19, 20, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:19, 20.
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:165, 166, 167 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:165, 166, 167 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:165, 166, 167 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:165, 166, 167 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:165, 166, 167 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:165, 166, 167 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:17, 18 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:165, 166, 167 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:165, 166, 167 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:165, 166, 167, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:165, 166, 167.
В одном варианте осуществления настоящее изобретение относится. к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO: 143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему.фрагменту любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183 или комплементарную ей последовательность, где два ортологических гена. сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183.
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:145, 122, 144, 178, 131, 179 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:145, 122, 144, 178, 131, 179 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:145, 122, 144, 178, 131, 179 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:145, 122, 144, 178, 131, 179, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:145, 122, 144, 178, 131, 179.
В одном варианте осуществления настоящее изобретение относится к растению или репродуктивному материалу, или материалу для размножения трансгенного растения, или культивируемой клетке трансгенного растения, которые экспрессируют или способны экспрессировать интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, где РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и где ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:128, 149, 184, 137 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:128, 149, 184, 137 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:128, 149, 184, 137 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:128, 149, 184, 137 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:128, 149, 184, 137 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:128, 149, 184, 137 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:128, 149, 184, 137 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:128, 149, 184, 137 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:128, 149, 184, 137 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:128, 149, 184, 137 или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:128, 149, 184, 137.
Применяемый в данном описании термин "ген-мишень" включает любой ген у насекомого-вредителя, который предполагается подавить. В предпочтительном варианте осуществления ген-мишень подавляется так, чтобы бороться с нападением вредителя, например путем нарушения жизненно важного биологического процесса, происходящего у вредителя, или путем уменьшения патогенности вредителя. Предпочтительные гены-мишени, следовательно, включают, но без ограничений, те, которые играют ключевую роль в регуляции питания, выживания, роста, развития, размножения, нападения и способности вызывать поражение. В соответствии с одним вариантом осуществления ген-мишень является таковым, что при подавлении или ингибировании его экспрессии насекомое-вредитель погибает. В соответствии с другим вариантом осуществления ген-мишень является таковым, что при подавлении или ингибировании его экспрессии рост вредителя предотвращается или замедляется, или останавливается, или задерживается, или затрудняется, размножение вредителя предотвращается, или предотвращается переход через жизненные циклы вредителя. В соответствии еще с другим вариантом осуществления настоящего изобретения ген-мишень является таковым, что при подавлении или ингибировании его экспрессии повреждение, вызванное вредителем и/или способностью вредителя поражать или нападать на местообитания, поверхности и/или растение, или сельскохозяйственную культуру уменьшается; или вредитель прекращает питаться из своих природных пищевых ресурсов, таких как растения и растительные продукты. Термины "нападать" и "поражать" или "нападение" и "поражение", как правило, используются взаимозаменяемо повсеместно.
Гены-мишени могут экспрессироваться во всех или некоторых клетках насекомых-вредителей. Кроме того, гены-мишени могут экспрессироваться насекомым-вредителем только на определенной стадии его жизненного цикла, например, зрелой стадии имаго, незрелой нимфы, или личиночной стадии, или стадии яйца.
Применяемое в данном описании виды "вредителей" предпочтительно являются видами насекомых, которые вызывают поражение или нападают, предпочтительно на растения.
Предпочтительными насекомыми, патогенными для растений, в соответствии с настоящим изобретением, являются вредители растений, выбранные из группы, включающей Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук), или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Laodelphax spp. (например, L. striatellus (малый коричневый дельфацид)); Nephotettix spp. (например, N. virescens или N. cincticeps (зеленая рисовая цикадка), или N. nigropictus (рисовая цикадка)); Sogatella spp. (или S. furcifera (белоспинный рисовый дельфацид)); Chilo spp. (например, С. suppressalis (огневка азиатская стеблевая), С. auricilius (огневка Chilo auricilius), или С. polychrysus (темноголовая рисовая огневка)); Sesamia spp. (например, S. inferens (розовый сверлильщик)); Tryporyza spp. (например, Т. innotata (огневка Tryporyza innotata), или Т. incertulas (желтый рисовый сверлильщик)); Anthonomus spp. (например, A. grandis (долгоносик хлопковый)); Phaedon spp. (например, Р. cochleariae (хреновый листоед)); Epilachna spp. (например, Е. varivetis (зерновка бобовая мексиканская)); Tribolium spp. (например, Т. castaneum (хрущак каштановый)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук), D. virgifera zeae (мексиканский кукурузный корнегрыз); Ostrinia spp. (например, О. nubilalis (огневка кукурузная)); Anaphothrips spp. (например, A. obscrurus (трипc злаковый)); Pectinophora spp. (например, Р. gossypiella (розовый коробочный червь хлопчатника)); Heliothis spp. (например, H. virescens (табачная листовертка)); Trialeurodes spp. (например, Т. abutiloneus (белокрылка Trialeurodes abutiloneus) T. vaporariorum (белокрылка тепличная)); Bemisia spp. (например, В. argentifolii (белокрылка магнолиевая)); Aphis spp. (например, А. gossypii (тля хлопковая)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Euschistus spp. (например, Е. conspersus (клоп Euschistus conspersus)); Chlorochroa spp. (например, С. sayi (клоп Сэя)); Nezara spp. (например, N. viridula (зеленый овощной клоп)); Thrips spp. (например, Т. tabaci (трипс луковый)); Frankliniella spp. (например, F. fusca (трипс табачный), или F. occidentalis (западный цветочный трипс)); Acheta spp. (например, A. domesticus (сверчок домовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Macrosiphum spp. (например, М. euphorbiae (тля картофельная большая)); Blissus spp. (например, В. leucopterus leucopterus (клоп-черепашка пшеничный североамериканский)); Acrosternum spp. (например, A. hilare (клоп-щитник Acrosternum hilare)); Chilotraea spp. (например, С. polychrysa (огневка Chilotraea polychrysa)); Lissorhoptrus spp. (например, L. oryzophilus (долгоносик рисовый водяной)); Rhopalosiphum spp. (например, R. maidis (тля кукурузная)); и Anuraphis spp. (например, А. maidiradicis (тля кукурузная корневая)).
В соответствии с более конкретными вариантами осуществления настоящее изобретение применимо к видам, принадлежащим к семейству Chrysomelidae или листоеды. Жуки листоеды, такие как колорадские жуки, блошки, кукурузные корневые жуки, и долгоносики, такие как долгоносик люцерновый, являются особенно важными вредителями. Конкретные виды рода Leptinotarsa, контролируемые в соответствии с настоящим изобретением, включают колорадского жука (Leptinotarsa decemlineata (Say) и ложного картофельного жука (Leptinotarsa juncta (Say). СРВ является (серьезным) вредителем нашего домашнего картофеля, других культурных и диких клубненосных и неклубненосных видов картофеля и других видов растений семейства Solanaceous (пасленовых), включая сельскохозяйственные культуры томатов, баклажанов, перцев, табака (виды рода Nicotiana, включая декоративные), физалиса, риса, кукурузы или хлопчатника; и виды сорняков/травянистых растений, паслен каролинский, черный паслен, дурман обыкновенный, белену и паслен клювообразный. Кукурузные корневые жуки включают виды, основывающие род Diabrotica (например, D. undecimpunctata undecimpunctata, D. undecimpunctata howardii, D. longicornis, D. virgifera и D. balteata). Кукурузные корневые жуки причиняют существенный вред кукурузе и тыквенным.
В соответствии с более конкретным вариантом осуществления настоящее изобретение применимо к видам, принадлежащим к отряду полужесткокрылые (Hemipterans) (семейство Aphidoidea), таким как Myzus persicae (тля персиковая зеленая), Aphis fabae (тля свекловичная), Acyrthosiphum pisum (тля гороховая), Brevicoryne brassicae (тля капустная), Sitobion avenae (тля большая злаковая), Cavariella aegopodii (тля зонтичная), Aphis craccivora (тля арахисовая), Aphis gossypii (тля хлопковая), Toxoptera aurantii (тля померанцевая), Cavariella spp (тля тминная), Chaitophorus spp (тли рода Chaitophorus), Cinara spp. (тли рода Cinara), Drepanosiphum platanoides (тля яворовая), Elatobium spp (тли рода Elatobium), которые вызывают повреждение растений, таких как деревья рода слива, в частности, персик, абрикос и слива; деревьев, которые в основном культивируются для производства древесины, таких как ивы и тополя, пропашных культур, таких как кукуруза, хлопчатник, соя, пшеница и рис, овощных культур из семейств Solanaceae, Chenopodiaceae, Compositae, Cruciferae и Cucurbitaceae, включая, но без ограничений, артишок, спаржу, фасоль, свеклу, брокколи, брюссельскую капусту, кочанную капусту, морковь, цветную капусту, мускусную дыню, сельдерей, кукурузу, огурцы, укроп, капусту кормовую, кольраби, репу, баклажан, салат, горчицу, бамию, петрушку, пастернак, горох, перец, картофель, редис, шпинат, тыкву, томат, репу, кресс водяной и арбуз; или полевых культур, таких как, но без ограничений, табак, сахарная свекла и подсолнечник; цветочных культур или других декоративных растений, таких как сосны и хвойные деревья. Другие полужесткокрылые (Hemipterans) относятся к Nilaparvata ssp (например N. lugens, Sogatella furcifera) и причиняют вред растениям риса. Другие полужесткокрылые (Hemipterans) относятся к Lygus ssp (например, Lygus hesperus, Lygus rugulipennis, Lygus lineolaris, Lygus sully) и другим видам растительноядных насекомых в семействе Miridae, и причиняют вред хлопчатнику, растениям картофеля, клубнике, хлопчатнику, люцерне, канола, персику, сливам, винограду, латуку, баклажану, луку, зеленым бобам. А также некоторым средиземноморским деревьям и некоторым декоративным деревьям, таким как вяз (Ulmus spp.), кедровый орех (Pinus Pinea), платан кленолистный (Platanus Acerifolia), яблоня Malus alba (Malus alba). Другие полужесткокрылые (Hemipterans) относятся к семейству Pentatomoidea, их обычно называют щитники, лесные клопы и клопы-вонючки (например; коричневый мраморный щитник (Halyomorpha halys), клоп-щитник Euschistus conspersus (Euschistus conspersus), зеленый овощной клоп (Nezara viridula), щитник красноногий (Pentatoma rufipes), клоп капустный (Murgantia histrionica), клоп Oebalus pugnax (Oebalus pugnax)), и они наносят ущерб плодам, включая яблоки, персики, инжир, шелковицу, плодам цитрусовых и хурме, ежевике, и овощам, включая сахарную кукурузу, томаты, соевые бобы, фасоль лимскую и паприку, капусту, цветную капусту, репу, хрен, капусту кормовую, горчицу, брюссельскую капусту, картофель, баклажаны, бамию, бобы, спаржу, свеклу, сорняки, фруктовые деревья и полевые культуры, такие как полевая кукуруза и соевые бобы. Щитники также являются вредителями трав, сорго и риса.
Растение для применения в способах по настоящему изобретению или трансгенное растение в соответствии с настоящим изобретением охватывает любое растение, но предпочтительно является растением, которое является восприимчивым к нападению насекомых, патогенных для растения.
Соответственно, настоящее изобретение распространяется на растения и на способы, описываемые в данном описании, где растение выбирают из следующей группы растений (или сельскохозяйственных культур): люцерна, яблоня, абрикос, артишок, спаржа, авокадо, банан, ячмень, бобы, свекла, ежевика, черника, брокколи, брюссельская капуста, капуста, канола, морковь, маниок, цветная капуста, зерновые, сельдерей, вишня, цитрусы, клементины, кофе, кукуруза, хлопчатник, огурцы, баклажаны, эндивий, эвкалипт, инжир, виноград, грейпфрут, арахис, физалис, киви, латук, лук-порей, лимон, лайм, сосна, кукуруза, манго, дыня, просо, грибы, орехи, овес, бамия, лук, апельсин, декоративное растение или цветок, или дерево, папайя, петрушка, горох, персики, арахис, торфяной мох, перец, хурма, ананас, подорожник, слива, гранат, картофель, тыква, красный эндивий, редис, рапсовое семя, малина, рис, рожь, сорго, соя, соевые бобы, шпинат, клубника, сахарная свекла, сахарный тростник, подсолнечник, сладкий картофель, мандарин, чай, табак, томаты, виноград культурный, арбуз, пшеница, ямc и цукини.
В конкретных вариантах осуществления настоящее изобретение предусматривает гены-мишени, которые кодируют белки, участвующие в функции белка wings up А (тропонина I), субъединицы II митохондриальной цитохром с-оксидазы или одного из рибосомных белков, как указано в таблице 1.
В предпочтительных вариантах осуществления настоящее изобретение предусматривает гены-мишени, выбранные из группы генов, (i) имеющих нуклеотидную последовательность, содержащей любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (ii) имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или (iii) имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000, или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iv) имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28,, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 или комплементарной ей последовательности, или (v) имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или (vi) ген которой у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389;
и где нуклеотидная последовательность указанного гена составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
Аминокислотные последовательности, кодируемые генами-мишенями по настоящему изобретению, представлены SEQ ID NO:79, 349, 405, 352, 356, 80, 326, 81, 327, 82, 83, 328, 84, 329, 85, 86, 359, 87-91, 330, 350, 353, 331, 351, 332-336, 337, 354, 338-344, 346, 345, 347, 348, 357, 355, 358, 390-393.
Применяемый в данном описании термин "обладающий" имеет такое же значение, как "содержащий".
Применяемый в данном описании термин "идентичность последовательностей" используется для описания сходства последовательностей между двумя или более нуклеотидными или аминокислотными последовательностями. Процент "идентичности последовательностей" между двумя последовательностями определяется путем сравнения двух оптимально выровненных последовательностей в окне сравнения (определенное число положений), при этом часть последовательности в окне сравнения может содержать присоединения или делеции (т.е. пробелы) по сравнению с контрольной последовательностью с целью достижения оптимального выравнивания. Процент идентичности последовательностей рассчитывается путем определения числа положений, в которых идентичное нуклеотидное основание или аминокислотный остаток встречается в обеих последовательностях, с получением числа 'совпадающих' положений, деления числа совпадающих положений на общее число положений в окне сравнения и умножения результата на 100. Способы и программное обеспечение для определения идентичности последовательностей доступны в данной области и включают программное обеспечение Blast и GAP-анализ. Для нуклеиновых кислот, процент идентичности рассчитывают предпочтительно с помощью инструмента выравнивания BlastN, посредством чего процент идентичности рассчитывают по всей длине запросной нуклеотидной последовательности.
Специалисту в данной области будет понятно, что гомологи или ортологи (гомологи, существующие у различных видов) генов-мишеней, представленные любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, можно идентифицировать. Данные гомологи и/или ортологи вредителей также находятся в рамках настоящего изобретения. Предпочтительные гомологи и/или ортологи являются генами, подобными в нуклеотидной последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов гомолог и/или ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80% или 85%, более предпочтительно по меньшей мере на 90% или 95%, и наиболее предпочтительно по меньшей мере приблизительно 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности. Аналогично, также предпочтительными гомологами и/или ортологами являются белки, которые подобны в аминокислотной последовательности до такой степени, что при оптимальном выравнивании и сравнении двух аминокислотных последовательностей гомолог и/или ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80% или 85%, более предпочтительно по меньшей мере на 90% или 95%, и наиболее предпочтительно по меньшей мере приблизительно 99% идентичной любой из SEQ ID NO:79-91, 326-359, 390-395.
Другие гомологи являются генами, которые являются аллелями гена, содержащего последовательность, которая представлена любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176 , 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389. Дополнительно предпочтительными гомологами являются гены, содержащие по меньшей мере один одиночный нуклеотидный полиморфизм (SNP) по сравнению с геном, содержащим последовательность, которая представлена любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
'Интерферирующая рибонуклеиновая кислота (РНК)' по настоящему изобретению охватывает любой тип молекулы РНК, способной подавлять или подвергать 'сайленсингу' экспрессию гена-мишени, включая, но без ограничений, смысловую РНК, антисмысловую РНК, короткую интерферирующую РНК (siPHК), микроРНК (miPHК), двухцепочечную РНК (dsPHК) шпилечную РНК (RNA) и тому подобное. Способы анализа на функциональные молекулы интерферирующих РНК хорошо известны в данной области и описаны в данном описании в других местах.
Молекулы интерферирующей РНК по настоящему изобретению обеспечивают последовательность-специфическое подавление экспрессии гена-мишени путем связывания с целевой нуклеотидной последовательностью в пределах гена-мишени. Связывание происходит в результате спаривания оснований между комплементарными областями интерферирующей РНК и целевой нуклеотидной последовательности. Применяемый в данном описании термин 'сайленсирующий элемент' относится к части или области интерферирующей РНК, содержащей или состоящей из последовательности нуклеотидов, которая комплементарна или по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и которая функционирует как активная часть интерферирующей РНК с непосредственным подавлением экспрессии указанного гена-мишени. В одном варианте осуществления настоящего изобретения сайленсирующий элемент содержит или состоит из последовательности по меньшей мере из 17 последовательных нуклеотидов, предпочтительно по меньшей мере 18 или 19 последовательных нуклеотидов, более предпочтительно по меньшей мере 21 последовательного нуклеотида, еще более предпочтительно по меньшей мере 22, 23, 24 или 25 последовательных нуклеотидов, комплементарных целевой нуклеотидной последовательности в пределах гена-мишени.
Применяемое в данном описании выражение "экспрессия гена-мишени" относится к транскрипции и накоплению РНК-транскрипта, кодируемого геном-мишенью, и/или трансляции mРНК в белок. Термин 'подавлять' предназначен для обозначения любого из способов, известных в данной области, с помощью которого молекулы интерферирующей РНК снижают уровень первичных транскриптов РНК, mРНК или белка, полученного из гена-мишени. В определенных вариантах осуществления подавление относится к ситуации, когда уровень РНК или белка, полученного из гена, уменьшается по меньшей мере на 10%, предпочтительно по меньшей мере на 33%, более предпочтительно по меньшей мере на 50%, еще более предпочтительно по меньшей мере на 80%. В особенно предпочтительных вариантах осуществления подавление относится к снижению уровня РНК или белка, полученного из гена по меньшей мере на 80%, предпочтительно по меньшей мере на 90%, более предпочтительно по меньшей мере на 95% и наиболее предпочтительно по меньшей мере на 99% в клетках насекомого-вредителя по сравнению с соответствующим контрольным насекомым-вредителем, которое, например, не подвергалось воздействию интерферирующей РНК или подвергалось воздействию контрольной молекулы интерферирующей РНК. Способы выявления снижения уровней РНК или белка хорошо известны в данной области и включают гибридизацию РНК в растворе, нозерн гибридизацию, обратную транскрипцию (например, анализ количественной ОТ-ПЦР (RT-PCR)), микроматричный анализ, связывание антител, иммуноферментный твердофазный анализ (ELISA) и вестерн-блоттинг. В другом варианте осуществления настоящего изобретения подавление относится к снижению уровней РНК или белка, достаточному для получения выявляемого изменения фенотипа вредителя, по сравнению с соответствующей мерой борьбы с вредителями, например, гибелью клеток, прекращением роста или тому подобное. Подавление, таким образом, можно измерить с помощью фенотипического анализа насекомого-вредителя с использованием методов, обычных в данной области.
В предпочтительном варианте осуществления настоящего изобретения интерферирующая РНК подавляет экспрессию гена с помощью РНК-интерференции или PHКi. PHКi является процессом последовательность - специфической генной регуляции, как правило, опосредованной молекулами двухцепочечной РНК, такими как короткие интерферирующие РНК (siPHК). siPHК содержат смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК. Смысловая цепь или 'направляющая цепь' молекулы siPHК содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенных в пределах РНК-транскрипта гена-мишени. Смысловая цепь siPHК, следовательно, способна соединяться с РНК-транскриптом посредством спаривания оснований по Уотсон-Крику, и направлять РНК на деградацию в пределах клеточного комплекса, известного как индуцируемый РНК сайленсирующий комплекс или RISC. Таким образом, в отношении предпочтительных молекул интерферирующей РНК по настоящему изобретению сайленсирующий элемент, как изложено в данном описании, может являться двухцепочечной областью, содержащей соединенные комплементарные цепи, из которых по меньшей мере одна цепь содержит или состоит из последовательности нуклеотидов, комплементарной или по меньшей мере частично комплементарную целевой нуклеотидной последовательности в пределах гена-мишени. В одном варианте осуществления двухцепочечная область составляет в длину по меньшей мере 21, 22, 23, 24, 25, 30, 35, 40, 50, 55, 60, 70, 80, 90, 100, 125, 150, 175, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 пар оснований.
Более длинные двухцепочечные молекулы РНК (dsPHК), содержащие один или более функциональных двухцепочечных сайленсирующих элементов, которые описаны в данном описании в других местах, и способные к сайленсингу генов посредством PHКi, также рассматриваются в рамках настоящего изобретения. Такие более длинные молекулы dsPHК содержат по меньшей мере 80, 200, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 пар оснований. Данные молекулы dsPHК могут служить в качестве предшественников активных молекул siPHК, которые направляют РНК-транскрипт к комплексу RISC для последующей деградации. Молекулы dsPHК, присутствующие в среде, окружающей организм или его клетки, могут поглощаться организмом и перерабатываться ферментом, называемым Dicer, с получением молекул siPHК. Альтернативна, dsPHК может быть получена in vivo, т.е. транскрибирована из полинуклеотида или полинуклеотидов, кодирующих dsPHК, присутствующих в клетке, например, бактериальной клетке или растительной клетке, и впоследствии обработана с помощью Dicer, либо в пределах клетки-хозяина, либо предпочтительно в пределах клеток насекомого-вредителя после поглощения более длинной dsРНК-предшественника. dsPHК может образовываться из двух отдельных (смысловой и антисмысловой) цепей РНК, которые соединяются посредством комплементарного спаривания оснований. Альтернативно, dsPHК может являться одной цепью, способной складываться назад на себя с образованием шпилечной РНК (РНК) или структуры стебель-петля. В случае РНК, двухцепочечная область или 'стебель' образована из двух областей или сегментов РНК, которые являются существенно инвертированными повторами друг друга и обладают достаточной комплементарностью для обеспечения образования двухцепочечной области. Один или более функциональных двухцепочечных сайленсирующих элементов могут присутствовать в данной 'области стебля' молекулы. Области инвертированных повторов, как правило, разделены областью или сегментом РНК, известным как область "петли". Данная область может содержать любую нуклеотидную последовательность, придающую достаточную гибкость для обеспечения осуществления самоспаривания между фланкирующими комплементарными областями РНК. В общем, область петли является по существу одноцепочечной и действует как разделительный элемент между инвертированными повторами.
Все молекулы интерферирующей РНК по настоящему изобретению обеспечивают последовательность - специфическое подавление экспрессии гена-мишени путем связывания с целевой нуклеотидной последовательностью в пределах гена-мишени. Связывание происходит в результате комплементарного спаривания оснований между сайленсирующим элементом интерферирующей РНК и целевой нуклеотидной последовательностью. Молекулы интерферирующей РНК по настоящему изобретению содержат по меньшей мере один или по меньшей мере два сайленсирующих элемента. В одном варианте осуществления настоящего изобретения целевая нуклеотидная последовательность содержит последовательность нуклеотидов, которая представлена РНК-транскриптом гена-мишени или его фрагментом, где фрагмент составляет предпочтительно по меньшей мере 17 нуклеотидов, более предпочтительно по меньшей мере 18, 19 или 20 нуклеотидов или наиболее предпочтительно по меньшей мере 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 нуклеотидов. В предпочтительном варианте осуществления настоящего изобретения целевая нуклеотидная последовательность содержит последовательность нуклеотидов, эквивалентную РНК-транскрипту, кодируемому любым из полинуклеотидов, который выбирают из группы, включающей (i) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100 или 1115 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, или (ii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью,
или (iii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, так, что при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид являлся по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iv) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 2-42-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 3.18-321, 386, 387, 388, 389 или ее комплементарной последовательностью, и где указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-2-29, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности,
или (v) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, и где указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (vi) полинуклеотид, кодирующий аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389. В более предпочтительном варианте осуществления указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
Предпочтительно, молекулы интерферирующей РНК по настоящему изобретению содержат по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsPHК содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени.
Сайленсирующий элемент или по меньшей мере одна его цепь, где сайленсирующий элемент является двухцепочечным, может являться полностью комплементарным или частично комплементарным целевой нуклеотидной последовательности гена-мишени. Применяемый в данном описании термин "полностью комплементарный" означает, что все основания нуклеотидной последовательности сайленсирующего элемента являются комплементарными или 'соответствуют' основаниям целевой нуклеотидной последовательности. Термин "по меньшей мере частично комплементарный" означает, что существует менее чем 100% совпадения между основаниями сайленсирующего элемента и основаниями целевой нуклеотидной последовательности. Специалисту в данной области будет понятно, что сайленсирующий элемент должен являться только по меньшей мере частично комплементарным целевой нуклеотидной последовательности для опосредования подавления экспрессии гена-мишени. Как известно в данной области, последовательности РНК со вставками, делениями и нарушениями комплементарности относительно целевой последовательности все же могут являться эффективными при PHКi. В соответствии с настоящим изобретением предпочтительно, чтобы сайленсирующий элемент и целевая нуклеотидная последовательность гена-мишени обладали по меньшей мере 80% или 85% идентичностью последовательности, предпочтительно по меньшей мере 90% или 95% идентичностью последовательности или более предпочтительно по меньшей мере 97% или 98% идентичностью последовательности, и еще более предпочтительно по меньшей мере 99% идентичностью последовательности. Альтернативно, сайленсирующий элемент может содержать 1, 2 или 3 нарушения комплементарности по сравнению с целевой нуклеотидной последовательностью на каждом отрезке из 24 частично комплементарных нуклеотида.
Специалисту в данной области будет очевидным, что степень комплементарности между сайленсирующим элементом и целевой нуклеотидной последовательностью может варьировать в зависимости от гена-мишени, подлежащего подавлению, или в зависимости от вида насекомого-вредителя, у которого экспрессия гена подлежит контролю.
В другом варианте осуществления настоящего изобретения сайленсирующий элемент содержит последовательность нуклеотидов, которая является эквивалентом РНК любого из полинуклеотидов, выбранных из группы, включающей полинуклеотид, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100 или 1115 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, или (ii) полинуклеотид, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности так, что при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iii) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере из 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100,. 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и где указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью такой, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, где указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину. Следует понимать, что в таких вариантах осуществления сайленсирующий элемент может содержать или состоять из области двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых, смысловая цепь, содержит последовательность нуклеотидов по меньшей мере частично комплементарную целевой нуклеотидной последовательности в пределах гена-мишени.
Целевую нуклеотидную последовательность можно выбирать из любой подходящей области или нуклеотидной последовательности гена-мишени или его РНК-транскрипта. Например, целевая нуклеотидная последовательность может располагаться в пределах 5'UTR или 3'UTR гена-мишени или РНК транскрипта или в пределах экзонной или интронной областей гена.
Специалисту в данной области известны способы идентифицирования наиболее подходящих целевых нуклеотидных последовательностей, в рамках полноразмерного гена-мишени. Например, множество сайленсирующих элементов, воздействующих на различные области гена-мишени, можно синтезировать и тестировать. Альтернативно, расщепление РНК-транскрипта с помощью ферментов, таких как РНКаза Н, можно использовать для определения сайтов в РНК, которые находятся в конформации, восприимчивой к сайленсингу гена. Сайты-мишени также можно идентифицировать с применением in silico подходов, например, использование компьютерных алгоритмов, предназначенных для прогнозирования эффективности сайленсинга генов на основе воздействия на различные сайты в пределах гена полной длины.
Интерферирующие РНК по настоящему изобретению могут содержать один сайленсирующий элемент или несколько сайленсирующих элементов, где каждый сайленсирующий элемент содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах- гена-мишени, и которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии указанного гена-мишени. Конкатемерные РНК-конструкции данного типа описаны в WO 2006/046148, который включен в данное описание посредством ссылки. В контексте настоящего изобретения термин 'несколько' означает по меньшей мере два, по меньшей мере три, по меньшей мере четыре и т.д., и по меньшей мере до 10, 15, 20 или по меньшей мере 30. В одном варианте осуществления интерферирующая РНК содержит нескольких копий одного сайленсирующего элемента, т.е. повторов сайленсирующего элемента, который связывается с конкретной целевой нуклеотидной последовательностью в пределах определенного гена-мишени. В другом варианте осуществления сайленсирующие элементы в пределах интерферирующей РНК содержат или состоят из различных последовательностей нуклеотидов, комплементарных различным целевым нуклеотидным последовательностям. Должно быть ясно, что комбинации нескольких копий одного и того же сайленсирующего элемента, комбинированные с сайленсирующими элементами, связывающимися с различными целевыми нуклеотидными последовательностями, находятся в рамках настоящего изобретения.
Различные целевые нуклеотидные последовательности могут происходить из одного гена-мишени у вида насекомого-вредителя для достижения улучшения подавления конкретного гена-мишени у вида насекомого-вредителя. В этом случае сайленсирующие элементы можно комбинировать в интерферирующей РНК в исходном порядке, в котором целевые нуклеотидные последовательности встречаются в гене-мишени, или сайленсирующие элементы можно смешивать и комбинировать случайно в любом порядке ранжирования в рамках интерферирующей РНК по сравнению с порядком целевых нуклеотидных последовательностей в гене-мишени.
Альтернативно, различные целевые нуклеотидные последовательности представляют один ген-мишень, но происходят из различных видов насекомых-вредителей.
Альтернативно, различные целевые нуклеотидные последовательности могут происходить из различных генов-мишеней. Если интерферирующая РНК предназначена для использования для предупреждении и/или борьбы с нападением вредителей предпочтительно выбирать различные гены-мишени из группы генов, регулирующих жизненно важные биологические функции вида насекомого-вредителя, включая, но без ограничений, выживание, рост, развитие, размножение и патогенность. Гены-мишени могут регулировать одинаковые или различные биологические пути или процессы. В одном варианте осуществления по меньшей мере один из сайленсирующих элементов содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, где ген-мишень выбран из группы генов, как описано выше.
В дополнительном варианте осуществления настоящего изобретения различные гены, на которые воздействуют различные сайленсирующие элементы, происходят из одного вида насекомого-вредителя. Данный подход предназначен для достижения усиленного воздействия на один вид насекомого-вредителя. В частности, различные гены-мишени могут экспрессироваться дифференциально на различных стадиях жизненного цикла насекомого, например, зрелого имаго, незрелой личиночной и стадии яйца. Интерферирующую РНК по настоящему изобретению можно использовать для предотвращения и/или борьбы с нападением насекомых-вредителей на более чем одной-стадии жизненного цикла насекомого.
В альтернативном варианте осуществления настоящего изобретения различные гены, на которые воздействуют различные сайленсирующие элементы, происходят из различных видов насекомых-вредителей. Интерферирующую РНК по настоящему изобретению можно, таким образом, использовать для предотвращения и/или борьбы с нападением более чем одного вида насекомых-вредителей одновременно.
Сайленсирующие элементы могут располагаться в виде одной непрерывной области интерферирующей РНК или могут разделяться присутствием линкерной последовательности. Линкерная последовательность может содержать короткую случайную нуклеотидную последовательность, которая не комплементарна ни одной из целевых нуклеотидных последовательностей или генов-мишеней. В одном варианте осуществления линкер является при определенных условиях самостоятельно расщепляющейся последовательностью РНК, предпочтительно рН-чувствительным линкером или гидрофобно-чувствительным Линкером. В одном варианте осуществления линкер содержит последовательность нуклеотидов, эквивалентную интронной последовательности. Линкерные последовательности по настоящему изобретению могут находиться в диапазоне длины от приблизительно 1 пары оснований до приблизительно 10000 пар оснований, при условии, что линкер не ухудшает способности интерферирующей РНК подавлять экспрессию гена-мишени (генов-мишеней).
В дополнение к сайленсирующему элементу (элементам) и любым линкерным последовательностям интерферирующая РНК по настоящему изобретению может содержать по меньшей мере одну дополнительную полинуклеотидную последовательность. В различных вариантах осуществления настоящего изобретения дополнительную последовательность выбирают из (i) последовательности, способной защитить интерферирующую РНК от РНК процессинга, (ii) последовательности, влияющей на стабильность интерферирующей РНК, (iii) последовательности, обеспечивающей связывание белков, например, для облегчения поглощения интерферирующей РНК клетками вида насекомого-вредителя, (iv) последовательности, облегчающей крупномасштабное производство интерферирующей РНК, (v) последовательности, которая является аптамером, который связывается с рецептором или молекулой на поверхности клеток насекомого-вредителя для облегчения поглощения, или (v) последовательности, которая катализирует процессинг интерферирующей РНК в клетках насекомого-вредителя и тем самым повышает эффективность интерферирующей РНК. Структуры для повышения стабильности молекулы РНК хорошо известны в данной области и описаны дополнительно в WO 2006/046148, которая включена в данное описание посредством ссылки.
Длина интерферирующей РНК по настоящему изобретению должна являться достаточной для поглощения клетками вида насекомого-вредителя и подавления генов-мишеней у вредителя, как описано в данном описании в других местах. Однако верхний предел длины может зависеть от (i) требования к интерферирующей РНК поглощаться клетками вредителя, и (ii) требования к интерферирующей РНК подвергаться процессингу в клетках вредителя для опосредования сайленсинга генов путем PHКi. Длина также может зависеть от способа получения и состава для доставки интерферирующей РНК к клеткам. Предпочтительно, интерферирующая РНК по настоящему изобретению составляет от 21 до 10000 нуклеотидов в длину, предпочтительно от 50 до 5000 нуклеотидов или от 100 до, 2500 нуклеотидов, более предпочтительно от 90 до 2000 нуклеотидов в длину.
Интерферирующая РНК может содержать основания ДНК, неприродные основания или неприродные связи остова, или модификации сахарофосфатного остова, например, для повышения стабильности во время хранения или повышения устойчивости к деградации нуклеазами. Дополнительно, интерферирующая РНК может быть получена химически или ферментативно специалистом в данной области с помощью управляемых вручную или автоматизированных реакций. Альтернативно, интерферирующая РНК может транскрибироваться из полинуклеотида, кодирующего таковую. Таким образом, в настоящем описании предусматривается выделенный полинуклеотид, кодирующий любую из интерферирующих РНК по настоящему изобретению.
Также в настоящем описании предусматривается выделенный полинуклеотид, выбранный из группы, включающей (i) полинуклеотид, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, или (ii) полинуклеотид, который содержит по меньшей мере 21 предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100 или 1115 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214.-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или ее комплементарной последовательностью, или (iii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450,- 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности так, что при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iv) полинуклеотид, который содержит фрагмент по меньшей мере из 21 предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и где указанный фрагмент или указанная комплементарная последовательность обладает такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 или комплементарной ей последовательности, или (v) полинуклеотид, который содержит фрагмент по меньшей мере из 21 предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и где указанный фрагмент или указанная комплементарная последовательность обладает такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (vi) полинуклеотид, кодирующий аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, и где указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
В предпочтительных вариантах осуществления выделенный полинуклеотид является частью молекулы интерферирующей РНК, как правило, частью сайленсирующего элемента, содержащего по меньшей мере одну двухцепочечную область, содержащую смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsPHК содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени. Смысловая цепь dsPHК, следовательно, способна соединяться с РНК-транскриптом и воздействовать на РНК для деградации в пределах PHКi-индуцируемого сайленсирующего комплекса или RISC.
Полинуклеотиды по настоящему изобретению можно вставлять с помощью рутинных методов молекулярного клонирования в ДНК-конструкции или векторы, известные в данной области. Следовательно, в соответствии с одним вариантом осуществления предусматривается ДНК-конструкция, содержащая любые из полинуклеотидов по настоящему изобретению. Предпочтительно, в данном описании предусматривается ДНК-конструкция, содержащая полинуклеотид, кодирующий по меньшей мере одну из интерферирующих РНК по настоящему изобретению. ДНК-Конструкция может являться рекомбинантным ДНК-вектором, например, бактериальным или дрожжевым вектором, или растительным вектором. В предпочтительном варианте осуществления настоящего изобретения ДНК-конструкция является экспрессирующей конструкцией, и полинуклеотид функционально связан с по меньшей мере одной регуляторной последовательностью, способной управлять экспрессией полинуклеотидной последовательности. Термин 'регуляторная последовательность' следует рассматривать в широком контексте, и он предназначен для обозначения любой нуклеотидной последовательности, способной воздействовать на экспрессию полинуклеотидов, с которыми она функционально связана, включая, но без ограничений, промоторы, энхансеры и другие природные или синтетические элементы-активаторы транскрипции. Регуляторная последовательность может располагаться на 5'- или 3'-конце полинуклеотидной последовательности. Термин 'функционально связанный' относится к функциональной связи между регуляторной последовательностью и полинуклеотидной последовательностью, такой, что регуляторная последовательность управляет экспрессией полинуклеотида. Функционально связанные элементы могут являться смежными или несмежными.
Предпочтительно, регуляторная последовательность является промотором, выбранным из группы, включающей, но без ограничений, конститутивные промоторы, индуцируемые промоторы, тканеспецифические промоторы и промоторы, специфичные для стадии роста/развития. В одном варианте полинуклеотид находится под контролем сильного конститутивного промотора, такого как любой, выбранный из группы, включающей CaMV35S промотор, двойной CaMV35S промотор, убиквитиновый промотор, актиновый промотор, rubisco промотор, GOS2 промотор, промотор 34S вируса мозаики норичника. В другом варианте осуществления регуляторная последовательность является растительным промотором для использования в регуляции экспрессии полинуклеотида в растениях. Растительные промоторы, в частности, тканеспецифические растительные промоторы, охватываемые объемом настоящего изобретения, описаны более подробно в данном описании в других местах.
Необязательно одну или более последовательностей терминации транскрипции можно включать в экспрессирующую конструкцию по настоящему изобретению. Термин 'последовательность терминации транскрипции' охватывает управляющую последовательность в конце транскрипционной единицы, которая сигнализирует о терминации транскрипции, 3' процессинге и полиаденилировании первичного транскрипта. Дополнительные регуляторные последовательности, включая, но без ограничений, энхансеры транскрипции или трансляции, можно включать в экспрессионную конструкцию, например, как с усиленным двойным промотором CaMV35S.
Настоящее изобретение также охватывает способ создания любой из интерферирующих РНК по настоящему изобретению, включающий стадии: (i) контакта полинуклеотида, кодирующего указанную интерферирующую РНК, или ДНК-конструкции, содержащей полинуклеотид, с бесклеточными компонентами; или (ii) введения в клетку (например, путем трансформации, трансфекции или инъекции) полинуклеотида, кодирующего указанную интерферирующую РНК, или ДНК-конструкции, содержащей полинуклеотид.
Настоящее изобретение, таким образом, также относится к любому двухцепочечному рибонуклеотиду, полученному при экспрессии полинуклеотида, описанного в данном описании.
Соответственно, также в данном описании предусматривается клетка-хозяин, трансформированная любым из полинуклеотидов, описанных в данном описании. Дополнительно охватываются настоящим клетки-хозяева, содержащие любую из интерферирующих РНК по настоящему изобретению, любой из полинуклеотидов по настоящему изобретению или ДНК-конструкцию, содержащую любой из полинуклеотидов. Клетка-хозяин может являться прокариотической клеткой, включая, но без ограничений, грамположительные и грамотрицательные бактериальные клетки, или эукариотической клеткой, включая, но без ограничений, клетки дрожжей или клетки растений. Предпочтительно, указанная клетка-хозяин является бактериальной клеткой или растительной клеткой. Бактериальную клетку можно выбирать из группы, включающей, но без ограничений, грамположительные и грамотрицательные клетки, включающие Escherichia spp. (например, Е. coli), Bacillus spp. (например, В. thuringiensis), Rhizoblum spp., Lactobacillus spp., Lactococcus spp., Pseudomonas spp. и Agrobacterium spp. Полинуклеотид или ДНК-конструкция по настоящему изобретению может существовать или поддерживаться в клетке-хозяине как в не хромосомный элемент, или может являться стабильно включенным в геном клетки-хозяина. Характеристики, представляющие особый интерес при выборе клетки-хозяина для целей настоящего изобретения, включают простоту, с которой полинуклеотид или ДНК-конструкцию, кодирующую интерферирующую РНК, можно ввести в хозяина, доступность совместимых экспрессионных систем, эффективность экспрессии и стабильность интерферирующей РНК в хозяине.
Предпочтительно, интерферирующие РНК по настоящему изобретению экспрессируются в растительных клетках-хозяевах. Предпочтительные растения, представляющие интерес, включают, но без ограничений, хлопчатник, картофель, рис, томат, канола, сою, подсолнечник, сорго, просо, кукурузу, люцерну, землянику, баклажаны, перец и табак.
В ситуациях, где интерферирующая РНК экспрессируется в клетке-хозяине и/или применяется для предотвращения и/или борьбы с нападением вредителя на организм-хозяин, предпочтительно, чтобы интерферирующая РНК не демонстрировала значительных 'нецелевых' эффектов, т.е. интерферирующая РНК не влияла на экспрессию генов в хозяине. Предпочтительно, сайленсирующий элемент не демонстрирует значительной комплементарности с нуклеотидными последовательностями, кроме заданной целевой нуклеотидной последовательности гена-мишени. В одном варианте осуществления настоящего изобретения сайленсирующий элемент демонстрирует менее 30%, более предпочтительно менее 20%, более предпочтительно менее 10% и еще более предпочтительно менее 5% идентичность последовательности с любым геном клетки-хозяина или организма. Если известны данные геномной последовательности для организма-хозяина, можно перепроверить идентичность с сайленсирующим элементом, применяя стандартные инструменты биоинформатики. В одном варианте осуществления отсутствует идентичность последовательности между сайленсирующим элементом и геном из клетки-хозяина или организма-хозяина в области из 17, более предпочтительно, в области из 18 или 19 и наиболее предпочтительно, в области из 20 или 21 последовательного нуклеотида.
При практическом применении настоящего изобретения интерферирующие РНК по настоящему изобретению можно использовать для предотвращения и/или борьбы с любым насекомым вредителем, принадлежащим к отрядам Coleoptera, Lepidoptera, Diptera, Dichyoptera, Orthoptera, Hemiptera и Siphonaptera.
Также в данном описании предусматривается способ предотвращения и/или борьбы с нападением вредителей, при котором вид насекомого-вредителя контактирует с эффективным количеством по меньшей мере одной интерферирующей РНК, где РНК функционирует при поглощении указанным вредителем с подавлением экспрессии жизненно важного гена-мишени вредителя. Жизненно важным геном-мишенью может являться любой ген вредителя, участвующий в регуляции жизненно важного биологического процесса, необходимого вредителю для инициирования или поддержания нападения, включая, но без ограничений, выживание, рост, развитие, размножение и патогенность. В частности, ген-мишень может являться любым из генов вредителя, как описано в данном описании в других местах.
Кроме того, в данном описании предусматривается способ предотвращения и/или борьбы с нападением вредителей в поле культурных растений, где указанный способ включает экспрессию в указанных растениях эффективного количества интерферирующей РНК, описываемой в данном описании.
В способе, предназначенном для борьбы с нападением вредителя, фраза 'эффективное количество' распространяется на количество или концентрацию интерферирующей РНК, необходимые для получения фенотипического эффекта у вредителя таким образом, чтобы уменьшались количества организмов-вредителей, поражающих организм-хозяин, и/или уменьшалось количество повреждений, вызванных вредителем. В одном варианте осуществления фенотипическим эффектом является гибель вредителя, и интерферирующая РНК используется для достижения по меньшей мере 20%, 30%, 40%, предпочтительно по меньшей мере 50%, 60%, 70%, более предпочтительно, по меньшей мере 80% или 90% смертности вредителей по сравнению с контрольными насекомыми-вредителями. В дополнительном варианте осуществления, фенотипические эффекты включают, но без ограничений, остановку роста вредителя, прекращение питания и уменьшение яйцекладки. Общее количество организмов-вредителей, поражающих организм-хозяин, можно таким образом уменьшить по меньшей мере на 20%, 30%, 40%, предпочтительно по меньшей мере на 50%, 60%, 70%, более предпочтительно по меньшей мере на 80% или 90% по сравнению с контрольными вредителями. Альтернативно, ущерб, наносимый насекомыми-вредителями, можно уменьшить по меньшей мере на 20%, 30%, 40%, предпочтительно по меньшей мере на 50%, 60%, 70%, более предпочтительно по меньшей мере на 80% или 90% по сравнению с контрольными насекомыми-вредителями. Таким образом, способ по настоящему изобретению можно применять для достижения по меньшей мере 20%, 30%, 40%, предпочтительно по меньшей мере 50%, 60%, 70%, более предпочтительно по меньшей мере 80% или 90% борьбы с вредителями.
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, или 310-313 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, или 310-313 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp.(например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук),
где ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO:79, 349, 405, 352 или 356 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:141, 11, 12, 47-50 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:141, 11, 12, 47-50 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), где ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO:328 или 84 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарный по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:17, 18, 59-62 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полуже.сткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:17, 18, 59-62 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. Persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), где ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в SEQ ID NO:87 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:19, 20, 63-66 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:19, 20, 63-66 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp.(например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. Persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), где ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в SEQ ID NO:88 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:165, 167, 166, 270-273 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. Persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук). В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:165, 167, 166, 270-273 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), где ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO:347 или 348 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, М. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук),
при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO:330, 350 или 353 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:145, 122, 144, 178, 131, 179, 210-213, 290-293 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:145, 122, 144, 178, 131, 179, 210-213, 290-293 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO:331 или 351 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:128, 149, 184, 137, 185, 234-237, 302-305 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном описании, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO:128, 149, 184, 137, 185, 234-237, 302-305 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO:337 или 354 (при оптимальном выравнивании указанных кодируемых белков).
В одном варианте осуществления растение, подлежащее обработке, сконструировано для экспрессии интерферирующей РНК внутриклеточно, посредством транскрипции с полинуклеотида, включенного в него. По мере того как вредитель питается тканями растения, клетки, содержащие интерферирующую РНК, расщепляются внутри пищеварительного тракта насекомого, и интерферирующая РНК, таким образом, распределится внутри тела насекомого, приводя к подавлению генов-мишеней.
Таким образом, в соответствии с другим аспектом настоящего изобретения предусматривается способ создания трансгенного растения, устойчивого к нападению вида насекомого-вредителя, включающий стадии: (а) трансформацию растительной клетки ДНК-конструкцией, содержащей полинуклеотидную последовательность, кодирующую интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного вида насекомого-вредителя, (b) регенерацию растения из трансформированной растительной клетки, и (с) выращивание трансформированного растения в условиях, подходящих для экспрессии интерферирующей РНК из рекомбинантной ДНК-конструкции, при этом указанное растение, таким образом, является устойчивым к указанному вредителю по сравнению с нетрансформированным растением.
Интерферирующая РНК, экспрессируемая растением или его частью, может являться любой из тех, которые раскрыты в других местах в данном описании. Предпочтительно, интерферирующая РНК содержит или состоит по меньшей мере из одного сайленсирующего элемента, и указанный сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых (смысловая цепь) содержит последовательность нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени. Там, где часть интерферирующей РНК является двухцепочечной, две цепи молекулы могут экспрессироваться по меньшей мере из двух отдельных полинуклеотидов, или могут кодироваться одним полинуклеотидом, кодирующим интерферирующую РНК с помощью, например, структуры типа стебель-петля или так называемой шпилечной структуры, описываемой в других местах в данном описании.
Интерферирующая РНК, экспрессируемая растением или его частью, может воздействовать на любые из генов вредителя, описываемые в других местах в данном описании. В частности, ген-мишень можно выбирать из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1-26, 121-205, 386-389, 394, 400 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325; 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204,16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401,- 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности. Ген-мишень также может являться у насекомого-вредителя ортологом гена, который обладает нуклеотидной последовательностью, содержащей любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 402, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
В одном варианте осуществления ген-мишень:
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233.
В одном варианте осуществления ген-мишень:
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%,. 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273.
В одном варианте осуществления ген-мишень:
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, или 401 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, что при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 или комплементарной ей последовательности, так, что при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или 401 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO:1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 401.
Предпочтительно, нуклеотидная последовательность указанного гена-мишени составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину. Кроме того, важно, чтобы интерферирующая РНК не нарушала экспрессии какого-либо гена растения-хозяина.
Применяемый в данном описании термин 'трансгенное растение' или 'трансгенная растительная клетка' относится к любому растению или растительной клетке, которые созданы методами генетической инженерии, или происходят от растения, которое создано методами генетической инженерии таким образом, чтобы нести экзогенную полинуклеотидную последовательность. 'Экзогенный' относится к тому факту, что полинуклеотид происходит из-за пределов растительной клетки. Как правило, экзогенный полинуклеотид не является присущим для трансгенного растения, т.е. он не обнаруживается в природных условиях в пределах генома растения.
Применяемый в данном описании термин 'трансформация' относится к введению экзогенных полинуклеотидных молекул в растение или его клетку. Методы введения полинуклеотидов в растения известны в данной области. В одном варианте осуществления настоящего изобретения растения являются 'стабильно трансформированными', с помощью полинуклеотида или ДНК-конструкции, содержащей полинуклеотид, т.е. полинуклеотид или ДНК-конструкция, введенная в растительную клетку, интегрируется в геном растения и способна наследоваться его потомством. Протоколы трансформации для введения полинуклеотидов или ДНК-конструкций в клетки растений могут варьировать в зависимости от вовлеченного вида растения. Подходящие способы трансформация включают, но без ограничений, микроинъекцию, электропорацию, Agrobacterium-опосредованную трансформацию и баллистическое ускорение частиц. Способы также известны в данной области для целенаправленного встраивания полинуклеотида или ДНК-конструкции в специфическом положении в геном растения с использованием сайт-специфических систем рекомбинации.
ДНК-конструкция, содержащая полинуклеотид, кодирующий активную молекулу интерферирующей РНК, может являться любым вектором, подходящим для трансформации растительных клеток. Подходящие векторы включают, но без ограничений, бактериальные плазмиды, например Ti-плазмида Agrobacterium tumefaciens, и вирусные векторные системы. ДНК-конструкция, вводимая в клетки растения, не должна являться вредной или токсичной для растения, и/или не должна являться вредной или токсичной для любых организмов выше по цепи питания, которые питаются указанными растениями.
В одном варианте осуществления ДНК-конструкция является экспрессирующей конструкцией, содержащей полинуклеотид, кодирующий интерферирующую РНК, функционально связанную с регуляторной последовательностью, способной управлять экспрессией полинуклеотидной последовательности в растениях, такой как любая, выбранная из группы, содержащей CaMV35S промотор, двойной CaMV35S промотор, убиквитиновый промотор, актиновый промотор, rubisco промотор, GOS2 промотор, промотор 34S вируса мозаики норичника и усиленный двойной CaMV35S промотор. Предпочтительно, регуляторная последовательность является растительным промотором, выбранным из тех, которые известны в данной области. В некоторых вариантах осуществления может являться предпочтительным, чтобы растение продуцировало молекулы интерферирующей РНК только в тех частях растения, которые вступают в контакт с и/или повреждаются видом насекомого-вредителя, например, надземные части растения, корни и т.д. Данный эффект можно достичь посредством использования тканеспецифических растительных промоторов, включая, но без ограничений, промоторы, специфические для листьев, промоторы, специфические для корней, промоторы, специфические для стебля, промоторы, специфические для цветка и промоторы, специфические для плода, известны в данной области. Подходящими примерами промоторов, специфических для корней, являются PsMTA и промотор хитиназы класса III. Примерами специфических для листьев и специфических для стебля, или специфических для фотосинтетических тканей промоторов, которые также являются фотоактивируемыми, являются промоторы двух хлорофилл-связывающих белков (cab1 и cab2) из сахарной свеклы, рибулозобисфосфаткарбоксилазы (Rubisco), кодируемой rbcS, A (gapА) и В (gapВ) субъединиц глицеральдегид-3-фосфатдегидрогеназы хлоропластов, промотор гена из Solanum tuberosum, кодирующего специфический для листьев и стебля (ST-LS1) белок, стебель-регулируемых (stem-regulated), индуцируемых защитой генов как, например, JAS промоторы, промоторы, специфические для цветка, такие как промотор халконсинтазы, и промоторы, специфические для плода, такие как RJ39 из земляники.
В других вариантах осуществления может являться предпочтительным, чтобы растение продуцировало молекулы интерферирующей РНК только на определенной стадии его роста. Данный эффект можно достичь посредством использования специфических для развития растительных промоторов, которые управляют экспрессией только в течение определенных периодов развития растения. В частности, является важным защищать растения от нападения вредителей на ранних стадиях роста растения или во время цветения (например, в случае риса), или во время плодоношения или созревания плодов, или залива зерна, так как это является временем, когда растение может наиболее серьезно повреждаться.
ДНК-конструкция для использования в трансформации растения в соответствии с настоящим способом, может содержать более одного полинуклеотида, кодирующего молекулу интерферирующей РНК по настоящему изобретению. В одном варианте осуществления различные полинуклеотиды могут кодировать молекулы интерферирующих РНК, воздействующие на различные нуклеотидные последовательности в пределах одного гена-мишени. В дополнительном варианте осуществления различные полинуклеотиды могут кодировать молекулы интерферирующих РНК, воздействующие на различные нуклеотидные последовательности в пределах различных генов-мишеней, где различные гены-мишени происходят из одного или различных видов насекомых-вредителей. Там, где ДНК-конструкция кодирует более одной интерферирующей РНК, данные РНК могут экспрессироваться дифференциально в различных тканях растения в силу того, что находятся под контролем различных тканеспецифических промоторных последовательностей, описываемых в других местах в данном описании. В одном варианте осуществления растение сконструировано для экспрессии интерферирующей РНК в листьях, которая подавляет экспрессию гена-мишени у насекомого, которое питается листьями, и для дополнительной экспрессии интерферирующей РНК в корнях, которая подавляет экспрессию гена-мишени у насекомого, которое заселяет почву и питается корнями растений.
ДНК-конструкция может также содержать по меньшей мере один дополнительный полинуклеотид, представляющий интерес, например, полинуклеотид, кодирующий дополнительную регуляторную молекулу РНК, полинуклеотид, кодирующий белок, токсичный для вида насекомого-вредителя, и/или полинуклеотид, кодирующий белок, придающий устойчивость или стойкость к гербицидам.
В соответствии с настоящим способом растение регенерируют из трансформированной растительной клетки с использованием методов, известных в данной области. Один из таких методов включает ферментативное расщепление стенки растительной клетки с получением протопластов растения, которые впоследствии могут подвергаться нескольким циклам деления и дифференцировки клеток с получением взрослого растения. Взрослые растения, полученные таким образом, можно впоследствии тестировать на устойчивость к нападению вредителей. Термин 'устойчивый', применяемый в данном описании, следует толковать широко, он относится к способности растения защищаться от нападения вредителя, который, как правило, способен нанести повреждения или ущерб растению. Под устойчивым может подразумеваться, что растение более не является восприимчивым к нападению вредителя, или что любые симптомы заболевания, обусловленные нападением вредителя, снижаются по меньшей мере примерно на 20%, предпочтительно по меньшей мере на 30%, 40% или 50%, более предпочтительно по меньшей мере на 60%, 70% или 80% и наиболее предпочтительно по меньшей мере на 90%. Методы измерения устойчивости растения к видам насекомых-вредителей общеизвестны в данной области и включают, но без ограничений, измерение с течением времени среднего диаметра поражения, биомассы или веса вредителей, выживаемость и/или смертность вредителей, и/или общий процент разрушенных тканей растения.
В одном варианте осуществления настоящий способ получения трансгенного растения также включает стадию, на котором получают отпрысков или потомство трансгенного растения и тестируют потомство на устойчивость к насекомым-вредителям. Можно получить два или более поколений для подтверждения того, что экспрессия признака устойчивости стабильно поддерживается и наследуется. Семена также можно собирать от родительского трансгенного растения и/или его потомства для тестирования на устойчивость к насекомым-вредителям.
Также настоящим изобретением охватывается способ получения трансгенных растений, устойчивых к нападению вида насекомого-вредителя, включающий стадии скрещивания первого трансгенного растения, несущего ДНК-конструкцию, кодирующую интерферирующую РНК, которая функционирует с подавлением экспрессии гена-мишени вредителя, со вторым растением, у которого отсутствует указанная ДНК-конструкция, и отбора потомства, устойчивого к указанному вредителю. Скрещивание можно осуществлять любыми способами, обычно используемыми в сфере селекции растений. Потомство, отобранное по устойчивости к вредителям, можно дополнительно самоопылять или 'подвергать самоопылению' для получения последующего поколения, устойчивого к вредителю потомства. В одном варианте осуществления несколько циклов самоопыления или самоопыления можно осуществлять для получения 2, 3, 4, 5 или более поколений потомства. Полученное потомство можно тестировать на устойчивость к вредителям для подтверждения того, что экспрессия признака устойчивости стабильно поддерживается и наследуется.
В дополнительном варианте осуществления любое устойчивое к вредителю потомство растений, полученное от скрещивания первого трансгенного родительского растения, несущего ДНК-конструкцию, представляющую интерес, со вторым родительским растением, у которого отсутствует указанная ДНК-конструкция, можно подвергать возвратному скрещиванию со вторым родительским растением и тестировать последующее потомство на устойчивость к нападению вредителя. Растения или их потомство можно тестировать на устойчивость к нападению вредителя, либо с помощью фенотипического анализа, описываемого в других местах в данном описании, либо с помощью стандартных молекулярных методов. Например, устойчивые к вредителям растения можно идентифицировать путем обнаружения присутствия полинуклеотидной последовательности, кодирующей интерферирующую РНК, которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени. Методы обнаружения присутствия специфических полинуклеотидных последовательностей в клетках известны в данной области и включают ПЦР, ферментативное расщепление и SNP анализ.
Способы по настоящему изобретению можно применять для получения 'многоуровневых трансгенных' растений, которые устойчивы к виду насекомого-вредителя, и которые необязательно обладают по меньшей мере одним дополнительным желательным признаком. Применяемое в данном описании 'многоуровневое' трансгенное растение относится к растению, которое несет более одной экзогенной полинуклеотидной последовательности. Фраза 'более одного' относится к возможности растения нести по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 экзогенных полинуклеотида. В одном варианте осуществления растительная клетка, трансформированная с помощью ДНК-конструкции, кодирующей интерферирующую РНК, воздействующую на ген вредителя, может являться предварительно сконструированной с содержанием отдельного экзогенного полинуклеотида. Альтернативно, способ получения трансгенного растения из растительной клетки, описываемый в данном описании, может включать протокол котрансформации, где ДНК-конструкция, кодирующая интерферирующую РНК по настоящему изобретению, доставляется в растительную клетку одновременно или последовательно с отдельным экзогенным полинуклеотидом.
Многоуровневые трансгенные растения, демонстрирующие устойчивость к вредителям, также можно получать с помощью стандартных методов селекции растений. В одном варианте осуществления первое устойчивое к вредителям трансгенное растение скрещивают со вторым растением, сконструированным для экспрессии экзогенного полинуклеотида или гетерологичного гена, придающего желаемый признак растения. Любое полученное потомство можно тестировать на устойчивость к вредителям и приобретение дополнительного желаемого признака. Альтернативно или в дополнение, любое устойчивое к вредителям потомство, полученное от скрещивания, можно подвергать возвратному скрещиванию со вторым родителем с целью получения дальнейшего потомства, которое можно отбирать по наследованию гетерологичного гена, переносимого вторым родителем, и тем самым, дополнительного желаемого признака растения. Экзогенные полинуклеотиды, которые несет многоуровневое трансгенное растение по настоящему изобретению, могут экспрессироваться в тех же частях растения или могут экспрессироваться дифференциально в силу того, что экспрессия каждого контролируется с помощью отличного тканеспецифического промотора.
В одном варианте осуществления экзогенный полинуклеотид или гетерологичный ген, придающий дополнительный желаемый признак, кодирует другую интерферирующую РНК, воздействующую на тот же самый или другой вид насекомого-вредителя. В дополнительном варианте осуществления гетерологичный ген кодирует белок, вредный или токсичный для вида насекомого-вредителя растения, например, инсектицидный белок, выбранный из группы, включающей, но без ограничений, инсектицидные белки Bacillus thuringlensis, инсектицидные белки Xenorhabdus, инсектицидные белки Photorhabdus, инсектицидные белки Bacillus laterosporous, инсектицидные белки Bacillus sphaericus иинсектицидные белки VIP, такие как белок, выбранный из группы, включающей, но без ограничений Cry1Ab, Cry1C, Cry2Aa, Cry3, CryET70, Cry22, CryЕТ33, CryET34, CryET80, CryET76, TIC100, TIC101, TIC851, TIC900, TIC901, TIC1201, TIC407, TIC417 и инсектицидные белки PS149B1. В еще одном дополнительном варианте осуществления гетерологичный ген кодирует белок, придающий устойчивость или стойкость к гербицидам. Примеры генов, придающих устойчивость или стойкость к гербицидам включают Bar, EPSPS, который придает устойчивость к глифосату, AL5, который придает устойчивость к имидазолинону, и сульфонилмочевине, и bxn, который придает устойчивость к бромоксинилу.
Также в данном описании предусматривается способ получения гибридных семян от любого из трансгенных растений, полученных с помощью способов по настоящему изобретению, где указанный способ включает стадии: (i) посадки семян, полученных от первого инбредного растения, и семян, полученных от второго инбредного растения, где по меньшей мере, одно из инбредных растений является трансгенным растением, устойчивым к нападению вредителя, (ii) выращивания семян в растения, которые имеют цветы, (iii) предотвращения самоопыления по меньшей мере одного из первого или второго взрослых растений, (iv) предоставления возможности перекрестного опыления между первым и вторым растениями; и (v) сбор семян, полученных в результате перекрестного опыления. Гибридное семя, полученное данным способом, и гибридные растения, полученные путем культивирования указанных семян, находятся в пределах объема настоящего изобретения. Гибридные растения, полученные данным способом, как правило, являются генетически однородными, и вероятно продемонстрируют гетерозис или гибридную силу. Таким образом, сельскохозяйственные культуры с потенциалом к повышению урожайности можно получить с помощью такого способа.
В группу трансгенных растений по настоящему изобретению включены трансгенные растения, полученные любым из способов, описываемых в данном описании. Таким образом, в одном варианте осуществления настоящего изобретения трансгенные растения включают многоуровневые трансгенные признаки, несущие первый экзогенный полинуклеотид, придающий устойчивость к вредителям, и по меньшей мере один дополнительный экзогенный полинуклеотид или гетерологичный ген, придающий дополнительный желаемый признак растения. Дополнительные гетерологичные гены могут содержать гены, кодирующие дополнительные пестицидные средства, гены, кодирующие белки, токсичные или вредные для видов насекомых-вредителей, и/или гены, кодирующие белки, придающие устойчивость к гербицидам, как описано в других местах в данном описании.
Предпочтительные трансгенные растения в соответствии с настоящим изобретением, включают, но без ограничений, хлопчатник, картофель, рис, томат, канола, сою, подсолнечник, сорго, просо, кукурузу, люцерну, землянику, баклажан, перец и табак.
Также в данном описании предусматривается применение интерферирующей рибонуклеиновой кислоты (РНК), описываемой в данном описании, или ДНК-конструкции, описываемой в данном описании, для предотвращения и/или борьбы с нападением насекомых-вредителей, предпочтительно нападением насекомых-вредителей на растения.
Настоящее изобретение будет дополнительно понятно со ссылкой на следующие неограничивающие примеры. Примеры
Пример 1 Идентификация генов-мишеней у видов насекомых вредителей
1.1. Lygus hesperus нормализованная библиотека сДНК и получение dsPHК в многолуночных планшетах для скрининговых исследований
Нуклеиновые кислоты выделяли из нимф Lygus hesperus различных стадий жизни, включая только что вылупившихся нимф 2, 4, 6 и 9 дневных нимф и взрослых. Библиотеку сДНК получали с использованием ПЦР-набора для синтеза сДНК SMARTer™, следуя инструкциям производителя (Clontech Cat. No 634925). Библиотеку сДНК нормализовали с использованием Trimmer kit (Evrogen Cat No NK001) и клонировали в PCR4-TOPO вектор (Invitrogen). Нормализация введенных в клоны М2 адаптеров (Trimmer Kit, Evrogen, SEQ ID NO:92: AAGCAGTGGTATCAACGCAG), противоположно ориентированных на каждом конце клонов. Рекомбинантные конструкции векторов трансформировали в клетки Escherichia coil штамма ТОР10 (Invitrogen). Трансформированные клетки затем разбавляли и высевали с целью получения отдельных колоний или клонов. Клоны проверяли для подтверждения того, что избыточность клонов для библиотеки не превышает 5%. Отдельные клоны отбирали в жидкую LB (бульон Лурия) среду, в 96-луночные планшеты с глубокими лунками, и выращивали в течение ночи при 37°С. Планшеты также включали положительные (Lh423) и отрицательные (FP) контрольные клоны.
Для создания dsPHК, смысловой и антисмысловой фрагменты ДНК, содержащие последовательность промотора Т7, создавали с помощью ПЦР. Кратко, на клон 1 мкл бактериальной суспензии помещали в многолуночные ПЦР-планшеты, содержащие REDTaq® (Sigma Cat No D4309) и праймеры OGCC2738 (SEQ ID NО:93: AAGCAGTGGTATCAACGCAG) и OGCC2739 (SEQ ID NО:94: GCGTAATACGACTCACTATAGGAAGCAGTGGTATCAACGCAG) на основе М2 и Т7-М2 последовательностей, соответственно. За ПЦР-реакцией следовала in vitro транскрипция, где на клон 6 мкл продукта ПЦР добавляли к 9 мкл RiboMAX™ Large Scale РНК Production System-T7 (Promega Cat No P1300) и инкубировали в течение ночи при 37°С. Конечный раствор dsPHК разбавляли в 2 раза в L. hesperus сахарозной пище, содержащей 15% сахарозы и 5 мкг/мкл дрожжевой tPHК (Invitrogen) Cat No 15401-029) и использовали для скрининга. dsPHК, соответствующей положительному Lh423 контрольному клону, является SEQ ID NО:101, и отрицательному FP контрольному клону, является SEQ ID NО:104 (см. таблицу 4).
1.2. Скрининг на новые и эффективные Lygus hesperus гены-мишени с использованием сДНК-библиотеки экспрессируемых dsPHК
Разработали новое скрининговое исследование на эффективные Lygus hesperus мишени. Постановка анализа являлась следующей: каждая лунка 96-луночного планшета содержит однодневную L. hesperus нимфу под действием саше из парафильма, содержащего сахарозную пищу, которая включает либо тестируемую dsPHК, либо контрольную dsPHК в присутствии tPHК. Каждый планшет содержал dsPHК из 90 различных клонов, 3×Lh423 (положительный контроль) и 3×FP (флуоресцентный белок; отрицательный контроль). Каждый клон (тестируемая dsPHК) реплицировали на трех планшетах. После трех дней воздействия зарегистрировали число выживших нимф и заменили пищу свежей пищей (комплексной) для выращивания, не содержащую dsPHК. Смертность оценивали в дни 4, 6 и 8. Идентичную постановку использовали для подтверждающих анализов первого и второго цикла, с 8 и 20 насекомыми соответственно, с одной нимфой на лунку.
Систему анализа проверяли с использованием dsPHK, соответствующей Lh423 мишени, в качестве положительного контроля и dsPHК флуоресцентного белка в качестве отрицательного контроля: более 90% являлись истинно положительными и менее 5% являлись ложноположительными, соответственно.
Протестировали двадцать 96-луночных планшетов, поименованных Lh001-Lh020 (см. нижнюю линию на фиг. 1 и 2), содержащих 1800 отдельных клонов. 205 кандидатов идентифицировали и протестировали в первом подтверждающем анализе. Устанавливая пороговую величину на показателе >50% смертности, 41 отдельный клон идентифицировали и продвинули во второй цикл подтверждения. В анализе клоны сравнивали с положительными контролями Lh423 (RpL19) и Lh105.2 (Sec23) и отрицательным контролем Pt (кодирующим флуоресцентный белок коралла). dsPHК, соответствующей положительному (Lh423) контрольному клону, является SEQ ID NО:101, соответствующей положительному Lh105.2 контрольному клону, является SEQ ID NО:102 и отрицательному (Pt) контрольному клону является SEQ ID NО:104 (см. таблицу 4).
Подтверждающие анализы второго цикла, тестирующие 20 насекомых/тестируемых dsPHК, начали для всех тестируемых dsPHК, показывающих ≥50% смертности в первом подтверждении (фиг. 1 и 2). Мишени-кандидаты, соответствующие подтвержденным тестируемым dsPHК, обозначили с помощью "Lhxxx число" (см. таблицу 1). Применяя то же пороговое значение при ≥50% смертности, 15 мишеней подтвердили в первом скрининге.
Выполняли второй скрининг для идентификации новых Lygus hesperus мишеней. Результаты подтверждающих анализов второго цикла представлены на фиг. 14, Применяя то же пороговое значение при ≥50% смертности, несколько мишеней подтвердили во втором скрининге (см. таблицу 1C).
1.3. Идентификация Lygus мишеней
Одновременно с подтверждающими анализами на насекомых, вставки, соответствующие положительным клонам, секвенировали, и BlastX поиск по сравнению с базами данных белков Drosophila и Tribolium использовали для подтверждения идентичности мишеней. В таблице 1 приведено краткое изложение биоинформационного анализа и текущая аннотация новых идентифицированных L. hesperus целевых последовательностей.
Пятнадцать новых L. hesperus мишеней идентифицировали в первом скрининге и 11 новых L. Hesperus мишеней идентифицировали во втором скрининге. Все мишени проявляют высокую эффективность против нимф L. hesperus, указывая на то, что сДНК, кодирующие двухцепочечные РНК, содержащиеся в них, являются существенно важными для выживания вредителя, и таким образом представляют гены-мишени, представляющие интерес в целях борьбы с вредителями. Определяли последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней, следовательно, как представлено в таблицах 2 и 3 соответственно.
Lh594, Lygus hesperus ортолог Drosophila тропонина I, участвующего в сокращении мышц и, следовательно, отсутствующего у растений, представляет новый класс мишеней, относящихся к специфическому для животных физиологическому пути, еще не изученному на GM-PHКi. У плодовой мушки тропонин I описан как гаплонедостаточный ген, проявляющий мутантный фенотип в гетерозиготном состоянии. Такие гены могут являться особенно восприимчивыми к снижению уровней экспрессии mРНК и таким образом могут рассматриваться как идеальные мишени для PHКi.
В данном Lh594 пути, отобрали восемь мишеней (см. таблицу 1В). Для каждой мишени до 4 пар вырожденных ПЦР праймеров сконструировали на основе выравнивания последовательностей различных насекомых, включая пчелу, Tribolium и тлю. Данные праймеры применяют для амплификации фрагментов из Lygus hesperus мишеней. Определяли последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней, как представлено в таблицах 2 и 3, соответственно.
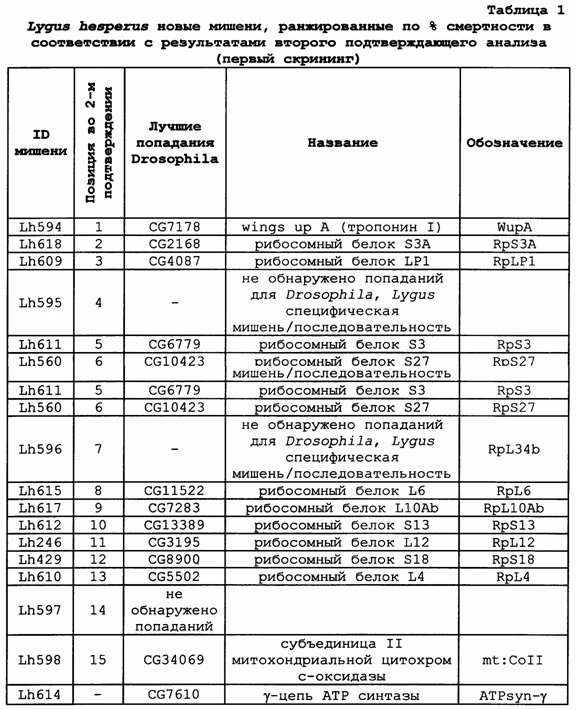
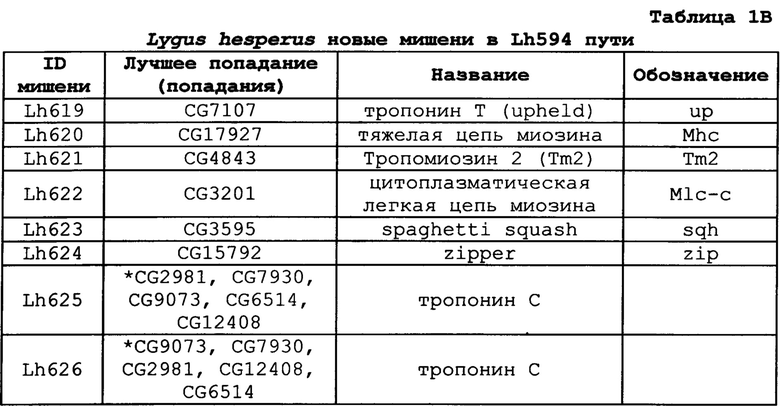
*неясно: несколько попаданий в семействе - ранжированы в соответствии с собственным значением (e-value)
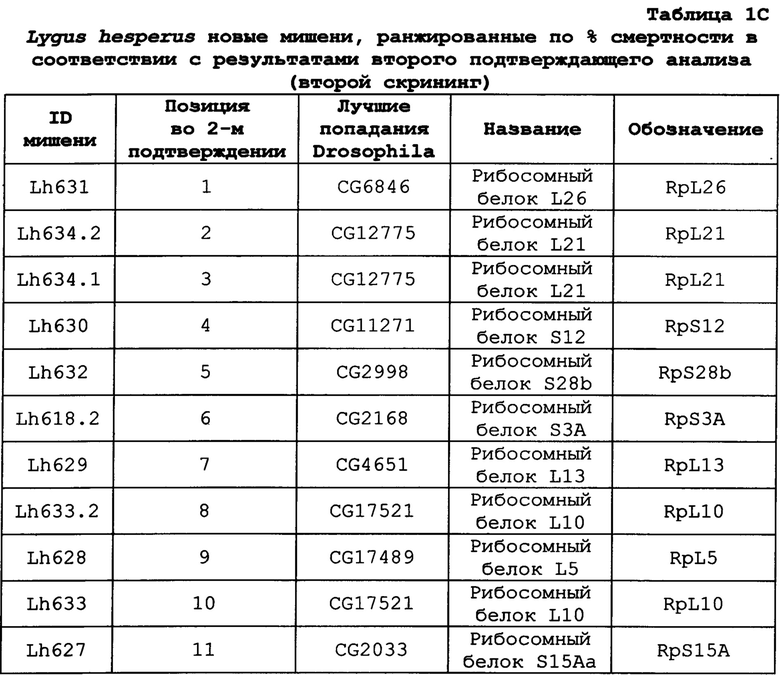
1.4. Клонирование полноразмерной сДНК с помощью RACE (быстрая амплификация концов сДНК)
Для клонирования полноразмерной сДНК, начиная от известного клона внутреннего фрагмента из самых эффективных мишеней, использовали набор 5'/3' RACE (Roche, Cat. No. 1734792; на основе Sambrook, J. & Russell, D.M). Следовали стандартному протоколу, описанному в руководстве по эксплуатации. Кратко, для 5' RACE сконструировали антисмысловой праймер, специфический для целевой последовательности, на известной последовательности, и применяли для синтеза первой цепи сДНК, применяя Lygus РНК в качестве матрицы. К первой цепи сДНК добавляли хвост и использовали в качестве якоря для синтеза второй цепи и амплификации неизвестной концевой части транскрипта. Для 3' RACE применяли якорный праймер олиго dT для синтеза первой цепи сДНК. Для 5' и 3' RACE применяли вложенные праймеры, специфические для целевой последовательности, во второй реакции ПЦР. Фрагменты ПЦР анализировали на агарозном геле, очищали, клонировали и секвенировали для подтверждения.
Последовательности полноразмерной сДНК, соответствующие мишеням, собрали в VectorNTi, полностью интегрированном пакете программного обеспечения для анализа последовательности, с целью анализа последовательности ДНК (Invitrogen).
Пример 2 In vitro получение двухцепочечных РНК для сайленсинг генов
2.1. Получение dsPHК, соответствующих частичным последовательностям Lygus hesperus генов-мишеней
Двухцепочечную РНК синтезировали в миллиграммовых количествах. Сначала две отдельные матрицы 5' Т7 РНК - полимеразного промотора (смысловая матрица и антисмысловая матрица) создавали с помощью ПЦР. ПЦР разрабатывали и проводили таким образом, чтобы получить смысловой и антисмысловой матричные полинуклеотиды, каждый из которых обладает Т7 промотором в различной ориентации по отношению к целевой последовательности, подлежащей транскрибированию.
Для каждого из генов-мишеней создавали смысловую матрицу, применяя мишень-специфический Т7 прямой праймер и мишень-специфический обратный праймер. Антисмысловые матрицы создавали, применяя мишень-специфический прямой праймер и мишень-специфический Т7 обратный праймер. Последовательности соответствующих праймеров для амплификации смысловой и антисмысловой матриц посредством ПЦР для каждого из генов-мишеней приведены в таблице 4. ПЦР-продукты анализировали с помощью электрофореза в агарозном геле и очистили. Полученные Т7 смысловую и антисмысловую матрицы смешали и транскрибировали путем добавления Т7 РНК-полимеразы. Одноцепочечным РНК, полученным путем транскрипции с матриц, предоставили возможность соединиться, обработали с помощью ДНКазы и РНКазы и очистили осаждением. Смысловая цепь образующейся в результате dsPHK, полученная от каждого из генов-мишеней, приведена в таблице 4.
2.2. Тестирование в анализе на выживаемость для новых Lygus hesperus мишеней
Для создания возможности ранжирования в соответствии с эффективностью, in vitro dsPHК, соответствующие новым мишеням, синтезировали и применили к L. hesperus в 10-дневных биопробах анализа на выживаемость. Кратко, однодневных нимф L. hesperus поместили в 96-луночные планшеты с сахарозными пленками, содержащими 0,5 мкг/мкл dsРНК-мишени, с добавлением 5 мкг/мкл дрожжевой tPHК. Планшеты инкубировали в течение 3 дней в стандартных Lygus условиях выращивания. В дни 3, 6 и 8, пищевые пленки обновляли пленками, содержащими Lygus только пищу. Lh423 (RpL19) использовали в качестве положительного контроля, и GFP dsPHК и сахарозную пищу использовали в качестве отрицательного контроля.
Результаты анализа на выживаемость подтвердили данные первого и второго подтверждающих анализов. Lh594 установили в качестве высокоэффективной мишени с активностью и скоростью поражения, более сильной, чем сильный контроль Lh423.
До сих пор Lygus скрининг на новые мишени идентифицировал новые мишени с активностями более высокими или находящимися в диапазоне положительного контроля Lh423, которые включают Lh429, Lh594, Lh609, Lh610, Lh611, Lh617 и Lh618. Смертность, вызванная данными мишенями, показана на фиг. 3 и 4.
Для обеспечения более точного ранжирования мишеней в соответствии с их активностью выполнили анализы концентрации в зависимости от дозы. Новые мишени тестировали в in vitro анализах при концентрации в диапазоне от 0,4 до 0,025 мкг/мкл. Согласно условию, 24-дневных нимф тестировали в постановке с 96-луночным планшетом, в сахарозной пище с добавлением dsPHК и носителя tPHК. Результаты представлены в виде % выживаемости в течение 10-дневного эксперимента (фиг. 7-11) и суммированы в таблице 5.
На основе анализа кривой концентрации мишени ранжировали путем сравнения с исходными контролями Lh423 и Lh105 (таблица 5).
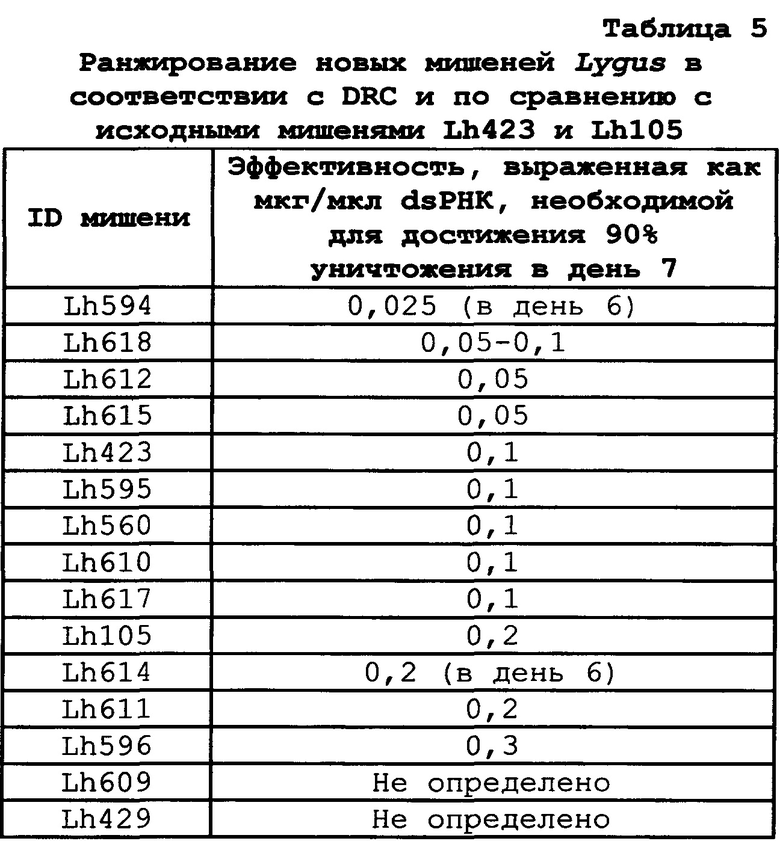
Эффективность Lh594 дополнительно подтверждали. Эффект данной мишени отчетливо наблюдается по меньшей мере за один день до эффекта других мишеней и исходного положительного контроля Lh105 и Lh423. Так как Lh594 являлась высокоэффективной, LD50 не достигали в стандартном DRC эксперименте с концентрацией в диапазоне от 0,4 до 0,025 мкг/мкл dsPHК (фиг. 8), Lh594 эксперимент, следовательно, повторяли, включая более низкие концентрации в диапазоне от 0,05 до 0,001 мкг/мкл dsPHК (фиг. 12). В заключение, активность Lh594 наблюдали при такой низкой концентрации, как 0,0025 мкг/мкл, и примерно 90% уничтожение (что соответствует примерно 10% выживаемости) получали в день 6 при 0,025 мкг dsPHК.
Для дополнительного изучения эффективности Lh594 и роли tPHК носителя в ответе на PHКi у Lygus hesperus, дополнительные in vitro анализы питания подготовляли в отсутствие носителя tPHК. Lh594, Lh423 (исходный контроль) и GFP (отрицательный контроль) dsPHК получали in vitro, применяя стандартный способ. dsPHК очистили и тестировали при 5 мкг/мкл в отсутствие tPHК (фиг. 13А).
В отсутствие tPHК, мишени Lh594 и Lh423 индуцировали высокую летальность у Lygus нимф. Результаты данного эксперимента были воспроизведены позднее. dsPHK-мишень обладала способностью индуцировать эффекты PHКi путем кормления у Lygus нимф в отсутствие tPHК.
Для исследования активности dsPHК при более низких концентрациях в отсутствие носителя tPHК, проводили дополнительные эксперименты, применяя уменьшение количеств dsPHК (фиг. 13В).
Аналогичный подход применялся для lygus мишеней, которые идентифицировали во втором скрининге. Для обеспечения ранжирования мишеней в соответствии с их активностью выполняли анализы концентрации в зависимости от дозы. Новые мишени тестировали в in vitro анализах при концентрациях в диапазоне от 0,5 до 0,05 мкг/мкл. Согласно условию, 24-дневных нимф тестировали в постановке с 96-луночным планшетом, в сахарозной пище с добавлением dsPHК и носителя tPHК. Результаты представлены в виде % выживаемости в течение 9-дневного эксперимента (фиг. 17 A-D). Lh594 и Lh423 включали в анализ в качестве эталонных мишеней. Результаты суммированы в таблице 6. На основе анализов кривой концентрации мишени ранжировали путем сравнения с исходным контролем Lh423.

Пример 3 Скрининг пути тропонина
Для создания возможности тестирования мишеней пути тропонина, in vitro полученные dsPHК, соответствующие Lh619, Lh620, Lh621, Lh622, Lh622, Lh623, Lh624, Lh625 и Lh626, синтезировали и применили к L. hesperus в 10-дневных биопробах анализа на выживаемость. Кратко, однодневных нимф L. hesperus помещали в 96-луночные планшеты с сахарозными пленками, содержащими 0,5 мкг/мкл dsРНК-мишени, с добавлением 5 мкг/мкл дрожжевой tPHК. Планшеты инкубировали в течение 3 дней в стандартных Lygus условиях выращивания. В дни 3, 6 и 8 пищевые пленки обновляли пленками, содержащими Lygus только пищу. Lh594 (тропонин I) использовали в качестве положительного контроля и GFP dsPHК и сахарозную пищу использовали в качестве отрицательных контролей (фиг. 15). Четыре мишени затем включили в анализы кривой дозозависимости в in vitro анализе, с концентрациями в диапазоне от 0,4 до 0,025 мкг/мкл. Согласно условию, 24-дневных нимф тестировали в постановке с 96-луночным планшетом, в сахарозной пище с добавлением dsPHК и носителя tPHК. Результаты представлены в виде % выживаемости в течение 10-дневного эксперимента (фиг. 16А-В).
Пример 4 Создание растений, устойчивых к видам насекомых-вредителей
4.1. Сборка растительных экспрессирующих векторов, содержащих Lygus hesperus шпилечную последовательность для трансформации картофеля или хлопчатника
Поскольку механизм РНК-интерференции работает посредством фрагментов dsPHК, целевые полинуклеотидные последовательности клонировали в антисмысловой и смысловой ориентации, разделенной спейсером
(SEQ ID NО:98:
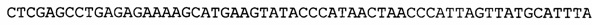
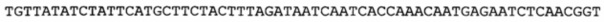
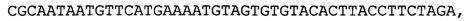 или
или
SEQ ID NО:385:


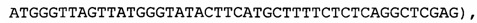 с формированием шпилечной конструкции dsPHК. Шпилечные конструкции dsPHК, кодирующие молекулы L. hesperus dsPHК, полученные из генов-мишеней, которые приведены в данном описании, субклонировали в растительный экспрессирующий вектор. Аналогично GUS dsPHК контрольную шпилечную конструкцию, где смысловую полинуклеотидную последовательность, кодирующую GUS (SEQ ID NО:97:
с формированием шпилечной конструкции dsPHК. Шпилечные конструкции dsPHК, кодирующие молекулы L. hesperus dsPHК, полученные из генов-мишеней, которые приведены в данном описании, субклонировали в растительный экспрессирующий вектор. Аналогично GUS dsPHК контрольную шпилечную конструкцию, где смысловую полинуклеотидную последовательность, кодирующую GUS (SEQ ID NО:97:

клонировали в антисмысловой и смысловой ориентации, разделенной тем же интроном (SEQ ID NО:98 или SEQ ID NО:385), субклонировали в растительный экспрессирующий вектор.
Растительный экспрессирующий вектор содержал также элементы, необходимые для поддержания плазмиды в бактериальной клетке. Шпилечная конструкция dsPHК расположена между левой границей (LB) и правой границей (RB), 3' по ходу транскрипции от 35S промотора вируса мозаики цветной капусты (P35S) и 5' против хода транскрипции от TNOS терминатора. GFP репортерную экспрессионную кассету, содержащую GFP последовательность в окружении P35S промотора и терминатора, субклонировали в растительный вектор трансформации, несущий L. hesperus шпилечную кассету. NPT II экспрессионную кассету, содержащую NPT II последовательность в окружении P35S промотора и терминатора, использовали для отбора растений, которые эффективно трансформировали. Правильность сборки генетических фрагментов в растительном векторе экспрессии подтвердили путем секвенирования (фиг. 5).
Растительные векторы экспрессии, содержащие отдельные L. hesperus шпильки-мишени впоследствии трансформировали в Agrobacterlum tumefaciens. Для всех L. hesperus генов-мишеней, приведенных в данном описании, фрагменты можно отобрать и клонировать в виде шпилек аналогичным образом.
4.2. Трансформация картофеля с помощью растительного вектора экспрессии, содержащего Lygus hesperus шпилечную последовательность, и тестирование трансформированных растений картофеля на устойчивость к L. hesperus
Пример, приведенный ниже, является иллюстрацией к выводу, что трансгенные растения картофеля, экспрессирующие шпилечные РНК, специфические для гена-мишени, неблагоприятно влияют на выживаемость и/или развитие видов насекомых-вредителей.
Lygus hesperus PHКi - путем кормления in planta
После положительных результатов, полученных в dsPHК экспериментах с питанием у L. hesperus, проводили контрольно-проверочные эксперименты in planta.
Анализ in planta разработали с in vitro проростками картофеля, которые могут поддерживать выживание насекомых по меньшей мере 8 дней, сохраняя исходную смертность низкой. L. hesperus нимфы выживают и питаются проростками картофеля дикого типа. Это подтверждается видимым повреждением, вызванным насекомыми, которое можно наблюдать на листьях и почках (фиг. 6).
В анализе, L. hesperus кормили трансгенным картофелем, экспрессирующим шпилечную dsPHК, воздействующую на L. hesperus мишени, идентифицированные в данном описании. Создавали плазмиды, несущие шпилечные конструкции и GUS контроль.
Проростки анализировали с помощью ПЦР для подтверждения интеграции Т-ДНК и наличия шпильки перед размножением. Получали избыточные экспланты с целью получения по меньшей мере 30 независимых событий для каждой конструкции.
Трансформации картофеля
Стабильно трансформированные растения картофеля получали, применяя адаптированные протоколы, полученные Julie Gilbert в NSF Potato Genome Project (http://www.potatogenome.org/nsf5). Экспланты стеблевых междоузлий картофеля 'Line V' (первоначально полученные из лаборатории селекции растений Laboratory of Plant Breeding при PRI Вагенинген, Нидерланды), который получен от восприимчивого диплоидного Solarium tuberosum 6487-9, использовали в качестве исходного материала для трансформации. В in vitro-полученные экспланты инокулировали Agrobactenum tumefaciens C58C1RifR, содержащими шпилечные конструкции. После трех дней совместного культивирования экспланты помещали на селективную среду, содержащую 100 мг/л канамицина и 300 мг/л тиментина. Через 6 недель после трансформации первые предполагаемые побеги удаляли и укореняли на селективной среде. Побеги, происходящие из различных эксплантов, рассматривались как независимые события, побеги происходящие из одного и того же каллуса, называют 'сиблинги' до тех пор, пока их клональный статус можно проверить с помощью саузерн-блоттинга, и узловые черенки побегов называют 'клоны'.
Трансгенный статус укореняющихся побегов проверяли либо с помощью GFP флуоресценции, либо с помощью плюс/минус ПЦР для вставленной целевой последовательности. Положительные побеги затем клонально размножали в культуре ткани для того, чтобы обеспечить достаточно копий, доступных для Lygus hesperus анализов. Данные побеги либо содержали в среде тканевой культуры, либо переносили в почву, предоставляя возможность большей гибкости для тестирования на устойчивость к L. hesperus нимфам/имаго. Первые растения являлись доступными для тестирования через четырнадцать недель после трансформации.
Биопроба
Молодые трансгенные растения картофеля, либо содержали в среде тканевой культуры, либо выращивали в почве в помещении камеры для выращивания растений со следующими условиями: 25±1°С, 50±5% относительной влажности, 16:8 часов светлый: темный фотопериод. В конструкции ряд событий (например, двадцать) создавали с помощью подходящего количества клонов (например, десять) на событие. Ряд молодых Lygus нимф/имаго помещали на содержащиеся в отдельных клетках молодые (например, на стадии 4-5 развернутых листьев) растения картофеля и оставляли по меньшей мере на семь дней перед оценкой устойчивости к Lygus hesperus с точки зрения снижения выживаемости нимф/личинок/имаго, задержки развития и остановки роста, и/или уменьшения повреждения растений при питании.
Применимость in planta PHКi для защиты сельскохозяйственных культур от Lygus тестировали в анализе с использованием трансгенного картофеля, экспрессирующего шпильки, соответствующие Lygus генам-мишеням. В таблице 7 суммированы количества полученного и протестированного трансгенного картофеля. Трансгенные события создавали с помощью шпилек, соответствующих Lygus мишеням Lh423 (шпилечная последовательность для Lh423 представлена в SEO ID NО:402; смысловая последовательность Lh423-мишени представлена в SEQ ID NО:101) и Lh594 (шпилечная последовательность для Lh594 представлена в SEQ ID N0:401; смысловая последовательность Lh594-мишени представлена в SEQ ID NO:2), и GUS в качестве контроля (шпилечная последовательность для GUS представлена в SEQ ID NO:403; смысловая последовательность GUS представлена в SEQ ID NO:97). В данном анализе SEQ ID NO:385 использовали в качестве интрона.

Проростки размножали сначала в ящиках, затем в отдельных вегетационных сосудах, содержащих MS среду для укоренения (4,4 г/л MS солей и витаминов, 30 г/л сахарозы, 10 г/л Gelrite, pH 5,7), при подготовке Lygus анализов с питанием. Два независимых GUS события отобрали из 8 независимых событий, протестированных в 2 независимых экспериментах (фиг. 18А-В). В анализе 20-30 трансгенных растений одного и того же события, каждое посаженное в отдельный вегетационный сосуд, тестировали и сравнивали с WT проростками. Для трансгенных линий, несущих Lh423 и Lh594 шпильки, 28 и 25 независимых событий протестировали соответственно, и для каждого независимого трансгенного события протестировали 20-30 проростков, каждый из которых был посажен в отдельный вегетационный сосуд (фиг. 6).
Как и ожидалось, в первичных трансформантах диапазон активности наблюдали для 28 независимых Lh423 трансгенных событий (фиг. 19); 6 независимых Р006 событий индуцировали свыше 60% летальности в день 9, и в одном событии летальность достигла 80% в день 9 (фиг. 20).
Как и ожидалось в первичных трансформантах, и как наблюдалось для Lh423 первичных трансформантов, диапазон активности также наблюдали для 25 независимых Lh594 трансгенных событий (фиг. 21); 6 независимых Р007 событий индуцировали свыше 60% летальности в день 9, ив одном событии летальность достигла 80% в день 9 (фиг. 22). В дополнение, задержку роста и остановку роста отчетливо наблюдали у выживших насекомых.
Результаты qRT-ПЦР питавшегося растениями Lygus
Для подтверждения того, что наблюдаемое снижение выживаемости Lygus, питавшегося трансгенными проростками картофеля, экспрессирующими шпильки, направленные против эндогенных генов, являлось истинным ответом на PHKi, измеряли уровень подавления mРНК-мишени (Lh423) с помощью количественной ПЦР в реальном времени (qRT-ПЦР). Насекомым предоставили возможность питаться 3-мя событиями, несущими Lh423 шпильку (Р006/59, /22 и /29), и одним событием, несущим контрольную GUS шпильку (Р001/28), в качестве контроля. Насекомых собирали через 5 дней и немедленно замораживали в жидком азоте. Суммарную РНК экстрагировали из 5 пулов 5 насекомых, применяя реагент TRI и в соответствии с инструкциями производителя (SIGMA). После обработки ДНКазой I (Promega) для удаления геномной ДНК с последующей экстракцией фенолом и хлороформом и осаждением этанолом, осажденные РНК растворяли в воде. Для каждого образца 1 мкг РНК подвергали обратной транскрипции с помощью смеси случайных гексамеров и якорных олиго dT праймеров. qRT-ПЦР ПЦР проводили на BioRad I-циклере, применяя iQ SYBR Green Supermix (Biorad), и с использованием рекомендованных производителем условий. qRT-ПЦР праймеры (таблица 8) разработали, применяя BeaconDesign; во избежание ПЦР-артефактов, предполагаемых в наличии экспрессируемой растением dsPHК, поглощаемой насекомыми, последовательности праймеров расположили на 3' dsPHК последовательности. GeNorm алгоритм использовали для нормализации уровня mРНК-мишени, применяя 2 конститутивных гена, Lh425 (SDHA) и Lh427 (rpl 11).
В GUS трансгенном контроле не наблюдали подавления Lh423 эндогенной mРНК-мишени насекомого. Но результаты ясно показали подавление эндогенной Lygus Lh423 mPHК, соответствующей dsPHК, поглощаемой животными, питающимися 3-мя различными событиями Lh423 трансгенных растений (фиг. 23).
4.3. Трансформация хлопчатника с помощью растительного вектора экспрессии, содержащего Lygus hesperus шпилечную последовательность, и тестирование трансформированного каллусного материала или растений хлопчатника на устойчивость к L. hesperus
Пример, приведенный ниже, является иллюстрацией к выводу, что трансгенные растения или каллус хлопчатника, экспрессирующие шпилечные РНК, специфические для генов-мишеней, неблагоприятно влияют на выживаемость и/или развитие видов насекомых-вредителей.
Трансформация хлопчатника
Поверхность семени Coker 312 стерилизовали с применением сначала промывания в течение 5 минут в 70% этаноле, и затем встряхивания в 20% отбеливающего (Clorox Co. США, 1% активного хлора) раствора плюс 10 капель неионного детергента, Tween® 20, на литр. Семя затем промывали 3 раза в стерильной дистиллированной воде, затем досуха промакивали стерильной фильтровальной бумагой. Стерильное семя проращивают на среде для прорастания (SG) в течение 4-6 дней, и в этот момент гипокотили собирают и нарезают по 0,5 см длиной, подготавливая для инокулирования Agrobacterium. Срезы помещали на стерильную фильтровальную бумагу, покрывая средой на основе среды Мурасиге-Скуга (Murashige and Skoog), не содержащей фитогормоны. Эксплантаты инкубировали при 16:8 часовом цикле день:ночь при 28°С +/-°С в течение 3 дней перед инокулированием.
Для инокуляции, Agrobacterium tumefaciens культуру (10 мл) в жидкой LB среде, штамм GV3101 (рМР90) GentR или штамм LBA4404, содержащие предпочтительную шпильку РНК-мишени и растительную селекционную кассету, кодирующую устойчивость к гигромицину, выращивают в течение ночи, и 5 мл применяют для инокуляции 100 мл культуры вечером накануне инокуляции. Культуру центрифугировали, ресуспендировали и разбавляли до OD 600 в 0,15 в бактериальной среде для разбавления.
В сегменты гипокотилей инокулировали Agrobacterium суспензию в течение 5 минут. После этого эксплантаты промокали стерильной фильтровальной бумагой для удаления избытка бактериальной суспензии. Эксплантаты инкубировали в темноте на среде для совместного культивирования хлопчатника (ССМ) в течение 48 часов. Эксплантаты затем помещали на селективную среду для индукции каллусов (SCIM), содержащую 10 мг/л гигромицина и 500 мг/л цефотаксима и включающую фитогормоны 2, 4-дихлорфеноксиуксусную кислоту (0,1 мкг/мл) и кинетин (0,1 мкг/мл). Через 4-6 недель наблюдали первые устойчивые каллусы, и их можно переместить на свежую SCIM и дополнительно амплифицировать для молекулярной оценки и биопробы на насекомых. Планшеты обновляли каждые 4-6 недель для поддержания выбора питательных веществ и антибиотиков.
Каллусы, которые, как показано, дают положительный результат в биопробе с питанием насекомых, выбирали для регенерации, и каллус переносили на неселективную среду для созревания соматических зародышей, рецептура основана на публикации Trolinder и Goodin, 1986 г. Как только эмбрионы достигали 4 мм в длину и обладали дифференцированными семядолями и корешками (через 2-3 месяца после переноса на среду для созревания), их можно было переносить на среду для роста корней в длину. Она включала стерилизованный вермикулит в больших пробирках, пропитанный жидкой средой на основе среды Стюарта и Хсу (Stewart & Hsu) (1977) с добавлением кинетина, гиберелловой кислоты, при этом и то, и другое добавляли в конечной концентрации 0,1 мг/л. Эмбрионы инкубировали при 28°С при 16:8 цикле дня/ночи, и как только они достигали стадии 2-3 листьев, проростки можно было закаливать в вегетационных сосудах вермикулита, заключенных в пропагаторе для поддержания влажности. Как только растения полностью были закалены (стадия 4-6 истинных листочков), их можно было высаживать в 50:50 смесь торфа : суглинка и выращивать при 14:10 световом цикле пои 30/20°С день/ночь.
Биопроба
L. hesperus нимф помещали в чашку Петри, содержащую недифференцированную каллусную ткань хлопчатника, экспрессирующую шпилечную РНК-мишень. В конструкции ряд трансформированных каллусов хлопчатника получали и тестировали в биопробе с питанием на снижение выживаемости нимф/имаго и/или задержку развития и остановку роста. Трансгенные каллусы, не экспрессирующие L. hesperus фрагмент гена шпилечной РНК-мишени, служили в качестве отрицательного контроля. Кроме того, молодые регенерированные растения хлопчатника из трансгенных каллусов выращивали в почве в помещении камеры для выращивания растений со следующими условиями: 30/20°С день/ночь, 50±5% относительной влажности, 14:10 часов светлого : темного фотопериода. В конструкции создавали ряд событий (например, двадцать). Ряд молодых L. hesperus нимф/имаго помещали на содержащиеся в отдельных клетках молодые (например, на стадии 4-5 развернутых листков) растения и оставляли по меньшей мере на семь дней перед оценкой устойчивости к L. hesperus с точки зрения снижения выживаемости нимф/имаго, задержки развития и остановки роста и/или уменьшения повреждения растений при питании. Растения хлопчатника, не трансформированные L. hesperus фрагментом гена шпилечной РНК-мишени, служили в качестве отрицательного контроля.
Пример 5. Идентификация генов-мишеней у Leptinotarsa decemlineata
5.1. Leptinotarsa decemlineata нормализованная сДНК библиотека и получение dsPHК в многолуночных планшетах для скрининговых исследований
Нуклеиновые кислоты выделяли из Leptinotarsa decemlineata личинок различных стадий. Библиотеку сДНК получали с использованием ПЦР-набора для синтеза сДНК SMARTer™, следуя инструкциям производителя (Clontech Cat. No 634925). Библиотеку сДНК нормализовали, применяя набор Trimmer (Evrogen Cat No NK001) и клонировали в PCR®-BLUNTII-TOPO® вектор (Invitrogen). Нормализация введенных в клоны М2 адаптеров (Trimmer Kit, Evrogen, SEQ ID N0:92: AAGCAGTGGTATCAACGCAG), противоположно ориентированных на каждом конце клонов. Рекомбинантные конструкции векторов трансформировали в клетки Escherichia coli strain ТОР10 (Invitrogen). Трансформированные клетки затем разбавляли и высевали с целью получения отдельных колоний или клонов. Клоны проверяли для подтверждения того, что избыточность клонов для библиотеки не превышает 5%. Отдельные клоны инокулировали в жидкую LB (бульон Лурия) среду, в 96-луночные планшеты, и выращивали в течение ночи при 37°С. Планшеты также включали положительный (Ld513) и отрицательный (FP) контрольные клоны.
Для создания dsPHК с помощью ПЦР создавали смысловой и антисмысловой фрагменты ДНК, содержащие последовательность промотора Т7. Кратко, на клон 1 мкл бактериальной суспензии помещали в многолуночные ПЦР-планшеты, содержащие REDTaq® (Sigma Cat No D4309) и праймеры oGCC2738 (SEQ ID NО:93: AAGCAGTGGTATCAACGCAG) и oGCC2739 (SEQ ID NО:94: GCGTAATACGACTCACTATAGGAAGCAGTGGTATCAACGCAG) на основе последовательностей М2 и Т7-М2, соответственно. За ПЦР реакцией следовала In vitro транскрипция, где на клон использовали 6 мкл ПЦР-продукта в 20 мкл реакционного объема, содержащего реагенты для транскрипции, обеспеченные набором RiboMAX™ Large Scale РНК Production System-T7 (Promega Cat No P1300), и инкубировали в течение ночи при 37°С. Конечный раствор dsPHК разбавляли стерильной водой Milli-Q и использовали для скрининга. dsPHК, соответствующей положительному Ld513 контрольному клону, является SEQ ID NО:400 (см. таблицу 11), и отрицательному FP контрольному клону является SEQ ID NО:104 (см. таблицу 4).
5.2. Скрининг на новые и эффективные Leptinotarsa decemlineata гены мишени с использованием сДНК-библиотеки экспрессируемых dsPHК
Каждая лунка 48-луночного планшета содержала 0,5 мл искусственной пищи, предварительно обработанной с помощью наружного покрытия 25 мкл (или 1 мкг) тестируемой или контрольной dsPHК. Одну L2 личинку помещали в каждую лунку, и на клон тестировали 3 личинки. Количества выживших СРВ оценивали в дни 4, 7 и 10.
Во второй биопробе личинок СРВ кормили пищей, обработанной наружно наносимой тестируемой dsPHК, полученной из клонов, полученных из нормализованной библиотеки сДНК. Одну личинку помещали в лунку 48-луночного мультипланшета, содержащего 0,5 мл пищи, предварительно обработанной с помощью наружного покрытия 25 мкл 40 нг/мкл dsPHК раствора. Всего на обработку (клон) тестировали двадцать четыре личинки. Количество выживших насекомых оценивали в дни 4, 5, 6, 7, 8 и 11. Процент смертности личинок рассчитывали относительно дня 0 (начало исследования) (см. фиг. 29).
5.3. Идентификация мишеней у L. decemlineata жуков
Новые целевые последовательности из скрининга в 5.2. и целевые последовательности, соответствующие мишеням пути тропонина, ортологическим Lygus Lh594, Lh619 и Lh620 последовательностям, идентифицировали у L. decemlineata. Праймеры, которые обеспечили соответствующие фрагменты сДНК для Ld594, перечислены в таблице 19. Определяли последовательности сДНК и предсказанные аминокислотные последовательности данных генов-мишеней, как представлено в таблицах 9 и 10, соответственно.
5.4. Получение dsPHК, соответствующих частичным последовательностям L. decemlineata генов-мишеней
dsPHК синтезировали, применяя праймеры, приведенные в таблице 11. Смысловая цепь полученной в результате dsPHК, полученная из генов-мишеней, приведена в таблице 11.
5.5. Тестирование в анализе на выживаемость для новых L. decemlineata мишеней
Ранний личиночный анализ
Синтетические dsPHK получали для 3 мишеней, Ld594, Ld619 и Ld620, и тестировали в анализе с питанием на личинках СРВ (см. фигуру 24). 10-дневный анализ проводили в 48-луночных планшетах, на искусственной пище (на основе Gelman et al, J Ins Sc, 1:7, 1-10: Artificial diets for rearing the Colorado Potato Beetle), с добавлением 1 мкг dsРНК/лунка с 12 личинками согласно условиям.
Наблюдали четкий эффект на развитие личинок. Второй анализ проводили для изучения эффекта данных dsPHК в течение окукливания и метаморфоза (см. анализ окукливания ниже).
Анализ окукливания
Анализ окукливания СРВ проводили для изучения эффекта PHКi нокдауна Ld594, Ld619 и Ld620 в течение окукливания и метаморфоза. Личинок четвертой возрастной стадии кормили 1 мкг in vitro синтезированной dsPHК, распределенной на листовой дисках картофеля, и затем переносили в коробку с необработанными свежими листьями картофеля. Через четыре дня выживших насекомых помещали на вермикулит для обеспечения окукливания. Обработанные с помощью Lh594 насекомые были медленными, более мелкими и в основном не способными пройти окукливание. Вылупление куколки оценивали в конце эксперимента. Для необработанного контроля 24 личинки окуклились, и все вылупились в здоровых имаго. Для Ld620 наблюдали снижение численности личинок, прогрессирующих в окукливание. Для трех протестированных мишеней отсутствовали личинки, развившиеся в здоровые куколки, и ни одна не вылупилась в имаго. Мертвые насекомые, извлеченные из вермикулита, демонстрировали различную степень пороков развития (фиг. 25).
Ld594, Ld619 и Ld620 вначале предстали в качестве несмертельных мишеней в анализе личинок СРВ, хотя снижение жизнеспособности отчетливо наблюдалась у насекомых, обработанных dsPHК. С другой стороны, в анализе окукливания все 3 мишени индуцировали сильные эффекты и ингибировали вхождение в окукливание и/или метаморфоз.
Анализ имаго
Для оценки активности Ld594, Ld619 и Ld620 у имаго СРВ проводили анализ листовых дисков. Листовые диски картофеля (1,7 см в диаметре) покрывали dsPHК или контролями и помещали в 3,5 см чашки Петри с одним взрослым жуком. На следующий день насекомым предоставили свежий обработанный листовой диск. На третий день имаго переносили в коробку, содержащую достаточно свежих, необработанных листьев картофеля для поддержания выживания необработанных контролей. На обработку тестировали 6 имаго и подсчитывали количества выживших и умирающих насекомых с регулярными интервалами от дня 6 до дня 13. Насекомые считались умирающими, если они не могли перевернуться после укладывания на спину.
Несмотря на относительно высокий уровень фона в отрицательном контроле в данном конкретном анализе, наблюдали четкие эффекты на насекомых, которых подвергли воздействию Ld594 или Ld.619 dsPHK (фиг. 26).
Пример 6 Идентификация генов-мишеней у Nilaparvata lugens
6.1. Идентификация Nilaparvata lugens мишеней
Новые целевые последовательности, соответствующие мишеням пути тропонина и названные N1594 (тропонин I), N1619 (тропонин Т) и N1626 (тропонин С), идентифицировали у коричневого дельфацида, Nilaparvata lugens. Ортологические последовательности Lygus генов, названные N1594 (тропонин I), N1619 (тропонин Т) и N1625/626 (тропонин С), клонировали посредством ПЦР с вырожденными праймерами с использованием ВРН сДНК в качестве матрицы. В дополнение, полноразмерную сДНК идентифицировали для N1594, применяя RACE (см. способ выше). ПЦР-систему AmpliTaq Gold (Applied Biosystems) применяли, следуя инструкциям производителя и в стандартных условиях для реакции ПЦР с вырожденными праймерами, как правило, следующим образом: 1 цикл в 10 минут при 95°С, с последующими 40 циклами по 30 секунд при 95°С, 1 минуту при 50°С и 1 минуту при 72°С, с последующими 10 минутами при 72°С. Для увеличения степени успеха до 10 различных вырожденных праймеров, прямых и обратных, разработали на основе выравниваний ортологических последовательностей у других видов и использовали в различных комбинациях. Полученные ПЦР-фрагменты очищали из геля с помощью набора для экстракции геля (Qiagen Cat. No 28706) и клонировали в ТОРО ТА вектор (Invitrogen). Клоны секвенировали и консенсусные последовательности использовали в Blast поисках в отношении различных доступных баз данных последовательностей насекомых для подтверждения значимости вставки. Вырожденные праймеры, которые привели к успешной амплификации, перечислены в таблице 20.
Определяли последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней и еще одного гена-мишени (N1537), как представлено в таблицах 12 и 13, соответственно.
6.2. Получение dsPHК, соответствующих частичным последовательностям Nilaparvata lugens генов-мишеней
dsPHК синтезировали с использованием праймеров, которые указаны в таблице 14. Смысловая цепь полученной в результате dsPHК, полученная из каждого из генов-мишеней, приведена в таблице 14.
6.3. Тестирование в анализе на выживаемость для новых Nilaparvata lugens мишеней
dsPHК синтезировали и тестировали в предварительно оптимизированных анализах PHКi путем кормления у ВРН, в присутствии цвиттерионного детергента, CHAPSO, при 0,1% конечной концентрации. dsPHК тестировали при 0,5 мкг/мкл конечной концентрации. N1537, эффективную мишень в анализах ВРН, использовали в качестве исходной мишени в данном анализе. Выживаемость насекомых оценивали на протяжении 9 дней.
Результаты биопробы показали, что у ВРН N1594, N1619 и N1626 также являлись эффективными мишенями PHКi у ВРН (фиг. 27).
Пример 7 Идентификация генов-мишеней у Acyrthosiphon pisum
7.1. Идентификация Acyrthosiphon pisum мишеней
Новые целевые последовательности идентифицировали у тлей и назвали Ар423, Ар537, Ар560 и Ар594, следуя той же номенклатуре: "Арххх", где "Ар" соответствует Acyrthosiphon pisum и "ххх" соответствует ID мишени. Праймеры разрабатывали на основе открытого источника предсказания генов в AphidBase (ref: http://www.aphidbase.com/) (таблица 15).
Определяли последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней, как представлено в таблицах 16 и 17, соответственно.
7.2. Получение dsPHК, соответствующих частичным последовательностям генов-мишеней тли
dsPHК синтезировали с использованием праймеров, которые указаны в таблице 18. Смысловая цепь полученной в результате dsPHК, полученная из каждого из генов-мишеней, приведена в таблице 18.
7.3. Тестирование в анализе на выживаемость для новых мишеней у тлей
PHКi путем кормления тестировали у Acyrthosiphon pisum (гороховая тля) с 4 мишенями Ар594, Ар423, Ар560, Ар537. Последовательности амплифицировали с помощью ПЦР с использованием праймеров, разработанных на основе открытого источника информации о последовательностях (http://www.aphidbase.com), и сДНК, полученной из тлей. Синтетические dsPHK получали и тестировали в конечной концентрации 0,5 мкг/мкл в присутствии 5 мкг/мкл дрожжевой tPHК в сахарозной пище. Десять новорожденных нимф гороховой тли поместили в маленькую чашку Петри (32 мм). Пятьдесят мкл пищи (с tPHК и dsPHК) наносили пипеткой на верхнюю часть первого слоя парафильма. Второй слой парафильма покрывал пищу и создавал пакетик для кормления, где тли могли питаться. На каждую мишень подготовили пять повторов 10 новорожденных нимф. GFP dsPHК использовали в качестве отрицательного контроля. Пищу обновляли в дни 4 и 7 анализов и оценивали выживаемость (фиг. 28).
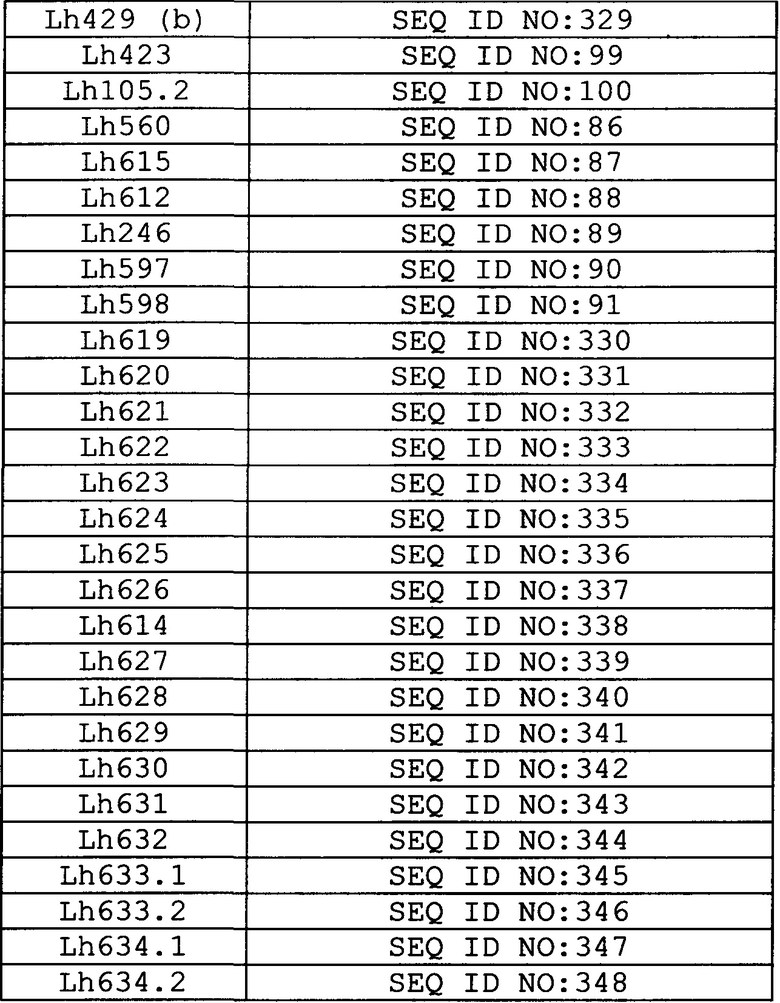
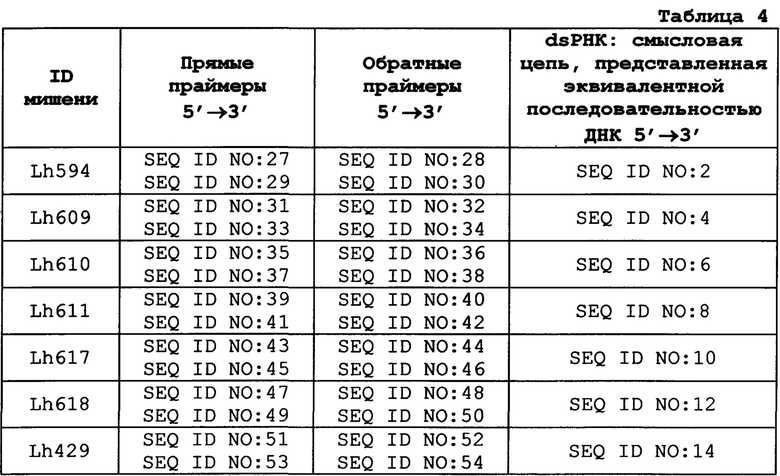
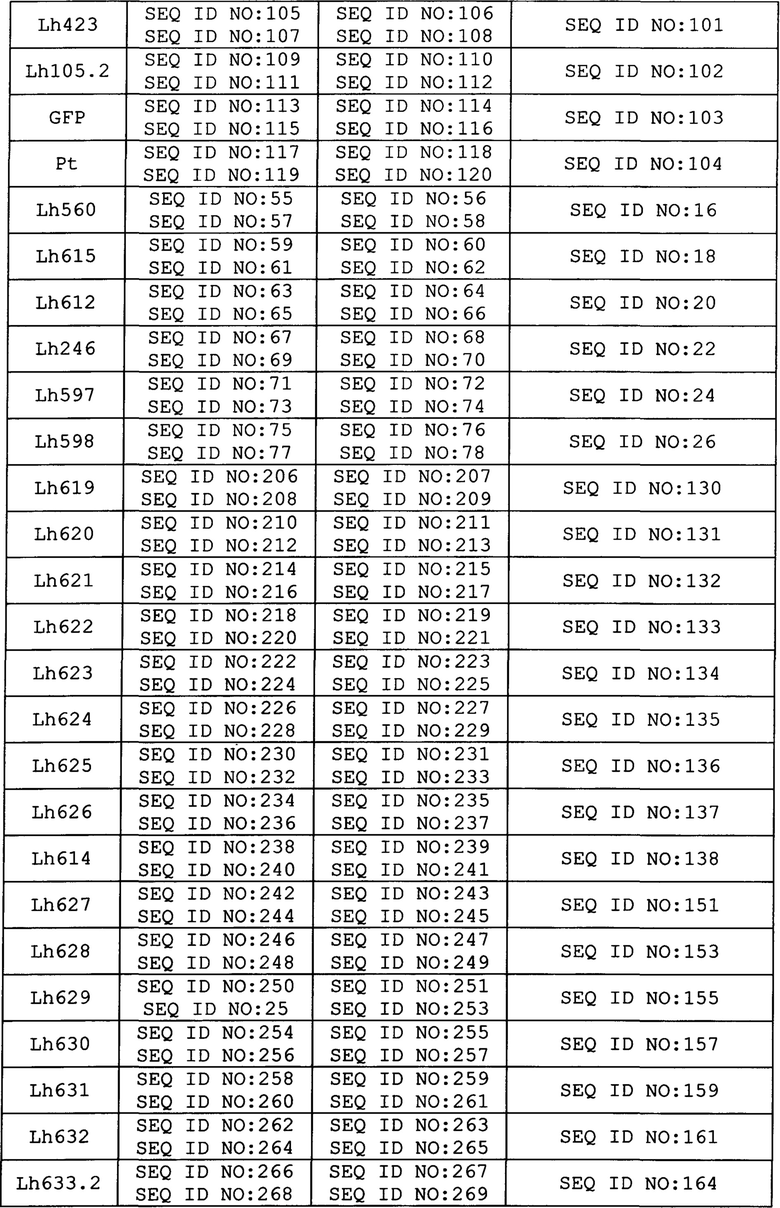


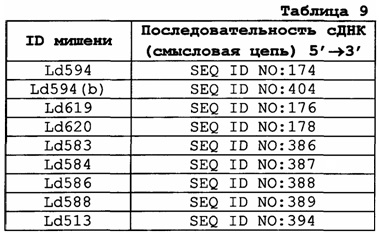

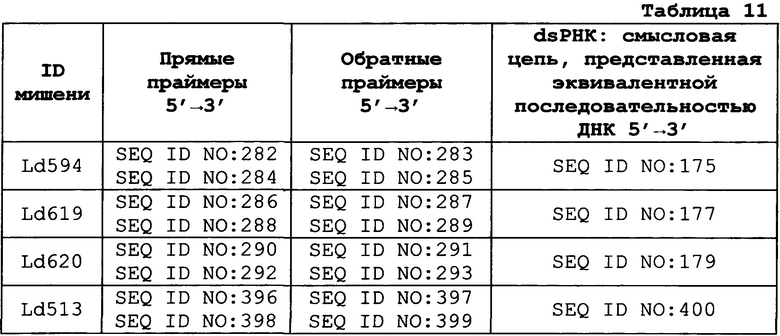


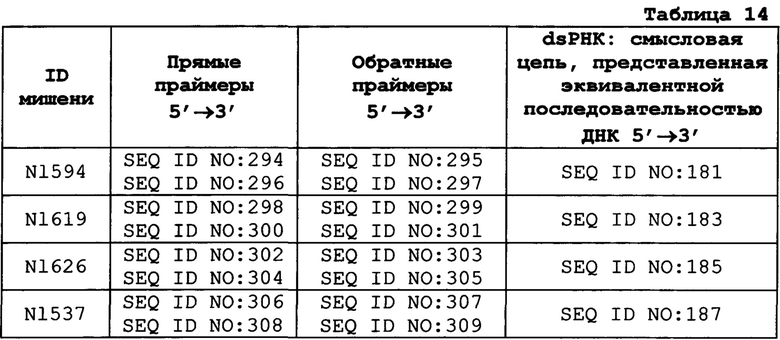
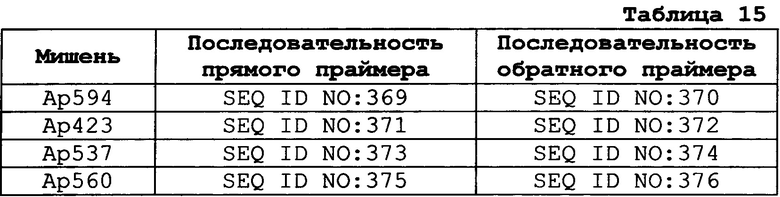
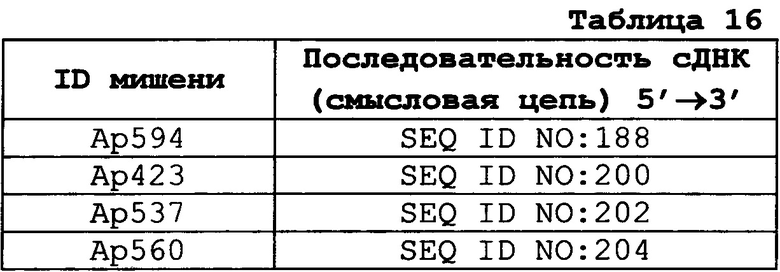
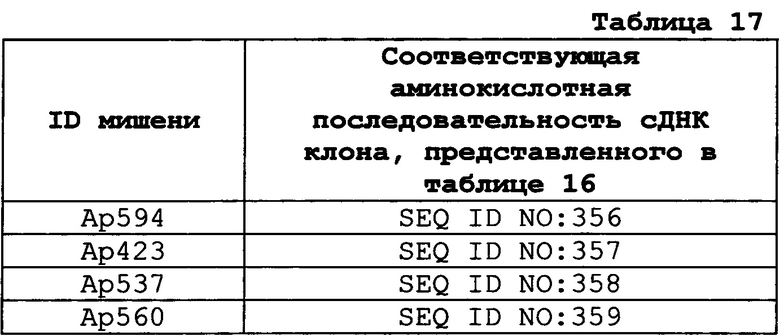
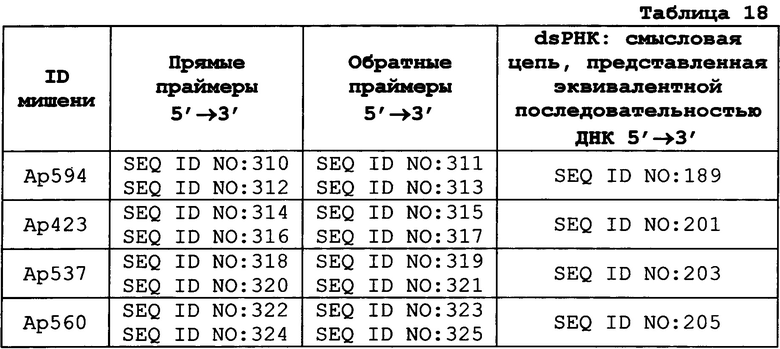

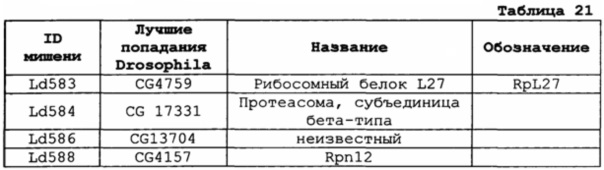
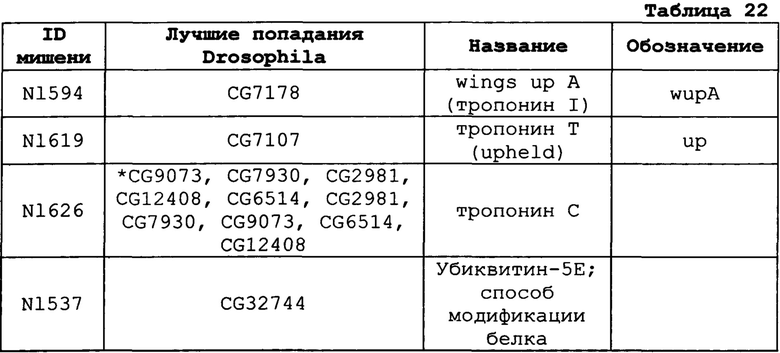
* неясно: несколько попаданий в семействе
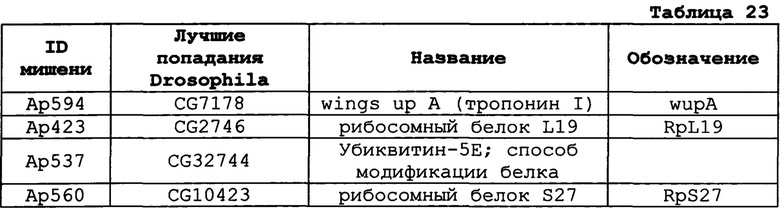
Специалистам в данной области будет очевидно, что многочисленные изменения и/или модификации можно внести в приведенные выше анализы без отступления от сущности или объемаданного анализа, как описано в общем. Специалисты в данной области обнаружат или смогут установить, применяя не более чем обычное экспериментирование, многие эквиваленты конкретных примеров, и такие эквиваленты охватываются настоящим изобретением. Настоящий пример, таким образом, следует рассматривать во всех отношениях как иллюстративный, а не ограничительный.
| название | год | авторы | номер документа |
|---|---|---|---|
| ПОДАВЛЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ | 2012 |
|
RU2665549C1 |
| РАСТЕНИЯ, УСТОЙЧИВЫЕ К НАСЕКОМЫМ-ВРЕДИТЕЛЯМ | 2012 |
|
RU2671143C2 |
| ПОДАВЛЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ | 2012 |
|
RU2619219C2 |
| ПОДАВЛЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ | 2012 |
|
RU2653752C2 |
| ПОДАВЛЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ | 2018 |
|
RU2725953C2 |
| УМЕНЬШЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ | 2011 |
|
RU2662995C2 |
| КОНТРОЛЬ ЖЕСТКОКРЫЛЫХ ВРЕДИТЕЛЕЙ С ПРИМЕНЕНИЕМ МОЛЕКУЛ РНК | 2017 |
|
RU2801251C2 |
| СПОСОБЫ ГЕНЕТИЧЕСКОГО КОНТРОЛЯ ПОРАЖЕНИЯ РАСТЕНИЙ НАСЕКОМЫМИ И ПРИМЕНЯЕМЫЕ ДЛЯ ЭТОГО КОМПОЗИЦИИ | 2006 |
|
RU2478710C2 |
| ПОЛИНУКЛЕОТИД И СПОСОБ, ИСПОЛЬЗУЕМЫЙ ДЛЯ ОСУЩЕСТВЛЕНИЯ КОНТРОЛЯ НАД НАШЕСТВИЕМ НАСЕКОМЫХ | 2019 |
|
RU2775717C1 |
| ПОЛИНУКЛЕОТИД И СПОСОБ ДЛЯ ОСУЩЕСТВЛЕНИЯ КОНТРОЛЯ НАД ПОРАЖЕНИЕМ НАСЕКОМЫМИ | 2019 |
|
RU2775943C1 |





































































































































































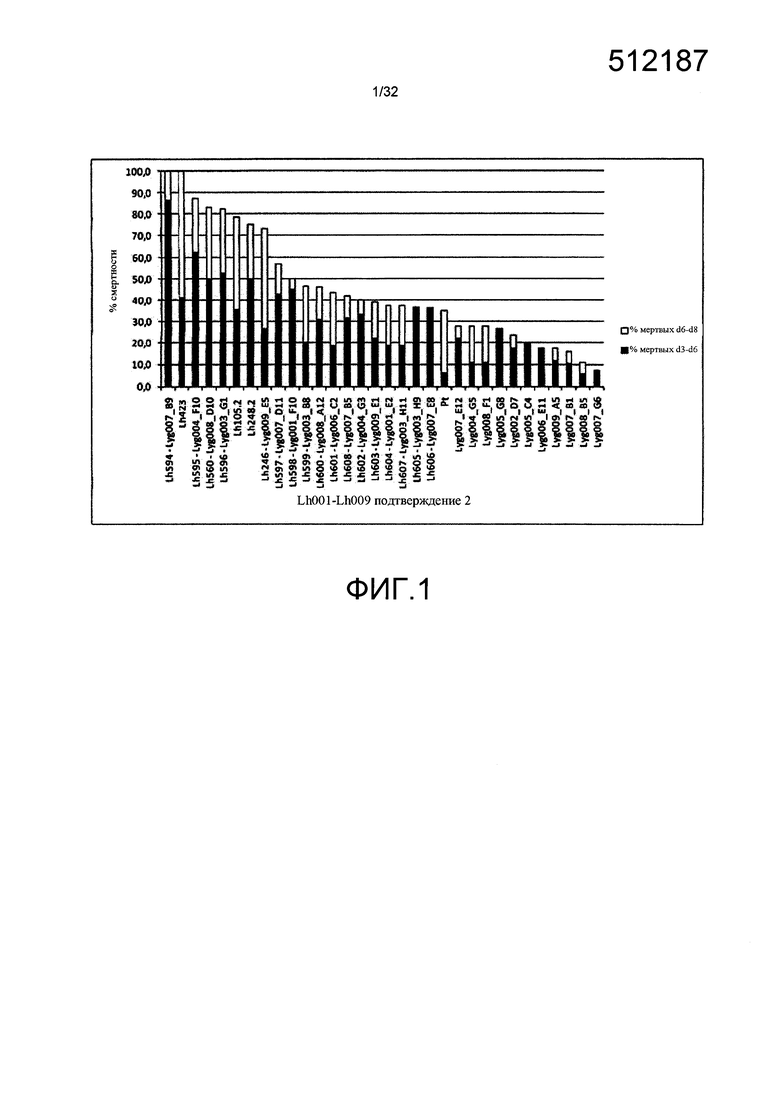
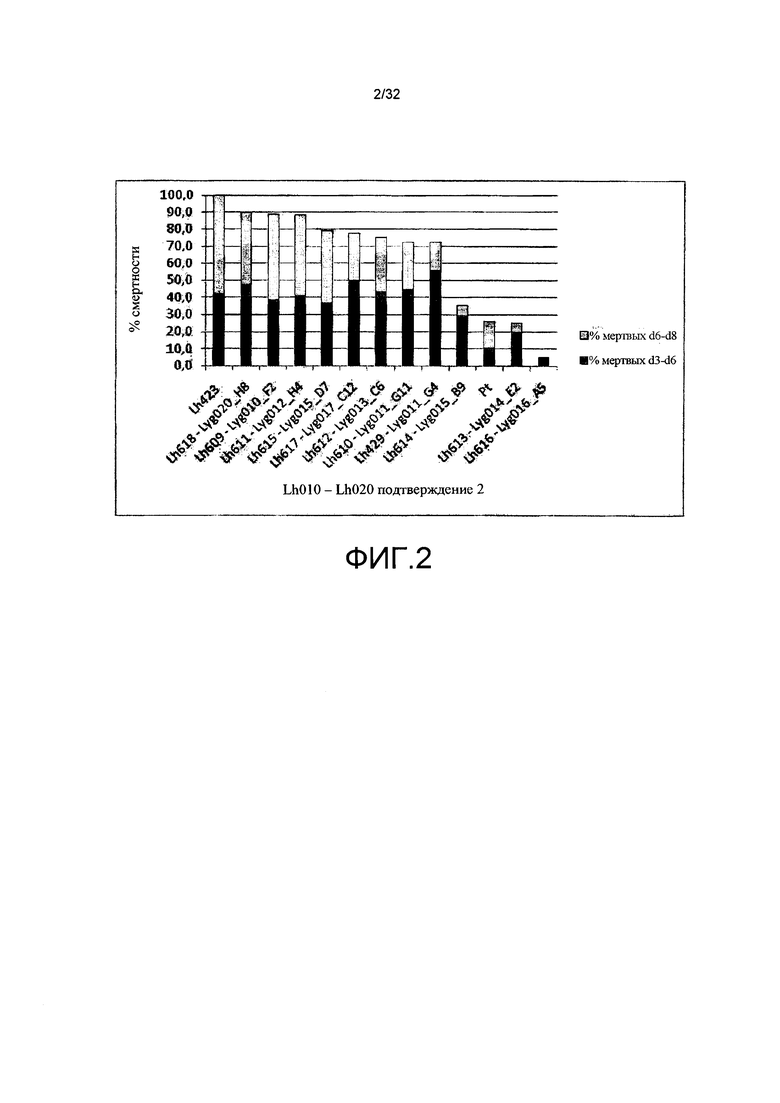
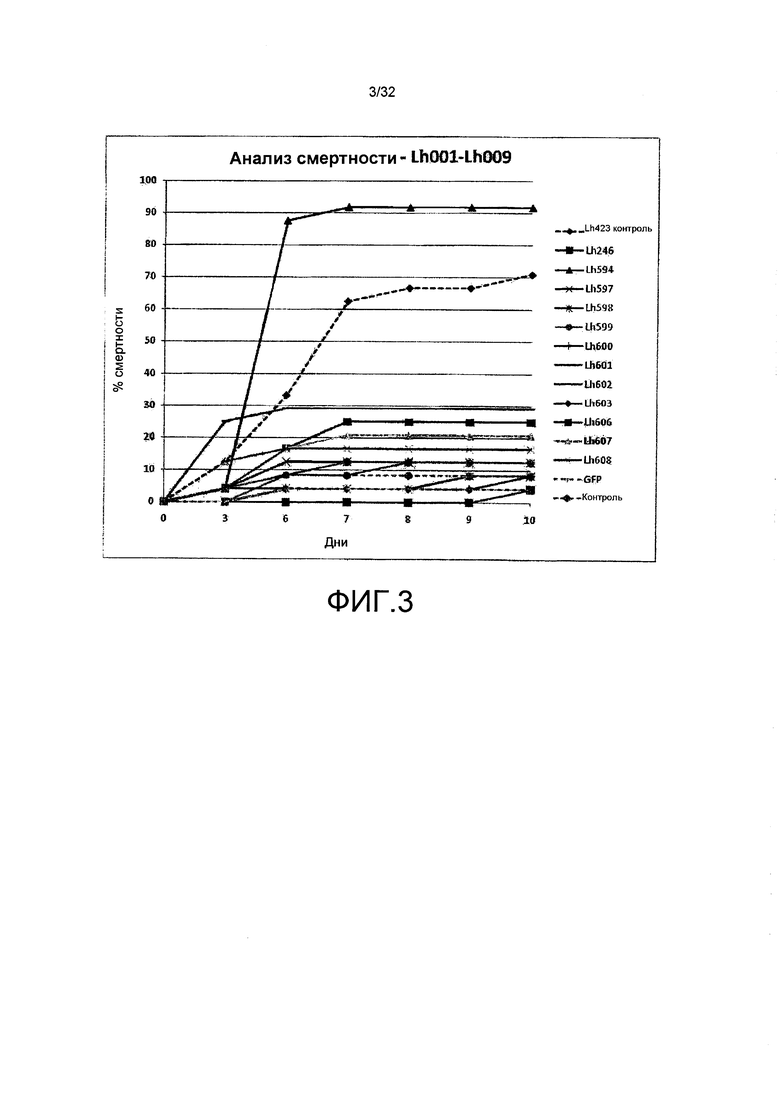
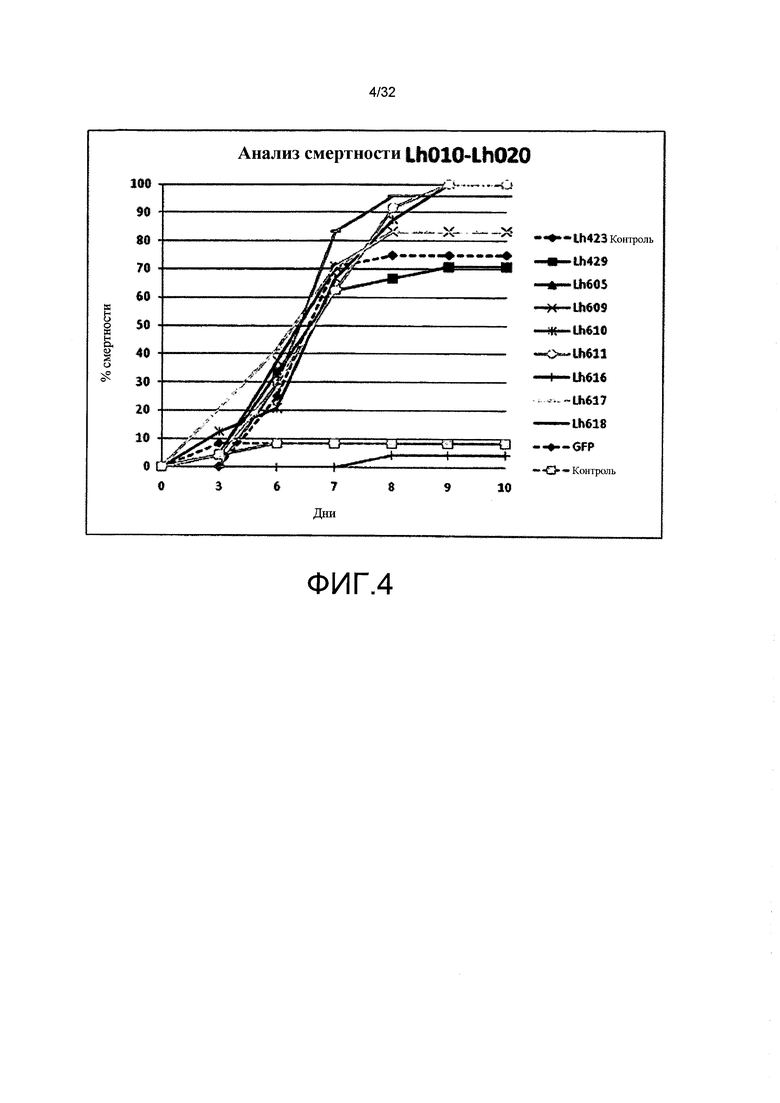
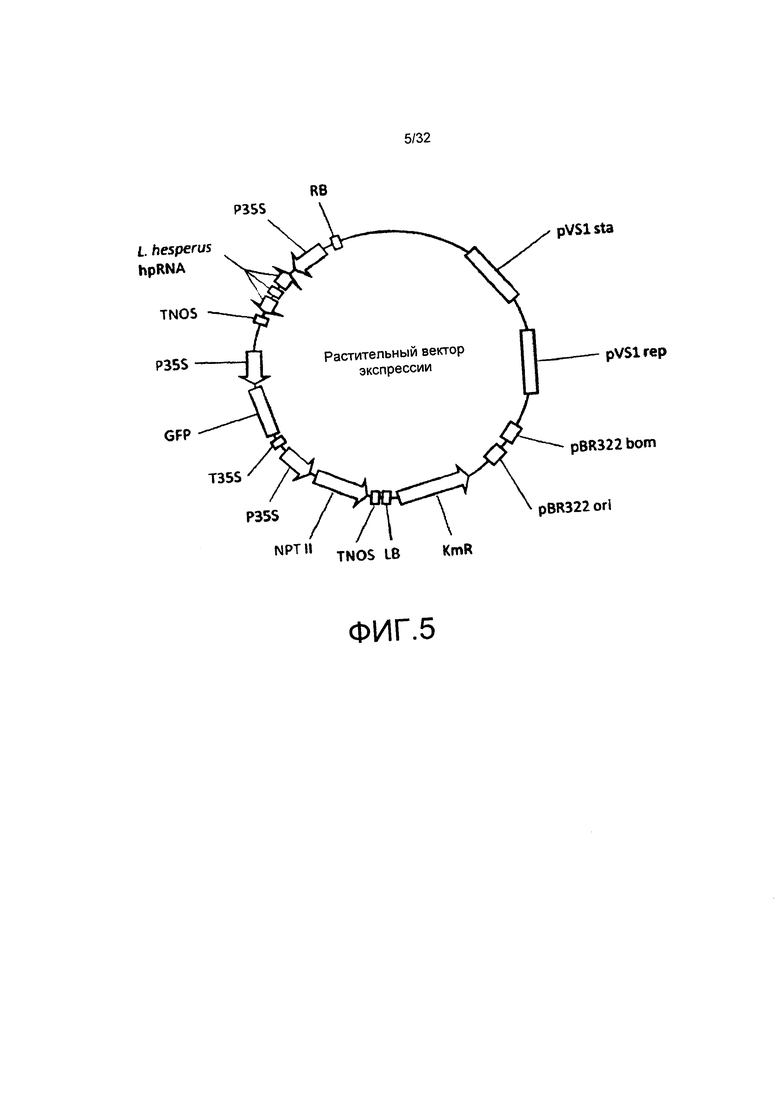
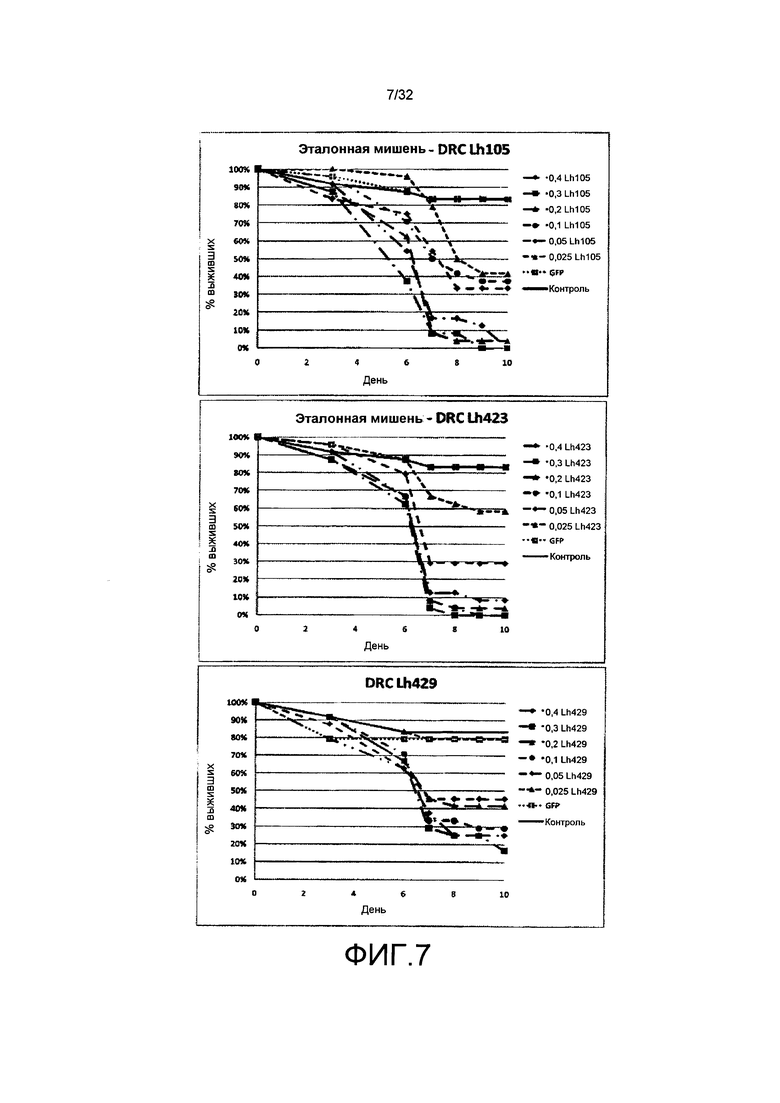
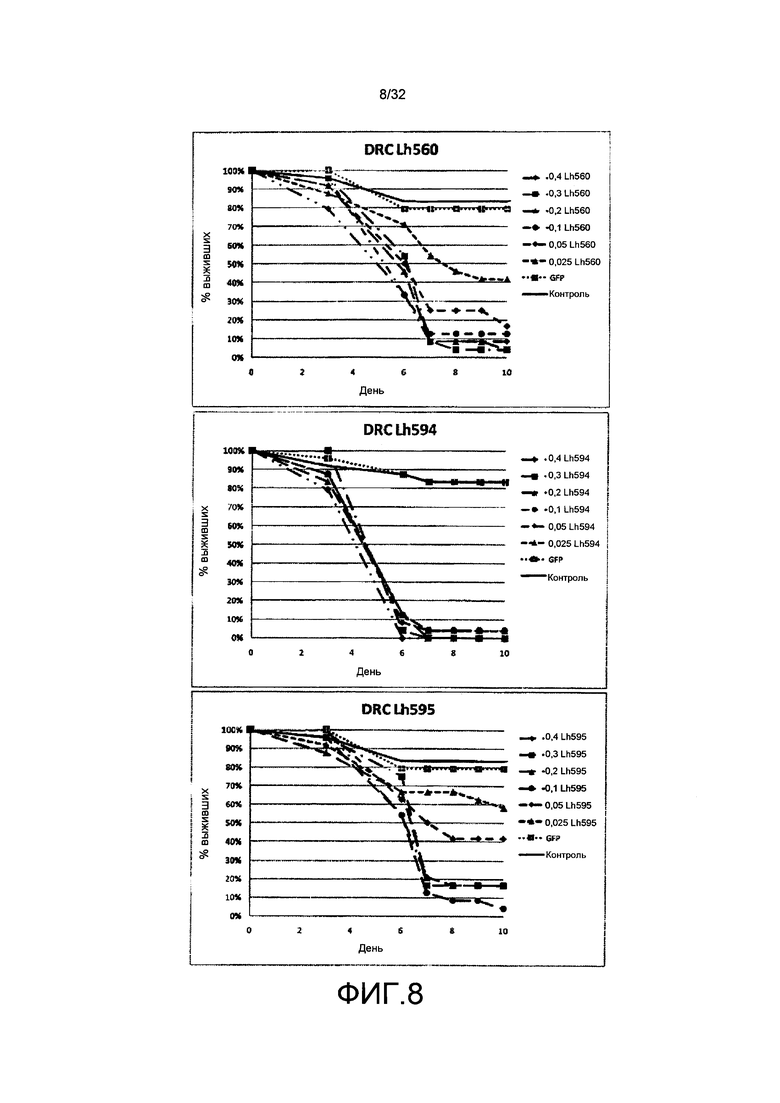
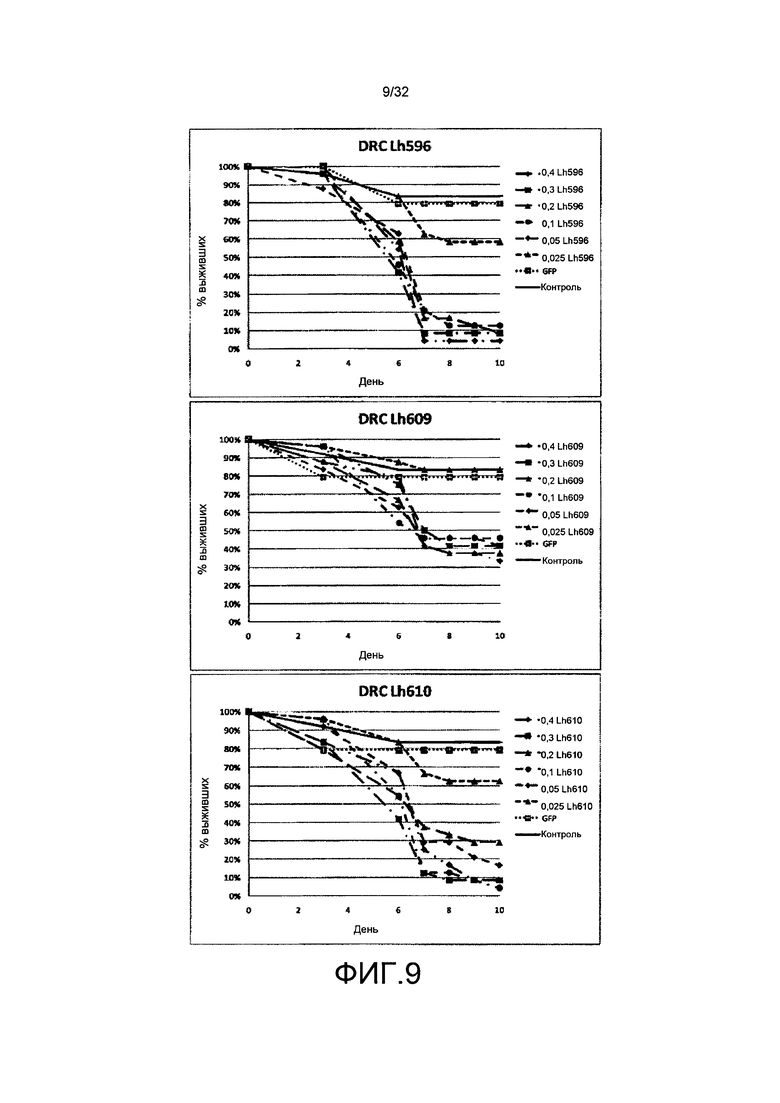
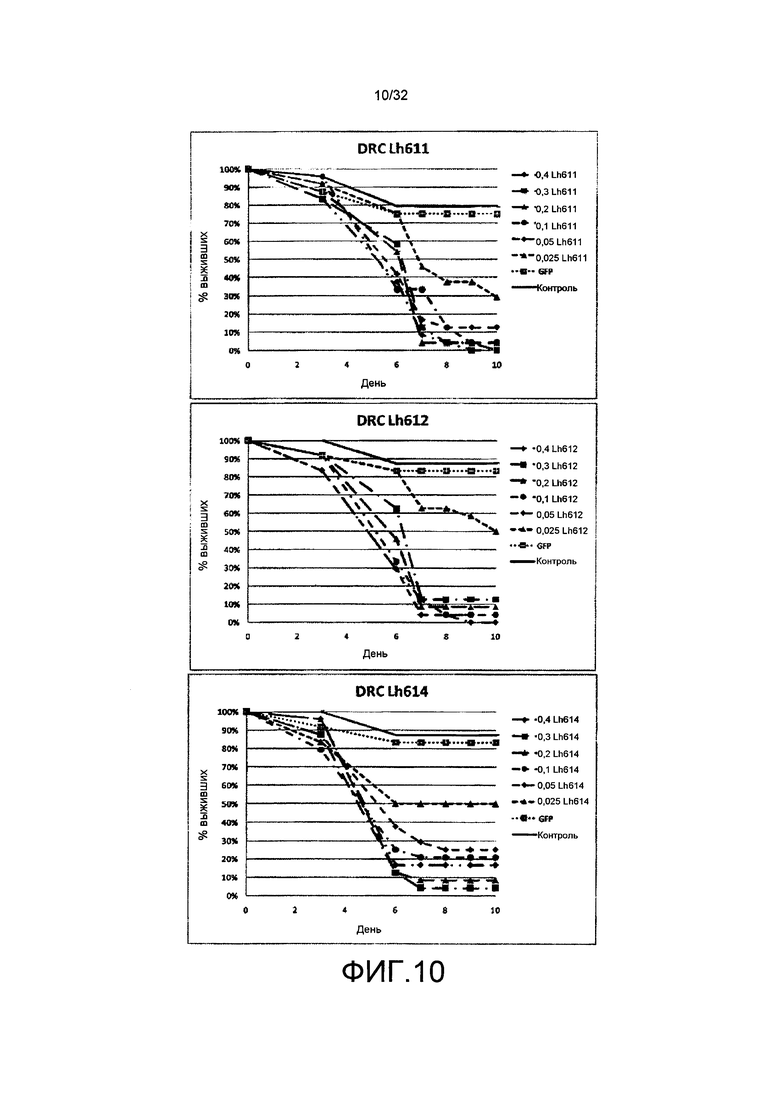
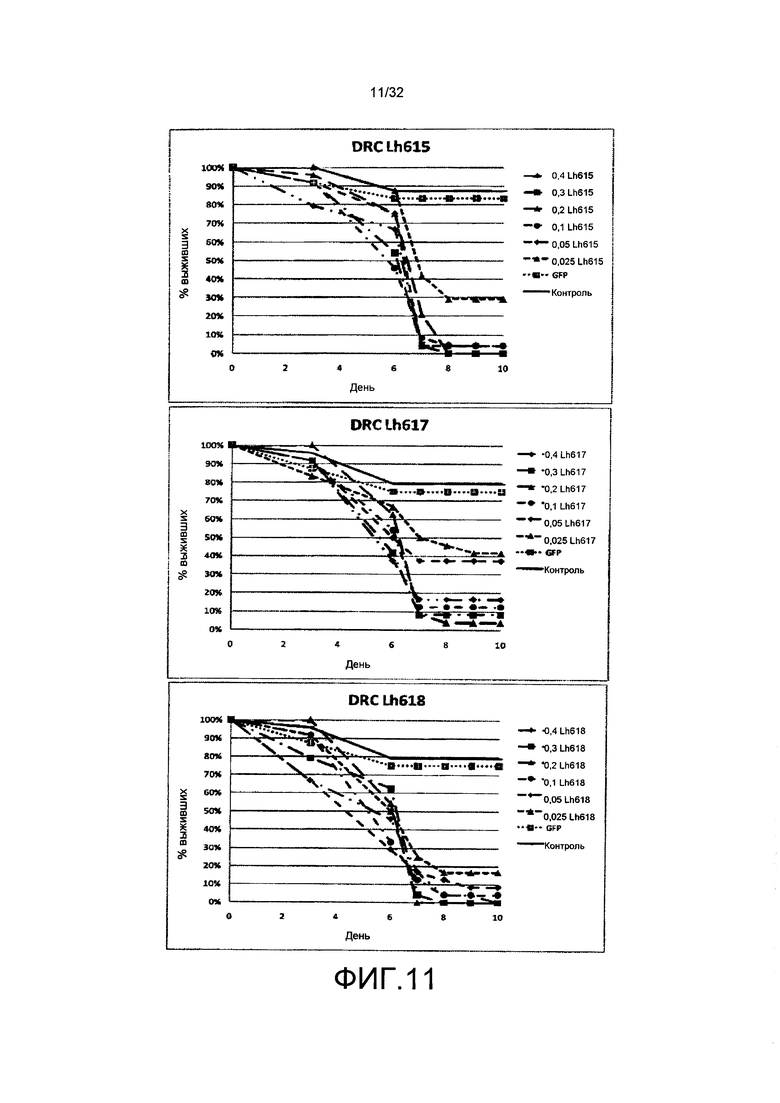
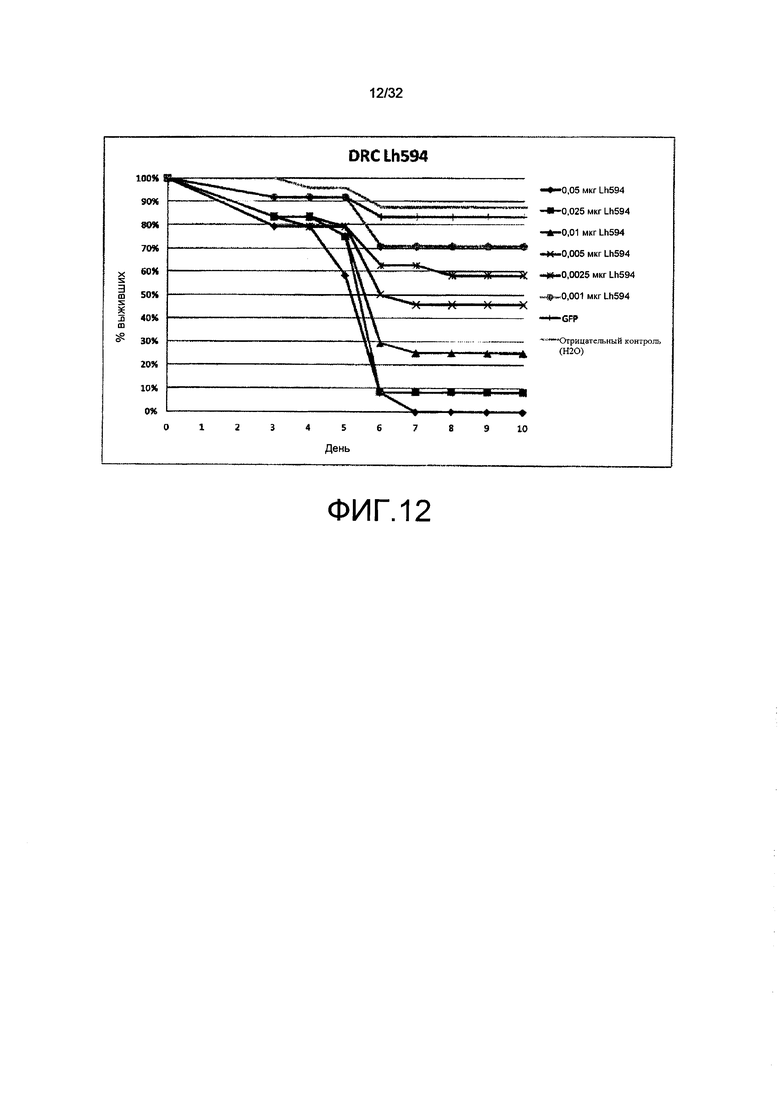
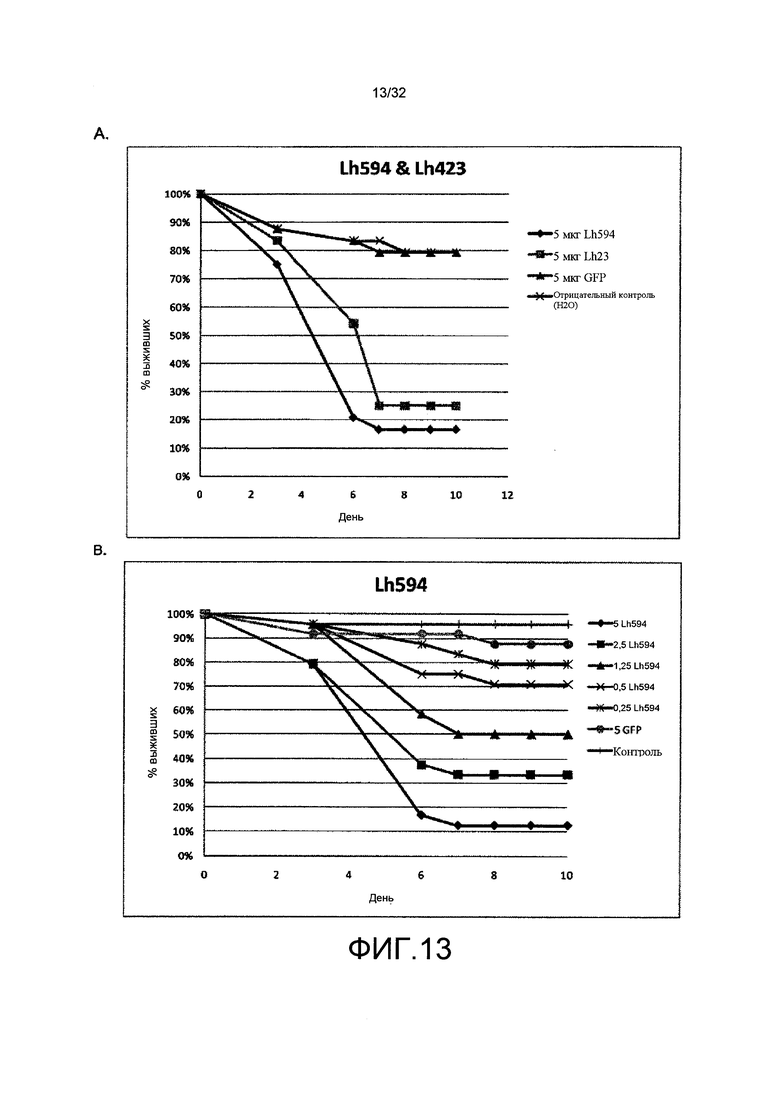

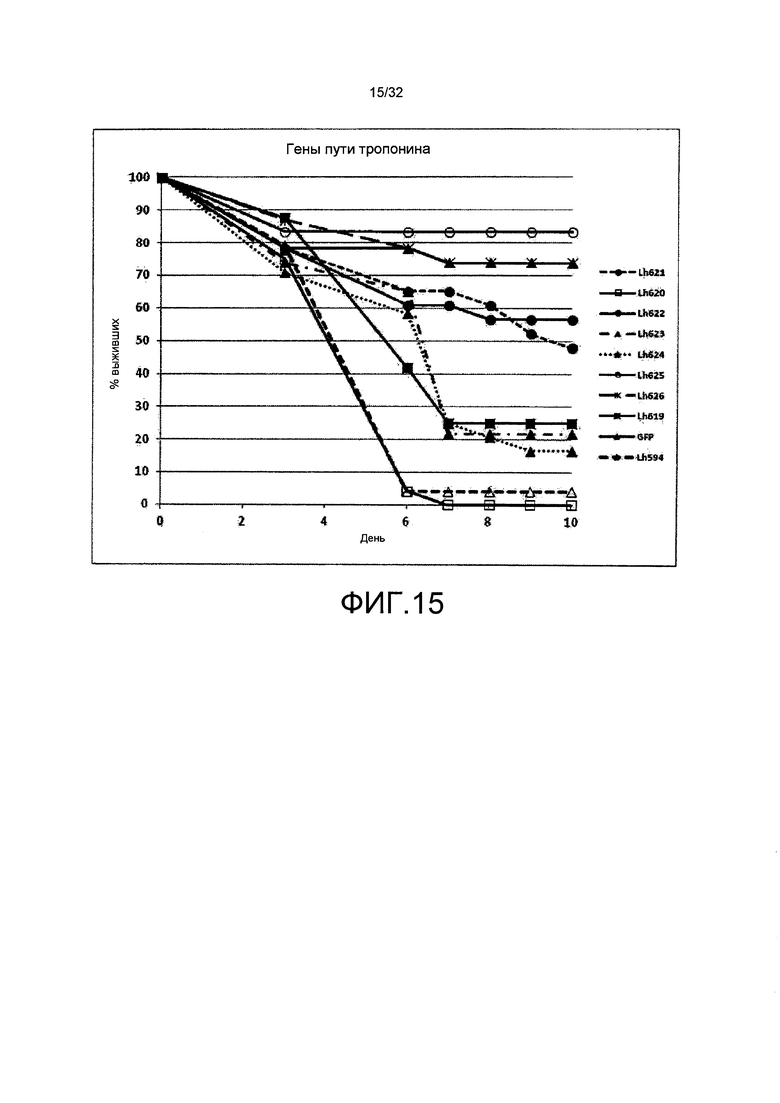
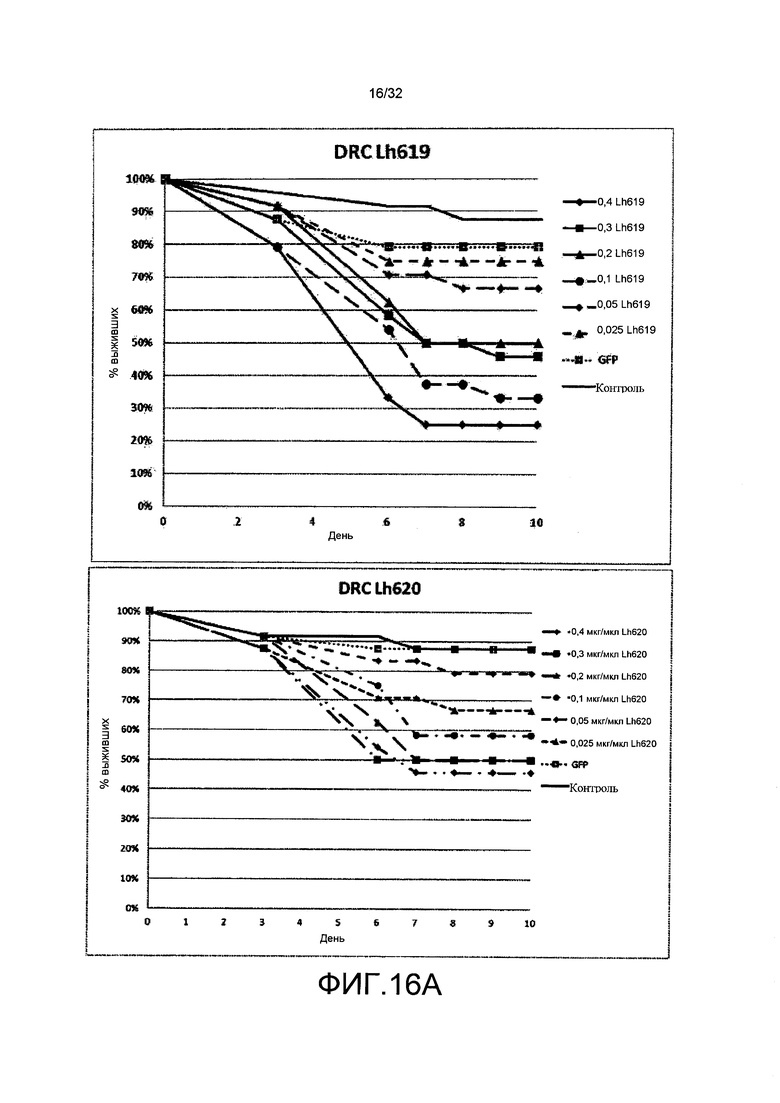
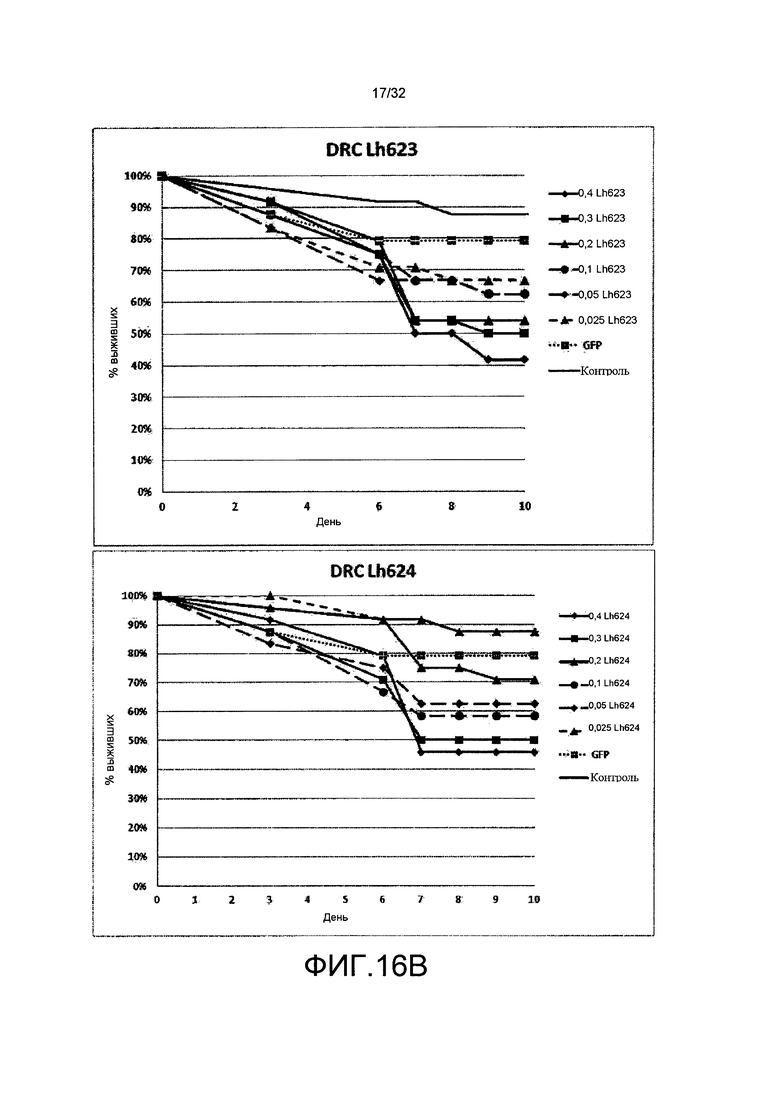

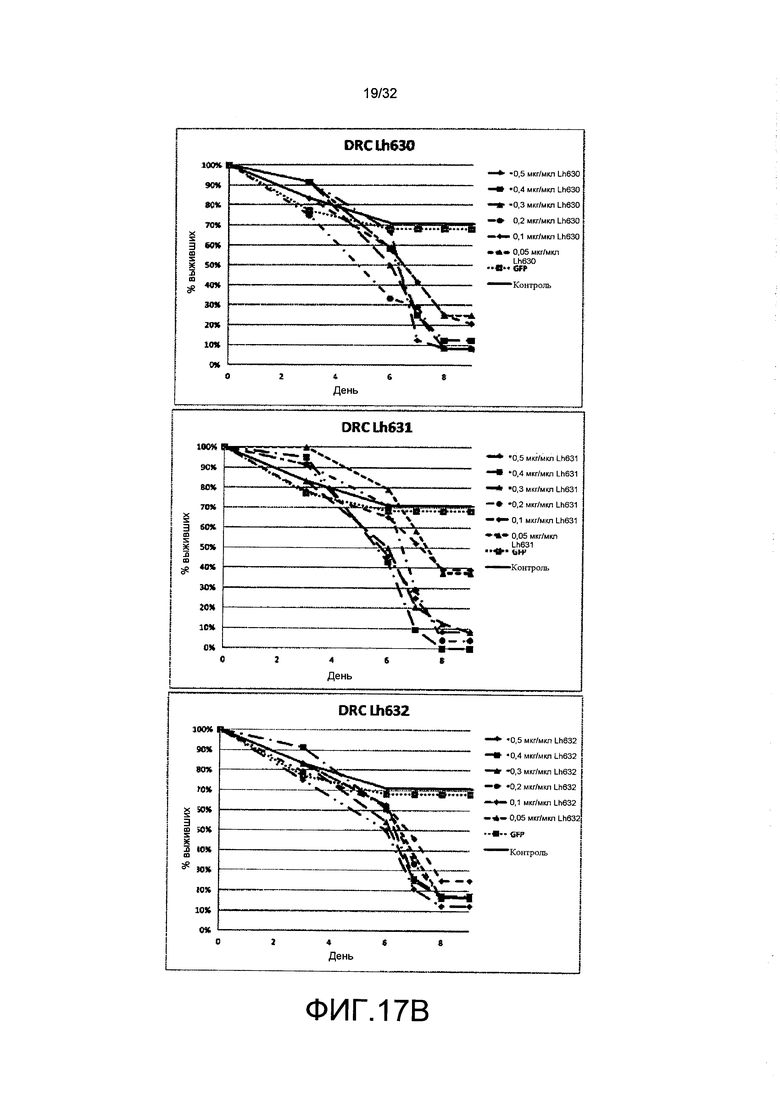
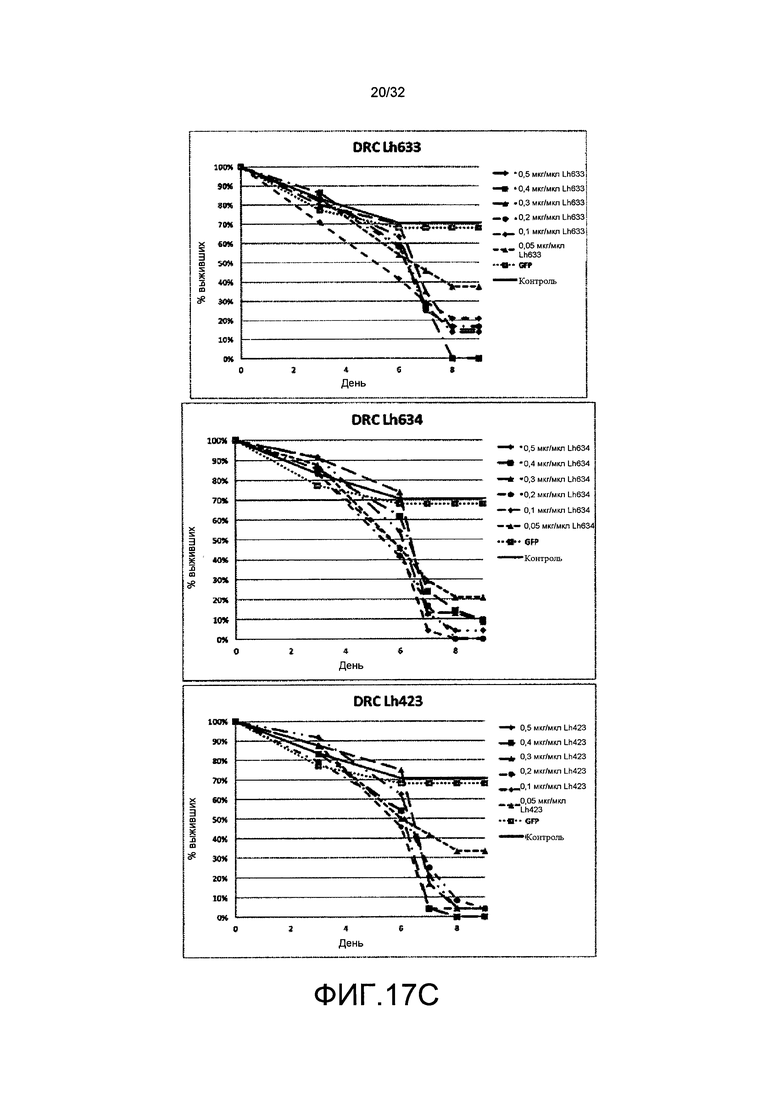
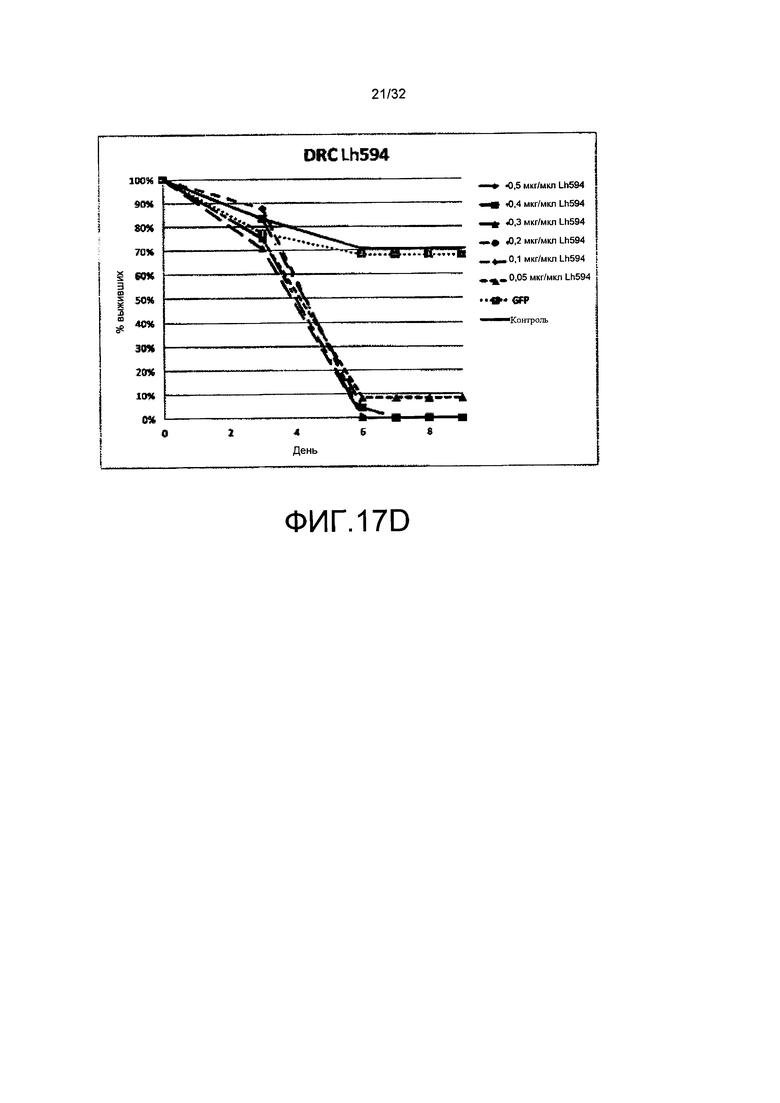
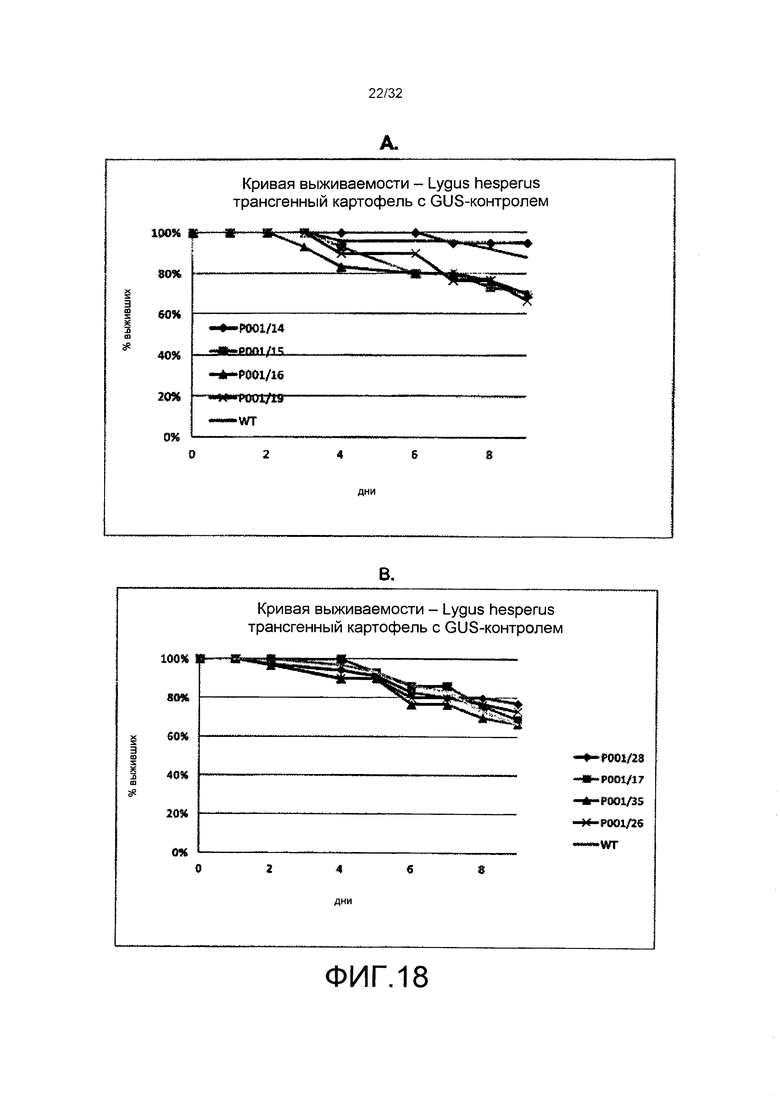
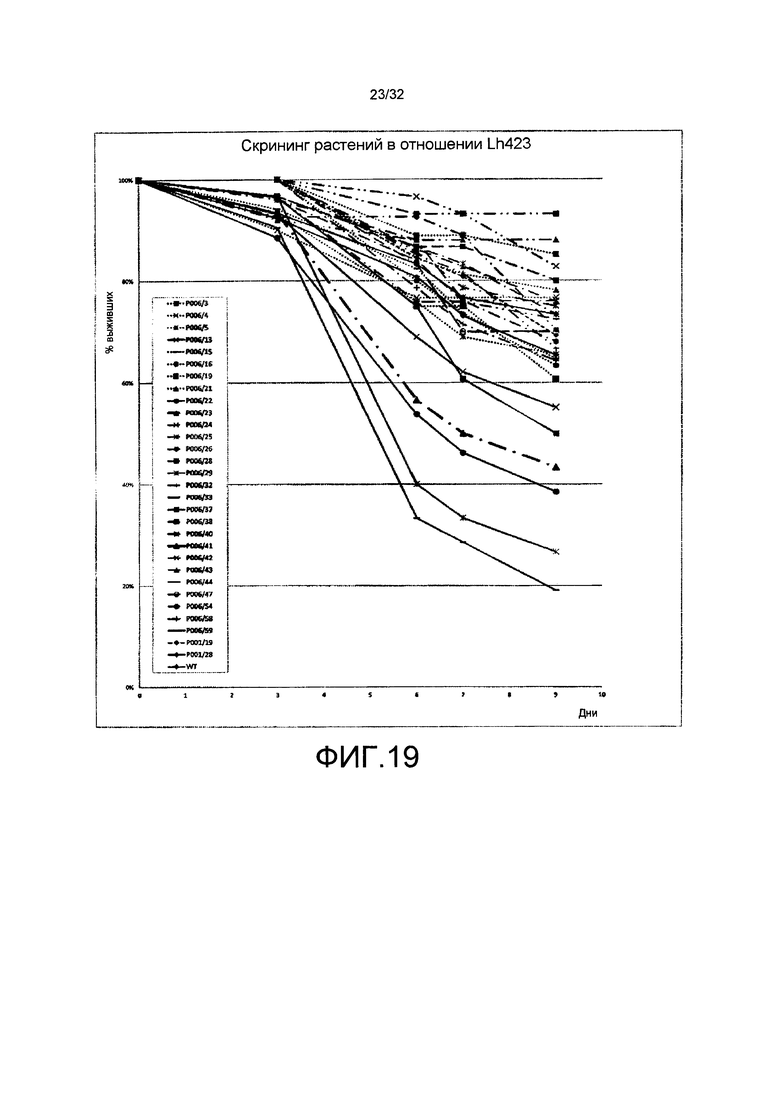
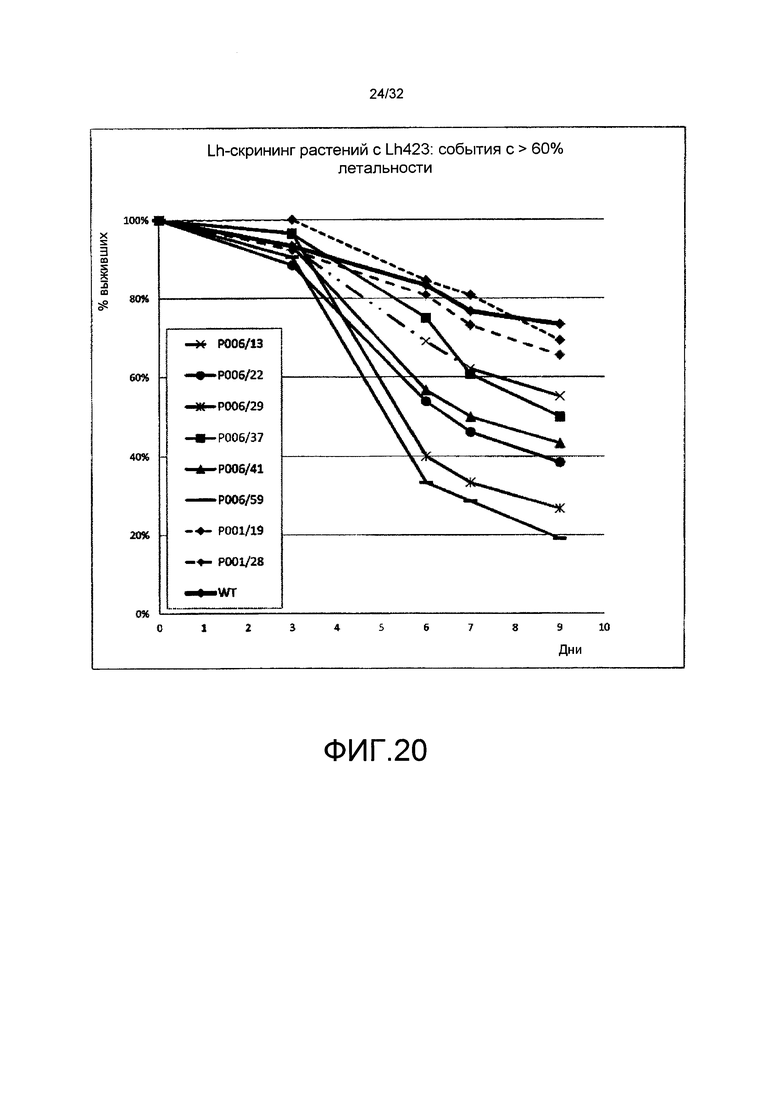
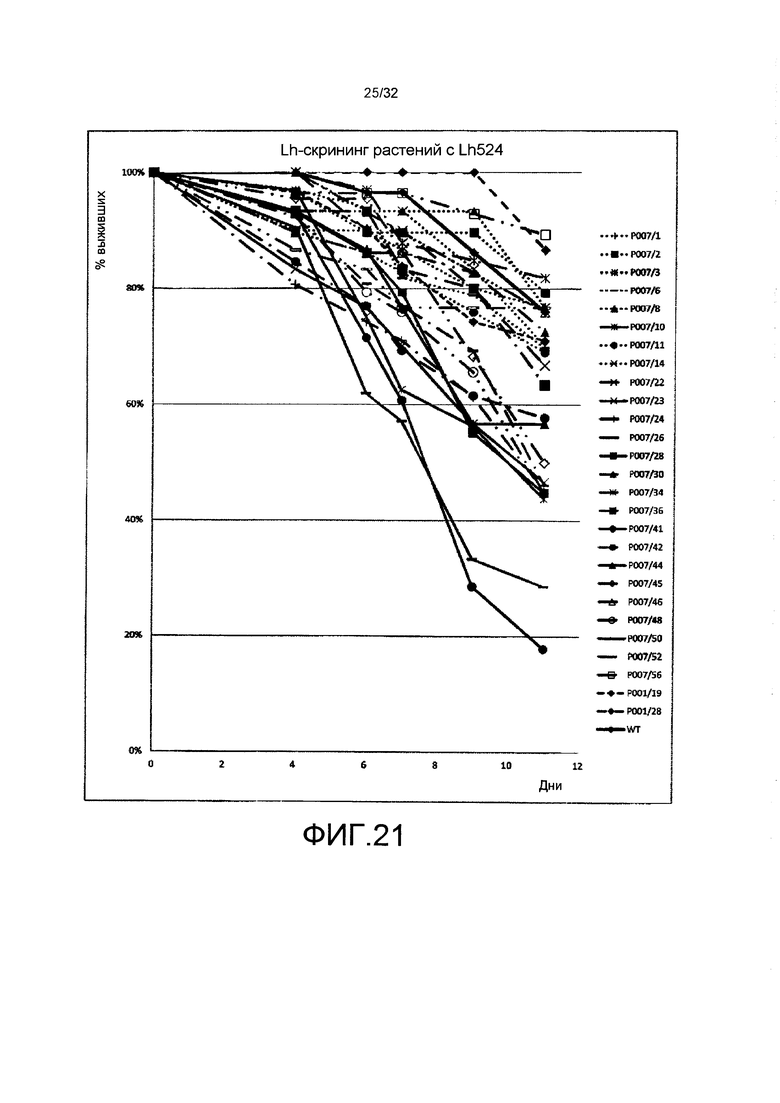
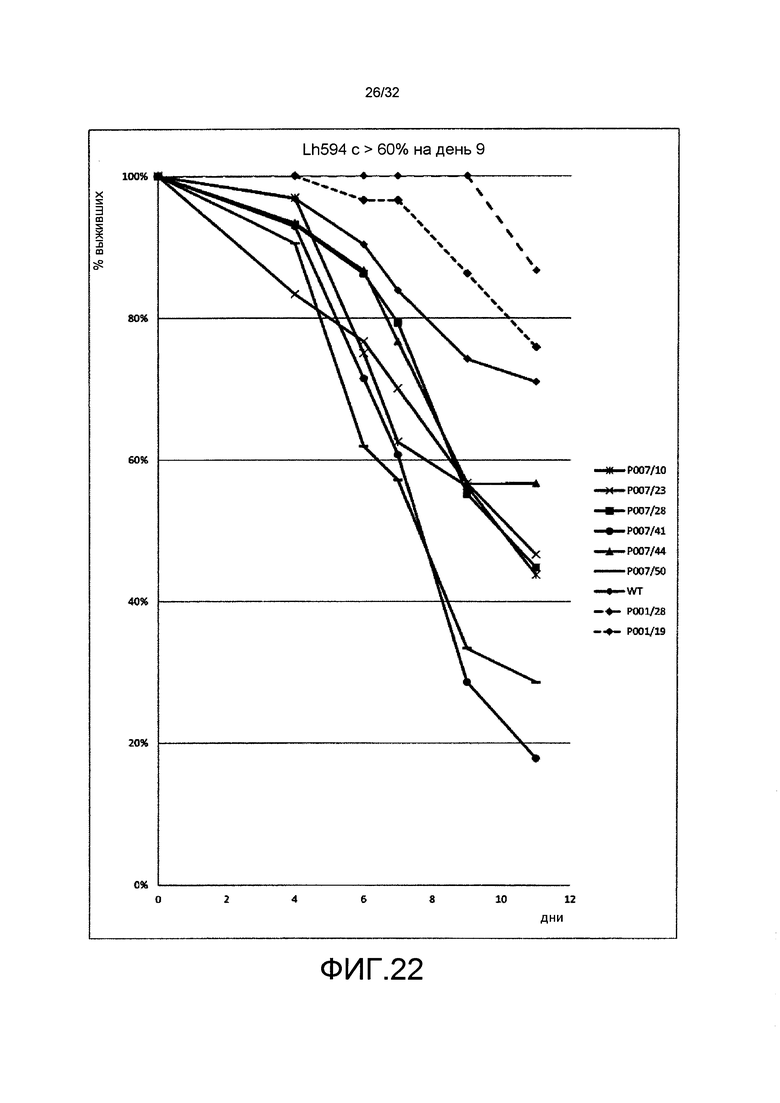
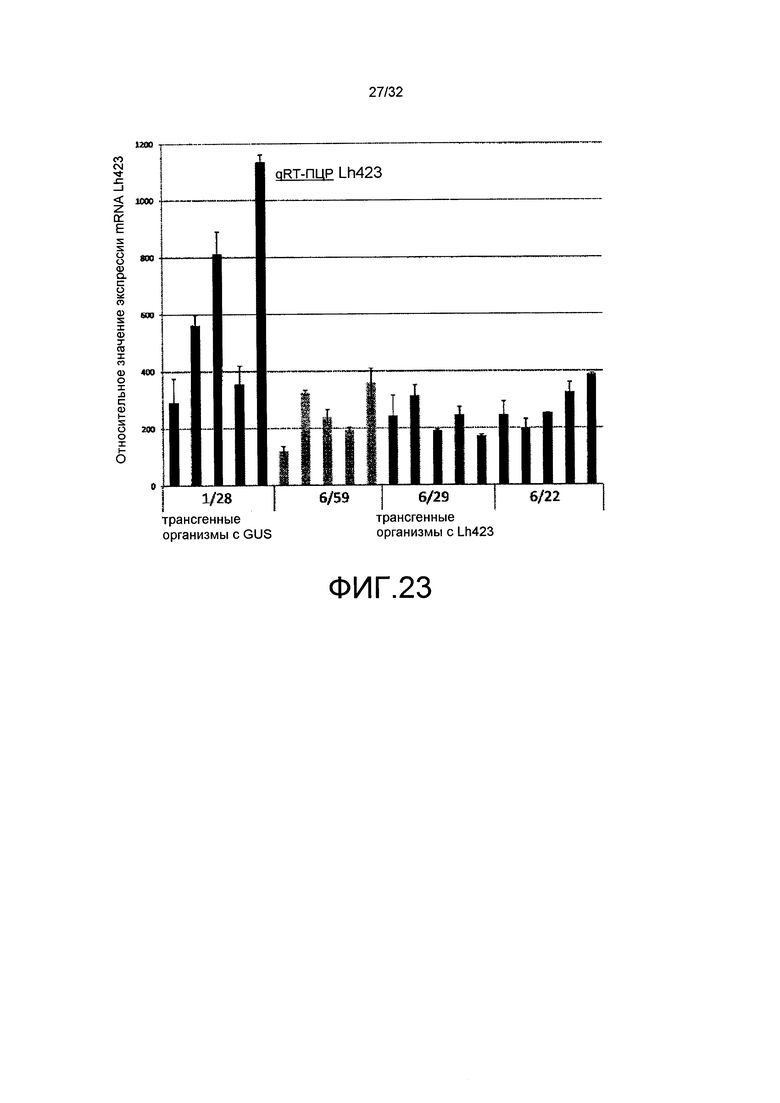
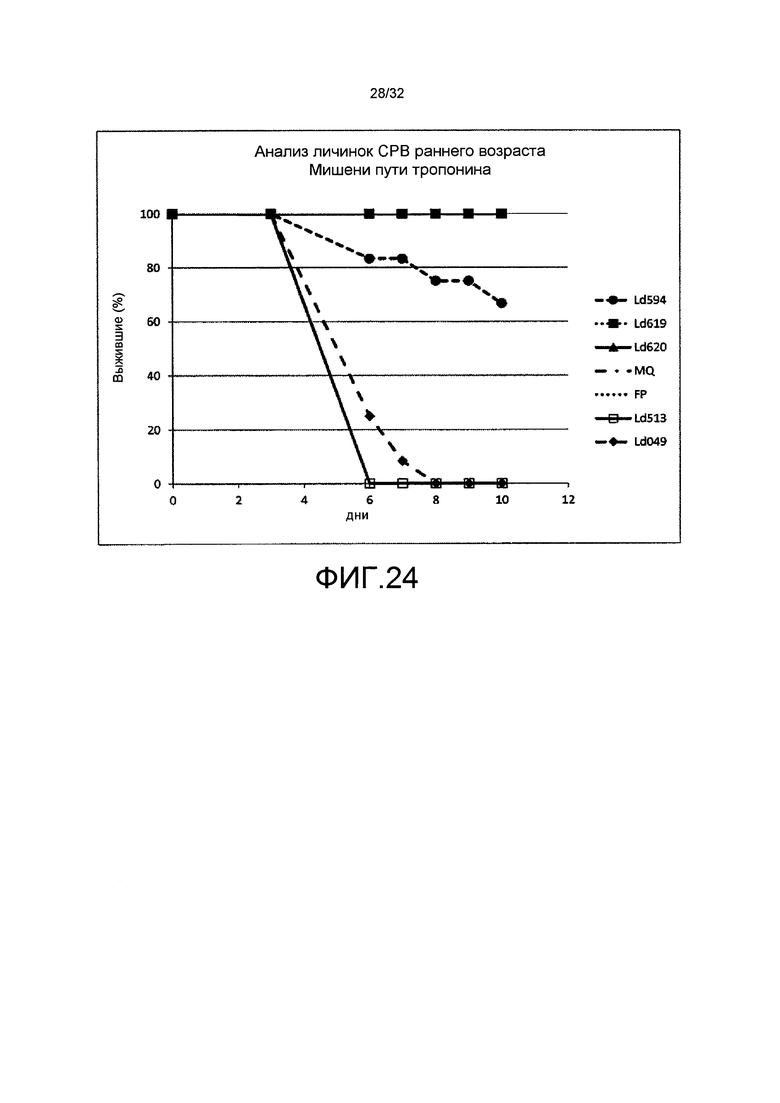

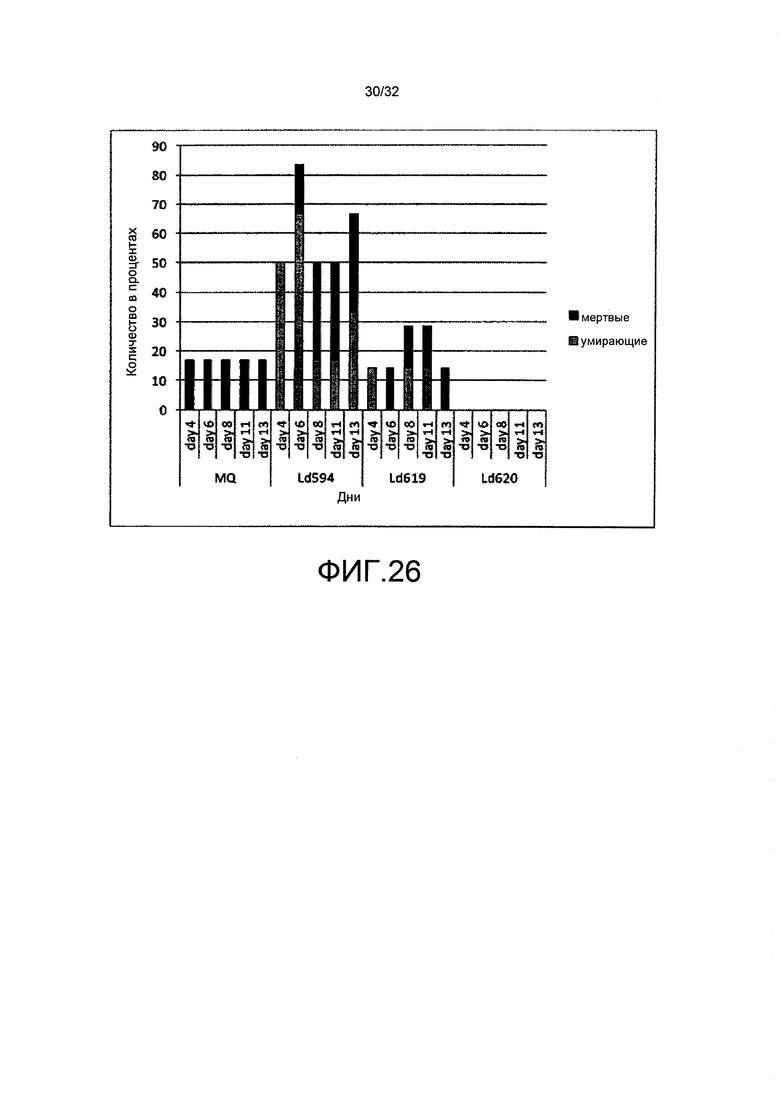
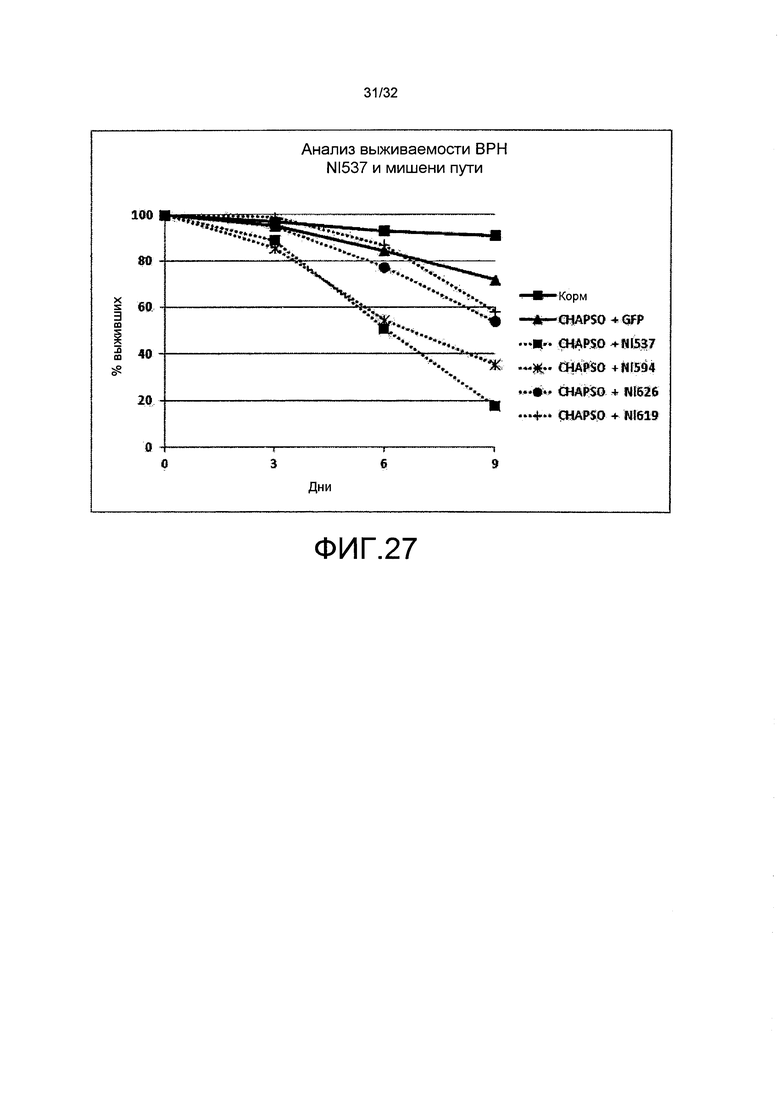

Изобретение относится к области биохимии, в частности к трансгенному растению, устойчивому к заражению насекомым-вредителем Leptinotarsa decemlineata. Также раскрыты трансгенное семя для получения указанного трансгенного растения, способ получения указанного трансгенного растения, способ идентификации трансгенного растения. Изобретение позволяет получить трансгенное растение, устойчивое к заражению насекомым-вредителем Leptinotarsa decemlineata. 6 н. и 4 з.п. ф-лы, 29 ил., 23 табл., 7 пр.
1. Трансгенное растение, устойчивое к заражению насекомым-вредителем, которое экспрессирует или способно экспрессировать одну рибонуклеиновую кислоту (РНК) для подавления гена-мишени у Leptinotarsa decemlineata, где РНК содержит по меньшей мере один сайленсирующий элемент, где сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности из по меньшей мере 200 последовательных нуклеотидов, которая идентична SEQ ID NO: 389.
2. Трансгенное растение по п. 1, где указанный ген-мишень кодирует белок rpn12.
3. Трансгенное семя для получения трансгенного растения, устойчивого к заражению насекомым-вредителем, где указанное трансгенное семя экспрессирует или способно экспрессировать одну рибонуклеиновую кислоту (РНК) для подавления экспрессии гена-мишени у Leptinotarsa decemlineata, где РНК содержит по меньшей мере один сайленсирующий элемент, где сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности из по меньшей мере 200 последовательных нуклеотидов, которая идентична SEQ ID NO: 389.
4. Способ получения трансгенного растения, устойчивого к нападению Leptinotarsa decemlineata, включающий:
(a) трансформацию растительной клетки с помощью ДНК-конструкции, содержащей полинуклеотидную последовательность, кодирующую рибонуклеиновую кислоту (РНК), для подавления гена-мишени у Leptinotarsa decemlineata, где РНК содержит по меньшей мере один сайленсирующий элемент, где сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности из по меньшей мере 200 последовательных нуклеотидов, которая идентична SEQ ID NO: 389;
(b) регенерацию растения из трансформированной растительной клетки; и
(с) выращивание трансформированного растения в условиях, подходящих для экспрессии РНК из ДНК-конструкции, при этом указанное растение является устойчивым к указанному вредителю по сравнению с нетрансформированным растением.
5. Способ по п. 4, отличающийся тем, что трансгенное растение является таким, как определено в п. 1 или 2.
6. Способ по п. 4 или 5, отличающийся тем, что способ дополнительно включает стадии получения одного или нескольких потомков от трансформированного растения и тестирования потомства на устойчивость к насекомому-вредителю.
7. Способ идентификации трансгенного растения по п. 1 или 2, где потомство, устойчивое к насекомым, идентифицируют с помощью обнаружения присутствия полинуклеотидной последовательности, кодирующей рибонуклеиновую кислоту (РНК), где РНК функционирует для подавления экспрессии гена-мишени у Leptinotarsa decemlineata, где ген-мишень идентичен SEQ ID NO: 389.
8. Способ по любому из пп. 4-7, отличающийся тем, что растение, устойчивое к нападению насекомого-вредителя, выбрано из хлопчатника, картофеля, риса, канолы, подсолнечника, сорго, проса, кукурузы, земляники, сои, люцерны, томата, баклажана, перца и табака.
9. Трансгенное растение, устойчивое к Leptinotarsa decemlineata, полученное способом по любому из пп. 4-8, где указанное трансгенное растение экспрессирует или способно экспрессировать одну рибонуклеиновую кислоту (РНК) для подавления экспрессии гена-мишени у Leptinotarsa decemlineata, где РНК содержит по меньшей мере один сайленсирующий элемент, где сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности из по меньшей мере 200 последовательных нуклеотидов, которая идентична SEQ ID NO: 389.
10. Семя для получения растений по п. 9, где указанное семя экспрессирует или способно экспрессировать одну рибонуклеиновую кислоту (РНК) для подавления экспрессии гена-мишени у Leptinotarsa decemlineata, где РНК содержит по меньшей мере один сайленсирующий элемент, где сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности из по меньшей мере 200 последовательных нуклеотидов, которая идентична SEQ ID NO: 389.
| WO 2010025320 A1, 04.03.2010 | |||
| US 0007812219 B2, 12.10.2010 | |||
| WO 2005049841 A1, 02.06.2005 | |||
| US 20060021087 A1, 26.01.2006 | |||
| РАСТИТЕЛЬНЫЕ КЛЕТКИ И ОРГАНИЗМЫ РАСТЕНИЙ С МОДИФИЦИРОВАННЫМ КЛЕТОЧНЫМ РОСТОМ, РАЗВИТИЕМ И ДИФФЕРЕНЦИРОВКОЙ | 2004 |
|
RU2349642C2 |
Авторы
Даты
2019-06-17—Публикация
2012-05-02—Подача