Область изобретения
Настоящее изобретение относится в целом к генетическому контролю нападения видов насекомых-вредителей, в частности, предотвращению и/или борьбе с нападением вредителей на растения. Более конкретно, настоящее изобретение относится к подавлению экспрессии генов-мишеней у видов насекомых-вредителей с помощью интерферирующих молекул рибонуклеиновой кислоты (РНК). Также предлагаются композиции и комбинации, содержащие интерферирующие молекулы РНК по настоящему изобретению для использования в местных применениях, например, в форме инсектицидов.
Предпосылки изобретения
Существует множество видов насекомых-вредителей, которые могут поражать или нападать на большое множество местообитаний и организмов-хозяев. Насекомые-вредители включают разнообразные виды из отрядов насекомых Hemiptera (клопы), Coleoptera (жуки), Siphonaptera (блохи), Dichyoptera (тараканы и богомолы), Lepidoptera (моли и бабочки), Orthoptera (например, кузнечики) и Diptera (настоящие мухи). Нападение вредителей может приводить к значительному ущербу. Насекомые-вредители, которые нападают на виды растений, вызывают особые проблемы в сельском хозяйстве, поскольку они могут приводить к серьезному повреждению сельскохозяйственных культур и значительно снижать урожайность растений. Большое разнообразие различных типов растений, включая товарные культуры, такие как рис, хлопчатник, соя, картофель и кукуруза, чувствительны к нападению вредителей.
Традиционно, нападение насекомых-вредителей предотвращают или контролируют посредством применения химических пестицидов. Однако данные химические вещества не всегда пригодны для применения в обработке сельскохозяйственных культур, так как они могут являться токсичными для других видов и могут наносить значительный ущерб окружающей среде. За последние десятилетия исследователи разработали более экологически безопасные способы контроля нападения вредителей. Например, применялись микроорганизмы, такие как бактерии Bacillus thuringiensis, которые в естественных условиях экспрессируют белки, токсичные для насекомых-вредителей. Ученые также выделили гены, кодирующие эти инсектицидные белки, и использовали их для создания трансгенных культур, устойчивых к насекомым-вредителям, например, растения кукурузы и хлопчатника, генетически сконструированные для продуцирования белков семейства Cry.
Хотя бактериальные токсины являлись весьма успешными в борьбе с определенными типами вредителей, они не эффективны против всех видов вредителей. Исследователи, следовательно, искали другие, более целенаправленные подходы к борьбе с вредителями и, в частности, к РНК-интерференции или “сайленсингу генов” в качестве средств для борьбы с вредителями на генетическом уровне.
РНК-интерференция или ʺRNAiʺ является процессом, посредством которого экспрессия генов в отношении клетки или всего организма подавляется последовательность-специфическим образом. RNAi является в настоящее время общепризнанным методом в данной области для ингибирования или подавления экспрессии генов у самых разнообразных организмов, включая организмы-вредители, такие как грибы, нематоды и насекомые. Кроме того, предыдущие исследования показали, что подавление экспрессии генов-мишеней у видов насекомых-вредителей можно использовать в качестве средства для борьбы с нападением вредителей.
WO2007/074405 описывает способы ингибирования экспрессии генов-мишеней у беспозвоночных вредителей, включая колорадского жука. Кроме того, WO2009/091864 описывает композиции и способы подавления генов-мишеней у видов насекомых-вредителей, включая вредителей из рода Lygus.
Хотя применение RNAi для подавления экспрессии генов у видов вредителей в данной области известно, успех данного метода в применении в качестве меры борьбы с вредителями зависит от выбора наиболее подходящих генов-мишеней, а именно тех, у которых потеря функции приводит к значительному нарушению жизненно важного биологического процесса и/или гибели организма. Настоящее изобретение, таким образом, направлено на подавление конкретных генов-мишеней у насекомых-вредителей в качестве средства достижения более эффективного предотвращения и/или борьбы с нападением насекомых-вредителей, в частности на растения.
Краткое описание изобретения
Авторы настоящего изобретения пытались идентифицировать улучшенные средства для предотвращения и/или борьбы с нападением насекомых-вредителей с применением генетических подходов. В частности, они исследовали применение RNAi для подавления генов таким образом, чтобы снизить способность насекомых-вредителей выживать, расти, колонизировать специфические местообитания и/или нападать на организмы-хозяева, и таким образом ограничить ущерб, наносимый вредителем.
Таким образом, в соответствии с одним аспектом настоящего изобретения предлагается интерферирующая рибонуклеиновая кислота (РНК или двухцепочечная РНК), которая функционирует при поглощении видами насекомых-вредителей с подавлением экспрессии гена-мишени у указанного насекомого-вредителя,
при этом РНК содержит по меньшей мере один сайленсирующий элемент, при этом сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющую такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59 -62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, таким образом, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной соответствующему указанному фрагменту любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(v) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43- 46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или
(vi) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
В конкретном аспекте настоящего изобретения молекулы интерферирующей РНК по настоящему изобретению содержат по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsRNA содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК или двухцепочечной РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233.
Эти гены-мишени кодируют белки, входящие в состав комплекса тропонин/миофиламент.
В дополнительном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК или двухцепочечной РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273.
Эти гены-мишени кодируют рибосомальные белки насекомых.
В некоторых вариантах осуществления настоящее изобретение относится к интерферирующей РНК, которая содержит по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент молекулы интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsRNA содержит последовательность по меньшей мере из 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98%, 99% или 100% комплементарной последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени из комплекса тропонин/миофиламент.
В одном варианте осуществления ген-мишень кодирует белок wings up A (тропонин I) насекомых (например, у насекомых ортолог белка CG7178 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 1, 2, 174, 404, 175, 180, 181, 188 и 189. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 79, 349, 405, 352 или 356.
В одном варианте осуществления ген-мишень кодирует белок upheld (например, у насекомых ортолог белка CG7107 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 121, 130, 142, 143, 176, 177, 182 и 183. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 330, 350 или 353.
В одном варианте осуществления ген-мишень кодирует белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm) или белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 123 и 132. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 332.
В одном варианте осуществления ген-мишень кодирует тяжелую цепь миозина (например, у насекомых ортолог белка CG17927 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 122, 131, 144, 145, 178 и 179. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 331 или 351.
В одном варианте осуществления ген-мишень кодирует цитоплазматический белок легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 124 и 133. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 333.
В одном варианте осуществления ген-мишень кодирует белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 125 и 134. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности с SEQ ID NO 334.
В одном варианте осуществления ген-мишень кодирует белок zipper (например, у насекомых ортолог белка CG15792 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 126 и 135. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности с SEQ ID NO 335.
В одном варианте осуществления ген-мишень кодирует тропонин C (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 127 и 136, или 128 и 137, или 184 и 185. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 336, 337 и 354.
Согласно другому варианту осуществления настоящее изобретение относится к интерферирующей РНК, которая содержит по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент молекулы интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsRNA содержит последовательность по меньшей мере из 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98%, 99% или 100% комплементарной последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени, который кодирует рибосомный белок насекомых.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 11, 12 и 141. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или обеими из SEQ ID NO 84 или 328.
В одном варианте осуществления ген-мишень кодирует рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), при этом указанный ген-мишень представлен посредством SEQ ID NO 3 и 4. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 80.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 7 и 8. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 82.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), представленный с помощью SEQ ID NO 9 и 10. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 83.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S18 (например, у насекомых ортолог белка CG8900 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 13 и 14. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 85.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 5 и 6. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 81.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 15 и 16, 204 и 205. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или обеими из SEQ ID NO 86 и 359.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 17 и 18. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 87.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 19 и 20. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 88.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 21 и 22. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 89.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 158 и 159. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 343.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 165, 166 и 167. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 347 и 348.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 156 и 157. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 342.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 160 и 161. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 344.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 154 и 155. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 341.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 163 и 164. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 345.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 152 и 153. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 340.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S15Aa (например, у насекомых ортолог белка CG2033 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 150 и 151. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 339.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 200 и 201. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 357.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 386. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 390.
В одном варианте осуществления ген-мишень кодирует белок субъединицы II митохондриальной цитохром с-оксидазы (например, у насекомых ортолог белка CG34069 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 25 и 26. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 91.
В одном варианте осуществления ген-мишень кодирует γ-цепь АТФ-синтазы (например, у насекомых ортолог белка CG7610 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 129 и 138. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 338.
В одном варианте осуществления ген-мишень кодирует убиквитин-5E (например, у насекомых ортолог белка CG32744 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 186 и 187, 202 и 203. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или обеими из SEQ ID NO 355 и 358.
В одном варианте осуществления ген-мишень кодирует протеасомную субъединицу бета-типа (например, у насекомых ортолог белка CG17331 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 387. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 391.
В одном варианте осуществления ген-мишень кодирует белок, который у насекомых является ортологом белка CG13704 Dm, при этом указанный ген-мишень представлен с помощью SEQ ID NO 388. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 392.
В одном варианте осуществления ген-мишень кодирует белок Rpn12 (например, у насекомых ортолог белка CG4157 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 389. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 393.
В соответствии со вторым аспектом настоящего изобретения предусматривается композиция для предотвращения и/или борьбы с нападением насекомых-вредителей, содержащая по меньшей мере одну интерферирующую рибонуклеиновую кислоту (РНК) и по меньшей мере один подходящий носитель, наполнитель или разбавитель, причем интерферирующая РНК функционирует при поглощении насекомым с подавлением экспрессии гена-мишени у указанного вредителя,
при этом РНК содержит по меньшей мере один сайленсирующий элемент, при этом сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 75% идентична любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющую такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59 -62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, таким образом, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75% идентичной любому соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% идентична любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
Композицию по настоящему изобретению можно применять для предотвращения и/или борьбы с нападением вредителя. В некоторых вариантах осуществления композицию можно применять в качестве пестицида для растения или для материала для размножения или репродуктивного материала растения. В дополнительном аспекте предусматривается комбинация для предотвращения и/или борьбы с нападением насекомых-вредителей, содержащая композицию по настоящему изобретению и по меньшей мере одно активное средство.
В дополнительном аспекте в данном документе предусматривается способ подавления экспрессии гена-мишени у вида насекомого-вредителя с целью предотвращения и/или борьбы с нападением вредителей, включающий приведение в контакт указанного вида вредителя с эффективным количеством по меньшей мере одной интерферирующей рибонуклеиновой кислоты (РНК), причем интерферирующая РНК функционирует при поглощении вредителем с подавлением экспрессии гена-мишени у указанного вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась бы по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, таким образом, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274 -277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента являлась бы по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, при этом два ортологических гена являются подобными в последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
В соответствии с дополнительным аспектом настоящего изобретения предусматривается выделенный полинуклеотид, выбранный из группы, состоящей из:
(i) полинуклеотида, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, которая представлена любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, или
(ii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, или
(iii) полинуклеотида, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, которая представлена в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, таким образом, чтобы при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид являлся по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(iv) полинуклеотида, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и при этом указанный фрагмент или указанная комплементарная последовательность обладают такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(v) полинуклеотида, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и при этом указанный фрагмент или указанная комплементарная последовательность обладают такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или
(vi) полинуклеотида, кодирующего аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, и
при этом указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
Аминокислотные последовательности, кодируемые генами-мишенями по настоящему изобретению, представлены SEQ ID NO 79, 349, 405, 352, 356, 80, 326, 81, 327, 82, 83, 328, 84, 329, 85, 86, 359, 87-91, 330, 350, 353, 331, 351, 332-336, 337, 354, 338-344, 346, 345, 347, 348, 357, 355, 358, 390-393, соответственно.
В конкретном аспекте по настоящему изобретению выделенный полинуклеотид является частью молекулы интерферирующей РНК, как правило, частью сайленсирующего элемента, содержащего по меньшей мере одну двухцепочечную область, содержащую смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, где смысловая цепь молекулы dsRNA содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени. Более конкретно, выделенный полинуклеотид клонирован в ДНК-конструкцию в смысловой и антисмысловой ориентации так, что при транскрипции смыслового и антисмыслового полинуклеотида формируется молекула dsRNA, которая функционирует при поглощении вредителем с ингибированием или подавлением экспрессии гена-мишени в указанном вредителе.
В одном варианте осуществления настоящее изобретение относится к выделенному полинуклеотиду, который клонирован в ДНК-конструкцию в смысловой и антисмысловой ориентации так, что при транскрипции смыслового и антисмыслового полинуклеотида формируется молекула dsRNA, которая функционирует при поглощении насекомым с ингибированием или подавлением экспрессии гена-мишени в пределах комплекса тропонин/миофиламент.
В одном варианте осуществления ген-мишень кодирует белок wings up A (тропонин I) насекомых (например, у насекомых ортолог белка CG7178 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 1, 2, 174, 404, 175, 180, 181, 188 и 189. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 79, 349, 405, 352 или 356.
В одном варианте осуществления ген-мишень кодирует белок upheld (например, у насекомых ортолог белка CG7107 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 121, 130, 142, 143, 176, 177, 182 и 183. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 330, 350 или 353.
В одном варианте осуществления ген-мишень кодирует белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm) или белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 123 и 132. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 332.
В одном варианте осуществления ген-мишень кодирует тяжелую цепь миозина (например, у насекомых ортолог белка CG17927 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 122, 131, 144, 145, 178 и 179. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 331 или 351.
В одном варианте осуществления ген-мишень кодирует цитоплазматический белок легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 124 и 133. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 333.
В одном варианте осуществления ген-мишень кодирует белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 125 и 134. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 334.
В одном варианте осуществления ген-мишень кодирует белок zipper (например, у насекомых ортолог белка CG15792 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 126 и 135. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности с SEQ ID NO 335.
В одном варианте осуществления ген-мишень кодирует тропонин C (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 127 и 136, или 128 и 137, или 184 и 185. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или несколькими из SEQ ID NO 336, 337 и 354.
В соответствии с другими вариантами осуществления настоящее изобретение относится к выделенному полинуклеотиду, который клонирован в ДНК-конструкцию в смысловой и антисмысловой ориентации так, что при транскрипции смыслового и антисмыслового полинуклеотида формируется молекула dsRNA, которая функционирует при поглощении насекомым с ингибированием или подавлением экспрессии гена-мишени, который кодирует рибосомный белок насекомых.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 11, 12 и 141. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или обеими из SEQ ID NO 84 или 328.
В одном варианте осуществления ген-мишень кодирует рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 3 и 4. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 80.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 7 и 8. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 82.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), представленный с помощью SEQ ID NO 9 и 10. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 83.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S18 (например, у насекомых ортолог белка CG8900 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 13 и 14. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 85.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 5 и 6. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 81.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 15 и 16, 204 и 205. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с одной или обеими из SEQ ID NO 86 и 359.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 17 и 18. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 87.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 19 и 20. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 88.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 21 и 22. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 89.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 158 и 159. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 343.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 165, 166 и 167. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 347 и 348.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 156 и 157. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 342.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 160 и 161. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 344.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 154 и 155. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 341.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 163 и 164. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 345.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 152 и 153. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 340.
В одном варианте осуществления ген-мишень кодирует рибосомный белок S15Aa (например, у насекомых ортолог белка CG2033 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 150 и 151. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 339.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 200 и 201. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 357.
В одном варианте осуществления ген-мишень кодирует рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm), при этом указанный ген-мишень представлен с помощью SEQ ID NO 386. В предпочтительном варианте осуществления ортолог у насекомого обладает по меньшей мере 85%, 90%, 92%, 94%, 96%, 98%, 99% или 100% идентичности аминокислотной последовательности с SEQ ID NO 390.
Предпочтительно способы по настоящему изобретению находят практическое применение в предотвращении и/или борьбе с нападением насекомых-вредителей, в частности, борьбе с нападением вредителей сельскохозяйственных культур, таких как, но без ограничений, хлопчатник, картофель, рис, клубника, люцерна, соя, томат, канола, подсолнечник, сорго, просо, кукуруза, баклажан, перец и табак. В дополнение, интерферирующую РНК по настоящему изобретению можно вводить в растения, подлежащие защите, с помощью обычных методов генной инженерии.
Во всех аспектах настоящего изобретения в предпочтительных вариантах осуществления ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233.
Эти гены-мишени кодируют белки, входящие в состав комплекса тропонин/миофиламент.
Во всех аспектах настоящего изобретения в предпочтительных вариантах осуществления ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266 -269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273.
Эти гены-мишени кодируют рибосомальные белки насекомых.
Во всех аспектах настоящего изобретения в предпочтительных вариантах осуществления ген-мишень (i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313.
Краткое описание таблиц и фигур
Таблица 1 Новые мишени Lygus hesperus, идентифицированные из первого анализа.
Таблица 1B Новые мишени Lygus hesperus в пути Lh594.
Таблица 1C Новые мишени Lygus hesperus, идентифицированные из второго цикла анализа.
Таблица 2 Полинуклеотидные последовательности генов-мишеней, идентифицированные у Lygus hesperus.
Таблица 3 Аминокислотные последовательности генов-мишеней, идентифицированные у Lygus hesperus.
Таблица 4 dsRNA (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие генам-мишеням Lygus hesperus и праймерам для продуцирования dsRNA.
Таблица 5 Ранжирование мишеней Lygus hesperus в соответствии с кривыми дозовой зависимости (DRC) и по сравнению с исходными мишенями Lh423 и Lh105.
Таблица 6 Мишени Lygus hesperus из второго цикла анализа-ранжирования в соответствии с DRC и по сравнению с исходными мишенями Lh423 и Lh594.
Таблица 7 Полинуклеотидные последовательности генов-мишеней, идентифицированные у колорадского жука (CPB).
Таблица 8 Аминокислотные последовательности генов-мишеней, идентифицированные у CPB.
Таблица 9 dsRNA (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие CPB генам-мишеням и праймерам для продуцирования dsRNA.
Таблица 10 Полинуклеотидные последовательности генов-мишеней, идентифицированные у коричневого дельфацида (BPH).
Таблица 11 Аминокислотные последовательности генов-мишеней, идентифицированные у BPH.
Таблица 12 dsRNA (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие BPH генам-мишеням и праймерам для продуцирования dsRNA.
Таблица 13 Праймеры, используемые для амплификации cDNA тлей, на основе геномной последовательности гороховой тли.
Таблица 14 Полинуклеотидные последовательности генов-мишеней, идентифицированные у тлей.
Таблица 15 Аминокислотные последовательности генов-мишеней, идентифицированные у тлей.
Таблица 16 dsRNA (смысловая цепь, представленная эквивалентной последовательностью ДНК), соответствующие генам-мишеням тлей и праймерам для продуцирования dsRNA.
Таблица 17 Вырожденные праймеры, используемые для амплификации CPB Ld594 cDNA.
Таблица 18 Вырожденные праймеры, используемые для амплификации BPH cDNA.
Таблица 19: Новые мишени Leptinotarsa decemlineata из данного теста.
Таблица 20: Новые идентифицированные мишени Nilaparvata lugens.
Таблица 21: Новые идентифицированные мишени Acyrthosiphon pisum.
Фигура 1: Полоски Lh001_009 второго подтверждающего анализа. Темные полосы: смертность в день 3-6, светлые полосы: смертность в день 6-8. Кандидатные клоны названы с применением “Lygxxx” скрининговых кодов и “Lhxxx” номенклатурных кодов мишеней.
Фигура 2: Полоски Lh010_020 второго подтверждающего анализа. Темные полосы: смертность в день 3-6, светлые полосы: смертность в день 6-8. Кандидатные клоны названы с применением “Lygxxx” скрининговых кодов и “Lhxxx” номенклатурных кодов мишеней.
Фигура 3: Анализ смертности новых мишеней Lygus из полосок Lh001-Lh009, выражен в % смертности за 10-дневный период. Контроли обозначены пунктирными линиями. Положительный контроль: Lh423 dsRNA (RpL19). Отрицательные контроли: GFP dsRNA и только корм (контроль).
Фигура 4: Анализ смертности новых мишеней Lygus из полосок Lh010-Lh020, выражен в % смертности за 10-дневный период. Контроли обозначены пунктирными линиями. Положительный контроль: Lh423 (RpL19). Отрицательные контроли: GFP и только корм (контроль).
Фигуры 5-9 Новые мишени Lygus hesperus-кривые дозовой зависимости при концентрациях очищенной синтетической dsRNA в диапазоне от 0,4 до 0,025 мкг/мкл (на фигуре единица измерения “мкг/мкл” не отображена). GFP dsRNA и воду milliQ применяли как отрицательные контроли. dsRNA мишеней получали с использованием праймеров, как описано в примерном разделе 1.1.
Фигура 10 Lh594 кривая дозовой зависимости, при концентрациях dsRNA в диапазоне от 0,05 до 0,001 мкг/мкл. GFP dsRNA и воду milliQ применяли как отрицательные контроли.
Фигура 11 A dsRNA активность в биопробе Lygus hesperus в отсутствие tRNA. Lh594 (5 мкг/мкл); положительный контроль: Lh423 (5 мкг/мкл); отрицательные контроли: GFP dsRNA (5 мкг/мкл) и вода milliQ; B Идентификация Lh594 предела активности с использованием уменьшающейся концентрации dsRNA (от 5 мкг до 0,25 мкг). Отрицательные контроли: GFP dsRNA (5 мкг/мкл) и вода milliQ.
Фигура 12 Полоски Lh010-Lh020 второго подтверждающего анализа мишеней второго теста. Темные полосы: смертность в день 4-8, светлые полосы: смертность в день 4-6. Кандидатные клоны названы с применением “Lygxxx” скрининговых кодов и “Lhxxx” номенклатурных кодов мишеней.
Фигура 13 Результаты анализа для мишеней Lygus пути тропонина, тестированных при 0,5 мкг/мкл фиксировано.
Фигура 14 A-B Новые мишени Lygus hesperus из пути тропонина-кривые дозовой зависимости при концентрациях очищенной синтетической dsRNA в диапазоне от 0,4 до 0,025 мкг/мкл (на фигуре единица измерения “мкг/мкл” не всегда отображена). GFP dsRNA и воду milliQ применяли как отрицательные контроли.
Фигура 15 A-D Новые мишени Lygus hesperus из мишеней второго теста - кривые дозовой зависимости при концентрациях очищенной синтетической dsRNA в диапазоне от 0,5 до 0,05 мкг/мкл. GFP dsRNA и воду milliQ применяли как отрицательные контроли.
Фигура 16 Анализ выживаемости личинок CPB, обработанных с помощью 1 мкг dsRNA Ld594, Ld619 и Ld620. Положительные контроли включали 1 мкг dsRNA исходных мишеней Ld513 и Ld049. Отрицательные контроли включали воду milliQ и FP.
Фигура 17 Эффекты Ld594, Ld619 и Ld620 dsRNA на окукливание CPB 4-й возрастной стадии личинок по сравнению с необработанным контролем (UTC). Жукам скармливали 1 мкг dsRNA, распределенной на листовых дисках картофеля, затем предоставляли возможность питаться на необработанных листьях картофеля (A) в течение 4 дней перед помещением на вермикулит. Для оценки эффекта dsRNA мертвых насекомых извлекали из вермикулита (из-за сильных эффектов, индуцированных с помощью Ld594 dsRNA, ни одну куколку невозможно восстановить из вермикулита и, следовательно, никакое изображение не доступно для данной dsRNA-мишени) (B).
Фигура 18 Эффект CPB Ld594, 619 и 620 dsRNA на выживаемость и приспособляемость имаго CPB. Оценки проводили в дни 4, 6, 7, 8, 11 и 13. Контроль MQ: вода milliQ.
Фигура 19 Активность dsRNA из Nl594 пути у коричневого дельфацида. DsRNA тестировали при 0,5 мкг/мкл в присутствии 0,1% CHAPSO. Положительный контроль: Nl537 dsRNA (0,5 мкг/мкл), отрицательные контроли: GFP dsRNA (0,5 мкг/мкл) и только корм.
Фигура 20 Активность dsRNA из Ap594, Ap423, Ap537 и Ap560 на A. pisum. DsRNA тестировали при 0,5 мкг/мкл в присутствии 5 мкг/мкл tRNA. Отрицательный контроль: GFP dsRNA (0,5 мкг/мкл).
Фигура 21 Проценты смертности личинок L. decemlineata на искусственном корме, обработанном с помощью dsRNA. Ld583, Ld584, Ld586 и Ld588 представляют клоны-мишени. Положительный контроль: Ld513; отрицательный контроль: FP.
Подробное описание настоящего изобретения
Авторы настоящего изобретения обнаружили, что подавление экспрессии конкретных генов-мишеней у видов насекомых-вредителей с помощью RNAi можно применять для эффективного предотвращения и/или борьбы с нападением указанного насекомого-вредителя.
Применяемый в данном документе термин “борьба” с нападением вредителей относится к любому воздействию на вредителя, которое служит для ограничения и/или уменьшения количества организмов вредителей и/или повреждения, вызываемого вредителем. Предпочтительные гены-мишени являются, следовательно, жизненно важными генами, которые контролируют или регулируют одну или более жизненно важные биологические функции у насекомых-вредителей, например, деление клеток, размножение, энергетический обмен, пищеварение, неврологическая функция и тому подобное. Подавление данных жизненно важных генов с помощью RNAi методов может вести к гибели насекомого или иным образом значительно замедлять рост и развитие, или нарушать способность вредителей колонизировать местообитание или нападать на организмы-хозяева.
Авторы настоящего изобретения в настоящее время идентифицировали лучшие гены-мишени видов насекомых-вредителей, принадлежащих к Lygus, Leptinotarsa, Nilaparvata и Acyrthosiphum родам, чьи мишени предусматриваются для применения в отдельности или в комбинации в качестве эффективных средств для RNAi-опосредованной борьбы с нападением насекомых на, например, агрономически важные культуры. Ортологи этих недавно идентифицированных генов-мишеней можно использовать у других видов насекомых для борьбы с нападением насекомых на соответствующие значимые культуры.
Более конкретно, авторы настоящего изобретения описывают в данном документе, что гены, кодирующие белки комплекса тропонин/миофиламент, образуют отличные гены-мишени для подавления с помощью методики РНК-ингибирования. Один из данных генов-мишеней кодировал белок тропонин I (wings up A) насекомых, который является ортологом белка CG7178 дрозофилы. Данный белок участвует в мышечных сокращениях и принадлежит к физиологическому пути, который еще не полностью исследован для борьбы с (насекомыми) вредителями посредством РНК-ингибирования. Кроме того, поскольку данный белковый комплекс является специфичным для животных, не известен ни один гомолог или ортолог генов растений, что снижает риск нетипичных фенотипов растений при экспрессии dsRNA-мишени в растениях. В дополнение, у дрозофилы тропонин I описывается как гаплонедостаточный ген, проявляющий мутантный фенотип в гетерозиготном состоянии. Такие гены особенно восприимчивы к снижению уровней экспрессии mRNA и как таковые могут считаться идеальными мишенями RNAi.
Дополнительные вызывающие интерес гены-мишени в данном комплексе тропонин/миофиламент перечислены ниже.
Таким образом, в соответствии с одним вариантом осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233.
В предпочтительном варианте осуществления ген-мишень кодирует белок насекомых, выбранный из комплекса тропонин/миофиламент, выбранный из группы, включающей тропонин I (например, у насекомых ортолог белка CG7178 Dm), белок upheld (например, у насекомых ортолог белка CG7107 Dm), белок тропомиозин 1 (например, у насекомых ортолог белка CG4898 Dm), белок тропомиозин 2 (например, у насекомых ортолог белка CG4843 Dm), тяжелую цепь миозина (например, у насекомых ортолог белка CG17927 Dm), цитоплазматический белок легкой цепи миозина (например, у насекомых ортолог белка CG3201 Dm), белок spaghetti squash (например, у насекомых ортолог белка CG3595 Dm), белок zipper (например, у насекомых ортолог белка CG15792 Dm), тропонин C (например, у насекомых ортолог белка CG2981, CG7930, CG9073, CG6514, CG12408, CG9073, CG7930, CG2981, CG12408 или CG6514 Dm).
В других вариантах осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75% предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273.
В предпочтительном варианте осуществления ген-мишень кодирует рибосомный белок насекомых, выбранный из группы, содержащей рибосомный белок S3A (например, у насекомых ортолог белка CG2168 Dm), рибосомный белок LP1 (например, у насекомых ортолог белка CG4087 Dm), рибосомный белок S3 (например, у насекомых ортолог белка CG6779 Dm), рибосомный белок L10Ab (например, у насекомых ортолог белка CG7283 Dm), рибосомный белок S18 (например, у насекомых ортолог белка CG8900 Dm), рибосомный белок L4 (например, у насекомых ортолог белка CG5502 Dm), рибосомный белок S27 (например, у насекомых ортолог белка CG10423 Dm), рибосомный белок L6 (например, у насекомых ортолог белка CG11522 Dm), рибосомный белок S13 (например, у насекомых ортолог белка CG13389 Dm), и рибосомный белок L12 (например, у насекомых ортолог белка CG3195 Dm), рибосомный белок L26 (например, у насекомых ортолог белка CG6846 Dm), рибосомный белок L21 (например, у насекомых ортолог белка CG12775 Dm), рибосомный белок S12 (например, у насекомых ортолог белка CG11271 Dm), рибосомный белок S28b (например, у насекомых ортолог белка CG2998 Dm), рибосомный белок L13 (например, у насекомых ортолог белка CG4651 Dm), рибосомный белок L10 (например, у насекомых ортолог белка CG17521 Dm), рибосомный белок L5 (например, у насекомых ортолог белка CG17489 Dm), рибосомный белок S15Aa (например, у насекомых ортолог белка CG2033 Dm), рибосомный белок L19 (например, у насекомых ортолог белка CG2746 Dm), рибосомный белок L27 (например, у насекомых ортолог белка CG4759 Dm).
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 141, 11, 12 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 141, 11, 12 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 141, 11, 12 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 141, 11, 12 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 141, 11, 12 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 141, 11, 12 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 141, 11, 12 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 141, 11, 12 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 141, 11, 12 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 141, 11, 12, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 141, 11, 12.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 17, 18 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 17, 18 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 17, 18 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 17, 18 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 17, 18 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 17, 18 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 17, 18 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 17, 18 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 17, 18 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 17, 18, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 17, 18.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 19, 20 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 19, 20 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 19, 20 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 19, 20 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 19, 20 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 19, 20 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 19, 20 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 19, 20 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 19, 20 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 19, 20, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 19, 20.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 165, 166, 167 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 165, 166, 167 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 165, 166, 167 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 165, 166, 167 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 165, 166, 167 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 165, 166, 167 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 17, 18 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 165, 165, 167 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 165, 166, 167 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 165, 166, 167, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 165, 166, 167.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 145, 122, 144, 178, 131, 179 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 145, 122, 144, 178, 131, 179 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 145, 122, 144, 178, 131, 179 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 145, 122, 144, 178, 131, 179, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 145, 122, 144, 178, 131, 179.
В одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, при этом РНК содержит по меньшей мере один сайленсирующий элемент, причем сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 128, 149, 184, 137 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 128, 149, 184, 137 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 128, 149, 184, 137 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 128, 149, 184, 137 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 128, 149, 184, 137 или комплементарной ей последовательности, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из любой из SEQ ID NO 128, 149, 184, 137 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 128, 149, 184, 137 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 128, 149, 184, 137 или комплементарной ей последовательности, или
(iv) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 128, 149, 184, 137 или комплементарную ей последовательность, где два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из последовательностей, представленных в SEQ ID NO 128, 149, 184, 137, или
(v) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85%, предпочтительно по меньшей мере на 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 128, 149, 184, 137.
В еще одном варианте осуществления настоящее изобретение относится к интерферирующей рибонуклеиновой кислоте (РНК или двухцепочечной РНК), которая подавляет экспрессию гена-мишени, кодирующего белок субъединицы II митохондриальной цитохром с-оксидазы (например, у насекомых ортолог белка CG34069 Dm).
Таким образом, в одном аспекте настоящее изобретение предлагает интерферирующую рибонуклеиновую кислоту (РНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя.
Применяемый в данном документе термин “ген-мишень” включает любой ген у насекомого-вредителя, который предполагается подавить. В предпочтительном варианте осуществления ген-мишень подавляется так, чтобы бороться с нападением вредителя, например, путем нарушения жизненно важного биологического процесса, происходящего у вредителя, или путем уменьшения патогенности вредителя. Предпочтительные гены-мишени, следовательно, включают, но без ограничений, те, которые играют ключевую роль в регуляции питания, выживания, роста, развития, размножения, нападения и способности вызывать поражение. В соответствии с одним вариантом осуществления ген-мишень является таковым, что при подавлении или ингибировании его экспрессии насекомое-вредитель погибает. В соответствии с другим вариантом осуществления ген-мишень является таковым, что при подавлении или ингибировании его экспрессии рост вредителя предотвращается или замедляется, или останавливается, или задерживается, или затрудняется, размножение вредителя предотвращается, или предотвращается переход через жизненные циклы вредителя. В соответствии еще с другим вариантом осуществления настоящего изобретения ген-мишень является таковым, что при подавлении или ингибировании его экспрессии повреждение, вызванное вредителем и/или способностью вредителя поражать или нападать на местообитания, поверхности и/или растение, или сельскохозяйственную культуру уменьшается; или вредитель прекращает питаться из своих природных пищевых ресурсов, таких как растения и растительные продукты. Термины “нападать” и “поражать” или “нападение” и “поражение”, как правило, используются взаимозаменяемо повсеместно.
Гены-мишени могут экспрессироваться во всех или некоторых клетках насекомых-вредителей. Кроме того, гены-мишени могут экспрессироваться насекомым-вредителем только на определенной стадии его жизненного цикла, например, зрелой стадии имаго, незрелой нимфы или личиночной стадии, или стадии яйца.
Применяемые в данном документе виды “вредителей” предпочтительно являются видами насекомых, которые вызывают поражение или нападают, предпочтительно на растения. Виды насекомых могут включать и виды, принадлежащие к отрядам Coleoptera, Lepidoptera, Diptera, Dichyoptera, Orthoptera, Hemiptera или Siphonaptera.
Предпочтительными насекомыми, патогенными для растений, в соответствии с настоящим изобретением, являются вредители растений, выбранные из группы, включающей Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук), или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Laodelphax spp. (например, L. striatellus (малый коричневый дельфацид)); Nephotettix spp. (например, N. virescens, или N. cincticeps (зеленая рисовая цикадка), или N.nigropictus (рисовая цикадка)); Sogatella spp. (или S. furcifera (белоспинный рисовый дельфацид)); Chilo spp. (например, C. suppressalis (огневка азиатская стеблевая), C. auricilius (огневка Chilo auricilius), или C. polychrysus (темноголовая рисовая огневка)); Sesamia spp. (например, S. inferens (розовый сверлильщик)); Tryporyza spp. (например, T. innotata (огневка Tryporyza innotata), или T. incertulas (желтый рисовый сверлильщик)); Anthonomus spp. (например, A. grandis (долгоносик хлопковый)); Phaedon spp. (например, P. cochleariae (хреновый листоед)); Epilachna spp. (например, E. varivetis (зерновка бобовая мексиканская)); Tribolium spp. (например, T. castaneum (хрущак каштановый)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук), D. virgifera zeae (мексиканский кукурузный корнегрыз); Ostrinia spp. (например, O. nubilalis (огневка кукурузная)); Anaphothrips spp. (например, A. obscrurus (трипс злаковый)); Pectinophora spp. (например, P. gossypiella (розовый коробочный червь хлопчатника)); Heliothis spp. (например, H. virescens (табачная листовертка)); Trialeurodes spp. (например, T. abutiloneus (белокрылка Trialeurodes abutiloneus) T. vaporariorum (белокрылка тепличная)); Bemisia spp. (например, B. argentifolii (белокрылка магнолиевая)); Aphis spp. (например, A. gossypii (тля хлопковая)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Euschistus spp. (например, E. conspersus (клоп Euschistus conspersus)); Chlorochroa spp. (например, C. sayi (клоп Сэя)); Nezara spp. (например, N. viridula (зеленый овощной клоп)); Thrips spp. (например, T. tabaci (трипс луковый)); Frankliniella spp. (например, F. fusca (трипс табачный), или F. occidentalis (западный цветочный трипс)); Acheta spp. (например, A. domesticus (сверчок домовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Macrosiphum spp. (например, M. euphorbiae (тля картофельная большая)); Blissus spp. (например, B. leucopterus leucopterus (клоп-черепашка пшеничный североамериканский)); Acrosternum spp. (например, A. hilare (клоп-щитник Acrosternum hilare)); Chilotraea spp. (например, C. polychrysa (огневка Chilotraea polychrysa)); Lissorhoptrus spp. (например, L. oryzophilus (долгоносик рисовый водяной)); Rhopalosiphum spp. (например, R. maidis (тля кукурузная)); и Anuraphis spp. (например, A. maidiradicis (тля кукурузная корневая)).
В соответствии с более конкретным вариантом осуществления настоящее изобретение применимо к видам, принадлежащим к семейству Chrysomelidae или листоеды. Жуки листоеды, такие как колорадские жуки, блошки, кукурузные корневые жуки, и долгоносики, такие как долгоносик люцерновый, являются особенно важными вредителями. Конкретные виды рода Leptinotarsa, контролируемые в соответствии с настоящим изобретением, включают колорадского жука (Leptinotarsa decemlineata (Say)) и ложного картофельного жука (Leptinotarsa juncta (Say)). CPB является (серьезным) вредителем нашего домашнего картофеля, других культурных и диких клубненосных и неклубненосных видов картофеля и других видов растений семейства Solanaceous (пасленовых), включая сельскохозяйственные культуры томатов, баклажанов, перцев, табака (виды рода Nicotiana, включая декоративные), физалиса, риса, кукурузы или хлопчатника; и виды сорняков/травянистых растений, паслен каролинский, черный паслен, дурман обыкновенный, белену и паслен клювообразный. Кукурузные корневые жуки включают виды, основывающие род Diabrotica (например, D. undecimpunctata undecimpunctata, D. undecimpunctata howardii, D. longicornis, D. virgifera и D. balteata). Кукурузные корневые жуки причиняют существенный вред кукурузе и тыквенным.
В соответствии с более конкретным вариантом осуществления настоящее изобретение применимо к видам, принадлежащим к отряду Hemipterans (семейство Aphidoidea), таким как Myzus persicae (тля персиковая зеленая), Aphis fabae (тля свекловичная), Acyrthosiphum pisum (тля гороховая), Brevicoryne brassicae (тля капустная), Sitobion avenae (тля большая злаковая), Cavariella aegopodii (тля зонтичная), Aphis craccivora (тля арахисовая), Aphis gossypii (тля хлопковая), Toxoptera aurantii (тля померанцевая), Cavariella spp (тля тминная), Chaitophorus spp (тли рода Chaitophorus), Cinara spp.(тли рода Cinara), Drepanosiphum platanoides (тля яворовая), Elatobium spp (тли рода Elatobium), которые вызывают повреждение растений, таких как деревья рода сливы, в частности, персик, абрикос и слива; деревьев, которые в основном культивируются для производства древесины, таких как ивы и тополя, пропашных культур, таких как кукуруза, хлопчатник, соя, пшеница и рис, овощных культур из семейств Solanaceae, Chenopodiaceae, Compositae, Cruciferae и Cucurbitaceae, включая, но без ограничений, артишок, спаржу, фасоль, свеклу, брокколи, брюссельскую капусту, кочанную капусту, морковь, цветную капусту, мускусную дыню, сельдерей, кукурузу, огурцы, укроп, капусту кормовую, кольраби, репу, баклажан, салат, горчицу, бамию, петрушку, пастернак, горох, перец, картофель, редис, шпинат, тыкву, томат, репу, кресс водяной и арбуз; или полевых культур, таких как, но без ограничений, табак, сахарная свекла и подсолнечник; цветочных культур или других декоративных растений, таких как сосны и хвойные деревья. Другие Hemipterans относятся к Nilaparvata ssp (например N. lugens, Sogatella furcifera) и причиняют вред растениям риса. Другие Hemipterans относятся к Lygus ssp (например, Lygus hesperus, Lygus rugulipennis, Lygus lineolaris, Lygus sully) и другим видам растительноядных насекомых в семействе Miridae, и причиняют вред хлопчатнику, растениям картофеля, клубнике, хлопчатнику, люцерне, канола, персику, сливам, винограду, латуку, баклажану, луку, зеленым бобам. А также некоторым средиземноморским деревьям и некоторым декоративным деревьям, таким как вяз (Ulmus spp.), кедровый орех (Pinus Pinea), платан кленолистный (Platanus Acerifolia), яблоня (Malus alba). Другие Hemipterans относятся к семейству Pentatomoidea, их обычно называют щитники, лесные клопы и клопы-вонючки (например, коричневый мраморный щитник (Halyomorpha halys), клоп-щитник (Euschistus conspersus), зеленый овощной клоп (Nezara viridula), щитник красноногий (Pentatoma rufipes), клоп капустный (Murgantia histrionica), клоп (Oebalus pugnax)), и они наносят ущерб плодам, включая яблоки, персики, инжир, шелковицу, плодам цитрусовых и хурме, ежевике, и овощам, включая сахарную кукурузу, томаты, соевые бобы, фасоль лимскую и паприку, капусту, цветную капусту, репу, хрен, капусту кормовую, горчицу, брюссельскую капусту, картофель, баклажаны, бамию, бобы, спаржу, свеклу, сорняки, фруктовые деревья и полевые культуры, такие как полевая кукуруза и соевые бобы. Щитники также являются вредителями трав, сорго и риса.
Растение для применения в способах по настоящему изобретению или трансгенное растение в соответствии с настоящим изобретением охватывает любое растение, но предпочтительно является растением, которое является восприимчивым к нападению насекомых, патогенных для растения.
Соответственно, настоящее изобретение распространяется на растения и на способы, описываемые в данном документе, где растение выбирают из следующей группы растений (или сельскохозяйственных культур): люцерна, яблоня, абрикос, артишок, спаржа, авокадо, банан, ячмень, бобы, свекла, ежевика, черника, брокколи, брюссельская капуста, капуста, канола, морковь, маниок, цветная капуста, зерновые, сельдерей, вишня, цитрусы, клементины, кофе, кукуруза, хлопчатник, огурцы, баклажаны, эндивий, эвкалипт, инжир, виноград, грейпфрут, арахис, физалис, киви, латук, лук-порей, лимон, лайм, сосна, кукуруза, манго, дыня, просо, грибы, орехи, овес, бамия, лук, апельсин, декоративное растение или цветок, или дерево, папайя, петрушка, горох, персики, арахис, торфяной мох, перец, хурма, ананас, подорожник, слива, гранат, картофель, тыква, красный эндивий, редис, рапсовое семя, малина, рис, рожь, сорго, соя, соевые бобы, шпинат, клубника, сахарная свекла, сахарный тростник, подсолнечник, сладкий картофель, мандарин, чай, табак, томаты, виноград культурный, арбуз, пшеница, ямс и кабачок.
В конкретных вариантах осуществления настоящее изобретение предусматривает гены-мишени, которые кодируют белки, участвующие в функции белка wings up A (тропонина I), субъединицы II митохондриальной цитохром с-оксидазы или одного из рибосомных белков, как указано в таблице 1.
В предпочтительных вариантах осуществления настоящее изобретение предусматривает гены-мишени, которые выбраны из группы генов (i) имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (ii) имеющих нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, или (iii) имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или имеющих такую нуклеотидную последовательность, чтобы при оптимальном выравнивании и сравнении указанного гена, содержащего указанный фрагмент, с любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iv) имеющих нуклеотидную последовательность, содержащую фрагмент из по меньшей мере 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, таким образом, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность указанного фрагмента являлась по меньшей мере на 75%, предпочтительно по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (v) имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, или (vi) ген которой у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую любую из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентична любой из последовательностей, представленных в SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389,
и где нуклеотидная последовательность указанного гена составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
Аминокислотные последовательности, кодируемые генами-мишенями по настоящему изобретению, представлены SEQ ID NO 79, 349, 405, 352, 356, 80, 326, 81, 327, 82, 83, 328, 84, 329, 85, 86, 359, 87-91, 330, 350, 353, 331, 351, 332-336, 337, 354, 338-344, 346, 345, 347, 348, 357, 355, 358, 390-393.
Применяемый в данном документе термин ʺобладающийʺ имеет такое же значение, как ʺсодержащийʺ.
Применяемый в данном документе термин ʺидентичность последовательностейʺ используется для описания сходства последовательностей между двумя или более нуклеотидными или аминокислотными последовательностями. Процент ʺидентичности последовательностейʺ между двумя последовательностями определяется путем сравнения двух оптимально выровненных последовательностей в окне сравнения (определенное число положений), при этом часть последовательности в окне сравнения может содержать присоединения или делеции (т.е. пробелы) по сравнению с контрольной последовательностью с целью достижения оптимального выравнивания. Процент идентичности последовательностей рассчитывается путем определения числа положений, в которых идентичное нуклеотидное основание или аминокислотный остаток встречается в обеих последовательностях, с получением числа ʺсовпадающихʺ положений, деления числа совпадающих положений на общее число положений в окне сравнения и умножения результата на 100. Способы и программное обеспечение для определения идентичности последовательностей доступны в данной области и включают программное обеспечение Blast и GAP-анализ. Для нуклеиновых кислот процент идентичности рассчитывают предпочтительно с помощью инструмента выравнивания BlastN, посредством чего процент идентичности рассчитывают по всей длине запросной нуклеотидной последовательности.
Специалисту в данной области будет понятно, что гомологи или ортологи (гомологи, существующие у различных видов) генов-мишеней, представленные любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, могут быть идентифицированы. Данные гомологи и/или ортологи вредителей также находятся в рамках настоящего изобретения. Предпочтительные гомологи и/или ортологи являются генами, подобными в нуклеотидной последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов гомолог и/или ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80% или 85%, более предпочтительно по меньшей мере на 90% или 95%, и наиболее предпочтительно по меньшей мере приблизительно 99% идентичной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности. Аналогично, также предпочтительными гомологами и/или ортологами являются белки, которые подобны в аминокислотной последовательности до такой степени, что при оптимальном выравнивании и сравнении двух аминокислотных последовательностей гомолог и/или ортолог имеет последовательность, которая является по меньшей мере на 75%, предпочтительно по меньшей мере на 80% или 85%, более предпочтительно по меньшей мере на 90% или 95%, и наиболее предпочтительно по меньшей мере приблизительно 99% идентичной любой из SEQ ID NO 79, 349, 405, 352, 356, 80, 326, 81, 327, 82, 83, 328, 84, 329, 85, 86, 359, 87-91, 330, 350, 353, 331, 351, 332-336, 337, 354, 338-344, 346, 345, 347, 348, 357, 355, 358, 390-393.
Другие гомологи являются генами, которые являются аллелями гена, содержащего последовательность, которая представлена любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389. Дополнительные предпочтительные гомологи являются генами, содержащими по меньшей мере один однонуклеотидный полиморфизм (SNP) по сравнению с геном, содержащим последовательность, которая представлена любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389.
ʺИнтерферирующая рибонуклеиновая кислота (РНК)ʺ по настоящему изобретению охватывает любой тип молекулы РНК, способной подавлять или подвергать ʺсайленсингуʺ экспрессию гена-мишени, включая, но без ограничений, смысловую РНК, антисмысловую РНК, короткую интерферирующую РНК (siRNA), микроРНК (miRNA), двухцепочечную РНК (dsRNA), шпилечную РНК (RNA) и тому подобное. Способы анализа функциональных молекул интерферирующих РНК хорошо известны в данной области и описаны в данном документе в других местах.
Молекулы интерферирующей РНК по настоящему изобретению обеспечивают последовательность-специфическое подавление экспрессии гена-мишени путем связывания с целевой нуклеотидной последовательностью в пределах гена-мишени. Связывание происходит в результате спаривания оснований между комплементарными областями интерферирующей РНК и целевой нуклеотидной последовательности. Применяемый в данном документе термин ʺсайленсирующий элементʺ относится к части или области интерферирующей РНК, содержащей или состоящей из последовательности нуклеотидов, которая комплементарна или по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и которая функционирует как активная часть интерферирующей РНК с непосредственным подавлением экспрессии указанного гена-мишени. В одном варианте осуществления настоящего изобретения сайленсирующий элемент содержит или состоит из последовательности по меньшей мере из 17 последовательных нуклеотидов, предпочтительно по меньшей мере 18 или 19 последовательных нуклеотидов, более предпочтительно по меньшей мере 21 последовательного нуклеотида, еще более предпочтительно по меньшей мере 22, 23, 24 или 25 последовательных нуклеотидов, комплементарных целевой нуклеотидной последовательности в пределах гена-мишени.
Применяемое в данном документе выражение ʺэкспрессия гена-мишениʺ относится к транскрипции и накоплению РНК-транскрипта, кодируемого геном-мишенью, и/или трансляции mRNA в белок. Термин ʺподавлятьʺ предназначен для обозначения любого из способов, известных в данной области, с помощью которого молекулы интерферирующей РНК снижают уровень первичных транскриптов РНК, mRNA или белка, полученного из гена-мишени. В определенных вариантах осуществления подавление относится к ситуации, когда уровень РНК или белка, полученного из гена, уменьшается по меньшей мере на 10%, предпочтительно по меньшей мере на 33%, более предпочтительно по меньшей мере на 50%, еще более предпочтительно по меньшей мере на 80%. В особенно предпочтительных вариантах осуществления подавление относится к снижению уровня РНК или белка, полученного из гена, по меньшей мере на 80%, предпочтительно по меньшей мере на 90%, более предпочтительно по меньшей мере на 95% и наиболее предпочтительно по меньшей мере на 99% в клетках насекомого-вредителя по сравнению с соответствующим контрольным насекомым-вредителем, которое, например, не подвергалось воздействию интерферирующей РНК или подвергалось воздействию контрольной молекулы интерферирующей РНК. Способы выявления снижения уровней РНК или белка хорошо известны в данной области и включают гибридизацию РНК в растворе, нозерн гибридизацию, обратную транскрипцию (например, количественный анализ ОТ-ПЦР (RT-PCR)), микроматричный анализ, связывание антител, иммуноферментный твердофазный анализ (ELISA) и вестерн-блоттинг. В другом варианте осуществления настоящего изобретения подавление относится к снижению уровней РНК или белка, достаточному для получения выявляемого изменения фенотипа вредителя, по сравнению с соответствующей мерой борьбы с вредителями, например, гибелью клеток, прекращением роста или тому подобное. Подавление, таким образом, можно измерить с помощью фенотипического анализа насекомого-вредителя с использованием методов, обычных в данной области.
В предпочтительном варианте осуществления настоящего изобретения интерферирующая РНК подавляет экспрессию гена с помощью РНК-интерференции или RNAi. RNAi является процессом последовательность-специфической генной регуляции, как правило, опосредованной молекулами двухцепочечной РНК, такими как короткие интерферирующие РНК (siRNA). siRNA содержат смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК. Смысловая цепь или “направляющая цепь” молекулы siRNA содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенных в пределах РНК-транскрипта гена-мишени. Смысловая цепь siRNA, следовательно, способна соединяться с РНК-транскриптом посредством спаривания оснований по Уотсон-Крику и направлять РНК на деградацию в пределах клеточного комплекса, известного как индуцируемый RNAi сайленсирующий комплекс или RISC. Таким образом, в отношении предпочтительных молекул интерферирующей РНК по настоящему изобретению сайленсирующий элемент, как изложено в данном документе, может являться двухцепочечной областью, содержащей соединенные комплементарные цепи, из которых по меньшей мере одна цепь содержит или состоит из последовательности нуклеотидов, комплементарной или по меньшей мере частично комплементарной целевой нуклеотидной последовательности в пределах гена-мишени. В одном варианте осуществления двухцепочечная область составляет в длину по меньшей мере 21, 22, 23, 24, 25, 30, 35, 40, 50, 55, 60, 70, 80, 90, 100, 125, 150, 175, 200, 300, 400, 500, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 пар оснований.
Более длинные двухцепочечные молекулы РНК (dsRNA), содержащие один или более функциональных двухцепочечных сайленсирующих элементов, которые описаны в данном документе в других местах, и способные к сайленсингу генов посредством RNAi, также рассматриваются в рамках настоящего изобретения. Такие более длинные молекулы dsRNA содержат по меньшей мере 80, 200, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 пар оснований. Данные молекулы dsRNA могут служить в качестве предшественников активных молекул siRNA, которые направляют РНК-транскрипт к комплексу RISC для последующей деградации. Молекулы dsRNA, присутствующие в среде, окружающей организм или его клетки, могут поглощаться организмом и перерабатываться ферментом, называемым Dicer, с получением молекул siRNA. Альтернативно, dsRNA может быть получена in vivo, т.е. транскрибирована из полинуклеотида или полинуклеотидов, кодирующих dsRNA, присутствующих в клетке, например, бактериальной клетке или растительной клетке, и впоследствии обработана с помощью Dicer, либо в пределах клетки-хозяина, либо предпочтительно в пределах клеток насекомого-вредителя после поглощения более длинной dsRNA-предшественника. dsRNA может образовываться из двух отдельных (смысловой и антисмысловой) цепей РНК, которые соединяются посредством комплементарного спаривания оснований. Альтернативно, dsRNA может являться одной цепью, способной складываться назад на себя с образованием шпилечной РНК (РНК) или структуры стебель-петля. В случае РНК, двухцепочечная область или ʺстебельʺ образована из двух областей или сегментов РНК, которые являются существенно инвертированными повторами друг друга и обладают достаточной комплементарностью для обеспечения образования двухцепочечной области. Один или более функциональных двухцепочечных сайленсирующих элементов могут присутствовать в данной ʺобласти стебляʺ молекулы. Области инвертированных повторов, как правило, разделены областью или сегментом РНК, известным как область ʺпетлиʺ. Данная область может содержать любую нуклеотидную последовательность, придающую достаточную гибкость для обеспечения осуществления самоспаривания между фланкирующими комплементарными областями РНК. В общем, область петли является по существу одноцепочечной и действует как разделительный элемент между инвертированными повторами.
Все молекулы интерферирующей РНК по настоящему изобретению обеспечивают последовательность-специфическое подавление экспрессии гена-мишени путем связывания с целевой нуклеотидной последовательностью в пределах гена-мишени. Связывание происходит в результате комплементарного спаривания оснований между сайленсирующим элементом интерферирующей РНК и целевой нуклеотидной последовательностью. Молекулы интерферирующей РНК по настоящему изобретению содержат по меньшей мере один или по меньшей мере два сайленсирующих элемента. В одном варианте осуществления настоящего изобретения целевая нуклеотидная последовательность содержит последовательность нуклеотидов, которая представлена РНК-транскриптом гена-мишени или его фрагментом, при этом фрагмент составляет предпочтительно по меньшей мере 17 нуклеотидов, более предпочтительно по меньшей мере 18, 19 или 20 нуклеотидов, или наиболее предпочтительно по меньшей мере 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 нуклеотидов. В предпочтительном варианте осуществления настоящего изобретения целевая нуклеотидная последовательность содержит последовательность нуклеотидов, эквивалентную РНК-транскрипту, кодируемому любым из полинуклеотидов, который выбирают из группы, включающей (i) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100 или 1115 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, или (ii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности,
или (iii) полинуклеотида, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, которая представлена в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, таким образом, чтобы при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид являлся по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iv) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, и при этом указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью, таким образом, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности,
или (v) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, и при этом указанный фрагмент или указанная комплементарная последовательность обладает нуклеотидной последовательностью, таким образом, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (vi) полинуклеотид, кодирующий аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389. В более предпочтительном варианте осуществления вышеупомянутого указанный полинуклеотид составляет в длину не более чем 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов.
Предпочтительно молекулы интерферирующей РНК по настоящему изобретению содержат по меньшей мере одну двухцепочечную область, как правило, сайленсирующий элемент интерферирующей РНК, содержащий смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, при этом смысловая цепь молекулы dsRNA содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени.
Сайленсирующий элемент или по меньшей мере одна его цепь, при этом сайленсирующий элемент является двухцепочечным, может являться полностью комплементарным или частично комплементарным целевой нуклеотидной последовательности гена-мишени. Применяемый в данном документе термин ʺполностью комплементарныйʺ означает, что все основания нуклеотидной последовательности сайленсирующего элемента являются комплементарными или ʺсоответствуютʺ основаниям целевой нуклеотидной последовательности. Термин ʺпо меньшей мере частично комплементарныйʺ означает, что существует менее чем 100% совпадения между основаниями сайленсирующего элемента и основаниями целевой нуклеотидной последовательности. Специалисту в данной области будет понятно, что сайленсирующий элемент должен являться лишь по меньшей мере частично комплементарным целевой нуклеотидной последовательности для опосредования подавления экспрессии гена-мишени. Как известно в данной области, последовательности РНК со вставками, делециями и нарушениями комплементарности относительно целевой последовательности все же могут являться эффективными при RNAi. В соответствии с настоящим изобретением предпочтительно, чтобы сайленсирующий элемент и целевая нуклеотидная последовательность гена-мишени обладали по меньшей мере 80% или 85% идентичности последовательности, предпочтительно по меньшей мере 90% или 95% идентичности последовательности или более предпочтительно по меньшей мере 97% или 98% идентичности последовательности, и еще более предпочтительно по меньшей мере 99% идентичности последовательности. Альтернативно, сайленсирующий элемент может содержать 1, 2 или 3 нарушения комплементарности по сравнению с целевой нуклеотидной последовательностью на каждом отрезке из 24 частично комплементарных нуклеотидов.
Специалисту в данной области будет очевидным, что степень комплементарности между сайленсирующим элементом и целевой нуклеотидной последовательностью может варьировать в зависимости от гена-мишени, подлежащего подавлению, или в зависимости от вида насекомого-вредителя, у которого экспрессия гена подлежит контролю.
В другом варианте осуществления настоящего изобретения сайленсирующий элемент содержит последовательность нуклеотидов, которая является эквивалентом РНК любого из полинуклеотидов, выбранных из группы, включающей полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100 или 1115 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, или (ii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, таким образом, чтобы при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид являлся по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iii) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и при этом указанный фрагмент или указанная комплементарная последовательность обладают такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, при этом указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину. Следует понимать, что в таких вариантах осуществления сайленсирующий элемент может содержать или состоять из области двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых, смысловая цепь, содержит последовательность нуклеотидов по меньшей мере частично комплементарную целевой нуклеотидной последовательности в пределах гена-мишени.
Целевую нуклеотидную последовательность можно выбирать из любой подходящей области или нуклеотидной последовательности гена-мишени или его РНК-транскрипта. Например, целевая нуклеотидная последовательность может располагаться в пределах 5’UTR или 3’UTR гена-мишени или РНК транскрипта или в пределах экзонной или интронной областей гена.
Специалисту в данной области известны способы идентифицирования наиболее подходящих целевых нуклеотидных последовательностей в рамках полноразмерного гена-мишени. Например, множество сайленсирующих элементов, воздействующих на различные области гена-мишени, можно синтезировать и тестировать. Альтернативно, расщепление РНК-транскрипта с помощью ферментов, таких как РНКаза Н, можно использовать для определения сайтов в РНК, которые находятся в конформации, восприимчивой к сайленсингу гена. Сайты-мишени также можно идентифицировать с применением in silico подходов, например, использование компьютерных алгоритмов, предназначенных для прогнозирования эффективности сайленсинга генов на основе воздействия на различные сайты в пределах гена полной длины.
Интерферирующие РНК по настоящему изобретению могут содержать один сайленсирующий элемент или несколько сайленсирующих элементов, при этом каждый сайленсирующий элемент содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии указанного гена-мишени. Конкатемерные РНК-конструкции данного типа описаны в WO2006/046148, который включен в данном документе в качестве ссылки. В контексте настоящего изобретения термин ʺнесколькоʺ означает по меньшей мере два, по меньшей мере три, по меньшей мере четыре и т.д., и по меньшей мере до 10, 15, 20 или по меньшей мере 30. В одном варианте осуществления интерферирующая РНК содержит несколько копий одного сайленсирующего элемента, т.е. повторов сайленсирующего элемента, который связывается с конкретной целевой нуклеотидной последовательностью в пределах определенного гена-мишени. В другом варианте осуществления сайленсирующие элементы в пределах интерферирующей РНК содержат или состоят из различных последовательностей нуклеотидов, комплементарных различным целевым нуклеотидным последовательностям. Должно быть ясно, что комбинации нескольких копий одного и того же сайленсирующего элемента, комбинированные с сайленсирующими элементами, связывающимися с различными целевыми нуклеотидными последовательностями, находятся в рамках настоящего изобретения.
Различные целевые нуклеотидные последовательности могут происходить из одного гена-мишени у вида насекомого-вредителя для достижения улучшения подавления конкретного гена-мишени у вида насекомого-вредителя. В этом случае сайленсирующие элементы можно комбинировать в интерферирующей РНК в исходном порядке, в котором целевые нуклеотидные последовательности встречаются в гене-мишени, или сайленсирующие элементы можно смешивать и комбинировать случайно в любом порядке ранжирования в рамках интерферирующей РНК по сравнению с порядком целевых нуклеотидных последовательностей в гене-мишени.
Альтернативно различные целевые нуклеотидные последовательности представляют один ген-мишень, но происходят из различных видов насекомых-вредителей.
Альтернативно различные целевые нуклеотидные последовательности могут происходить из различных генов-мишеней. Если интерферирующая РНК предназначена для использования для предупреждении и/или борьбы с нападением вредителей, предпочтительно выбирать различные гены-мишени из группы генов, регулирующих жизненно важные биологические функции вида насекомого-вредителя, включая, но без ограничений, выживание, рост, развитие, размножение и патогенность. Гены-мишени могут регулировать одинаковые или различные биологические пути или процессы. В одном варианте осуществления по меньшей мере один из сайленсирующих элементов содержит или состоит из последовательности нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, при этом ген-мишень выбран из группы генов, которая описана ранее.
В дополнительном варианте осуществления настоящего изобретения различные гены, на которые воздействуют различные сайленсирующие элементы, происходят из одного вида насекомого-вредителя. Данный подход предназначен для достижения усиленного воздействия на один вид насекомого-вредителя. В частности, различные гены-мишени могут экспрессироваться дифференциально на различных стадиях жизненного цикла насекомого, например, зрелого имаго, незрелой личиночной и стадии яйца. Интерферирующую РНК по настоящему изобретению можно использовать для предотвращения и/или борьбы с нападением насекомых-вредителей на более чем одной стадии жизненного цикла насекомого.
В альтернативном варианте осуществления настоящего изобретения различные гены, на которые воздействуют различные сайленсирующие элементы, происходят из различных видов насекомых-вредителей. Интерферирующую РНК по настоящему изобретению можно, таким образом, использовать для предотвращения и/или борьбы с нападением более чем одного вида насекомых-вредителей одновременно.
Сайленсирующие элементы могут располагаться в виде одной непрерывной области интерферирующей РНК или могут разделяться присутствием линкерной последовательности. Линкерная последовательность может содержать короткую случайную нуклеотидную последовательность, которая не комплементарна ни одной из целевых нуклеотидных последовательностей или генов-мишеней. В одном варианте осуществления линкер является при определенных условиях самостоятельно расщепляющейся последовательностью РНК, предпочтительно pH-чувствительным линкером или гидрофобно-чувствительным линкером. В одном варианте осуществления линкер содержит последовательность нуклеотидов, эквивалентную интронной последовательности. Линкерные последовательности по настоящему изобретению могут находиться в диапазоне длины от приблизительно 1 пары оснований до приблизительно 10000 пар оснований, при условии, что линкер не ухудшает способности интерферирующей РНК подавлять экспрессию гена(генов)-мишени.
В дополнение к сайленсирующему элементу(элементам) и любым линкерным последовательностям интерферирующая РНК по настоящему изобретению может содержать по меньшей мере одну дополнительную полинуклеотидную последовательность. В различных вариантах осуществления настоящего изобретения дополнительную последовательность выбирают из (i) последовательности, способной защитить интерферирующую РНК от РНК процессинга, (ii) последовательности, влияющей на стабильность интерферирующей РНК, (iii) последовательности, обеспечивающей связывание белков, например, для облегчения поглощения интерферирующей РНК клетками вида насекомого-вредителя, (iv) последовательности, облегчающей крупномасштабное производство интерферирующей РНК, (v) последовательности, которая является аптамером, который связывается с рецептором или молекулой на поверхности клеток насекомого-вредителя для облегчения поглощения, или (vi) последовательности, которая катализирует процессинг интерферирующей РНК в клетках насекомого-вредителя и тем самым повышает эффективность интерферирующей РНК. Структуры для повышения стабильности молекулы РНК хорошо известны в данной области и описаны дополнительно в WO2006/046148, который включен в данном документе в качестве ссылки.
Длина интерферирующей РНК по настоящему изобретению должна являться достаточной для поглощения клетками вида насекомого-вредителя и подавления генов-мишеней у вредителя, как описано в данном документе в других местах. Однако верхний предел длины может зависеть от (i) требования к интерферирующей РНК поглощаться клетками вредителя и (ii) требования к интерферирующей РНК подвергаться процессингу в клетках вредителя для опосредования сайленсинга генов путем RNAi. Длина также может зависеть от способа получения и состава для доставки интерферирующей РНК к клеткам. Предпочтительно интерферирующая РНК по настоящему изобретению составляет от 21 до 10000 нуклеотидов в длину, предпочтительно от 50 до 5000 нуклеотидов или от 100 до 2500 нуклеотидов, более предпочтительно от 80 до 2000 нуклеотидов в длину.
Интерферирующая РНК может содержать основания ДНК, неприродные основания или неприродные связи остова, или модификации сахарофосфатного остова, например, для повышения стабильности во время хранения или повышения устойчивости к деградации нуклеазами. Дополнительно, интерферирующая РНК может быть получена химически или ферментативно специалистом в данной области с помощью управляемых вручную или автоматизированных реакций. Альтернативно, интерферирующая РНК может транскрибироваться из полинуклеотида, кодирующего таковую. Таким образом, в настоящем документе предусматривается выделенный полинуклеотид, кодирующий любую из интерферирующих РНК по настоящему изобретению.
Также в настоящем документе предусматривается выделенный полинуклеотид, выбранный из группы, включающей (i) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, или (ii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23, 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100 или 1115 последовательных нуклеотидов нуклеотидной последовательности, представленной любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательностью, или (iii) полинуклеотид, который содержит по меньшей мере 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности так, что при оптимальном выравнивании и сравнении двух последовательностей указанный полинуклеотид является по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичным любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (iv) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотидной последовательности, представленной в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и при этом указанный фрагмент или указанная комплементарная последовательность обладают такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (v) полинуклеотид, который содержит фрагмент по меньшей мере из 21, предпочтительно по меньшей мере 22, 23 или 24, 25, 26, 27, 28, 29, 30, 35, 40, 45, 50, 55, 60, 70, 80, 90, 100, 110, 125, 150, 175, 200, 225, 250, 300, 350, 400, 450, 500, 550, 600, 700, 800, 900, 1000, 1100, 1200, 1300, 1400, 1500, 2000 или 3000 последовательных нуклеотидов нуклеотида, представленного в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, и при этом указанный фрагмент или указанная комплементарная последовательность обладают такой нуклеотидной последовательностью, чтобы при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 указанная нуклеотидная последовательность являлась по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, 90%, 95%, 98% или 99% идентичной указанному соответствующему фрагменту из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389 или комплементарной ей последовательности, или (vi) полинуклеотид, кодирующий аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, 80%, 85%, 90%, 95%, 98% или 99% идентичной аминокислотной последовательности, кодируемой любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, 310-313, 3, 4, 31-34, 139, 5, 6, 35-38, 140, 7, 8, 39-42, 9, 10, 43-46, 141, 11, 12, 47-50, 13, 14, 51-54, 15, 204, 16, 205, 55-58, 322-325, 17, 18, 59-62, 19, 20, 63-66, 21, 22, 67-70, 23, 24, 71-74, 25, 26, 75-78, 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301, 145, 122, 144, 178, 131, 179, 210-213, 290-293, 123, 132, 214-217, 124, 133, 218-221, 146, 125, 134, 222-225, 147, 126, 135, 226-229, 127, 148, 136, 230-233, 128, 149, 184, 137, 185, 234-237, 302-305, 129, 138, 238-241, 150, 151, 242-245, 152, 153, 246-249, 154, 155, 250-253, 156, 157, 254-257, 158, 159, 258-261, 160, 161, 262-265, 163, 162, 164, 266-269, 165, 167, 166, 270-273, 168, 170, 169, 274-277, 172, 173, 278-281, 200, 201, 314-317, 186, 202, 187, 203, 306-309, 318-321, 386, 387, 388, 389, и при этом указанный полинуклеотид составляет не более 10000, 9000, 8000, 7000, 6000, 5000, 4000, 3000, 2000 или 1500 нуклеотидов в длину.
В предпочтительных вариантах осуществления выделенный полинуклеотид является частью молекулы интерферирующей РНК, как правило, частью сайленсирующего элемента, содержащего по меньшей мере одну двухцепочечную область, содержащую смысловую цепь РНК, соединенную путем комплементарного спаривания оснований с антисмысловой цепью РНК, при этом смысловая цепь молекулы dsRNA содержит последовательность нуклеотидов, комплементарную последовательности нуклеотидов, расположенной в пределах РНК-транскрипта гена-мишени. Смысловая цепь dsRNA, следовательно, способна соединяться с РНК-транскриптом и воздействовать на РНК для деградации в пределах RNAi-индуцируемого сайленсирующего комплекса или RISC.
Полинуклеотиды по настоящему изобретению можно вставлять с помощью рутинных методов молекулярного клонирования в ДНК-конструкции или векторы, известные в данной области. Следовательно, в соответствии с одним вариантом осуществления предусматривается ДНК-конструкция, содержащая любые из полинуклеотидов по настоящему изобретению. Предпочтительно в данном документе предусматривается ДНК-конструкция, содержащая полинуклеотид, кодирующий по меньшей мере одну из интерферирующих РНК по настоящему изобретению. ДНК-Конструкция может являться рекомбинантным ДНК-вектором, например, бактериальным, вирусным или дрожжевым вектором. В предпочтительном варианте осуществления настоящего изобретения ДНК-конструкция является экспрессирующей конструкцией, и полинуклеотид функционально связан с по меньшей мере одной регуляторной последовательностью, способной управлять экспрессией полинуклеотидной последовательности. Термин ʺрегуляторная последовательностьʺ следует рассматривать в широком контексте, и он предназначен для обозначения любой нуклеотидной последовательности, способной воздействовать на экспрессию полинуклеотидов, с которыми она функционально связана, включая, но без ограничений, промоторы, энхансеры и другие природные или синтетические элементы-активаторы транскрипции. Регуляторная последовательность может располагаться на 5'-или 3'-конце полинуклеотидной последовательности. Термин ʺфункционально связанныйʺ относится к функциональной связи между регуляторной последовательностью и полинуклеотидной последовательностью, такой, что регуляторная последовательность управляет экспрессией полинуклеотида. Функционально связанные элементы могут являться смежными или несмежными.
Предпочтительно регуляторная последовательность является промотором, выбранным из группы, включающей, но без ограничений, конститутивные промоторы, индуцируемые промоторы, тканеспецифические промоторы и промоторы, специфичные для стадии роста/развития. В одном варианте осуществления полинуклеотид находится под контролем сильного конститутивного промотора, такого как любой, выбранный из группы, включающей CaMV35S промотор, двойной CaMV35S промотор, убиквитиновый промотор, актиновый промотор, rubisco промотор, GOS2 промотор, промотор 34S вируса мозаики норичника.
Необязательно одну или более последовательностей терминации транскрипции можно включать в экспрессирующую конструкцию по настоящему изобретению. Термин ʺпоследовательность терминации транскрипцииʺ охватывает управляющую последовательность в конце транскрипционной единицы, которая сигнализирует о терминации транскрипции, 3’ процессинге и полиаденилировании первичного транскрипта. Дополнительные регуляторные последовательности, включая, но без ограничений, энхансеры транскрипции или трансляции, можно включать в экспрессионную конструкцию, например, как с усиленным двойным промотором CaMV35S.
Настоящее изобретение также охватывает способ создания любой из интерферирующих РНК по настоящему изобретению, включающий этапы: (i) контакта полинуклеотида, кодирующего указанную интерферирующую РНК, или ДНК-конструкции, содержащей полинуклеотид, с бесклеточными компонентами; или (ii) введения в клетку (например, путем трансформации, трансфекции или инъекции) полинуклеотида, кодирующего указанную интерферирующую РНК, или ДНК-конструкции, содержащей полинуклеотид.
Настоящее изобретение, таким образом, также относится к любому двухцепочечному рибонуклеотиду, полученному при экспрессии полинуклеотида, описанного в данном документе.
Соответственно, также в данном документе предусматривается клетка-хозяин, трансформированная любым из полинуклеотидов, описанных в данном документе. Дополнительно настоящим изобретением охватываются клетки-хозяева, содержащие любую из интерферирующих РНК по настоящему изобретению, любой из полинуклеотидов по настоящему изобретению или ДНК-конструкцию, содержащую любой из полинуклеотидов. Клетка-хозяин может являться прокариотической клеткой, включая, но без ограничений, грамположительные и грамотрицательные бактериальные клетки, или эукариотической клеткой, включая, но без ограничений, клетки дрожжей или клетки растений. Предпочтительно указанная клетка-хозяин является бактериальной клеткой или растительной клеткой. Бактериальную клетку можно выбирать из группы, включающей, но без ограничений, грамположительные и грамотрицательные клетки, включающие Escherichia spp. (например, E. coli), Bacillus spp. (например, B. thuringiensis), Rhizobium spp., Lactobacillus spp., Lactococcus spp., Pseudomonas spp. и Agrobacterium spp. Полинуклеотид или ДНК-конструкция по настоящему изобретению может существовать или поддерживаться в клетке-хозяине как внехромосомный элемент, или может являться стабильно включенным в геном клетки-хозяина. Характеристики, представляющие особый интерес при выборе клетки-хозяина для целей настоящего изобретения, включают простоту, с которой полинуклеотид или ДНК-конструкцию, кодирующую интерферирующую РНК, можно ввести в хозяина, доступность совместимых экспрессионных систем, эффективность экспрессии и стабильность интерферирующей РНК в хозяине.
Предпочтительно интерферирующие РНК по настоящему изобретению экспрессируются в растительных клетках-хозяевах. Предпочтительные растения, представляющие интерес, включают, но без ограничений, хлопчатник, картофель, рис, томат, канолу, сою, подсолнечник, сорго, просо, кукурузу, люцерну, землянику, баклажаны, перец и табак.
В ситуациях, где интерферирующая РНК экспрессируется в клетке-хозяине и/или применяется для предотвращения и/или борьбы с нападением вредителя на организм-хозяин, предпочтительно, чтобы интерферирующая РНК не демонстрировала значительных ʺнецелевыхʺ эффектов, т.е. интерферирующая РНК не влияла на экспрессию генов в хозяине. Предпочтительно сайленсирующий элемент не демонстрирует значительной комплементарности с нуклеотидными последовательностями, кроме заданной целевой нуклеотидной последовательности гена-мишени. В одном варианте осуществления настоящего изобретения сайленсирующий элемент демонстрирует менее 30%, более предпочтительно менее 20%, более предпочтительно менее 10% и еще более предпочтительно менее 5% идентичности последовательности с любым геном клетки-хозяина или организма. Если известны данные геномной последовательности для организма-хозяина, можно перепроверить идентичность с сайленсирующим элементом, применяя стандартные инструменты биоинформатики. В одном варианте осуществления отсутствует идентичность последовательности между сайленсирующим элементом и геном из клетки-хозяина или организма-хозяина в области из 17, более предпочтительно в области из 18 или 19 и наиболее предпочтительно в области из 20 или 21 последовательных нуклеотидов.
При практическом применении настоящего изобретения интерферирующие РНК по настоящему изобретению можно использовать для предотвращения и/или борьбы с любым насекомым вредителем, принадлежащим к отрядам Coleoptera, Lepidoptera, Diptera, Dichyoptera, Orthoptera, Hemiptera и Siphonaptera.
Кроме того, в соответствии с другим аспектом настоящего изобретения в настоящем документе предусматривается композиция для предотвращения и/или борьбы с нападением насекомых-вредителей, содержащая по меньшей мере одну интерферирующую рибонуклеиновую кислоту (РНК) и необязательно по меньшей мере один подходящий носитель, наполнитель или разбавитель, причем интерферирующая РНК функционирует при поглощении насекомым с подавлением экспрессии гена-мишени у указанного вредителя. Интерферирующей РНК может являться любая из тех, которые раскрыты в других разделах данного документа. Предпочтительно интерферирующая РНК содержит или состоит по меньшей мере из одного сайленсирующего элемента, при этом указанный сайленсирующий элемент является областью двухцепочечной РНК, содержащей соединенные комплементарные цепи, одна цепь из которых (смысловая цепь) содержит последовательность нуклеотидов, которая по меньшей мере частично комплементарна целевой нуклеотидной последовательности в пределах гена-мишени. ʺГеном-мишеньюʺ может являться любой ген-мишень, раскрываемый в разделах настоящего документа, включая, но без ограничений, гены, участвующие в регуляции выживания, росте, развитии, размножении и патогенности вредителя. Альтернативно, композиция содержит по меньшей мере одну клетку-хозяина, содержащую по меньшей мере одну интерферирующую молекулу РНК или конструкцию ДНК, кодирующую тот же самый и необязательно по меньшей мере один подходящий носитель, наполнитель или разбавитель, причем интерферирующая РНК функционирует при поглощении клетки-хозяина насекомым с подавлением экспрессии гена-мишени у указанного вредителя.
При практическом применении настоящего изобретения композицию можно использовать для предотвращения и/или борьбы с любым насекомым вредителем, принадлежащим к отрядам Coleoptera, Lepidoptera, Diptera, Dichyoptera, Orthoptera, Hemiptera и Siphonaptera. Таким образом, композиция может быть представлена в любой форме, подходящей для применения по отношению к насекомым-вредителям или для применения к субстратам и/или организмам, в частности растений, чувствительных к нападению указанного насекомого-вредителя. В одном варианте осуществления композиция предназначена для применения в целях предотвращения и/или борьбы с нападением вредителей растений, или материала для размножения, или репродуктивного материала растения и, таким образом, направлена на виды насекомых-вредителей, которые нападают на растения. Композиция по настоящему изобретению особенно эффективна, если насекомое-вредитель относится к категории “грызущих” насекомых, которые наносят значительное повреждение растениям, поедая растительные ткани, такие как корни, листья, цветы, почки, ветки и т.п. Примеры из этой большой категории насекомых включают жуков и их личинок.
Композицию по настоящему изобретению можно применять для борьбы с насекомыми-вредителями на всех этапах их жизненного цикла, например, этапе половозрелой особи, этапах личинки и яйца.
В контексте композиции по настоящему изобретению интерферирующая РНК может быть получена из ДНК-конструкции, в частности экспрессионной конструкции, которые описаны в других разделах данного документа, содержащей полинуклеотид, который ее кодирует. В предпочтительных вариантах осуществления интерферирующая РНК может быть произведена внутри клетки-хозяина или организма, сконструированного для экспрессии указанной интерферирующей РНК с полинуклеотидом, который ее кодирует.
Подходящие организмы-хозяева для применения в композициях по настоящему изобретению включают, но без ограничений, композиции, которые известны как колонизирующие среду на и/или вокруг вызывающих интерес растений или культурных растений, т.е. растений или культурных растений, чувствительных к нападению видов насекомых-вредителей. Такие микроорганизмы включают, но без ограничений, микроорганизмы, которые заселяют филлоплану (поверхность листьев растений) и/или ризосферу (почву, окружающую корни растений). Эти микроорганизмы отбирают так, чтобы они были способны успешно конкурировать с организмами дикого типа, присутствующими в среде растения. Подходящие микроорганизмы для применения в качестве хозяев включают различные виды бактерий, дрожжей и грибов. Понятно, что выбираемые микроорганизмы не должны быть токсичными для растений. Такие композиции, применяемые по отношению к растениям, чувствительным к нападению видов насекомых-вредителей, будут поглощаться насекомыми-вредителями при кормлении на обработанных растениях.
Организмы-хозяева, которые в естественных условиях не заселяют растение и/или их среду, также находятся в рамках настоящего изобретения. Такие организмы могут служить лишь в качестве средства для получения интерферирующей РНК композиции. Например, в одном варианте осуществления интерферирующая РНК ферментируется/продуцируется в бактериальном хозяине и бактерии после этого инактивируются/погибают. Полученные бактерии могут быть обработаны и использованы в качестве инсектицидного спрея таким же способом, как были использованы штаммы Bacillus thuringiensis в качестве инсектицида для нанесения распылением. В некоторых вариантах осуществления бактериальный экстракт или лизат может быть подходящим образом очищен с тем, чтобы оставить экстракт, содержащий, по сути, чистую интерферирующую РНК, который затем составляют в одну из композиций по настоящему изобретению. Стандартные методики экстракции/очистки будут известны специалисту в данной области.
Композиции по настоящему изобретению могут находиться в любой подходящей физической форме для применения по отношению к насекомым. Например, композиция может быть в твердой форме (порошок, таблетка или приманка), жидкой форме (в том числе форма, вводимая в качестве распыляемого инсектицида) или форме геля. В конкретном варианте осуществления композиция может представлять собой покрытие, пасту или порошок, который можно нанести на субстрат с целью защиты указанного субстрата от нападения насекомых. В данном варианте осуществления композицию можно применять для защиты любого субстрата или материала, который чувствителен к нападению насекомых или повреждению, наносимому насекомыми.
Природа наполнителей и физической формы композиции может варьировать в зависимости от природы субстрата, который необходимо обрабатывать. Например, композиция может представлять собой жидкость, которую наносят кистью или распыляют на подлежащий обработке материал или субстрат или впечатывают в него, или покрытие или порошок, который наносят на подлежащий обработке материал или субстрат.
В одном варианте осуществления композиция находится в форме приманки. Приманка предназначена для привлечения насекомого войти в контакт с композицией. После вхождения в контакт с нею композиция поглощается насекомым путем, например, переваривания, и опосредует RNAi, таким образом убивая насекомое. Указанная приманка может содержать пищевой продукт, такой как белковый продукт питания, например, рыбную муку. В качестве приманки также можно применять борную кислоту. Приманка может зависеть от вида, являющегося мишенью. Также можно применять аттрактант. Аттрактантом может быть феромон, такой как, например, феромон самца или самки. Как, например, в настоящем изобретении можно применять феромоны, приведенные в книге ʺInsect Pheremones and their use in Pest Managementʺ (Howse et al., Chapman and Hall, 1998). Аттрактант действует как привлекающий насекомого к приманке и может быть направлен на конкретное насекомое или может привлекать целый спектр насекомых. Приманка может быть в любой подходящей форме, такой как твердая, пастообразная форма, форма таблетки или порошковая форма.
Приманка также может быть принесена насекомым обратно в колонию. Затем приманка может играть роль источника питания для других членов колонии, обеспечивая, таким образом, эффективную борьбу с большим количеством насекомых и, потенциально, всей колонией насекомых-вредителей. Это является преимуществом, связанным с применением двухцепочечной РНК по настоящему изобретению, поскольку замедленное действие RNAi-опосредованных эффектов на вредителей делает возможным перенос приманки обратно в колонию, таким образом доставляя максимальный эффект в смысле воздействия на насекомых.
Кроме того, композиции, которые контактируют с насекомыми, могут оставаться на кутикуле насекомого. При очистке, независимо от того, чистится ли отдельное насекомое самостоятельно, или насекомые чистят друг друга, композиции могут быть поглощены и, таким образом, могут опосредовать свои эффекты на насекомых. Для этого необходимо, чтобы композиция была достаточно стабильной с тем, чтобы интерферирующая РНК оставалась интактной и была способна опосредовать RNAi даже при воздействии внешних условий среды в течение длительного периода времени, который может представлять собой, например, период длительностью несколько дней.
Приманки могут быть представлены в подходящем ʺкорпусеʺ или ʺловушкеʺ. Такие корпуса и ловушки коммерчески доступны, а существующие ловушки могут быть адаптированы для включения композиций по настоящему изобретению. Любой корпус или ловушка, которые могут заманивать насекомого зайти, включены в объем настоящего изобретения. Корпус или ловушка могут иметь форму, например, коробки, и могут быть представлены в заранее сформированном состоянии или могут быть, например, в форме складываемого картона. Подходящие материалы для корпуса или ловушки включают пластмассу и картон, в частности, гофрированный картон. Подходящими размерами для такого корпуса или ловушки являются, например, ширина 7-15 см, длина 15-20 см и высота 1-5 см. Внутренние поверхности ловушек могут быть оклеены липким веществом с тем, чтобы ограничить движение насекомого после попадания в ловушку. Корпус или ловушка могут иметь внутри подходящее углубление, которое может удерживать на месте приманку. Ловушка отличается от корпуса, поскольку насекомое после входа не может легко покинуть ловушку, в то время как корпус играет роль ʺкормушкиʺ, которая обеспечивает насекомому предпочтительную среду, в которой они могут кормиться и чувствовать себя в безопасности от хищников.
Соответственно, в дополнительном аспекте настоящее изобретение предлагает корпус или ловушку для насекомых, которые содержат композицию по настоящему изобретению, которая может включать любой из признаков описываемой в данном документе композиции.
В дополнительном альтернативном варианте осуществления композиция может быть представлена в форме спрея. Таким образом, человек-пользователь может непосредственно опрыскивать композицией вредителя. Композиция затем усваивается насекомым, откуда она может опосредовать РНК-интерференцию, таким образом борясь с насекомым. Предпочтительно спрей представляет собой распыляемый под давлением спрей/спрей в виде аэрозоля или спрей для пульверизатора. Частицы могут иметь подходящий размер так, чтобы они прикреплялись к насекомому, например к экоскелету, и могли поглощаться оттуда. Размер частиц может быть измерен известными способами, такими как применение Mastersizer, который является коммерчески доступным устройством.
В еще одном дополнительном варианте осуществления носитель представляет собой электростатически заряженный порошок или частицу, которая прикрепляется к насекомому. Подходящие порошки и частицы, которые могут прикрепляться к насекомому и, таким образом, доставлять РНК-конструкции по настоящему изобретению, описаны подробно в WO 94/00980 и WO 97/33472, обе из которых включены в данный документ в качестве ссылки.
Альтернативно, носитель может содержать магнитные частицы, которые прикрепляются к кутикуле насекомого. Подходящие магнитные частицы, которые могут прикрепляться к насекомому и, таким образом, доставлять РНК-конструкции по настоящему изобретению, описаны подробно в WO 00/01236, ссылка на которую включена в данный документ.
В еще одном дополнительном варианте осуществления носитель композиции содержит металлические частицы, которые изначально не намагничены, но которые могут стать магнитно поляризованными под воздействием электрического поля, производимым организмом насекомого. Этот механизм действия подробно описан в WO 2004/049807 и включен в данный документ в качестве ссылки.
Предпочтительно композиция включает носитель, который увеличивает поглощение интерферирующей РНК насекомым-вредителем. Такой носитель может представлять собой липидный носитель, предпочтительно содержащий одно или несколько из следующего: эмульсии типа масла в воде, мицеллы, холестерин, липополиамины и липосомы. Другие средства, которые способствуют поглощению конструкций по настоящему изобретению, хорошо известны специалистам в данной области и включают поликатионы, декстраны и (трис)-катионные липиды, такие как CS096, CS102 и т.п. Коммерчески доступные липосомы включают LIPOFECTIN® и CELLFECTIN® и т.п. Ряд подходящих носителей перечислены под заголовком ʺУсиливающее трансфекцию средствоʺ в патентной заявке WO 03/004644, а каждый из приведенных примеров, таким образом, включен в качестве ссылки.
В дополнительном предпочтительном варианте осуществления носитель представляет собой конденсирующее нуклеиновую кислоту средство. Предпочтительно конденсирующее нуклеиновую кислоту средство включает спермидин или протамина сульфат или его производное.
Если композиция по настоящему изобретению предназначена для применения в предотвращении и/или борьбе с нападением насекомых на растение, то композиция может содержать подходящий с точки зрения сельского хозяйства носитель. Таким носителем может быть любой материал, который переносит подлежащее обработке растение, который не причиняет ненадлежащий вред для среды или других находящихся в ней организмов и который позволяет интерферирующей РНК оставаться эффективной по отношению к видам насекомых-вредителей. В частности, композиции по настоящему изобретению могут быть составлены для доставки растениям согласно установившимся сельскохозяйственным методикам, применяемым в биоинсектицидной промышленности. Композиция может содержать дополнительные компоненты, способные осуществлять другие функции, включая, но без ограничений, (i) усиление или повышение поглощения интерферирующей РНК клетками вредителя и (ii) стабилизацию активных компонентов композиции. Конкретными примерами таких дополнительных компонентов, содержащихся в композиции, включающей интерферирующую РНК, являются tRNA дрожжей или общая РНК дрожжей.
Композиции могут быть составлены для непосредственного применения или в виде концентрата основной композиции, который необходимо разбавить перед применением. Альтернативно, композиция может поставляться в виде набора, содержащего интерферирующую РНК или клетку-хозяина, ее содержащую или экспрессирующую, в одной емкости и подходящий разбавитель или носитель для РНК или клетки-хозяина в отдельной емкости. При конкретном применении настоящего изобретения композицию можно применять по отношению к растению или любой части растения на любом этапе развития растения. В одном варианте осуществления композицию применяют по отношению к надземным частям растения, например, при культивировании растительных культур в полевых условиях. В дополнительном варианте осуществления композицию применяют по отношению к семенам растения либо пока они находятся в хранилище, либо сразу после высадки в почву. Обычно важно хорошо осуществлять борьбу с вредителями на ранних стадиях роста растения, поскольку это время, когда растение может быть наиболее тяжело повреждено видами вредителей.
Композицию можно вносить в среду насекомых-вредителей при помощи различных методик, включая, но без ограничений, опрыскивание, распыление, опыление, рассеивание, полив, нанесение покрытия на семена, обработка семян, внесение в почву и введение в поливную воду. При обработке растений, чувствительных к нападению вредителей, композиция может быть доставлена на растение или часть растения до появления вредителя (с целью предотвращения) или после начала появления признаков нападения вредителя (с целью борьбы с вредителем).
В дополнительном варианте осуществления настоящего изобретения композиции по настоящему изобретению могут быть составлены так, чтобы они содержали по меньшей мере одно дополнительное активное средство. Таким образом, композиция может быть представлена в виде ʺнабора частейʺ, включающего композицию, содержащую интерферирующую РНК, в одной емкости и один или несколько подходящих активных ингредиентов, например химический или биологический пестицид, в отдельной емкости. Альтернативно, композиции могут быть представлены в виде смесей, которые стабильны и подлежат применению в сочетании друг с другом.
Подходящие активные ингредиенты, которые могут действовать дополняющим образом по отношению к интерферирующим молекулам РНК по настоящему изобретению, включают, но без ограничений, следующие: хлорпирофос, аллетрин, ресметрин, тетрабромэтил, диметол-циклопропанкарбоновая кислота (которую обычно включают в жидкие композиции); и гидраметилнон, авермектин, хлорпирофос, сульфурамид, гидропрен, фипронил (рецептор GABA), изопропилфенилметилкарбамат, индоксакарб (PARA), новифлумурон (ингибитор синтеза хитина), имипротрин (PARA), абамектин (глутамат-зависимый канал для ионов хлора), имидаклоприд (ацетилхолиновый рецептор) (которые включены в композиции приманок).
В предпочтительном варианте осуществления активный ингредиент известен как предпочтительный инсектицид в плане проблем, связанных со здравоохранением и окружающей средой, такой как, например, гидраметилнон и авермектин.
В следующем варианте осуществления настоящего изобретения композицию составляют так, чтобы она содержала по меньшей мере одно агрономическое средство, например, гербицид или дополнительный пестицид. Применяемое в данном документе выражение ʺвторой пестицидʺ или ʺдополнительный пестицидʺ относится к пестициду, отличному от первой или изначальной интерферирующей молекулы РНК композиции. Альтернативно, композиция по настоящему изобретению может быть доставлена в комбинации по меньшей мере с одним другим агрономическим средством, например, гербицидом или вторым пестицидом. В одном варианте осуществления композиция представлена в комбинации с гербицидом, выбранным из любого известного из уровня техники, например, глифосатом, имидазолиноном, сульфонилмочевиной и бромоксинилом. В следующем варианте осуществления композиция представлена в комбинации по меньшей мере с одним дополнительным пестицидом. Дополнительный пестицид может быть выбран из любых известных из уровня техники пестицидов и/или может включать интерферирующую рибонуклеиновую кислоту, которая функционирует при поглощении вредителем с подавлением экспрессии гена-мишени указанного вида вредителя. В одном варианте осуществления целевым вредителем является вид насекомого-вредителя, а интерферирующая РНК выбрана из любой из описанных в данном документе интерферирующих РНК. В следующем варианте осуществления дополнительный пестицид включает интерферирующую РНК, которая функционирует с подавлением экспрессии известного гена у любого целевого вида вредителя, но не ограничиваясь вредителями-насекомыми. Изначальная интерферирующая молекула РНК композиции и второй или дополнительный пестицид(ы) могут целенаправленно воздействовать на один вид насекомого-вредителя или могут быть направлены на воздействие на различные виды насекомых-вредителей. Например, изначальная интерферирующая РНК и второй пестицид могут целенаправленно воздействовать на различные виды насекомых-вредителей или может целенаправленно воздействовать на различные семейства или классы организмов-вредителей, например, грибы, или нематоды, или насекомые. Специалисту в данной области будет понятно, как протестировать комбинации интерферирующих молекул РНК и других агрономических средств в отношении синергических эффектов. В предпочтительном варианте осуществления композиция содержит первую интерферирующую молекулу РНК, описанную в других разделах данного документа, и один или несколько дополнительных пестицидов, каждый из которых токсичен для одного и того же насекомого-вредителя, причем один или несколько дополнительных пестицидов выбраны из пататина, инсектицидного белка Bacillus thuringiensis, инсектицидного белка Xenorhabdus, инсектицидного белка Photorhabdus, инсектицидного белка Bacillus laterosporous, инсектицидного белка Bacillus spaericus и лигнина, и при этом указанный инсектицидный белок Bacillus thuringiensis выбран из группы, состоящей из Cry1Ab, Cry1C, Cry2Aa, Cry3, TIC851, CryET70, Cry22, VIP, TIC901, TIC1201, TIC407, TIC417, бинарного инсектицидного белка, выбранного из CryET33 и CryET34, CryET80 и CryET76, TIC100 и TIC101 и PS149B1, и инсектицидных химер любого из перечисленных инсектицидных белков.
Различные компоненты описанных в данном документе комбинаций можно вводить, например, организму-хозяину, чувствительному к нападению вредителя, в любом порядке. Компоненты на подлежащую обработке территорию или организм можно доставлять одновременно или последовательно.
Также в данном документе предусматривается способ предотвращения и/или борьбы с нападением вредителей, при котором вид насекомого-вредителя контактирует с эффективным количеством по меньшей мере одной интерферирующей РНК, при этом РНК функционирует при поглощении указанным вредителем с подавлением экспрессии жизненно важного гена-мишени вредителя. Жизненно важным геном-мишенью может являться любой ген вредителя, участвующий в регуляции жизненно важного биологического процесса, необходимого вредителю для инициирования или поддержания нападения, включая, но без ограничений, выживание, рост, развитие, размножение и патогенность. В частности, ген-мишень может являться любым из генов вредителя, как описано в данном документе в других местах.
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, или 310-313, или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 1, 174, 404, 180, 188, 2, 175, 181, 189, 27-30, 282-285, 294-297, или 310-313, или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук),
где ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO 79, 349, 405, 352 или 356 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 141, 11, 12, 47-50 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 141, 11, 12, 47-50 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO 328 или 84 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарный по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO 17, 18, 59-62 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 17, 18, 59-62 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в SEQ ID NO 87 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 19, 20, 63-66 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарный по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO 19, 20, 63-66 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в SEQ ID NO 88 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 165, 167, 166, 270-273 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук). В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 165, 167, 166, 270-273 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO 347 или 348 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарный по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 143, 121, 142, 176, 182, 130, 177, 183, 206-209, 286-289, 298-301 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук),
при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO 330, 350 или 353 (при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 145, 122, 144, 178, 131, 179, 210-213, 290-293 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду в любой из SEQ ID NO 145, 122, 144, 178, 131, 179, 210-213, 290-293 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в SEQ ID NO 331 или 351(при оптимальном выравнивании указанных кодируемых белков).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 128, 149, 184, 137, 185, 234-237, 302-305 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук).
В способах, описанных в данном документе, для подавления экспрессии гена-мишени у вида насекомого-вредителя, молекулы двухцепочечной РНК, содержащей по меньшей мере 21 пару оснований, одна цепь которой содержит или состоит из последовательности нуклеотидов, комплементарной по меньшей мере 21 последовательному нуклеотиду из любой из SEQ ID NO 128, 149, 184, 137, 185, 234-237, 302-305 или комплементарной ей последовательности, можно применять для подавления экспрессии ортологического гена-мишени у жесткокрылых, полужесткокрылых, чешуекрылых или двукрылых насекомых, выбранных из группы, включающей, но без ограничений, Leptinotarsa spp. (например, L. decemlineata (колорадский жук), L. juncta (ложный картофельный жук) или L. texana (картофельный жук Leptinotarsa texana)); Nilaparvata spp. (например, N. lugens (коричневый дельфацид)); Lygus spp. (например, L. lineolaris (клоп-слепняк Lygus lineolaris) или L. hesperus (слепняк западный матовый)); Myzus spp. (например, M. persicae (тля персиковая зеленая)); Diabrotica spp. (например, D. virgifera virgifera (западный кукурузный жук), D. barberi (северный кукурузный жук), D. undecimpunctata howardi (южный кукурузный жук) или D. virgifera zeae (мексиканский кукурузный жук), при этом ортологические гены кодируют белок, обладающий аминокислотной последовательностью, которая является по меньшей мере на 85%, 90%, 92%, 94%, 96%, 98%, 99% идентичной аминокислотной последовательности, представленной в любой из SEQ ID NO 337 или 354 (при оптимальном выравнивании указанных кодируемых белков).
Кроме того, в данном документе предусматривается способ предотвращения и/или борьбы с нападением вредителей в поле культурных растений, при этом указанный способ включает экспрессию в указанных растениях эффективного количества интерферирующей РНК, описываемой в данном документе.
В способе, предназначенном для борьбы с нападением вредителя, фраза ʺэффективное количествоʺ распространяется на количество или концентрацию интерферирующей РНК, необходимые для получения фенотипического эффекта у вредителя таким образом, чтобы уменьшались количества организмов-вредителей, поражающих организм-хозяин, и/или уменьшалось количество повреждений, вызванных вредителем. В одном варианте осуществления фенотипическим эффектом является гибель вредителя, и интерферирующая РНК используется для достижения по меньшей мере 20%, 30%, 40%, предпочтительно по меньшей мере 50%, 60%, 70%, более предпочтительно по меньшей мере 80% или 90% смертности вредителей по сравнению с контрольными насекомыми-вредителями. В дополнительном варианте осуществления фенотипические эффекты включают, но без ограничений, остановку роста вредителя, прекращение питания и уменьшение яйцекладки. Общее количество организмов-вредителей, поражающих организм-хозяин, можно таким образом уменьшить по меньшей мере на 20%, 30%, 40%, предпочтительно по меньшей мере на 50%, 60%, 70%, более предпочтительно по меньшей мере на 80% или 90% по сравнению с контрольными вредителями. Альтернативно, ущерб, наносимый насекомыми-вредителями, можно уменьшить по меньшей мере на 20%, 30%, 40%, предпочтительно по меньшей мере на 50%, 60%, 70%, более предпочтительно по меньшей мере на 80% или 90% по сравнению с контрольными насекомыми-вредителями. Таким образом, способ по настоящему изобретению можно применять для достижения по меньшей мере 20%, 30%, 40%, предпочтительно по меньшей мере 50%, 60%, 70%, более предпочтительно по меньшей мере 80% или 90% эффективности при борьбе с вредителями.
Применяемый в данном документе термин ʺрастениеʺ может содержать любой репродуктивный материал или материал для размножения растения. Ссылка на растение также может включать клетки растений, протопласты растений, культуры тканей растений, растительные каллусы, маточные корневища растений и растительные клетки, которые являются интактными в растениях или частях растений, такие как зародыши, пыльца, семяпочки, семена, листья, цветы, ветки, фрукты, ядра, колосья, початки, шелуха, стебли, корни, кончики корней и тому подобное.
Также в данном документе предусматривается применение интерферирующей рибонуклеиновой кислоты (РНК), описываемой в данном документе, или ДНК-конструкции, описываемой в данном документе, для предотвращения и/или борьбы с нападением насекомых-вредителей, предпочтительно нападением насекомых-вредителей на растения.
Настоящее изобретение будет дополнительно понятно со ссылкой на следующие неограничивающие примеры.
Примеры
Пример 1 Идентификация генов-мишеней у видов насекомых вредителей
1.1. Нормализованная cDNA-библиотека Lygus hesperus и получение dsRNA в многолуночных планшетах для скрининговых исследований
Нуклеиновые кислоты выделили из нимф Lygus hesperus различных стадий жизни, включая только что вылупившихся нимф 2, 4, 6 и 9 дневных нимф и взрослых. Библиотеку cDNA получали с использованием ПЦР-набора для синтеза cDNA SMARTer™, следуя инструкциям производителя (Clontech кат. № 634925). Библиотеку cDNA нормализовали с использованием Trimmer kit (Evrogen кат. № NK001) и клонировали в векторе PCR4-TOPO (Invitrogen). Нормализация введенных в клоны адаптеров M2 (Trimmer Kit, Evrogen, SEQ ID NO 92: AAGCAGTGGTATCAACGCAG), противоположно ориентированных на каждом конце клонов. Рекомбинантные конструкции векторов трансформировали в клетки Escherichia coli, штамм TOP10 (Invitrogen). Трансформированные клетки затем разбавляли и высевали с целью получения отдельных колоний или клонов. Клоны проверяли для подтверждения того, что избыточность клонов для библиотеки не превышает 5%. Отдельные клоны отбирали в жидкую LB (бульон Лурия) среду, в 96-луночные планшеты с глубокими лунками, и выращивали в течение ночи при 37°C. Планшеты также включали положительные (Lh423) и отрицательные (FP) контрольные клоны.
Для создания dsRNA с помощью ПЦР создали смысловой и антисмысловой фрагменты ДНК, содержащие последовательность промотора T7. Вкратце, помещали по 1 мкл бактериальной суспензии на клон в многолуночные ПЦР-планшеты, содержащие REDTaq® (Sigma кат. № D4309) и праймеры oGCC2738 (SEQ ID NO 93: AAGCAGTGGTATCAACGCAG) и oGCC2739 (SEQ ID NO 94: GCGTAATACGACTCACTATAGGAAGCAGTGGTATCAACGCAG) на основе M2 и T7-M2 последовательностей, соответственно. За ПЦР-реакцией следовала in vitro транскрипция, где добавляли 6 мкл продукта ПЦР на клон к 9 мкл RiboMAX™ Large Scale RNA Production System-T7 (Promega кат. № P1300) и инкубировали в течение ночи при 37°C. Конечный раствор dsRNA разбавляли в 2 раза в L. hesperus сахарозной пище, содержащей 15% сахарозы и 5 мкг/мкл дрожжевой tRNA (Invitrogen кат. № 15401-029) и использовали для скрининга. dsRNA, соответствующей положительному контрольному клону Lh423, является SEQ ID NO 101, и отрицательному FP контрольному клону является SEQ ID NO 104 (см. таблицу 4).
1.2. Скрининг на новые и эффективные гены-мишени Lygus hesperus с использованием cDNA-библиотеки экспрессируемых dsRNA
Разработали новое скрининговое исследование на эффективные мишени Lygus hesperus. Постановка анализа являлась следующей: каждая лунка 96-луночного планшета содержит однодневную L. hesperus нимфу под действием саше из парафильма, содержащего сахарозную пищу, которая включает либо тестируемую dsRNA, либо контрольную dsRNA в присутствии tRNA. Каждый планшет содержал dsRNA из 90 различных клонов, 3×Lh423 (положительный контроль) и 3×FP (флуоресцентный белок; отрицательный контроль). Каждый клон (тестируемая dsRNA) реплицировали на трех планшетах. После трех дней воздействия зарегистрировали число выживших нимф и заменили пищу на свежую пищу (комплексной) для выращивания, не содержащую dsRNA. Смертность оценивали в дни 4, 6 и 8. Идентичную постановку использовали для подтверждающих анализов первого и второго цикла, соответственно с 8 и 20 насекомыми, с одной нимфой на лунку.
Систему анализа проверяли с использованием dsRNA, соответствующей мишени Lh423, в качестве положительного контроля и dsRNA флуоресцентного белка в качестве отрицательного контроля: более 90% являлись истинно положительными и менее 5% являлись ложноположительными, соответственно.
Протестировали двадцать 96-луночных планшетов, поименованных Lh001-Lh020 (см. нижнюю линию на фигурах 1 и 2), содержащих 1800 отдельных клонов. 205 кандидатов идентифицировали и протестировали в первом подтверждающем анализе. Устанавливая пороговую величину на показателе ≥50% смертности, 41 отдельный клон идентифицировали и продвинули во второй цикл подтверждения. В анализе клоны сравнивали с положительными контролями Lh423 (RpL19) и Lh105.2 (Sec23) и отрицательным контролем Pt (кодирующим флуоресцентный белок коралла). dsRNA, соответствующей положительному контрольному клону(Lh423), является SEQ ID NO 101, соответствующей положительному контрольному клону Lh105.2 является SEQ ID NO 102 и отрицательному контрольному клону (Pt), является SEQ ID NO 104 (см. таблицу 4).
Подтверждающие анализы второго цикла, тестирующие 20 насекомых/тестируемых dsRNA, начали для всех тестируемых dsRNA, показывающих ≥50% смертности в первом подтверждении (фигуры 1 и 2). Подходящие мишени, соответствующие подтвержденным тестируемым dsRNA, обозначили с помощью ʺLhxxx числоʺ (см. таблицу 1). Применяя то же пороговое значение при ≥50% смертности, в первом скрининге подтвердили 15 мишеней.
Выполнили второй скрининг для идентификации новых мишеней Lygus hesperus. Результаты подтверждающих анализов второго цикла представлены на фигуре 12. Применяя то же пороговое значение при ≥50% смертности, несколько мишеней подтвердили во втором скрининге (см. таблицу 1С).
1.3. Идентификация мишеней Lygus
Одновременно с подтверждающими анализами на насекомых, вставки, соответствующие положительным клонам, секвенировали и для подтверждения идентичности мишеней использовали BlastX поиски по сравнению с базами данных белков Drosophila и Triboliu. В таблице 1 приводится краткое изложение биоинформационного анализа и текущая аннотация новых идентифицированных целевых последовательностей L. hesperus.
Пятнадцать новых мишеней L. hesperus идентифицировали в первом скрининге и 11 новых мишеней L. Hesperus идентифицировали во втором скрининге. Все мишени проявляют высокую эффективность против нимф L. hesperus, указывая на то, что cDNA, кодирующие двухцепочечные РНК, содержащиеся в них, являются существенно важными для выживания вредителя и, таким образом, представляют гены-мишени, представляющие интерес в целях борьбы с вредителями. Последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней, следовательно, определили и представили в таблицах 2 и 3, соответственно.
Lh594, ортолог Lygus hesperus к тропонину I Drosophila, участвующему в сокращении мышц и, следовательно, отсутствующему у растений, представляет новый класс мишеней, относящихся к специфическому для животных физиологическому пути, еще не изученному на GM-RNAi. У плодовой мушки тропонин I описан как гаплонедостаточный ген, проявляющий мутантный фенотип в гетерозиготном состоянии. Такие гены могут являться особенно восприимчивыми к снижению уровней экспрессии mRNA и, таким образом, могут рассматриваться как идеальные мишени для RNAi.
В данном пути Lh594 отобрали восемь мишеней (см. таблицу 1B). Для каждой мишени до 4 пар вырожденных праймеров ПЦР сконструировали на основе выравнивания последовательностей различных насекомых, включая пчелу, Tribolium и тлю. Данные праймеры применяют для амплификации фрагментов из мишеней Lygus hesperus. Последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней определили и представили в таблицах 2 и 3, соответственно.
новые мишени Lygus hesperus, ранжированные по % смертности в соответствии с результатами второго подтверждающего анализа (первый скрининг)
последовательность Lygus
новые мишени Lygus hesperus в пути Lh594
новые мишени Lygus hesperus, ранжированные по % смертности в соответствии с результатами второго подтверждающего анализа (второй скрининг)
во 2-м подтверждении
1.4. Клонирование полноразмерной cDNA с помощью RACE (быстрая амплификация концов cDNA)
Для клонирования полноразмерной cDNA, начиная от известного клона внутреннего фрагмента из самых эффективных мишеней, использовали набор 5’/3’ RACE (Roche, кат. № 1734792; на основе Sambrook, J. & Russell, D.M). Следовали стандартному протоколу, описанному в руководстве по эксплуатации. Вкратце, для 5’ RACE сконструировали антисмысловой праймер, специфический для целевой последовательности, на известной последовательности и применяли для синтеза первой цепи cDNA, применяя РНК Lygus в качестве матрицы. К первой цепи cDNA добавили хвост и использовали в качестве якоря для синтеза второй цепи и амплификации неизвестной концевой части транскрипта. Для 3’ RACE применяли якорный олиго-dT праймер для синтеза первой цепи cDNA. Для 5’ и 3’ RACE во второй реакции ПЦР применяли вложенные праймеры, специфические для целевой последовательности. Фрагменты ПЦР анализировали на агарозном геле, очищали, клонировали и секвенировали для подтверждения.
Последовательности полноразмерной cDNA, соответствующие мишеням, собрали в VectorNTi, полностью интегрированном пакете программного обеспечения для анализа последовательности, с целью анализа последовательности ДНК (Invitrogen).
Пример 2. In vitro получение двухцепочечных РНК для сайленсинга генов
2.2. Получение dsRNA, соответствующих частичным последовательностям генов-мишеней Lygus hesperus
Двухцепочечную РНК синтезировали в миллиграммовых количествах. Сначала две отдельные матрицы 5' T7 РНК-полимеразного промотора (смысловая матрица и антисмысловая матрица) создали с помощью ПЦР. ПЦР разрабатывали и проводили таким образом, чтобы получить смысловой и антисмысловой матричные полинуклеотиды, каждый из которых обладает T7 промотором в различной ориентации по отношению к целевой последовательности, подлежащей транскрибированию.
Для каждого из генов-мишеней создали смысловую матрицу, применяя мишень-специфический T7 прямой праймер и мишень-специфический обратный праймер. Антисмысловые матрицы создали, применяя мишень-специфический прямой праймер и мишень-специфический T7 обратный праймер. Последовательности соответствующих праймеров для амплификации смысловой и антисмысловой матриц посредством ПЦР для каждого из генов-мишеней приведены в таблице 4. ПЦР-продукты анализировали с помощью электрофореза в агарозном геле и очистили. Полученные смысловую и антисмысловую T7 матрицы смешали и транскрибировали путем добавления T7 РНК-полимеразы. Одноцепочечным РНК, полученным путем транскрипции с матриц, предоставили возможность соединиться, обработали с помощью ДНКазы и РНКазы и очистили осаждением. Смысловая цепь образующейся в результате dsRNA, полученная от каждого из генов-мишеней, приведена в таблице 4.
2.2. Испытания анализа выживаемости для новых мишеней Lygus hesperus
Для создания возможности ранжирования в соответствии с эффективностью, in vitro dsRNA, соответствующие новым мишеням, синтезировали и применили к L. hesperus в 10-дневных биопробах анализа выживаемости. Вкратце, однодневных нимф L. hesperus поместили в 96-луночные планшеты с сахарозными пленками, содержащими 0,5 мкг/мкл dsRNA-мишени, с добавлением 5 мкг/мкл дрожжевой tRNA. Планшеты инкубировали в течение 3 дней в стандартных условиях выращивания Lygus. В дни 3, 6 и 8 пищевые пленки обновляли пленками, содержащими только пищу Lygus. Lh423 (RpL19) использовали в качестве положительного контроля, а GFP dsRNA и сахарозную пищу использовали в качестве отрицательного контроля.
Результаты анализа выживаемости подтвердили данные первого и второго подтверждающих анализов. Lh594 установили в качестве высокоэффективной мишени с активностью и скоростью поражения, более сильной, чем сильный контроль Lh423.
До сих пор скрининг Lygus на новые мишени идентифицировал новые мишени с активностями более высокими или находящимися в диапазоне положительного контроля Lh423, которые включают Lh429, Lh594, Lh609, Lh610, Lh611, Lh617 и Lh618. Смертность, вызванная данными мишенями, показана на фигурах 3 и 4.
Для обеспечения более точного ранжирования мишеней в соответствии с их активностью выполнили анализы концентрации в зависимости от дозы. Новые мишени тестировали в in vitro анализах при концентрации в диапазоне от 0,4 до 0,025 мкг/мкл. Согласно условию, 24-дневных нимф тестировали в постановке с 96-луночным планшетом, в сахарозной пище с добавлением dsRNA и носителя tRNA. Результаты представлены в виде % выживаемости в течение 10-дневного эксперимента (фигуры 5-9) и суммированы в таблице 5.
На основе анализа кривой концентрации мишени ранжировали путем сравнения с исходными контролями Lh423 и Lh105 (таблица 5).
ранжирование новых мишеней Lygus в соответствии с DRC и по сравнению с исходными мишенями Lh423 и Lh105
мишени
Эффективность Lh594 подтвердили дополнительно. Эффект данной мишени отчетливо наблюдается по меньшей мере за один день до эффекта других мишеней и исходного положительного контроля Lh105 и Lh423. Так как Lh594 являлась высокоэффективной, в стандартном DRC эксперименте с концентрацией в диапазоне от 0,4 до 0,025 мкг/мкл dsRNA LD50 не достигли (фигура 6), следовательно, Lh594 эксперимент повторили, включая более низкие концентрации в диапазоне от 0,05 до 0,001 мкг/мкл dsRNA (фигура 10). В заключение, активность Lh594 наблюдали при такой низкой концентрации, как 0,0025 мкг/мкл, и получили примерно 90% уничтожение (что соответствует примерно 10% выживаемости) в день 6 при 0,025 мкг dsRNA.
Для дополнительного изучения эффективности Lh594 и роли носителя tRNA в ответе на RNAi у Lygus hesperus дополнительные in vitro анализы питания подготовили в отсутствие носителя tRNA. Lh594, Lh423 (исходный контроль) и GFP (отрицательный контроль) dsRNA получили in vitro, применяя стандартный способ. dsRNA очистили и тестировали при 5 мкг/мкл в отсутствие tRNA (фигура 11 A).
В отсутствие tRNA, мишени Lh594 и Lh423 индуцировали высокую летальность у нимф Lygus. Результаты данного эксперимента были воспроизведены позднее. dsRNA-мишень обладала способностью индуцировать эффекты RNAi-путем-кормления у нимф Lygus в отсутствие tRNA.
Для исследования активности dsRNA при более низких концентрациях в отсутствие носителя tRNA, поставили дополнительные эксперименты, применяя уменьшение количеств dsRNA (фигура 11 B).
Аналогичный подход применялся для мишеней Lygus, которые идентифицировали во втором скрининге. Для обеспечения ранжирования мишеней в соответствии с их активностью выполнили анализы концентрации в зависимости от дозы. Новые мишени тестировали в in vitro анализах при концентрациях в диапазоне от 0,5 до 0,05 мкг/мкл. Согласно условию, 24-дневных нимф тестировали в постановке с 96-луночным планшетом, в сахарозной пище с добавлением dsRNA и носителя tRNA. Результаты представлены в виде % выживаемости в течение 9-дневного эксперимента (фигуры 15 A-D). Lh594 и Lh423 включили в анализ в качестве эталонных мишеней. Результаты суммированы в таблице 6. На основе анализа кривой концентрации мишени ранжировали путем сравнения с исходным контролем Lh423.
новые мишени Lygus из второго скрининга, ранжирование в соответствии с DRC и по сравнению с исходными мишенями Lh423 и Lh594
Пример 3. Скрининг пути тропонина
Для создания возможности тестирования мишеней пути тропонина, in vitro полученные dsRNA, соответствующие Lh619, Lh620, Lh621, Lh622, Lh622, Lh623, Lh624, Lh625 и Lh626, синтезировали и применили к L. hesperus в 10-дневных биопробах анализа выживаемости. Вкратце, однодневных нимф L. hesperus поместили в 96-луночные планшеты с сахарозными пленками, содержащими 0,5 мкг/мкл dsRNA-мишени, с добавлением 5 мкг/мкл дрожжевой tRNA. Планшеты инкубировали в течение 3 дней в стандартных условиях выращивания Lygus. В дни 3, 6 и 8 пищевые пленки обновляли пленками, содержащими только пищу Lygus. Lh594 (тропонин I) использовали в качестве положительного контроля и GFP dsRNA и сахарозную пищу использовали в качестве отрицательных контролей (фигура 13). Четыре мишени затем включили в анализы кривой дозовой зависимости в in vitro анализе, с концентрациями в диапазоне от 0,4 до 0,025 мкг/мкл. Согласно условию, 24-дневных нимф тестировали в постановке с 96-луночным планшетом, в сахарозной пище с добавлением dsRNA и носителя tRNA. Результаты представлены в виде % выживаемости в течение 10-дневного эксперимента (фигуры 14 A-B).
Пример 4. Идентификация генов-мишеней у Leptinotarsa decemlineata
4.1. Нормализованная cDNA-библиотека Leptinotarsa decemlineata и получение dsRNA в многолуночных планшетах для скрининговых исследований
Нуклеиновые кислоты выделили из личинок Leptinotarsa decemlineata различных стадий. Библиотеку cDNA получали с использованием ПЦР-набора для синтеза cDNA SMARTer™, следуя инструкциям производителя (Clontech кат. № 634925). Библиотеку cDNA нормализовали, применяя набор Trimmer kit (Evrogen кат. № NK001), и клонировали в PCR®-BLUNTII-TOPO® вектор (Invitrogen). При нормализации клонов вводили M2 адаптеры (Trimmer Kit, Evrogen, SEQ ID NO 92: AAGCAGTGGTATCAACGCAG), противоположно ориентированных на каждом конце клонов. Рекомбинантные конструкции векторов трансформировали в клетки Escherichia coli, штамм TOP10 (Invitrogen). Трансформированные клетки затем разбавляли и высевали с целью получения отдельных колоний или клонов. Клоны проверяли для подтверждения того, что избыточность клонов для библиотеки не превышает 5%. Отдельные клоны инокулировали в жидкую LB (бульон Лурия) среду, в 96-луночные планшеты, и выращивали в течение ночи при 37°C. Планшеты также включали положительный (Ld513) и отрицательный (FP) контрольные клоны.
Для создания dsRNA с помощью ПЦР создали смысловой и антисмысловой фрагменты ДНК, содержащие последовательность T7 промотора. Вкратце, на клон, 1 мкл бактериальной суспензии помещали в многолуночные ПЦР-планшеты, содержащие REDTaq® (Sigma кат. № D4309) и праймеры oGCC2738 (SEQ ID NO 93: AAGCAGTGGTATCAACGCAG) и oGCC2739 (SEQ ID NO 94: GCGTAATACGACTCACTATAGGAAGCAGTGGTATCAACGCAG) на основе последовательностей M2 и T7-M2, соответственно. За ПЦР реакцией следовала in vitro транскрипция, при этом на клон использовали 6 мкл ПЦР-продукта в 20 мкл реакционного объема, содержащего реагенты для транскрипции, обеспеченные набором RiboMAX™ Large Scale RNA Production System-T7 kit (Promega кат. № P1300), и инкубировали в течение ночи при 37°C. Конечный раствор dsRNA разбавляли стерильной водой Milli-Q и использовали для скрининга. dsRNA, соответствующей положительному Ld513 контрольному клону, является SEQ ID NO 400 (см. таблицу 9), и отрицательному FP контрольному клону, является SEQ ID NO 104 (см. таблицу 4).
4.2. Скрининг на новые и эффективные гены мишени Leptinotarsa decemlineata с использованием cDNA-библиотеки экспрессируемых dsRNA
Каждая лунка 48-луночного планшета содержала 0,5 мл искусственной пищи, предварительно обработанной с помощью наружного покрытия 25 мкл (или 1 мкг) тестируемой или контрольной dsRNA. Одну L2 личинку помещали в каждую лунку и на клон тестировали 3 личинки. Количества выживших CPB оценивали в дни 4, 7 и 10.
Во второй биопробе личинок CPB кормили пищей, обработанной наружно наносимой тестируемой dsRNA, полученной из клонов, полученных из нормализованной библиотеки cDNA. Одну личинку помещали в лунку 48-луночного мультипланшета, содержащего 0,5 мл пищи, предварительно обработанной с помощью наружного покрытия 25 мкл 40 нг/мкл dsRNA раствора. Всего на обработку (клон) тестировали двадцать четыре личинки. Количество выживших насекомых оценивали в дни 4, 5, 6, 7, 8 и 11. Процент смертности личинок рассчитывали относительно дня 0 (начало исследования) (см. фигуру 21).
4.3. Идентификация мишеней у L. decemlineata жуков
Новые целевые последовательности из скрининга в 5.2. и целевые последовательности, соответствующие мишеням пути тропонина, ортологическим Lygus Lh594, Lh619 и Lh620 последовательностям, идентифицировали у L. decemlineata. Праймеры, которые обеспечили соответствующие фрагменты cDNA для Ld594, перечислены в таблице 17. Последовательности cDNA и предсказанные аминокислотные последовательности данных генов-мишеней определили и представили в таблицах 7 и 9, соответственно.
4.4. Получение dsRNA, соответствующих частичным последовательностям L. decemlineata генов-мишеней
dsRNA синтезировали с использованием праймеров, которые указаны в таблице 9. Смысловая цепь полученной в результате dsRNA, полученная из генов-мишеней, приведена в таблице 9.
4.5. Испытания анализа выживаемости для новых L. decemlineata мишеней
Ранний личиночный анализ
Синтетические dsRNA получили для 3 мишеней, Ld594, Ld619 и Ld620, и тестировали в анализе с питанием на личинках CPB (см. фигуру 16). 10-дневный анализ проводили в 48-луночных планшетах, на искусственной пище (на основе Gelman et al, J Ins Sc,1:7, 1-10: Artificial diets for rearing the Colorado Potato Beetle), с добавлением 1 мкг dsRNA/лунку с 12 личинками согласно условиям.
Наблюдали четкий эффект на развитие личинок. Второй анализ поставили для изучения эффекта данных dsRNA в течение окукливания и метаморфоза (см. анализ окукливания ниже).
Анализ окукливания
Анализ окукливания CPB поставили для изучения эффекта RNAi нокдауна Ld594, Ld619 и Ld620 в течение окукливания и метаморфоза. Личинок четвертой возрастной стадии кормили 1 мкг in vitro синтезированной dsRNA, распределенной на листовых дисках картофеля, и затем переносили в коробку с необработанными свежими листьями картофеля. Через четыре дня выживших насекомых помещали на вермикулит для обеспечения окукливания. Обработанные с помощью Lh594 насекомые были медленными, более мелкими и, в основном, не способными пройти окукливание. Вылупление куколки оценивали в конце эксперимента. Для необработанного контроля 24 личинки окуклились и все вылупились в здоровых имаго. Для Ld620 наблюдали снижение численности личинок, прогрессирующих в окукливание. Для трех протестированных мишеней отсутствовали личинки, развившиеся в здоровые куколки, и ни одна не вылупилась в имаго. Мертвые насекомые, извлеченные из вермикулита, демонстрировали различную степень пороков развития (фиг. 17).
Ld594, Ld619 и Ld620 вначале предстали в качестве несмертельных мишеней в анализе личинок CPB, хотя снижение жизнеспособности отчетливо наблюдалось у насекомых, обработанных dsRNA. С другой стороны, в анализе окукливания все 3 мишени индуцировали сильные эффекты и ингибировали вхождение в окукливание и/или метаморфоз.
Анализ имаго
Для оценки активности Ld594, Ld619 и Ld620 у имаго CPB поставили анализ листовых дисков. Листовые диски картофеля (1,7 см в диаметре) покрыли dsRNA или контролями и поместили в 3,5 см чашки Петри с одним взрослым жуком. На следующий день насекомым предоставили свежий обработанный листовой диск. На третий день имаго перенесли в коробку, содержащую достаточно свежих, необработанных листьев картофеля для поддержания выживания необработанных контролей. На обработку тестировали 6 имаго и подсчитывали количества выживших и умирающих насекомых с регулярными интервалами от дня 6 до дня 13. Насекомые считались умирающими, если они не могли перевернуться после укладывания на спину.
Несмотря на относительно высокий уровень фона в отрицательном контроле в данном конкретном анализе, наблюдали четкие эффекты на насекомых, которых подвергли воздействию Ld594 или Ld619 dsRNA (фиг. 18).
Пример 5. Идентификация генов-мишеней у Nilaparvata lugens
5.1. Идентификация мишеней Nilaparvata lugens
Новые целевые последовательности, соответствующие мишеням пути тропонина и названные Nl594 (тропонин I), Nl619 (тропонин T) и Nl626 (тропонин C), идентифицировали у коричневого дельфацида, Nilaparvata lugens. Ортологические последовательности генов Lygus, названные Nl594 (тропонин I), Nl619 (тропонин T) и Nl625/626 (тропонин C), клонировали посредством ПЦР с вырожденными праймерами с использованием BPH cDNA в качестве матрицы. В дополнение, полноразмерную cDNA идентифицировали для Nl594, применяя RACE (см. способ выше). ПЦР-систему AmpliTaq Gold (Applied Biosystems) применяли, следуя инструкциям производителя и в стандартных условиях для реакции ПЦР с вырожденными праймерами, как правило, следующим образом: 1 цикл в 10 минут при 95°C, с последующими 40 циклами по 30 секунд при 95°C, 1 минуту при 50°C и 1 минуту при 72°C, с последующими 10 минутами при 72°C. Для увеличения степени успеха до 10 различных вырожденных праймеров, прямых и обратных, разработали на основе выравниваний ортологических последовательностей у других видов и использовали в различных комбинациях. Полученные ПЦР-фрагменты очищали из геля с помощью набора для экстракции геля (Qiagen Cat. No 28706) и клонировали в TOPO TA вектор (Invitrogen). Клоны секвенировали и консенсусные последовательности использовали в Blast поисках в отношении различных доступных баз данных последовательностей насекомых для подтверждения значимости вставки. Вырожденные праймеры, которые привели к успешной амплификации, перечислены в таблице 18.
Последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней и еще одного гена-мишени (Nl537) определили и представили в таблицах 10 и 11, соответственно.
5.2 Получение dsRNA, соответствующих частичным последовательностям генов-мишеней Nilaparvata lugens
dsRNA синтезировали с использованием праймеров, которые указаны в таблице 12. Смысловая цепь образующейся в результате dsRNA, полученная от каждого из генов-мишеней, приведена в таблице 12.
5.3. Испытания анализа выживаемости для новых мишеней Nilaparvata lugens
dsRNA синтезировали и тестировали в предварительно оптимизированных анализах RNAi-путем-кормления у BPH, в присутствии цвиттерионного детергента, CHAPSO, при 0,1% конечной концентрации. dsRNA тестировали при 0,5 мкг/мкл конечной концентрации. Nl537, эффективную мишень в анализах BPH, использовали в качестве исходной мишени в данном анализе. Выживаемость насекомых оценивали на протяжении 9 дней.
Результаты биопробы показали, что у BPH Nl594, Nl619 и Nl626 также являлись эффективными мишенями RNAi у BPH (фиг. 19).
Пример 6. Идентификация генов-мишеней у Acyrthosiphon pisum
6.1 Идентификация мишеней Acyrthosiphon pisum
Новые целевые последовательности идентифицировали у тлей и назвали Ap423, Ap537, Ap560 и Ap594, следуя той же номенклатуре: “Apxxx”, где “Ap” соответствует Acyrthosiphon pisum и “xxx” соответствует ID мишени. Праймеры разрабатывали на основе открытого источника предсказания генов в AphidBase (ref: http://www.aphidbase.com/) (таблица 13).
Последовательности ДНК и предсказанные аминокислотные последовательности данных генов-мишеней определили и представили в таблицах 14 и 15, соответственно.
6.2 Получение dsRNA, соответствующих частичным последовательностям генов-мишеней тли
dsRNA синтезировали с использованием праймеров, которые указаны в таблице 16. Смысловая цепь образующейся в результате dsRNA, полученная от каждого из генов-мишеней, приведена в таблице 16.
6.3 Испытания анализа выживаемости для новых мишеней у тлей
RNAi-путем-кормления тестировали у Acyrthosiphon pisum (гороховая тля) с 4 мишенями Ap594, Ap423, Ap560, Ap537. Последовательности амплифицировали с помощью ПЦР с использованием праймеров, разработанных на основе открытого источника информации о последовательностях (http://www.aphidbase.com), и cDNA, полученной из тлей. Синтетические dsRNA получали и тестировали в конечной концентрации 0,5 мкг/мкл в присутствии 5 мкг/мкл дрожжевой tRNA в сахарозной пище. Десять новорожденных нимф гороховой тли поместили в маленькую чашку Петри (32 мм). Пятьдесят мкл пищи (с tRNA и dsRNA) наносили пипеткой на верхнюю часть первого слоя парафильма. Второй слой парафильма покрывал пищу и создавал пакетик для кормления, где тли могли питаться. На каждую мишень подготовили пять повторов 10 новорожденных нимф. GFP dsRNA использовали в качестве отрицательного контроля. Пищу обновляли в дни 4 и 7 анализов и оценивали выживаемость (фиг. 20).
cDNA клона, представленного в таблице 2
5’→3’
5’→3’
5’→3’
SEQ ID NO 29
SEQ ID NO 30
SEQ ID NO 33
SEQ ID NO 34
SEQ ID NO 37
SEQ ID NO 38
SEQ ID NO 41
SEQ ID NO 42
SEQ ID NO 45
SEQ ID NO 46
SEQ ID NO 49
SEQ ID NO 50
SEQ ID NO 53
SEQ ID NO 54
SEQ ID NO 107
SEQ ID NO 108
SEQ ID NO 111
SEQ ID NO 112
SEQ ID NO 115
SEQ ID NO 116
SEQ ID NO 119
SEQ ID NO 120
SEQ ID NO 57
SEQ ID NO 58
SEQ ID NO 61
SEQ ID NO 62
SEQ ID NO 65
SEQ ID NO 66
SEQ ID NO 69
SEQ ID NO 70
SEQ ID NO 73
SEQ ID NO 74
SEQ ID NO 77
SEQ ID NO 78
SEQ ID NO 208
SEQ ID NO 209
SEQ ID NO 212
SEQ ID NO 213
SEQ ID NO 216
SEQ ID NO 217
SEQ ID NO 220
SEQ ID NO 221
SEQ ID NO 224
SEQ ID NO 225
SEQ ID NO 228
SEQ ID NO 229
SEQ ID NO 232
SEQ ID NO 233
SEQ ID NO 236
SEQ ID NO 237
SEQ ID NO 240
SEQ ID NO 241
SEQ ID NO 244
SEQ ID NO 245
SEQ ID NO 248
SEQ ID NO 249
SEQ ID NO 25
SEQ ID NO 253
SEQ ID NO 256
SEQ ID NO 257
SEQ ID NO 260
SEQ ID NO 261
SEQ ID NO 264
SEQ ID NO 265
SEQ ID NO 268
SEQ ID NO 269
SEQ ID NO 272
SEQ ID NO 273
SEQ ID NO 276
SEQ ID NO 277
SEQ ID NO 280
SEQ ID NO 281
5’→3’
5’→3’
5’→3’
ДНК 5’→3’
SEQ ID NO 284
SEQ ID NO 285
SEQ ID NO 288
SEQ ID NO 289
SEQ ID NO 292
SEQ ID NO 293
SEQ ID NO 398
SEQ ID NO 399
5’→3’
5’→3’
5’→3’
5’→3’
SEQ ID NO 296
SEQ ID NO 297
SEQ ID NO 300
SEQ ID NO 301
SEQ ID NO 304
SEQ ID NO 305
SEQ ID NO 308
SEQ ID NO 309
5’→3’
5’→3’
5’→3’
SEQ ID NO 312
SEQ ID NO 313
SEQ ID NO 316
SEQ ID NO 317
SEQ ID NO 320
SEQ ID NO 321
SEQ ID NO 324
SEQ ID NO 325
Специалистам в данной области будет очевидно, что многочисленные изменения и/или модификации можно внести в вышеупомянутые анализы без отступления от сущности или объема данного анализа, который описан в общем. Специалисты в данной области обнаружат или смогут установить, применяя не более чем обычное экспериментирование, многие эквиваленты конкретных примеров, и такие эквиваленты охватываются настоящим изобретением. Настоящий пример, таким образом, следует рассматривать во всех отношениях как иллюстративный, а не ограничительный.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> ДЕВГЕН НВ
<120> ПОДАВЛЕНИЕ ЭКСПРЕССИИ ГЕНОВ У НАСЕКОМЫХ-ВРЕДИТЕЛЕЙ
<130> NLW/P122274WO01
<160> 405
<170> PatentIn версия 3.5
<210> 1
<211> 1096
<212> ДНК
<213> Lygus hesperus
<400> 1
gcgatctaag gcaggtggca gacagctcga tgacggcagt gggccaagca ataatggata 60
gtcattcata gcaccccagc tttactaagc tctgccgtag tgttggattg ggagcggata 120
caattcacca cagaacagct atgacatgat acgcagtccg aataccctca taaaggacta 180
gtctgcaggt ttaacgatcg cgtagcagtg tatcacgcag agtacatggg gagtgactgt 240
gtgaacctgc tgggtacatc atcacccctc tccttcttca gttatataag acacagtccc 300
taaaggacac cagcaaaaat ggcggatgat gaggcgaaga aggccaaaca ggccgaaatc 360
gagaggaagc gcgctgaagt gcgcaagagg atggaggaag cctctaaggc gaagaaagcc 420
aagaagggtt tcatgacccc ggaaaggaag aagaaactcc gactcctgct gaggaaaaaa 480
gccgctgagg aactgaagaa ggagcaggaa cgcaaagcag ctgagaggag gcgaacgatt 540
gaggagcgct gcgggcaaat tgccgacgtc gacaacgcca atgaagcaac cttgaagaaa 600
ctctgcacag actaccataa gcgaattgac gctctggaga ggagtaaaat tgacatcgaa 660
ttcgaagtgg agagacgtga ccttgagatc gccgacctca acagccaggt caacgacctc 720
cgtggtaaat tcgtcaaacc taccttgaaa aaggtttcca agtacgaaaa caaattcgcc 780
aagctccaga agaaggctgc cgagttcaac ttcagaaacc aactcaaggt cgtcaaaaag 840
aaagaattca ccctggaaga agaagacaaa gagccgaaga aatcggaaaa ggcggagtgg 900
cagaagaaat gaagggaaaa caagcacacc atctcacaaa ataaaataaa cgaaaatctt 960
tcacacgttt accaatttta taacacggtc ctcacaaatt atgttcctta aataatttgt 1020
ataatccatc ctcgcactac aatcaatatt aatatttaaa tacaaaacca aaaaaaaaaa 1080
aaaaaaaaaa aaaaaa 1096
<210> 2
<211> 491
<212> ДНК
<213> Lygus hesperus
<400> 2
caaacaggcc gaaatcgaga ggaagcgcgc tgaagtgcgc aagaggatgg aggaagcctc 60
taaggcgaag aaagccaaga agggtttcat gaccccggaa aggaagaaga aactccgact 120
cctgctgagg aaaaaagccg ctgaggaact gaagaaggag caggaacgca aagcagctga 180
gaggaggcga acgattgagg agcgctgcgg gcaaattgcc gacgtcgaca acgccaatga 240
agcaaccttg aagaaactct gcacagacta ccataagcga attgacgctc tggagaggag 300
taaaattgac atcgaattcg aagtggagag acgtgacctt gagatcgccg acctcaacag 360
ccaggtcaac gacctccgtg gtaaattcgt caaacctacc ttgaaaaagg tttccaagta 420
cgaaaacaaa ttcgccaagc tccagaagaa ggctgccgag ttcaacttca gaaaccaact 480
caaggtcgtc a 491
<210> 3
<211> 431
<212> ДНК
<213> Lygus hesperus
<400> 3
atgggcatca tgtcgaaagc tgaactcgct tgtgtttact ccgctctcat cctcatcgac 60
gacgatgtcg ccgtgacggg tgagaagatt caaaccatcc tgaaggctgc cagtgtcgac 120
atcgagccgt actggcccgg tctgttcgcc aaggccctcg agggtatcaa ccccaaagac 180
ctcatctcct ccattggaag cggagttggt gctggagcgc cggctgtcgg tggagctgca 240
cctgccgccg ctgctgcccc tgccgctgag gctaagaagg aagagaagaa gaaggtcgaa 300
agcgatccag aatccgatga tgacatgggc ttcggtcttt tcgactaaga gcattccaca 360
gcgggttctc atttgttttt aagattttct tttaaaaaat aaaacttcca aaaaaaaaaa 420
aaaaaaaaaa g 431
<210> 4
<211> 332
<212> ДНК
<213> Lygus hesperus
<400> 4
gggcatcatg tcgaaagctg aactcgcttg tgtttactcc gctctcatcc tcatcgacga 60
cgatgtcgcc gtgacgggtg agaagattca aaccatcctg aaggctgcca gtgtcgacat 120
cgagccgtac tggcccggtc tgttcgccaa ggccctcgag ggtatcaacc ccaaagacct 180
catctcctcc attggaagcg gagttggtgc tggagcgccg gctgtcggtg gagctgcacc 240
tgccgccgct gctgcccctg ccgctgaggc taagaaggaa gagaagaaga aggtcgaaag 300
cgatccagaa tccgatgatg acatgggctt cg 332
<210> 5
<211> 468
<212> ДНК
<213> Lygus hesperus
<400> 5
atgggggcag gtcttctcca taaccataga ttatcttcgt gtatcgtgtc gggctttcgg 60
ctgaggtcct aattagtaaa taatgattcc gcctacgtcg cggcctcagg tcactgtcta 120
cagtgacaaa aatgaggcca ccgggactct cctcaacctc ccggctgtct tcaacgcccc 180
cattcgcccc gatgttgtga acttcgttca ccaaaatgtc gctaaaaacc acaggcagcc 240
ctactgtgtc tccgctcaag ctggtcatca gacttcagct gagtcctggg gtaccggtcg 300
tgctgtggct cgtatccccc gtgttcgcgg aggtggtact caccgctcag gtcagggtgc 360
ttttggcaac atgtgtcgcg gcggtaggat gttcgctccc actcgcccat ggcgtcgttg 420
gcaccgcaaa atcaacgtta accaaaaaaa aaaaaaaaaa aaaaaaaa 468
<210> 6
<211> 429
<212> ДНК
<213> Lygus hesperus
<400> 6
gggcaggtct tctccataac catagattat cttcgtgtat cgtgtcgggc tttcggctga 60
ggtcctaatt agtaaataat gattccgcct acgtcgcggc ctcaggtcac tgtctacagt 120
gacaaaaatg aggccaccgg gactctcctc aacctcccgg ctgtcttcaa cgcccccatt 180
cgccccgatg ttgtgaactt cgttcaccaa aatgtcgcta aaaaccacag gcagccctac 240
tgtgtctccg ctcaagctgg tcatcagact tcagctgagt cctggggtac cggtcgtgct 300
gtggctcgta tcccccgtgt tcgcggaggt ggtactcacc gctcaggtca gggtgctttt 360
ggcaacatgt gtcgcggcgg taggatgttc gctcccactc gcccatggcg tcgttggcac 420
cgcaaaatc 429
<210> 7
<211> 523
<212> ДНК
<213> Lygus hesperus
<400> 7
atgggatctc tatgctgaaa aggtcgccac cagaggtttg tgtgctattg cacaagctga 60
atccctccgt tacaaactca ttggcggtct tgctgtccga ggggcttgct atggtgtcct 120
tcgcttcatc atggaaaatg gtgccaaggg ttgcgaagtc gtagtatctg gaaaactgcg 180
tggtcagaga gccaagtcaa tgaagttcgt ggatggtttg atgatccaca gtggggatcc 240
ctgtaacgaa tatgttgata ctgctacccg acatgtgctc cttagacaag gtgtcctggg 300
aataaaggtg aagattatgt tgccgtggga cgttaccggc aaaaatgggc cgaagaaccc 360
tcttcccgac cacgtcagcg ttctcttacc taaggaggag ctaccaaatt tggccgttag 420
tgtgcctgga tccgacatca aaccaaagcc tgaagtacca gcacccgctt tgtgaatata 480
aacttctttt ttgtaaaaaa aaaaaaaaaa aaaaaaaaaa aaa 523
<210> 8
<211> 431
<212> ДНК
<213> Lygus hesperus
<400> 8
attgcacaag ctgaatccct ccgttacaaa ctcattggcg gtcttgctgt ccgaggggct 60
tgctatggtg tccttcgctt catcatggaa aatggtgcca agggttgcga agtcgtagta 120
tctggaaaac tgcgtggtca gagagccaag tcaatgaagt tcgtggatgg tttgatgatc 180
cacagtgggg atccctgtaa cgaatatgtt gatactgcta cccgacatgt gctccttaga 240
caaggtgtcc tgggaataaa ggtgaagatt atgttgccgt gggacgttac cggcaaaaat 300
gggccgaaga accctcttcc cgaccacgtc agcgttctct tacctaagga ggagctacca 360
aatttggccg ttagtgtgcc tggatccgac atcaaaccaa agcctgaagt accagcaccc 420
gctttgtgaa t 431
<210> 9
<211> 823
<212> ДНК
<213> Lygus hesperus
<400> 9
catggggaca ctctcttttt cttcatcgcg tggctcgctg ccgtgtggtt agggagtttc 60
ctactttaat tttttagtgt aattcatctt caaaatgacg tcgaaggttt ctcgtgagac 120
cctctacgag tgcatcaatg gagtcatcca gtcctcccag gagaagaaga ggaacttcgt 180
ggagactgtg gagatccaga tcggtctgaa gaactacgat ccccagaagg acaagcgttt 240
ctcgggaact gtcaagctga agcacattcc aaggcctaaa atgcaggttt gcatcctcgg 300
agatcaacag cattgcgacg aggccaaagc caacaacgtg ccctacatgg acgtcgaggc 360
tctgaagaag ctcaacaaaa acaagaagct cgtcaagaaa ttggccaaga aatacgacgc 420
tttcctcgcc tcagaagccc tcatcaagca gatccccagg ctcctcggac ccggtctcaa 480
caaggcgggc aagttccctg gtctcctctc tcaccaggag tccatgatga tgaagatcga 540
cgaagtcaag gccaccatca agttccaaat gaagaaggtg ttgtgcctct cagtggctgt 600
cggtcacgtc ggcatgactg ctgatgagct cgtccagaac gtgcacttgt cggtcaactt 660
cctcgtttcg ctcctcaaga agcactggca gaacgtcagg tctctccacg tcaaatccac 720
gatgggacct ccccagaggc tttactaaac atcttgtttt ttacttttga cgaataaaat 780
tcgttttatt ctcgaaaaaa aaaaaaaaaa aaaaaaaaaa aaa 823
<210> 10
<211> 607
<212> ДНК
<213> Lygus hesperus
<400> 10
ccctctacga gtgcatcaat ggagtcatcc agtcctccca ggagaagaag aggaacttcg 60
tggagactgt ggagatccag atcggtctga agaactacga tccccagaag gacaagcgtt 120
tctcgggaac tgtcaagctg aagcacattc caaggcctaa aatgcaggtt tgcatcctcg 180
gagatcaaca gcattgcgac gaggccaaag ccaacaacgt gccctacatg gacgtcgagg 240
ctctgaagaa gctcaacaaa aacaagaagc tcgtcaagaa attggccaag aaatacgacg 300
ctttcctcgc ctcagaagcc ctcatcaagc agatccccag gctcctcgga cccggtctca 360
acaaggcggg caagttccct ggtctcctct ctcaccagga gtccatgatg atgaagatcg 420
acgaagtcaa ggccaccatc aagttccaaa tgaagaaggt gttgtgcctc tcagtggctg 480
tcggtcacgt cggcatgact gctgatgagc tcgtccagaa cgtgcacttg tcggtcaact 540
tcctcgtttc gctcctcaag aagcactggc agaacgtcag gtctctccac gtcaaatcca 600
cgatggg 607
<210> 11
<211> 435
<212> ДНК
<213> Lygus hesperus
<400> 11
atgggaccaa taaagatcaa ctttcccaga gaaagacttg ctatgcccag cataatcagg 60
tccgagaaat ccgcaaaaag atggttaaaa acatcagtga cagcatttcc agctgtgatt 120
tgaggagtgt tgtgaacaag ctgatcccag actccatcgc taaagatata gaaaagaatt 180
gccaaggaat ctacccactc cacgatgtgt acattcggaa ggtgaaggtg ttgaagaagc 240
cgaggttcga gctcagcaag ctccttgagc ttcacgtcga tggcaaaggg atcgacgaac 300
ccggcgcgaa agtgacgagg actgacgctt acgagcctcc agttcaagag tctgtctaag 360
taaacatttt atataaagtt aacaaaaaat aaaggtgtct cgcctgacta aaaaaaaaaa 420
aaaaaaaaaa aaaaa 435
<210> 12
<211> 353
<212> ДНК
<213> Lygus hesperus
<400> 12
ccaataaaga tcaactttcc cagagaaaga cttgctatgc ccagcataat caggtccgag 60
aaatccgcaa aaagatggtt aaaaacatca gtgacagcat ttccagctgt gatttgagga 120
gtgttgtgaa caagctgatc ccagactcca tcgctaaaga tatagaaaag aattgccaag 180
gaatctaccc actccacgat gtgtacattc ggaaggtgaa ggtgttgaag aagccgaggt 240
tcgagctcag caagctcctt gagcttcacg tcgatggcaa agggatcgac gaacccggcg 300
cgaaagtgac gaggactgac gcttacgagc ctccagttca agagtctgtc taa 353
<210> 13
<211> 474
<212> ДНК
<213> Lygus hesperus
<400> 13
catgggtacg aatatcgacg gtaaaagaaa ggtgatgttc gccatgaccg ccatcaaagg 60
tgtcggcaga cggtacgcca acattgtcct caagaaggcc gatgtcaact tggacaagag 120
ggccggcgaa tgctccgaag aagaagttga aaagatcgtt accatcatgc aaaaccctag 180
gcaatacaaa attcccaact ggttcctcaa cagacaaaaa gacaccgtcg agggcaaata 240
ctctcagttg acttcctccc tgctggattc caagctccgt gacgaccttg agcgactcaa 300
gaagatcagg gcccacagag gcatgaggca ctactggggt ttgagggtgc gtggtcaaca 360
cacgaagacc accggaagga gaggacgaac tgttggtgtg tccaagaaga agtaatttta 420
atttcctaat aaattggttt tttcaaaaaa aaaaaaaaaa aaaaaaaaaa aaaa 474
<210> 14
<211> 332
<212> ДНК
<213> Lygus hesperus
<400> 14
gaaaggtgat gttcgccatg accgccatca aaggtgtcgg cagacggtac gccaacattg 60
tcctcaagaa ggccgatgtc aacttggaca agagggccgg cgaatgctcc gaagaagaag 120
ttgaaaagat cgttaccatc atgcaaaacc ctaggcaata caaaattccc aactggttcc 180
tcaacagaca aaaagacacc gtcgagggca aatactctca gttgacttcc tccctgctgg 240
attccaagct ccgtgacgac cttgagcgac tcaagaagat cagggcccac agaggcatga 300
ggcactactg gggtttgagg gtgcgtggtc aa 332
<210> 15
<211> 440
<212> ДНК
<213> Lygus hesperus
<400> 15
gtgagttctt ctgttgatta gtttttcctt ccctgaaatt atttcgttga agttaatttg 60
gattaccctg aaagaatccg ctgctttttc tctcgctaaa aatcttttac acccgtcacc 120
acggccccct gtgggcaggc acaagctgaa gcacctgccc gtgcacccta actcgcactt 180
catggacgtc aactgccctg ggtgttataa aatcccaacg gtgttctccc ccgcccagaa 240
cgacttcggc tgctggacct gttccaccat cctctgcctg cccacagggg gccgtgccga 300
cctcaccaaa agatgctcgt ttaggagaaa tcaacattat tattcttggt gggaacactt 360
attttttttg taattaaatt tcaaactaca aaataacttt tccgaaaaac actacaaaaa 420
aaattaaaaa caaaaaaaaa 440
<210> 16
<211> 324
<212> ДНК
<213> Lygus hesperus
<400> 16
cttccctgaa attatttcgt tgaagttaat ttggattacc ctgaaagaat ccgctgcttt 60
ttctctcgct aaaaatcttt tacacccgtc accacggccc cctgtgggca ggcacaagct 120
gaagcacctg cccgtgcacc ctaactcgca cttcatggac gtcaactgcc ctgggtgtta 180
taaaatccca acggtgttct cccccgccca gaacgacttc ggctgctgga cctgttccac 240
catcctctgc ctgcccacag ggggccgtgc cgacctcacc aaaagatgct cgtttaggag 300
aaatcaacat tattattctt ggtg 324
<210> 17
<211> 357
<212> ДНК
<213> Lygus hesperus
<400> 17
atgggttcaa gagagttaaa gccaagaggg ccaagaagga cgacggtgag atatttgccg 60
ctaaaaagga agtctacaag ccctctgagc agaggaaagc agaccagaaa aacattgaca 120
aacagaccct gaaagccatc aagcgactca agggagacgc ttgcctcatg aggaaatacc 180
tttgcaccat gttcggattc aggagcagtc aatatcccca ccgtatgaag ttttaatatg 240
ttttcagcca ataaataagt gaaagtttct cttttttatt actacagact caaattttta 300
ttttctgaaa attattaaaa attcttaatg gcaaaaaaaa aaaaaaaaaa aaaaaaa 357
<210> 18
<211> 223
<212> ДНК
<213> Lygus hesperus
<400> 18
gttcaagaga gttaaagcca agagggccaa gaaggacgac ggtgagatat ttgccgctaa 60
aaaggaagtc tacaagccct ctgagcagag gaaagcagac cagaaaaaca ttgacaaaca 120
gaccctgaaa gccatcaagc gactcaaggg agacgcttgc ctcatgagga aatacctttg 180
caccatgttc ggattcagga gcagtcaata tccccaccgt atg 223
<210> 19
<211> 632
<212> ДНК
<213> Lygus hesperus
<400> 19
atgggacctt ttttccgtgt gtctggctta ggcctcgcgt gttcttgtat ttttacggga 60
aatttagtga aaaagtgtaa atttaacgcg taaaaatggg tcgtatgcac gcacctggta 120
agggtatttc ccagtcagct ctcccctatc gtcgtagcgt cccaacatgg ctgaagctca 180
ctcctgacga cgtcaaggat cagattttca aactcaccaa gaaaggactg actccatctc 240
agatcggtgt catcctcagg gattctcacg gtgtggctca agtcagattc gtcaccgggt 300
cgaagatcct caggatcatg aaagccatcg gcctcgctcc tgacctccca gaggacctct 360
acttcctcat caaaaaagcc gttgctatca ggaaacatct tgaaagaaat aggaaagaca 420
aagactctaa attcggactt atccccgtcg agtccaggat ccacaggttg gcaagatact 480
acaaaaccaa gggcaccctt ccacccacct ggaaatacga gtccagcacc gcctctgctc 540
tggtggcttg aatattcaac tttttatttg tctactgttt aattaatata atgtgattta 600
gcaaaaaaaa aaaaaaaaaa aaaaaaaaaa aa 632
<210> 20
<211> 457
<212> ДНК
<213> Lygus hesperus
<400> 20
gggtcgtatg cacgcacctg gtaagggtat ttcccagtca gctctcccct atcgtcgtag 60
cgtcccaaca tggctgaagc tcactcctga cgacgtcaag gatcagattt tcaaactcac 120
caagaaagga ctgactccat ctcagatcgg tgtcatcctc agggattctc acggtgtggc 180
tcaagtcaga ttcgtcaccg ggtcgaagat cctcaggatc atgaaagcca tcggcctcgc 240
tcctgacctc ccagaggacc tctacttcct catcaaaaaa gccgttgcta tcaggaaaca 300
tcttgaaaga aataggaaag acaaagactc taaattcgga cttatccccg tcgagtccag 360
gatccacagg ttggcaagat actacaaaac caagggcacc cttccaccca cctggaaata 420
cgagtccagc accgcctctg ctctggtggc ttgaata 457
<210> 21
<211> 407
<212> ДНК
<213> Lygus hesperus
<400> 21
atgggaccgt ttgcctcaca atccagaaca gacaggctgc catatccgtc gtcccctctg 60
cagcctccct cgtaatcaag gccctcaaag agcccccgag ggacaggaag aagaacaaga 120
acatcaaaca cgacggtaac ctgagtatgg atgacattct cggaattgcc aaaaccatga 180
ggccgaggtc gatgtccagg aaactggaag gaaccgtcaa ggaaatcctt gggacagctc 240
agtctgtcgg atgcacgatc gaaggccgag ctccccacga cgtcatcgac tccatcaaca 300
acggcgaaat ggaaatccct gacgaataaa ctgttcatga gtttatggat tttatataaa 360
aaataaaaag ttgaaaaatc caaaaaaaaa aaaaaaaaag aaaaaaa 407
<210> 22
<211> 302
<212> ДНК
<213> Lygus hesperus
<400> 22
accgtttgcc tcacaatcca gaacagacag gctgccatat ccgtcgtccc ctctgcagcc 60
tccctcgtaa tcaaggccct caaagagccc ccgagggaca ggaagaagaa caagaacatc 120
aaacacgacg gtaacctgag tatggatgac attctcggaa ttgccaaaac catgaggccg 180
aggtcgatgt ccaggaaact ggaaggaacc gtcaaggaaa tccttgggac agctcagtct 240
gtcggatgca cgatcgaagg ccgagctccc cacgacgtca tcgactccat caacaacggc 300
ga 302
<210> 23
<211> 794
<212> ДНК
<213> Lygus hesperus
<400> 23
catggggagt caatttggat ctatcgccag atgaagatgt ctcctgccgt gttcgctgtt 60
ctgctggtac tttcagcttc ccaggtcttg ggagatgatg catccaagtt ccaacacgag 120
gaaatcatgg aagtcctcag ctcggtcaac aaaaccgtca acaaattgta cgacttgatg 180
tccacgcaga aggaaagaga tattgacttt atcgagaaga aaatggatga gacgtaccag 240
caactcagga acaagaggga ggcgccggct gagaaccctg aagccattga caagatccaa 300
aacgcgttca aaagctttca agacggcgtc aaggacttcg tcaagtccgc ttcttcctcg 360
gacctctaca agaaggttca ggaaatcggc gaggacctgt agaacaaagg caaagagctc 420
ggagagaagc tgcaagaaac catcaataac gccagaacga aaaactcaga cgagaagaag 480
gactaaactg aggattttga ctctgcacaa acgcccgttg gtgtttaaac gtatttctta 540
cgtttattat catcggggtt catgaaatca aaaatacacc atcgcatacc acctcgaaaa 600
gaacataata tatgtgaaaa gacaagaaaa ggtgttcaat tgtgtcttta actggtggtt 660
atcacgattc acatgaaata ctactaagaa aacccaaaaa ccgtcatgaa acccgaagta 720
tgcttctgta ttacctaatt gtgctgataa ttcttaataa aatattatac tgagaaaaaa 780
aaaaaaaaaa aaaa 794
<210> 24
<211> 278
<212> ДНК
<213> Lygus hesperus
<400> 24
ttctgctggt actttcagct tcccaggtct tgggagatga tgcatccaag ttccaacacg 60
aggaaatcat ggaagtcctc agctcggtca acaaaaccgt caacaaattg tacgacttga 120
tgtccacgca gaaggaaaga gatattgact ttatcgagaa gaaaatggat gagacgtacc 180
agcaactcag gaacaagagg gaggcgccgg ctgagaaccc tgaagccatt gacaagatcc 240
aaaacgcgtt caaaagcttt caagacggcg tcaaggac 278
<210> 25
<211> 437
<212> ДНК
<213> Lygus hesperus
<400> 25
atgggatcca ataataacca ttaaggcaat tggacatcaa tgatactgaa catatgaata 60
ttcagatatc aaaaatatcg aaatagaatc atatataaaa ccaactaacg cattagaaaa 120
taacgaattc cgattacttg aagtagacaa tcgaatcgta ttacctataa aatcaactat 180
ccgaattcta gttacatcat ctgatgtaat tcattcatga accatcccaa gtttgggaat 240
caaaattgat ggcacaccag gacgattaaa tcaagggaga ataaacataa accgaccagg 300
actaatatat gggcaatgtt ctgaaatttg tggagcaaac cacagattta taccaatcgt 360
aattgaaaga gtttcaatta atcaatttat aaactgatta aattcaaaat aaaaaaaaaa 420
aaaaaaaaaa aaaaaaa 437
<210> 26
<211> 327
<212> ДНК
<213> Lygus hesperus
<400> 26
aacgcagagt acatgggatc caataataac cattaaggca attggacatc aatgatactg 60
aacatatgaa tattcagata tcaaaaatat cgaaatagaa tcatatataa aaccaactaa 120
cgcattagaa aataacgaat tccgattact tgaagtagac aatcgaatcg tattacctat 180
aaaatcaact atccgaattc tagttacatc atctgatgta attcattcat gaaccatccc 240
aagtttggga atcaaaattg atggcacacc aggacgatta aatcaaggga gaataaacat 300
aaaccgacca ggactaatat atgggca 327
<210> 27
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 27
gcgtaatacg actcactata ggcaaacagg ccgaaatcga ga 42
<210> 28
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 28
tgacgacctt gagttggttt ctg 23
<210> 29
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 29
caaacaggcc gaaatcgaga 20
<210> 30
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 30
gcgtaatacg actcactata ggtgacgacc ttgagttggt ttctg 45
<210> 31
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 31
gcgtaatacg actcactata gggggcatca tgtcgaaagc tg 42
<210> 32
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 32
cgaagcccat gtcatcatcg 20
<210> 33
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 33
gggcatcatg tcgaaagctg 20
<210> 34
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 34
gcgtaatacg actcactata ggcgaagccc atgtcatcat cg 42
<210> 35
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 35
gcgtaatacg actcactata gggggcaggt cttctccata acca 44
<210> 36
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 36
gattttgcgg tgccaacgac 20
<210> 37
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 37
gggcaggtct tctccataac ca 22
<210> 38
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 38
gcgtaatacg actcactata gggattttgc ggtgccaacg ac 42
<210> 39
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 39
gcgtaatacg actcactata ggattgcaca agctgaatcc ctcc 44
<210> 40
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 40
attcacaaag cgggtgctgg 20
<210> 41
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 41
attgcacaag ctgaatccct cc 22
<210> 42
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 42
gcgtaatacg actcactata ggattcacaa agcgggtgct gg 42
<210> 43
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 43
gcgtaatacg actcactata ggccctctac gagtgcatca atgg 44
<210> 44
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 44
cccatcgtgg atttgacgtg 20
<210> 45
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 45
ccctctacga gtgcatcaat gg 22
<210> 46
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 46
gcgtaatacg actcactata ggcccatcgt ggatttgacg tg 42
<210> 47
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 47
gcgtaatacg actcactata ggccaataaa gatcaacttt cccagag 47
<210> 48
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 48
ttagacagac tcttgaactg gaggc 25
<210> 49
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 49
ccaataaaga tcaactttcc cagag 25
<210> 50
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 50
gcgtaatacg actcactata ggttagacag actcttgaac tggaggc 47
<210> 51
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 51
gcgtaatacg actcactata gggaaaggtg atgttcgcca tgac 44
<210> 52
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 52
ttgaccacgc accctcaaac 20
<210> 53
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 53
gaaaggtgat gttcgccatg ac 22
<210> 54
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 54
gcgtaatacg actcactata ggttgaccac gcaccctcaa ac 42
<210> 55
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 55
gcgtaatacg actcactata ggcttccctg aaattatttc gttgaag 47
<210> 56
<211> 28
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 56
caccaagaat aataatgttg atttctcc 28
<210> 57
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 57
cttccctgaa attatttcgt tgaag 25
<210> 58
<211> 50
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 58
gcgtaatacg actcactata ggcaccaaga ataataatgt tgatttctcc 50
<210> 59
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 59
gcgtaatacg actcactata gggttcaaga gagttaaagc caagagg 47
<210> 60
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 60
catacggtgg ggatattgac tg 22
<210> 61
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 61
gttcaagaga gttaaagcca agagg 25
<210> 62
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 62
gcgtaatacg actcactata ggcatacggt ggggatattg actg 44
<210> 63
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 63
gcgtaatacg actcactata gggggtcgta tgcacgcacc tg 42
<210> 64
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 64
tattcaagcc accagagcag agg 23
<210> 65
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 65
gggtcgtatg cacgcacctg 20
<210> 66
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 66
gcgtaatacg actcactata ggtattcaag ccaccagagc agagg 45
<210> 67
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 67
gcgtaatacg actcactata ggaccgtttg cctcacaatc ca 42
<210> 68
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 68
tcgccgttgt tgatggagtc 20
<210> 69
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 69
accgtttgcc tcacaatcca 20
<210> 70
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 70
gcgtaatacg actcactata ggtcgccgtt gttgatggag tc 42
<210> 71
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 71
gcgtaatacg actcactata ggggtatcaa cgcagagtac atggg 45
<210> 72
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 72
gtccttgacg ccgtcttgaa 20
<210> 73
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 73
ggtatcaacg cagagtacat ggg 23
<210> 74
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 74
gcgtaatacg actcactata gggtccttga cgccgtcttg aa 42
<210> 75
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 75
gcgtaatacg actcactata ggtgcccata tattagtcct ggtc 44
<210> 76
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 76
aacgcagagt acatgggatc 20
<210> 77
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 77
tgcccatata ttagtcctgg tc 22
<210> 78
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 78
gcgtaatacg actcactata ggaacgcaga gtacatggga tc 42
<210> 79
<211> 197
<212> БЕЛОК
<213> Lygus hesperus
<400> 79
Met Ala Asp Asp Glu Ala Lys Lys Ala Lys Gln Ala Glu Ile Glu Arg
1 5 10 15
Lys Arg Ala Glu Val Arg Lys Arg Met Glu Glu Ala Ser Lys Ala Lys
20 25 30
Lys Ala Lys Lys Gly Phe Met Thr Pro Glu Arg Lys Lys Lys Leu Arg
35 40 45
Leu Leu Leu Arg Lys Lys Ala Ala Glu Glu Leu Lys Lys Glu Gln Glu
50 55 60
Arg Lys Ala Ala Glu Arg Arg Arg Thr Ile Glu Glu Arg Cys Gly Gln
65 70 75 80
Ile Ala Asp Val Asp Asn Ala Asn Glu Ala Thr Leu Lys Lys Leu Cys
85 90 95
Thr Asp Tyr His Lys Arg Ile Asp Ala Leu Glu Arg Ser Lys Ile Asp
100 105 110
Ile Glu Phe Glu Val Glu Arg Arg Asp Leu Glu Ile Ala Asp Leu Asn
115 120 125
Ser Gln Val Asn Asp Leu Arg Gly Lys Phe Val Lys Pro Thr Leu Lys
130 135 140
Lys Val Ser Lys Tyr Glu Asn Lys Phe Ala Lys Leu Gln Lys Lys Ala
145 150 155 160
Ala Glu Phe Asn Phe Arg Asn Gln Leu Lys Val Val Lys Lys Lys Glu
165 170 175
Phe Thr Leu Glu Glu Glu Asp Lys Glu Pro Lys Lys Ser Glu Lys Ala
180 185 190
Glu Trp Gln Lys Lys
195
<210> 80
<211> 115
<212> БЕЛОК
<213> Lygus hesperus
<400> 80
Met Gly Ile Met Ser Lys Ala Glu Leu Ala Cys Val Tyr Ser Ala Leu
1 5 10 15
Ile Leu Ile Asp Asp Asp Val Ala Val Thr Gly Glu Lys Ile Gln Thr
20 25 30
Ile Leu Lys Ala Ala Ser Val Asp Ile Glu Pro Tyr Trp Pro Gly Leu
35 40 45
Phe Ala Lys Ala Leu Glu Gly Ile Asn Pro Lys Asp Leu Ile Ser Ser
50 55 60
Ile Gly Ser Gly Val Gly Ala Gly Ala Pro Ala Val Gly Gly Ala Ala
65 70 75 80
Pro Ala Ala Ala Ala Ala Pro Ala Ala Glu Ala Lys Lys Glu Glu Lys
85 90 95
Lys Lys Val Glu Ser Asp Pro Glu Ser Asp Asp Asp Met Gly Phe Gly
100 105 110
Leu Phe Asp
115
<210> 81
<211> 121
<212> БЕЛОК
<213> Lygus hesperus
<400> 81
Met Ile Pro Pro Thr Ser Arg Pro Gln Val Thr Val Tyr Ser Asp Lys
1 5 10 15
Asn Glu Ala Thr Gly Thr Leu Leu Asn Leu Pro Ala Val Phe Asn Ala
20 25 30
Pro Ile Arg Pro Asp Val Val Asn Phe Val His Gln Asn Val Ala Lys
35 40 45
Asn His Arg Gln Pro Tyr Cys Val Ser Ala Gln Ala Gly His Gln Thr
50 55 60
Ser Ala Glu Ser Trp Gly Thr Gly Arg Ala Val Ala Arg Ile Pro Arg
65 70 75 80
Val Arg Gly Gly Gly Thr His Arg Ser Gly Gln Gly Ala Phe Gly Asn
85 90 95
Met Cys Arg Gly Gly Arg Met Phe Ala Pro Thr Arg Pro Trp Arg Arg
100 105 110
Trp His Arg Lys Ile Asn Val Asn Gln
115 120
<210> 82
<211> 133
<212> БЕЛОК
<213> Lygus hesperus
<400> 82
Trp Asp Leu Tyr Ala Glu Lys Val Ala Thr Arg Gly Leu Cys Ala Ile
1 5 10 15
Ala Gln Ala Glu Ser Leu Arg Tyr Lys Leu Ile Gly Gly Leu Ala Val
20 25 30
Arg Gly Ala Cys Tyr Gly Val Leu Arg Phe Ile Met Glu Asn Gly Ala
35 40 45
Lys Gly Cys Glu Val Val Val Ser Gly Lys Leu Arg Gly Gln Arg Ala
50 55 60
Lys Ser Met Lys Phe Val Asp Gly Leu Met Ile His Ser Gly Asp Pro
65 70 75 80
Cys Asn Glu Tyr Val Asp Thr Ala Thr Arg His Val Leu Leu Arg Gln
85 90 95
Gly Val Leu Gly Ile Lys Val Lys Ile Met Leu Pro Trp Asp Val Thr
100 105 110
Gly Lys Asn Gly Pro Lys Asn Pro Leu Pro Asp His Val Ser Val Leu
115 120 125
Leu Pro Lys Glu Glu
130
<210> 83
<211> 217
<212> БЕЛОК
<213> Lygus hesperus
<400> 83
Met Thr Ser Lys Val Ser Arg Glu Thr Leu Tyr Glu Cys Ile Asn Gly
1 5 10 15
Val Ile Gln Ser Ser Gln Glu Lys Lys Arg Asn Phe Val Glu Thr Val
20 25 30
Glu Ile Gln Ile Gly Leu Lys Asn Tyr Asp Pro Gln Lys Asp Lys Arg
35 40 45
Phe Ser Gly Thr Val Lys Leu Lys His Ile Pro Arg Pro Lys Met Gln
50 55 60
Val Cys Ile Leu Gly Asp Gln Gln His Cys Asp Glu Ala Lys Ala Asn
65 70 75 80
Asn Val Pro Tyr Met Asp Val Glu Ala Leu Lys Lys Leu Asn Lys Asn
85 90 95
Lys Lys Leu Val Lys Lys Leu Ala Lys Lys Tyr Asp Ala Phe Leu Ala
100 105 110
Ser Glu Ala Leu Ile Lys Gln Ile Pro Arg Leu Leu Gly Pro Gly Leu
115 120 125
Asn Lys Ala Gly Lys Phe Pro Gly Leu Leu Ser His Gln Glu Ser Met
130 135 140
Met Met Lys Ile Asp Glu Val Lys Ala Thr Ile Lys Phe Gln Met Lys
145 150 155 160
Lys Val Leu Cys Leu Ser Val Ala Val Gly His Val Gly Met Thr Ala
165 170 175
Asp Glu Leu Val Gln Asn Val His Leu Ser Val Asn Phe Leu Val Ser
180 185 190
Leu Leu Lys Lys His Trp Gln Asn Val Arg Ser Leu His Val Lys Ser
195 200 205
Thr Met Gly Pro Pro Gln Arg Leu Tyr
210 215
<210> 84
<211> 118
<212> БЕЛОК
<213> Lygus hesperus
<400> 84
Gly Thr Asn Lys Asp Gln Leu Ser Gln Arg Lys Thr Cys Tyr Ala Gln
1 5 10 15
His Asn Gln Val Arg Glu Ile Arg Lys Lys Met Val Lys Asn Ile Ser
20 25 30
Asp Ser Ile Ser Ser Cys Asp Leu Arg Ser Val Val Asn Lys Leu Ile
35 40 45
Pro Asp Ser Ile Ala Lys Asp Ile Glu Lys Asn Cys Gln Gly Ile Tyr
50 55 60
Pro Leu His Asp Val Tyr Ile Arg Lys Val Lys Val Leu Lys Lys Pro
65 70 75 80
Arg Phe Glu Leu Ser Lys Leu Leu Glu Leu His Val Asp Gly Lys Gly
85 90 95
Ile Asp Glu Pro Gly Ala Lys Val Thr Arg Thr Asp Ala Tyr Glu Pro
100 105 110
Pro Val Gln Glu Ser Val
115
<210> 85
<211> 128
<212> БЕЛОК
<213> Lygus hesperus
<400> 85
Lys Val Met Phe Ala Met Thr Ala Ile Lys Gly Val Gly Arg Arg Tyr
1 5 10 15
Ala Asn Ile Val Leu Lys Lys Ala Asp Val Asn Leu Asp Lys Arg Ala
20 25 30
Gly Glu Cys Ser Glu Glu Glu Val Glu Lys Ile Val Thr Ile Met Gln
35 40 45
Asn Pro Arg Gln Tyr Lys Ile Pro Asn Trp Phe Leu Asn Arg Gln Lys
50 55 60
Asp Thr Val Glu Gly Lys Tyr Ser Gln Leu Thr Ser Ser Leu Leu Asp
65 70 75 80
Ser Lys Leu Arg Asp Asp Leu Glu Arg Leu Lys Lys Ile Arg Ala His
85 90 95
Arg Gly Met Arg His Tyr Trp Gly Leu Arg Val Arg Gly Gln His Thr
100 105 110
Lys Thr Thr Gly Arg Arg Gly Arg Thr Val Gly Val Ser Lys Lys Lys
115 120 125
<210> 86
<211> 122
<212> БЕЛОК
<213> Lygus hesperus
<400> 86
Val Leu Leu Leu Ile Ser Phe Ser Phe Pro Glu Ile Ile Ser Leu Lys
1 5 10 15
Leu Ile Trp Ile Thr Leu Lys Glu Ser Ala Ala Phe Ser Leu Ala Lys
20 25 30
Asn Leu Leu His Pro Ser Pro Arg Pro Pro Val Gly Arg His Lys Leu
35 40 45
Lys His Leu Pro Val His Pro Asn Ser His Phe Met Asp Val Asn Cys
50 55 60
Pro Gly Cys Tyr Lys Ile Pro Thr Val Phe Ser Pro Ala Gln Asn Asp
65 70 75 80
Phe Gly Cys Trp Thr Cys Ser Thr Ile Leu Cys Leu Pro Thr Gly Gly
85 90 95
Arg Ala Asp Leu Thr Lys Arg Cys Ser Phe Arg Arg Asn Gln His Tyr
100 105 110
Tyr Ser Trp Trp Glu His Leu Phe Phe Leu
115 120
<210> 87
<211> 77
<212> БЕЛОК
<213> Lygus hesperus
<400> 87
Gly Phe Lys Arg Val Lys Ala Lys Arg Ala Lys Lys Asp Asp Gly Glu
1 5 10 15
Ile Phe Ala Ala Lys Lys Glu Val Tyr Lys Pro Ser Glu Gln Arg Lys
20 25 30
Ala Asp Gln Lys Asn Ile Asp Lys Gln Thr Leu Lys Ala Ile Lys Arg
35 40 45
Leu Lys Gly Asp Ala Cys Leu Met Arg Lys Tyr Leu Cys Thr Met Phe
50 55 60
Gly Phe Arg Ser Ser Gln Tyr Pro His Arg Met Lys Phe
65 70 75
<210> 88
<211> 151
<212> БЕЛОК
<213> Lygus hesperus
<400> 88
Met Gly Arg Met His Ala Pro Gly Lys Gly Ile Ser Gln Ser Ala Leu
1 5 10 15
Pro Tyr Arg Arg Ser Val Pro Thr Trp Leu Lys Leu Thr Pro Asp Asp
20 25 30
Val Lys Asp Gln Ile Phe Lys Leu Thr Lys Lys Gly Leu Thr Pro Ser
35 40 45
Gln Ile Gly Val Ile Leu Arg Asp Ser His Gly Val Ala Gln Val Arg
50 55 60
Phe Val Thr Gly Ser Lys Ile Leu Arg Ile Met Lys Ala Ile Gly Leu
65 70 75 80
Ala Pro Asp Leu Pro Glu Asp Leu Tyr Phe Leu Ile Lys Lys Ala Val
85 90 95
Ala Ile Arg Lys His Leu Glu Arg Asn Arg Lys Asp Lys Asp Ser Lys
100 105 110
Phe Gly Leu Ile Pro Val Glu Ser Arg Ile His Arg Leu Ala Arg Tyr
115 120 125
Tyr Lys Thr Lys Gly Thr Leu Pro Pro Thr Trp Lys Tyr Glu Ser Ser
130 135 140
Thr Ala Ser Ala Leu Val Ala
145 150
<210> 89
<211> 108
<212> БЕЛОК
<213> Lygus hesperus
<400> 89
Gly Thr Val Cys Leu Thr Ile Gln Asn Arg Gln Ala Ala Ile Ser Val
1 5 10 15
Val Pro Ser Ala Ala Ser Leu Val Ile Lys Ala Leu Lys Glu Pro Pro
20 25 30
Arg Asp Arg Lys Lys Asn Lys Asn Ile Lys His Asp Gly Asn Leu Ser
35 40 45
Met Asp Asp Ile Leu Gly Ile Ala Lys Thr Met Arg Pro Arg Ser Met
50 55 60
Ser Arg Lys Leu Glu Gly Thr Val Lys Glu Ile Leu Gly Thr Ala Gln
65 70 75 80
Ser Val Gly Cys Thr Ile Glu Gly Arg Ala Pro His Asp Val Ile Asp
85 90 95
Ser Ile Asn Asn Gly Glu Met Glu Ile Pro Asp Glu
100 105
<210> 90
<211> 133
<212> БЕЛОК
<213> Lygus hesperus
<400> 90
His Gly Glu Ser Ile Trp Ile Tyr Arg Gln Met Lys Met Ser Pro Ala
1 5 10 15
Val Phe Ala Val Leu Leu Val Leu Ser Ala Ser Gln Val Leu Gly Asp
20 25 30
Asp Ala Ser Lys Phe Gln His Glu Glu Ile Met Glu Val Leu Ser Ser
35 40 45
Val Asn Lys Thr Val Asn Lys Leu Tyr Asp Leu Met Ser Thr Gln Lys
50 55 60
Glu Arg Asp Ile Asp Phe Ile Glu Lys Lys Met Asp Glu Thr Tyr Gln
65 70 75 80
Gln Leu Arg Asn Lys Arg Glu Ala Pro Ala Glu Asn Pro Glu Ala Ile
85 90 95
Asp Lys Ile Gln Asn Ala Phe Lys Ser Phe Gln Asp Gly Val Lys Asp
100 105 110
Phe Val Lys Ser Ala Ser Ser Ser Asp Leu Tyr Lys Lys Val Gln Glu
115 120 125
Ile Gly Glu Asp Leu
130
<210> 91
<211> 118
<212> БЕЛОК
<213> Lygus hesperus
<400> 91
Thr Tyr Glu Tyr Ser Asp Ile Lys Asn Ile Glu Ile Glu Ser Tyr Ile
1 5 10 15
Lys Pro Thr Asn Ala Leu Glu Asn Asn Glu Phe Arg Leu Leu Glu Val
20 25 30
Asp Asn Arg Ile Val Leu Pro Ile Lys Ser Thr Ile Arg Ile Leu Val
35 40 45
Thr Ser Ser Asp Val Ile His Ser Thr Ile Pro Ser Leu Gly Ile Lys
50 55 60
Ile Asp Gly Thr Pro Gly Arg Leu Asn Gln Gly Arg Ile Asn Ile Asn
65 70 75 80
Arg Pro Gly Leu Ile Tyr Gly Gln Cys Ser Glu Ile Cys Gly Ala Asn
85 90 95
His Arg Phe Ile Pro Ile Val Ile Glu Arg Val Ser Ile Asn Gln Phe
100 105 110
Ile Asn Leu Asn Ser Lys
115
<210> 92
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Адаптер
<400> 92
aagcagtggt atcaacgcag 20
<210> 93
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 93
aagcagtggt atcaacgcag 20
<210> 94
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 94
gcgtaatacg actcactata ggaagcagtg gtatcaacgc ag 42
<210> 95
<211> 717
<212> ДНК
<213> Lygus hesperus
<400> 95
aaagtgtggt tctcttcgtc cgaccatgag ttcgctcaaa ctgcagaaga ggctcgccgc 60
ctcggtgatg agatgcggca agaagaaagt gtggttggac cctaatgaaa tcaacgaaat 120
cgccaacacc aactctaggc aaaacatccg taagctgatc aaggatggtt tgatcatcaa 180
aaagcctgtg gctgtccact ccagagcccg cgtccgtaaa aacacagaag ccagacggaa 240
gggtcgtcat tgtggcttcg gtaagaggaa gggtaccgcc aacgccagaa tgcctgtgaa 300
ggtcctgtgg gtcaacagaa tgagagtcct gcgacggctc cttaaaaaat acagagaagc 360
caagaagatc gataggcaaa tgtaccacga cctttacatg aaagccaaag gtaacgtctt 420
caaaaacaag agggtactga tggacttcat tcacaagaag aaggctgaaa aggcgagatc 480
aaagatgttg aaggaccagg cagaggcgag acgtttcaag gtcaaggagg cgaagaagag 540
gcgcgaggag aggatcgcca ccaagaagca agagatcatg caggcgtacg cccgagaaga 600
cgaggctgcc gtcaaaaagt gatctcgccc cctccgtttt taaattttaa acaaaaaacg 660
tattttgtac aaaaatttac aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aaaaaaa 717
<210> 96
<211> 2304
<212> ДНК
<213> Lygus hesperus
<400> 96
atgacgacct acgaggagtt cattcaacag agcgaggagc gcgacggtat caggttcact 60
tggaacgtct ggccatcaag tcgcatcgaa gccaccaggt tggtcgtacc cgtaggatgt 120
ctctatcaac cactaaaaga acgcacggat cttccagcta ttcaatacga tcccgttcta 180
tgcactagga atacctgtag agccatactc aacccgatgt gccaagtaaa ctatagggca 240
aagttgtggg tgtgtaactt ctgtttccag aggaatccgt tcccaccaca atacgccgca 300
atttccgagc agcatcagcc tgctgagttg attccatcat tctcaactat agagtatact 360
atatctagag ctcaattttt gcctcctata ttcctattgg tggtggatac gtgtttggat 420
gatgacgagc taggagctct gaaagattcg ttacaaacgt ctctatcttt gctaccaacc 480
aactccctag ttggtctgat cacgtttggt aaaatggtcc aagttcacga acttgggtgt 540
gaaggttgtt cccggagcta cgtgttcaga ggcaccaagg atttgacgtc caagcaagta 600
caggacatgc ttgggatcgg aaaggtttcc gcttctcctc agcaacagca gcaaagggca 660
atgggcggtc agcagccatt ccccaccaat cggttcattc agccgattca aagttgtgac 720
atgagcctca ccgacttgtt gggcgaaatg cagcgtgatc catggccagt gggtcagggt 780
aagcgacctc ttagatcaac gggtgctgct ctagctattg ccattgggtt gttggagtgc 840
tcctacccca acacgggagc aaaagtcatg ttgttccttg gtggcccttg ttcccaaggg 900
cctggtcaag ttgtcaatga tgacctgagg gaacctatcc gctctcatca tgacatccag 960
aaagataatg cccgctacat gaaaaaagcc attaaacatt acgattcttt ggcattgaga 1020
gcagccacta atgggcattc agtagacatt tattcctgtg ctttagatca gacaggtttg 1080
gcggaaatga agcaatgttg caattctact gggggtcata tggtgatggg tgacaccttc 1140
aactccactt tgttcaaaca gacgttccag agggtgctct cccgtgatca aaaaggcgaa 1200
ttcaaaatgg ctttcaatgg cgtagttgaa gtcaaaacct cccgagagct aaaagttatg 1260
ggagccattg ggccttgcgt ttcattgaat acgaaaggtc cgtgtgttag tgaaactgac 1320
atagggcttg gaggaacttg ccagtggaag ttctgcacat ttaaccaaaa taccactgct 1380
gccatgttct ttgaggtagt aaaccaacac gctgctccta tccctcaagg tggaagagga 1440
tgtatacagt tcataactca ataccagcat gcgtcgggcc aaaggcgcat ccgagtaacc 1500
actgtagcca ggaattgggc tgatgcgact accaacatgc accatgttag tgcaggattt 1560
gatcaggaag ctggagcggt actcatggcc aggatggtcg ttcacagagc tgaaactgat 1620
gatggacctg atgtcatgag atgggctgat cgcatgttga ttcgtctttg ccagaaattc 1680
ggcgagtaca acaaggatga tccaaatagt ttccgcctcc cagaaaactt ctcgctttac 1740
ccacagttca tgtatcactt gagaaggtcc caattcttgc aggtattcaa caacagccca 1800
gacgaaacgt cgtactatcg tcacatcttg atgcgggaag atttgtcgca gagcttgatc 1860
atgattcagc cgatcctgta cagttacagt ttcaacggtc cagaaccagt ccttttggac 1920
acttccagca ttcaacctga tcggatcctg ctgatggaca ccttcttcca aatcctcatc 1980
ttccacggcg agaccatcgc ccagtggcgt gcccaaaggt accaggacct acctgaatat 2040
gagaacttca agcagctcct acaggctcct gtagacgatg ctaaggaaat cctgcacact 2100
cggttcccca tgccgaggta cattgacacc gaacagggcg gatcacaagc tagattcctt 2160
ctctccaaag tcaacccatc ccaaactcac aacaacatgt acggctatgg aggggaattt 2220
ggagcccctg tgctcactga tgatgtttcc ctccaagtct tcatggaaca ccttaaaaag 2280
ctagccgttt catttactgc ctag 2304
<210> 97
<211> 311
<212> ДНК
<213> Искусственная
<220>
<223> GUS
<400> 97
ccagcgtatc gtgctgcgtt tcgatgcggt cactcattac ggcaaagtgt gatggagcat 60
cagggcggct atacgccatt tgaagccgat gtcacgccgt atgttattgc cgggaaaagt 120
gtacgtatct gaaatcaaaa aactcgacgg cctgtgggca ttcagtctgg atcgcgaaaa 180
ctgtggaatt gatccagcgc cgtcgtcggt gaacaggtat ggaatttcgc cgattttgcg 240
acctcgcaag gcatattcgg gtgaaggtta tctctatgaa ctgtgcgtca cagccaaaag 300
ccagacagag t 311
<210> 98
<211> 170
<212> ДНК
<213> Искусственная
<220>
<223> Интрон
<400> 98
ctcgagcctg agagaaaagc atgaagtata cccataacta acccattagt tatgcattta 60
tgttatatct attcatgctt ctactttaga taatcaatca ccaaacaatg agaatctcaa 120
cggtcgcaat aatgttcatg aaaatgtagt gtgtacactt accttctaga 170
<210> 99
<211> 198
<212> БЕЛОК
<213> Lygus hesperus
<400> 99
Met Ser Ser Leu Lys Leu Gln Lys Arg Leu Ala Ala Ser Val Met Arg
1 5 10 15
Cys Gly Lys Lys Lys Val Trp Leu Asp Pro Asn Glu Ile Asn Glu Ile
20 25 30
Ala Asn Thr Asn Ser Arg Gln Asn Ile Arg Lys Leu Ile Lys Asp Gly
35 40 45
Leu Ile Ile Lys Lys Pro Val Ala Val His Ser Arg Ala Arg Val Arg
50 55 60
Lys Asn Thr Glu Ala Arg Arg Lys Gly Arg His Cys Gly Phe Gly Lys
65 70 75 80
Arg Lys Gly Thr Ala Asn Ala Arg Met Pro Val Lys Val Leu Trp Val
85 90 95
Asn Arg Met Arg Val Leu Arg Arg Leu Leu Lys Lys Tyr Arg Glu Ala
100 105 110
Lys Lys Ile Asp Arg Gln Met Tyr His Asp Leu Tyr Met Lys Ala Lys
115 120 125
Gly Asn Val Phe Lys Asn Lys Arg Val Leu Met Asp Phe Ile His Lys
130 135 140
Lys Lys Ala Glu Lys Ala Arg Ser Lys Met Leu Lys Asp Gln Ala Glu
145 150 155 160
Ala Arg Arg Phe Lys Val Lys Glu Ala Lys Lys Arg Arg Glu Glu Arg
165 170 175
Ile Ala Thr Lys Lys Gln Glu Ile Met Gln Ala Tyr Ala Arg Glu Asp
180 185 190
Glu Ala Ala Val Lys Lys
195
<210> 100
<211> 767
<212> БЕЛОК
<213> Lygus hesperus
<400> 100
Met Thr Thr Tyr Glu Glu Phe Ile Gln Gln Ser Glu Glu Arg Asp Gly
1 5 10 15
Ile Arg Phe Thr Trp Asn Val Trp Pro Ser Ser Arg Ile Glu Ala Thr
20 25 30
Arg Leu Val Val Pro Val Gly Cys Leu Tyr Gln Pro Leu Lys Glu Arg
35 40 45
Thr Asp Leu Pro Ala Ile Gln Tyr Asp Pro Val Leu Cys Thr Arg Asn
50 55 60
Thr Cys Arg Ala Ile Leu Asn Pro Met Cys Gln Val Asn Tyr Arg Ala
65 70 75 80
Lys Leu Trp Val Cys Asn Phe Cys Phe Gln Arg Asn Pro Phe Pro Pro
85 90 95
Gln Tyr Ala Ala Ile Ser Glu Gln His Gln Pro Ala Glu Leu Ile Pro
100 105 110
Ser Phe Ser Thr Ile Glu Tyr Thr Ile Ser Arg Ala Gln Phe Leu Pro
115 120 125
Pro Ile Phe Leu Leu Val Val Asp Thr Cys Leu Asp Asp Asp Glu Leu
130 135 140
Gly Ala Leu Lys Asp Ser Leu Gln Thr Ser Leu Ser Leu Leu Pro Thr
145 150 155 160
Asn Ser Leu Val Gly Leu Ile Thr Phe Gly Lys Met Val Gln Val His
165 170 175
Glu Leu Gly Cys Glu Gly Cys Ser Arg Ser Tyr Val Phe Arg Gly Thr
180 185 190
Lys Asp Leu Thr Ser Lys Gln Val Gln Asp Met Leu Gly Ile Gly Lys
195 200 205
Val Ser Ala Ser Pro Gln Gln Gln Gln Gln Arg Ala Met Gly Gly Gln
210 215 220
Gln Pro Phe Pro Thr Asn Arg Phe Ile Gln Pro Ile Gln Ser Cys Asp
225 230 235 240
Met Ser Leu Thr Asp Leu Leu Gly Glu Met Gln Arg Asp Pro Trp Pro
245 250 255
Val Gly Gln Gly Lys Arg Pro Leu Arg Ser Thr Gly Ala Ala Leu Ala
260 265 270
Ile Ala Ile Gly Leu Leu Glu Cys Ser Tyr Pro Asn Thr Gly Ala Lys
275 280 285
Val Met Leu Phe Leu Gly Gly Pro Cys Ser Gln Gly Pro Gly Gln Val
290 295 300
Val Asn Asp Asp Leu Arg Glu Pro Ile Arg Ser His His Asp Ile Gln
305 310 315 320
Lys Asp Asn Ala Arg Tyr Met Lys Lys Ala Ile Lys His Tyr Asp Ser
325 330 335
Leu Ala Leu Arg Ala Ala Thr Asn Gly His Ser Val Asp Ile Tyr Ser
340 345 350
Cys Ala Leu Asp Gln Thr Gly Leu Ala Glu Met Lys Gln Cys Cys Asn
355 360 365
Ser Thr Gly Gly His Met Val Met Gly Asp Thr Phe Asn Ser Thr Leu
370 375 380
Phe Lys Gln Thr Phe Gln Arg Val Leu Ser Arg Asp Gln Lys Gly Glu
385 390 395 400
Phe Lys Met Ala Phe Asn Gly Val Val Glu Val Lys Thr Ser Arg Glu
405 410 415
Leu Lys Val Met Gly Ala Ile Gly Pro Cys Val Ser Leu Asn Thr Lys
420 425 430
Gly Pro Cys Val Ser Glu Thr Asp Ile Gly Leu Gly Gly Thr Cys Gln
435 440 445
Trp Lys Phe Cys Thr Phe Asn Gln Asn Thr Thr Ala Ala Met Phe Phe
450 455 460
Glu Val Val Asn Gln His Ala Ala Pro Ile Pro Gln Gly Gly Arg Gly
465 470 475 480
Cys Ile Gln Phe Ile Thr Gln Tyr Gln His Ala Ser Gly Gln Arg Arg
485 490 495
Ile Arg Val Thr Thr Val Ala Arg Asn Trp Ala Asp Ala Thr Thr Asn
500 505 510
Met His His Val Ser Ala Gly Phe Asp Gln Glu Ala Gly Ala Val Leu
515 520 525
Met Ala Arg Met Val Val His Arg Ala Glu Thr Asp Asp Gly Pro Asp
530 535 540
Val Met Arg Trp Ala Asp Arg Met Leu Ile Arg Leu Cys Gln Lys Phe
545 550 555 560
Gly Glu Tyr Asn Lys Asp Asp Pro Asn Ser Phe Arg Leu Pro Glu Asn
565 570 575
Phe Ser Leu Tyr Pro Gln Phe Met Tyr His Leu Arg Arg Ser Gln Phe
580 585 590
Leu Gln Val Phe Asn Asn Ser Pro Asp Glu Thr Ser Tyr Tyr Arg His
595 600 605
Ile Leu Met Arg Glu Asp Leu Ser Gln Ser Leu Ile Met Ile Gln Pro
610 615 620
Ile Leu Tyr Ser Tyr Ser Phe Asn Gly Pro Glu Pro Val Leu Leu Asp
625 630 635 640
Thr Ser Ser Ile Gln Pro Asp Arg Ile Leu Leu Met Asp Thr Phe Phe
645 650 655
Gln Ile Leu Ile Phe His Gly Glu Thr Ile Ala Gln Trp Arg Ala Gln
660 665 670
Arg Tyr Gln Asp Leu Pro Glu Tyr Glu Asn Phe Lys Gln Leu Leu Gln
675 680 685
Ala Pro Val Asp Asp Ala Lys Glu Ile Leu His Thr Arg Phe Pro Met
690 695 700
Pro Arg Tyr Ile Asp Thr Glu Gln Gly Gly Ser Gln Ala Arg Phe Leu
705 710 715 720
Leu Ser Lys Val Asn Pro Ser Gln Thr His Asn Asn Met Tyr Gly Tyr
725 730 735
Gly Gly Glu Phe Gly Ala Pro Val Leu Thr Asp Asp Val Ser Leu Gln
740 745 750
Val Phe Met Glu His Leu Lys Lys Leu Ala Val Ser Phe Thr Ala
755 760 765
<210> 101
<211> 511
<212> ДНК
<213> Lygus hesperus
<400> 101
ggtgatgaga tgcggcaaga agaaagtgtg gttggaccct aatgaaatca acgaaatcgc 60
caacaccaac tctaggcaaa acatccgtaa gctgatcaag gatggtttga tcatcaaaaa 120
gcctgtggct gtccactcca gagcccgcgt ccgtaaaaac acagaagcca gacggaaggg 180
tcgtcactgt ggcttcggta agaggaaggg taccgccaac gccagaatgc ctgtgaaggt 240
cctgtgggtc aacagaatga gagtcctgcg acggctcctt aaaaaataca gagaagccaa 300
gaagatcgat aggcaaatgt accacgacct ttacatgaaa gccaaaggta acgtcttcaa 360
aaacaagagg gtactgatgg acttcattca caagaagaag gctgaaaagg cgagatcaaa 420
gatgttgaag gaccaggcag aggcgagacg tctcaaggtc aaggaggcga agaagaggcg 480
cgaggagagg atcgccacca agaagcaaga g 511
<210> 102
<211> 1145
<212> ДНК
<213> Lygus hesperus
<400> 102
tgggttgttg gagtgctcct accccaacac gggagcaaaa gtcatgttgt tccttggtgg 60
cccttgttcc caagggcctg gtcaagttgt caatgatgac ctgagggaac ctatccgctc 120
tcatcatgac atccagaaag ataatgcccg ctacatgaaa aaagccatta aacattacga 180
ttctttggca ttgagagcag ccactaatgg gcattcagta gacatttatt cctgtgcttt 240
agatcagaca ggtttggcgg aaatgaagca atgttgcaat tctactgggg gtcatatggt 300
gatgggtgac accttcaact ccactttgtt caaacagacg ttccagaggg tgctctcccg 360
tgatcaaaaa ggcgaattca aaatggcttt caatggcgta gttgaagtca aaacctcccg 420
agagctaaaa gttatgggag ccattgggcc ttgcgtttca ttgaatacga aaggtccgtg 480
tgttagtgaa actgacatag ggcttggagg aacttgccag tggaagttct gcacatttaa 540
ccaaaatacc actgctgcca tgttctttga ggtagtaaac caacacgctg ctcctatccc 600
tcaaggtgga agaggatgta tacagttcat aactcaatac cagcatgcgt cgggccaaag 660
gcgcatccga gtaaccactg tagccaggaa ttgggctgat gcgactacca acatgcacca 720
tgttagtgca ggatttgatc aggaagctgg agcggtactc atggccagga tggtcgttca 780
cagagctgaa actgatgatg gacctgatgt catgagatgg gctgatcgca tgttgattcg 840
tctttgccag aaattcggcg agtacaacaa ggatgatcca aatagtttcc gcctcccaga 900
aaacttctcg ctttacccac agttcatgta tcacttgaga aggtcccaat tcttgcaggt 960
attcaacaac agcccagacg aaacgtcgta ctatcgtcac atcttgatgc gggaagattt 1020
gtcgcagagc ttgatcatga ttcagccgat cctgtacagt tacagtttca acggtccaga 1080
accagtcctt ttggacactt ccagcattca acctgatcgg atcctgctga tggacacctt 1140
cttcc 1145
<210> 103
<211> 258
<212> ДНК
<213> Искусственная
<220>
<223> GFP
<400> 103
agatacccag atcatatgaa acggcatgac tttttcaaga gtgccatgcc cgaaggttat 60
gtacaggaaa gaactatatt tttcaaagat gacgggaact acaagacacg taagtttaaa 120
cagttcggta ctaactaacc atacatattt aaattttcag gtgctgaagt caagtttgaa 180
ggtgataccc ttgttaatag aatcgagtta aaaggtattg attttaaaga agatggaaac 240
attcttggac acaaattg 258
<210> 104
<211> 745
<212> ДНК
<213> Искусственная
<220>
<223> Флуоресцентный белок коралла Pt
<400> 104
agtgtaataa cttactttga gtctaccgtc atgagtgcaa ttaaaccagt catgaagatt 60
gaattggtca tggaaggaga ggtgaacggg cacaagttca cgatcacggg agagggacaa 120
ggcaagcctt acgagggaac acagactcta aaccttacag tcactaaagg cgtgcccctt 180
cctttcgctt tcgatatctt gtcaacagca ttccagtatg gcaacagggt atttaccaaa 240
tacccagatg atataccgga ctatttcaag cagacctttc cggaaggata ttcgtgggaa 300
agaactttca aatatgaaga gggcgtttgc accacaaaga gtgacataag cctcaagaaa 360
ggccaaccag actgctttca atataaaatt aactttaaag gggagaagct tgaccccaac 420
ggcccaatta tgcagaagaa gaccctgaaa tgggagccat ccactgagag gatgtacatg 480
gacgtggata aagacggtgc aaaggtgctg aagggcgatg ttaatgcggc cctgttgctt 540
gaaggaggtg gccattatcg ttgtgacttt aacagtactt acaaggcgaa gaaaactgtg 600
tccttcccag catatcactt tgtggaccac cgcattgaga ttttgagcca caatacggat 660
tacagcaagg ttacactgta tgaagttgcc gtggctcgca attctcctct tcagattatg 720
gcgccccagt aaaggcttaa cgaaa 745
<210> 105
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 105
gcgtaatacg actcactata ggtgatgaga tgcggcaaga ag 42
<210> 106
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 106
ctcttgcttc ttggtggcga tc 22
<210> 107
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 107
ggtgatgaga tgcggcaaga ag 22
<210> 108
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 108
gcgtaatacg actcactata ggctcttgct tcttggtggc gatc 44
<210> 109
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 109
gcgtaatacg actcactata ggtgggttgt tggagtgctc ctac 44
<210> 110
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 110
ggaagaaggt gtccatcagc ag 22
<210> 111
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 111
tgggttgttg gagtgctcct ac 22
<210> 112
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 112
gcgtaatacg actcactata ggggaagaag gtgtccatca gcag 44
<210> 113
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 113
gcgtaatacg actcactata ggagataccc agatcatatg aaacgg 46
<210> 114
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 114
caatttgtgt ccaagaatgt ttcc 24
<210> 115
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 115
agatacccag atcatatgaa acgg 24
<210> 116
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 116
gcgtaatacg actcactata ggcaatttgt gtccaagaat gtttcc 46
<210> 117
<211> 43
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 117
gcgtaatacg actcactata ggagtgtaat aacttacttt gag 43
<210> 118
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 118
tttcgttaag cctttactgg 20
<210> 119
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 119
agtgtaataa cttactttga g 21
<210> 120
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 120
gcgtaatacg actcactata ggtttcgtta agcctttact gg 42
<210> 121
<211> 473
<212> ДНК
<213> Lygus hesperus
<400> 121
tgccgggccg ctcgccgaac catctgggaa gcttggaatg ggctcgactg ccgaactgat 60
caactttttc ggtccacacc ttttctatca actccttata ccgctccagg atgccgcctt 120
caaacagttt tttcttgtcg tcataagatc tggtgtcaac tcttcgttca tatttggagg 180
cgacttggat tttgggtggg tacttgccgg tgagggcctc agggtccaag ccttttttca 240
aggctttgtg ccgaagttgt tgcttctgtc tttccttcag ttctttaaga tcgtagtctt 300
gcctcttttg cctttcctca agatcgtatt tctcggtctc aagtttgaca atggcttccc 360
agagttcctg agctttgatg cgtagcctgt ctatgctcat attttctatc gccaggggct 420
tgagcctaat gctgagggag atacgtttct cttcctccag ctgctccttg gtc 473
<210> 122
<211> 773
<212> ДНК
<213> Lygus hesperus
<400> 122
gctgctcgcc gtccagttcg ttttcgagtt ccctgacacg ttgttccagc ttggcgatgg 60
ccttcttgcc tcccttgagg gcgttgtttt cggcttcgtc caacctgact tggagttcct 120
tgatttgcgt ttccagagcc ttgcggagct tctcctgggt ctgagcgtgg tcctgttctg 180
ccctgagttc atcagctaac ctagcggcat caaccattgc cttcttggcc ttctcttcgg 240
agttcttggc ttcgttgaga agttcgtcga ggtcagcatg aagtgtctgc aactctccct 300
caagcttgcg tttggcggct gaggcgctgg tagcttgggc agccaactcg ttgatctgtt 360
cgtgggcatc tccaagttct tgttcggctt ggcgcctgcc cctgtcggcc tgttcgagga 420
gagtgcgcga ctcctcgagc tcgtttccga gagcgttggc cctcctttcg gcgattccga 480
gttgttcacg agcatcgtcg cgtgcccttt gttcttcctc aagagcggtc tgtacgtcct 540
tgagttgttg ttggtatttc ttgatggtct tctgggcttc ggcgttagcc ttgttggcgt 600
ggtcgagagc gatttcgagt tcgttgatgt cggcttcaag cttcttcttc atgcgaagag 660
cctcagcctt acccttggct tcagcctcca agctggcttg catggagtcg agtgcccgtt 720
ggtggttctt cctggtgttc tcgaactcct cctccttttc ctggatccgc cgg 773
<210> 123
<211> 771
<212> ДНК
<213> Lygus hesperus
<400> 123
tggacgccat caagaagaaa atgcaggcga tgaagatgga gaaggacacg gccatggaca 60
aggccgacac ctgcgagggg caggccaagg acgctaacac ccgcgccgac aaaatccttg 120
aagatgtgag ggacctccaa aagaaactca accaggtaga aagtgatctc gaaaggacca 180
agagggaact cgagacgaaa accaccgaac tcgaagagaa ggagaaggcc aacaccaacg 240
ctgagagcga ggtcgcctcc ctcaacagga aagtccagat ggttgaagag gacttggaaa 300
gatctgaaga aaggtccggc accgcacaac aaaaactgtc cgaagcctcc cacgccgctg 360
atgaagcctc tcgtatgtgc aaagtattgg agaacaggtc acaacaggat gaggagagga 420
tggaccagct caccaaccag ctgaaagaag cccgactcct cgctgaagac gccgacggca 480
aatcggatga ggtatcaagg aagctggcct tcgttgaaga cgaactggaa gtagctgaag 540
atcgtgtcaa atctggagac tcgaagatca tggagcttga ggaggagttg aaagttgtcg 600
gtaacagctt gaaatctctc gaagtttcag aggagaaggc caaccagcga gtcgaagagt 660
acaaacgtca aatcaagcaa ctgactgtca agttgaagga ggctgaagct cgcgctgagt 720
tcgccgaaaa gacagtcaag aagttgcaga aagaggtgga ccggctggag g 771
<210> 124
<211> 257
<212> ДНК
<213> Lygus hesperus
<400> 124
tgcgggccct ggggcagaat cccacagaat ctgacgtgaa gaagttcacc caccagcaca 60
aaccagatga aagaatcagc ttcgaggtgt ttctcccgat ataccaagcc atatcgaagg 120
gtaggacgtc agacacagct gaagacttca tcgagggtct cagacacttt gacaaagatg 180
gaaatggctt catttcaaca gctgagcttc gccacttgct cacaactttg ggcgaaaaac 240
tgaccgacga cgaggtg 257
<210> 125
<211> 410
<212> ДНК
<213> Lygus hesperus
<400> 125
gccacctcca acgtgtttgc catgttcgat caggctcaga ttcaagaatt caaggaggca 60
ttcaacatga tcgaccagaa cagggacggc ttcgtggata aggaagacct ccatgacatg 120
ctcgcttccc taggtaagaa cccctcagac gagtatctcg aggggatgat gaacgaggcg 180
cctggtccca tcaacttcac aatgttcctc accctcttcg gtgagcggct tcagggaact 240
gatccggagg aggttatcaa gaacgcattt gggtgttttg acgaagacaa caacggattc 300
atcaacgagg aaagactgcg cgagctgctc acctccatgg gggacaggtt cactgatgaa 360
gacgtggacg aaatgtaccg agaggccccc atcaagaacg gcatgttcga 410
<210> 126
<211> 1021
<212> ДНК
<213> Lygus hesperus
<400> 126
tgttcatcct ggagcaggag gagtatcaga gagaaggtat tgaatggaag ttcatcgact 60
tcggacttga tcttcagccg accattgatc tcattgataa gccaatggga gtcatggctc 120
tcctggatga agaatgttgg ttccccaaag ccactgacaa gaccttcgtt gagaagctgg 180
tcggtgctca cagcgttcac cccaaattca tcaaaactga tttccgtgga gtcgccgact 240
ttgctgtcgt ccattatgcc ggaaaagtcg attattcggc ggcgcagtgg ctgatgaaga 300
acatggaccc tctgaacgaa aacgtcgtgc agctcctcca gaactcgcaa gatccgttcg 360
tcatccacat ctggaaggac gcagagatcg tcggcatggc tcaccaagct ctcagcgaca 420
ctcagtttgg agctcgtacc aggaagggta tgttccgaac cgtgtctcaa ctctacaaag 480
accagctgtc caaactcatg atcacacttc gcaacacgaa ccccaacttc gtccgttgca 540
tcctccccaa ccacgagaag agagctggca agatcgatgc tcctttggtg ctggatcagc 600
tcagatgcaa cggtgtgttg gaaggcatca gaatttgcag acaaggtttc ccgaatagaa 660
tcccattcca ggaattccgg caaagatacg agctcttaac tcccaatgtc atccccaaag 720
ggttcatgga cggtaaaaag gcttgcgaga agatgatcaa cgctctcgaa ctggacccta 780
atctctacag agttggtcag tccaagatat tcttcagagc tggagtctta gctcatctag 840
aagaagagcg cgactataag attactgatc tgatagccaa tttccgggct ttctgtaggg 900
gatatcttgc ccgaaggaac taccaaaagc gtcttcagca gctcaacgcc attcgtatta 960
tccagcgaaa ttgctcagct tacttgaagt tgaggaactg gcaatggtgg cggctgtaca 1020
c 1021
<210> 127
<211> 325
<212> ДНК
<213> Lygus hesperus
<400> 127
cggtcatcat ctccatgaac tcgtcgaagt caacagttcc ggaaccgtca gaatcaattt 60
cagcaatcat catgtcaagt tcttgggagg tgattttgtc gtcgagttcc ttcaggattt 120
ccctcaagac gtcagtggta atgtaaccgt tcccttcctt gtcgtagagc ctgaaggcct 180
ccctcagttc ttgctgcatg gcctcagcat cttgtgtctc atcttctgtc aggaaaccgg 240
cagccaaggc tacgaactcc tcaaattcaa gttgtccaga gccatcagcg tcgacctccg 300
caatgatctc ctccaggatc ttctt 325
<210> 128
<211> 463
<212> ДНК
<213> Lygus hesperus
<400> 128
cggtcatcat ctccatgaac tcgtcgaagt cgacagttcc ggatccgtca gagtcgatct 60
cctcgatgat catgtccagc tcctcgttgg tcagctgctc gtccaattca tgaaggattt 120
ctttgaggca ggaggtcggg atgtagccat taccttcttt gtcgtagaga cggaaggctt 180
ctcgcagctc tttctgcatg gcttcatcgt cttcctcaac aatgaacttg gctgccaacg 240
tgatgaactc ttcaaactcc agccttcccg atttgtcagc gtcaacttct tcgatgagtt 300
catcgagaat cttcttgttg aagggttgac ccatgagtct gaggatgtcg gccaccatgt 360
ccgtcgggat ggaacccgag tgatcccggt cgaaagcgtt caacgcgatg gtcatgatgg 420
ggataattcg gttaattctg ttagaccagt ccgattagtg acg 463
<210> 129
<211> 413
<212> ДНК
<213> Lygus hesperus
<400> 129
atgggtgaag gagggtgcct gctcagagca gtcctccagg atgacggcta tggacaacgc 60
ctcgaagaac gccgctgaga tgatcgacaa gctgaccttg acgttcaaca ggactcggca 120
agccgtcatc accagggagc tcatcgaaat catctccggt gcctctgctt tggagtaacg 180
tctcagctca cccagccacc tcccgtagat ccactagtgc tgcgagagac cgagtacctc 240
gttctattca ccctgtacat ttcttaatca atattattgg aattcgattc gatagtcgta 300
tgctgggaaa tatcttgttc atattcatga tacttgttca acattgttct ggtaaataat 360
ttatgtaata caggttgagt taccaaaaaa aaaaaaaaaa aaaaaaaaaa aaa 413
<210> 130
<211> 449
<212> ДНК
<213> Lygus hesperus
<400> 130
gcagctggag gaagagaaac gtatctccct cagcattagg ctcaagcccc tggcgataga 60
aaatatgagc atagacaggc tacgcatcaa agctcaggaa ctctgggaag ccattgtcaa 120
acttgagacc gagaaatacg atcttgagga aaggcaaaag aggcaagact acgatcttaa 180
agaactgaag gaaagacaga agcaacaact tcggcacaaa gccttgaaaa aaggcttgga 240
ccctgaggcc ctcaccggca agtacccacc caaaatccaa gtcgcctcca aatatgaacg 300
aagagttgac accagatctt atgacgacaa gaaaaaactg tttgaaggcg gcatcctgga 360
gcggtataag gagttgatag aaaaggtgtg gaccgaaaaa gttgatcagt tcggcagtcg 420
agcccattcc aagcttccca gatggttcg 449
<210> 131
<211> 719
<212> ДНК
<213> Lygus hesperus
<400> 131
aggagttcga gaacaccagg aagaaccacc aacgggcact cgactccatg caagccagct 60
tggaggctga agccaagggt aaggctgagg ctcttcgcat gaagaagaag cttgaagccg 120
acatcaacga actcgaaatc gctctcgacc acgccaacaa ggctaacgcc gaagcccaga 180
agaccatcaa gaaataccaa caacaactca aggacgtaca gaccgctctt gaggaagaac 240
aaagggcacg cgacgatgct cgtgaacaac tcggaatcgc cgaaaggagg gccaacgctc 300
tcggaaacga gctcgaggag tcgcgcactc tcctcgaaca ggccgacagg ggcaggcgcc 360
aagccgaaca agaacttgga gatgcccacg aacagatcaa cgagttggct gcccaagcta 420
ccagcgcctc agccgccaaa cgcaagcttg agggagagtt gcagacactt catgctgacc 480
tcgacgaact tctcaacgaa gccaagaact ccgaagagaa ggccaagaag gcaatggttg 540
atgccgctag gttagctgat gaactcaggg cagaacagga ccacgctcag acccaggaga 600
agctccgcaa ggctctggaa acgcaaatca aggaactcca agtcaggttg gacgaagccg 660
aaaacaacgc cctcaaggga ggcaagaagg ccatcgccaa gctggaacaa cgtgtcagg 719
<210> 132
<211> 737
<212> ДНК
<213> Lygus hesperus
<400> 132
gcaggcgatg aagatggaga aggacacggc catggacaag gccgacacct gcgaggggca 60
ggccaaggac gctaacaccc gcgccgacaa aatccttgaa gatgtgaggg acctccaaaa 120
gaaactcaac caggtagaaa gtgatctcga aaggaccaag agggaactcg agacgaaaac 180
caccgaactc gaagagaagg agaaggccaa caccaacgct gagagcgagg tcgcctccct 240
caacaggaaa gtccagatgg ttgaagagga cttggaaaga tctgaagaaa ggtccggcac 300
cgcacaacaa aaactgtccg aagcctccca cgccgctgat gaagcctctc gtatgtgcaa 360
agtattggag aacaggtcac aacaggatga ggagaggatg gaccagctca ccaaccagct 420
gaaagaagcc cgactcctcg ctgaagacgc cgacggcaaa tcggatgagg tatcaaggaa 480
gctggccttc gttgaagacg aactggaagt agctgaagat cgtgtcaaat ctggagactc 540
gaagatcatg gagcttgagg aggagttgaa agttgtcggt aacagcttga aatctctcga 600
agtttcagag gagaaggcca accagcgagt cgaagagtac aaacgtcaaa tcaagcaact 660
gactgtcaag ttgaaggagg ctgaagctcg cgctgagttc gccgaaaaga cagtcaagaa 720
gttgcagaaa gaggtgg 737
<210> 133
<211> 205
<212> ДНК
<213> Lygus hesperus
<400> 133
cagaatccca cagaatctga cgtgaagaag ttcacccacc agcacaaacc agatgaaaga 60
atcagcttcg aggtgtttct cccgatatac caagccatat cgaagggtag gacgtcagac 120
acagctgaag acttcatcga gggtctcaga cactttgaca aagatggaaa tggcttcatt 180
tcaacagctg agcttcgcca cttgc 205
<210> 134
<211> 326
<212> ДНК
<213> Lygus hesperus
<400> 134
ggaggcattc aacatgatcg accagaacag ggacggcttc gtggataagg aagacctcca 60
tgacatgctc gcttccctag gtaagaaccc ctcagacgag tatctcgagg ggatgatgaa 120
cgaggcgcct ggtcccatca acttcacaat gttcctcacc ctcttcggtg agcggcttca 180
gggaactgat ccggaggagg ttatcaagaa cgcatttggg tgttttgacg aagacaacaa 240
cggattcatc aacgaggaaa gactgcgcga gctgctcacc tccatggggg acaggttcac 300
tgatgaagac gtggacgaaa tgtacc 326
<210> 135
<211> 944
<212> ДНК
<213> Lygus hesperus
<400> 135
gacttgatct tcagccgacc attgatctca ttgataagcc aatgggagtc atggctctcc 60
tggatgaaga atgttggttc cccaaagcca ctgacaagac cttcgttgag aagctggtcg 120
gtgctcacag cgttcacccc aaattcatca aaactgattt ccgtggagtc gccgactttg 180
ctgtcgtcca ttatgccgga aaagtcgatt attcggcggc gcagtggctg atgaagaaca 240
tggaccctct gaacgaaaac gtcgtgcagc tcctccagaa ctcgcaagat ccgttcgtca 300
tccacatctg gaaggacgca gagatcgtcg gcatggctca ccaagctctc agcgacactc 360
agtttggagc tcgtaccagg aagggtatgt tccgaaccgt gtctcaactc tacaaagacc 420
agctgtccaa actcatgatc acacttcgca acacgaaccc caacttcgtc cgttgcatcc 480
tccccaacca cgagaagaga gctggcaaga tcgatgctcc tttggtgctg gatcagctca 540
gatgcaacgg tgtgttggaa ggcatcagaa tttgcagaca aggtttcccg aatagaatcc 600
cattccagga attccggcaa agatacgagc tcttaactcc caatgtcatc cccaaagggt 660
tcatggacgg taaaaaggct tgcgagaaga tgatcaacgc tctcgaactg gaccctaatc 720
tctacagagt tggtcagtcc aagatattct tcagagctgg agtcttagct catctagaag 780
aagagcgcga ctataagatt actgatctga tagccaattt ccgggctttc tgtaggggat 840
atcttgcccg aaggaactac caaaagcgtc ttcagcagct caacgccatt cgtattatcc 900
agcgaaattg ctcagcttac ttgaagttga ggaactggca atgg 944
<210> 136
<211> 318
<212> ДНК
<213> Lygus hesperus
<400> 136
atcctggagg agatcattgc ggaggtcgac gctgatggct ctggacaact tgaatttgag 60
gagttcgtag ccttggctgc cggtttcctg acagaagatg agacacaaga tgctgaggcc 120
atgcagcaag aactgaggga ggccttcagg ctctacgaca aggaagggaa cggttacatt 180
accactgacg tcttgaggga aatcctgaag gaactcgacg acaaaatcac ctcccaagaa 240
cttgacatga tgattgctga aattgattct gacggttccg gaactgttga cttcgacgag 300
ttcatggaga tgatgacc 318
<210> 137
<211> 423
<212> ДНК
<213> Lygus hesperus
<400> 137
atccccatca tgaccatcgc gttgaacgct ttcgaccggg atcactcggg ttccatcccg 60
acggacatgg tggccgacat cctcagactc atgggtcaac ccttcaacaa gaagattctc 120
gatgaactca tcgaagaagt tgacgctgac aaatcgggaa ggctggagtt tgaagagttc 180
atcacgttgg cagccaagtt cattgttgag gaagacgatg aagccatgca gaaagagctg 240
cgagaagcct tccgtctcta cgacaaagaa ggtaatggct acatcccgac ctcctgcctc 300
aaagaaatcc ttcatgaatt ggacgagcag ctgaccaacg aggagctgga catgatcatc 360
gaggagatcg actctgacgg atccggaact gtcgacttcg acgagttcat ggagatgatg 420
acc 423
<210> 138
<211> 252
<212> ДНК
<213> Lygus hesperus
<400> 138
ggtgaaggag ggtgcctgct cagagcagtc ctccaggatg acggctatgg acaacgcctc 60
gaagaacgcc gctgagatga tcgacaagct gaccttgacg ttcaacagga ctcggcaagc 120
cgtcatcacc agggagctca tcgaaatcat ctccggtgcc tctgctttgg agtaacgtct 180
cagctcaccc agccacctcc cgtagatcca ctagtgctgc gagagaccga gtacctcgtt 240
ctattcaccc tg 252
<210> 139
<211> 1110
<212> ДНК
<213> Lygus hesperus
<400> 139
gtctccgctc aagctggtca tcagacttca gctgagtcct ggggtaccgg tcgtgctgtg 60
gctcgtatcc cccgtgttcg cggaggtggt actcaccgct caggtcaggg tgcttttggc 120
aacatgtgtc gcggcggtag gatgttcgct cccactcgcc catggcgtcg ttggcaccgc 180
aagatcaacg ttaaccaaaa acgttatgcc gtcgtgtccg ccatcgctgc atccggcgtc 240
ccagccctcg tcatgtccaa aggacacatg gtgcaaagcg tccctgaatt cccccttgtt 300
gtgtctgaca aagttcagga atacactaaa accaaacagg ctgtcatctt ccttcaccgc 360
atcaaagcct ggcaagacat ccagaaagtg tacaagtcga agaggttccg tgctggtaag 420
ggtaaaatga ggaaccgcag gaggatccag aggcgtggac ccctcatcat ctacgaccag 480
gatcagggtc tgaacagggc tttccgtaac attcccggcg tcgatttgat cgaagtgagc 540
cgcctcaact tgctgaagct cgctccagga ggtcacatcg gccggttcgt catctggact 600
cagtcggcct tcgagaagtt ggacgccctc tacggcacct ggaagaagaa gtccaccctc 660
aaggctggat acaatctccc catgcccaag atggccaaca ccgacctttc ccgcctcttc 720
aaggccccgg agatcaaggc tgtcctcagg aatcccaaga agaccatcgt acgacgagtg 780
cgcaaactga accctctccg caacaccagg gctatgctgc gtctcaaccc atacgctgct 840
gtcctcaaga ggaaggccat ccttgatcaa aggaagttga aactccagaa gctcgtagaa 900
gctgccaaga agggagatac caagctgtcg ccccgcgtcg agcgtcacct gaagatgatc 960
gagagaagga aagccctgat caagaaagcc aaggctgcca agcccaagaa gcccaaaacg 1020
gccaagaaac ccaagaccgc cgagaaggca ccagcacccg ccaagaaggc ggcagcgccc 1080
aaaaaggcca ccacccctgc caagaaatga 1110
<210> 140
<211> 729
<212> ДНК
<213> Lygus hesperus
<400> 140
atggccaatg ctaagcctat ttctaagaag aagaagtttg tgtctgacgg tgtcttcaaa 60
gccgaattga acgaatttct taccagagaa ctcgctgaag aggggtactc aggtgttgag 120
gtccgagtga cccccaacaa gacagaaatt atcatcatgg cgacaaggac acaaagcgtt 180
cttggtgata agggccgccg aatcagggag ctcacgtctg tagttcagaa aagattcaat 240
ttcaagcctc agactttgga tctctatgct gaaaaggtcg ccaccagagg tttgtgtgct 300
attgcacaag ctgaatccct ccgttacaaa ctcattggcg gtcttgctgt ccgaggggct 360
tgctatggtg tccttcgctt catcatggaa aatggtgcca agggttgcga agtcgtagta 420
tctggaaaac tgcgtggtca gagagccaag tcaatgaagt tcgtggatgg tttgatgatc 480
cacagtgggg atccctgtaa cgaatatgtt gatactgcta cccgacatgt gctccttaga 540
caaggtgtcc tgggaataaa ggtgaagatt atgttgccgt gggacgttac cggcaaaaat 600
gggccgaaga accctcttcc cgaccacgtc agcgttctct tacctaagga ggagctacca 660
aatttggccg ttagtgtgcc tggatccgac atcaaaccaa agcctgaagt accagcaccc 720
gctttgtga 729
<210> 141
<211> 789
<212> ДНК
<213> Lygus hesperus
<400> 141
atggctgttg gtaaaaataa gggtctatcg aaaggaggaa agaagggagt taaaaaaaag 60
gtagtggacc ctttcaccag gaaggattgg tacgatgtta aggctccttc catgttcaaa 120
aagcgtcaag ttggcaaaac tttggtcaac cgaactcagg gaaccaagat tgcttctgaa 180
gggttgaaag gacgagtttt cgaagtttcg ctcgctgata tccaggagga cactgatgcc 240
gagcgctcct tcaggaaatt caggctcatc gctgaagatg tccaagccag aaacgtcctt 300
accaatttcc acggtatgga tttgaccact gacaaactcc ggagcatggt caagaagtgg 360
cagactctca tcgaagccaa cgttgacgtc aagaccaccg acggctacct cctgcgcgtc 420
ttctgcatag gattcaccaa taaagatcaa ctttcccaga gaaagacttg ctatgcccag 480
cataatcagg tccgagaaat ccgcaaaaag atggttaaaa acatcagtga cagcatttcc 540
agctgtgatt tgaggagtgt tgtgaacaag ctgatcccag actccatcgc taaagatata 600
gaaaagaatt gccaaggaat ctacccactc cacgatgtgt acattcggaa ggtgaaggtg 660
ttgaagaagc cgaggttcga gctcagcaag ctccttgagc ttcacgtcga tggcaaaggg 720
atcgacgaac ccggcgcgaa agtgacgagg actgacgctt acgagcctcc agttcaagag 780
tctgtctaa 789
<210> 142
<211> 473
<212> ДНК
<213> Lygus hesperus
<400> 142
gaccaaggag cagctggagg aagagaaacg tatctccctc agcattaggc tcaagcccct 60
ggcgatagaa aatatgagca tagacaggct acgcatcaaa gctcaggaac tctgggaagc 120
cattgtcaaa cttgagaccg agaaatacga tcttgaggaa aggcaaaaga ggcaagacta 180
cgatcttaaa gaactgaagg aaagacagaa gcaacaactt cggcacaaag ccttgaaaaa 240
aggcttggac cctgaggccc tcaccggcaa gtacccaccc aaaatccaag tcgcctccaa 300
atatgaacga agagttgaca ccagatctta tgacgacaag aaaaaactgt ttgaaggcgg 360
catcctggag cggtataagg agttgataga aaaggtgtgg accgaaaaag ttgatcagtt 420
cggcagtcga gcccattcca agcttcccag atggttcggc gagcggcccg gca 473
<210> 143
<211> 1463
<212> ДНК
<213> Lygus hesperus
<400> 143
gggtctcagc tgaggcacat tccatctcgt cgcaaatctt tcctgcatct ctcctgggtg 60
acctttaggt gaccaatcac atccatcatg tcggacgagg agtattcgga gtcggaggaa 120
gagacccagc cggaaccaca gaaaaaacca gaggctgaag gaggcggcga cccagaattc 180
gtcaagcgta aggaagccca gacctcagcc ttagacgagc agcttaaaga ctatatcgca 240
gaatggagga aacaaagagc tcgcgaagaa gaagacctca agaagctgaa ggagaagcaa 300
gccaagcgca aggtcgctcg ggcagaagaa gaaaagagat tggcggaaaa gaagaagcag 360
gaagaagaac gacgtgtgag ggaagcagaa gagaagaaac agagggaaat cgaagagaag 420
aggcgaaggc ttgaagaggc cgagaagaag agacaagcca tgatggctgc tctcaaggac 480
cagagcaaaa cgaagggacc caattttgtc gttaataaga aagccgaaac ccttggcatg 540
tcctccgctc aaattgagcg caacaagact aaggaacagc ttgaggaaga aaaacgtatc 600
tccctcagca ttaggctcaa gcccctggcg atagaaaata tgagcataga caggctacgc 660
ataaaagctc aggaactctg ggaagccatt gtcaaacttg agaccgagaa atacgatctt 720
gaggaaaggc aaaagaggca agactacgat cttaaagaac tgaaggaaag acagaagcaa 780
caacttcggc acaaagcctt gaaaaaaggc ttggaccctg aggccctcac cggcaagtac 840
ccacccaaaa tccaagtcgc ctccaaatat gaacgaagag ttgacaccag atcttatgac 900
gacaagaaaa aactgtttga aggcggcatc ctggagcggt ataaggagtt gatagaaaag 960
gtgtggaccg aaaaagttga tcagttcggc agtcgagccc attccaagct tcccagatgg 1020
ttcggcgagc ggcccggcaa gaagaaggat gcccctgaaa gcccggaaga agaggaagtg 1080
aaggtagaag atgaacctga agctgaacca agcttcatgc tcgacgaaga agaagaagaa 1140
gcggaagaag aggaggcgga agaggaagag gaagccgagg aagaggagga agaagaagag 1200
gaagaggaag aggaggagga ggaagaagaa taggtctttt tcaacatttc actgcaccca 1260
cagttccacg gtctttccgc ccacaaactc aatctgtgct cacgagatct tagcaggaaa 1320
agtattgcga cccgataaga acaaattaaa ttatttttgg aatatctcgt tcagttattt 1380
cgtgagaaac aattttattc atgtaaacga ttaaaagatc ccatacattt ccaaaaaaaa 1440
aaaaaaaaaa aaaaaaaaaa aaa 1463
<210> 144
<211> 773
<212> ДНК
<213> Lygus hesperus
<400> 144
ccggcggatc caggaaaagg aggaggagtt cgagaacacc aggaagaacc accaacgggc 60
actcgactcc atgcaagcca gcttggaggc tgaagccaag ggtaaggctg aggctcttcg 120
catgaagaag aagcttgaag ccgacatcaa cgaactcgaa atcgctctcg accacgccaa 180
caaggctaac gccgaagccc agaagaccat caagaaatac caacaacaac tcaaggacgt 240
acagaccgct cttgaggaag aacaaagggc acgcgacgat gctcgtgaac aactcggaat 300
cgccgaaagg agggccaacg ctctcggaaa cgagctcgag gagtcgcgca ctctcctcga 360
acaggccgac aggggcaggc gccaagccga acaagaactt ggagatgccc acgaacagat 420
caacgagttg gctgcccaag ctaccagcgc ctcagccgcc aaacgcaagc ttgagggaga 480
gttgcagaca cttcatgctg acctcgacga acttctcaac gaagccaaga actccgaaga 540
gaaggccaag aaggcaatgg ttgatgccgc taggttagct gatgaactca gggcagaaca 600
ggaccacgct cagacccagg agaagctccg caaggctctg gaaacgcaaa tcaaggaact 660
ccaagtcagg ttggacgaag ccgaaaacaa cgccctcaag ggaggcaaga aggccatcgc 720
caagctggaa caacgtgtca gggaactcga aaacgaactg gacggcgagc agc 773
<210> 145
<211> 5446
<212> ДНК
<213> Lygus hesperus
<400> 145
tcaggaaaac tggctggtgc tgatattgag acctatctgc tggagaaggc tcgtgtcatc 60
tcccaacaaa cactcgagag atcctaccac attttctacc agatgatgtc tggagctgtc 120
aagggcgtca aggaaatgtg cttgctggtc gacgatatct atacgtacaa cttcatatcc 180
cagggtaaag tcagcattgc aggcgttgat gacggagagg aaatggttct gaccgatcaa 240
gccttcgaca tcttgggttt caccaagcaa gagaaggaag acatctacaa gatcaccgcc 300
gctgtcattc acatgggtac catgaagttc aagcaaaggg gtcgtgaaga gcaggctgaa 360
gccgatggaa ctgaggaagg cggtaaggtc ggtgtgctcc tcggtatcga cggtgacgac 420
ttgtacaaga atatgtgcaa gcccagaatc aaggtcggaa ctgagttcgt gacccaggga 480
aagaacgtca accaggtctc atactctctc ggtgccatgt ccaagggtat gttcgatcgt 540
ctcttcaaat tcttggtcaa gaaatgtaac gaaactctgg acaccaaaca gaagagacag 600
cacttcattg gtgtactgga tattgccggg ttcgaaattt tcgacttcaa cggttttgag 660
caactgtgta tcaacttcac caacgagaaa ttgcaacaat tcttcaacca ccacatgttc 720
gtactcgagc aagaagagta caagagggaa ggcattaact gggctttcat tgatttcgga 780
atggacttgc tcgcttgtat tgaactgatt gagaagccca tgggtatctt gtccatcctt 840
gaagaagagt ctatgttccc caaggctact gacaagacct ttgaggacaa actcatcacc 900
aaccacttgg gcaaatctcc caacttcagg aagcccgccg ttccaaagcc tggccaacaa 960
gctggtcact tcgccatcgc tcactacgct ggttgcgtgt catacaacat caccggctgg 1020
cttgagaaga acaaggatcc gttgaacgac actgttgtcg atcagtacaa gaagggaacc 1080
aacaaactgt tgtgcgagat cttcgctgat catcctggcc aatctggtgc ccctggtggt 1140
gatgctggtg gcaagggtgg tcgtggcaag aaaggtggtg gcttcgccac tgtgtcatct 1200
tcctacaagg aacaattgaa caacttgatg accactttga agagcacaca gcctcacttc 1260
gtccgttgta tcatccccaa cgaattgaaa cagcccggtg ttattgattc tcacttggtc 1320
atgcaccagc tgacttgtaa cggtgtactt gaaggcatcc gtatttgccg taaaggcttc 1380
cccaacagga tgaactaccc tgacttcaag ctccgataca agatccttaa ccccgctgcc 1440
gtggacagag agagtgatat cctcaaggct gctggtctcg tccttgagtc aactgggctc 1500
gaccctgata tgtaccgtct cggccacacc aaggtgttct tcagggccgg agttttgggt 1560
caacttgaag aattgcgtga cgacaggctt agcaagatca tcggatggat gcaggccttc 1620
atgcgcggtt acctcgtcag gaaggagtac aagaagctcc aggaacagag gttagccctc 1680
caagttgtcc agcgcaactt gagaaggtac ctccaactga ggacctggcc ctggtggaag 1740
atgtggtcca gggtcaagcc cctcctcaac gtcgccaacg tcgaagagga gatgcggaaa 1800
ctcgaagagt tggtcgccga gacccaggcc gctttggaga aggaggagaa gctgaggaag 1860
gaggccgaag cccttaacgc caagcttctc caagagaaga ccgaccttct caggaacttg 1920
gaaggagaga agggatccat cagcggtatc caggaacgat gtgccaagct gcaagcccaa 1980
aaggccgatc ttgagtctca actcatggac acccaagaaa ggctgcagaa cgaagaagat 2040
gccaggaacc agctcttcca acagaagaag aaattggaac aagaagccgc tgccctcaag 2100
aaggacatcg aagatctcga actctccaac caaaagaccg accaagataa ggccagcaag 2160
gaacaccaaa tcagaaacct caatgacgag atcgctcacc aagatgactt gatcaacaag 2220
ctcaacaagg agaagaaaat ccagagcgaa ctcaaccaaa agactgctga agaacttcag 2280
gccgctgaag acaaaatcaa ccacctcacc aaggttaagg tcaagcttga acagaccttg 2340
gatgaactcg aagacaccct cgaacgtgaa aagaaactcc gaggagatgt cgaaaaggcc 2400
aagaggaaga ctgaaggcga cctcaagctc actcaggaag ccgttgccga tcttgaaagg 2460
aacaagaaag aactcgaaca gaccatccag aggaaagaca aggaaattgc ttccctcacc 2520
gccaagctcg aagacgaaca atccatcgtc aacaagactg gcaaacagat caaggaactc 2580
cagagccgca ttgaagagct cgaggaggaa gtcgaggctg agaggcaagc ccgcggaaag 2640
gctgagaagc aacgtgctga cctcgcccgc gaacttgagg aactcggcga gaggttagag 2700
gaagctggtg gtgccacctc tgcccagatc gagctcaaca agaagcgtga agctgagatg 2760
agcaaactca ggagggacct ggaagaagcc aacatccagc acgaaggcac gctcgccaac 2820
ctccgcaaga agcacaacga tgctgtcagt gagatgggag accaaatcga ccagctcaac 2880
aaacttaaga ccaaggttga aaaggagaag tctcaatacc tcggtgaact caacgacgtc 2940
cgcgcctcca ttgaccactt gaccaacgag aaggctgcca ctgaaaaggt tgccaagcaa 3000
ctgcaacacc aaatcaatga agttcaaggc aaacttgatg aagctaacag gacgctcaac 3060
gacttcgatg ctgccaagaa gaagttgtct attgagaact ctgacctcct cagacagttg 3120
gaggaagctg agagccaagt ttctcaactt agcaagatca agatctccct caccactcaa 3180
ctcgaggaca ctaagcgtct cgccgatgag gaagctaggg aacgcgcaac ccttcttggc 3240
aagttccgca acttggaaca cgaccttgac aacctgaggg aacaggtgga ggaagaagcc 3300
gaagctaagg ctgatatcca acgtcaactc agcaaggcca acgctgaagc tcagttgtgg 3360
cgcagcaagt acgaaagcga gggtgttgcc cgcgctgagg agcttgagga ggccaagagg 3420
aaactccagg cccgtttggc tgaggctgag gagaccattg agtccctcaa ccagaaggtt 3480
atcgcccttg agaagacgaa gcagcgcctt gccactgaag tcgaggatct gcagctcgag 3540
gtcgaccgtg ccaacgccat tgccaatgcc gctgaaaaga aggctaaggc tattgacaag 3600
atcattggtg aatggaaact caaggttgat gaccttgctg ctgagcttga tgctagtcaa 3660
aaggaatgca gaaactactc cactgagctc ttcaggctca agggagctta tgaagaagga 3720
caggaacaac ttgaagctgt ccgcagggag aacaagaacc ttgctgatga agtcaaggac 3780
ttgctcgacc agatcggtga gggtggccgc aacatccacg aaattgagaa gcagcgcaag 3840
aggctcgaag ttgagaagga cgaacttcag gccgctcttg aggaggctga agccgctctt 3900
gaacaggagg agaacaaagt actcagggct caacttgagc tcagccaggt gcgtcaagaa 3960
attgaccgcc gcatccagga gaaggaagag gagttcgaga acaccaggaa gaaccaccaa 4020
cgggcactcg actccatgca agccagcttg gaggctgaag ccaagggtaa ggctgaggct 4080
cttcgcatga agaagaagct tgaagccgac atcaacgaac tcgaaatcgc tctcgaccac 4140
gccaacaagg ctaacgccga agcccagaag accatcaaga aataccaaca acaactcaag 4200
gacgtacaga ccgctcttga ggaagaacaa agggcacgcg acgatgctcg tgaacaactc 4260
ggaatcgccg aaaggagggc caacgctctc ggaaacgagc tcgaggagtc gcgcactctc 4320
ctcgaacagg ccgacagggg caggcgccaa gccgaacaag aacttggaga tgcccacgaa 4380
cagatcaacg agttggctgc ccaagctacc agcgcctcag ccgccaaacg caagcttgag 4440
ggagagttgc agacacttca tgctgacctc gacgaacttc tcaacgaagc caagaactcc 4500
gaagagaagg ccaagaaggc aatggttgat gccgctaggt tagctgatga actcagggca 4560
gaacaggacc acgctcagac ccaggagaag ctccgcaagg ctctggaaac gcaaatcaag 4620
gaactccaag tcaggttgga cgaagccgaa aacaacgccc tcaagggagg caagaaggcc 4680
atcgccaagc tggaacaacg tgtcagggaa ctcgaaaacg aactggacgg cgagcagagg 4740
agacacgccg acgcacaaaa gaacctccgt aaatccgagc gtagaattaa ggagctcagt 4800
ttccagtccg acgaggaccg taagaaccac gaacgcatgc aagacctcgt agacaaactg 4860
caacagaaga tcaagactta caagaggcag attgaagaag ccgaagaaat cgcggccctt 4920
aacctcgcca aattccgcaa agcacaacaa gaactcgaag aagctgaaga acgcgctgat 4980
ctcgctgaac aggctgtttc caaattcaga acaaagggtg gacgcgcagg atctgctgcc 5040
agagcgatga gccctgtcgg ccagaagtga aggaacgaat aagcggacgt ataagctatc 5100
aatacctcgc acacaaacct gccaggcctc aatttgacgg caatgccttc ccaccacgat 5160
tcgatctaca tcccgacgac ttttaagatc tttgatagca acgcaaaaca tcaaatgaaa 5220
atcttttaaa ttttatgtat ttattttgac ctattttatt aagttattgt taatacaaac 5280
ataattccat gagctagata tctagccaac gaaccatcac aatcacgatt attcgaactg 5340
tacgatagaa gcattatttg tacagctgga ccatttacaa aatatttttg cttcgaataa 5400
taaagagttt atatcgcgaa aaaaaaaaaa aaaaaaaaaa aaaaaa 5446
<210> 146
<211> 964
<212> ДНК
<213> Lygus hesperus
<400> 146
tcctcctctg gtgcccgact cttcaaatac ccaaatccag tcatgtcttc ccgtaaaacc 60
gctggccgca gggcgaccac caagaagcgc gctcagcgtg cgacgtcaaa cgtattcgcc 120
atgttcgatc aggctcagat tcaagaattc aaggaggcat tcaacatgat cgaccagaac 180
agggacggct tcgtggataa ggaagacctc catgacatgc tcgcttccct aggtaagaac 240
ccctcagacg agtatctcga ggggatgatg aacgaggcgc ctggtcccat caacttcaca 300
atgttcctca ccctcttcgg tgagcggctt cagggaactg atccggagga ggttatcaag 360
aacgcatttg ggtgttttga cgaagacaac aacggattca tcaacgagga aagactgcgc 420
gagctgctca cctccatggg ggacaggttc actgatgaag acgtggacga aatgtaccga 480
gaggccccca tcaagaacgg catgttcgac tacatcgaat tcactcggat cctcaagcac 540
ggagccaaag acaaagacga gcagtgacct atcaaatcct cgtcaacctc ccttcagtaa 600
tttgaaacca atccatcaaa ttttgtttaa aactcttact taaaatccga tcatctacgt 660
cactttgcca ccaatcggta ttattttttg agccgttcct acataaatcg aattaatttt 720
atacctacga atcatattgt tggaaatttc tctcttgtac ttatactttc tgttatttcc 780
taatttttct aactaaccaa gttagtcgtt agtttttatt cattccttta taaattatta 840
gttatccatt tttaatcatc ttgaagttat ttgtttttcg agtggtagaa tatttataca 900
ttttccaata tataatggtt tattcattct taaaaaacga aaaaaaagaa aaaaaaaaaa 960
aaaa 964
<210> 147
<211> 5872
<212> ДНК
<213> Lygus hesperus
<400> 147
gatcttacct gcctgaacga ggcgtccgtt cttcacaaca tcaaggacag atattactcc 60
ggattgattt atacgtattc gggactcttc tgcgtggtgg tcaaccctta caagaaactg 120
ccaatctaca cagagagaat catggagaaa tacaaaggcg tcaaaagaca cgacctccct 180
ccacacgtat tcgccatcac agacacagct taccgttcta tgctgcaaga tagggaagat 240
caatcgatac tctgcaccgg cgaatcgggt gcggggaaaa ccgaaaacac gaaaaaagta 300
atccagtact tggcctacgt tgcagcctcg aaacccaaat cttccgcatc cccacatacg 360
gcccagagtc aagctctgat cattggagaa ctcgaacaac agctgcttca agctaaccca 420
attttggaag cattcggaaa cgccaagact gttaaaaacg ataattcttc tcgattcggt 480
aaattcattc gtatcaattt cgacgcatca ggctacatcg caggagccaa catagaaacg 540
tatcttctag agaaatctag ggccatcaga caagcgaaag atgagcgaac gttccacatc 600
ttttaccaac ttctggccgg agcatctgca gaacaaagaa aggagttcat cctcgaagat 660
ccgaaaaact accctttcct cagcagcggg atggtgtctg tgcctggagt tgacgatggt 720
gttgatttcc aagcaactat cgcctccatg tccatcatgg gcatgaccaa cgacgatctt 780
tccgctctct tccgcatcgt cagtgccgtc atgctgttcg gcagcatgca gttcaagcag 840
gagcgaaaca gcgaccaggc gacgctccca gacaacactg tagcgcaaaa aatcgcccac 900
ctccttggtc tctcaatcac agagatgacc aaagcgttcc tcaggcctag aatcaaagta 960
ggacgggatt tcgtcaccaa ggctcaaact aaggaacaag ttgagttcgc agtggaagcc 1020
atttcgaaag cctgctacga acgtatgttc cgatggctcg tcaacagaat caaccgctcc 1080
ctggatcgta ccaaaaggca gggagcatct ttcattggta ttcttgatat ggctggtttc 1140
gaaatctttg agatcaactc cttcgagcag ctttgtatca attacaccaa tgagaaactt 1200
caacaactct tcaaccacac catgttcatt ttggagcaag aggagtacca gagagaaggt 1260
attgaatgga agttcatcga cttcggactt gatcttcagc cgaccattga tctcattgat 1320
aagccaatgg gagtcatggc tctcctggat gaagaatgtt ggttccccaa agccactgac 1380
aagaccttcg ttgagaagct ggtcggtgct cacagcgttc accccaaatt catcaaaact 1440
gatttccgtg gagtcgccga ctttgctgtc gtccattatg ccggaaaagt cgattattcg 1500
gcggcgcagt ggctgatgaa gaacatggac cctctgaacg aaaacgtcgt gcagctcctc 1560
cagaactcgc aagatccgtt cgtcatccac atctggaagg acgcagagat cgtcggcatg 1620
gctcaccaag ctctcagcga cactcagttt ggagctcgta ccaggaaggg tatgttccga 1680
accgtgtctc aactctacaa agaccagctg tccaaactca tgatcacact tcgcaacacg 1740
aaccccaact tcgtccgttg catcctcccc aaccacgaga agagagctgg caagatcgat 1800
gctcctttgg tgctggatca gctcagatgc aacggtgtgt tggaaggcat cagaatttgc 1860
agacaaggtt tcccgaatag aatcccattc caggaattcc ggcaaagata cgagctctta 1920
actcccaatg tcatccccaa agggttcatg gacggtaaaa aggcttgcga gaagatgatc 1980
aacgctctcg aactggaccc taatctctac agagttggtc agtccaagat attcttcaga 2040
gctggagtct tagctcatct agaagaagag cgcgactata agattactga tctgatagcc 2100
aatttccggg ctttctgtag gggatatctt gcccgaagga actaccaaaa gcgtcttcag 2160
cagctcaacg ccattcgtat tatccagcga aattgctcag cttacttgaa gttgaggaac 2220
tggcaatggt ggcggctgta caccaaggtc aaacctctgc ttgaagtgac gaaacaagaa 2280
gagaagctga cgcaaaagga agacgaactg aagcaggtcc gcgagaaact ggacaaccag 2340
gtgaggtcca aggaagagta tgaaaagagg cttcaggacg ctttggagga gaaagctgct 2400
ctggcagagc aacttcaggc agaagtagag ctgtgtgcgg aagccgaaga aatgagagcc 2460
aggctcgctg tgaggaagca agaactagag gaaattctcc acgatctaga agccagaata 2520
gaggaagaag agcaacgaaa cacggtcctc atcaacgaaa agaagaagtt gaccctcaac 2580
atcgccgacc tcgaagaaca actggaagag gaagaaggag ctcgacagaa actccaactc 2640
gaaaaagtcc agatcgaagc tcggctgaag aaaatggaag aggacctcgc tctggccgaa 2700
gacaccaaca ccaaagtcgt aaaggagaag aaagtgttgg aagagagggc tagtgacttg 2760
gcccagaccc tcgctgagga agaagaaaaa gctaaacacc tcgcgaagct caagaccaag 2820
cacgagacga cgatagcgga attggaagag aggttgctca aagacaatca gcagaggcag 2880
gaaatggata ggaacaagag gaagatcgaa tcagaggtga atgatttgaa agaacaaatt 2940
aacgagaaga aggtccaagt agaggagctt cagttgcaac tcgggaagag ggaagaggaa 3000
atcgctcaag ctctgatgag aattgacgag gaaggagcag gcaaagctca gactcaaaag 3060
gctctcaggg aattggagtc tcagctggct gagctacaag aggatctaga ggctgaaaag 3120
gccgctcgcg ccaaggccga aaagcagaag cgcgacctca acgaagaact cgagtccctc 3180
aagaatgaac ttcttgactc actggacacg acagcagctc aacaggaatt gaggaccaag 3240
agagaacacg aactggcaac gctcaagaaa acattagaag aggaaacgca cattcacgaa 3300
gtatctctca ccgaaatgag gcacaaacac actcaagaag tcgctgcact caacgaacag 3360
ttggagcaac tcaaaaaggc caaatctgca ctcgaaaaat cgaaagcaca acttgaaggg 3420
gaagctgctg agctcgccaa cgaactggaa acagcaggaa cgagcaaggg cgagagtgaa 3480
aggaaacgga agcaggccga atcgtctctg caggagctct cgtcgcgact cttggaaatg 3540
gagagaacca aagccgagct ccaagagagg gtccagaaac tgtctgcaga agccgactct 3600
gtcaatcagc agttggaagc agcggaactg aaagcatcag cagccctcaa ggcatctggt 3660
accttggaga ctcagctcca ggaggcgcaa gtgctcctgg aagaggaaac tcggcagaag 3720
ctgtcgttga ccaccaaact gaaaggcctc gaaagcgaaa gagatgctct caaagagcaa 3780
ctctacgaag aggacgaggg taggaagaac ctagaaaaac agatggcgat actcaatcaa 3840
caagtagctg aaagcaagaa gaagtctgaa gaagaaacgg aaaaaataac tgaactcgaa 3900
gaaagtcgca aaaaattgct caaagacata gaaattcttc aaaggcaagt cgaagaactt 3960
caagttacca acgacaaatt agagaaaggc aagaagaagc tgcagtcaga actggaagac 4020
ctcaccatcg acctggagtc tcagagaaca aaggtggtcg agctcgagaa gaaacaaaga 4080
aatttcgaca aagttttggc cgaagaaaaa gcgttgtcgc aacaaatcac gcacgagagg 4140
gatgcggctg aaagagaagc ccgtgaaaag gaaactagag tactgtcgct gacgcgagaa 4200
ctcgatgaat tcatggagaa aatcgaggaa ctggagagaa gcaaacggca actccaggct 4260
gaactagacg agctggtcaa caaccaaggc accaccgaca aaagcgtgca cgaattggaa 4320
agggcgaaac gagttctgga gtcacaactt gcagagcaga aagcacaaaa tgaagagctt 4380
gaagatgaac tccaaatgac ggaagacgcc aaattgaggc tcgaagtcaa catgcaagct 4440
ctgagagctc aattcgaaag agatctacag ggcaaagaag agtcgggaga agaaaagagg 4500
agaggattgc tgaaacagct gagggacatt gaggctgaac ttgaagacga gagaaaacaa 4560
aggaccgctg ctgttgcctc tagaaagaag attgaagcgg atttcaaaga tgtagaacag 4620
caactggaaa tgcacactaa ggtaaaggaa gatcttcaga agcaactgaa gaaatgccag 4680
gtccaactga aggacgcaat cagagacgcg gaagaggctc ggctcggtcg ggaagagctg 4740
caggctgccg ctaaagaggc cgaaaggaag tggaagggtt tggaaacgga gctcattcaa 4800
gtgcaagagg atttgatggc gagcgaaagg cagcggcggg cagcggaagc cgaaagggat 4860
gaagtcgttg aagaagccaa caagaatgtc aagagcttat cgaatcttct cgacgaaaag 4920
aagaggctcg aagcccaatg ctcaggcctg gaagaggaac tcgaagaaga acttagcaac 4980
aatgaggccc tccaagacaa agcgagaaaa gcacaactca gcgttgagca acttaatgca 5040
gaacttgctg ccgaacggag taatgtgcag aaacttgagg gaacgagatt gtcgatggaa 5100
aggcaaaaca aggaactgaa ggccaaactg aacgaactgg aaacgttaca acgcaacaag 5160
ttcaaggcca atgcgtctct ggaggctaag attaccaatc ttgaagagca actggaaaat 5220
gaagccaagg aaaagctact tctccagaaa ggcaacagga agctcgacaa gaaaatcaaa 5280
gacctcctcg ttcaattgga ggatgaaagg aggcatgccg accagtataa agaacaagtc 5340
gagaagatca acgtcagggt gaagacgcta aagcgaactt tggacgacgc cgaagaagaa 5400
atgagtaggg agaagaccca gaagaggaaa gcacttcgcg aattggaaga cctcagggag 5460
aactacgatt ccctactccg agagaacgat aacctcaaaa acaaactcag gcggggcggc 5520
ggtatttccg ggatctcgag caggctcgga ggctccaagc gaggttccat ccccggagag 5580
gattcccagg gtctcaacaa caccacagac gaatcagtcg atggtgacga tatctcgaat 5640
ccttaaacgc tacttggatt taccagccag catccaactt tccactgaag acgtctccca 5700
taaacgttga aagagacccg tcgaggaaga aaaaaaggct ctttaagaaa aactattctg 5760
cctttttcaa aactttgtac ttaaaagtac tttcgcttaa caatgaaaga agaataaaaa 5820
tgtaaagttt tcatttatac aaaaaaaaaa aaaaaaaaaa aaaaaaaaaa aa 5872
<210> 148
<211> 325
<212> ДНК
<213> Lygus hesperus
<400> 148
aagaagatcc tggaggagat cattgcggag gtcgacgctg atggctctgg acaacttgaa 60
tttgaggagt tcgtagcctt ggctgccggt ttcctgacag aagatgagac acaagatgct 120
gaggccatgc agcaagaact gagggaggcc ttcaggctct acgacaagga agggaacggt 180
tacattacca ctgacgtctt gagggaaatc ctgaaggaac tcgacgacaa aatcacctcc 240
caagaacttg acatgatgat tgctgaaatt gattctgacg gttccggaac tgttgacttc 300
gacgagttca tggagatgat gaccg 325
<210> 149
<211> 428
<212> ДНК
<213> Lygus hesperus
<400> 149
aattatcccc atcatgacca tcgcgttgaa cgctttcgac cgggatcact cgggttccat 60
cccgacggac atggtggccg acatcctcag actcatgggt caacccttca acaagaagat 120
tctcgatgaa ctcatcgaag aagttgacgc tgacaaatcg ggaaggctgg agtttgaaga 180
gttcatcacg ttggcagcca agttcattgt tgaggaagac gatgaagcca tgcagaaaga 240
gctgcgagaa gccttccgtc tctacgacaa agaaggtaat ggctacatcc cgacctcctg 300
cctcaaagaa atccttcatg aattggacga gcagctgacc aacgaggagc tggacatgat 360
catcgaggag atcgactctg acggatccgg aactgtcgac ttcgacgagt tcatggagat 420
gatgaccg 428
<210> 150
<211> 538
<212> ДНК
<213> Lygus hesperus
<400> 150
gcttctttta caaatcgcac cacgccgact taattcattc ccggagggtt taaattttat 60
cgaagcagca tggtgcggat gaatgtgctg agcgatgctc tgaaaagcat caacaatgct 120
gagaagaggg gcaaaaggca ggtgctcctg aggccttgtt ccaaagtcat cattaaattc 180
cttacagtga tgatgaagaa aggttatatc ggcgaattcg aaatagtaga tgatcacaga 240
tctggtaaaa tcgtcgtcaa cctcaacggc agattgaaca aatgtggagt tatatcgccc 300
agattcgacg tacccatcac acaaatcgaa aaatggacga acaacctcct gccttcccga 360
cagttcggtt atgtcgtact caccactagt ggagggatca tggatcacga agaagccagg 420
cgaaaacatc ttgggggtaa aatattaggg tttttctttt aataaaaaaa gacgagatgt 480
aaattaataa aactctttta cgtttcgcta aaaaaaaaaa aaaaaaaaaa aaaaaaaa 538
<210> 151
<211> 405
<212> ДНК
<213> Lygus hesperus
<400> 151
ccacgccgac ttaattcatt cccggagggt ttaaatttta tcgaagcagc atggtgcgga 60
tgaatgtgct gagcgatgct ctgaaaagca tcaacaatgc tgagaagagg ggcaaaaggc 120
aggtgctcct gaggccttgt tccaaagtca tcattaaatt ccttacagtg atgatgaaga 180
aaggttatat cggcgaattc gaaatagtag atgatcacag atctggtaaa atcgtcgtca 240
acctcaacgg cagattgaac aaatgtggag ttatatcgcc cagattcgac gtacccatca 300
cacaaatcga aaaatggacg aacaacctcc tgccttcccg acagttcggt tatgtcgtac 360
tcaccactag tggagggatc atggatcacg aagaagccag gcgaa 405
<210> 152
<211> 470
<212> ДНК
<213> Lygus hesperus
<400> 152
tgtcgatggc ggtcttaaca tcccccattc caccaagagg ttccctgggt acgacagtga 60
gtctaaggaa ttcaacgctg aggtccacag gaagcacatt ttcggcattc acgtcgctga 120
ctacatgcgt cagctggctg aagaggatga cgatgcttac aagaagcagt tctcgcagta 180
tgtcaagaac ggagtcactg ctgacagcat tgaaagtatc tacaagaagg ctcacgaagc 240
aatccgagct gatccaactc gcaaaccact tgagaagaag gaagtcaaga agaagaggtg 300
gaaccgcgcc aagctttcct tgtctgaaag gaagaacacc atcaaccaaa agaaggcaac 360
ttatctcaag aaagtggaag ctggagaaat cgaataagtt tttatattcc tgacattacc 420
cattaaaggt ttcgttttaa cctaaaaaaa aaaaaaaaaa aaaaaaaaaa 470
<210> 153
<211> 387
<212> ДНК
<213> Lygus hesperus
<400> 153
tgtcgatggc ggtcttaaca tcccccattc caccaagagg ttccctgggt acgacagtga 60
gtctaaggaa ttcaacgctg aggtccacag gaagcacatt ttcggcattc acgtcgctga 120
ctacatgcgt cagctggctg aagaggatga cgatgcttac aagaagcagt tctcgcagta 180
tgtcaagaac ggagtcactg ctgacagcat tgaaagtatc tacaagaagg ctcacgaagc 240
aatccgagct gatccaactc gcaaaccact tgagaagaag gaagtcaaga agaagaggtg 300
gaaccgcgcc aagctttcct tgtctgaaag gaagaacacc atcaaccaaa agaaggcaac 360
ttatctcaag aaagtggaag ctggaga 387
<210> 154
<211> 745
<212> ДНК
<213> Lygus hesperus
<400> 154
gtcctacgtg tttccggaaa aacgtgcatt tcgcgtaccc ctcgtggtga tccgttttca 60
tagaaataat ccaaaatggc tcccaagggg aataatatga ttcccaatgg ccatttccac 120
aaggattggc agaggttcat caaaacctgg ttcaaccagc ctgcccgcaa gttgaggagg 180
agaaacaaga ggttggagaa ggcccaacgg ctcgcgcccc gccccgcggg acctcttcgc 240
cccgctgtca gatgtcccac cgtcaggtac cacaccaagc tacgacctgg acgtggcttc 300
accttggaag aaatcaagag agccggtctg tgcaaaggat tcgcgatgtc catcggaatc 360
gctgtcgacc ccagaagaag gaataaatcc atcgagtccc tccaactcaa tgtacagaga 420
ctcaaggagt acagggctaa gcttatcctc ttcccacaca agaatgccaa gaaactgaag 480
aagggagaag ctactgagga agagaggaag gtggccaccc aacagcccct gccagttatg 540
cccatcaagc aaccagtcat caaattcaag gctcgcgtca ttacagacga tgagaagaaa 600
tactctgcct tcaccgccct ccgcaaggga cgagcagacc aaaggttggt cggtatccgt 660
gctaagcgcg caaaggaagc cgcagaaaac gccgaagacc cctctaaagc tcctaaaaaa 720
aaaaaaaaaa aaaaaaaaaa aaaaa 745
<210> 155
<211> 527
<212> ДНК
<213> Lygus hesperus
<400> 155
aacgtgcatt tcgcgtaccc ctcgtggtga tccgttttca tagaaataat ccaaaatggc 60
tcccaagggg aataatatga ttcccaatgg ccatttccac aaggattggc agaggttcat 120
caaaacctgg ttcaaccagc ctgcccgcaa gttgaggagg agaaacaaga ggttggagaa 180
ggcccaacgg ctcgcgcccc gccccgcggg acctcttcgc cccgctgtca gatgtcccac 240
cgtcaggtac cacaccaagc tacgacctgg acgtggcttc accttggaag aaatcaagag 300
agccggtctg tgcaaaggat tcgcgatgtc catcggaatc gctgtcgacc ccagaagaag 360
gaataaatcc atcgagtccc tccaactcaa tgtacagaga ctcaaggagt acagggctaa 420
gcttatcctc ttcccacaca agaatgccaa gaaactgaag aagggagaag ctactgagga 480
agagaggaag gtggccaccc aacagcccct gccagttatg cccatca 527
<210> 156
<211> 576
<212> ДНК
<213> Lygus hesperus
<400> 156
gccttattga acgtggtcga cagaaaactc ggtttctgag ctcatctcaa catggatatc 60
gaagaaccgg ccgcggcccc tacggagccc tcggacgtca acaccgccct tcaagaggtc 120
ctcaaggccg cccttcaaca cggagtcgtc gtccacggta tccacgagtc cgccaaggcc 180
ctcgacaaga ggcaagcttt gttgtgcgtc ctcgctgaga actgcgacga gccgatgtac 240
aagaagctgg tacaagccct ctgctcagag caccacatcc ccctcgtcaa agtagattcc 300
aataagaaac tcggcgaatg gacgggcctt tgcaagatcg acaagaccgg caaatctagg 360
aaaatcgtcg gctgctcttg tgtcgtcatc aaggactggg gtgaggacac gccccacttg 420
gacctcctca aggactacat cagggacgtc ttctaagaag tttctcctca atttcctttt 480
tataatgatt taacaactga gaattaataa taaaaatgtt aaattaaaca aaaaaatctc 540
aaaactgtaa aaaaaaagaa gaaaaaaaaa aaaaaa 576
<210> 157
<211> 442
<212> ДНК
<213> Lygus hesperus
<400> 157
ccttattgaa cgtggtcgac agaaaactcg gtttctgagc tcatctcaac atggatatcg 60
aagaaccggc cgcggcccct acggagccct cggacgtcaa caccgccctt caagaggtcc 120
tcaaggccgc ccttcaacac ggagtcgtcg tccacggtat ccacgagtcc gccaaggccc 180
tcgacaagag gcaagctttg ttgtgcgtcc tcgctgagaa ctgcgacgag ccgatgtaca 240
agaagctggt acaagccctc tgctcagagc accacatccc cctcgtcaaa gtagattcca 300
ataagaaact cggcgaatgg acgggccttt gcaagatcga caagaccggc aaatctagga 360
aaatcgtcgg ctgctcttgt gtcgtcatca aggactgggg tgaggacacg ccccacttgg 420
acctcctcaa ggactacatc ag 442
<210> 158
<211> 601
<212> ДНК
<213> Lygus hesperus
<400> 158
ctttcatttg tatagtacgg acgggtagtt tagttgtgtc ggttcatcgt aattcatcgg 60
ctgaatcatg aagatgaata aattggtcac ttcctcgagg aggaagaaca ggaagaggca 120
cttcaccgcc ccatcccaca tccgtagaaa gttgatgtcg gcaccactgt ccaaagaact 180
taggcagaag tacaacgtcc gaactatgcc tgtgaggaag gacgatgaag tccaggttgt 240
acgaggacac tacaaaggcc aacaggttgg caaagtcctc caggtgtaca ggaagaagtt 300
cattatttac attgagcgga tccaaagaga aaaagccaat ggtgccagcg tttacgttgg 360
cattcacccc tcaaagtgtg tgatcgtcaa attgaaggtc gacaaggata ggaaagaaat 420
ccttgacaga agatccaaag gacgtgactt ggcacttggc aaggacaagg gcaaatacac 480
cgaagacagt acgactgcta tggacacgtc ttaaattaat ttggtttatt tggttcctta 540
actccgttct tctttaataa tgactttttt aaagcaaaaa aaaaaaaaaa aaaaaaaaaa 600
a 601
<210> 159
<211> 448
<212> ДНК
<213> Lygus hesperus
<400> 159
gtacggacgg gtagtttagt tgtgtcggtt catcgtaatt catcggctga atcatgaaga 60
tgaataaatt ggtcacttcc tcgaggagga agaacaggaa gaggcacttc accgccccat 120
cccacatccg tagaaagttg atgtcggcac cactgtccaa agaacttagg cagaagtaca 180
acgtccgaac tatgcctgtg aggaaggacg atgaagtcca ggttgtacga ggacactaca 240
aaggccaaca ggttggcaaa gtcctccagg tgtacaggaa gaagttcatt atttacattg 300
agcggatcca aagagaaaaa gccaatggtg ccagcgttta cgttggcatt cacccctcaa 360
agtgtgtgat cgtcaaattg aaggtcgaca aggataggaa agaaatcctt gacagaagat 420
ccaaaggacg tgacttggca cttggcaa 448
<210> 160
<211> 456
<212> ДНК
<213> Lygus hesperus
<400> 160
ggctgttgtc ggctggtcat atcccgtttt ccacgtggtg tgtcgagtta tttttcttgt 60
aaattcgcat ttaaaatcgg atttataacc gaaattcatt atggaaaagc cagtagtttt 120
ggcccgtgtc atcaaaatcc tcggacgtac cggctcacag ggccaatgta cgcaagtgaa 180
ggtggagttc attggtgagc agaaccgaca gatcatcagg aacgtgaaag gaccagttag 240
agaaggcgac atcctcacac tcctagagtc tgaaagagaa gcgagaagac tgaggtagtg 300
ggaggtggcg atgcgttacg ttattttact tcattcaaca tttgaaaaaa accatcttcg 360
tgacaaaaaa catcttcacg caactatttg tattacctat gtttcgtaaa taaagtaacc 420
tcgttactta aaaaaaaaaa aaaaaaaaaa aaaaaa 456
<210> 161
<211> 321
<212> ДНК
<213> Lygus hesperus
<400> 161
ctgttgtcgg ctggtcatat cccgttttcc acgtggtgtg tcgagttatt tttcttgtaa 60
attcgcattt aaaatcggat ttataaccga aattcattat ggaaaagcca gtagttttgg 120
cccgtgtcat caaaatcctc ggacgtaccg gctcacaggg ccaatgtacg caagtgaagg 180
tggagttcat tggtgagcag aaccgacaga tcatcaggaa cgtgaaagga ccagttagag 240
aaggcgacat cctcacactc ctagagtctg aaagagaagc gagaagactg aggtagtggg 300
aggtggcgat gcgttacgtt a 321
<210> 162
<211> 865
<212> ДНК
<213> Lygus hesperus
<400> 162
aatcccggat tcatcgtttt attgaattgt ttttcgaagt ttctggtatt atcgttaaat 60
tagtctgtta agccctcatc cgtgatttgg caagttgttg attgttctat tttccttttt 120
ccagaaaatg gggagacgtc cagcgaggtg ttatcggtac tgtaaaaaca agccataccc 180
ccaaatcccg gttctgtcgt ggtgtccccg accccaagat caggatcttc gatctgggaa 240
agaagaaggc ccgcgtggaa gacttccccc tctgcgttca cctcgtctcc gatgagtacg 300
agcagctgtc ctccgaagcc ctcgaggcag gacgtatctg ctgcaacaag tacctcgtca 360
agaactgcgg caaggaccag ttccacatca ggatgaggct ccaccccttc cacgtcatta 420
ggatcaacaa aatgttatcg tgcgctggag ctgataggct ccagacaggg atgagaggag 480
cgttcggaaa gccgcaagga accgtcgctc gcgtccgcat cggtcagccc atcatgagcg 540
tccgctcgtc cgacaggtac aaggccgccg tcatcaaggc tctgaggaga gccaaattca 600
agttccctgg tcgccagaag atctacgttt ccaagaaatg gggcttcacc aagttcgacc 660
gcgaagagta cgagggcctt aggaacgaca acaaactagc gaatgacggc tgcaacgtca 720
aattgaggcc ggatcacgga cctttgcagg cgtggaggaa ggctcagctt gacatcgctg 780
ctggcctcta aattactttc caatggtttt ataaatcaac aaataaaact cgttttatgt 840
aaaaaaaaaa aaaaaaaaaa aaaaa 865
<210> 163
<211> 792
<212> ДНК
<213> Lygus hesperus
<400> 163
ggttcctttc tcagattttg actttgccgt gttgtctctc ccaattttcc aaaatgggga 60
gacgtccagc gaggtgttat cggtactgta aaaacaagcc ataccccaaa tcccggttct 120
gtcgtggtgt ccccgacccc aagatcagga tcttcgatct gggaaagaag aaggcccgcg 180
tggaagactt ccccctctgc gttcacctcg tctccgatga gtacgagcag ctgtcctccg 240
aagccctcga ggcaggacgt atctgctgca acaagtacct cgtcaagaac tgcggcaagg 300
accagttcca catcaggatg aggctccacc ccttccacgt cattaggatc aacaaaatgt 360
tatcgtgcgc tggagctgat aggctccaga cagggatgag aggagcattc ggaaagccgc 420
aaggaaccgt cgctcgcgtc cgcatcggtc agcccatcat gagcgtccgc tcgtccgaca 480
ggtacaaggc cgccgtcatc gaggctctga ggagagccaa attcaagttc cctggtcgcc 540
agaagatcta cgtttccaag aaatggggct tcaccaagtt cgaccgcgaa gagtacgagg 600
gccttaggaa cgacaacaaa ctagcgaatg gcggctgcaa cgtcaaattg aggccggatc 660
acggaccttt gcaggcgtgg aggaaggctc agcttgacat cgctgctggc ctctaaatta 720
ctttccaatg gttttataaa tcaacaaata aaactcgttt tatctaaaaa aaaaaaaaaa 780
aaaaaaaaaa aa 792
<210> 164
<211> 645
<212> ДНК
<213> Lygus hesperus
<400> 164
agccctcatc cgtgatttgg caagttgttg attgttctat tttccttttt ccagaaaatg 60
gggagacgtc cagcgaggtg ttatcggtac tgtaaaaaca agccataccc ccaaatcccg 120
gttctgtcgt ggtgtccccg accccaagat caggatcttc gatctgggaa agaagaaggc 180
ccgcgtggaa gacttccccc tctgcgttca cctcgtctcc gatgagtacg agcagctgtc 240
ctccgaagcc ctcgaggcag gacgtatctg ctgcaacaag tacctcgtca agaactgcgg 300
caaggaccag ttccacatca ggatgaggct ccaccccttc cacgtcatta ggatcaacaa 360
aatgttatcg tgcgctggag ctgataggct ccagacaggg atgagaggag cgttcggaaa 420
gccgcaagga accgtcgctc gcgtccgcat cggtcagccc atcatgagcg tccgctcgtc 480
cgacaggtac aaggccgccg tcatcaaggc tctgaggaga gccaaattca agttccctgg 540
tcgccagaag atctacgttt ccaagaaatg gggcttcacc aagttcgacc gcgaagagta 600
cgagggcctt aggaacgaca acaaactagc gaatgacggc tgcaa 645
<210> 165
<211> 619
<212> ДНК
<213> Lygus hesperus
<400> 165
gctttaccga ttccgttctt gtttagtcca cgtttctctg ctcattcgtg cagattttaa 60
aacatgacca actccaaagg ttatcgtcgc ggaacgaggg atctcttctc gaggcccttc 120
cgtcaccatg gtgtcatccc actctcaacg tacatgaaag tataccgagt aggagacatc 180
gtatctatca aaggtaatgg agcagtgcaa aaaggtatgc cccacaaagt ttaccacggc 240
aagaccggac gagtctacaa tgttacacct cgcgcccttg gtgttattgt caacaagagg 300
gttcgtggaa aaatccttcc caagaggatc aacatcagga ttgaacacgt caaccacagt 360
aaatgcagag aagatttctt gaagcgagtg cgagaaaatg aaaggctccg caaattcgcc 420
aaagaaactg gcaccagggt tgaactcaaa agacagcctg ctcagccacg ccctgcacac 480
tttgtacaag ctaaagaagt cccagagctg ctggccccca taccttacga gttcatcgct 540
taaaaaattt tcaattccat cttaacttta tatatttgaa taaaattgtg ttctcaaaaa 600
aaaaaaaaaa aaaaaaaaa 619
<210> 166
<211> 461
<212> ДНК
<213> Lygus hesperus
<400> 166
acgtttctct gctcattcgt gcagatttta aaacatgacc aactccaaag gttatcgtcg 60
cggaacgagg gatctcttct cgaggccctt ccgtcaccat ggtgtcatcc cactctcaac 120
gtacatgaaa gtataccgag taggagacat cgtatctatc aaaggtaatg gagcagtgca 180
aaaaggtatg ccccacaaag tttaccacgg caagaccgga cgagtctaca atgttacacc 240
tcgcgccctt ggtgttattg tcaacaagag ggttcgtgga aaaatccttc ccaagaggat 300
caacatcagg attgaacacg tcaaccacag taaatgcaga gaagatttct tgaagcgagt 360
gcgagaaaat gaaaggctcc gcaaattcgc caaagaaact ggcaccaggg ttgaactcaa 420
aagacagcct gctcagccac gccctgcaca ctttgtacaa g 461
<210> 167
<211> 481
<212> ДНК
<213> Lygus hesperus
<400> 167
caacgtacat gaaagtatac cgagtaggag acatcgtatc tatcaaaggt aatggagcag 60
tgcaaaaagg tatgccccac aaagtttacc acggcaagac cggacgagtc tataatgtta 120
cacctcgcgc ccttggtgtt attgtcaaca agagggttcg tggaaaaatc cttcccaaga 180
ggatcaacat caggattgaa cacgtcaacc acagtaaatg cagagaagat ttcttgaagc 240
gagtgcgaga aaatgagagg ctccgcaaat tcgccaaaga aactggcacc agggttgaac 300
tcaaaagaca gcctgctcag ccacgccctg cacactttgt acaagctaaa gaagtcccag 360
agctgctggc ccccatacct tacgagttca tcgcttaaac aattttcaat tccatcttaa 420
ctttatatat ttgaataaaa ttgtgttccc taaaaaaaaa aaaaaaaaaa aaaaaaaaaa 480
a 481
<210> 168
<211> 747
<212> ДНК
<213> Lygus hesperus
<400> 168
gcataaatat atagggcgat tgatttagcg gccgcgaatt cgcccttaag cagtggtatc 60
aacgcagagg gggggtcttc tctcccggtt ttcttcttgc ccgaatcgtc catcctgatg 120
ttggggtcac tgtcaccacg accatacccc aatttggggt atggcttggt tgtcccctac 180
ccataaatcc tgattggaca tctccccatt atgaaagact gcgagaaaca cccctgcccc 240
cggctttaaa cccacggcta aggggggatt cgcgggcggc aaatttcatt cggcccatag 300
tgagtcgtat tacaattcac tgggcgtcct ttttacacct tcggaccggg aaaaacctgg 360
cggttaccca aaatccgtta tttgccacat ccccctttac tccactgggt tatataacaa 420
agaggcccct tccaatgtcc tttcccaaaa gtgcgcagcc ctatactaat ggcctttaaa 480
ggaaccccta ttaaaaaaaa aacccttaac cacaggttgg tgatgtaacc aaggaaaata 540
atgaacacac cgggccaaag aaggtgatac ccctggtctt ggcgaccgcc tgtcaaatct 600
tcctcccgga acgaaacccg tagtggcatc gaggaataac cttgcgcatc atagactcca 660
aatggccact gtggccgctc tcgattcatg gaagaaatga gatgacccct accccgcgca 720
aaaggattca gaaccaatac cagaatc 747
<210> 169
<211> 1052
<212> ДНК
<213> Lygus hesperus
<220>
<221> иной_признак
<222> (662)..(665)
<223> n представляет собой a, c, g или t
<400> 169
ggttttcttc ttgcccgaat cgtccatcct gatgttgggg tcactgtcac cacgaccata 60
ccccaatttg gggtatggct tggttgtccc ctacccataa atcctgattg gacatctccc 120
cattatgaaa gactgcgaga aacacccctg cccccggctt taaacccacg gctaaggggg 180
gattcgcggg cggcaaattt cattcggccc atagtgagtc gtattacaat tcactgggcg 240
tcctttttac accttcggac cgggaaaaac ctggcggtta cccaaaatcc gttatttgcc 300
acatccccct ttactccact gggttatata acaaagaggc cccttccaat gtcctttccc 360
aaaagtgcgc agccctatac taatggcctt taaaggaacc cctattaaaa aaaaaaccct 420
taaccacagg ttggtgatgt aaccaaggaa aataatgaac acaccgggcc aaagaaggtg 480
atacccctgg tcttggcgac cgcctgtcaa atcttcctcc cggaacgaaa cccgtagtgg 540
catcgaggaa taaccttgcg catcatagac tccaaatggc cactgtggcc gctctcgatt 600
catggaagaa atgagatgac ccctaccccg cgcaaaagga ttcagaacca ataccagaat 660
cnnnntagca aaacggctat ttcccggttc tttgtcggat tcttttgcca gggccatgcc 720
ttttcccgga atggaaggcg ggctgtttga gaaacgcatt aaatgggatt agtccattca 780
taggccaccc aaggaaacca ctttaatttc gggttggtag gttgagagaa atggtgaggg 840
gtaacaattt tacaccggga accgtttatg cccagaatta ccccagcttc gaattaaccc 900
cccctaaagg ggatagttcc gccgggttaa aagaaattcg ccttaaacca gtgttttaaa 960
gcaggagaca gaagtgtttc tcgcaagctt tcaaaatggg gagatgtcca atcaggattt 1020
atgggtaggg tacaaccaag ccgaacccca aa 1052
<210> 170
<211> 555
<212> ДНК
<213> Lygus hesperus
<400> 170
tagcaaaacg gctatttccc ggttctttgt cggattcttt tgccagggcc atgccttttc 60
ccggaatgga aggcgggctg tttgagaaac gcattaaatg ggattagtcc attcataggc 120
cacccaagga aaccacttta atttcgggtt ggtaggttga gagaaatggt gaggggtaac 180
aattttacac cgggaaccgt ttatgcccag aattacccca gcttcgaatt aaccccccct 240
aaaggggata gttccgccgg gttaaaagaa attcgcctta aaccagtgtt ttaaagcagg 300
agacagaagt gtttctcgca agctttcaaa atggggagat gtccaatcag gatttatggg 360
tagggtacaa ccaagccgaa ccccaaatcc ctgttctgtc gtggtgacag tgaccccaag 420
atctggatgt tcgttttggg aaagaagaaa accgggaggg accacttcct cctctgcgtt 480
gataccactg cttaagggcg aattcgttta aacctgcagg actagtccct tagtgagggt 540
aatctagcag cccac 555
<210> 171
<211> 1052
<212> ДНК
<213> Lygus hesperus
<220>
<221> иной_признак
<222> (662)..(665)
<223> n представляет собой a, c, g или t
<400> 171
ggttttcttc ttgcccgaat cgtccatcct gatgttgggg tcactgtcac cacgaccata 60
ccccaatttg gggtatggct tggttgtccc ctacccataa atcctgattg gacatctccc 120
cattatgaaa gactgcgaga aacacccctg cccccggctt taaacccacg gctaaggggg 180
gattcgcggg cggcaaattt cattcggccc atagtgagtc gtattacaat tcactgggcg 240
tcctttttac accttcggac cgggaaaaac ctggcggtta cccaaaatcc gttatttgcc 300
acatccccct ttactccact gggttatata acaaagaggc cccttccaat gtcctttccc 360
aaaagtgcgc agccctatac taatggcctt taaaggaacc cctattaaaa aaaaaaccct 420
taaccacagg ttggtgatgt aaccaaggaa aataatgaac acaccgggcc aaagaaggtg 480
atacccctgg tcttggcgac cgcctgtcaa atcttcctcc cggaacgaaa cccgtagtgg 540
catcgaggaa taaccttgcg catcatagac tccaaatggc cactgtggcc gctctcgatt 600
catggaagaa atgagatgac ccctaccccg cgcaaaagga ttcagaacca ataccagaat 660
cnnnntagca aaacggctat ttcccggttc tttgtcggat tcttttgcca gggccatgcc 720
ttttcccgga atggaaggcg ggctgtttga gaaacgcatt aaatgggatt agtccattca 780
taggccaccc aaggaaacca ctttaatttc gggttggtag gttgagagaa atggtgaggg 840
gtaacaattt tacaccggga accgtttatg cccagaatta ccccagcttc gaattaaccc 900
cccctaaagg ggatagttcc gccgggttaa aagaaattcg ccttaaacca gtgttttaaa 960
gcaggagaca gaagtgtttc tcgcaagctt tcaaaatggg gagatgtcca atcaggattt 1020
atgggtaggg tacaaccaag ccgaacccca aa 1052
<210> 172
<211> 1175
<212> ДНК
<213> Lygus hesperus
<400> 172
ctcagcgaga tccctaagac aacgcctgcc acgtgggaga atatcggaca cgcctcccca 60
gagtgcggaa aggggaacgg cgttccgtat cggtcaaggt gcaagcttcg gaaccggagg 120
acgaccgttg caaggtgcaa ggggcaggta tcttgtattt tcattgtgcg tgtcgacatc 180
taccaaactg agacttggag ttcgatattt tgacgatggg gccgggggcc ggaggcaaaa 240
cgacaaacac aggcaccgtg accgtgttcc ggtccctggc ctgcgttgcc ttacgttcac 300
atcttgttct tgcgctttct ctggttttac gataacccta ctacgagttt agtagagccg 360
atcccgtagc cgaagccaaa gcccaagcgc tccgtatccg agaacgcgga agagcacgaa 420
ctccccaaac ccctccgccc ctcccccgcg cgtatccgaa acacaaatgc agcgggcagt 480
acaggttttg gaaggggacg cgggcagtga gcgcaatgca agtaaatgtg attagctcat 540
ggctacgcag ccctgctttt tcagtttcgg ttcggatcgt tagggggtgt gggattggga 600
gcggattcaa tctggacagg aaacagctat gaccaaggtc acgccaagct ctgaattaac 660
cctcaggaaa gggactagtc cggcaggttg aaacgaactc gcccctaagc agtggtatca 720
gagcacagtg gttttttttt tttgtttttt ttcgtagaaa aaaatatgta ttaagtcaat 780
taattaaatc attggttttc tggcttcaca acaggtggca cgtgctgtgc tcggagaaat 840
ttatgaacta tgttctgttc ttcaatgagg aaagatgaga tgatccattc tcagacacat 900
tcagacagag gacaccaccg taagccctat ccacagtctg tccacgtaag gggatcgtgt 960
ccccttccat gggcagagca gggagagggc cgtaagcttg ttcttgcgtc atcaacatgt 1020
gggggtaatg ttggtcatag cgatgttcgg tacacaagag aaccacctgg tgtaatcatt 1080
acagcacagc aatactctgt gttttgtaag ataacaaaaa aggtacttaa gacgctgaac 1140
cattttctac gatcggaaaa caaaaaaaaa gaaaa 1175
<210> 173
<211> 1023
<212> ДНК
<213> Lygus hesperus
<400> 173
tcagcgagat ccctaagaca acgcctgcca cgtgggagaa tatcggacac gcctccccag 60
agtgcggaaa ggggaacggc gttccgtatc ggtcaaggtg caagcttcgg aaccggagga 120
cgaccgttgc aaggtgcaag gggcaggtat cttgtatttt cattgtgcgt gtcgacatct 180
accaaactga gacttggagt tcgatatttt gacgatgggg ccgggggccg gaggcaaaac 240
gacaaacaca ggcaccgtga ccgtgttccg gtccctggcc tgcgttgcct tacgttcaca 300
tcttgttctt gcgctttctc tggttttacg ataaccctac tacgagttta gtagagccga 360
tcccgtagcc gaagccaaag cccaagcgct ccgtatccga gaacgcggaa gagcacgaac 420
tccccaaacc cctccgcccc tcccccgcgc gtatccgaaa cacaaatgca gcgggcagta 480
caggttttgg aaggggacgc gggcagtgag cgcaatgcaa gtaaatgtga ttagctcatg 540
gctacgcagc cctgcttttt cagtttcggt tcggatcgtt agggggtgtg ggattgggag 600
cggattcaat ctggacagga aacagctatg accaaggtca cgccaagctc tgaattaacc 660
ctcaggaaag ggactagtcc ggcaggttga aacgaactcg cccctaagca gtggtatcag 720
agcacagtgg tttttttttt ttgttttttt tcgtagaaaa aaatatgtat taagtcaatt 780
aattaaatca ttggttttct ggcttcacaa caggtggcac gtgctgtgct cggagaaatt 840
tatgaactat gttctgttct tcaatgagga aagatgagat gatccattct cagacacatt 900
cagacagagg acaccaccgt aagccctatc cacagtctgt ccacgtaagg ggatcgtgtc 960
cccttccatg ggcagagcag ggagagggcc gtaagcttgt tcttgcgtca tcaacatgtg 1020
ggg 1023
<210> 174
<211> 454
<212> ДНК
<213> leptinotarsa decemlineata
<400> 174
ccaagaaggc caagaagggg tttatgaccc ctgagaggaa gaagaaactt aggttattgc 60
tgagaaagaa agcagcagaa gaactgaaaa aagaacaaga acgcaaagct gccgaaagga 120
gacgtattat tgaagagaga tgcggaaaac caaaactcat tgatgaggca aatgaagagc 180
aggtgaggaa ctattgcaag ttatatcacg gtagaatagc taaactggag gaccagaaat 240
ttgatttgga ataccttgtc aaaaagaaag acatggagat cgccgaattg aacagtcaag 300
tcaacgacct caggggtaaa ttcgtcaaac ccactctcaa gaaagtatcc aaatacgaga 360
acaaatttgc taaactccaa aagaaagcag cagaattcaa tttccgtaat caactgaaag 420
ttgtaaagaa gaaggagttc accctggagg agga 454
<210> 175
<211> 431
<212> ДНК
<213> leptinotarsa decemlineata
<400> 175
ggtttatgac ccctgagagg aagaagaaac ttaggttatt gctgagaaag aaagcagcag 60
aagaactgaa aaaagaacaa gaacgcaaag ctgccgaaag gagacgtatt attgaagaga 120
gatgcggaaa accaaaactc attgatgagg caaatgaaga gcaggtgagg aactattgca 180
agttatatca cggtagaata gctaaactgg aggaccagaa atttgatttg gaataccttg 240
tcaaaaagaa agacatggag atcgccgaat tgaacagtca agtcaacgac ctcaggggta 300
aattcgtcaa acccactctc aagaaagtat ccaaatacga gaacaaattt gctaaactcc 360
aaaagaaagc agcagaattc aatttccgta atcaactgaa agttgtaaag aagaaggagt 420
tcaccctgga g 431
<210> 176
<211> 888
<212> ДНК
<213> leptinotarsa decemlineata
<400> 176
agcagtggta tcaacgcaga gtacgcgggg acatcgagga gaagaggcaa cgcctcgaag 60
aggctgaaaa gaaacgccag gccatgatgc aggccctcaa ggaccagaac aagaacaagg 120
ggcccaactt caccatcacc aagagggatg cttcatctaa cctttctgcc gctcagttgg 180
aacgcaacaa gaccaaggag caactcgagg aagagaagaa aatttccctt tccatccgca 240
tcaagccctt ggtcgttgat ggtctgggcg tagataaact ccgtctgaaa gcacaagaac 300
tttgggaatg catcgtcaag ttggagactg aaaagtacga cttggaagag aggcagaaac 360
gtcaagacta cgatctcaaa gagctgaaag aaagacagaa acaacagctg agacacaaag 420
ccttgaagaa gggtctagac ccagaagccc taaccggcaa atacccgcct aaaatccaag 480
tagcctccaa atatgaacgt cgtgttgaca cgaggtcgta tggagacaaa aagaagctat 540
tcgaaggggg attagaagaa atcattaaag agaccaatga aaagagctgg aaagagaaat 600
ttggacagtt cgattccaga caaaaggcaa gacttcccaa gtggttcggt gaacgtcctg 660
gcaaaaaacc tggagatccc gaaactccag aaggcgagga ggagggcaaa caagtcattg 720
atgaggatga cgacctcaag gagcctgtaa tcgaagctga aattgaagaa gaggaggaag 780
aagaggaagt cgaggtcgat gaagaagaag aggatgacga agaagaagaa gaagaagagt 840
gaatgccaaa ggcagaagat aatcatgaaa tcaacattag ataacgtc 888
<210> 177
<211> 404
<212> ДНК
<213> leptinotarsa decemlineata
<400> 177
caaggaccag aacaagaaca aggggcccaa cttcaccatc accaagaggg atgcttcatc 60
taacctttct gccgctcagt tggaacgcaa caagaccaag gagcaactcg aggaagagaa 120
gaaaatttcc ctttccatcc gcatcaagcc cttggtcgtt gatggtctgg gcgtagataa 180
actccgtctg aaagcacaag aactttggga atgcatcgtc aagttggaga ctgaaaagta 240
cgacttggaa gagaggcaga aacgtcaaga ctacgatctc aaagagctga aagaaagaca 300
gaaacaacag ctgagacaca aagccttgaa gaagggtcta gacccagaag ccctaaccgg 360
caaatacccg cctaaaatcc aagtagcctc caaatatgaa cgtc 404
<210> 178
<211> 1155
<212> ДНК
<213> leptinotarsa decemlineata
<400> 178
gctcttcaga atgaacttga agaatctcgt acactgttgg aacaagctga ccgtgcccgt 60
cgccaagcag aacaagaatt gggagatgct cacgaacaat tgaatgatct tggtgcacag 120
aatggttctc tgtctgccgc caagaggaaa ctggaaactg aactccaaac tctccattcc 180
gatcttgatg aacttctcaa tgaagccaag aactctgagg agaaggctaa gaaagccatg 240
gtcgatgcag ctcgtcttgc agatgaactg agagcagaac aagatcatgc acaaactcag 300
gagaaacttc gtaaagcctt agaatcacaa atcaaggacc ttcaagttcg tctcgacgag 360
gctgaagcta acgccctcaa aggaggtaag aaagcaatcg ctaaacttga acaacgcgtc 420
agggaattgg agaatgagtt agatggtgaa caaagacgac acgccgatgc tcaaaagaat 480
ttgagaaagt ccgaacgtcg catcaaggag ctcagcctcc aagctgaaga agaccgtaag 540
aaccacgaaa aaatgcaaga cttagtcgac aaacttcaac agaaaatcaa gacccacaag 600
aggcaaatag aagaagctga agaaatagcg gctctcaatt tggccaaatt ccgtaaagca 660
caacaggaat tggaagaagc agaagagcgt gcagaccttg ctgaacaagc aattgtcaaa 720
ttccgtacca agggacgttc tggatcagca gctaggggag ccagccctgc gcctcagcga 780
cagcgtccca cattcggaat gggagattca cttggaggtg ccttccctcc aaggttcgat 840
cttgcacccg actttgaatg aatctgacat tgtgttataa gtgtaaggtg aacattctat 900
cgcagtgtaa atatcatccc aatgcgaatc aattctacat tcagtttaag tcattctatc 960
tctcaaaata ataatagtgt catccattct cactatcaaa tcaagacaag agatgatgat 1020
cagagaacac gtatcacatc tacagcaaac cctcagtcct cggcatctct gataatattt 1080
tcaattatcg agattgatga tatcgggtgt tgaatgctga tgaatagaag gcgccctatg 1140
gaaataagag agaag 1155
<210> 179
<211> 523
<212> ДНК
<213> leptinotarsa decemlineata
<400> 179
gaatctcgta cactgttgga acaagctgac cgtgcccgtc gccaagcaga acaagaattg 60
ggagatgctc acgaacaatt gaatgatctt ggtgcacaga atggttctct gtctgccgcc 120
aagaggaaac tggaaactga actccaaact ctccattccg atcttgatga acttctcaat 180
gaagccaaga actctgagga gaaggctaag aaagccatgg tcgatgcagc tcgtcttgca 240
gatgaactga gagcagaaca agatcatgca caaactcagg agaaacttcg taaagcctta 300
gaatcacaaa tcaaggacct tcaagttcgt ctcgacgagg ctgaagctaa cgccctcaaa 360
ggaggtaaga aagcaatcgc taaacttgaa caacgcgtca gggaattgga gaatgagtta 420
gatggtgaac aaagacgaca cgccgatgct caaaagaatt tgagaaagtc cgaacgtcgc 480
atcaaggagc tcagcctcca agctgaagaa gaccgtaaga acc 523
<210> 180
<211> 865
<212> ДНК
<213> nilaparvata lugens
<400> 180
ctaggagtat ctcctacgta attcggtgct tgagccaact gcagctactc acttttttcc 60
aggttcagtg gtagggacgc aaacacagct aaaatggcgg acgatgaggc aaagaaggca 120
aagcaggcgg aaatcgaccg caagagagcc gaggtccgca agcggatgga ggaagcctcc 180
aaggccaaga aggccaagaa aggtttcatg acgcctgaca gaaagaagaa gctcaggttg 240
ttgctgagga aaaaggctgc tgaggaattg aagaaggaac aggagaggaa agccgcggaa 300
aggagaagga tcatcgagga gaggtgtggc aaggctgttg atctcgatga cggaagtgaa 360
gagaaagtca aggcaacttt aaaaacctat cacgacagaa ttggaaaatt ggaggatgaa 420
aaatttgacc tggaatatat tgtaaaaaag aaagacttcg agatcgctga cctcaacagc 480
caggtgaatg acctccgtgg taaatttgtc aagccaacct tgaaaaaagt ctccaaatat 540
gagaacaaat tcgccaagct ccagaagaaa gcagctgaat tcaatttcag aaatcagctc 600
aaagttgtca agaagaagga attcaccttg gaagaagaag acaaggagcc gaagaaatcg 660
gagaaagccg aatggcagaa gaaatgaact cacatcacct cttcataata ttgtcccaca 720
cttctacaac cttcatcaaa taacttttat tcgagtaaac ttactgttac taacaaaatt 780
acaaaaccaa actcttatca tcaacgtagg caatgtgctc aacttatttc ttaaacatat 840
tgtccagcta tttattgaaa ttaaa 865
<210> 181
<211> 269
<212> ДНК
<213> nilaparvata lugens
<400> 181
aagaagaagc tcaggttgtt gctgaggaaa aaggctgctg aggaattgaa gaaggaacag 60
gagaggaaag ccgcggaaag gagaaggatc atcgaggaga ggtgtggcaa ggctgttgat 120
ctcgatgacg gaagtgaaga gaaagtcaag gcaactttaa aaacctatca cgacagaatt 180
ggaaaattgg aggatgaaaa atttgacctg gaatatattg taaaaaagaa agacttcgag 240
atcgctgacc tcaacagcca ggtgaatga 269
<210> 182
<211> 553
<212> ДНК
<213> nilaparvata lugens
<400> 182
aatgatggcg gctctcaagg accagagcaa atcgaaagga cccaacttca ccgtaaacaa 60
gaaaacagac ttgaacatga cgtcagctca aatggaaagg aacaagacta aggagcagct 120
ggaggaggag aagaagatct ctctgtcgtt ccgcatcaag ccgttggcca tcgagaacat 180
gagcatcaac gcactgcgcg ccaaggccca ggaactgtgg gactgcatcg tcaagctcga 240
aactgagaag tacgatctgg aggaacgcca gaagaggcag gactacgatc tcaaagaatt 300
gaaagaaaga caaaagcaac agctgaggca taaagccctc aaaaaaggtc tagaccctga 360
ggctctcaca ggaaagtacc caccaaaaat ccaagttgcc tccaaatatg aaagacgtgt 420
agatacaagg tcatacgacg acaagaagaa gctcttcgaa ggtggctggg acacattaac 480
atcagaaacc aatgagaaaa tatggaagag cagaaacgat cagttttcaa atcgtagcaa 540
ggctaaactg cca 553
<210> 183
<211> 470
<212> ДНК
<213> nilaparvata lugens
<400> 183
atgatggcgg ctctcaagga ccagagcaaa tcgaaaggac ccaacttcac cgtaaacaag 60
aaaacagact tgaacatgac gtcagctcaa atggaaagga acaagactaa ggagcagctg 120
gaggaggaga agaagatctc tctgtcgttc cgcatcaagc cgttggccat cgagaacatg 180
agcatcaacg cactgcgcgc caaggcccag gaactgtggg actgcatcgt caagctcgaa 240
actgagaagt acgatctgga ggaacgccag aagaggcagg actacgatct caaagaattg 300
aaagaaagac aaaagcaaca gctgaggcat aaagccctca aaaaaggtct agaccctgag 360
gctctcacag gaaagtaccc accaaaaatc caagttgcct ccaaatatga aagacgtgta 420
gatacaaggt catacgacga caagaagaag ctcttcgaag gtggctggga 470
<210> 184
<211> 367
<212> ДНК
<213> nilaparvata lugens
<400> 184
tgccttcgac cgtgaaaggt ctggaagtat cccaacagac atggtcgccg acatcctcag 60
gctcatggga cagcctttca acaagaagat cctcgacgaa ctcattgagg aagttgatgc 120
tgacaaatct ggccgtcttg agtttgacga attcgtgact ctggccgcca aattcattgt 180
tgaggaagac gatgaggcaa tgcagaagga attgaaggaa gctttcagat tatacgacaa 240
ggaaggtaac ggctacatcc ccacatcatg tctgaaggaa atcttaaggg aacttgacga 300
tcagctgaca aacgaggaac tcaacatgat gattgatgag atcgactctg acggatcagg 360
aactgtt 367
<210> 185
<211> 204
<212> ДНК
<213> nilaparvata lugens
<400> 185
acatcctcag gctcatggga cagcctttca acaagaagat cctcgacgaa cttattgagg 60
aggttgatgc tgacaagtct ggccgtctag agtttgacga attcgtgact ctggccgcca 120
aattcattgt tgaggaagac gatgaggcaa tgcagaagga attgaaggaa gctttcagat 180
tatacgacaa ggaaggtaac ggct 204
<210> 186
<211> 221
<212> ДНК
<213> nilaparvata lugens
<400> 186
cgtaaaaact ctgaccggca agaccatcac cttggaagtg gagccttccg ataccattga 60
aaacgtgaag gccaagatcc aagacaagga gggaattcct cccgaccagc agagacttat 120
cttcgctgga aagcaactgg aggatggcag aaccctgtcc gactacaaca tccaaaaaga 180
atctacactc cacttggttc tcagacttcg tggtggaact a 221
<210> 187
<211> 221
<212> ДНК
<213> nilaparvata lugens
<400> 187
cgtaaaaact ctgaccggca agaccatcac cttggaagtg gagccttccg ataccattga 60
aaacgtgaag gccaagatcc aagacaagga gggaattcct cccgaccagc agagacttat 120
cttcgctgga aagcaactgg aggatggcag aaccctgtcc gactacaaca tccaaaaaga 180
atctacactc cacttggttc tcagacttcg tggtggaact a 221
<210> 188
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 188
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 189
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 189
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 190
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 190
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 191
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 191
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 192
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 192
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 193
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 193
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 194
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 194
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 195
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 195
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 196
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 196
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 197
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 197
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 198
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 198
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 199
<211> 759
<212> ДНК
<213> Acyrthosiphon pisum
<400> 199
atggccgacg atgaagctaa gaaagcaaaa caggcggaaa tcgaccgcaa gagggccgaa 60
gtgcgcaagc gtatggaaga ggcgtccaag gccaagaagg ccaagaaggg tttcatgacc 120
ccagacagaa agaagaaact ccgtctgttg ttgaaaaaaa aggcggccga agagttgaag 180
aaagaacaag aacgcaaagc tgccgaacga aggcggatca tcgaagagcg gtgcggacaa 240
ccgaagaaca tcgacgacgc cggcgaagag gagcttgcgg aaatctgcga agaactatgg 300
aaacgggttt acaccgtaga gggcataaaa tttgacttgg aaagggatat caggatgaaa 360
gttttcgaga tcagcgaatt gaacagccaa gtcaatgact tacgaggaaa attcgtcaaa 420
ccaacattga agaaggtttc caaatacgaa aacaaattcg caaaactcca aaagaaagcg 480
gcggagttca acttcagaaa ccaactgaaa gtagtgaaga aaaaggagtt caccttggaa 540
gaagaagaca aagagaaaaa acccgattgg tccaaaaagg gagacgaaaa gaagggcgaa 600
ggagaagacg gcgacggtac cgaagacgaa aagaccgacg acggtttgac caccgaaggc 660
gaatcggtcg cgggcgatct aacggacgcg acggaagacg cgcagagcga caacgagata 720
ctcgaaccag aacccgtggt tgaacccgaa ccagaacca 759
<210> 200
<211> 541
<212> ДНК
<213> Acyrthosiphon pisum
<400> 200
cggtaatgcg atgcggtaag aagaaggtat ggttggatcc aaacgaaata aatgaaattg 60
ccaacaccaa ttccagacaa aatattcgta agttgatcaa agatggtttg atcattaaaa 120
agccagtagc tgtacactct agggctcgtg cacgtaaaaa tgcagatgcc agaagaaaag 180
gtcgtcattg tggttttggt aaaaggaagg gtactgctaa tgctcgaaca cctcaaaaag 240
acctttgggt gaaaagaatg cgagtattaa ggcggttgct taaaaaatac cgtgaagcaa 300
agaaaattga caaccatctt taccatcagt tatacatgaa ggctaagggt aatgttttca 360
agaacaaacg tgtattgatg gagttcatcc acaaaaagaa ggcagagaag gcccgtgcca 420
agatgttgag tgatcaagct gaagctagac gtcaaaaggt taaggaagct aggaaacgta 480
aagaagcaag atttttacaa aataggaagg aacttttggc tgcatacgcc cgagaagatg 540
a 541
<210> 201
<211> 541
<212> ДНК
<213> Acyrthosiphon pisum
<400> 201
cggtaatgcg atgcggtaag aagaaggtat ggttggatcc aaacgaaata aatgaaattg 60
ccaacaccaa ttccagacaa aatattcgta agttgatcaa agatggtttg atcattaaaa 120
agccagtagc tgtacactct agggctcgtg cacgtaaaaa tgcagatgcc agaagaaaag 180
gtcgtcattg tggttttggt aaaaggaagg gtactgctaa tgctcgaaca cctcaaaaag 240
acctttgggt gaaaagaatg cgagtattaa ggcggttgct taaaaaatac cgtgaagcaa 300
agaaaattga caaccatctt taccatcagt tatacatgaa ggctaagggt aatgttttca 360
agaacaaacg tgtattgatg gagttcatcc acaaaaagaa ggcagagaag gcccgtgcca 420
agatgttgag tgatcaagct gaagctagac gtcaaaaggt taaggaagct aggaaacgta 480
aagaagcaag atttttacaa aataggaagg aacttttggc tgcatacgcc cgagaagatg 540
a 541
<210> 202
<211> 823
<212> ДНК
<213> Acyrthosiphon pisum
<400> 202
gttgtagtcg gaaagggtac gtccgtcttc aagttgtttt ccggcaaaga tcaaacgttg 60
ttggtctggt gggatacctt ctttgtcttg gatcttggct tttacatttt caatggaatc 120
agatgattcc acctccaatg taatggtctt tccagtgagg gtctttacaa agatttgcat 180
accaccacgg agacgcaaca ctaagtgaag ggtagattct ttctggatgt tgtagtcaga 240
aagtgtgcgt ccgtcttcaa gttgctttcc ggcaaagatc aaacgttgtt ggtcaggtgg 300
aataccttct ttgtcttgga tcttagcttt tacattttca atggaatctg atgactcaac 360
ttccaatgta atggtctttc cagtgagggt ctttacaaag atttgcatac caccacggag 420
acgcaacact aagtgaaggg tagattcttt ctggatgttg tagtcggaaa gggtacgtcc 480
gtcttcaagt tgctttccgg caaagatcaa acgttgttgg tctggtggga taccttcttt 540
gtcttggatc ttggctttta cattttcaat ggaatcagat gattccacct ccaatgtaat 600
ggtctttcca gtgagggtct ttacaaagat ttgcatacca ccacggagac gcaacactaa 660
gtgaagggta gattctttct ggatgttgta gtcggaaagg gtacgtccgt cttcaagttg 720
ctttccagca aagatcaaac gttgctggtc tggtgggata ccttccttgt cttggatctt 780
ggccttaaca ttttcaatgg aatctgatga ctcaacttcc aaa 823
<210> 203
<211> 823
<212> ДНК
<213> Acyrthosiphon pisum
<400> 203
gttgtagtcg gaaagggtac gtccgtcttc aagttgtttt ccggcaaaga tcaaacgttg 60
ttggtctggt gggatacctt ctttgtcttg gatcttggct tttacatttt caatggaatc 120
agatgattcc acctccaatg taatggtctt tccagtgagg gtctttacaa agatttgcat 180
accaccacgg agacgcaaca ctaagtgaag ggtagattct ttctggatgt tgtagtcaga 240
aagtgtgcgt ccgtcttcaa gttgctttcc ggcaaagatc aaacgttgtt ggtcaggtgg 300
aataccttct ttgtcttgga tcttagcttt tacattttca atggaatctg atgactcaac 360
ttccaatgta atggtctttc cagtgagggt ctttacaaag atttgcatac caccacggag 420
acgcaacact aagtgaaggg tagattcttt ctggatgttg tagtcggaaa gggtacgtcc 480
gtcttcaagt tgctttccgg caaagatcaa acgttgttgg tctggtggga taccttcttt 540
gtcttggatc ttggctttta cattttcaat ggaatcagat gattccacct ccaatgtaat 600
ggtctttcca gtgagggtct ttacaaagat ttgcatacca ccacggagac gcaacactaa 660
gtgaagggta gattctttct ggatgttgta gtcggaaagg gtacgtccgt cttcaagttg 720
ctttccagca aagatcaaac gttgctggtc tggtgggata ccttccttgt cttggatctt 780
ggccttaaca ttttcaatgg aatctgatga ctcaacttcc aaa 823
<210> 204
<211> 172
<212> ДНК
<213> Acyrthosiphon pisum
<400> 204
aagacttgct tcatcctact gcaattgaag aacgcaggaa acacaaatta aagcgccttg 60
ttcaacaccc aaactctttt ttcatggatg tcaaatgccc tggatgttat aaaattacaa 120
ctgtattcag tcacgctcag agtgtagtta tatgtaccgg atgttccaca at 172
<210> 205
<211> 172
<212> ДНК
<213> Acyrthosiphon pisum
<400> 205
aagacttgct tcatcctact gcaattgaag aacgcaggaa acacaaatta aagcgccttg 60
ttcaacaccc aaactctttt ttcatggatg tcaaatgccc tggatgttat aaaattacaa 120
ctgtattcag tcacgctcag agtgtagtta tatgtaccgg atgttccaca at 172
<210> 206
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 206
cgaaccatct gggaagcttg gaatg 25
<210> 207
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 207
gcagctggag gaagagaaac gtatc 25
<210> 208
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 208
gcgtaatacg actcactata ggcgaaccat ctgggaagct tggaatg 47
<210> 209
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 209
gcgtaatacg actcactata ggcagctgga ggaagagaaa cgtatc 46
<210> 210
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 210
agttcgagaa caccaggaag 20
<210> 211
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 211
cctgacacgt tgttccagct tg 22
<210> 212
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 212
gcgtaatacg actcactata ggaggagttc gagaacacca ggaag 45
<210> 213
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 213
gcgtaatacg actcactata ggcctgacac gttgttccag cttg 44
<210> 214
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 214
gcaggcgatg aagatggaga 20
<210> 215
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 215
ccacctcttt ctgcaacttc ttga 24
<210> 216
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 216
gcgtaatacg actcactata gggcaggcga tgaagatgga ga 42
<210> 217
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 217
gcgtaatacg actcactata ggccacctct ttctgcaact tcttga 46
<210> 218
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 218
cagaatccca cagaatctga cgtga 25
<210> 219
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 219
gcaagtggcg aagctcagct 20
<210> 220
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 220
gcgtaatacg actcactata ggcagaatcc cacagaatct gacgtga 47
<210> 221
<211> 41
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 221
gcgtaatacg actcactata ggcaagtggc gaagctcagc t 41
<210> 222
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 222
cgtgtttgcc atgttcgatc a 21
<210> 223
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 223
ggtacatttc gtccacgtct tca 23
<210> 224
<211> 43
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 224
gcgtaatacg actcactata ggcgtgtttg ccatgttcga tca 43
<210> 225
<211> 43
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 225
gcgtaatacg actcactata ggtacatttc gtccacgtct tca 43
<210> 226
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 226
gacttgatct tcagccgacc att 23
<210> 227
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 227
ccattgccag ttcctcaact tca 23
<210> 228
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 228
gcgtaatacg actcactata ggacttgatc ttcagccgac catt 44
<210> 229
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 229
gcgtaatacg actcactata ggccattgcc agttcctcaa cttca 45
<210> 230
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 230
cgcaatgatc tcctccagga t 21
<210> 231
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 231
ggtcatcatc tccatgaact cgtc 24
<210> 232
<211> 43
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 232
gcgtaatacg actcactata ggcgcaatga tctcctccag gat 43
<210> 233
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 233
gcgtaatacg actcactata gggtcatcat ctccatgaac tcgtc 45
<210> 234
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 234
cgtcactaat cggactggtc taacag 26
<210> 235
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 235
ggtcatcatc tccatgaact cgtc 24
<210> 236
<211> 48
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 236
gcgtaatacg actcactata ggcgtcacta atcggactgg tctaacag 48
<210> 237
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 237
gcgtaatacg actcactata gggtcatcat ctccatgaac tcgtc 45
<210> 238
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 238
ggtgaaggag ggtgcctgct cag 23
<210> 239
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 239
cagggtgaat agaacgaggt actcg 25
<210> 240
<211> 40
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 240
aatacgactc actatagggc gctatgaaat tccaagcaca 40
<210> 241
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 241
gcgtaatacg actcactata ggcagggtga atagaacgag gtactcg 47
<210> 242
<211> 29
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 242
ctcaacgaag gtcttgtcag tggctttgg 29
<210> 243
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 243
ttcgcctggc ttcttcgtga 20
<210> 244
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 244
gcgtaatacg actcactata ggccacgccg acttaattca ttcc 44
<210> 245
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 245
gcgtaatacg actcactata ggttcgcctg gcttcttcgt ga 42
<210> 246
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 246
tgtcgatggc ggtcttaaca tc 22
<210> 247
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 247
tctccagctt ccactttctt gaga 24
<210> 248
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 248
gcgtaatacg actcactata ggtgtcgatg gcggtcttaa catc 44
<210> 249
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 249
gcgtaatacg actcactata ggtctccagc ttccactttc ttgaga 46
<210> 250
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 250
aacgtgcatt tcgcgtaccc 20
<210> 251
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 251
tgatgggcat aactggcagg 20
<210> 252
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 252
gcgtaatacg actcactata ggaacgtgca tttcgcgtac cc 42
<210> 253
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 253
gcgtaatacg actcactata ggtgatgggc ataactggca gg 42
<210> 254
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 254
ccttattgaa cgtggtcgac ag 22
<210> 255
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 255
ctgatgtagt ccttgaggag 20
<210> 256
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 256
gcgtaatacg actcactata ggccttattg aacgtggtcg acag 44
<210> 257
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 257
gcgtaatacg actcactata ggctgatgta gtccttgagg ag 42
<210> 258
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 258
gtacggacgg gtagtttagt tgtgtc 26
<210> 259
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 259
ttgccaagtg ccaagtcacg 20
<210> 260
<211> 48
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 260
gcgtaatacg actcactata gggtacggac gggtagttta gttgtgtc 48
<210> 261
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 261
gcgtaatacg actcactata ggttgccaag tgccaagtca cg 42
<210> 262
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 262
ctgttgtcgg ctggtcatat cc 22
<210> 263
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 263
taacgtaacg catcgccacc 20
<210> 264
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 264
gcgtaatacg actcactata ggctgttgtc ggctggtcat atcc 44
<210> 265
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 265
gcgtaatacg actcactata ggtaacgtaa cgcatcgcca cc 42
<210> 266
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 266
agccctcatc cgtgatttgg 20
<210> 267
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 267
gatccggcct caatttgacg 20
<210> 268
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 268
gcgtaatacg actcactata ggagccctca tccgtgattt gg 42
<210> 269
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 269
gcgtaatacg actcactata gggatccggc ctcaatttga cg 42
<210> 270
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 270
acgtttctct gctcattcgt gc 22
<210> 271
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 271
cttgtacaaa gtgtgcaggg 20
<210> 272
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 272
gcgtaatacg actcactata ggacgtttct ctgctcattc gtgc 44
<210> 273
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 273
gcgtaatacg actcactata ggcttgtaca aagtgtgcag gg 42
<210> 274
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 274
ggttttcttc ttgcccgaat cg 22
<210> 275
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 275
tttggggttc ggcttggttg 20
<210> 276
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 276
gcgtaatacg actcactata ggggttttct tcttgcccga atcg 44
<210> 277
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 277
gcgtaatacg actcactata ggtttggggt tcggcttggt tg 42
<210> 278
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 278
tcagcgagat ccctaagaca acg 23
<210> 279
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 279
ccccacatgt tgatgacgca 20
<210> 280
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 280
gcgtaatacg actcactata ggtcagcgag atccctaaga caacg 45
<210> 281
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 281
gcgtaatacg actcactata ggccccacat gttgatgacg ca 42
<210> 282
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 282
ggtttatgac ccctgagagg aag 23
<210> 283
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 283
ctccagggtg aactccttct tc 22
<210> 284
<211> 43
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 284
gcgtaatacg actcactata ggtttatgac ccctgagagg aag 43
<210> 285
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 285
gcgtaatacg actcactata ggctccaggg tgaactcctt cttc 44
<210> 286
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 286
caaggaccag aacaagaaca aggg 24
<210> 287
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 287
gacgttcata tttggaggct acttgg 26
<210> 288
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 288
gcgtaatacg actcactata ggcaaggacc agaacaagaa caaggg 46
<210> 289
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 289
gcgtaatacg actcactata ggacgttcat atttggaggc tacttgg 47
<210> 290
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 290
aatctcgtac actgttggaa caagc 25
<210> 291
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 291
ggttcttacg gtcttcttca gcttg 25
<210> 292
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 292
gcgtaatacg actcactata ggaatctcgt acactgttgg aacaagc 47
<210> 293
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 293
gcgtaatacg actcactata ggttcttacg gtcttcttca gcttg 45
<210> 294
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 294
aagaagaagc tcaggttgtt gc 22
<210> 295
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 295
tcattcacct ggctgttgag 20
<210> 296
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 296
gcgtaatacg actcactata ggaagaagaa gctcaggttg ttgc 44
<210> 297
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 297
gcgtaatacg actcactata ggtcattcac ctggctgttg ag 42
<210> 298
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 298
acatcctcag gctcatggga 20
<210> 299
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 299
agccgttacc ttccttgtcg 20
<210> 300
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 300
gcgtaatacg actcactata ggacatcctc aggctcatgg ga 42
<210> 301
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 301
gcgtaatacg actcactata ggagccgtta ccttccttgt cg 42
<210> 302
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 302
acatcctcag gctcatggga 20
<210> 303
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 303
agccgttacc ttccttgtcg 20
<210> 304
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 304
gcgtaatacg actcactata ggacatcctc aggctcatgg ga 42
<210> 305
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 305
gcgtaatacg actcactata ggagccgtta ccttccttgt cg 42
<210> 306
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 306
cgtaaaaact ctgaccggca agac 24
<210> 307
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 307
tagttccacc acgaagtctg agaacc 26
<210> 308
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 308
gcgtaatacg actcactata ggcgtaaaaa ctctgaccgg caagac 46
<210> 309
<211> 48
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 309
gcgtaatacg actcactata ggtagttcca ccacgaagtc tgagaacc 48
<210> 310
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 310
atggccgacg atgaagctaa g 21
<210> 311
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 311
tggttgtggt tctggttcgg 20
<210> 312
<211> 43
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 312
gcgtaatacg actcactata ggatggccga cgatgaagct aag 43
<210> 313
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 313
gcgtaatacg actcactata ggtggttctg gttcgggttc aa 42
<210> 314
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 314
cggtaatgcg atgcggtaag 20
<210> 315
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 315
tcatcttctc gggcgtatgc 20
<210> 316
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 316
gcgtaatacg actcactata ggcggtaatg cgatgcggta ag 42
<210> 317
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 317
gcgtaatacg actcactata ggtcatcttc tcgggcgtat gc 42
<210> 318
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 318
tttggaagtt gagtcatcag attcc 25
<210> 319
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 319
gttgtagtcg gaaagggtac gtcc 24
<210> 320
<211> 47
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 320
gcgtaatacg actcactata ggtttggaag ttgagtcatc agattcc 47
<210> 321
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 321
gcgtaatacg actcactata gggttgtagt cggaaagggt acgtcc 46
<210> 322
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 322
aagacttgct tcatcctact gca 23
<210> 323
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 323
attgtggaac atccggtaca 20
<210> 324
<211> 45
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 324
gcgtaatacg actcactata ggaagacttg cttcatccta ctgca 45
<210> 325
<211> 42
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 325
gcgtaatacg actcactata ggattgtgga acatccggta ca 42
<210> 326
<211> 424
<212> БЕЛОК
<213> Lygus hesperus
<400> 326
Met Ile Pro Pro Thr Ser Arg Pro Gln Val Thr Val Tyr Ser Asp Lys
1 5 10 15
Asn Glu Ala Thr Gly Thr Leu Leu Asn Leu Pro Ala Val Phe Asn Ala
20 25 30
Pro Ile Arg Pro Asp Val Val Asn Phe Val His Gln Asn Val Ala Lys
35 40 45
Asn His Arg Gln Pro Tyr Cys Val Ser Ala Gln Ala Gly His Gln Thr
50 55 60
Ser Ala Glu Ser Trp Gly Thr Gly Arg Ala Val Ala Arg Ile Pro Arg
65 70 75 80
Val Arg Gly Gly Gly Thr His Arg Ser Gly Gln Gly Ala Phe Gly Asn
85 90 95
Met Cys Arg Gly Gly Arg Met Phe Ala Pro Thr Arg Pro Trp Arg Arg
100 105 110
Trp His Arg Lys Ile Asn Val Asn Gln Lys Arg Tyr Ala Val Val Ser
115 120 125
Ala Ile Ala Ala Ser Gly Val Pro Ala Leu Val Met Ser Lys Gly His
130 135 140
Met Val Gln Ser Val Pro Glu Phe Pro Leu Val Val Ser Asp Lys Val
145 150 155 160
Gln Glu Tyr Thr Lys Thr Lys Gln Ala Val Ile Phe Leu His Arg Ile
165 170 175
Lys Ala Trp Gln Asp Ile Gln Lys Val Tyr Lys Ser Lys Arg Phe Arg
180 185 190
Ala Gly Lys Gly Lys Met Arg Asn Arg Arg Arg Ile Gln Arg Arg Gly
195 200 205
Pro Leu Ile Ile Tyr Asp Gln Asp Gln Gly Leu Asn Arg Ala Phe Arg
210 215 220
Asn Ile Pro Gly Val Asp Leu Ile Glu Val Ser Arg Leu Asn Leu Leu
225 230 235 240
Lys Leu Ala Pro Gly Gly His Ile Gly Arg Phe Val Ile Trp Thr Gln
245 250 255
Ser Ala Phe Glu Lys Leu Asp Ala Leu Tyr Gly Thr Trp Lys Lys Lys
260 265 270
Ser Thr Leu Lys Ala Gly Tyr Asn Leu Pro Met Pro Lys Met Ala Asn
275 280 285
Thr Asp Leu Ser Arg Leu Phe Lys Ala Pro Glu Ile Lys Ala Val Leu
290 295 300
Arg Asn Pro Lys Lys Thr Ile Val Arg Arg Val Arg Lys Leu Asn Pro
305 310 315 320
Leu Arg Asn Thr Arg Ala Met Leu Arg Leu Asn Pro Tyr Ala Ala Val
325 330 335
Leu Lys Arg Lys Ala Ile Leu Asp Gln Arg Lys Leu Lys Leu Gln Lys
340 345 350
Leu Val Glu Ala Ala Lys Lys Gly Asp Thr Lys Leu Ser Pro Arg Val
355 360 365
Glu Arg His Leu Lys Met Ile Glu Arg Arg Lys Ala Leu Ile Lys Lys
370 375 380
Ala Lys Ala Ala Lys Pro Lys Lys Pro Lys Thr Ala Lys Lys Pro Lys
385 390 395 400
Thr Ala Glu Lys Ala Pro Ala Pro Ala Lys Lys Ala Ala Ala Pro Lys
405 410 415
Lys Ala Thr Thr Pro Ala Lys Lys
420
<210> 327
<211> 242
<212> БЕЛОК
<213> Lygus hesperus
<400> 327
Met Ala Asn Ala Lys Pro Ile Ser Lys Lys Lys Lys Phe Val Ser Asp
1 5 10 15
Gly Val Phe Lys Ala Glu Leu Asn Glu Phe Leu Thr Arg Glu Leu Ala
20 25 30
Glu Glu Gly Tyr Ser Gly Val Glu Val Arg Val Thr Pro Asn Lys Thr
35 40 45
Glu Ile Ile Ile Met Ala Thr Arg Thr Gln Ser Val Leu Gly Asp Lys
50 55 60
Gly Arg Arg Ile Arg Glu Leu Thr Ser Val Val Gln Lys Arg Phe Asn
65 70 75 80
Phe Lys Pro Gln Thr Leu Asp Leu Tyr Ala Glu Lys Val Ala Thr Arg
85 90 95
Gly Leu Cys Ala Ile Ala Gln Ala Glu Ser Leu Arg Tyr Lys Leu Ile
100 105 110
Gly Gly Leu Ala Val Arg Gly Ala Cys Tyr Gly Val Leu Arg Phe Ile
115 120 125
Met Glu Asn Gly Ala Lys Gly Cys Glu Val Val Val Ser Gly Lys Leu
130 135 140
Arg Gly Gln Arg Ala Lys Ser Met Lys Phe Val Asp Gly Leu Met Ile
145 150 155 160
His Ser Gly Asp Pro Cys Asn Glu Tyr Val Asp Thr Ala Thr Arg His
165 170 175
Val Leu Leu Arg Gln Gly Val Leu Gly Ile Lys Val Lys Ile Met Leu
180 185 190
Pro Trp Asp Val Thr Gly Lys Asn Gly Pro Lys Asn Pro Leu Pro Asp
195 200 205
His Val Ser Val Leu Leu Pro Lys Glu Glu Leu Pro Asn Leu Ala Val
210 215 220
Ser Val Pro Gly Ser Asp Ile Lys Pro Lys Pro Glu Val Pro Ala Pro
225 230 235 240
Ala Leu
<210> 328
<211> 262
<212> БЕЛОК
<213> Lygus hesperus
<400> 328
Met Ala Val Gly Lys Asn Lys Gly Leu Ser Lys Gly Gly Lys Lys Gly
1 5 10 15
Val Lys Lys Lys Val Val Asp Pro Phe Thr Arg Lys Asp Trp Tyr Asp
20 25 30
Val Lys Ala Pro Ser Met Phe Lys Lys Arg Gln Val Gly Lys Thr Leu
35 40 45
Val Asn Arg Thr Gln Gly Thr Lys Ile Ala Ser Glu Gly Leu Lys Gly
50 55 60
Arg Val Phe Glu Val Ser Leu Ala Asp Ile Gln Glu Asp Thr Asp Ala
65 70 75 80
Glu Arg Ser Phe Arg Lys Phe Arg Leu Ile Ala Glu Asp Val Gln Ala
85 90 95
Arg Asn Val Leu Thr Asn Phe His Gly Met Asp Leu Thr Thr Asp Lys
100 105 110
Leu Arg Ser Met Val Lys Lys Trp Gln Thr Leu Ile Glu Ala Asn Val
115 120 125
Asp Val Lys Thr Thr Asp Gly Tyr Leu Leu Arg Val Phe Cys Ile Gly
130 135 140
Phe Thr Asn Lys Asp Gln Leu Ser Gln Arg Lys Thr Cys Tyr Ala Gln
145 150 155 160
His Asn Gln Val Arg Glu Ile Arg Lys Lys Met Val Lys Asn Ile Ser
165 170 175
Asp Ser Ile Ser Ser Cys Asp Leu Arg Ser Val Val Asn Lys Leu Ile
180 185 190
Pro Asp Ser Ile Ala Lys Asp Ile Glu Lys Asn Cys Gln Gly Ile Tyr
195 200 205
Pro Leu His Asp Val Tyr Ile Arg Lys Val Lys Val Leu Lys Lys Pro
210 215 220
Arg Phe Glu Leu Ser Lys Leu Leu Glu Leu His Val Asp Gly Lys Gly
225 230 235 240
Ile Asp Glu Pro Gly Ala Lys Val Thr Arg Thr Asp Ala Tyr Glu Pro
245 250 255
Pro Val Gln Glu Ser Val
260
<210> 329
<211> 152
<212> БЕЛОК
<213> Lygus hesperus
<400> 329
Met Ser Leu Met Leu Pro Glu Lys Phe Gln His Ile Leu Arg Ile Met
1 5 10 15
Gly Thr Asn Ile Asp Gly Lys Arg Lys Val Met Phe Ala Met Thr Ala
20 25 30
Ile Lys Gly Val Gly Arg Arg Tyr Ala Asn Ile Val Leu Lys Lys Ala
35 40 45
Asp Val Asn Leu Asp Lys Arg Ala Gly Glu Cys Ser Glu Glu Glu Val
50 55 60
Glu Lys Ile Val Thr Ile Met Gln Asn Pro Arg Gln Tyr Lys Ile Pro
65 70 75 80
Asn Trp Phe Leu Asn Arg Gln Lys Asp Thr Val Glu Gly Lys Tyr Ser
85 90 95
Gln Leu Thr Ser Ser Leu Leu Asp Ser Lys Leu Arg Asp Asp Leu Glu
100 105 110
Arg Leu Lys Lys Ile Arg Ala His Arg Gly Met Arg His Tyr Trp Gly
115 120 125
Leu Arg Val Arg Gly Gln His Thr Lys Thr Thr Gly Arg Arg Gly Arg
130 135 140
Thr Val Gly Val Ser Lys Lys Lys
145 150
<210> 330
<211> 381
<212> БЕЛОК
<213> Lygus hesperus
<400> 330
Met Ser Asp Glu Glu Tyr Ser Glu Ser Glu Glu Glu Thr Gln Pro Glu
1 5 10 15
Pro Gln Lys Lys Pro Glu Ala Glu Gly Gly Gly Asp Pro Glu Phe Val
20 25 30
Lys Arg Lys Glu Ala Gln Thr Ser Ala Leu Asp Glu Gln Leu Lys Asp
35 40 45
Tyr Ile Ala Glu Trp Arg Lys Gln Arg Ala Arg Glu Glu Glu Asp Leu
50 55 60
Lys Lys Leu Lys Glu Lys Gln Ala Lys Arg Lys Val Ala Arg Ala Glu
65 70 75 80
Glu Glu Lys Arg Leu Ala Glu Lys Lys Lys Gln Glu Glu Glu Arg Arg
85 90 95
Val Arg Glu Ala Glu Glu Lys Lys Gln Arg Glu Ile Glu Glu Lys Arg
100 105 110
Arg Arg Leu Glu Glu Ala Glu Lys Lys Arg Gln Ala Met Met Ala Ala
115 120 125
Leu Lys Asp Gln Ser Lys Thr Lys Gly Pro Asn Phe Val Val Asn Lys
130 135 140
Lys Ala Glu Thr Leu Gly Met Ser Ser Ala Gln Ile Glu Arg Asn Lys
145 150 155 160
Thr Lys Glu Gln Leu Glu Glu Glu Lys Arg Ile Ser Leu Ser Ile Arg
165 170 175
Leu Lys Pro Leu Ala Ile Glu Asn Met Ser Ile Asp Arg Leu Arg Ile
180 185 190
Lys Ala Gln Glu Leu Trp Glu Ala Ile Val Lys Leu Glu Thr Glu Lys
195 200 205
Tyr Asp Leu Glu Glu Arg Gln Lys Arg Gln Asp Tyr Asp Leu Lys Glu
210 215 220
Leu Lys Glu Arg Gln Lys Gln Gln Leu Arg His Lys Ala Leu Lys Lys
225 230 235 240
Gly Leu Asp Pro Glu Ala Leu Thr Gly Lys Tyr Pro Pro Lys Ile Gln
245 250 255
Val Ala Ser Lys Tyr Glu Arg Arg Val Asp Thr Arg Ser Tyr Asp Asp
260 265 270
Lys Lys Lys Leu Phe Glu Gly Gly Ile Leu Glu Arg Tyr Lys Glu Leu
275 280 285
Ile Glu Lys Val Trp Thr Glu Lys Val Asp Gln Phe Gly Ser Arg Ala
290 295 300
His Ser Lys Leu Pro Arg Trp Phe Gly Glu Arg Pro Gly Lys Lys Lys
305 310 315 320
Asp Ala Pro Glu Ser Pro Glu Glu Glu Glu Val Lys Val Glu Asp Glu
325 330 335
Pro Glu Ala Glu Pro Ser Phe Met Leu Asp Glu Glu Glu Glu Glu Ala
340 345 350
Glu Glu Glu Glu Ala Glu Glu Glu Glu Glu Ala Glu Glu Glu Glu Glu
355 360 365
Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu
370 375 380
<210> 331
<211> 1689
<212> БЕЛОК
<213> Lygus hesperus
<400> 331
Ser Gly Lys Leu Ala Gly Ala Asp Ile Glu Thr Tyr Leu Leu Glu Lys
1 5 10 15
Ala Arg Val Ile Ser Gln Gln Thr Leu Glu Arg Ser Tyr His Ile Phe
20 25 30
Tyr Gln Met Met Ser Gly Ala Val Lys Gly Val Lys Glu Met Cys Leu
35 40 45
Leu Val Asp Asp Ile Tyr Thr Tyr Asn Phe Ile Ser Gln Gly Lys Val
50 55 60
Ser Ile Ala Gly Val Asp Asp Gly Glu Glu Met Val Leu Thr Asp Gln
65 70 75 80
Ala Phe Asp Ile Leu Gly Phe Thr Lys Gln Glu Lys Glu Asp Ile Tyr
85 90 95
Lys Ile Thr Ala Ala Val Ile His Met Gly Thr Met Lys Phe Lys Gln
100 105 110
Arg Gly Arg Glu Glu Gln Ala Glu Ala Asp Gly Thr Glu Glu Gly Gly
115 120 125
Lys Val Gly Val Leu Leu Gly Ile Asp Gly Asp Asp Leu Tyr Lys Asn
130 135 140
Met Cys Lys Pro Arg Ile Lys Val Gly Thr Glu Phe Val Thr Gln Gly
145 150 155 160
Lys Asn Val Asn Gln Val Ser Tyr Ser Leu Gly Ala Met Ser Lys Gly
165 170 175
Met Phe Asp Arg Leu Phe Lys Phe Leu Val Lys Lys Cys Asn Glu Thr
180 185 190
Leu Asp Thr Lys Gln Lys Arg Gln His Phe Ile Gly Val Leu Asp Ile
195 200 205
Ala Gly Phe Glu Ile Phe Asp Phe Asn Gly Phe Glu Gln Leu Cys Ile
210 215 220
Asn Phe Thr Asn Glu Lys Leu Gln Gln Phe Phe Asn His His Met Phe
225 230 235 240
Val Leu Glu Gln Glu Glu Tyr Lys Arg Glu Gly Ile Asn Trp Ala Phe
245 250 255
Ile Asp Phe Gly Met Asp Leu Leu Ala Cys Ile Glu Leu Ile Glu Lys
260 265 270
Pro Met Gly Ile Leu Ser Ile Leu Glu Glu Glu Ser Met Phe Pro Lys
275 280 285
Ala Thr Asp Lys Thr Phe Glu Asp Lys Leu Ile Thr Asn His Leu Gly
290 295 300
Lys Ser Pro Asn Phe Arg Lys Pro Ala Val Pro Lys Pro Gly Gln Gln
305 310 315 320
Ala Gly His Phe Ala Ile Ala His Tyr Ala Gly Cys Val Ser Tyr Asn
325 330 335
Ile Thr Gly Trp Leu Glu Lys Asn Lys Asp Pro Leu Asn Asp Thr Val
340 345 350
Val Asp Gln Tyr Lys Lys Gly Thr Asn Lys Leu Leu Cys Glu Ile Phe
355 360 365
Ala Asp His Pro Gly Gln Ser Gly Ala Pro Gly Gly Asp Ala Gly Gly
370 375 380
Lys Gly Gly Arg Gly Lys Lys Gly Gly Gly Phe Ala Thr Val Ser Ser
385 390 395 400
Ser Tyr Lys Glu Gln Leu Asn Asn Leu Met Thr Thr Leu Lys Ser Thr
405 410 415
Gln Pro His Phe Val Arg Cys Ile Ile Pro Asn Glu Leu Lys Gln Pro
420 425 430
Gly Val Ile Asp Ser His Leu Val Met His Gln Leu Thr Cys Asn Gly
435 440 445
Val Leu Glu Gly Ile Arg Ile Cys Arg Lys Gly Phe Pro Asn Arg Met
450 455 460
Asn Tyr Pro Asp Phe Lys Leu Arg Tyr Lys Ile Leu Asn Pro Ala Ala
465 470 475 480
Val Asp Arg Glu Ser Asp Ile Leu Lys Ala Ala Gly Leu Val Leu Glu
485 490 495
Ser Thr Gly Leu Asp Pro Asp Met Tyr Arg Leu Gly His Thr Lys Val
500 505 510
Phe Phe Arg Ala Gly Val Leu Gly Gln Leu Glu Glu Leu Arg Asp Asp
515 520 525
Arg Leu Ser Lys Ile Ile Gly Trp Met Gln Ala Phe Met Arg Gly Tyr
530 535 540
Leu Val Arg Lys Glu Tyr Lys Lys Leu Gln Glu Gln Arg Leu Ala Leu
545 550 555 560
Gln Val Val Gln Arg Asn Leu Arg Arg Tyr Leu Gln Leu Arg Thr Trp
565 570 575
Pro Trp Trp Lys Met Trp Ser Arg Val Lys Pro Leu Leu Asn Val Ala
580 585 590
Asn Val Glu Glu Glu Met Arg Lys Leu Glu Glu Leu Val Ala Glu Thr
595 600 605
Gln Ala Ala Leu Glu Lys Glu Glu Lys Leu Arg Lys Glu Ala Glu Ala
610 615 620
Leu Asn Ala Lys Leu Leu Gln Glu Lys Thr Asp Leu Leu Arg Asn Leu
625 630 635 640
Glu Gly Glu Lys Gly Ser Ile Ser Gly Ile Gln Glu Arg Cys Ala Lys
645 650 655
Leu Gln Ala Gln Lys Ala Asp Leu Glu Ser Gln Leu Met Asp Thr Gln
660 665 670
Glu Arg Leu Gln Asn Glu Glu Asp Ala Arg Asn Gln Leu Phe Gln Gln
675 680 685
Lys Lys Lys Leu Glu Gln Glu Ala Ala Ala Leu Lys Lys Asp Ile Glu
690 695 700
Asp Leu Glu Leu Ser Asn Gln Lys Thr Asp Gln Asp Lys Ala Ser Lys
705 710 715 720
Glu His Gln Ile Arg Asn Leu Asn Asp Glu Ile Ala His Gln Asp Asp
725 730 735
Leu Ile Asn Lys Leu Asn Lys Glu Lys Lys Ile Gln Ser Glu Leu Asn
740 745 750
Gln Lys Thr Ala Glu Glu Leu Gln Ala Ala Glu Asp Lys Ile Asn His
755 760 765
Leu Thr Lys Val Lys Val Lys Leu Glu Gln Thr Leu Asp Glu Leu Glu
770 775 780
Asp Thr Leu Glu Arg Glu Lys Lys Leu Arg Gly Asp Val Glu Lys Ala
785 790 795 800
Lys Arg Lys Thr Glu Gly Asp Leu Lys Leu Thr Gln Glu Ala Val Ala
805 810 815
Asp Leu Glu Arg Asn Lys Lys Glu Leu Glu Gln Thr Ile Gln Arg Lys
820 825 830
Asp Lys Glu Ile Ala Ser Leu Thr Ala Lys Leu Glu Asp Glu Gln Ser
835 840 845
Ile Val Asn Lys Thr Gly Lys Gln Ile Lys Glu Leu Gln Ser Arg Ile
850 855 860
Glu Glu Leu Glu Glu Glu Val Glu Ala Glu Arg Gln Ala Arg Gly Lys
865 870 875 880
Ala Glu Lys Gln Arg Ala Asp Leu Ala Arg Glu Leu Glu Glu Leu Gly
885 890 895
Glu Arg Leu Glu Glu Ala Gly Gly Ala Thr Ser Ala Gln Ile Glu Leu
900 905 910
Asn Lys Lys Arg Glu Ala Glu Met Ser Lys Leu Arg Arg Asp Leu Glu
915 920 925
Glu Ala Asn Ile Gln His Glu Gly Thr Leu Ala Asn Leu Arg Lys Lys
930 935 940
His Asn Asp Ala Val Ser Glu Met Gly Asp Gln Ile Asp Gln Leu Asn
945 950 955 960
Lys Leu Lys Thr Lys Val Glu Lys Glu Lys Ser Gln Tyr Leu Gly Glu
965 970 975
Leu Asn Asp Val Arg Ala Ser Ile Asp His Leu Thr Asn Glu Lys Ala
980 985 990
Ala Thr Glu Lys Val Ala Lys Gln Leu Gln His Gln Ile Asn Glu Val
995 1000 1005
Gln Gly Lys Leu Asp Glu Ala Asn Arg Thr Leu Asn Asp Phe Asp
1010 1015 1020
Ala Ala Lys Lys Lys Leu Ser Ile Glu Asn Ser Asp Leu Leu Arg
1025 1030 1035
Gln Leu Glu Glu Ala Glu Ser Gln Val Ser Gln Leu Ser Lys Ile
1040 1045 1050
Lys Ile Ser Leu Thr Thr Gln Leu Glu Asp Thr Lys Arg Leu Ala
1055 1060 1065
Asp Glu Glu Ala Arg Glu Arg Ala Thr Leu Leu Gly Lys Phe Arg
1070 1075 1080
Asn Leu Glu His Asp Leu Asp Asn Leu Arg Glu Gln Val Glu Glu
1085 1090 1095
Glu Ala Glu Ala Lys Ala Asp Ile Gln Arg Gln Leu Ser Lys Ala
1100 1105 1110
Asn Ala Glu Ala Gln Leu Trp Arg Ser Lys Tyr Glu Ser Glu Gly
1115 1120 1125
Val Ala Arg Ala Glu Glu Leu Glu Glu Ala Lys Arg Lys Leu Gln
1130 1135 1140
Ala Arg Leu Ala Glu Ala Glu Glu Thr Ile Glu Ser Leu Asn Gln
1145 1150 1155
Lys Val Ile Ala Leu Glu Lys Thr Lys Gln Arg Leu Ala Thr Glu
1160 1165 1170
Val Glu Asp Leu Gln Leu Glu Val Asp Arg Ala Asn Ala Ile Ala
1175 1180 1185
Asn Ala Ala Glu Lys Lys Ala Lys Ala Ile Asp Lys Ile Ile Gly
1190 1195 1200
Glu Trp Lys Leu Lys Val Asp Asp Leu Ala Ala Glu Leu Asp Ala
1205 1210 1215
Ser Gln Lys Glu Cys Arg Asn Tyr Ser Thr Glu Leu Phe Arg Leu
1220 1225 1230
Lys Gly Ala Tyr Glu Glu Gly Gln Glu Gln Leu Glu Ala Val Arg
1235 1240 1245
Arg Glu Asn Lys Asn Leu Ala Asp Glu Val Lys Asp Leu Leu Asp
1250 1255 1260
Gln Ile Gly Glu Gly Gly Arg Asn Ile His Glu Ile Glu Lys Gln
1265 1270 1275
Arg Lys Arg Leu Glu Val Glu Lys Asp Glu Leu Gln Ala Ala Leu
1280 1285 1290
Glu Glu Ala Glu Ala Ala Leu Glu Gln Glu Glu Asn Lys Val Leu
1295 1300 1305
Arg Ala Gln Leu Glu Leu Ser Gln Val Arg Gln Glu Ile Asp Arg
1310 1315 1320
Arg Ile Gln Glu Lys Glu Glu Glu Phe Glu Asn Thr Arg Lys Asn
1325 1330 1335
His Gln Arg Ala Leu Asp Ser Met Gln Ala Ser Leu Glu Ala Glu
1340 1345 1350
Ala Lys Gly Lys Ala Glu Ala Leu Arg Met Lys Lys Lys Leu Glu
1355 1360 1365
Ala Asp Ile Asn Glu Leu Glu Ile Ala Leu Asp His Ala Asn Lys
1370 1375 1380
Ala Asn Ala Glu Ala Gln Lys Thr Ile Lys Lys Tyr Gln Gln Gln
1385 1390 1395
Leu Lys Asp Val Gln Thr Ala Leu Glu Glu Glu Gln Arg Ala Arg
1400 1405 1410
Asp Asp Ala Arg Glu Gln Leu Gly Ile Ala Glu Arg Arg Ala Asn
1415 1420 1425
Ala Leu Gly Asn Glu Leu Glu Glu Ser Arg Thr Leu Leu Glu Gln
1430 1435 1440
Ala Asp Arg Gly Arg Arg Gln Ala Glu Gln Glu Leu Gly Asp Ala
1445 1450 1455
His Glu Gln Ile Asn Glu Leu Ala Ala Gln Ala Thr Ser Ala Ser
1460 1465 1470
Ala Ala Lys Arg Lys Leu Glu Gly Glu Leu Gln Thr Leu His Ala
1475 1480 1485
Asp Leu Asp Glu Leu Leu Asn Glu Ala Lys Asn Ser Glu Glu Lys
1490 1495 1500
Ala Lys Lys Ala Met Val Asp Ala Ala Arg Leu Ala Asp Glu Leu
1505 1510 1515
Arg Ala Glu Gln Asp His Ala Gln Thr Gln Glu Lys Leu Arg Lys
1520 1525 1530
Ala Leu Glu Thr Gln Ile Lys Glu Leu Gln Val Arg Leu Asp Glu
1535 1540 1545
Ala Glu Asn Asn Ala Leu Lys Gly Gly Lys Lys Ala Ile Ala Lys
1550 1555 1560
Leu Glu Gln Arg Val Arg Glu Leu Glu Asn Glu Leu Asp Gly Glu
1565 1570 1575
Gln Arg Arg His Ala Asp Ala Gln Lys Asn Leu Arg Lys Ser Glu
1580 1585 1590
Arg Arg Ile Lys Glu Leu Ser Phe Gln Ser Asp Glu Asp Arg Lys
1595 1600 1605
Asn His Glu Arg Met Gln Asp Leu Val Asp Lys Leu Gln Gln Lys
1610 1615 1620
Ile Lys Thr Tyr Lys Arg Gln Ile Glu Glu Ala Glu Glu Ile Ala
1625 1630 1635
Ala Leu Asn Leu Ala Lys Phe Arg Lys Ala Gln Gln Glu Leu Glu
1640 1645 1650
Glu Ala Glu Glu Arg Ala Asp Leu Ala Glu Gln Ala Val Ser Lys
1655 1660 1665
Phe Arg Thr Lys Gly Gly Arg Ala Gly Ser Ala Ala Arg Ala Met
1670 1675 1680
Ser Pro Val Gly Gln Lys
1685
<210> 332
<211> 256
<212> БЕЛОК
<213> Lygus hesperus
<400> 332
Asp Ala Ile Lys Lys Lys Met Gln Ala Met Lys Met Glu Lys Asp Thr
1 5 10 15
Ala Met Asp Lys Ala Asp Thr Cys Glu Gly Gln Ala Lys Asp Ala Asn
20 25 30
Thr Arg Ala Asp Lys Ile Leu Glu Asp Val Arg Asp Leu Gln Lys Lys
35 40 45
Leu Asn Gln Val Glu Ser Asp Leu Glu Arg Thr Lys Arg Glu Leu Glu
50 55 60
Thr Lys Thr Thr Glu Leu Glu Glu Lys Glu Lys Ala Asn Thr Asn Ala
65 70 75 80
Glu Ser Glu Val Ala Ser Leu Asn Arg Lys Val Gln Met Val Glu Glu
85 90 95
Asp Leu Glu Arg Ser Glu Glu Arg Ser Gly Thr Ala Gln Gln Lys Leu
100 105 110
Ser Glu Ala Ser His Ala Ala Asp Glu Ala Ser Arg Met Cys Lys Val
115 120 125
Leu Glu Asn Arg Ser Gln Gln Asp Glu Glu Arg Met Asp Gln Leu Thr
130 135 140
Asn Gln Leu Lys Glu Ala Arg Leu Leu Ala Glu Asp Ala Asp Gly Lys
145 150 155 160
Ser Asp Glu Val Ser Arg Lys Leu Ala Phe Val Glu Asp Glu Leu Glu
165 170 175
Val Ala Glu Asp Arg Val Lys Ser Gly Asp Ser Lys Ile Met Glu Leu
180 185 190
Glu Glu Glu Leu Lys Val Val Gly Asn Ser Leu Lys Ser Leu Glu Val
195 200 205
Ser Glu Glu Lys Ala Asn Gln Arg Val Glu Glu Tyr Lys Arg Gln Ile
210 215 220
Lys Gln Leu Thr Val Lys Leu Lys Glu Ala Glu Ala Arg Ala Glu Phe
225 230 235 240
Ala Glu Lys Thr Val Lys Lys Leu Gln Lys Glu Val Asp Arg Leu Glu
245 250 255
<210> 333
<211> 85
<212> БЕЛОК
<213> Lygus hesperus
<400> 333
Arg Ala Leu Gly Gln Asn Pro Thr Glu Ser Asp Val Lys Lys Phe Thr
1 5 10 15
His Gln His Lys Pro Asp Glu Arg Ile Ser Phe Glu Val Phe Leu Pro
20 25 30
Ile Tyr Gln Ala Ile Ser Lys Gly Arg Thr Ser Asp Thr Ala Glu Asp
35 40 45
Phe Ile Glu Gly Leu Arg His Phe Asp Lys Asp Gly Asn Gly Phe Ile
50 55 60
Ser Thr Ala Glu Leu Arg His Leu Leu Thr Thr Leu Gly Glu Lys Leu
65 70 75 80
Thr Asp Asp Glu Val
85
<210> 334
<211> 174
<212> БЕЛОК
<213> Lygus hesperus
<400> 334
Met Ser Ser Arg Lys Thr Ala Gly Arg Arg Ala Thr Thr Lys Lys Arg
1 5 10 15
Ala Gln Arg Ala Thr Ser Asn Val Phe Ala Met Phe Asp Gln Ala Gln
20 25 30
Ile Gln Glu Phe Lys Glu Ala Phe Asn Met Ile Asp Gln Asn Arg Asp
35 40 45
Gly Phe Val Asp Lys Glu Asp Leu His Asp Met Leu Ala Ser Leu Gly
50 55 60
Lys Asn Pro Ser Asp Glu Tyr Leu Glu Gly Met Met Asn Glu Ala Pro
65 70 75 80
Gly Pro Ile Asn Phe Thr Met Phe Leu Thr Leu Phe Gly Glu Arg Leu
85 90 95
Gln Gly Thr Asp Pro Glu Glu Val Ile Lys Asn Ala Phe Gly Cys Phe
100 105 110
Asp Glu Asp Asn Asn Gly Phe Ile Asn Glu Glu Arg Leu Arg Glu Leu
115 120 125
Leu Thr Ser Met Gly Asp Arg Phe Thr Asp Glu Asp Val Asp Glu Met
130 135 140
Tyr Arg Glu Ala Pro Ile Lys Asn Gly Met Phe Asp Tyr Ile Glu Phe
145 150 155 160
Thr Arg Ile Leu Lys His Gly Ala Lys Asp Lys Asp Glu Gln
165 170
<210> 335
<211> 1881
<212> БЕЛОК
<213> Lygus hesperus
<400> 335
Asp Leu Thr Cys Leu Asn Glu Ala Ser Val Leu His Asn Ile Lys Asp
1 5 10 15
Arg Tyr Tyr Ser Gly Leu Ile Tyr Thr Tyr Ser Gly Leu Phe Cys Val
20 25 30
Val Val Asn Pro Tyr Lys Lys Leu Pro Ile Tyr Thr Glu Arg Ile Met
35 40 45
Glu Lys Tyr Lys Gly Val Lys Arg His Asp Leu Pro Pro His Val Phe
50 55 60
Ala Ile Thr Asp Thr Ala Tyr Arg Ser Met Leu Gln Asp Arg Glu Asp
65 70 75 80
Gln Ser Ile Leu Cys Thr Gly Glu Ser Gly Ala Gly Lys Thr Glu Asn
85 90 95
Thr Lys Lys Val Ile Gln Tyr Leu Ala Tyr Val Ala Ala Ser Lys Pro
100 105 110
Lys Ser Ser Ala Ser Pro His Thr Ala Gln Ser Gln Ala Leu Ile Ile
115 120 125
Gly Glu Leu Glu Gln Gln Leu Leu Gln Ala Asn Pro Ile Leu Glu Ala
130 135 140
Phe Gly Asn Ala Lys Thr Val Lys Asn Asp Asn Ser Ser Arg Phe Gly
145 150 155 160
Lys Phe Ile Arg Ile Asn Phe Asp Ala Ser Gly Tyr Ile Ala Gly Ala
165 170 175
Asn Ile Glu Thr Tyr Leu Leu Glu Lys Ser Arg Ala Ile Arg Gln Ala
180 185 190
Lys Asp Glu Arg Thr Phe His Ile Phe Tyr Gln Leu Leu Ala Gly Ala
195 200 205
Ser Ala Glu Gln Arg Lys Glu Phe Ile Leu Glu Asp Pro Lys Asn Tyr
210 215 220
Pro Phe Leu Ser Ser Gly Met Val Ser Val Pro Gly Val Asp Asp Gly
225 230 235 240
Val Asp Phe Gln Ala Thr Ile Ala Ser Met Ser Ile Met Gly Met Thr
245 250 255
Asn Asp Asp Leu Ser Ala Leu Phe Arg Ile Val Ser Ala Val Met Leu
260 265 270
Phe Gly Ser Met Gln Phe Lys Gln Glu Arg Asn Ser Asp Gln Ala Thr
275 280 285
Leu Pro Asp Asn Thr Val Ala Gln Lys Ile Ala His Leu Leu Gly Leu
290 295 300
Ser Ile Thr Glu Met Thr Lys Ala Phe Leu Arg Pro Arg Ile Lys Val
305 310 315 320
Gly Arg Asp Phe Val Thr Lys Ala Gln Thr Lys Glu Gln Val Glu Phe
325 330 335
Ala Val Glu Ala Ile Ser Lys Ala Cys Tyr Glu Arg Met Phe Arg Trp
340 345 350
Leu Val Asn Arg Ile Asn Arg Ser Leu Asp Arg Thr Lys Arg Gln Gly
355 360 365
Ala Ser Phe Ile Gly Ile Leu Asp Met Ala Gly Phe Glu Ile Phe Glu
370 375 380
Ile Asn Ser Phe Glu Gln Leu Cys Ile Asn Tyr Thr Asn Glu Lys Leu
385 390 395 400
Gln Gln Leu Phe Asn His Thr Met Phe Ile Leu Glu Gln Glu Glu Tyr
405 410 415
Gln Arg Glu Gly Ile Glu Trp Lys Phe Ile Asp Phe Gly Leu Asp Leu
420 425 430
Gln Pro Thr Ile Asp Leu Ile Asp Lys Pro Met Gly Val Met Ala Leu
435 440 445
Leu Asp Glu Glu Cys Trp Phe Pro Lys Ala Thr Asp Lys Thr Phe Val
450 455 460
Glu Lys Leu Val Gly Ala His Ser Val His Pro Lys Phe Ile Lys Thr
465 470 475 480
Asp Phe Arg Gly Val Ala Asp Phe Ala Val Val His Tyr Ala Gly Lys
485 490 495
Val Asp Tyr Ser Ala Ala Gln Trp Leu Met Lys Asn Met Asp Pro Leu
500 505 510
Asn Glu Asn Val Val Gln Leu Leu Gln Asn Ser Gln Asp Pro Phe Val
515 520 525
Ile His Ile Trp Lys Asp Ala Glu Ile Val Gly Met Ala His Gln Ala
530 535 540
Leu Ser Asp Thr Gln Phe Gly Ala Arg Thr Arg Lys Gly Met Phe Arg
545 550 555 560
Thr Val Ser Gln Leu Tyr Lys Asp Gln Leu Ser Lys Leu Met Ile Thr
565 570 575
Leu Arg Asn Thr Asn Pro Asn Phe Val Arg Cys Ile Leu Pro Asn His
580 585 590
Glu Lys Arg Ala Gly Lys Ile Asp Ala Pro Leu Val Leu Asp Gln Leu
595 600 605
Arg Cys Asn Gly Val Leu Glu Gly Ile Arg Ile Cys Arg Gln Gly Phe
610 615 620
Pro Asn Arg Ile Pro Phe Gln Glu Phe Arg Gln Arg Tyr Glu Leu Leu
625 630 635 640
Thr Pro Asn Val Ile Pro Lys Gly Phe Met Asp Gly Lys Lys Ala Cys
645 650 655
Glu Lys Met Ile Asn Ala Leu Glu Leu Asp Pro Asn Leu Tyr Arg Val
660 665 670
Gly Gln Ser Lys Ile Phe Phe Arg Ala Gly Val Leu Ala His Leu Glu
675 680 685
Glu Glu Arg Asp Tyr Lys Ile Thr Asp Leu Ile Ala Asn Phe Arg Ala
690 695 700
Phe Cys Arg Gly Tyr Leu Ala Arg Arg Asn Tyr Gln Lys Arg Leu Gln
705 710 715 720
Gln Leu Asn Ala Ile Arg Ile Ile Gln Arg Asn Cys Ser Ala Tyr Leu
725 730 735
Lys Leu Arg Asn Trp Gln Trp Trp Arg Leu Tyr Thr Lys Val Lys Pro
740 745 750
Leu Leu Glu Val Thr Lys Gln Glu Glu Lys Leu Thr Gln Lys Glu Asp
755 760 765
Glu Leu Lys Gln Val Arg Glu Lys Leu Asp Asn Gln Val Arg Ser Lys
770 775 780
Glu Glu Tyr Glu Lys Arg Leu Gln Asp Ala Leu Glu Glu Lys Ala Ala
785 790 795 800
Leu Ala Glu Gln Leu Gln Ala Glu Val Glu Leu Cys Ala Glu Ala Glu
805 810 815
Glu Met Arg Ala Arg Leu Ala Val Arg Lys Gln Glu Leu Glu Glu Ile
820 825 830
Leu His Asp Leu Glu Ala Arg Ile Glu Glu Glu Glu Gln Arg Asn Thr
835 840 845
Val Leu Ile Asn Glu Lys Lys Lys Leu Thr Leu Asn Ile Ala Asp Leu
850 855 860
Glu Glu Gln Leu Glu Glu Glu Glu Gly Ala Arg Gln Lys Leu Gln Leu
865 870 875 880
Glu Lys Val Gln Ile Glu Ala Arg Leu Lys Lys Met Glu Glu Asp Leu
885 890 895
Ala Leu Ala Glu Asp Thr Asn Thr Lys Val Val Lys Glu Lys Lys Val
900 905 910
Leu Glu Glu Arg Ala Ser Asp Leu Ala Gln Thr Leu Ala Glu Glu Glu
915 920 925
Glu Lys Ala Lys His Leu Ala Lys Leu Lys Thr Lys His Glu Thr Thr
930 935 940
Ile Ala Glu Leu Glu Glu Arg Leu Leu Lys Asp Asn Gln Gln Arg Gln
945 950 955 960
Glu Met Asp Arg Asn Lys Arg Lys Ile Glu Ser Glu Val Asn Asp Leu
965 970 975
Lys Glu Gln Ile Asn Glu Lys Lys Val Gln Val Glu Glu Leu Gln Leu
980 985 990
Gln Leu Gly Lys Arg Glu Glu Glu Ile Ala Gln Ala Leu Met Arg Ile
995 1000 1005
Asp Glu Glu Gly Ala Gly Lys Ala Gln Thr Gln Lys Ala Leu Arg
1010 1015 1020
Glu Leu Glu Ser Gln Leu Ala Glu Leu Gln Glu Asp Leu Glu Ala
1025 1030 1035
Glu Lys Ala Ala Arg Ala Lys Ala Glu Lys Gln Lys Arg Asp Leu
1040 1045 1050
Asn Glu Glu Leu Glu Ser Leu Lys Asn Glu Leu Leu Asp Ser Leu
1055 1060 1065
Asp Thr Thr Ala Ala Gln Gln Glu Leu Arg Thr Lys Arg Glu His
1070 1075 1080
Glu Leu Ala Thr Leu Lys Lys Thr Leu Glu Glu Glu Thr His Ile
1085 1090 1095
His Glu Val Ser Leu Thr Glu Met Arg His Lys His Thr Gln Glu
1100 1105 1110
Val Ala Ala Leu Asn Glu Gln Leu Glu Gln Leu Lys Lys Ala Lys
1115 1120 1125
Ser Ala Leu Glu Lys Ser Lys Ala Gln Leu Glu Gly Glu Ala Ala
1130 1135 1140
Glu Leu Ala Asn Glu Leu Glu Thr Ala Gly Thr Ser Lys Gly Glu
1145 1150 1155
Ser Glu Arg Lys Arg Lys Gln Ala Glu Ser Ser Leu Gln Glu Leu
1160 1165 1170
Ser Ser Arg Leu Leu Glu Met Glu Arg Thr Lys Ala Glu Leu Gln
1175 1180 1185
Glu Arg Val Gln Lys Leu Ser Ala Glu Ala Asp Ser Val Asn Gln
1190 1195 1200
Gln Leu Glu Ala Ala Glu Leu Lys Ala Ser Ala Ala Leu Lys Ala
1205 1210 1215
Ser Gly Thr Leu Glu Thr Gln Leu Gln Glu Ala Gln Val Leu Leu
1220 1225 1230
Glu Glu Glu Thr Arg Gln Lys Leu Ser Leu Thr Thr Lys Leu Lys
1235 1240 1245
Gly Leu Glu Ser Glu Arg Asp Ala Leu Lys Glu Gln Leu Tyr Glu
1250 1255 1260
Glu Asp Glu Gly Arg Lys Asn Leu Glu Lys Gln Met Ala Ile Leu
1265 1270 1275
Asn Gln Gln Val Ala Glu Ser Lys Lys Lys Ser Glu Glu Glu Thr
1280 1285 1290
Glu Lys Ile Thr Glu Leu Glu Glu Ser Arg Lys Lys Leu Leu Lys
1295 1300 1305
Asp Ile Glu Ile Leu Gln Arg Gln Val Glu Glu Leu Gln Val Thr
1310 1315 1320
Asn Asp Lys Leu Glu Lys Gly Lys Lys Lys Leu Gln Ser Glu Leu
1325 1330 1335
Glu Asp Leu Thr Ile Asp Leu Glu Ser Gln Arg Thr Lys Val Val
1340 1345 1350
Glu Leu Glu Lys Lys Gln Arg Asn Phe Asp Lys Val Leu Ala Glu
1355 1360 1365
Glu Lys Ala Leu Ser Gln Gln Ile Thr His Glu Arg Asp Ala Ala
1370 1375 1380
Glu Arg Glu Ala Arg Glu Lys Glu Thr Arg Val Leu Ser Leu Thr
1385 1390 1395
Arg Glu Leu Asp Glu Phe Met Glu Lys Ile Glu Glu Leu Glu Arg
1400 1405 1410
Ser Lys Arg Gln Leu Gln Ala Glu Leu Asp Glu Leu Val Asn Asn
1415 1420 1425
Gln Gly Thr Thr Asp Lys Ser Val His Glu Leu Glu Arg Ala Lys
1430 1435 1440
Arg Val Leu Glu Ser Gln Leu Ala Glu Gln Lys Ala Gln Asn Glu
1445 1450 1455
Glu Leu Glu Asp Glu Leu Gln Met Thr Glu Asp Ala Lys Leu Arg
1460 1465 1470
Leu Glu Val Asn Met Gln Ala Leu Arg Ala Gln Phe Glu Arg Asp
1475 1480 1485
Leu Gln Gly Lys Glu Glu Ser Gly Glu Glu Lys Arg Arg Gly Leu
1490 1495 1500
Leu Lys Gln Leu Arg Asp Ile Glu Ala Glu Leu Glu Asp Glu Arg
1505 1510 1515
Lys Gln Arg Thr Ala Ala Val Ala Ser Arg Lys Lys Ile Glu Ala
1520 1525 1530
Asp Phe Lys Asp Val Glu Gln Gln Leu Glu Met His Thr Lys Val
1535 1540 1545
Lys Glu Asp Leu Gln Lys Gln Leu Lys Lys Cys Gln Val Gln Leu
1550 1555 1560
Lys Asp Ala Ile Arg Asp Ala Glu Glu Ala Arg Leu Gly Arg Glu
1565 1570 1575
Glu Leu Gln Ala Ala Ala Lys Glu Ala Glu Arg Lys Trp Lys Gly
1580 1585 1590
Leu Glu Thr Glu Leu Ile Gln Val Gln Glu Asp Leu Met Ala Ser
1595 1600 1605
Glu Arg Gln Arg Arg Ala Ala Glu Ala Glu Arg Asp Glu Val Val
1610 1615 1620
Glu Glu Ala Asn Lys Asn Val Lys Ser Leu Ser Asn Leu Leu Asp
1625 1630 1635
Glu Lys Lys Arg Leu Glu Ala Gln Cys Ser Gly Leu Glu Glu Glu
1640 1645 1650
Leu Glu Glu Glu Leu Ser Asn Asn Glu Ala Leu Gln Asp Lys Ala
1655 1660 1665
Arg Lys Ala Gln Leu Ser Val Glu Gln Leu Asn Ala Glu Leu Ala
1670 1675 1680
Ala Glu Arg Ser Asn Val Gln Lys Leu Glu Gly Thr Arg Leu Ser
1685 1690 1695
Met Glu Arg Gln Asn Lys Glu Leu Lys Ala Lys Leu Asn Glu Leu
1700 1705 1710
Glu Thr Leu Gln Arg Asn Lys Phe Lys Ala Asn Ala Ser Leu Glu
1715 1720 1725
Ala Lys Ile Thr Asn Leu Glu Glu Gln Leu Glu Asn Glu Ala Lys
1730 1735 1740
Glu Lys Leu Leu Leu Gln Lys Gly Asn Arg Lys Leu Asp Lys Lys
1745 1750 1755
Ile Lys Asp Leu Leu Val Gln Leu Glu Asp Glu Arg Arg His Ala
1760 1765 1770
Asp Gln Tyr Lys Glu Gln Val Glu Lys Ile Asn Val Arg Val Lys
1775 1780 1785
Thr Leu Lys Arg Thr Leu Asp Asp Ala Glu Glu Glu Met Ser Arg
1790 1795 1800
Glu Lys Thr Gln Lys Arg Lys Ala Leu Arg Glu Leu Glu Asp Leu
1805 1810 1815
Arg Glu Asn Tyr Asp Ser Leu Leu Arg Glu Asn Asp Asn Leu Lys
1820 1825 1830
Asn Lys Leu Arg Arg Gly Gly Gly Ile Ser Gly Ile Ser Ser Arg
1835 1840 1845
Leu Gly Gly Ser Lys Arg Gly Ser Ile Pro Gly Glu Asp Ser Gln
1850 1855 1860
Gly Leu Asn Asn Thr Thr Asp Glu Ser Val Asp Gly Asp Asp Ile
1865 1870 1875
Ser Asn Pro
1880
<210> 336
<211> 108
<212> БЕЛОК
<213> Lygus hesperus
<400> 336
Lys Lys Ile Leu Glu Glu Ile Ile Ala Glu Val Asp Ala Asp Gly Ser
1 5 10 15
Gly Gln Leu Glu Phe Glu Glu Phe Val Ala Leu Ala Ala Gly Phe Leu
20 25 30
Thr Glu Asp Glu Thr Gln Asp Ala Glu Ala Met Gln Gln Glu Leu Arg
35 40 45
Glu Ala Phe Arg Leu Tyr Asp Lys Glu Gly Asn Gly Tyr Ile Thr Thr
50 55 60
Asp Val Leu Arg Glu Ile Leu Lys Glu Leu Asp Asp Lys Ile Thr Ser
65 70 75 80
Gln Glu Leu Asp Met Met Ile Ala Glu Ile Asp Ser Asp Gly Ser Gly
85 90 95
Thr Val Asp Phe Asp Glu Phe Met Glu Met Met Thr
100 105
<210> 337
<211> 141
<212> БЕЛОК
<213> Lygus hesperus
<400> 337
Ile Pro Ile Met Thr Ile Ala Leu Asn Ala Phe Asp Arg Asp His Ser
1 5 10 15
Gly Ser Ile Pro Thr Asp Met Val Ala Asp Ile Leu Arg Leu Met Gly
20 25 30
Gln Pro Phe Asn Lys Lys Ile Leu Asp Glu Leu Ile Glu Glu Val Asp
35 40 45
Ala Asp Lys Ser Gly Arg Leu Glu Phe Glu Glu Phe Ile Thr Leu Ala
50 55 60
Ala Lys Phe Ile Val Glu Glu Asp Asp Glu Ala Met Gln Lys Glu Leu
65 70 75 80
Arg Glu Ala Phe Arg Leu Tyr Asp Lys Glu Gly Asn Gly Tyr Ile Pro
85 90 95
Thr Ser Cys Leu Lys Glu Ile Leu His Glu Leu Asp Glu Gln Leu Thr
100 105 110
Asn Glu Glu Leu Asp Met Ile Ile Glu Glu Ile Asp Ser Asp Gly Ser
115 120 125
Gly Thr Val Asp Phe Asp Glu Phe Met Glu Met Met Thr
130 135 140
<210> 338
<211> 58
<212> БЕЛОК
<213> Lygus hesperus
<400> 338
Trp Val Lys Glu Gly Ala Cys Ser Glu Gln Ser Ser Arg Met Thr Ala
1 5 10 15
Met Asp Asn Ala Ser Lys Asn Ala Ala Glu Met Ile Asp Lys Leu Thr
20 25 30
Leu Thr Phe Asn Arg Thr Arg Gln Ala Val Ile Thr Arg Glu Leu Ile
35 40 45
Glu Ile Ile Ser Gly Ala Ser Ala Leu Glu
50 55
<210> 339
<211> 130
<212> БЕЛОК
<213> Lygus hesperus
<400> 339
Met Val Arg Met Asn Val Leu Ser Asp Ala Leu Lys Ser Ile Asn Asn
1 5 10 15
Ala Glu Lys Arg Gly Lys Arg Gln Val Leu Leu Arg Pro Cys Ser Lys
20 25 30
Val Ile Ile Lys Phe Leu Thr Val Met Met Lys Lys Gly Tyr Ile Gly
35 40 45
Glu Phe Glu Ile Val Asp Asp His Arg Ser Gly Lys Ile Val Val Asn
50 55 60
Leu Asn Gly Arg Leu Asn Lys Cys Gly Val Ile Ser Pro Arg Phe Asp
65 70 75 80
Val Pro Ile Thr Gln Ile Glu Lys Trp Thr Asn Asn Leu Leu Pro Ser
85 90 95
Arg Gln Phe Gly Tyr Val Val Leu Thr Thr Ser Gly Gly Ile Met Asp
100 105 110
His Glu Glu Ala Arg Arg Lys His Leu Gly Gly Lys Ile Leu Gly Phe
115 120 125
Phe Phe
130
<210> 340
<211> 131
<212> БЕЛОК
<213> Lygus hesperus
<400> 340
Val Asp Gly Gly Leu Asn Ile Pro His Ser Thr Lys Arg Phe Pro Gly
1 5 10 15
Tyr Asp Ser Glu Ser Lys Glu Phe Asn Ala Glu Val His Arg Lys His
20 25 30
Ile Phe Gly Ile His Val Ala Asp Tyr Met Arg Gln Leu Ala Glu Glu
35 40 45
Asp Asp Asp Ala Tyr Lys Lys Gln Phe Ser Gln Tyr Val Lys Asn Gly
50 55 60
Val Thr Ala Asp Ser Ile Glu Ser Ile Tyr Lys Lys Ala His Glu Ala
65 70 75 80
Ile Arg Ala Asp Pro Thr Arg Lys Pro Leu Glu Lys Lys Glu Val Lys
85 90 95
Lys Lys Arg Trp Asn Arg Ala Lys Leu Ser Leu Ser Glu Arg Lys Asn
100 105 110
Thr Ile Asn Gln Lys Lys Ala Thr Tyr Leu Lys Lys Val Glu Ala Gly
115 120 125
Glu Ile Glu
130
<210> 341
<211> 214
<212> БЕЛОК
<213> Lygus hesperus
<400> 341
Met Ala Pro Lys Gly Asn Asn Met Ile Pro Asn Gly His Phe His Lys
1 5 10 15
Asp Trp Gln Arg Phe Ile Lys Thr Trp Phe Asn Gln Pro Ala Arg Lys
20 25 30
Leu Arg Arg Arg Asn Lys Arg Leu Glu Lys Ala Gln Arg Leu Ala Pro
35 40 45
Arg Pro Ala Gly Pro Leu Arg Pro Ala Val Arg Cys Pro Thr Val Arg
50 55 60
Tyr His Thr Lys Leu Arg Pro Gly Arg Gly Phe Thr Leu Glu Glu Ile
65 70 75 80
Lys Arg Ala Gly Leu Cys Lys Gly Phe Ala Met Ser Ile Gly Ile Ala
85 90 95
Val Asp Pro Arg Arg Arg Asn Lys Ser Ile Glu Ser Leu Gln Leu Asn
100 105 110
Val Gln Arg Leu Lys Glu Tyr Arg Ala Lys Leu Ile Leu Phe Pro His
115 120 125
Lys Asn Ala Lys Lys Leu Lys Lys Gly Glu Ala Thr Glu Glu Glu Arg
130 135 140
Lys Val Ala Thr Gln Gln Pro Leu Pro Val Met Pro Ile Lys Gln Pro
145 150 155 160
Val Ile Lys Phe Lys Ala Arg Val Ile Thr Asp Asp Glu Lys Lys Tyr
165 170 175
Ser Ala Phe Thr Ala Leu Arg Lys Gly Arg Ala Asp Gln Arg Leu Val
180 185 190
Gly Ile Arg Ala Lys Arg Ala Lys Glu Ala Ala Glu Asn Ala Glu Asp
195 200 205
Pro Ser Lys Ala Pro Lys
210
<210> 342
<211> 134
<212> БЕЛОК
<213> Lygus hesperus
<400> 342
Met Asp Ile Glu Glu Pro Ala Ala Ala Pro Thr Glu Pro Ser Asp Val
1 5 10 15
Asn Thr Ala Leu Gln Glu Val Leu Lys Ala Ala Leu Gln His Gly Val
20 25 30
Val Val His Gly Ile His Glu Ser Ala Lys Ala Leu Asp Lys Arg Gln
35 40 45
Ala Leu Leu Cys Val Leu Ala Glu Asn Cys Asp Glu Pro Met Tyr Lys
50 55 60
Lys Leu Val Gln Ala Leu Cys Ser Glu His His Ile Pro Leu Val Lys
65 70 75 80
Val Asp Ser Asn Lys Lys Leu Gly Glu Trp Thr Gly Leu Cys Lys Ile
85 90 95
Asp Lys Thr Gly Lys Ser Arg Lys Ile Val Gly Cys Ser Cys Val Val
100 105 110
Ile Lys Asp Trp Gly Glu Asp Thr Pro His Leu Asp Leu Leu Lys Asp
115 120 125
Tyr Ile Arg Asp Val Phe
130
<210> 343
<211> 148
<212> БЕЛОК
<213> Lygus hesperus
<400> 343
Met Lys Met Asn Lys Leu Val Thr Ser Ser Arg Arg Lys Asn Arg Lys
1 5 10 15
Arg His Phe Thr Ala Pro Ser His Ile Arg Arg Lys Leu Met Ser Ala
20 25 30
Pro Leu Ser Lys Glu Leu Arg Gln Lys Tyr Asn Val Arg Thr Met Pro
35 40 45
Val Arg Lys Asp Asp Glu Val Gln Val Val Arg Gly His Tyr Lys Gly
50 55 60
Gln Gln Val Gly Lys Val Leu Gln Val Tyr Arg Lys Lys Phe Ile Ile
65 70 75 80
Tyr Ile Glu Arg Ile Gln Arg Glu Lys Ala Asn Gly Ala Ser Val Tyr
85 90 95
Val Gly Ile His Pro Ser Lys Cys Val Ile Val Lys Leu Lys Val Asp
100 105 110
Lys Asp Arg Lys Glu Ile Leu Asp Arg Arg Ser Lys Gly Arg Asp Leu
115 120 125
Ala Leu Gly Lys Asp Lys Gly Lys Tyr Thr Glu Asp Ser Thr Thr Ala
130 135 140
Met Asp Thr Ser
145
<210> 344
<211> 65
<212> БЕЛОК
<213> Lygus hesperus
<400> 344
Met Glu Lys Pro Val Val Leu Ala Arg Val Ile Lys Ile Leu Gly Arg
1 5 10 15
Thr Gly Ser Gln Gly Gln Cys Thr Gln Val Lys Val Glu Phe Ile Gly
20 25 30
Glu Gln Asn Arg Gln Ile Ile Arg Asn Val Lys Gly Pro Val Arg Glu
35 40 45
Gly Asp Ile Leu Thr Leu Leu Glu Ser Glu Arg Glu Ala Arg Arg Leu
50 55 60
Arg
65
<210> 345
<211> 229
<212> БЕЛОК
<213> Lygus hesperus
<400> 345
Leu Phe Tyr Phe Pro Phe Ser Arg Lys Trp Gly Asp Val Gln Arg Gly
1 5 10 15
Val Ile Gly Thr Val Lys Thr Ser His Thr Pro Lys Ser Arg Phe Cys
20 25 30
Arg Gly Val Pro Asp Pro Lys Ile Arg Ile Phe Asp Leu Gly Lys Lys
35 40 45
Lys Ala Arg Val Glu Asp Phe Pro Leu Cys Val His Leu Val Ser Asp
50 55 60
Glu Tyr Glu Gln Leu Ser Ser Glu Ala Leu Glu Ala Gly Arg Ile Cys
65 70 75 80
Cys Asn Lys Tyr Leu Val Lys Asn Cys Gly Lys Asp Gln Phe His Ile
85 90 95
Arg Met Arg Leu His Pro Phe His Val Ile Arg Ile Asn Lys Met Leu
100 105 110
Ser Cys Ala Gly Ala Asp Arg Leu Gln Thr Gly Met Arg Gly Ala Phe
115 120 125
Gly Lys Pro Gln Gly Thr Val Ala Arg Val Arg Ile Gly Gln Pro Ile
130 135 140
Met Ser Val Arg Ser Ser Asp Arg Tyr Lys Ala Ala Val Ile Lys Ala
145 150 155 160
Leu Arg Arg Ala Lys Phe Lys Phe Pro Gly Arg Gln Lys Ile Tyr Val
165 170 175
Ser Lys Lys Trp Gly Phe Thr Lys Phe Asp Arg Glu Glu Tyr Glu Gly
180 185 190
Leu Arg Asn Asp Asn Lys Leu Ala Asn Asp Gly Cys Asn Val Lys Leu
195 200 205
Arg Pro Asp His Gly Pro Leu Gln Ala Trp Arg Lys Ala Gln Leu Asp
210 215 220
Ile Ala Ala Gly Leu
225
<210> 346
<211> 220
<212> БЕЛОК
<213> Lygus hesperus
<400> 346
Met Gly Arg Arg Pro Ala Arg Cys Tyr Arg Tyr Cys Lys Asn Lys Pro
1 5 10 15
Tyr Pro Lys Ser Arg Phe Cys Arg Gly Val Pro Asp Pro Lys Ile Arg
20 25 30
Ile Phe Asp Leu Gly Lys Lys Lys Ala Arg Val Glu Asp Phe Pro Leu
35 40 45
Cys Val His Leu Val Ser Asp Glu Tyr Glu Gln Leu Ser Ser Glu Ala
50 55 60
Leu Glu Ala Gly Arg Ile Cys Cys Asn Lys Tyr Leu Val Lys Asn Cys
65 70 75 80
Gly Lys Asp Gln Phe His Ile Arg Met Arg Leu His Pro Phe His Val
85 90 95
Ile Arg Ile Asn Lys Met Leu Ser Cys Ala Gly Ala Asp Arg Leu Gln
100 105 110
Thr Gly Met Arg Gly Ala Phe Gly Lys Pro Gln Gly Thr Val Ala Arg
115 120 125
Val Arg Ile Gly Gln Pro Ile Met Ser Val Arg Ser Ser Asp Arg Tyr
130 135 140
Lys Ala Ala Val Ile Glu Ala Leu Arg Arg Ala Lys Phe Lys Phe Pro
145 150 155 160
Gly Arg Gln Lys Ile Tyr Val Ser Lys Lys Trp Gly Phe Thr Lys Phe
165 170 175
Asp Arg Glu Glu Tyr Glu Gly Leu Arg Asn Asp Asn Lys Leu Ala Asn
180 185 190
Gly Gly Cys Asn Val Lys Leu Arg Pro Asp His Gly Pro Leu Gln Ala
195 200 205
Trp Arg Lys Ala Gln Leu Asp Ile Ala Ala Gly Leu
210 215 220
<210> 347
<211> 159
<212> БЕЛОК
<213> Lygus hesperus
<400> 347
Met Thr Asn Ser Lys Gly Tyr Arg Arg Gly Thr Arg Asp Leu Phe Ser
1 5 10 15
Arg Pro Phe Arg His His Gly Val Ile Pro Leu Ser Thr Tyr Met Lys
20 25 30
Val Tyr Arg Val Gly Asp Ile Val Ser Ile Lys Gly Asn Gly Ala Val
35 40 45
Gln Lys Gly Met Pro His Lys Val Tyr His Gly Lys Thr Gly Arg Val
50 55 60
Tyr Asn Val Thr Pro Arg Ala Leu Gly Val Ile Val Asn Lys Arg Val
65 70 75 80
Arg Gly Lys Ile Leu Pro Lys Arg Ile Asn Ile Arg Ile Glu His Val
85 90 95
Asn His Ser Lys Cys Arg Glu Asp Phe Leu Lys Arg Val Arg Glu Asn
100 105 110
Glu Arg Leu Arg Lys Phe Ala Lys Glu Thr Gly Thr Arg Val Glu Leu
115 120 125
Lys Arg Gln Pro Ala Gln Pro Arg Pro Ala His Phe Val Gln Ala Lys
130 135 140
Glu Val Pro Glu Leu Leu Ala Pro Ile Pro Tyr Glu Phe Ile Ala
145 150 155
<210> 348
<211> 131
<212> БЕЛОК
<213> Lygus hesperus
<400> 348
Thr Tyr Met Lys Val Tyr Arg Val Gly Asp Ile Val Ser Ile Lys Gly
1 5 10 15
Asn Gly Ala Val Gln Lys Gly Met Pro His Lys Val Tyr His Gly Lys
20 25 30
Thr Gly Arg Val Tyr Asn Val Thr Pro Arg Ala Leu Gly Val Ile Val
35 40 45
Asn Lys Arg Val Arg Gly Lys Ile Leu Pro Lys Arg Ile Asn Ile Arg
50 55 60
Ile Glu His Val Asn His Ser Lys Cys Arg Glu Asp Phe Leu Lys Arg
65 70 75 80
Val Arg Glu Asn Glu Arg Leu Arg Lys Phe Ala Lys Glu Thr Gly Thr
85 90 95
Arg Val Glu Leu Lys Arg Gln Pro Ala Gln Pro Arg Pro Ala His Phe
100 105 110
Val Gln Ala Lys Glu Val Pro Glu Leu Leu Ala Pro Ile Pro Tyr Glu
115 120 125
Phe Ile Ala
130
<210> 349
<211> 150
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 349
Lys Lys Ala Lys Lys Gly Phe Met Thr Pro Glu Arg Lys Lys Lys Leu
1 5 10 15
Arg Leu Leu Leu Arg Lys Lys Ala Ala Glu Glu Leu Lys Lys Glu Gln
20 25 30
Glu Arg Lys Ala Ala Glu Arg Arg Arg Ile Ile Glu Glu Arg Cys Gly
35 40 45
Lys Pro Lys Leu Ile Asp Glu Ala Asn Glu Glu Gln Val Arg Asn Tyr
50 55 60
Cys Lys Leu Tyr His Gly Arg Ile Ala Lys Leu Glu Asp Gln Lys Phe
65 70 75 80
Asp Leu Glu Tyr Leu Val Lys Lys Lys Asp Met Glu Ile Ala Glu Leu
85 90 95
Asn Ser Gln Val Asn Asp Leu Arg Gly Lys Phe Val Lys Pro Thr Leu
100 105 110
Lys Lys Val Ser Lys Tyr Glu Asn Lys Phe Ala Lys Leu Gln Lys Lys
115 120 125
Ala Ala Glu Phe Asn Phe Arg Asn Gln Leu Lys Val Val Lys Lys Lys
130 135 140
Glu Phe Thr Leu Glu Glu
145 150
<210> 350
<211> 279
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 350
Gln Trp Tyr Gln Arg Arg Val Arg Gly Asp Ile Glu Glu Lys Arg Gln
1 5 10 15
Arg Leu Glu Glu Ala Glu Lys Lys Arg Gln Ala Met Met Gln Ala Leu
20 25 30
Lys Asp Gln Asn Lys Asn Lys Gly Pro Asn Phe Thr Ile Thr Lys Arg
35 40 45
Asp Ala Ser Ser Asn Leu Ser Ala Ala Gln Leu Glu Arg Asn Lys Thr
50 55 60
Lys Glu Gln Leu Glu Glu Glu Lys Lys Ile Ser Leu Ser Ile Arg Ile
65 70 75 80
Lys Pro Leu Val Val Asp Gly Leu Gly Val Asp Lys Leu Arg Leu Lys
85 90 95
Ala Gln Glu Leu Trp Glu Cys Ile Val Lys Leu Glu Thr Glu Lys Tyr
100 105 110
Asp Leu Glu Glu Arg Gln Lys Arg Gln Asp Tyr Asp Leu Lys Glu Leu
115 120 125
Lys Glu Arg Gln Lys Gln Gln Leu Arg His Lys Ala Leu Lys Lys Gly
130 135 140
Leu Asp Pro Glu Ala Leu Thr Gly Lys Tyr Pro Pro Lys Ile Gln Val
145 150 155 160
Ala Ser Lys Tyr Glu Arg Arg Val Asp Thr Arg Ser Tyr Gly Asp Lys
165 170 175
Lys Lys Leu Phe Glu Gly Gly Leu Glu Glu Ile Ile Lys Glu Thr Asn
180 185 190
Glu Lys Ser Trp Lys Glu Lys Phe Gly Gln Phe Asp Ser Arg Gln Lys
195 200 205
Ala Arg Leu Pro Lys Trp Phe Gly Glu Arg Pro Gly Lys Lys Pro Gly
210 215 220
Asp Pro Glu Thr Pro Glu Gly Glu Glu Glu Gly Lys Gln Val Ile Asp
225 230 235 240
Glu Asp Asp Asp Leu Lys Glu Pro Val Ile Glu Ala Glu Ile Glu Glu
245 250 255
Glu Glu Glu Glu Glu Glu Val Glu Val Asp Glu Glu Glu Glu Asp Asp
260 265 270
Glu Glu Glu Glu Glu Glu Glu
275
<210> 351
<211> 286
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 351
Ala Leu Gln Asn Glu Leu Glu Glu Ser Arg Thr Leu Leu Glu Gln Ala
1 5 10 15
Asp Arg Ala Arg Arg Gln Ala Glu Gln Glu Leu Gly Asp Ala His Glu
20 25 30
Gln Leu Asn Asp Leu Gly Ala Gln Asn Gly Ser Leu Ser Ala Ala Lys
35 40 45
Arg Lys Leu Glu Thr Glu Leu Gln Thr Leu His Ser Asp Leu Asp Glu
50 55 60
Leu Leu Asn Glu Ala Lys Asn Ser Glu Glu Lys Ala Lys Lys Ala Met
65 70 75 80
Val Asp Ala Ala Arg Leu Ala Asp Glu Leu Arg Ala Glu Gln Asp His
85 90 95
Ala Gln Thr Gln Glu Lys Leu Arg Lys Ala Leu Glu Ser Gln Ile Lys
100 105 110
Asp Leu Gln Val Arg Leu Asp Glu Ala Glu Ala Asn Ala Leu Lys Gly
115 120 125
Gly Lys Lys Ala Ile Ala Lys Leu Glu Gln Arg Val Arg Glu Leu Glu
130 135 140
Asn Glu Leu Asp Gly Glu Gln Arg Arg His Ala Asp Ala Gln Lys Asn
145 150 155 160
Leu Arg Lys Ser Glu Arg Arg Ile Lys Glu Leu Ser Leu Gln Ala Glu
165 170 175
Glu Asp Arg Lys Asn His Glu Lys Met Gln Asp Leu Val Asp Lys Leu
180 185 190
Gln Gln Lys Ile Lys Thr His Lys Arg Gln Ile Glu Glu Ala Glu Glu
195 200 205
Ile Ala Ala Leu Asn Leu Ala Lys Phe Arg Lys Ala Gln Gln Glu Leu
210 215 220
Glu Glu Ala Glu Glu Arg Ala Asp Leu Ala Glu Gln Ala Ile Val Lys
225 230 235 240
Phe Arg Thr Lys Gly Arg Ser Gly Ser Ala Ala Arg Gly Ala Ser Pro
245 250 255
Ala Pro Gln Arg Gln Arg Pro Thr Phe Gly Met Gly Asp Ser Leu Gly
260 265 270
Gly Ala Phe Pro Pro Arg Phe Asp Leu Ala Pro Asp Phe Glu
275 280 285
<210> 352
<211> 197
<212> БЕЛОК
<213> nilaparvata lugens
<400> 352
Met Ala Asp Asp Glu Ala Lys Lys Ala Lys Gln Ala Glu Ile Asp Arg
1 5 10 15
Lys Arg Ala Glu Val Arg Lys Arg Met Glu Glu Ala Ser Lys Ala Lys
20 25 30
Lys Ala Lys Lys Gly Phe Met Thr Pro Asp Arg Lys Lys Lys Leu Arg
35 40 45
Leu Leu Leu Arg Lys Lys Ala Ala Glu Glu Leu Lys Lys Glu Gln Glu
50 55 60
Arg Lys Ala Ala Glu Arg Arg Arg Ile Ile Glu Glu Arg Cys Gly Lys
65 70 75 80
Ala Val Asp Leu Asp Asp Gly Ser Glu Glu Lys Val Lys Ala Thr Leu
85 90 95
Lys Thr Tyr His Asp Arg Ile Gly Lys Leu Glu Asp Glu Lys Phe Asp
100 105 110
Leu Glu Tyr Ile Val Lys Lys Lys Asp Phe Glu Ile Ala Asp Leu Asn
115 120 125
Ser Gln Val Asn Asp Leu Arg Gly Lys Phe Val Lys Pro Thr Leu Lys
130 135 140
Lys Val Ser Lys Tyr Glu Asn Lys Phe Ala Lys Leu Gln Lys Lys Ala
145 150 155 160
Ala Glu Phe Asn Phe Arg Asn Gln Leu Lys Val Val Lys Lys Lys Glu
165 170 175
Phe Thr Leu Glu Glu Glu Asp Lys Glu Pro Lys Lys Ser Glu Lys Ala
180 185 190
Glu Trp Gln Lys Lys
195
<210> 353
<211> 184
<212> БЕЛОК
<213> nilaparvata lugens
<400> 353
Met Met Ala Ala Leu Lys Asp Gln Ser Lys Ser Lys Gly Pro Asn Phe
1 5 10 15
Thr Val Asn Lys Lys Thr Asp Leu Asn Met Thr Ser Ala Gln Met Glu
20 25 30
Arg Asn Lys Thr Lys Glu Gln Leu Glu Glu Glu Lys Lys Ile Ser Leu
35 40 45
Ser Phe Arg Ile Lys Pro Leu Ala Ile Glu Asn Met Ser Ile Asn Ala
50 55 60
Leu Arg Ala Lys Ala Gln Glu Leu Trp Asp Cys Ile Val Lys Leu Glu
65 70 75 80
Thr Glu Lys Tyr Asp Leu Glu Glu Arg Gln Lys Arg Gln Asp Tyr Asp
85 90 95
Leu Lys Glu Leu Lys Glu Arg Gln Lys Gln Gln Leu Arg His Lys Ala
100 105 110
Leu Lys Lys Gly Leu Asp Pro Glu Ala Leu Thr Gly Lys Tyr Pro Pro
115 120 125
Lys Ile Gln Val Ala Ser Lys Tyr Glu Arg Arg Val Asp Thr Arg Ser
130 135 140
Tyr Asp Asp Lys Lys Lys Leu Phe Glu Gly Gly Trp Asp Thr Leu Thr
145 150 155 160
Ser Glu Thr Asn Glu Lys Ile Trp Lys Ser Arg Asn Asp Gln Phe Ser
165 170 175
Asn Arg Ser Lys Ala Lys Leu Pro
180
<210> 354
<211> 122
<212> БЕЛОК
<213> nilaparvata lugens
<400> 354
Ala Phe Asp Arg Glu Arg Ser Gly Ser Ile Pro Thr Asp Met Val Ala
1 5 10 15
Asp Ile Leu Arg Leu Met Gly Gln Pro Phe Asn Lys Lys Ile Leu Asp
20 25 30
Glu Leu Ile Glu Glu Val Asp Ala Asp Lys Ser Gly Arg Leu Glu Phe
35 40 45
Asp Glu Phe Val Thr Leu Ala Ala Lys Phe Ile Val Glu Glu Asp Asp
50 55 60
Glu Ala Met Gln Lys Glu Leu Lys Glu Ala Phe Arg Leu Tyr Asp Lys
65 70 75 80
Glu Gly Asn Gly Tyr Ile Pro Thr Ser Cys Leu Lys Glu Ile Leu Arg
85 90 95
Glu Leu Asp Asp Gln Leu Thr Asn Glu Glu Leu Asn Met Met Ile Asp
100 105 110
Glu Ile Asp Ser Asp Gly Ser Gly Thr Val
115 120
<210> 355
<211> 73
<212> БЕЛОК
<213> nilaparvata lugens
<400> 355
Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Pro Ser
1 5 10 15
Asp Thr Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile
20 25 30
Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp
35 40 45
Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His
50 55 60
Leu Val Leu Arg Leu Arg Gly Gly Thr
65 70
<210> 356
<211> 253
<212> БЕЛОК
<213> Acyrthosiphon pisum
<400> 356
Met Ala Asp Asp Glu Ala Lys Lys Ala Lys Gln Ala Glu Ile Asp Arg
1 5 10 15
Lys Arg Ala Glu Val Arg Lys Arg Met Glu Glu Ala Ser Lys Ala Lys
20 25 30
Lys Ala Lys Lys Gly Phe Met Thr Pro Asp Arg Lys Lys Lys Leu Arg
35 40 45
Leu Leu Leu Lys Lys Lys Ala Ala Glu Glu Leu Lys Lys Glu Gln Glu
50 55 60
Arg Lys Ala Ala Glu Arg Arg Arg Ile Ile Glu Glu Arg Cys Gly Gln
65 70 75 80
Pro Lys Asn Ile Asp Asp Ala Gly Glu Glu Glu Leu Ala Glu Ile Cys
85 90 95
Glu Glu Leu Trp Lys Arg Val Tyr Thr Val Glu Gly Ile Lys Phe Asp
100 105 110
Leu Glu Arg Asp Ile Arg Met Lys Val Phe Glu Ile Ser Glu Leu Asn
115 120 125
Ser Gln Val Asn Asp Leu Arg Gly Lys Phe Val Lys Pro Thr Leu Lys
130 135 140
Lys Val Ser Lys Tyr Glu Asn Lys Phe Ala Lys Leu Gln Lys Lys Ala
145 150 155 160
Ala Glu Phe Asn Phe Arg Asn Gln Leu Lys Val Val Lys Lys Lys Glu
165 170 175
Phe Thr Leu Glu Glu Glu Asp Lys Glu Lys Lys Pro Asp Trp Ser Lys
180 185 190
Lys Gly Asp Glu Lys Lys Gly Glu Gly Glu Asp Gly Asp Gly Thr Glu
195 200 205
Asp Glu Lys Thr Asp Asp Gly Leu Thr Thr Glu Gly Glu Ser Val Ala
210 215 220
Gly Asp Leu Thr Asp Ala Thr Glu Asp Ala Gln Ser Asp Asn Glu Ile
225 230 235 240
Leu Glu Pro Glu Pro Val Val Glu Pro Glu Pro Glu Pro
245 250
<210> 357
<211> 179
<212> БЕЛОК
<213> Acyrthosiphon pisum
<400> 357
Val Met Arg Cys Gly Lys Lys Lys Val Trp Leu Asp Pro Asn Glu Ile
1 5 10 15
Asn Glu Ile Ala Asn Thr Asn Ser Arg Gln Asn Ile Arg Lys Leu Ile
20 25 30
Lys Asp Gly Leu Ile Ile Lys Lys Pro Val Ala Val His Ser Arg Ala
35 40 45
Arg Ala Arg Lys Asn Ala Asp Ala Arg Arg Lys Gly Arg His Cys Gly
50 55 60
Phe Gly Lys Arg Lys Gly Thr Ala Asn Ala Arg Thr Pro Gln Lys Asp
65 70 75 80
Leu Trp Val Lys Arg Met Arg Val Leu Arg Arg Leu Leu Lys Lys Tyr
85 90 95
Arg Glu Ala Lys Lys Ile Asp Asn His Leu Tyr His Gln Leu Tyr Met
100 105 110
Lys Ala Lys Gly Asn Val Phe Lys Asn Lys Arg Val Leu Met Glu Phe
115 120 125
Ile His Lys Lys Lys Ala Glu Lys Ala Arg Ala Lys Met Leu Ser Asp
130 135 140
Gln Ala Glu Ala Arg Arg Gln Lys Val Lys Glu Ala Arg Lys Arg Lys
145 150 155 160
Glu Ala Arg Phe Leu Gln Asn Arg Lys Glu Leu Leu Ala Ala Tyr Ala
165 170 175
Arg Glu Asp
<210> 358
<211> 275
<212> БЕЛОК
<213> Acyrthosiphon pisum
<400> 358
Gly Leu Glu Val Glu Ser Ser Asp Ser Ile Glu Asn Val Lys Ala Lys
1 5 10 15
Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe
20 25 30
Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile
35 40 45
Gln Lys Glu Ser Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met
50 55 60
Gln Ile Phe Val Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val
65 70 75 80
Glu Ser Ser Asp Ser Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys
85 90 95
Glu Gly Ile Pro Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln
100 105 110
Leu Glu Asp Gly Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser
115 120 125
Thr Leu His Leu Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val
130 135 140
Lys Thr Leu Thr Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp
145 150 155 160
Ser Ile Glu Asn Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro
165 170 175
Pro Asp Gln Gln Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly
180 185 190
Arg Thr Leu Ser Asp Tyr Asn Ile Gln Lys Glu Ser Thr Leu His Leu
195 200 205
Val Leu Arg Leu Arg Gly Gly Met Gln Ile Phe Val Lys Thr Leu Thr
210 215 220
Gly Lys Thr Ile Thr Leu Glu Val Glu Ser Ser Asp Ser Ile Glu Asn
225 230 235 240
Val Lys Ala Lys Ile Gln Asp Lys Glu Gly Ile Pro Pro Asp Gln Gln
245 250 255
Arg Leu Ile Phe Ala Gly Lys Gln Leu Glu Asp Gly Arg Thr Leu Ser
260 265 270
Asp Tyr Asn
275
<210> 359
<211> 56
<212> БЕЛОК
<213> Acyrthosiphon pisum
<400> 359
Asp Leu Leu His Pro Thr Ala Ile Glu Glu Arg Arg Lys His Lys Leu
1 5 10 15
Lys Arg Leu Val Gln His Pro Asn Ser Phe Phe Met Asp Val Lys Cys
20 25 30
Pro Gly Cys Tyr Lys Ile Thr Thr Val Phe Ser His Ala Gln Ser Val
35 40 45
Val Ile Cys Thr Gly Cys Ser Thr
50 55
<210> 360
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 360
atcatgcagg cgtacgcccg 20
<210> 361
<211> 19
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 361
cggagggggc gagatcact 19
<210> 362
<211> 62
<212> ДНК
<213> Искусственная
<220>
<223> Ампликон
<400> 362
atcatgcagg cgtacgcccg agaagacgag gctgccgtca aaaagtgatc tcgccccctc 60
cg 62
<210> 363
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 363
tgtgttggct actggtggct ac 22
<210> 364
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 364
tcggatggaa ctggacaaat tcaag 25
<210> 365
<211> 137
<212> ДНК
<213> Искусственная
<220>
<223> Ампликон
<400> 365
tgtgttggct actggtggct acggcagagc ttacttttca tgcacttcag ctcacacttg 60
cacgggagat ggccaagcaa tggtttcacg agctgggctt cccaacgaag atcttgaatt 120
tgtccagttc catccga 137
<210> 366
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 366
gcaacccgtg ttctccaaag c 21
<210> 367
<211> 28
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 367
tcaactcgta ttctcgtact ttcaaacc 28
<210> 368
<211> 146
<212> ДНК
<213> Искусственная
<220>
<223> Ампликон
<400> 368
gcaacccgtg ttctccaaag ccagatacac tgtgcgatcc ttcggtatca ggcgtaacga 60
aaaaatcgcc gttcactgca ctgtcagggg cgccaaagca gaggaaattc tggagcgtgg 120
tttgaaagta cgagaatacg agttga 146
<210> 369
<211> 21
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 369
atggccgacg atgaagctaa g 21
<210> 370
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 370
tggttctggt tcgggttcaa 20
<210> 371
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 371
cggtaatgcg atgcggtaag 20
<210> 372
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 372
tcatcttctc gggcgtatgc 20
<210> 373
<211> 25
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 373
tttggaagtt gagtcatcag attcc 25
<210> 374
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 374
gttgtagtcg gaaagggtac gtcc 24
<210> 375
<211> 20
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 375
attgtggaac atccggtaca 20
<210> 376
<211> 23
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 376
aagacttgct tcatcctact gca 23
<210> 377
<211> 28
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<220>
<221> иной_признак
<222> (20)..(20)
<223> n представляет собой a, c, g или t
<400> 377
ccaagaaggc caagaagggn ttyatgac 28
<210> 378
<211> 29
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 378
tcctcctcca gggtgaactc yttyttytt 29
<210> 379
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<220>
<221> иной_признак
<222> (21)..(21)
<223> n представляет собой a, c, g или t
<220>
<221> иной_признак
<222> (24)..(24)
<223> n представляет собой a, c, g или t
<400> 379
gccaagaagg gcttcatgac nccnga 26
<210> 380
<211> 27
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 380
gaagttgaac tcggcggcyt tyttytg 27
<210> 381
<211> 26
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<220>
<221> иной_признак
<222> (24)..(24)
<223> n представляет собой a, c, g или t
<400> 381
ctggaggagg ccgagaaraa rmgnca 26
<210> 382
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<220>
<221> иной_признак
<222> (14)..(14)
<223> n представляет собой a, c, g или t
<400> 382
tgccgggccg ctcnccraac ca 22
<210> 383
<211> 28
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<220>
<221> иной_признак
<222> (20)..(20)
<223> n представляет собой a, c, g или t
<220>
<221> иной_признак
<222> (23)..(23)
<223> n представляет собой a, c, g или t
<400> 383
agatcgccat cctgaggaan gcnttyra 28
<210> 384
<211> 31
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 384
cggtcatcat ctccatgaac tcrtcraart c 31
<210> 385
<211> 170
<212> ДНК
<213> Искусственная
<220>
<223> Интрон
<400> 385
tctagaaggt aagtgtacac actacatttt catgaacatt attgcgaccg ttgagattct 60
cattgtttgg tgattgatta tctaaagtag aagcatgaat agatataaca taaactagta 120
actaatgggt tagttatggg tatacttcat gcttttctct caggctcgag 170
<210> 386
<211> 521
<212> ДНК
<213> leptinotarsa decemlineata
<400> 386
tcgatttttc atttttcttt tattatttgg agtgggcctg ttgtggtcgt tatcaaaatg 60
ggtaaaataa tgaaatctgg taaagtcgta ttggtccttg gaggccgata cgctggaaga 120
aaggcagtag tcataaaaaa ttacgatgat gggacgtcag ataaacaata tggacatgcc 180
gtggtggctg gaatcgatag gtaccctaga aaaatccaca aacgtatggg caaaggaaaa 240
atgcacaaga ggtccaaaat caagcccttc cttaaggtgc tcaactataa ccatttgatg 300
cctacaagat attcagtgga tttgacttcg gacttgaaag tggcgcccaa agacctcaag 360
gatccagtga agaggaagaa gattaggttc caaaccagag ttaaattcga agagagatac 420
aagcaaggaa aacacaaatg gtttttccag aaattgaggt tctagattct ataaatttaa 480
ccattttgta atccacccac ctttttgttc aaataaattg t 521
<210> 387
<211> 475
<212> ДНК
<213> leptinotarsa decemlineata
<400> 387
tatcgcgaaa aatatacaac ttacaaaatg aggaacacgt atgagttgag ccctaaagaa 60
gcagcaaatt tcactcgtcg aaatttagca gatactcttc gaagcaggag tccatatcat 120
gttaatcttc tcttggctgg atatgacaag aaagacgggg ctcagttgta ttacatggat 180
tatctagcgt ctgttgctag tgttgattac gctgcccatg gatacggagg atatttctcc 240
ctttccataa tggatcgcaa ttatttgaaa accctgtcga aagatcaagg atacgaactt 300
ctgaaggaat gtgttaaaga agttcaaaag agacttgcta taaatttacc aaatttcaaa 360
gttcaggtta ttgataaaga tggtattaag gatatgccta atataacttc aaaaggtttg 420
aattgattaa gcaacttcag tttcagattt ttttctaaat aaacatttaa agtgt 475
<210> 388
<211> 467
<212> ДНК
<213> leptinotarsa decemlineata
<400> 388
gcggggactg gatacatctc taaaacacag aaaaatgaaa ttcttcaagt caggaatata 60
ttctgttgta tttttggcaa ttatattttc tttggtcact gaggaagtgg aaggtcgaag 120
gactatttta agagggcgta aaacactgac gagaacctat tttcgtgaca atgcagtccc 180
agcatacgtc atagtgatac tcgttggaat aggagaaatc attttgggag ctatcctgta 240
tgttataatg aggaaaacga taatagattt tcctttatca gggagttacg cagtggcccc 300
tactcaagaa gcataaatcc cattgaaatt gtgactgttt actttctttg gaaaaatgtg 360
tataataaat acaattcatt tataatattt atatttggaa cttaaaatac ttacaaaatt 420
accatttaca tgatcaaata actaataaag ttctgtctca attataa 467
<210> 389
<211> 906
<212> ДНК
<213> leptinotarsa decemlineata
<400> 389
ggattggaag taaaaatata caattcatgc tgtagctgta gtgtaaaaac tgaactgaaa 60
gccataaaat aaagaccttg caagaaacat gtccaagatt aatgaggtgt ctaatttgta 120
caaacaactg aaatcagaat ggaacacatc caatccaaat ttaagcaaat gtgaaaagct 180
tttgtcagat ttgaagcttg agctaacaca cttaatgttc cttccaactt caaacgccac 240
tgcttcaaaa caagaacttc ttctggcaag agatgttctg gaaattgggg tacaatggag 300
tatagctgca aatgatatac ctgcctttga aagatacatg gcacagttga aatgttatta 360
tttcgattat aagaatcaac ttcccgaatc ttctttcaaa tatcagttac tgggtctgaa 420
tttactattt ttgttatcac aaaatagagt ggcagagttc cacacagaat tagaattgtt 480
gcctgctgac cacattcaga atgatgtata catcaggcac cctccatcta ttgaacagta 540
ccttatggaa ggaagttata ataagatatt tctggcaaag ggaaatgtcc cagcaacaaa 600
ttacaatttt tttatggata tacttctaga tactatcaga ggggagattg cagattgtct 660
agagaaagca tatgaaaaaa tatcaattaa agatgttgct aggatgctat acttgggcag 720
tgaagaatcg gccaaggcct ttgtaacaaa gagtaagaca tggaaattag aaaaggacaa 780
cttctttcac ttcacgcccg aggttaaaaa gacacatgag ccaattctat ccaaagaatt 840
ggcacaacaa gctattgaat atgcaaaaga actggaaatg attgtttaaa gtaataaagt 900
ttttca 906
<210> 390
<211> 135
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 390
Met Gly Lys Ile Met Lys Ser Gly Lys Val Val Leu Val Leu Gly Gly
1 5 10 15
Arg Tyr Ala Gly Arg Lys Ala Val Val Ile Lys Asn Tyr Asp Asp Gly
20 25 30
Thr Ser Asp Lys Gln Tyr Gly His Ala Val Val Ala Gly Ile Asp Arg
35 40 45
Tyr Pro Arg Lys Ile His Lys Arg Met Gly Lys Gly Lys Met His Lys
50 55 60
Arg Ser Lys Ile Lys Pro Phe Leu Lys Val Leu Asn Tyr Asn His Leu
65 70 75 80
Met Pro Thr Arg Tyr Ser Val Asp Leu Thr Ser Asp Leu Lys Val Ala
85 90 95
Pro Lys Asp Leu Lys Asp Pro Val Lys Arg Lys Lys Ile Arg Phe Gln
100 105 110
Thr Arg Val Lys Phe Glu Glu Arg Tyr Lys Gln Gly Lys His Lys Trp
115 120 125
Phe Phe Gln Lys Leu Arg Phe
130 135
<210> 391
<211> 141
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 391
Tyr Arg Glu Lys Tyr Thr Thr Tyr Lys Met Arg Asn Thr Tyr Glu Leu
1 5 10 15
Ser Pro Lys Glu Ala Ala Asn Phe Thr Arg Arg Asn Leu Ala Asp Thr
20 25 30
Leu Arg Ser Arg Ser Pro Tyr His Val Asn Leu Leu Leu Ala Gly Tyr
35 40 45
Asp Lys Lys Asp Gly Ala Gln Leu Tyr Tyr Met Asp Tyr Leu Ala Ser
50 55 60
Val Ala Ser Val Asp Tyr Ala Ala His Gly Tyr Gly Gly Tyr Phe Ser
65 70 75 80
Leu Ser Ile Met Asp Arg Asn Tyr Leu Lys Thr Leu Ser Lys Asp Gln
85 90 95
Gly Tyr Glu Leu Leu Lys Glu Cys Val Lys Glu Val Gln Lys Arg Leu
100 105 110
Ala Ile Asn Leu Pro Asn Phe Lys Val Gln Val Ile Asp Lys Asp Gly
115 120 125
Ile Lys Asp Met Pro Asn Ile Thr Ser Lys Gly Leu Asn
130 135 140
<210> 392
<211> 104
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 392
Arg Gly Leu Asp Thr Ser Leu Lys His Arg Lys Met Lys Phe Phe Lys
1 5 10 15
Ser Gly Ile Tyr Ser Val Val Phe Leu Ala Ile Ile Phe Ser Leu Val
20 25 30
Thr Glu Glu Val Glu Gly Arg Arg Thr Ile Leu Arg Gly Arg Lys Thr
35 40 45
Leu Thr Arg Thr Tyr Phe Arg Asp Asn Ala Val Pro Ala Tyr Val Ile
50 55 60
Val Ile Leu Val Gly Ile Gly Glu Ile Ile Leu Gly Ala Ile Leu Tyr
65 70 75 80
Val Ile Met Arg Lys Thr Ile Ile Asp Phe Pro Leu Ser Gly Ser Tyr
85 90 95
Ala Val Ala Pro Thr Gln Glu Ala
100
<210> 393
<211> 266
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 393
Met Ser Lys Ile Asn Glu Val Ser Asn Leu Tyr Lys Gln Leu Lys Ser
1 5 10 15
Glu Trp Asn Thr Ser Asn Pro Asn Leu Ser Lys Cys Glu Lys Leu Leu
20 25 30
Ser Asp Leu Lys Leu Glu Leu Thr His Leu Met Phe Leu Pro Thr Ser
35 40 45
Asn Ala Thr Ala Ser Lys Gln Glu Leu Leu Leu Ala Arg Asp Val Leu
50 55 60
Glu Ile Gly Val Gln Trp Ser Ile Ala Ala Asn Asp Ile Pro Ala Phe
65 70 75 80
Glu Arg Tyr Met Ala Gln Leu Lys Cys Tyr Tyr Phe Asp Tyr Lys Asn
85 90 95
Gln Leu Pro Glu Ser Ser Phe Lys Tyr Gln Leu Leu Gly Leu Asn Leu
100 105 110
Leu Phe Leu Leu Ser Gln Asn Arg Val Ala Glu Phe His Thr Glu Leu
115 120 125
Glu Leu Leu Pro Ala Asp His Ile Gln Asn Asp Val Tyr Ile Arg His
130 135 140
Pro Pro Ser Ile Glu Gln Tyr Leu Met Glu Gly Ser Tyr Asn Lys Ile
145 150 155 160
Phe Leu Ala Lys Gly Asn Val Pro Ala Thr Asn Tyr Asn Phe Phe Met
165 170 175
Asp Ile Leu Leu Asp Thr Ile Arg Gly Glu Ile Ala Asp Cys Leu Glu
180 185 190
Lys Ala Tyr Glu Lys Ile Ser Ile Lys Asp Val Ala Arg Met Leu Tyr
195 200 205
Leu Gly Ser Glu Glu Ser Ala Lys Ala Phe Val Thr Lys Ser Lys Thr
210 215 220
Trp Lys Leu Glu Lys Asp Asn Phe Phe His Phe Thr Pro Glu Val Lys
225 230 235 240
Lys Thr His Glu Pro Ile Leu Ser Lys Glu Leu Ala Gln Gln Ala Ile
245 250 255
Glu Tyr Ala Lys Glu Leu Glu Met Ile Val
260 265
<210> 394
<211> 750
<212> ДНК
<213> Leptinotarsa decemlineata
<400> 394
cgcccagcag tggtatcaac gcagagtacg cgggagacat tcaagtcttg tgatagtgca 60
ggcacggcag ttcaaataaa ctggtgcctt caatttattt atatatttat acttttttac 120
tagaaaccaa atactaacca atcaacatgt gtgacgaaga ggttgccgca ttagtcgtag 180
acaatggatc tggtatgtgc aaagctggat ttgctgggga tgatgccccc cgtgcagttt 240
tcccatccat tgttggtcgt ccaagacatc aaggagttat ggtaggaatg ggccaaaagg 300
actcgtatgt aggagatgaa gcccaaagca aaagaggtat ccttaccttg aaatacccca 360
ttgaacacgg tattgtcaca aactgggatg atatggagaa aatctggcac cataccttct 420
acaatgaact tcgagttgcc cccgaagagc accctgtttt gttgacagag gcaccattga 480
accccaaagc caacagggag aagatgaccc agatcatgtt tgaaaccttc aatacccccg 540
ccatgtacgt cgccatccaa gctgtattgt ctctgtatgc ttctggtcgt acaactggta 600
ttgtgctgga ttctggagat ggtgtttctc acacagtacc aatctatgaa ggttatgccc 660
ttcctcatgc catccttcgt ttggacttgg ctggtagaga cttgactgat taccttatga 720
aaattctgac tgaacgtggt tactctttca 750
<210> 395
<211> 204
<212> БЕЛОК
<213> Leptinotarsa decemlineata
<400> 395
Pro Ile Asn Met Cys Asp Glu Glu Val Ala Ala Leu Val Val Asp Asn
1 5 10 15
Gly Ser Gly Met Cys Lys Ala Gly Phe Ala Gly Asp Asp Ala Pro Arg
20 25 30
Ala Val Phe Pro Ser Ile Val Gly Arg Pro Arg His Gln Gly Val Met
35 40 45
Val Gly Met Gly Gln Lys Asp Ser Tyr Val Gly Asp Glu Ala Gln Ser
50 55 60
Lys Arg Gly Ile Leu Thr Leu Lys Tyr Pro Ile Glu His Gly Ile Val
65 70 75 80
Thr Asn Trp Asp Asp Met Glu Lys Ile Trp His His Thr Phe Tyr Asn
85 90 95
Glu Leu Arg Val Ala Pro Glu Glu His Pro Val Leu Leu Thr Glu Ala
100 105 110
Pro Leu Asn Pro Lys Ala Asn Arg Glu Lys Met Thr Gln Ile Met Phe
115 120 125
Glu Thr Phe Asn Thr Pro Ala Met Tyr Val Ala Ile Gln Ala Val Leu
130 135 140
Ser Leu Tyr Ala Ser Gly Arg Thr Thr Gly Ile Val Leu Asp Ser Gly
145 150 155 160
Asp Gly Val Ser His Thr Val Pro Ile Tyr Glu Gly Tyr Ala Leu Pro
165 170 175
His Ala Ile Leu Arg Leu Asp Leu Ala Gly Arg Asp Leu Thr Asp Tyr
180 185 190
Leu Met Lys Ile Leu Thr Glu Arg Gly Tyr Ser Phe
195 200
<210> 396
<211> 44
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 396
gcgtaatacg actcactata ggatgtgtga cgaagaggtt gccg 44
<210> 397
<211> 24
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 397
gtcaacaaaa cagggtgctc ttcg 24
<210> 398
<211> 22
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 398
atgtgtgacg aagaggttgc cg 22
<210> 399
<211> 46
<212> ДНК
<213> Искусственная
<220>
<223> Праймер
<400> 399
gcgtaatacg actcactata gggtcaacaa aacagggtgc tcttcg 46
<210> 400
<211> 320
<212> ДНК
<213> Leptinotarsa decemlineata
<400> 400
atgtgtgacg aagaggttgc cgcattagtc gtagacaatg gatctggtat gtgcaaagct 60
ggatttgctg gggatgatgc cccccgtgca gttttcccat ccattgttgg tcgtccaaga 120
catcaaggag ttatggtagg aatgggccaa aaggactcgt atgtaggaga tgaagcccaa 180
agcaaaagag gtatccttac cttgaaatac cccattgaac acggtattgt cacaaactgg 240
gatgatatgg agaaaatctg gcaccatacc ttctacaatg aacttcgagt tgcccccgaa 300
gagcaccctg ttttgttgac 320
<210> 401
<211> 1152
<212> ДНК
<213> Искусственная
<220>
<223> Шпилька
<400> 401
tgacgacctt gagttggttt ctgaagttga actcggcagc cttcttctgg agcttggcga 60
atttgttttc gtacttggaa acctttttca aggtaggttt gacgaattta ccacggaggt 120
cgttgacctg gctgttgagg tcggcgatct caaggtcacg tctctccact tcgaattcga 180
tgtcaatttt actcctctcc agagcgtcaa ttcgcttatg gtagtctgtg cagagtttct 240
tcaaggttgc ttcattggcg ttgtcgacgt cggcaatttg cccgcagcgc tcctcaatcg 300
ttcgcctcct ctcagctgct ttgcgttcct gctccttctt cagttcctca gcggcttttt 360
tcctcagcag gagtcggagt ttcttcttcc tttccggggt catgaaaccc ttcttggctt 420
tcttcgcctt agaggcttcc tccatcctct tgcgcacttc agcgcgcttc ctctcgattt 480
cggcctgttt gtctagaagg taagtgtaca cactacattt tcatgaacat tattgcgacc 540
gttgagattc tcattgtttg gtgattgatt atctaaagta gaagcatgaa tagatataac 600
ataaactagt aactaatggg ttagttatgg gtatacttca tgcttttctc tcaggctcga 660
gcaaacaggc cgaaatcgag aggaagcgcg ctgaagtgcg caagaggatg gaggaagcct 720
ctaaggcgaa gaaagccaag aagggtttca tgaccccgga aaggaagaag aaactccgac 780
tcctgctgag gaaaaaagcc gctgaggaac tgaagaagga gcaggaacgc aaagcagctg 840
agaggaggcg aacgattgag gagcgctgcg ggcaaattgc cgacgtcgac aacgccaatg 900
aagcaacctt gaagaaactc tgcacagact accataagcg aattgacgct ctggagagga 960
gtaaaattga catcgaattc gaagtggaga gacgtgacct tgagatcgcc gacctcaaca 1020
gccaggtcaa cgacctccgt ggtaaattcg tcaaacctac cttgaaaaag gtttccaagt 1080
acgaaaacaa attcgccaag ctccagaaga aggctgccga gttcaacttc agaaaccaac 1140
tcaaggtcgt ca 1152
<210> 402
<211> 1192
<212> ДНК
<213> Искусственная
<220>
<223> Шпилька
<400> 402
ctcttgcttc ttggtggcga tcctctcctc gcgcctcttc ttcgcctcct tgaccttgag 60
acgtctcgcc tctgcctggt ccttcaacat ctttgatctc gccttttcag ccttcttctt 120
gtgaatgaag tccatcagta ccctcttgtt tttgaagacg ttacctttgg ctttcatgta 180
aaggtcgtgg tacatttgcc tatcgatctt cttggcttct ctgtattttt taaggagccg 240
tcgcaggact ctcattctgt tgacccacag gaccttcaca ggcattctgg cgttggcggt 300
acccttcctc ttaccgaagc cacagtgacg acccttccgt ctggcttctg tgtttttacg 360
gacgcgggct ctggagtgga cagccacagg ctttttgatg atcaaaccat ccttgatcag 420
cttacggatg ttttgcctag agttggtgtt ggcgatttcg ttgatttcat tagggtccaa 480
ccacactttc ttcttgccgc atctcatcac ctctagaagg taagtgtaca cactacattt 540
tcatgaacat tattgcgacc gttgagattc tcattgtttg gtgattgatt atctaaagta 600
gaagcatgaa tagatataac ataaactagt aactaatggg ttagttatgg gtatacttca 660
tgcttttctc tcaggctcga gggtgatgag atgcggcaag aagaaagtgt ggttggaccc 720
taatgaaatc aacgaaatcg ccaacaccaa ctctaggcaa aacatccgta agctgatcaa 780
ggatggtttg atcatcaaaa agcctgtggc tgtccactcc agagcccgcg tccgtaaaaa 840
cacagaagcc agacggaagg gtcgtcactg tggcttcggt aagaggaagg gtaccgccaa 900
cgccagaatg cctgtgaagg tcctgtgggt caacagaatg agagtcctgc gacggctcct 960
taaaaaatac agagaagcca agaagatcga taggcaaatg taccacgacc tttacatgaa 1020
agccaaaggt aacgtcttca aaaacaagag ggtactgatg gacttcattc acaagaagaa 1080
ggctgaaaag gcgagatcaa agatgttgaa ggaccaggca gaggcgagac gtctcaaggt 1140
caaggaggcg aagaagaggc gcgaggagag gatcgccacc aagaagcaag ag 1192
<210> 403
<211> 792
<212> ДНК
<213> Искусственная
<220>
<223> Шпилька
<400> 403
actctgtctg gcttttggct gtgacgcaca gttcatagag ataaccttca cccgaatatg 60
ccttgcgagg tcgcaaaatc ggcgaaattc catacctgtt caccgacgac ggcgctggat 120
caattccaca gttttcgcga tccagactga atgcccacag gccgtcgagt tttttgattt 180
cagatacgta cacttttccc ggcaataaca tacggcgtga catcggcttc aaatggcgta 240
tagccgccct gatgctccat cacactttgc cgtaatgagt gaccgcatcg aaacgcagca 300
cgatacgctg gtctagaagg taagtgtaca cactacattt tcatgaacat tattgcgacc 360
gttgagattc tcattgtttg gtgattgatt atctaaagta gaagcatgaa tagatataac 420
ataaactagt aactaatggg ttagttatgg gtatacttca tgcttttctc tcaggctcga 480
gccagcgtat cgtgctgcgt ttcgatgcgg tcactcatta cggcaaagtg tgatggagca 540
tcagggcggc tatacgccat ttgaagccga tgtcacgccg tatgttattg ccgggaaaag 600
tgtacgtatc tgaaatcaaa aaactcgacg gcctgtgggc attcagtctg gatcgcgaaa 660
actgtggaat tgatccagcg ccgtcgtcgg tgaacaggta tggaatttcg ccgattttgc 720
gacctcgcaa ggcatattcg ggtgaaggtt atctctatga actgtgcgtc acagccaaaa 780
gccagacaga gt 792
<210> 404
<211> 839
<212> ДНК
<213> leptinotarsa decemlineata
<400> 404
cagtcagcac cgagtccttg ttgactgctc acattttcca tcgtttctac cagaacaaca 60
gcaacaactt tcatcatggc ggacgacgag gaaaagagga ggaaacaagc ggaaattgaa 120
cgcaagaggg ctgaggtcag ggctcgcatg gaagaggcct ccaaggccaa aaaagccaag 180
aaaggtttca tgacccctga gaggaagaag aaacttaggt tattgctgag aaagaaagca 240
gcagaagaac tgaaaaaaga acaagaacgc aaagctgccg aaaggcgtat tattgaagag 300
agatgcggaa aaccaaaact cattgatgag gcaaatgaag agcaggtgag gaactattgc 360
aagttatatc acggtagaat agctaaactg gaggaccaga aatttgattt ggaatacctt 420
gtcaaaaaga aagacatgga gatcgccgaa ttgaacagtc aagtcaacga cctcaggggt 480
aaattcgtca aacccactct caagaaagta tccaaatacg agaacaaatt tgctaaactc 540
caaaagaaag cagcagaatt caatttccgt aatcaactga aagttgtaaa gaagaaggag 600
ttcaccctgg aggaggaaga caaagaaaag aagcccgatt ggtcgaagaa gggagacgaa 660
aagaaggtac aagaagtgga agcatgatct gtccctacaa tttaatattt cccttcgtcc 720
gtggaaattt tacaacttaa gatatattta ttttattcgc ttcttatgag actatgaaag 780
tgatgtctgc atgtatatta ttcgttttat gtatgtatta aaaaaagaac ttgattgaa 839
<210> 405
<211> 203
<212> БЕЛОК
<213> leptinotarsa decemlineata
<400> 405
Met Ala Asp Asp Glu Glu Lys Arg Arg Lys Gln Ala Glu Ile Glu Arg
1 5 10 15
Lys Arg Ala Glu Val Arg Ala Arg Met Glu Glu Ala Ser Lys Ala Lys
20 25 30
Lys Ala Lys Lys Gly Phe Met Thr Pro Glu Arg Lys Lys Lys Leu Arg
35 40 45
Leu Leu Leu Arg Lys Lys Ala Ala Glu Glu Leu Lys Lys Glu Gln Glu
50 55 60
Arg Lys Ala Ala Glu Arg Arg Ile Ile Glu Glu Arg Cys Gly Lys Pro
65 70 75 80
Lys Leu Ile Asp Glu Ala Asn Glu Glu Gln Val Arg Asn Tyr Cys Lys
85 90 95
Leu Tyr His Gly Arg Ile Ala Lys Leu Glu Asp Gln Lys Phe Asp Leu
100 105 110
Glu Tyr Leu Val Lys Lys Lys Asp Met Glu Ile Ala Glu Leu Asn Ser
115 120 125
Gln Val Asn Asp Leu Arg Gly Lys Phe Val Lys Pro Thr Leu Lys Lys
130 135 140
Val Ser Lys Tyr Glu Asn Lys Phe Ala Lys Leu Gln Lys Lys Ala Ala
145 150 155 160
Glu Phe Asn Phe Arg Asn Gln Leu Lys Val Val Lys Lys Lys Glu Phe
165 170 175
Thr Leu Glu Glu Glu Asp Lys Glu Lys Lys Pro Asp Trp Ser Lys Lys
180 185 190
Gly Asp Glu Lys Lys Val Gln Glu Val Glu Ala
195 200
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБЫ И СООТВЕТСТВУЮЩИЕ КОМПОЗИЦИИ ДЛЯ ИЗГОТОВЛЕНИЯ ПРОДУКТОВ ПИТАНИЯ И КОРМОВ | 2018 |
|
RU2780586C2 |
| КОМПОЗИЦИИ И СООТВЕТСТВУЮЩИЕ СПОСОБЫ ДЛЯ СЕЛЬСКОГО ХОЗЯЙСТВА | 2018 |
|
RU2805081C2 |
| КОНТРОЛЬ ЖЕСТКОКРЫЛЫХ ВРЕДИТЕЛЕЙ С ПРИМЕНЕНИЕМ МОЛЕКУЛ РНК | 2017 |
|
RU2783144C2 |
| КОМПОЗИЦИИ И СООТВЕТСТВУЮЩИЕ СПОСОБЫ КОНТРОЛЯ ЗАБОЛЕВАНИЙ, ПЕРЕДАВАЕМЫХ ПЕРЕНОСЧИКАМИ | 2018 |
|
RU2777518C2 |
| ИНСЕКТИЦИДНЫЕ БЕЛКИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2740312C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ, НАЦЕЛЕННЫЕ НА FLT3 | 2018 |
|
RU2820859C2 |
| КОМПОЗИЦИИ И СООТВЕТСТВУЮЩИЕ СПОСОБЫ КОНТРОЛЯ ЗАБОЛЕВАНИЙ, ПЕРЕДАВАЕМЫХ ПЕРЕНОСЧИКАМИ | 2018 |
|
RU2804136C2 |
| БЕЛОК И ВАКЦИНА ПРОТИВ ИНФЕКЦИИ SARS-CoV-2 | 2020 |
|
RU2815060C1 |
| Химерные антигенные рецепторы, нацеливающиеся на CD70 | 2019 |
|
RU2801824C2 |
| КОМПОЗИЦИИ И СООТВЕТСТВУЮЩИЕ СПОСОБЫ ДЛЯ СЕЛЬСКОГО ХОЗЯЙСТВА | 2018 |
|
RU2783258C2 |
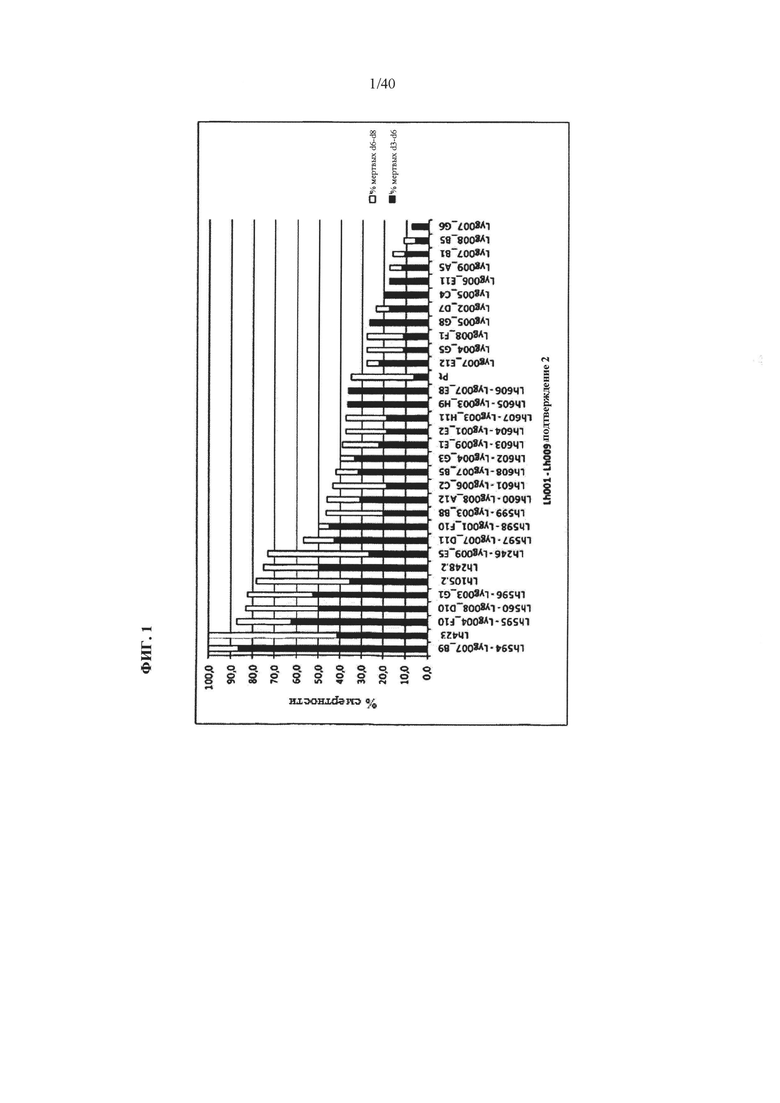

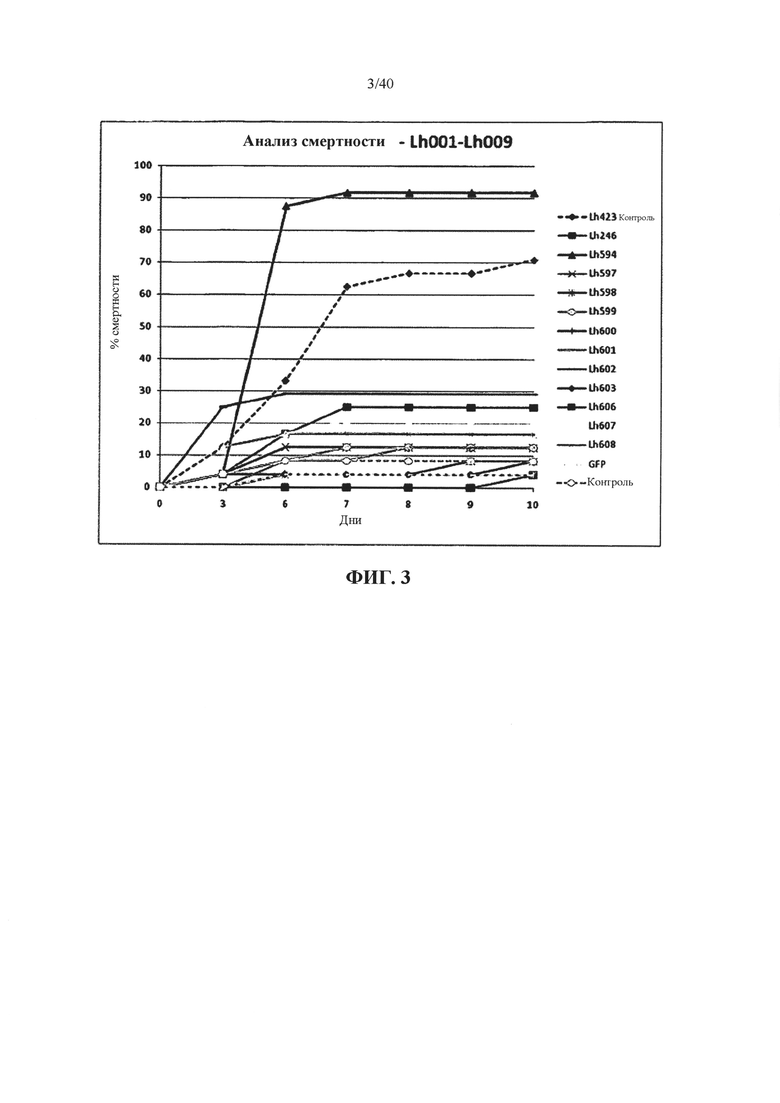
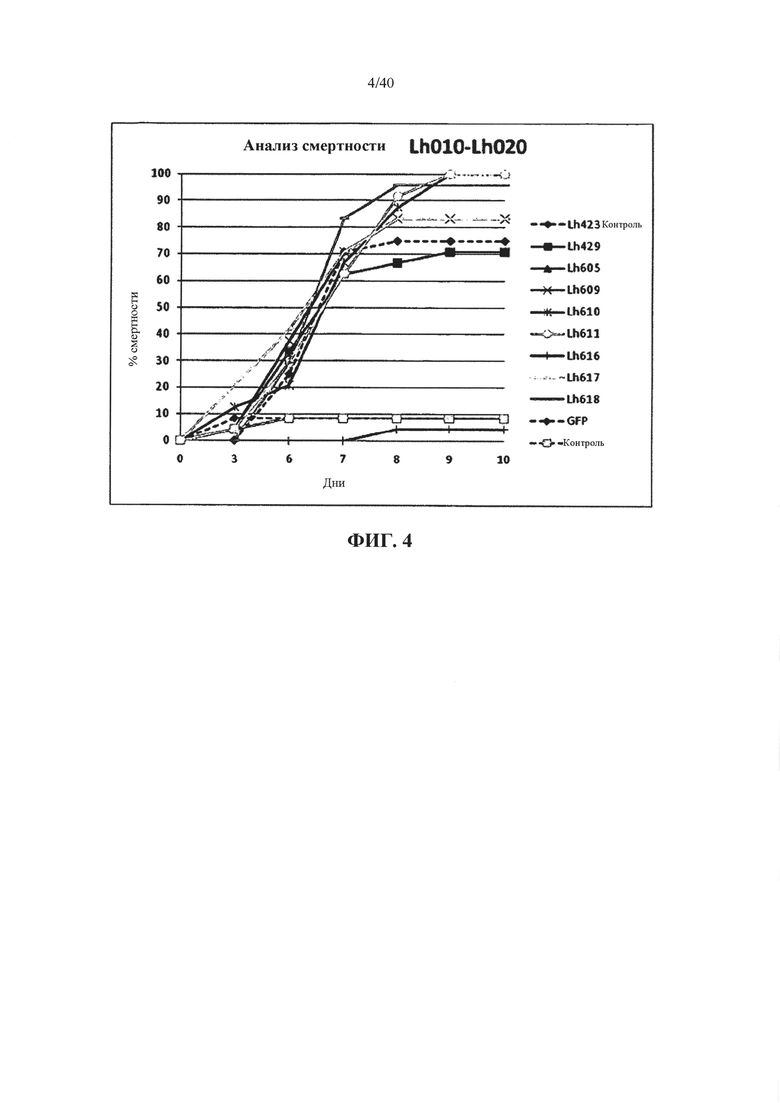
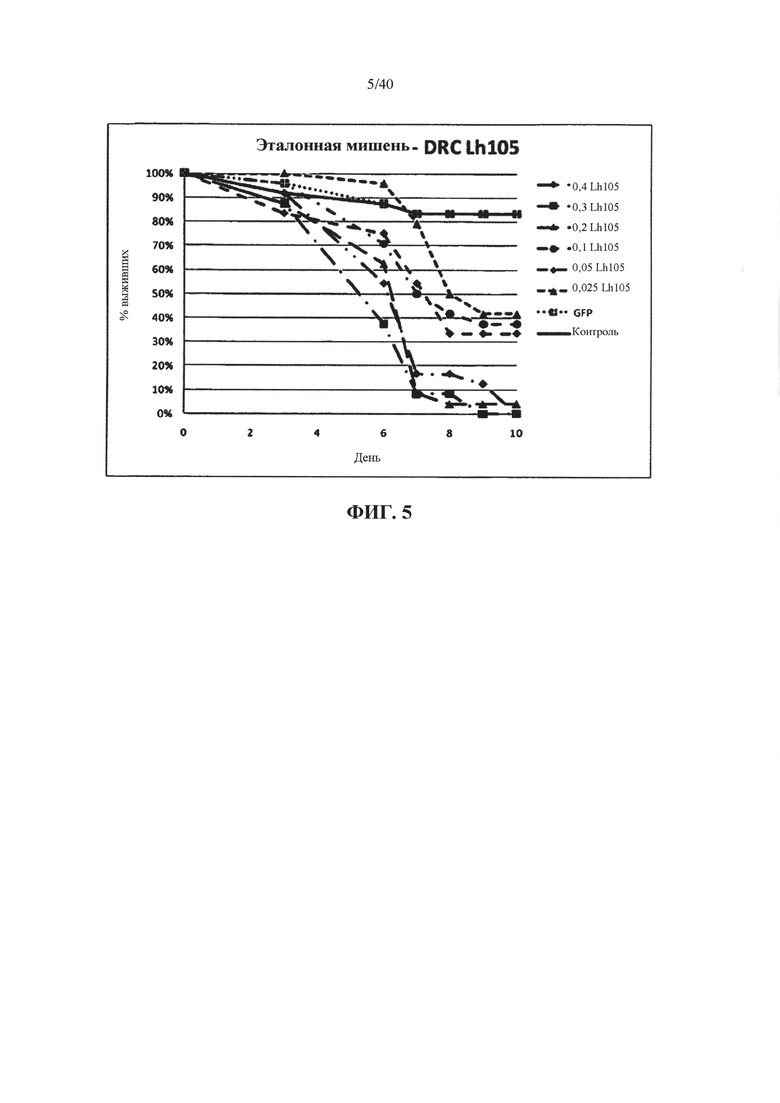
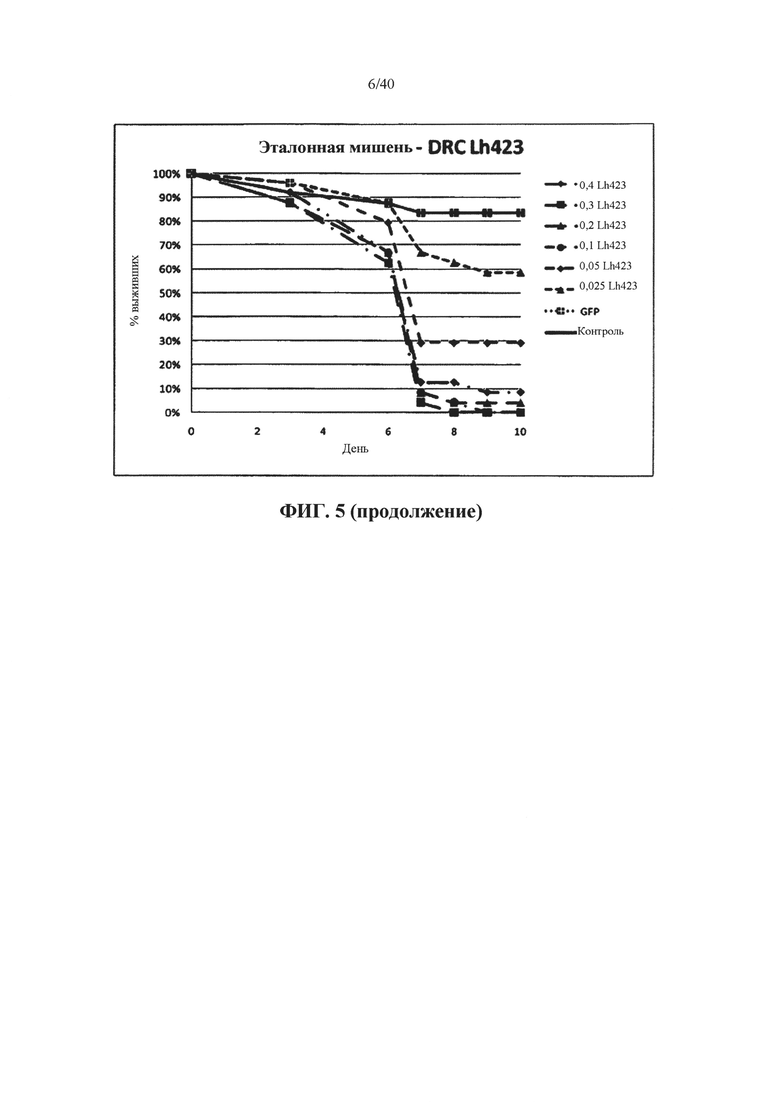
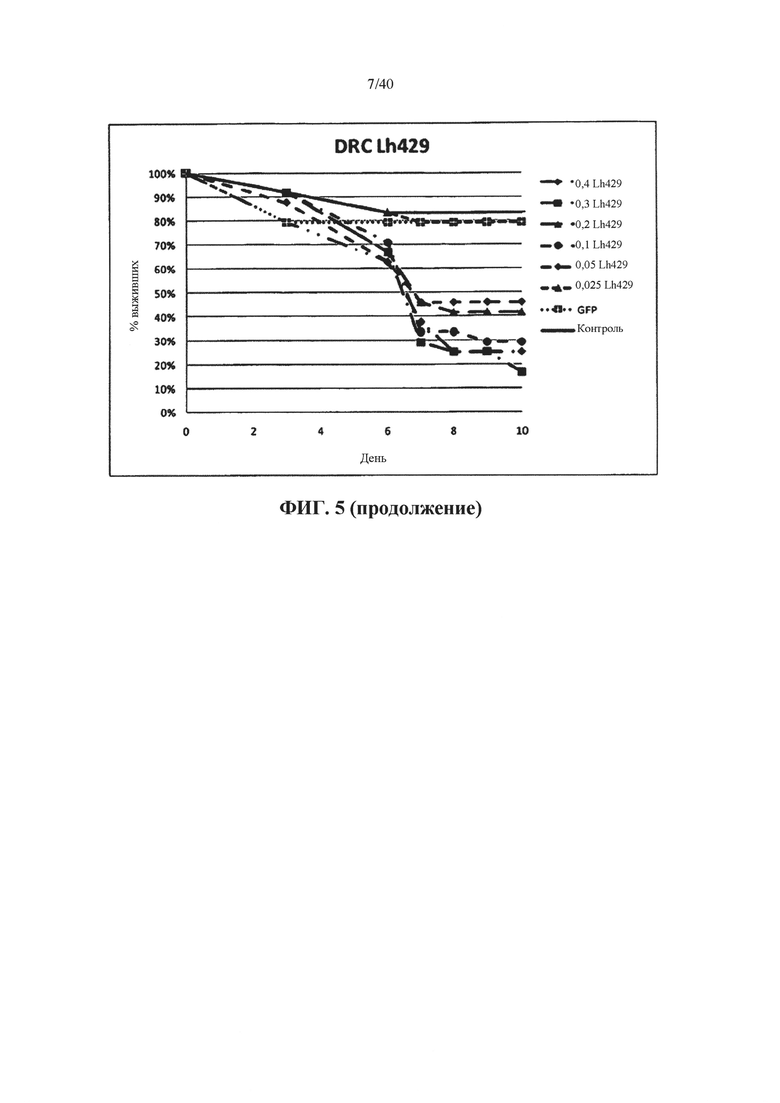
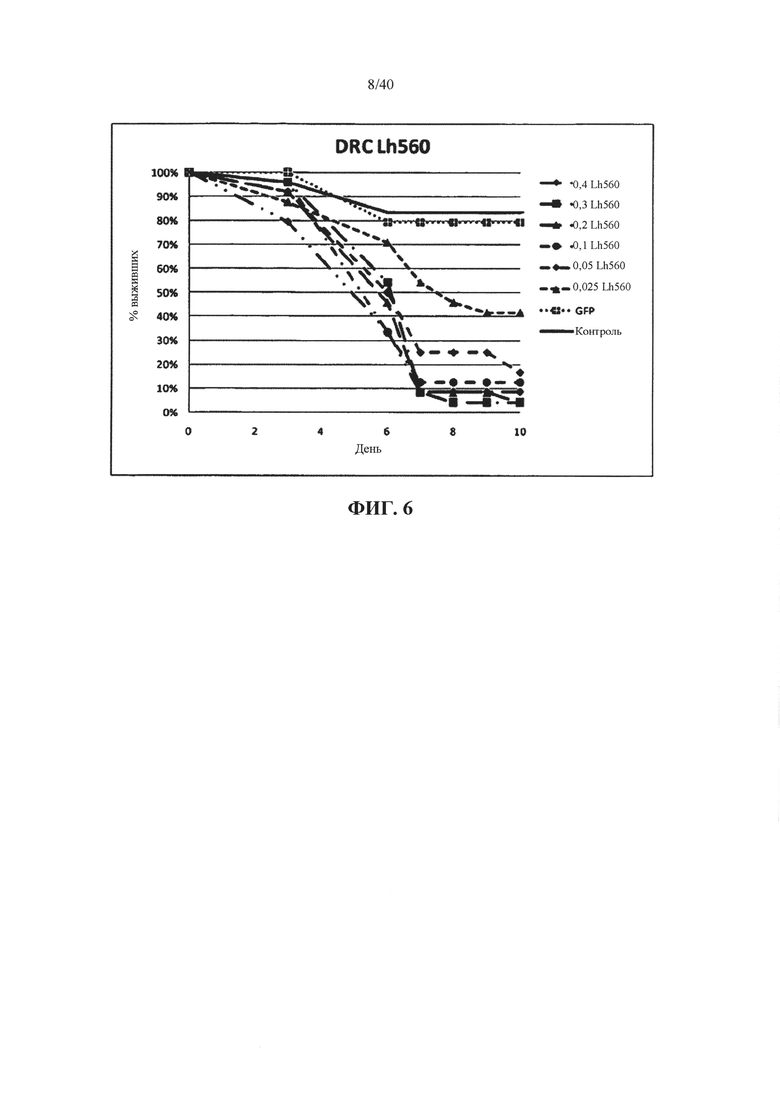
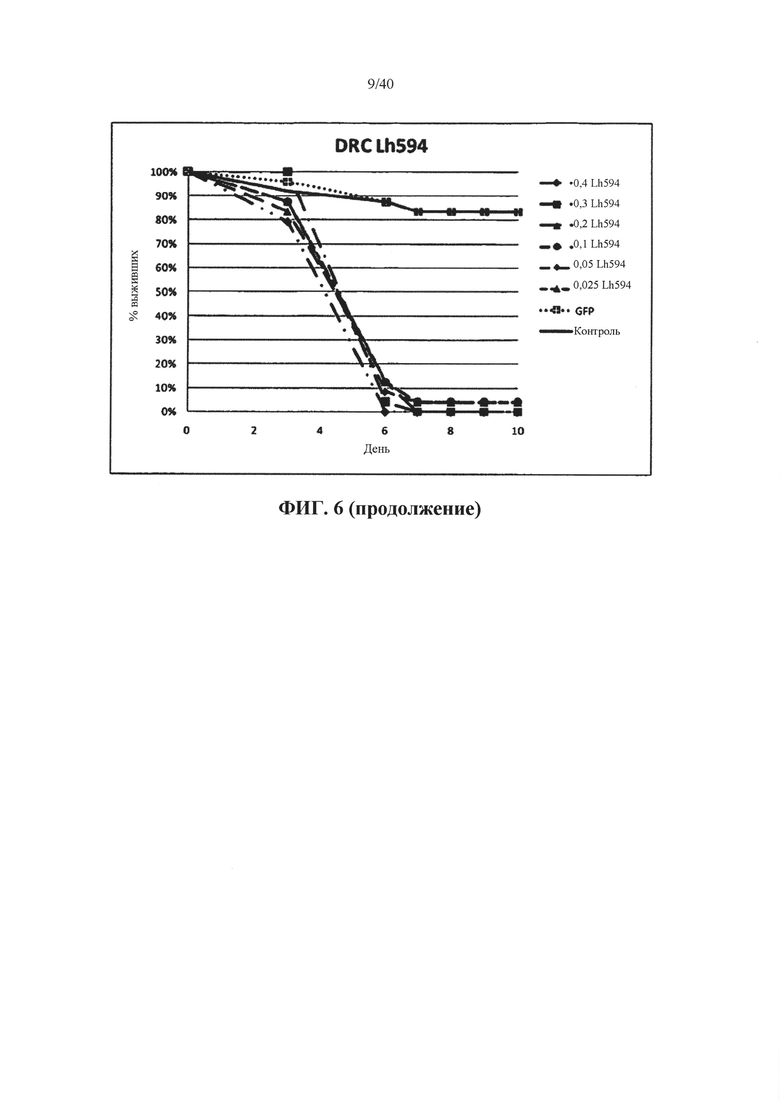
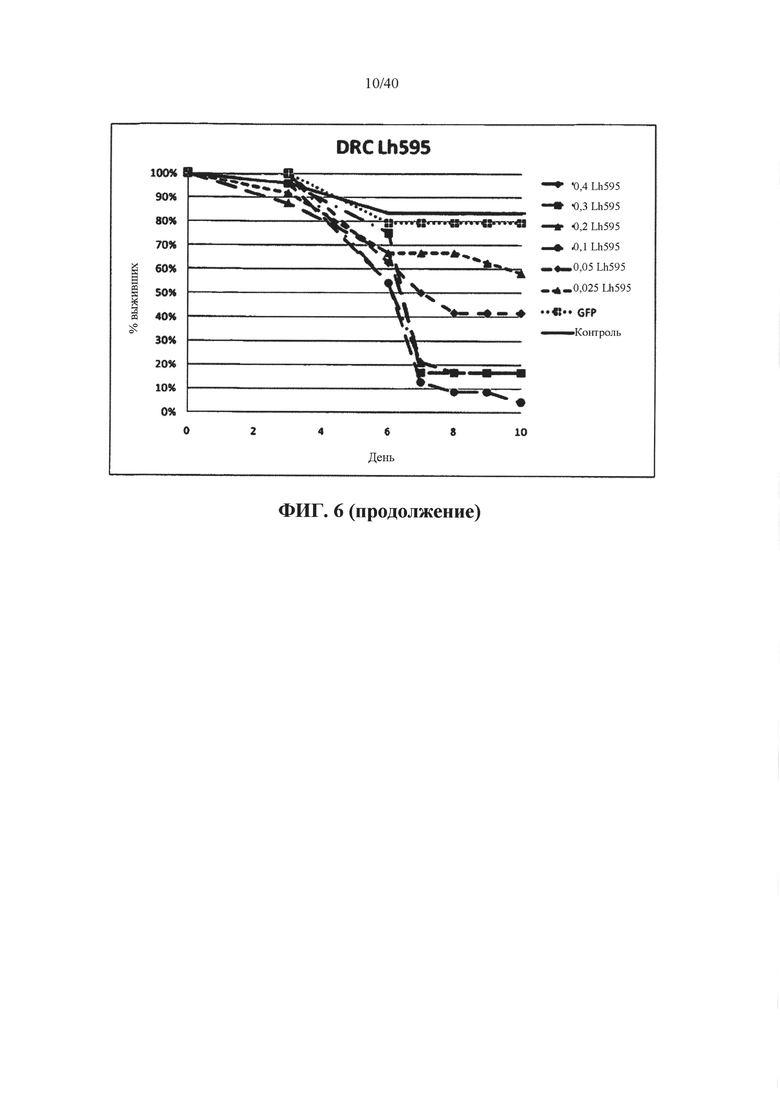
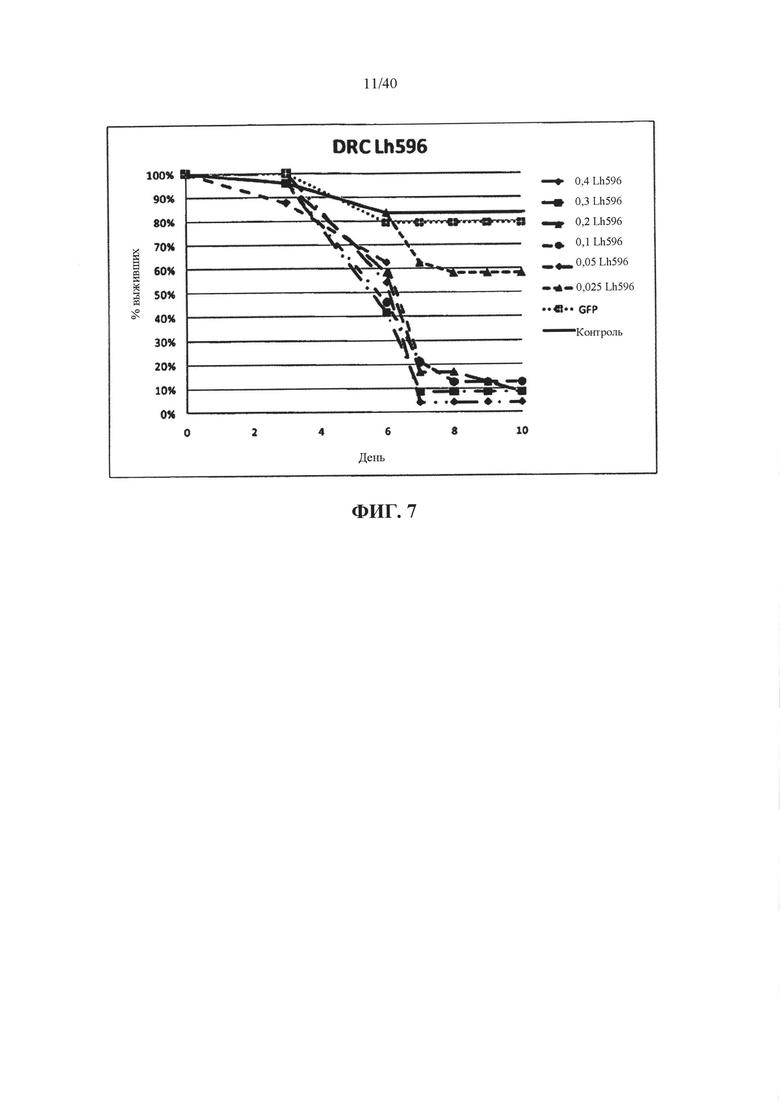
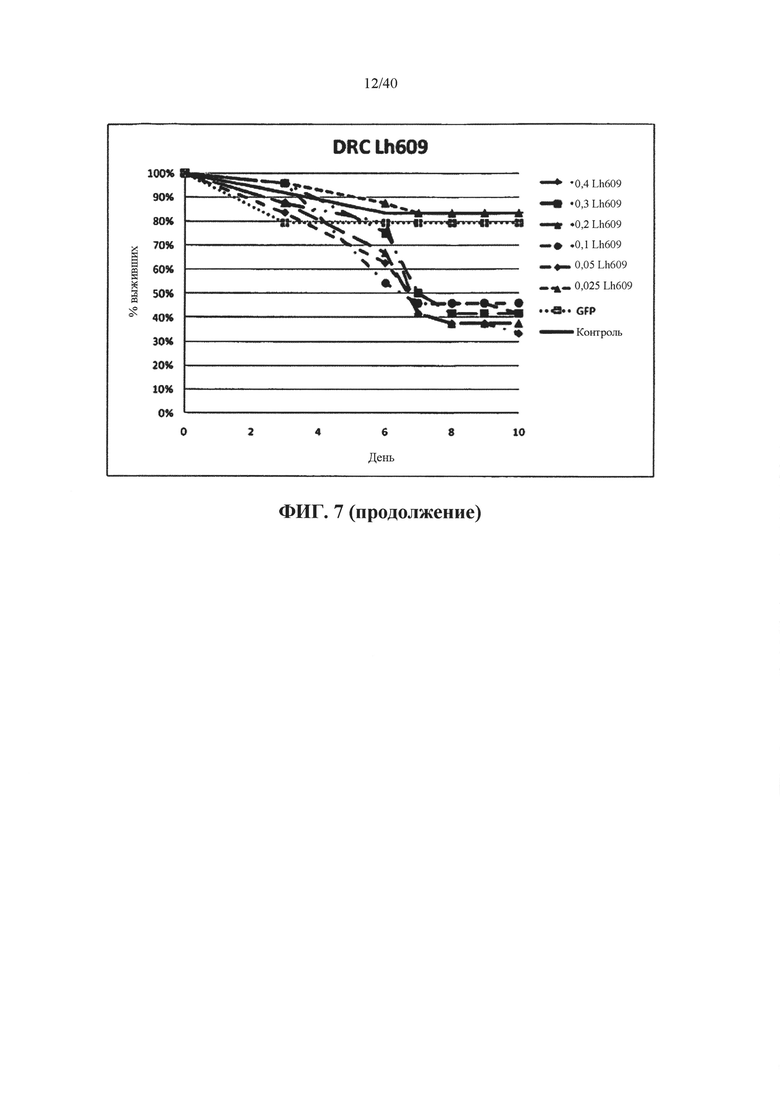

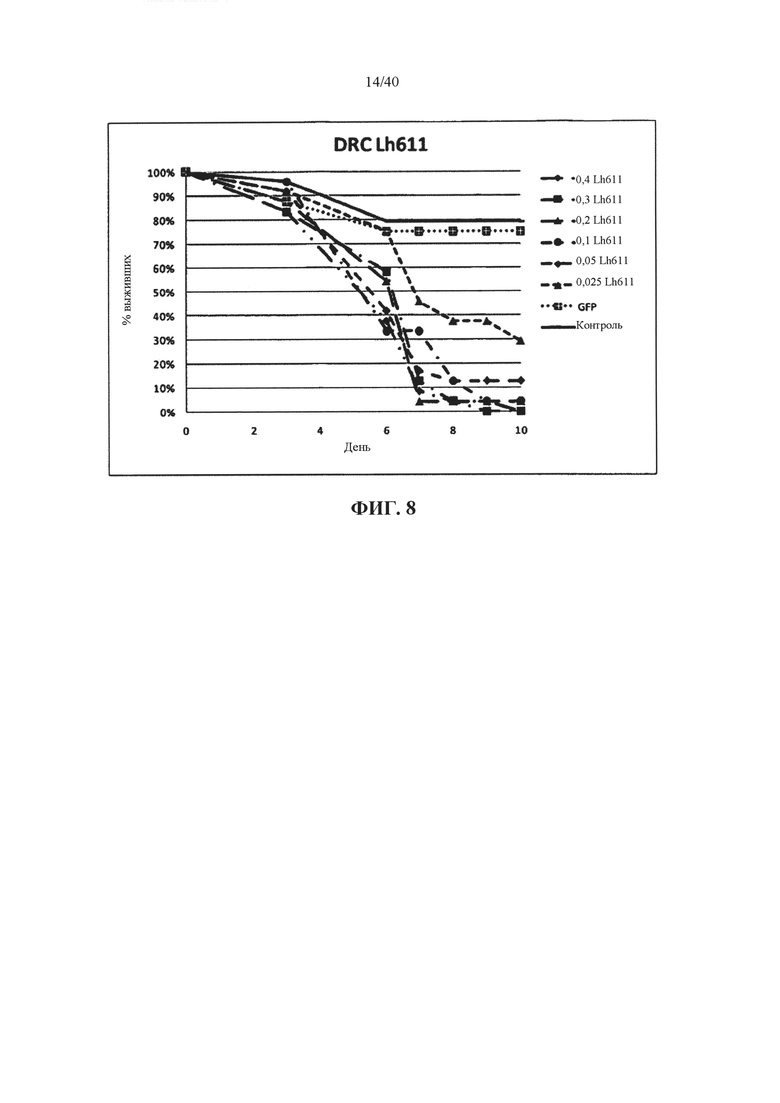
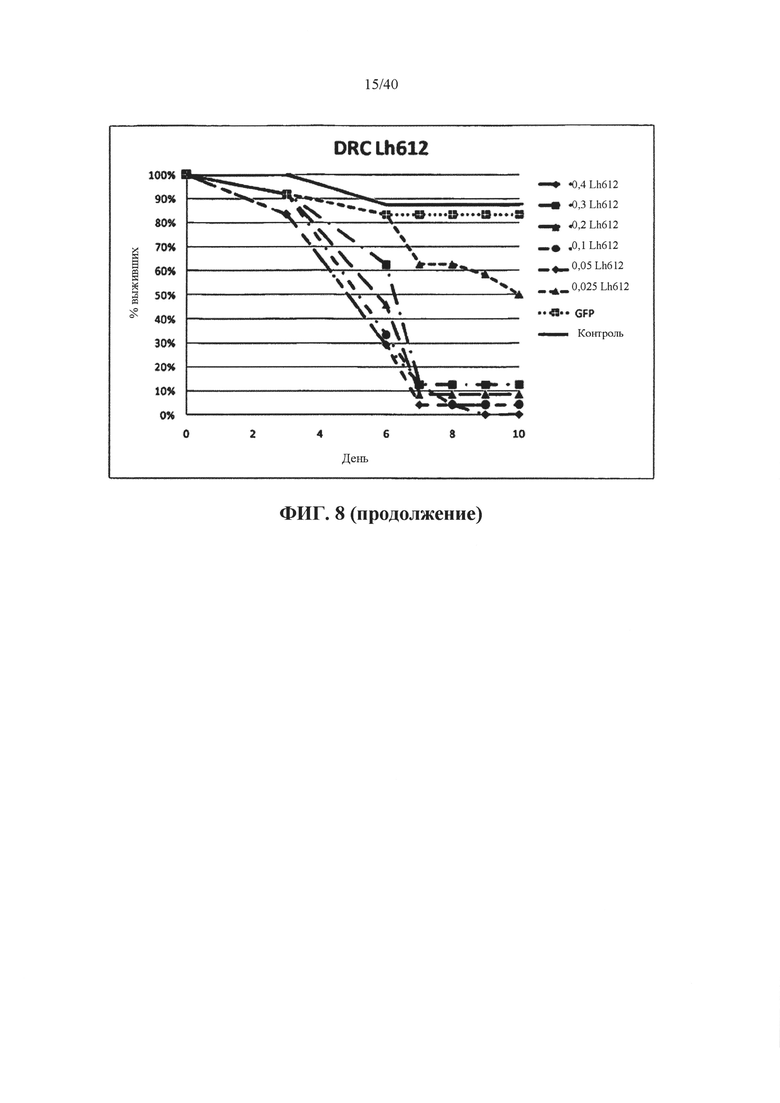

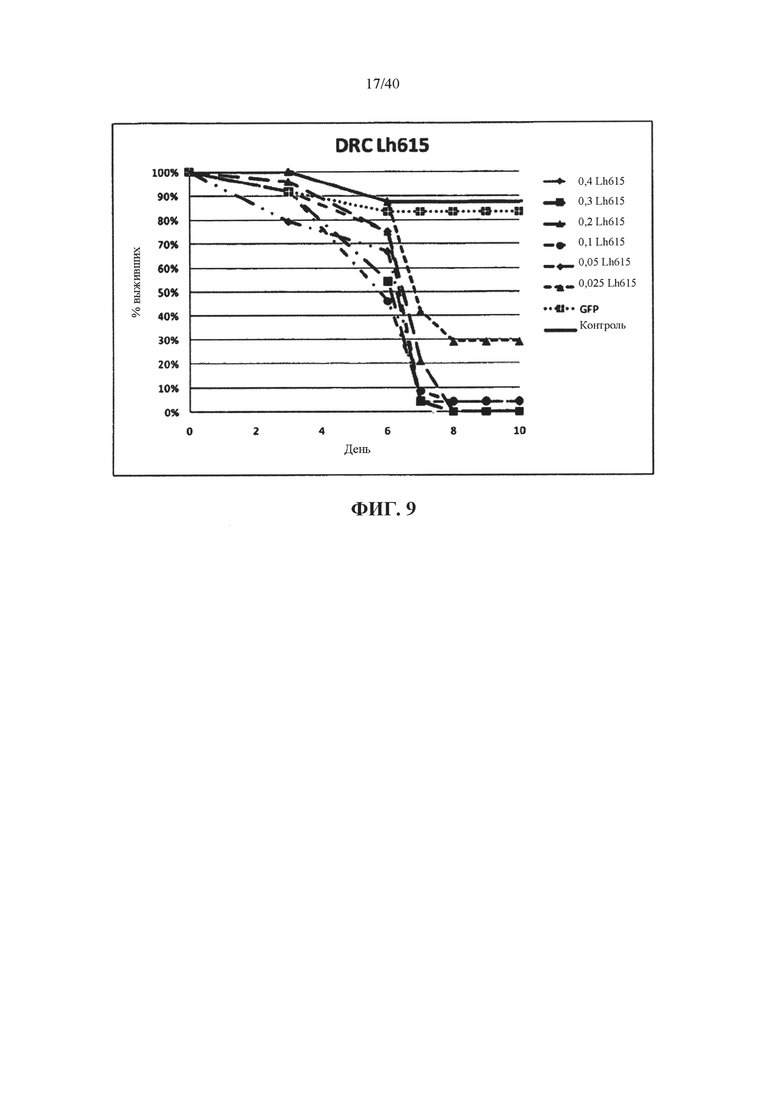

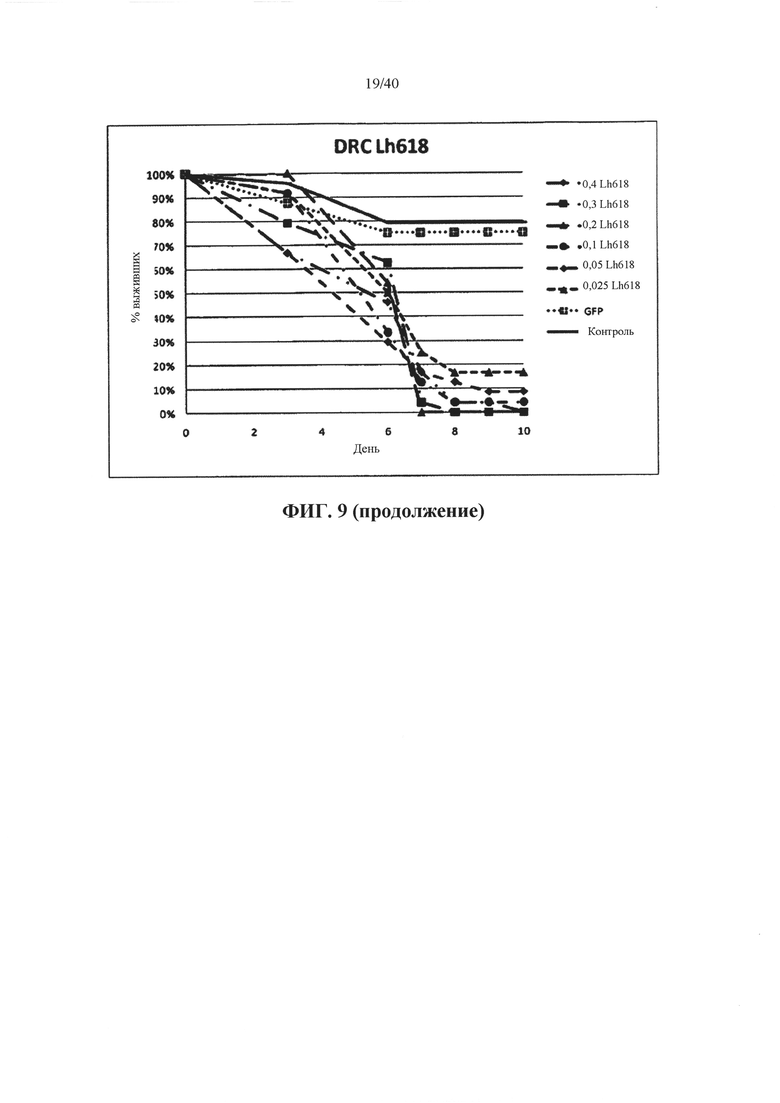
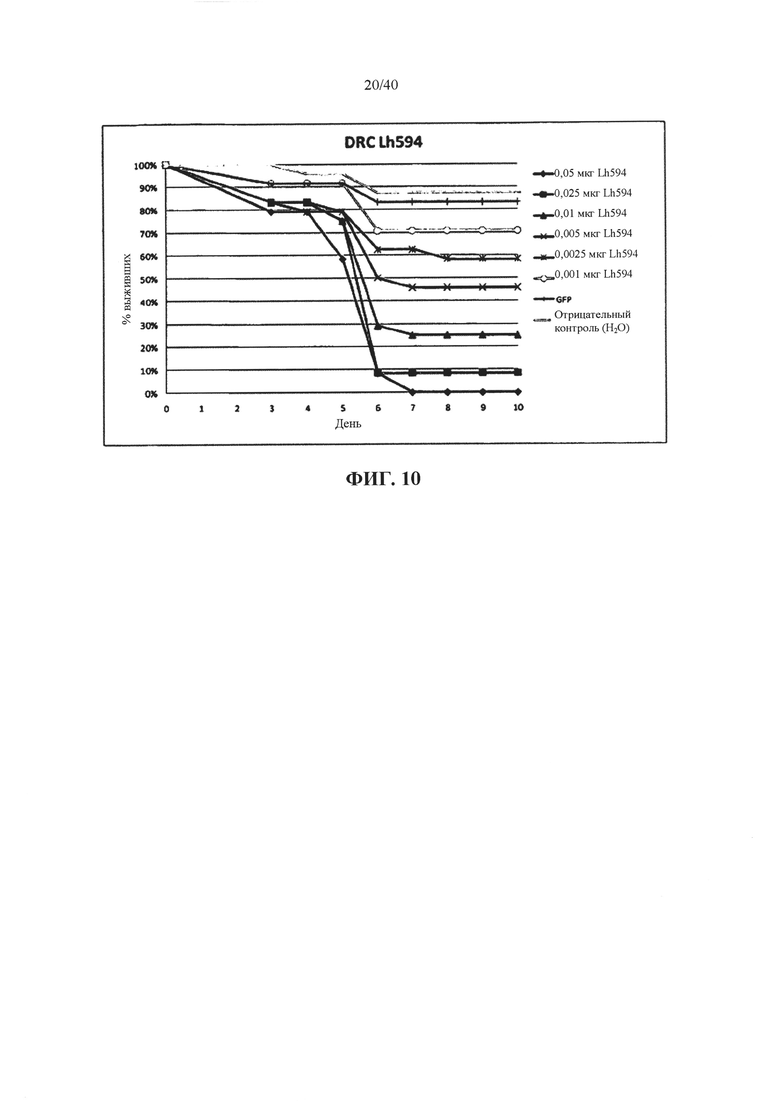
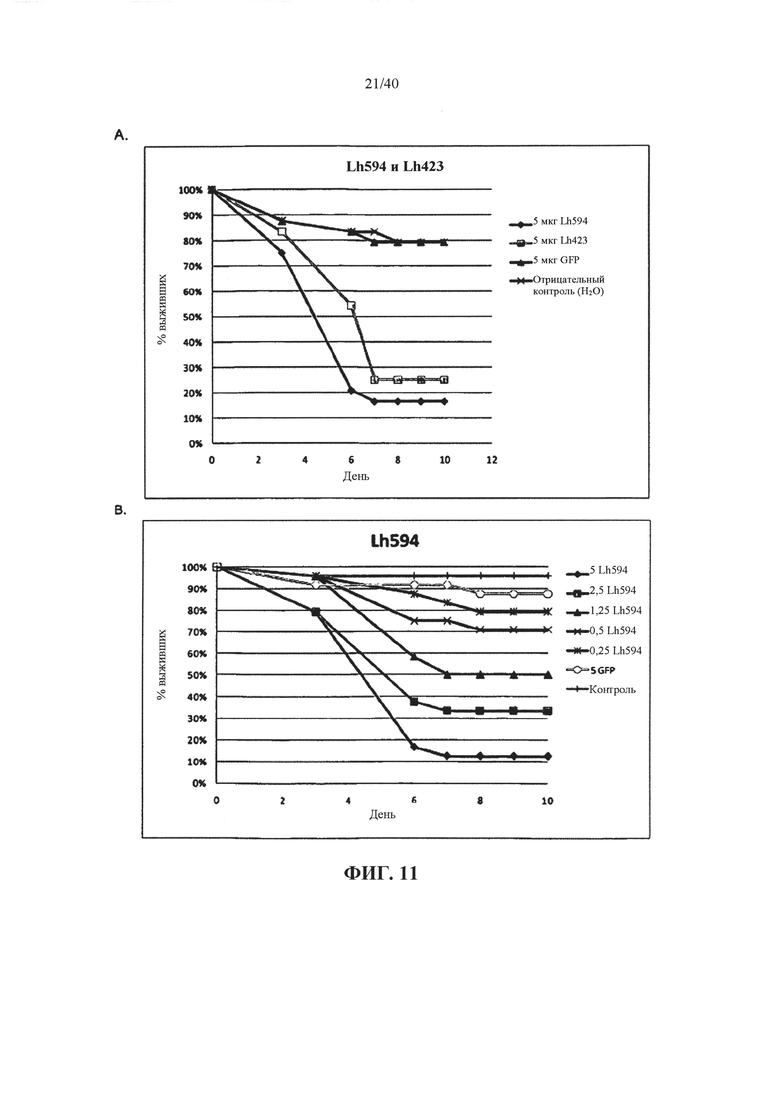

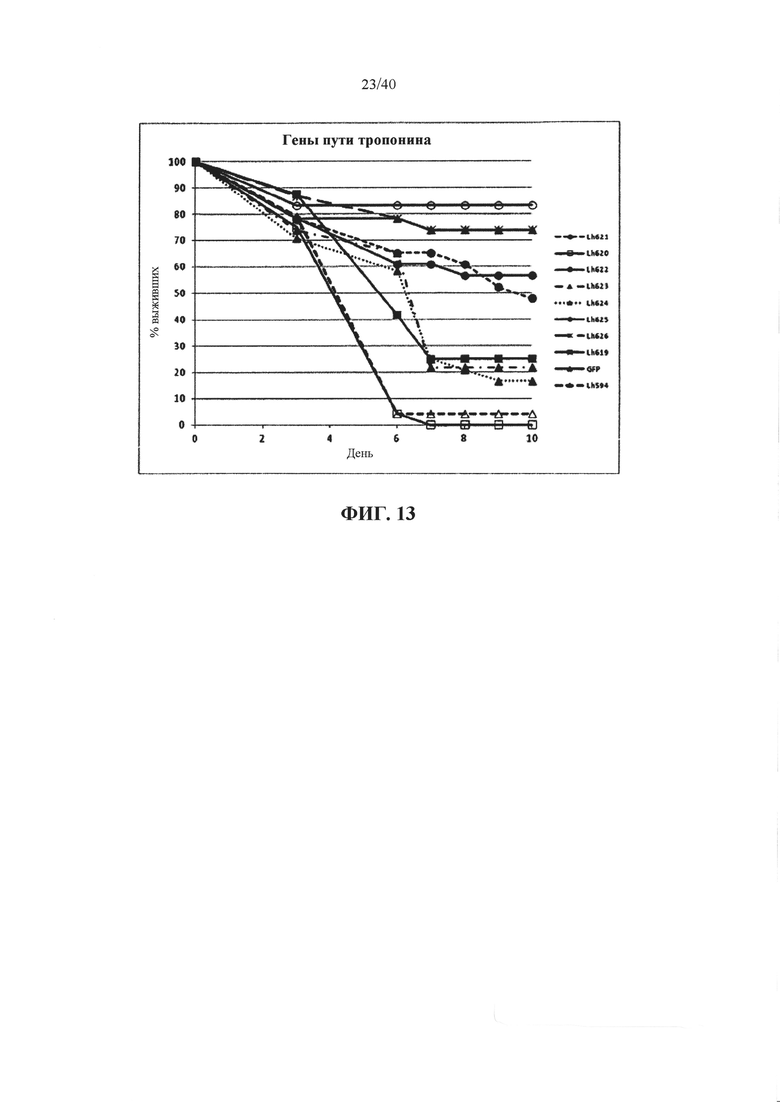
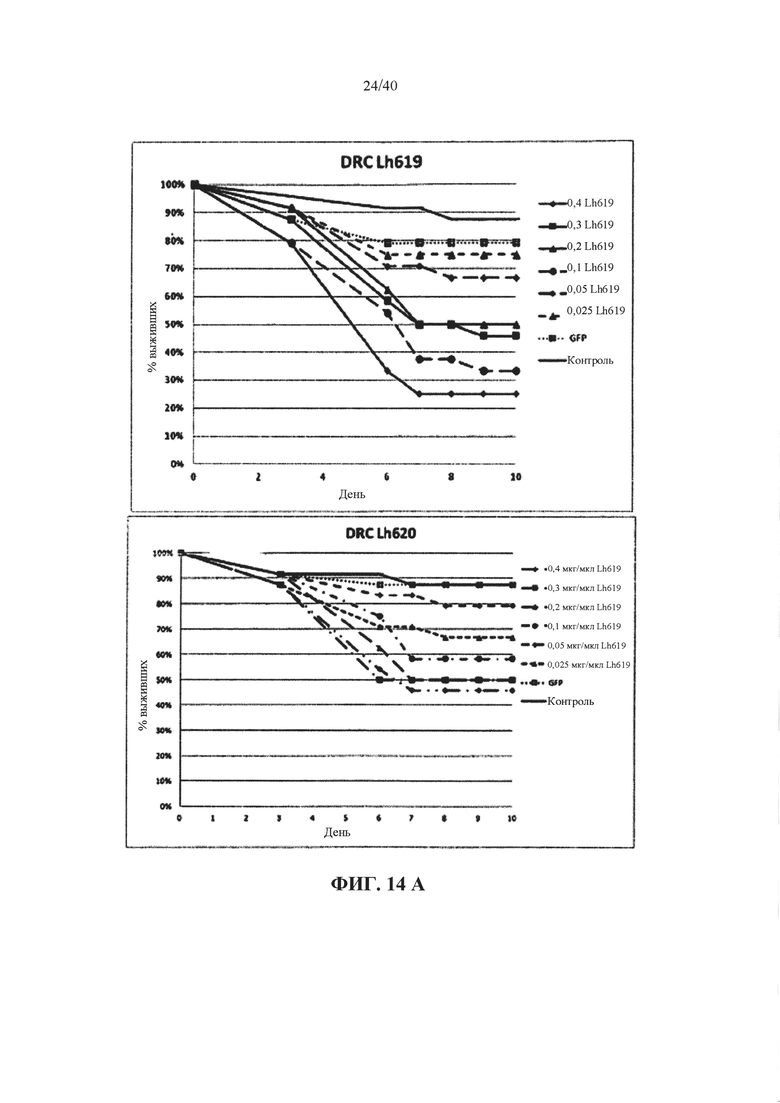
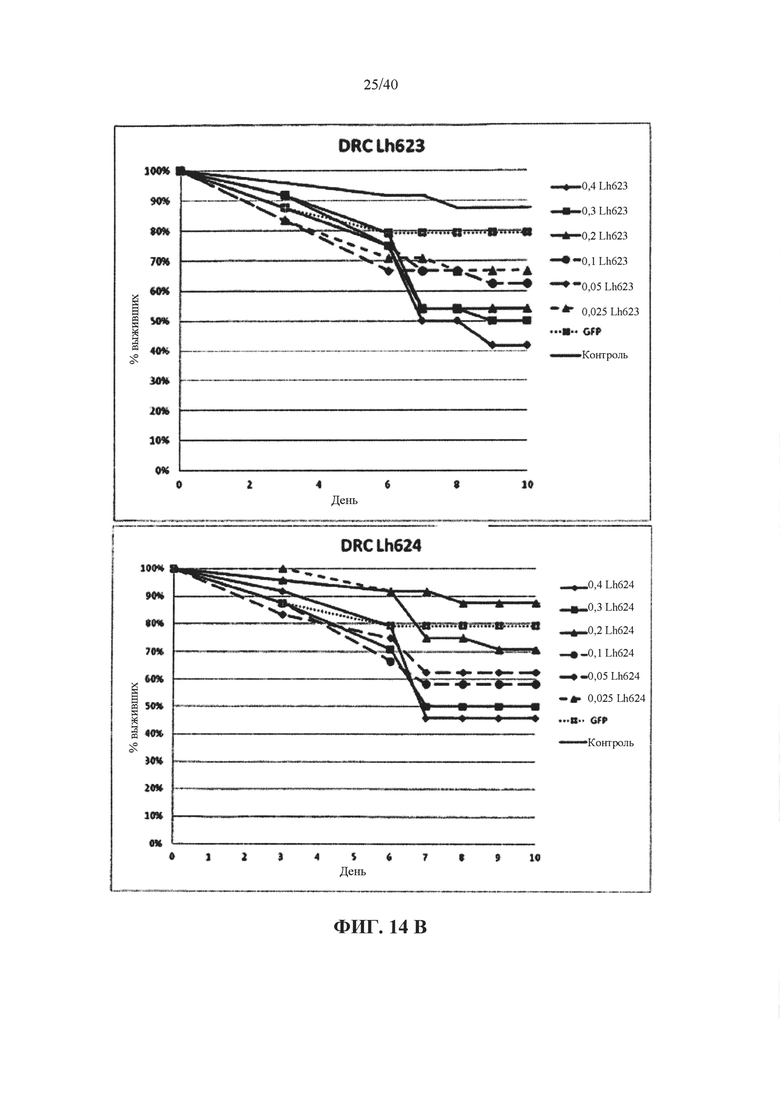
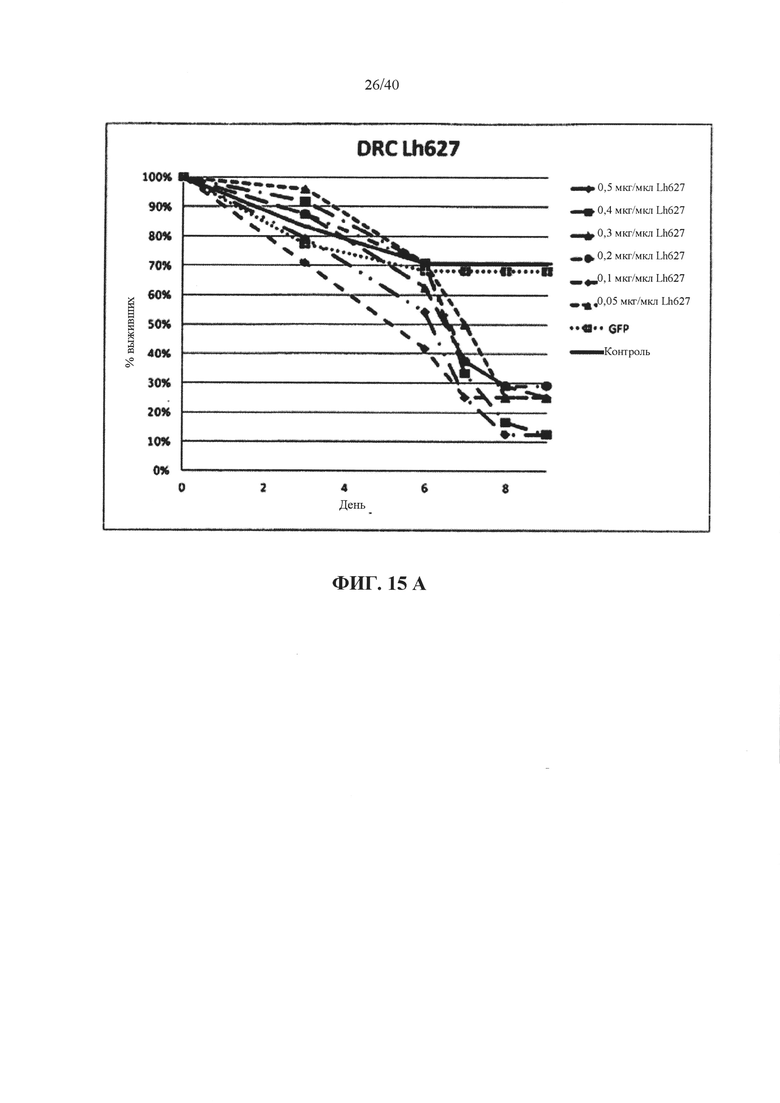
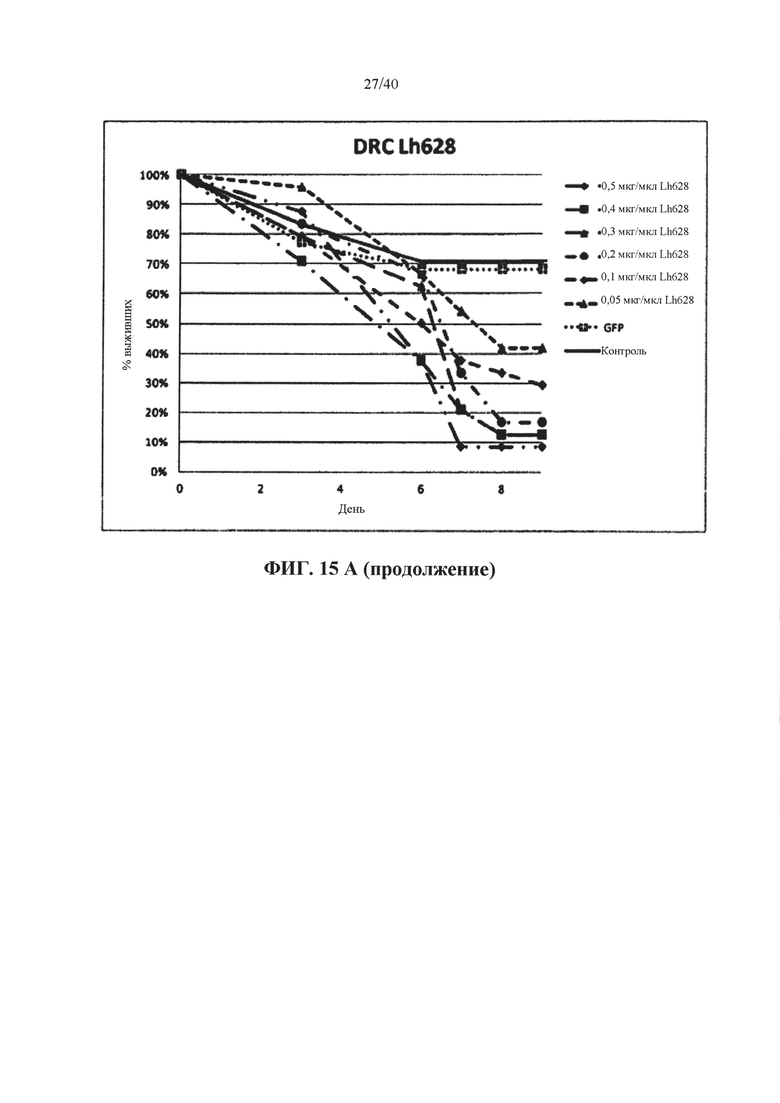

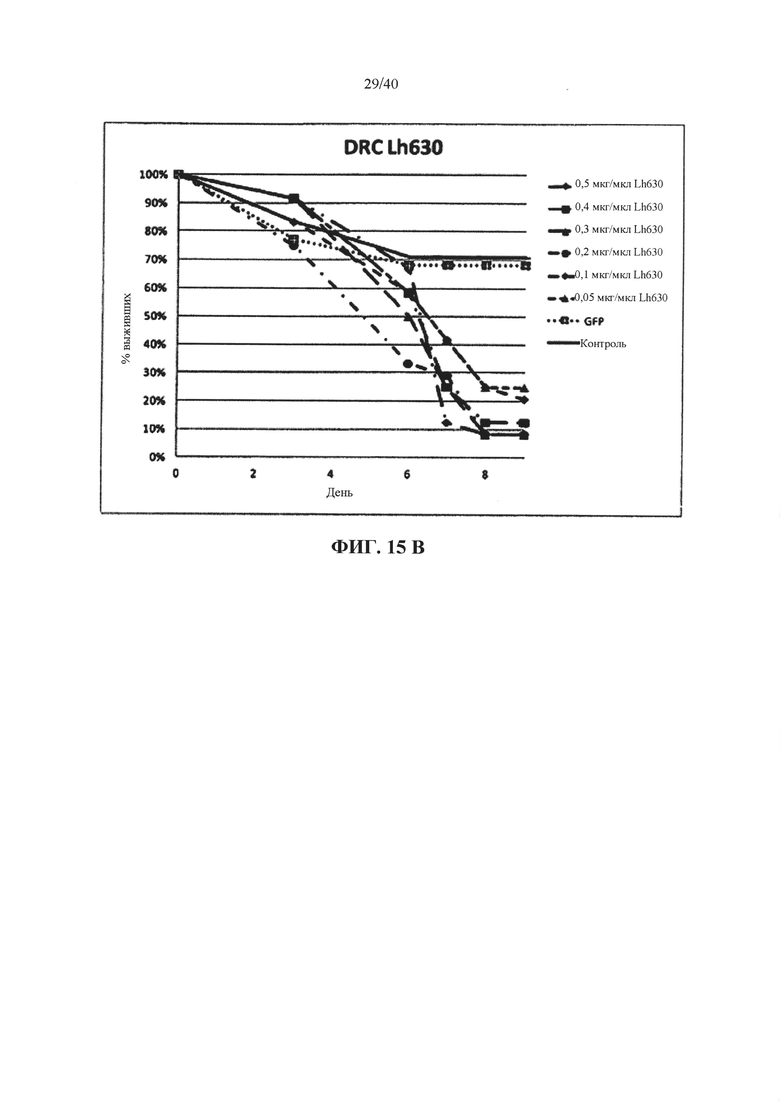
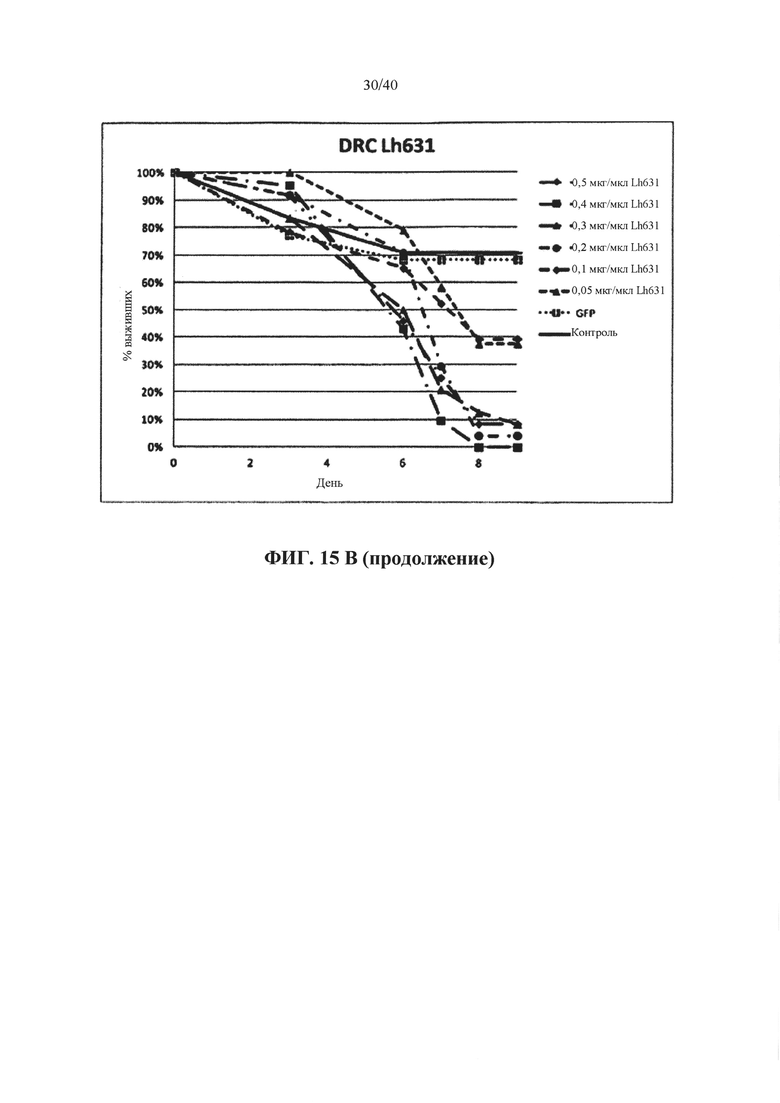
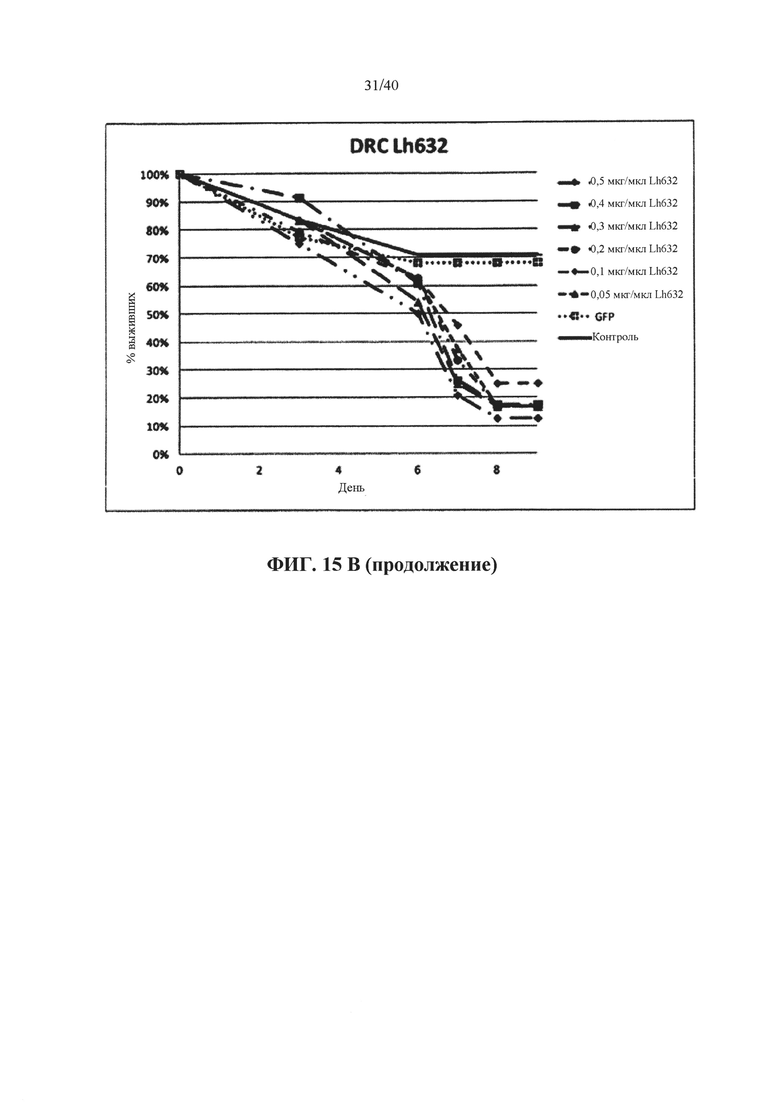



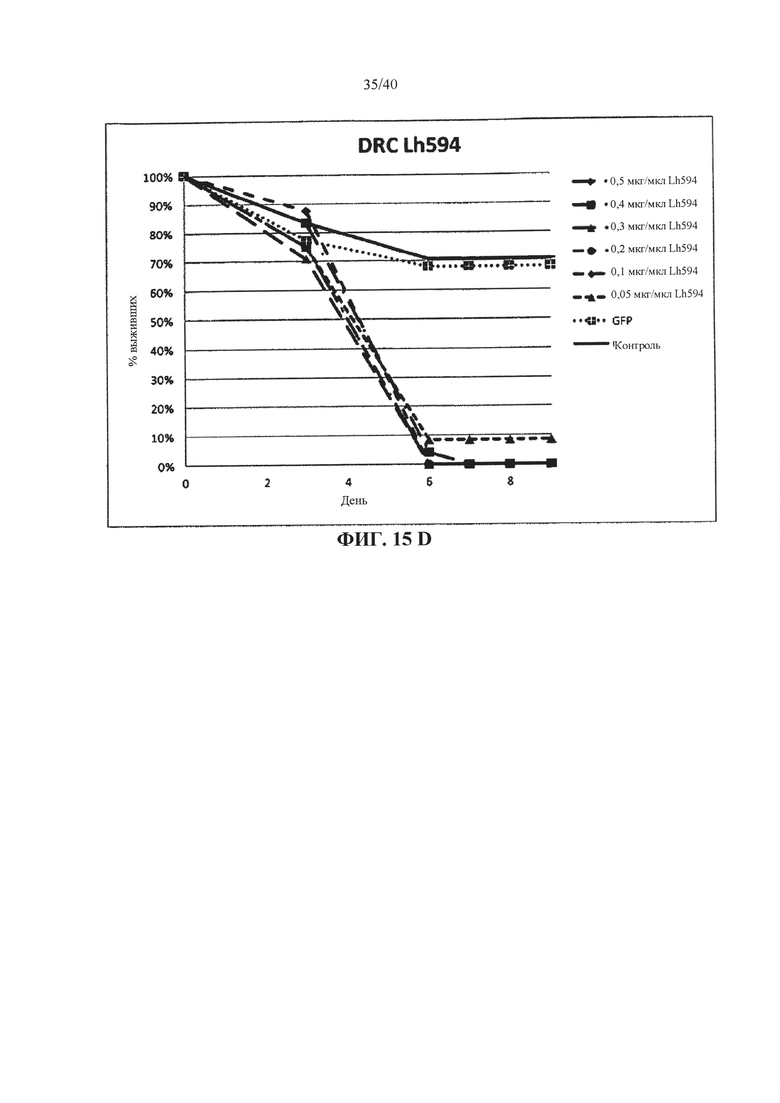
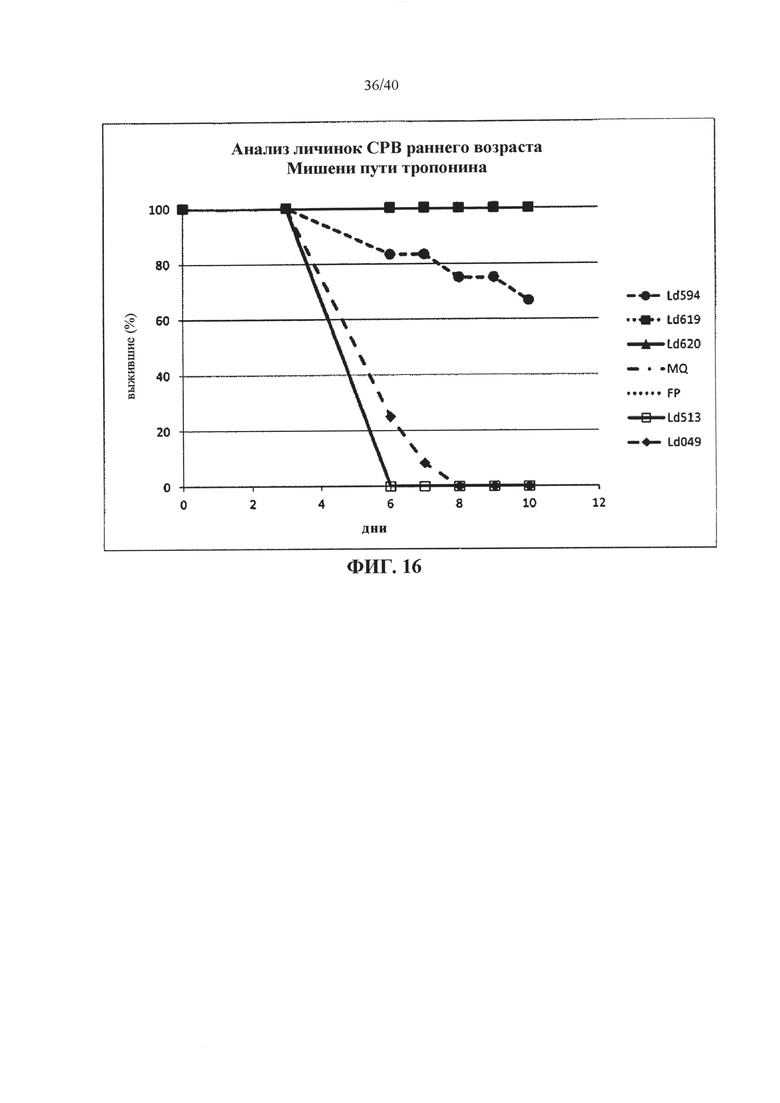



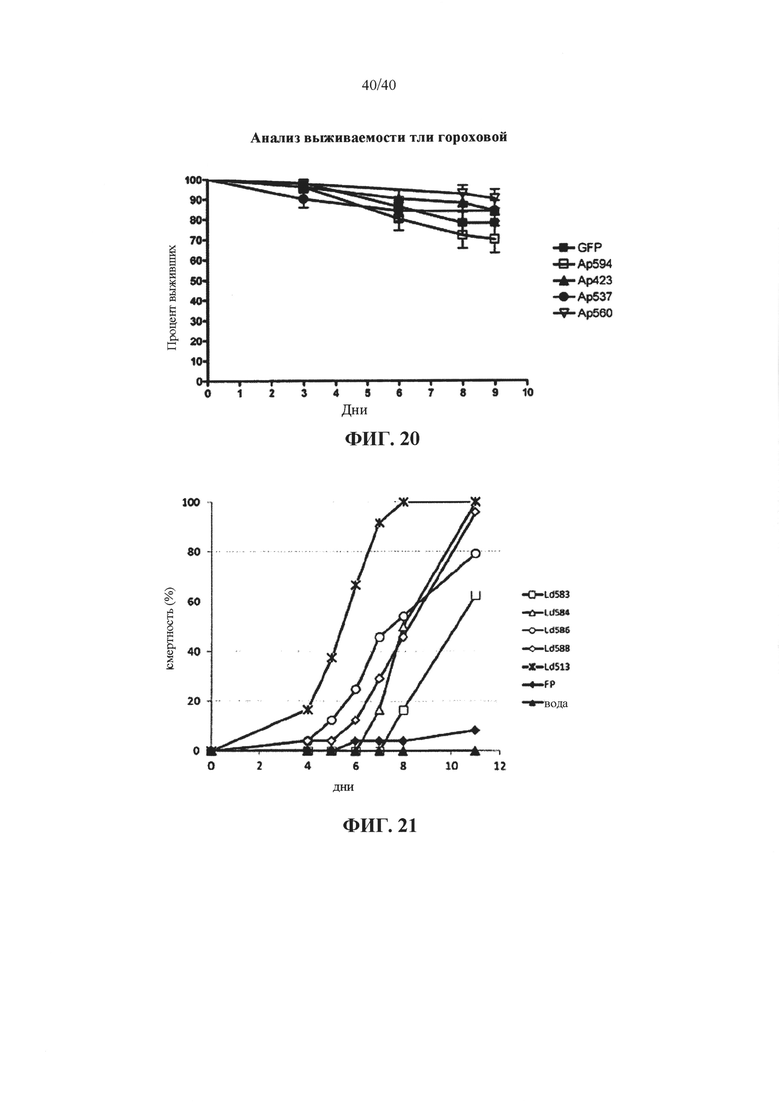
Изобретение относится к области биотехнологии. Описана группа изобретений, включающая двухцепочечную рибонуклеиновую кислоту (дцРНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя, ДНК-конструкцию для экспрессии вышеуказанной дцРНК, клетку-хозяин, композицию для предотвращения или борьбы с нападением насекомых-вредителей, применение композиции для предотвращения и/или борьбы с нападением вредителя, применение композиции в качестве пестицида для растений, способ подавления экспрессии гена-мишени у вида насекомого- вредителя с целью предотвращения и/или борьбы с нападением вредителей, применение дцРНК, ДНК-конструкции или композиции для предотвращения или борьбы с нападением насекомого-вредителя, и набор, содержащий дцРНК, ДНК-конструкцию или композицию, для предотвращения или борьбы с нападением насекомого-вредителя и один подходящий разбавитель или носитель. Изобретение расширяет арсенал средств для подавления экспрессии гена-мишени у насекомых-вредителей. 9 н. и 17 з.п. ф-лы, 21 табл., 6 пр., 21 ил.
1. Двухцепочечная рибонуклеиновая кислота (дцРНК), которая функционирует при поглощении видом насекомого-вредителя с подавлением экспрессии гена-мишени у указанного насекомого-вредителя,
при этом дцРНК содержит соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности по меньшей мере 21 последовательного нуклеотида, которая комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и
при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую SEQ ID NO 387 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 85% идентична SEQ ID NO 387 или комплементарной ей последовательности, или
(ii) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую SEQ ID NО 387 или
комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 85% идентична последовательности, представленной в SEQ ID NО 387, или
(iii) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85% идентичной аминокислотной последовательности, кодируемой SEQ ID NО 387;
где видами указанных насекомых-вредителей являются Leptinotarsa
spp., Nilaparvata spp. или Lygus spp.
2. ДцРНК по п.1, где насекомое-вредитель растения выбрано из группы, состоящей из Leptinotarsa decemlineata (колорадский жук), Nilaparvata lugens (коричневый дельфацид) или Lygus hesperus (слепняк западный матовый).
3. ДцРНК по пп. 1, 2, где ген-мишень кодирует субъединицу протеасомного белка бета-типа или ортолог белка насекомых CG17331 Dm.
4. ДцРНК по любому из пп. 1-3, где подавление экспрессии гена-мишени вызывает снижение роста, развития, размножения или выживания вредителя по сравнению с видом вредителя, подверженного воздействию интерферирующей рибонуклеиновой кислоты, воздействующей на несущественный ген, или интерферирующей рибонуклеиновой кислоты, которая не подавляет какие-либо гены у вида-вредителя.
5. ДНК-конструкция для экспрессии дцРНК по любому из пп.1-4, где ДНК-конструкция является экспрессирующей конструкцией, в которой полинуклеотидная последовательность, кодирующая дцРНК, функционально связана по меньшей мере с одной регуляторной последовательностью, способной управлять экспрессией полинуклеотидной последовательности.
6. Клетка-хозяин для экспрессии дцРНК по любому из пп.1-4, содержащая дцРНК по любому из пп.1-4 или ДНК-конструкцию по п. 5.
7. Клетка-хозяин по п.6, где клетка-хозяин является прокариотической или эукариотической клеткой.
8. Клетка-хозяин по п.7, где клетка-хозяин является бактериальной клеткой.
9. Композиция для предотвращения или борьбы с нападением насекомых-вредителей, содержащая по меньшей мере одну дц рибонуклеиновую кислоту (дцРНК) и по меньшей мере один подходящий носитель, наполнитель или разбавитель, причем дцРНК функционирует при поглощении вредителем с подавлением экспрессии гена-мишени у указанного вредителя,
при этом дцРНК содержит соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности по меньшей мере 21 последовательного нуклеотида, которая комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и
при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую SEQ ID NO 387 или комплементарную ей последовательность, или имеющих такую нуклеотидную последовательность, которая при оптимальном выравнивании и сравнении двух последовательностей по меньшей мере на 85% идентична SEQ ID NO 387 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из SEQ ID NО 387 или комплементарную ей последовательность, при этом при оптимальном выравнивании указанного фрагмента и сравнении c соответствующим фрагментом в SEQ ID NО 387 указанная нуклеотидная последовательность указанного фрагмента по меньшей мере на 85% идентична указанному соответствующему фрагменту SEQ ID NO 387 или комплементарной ей последовательности, или
(iii) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую SEQ ID NО 387 или
комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 85% идентична последовательности, представленной в SEQ ID NО 387, или
(iv) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85% идентичной аминокислотной последовательности, кодируемой SEQ ID NО 387.
10. Композиция по п.9, где дцРНК является такой, как определено в любом из пп.1-4.
11. Композиция по п. 9 или 10, где дцРНК кодируется ДНК- конструкцией по п. 5.
12. Композиция по любому из пп.9-11, где дцРНК продуцируется клеткой-хозяином по любому из пп. 6-8.
13. Композиция по п.12, где клетка-хозяин является бактериальной клеткой.
14. Композиция по любому из пп.9-13, где композиция находится в форме, подходящей для поглощения насекомым.
15. Композиция по любому из пп.9-14, где композиция находится в твердой, жидкой или гелеобразной форме.
16. Композиция по любому из пп. 9-15, где композиция составлена в виде инсектицидного спрея.
17. Композиция по п.16, где спрей представляет собой распыляемый под давлением спрей/спрей в виде аэрозоля или спрей для пульверизатора.
18. Применение композиции по любому из пп.9-17 для предотвращения и/или борьбы с нападением вредителя.
19. Применение композиции по любому из пп.9-17 в качестве пестицида для растений.
20. Способ подавления экспрессии гена-мишени у вида насекомого-вредителя с целью предотвращения и/или борьбы с нападением вредителей, включающий приведение в контакт указанного вида вредителя с эффективным количеством по меньшей мере одной дц рибонуклеиновой кислоты (дцРНК), где дцРНК функционирует при поглощении вредителем с подавлением экспрессии гена-мишени у указанного вредителя,
при этом дцРНК содержит соединенные комплементарные цепи, одна цепь из которых содержит или состоит из последовательности по меньшей мере 21 последовательного нуклеотида, которая комплементарна целевой нуклеотидной последовательности в пределах гена-мишени, и
при этом ген-мишень
(i) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую SEQ ID NО 387 или комплементарную ей последовательность, или имеющих нуклеотидную последовательность, такую, которая при оптимальном выравнивании и сравнении двух последовательностей является по меньшей мере на 85% идентичной SEQ ID NО 387 или комплементарной ей последовательности, или
(ii) выбран из группы генов, имеющих нуклеотидную последовательность, содержащую фрагмент по меньшей мере из 21 последовательного нуклеотида из SEQ ID NO 387 или комплементарной ей последовательности, и при этом при оптимальном выравнивании и сравнении указанного фрагмента с соответствующим фрагментом в SEQ ID NO 387 указанная нуклеотидная последовательность указанного фрагмента является по меньшей мере на 85% идентичной указанному соответствующему фрагменту SEQ ID NO 387 или комплементарной ей последовательности, или
(iii) у насекомого-вредителя является ортологом гена, имеющего нуклеотидную последовательность, содержащую SEQ ID NO 387 или комплементарную ей последовательность, при этом два ортологических гена сходны по последовательности до такой степени, что при оптимальном выравнивании и сравнении двух генов ортолог имеет последовательность, которая по меньшей мере на 85% идентична последовательности, представленной в SEQ ID NO 387, или
(iv) выбран из группы генов, имеющих нуклеотидную последовательность, кодирующую аминокислотную последовательность, которая при оптимальном выравнивании и сравнении двух аминокислотных последовательностей является по меньшей мере на 85% идентичной аминокислотной последовательности, кодируемой SEQ ID NО 387.
21. Способ по п.20, где дцРНК является такой, как определено в любом из пп.1-4.
22. Способ по п.20 или 21, где подавление экспрессии гена-мишени у вида насекомого-вредителя применяют с целью получения по меньшей мере 20% эффективности борьбы с вредителями или по меньшей мере 20% смертности вредителей по сравнению с контрольными насекомыми-вредителями, проконтактировавшими с дц рибонуклеиновой кислотой (дцРНК), воздействующей на несущественный ген вредителя или ген-мишень, не экспрессирующийся у указанного вредителя.
23. Способ по любому из пп.20-22, где способ применяют для предотвращения и/или борьбы с нападением вредителя на растение.
24. Способ по п.23, где растение выбирают из группы, включающей хлопчатник, картофель, рис, канолу, подсолнечник, сорго, просо, кукурузу, землянику, сою, люцерну, томат, баклажан, перец и табак.
25. Применение дц рибонуклеиновой кислоты (дцРНК) по любому из пп.1-4, ДНК-конструкции по п. 5 или композиции по любому из пп. 9-17 для предотвращения или борьбы с нападением насекомого-вредителя.
26. Набор, содержащий дц рибонуклеиновую кислоту (дцРНК) по любому из пп.1-4, ДНК-конструкцию по п. 5 или композицию по любому из пп. 9-17, для предотвращения или борьбы с нападением насекомого-вредителя и один подходящий разбавитель или носитель.
| WO 2007035650 A3, 29.03.2007 | |||
| WO 2005110068 A3, 24.11.2005 | |||
| RU 2008114846 A, 27.10.2009 | |||
| P | |||
| Chellappan, J | |||
| Xia, X | |||
| Zhou, S | |||
| Gao, X | |||
| Zhang, G | |||
| Coutino, F | |||
| Vazquez, W | |||
| Zhang, H | |||
| Jin "siRNAs from miRNA sites mediate DNA methylation of target genes" Nucleic Acids Research, 2010, Vol | |||
| Способ сужения чугунных изделий | 1922 |
|
SU38A1 |
| Прибор для промывания газов | 1922 |
|
SU20A1 |
Авторы
Даты
2020-07-07—Публикация
2018-08-20—Подача