Изобретение относится к отрасли питомниководства, в частности к методам генетической идентификации подвоев яблони.
Существует пять групп подвоев по силе роста: очень слаборослые, карликовые, полукарликовые, среднерослые, сильнорослые [Трусевич Г.В., Плодоводство. М., «Колос», 1975 - 133 с.]. Невозможно однозначно отличить очень слаборослый подвой от карликового, как и карликовый от полукарликового, который может быть также ошибочно идентифицирован как среднерослый. В первую очередь это обусловлено сортоспецифичным взаимодействием подвой/привой, а также применяемыми в питомнике агротехническими приемами.
Идентификация подвоев, использовавшихся при формировании саженцев яблони, актуальна по причине существенного влияния подвоя на характеристики растущего дерева. Под его воздействием изменяются сила роста, время вступления в плодоношение, выносливость, долговечность и урожайность растения. [Трусевич Г.В., Плодоводство. М., «Колос», 1975. - 130 с.]
Известно, что для идентификации подвоя проводится апробация посадочного материала. Следует, однако, отметить, что она проводится по комплексу фенотипических признаков сорта, при этом зачастую отсутствует возможность контроля соответствия использованного подвойного материала [Егоров Е.А., Еремин Г.В., Ульяновская Е.В. и др. Современные методологические аспекты организации селекционного процесса в садоводстве и виноградарстве. - Краснодар: СКЗНИИСиВ, 2012. - 569 с.]
Недостатком данного метода является затруднение в достоверности определения подвоя внутри групп. Но самым главным фактором является то, что данную апробацию можно провести лишь спустя 1-2 года после посадки саженцев. Только спустя этот период времени можно с определенной степенью достоверности судить о силе роста дерева и предположить, к какой группе принадлежит подвой. Необходимо еще раз отметить, что точно идентифицировать какой тип подвоя при этом использовался, практически невозможно.
Наиболее близким к заявляемому способу техническим решением является способ ДНК-паспортизации подвоев яблони серии СК (СК 1, СК 2У, СК 4, СК 5, СК 7) по трем STR-маркерам (СН03а04, CN445290, CN581493-SSR). [И.И. Супрун, Я.И. Алексеев, О.П. Малюченко, А.В. Бабаков, Я.В. Ушакова. Генотипирование подвоев яблони отечественной селекции с использованием мультиплексного STR-анализа // Селекция и сортоиспытание. - 2012 - №4- С. 20-22].
Недостатки способа: 3 маркера недостаточно для формирования уникального ДНК-паспорта. Техническим результатом при использовании предлагаемого изобретения является экспрессность и достоверность генетической идентификации подвоев яблони.
Технический результат достигается за счет использования 12-ти микросателлитных ДНК-маркеров, позволяющих получить ДНК-паспорт, который впоследствии будет использован для точной, безошибочной идентификации образцов. Формирование мультиплексных наборов ДНК-маркеров позволит повысить скорость выполнения ДНК-маркерного анализа, т.к. в один мультиплексный набор будет входить 3-4 маркера, что позволит получать результаты генотипирования по 3-4 маркерам одновременно. Точность идентификации размера фрагмента ПЦР (Полимеразная цепная реакция) амплификации по используемым ДНК-маркерам будет составлять не менее 1 пары нуклеотидов.
На начальном этапе осуществления способа проводят экстракцию ДНК методом с применением цетилтриметиламмония бромида (ЦТАБ).
ПЦР-анализ проводили по стандартным методикам [Шибата Д.К. Полимеразная цепная реакция и молекулярно-генетический анализ биоптатов / Д.К. Шибата. М.: Мир, 1999. - С. 395-425]. ПЦР-смесь включала: 1 мкл пробы ДНК, полученной с использованием указанного протокола, 0,05 мМ dNTPs, 0,3 мкМ каждого праймера, входящего в праймерную пару используемого ДНК-маркера (2,5 мкл 10xSE ПЦР-буфера (ООО «Сибэнзим), 1 единица активности Taq-ДНК полимеразы, в общем объеме реакционной смеси 25 мкл. Постановку ПЦР проводили по следующей программе: 1 мин. при 94°С для начальной денатурации; следующие 35 циклов: денатурация 30 сек. при 94°С; 30 сек. отжиг праймеров при температуре 58°С; 30 сек. синтез при 72°С. Финальный цикл синтеза при 72°С - 5 мин.
Для проведения ПЦР-реакции использовали набор из 12-ти высокополиморфных SSR-маркеров для генетической паспортизации подвоев, распределенных в четыре мультиплексных набора. Каждый из мультиплексных наборов включает 3 маркера (таблица 1).
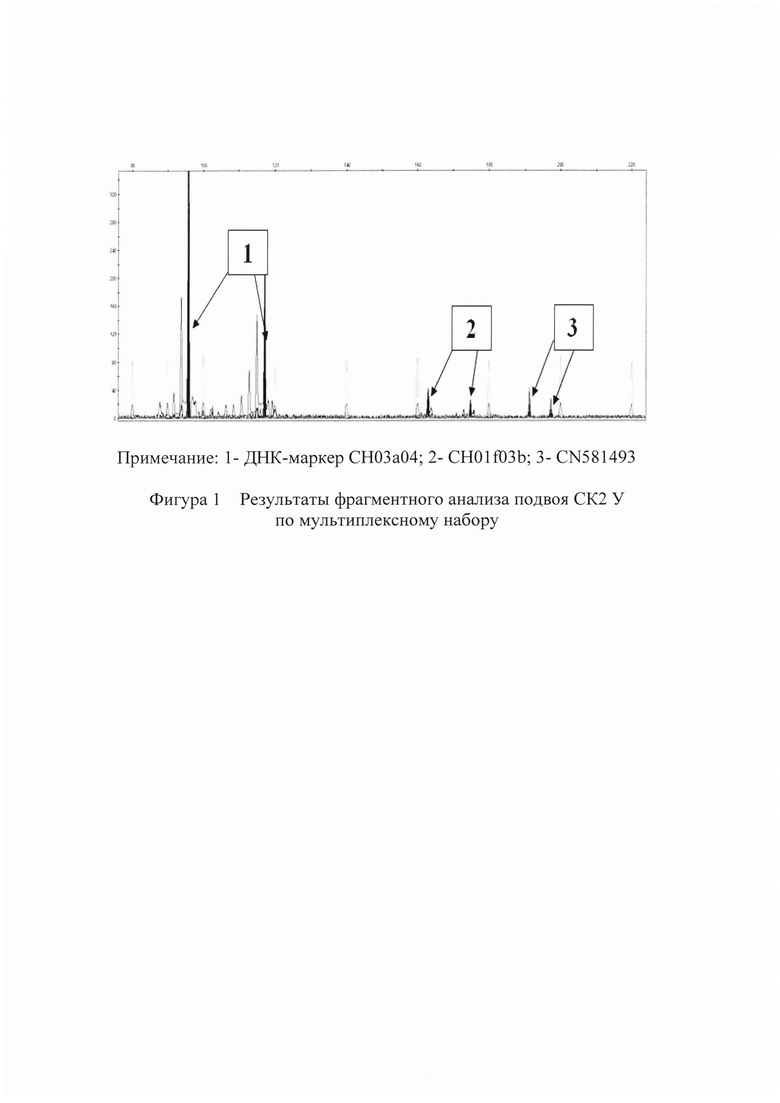
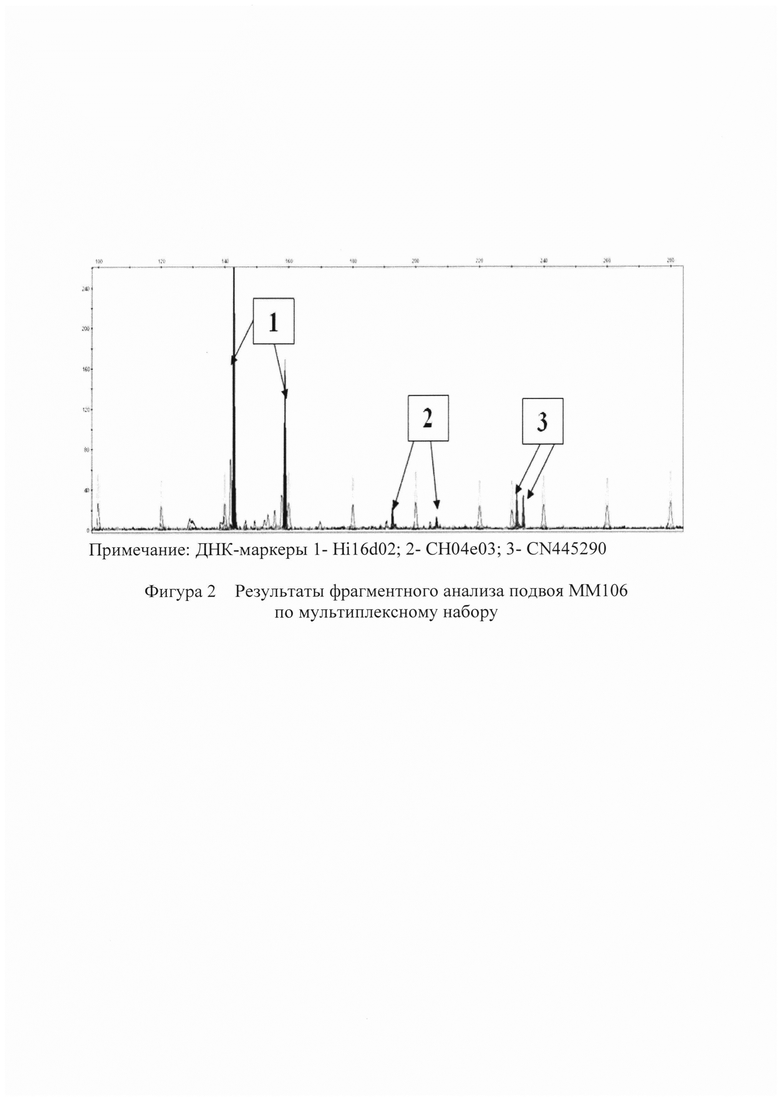
При распределении ДНК-маркеров по мультиплексным наборам учитывался такой параметр ДНК-маркеров как диапазон размеров продуктов ПЦР, синтезируемых с их использованием. Это позволяет при проведении электрофореза продуктов ПЦР на генетическом анализаторе ABIprism3130 получать на электрофореграммах пики (фигура 1, 2), не перекрывающиеся и не накладывающиеся друг на друга, что значительно повышает эффективность и достоверность анализа.
Анализ размеров амплифицированных фрагментов проводили на автоматическом генетическом анализаторе ABI prism 3130. Обработку данных осуществляли в программе Gene Mapper 4.1.
Пример 1. Формирование ДНК-паспорта полукарликового подвоя яблони СК 2У
По заявляемому способу использовали 12 указанных в таблице 1 микросателлитных ДНК-маркеров, объединенных в мультиплексные наборы по 3 ДНК-маркера в каждом.
Пример 2. Формирование ДНК-паспорта полукарликового подвоя яблони ММ106
Аналогично примеру 1, кроме того, что ДНК-паспорт формировали для подвоя ММ106. Результаты, полученные при ДНК-паспортизации подвоев СК2У и ММ106, приведены в таблице 2.
*Для каждого подвоя указан размер амплифицированных фрагментов по микросателлитным ДНК-маркерам в парах нуклеотидов. При выявлении двух фрагментов их размер указывается друг за другом, с разделением двоеточием.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ ПРЕДВАРИТЕЛЬНОГО ОТБОРА НОВЫХ ГЕНОТИПОВ КЛОНОВЫХ ПОДВОЕВ ЯБЛОНИ ПО КОЛИЧЕСТВЕННОМУ ОПРЕДЕЛЕНИЮ В ПИТОМНИКЕ ПОКАЗАТЕЛЯ СИЛЫ РОСТА ПРИВОЙНО-ПОДВОЙНЫХ КОМБИНАЦИЙ, ПОЛУЧЕННЫХ НА ИХ ОСНОВЕ | 2022 |
|
RU2789754C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ СОРТОВ И ЛИНИЙ СОИ | 2024 |
|
RU2839697C1 |
| СПОСОБ ДИАГНОСТИКИ СИЛЫ РОСТА ПОДВОЕВ ЯБЛОНИ | 2000 |
|
RU2192118C2 |
| СПОСОБ ВЫРАЩИВАНИЯ ИНТЕНСИВНОГО ЯБЛОНЕВОГО САДА | 2014 |
|
RU2566441C1 |
| Способ диагностики силы роста подвоев плодовых культур | 1988 |
|
SU1576040A1 |
| СПОСОБ ДНК-ИДЕНТИФИКАЦИИ И ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА ПАСТБИЩНОГО И ОДНОЛЕТНЕГО НА ОСНОВЕ СИСТЕМ SSR- и SCoT-МАРКИРОВАНИЯ | 2023 |
|
RU2826148C1 |
| СПОСОБ МОЛЕКУЛЯРНОГО МАРКИРОВАНИЯ, ОСНОВАННЫЙ НА МИКРОСАТЕЛЛИТНЫХ ЛОКУСАХ, ПРЕДНАЗНАЧЕННЫЙ ДЛЯ ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СЕЛЕКЦИОННЫХ ДОСТИЖЕНИЙ РАСТЕНИЙ РОДА RUBUS | 2018 |
|
RU2732922C2 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| Способ определения допустимого уплотнения деревьев в ряду при создании скороплодных насаждений яблони | 2021 |
|
RU2765239C1 |
| СПОСОБ ВЫРАЩИВАНИЯ САЖЕНЦЕВ ПЛОДОВЫХ КУЛЬТУР С ПРОМЕЖУТОЧНОЙ ВСТАВКОЙ | 2012 |
|
RU2506739C2 |
Изобретение относится к области биотехнологии. Представлен способ генетической идентификации подвоев яблони на основе анализа микросателлитных маркеров генома. Способ включает экстракцию ДНК, ПЦР-анализ, фрагментный анализ на генетическом анализаторе. Для формирования универсального ДНК-паспорта используются 12 микросателлитных ДНК-маркеров, распределенных в четыре мультиплексных набора, а именно: СН03а04, CH01f03b и CN581493 в первом наборе, Hi16d02, СН04е03 и CN445290 во втором наборе, СН04С07, GD 147 и СН04а12 в третьем наборе, NzmsEB146613, СН04е05 и СН02С09 в четвертом наборе. Изобретение позволяет повысить скорость выполнения ДНК-маркерного анализа и снизить его себестоимость, получить ДНК-паспорт, который может быть использован для точной, безошибочной идентификации образцов. Точность идентификации размера фрагмента ПЦР-амплификации по используемым ДНК-маркерам будет составлять не менее 1 пары нуклеотидов. 2 ил., 2 табл., 2 пр.
Способ генетической идентификации подвоев яблони, включающий экстракцию ДНК, ПЦР-анализ, фрагментный анализ на генетическом анализаторе, отличающийся тем, что для формирования универсального ДНК-паспорта использовали 12 микросателлитных ДНК-маркеров, распределенных в четыре мультиплексных набора, включающих по 3 маркера: СН03а04, CH01f03b и CN581493 в первом наборе, Hi16d02, СН04е03 и CN445290 во втором наборе, СН04С07, GD 147 и СН04а12 в третьем наборе, NzmsEB146613, СН04е05 и СН02С09 в четвертом наборе.
| СУПРУН И.И | |||
| и др., Генотипирование подвоев яблони отечественной селекции с использованием мультиплексного STR-анализа, САДОВОДСТВО И ВИНОГРАДАРСТВО, 2012, N4, с.20-22 | |||
| СУПРУН И.И., ТОКМАКОВ С.В | |||
| и др., Генотипирование сортов яблони российской селекции с использованием микросателлитных маркеров, Известия ТСХА, 2011, выпуск 6, с.162-166 |
Авторы
Даты
2020-07-30—Публикация
2019-03-21—Подача