Область техники, к которой относится изобретение
Изобретение относится к области биотехнологии и биохимии, в частности к способу молекулярно-генетической идентификации сортов, биотипов и гибридов сои на основе микросателлитных (SSR) маркеров.
Предшествующий уровень техники
Соя (Glicine max (L) Merr.) является одной из ключевых белково-масличных культур в мире. Ежегодно площади посевов сои увеличиваются, создаются новые сорта. Постоянный рост значимости сои в мировой экономике обусловлен комплексом ценных свойств культуры и ее использованием в различных отраслях промышленности. Сохранение генетического разнообразия и использование сортов с определенными характеристиками в качестве родителей для скрещивания имеет важное значение для создания новых сортов, адаптированных к изменяющимся климатическим условиям и устойчивых к болезням и вредителям. Изучение генетического разнообразия и паспортизация ценных сортов и форм важных сельскохозяйственных культур являются обязательными условиями успешного сохранения и использования различных сортов сельскохозяйственных видов растений и проводятся во многих научных центрах мира. Идентификация сортов помогает обеспечить подлинность семенного материала и его соответствие заявленным характеристикам сорта, и является широко распространенной задачей, которой занимаются во всем мире (Amiteye, 2021).
Наиболее удобными для идентификации генотипов в настоящее время являются молекулярно-генетические маркеры. В связи с этим большое значение имеет идентификация и паспортизация генотипов сои с использованием молекулярных маркеров.
Ранее, в целях генотипирования генетических ресурсов растений, широко использовались неспецифичные типы ДНК-маркеров, такие как RAPD (Xu and Gai, 2003). Однако, информативность этих сравнительно недорогих типов ДНК-маркеров является достаточно низкой, а использование их для идентификации сортов является неэффективным в связи с их неспецифичностью и непостоянной воспроизводимостью результатов в различных лабораториях. Кроме того, для подобного типа маркеров в большинстве случаев отсутствует информация по хромосомной локализации, они не являются кодоминантными типами маркеров, что затрудняет их использование в генетических исследованиях. Одним из наиболее используемых и эффективных типов маркеров являются микросателлитные ДНК-маркеры, или SSR (Simple Sequence Repeats - простые повторяющиеся последовательности). Микросателлитные локусы представляют собой участки дезоксирибонуклеиновой кислоты (ДНК), содержащие простые короткие повторяющиеся моно-, ди-, три-, тетра- или пентануклеотидные мотивы (тандемы), локализованные в геномах большинства видов эукариот.SSR-маркеры - кодоминантные маркеры, обладающие рядом таких преимуществ, как высокий уровень полиморфизма, воспроизводимости, случайное распределение по геному, относительно невысокая стоимость. Благодаря своим уникальным качествам микросателлитные маркеры стали востребованными в молекулярно-генетическом картировании и наилучшим образом подходят для паспортизации и дискриминации индивидуальных генотипов, так как микросателлитные локусы содержат большое число аллелей, кроме того возможность использования наборов локусов позволяет получать уникальные генетические паспорта с крайне низкой вероятностью случайного повторения (Brown et al., 1996; Varshney et al., 2005).
Известен способ молекулярного маркирования, основанный на микросателлитных локусах, предназначенный для генетической паспортизации селекционных достижений растений рода Rubus (RU 2732922 С2 от 24.09.2020), позволяющий осуществлять надежную и достоверную паспортизацию различных сортов малины с помощью 13 SSR-праймеров.
У сои также удалось выявить высокий уровень полиморфизма по микросателлитным локусам (Abe et al., 2003; Diwan and Cregan, 1997; Guo Juan et al., 2012; Tantasawat et al., 2011). В работе С.А. Рамазановой с соавторов (2020) было установлено, что система маркеров, идентифицирующая 13 SSR-локусов (Sat002, Satt005, Satt009, Soyprl, Sat36, Sat43, Soyhspl76, Soysc514, Satt141, Satt681, Satt181, Satt161 и Sat_263), обладает высоким дискриминирующим потенциалом для того, чтобы использовать ее для идентификации и паспортизации сортов сои (Рамазанова and Коломыцева, 2020).
В другом исследовании было проведено изучение генетического разнообразия 15 сортов и 22 перспективных линий сои казахстанской селекции с использованием 50 полиморфных SSR-маркеров, локализованных во всех 20 хромосомах генома сои, что позволило идентифицировать 167 аллелей, что в среднем составило 3,4 аллеля на маркер. Также было показано, что для паспортизации сортового генофонда, рекомендуются использовать SSR-маркеры с индексом полиморфного информационного содержания изучаемых маркеров -PIC (polymorphic information content) выше 0,5, так из 50 проанализированных маркеров наиболее информативными и надежными оказались 20 SSR-маркеров, на основе которых был разработан генетический паспорт для каждого сорта сои Казахстана (Абугалиева et al., 2013).
Другой способ описывает идентификацию сортов сои на основе SSR маркеров (RU 2388828 C1 от 10.05.2010), по следующему набору микросателлитных локусов - SATT 1, SATT 2, SATT 5, SATT 9, SOYPR 1, SOYGY 2, SAT 1, SAT 36, SOYHSP 176. Данный способ основан на проведении ПЦР-амплификации с последующей визуализацией с помощью электрофореза в агарозном геле. Заявляемый способ позволяет идентифицировать сорта сои, однако, зачастую приведенного набора маркеров бывает недостаточно для эффективной дискриминации большого числа близкородственных сортов и индивидуальных генотипов, кроме того, способ трудоемкий и требует большого количества времени, так как предполагает поочередное выполнение ПЦР с каждой парой праймеров при определенных температурах отжига с последующим разделением методом гель-электрофореза.
Методики, используемые в приведенных выше исследованиях, основывались на проведении ПЦР-амплификации с маркерами для каждого локуса отдельно, с последующей визуализацией с помощью гель-электрофореза, что весьма долго и трудоемко при анализе больших коллекций образцов.
Примеры использования мультиплексной ПЦР и фрагментного анализа можно встретить в основном в исследованиях, связанных с человеком (Ruitberg et al., 2001; Xiong et al., 2022) или животными (Dang et al., 2020; Shang et al., 2021, 2024). Ближайший пример использования мультиплексной ПЦР с последующей визуализацией на фрагментном анализе представлен для идентификации сортов растения вида Cannabis sativa (Xia et al., 2022), где с помощью мультиплекса из 19 маркеров было протестировано 85 образцов. В настоящее время для сои (Glycine max) подобных мультиплексных систем маркеров не создано.
Задача, решаемая заявляемым изобретением, состоит в повышении уровня идентификации сортов сои за счет увеличения числа используемых микросателлитных локусов, а также увеличение производительности анализа путем применения мультиплексных реакций (амплификация нескольких локусов с меченными флуоресцентными метками праймерами в одной пробирке) и автоматизации скрининга, в том числе за счет использования фрагментного анализа для разделения и определения размеров полученных фрагментов. Предложенный способ позволит ускорить и повысить эффективность идентификации сортов сои с помощью мультиплексной ПЦР, а также за счет использования фрагментного анализа увеличить точность получаемых результатов и упростить анализ для пользователей.
Целью предлагаемого изобретения является обеспечение достоверной паспортизации сортов и биотипов сои на основе применения высокополиморфных микросателлитных локусов, а также использование мультиплексной ПЦР и фрагментного анализа для ускорения и автоматизации процесса получения данных.
Сопоставительный анализ заявляемого технического решения со способом, зарегистрированным ранее, позволяет сделать вывод, что заявляемый способ идентификации сортов сои на основе микросателлитных (SSR) маркеров отличается от известного условиями осуществления идентификации аллельных вариантов SSR-локусов, а именно:
- используемыми веществами - праймерами,
- разработкой мультиплексной системы маркеров (амплификация нескольких локусов с меченными флуоресцентными метками праймерами в одной пробирке),
- визуализацией продуктов амплификации методом капиллярного электрофореза с помощью генетического анализатора.
Все вышеперечисленные пункты подтверждают новизну разработанного способа. Поставленная цель по созданию способа идентификации сортов сои достигается за счет использования набора эффективных и стабильных молекулярных маркеров, позволяющих выявить высокий уровень полиморфизма ДНК и получить воспроизводимые результаты.
Сущность изобретения
Задачей изобретения является разработка способа идентификации сортов сои, пригодного для паспортизации, за счет увеличения числа используемых микросателлитных локусов, а также увеличение производительности анализа и снижение его трудоемкости.
Для выполнения поставленной задачи нами были реализованы следующие этапы разработки:
- дизайн набора меченных флуоресцентными метками олигонуклеотидных праймеров (олигонуклеотидов) на SSR-локусы, наиболее часто используемые для идентификации и паспортизации сортов сои и дополнительные высокополиморфные SSR-локусы,
- оптимизация условий амплификации и реагентов для использования в мультиплексной ПЦР,
- последующая единовременная детекциея результатов 1ТЦР с помощью капиллярного электрофореза.
Нуклеотидные последовательности разработанных праймеров представлены в Таблице 1 и в Перечне последовательностей.
Способ идентификации сортов осуществляют следующим образом и включает следующие этапы:
выделение геномной ДНК, постановка двух мультиплексных ПЦР - амплификации с двумя смесями праймеров отдельно, где смесь праймеров №1 (далее СП1) включает в себя следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16 для идентификации локусов Sct001, Sat090, Satt141, Satt681, Sat084, Satt264, Satt440, Sat_177, и смесь праймеров №2 (далее СП2) включает в себя следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30 для идентификации локусов Satt245, Sattl81, Soysc514, Satt009, Soyhsp176, Soyprl, Satt431,
совместная визуализация (в одной лунке) методом капиллярного электрофореза продуктов амплификации, полученных при проведении двух раздельных мультиплексных ПЦР, с помощью генетического анализатора Нанофор 05 в присутствии маркера молекулярного веса СД-600,
программный расчет размера фрагмента относительно маркера молекулярного веса (размерного стандарта),
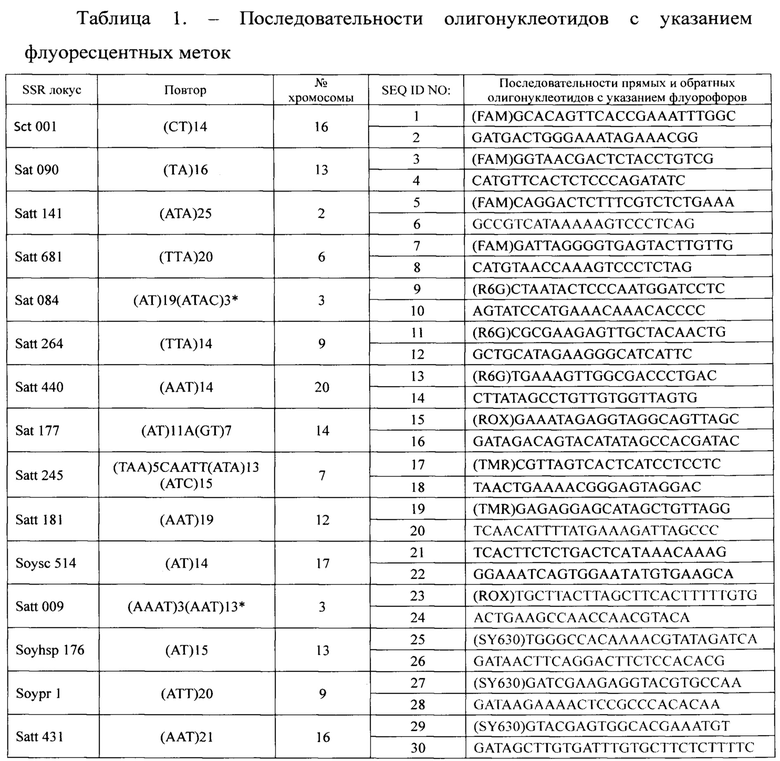
калибровка относительно положительного контрольного образца, представляющего собой ДНК сорта Бинго, использование которого необходимо при каждом анализе, для проверки прохождения ПЦР-амплификации и в качестве аналога аллельной лестницы в фрагментном анализе (Фиг. 1).
Краткое описание чертежей
Фиг. 1. Электрофореграмма продуктов ПЦР по 15 SSR-локусам на примере сорта сои Бинго со стандартом длины СД-600.
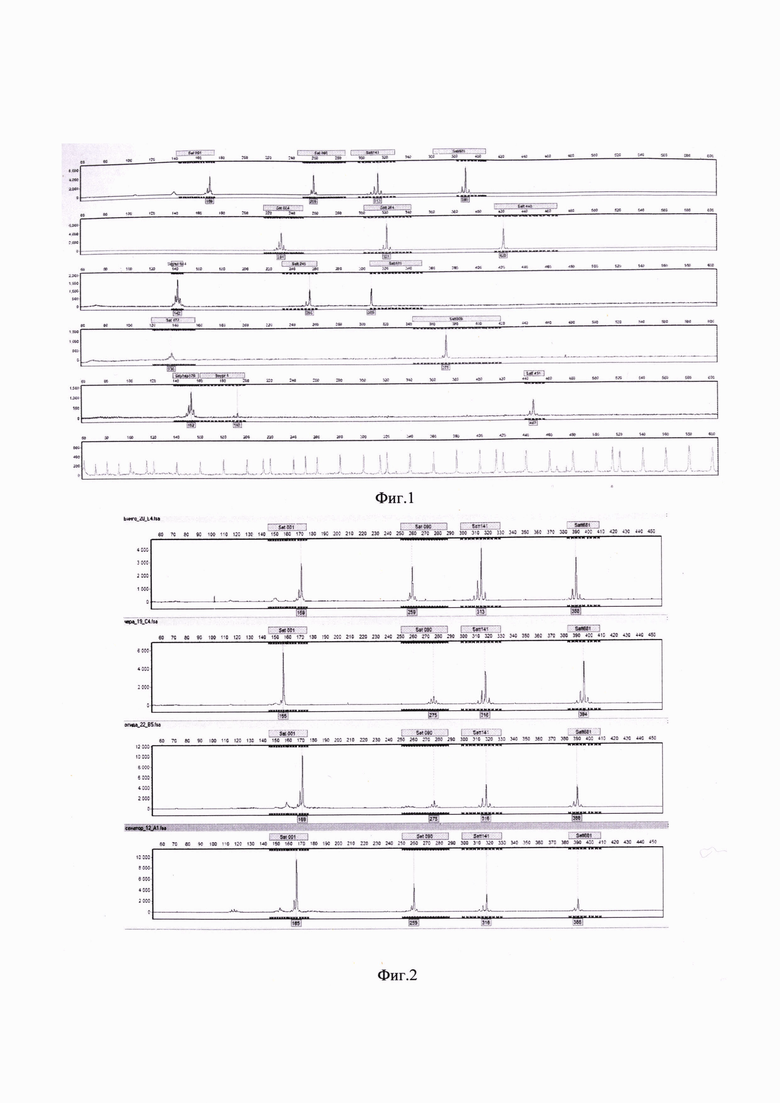
Фиг. 2. Электрофореграмма продуктов ПЦР на примере сортов сои Бинго, Чера, Эгида и Сенатор в канале детекции FAM.
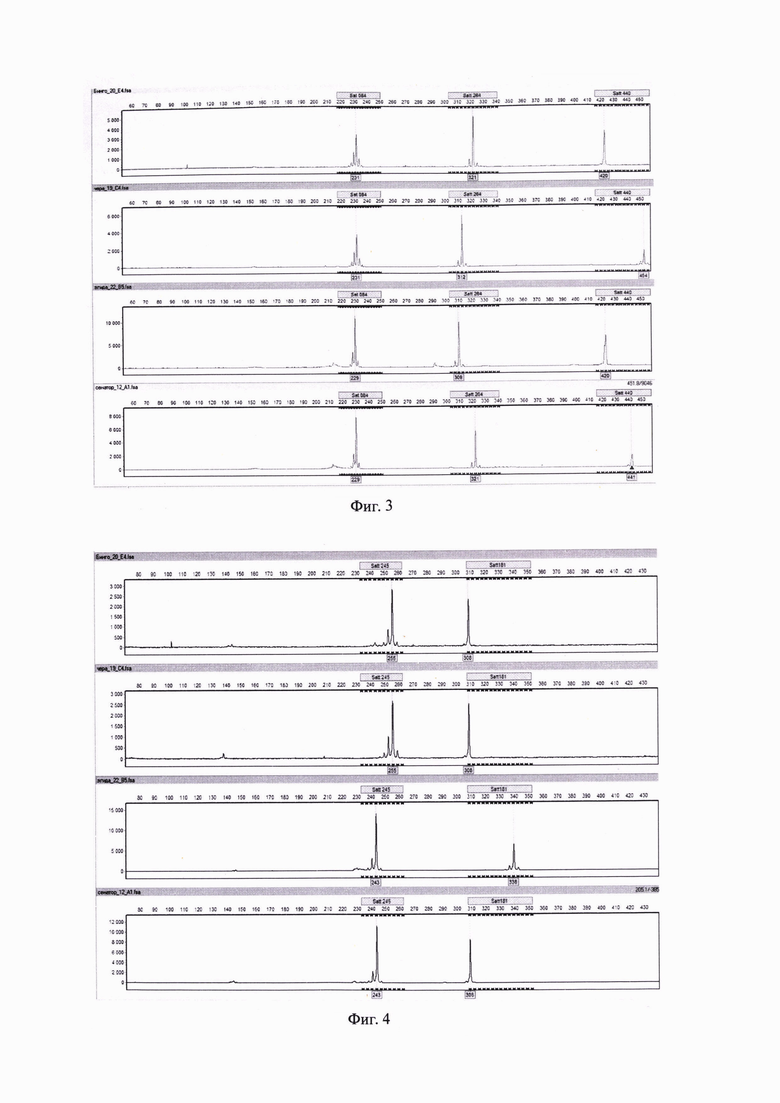
Фиг. 3. Электрофореграмма продуктов ПЦР на примере сортов сои Бинго, Чера, Эгида и Сенатор в канале детекции R6G.
Фиг. 4. Электрофореграмма продуктов ПЦР на примере сортов сои Бинго, Чера, Эгида и Сенатор в канале детекции TAMRA.
Фиг. 5. Электрофореграмма продуктов ПЦР на примере сортов сои Бинго, Чера, Эгида и Сенатор в канале детекции ROX.
Фиг. 6. Электрофореграмма продуктов ПЦР на примере сортов сои Бинго, Чера, Эгида и Сенатор в канале детекции Sy630.
Осуществление изобретения (Пример)
Для идентификации сортов сои были использованы растения 40 сортов сои. Пробу отбирали стандартным способом, каждую пробу, состоящую из 30 семян, измельчали в муку с использованием электрического гомогенизатора (Qiagen TissueLyser II, США). Выделение ДНК проводили из навески муки из 30-ти зерен - 100 мг с помощью станции для автоматического выделения нуклеиновых кислот Auto-Pure 96 (Allsheng, Китай) набором «МагноПрайм ГМО» (Интерлабсервис, Россия) согласно протоколу производителя.
Следующий этап заключался в постановке двух мультиплексных ПЦР-амплификаций с разработанными праймерами в составе СП1 и СП2, где смесь праймеров СП1 включает в себя следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16 для идентификации локусов Sct001, Sat090, Satt141, Satt681, Sat084, Satt264, Satt440, Sat_177 и смесь праймеров СП2 включает в себя следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30 для идентификации локусов Satt245, Satt181, Soysc514, Satt009, Soyhsp176, Soyprl, Satt431.
Мультиплексную ПЦР проводили на приборе "ДНК-амплификатор Т100 Thermal Cycler" (Bio-Rad, США) в 25 мкл стандартной ПЦР-смеси (Mg2+2,5 мМ, dNTP 0,25 мМ) с добавлением дополнительных компонентов, таких как 2 мкл BSA (бычий сывороточный альбумин) 40 мг/мкл и 2,5 мМ Сорбитола, в присутствии 1 мкл ДНК с концентрацией 5-10 нг/мкл.
Циклограмма: первичная денатурация при 95°С в течение 5 мин; затем 32 цикла: денатурация при 95°С - 20 сек, отжиг при 61°С - в течение 30 сек, элонгация при 68°С в течение 40 сек; финальная элонгация 68°С - 10 мин.
Продукты амплификации визуализировали методом капиллярного электрофореза с помощью генетического анализатора Нанофор 05 (Синтол, Россия) в присутствии маркера молекулярного веса СД-600. Капиллярный электрофорез на генетическом анализаторе проводился в соответствии с руководством пользователя, предоставляемым его производителем (Синтол, Россия). Параметры электрофореза зависели от длины капилляров и типа полимера, рекомендуемые параметры инжекции 3000 В, 5 секунд. Перед постановкой капиллярного электрофореза была проведена спектральная калибровка прибора, в соответствии с используемыми в системе красителями, калибратором СК-6 (Синтол, Россия).
Далее проводилась калибровка относительно положительного контрольного образца, представляющего собой ДНК сорта Бинго, использование которого необходимо при каждом анализе, для проверки прохождения ПЦР-амплификации и в качестве аналога аллельной лестницы в фрагментном анализе (Фиг. 1). Данный этап необходим, так как расчет длинны фрагмента от прибора к прибору может незначительно меняться, в связи с используемым полимером или линейкой капилляров.
По каждому локусу определялся размер фрагмента, соответствующий тому или иному аллелю. Локусы были распределены по диапазонам размера и каналам флюоресценции следующим образом:
детекция локусов Sct001, Sat090, Satt141, Satt681 проходила по каналу FAM в диапазонах 143 - 173, 251 - 287, 298 - 328, 382 - 406, соответственно (Фиг. 2),
детекция локусов Sat084, Satt264, Satt440 проходила по каналу R6G в диапазонах 217 - 249, 303 - 343, 414 - 466 соответственно (Фиг. 3),
детекция локусов Soysc514, Satt245, Satt181 проходила по каналу TAMRA в диапазонах 138 - 146, 234 - 261, 308 - 350 соответственно (Фиг. 4),
детекция локусов Sat177, Satt009 проходила по каналу ROX в диапазонах 122 - 156, 344 - 418 соответственно (Фиг. 5),
детекция локусов Soyhsp176, Soyprl, Satt431 проходила по каналу Sy630 в диапазонах 146 - 158, 186 - 198, 440 - 453, соответственно (Фиг. 6).
Анализ и сравнение профилей анализируемых сортов осуществляли с помощью программного обеспечения GeneMarker V3.0.1 (SoftGenetics, США). Данная программа проводит расчет длин фрагментов - аллелей относительно размерного стандарта, поэтому для корректной работы, в программе был создан файл, содержащий информацию о используемом размерном стандарте и длинах входящих в него фрагментов. Также для автоматического определения программой локусов и аллелей была создана панель, включающая детектируемые локусы и прописаны бины - диапазоны, соответствующие аллелям. Для этого была проведена постановка положительного контрольного образца, на размеры которого ориентировались как на стандарт (Фиг. 1). Анализ проводился при стандартных настройках программы с использованием размерного стандарта СД-600 и созданной нами панели.
Список литературы
1. Abe, J. et al. Soybean germplasm pools in Asia revealed by nuclear SSRs / J. Abe, D. Xu, Y. Suzuki, A. Kanazawa, Y. Shimamoto // 2003 - P. 445-453.
2. Amiteye, S. Basic concepts and methodologies of DNA marker systems in plant molecular breeding / S. Amiteye // 2021 - №10.
3. Brown, S.M. et al. Methods of genome analysis in plants / S.M. Brown, A. Szewc-McFadden, S. Kresovich // 1996 - P. 147-158.
4. Dang, W. et al. A novel 13-plex STR typing system for individual identification and parentage testing of donkeys (Equus asinus) / W. Dang, S. Shang, X. Zhang, Y Yu, D. Irwin, Z. Wang, S. Zhang // 2020 - №2 - P. 290-297.
5. Diwan, N. et al. Automated sizing of fluorescent-labeled simple sequence repeat (SSR) markers to assay genetic variation in soybean / N. Diwan, P. Cregan // 1997 -P. 723-733.
6. Guo Juan, G.J. et al. Population structure of the wild soybean (Glycine soja) in China: implications from microsatellite analyses / G.J. Guo Juan, L.Y Liu YiFei, W.Y Wang YunSheng, C.J. Chen JianJun, L.Y. Li YingHui, H.H. Huang Hong Wen, Q.L. Qiu LiJuan, WY Wang Ying // 2012.
7. Ruitberg, С.M. et al. STRBase: a short tandem repeat DNA database for the human identity testing community / CM. Ruitberg, D.J. Reeder, J.M. Butler // 2001 - №1 - P. 320-322.
8. Shang, S. et al. Development of a 19-plex short tandem repeat typing system for individual identification and parentage testing of horses (Equus caballus) / S. Shang, R. Jiang, R. Luo, S. Jia, D. Irwin, Z. Wang, S. Zhang // 2021 - №5 - P. 754-758.
9. Shang, S. et al. Development of a 17-plex STR typing system for the identification of individuals and parentage testing in cattle / S. Shang, Y. Wang, X. Yu, D. Zhang, R. Luo, R. Jiang, G. Zhao, X. Du, J. Zhang, D.M. Irwin, others // 2024 - №1 - P. 1-10.
10. Tantasawat, P. et al. SSR analysis of soybean ('glycine max'(L.) merr.) genetic relationship and variety identification in thailand / P. Tantasawat, J. Trongchuen, T. Prajongjai, S. Jenweerawat, W. Chaowiset // 2011 - №3 - P. 283-290.
11. Varshney, R.K. et al. Genie microsatellite markers in plants: features and applications / R.K. Varshney, A. Graner, M.E. Sorrells // 2005 - №1 - P. 48-55.
12. Xia, R. et al. Development and validation of a novel and fast detection method for cannabis sativa: a 19-plex short tandem repeat typing system / R. Xia, R. Tao, Y. Qu, X. Zhang, H. Yu, C. Yuan, S. Zhang, C. Li // 2022 - P. 837945.
13. Xiong, С. et al. Development and validation of a multiplex typing system with 32 Y-STRs for forensic application / C. Xiong, C. Yang, W. Wu, Y. Zeng, T. Lin, L. Chen, H. Liu, C. Liu, W. Du, M. Wang, others // 2022 - P. 111409.
14. Xu, D. et al. Genetic diversity of wild and cultivated soybeans growing in China revealed by RAPD analysis / D. Xu, J. Gai // 2003 - №6 - P. 503-506.
15. Абугалиева, С. et al. ДНК-фингерпринтинг сортов сои Казахстана с использованием SSR маркеров / С.Абугалиева, Л. Волкова, А. Нурланова, А. Жанпеисова, Е. Туруспеков // 2013 - №3 - Р. 26-34.
16. Рамазанова, С.et al. Оптимизация технологии генотипирования сои на основе анализа полиморфизма SSR-локусов ДНК / С.Рамазанова, А. Коломыцева // 2020 - №1 (181) - Р. 42-48.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Приложение МАТ
A29L-7 и ИФА тест-система для выявления ВОО.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2024-10-08">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2024-10-08</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>123</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>12345</ApplicationNumberText>
<FilingDate>2024-10-01</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Плазмидная генетическая
конструкция pET28b-MozAg, обеспечивающая экспрессию и секрецию
мозаичного белка MozAg в прокариотической системе, мозаичный белок
MozAg, используемый для иммунизации мыши с целью получения на основе
мышиных спленоцитов гибридомы, продуцирующей моноклональное антитело
A29L-7, моноклональное антитело A29L-7 и тест-система для выявления
антигена вируса оспы обезьян с его использованием</InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>6734</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6734</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgctatcatgccataccgcgaaaggttttgcgccattcgatggtgtccg
ggatctcgacgctctcccttatgcgactcctgcattaggaagcagcccagtagtaggttgaggccgttga
gcaccgccgccgcaaggaatggtgcatgcaaggagatggcgcccaacagtcccccggccacggggcctgc
caccatacccacgccgaaacaagcgctcatgagcccgaagtggcgagcccgatcttccccatcggtgatg
tcggcgatataggcgccagcaaccgcacctgtggcgccggtgatgccggccacgatgcgtccggcgtaga
ggatcgagatctcgatcccgcgaaattaatacgactcactataggggaattgtgagcggataacaattcc
cctctagaaataattttgtttaactttaagaaggagatatacatatggcgcatcatcaccaccatcatca
tcacggtagcggtgaaaacctctattttcagagcggatccggtagcgcatatattctgcatagcgattac
aaaagcttcgaagatgcaaaagcaaattgtgcagcagaaagcggtccgggtctgagcggtagcacaccgg
aaaccattagcgaaaaaccggaagatattgataatagcaattgtagcagtggtccgggtgcagccaaaaa
tccggaaacaaaacgtgaagcaattgtgaaagcatatggcgacgataatggtcctggtctgccgagcagc
accgcaccggttctgaaaccgcgtcagcagaccaatggtccaggtgaaaccaaaaaggcaattagcgata
cccgtctgaaaaccctggatattcattacaatgaatcaggaccgggtgtgaaagataatgaagtgatgca
agaaaaacgcgacgtggtgattgttaatgatgacccggatcattataaaggtcctggcgcactgtgggat
agcaaatttttcattgaactggaaaacaaaaacgtggaatatgttggccctggtgttcgtcagtatatta
ccgcacaggatcagcctcgttttgatattacctataacattaccgatgcagcacgtcatggtccgggaat
tatgtatgaacagtatttcgtgaacgattatgatcgcgtgccgatttattataatggtaaccgcgtgatc
tataacgatgaaattggtccaggcgaactgctgattagctatgataaagcatgtgcctgcattaagctga
acctgtataaagttgcaattctgccaggtccgggttatatcctgcattcagactataaaagtttcgagga
cgccaaagccaactgcgcagcagaatcaggtccaggcctgccgagttcaaccgctcctgtgctgaaacct
cgccagcaaacaaacggacctggtgcgctgtgggactcaaaattctttattgagcttgagaataaaaacg
tcgagtatgtaggtccaggtctgatcagttatgacaaagcctgcgcatgtattaaactgaatttatacaa
agtggccatcctgcctggtcctggcagcggtagtacccctgaaacaatttcagaaaaacctgaggatatc
gacaattcaaatggaccgggtaagaaagccatttcagatacacgcttaaaaacgctggacatccactata
atggccctggccagtatatcacagcccaggaccaaccgagatttgatatcacgtataatatcacagatgc
cggaccaggggctgcgaaaaatcctgagactaaacgcgaggccatcgttaaagcctatggtgatgataac
ggaccaggtgataacgaggttatgcaagagaagcgtgatgttgttattgtgaacgatgatcctgatcatg
gcccaggtatcatgtatgagcaatactttgtcaatgattatgatagagtcccgatctactacaatggcaa
tcgtgtgatttacaacgatgagatataatgactcgagactgctaacaaagcccgaaaggaagctgagttg
gctgctgccaccgctgagcaataactagcataaccccttggggcctctaaacgggtcttgaggggttttt
tgctgaaaggaggaactatatccggattggcgaatgggacgcgccctgtagcggcgcattaagcgcggcg
ggtgtggtggttacgcgcagcgtgaccgctacacttgccagcgccctagcgcccgctcctttcgctttct
tcccttcctttctcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggtt
ccgatttagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggcca
tcgccctgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgttcc
aaactggaacaacactcaaccctatctcggtctattcttttgatttataagggattttgccgatttcggc
ctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattttaacaaaatattaacgtttaca
atttcaggtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacattcaa
atatgtatccgctcatgaattaattcttagaaaaactcatcgagcatcaaatgaaactgcaatttattca
tatcaggattatcaataccatatttttgaaaaagccgtttctgtaatgaaggagaaaactcaccgaggca
gttccataggatggcaagatcctggtatcggtctgcgattccgactcgtccaacatcaatacaacctatt
aatttcccctcgtcaaaaataaggttatcaagtgagaaatcaccatgagtgacgactgaatccggtgaga
atggcaaaagtttatgcatttctttccagacttgttcaacaggccagccattacgctcgtcatcaaaatc
actcgcatcaaccaaaccgttattcattcgtgattgcgcctgagcgagacgaaatacgcgatcgctgtta
aaaggacaattacaaacaggaatcgaatgcaaccggcgcaggaacactgccagcgcatcaacaatatttt
cacctgaatcaggatattcttctaatacctggaatgctgttttcccggggatcgcagtggtgagtaacca
tgcatcatcaggagtacggataaaatgcttgatggtcggaagaggcataaattccgtcagccagtttagt
ctgaccatctcatctgtaacatcattggcaacgctacctttgccatgtttcagaaacaactctggcgcat
cgggcttcccatacaatcgatagattgtcgcacctgattgcccgacattatcgcgagcccatttataccc
atataaatcagcatccatgttggaatttaatcgcggcctagagcaagacgtttcccgttgaatatggctc
ataacaccccttgtattactgtttatgtaagcagacagttttattgttcatgaccaaaatcccttaacgt
gagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttc
tgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaaga
gctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttctagtg
tagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgt
taccagtggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccgga
taaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacacc
gaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggt
atccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatct
ttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcaggggggcgg
agcctatggaaaaacgccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcaca
tgttctttcctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgc
tcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcctgatgcggtat
tttctccttacgcatctgtgcggtatttcacaccgcatatatggtgcactctcagtacaatctgctctga
tgccgcatagttaagccagtatacactccgctatcgctacgtgactgggtcatggctgcgccccgacacc
cgccaacacccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaagctgtgac
cgtctccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgcgcgaggcagctgcggtaa
agctcatcagcgtggtcgtgaagcgattcacagatgtctgcctgttcatccgcgtccagctcgttgagtt
tctccagaagcgttaatgtctggcttctgataaagcgggccatgttaagggcggttttttcctgtttggt
cactgatgcctccgtgtaagggggatttctgttcatgggggtaatgataccgatgaaacgagagaggatg
ctcacgatacgggttactgatgatgaacatgcccggttactggaacgttgtgagggtaaacaactggcgg
tatggatgcggcgggaccagagaaaaatcactcagggtcaatgccagcgcttcgttaatacagatgtagg
tgttccacagggtagccagcagcatcctgcgatgcagatccggaacataatggtgcagggcgctgacttc
cgcgtttccagactttacgaaacacggaaaccgaagaccattcatgttgttgctcaggtcgcagacgttt
tgcagcagcagtcgcttcacgttcgctcgcgtatcggtgattcattctgctaaccagtaaggcaaccccg
ccagcctagccgggtcctcaacgacaggagcacgatcatgcgcacccgtggggccgccatgccggcgata
atggcctgcttctcgccgaaacgtttggtggcgggaccagtgacgaaggcttgagcgagggcgtgcaaga
ttccgaataccgcaagcgacaggccgatcatcgtcgcgctccagcgaaagcggtcctcgccgaaaatgac
ccagagcgctgccggcacctgtcctacgagttgcatgataaagaagacagtcataagtgcggcgacgata
gtcatgccccgcgcccaccggaaggagctgactgggttgaaggctctcaagggcatcggtcgagatcccg
gtgcctaatgagtgagctaacttacattaattgcgttgcgctcactgcccgctttccagtcgggaaacct
gtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgccagggt
ggtttttcttttcaccagtgagacgggcaacagctgattgcccttcaccgcctggccctgagagagttgc
agcaagcggtccacgctggtttgccccagcaggcgaaaatcctgtttgatggtggttaacggcgggatat
aacatgagctgtcttcggtatcgtcgtatcccactaccgagatatccgcaccaacgcgcagcccggactc
ggtaatggcgcgcattgcgcccagcgccatctgatcgttggcaaccagcatcgcagtgggaacgatgccc
tcattcagcatttgcatggtttgttgaaaaccggacatggcactccagtcgccttcccgttccgctatcg
gctgaatttgattgcgagtgagatatttatgccagccagccagacgcagacgcgccgagacagaacttaa
tgggcccgctaacagcgcgatttgctggtgacccaatgcgaccagatgctccacgcccagtcgcgtaccg
tcttcatgggagaaaataatactgttgatgggtgtctggtcagagacatcaagaaataacgccggaacat
tagtgcaggcagcttccacagcaatggcatcctggtcatccagcggatagttaatgatcagcccactgac
gcgttgcgcgagaagattgtgcaccgccgctttacaggcttcgacgccgcttcgttctaccatcgacacc
accacgctggcacccagttgatcggcgcgagatttaatcgccgcgacaatttgcgacggcgcgtgcaggg
ccagactggaggtggcaacgccaatcagcaacgactgtttgcccgccagttgttgtgccacgcggttggg
aatgtaattcagctccgccatcgccgcttccactttttcccgcgttttcgcagaaacgtggctggcctgg
ttcaccacgcgggaaacggtctgataagagacaccggcatactctgcgacatcgtataacgttactggtt
tcacattcaccaccctgaattgactctcttccggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>514</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..514</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAGSGENLYFQSGSGSAYILHSDYKSFEDAKANCAAESGPGLSGSTPET
ISEKPEDIDNSNCSSGPGAAKNPETKREAIVKAYGDDNGPGLPSSTAPVLKPRQQTNGPGETKKAISDTR
LKTLDIHYNESGPGVKDNEVMQEKRDVVIVNDDPDHYKGPGALWDSKFFIELENKNVEYVGPGVRQYITA
QDQPRFDITYNITDAARHGPGIMYEQYFVNDYDRVPIYYNGNRVIYNDEIGPGELLISYDKACACIKLNL
YKVAILPGPGYILHSDYKSFEDAKANCAAESGPGLPSSTAPVLKPRQQTNGPGALWDSKFFIELENKNVE
YVGPGLISYDKACACIKLNLYKVAILPGPGSGSTPETISEKPEDIDNSNGPGKKAISDTRLKTLDIHYNG
PGQYITAQDQPRFDITYNITDAGPGAAKNPETKREAIVKAYGDDNGPGDNEVMQEKRDVVIVNDDPDHGP
GIMYEQYFVNDYDRVPIYYNGNRVIYNDEIENLYFQSHHHHHHHH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>6127</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6127</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcttttccaacctaaatagaacttcatcgttgcgtttacaacactttt
ctatttgttcaaactttgttgttatattagtaatctttttttccaaattagttagccgttgtttgagagt
ttcctcattatcgtctccataggctttaacaattgcttcgcgtttagtctctggatttttagcagccttt
gtagagaaaaattcagttgctggaattgcaagatcgtcatctccggggaaaagagttccgtcggatccga
tatcagccatggccttgtcgtcgtcgtcggtacccagatctgggctgtccatgtgctggcgttcgaattt
agcagcagcggtttctttcataccagaaccgcgtggcaccagaccagaagaatgatgatgatgatggtgc
atatggccagaaccagaaccggccaggttagcgtcgaggaactctttcaactgacctttagacagtgcac
ccactttggttgccgccacttcaccgtttttgaacagcagcagagtcgggataccacggatgccatattt
cggcgcagtgccagggttttgatcgatgttcagttttgcaacggtcagtttgccctgatattcgtcagcg
atttcatccagaatcggggcgatcattttgcacggaccgcaccactctgcccagaaatcgacgaggatcg
ccccgtccgctttgagtacatccgtgtcaaaactgtcgtcagtcaggtgaataattttatcgctcatatg
tatatctccttcttaaagttaaacaaaattatttctagaggggaattgttatccgctcacaattccccta
tagtgagtcgtattaatttcgcgggatcgagatcgatctcgatcctctacgccggacgcatcgtggccgg
catcaccggcgccacaggtgcggttgctggcgcctatatcgccgacatcaccgatggggaagatcgggct
cgccacttcgggctcatgagcgcttgtttcggcgtgggtatggtggcaggccccgtggccgggggactgt
tgggcgccatctccttgcatgcaccattccttgcggcggcggtgctcaacggcctcaacctactactggg
ctgcttcctaatgcaggagtcgcataagggagagcgtcgagatcccggacaccatcgaatggcgcaaaac
ctttcgcggtatggcatgatagcgcccggaagagagtcaattcagggtggtgaatgtgaaaccagtaacg
ttatacgatgtcgcagagtatgccggtgtctcttatcagaccgtttcccgcgtggtgaaccaggccagcc
acgtttctgcgaaaacgcgggaaaaagtggaagcggcgatggcggagctgaattacattcccaaccgcgt
ggcacaacaactggcgggcaaacagtcgttgctgattggcgttgccacctccagtctggccctgcacgcg
ccgtcgcaaattgtcgcggcgattaaatctcgcgccgatcaactgggtgccagcgtggtggtgtcgatgg
tagaacgaagcggcgtcgaagcctgtaaagcggcggtgcacaatcttctcgcgcaacgcgtcagtgggct
gatcattaactatccgctggatgaccaggatgccattgctgtggaagctgcctgcactaatgttccggcg
ttatttcttgatgtctctgaccagacacccatcaacagtattattttctcccatgaagacggtacgcgac
tgggcgtggagcatctggtcgcattgggtcaccagcaaatcgcgctgttagcgggcccattaagttctgt
ctcggcgcgtctgcgtctggctggctggcataaatatctcactcgcaatcaaattcagccgatagcggaa
cgggaaggcgactggagtgccatgtccggttttcaacaaaccatgcaaatgctgaatgagggcatcgttc
ccactgcgatgctggttgccaacgatcagatggcgctgggcgcaatgcgcgccattaccgagtccgggct
gcgcgttggtgcggacatctcggtagtgggatacgacgataccgaagacagctcatgttatatcccgccg
ttaaccaccatcaaacaggattttcgcctgctggggcaaaccagcgtggaccgcttgctgcaactctctc
agggccaggcggtgaagggcaatcagctgttgcccgtctcactggtgaaaagaaaaaccaccctggcgcc
caatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctggcacgacaggtttcccga
ctggaaagcgggcagtgagcgcaacgcaattaatgtaagttagctcactcattaggcaccgggatctcga
ccgatgcccttgagagccttcaacccagtcagctccttccggtgggcgcggggcatgactatcgtcgccg
cacttatgactgtcttctttatcatgcaactcgtaggacaggtgccggcagcgctctgggtcattttcgg
cgaggaccgctttcgctggagcgcgacgatgatcggcctgtcgcttgcggtattcggaatcttgcacgcc
ctcgctcaagccttcgtcactggtcccgccaccaaacgtttcggcgagaagcaggccattatcgccggca
tggcggccccacgggtgcgcatgatcgtgctcctgtcgttgaggacccggctaggctggcggggttgcct
tactggttagcagaatgaatcaccgatacgcgagcgaacgtgaagcgactgctgctgcaaaacgtctgcg
acctgagcaacaacatgaatggtcttcggtttccgtgtttcgtaaagtctggaaacgcggaagtcagcgc
cctgcaccattatgttccggatctgcatcgcaggatgctgctggctaccctgtggaacacctacatctgt
attaacgaagcgctggcattgaccctgagtgatttttctctggtcccgccgcatccataccgccagttgt
ttaccctcacaacgttccagtaaccgggcatgttcatcatcagtaacccgtatcgtgagcatcctctctc
gtttcatcggtatcattacccccatgaacagaaatcccccttacacggaggcatcagtgaccaaacagga
aaaaaccgcccttaacatggcccgctttatcagaagccagacattaacgcttctggagaaactcaacgag
ctggacgcggatgaacaggcagacatctgtgaatcgcttcacgaccacgctgatgagctttaccgcagct
gcctcgcgcgtttcggtgatgacggtgaaaacctctgacacatgcagctcccggagacggtcacagcttg
tctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtcagcgggtgttggcgggtgtcggggc
gcagccatgacccagtcacgtagcgatagcggagtgtatactggcttaactatgcggcatcagagcagat
tgtactgagagtgcaccatatatgcggtgtgaaataccgcacagatgcgtaaggagaaaataccgcatca
ggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagc
tcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaa
ggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctg
acgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggc
gtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcc
tttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcg
ttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaacta
tcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattagc
agagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctacactagaagga
cagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccgg
caaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaagga
tctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcacgttaaggga
ttttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatc
aatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctca
gcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacgatacgggagg
gcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcagc
aataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtct
attaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgccattg
ctgcaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaag
gcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcaga
agtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttactgtcatgccat
ccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgacc
gagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcatc
attggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaac
ccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacagg
aaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactcttccttttt
caatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaaa
ataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgaaattgtaaacgttaatatttt
gttaaaattcgcgttaaatttttgttaaatcagctcattttttaaccaataggccgaaatcggcaaaatc
ccttataaatcaaaagaatagaccgagatagggttgagtgttgttccagtttggaacaagagtccactat
taaagaacgtggactccaacgtcaaagggcgaaaaaccgtctatcagggcgatggcccactacgtgaacc
atcaccctaatcaagttttttggggtcgaggtgccgtaaagcactaaatcggaaccctaaagggagcccc
cgatttagagcttgacggggaaagccggcgaacgtggcgagaaaggaagggaagaaagcgaaaggagcgg
gcgctagggcgctggcaagtgtagcggtcacgctgcgcgtaaccaccacacccgccgcgcttaatgcgcc
gctacagggcgcgtcccattcgccaatccggatatagttcctcctttcagcaaaaaacccctcaagaccc
gtttagaggccccaaggggttatgctagttattgctcagcggtggcagcagccaactcagcttcctttcg
ggctttgttagcagccggatctcagtggtggtggtggtggtgctcgagtgcggccgca</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>260</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..260</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSDKIIHLTDDSFDTDVLKADGAILVDFWAEWCGPCKMIAPILDEIADE
YQGKLTVAKLNIDQNPGTAPKYGIRGIPTLLLFKNGEVAATKVGALSKGQLKEFLDANLAGSGSGHMHHH
HHHSSGLVPRGSGMKETAAAKFERQHMDSPDLGTDDDDKAMADIGSDGTLFPGDDDLAIPATEFFSTKAA
KNPETKREAIVKAYGDDNEETLKQRLTNLEKKITNITTKFEQIEKCCKRNDEVLFRLEKLAAALEHHHHH
H</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>106</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..106</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QFTASLAVSLGQRATISCRASESVDSYGNSFMHWYQQKPGQPPKLLIYR
ASNLESGIPARFSGSGSRTDFTLTINPVEADDVATYYCQQSNEDPRAFGGGTKLEIK</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>204</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..204</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VQLKESGPGLVAPSQSLSITCTVSGFSLTDYGVSWIRQPPGKGLEWLGV
IWGGGSTYYNSALKSRLSISKDNSKSQVFLKMNSLQTDDTCAGCTCTCAAATCCAGACTGAGCATCAGCA
AGGACAACTCCAAGAGCCAAGTTTTCTTAAAAATGAACAGTCTGCAAACTGATGACACAGAMYYCAKPKR
YYGSGVKEPQSPSPQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Настоящее изобретение относится к области биотехнологии и биохимии. Предложен способ молекулярно-генетической идентификации сортов и линий сои. Способ включает выделение геномной ДНК, постановку двух мультиплексных ПЦР - амплификации с двумя смесями праймеров. Смесь праймеров №1 включает следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16 для идентификации локусов Sct001, Sat090, Satt141, Satt681, Sat084, Satt264, Satt440, Sat_177. Смесь праймеров №2 включает следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30 для идентификации локусов Satt245, Satt181, Soysc514, Satt009, Soyhsp176, Soyprl, Satt431. Далее проводят визуализацию продуктов амплификации с помощью капиллярного электрофореза и идентификацию сортов и линий сои. Изобретение позволяет эффективно идентифицировать сорта и гибриды сои и может быть использовано в селекции и семеноводстве сои. 6 ил., 1 табл.
Способ молекулярно-генетической идентификации сортов и линий сои, включающий в себя следующие этапы:
выделение геномной ДНК,
постановка двух мультиплексных ПЦР - амплификации с двумя смесями праймеров, где смесь праймеров №1 включает в себя следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 9, SEQ ID NO: 10, SEQ ID NO: 11, SEQ ID NO: 12, SEQ ID NO: 13, SEQ ID NO: 14, SEQ ID NO: 15, SEQ ID NO: 16 для идентификации локусов Sct001, Sat090, Satt141, Satt681, Sat084, Satt264, Satt440, Sat_177, и смесь праймеров №2 включает в себя следующие пары олигонуклеотидных последовательностей: SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26, SEQ ID NO: 27, SEQ ID NO: 28, SEQ ID NO: 29, SEQ ID NO: 30 для идентификации локусов Satt245, Satt181, Soysc514, Satt009, Soyhsp176, Soyprl, Satt431,
визуализация продуктов амплификации с помощью капиллярного электрофореза,
идентификация сортов и линий сои путем получения для каждого образца уникального профиля - набора аллелей в анализируемых локусах, и дальнейшего сравнения полученных профилей исследуемых образцов друг с другом для их последующей дифференциации.
| СПОСОБ ДНК-ИДЕНТИФИКАЦИИ И ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА ПАСТБИЩНОГО И ОДНОЛЕТНЕГО НА ОСНОВЕ СИСТЕМ SSR- и SCoT-МАРКИРОВАНИЯ | 2023 |
|
RU2826148C1 |
| Машина для обрывания головок льна | 1931 |
|
SU30796A1 |
| Устройство для подвешивания самоспекающихся электродов электрических печей | 1929 |
|
SU26271A1 |
| CN 109338008 A, 15.02.2019 | |||
| CN 102443583 A, 09.05.2012 | |||
| СПОСОБ ИДЕНТИФИКАЦИИ СОРТОВ СОИ НА ОСНОВЕ МИКРОСАТЕЛЛИТНЫХ (SSR) МАРКЕРОВ | 2008 |
|
RU2388828C1 |
| РАМАЗАНОВА С.А | |||
| Идентификация сортов сои с использованием молекулярно-генетических методов, автореферат диссертации, Краснодар, 2008, с | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Бондаренко О.Н | |||
| и др | |||
| Подбор микросателлитных | |||
Авторы
Даты
2025-05-12—Публикация
2024-11-01—Подача