Изобретение относится к области молекулярной биологии и генетики растений, в частности, к способу маркирования и паспортизации сортов райграса на основе генетических дескрипторов - ДНК-маркеров.
Известен способ идентификации сортов райграса пастбищного (Lolium perenne L.) и райграса гибридного (Lolium Blolium x boucheanum Kunth.), основанный на использовании специфических праймеров, фланкирующих области микросателлитных локусов в геномах этих видов [1]. Прямой и обратный праймеры для идентификации 9 сортов разрабатывали с учетом информации о нуклеотидных последовательностях маркеров, применявшихся ранее для амплификации геномной ДНК райграса итальянского и гибридного. Базовую последовательность маркера, специфичного для сорта, получали путем объединения последовательностей генов, присутствующих в геноме Lolium perenne L. и Lolium Blolium x boucheanum Kunth. Дифференциацию сортов осуществляли на основе определения различий в длине амплифицированных фрагментов. Недостатком данного способа является сложность разработки корректно функционирующего праймера к специфичному маркеру для идентификации сорта. Это кропотливая и дорогостоящая процедура, затруднительная для проведения рутинного анализа по определению, например, сортовой чистоты большой партии семян.
Известен способ применения набора специфических молекулярных SSR-маркеров для определения подлинности семян райграса пастбищного [2]. Из тестируемых партий семян случайно отбирали по 10 штук, получали проростки для выделения ДНК и проведения ПНР с целью выявить принадлежность к определенному сорту. От других известных методик данный способ отличается набором праймеров и условиями амплификации. Основным недостатком является небольшая выборка семян от каждого образца, недостаточная для оценки генетического полиморфизма высокогетерогенных сортов-популяций райграса [3]. Кроме того, эффективность метода снижает канцерогенность используемого в работе 8% полиакриламидного геля и трудоемкость его приготовления.
Известен способ оценки генетического разнообразия и идентификации сортов райграса однолетнего на основе SSR-маркеров [4]. Геномную ДНК для анализа 6 тетраплоидных сортов, зарегистрированных в Китае, выделяли с помощью «мульти-балк-стратегии». Для этого из листьев 10 индивидуальных генотипов от каждого сорта формировали 10 «балк-образцов», при генотипировании которых определили стандартный набор из 12 пар SSR-праймеров для выявления аллелей, встречающихся суммарно во всех образцах с частотой не менее 40%. Недостатки метода: сложность и многоступенчатость анализа и статистической обработки результатов, высокая стоимость реагентов для всех процедур, в частности дорогостоящего коммерческого KITa для ДНК-экстракции (производитель - «Tiangen R, Beijing», China) и раствора серебра (AgNO3) для окрашивания продуктов ПЦР после электрофореза в 6% ПААГ (полиакриламидном геле). Затратна по времени и труду подготовка материала для ДНК-экстракции (проращивание семян в ростовых камерах, пересадка проростков в сосуды и доращивание в теплице в течение двух недель для сбора молодых листьев), а также процедура детекции продуктов ПЦР.
Известен метод ДНК-паспортизации сортообразцов и гибридов многолетних злаковых трав с использованием маркеров 3-х типов [5]. Авторы изучали генетический полиморфизм лисохвоста лугового, житняка гребенчатого, гибридов фестулолиума с помощью мультилокусных маркеров, маркирующих произвольные участки ДНК (набор из 12 ISSR- и 4-х RAPD-праймеров соответственно). Для оценки генетического полиморфизма образцов райграса использовали 5 праймеров к SCoT-маркерам, маркирующим ДНК, ассоциированную с кодирующими участками генома. Полученные ПЦР-продукты разделяли в агарозном геле и выявляли сортоспецифичные мультилокусные ДНК-спектры для разработки молекулярно-генетических формул-паспортов. На основе генотипирования с использованием SCoT-маркеров получены паспорта для 1 сорта райграса многоцветкового (Lolium multiflorum L.) и 1 межвидового гибрида (Lolium multiflorum × Lolium perenne). Недостатки данного способа: отсутствие репрезентативной выборки (анализ каждого образца проводится на уровне отдельного генотипа); в СТАБ-методе выделения ДНК используется жидкий азот, требующий особых условий перевозки и хранения, а также небезопасные для здоровья персонала меркаптоэтанол и хлороформ. Существенно снижает точность анализа и статистической обработки а, следовательно, информативность и корректность составленных паспортов методика определения размеров полученных ПЦР-продуктов: по результатам детекции в агарозном геле без последующей верификации данных.
Цель изобретения - разработка достоверного и надежного способа ДНК-идентификации и генетической паспортизации сортов райграса на основе ПЦР-технологии с использованием специфических микросателлитных (SSR) локусов и мультилокусных SCoT-маркеров.
Поставленная цель достигается тем, что способ ДНК-идентификации и генетической паспортизации сортов райграса пастбищного и однолетнего включает: использование специфических SSR-локусов и мультилокусных SCoT-маркеров, маркирующих ДНК, ассоциированную с кодирующими участками генома, для оценки генетического полиморфизма, детекцию продуктов ПЦР путем предварительного электрофореза в агарозном геле, определение размеров сортоспецифичных фрагментов в ДНК-спектрах с последующей валидацией полученных результатов и разработку молекулярно-генетических формул на основе выявленного аллельного разнообразия. Выделение ДНК осуществляют модифицированным SDS-методом из пророщенных семян, взятых в количестве не менее тридцати от каждого сорта, ДНК-спектры для исследуемых сортов получают с использованием двух систем маркирования (7 микросателлитных SSR-локусов и 9 SCoT-маркеров), размеры уникальных для образца исследуемой коллекции ДНК-фрагментов верифицируют методом высокоразрешающего капиллярного электрофореза и/или выборочным секвенированием, а в паспорте, наряду с молекулярно-генетической формулой, указывают и важнейшие морфо-биологические и хозяйственные характеристики сорта.
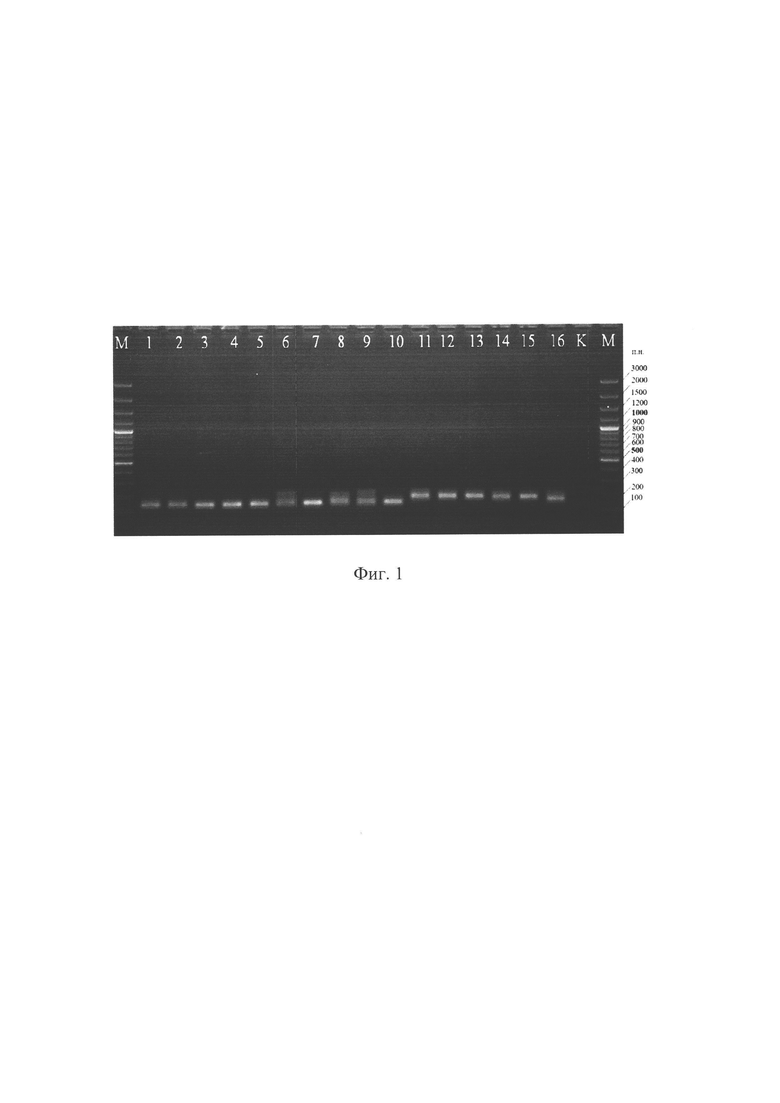
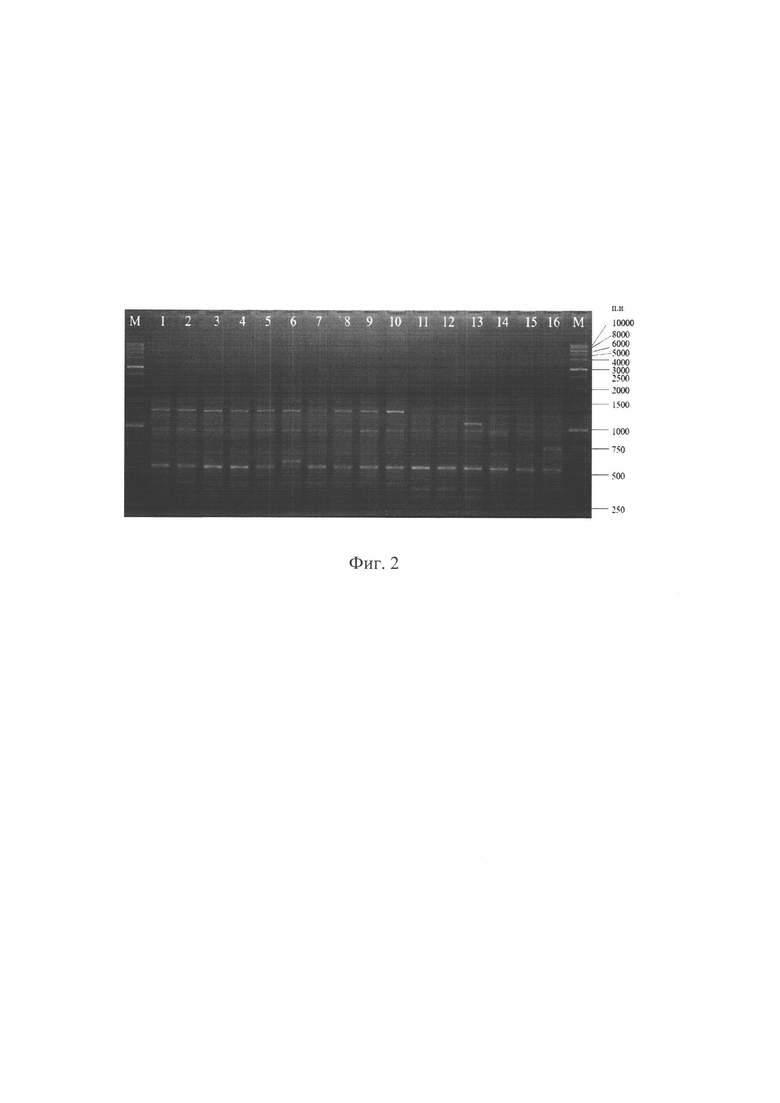
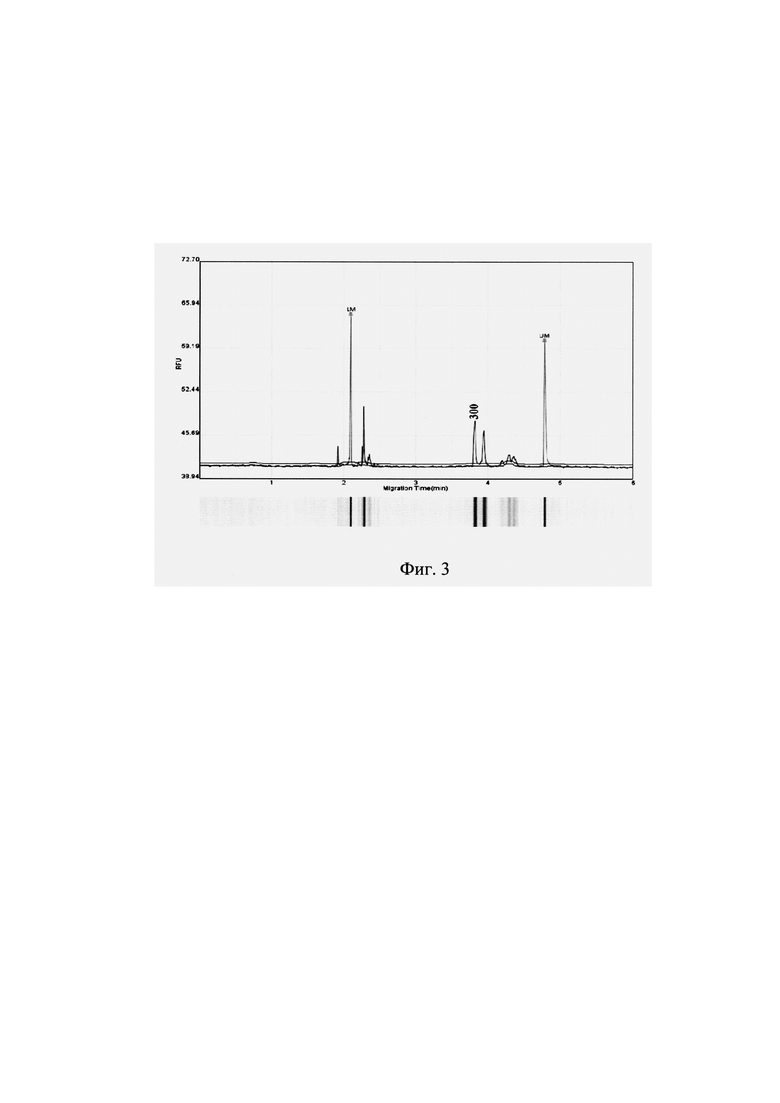
На фиг. 1 изображена электрофореграмма продуктов ЦЦР образцов райграса с SSR-праймером LPSSRh01h06, К - контроль (вода); М - маркер молекулярной массы 100 bp Ladder, GeneRuler («Thermo Fisher Scientific)), USA); на фиг. 2 - электрофореграмма продуктов ПЦР образцов райграса, полученных с использованием маркера SCoT 35, М - маркер молекулярной массы 1kB DNA Ladder («Евроген Лаб», Россия). Последовательность загрузки образцов в лунки геля на фигурах 1 и 2: 1-сорт Агат; 2-Дуэт; 3-ВИК 66; 4-Карат; 5-Феникс; 6-ВИК 22; 7-Ленинградский 809; 8-Вея; 9-Веймар; 10-Выль; 11-Рапид; 12-Московский 74; 13-Roznovsky; 14-Sprint; 15-Изорский; 16-образец Дикорастущий; на фиг. 3 изображен генетический профиль райграса пастбищного сорта Агат с SSR-маркером G05_044 по результатам капиллярного электрофореза. Цифровым показателем (300) обозначен размер уникального для сорта ДНК-фрагмента; на фиг. 4 представлен образец выравнивания относительно генома L. perenne (gramene) нуклеотидной последовательности уникального для сорта ВИК 22 ДНК-фрагмента, полученного с использованием праймера к маркеру SCoT 35; на фиг. 5 представлен образец генетического паспорта райграса пастбищного сорта Агат, разработанного на основе SSR-маркеров; на фиг. 6 изображен образец генетического паспорта райграса пастбищного сорта ВИК 22 - на основе SCoT-маркеров; фиг. 7 - скриншот интернет-страницы международной БД GenBank ресурса NCBI с данными по секвенированию специфичной нуклеотидной последовательности сорта райграса пастбищного Карат.
Райграс используется в мировой практике луговодства для производства кормов, в фитомелиоративных целях для повышения плодородия почв и рекультивации земель, при создании газонов различного типа. В северозападных, западных и центральных регионах России наиболее распространен райграс пастбищный или многолетний (Lolium perenne L.) и райграс однолетний (многоцветковый) (Lolium multiflorum Lam.). Ежегодно создаются новые улучшенные сорта, для идентификации и паспортизации которых, как и уже существующих, необходимы надежные системы маркирования. Это особенно актуально для райграса,- культуры с многолетним циклом развития и высокой внутрисортовой генетической гетерогенностью, обусловленной перекрестным типом опыления, и при этом характеризующейся выраженным сходством между сортами по морфологическим признакам.
В последние годы для оценки генетического разнообразия и дифференциации сортов, линий, генотипов успешно применяют микросателлитные локусы или SSR-маркеры (Simple Sequence Repeat) [6]. Они представляют собой простые короткие повторяющиеся повторы последовательностей нуклеотидов, которые тандемно распространены в геномах большинства видов эукариот.В качестве экономических преимуществ анализа на основе микросателлитных маркеров можно отметить, что он не требует большого количества и высокого качества матричной ДНК для ПЦР, несложен для выполнения и относительно недорог, если использовать информацию о сиквенсах ранее разработанных маркеров. Однако при исследовании близкородственного материала не всегда удается выявить полиморфизм на межсортовом уровне. В этом случае для повышения эффективности оценки генетической изменчивости оправданным является применение дополнительной системы маркирования, позволяющей проанализировать другие области генома, не связанные с изменениями в микросателлитных локусах.
В нашем изобретении наряду с SSR-маркерами использовали анализ на основе SCoT-маркеров (Start Codon Targeted) [7]. Данный метод является низкозатратным, характеризуется высокой степенью выявляемого полиморфизма с помощью одного праймера большой длины (до 18 нуклеотидов), что обеспечивает стабильную воспроизводимость результатов. SCoT-маркеры считаются «молекулярными маркерами генов», т.к. позволяют маркировать ДНК в кодирующих участках генома, в отличие от рандомных RAPD и ISSR. В связи с этим информация, полученная при изучении генетического разнообразия, может быть полезной для селекционных целей.
Поскольку ни одна из существующих систем молекулярного маркирования не является универсальной, паспортизация сортов, основанная на комбинации методов с использованием маркеров двух типов (SSR и SCoT) и последующего выборочного секвенирования для валидации выявленных генетических дескрипторов, является наиболее эффективным и экономичным подходом.
Способ осуществляется следующим образом. Из партии семян исследуемых сортов случайным образом отбирают по 100 штук для каждого образца, проращивают их на фильтровальной бумаге в чашках Петри в течение 7 суток, увлажняя по мере необходимости. Из эпикотилей полученных проростков формируют суммарную навеску в количестве 30 генотипов от сорта («балк-образец») и выделяют геномную ДНК с помощью SDS-метода [8], модифицированного в условиях лаборатории с учетом специфики данной части растительной ткани. Модификации заключаются в следующем. Токсичные хлороформ, фенол, изоамиловый спирт заменены дешевым, простым в подготовке и применении, безопасным для здоровья персонала, 5 М ацетатом аммония в качестве коагулянта белков. Упрощена процедура ДНК-экстракции за счет сокращения этапов дополнительной очистки и промывания растворов этанолом, что позволяет сэкономить время, уменьшить расход пробирок и одноразовых наконечников. Объем экстракционного SDS-буфера определяют индивидуально на каждый образец, увеличивая или уменьшая, в зависимости от количества доступной растительной массы и требуемого количества ДНК для исследований. Внесенные в протокол изменения обеспечивают получение качественных образцов ДНК из суммарной навески проростков при относительно низкой стоимости, доступности и безопасности используемых реагентов, простоте и оперативности выполнения всех процедур. Образцы ДНК амплифицируют с набором предварительно отобранных, информативных для культуры, праймеров с последующей детекцией ПНР-продуктов в агарозном геле. Размеры выявленных сортоспецифичных аллелей верифицируют путем высокоразрешающего капиллярного электрофореза и/или выборочным секвенированием на автоматическом анализаторе. После статистической обработки результатов генотипирования составляют молекулярно-генетические формулы (основу ДНК-паспортов); секвенированные нуклеотидные последовательности, идентифицирующие сорта райграса, размещают в базах данных для хранения, систематизации и рационального использования информации в фундаментальных исследованиях и в прикладных целях.
Заявляемый способ иллюстрируется примерами и чертежами, где представлены последовательность выполнения экспериментальных работ и образцы получаемых результатов.
Пример 1. Выделение тотальной геномной ДНК для ПЦР-анализа из проростков генотипов анализируемых сортов.
SDS-буфер для ДНК-экстракции содержит 200 mM трис HCl рН 7,5; 250 mM NaCl; 25 mM ЭДТА; 0,5% SDS. Гомогенизацию растительной ткани осуществляют вручную в ступке с пестиком или на гомогенизаторе «TissueLyser LT» («QIAGEN», Германия) с помощью металлических шариков. После этапа лизиса клеточных оболочек к экстракту добавляют 3 мкл фермента РНКазы для разрушения молекул РНК и инкубируют смесь в термостате при 60°С в течение 1 часа. От белковых примесей освобождаются с помощью 5 М ацетата аммония. Проводят осаждение изопропанолом и однократное промывание осадка охлажденным 70%-ным этиловым спиртом с последующим подсушиванием на воздухе, а затем растворяют его в 50 мкл ТЕ буфера (0,1 М Трис-HCl, 1 мМ ЭДТА, рН 8,0). Количественные и качественные показатели образцов ДНК определяют методом электрофореза в 1,5%-ном агарозном геле и на спектрофотометре «Nano Spectrophotometer Nabi» («MicroDigital», Корея). Конечную концентрацию образцов ДНК для использования в ПЦР доводят до 30 нг/мкл.
Пример 2. ПЦР-анализ по выявлению генетического полиморфизма в сортах райграса пастбищного и однолетнего с использованием SSR-маркеров.
Реактивы для анализа по двум системам маркирования получали от компании ЗАО «Евроген Лаб» (Россия). Полимеразная цепная реакция проводится в амплификаторе «Т100 Thermal Cycler» («Bio-Rad», США) с использованием реакционной смеси объемом 20 мкл, содержащей 3 мкл 10x Taq Turbo buffer, 0,4 мкл 50х dNTP mix (10 мМ каждого из дезоксинуклеотидов), 0,3 мкл Taq-ДНК полимеразы (5 е.а./мкл), 1 мкл образца ДНК (30 нг/мкл), по 1 мкл (10 мкМ) прямого и обратного праймеров и 13 мкл деионизированной воды. Режимы амплификации выбирают в соответствии с рекомендациями разработчиков маркеров [9, 10, 11]. Для праймеров к маркерам LPSSRh01h06 и LPSSRk03b03 температура отжига подбиралась индивидуально с использованием онлайн-инструмента «Tm Calculator)). Продукты ПЦР разделяют с помощью предварительного электрофореза в 1,6%-ном агарозном геле при 90 V в течение 90 минут для выявления полиморфных фрагментов амплификации (фиг. 1).
Пример 3. ПЦР-анализ по выявлению генетического полиморфизма в сортах райграса пастбищного и однолетнего с использованием SCoT-маркеров.
Реакционная смесь общим объемом 20 мкл содержит 2 мкл 10×Taq Turbo buffer, 0,4 мкл 50х dNTP mix (10 мМ каждого из дезоксинуклеотидов), 0,2 мкл Taq-ДНК полимеразы (5 е.а./мкл), 1 мкл ДНК (концентрация 30 нг/мкл), 1 мкл праймера (8 мкМ), 15,4 мкл деионизированной воды. Условия амплификации в термоциклере «Т100 Thermal Cycler»: 3 мин - 94°С; 1 мин - 94°С, 1 мин - 50°С и 1 мин - 72°С (35 циклов); финальная элонгация - 5 мин при 72°С. Электрофорез фрагментов амплификации проводится в 2 этапа: при 90 V в течение 120 минут и дополнительно 60 минут при 50 V (фиг. 2).
Размеры продуктов ПЦР, выявленных по двум системам маркирования, определяют с помощью пакета программ Image Lab version 6.0.1 на приборе «GelDoc XR Plus» («Bio-Rad», США) в сравнении с маркерами молекулярной массы 1kb DNA Ladder («Евроген Лаб», Россия) и 100 bp Ladder, GeneRuler («Thermo Fisher Scientific)), США). Для коллекции из 15 сортов райграса выделены 7 SSR и 9 SCoT-маркеров, с которыми получены воспроизводимые фрагменты амплификации с полиморфизмом между отдельными образцами (табл. 1).
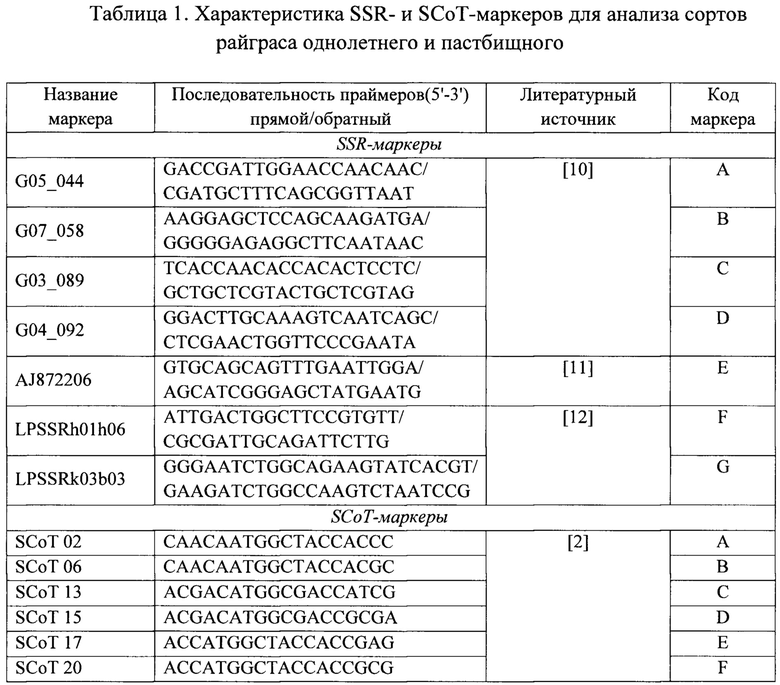
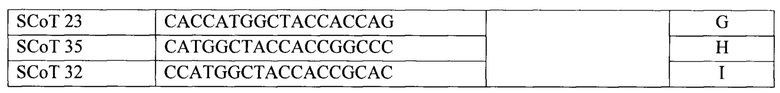
Пример 4. Валидация размеров сортоспецифичных аллелей и разработка молекулярно-генетических формул, как основы генетического паспорта сорта.
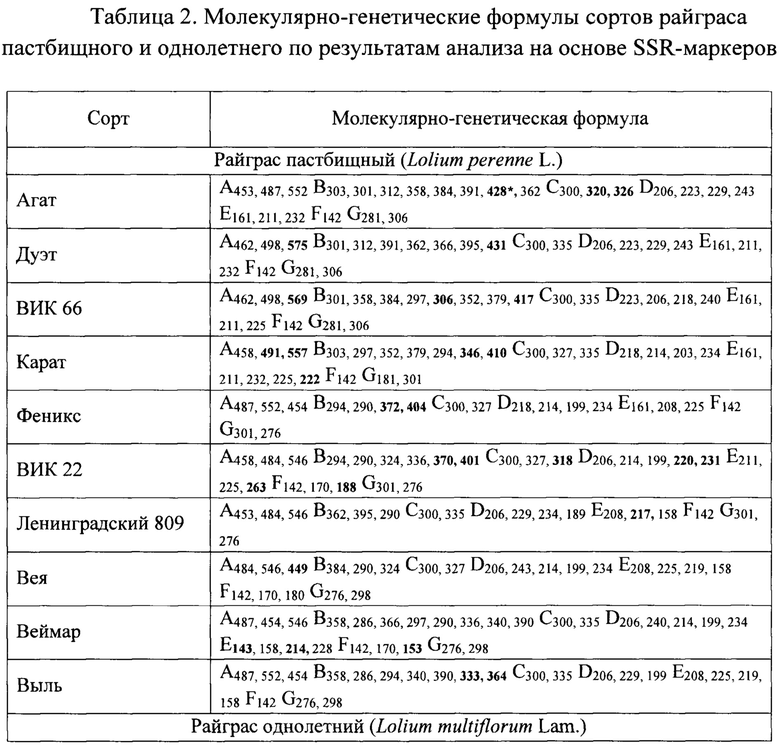
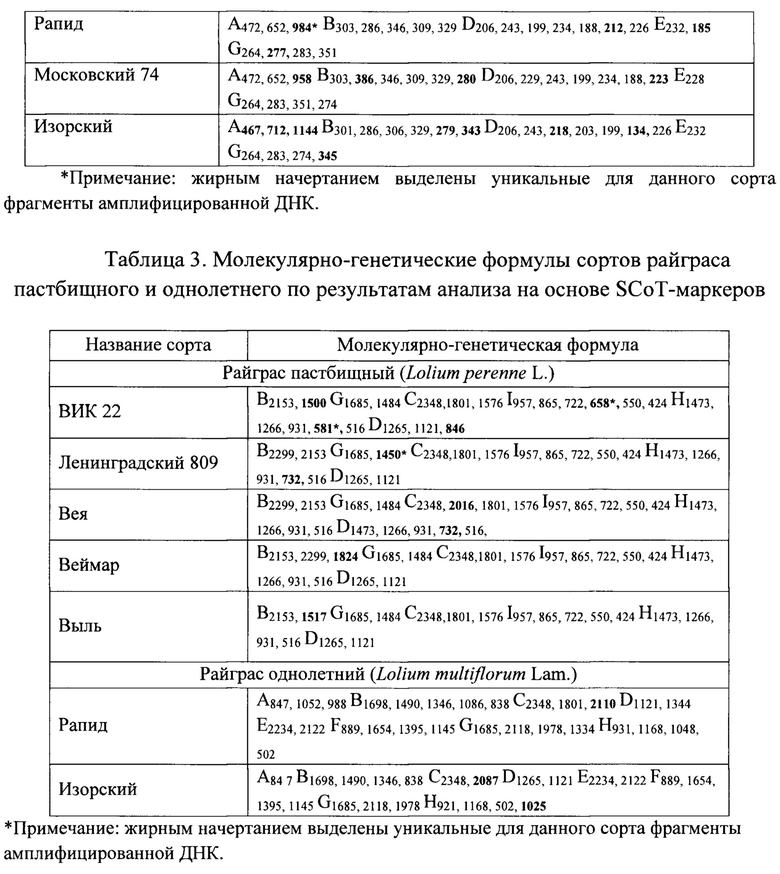
Воспроизводимые, уникальные для сорта в исследуемой коллекции, фрагменты ДНК верифицируют методом капиллярного электрофореза и/или передают для клонирования и секвенирования (ЗАО «Евроген Лаб», Россия) (фиг. 3, 4). Молекулярно-генетические формулы, которые в нашем способе служат основой для разработки генетических паспортов, составлены для 5 сортов райграса пастбищного и 2 сортов райграса однолетнего по двум панелям маркеров (SSR и SCoT), а также для 5 сортов Lolium perenne L. и 1 сорта Lolium multiflorum Lam. - только по SSR-локусам (табл. 2, 3).
Пример 5. Разработка дизайна генетического паспорта.
Для сортов, выделившихся из общей коллекции уникальным фрагментом в ДНК-спектре, разрабатывают генетические паспорта, в которые для полноты характеристики включают информацию по происхождению сорта, регионам возделывания, основным морфологическим признакам и хозяйственно-биологическим свойствам (фиг. 5, 6).
Пример 6. Размещение сортоспецифичных ДНК-фрагментов, полученных при разработке способа, в базах данных.
Нуклеотидные последовательности, идентифицирующие сорта райграса, после верификации секвенированием и «выравнивания» относительно генома L. perenne (gramene), размещают в специализированной базе данных [12] (табл. 4), а также депонируют в международной БД GenBank NCBI (https://www.ncbi.nlm.nih.gov) (фиг. 7). Эта информация может использоваться в исследованиях по генетике и филогенетике райграса разных видов, а также в работе селекционеров по подбору перспективных пар для скрещивания, контролю результатов гибридизации и структуры сортов-популяций при решении вопросов сортообновления и сортосмены.

Сопоставительный анализ позволяет сделать вывод, что заявляемый способ отличается от известных используемыми веществами (реагенты для ДНК-экстракции, наборы праймеров к SSR и SCoT-маркерам), а также методами и условиями выполнения процедур анализа (формирование репрезентативной выборки генотипов - не менее 30 на 1 сорт/сортообразец, операции по выделению ДНК, проведению ПЦР и верификации полученных результатов). Предложен оригинальный дизайн генетического паспорта, включающий не только молекулярно-генетическую формулу образца, но и его важнейшие морфо-биологические и хозяйственные характеристики.
Преимущества заявляемого способа заключаются в улучшенной воспроизводимости результатов за счет большей длины SCoT-праймеров (в сравнении с RAPD) и высокой температуры их отжига; в простоте, безопасности для персонала и скорости выполнения всех процедур; повышенной надежности и достоверности идентификации сортов, обусловленной использованием объемных образцов для анализа, применением двух систем маркирования, верификацией данных с помощью выборочного секвенирования; в снижении экономических затрат за счет эффективного и дешевого модифицированного метода выделения ДНК и универсальных праймеров для ПЦР. Размещенную в базах данных информацию по специфичным нуклеотидным последовательностям для райграса пастбищного и однолетнего можно использовать в системе сортоиспытания и в селекционных программах, что позволит удешевить, сократить сроки и повысить точность оценки исходного материала.
Список литературы
1. JP2011211933A. 2011-10-27. SSR Primer Pair Useful for Identification of Cultivars of Lolium perenne L. and Lolium X Boucheanum Kunth, and Use Thereof. U. Nana, S. Koi, F. Akihiko, H. Kuniaki, O. Yasuko. Japan Grassland farming Forage Seed Association; Nat. Livestock Breeding Center.
2. CN103773868A 2014-05-07 Specific molecular marker kit for identifying authenticity of perennial ryegrass seed and method thereof. L. Zhipeng, L. Peng, L. Dong, W. Yanrong, T. Jianhui, W. Yu. Langzhov University.
3. Liu S., Feuerstein Ulf, Luesink W., Schulze S., Asp Т., Studer В., Becker H.C. and Dehmer K. DArT, SNP, and SSR analyses of genetic diversity in Lolium perenne L. using bulk sampling. BMC Genetics (2018) 19:10. DOI 10.1186/s 12863-017-0589.
4. Nie G, Huang T, Ma X, Huang L, Peng Y, Yan Y, Li Z, Wang X, Zhang X. Genetic variability evaluation and cultivar identification of tetraploid annual ryegrass using SSR markers. 2019. Peer J 7:e7742. http://doi.org/10.7717/peerj.7742.
5. Кондрацкая И.П., Юхимук А.Н., Чижик О.В., Решетников В.Н., Столепченко В.А., Васько П.П. ДНК паспортизация сортообразцов и гибридов многолетних злаковых трав. Фактори експериментальноï еволюцiï органiзмiв. 2018. Том 22. С. 267-273. ISSN 2415-3826.
6. Tautz D., Renz M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes. Nucleic Acids Research. 1984. 12(10):4127-4138. DOI: 10.1093nar/12.10.4127.
7. Collard BCY, Mackill DJ. Start codon targeted (SCoT) polymorphism: a simple, novel DNA marker technique for generating gene-targeted markers in plants. Plant Molecular Biology Reporter. 2009. 27(1):86-93. DOI: 10.1007/s11105-008-0060.
8. Kirby K.S. and Cook E.A. Isolation of deoxyribonucleic acid from mammalian tissues. Biochemical Journal. 1967. V. 104 (1). P. 254-257. DOI: 10.1042/bj 1040254.
9. Studer В., Asp Т., Frei U., Hentrup S., Meally H., Guillard A., et al. Expressed sequence tag-derived microsatellite markers of perennial ryegrass (Lolium perenne L.). Molecular Breeding. 2008. 21:533-548. DOI: 10.1007/s11032-007-9148-0.
10. Lauvergeat V., Barre P., Bonnet M., Ghesquiere M. Sixty simple sequence repeat markers for use in the Festuca-Lolium complex of grasses. Molecular Ecology Notes. 2005. 5(2):401-405. DOI: 10.1111/j.1471-8286.2005.00941.x.
11. Wang J., Dobrowolski M.P., Cogan N.O., Forster J.W., Smith K.F. Assignment of individual genotypes to specific forage cultivars of perennial ryegrass based on SSR markers. Crop Science. 2009. 49(l):49-58. DOI: 10.2135/cropsci2008.03.0177.
12. Клименко И.А., Шамустакимова A.O., Мавлютов Ю.М., Душкин В. А., Антонов А. А. База данных нуклеотидных последовательностей, идентифицирующих сорта кормовых культур. Свидетельство о государственной регистрации базы данных №2023622067. 22.06.2023.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="СПОСОБ
ДНК-ИДЕНТИФИКАЦИИ И ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА
ПАСТБИЩНОГО И ОДНОЛЕТНЕГО НА ОСНОВЕ СИСТЕМЫ SSR- И
SCoT-МАРКИРОВАНИЯ.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-11-13">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023124594</ApplicationNumberText>
<FilingDate>2023-09-25</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">ФНЦ "ВИК им. В.Р.
Вильямса"</ApplicantName>
<ApplicantNameLatin>FNC "VIK im. V.R.
Vil'yamsa"</ApplicantNameLatin>
<InventionTitle languageCode="ru">СПОСОБ ДНК-ИДЕНТИФИКАЦИИ И
ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА ПАСТБИЩНОГО И ОДНОЛЕТНЕГО
НА ОСНОВЕ СИСТЕМЫ SSR- И SCoT-МАРКИРОВАНИЯ</InventionTitle>
<SequenceTotalQuantity>26</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaccgattggaaccaacaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgatgctttcagcggttaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaggagctccagcaagatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggggagaggcttcaataac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaccaacaccacactcctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgctcgtactgctcgtag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggacttgcaaagtcaatcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcgaactggttcccgaata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcagcagtttgaattgga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcatcgggagctatgaatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attgactggcttccgtgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcgattgcagattcttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggaatctggcagaagtatcacgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaagatctggccaagtctaatccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacaatggctaccaccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacaatggctaccacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgacatggcgaccatcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgacatggcgaccgcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatggctaccaccgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatggctaccaccgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caccatggctaccaccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catggctaccaccggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccatggctaccaccgcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>300</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..300</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaccaacaccacactcctccacccgctcgctccctccctcccgctcgc
cccgctgccggccggcgaatccgagcaatggtgcgcgccactcgctctccgctctccctttctctccccc
ttccccttccccgccgccgcccgatcatcatctcgggttctccgccgtcttttgcaggatttcaaggggt
tctgggagtccaagttcgggggcaagaaggagccggagcgggacggggagcccaacggcagcgtcaagaa
gcggaccgccgatttggccatctacgagcagtacgagcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>142</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..142</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attgactggcttccgtgttcattgatcattgccctgaaaattattgtca
gcaacaaaaccatgatgaacatcagacacaacacaattcaaatgtccaaagaagatgacacacacacacc
tgtaacaagaatctgcaatcgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>581</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..581</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Lolium perenne</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catggctaccaccggccccaaaatatgaccttatgtgcaacttatttag
tttcttatgcactacctgtcgggggtgctccccggcaatgcacaccatgaggggcttagggtttacagct
gcaggctagcacgagacatcggtaaccagacaaatggggagagagatttacccaggcccggggccctcga
tgaggtaaaacccttacttcttgcttgtctgatctcgattatcgaaagtataccgggttagaatggggta
gccgatgggctgcggtgatcacaccgagaggcaagggttctaagctctaggttttatcttgcaatggttc
tatgcgtgtaagttgtgttccggcagatcctctcctagcctttatatagcatgccaggtctcgtgagatc
tgtccagatttgactaaggtacaaagaggaatagtctaaatcttccttctatcatatcttatttccttgt
ccatcaagtatctttccttcggatgagtcttcttccccaacgtcgacgtgcaagcccacttcagatatta
ggcaatcttcatggacccctggttgggccggtggtagccatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ СОРТОВ И ЛИНИЙ СОИ | 2024 |
|
RU2839697C1 |
| Способ генетической паспортизации сортов розы эфиромасличной с использованием ISSR-маркеров | 2023 |
|
RU2802945C1 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| СПОСОБ МОЛЕКУЛЯРНОГО МАРКИРОВАНИЯ, ОСНОВАННЫЙ НА МИКРОСАТЕЛЛИТНЫХ ЛОКУСАХ, ПРЕДНАЗНАЧЕННЫЙ ДЛЯ ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СЕЛЕКЦИОННЫХ ДОСТИЖЕНИЙ РАСТЕНИЙ РОДА RUBUS | 2018 |
|
RU2732922C2 |
| Способ подбора ДНК-маркеров для получения штаммспецифичных олигонуклеотидов к промышленным микроорганизмам и наборы штаммспецифичных олигонуклеотидов, разработанных этим способом для бактерий из видов Agrobacterium radiobacter и Paenibacillus polymyxa | 2023 |
|
RU2822667C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ И СПОСОБ ОБОГАЩЕНИЯ ГЕНОМНОЙ КДНК ВИРУСА КРЫМСКОЙ-КОНГО ГЕМОРРАГИЧЕСКОЙ ЛИХОРАДКИ ГЕНОТИПА ЕВРОПА-1 (V) МЕТОДОМ ПОЛИМЕРАЗНОЙ ЦЕПНОЙ РЕАКЦИИ | 2023 |
|
RU2839313C1 |
| Способ мультиплексной идентификации 32 генетических маркеров льна | 2022 |
|
RU2804939C1 |
| Способ мультиплексной идентификации 52 генетических маркеров хозяйственно ценных признаков льна | 2023 |
|
RU2826718C1 |
| Способ идентификации SNP-генотипов возбудителя чумы средневекового биовара филогенетических ветвей 2.MED1 и 2.MED4 из очагов Северного и Северо-Западного Прикаспия методом секвенирования по Сэнгеру | 2024 |
|
RU2831366C1 |
| СПОСОБ ОПРЕДЕЛЕНИЯ СУБЛИНИЙ ВОЗБУДИТЕЛЯ ТУБЕРКУЛЕЗА ЛИНИИ L2 Beijing НА БИОЛОГИЧЕСКИХ МИКРОЧИПАХ | 2022 |
|
RU2790296C1 |




Изобретение относится к области молекулярной биологии и генетики растений. Описан способ ДНК-идентификации и генетической паспортизации сортов райграса пастбищного и однолетнего на основе систем SSR- и SCoT-маркирования. Способ включает использование мультилокусных SCoT-маркеров, маркирующих ДНК, ассоциированную с кодирующими участками генома, для оценки генетического полиморфизма, детекцию продуктов ПЦР путем предварительного электрофореза в агарозном геле и составление молекулярно-генетических формул по результатам выявленного аллельного разнообразия. При этом выделение ДНК осуществляют модифицированным SDS-методом, в котором в качестве коагулянта белков используют 5 М ацетат аммония, ДНК-экстракцию осуществляют без этапов дополнительной очистки и промывания растворов этанолом, из пророщенных семян, взятых в количестве не менее тридцати от каждого сорта, сортоспецифичные ДНК-спектры получают с использованием двух систем маркирования: 7 микросателлитных SSR-локусов и 9 SCoT-маркеров, выявляют уникальные для образца исследуемой коллекции ДНК-фрагменты, размеры которых верифицируют методом высокоразрешающего капиллярного электрофореза и/или выборочным секвенированием, в паспорт вносят молекулярно-генетическую формулу сорта, а также морфобиологические и хозяйственные характеристики сорта. Способ позволяет осуществлять надежную и достоверную дифференциацию и идентификацию сортов, включенных в Государственный реестр РФ, и может быть использован в селекции, семеноводстве, в системе сортоиспытания и регистрации, в работе контрольно-испытательных лабораторий, в решении правовых вопросов по защите прав интеллектуальной собственности. 7 ил., 4 табл., 6 пр.
Способ ДНК-идентификации и генетической паспортизации сортов райграса пастбищного и однолетнего на основе систем SSR- и SCoT-маркирования, включающий использование мультилокусных SCoT-маркеров, маркирующих ДНК, ассоциированную с кодирующими участками генома, для оценки генетического полиморфизма, детекцию продуктов ПЦР путем предварительного электрофореза в агарозном геле и составление молекулярно-генетических формул по результатам выявленного аллельного разнообразия, при этом выделение ДНК осуществляют модифицированным SDS-методом, в котором в качестве коагулянта белков используют 5 М ацетат аммония, ДНК-экстракцию осуществляют без этапов дополнительной очистки и промывания растворов этанолом, из пророщенных семян, взятых в количестве не менее тридцати от каждого сорта, сортоспецифичные ДНК-спектры получают с использованием двух систем маркирования: 7 микросателлитных SSR-локусов и 9 SCoT-маркеров, выявляют уникальные для образца исследуемой коллекции ДНК-фрагменты, размеры которых верифицируют методом высокоразрешающего капиллярного электрофореза и/или выборочным секвенированием, в паспорт вносят молекулярно-генетическую формулу сорта, а также морфобиологические и хозяйственные характеристики сорта.
| И | |||
| А | |||
| Клименко, Н | |||
| Н | |||
| Козлов, ОЦЕНКА СОРТООБРАЗЦОВ КЛЕВЕРА ЛУГОВОГО НА ОСНОВЕ МИКРОСАТЕЛЛИТНОГО АНАЛИЗА, МНОГОФУНКЦИОНАЛЬНОЕ АДАПТИВНОЕ КОРМОПРОИЗВОДСТВО, Сборник научных трудов, выпуск 19 (67), Москва 2018, стр.29-34 | |||
| Способ генетической паспортизации сортов розы эфиромасличной с использованием ISSR-маркеров | 2023 |
|
RU2802945C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ ПОПУЛЯЦИЙ ДРЕВЕСНЫХ ВИДОВ РАСТЕНИЙ | 2012 |
|
RU2505956C2 |
| Batoul Kobeissi, Abbas Saidi et al., Applicability of SCoT and SSR | |||
Авторы
Даты
2024-09-04—Публикация
2023-09-25—Подача