Область изобретения
Настоящее изобретение относится к семенам культивированного растения салат-латук (Lactuca sativa), самим растениям и частям растений, содержащим один или более фрагментов интрогрессии из растения салат-латук дикого типа, такого как Lactuca virosa, на хромосоме 6 и/или хромосоме 7, причем фрагмент интрогрессии содержит локус количественных признаков (QTL) для резистентности против Nasonovia ribisnigri биотипа 1 (также называемого в настоящей заявке Nr:1 или биотип Nr:1), обозначаемый как QTL6.1 (для QTL на хромосоме 6) и QTL7.1 и 7.2 (для QTL на хромосоме 7). Настоящее изобретение также относится к семенам культивированного растения салат-латук (Lactuca sativa), самим растениям и частям растений, выращенным из этих семян, которые являются резистентными к Nasonovia ribisnigri биотипа 1 благодаря присутствию фрагмента интрогрессии из L. virosa, содержащего QTL6.1 и/или QTL7.1 (и/или QTL7.2), а также к потомству растений и материалу для размножения для получения растений. Настоящее изобретение также относится к источникам растения салат-латук дикого типа для обеспечивающих резистентность QTL для применения при разведении Nr:1 резистентных растений салат-латук.
Уровень техники
Тля растений салат-латук (Nasonovia ribisnigri (Mosley)) является основным вредителем, встречающимся в растениях салат-латук во всем мире. Проблема стала серьезной для получения растений салат-латук в 1970-х годах в Северо-Западной Европе и быстро распространилась по всей Европе. Затем, в 1980-х годах, тля была обнаружена в Канаде. Позднее эта проблема появилась в США (Калифорния и Аризона). Совсем недавно тля растений салат-латук была обнаружена в Новой Зеландии и Австралии.
Тля растений салат-латук может колонизировать растения салат-латук на любой стадии растения и питаться предпочтительно более молодыми листьями. Большое количество тлей на растении способно уменьшить рост растений и деформировать форму головки, так что головки растений салат-латук тогда не могут поступать на рынок. Наличие высоких количеств тлей в головках растений салат-латук является причиной того, что розничные торговцы отказываются покупать растения салат-латук у производителей. На стадии молодого растения можно контролировать растения салат-латук с помощью инсектицида. Сообщалось, что некоторые продукты эффективны для борьбы с популяциями тли. Однако резистентность к химическим веществам была отмечена у некоторых популяций тли. Кроме того, на более ранних этапах развития невозможно бороться с тлей с помощью инсектицидов, поскольку химические продукты не могут поступить в головку растения салат-латук.
С 2007 года в Европе известны два биотипа тли растений салат-латуковой, которые были обозначены биотипом Nr:0 и Nr:1. Полная и частичная резистентность против Nasisovia ribisnigri биотипа Nr:0 были обнаружены у Lactuca virosa, дикого родственника растения салат-латук (Eenink and Dieleman, Euphytica 32(3), 691-695 (1982)). Полная резистентность была обусловлена одним доминантным геном, обозначаемым геном Nr. Ген Nr был перенесен из L. virosa под номером доступа IVT280 в культивированное растение L. sativa и оказался высокоэффективным (Arend et al. 1999, Eucarpia Leafy Vegetables '99. Palacky University, Olomouc, Czech Republic, p149-157).
Однако селекционеры осознали, что выпуск сортов, резистентных к тли растения салат-латук, не был прямым. Ген резистентности Nr был обнаружен тесно связанным с рецессивными генами, которые обладают сильными негативными побочными эффектами. Растения, гомозиготные по гену Nr, показали более низкий рост, более светлый зеленый цвет и ускоренное разложение хлорофилла в более старых листьях. Этот негативный фенотип был также обозначен как фенотип «Компактный рост и быстрое старение» или «фенотип CRA», и было возможным найти рекомбинантные растения салат-латук, в которых ген Nr присутствовал в гомозиготной форме, но в которых фенотип CRA не был выражены (см., например, ЕР 0921720 В1). Эти рекомбинантные растения, в которых имело место событие рекомбинации (т.е. мейотический кроссинговер) между геном Nr и связанными рецессивными генами, служили источником гена резистентности Nr, который не был связан с фенотипом отрицательных побочных эффектов.
Ген Nr резистентности из IVT280 (CGNO4683) широко применяется в коммерческих культиварах растения салат-латук, таких как сорта 'Barcelona', 'Mafalda' (оба Nunhems B.V.) и многие другие.
Другие источники резистентности против биотипа Nr:0 все еще пытаются найти, так как широкомасштабное применение одного гена резистентности сталкивается с угрозой нарушения резистентности. Гены, которые имеют различные механизмы резистентности, могут эффективно применяться в таких обстоятельствах. Например, гены резистентности против нового биотипа Nr:0 были обнаружены в образцах L. serriola PI 491093 и в образце L. virosa PI 274378 (Мс Creight 2008 HortScience 43:1355-1358; McCreight and Liu (2012), HortScience 47(2):179-184). Было обнаружено, резистентность, найденная в PI274378, является полной и аллельной по отношению к гену Nr из IVT280. В PI49093 была обнаружена частичная резистентность, и авторы предложили обозначить аллель этой резистентности Nr0P (в отличие от Nr0C для аллеля полной резистентности, найденного в PI274378). Они предложили применять этот аллель частичной резистентности в областях, где аллель полной резистентность еще не был широко использован, для того чтобы задержать или предотвратить появление биотипов тли, которые преодолевают Nr резистентность.
Также резистентность против Nasonovia ribisnigri биотипа Nr:1 является востребованной. Различные гены резистентности и механизмы резистентности также желательны в отношении Nr:1, чтобы продлить применение генов резистентности. При крупномасштабном использовании одного гена резистентности, обладающего определенным механизмом резистентности, шансы высоки, что резистентность будет преодолена популяцией тли, как это случилось в 2007 году для гена Nr в Европе, когда появилась новая Nasisovia ribisnigri биотипа Nr :1. Таким образом, ситуация на сегодняшний день такова, что тля биотипа Nr:0 все еще может контролироваться одним геном Nr, полученным из IVT L. virosa экземпляра (например в США, где биотип Nr:1 еще не встречается), но что этот ген является неэффективным против тли биотипа Nr: 1, обнаруженной в Европе. Биотип Nr:1 впервые был обнаружен только в Центральной Европе, но теперь распространяется на другие районы, а в 2010 году также был обнаружен на полях в Испании (Cid et al. 2012, Arthropod-Plant Interactions 6: 655-669).
В некоторых публикациях описываются источники, которые, как предполагается, содержат ген резестентности против тли растения салат-латук биотипа Nr:1. Например, три экземпляра (CGN13361, CGN16266, CGN16272) описаны как резистентные против обоих типов тли биотипов Nr:0 и Nr:1 (Anonymous, 4. Nov. 2008, IP.COM document 000176078) и были предложены для применения для разведения растения салат-латук, резистентного против Nr:0 и Nr:1. В этой статье экземпляры L. virosa CGN16272 и CGN16266 указываются для применения в программах обратного скрещивания с культиваром Daguan (Syngenta), и культивар Funly (Syngenta) соответственно (у обоих этих культиваров отсутствуют гены резистентности Nasonovia), и потомство, как указано, проявляет резистентность, подобную резистентности донорских образцов. Также маркеры гена резистентности CGN16272, как указано, разработаны из кроссов CGN16272 с культиваром Cobham Green (Anonymous, 4. Nov. 2008, выше). Эти три экземпляра также были проанализированы в Cid et al. (2012, выше) и, как было обнаружено, имеют высокую резистентность против Nr:0, но только частичную резистентность против биотипа Nr:1.
Cid et al. (2012, выше) также идентифицировали три экземпляра L. virosa с некоторой резистентностью против обоих биотипов Nr:1 и Nr:0, а именно CGN16274, CGN21399 и CGNO5148. В этом исследовании авторы ставили своей целью найти резистентность против обоих биотипов тли растения салат-латук, Nr:0 и Nr:1, в одном экземпляре Lactuca дикого типа. Однако тля биотипа Nr:1 все еще способна питаться и размножать на этих экземплярах дикого типа, хотя и в меньшей степени, чем на восприимчивых контролях (смотрите Фиг. 4).
В WO 2011/058192 раскрывается L. serriola 10G.913571 как резистентный против биотипа Nr:1, Хотя никаких данных для обоснования этого заявления не представлено и не указывается уровень резистентности и методы его определения.
В WO 2012/066008 и WO 2012/065629 также раскрывается Nr:1 резистентность из экземпляра L. serriola для переноса в образец семян, обозначенный 10G.913569. Опять же, никаких данных не представлено и не указан уровень резистентности и методы определения резистентности.
Таким образом, в уровне техники, таком как Anonymous 2008, выше, и Cid et al. 2012, выше, идентифицированы только некоторые экземпляры дикого типа, на которых число тли Nr:1 уменьшилось до некоторой степени, и генетическая основа не раскрыта.
Сохраняется потребность в идентификации генов, которые могут обеспечить резистентность против биотипа Nr:1, чтобы разработать культивированное растение салат-латук, имеющее Nr:1 резистентность. Авторы настоящего изобретения искали образцы, которые считались восприимчивыми к тли биотипа Nr:0, чтобы идентифицировать (новые) гены резистентности против биотипа Nr:1 в этих экземплярах. Кроме того, они также ставили задачу идентификации генов, которые могут обеспечить как резистентность как свободного выбора, так и без выбора. Они обнаружили экземпляр L. virosa (представительный экземпляр семян которого был депонирован под номером NCIMB42086), имеющий высокие уровни резистентности против биотипа Nr:1, как в условиях свободного выбора, так и без выбора, и решили попытаться нанести на генетическую карту резистентность, чтобы идентифицировать как много и какие геномные области L. virosa ответственны за обеспечение Nr:1 резистентности.
При попытке нанести на генетическую карту резистентность, авторы настоящего изобретения столкнулись с серьезными проблемами в создании популяции растений, пригодных для картирования QTL (т.е. картирования популяции, состоящей из особей, подвергшихся хромосомной мейотической рекомбинации между геномами L. sativa и L. virosa). Причиной вероятно является то, что хромосомы L. virosa и L. saliva, которые являются двумя различными видами, весьма различны, что приводит к барьерам скрещивания и бесплодию, а также к потенциальным проблемам в ходе мейоза и кроссовинга. Никаких полезных популяций F2 не могло быть создано, и только после многих скрещиваний с различными рекуррентными родителями авторам настоящего изобретения удалось создать достаточно большие семейства обратных скрещиваний, которые могли бы применяться для нанесения на генетическую карту. Эти популяции для картирования также нелегко анализировать с использованием молекулярных маркеров и фенотипирования, т.е. было довольно удивительно, что авторам настоящего изобретения удалось создать генетическую карту с маркерами SNP, которые являются полиморфными между рекуррентным родителем и экземпляром L. virosa, и также смогли нанести резистентность против Nr:1 Nasonovia на эту карту.
Неожиданно, в исследования первого картирования QTL (применяя популяцию ВС1) не обнаружили один ген, а три геномные области (из которых только две позднее также обнаружили в другой беккросс популяции) на двух различных хромосомах L. Virosa, которые вносят свой вклад в Nr:1 резистентность. Обе инокуляции с контролируемой средой (свободного выбора и без выбора) и данные для поля (также свободного выбора и без выбора) показали высокие уровни Nr:1 резистентности в отношении трех географически различных биотипов Nr:1 (Германия, Франция и Испания). На самом деле, при полевых испытаниях в Испании (Murcia) полувзрослые и взрослые растения экземпляра NCIMB42086 не несли какой-либо тли Nr:1 на своих листьях как в испытаниях свободного выбора, так и в испытаниях без выбора.
В исследовании позднего картирования, два QTL (QTL6.1 и QTL7.1) снова были обнаружены, и область QTL могла быть уменьшена. Это второе исследование по нанесению на генетическую карту не аннулировало результаты первого исследования, и три QTL охватываются настоящим изобретение.
Задачей настоящего изобретения является обеспечение трех QTL (обозначены как QTL6.1, QTL7.1 и/или QTL7.2) из L. Virosa, которые могут применяться для получения культивированных растений салат-латук, имеющих резистентность против биотипа Nr:1.
Задачей настоящего изобретения также является обеспечение культивированных растений салат-латук, содержащих один или два или три QTL (QTL6.1 и/или QTL7.1 и/или QTL7.2), интрогрессированных из растения салат-латук дикого типа, такого как L. virosa, в геном L. sativa, причем интрогрессии обеспечивают резистентность против биотипа Nr:1.
Таким образом, различные культивированные растения салат-латук охватываются согласно настоящему изобретению: а) культивированные растения салат-латук, содержащие только один QTL, обеспечивающий Nr:1 резистентность, выбранный из QTL6.1, QTL7.1 и QTL7.2; b) культивированные растения салат-латук, содержащие два QTL, обеспечивающие Nr:1 резистентность, выбранные из QTL6.1 и QTL7.1 и QTL7.2 (в одном варианте растение, содержащее как QTL6.1, так и QTL7.1, является конкретным вариантом выполнения настоящего изобретения); с) культивированные растения салат-латук, содержащие три QTL, обеспечивающие Nr:1 резистентность, выбранные из QTL6.1, QTL7.1 и QTL7.2.
Задачей настоящего изобретения также является обеспечение культивированных растений салат-латук, содержащих один или два QTL, выбранные из QTL6.1 и QTL7.1, интрогрессированные из растения салат-латук дикого типа, такого как L. virosa, в геном L. sativa genome, причем интрогрессии обеспечивают резистентность против биотипа Nr:1.
В одном варианте QTL являются получаемыми из (являются как в) семян, депонированных под номером доступа NCIMB42086. Фрагмент (фрагменты) интрогрессии, содержащий/содержащие QTL, обнаруживаемый/обнаруживаемые анализом молекулярных маркеров, который обнаруживает по меньшей мере 1, 2, 3, 4, или более маркеров. В другом варианте QTL являются получаемыми из (являются как в) других Nr:1 резистентных экземпляров растения салат-латук дикого типа, особенно экземпляров L. virosa, причем фрагмент (фрагменты интрогрессии является/являются обнаруживаемыми анализом молекулярных маркеров, который обнаруживает по меньшей мере 1, 2, 3, 4, или более (т.е. 5, 6, 7, 8, 9, 10, 11, 12 или более) маркеров, раскрытых в настоящей заявке. В одном варианте экземпляр L. virosa является одним из двух типов экземпляров, и фрагмент интрогрессии содержит специфичный для экземпляра L. virosa SNP маркер (называемый VSP для конкретного Virosa), выбранный из VSP1 и VSP2, оба специфичны для одного экземпляра L. Virosa, и VSP3 и VSP4, оба специфичны для другого экземпляра L. virosa.
Несмотря на проблемы нанесения на генетическую карту межвидового QTL, указанные выше, такие как неплодородие, нарушение сегрегации и т.д., первоначально обнаруженные области QTL (которые были первоначально нанесены на генетическую карту физической области в диапазоне от 60 до 240 Мб на хромосоме 6; и от 170 до 235 Мб на хромосоме 7 для QTL7.1; и от 70 до 150 Мб для QTL 7.2) могли бы быть нанесены на генетическую карту области, в диапазоне от 77 Мб до 161 Мб на хромосоме 6 (содержащей QTL6.1) и от 203 Мб до 219 Мб на хромосоме 7, содержащей QTL7.1.
Таким образом, в одном варианте обеспечивается культивированное растение Lactuca sativa, содержащее фрагмент интрогрессии на хромосоме 6 (содержащей QTL6.1) и/или на хромосоме 7 (содержащей QTL7.1), причем каждый в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1). В одном варианте фрагмент интрогрессии содержит всю или часть области, начинающейся в месте 77 Мб на хромосоме 6 и заканчивающейся в месте 161 Мб на хромосоме 6, и/или фрагмент интрогрессии содержит всю или часть области, начинающейся в месте 203 Мб на хромосоме 7 и заканчивающейся в месте 219 Мб на хромосоме 7. Смотрите например, Фиг. 3В, где показаны хромосомы 6 и 7 L. sativa, где серые полосы иллюстрируют фрагменты интрогрессии из Nr:1 резистентного экземпляра дикого типа, как например экземпляр L. virosa (например, NCIMB42086), содержащие обеспечивающие резистентность QTL QTL6.1 и QTL7.1, или их варианты. В одном варианте фрагмент интрогрессии, содержащий QTL7.2, может также необязательно присутствовать в культивированном растении салат-латук.
Также понятно, что более маленький фрагмент интрогрессии (т.е. содержащий часть, придающую резистентность, вышеупомянутой области в диапазоне от 77 Мб до 161 Мб хромосомы 6), который сохраняет QTL6.1 (или вариант) может быть фрагментом, имеющим размер, равный 80 Мб, 70 Мб, 60 Мб, 50 Мб, 40 Мб, 30 Мб, 20 Мб, 10 Мб, 5 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 100 тысяч пар оснований, 50 тысяч пар оснований или менее, и содержит QTL6.1 или его вариант. В одном варианте часть составляет по меньшей мере 5 тысяч пар оснований, 10 тысяч пар оснований, 20 тысяч пар оснований по размеру, или более.
Также понятно, что более маленький фрагмент интрогрессии (т.е. содержащий часть, придающую резистентность, вышеупомянутой области в диапазоне от 203 Мб до 219 Мб хромосомы 7), который сохраняет QTL7.1 (или вариант), может быть фрагментом, имеющим размер, равный 15 Мб, 10 Мб, 5 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 100 тысяч пар оснований, 50 тысяч пар оснований или менее, и содержит QTL7.1 или его вариант. В одном варианте часть составляет по меньшей мере 5 тысяч пар оснований, 10 тысяч пар оснований, 20 тысяч пар оснований по размеру, или более.
В одном варианте фрагмент интрогрессии на хромосоме 6 является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, предпочтительно по меньшей мере 2 или 3 или 4 или 5 (или более) маркеров, выбранных из группы, состоящей из:
а) Генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 24);
генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP1.23 и SNP_03 (например, между SNP1.23 и SNP2.24, SNP1.23 и SNP_02); или между SNP_02 и SNP_03 (например, между SNP_02 и SNP2.24); или между SNP2.24 и SNP_03;
e) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный в пределах расстояния, равного 10 Мб, предпочтительно в пределах 5 Мб, любого маркера, выбранного из SNP1.23, SNP_02, SNP2.24, или SNP_03.
Необязательно, в одном варианте фрагмент интрогрессии содержит (и обнаруживается с его помощью) маркер, специфичный для экземпляра L. virosa, выбранный из генотипа GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 26) и генотипа АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 27). Применяя SNP маркеры VSP1 и VSP3, фрагменты интрогрессии, содержащие QTL6.1, из экземпляров двух различных типов L. virosa, можно отличить.
В другом варианте, фрагмент интрогрессии на дальнем конце хромосомы 7, содержащей QTL 7.1 (в физическом положении между 203 Мб и 219 Мб хромосомы 7), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, предпочтительно по меньшей мере 2 или 3 или 4 или 5 (или более) маркеров, выбранных из группы, состоящей из:
a. генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
b. генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP 17.25 в SEQ ID NO: 25 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 25);
c. генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
d. генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
e. любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_17 и SNP_19 (например, между SNP_17 и SNP_18, между SNP_17 и SNP17.25; или между SNP17.25 и SNP_19, или между SNP17.25 и SNP_18, или между SNP_18 и SNP_19);
f. любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный в пределах расстояния, равного 12 Мб, 10 Мб, предпочтительно в пределах 5 Мб, любого маркера, выбранного из SNP_17, SNP_17.25, SNP_18 и SNP_19.
Необязательно, в одном варианте фрагмент интрогрессии содержит (и обнаруживается с его помощью) маркер, специфичный для экземпляра L. virosa, выбранный из генотипа СС или АС для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 28) и генотипа GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 29). Применяя SNP маркеры VSP2 и VSP4 фрагменты интрогрессии, содержащие QTL7.1, из экземпляров двух различных типов L. virosa, можно отличить.
При упоминании, что фрагмент интрогрессии является "обнаруживаемым анализом молекулярных маркеров, который обнаруживает" один или более маркеров, это означает, что фрагмент интрогрессии содержит генотип резистентности этого маркера.
В другом варианте обеспечиваются семена, растения и части растения или культивированное растение салат-латук, содержащие фрагмент интрогрессии из растения салат-латук дикого типа, как например из L. virosa, содержащий QTL6.1 и/или QTL7.1 (и необязательно QTL7.2), причем фрагмент интрогрессии обеспечивает резистентность против Nasonovia ribisnigri биотипа Nr:1. В одном варианте фрагментом интрогрессии является фрагмент из L. virosa, особенно из экземпляров L. Virosa, имеющих Nr:1 резистентность как в тесте свободного выбора, так и в испытании без выбора, как описано в настоящей заявке. В одном варианте фрагментом интрогрессии является фрагмент из экземпляра NCIMB42086, или он является получаемым из экземпляра NCIMB42086, или его потомства или потомков.
В другом варианте фрагмент интрогрессии из экземпляра L. virosa, который содержит следующие специфичные для экземпляра L. virosa SNP маркеры: генотип GG (гомозиготный) или генотип GT (гетерозиготный) при нуклеотиде 71 последовательности SEQ ID NO: 26 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 26), называемый VSP1 маркер; и генотип СС (гомозиготный) или генотип АС (гетерозиготный) при нуклеотиде 71 последовательности SEQ ID NO: 28 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 28), называемый VSP2. VSP1 и VSP2 обнаруживаются в других Nr:1 резистентных экземплярах, таких как NCIMB42086.
В другом варианте фрагментом интрогрессии является фрагмент из экземпляра L. virosa, который содержит следующие специфичные для экземпляра L. virosa SNP маркеры: генотип АА (гомозиготный) или генотип АС (гетерозиготный) при нуклеотиде 71 последовательности SEQ ID NO: 27 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 27), называемый VSP3 маркер; и генотип GG (гомозиготный) или генотип AG (гетерозиготный) при нуклеотиде 71 последовательности SEQ ID NO: 29 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 29), называемый VSP4. VSP3 и VSP4 обнаруживаются в других Nr:1 резистентных экземплярах.
Также обеспечиваются способы получения и/или идентификации и/или отбора культивированных растений салат-латук, содержащих интрогрессию из растения салат-латук дикого типа, такого как L. virosa, на хромосоме 6 (содержащей QTL6.1) и/или хромосоме 7 (содержащей QTL 7.1 и/или QTL7.2), как и способы переноса QTL в различные линии или сорта культивированных растений, особенно в Nr:1 восприимчивые линии или сорта растений салат-латук.
Общие определения
Форма единственного числа не исключает возможность, что присутствует более чем один элемент, если исходя из контекста не следует, что присутствует один и только один из элементов. Форма единственного числа, таким образом, как правило, означает "по меньшей мере один".
«Сорт растения» представляет собой группу растений в пределах одного и того же ботанического таксона наименьшей известной степени, которая (независимо от того, выполнены ли условия для признания прав селекционера растений или нет) может быть определена на основе выражения признаков, которую в результате определенного генотипа или комбинации генотипов можно отличить от любой другой группы растений посредством выражения по меньшей мере одного из этих признаков, и можно рассматривать как единое целое, потому что можно размножать без какого-либо изменения. Поэтому термин «сорт растения» не может применяться для обозначения группы растений, даже если они одного вида, если все они характеризуются наличием одного или двух локусов или генов (или фенотипических характеристик, обусловленных этими конкретными локусами или генами), но которые могут сильно отличаться друг от друга в отношении других локусов или генов в геноме.
"Салат-латук" или "культивированное растение салат-латук" или "культивированное Lactuca sativa" относятся в контексте настоящего изобретения к растениям вида Lactuca sativa L. (или семенам, из которых растения могут быть выращены), и частям таких растений, выращенным людьми для питания и имеющим хорошие агрономические характеристики. Это включает любое культивированное растение салат-латук, такое как линии разведения (например, беккросс линии, инбредные линии), культивары и сорта любого типа. В общем выделяют типы с головками и без головок растений салат-латук. Типы с головками включают, например, типы салат Crisphead, салат кочанный и салат-ромен (салат кос), тогда как типы без головок включают листовые типы. Культивированные растения салат-латук не являются растениями "салат-латук дикого типа" или растениями "Lactuca дикого типа", т.е. растениями, которые, в общем, имеют намного более плохие урожаи и более плохие агрономические характеристики, чем культивированные растения, и, например, растут естественным образом в диких популяциях.
Экземпляры "растения салат-латук дикого типа" или "Lactuca дикого типа" относятся к растениям видов, отличных от культивированного Lactuca sativa, как например Lactuca virosa, Lactuca serriola, Lactuca saligna, Lactuca perennis, и другие. Предпочтительно, такое растение салат-латук дикого типа содержит или состоит из видов Lactuca, которые являются взаимообогащенными с L. sativa, необязательно с помощью методики спасения эмбрионов (смотрите Maisonneuve 1987, Agronomique 7: 313-319 и Maisonneuve et al. 1995, Euphytica 85:281-285) и/или методики удвоения хромосом (Thompson and Ryder 1961, US Dept Agric Tech Bul. 1224), или способов, посредством которых гены могут быть перенесены в L. sativa посредством видов-посредников, таких как L. serriola (Eenink et al. 1982, выше).
Как применяется в настоящей заявке, термин "растение" включает семя (из которого растение может быть выращено), все растение или любые его части, такие как растительные органы (например, собранные или не собранные листья и т.д.), растительные клетки, протопласты растений, растительные клеточные или тканевые культуры, каллус растений, скопления растительных клеток, трансплантаты растений, сеянцы, растительные клетки, которые являются интактными в растениях, клоны растений или микроразмножения, или части растений (например, собранные ткани или органы), такие как растительные черенки, вегетативные размножения, зародыши, пыльца, семяпочки, цветы, листья, головки, семена (полученные из растений после самооплодотворения или перекрестного оплодотворения), клонально размноженные растения, корни, стволы, стебли, кончики корней, черенки, части любого из них и тому подобное, или их производные, предпочтительно имеющие такое же генетическое строение (или очень подобное генетическое строение) как растение, из которого их получают. Также включена любая стадия развития, такая как саженцы, черенки до или после укоренения, зрелые и/или незрелые растения или зрелые и/или незрелые листья. Когда упоминаются «семена растения», они либо относятся к семенам, из которых растение может быть выращено, или к семенам, полученным из растения после самооплодотворения или перекрестного оплодотворения.
"Соматические клетки" и "репродуктивные клетки" можно различать, при этом соматическими клетками являются клетки, отличные от гамет (например, семяпочки и пыльца), зародышевых половых клеток и гаметоцитов. Гаметы, зародышевые клетки и гаметоциты являются "репродуктивными клетками".
"Тканевая культура" или "клеточная культура" относится к in vitro композиции, содержащей выделенные клетки одного того же или различного типа или набор таких клеток, организованный в растительную ткань. Тканевые культуры и клеточные культуры растения салат-латук и регенерация растений салат-латук из них хорошо известна и опубликована для общего доступа (смотрите, например, Teng et al., HortScience. 1992, 27(9): 1030-1032 Teng et al., HortScience. 1993, 28(6): 669-1671, Zhang et al., Journal of Genetics and Breeding. 1992, 46(3): 287-290).
"Собранный растительный материал" относится согласно настоящему изобретению к частям растений (например, листья, части листьев или головки, отделенные от всего растения), которые были собраны для дальнейшего хранения и/или дальнейшего применения.
"Собранные семена" относится к семенам, собранным с лини или сорта, например, полученным после самооплодотворения или перекрестного оплодотворения и собранным.
"Собранные листья" или "собранные головки", как применяется в настоящей заявке, относится к листьям растения салат-латук или частям листьев или головкам, т.е. растению без корневой системы, например, по существу ко всем (собранным) листьям. Листья могут быть целыми или порезанными на части.
"Потомство" или "потомства" или "потомки", как применяется в настоящей заявке, относится к потомству или первому и всем последующим потомкам, полученным из (произведенным из) (получаемым из или производимым из) растения согласно настоящему изобретению, которое содержит (сохраняет) один или более обеспечивающие Nr:1 резистентность QTL в гомозиготной или гетерозиготной форме и/или фенотип Nr:1 резистентности, как описано в настоящей заявке. Потомство может быть получено путем регенерации клеточной культуры или тканевой культуры, или частей растения, или самоопыления растения, или путем получения семян растения. В других вариантах выполнения настоящего изобретения, потомство может также охватывать растения салат-латук, полученные в результате скрещивания по меньшей мере одного растения салат-латук с другим растением салат-латук того же или другого сорта или линии (разведения), или с другими растениями Lactuca дикого типа, обратного скрещивания, вставки локуса в растение или мутации. Потомством является, например, потомство первого поколения, т.е. потомство, непосредственно полученное из, полученное из, получаемое из или произведенное из родительского растения, посредством, например, традиционных методов разведения (самоопыление и/или скрещивание) или регенерации или трансформации. Однако термин "потомство", в общем, охватывает другие поколения, такие как второе, третье, четвертое, пятое, шестое, седьмое или более поколения, т.е. поколения растений, которые производят из, получают из, являются получаемыми из или являются производимыми из предыдущего поколения, посредством, например, традиционных методов разведения, регенерации или методов генетической трансформации. Например, поколение второй генерации может быть получено из поколения первой генерации посредством любого из способов, указанных выше. Также двойные гаплоидные растения являются потомством.
Термин "линия растения" или "линия скрещивания" относится к растению и его потомству, являющемуся весьма однородным по фенотипу растения. Как применяется в настоящей заявке, термин "инбредная линия" относится к линии растений, которая неоднократно самоопылялась и является почти гомозиготной для всех аллелей. Таким образом, термин "инбредная линия" или "родительская линия" относится к растению, которое подвергалось нескольким генерациям (например, по меньшей мере 4, 5, 6, 7 или более) инбридинга, приводя к линии растения с высокой однородностью.
"F1, F2, F3, и т.д." относится к последовательным родственным поколениям после кросса между двумя родительскими растениями или родительскими линиями. Растения, выращенные из семян, полученных скрещиванием двух растений или линий, называют поколением F1. Самоопыление растений F1 приводит к поколению F2 и т.д.
"Гибрид" относится к семенам, собранным в результате скрещивания одной линии или сорта растений с другой линией или сортом растений, и растениям или частям растений, выращенным из этих семян.
Растение "F1 гибрид" (или F1 гибридное семя) представляет собой поколение, полученное скрещиванием двух неизогенных инбредных родительских линий. Таким образом, F1 гибридные семена представляют собой семена, из которых вырастают F1 гибридные растения.
"Межвидовый гибрид" относится к гибриду, полученному в результате скрещивания одного вида, например, L. sativa, с растением другого вида, например, L. virosa.
"Скрещивание" относится к скрещиванию двух исходных растений. В равной степени "Перекрестное опыление" означает оплодотворение путем объединения двух гамет из разных растений.
"Самоопыление" относится к самооплодотворению растения, т.е. к союзу гамет из одного и того же растения.
"Обратное скрещивание" относится к методу селекции, с помощью которого признак, такой как один или более Nr:1 резистентность обеспечивающие QTL, может быть перенесен из более низшего генетического фона (например, растение салат-латук дикого типа; также обозначается как "донор") в высший генетический фон (также обозначается как "рекуррентный родитель"), например культивированное растение салат-латук. Потомство кросса (например, растение F1, полученное скрещиванием дикого Nr:1-резистентного растения салат-латук с культивированным Nr:1-восприимчивым растением салат-латук или растение F2 или растение F3, и т.д., полученное самоопылением F1) является "обратно скрещенным" до родителя с высшим генетическим фоном, например культивированного Nr:1-восприимчивого родителя. После повторного обратного скрещивания, признак низшего генетического фона будет включен в высший генетический фон. Термины "трансформированный ген" или "трансформированное растение" или "единичная (или двойная или тройная) трансформация локусом " в этом контексте относится к растениям, которые разрабатывают путем обратного скрещивания, в котором по существу все желательные морфологические и/или физиологические характеристики рекуррентного родителя воспроизводятся в дополнение к одному или более QTL (например, Nr:1 резистентность обеспечивающий QTL6.1, QTL7.1 и/или QTL7.2), перенесенным из родителя-донора.
Термин "традиционные методы разведения" согласно настоящему изобретению охватывает скрещивание, обратное скрещивание, самоопыление, селекцию, удвоение хромосом, получение двойного галоида, эмбриональное спасение, применение вида посредника, слияние протопластов, выбор с помощью маркера, мутационную селекцию и т.д. как известно селекционеру (т.е. методы, отличные от методов генетической модификации/трансформации/трансгенных методов), посредством которых, например, один или более обеспечивающих Nr:1-резистентность QTL, обозначаемые в настоящей заявке как QTL6.1, QTL7.1 и/или QTL7.2, могут быть получены, идентифицированы, отобраны и/или перенесены.
"Регенерация" относится к развитию растения из in vitro клеточной культуры или тканевой культуры или вегетативному размножению.
"Вегетативное размножение", "вегетативная репродукция" или "клональное размножение" применяются в настоящей заявке взаимозаменяемо и означают способ, в котором берут часть растения и позволяют части растения образовать по меньшей мере корни, где часть растения, например, определяется как или происходит из (например, путем разрезания) листа, пыльцы, зародыша, семядолей, гипокотиля, клеток, протопласт, меристематической клетки, корня, кончика корня, пестика, пыльника, цветка, кончика побега, побега, стебля, плода и черешка. Когда целое растение регенерируется путем вегетативного размножения, это также обозначается как "вегетативное размножение" или "вегетативно размножающееся растение".
"Растение с единично (или дважды или трижды) трансформированным локусом" относится к растениям, которые разрабатывают методами селекции растений, содержащими или состоящими из обратного скрещивания, где практически все желаемые морфологические и/или физиологические характеристики растения салат-латук восстанавливаются в дополнение к характеристикам одного локуса (или двух или трех локусов), перенесенного в растение посредством, например, методики обратного скрещивания.
"Трансген" или "химерный ген" относится к генетическому локусу, содержащему последовательность ДНК, который был введен в геном растения салат-латук путем трансформации. Растение, содержащее трансген, стабильно интегрированный в его геном, обозначается как "трансгенное растение".
«По существу эквивалентный» или «существенно не отличающийся» или «не статистически значимо отличающийся» относится к признаку, который при сравнении, например, между двумя линиями или сортами растений, является эквивалентным или почти идентичным. Другими словами, признак, являющийся «по существу эквивалентным» между двумя линиями или сортами растений, означает, что среднее значение для указанного признака отличается менее чем на 10% (например, 9, 8, 7, 6, 5, 4, 3, 2, 1% или менее), и статистическая значимость этого различия не имеет р≥0,05, применяя ANOVA.
Термин "среднее значение" относится согласно настоящему изобретению к среднему арифметическому значению. Термин "среднее" относится к среднему арифметическому значению нескольких измерений. Специалисту в данной области техники понятно, что фенотип линии растения или сорта зависит в некоторой степени от условий произрастания, и поэтому измеряются средние арифметические значения для по меньшей мере 10, 15, 20, 30, 40, 50 или более растений, предпочтительно в рандомизированных экспериментальных моделях с несколькими повторениями и подходящими контрольными растениями, выращенными при тех же условиях в том же эксперименте.
"Статистически значимый" или "статистически значимо" отличный или "значительно" отличный относится к характеристике линии растения или сорта, такой как Nr:1 резистентность, которые, при сравнении с подходящим контролем (например, согласно настоящему изобретению генетической контрольной линией, не содержащей QTL, или Nr:1 восприимчивым контрольным сортом, таким как Mafalda) показывают статистически значимое отличие их характеристик (например, р-значение составляет менее 0.05, р<0.05, применяя ANOVA) от (среднего значения) контроля. Так, например, линия или сорт или генотип растении, которые имеют в среднем «статистически значительно меньшее количество» тлей или «значительно меньше» тлей, чем контроль, представляет собой растение, у которого разница в среднем числе тлей статистически значима по сравнению с контрольным растением.
"Тля растения салат-латук" относится к тле вида Nasonovia ribisnigri.
"Биотип Nr:0" относится к биотипу тли растения салат-латук, против которого ген Nr из IVT280 обеспечивает резистентность, т.е. тля растений салат-латук этого биотипа не способна питаться и размножаться на сортах, содержащих ген Nr, такой как Mafalda (Nunhems), Barcelona (Nunhems), или другие.
"Биотип Nr:1" относится к биотипу тли растения салат-латук, против которого ген Nr из IVT280 не обеспечивает резистентность. Таким образом, тля растений салат-латук этого биотипа может питаться и размножаться на сортах и линиях, содержащих ген Nr, такой как Mafalda (Nunhems), Barcelona (Nunhems), или другие.
При ссылке на тесты на резистентность, проводимые в контролируемых условиях окружающей среды (например, в климатических ячейках), предпочтительно делается ссылка на клональные колонии единичных, выбранных тлей биотипа Nr:0 или Nr:1. Это также обозначается в настоящей заявке как "изоляты" тли.
Линия, сорт или экземпляры дикого или культивированного растения салат-латук указывается как "Nr:0 резистентное растение или "растение, резистентное против биотипа Nr:0", или растение, имеющее "Nr:0 резистентность", или "фенотип Nr:0 резистентности", если размножение (среднее число тлей на растение, высчитываемое периодически, например, через около 7, 14, 21, 28, 35 и/или более дней после заражения тлей биотипа Nr:0) биотипа Nr:0 является статистически значительно более низким по сравнению с контрольными растениями, не содержащими гены резистентности против тли растения салат-латук, такими как восприимчивые сорта Salinas (синоним Saladin; первоначально разработанный селекционером Ryder E.J., USDA, ARS, California, USA), или другие. Значительное уменьшение размножения биотипа Nr:0 может быть определено, применяя, например, испытание в теплице или закрытый полевой тест, как известно в данной области техники, например, как описано в McCreight and Liu, 2012, HortScience 47(2), в Materials and Methods, или в других источниках. Испытание в теплице или закрытый полевой тест могут быть с выбором (тля способна питаться и размножаться на нескольких линиях или сортах растений) и/или без выбора (тля способна питаться и размножаться только на одной линии или сорте растений). Альтернативно могут проводиться тесты в открытом поле, причем естественное заражение восприимчивых контролей, таких как сорт Salinas (синоним Saladin), контролируется, и когда зарожение контроля становится многочисленным, число тли подсчитывают на тестовых растениях и контролях. Термин охватывает как "частичную Nr:0 резистентность" (как в PI491093, описанном в McCreight and Liu, 2012), так и "полную Nr:0 резистентность" (как в IVT280, смотрите тот же McCreight and Liu 2012). На полностью Nr:0 резистентных растениях практически отсутствует тля биотипа Nr:0, которая питается и размножается на нем, при измерении в еженедельные моменты времени после инокуляции или после заражения.
Линия, сорт или экземпляры дикого или культивированного растения салат-латук указывается как "Nr:1 резистентное растение", или "растение, резистентное против биотипа Nr:1", или растение, имеющее "Nr:1 резистентность", или "фенотип Nr:1 резистентности", или "растение, имеющее значительно уменьшенную восприимчивость", или с "значительно усиленной резистентностью", если среднее число тлей биотипа Nr:1 на растение, вычисленное в один или более моментов времени после заражения тлей биотипа Nr:1 (например, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или более недель после переноса саженцев в поле; и/или после того, как растения достигли стадии 3-4 настоящих листьев и после этого т.е. когда растения достигли 30%, 40%, 50%, 60%, 70%, 80%, 90% или 100% от конечного размера взрослого растения) является статистически значительно уменьшенным по сравнению с контрольными растениями, не содержащими гены резистентности против тли растения салат-латук (как например сорт Salinas (синоним Saladin)) и/или по сравнению с контрольными растениями, имеющими ген Nr резистентности, как например сорт Mafalda (Nunhems) и/или генетическим контролем, не содержащим фрагмент (фрагменты) интрогрессии, но который в остальном является генетически идентичным или генетически весьма подобным растению, содержащему фрагмент (фрагменты) интрогрессии. В одном варианте значительное уменьшение относится к среднему числу тлей на Nr:1 резистентное растение, составляющему самое большое 50%, 49%, 48%, 47%, 45%, предпочтительно самое большое 40%, предпочтительно самое большое 30%, 20% или 10%, более предпочтительно самое большое 5%, 3%, 2%, или 1% от среднего числа тлей, обнаруженных на Nr:1 восприимчивом сорте, как например сорт Mafalda (или другие Nr:1 восприимчивые сорта, содержащие ген Nr), или на генетическом контроле, при выращивании при одних и тех же условиях. В одном варианте выполнения настоящего изобретения, растения являются свободными или почти свободными (менее чем в среднем 10, 9, 8, 7, 6, 5, 4, 3 тлей на сорт или линию растения) от тли Nr:1. Nr:1 резистентность может быть определена в ходе анализов на Nr:1 резистентность, как определено и описано в части «Примеры».
"Анализ Nr:1-резистентности" «Анализ Nr:1-резистентности» может быть либо анализом без выбора (тли могут питаться и размножаться на одной линии или сорте растения) и/или анализом (свободного) выбора, как например, описанный в Примерах. Анализы с выбором или без выбора могут проводиться в контролируемых средах, таких как климатические ячейки или в поле (открытое поле для анализа с выбором или закрытое поле для анализов без выбора). Анализы с выбором на резистентность относятся к анализам, где тли могут выбирать среди разных генотипов растений для кормления и размножения. Как правило, анализы с выбором применяются для идентификации антиксенозной (без предпочтения) резистентности, т.е. резистентности, вызванной факторами, которые делают генотип растения менее привлекательным. Анализы без выбора на резистентность относятся к анализам, где тли не могут выбирать среди разных генотипов растений для кормления и размножения, но им разрешено питаться и размножаться только на одном генотипе. Это позволяет обнаружить антибиоз. На Nr:1 восприимчивых контрольных растениях, таких как Mafalda, тля способна размножаться до более чем около 50, 100, 150, 200, 250, 300 или более тлей. Резистентность, эффективная в условиях отсутствия выбора, действует на самих насекомых, например, они умирают, производят меньше потомства или растут более медленно.
Генетический элемент, фрагмент интрогрессии или ген или аллель, сообщающий признак (такой как один или более QTL, обеспечивающих резистентность против N. ribisnigri билтипа Nr:1) "получаемый из" или может быть "получен из" или "происходящий из" или может "происходить из" или "как присутствует в" или "как обнаружено в" растении или семени, если он может быть перенесен из растения или семени, в котором он присутствует, в другое растение или семя, в котором он не присутствует (как например линия или сорт), применяя традиционные методы разведения, не приводящие к фенотипическому изменению растения-реципиента, за исключением добавления признака (Nr:1 резистентность), сообщаемого генетическим элементов, локусом, фрагментом интрогрессии, геном или аллелем. Термины используются взаимозаменяемо, и генетический элемент, локус, фрагмент интрогрессии, ген или аллель могут, таким образом, быть перенесены в любой другой генетический фон, лишенный этого признака. Могут применяться не только семена, депонированные и содержащие генетический элемент, локус, фрагмент интрогрессии, ген или аллель, но также потомство/потомки таких семян, которые были отобраны для сохранения генетического элемента, локуса, фрагмента интрогрессии, гена или аллеля, могут применяться и охватываются настоящим изобретением, как например коммерческие сорта, разработанные из депонированных семян или из их потомства. Содержит ли растение (ли геномная ДНК, клетка или ткань растения) тот же самый генетический элемент, локус, фрагмент интрогрессии, ген или аллель, как получено из депонированных семян, может быть определено специалистом в данной области техники, применяя одну или более методик, известных в данной области, таких как фенотипические анализы, секвенирование всего генома, анализ молекулярного маркера, нанесение признаков на генетическую карту, роспись хромосомы, тесты аллеморфизма и тому подобное, или комбинации методик.
Термин "аллель (аллели)" означает любую одну или более альтернативные формы гена в конкретном локусе, все аллели которого относятся к одному признаку или характеристике в конкретном локусе. В диплоидной клетке организма аллели данного гена расположены в определенном месте или локусе (множество локусов) на хромосоме. Один аллель присутствует на каждой хромосоме пары гомологичных хромосом. Диплоидные виды растений могут содержать большое количество различных аллелей в определенном локусе. Это могут быть одинаковые аллели гена (гомозиготные) или два разных аллеля (гетерозиготные)
Термин «ген» означает последовательность (геномную) ДНК, содержащую область (транскрибируемый участок), которая транскрибируется в молекулу информационной РНК (мРНК) в клетке и функционально связанную регуляторную область (например, промотор). Различные аллели гена представляют собой, таким образом, различную альтернативную форму гена, которая может быть в виде, например, различий в один или более нуклеотидов в геномной последовательности ДНК (например, в промоторной последовательности, экзонных последовательностях, интронных последовательностях и т.д.), мРНК и/или аминокислотной последовательности кодируемого белка.
"Тест на аллелизм" относится к генетическому тесту, посредством которого можно протестировать могут ли два фенотипа, например, два Nr:1 резистентности, встречающиеся в двух линиях или сортах растений, определяться одним и тем же геном или двумя различными генами. Например, тестируемые растения скрещиваются друг с другом, F1 самоопыляется, и определяется сегрегация фенотипов среди потомства F2. Отношение сегрегации указывает, являются ли гены аллельными.
Термин "локус" (множество локусов) означает определенное место или места или участок на хромосоме, где обнаруживается, например, ген или генетический маркер. Локус Nr:1 резистентности (или локус/локусы, обеспечивающие резистентность) является, таким образом, местом в геноме растения салат-латук дикого типа, особенно в экземпляре Lactuca virosa, таком как (но без ограничения к этому) NCIMB 42086, где обеспечивающий (обеспечивающие) Nr:1-резистентность QTL обнаруживается/обнаруживаются на хромосоме 6 (QTL6.1) и/или на хромосоме 7 (QTL7.1 и/или QTL7.2). В культивированном растении салат-латук согласно настоящему изобретению один или более QTL, обеспечивающих Nr:1 резистентность, интрогрессируются из экземпляра растения салат-латук дикого типа, которое содержит один или более из QTL, как например экземпляр L. virosa дикого типа, депорнированный под номером NCIMB 42086.
Термин "локус количественных признаков" или "QTL" представляет собой хромосомный локус, который кодирует один или более аллелей, которые влияют на экспрессивность непрерывно распределенного (количественного) фенотипа. Локусы количественных признаков, обеспечивающие резистентность называют в настоящей заявке QTL6.1, QTL7.1 и QTL7.2.
"Геном растения салат-латук" и "физическое положение в геноме растения салат-латук " и на "хромосоме 6" и/или на " хромосоме 7" относится к физическому геному культивированного растения салат-латук, смотрите сайт lgr.genomecenter.ucdavis.edu (база данных по растениям салат-латук version 3.2, содержащих хромосому 6, как обозначено Lsat_1_v4_lg6, и хромосому 7, как обозначено Lsat_1_v4_lg7), и физическим хромосомам и физическому положению на хромосомах. Поэтому, например, SNP_01 локализуется при нуклеотиде (или 'основании'), расположенном физически при нуклеотиде 60,688,939 хромосомы 6, которая имеет физический размер от 0 до 244.7 Мб. Подобным образом, SNP_08 локализуется при нуклеотиде (или 'основании'), расположенном в месте 72,772,104 хромосомы 7, где хромосома имеет физический размер от 0 до 242.9 Мб.
«Физическое расстояние» между локусами (например, между молекулярными маркерами и/или между фенотипическими маркерами) на одной и той же хромосоме представляет собой фактически физическое расстояние, выраженное в основаниях или парах оснований (п.о.), килооснованиях или парах килооснований (тысяча пар оснований) или мегабазах или парах мегабаз (Мб).
«Генетическое расстояние» между локусами (например, между молекулярными маркерами и/или между фенотипическими маркерами) на одной и той же хромосоме измеряется частотой кроссинговера или частотой рекомбинации (RF) и указывается в сантиморганах (сМ). Одному сМ соответствует частота рекомбинации 1%. Если никакие рекомбинанты не могут быть найдены, RF равна нулю, и локусы либо очень близки друг к другу физически, либо идентичны. Чем дальше расположены два локуса, тем выше RF.
"Фрагмент интрогрессии" или "сегмент интрогрессия" или "область интрогрессия" относится к фрагменту хромосомы (или части или области хромосомы), который был введен в другое растение того же или родственного вида путем скрещивания или традиционных методов разведения, таких как обратное скрещивание, т.е. интрогрессированный фрагмент является результатом методов размножения, ссылающихся на глагол «интрогрессировать» (например, обратное скрещивание). В отношении растений салат-латук, экземпляры растения салат-латук дикого типа могут применяться для интрогрессии фрагментов дикого генома (например, L. virosa) в геном культивированного растения салат-латук, L. sativa. Такое культивированное растение салат-латук, таким образом, имеет "геном культивированного растения L. sativa", но содержит в геноме фрагмент (или два или три фрагмента) растения салат-латук дикого типа, например, фрагмент интрогрессии (или два или три) генома родственника дикого типа Lactuca, такого как L. virosa. Понятно, что термин "фрагмент интрогрессии" не включает хромосому целиком, а только часть хромосомы. Фрагменты интрогрессии могут быть большими, например, составлять даже половину хромосомы, но предпочтительно он меньше, как например, около 80 Мб, 74 Мб, 73 Мб, 70 Мб, 50 Мб, 30 Мб, 20 Мб, 15 Мб или менее, как например, около 10 Мб или менее, около 9 Мб или менее, около 8 Мб или менее, около 7 Мб или менее, около 6 Мб или менее, около 5 Мб или менее, около 4 Мб или менее, около 3 Мб или менее, около 2 Мб или менее, около 1 Мб (равно 1,000,000 пар оснований) или менее, или около 0.5 Мб (равно 500,000 пар оснований) или менее, как например около 200,000 пар оснований (равно 200 пар килооснований) или менее, около 100,000 пар оснований (100 тысяч пар оснований) или менее, около 50,000 пар оснований (50 тысяч пар оснований) или менее, около 25,000 пар оснований (25 тысяч пар оснований) или менее.
"Однородность" или "однородный" относится к генетическим и фенотипическим характеристикам линии или сорта растений. Инбредные линии являются генетически весьма однородными, поскольку их получают путем нескольких поколений инбридинга.
Термин "аллель Nr:1" или "аллель Nr:1 резистентности" относится к аллелю, обнаруженному в локусе QTL6.1 или QTL7.1 или QTL7.2, который в одном варианте выполнения настоящего изобретения является интрогрессированным в культивированное растение салат-латук (на хромосому 6 и/или 7 культивированного L. saliva) из растения салат-латук дикого типа, особенно из экземпляра L. virosa. Термин "Nr:1-аллель", таким образом, также охватывает Nr:1-аллели, получаемые из различных экземпляров растения салат-латук дикого типа. Когда один или два №:1-аллеля присутствуют на конкретном локусе в геноме (т.е. в гетерозиготной или гомозиготной форме, соответственно), линия или сорт растения имеют значительно усиленную Nr:1 резистентность по сравнению с генетическим контролем, не содержащим QTL. В культивированном растении салат-латук, не содержащим фрагмент интрогрессии, аллель L. Sativa, обнаруженный в том же локусе на хромосоме 6 и/или 7, в настоящей заявке обозначается как аллель "дикого типа" (wt). Таким образом, культивированное растение салат-латук, восприимчивое к Nr:1 и не содержащее QTL на хромосоме 6 и 7, обозначается как wt/wt, тогда как QTL6.1/wt и/или QTL7.1/wt и/или QTL7.2/wt растения, и QTL6.1/ QTL6.1 и/или QTL7.1/ QTL7.1 и/или QTL7.2/ QTL7.2 растения представляют собой культивированные растения салат-латук, которые имеют QTL в гетерозиготной или гомозиготной форме, соответственно. Генотип SNP маркеров, раскрытых в настоящей заявке, также указывает на генотип дикого типа или фрагмент интрогрессии, содержащий QTL, находящиеся в гомозиготной или гетерозиготной форме. Например, генотипом SNP_01, указывающим на QTL6.1, является 'AT' (указывающий на QTL6.1/wt) или 'АА' (указывающий на QTL6.1/ QTL6.1), тогда как генотипом, указывающим на дикий тип является 'ТТ' (wt/wt). В настоящее заявке приводится подобное описание для всех других SNP. Таким образом, когда делается ссылка на SNP маркер в настоящей заявке или SNP генотип, это означает ссылку на генотип маркера, указывающий на фрагмент интрогрессии, содержащий QTL, обеспечивающий Nr:1 резистентность (в гомозиготной или гетерозиготной форме).
"Вариантные" или "ортологичные" последовательности или "вариант QTL6.1, QTL7.1 или QTL7.2" относится к QTL (QTL6.1, QTL7.1 или QTL7.2), или фрагменту (фрагментам) интрогрессии, содержащему их, которые получены из других растений салат-латук дикого типа (особенно других растений L. virosa или экземпляров), чем QTL6.1, QTL7.1 и QTL7.2 (и геномная область, содержащая их), присутствующие в NCIMB42086, но варианты которых содержат один или более или все SNP, связанные с QTL6.1, QTL7.1 и/или QTL7.2, и где вариантная геномная последовательность имеет существенную идентичность последовательности с SEQ ID NO: содержащей SNP (любой одной последовательностью из SEQ ID NO: 1-22, SNP1.23, SNP2.24, SNP17.25, VSP1 - VSP4), имеет идентичность последовательности по меньшей мере 85%, 90%, 95%, 98%, 99% или более. Таким образом, когда в настоящем описании делается ссылка на определенный генотип SNP в определенной геномной последовательности (выбранной из SEQ ID NO: 1 - SEQ ID NO: 22, SNP1.23, SNP2.24, SNP17.25, VSP1 - VSP4), это охватывает также генотип SNP в вариантах геномной последовательности, т.е. генотип SNP в геномной последовательности, имеющей идентичность последовательности по меньшей мере 85%, 90%, 95%, 98%, 99% с последовательностью, на которую ссылаются (выбранной из SEQ ID NO: 1 - SEQ ID NO: 22, SNP 1.23, SNP2.24, SNP 17.25, VSP1 - VSP4). Таким образом, любая ссылка в настоящей заявке на любую одну из последовательностей SEQ ID NO: 1 - 22, SNP1.23, SNP2.24, SNP17.25, VSP1 -VSP4, в одном варианте также охватывает вариант любой одной из SEQ ID NO: 1 - 22, SNP1.23, SNP2.24, SNP17.25, VSP1 - VSP4, причем указанный вариант имеет идентичность последовательности по меньшей мере 85%, 90%, 95%, 98%, 99% или более с указанной последовательностью.
"Генетическим контролем" является линия, сорт или гибрид растения салат-латук, которые имеют такой же или очень подобный культивированный геном, как культивированное растение салат-латук, содержащее интрогрессию на хромосоме 6 (QTL6.1) и/или 7 (QTL7.1 и/или QTL7.2), за исключением того, что не содержит интрогрессию на хромосоме 6 и 7, т.е. хромосомы 6 и 7 относятся к "дикому типу" (wt/wt), т.е. геном культивированного растения салат-латук.
Термин "анализ маркеров" относится к анализу маолекулярных маркеров, которые могут применяться, чтобы протестировать присутствует ли на хромосоме 6 и/или 7 L. sativa интрогрессия из растения салат-латук дикого типа, фрагмент интрогрессии которого содержит обеспечивающий Nr:1 резистентность QTL (QTL6.1 и/или QTL7.1 и/или QTL7.2, или их вариант) (или содержит ли растение салат-латук дикого типа QTL6.1 и/или QTL7.1 и/или QTL7.2 или их варианты в своем геноме), путем обнаружения генотипа любого одного или более маркеров, связанных с QTL6.1 (или вариант), например, генотипа одного или более SNP маркеров, выбранных из SNP_01 - SNP_07, и/или любого специфичного для генома растения салат-латук дикого типа (особенно L. virosa) маркера между SNP маркерами SNP_01 и SNP_07, и/или в пределах 7 сМ или в пределах 5 сМ любого одного из этих маркеров, и/или в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований или менее любого одного из этих маркеров; альтернативно путем обнаружения генотипа любого одного или более маркеров, связанных с QTL6.1 (или вариантом), например, генотипа одного или более SNP маркеров, выбранных из SNP1.23, SNP_02, SNP2.24 и SNP_03 (необязательно также VSP1 или VSP3), и/или любого специфичного для генома растения салат-латук дикого типа (особенно L. virosa) маркера между SNP маркерами SNP1.23, SNP_02, SNP2.24 и SNP_03 (необязательно также VSP1 или VSP3), и/или в пределах 7 сМ или в пределах 5 сМ любого одного из этих маркеров, и/или в пределах 12 Мб, 10 Мб, 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований или менее любого одного из этих маркеров; и/или генотипа любого одного или более маркеров, связанных с QTL7.1 (или вариант), например, генотипа одного или более SNP маркеров, выбранных из SNP_15 - SNP_22, и/или любого специфичного для генома растения салат-латук дикого типа (особенно L. virosa) маркера между SNP маркерами SNP_15 и SNP_22, и/или в пределах 7 сМ или в пределах 5 сМ любого одного из этих маркеров, и/или в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований или менее любого одного из этих маркеров; альтернативно путем обнаружения генотипа любого одного или более маркеров, связанных с QTL7.1 (или вариант), например, генотипа одного или более SNP маркеров, выбранных из SNP_17, SNP_17.25, SNP_18 и SNP_19 (необязательно также VSP2 или VSP4), и/или любого специфичного для генома растения салат-латук дикого типа (особенно L. virosa) маркера между SNP маркерами SNP_17, SNP_17.25, SNP_18 и SNP_19 (необязательно также VSP2 или VSP4), и/или в пределах 7 сМ или в пределах 5 сМ любого одного из этих маркеров, и/или в пределах 12 Мб, 10 Мб, 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований или менее любого одного из этих маркеров; и/или генотипа любого одного или более маркеров, связанных с QTL7.2 (или вариант), например, генотипа одного или более SNP маркеров, выбранных из SNP_08 - SNP_14, и/или любой маркера, специфичного для генома растения салат-латук, между SNP маркерами SNP_08 и SNP_14, и/или в пределах 7 сМ или в пределах 5 сМ любого одного из этих маркеров, и/или в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований или менее любого одного из этих маркеров. Маркер "между" двумя маркерами является физически расположенным между маркерами на хромосоме.
"Рекомбинантная хромосома" относится к хромосоме, имеющей новый генетический облик, возникающий в результате кроссинговера между гомологичными хромосомами, например "рекомбинантная хромосома 6" или "рекомбинантная хромосома 7", т.е. хромосома 6 или 7, которая не присутствует ни в одном из родительских растений и возникла в результате редкого события кроссинговера между гомологичными хромосомами пары хромосом 6 или 7, соответственно. Согласно настоящему изобретению обеспечиваются, например, рекомбинантные хромосомы огурца 6 и 7, причем каждая содержит QTL Nr:1 резистентности.
"Выбор с помощью маркера" или "MAS" представляет собой процесс использования присутствия молекулярных маркеров, которые генетически связаны с определенным локусом или с определенной областью хромосомы (например, фрагмент интрогрессии), для выбора растений по присутствию конкретного локуса или области (фрагмент интрогрессии). Например, молекулярный маркер, генетически связанный с Nr:1 QTL, может применяться для обнаружения и/или выбора растений салат-латук, содержащих Nr:1 QTL на хромосоме 6 и/или 7. Чем теснее генетическая связь молекулярного маркера с локусом (например, около 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3сМ, 2 сМ, 1 сМ, 0.5 сМ или менее), тем менее вероятно, что маркер диссоциирует от локуса в ходе рекомбинации в мейозе. Подобным образом, чем теснее два маркера связаны друг с другом (например, в пределах 7 сМ или 5 сМ, 4 сМ, 3сМ, 2 сМ, 1 сМ или менее), тем менее вероятно, что два маркера будут отделены друг от друга (и более вероятно, что они совместно отделяться в качестве единого целого).
Маркер "в пределах 7 сМ или в пределах 5 сМ" другого маркера относится к маркеру, который генетически привязывается к области, фланкирующей маркер, в пределах 7 сМ или 5 сМ (т.е. с обеих сторон маркера). Подобным образом, маркер в пределах 10 Мб, 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований, 10 тысяч пар оснований, 5 тысяч пар оснований или менее другого маркера относится к маркеру, который физически находится в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Mб, 0.2 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований, 10 тысяч пар оснований, 5 тысяча пар оснований или менее, области геномной ДНК, фланкирующей маркер (т.е. с обеих сторон маркера).
"LOD-оценка" (логарифм (основание 10) расхождений) относится к статистическому тесту, часто используемому для анализа сцепления в популяциях животных и растений. Оценка LOD сравнивает вероятность получения тестовых данных, если эти два локуса (локусы молекулярных маркеров и/или локусы фенотипических признаков) действительно сцеплены, с вероятностью наблюдения одних и тех же данных чисто случайно. Положительные оценки LOD свидетельствуют о наличие сцепления, и оценка LOD выше 3.0 рассматривается как очевидное сцепление. Оценка LOD, равная +3, указывает на вероятность от 1000 до 1, что наблюдаемое сцепление не происходит случайно.
"Выделенная последовательность нуклеиновой кислоты" или "выделенная ДНК" относится к последовательности нуклеиновой кислоты, которая больше не находится в природной среде, из которой она была выделена, например, последовательности нуклеиновой кислоты в бактериальной клетке-хозяине или в ядерном или пластидном геноме растений.
"Клетка-хозяин" или "рекомбинантная клетка-хозяин" или "трансформированная клетка" являются терминами, относящимися к новой индивидуальной клетке (или организму), возникающей в результате по меньшей мере одной молекулы нуклеиновой кислоты, введенной в указанную клетку. Клетка-хозяин предпочтительно является растительной клеткой или бактериальной клеткой. Клетка-хозяин может содержать нуклеиновую кислоту в качестве экстрахромосомно (эписомальной) реплицирующейся молекулы, или содержать нуклеиновую кислоту, интегрированную в ядерный или пластидный геном клетки-хозяина, или в качестве введенной хромосомы, например минихромосомы.
"Идентичность последовательности" и "сходство последовательности" можно определить выравниванием двух пептидных или двух нуклеотидных последовательностей с использованием глобальных или локальных алгоритмов выравнивания. Последовательности могут затем обозначаться как «по существу идентичные" или "весьма подобные", когда они оптимально выровнены, например, с помощью программы GAP или BESTFIT или фильтрующей программы "Needle" (с использованием параметров по умолчанию, см. ниже), определяя по меньшей мере определенный минимальный процент идентичности последовательности (как определено ниже). Эти программы используют глобальный алгоритм выравнивания Нидлмана-Вунша для выравнивания двух последовательностей по всей длине, получая максимальное количество совпадений и сводя к минимуму количество гэпов. Обычно параметры по умолчанию используется штраф за открытие гэпа = 10 и штраф за расширение гэпа = 0,5 (как для нуклеотидного, так и белкового выравнивания). Для нуклеотидов применяемой матрицей весов замен является DNAFULL, и для белков матрицей весов замен является Blosum62 (Henikoff & Henikoff, 1992, PNAS 89, 915-919). Выравнивания последовательности и показатели процентной идентичности последовательности, например, могут быть определены с помощью компьютерных программ, таких как EMBOSS, как доступно во всемирной сети по адресу ebi.ac.uk/Tools/psa/emboss_needle/. Альтернативно подобие или идентичность последовательности могут быть определены с помощью функции поиска в базах данных, таких как FASTA, BLAST и т.д., но совпадения должны быть получены и приведены в соответствие попарно, чтобы сравнить идентичность последовательности. Два протеина или два белковых домена или две последовательности нуклеиновых кислот имеют «существенную идентичность последовательности», если идентичность последовательности в процентах составляет по меньшей мере 85%, 90%, 95%, 98%, 99% или более (например по меньшей мере 99.1, 99.2 99.3 99.4, 99.5, 99.6, 99.7, 99.8, 99.9 или более (как определено с помощью Emboss "needle", применяя параметры по умолчанию, т.е. штраф за открытие гэпа = 10, штраф за расширение гэпа = 0.5, применяя матрицу весов замен DNAFULL для нуклеиновых кислот, и Blosum62 для белков).
Когда делается ссылка на последовательность нуклеиновой кислоты (например ДНК или геномную ДНК), имеющую "последовательность, по существу идентичную" ссылочной последовательности или имеющую последовательность, идентичную на по меньшей мере 80%, например по меньшей мере 85%, 90%, 95%, 98%, 99%, 99.2%, 99.5%, 99.9% ссылочной последовательности, в одном варианте выполнения настоящего изобретения указанная нуклеотидная последовательность рассматривается как по существу идентичная данной нуклеотидной последовательности и может быть идентифицирована, применяя строгие условия гибридизации. В другом варианте выполнения настоящего изобретения, последовательность нуклеиновой кислоты содержит одну или более мутаций, по сравнению с данной нуклеотидной последовательностью, но все еще может быть идентифицирована, применяя строгие условия гибридизации.
"Строгие условия гибридизации" могут применяться для идентификации нуклеотидных последовательностей, которые по существу идентичны данной нуклеотидной последовательности. Строгие условия зависят от последовательности и будут отличаться в разных обстоятельствах. Как правило, строгие условия выбираются так, что составляют на около 5°С точки плавления (Tm) для конкретных последовательностей при определенной ионной силе и рН. Tm представляет собой температуру (при определенной ионной силе и рН), при которой 50% целевой последовательности гибридизуется идеально подобранным зондом. Обычно выбирают такие строгие условия, при которых концентрация соли составляет около 0,02 моля при рН 7, а температура составляет по меньшей мере 60°С. Уменьшение концентрации соли и/или повышение температуры увеличивает жесткость условий. Строгие условия для гибридизаций РНК-ДНК (нозерн-блоты с использованием зонда, например, 100 нт) являются, например, теми, которые включают по меньшей мере одну промывку в 0.2Х SSC при 63°С в течение 20 мин, или эквивалентные условия. Строгие условия для гибридизации ДНК-ДНК (Саузерн-блоты с использованием зонда, например, 100нт) являются, например, теми, которые включают по меньшей мере одну промывку (как правило 2) в 0.2Х SSC при температуре, равной по меньшей мере 50°С, как правило около 55°С, в течение 20 мин, или эквивалентные условия. Смотрите также Sambrook et al. (1989) и Sambrook and Russell (2001).
Краткое описание чертежей
Фиг. 1: А. График, показывающий среднее число тли (немецкий изолят Nr:1) на восприимчивом контроле сорта Mafalda и на NCIMB 42086 в тесте без выбора, на 7, 14, 21, 28 и 35 дни после инокуляции. В. показывает тот же самый график как А., но с более детальной шкалой.
Фиг. 2: А. График, показывающий среднее число тли на NCIMB 42086 в тесте со свободным выбором, на 7, 14, 21, 28 и 35 дни после инокуляции. А. относится к немецкому изоляту Nr:1 из области Pfalz, тогда как В. относится к французскому изоляту Nr:1 из области Perpignan.
Фиг. 3А: Схематический график (не в масштабе) хромосомы 6 и 7 растения L. sativa, содержащего фрагмент интрогрессии (например, толстая полоса, которая может быть длиннее или короче, чем нарисовано здесь) из экземпляра L. virosa NCIMB42086 на хромосоме 6 (содержащий QTL6.1) и два фрагмента интрогрессии из экземпляра L. virosa NCIMB42086 на хромосоме 7 (содержащие QTL7.1 и QTL7.2), а также SNP маркеры, указывающие на фрагменты интрогрессии и их физическое положение в геноме растения салат-латук. Число звезд (*) указывает на LOD-оценку, являющуюся значимой (одна звезда) или весьма значимой (две или три звезды).
Фиг. 3В: Схематический график (не в масштабе) хромосомы 6 и 7 растения L. sativa, содержащего фрагмент интрогрессии (серая полоса) из L. virosa дикого типа (например, из экземпляра NCIMB42086) на хромосоме 6 (содержащий QTL6.1 или его вариант) и на хромосоме 7 (содержащий QTL7.1 или его вариант). Знак * указывает на SNP маркеры, которые являются специфичными для двух экземпляров L. virosa дикого типа, SNP маркеры VSP1 и VSP3 на хромосоме 6 и VSP2 и VSP4 на хромосоме 7 (причем VSP1 и VSP2 являются специфичными для одного из экземпляров, и VSP3 и VSP4 являются специфичными для другого экземпляра.
Фиг. 4: Результаты полевого анализа без выбора, проведенного в Мурсии, Испания, показали ноль тли на NCIMB42086 по сравнению с >300 тлей на Nr:0 резистентном сорте Mafalda.
Подробное описание изобретения
Нестоящее изобретение относится к культивированному растению салат-латук, Lactuca sativa, содержащему один или два или три QTL, интрогрессированных из растения салат-латук дикого типа, где указанные QTL обеспечивают резистентность против N. ribisnigri биотипа 1 (Nr:1). В частности, Nr:1 резистентность обеспечивается фрагментом интрогрессии на хромосоме 6 (содержащий QTL6.1) и/или 7 (содержащий QTL 7.1 и/или QTL7.2) культивированного растения салат-латук, где указанным фрагментом интрогрессии является фрагмент из растения салат-латук дикого типа, в частности растение вида Lactuca virosa.
Таким образом, в одном варианте обеспечивается растение Lactuca sativa, содержащее фрагмент интрогрессии из растения салат-латук дикого типа на хромосоме 6 и/или на хромосоме 7, где указанный фрагмент интрогрессии на хромосоме 6 содержит локус количественных признаков (QTL), обозначается как QTL6.1, и где указанный фрагмент интрогрессии на хромосоме 7 содержит локус количественных признаков обозначается как QTL7.1 и/или QTL7.2, и где указанный фрагмент интрогрессии на хромосоме 6 и/или 7 обеспечивает резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1). Каждый из фрагментов интрогрессии может быть в гомозиготной или гетерозиготной форме. В одном варианте фрагмент интрогрессии (фрагменты) находится/находятся в гомозиготной форме. В одном варианте, когда растение содержит более одного фрагмента интрогрессии, т.е. два или три фрагмента, они происходят из одного и того же экземпляра растения салат-латук дикого типа. В одном варианте выполнения настоящего изобретения этим экземпляром растения салат-латук дикого типа является L. virosa (такой как NCIMB 42086 или его потомство). В одном варианте экземпляром дикого типа является экземпляр L. virosa, выбранный из экземпляра, содержащего специфичные для экземпляра L. virosa маркеры VSP1 и VSP2; или экземпляр, содержащий специфичные для экземпляра L. virosa маркеры VSP3 и VSP4.
Когда в настоящей заявке делается ссылка на фрагмент интрогрессии на хромосоме 6 и/или 7, имеющий обеспечивающий Nr:1 резистентность QTL, это охватывает различные размеры фрагментов интрогрессии, например, фрагмент, содержащий все SNP маркеры (SNP_01 - SNP_07, или альтернативно SNP1.23, SNP_02, SNP2.24 и SNP_03, или любой маркер между ними, для фрагмента на хромосоме 6 (содержащего QTL6.1); SNP_08 - SNP_14, или любой маркер между ними, для фрагмента на хромосоме 7 (содержащего QTL7.2); SNP_15 - SNP_22, или альтернативно SNP_17, SNP17.25, SNP_18 и SNP_19, или любой маркер между ними, для фрагмента на хромосоме 7, содержащего QTL7.1), но также более маленькие фрагменты интрогрессии (содержащие, например, 1, 2, 3 или 4 из SNP маркеров), где, однако, фрагмент остается достаточно большим, чтобы обеспечить Nr:1 резистентность, когда фрагмент интрогрессии (фрагменты) находится/находятся в гетерозиготной или гомозиготной форме в геноме культивированного растения салат-латук.
Когда в настоящей заявке делается ссылка на SNP маркеры, которые указывают на присутствие фрагмента интрогрессии (и Nr:1 QTL, присутствующий на фрагменте интрогрессии), понятно, что делается ссылка на генотип SNP, который указывает на фрагмент интрогрессии, т.е. генотип SNP, как представлено в Таблице 1, 2 и 3, или в Таблице 4, 5, 6 и 7, и далее в настоящей заявке. Отмечается, что генотип маркера SNP может различаться между фрагментом интрогрессии, находящимся в гомозиготной или гетерозиготной форме, как показано в этих Таблицах. В гомозиготной форме нуклеотид является идентичным, тогда как в гетерозиготной форме нуклеотид является идентичным. Генотипом SNP хромосомы 'дикого типа', не содержащей фрагмент интрогрессии, является другим генотипом, также перечисленным в Таблицах 1-3 и Таблицах 4-7 (под генотипом родителя L. sativa). Поэтому, например, генотипом SNP_01, указывающий на фрагмент интрогрессии, содержащий QTL6.1, является 'AT' (QTL6.1/wt, т.е. гомозиготным для обеспечивающего резистентность QTL) или 'АА' (QTL6.1/ QTL6.1, т.е. гомозиготным для обеспечивающего резистентность QTL), тогда как генотипом SNP, указывающим на дикий тип/генетический контроль (не содержащим фрагмент интрогрессии) является 'ТТ' (wt/wt). Таким образом, при ссылке на растение или часть растения (например, клетку), содержащие фрагмент интрогрессии в гомозиготной или гетерозиготной форме, понимается, что SNP маркеры, связанные с фрагментом интрогрессии, имеют соответствующий генотип SNP. Например, растение согласно изобретению, которое является гомозиготным для фрагмента интрогрессии, содержащего QTL6.1, содержит SNP маркеры в гомозиготной форме.
Поэтому, в одном варианте обеспечивается культивированное растение L. sativa, содержащее фрагмент интрогрессии на хромосоме 6 и/или на хромосоме 7 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает резистентность против биотипа Nr:1. В предпочтительном варианте, один, два или все три фрагмента интрогрессии находятся в гомозиготной форме (и SNP маркер (маркеры), указывающие на QTL, являются гомозиготными для упомянутого нуклеотида).
Резистентность против Nr:1 фенотипически выражается как (статистически) значительно более низкое среднее число тли на биотип Nr:1 на растения линии или сорта культивированного растения салат-латук, содержащих фрагмент (фрагменты) интрогрессии на хромосоме 6 (содержащей QTL6.1) и/или 7 (содержащей QTL7.1 и/или 7.2) в гомозиготной или гетерозиготной форме, по сравнению с контрольной линией или сортом, не содержащими фрагмент интрогрессии на хромосоме 6 и 7, при выращивании при тех же условиях окружающей среды. Контрольной линией или сортом является линия или сорт культивированного растения салат-латук, которое является восприимчивым против Nr:1. В одном варианте их выбирают из сорта, который является восприимчивым против Nr:0 и Nr:1 (т.е. не имеет N. ribisnigri резистентность). В другом предпочтительном варианте это линия или сорт, имеющие Nr:0 резистентность, обеспеченную доминантным геном Nr, как например сорт Mafalda (или другие, такие как Susana, Sylvesta, Veronique, и многие другие, смотрите в интернете на nunhems.nl, где сорта с Nr:0 резистентностью обозначаются как 'HR'). В другом варианте это линия или сорт в качестве генетического контроля.
Таким образом, согласно настоящему изобретению обеспечиваются различные культивированные растения салат-латук, которые содержат либо фрагмент интрогрессии на хромосоме 6 (содержащий QTL6.1 или его вариант) в гомозиготной или гетерозиготной форме; или которые содержат фрагмент интрогрессии на хромосоме 7 (содержащий QTL7.1 или его вариант) в гомозиготной или гетерозиготной форме; или которые содержат фрагмент интрогрессии на хромосоме 7 (содержащий QTL7.2 или его вариант) в гомозиготной или гетерозиготной форме; или которые содержат два фрагмента интрогрессии (QTL6.1 и QTL7.1 или варианты любого из них; или QTL6.1 и QTL7.2 или варианты любого из них; или QTL7.1 и QTL7.2 или варианты любого из них; где два QTL могут независимо друг от друга находиться в гомозиготной или гетерозиготной форме), или все три фрагмента интрогрессии (QTL6.1, QTL7.1 и QTL7.2 или варианты любого из них), причем любой один из этих трех QTL независимо находится в гомозиготной или в гетерозиготной форме.
Растения согласно настоящему изобретению содержат геном культивированного растения салат-латук, с одной или двумя рекомбинантными хромосомами 6 и/или с одной или двумя рекомбинантными хромосомами 7. Рекомбинантные хромосомы содержат фрагмент растения салат-латук дикого типа (особенно растения L. virosa; в одном варианте экземпляра L. virosa NCIMB42086 или его потомства, или из другого L. virosa, такого как экземпляр, содержащий VSP1 и VSP2 или содержащий VSP3 и VSP4), который легко отличим от генома культивированного растения салат-латук посредством анализа молекулярного маркера (применяя, например, маркеры, обеспеченные согласно настоящему изобретения), секвенирования всего генома, «росписи» хромосомы и подобных методик.
В одном варианте присутствие фрагмента (фрагментов) интрогрессии на хромосомах 6 и/или 7 в геноме растения или растительной клетки или растительной ткани (или в ДНК, экстрагированной из него) является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает один или более молекулярных маркеров фрагмента (фрагментов) интрогрессии. Однако, как упомянуто, могут применяться другие методики, например, маркеры генотипа SNP также могут быть определены путем секвенирования или применяя альтернативные маркеры, расположенные между SNP маркерами, раскрытыми в настоящей заявке, или в пределах 7 сМ, или в пределах 5 сМ, маркера, раскрытого в настоящей заявке; или в пределах 10 Мб, 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований, 10 тысяч пар оснований, 5 тысяч пар оснований или менее маркера, раскрытого в настоящей заявке.
Растения салат-латук, содержащие фрагмент интрогрессии на хромосоме 6 (содержащий QTL 6.1 или его вариант)
На основе результатов первого картирования QTL следующие культивированные растения салат-латук охватываются настоящим изобретением.
В одном варианте фрагмент интрогрессии на хромосоме 6 является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, или 7 маркеров, выбранных из группы, состоящей из:
a) генотип АА или AT для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4);
e) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5);
f) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6);
g) генотип GG или GT для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7);
h) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_01 и SNP_07 (например, между SNP_01 и SNP_06, SNP_01 и SNP_05, SNP_01 и SNP_04, SNP_01 и SNP_03, SNP_01 и SNP_02); или между SNP_02 и SNP_07 (например, между SNP_02 и SNP_06, SNP_02 и SNP_05, SNP_02 и SNP_04, SNP_02 и SNP_03); или между SNP_03 и SNP_07 (например, между SNP_03 и SNP_06, SNP_03 и SNP_05, SNP_03 и SNP_04); или между SNP_04 и SNP_7 (например, между SNP_04 и SNP_06, SNP_04 и SNP_05); или между SNP_05 и SNP_07 (например, между SNP_05 и SNP_06); или между SNP_06 и SNP_07.
Как упомянуто, специалист в данной области техники может разработать другие молекулярные маркеры, например, маркер, специфичный для генома L. virosa дикого типа, между маркером SNP_01 и SNP_07 и/или в пределах 7 сМ или в пределах 5 сМ любого одного из SNP_01 - SNP_07, и/или в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований, 10 тысяч пар оснований, 5 тысяч пар оснований или менее любого одного из SNP_01 - SNP_07. - Такими маркерами также могут быть удлинение нуклеотида, CAPS маркеры, INDEL и т.д. Специалист в данной области техники может, например, секвенировать фрагмент интрогрессии или область QTL и применять информацию о последовательности для разработки новых маркеров и анализов маркеров.
В другом варианте фрагмент интрогрессии на хромосоме 6 (содержащий QTL6.1 или вариант) является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, или все 7 маркеров, выбранных из группы, состоящей из:
а) генотип АА или AT для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1);
b) генотип CC или CT для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4);
e) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5);
f) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6);
g) генотип GG или GT для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7).
В другом варианте обеспечивается культивированное растение L. sativa, содержащее фрагмент интрогрессии на хромосоме 6 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии придает Nr:1 резистентность, и где указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2, 3 или 4 (или по меньшей мере 5, 6 или все 7) последовательных маркеров, выбранных из группы, состоящей из:
a) генотип АА или AT для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4);
e) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5);
f) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6);
g) генотип GG или GT для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7).
SNP маркеры SNP_01 - SNP_07 располагаются в данном порядке на фрагменте интрогрессии. Последовательные маркеры означают маркеры в том же последовательном порядке, поэтому, например, двумя последовательными маркерами могут быть SNP_01 и SNP_02; SNP_02 и SNP_03; SNP_03 и SNP_04 и т.д., и тремя последовательными маркерами могут быть SNP_01 и SNP_02 и SNP_03; SNP_02 и SNP_03 и SNP_04; и т.д.
Фрагмент, таким образом, может быть меньше и не содержать 1, 2, 3, 4, 5 или даже 6 маркеров, но он может все еще обеспечивать Nr:1 резистентность культивированному растению салат-латук, т.е. он может все еще содержать Nr:1 аллель. Такие более маленькие фрагменты интрогрессии являются вариантом выполнения настоящего изобретения.
Растения, содержащие более маленькие фрагменты интрогрессии, могут быть получены, например, исходя из растения, содержащего большой фрагмент интрогрессии и скрещиванием такого растения с другим культивированным растением салат-латук и самоопылением потомства указанного кросса с получением популяции растений, которые могут содержать рекомбинанты, имеющие более маленький фрагмент интрогрессии на хромосоме 6. Анализы маркеров могут применяться для определения размера более маленького фрагмента интрогрессии. Один или более из SNP маркеров SNP_01 - SNP_07 могут отсутствовать (т.е. растение может содержать только 1, 2, 3, 4, 5, 6 SNP маркеров). Фенотип Nr:1 резистентности растений, содержащих такой более маленький фрагмент интрогрессии, может затем быть определен, как описано в настоящей заявке, т.е. выращивая множество растений, содержащих более маленький фрагмент интрогрессии в контролируемой окружающей среде или полевых испытаниях, вместе с подходящими контрольными растениями, не содержащими фрагмент интрогрессии. Анализом может быть анализ свободного выбора или анализ без выбора, так как резистентность, идентифицированная согласно настоящему изобретению, обеспечивает оба типа резистентности. Контрольными растениями являются предпочтительно генетический контроль или доступный сорт, такой как Mafalda. Если Nr:1 резистентность остается значительно выше, чем в контроле, тогда более маленький фрагмент интрогрессии сохранил QTL6.1 (или его вариант).
Альтернативно, тот же самый или вариант QTL (QTL6.1 или вариант QTL6.1) может быть интрогрессирован из другого источника дикого типа, причем необязательно не все SNP маркеры, раскрытые в настоящей заявке, могут присутствовать. Такими альтернативными источниками дикого типа являются предпочтительно экземпляры L. virosa. Они могут быть идентифицированы, применяя SNP маркеры согласно настоящему изобретению, путем анализа зародышевой плазмы (например, экземпляры L. virosa), применяя анализ маркеров для обнаружения генотипа маркеров SNP_01 - SNP_07 или любого маркера между SNP_01 и SNP_07. Альтернативно такие источники дикого типа могут быть идентифицированы фенотипически и необязательно проанализированы на поздней стадии на присутствие одного или более описанных маркеров, или необязательно потомство из кроссов с такими экземплярами может быть проанализировано на маркеры. Растения, содержащие тот же самый или вариант QTL6.1 из других источников, также являются вариантом выполнения настоящего изобретения. Пока по меньшей мере 1, 2, 3, 4, 5, 6 или более SNP, предпочтительно по меньшей мере 2, 3, 4, 5, 6 или более последовательных SNP маркеров SNP_01 - SNP_07 также имеют генотип SNP, указывающий на QTL, растение содержит QTL6.1 (или его вариант). Специалист в данной области техники может интрогрессировать QTL6.1 (или его вариант) в культивированный салат-латук, что придать Nr:1 резистентность, как описано в настоящей заявке.
В конкретном варианте выполнения настоящего изобретения растение согласно настоящему изобретению содержит фрагмент интрогрессии, содержащий по меньшей мере подгруппу SNP маркеров, т.е. по меньшей мере 1, 2, 3, 4, или все 5 из следующих маркеров, выбранных из группы, состоящей из:
a) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
b) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
c) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4);
d) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5);
e) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6); и необязательно
f) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно L. virosa, между SNP_02 и SNP_06.
Предпочтительно, растение согласно настоящему изобретению содержит фрагмент интрогрессии, содержащий по меньшей мере SNP маркеры SNP_03, SNP_04, SNP_05 и/или SNP_06 (или любой маркер между любыми из них), особенно по меньшей мере SNP_04, SNP_05 и/или SNP_06 (или любой маркер между любыми из них).
Таким образом, фрагмент интрогрессии (и культивированное растение салат-латук или часть растения, например, клетка, содержащие фрагмент интрогрессии) может быть обнаружен посредством анализа маркеров путем обнаружения генотипа SNP фрагмента интрогрессии (т.е. растения салат-латук дикого типа, например, зародышевая плазма L. virosa) одного или более или всех вышеуказанных маркеров.
В другом варианте, растение согласно настоящему изобретению содержит фрагмент интрогрессии, содержащий по меньшей мере SNP_04, т.е. фрагмент интрогрессии обнаруживается посредство анализа маркеров, обнаруживающего генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4. Необязательно также фланкирующие маркеры, SNP_03 и/или SNP_05 обнаруживаются, т.е. фрагмент интрогрессии обнаруживается посредство анализа маркеров, обнаруживающего по меньшей мере SNP_04 и необязательно также по меньшей мере один из следующих маркеров:
- генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5); и/или
- генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3); и необязательно
- любой маркер, специфичный для генома растения салат-латук дикого типа, особенно L. virosa, между SNP_03 и SNP_05.
Растения салат-латук, содержащие фрагмент интрогрессии на хромосоме 6 (содержащий QTL 6.1 или его вариант)
На основе данных позднего картирования QTL область QTL могла бы быть уточнена, и обеспечиваются культивированные растения салат-латук, содержащие фрагмент интрогрессии из Lactuca virosa, где фрагмент интрогрессии содержит QTL6.1 (или его вариант), причем фрагмент интрогрессии содержит всю или часть области, начинающейся в месте 77 Мб на хромосоме 6 и заканчивающейся в месте 161 Мб на хромосоме 6.
Таким образом, в одном варианте обеспечивается растение Lactuca sativa, содержащее фрагмент интрогрессии из Lactuca virosa на хромосоме 6, которое содержит локус количественных признаков, который придает резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1), и где фрагмент интрогрессии на хромосоме 6 содержит всю или часть области, начинающейся в месте 77 Мб и заканчивающейся в месте 161 Мб хромосомы 6.
Понятно, что более маленький фрагмент интрогрессии (т.е. содержащий часть, придающую резистентность, вышеупомянутой области, начинающейся в месте 77 Мб - 161 Мб хромосомы 6), который сохраняет QTL6.1 (или вариант), может быть фрагментом, имеющим размер, равный 80 Мб, 70 Мб, 60 Мб, 50 Мб, 40 Мб, 30 Мб, 20 Мб, 10 Мб, 5 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 100 тысяч пар оснований, 50 тысяч пар оснований или менее, и содержит QTL6.1 или его вариант. В одном варианте часть составляет по меньшей мере 5 тысяч пар оснований, 10 тысяч пар оснований, 20 тысяч пар оснований по размеру, или более.
В одном варианте фрагмент интрогрессии на хромосоме 6 содержится и является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, предпочтительно по меньшей мере 2 или 3 или 4 или 5 (или более) маркеров, выбранных из группы, состоящей из:
a) Генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 24);
генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP1.23 и SNP_03 (например, между SNP1.23 и SNP2.24, SNP1.23 и SNP_02); или между SNP_02 и SNP_03 (например, между SNP_02 и SNP2.24); или между SNP2.24 и SNP_03;
е) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный в пределах расстояния, равного 10 Мб, предпочтительно в пределах 5 Мб, любого маркера, выбранного из SNP1.23, SNP_02, SNP2.24, или SNP_03.
Необязательно, в одном варианте фрагмент интрогрессии содержит (и обнаруживается с его помощью) маркер, специфичный для экземпляра L. virosa, выбранный из генотипа GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 26) и генотипа АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 27). Применяя SNP маркеры VSP1 и VSP3 фрагменты интрогрессии, содержащие QTL6.1 из экземпляров различных типов L. virosa, можно отличить.
Фрагмент интрогрессии может быть в гетерозиготной или гомозиготной форме, как указывается генотипом SNP. Поэтому, в одном варианте фрагмент интрогрессии находится в гомозиготной форме, и генотип маркера SNP является гомозиготным генотипом.
Как упомянуто, варианты QTL6.1 могут быть идентифицированы и интрогрессированы из различных Nr:1 резистентных Lactuca virosa экземпляров. Такие варианты могут содержать геномную последовательность, которая на 100% не идентична последовательностям, раскрытым в настоящей заявке, но может все еще иметь существенную идентичность последовательности (как например, по меньшей мере 85%, 90% или более), когда выравниваются геномные последовательности одной длины. То что существует вариация в области QTL, где локализован QTL6.1, можно увидеть благодаря тому факту, что специфичные для экземпляра SNP маркеры могут быть идентифицированы в интрогрессиях QTL6.1 из двух различных экземпляров L. virosa, интрогрессии которых, однако, обе имеют резистентность, обеспечиваемую QTL6.1. Поэтому фрагмент интрогрессии, содержащий VSP1, является фрагментом интрогрессии, отличным от содержащего VSP3, но оба содержат QTL6.1. В пределах экземпляров L. virosa дикого типа, которые содержат QTL6.1, может иметься, таким образом, геномная вариация в области, в интервале 77 Мб - 161 Мб на хромосоме 6. Однако такие экземпляры могут в равной степени применяться для интрогрессии всей или части области, начинающейся в месте 77 Мб и заканчивающейся в месте 161 Мб хромосомы 6, в Nr:1 чувствительный культивированный салат-латук, чтобы получить растения согласно настоящему изобретению. Исходя из сведений, раскрытых в настоящей заявке, что область содержит QTL, специалист в данной области техники может интрогрессировать такую же область или более маленькую часть, обеспечивающую резистентность, в культивированный салат-латук.
В одном варианте фрагмент интрогрессии на хромосоме 6 содержится и является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает, по меньшей мере один, предпочтительно по меньшей мере 2 или 3 или 4 маркера, выбранных из группы, состоящей из:
a) Генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23;
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2;
c) генотип ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 24;
d) генотип AA или AC для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3.
В одном варианте фрагмент интрогрессии происходит из семян, депонированных под номером NCIMB42086, или их потомства.
В одном варианте фрагментом интрогрессии является фрагмент из другого Nr:1 резистентного экземпляра L. virosa, как например экземпляр, содержащий генотип АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 27).
В одном варианте фрагментом интрогрессии является фрагмент из другого Nr:1 резистентного экземпляра L. virosa, как например экземпляр, содержащий генотип GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 26).
Другие варианты, описанные в настоящей заявке, на основе первого анализа QTL и маркеры SNP_01 - SNP_07 в равной степени относятся к маркерам и интрогрессии, идентифицированным в этом более позднем анализе. Поэтому, например, QTL6.1 может быть интрогрессирован в любой культивированный салат-латук, особенно Nr:1 чувствительные линии или варианты, с помощью, например, обратного скрещивания. Он также может быть объединен в культивированном растении салат-латук с рекомбинантной хромосомой 7, содержащей QTL7.1 и/или QTL7.2.
Растения салат-латук, содержащие фрагмент интрогрессии на хромосоме 7 (QTL 7.1 и/или QTL7.2 или их варианты)
На основе результатов первого картирования QTL следующие культивированные растения салат-латук охватываются настоящим изобретением.
В одном варианте фрагмент интрогрессии, содержащий QTL 7.2 или его вариант (и культивированное растение салат-латук или часть растения, содержащие фрагмент интрогрессии) на хромосоме 7, является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, или 7 маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9);
c) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10);
d) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11);
e) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12);
f) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 13);
g) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 14);
h) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_08 и SNP_14 (например, между SNP_08 и SNP_13, SNP_08 и SNP_12, SNP_08 и SNP_11, SNP_08 и SNP_10, SNP_08 и SNP_09); или между SNP_09 и SNP_14 (например, между SNP_09 и SNP_13, SNP_09 и SNP_12, SNP_09 и SNP_11, SNP_09 и SNP_10); или между SNP_10 и SNP_14 (например, между SNP_10 и SNP_13, SNP_10 и SNP_12, SNP_10 и SNP_11); или между SNP_11 и SNP_14 (например, между SNP_11 и SNP_13, SNP_11 и SNP_12); или между SNP_12 и SNP_14 (например, между SNP_12 и SNP_13); или между SNP_13 и SNP_14.
Как упомянуто, специалист в данной области техники может разработать другие молекулярные маркеры, например, маркер, специфичный для генома растения салат-латук дикого типа, например, маркеры, специфичные для генома L. virosa, между маркером SNP_08 и SNP_14 и/или в пределах 7 сМ или в пределах 5 сМ любого одного из SNP_08 - SNP_14, и/или в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований, 10 тысяч пар оснований, 5 тысяч пар оснований или менее любого одного из SNP_08 - SNP_14. Такими маркерами также могут быть удлинение нуклеотида, CAPS маркеры, INDEL и т.д.
В другом варианте фрагмент интрогрессии на хромосоме 7 (содержащий QTL 7.2 или вариант) является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, или все 7 маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9);
c) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10);
d) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11);
e) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12);
f) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 13);
g) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 14).
В другом варианте обеспечивается культивированное растение салат-латук, содержащее фрагмент интрогрессии на хромосоме 7 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии содержит QTL7.2, придающий Nr:1 резистентность, и где указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2, 3 или 4 (или по меньшей мере 5, 6, 7) последовательных маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9);
c) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10);
d) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11);
e) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12);
f) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 13);
g) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 14).
SNP маркеры SNP_08 - SNP_14 располагаются в данном порядке на фрагменте интрогрессии. Последовательные маркеры означают маркеры в том же последовательном порядке, поэтому, например, двумя последовательными маркерами могут быть SNP_08 и SNP_09; SNP_09 и SNP_10; SNP_10 и SNP_11 и т.д., и тремя последовательными маркерами могут быть SNP_08 и SNP_09 и SNP_10; SNP_09 и SNP_10 и SNP_11; и т.д.
Фрагмент, таким образом, может быть меньше и не содержать 1, 2, 3, 4, 5, 6 маркеров, но он может все еще придавать Nr:1 резистентность культивированному растению салат-латук, т.е. он может все еще содержать Nr:1 аллель. Такие более маленькие фрагменты интрогрессии являются вариантом выполнения настоящего изобретения. Растения, содержащие более маленькие фрагменты интрогрессии, могут быть получены, например, исходя из растения, содержащего большой фрагмент интрогрессии и скрещиванием такого растения с другим культивированным растением салат-латук и самоопылением потомства указанного кросса с получением популяции растений, которые могут содержать рекомбинанты, имеющие более маленький фрагмент интрогрессии на хромосоме 7. Анализы маркеров могут применяться для определения размера более маленького фрагмента интрогрессии. Один или более из SNP маркеров SNP_08 - SNP_14 могут отсутствовать (т.е. растение может содержать только 1, 2, 3, 4, 5, или 6 SNP маркеров). Nr:1 резистентность растений, содержащих такой более маленький фрагмент интрогрессии, можно затем сравнить в анализах Nr:1, как описано в настоящей заявке, т.е. выращивая множество растений, содержащих более маленький фрагмент интрогрессии в полевых испытаниях вместе с подходящими контрольными растениями, не содержащими фрагмент интрогрессии. Контрольными растениями предпочтительно являются генетический контроль или восприимчивый контроль, такой как Mafalda. Если Nr:1 резистентность остается значительно выше, чем в контроле, тогда более маленький фрагмент интрогрессии сохранил QTL7.2 (или вариант).
Альтернативно, тот же самый или вариант QTL (QTL7.2 или вариант QTL7.2) может быть интрогрессирован из другого источника дикого типа, как например различные экземпляры L.virosa, причем необязательно не все SNP маркеры, раскрытые в настоящей заявке, могут присутствовать. Такие альтернативные источники дикого типа могут быть идентифицированы, применяя SNP маркеры, раскрытые в настоящей заявке, путем скрининга зародышевой плазмы дикого типа, например, экземпляров L. virosa, применяя анализ маркеров для обнаружения генотипа маркеров SNP_08 - SNP_14, или маркера между ними. Альтернативно такие источники дикого типа могут быть идентифицированы фенотипически и необязательно проанализированы на поздней стадии на присутствие одного или более описанных маркеров, или необязательно потомство из кроссов с такими экземплярами может быть проанализировано на маркеры. Растения, содержащие QTL7.2 или вариант QTL7.2 из других источников, также являются вариантом выполнения настоящего изобретения. Пока по меньшей мере 1, 2, 3, 4, 5, 6, или 7 или более SNP, предпочтительно по меньшей мере 2, 3, 4, 5, 6 или 7 последовательных SNP маркеров SNP_08 - SNP_14 также имеют генотип SNP, указывающий на QTL, растение содержит QTL7.2 (или его вариант). Специалист в данной области техники может интрогрессировать QTL7.2 (или его вариант) в культивированный салат-латук, чтобы получить Nr:1 резистентность, как описано в настоящей заявке.
В конкретном варианте выполнения настоящего изобретения растение согласно настоящему изобретению содержит фрагмент интрогрессии, содержащий по меньшей мере подгруппу SNP маркеров, т.е. по меньшей мере 1, 2, 3, 4 или все 5 из следующих маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9);
c) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10);
d) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11);
e) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12); и необязательно
f) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_08 и SNP_12.
Особенно, в одном варианте культивированное растение салат-латук согласно настоящему изобретению содержит по меньшей мере 1, 2 или 3 маркера, выбранные из группы, состоящей из:
a) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9);
b) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10);
c) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11); и необязательно
d) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_09 и SNP_11.
Таким образом, фрагмент интрогрессии (и культивированное растение салат-латук или часть растения, например, клетка, содержащие фрагмент интрогрессии) может быть обнаружен посредством анализа маркеров путем обнаружения генотипа SNP фрагмента интрогрессии (т.е. зародышевой плазмы растения салат-латук дикого типа) одного или более или всех вышеуказанных маркеров.
В одном варианте фрагмент интрогрессии, содержащий QTL 7.1 (и культивированное растение салат-латук или часть растения, содержащие фрагмент интрогрессии) на хромосоме 7, является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7 или 8 маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТА для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15);
b) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 16);
c) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
d) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
e) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
f) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20);
g) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21);
h) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22);
i) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_15 и SNP_22 (например, между SNP_15 и SNP_21, SNP_15 и SNP_20, SNP_15 и SNP_19, SNP_15 и SNP_18, SNP_15 и SNP_17, SNP_15 и SNP_16); или между SNP_16 и SNP_22 (например, между SNP_16 и SNP_21, SNP_16 и SNP_20, SNP_16 и SNP_19, SNP_16 и SNP_18, SNP_16 и SNP_17); или между SNP_17 и SNP_22 (например, между SNP_17 и SNP_21, SNP_17 и SNP_20, SNP_17 и SNP_19, SNP_17 и SNP_18); или между SNP_18 и SNP_22 (например, между SNP_18 и SNP_21, SNP_18 и SNP_20, SNP_18 и SNP_19); или между SNP_19 и SNP_22 (например, между SNP_19 и SNP_21, SNP_19 и SNP_20); или между SNP_20 и SNP_22; или между SNP_21 и SNP_22.
Как упомянуто, специалист в данной области техники может разработать другие молекулярные маркеры, например, маркер, специфичный для генома растения салат-латук дикого типа, например, маркеры, специфичные для генома L. virosa, между маркером SNP_15 и SNP_22 и/или в пределах 7 сМ или в пределах 5 сМ любого одного из SNP_15 - SNP_22, и/или в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 тысяч пар оснований, 20 тысяч пар оснований, 10 тысяч пар оснований, 5 тысяч пар оснований или менее любого одного из SNP_15 - SNP_22. Такими маркерами также могут быть удлинение нуклеотида, CAPS маркеры, INDEL и т.д.
В другом варианте фрагмент интрогрессии на хромосоме 7 (содержащий QTL 7.1 или вариант) является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7 или все 8 маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТА для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15);
b) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 16);
c) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
d) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
e) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
f) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20);
g) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21);
h) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22).
В другом варианте обеспечивается культивированное растение салат-латук, содержащее фрагмент интрогрессии на хромосоме 7 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии содержит QTL7.1, придающий Nr:1 резистентность, и где указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2, 3 или 4 (или по меньшей мере 5, 6, 7, 8) последовательных маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТА для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15);
b) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 16);
c) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
d) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
e) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
f) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20);
g) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21);
h) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22).
SNP маркеры SNP_15 - SNP_22 располагаются в данном порядке на фрагменте интрогрессии. Последовательные маркеры означают маркеры в том же последовательном порядке, поэтому, например, двумя последовательными маркерами могут быть SNP_12 и SNP_13; SNP_13 и SNP_14; SNP_14 и SNP_15 и т.д., и тремя последовательными маркерами могут быть SNP_12 и SNP_13 и SNP_14; SNP_13 и SNP_14 и SNP_15; и т.д.
Фрагмент, таким образом, может быть меньше и не содержать 1, 2, 3, 4, 5, 6 или 7 маркеров, но он может все еще обеспечивать Nr:1 резистентность культивированному растению салат-латук, т.е. он может все еще содержать Nr:1 аллель. Такие более маленькие фрагменты интрогрессии являются вариантом выполнения настоящего изобретения. Растения, содержащие более маленькие фрагменты интрогрессии могут быть получены, например, исходя из растения, содержащего большой фрагмент интрогрессии и скрещиванием такого растения с другим культивированным растением салат-латук и самоопылением потомства указанного кросса с получением популяции растений, которые могут содержать рекомбинанты, имеющие более маленький фрагмент интрогрессии на хромосоме 7 (содержащий QTL 7.1 или вариант). Анализы маркеров могут применяться для определения размера более маленького фрагмента интрогрессии. Один или более из SNP маркеров SNP_15 - SNP_22 могут отсутствовать (т.е. растение может содержать только 1, 2, 3, 4, 5, 6 или 7 SNP маркеров). Nr:1 резистентность растений, содержащих такой более маленький фрагмент интрогрессии, можно сравнить в анализах Nr:1, как описано в настоящей заявке, т.е. выращивая множество растений, содержащих более маленький фрагмент интрогрессии в полевых испытаниях вместе с подходящими контрольными растениями, не содержащими фрагмент интрогрессии. Контрольными растениями предпочтительно являются генетический контроль или восприимчивый контроль, такой как Mafalda. Если Nr:1 резистентность остается значительно выше, чем в контроле, тогда более маленький фрагмент интрогрессии сохранил QTL7.1 (или вариант).
Альтернативно, тот же самый или вариант QTL (QTL7.1 или вариант QTL7.1) может быть интрогрессирован из другого источника дикого типа, как например различные экземпляры L.virosa, причем необязательно не все SNP маркеры, раскрытые в настоящей заявке, могут присутствовать. Такие альтернативные источники дикого типа могут быть идентифицированы, применяя SNP маркеры, раскрытые в настоящей заявке, путем скрининга зародышевой плазмы дикого типа, например, L. virosa экземпляров, применяя анализ маркеров для обнаружения генотипа маркеров SNP_15 - SNP_22, или маркеров между ними. Альтернативно такие источники дикого типа могут быть идентифицированы фенотипически и необязательно проанализированы на поздней стадии на присутствие одного или более описанных маркеров, или необязательно потомство из кроссов с такими экземплярами может быть проанализировано на маркеры. Растения, содержащие QTL7.1 или вариант QTL7.1 из других источников, также являются вариантом выполнения настоящего изобретения. Пока по меньшей мере 1, 2, 3, 4, 5, 6, 7 или 8 или более SNP, предпочтительно по меньшей мере 2, 3, 4, 5, 6, 7 или 8 последовательных SNP маркеров SNP_15 - SNP_22 также имеют генотип SNP, указывающий на QTL, растение содержит QTL7.1 (или его вариант). Специалист в данной области техники может интрогрессировать QTL7.1 (или его вариант) в культивированное растение салат-латук, чтобы получить Nr:1 резистентность, как описано в настоящей заявке.
В конкретном варианте выполнения настоящего изобретения растение согласно настоящему изобретению содержит фрагмент интрогрессии, содержащий по меньшей мере подгруппу SNP маркеров, т.е. по меньшей мере 1, 2, 3, 4 или все 5 из следующих маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
b) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
c) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
d) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20);
e) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21); и необязательно
f) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_19 и SNP_21.
Особенно, в одном варианте культивированное растение салат-латук согласно настоящему изобретению содержит по меньшей мере 1, 2 или 3 маркеры, выбранные из группы, состоящей из
a) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
b) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20);
c) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21); и необязательно
d) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_19 и SNP_21.
Таким образом, фрагмент интрогрессии (и культивированное растение салат-латук или часть растения, например, клетка, содержащие фрагмент интрогрессии) может быть обнаружен посредством анализа маркеров путем обнаружения генотипа SNP фрагмента интрогрессии (т.е. зародышевой плазмы растения салат-латук дикого типа) одного или более или всех вышеуказанных маркеров.
Растения салат-латук, содержащие фрагмент интрогрессии на хромосоме 7 (QTL7.1 или его вариант)
На основе данных позднего картирования QTL область QTL7.1 могла бы быть уточнена, и обеспечиваются культивированные растения салат-латук, содержащие фрагмент интрогрессии из Lactuca virosa, где фрагмент интрогрессии содержит QTL7.1 (или его вариант), причем фрагмент интрогрессии содержит всю или часть области, начинающейся в месте 203 Мб на хромосоме 7 и заканчивающейся в месте 219 Мб на хромосоме 7.
Таким образом, в одном варианте обеспечивается растение Lactuca sativa, содержащее фрагмент интрогрессии из Lactuca virosa на хромосоме 7, которое содержит локус количественных признаков, который придает резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1), и где фрагмент интрогрессии на хромосоме 7 содержит всю или часть области, начинающейся в месте 203 Мб на хромосоме 7 и заканчивающейся в месте 219 Мб хромосомы 7.
Понятно, что более маленький фрагмент интрогрессии (т.е. содержащий часть, придающую резистентность, вышеупомянутой области в диапазоне 203 Мб - 219 Мб хромосомы 7), который содержит QTL7.1 (или вариант), может быть фрагментом, имеющим размер, равный 15 Мб, 10 Мб, 5 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 100 тысяч пар оснований, 50 тысяч пар оснований или менее и содержит QTL7.1 или его вариант. В одном варианте часть составляет по меньшей мере 5 тысяч пар оснований, 10 тысяч пар оснований, 20 тысяч пар оснований по размеру или более.
В одном варианте фрагмент интрогрессии на хромосоме 7 содержится и является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, предпочтительно по меньшей мере 2 или 3 или 4 или 5 (или более) маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
b) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 25);
c) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
e) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_17 и SNP_19 (например, между SNP_17 и SNP_18, между SNP_17 и SNP17.25; или между SNP17.25 и SNP_19, или между SNP17.25 и SNP_18, или между SNP_18 и SNP_19);
f) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный в пределах расстояния, равного 12 Мб или 10 Мб, предпочтительно в пределах 5 Мб, любого маркера, выбранного из SNP_17, SNP_17.25, SNP_18 и SNP_19.
Необязательно, в одном варианте фрагмент интрогрессии содержит (и обнаруживается с его помощью) маркер, специфичный для экземпляра L. virosa, выбранный из генотипа СС или АС для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 28) и генотипа GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 29). Применяя SNP маркеры VSP2 и VSP4, фрагменты интрогрессии, содержащие QTL7.1 из экземпляров двух различных типов L. virosa, можно отличить.
Фрагмент интрогрессии может быть в гетерозиготной или гомозиготной форме, как указывается генотипом SNP. Поэтому, в одном варианте фрагмент интрогрессии находится в гомозиготной форме, и генотип маркера SNP является гомозиготным генотипом.
Как упомянуто, варианты QTL7.1 могут быть идентифицированы и интрогрессированы из различных Nr:1 резистентных экземпляров Lactuca virosa. Такие варианты могут содержать геномную последовательность, которая на 100% не идентична последовательностям, раскрытым в настоящей заявке, но может все еще иметь существенную идентичность последовательности (как например, по меньшей мере 85%, 90% или более), когда выравниваются геномные последовательности одной длины. То, что существует вариация в области QTL, где локализован QTL7.1, можно увидеть благодаря тому факту, что специфичные для экземпляра SNP маркеры могут быть идентифицированы в интрогрессиях QTL7.1 из двух различных экземпляров L. virosa, интрогрессии которых, однако, обе имеют резистентность, обеспечиваемую QTL6.1. Поэтому, фрагмент интрогрессии, содержащий VSP1, является фрагментом интрогрессии, отличным от содержащего VSP3, но оба содержат QTL7.1. В пределах экземпляров L. virosa дикого типа, которые содержат QTL7.1, может иметься, таким образом, геномная вариация в области, в интервале 203 Мб - 219 Мб на хромосоме 7. Однако такие экземпляры могут в равной степени применяться для интрогрессии всей или части области, начинающейся в месте 203 Мб и заканчивающейся в месте 219 Мб хромосомы 7, в Nr:1 чувствительный культивированный салат-латук, чтобы получить растения согласно настоящему изобретению. Исходя из сведений, раскрытых в настоящей заявке, что область содержит QTL, специалист в данной области техники может интрогрессировать такую же область или более маленькую часть, обеспечивающую резистентность, в культивированный салат-латук.
В одном варианте фрагмент интрогрессии на хромосоме 7 содержится и является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает, по меньшей мере один, предпочтительно по меньшей мере 2 или 3 или 4 маркера, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
b) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 25);
c) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
В одном варианте фрагмент интрогрессии происходит из семян, депонированных под номером NCIMB42086 или их потомства.
В одном варианте фрагментом интрогрессии является фрагментом из другого Nr:1 резистентного экземпляра L. virosa, такого как экземпляр, имеющий генотип GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 29).
В одном варианте фрагментом интрогрессии является фрагментом из другого Nr:1 резистентного экземпляра L. virosa, такого как экземпляр, имеющий генотип СС или СА для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 28).
Другие варианты, описанные в настоящей заявке на основе первого анализа QTL для QTL7.1 и маркеров SNP_15 - SNP_22, в равной степени относятся к маркерам и интрогрессии, идентифицированным в этом более позднем анализе. Так, например, QTL7.1 можно интрогрессировать в любое культивированное растение салат-латук, особенно Nr:1 восприимчивые линии или сорта, посредством, например, обратного скрещивания. Он может также быть комбинирован в культивированном растении салат-латук с рекомбинантной хромосомой 6, содержащей QTL6.1, и/или со вторым QTL на хромосоме 7, а именно QTL7.2.
Таким образом, в одном варианте три локуса количественных признаков (QTL6.1 и/или QTL7.1 и/или QTL7.2), как было обнаружено, присутствуют на хромосоме 6 и 7 растения салат-латук дикого типа (особенно L. virosa, такой как экземпляр NCIMB42086), которые, при переносе (интрогрессии) в сорт или линию разведения культивированного растения салат-латук по отдельности или в комбинации, и при присутствии в гетерозиготной или гомозиготной форме, обеспечивают Nr:1 резистентность культивированному растению салат-латук. В одном варианте культивированное растение салат-латук содержит фрагмент (фрагменты) интрогрессии только на одной из хромосомы 6 и/или 7, тогда как гомологичными хромосомами 6 и 7 пары могут быть нерекомбинантная хромосома 6 и/или 7 культивированного растения салат-латук, не содержащего фрагмент (фрагменты) интрогрессии. В другом варианте культивированное растение салат-латук содержит фрагмент (фрагменты) интрогрессии на обеих хромосомах 6 и/или 7 гомологичной пары (интрогрессия находится в гомозиготной форме). Генотипами культивированных растений салат-латук для растений, содержащих один фрагмент интрогрессии, могут, таким образом, быть: QTL6.1/wt, QTL6.1/QTL6.1, QTL7.1/wt, QTL7.1/QTL7.1, QTL7.2/wt, QTL7.2/QTL7.2; для растений, содержащих два фрагмента интрогрессии, один на хромосоме 6 и один на хромосоме 7: QTL6.1/wt плюс QTL7.1/wt, QTL6.1/QTL6.1 плюс QTL7.1/wt, QTL6.1/QTL6.1 плюс QTL7.1/QTL7.1, QTL6.1/wt плюс QTL7.1/QTL7.1; QTL6.1/wt плюс QTL7.2/wt, QTL6.1/QTL6.1 плюс QTL7.2/wt, QTL6.1/QTL6.1 плюс QTL7.2/QTL7.2, QTL6.1/wt плюс QTL7.2/QTL7.2; для растений, содержащих два фрагмента интрогрессии на хромосоме 7: QTL7.2/wt плюс QTL7.1/wt, QTL7.2/QTL7.2 плюс QTL7.1/wt, QTL7.2/QTL7.2 плюс QTL7.1/QTL7.1, QTL7.2/wt плюс QTL7.1/QTL7.1. Растения, содержащие два фрагмента интрогрессии, один на хромосоме 6 и один на хромосоме 7, как описано выше, могут дополнительно содержать третий QTL на хромосоме 7 в гетерозиготной или гомозиготной форме.
Фрагменты интрогрессии могут происходить из одного и того же экземпляра, но они могут также происходить из различных экземпляров. В одном варианте фрагменты интрогрессии на хромосоме 6 происходят из того же экземпляра, как и фрагменты интрогрессии на хромосоме 7. Однако, также можно комбинировать фрагменты интрогрессии из различных экземпляров, например, фрагменты на хромосоме 6 могут происходить из одного экземпляра, и фрагменты на хромосоме 7 могут происходить из другого экземпляра. Например, маркеры VSP1 и VSP2 происходят из одного экземпляра, тогда как маркеры VSP3 и VSP4 происходят из другого экземпляра. В одном варианте фрагмент интрогрессии на хромосоме 6 содержит VSP1 маркер, и фрагмент интрогрессии на хромосоме 7 содержит VSP2 маркер. В другом варианте фрагмент интрогрессии на хромосоме 6 содержит VSP3 маркер, и фрагмент интрогрессии на хромосоме 7 содержит VSP4 маркер. Но фрагмент интрогрессии на хромосоме 6 и 7 может также происходить из различных экземпляров, потому, например, фрагмент на хромосоме 6 может содержать VSP1, тогда как фрагмент на хромосоме 7 может содержать VSP4, или фрагмент на хромосоме 6 может содержать VSP3 маркер, и фрагмент на хромосоме 7 может содержать VSP2 маркер. Подобным образом, растение, гомозиготное для фрагмента интрогрессии, например, содержащего QTL6.1, может содержать тот же фрагмент в гомозиготной форме, но может также содержать два различных фрагмента интрогрессии.
Хотя согласно настоящему изобретению источниками трех QT являются специфические источники дикого типа, вероятно, имеются другие экземпляры растений салат-латук дикого типа (особенно экземпляры L. virosa), которые содержат QTL6.1 и/или QTL7.1 и/или QTL7.2 в том же самом локусе/локусах на хромосоме 6 и/или 7. Такие локусы могут содержать Nr:1 аллели, которые имеют слегка различные нуклеотидные последовательности, т.е. варианты аллелей (QTL), раскрытых в настоящей заявке. Такие вариантные QTL могут также быть идентифицированы и интрогрессированы в культивированное растение салат-латук, как описано в настоящей заявке, с получением культивированного растения салат-латук, содержащего геном культивированного растения L. sativa и рекомбинантную хромосому 6 и/или 7, причем рекомбинантная хромосома 6 и/или 7 содержит фрагмент интрогрессии диких видов Lactuca (особенно L. virosa), который обеспечивает Nr:1 резистентность культивированному растению салат-латук, когда присутствует в гомозиготной или гетерозиготной форме.
Чтобы идентифицировать такие растения салат-латук дикого типа, содержащие QTL6.1 и/или QTL7.1 и/или QTL7.2 (или варианты QTL), экземпляры дикого типа могут быть скринированы, например, в анализе маркеров или путем сравнения последовательностей или другими способами, на присутствие одного или более из SNP маркеров, раскрытых в настоящей заявке. Предполагаемые обеспечивающие Nr:1 резистентность QTL (или варианты QTL) могут затем быть интрогрессированы в культивированное растение салат-латук, например, необязательно, применяя MAS (выбор с помощью маркера), т.е. применяя один или более (или все) из SNP маркеров, раскрытых в настоящей заявке (или маркеров между ними), для обнаружения и/или выбора растений потомства (например, беккросс потомства), содержащих рекомбинантную хромосому 6 и/или 7. Выбранные растения, т.е. культивированные растения салат-латук, содержащие фрагмент интрогрессии на хромосоме 6 и/или 7, где фрагмент интрогрессии на хромосоме 6 обнаруживается одним или более из SNP маркеров SNP_01 - SNP_07 или альтернативно одним или более из SNP маркеров SNP1.23, SNP_02, SNP2.24 или SNP_03, или маркерами между любыми из них (как описано в настоящей заявке), и/или где фрагмент интрогрессии на хромосоме 7 обнаруживается одним или более из SNP маркеров SNP_08 - SNP_14, или маркерами между ними (как описано в настоящей заявке), обнаруживая QTL7.2 или его вариант, и/или одним или более из SNP маркеров SNP_15 - SNP_22 или альтернативно SNP_17, SNP17.25, SNP_18 или SNP_19, или маркерами между любыми из них (как описано в настоящей заявке), обнаруживая QTL7.1 или его вариант, могут затем быть фенотипированы на Nr:1 резистентность вместе с подходящими контрольными растениями, предпочтительно по меньшей мере генетическим контролем и/или Nr:1 восприимчивым растением, таким как Mafalda, чтобы определить действительно ли фрагмент интрогрессии обеспечивает Nr:1 резистентность. Могут применяться один или более анализов на Nr:1 резистентность, как описано.
Экземпляры растений салат-латук дикого типа, таких как L. virosa, получают из системной коллекции зародышевой плазмы USDA National Plant или из других коллекций семян, таких как CGN в Wageningen, и могут, таким образом, быть скринированы на присутствие QTL6.1 (или вариант) и/или QTL7.1 (или вариант) и/или QTL7.2 (или вариант), применяя, например, анализ маркеров, как описано в настоящей заявке, и экземпляры, содержащие один или более из SNP маркеров, указывающих на QTL 6.1 или вариант; и/или содержащие один или более из SNP маркеров, указывающих на QTL7.2 или вариант; и/или содержащие один или более из SNP маркеров, указывающих на QTL7.1 или вариант, могут быть скрещены с культивированным растением салат-латук, имеющим нерекомбинантные хромосомы 6 и 7 нормального дикого типа. Потомство F2 (или следующее потомство, такое как F3, или предпочтительно беккросс потомство, такое как ВС1, ВС2, ВС3 или BC1S1 и т.д.,) может затем быть скринировано на рекомбинантные растения, имеющие фрагмент интрогрессии или его часть, обеспечивающую резистентность, применяя анализы молекулярных маркеров, как описано в настоящей заявке. Альтернативно, экземпляры дикого типа могут быть скринированы фенотипически, применяя анализ на Nr:1 резистентность, и только потомственные растения, полученные из кроссов с такими экземплярами дикого типа, могут быть скринированы на присутствие маркеров (и фрагментов интрогрессии). Такие потомственные растения также находятся в пределах объема настоящего изобретения.
В конкретном варианте выполнения настоящего изобретения, фрагмент интрогрессии, содержащий обеспечивающий Nr:1 резистентность QTL6.1 и/или обеспечивающий Nr:1 резистентность QTL7.1 и/или обеспечивающий Nr:1 резистентность QTL7.2, происходит из (или получен из) или является получаемым из (или полученный из; или как представлено в) семян, представительный пример которых был депонирован под номером NCIMB42086, или из их потомства. Потомством может быть любое потомство, которое сохранило один или более (или все) SNP маркеры, указывающие на QTL, как описано. Таким образом, потомство не ограничивается до F1 или F2 потомства депозита, но может быть любым потомством, полученным ли самоопылением и/или скрещиванием с другим растением салат-латук.
В одном варианте выполнения настоящего изобретения фрагмент интрогрессии, идентифицируемый одним или более маркерами, описанными в настоящей заявке, особенно маркерами SNP_01 - SNP_07 (или любым маркером между ними) для фрагмента интрогрессии на хромосоме 6 (QTL6.1 или вариант) или альтернативно SNP1.23, SNP_02, SNP2.24 и/или SNP_03 (или любым маркером между ними), необязательно также VSP1 или VSP3; и SNP_08 - SNP_14 (или любым маркером между ними) для фрагмента интрогрессии на хромосоме 7, обозначается как QTL7.2 (или вариант), и SNP_15 - SNP_22 (или любым маркером между ними) или альтернативно SNP_17, SNP17.25, SNP_18 и/или SNP_19 (или любым маркером между ними), необязательно VSP2 или VSP4, для фрагмента интрогрессии на хромосоме 7, обозначается как QTL7.1 (или вариант).
В одном варианте настоящее изобретение обеспечивает линию или сорт культивированного растения салат-латук, имеющие геном L. sativa, линия или сорт которого имеют Nr:1 резистентность, где Nr:1 резистентность обеспечивается фрагментом интрогрессии на хромосоме 6 и/или хромосоме 7 культивированного растения салат-латук, где указанный фрагмент интрогрессии получают (или является получаемым) скрещиванием Nr:1 резистентного растения L. virosa (которое содержит один или более маркеров, раскрытых в настоящей заявке, связанных с QTL) с культивированным растением салат-латук.
варианте настоящее изобретение обеспечивает линию или сорт культивированного растения салат-латук, имеющие геном L. sativa, линия или сорт которого имеют Nr:1 резистентность, где Nr:1 резистентность обеспечивается фрагментом интрогрессии на хромосоме 6 и/или хромосоме 7 культивированного растения салат-латук, где указанный фрагмент интрогрессии получают (или является получаемым) скрещиванием растения, выращенного из семян, депонированных под номером NCIMB 42086, или потомства этого растения (которое содержит один или более маркеров, раскрытых в настоящей заявке, связанных с QTL) с культивированным растением салат-латук.
В другом варианте выполнения настоящее изобретение относится к растению согласно настоящему изобретению т.е. культивированному растению L. sativa, содержащему фрагмент интрогрессии из растения салат-латук дикого типа на хромосоме 6 и/или 7 в гомозиготной или гетерозиготной форме, и где указанный фрагмент интрогрессии является вариантом геномной последовательности, содержащей QTL, как обнаруживается в семенах, депонированных под номером NCIMB 42086, т.е. он содержит QTL Nr:1, но геномная последовательность может быть отличной. Так как экземпляры дикого типа будут генетически расходящимися, геномная последовательность фрагмента интрогрессии, содержащего QTL6.1 или QTL7.1 или QTL7.2 (эти QTL согласно настоящему изобретению также обозначаются как варианты или ортологи QTL6.1, QTL7.1 и QTL7.2), из других экземпляров салат-латук дикого типа (например, другие экземпляры L. virosa, чем депонированный под номером доступа NCIMB42086) наиболее вероятно не будет идентичной с геномной последовательностью, и даже обеспечивающий Nr:1 резистентность ген (содержащий промотор, интроны и экзоны) может отличаться по нуклеотидной последовательности, но функция будет такой же, т.е. обеспечение Nr:1 резистентности. Расхождение можно увидеть в том, что некоторые маркеры SNP, связанные с (вариантом) QTL6.1 и/или (вариантом) QTL7.1 и/или (вариантом) QTL7.2 могут быть в общем обнаружены в различных экземплярах, тогда как другие SNP маркеры могут быть обнаружены только в определенных экземплярах. Поэтому, например, не все из SNP_01 - SNP_7, или не все из SNP1.23, SNP_02, SNP2.24 или SNP_03, и/или SNP_08 - SNP_14 и/или SNP_15 - SNP_22, или SNP_17, SNP17.25, SNP_18 или SNP_19, можно обнаружить в других Nr:1 резистентных растениях салат-латук дикого типа (например, экземпляры L. virosa), тогда как эти экземпляры могут все еще содержать QTL варианты в той же области. Подобным образом, геномная последовательность, содержащая каждый из SNP маркеров, может не быть на 100% идентична последовательности, раскрытой в настоящей заявке, но может иметь идентичность последовательности только (по меньшей мере) 85%, 90%, 95%, 98% или 99% с последовательностью, раскрытой в настоящей заявке, т.е. с любой одной последовательностью из SEQ ID NO: 1 - SEQ ID NO: 22 и SEQ ID NO: 23-29. Однако обеспечивающий Nr:1 резистентность QTL6.1 (или вариант) и QTL7.1 (или вариант) и QTL7.2 (или вариант, содержащий, например, вариант или ортолог аллеля Nr:1) может все еще присутствовать в таких экземплярах дикого типа. Специалист в данной области техники способен идентифицировать и интрогрессировать (вариант) QTL 6.1 и/или (вариант) QTL 7.1 и/или (вариант) QTL7.2, содержащие область, обнаруживаемую в других экземплярах растений салат-латук дикого типа (особенно экземпляры L. virosa; в частности экземпляры L. virosa, имеющие резистентность против Nr:1 как свободного выбора, так и без выбора) в культивированном растении салат-латук без ненужного груза.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии, или области хромосомы 6 (или области варианта или ортологичной хромосомы 6), содержащей QTL6.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3, 4, 5, 6, или более (или все 7) маркеров однонуклеотидного полиморфизма (SNP), выбранных из группы, состоящей из:
a) генотип АА или AT для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4);
e) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5);
f) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6);
g) генотип GG или GT для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7);
h) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_01 и SNP_07.
Таким образом, в одном варианте выполнения настоящего изобретения растения согласно настоящему изобретению содержат по меньшей мере аденин (А) (т.е. генотип АА или AT) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 1 (обозначается как SNP_01) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 2 (обозначается как SNP_02) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2; и/или по меньшей мере аденин (А) (т.е. генотип АА или АС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 3 (обозначается как SNP_03) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 71 последовательности SEQ ID NO: 4 (обозначается как SNP_04) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4; и/или по меньшей мере тимин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 5 (обозначается как SNP_05) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СА) вместо двух аденинов (АА) при нуклеотиде 71 последовательности SEQ ID NO: 6 (обозначается как SNP_06) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GT) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 7 (обозначается как SNP_07) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7.
В другом варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии, или области хромосомы 6 (или области варианта или ортологичной хромосомы 6), содержащей QTL6.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3 или 4 из маркеров однонуклеотидного полиморфизма (SNP), выбранных из группы, состоящей из:
а) Генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23;
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2;
c) генотип ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 24;
d) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3;
e) любой маркер, специфичный для генома L. virosa, между маркером SNP1.23 и SNP_03.
Таким образом, в одном варианте выполнения настоящего изобретения растения согласно настоящему изобретению содержат по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 23 (обозначается как SNP1.23) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 2 (обозначается как SNP_02) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2; и/или по меньшей мере тимин (Т) (т.е. генотип ТТ или СТ) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 24 (обозначается как SNP2.24) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 24; и/или по меньшей мере аденин (А) (т.е. генотип АА или АС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 3 (обозначается как SNP_03) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии, или области хромосомы 7 (или области варианта или ортологичной хромосомы 7), содержащей QTL7.2 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3, 4, 5, 6, или более (или все 7) маркеров однонуклеотидного полиморфизма (SNP), выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9);
c) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10);
d) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11);
e) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12);
f) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 13);
g) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 14);
h) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_08 и SNP_14.
Таким образом, в одном варианте выполнения настоящего изобретения растения согласно настоящему изобретению содержат по меньшей мере тимин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 8 (обозначается как SNP_08) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 9 (обозначается как SNP_09) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9; и/или по меньшей мере аденин (А) (т.е. генотип АА или AG) вместо двух гуанинов (GG) при нуклеотиде 71 последовательности SEQ ID NO: 10 (обозначается как SNP_10) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СА) вместо двух аденинов (АА) при нуклеотиде 71 последовательности SEQ ID NO: 11 (обозначается как SNP_11) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 12 (обозначается как SNP_12) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 136 последовательности SEQ ID NO: 13 (обозначается как SNP_13) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 13; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 14 (обозначается как SNP_14) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности c SEQ ID NO: 14.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии, или области хромосомы 7 (или области варианта или ортологичной хромосомы 7), содержащей QTL7.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3, 4, 5, 6, 7 или более (или все 8) маркеров однонуклеотидного полиморфизма (SNP), выбранных из группы, состоящей из:
a) генотип ТТ или ТА для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15);
b) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 16);
c) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
d) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
e) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
f) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20);
g) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21);
h) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22);
i) любой маркер, специфичный для генома растения салат-латук, такой как маркер, специфичный для генома L. virosa, между маркером SNP_15 и SNP_22.
Таким образом, в одном варианте выполнения настоящего изобретения растения согласно настоящему изобретению содержат по меньшей мере тимин (Т) (т.е. генотип ТТ или ТА) вместо двух аденинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 15 (обозначается как SNP_15) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 71 последовательности SEQ ID NO: 16 (обозначается как SNP_16) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 16; и/или по меньшей мере тимин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 17 (обозначается как SNP_17) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GC) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 18 (обозначается как SNP_18) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 71 последовательности SEQ ID NO: 19 (обозначается как SNP_19) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19; и/или по меньшей мере тимин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 72 последовательности SEQ ID NO: 20 (обозначается как SNP_20) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СА) вместо двух аденинов (АА) при нуклеотиде 41 последовательности SEQ ID NO: 21 (обозначается как SNP_21) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21; и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 71 последовательности SEQ ID NO: 22 (обозначается как SNP_22) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22.
В другом варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии, или области хромосомы 7 (или области варианта или ортологичной хромосомы 7), содержащей QTL7.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3, 4 маркеров однонуклеотидного полиморфизма (SNP), выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
b) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 25);
c) генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
e) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_17 и SNP_19.
Таким образом, в одном варианте выполнения настоящего изобретения растения согласно настоящему изобретению содержат по меньшей мере тимин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 17 (обозначается как SNP_17) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17; по меньшей мере тимин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 25 (обозначается как SNP17.25) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 25; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GC) вместо двух цитозинов (СС) при нуклеотиде 71 последовательности SEQ ID NO: 18 (обозначается как SNP_18) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18; и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 71 последовательности SEQ ID NO: 19 (обозначается как SNP_19) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19.
Генотип SNP относится к двум нуклеотидам и геномным последовательностям, содержащим один из этих двух нуклеотидов, один на каждой хромосоме 6 (для SNP_01 - SNP_07 или для SNP1.23, SNP_02, SNP2.24 или SNP_03) или 7 (для SNP_08 - SNP_14 и SNP_15 - SNP_22 или для SNP_17, SNP17.25, SNP_18 или SNP_19). Поэтому растение, имеющее генотип СС для SNP_22 имеет идентичный нуклеотид (С) на обеих хромосомах, тогда как растение, имеющее генотип СТ для SNP_22, имеет одну хромосому с С при нуклеотиде 71 последовательности SEQ ID NO: 22 (или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22) и на хромосоме с Т при нуклеотиде 71 последовательности SEQ ID NO: 22 (или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22). Поскольку геномные последовательности вокруг SNP маркеров, раскрытых в настоящей заявке, могут слегка варьироваться в фрагментах интрогрессии от других экземпляров растения салат-латук дикого типа (т.е. варианты или ортологичные области хромосомы 6 или 7), ясно, что нуклеотидные последовательности до и после SNP могут не иметь 100% идентичность с последовательностями, раскрытыми в настоящей заявке. Поэтому последовательности, имеющие существенную идентичность последовательностей с представленными здесь последовательностями, но которые содержат один и тот же SNP, охватываются настоящим изобретением. Также ясно, что в определенных вариантах интрогрессия находится в гомозиготной форме, и генотипом маркера SNP тогда является генотип, гомозиготный для QTL.
Фрагмент интрогрессии может быть большим, даже составлять половину хромосомы, или маленьким, пока сохраняется часть, обеспечивающая Nr:1 резистентность. В одном варианте фрагмент интрогрессии на хромосоме 6 и/или 7 равен или составляет менее 120 Мб, 100 Мб, 84 Мб, 80 Мб, 75 Мб, 74 Мб, 73 Мб, 60 Мб, 50 Мб, 40 Мб, 30 Мб, 20 Мб, 16 Мб, 15 Мб, 10 Мб по размеру, предпочтительно равен или составляет менее 8 Мб по размеру, более предпочтительно равен или составляет менее 6, 5, 4, 3 или 2.5 Мб по размеру, например, равен или составляет менее 2Mb. В одном варианте фрагмент интрогрессии составляет по меньшей мере 0.2 Мб, 0.5 Мб, 1.0 Мб, 1.5 Мб, 1.9 Мб, 2.0 Мб, 2.5 Мб или 3 Мб по размеру. Таким образом, различные диапазоны размеров интрогрессии охватываются настоящим изобретением. Размер может быть легко определен, например, секвенированием целого генома или секвенированием следующего поколения, например, как описано в Qi et al. 2013 (Nature Genetics December 2013, Vol 45, No. 12, pages 1510-1518) или в Huang et al. 2009 (Nature Genetics, Volume 41, Number 12, p1275-1283). В частности, области интрогрессия могут легко отличаться от культивированных геномных областей, благодаря большому числу генетической изменчивости (SNP, INDEL и т.д.) в области интрогрессии.
Специалисту известно как скринировать и идентифицировать растение салат-латук дикого типа, например, L. virosa, на присутствие любого одного из QTL или ортологов или вариантов, как описано в настоящей заявке. Например, различные экземпляры L. virosa могут сначала быть выбраны фенотипически путем анализа их Nr:1 резистентности, особенно резистентности свободного выбора и/или без выбора, и в одном варианте выбирают те экземпляры, которые содержат как резистентность как свободного выбора, так и без выбора. Альтернативно, различные экземпляры L. virosa могут быть скринированы непосредственно на присутствие одного или более из SNP маркеров (или маркеров между SNP маркерами), описанными в настоящей заявке. Как только кандидатное растение салат-латук дикого типа, например, L. virosa, было идентифицировано, специалисту известно как перенести один, два или все три из QTL согласно изобретению из растения салат-латук дикого типа в культивированное растение салат-латук, применяя традиционные методы разведения. Например, растения, выращенные из экземпляров дикого типа, такие как растения, выращенные из депонированных семян (NCIMB42086), могут быть скрещены с культивированным растением салат-латук с получением F1 семян. Растения F1 могут быть самоопылены один или более раз с получением F2 или F3 растений (или потомства дальнейшего самоопыления), и/или F1, F2 растения или F3 растения и т.д., могут быть обратно скрещены с культивированным родителем салат-латук. Потомственные растения, которые содержат QTL6.1 (или вариант) и/или QTL7.1 (или вариант) и/или QTL7.2 (или вариант) может быть скринировано по и отобрано по присутствию одного или более или всех из вышеописанных SNP маркеров (или маркеров между любыми из этих маркеров), чтобы идентифицировать растения, содержащие рекомбинантную хромосому 6 и/или 7, содержащую QTL. Методики, такие как эмбриональное спасение, могут должны быть применены для получения потомства из межвидовых кроссов (например, между L. sativa и L. virosa).
В другом варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растение салат-латук, или области хромосомы 6 (или ортологичной области хромосомы 6), содержащей QTL6.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип АА или AT для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1(или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1);
b) генотип GG или GT для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7);
c) любой маркер, специфичный для генома растения салат-латук, между маркером SNP_01 и SNP_07;
d) любой маркер, специфичный для генома растения салат-латук, который генетически связан в пределах 7 сМ, 5 сМ, 3 сМ или менее любого одного из маркеров SNP_01 - SNP_07; и
е) любой маркер, специфичный для генома растения салат-латук, который физически связан в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее любого одного из маркеров SNP_01 - SNP_07.
В одном варианте маркерами согласно с) являются один или более из SNP_02 - SNP_06.
В одном варианте по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b) и/или с) выше. В другом варианте, по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте по меньшей мере 1, 2, или 3 маркера согласно с) обнаруживаются, особенно по меньшей мере SNP_04, SNP_05 и/или SNP_06.
Любой маркер, специфичный для генома растения салат-латук, (например, специфичный для генома L. virosa) между маркер согласно а) и b), который генетически наносится на карту области хромосомы 6 между маркером SNP_01 и SNP_07 и/или который физически лежит между маркером SNP_01 и SNP_07, и который указывает на область хромосомы 6 растения салат-латук дикого типа. Это означает, что маркер является полиморфным между геномом культивированного растения салат-латук и геномом растения салат-латук дикого типа. В одном варианте маркером является однонуклеотидный полиморфизм (SNP), но другие молекулярные маркеры, такие как RFLP, AFLP, RAPD, ДНК севенирование и т.д., могут применяться в равной степени.
В альтернативном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растение салат-латук, или области хромосомы 6 (или ортологичной области хромосомы 6), содержащей QTL6.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23);
b) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3;
c) любой маркер, специфичный для генома L. virosa, физически расположенный между маркером SNP1.23 и SNP_03.
d) любой маркер, специфичный для генома L. virosa, который физически связан в пределах 10 Мб, 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее любого одного из маркеров SNP1.23 - SNP_03.
В одном варианте маркерами согласно с) являются один или более из SNP_02, SNP2.24, необязательно VSP1 и/или VSP3.
В одном варианте по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b) и/или с) выше. В другом варианте, по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b), с), и/или d) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с) и/или d) обнаруживаются. В одном варианте по меньшей мере 1, 2, или 3 маркера согласно с) обнаруживаются, особенно по меньшей мере SNP_02 и/или SNP2.24.
Любой маркер, специфичный для генома L. virosa, означает, что маркер указывает на фрагмент интрогрессии и присутствие генома L. virosa, т.е. маркер является полиморфным между геномом культивированного растения салат-латук и геномом растения салат-латук L. virosa дикого типа. В одном варианте маркером является однонуклеотидный полиморфизм (SNP), но другие молекулярные маркеры, такие как RFLP, AFLP, RAPD, ДНК севенирование и т.д., могут применяться в равной степени.
Подобным образом, в одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растение салат-латук, или области хромосомы 7 (или ортологичной области хромосомы 7), содержащей QTL7.2 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8);
c) любой маркер, специфичный для генома растения салат-латук, между маркером SNP_08 и SNP_14;
d) любой маркер, специфичный для генома растения салат-латук, который генетически связан в пределах 7 сМ, 5 сМ, 3 сМ или менее любого одного из маркеров SNP_08 - SNP_14; и
e) любой маркер, специфичный для генома растения салат-латук, который физически связан в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее любого одного из маркеров SNP_08 - SNP_14.
В одном варианте маркерами согласно с) являются один или более из SNP_09 - SNP_13.
В одном варианте по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b) и/или с) выше. В другом варианте, по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте по меньшей мере 1, 2 или 3 маркера согласно с) обнаруживаются, особенно SNP_09, SNP_10 и/или SNP_11.
Любой маркер, специфичный для генома растения салат-латук, (например, специфичный для генома L. virosa) между маркером согласно а) и b) который генетически наносится на карту области хромосомы 6 между маркером SNP_08 и SNP_14 и/или который физически лежит между маркером SNP_08 и SNP_14, и который указывает на область хромосомы 7 растения салат-латук дикого типа. Это означает, что маркер является полиморфным между геномом культивированного растения салат-латук и геномом дикого растения салат-латук. В одном варианте маркером является однонуклеотидный полиморфизм (SNP), но другие молекулярные маркеры, такие как RFLP, AFLP, RAPD, ДНК севенирование и т.д., могут применяться в равной степени.
Подобным образом, в одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растение салат-латук, или области хромосомы 7 (или ортологичной области хромосомы 7), содержащей QTL7.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере одного из маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТА для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 15 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 22 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22);
c) любой маркер, специфичный для генома растения салат-латук, между маркером SNP_15 и SNP_22;
d) любой маркер, специфичный для генома растения салат-латук, который генетически связан в пределах 7 сМ, 5 сМ, 3 сМ или менее любого одного из маркеров SNP_15 - SNP_22; и
e) любой маркер, специфичный для генома растения салат-латук, который физически связан в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее любого одного из маркеров SNP_15 - SNP_22.
В одном варианте маркерами согласно с) являются один или более из SNP_16 - SNP_21.
В одном варианте по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b) и/или с) выше. В другом варианте, по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте по меньшей мере 1, 2 или 3 маркера согласно с) обнаруживаются, особенно SNP_19, SNP_20 и/или SNP_21.
Любой маркер, специфичный для генома растения салат-латук, (например, специфичный для генома L. virosa) между маркером согласно а) и b), относится к любому маркеру, который генетически находится на карте области хромосомы 6 между маркером SNP_15 и SNP_22 и/или который физически лежит между маркером SNP_15 и SNP_22, и который указывает на область хромосомы 7 растения салат-латук дикого типа. Это означает, что маркер является полиморфным между геномом культивированного растения салат-латук и геномом растения салат-латук дикого типа. В одном варианте маркером является однонуклеотидный полиморфизм (SNP), но другие молекулярные маркеры, такие как RFLP, AFLP, RAPD, секвенирование ДНК и т.д., могут применяться в равной степени.
В альтернативном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растение салат-латук, или области хромосомы 7 (или ортологичной области хромосомы 7), содержащей QTL7.1 (или вариант), является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
b) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
c) любой маркер, специфичный для генома L. virosa, физически расположенный между маркером SNP_17 и SNP_19.
d) любой маркер, специфичный для генома L. virosa, который физически связан в пределах 12 Мб, 10 Мб, 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее любого одного из маркеров SNP_17 - SNP_19.
В одном варианте маркерами согласно с) являются один или более из SNP17.25, SNP_18, необязательно VSP4.
В одном варианте маркером согласно d) является VSP2 или SNP_16.
В одном варианте по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b) и/или с) выше. В другом варианте, по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров обнаруживаются из маркеров согласно а), b), с), и/или d) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с) и/или d) обнаруживаются. В одном варианте по меньшей мере 1, 2, или 3 маркера согласно с) обнаруживаются, особенно по меньшей мере SNP17.25 и/или SNP_18.
Любой маркер, специфичный для генома L. virosa, означает, что маркер указывает на фрагмент интрогрессии и присутствие генома L. virosa, т.е. маркер является полиморфным между геномом культивированного растения салат-латук и геномом растения салат-латук L. virosa дикого типа. В одном варианте маркером является однонуклеотидный полиморфизм (SNP), но другие молекулярные маркеры, такие как RFLP, AFLP, RAPD, ДНК севенирование и т.д., могут применяться в равной степени.
Также обеспечиваются семена, из которых растение согласно настоящему изобретению может быть выращено, как и листья растения салат-латук (или его части), и головки, собранные с растения согласно настоящему изобретению и содержащие рекомбинантную хромосому 6 и/или 7 в их геноме. Подобным образом, обеспечивается растительная клетка, ткань или часть растения растения или семени, содержащие по меньшей мере одну рекомбинантную хромосому 6 и/или 7, где указанная рекомбинантная хромосома 6 и/или 7 содержит фрагмент интрогрессии из растения салат-латук дикого типа, и где указанный фрагмент интрогрессии содержит QTL, придающий Nr:1 резистентность.
Молекулярные маркеры, описанные в настоящей заявке, могут быть обнаружены согласно стандартному методу. Например, SNP маркеры могут быть легко обнаружены, применяя анализ KASP (смотрите www.kpbioscience.co.uk) или другие анализы. Для разработки анализов KASP, например, 70 пар оснований против хода транскрипции и 70 пар оснований по ходу трнаскрипции SNP можно выделить, и два аллель-специфичных прямых праймера и один аллель-специфичный обратный праймер могут быть разработаны. Смотрите, например, Allen et al. 2011, Plant Biotechnology J. 9, 1086-1099, в частности p097-1098 для способа на основе анализа KASP.
Таким образом, в одном варианте, SNP маркеры и присутствие/отсутствия маркера, связанного с QTL, определяют, применяя анализ KASP, но также в равной степени могут применяться и другие анализы. Например, необязательно также может применяться ДНК секвенирование.
Физический размер фрагмента интрогрессии можно определить различными способами, такими как физическое картирование, секвенирование или путем визуализации интрогрессии, применяя флуоресцентные изображения in situ гибридизации (FISH) (Verlaan et al. 2011, Plant Journal 68: 1093-1103).
Растения с различными размерами фрагментов интрогрессии на хромосоме 6 и/или 7 могут быть получены, путем генерации рекомбинантных растений из популяции растений, полученных из кросса между культивированным растением салат-латук (не содержащим интрогрессии) и Nr:1 резистентным растением L. virosa или между культивированным растением салат-латук и растением согласно настоящему изобретению (культивированное растение салат-латук, содержащее рекомбинантную хромосому 6 и/или 7), и отбора потомства, имеющего различные размеры интрогрессии.
Способы
Маркеры и геномные области, идентифицированные в настоящей заявке, могут применяться в различных способах, и это применяется к первым идентифицированным областям и маркерам (смотрите, например, Фиг. 3А), а также к последним идентифицированным областям и маркерам (смотрите, например, Фиг. 3В), потому что QTL являются такими же. Нестоящее изобретение обеспечивает ряд способов, а именно:
1) способ идентификации растения салат-латук дикого типа, особенно экземпляра L. virosa, содержащего один или более из QTL6.1, QTL7.1 и/или QTL7.2 (или вариантов любого из них);
2) способ переноса одного или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или вариантов любого из них), из растения салат-латук дикого типа (например, L. virosa) в культивированное растение салат-латук (L. sativa) с получением Nr:1 резистентного культивированного растения салат-латук;
3) способ скрининга линий или сортов культивированного растения салат-латук на присутствие одного или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или вариантов любого из них); и
4) способ переноса одного или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или вариантов любого из них) из культивированного растения салат-латук (L. sativa) в другое культивированное растение салат-латук, например, в Nr:1 восприимчивую линию или сорт растения салат-латук;
5) способ применения семян, депонированных под номером доступа NCIMB42086, или их потомства, для получения Nr:1 резистентного культивированного растения салат-латук;
6) способ культивирования растений согласно настоящему изобретению, т.е. Nr:1 резистентного L. sativa растения, содержащие один или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или вариантов любого из них), в областях, где присутствует N. ribisnigri биотипа Nr:1.
Способ идентификации растения салат-латук дикого типа, содержащего один или более из QTL6.1, QTL7.1 и/или QTL7.2 (или вариантов любого из них)
В одном варианте обеспечивается способ идентификации растений салат-латук дикого типа, содержащих один или более из QTL6.1, QTL7.1 и/или QTL7.2 (или их вариантов), включающий стадии:
a) обеспечение растения салат-латук дикого типа или множества растений салат-латук дикого типа;
b) необязательно тестирование растения салат-латук дикого типа или множества растений на Nr:1 резистентность в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL6.1 или его вариант и/или указывающих на QTL7.1 или его вариант и/или указывающих на QTL7.2 или его вариант; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
f) необязательно скрещивание растение салат-латук дикого типа согласно d) с культивированным растением салат-латук.
Необязательно способ дополнительно содержит интрогрессинг QTL6.1, QTL7.1 и/или QTL7.2 (или их вариантов) в культивированное растение салат-латук, особенно Nr:1 восприимчивый салат-латук, и получение L. sativa растения, имеющего Nr:1 резистентность, обеспеченную одним или более из фрагментов интрогрессии. Это может, например, быть сделано путем обратного скрещивания. Необязательно может применяться отбор, связанный с маркером.
Растением или растениями на стадии а) являются предпочтительно L. virosa, например, происходящие из различных географических областей.
На стадии b) или е) анализ фенотипической Nr: резистентности (например, полевое испытание или тест в контролируемой окружающей среде) может осуществляться, чтобы отобрать растения, которые резистентны против Nr:1, и поэтому предположительно содержат один или более из QTL. Анализом на Nr:1 резистентность может быть анализ свободного выбора и/или анализ без выбора.
Nr:1 резистентное растение L. sativa, полученное этим способом, также является вариантом выполнения настоящего изобретения.
Геномная ДНК на стадии с) может быть проанализирована на присутствие одного или более маркеров, указывающих на QTL6.1 или его вариант, как описано выше, например, путем определения присутствия одного или более маркеров, выбранных из группы, состоящей из:
a) генотип АА или AT для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 1; и/или
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2; и/или
c) генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3; и/или
d) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 4; и/или
e) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 5; и/или
f) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 6; и/или
g) генотип GG или GT для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 7; и/или
h) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_01 и SNP_07, например, между любыми двумя маркерами из SNP_01 - SNP_07.
Альтернативно, геномная ДНК на стадии с) может быть проанализирована на присутствие одного или более маркеров, указывающих на QTL6.1 или его вариант, как описано выше, например, путем определения присутствия одного или более маркеров, выбранных из группы, состоящей из:
a) Генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 23);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 2);
c) генотип ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 24);
генотип АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 3);
d) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP1.23 и SNP_03 (например, между SNP1.23 и SNP2.24, SNP1.23 и SNP_02); или между SNP_02 и SNP_03 (например, между SNP_02 и SNP2.24); или между SNP2.24 и SNP_03;
e) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный в пределах расстояния, равного 10 Мб, предпочтительно в пределах 5 Мб, любого маркера, выбранного из SNP1.23, SNP_02, SNP2.24, или SNP_03.
Необязательно также L. virosa специфические маркеры (VSP1 или VSP3) могут быть проанализированы, чтобы идентифицировать и/или отличить типы экземпляров, содержащих QTL.
Геномная ДНК на стадии с) может быть проанализирована на присутствие одного или более маркеров, указывающих на QTL7.2 или его вариант, как описано выше, например, путем определения присутствия одного или более маркеров, выбранных из группы, состоящей из:
а) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 8;
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 9;
c) генотип АА или AG для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 10;
d) генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 11;
e) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 12;
f) генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 13;
g) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 14;
h) любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_08 и SNP_14, например, между любыми двумя маркерами из SNP_08 - SNP_14.
Геномная ДНК на стадии с) может быть проанализирована на присутствие одного или более маркеров, указывающих на QTL7.1 или его вариант, как описано выше, например, путем определения присутствия одного или более маркеров, выбранных из группы, состоящей из:
i. генотип ТТ или ТА для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 15;
ii. генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 16;
iii. генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17;
iv. генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18;
v. генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19;
vi. генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 20;
vii. генотип СС или СА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 21;
viii. генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 22;
ix. любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_15 и SNP_22, например, между любыми двумя маркерами из SNP_15 - SNP_22.
Альтернативно, геномная ДНК на стадии с) может быть проанализирована на присутствие одного или более маркеров, указывающих на QTL7.1 или его вариант, как описано выше, например, путем определения присутствия одного или более маркеров, выбранных из группы, состоящей из:
- генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 17);
- генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 25);
- генотип GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 18);
- генотип GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в последовательности, имеющей существенную идентичность последовательности с SEQ ID NO: 19);
- любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный физически между SNP_17 и SNP_19 (например, между SNP_17 и SNP_18, между SNP_17 и SNP17.25; или между SNP17.25 и SNP_19, или между SNP17.25 и SNP_18, или между SNP_18 и SNP_19);
- любой маркер, специфичный для генома растения салат-латук дикого типа, особенно маркер, специфичный для генома L. virosa, расположенный в пределах расстояния, равного 12 Мб, 10 Мб, предпочтительно в пределах 5 Мб, любого маркера, выбранного из SNP_17, SNP_17.25, SNP_18 и SNP_19.
Необязательно также специфичные для экземпляра L. virosa маркеры могут быть скринированы (VSP2 или VSP4), чтобы определить типы экземпляров, содержащих QTL.
Скрининг маркеров может быть сделан любой подходящей методикой или комбинацией методик, известными специалисту в данной области техники, например, на основе ПЦР, на основе секвенирования и т.д. Понятно, что скрининг геномной ДНК может быть сделан для растений, частей растения, семян или геномной ДНК, выделенной из них.
На стадии d) способа идентифицируется растение, содержащее один или более маркеров, например, по меньшей мере 1, 2, 3, 4, 5, 6 или все 7 из SNP_01 - SNP_07 и/или любой маркер между SNP_01 и SNP_07; или альтернативно один или более маркеров из SNP1.23, SNP_02, SNP2.24 или SNP_03 (и необязательно VSP1 или VSP3) и/или любой маркер между SNP1.23 и SNP_03; по меньшей мере по меньшей мере 1, 2, 3, 4, 5, 6 или все 7 из SNP_08 - SNP_14 и/или любой маркер между SNP_08 и SNP_14; по меньшей мере 1, 2, 3, 4, 5, 6, 7 или все 8 из SNP_15 - SNP_22 и/или любой маркер между SNP_15 и SNP_22; или альтернативно SNP_17, SNP17.25, SNP_18 или SNP_19 (и необязательно VSP2 или VSP4) или любой маркер между SNP_17 и SNP_19.
Таким образом, в одном варианте обеспечивается способ получения культивированного растения салат-латук, имеющего Nr:1 резистентность, включающий стадии:
а) Обеспечение растения салат-латук дикого типа, особенно растения Lactuca virosa, содержащего 1, 2, 3, 4, 5, или более SNP маркеров, указывающих на QTL6.1 (или вариант); и/или 1, 2, 3, 4, 5, или более SNP маркеров, указывающих на QTL7.2 (или вариант) и/или 1, 2, 3, 4, 5, или более SNP маркеров, указывающих на QTL7.1 (или вариант);
b) Скрещивание указанного растения салат-латук дикого типа, особенно указанного растения Lactuca virosa с культивированным растением салат-латук, которое восприимчиво к тли растения салат-латук Nr:1, с получением семян F1;
c) Необязательно самоопыление растений, выращенных из семян F1 один или более раз с получением F2, F3 или потомства дальнейшего самоопыления;
d) Скрещивание указанного F1 или потомства дальнейшего самоопыления с культивированным растением салат-латук согласно стадии b), с получением беккросс потомства;
e) Отбор беккросс потомства, которое содержит резистентность против биотипа Nr:1.
Растение салат-латук, полученное этим способом, также охватывается настоящим изобретением.
Способ переноса одного или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или их вариантов), из растения салат-латук дикого типа (например. L. virosa) в культивированное растение салат-латук (L. sativa) с получением Nr:1 резистентного культивированного растения салат-латук
В другом варианте обеспечивается способ переноса одного или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или их вариантов), из растения салат-латук дикого типа (например, L. virosa) в культивированное растение салат-латук (L. sativa) с получением Nr:1 резистентного культивированного растения салат-латук, включающий:
a) обеспечение растения салат-латук дикого типа, содержащего QTL6.1, QTL7.1 и/или QTL7.2 (или их варианты);
b) скрещивание растение салат-латук дикого типа согласно а) с культивированным растением салат-латук с получением F1;
c) необязательно самоопыление F1 один или более раз с получением дальнейшего потомства самоопыления;
d) обратное скрещивание F1 или дальнейшего потомства самоопыления один или более раз до культивированного растения салат-латук согласно стадии b) (рекуррентный родитель);
e) идентификация и/или отбор беккросс потомства, содержащего геном культивированного растения салат-латук согласно стадии b) (рекуррентный родитель), содержащего фрагмент интрогрессии из растения салат-латук дикого типа согласно а) (родитель-донор) на хромосоме 6 и/или на хромосоме 7.
В одном варианте растением салат-латук дикого типа согласно а) является L. virosa. В одном варианте L. virosa является Nr:1 резистентным, при тестировании в анализе на Nr:1 резистентность. В одном варианте растением L. virosa согласно а) является NCIMB42086, или его потомство, полученное самоопылением и/или скрещиванием, где потомство содержит QTL6.1, QTL7.1 и/или QTL7.2. В другом варианте растением согласно а) является экземпляр L. virosa, содержащий virosa специфичные маркеры VSP1 и/или VSP2 или содержащий virosa специфичные маркеры VSP3 и/или VSP4 (как показано в Таблице 6 и 7).
Культивированное растением салат-латук согласно стадии b) в способе выше (и в любом другом способе согласно настоящему изобретению) может быть любое L. sativa, такое как инбредная линия или сорт. Оно может быть любого типа, такого как листовой или рассыпной салат, салат кочанный или салат Bibb, салат-ромен или салат кос, салат Crisphead или салат айсберг, салат Celtuce или спаржевый салат. Это предпочтительно Nr:1 восприимчивое растение, хотя это также может быть растение, имеющее Nr:1 резистентность, обеспеченную различными локусами, чтобы уложить локусы Nr:1 резистентности в одну линию или сорт растений. Это может быть Nr:0 резистентное растение. Оно может содержать доминантный Nr-ген.
При ссылке на обратное скрещивание и беккросс потомство, это может также включать потомство, полученное обратным скрещиванием (ВС) и самоопылением (S), например, BC1S1, BC1S2, BC2S1 и т.д.,
На стадии е) могут применяться любые маркеры и анализы маркеров, описанные в настоящей заявке.
Растения, полученные способом, также являются вариантом выполнения настоящего изобретения, как описано в настоящей заявке. Этими растениями являются, таким образом, культивированные растения L. sativa (любого типа), содержащие один или более из QTL согласно настоящему изобретению на хромосоме 6 (QTL6.1 или вариант) и/или 7 (QTL7.1 и/или QTL7.2 или варианты) в гомозиготной или гетерозиготной форме.
Так как барьеры стерильности могут существовать между L. sativa и растениями салат-латук дикого типа, как например L. virosa, растение L. virosa (например, растения, выращенные из семян, имеющих номер доступа NCIMB 42086 или другие экземпляры L. virosa) может быть скрещено с видами-посредниками, такими как L. serriola (Eenink et al. 1982, Euphytica 31:291-299), и/или другие способы, такие как тетраплоидизация, могут применяться для преодоления барьеров стерильности. Thompson и Ryder (1961; US Department of Agriculture, Tech. Bulletin no. 1244), например, скрестили L. virosa с (L. serriola x L. sativa) гибридом, что дало стерильный межвидовый гибрид F1. Однако тетраплоидизация F1 и последующее скрещивание и диплоидизация позволили интрогрессировать признаки из L. virosa в L. sativa. Также эмбриональное спасение может применяться для восстановления жизнеспособных зародышей из межвидовых кроссов (Maisonneuve et al. 1995, Euphytica 85: 281-285). Таким образом, когда где-либо в описании делается ссылка на культивированные растения салат-латук (Z. sativa), содержащие один или более QTL, обеспечивающие Nr:1 резистентность, получаемые скрещиванием растения L. virosa с культивированным растением салат-латук, это может включать (но без ограничения к этому) стадии, которые преодолевают барьеры стерильности, такие как применение видов-посредников, эмбриональное спасение и/или обработка колхицином (удвоение хромосом).
Способ скрининга линий или сортов культивированного растения салат-латук на присутствие одного или более из QTL, выбранных из QTL6.1. OTL7.1 и/или QTL7.2 (или вариантов любого из них)
Способ подобен способу идентификации растения салат-латук дикого типа, содержащего один или более из QTL, но в данном способе культивированные растения салат-латук, т.е. растения L. sativa, скринируются.
Способ, таким образом, включает следующие стадии:
a) обеспечение культивированное растение салат-латук или множества культивированных растений салат-латук;
b) необязательно тестирование культивированного растения салат-латук или множества растений на Nr:1 резистентность в анализе на Nr:1 резистентность (например, свободного выбора и/или без выбора);
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных на стадии b), на присутствие одного или более маркеров, указывающих на QTL6.1 или его вариант и/или указывающих на QTL7.1 или его вариант и/или указывающих на QTL7.2 или его вариант; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
е) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе на№:1 резистентность.
Применяя этот метод, например, коммерческие конкурентные сорта могут быть проанализированы, чтобы определить содержат ли они один или более из QTL согласно настоящему изобретению.
Способ переноса одного или более из QTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или их варианты) из культивированного растения салат-латук (L. sativa) в другое культивированное растение салат-латук, например, в Nr:1 восприимчивую линию или сорт растения салат-латук
QTL согласно настоящему изобретению могут, конечно, также быть перенесены из одного культивированного растения L. sativa в другое культивированное растение L. sativa, чтобы получить различные типы и различные сорта растения салат-латук, которые являются Nr:1 резистентными.
Этот способ включает стадии:
a) обеспечение растения L. sativa, содержащего один или более или все из QTL6.1, QTL7.1 и/или QTL7.2 или вариант любого из них;
b) скрещивание растения L. sativa согласно а) со вторым растением L. sativa;
c) сбор F1 семян из указанного кросса и необязательно самоопыление указанных F1 растений один или более раз с получением F2 или F3 или популяции дальнейшего самоопыления,
d) необязательно обратное скрещивание растения F1 или F2 или F3 или растения дальнейшего самоопыления со вторым растением L. sativa согласно b) с получением беккросс популяции,
e) необязательно самоопыление беккросс популяции один или более раз,
f) идентификация F1, F2, F3, растения дальнейшего самоопыления или беккросс растения, которые содержат один или более или все из маркеров генотип SNP, указывающих на фрагмент интрогрессии на хромосоме 6 (QTL6.1) и/или указывающих на фрагмент интрогрессии на хромосоме 7 (QTL7.1 и/или QTL7.2).
Фрагменты интрогрессии, содержащие QTL6.1, QTL7.1 и QTL7.2 (или варианты любого из них) могут быть перенесены все вместе или по отдельности в другое культивированное растение салат-латук.
В одном варианте второе культивированное растение салат-латук согласно b) является Nr:1 восприимчивым растением салат-латук, или по меньшей мере растением, не содержащим QTL, которые оно должно получить у донора согласно а).
Новое полученное растение салат-латук может снова быть любого типа и любой линии или разновидности. Таким образом, QTL могут быть перенесены путем традиционного разведения из одного культивированного растения салат-латук в другое, например, из кочанного салата в салат-ромен, из спаржевого салата в салат Bibb, из салата ромен в рассыпной салат и т.д., в ходе передачи размер фрагмента интрогрессии может быть уменьшен в результате рекомбинации, и некоторые маркеры могут, таким образом, не присутствовать в полученном растении.
Растения, полученные этим способом, также являются вариантом выполнения настоящего изобретения.
Таким образом, обеспечивается способ получения культивированного растения салат-латук, имеющего Nr:1 резистентность, включает стадии:
a) Обеспечение культивированного растения салат-латук, содержащего 1, 2, 3, 4, 5, или более SNP маркеров, указывающих на QTL6.1 (или вариант); и/или 1, 2, 3, 4, 5, или более SNP маркеров, указывающих на QTL7.2 (или вариант)и/или 1, 2, 3, 4, 5, или более SNP маркеров, указывающих на QTL7.1 (или вариант);
b) Скрещивание указанного культивированного растения салат-латук с другим культивированным растением салат-латук, которое восприимчиво к тли растения салат-латук Nr:1 с получением F1 семян;
c) Необязательно самоопыление растений, выращенных из семян F1 один или более раз с получением F2, F3 или потомства дальнейшего самоопыления;
d) Идентификация растений салат-латук, выращенных из F1, F2, F3, или следующего потомства самоопыления, которое имеет фенотип Nr:1 резистентности и/или которое содержит фрагмент интрогрессии или резистентность-обеспечивающую часть фрагмента интрогрессии;
e) Необязательно скрещивание указанного идентифицированного F1 потомства или самоопыление потомства до культивированного растения салат-латук согласно стадии b), с получением беккросс потомства;
f) Необязательно отбор беккросс потомства, которое имеет резистентность против биотипа Nr:1 и/или которое содержит фрагмент интрогрессии или резистентность-обеспечивающую часть фрагмента интрогрессии.
На стадии d) и/или f) маркеры, описанные в настоящей заявке, могут применяться.
Растение салат-латук, полученное этим способом, также охватывается настоящим изобретением.
Способ применения семян, депонированных под номером доступа NCIMB42086. или их потомства, для получения Nr:1 резистентного культивированного растения салат-латук
NCIMB42086 содержит все три QTL в гомозиготной форме и может, таким образом, применяться для получения линий или сортов культивированного растения салат-латук, содержащих один или более из QTL, как уже описано. Подобным образом, потомства NCIMB42086, которые сохраняют один или более из QTL, могут применяться для получения линий или сортов культивированного растения салат-латук, содержащих один или более из QTL.
Способ культивирования растений согласно настоящему изобретению, т.е. Nr:1 резистентных L. sativa растений, содержащих один или более из OTL, выбранных из QTL6.1, QTL7.1 и/или QTL7.2 (или их вариантов), в областях, где присутствует N. ribisnigri биотипа Nr:1
Растения согласно настоящему изобретению могут быть культивированы в областях естественного заражения Nr:1. Поскольку идентифицированные в настоящей заявке QTL обеспечиваются резистентность против N. ribisnigri биотипа Nr:1 не только в условиях свободного выбора, но также в условиях без выбора, резистентность является очень эффективной в поле, так как можно ожидать хорошие урожая даже в случаях, когда у насекомых нет другого выбора для кормления и размножения. Резистентность, которая присутствует только в условиях свободного выбора, рискованно применять, тля по-прежнему может выбирать растения для кормления и размножения в ситуациях, когда нет другого предпочтительного выбора.
Кроме того, QTL согласно изобретению, как показано, обеспечивают резистентность против различных изолятов биотипа Nr:1, происходящих из различных стран, как например Германия, Франция и Испания. Поэтому ожидается, что резистентность будет эффективной и долговечной в Германии, Франции, Испании, Великобритании и других европейских странах, а также в других странах мира, где биотип Nr:1 может быть найден.
В одном варианте QTL согласно изобретению также обеспечивают резистентность против биотипа Nr:0, по меньшей мере против европейских биотипов Nr:0 (т.е. по меньшей мере против немецкого, французского и испанского биотипов Nr:1). Таким образом, в одном варианте культивированные растения салат-латук согласно настоящему изобретению (содержащие один или более из QTL) являются резистентными против Nr:1 и по меньшей мере также против европейских биотипов Nr:0. В одном варианте растения являются восприимчивыми против US или Калифорнийских биотипов Nr:0, хотя это еще предстоит проверить.
Полевая резистентность растений согласно настоящему изобретению (содержащих один или более из QTL) против биотипа Nr:1, значительно выше (т.е. как измерено по значительно меньшими средними числами тлей), чем для восприимчивых контролей, таких как Mafalda. Это может быть, например, проверено в открытом поле на участках естественного заражения Nr:1 (например, в Murcia, Испания) путем посева или посадки растений салат-латук линии или сорта, содержащих один или более из фрагменты интрогрессии в их геноме, в поле, вместе с подходящими контролями, такими как восприимчивый сорт Mafalda и/или линия генетического контроля. Предпочтительно по меньшей мере около 10, 15, 20 или более растений на линию или сорт включены, а также по меньшей мере два или предпочтительно три репликата. Растения могут контролироваться еженедельно и после того, как на восприимчивом контроле будет обнаружено достаточное заражение (например, по мере мере 100 или более тли), можно подсчитать количество тлей растения салат-латук на репрезентативном числе растений каждой линии или сорта Предпочтительно, в полевых условиях среднее количество тлей биотипа Nr:1 на растениях согласно настоящему изобретению значительно меньше по сравнению с восприимчивыми контролями, такими как Mafalda. Среднее число тлей предпочтительно не определяется на молодых сеянцах (ниже стадии 3-4 настоящих листов), так как в Примерах было обнаружено, что у таких молодых растениях резистентность еще не полностью выражена.
Хотя растения NCIMB42086 (содержащие все три QTL в гомозиготной форме), как обнаружено, полностью свободны от тли Nr:1 в полевых испытаниях со свободным выбором и без выбора, проведенных в Испании, может быть (без ограничения рассуждением), что культивированное растение салат-латук, содержащее только один или два из QTL (или вариантов), или не все три интрогрессии, содержащие QTL (или варианты) в гомозиготной форме, могут быть не полностью резистентны против Nr:1. Поэтому, в одном варианте культивированные растения салат-латук согласно настоящему изобретению, содержащие один или более из QTL (QTL6.1, QTL7.1 и/или QTL7.2 или варианты любого из них) в гомозиготной или гетерозиготной форме, несут среднее число тлей биотипа Nr:1, равное или менее 50%, 49%, 48%, 47%, 46%, 45%, 44%, 43% 40%, 30%, 20%, 10%, 5%, 3%, 2% или 1%, от среднего числа тлей, обнаруженных на сорте Mafalda (или другом Nr:1 восприимчивом сорте, предпочтительно содержащем ген Nr), или на генетическом контроле, при выращивании при тех же условиях. Например, испытание свободного выбора или без выбора можно выполнить в поле, как описано в примерах, для определения среднего количества тлей Nr:1 на растениях по изобретению и на контрольных растениях.
В одном варианте культивированные растения салат-латук согласно настоящему изобретению содержат в среднем 50 или менее тлей N. ribisnigri биотипа Nr:1, или в среднем 40, 30, 20, или 10 или менее Nr:1 тлей. В другом варианте культивированные растения салат-латук согласно настоящему изобретению содержат (в среднем) ноль тлей Nr:1 или по существу ноль Nr:1 тлей (в среднем 5 тлей или менее), при выращивании в поле, тогда как контрольное растение, такое как Mafalda, содержит значительное число тлей биотипа Nr:1. Значительное число составляет по меньшей мере 100 тлей, в среднем, 150, 200, 250 или более.
В другом варианте культивированные растения салат-латук согласно настоящему изобретению содержат в среднем 50 или менее тлей N. ribisnigri (любого биотипа, т.е. биотипа Nr:0 и биотипа Nr:1), или в среднем 40, 30, 20, или 10 или менее тлей N. ribisnigri. В другом варианте культивированные растения салат-латук согласно настоящему изобретению содержат (в среднем) ноль тлей N. ribisnigri или по существу ноль тлей (в среднем 5 тлей или менее), при выращивании в поле в области, где как Nr:1, так и Nr:0 присутствуют, при этом контрольное растение, такое как Mafalda, содержит значительное число тлей биотипа Nr:1, и тогда как Nr:0 восприимчивый сорт содержит значительно число тли биотипа Nr:0. Значительно число составляет по меньшей мере 100 тлей, в среднем, 150, 200, 250 или более.
Культивированные растения салат-латук согласно настоящему изобретению могут быть любого типа. Они могут представлять собой типы зеленый салат или красный салат, зеленый и красный салат (например, пятнистый), салат babyleaf, салат типа Литтл Джем, рассыпной салат (также обозначается как разрезанный салат или салат в пучках), салат кочанный, салат Bibb, Салат Батавия (или Summercrisp), салат кочанный, салат-ромен (или салат кос) салат-латук, салат Crisphead (или салат айсберг) салат-латук, многолистный салат, салат группы Great Lakes, салат группы Vanguard, салат группы Salinas, салат группы Eastern (Ithaca), салат Celtuce или Stem или Latin и т.д. Они могут также быть межрыночного типа, например, признаки салата кос и салата айсберг, или признаки салата айсберг и салата кос и т.д. Это могут быть инбредные линии, F1 гибриды, двойные гаплоиды, трансгенные растения, мутантные растения и т.д.
В одном варианте фрагмент (фрагменты) интрогрессии, содержащие один или более из QTL 6.1, 7.1 и/или 7.2 (или вариантов) находится в гомозиготной форме в культивированном растении салат-латук согласно настоящему изобретению. Самоопыление один или более раз обеспечивает то, что фрагменты интрогрессии находятся в гомозиготной форме, и SNP маркер (маркеры) затем также показывают гомозиготный генотип.
В другом варианте культивированное растение салат-латук согласно настоящему изобретению обладает хорошей плодовитостью и легко кроссируется с другими культивированными сортами или сортами растения салат-латук. Предпочтительно фрагменты любого дикого генома (например, L. virosa), которые совместно вводятся с QTL и которые придают любые отрицательные характеристики культивированному растению, такие как низкая плодовитость и/или карликовый рост, удаляются. Это можно сделать, выбирая рекомбинанты, имеющие более короткий фрагмент интрогрессии, но сохраняющие часть Nr:1-резистентности.
Растения согласно настоящему изобретению могут применяться для получения потомства (или потомков), которые содержат или сохраняют QTL (или варианты) и фенотип Nr:1 резистентности. Для получения потомства культивированное растение салат-латук согласно настоящему изобретению могут быть самоопылены и/или скрещены один или более раз с другим растением салат-латук, и семена могут быть собраны.
Также обеспечиваются семена, из которых можно выращивать любое из растений согласно настоящему состоянию.
В одном варианте выполнения настоящего изобретения обеспечивается применение растения салат-латук, представительные семена которого были депонированы под номером доступа NCIMB 42086, или его потомства (например, полученного путем самоопыления), для получения Nr:1 резистентного культивированного растения салат-латук.
В другом варианте выполнения настоящего изобретения обеспечивается применение культивированного растения салат-латук, содержащего фенотип Nr:1 резистентности, обеспеченный одним или более QTL, как обнаружено в/как получают из семян, депонированных под номером доступа NCIMB 42086, или из их потомства (например, полученного самоопылением), для получения Nr:1 резистентного культивированного растения салат-латук.
Семена
Также обеспечиваются семена, из которых можно выращивать любое из растений согласно настоящему изобретению, как и контейнеры или упаковки, включающие или содержащие такие семена. Семена можно отличить от других семян благодаря присутствию одного или более QTL (как можно проверить с использованием анализов молекулярных маркерных, описанных в настоящей заявке) и фенотипически.
В одном варианте выполнения семена упаковывают в маленькие и/или большие контейнеры (например, мешки, коробки, банки и т.д.). Семена можно гранулировать перед упаковкой (и формировать пилюли или гранулы) и/или обрабатывать различными соединениями, как например покрытие семян.
Гранулирование семян можно объединять с покрытием пленкой (Halmer, Р. 2000. Commercial seed treatment technology. In: Seed technology and its biological basis. Eds: Black, M. and Bewley, J. D., pages 257-286). Гранулирование создает круглые или округленные формы, которые легко высеваются на современных посевных машинах. Смесь для гранулирования обычно содержит семена и по меньшей мере склеивающее вещество и наполнитель. Последним может быть, например, глина, слюда, мел или целлюлоза. Кроме того, некоторые добавки могут быть включены для улучшения конкретных свойств пеллеты, например, композиция для обработки семян, содержащая по меньшей мере один инсектицидное, акарицидное, нематоцидное или фунгицидное соединение может быть добавлено непосредственно в смесь для гранулирования или в отдельные слои. Композиция для обработки семян может включать только один из этих типов соединений, смесь двух или более одного и того же типов соединений или смесь одного или более из одного и того же типов соединений с по меньшей мере одним другим инсектицидом, акарицидом, нематоцидом или фунгицидом.
Композиции, особенно подходящие для применения для обработки семян могут быть добавлены к семенам в виде пленочного покрытия, включая также возможность использования покрытия в или на пеллете, а также включая композицию для обработки семян непосредственно в смесь пеллет. Характерно, что пленочное покрытие представляет собой однородную, без пыли, водопроницаемую пленку, равномерно покрывающую поверхность всех отдельных семян (Halmer, Р. 2000. Commercial seed treatment technology. In: Seed technology and its biological basis. Eds: Black, M. and Bewley, J.D., pages 257-286). Помимо композиции, покрывающая смесь обычно также содержит другие ингредиенты, такие как вода, склеивающее вещество (обычно полимер), наполнительные материалы, пигменты и определенные добавки для улучшения конкретных свойств покрытия. Несколько покрытий можно комбинировать на одном семени.
Кроме того, возможны несколько комбинаций с пленочным покрытием: пленочное покрытие может быть добавлено на внешней стороне пеллеты, между двумя слоями материала для гранулирования и непосредственно на семена перед добавлением материала для гранулирования. Также более одного слоя пленочного покрытия могут быть включены в одну пеллету. Конкретным типом гранулирования является инкрустация. В этой методике применяется меньше наполнителя, и результатом является 'минипеллета'.
Семена также могут быть загрунтованы. Из всех коммерчески посаженных семян овощей салат-латук наиболее часто грунтуется. Грунтование представляет собой процесс на водной основе, который выполняется на семенах для повышения однородности прорастания и выхода из почвы и, таким образом, для укрепления растительного слоя. Грунтование уменьшает промежуток времени между появлением первого и последнего саженцев. Способы приготовления семян растения салат-латук хорошо известны в данной области техники (смотрите, например, Hill et al HortScience 42(6): 1436, 2007).
Части растения и вегетативные размножения
В другом варианте части растения, полученные из (получаемые из) растения согласно настоящему изобретению обеспечиваются в настоящей заявке, и контейнеры или упаковки, содержащие указанные части растения. Любые части растения могут отличаться от других частей растения салат-латук часть растения присутствием рекомбинантной хромосомы 6 и/или 7, т.е. присутствием фрагмента интрогрессии из растения салат-латук дикого типа, например, из L. virosa, на хромосоме 6 и/или 7. Это можно легко проверить по присутствию одного или более или всех маркеров, описанных в настоящей заявке.
В предпочтительном варианте выполнения настоящего изобретения частями растения являются листья или головки культивированных растений салат-латук согласно настоящему изобретению, предпочтительно собранные листья или головки, или их части.
Другие части растений согласно настоящему изобретению включают стебли, черенки, черешки, семядоли, цветы, пыльники, пыльцу, завязи, корни, кончики корней, протопласты, каллюс, микроспоры, цветоножки, семечки, побеги, семена, зародыши, зародышевые мешки, клетки, меристемы, почки и т.д. Семена включают, например, семена, полученные на растении согласно изобретению после самоопыления или после опыления пыльцой с другого растения салат-латук.
В другом варианте частью растения является растительная клетка или растительная ткань. В еще другом варианте, частью растения нерегенерируемая клетка или регенерируемая клетка. В другом варианте растительной клеткой является соматическая клетка. В другом варианте растительной клеткой является репродуктивная клетка, такая как семяпочка или пыльца. Эти клетки являются гаплоидными. Когда они регенерируются в целые растения, они включают гаплоидный геном исходного растения. Если происходит удвоение хромосом (например, посредством химической обработки), двойное гаплоидное растение может быть регенерировано. В одном варианте растением согласно настоящему изобретению является гаплоидное или двойное гаплоидное растение салат-латук.
Более того, согласно настоящему изобретению обеспечивается in vitro клеточная культура или тканевая культура растений салат-латук согласно настоящему изобретению, где клеточная или тканевая культура происходит из частей растения, описанных выше, таких как, например и без ограничения к этому, листья, пыльца, зародыши, семядоли, гипокотилии, меристематические клетки, корни, кончики корней, пыльники, цветы, семена или стебли, соматические клетки, репродуктивные клетки. Например, отрезанные части листа, гипокотили или ствола могут применяться в качестве тканевой культуры.
В конкретном варианте обеспечивается in vitro клеточная культура или тканевая культура растений салат-латук согласно настоящему изобретению, где клеточная или тканевая культура происходит из частей растения, описанных выше, где такие части растения не содержат репродуктивные клетки. В другом варианте выполнения настоящего изобретения, клеточная культура или тканевая культура не содержит регенерируемые клетки. В одном варианте обеспечиваются неразмножающиеся клетки и клеточная культура или тканевая культура, содержащая или состоящая из неразмножающихся клеток согласно настоящему изобретению.
Также обеспечиваются растения салат-латук, регенерированные из вышеописанных частей растения или регенерированные из вышеописанных клеточных или тканевых культур, причем указанное регенерированное растение имеет Nr:1 резистентность, т.е. сохраняет фрагмент (фрагменты) интрогрессии, придающие Nr:1 резистентность. Эти растения также могут обозначаться как вегетативно размножающиеся растения согласно настоящему изобретению.
Также обеспечиваются собранные листья и/или головки растений согласно настоящему изобретению и упаковки, содержащие множество листьев и/или головок растений согласно настоящему изобретению, как например 1, 2, 3, 4, 5, 10, 12, 20 головок.
Настоящее изобретение также обеспечивает пищевой или кормовой продукт, содержащий или состоящий из части растения, описанной в настоящей заявке. Пищевой или кормовой продукт может быть свежим или обработанным, например, консервированным, приготовленным на пару, вареным, жареным, бланшированным и/или замороженным и т.д. Примерами являются салаты или салатные смеси, содержащие листья или части листьев растений согласно настоящему изобретению.
Растение салат-латук согласно настоящему изобретению или его потомство, сохраняющее фенотип Nr:1 резистентности и фрагмент (фрагменты) интрогрессии, и части вышеуказанных растений, могут быть упакованы подходящим образом для, например, транспортировки и/или проданы свежими. Такие части охватывают любые клетки, ткани и органы, получаемые из саженцев или растений, таких как, но не ограничиваясь ими: головки, черенки, пыльца, листья, части листьев и подобное. Головки и листья могут быть собраны в виде маленького листа или позже. Растение, растения или их части могут быть упакованы в контейнер (например, мешки, коробки, банки и т.д.) отдельно или вместе с другими растениями или материалами. Части можно хранить и/или далее обрабатывать. Поэтому также охватываются пищевые или кормовые продукты, содержащие одну или более из таких частей, таких листьев или их частей, получаемых из растения согласно настоящему изобретению, его потомства и частей вышеуказанных растений. Например, настоящее изобретение также обеспечивает контейнеры, такие как банки, коробки, ящики, сумки, картонные коробки, упаковка с модифицированной атмосферой, пленки (например, биоразлагаемые пленки), и т.д., содержащие части растений (свежие и/или обработанные) согласно настоящему изобретению.
Растения и потомства (потомки)
В другом варианте выполнения настоящего изобретения обеспечиваются растения и части растений салат-латук согласно настоящему изобретению, и потомство растений салат-латук согласно настоящему изобретению, например, выращенные из семян, полученные путем полового или вегетативного размножения, регенерированные из вышеуказанных частей растений, или регенерированные из клеточной или тканевой культуры, где репродуцированное (размножение семенами или вегетативное размножение) растение содержит фенотип Nr:1 резистентности (и, таким образом, фрагмент (фрагменты) интрогрессии, придающие Nr:1 резистентность).
Как упомянуто выше, содержит или нет растение, потомство или потомство вегетативного размножения фенотип Nr:1 резистентности может быть протестировано фенотипически, применяя например, тест с выбором и/или тест без выбора, либо полевые испытания, либо испытания с контролируемой средой, как описано выше или в Примерах, и/или применяя молекулярные методики, такие как анализ молекулярных маркеров (применяя один или более или все маркеры, описанные в настоящей заявке), ДНК севенирование (например, секвенирование целого генома для идентификации L. virosa интрогрессии), роспись хромосомы и т.д.
В одном варианте выполнения настоящего изобретения, QTL Nr:1 резистентность, получаемый (получаемые) из (полученные из) NCIMB42086 или из других растений салат-латук дикого типа (например, других Nr:1 резистентных L. virosa экземпляров) может (могут) быть (комбинирован) комбинированы с другими генами, такими как другие гены Nr:1 резистентности (например, отдельные гены или QTL на различных хромосомах), с генами Nr:0 резистентности (например, ген Nr) или с другими признаками, как например резистентность против ложномучнистой росы, прель Sclerotinia, серая гниль Botrytis, настоящая мучнистая роса, антракноз, прикорневое побурение пасленовых, корневая гниль, мозаичный вирус салата, разрастание жилок салата, тля салата, вирус западной желтухи свеклы и желтуха астр. Резистентность против одного или более из следующих вредителей также может присутствовать или вводиться в растения согласно изобретению: резистентность против Sclerotinia minor (преждевременное осыпание листьев), Sclerotinia sclerotiorum (преждевременное осыпание листьев), Rhizoctonia solani (осыпание), Erysiphe cichoracearum (настоящая мучнистая роса), Fusarium oxysporum f. sp. lactucae (фузариозный вилт). Другие гены резистентности против патогенных вирусов (например, вирус инфекционной желтухи растения салат-латук(LIYV), мозаичный вирус растения салат-латук (LMV), мозаичный вирус огурца (CMV), вирус западной желтухи свеклы (BWYV), мозаичный вирус люцерны (AMV)), грибов, бактерий или вредителей растения салат-латук также могут вводиться.
Кроме того, настоящее изобретение обеспечивает потомство (или потомков), содержащих или сохраняющих обеспечивающий (обеспечивающие) Nr:1 резистентность QTL согласно настоящему изобретению, как например потомство, полученное, например, самоопылением один или более раз и/или перекрестным опылением растения согласно настоящему изобретению с другим растением салат-латук другого сорта или линии разведения, или с растением салат-латук согласно настоящему изобретению один или более раз. В частности, настоящее изобретение обеспечивает потомство, которое сохраняет QTL6.1, QTL7.1 и/или QTL7.2, как обнаружено в NCIMB 42086. В одном варианте настоящее изобретение обеспечивает потомственное растение, имеющее Nr:1 резистентность, такое как потомственное растение, которое получено из культивированного растения салат-латук согласно настоящему изобретению, имеющего Nr:1 резистентность, одним или более методами, выбранными из группы, состоящей из: самоопыление, скрещивание, мутация, получение двойного гаплоида или трансформация.
Мутацией могут быть спонтанные мутации или индуцированными человеком мутации или сомаклональные мутации. Смотрите, например, Моu 2011, Mutations in lettuce improvement, International Journal of Plant Genomics Volume 2011, Article ID 723518.
В одном варианте выполнения настоящего изобретения, растения или семена согласно настоящему изобретению могут быть мутированы (путем, например, облучения, химического мутагенеза, тепловая обработка, TILLING и т.д.) и/или мутированные семена или растения могут быть отобраны (например, природные варианты, сомаклональные варианты и т.д.), чтобы заменить одну или более характеристик растений. Подобным образом, растения согласно настоящему изобретению могут быть трансформированы и регенерированы, причем один или более химерных генов вводятся в растения. Трансформация может осуществляться, применяя стандартные методы, такие как трансформация, опосредованная Agrobacterium tumefaciens, или биолистическая трансформация, с последующим отбором трансформированных клеток и регенерацией в растения. Целевой признак (например, гены, обеспечивающие резистентность против вредителей или заболеваний, гербицидную, фунгицидную или инсектицидную толерантность и т.д.) может быть введен в растения, или его потомство, путем трансформации растения согласно настоящему изобретению или его потомства трансгеном, который обеспечивает целевой признак, где трансформированное растение сохраняет обеспечивающую Nr:1 резистентность интрогрессию (интрогрессии) и имеет целевой признак.
Обеспечивающий (обеспечивающие) Nr:1 резистентность QTL могут быть перенесены в потомство путем дальнейшего разведения, особенно в другие культивированные растения салат-латук. В одном варианте потомством является F1 потомство, полученное скрещиванием культивированного растения салат-латук согласно настоящему изобретению с другим растением, или S1 потомство, полученное самоопылением растения согласно настоящему изобретению. Также охватывается F2 потомство, полученное самоопылением F1 растений, F3, F4 или другие потомки, полученные самоопылением и/или обратным скрещиванием, которые сохраняют один или более QTL. "Дальнейшее разведение" охватывает традиционные методы разведения (например, самоопыление, скрещивание, обратное скрещивание), разведение, связанное с маркерами, и/или разведение с мутациями. В одном варианте выполнения настоящего изобретения, потомство включает QTL6.1, QTL7.1 и/или QTL7.2, как описано в NCIMB 42086.
В одном варианте настоящее изобретение охватывает гаплоидные растения и/или двойные гаплоидные растения согласно настоящему изобретению. Гаплоидные и двойные гаплоидные (DH) растения могут быть, например, получены посредством культивирования пыльника или микроспоры и регенерации в целое растение. Для получение DH может быть индуцировано удвоение хромосом с использованием известных методов, таких как обработка колхицином или тому подобное. Так, в одном варианте обеспечивается культивированное растение салат-латук, содержащее один или более обеспечивающих Nr:1 резистентность QTL, как описано, где растением является двойное гаплоидное растение.
В другом варианте выполнения настоящее изобретение относится к способу получения семени растения салат-латук, включающему скрещивание культивированного растения салат-латук согласно настоящему изобретению с самим растением или с другим растением салат-латук и сбор полученных семян. В другом варианте выполнения настоящее изобретение относится к семени, полученному согласно этому способу и/или к растению салат-латук, полученному путем выращивания таких семян.
Таким образом, в одном варианте обеспечивается потомство культивированного растения салат-латук согласно настоящему изобретению, где потомственное растение получают путем самоопыления, скрещивания, мутации, получения двойного гаплоида или трансформации, и где потомство сохраняет Nr:1 резистентность, описанную в настоящей заявке.
В еще одном варианте выполнения настоящее изобретение обеспечивает способ определения генотипа растения согласно настоящему изобретению, включающий обнаружение в геноме растения по меньшей мере первого полиморфизма (например, одного или более маркеров, описанных в настоящей заявке). Способ может, в определенных вариантах выполнения настоящего изобретения, включать обнаружение множества полиморфизмов в геноме растения (например, двух или более маркеров, описанных в настоящей заявке, указывающих на QTL6.1, QTL7.1, QTL7.2 или варианты любого из них). Например, образец нуклеиновой кислоты получают из растения и полиморфизма, или множество полиморфизмов обнаруживают в указанных нуклеиновых кислотах. Способ может также включать сохранение результатов стадии обнаружения множества полиморфизмов на машиночитаемом носителе.
Помимо представленных в настоящей заявке маркеров SNP, также больше молекулярных маркеров может быть разработано специалистом, например, между любыми маркеров, представленными в настоящей заявке, или другие новые маркеры, например, маркеры, связанные с вариантами QTL6.1, QTL7.1 и/или QTL7. 2. Специалисту в данной области техники известно как разработать молекулярные маркеры. Например, это может быть сделано путем скрещивания Nr:1 резистентного растения салат-латук согласно настоящему изобретению (или культивированного растения салат-латук или растения салат-латук дикого типа, такого как NCIMB42086) с Nr:1 восприимчивым растением салат-латук, и путем разработки расщепляющейся популяции (например, F2 или беккросс популяция) из этого кросса. Расщепляющаяся популяции может затем быть фенотипирована на Nr:1 резистентность и генотипирована с использованием, например, молекулярных маркеров, таких как SNP (однонуклеотидные полиморфизмы), AFLP (полиморфизм длин амплифицированных фрагментов; смотрите, например, ЕР 534858), или других, и с помощью программного обеспечения для анализа могут быть идентифицированы молекулярные маркеры, которые совместно расщепляются с фенотипом Nr:1 резистентности в расщепляющейся популяции.
В одном варианте экземпляр L. virosa дикого типа, из которого любой из QTL QTL6.1 или вариант, QTL7.1 или вариант и/или QTL7.2 или вариант интрогрессируются в культивированное растение салат-латук, не является следующими экземплярами: CGN13361, CGN16266, CGN16272, CGN04757, CGN04930, CGN04973, CGN16274, CGN21399 или CGN05148.
Информация по депонированию
QTL согласно настоящему изобретению были идентифицирован в, и получены из, потомстве образца экземпляра L. virosa дикого типа, PI273597 (источник US NGRP; National Genetic Resource Program, www.ars-grin.gov), происходящего из Германии (Baden  ). Nr:1 резистентное растение, идентифицированное в Примерах, приведенных далее, было выращено и размножено, и представительные примеры семян были депонированы Nunhems B.V. 5 декабря 2012 под номером доступа NCIMB 42086. Таким образом, всего 2500 семян L. virosa NCIMB 42086 было депонировано Nunhems B.V. 5 декабря 2012, в NCIMB Ltd., Ferguson Building, Craibstone Estate, Bucksburn, Aberdeen AB21 9YA, United Kingdom (NCIMB). Доступ к депозиту будет доступен в течение срока действия этой заявки лицам, определенным Директором Патентного ведомства США и имеющим на это право по запросу. Согласно 37 C.F.R. § 1.808(b), все ограничения, налагаемые хозяином депозита на доступность публикуемого материала, будут безвозвратно удалены после выдачи патента. Депозит будет храниться в течение 30 лет или 5 лет после последнего запроса или для продлеваемого срока действия патента, в зависимости от того, что больше, и будет заменен, если он когда-либо станет нежизнеспособным в течение этого периода. Заявитель не отказывается от каких-либо прав, предоставленных в соответствии с настоящим патентом согласно настоящей заявке или в соответствии с законом о защите сортов растений (7 USC 2321 et seq.).
). Nr:1 резистентное растение, идентифицированное в Примерах, приведенных далее, было выращено и размножено, и представительные примеры семян были депонированы Nunhems B.V. 5 декабря 2012 под номером доступа NCIMB 42086. Таким образом, всего 2500 семян L. virosa NCIMB 42086 было депонировано Nunhems B.V. 5 декабря 2012, в NCIMB Ltd., Ferguson Building, Craibstone Estate, Bucksburn, Aberdeen AB21 9YA, United Kingdom (NCIMB). Доступ к депозиту будет доступен в течение срока действия этой заявки лицам, определенным Директором Патентного ведомства США и имеющим на это право по запросу. Согласно 37 C.F.R. § 1.808(b), все ограничения, налагаемые хозяином депозита на доступность публикуемого материала, будут безвозвратно удалены после выдачи патента. Депозит будет храниться в течение 30 лет или 5 лет после последнего запроса или для продлеваемого срока действия патента, в зависимости от того, что больше, и будет заменен, если он когда-либо станет нежизнеспособным в течение этого периода. Заявитель не отказывается от каких-либо прав, предоставленных в соответствии с настоящим патентом согласно настоящей заявке или в соответствии с законом о защите сортов растений (7 USC 2321 et seq.).
Различные модификации и варианты описанных продуктов и способов согласно настоящему изобретению будут очевидны специалистам в данной области техники без отхода от объема и сущности изобретения. Хотя изобретение было описано в связи с конкретными предпочтительными вариантами выполнения, должно быть понятно, что изобретение как заявлено не должно быть чрезмерно ограничено такими конкретными вариантами выполнения. Действительно, различные модификации описанных режимов для осуществления настоящего изобретения, которые очевидны для специалистов в селекции растений, химии, биологии, патологии растений или связанных с ними областях, должны быть в пределах объема следующей формулы изобретения.
Настоящее изобретение далее дополнительно проиллюстрировано в следующих примерах, которые приведены только для иллюстрации и никоим образом не предназначены для ограничения изобретения.
Примеры
Пример 1 - Nr:1 резистентность в испытании со свободным выбором
1.1 Растительный материал
Семена высевали в пластиковые лотки (4×7 горшков). Пластиковые лотки заполнялись смесью грунта, состоящей из двух разных источников торфа. После высева лотки помещают в пластиковые коробки. Затем эти ящики помещались на полки в климатических камерах (цикл 16/8 часов с дневным и ночным циклом с использованием флуоресцентных ламп, интенсивность света составляла приблизительно 50 мкмоль.м-2.с-1 PAR (фотосинтетически активное излучение), температура для дневного/ночного периодов составляли 20/16°С; относительная влажность была постоянной до 80%). Во время посева в каждом горшке помещалось от одного до двух семян. Примерно через неделю после прорастания проращенные проростки разрежались по мере необходимости, т.е. на каждый горшок сохранялся только один проросток. Растения хранили в климатических камерах (условия окружающей среды, описанные выше, сохраняются на протяжении всего исследования) до конца эксперимента. Полив проводили по мере необходимости.
1.2 Материал многозачаткового растения
Многозачатковыми растениями являются растения, используемые с получением популяции тли, необходимой для испытания. Многозачатковые растения высевали на неделе 1. Семена проращивали и растения поддерживали так же, как тестируемый растительный материал. Выращенный в открытом поле салат кочанный сорта Mafalda (Nunhems / BayerCropScience Vegetable Seeds) применяли в качестве многозачаткового растения, но и другие Nr:0 резистентные растения могли применяться в равной степени.
1.3 Опытный образец
Климатическая камера может вмещать до шести передвижных стеллажей (MSU). Каждый блок состоял из трех полок/плато. На одной полке можно было хранить не более трех пластиковых ящиков, каждый из которых содержал до 28 растений.
Было проведено сравнение дикого экземпляра NCIMB42086 вместе с 13 другими экземплярами дикого вида на уровень резистентности против биотипа Nr:1. В климатической ячейке было использовано 36 коробок. Каждая коробка содержала два растения каждой линии/экземпляра растений (положения растений в каждой коробке были рандомизированы). Затем 36 ящиков были помещены на два нижних плато из шести MSU. Не были включены какие-либо известные восприимчивые кандидаты.
1.4 Насекомое
Один изолят Nr:1 тли первоначально происходил из поля с растениями салат-латук в области Пфальц в Германии. Другой изолят Nr:1 тли первоначально происходил из поля растений салат-латук около Перпиньяна, Франция.
Когда тля не нужна в целях испытания, небольшая колония насекомых была сохранена на «поддерживающих» растениях Mafalda (от 5 до 10 отдельных растений) в климатической ячейке. Растения хранились в одном ящике.
1.5 Получение инокулятов
Взращивание популяции тли, используемой для испытания (т.е. «инокулята»), было начато на 4-й неделе планирования. Взращивание было начато путем сбрасывания нимф и взрослых (бескрылых) тлей на вершине 3-недельных растений Mafalda. На коробку, содержащую 28 растений, было использовано около 500 тлей. Растения и тлю хранили в ящиках и хранили открытыми на полках в климатической камере с использованием описанных выше условий окружающей среды. Сразу после инокуляции популяцию тли оставляют развиваться на многозачатковых растениях в течение трех недель, т.е. до тех пор, пока они не потребуются для инокуляции согласно испытанию.
1.6 Инокуляция согласно испытанию
Тестируемые растения инокулировали на 7-й неделе, т.е. когда растениям было 3 недели.
Точно 20 тлей были перенесены на вершину каждого тестируемого растения. Это было достигнуто с помощью кисти, чтобы забрать насекомых с многозачатковых растений и поместить их на сердцевину тестируемых растений. Использовались только неопределенные нимфы и взрослые особи. Тли, которые были явно крылатыми, не были включены.
1.7 План испытания


1.8 Сбор и анализ результатов
Было подсчитано количество тлей, присутствующих на каждом отдельном растении. Подсчет производился еженедельно в течение 5 недель после инокуляции (смотрите план испытания выше).
1.9 Результаты испытания со свободным выбором
Результаты испытания с (свободным) выбором показаны на Фиг. 2А и 2В. Результаты как с немецкими (Фиг. 2А), так и с французскими (Фиг. 2В) Nr:1 изолятами показывают, что растения NCIMB 42086 несли очень мало тлей и, следовательно, имели высокий уровень Nr:1 резистентности. Первоначально средняя численность тли на NCIMB 42086 увеличивалась, но только до среднего числа 22 (немецкий изолят) и 22,5 (французский изолят). После первоначального увеличения числа тлей количество постоянно снижалось, пока они не достигли в среднем 1,95 (немецкий изолят) и 2,79 (французский изолят) через 5 недель на NCIMB 42086.
Таким образом, у молодых растений в возрасте около 8 недель практически не было обнаружено Nr:1 тли на NCIMB42086 в условиях свободного выбора.
Пример 2. Nr:1 резистентность в испытании без выбора
2.1 Описание протокола испытания Nr:1 без выбора
Протокол, используемый во время тестов без выбора, был почти идентичен протоколу в испытании с выбором в примере 1. Тем не менее различия присутствовали в параметрах испытуемого растительного материала, в климатической ячейке и в самой модели испытания.
В отличие от испытания с выбором, в испытании без выбора коробки индивидуально содержались в пластмассовых шатрах.
Кроме того, в каждой коробке/клетке использовался один растительный генотип (одиночные линии/экземпляры): другими словами, ни одна из коробок не содержала комбинации линий тестируемых растений.
Уровни резистентности, имеющиеся в NCIMB 42086 и трех других экземплярах, сравнивались в тесте без выбора. Сорт Mafalda использовался в качестве отрицательного контроля.
Для каждой линии растений использовались четыре клетки. Коробку, содержащую 18 растений одной линии, помещали в клетки. Затем клетки были помещены в климатическую ячейку полурандомизированным образом. Всего было использовано 20 клеток и 360 растений (72 растения на линию или экземпляр).
Инокуляцию тли и сбор результатов проводили, как описано в примере 1, за исключением того, что испытывали только немецкий Nr:1 изолят.
2.2 Результаты испытания без выбора
Результаты испытания без выбора показаны на Фиг. 1А и Фиг. 1 В, которые показывают больший масштаб.
Как можно видеть, NCIMB 42086 имеет резистентность против биотипа Nr:1, т.е. среднее число тлей значительно ниже, чем у восприимчивого контрольного сорта Mafalda. Первоначально среднее число тли слегка увеличилось, но не до выше 30 (для NCIMB 42086 оно составляло 28 через две недели), после чего оно постоянно снижалось до нуля.
Таким образом, у молодых растений в возрасте около 8 недель не было найдено Nr:1 тлей на NCIMB 42086 в условиях отсутствия выбора.
Пример 2. Полевые испытания полувзрослых и взрослых растений (с выбором и без выбора)
2.1 Полевое испытание со свободным выбором
2.1.1 Рост растения
Для каждого испытания семена высевали в отдельных смешанных компостных/торфяных «пробках» и проращивали в неотапливаемых теплицах. Примерно через 7-9 недель после посева пророщенные проростки были пересажены на приподнятые слои в поле. Поле находилось в провинции Мурсия, Испания. Полив проводили с использованием водопроводных труб, расположенных на верхушке поднятых слоев, среди рядов растений. Растения содержались в этом месте до окончания испытаний.
2.1.2 Модель испытания
Два независимых испытания были проведены в течение двух лет подряд, обозначается как Год 1 и Год 2 (Y1 и Y2 соответственно). Одну и ту же модель испытания применяли для обоих испытаний, и она подробно описана далее.
В поле были испытаны пять различных линий растений, среди которых NCIMB 42086 и контрольные сорта Mafalda и Scala (оба сорта семян овощей Nunhems/ Bayer CropScience). Как Mafalda, так и Scala являются Nr:0 резистентными, но Nr:1 восприимчивы. Scala относится к виду салат ко /салат-ромен типа.
Для каждого исследования использовали приподнятые слои. На каждом из поднятых слоев растения высевали с репликатами для 16 растений, и 5 репликатов высевали на каждый приподнятый слой. Тест был частично рандомизирован, т.е. в ходе испытания осуществляли пересаживание для обеспечения того, чтобы каждая из линий растений была представлена на каждом поднятом слое, но для каждого поднятого слоя порядок репликатов был рандомизирован.
2.1.3 Насекомое и инокуляция
Целью испытаний было исследование резистентности против N. ribisnigri Nr:1 в полевых условиях. Растения не целенаправленно инокулировали, но оставляли для заражения встречающейся в природе популяцией Nr:1 тли. Образцы насекомых были собраны в конце испытаний для идентификации под микроскопами.
2.1.4 Сбор и анализ результатов
Резистентность различных тестируемых линий растений оценивалась путем подсчета количества тлей, присутствующей на каждом отдельном растении. Подсчет был сделан через 14 недель после пересаживания в ходе испытания Y1 и через 11 недель после пересаживания в ходе испытания Y2
2.1.5 - Результаты полевого испытания со свободным выбором Год 1 и Год 2
В обоих испытаниях у взрослых растений (от 18 до 20 недель) NCIMB42086 вообще не было обнаружено тлей, тогда как контрольный сорт Mafalda и Nr:1 восприимчивый сорт Scala оба несли значительное количество тлей биотипа Nr:1.
2.2 Полевое испытание без выбора
2.2.1 - Рост растения
В течение испытания Год 2 в полевых условиях проводилось исследование без выбора, в клетке, наряду с испытанием со свободным выбором, описанным выше. Как описано выше, растения проращивали в неотапливаемой теплице перед переносом на приподнятые слои в полях через около 7-9 недель после посева. Вода также была предоставлена через трубки, расположенные среди растений на вершине поднятых слоев. Однако, в отличие от испытаний со свободным выбором, растения были заключены в клетки (примерно по 10 м3 каждая), оборудованные сеткой, защищающей от насекомых (размер сетки был достаточным для предотвращения проникновения в нее трипсов).
2.2.2 - Модель испытания
В поле использовались две отдельные клетки. В каждой клетке использовались два приподнятых слоя. На каждом слое помещали 20 растений, т.е. 40 растений в клетке.
В каждую клетку была пересажена одна линия растений, т.е. NCIMB42086 или Mafalda.
2.2.3 Насекомое и инокуляция
N. ribisnigri тля, необходимая для инокуляции тестируемого растительного материала, была получена на участках растений Mafalda, произрастающих в близлежащем поле. Участки пассивно заражали естественной популяцией Nr:1 тли.
В день инокуляции (растениям было около 8 недель) с растений Mafalda собирали бескрылых взрослых особей. Собранные тли затем переносили индивидуально на верхнюю часть каждого тестируемого растения с помощью кисти. Всего на каждое тестируемое растение было нанесено 10 насекомых.
2.2.4 - Сбор и анализ данных
Резистентность различных тестируемых линий растений оценивали путем подсчета количества тлей, присутствующих на каждом отдельном растении. Подсчет был сделан приблизительно через 3 недели после инокуляции.
2.2.5 Результаты полевого испытания без выбора полувзрослых растений (растения в возрасте около 11 недель)
Результаты показаны на Фиг. 4. NCIMB42086 вообще не нес тлю, тогда как контрольный сорт Mafalda нес значительное количество тлей биотипа Nr:1 (>300)
2.2.6 Выводы для полевых испытаний свободного выбора и без выбора полувзрослых и взрослых растений
Результаты показывают, что резистентность Nr:1, обнаруженная на молодых растениях (Пример 1, растения в возрасте около 8 недель) NCIMB 42086 в климатических ячейках, является высокоэффективной в полевых условиях как в испытаниях свободного выбора, так и в испытаниях без выбора полувзрослых и взрослых растений. Резистентность в поле оказалась полной (вообще нет тли), как в условиях свободного выбора, так и без выбора. Таким образом, NCIMB42086 является резистентным к различным популяциям Nr:1, немецким, французским и испанским.
Пример 3. QTL картирование Nr:1 резистентности
Чтобы определить, сколько генетических локусов и какие генетические локусы ответственны за Nr:1 резистентность NCIMB42086, NCIMB42086 был скрещен с восприимчивым растением салат-латук N. ribisnigri. F1 был обратно скрещен с родителем L. sativa, чтобы генерировать популяцию ВС1, которая использовалась для картирования QTL.
Данные фенотипирования (общее число тлей биотипа Nr:1 на растение) собирали в два момента времени после инокуляции, две недели и три недели после инокуляции тлями биотипа Nr:1.
Результаты
Были идентифицированы три QTL, один на хромосоме 6 и два на хромосоме 7, как показано ниже и на Фиг. 3. Физическое положение на хромосомах основано на опубликованном геноме растения салат-латук, lgr.genomecenter.ucdavis.edu.
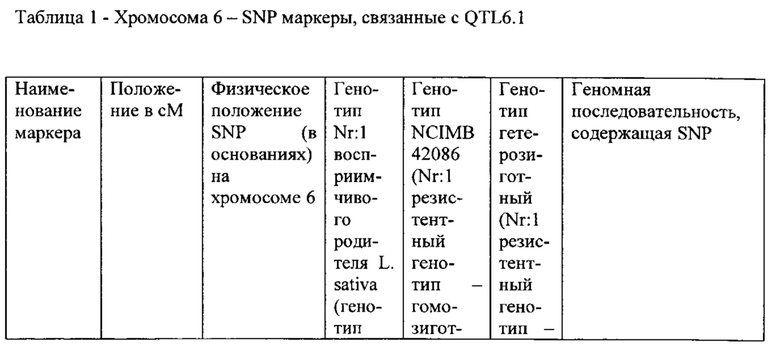
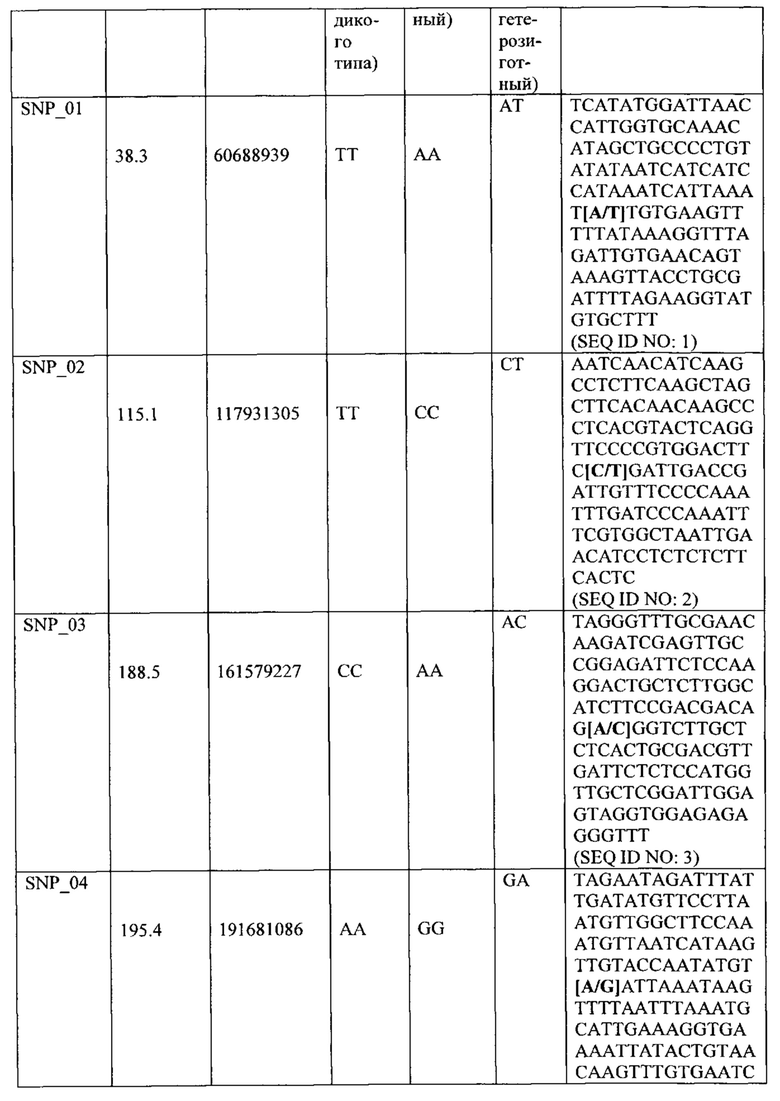
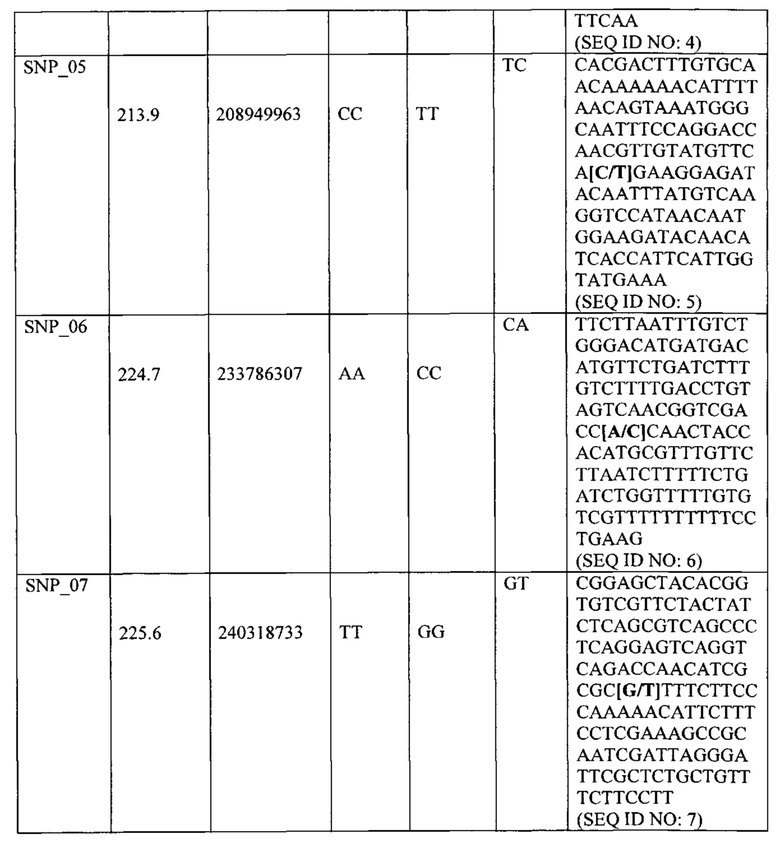

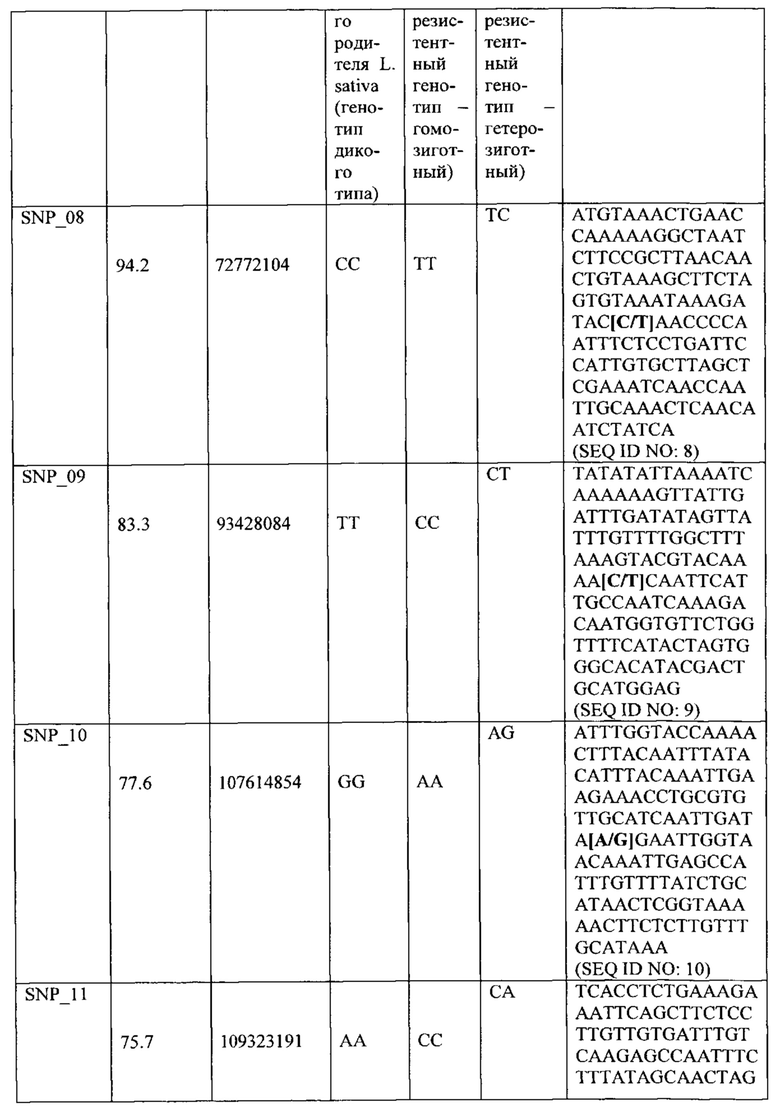
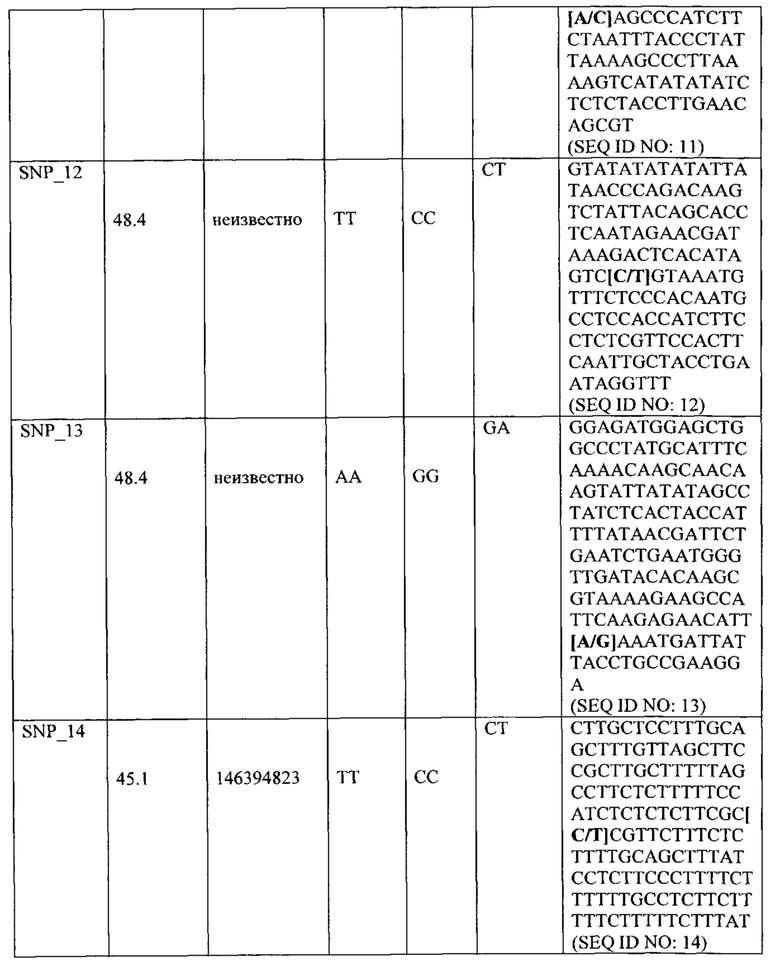
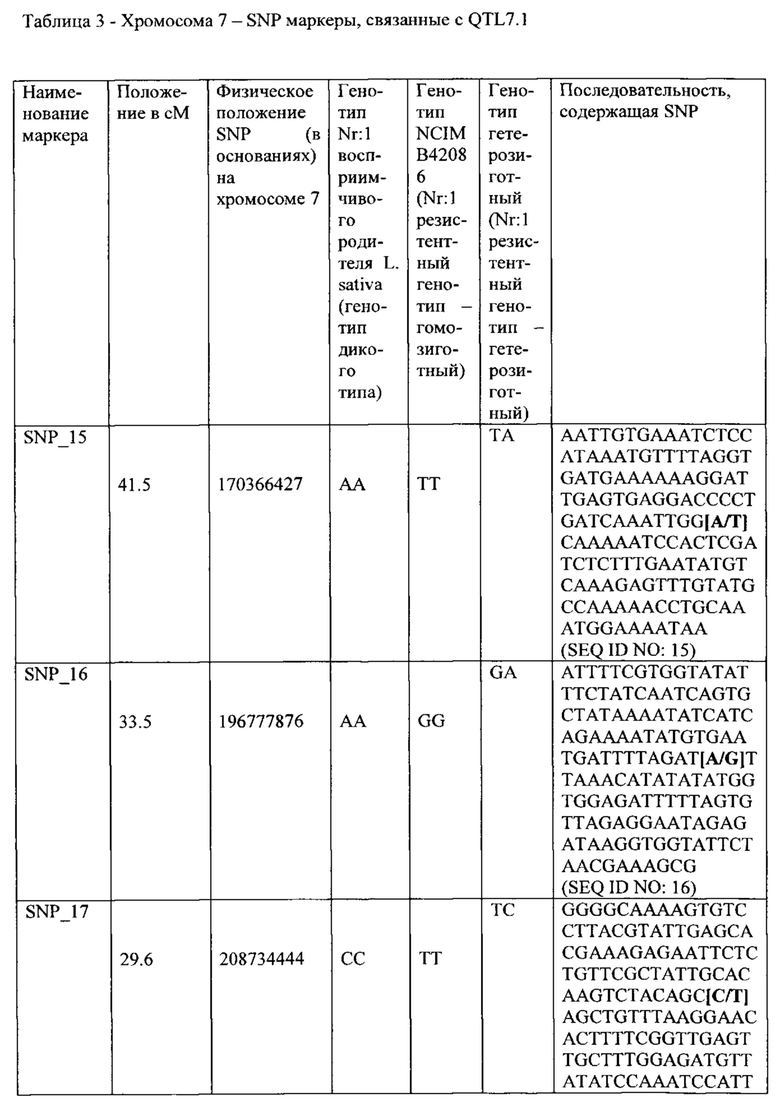
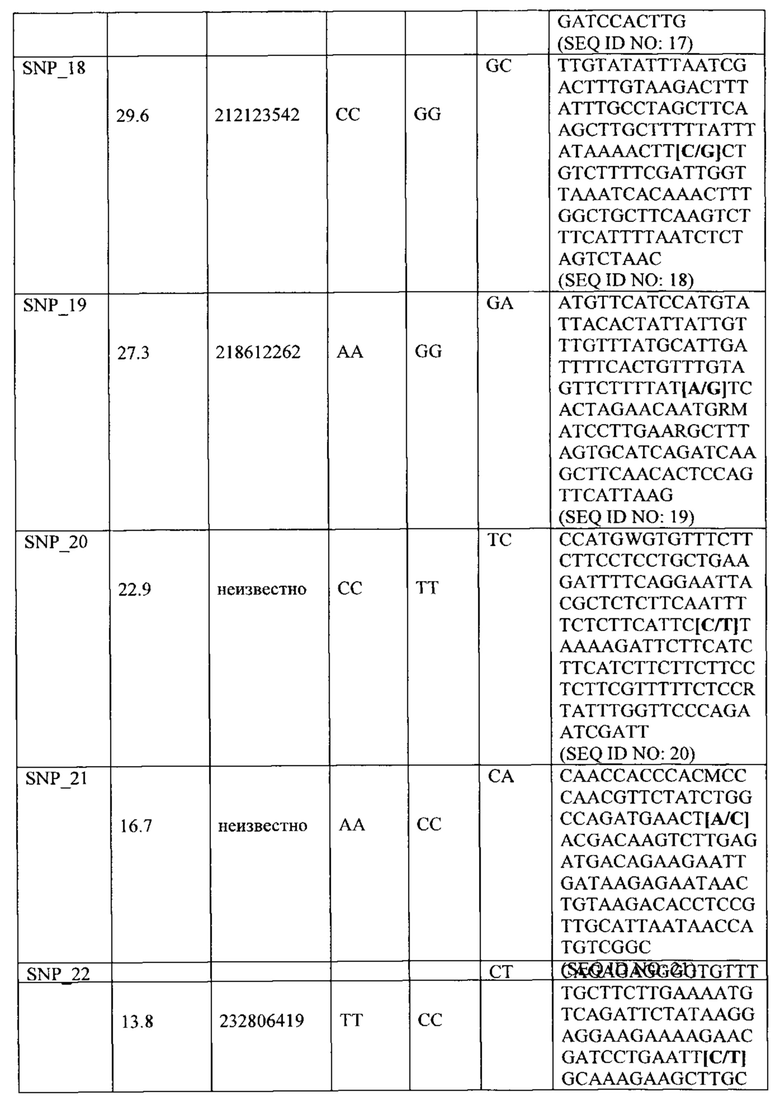
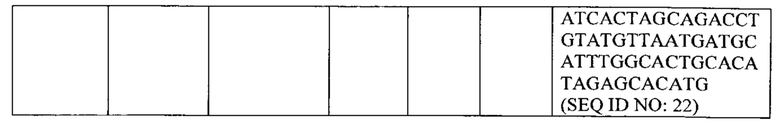
Пример 4 - картирование QTL Nr:1 резистентности
Новое фенотипирование и анализ маркеров провели в двух бэккросс популяциях, чтобы нанести на генетическую карту Nr:1 резистентность. Фенотипирование проводили, как описано выше.
Два QTL со значительной оценкой LOD были нанесены на генетическую карту на хромосоме 6 и на хромосоме 7, содержащих QTL6.1 и QTL7.1. Результаты также показаны на Фиг. 3В.
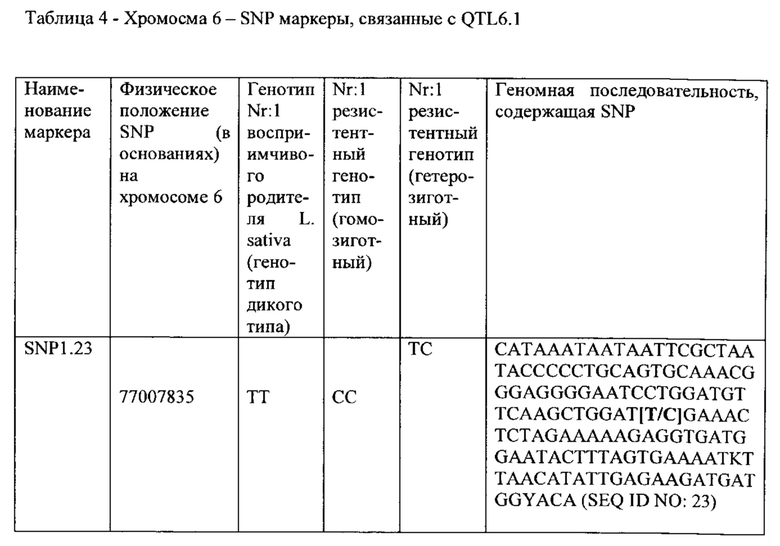
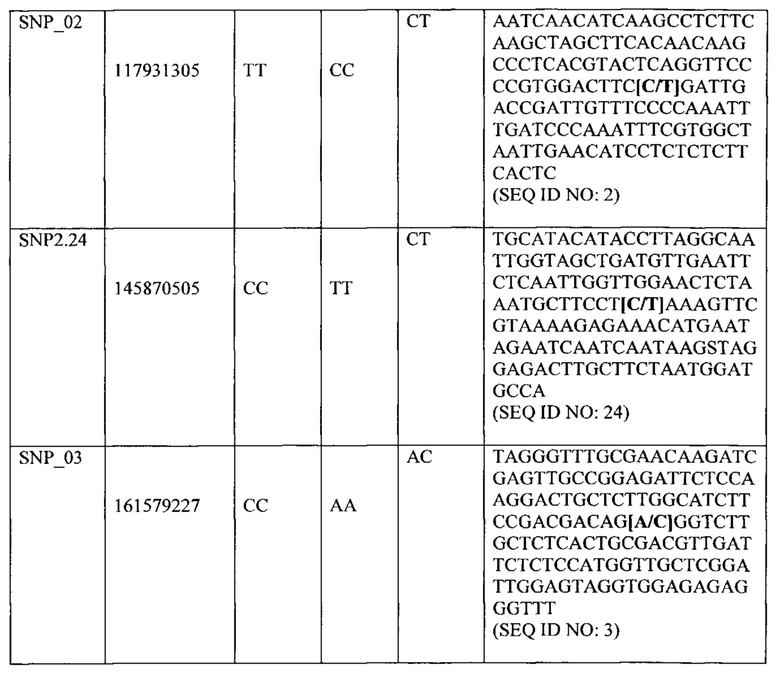
QTL6.1 и QTL7.1 были идентифицированы в двух разных типах Nr:1 резистентных экземпляров L. virosa, и были идентифицированы специфичные для экземпляров маркеры, которые могут различать интрогрессии и происходить из интрогрессии. Маркеры VSP1 и VSP2 идентифицируются в одном типе экземпляра, представленном NCIMB42086, и маркеры VSP3 и VSP4 в другом типе экземпляра.

Пример 5 - эксперимент в клетке
Два экземпляра L. virosa дикого типа, выше, имеют одинаково высокие уровни Nr:1 резистентности, но NCIMB 42086 обладает еще лучшей резистентностью, поскольку развитие нимф Nr:1 остановлено ранее на растениях NCIMB 42086 по сравнению с другим экземпляром L. Virosa, как это было сделано в эксперименте с клеткой.
Эксперимент проводился с использованием нимф в возрасте 1 день. На одно растение была помещена одна клетка, содержащая по меньшей мере 3 взрослых тли. Через один день после инокуляции все взрослые особи удаляли, оставляя только 3 нимфы на клетку. Через двенадцать дней после удаления взрослых особей подсчитывали количество взрослых (выживших) особей, количество новых нимф и кожи. Эксперимент проводился согласно полностью рандомизированной схеме с 28 репликатами (1 клетка/растение). Переменные были трансформированы и проанализированы с помощью ANOVA с последующим тестированием LSD.
Выживаемость и количество новых нимф было равным нулю для обоих экземпляров L. virosa дикого типа, тогда как для восприимчивого контроля составляли 0,86 (средняя выживаемость) и 17,57 (среднее число новых нимф). Однако экземпляры дикого типа отличались средним числом сброшенной кожи (среднее число для NCIMB42086 составляло 0,85, тогда как среднее число для другого экземпляра составляло 2,11, а для восприимчивого контроля 5,85), причем NCIMB42086 имел значительно более низкое среднее число сброшенной кожи, показывая, что развитие нимфы остановлено ранее для NCIMB42086.
--->
Перечень последовательностей
<110> Nunhems B.V.
<120> РАСТЕНИЯ САЛАТ-ЛАТУК, ИМЕЮЩИЕ РЕЗИСТЕНТНОСТЬ ПРОТИВ NASONOVIA RIBISNIGRI БИОТИПА 1
<130> BCS14-8019
<150> 14191135.4
<151> 2014-10-30
<160> 29
<170> PatentIn version 3.5
<210> 1
<211> 141
<212> ДНК
<213> Lettuce
<400> 1
tcatatggat taaccattgg tgcaaacata gctgcccctg tatataatca tcatccataa 60
atcattaaat atgtgaagtt tttataaagg tttagattgt gaacagtaaa gttacctgcg 120
attttagaag gtatgtgctt t 141
<210> 2
<211> 141
<212> ДНК
<213> lettuce
<400> 2
aatcaacatc aagcctcttc aagctagctt cacaacaagc cctcacgtac tcaggttccc 60
cgtggacttc cgattgaccg attgtttccc caaatttgat cccaaatttc gtggctaatt 120
gaacatcctc tctcttcact c 141
<210> 3
<211> 141
<212> ДНК
<213> lettuce
<400> 3
tagggtttgc gaacaagatc gagttgccgg agattctcca aggactgctc ttggcatctt 60
ccgacgacag aggtcttgct ctcactgcga cgttgattct ctccatggtt gctcggattg 120
gagtaggtgg agagagggtt t 141
<210> 4
<211> 141
<212> ДНК
<213> lettuce
<400> 4
tagaatagat ttattgatat gttccttaat gttggcttcc aaatgttaat cataagttgt 60
accaatatgt gattaaataa gttttaattt aaatgcattg aaaggtgaaa attatactgt 120
aacaagtttg tgaatcttca a 141
<210> 5
<211> 141
<212> ДНК
<213> lettuce
<400> 5
cacgactttg tgcaacaaaa aacattttaa cagtaaatgg gcaatttcca ggaccaacgt 60
tgtatgttca tgaaggagat acaatttatg tcaaggtcca taacaatgga agatacaaca 120
tcaccattca ttggtatgaa a 141
<210> 6
<211> 141
<212> ДНК
<213> lettuce
<400> 6
ttcttaattt gtctgggaca tgatgacatg ttctgatctt tgtcttttga cctgtagtca 60
acggtcgacc ccaactacca catgcgtttg ttcttaatct ttttctgatc tggtttttgt 120
gtcgtttttt ttttcctgaa g 141
<210> 7
<211> 141
<212> ДНК
<213> lettuce
<400> 7
cggagctaca cggtgtcgtt ctactatctc agcgtcagcc ctcaggagtc aggtcagacc 60
aacatcgcgc gtttcttccc aaaaacattc tttcctcgaa agccgcaatc gattagggat 120
tcgctctgct gtttcttcct t 141
<210> 8
<211> 141
<212> ДНК
<213> lettuce
<400> 8
atgtaaactg aaccaaaaag gctaatcttc cgcttaacaa ctgtaaagct tctagtgtaa 60
ataaagatac taaccccaat ttctcctgat tccattgtgc ttagctcgaa atcaaccaat 120
tgcaaactca acaatctatc a 141
<210> 9
<211> 141
<212> ДНК
<213> lettuce
<400> 9
tatatattaa aatcaaaaaa gttattgatt tgatatagtt atttgttttg gctttaaagt 60
acgtacaaaa ccaattcatt gccaatcaaa gacaatggtg ttctggtttt catactagtg 120
ggcacatacg actgcatgga g 141
<210> 10
<211> 141
<212> ДНК
<213> lettuce
<400> 10
atttggtacc aaaactttac aatttataca tttacaaatt gaagaaacct gcgtgttgca 60
tcaattgata agaattggta acaaattgag ccatttgttt tatctgcata actcggtaaa 120
aacttctctt gtttgcataa a 141
<210> 11
<211> 141
<212> ДНК
<213> lettuce
<400> 11
tcacctctga aagaaattca gcttctcctt gttgtgattt gtcaagagcc aatttcttta 60
tagcaactag cagcccatct tctaatttac cctattaaaa gcccttaaaa gtcatatata 120
tctctctacc ttgaacagcg t 141
<210> 12
<211> 142
<212> ДНК
<213> lettuce
<400> 12
gtatatatat attataaccc agacaagtct attacagcac ctcaatagaa cgataaagac 60
tcacatagtc cgtaaatgtt tctcccacaa tgcctccacc atcttcctct cgttccactt 120
caattgctac ctgaataggt tt 142
<210> 13
<211> 160
<212> ДНК
<213> lettuce
<400> 13
ggagatggag ctggccctat gcatttcaaa acaagcaaca agtattatat agcctatctc 60
actaccattt tataacgatt ctgaatctga atgggttgat acacaagcgt aaaagaagcc 120
attcaagaga acattgaaat gattattacc tgccgaagga 160
<210> 14
<211> 141
<212> ДНК
<213> lettuce
<400> 14
cttgctcctt tgcagctttg ttagcttccg cttgcttttt agccttctct ttttccatct 60
ctctcttcgc ccgttctttc tcttttgcag ctttatcctc ttcccttttc ttttttgcct 120
cttctttttc tttttcttta t 141
<210> 15
<211> 141
<212> ДНК
<213> lettuce
<400> 15
aattgtgaaa tctccataaa tgttttaggt gatgaaaaaa ggattgagtg aggacccctg 60
atcaaattgg tcaaaaatcc actcgatctc tttgaatatg tcaaagagtt tgtatgccaa 120
aaacctgcaa atggaaaata a 141
<210> 16
<211> 141
<212> ДНК
<213> lettuce
<400> 16
attttcgtgg tatatttcta tcaatcagtg ctataaaata tcatcagaaa atatgtgaat 60
gattttagat gttaaacata tatatggtgg agatttttag tgttagagga atagagataa 120
ggtggtattc taacgaaagc g 141
<210> 17
<211> 141
<212> ДНК
<213> lettuce
<400> 17
ggggcaaaag tgtccttacg tattgagcac gaaagagaat tctctgttcg ctattgcaca 60
agtctacagc tagctgttta aggaacactt ttcggttgag ttgctttgga gatgttatat 120
ccaaatccat tgatccactt g 141
<210> 18
<211> 141
<212> ДНК
<213> lettuce
<400> 18
ttgtatattt aatcgacttt gtaagacttt atttgcctag cttcaagctt gctttttatt 60
tataaaactt gctgtctttt cgattggtta aatcacaaac tttggctgct tcaagtcttt 120
cattttaatc tctagtctaa c 141
<210> 19
<211> 141
<212> ДНК
<213> lettuce
<400> 19
atgttcatcc atgtattaca ctattattgt ttgtttatgc attgattttc actgtttgta 60
gttcttttat gtcactagaa caatgrmatc cttgaargct ttagtgcatc agatcaagct 120
tcaacactcc agttcattaa g 141
<210> 20
<211> 142
<212> ДНК
<213> lettuce
<400> 20
ccatgwgtgt ttcttcttcc tcctgctgaa gattttcagg aattacgctc tcttcaattt 60
tctcttcatt cttaaaagat tcttcatctt catcttcttc ttcctcttcg tttttctccr 120
tatttggttc ccagaatcga tt 142
<210> 21
<211> 121
<212> ДНК
<213> lettuce
<400> 21
caaccaccca cmcccaacgt tctatctggc cagatgaact cacgacaagt cttgagatga 60
cagaagaatt gataagagaa taactgtaag acacctccgt tgcattaata accatgtcgg 120
c 121
<210> 22
<211> 141
<212> ДНК
<213> lettuce
<400> 22
cagagagggg tgttttgctt cttgaaaatg tcagattcta taaggaggaa gaaaagaacg 60
atcctgaatt cgcaaagaag cttgcatcac tagcagacct gtatgttaat gatgcatttg 120
gcactgcaca tagagcacat g 141
<210> 23
<211> 141
<212> ДНК
<213> lettuce
<400> 23
cataaataat aattcgctaa taccccctgc agtgcaaacg ggaggggaat cctggatgtt 60
caagctggat cgaaactcta gaaaaagagg tgatggaata ctttagtgaa aatkttaaca 120
tattgagaag atgatggyac a 141
<210> 24
<211> 141
<212> ДНК
<213> lettuce
<400> 24
tgcatacata ccttaggcaa ttggtagctg atgttgaatt ctcaattggt tggaactcta 60
aatgcttcct taaagttcgt aaaagagaaa catgaataga atcaatcaat aagstaggag 120
acttgcttct aatggatgcc a 141
<210> 25
<211> 141
<212> ДНК
<213> lettuce
<400> 25
cgcatcatcg acctctcatt taattcattt tctggtgatc tgccacatca gtacttccag 60
gattggtcag taatgaagga gacaaaacaa aatgcagcat atatgcaakc aaatgttgat 120
attttggggg aaargtacat c 141
<210> 26
<211> 141
<212> ДНК
<213> lettuce
<400> 26
ttcatgcttc tcactccatg tgtaagtagc tcctttatgg gtaaatggtg tcaacggaac 60
aacaactgaa gaaaatcctt agataaacct tttgtaacat ccaactaatc ctaggatact 120
acaagtctca gtgggacttt t 141
<210> 27
<211> 141
<212> ДНК
<213> lettuce
<400> 27
aaagtgcata gtcttgtgag cttcttccat gagaagttct ctcattcctc ctaccttggg 60
caccaaaatc atattttgga attcgtttaa tcctttattg tttgtactaa atgctaacat 120
tttgcctaaa cgttccacct t 141
<210> 28
<211> 141
<212> ДНК
<213> lettuce
<400> 28
tataaatgtg ttgcaagaaa actgaatttc aagaaagcaa atgtaatcac tcttttttaa 60
ttatttgtag cacatcgtac tgatcatttt gagaagttcg atcaaaagtt tatctattat 120
cccaatttga tcactttgaa a 141
<210> 29
<211> 141
<212> ДНК
<213> lettuce
<400> 29
gttcatcaat ttcctttagc tctttatcaa ggaaatattc tttttcctcg tagcgaaggg 60
ccatatgaat gtttcagatc caatcgttga agtttgaccc atcgaagata attctcccaa 120
ccaatttcat gagtgagacc a 141
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ИНТРОГРЕССИЯ ДВУХ QTL УРОЖАЯ В РАСТЕНИЯ CUCUMIS SATIVUS | 2017 |
|
RU2746936C2 |
| ИНТРОГРЕССИЯ QTL УРОЖАЯ В РАСТЕНИЯ CUCUMIS SATIVUS | 2016 |
|
RU2741964C2 |
| QTL УРОЖАЯ В РАСТЕНИЯХ ОГУРЦА | 2015 |
|
RU2723586C2 |
| QTL УРОЖАЯ В РАСТЕНИЯХ ОГУРЦА | 2015 |
|
RU2723581C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| РАСТЕНИЯ ОГУРЦА, УСТОЙЧИВЫЕ К ЗАБОЛЕВАНИЯМ | 2006 |
|
RU2418405C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ НИЗКОГО СОДЕРЖАНИЯ ПАЛЬМИТИНОВОЙ КИСЛОТЫ В ПОДСОЛНЕЧНИКЕ (HELIANTHUS ANNUS) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2013 |
|
RU2670517C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| ГЕНЕТИЧЕСКИЕ ЛОКУСЫ, СВЯЗАННЫЕ С УСТОЙЧИВОСТЬЮ К ПЫЛЬНОЙ ГОЛОВНЕ У МАИСА | 2009 |
|
RU2562864C2 |
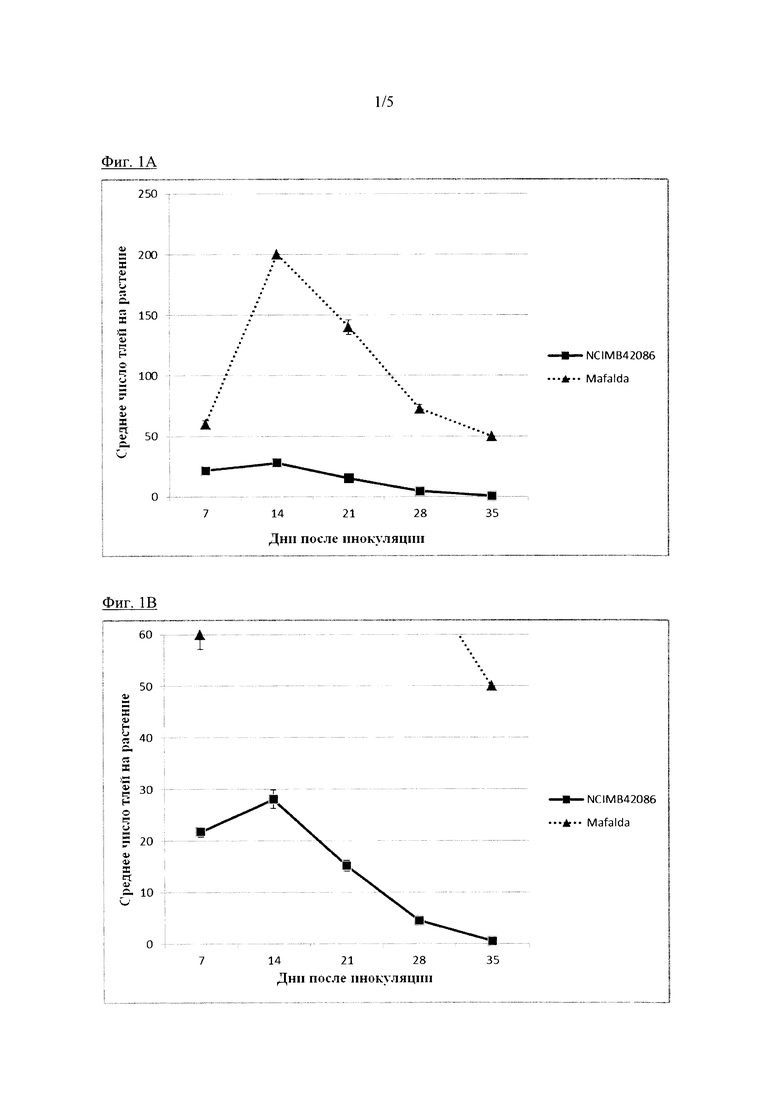
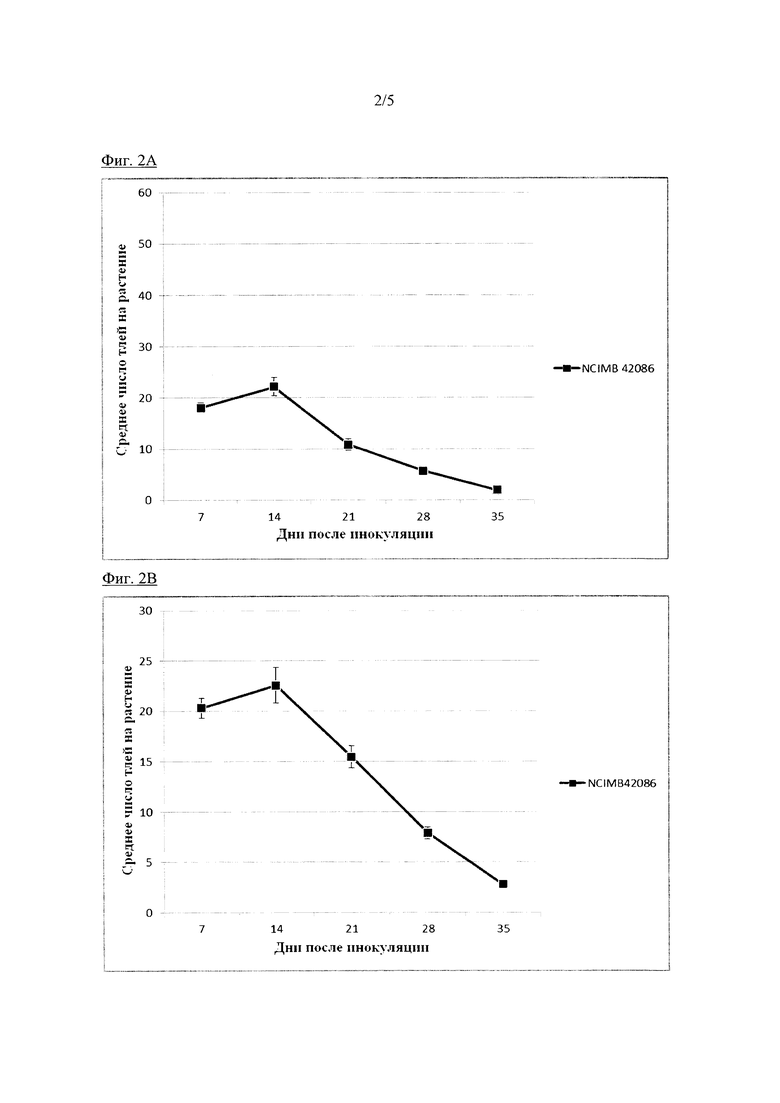

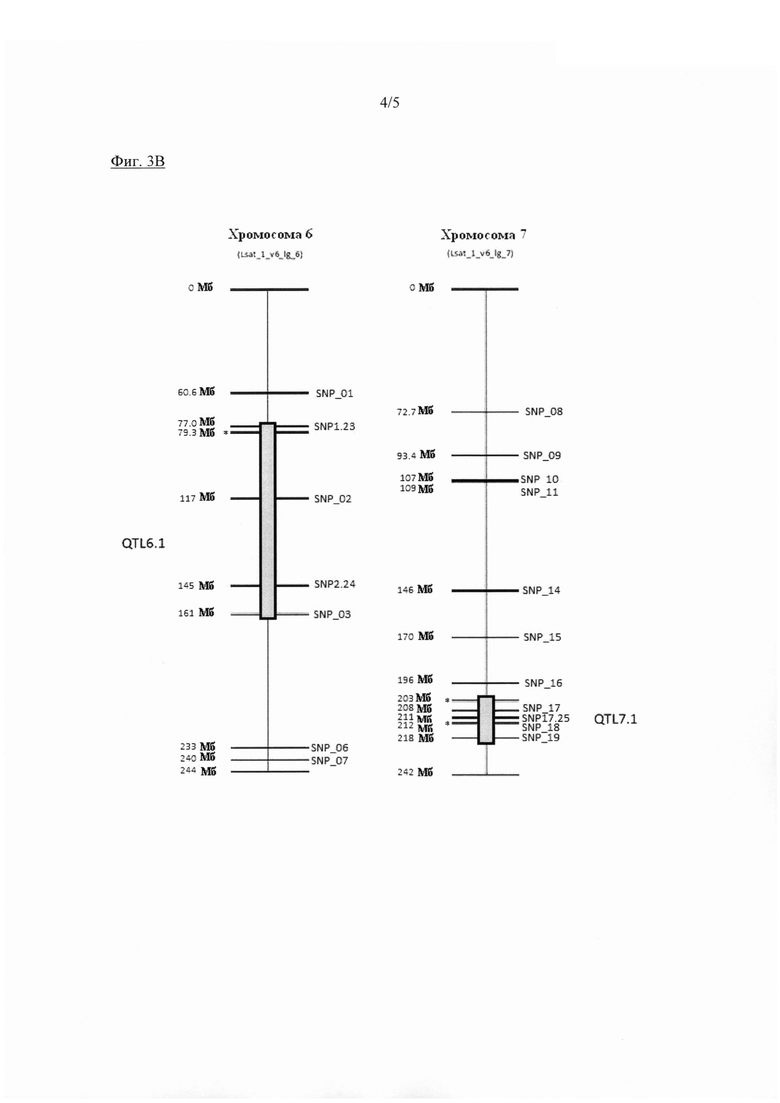

Изобретение относится к области биохимии, в частности к растению Lactuca sativa, содержащему фрагмент интрогрессии из Lactuca virosa на хромосоме 6 и на хромосоме 7, которое обладает большей резистентностью против Nasonovia ribisnigri биотипа 1 (Nr:1) по сравнению с растением, не содержащим указанные фрагменты интрогрессии, а также к его семени, листку, части и головке. Также раскрыт способ идентификации растения салат-латук дикого типа, содержащего QTL6.1, а также способ идентификации растения салат-латук дикого типа, содержащего QTL7.1, и способ идентификации растения салат-латук дикого типа, содержащего QTL6.1 и QTL7.1. Изобретение также относится к способу скрининга линий культивированного растения салат-латук или вариантов на присутствие QTL6.1, способу скрининга линий культивированного растения салат-латук или вариантов на присутствие QTL7.1, а также к способу скрининга линий культивированного растения салат-латук или вариантов на присутствие QTL6.1 и QTL7.1. Изобретение позволяет эффективно получать растение Lactuca sativa с повышенной резистентностью против Nasonovia ribisnigri биотипа 1 (Nr:1). 11 н. и 6 з.п. ф-лы, 7 табл., 5 пр., 4 ил.
1. Растение Lactuca sativa, содержащее фрагмент интрогрессии из Lactuca virosa на хромосоме 6 и на хромосоме 7, каждый из которых содержит локус количественных признаков, обозначаемый QTL6.1 и QTL7.1, где QTL6.1 и QTL7.1 являются получаемыми из семян, депонированных под номером доступа NCIMB 42086, и где указанное растение обладает большей резистентностью против Nasonovia ribisnigri биотипа 1 (Nr:1) по сравнению с растением, не содержащим указанные фрагменты интрогрессии, и где указанный фрагмент интрогрессии на хромосоме 6 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из маркеров, выбранных из группы, состоящей из:
a) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
b) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
c) генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
d) генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
и где указанный фрагмент интрогрессии на хромосоме 7 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из маркеров, выбранных из группы, состоящей из:
a) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
b) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
c) генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
d) генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19.
2. Растение по п. 1, где указанный фрагмент интрогрессии на хромосоме 6 дополнительно содержит следующие маркеры:
a) генотип GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 или
b) генотип АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27.
3. Растение по п. 1, где указанный фрагмент интрогрессии на хромосоме 7 дополнительно содержит следующие маркеры:
a) генотип СС или СА для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28 или
b) генотип GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29.
4. Растение по п. 1, где указанное растение имеет резистентность против Nasonovia ribisnigri биотипа 1 в условиях как испытания со свободным выбором, так и испытания без выбора в анализе резистентности Nr:1, при этом в испытании со свободным выбором тле предоставляется выбор питания и размножения на нескольких различных линиях растений, а в испытании без выбора тле предоставляется для питания и размножения только одна линия растения.
5. Растение по п. 1, где среднее число тлей биотипа 1 на указанном растении составляет не более 50% от среднего числа тлей биотипа 1, обнаруженного на Nr:1 восприимчивом растении, не содержащем фрагмент интрогрессии на хромосоме 6 и на хромосоме 7, при выращивании при одних и тех же условиях.
6. Растение по п. 1, где среднее число тлей биотипа 1 на указанном растении составляет менее 10 тлей, тогда как среднее число тлей биотипа 1 на Nr:1 восприимчивом растении, не содержащем фрагмент интрогрессии на хромосоме 6 и на хромосоме 7, составляет по меньшей мере 100 тлей, при выращивании при одних и тех же условиях.
7. Растение по п. 1, где растение Lactuca sativa выбирается из группы, состоящей из: листовой салат, салат-ромен, салат Crisphead, салат кочанный, Салат Батавия и спаржевый салат.
8. Семя, предназначенное для выращивания растения по любому из пп. 1-7, содержащего фрагмент интрогрессии на хромосоме 6 и на хромосоме 7, и где указанный фрагмент интрогрессии на хромосоме 6 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
a) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
b) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
c) генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
d) генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
и где указанный фрагмент интрогрессии на хромосоме 7 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
a) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
b) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
c) генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
d) генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19.
9. Листок растения по любому из пп. 1-7, предназначенный для использования в качестве продукта или кормового продукта, и содержащий фрагмент интрогрессии на хромосоме 6 и на хромосоме 7, и где указанный фрагмент интрогрессии на хромосоме 6 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
а) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
b) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
c) генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
d) генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
и где указанный фрагмент интрогрессии на хромосоме 7 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
a) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
b) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
c) генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
d) генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19.
10. Часть растения салат-латук по любому из пп. 1-7, где часть выбирается из группы, состоящей из: стеблей, черенков, черешков, семядолей, цветов, пыльников, пыльцы, завязей, корней, корневых кончиков, протопластов, каллюса, микроспор, цветоножек, семяпочек, побегов, семян, зародышей, зародышевых мешков, клеток, почек, листьев, меристемы, причем указанная часть содержит фрагмент интрогрессии на хромосоме 6 и на хромосоме 7, пригодная для обеспечения большей резистентности против Nasonovia ribisnigri биотипа 1 (Nr:1) у растения, полученного из нее, по сравнению с растением, не содержащим указанные фрагменты интрогрессии, и где указанный фрагмент интрогрессии на хромосоме 6 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
а) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
b) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
c) генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
d) генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
и где указанный фрагмент интрогрессии на хромосоме 7 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
a) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
b) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ IDNO: 25;
c) генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
d) генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19.
11. Головка растения по любому из пп. 1-7, предназначенная для использования в качестве продукта или кормового продукта, и содержащая фрагмент интрогрессии на хромосоме 6 и на хромосоме 7, и где указанный фрагмент интрогрессии на хромосоме 6 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
a) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
b) генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
c) генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
d) генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
и где указанный фрагмент интрогрессии на хромосоме 7 содержит по меньшей мере один, предпочтительно по меньшей мере 2, или 3, или 4 из следующих маркеров:
a) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
b) генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
c) генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
d) генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19.
12. Способ идентификации растения салат-латук дикого типа, содержащего QTL6.1, включающий стадии:
a) обеспечение растения салат-латук дикого типа или множества растений салат-латук дикого типа;
b) необязательно тестирование растения салат-латук дикого типа или множества растений на резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1) в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL6.1; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
причем маркеры, указывающие на QTL6.1, выбраны из группы, состоящей из:
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ IDNO: 23;
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
генотипа GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 или
генотипа АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27.
13. Способ скрининга линий культивированного растения салат-латук или вариантов на присутствие QTL6.1, включающего стадии:
a) обеспечение культивированного растения салат-латук или множества культивированных растений салат-латук;
b) необязательно тестирование культивированного растения салат-латук или множества растений на резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1) в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL6.1; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
причем маркеры, указывающие на QTL6.1, выбраны из группы, состоящей из:
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
генотипа GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 или
генотипа АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27.
14. Способ идентификации растения салат-латук дикого типа, содержащего QTL7.1, включающий стадии:
a) обеспечение растения салат-латук дикого типа или множества растений салат-латук дикого типа;
b) необязательно тестирование растения салат-латук дикого типа или множества растений на резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1) в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL7.1; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
и где маркеры, указывающие на QTL7.1, выбраны из группы, состоящей из:
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19;
генотипа СС или СА для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28;
генотипа GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29.
15. Способ идентификации растения салат-латук дикого типа, содержащего QTL6.1 и QTL7.1, включающий стадии:
a) обеспечение растения салат-латук дикого типа или множества растений салат-латук дикого типа;
b) необязательно тестирование растения салат-латук дикого типа или множества растений на резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1) в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL6.1 и указывающих на QTL7.1; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
причем маркеры, указывающие на QTL6.1, выбраны из группы, состоящей из:
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
генотипа GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 или
генотипа АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27;
и где маркеры, указывающие на QTL7.1, выбраны из группы, состоящей из:
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ IDNO: 18;
генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ IDNO: 19;
генотипа СС или СА для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28;
генотипа GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29.
16. Способ скрининга линий культивированного растения салат-латук или вариантов на присутствие QTL7.1, включающего стадии:
a) обеспечение культивированного растения салат-латук или множества культивированных растений салат-латук;
b) необязательно тестирование культивированного растения салат-латук или множества растений на резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1) в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL7.1; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
причем маркеры, указывающие на QTL7.1, выбраны из группы, состоящей из:
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19;
генотипа СС или СА для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28;
генотипа GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29.
17. Способ скрининга линий культивированного растения салат-латук или вариантов на присутствие QTL6.1 и QTL7.1, включающего стадии:
a) обеспечение культивированного растения салат-латук или множества культивированных растений салат-латук;
b) необязательно тестирование культивированного растения салат-латук или множества растений на резистентность против Nasonovia ribisnigri биотипа 1 (Nr:1) в анализе Nr:1 резистентности;
c) скрининг геномной ДНК растения или множества растений согласно а), или необязательно только Nr:1 резистентного растения или растений, идентифицированных в b), на присутствие одного или более маркеров, указывающих на QTL6.1 и указывающих на QTL7.1; и
d) идентификация растения, содержащего один или более из указанных маркеров согласно с);
e) необязательно тестирование растения согласно d) на Nr:1 резистентность в анализе Nr:1 резистентности;
причем маркеры, указывающие на QTL6.1, выбраны из группы, состоящей из:
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP1.23 в SEQ ID NO: 23;
генотипа СС или СТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
генотипа ТТ или СТ для маркера однонуклеотидного полиморфизма SNP2.24 в SEQ ID NO: 24;
генотипа АА или АС для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
генотипа GG или GT для маркера однонуклеотидного полиморфизма VSP1 в SEQ ID NO: 26 или
генотипа АА или АС для маркера однонуклеотидного полиморфизма VSP3 в SEQ ID NO: 27;
и где маркеры, указывающие на QTL7.1, выбраны из группы, состоящей из:
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
генотипа ТТ или ТС для маркера однонуклеотидного полиморфизма SNP17.25 в SEQ ID NO: 25;
генотипа GG или GC для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
генотипа GG или GA для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19;
генотипа СС или СА для маркера однонуклеотидного полиморфизма VSP2 в SEQ ID NO: 28;
генотипа GG или GA для маркера однонуклеотидного полиморфизма VSP4 в SEQ ID NO: 29.
| CID M | |||
| et al., New sources of resistance to lettuce aphids in Lactuca spp., Arthropod-Plant Interactions, 2012, Vol.6, pp.655-669 | |||
| US2014289883 A1, 25.09.2014 | |||
| US2012144513 A1, 07.06.2012 | |||
| WO2011058192 A1, 19.05.2011 | |||
| РАСТЕНИЯ ОГУРЦА, УСТОЙЧИВЫЕ К ЗАБОЛЕВАНИЯМ | 2006 |
|
RU2418405C2 |
Авторы
Даты
2020-09-07—Публикация
2015-10-29—Подача