Область изобретения
Настоящее изобретение относится к области разведения огурцов. Присутствует локус количественных признаков (QTL), расположенный на хромосоме 3 генома огурца, который может применяться для увеличения урожая культивированных огурцов (Cucumis sativus var. sativus), таких как корнишоны (например, виды American pickling, European pickling), салатные огурцы (например, American slicing), длинные огурцы, короткие огурцы. Европейские тепличные огурцы, огурцы типа Бейт-Альфа, огурцы типа oriental trellis (также обозначаемые как 'burpless'), Азиатские огурцы (которые могут быть далее подразделены на различные типы, такие как индийский пестрый огурец, китайский длинный огурец, корейский огурец и японский огурец, причем первый относится к индийской группе огурцов, а последние три к восточноазиатской группе огурцов).
QTL обозначается в настоящей заявке как QTL3.1. Также обеспечиваются культивированные растения огурца, содержащие фрагмент интрогрессии на хромосоме 3, содержащей QTL3.1, причем фрагмент интрогрессии значительно увеличивают урожай плодов культивированного огурца, содержащего интрогрессию по сравнению с таким же культивированным огурцом, не содержащим интрогрессию. Фрагмент интрогрессии, содержащий QTL3.1, происходит из дикого родственника огурца. Одно растение дикого родственника огурца было использовано для создания двойной гаплоидной популяции, которая затем использовалась для картирования и для интрогрессии QTL в европейский длинный тип огурца. Из этого типа QTL легко переносится в любой другой культивированный тип огурца, такой как короткие типы огурцов, или в другие линии разведения или сорта длинного огурца. Семена, содержащие фрагмент интрогрессии, депонировали под номером доступа NCIMB 42346.
В одном варианте выполнения настоящего изобретения культивированное растение огурца, содержащее фрагмент интрогрессии на хромосоме 3 обеспечивается, содержащий QTL3.1, в результате чего фрагмент интрогрессии значительно увеличивает выход плодов культивированного огурца, содержащего интрогрессию, по сравнению с тем же культивированным огурцом, у которого нет интрогрессии. Также один или более молекулярных маркеров (особенно однонуклеотидные полиморфизмы или SNP), которые присутствуют на фрагменте интрогрессии и которые указывают на присутствие фрагмента интрогрессии, и способы применения таких маркеров обеспечиваются настоящим изобретением. Подобным образом получают семена, части растений, клетки и/или ткани, содержащие QTL3.1 в их геноме и имеющие, в противном случае, геном культивированного огурца в их геноме. Отмечают, что термин «геном культивированного огурца» не исключает, что существуют другие фрагменты интрогрессии во всем геноме, например, на других хромосомах и/или для других признаков.
Подобным образом, семена, части растения, семена и/или ткани, содержащие QTL3.1 на хромосоме 3 и содержащие, в противном случае, хромосому 3 культивированного огурца в их геноме обеспечиваются. В одном варианте выполнения настоящего изобретения растения, семена, части растения, клетки и/или ткани содержат фрагмент интрогрессии из дикого родственника огурца, причем фрагмент интрогрессии содержит QTL3.1, где QTL локализуется физически в области, начинающейся при 21.5 Мб и заканчивающейся при 27.23 Мб хромосомы 3. Таким образом, в одном варианте выполнения настоящего изобретения вся или часть области, начинающейся при 21.5 Мб и заканчивающейся при 27.23 Мб хромосомы 3, происходит из дикого родственника огурца и содержит положительный QTL урожая (QTL3.1 или его вариант). В одном варианте выполнения настоящего изобретения другие области хромосомы 3, т.е. от 0 Мб до 21.5 Мб и/или от 27.23 Мб до конца хромосомы 3, содержат или состоят из хромосомных областей культивированного огурца.
В одном варианте выполнения настоящего изобретения культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии из дикого родственника огурца на хромосоме 3 в гомозиготной или гетерозиготной форме, обеспечивается, где указанный фрагмент интрогрессии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1 (или варианта последовательности SEQ ID NO: 1) и маркером однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27 (или варианта последовательности SEQ ID NO: 1), где QTL обеспечивает увеличение урожая плодов огурца. В одном варианте выполнения настоящего изобретения QTL локализуется между основанием 21,507,892 (SNP_01) и основанием 27,233,985 (SNP_27) хромосомы 3.
В одном варианте выполнения настоящего изобретения QTL3.1 (т.е. фрагмент интрогрессии, содержащий QTL) присутствует в гетерозиготной форме в культивированном растении огурца, клетке или ткани, особенно в длинном огурце. В другом варианте выполнения настоящего изобретения QTL3.1 (т.е. фрагмент интрогрессии, содержащий QTL) присутствует в гомозиготной форме в культивированном растении огурца, клетке или ткани, особенно в длинном огурце. В конкретном варианте выполнения настоящего изобретения культивированное растение огурца представляет собой F1 гибрид, особенно F1 гибрид, полученный скрещиванием двух инбредных родительских линий, причем по меньшей мере одна из родительских линий содержит QTL3.1 (т.е. фрагмент интрогрессии, содержащий QTL) в гомозиготной форме. В конкретном варианте выполнения настоящего изобретения культивированное растение огурца не содержит какие-либо другие фрагменты интрогрессии на хромосоме 3 генома огурца, которые влияют на урожай. В другом варианте выполнения настоящего изобретения, могут присутствовать другие фрагменты интрогрессии на хромосоме 3, например, на различных областях хромосомы.
Уровень техники
Культивированный огурец (Cucumis sativus var. sativus L.) является важной овощной культурой во всем мире. Он принадлежит к семейству Cucurbitaceae. Предполагается, что он происходит из Юго-Восточной Азии от диких предков с небольшими горькими плодами, такими как Cucumis sativus сорт hardwickii.
Геном культивированного огурца имеет семь пар хромосом (n=7), и размер гаплоидного генома составляет около 367 Мб (мегабазы) с общей оценкой около 26,682 генов. Геном огурца стал первым геномом овощной культуры, который был секвенирован (Huang et al. 2009, Nature Genetics, Volume 41, Number 12, p 1275-1283 и http://www.icugi.org/cgi-bin/gb2/gbrowse/cucumber_v2/).
Урожай культивированного огурец в последние десятилетия не увеличился. Shetty и Wehner 2002 (CropSci. 42: 2174-2183) провели скрининг USDA коллекции зародышевой плазмы огурца по качеству плодов и урожайности плодов в полевых условиях в Северной Каролине (США) и предположили, что культурные сорта с высокой урожайностью, выявленные в их исследовании, могут быть использованы для разработки культурных сортов с высокой урожайностью.
В WO 2009/082222 применялись сведения, полученные Shetty и Wehner в 2002 (выше) для экземпляров Turkish Beit-Alpha местный сорт PI 169383, для идентификации QTL для массы плодов огурцов на стадии сбора урожая на группе сцепления 3 и/или 4 для PI 69383. Этим донором является местный сорт огурца Turkish Beit-Alpha (который не является диким родственником огурца) и не содержит маркеры SNP согласно настоящему изобретению. Кроме того, в патентной заявке описываются растения огурца, содержащие замещение всей хромосомы 3, т.е. вся хромосома 3 взята из донора PI 169383.
Wei et al. 2014 (BMC Genomics 15: 1158, p 1-10) раскрывают генетическую карту длины незрелого и зрелого плода и массы незрелого плода в популяции, полученной в результате скрещивания между китайской инбредной линией огурца (СС3) и NC76. NC76 получен из местного сорта Cucumis sativus var. sativus из Афганистана (PI 246930) (который не является диким родственником огурца) и имеет короткие плоды (7~10 см). Посредством скрещивания NC76 с короткими плодами с СС3 с длинными плодами, они получили генетическую карту QTL в сегрегации популяции F2 для длины плода (незрелого и зрелого) и массы незрелого плода, как представлено в дополнительном файле 6. Они обнаружили два QTL для длины незрелого плода (f13.1 и f13.2), два QTL для массы незрелого плода (fw3.1 и fw3.2) и один QTL для длины зрелого плода (mf13.1) на группе сцепления 3 (LG3), в дополнение к другому QTL для длины незрелого плода на LG 1 (f11.l) и LG6 (f16.1), но они не раскрывают какой-либо информации в отношении урожая плодов в общем.
Fazio et al. 2003 (Theor Appi Genet 107: 864-874) нанесли на генетическую карту признаки, включая накопленные плоды на растение за три урожая и морфологические признаки, такие как мелколистность ('11'). Их группа сцепления 6 как оказалось, соответствует физической хромосоме 3. Они нанесли на генетическую карту один QTL, называемый fp16.1, который оказал положительный эффект на кумулятивное число плодов на растение за три сбора урожая (число плодов на растение измеряли при трех моментах времени сбора урожая) к маркеру OP-AG1-2 при положении 40.8 сМ. Его обнаружили только в одном положении. Они также нанесли на генетическую карту другой QTL, называемый nfp6.2, который оказывал положительный эффект на число плодов на растение, к маркеру AK5-SCAR в положении 38.6 сМ. Этот QTL обнаружили в одном положении и обнаружили при всех трех сборах урожая в этом положении. Эти QTL физически локализуются ниже половины хромосомы 3 (около 3.0 Мб), тогда как QTL согласно настоящему изобретению локализуется выше половины хромосомы 3, между 2 1.50 Мб и 27.23 Мб хромосомы 3.
Тем не менее, по-прежнему существует потребность в определении QTL для урожая плодов огурца, чтобы иметь возможность увеличить урожайность плодов современных сортов огурца.
Общие определения
Форма единственного числа не исключает возможность, что присутствует более, чем один элемент, если исходя из контекста не следует, что присутствует один и только один из элементов. Форма единственного числа, таким образом, как правило, означает "по меньшей мере один".
Как применяется в настоящей заявке, "растение" включает в себя целое растение или любые части или их производные, такие как органы растений (например, собранные или несобранные органы накопления запасных питательных веществ, клубни, плоды, листья, семена, и т.д.), растительные клетки, протопласты растений, культуры растительных клеток или тканей, из которых могут быть регенерированы целые растения, каллусы растений, скопления клеток растений и растительные клетки, которые являются интактными в растениях, или части растений, такие как зародыши, пыльца, семяпочки, завязи, плоды (например, собранные ткани или органы, такие как собранные плоды огурца или их части), цветы, листья, семена, клубни, луковицы, размноженные клонами растения, корни, корнеплоды, стебли, верхушки корней и тому подобное. Также включена любая стадия развития, такая как саженцы, незрелые и зрелые, и т.д. Когда упоминаются «семена растения», они либо относятся к семенам, из которых растение может быть выращено, или к семенам, произведенным на растении, после самоопыления или перекрестного опыления.
«Сорт растения» представляет собой группу растений в пределах одного и того же ботанического таксона наименьшей известной степени, которая (независимо от того, выполнены ли условия для признания прав селекционера растений или нет) может быть определена на основе выражения признаков, которую в результате определенного генотипа или комбинации генотипов можно отличить от любой другой группы растений посредством выражения по меньшей мере одного из этих признаков, и можно рассматривать как единое целое, потому что можно размножать без какого-либо изменения. Поэтому термин «сорт растения» не может применяться для обозначения группы растений, даже если они одного вида, если все они характеризуются наличием одного или двух локусов или генов (или фенотипических характеристик, обусловленных этими конкретными локусами или генами), но которые могут сильно отличаться друг от друга в отношении других локусов или генов.
"F1, F2, F3, и т.д." относится к последовательным родственным поколениям после кросса между двумя родительскими растениями или родительскими линиями. Растения, выращенные из семян, полученных скрещиванием двух растений или линий, называют поколением F1. Самоопыление растений F1 приводит к поколению F2 и т.д.
Растение "F1 гибрид" (или F1 гибридное семя) представляет собой поколение, полученное скрещиванием двух инбредных родительских линий. Таким образом, F1 гибридные семена представляют собой семена, из которых вырастают F1 гибридные растения. F1 гибриды являются более сильными и дают более высокий урожай, благодаря гетерозису. Инбредные линии, по существу, являются гомозиготными в большинстве локусов генома.
Термин "линия растения" или "линия скрещивания" относится к растению и его потомству. Как применяется в настоящей заявке, термин "инбредная линия" относится к линии растений, которая неоднократно самоопылялась и является почти гомозиготной. Таким образом, термин "инбредная линия" или "родительская линия" относится к растению, которое подвергалось нескольким генерациям (например, по меньшей мере 5, 6, 7 или более) инбридинга, приводя к линии растения с высокой однородностью.
Термин "аллель (аллели)" означает любую одну или более альтернативные формы гена в конкретном локусе, все аллели которого относятся к одному признаку или характеристике в конкретном локусе. В диплоидной клетке организма аллели данного гена расположены в определенном месте или локусе (множество локусов) на хромосоме. Один аллель присутствует на каждой хромосоме пары гомологичных хромосом. Диплоидные виды растений могут содержать большое количество различных аллелей в определенном локусе. Это могут быть одинаковые аллели гена (гомозиготные) или два разных аллеля (гетерозиготные). Таким образом, например, в настоящей заявке может быть сделана ссылка на "аллель урожая" локуса урожая QTL3.1.
Термин «ген» означает последовательность (геномную) ДНК, содержащую область (транскрибируемый участок), которая транскрибируется в молекулу информационной РНК (мРНК) в клетке и функционально связанную регуляторную область (например, промотор). Различные аллели гена представляют собой, таким образом, различную альтернативную форму гена. которая может быть в виде, например, различий в один или более нуклеотидов в геномной последовательности ДНК (например, в промоторной последовательности, экзонных последовательностях, интронных последовательностях и т.д.). мРНК и/или аминокислотной последовательности кодируемого белка.
Термин "локус" (множество локусов) означает определенное место или места или участок на хромосоме, где обнаруживается, например QTL, ген или генетический маркер. Таким образом, локус урожая (локус увеличения урожая) является местом в геноме огурца, где QTL3.1 обнаруживается. В культивированном огурце QTL находятся на хромосоме 3 (применяя хромосомное ассигнование из Huang et al. 2009, Nature Genetics, Volume 41, Number 12, p 1275-1283 и http://www.icugi.org/cgi-bin/gb2/gbrowse/cucumber_v2/) т.е. они интрогрессированы в геном культивированного огурца (т.е. на хромосому 3) из дикого родственника огурца.
Термин "локус количественных признаков", или "QTL" представляет собой хромосомный локус, который кодирует один или более аллелей, которые влияют на экспрессивность непрерывно распределенного (количественного) фенотипа. Локус количественных признаков, обеспечивающий увеличенный урожай (или "QTL урожая"), называют в настоящей заявке QTL3.1.
"Геном огурца" и "физическое положение в геноме огурца" и "хромосома 3" относятся к физическому геному культивированного огурца, во всемирном интернет-пространстве на icugi.org/cgi-bin/gb2/gbrowse/cucumber_v2/, и физическим хромосомам и физическому положению на хромосомах. Поэтому, например, SNP_01 расположен на нуклеотиде (или 'основании'), физически расположенном в месте нуклеотида 21,507,892 хромосомы 3, которая имеет физический размер от 0 до 39,47 Мб (т.е. 39,474,669 оснований).
«Физическое расстояние» между локусами (например, между молекулярными маркерами и/или между фенотипическими маркерами) на одной и той же хромосоме представляет собой фактически физическое расстояние, выраженное в основаниях или парах оснований (пары оснований), килооснованиях или парах килооснований (тысяча пар оснований) или мегабазах или парах мегабаз (Мб).
«Генетическое расстояние» между локусами (например, между молекулярными маркерами и/или между фенотипическими маркерами) на одной и той же хромосоме измеряется частотой кроссинговера или частотой рекомбинации (RF) и указывается в сантиморганах (сМ). Одному сМ соответствует частота рекомбинации 1%. Если никакие рекомбинанты не могут быть найдены, RF равна нулю, и локусы либо очень близки друг к другу физически, либо идентичны. Чем дальше расположены два локуса, тем выше RF.
"Фрагмент интрогрессии" или "сегмент интрогрессия" или "область интрогрессия" относится к фрагменту хромосомы (или части или области хромосомы), который был введен в другое растение того же или родственного вида путем скрещивания или традиционных методов разведения, таких как обратное скрещивание, т.е. интрогрессированный фрагмент является результатом методов размножения, ссылающихся на глагол «интрогрессировать» (например, обратное скрещивание). В отношении огурцов дикие или примитивные огурцы (например, местные сорта) или дикие родственники культивированного огурца могут применяться для интрогрессии фрагментов дикого генома в геном культивированного огурца, Cucumis sativus var. sativus L. Такое культивированное растение огурца, таким образом, имеет "геном культивированного Cucumis sativus var. sativus", но содержит в геноме фрагмент дикого или примитивного огурца (например, местный сорт) или дикого родственника огурца, например, фрагмент интрогрессии генома дикого родственника Cucumis sativus, такого как Cucumis sativus сорт hardwickii, С.sativus сорт sikkimensis Cucumis sativus сорт xishuangbannesis, или другого дикого родственника огурца. Поэтому, например, согласно настоящему изобретению обеспечивается культивированный огурец, содержащий геном культивированного огурца, и в этом геноме один фрагмент интрогрессии на хромосоме 3 культивированного огурца, которые сообщают увеличенный урожай по сравнению с геномом культивированного огурца, не содержащим фрагменты интрогрессии (и имеющим хромосому 3 культивированного огурца без фрагмента интрогрессии). Понятно, что термин "фрагмент интрогрессии" не включает хромосому целиком, а только часть хромосомы. Фрагменты интрогрессии могут быть большими, например, составлять три четверти или половину хромосомы, но предпочтительно он меньше, как например, около 15 Мб или менее, как например, около 10 Мб или менее, около 9 Мб или менее, около 8 Мб или менее, около 7 Мб или менее, около 6 Мб или менее, около 5 Мб или менее, около 4 Мб или менее, около 3 Мб или менее, около 2.5 Мб или 2 Мб или менее, около 1 Мб (равно 1,000,000 пар оснований) или менее, или около 0.5 Мб (равно 500,000 пар оснований) или менее, как например, около 200,000 пар оснований (равно 200 пар килооснований) или менее, около 100,000 пар оснований (100 тысяч пар оснований) или менее, около 50,000 пар оснований (50 тысяч пар оснований) или менее, около 25.000 пар оснований (25 тысяч пар оснований) или менее.
"Культивированный огурец" или "одомашненный огурец" относится к растениям Cucumis sativus var. sativus т.е. сорта, линии скрещивания или культивары, культивированные людьми и имеющие хорошие агрономические характеристики, в частности производящих съедобные и маркируемые плоды хорошего качества и однородности; такие растения не являются растением "дикого родственника огурца" или "примитивного огурца", т.е. растениями, которые в общем дают намного более плохие урожаи и имеют более плохие агрономические характеристики, чем культивированные растения, и являются менее однородными генетически и по их физиологическим и/или морфологическим характеристикам. "Дикие растения" включают, например, экотипы, местные сорта или дикие экземпляры или дикие родственники видов. Культивированные растения огурца (линии или сорта) также могут отличаться от дикого или примитивного огурца значительно более низким количеством SNP (менее 2,000,000 SNP) и INDEL (вставки/делеции более короткие, чем 5 пар оснований; менее 150,000 INDEL) в геноме и их значительно более низкой нуклеотидной плотностью (равна или менее 2.3×10-3 π), как описано в Таблица 1 в Qi et al, Nature Genetics December 2013.Vol 45.No. 12, pages 1510-1518. Число SNP, число INDEL и нуклеотидную плотность можно определить, как описано в настоящей заявке, в частности в части 'Способы'.
«Индийская группа огурцов» относится к диким огурцам или диким родственникам огурцов из Индии, имеющим большое числом SNP (более 3000000 SNP) и INDEL (вставки/делеции короче 5 пар оснований, более 200000 INDEL) в геноме и высокую нуклеотидную плотность (более 3.0×10-3 π или даже более 4.0×10-3 π).
"Евразийская группа огурцов" относится к культивированным огурцам из центральной или западной Азии, Европы и Соединенных Штатов, имеющим низкое число SNP (менее 2.000,000 SNP. или менее 1,500.000 SNP) и INDEL (вставки/делеции короче 5 пар оснований; менее 150,000 INDEL, предпочтительно менее 100.000) в геноме и низкую нуклеотидную плотность (равна или менее 2.3×10-3 π, предпочтительно менее 2.0×10-3 π).
"Восточноазиатская группа огурцов" относится к культивированным огурцам из Восточной Азии, как например, Китай, Корея и Япония, имеющим низкое число SNP (менее 2,000,000 SNP, или менее 1,500,000 SNP) и INDEL (вставки/делении короче 5 пар оснований; менее 150,000 INDEL, предпочтительно менее 100,000) в геноме и низкую нуклеотидную плотность (равна или менее 2.3×10-3 π, предпочтительно менее 2.0×10-3 π или даже менее 1.5×10-3 π).
"Группа огурцов Xishuangbanna" относится к огурцам из области Xishuangbanna в Китае, имеющим низкое число SNP (менее 2,000,000 SNP, или менее 1,500,000 SNP или даже менее 100,000 SNP) и INDEL (вставки/делеции короче 5 пар оснований; менее 150,000 INDEL, предпочтительно менее 100,000) в геноме и низкую нуклеотидную плотность (равен или менее 2.3×10-3 π, предпочтительно менее 2.0×10-3 π или даже менее 1.5×10-3 π).
"Дикий огурец" или "примитивный огурец" относится к С. sativus var. sativus, которые обычно дают гораздо более плохие урожаи и имеют более плохие агрономические характеристики, чем культивированные растения и менее однородны генетически и по своим физиологическим и/или морфологическим характеристикам. Дикие растения включают, например, экотипы, местные сорта или дикие экземпляры или дикие родственники вида.
"Дикий родственник (родственники) огурца" относятся к Cucumis sativus сорт hardwickii, С. sativus сорт sikkimensis, Cucumis sativus сорт xishuangbannesis.
"Местный сорт (сорта)" относится к примитивным культиварам Cucumis sativus var. sativus, разработанным в местных географических регионах, которые часто демонстрируют высокую степень генетической изменчивости в их геноме и проявляют высокую степень морфологической и/или физиологической изменчивости в пределах местного сорта (например, большая вариация в размере плода, и т.д.), т.е. являются значительно менее однородными, чем культивированный огурец. Местные сорта, поэтому, согласно настоящему изобретению включены в группу "дикий огурец, которая отличается от "культивированного огурца.
"Однородность" или "однородные" относится к генетическим и фенотипическим характеристикам линии растения или сорта. Инбредные линия являются весьма генетически однородными, поскольку их получают путем нескольких генераций инбридинга. Подобным образом, и F1 гибриды, которые получают из таких инбредных линий, являются весьма однородными по своим генотипическим и фенотипическим характеристикам и производительности.
Термин "аллель урожая" относится к аллелю, обнаруживаемому в локусе урожая QTL3.1, интрогрессированному в культивированный огурец (на хромосоме 3 культивированного С.sativus var. sativus) из дикого родственника огурца. Термин "аллель урожая", таким образом, также охватывает аллели урожая, получаемые из других экземпляров Cucumis. Когда один или два аллеля урожая присутствуют в локусе в геноме (т.е. в гетерозиготной или гомозиготной форме), линия или сорт растения дает значительно более высокий урожай плодов, чем генетический контроль, не содержащий QTL. В культивированном растении огурца, не содержащем фрагмент интрогрессии, аллель С. sativus var. sativus, обнаруживаемая в том же локусе на хромосоме 3, согласно настоящему изобретению обозначается как аллель "дикого типа" (wt). Так как QTL урожая являются доминантными, растения wt/wt показывают стандартный урожай, тогда как растения QTL3.1/wt и растения QTL3.1/QTL3.1 представляют собой растения, которые обладают фенотипом увеличенного урожая, обеспечиваемым аллелем (аллелями) урожая. Генотип SNP маркеров, раскрытых в настоящей заявке, также указывает на дикий тип или QTL либо в гомозиготной, либо в гетерозиготной форме. Например, генотип SNP_01, указывающий на QTL3.1 представляет собой 'ТС' (QTL3.l/wt) или 'ТТ' (QTL3.1/QTL3.1), тогда как генотипом, указывающим на дикий тип, является 'СС' (wt/wt).
Генетический элемент, фрагмент интрогрессии или ген или аллель, сообщающий признак (такой как урожай) считается "получаемый из" или может быть "получен из" или "происходящий из" или может "происходить из" или "как присутствует в" или "как обнаружено в" растении или семени или ткани или клетки, если он может быть перенесен из растения или семени, в котором он присутствует, в другое растение или семя, в котором он не присутствует (как например линия или сорт), используя традиционные методы разведения, не приводящие к фенотипическому изменению растения-реципиента, за исключением добавления признака, сообщаемого генетическим элементов, локусом, фрагментом интрогрессии, геном или аллелем. Термины используются взаимозаменяемо, и генетический элемент, локус, фрагмент интрогрессии, ген или аллель могут, таким образом, быть перенесены в любой другой генетический фон, лишенный этого признака. Могут применяться не только семена, депонированные и содержащие генетический элемент, локус, фрагмент интрогрессии, ген или аллель, но также потомство/потомки таких семян, которые были отобраны для сохранения генетического элемента, локуса, фрагмента интрогрессии, гена или аллеля, могут применяться и охватываются настоящим изобретением, как например коммерческие сорта, разработанные из депонированных семян или из их потомства. Содержит ли растение (ли геномная ДНК, клетка или ткань растения) тот же самый генетический элемент, локус, фрагмент интрогрессии, ген или аллель, как получено из депонированных семян, может быть определено специалистом в данной области техники, используя одну или более методик, известных в данной области, таких как фенотипические анализы, секвенирование всего генома, анализ молекулярного маркера, нанесение признаков на генетическую карту, роспись хромосомы, тесты аллеморфизма и тому подобное, или комбинации методик.
"Вариант" или "ортолог" последовательности (вариантная или ортологическая последовательность) или "вариант QTL3.1" относится к QTL урожая (QTL3.1), или фрагменту интрогрессии, содержащему QTL, которые получены из других растений диких родственников огурца, чем QTL3.1, присутствующий в NCIMB 42346, но вариант которых содержит один или более SNP, связанных с QTL3.1. и где вариантная геномная последовательность имеет существенную идентичность последовательности с SEQ ID NO: содержащей SNP (любой одной последовательностью из SEQ ID NO: 1-27), т.е. имеет идентичность последовательности по меньшей мере 85%, 90%, 95%, 98%, 99% или более. Таким образом, когда в настоящем описании делается ссылка на определенный генотип SNP в определенной геномной последовательности (выбранной из SEQ ID NO: 1 - SEQ ID NO: 27), это охватывает также генотип SNP в вариантах геномной последовательности, т.е. генотип SNP в геномной последовательности, имеющей идентичность последовательности по меньшей мере 85%, 90%, 95%, 98%, 99% с последовательностью, на которую ссылаются (выбранной из SEQ ID NO: 1 - SEQ ID NO: 27). Таким образом, любая ссылка в настоящей заявке на любую одну из последовательностей SEQ ID NO: 1-27 в одном варианте также охватывает вариант любой одной из SEQ ID NO: 1-27, причем указанный вариант имеет идентичность последовательности по меньшей мере 85%, 90%, 95%, 98%, 99% или более с указанной последовательностью.
"Урожай" или "урожай плодов" или "средний урожай" относится к среднему числу плодов на растение (FrPP) и/или средней массе плода (граммы) на растение (GrPP). Их определяют для каждой линии растения, гибрида или сорта, выращенных при таких же условиях (например, линия, гибрид или сорт с QTL и генетический контроль без QTL), и среднее FrPP и/или GrPP каждой линии, гибрида или сорта вычисляют. В зависимости от типа огурца, урожай плодов измеряют различными путями. Так, например, типы, которые производят плоды непрерывно в течение определенного периода времени, такие как поступающие на рынок в свежем виде (например, длинные типы огурцов, такие как Европейский тепличный огурец, мини-или миди-типы), плоды собираются, когда они достигают товарного размера, и сбор урожая производится в течение определенного периода времени упоминается как «период сбора урожая» (например, период сбора урожая начинается, когда первые плоды достигают товарного размера и могут иметь длину, соответствующую по меньшей мере 10. 11, 12 или более неделям). Так, например, среднее FrPP и/или GrPP на линию измеряется на день и складывается в течение всех дней в конце периода сбора, чтобы рассчитать кумулятивное FrPP и/или GrPP для каждой линии или сорта (смотрите также примеры). «Товарный размер» относится к плодам, которые являются достаточно длинными и достаточно тяжелыми для продажи.
Таким образом, плоды товарного размера собирают в момент времени, который является оптимальным или около оптимального для товарного плода и плода для продажи. Для длинных типов огурцов, таких как Европейский тепличный огурец, товарный размер достигается, когда плод составляет по меньшей мере около 26 или 27 см в длину и имеет минимальную массу 250 грамм. Для типов огурцов, которые собирают только в один момент времени, таких как корнишоны, "урожай" или "урожай плодов" или "средний урожай" относится к среднему числу плодов, составляющих или выше 1.5 см по диаметру, на растение (FrPP) и/или среднюю массу плодов (граммы), составляющих или выше 1.5 см по диаметру, на растение (GrPP) в единый момент времени сбора урожая. Единый момент времени сбора урожая соответствует практике выращивания и выбран для максимального увеличения количества плодов, имеющих диаметр 1,5 см и 5,0 см. В зависимости от желаемого размера плода момент времени, как правило, достигается, когда около 5%, около 10%, около 15% или около 20% плодов больше по размеру (т.е. имеют диаметр плода, равный 5.0 см или более). Уборка производится вручную или машинным сбором. Таким образом, в одном варианте собирают все плоды на растение и подсчитывают и/или взвешивают только те, которые имеют диаметр по меньше мере 1,5 см (т.е. подсчитываются и/или взвешиваются все плоды с диаметром по меньше мере 1,5 см, в том числе и негабаритные).
"Увеличенный урожай плодов" или "значительно увеличенный урожай плодов" относится к линии или сорту культивированного растения огурца, содержащим фрагмент интрогрессии на хромосоме 3, содержащий QTL3.1, дающим (благодаря QTL) статистически значительно более высокое среднее число плодов на растение (FrPP) и/или значительно более высокую среднюю массу плода на растение (GrPP), по сравнению с растением генетического контроля, не содержащим фрагменты интрогрессии на хромосоме 3, при выращивании в экспериментах на урожай в тех же условиях. Предпочтительно полевые условия проводятся с несколькими повторениями (2, 3. или предпочтительно 3, 4, 5, 6, 7, 8, или более) с достаточным количеством растений (например, по меньшей мере 8, 9, 10, 15, 20, 30, 40, или более растений на линию), содержащих фрагмент интрогрессии на хромосоме 3 и не содержащих фрагмент интрогрессии на хромосоме 3 (т.е. генетические контроли).
"Генетический контроль" представляет собой линию, сорт или гибрид огурца, который имеет такой же или очень подобный культурный геном, как и растение огурца, содержащее интрогрессию на хромосоме 3, за исключением того, что не содержит интрогрессии на хромосоме 3, т.е. хромосома 3 является "дикого типа", т.е. геном культивированного огурца. Например, семена, депонированные под номером доступа NCIMB 42346 представляют собой семена тестового гибрида F1, полученного между линией интрогрессии (огурца длинного типа), содержащей QTL3.1 на хромосоме 3, и элитной линией скрещивания огурца (огурца длинного типа), тогда как генетическим контролем, депонированным под номером NCIMB 42345, являются семена рекуррентного родителя линии интрогрессии (не содержащей QTL3.1) и той же элитной линия скрещивания огурца.
Термин "анализ маркера" относится к анализу молекулярного маркера, который может применяться, чтобы протестировать присутствует ли на хромосоме 3 культивированного С. sativus var. sativus интрогрессия из дикого родственника огурца, фрагмент интрогрессии которых содержит QTL урожая (QTL3.1) (или содержит ли дикий родственник огурца QTL3.1 в его геноме), путем определения генотипа любого одного или более маркеров, связанных с QTL3.1, например, например, генотипа одного или более SNP маркеров, выбранных из SNP_01 -SNP_27, и/или любого специфичного для генома дикого родственника огурца маркера между SNP маркерами SNP_01 и SNP_27, и/или в пределах 7 сМ или в пределах 5 сМ, 3 сМ, 2 сМ, 1 сМ любого одного из этих маркеров, и/или в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.1 Мб, 50 кб, 20 кб, 10 кб, 5 кб, 2 кб, 1 кб или менее любого одного из этих маркеров. Маркер "между" двумя маркерами физически располагается между маркерами на хромосоме.
SNP маркеры, приведенные в настоящей заявке, т.е. SNP_01 - SNP_27, локализуются в данном порядке на фрагменте интрогрессии. "Последовательные" маркеры относятся к маркерам в том же порядке последовательности, так например, двумя последовательными маркерами могут быть SNP_01 и SNP_02; SNP_02 и SNP_03; SNP_03 и SNP_04, и т.д.. и тремя последовательными маркерами могут быть SNP_01 и SNP_02 и SNP_03; SNP_02 и SNP_03 и SNP_04; и т.д.
"Среднее значение" или "среднее" относится согласно настоящему изобретению к среднему арифметическому значению, и оба термина используются взаимозаменяемо. Термин "среднее значение" или "среднее", таким образом, относится к среднему арифметическому значению для нескольких измерений. Специалисту в данной области техники понятно, что фенотип линии растения или сорта зависит в некоторой степени от условий произрастания, и поэтому измеряются средние арифметические значения для по меньшей мере 8, 9, 10, 15, 20, 30, 40, 50 или более растений (или частей растений), предпочтительно в рандомизированных экспериментальных моделях с несколькими повторениями и подходящими контрольными растениями, выращенными при тех же условиях в том же эксперименте. "Статистически значимый" или "статистически значимо" отличный или "значительно" отличный относится к характеристике линии растения или сорта, которые, при сравнении с подходящим контролем (например, в настоящей заявке, генетическим контролем), показывают статистически значимое отличие их характеристик (например, р-значение составляет менее 0.05, р<0.05, применяя ANOVA) от (среднего значения) контроля.
"Рекомбинантная хромосома" относится к хромосоме, имеющей новый генетический облик, возникающий в результате кроссинговера между гомологичными хромосомами, например "рекомбинантная хромосома 3", т.е. хромосома 3, которая не присутствует ни в одном из родительских растений и возникла в результате редкого события кросеинговера между гомологичными хромосомами пары хромосомы 3. Согласно настоящему изобретению обеспечиваются, например, рекомбинантная хромосома огурца 3, содержащая интрогрессию из дикого родственника огурца.
Термин "традиционные методы разведения" согласно настоящему изобретению охватывает скрещивание, обратное скрещивание, самоопыление, селекцию, получение двойного галоида, эмбриональное спасение, слияние протопластов, выбор с помощью маркера, мутационную селекцию и т.д. как известно селекционеру (т.е. методы, отличные от методов генетической модификации/трансформации/трансгенных методов), посредством которых, например, рекомбинантную хромосому 3 можно получить, идентифицировать и/или переносить.
"Обратное скрещивание" относится к методу селекции, с помощью которого (одиночный) признак, такой как QTL урожая, может быть перенесен из более низшего генетического фона (например, дикого родственника огурца или огурца дикого типа; также обозначается как «донор») в высший генетический фон (также обозначается как "рекуррентный родитель"), например культивированный огурец. Потомство кросса (например, растение F1, полученное скрещиванием дикого родственника огурца с культивированным огурцом; или растение F2 или растение F3, и т.д., полученное самоопылением F1) является "обратно скрещенным" до родителя с высшим генетическим фоном, например культивированного родителя. После повторного обратного скрещивания, признак низшего генетического фона будет включен в высший генетический фон.
"Выбор с помощью маркера" или "MAS" представляет собой процесс использования присутствия молекулярных маркеров, которые генетически связаны с определенным локусом или с определенной областью хромосомы (например, фрагмент интрогрессии), для выбора растений по присутствию конкретного локуса или области (фрагмент интрогрессии). Например, молекулярный маркер, генетически связанный с QTL урожая, может применяться для обнаружения и/или выбора растения огурца, содержащего QTL урожая на хромосоме 2 и/или 6. Чем ближе генетическая связь молекулярного маркера с локусом (например около 7 сМ, 6 сМ, 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ, 0.5 сМ или менее), тем менее вероятно, что маркер диссоциирует от локуса посредством мейотической рекомбинации. Аналогично, чем ближе два маркера связаны друг с другом (например, в пределах 7 сМ или 5 сМ, 4 сМ, 3 сМ, 2 сМ, 1 сМ или менее), тем менее вероятно, что два маркера будут отделены друг от друга (и, скорее всего, они будут совместно выделяться как единое целое).
Маркер "в пределах 7 сМ или в пределах 5 сМ, 3 сМ, 2 сМ, или 1 сМ" другого маркера относится к маркеру, который генетически привязывается к области, фланкирующей маркер, в пределах 7 сМ или 5 сМ, 3 сМ, 2 сМ, или 1 сМ (т.е. с обеих сторон маркера). Подобным образом, маркер в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 кб, 20 кб, 10 кб, 5 кб, 2 кб, 1 кб или менее, области геномной ДНК, фланкирующей маркер (т.е. с обеих сторон маркера).
"LOD-оценка" (логарифм (основание 10) расхождений) относится к статистическому тесту, часто используемому для анализа сцепления в популяциях животных и растений. Оценка LOD сравнивает вероятность получения тестовых данных, если эти два локуса (локусы молекулярных маркеров и/или локусы фенотипических признаков) действительно сцеплены, с вероятностью наблюдения одних и тех же данных чисто случайно. Положительные оценки LOD свидетельствуют о наличие сцепления, и оценка LOD выше 3.0 рассматривается как очевидное сцепление. Оценка LOD, равная +3, указывает на вероятность от 1000 до 1, что наблюдаемое сцепление не происходит случайно.
"Вегетативное размножение", "вегетативная репродукция" или "клональное размножение" применяются в настоящей заявке взаимозаменяемо и означают способ, в котором берут часть растения и позволяют части растения образовать по меньшей мере корни, где часть растения, например, определяется как или происходит из (например, путем разрезания) листа, пыльцы, зародыша, семядолей, гипокотиля, клеток, протопласт, меристематической клетки, корня, кончика корня, пестика, пыльника, цветка, кончика побега, побега, стебля, плода, черешка и т.д. Когда целое растение регенерируется путем вегетативного размножения, это также обозначается как вегетативное размножение.
"Клеточная культура" или "тканевая культура" относится к in vitro культуре клеток или тканей растения.
"Регенерация" относится к развитию растения из клеточной культуры или тканевой культуры или вегетативному размножению.
Термин "неразмножающаяся клетка" относится к клетке, которая не может регенерироваться в целое растение.
"Трансген" или "химерный ген" относится к генетическому локусу, содержащему последовательность ДНК, такую как рекомбинантный ген, который был введен в геном растения путем трансформации, такой как трансформация, опосредованная Agrobacterium. Растение, содержащее трансген, стабильно интегрированный в его геном, обозначается как "трансгенное растение".
"Выделенная последовательность нуклеиновой кислоты" или "выделенная ДНК" относится к последовательности нуклеиновой кислоты, которая больше не находится в природной среде, из которой она была выделена, например, последовательности нуклеиновой кислоты в бактериальной клетке-хозяине или в ядерном или пластидном геноме растении.
"Клетка-хозяин" или "рекомбинантная клетка-хозяин" или "трансформированная клетка" являются терминами, относящимися к новой индивидуальной клетке (или организму), возникающей в результате по меньшей мере одной молекулы нуклеиновой кислоты, введенной в указанную клетку. Клетка-хозяин предпочтительно является растительной клеткой или бактериальной клеткой. Клетка-хозяин может содержать нуклеиновую кислоту в качестве экстрахромосомно (эписомальной) реплицирующейся молекулы, или содержать нуклеиновую кислоту, интегрированную в ядерный или пластидный геном клетки-хозяина, или в качестве введенной хромосомы, например минихромосомы.
"Идентичность последовательности" и "сходство последовательности" можно определить выравниванием двух пептидных или двух нуклеотидных последовательностей с использованием глобальных или локальных алгоритмов выравнивания. Последовательности могут затем обозначаться как «по существу идентичные" или "весьма подобные", когда они оптимально выровнены, например, с помощью программы GAP или BESTFIT или фильтрующей программы "Needle" (с использованием параметров по умолчанию, см. ниже), определяя по меньшей мере определенный минимальный процент идентичности последовательности (как определено ниже). Эти программы используют глобальный алгоритм выравнивания Нидлмана-Вунша для выравнивания двух последовательностей по всей длине, получая максимальное количество совпадений и сводя к минимуму количество гэпов. Обычно параметры по умолчанию используется штраф за открытие гэпа = 10 и штраф за расширение гэпа = 0,5 (как для нуклеотидного, так и белкового выравнивания). Для нуклеотидов применяемой матрицей весов замен является DNAFULL, и для белков матрицей весов замен является Blosum 62 (Henikoff & Henikoff, 1992, PNAS 89. 915-919). Выравнивания последовательности и показатели процентной идентичности последовательности, например, могут быть определены с помощью компьютерных программ, таких как EMBOSS, как доступно во всемирной сети по адресу ebi.ac.uk/Tools/psa/emboss_needle/. Альтернативно подобие или идентичность последовательности могут быть определены с помощью функции поиска в базах данных, таких как FASTA, BLAST и т.д., но совпадения должны быть получены и приведены в соответствие попарно, чтобы сравнить идентичность последовательности. Два протеина или два белковых домена или две последовательности нуклеиновых кислот имеют «существенную идентичность последовательности», если идентичность последовательности в процентах составляет по меньшей мере 85%, 90%, 95%, 98%, 99% или более (например по меньшей мере 99.1, 99.2 99.3 99.4, 99.5, 99.6, 99.7, 99.8, 99.9 или более (как определено с помощью Emboss "needle", используя параметры по умолчанию, т.е. штраф за открытие гэпа = 10. штраф за расширение гэпа = 0.5, используя матрицу весов замен DNAFULL для нуклеиновых кислот, и Blosum62 для белков).
Когда делается ссылка на последовательность нуклеиновой кислоты (например, ДНК или геномную ДНК), имеющую "последовательность, по существу идентичную" ссылочной последовательности или имеющую последовательность, идентичную на по меньшей мере 80%, например по меньшей мере 85%, 90%, 95%, 98%, 99%, 99.2%, 99.5%, 99.9% ссылочной последовательности, в одном варианте выполнения настоящего изобретения указанная нуклеотидная последовательность рассматривается как по существу идентичная данной нуклеотидной последовательности и может быть идентифицирована, используя строгие условия гибридизации. В другом варианте выполнения настоящего изобретения, последовательность нуклеиновой кислоты содержит одну или более мутаций, по сравнению с данной нуклеотидной последовательностью, но все еще может быть идентифицирована, используя строгие условия гибридизации.
"Строгие условия гибридизации" могут применяться для идентификации нуклеотидных последовательностей, которые по существу идентичны данной нуклеотидной последовательности. Строгие условия зависят от последовательности и будут отличаться в разных обстоятельствах. Как правило, строгие условия выбираются так, что составляют на около 5°С точки плавления (Tm) для конкретных последовательностей при определенной ионной силе и рН. Tm представляет собой температуру (при определенной ионной силе и рН), при которой 50% целевой последовательности гибридизуется идеально подобранным зондом. Обычно выбирают такие строгие условия, при которых концентрация соли составляет около 0,02 моля при рН 7, а температура составляет по меньшей мере 60°С. Уменьшение концентрации соли и/или повышение температуры увеличивает жесткость условий. Строгие условия для гибридизаций РНК-ДНК (нозерн-блоты с использованием зонда, например, 100 нт) являются, например, теми, которые включают по меньшей мере одну промывку в 0.2Х SSC при 63°С в течение 20 мин, или эквивалентные условия. Строгие условия для гибридизации ДНК-ДНК (Саузерн-блоты с использованием зонда, например, 100 нт) являются, например, теми, которые включают по меньшей мере одну промывку (как правило 2) в 0.2Х SSC при температуре, равной по меньшей мере 50°С, как правило около 55°С, в течение 20 мин, или эквивалентные условия. Смотрите также Sambrook et al. (1989) и Sambrook и Russell (2001).
"Тонкое картирование" относится к способам, посредством которых положение QTL может быть определено более точно (сужено), и посредством которых размер фрагмента интрогрессии, содержащего QTL, сокращается. Например, почти изогенная линия для QTL (QTL-NIL) может быть получена, которая содержит различные, перекрывающиеся фрагменты фрагменты интрогрессии в пределах иной однородной генетической среды рекуррентного родителя. Такие линии могут затем применяться для нанесения на генетическую карту места локализации QTL и для идентификации линии, имеющей более короткий фрагмент интрогрессии, содержащий QTL.
Подробное описание изобретения
Настоящее изобретение относится к культивированному растению Cucumis sativus var. sativus, содержащему QTL урожая на хромосоме 3, интрогрессированного из дикого родственника огурца. В частности, повышенная урожайность обеспечивается (сообщается) фрагментом интрогрессии на хромосоме культивированного огурца 3 (содержащей QTL3.1 или его вариант), где указанный фрагмент интрогрессии происходит из дикого родственника огурца.
Когда в настоящей заявке делается ссылка на фрагмент интрогрессии на хромосоме 3, имеющий QTL урожая, это охватывает различные размеры фрагментов интрогрессии, например, фрагмент, как обнаружено в NCIMB 42346, содержащий все маркеры SNP (SNP_01 - SNP_27, или любой маркер между ними, для фрагмента на хромосоме 3), но также более маленький фрагмент интрогрессии (содержащий менее 27 SNP маркеров, как например, только 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25 или 26 из SNP маркеров), где, однако, фрагмент остается достаточно большим для сообщения значительно увеличенного урожая (по сравнению с генетическим контролем), когда фрагмент интрогрессии находится в гетерозиготной или гомозиготной форме в геноме культивированного огурца. Другими словами, фрагмент сохраняет QTL3.1 или его вариант, т.е. все еще обеспечивает значительно увеличенный урожай (по сравнению с контролем, например, генетическим контролем), когда фрагмент интрогрессии находится в гетерозиготной или гомозиготной форме в геноме культивированного огурца.
Таким образом, в одном варианте выполнения настоящего изобретения обеспечивается культивированное растение огурца, содержащее фрагмент интрогрессии из дикого родственника огурца, где фрагмент интрогрессии содержит QTL3.1, или его вариант, и где фрагмент интрогрессии содержит всю или часть области, начинающейся при нуклеотиде (или основании) 21,507,892 хромосомы 3 и заканчивающейся при нуклеотиде (или основании) 27,233,985 хромосомы 3. Другими словами, вся или часть области, начинающейся при нуклеотиде 21,507,892 хромосомы 3 и заканчивающейся при нуклеотиде 27,233,985 хромосомы 3, происходит, в одном варианте выполнения настоящего изобретения, из дикого родственника огурца и содержит QTL3.1 или его вариант. Какая подобласть содержит QTL3.1 можно определить посредством, например, тонкого картирования. Поэтому, например, если QTL3.1, как обнаружено, находится между SNP_01 и SNP_10, тогда растение согласно настоящему изобретению должно содержать только область интрогрессии, начинающуюся при нуклеотиде 21,507,892 хромосомы 3 (SNP_01) и заканчивающуюся при нуклеотиде 23,706,444 (SNP_10) хромосомы 3.
В одном варианте выполнения настоящего изобретения QTL3.1 (или его вариант) локализуется между маркером SNP_01 в SEQ ID NO: 1 (или в варианте последовательности SEQ ID NO: 1) и маркер SNP_27 в SEQ ID NO: 27 (или в варианте последовательности SEQ ID NO: 27). В другом варианте выполнения настоящего изобретения QTL3.1 (или его вариант) локализуется между маркером SNP_01 в SEQ ID NO: 1 (или в варианте последовательности SEQ ID NO: 1) и маркер SNP_10 в SEQ ID NO: 10 (или в варианте последовательности SEQ ID NO: 10). В другом варианте выполнения настоящего изобретения QTL3.1 (или его вариант) локализуется между маркером SNP_10 в SEQ ID NO: 10 (или в варианте последовательности SEQ ID NO: 10) и маркер SNP_20 в SEQ ID NO: 20 (или в варианте последовательности SEQ ID NO: 20). В другом варианте выполнения настоящего изобретения QTL3.1 (или его вариант) локализуется между маркером SNP_20 в SEQ ID NO: 20 (или в варианте последовательности SEQ ID NO: 20) и маркер SNP_27 в SEQ ID NO: 27 (или в варианте последовательности SEQ ID NO: 27). В другом варианте выполнения настоящего изобретения QTL3.1 (или его вариант) локализуется между маркером SNP_06 в SEQ ID NO: 06 (или в варианте последовательности SEQ ID NO: 06) и маркер SNP_23 в SEQ ID NO: 23 (или в варианте последовательности SEQ ID NO: 23).
В другом варианте выполнения настоящего изобретения фрагмент интрогрессии согласно настоящему изобретению (содержащий QTL3.1 или его вариант) представляет собой фрагмент, содержащий более маленький фрагмент (часть) области, начинающейся при нуклеотиде (или основании) 21,507,892 хромосомы 3 и заканчивающейся при нуклеотиде (или основании) 27,233,985 хромосомы 3, например, имеющий размер, например, 5.0 Мб, 4.0 Мб, 3.0 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 100 кб, 50 кб, 35 кб, 30 кб, 20 кб, или менее и содержащий QTL или его вариант. В одном варианте выполнения настоящего изобретения часть составляет по меньшей мере 5 кб, 10 кб, 20 кб по размеру, или более.
В одном варианте выполнения настоящего изобретения культивированное растение огурца согласно настоящему изобретению содержит фрагмент интрогрессии из дикого родственника огурца, фрагмент интрогрессии которого содержит QTL3.1 или его вариант, где фрагмент интрогрессии содержит все части области, начинающейся при 21.50 Мб и заканчивающейся при 27.3 Мб физической хромосомы 3.
В одном варианте выполнения настоящего изобретения фрагмент интрогрессии на хромосоме 3, содержащий QTL3.1, или его вариант, является получаемым скрещиванием растения, выращенного из NCIMB42346 с другим растением огурца, особенно культивированного растения огурца, в одном варианте выполнения настоящего изобретения огурцом длинного типа.
В одном варианте выполнения настоящего изобретения культивированное растение огурца согласно настоящему изобретению, содержащее QTL3.1, или его вариант, представляет собой растение, в котором указанный фрагмент интрогрессии на хромосоме 3 является получаемым скрещиванием растения, выращенного из семян, депонированных под номером доступа NCIMB 42345, с другим растением огурца. Таким образом, в одном варианте выполнения настоящего изобретения QTL представляет собой QTL, присутствующий в семенах, депонированных под номером доступа NCIMB 42345.
Когда делается ссылка на маркеры SNP в настоящей заявке, которые указывают на присутствие фрагмента интрогрессии (и QTL урожая, присутствующий на фрагменте интрогрессии), понятно, что упоминается генотип SNP, который является показателем фрагмента интрогрессии, т.е. генотип SNP, как представлено в Таблице 5 далее. Отмечается, что генотип маркера SNP может отличать фрагмент интрогрессии, находящийся в гомозиготной или гетерозиготной форме, как показано в таблице. В гомозиготной форме нуклеотид идентичен, а в гетерозиготной форме нуклеотид не идентичен. SNP-генотип хромосомы «дикого типа» без фрагмента интрогрессии это другой генотип, также приведенный в Таблице 5 (под генотипом рецидивирующего родителя). Так, например, генотип SNP_01 указывающий на фрагмент интрогрессии, содержащий QTL3.1, представляет собой 'ТС' (QTL3.1/wt) или 'ТТ' (QTL3./QTL3.1), тогда как генотип SNP, указывающий на дикий тип/генетический контроль (отсутствие фрагмента интрогрессии), представляет собой 'СС' (wt/wt). Таким образом, при ссылке на растение или часть растения (например, клетка), содержащие фрагмент интрогрессии в гомозиготной или гетерозиготной форме, понятно, что маркеры SNP, связанные с фрагментом интрогрессии, имеют соответствующий генотип SNP.
Поэтому, в одном варианте выполнения настоящего изобретения, обеспечивает культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает увеличение урожая плодов огурца по сравнению с растением огурца, не содержащим фрагмент интрогрессии на хромосоме 3, например, генетическим контролем или контрольным сортом, при выращивании при тех же условиях.
Увеличение урожая плодов огурца фенотипически выражается как (статистически) значительно более высокое среднее число плодов на растение (FrPP) линии или разновидности культивированного растения огурца, содержащей фрагмент интрогрессии на хромосоме 3 в гомозиготной или гетерозиготной форме, по сравнению с линией или разновидностью генетического контроля, не содержащей фрагмент интрогрессии на хромосоме 3, при выращивании в той же среде, и/или значительно более высокая средняя масса плода на растение (GrPP) линии или разновидности растений, содержащей фрагмент интрогрессии, по сравнению с линией или разновидностью генетического контроля, не содержащей фрагмент интрогрессии, при выращивании в той же среде.
Урожай плодов (общее среднее FrPP и/или GrPP) предпочтительно составляет у растения огурца, содержащего QTL3.1 (или вариант) по меньшей мере на 3%, 4%, 5%, 6%, 7%, 8%, 9%, 10%, 11%, 12%, 13%, 14%, 15% выше, чем у контроля, предпочтительно чем у генетического контроля, при выращивании при тех же условиях.
Растения согласно настоящему изобретению поэтому содержат геном культивированного огурца, с по меньшей мере одной или двумя рекомбинантными хромосомами, а именно одной или двумя рекомбинантными хромосомами 3 (т.е. гетерозиготной или гомозиготной). Рекомбинантные хромосомы содержат фрагмент дикого родственника огурца, который легко отличить от генома культивированного огурца посредством анализа молекулярного маркера, секвенирования всего генома, "росписи" хромосомы и подобных методик.
В одном варианте выполнения настоящего изобретения фрагмент интрогрессии на хромосоме 3 происходит из дикого родственника огурца, содержит положительный QTL3.1 урожая, или его вариант, и содержит всю или часть области, начинающейся при нуклеотиде 21,507,892 хромосомы 3 и заканчивающейся при нуклеотиде 27,233,985 хромосомы 3. Таким образом, фрагмент интрогрессии содержит QTL3.1 урожая или его вариант и один или более или все (например, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27) SNP маркеры дикого родственника огурца, выбранного SNP_01 - SNP_27, как показано в Таблице 5.
Понятно, что для маркеров, которые представляют собой более маленькие фрагменты интрогрессии, описанные в настоящей заявке, т.е. не содержащие 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15 или более маркеров из маркеров, перечисленных в настоящей заявке (например, SNP_01 to SNP_27), или подгрупп этих маркеров, SNP генотип дикого родственника огурца отсутствует (т.е. донорный генотип не обнаруживается), и, вместо этого, SNP генотип культивированного огурца обнаруживается для "пропущенного" SNP маркера.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии на хромосоме 3 в геноме растения или клетки растения или ткани растения (или в ДНК, экстрагированной из них) обнаруживается посредством анализа молекулярного маркера, который обнаруживает один или более молекулярных маркеров фрагмента интрогрессии. Однако, как упомянуто, могут применяться другие методики, например, SNP генотип маркеров также может быть обнаружен посредством секвенирования или применяя альтернативные маркеры, локализованные между SNP маркерами, раскрытыми в настоящей заявке, или в пределах 7 сМ, или в пределах 5 сМ, маркера, раскрытого в настоящей заявке; или в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 кб, 20 кб, 10 кб, 5 кб, 2 кб, 1 кб или менее маркера, раскрытого в настоящей заявке.
Когда ссылка делается на один или более молекулярные маркеры, являющиеся "обнаруживаемыми" посредством анализа молекулярного маркера, это означает, конечно, что растение или часть растения содержит один или более маркеров в его геноме, так как, в противном случае, маркеры были бы не обнаруживаемыми.
Растения огурца, содержащие фрагмент интрогрессии на хромосоме 3 (QTL 3.1 урожая)
В одном варианте выполнения настоящего изобретения обеспечивается культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии из дикого родственника огурца на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогресеии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1 (или варианта последовательности SEQ ID NO: 1) и маркером однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27 (или варианта последовательности SEQ ID NO: 1), где QTL обеспечивает увеличение урожая плодов огурца. В одном варианте выполнения настоящего изобретения QTL локализован между основанием 21,507,892 (SNP_01) и основанием 27,233,985 (SNP_27) хромосомы 3.
Таким образом, в одном варианте выполнения настоящего изобретения QTL3.1 локализуется в области между SNP_01 в SEQ ID NO: 1 (или в ее варианте) и SNP_27 в SEQ ID NO: 27 (или его вариант).
Поэтому, в одном варианте выполнения настоящего изобретения обеспечивается культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает увеличение урожая плодов огурца (по сравнению с растением, не содержащим фрагмент интрогрессии, например, генетический контроль), и где указанный фрагмент интрогрессии обнаруживается посредством анализа молекулярного маркера (т.е. растение содержит один или более молекулярных маркеров), который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26 или 27 маркеров, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в ее варианте);
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в ее варианте);
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в ее варианте);
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в ее варианте);
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в ее варианте);
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6(или в ее варианте);
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7(или в ее варианте);
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8(или в ее варианте);
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в ее варианте);
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в ее варианте);
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в ее варианте);
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в ее варианте);
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в ее варианте);
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в ее варианте);
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в ее варианте);
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в ее варианте);
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в ее варианте);
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в ее варианте);
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
u) генотип AC или АА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в ее варианте);
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в ее варианте);
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23 (или в ее варианте);
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 в SEQ ID NO: 24 (или в ее варианте);
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 в SEQ ID NO: 25 (или в ее варианте);
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 в SEQ ID NO: 26 (или в ее варианте);
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27 (или в ее варианте); и необязательно
bb) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_01 и SNP_27.
Как упомянуто ранее, при ссылке SNP в вариантной последовательности (в варианте последовательности), эта вариантная последовательность на по меньшей мере 85% идентична упомянутой последовательности.
В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17,18, 19, 20, 21, 22, 23, 24, 25, 26 или 27 маркеров выбираются из группы, состоящей из маркеров а)-аа). В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26 или 27 маркеров являются последовательными маркерами.
Как упомянуто, специалист в данной области техники также может разработать другие молекулярные маркеры, например, маркер, специфичный для генома дикого родственника огурца, между маркером SNP_01 и SNP_27 и/или в пределах 7 сМ или в пределах 5 сМ любого из SNP_01 - SNP_27, и/или в пределах 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1 Мб, 0.5 Мб, 0.4 Мб, 0.3 Мб, 0.2 Мб, 0.1 Мб, 50 кб, 20 кб, 10 кб, 5 kb или менее любого из SNP_01 - SNP_27. Такими маркерами также могут быть удлинение нуклеотида, CAPS маркеры, INDEL и т.д. Специалист в данной области техники может, например, секвенировать фрагмент интрогрессии, обнаруженный в семенах, депонированных под номером доступа NCIMB 42346 и применять информацию о последовательности для разработки новых маркеров и анализов маркеров.
В другом варианте выполнения настоящего изобретения QTL3.1 локализуется в области между SNP_01 в SEQ ID NO: 1 (или в ее варианте) и SNP_10 в SEQ ID NO: 10 (или его вариант).
Таким образом, в другом варианте выполнения настоящего изобретения обеспечивается культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает увеличение урожая плодов огурца (по сравнению с растением, не содержащим фрагмент интрогрессии, например, генетический контроль), и где указанный фрагмент интрогрессии обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10 маркеров, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в ее варианте);
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в ее варианте);
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в ее варианте);
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в ее варианте);
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в ее варианте);
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в ее варианте);
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в ее варианте);
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в ее варианте);
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в ее варианте);
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте); и необязательно
k) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_01 и SNP_10.
В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, или 10 маркеров выбираются из группы, состоящей из маркеров a)-j). В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, или 10 маркеров являются последовательными маркерами.
В другом варианте выполнения настоящего изобретения QTL3.1 локализуется в области между SNP_10 в SEQ ID NO: 10 (или в ее варианте) и SNP_20 в SEQ ID NO: 20 (или его вариант).
Поэтому в другом варианте выполнения настоящего изобретения обеспечивается культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает увеличение урожая плодов огурца (по сравнению с растением, не содержащим фрагмент интрогрессии, например, генетический контроль), и где указанный фрагмент интрогрессии обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10 или 11 маркеров, выбранных из группы, состоящей из:
1) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
2) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в ее варианте);
3) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в ее варианте);
4) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в ее варианте);
5) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в ее варианте);
6) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в ее варианте);
7) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в ее варианте);
8) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в ее варианте);
9) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в ее варианте);
10) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в ее варианте);
11) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте); и необязательно
12) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_10 и SNP_20.
В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10 или 11 маркеров выбираются из группы, состоящей из маркеров 1)-11). В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, или 11 маркеров являются последовательными маркерами.
В другом варианте выполнения настоящего изобретения QTL3.1 локализуется в области между SNP_20 в SEQ ID NO: 20 (или в ее варианте) и SNP_27 в SEQ ID NO: 27 (или его вариант).
Поэтому в другом варианте выполнения настоящего изобретения обеспечивается культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогрессии на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогрессии обеспечивает увеличение урожая плодов огурца (по сравнению с растением, не содержащим фрагмент интрогрессии, например, генетический контроль), и где указанный фрагмент интрогрессии обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7 или 8 маркеров, выбранных из группы, состоящей из:
1) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
2) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в ее варианте);
3) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в ее варианте);
4) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23 (или в ее варианте);
5) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 в SEQ ID NO: 24 (или в ее варианте);
6) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 в SEQ ID NO: 25 (или в ее варианте);
7) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 в SEQ ID NO: 26 (или в ее варианте);
8) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27 (или в ее варианте); и необязательно
9) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_20 и SNP_27.
В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7 или 8 маркеров выбираются из группы, состоящей из маркеров 1)-8). В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, или 8 маркеров являются последовательными маркерами.
В другом варианте выполнения настоящего изобретения QTL3.1 локализуется в области между SNP_06 в SEQ ID NO: 06 (или в ее варианте) и SNP_23 в SEQ ID NO: 23 (или его вариант).
Таким образом, обеспечивается культивированное растение Cucumis sativus var. sativus, содержащее фрагмент интрогресеии на хромосоме 3 в гомозиготной или гетерозиготной форме, где указанный фрагмент интрогресеии обеспечивает увеличение урожая плодов огурца (по сравнению с растением, не содержащим фрагмент интрогресеии, например, генетический контроль), и где указанный фрагмент интрогрессии обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18 маркеров, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в ее варианте);
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в ее варианте);
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в ее варианте);
d) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в ее варианте);
e) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
f) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в ее варианте);
g) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в ее варианте);
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в ее варианте);
i) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в ее варианте);
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в ее варианте);
k) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в ее варианте);
l) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в ее варианте);
m) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в ее варианте);
n) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в ее варианте);
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
р) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в ее варианте);
q) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в ее варианте);
r) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23 (или в ее варианте); и необязательно
s) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_06 и SNP_23.
В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 или 18 маркеров выбираются из группы, состоящей из маркеров а)-r). В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, или 18 маркеров являются последовательными маркерами.
Фрагмент, содержащий QTL, может, таким образом, быть большим (содержащий SNP_01 - SNP_27), или может быть меньше и не содержать маркеры, имеющие генотип дикого родственника огурца (т.е. маркеры имеют, вместо этого, генотип культивированного огурца, смотрите также генотип рекуррентного родителя в Таблице 5), но он может все еще обеспечивать увеличенный урожай культивированному растению огурца, т.е. он может все еще содержать аллель урожая (QTL3.1 или вариант). Такие более маленькие фрагменты интрогрессии представляют собой вариант выполнения настоящего изобретения. Растения, имеющие более маленькие фрагменты интрогрессии, которые все еще обеспечивают увеличенный урожай (т.е. содержат аллель урожая), могут быть получены используя известные методики, такие как тонкое картирование или подобные методики. Например, начиная с растения, содержащего фрагмент интрогрессии, как обнаруживается в семенах, депонированных под номером доступа NCIMB 42346, и скрещивая такое растение с другим культивированным растением огурца, и посредством самоопыления потомства указанного кросса, и/или обратного скрещивания потомства, с получением популяции растения, которая может содержать рекомбинанты, имеющие более маленький фрагмент интрогрессии на хромосоме 3, фрагменты которой все еще обеспечивают увеличенный выход относительно растения, не содержащего фрагмент интрогрессии (такого как генетический контроль, например, растения, выращенные из семян, депонированных под номером NCIMB 42345), например, фрагмент, содержащий маркеры SNP_01 - SNP_10 (или более маленький, например, содержащий только 9, 8, 7, 6, 5, 4, 3, 2 или 1 из SNP маркеров), SNP_10 - SNP_20 (или более маленький, например, содержащий только 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 из SNP маркеров), SNP_20 - SNP_27 (или более маленький, например, содержащий только 7, 6, 5, 4, 3, 2 или 1 из SNP маркеров) или SNP_06 - SNP_23 (или более маленький, например, содержащий только 17, 16, 15, 14, 13, 12, 11, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 из SNP маркеров), анализы маркера могут применяться для определения размера более маленького фрагмента интрогрессии. Один или более SNP маркеров с генотипом дикого родственника огурца могут отсутствовать. Генотип культивированного огурца затем обнаруживается по этим SNP маркерам. Урожай растений, содержащих такой более маленький фрагмент интрогрессии, можно затем сравнить в экспериментах на урожайность, как описано в настоящей заявке, т.е. выращивая множество растений, содержащих более маленький фрагмент интрогрессии, в полевых экспериментах вместе с подходящими контрольными растениями, не содержащими фрагмент интрогрессии. Контрольные растения предпочтительно являются генетическим контролем, таким как NCIMB 42345. Если средний урожай остается значительно выше, чем в контроле, то меньший фрагмент интрогрессии сохранил QTL3.1.
Альтернативно, тот же или сорт QTL (QTL3.1 или вариант QTL3.1) может быть интрогрессирован из другого источника дикого типа, причем необязательно не все маркеры SNP, раскрытые в настоящей заявке, могут присутствовать. Такие альтернативные источники дикого родственника огурца могут быть идентифицированы с использованием маркеров SNP, представленных в настоящей заявке, посредством скрининга зародышевой плазмы (т.е. экземпляров) диких родственников огурца, применяя анализ маркера для обнаружения генотипа маркеров SNP_01 - SNP_27, или маркеров SNP_01 - SNP_10, SNP_10 - SNP_20, SNP_20 - SNP_27, или SNP_06 - SNP_23, или даже только более маленькой подгруппы этих маркеров (например, 2, 3, 4, 5, 6, 7, 8, или более). Растения, содержащие QTL3.1 или вариант QTL3.1 из других источников, также являются вариантом выполнения настоящего изобретения. До тех пор, пока по меньшей мере один или более (или все) из SNP из SNP_01 - SNP_27, или из SNP из SNP_01 - SNP_10, или из SNP из SNP_10 - SNP_20, или из SNP из SNP_20 - SNP_27, или из SNP из SNP_06 - SNP_23, присутствует, растение имеет урожай-повышающий генотип, т.е. растение содержит QTL3.1 (или его вариант). Специалист в данной области техники может интрогрессировать QTL3.1 (или его вариант) в культивированный огурец, чтобы увеличить урожай плодов, как описано в настоящей заявке, и чтобы подтвердить, что QTL увеличивает урожай, когда присутствует в культивированном огурце.
Как описано выше, в одном варианте выполнения настоящего изобретения культивированное растение огурца согласно настоящему изобретению содержит фрагмент интрогрессии, содержащий по меньшей мере подряд SNP маркеров с генотипом дикого родственника огурца, т.е. по меньшей мере 1, 2, 3, 4, или 5 маркеров из SNP_01 - SNP_27, или SNP_01 - SNP_10, или из SNP_10 - SNP_20, или из SNP_20 - SNP_27, или из SNP_06 - SNP_23. В одном варианте выполнения настоящего изобретения культивированное растение огурца содержит все, или все за исключением 1 или 2 маркеров из SNP_01 - SNP_27, или из SNP_01 - SNP_10, или из SNP_10 - SNP_20, или из SNP_20 - SNP_27, или из SNP_06 - SNP_23.
Таким образом, фрагмент интрогрессии (и культивированное растение огурца или часть растения, например, клетка, содержащие фрагмент интрогрессии) может быть обнаружен в анализе маркера посредством обнаружения SNP генотипа фрагмента интрогрессии (т.е. зародышевой плазмы дикого родственника огурца) одного или более или всех маркеров, указанных выше.
Таким образом, в одном варианте выполнения настоящего изобретения, локус количественных признаков (QTL3.1), как обнаружено, присутствует на хромосоме 3 дикого родственника огурца, который, при переносе (интрогрессии) в сорт или линию разведения культивированного огурца, и когда присутствует в гетерозиготной или гомозиготной форме, придает значительно увеличенный урожай плодов культивированному растению огурца. QTL, или фрагмент интрогрессии, содержащий QTL (содержащий аллель урожая), является, таким образом, доминантным, т.е. он является достаточным, чтобы иметь фрагмент интрогрессии на одной из хромосом 3 (одна рекомбинантная хромосома 3), тогда как гомологичной хромосомой 3 пары может быть (нерекомбинантная) хромосома 3 культивированного С.sativus var. sativus, не содержащего фрагмент интрогрессии.
Хотя источником QTL урожая в контексте настоящего изобретения является единственным специфическим диким источником, вероятно, есть и другие дикие виды Cucumis, которые содержат QTL3.1 в том же локусе на хромосоме 3. Такие локусы могут содержать аллели урожая, которые имеют несколько другие нуклеотидные последовательности, т.е. варианты аллелей (QTL), обнаруживаемых в настоящей заявке. Такие варианты QTL также могут быть идентифицированы и интрогрессированы в культивированный огурец, как описано в настоящей заявке, с получением культивированного растения огурца, содержащего геном культивированного С. sativus var. sativus и рекомбинантную хромосому 3, причем рекомбинантная хромосома 3 содержит фрагмент интрогрессии дикого вида Cucumis sativus, который сообщает увеличенный урожай фенотипу культивированного растения огурца, когда присутствует в гомозиготной или гетерозиготной форме. Чтобы идентифицировать такой дикий огурец или дикие родственники огурца, содержащие QTL3.1, дикие образцы могут быть проанализированы, например, в анализе маркера путем сравнения последовательностей или другими методами, на присутствие одного или более SNP маркеров, приведенных в настоящей заявке. Предполагаемые QTL урожая (или варианты QTL) могут затем быть интрогрессированы в культивированный огурец, например, применяя MAS, т.е. применяя один или более (или все) из SNP маркеров, приведенных в настоящей заявке для обнаружения и/или отбора потомственных растений (например, беккросс-растений), содержащих рекомбинантную хромосому 3. Отобранные растения, т.е. культивированные растения огурца, содержащие фрагмент интрогрессии на хромосоме 3, где фрагмент интрогрессии на хромосоме 3 обнаруживается одним или более из SNP маркеров SNP_01 - SNP_27, одним или более из SNP маркеров SNP_01 - SNP_10, одним или более из SNP маркеров SNP_10 - SNP_20, одним или более из SNP маркеров SNP_20 - SNP_27, или одним или более из SNP маркеров SNP_06 - SNP_23 (как описано в настоящей заявке), могут затем подвергаться определению фенотипа в экспериментах на урожай вместе с подходящими контрольными растениями, предпочтительно по меньшей мере генетическим контролем, чтобы определить действительно ли фрагмент интрогрессии вызывает значительное увеличение урожая.
Образцы диких родственников огурца получают из системной коллекции зародышевой плазмы USDA National Plant или других коллекций семян, и могут, таким образом, быть протестированы на присутствие QTL3.1, применяя, например, анализ маркеров, как описано в настоящей заявке, и образцы, содержащие один или более из SNP маркеров (например, по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26 или все 27 SNP маркеров, указывающих на QTL3.1) могут быть скрещены с культивированным растением огурца, имеющим нормальные нерекомбинантные хромосомы 3 дикого типа. F1 или F2 поколение (или последующее поколение, такое как F3 или беккросс-поколение) может затем быть проанализировано на рекомбинантные растения, имеющие фрагмент интрогрессии или его часть, применяя анализы молекулярных маркеров, описанные в настоящей заявке.
В одном варианте выполнения настоящего изобретения, фрагмент интрогрессии происходит из дикого родственника огурца, который принадлежит к Индийской группе огурцов, и который переносится на хромосому 3 Евразийской группы хромосом, таким образом создавая культивированное растение огурца, содержащее QTL3.1 урожая или его вариант. Таким образом, в одном варианте выполнения настоящего изобретения фрагмент интрогрессии, содержащий QTL3.1 урожая является происходящим из (или происходит из) или является получаемым из (или происходит из; или как присутствует в) диком родственнике огурца, который принадлежит к Индийской группе огурцов.
В конкретном варианте выполнения настоящего изобретения, фрагмент интрогрессии, содержащий QTL3.1 урожая является происходящим из (или происходит из) или является получаемым из (или происходит из; или как присутствует в) семян, представительный пример которых был депонирован под номером доступа NCIMB 42346, или из их потомства. Потомством может быть любое потомство, которое содержит один или более (или все) SNP маркеры, указывающие на (и связанные с) QTL, как описано. Таким образом, потомство не ограничено до F1 или F2 потомства депозита, но может быть получено любым потомством, полученным либо самоопылением и/или скрещиванием с другим растением огурца.
В одном варианте выполнения настоящего изобретения фрагмент интрогрессии идентифицируется одним или более из маркеров, описанных в настоящей заявке, особенно маркеров SNP_01 - SNP_27 для фрагмента интрогрессии на хромосоме 3, или подряда маркеров, таких как один или более из маркеров, выбранных из SNP маркеров SNP_01 - SNP_10, или из SNP маркеров SNP_10 - SNP_20, или из SNP маркеров SNP_20 - SNP_27, или из SNP маркеров SNP_06 - SNP_23. В одном варианте выполнения настоящего изобретения изобретение обеспечивает культивированное растение огурца, имеющее геном культивированного (одомашненного) огурца, который имеет увеличенный урожай плодов, где увеличенный урожай плодов обеспечивается фрагментом интрогрессии на хромосоме 3 культивированного огурца, где указанный фрагмент интрогрессии получают посредством (или является получаемым посредством) скрещивания культивированного растения, выращенного из семян, депонированных под номером NCIMB 42346, или потомства этого растения (которое содержит один или более маркеров, раскрытых в настоящей заявке, связанных с QTL) с культивированным растением огурца. Таким образом, в одном варианте выполнения настоящего изобретения культивированное растение огурца согласно настоящему изобретению содержит тот же фрагмент интрогрессии и ту же рекомбинантную хромосому 3, как присутствует в NCIMB 42346 (содержащий все из маркеров SNP генотипа дикого родственника огурца SNP_01 - SNP_27), или оно содержит более короткий фрагмент этого фрагмента интрогрессии, причем более короткий фрагмент сохраняет генетический элемент, обеспечивающий увеличенный урожай плодов (QTL3.1).
Таким образом, в одном варианте выполнения настоящего изобретения изобретение относится к растению согласно настоящему изобретению т.е. культивированному растению Cucumis sativus var. sativus, содержащему фрагмент интрогрессии из дикого родственника огурца на хромосоме 3 в гомозиготной или гетерозиготной форме, и где указанный фрагмент интрогрессии представляет собой фрагмент интрогрессии "как в" / являющийся "идентичным с" / являющийся "таким же как в" семенах, депонированных под номером NCIMB 42346, или представляет его более короткий фрагмент, но все еще обеспечивает увеличенный урожай плодов благодаря присутствию QTL3.1.
В другом варианте выполнения настоящего изобретения изобретение относится к растению согласно настоящему изобретению т.е. культивированному растению Cucumis sativus var. sativus, содержащему фрагмент интрогрессии из дикого родственника огурца на хромосоме 3 в гомозиготной или гетерозиготной форме, и где указанный фрагмент интрогрессии представляет собой фрагмент интрогрессии или вариант фрагмента интрогрессии из семян, депонированных под номером NCIMB 42346, т.е. он содержит QTL 3.1 урожая, но геномная последовательность может быть другой. Поскольку дикие образцы будут генетически различными, геномная последовательность фрагмента интрогрессии, содержащего QTL3.1 из других диких родственников огурца, наиболее вероятно не будет идентична геномной последовательности, так как интрогрессирована в NCIMB 42346, и даже ген, обеспечивающий урожай (содержащий промотор, интроны и экзоны), может отличаться по нуклеотидной последовательности, но функция будет такой же, т.е. обеспечение увеличенного урожая плодов. Расхождение можно увидеть в том, что некоторые маркеры SNP, связанные с QTL3.1, могут быть в общем обнаружены в различных экземплярах, тогда как другие SNP маркеры могут быть обнаружены только в определенных экземплярах. Поэтому, например, не все из SNP_01 - SNP_27 можно обнаружить в других диких родственниках огурца. Однако повышающая урожай QTL3.1 (содержащая, например, вариант или ортолог аллеля урожая) все еще может присутствовать в таких экземплярах дикого типа. Специалист в данной области техники способен идентифицировать и интрогрессировать область, содержащую QTL 3.1, обнаруженную в других диких родственниках огурца, в культивированный огурец, например, обнаруживая диких родственников, содержащих SNP маркеры или их подряд, и перенося эти SNP маркеры (или подряд) в линию или сорт культивированного огурца, и оценивая урожай плодов культивированной линии или сорта по сравнению с линией или сортом, не содержащим SNP маркеры (или подряд), т.е. не содержащим фрагмент интрогрессии.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии или области хромосомы 3 (или вариантная или ортологическая область хромосомы 3), содержащих QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26 или более (или всеми 27) маркерами однонуклеотидного полиморфизма (SNP), выбранными из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в ее варианте);
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в ее варианте);
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в ее варианте);
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в ее варианте);
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в ее варианте);
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в ее варианте);
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в ее варианте);
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в ее варианте);
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в ее варианте);
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в ее варианте);
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в ее варианте);
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в ее варианте);
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в ее варианте);
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в ее варианте);
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в ее варианте);
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в ее варианте);
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в ее варианте);
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в ее варианте);
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в ее варианте);
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в ее варианте);
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23 (или в ее варианте);
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 в SEQ ID NO: 24 (или в ее варианте);
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 в SEQ ID NO: 25 (или в ее варианте);
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 в SEQ ID NO: 26 (или в ее варианте);
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27 (или в ее варианте); и
bb) необязательно любой специфичный для генома дикого родственника огурца маркер между маркером SNP_01 и SNP_27.
В одном варианте выполнения настоящего изобретения указанный по меньшей мере 1, предпочтительно по меньшей мере 2 или 3, или по меньшей мере 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26 или 27 маркеров, которые обнаруживаются, представляют собой последовательные маркеры.
Таким образом, в одном варианте выполнения настоящего изобретения растения согласно настоящему изобретению содержат по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 1 (упоминается как SNP_01) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 1 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 2 (упоминается как SNP_02) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 2 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 3 (упоминается как SNP_03) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 3 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 4 (упоминается как SNP_04) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 4 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 75 последовательности SEQ ID NO: 5 (упоминается как SNP_05) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 5 (другими словами гуанин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 6 (упоминается как SNP_06) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 6 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 7 (упоминается как SNP_07) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 7 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 8 (упоминается как SNP_08) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 8 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 9 (упоминается как SNP_09) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 9 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 10 (упоминается как SNP_10) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 10 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или TG) вместо двух гуанинов (GG) при нуклеотиде 75 последовательности SEQ ID NO: 11 (упоминается как SNP_11) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 11 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере аденин (А) (т.е. генотип АА или AG) вместо двух гуанинов (GG) при нуклеотиде 75 последовательности SEQ ID NO: 12 (упоминается как SNP_12) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 12 (другими словами аденин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 13 (упоминается как SNP_13) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 13 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере аденин (А) (т.е. генотип АА или AG) вместо двух гуанинов (GG) при нуклеотиде 75 последовательности SEQ ID NO: 14 (упоминается как SNP_14) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 14 (другими словами аденин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 15 (упоминается как SNP_15) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 15 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 16 (упоминается как SNP_16) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 16 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 75 последовательности SEQ ID NO: 17 (упоминается как SNP_17) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 17 (другими словами гуанин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 18 (упоминается как SNP_18) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 18 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 75 последовательности SEQ ID NO: 19 (упоминается как SNP_19) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 19 (другими словами гуанин находится в физическом положении хромосомы 3, показанной в Таблице 5); и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 20 (упоминается как SNP_20) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 20 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере аденин (А) (т.е. генотип АА или АС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 21 (упоминается как SNP_21) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 21 (другими словами аденин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере тиамин (Т) (т.е. генотип ТТ или ТС) вместо двух цитозинов (СС) при нуклеотиде 75 последовательности SEQ ID NO: 22 (упоминается как SNP_22) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 22 (другими словами тиамин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере цитозин (С) (т.е. генотип СС или СТ) вместо двух тиминов (ТТ) при нуклеотиде 75 последовательности SEQ ID NO: 23 (упоминается как SNP_23) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 23 (другими словами цитозин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 75 последовательности SEQ ID NO: 24 (упоминается как SNP_24) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 24 (другими словами гуанин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 75 последовательности SEQ ID NO: 25 (упоминается как SNP_25) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 25 (другими словами гуанин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере гуанин (G) (т.е. генотип GG или GA) вместо двух аденинов (АА) при нуклеотиде 251 последовательности SEQ ID NO: 26 (упоминается как SNP_26) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 26 (другими словами гуанин находится в физическом положении хромосомы 3, показанной в Таблице 5);
и/или по меньшей мере аденин (А) (т.е. генотип АА или AG) вместо двух гуанинов (GG) при нуклеотиде 75 последовательности SEQ ID NO: 27 (упоминается как SNP_27) или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 27 (другими словами аденин находится в физическом положении хромосомы 3, показанной в Таблице 5).
В другом варианте выполнения настоящего изобретения, присутствие фрагмента интрогрессии или области хромосомы 3 (или вариантная или ортологическая область хромосомы 3), содержащих QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 1, предпочтительно по меньшей мере 2, 3, 4, 5, 6, 7, 8, или более маркеров однонуклеотидного полиморфизма (SNP) из подгрупп, состоящих из: SNP_01 - SNP10 или любого маркера, специфичного для генома дикого родственника огурца, локализованного между маркером SNP_01 и SNP_10; SNP_10 - SNP_20 или любого маркера, специфичного для генома дикого родственника огурца, локализованного между маркером SNP_10 и SNP_20; SNP_20 - SNP_27 или любого маркера, специфичного для генома дикого родственника огурца, локализованного между маркером SNP_20 и SNP_27; или SNP_06 - SNP_23 или любого маркера, специфичного для генома дикого родственника огурца, локализованного между маркером SNP_06 и SNP_23.
Генотип SNP ссылается на два нуклеотида, и геномные последовательности, содержащие один из этих двух нуклеотидов, по одному на каждую хромосому 3. Поэтому растение, имеющее генотип ТТ для SNP_01 имеет идентичный нуклеотид (Т) на обеих хромосомах (т.е. является гомозиготным), тогда как растение, имеющее генотип ТС для SNP_01 имеет одну хромосому с Т при нуклеотиде 75 последовательности SEQ ID NO: 1 (или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 1) и одну хромосому с С при нуклеотиде 75 последовательности SEQ ID NO: 1 (или при эквивалентном нуклеотиде геномной последовательности, имеющей существенную идентичность последовательности SEQ ID NO: 1) и является гетерозиготным. Так как геномные последовательности вокруг маркеров SNP, представленные в настоящей заявке, могут незначительно отличаться в фрагментах интрогрессии, от других диких родственников огурца (т.е. вариантных или ортологических областях хромосом 3), ясно, что нуклеотидные последовательности до и после SNP могут не быть на 100% идентичными последовательностям, представленным в настоящей заявке. Поэтому последовательности, имеющие существенную идентичность последовательности последовательностям, представленным в настоящей заявке (при выравнивании по всей длине, как обнаружено), но которые содержат тот же SNP, охватываются настоящим изобретением.
В одном варианте выполнения настоящего изобретения, фрагмент интрогрессии, или область хромосомы 3 (или вариантная или ортологическая область хромосомы 3), содержащие QTL (QTL3.1 или вариант), который является обнаруживаемым одним или более маркерами, происходит из дикого родственника огурца, и в одном варианте выполнения настоящего изобретения дикий родственник является членом Индийской группы огурцов. В одном варианте выполнения настоящего изобретения он представляет собой тот же фрагмент интрогрессии как обнаруживается на хромосоме 3 в семенах, депонированных под номером доступа NCIMB42346, или более маленький фрагмент, содержащий QTL. SNP маркеры SNP_01 - SNP_27 занимают область 5.7 Мб. В одном варианте выполнения настоящего изобретения фрагмент интрогрессии на хромосоме 3 равен или составляет менее 10 Мб по размеру, предпочтительно равен или составляет менее 8 Мб по размеру, более предпочтительно равен или составляет менее 6, 5.7, 5, 4, 3 или 2.5 Мб по размеру, например, равен или составляет менее 2 Мб. В одном варианте выполнения настоящего изобретения фрагмент интрогрессии составляет по меньшей мере 0.2 Мб, 0.5 Мб, 1.0 Мб, 1.5 Мб, 1.9 Мб, 2.0 Мб, 2.5 Мб, 2.7 Mb или 3 Мб по размеру. Таким образом, различные диапазоны размеров интрогрессия охватываются настоящим изобретением, такие как фрагменты менее 10 Мб, но более 0.2 Мб, менее 6 Мб или 3 Мб, но более 0.2 Мб, 0.5 MB или 1 Мб, и т.д., которые сохраняют QTL3.1 и один или более из SNP маркеров SNP_01 - SNP_27, или подгрупп SNP_01 - SNP_10; SNP_10 - SNP_20; SNP_20 - SNP_27 или SNP_06 - SNP_23. Как упомянуто ранее, локализация QTL3.1 в области SNP_01 - SNP_27 может быть определена посредством тонкого картирования, и рекомбинанты, содержащие QTL3.1 на более маленьком фрагменте интрогрессии, могут быть получены Размер может быть легко определен посредством, например, секвенирования всего генома или секвенирования нового поколения, например, как описано в Qi et al. 2013 {выше) или в Huang et al. 2009 {выше). В частности, области интрогрессии могут легко отличаться от культивированных геномных областей, из-за большего числа геномной изменчивости (SNP, INDEL и т.д.) в области интрогрессии.
Чтобы получить фрагмент интрогрессии, присутствующий на хромосоме 3, из депонированных семян (NCIMB42346), т.е. перенести один или оба фрагмента интрогрессии, содержащих QTL в другое культивированное растение огурца, растение выращивают из семени, и растение скрещивают с культивированным растением огурца, чтобы получить F1 семена. Так как NCIMB42346 содержит только одну рекомбинантную хромосому 3 (содержащую фрагмент интрогрессии), только около половины (50%) семян F1 и растений, выращенных из них, содержит одну рекомбинантную хромосому 3 из родителя NCIMB42346 и одну нерекомбинантную хромосому 3 из другого культивированного растения. Другая половина F1 семян не содержит рекомбинантную хромосому 3, но только две копии нерекомбинантной хромосомы 3. Таким образом, посредством традиционного разведения можно перенести рекомбинантную хромосому 3 из NCIMB42346 в другие линии или сорта культивированного огурца. Растения, которые содержат QTL3.1, могут быть скринированы на и выбраны по присутствию одного или более из вышеуказанных SNP маркеров, чтобы идентифицировать растения, содержащие рекомбинантную хромосому 3.
Чтобы получить более короткие фрагменты интрогрессии мейоз должен иметь место, и растения, содержащие рекомбинантные хромосомы 3, и особенно новые события рекомбинации в пределах фрагмента интрогрессии, должны быть идентифицированы. Например, семена NCIMB42346 могут самоопыляться один или более раз для получения F1, F2 или F3 растений (или других поколений самоопыления), и/или F1, F2 или F3 растения (и т.д.), содержащие рекомбинантную хромосому 3 могут быть обратно скрещены с культивированным растением. Растения, которые содержат рекомбинантную хромосому 3, могут быть проанализированы и отобраны по присутствию одного или более из вышеуказанных SNP маркеров, чтобы идентифицировать растения, содержащие более маленький фрагмент интрогрессии. Такие новые рекомбинанты могут затем быть протестированы на присутствие QTL3.1 на более маленьком фрагменте интрогрессии посредством определения среднего урожая плодов по сравнению с (генетическим) контролем, не содержащим фрагмент интрогрессии.
Подобным образом, культивированные растения огурца, содержащие QTL3.1 (или его вариант), могут быть получены и/или идентифицированы, применяя различные способы. Например, чтобы получить культивированное растение огурца, содержащее фрагмент интрогрессии из дикого родственника огурца, идентифицируют первый дикий родственник огурца, который содержит один или более из SNP маркеров, связанных с QTL3.1, раскрытых в настоящей заявке, например, любой один, или более, или все маркеры, описанные в настоящей заявке выше. Идентифицированное растение скрещивают с культивированным растением огурца, чтобы получить F1 семена. F1 могут быть самоопылены с получением F2, F3, и т.д. растений, и/или F2 растения или F3 растения, и т.д., могут быть обратно скрещены с родительским культивированным огурцом. Растения, которые содержат QTL3.1 (или его вариант) могут быть проанализированы и/или отобраны по присутствию одного или более из вышеуказанных SNP маркеров, и/или проанализированы и/или отобраны по фенотипу увеличенного урожая по сравнению с первоначальным культивированным родителем (не содержащим интрогрессии). Альтернативно или дополнительно, нанесение QTL на генетическую карту может осуществляться, чтобы идентифицировать другие молекулярные маркеры, связанные с QTL3.1 (или его вариантом), и/или чтобы получить культивированные растения огурца, содержащие фрагмент интрогрессии на хромосоме 3, который увеличивает значительно увеличенный урожай.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растении огурца или области хромосомы 3 (или ортологической области хромосомы 3), содержащей QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, два, три, четыре, пять или более маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в ее варианте);
b) генотип АА или GA для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27 (или в ее варианте);
c) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_01 и SNP_27;
d) любой специфичный для генома дикого родственника огурца маркер, который генетически сцеплен в пределах 7 сМ, 5 сМ, 3 сМ или менее маркера SNP_01 или SNP_27; и
e) любой специфичный для генома дикого родственника огурца маркер, который физически сцеплен в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее маркера SNP_01 или SNP_27.
В одном варианте выполнения настоящего изобретения маркеры согласно с) представляют собой один или более из SNP_02 - SNP_26. В одном варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b) и/или с) выше. В другом варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте выполнения настоящего изобретения обнаруживаемыми маркерами являются последовательные маркеры.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растении огурца или области хромосомы 3 (или ортологической области хромосомы 3), содержащей QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, два, три, четыре, пять или более маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в ее варианте);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
c) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_01 и SNP_10;
d) любой специфичный для генома дикого родственника огурца маркер, который генетически сцеплен в пределах 7 сМ, 5 сМ, 3 сМ или менее маркера SNP_01 или SNP_10; и
e) любой специфичный для генома дикого родственника огурца маркер, который физически сцеплен в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее маркера SNP_01 или SNP_10.
В одном варианте выполнения настоящего изобретения маркеры согласно с) представляют собой один или более из SNP_02 - SNP_09. В одном варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b) и/или с) выше. В другом варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте выполнения настоящего изобретения обнаруживаемыми маркерами являются последовательные маркеры.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растении огурца или области хромосомы 3 (или ортологической области хромосомы 3), содержащей QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, два, три, четыре, пять или более маркеров, выбранных из группы, состоящей из:
a) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
c) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_10 и SNP_20;
d) любой специфичный для генома дикого родственника огурца маркер, который генетически сцеплен в пределах 7 сМ, 5 сМ, 3 сМ или менее маркера SNP_10 или SNP_20; и
e) любой специфичный для генома дикого родственника огурца маркер, который физически сцеплен в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее маркера SNP_10 или SNP_20.
В одном варианте выполнения настоящего изобретения маркеры согласно с) представляют собой один или более из SNP_11 - SNP_19. В одном варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b) и/или с) выше. В другом варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте выполнения настоящего изобретения обнаруживаемыми маркерами являются последовательные маркеры.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растении огурца или области хромосомы 3 (или ортологической области хромосомы 3), содержащей QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, два, три, четыре, пять или более маркеров, выбранных из группы, состоящей из:
a) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
b) генотип АА или GA для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27 (или в ее варианте);
c) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_20 и SNP_27;
d) любой специфичный для генома дикого родственника огурца маркер, который генетически сцеплен в пределах 7 сМ, 5 сМ, 3 сМ или менее маркера SNP_20 или SNP_27; и
e) любой специфичный для генома дикого родственника огурца маркер, который физически сцеплен в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее маркера SNP_20 или SNP_27.
В одном варианте выполнения настоящего изобретения маркеры согласно с) представляют собой один или более из SNP_21 - SNP_26. В одном варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b) и/или с) выше. В другом варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте выполнения настоящего изобретения обнаруживаемыми маркерами являются последовательные маркеры.
В одном варианте выполнения настоящего изобретения присутствие фрагмента интрогрессии в культивированном растении огурца или области хромосомы 3 (или ортологической области хромосомы 3), содержащей QTL3.1, обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один, два, три, четыре, пять или более маркеров, выбранных из группы, состоящей из:
a) генотип ТТ или ТС для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 06 (или в ее варианте);
b) генотип СС или СТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23 (или в ее варианте);
c) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_06 и SNP_23;
d) любой специфичный для генома дикого родственника огурца маркер, который генетически сцеплен в пределах 7 сМ, 5 сМ, 3 сМ или менее маркера SNP_06 или SNP_23; и
e) любой специфичный для генома дикого родственника огурца маркер, который физически сцеплен в пределах 5 Мб, 3 Мб, 2 Мб, 1 Мб, 0.5 Мб или 0.2 Мб или менее маркера SNP_06 или SNP_23.
В одном варианте выполнения настоящего изобретения маркеры согласно с) представляют собой один или более из SNP_07 - SNP_22. В одном варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b) и/или с) выше. В другом варианте выполнения настоящего изобретения, обнаруживаются по меньшей мере один, два, по меньшей мере три, по меньшей мере четыре или более маркеров из маркеров согласно а), b), с), d) и/или е) выше. В одном варианте выполнения настоящего изобретения по меньшей мере маркер согласно а) и/или b) обнаруживается, и необязательно по меньшей мере один, два, три или более маркеров согласно с), d) и/или е) обнаруживаются. В одном варианте выполнения настоящего изобретения обнаруживаемыми маркерами являются последовательные маркеры.
Любой маркер, специфичный для генома дикого родственника огурца, между двумя маркерами относится к любому молекулярному маркеру, который наносится на генетическую карту в области хромосомы 3 между двумя маркерами и/или который физически лежит между двумя маркерами, и который указывает на область хромосомы 3 дикого родственника огурца. Это означает, что маркер является полиморфным между геном культивированного огурца и геном дикого родственника огурца. В одном варианте, маркером является однонуклеотидный полиморфизм (SNP), но и другие молекулярные маркеры, такие как RFLP, AFLP, RAPD, ДНК секвенирования и т.д., могут применяться эквивалентным образом.
Фрагмент интрогрессии в растениях согласно настоящему изобретению в одном варианте представляет собой фрагмент хромосомы 3, который присутствует в семенах, депонированных под номером доступа NCIMB42346, или более маленький вариант этого фрагмента, сохраняющий QTL (полученный, например, посредством рекомбинации в пределах фрагмента интрогрессии).
Фрагмент интрогрессии в одном варианте равен или менее 10 Мб по размеру, предпочтительно равен или менее 8 Мб, 5 Мб, 3 Мб, 2.5 Мб, 2 Мб, 1.5 Мб, 1 Мб по размеру. В другом варианте фрагмент интрогрессии составляет по меньшей мере 0.5 Мб или по меньшей мере 1 Мб по размеру.
Также обеспечиваются семена, из которых растение согласно настоящему изобретению может быть выращено, как и плоды огурца, собранные с растения согласно настоящему изобретению и содержащие рекомбинантную хромосому 3 в их геноме. Подобным образом, обеспечивается растительная клетка, ткань или часть растения или семени, содержащего по меньшей мере одну рекомбинантную хромосому 3, где указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, и где указанный фрагмент интрогрессии содержит аллель, обеспечивающий значительно увеличенный урожай плодов.
Молекулярные маркеры, описанные в настоящей заявке, могут быть обнаружены согласно стандартному методу. Например, SNP маркеры могут быть легко обнаружены, применяя анализ KASP (смотрите www.kpbioscience.co.uk) или другие анализы. Для разработки анализы KASP, например, 70 пар оснований против хода транскрипции и 70 пар оснований по ходу транскрипции SNP можно выделить, и два аллель-специфичных прямых праймера и один аллель-специфичный обратный праймер могут быть разработаны. Смотрите, например, Allen et al. 2011, Plant Biotechnology J. 9, 1086-1099, в частности p097-1098 для способа на основе анализа KASP.
Таким образом, в одном варианте выполнения настоящего изобретения, SNP маркеры и присутствие/отсутствие маркера, связанного с QTL урожая, определяется, применяя анализ KASP, но в равной степени могут применяться другие анализы SNP генотипирования. Например, анализы SNP генотипирования TaqMan, высокоточный анализ кинетики плавления продуктов амплификации (HRM), SNP - массивы генотипирования (например, Fluidigm, Illumina, etc.) или ДНК секвенирование может применяться в равной степени.
Физический размер фрагмента интрогрессии можно определить различными способами, такими как физическое картирование, секвенирование или путем визуализации интрогрессии, применяя флуоресцентные изображения in situ гибридизации (FISH) (Verlaan et al. 2011, Plant Journal 68: 1093-1103).
Культивированные растения огурца с более маленькими фрагментами интрогрессии на хромосоме 3 могут быть получены путем генерации новых рекомбинантных растений из популяции растений, полученных из кросса между культивированным растением огурца (не содержащим интрогрессии) и растением согласно настоящему изобретению, и отбора рекомбинантного потомство, имеющего более маленькие размеры интрогрессии. Такие растения, таким образом, в одном варианте выполнения настоящего изобретения представляют собой растения, полученные из (потомство или потомки) рекомбинантной хромосомы 3, присутствующей в растениях, семена которых были депонированы под номером NCIMB42346. Такое потомство и потомки, которые сохраняют QTL3.1 и, таким образом, имеют более высокий урожай, по сравнению с растениями, не содержащими интрогрессию, как описано в настоящей заявке, охватываются настоящим изобретением.
В томатах, например, большой S. chilense фрагмент интрогрессии на хромосоме 6 (около 27 сМ), который содержит Ту-3 аллель, был уменьшен путем селекции рекомбинантной линии потомства (LA1931-AL-F2), которая содержит намного более маленький S. chilense фрагмент интрогрессии (около 6 сМ), содержащий Ту-3 (смотрите Ji et al. 2007, Mol. Breeding 20: 271-284).
Культивированное растение огурца согласно настоящему изобретению может быть инбредным ОР (перекрестноопыляющийся сортом) или F1 гибридом. В одном варианте выполнения настоящего изобретения F1 гибрид содержит только одну рекомбинантную хромосому 3 (содержащую фрагмент интрогрессии с QTL), т.е. F1 гибрид является гетерозиготным для фрагмента интрогрессии, и SNP маркер, описанный в настоящей заявке. Такой F1 гибрид получают скрещиванием двух инбредных родительских линий, одна из которых обладает фрагментом интрогрессии (предпочтительно в гомозиготной форме, хотя необязательно), и сбором F1 гибридных семян указанного кросса. В другом варианте выполнения настоящего изобретения F1 гибрид может содержать фрагмент интрогрессии в гомозиготной форме, т.е. полученный скрещиванием двух инбредных родительских линий, причем каждая содержит фрагмент интрогрессии в гомозиготной или гетерозиготной форме.
Культивированное растение огурца может быть любого типа. Предпочтительно оно имеет хорошие агрономические характеристики и хорошие характеристики качества плодов. Культивированное растение огурца является в одном варианте однородным, как генетически, так и фенотипически. В частности характеристики плода одинаковы, например, по форме, цвету кожи, толщине кожи, ребрам кожи, вязкости кожи, колючкам (цвет колючек, плотность колючек и т.д.), наличию/отсутствию бородавок, длине и диаметру на стадии зрелости, пригодной в пищу, цветкам, и т.д. Подобным образом, характеристики семян (т.е. характеристики семян, из которых растение выращивают) являются однородными, например, размер семени, цвет семени и т.д. Таким образом, растения линии или сорта, содержащие QTL в гомозиготной или гетерозиготной форме, дают однородные плоды, что означает небольшую вариацию между плодами растений, выращенных в тех же условиях окружающей среды и когда фрукты находятся на одной и той же стадии развития (например, для качественных характеристик по меньшей мере 98%, 99% или предпочтительно 100% всех растений или частей растений, плоды или семена идентичны по характеристикам; для количественных характеристик по меньшей мере 90%, 95%, 98% всех растений или частей растений, плоды или семена идентичны по характеристикам).
Культивированное растение огурца, содержащее QTL3.1 (или варианты любого из них) согласно настоящему изобретению могут быть любого типа, например, оно может быть любого из следующих видов огурца: корнишоны (например, вид American pickling, European pickling), салатные огурцы (например American slicing), длинные огурцы, короткие огурцы, Европейские тепличные огурцы, огурцы типа Бейт-Альфа, огурцы вида oriental trellis, Азиатские огурцы (например, выбранные из видов индийский пестрый огурец, китайский длинный огурец, корейский огурец и японский огурец). В одном варианте культивированный огурец согласно настоящему изобретению представляет собой инбредную линию или F1 гибрид корнишона, салатного огурца, длинного огурца, короткого огурца, Европейского тепличного огурца, огурца типа Бейт-Альфа, огурцов вида oriental trelliss, китайского длинного огурца, корейского огурца или японского огурца. В конкретном варианте выполнения настоящего изобретения огурцом является инбредная линия или F1 гибрид Европейского тепличного огурца.
Растением может быть простой гибрид F1 или инбредная линия, содержащая один или оба QTL в гомозиготной или гетерозиготной форме. В одном варианте выполнения настоящего изобретения это F1 гибрид, полученный путем скрещивания (инбредного) родительского растения, содержащего QTL3.1 (или варианты любого из них) в гомозиготной форме, с (инбредным) родительским растением, не содержащим QTL3.1 (т.е. не содержащим фрагменты интрогрессии, содержащие указанные QTL). Таким образом, в одном варианте F1 гибрид является гетерозиготным для QTL3.1.
В другом варианте выполнения настоящего изобретения это F1 гибрид, полученный путем скрещивания (инбредного) родительского растения, содержащего QTL3.1 (или варианты любого из них) в гомозиготной форме, с (инбредным) родительским растением, которое также содержит QTL3.1 (или варианты любого из них) в гомозиготной форме. Таким образом, в одном варианте выполнения настоящего изобретения F1 гибрид является гомозиготным для QTL3.1.
В одном варианте выполнения настоящего изобретения F1 гибрид представляет собой Европейский тепличный огурец, подходящий для традиционного выращивания в теплице или для выращивания с высокой проволокой. При способе традиционного выращивания в теплице основной ствол растения стремится к горизонтальной железной проволоке, которая подвешена на высоте около двух метров над землей. Когда растение достигает этой высоты и присоединяется к проволоке, это «завершается» удалением его точки роста, чтобы прекратить дальнейшее разрастание, после чего начинают развиваться боковые побеги. Эти боковые побеги позволяют расти вниз на высоте около 1 метра над землей, и затем точки роста удаляются от них. За этим следует цветение и развитие плодов как на стебле, так и на боковых побегах или усиках, но плоды на усиках развиваются позже, чем на стебле. Плоды собирают через около 6 недель после посева. При выращивании с высокой проволокой боковые усики не могут расти, и весь урожай происходит на стебле. Конкретные сорта были разработаны Nunhems, которые очень подходят для выращивания с подвешиванием на проволоке, поскольку они обеспечивают ген, называемый «compact», смотрите WO 2009/059777, например, сорта High-Jack, Hi-Power, Hi-Lisa.
Таким образом, в одном варианте выполнения настоящего изобретения согласно настоящему изобретению культивированное растение огурца содержит дополнительный ген compact, описанный в WO 2009/059777. Ген compact предпочтительно присутствует в гетерозиготной форме.
В другом варианте выполнения настоящего изобретения фрагмент интрогрессии согласно настоящему изобретению присутствует в огурце длинного типа, таком как сорт Kasja (Nunhems), который представляет собой сорт длинного огурца, дающий плоды 27-38 см. "Огурец длинного типа" или "длинные растения огурца" представляют собой тепличные огурцы, характеризующиеся плодами, составляющими по меньшей мере около 26 см или 27 см - 37 или 38 см в длину или длиннее (например, 40 см, 42 см или более), предпочтительно с образованием партенокарпического плода. Примерами огурцов длинного типа являются сорта Sabrina и Korinda, или растения огурца, которым присваивается оценка 7-9 для длины плода согласно протоколу CPVO (смотрите пункт 19 в главе 1 этого протокола). Другими сортами длинного огурца являются, например, Bodega, Bologna, Kamaro, Flamingo, Discover, Kalunga, Kasja, Logica, Millagon. Nicola, Milika, Manuela, Frida, Activa, Alaya, Savanna, Sienna, Bella, Sheila, Bornand.
В одном варианте выполнения настоящего изобретения Европейский тепличный огурец представляет собой растение, семена которого были депонированы под номером доступа NCIMB42346, или его потомство, причем потомство сохраняет QTL3.1 (как обнаружено по присутствию одного или более маркеров, как описано в настоящей заявке).
В другом варианте выполнения настоящего изобретения растение согласно настоящему изобретению не представляет собой растение огурца дикого типа или дикий родственник огурца или местный сорт.
В другом варианте выполнения настоящего изобретения растение согласно настоящему изобретению представляет собой культивированный огурец Евразийской группы огурцов, Восточноазиатской группы огурцов или группы огурцов Xishuangbanna. В другом варианте выполнения настоящего изобретения растение согласно настоящему изобретению не является огурцом индийской группы огурцов.
В одном варианте выполнения настоящего изобретения согласно настоящему изобретению культивированное растение огурца, содержащее QTL3.1 (или вариант) дает бессемянные плоды без опыления, т.е. является партенокарпическим. Большинство Европейских тепличных огурцов являются партенокарпическими, т.е. женские цветки дают плоды без опыления, причем плоды остаются бессемянными, партенокарпия генетически контролируется, и специалистам по разведению известно как ввести признак партенокарпии в линию или сорт огурца (смотрите, например, Chapter 13 entitled "Cucumber" by Т. tatlioglu, page 207-209 in the book Genetic Improvement of Vegetable Crops, Editors G. Kalloo and BO Bergh, Pergamon Press, 2012, ISBN0080408265).
В другом варианте выполнения настоящего изобретения культивированное растение огурца, содержащее QTL3.1 (или вариант), главным образом является женским или полностью женским (дает 100% женских цветков). Это означает, что главным образом или только женские цветки продуцируются. Этот признак также генетически контролируется, и специалистам по разведению известно как ввести женский признак в линию или сорт огурца (смотрите, например, Chapter 13 entitled "Cucumber" by Т. tatlioglu, page 207-209 in the book Genetic Improvement of Vegetable Crops, Editors G. Kalloo and BO Bergh, Pergamon Press, 2012, ISBN0080408265).
В одном варианте выполнения настоящего изобретения огурец согласно настоящему изобретению является как партенокарпическим, так и женским. Таким образом, растение дает главным образом или только женские цветки, которые дают бессемянные плоды без опыления. В женских огурцах мужские цветки могут быть индуцированы посредством обработки нитрата серебра. Этот способ применяется для получения пыльцы и для самоопыления инбредной женской линии огурца.
В другом варианте выполнения настоящего изобретения растение огурца согласно настоящему изобретению является гермафродитным (дает как мужские, так и женские цветки), необязательно партенокарпическим и гермафродитным.
В другом варианте выполнения настоящего изобретения культивированное растение огурца, содержащее QTL3.1 (или вариант) является однородным и генетически стабильным с точки зрения морфологических характеристик плодов, которые дает указанное растение, например, с точки зрения формы плода, цвета плода, толщины кожи, бородавок и т.д.
Характеристики плода, такие как средняя длина плода, средний диаметр плода, толщина кожи, наличие/отсутствие бородавок, иглистость, вязкость кожи, цвет кожи, форма шейки плода, коничность плода, форма срединного кросс-сечения, наличие или отсутствие семян (партенокарпия), и т.д. зависят от типа огурца, т.е. культивированного генетического фона (генофонд), в который QTL интрогрессируется/интрогрессируются. Таким образом, в зависимости от типа огурца настоящее изобретение охватывает различные формы, размеры и виды плодов. В одном варианте выполнения настоящего изобретения плоды являются бессемянными.
Двумя основными типами плодов огурца, которые сегодня коммерчески продаются в Соединенных Штатах, являются свежий (нарезаемый) тип и обрабатываемый (маринуемый) тип. Сорта и методы производства обычно адаптируются к конечному использованию. Салатные огурцы часто более длинные, крупные и имеют более темную и более толстую кожу, тогда как маринуемые/обрабатываемые огурцы имеют более короткие плоды, более тонкую кожу с внутренней мякотью, что делает их более пригодными для маринования. Бессемянные сорта обычно предпочтительны как для свежего рынка, так и для маринования, так как развивающиеся и крупные семена непривлекательны.
В одном варианте выполнения настоящего изобретения растение согласно настоящему изобретению относится к маринуемому типу (обрабатываемому типу) производит плоды со средней длиной плода на стадии съедобного плода, равной по меньшей мере 10 см, или по меньшей мере 11 см, или по меньшей мере 12 см, или по меньшей мере 13 см, и/или отношение длины плода к диаметру, равное по меньшей мере 2, по меньшей мере 2.5, по меньшей мере 3, или более.
В другом варианте выполнения настоящего изобретения растение согласно настоящему изобретению относится к свежему типу, например типу длинного огурца или нарезаемому типу, и производит плоды со средней длиной плода на стадии съедобного плода, которая длиннее, чем у маринованного типа, например по меньшей мере 15 см, 16 см, 17 см, 18 см, 19 см, 20 см, 25 см, 26 см, 27 см, 28 см, 29 см, 30 см, 32 см, 40 см, или более.
В предпочтительном варианте выполнения настоящего изобретения растение согласно настоящему изобретению представляет собой огурец длинного типа, дающий плоды товарного размера, особенно бессемянные плоды. Плоды товарного размера, и их части, и пищевые или кормовые продукты, содержащие их, также охватываются настоящим изобретением. В одном варианте выполнения настоящего изобретения SNP маркеры являются обнаруживаемыми в плодах, частях плодов или пищевых или кормовых продуктах, содержащих их.
В одном варианте выполнения настоящего изобретения растением является неопределенный огурец. В другом варианте выполнения настоящего изобретения огурец является определенным.
Также семена, из которых растение согласно настоящему изобретению может быть выращено, обеспечивается согласно настоящему изобретению, как и плоды огурца, собранные с растения согласно настоящему изобретению. Они содержат QTL в их геноме и поэтому могут отличаться от других плодов присутствием одного или более из SNP маркеров, приведенных в настоящей заявке.
В одном варианте выполнения настоящего изобретения плоды не имеют горечи (выбранные из групп горьких и не горьких) на стадии съедобного плода и/или размера, подходящего для продажи.
В одном варианте выполнения настоящего изобретения плод имеет тонкую кожу (выбранный из групп толстых и тонких) на стадии съедобного плода и/или размера, подходящего для продажи.
В другом варианте выполнения настоящего изобретения QTL интрогрессируются в тип огурца, называемый 'Compact', как описано в US 8710303 B2. Таким образом, растения огурца согласно настоящему изобретению содержат ген compact, как описано в US 8710303 B2 в гомозиготной или гетерозиготной форме, например, как присутствует в сортах Hi-Jack и Hi-Lisa (оба Nunhems).
Другим вариантом выполнения настоящего изобретения является растительная клетка, ткань или часть растения или семени согласно настоящему изобретению, содержащие по меньшей мере одну рекомбинантную хромосому 3, где указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, и где указанный фрагмент интрогрессии содержит QTL, обеспечивающий увеличенный урожай плодов.
Также настоящим изобретением охватывается применение рекомбинантной хромосомы 3, содержащей фрагмент интрогрессии из дикого родственника огурца (причем указанный фрагмент интрогрессии содержит аллель, обеспечивающий увеличенный урожай плодов) для разведения сортов огурца, дающих увеличенный урожай плодов. В одном варианте выполнения настоящего изобретения указанная рекомбинантная хромосома 3 представляет собой рекомбинантную хромосому 3, как обнаружено в семенах, депонированных под номером доступа NCIMB42346, или происходят из указанной рекомбинантной хромосомы 3 (например, представляет собой более маленький фрагмент фрагмента интрогрессии, обнаруженного в указанных семенах).
Подобным образом, настоящее изобретение охватывает применение хромосомы 3, как обнаружено в семенах, депонированных под номером доступа NCIMB42346, или в их потомстве, для получения культивированного растения огурца, содержащего фрагмент интрогрессии на указанной хромосоме 3, где указанный фрагмент интрогрессии обеспечивает увеличенный урожай плодов, по сравнению с растением огурца в качестве генетического контроля, не содержащим указанный фрагмент интрогрессии (как например растения, выращенные из семян, депонированных под номером доступа NCIMB42345).
Подобным образом, настоящее изобретение охватывает применение растений, выращенных из семян, депонированных под номером доступа NCIMB42346, или их потомства, для получения культивированного растения огурца, имеющего увеличенный урожай плодов, где указанный увеличенный урожай плодов обеспечивается фрагментом интрогрессии, полученным из хромосомы 3 указанных растений, или их потомства.
Также обеспечивается применение растений, выращенных из семян, депонированных под номером доступа NCIMB42346, или их потомства, для переноса QTL3.1 или фрагмента интрогрессии или его подфрагмента, содержащих QTL3.1, в другое растение огурца.
Также обеспечивается способ идентификации (обнаружения) культивированного растения С. sativus var. sativus, содержащего фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии представляет собой, как обнаружено в NCIMB42346, или более маленький фрагмент, производный от него, включающий:
a) обеспечение культивированного растения или частей растения С. sativus var. sativus или ДНК такого растения или части растения,
b) скрининг указанного растения, части растения или ДНК, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, выбранный из группы, состоящей из
SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; и
c) идентификацию и/или отбор растения, содержащего:
i) по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более из SNP маркеров SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; или
ii) по меньшей мере 2, 3, 4 5, 6, 7, 8, 9, 10 или более последовательных маркеров, выбранных из SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; или
iii) по меньшей мере 1, 2, 3, 4 5, 6, 7, 8 или более маркеров из группы, причем указанная группа состоит из SNP_01 - SNP_10; SNP_10 - SNP_20; SNP_20 - SNP_27; SNP_06 - SNP_23; или
vi) по меньшей мере 2, 3, 4 5, 6, 7, 8 или более последовательных маркеров из группы, причем указанная группа состоит из SNP_01 - SNP_10; SNP_10 - SNP_20; SNP_20 - SNP_27; SNP_06 - SNP_23.
Кроме того, обеспечивается способ получения F1 гибридных растений С. sativus, содержащих фрагмент интрогрессии, обеспечивающий увеличенный урожай плодов, включающий:
a) обеспечение первого инбредного растения огурца, содержащего рекомбинантную хромосому 3 в гомозиготной форме, имеющую фрагмент интрогрессии, содержащий аллель, обеспечивающий увеличенный урожай, где указанный фрагмент интрогрессии представляет собой такой как в NCIMB42346 или более маленький фрагмент,
b) обеспечение второго инбредного растения огурца,
c) скрещивание указанного растения огурца согласно а) с указанным растением огурца согласно b),
d) сбор F1 гибридных семян из указанного кросса.
Собранные F1 гибридные семена также являются вариантом выполнения настоящего изобретения.
В другом варианте выполнения настоящего изобретения обеспечивается способ получения потомства NCIMB42346, причем указанный способ включает:
a) выращивание растения из семян, депонированных под номером доступа NCIMB42346;
b) самоопыление указанного растения один или более раз или скрещивание указанного растения один или более раз с другим растением огурца с получением семян потомства;
c) скрининг указанных семян или растений потомства, выращенных из указанных семян, или частей семян или растений, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, выбранный из группы, состоящей из:
SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3;
d) идентификацию и/или отбор потомственного растения, содержащего:
i) по меньшей мере 1 из SNP маркеров SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; или
ii) по меньшей мере 2, 3, или 4 последовательных маркеров, выбранных из SNP_1 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; или
iii) по меньшей мере 1, 2, или 3 маркеров из группы маркеров, состоящей из SNP_1 - SNP_10; SNP_10 - SNP_20; SNP_20 - SNP_27; SNP_06 - SNP_23 для обнаружения фрагмента интрогрессии на хромосоме 3; или
iv) по меньшей мере 2, 3 или 4 последовательных маркеров из группы маркеров, состоящей из SNP_1 - SNP_10; SNP_10 - SNP_20; SNP_20 - SNP_27; SNP_06 - SNP_23 для обнаружения фрагмента интрогрессии на хромосоме 3.
Растение огурца на стадии b предпочтительно представляет собой культивированный огурец, такой как Европейский тепличный огурец или огурец длинного типа.
Способ необязательно дополнительно включает стадию идентификации потомственного растения, имеющего увеличенный урожай плодов по сравнению с контролем.
Потомственное растение, полученное вышеуказанным способом, также является объектом настоящего изобретения. Потомственное растение может содержать более короткий фрагмент интрогрессии, чем обнаруживаемый в NCIMB42346, который сохраняет QTL3.1.
Также настоящее изобретение обеспечивает контейнеры и упаковки, содержащие или содержащие семена, из которых растения согласно настоящему изобретению могут быть выращены. Они могут быть помечены как содержащие семена культивированного огурца, обеспечивающие увеличенный или высокий урожай плодов.
Также обеспечиваются потомственные семена и потомственные растения согласно настоящему изобретению, которые сохраняют интрогрессию на хромосоме 3, содержащую QTL3.1 (или вариант), или которые содержат более маленькую интрогрессию (например, происходящую из фрагмента, представлено в NCIMB42346), которая все еще обеспечивает увеличенный урожай, т.е. которая все еще содержит QTL3.1. Потомством может быть любое поколение, полученное самоопылением растения огурца согласно настоящему воздействию и/или скрещиванием растения огурца согласно настоящему соединению с другим растением огурца один или более раз. Потомством являются, поэтому, либо поколение (семена), полученные из первого кросса (F1) или самоопыления (S1), либо любое другое поколение, полученное путем скрещивания и/или самоопыления (F2, F3, и т.д.) и/или обратного скрещивания (ВС1, ВС2, и т.д.) одного или более выбранных растений поколения F1 и/или S1 и/или ВС1 (или растений любого другого поколения, например, F2) с другим растением огурца (и/или с диким родственником огурца). Потомство предпочтительно отбирают по сохранению рекомбинантной хромосомы 3, содержащей фрагмент интрогрессии из дикого родственника огурца. Таким образом, потомство также имеет фенотип увеличенного урожая, предпочтительно такой же урожай, как растение, как растение, применяемое в первоначальном кроссе или самоопыление. Присутствие (или сохранение) фрагмента интрогрессии, содержащего QTL, может быть определено фенотипически и/или применяя анализ (анализы) молекулярного маркера, описанные в настоящей заявке. Что касается фенотипической оценки, то, разумеется, необходимо учитывать характер доминирования QTL.
Другим объектом настоящего изобретения являются части растения огурца согласно настоящему изобретению. Части включают, например, клетки и культуры клеток, культуры тканей, вегетативные ткани растения (листья, корни и т.д.), цветы, пыльцу, зародыши, плоды, части плодов и т.д. Части растения содержат фрагмент интрогрессии на хромосоме 3, как описано, и как можно обнаружить, применяя один или более описанных маркеров. Также, когда целые растения регенерируются из таких частей огурца, таких как клетки, клеточные или тканевые культуры, регенерированные растения содержат рекомбинантную хромосому 3, и фенотип урожая.
Таким образом, также обеспечивается растительная клетка, ткань или часть растения или семени согласно изобретению, содержащие по меньшей мере одну рекомбинантную хромосому 3, где указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из растения дикого родственника огурца, и где указанный фрагмент интрогрессии содержит аллель, обеспечивающий увеличенный урожай плодов.
Также in vitro клеточные культуры и in vitro тканевые культуры охватываются настоящим изобретением, описанной клетки или ткани, содержащей рекомбинантную хромосому 3. Предпочтительно клетки или ткани могут быть регенерированы в целое растение огурца, т.е. клетки являются регенерируемыми клетками, и ткани содержат регенерируемые клетки. Таким образом, также вегетативные размножения растений согласно настоящему изобретению являются вариантом выполнения настоящего изобретения. Таким образом, обеспечивается вегетативно размножаемое культивированное растение огурца, которое содержит рекомбинантную хромосому 3, как описано в настоящей заявке. В другом варианте настоящее изобретение охватывает неразмножающиеся клетки, содержащие QTL3.1, как и ткани, содержащие такие клетки.
Другим объектом настоящего изобретения является плод огурца, собранный с растения согласно настоящему изобретению. Плоды огурца для продажи, в частности для свежего рынка (нарезка), обычно сортируются в зависимости от размера и качества плодов после сбора урожая. Смотрите, например, the United States Standards for Grades of Cucumbers, US Department of Agriculture, Effective March 1, 1985 и переиздание January 1997. Различают различные марки огурцов. Таким образом, в одном варианте собранные плоды обозначают как класс U.S. Fancy, класс U.S. Extra No. 1, класс U.S. No. 1, малый класс U.S. No. 1, большой класс U.S. No. 1, класс U.S. No. 2. Также предусмотрены контейнеры или упаковки, содержащие или состоящие из собранных плодов огурца. Аналогичным образом, клетки плодов отличаются от других плодов огурца наличием рекомбинантной хромосомы 3 (как определено в одном или более анализах молекулярного маркера).
В другом варианте выполнения настоящего изобретения огурец представляет собой огурец длинного типа, и собранные и необязательно обработанные плоды (например, нарезанные или разрезанные) обеспечиваются.
В другом варианте огурец является огурцом типа огурца для маринования, и обеспечиваются собранные и необязательно маринованные плоды.
Настоящее изобретение также относится к пищевому продукту или кормовому продукту, содержащему или состоящему из частей растения, описанных в настоящей заявке, предпочтительно плод огурца или его часть и/или экстракт из части растения, описанного в настоящей заявке. Пищевой или кормовой продукт может быть свежим или обработанным, например, маринованным, консервированным, пропаренным, вареным, жареным, бланшированным и/или замороженным и т.д. Например, контейнеры, такие как банки, коробки, ящики, сумки, картонные коробки, упаковка с модифицированной атмосферой, пленки (например, биоразлагаемые пленки), и т.д., содержащие части растений, такие как плоды или части плодов (свежих и/или обработанные), описанных в настоящей заявке, также обеспечиваются настоящим изобретением.
Способы и применения согласно настоящему изобретению
В другом варианте выполнения настоящего изобретения, изобретение обеспечивает способ получения нового культивированного растения огурца, которое содержит фрагмент интрогрессии на хромосоме 3 (который обеспечивает увеличенный урожай) в гомозиготной или гетерозиготной форме, как описано. Способ содержит скрещивание растения согласно настоящему изобретению, или его потомственного растения, либо в качестве мужского, либо в качестве женского родителя со вторым растением огурца (или диким родственником огурца) один или более раз, и/или самоопыление растения огурца согласно настоящему изобретению, или его потомственного растения, один или более раз, и отбор потомства после указанного скрещивания и/или самоопыления.
Таким образом, обеспечивается способ переноса рекомбинантной хромосомы 3, содержащей QTL урожая, из одного (культивированного) растения огурца в другое (культивированное) растение огурца, в частности в сорта огурца или линии скрещивания, для которых урожай плодов должен быть увеличен.
Способ включает стадии:
a) обеспечение первого культивированного растения огурца, содержащего рекомбинантную хромосому 3, имеющую фрагмент интрогрессии, содержащий аллель, обеспечивающий увеличенный урожай плодов, в гомозиготной форме,
b) обеспечение второго культивированного растения огурца, в частности растения, имеющего хромосому 3 дикого типа (нерекомбинантную),
c) скрещивание указанного растения огурца согласно а) с указанным растением огурца согласно b),
d) сбор F1 гибридных семян из указанного кросса и
e) необязательно самоопыление растения, выращенного из указанных F1 гибридных семян, с получением семян F2 или поколений дальнейшего самоопыления, и необязательно отбор F2 семена или семян поколения дальнейшего самоопыления, имеющих рекомбинантную хромосому 3, и
f) необязательно разведение далее с растениями, выращенными из указанных семян F1 или F2 или поколения дальнейшего самоопыления с получением растения огурца, имеющего хорошие агрономические характеристики и содержащего один или оба фрагмента интрогрессии в гомозиготной или гетерозиготной форме.
Присутствие или отсутствие рекомбинантной хромосомы 3, и фрагмента интрогрессии, может быть определено одним или более из описанных в настоящей заявке анализов молекулярных маркерных и/или путем определения, значительно ли повышается урожай по сравнению с растением согласно b). Дальнейшее разведение на стадии f) может содержать самоопыление, скрещивание, получение двойного гаплоида, обратное скрещивание и их комбинацию (например обратное скрещивание и самоопыление), и т.д. Растения и семена, получаемые вышеуказанным способом, охватываются настоящим изобретением.. В одном варианте выполнения настоящего изобретения растением согласно стадии а) может быть растением, выращенным из семян, депонированных под NCIMB42346, или их потомства, или растением, содержащим фрагмент интрогрессии на хромосоме 3, как присутствует в семенах, депонированных под NCIMB42346, или более короткий фрагмент этого фрагмента.
Также обеспечивается способ получения F1 гибридного растения культивированного огурца, содержащего QTL урожая на хромосоме 3, включающий:
a) обеспечение первого инбредного растения огурца, содержащего по меньшей мере одну рекомбинантную хромосому 3, содержащую фрагмент интрогрессии, содержащий QTL урожая, выбранный из QTL3.1 или его варианта
b) обеспечение второго инбредного растения огурца, содержащего по меньшей мере одну рекомбинантную хромосому 3, содержащую фрагмент интрогрессии, содержащий QTL урожая, выбранный из QTL3.1 или его варианта,
c) скрещивание указанного растения огурца согласно а) с указанным растением огурца согласно b),
d) сбор F1 гибридных семян из указанного кросса.
Инбредное растение огурца согласно а) и b) может быть гомозиготным и/или гетерозиготным в отношении фрагмента интрогрессии на хромосоме 3, и они могут содержать фрагменты интрогрессии различных размеров и/или различного происхождения, т.е. из различных диких родственников огурца. Поэтому, например, фрагмент интрогрессии в а) может быть таким же или отличным от фрагмента интрогрессии в b). В одном варианте инбредное растение огурца согласно а) содержит QTL3.1 или его вариант в гомозиготной форме, и/или инбредное растение огурца согласно b) содержит QTL3.1 или его вариант в гомозиготной форме. В одном варианте фрагмент интрогрессии, содержащий QTL3.1, представляет собой фрагмент, как обнаружено в NCIMB42346, или его более маленький фрагмент.
F1 гибридные семена предпочтительно содержат по меньшей мере одну рекомбинантную хромосому 3, и поэтому F1 растения, выращенные из семян, действительно дают увеличенный урожай плодов по сравнению с генетическим контролем.
Растения и семена, получаемые вышеуказанным способом, охватываются настоящим изобретением.
Другим объектом настоящего изобретения является способ получения культивированного растения огурца, содержащего фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии содержит QTL урожая, причем указанный способ включает стадии:
a) обеспечение первого культивированного растения огурца,
b) обеспечение второго растения дикого родственника огурца, где указанное растение содержит QTL3.1 (или его вариант), как определено присутствием одного или более SNP маркеров, как описано в настоящей заявке,
c) скрещивание указанного растения огурца согласно а) с указанным растением огурца согласно b),
d) сбор F1 семян из указанного кросса и обратное скрещивание F1 растения с растением огурца согласно а) с получением беккросс (ВС1) популяции, или самоопыление указанных F1 растений один или более раз с получением F2 или F3 или более нового поколения популяции самоопыления,
e) необязательно обратное скрещивание растения согласно d) один или более раз с растением огурца согласно а) с получением более нового поколения беккросс-популяции, и
f) идентификацию F2, F3, или более нового поколения самоопыления, или ВС1 или беккросс-растения более нового поколения, которые содержат интрогрессию на хромосоме 3, где указанный фрагмент интрогрессии содержит 3.1 (или его вариант).
Когда речь идет о беккросс-популяциях в способе, беккросс-популяции также быть самоопыленными, т.е. BC1S1, BC1S2, BC2S1, BC2S2 или другие.
На одной или более из стадий b)-f) присутствие QTL (или фрагмента интрогрессии, содержащего QTL) может быть протестировано (и растения могут быть выбраны) путем осуществления анализа молекулярного маркера, как описано в настоящей заявке, например путем определения содержит ли растение один или более SNP маркеров (например один или более из SNP_01 - SNP_27; или один или более из SNP_01 - SNP_10; или один или более из SNP_10 - SNP_20; или один или более из SNP_20 - SNP_27; или один или более из SNP_06 - SNP_23; и/или любой маркер, специфичный для генома дикого родственника огурца, между любым из этих маркеров).
Применяя этот способ можно получить и/или отобрать новые культивированные растения огурца, содержащие интрогрессию с QTL3.1 (или вариант) из источника дикого типа, такого как дикий родственник огурца.
В одном варианте выполнения настоящего изобретения способ получения культивированного растения огурца, содержащего фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии содержит QTL урожая, включает стадии:
a) обеспечение первого культивированного растения огурца,
b) обеспечение второго растения дикого родственника огурца, содержащего один или более маркеров SNP согласно настоящему изобретению,
c) скрещивание указанного растения согласно а) с указанным растением согласно b),
d) сбор F1 семян из указанного кросса и обратное скрещивание F1 растения с растением огурца согласно а) с получением беккросс (ВС1) популяции, или самоопыление указанного F1 растения один или более раз с получением F2 или F3 популяции,
e) необязательно самоопыление беккросс-популяции с получением, например, BC1S1 или BC1S2 популяции,
f) идентификацию F2, F3, ВС1 BC1S1 или BC1S2 растения, которое содержит (один или более) SNP маркеров и/или любой специфичный для генома дикого родственника огурца маркер между SNP маркерами.
Также обеспечивается способ идентификации растения дикого родственника огурца, содержащего QTL урожая на хромосоме 3, причем указанный способ включает:
A) обеспечение экземпляра дикого родственника огурца или нескольких экземпляров;
B) скрининг указанного экземпляра (экземпляров), применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один (или по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10 или более) SNP маркер, выбранный из группы, состоящей из: SNP_01 - SNP_27 (или подгрупп SNP маркеров, таких как SNP_01 - SNP_10; SNP_10 - SNP_20; SNP_20 - SNP_27; SNP_06 - SNP_23);
C) идентификацию и/или селекцию экземпляра согласно b), содержащего по меньшей мере один или более следующих маркеров:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1 (или в ее варианте);
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2 (или в ее варианте);
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3 (или в ее варианте);
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4 (или в ее варианте);
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5 (или в ее варианте);
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6 (или в ее варианте);
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7 (или в ее варианте);
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8 (или в ее варианте);
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9 (или в ее варианте);
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10 (или в ее варианте);
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11 (или в ее варианте);
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12 (или в ее варианте);
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13 (или в ее варианте);
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14 (или в ее варианте);
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15 (или в ее варианте);
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16 (или в ее варианте);
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17 (или в ее варианте);
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18 (или в ее варианте);
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19 (или в ее варианте);
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20 (или в ее варианте);
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21 (или в ее варианте);
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22 (или в ее варианте);
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23 (или в ее варианте);
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 в SEQ ID NO: 24 (или в ее варианте);
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 в SEQ ID NO: 25 (или в ее варианте);
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 в SEQ ID NO: 26 (или в ее варианте);
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27 (или в ее варианте);
bb) любой специфичный для генома дикого родственника огурца маркер между маркером SNP_01 и SNP_27, и необязательно
D) интрогрессинг указанного QTL из указанного дикого экземпляра в культивированный огурец (например путем обратного скрещивания).
На стадии В), С) и D) также могут применяться другие тесты на основе молекулярных маркеров, описанные в настоящей заявке. С помощью этого теста можно, таким образом, провести скрининг дикого родственника огурца на присутствие одного или более маркеров и, таким образом, присутствие QTL3.1 (или их вариантов) и интрогрессировать QTL в культивированные растения огурца. Растения и семена, полученные этим способом, также являются вариантом выполнения настоящего изобретения.
Еще одним объектом настоящего изобретения является способ идентификации культивированного растения огурца, содержащего фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии содержит QTL урожая, причем указанный способ включает: скрининг культивированного растения огурца или популяции культивированного растения огурца или частей таких растений огурца (например, плоды, клетки, ДНК), применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер (предпочтительно 2, 3, 4, 5 или более; предпочтительно последовательных SNP маркеров), указывающий на (сцепленный с) QTL3.1, как описано в настоящей заявке.
В этом способе могут применяться другие тесты на основе молекулярных маркеров, описанные в настоящей заявке. Таким образом, применяя этот способ можно обнаружить присутствие фрагмента интрогрессии на хромосоме 3, содержащего QTL3.1, в культивированные растения огурца или части растений.
Еще одним объектом настоящего изобретения является способ обнаружения содержит ли культивированное растение огурца фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии содержит QTL3.1, причем указанный способ включает:
a) обеспечение культивированного растения огурца или части растения,
b) скрининг указанного растения или указанной части растения (или ДНК, полученной из указанного растения или части растения), применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один (предпочтительно по меньшей мере 2, 3, 4, 5 или более) SNP маркеров, выбранных из группы, состоящей из:
SNP_01 - SNP_27 и/или любого маркера, специфичного для генома дикого родственника огурца, между маркером SNP_01 и SNP_27.
Скрининг молекулярных маркеров очевидно включает получение материала растения и анализ геномной ДНК материала на генотип маркера.
В этом способе могут также применяться другие тесты на основе молекулярных маркеров, описанные в настоящей заявке.
Настоящее изобретение также охватывает способ получения культивированного растения огурца, содержащего фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии содержит QTL3.1, включающий:
a) обеспечение первого культивированного растения огурца, не содержащего фрагмент интрогрессии, включающий QTL3.1,
b) обеспечение второго культивированного растения огурца, выбранного из растения, выращенного из семян, депонированных под номером доступа NCIMB42346, или их потомства;
c) скрещивание указанного растения согласно а) с указанным растением согласно b),
d) сбор F1 семян из указанного кросса и необязательно самоопыление указанного F1 растения один или более раз с получением F2 или F3 или другой популяции самоопыления,
e) необязательно обратное скрещивание F1 растения или F2 или F3 или другого самоопыленного растения с растением согласно а) с получением беккросс-популяции,
f) необязательно самоопыление беккросс популяции один или более раз,
g) идентификацию F1, F2, F3, дальнейшее самоопыление или беккросс растения, которое содержит один или более или все генотипы SNP маркера, указывающие на фрагмент интрогрессии на хромосоме 3.
Другим объектом настоящего изобретения является способ получения F1 гибридного растения, включающий:
а) обеспечение первого инбредного растения огурца, содержащего по меньшей мере одну рекомбинантную хромосому 3, имеющую фрагмент интрогрессии, содержащий QTL3.1, где указанный фрагмент интрогрессии представляет собой фрагмент, как обнаружено в NCIMB42346, или более короткий фрагмент этого фрагмента интрогрессии,
b) обеспечение второго инбредного растения огурца с или без рекомбинантной хромосомой 3,
c) скрещивание указанного растения согласно а) с указанным растением согласно b),
d) сбор F1 гибридных семян из указанного кросса.
Другим объектом настоящего изобретения является способ получения потомства NCIMB42346, сохранившего QTL3.1, причем указанный способ включает:
a) выращивание растения из семян, депонированных под номером доступа NCIMB42346;
b) самоопыление указанного растения один или более раз или скрещивание указанного растения один или более раз с другим культивированным растением огурца с получением семян потомства;
c) скрининг указанных семян потомства или растений, выращенных из указанных семян, или частей семян или растений, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, раскрытый в настоящей заявке;
d) идентификацию и/или отбор потомственного растения, содержащего по меньшей мере один, два, три или более маркеров SNP, указывающих фрагмент интрогрессии, содержащий QTL3.1 (как описано в настоящей заявке); и
е) необязательно подтверждение увеличенного урожая плодов указанного потомственного растения.
В одном варианте выполнения настоящего изобретения урожай согласно е) является предпочтительно по меньшей мере таким же урожаем, как для растений, выращенных из NCIMB43246, при выращивании при тех же условиях.
Обеспечивается способ получения потомства NCIMB42346, причем указанный способ включает:
a) выращивание растения из семян, депонированных под номером доступа NCIMB42346;
b) самоопыление указанного растения один или более раз или скрещивание указанного растения один или более раз с другим культивированным растением огурца с получением семян потомства;
c) скрининг указанных семян потомства или растений, выращенных из указанных семян, или частей семян или растений, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, выбранный из группы, состоящей из:
SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3;
d) идентификацию и/или отбор потомственного растения, содержащего:
i) по меньшей мере 1 из SNP маркеров SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; или
ii) по меньшей мере 2, 3, или 4 последовательных маркера, выбранных из SNP_01 - SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3, и
е) необязательно подтверждение увеличенного урожая плодов указанного потомственного растения.
В одном варианте выполнения настоящего изобретения урожай согласно е) является предпочтительно по меньшей мере таким же урожаем, как для растений, выращенных из NCIMB43246, при выращивании при тех же условиях.
Потомственное растение, полученное любым из вышеуказанных способов также является объектом настоящего изобретения.
Также можно применять способы и маркеры, описанные в настоящей заявке, для уменьшения размера фрагмента интрогрессии, содержащего QTL, т.е. чтобы получить и отобрать рекомбинанты, имеющие более маленький фрагмент интрогрессии на хромосоме 3, но который сохраняет часть, увеличивающую урожай, фрагмента интрогрессии.
В одном варианте выполнения настоящего изобретения изобретение охватывает применение рекомбинантной хромосомы 3, содержащей фрагмент интрогрессии из дикого родственника огурца, причем указанный фрагмент интрогрессии содержит QTL урожая, для разведения сортов огурца, имеющих увеличенный урожай плодов.
Также обеспечивается применение хромосомы 3, как обнаружено в семенах, депонированных под номером доступа NCIMB42346, или их потомства для получения культивированного растения огурца, содержащего фрагмент интрогрессии указанной хромосомы 3.
Также обеспечивается применение растений, выращенных из семян, депонированных под номером доступа NCIMB42346, или их потомства, для получения культивированного растения огурца, имеющего увеличенный урожай плодов, где указанный увеличенный урожай плодов обеспечивается фрагментом интрогрессии, полученным из хромосомы 3 указанных растений или потомства.
ДНК и хромосомы согласно настоящему изобретению
В одном варианте выполнения настоящего изобретения модифицированная (рекомбинантная) хромосома 3 культивированного огурца обеспечивается согласно настоящему изобретению, которая содержит фрагмент интрогрессии дикого родственника огурца, как описано в описании настоящего изобретения. В одном варианте выполнения настоящего изобретения рекомбинантную хромосому выделяют из ее природной окружающей среды. В другом варианте выполнения настоящего изобретения она находится в растительной клетке, в частности в клетке огурца, в частности в клетке культивированного огурца. Также согласно настоящему изобретению обеспечивается выделенная часть рекомбинантной хромосомы, содержащей QTL.
В другом варианте выполнения настоящего изобретения обеспечивается рекомбинантная молекула нуклеиновой кислоты, в частности рекомбинантная молекула ДНК, которая содержит аллель урожая согласно настоящему изобретению. В одном варианте выполнения настоящего изобретения аллель урожая обнаруживается посредством одного или более анализов молекулярных маркеров, описанных в настоящей заявке. Также обеспечивается вектор ДНК, содержащий рекомбинантную ДНК. Рекомбинантной молекулой ДНК или вектором ДНК может быть выделенная молекула нуклеиновой кислоты. ДНК, содержащая аллель урожая, может быть в микроорганизмах, таких как бактерии (например Agrobacterium).
Применение такой (выделенной или экстрагированной) молекулы нуклеиновой кислоты и/или такой рекомбинантной хромосомы или ее части для получения растительной клетки и растения, содержащего аллель урожая, охватывается настоящим изобретением. В одном варианте выполнения настоящего изобретения это может применяться для получения трансгенной растительной клетки и трансгенного растения, например клетки огурца, растений огурца и частей (например, плоды), содержащих аллель урожая, и растения, имеющего фенотип увеличенного урожая плодов.
Таким образом, трансгенные растительные клетки, например трансгенные клетки огурца, содержащие в их геноме рекомбинантную хромосому 3, как описано, и/или рекомбинантную молекулу нуклеиновой кислоты, содержащую аллель урожая, также являются вариантом выполнения настоящего изобретения. В одном варианте выполнения настоящего изобретения молекула ДНК, содержащая аллель урожая стабильно интегрируется в геном огурца.
Аллель урожая может также быть клонирована, и химерный ген может быть получен, например, функционально связывая растительный экспрессируемый промотор с аллелем урожая. Такой химерный ген может быть введен в растительную клетку, и растительная клетка может быть регенерирована в целое растение с получением трансгенного растения. В одном варианте трансгенным растением является растение огурца или растение дыни.
Таким образом, трансгенное растения, в частности трансгенный культивированный огурец или растения дыни, содержащие аллель урожая и дающие увеличенный урожай плодов, обеспечиваются согласно настоящему изобретению.
В частности клетки или клеточные культуры, содержащие рекомбинантную хромосому 3 согласно настоящему изобретению, являются вариантом выполнения настоящего изобретения, независимо от того вводится ли рекомбинантная хромосома 3 с помощью трансгенных способов или с помощью способов разведения. Клетки являются, например, in vitro и являются регенерируемыми в растения, содержащие рекомбинантную хромосому 3 согласно настоящему изобретению.
Также последовательности молекулярных маркеров (и выделенные молекулы нуклеиновых кислот, содержащие указанную последовательность), раскрытые в настоящей заявке, и молекулярные маркеры между любыми из упомянутых молекулярных маркеров, раскрытые в настоящей заявке, связанные с QTL3.1 урожая, и их применение для обнаружения и/или получения растений огурца, содержащих QTL, охватываются настоящим изобретением.
Депонирования семян
Представительный образец семян гибрида Cucumis sativus var. sativus длинного типа огурца, обозначенный CUCYLD-3, содержащий фрагмент интрогрессии, содержащий QTL3.1 в гетерозиготной форме, и генетический контроль (GC), не содержащий фрагмент интрогрессии и QTL, обозначенный CUYLD-GC, были депонированы Nunhems B.V. 17 декабря 2014 в NCIMB Ltd. (Ferguson Building, Craibstone Estate, Bucksburn Aberdeen, Scotland AB21 9YA, UK) в соответствии с Будапештским договором в рамках Экспертного решения (ЕРС 2000, Rule 32(1)). Семенам присвоили следующие номера депонирования NCIMB42346 (CUCYLD-3) и NCIMB42345 (CUYLD-GC).
Заявитель просит, чтобы образцы биологического материала и любого материала, полученного из него, были предоставлены только назначенному эксперту в соответствии с Правилом 32 (1) ЕРС или в соответствии с соответствующим законодательством стран или договорами, имеющими аналогичные правила и положения, до упоминания о выдачи патента или в течение 20 лет с даты подачи заявки, если заявка будет отклонена, отозвана или признана отозванной.
Доступ к депозиту будет доступен в течение срока действия этой заявки лицам, определенным Директором Патентного ведомства США, имеющим на это право при запросе. С учетом 37 C.F.R. § 1.808 (b), все ограничения, налагаемые владельцем депозита на широкую доступность материала, будут безвозвратно сняты после выдачи патента. Депозит будет храниться в течение 30 лет, или 5 лет после последнего запроса, или в течение срока действия патента, в зависимости от того, что больше, и будет заменен, если он станет нежизнеспособным в течение этого периода. Заявитель не отказывается от любых прав, предоставляемых в соответствии с настоящим патентом по этой заявке или в соответствии с Законом о защите сортов (7 USC 2321 et seq.).
Следующие неограничивающие примеры описывают, как можно получать растения согласно настоящему изобретению, содержащие QTL3. Если иного не указано в Примерах, все методики молекулярной ДНК осуществляются согласно стандартным протоколам, как описано в Sambrook et al. (1989) Molecular Cloning: A Laboratory Manual, Second Edition, Cold Spring Harbor Laboratory Press, and Sambrook and Russell (2001) Molecular Cloning: A Laboratory Manual, Third Edition, Cold Spring Harbor Laboratory Press, NY; и в Volumes 1 and 2 of Ausubel et al. (1994) Current Protocols in Molecular Biology, Current Protocols, USA. Стандартные материалы и способы для молекулярной работы с растением описываются в Plant Molecular Biology Labfax (1993) by R.D.D. Croy, совместно опубликованным BIOS Scientific Publications Ltd (UK) и Blackwell Scientific Publications, UK. Стандартные методы разведения описываются в 'Principles of Plant breeding', Second Edition, Robert W. Allard (ISBN 0-471-02309-4).
Примеры
Пример 1 - Идентификация QTL урожая
Разработка популяции
Экземпляр дикого родственника огурца, полученный из США, был скрещен с линией скрещивания длинного огурца, являющейся собственностью, HMRKC, в ходе программы по разведению для североевропейского и североамериканского рынка тепличных огурцов. HMRKC представляет собой элитную линию для программы на основе длинного тепличного огурца.
Популяцию QTL-обнаружения разработали из кросса между HMRKC и диким типом. В ходе разработки популяции только женские цветущие растения сохранили, чтобы облегчить измерения урожая.
SNP маркеры применялись в ходе нескольких генераций, чтобы выбрать длинные плоды и оптимизировать перекрытие генома и гомозиготность. BC2S2 популяцию применяли для составления генетической карты.
220 BC2S2 растения самоопылялись для получения BC2S3. BC2S2 растения также были скрещены с элитной линией из программы разведения, линией CUZL0176, для создания тестовых гибридов. Также получили генетический контроль посредством скрещивания HMRKC с CUZL0176. В испытаниях на урожай применяли 220 тестовых гибридов и генетический контроль.
Эксперименты на урожай
Целью эксперимента на урожай было измерение урожайности для длинных тепличных огурцов в ходе летне-осеннего периода в Нидерландах. Эксперимент состоял из 220 тестовых гибридов и 30 повторений генетического контроля.
Таким образом, в июне 2009 года было засеяно 250 участков в ручную в кассетах для рассады с помощью пробок из минеральной ваты. Кассеты выдерживали в течение 4 дней при температуре по меньшей мере 24°С. Через 4 дня после посева пробки с проросшими семенами были пересажены на блоки из минеральной ваты. В течение приблизительно 3 недель горшки из минеральной ваты содержались в определенном отсеке теплицы, в зоне выращивания растений. В этой области растения будут расти, пока они не будут готовы к посадке в теплице.
Растения высотой примерно 30 см транспортировались к садоводу примерно через 4 недели после посева. У садовода поддерживалось 8 растений на участок. Всего эксперимент состоял из 250 участков*8 растений. Было зарегистрировано точное количество растений на участок. Растения выращивались традиционным голландским способом. Это означает, что растения выращивали вертикально, поддерживая проволоку на высоте примерно до 220 см. На этой высоте верхняя часть растения удаляется, и растение продолжает расти на боковых поверхностях. Примерно через 3 недели после трансплантации можно собрать первые плоды (т.е. они имеют товарный размер). Период сбора урожая начался в августе и продолжался до конца октября. Растения собирали вручную 3-7 раз в неделю путем сбора товарных плодов.
Урожай был измерен двумя различными способами. Общее количество плодов на участок было подсчитано и разделено на количество растений этого участка. Это дает урожай, выраженный в среднем количестве плодов на растение (FrPP). Второе измерение состояло в том, чтобы получить массу плода на участок и разделить ее на количество растений, чтобы получить средний урожай в граммах на растение (GrPP).
В 2009 оду в Нидерландах было проведено два эксперимента.
Данные об урожае были использованы для анализа обнаружения QTL. QTL на хромосоме 3 был обнаружен с пиком (показатель LOD 6.3) при 89.347 сМ на генетической карте (интервал LOD начался в положении 76.214 сМ и закончился при 93.377 сМ хромосомы 3).
Таблица 1 и Таблица 2 показывают эффективность тестовых гибридов с интрогрессией из дикого родственника огурца на хромосоме 3 по сравнению с генетическим контролем, не содержащим интрогрессию на хромосоме 3. Увеличение урожая благодаря QTL3.1 составляло в среднем, для двух испытаний, 6% при выражении в GrPP и 16% при выражении в FrPP.
Таблица 1 - Урожай тестовых гибридов, содержащих интрогрессию на хромосоме 3 (QTL3.1), по сравнению с гибридом генетическим контролем (полученным путем скрещивания HMRKC с CUZL0176), не содержащим интрогрессию на хромосоме 3. Данные по урожаю основаны на двух испытаниях в Нидерландах (NL)
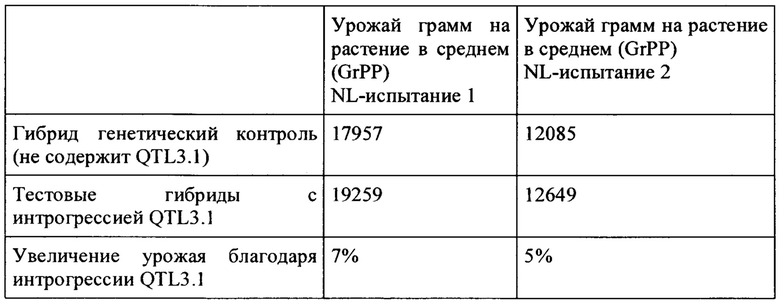
Таблица 2 - Урожай тестовых гибридов, содержащих интрогрессию на хромосоме 3 (QTL3.1) по сравнению с гибридом генетическим контролем (полученным скрещиванием HMRKC с CUZL0176), не содержащим интрогрессию на хромосоме 3. Данные по урожаю основаны на 2 испытаниях в Нидерландах (NL)
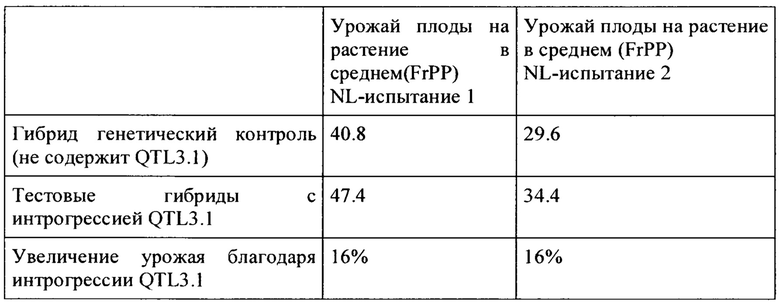
На основании результатов испытаний QTL-обнаружения была выбрана одна конкретная линия BC2S3, содержащая интрогрессию на хромосоме 3. Эта линия BC2S3 была скрещена с элитной линией CUZL0176 для создания тестового гибрида под названием PRE.N1.1168. Линия BC2S3 также была обратно скрещена с элитной линией HMRKC для создания ВС3. ВС3 был самоопылен дважды, чтобы создать линию BC3S2, содержащую только интрогрессии на хромосоме 3 из дикого родственника огурца. Эта линия BC3S2 была скрещена с элитной линией CUZL0176, чтобы создать новый тестовый гибрид, семена которого были депонированы под номером доступа NCIMB42346. NCIMB42346 имеет интрогрессию из дикого родственника огурца, содержащего QTL3.1 на хромосоме 3. Таким образом, NCIMB42346 гетерозиготно для QTL3.1.
Для сравнения элитная линия HMRKC была скрещена с элитной линией CUZL0176, создавая гибрид генетический контроль, семена которого были депонированы под номером доступа NCIMB42345.
В 2011 году было проведено два испытания на урожайность с PRE.N1.1168. Оба испытания были проведены с июня и заканчивались в октябре 2011 года. Сбор урожая в ходе обоих испытаний проводился с 31 недели до 43 недели. Таблица 3 показывает, что среднее увеличение урожайности для двух испытаний составило 20%, как выражается в плодах на растение (в среднем). Среднее увеличение урожайности для двух испытаний, как выражается в граммах на растение (в среднем), составляло 10% (данные не показаны).
Таблица 3 - Испытание урожайности 2011, 2 испытания и 4 повторений на испытание

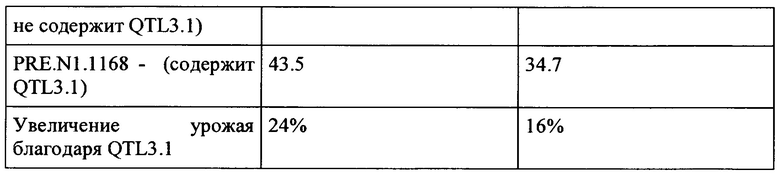
Другое испытание урожайности было проведено в 2014 году с NCIMB42346, включающим QTL3.1, и генетическим контролем NCIMB42345. Это испытание было проведено в период с июня по сентябрь 2014 года в Нидерландах. Плоды собирали с 29 недели до 39 недели. NCIMB42346 и генетический контроль NCIMB42345 были посажены при 8 повторах.
Таблица 4 показывает, что увеличение урожая благодаря QTL3.1, выраженное в плодах на растение в среднем, составляло 3,4%. Увеличение урожайности граммах на растение в среднем составило 2.2%.
Таблица 4 - Испытание урожайности 2014, 8 повторений. Урожайность выражалась в собранных плодах на растение в среднем (FrPP) и собранных граммах на растение в среднем (GrPP)
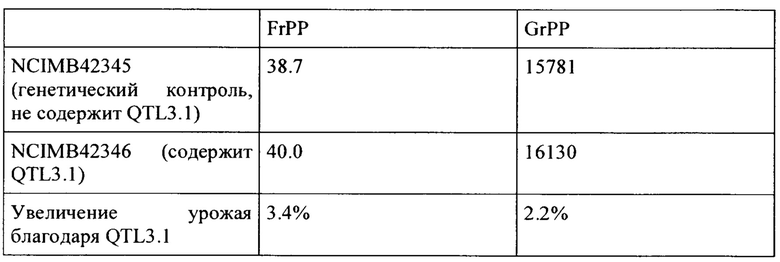
Пример 2
Маркеры однонуклеотидного полиморфизма (SNP) были идентифицированы, охватывающие фрагменты интрогрессии, и их положение на физической карте С. sativus было определено.
Таблица 5 - SNP маркеры для QTL3.1 фрагмента интрогрессии
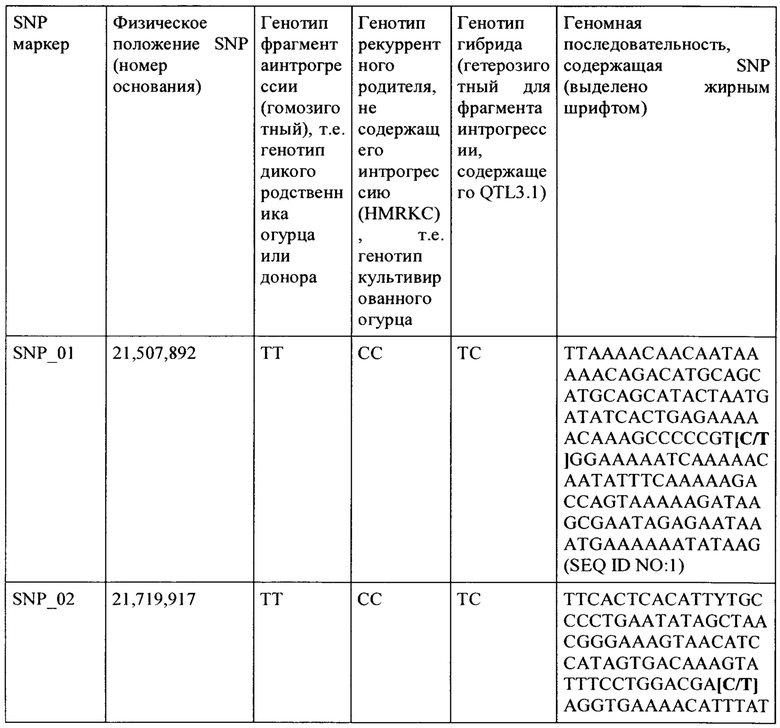
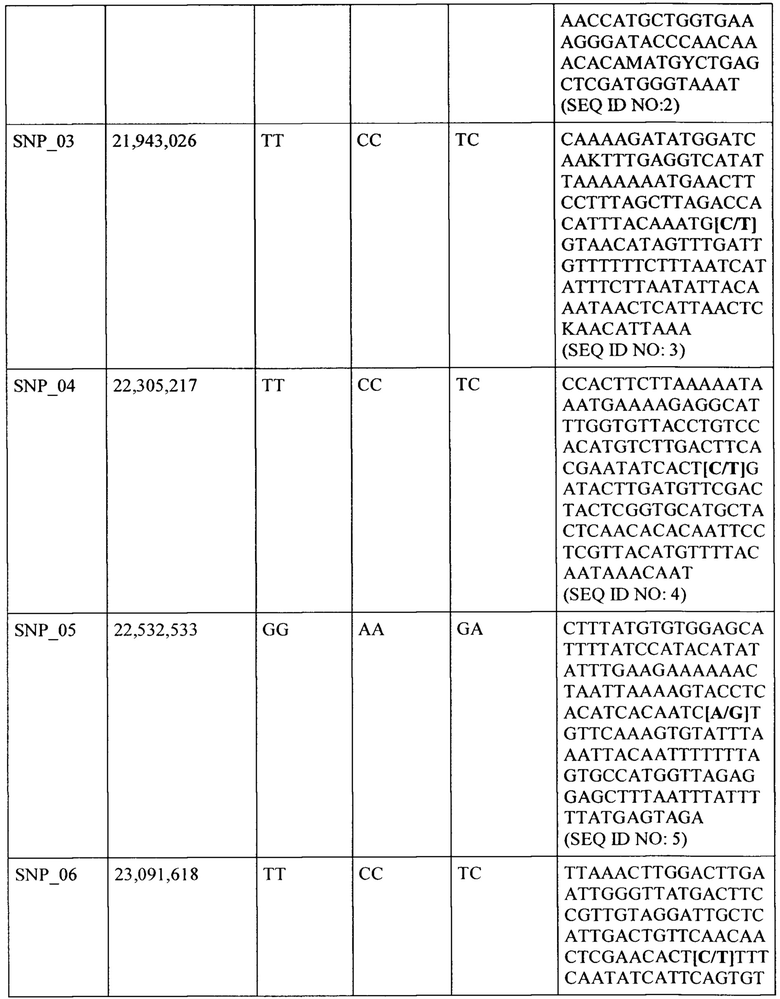
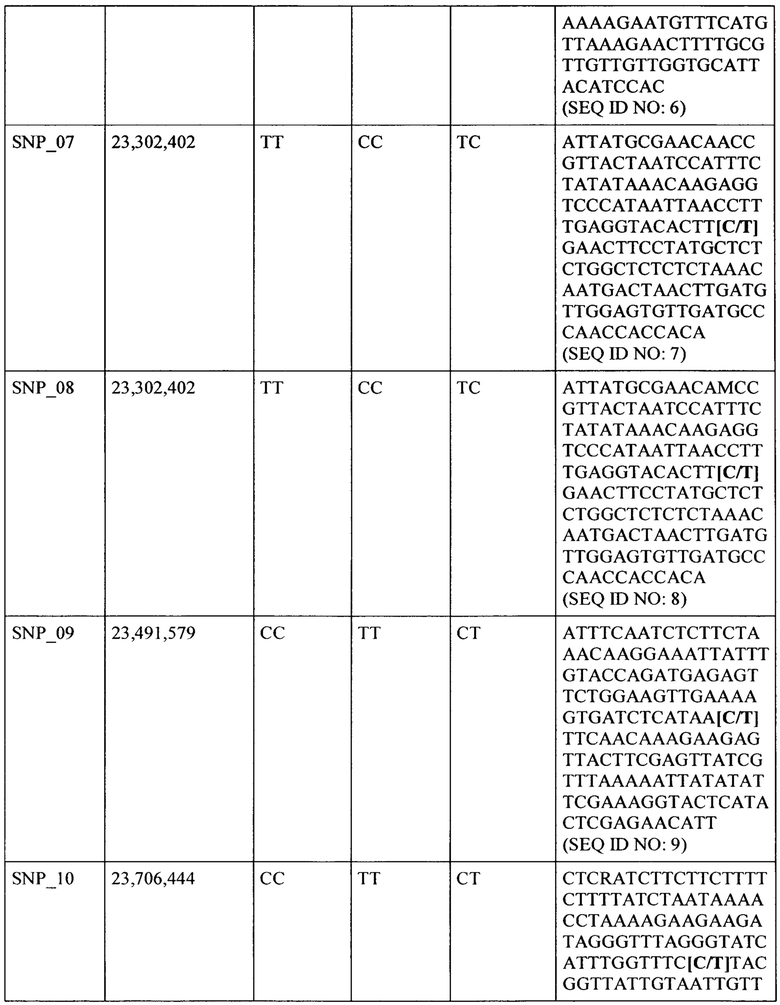
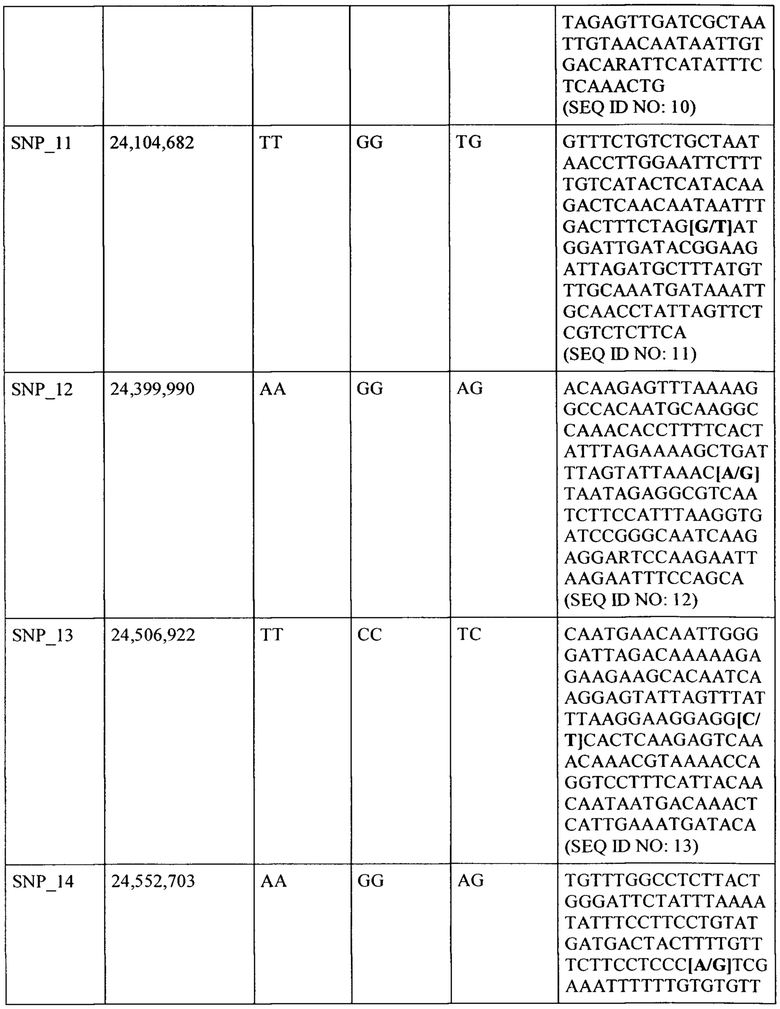
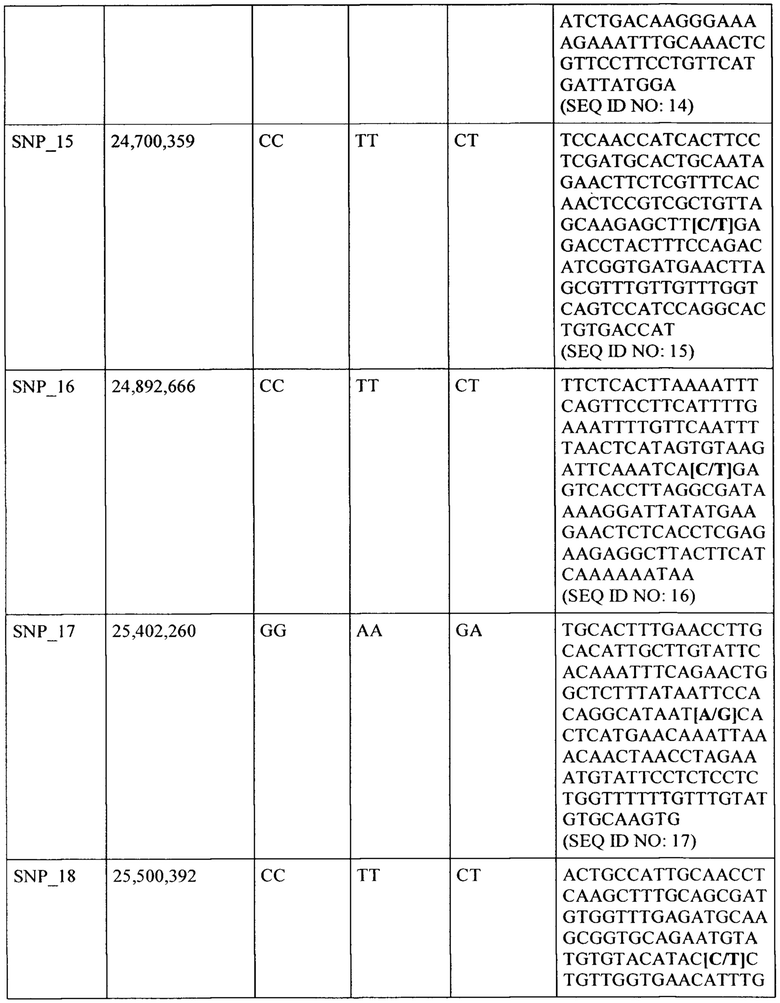
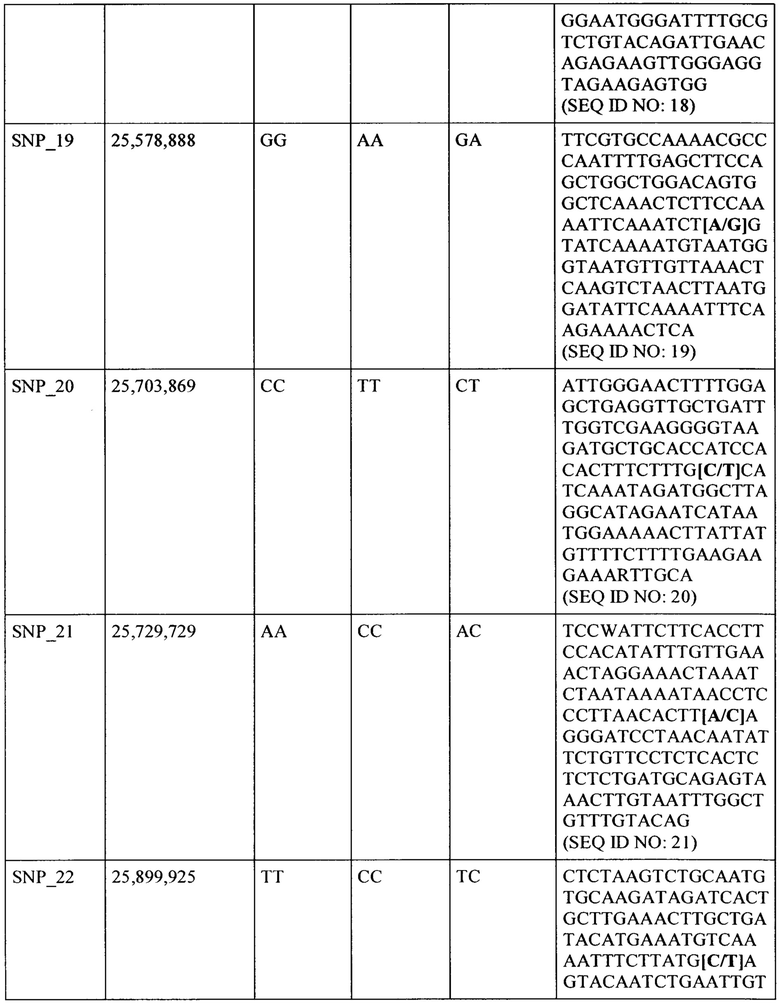
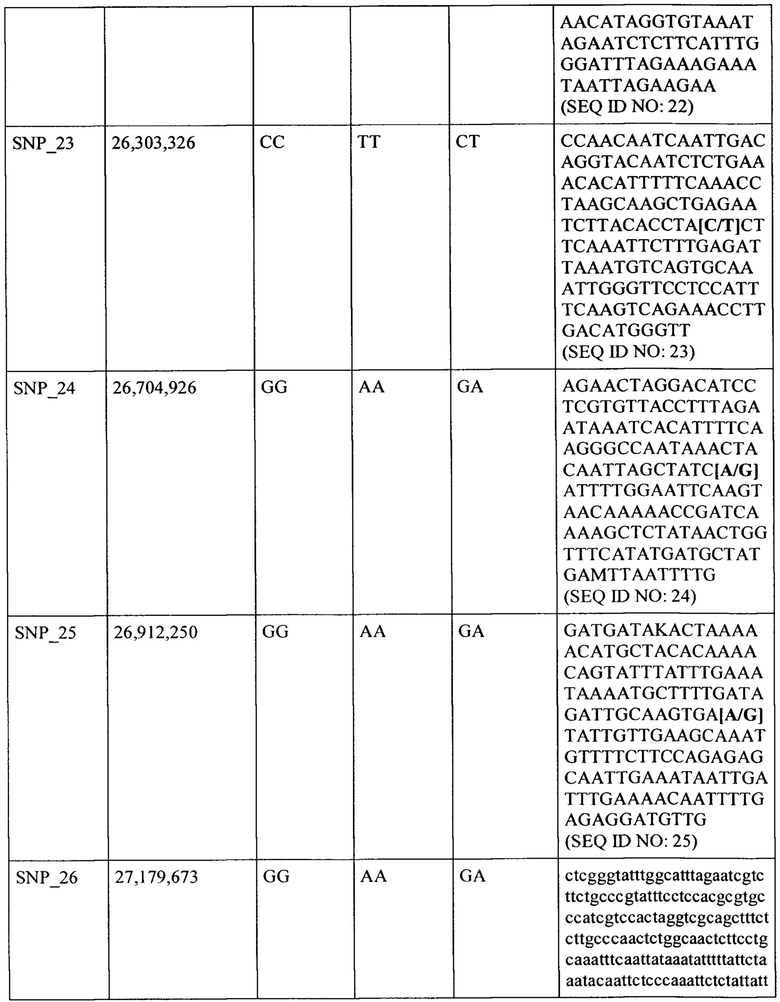
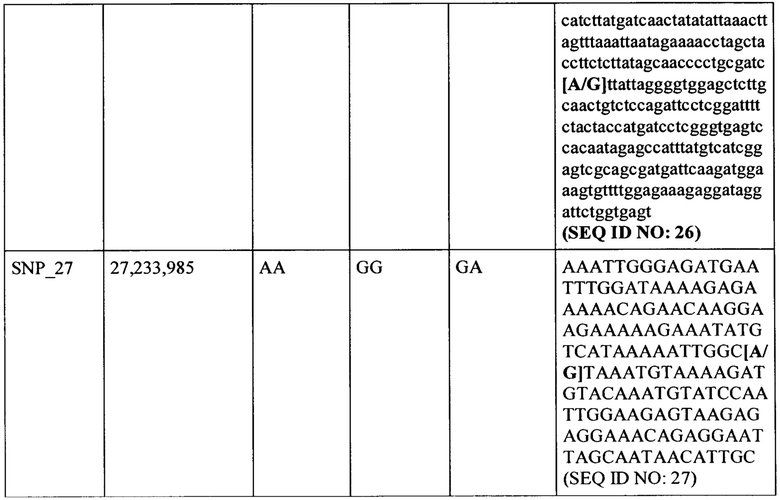
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Nunhems B.V.
<120> ИНТРОГРЕССИЯ QTL УРОЖАЯ В РАСТЕНИЯ CUCUMIS SATIVUS
<130> BCS15-8020
<150> EP15166819.1
<151> 2015-05-07
<160> 27
<170> PatentIn version 3.5
<210> 1
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP1
<222> (75)..(75)
<400> 1
ttaaaacaac aataaaaaca gacatgcagc atgcagcata ctaatgatat cactgagaaa 60
aacaaagccc ccgttggaaa aatcaaaaac aatatttcaa aaagaccagt aaaaagataa 120
gcgaatagag aataaatgaa aaaatataag 150
<210> 2
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP2
<222> (75)..(75)
<400> 2
ttcactcaca ttytgcccct gaatatagct aacgggaaag taacatccat agtgacaaag 60
tatttcctgg acgataggtg aaaacattta taaccatgct ggtgaaaggg atacccaaca 120
aacacamatg yctgagctcg atgggtaaat 150
<210> 3
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP3
<222> (75)..(75)
<400> 3
caaaagatat ggatcaaktt tgaggtcata ttaaaaaaat gaacttcctt tagcttagac 60
cacatttaca aatgtgtaac atagtttgat tgttttttct ttaatcatat ttcttaatat 120
tacaaataac tcattaactc kaacattaaa 150
<210> 4
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP4
<222> (75)..(75)
<400> 4
ccacttctta aaaataaatg aaaagaggca tttggtgtta cctgtccaca tgtcttgact 60
tcacgaatat cacttgatac ttgatgttcg actactcggt gcatgctact caacacacaa 120
ttcctcgtta catgttttac aataaacaat 150
<210> 5
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP5
<222> (75)..(75)
<400> 5
ctttatgtgt ggagcatttt atccatacat atatttgaag aaaaaactaa ttaaaagtac 60
ctcacatcac aatcgtgttc aaagtgtatt taaattacaa tttttttagt gccatggtta 120
gaggagcttt aatttatttt tatgagtaga 150
<210> 6
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP6
<222> (75)..(75)
<400> 6
ttaaacttgg acttgaattg ggttatgact tccgttgtag gattgctcat tgactgttca 60
acaactcgaa cactttttca atatcattca gtgtaaaaga atgtttcatg ttaaagaact 120
tttgcgttgt tgttggtgca ttacatccac 150
<210> 7
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP7
<222> (75)..(75)
<400> 7
attatgcgaa caaccgttac taatccattt ctatataaac aagaggtccc ataattaacc 60
tttgaggtac actttgaact tcctatgctc tctggctctc tctaaacaat gactaacttg 120
atgttggagt gttgatgccc aaccaccaca 150
<210> 8
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP8
<222> (75)..(75)
<400> 8
attatgcgaa camccgttac taatccattt ctatataaac aagaggtccc ataattaacc 60
tttgaggtac actttgaact tcctatgctc tctggctctc tctaaacaat gactaacttg 120
atgttggagt gttgatgccc aaccaccaca 150
<210> 9
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP9
<222> (75)..(75)
<400> 9
atttcaatct cttctaaaca aggaaattat ttgtaccaga tgagagttct ggaagttgaa 60
aagtgatctc ataacttcaa caaagaagag ttacttcgag ttatcgttta aaaattatat 120
attcgaaagg tactcatact cgagaacatt 150
<210> 10
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP10
<222> (75)..(75)
<400> 10
ctcratcttc ttcttttctt ttatctaata aaacctaaaa gaagaagata gggtttaggg 60
tatcatttgg tttcctacgg ttattgtaat tgtttagagt tgatcgctaa ttgtaacaat 120
aattgtgaca rattcatatt tctcaaactg 150
<210> 11
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP11
<222> (75)..(75)
<400> 11
gtttctgtct gctaataacc ttggaattct tttgtcatac tcatacaaga ctcaacaata 60
atttgacttt ctagtatgga ttgatacgga agattagatg ctttatgttt gcaaatgata 120
aattgcaacc tattagttct cgtctcttca 150
<210> 12
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP12
<222> (75)..(75)
<400> 12
acaagagttt aaaaggccac aatgcaaggc caaacacctt ttcactattt agaaaagctg 60
atttagtatt aaacataata gaggcgtcaa tcttccattt aaggtgatcc gggcaatcaa 120
gaggartcca agaattaaga atttccagca 150
<210> 13
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP13
<222> (75)..(75)
<400> 13
caatgaacaa ttggggatta gacaaaaaga gaagaagcac aatcaaggag tattagttta 60
tttaaggaag gaggtcactc aagagtcaaa caaacgtaaa accaggtcct ttcattacaa 120
caataatgac aaactcattg aaatgataca 150
<210> 14
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP14
<222> (75)..(75)
<400> 14
tgtttggcct cttactggga ttctatttaa aatatttcct tcctgtatga tgactacttt 60
tgtttcttcc tcccatcgaa attttttgtg tgttatctga caagggaaaa gaaatttgca 120
aactcgttcc ttcctgttca tgattatgga 150
<210> 15
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP15
<222> (75)..(75)
<400> 15
tccaaccatc acttcctcga tgcactgcaa tagaacttct cgtttcacaa ctccgtcgct 60
gttagcaaga gcttcgagac ctactttcca gacatcggtg atgaacttag cgtttgttgt 120
ttggtcagtc catccaggca ctgtgaccat 150
<210> 16
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP16
<222> (75)..(75)
<400> 16
ttctcactta aaatttcagt tccttcattt tgaaattttg ttcaatttta actcatagtg 60
taagattcaa atcacgagtc accttaggcg ataaaaggat tatatgaaga actctcacct 120
cgagaagagg cttacttcat caaaaaataa 150
<210> 17
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP17
<222> (75)..(75)
<400> 17
tgcactttga accttgcaca ttgcttgtat tcacaaattt cagaactggc tctttataat 60
tccacaggca taatgcactc atgaacaaat taaacaacta acctagaaat gtattcctct 120
cctctggttt tttgtttgta tgtgcaagtg 150
<210> 18
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP18
<222> (75)..(75)
<400> 18
actgccattg caacctcaag ctttgcagcg atgtggtttg agatgcaagc ggtgcagaat 60
gtatgtgtac ataccctgtt ggtgaacatt tgggaatggg attttgcgtc tgtacagatt 120
gaacagagaa gttgggaggt agaagagtgg 150
<210> 19
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP19
<222> (75)..(75)
<400> 19
ttcgtgccaa aacgcccaat tttgagcttc cagctggctg gacagtggct caaactcttc 60
caaaattcaa atctggtatc aaaatgtaat gggtaatgtt gttaaactca agtctaactt 120
aatggatatt caaaatttca agaaaactca 150
<210> 20
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP20
<222> (75)..(75)
<400> 20
attgggaact tttggagctg aggttgctga tttggtcgaa ggggtaagat gctgcaccat 60
ccacactttc tttgccatca aatagatggc ttaggcatag aatcataatg gaaaaactta 120
ttatgttttc ttttgaagaa gaaarttgca 150
<210> 21
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP21
<222> (75)..(75)
<400> 21
tccwattctt caccttccac atatttgttg aaactaggaa actaaatcta ataaaataac 60
ctcccttaac acttaaggga tcctaacaat attctgttcc tctcactctc tctgatgcag 120
agtaaacttg taatttggct gtttgtacag 150
<210> 22
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP22
<222> (75)..(75)
<400> 22
ctctaagtct gcaatgtgca agatagatca ctgcttgaaa cttgctgata catgaaatgt 60
caaaatttct tatgtagtac aatctgaatt gtaacatagg tgtaaataga atctcttcat 120
ttgggattta gaaagaaata attagaagaa 150
<210> 23
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP23
<222> (75)..(75)
<400> 23
ccaacaatca attgacaggt acaatctctg aaacacattt ttcaaaccta agcaagctga 60
gaatcttaca cctaccttca aattctttga gattaaatgt cagtgcaaat tgggttcctc 120
catttcaagt cagaaacctt gacatgggtt 150
<210> 24
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP24
<222> (75)..(75)
<400> 24
agaactagga catcctcgtg ttacctttag aataaatcac attttcaagg gccaataaac 60
tacaattagc tatcgatttt ggaattcaag taacaaaaac cgatcaaaag ctctataact 120
ggtttcatat gatgctatga mttaattttg 150
<210> 25
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP25
<222> (75)..(75)
<400> 25
gatgatakac taaaaacatg ctacacaaaa cagtatttat ttgaaataaa atgcttttga 60
tagattgcaa gtgagtattg ttgaagcaaa tgttttcttc cagagagcaa ttgaaataat 120
tgatttgaaa acaattttga gaggatgttg 150
<210> 26
<211> 413
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP26
<222> (251)..(251)
<400> 26
ctcgggtatt tggcatttag aatcgtcttc tgcccgtatt tcctccacgc gtgcccatcg 60
tccactaggt cgcagctttc tcttgcccaa ctctggcaac tcttcctgca aatttcaatt 120
ataaatattt ttattctaaa tacaattctc ccaaattctc tattattcat cttatgatca 180
actatatatt aaacttagtt taaattaata gaaaacctag ctaccttctc ttatagcaac 240
ccctgcgatc gttattaggg gtggagctct tgcaactgtc tccagattcc tcggattttc 300
tactaccatg atcctcgggt gagtccacaa tagagccatt tatgtcatcg gagtcgcagc 360
gatgattcaa gatggaaagt gttttggaga aagaggatag gattctggtg agt 413
<210> 27
<211> 150
<212> ДНК
<213> Cucumis sativus
<220>
<221> SNP27
<222> (75)..(75)
<400> 27
aaattgggag atgaatttgg ataaaagaga aaaacagaac aaggaagaaa aagaaatatg 60
tcataaaaat tggcataaat gtaaaagatg tacaaatgta tccaattgga agagtaagag 120
aggaaacaga ggaattagca ataacattgc 150
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ИНТРОГРЕССИЯ ДВУХ QTL УРОЖАЯ В РАСТЕНИЯ CUCUMIS SATIVUS | 2017 |
|
RU2746936C2 |
| QTL УРОЖАЯ В РАСТЕНИЯХ ОГУРЦА | 2015 |
|
RU2723586C2 |
| QTL УРОЖАЯ В РАСТЕНИЯХ ОГУРЦА | 2015 |
|
RU2723581C2 |
| РАСТЕНИЯ САЛАТ-ЛАТУК, ИМЕЮЩИЕ РЕЗИСТЕНТНОСТЬ ПРОТИВ NASONOVIA RIBISNIGRI БИОТИПА 1 | 2015 |
|
RU2731639C2 |
| РАСТЕНИЯ ОГУРЦА, УСТОЙЧИВЫЕ К ЗАБОЛЕВАНИЯМ | 2006 |
|
RU2418405C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM4 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2718584C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ ГЕНА RLM2 РЕЗИСТЕНТНОСТИ К ЧЕРНОЙ НОЖКЕ BRASSICA NAPUS И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2014 |
|
RU2717017C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ НИЗКОГО СОДЕРЖАНИЯ ПАЛЬМИТИНОВОЙ КИСЛОТЫ В ПОДСОЛНЕЧНИКЕ (HELIANTHUS ANNUS) И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2013 |
|
RU2670517C2 |
| МОЛЕКУЛЯРНЫЕ МАРКЕРЫ, АССОЦИИРОВАННЫЕ С УСТОЙЧИВОСТЬЮ ПОДСОЛНЕЧНИКА К OROBANCHE | 2018 |
|
RU2776361C2 |
| SNP-МАРКЕРЫ И ОТБОР В ОТНОШЕНИИ НИЗКОГО СОДЕРЖАНИЯ КЛЕТЧАТКИ В ПРЕДСТАВИТЕЛЯХ РОДА BRASSICA | 2019 |
|
RU2834673C2 |
Изобретение относится к области биохимии, в частности к культивированному растению Cucumis sativus var. sativus, обладающему увеличенным урожаем плодов, к его части, семени, плоду, а также к его клетке. Также раскрыт способ идентификации культивированного растения С.sativus var. sativus, включающего рекомбинантную хромосому 3, а также к способу обнаружения содержит ли культивированное растение огурца фрагмент интрогрессии на хромосоме 3. Изобретение также относится к способу идентификации растения дикого родственника огурца, содержащего QTL урожая на хромосоме 3. Изобретение позволяет эффективно получать растение Cucumis sativus var. sativus, обладающее увеличенным урожаем плодов. 8 н. и 13 з.п. ф-лы, 5 табл., 2 пр.
1. Культивированное растение Cucumis sativus var. sativus, включающее рекомбинантную хромосому 3, причем указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, где указанный фрагмент интрогрессии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при основании 21,507,892 хромосомы 3 и маркером однонуклеотидного полиморфизма SNP_27 при основании 27,233,985 хромосомы 3, где QTL обеспечивает увеличение урожая плодов огурца, причем QTL представляет собой QTL, присутствующий в семенах, депонированных под номером доступа NCIMB42346, причем растение обладает увеличенным урожаем плодов по сравнению с растением, не содержащим указанный фрагмент интрогрессии, и причем указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
2. Растение по п. 1, где указанное увеличение урожая плодов огурца фенотипически выражается как значительно более высокое среднее количество плодов на растение (FrPP) растения, содержащего фрагмент интрогрессии, по сравнению с растением, не содержащим фрагмент интрогрессии, при выращивании в той же среде, и/или значительно более высокая средняя масса плода на растение (GrPP) растения, содержащего фрагмент интрогрессии, по сравнению с растением, не содержащим фрагмент интрогрессии, при выращивании в той же среде.
3. Растение по п. 1, где указанный фрагмент интрогрессии на хромосоме 3 обнаруживается посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2 или 3 маркера, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
4. Растение по п. 1, содержащее QTL и содержащее по меньшей мере 1, 2, 3, 4 или 5 маркеров, выбранных из одной из групп а), b), с) или d):
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10; или
b) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10; генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11; генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13; генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20; или
c) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20; генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22; генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26; генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27; или
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10; генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11; генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13; генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18; генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19; генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20; генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21; генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22; генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23.
5. Растение по п. 1, где фрагмент интрогрессии находится в гетерозиготной форме и один или более SNP маркеров имеют гетерозиготный генотип SNP.
6. Растение по п. 1, где фрагмент интрогрессии находится в гомозиготной форме и один или более SNP маркеров имеют гомозиготный генотип SNP.
7. Растение по п. 1, где растение относится к одному из следующих типов огурца: салатный огурец, длинный огурец, Европейский тепличный огурец.
8. Растение по п. 1, где растением является культивированный огурец Евразийской группы огурцов и где фрагмент интрогрессии происходит из Cucumis sativus var. hardwickii, С. sativus var. sikkimensis или Cucumis sativus var. xishuangbannesi.
9. Растение по п. 1, где растение является партенокарпическим.
10. Растение по любому из пп. 1-9, где указанный фрагмент интрогрессии на хромосоме 3 является получаемым скрещиванием растения, выращенного из семян, депонированных под номером доступа NCIMB42346, с другим растением огурца.
11. Семя для выращивания культивированного растения Cucumis sativus var. sativus, включающее рекомбинантную хромосому 3, причем указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, где указанный фрагмент интрогрессии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при основании 21,507,892 хромосомы 3 и маркером однонуклеотидного полиморфизма SNP_27 при основании 27,233,985 хромосомы 3, где QTL обеспечивает увеличение урожая плодов огурца, причем QTL представляет собой QTL, присутствующий в семенах, депонированных под номером доступа NCIMB42346, причем растение обладает увеличенным урожаем плодов по сравнению с растением, не содержащим указанный фрагмент интрогрессии, и причем указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
12. Семя по п. 11, где указанный фрагмент интрогрессии на хромосоме 3 является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2 или 3 маркера, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
13. Плод огурца для получения пищевого продукта или кормового продукта, собранный с культивированного растения Cucumis sativus var. sativus, включающий рекомбинантную хромосому 3, причем указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, где указанный фрагмент интрогрессии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при основании 21,507,892 хромосомы 3 и маркером однонуклеотидного полиморфизма SNP_27 при основании 27,233,985 хромосомы 3, где QTL обеспечивает увеличение урожая плодов огурца, причем QTL представляет собой QTL, присутствующий в семенах, депонированных под номером доступа NCIMB42346, причем растение обладает увеличенным урожаем плодов по сравнению с растением, не содержащим указанный фрагмент интрогрессии, и причем указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
14. Плод огурца по п. 13, где указанный фрагмент интрогрессии на хромосоме 3 является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2 или 3 маркера, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
15. Растительная клетка культивированного растения Cucumis sativus var. sativus, включающая рекомбинантную хромосому 3, для обеспечения увеличенного урожая плодов у растения, полученного из нее, по сравнению с растением, не содержащим указанную рекомбинантную хромосому 3, причем указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, где указанный фрагмент интрогрессии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при основании 21,507,892 хромосомы 3 и маркером однонуклеотидного полиморфизма SNP_27 при основании 27,233,985 хромосомы 3, где QTL обеспечивает увеличение урожая плодов огурца, причем QTL представляет собой QTL, присутствующий в семенах, депонированных под номером доступа NCIMB42346, причем растение обладает увеличенным урожаем плодов по сравнению с растением, не содержащим указанный фрагмент интрогрессии, и причем указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
16. Растительная клетка по п. 15, где указанный фрагмент интрогрессии на хромосоме 3 является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2 или 3 маркера, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
17. Способ идентификации культивированного растения С. sativus var. sativus, включающего рекомбинантную хромосому 3, причем указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, содержащий локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при основании 21,507,892 хромосомы 3 и маркером однонуклеотидного полиморфизма SNP_27 при основании 27,233,985 хромосомы 3, и обладающего увеличенной урожайностью плодов по сравнению с растением, не содержащим указанный фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии представляет собой такой, как обнаружено в NCIMB42346, или более маленький фрагмент, производный от него, причем указанный способ включает:
a) обеспечение популяции культивированных растений С. sativus var. sativus,
b) скрининг указанной популяции, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, выбранный из группы, состоящей из:
SNP_01-SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3;
c) идентификацию и/или отбор растения, содержащего:
i) по меньшей мере 1 из SNP маркеров SNP_01-SNP_27 для обнаружения фрагмента интрогрессии на хромосоме 3; или
ii) по меньшей мере 2, 3 или 4 последовательных маркеров, выбранных из SNP_01-SNP_27, для обнаружения фрагмента интрогрессии на хромосоме 3,
причем указанные маркеры SNP_01-SNP_27 представляют собой:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
18. Способ обнаружения содержит ли культивированное растение огурца фрагмент интрогрессии на хромосоме 3, где указанный фрагмент интрогрессии содержит QTL, где QTL обеспечивает увеличение урожая плодов огурца, причем указанный способ включает:
a) обеспечение культивированного растения огурца или части растения,
b) скрининг указанного растения или указанной части растения или ДНК, полученной из указанного растения или части растения, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, выбранный из группы, состоящей из SNP_01-SNP_27,
причем указанные маркеры SNP_01-SNP_27 представляют собой:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип AC или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
19. Способ идентификации растения дикого родственника огурца, содержащего QTL урожая на хромосоме 3, причем указанный способ включает:
A) обеспечение экземпляра дикого родственника огурца или нескольких экземпляров;
B) скрининг указанного экземпляра или указанных экземпляров, применяя анализ молекулярного маркера, который обнаруживает по меньшей мере один SNP маркер, выбранный из группы, состоящей из SNP_01-SNP_27;
C) идентификацию и/или селекцию экземпляра согласно В), содержащего по меньшей мере один или более следующих маркеров:
а) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 в SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 в SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 в SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 в SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 в SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 в SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 в SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 в SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 в SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 в SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 в SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 в SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 в SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 в SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 в SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 в SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 в SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 в SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 в SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 в SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 в SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 в SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 в SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 в SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 в SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 в SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 в SEQ ID NO: 27.
20. Часть растения культивированного растения Cucumis sativus var. sativus, включающая рекомбинантную хромосому 3, для обеспечения увеличенного урожая плодов у растения, полученного из нее, по сравнению с растением, не содержащим указанную рекомбинантную хромосому 3, причем указанная рекомбинантная хромосома 3 содержит фрагмент интрогрессии из дикого родственника огурца, где указанный фрагмент интрогрессии содержит локус количественных признаков (QTL), локализованный между маркером однонуклеотидного полиморфизма SNP_01 при основании 21,507,892 хромосомы 3 и маркером однонуклеотидного полиморфизма SNP_27 при основании 27,233,985 хромосомы 3, где QTL обеспечивает увеличение урожая плодов огурца, причем QTL представляет собой QTL, присутствующий в семенах, депонированных под номером доступа NCIMB42346, причем растение обладает увеличенным урожаем плодов по сравнению с растением, не содержащим указанный фрагмент интрогрессии, и причем указанный фрагмент интрогрессии является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере один маркер, выбранный из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
21. Часть растения по п. 20, где указанный фрагмент интрогрессии на хромосоме 3 является обнаруживаемым посредством анализа молекулярного маркера, который обнаруживает по меньшей мере 2 или 3 маркера, выбранных из группы, состоящей из:
a) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_01 при нуклеотиде 75 последовательности SEQ ID NO: 1;
b) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_02 при нуклеотиде 75 последовательности SEQ ID NO: 2;
c) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_03 при нуклеотиде 75 последовательности SEQ ID NO: 3;
d) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_04 при нуклеотиде 75 последовательности SEQ ID NO: 4;
e) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_05 при нуклеотиде 75 последовательности SEQ ID NO: 5;
f) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_06 при нуклеотиде 75 последовательности SEQ ID NO: 6;
g) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_07 при нуклеотиде 75 последовательности SEQ ID NO: 7;
h) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_08 при нуклеотиде 75 последовательности SEQ ID NO: 8;
i) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_09 при нуклеотиде 75 последовательности SEQ ID NO: 9;
j) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_10 при нуклеотиде 75 последовательности SEQ ID NO: 10;
k) генотип TG или ТТ для маркера однонуклеотидного полиморфизма SNP_11 при нуклеотиде 75 последовательности SEQ ID NO: 11;
l) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_12 при нуклеотиде 75 последовательности SEQ ID NO: 12;
m) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_13 при нуклеотиде 75 последовательности SEQ ID NO: 13;
n) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_14 при нуклеотиде 75 последовательности SEQ ID NO: 14;
о) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_15 при нуклеотиде 75 последовательности SEQ ID NO: 15;
р) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_16 при нуклеотиде 75 последовательности SEQ ID NO: 16;
q) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_17 при нуклеотиде 75 последовательности SEQ ID NO: 17;
r) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_18 при нуклеотиде 75 последовательности SEQ ID NO: 18;
s) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_19 при нуклеотиде 75 последовательности SEQ ID NO: 19;
t) генотип СТ или СС для маркера однонуклеотидного полиморфизма SNP_20 при нуклеотиде 75 последовательности SEQ ID NO: 20;
u) генотип АС или АА для маркера однонуклеотидного полиморфизма SNP_21 при нуклеотиде 75 последовательности SEQ ID NO: 21;
v) генотип ТС или ТТ для маркера однонуклеотидного полиморфизма SNP_22 при нуклеотиде 75 последовательности SEQ ID NO: 22;
w) генотип СТ или ТТ для маркера однонуклеотидного полиморфизма SNP_23 при нуклеотиде 75 последовательности SEQ ID NO: 23;
х) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_24 при нуклеотиде 75 последовательности SEQ ID NO: 24;
у) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_25 при нуклеотиде 75 последовательности SEQ ID NO: 25;
z) генотип GA или GG для маркера однонуклеотидного полиморфизма SNP_26 при нуклеотиде 251 последовательности SEQ ID NO: 26;
аа) генотип AG или АА для маркера однонуклеотидного полиморфизма SNP_27 при нуклеотиде 75 последовательности SEQ ID NO: 27.
| WO 2009082222 A1, 02.07.2009 | |||
| QINGZHEN WEI et al., An SNP-based saturated genetic map and QTL analysis of fruit-related traits in cucumber using specific-length amplified fragment (SLAF) sequencing, BMC Genomics, 2014, 15:1158 | |||
| FAZIO G | |||
| et al., Genetic mapping and QTL analysis of horticultural traits in cucumber (Cucumis sativus L.) using |
Авторы
Даты
2021-02-01—Публикация
2016-05-03—Подача