ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
[1] Настоящая заявка испрашивает приоритет по предварительной заявке на патент США с порядковым № 62/323589, поданной 15 апреля 2016 года, которая настоящим включена посредством ссылки во всей своей полноте.
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
[2] Раскрытые изобретения относятся, в целом, к способам выявления мутаций и слияний нуклеиновой кислоты с применением способов амплификации, таких как полимеразная цепная реакция (ПЦР).
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
[3] Выявление мутаций, ассоциированных с типами рака, независимо от того, происходит ли это перед диагностированием, при диагностировании, для определения стадии заболевания или для контроля эффективности лечения, традиционно основывается на образцах биопсии солидных опухолей. Получение таких образцов является очень инвазивным и сопряжено с риском потенциального содействия образованию метастазов или послеоперационных осложнений. Необходимы лучшие и менее инвазивные способы выявления мутаций, ассоциированных с раком.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[4] В одном варианте осуществления в данном документе предусматривается способ определения однонуклеотидных вариантов, присутствующих в плоскоклеточной карциноме легкого. Способ по этому варианту осуществления предусматривает получение набора ампликонов путем осуществления мультиплексной реакции амплификации в отношении нуклеиновых кислот, выделенных из образца крови или ее фракции от индивидуума с подозрением на плоскоклеточную карциному легкого, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, которые, как известно, ассоциированы с раком легкого; и
[5] определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов, где сегмент содержит локус однонуклеотидного варианта, за счет чего осуществляется определение однонуклеотидных вариантов, присутствующих в плоскоклеточной карциноме.
[6] В другом варианте осуществления в данном документе предусматривается способ подтверждения диагноза рака легкого у индивидуума с подозрением на рак легкого на основании образца крови или ее фракции от индивидуума. Способ предусматривает получение набора ампликонов путем осуществления мультиплексной реакции амплификации в отношении нуклеиновых кислот, выделенных из образца, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, которые, как известно, ассоциированы с раком легкого; и
[7] определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов, где сегмент содержит локус однонуклеотидного варианта, за счет чего осуществляется определение того, присутствует ли один или более однонуклеотидных вариантов в нескольких локусах однонуклеотидных вариантов. Согласно иллюстративным вариантам осуществления
[8] отсутствие однонуклеотидного варианта подтверждает диагноз аденокарциномы стадии 1a, 2a или 2b,
[9] присутствие однонуклеотидного варианта подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 2b или 3a, и/или
[10] присутствие 5, 10, 15 или более однонуклеотидных вариантов подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 2b или 3.
[11] В определенных вариантах осуществления присутствие 5, 10 или 15 или более однонуклеотидных вариантов подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 3.
[12] В иллюстративных примерах любого из вариантов осуществления способа, представленных в данном документе, который включает стадию амплификации, реакция амплификации представляет собой ПЦР-реакцию, при этом температура отжига на 1—15°C превышает температуру плавления по меньшей мере 50, 60, 70, 85, 80, 90, 95 или 100% праймеров из набора праймеров, продолжительность стадии отжига в ПЦР-реакции составляет 15—60 минут, концентрация праймеров в реакции амплификации составляет 1—10 нМ, и праймеры в наборе праймеров разработаны для сведения к минимуму образования димеров из праймеров.
[13] В любом из вариантов осуществления способа по настоящему изобретению, который предусматривает определение или выявление присутствия SNV с применением способа амплификации, эффективность и частоту ошибок на цикл можно определять для каждой реакции амплификации из мультиплексной реакции амплификации набора локусов однонуклеотидной изменчивости, и эффективность и частоту ошибок можно применять для определения того, присутствует ли в образце однонуклеотидный вариант из набора локусов однонуклеотидных вариантов. В некоторых из этих приводимых в качестве примера вариантах осуществления определяют достоверность и распознавание SNV осуществляют, если превышено предельное значение достоверности, такое как 90%, 95% или 98% достоверность.
[14] В других вариантах осуществления в данном документе представлены композиции и твердые подложки по настоящему изобретению.
[15] Другие варианты осуществления, а также признаки и преимущества раскрытых изобретений будут очевидны из следующего подробного описания и формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
[16] Комплект материалов патента или заявки содержит по меньшей мере один графический материал, выполненный в цвете. Копии публикации настоящего патента или патентной заявки с цветным графическим материалом(-ами) будут предоставлены Ведомством после запроса и оплаты необходимого сбора.
[17] Варианты осуществления, раскрытые в настоящем документе, будут дополнительно объяснены со ссылкой на приложенные графические материалы, где ссылка на аналогичные структуры приводится с помощью аналогичных чисел на всех нескольких изображениях. Показанные графические материалы не обязательно изображены в масштабе, вместо этого акцент, в основном, сделан на иллюстрации принципов вариантов осуществления, раскрытых в настоящем изобретении.
[18] ФИГ. 1 представляет собой диаграмму рабочего протокола.
[19] ФИГ. 2. Верхняя панель: число SNV на образец; нижняя панель: рабочие анализы, отсортированные в соответствии с категорией драйверных мутаций.
[20] Фигура 3. Измеренная концентрация cfDNA. Каждая точка на графике обозначает образец плазмы крови.
[21] Фигура 4. Образцы, демонстрирующие хорошую корреляцию между измерениями VAF ткани, определенными ранее (ось x) и в данном документе с применением mPCR-NGS (ось y). Каждый образец показан в виде отдельного блока, а точки VAF на графике окрашены в соответствии с фрагментом ткани.
[22] Фигура 5. Образцы, демонстрирующие слабую корреляцию между измерениями VAF ткани, определенными ранее (ось x) и в данном документе с применением mPCR-NGS (ось y). Каждый образец показан в виде отдельного блока, а точки VAF на графике окрашены в соответствии с фрагментом ткани.
[23] Фигура 6A. Гистограмма глубины прочтения в зависимости от полученного в результате распознавания. Вверху: в анализе не выявили ожидаемые SNV в плазме крови. Внизу: в анализе выявили ожидаемые SNV в плазме крови.
[24] Фигура 7. Число SNV, выявленных в плазме крови, в соответствии с гистологическим типом.
[25] Фигура 8. Выявление SNV (слева) и выявление образца (справа) в плазме крови в соответствии со стадией опухоли.
[26] Фигура 9. VAF плазмы крови в зависимости от стадии опухоли и клональности SNV.
[27] Фигура 10. Число SNV, выявленных в плазме крови из каждого образца, в зависимости от количества вводимой cfDNA.
[28] Фигура 11. VAF плазмы крови в зависимости от среднего VAF опухоли. Среднюю VAF опухоли рассчитывали для всех фрагментов опухоли, проанализированных из каждой опухоли.
[29] На ФИГ. 12 показаны клональные соотношения (от красного до синего) и частота мутантного вариантного аллеля (MutVAF) для каждого выявленного SNV. Общее число SNV, выявленных в каждом образце, помещено в одну колонку, а образцы разделены по категориям в соответствии со стадией опухоли (стадия pTNM). Включены образцы с отсутствием выявления SNV. Клональное соотношение определяется как соотношение числа фрагментов опухоли, в которых выявили SNV, и общего числа фрагментов, проанализированных из данной опухоли.
[30] На ФИГ. 13 показан клональный статус (синий для клональных и красный для субклональных) и частота мутантного вариантного аллеля (MutVAF) для каждого выявленного SNV. Общее число SNV, выявленных в каждом образце, помещено в одну колонку, а образцы разделены по категориям в соответствии со стадией опухоли (стадия pTNM). Включены образцы с отсутствием выявления SNV. Клональный статус определяли с помощью PyCloneCluster с применением данных полноэкзомного секвенирования опухолевой ткани.
[31] На ФИГ. 14 показан клональный статус (синий для клональных и красный для субклональных) и частота мутантного вариантного аллеля (MutVAF) для каждого выявленного SNV, где на верхней панели показаны только клональные SNV, а на нижней панели показаны только субклональные SNV. Общее число SNV, выявленных в каждом образце, помещено в одну колонку, а образцы разделены по категориям в соответствии со стадией опухоли (стадия pTNM). Включены образцы с отсутствием выявления SNV. Клональный статус определяли с помощью PyCloneCluster с применением данных полноэкзомного секвенирования опухолевой ткани.
[32] На ФИГ. 15 показано число SNV, выявленных в плазме крови, в зависимости от гистологического типа и размера опухоли. Гистологический тип и стадию опухоли определяли в соответствии с гистопатологическим заключением. Каждая точка на графике окрашена в соответствии с размером, при этом красный обозначает самым крупный размер опухоли, а синий обозначает самый маленький размер опухоли.
[33] ФИГ. 16 представляет собой таблицу анализа cfDNA, показывающего концентрацию ДНК, эквиваленты геномных копий в препаратах библиотеки, степень гемолиза плазмы крови и профиль cDNA для всех образцов.
[34] ФИГ. 17 представляет собой таблицу SNV, выявленных в плазме крови, для каждого образца.
[35] ФИГ. 18 представляет собой таблицу дополнительных SNV, выявленных в плазме крови.
[36] ФИГ. 19 представляет собой таблицу числа аналитов, исходя из генов в экспериментах из примера 1.
[37] ФИГ. 20 представляет собой таблицу с информацией, касающейся образцов, проанализированных в исследовании из примера 1, а также данных, полученных на основании эксперимента, представленного в примере 1.
[38] На ФИГ. 21 представлен пример выявленных аналитов и их фоновые доли аллелей у образца плазмы крови, полученного во время рецидива (LTX103).
[39] Определенные выше фигуры представлены в целях представления сведений, а не ограничения.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[40] Способы и композиции, представленные в данном документе, улучшают выявление, диагностирование, определение стадии, скрининг, лечение и контроль течения рака легкого. В иллюстративных вариантах осуществления посредством способов, представленных в данном документе, анализируют мутации, представляющие собой однонуклеотидные варианты (SNV), в циркулирующих жидкостях организма, в частности, содержащих циркулирующую опухолевую ДНК. Способы обеспечивают преимущество идентификации большего числа мутаций, которые обнаружены в опухоли, и клональных, а также субклональных мутаций в ходе одного теста, а не множественных тестов, которые потребовались бы, если бы они вообще были эффективны, в которых используются образцы опухоли. Способы и композиции могут быть полезны сами по себе, или они могут быть полезны при применении наряду с другими способами выявления, диагностирования, определения стадии, скрининга, лечения и контроля течения рака легкого, например, чтобы помочь подтвердить результаты этих других способов для обеспечения более достоверного и/или определенного результата.
[41] Соответственно, в одном варианте осуществления в данном документе представлен способ определения однонуклеотидных вариантов, присутствующих в плоскоклеточной карциноме легкого, путем определения однонуклеотидных вариантов, присутствующих в образце ctDNA от индивидуума, такого как индивидуум, имеющий плоскоклеточную карциному или с подозрением на нее, с применением рабочего протокола амплификации/секвенирования ctDNA SNV, представленного в данном документе.
[42] В другом варианте осуществления в данном документе представлен способ выявления плоскоклеточной карциномы легкого в образце крови или ее фракции от индивидуума, такого как индивидуум с подозрением на рак, который предусматривает определение однонуклеотидных вариантов, присутствующих в образце, путем определения однонуклеотидных вариантов, присутствующих в образце ctDNA, с применением рабочего протокола амплификации/секвенирования ctDNA SNV, представленного в данном документе. Присутствие в образце на нижнем пределе диапазона 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15 SNV и на верхнем пределе диапазона 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 30, 40 или 50 SNV в нескольких однонуклеотидных локусах является показателем присутствия плоскоклеточной карциномы.
[43] В другом варианте осуществления в данном документе представлен способ выявления клонального однонуклеотидного варианта в опухоли легкого у индивидуума. Способ предусматривает осуществление рабочего протокола амплификации/секвенирования ctDNA SNV, как представлено в данном документе, и определение частоты вариантного аллеля для каждого из локусов SNV, исходя из последовательности нескольких копий из серии ампликонов. Более высокая относительная частота аллеля по сравнению с другими однонуклеотидными вариантами в нескольких локусах однонуклеотидных вариантов является показателем клонального однонуклеотидного варианта в опухоли. Значения частоты вариантных аллелей хорошо известны в области секвенирования. Поддержка этого варианта осуществления представлена, например, на ФИГ. 12—14.
[44] В определенных вариантах осуществления способ дополнительно предусматривает определение плана лечения, терапевтического средства и/или введение индивидууму соединения, которое нацеливается на один или более клональных однонуклеотидных вариантов. В определенных примерах терапевтическое средство не нацеливается на субклональные и/или другие клональные SNV. Конкретные терапевтические средства и ассоциированные мутации представлены в других разделах настоящего описания и известны из уровня техники. Соответственно, в определенных примерах способ дополнительно предусматривает введение индивидууму соединения, при этом известно, что соединение является особенно эффективным в лечении плоскоклеточной карциномы легкого c одним или более определенными однонуклеотидными вариантами.
[45] В определенных аспектах этого варианта осуществления частота вариантного аллеля, превышающая 0,25%, 0,5%, 0,75%, 1,0%, 5% или 10%, является показателем клонального однонуклеотидного варианта. Эти предельные значения подтверждаются данными в табличной форме на ФИГ. 20.
[46] В определенных примерах этого варианта осуществления плоскоклеточная карцинома представляет собой плоскоклеточную карциному стадии 1a, 1b или 2a. В определенных примерах этого варианта осуществления плоскоклеточная карцинома представляет собой плоскоклеточную карциному стадии 1a или 1b. В определенных примерах варианта осуществления индивидуум не подвергается хирургическому вмешательству. В определенных примерах варианта осуществления индивидуум не подвергается биопсии.
[47] В некоторых примерах этого варианта осуществления клональный SNV идентифицируют или дополнительно идентифицируют, если другое тестирование, такое как прямое тестирование опухоли, свидетельствует, что исследуемый SNV представляет собой клональный SNV, в случае любого исследуемого SNV, который характеризуется частотой вариабельного аллеля, превышающей частоту по меньшей мере одной четверти, одной трети, половины или трех четвертей других однонуклетидных вариантов, которые были определены.
[48] В некоторых вариантах осуществления в данном документе способы выявления SNV в ctDNA можно применять вместо прямого анализа ДНК из опухоли. Результаты, представленные в данном документе, демонстрируют, что SNV, которые с намного большей вероятностью являются клональными SNV, характеризуются более высокими VAF (см., например, ФИГ. 12—14).
[49] В определенных примерах любого из вариантов осуществления способа, представленных в данном документе, перед осуществлением целевой амплификации в отношении ctDNA от индивидуума, получают данные в отношении SNV, которые обнаружены в опухоли от индивидуума. Соответственно, в этих вариантах осуществления реакцию амплификации/секвенирования SNV осуществляют в отношении одного или более образцов опухоли от индивидуума. В этих способах реакция амплификации/секвенирования ctDNA SNV, представленная в данном документе, все также является преимущественной, поскольку она обеспечивает жидкую биопсию для выявления клональных и субклональных мутаций. Кроме того, как представлено в данном документе, клональные мутации можно более однозначно идентифицировать у индивидуума с раком легкого, если для SNV определено высокое процентное значение VAF, например, больше 1, 2, 3, 4, 5, 6, 7, 8, 9, 10% VAF в образце ctDNA от индивидуума.
[50] В определенном варианте осуществления способ, представленный в данном документе, можно применять для определения того, стоит ли выделять и анализировать ctDNA из циркулирующих свободных нуклеиновых кислот от индивидуума с раком легкого. Во-первых, с помощью него определяют, является ли рак легкого аденокарциномой или плоскоклеточной карциномой. Если рак легкого представляет собой плоскоклеточную карциному, у индивидуума выделяют циркулирующие свободные нуклеиновые кислоты. В некоторых примерах способ дополнительно предусматривает определение стадии рака легкого, где, если рак легкого представляет собой плоскоклеточную карциному или аденокарциному стадии 3a, у индивидуума выделяют циркулирующие свободные нуклеиновые кислоты. Результаты, представленные на ФИГ. 15 и в табличной форме на ФИГ. 20, демонстрируют, что SNV широко распространены в плоскоклеточной карциноме или аденокарциноме стадии 3a, однако, SNV намного менее распространены в ADC более ранней стадии. Соответственно, можно достичь важной экономии расходов на медицинское обслуживание за счет избавления от тестирования в отношении SNV у пациентов с ADC стадии 1a, 1b и/или 2a.
[51] В примерах, если рак легкого представляет собой плоскоклеточную карциному или аденокарциному стадии 3a, циркулирующие нуклеиновые кислоты выделяют у индивидуума. Кроме того, в некоторых примерах, если
[52] рак легкого представляет собой стадию плоскоклеточной карциномы или аденокарциному стадии 3a нуклеиновые кислоты не выделяют из опухоли легкого индивидуума.
[53] Для некоторых способов в данном документе представлены композиции и/или твердые подложки по настоящему изобретению. F1. Композиция, содержащая фрагменты циркулирующих опухолевых нуклеиновых кислот, содержащие универсальный адаптер, где циркулирующие опухолевые нуклеиновые кислоты происходят из опухоли, представляющей собой плоскоклеточную карциному легкого.
[54] В некоторых вариантах осуществления в данном документе представлена композиция по настоящему изобретению, которая содержит фрагменты циркулирующих опухолевых нуклеиновых кислот, содержащие универсальный адаптер, где циркулирующие опухолевые нуклеиновые кислоты происходят из образца крови или ее фракции от индивидуума с плоскоклеточной карциномой легкого. Результаты, представленные в примере 1, демонстрируют неожиданное преимущество способов тестирования на основе амплификации/секвенирования ctDNA SNV. Эти способы, как правило, предусматривают образование фрагмента ctDNA, который содержит универсальный адаптер. Кроме того, такие способы, как правило, предусматривают образование твердой подложки, в частности, твердой подложки для высокопроизводительного секвенирования, которая содержит несколько клональных популяций нуклеиновых кислот, где клональные популяции содержат ампликоны, полученные из образца циркулирующих свободных нуклеиновых кислот, где ctDNA. В иллюстративных вариантах осуществления, основанных на неожиданных результатах, представленных в данном документе, ctDNA происходит из опухоли, представляющей собой плоскоклеточную карциному легкого.
[55] Аналогично, в качестве варианта осуществления настоящего изобретения в данном документе представлена твердая подложка, содержащая несколько клональных популяций нуклеиновых кислот, где клональные популяции содержат фрагменты нуклеиновой кислоты, полученные из образца циркулирующих свободных нуклеиновых кислот из образца крови или ее фракции от индивидуума с плоскоклеточной карциномой легкого.
[56] В определенных вариантах осуществления фрагменты нуклеиновой кислоты в различных клональных популяциях содержат один и тот же универсальный адаптер. Такая композиция, как правило, образуется во время реакции высокопроизводительного секвенирования в способах по настоящему изобретению, осуществляемых в примере 1.
[57] Клональные популяции нуклеиновых кислот могут быть получены из фрагментов нуклеиновой кислоты, полученных из набора образцов от двух или более индивидуумов. В этих вариантах осуществления фрагменты нуклеиновой кислоты содержат один из серии молекулярных штрихкодов, соответствующих образцу в наборе образцов.
[58] Подробные аналитические способы представлены в данном документе как SNV-способ 1 и SNV-способ 2 в аналитическом разделе в данном документе. Любой из способов, представленных в данном документе, может дополнительно включать аналитические стадии, представленные в данном документе. Соответственно, в определенных примерах способы определения того, присутствует ли в образце однонуклеотидный вариант, предусматривают идентификацию значения достоверности для каждого определения аллеля в каждом из наборов локусов однонуклеотидной изменчивости, которое может быть основано по меньшей мере частично на глубине прочтения локуса. Предел достоверности может быть установлен на уровне по меньшей мере 75%, 80%, 85%, 90%, 95%, 96%, 96%, 98% или 99%. Предел достоверности может быть установлен на различных уровнях для различных типов мутаций.
[59] Способ можно осуществлять при глубине прочтения для набора локусов однонуклеотидной изменчивости, составляющей по меньшей мере 5, 10, 15, 20, 25, 50, 100, 150, 200, 250, 500, 1000, 10000, 25000, 50000, 100000, 250000, 500000 или 1 миллион. На ФИГ. 20 представлены данные о глубине прочтения для локусов SNV, успешно проанализированных в примере 1.
[60] В определенных вариантах осуществления способ по любому из вариантов осуществления в данном документе предусматривает определение эффективности и/или частоты ошибок на цикл, которые определяют для каждой реакции амплификации из мультиплексной реакции амплификации локуса однонуклеотидной изменчивости. Затем эффективность и частоту ошибок можно применять для определения того, присутствует ли в образце однонуклеотидный вариант из набора локусов однонуклеотидных вариантов. В некоторых вариантах осуществления также могут быть включены более подробно описанные аналитические стадии, представленные в SNV-способе 2, представленном в аналитическом способе.
[61] В иллюстративных вариантах осуществления любого из способов в данном документе набор локусов однонуклеотидной изменчивости включает все локусы однонуклеотидной изменчивости, идентифицированные в наборах данных TCGA и COSMIC для рака легкого, или для аденокарциномы легкого, и/или, в частности, для плоскоклеточной карциномы легкого.
[62] В определенных вариантах осуществления любого из способов в данном документе набор локусов однонуклеотидных вариантов включает на нижнем пределе диапазона 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 40, 50, 75, 100, 250, 500, 1000, 2500, 5000 или 10000 локусов однонуклеотидной изменчивости, которые, как известно, ассоциированы с раком легкого, ADC легкого и/или, в частности, SCC легкого, и на верхнем пределе диапазона 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 40, 50, 75, 100, 250, 500, 1000, 2500, 5000, 10000, 20000 и 25000 локусов.
[63] В любом из способов выявления SNV в данном документе, который включает рабочий протокол амплификации/секвенирования ctDNA SNV, для мультиплексной ПЦР можно использовать улучшенные параметры амплификации. Например, где реакция амплификации представляет собой ПЦР-реакцию, и температура отжига превышает температуру плавления по меньшей мере 10, 20, 25, 30, 40, 50, 06, 70, 75, 80, 90, 95 или 100% праймеров из набора праймеров на нижнем пределе диапазона на 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10°C и на верхнем пределе диапазона на 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14 или 15°.
[64] В определенных вариантах осуществления, где реакция амплификации представляет собой ПЦР-реакцию, продолжительность стадии отжига в ПЦР-реакции составляет от 10, 15, 20, 30, 45 и 60 минут на нижнем пределе диапазона до 15, 20, 30, 45, 60, 120, 180 или 240 минут на верхнем пределе диапазона. В определенных вариантах осуществления концентрация праймеров при амплификации, такой как ПЦР-реакция, составляет 1—10 нМ. Кроме того, в приводимых в качестве примера вариантах осуществления праймеры в наборе праймеров разработаны для сведения к минимуму образования димеров из праймеров.
[65] Соответственно, в примере любого из способов в данном документе, который включает стадию амплификации, реакция амплификации представляет собой ПЦР-реакцию, температура отжига на 1—10°C превышает температуру плавления по меньшей мере 90% праймеров из набора праймеров, продолжительность стадии отжига в ПЦР-реакции составляет 15—60 минут, концентрация праймеров в реакции амплификации составляет 1—10 нМ, и праймеры в наборе праймеров разработаны для сведения к минимуму образования димеров из праймеров. В дополнительном аспекте этого примера мультиплексную реакцию амплификации осуществляют в условиях ограничения праймеров.
[66] В другом варианте осуществления в данном документе представлен способ подтверждения диагноза рака легкого у индивидуума, такого как индивидуум с подозрением на рак легкого, на основании образца крови или ее фракции от индивидуума, который предусматривает осуществление рабочего протокола амплификации/секвенирования ctDNA SNV, как представлено в данном документе, для определения того, присутствует ли один или более однонуклеотидных вариантов в нескольких локусах однонуклеотидных вариантов. В этом варианте осуществления применимы следующие элементы, утверждения, рекомендации или правила:
[67] отсутствие однонуклеотидного варианта подтверждает диагноз аденокарциномы стадии 1a, 1b или 2a,
[68] присутствие однонуклеотидного варианта подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 2b или 3a, и/или
[69] присутствие десяти или более однонуклеотидных вариантов подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 2b или 3.
[70] Вышеуказанные элементы, утверждения, рекомендации или правила подтверждаются результатами из примера 1 (см., например, табличные данные на ФИГ. 20). Эти результаты идентифицируют анализ с применением рабочего протокола амплификации/секвенирования ctDNA SNV из образцов ADC и SCC легкого от индивидуума в качестве ценного способа идентификации SNV, обнаруженных в опухоли ADC, в частности, в опухолях ADC стадии 2b и 3a, и, в частности, опухоли SCC на любой стадии (см., например, ФИГ. 15 и ФИГ. 20).
[71] В определенных примерах этот вариант осуществления дополнительно включает определение стадии патологического изменения при раке легкого с помощью неинвазивного способа, например, размер опухоли можно определять с помощью неинвазивных способов.
[72] В определенных вариантах осуществления способы выявления SNV в данном документе можно применять для назначения схемы лечения. Существуют доступные и находящие в разработке терапевтические средства, которые нацеливаются на специфические мутации, ассоциированные с ADC и SCC (Nature Review Cancer. 14:535—551 (2014). Например, выявление мутации EGFR в L858R или T790M может быть информативным для выбора терапевтического средства. Эрлотиниб, гефитиниб, афатиниб, AZK9291, CO-1686 и HM61713 представляют собой современные терапевтические средства, которые одобрены в США или применяются в клинических исследованиях, которые нацеливаются на специфические мутации EGFR. В другом примере мутацию G12D, G12C или G12V в KRAS можно использовать для назначения индивидууму терапевтического средства из комбинации селуметиниба плюс доцетаксела. В качестве другого примера мутацию V600E в BRAF можно использовать для назначения субъекту лечения с помощью вемурафениба, дабрафениба и траметиниба.
[73] В определенных иллюстративных вариантах осуществления образец, анализируемый в способах по настоящему изобретению, представляет собой образец крови или ее фракции. В определенных вариантах осуществления способы, представленные в данном документе, специально адаптированы для амплификации фрагментов ДНК, в частности, фрагментов опухолевой ДНК, которые обнаружены в циркулирующей опухолевой ДНК (ctDNA). Длина такие фрагментов, как правило, составляет приблизительно 160 нуклеотидов.
[74] Из уровня техники известно, что внеклеточная нуклеиновая кислота (cfNA), например cfDNA, может высвобождаться в кровоток за счет различных форм клеточной гибели, таких как апоптоз, некроз, аутофагия и некроптоз. cfDNA является фрагментированной и распределение фрагментов по размеру варьирует от 150—350 п. о. до > 10000 п. о. (см. Kalnina et al. World J Gastroenterol. 2015 Nov 7; 21(41): 11636—11653). Например, распределения фрагментов ДНК плазмы по размеру у пациентов с гепатоклеточной карциномой (HCC) охватывали диапазон 100—220 п. о. в длину, при этом пик по частоте подсчета составлял приблизительно 166 п. о., а самая высокая концентрация опухолевой ДНК была у фрагментов длиной 150—180 п. о. (см.: Jiang et al. Proc Natl Acad Sci USA 112:E1317–E1325).
[75] В иллюстративном варианте осуществления циркулирующую опухолевую ДНК (ctDNA) выделяют из крови с применением пробирки с EDTA-2Na после удаления клеточного дебриса и тромбоцитов с помощью центрифугирования. Образцы плазмы можно хранить при -80°C до выделения ДНК с применением, например, набора QIAamp DNA Mini (Qiagen, Хильден, Германия), (например, Hamakawa et al., Br J Cancer. 2015; 112:352—356). Hamakava et al. сообщали, что средняя концентрация внеклеточной ДНК, выделенной из всех образцов, составляет 43,1 нг на мл плазмы (диапазон 9,5—1338 нг/мл), а диапазон мутантной фракции составляет 0,001—77,8% со средним значением 0,90%.
[76] В некоторых иллюстративных вариантах осуществления образцом является опухоль. Из уровня техники известны способы выделения нуклеиновой кислоты из опухоли и способы создания библиотеки нуклеиновых кислот на основании такого образца ДНК, принимая во внимание изложенные в данном документе идеи. Кроме того, принимая во внимание изложенные в данном документе идеи, специалист в данной области техники поймет, как создать библиотеку нуклеиновых кислот, подходящую для способов, изложенных в данном документе, из других образцов, таких как образцы других биологических жидкостей, в которых свободно плавает ДНК, в дополнение к образцам ctDNA.
[77] В определенных вариантах осуществления способы по настоящему изобретению, как правило, включают стадию получения и амплификации библиотеки нуклеиновых кислот из образца (т. е. построения библиотеки). Во время стадии построения библиотеки нуклеиновые кислоты из образца могут иметь адаптеры для лигирования, зачастую упоминаемые как метки библиотеки или адапторные метки для лигирования (LT), прикрепляемые, если адаптеры для лигирования содержат универсальную праймерную последовательность, после универсальной амплификации. В варианте осуществления это можно выполнять с применением стандартного протокола, разработанного для создания библиотек секвенирования после фрагментации. В варианте осуществления образец ДНК может иметь тупой конец, и затем на 3′-конец можно добавлять A. Можно добавлять или лигировать Y-адаптор с T-выступающим концом. В некоторых вариантах осуществления могут использоваться другие липкие концы, отличные от A или T-выступающего конца. В некоторых вариантах осуществления можно добавлять другие адапторы, например, адапторы для лигирования, содержащие петлю. В некоторых вариантах осуществления адапторы могут иметь метку, разработанную для ПЦР-амплификации.
[78] Ряд вариантов осуществления, представленных в данном документе, включает выявление SNV в образце ctDNA. В иллюстративных вариантах осуществления такие способы включают стадию амплификации и стадию секвенирования (иногда упоминаемую в данном документе как «рабочий протокол амплификации/секвенирования ctDNA SNV»). В иллюстративном примере рабочий протокол амплификации/секвенирования ctDNA может включать получение набора ампликонов путем осуществления мультиплексной реакции амплификации в отношении нуклеиновых кислот, выделенных из образца крови или ее фракции от индивидуума, такого как индивидуум с подозрением на рак легкого, например, плоскоклеточную карциному, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, таких как локус SNV, как известно, ассоциированный с раком легкого; и
[79] определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов, где сегмент содержит локус однонуклеотидного варианта. Таким образом, в этом приводимом в качестве примера способе определяют однонуклеотидные варианты, присутствующие в образце.
[80] Более подробно, приводимый в качестве примера рабочий протокол амплификации/секвенирования ctDNA SNV может включать образование реакционной смеси для амплификации путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученной на основании образца, и набора праймеров, каждый из которых связывается на эффективном расстоянии от локуса однонуклеотидного варианта, или набора пар праймеров, каждый из которых перекрывает эффективный участок, который содержит локус однонуклеотидного варианта. В приводимых в качестве примера вариантах осуществления локус однонуклеотидного варианта представляет собой локус, который, как известно, ассоциирован с раком легкого, например, аденокарциномой легкого и/или, в особенно иллюстративных вариантах осуществления, плоскоклеточной карциномой. Затем предусмотрено воздействие на реакционную смесь для амплификации c помощью условий амплификации для получения набора ампликонов, содержащих по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, предпочтительно, как известно, ассоциированных с раком легкого; и
[81] определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов, где сегмент содержит локус однонуклеотидного варианта.
[82] Эффективное расстояние связывания праймеров может располагаться в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 20, 25, 30, 35, 40, 45, 50, 75, 100, 125 или 150 пар оснований от локуса SNV. Эффективный диапазон, который перекрывает пара праймеров, как правило, включает SNV и составляет, как правило, 160 пар оснований или меньше, и может составлять 150, 140, 130, 125, 100, 75, 50 или 25 пар оснований или меньше. В других вариантах осуществления эффективный диапазон, который перекрывает пара праймеров, составляет на нижнем пределе диапазона 20, 25, 30, 40, 50, 60, 70, 75, 100, 110, 120, 125, 130, 140 или 150 нуклеотидов от локуса SNV и на верхнем пределе диапазона 25, 30, 40, 50, 60, 70, 75, 100, 110, 120, 125, 130, 140 или 150, 160, 170, 175 или 200 нуклеотидов.
[83] Дополнительные подробности, касающиеся способов амплификации, которые можно использовать в рабочем протоколе амплификации/секвенирования ctDNA SNV для выявления SNV для применения в способах по настоящему изобретению, представлены в других разделах настоящего описания.
[84] Аналитические методы распознавания SNV
[85] Во время осуществления способов, представленных в данном документе, для ампликонов, созданных с помощью мозаичной мультиплексной ПЦР получают данные секвенирования. Существуют доступные инструменты разработки алгоритмов, которые можно использовать и/или адаптировать для анализа этих данных, чтобы определить с определенными пределами достоверности, присутствует ли мутация, такая как SNV, в целевом гене, как проиллюстрировано в примере 1 в данном документе.
[86] Риды секвенирования можно демультиплексировать с применением внутрилабораторного инструмента и картировать с применением программного обеспечения для выравнивания на основе алгоритма Барроуза-Уилера, функции Bwa-MEM (BWA, программное обеспечение для выравнивания на основе алгоритма Барроуза-Уилера (см. Li H. and Durbin R. (2010) Fast and accurate long-read alignment with Burrows-Wheeler Transform. Bioinformatics, Epub. [PMID: 20080505]) в режиме одного конца с применением ридов, ограниченных с помощью PEAR, на геноме hg19. Статистику QC для амплификации можно осуществлять путем анализа общего числа ридов, числа картированных ридов, числа картированных ридов на мишени и числа подсчитанных ридов.
[87] В определенных вариантах осуществления вместе со способами по настоящему изобретению можно применять любой аналитический способ выявления SNV на основании данных секвенирования нуклеиновой кислоты, который включает стадию выявления SNV или определения того, присутствует ли SNV. В определенных иллюстративных вариантах осуществления применяют способы по настоящему изобретению, в которых используется SNV-СПОСОБ 1, приведенный ниже. В других, даже более иллюстративных вариантах осуществления, в способах по настоящему изобретению, которые включают стадию выявления SNV или определения того, присутствует ли SNV в локусе SNV, используется SNV-СПОСОБ 2, приведенный ниже.
[88] SNV-СПОСОБ 1. Для этого варианта осуществления конструируют модель фоновой ошибки с применением образцов нормальной плазмы крови, которые секвенировали в том же прогоне секвенирования, для внесения поправки на специфические для прогона артефакты. В определенных вариантах осуществления в том же прогоне секвенирования анализируют 5, 10, 15, 20, 25, 30, 40, 50, 100, 150, 200, 250 или более 250 образцов нормальной плазмы крови. В определенных иллюстративных вариантах осуществления в том же прогоне секвенирования анализируют 20, 25, 40 или 50 образцов нормальной плазмы крови. Удаляют положения шумов с нормальной средней частотой вариантного аллеля, превышающей предельное значение. Например, в определенных вариантах осуществления это предельное значение составляет > 0,1%, 0,2%, 0,25%, 0,5%, 1%, 2%, 5% или 10%. В определенных иллюстративных вариантах осуществления удаляют положения шумов с нормальной средней частотой вариантного аллеля, превышающей 0,5%. Образцы с выпадающими значениями итерационно удаляли из модели для внесения поправки на шум и загрязнение. В определенных вариантах осуществления из анализа данных удаляли образцы с Z-баллом, превышающим 5, 6, 7, 8, 9 или 10. Для каждой замены основания в каждом геномном локусе рассчитывали взвешенное среднее значение глубины прочтения и стандартное отклонение ошибки. Положения образцов опухоли или плазмы крови с внеклеточной ДНК по меньшей мере с 5 вариантами ридов и Z-баллом, составляющим 10, в сопоставлении с моделью фоновой ошибки, например, можно распознавать как кандидатную мутацию.
[89] SNV-СПОСОБ 2. В случае этого варианта осуществления однонуклеотидные варианты (SNV) определяют с применением данных ctDNA плазмы крови. Процесс ПЦР моделируют как стохастический процесс, оценивая параметры с применением обучающего набора и делая конечные распознавания SNV для отдельного тестируемого набора. Определяют распространение ошибки по всем множественным циклам ПЦР и рассчитывают среднее значение и дисперсию фоновой ошибки, и в иллюстративных вариантах осуществления фоновую ошибку разграничивают от настоящих мутаций.
[90] Для каждого основания оценивают следующие параметры:
[91] p = эффективность (вероятность того, что каждый рид реплицируется в каждом цикле)
[92] pe = частота ошибок на цикл для мутации типа e (вероятность того, что происходит ошибка типа e)
[93] X0 = начальное число молекул
[94] По мере того, как рид реплицируется в ходе процесса ПЦР, происходит все больше ошибок. Следовательно, профиль ошибок ридов определяется степенями расхождения с оригинальным ридом. Авторы настоящего изобретения обозначают рид как относящийся к k-му поколению, если он прошел через k репликаций, прежде чем он был получен.
[95] Авторы настоящего изобретения определяют следующие переменные для каждого основания:
[96] Xij = число ридов поколения i, полученных в цикле j ПЦР
[97] Yij = общее число ридов поколения i в конце цикла j
[98] Xije = число ридов поколения i с мутацией e, полученной в цикле j ПЦР
[99] Более того, в дополнение к нормальным молекулам X0, если в начале процесса ПЦР имеются дополнительные молекулы feX0 с мутацией e (следовательно fe/(1+fe) будет долей подвергнутых мутации молекул в начальной смеси).
[100] С учетом общего числа ридов поколения i-1 в цикле j-1, число ридов поколения i, полученных в цикле j, имеет биномиальное распределение с размером выборки Yi-1,j-1 и параметром вероятности p. Следовательно, E(Xij, |Yi-1,j-1, p) = p Yi-1,j-1 и Var(Xij, |Yi-1,j-1, p)= p(1-p) Yi-1,j-1.
[101] Авторы настоящего изобретения также имеют 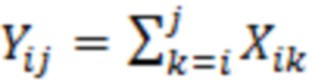 . Следовательно, с помощью рекурсии, симуляции или аналогичных способов авторы настоящего изобретения могут определить E(Xij,). Аналогично, авторы настоящего изобретения могут определить Var(Xij) = E(Var(Xij, | p)) + Var(E(Xij, | p)) с применением распределения p.
. Следовательно, с помощью рекурсии, симуляции или аналогичных способов авторы настоящего изобретения могут определить E(Xij,). Аналогично, авторы настоящего изобретения могут определить Var(Xij) = E(Var(Xij, | p)) + Var(E(Xij, | p)) с применением распределения p.
[102] Наконец, E(Xije|Yi-1,j-1, pe) = peYi-1,j-1 и Var(Xije|Yi-1,j-1, p)= pe(1- pe) Yi-1,j-1, и авторы настоящего изобретения могут применять их для вычисления E(Xije) и Var(Xije).
[103] В определенных вариантах осуществления SNV-способ 2 осуществляют следующим образом:
[104] a) оценить эффективность ПЦР и частоту ошибок на цикл с применением обучающего набора данных;
[105] b) оценить число исходных молекул для набора экспериментальных данных по каждому основанию с применением распределения эффективности, оцененной на стадии (a);
[106] c) при необходимости, обновить оценку эффективности для набора экспериментальных данных с применением исходного числа молекул, оцененного на стадии (b);
[107] d) оценить среднее значение и дисперсию для общего числа молекул, молекул фоновой ошибки и молекул с реальными мутациями (для области поиска, состоящей из начального процентного значения молекул с реальными мутациями) с применением данных тестируемого набора и параметров, оцененных на стадиях (a), (b) и (c);
[108] e) согласовать распределение с общим числом молекул с ошибкой (фоновая ошибка и реальная мутация) у общего числа молекул, и рассчитать правдоподобие для процентного значения каждой реальной мутации в области поиска; и
[109] f) определить процентное значение наиболее вероятной реальной мутации и рассчитать достоверность с применением данных из стадии (e).
[110] Предельное значение достоверности можно применять для идентификации SNV в локусе SNV. Например, для распознавания SNV можно применять предельное значение достоверности 90%, 95%, 96%, 97%, 98% или 99%.
[111] Алгоритм приводимого в качестве примера SNV-СПОСОБА 2
[112] Алгоритм начинается путем оценки эффективности и частоты ошибок на цикл с применением обучающего набора. Пусть n обозначает общее число циклов ПЦР.
[113] Число ридов Rb по каждому основанию b можно приближенно описать (1+pb) n X0, где pb представляет собой эффективность по основанию b. Затем (Rb/ X0)1/n можно применять для приближенного расчета 1+pb. Затем мы можем определить среднее и стандартное отклонение pb для всех обучающих образцов, чтобы оценить параметры распределения вероятности (такое как нормальное распределение, бета-распределение или аналогичные распределения) для каждого основания.
[114] Аналогично, число ридов с ошибкой e, Rbe, по каждому основанию b можно применять для оценки 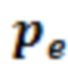 . После определения среднего и стандартного отклонения частоты ошибок для всех обучающих образцов, авторы настоящего изобретения приближенно рассчитали ее распределение вероятности (такое как нормальное распределение, бета-распределение или аналогичные распределения), параметры которого оценивают с применением значений данного среднего и стандартного отклонения.
. После определения среднего и стандартного отклонения частоты ошибок для всех обучающих образцов, авторы настоящего изобретения приближенно рассчитали ее распределение вероятности (такое как нормальное распределение, бета-распределение или аналогичные распределения), параметры которого оценивают с применением значений данного среднего и стандартного отклонения.
[115] После этого, для экспериментальных данных авторы настоящего изобретения оценивают первоначальную исходную копию по каждому основанию как 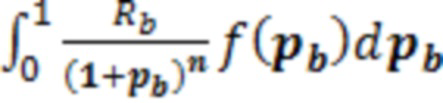 , где f(.) представляет собой распределение, оцененное на основе обучающего набора.
, где f(.) представляет собой распределение, оцененное на основе обучающего набора.
[116]
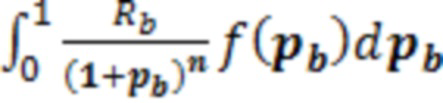 , где f(.) представляет собой распределение, оцененное на основании обучающего набора.
, где f(.) представляет собой распределение, оцененное на основании обучающего набора.
[117] Следовательно, авторы настоящего изобретения оценили параметры, которые будут использоваться в стохастическом процессе. Затем, с применением этих оценок авторы настоящего изобретения могут оценить среднее значение и дисперсию молекул, созданных в ходе каждого цикла (следует отметить, что авторы настоящего изобретения делают это отдельно для нормальных молекул, молекул с ошибками и молекул с мутациями).
[118] Наконец, путем применения вероятностного способа (такого как способ максимального правдоподобия или аналогичные способы), авторы настоящего изобретения могут определить наилучшее значение fe, которое согласуется с распределением молекул с ошибкой, молекул с мутацией и нормальных молекул наилучшим образом. Более конкретно, авторы настоящего изобретения оценивают ожидаемое соотношение молекул с ошибкой и общего числа молекул для различных значений fe в конечных ридах, и определяют правдоподобие их данных в отношении каждого из этих значений, и затем выбирают значение с самым высоким правдоподобием.
[119] Праймерные «хвосты» могут улучшать выявление фрагментированной ДНК из библиотек с универсальными метками. Гибридизация может быть улучшена (например, снижена температура плавления (Tm)), если метка библиотеки и праймерные «хвосты» содержат гомологичную последовательность, и праймеры могут элонгироваться, только если часть целевой последовательности праймера находится в фрагменте ДНК образца. В некоторых вариантах осуществления можно использовать 13 или более пар оснований, специфических в отношении мишени. В некоторых вариантах осуществления можно использовать 10—12 пар оснований, специфических в отношении мишени. В некоторых вариантах осуществления можно использовать 8—9 пар оснований, специфических в отношении мишени. В некоторых вариантах осуществления можно использовать 6—7 пар оснований, специфических в отношении мишени.
[120] В одном варианте осуществления библиотеки получают на основе образцов, описанных выше, путем лигирования адапторов к концам фрагментов ДНК в образцах или к концам фрагментов ДНК, полученных из ДНК, выделенной из образцов. Затем фрагменты можно амплифицировать с применением ПЦР, например, согласно следующему, приводимому в качестве примера протоколу:
[121] 95°C, 2 мин.; 15 x [95°C, 20 сек., 55°C, 20 сек., 68°C, 20 сек.], 68°C 2 мин., 4°C удержание.
[122] Из уровня техники известно множество наборов и способов получения библиотек нуклеиновых кислот, которые включают сайты связывания универсального праймера для последующей амплификации, например клональной амплификации, и для последующего секвенирования. Чтобы содействовать облегчению лигирования адаптеров, построение и амплификация библиотеки могут включать репарацию концов и аденилирование (т. е. добавление A-хвоста). Наборы, в частности адаптированные для построения библиотек из небольших фрагментов нуклеиновой кислоты, в частности циркулирующей свободной ДНК, могут быть пригодны для практического осуществления способов, представленных в данном документе. Например, наборы NEXTflex Cell Free, которые доступны от Bioo Scientific (), или набор Natera Library Prep Kit (доступен от Natera, Inc. Сан-Карлос, Калифорния). Однако, такие наборы, как правило, будут модифицировать для включения адапторов, которые приспособлены для стадий амплификации и секвенирования по способам, представленным в данном документе. Лигирование адапторов можно осуществлять с применением коммерчески доступных наборов, таких как набор для лигирования, который находится в наборе AGILENT SURESELECT (Agilent, Калифорния).
[123] В случае способов по настоящему изобретению целевые участки из библиотеки нуклеиновых кислот, полученной на основе ДНК, выделенной из образца, в частности, образца циркулирующей свободной ДНК, затем амплифицируют. Для этой амплификации используют серию праймеров или пар праймеров, которые могут включать от 5, 10, 15, 20, 25, 50, 100, 125, 150, 250, 500, 1000, 2500, 5000, 10000, 20000, 25000 или 50000 на нижнем пределе диапазона до 15, 20, 25, 50, 100, 125, 150, 250, 500, 1000, 2500, 5000, 10000, 20000, 25000, 50000, 60000, 75000 или 100000 праймеров на верхнем пределе диапазона, каждый из которых связывается с одним из серии сайтов связывания праймера.
[124] Дизайны праймеров можно создавать с помощью Primer3 (Untergrasser A, Cutcutache I, Koressaar T, Ye J, Faircloth BC, Remm M, Rozen SG (2012) «Primer3 - new capabilities and interfaces.» Nucleic Acids Research 40(15):e115 и Koressaar T, Remm M (2007) «Enhancements and modifications of primer design program Primer3.» Bioinformatics 23(10):1289—91) код источника доступен на primer3.sourceforge.net). Специфичность праймеров можно оценивать с помощью BLAST и добавлять к существующим критериям схемы процесса дизайна праймеров.
[125] Специфичность праймеров можно определять с применением программы BLASTn из пакета ncbi-blast-2.2.29+. Параметр задачи «blastn-short» можно применять для картирования праймеров относительно генома человека hg19. Дизайны праймеров можно определять как «специфические», если праймер имеет менее 100 совпадений в геноме, и главное совпадение представляет собой участок связывания праймера, комплементарного мишени, в геноме, и он по меньшей мере на два балла превышает другие совпадения (балл определяют с помощью программы BLASTn). Это может быть сделано, чтобы обеспечить уникальное совпадение в геноме и не иметь множества других совпадений на всем протяжении генома.
[126] Конечные отобранные праймеры можно визуализировать в IGV (James T. Robinson, Helga Thorvaldsdуttir, Wendy Winckler, Mitchell Guttman, Eric S. Lander, Gad Getz, Jill P. Mesirov. Integrative Genomics Viewer. Nature Biotechnology 29, 24—26 (2011)) и программе просмотра UCSC (Kent WJ, Sugnet CW, Furey TS, Roskin KM, Pringle TH, Zahler AM, Haussler D. The human genome browser at UCSC. Genome Res. 2002 Jun;12(6):996—1006) с применением файлов в формате BED и карт покрытия для подтверждения.
[127] В определенных вариантах осуществления способы по настоящему изобретению предусматривают образование реакционной смеси для амплификации. Реакционная смесь, как правило, образуется путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученной на основе образца, набора прямых и обратных праймеров, специфических в отношении целевых участков, которые содержат SNV. Реакционные смеси, представленные в данном документе, сами по себе образуют иллюстративные варианты осуществления, отдельный аспект настоящего изобретения.
[128] Реакционная смесь для амплификации, пригодная в настоящем изобретении, содержит компоненты, известные в области амплификации нуклеиновых кислот, в частности ПЦР-амплификации. Например, реакционная смесь, как правило, содержит нуклеотидтрифосфаты, полимеразу и магний. Полимеразы, которые применимы в настоящем изобретении, могут включать любую полимеразу, которая может применяться в реакции амплификации, в частности, полимеразы, которые пригодны в ПЦР-реакциях. В определенных вариантах осуществления особенно применимы Taq-полимеразы с горячим стартом. Реакционные смеси для амплификации, пригодные в практическом осуществлении способов, представленных в данном документе, такие как мастер-микс AmpliTaq Gold (Life Technologies, Карлсбад, Калифорния), являются коммерчески доступными.
[129] Условия амплификации (например, термоциклирование) для ПЦР хорошо известны из уровня техники. Способы, представленные в данном документе, могут предусматривать любые условия ПЦР-циклирования, которые приводят в результате к амплификации целевых нуклеиновых кислот, таких как целевые нуклеиновые кислоты из библиотеки. Неограничивающие приводимые в качестве примера условия циклирования представлены в разделе примеров в данном документе.
[130] Существует множество рабочих протоколов, которые возможны при проведении ПЦР; некоторые рабочие протоколы, типичные для способов, раскрытых в данном документе, представлены в данном документе. Стадии, кратко изложенные в данном документе, не означают исключение других возможных стадий, и также не подразумевается, что любая из стадий, описанных в данном документе, требуется для того, чтобы способ выполнялся надлежащим образом. Из литературы известно огромное число вариаций параметров или других модификаций, и их можно выполнять без воздействия на сущность настоящего изобретения.
[131] В определенных вариантах осуществления способа, представленного в данном документе, определяют по меньшей мере часть, а в иллюстративный примерах, целую последовательность ампликона, такого как целевой ампликон внешнего праймера. Способы определения последовательности ампликона известны из уровня техники. Для такого определения последовательности можно применять любой из способов секвенирования, известных из уровня техники, например, секвенирование по Сэнгеру. В иллюстративный вариантах осуществления методики высокопроизводительного секвенирования нового поколения (также обозначаемые в данном документе как методики широкомасштабного параллельного секвенирования), такие как без ограничений используемые в MYSEQ (ILLUMINA), HISEQ (ILLUMINA), ION TORRENT (LIFE TECHNOLOGIES), GENOME ANALYZER ILX (ILLUMINA), GS FLEX+ (ROCHE 454), можно применять для секвенирования ампликонов, полученных с помощью способов, представленных в данном документе.
[132] Высокопроизводительные генетические секвенаторы допускают применение штрихкодирования (т. е. мечения образца отличительными последовательностями нуклеиновой кислоты), чтобы идентифицировать конкретные образцы от индивидуумов, за счет чего обеспечивая возможность одновременного анализа множества образцов за один прогон секвенатора ДНК. Число раз, которое секвенируется данный участок генома в препарате библиотеки (или другом представляющем интерес препарате нуклеиновой кислоты) (число ридов), будет пропорционально числу копий такой последовательности в представляющем интерес геноме (или уровню экспрессии в случае препаратов, содержащих cDNA). При таких количественных определениях можно учитывать систематические ошибки в эффективности амплификации.
[133] Целевые гены
[134] В приводимых в качестве примера вариантах осуществления целевые гены по настоящему изобретению представляют собой гены, связанные с раком, и во многих иллюстративных вариантах осуществления - гены, связанные с раком легкого. Ген, связанный с раком (например, ген, связанный с раком легкого, или ген, связанный с SCC легкого, или ген, связанный с ADC легкого), обозначает ген, ассоциированный с измененным риском развития рака (например, рака легкого, или SCC легкого, или ADC легкого соответственно) или измененным прогнозом в отношении рака. Приводимые в качестве примера гены, связанные с раком, которые содействуют развитию рака, включают онкогены; гены, которые усиливают пролиферацию клеток, инвазию или метастазирование; гены, которые подавляют апоптоз, и проангиогенные гены. Гены, связанные с раком, которые подавляют развитие рака включают без ограничений гены-супрессоры опухоли; гены, которые подавляют пролиферацию клеток, инвазию или метастазирование; гены, которые содействуют апоптозу, и антиангиогенные гены.
[135] В варианте осуществления способ выявления мутации начинается выбором участка гена, который становится мишенью. Участок с известными мутациями применяют для разработки праймеров для mPCR-NGS, служащей для амплификации и выявления мутации.
[136] Способы, представленные в данном документе, можно применять для выявления фактически любого типа мутации, в частности мутаций, которые, как известно, ассоциированы с раком, и, наиболее конкретно, способы, представленные в данном документе, направлены на мутации, в частности SNV, ассоциированные с раком легкого, в частности, аденокарциномой и плоскоклеточной карциномой. Приводимые в качестве примера SNV могут находиться в одном или более из следующих генов: EGFR, FGFR1, FGFR2, ALK, MET, ROS1, NTRK1, RET, HER2, DDR2, PDGFRA, KRAS, NF1, BRAF, PIK3CA, MEK1, NOTCH1, MLL2, EZH2, TET2, DNMT3A, SOX2, MYC, KEAP1, CDKN2A, NRG1, TP53, LKB1 и PTEN, которые были идентифицированы в образцах различных типов рака легкого, как подвергнутые мутации, характеризующиеся увеличенным числом копий или слитые с другими генами и их комбинациями (Non-small-cell lung cancers: a heterogeneous set of diseases. Chen et al. Nat. Rev. Cancer. 2014 Aug 14(8):535—551). В другом примере в перечень генов входят перечисленные выше гены, для которых сообщалось наличие SNV, такие как в процитированном документе Chen et al. В другом варианте осуществления SNV могут включать SNV, обнаруженные в одном из генов, находящихся в таблице 19 в данном документе. SNV в генах, перечисленных в таблице 19, анализировали в эксперименте из примера 1. SNV в этих генах выявляли в образцах опухоли, соответствующих образцам ctDNA из примера 1. В некоторых вариантах осуществления SNV, которые анализируют в способах, представленных в данном документе, могут включать любой из генов, перечисленных в данном абзаце выше, или любой из генов в таблице 19, которые не были перечислены выше. В данном документе представлены способы, в которых используется специфическое определение конкретного SNV в конкретном гене для управления терапией с применением лекарственных средств нацеленного действия.
[137] Реакционные смеси для амплификации (например, ПЦР)
[138] В определенных вариантах осуществления способы по настоящему изобретению предусматривают образование реакционной смеси для амплификации. Реакционная смесь, как правило, образуется путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученных на основе образца, серии прямых мишень-специфических внешних праймеров и обратного внешнего универсального праймера для синтеза первой нити. Другой иллюстративный вариант осуществления представляет собой реакционную смесь, которая содержит прямые мишень-специфические внутренние праймеры вместо прямых мишень-специфических внешних праймеров и ампликоны из первой ПЦР-реакции с применением внешних праймеров вместо фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот. Реакционные смеси, представленные в данном документе, сами по себе образуют иллюстративные варианты осуществления, отдельный аспект настоящего изобретения. В иллюстративных вариантах осуществления реакционные смеси представляют собой смеси для ПЦР-реакции. Смеси для ПЦР-реакции, как правило, включают магний.
[139] В некоторых вариантах осуществления реакционная смесь содержит этилендиаминтетрауксусную кислоту (EDTA), магний, хлорид тетраметиламмония (TMAC) или любую их комбинацию. В некоторых вариантах осуществления концентрация TMAC составляет 20—70 мМ включительно. Без ограничения какой-либо конкретной теорией, полагают, что TMAC связывается с ДНК, стабилизирует дуплексы, повышает специфичность праймеров и/или выравнивает значения температуры плавления различных праймеров. В некоторых вариантах осуществления TMAC повышает однородность количества амплифицированных продуктов для различных мишеней. В некоторых вариантах осуществления концентрация магния (такого как магний из хлорида магния) составляет 1—8 мМ.
[140] Большое число праймеров, применяемых для мультиплексной ПЦР с большим числом мишеней, может образовывать хелатный комплекс с большим количеством магния (2 фосфата в праймерах образуют хелатный комплекс с 1 атомом магния). Например, когда используется достаточное количество праймеров, так что концентрация фосфата из праймеров составляет ~9 мМ, тогда праймеры могут снижать эффективную концентрацию магния на ~4,5 мМ. В некоторых вариантах осуществления EDTA применяют для снижения количества магния, доступного в качестве кофактора для полимеразы, поскольку высокие концентрации магния могут приводить к ошибкам в ПЦР, таким как амплификация нецелевого локуса. В некоторых вариантах осуществления определенная концентрация EDTA снижает количество доступного магния до 1—5 мМ (как, например, до 3—5 мМ).
[141] В некоторых вариантах осуществления pH составляет 7,5—8,5, как, например, 7,5—8, 8—8,3 или 8,3—8,5 включительно. В некоторых вариантах осуществления, например, Tris используют при концентрации, составляющей 10—100 мМ, как, например, 10—25 мМ, 25—50 мМ, 50—75 мМ или 25—75 мМ включительно. В некоторых вариантах осуществления любую из этих концентраций Tris используют при pH 7,5—8,5. В некоторых вариантах осуществления используют комбинацию KCl и (NH4)2SO4, как, например, 50—150 мМ KCl и 10—90 мМ (NH4)2SO4 включительно. В некоторых вариантах осуществления концентрация KCl составляет 0—30 мМ, 50—100 мМ или 100—150 мМ включительно. В некоторых вариантах осуществления концентрация (NH4)2SO4 составляет 10—50 мМ, 50—90 мМ, 10—20 мМ, 20—40 мМ, 40—60 мМ или 60—80 мМ (NH4)2SO4 включительно. В некоторых вариантах осуществления концентрация аммония [NH4+] составляет 0—160 мМ, как, например, 0—50, 50—100 или 100—160 мМ включительно. В некоторых вариантах осуществления суммарная концентрация калия и аммония ([K+] + [NH4+]) составляет 0—160 мМ, как, например, 0—25, 25—50, 50—150, 50—75, 75—100, 100—125 или 125—160 мМ включительно. Приводимый в качестве примера буфер с [K+] + [NH4+] = 120 мМ содержит 20 мМ KCl и 50 мМ (NH4)2SO4. В некоторых вариантах осуществления буфер включает 25—75 мМ Tris, pH 7,2—8, 0—50 мМ KCl, 10—80 мМ сульфата аммония и 3—6 мМ магния включительно. В некоторых вариантах осуществления буфер включает 25—75 мМ Tris, pH 7—8,5, 3—6 мМ MgCl2, 10—50 мМ KCl и 20—80 мМ (NH4)2SO4 включительно. В некоторых вариантах осуществления используют 100—200 Ед./мл полимеразы. В некоторых вариантах осуществления используют 100 мМ KCl, 50 мМ (NH4)2SO4, 3 мМ MgCl2, по 7,5 нМ каждого праймера в библиотеке, 50 мМ TMAC и 7 мкл матрицы ДНК в конечном объеме 20 мл при pH 8,1.
[142] В некоторых вариантах осуществления используют загущающее средство, такое как полиэтиленгликоль (PEG, такой как PEG 8000) или глицерин. В некоторых вариантах осуществления количество PEG (такого как PEG 8000) составляет 0,1—20%, как, например, 0,5—15%, 1—10%, 2—8% или 4—8% включительно. В некоторых вариантах осуществления количество глицерина составляет 0,1—20%, как, например, 0,5—15%, 1—10%, 2—8% или 4—8% включительно. В некоторых вариантах осуществления загущающее средство позволяет использовать либо низкую концентрацию полимеразы и/или более короткое время отжига. В некоторых вариантах осуществления загущающее средство увеличивает однородность DOR и/или снижает число выпадений (невыявленных аллелей). Полимеразы. В некоторых вариантах осуществления используют полимеразу с корректирующей активностью, полимеразу без (или с несущественной) корректирующей активностью или смесь полимеразы с корректирующей активностью и полимеразы без (или с несущественной) корректирующей активностью. В некоторых вариантах осуществления используют полимеразу с горячим стартом, полимеразу без горячего старта или смесь полимеразы с горячим стартом и полимеразы без горячего старта. В некоторых вариантах осуществления используют ДНК-полимеразу HotStarTaq (см., например, QIAGEN, № по каталогу 203203). В некоторых вариантах осуществления используют ДНК-полимеразу AmpliTaq Gold®. В некоторых вариантах осуществления используют ДНК-полимеразу PrimeSTAR GXL, высокоточную полимеразу, которая обеспечивает эффективную ПЦР-амплификация при избытке матрицы в реакционной смеси и при амплификации длинных продуктов (Takara Clontech, Маунтин-Вью, Калифорния). В некоторых вариантах осуществления используют ДНК-полимеразу KAPA Taq или ДНК-полимеразу KAPA Taq HotStart; они основаны на односубъединичной ДНК-полимеразе Taq дикого типа от термофильной бактерии Thermus aquaticus. ДНК-полимеразы KAPA Taq и KAPA Taq HotStart характеризуются 5′-3′ полимеразной и 5′-3′ экзонуклеазной активностями, но отсутствием 3′-5′ экзонуклеазной (корректирующей) активности (см., например, KAPA BIOSYSTEMS, № по каталогу BK1000). В некоторых вариантах осуществления используют ДНК-полимеразу Pfu; она представляет собой крайне термостабильную ДНК-полимеразу от гипертермофильной археи Pyrococcus furiosus. Фермент катализирует матрица-зависимую полимеризацию нуклеотидов в дуплексную ДНК в направлении 5′→3′. ДНК-полимераза Pfu также проявляет 3′→5′ экзонуклеазную (корректирующую) активность, которая позволяет полимеразе исправлять ошибки вставки нуклеотидов. Она не обладает 5′→3′ экзонуклеазной активностью (см., например, Thermo Scientific, № по каталогу EP0501). В некоторых вариантах осуществления используют Klentaq1; она представляет собой аналог фрагмента Кленова из ДНК-полимеразы Taq, она не обладает экзонуклеазной или эндонуклеазной активностью (см., например, DNA POLYMERASE TECHNOLOGY, Inc, Сент-Луис, Миссури, № по каталогу 100). В некоторых вариантах осуществления полимераза представляет собой ДНК-полимеразу PHUSION, такую как высокоточная ДНК-полимераза PHUSION (M0530S, New England BioLabs, Inc.) или ДНК-полимераза PHUSION Hot Start Flex (M0535S, New England BioLabs, Inc.). В некоторых вариантах осуществления полимераза представляет собой ДНК-полимеразу Q5®, такую как высокоточная ДНК-полимераза Q5® (M0491S, New England BioLabs, Inc.) или высокоточную ДНК-полимеразу с горячим стартом Q5® (M0493S, New England BioLabs, Inc.). В некоторых вариантах осуществления полимераза представляет собой ДНК-полимеразу T4 (M0203S, New England BioLabs, Inc.).
[143] В некоторых вариантах осуществления используют 5—600 Ед./мл (единицы на 1 мл объема реакционной смеси) полимеразы, как, например, 5—100, 100—200, 200—300, 300—400, 400—500 или 500—600 Ед./мл включительно.
Способы ПЦР
[144] В некоторых вариантах осуществления ПЦР с горячим стартом используют для снижения или предотвращение полимеризации до термоциклирования при ПЦР. Приводимые в качестве примера способы ПЦР с горячим стартом включают начальное ингибирование ДНК-полимеразы или физическое разделение компонентов реакции, до тех пор пока реакционная смесь не достигнет более высоких значений температуры. В некоторых вариантах осуществления используют медленное высвобождение магния. Чтобы обладать активностью, ДНК-полимеразе требуются ионы магния, следовательно магний химически отделяют от реакционной смеси путем связывания с химическим соединением, и он высвобождается в раствор только при высокой температуре. В некоторых вариантах осуществления используется нековалентное связывание ингибитора. В этом способе пептид, антитело или аптамер нековалентно связываются с ферментом при низкой температуре и подавляют его активность. После инкубации при повышенной температуре ингибитор высвобождается и реакция начинается. В некоторых вариантах осуществления используется холодочувствительная Taq-полимераза, такая как модифицированная ДНК-полимераза с практическим полным отсутствием активности при низкой температуре. В некоторых вариантах осуществления используется химическая модификация. В этом способе молекула ковалентно связана с боковой цепью аминокислоты в активном центре ДНК-полимеразы. Молекула высвобождается из фермента при инкубации реакционной смеси при повышенной температуре. После высвобождения молекулы фермент активируется.
[145] В некоторых вариантах осуществления количество нуклеиновых кислот-матриц (таких как образец РНК или ДНК) составляет 20—5000 нг, как, например, 20—200, 200—400, 400—600, 600—1000; 1000—1500 или 2000—3000 нг включительно.
[146] В некоторых вариантах осуществления используют набор для мультиплексной ПЦР от QIAGEN (QIAGEN, № по каталогу 206143). В случае 100 x 50 мкл мультиплексных ПЦР-реакций набор включает 2x мастер-микс для мультиплексной ПЦР от QIAGEN (обеспечивающий конечную концентрацию 3 мМ MgCl2, 3 x 0,85 мл), 5x Q-раствор (1 x 2,0 мл) и воду, не содержащую РНКаз (2 x 1,7 мл). Мастер-микс (MM) для мультиплексной ПЦР от QIAGEN содержит комбинацию KCl и (NH4)2SO4, а также добавку для ПЦР, фактор MP, который повышает локальную концентрацию праймеров на матрице. Фактор MP стабилизирует специфически связанные праймеры, обеспечивая эффективную элонгацию праймеров под действием ДНК-полимеразы HotStarTaq. ДНК-полимераза HotStarTaq представляет собой модифицированную форму ДНК-полимеразы Taq и не обладает полимеразной активностью при комнатной температуре. В некоторых вариантах осуществления ДНК-полимеразу HotStarTaq активируют путем 15-минутной инкубации при 95°C, которую можно включать в любую существующую программу термоциклера.
[147] В некоторых вариантах осуществления используют 1x конечную концентрацию MM от QIAGEN (рекомендованную концентрацию), по 7,5 нМ каждого праймера в библиотеке, 50 мМ TMAC и 7 мкл ДНК-матрицы в конечном объеме 20 мкл. В некоторых вариантах осуществления условия термоциклирования при ПЦР включают 95°C в течение 10 минут (горячий старт); 20 циклов при 96°C в течение 30 секунд; 65°C в течение 15 минут и 72°C в течение 30 секунд; после чего следует 72°C в течение 2 минут (финальная элонгация) и затем удержание при 4°C.
[148] В некоторых вариантах осуществления используют 2x конечную концентрацию MM от QIAGEN (двойную дозу рекомендованной концентрации), по 2 нМ каждого праймера в библиотеке, 70 мМ TMAC и 7 мкл ДНК-матрицы в общем объеме 20 мкл. В некоторых вариантах осуществления также включают до 4 мМ EDTA. В некоторых вариантах осуществления условия термоциклирования при ПЦР включают 95°C в течение 10 минут (горячий старт); 25 циклов при 96°C в течение 30 секунд; 65°C в течение 20, 25, 30, 45, 60, 120 или 180 минут; и необязательно 72°C в течение 30 секунд); после чего следует 72°C в течение 2 минут (финальная элонгация) и затем удержание при 4°C.
[149] Другой приводимый в качестве примера набор условий включает подход полугнездовой ПЦР. В первой ПЦР-реакции используется объем реакционной смеси 20 мкл с 2x конечной концентрацией MM от QIAGEN, по 1,875 нМ каждого праймера в библиотеке (внешний прямой и обратный праймеры) и ДНК-матрица. Параметры термоциклирования включают 95°C в течение 10 минут; 25 циклов при 96°C в течение 30 секунд, 65°C в течение 1 минуты, 58°C в течение 6 минут, 60°C в течение 8 минут, 65°C в течение 4 минут и 72°C в течение 30 секунд; а затем 72°C в течение 2 минут и затем удержание при 4°C. Далее 2 мкл полученного в результате продукта, разведенного 1:200, используют в качестве вводимого материала во второй ПЦР-реакции. Для этой реакции использовали объем реакционной смеси 10 мкл с 1x конечной концентрацией MM от QIAGEN, по 20 нМ каждого внутреннего прямого праймера и 1 мМ обратной праймерной метки. Параметры термоциклирования включают 95°C в течение 10 минут; 15 циклов при 95°C в течение 30 секунд, 65°C в течение 1 минуты, 60°C в течение 5 минут, 65°C в течение 5 минут и 72°C в течение 30 секунд; а затем 72°C в течение 2 минут и затем удержание при 4°C. Необязательно температура отжига может превышать значения температуры плавления некоторых или всех праймеров, обсуждаемых в данном документе (см. заявку на патент США № 14/918544, поданную 20 октября 2015 года, которая включена в данный документ посредством ссылки во всей своей полноте).
[150] Температура плавления (Tm) представляет собой температуру, при которой половина (50%) ДНК-дуплексов из олигонуклеотида (такого как праймер) и полностью комплементарной ему последовательности диссоциирует и превращаются в однонитевую ДНК. Температура отжига (TA) представляет собой температуру, при которой выполняется протокол ПЦР. В случае вышеописанных способов она обычно на 5°C ниже самой низкой Tm используемых праймеров, вследствие чего образуются почти все возможные дуплексы (так что практически все праймерные молекулы связываются с нуклеиновой кислотой-матрицей). Хотя это является очень эффективным, при низких температурах происходит больше реакций неспецифического связывания. Одним следствием слишком низкой TA является то, что праймеры могут отжигаться с последовательностями, отличными от настоящей мишени, поскольку могут допускаться внутренние несовпадения одного основания или частичный отжиг. В некоторых вариантах осуществления настоящего изобретения используют TA выше Tm, при этом в данный момент времени праймер отжигается только на небольшой доле мишеней (как, например, только ~1—5%). Когда эти праймеры уже элонгировались, они удаляются из равновесия отжигающихся и диссоциировавших праймеров и мишени (поскольку элонгация быстро увеличивает Tm до более 70°C), и новые ~1—5% мишеней получают праймеры. Таким образом, давая реакции длительное время на отжиг, можно получить ~100% мишеней, копированных за цикл.
[151] В различных вариантах осуществления температура отжига на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 °C и 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13 или 15°C на нижнем пределе диапазона превышает температуру плавления (такую как эмпирически измеренная или рассчитанная Tm) по меньшей мере 25, 50, 60, 70, 75, 80, 90, 95 или 100% неидентичных праймеров. В различных вариантах осуществления температура отжига на 1—15°C (как, например, 1—10, 1—5, 1—3, 3—5, 5—10, 5—8, 8—10, 10—12 или 12—15°C включительно) превышает температуру плавления (такую как эмпирически измеренная или рассчитанная Tm) по меньшей мере 25; 50; 75; 100; 300; 500; 750; 1000; 2000; 5000; 7500; 10000; 15000; 19000; 20000; 25000; 27000; 28000; 30000; 40000; 50000; 75000; 100000 или всех неидентичных праймеров. В различных вариантах осуществления температура отжига на 1—15°C (например, 1—10, 1—5, 1—3, 3—5, 3—8, 5—10, 5—8, 8—10, 10—12 или 12—15°C включительно) превышает температуру плавления (такую как эмпирически измеренная или рассчитанная Tm) по меньшей мере 25%, 50%, 60%, 70%, 75%, 80%, 90%, 95% или всех неидентичных праймеров, и продолжительность стадии отжига (на цикл ПЦР) составляет 5—180 минут, как, например, 15—120 минут, 15—60 минут, 15—45 минут или 20—60 минут включительно.
[152] Приводимые в качестве примера способы мультиплексной ПЦР
[153] В различных вариантах осуществления используют длительное время отжига (как обсуждается в данном документе и приведено в качестве примера в примере 12) и/или низкие концентрации праймеров. В действительности, в определенных вариантах осуществления используют ограничивающие концентрация праймеров и/или условия. В различных вариантах осуществления продолжительность стадии отжига составляет от 15, 20, 25, 30, 35, 40, 45 или 60 минут на нижнем пределе диапазона до 20, 25, 30, 35, 40, 45, 60, 120 или 180 минут на верхнем пределе диапазона. В различных вариантах осуществления продолжительность стадии отжига (на цикл ПЦР) составляет 30—180 минут. Например, стадия отжига может составлять 30—60 минут, а концентрация каждого праймера может составлять менее 20, 15, 10 или 5 нМ. В других вариантах осуществления концентрация праймеров составляет 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20 или 25 нМ на нижнем пределе диапазона и 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25 и 50 на верхнем пределе диапазона.
[154] При высоком уровне мультиплексирования раствор может становиться вязким из-за большого количества праймеров в растворе. Если раствор является слишком вязким, можно снизить концентрацию праймеров до количества, которое все еще достаточно для того, чтобы праймеры связывались с ДНК-матрицей. В различных вариантах осуществления используют от 1000 до 100000 различных праймеров, и концентрация каждого праймера составляет менее 20 нМ, как, например, менее 10 нМ или 1—10 нМ включительно.
[155] Следующие примеры предложены, чтобы предоставить средним специалистам в данной области техники полное раскрытие и описание того, как применять варианты осуществления, представленные в данном документе, и они не подразумевают ограничение объема настоящего раскрытия, или они не подразумевают представление того, что примеры, приведенные ниже, являются всеми или единственными осуществляемыми экспериментами. Были приложены усилия по обеспечению точности с точки зрения применяемых чисел (например, количеств, температуры и т. д.), но должны учитываться некоторые экспериментальные ошибки и отклонения. Если не указано иное, части представляют собой части по объему, а температура представлена в градусах Цельсия. Следует понимать, что вариации описанных способов могут быть созданы без изменения фундаментальных аспектов, которые, как предусмотрено, иллюстрируют примеры.
ПРИМЕРЫ
[156] ПРИМЕР 1. Анализ однонуклеотидных вариантов (SNV) в циркулирующей опухолевой ДНК (ctDNA), полученной от пациентов с раком легкого
[157] Предварительное пилотное исследование продемонстрировало успешное выявление имеющих отношение к раку точковых мутаций в плазме крови пациентов с раком. В этом исследовании профиль мутаций 4 опухолей рака легкого определяли с помощью полноэкзомного секвенирования (WES) или Ampliseq (Life Technologies, Карлсбрад, Калифорния) и подгруппу этих мутаций успешно выявляли в соответствующих образцах плазмы крови с применением способа мультиплексного ПЦР-секвенирования следующего поколения (mPCR-NGS). В этом эксперименте под названием TRACERx способ mPCR-NGS применяли для выявления и отслеживания во времени специфических в отношении рака мутаций в плазме крови пациентов с раком и для оценки пригодности способа в контроле прогрессирования заболевания на протяжении лечения. Общая схема проекта показан на ФИГ. 1. Первой фазой проекта было определение профиля мутаций на исходном уровне в плазме крови 50 пациентов, не получавших лечения рака легкого. Образцы очищенной геномной ДНК из нескольких участков опухоли (2—7 участков на опухоль), образцы очищенной зародышевой ДНК и образцы интактной плазмы крови получали от 50 пациентов. Профиль мутаций всех участков опухоли предварительно определяли с помощью WES и AmpliSeq и подгруппу мутаций на пациента анализировали с помощью mPCR-NGS. Эти мутации включали как драйверные мутации, так и мутации-пассажиры и как клональные, так и субклональные мутации. На основании этих данных авторы настоящего изобретения разрабатывали анализы на основе мультиплексной ПЦР, подготавливали пулы праймеров (праймеры получали от IDT, Коралвилль, Айова), подвергали пулы праймеров QC и оптимизировали протокол mPCR для каждого пула. cfDNA плазмы крови очищали, проводили количественную оценку и преобразовывали в библиотеки. Библиотеки затем применяли в качестве вводимого материала в mPCR и продукты секвенировали и анализировали. Подобный протокол применяли в отношении геномной ДНК из опухоли и сопоставимых образцов нормы.
Описание образцов
[158] Образцы. Для каждого из первых 50 пациентов TRACERx выделяли 4—5 мл плазмы крови, полученной перед иссечением опухоли и перед какой-либо терапией. Образцы плазмы крови делили на аликвоты в пробирках объемом 2 мл и транспортировали замороженными на сухом льду. Очищенную геномную ДНК из не более 7 фрагментов опухоли из пораженных лимфатических узлов (если таковые имеются) и из фракции белых кровяных клеток (так называемой сопоставимой нормы) очищали и анализировали 500 нг очищенной ДНК из каждого образца, нормализованных до 10 нг/мкл. Образцы очищенной ДНК замораживали и транспортировали на сухом льду.
[159] Информация относительно SNV. Профиль мутаций, включая однонуклеотидные варианты (SNV) и варианты числа копий (CNV), каждого фрагмента опухоли определяли с помощью TRACERx с применением WES. Полный профиль мутаций каждой опухоли применяли для выявления клональной структуры и для воссоздания филогенетического древа каждой опухоли. PyClone (PyClone: statistical inference of clonal population structure in cancer. Roth et al, Nature Methods 11, 396—398 (2014)) применяли для выявления клональной структуры. PyClone обеспечивает идентификацию перечня субклонов c SNV и рассчитывает долю раковых клеток в них. Он также обеспечивает разделение SNV на категории клональных или субклональных. Категорию драйверности каждого SNV определяли и представляли как драйверную категорию (1—4, где 1 наиболее вероятно является драйверной мутацией, и 4 характеризуются наименьшей вероятностью). Для каждого пациента анализировали до 108 SNV, охватывающих все драйверные категории и включающих клональные и субклональные мутации. Выявленные доли аллелей каждого SNV в каждом фрагменте опухоли, лимфатическом узле и сопоставимом образце нормальной ДНК сравнивали с информацией клонального/субклонального кластера PyClone.
[160] Дополнительная информация. Для каждого пациента была доступна следующая информация: размер опухоли (мм), расположение опухоли (доля легкого), стадия опухоли, патологический тип опухоли, число пораженных лимфатических узлов, статус прорастания сосудов, а также деидентифицированная информация о распределительном госпитале.
Схема анализа и оптимизация протокола.
[161] Схема анализа. Процесс разработки схемы стандартного анализа Natera применяли для разработки правого и левого праймеров для ПЦР для всех данных SNV. Пару правого и левого праймеров для ПЦР, нацеливающихся на SNV, определяют как аналит для этого конкретного SNV. Следует учесть, что для одного аналита является возможным охватывать более чем 1 целевой SNV, если они находятся в непосредственной близости. Для каждой пары аналитов рассчитывали вероятность образования димеров праймеров. Данные о доле аллеля SNV в каждой опухоли применяли для воссоздания филогенетических древ с применением Lichee (Fast and scalable inference of multi-sample cancer lineages. Popic et al. Genome Biol. 2015 May 6;16:91). Перечень аналитов для каждого образца отфильтровывали для удаления праймеров, которые, как прогнозировалось, образуют димеры праймеров, уделяя первостепенное внимание аналитам, охватывающим драйверные SNV 1 и 2. Остальные анализы применяли для построения 5 сбалансированных пулов. Все аналиты, объединенные в пул, были сравнимы, что означает, что не было праймеров, которые, как прогнозировалось, образуют димеры праймеров в пуле. На каждой стадии аналиты отбирали таким образом, что аналиты, охватывающие драйверные SNV 1 и 2, имеют первостепенное значение, и для каждого пациента число отобранных SNV на ветвь было пропорционально общему числу SNV этой ветви из воссозданного филогенетического древа. Более конкретно, авторы настоящего изобретения пытались получить равномерную выборку SNV из ветвей в воссозданном филогенетическом древе, чтобы убедиться в том, что отобранные аналиты обеспечивают хорошее покрытие воссозданного древа. Конечная схема состояла из 972 аналитов, равномерно распределенных среди 5 пулов, и содержала 15—20 аналитов для каждого образца. Число SNV и число аналитов на образец в соответствии с категорией драйверных мутаций показаны на ФИГ. 2. Гены, в которых обнаружены SNV, и число SNV, которые анализировали на ген, находятся на ФИГ. 19.
[162] QC и оптимизация пула. 972 пар праймеров получали (IDT, Коралвилль, Айова) в отдельных лунках, обессоливали и нормализовали до 100 мкM. Аналиты объединяли в пул в соответствии со схемой объединения в пул и каждый пул применяли в комбинированном эксперименте QC/оптимизация. Для эксперимента оптимизации изменяли несколько параметров ПЦР и по данным последовательности оценивали эффекты на производительность секвенирования, а также число выпавших аналитов. Определяли условия ПЦР, которые приводили к наилучшим процентному значению целевых ридов, однородности глубины прочтения и наименьшему уровню ошибок. Праймеры, обусловливающие преобладающее большинство димеров праймеров, идентифицировали и удаляли из каждого пула (для каждого удаленного праймера удаляли также соответствующего партнера). После этой стадии в общей сложности оставалось 908 аналитов, равномерно распределенных среди 5 пулов.
Получение образца
[163] Выделение ДНК и QC. Все аликвоты плазмы крови от каждого пациента объединяли в пул перед выделением cfDNA и визуально оценивали степень гемолиза каждого объединенного в пул образца плазмы крови (отсутствие гемолиза, слабый гемолиз или сильный гемолиз). cfDNA выделяли с применением набора Qiagen NA (Валенсия, Калифорния), следуя протоколу, оптимизированному для 5 мл плазмы крови. Все образцы cfDNA подвергали QC на высокочувствительных чипах биоанализатора (Agilent, Санта-Клара, Калифорния). Те же циклы на высокочувствительном биоанализаторе также применяли для подсчета образцов cfDNA путем интерполяции высоты мононуклеосомальных пиков на калибровочную кривую, полученную для образца чистой cfDNA, количественную оценку которого предварительно проводили. Это было необходимым, поскольку cfDNA иногда содержит долю интактной ДНК, которая перекрывается с маркером большого размера на чипе, что делает количественную оценку мононуклеосомального пика недостоверной. Проводили количественную оценку репрезентативной подгруппы образцов очищенной геномной ДНК (из фрагментов опухоли, лимфатических узлов и белых кровяных клеток) с применением Nanodrops (Уилмингтон, Делавэр). Проводили количественную оценку всех образцов в ожидаемом диапазоне (~10 нг/мкл).
[164] Построение библиотеки cfDNA. Общее количество cfDNA из каждого образца плазмы крови применяли в качестве вводимого материала в Library Prep с применением набора Natera library prep и следуя инструкциям в наборе. Для двух образцов с чрезвычайно большими количествами cfDNA вводимое количество в Library Prep ограничивали до ~50000 геномных эквивалентов (165 нг). Библиотеки амплифицировали до получения плато и затем очищали с применением гранул Ampure (Beckman Coulter, Бреа, Калифорния), следуя инструкциям производителя. Очищенные библиотеки подвергали QC на LabChip.
[165] Мультиплексная ПЦР и секвенирование cfDNA. Материал библиотек из каждого образца плазмы крови применяли в качестве вводимого материала в мультиплексной ПЦР (mPCR) с применением соответствующего пула для анализа и оптимизированного протокола для mPCR плазмы крови. В протоколе использовали время отжига 15 минут при температуре 60C или 62,5C, которая была выше, чем Tm праймеров. Tm праймеров с применением теоретических расчетов составляла от 53 до 59C. Применяли концентрацию праймеров 10 нM. Продуктам mPCR присваивали штрихкод на отдельной стадии ПЦР, и продукты ПЦР со штрихкодами объединяли в 5 пулов в соответствии с информацией объединения в пул для анализа (см. раздел выше). Пулы очищали с применением гранул Ampure, следуя инструкциям производителя, подвергали QC на чипе биоанализатора DNA1000 (Agilent, Санта-Клара, Калифорния) и проводили количественную оценку с применением набора Qubit для определения dsDNA в широком диапазоне (Thermo Fisher Scientific, Уолтем, Массачусетс). Каждый пул содержал библиотеки, полученные как раскрыто выше, из 10 образцов плазмы крови пациентов с раком и 20 отрицательных контролей (полученных из cfDNA, взятой у предполагаемо здоровых добровольцев). Образцы отрицательного контроля получали, следуя необходимым нормативным процедурам. Каждый пул секвенировали на отдельном HiSeq 2500 с режимом быстрого прогона (Illumina, Сан-Диего, Калифорния) с 50 циклами одноиндексных ридов со спаренными концами.
[166] Мультиплексная ПЦР и секвенирование gDNA. Образцы геномной ДНК применяли в качестве вводимого материала в подобную mPCR с применением соответствующих пулов для анализа и оптимизированного протокола для ПЦР геномной ДНК. Продуктам mPCR присваивали штрихкод на отдельной стадии ПЦР и все продукты со штрихкодом объединяли в один пул. Пул очищали с применением гранул Ampure, следуя инструкциям производителя, подвергали QC на чипе биоанализатора DNA1000 и проводили количественную оценку с применением набора Qubit для определения dsDNA в широком диапазоне. Пул секвенировали на отдельном HiSeq2500 с режимом быстрого прогона с 50 циклами одноиндексных ридов со свободными концами.
Результаты
[167] ФИГ. 20 представляет собой таблицу, демонстрирующую подробные результаты анализа и подробную информацию в отношении образцов, которые анализировали в данном исследовании.
[168] Выделение и анализ cfDNA. Распределение концентраций cfDNA для 50 образцов плазмы крови (фигура 3) с последующим ожидаемым распределением на основе 5 мл плазмы крови (медианное значение 2200 эквивалентов геномных копий на мл плазмы крови). Концентрации cfDNA, степень гемолиза (визуально оцененная) и качественная оценка профиля размера cfDNA (визуально оцененная из следов биоанализатора) показаны в табличной форме на ФИГ. 16
[169] Анализ cfDNA. Концентрация очищенной cfDNA, степень гемолиза плазмы крови и профиль cfDNA показаны на фигуре 16. Концентрация cfDNA относится только к мононуклеосомальному пику и была определена по высоте мононуклеосомального пика с применением калибровочной кривой. Эквиваленты геномных копий рассчитывали с применением коэффициента перехода 3,3 пг/геном; 40 мкл очищенной cfDNA применяли в качестве вводимого материала в Library Prep; выделено зеленым цветом: для этих образцов ввод в library prep ограничивали до 50000 геномных эквивалентов. Профиль размера cfDNA: 1: большая часть cfDNA находится в мононуклеосомальном пике; 2: большая часть cfDNA находится в мононуклеосомальном пике, но видны другие размеры; 3: большой пик интактной ДНК (>1000 п.о.) видно наравне с мононуклеосомальным пиком, а также некоторые пики большей молекулярной массы. Гемолиз визуально оценивали на основе цвета плазмы крови. 0: отсутствие гемолиза (плазма крови желтого цвета); 1: слабый гемолиз (плазма крови светло-розового цвета); 2: сильный гемолиз (плазма крови ярко-розового или красного цвета).
[170] Анализ VAF в фрагментах опухоли. Данные последовательности из каждого из фрагментов опухоли анализировали для определения частоты вариантного аллеля (VAF) каждого SNV в каждом фрагменте опухоли, лимфатическом узле и сопоставимом образце нормы. Эти данные сравнивали с сопоставимыми данными, предоставленными отдельно из другого проверяемого сайта с применением других тестовых способов, таких как полногеномное секвенирование и экзомное секвенирование. Для большинства образцов предварительно определяли значения VAF ткани из каждого фрагмента опухоли, близко сопоставимые с недавно полученными значениями VAF ткани (фигура 4). Однако имелось большое количество образцов, в которых наблюдались значительные несоответствия (фигура 5). Наблюдали три типа несоответствий: (i) для одного или двух фрагментов все VAF равняются 0 или являются близкими к нулю 0 в предыдущем анализе, но не равняются нулю (и охватывают диапазон VAF, наблюдаемый в других фрагментах того же образца) в анализе mPCR-NGS (например, LTX041, LTX111); (ii) для нескольких анализов VAF равняются 0 в анализе mPCR-NGS, но не равняются нулю (и охватывают диапазон VAF, наблюдаемых в других фрагментах того же образца) в предыдущем анализе, и не наблюдали образования кластеров по фрагменту в данном режиме несоответствий (например, LTX093, LTX074); (iii) для нескольких анализов или участков ни один из анализов не потерпел неудачу, но соответствие между VAF, полученными в двух анализах, в целом было слабым (например, LTX063, LTX059).
[171] Также идентифицировали 16 соматических SNV из образцов ткани, о которых не сообщалось в распознаваниях SNV TRACERx. Среди этих новых соматических SNV 7 распознавали также в их соответствующих cfDNA плазмы крови. См. перечень на ФИГ. 18.
[172] Один образец (U_LTX206 с 19 аналитами) не прошел секвенирование и был исключен из анализа. Анализировали 889 аналитов, охватывающих 911 SNV. Анализы с глубиной прочтения менее 1000 рассматривали как потерпевшие неудачу, и их соответствующие SNV отмечали также как «не распознанные». В общей сложности 21 «не распознанный» SNV удаляли из анализа; в общей сложности анализировали 890 SNV.
[173] Каждый прогон принадлежал к одному пулу для анализа и содержал 10 образцов рака, а также 20 контрольных образцов. Набор SNV, преобразованный с помощью анализа в пул, рассматривали в качестве целевых SNV для связанного прогона. Для осуществления распознавания SNV в конкретном положении образца рака, прежде всего, для этого положения создавали модель фоновой ошибки. Модель ошибки конструировали на основе 20 отрицательных образцов и остальных образцов рака (8 или 9), которые, как ожидалось, не содержали SNV в данном положении, на основе предоставленной информации. Положения с VAF > 20% исключали из модели фоновой ошибки. Положительное распознавание SNV плазмы крови осуществляли, если достоверность для данной мутации в соответствующем образце плазмы крови превышала порог достоверности, составляющий от 95 до 98%.
[174] Общий уровень выявления SNV в плазме крови составляет 35,5% (310 из 890), подобный таковому предварительного пилотного исследования. В то время как алгоритм обеспечивал наиболее достоверные истинно положительные распознавания, число ложноположительных распознаваний находятся на приемлемом уровне (<0,25%). Средняя частота мутантного аллеля для SNV, выявленная с высокой достоверностью, составляет 0,875%, находясь в диапазоне от 0,011% до 13,93%. Образец рассматривали как «выявленный в плазме крови», если по меньшей мере один SNV, который, как ожидалось, присутствовал в этом образце, был достоверно выявлен в плазме крови. С применением данного определения общий уровень выявления образца в плазме крови составлял 69% (34 из 49 образцов), и для этого среднее число SNV, выявленных в плазме крови, составляло 9,1 (в диапазоне от 1 до 19). Число SNV, выявленное в плазме крови для каждого образца, показано в табличной форме на ФИГ. 17.
[175] Анализ SNV, которые не выявлены в плазме крови. Несколько наборов данных поддерживают вывод о том, что неспособность выявления >60% (580 из 911) ожидаемых SNV в плазме крови связана с тем фактом, что отсутствует достаточное количество сведений присутствия для данных мутаций в образце cfDNA, в отличие от какой-либо неэффективности способа mPCR-NGS: распределение глубины прочтения (DOR) подобно таковым для анализов, которые обеспечивали выявление ожидаемых SNV плазмы крови и таковым, которые не обеспечивали выявление ожидаемых SNV (фигура 6a) (среднее значение DOR 45,551 для анализов, которые обеспечивали выявление ожидаемых SNV, по сравнению с 45,133 для таковых, которые не обеспечивали этого). Это дает основание полагать, что анализы, соответствующие ложноотрицательным распознаваниям SNV, являются такими же по эффективности, как и для ложноположительных распознаваний. Кроме того, несмотря на высокий показатель DOR в целевом положении SNV, число мутантных ридов является практически несущественным. Фактически, 36% из них имеют 0 мутантных ридов, 75% из них имеют более чем 5 мутантных ридов, и остальные 25% ложноотрицательных распознаваний характеризуются VAF < ,1%.
Факторы, влияющие на выявление SNV в плазме крови.
[176] Оценивали несколько факторов, которые влияют на возможность выявления SNV в плазме крови. Информацию о количестве cfDNA и определении стадии опухоли, размере опухоли и значениях частоты SNV в фрагментах опухоли определяли в отдельных местоположениях.
[177] Гистологический тип. Наиболее важным прогностическим фактором того, выявят ли опухоль в плазме крови, по-видимому, является гистологический тип: 100% опухолей плоскоклеточной карциномы (SQCC) выявляли в плазме крови, в то время как только 50% (15/29) из опухолей аденокарциномы (ADC) выявляли в плазме крови в данном исследовании (фигура 7). Более того, среднее число выявленных SNV на образец составляло 12,7 (медианное значение = 13) для SQCC и 2,6 (медианное значение = 1) для ADC. В данной когорте была только одна опухоль карциносаркомы и одна аденосквамозная опухоль, так что на данном этапе нельзя сделать выводов о данных типах опухоли относительно их общей возможности выявления в плазме крови.
[178] Стадия и размер опухоли. Стадия и размер опухоли были одними из наиболее важных идентифицированных факторов, которые влияют на число SNV, выявленных в соответствующем образце плазмы крови (фигура 8). Опухоли стадии 1a имели наиболее низкие шансы выявления по меньшей мере одного SNV, а также наиболее низкий показатель успеха выявления SNV в плазме крови. Распределение VAF для SNV, которые выявляли из опухолей стадии 1a, был также ниже, чем для остальных опухолей (фигура 9). Поскольку размер и стадия опухоли коррелируют, подобную тенденцию наблюдали для размера опухоли. Поскольку это не было связано с неэффективностью анализа или предельными значениями чувствительности (см. ниже), наиболее вероятным объяснением этому является то, что такие опухоли, как правило, не характеризуются наличием cfDNA в плазме крови в количествах, которые можно обнаружить в объемах плазмы крови, используемых в данном исследовании. Эффект стадии и размера опухоли на число SNV, выявленных в ctDNA, варьировал у образцов ADC и SQCC. Образцы ADC характеризовались большей зависимостью от этих факторов с общей тенденцией намного меньшего числа выявленных SNV в ctDNA, чем выявленных в ctDNA образцов SCC. Фактически, SNV выявляли в ctDNA всех образцов SQCC, независимо от стадии: три SNV выявляли в ctDNA одного из образцов SCC и по меньшей мере 5 SNV выявляли в ctDNA остальных образцов SCC (фигура 15). Фактически, от 3 до 19 SNV выявляли в ctDNA образцов SCC. В 6 образцах ADC, которые относились к стадии 1a, SNV выявляли только в одном из образцов ctDNA, и в этом образце выявили только один SNV. Ни в одном из образцов ADC стадии 1a не выявляли более 1 SNV в ctDNA. В образцах ADC стадии 1b менее чем 5 SNV идентифицировали во всех за исключением двух образцов, с 7 SNV, идентифицированными в одном из образцов ADC стадии 1b, и 18 SNV, идентифицированными в одном из образцов стадии 1b.
[179] VAF и клональность опухоли. Соотношение клональности рассчитывали для каждой мутации как (число фрагментов опухоли, в которых выявлена мутация) / (общее число анализированных фрагментов этой опухоли). Мутации, которые наблюдали во всех анализированных срезах опухоли, рассматривали как «клональные», все другие рассматривали как «субклональные». VAF SNV, выявленных в плазме крови, коррелирует с «клональностью» мутаций, с большим числом клональных мутаций, отвечающих за наиболее высокие значения VAF в плазме крови (фигуры 9 и 12); подобным образом, SNV, присутствующие в нескольких фрагментах опухоли, как правило, отвечают за более высокие значения VAF плазмы крови в соответствующих образцах плазмы крови. В дополнение к соотношению клональности клональный статус каждого SNV разделяли по категориям с помощью PyCloneCluster на основе данных WES, полученных из опухолевой ткани. Клональные SNV, как правило, характеризуются более высокими значениями VAF (фигуры 13 и 14).
[180] Вводимая cfDNA и VAF опухоли. Корреляция между количеством cfDNA и числом и относительным числом SNV, выявленных в образцах плазмы крови, отсутствовала. Число SNV, выявленных в плазме крови, не прогнозируется по вводимому количеству cfDNA; однако все образцы с высоким значением ввода (>25000 копий) характеризовались по меньшей мере одним SNV, выявленным в плазме крови (фигура 10). VAF SNV плазмы крови также коррелирует с VAF SNV опухоли (фигура 11).
[181] Мультивариантный анализ. Регрессионный анализ выполняли для определения переменных, которые можно применять для прогнозирования выявления мутаций. Более конкретно, 0/1 переменных отклика применяли для аннотирования мутаций, которые авторы настоящего изобретения называли как присутствующие или отсутствующие. В модель включали следующие независимые переменные:
1. VAF опухоли
2. кластер PyClone (категориальная переменная)
3. стадия рака (категориальная переменная)
4. размер опухоли
5. количество вводимой ДНК
6. патологический тип (категориальная переменная)
7. число пораженных лимфатических узлов
8. прорастание сосудов (категориальная переменная)
9. пораженная доля
Логистическая регрессия показала, что следующие переменные были статистически значимо связаны с выявлением мутации (с p-значениями < 5%):
1. VAF опухоли (p = 4,3e-6)
2. кластер PyClone (p = 1,6e-4)
3. размер опухоли (p = 3,5e-4)
4. патологический тип (p = 8,3e-30)
[182] Выводы. Авторы настоящего изобретения продемонстрировали в данных примерах успешное выявление связанных с раком легкого SNV в образцах плазмы крови, полученных от пациентов с раком легкого. С применением персонализированной панели мультиплексной ПЦР, оптимизированной для данной когорты образцов, выявляли SNV с долей вариантного аллеля всего 0,01%. Из тестированных SNV 35% выявляли в образцах плазмы крови, и 67% из анализированных образцов характеризовались по меньшей мере одним выявленным SNV в плазме крови. Авторы настоящего изобретения также идентифицировали некоторые из факторов, которые содействуют успешному выявлению SNV плазмы крови. Они включают тип опухоли, стадию опухоли, размер опухоли, частоту аллеля SNV в опухоли и, в меньшей степени, количество анализированной cfDNA. Вывод от том, что не все SNV выявляли в плазме крови, и что не все образцы характеризуются обнаруживаемыми SNV в плазме крови, по-видимому, не обусловлен анализом или ограничениями протокола, поскольку эти анализы являлись эффективными (как свидетельствует их глубина прочтения при секвенировании), и их предел выявления был достаточным для выявления любых SNV, при их наличии в образце cfDNA. Скорее, неспособность выявления этих SNV была связана с тем фактом, что они не присутствуют в образце. Образцы из опухолей низкой степени дифференцировки и опухолей небольшого размера, наиболее вероятно, характеризовались ограниченными количествами циркулирующей опухолевой ДНК. Подобным образом, SNV, которые присутствовали в опухоли при низкой частоте аллеля, с меньшей вероятностью присутствовали в плазме крови. Однако даже опухоли с высокой степенью дифференцировки и относительно большим размером могут характеризоваться отсутствием SNV, выявленных в плазме крови. Возможно, за это отвечают другие причины биологического характера (такие как количество ctDNA, выделенной из опухоли), и что анализирование большего количества SNV на образец будет повышать шанс выявления некоторых из них. 0,4 мM dNTP (см. ФИГ. 12—3C).
[183] Специалисты в данной области техники могут разработать много модификаций и других вариантов осуществления в пределах объема и сути раскрытого в данном документе изобретения. Действительно, вариации описанных материалов, способов, графических материалов, экспериментальных примеров и вариантов осуществления могут быть созданы специалистами в данной области техники без изменения фундаментальных аспектов раскрытого изобретения. Любые из раскрытых вариантов осуществления можно применять в комбинации с любым другим раскрытым вариантом осуществления.
[184] Раскрытые варианты осуществления, примеры и эксперименты не предусмотрены ни для ограничения объема настоящего описания, ни для представления того, что представленные ниже эксперименты являются всеми или единственными выполняемыми экспериментами. Были приложены усилия по обеспечению точности в отношении применяемых чисел (например, количеств, температуры и т. д.), но должны учитываться некоторые экспериментальные ошибки и отклонения. Следует понимать, что вариации описанных способов могут быть созданы без изменения фундаментальных аспектов, которые, как предусмотрено, иллюстрируют эксперименты.
[185] ПРИМЕР 2. Смоделированный протокол мультиплексной ПЦР для отслеживания опухолевых мутаций в плазме крови
[186] Смоделированный протокол мультиплексной ПЦР (mPCR) от Natera разработан для оценки уровня ctDNA в плазме крови путем отслеживания набора специфических в отношении пациента мутаций, идентифицированных из образца(образцов) опухолевой ткани. С учетом данного специфического в отношении пациента профиля мутаций авторы настоящего изобретения разрабатывали персонализированные панели mPCR, которые можно применять относительно образцов плазмы крови соответствующего пациента в последовательном режиме.
[187] SNV-мишени. Профиль мутаций, который включает однонуклеотидные варианты (SNV) для каждого фрагмента опухоли, определяли на основе анализов секвенирования опухоли. Полный профиль мутаций каждой опухоли применяли для воссоздания филогенетического древа каждой опухоли. PyClone (Roth, et al. (2014). PyClone: Statistical inference of clonal population structure in cancer. Nature Methods 11: 396—398) применяли для идентификации кластеров SNV и расчета у них доли принадлежащих к раковым клеткам. Его использовали для разделения SNV на категории, или клональные, или субклональные. Определяли категорию драйверности каждого SNV (1—4, где 1 наиболее вероятно являлся драйверной мутацией, и 4 характеризовался наименьшей вероятностью).
[188] Схема анализа. Процесс разработки схемы стандартного анализа Natera применяли для разработки ПЦР-праймеров для всех данных SNV со следующими параметрами:
• Оптимальная температура плавления [Tm] 56°C, допустимый диапазон, 53°C‒59°C
• Длина ампликона, 50‒70 п.о.
• Содержание GC, 30‒70%
[189] Имеется в виду пара правого и левого ПЦР-праймеров, нацеливающихся на SNV, в качестве аналитов для данного определенного SNV. Для одного аналита является возможным охватывать более чем 1 целевой SNV, если они находились в непосредственной близости. Для каждой пары аналитов вероятность образования димера праймеров рассчитывали с применением термодинамического подхода (SantaLucia JR (1998) «A unified view of polymer, dumbbell and oligonucleotide DNA nearest-neighbor thermodynamics», Proc Natl Acad Sci 95:1460—65) для оценки стабильности структуры гибридизации, представляющей собой соединение пары праймеров. Аналиты объединяли в пул для сведение к минимуму числа праймеров с высокой вероятностью образования димера праймеров в том же пуле. Для каждого пациента устанавливали приоритеты в отношении аналитов таким образом, что 1) аналиты, охватывающие драйверные SNV, являлись наиболее приоритетными, и 2) выборка филогенетического древа являлась равномерной.
[190] QC и оптимизация пула. Праймеры заказывали у IDT в отдельных лунках, обессоленные и нормализованные до 100 мкМ. Аналиты объединяли в пул в Natera в соответствии со схемой объединения в пул для создания пулов для анализа, где каждый праймер находился в концентрации 250 нМ в воде. Каждый пул применяли в комбинированном эксперименте QC/оптимизация. Для эксперимента оптимизации параметры ПЦР (концентрация праймеров и температура отжига) отличались, и эффекты в отношении процентного значения целевых ридов, однородности глубины прочтения (измеренной как отношение 80-го процентиля/20-го процентиля), и число выпавших аналитов (определенных как аналиты с <1000 ридов) оценивали по данным секвенирования. Определяли условия ПЦР, которые приводят к получению наилучшего процентного значения целевых ридов, однородности глубины прочтения и самого низкого числа выпавших значений. Для всех пулов оптимальными условиями были 10 нМ праймеров и температура отжига 60°C или 62,5°C.
[191] Праймеры, обусловливающие преобладающее большинство димеров праймеров, идентифицировали и удаляли из каждого пула (для каждого удаленного праймера удаляли также соответствующего партнера).
[192] Выделение ДНК и QC. Аликвоты плазмы крови от каждого пациента объединяли в пул перед выделением cfDNA и визуально оценивали степень гемолиза каждого объединенного в пул образца плазмы крови и делали пометки (отсутствие гемолиза, слабый гемолиз или сильный гемолиз). cfDNA выделяли на Natera с применением набора Qiagen NA (Валенсия, Калифорния), следуя протоколу, оптимизированному для 5 мл плазмы крови. Все образцы cfDNA подвергали QC на высокочувствительных чипах биоанализатора. Те же циклы на высокочувствительном анализаторе также применяли также для подсчета образцов cfDNA путем интерполяции высоты мононуклеосомального пика на калибровочную кривую, полученную для образца чистой cfDNA, количественную оценку которого проводили предварительно. Это является необходимым, поскольку cfDNA иногда содержит долю интактной ДНК, которая перекрывается с маркером большого размера на чипе, делая количественную оценку мононуклеосомального пика недостоверной.
[193] Проводили количественную оценку образцов геномной ДНК (из фрагментов опухоли, лимфатических узлов и белых кровяных клеток) на Nanodrop.
[194] Построение библиотеки cfDNA. Общее количество cfDNA из каждого образца плазмы крови применяли в качестве вводимого материала в Library Prep с применением набора Natera library prep и следуя инструкциям в наборе. Для двух образцов с чрезвычайно большими количествами cfDNA вводимое количество в Library Prep ограничивали до ~50000 геномных эквивалентов (165 нг). Вкратце, для 40 мкл ДНК, экстрагированной из плазмы крови, которая присутствовала в фрагментах мононуклеосомальной и полинуклеосомальной длин, обеспечивали репарацию концов и добавление A-хвоста и лигировали персонализированные адапетры Natera. Библиотеки амплифицировали на протяжении 15 циклов до получения плато и затем очищали с применением гранул Ampure, следуя инструкциям производителя. Очищенные библиотеки подвергали QC на LabChip.
[195] Мультиплексная ПЦР и секвенирование cfDNA. Материал библиотек из каждого образца плазмы крови применяли в качестве вводимого материала в мультиплексной ПЦР с применением соответствующего пула для анализа и оптимизированного протокола для mPCR плазмы крови. Композиция для ПЦР представляла собой 1 x внутрилабораторного мастер-микса для ПЦР, 10 нМ праймеров, 3 мкл библиотек cfDNA (соответствующих ~600 нг ДНК) в общем объеме реакционной смеси 10 мкл. Условия термоциклирования представляли собой 95°C, 10 минут; 10 циклов (95°C, 30 секунд; 60°C или 62,5°C, 15 минут; 72°C, 30 секунд); 72°C, 2 минуты, удерживание при 4°C.
[196] Продуктам mPCR присваивали штрихкод на отдельной стадии ПЦР, и продукты ПЦР со штрихкодами объединяли в пул в соответствии с информацией объединения в пул для анализа.
[197] Пулы очищали с применением гранул Ampure, следуя инструкциям производителя, подвергали QC на чипе биоанализатора DNA1000 и проводили количественную оценку с применением набора Qubit для определения dsDNA в широком диапазоне. Каждый пул содержал продукты mPCR со штрихкодами из 10 библиотек плазмы крови пациента с раком и 20 отрицательных контролей (полученных из cfDNA, взятой у здоровых добровольцев). Образцы отрицательного контроля получали, следуя необходимым нормативным процедурам. Каждый пул секвенировали на отдельном HiSeq2500 с режимом быстрого прогона с 50 циклами одноиндексных ридов со спаренными концами.
[198] Мультиплексная ПЦР и секвенирование геномной ДНК. Образцы геномной ДНК (gDNA) применяли в качестве вводимого материала в подобную mPCR с применением соответствующих пулов для анализа и оптимизированного протокола для ПЦР геномной ДНК; 50 нг gDNA применяли в качестве вводимого материала. Продуктам mPCR присваивали штрихкод на отдельной стадии ПЦР и все продукты со штрихкодом объединяли в один пул. Пул очищали с применением гранул Ampure, следуя инструкциям производителя, подвергали QC на чипе биоанализатора DNA1000 и проводили количественную оценку с применением набора Qubit для определения dsDNA в широком диапазоне. Пул секвенировали на отдельном HiSeq2500 с режимом быстрого прогона с 50 циклами одноиндексных ридов со свободными концами.
[199] Процесс биоинформатических вычислений. Риды со спаренными концами картировали на эталонном геноме hg19 с помощью Novoalign v2.08.02, и сортировали и индексировали с применением SAMtools (Li H.*, Handsaker B.*, Wysoker A., Fennell T., Ruan J., Homer N., Marth G., Abecasis G., Durbin R. and 1000 Genome Project Data Processing Subgroup (2009) The Sequence alignment/map (SAM) format and SAMtools. Bioinformatics, 25, 2078—9). Все риды со спаренными концами ограничивали с применением PEAR (J. Zhang, K. Kobert, T. Flouri, A. Stamatakis. PEAR: A fast and accurate Illumina Paired-End reAd mergeR. Bioinformatics 30(5): 614—620, 2014) (с применением установленных по умолчанию параметров). Поскольку все ампликоны имеют длину менее 70 оснований, со спаренными ридами 50 п.о., полученными с помощью Illumina HiSeq 2500, все целевые риды сливались с перекрыванием минимум 30 п.о. Риды в несобранном виде являются нецелевыми, и их отфильтровывали на этой стадии. Ампликоны разрабатывали таким образом, что целевые положения SNV были размещены в перекрывающемся участке. Основания, которые не соответствовали в прямом и обратном ридах или которые имеют балла качества Phred менее 20, отфильтровывали для сведения к минимуму ошибок секвенирования на последующих стадиях. Слитые риды с качеством картирования выше 30 и самое большее одним несоответствием с последовательностью праймеров отмечали как целевые. Мишени с менее чем 1000 ридами рассматривали как не прошедшие испытание и отфильтровывали в дальнейших анализах. Контроль качества (QC) выполняли с применением внутрилабораторной проверки программы Java в отношении большого перечня статистических методов на образец, который предусматривал общее число ридов, картированные риды, целевые риды, число не прошедших испытание мишеней и средний уровень ошибок. Образец с менее чем 90% картированными ридами и более чем 3 не прошедшими испытания мишенями не проходил дальше и требовал повторного секвенирования.
[200] Статистическая модель. Процесс ПЦР моделировали как стохастический процесс, оценивающий параметры ошибки с применением набора из 29 контрольных образцов плазмы крови и обеспечивающий конечные распознавания SNV на целевых образцах с раком. Для каждого целевого SNV строили мишень-специфическую модель фоновой ошибки путем оценивания следующих параметров из контрольных образцов.
• Эффективность ПЦР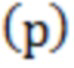 : вероятность того, что каждая молекула реплицировалась в цикле ПЦР.
: вероятность того, что каждая молекула реплицировалась в цикле ПЦР.
• Уровень ошибок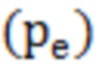 : частота ошибок на цикл для мутации типа e (например, соотношение аллеля A дикого типа и мутантного аллеля G).
: частота ошибок на цикл для мутации типа e (например, соотношение аллеля A дикого типа и мутантного аллеля G).
• Начальное число молекул 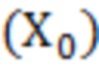
[201] Мишень-специфическую модель распространения ошибок применяли для определения характеристик распределения молекул с ошибкой. По мере того, как молекула реплицируется на протяжении процесса ПЦР, все больше ошибок происходит. Если ошибка происходит в цикле  и в системе находятся молекулы дикого типа
и в системе находятся молекулы дикого типа 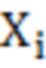 , эта молекула с ошибкой дублируется в следующем цикле с вероятностью
, эта молекула с ошибкой дублируется в следующем цикле с вероятностью 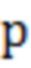 , и новые молекулы с ошибкой продуцируются из фоновых молекул дикого типа соответственно процессу с биномиальным распределением
, и новые молекулы с ошибкой продуцируются из фоновых молекул дикого типа соответственно процессу с биномиальным распределением . Используя рекурсивное соотношение, вычисляли среднее значение и дисперсию общего числа молекул
. Используя рекурсивное соотношение, вычисляли среднее значение и дисперсию общего числа молекул 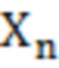 и числа молекул с ошибкой
и числа молекул с ошибкой  после
после 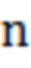 циклов ПЦР, как показано на ФИГ. 21.
циклов ПЦР, как показано на ФИГ. 21.
[202] Стадии алгоритма:
Оценивание эффективности ПЦР и уровня ошибок на цикл с применением нормальных контрольных образцов.
С применением оценки эффективности вычисление исходного числа молекул в тестовом наборе.
Применение данного исходного числа копий и предварительное распределение эффективности из обучающего набора для оценки эффективности ПЦР в тестовом образце.
Для диапазона значений и от 0 до 1 потенциальной фракции с реальными мутантами (используют 0,15 в качестве верхней границы) оценивают среднее значение и дисперсию для общего числа молекул, молекул с фоновой ошибкой и молекул с реальной мутацией с применением модели распространения ошибок, описанной в последнем разделе, и параметров, оцененных на стадиях a–c.
Применение среднего значения и дисперсии, оцененных на стадии d для вычисления вероятности L(и), в отношении фракции каждого потенциального реального мутанта. Отбор значения и, которое сводит к максимуму данную вероятность, (обозначенную как 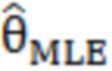 ) и вычисление балла достоверности (как
) и вычисление балла достоверности (как 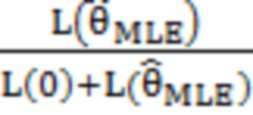 ).
).
Распознавание мутации, если балл достоверности составляет ≥≥95% для транзиций и ≥98% для трансверсий.
| название | год | авторы | номер документа |
|---|---|---|---|
| СПОСОБЫ ВЫЯВЛЕНИЯ И МОНИТОРИНГА РАКА ПУТЕМ ПЕРСОНАЛИЗИРОВАННОГО ВЫЯВЛЕНИЯ ЦИРКУЛИРУЮЩЕЙ ОПУХОЛЕВОЙ ДНК | 2019 |
|
RU2811503C2 |
| ОБНАРУЖЕНИЕ МУТАЦИЙ И ПЛОИДНОСТИ В ХРОМОСОМНЫХ СЕГМЕНТАХ | 2015 |
|
RU2717641C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ВЫСОКОМУЛЬТИПЛЕКСНОЙ ПЦР | 2012 |
|
RU2650790C2 |
| СПОСОБЫ НЕИНВАЗИВНОГО ПРЕНАТАЛЬНОГО УСТАНОВЛЕНИЯ ПЛОИДНОСТИ | 2011 |
|
RU2671980C2 |
| СПОСОБЫ АМПЛИФИКАЦИИ ДНК ДЛЯ СОХРАНЕНИЯ СТАТУСА МЕТИЛИРОВАНИЯ | 2018 |
|
RU2754038C2 |
| СПОСОБЫ НЕИНВАЗИВНОГО ПРЕНАТАЛЬНОГО УСТАНОВЛЕНИЯ ОТЦОВСТВА | 2011 |
|
RU2620959C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ДНК-ПРОФИЛИРОВАНИЯ | 2015 |
|
RU2708337C2 |
| ПАНЕЛЬ БИОМАРКЕРОВ И СПОСОБЫ ВЫЯВЛЕНИЯ МИКРОСАТЕЛЛИТНОЙ НЕСТАБИЛЬНОСТИ ПРИ РАЗНЫХ ВИДАХ РАКА | 2019 |
|
RU2795410C2 |
| ПОЛИНУКЛЕОТИДНЫЕ ПРАЙМЕРЫ | 2008 |
|
RU2491289C2 |
| ПРЯМОЙ ЗАХВАТ, АМПЛИФИКАЦИЯ И СЕКВЕНИРОВАНИЕ ДНК-МИШЕНИ С ИСПОЛЬЗОВАНИЕМ ИММОБИЛИЗИРОВАННЫХ ПРАЙМЕРОВ | 2011 |
|
RU2565550C2 |


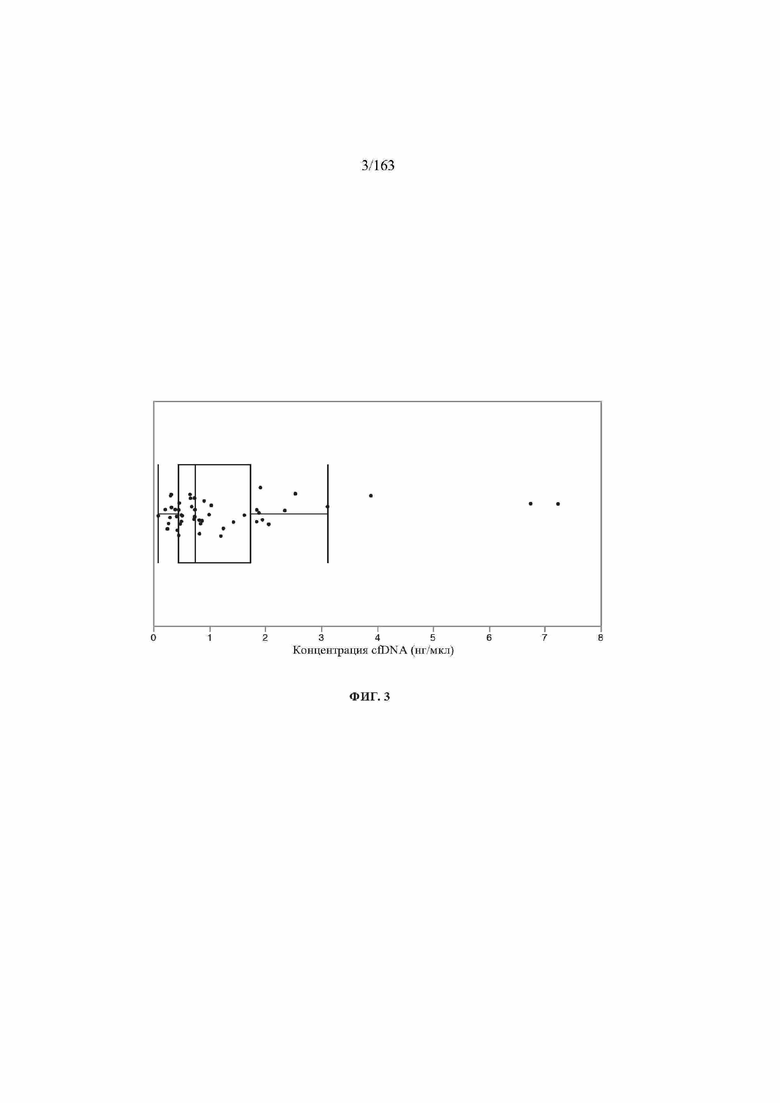






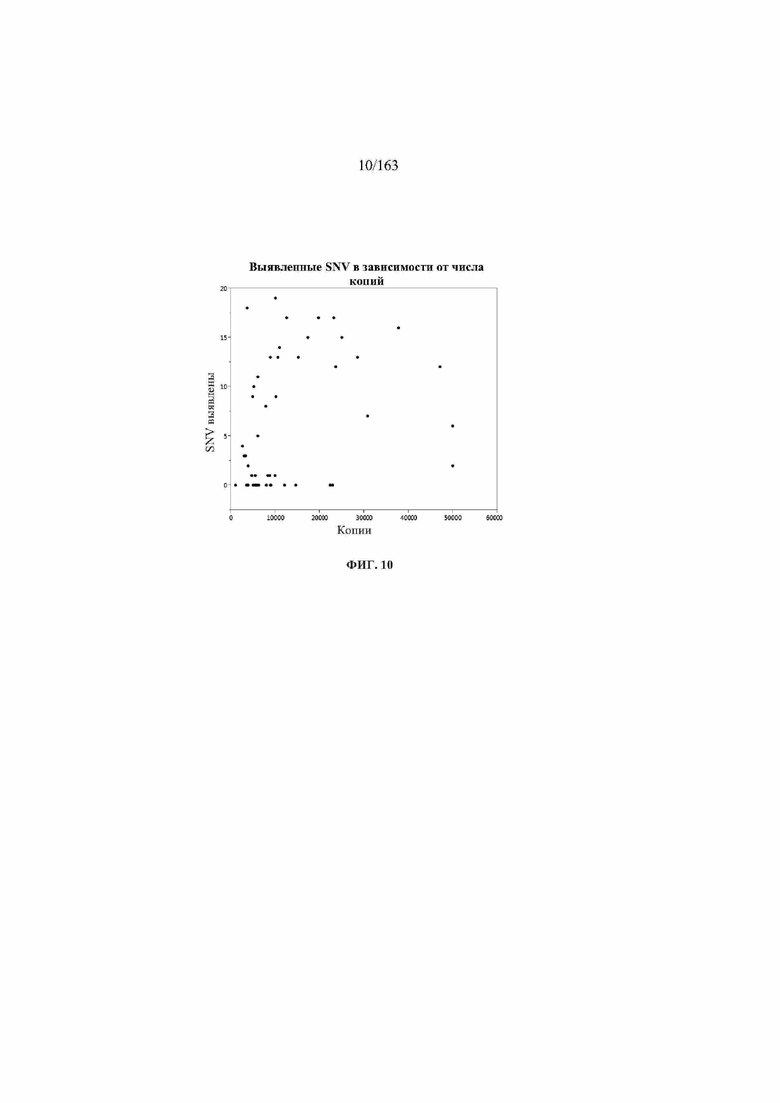
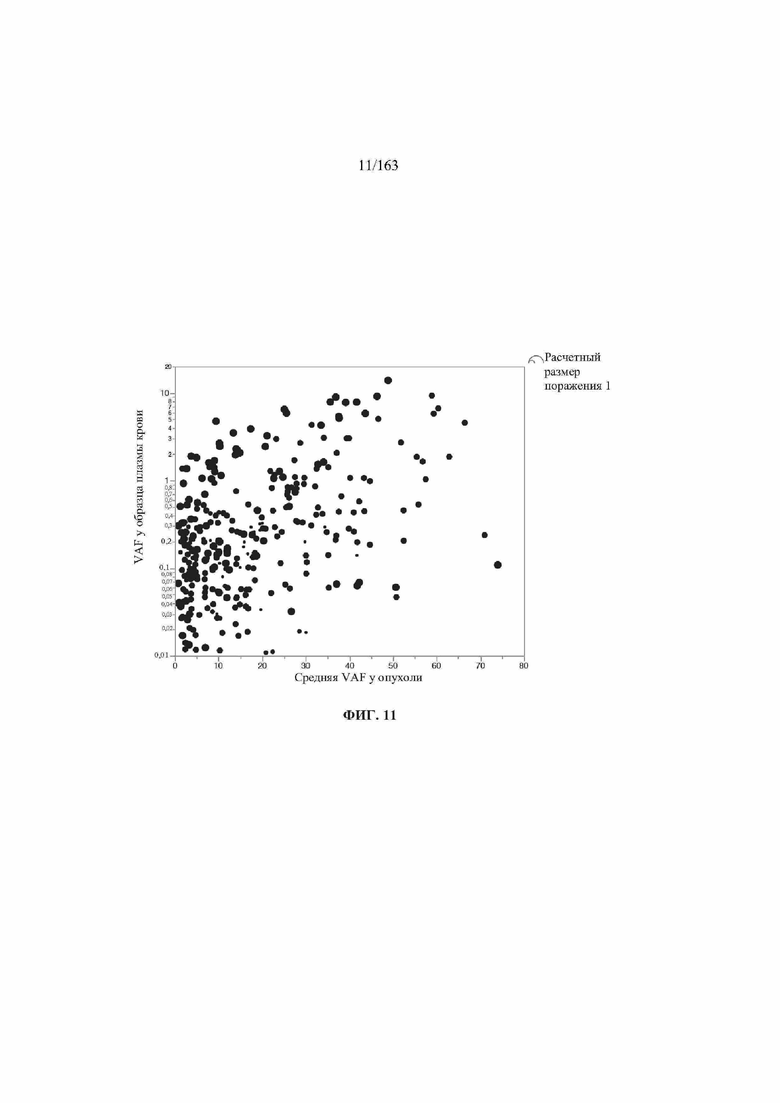




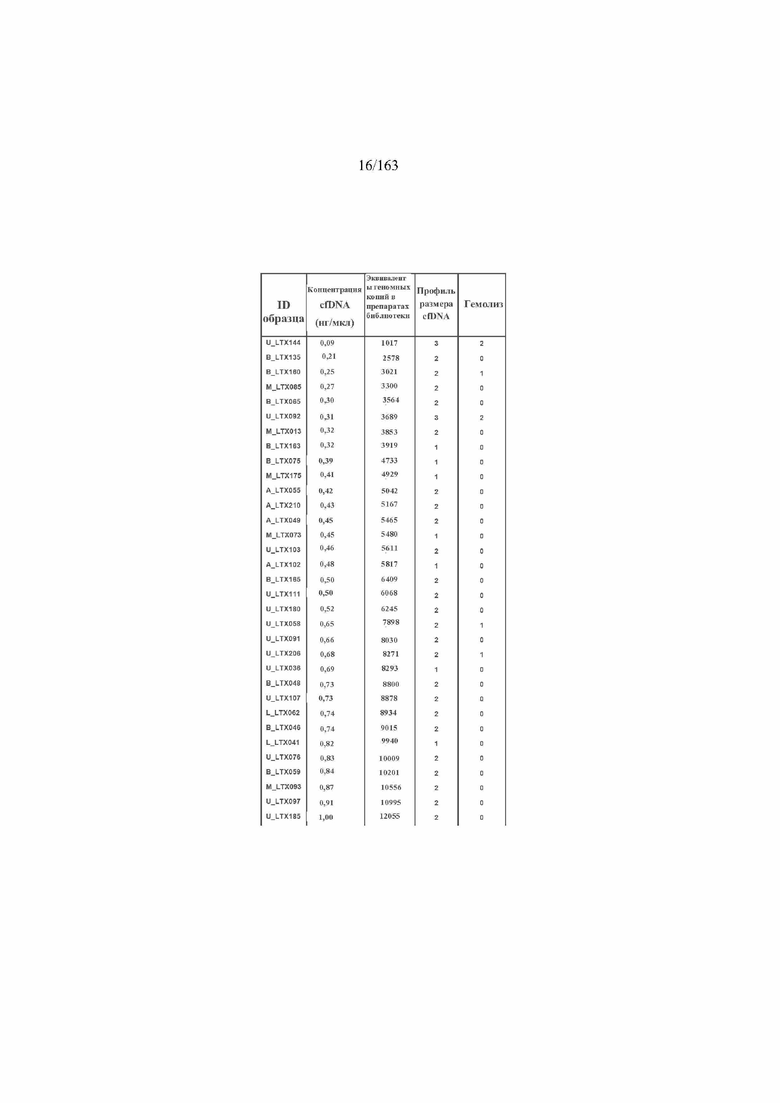


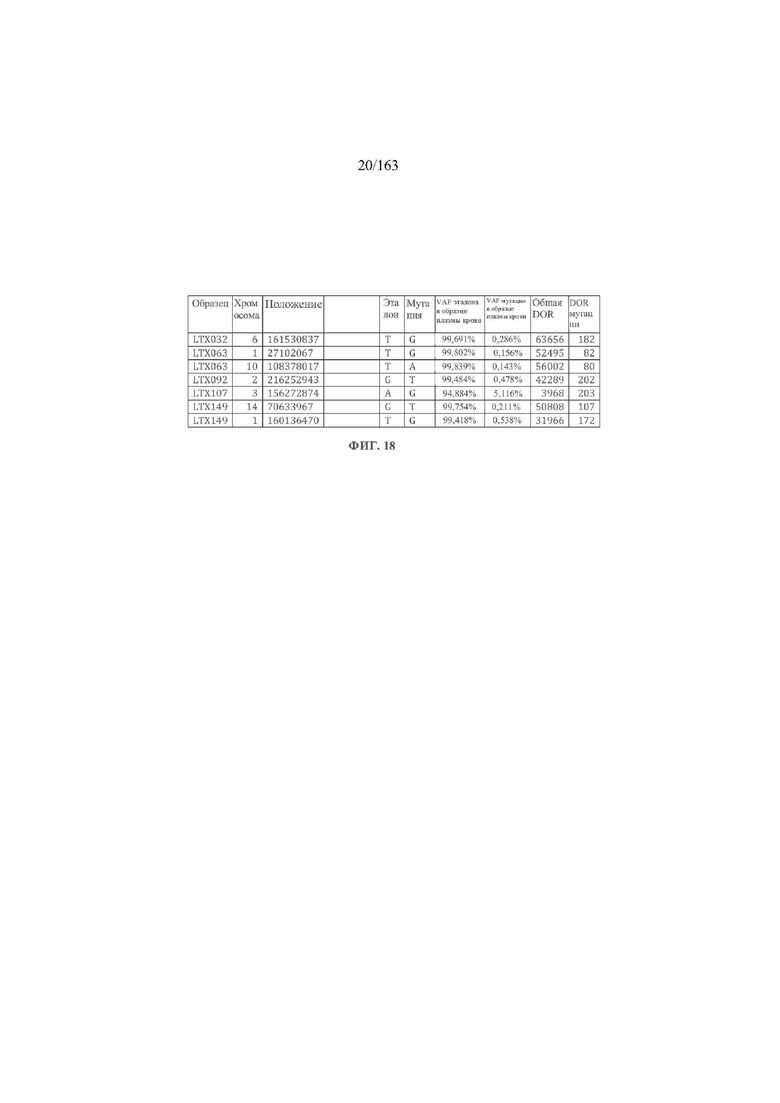
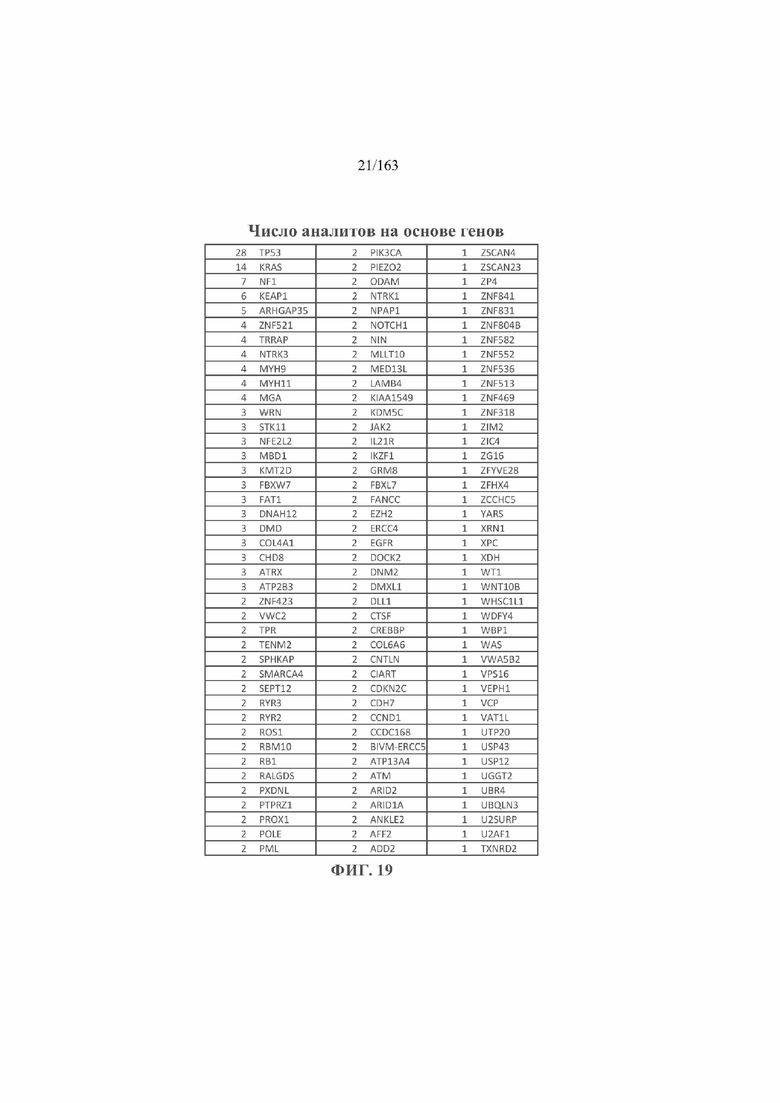

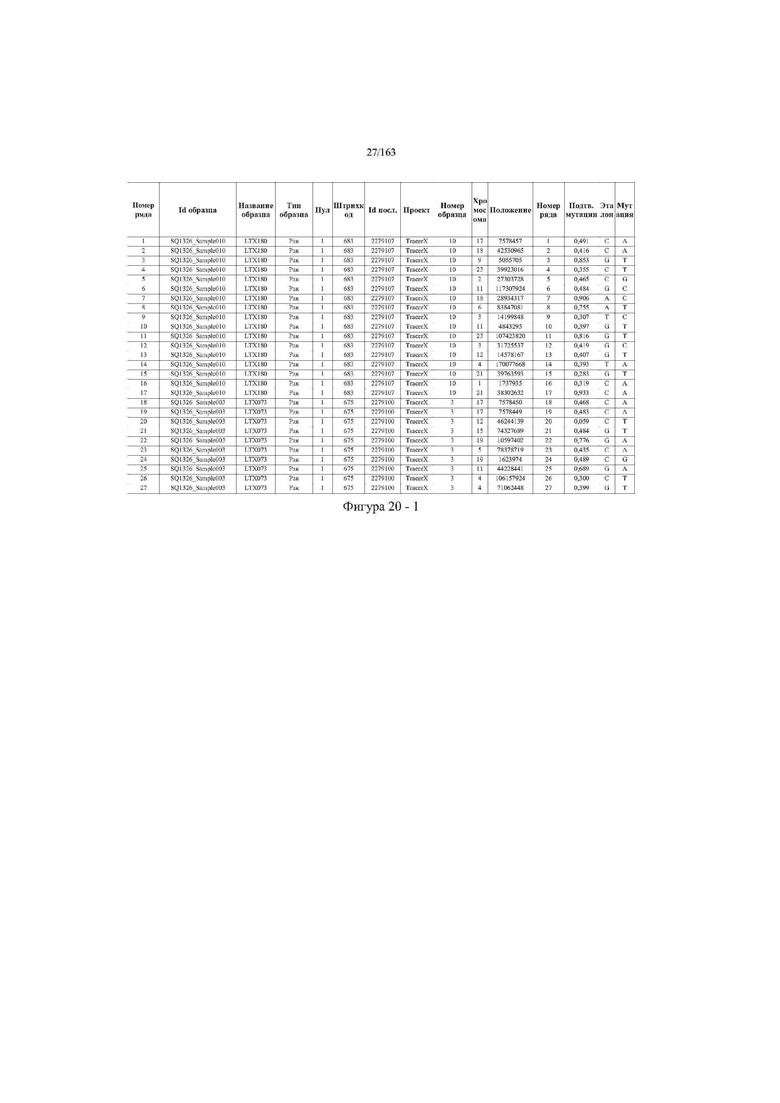
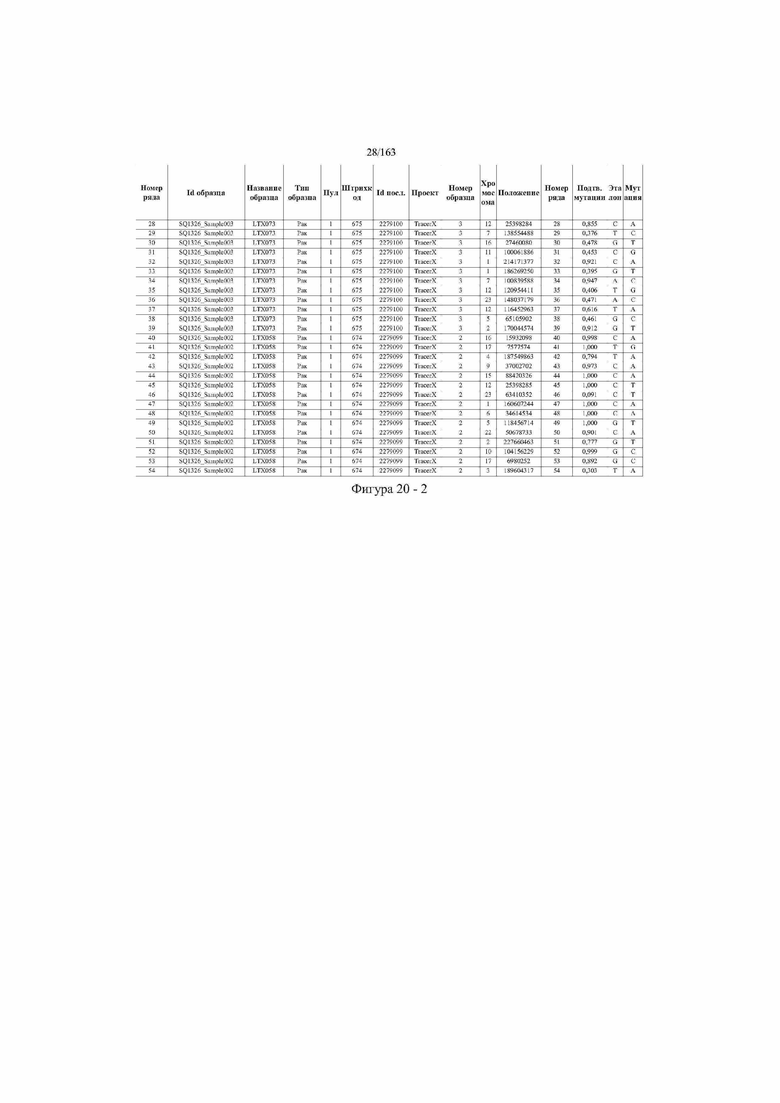
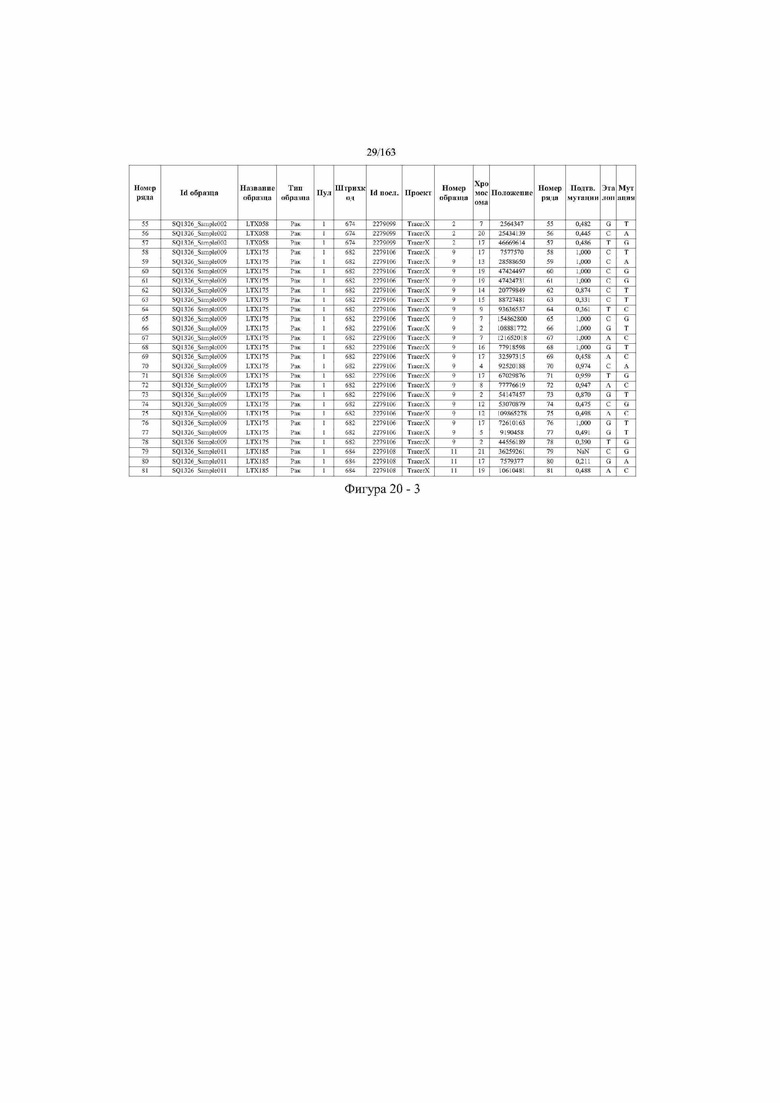
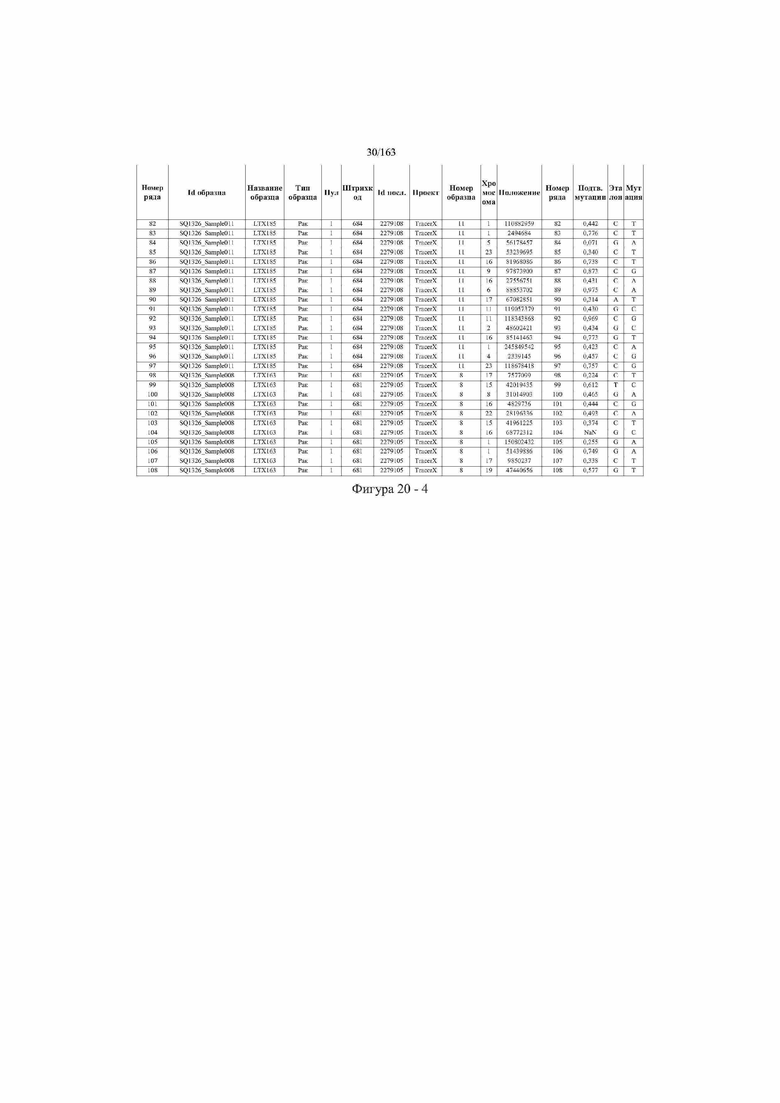
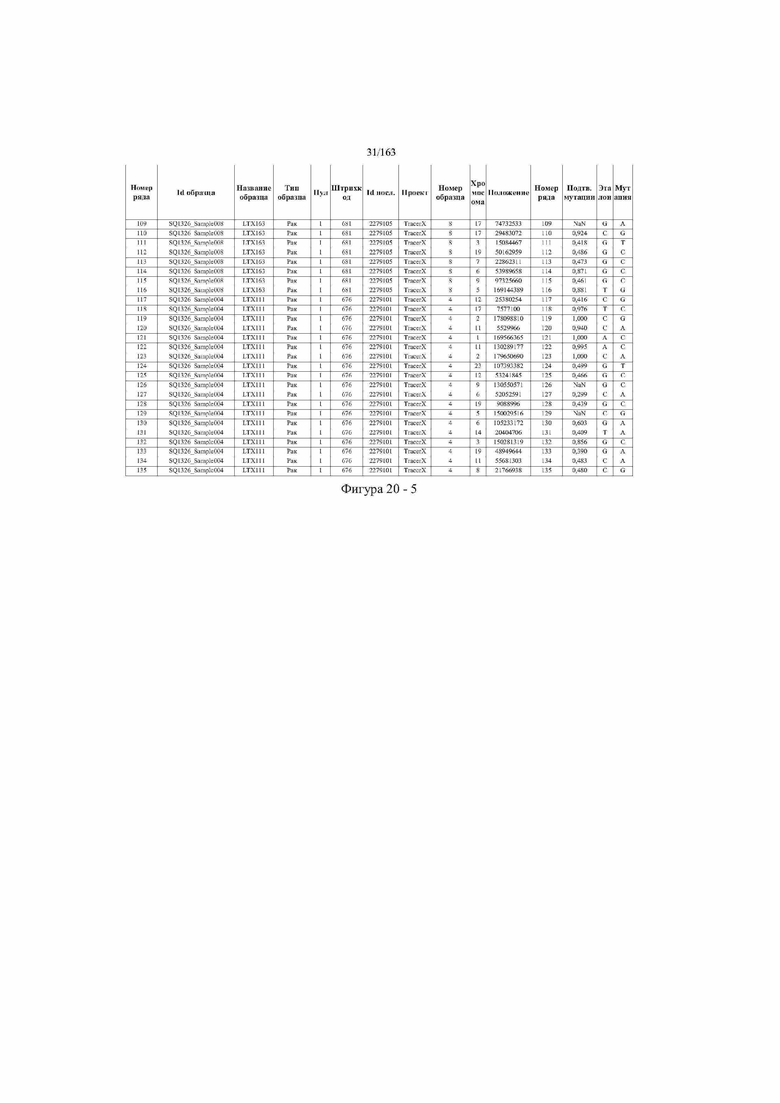
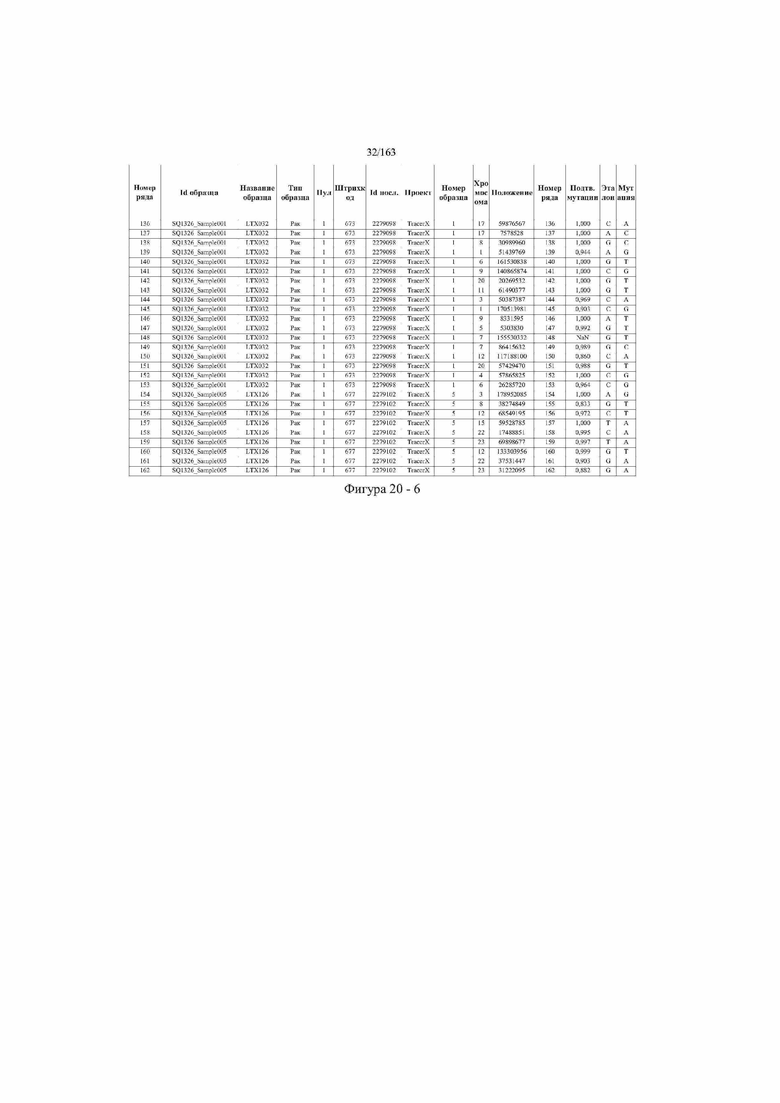

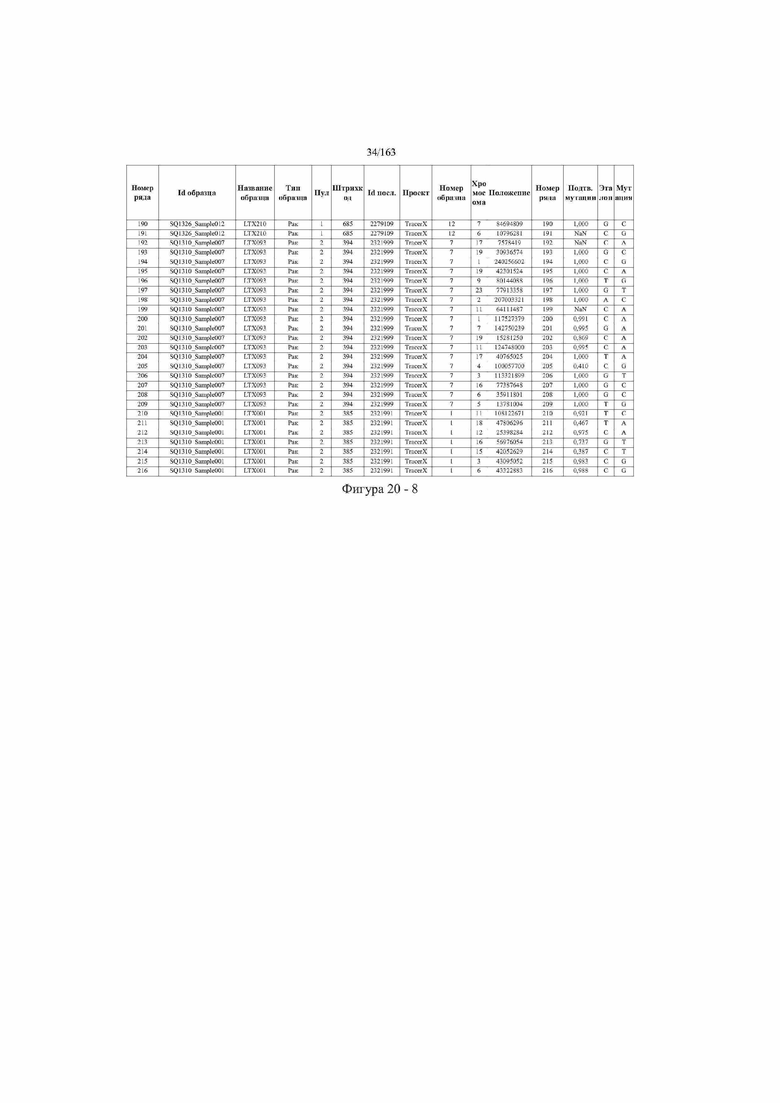
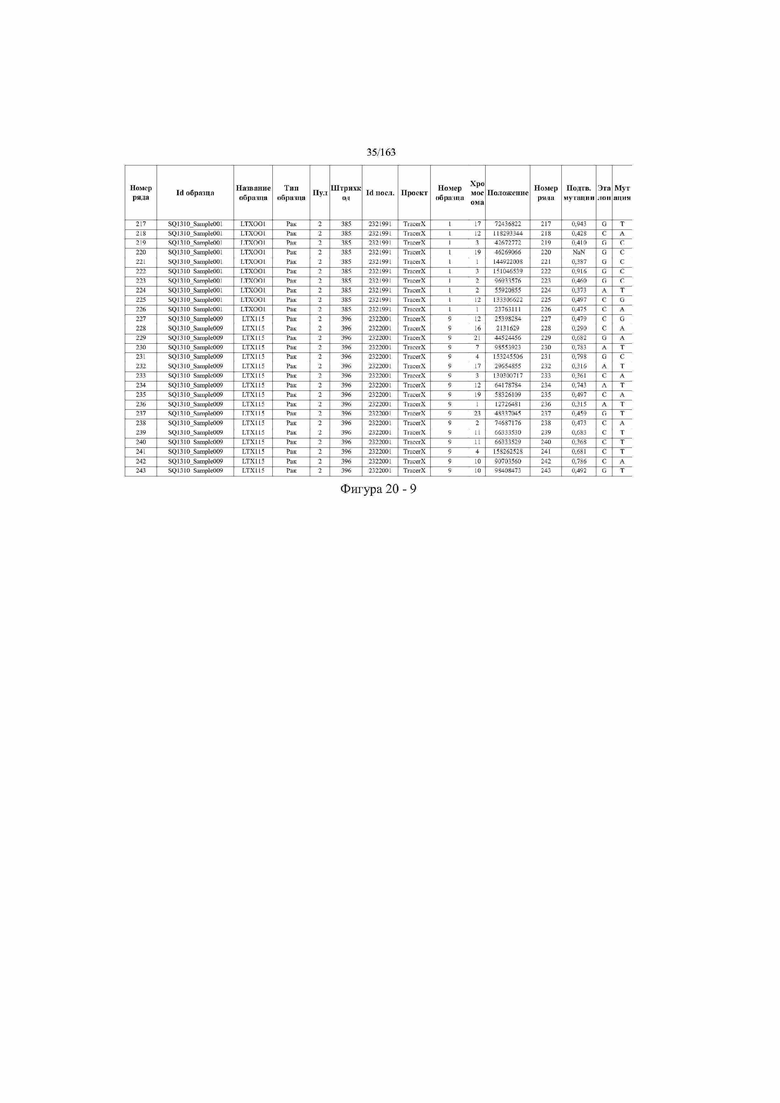
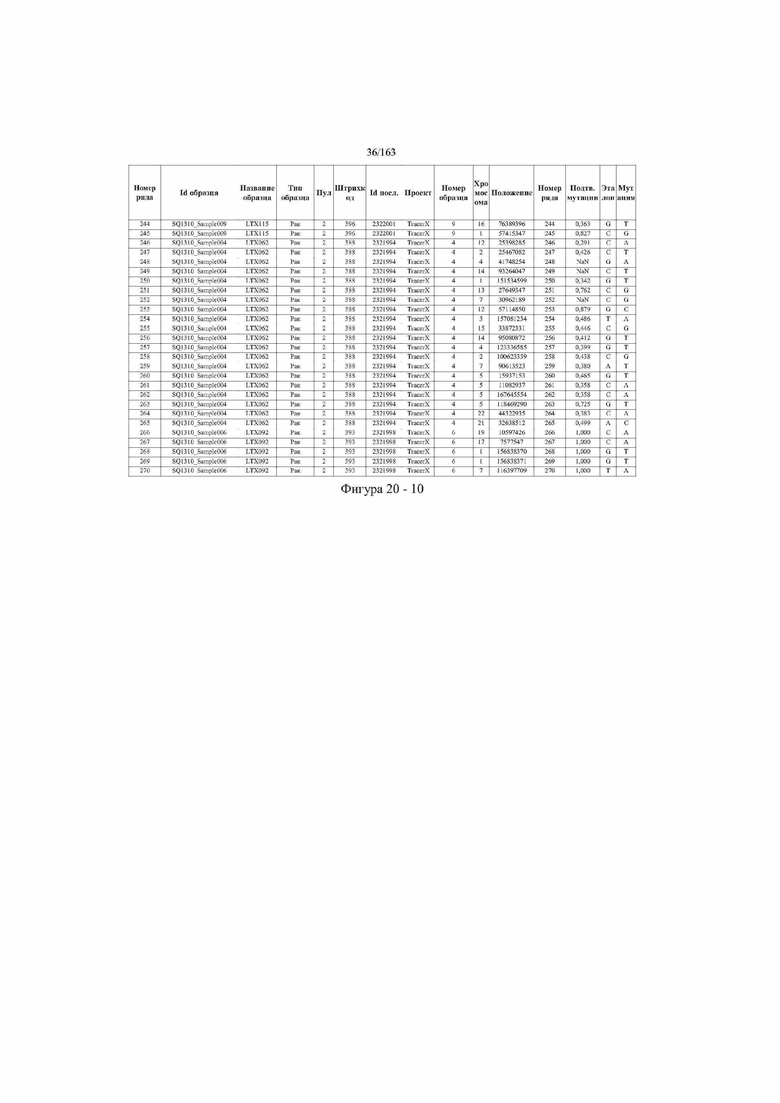
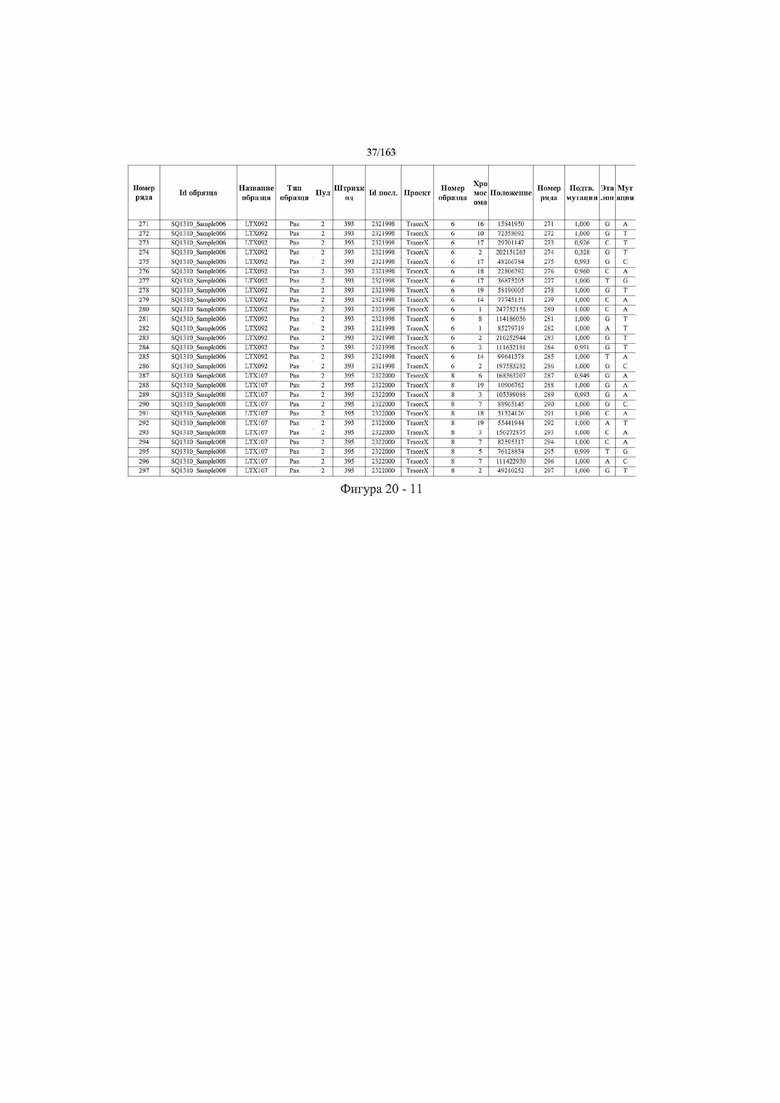
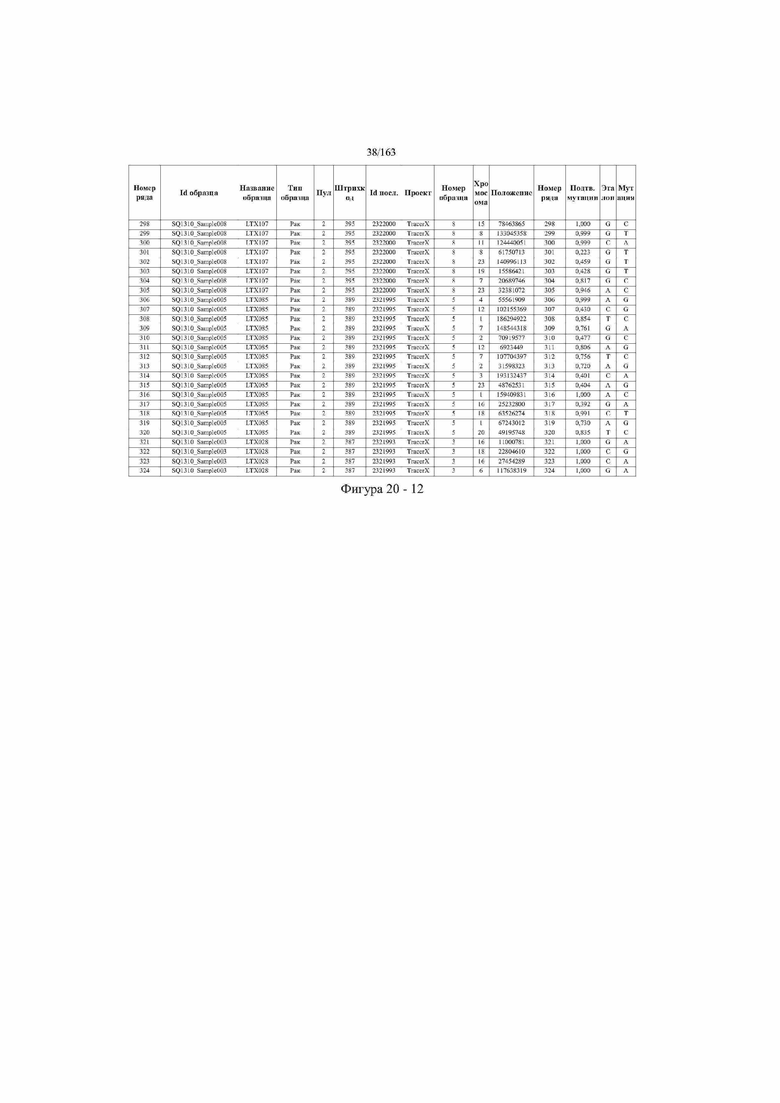
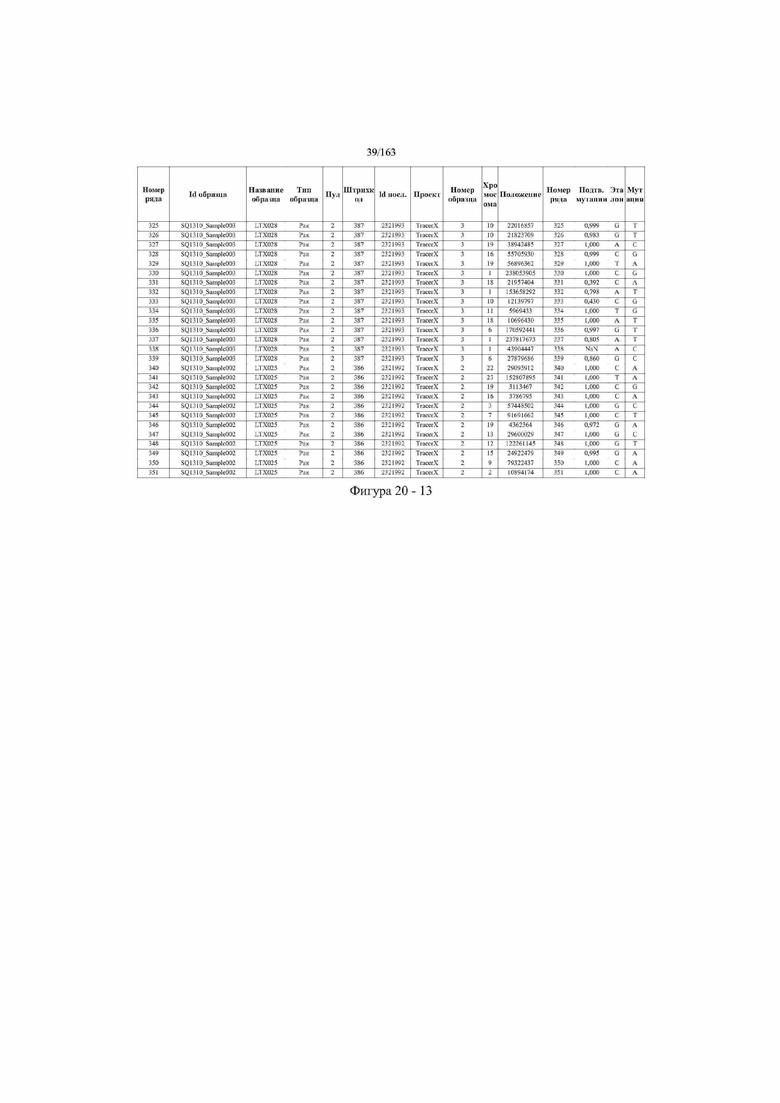
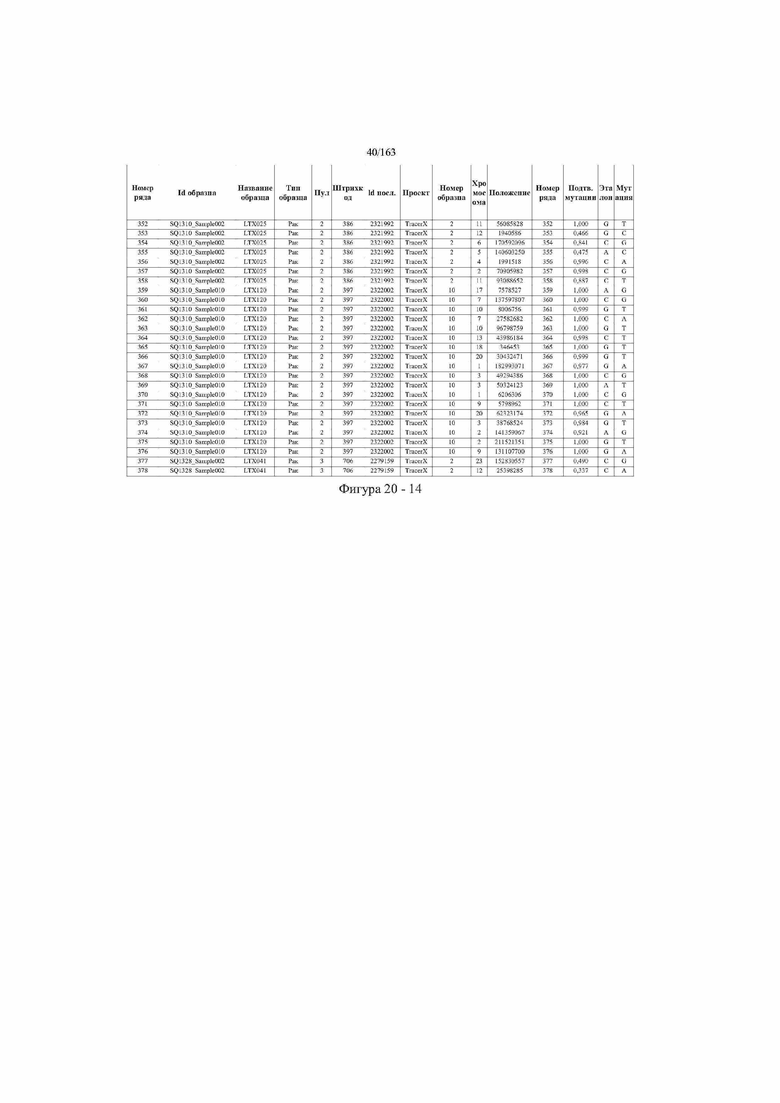
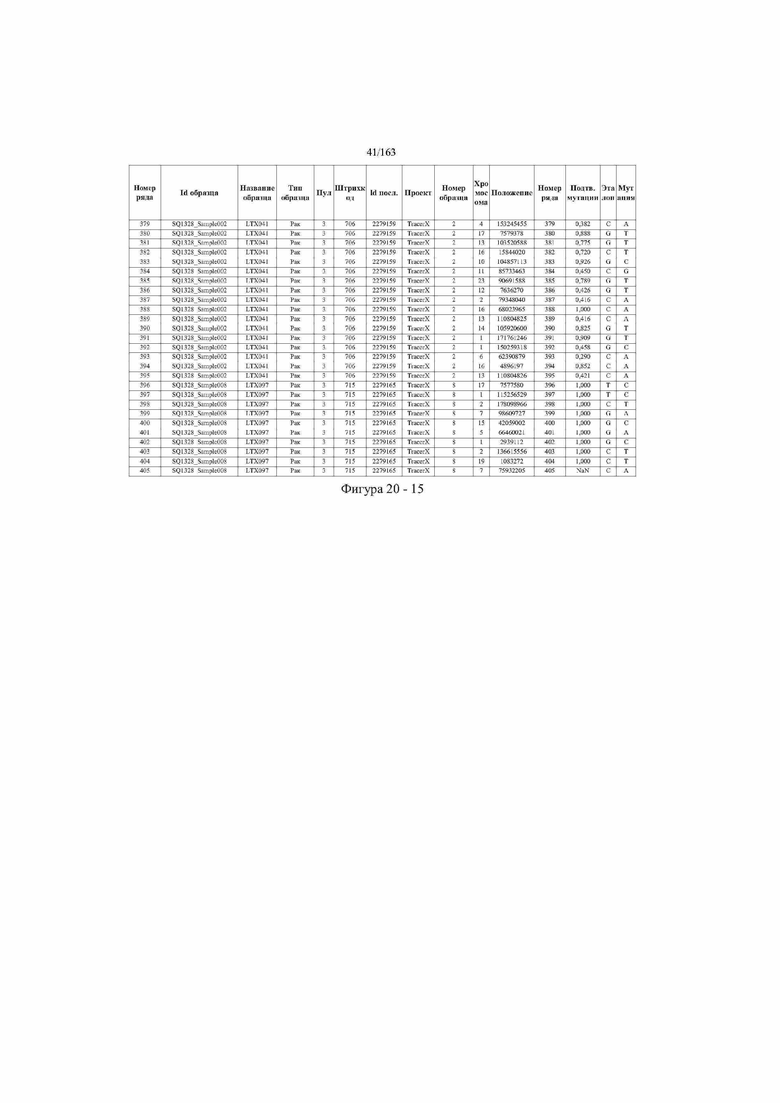
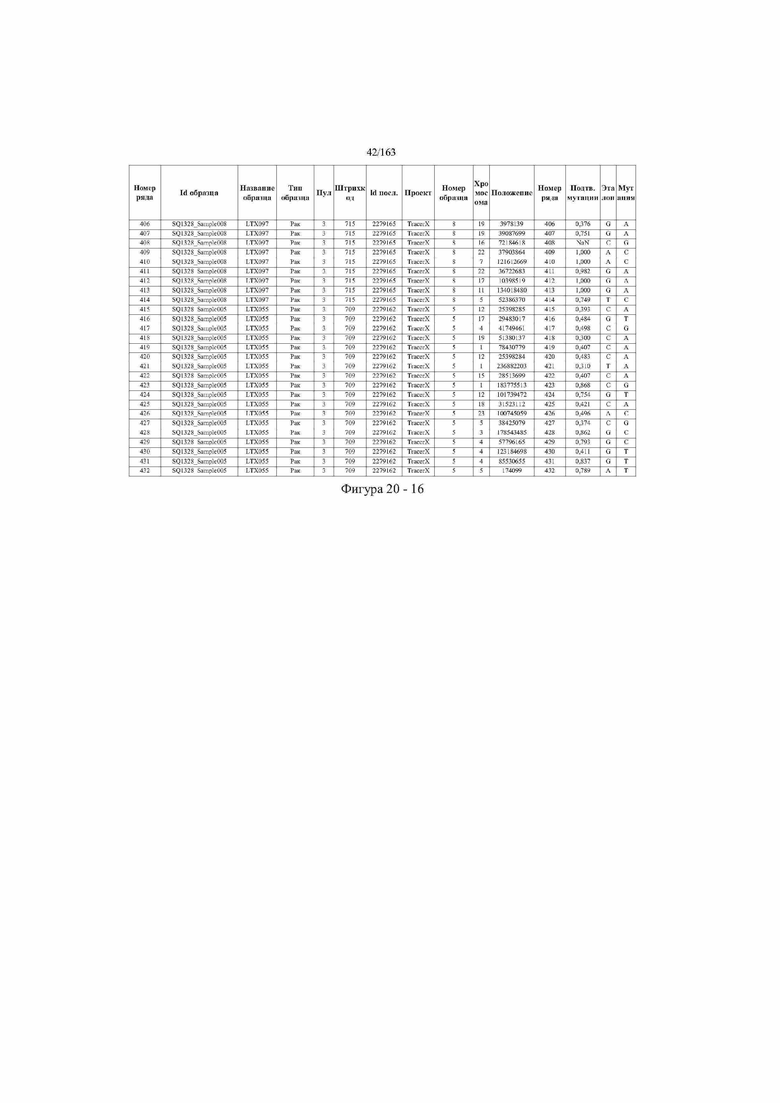
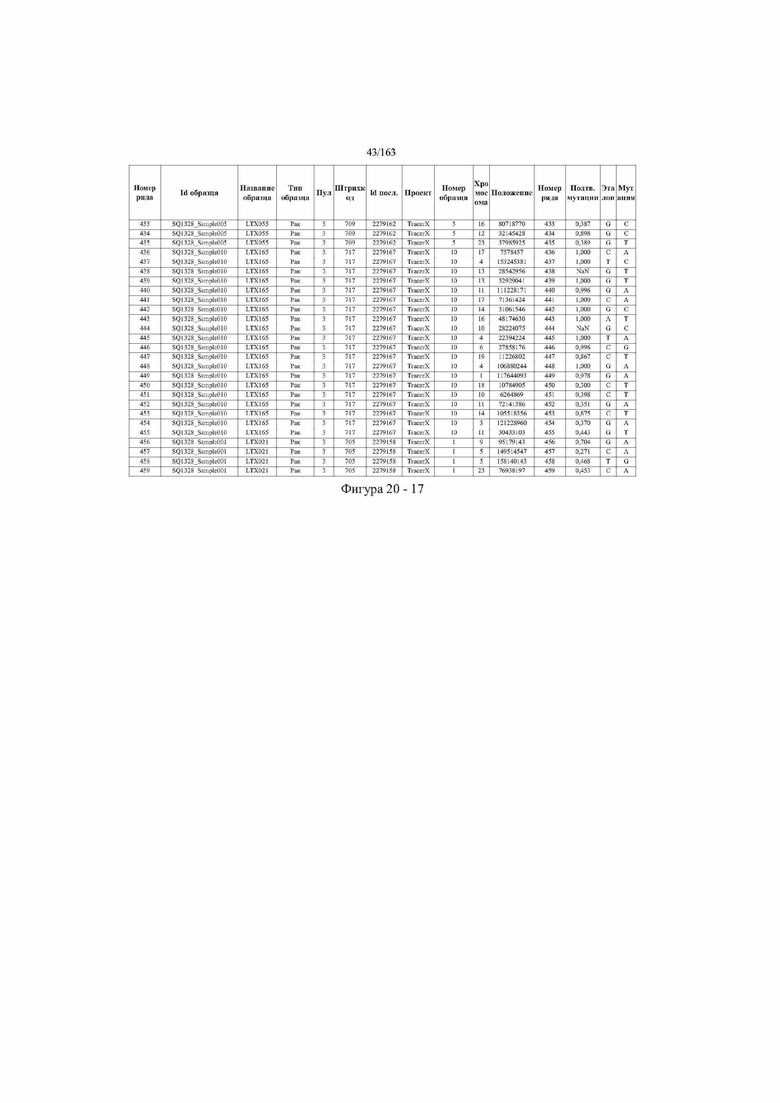
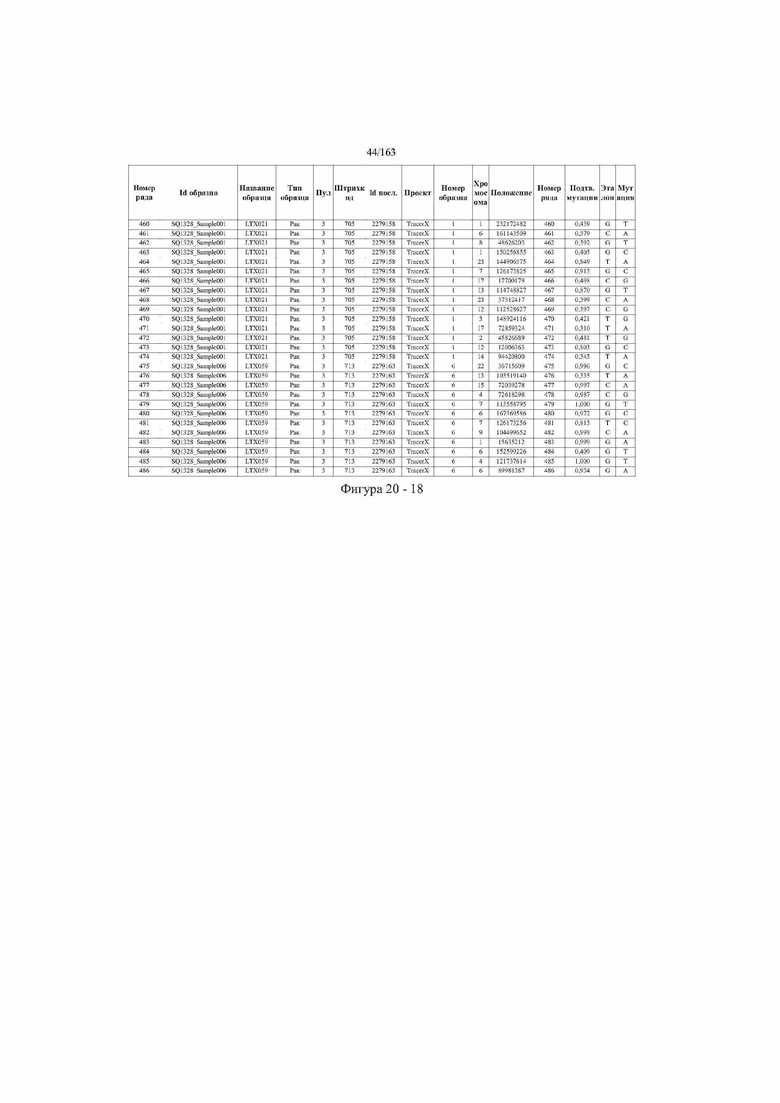
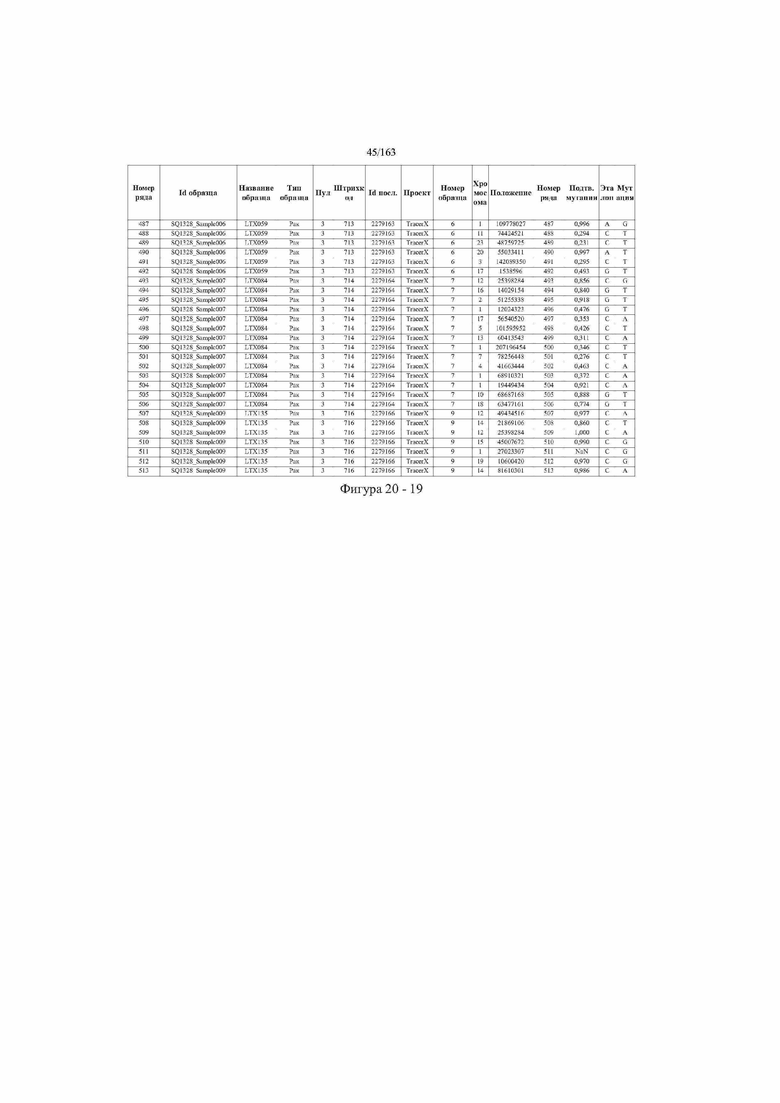

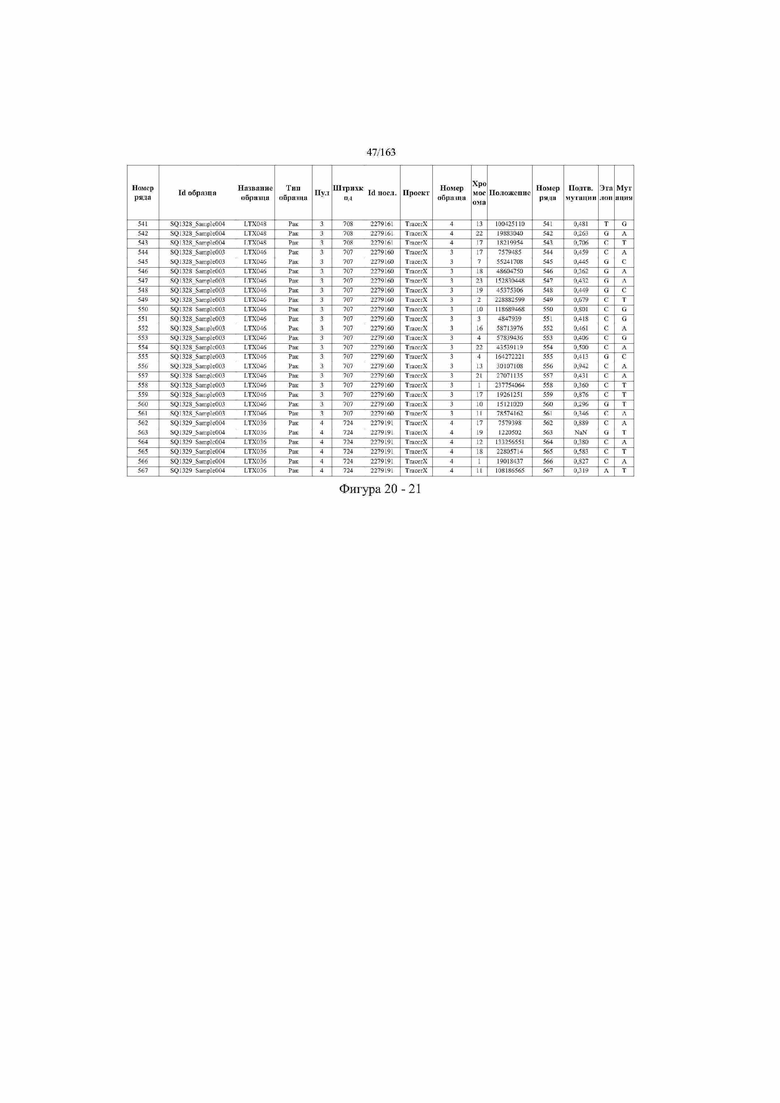
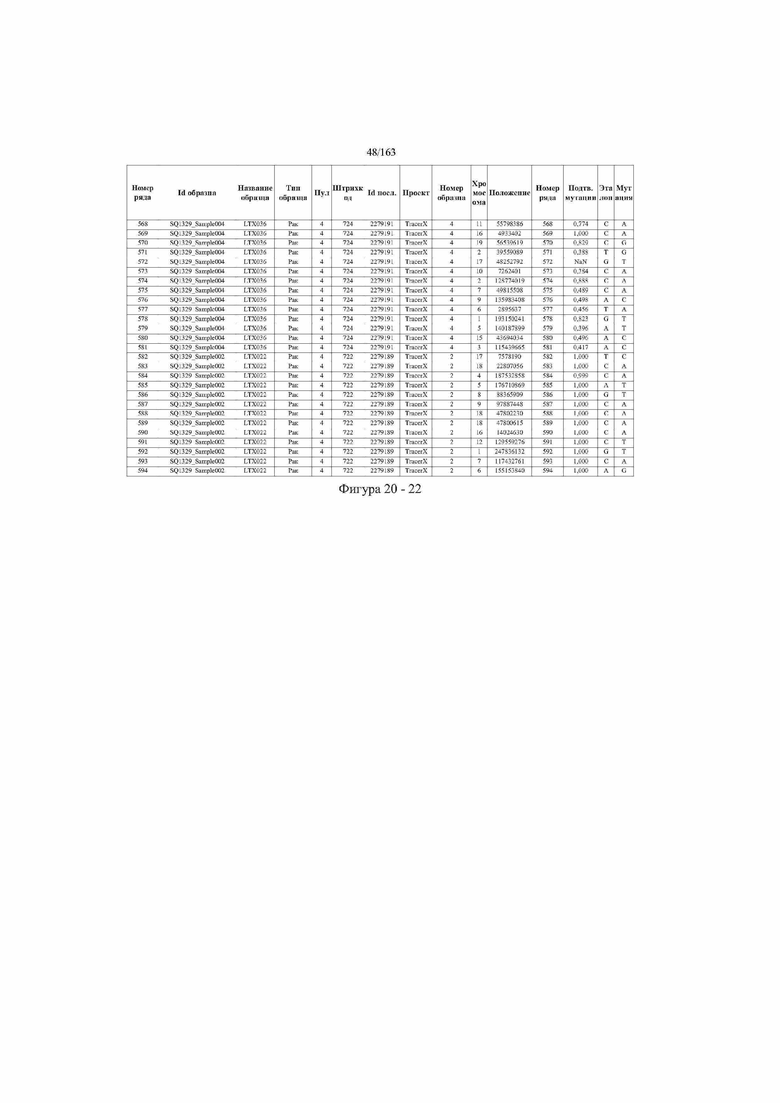
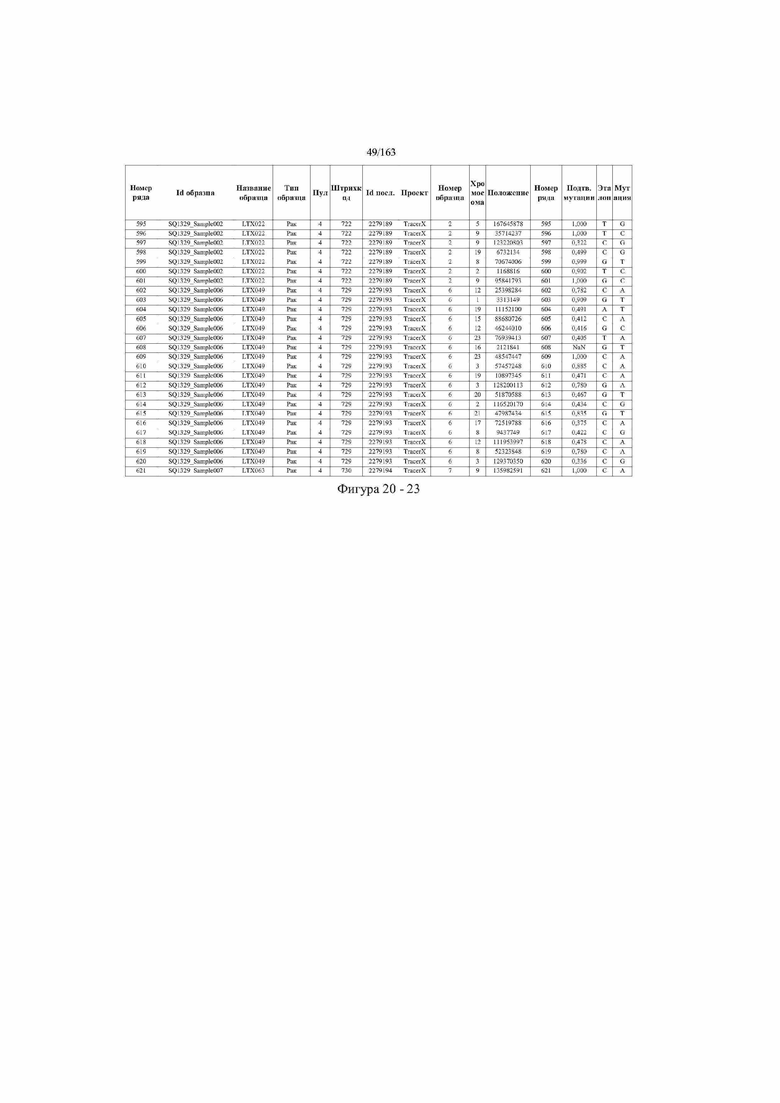
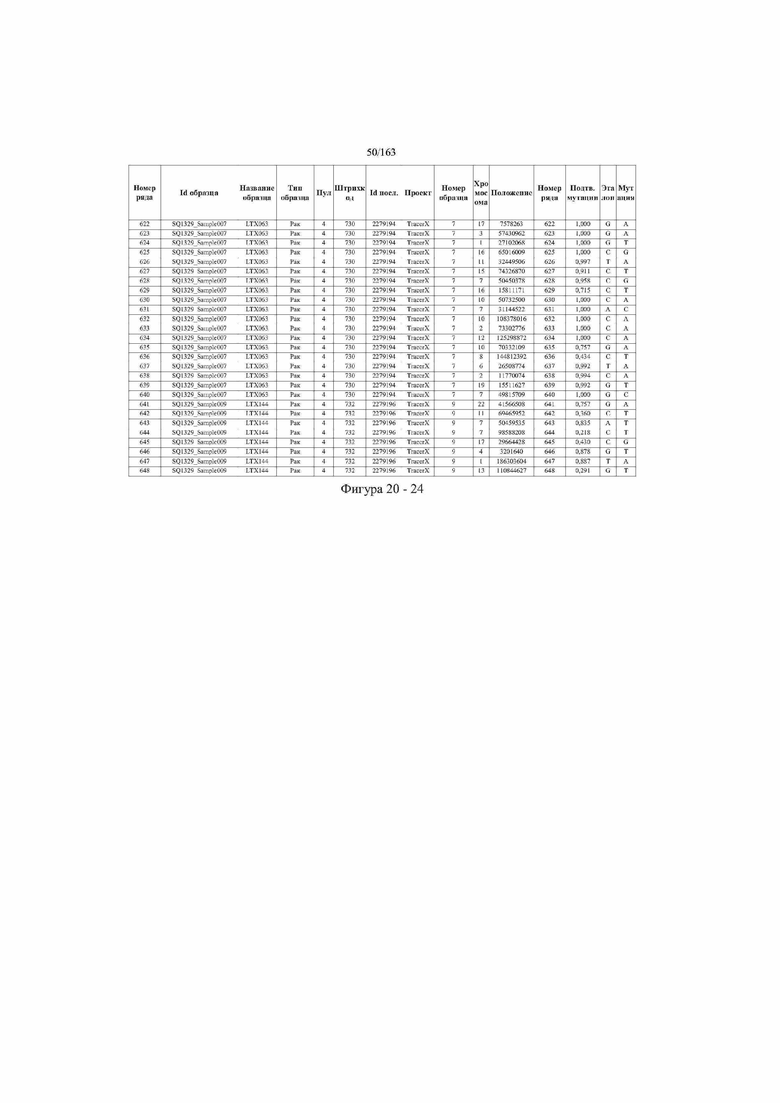
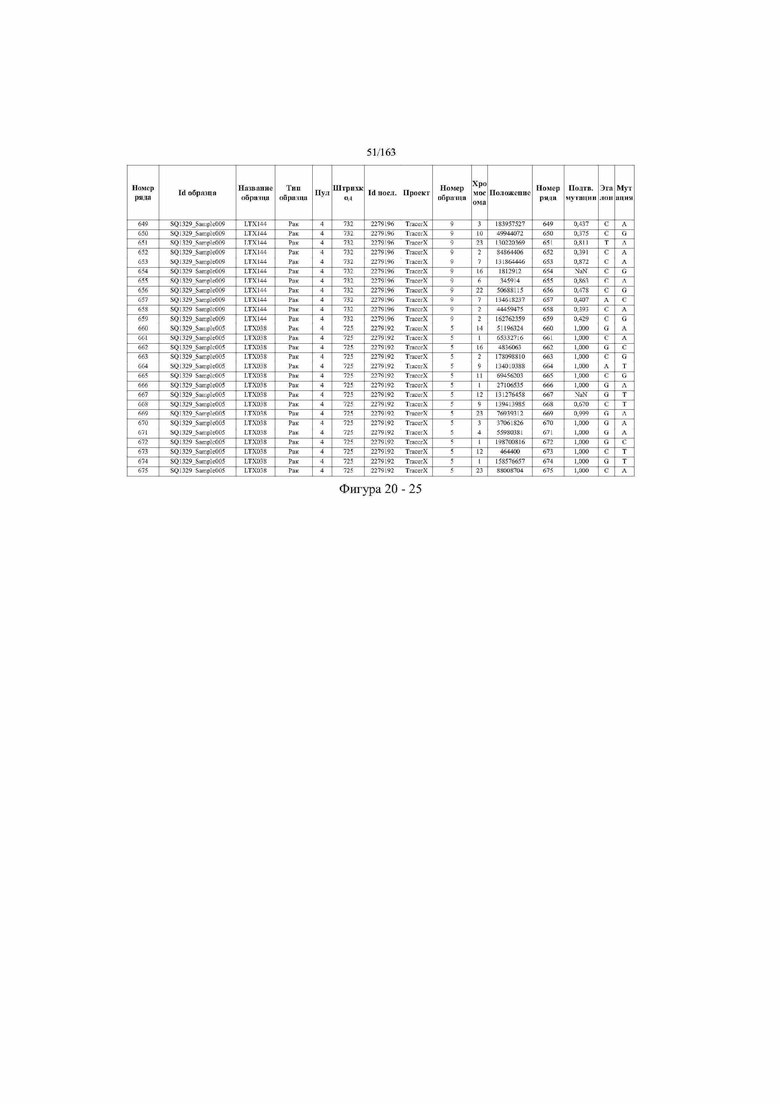
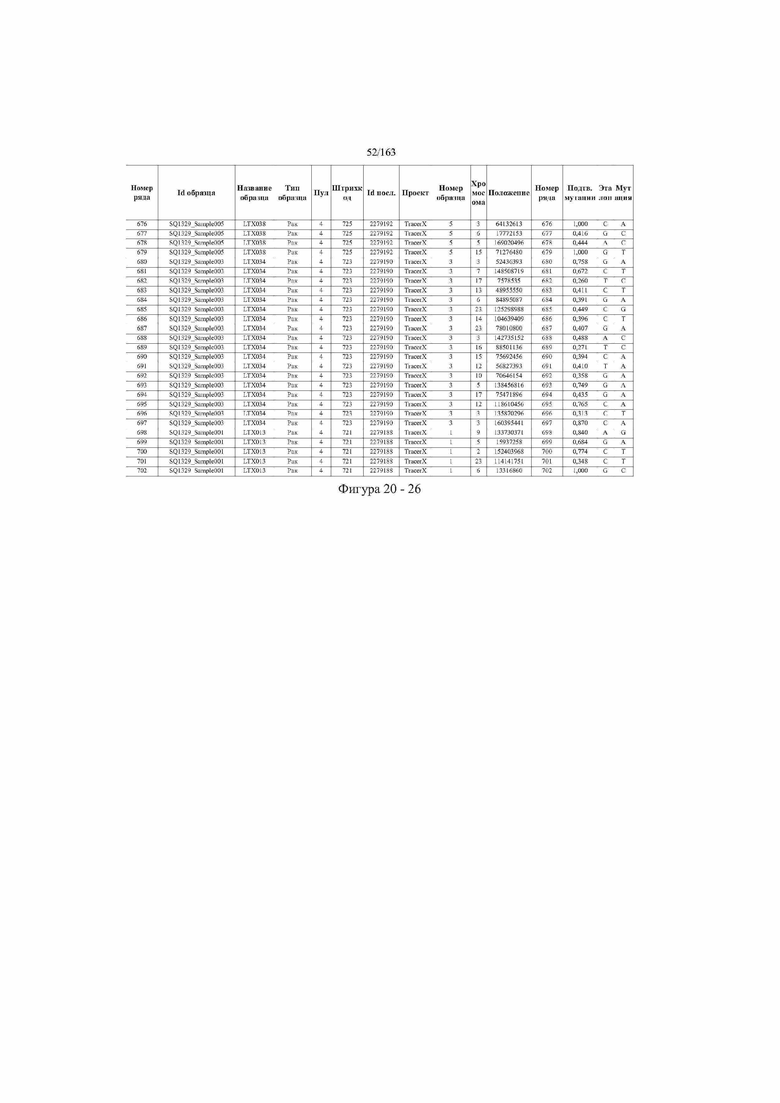
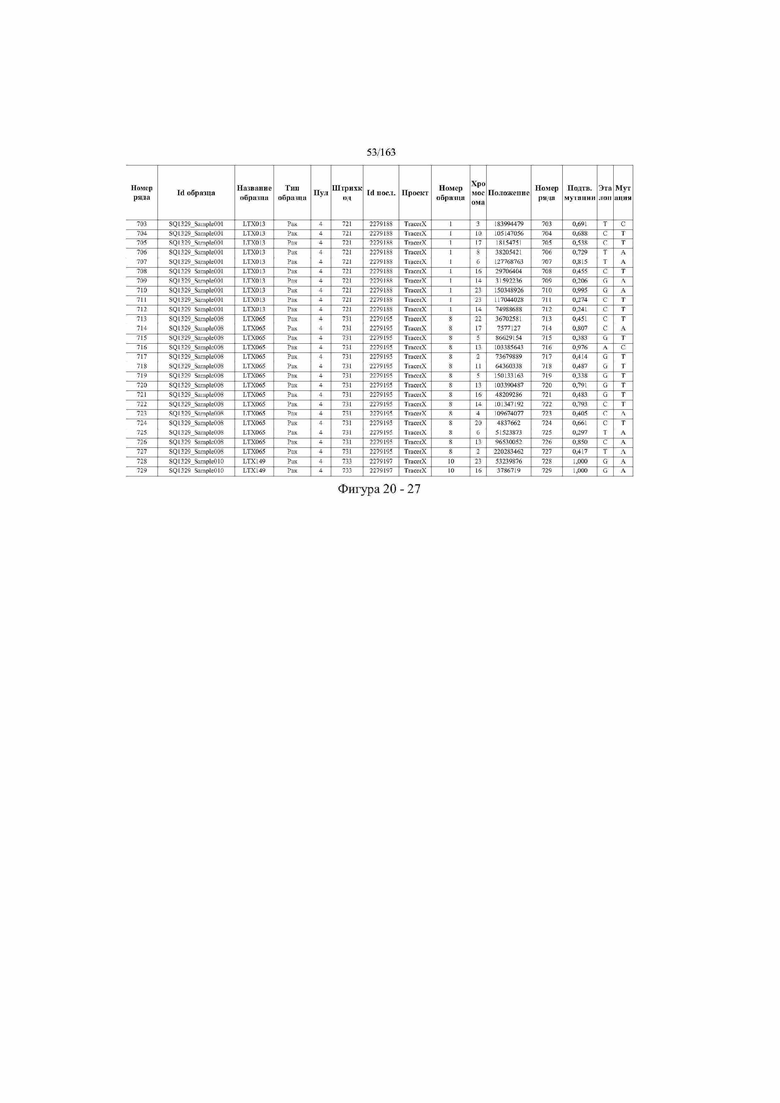
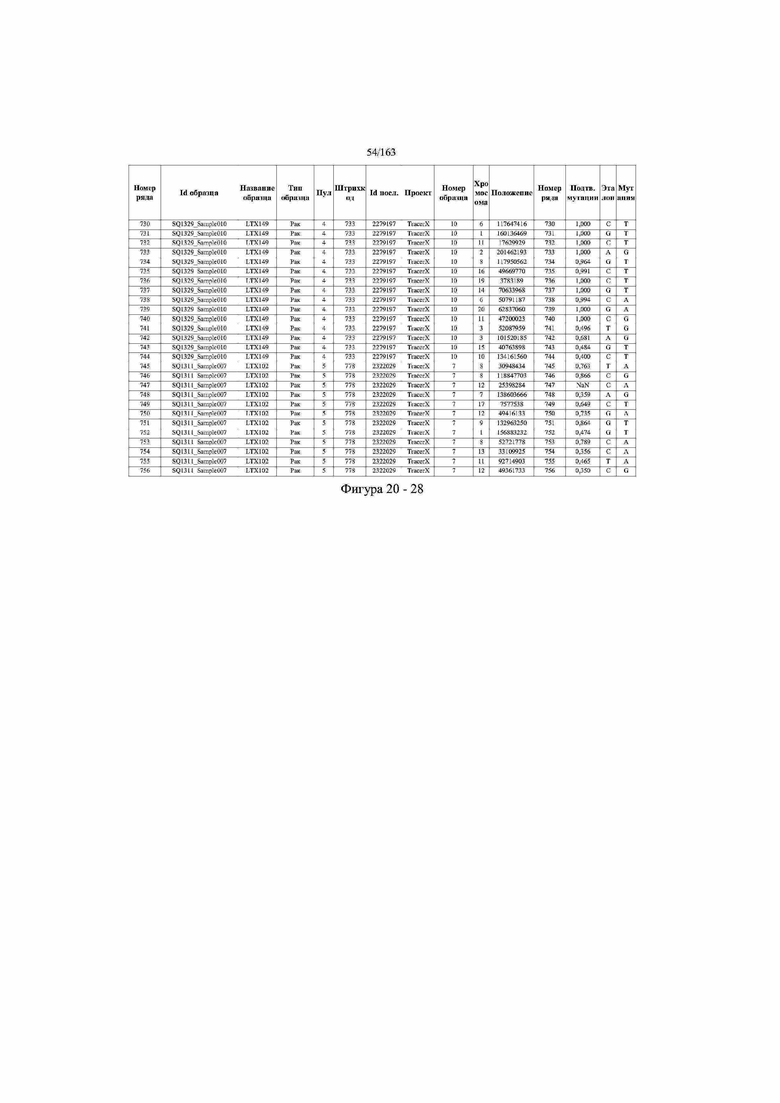

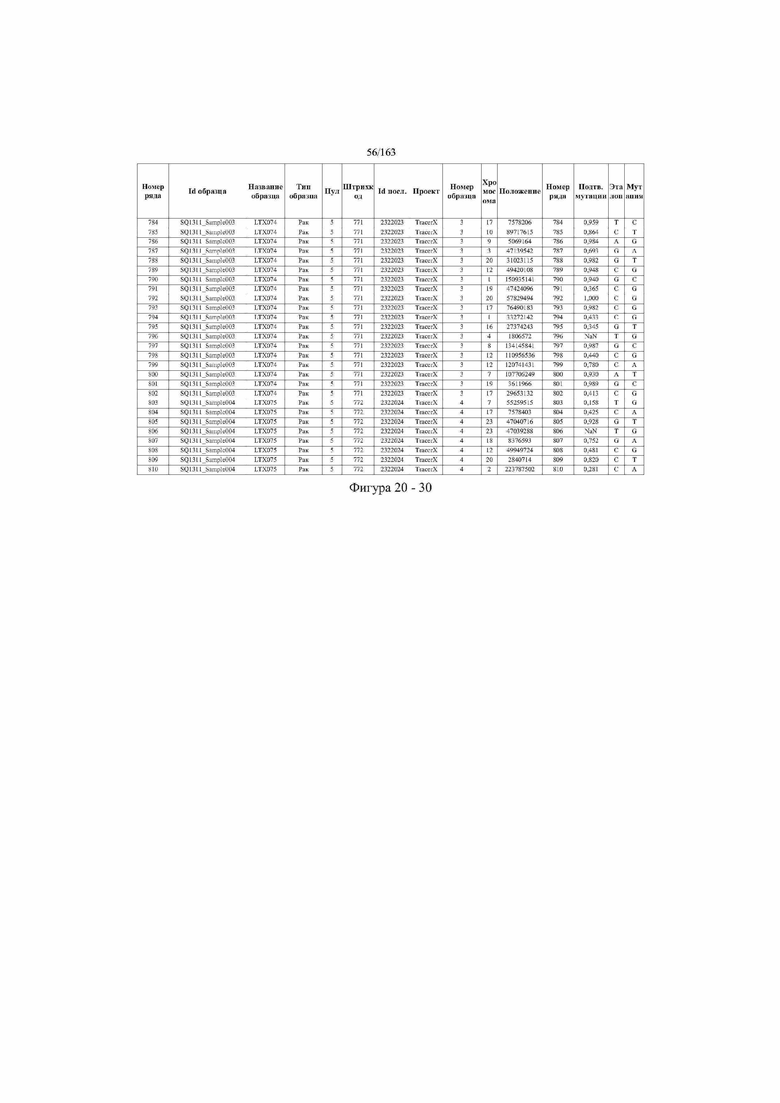

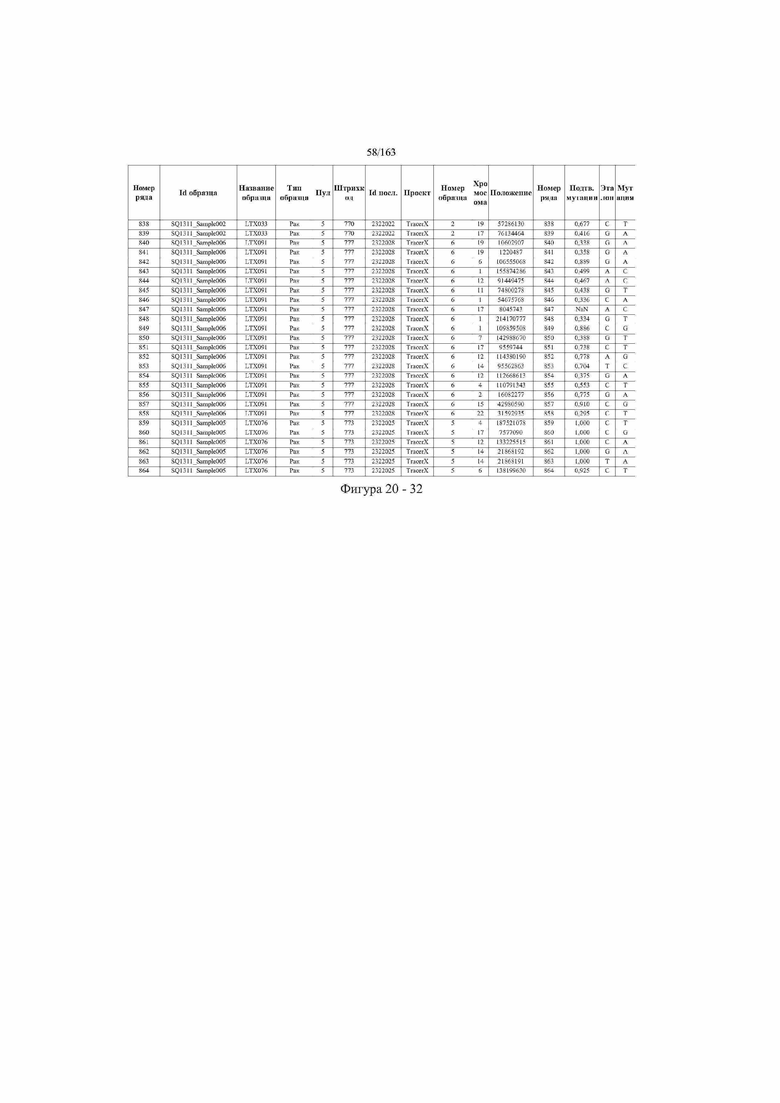
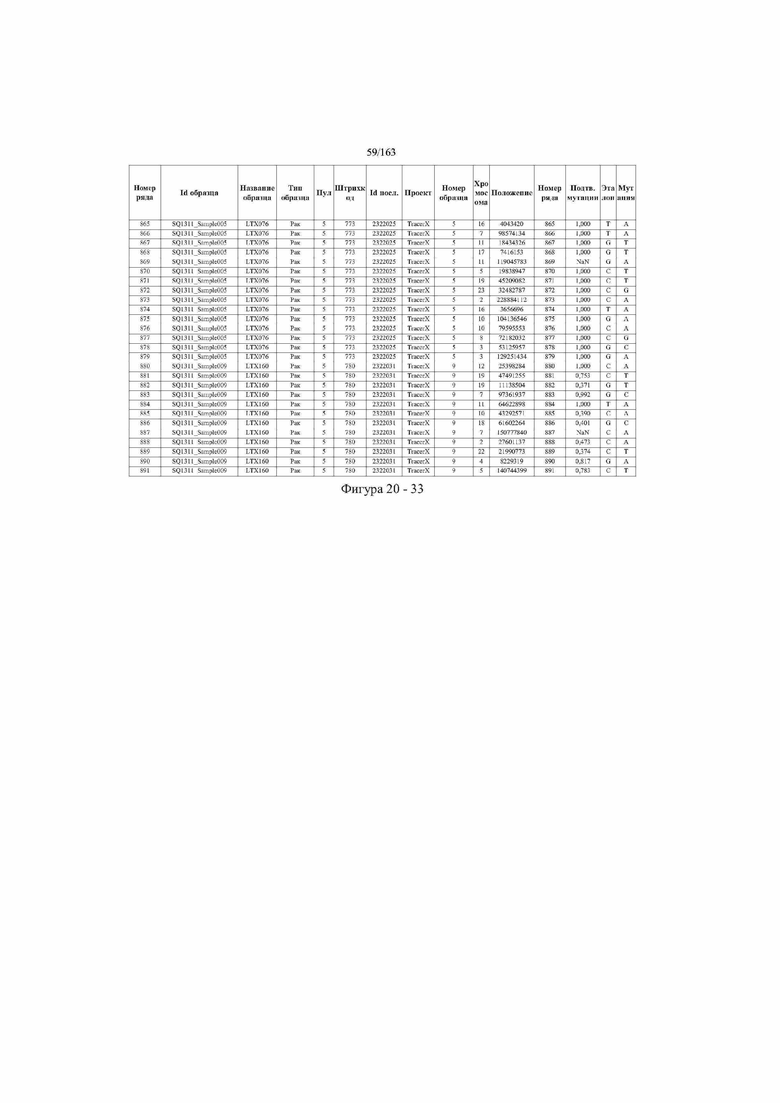
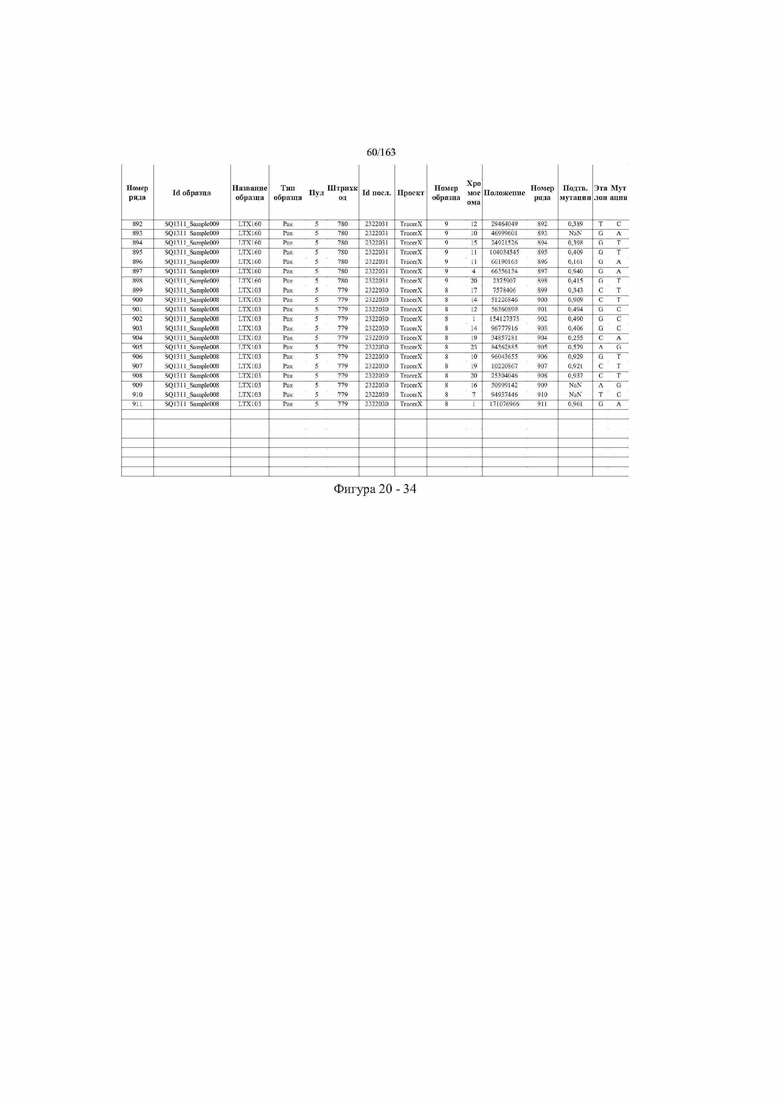
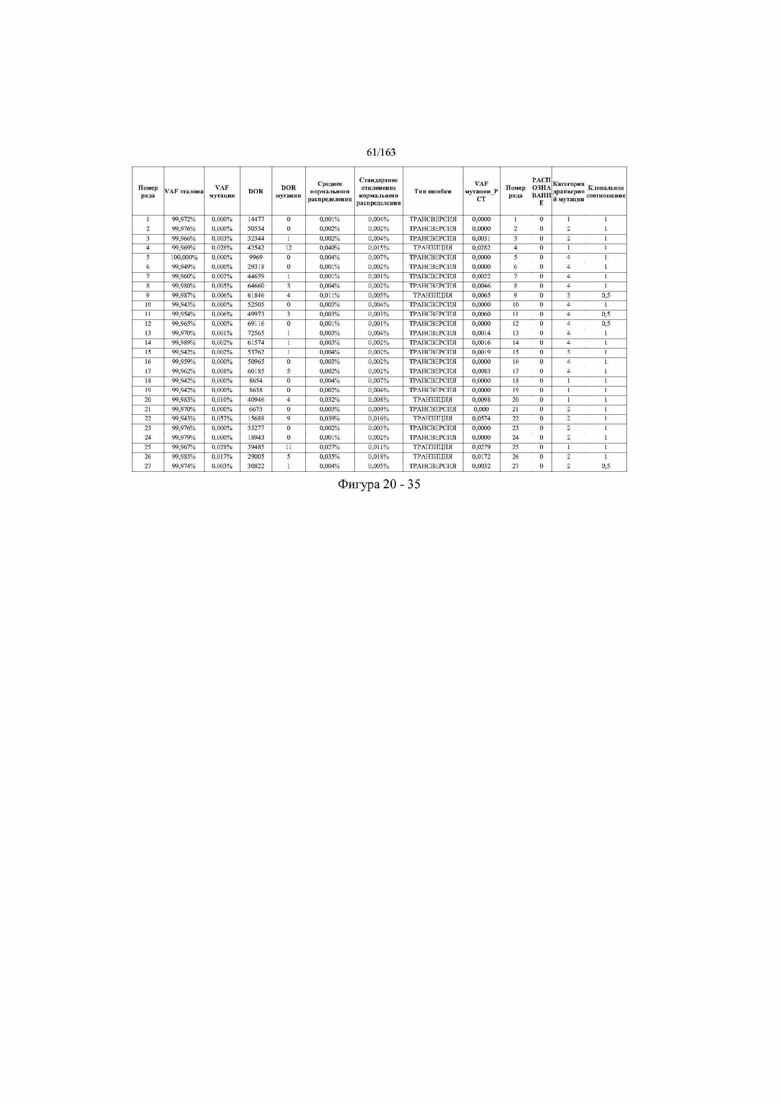

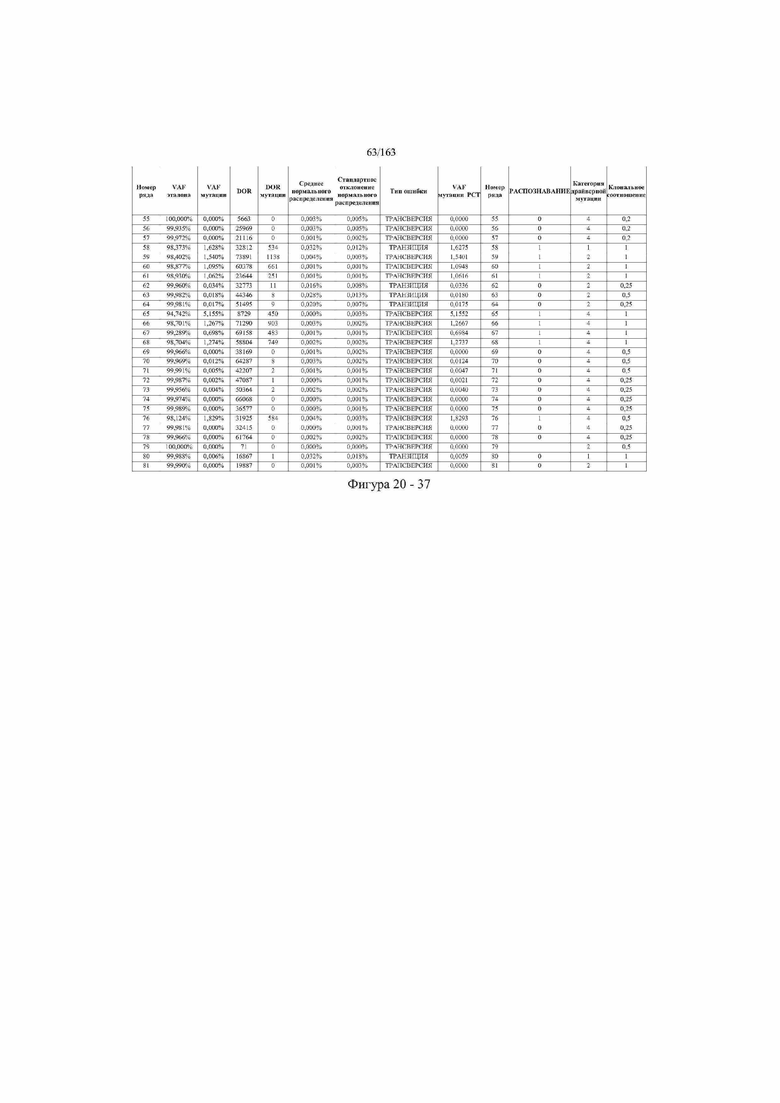
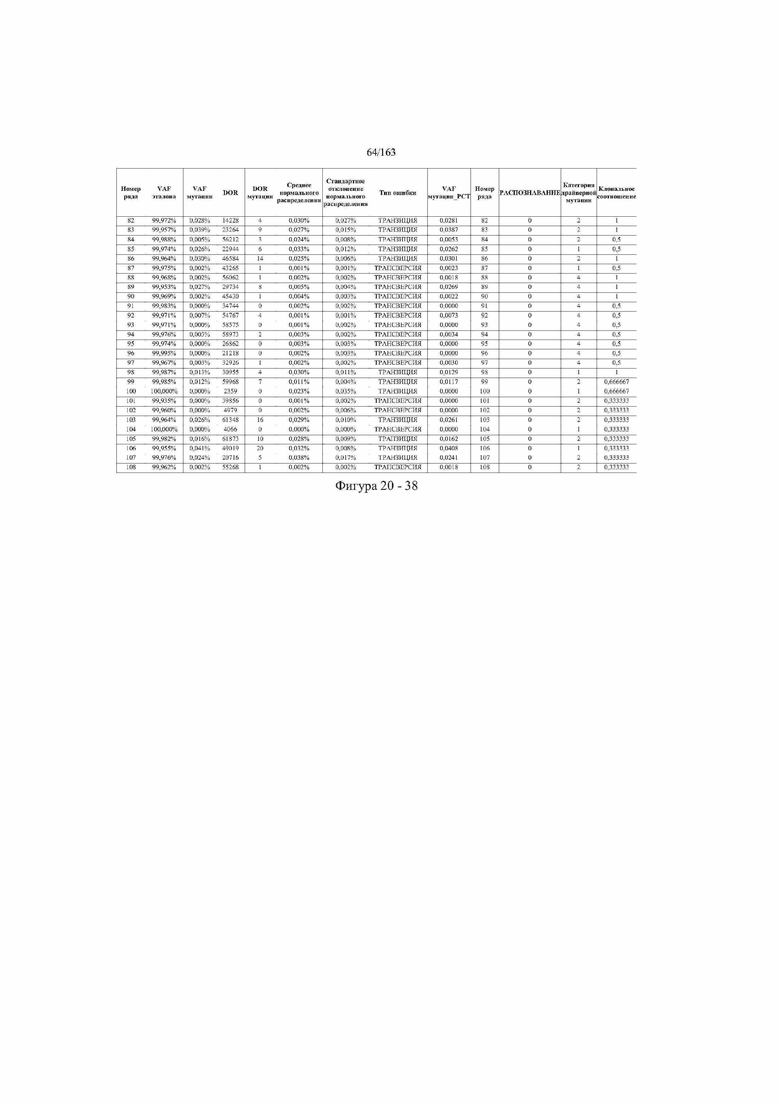
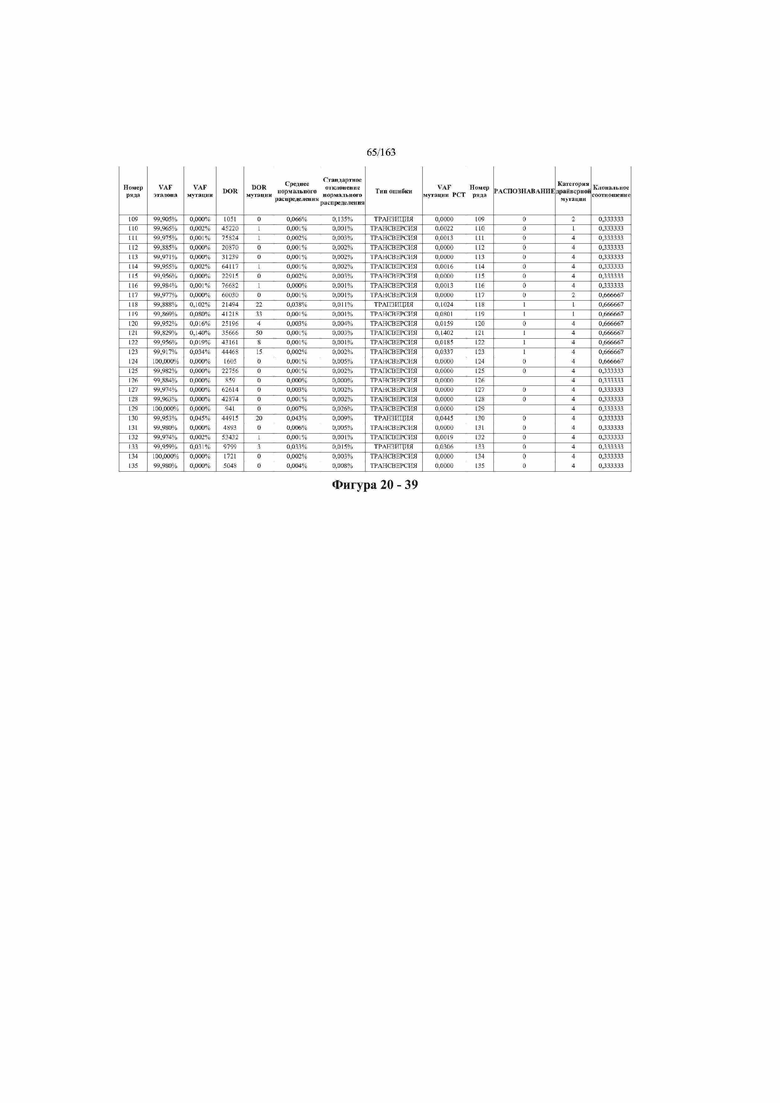

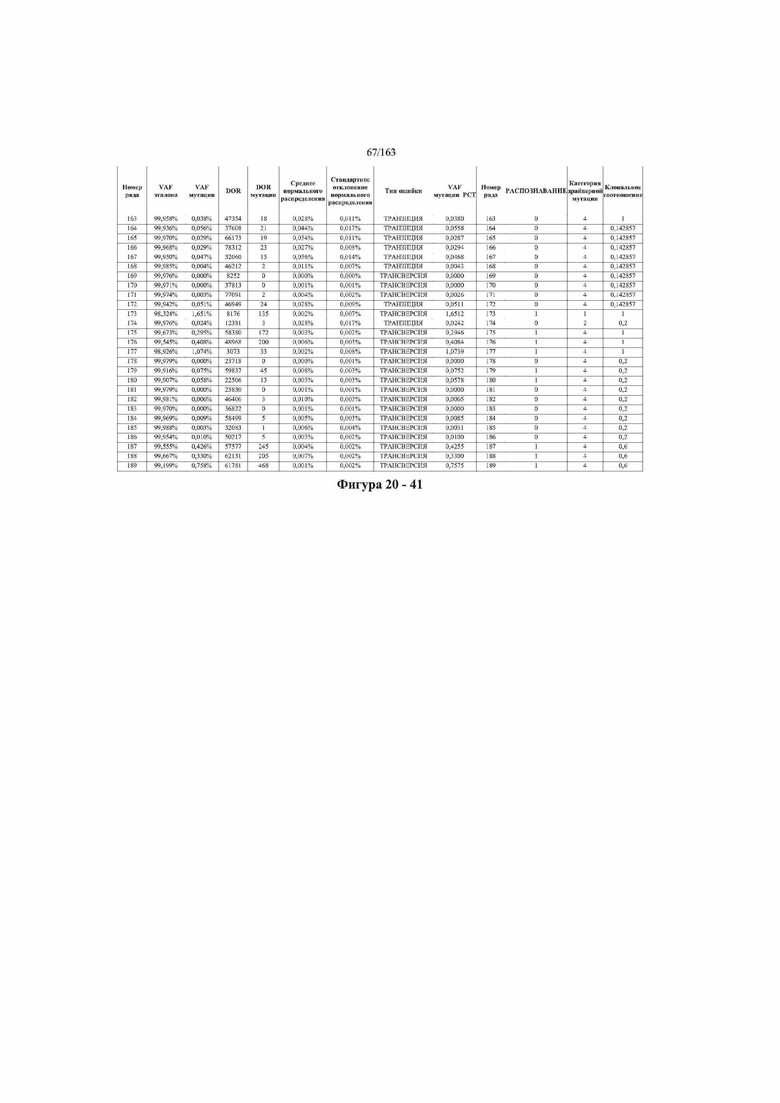
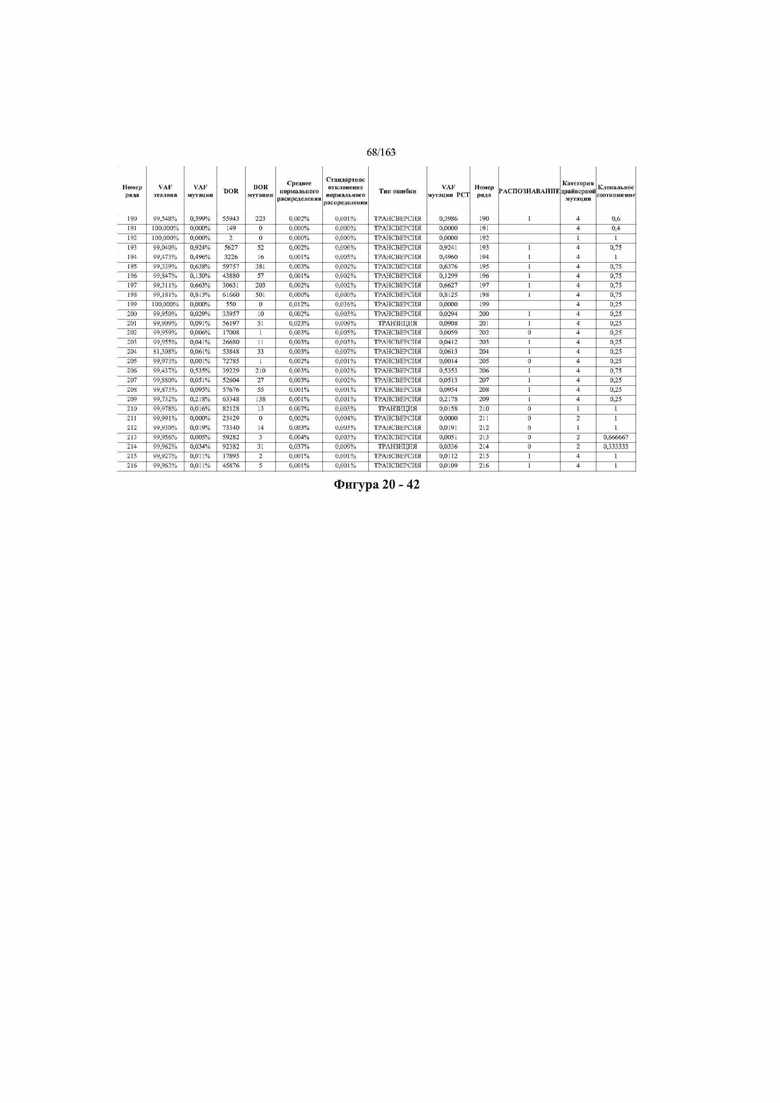
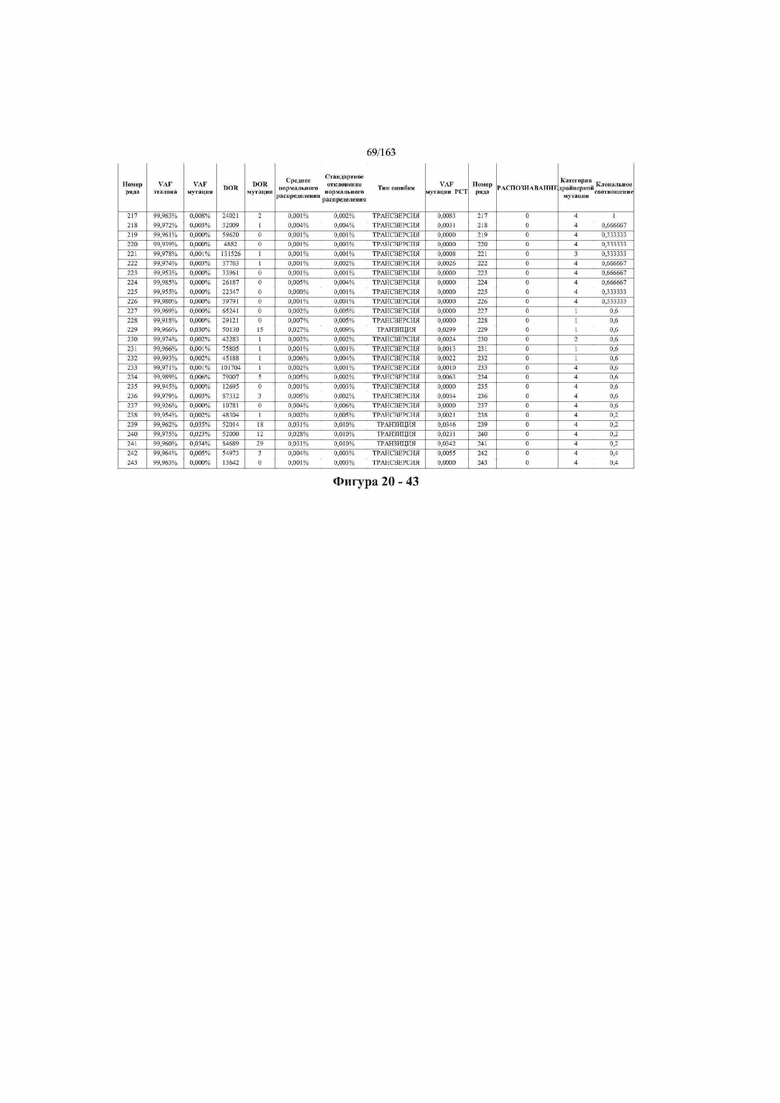
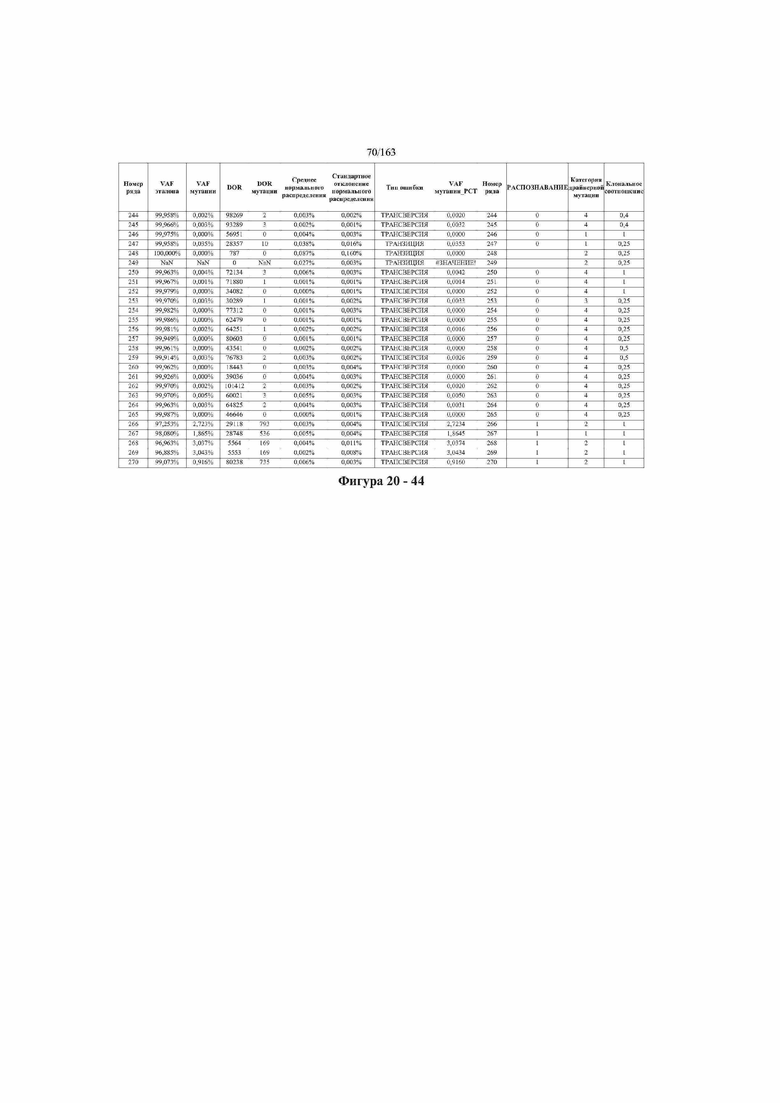




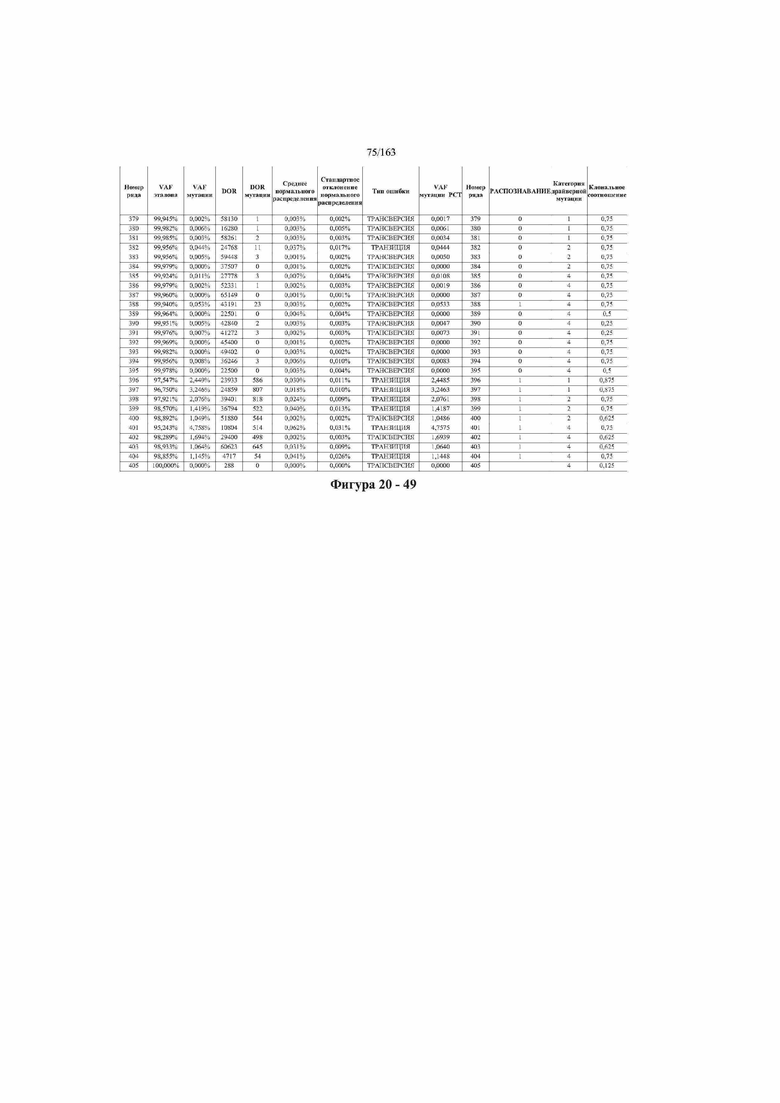

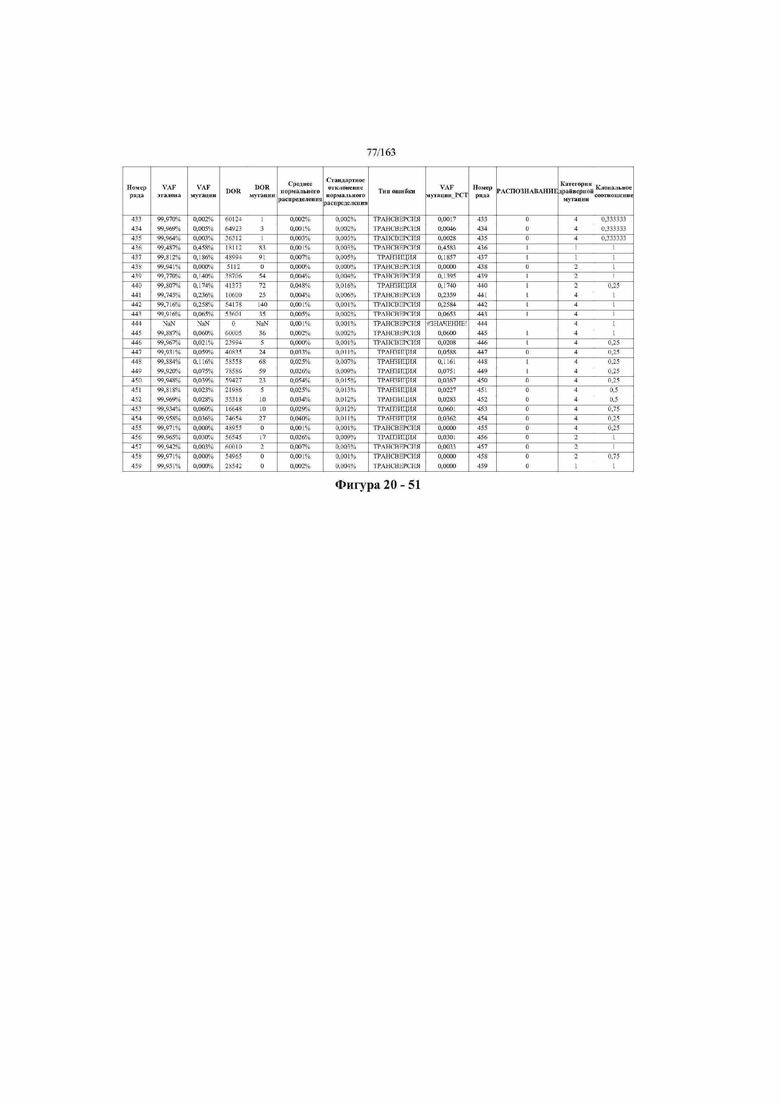
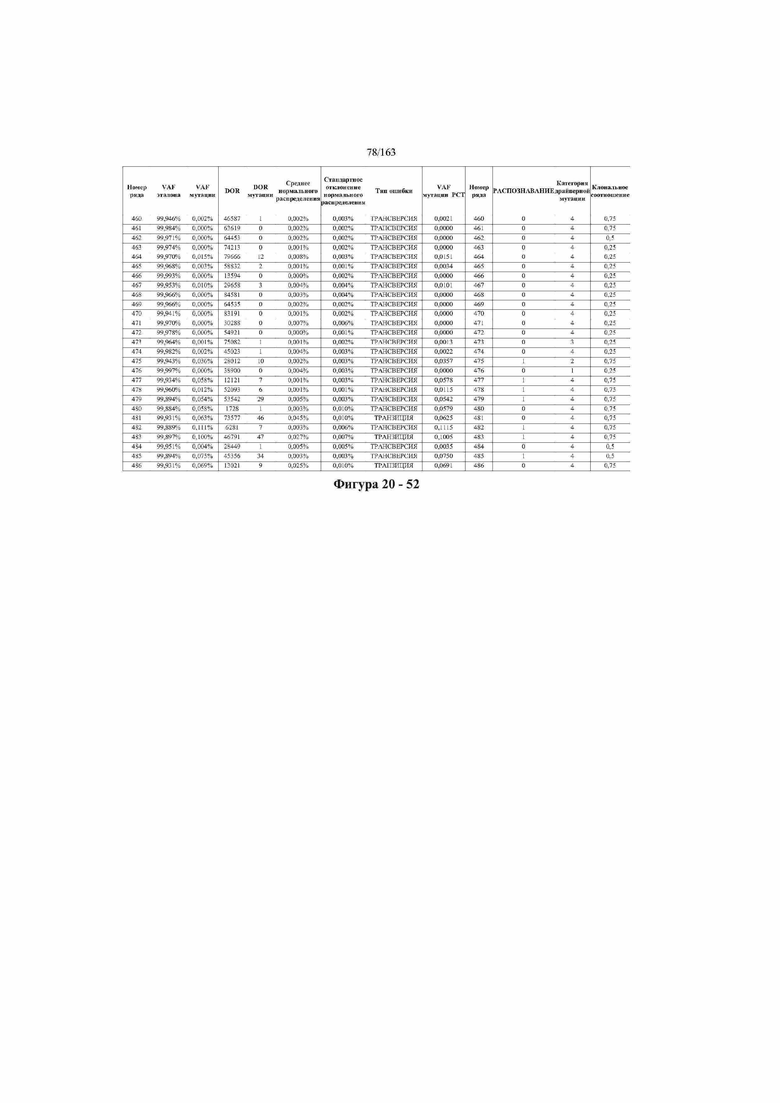
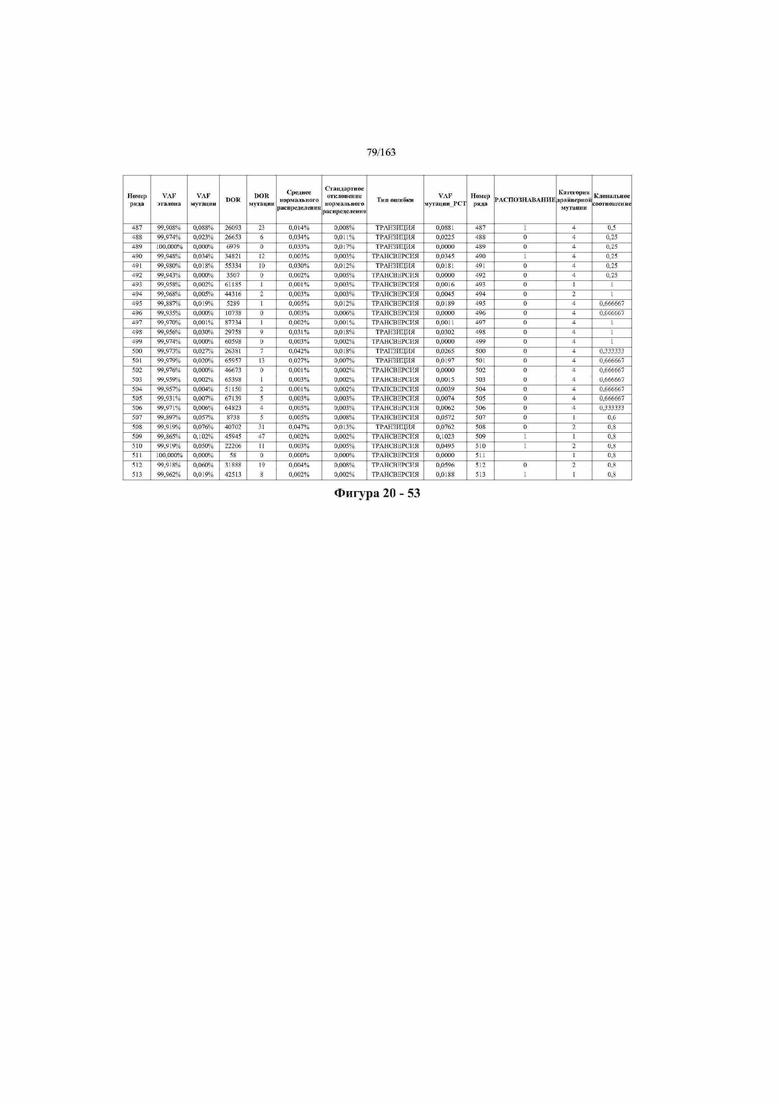
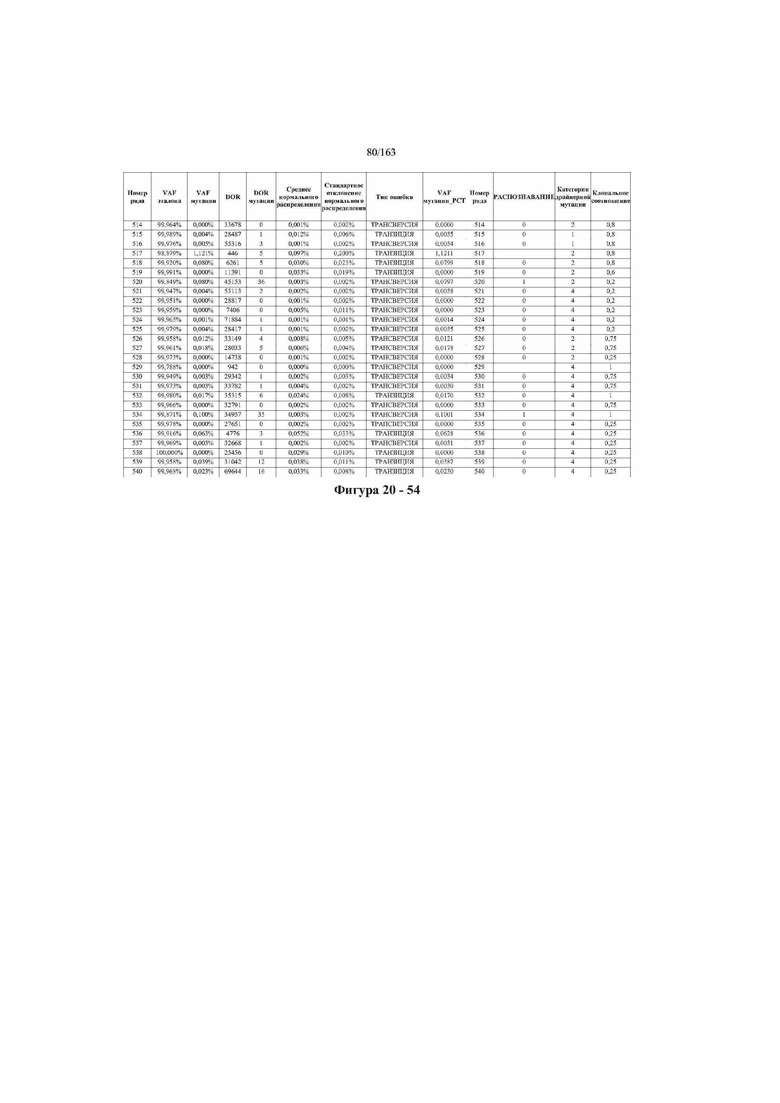
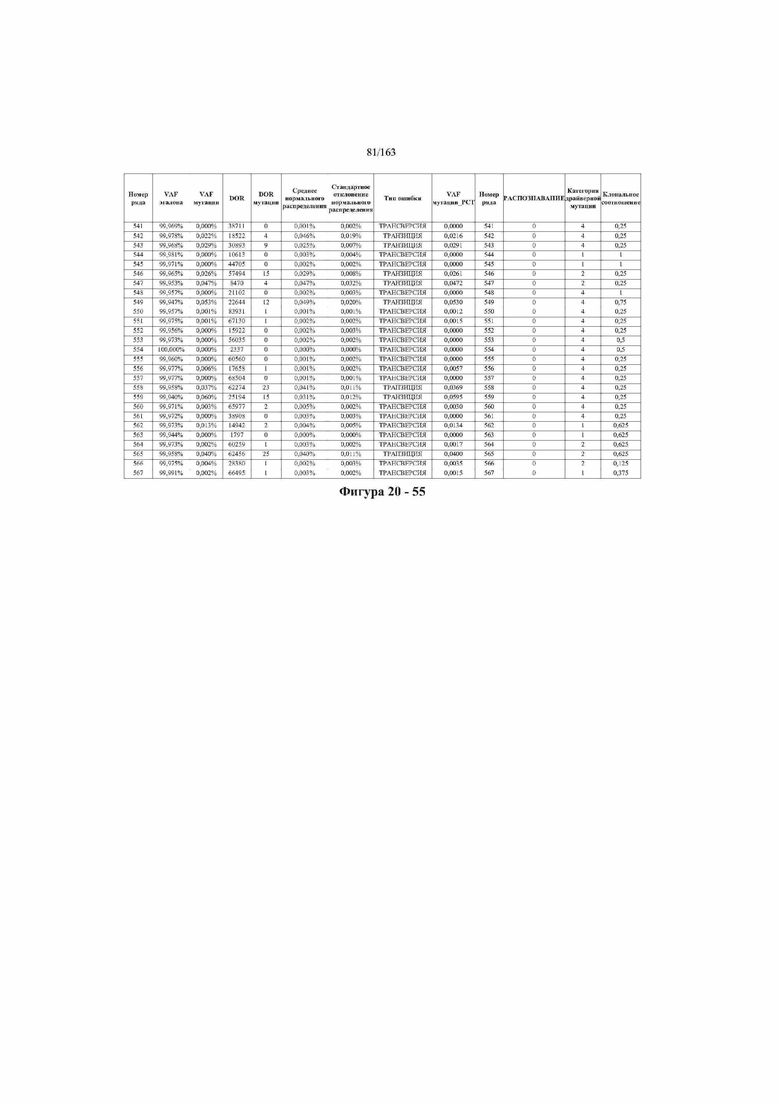
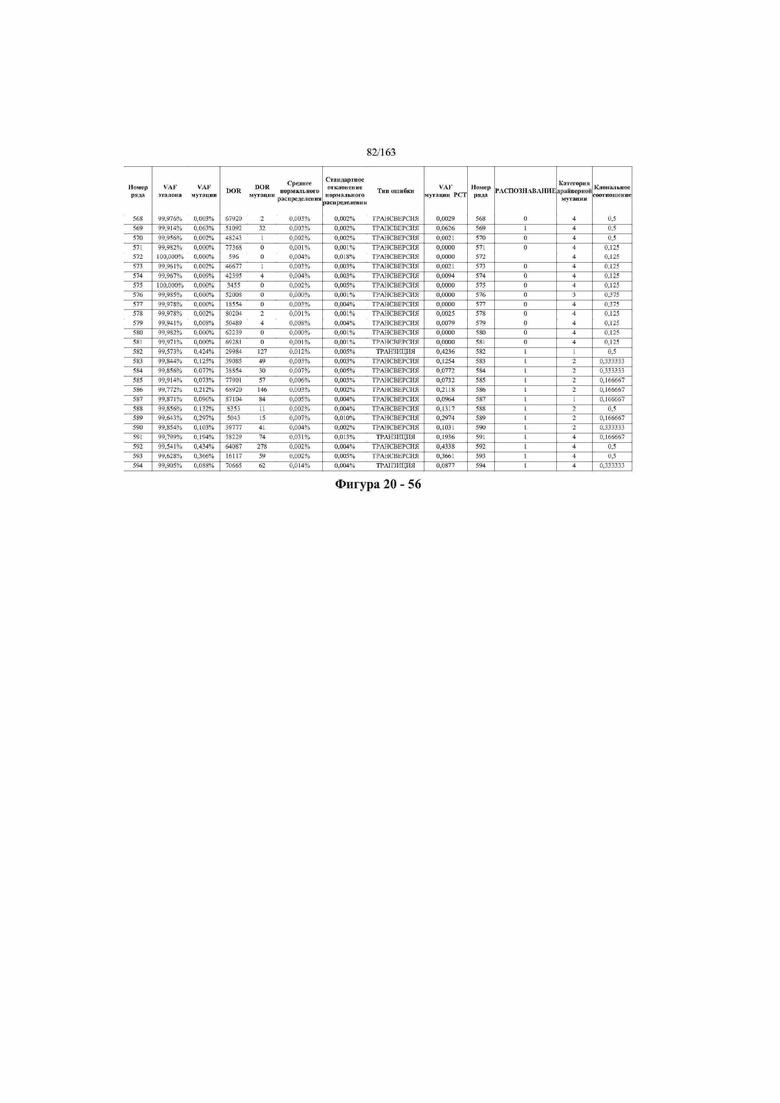

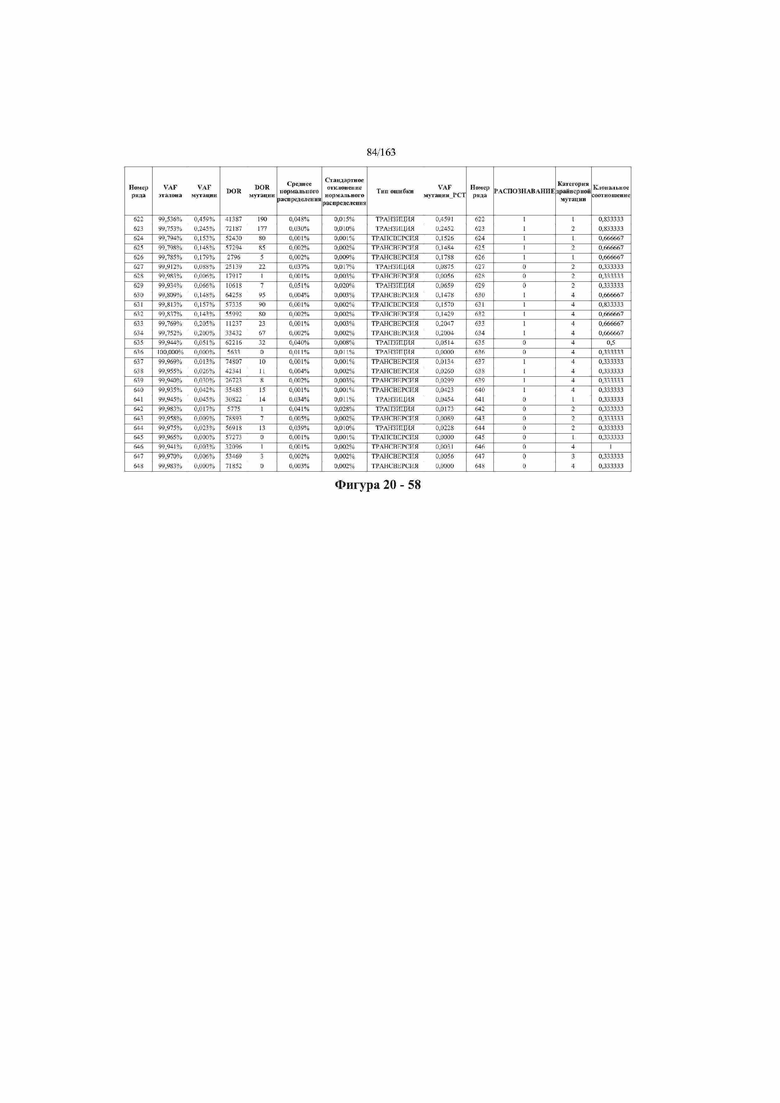
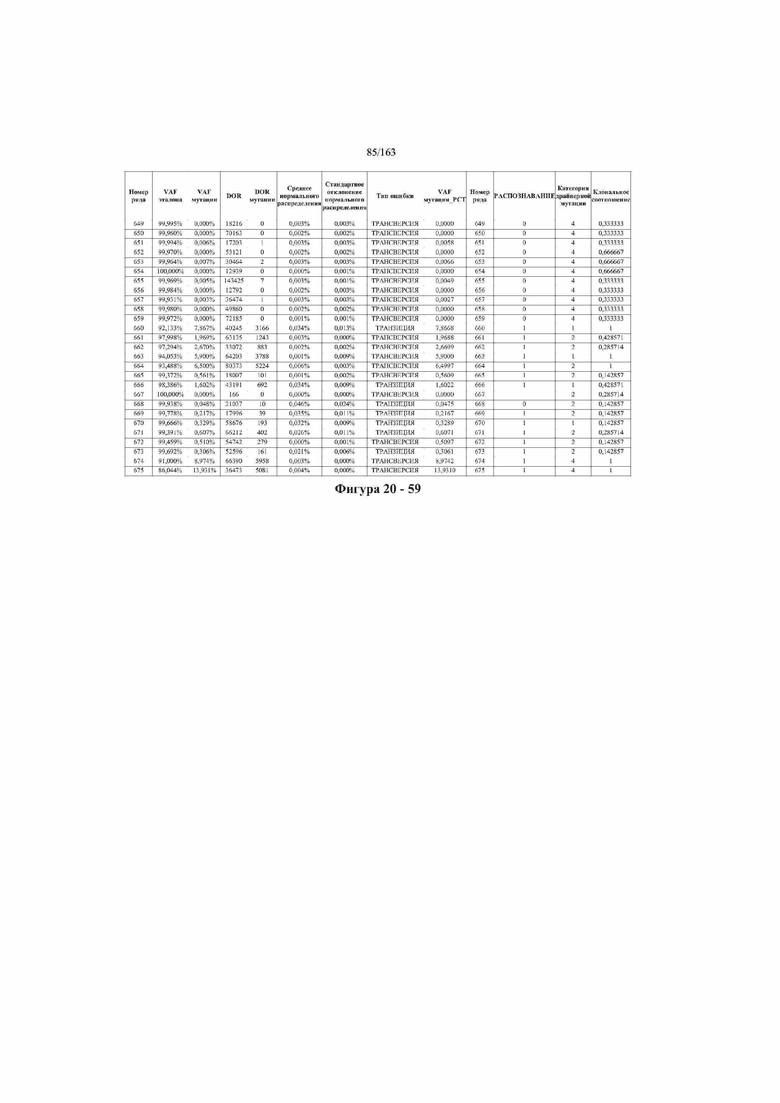
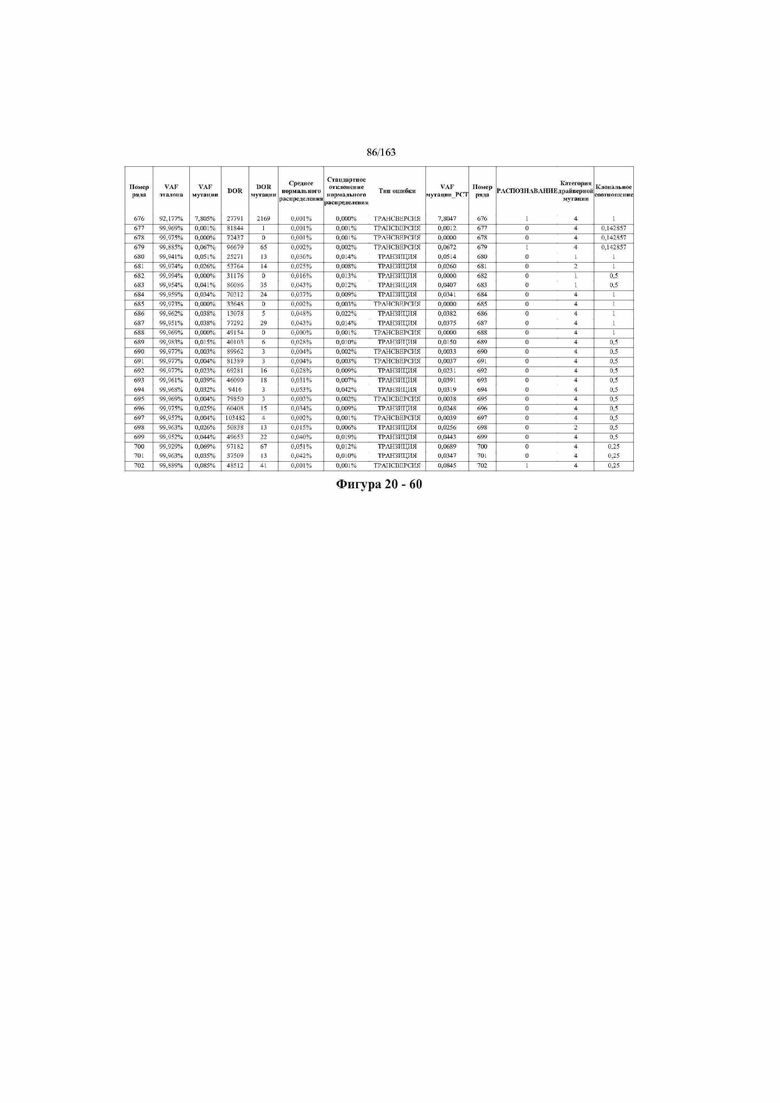
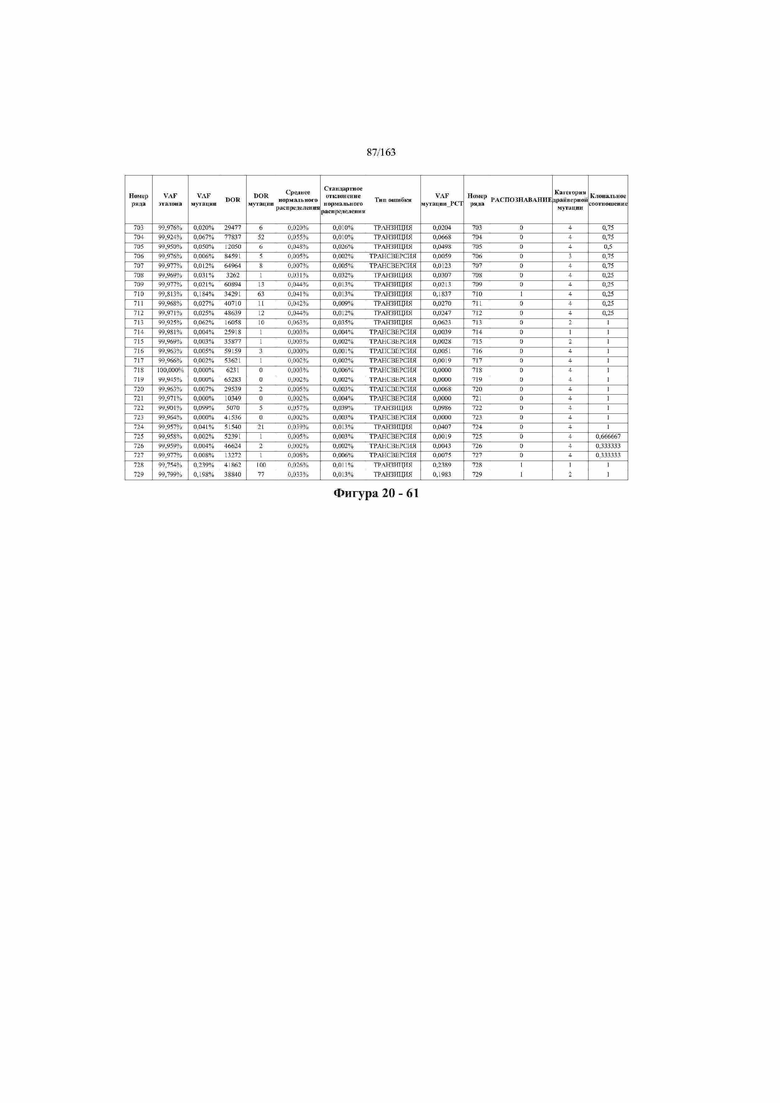
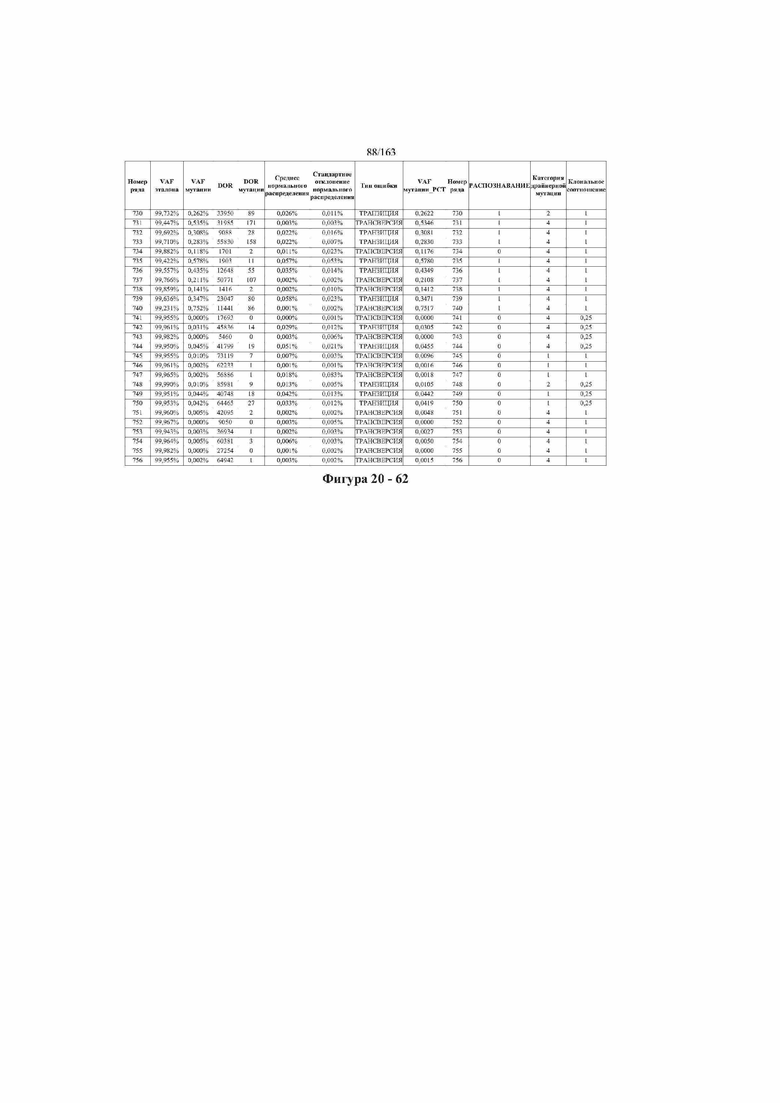
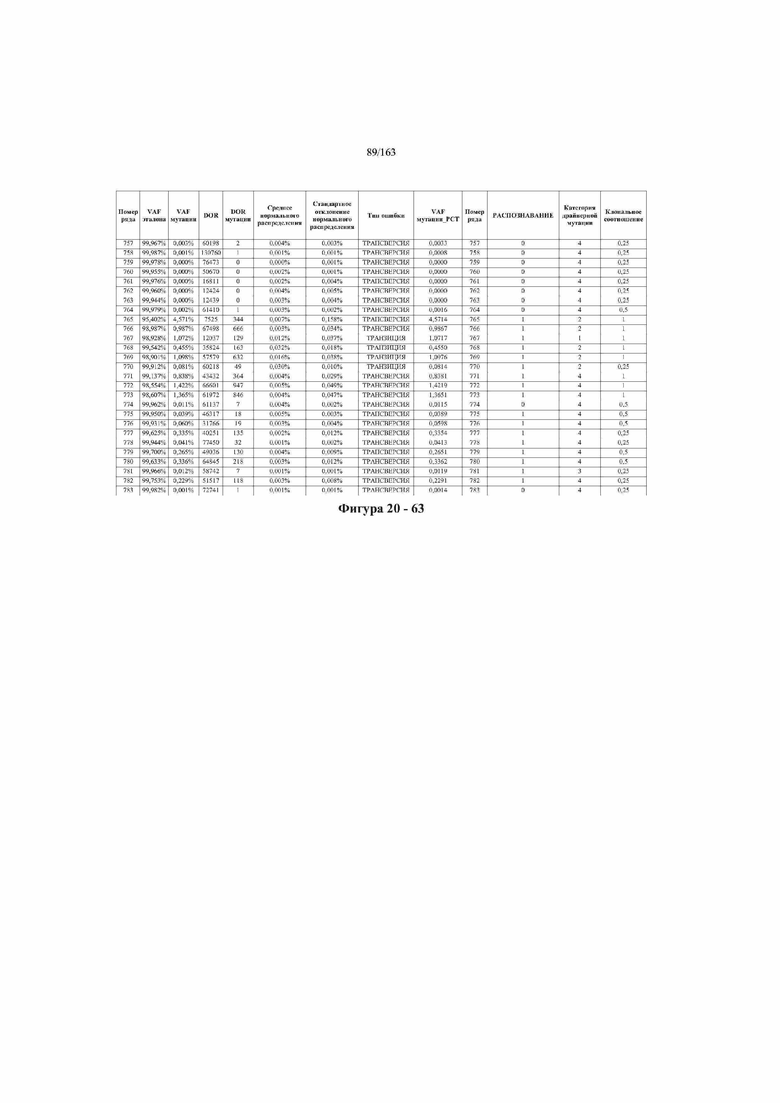
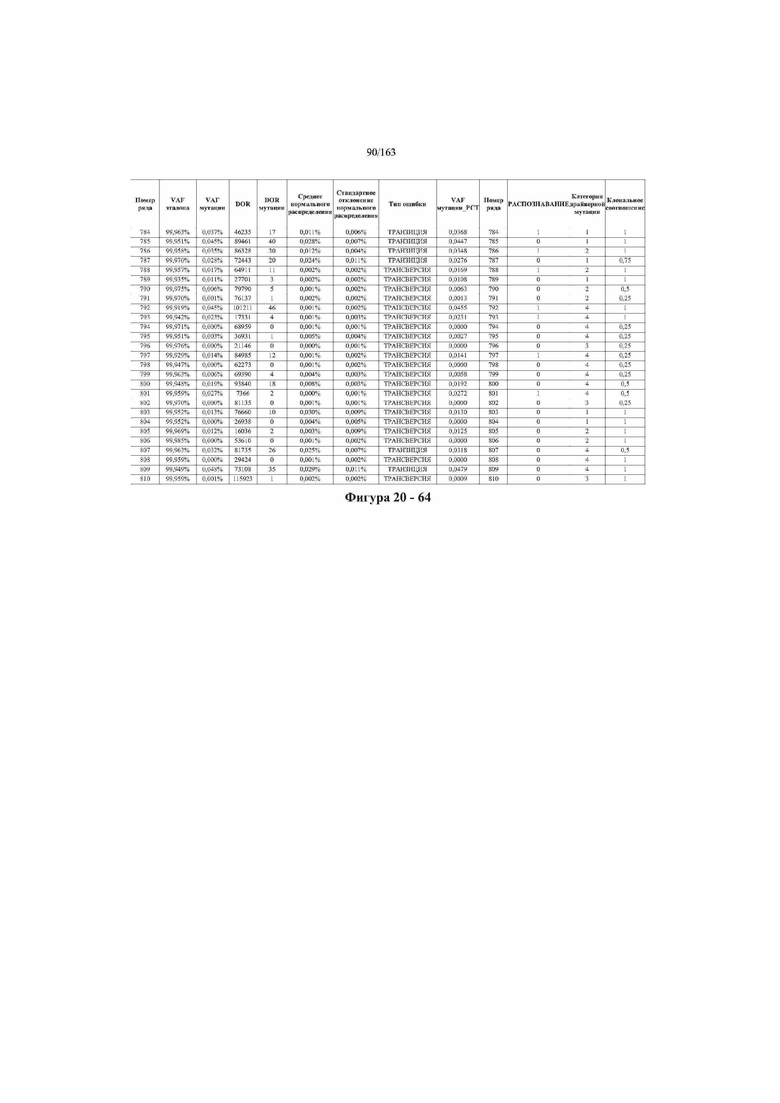
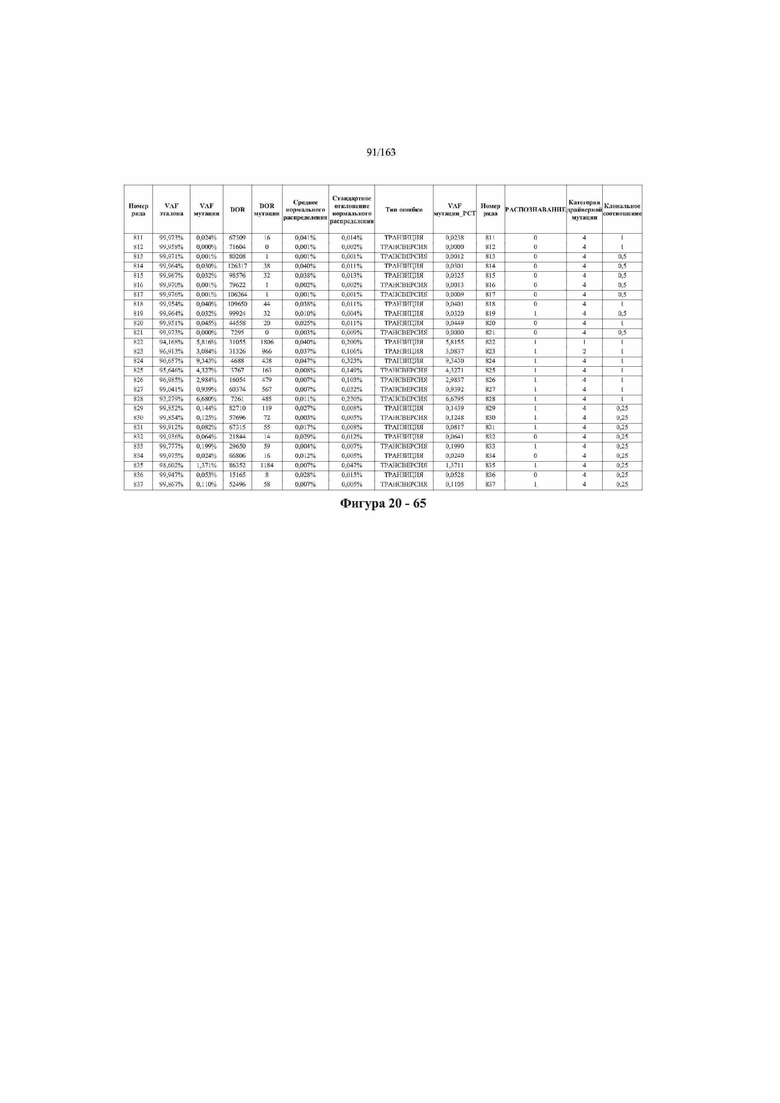
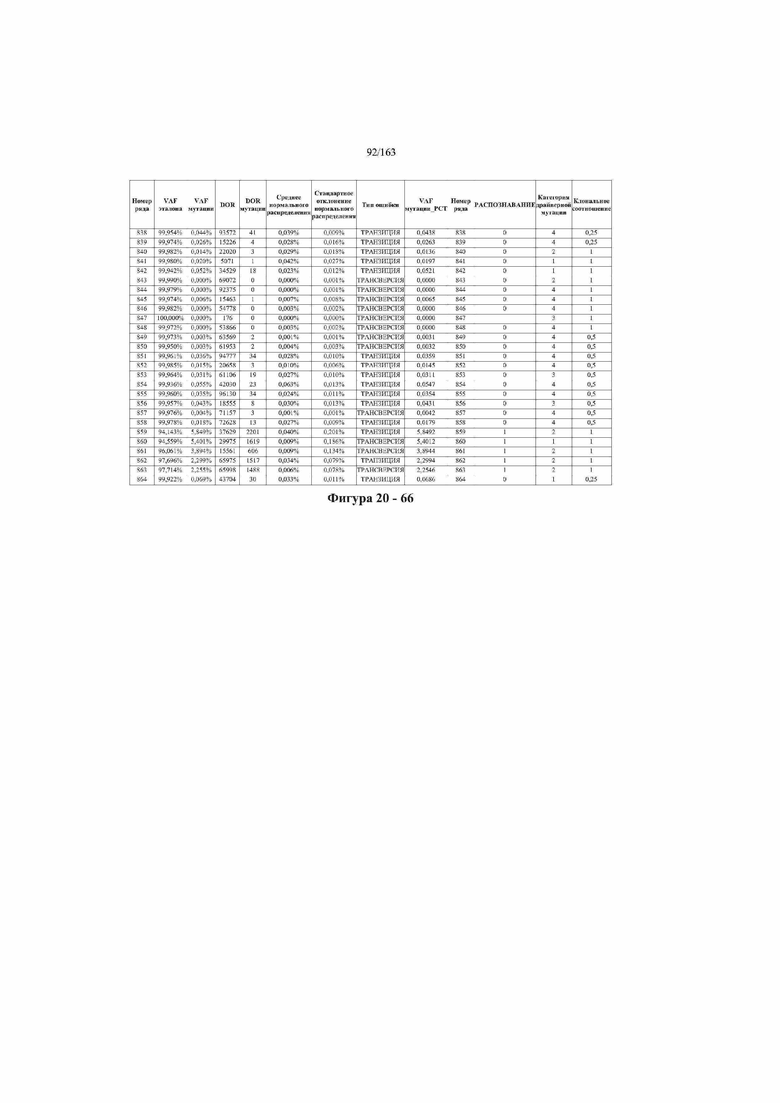
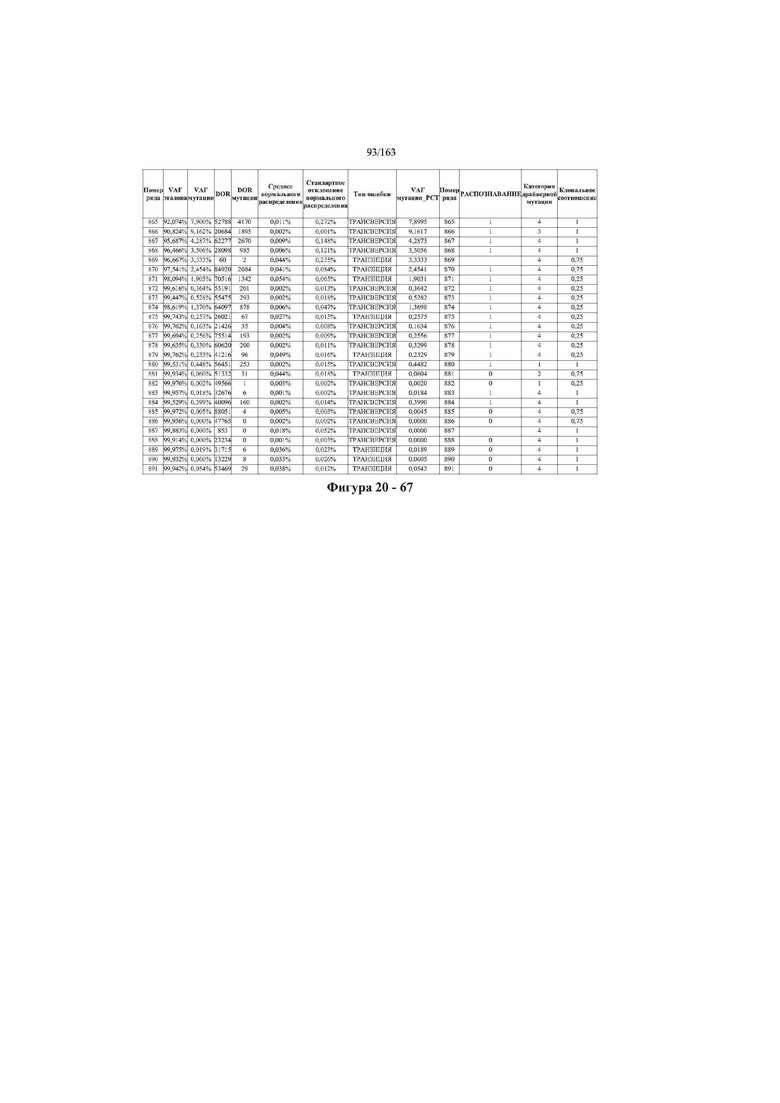

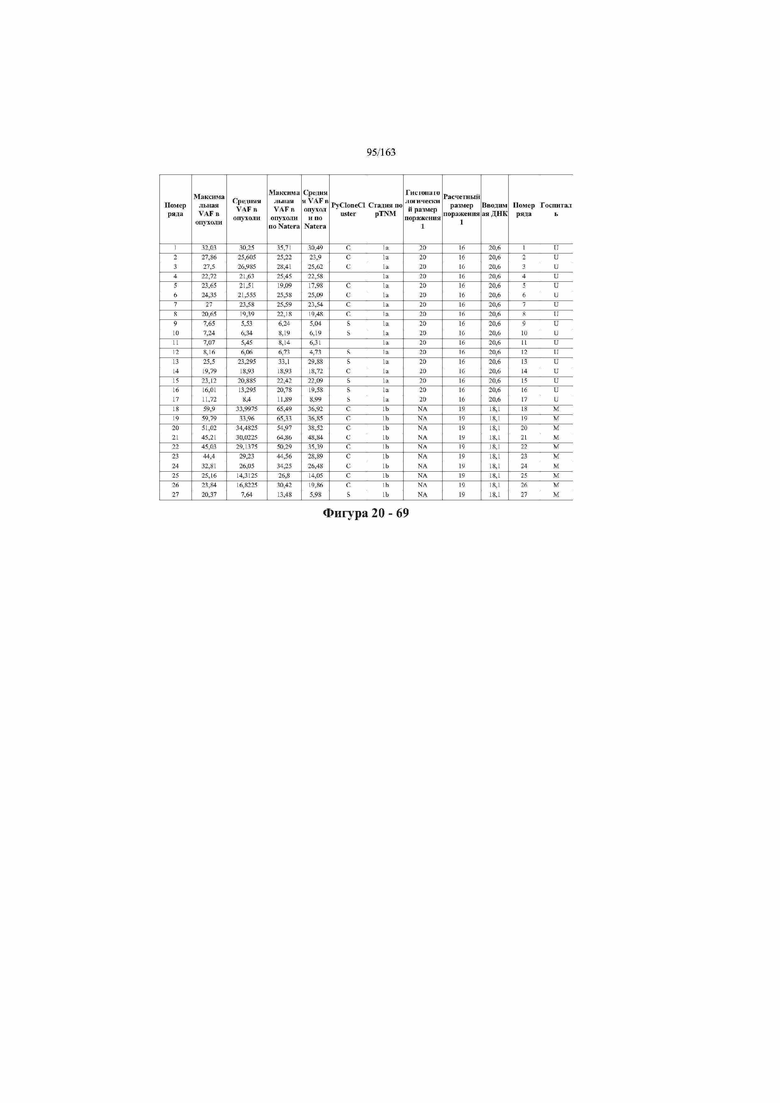
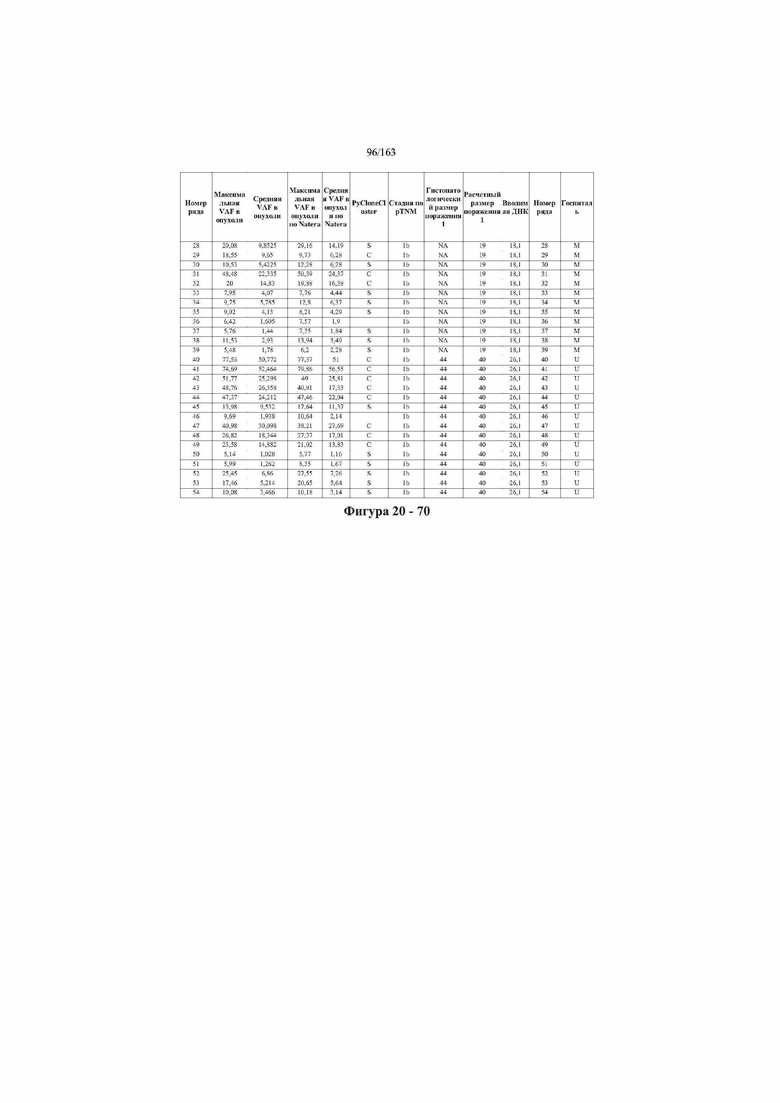
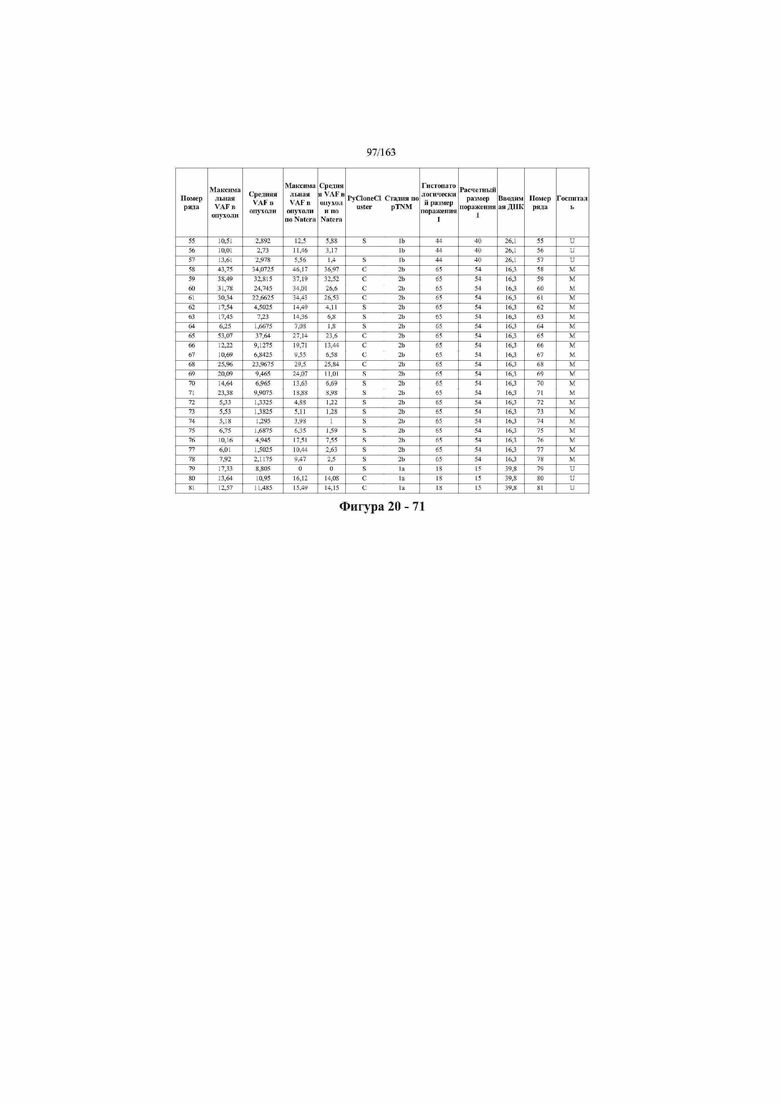

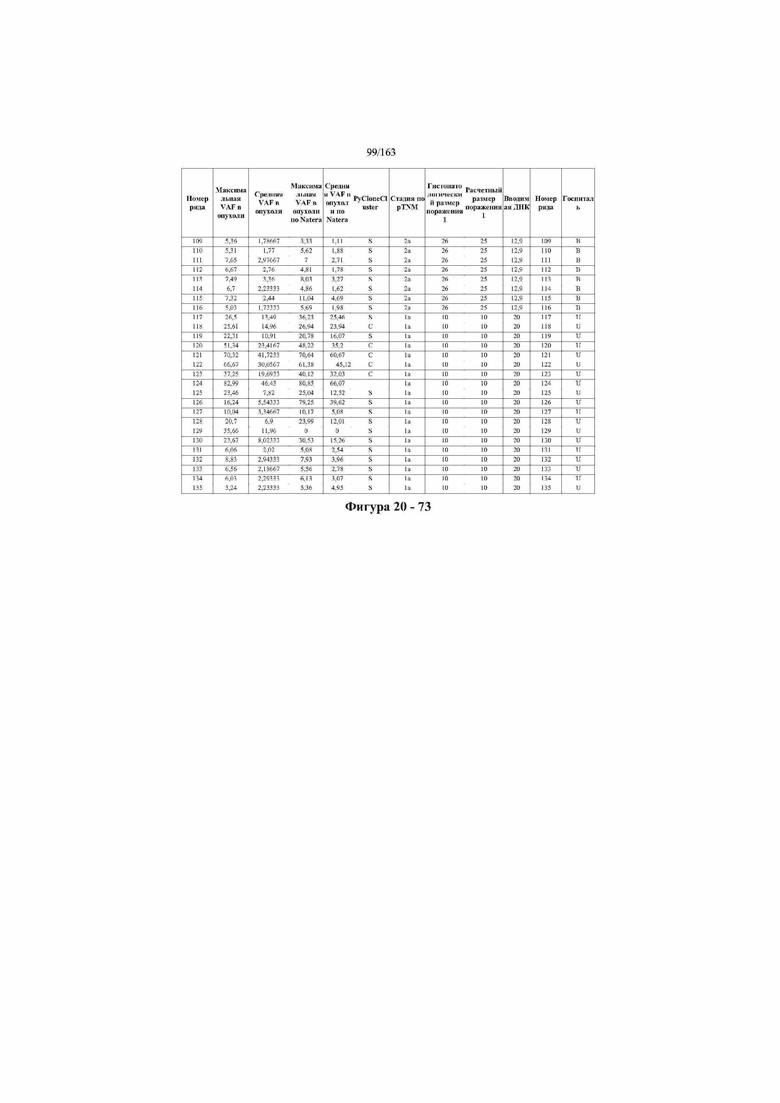
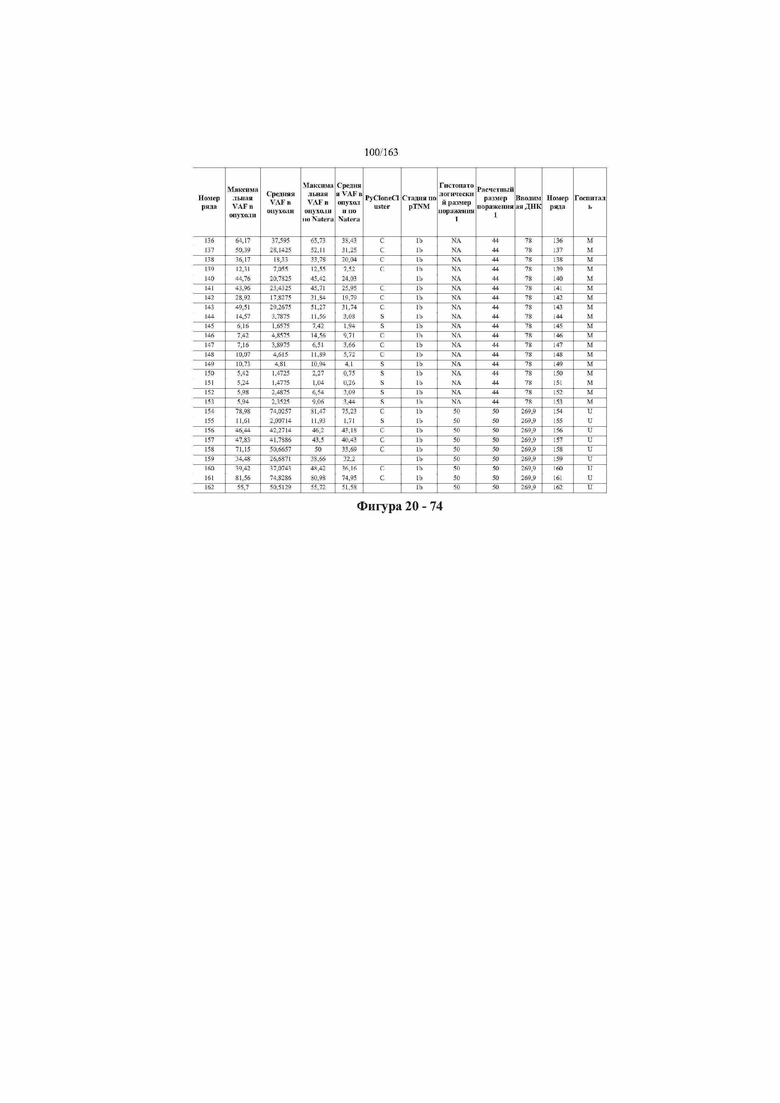
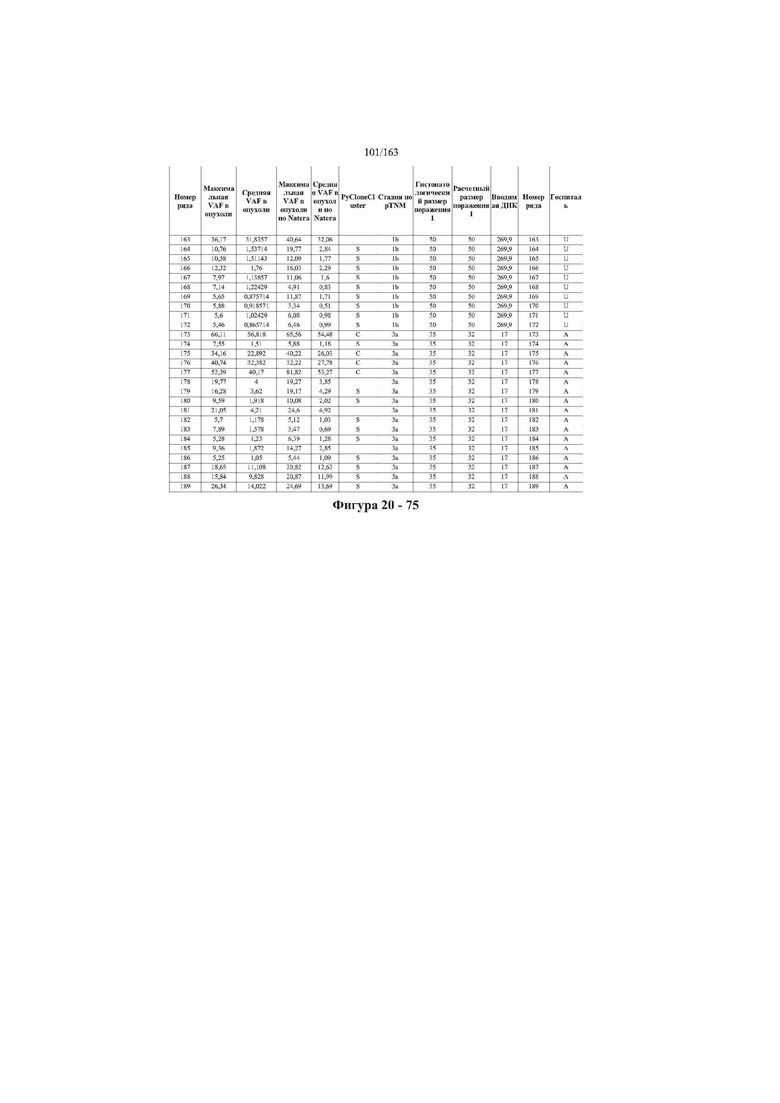
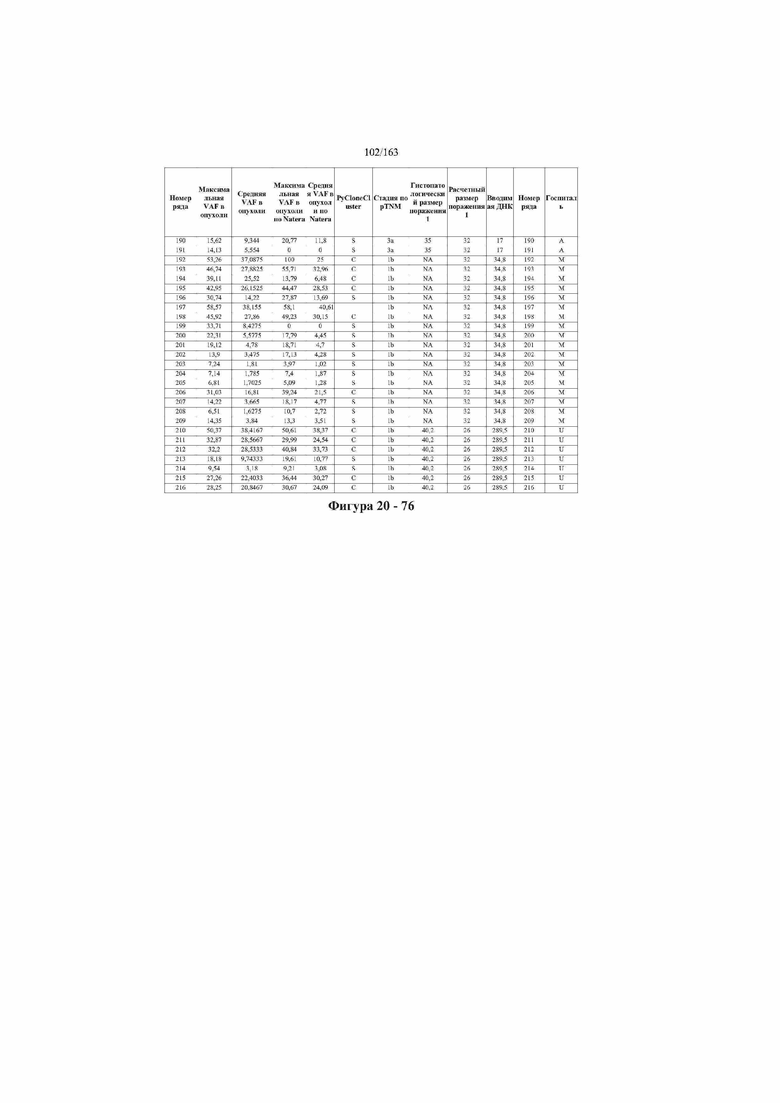

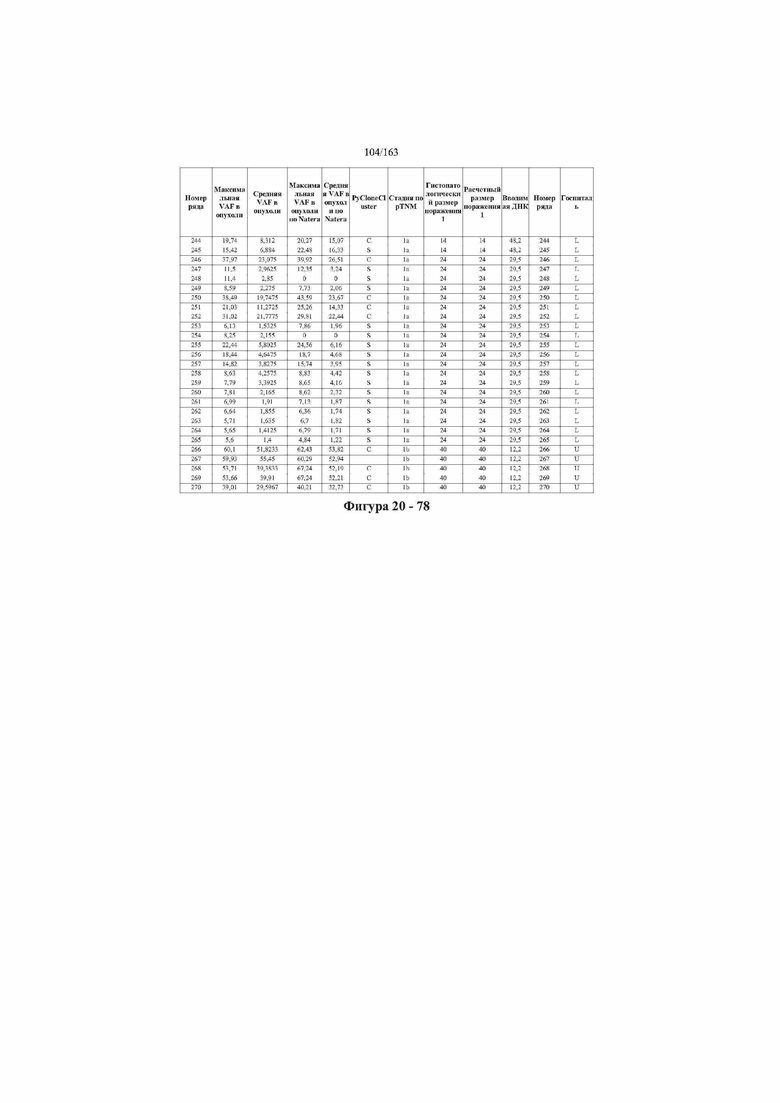
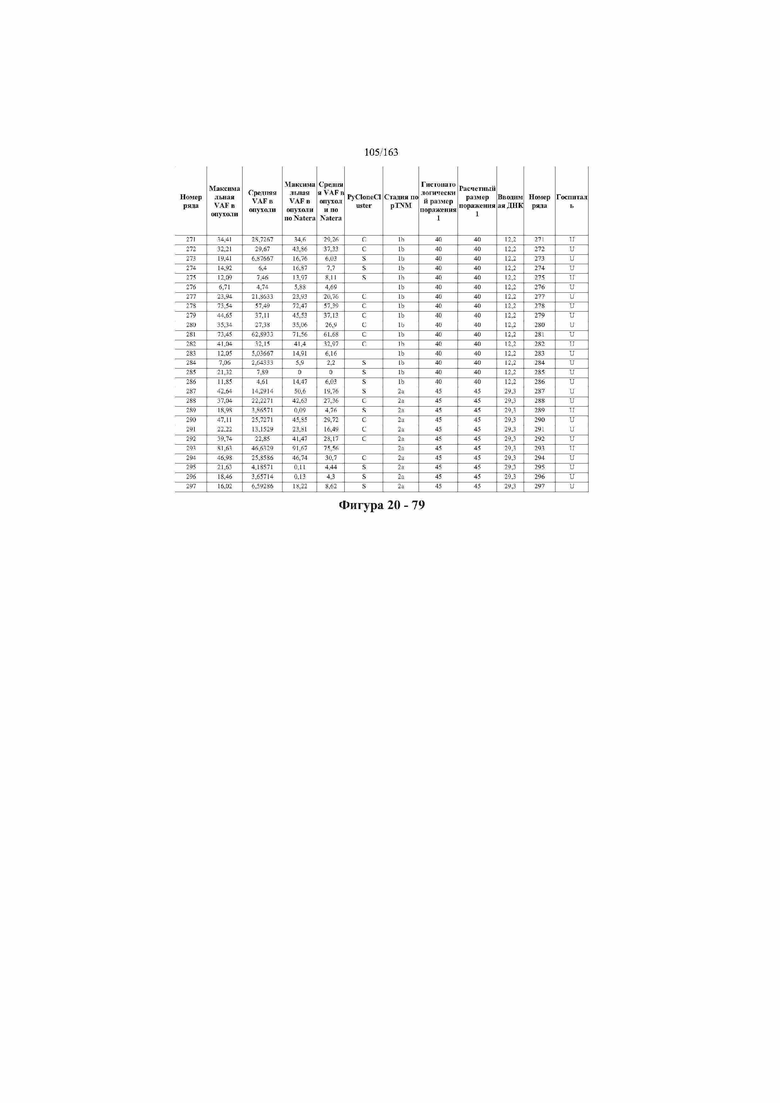
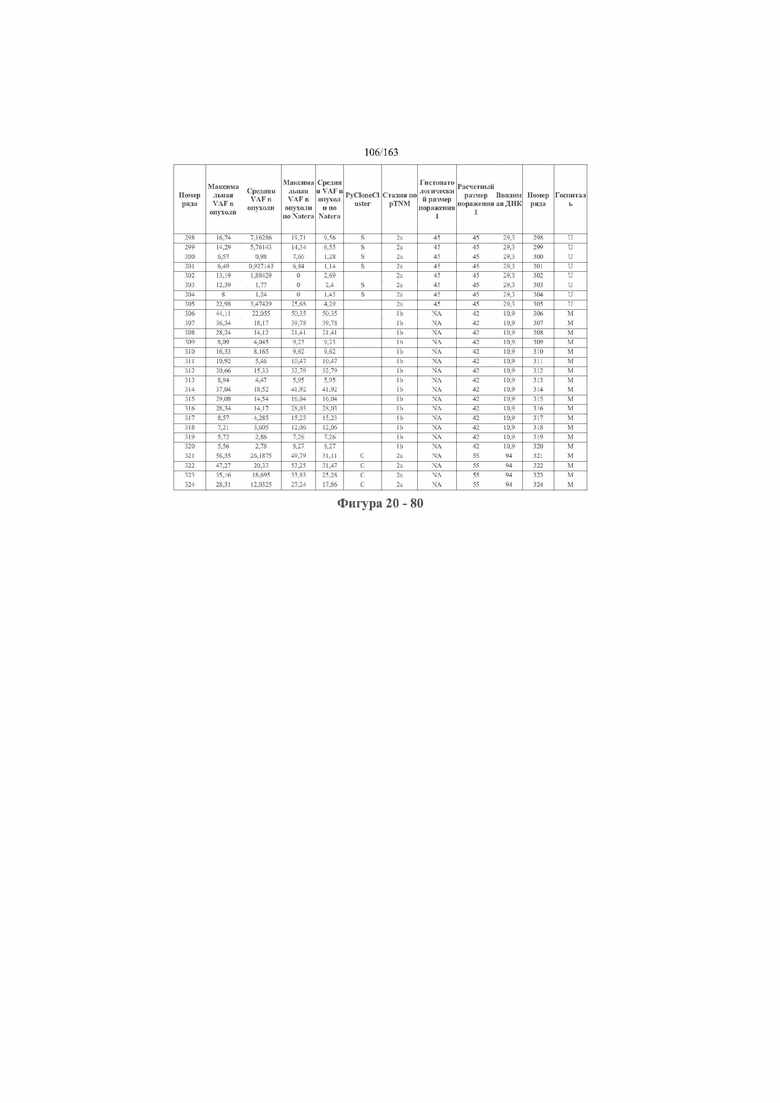
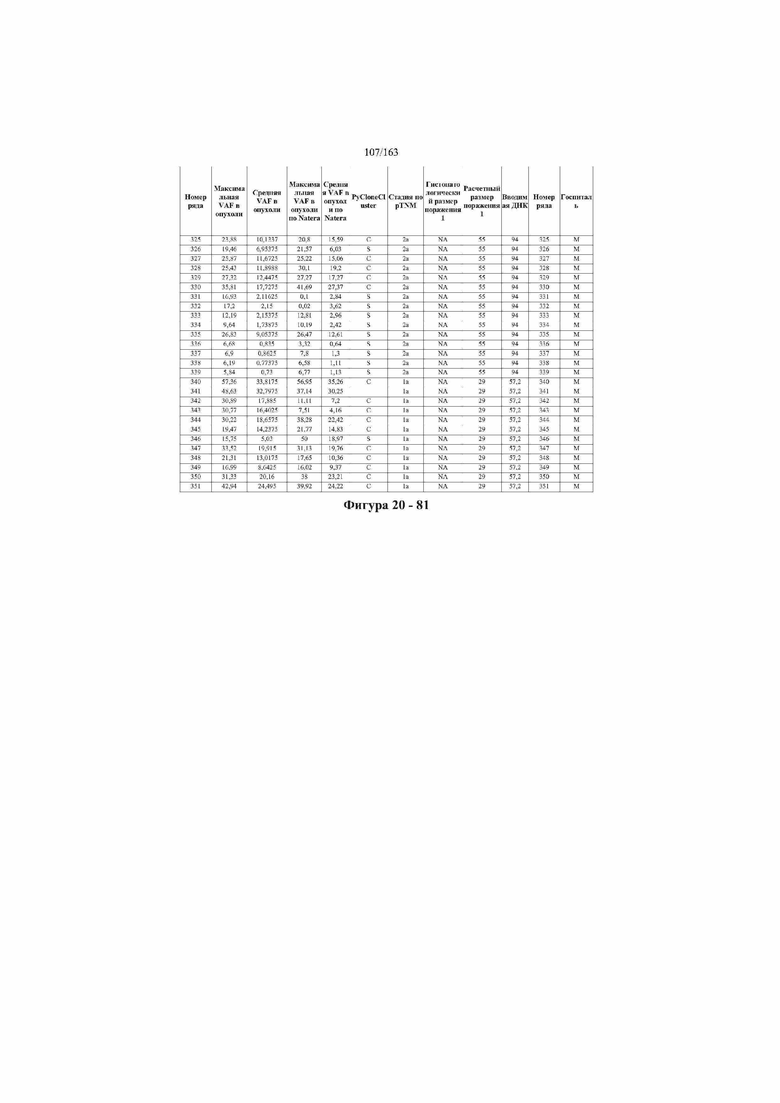
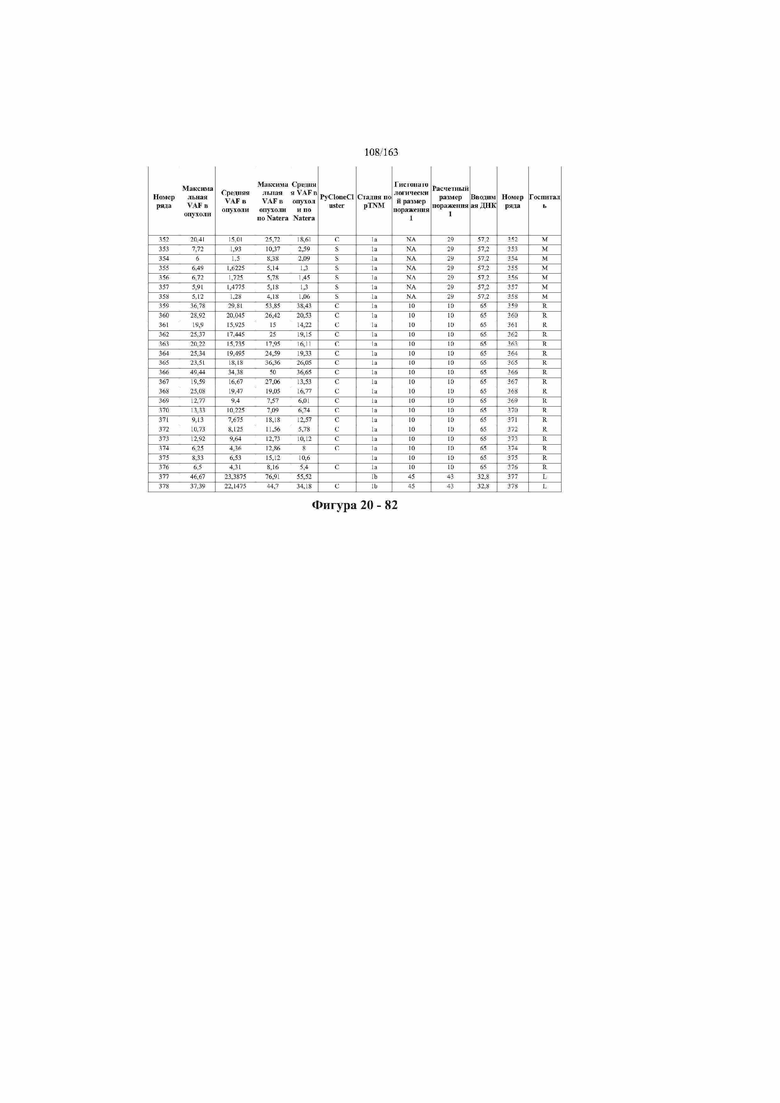

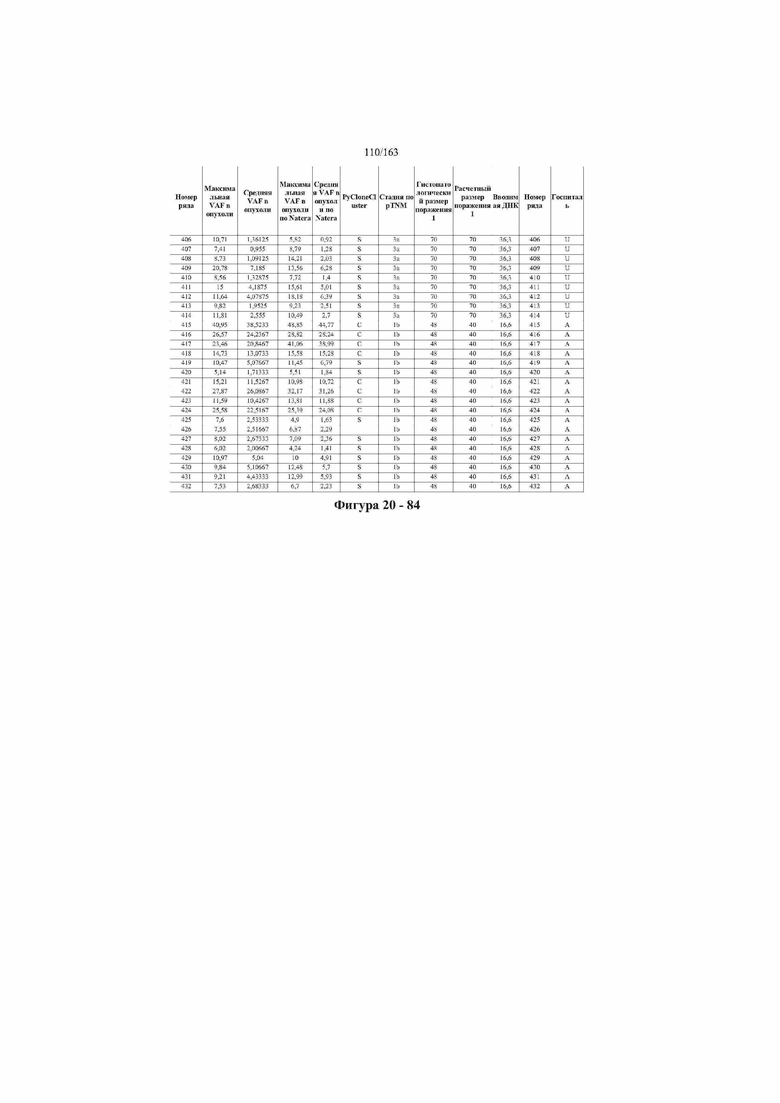
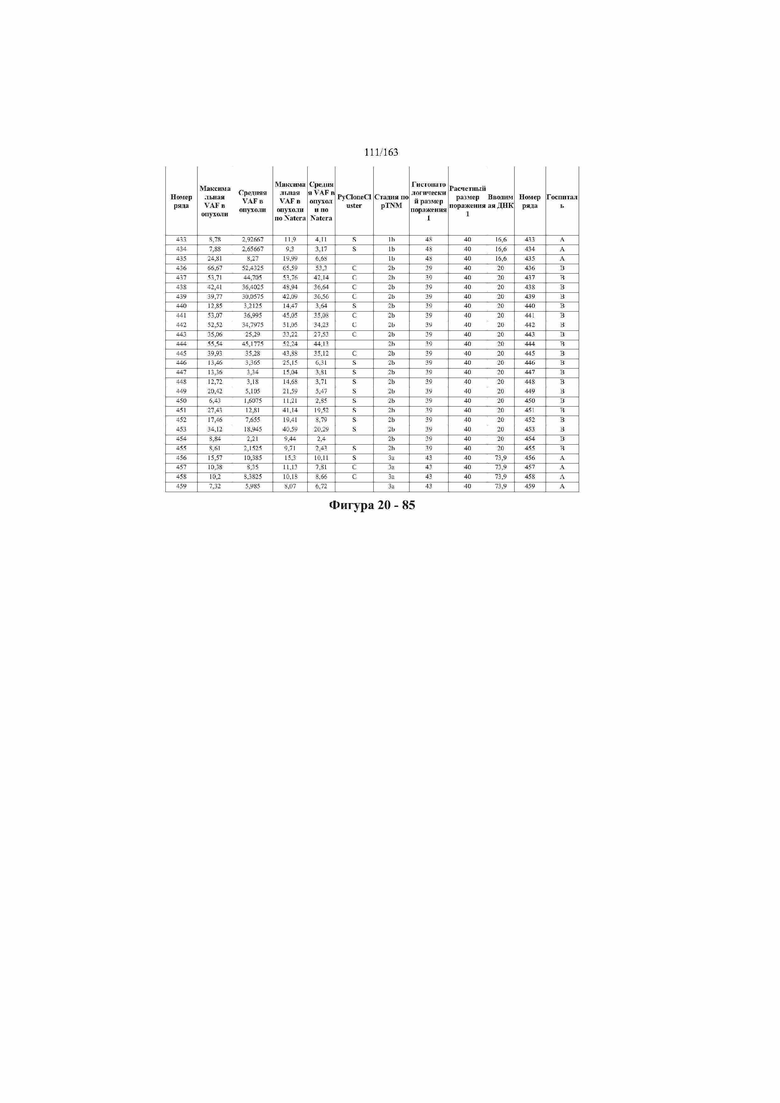
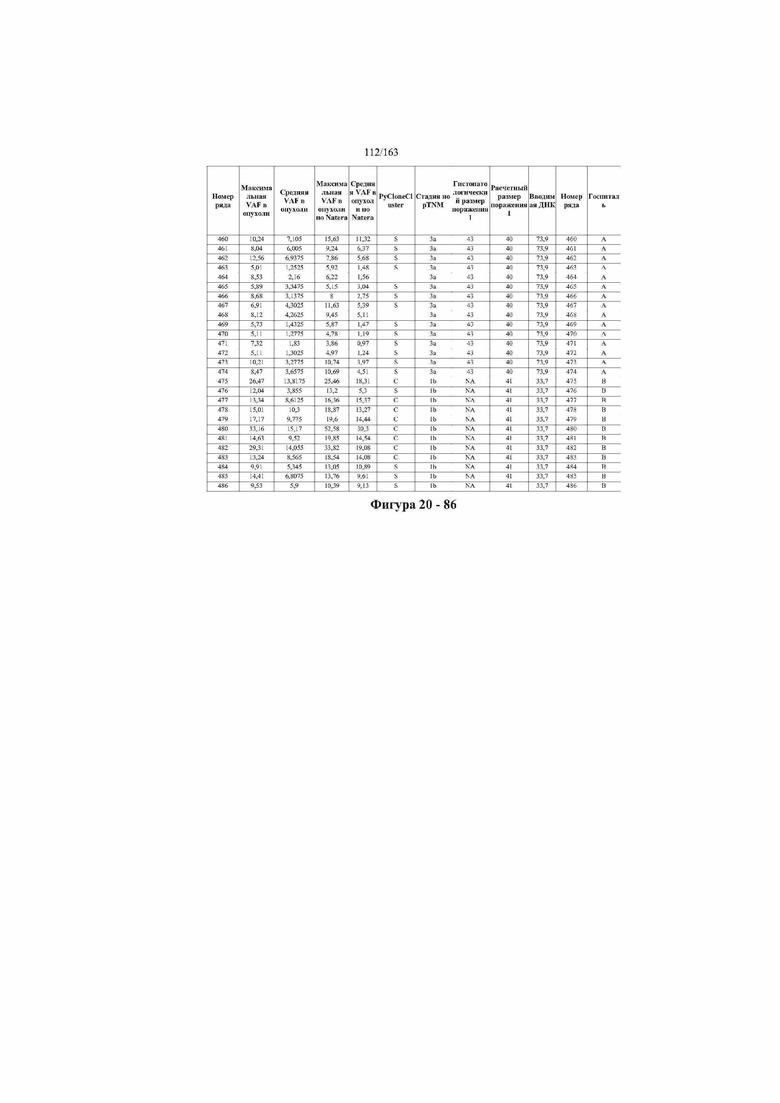

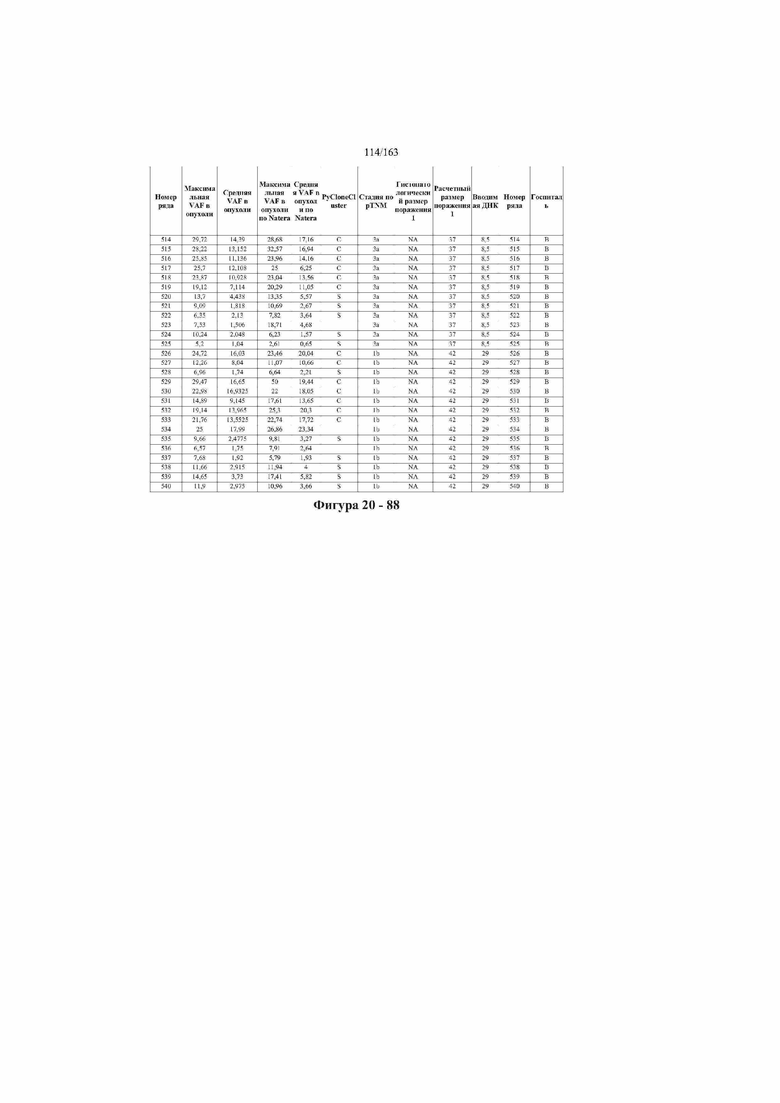
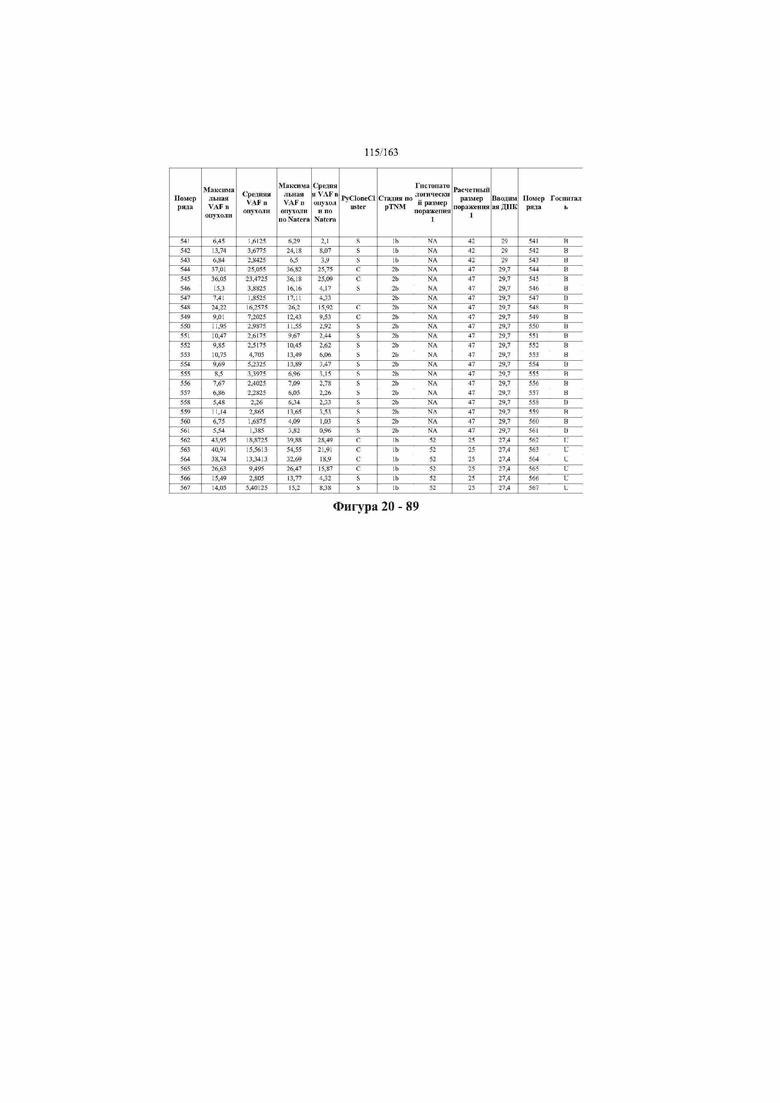
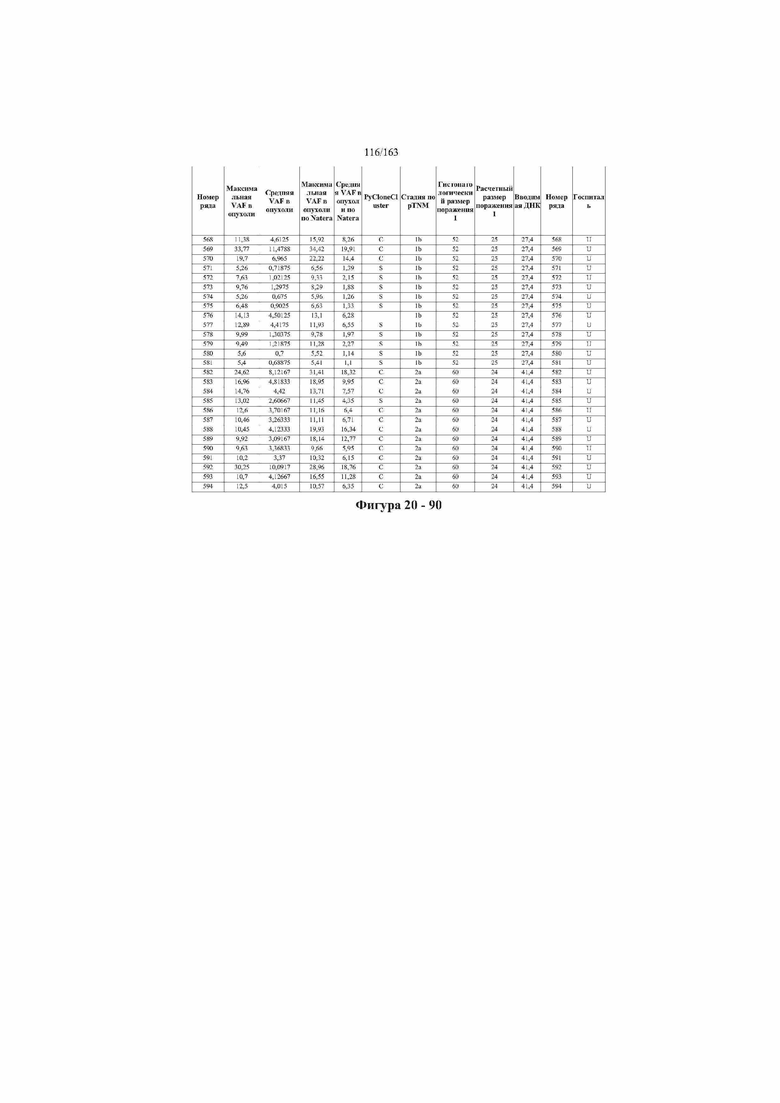
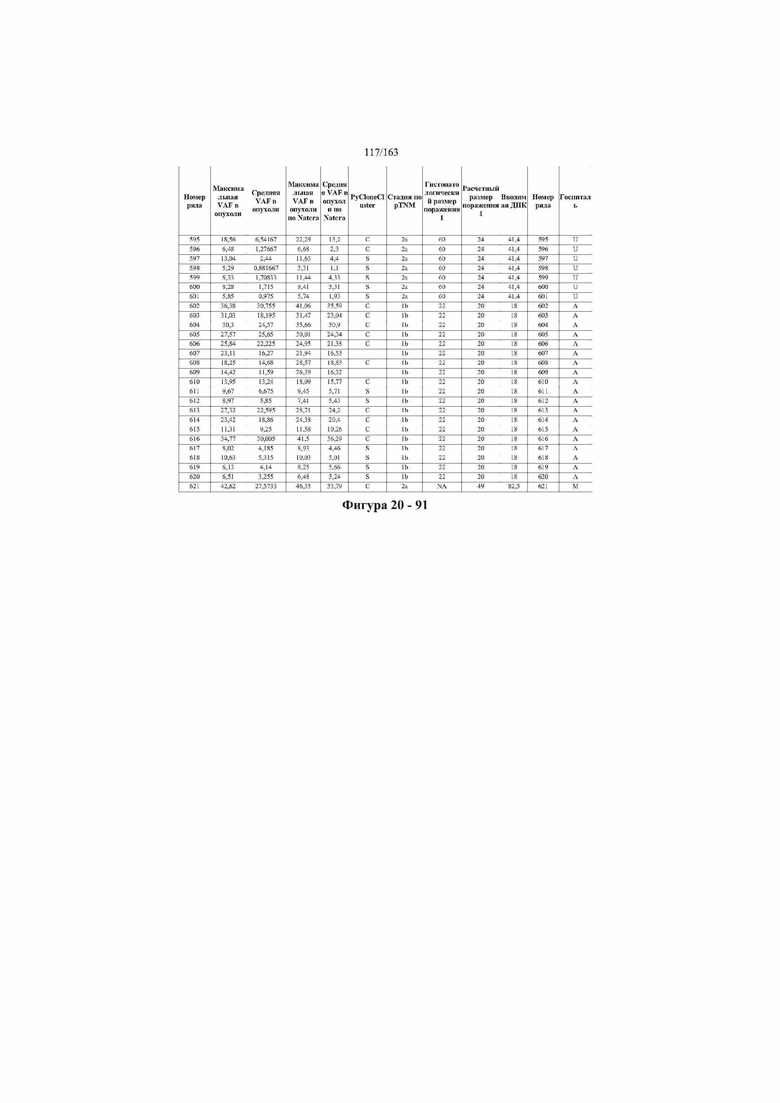

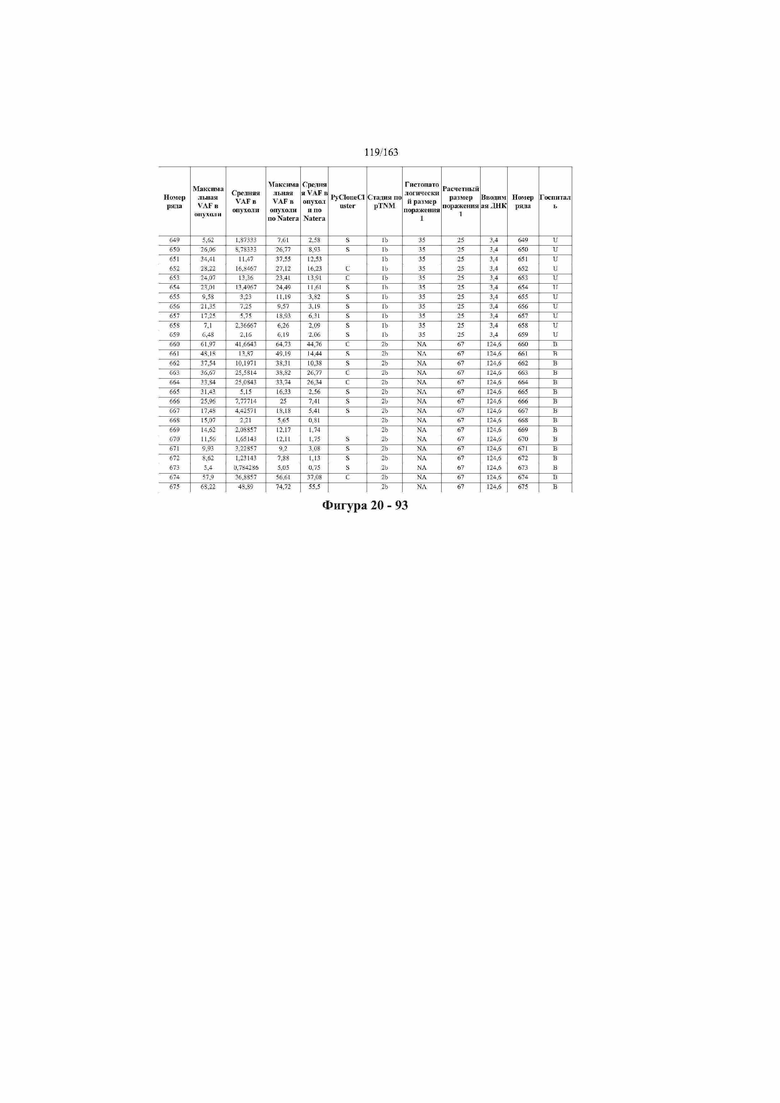

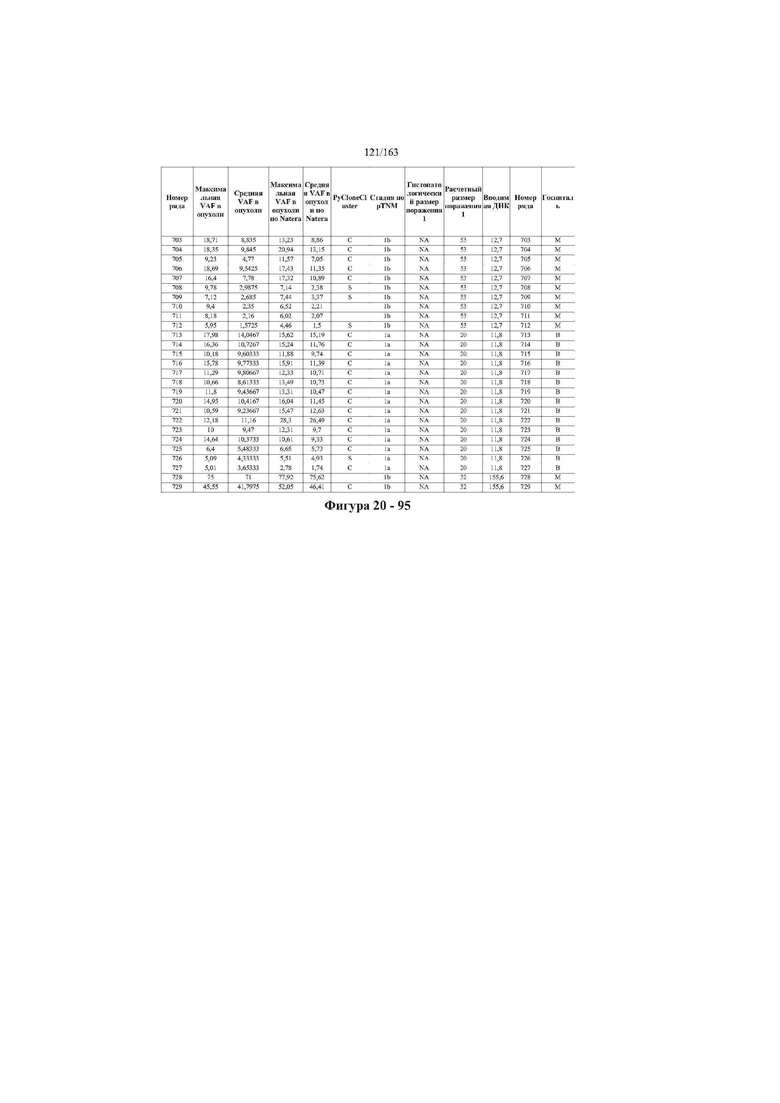
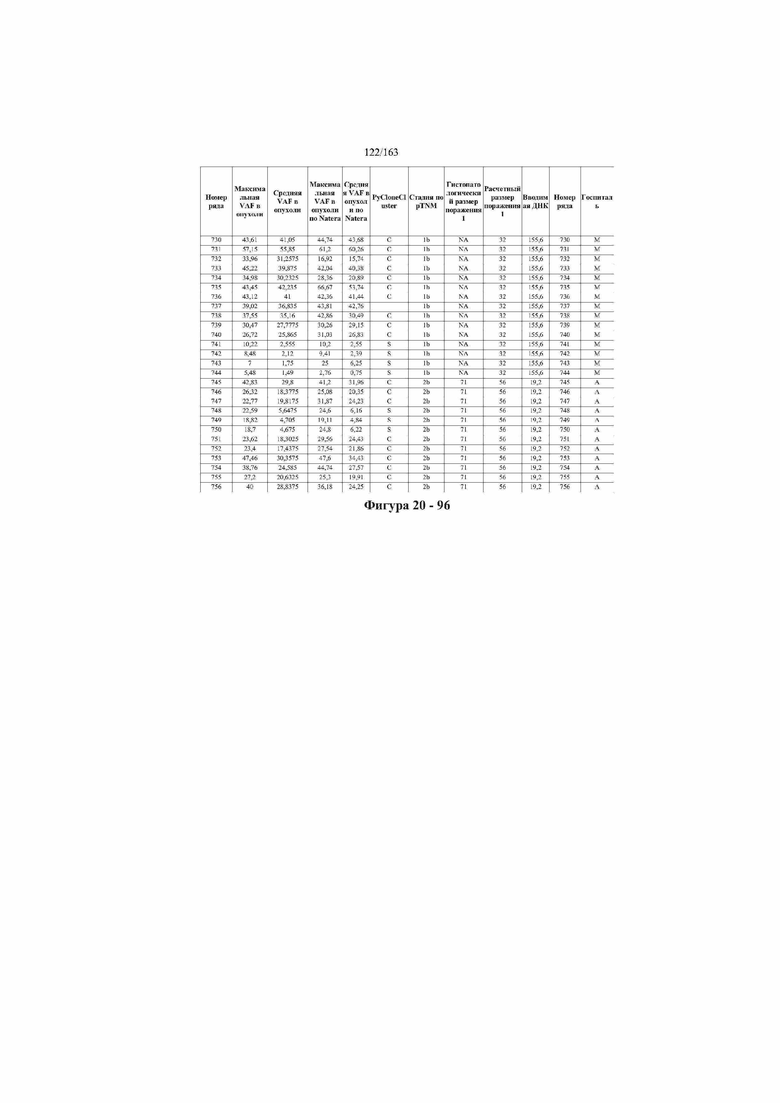
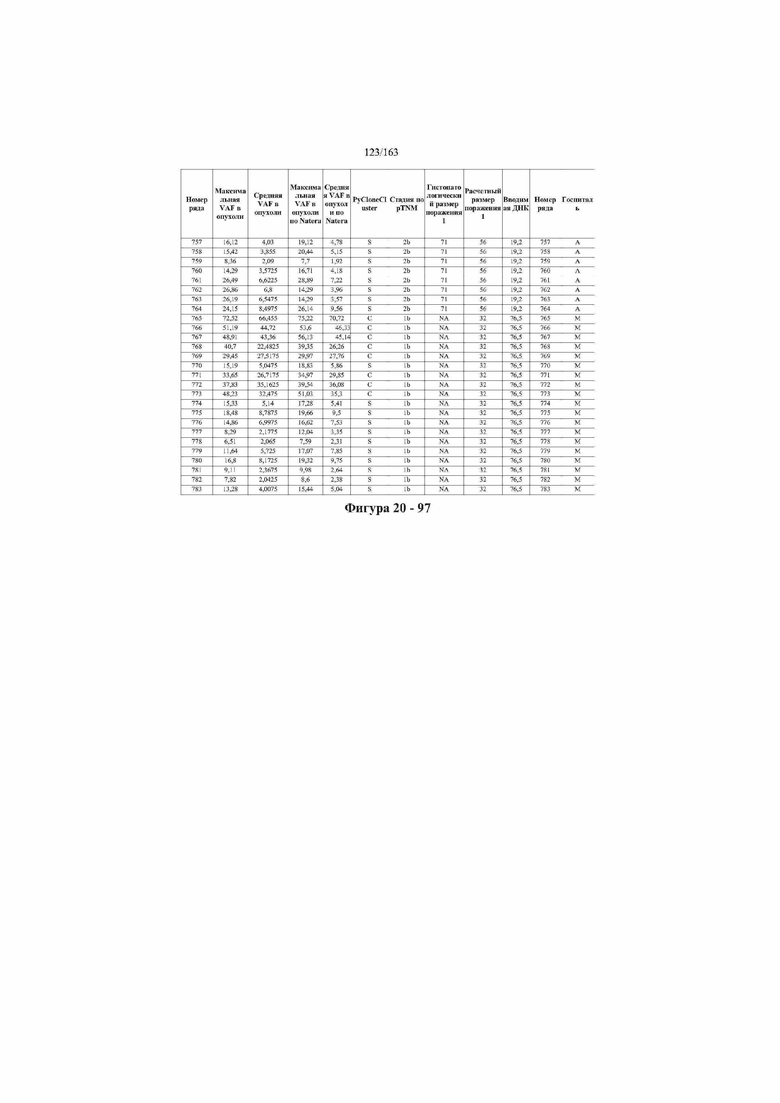
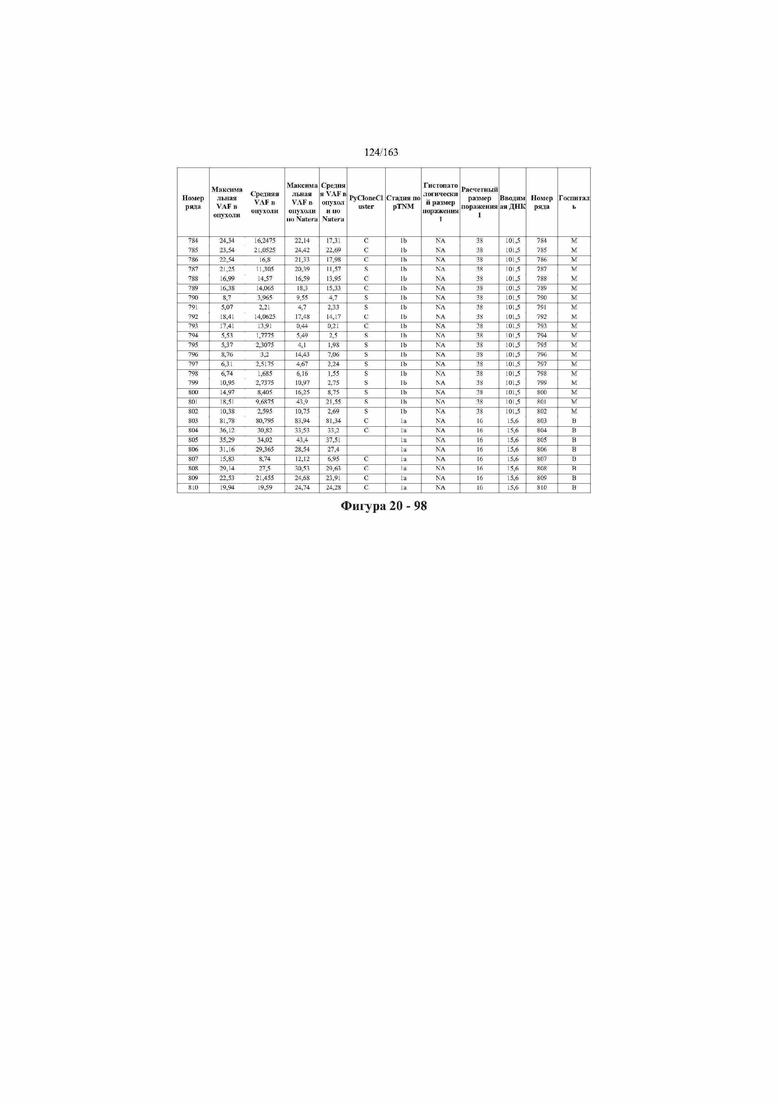

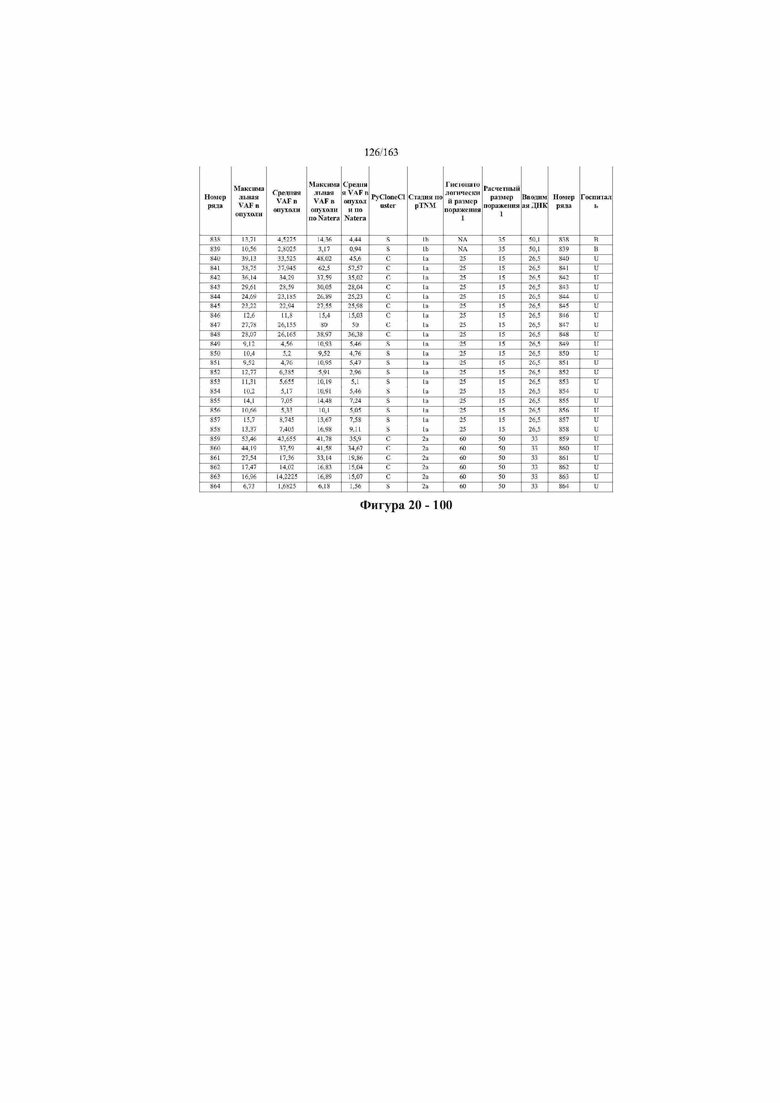
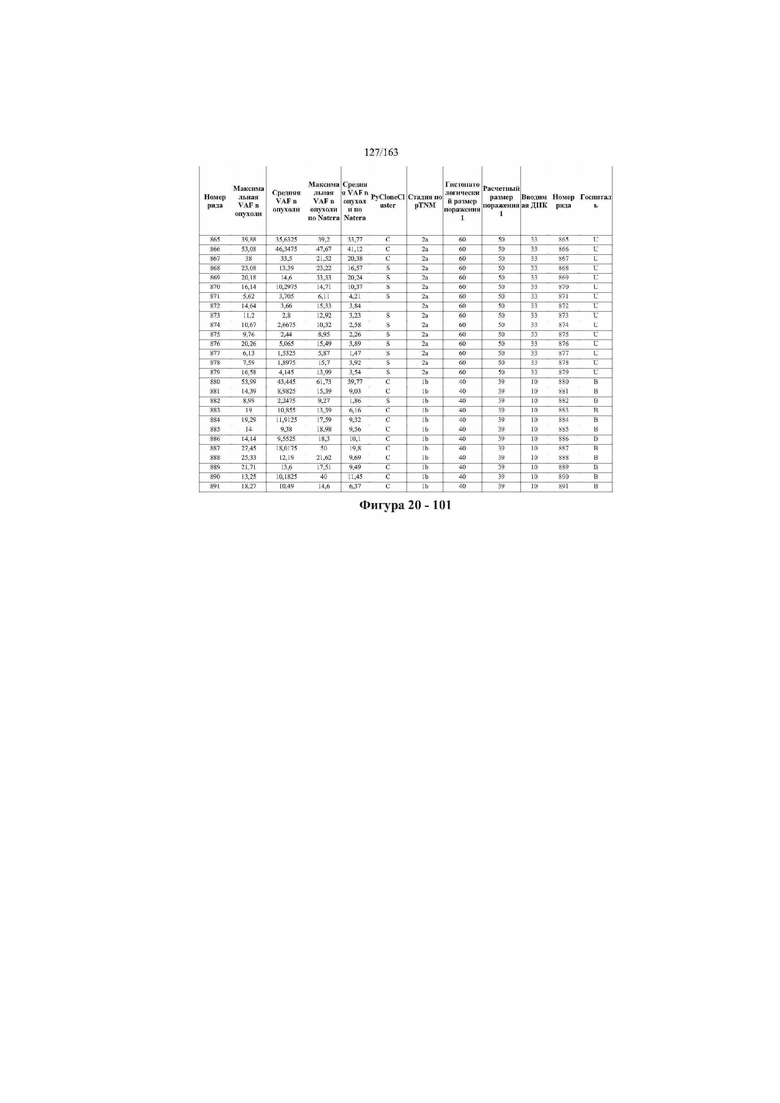
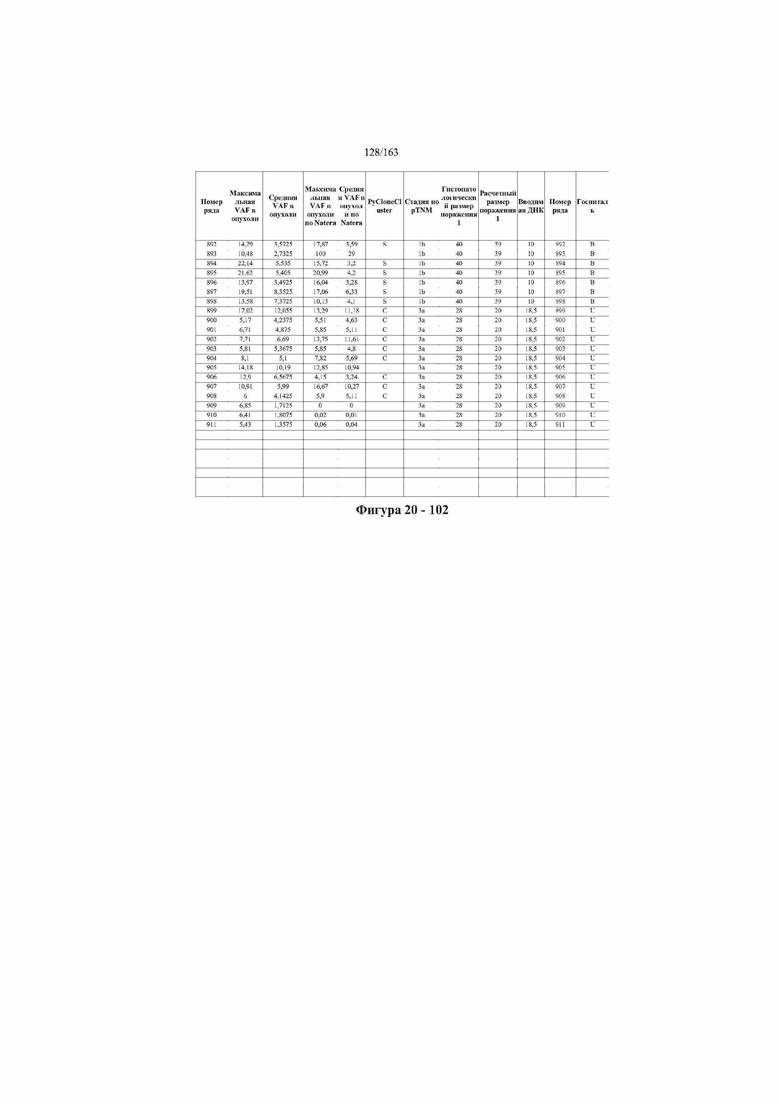
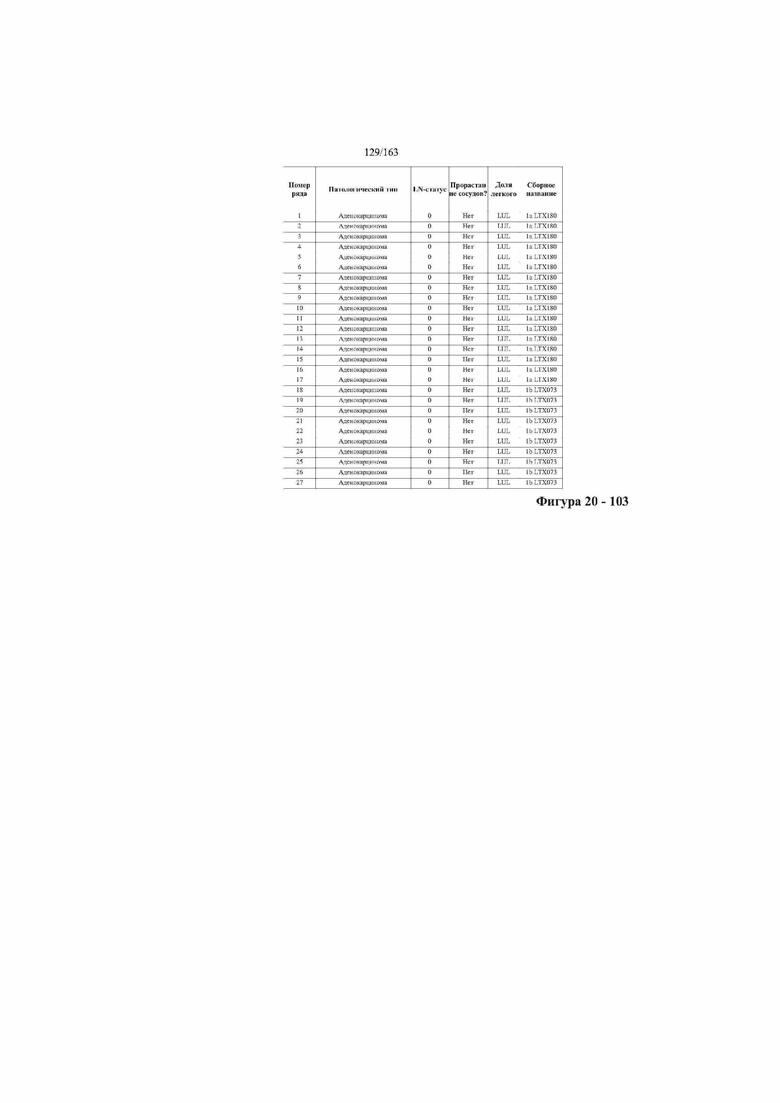
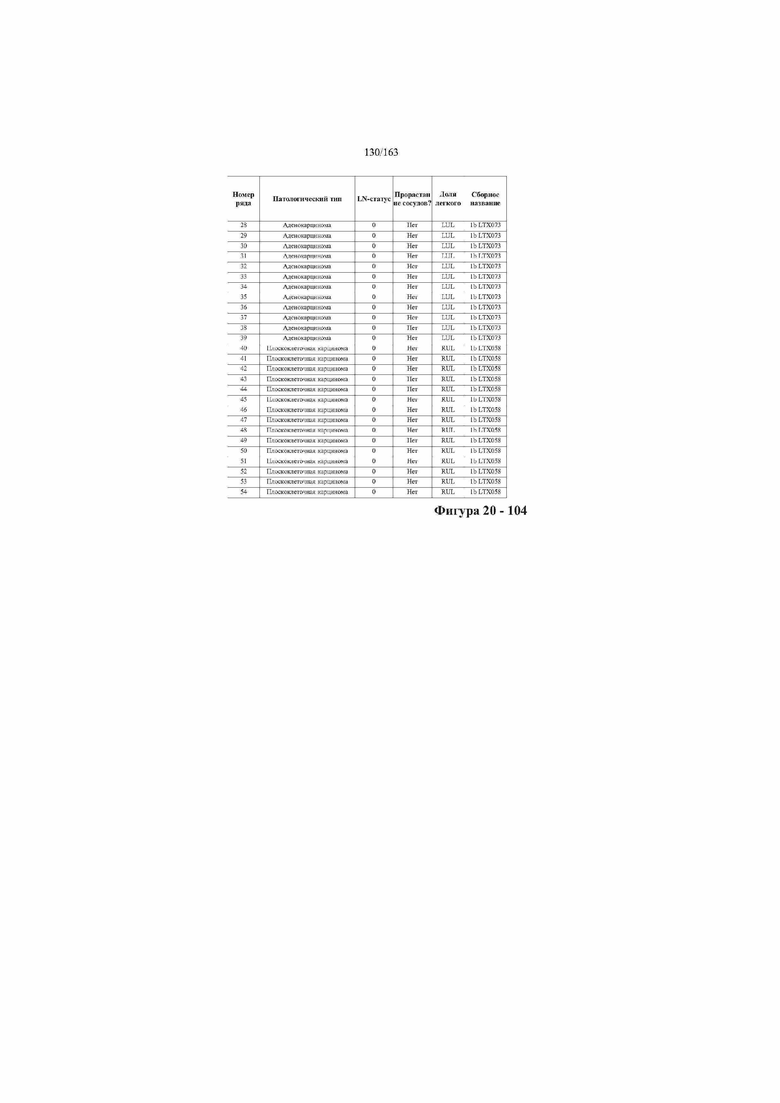
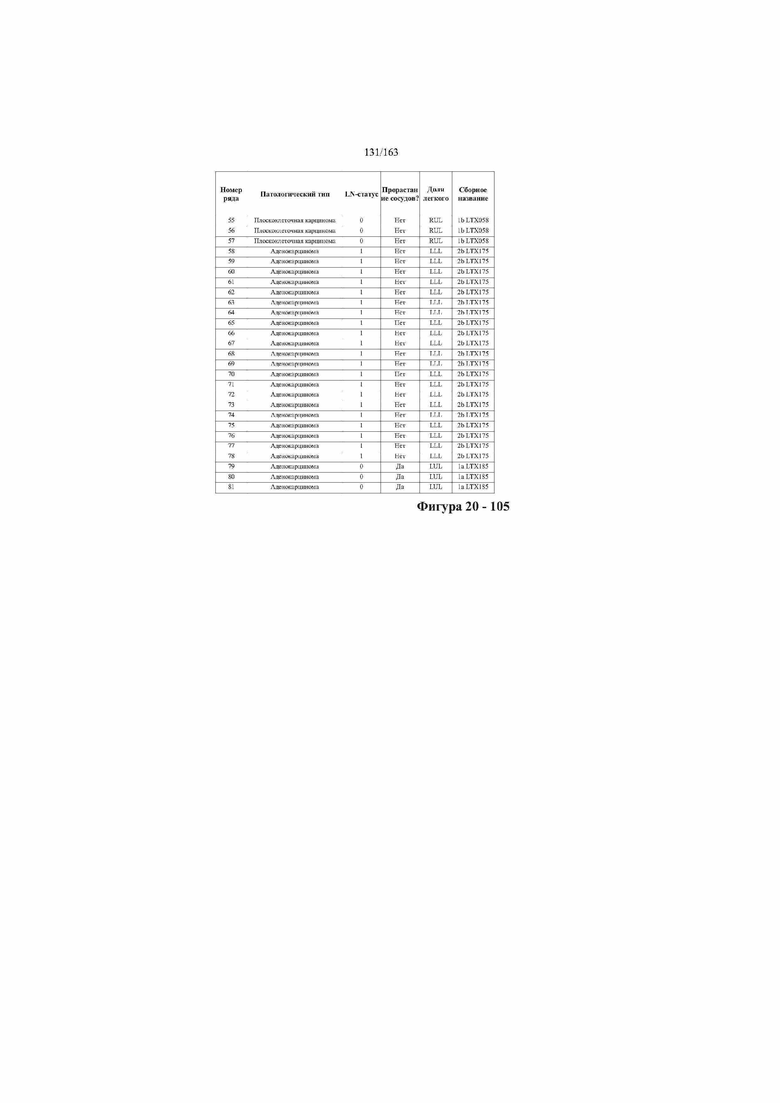
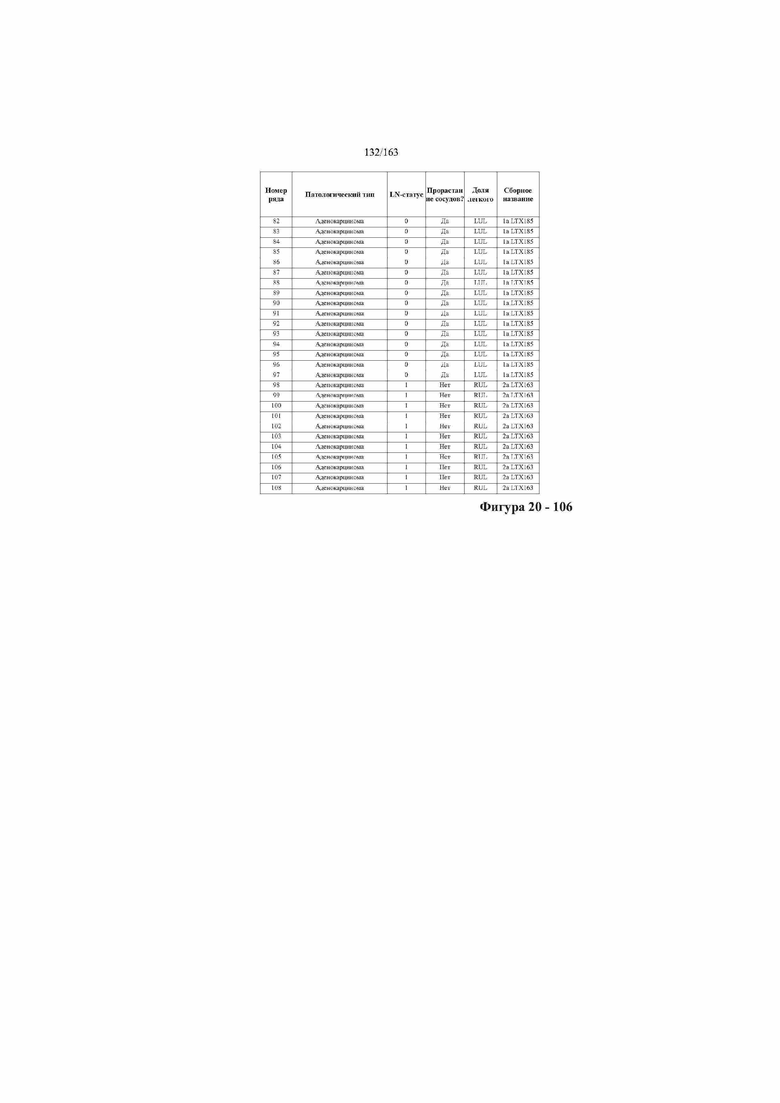
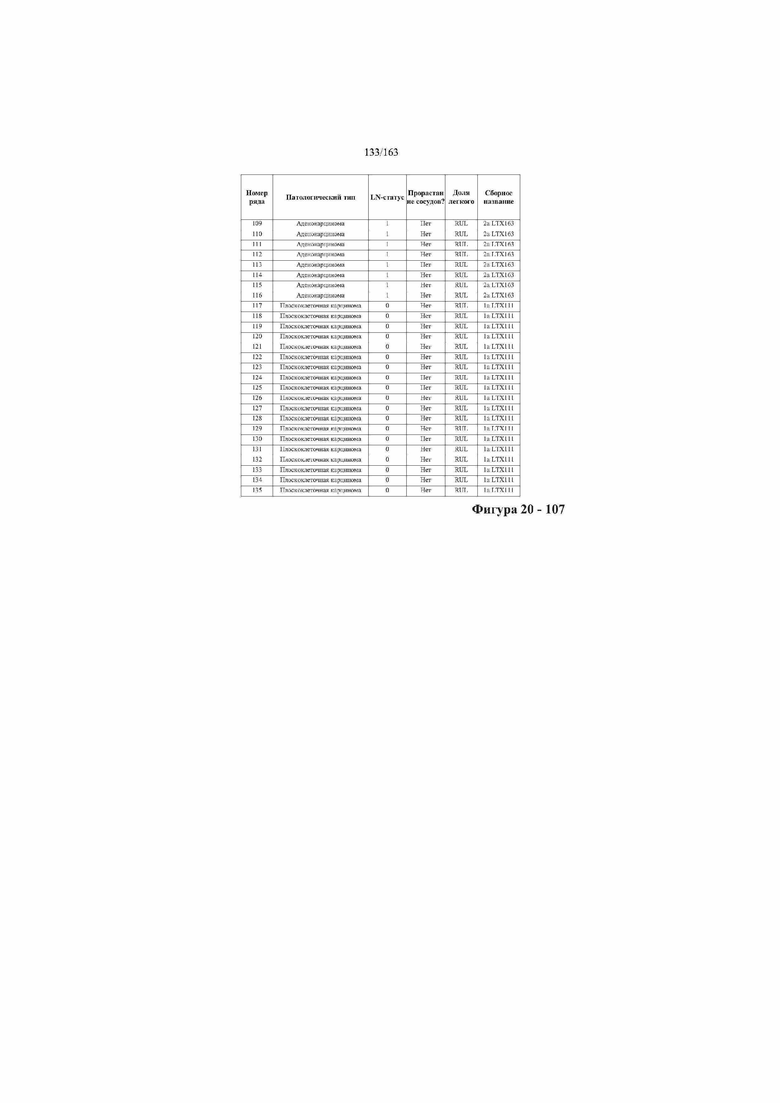

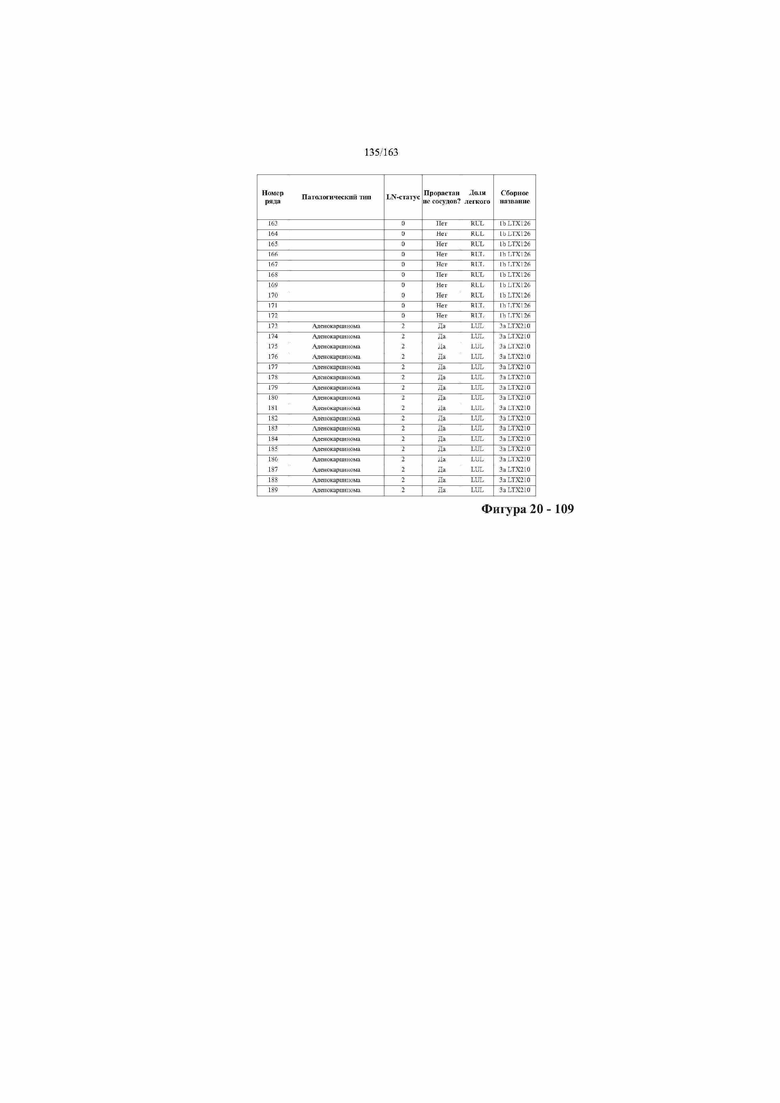

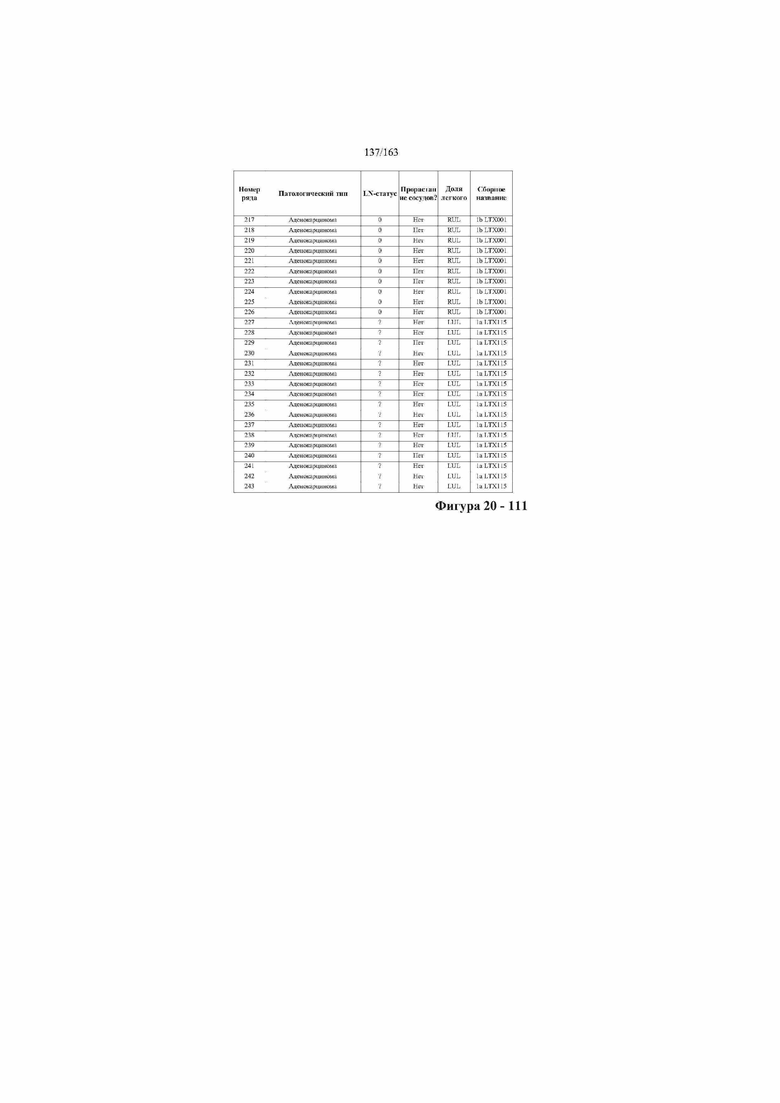
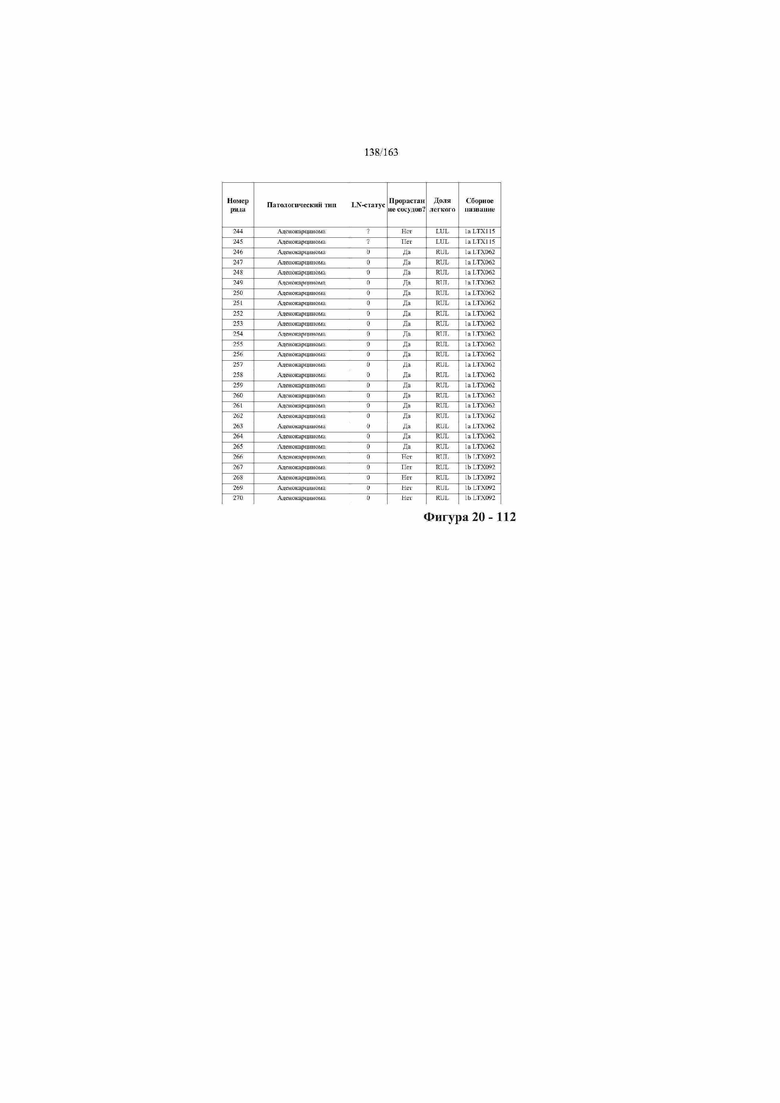


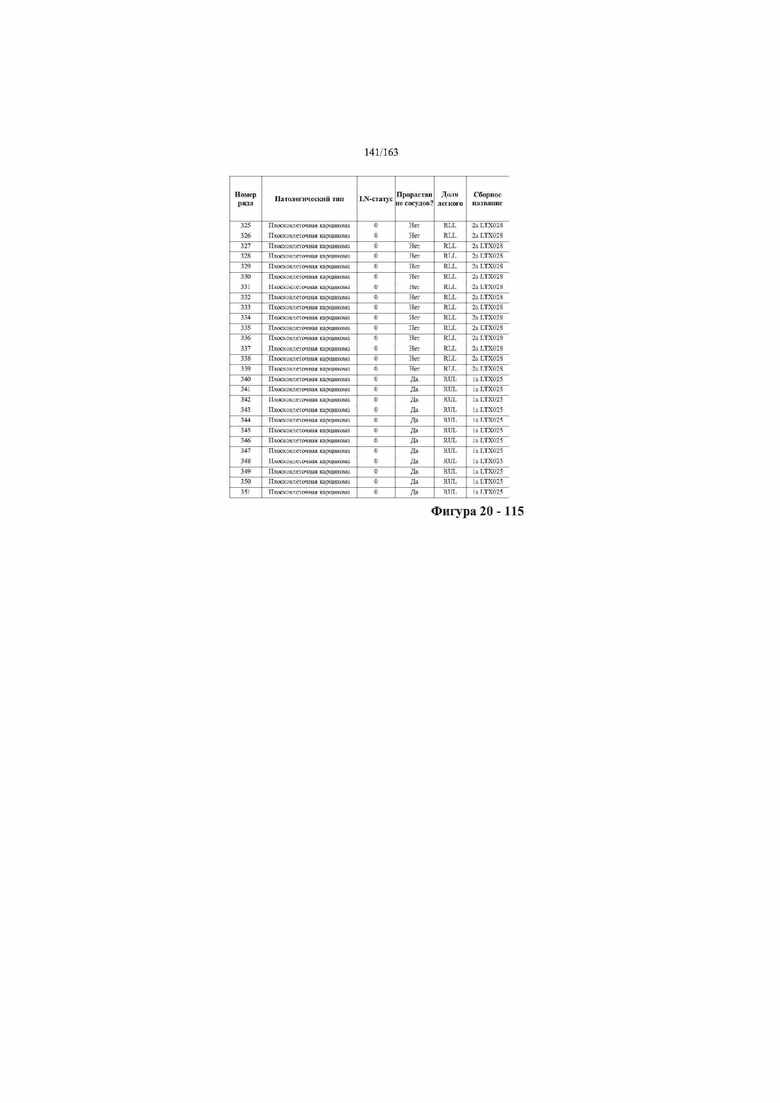


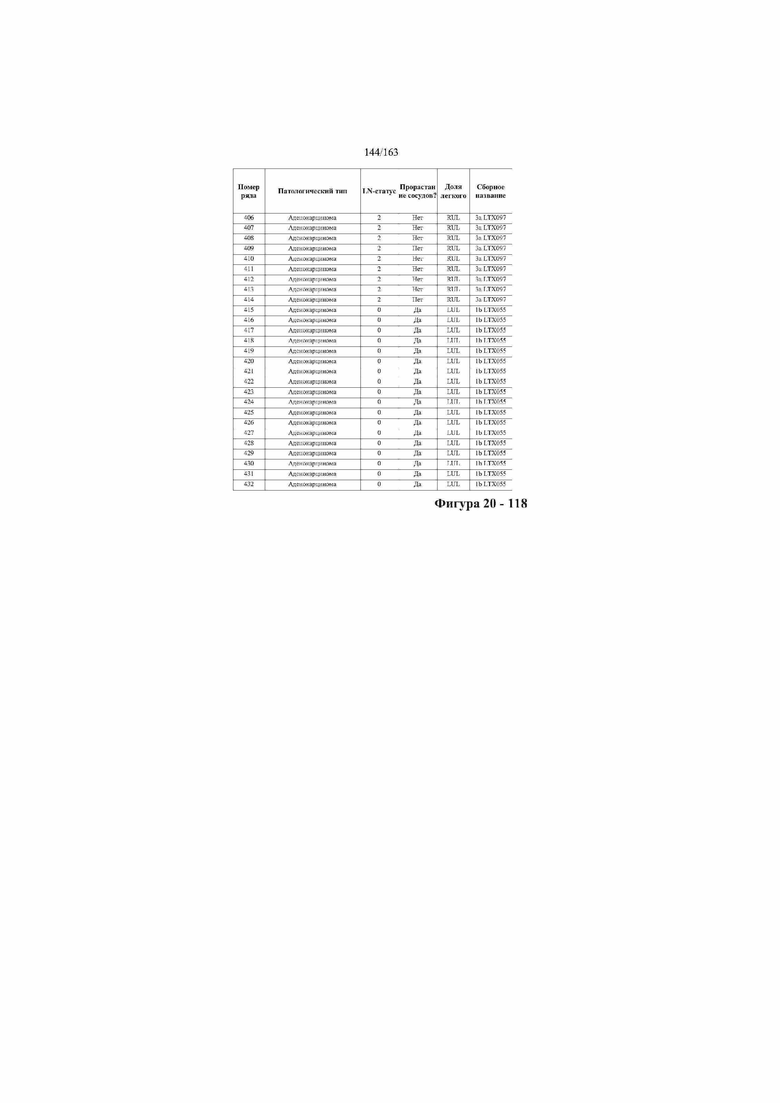

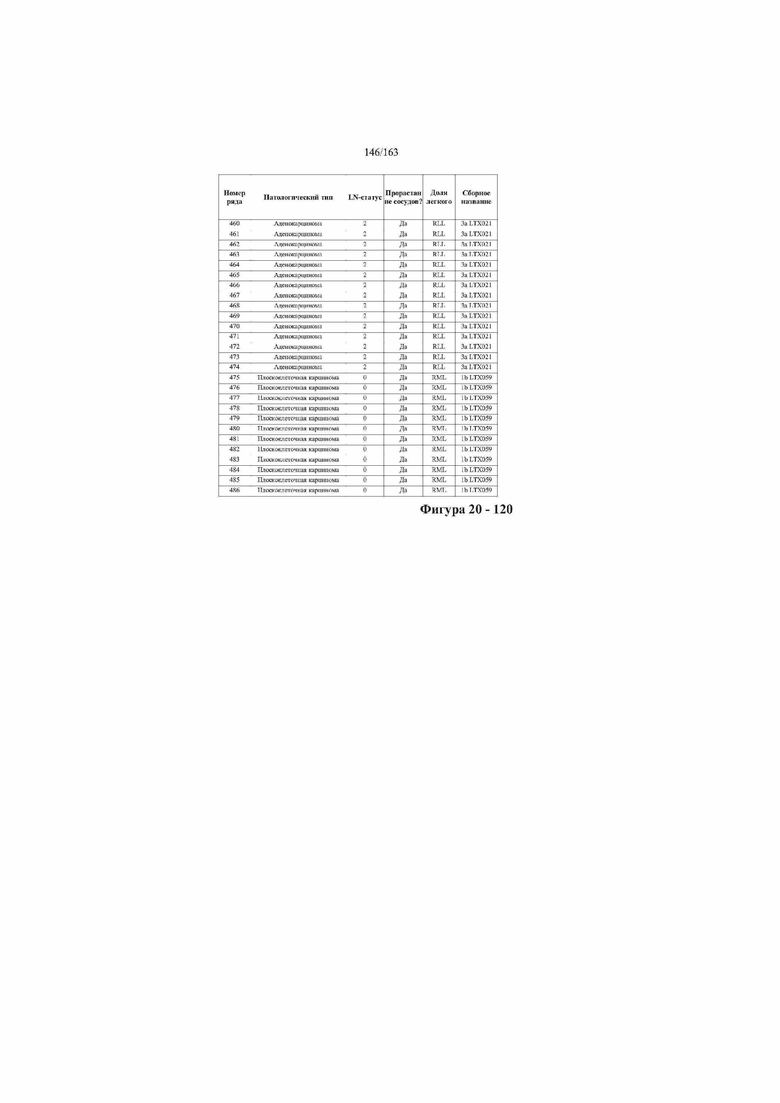
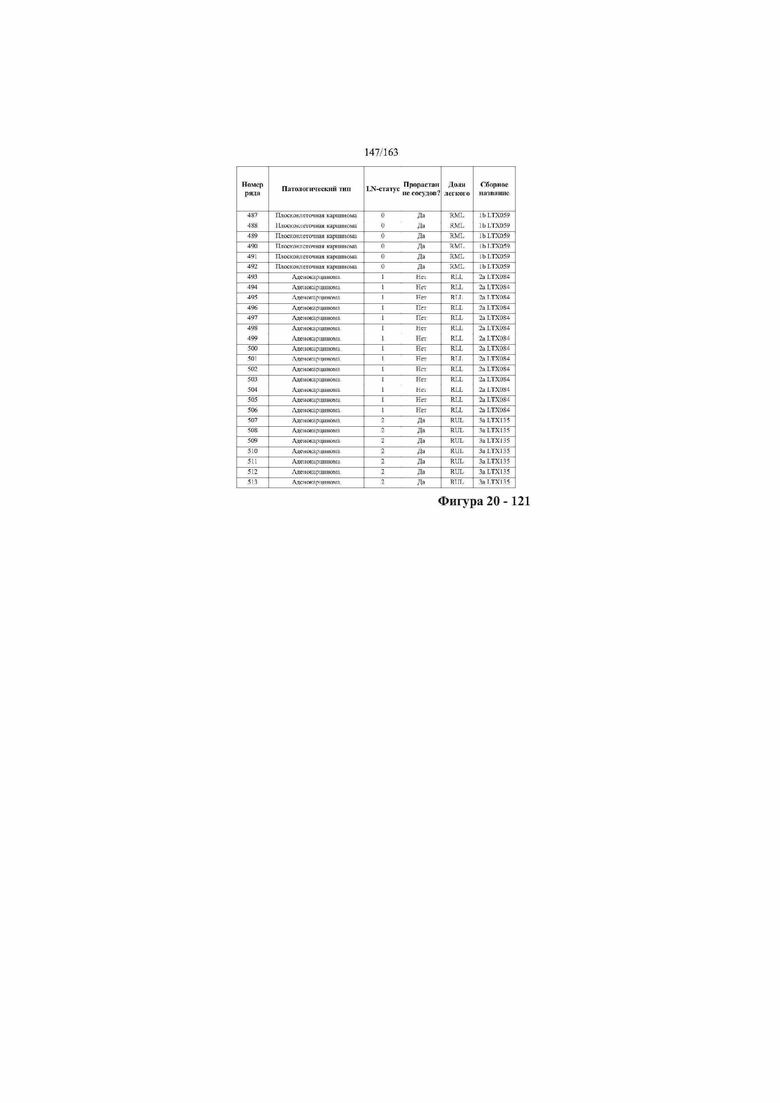
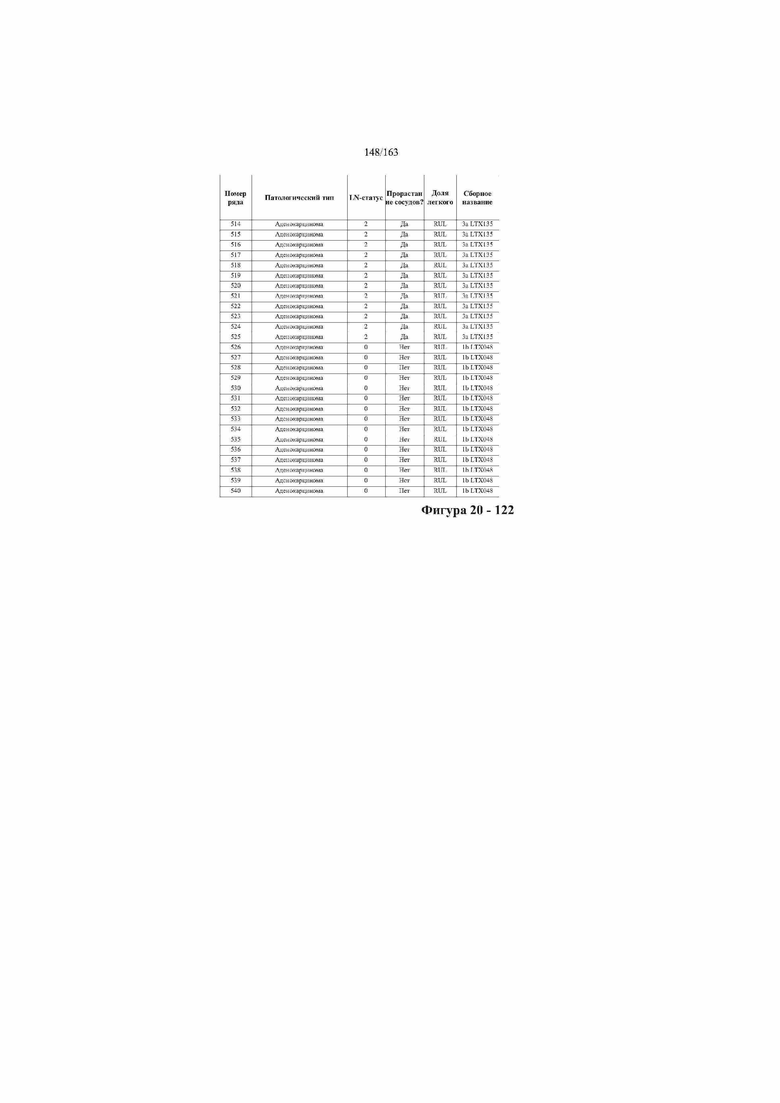
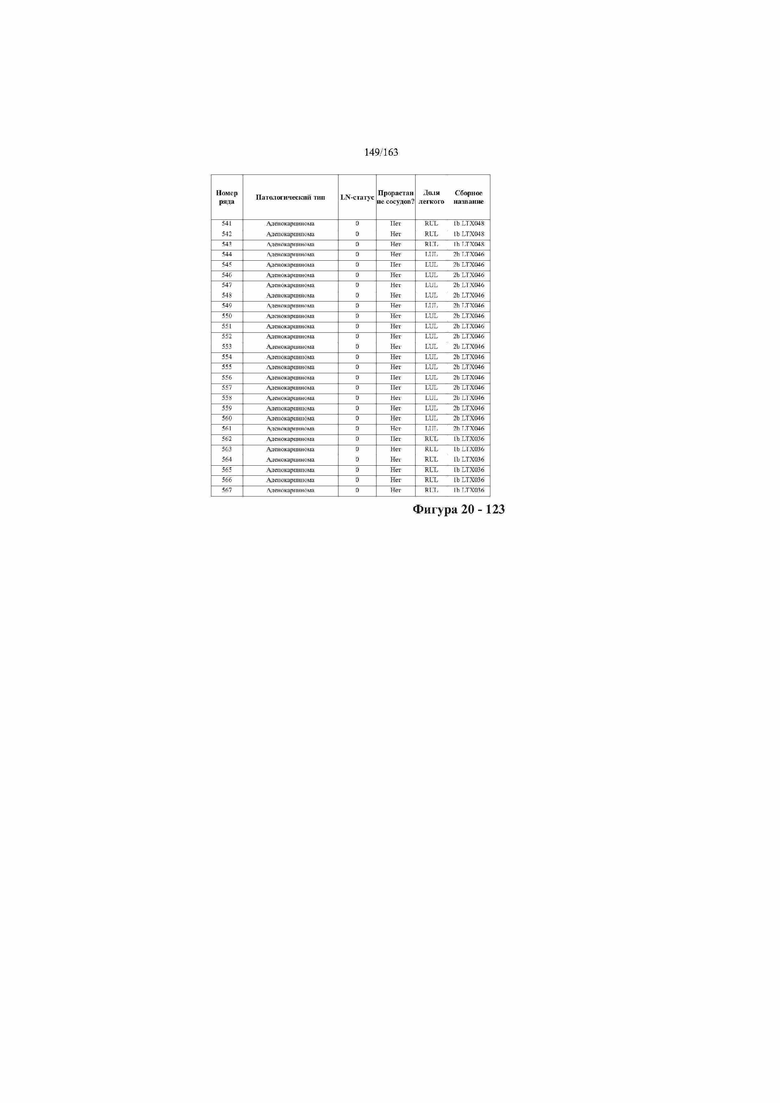
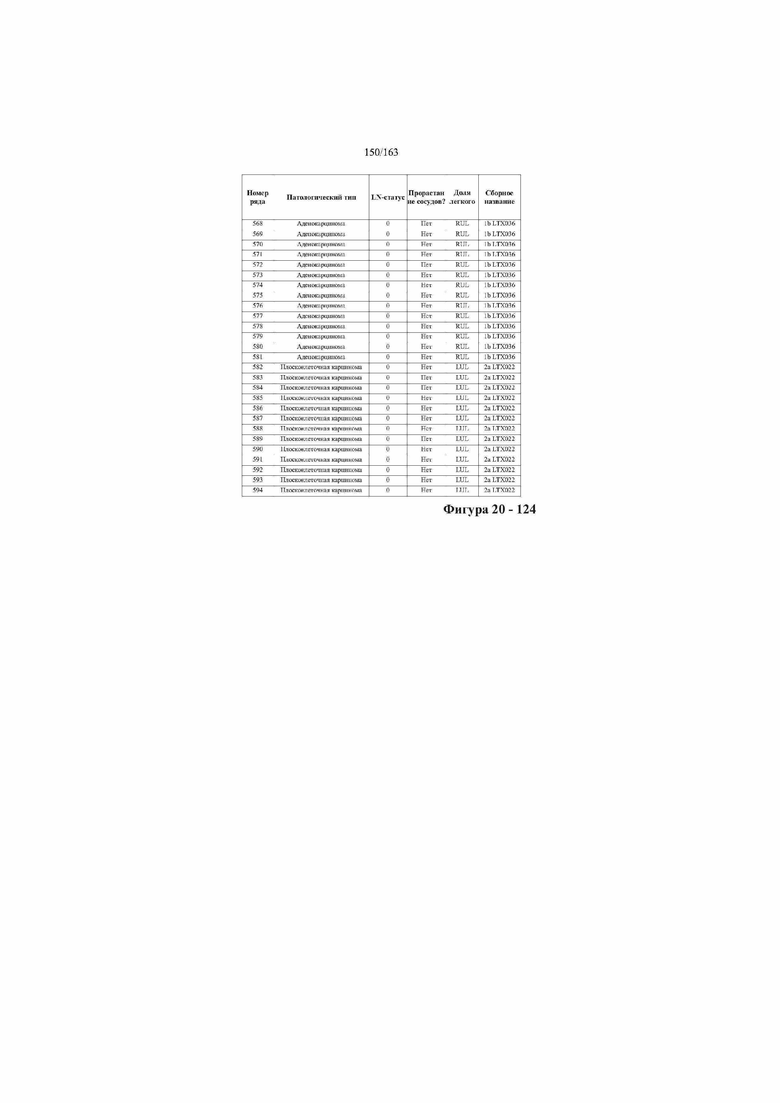
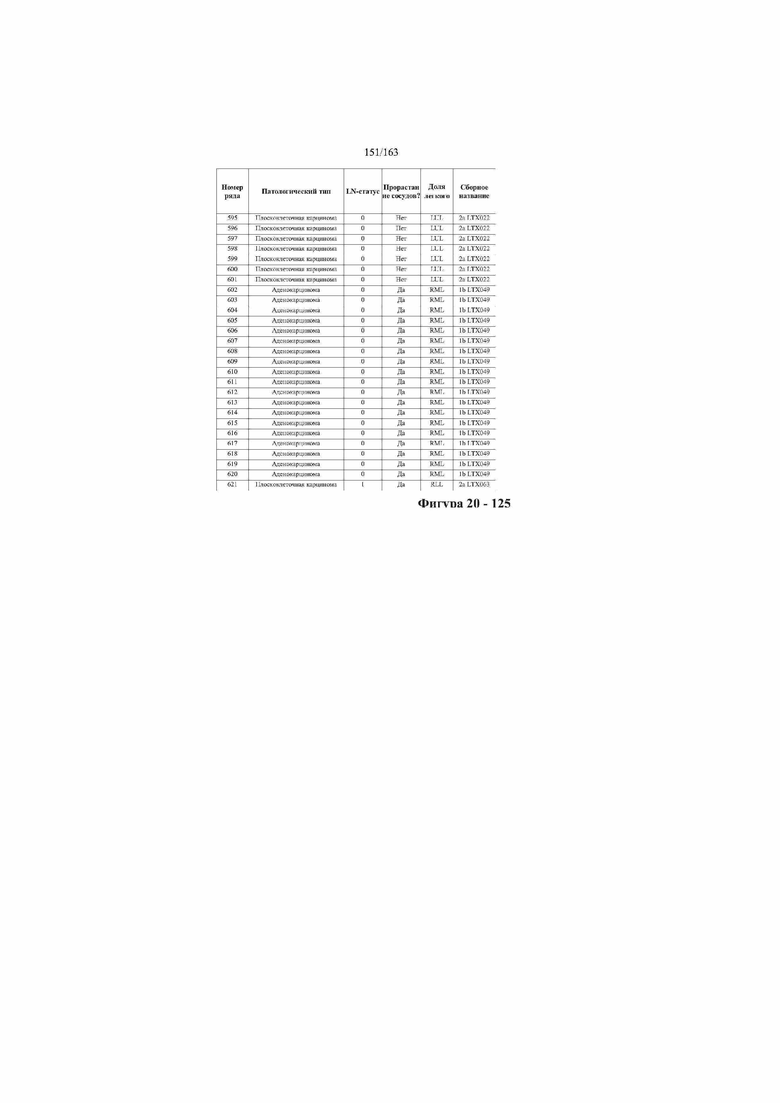






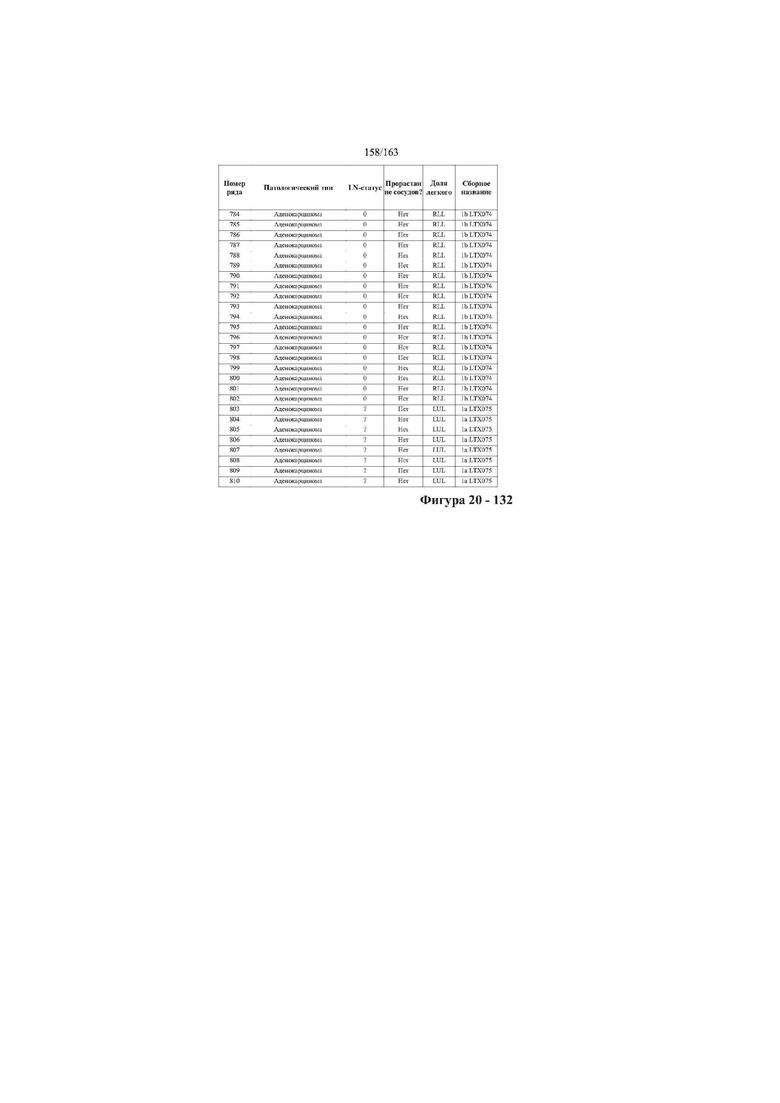





Изобретение относится к биотехнологии, в частности предусматриваются способы выявления однонуклеотидных вариантов при раке легкого, в частности аденокарциноме легкого стадии 3a и плоскоклеточной карциноме легкого. Предусматриваются дополнительные способы и композиции, такие как реакционные смеси и твердые подложки, содержащие клональные популяции нуклеиновых кислот. 4 н. и 35 з.п. ф-лы, 21 ил., 2 пр.
1. Способ определения присутствия однонуклеотидных вариантов, ассоциированных с плоскоклеточной карциномой легкого, предусматривающий:
создание и амплификацию библиотеки нуклеиновых кислот из нуклеиновых кислот, выделенных из образца опухоли от индивидуума, присоединение адаптеров для лигирования, содержащих петлю, к нуклеиновым кислотам, где адаптеры лигирования содержат универсальную праймерную последовательность, и амплификацию нуклеиновых кислот с помощью универсальной амплификации, где образец опухоли содержит образец ДНК с тупыми концами;
получение набора ампликонов путем
осуществления мультиплексной реакции ПЦР амплификации в отношении нуклеиновых кислот, выделенных из образца крови или ее фракции от индивидуума с подозрением на плоскоклеточную карциному легкого, для получения набора ампликонов, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, которые, как известно, ассоциированы с раком легкого,
где реакция мультиплексной ПЦР амплификации включает образование реакционной смеси для амплификации путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученной на основании взятого из опухоли образца крови или ее фракции, и набора прямых и обратных праймеров, каждый из которых связывается в пределах 25 пар оснований от локуса однонуклеотидного варианта, или набора пар праймеров, каждый из которых перекрывает участок из 25 пар оснований или меньше, содержащий локус однонуклеотидного варианта;
определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов для определения присутствия однонуклеотидных вариантов в плоскоклеточной карциноме; и
определение присутствия однонуклеотидных вариантов, если обнаружено по меньшей мере пять однонуклеотидных вариантов и Z-оценка, равная 10, против модели фоновой ошибки.
2. Способ по п. 1, где плоскоклеточная карцинома представляет собой плоскоклеточную карциному стадии 1a, 1b или 2a.
3. Способ по п. 1, где индивидуум не подвергается хирургическому вмешательству.
4. Способ по п. 1, где индивидуум не подвергается биопсии.
5. Способ по п. 1, дополнительно предусматривающий введение индивидууму соединения, при этом известно, что соединение является особенно эффективным в лечении плоскоклеточной карциномы легкого c одним или более определенными однонуклеотидными вариантами.
6. Способ по п. 1, где способ дополнительно предусматривает определение частоты вариантного аллеля для каждого из однонуклеотидных вариантов на основании определения последовательности.
7. Способ по п. 6, где план лечения рака легкого идентифицируют, исходя из определений частоты вариантного аллеля.
8. Способ по п. 6, дополнительно предусматривающий введение индивидууму соединения, при этом известно, что соединение является особенно эффективным в лечении плоскоклеточной карциномы легкого, характеризующейся одним из однонуклеотидных вариантов с частотой вариабельного аллеля, превышающей частоту по меньшей мере половины других однонуклеотидных вариантов, которые были определены.
9. Способ по п. 1, где нуклеиновые кислоты выделены из опухоли индивидуума, а однонуклеотидные варианты идентифицированы в опухоли из набора локусов однонуклеотидных вариантов перед определением последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов для образца крови или ее фракции.
10. Способ подтверждения диагноза рак легкого у индивидуума с подозрением на рак легкого на основании образца крови или ее фракции от индивидуума, предусматривающий:
создание и амплификацию библиотеки нуклеиновых кислот из нуклеиновых кислот, выделенных из образца опухоли от индивидуума, присоединение адаптеров для лигирования, содержащих петлю, к нуклеиновым кислотам, где адаптеры лигирования содержат универсальную праймерную последовательность, и амплификацию нуклеиновых кислот с помощью универсальной амплификации, где образец опухоли содержит образец ДНК с тупыми концами;
получение набора ампликонов путем
осуществления мультиплексной реакции ПЦР амплификации в отношении нуклеиновых кислот, выделенных из образца крови или ее фракции от индивидуума с подозрением на плоскоклеточную карциному легкого, для получения набора ампликонов, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, которые, как известно, ассоциированы с раком легкого,
где реакция мультиплексной ПЦР амплификации включает образование реакционной смеси для амплификации путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученной на основании взятого из опухоли образца крови или ее фракции, и набора прямых и обратных праймеров, каждый из которых связывается в пределах 25 пар оснований от локуса однонуклеотидного варианта, или набора пар праймеров, каждый из которых перекрывает участок из 25 пар оснований или меньше, содержащий локус однонуклеотидного варианта;
определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов для определения того, присутствуют ли однонуклеотидные варианты в плоскоклеточной карциноме, и
определение присутствия однонуклеотидных вариантов, если обнаружено по меньшей мере пять однонуклеотидных вариантов и Z-оценка, равная 10, против модели фоновой ошибки,
где:
a) отсутствие однонуклеотидного варианта подтверждает диагноз аденокарциномы стадии 1a, 2a или 2b,
b) присутствие однонуклеотидного варианта подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 2b или 3a или
c) присутствие десяти или более однонуклеотидных вариантов подтверждает диагноз плоскоклеточной карциномы или аденокарциномы стадии 2b или 3а.
11. Способ по п. 10, где способ дополнительно предусматривает определение стадии патологического изменения при раке легкого с помощью неинвазивного способа.
12. Способ выявления плоскоклеточной карциномы легкого в образце крови или ее фракции от индивидуума с подозрением на рак легкого, предусматривающий:
создание и амплификацию библиотеки нуклеиновых кислот из нуклеиновых кислот, выделенных из образца опухоли от индивидуума, присоединение адаптеров для лигирования, содержащих петлю, к нуклеиновым кислотам, где адаптеры лигирования содержат универсальную праймерную последовательность, и амплификацию нуклеиновых кислот с помощью универсальной амплификации, где образец опухоли содержит образец ДНК с тупыми концами;
получение набора ампликонов путем
осуществления мультиплексной реакции ПЦР амплификации в отношении нуклеиновых кислот, выделенных из образца крови или его фракции от индивидуума с подозрением на плоскоклеточную карциному легкого, для получения набора ампликонов, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, которые, как известно, ассоциированы с раком легкого,
где реакция мультиплексной ПЦР амплификации включает образование реакционной смеси для амплификации путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученной на основании взятого из опухоли образца крови или ее фракции, и набора прямых и обратных праймеров, каждый из которых связывается в пределах 25 пар оснований от локуса однонуклеотидного варианта, или набора пар праймеров, каждый из которых перекрывает участок из 25 пар оснований или меньше, содержащий локус однонуклеотидного варианта;
определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов, определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов для определения того, присутствуют ли однонуклеотидные варианты в плоскоклеточной карциноме; и
определение присутствия однонуклеотидных вариантов, если обнаружено по меньшей мере пять однонуклеотидных вариантов и Z-оценка, равная 10, против модели фоновой ошибки,
где присутствие однонуклеотидного варианта в образце для любого из нескольких однонуклеотидных локусов является показателем присутствия плоскоклеточной карциномы.
13. Способ выявления клонального однонуклеотидного варианта в опухоли легкого у индивидуума, предусматривающий:
создание и амплификацию библиотеки нуклеиновых кислот из нуклеиновых кислот, выделенных из образца опухоли от индивидуума, присоединение адаптеров для лигирования, содержащих петлю, к нуклеиновым кислотам, где адаптеры лигирования содержат универсальную праймерную последовательность, и амплификацию нуклеиновых кислот с помощью универсальной амплификации, где образец опухоли содержит образец ДНК с тупыми концами;
получение набора ампликонов путем
осуществления мультиплексной реакции ПЦР амплификации в отношении нуклеиновых кислот, выделенных из образца крови или ее фракции от индивидуума с подозрением на плоскоклеточную карциному легкого, для получения набора ампликонов, где каждый ампликон из набора ампликонов перекрывает по меньшей мере один локус однонуклеотидного варианта из набора локусов однонуклеотидных вариантов, которые, как известно, ассоциированы с раком легкого,
где реакция мультиплексной ПЦР амплификации включает образование реакционной смеси для амплификации путем объединения полимеразы, нуклеотидтрифосфатов, фрагментов нуклеиновой кислоты из библиотеки нуклеиновых кислот, полученной на основании взятого из опухоли образца крови или ее фракции, и набора прямых и обратных праймеров, каждый из которых связывается в пределах 25 пар оснований от локуса однонуклеотидного варианта, или набора пар праймеров, каждый из которых перекрывает участок из 25 пар оснований или меньше, содержащий локус однонуклеотидного варианта;
определение последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов для определения того, присутствуют ли однонуклеотидные варианты в плоскоклеточной карциноме;
определение присутствия однонуклеотидных вариантов, если обнаружено по меньшей мере пять однонуклеотидных вариантов и Z-оценка, равная 10, против модели фоновой ошибки; и
определение частоты вариантного аллеля для каждого из локусов однонуклеотидных вариантов (SNV), исходя из последовательности нескольких копий из серий ампликонов, где более высокая относительная частота аллеля по сравнению с другими однонуклеотидными вариантами из нескольких локусов однонуклеотидных вариантов является показателем клонального однонуклеотидного варианта в опухоли.
14. Способ по п. 13, дополнительно предусматривающий введение индивидууму соединения, которое нацеливается на один или более клональных однонуклеотидных вариантов, но не на другие однонуклеотидные варианты.
15. Способ по п. 13, где частота вариантного аллеля, превышающая 1,0%, является показателем клонального однонуклеотидного варианта.
16. Способ по п. 13, где плоскоклеточная карцинома представляет собой плоскоклеточную карциному стадии 1a, 1b или 2a.
17. Способ по п. 13, где плоскоклеточная карцинома представляет собой плоскоклеточную карциному стадии 1a или 1b.
18. Способ по п. 13, где индивидуум не подвергается хирургическому вмешательству.
19. Способ по п. 13, где индивидуум не подвергается биопсии.
20. Способ по п. 13, дополнительно предусматривающий введение индивидууму соединения, при этом известно, что соединение является особенно эффективным в лечении плоскоклеточной карциномы легкого c одним или более определенными однонуклеотидными вариантами.
21. Способ по п. 13, где способ дополнительно предусматривает определение частоты вариантного аллеля для каждого из однонуклеотидных вариантов на основании определения последовательности.
22. Способ по п. 13, дополнительно предусматривающий разработку плана лечения рака легкого, исходя из определений частоты вариантного аллеля.
23. Способ по п. 13, дополнительно предусматривающий введение индивидууму соединения, при этом известно, что соединение является особенно эффективным в лечении плоскоклеточной карциномы легкого, характеризующейся одним из однонуклеотидных вариантов с частотой вариабельного аллеля, превышающей частоту по меньшей мере половины других однонуклеотидных вариантов, которые были определены.
24. Способ по п. 13, где нуклеиновые кислоты выделены из опухоли индивидуума, а однонуклеотидные варианты идентифицированы в опухоли из набора локусов однонуклеотидных вариантов перед определением последовательности по меньшей мере сегмента каждого ампликона из набора ампликонов для образца крови или ее фракции.
25. Способ по любому из предыдущих пунктов, где выявляются по меньшей мере 5 SNV и где присутствие по меньшей мере 5 SNV является показателем плоскоклеточной карциномы.
26. Способ по любому из пп. 1-25, где определение того, присутствует ли в образце однонуклеотидный вариант, предусматривает идентификацию значения достоверности для каждого определения аллеля в каждом из наборов локусов однонуклеотидной изменчивости, исходя по меньшей мере частично из глубины прочтения для локуса.
27. Способ по любому из пп. 1-25, где распознавание однонуклеотидного варианта осуществляют, если значение достоверности в отношении присутствия однонуклеотидного варианта превышает 90%.
28. Способ по любому из пп. 1-25, где распознавание однонуклеотидного варианта осуществляют, если значение достоверности в отношении присутствия однонуклеотидного варианта превышает 95%.
29. Способ по любому из пп. 1-25, где набор локусов однонуклеотидной изменчивости содержит все локусы однонуклеотидной изменчивости, идентифицированные в наборах данных TCGA и COSMIC для рака легкого.
30. Способ по любому из пп. 1-25, где набор сайтов однонуклеотидной изменчивости содержит все сайты однонуклеотидной изменчивости, идентифицированные в наборах данных TCGA и COSMIC для плоскоклеточной карциномы легкого.
31. Способ по любому из пп. 1-25, где способ осуществляют при глубине прочтения для набора локусов однонуклеотидной изменчивости, составляющей по меньшей мере 1000.
32. Способ по любому из пп. 1-25, где набор локусов однонуклеотидных вариантов содержит 25—1000 локусов однонуклеотидной изменчивости, которые, как известно, ассоциированы с раком легкого.
33. Способ по любому из пп. 1-25, где эффективность и частоту ошибок на цикл определяют для каждой реакции амплификации из мультиплексной реакции амплификации локусов однонуклеотидной изменчивости и эффективность и частоту ошибок применяют для определения того, присутствует ли в образце однонуклеотидный вариант из набора локусов однонуклеотидных вариантов.
34. Способ по любому из пп. 1-25, где реакция амплификации представляет собой ПЦР-реакцию, а температура отжига на 1—15°C превышает температуру плавления по меньшей мере 50% праймеров из набора праймеров.
35. Способ по любому из пп. 1-25, где реакция амплификации представляет собой ПЦР-реакцию и продолжительность стадии отжига в ПЦР-реакции составляет 15—120 минут.
36. Способ по любому из пп. 1-25, где концентрация праймеров в реакции амплификации составляет 1—10 нМ.
37. Способ по любому из пп. 1-29, где праймеры в наборе праймеров разработаны для сведения к минимуму образования димеров из праймеров.
38. Способ по любому из пп. 1-25, где реакция амплификации представляет собой ПЦР-реакцию, причем температура отжига на 1—15°C превышает температуру плавления по меньшей мере 50% праймеров из набора праймеров, продолжительность стадии отжига в ПЦР-реакции составляет 15—120 минут, концентрация праймеров в реакции амплификации составляет 1—10 нМ и праймеры в наборе праймеров разработаны для сведения к минимуму образования димеров из праймеров.
39. Способ по любому из пп. 1-25, где мультиплексную реакцию амплификации осуществляют в условиях ограничения праймеров.
| M.Jamal-Hanjani et al.,Detection of ubiquitous and heterogeneous mutations in cell-free DNA from patients with early-stage non-small-cell lung cancer, Annals of Oncology, Volume 27, Issue 5,2016, pp 862-867 | |||
| Маршутина Н | |||
| В | |||
| и др., Сравнительная клинико-диагностическая значимость некоторых серологических опухолеассоциированных маркеров для |
Авторы
Даты
2021-12-01—Публикация
2017-04-17—Подача