Изобретение относится к области биотехнологии, в частности к определению мутации S:P681R SARS-CoV-2 с использованием полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) и может применяться для идентификации геновариантов SARS-CoV-2 при эпидемиологических исследованиях.
Коронавирусная инфекция (COVID-19) это инфекционное заболевание, вызванное SARS-CoV-2. Вирус передается от человека к человеку воздушно-капельным путем. Изменения в структуре генома SARS-CoV-2 приводят к возникновению вариантов, которые ВОЗ обозначила как вызывающие интерес (VOIs, Variants of Interest), вызывающие озабоченность (VOCs, Variants of Concern) и линии VOC под мониторингом (VOC lineages under monitoring (VOC-LUM). Определение мутаций в геноме SARS-CoV-2 и их классификация на VOIs и VOCs и VOC-LUM является важным элементом молекулярно-генетического мониторинга штаммов новой коронавирусной инфекции. Мутации, детекция которых необходима, перечислены в докладах Технической консультативной группы по эволюции вируса SARS-CoV-2 (TAG-VE) ВОЗ [https://www.who.int/publications/m/item/terms-of-reference-for-the-technical-advisory-group-on-sars-cov-2-virus-evolution-(tag-ve)].
Для определения геновариантов вируса и проведения молекулярно-генетического мониторинга SARS-CoV-2 используются методы полногеномного и фрагментного секвенирования, которые являются дорогостоящими и трудоемкими. С целью выявления SARS-CoV-2 Всемирной организацией здравоохранения предложено выделение РНК возбудителя с последующим проведением полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР) [https://www.who.int/emergencies/diseases/novel-coronavirus-2019/technical-guidance/laboratory-guidance].
Известные протоколы исследования направлены на выявление специфических нуклеотидных последовательностей в генах и межгенных промежутках:
- ORF1ab, N [http://ivdc.chinacdc.cn/kyjz/202001/t20200121_211337.html];
- RdRP, Е, N [https://www.who.int/docs/default-source/coronaviruse/protocol-v2-1.pdf?sfvrsn=a9ef618 с_2];
- ORF1b-nspH, N [https://www.who.int/docs/default-source/coronaviruse/peiris-protocol-16-l-20.pdf?sfvrsn=af1aac73_4];
- S, N [https://www.who.int/docs/default-source/coronaviruse/method-niid-20200123-2.pdf?sfvrsn=fbf75320_7];
- N [https://www.who.int/docs/default-source/coronaviruse/conventional-rt-pcr-followed-by-sequencing-for-detection-of-ncov-rirl-nat-inst-health-t.pdf?sfvrsn=42271c6d_4];
- N [https://www.fda.gov/media/134922/download, https://www.who.int/docs/default-source/coronaviruse/uscdcrt-pcr-panel-primer-probes.pdf?sfvrsn=fa29cb4b_2];
- RdRP [https://www.who.int/docs/default-source/coronaviruse/real-time-rt-pcr-assays-for-the-detection-of-sars-cov-2-institut-pasteur-paris.pdf?sfvrsn=3662fcb6_2].
При этом, проведенный анализ полногеномных нуклеотидных последовательностей SARS-CoV-2 показал частое наличие мутаций (в особенности делеций) в геномах данного коронавируса. В связи с этим основным недостатком приведенных выше аналогов является риск получения ложно отрицательных результатов ОТ-ПЦР, обусловленных блокированием реакции при наличии мутаций в области амплифицируемого участка.
Из уровня техники известен набор синтетических олигонуклеотидов для выявления РНК коронавируса и генетического типирования и выявления мутантных участков генома SARS-CoV-2 [Патент RU 2761025, дата подачи 26.07.2021]. Набор включает в себя от одной до четырнадцати комбинаций праймеров, при мультиплексной реакции фланкирующих с обеих сторон нуклеотидные последовательности, специфичные для вызывающих озабоченность генетических вариантов SARS-CoV-2: Wul-fw, E484K-fw, K417N-fw, K417T-fw, T478K-fw, Wul-rv, N501Y-rv, P681H-rv и P681R-rv.
Также из уровня техники известен реагент обнаружения нуклеиновых кислот, комплект и метод обнаружения для нового варианта Delta коронавируса 2019 года [Патент CN 113774169, 18.09.2021, 10.12.2021]. Способ определяет применение специфических праймеров и зондов, которые предназначены для детекции Delta-варианта (Delta (В. 1.617.2)) S-белка, кодирующего гены T19R, L452R, T478K и P681R, а также может содержать одностадийный метод ОТ-ПЦР, положительный и отрицательный контрольные образцы.
Мультиплексные наборы реагентов для определения геновариантов SARS-CoV-2 методом ПЦР, разработаны несколькими компаниями-производителями. Предложенная компанией ThermoFisher Scientific методика основана на использовании зондов типа TaqMan для определения сразу нескольких мутаций, специфичных для разных геновариантов [Enhanced fusogenicity and pathogenicity of SARS-CoV-2 Delta P681R mutation. Akatsuki Saito, Takashi Irie, Rigel Suzuki, Tadashi Maemura, Hesham Nasser, Keiya Uriu, Yusuke Kosugi, Kotaro Shirakawa, Kenji Sadamasu, Izumi Kimura, Jumpei Ito, Jiaqi Wu, Kiyoko Iwatsuki-Horimoto, Mutsumi Ito, Seiya Yamayoshi, Samantha Loeber, Masumi Tsuda, Lei Wang, Seiya Ozono, Erika P. Butlertanaka, Yuri L. Tanaka, Ryo Shimizu, Kenta Shimizu, Kumiko Yoshimatsu, Ryoko Kawabata, Takemasa Sakaguchi, Kenzo Tokunaga, Isao Yoshida, Hiroyuki Asakura, Mami Nagashima, Yasuhiro Kazuma, Ryosuke Nomura, Yoshihito Horisawa, Kazuhisa Yoshimura, Akifumi Takaori-Kondo, Masaki Imai, The Genotype to Phenotype Japan (G2P-Japan) Consortium, Shinya Tanaka, So Nakagawa, Terumasa Ikeda, Takasuke Fukuhara, Yoshihiro Kawaoka & Kei Sato, https://doi.org/l0.1038/s41586-021-04266-91.
Наборы, в которых предусмотрена мультиплексная ПЦР-РВ после проведения обратной транскрипции отличаются удобством для пользователя [Emergency SARS-CoV-2 Variants of Concern: Novel Multiplex Real-Time RT-PCR Assay for Rapid Detection and Surveillance. Chung HY, Jian MJ, Chang CK, Lin JC, Yeh KM, Chen CW, Hsieh SS, Hung KS, Tang SH, Perng CL, Chang FY, Wang CH, Shang HS. Microbiol Spectr. 2022 Feb 23;10(l):e0251321. doi: 10.1128/spectrum.02513-21. Epub 2022 Feb 23. PMID: 35196812; PMCID: PMC8865422; Rapid Automated Screening for SARS-CoV-2 B.1.617 Lineage Variants (Delta/Kappa) through a Versatile Toolset of qPCR-Based SNP Detection. Nörz D, Grunwald M, Tang HT, Olearo F, Günther T, Robitaille A, Fischer N, Grundhoff A, Aepfelbacher M, Pfefferle S, Lütgehetmann M. Diagnostics (Basel). 2021 Oct 1; 11(10): 1818. doi: 10.3390/diagnostics11101818. PMID: 34679517; PMCID: РМС8534837]. Однако, использование данных наборов не предполагает быстрой возможности замены мишеней для детекции новых геновариантов, содержащих миссенс мутации или замены.
Детекция дополнительных специфичных для возникающих геновариантов мишеней, так же, как и исключение из анализа мишеней для циркулирующих геновариантов позволяет повысить эффективность лабораторного исследования в целом. В связи с этим существует потребность в разработке олигонуклеотидов для выявления мутации S:P681R в биологических образцах с подтвержденным наличием РНК SARS-CoV-2: праймеров и флуоресцентно-меченого зонда для проведения ПЦР-РВ.
Технический результат заявляемого изобретения направлен на выявление мутации S:P681R в биологических образцах с подтвержденным наличием РНК SARS-CoV-2 с высокой степенью специфичности посредством таких олигонуклеотидов - праймеров и флуоресцентно-меченого зонда, которые позволяют эффективно определять мутацию S:P681R SARS-CoV-2 с использованием широко доступных методик и доступных материалов.
Технический результат достигается за счет применения химически-синтезированных олигонуклеотидов для определения наличия в биологическом образце с подтвержденным наличием РНК SARS-CoV-2 мутации S:P681R, имеющих следующий олигонуклеотидный состав:
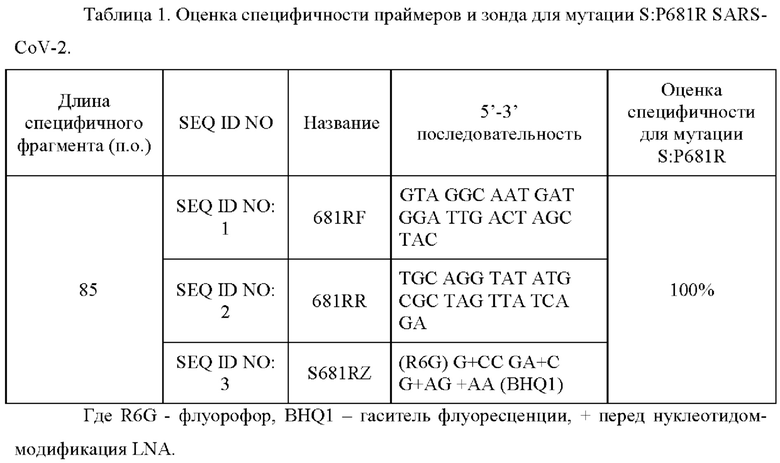
прямой праймер 681RF - SEQ ID NO: 1,
обратный праймер 681RR - SEQ ID NO: 2,
флуоресцентный зонд S681RZ - SEQ ID NO: 3.
Праймеры представляют собой последовательности олигонуклеотидов для амплификации фрагмента, содержащего мутацию S:P681R в биологических образцах с подтвержденным наличием РНК SARS-CoV-2, флуоресцентно-меченый конформационно-блокированный зонд является олигонуклеотидом, содержащим флуорофор и гаситель флуоресценции, позволяющим детектировать амплифицированный фрагмент.
Заявляемые олигонуклеотиды разработаны на основе известных последовательностей гена, кодирующего S-белок SARS-CoV-2, взятых из базы данных NCBI (https://www.ncbi.nlm.nih.gov/), в результате выбран фрагмент для детекции мутации S:P681R. К выбранному фрагменту подобраны праймеры и зонд для амплификации 85 пар оснований: прямой праймер 681RF - SEQ ID NO: 1; обратный праймер 681RR - SEQ ID NO: 2 и флуоресцентный зонд S681RZ - SEQ ID NO: 3.
Заявляемое изобретение является результатом работы в рамках совершенствования молекулярно-генетического мониторинга вариантов вируса SARS-CoV-2, проведенной в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия).
Для подбора целевых последовательностей - мест посадки олигонуклеотидов, используют фрагменты референсных геномов из базы данных NCBI (https://www.ncbi.nlm.nih.gov/), как вируса SARS-CoV-2 дикого типа, так и тех геновариантов, в которых встречается мутация S:P681R (Delta (В.1.617.2), Kappa (B.1.617.1), (B.1.617.3). Для поиска консервативных последовательностей применяют современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая AlignX, SnapGene Viewer, MEGA, Unipro UGENE. Составляют перечень мутаций, характерных для геновариантов. Затем к значимым мутациям подбирают олигонуклеотидные последовательности прямого и обратного праймеров, а также флуоресцентно-меченого зонда. Для детекции образцов, содержащих мутацию S:P681R, используют канал для детекции флуорофора R6G. Упомянутые олигонуклеотидные последовательности приведены в Таблице 1.
Анализ заявляемых последовательностей с использованием ресурса Primer BLAST продемонстрировал, что прямой праймер 681RF (SEQ ID NO: 1) и обратный праймер 681RR (SEQ ID NO: 2) амплифицируют участок, в котором располагается мутация S:P681R со 100% специфичностью.
В качестве биологического материала используются мазки/отделяемое носоглотки и ротоглотки, с подтвержденным наличием РНК SARS-CoV-2 (например, после проведения анализа на наборе реагентов АмплиСенс® COVID-19-FL).
Выделение РНК из биологического материала проводят в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Выделение РНК из биологического материала производят с помощью комплекта реагентов в соответствии с инструкцией производителя. Для выделения РНК может быть использован комплект реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный набор для выделения РНК. Оптимальная концентрация РНК - 103-105 копий в 10 мкл. Реакцию обратной транскрипции (ОТ) проводят с помощью комплекта реагентов в соответствии с инструкцией производителя. Для проведения реакции ОТ может быть использован комплект реагентов «РЕВЕРТА-L» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любой аналогичный набор. Готовый препарат кДНК может храниться при температуре от 2 до 8°С в течение недели, при температуре от минус 24 до минус 16°С в течение 6 мес.и при температуре не выше минус 68°С в течение года.
Полимеразная цепная реакция (ПЦР) - это эффективный способ получения in vitro большого числа копий специфических нуклеотидных последовательностей. Их амплификация осуществляется в ходе трехэтапного циклического процесса.
Процесс ПЦР-амплификации заключается в многократном повторении процессов денатурации, ренатурации и синтеза. Денатурация (95°С) - термическое воздействие на молекулу ДНК с целью получения одноцепочечной структуры. Ренатурация (55-60°С) - праймеры, добавленные в реакцию, спариваются с разделенными цепями. Синтез (70-75°С) - синтез второй цепи ДНК. Каждый цикл длится 3-5 мин.
Анализ данных проводится на основе детекции амплификатором уровня флуоресцентного сигнала испускаемого интеркалирующим красителем. При увеличении числа копий анализируемого участка детектируется экспоненциальный рост флуоресцентного сигнала. В результате наблюдается S-образная кривая в случае наличия специфичного флуоресцентному зонду сигнала, или ее отсутствие, в случае неспецифичной для зонда последовательности. Анализ кривых позволяет судить об отсутствии или наличии мутации в исследуемых образцах.
ПЦР-РВ проводится с применением заявляемых представленных в Таблице 1 олигонуклеотидов - праймеров и зонда, для детекции мутации S:P681R SARS-CoV-2.
ПЦР-РВ проводят при следующих условиях:
Общий объем реакционной смеси - 25 мкл.
Компоненты ПЦР смешиваются следующим образом:
(a) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры SEQ ID NO: l, SEQ ID NO: 2 - по 0,7 мМ; - флуоресцентный зонд SEQ ID NO: 3 - 0,3 мМ; -dNTPs - 0,44 мМ.
(b) реактив, содержащий рекомбинантный фермент Taq ДНК-полимеразу, например, 0,5 мкл «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любой аналогичный коммерческий набор в соответствии с инструкцией производителя.
(c) ПЦР-буфер, содержащий MgCl2, например, 5,0 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) или любого аналогичного коммерческого набора в соответствии с инструкцией производителя.
(d) полученная методом обратной транскрипции кДНК - 10 мкл. Амплификацию проводят на приборе с возможностью флуоресцентной детекции,
например, «Rotor-Gene Q» («Qiagen», Германия) или на любом другом приборе с 2-5 каналами детекции в соответствии с инструкцией производителя.
Амплификацию проводят по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресценции проводится на этапе 60°С по каналу для флуорофора R6G.
Для анализа результатов задают пороговую линию, соответствующую величине 10% от среднего сигнала флуоресценции образца с наличием мутации S:Ins214EPE. Образцы, для которых кривые флуоресценции пересекают пороговую линию, и, при этом кинетика накопления флуоресцентного сигнала является экспоненциальной, являются положительными, то есть содержат мутацию S:P681R SARS-CoV-2.
Реализация заявляемого изобретения поясняется следующими примерами: Пример 1. Получение олигонуклеотидов для определения мутации S:P681R SARS-CoV-2.
Для подбора целевых последовательностей - мест посадки олигонуклеотидов, используют фрагменты референсных геномов из базы данных NCBI (https://www.ncbi.nlm.nih.gov/), как SARS-CoV-2 дикого типа, так и тех геновариантов, в которых встречается мутация S:P681R (Delta (B.1.617.2), refseq MZ359841 (NCBI); Kappa (B.1.617.1), EPI_ISL_1544071 (GISAID (https://gisaid.org/)). Для поиска мутаций использованы современные алгоритмы in silico анализа нуклеотидных последовательностей и программы, находящиеся в открытом доступе, включая AlignX, Unipro UGENE, олигокалькулятор (Oligo Calculators) Integrated DNA Technologies, Inc. (Oligo Analyzer (idtdna.com)). Был составлен перечень консервативных участков, характерных только мутации S:P681R SARS-CoV-2, к которым были подобраны олигонуклеотидные последовательности прямого 681RF и обратного 681RR праймеров, а также флуоресцентный зонд S681RZ.
Анализ упомянутых последовательностей с использованием ресурса Primer BLAST показал, что прямой 681RF и обратный 681RR праймеры амлифицируют участок с мутацией S:P681R со 100% специфичностью.
Олигонуклеотиды для определения мутации S:P681R SARS-CoV-2- прямой праймер 681RF, обратный праймер 681RR, флуоресцентный зонд S681RZ, представлены уникальными последовательностями SEQ ID NO: 1, SEQ ID NO: 2 и SEQ ID NO: 3 соответственно.
Пример 2. Детекция мутации S:P681R SARS-CoV-2 методом ПЦР-РВ.
Определение мутации S:P681R SARS-CoV-2 проводят методом ПЦР-РВ при следующих условиях:
Общий объем реакционной смеси - 25 мкл. Компоненты ПЦР смешиваются следующим образом:
(а) 10 мкл смеси, содержащей:
- олигонуклеотидные праймеры: SEQ ID NO: 1, SEQ ID NO: 2 - по 0,7 мМ;
- флуоресцентный зонд: SEQ ID NO: 3 - 0,3 мМ;
- dNTPs - 0,44 мМ.
(b) 0,5 мкл реактива «Полимераза TaqF» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего рекомбинантный фермент Taq ДНК-полимеразу.
(c) 5,0 мкл ПЦР-буфера «ОТ-ПЦР-смесь-2 FEP/FRT» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), содержащего MgCl2.
(d) полученная после реакции обратной транскрипции («РЕВЕРТА-L» ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) кДНК 10 мкл.
ПЦР-РВ проводили с флуоресцентной детекцией на приборе с 5 каналами детекции - «Rotor-Gene Q» («Qiagen», Германия).
Подготовленный описанным способом материал, содержащий уникальные олигонуклеотидные последовательности SEQ ID NO NO: 1-3, используют для определения мутации S:P681R SARS-CoV-2 в образцах биологического материала.
Пример 3. Обнаружение мутации S:P681R SARS-CoV-2 в образцах биологического материала.
Для определения мутации S:P681R SARS-CoV-2 выбрано 50 образцов, с подтвержденным наличием РНК SARS-CoV-2.
Для исследования использовали клинический материал - мазки/отделяемое носоглотки и ротоглотки, с подтвержденным наличием РНК SARS-CoV-2 набором реагентов АмплиСенс® COVID-19-FL (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия). Выделение РНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Выделение РНК осуществляли методом нуклеопреципитации с применением набора реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) в соответствии с инструкцией к набору. Обратную транскрипцию проводили с применением набора реагентов «РЕВЕРТА-L» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) в соответствии с инструкцией к набору.
ПЦР-РВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO NO: 1-3.
Амплификацию проводили на приборе «Real-time CFX96 Touch» («Bio-Rad», США) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресценции проводилась на этапе 60°С по каналу для флуорофора R6G. Для анализа результатов задали пороговую линию, соответствующую величине 10% от среднего сигнала образца с мутацией S:P681R.
Для 47 образцов кривая флуоресценции пересекла пороговую линию и, при этом кинетика накопления флуоресцентного сигнала была экспоненциальной, что свидетельствует о том, что данные образцы содержат мутацию S:P681R SARS-CoV-2.
Для данных 47 образцов наличие мутации S:P681R SARS-CoV-2 подтверждено фрагментным секвенированием с использованием метода секвенирования по Сэнгеру. Фрагментное секвенирование выполнялось на генетическом анализаторе ABI 3500xL (Applied Biosystems, США), выравнивание и анализ полученных последовательностей выполнялось с помощью программы AlignX («Thermo Fisher Scientific)), США). Отсутствие мутации в 3 образцах без флуоресцентного сигнала также подтверждено фрагментным секвенированием.
Таким образом, из всей выборки выявлено 47 образцов, содержащих мутацию S:P681R SARS-CoV-2.
Пример 4. Обнаружение мутации S:P681R SARS-CoV-2 в образцах биологического материала.
Для определения мутации S:P681R SARS-CoV-2 выбрано 12 образцов, с подтвержденным наличием РНК SARS-CoV-2.
Выделение РНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Выделение РНК осуществляли методом нуклеопреципитации с применением набора реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) в соответствии с инструкцией к набору. Обратную транскрипцию проводили с применением набора реагентов «РЕВЕРТА-L» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) в соответствии с инструкцией к набору.
ПЦР-РВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO NO: 1-3.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресценции проводилась на этапе 60°С по каналу для флуорофора R6G. Для анализа результатов задали пороговую линию, соответствующую величине 10% от среднего сигнала образца с мутацией S:P681R. Для 7 образцов кривая флуоресценции пересекла пороговую линию и, при этом кинетика накопления флуоресцентного сигнала экспоненциальная, что свидетельствует о том, что данные образцы содержат мутацию S:P681R SARS-CoV-2.
Для данных 7 образцов наличие мутации S:P681R SARS-CoV-2 подтверждено фрагментным секвенированием с использованием метода секвенирования по Сэнгеру. Фрагментное секвенирование выполнялось на генетическом анализаторе ABI 3500xL (Applied Biosystems, США), выравнивание и анализ полученных последовательностей выполнялось с помощью программы AlignX («Thermo Fisher Scientific)), США). Отсутствие мутации в 5 образцах без флуоресцентного сигнала также подтверждено фрагментным секвенированием.
Таким образом, из всей выборки выявлено 7 образцов, содержащих мутацию S:P681R SARS-CoV-2.
Пример 5. Обнаружение мутации S:P681R SARS-CoV-2 в образцах биологического материала.
Для определения мутации S:P681R SARS-CoV-2 выбрано 8 образцов, с подтвержденным наличием РНК SARS-CoV-2.
В работе исследовали пробы клинического материала, мазки/отделяемое носоглотки и ротоглотки, с подтвержденным наличием РНК SARS-CoV-2 набором реагентов АмплиСенс® COVID-19-FL (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия). Выделение РНК из биологического материала проводили в соответствии с МУ 1.3.2569-09 «Организация работ лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности)). Выделение РНК осуществляли методом нуклеопреципитации с применением набора реагентов «РИБО-преп» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) в соответствии с инструкцией к набору. Обратную транскрипцию проводили с применением набора реагентов «РЕВЕРТА-L» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Москва, Россия) в соответствии с инструкцией к набору.
ПЦР-РВ проводили в условиях, описанных в Примере 2, с использованием уникальных олигонуклеотидных последовательностей SEQ ID NO NO: 1-3.
Амплификацию проводили на приборе «Rotor-Gene Q» («Qiagen», Германия) по следующей программе: 1 цикл 95°С в течение 15 минут, 45 циклов при температуре 95°С - 10 секунд / 60°С - 20 секунд. Детекция флуоресценции проводилась на этапе 60°С по каналу для флуорофора R6G. Для анализа результатов задали пороговую линию, соответствующую величине 10% от среднего сигнала флуоресценции образца с наличием мутации S:P681R. Кривая флуоресценции не пересекла пороговую линию, что свидетельствовало о том, что в выборке отсутствуют образцы, содержащие мутацию S:P681R SARS-CoV-2.
Наличие других мутаций SARS-CoV-2 в данных 5 образцах, так же как и отсутствие мутации S:P681R, подтверждено фрагментным секвенированием с использованием метода секвенирования по Сэнгеру. Фрагментное секвенирование выполнялось на генетическом анализаторе ABI 3500xL (Applied Biosystems, США), выравнивание и анализ полученных последовательностей выполнялось с помощью программы AlignX («Thermo Fisher Scientific», США).
Заявляемое изобретение позволяет выявлять мутацию S:P681R в биологических образцах с подтвержденным наличием РНК SARS-CoV-2. Синтезированные олигонуклеотиды SEQ ID NO NO: 1-3 не дают перекрестных реакций с другими протестированными образцами, амлифицируют заданный участок со 100% специфичностью и позволяют определять наличие или отсутствие в образцах биологического материала значимой мишени S:P681R.
| название | год | авторы | номер документа |
|---|---|---|---|
| Олигонуклеотиды для определения мутации S:Ins214EPE SARS-CoV-2 | 2022 |
|
RU2795017C1 |
| Олигонуклеотиды для определения мутации S:delVYY143-145 SARS-CoV-2 | 2022 |
|
RU2795016C1 |
| Олигонуклеотиды для определения мутации S:N501Y SARS-CoV-2 | 2022 |
|
RU2791958C1 |
| Олигонуклеотиды для определения мутации S:L452R SARS-CoV-2 | 2022 |
|
RU2795018C1 |
| Олигонуклеотиды для определения мутации S:delHV69-70 SARS-CoV-2 | 2022 |
|
RU2795014C1 |
| Способ выявления и количественного определения вируса SARS-COV-2 с помощью цифровой капельной ПЦР | 2024 |
|
RU2839700C1 |
| Способ генотипирования гена TLR8 по полиморфизму rs3764880 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805864C1 |
| Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2 и олигонуклеотидные праймеры для его реализации | 2021 |
|
RU2762759C1 |
| Способ обнаружения РНК вируса SARS-CoV-2 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе | 2021 |
|
RU2764021C1 |
| Способ генотипирования гена TLR4 по полиморфизму rs4986790 и набор олигонуклеотидных праймеров и зондов для его реализации | 2023 |
|
RU2805862C1 |
Изобретение относится к области биотехнологии, в частности к определению мутации S:P681R SARS-CoV-2 методом полимеразной цепной реакции, проводимой в режиме реального времени. Представлен набор праймеров и зонда для определения мутации S:P681R SARS-CoV-2, включающий прямой праймер 681RF, обратный праймер 681RR, флуоресцентный зонд S681RZ, которые представлены уникальными последовательностями SEQ ID NO: 1-3 соответственно. При этом для создания прямого и обратного праймеров, флуоресцентного зонда используют фрагменты референсных геномов SARS-CoV-2 дикого типа, Delta (В.1.617.2), Карра (В.1.617.1), (В.1.617.3). Синтезированные олигонуклеотиды SEQ ID NO NO: 1-3 не дают перекрестных реакций с другими протестированными образцами, амплифицируют заданный участок со 100% специфичностью и позволяют определять наличие или отсутствие в образцах биологического материала значимой мишени S:P681R. Изобретение может применяться для идентификации геновариантов SARS-CoV-2 при эпидемиологических исследованиях. 2 з.п. ф-лы, 1 табл.
1. Набор праймеров и зонда для определения мутации S:P681R SARS-CoV-2, имеющий следующий состав:
прямой праймер 681RF - SEQ ID NO: 1;
обратный праймер 681RR - SEQ ID NO: 2;
флуоресцентный зонд S681RZ - SEQ ID NO: 3.
2. Набор праймеров и зонда для определения мутации S:P681R SARS-CoV-2 по п. 1, где для создания прямого и обратного праймеров, флуоресцентного зонда используют фрагменты референсных геномов SARS-CoV-2 дикого типа, Delta (B.1.617.2), Kappa (B.1.617.1), (B.1.617.3).
3. Набор праймеров и зонда для определения мутации S:P681R SARS-CoV-2 по п. 1, где флуоресцентный зонд является олигонуклеотидом, содержащим флуорофор R6G и гаситель флуоресценции BHQ1, позволяющий детектировать амплифицированный фрагмент.
| НАБОР СИНТЕТИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ РНК КОРОНАВИРУСА | 2021 |
|
RU2761025C1 |
| WO 2021191795 A1, 30.09.2021 | |||
| WO 2021176099 A1, 10.09.2021. | |||
Авторы
Даты
2023-04-27—Публикация
2022-10-06—Подача