Изобретение относится к медицинской микробиологии, молекулярной биологии, в частности применимо для выявления в образцах геновариантов SARS-CoV-2 с помощью цифровой капельной ПЦР (цкПЦР), его абсолютной количественной оценки в биологических жидкостях и для наблюдения динамики вирусной нагрузки у пациента в процессе заболевания COVID-19.
SARS-CoV-2 - это оболочечный одноцепочный РНК-вирус, относящийся к подроду Sarbecovirus рода Betacoronavirus. Вызывает опасное инфекционное заболевание COVID-19. Передается воздушно-капельным, воздушно-пылевым и контактным путем. Инкубационный период может достигать 14 дней, часто протекает бессимптомно, что не препятствует распространению инфекции, но затрудняет своевременную диагностику.
Современный уровень молекулярно-генетических методов исследования позволил разработать быстрые и эффективные способы выявления и дифференциации SARS-CoV-2, обладающие высокой эффективностью, чувствительностью и надежностью. Чаще всего для этих целей используют ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени. Предложенные подходы обладают чувствительностью 100-1000 ГЭ (копий)/мл и, в большинстве случаев, направлены на качественную детекцию РНК патогена. В тоже время количественное определение патогена особенно актуально.
В настоящее время для прямого подсчета числа мишеней нуклеиновых кислот применяется цкПЦР - усовершенствованный метод ПЦР с гибридизационно-флуоресцентным (либо флуоресцентным) учетом результатов. Цифровая капельная ПЦР позволяет определять абсолютное количество патогена с высокой точностью и способна выявлять единичные копии искомого гена в образце при низких концентрациях недоступных для ПЦР с электрофоретическим и гибридизационно-флуоресцентным учетом результатов.
Возможности заявляемого способа могут быть полезны в случаях быстротечных вирусных инфекций и сочетанных инфекционных заболеваний, когда требуется быстрая оценка эффективности терапии и соответствия клинических проявлений заболевания изменению вирусной нагрузки, что позволяет оценить правильность выбора терапевтической схемы.
Зарегистрированная в России тест-система ФБУН ЦНИИ Эпидемиологии Роспотребнадзора «Набор реагентов для выявления и количественного определения РНК SARS-CoV-2 методом ОТ-ПЦР АмплиСенс® COVID-19-FL» (РЗН 2021/14026) [1] имеет нижний предел обнаружения РНК вируса SARS-CoV-2 до 5×102 ГЭ (копий)/мл. Точность определения концентрации копий вируса с помощью предложенного набора напрямую связана с точностью определения концентрации положительного контрольного образца (ПКО) для чего требуется конструирование генно-инженерных ДНК-калибраторов: проб с заведомо известной концентрацией ГЭ (копий)/мл. Количественное определение РНК в данной тест-системе основано на относительном определении количества РНК, в отличие от цкПЦР, которая позволяет проводить определение абсолютного количества целевых НК.
Патенты RU2764021C1, опубл. 01.12.2022, RU2764022C1, опубл. 01.12.2022 и RU2764023C1, опубл. 01.12.2022 [2, 3, 4] предлагают способы выявления РНК вируса SARS-CoV-2 в сверхнизких концентрациях. Повышение эффективности обнаружения РНК вируса SARS-CoV-2 в этих способах происходит за счет проведения предварительной специфической амплификации и учета результатов посредством рибонуклеопротеинового комплекса системы CRISPR/Cas, что обеспечивает возможность выявления РНК SARS-CoV-2 до единичных (1,64) копий РНК в одной реакции. Изобретения обеспечивают повышение эффективности выявления РНК до 10 раз в сравнении с классическими методами ПЦР. Но данные способы не предназначены для количественного выявления РНК вируса в образце.
Одним из подходов, совмещающих возможность повышения чувствительности анализа и количественной оценки содержания патогена может быть цкПЦР, основанная на генерации микрокапель из реакционной смеси (содержащей в своем составе праймеры, флуоресцентную метку (зонд), либо интеркалирующий флуорофор, исследуемую кДНК и необходимые для проведения цкПЦР вещества и ферменты) с последующим учетом флуоресцентного сигнала в каждой микрокапле после завершения программы амплификации.
Компанией Bio-Rad Laboratories, Inc., США (далее Bio-Rad) предложен подход для выполнения таких исследований с помощью цкПЦР. Комплекс «QX200 Droplet Digital PCR System» компании Bio-Rad может применяться для выявления вируса SARS-CoV-2. Применение данного комплекса описано в работе Tao Suo at al. (2020) [5], разработаны праймеры и зонды, нацеленные на области генома ORFlab и N SARS-CoV-2. Предложенный подход обладает высокой чувствительностью (вплоть до единичных копий искомого гена в образце) и высокой специфичностью, но не выявляет все известные геноварианты SARS-CoV-2.
Тест-система «2019-nCoV CDC ddPCR Triplex Probe Assay» (Bio-Rad) [6] предназначена для качественного обнаружения РНК вируса SARS-CoV-2 в мазках из носоглотки и ротоглотки методом цкПЦР с помощью комплекса приборов «QX200 Droplet Digital PCR System». В данной тест-системе компанией Bio-Rad реализован одноэтапный процесс получения кДНК и последующей амплификации с помощью разработанных реагентов «One-Step RT-ddPCR Advanced Kit for Probes». Праймеры данной тест-системы подобраны на участки гена N, кодирующие нуклеокапсид вируса. Производитель позиционирует данную тест-систему как инструмент для качественного выявления единичных копий фрагмента гена N SARS-CoV-2 в исследуемом образце.
Тест-система «COVID-19 RT-Digital PCR Detection Kit» компании «Gnomegen LLC, США» [7] для качественного обнаружения РНК вируса SARS-CoV-2 в мазках из носоглотки и ротоглотки методом цифровой капельной ПЦР является аналогом тест-системы «2019-nCoV CDC ddPCR Triplex Probe Assay» компании Bio-Rad. Тест-система основана на проведении реакции амплификации в чипе, содержащем массив из 20000 реакционных микролунок. Гидрофобное покрытие на поверхности чипа позволяет осуществлять независимые реакции амплификации в каждой микролунке с последующим учетом флуоресцентного сигнала в каждой из них. Праймеры данной тест-системы также подобраны на участки гена N, кодирующие нуклеокапсид COVID-19. Обратная транскрипция и ПЦР-амплификация проходят в один этап.
Описанные тест-системы используют в качестве мишени ген N, в котором с высокой частотой происходят мутации. Последовательность праймеров для этих тест-систем не указана. Поэтому нельзя предугадать будут ли данные праймеры выявлять новые гено-варианты вируса.
На данный момент в научной и специальной литературе описано применение цкПЦР, в основном, для выявления SARS-CoV-2 в образцах с низкой концентрацией вируса (например, в сточных водах). Ввиду высокой частоты мутаций в геноме вируса SARS-CoV-2 для достоверности получаемых результатов обычно используют наборы праймеров для выявления конкретного геноварианта вируса. В работе Steven С.Holland at al. (2023) [8] описано применение цкПЦР для выявления различных геновариантов вируса SARS-CoV-2 с помощью набора праймеров на S ген, где каждая пара подобрана на конкретную мутацию в геноме. В работе Sang-Soo Lee at al. (2024) [9] предписывается для достоверного измерения концентрации РНК вируса SARS-CoV-2 в исследуемом образце с помощью цкПЦР использовать несколько наборов праймеров на различные участки генома: Е, S, N. В работе Margaret G. Mills at al. (2022) [10] для точной идентификации различных геновариантов SARS-CoV-2 предложено использовать набор из несколько пар праймеров на ген S.
Технический результат изобретения заключается в обеспечении быстрого и надежного обнаружения стабильного фрагмента гена S SARS-CoV-2 в исследуемом образце с одновременным абсолютным подсчетом количества обнаруженных копий гена в динамике развития инфекционного процесса с помощью цкПЦР.
Технический результат достигается способом выявления и количественного определения геновариантов SARS-CoV-2 в динамике развития инфекционного процесса с помощью цкПЦР, которая предусматривает выделение из исследуемого биологического материала тотальной РНК, получение на ее основе комплементарной ДНК (кДНК) и использование разработанных нами праймеров и зонда на фрагмент гена S SARS-CoV-2 (SEQ ID NO:1-3). Из реакционной смеси, содержащей в своем составе кДНК, праймеры, зонд и необходимые для проведения цкПЦР реагенты, прибор для цкПЦР генерирует до 20000 микрокапель (обычно 10000-15000), разделяя, таким образом, реакционную смесь на тысячи частей, и проводит учет результатов ПЦР в каждой из них. Наличие флуоресценции в микрокапле говорит о том, что в ней находилась как минимум одна копия искомого гена. Подсчет количества микрокапель с флуоресценцией и соотнесение их с общим числом микрокапель, дает возможность рассчитать, сколько было копий искомого гена в реакционной смеси.
Отличительной особенностью заявляемого способа является использование сконструированных праймеров и зонда в формате TaqMan меченного интеркалирующим флуорофором карбоксифлуоресцеином (FAM) и гасителем флуоресценции real-time quenchers - BHQ1 (Синтол, Россия), имеющие последовательности:
SEQ ID NO: 1 PR 55 F - 5'-TTGCCACCTTTGCTCACAGA-3';
SEQ ID NO: 2 PR 56 R - 5'-AGGTCCAACCAGAAGTGA-3'
SEQ ID NO: 3 Probe - 5'-FAM-ACACTTCTGCACTGTTAGCGGGTACA-BHQ1-3'
Данные праймеры комплементарны гену S, кодирующему S-белок (спайк-белок), отвечающему за вирулентность SARS-CoV-2. Ген S в высокой степени подвержен мутациям, причем какие-то из них приводят к повышению вирулентных свойств вируса. Многие предложенные ранее праймеры на этот ген не выявляют новые геноварианты вируса, что приводит к ложноотрицательным ответам ПЦР, или содержат наборы праймеров, идентифицирующих мутации, характерные для отдельных геновариантов, что делает практически невозможным их использование для наблюдения динамики инфекционного процесса.
Поэтому конструирование праймеров для цкПЦР определялось такими параметрами, как специфичность по отношению к геномным последовательностям разных геновариантов вируса SARS-CoV-2, высокая эффективность ПЦР, температура плавления 55-60°С, соотношение G/C в пределах от 40 до 65%.
Заявляемый способ осуществляют следующим образом.
Выделение тотальной РНК из исследуемого материала (назофарингеальных мазков) осуществляют с помощью коммерческого набора «РИБО-преп» (ФБУН ЦНИИ эпидемиологии Роспотребнадзора). Получение кДНК осуществляют с помощью коммерческого набора «РЕВЕРТА-L» (ФБУН ЦНИИ эпидемиологии Роспотребнадзора). Набор позволяет получить раствор кДНК объемом 20 мкл (из которого 10 мкл приходится на исходный раствор РНК, полученный в результате выделения нуклеиновых кислот и 10 мкл на реагенты, необходимые для проведения обратной транскрипции). К полученной в реакции обратной транскрипции кДНК объемом 20 мкл добавляют 20 мкл ДНК-буфера для достижения суммарного объема 40 мкл, в соответствии с инструкцией производителя коммерческого набора «РЕВЕРТА-L», что соответствует 4-х кратному разведению исходного образца. Для получения оптимальной концентрации кДНК, обеспечивающей возможность подсчета количества копий искомого гена в исследуемом образце, полученный раствор подвергают серии десятикратных разведений ТЕ-буфером (ЗАО «Евроген», РФ) в 10-100000 раз относительно концентрации исходного образца. Зная, во сколько раз был разведен исследуемый образец, выполняют перерасчет количества копий искомого гена на 1 мл исследуемого образца.
Определение чувствительности цифровой капельной ПЦР иллюстрировано исследованием различных разведений кДНК, полученных из назофарингеального мазка пациента N, с заболеванием COVID-19. Результаты представлены в таблице 1.
Данные таблицы показывают, что при очень высокой концентрации кДНК (разведение №1-2) прибор не может точно отражать количество ГЭ в образце. В этих случаях детектируется флуоресценция во всех сгенерированных прибором микрокаплях. Снижение концентрации ГЭ (разведения №3-5) позволяет проводить измерения. Наиболее точное определение концентрации происходит, когда количество копий гена в исследуемом образце находится в диапазоне от 104 до 106 на мл (разведения №3-4). Если количество копий гена в исследуемом образце больше, чем общее количество сгенерированных прибором микрокапель (обычно 10000-15000 на одну реакцию), точный подсчет становится невозможным, так как наблюдается амплификация в каждой сгенерированной прибором микрокапле. При последующем разведении пробы, когда количество копий гена в образце измеряется единицами (столбец 3, разведение №5 таблицы) точное определение концентрации также становится невозможным. Это связано со случайным неравномерным распределением малого количества кДНК в исследуемых пробах, что приводит к большой вариабельности измеренных значений.
Затем проводят цкПЦР с помощью комплекса приборов «QX200 Droplet Digital PCR System» (Bio-Rad) или аналогичных приборов других фирм. Реакционная смесь объемом 20 мкл содержит: 10 мкл «ddPCR Supermix for Probes» и 10 мкл смеси праймеров, зонда и раствора кДНК. Рекомендованный объем праймеров и зонда (8 пМоль каждого праймера и 4 пМоль зонда на одну реакцию) составляет 0,5-2 мкл, рекомендованный объем исследуемого образца кДНК составляет 8-9,5 мкл. Уменьшение исследуемого объема кДНК менее 8 мкл не рекомендуется, так как приводит к снижению чувствительности. Для вычисления количества копий искомого гена на 10 мкл исследуемого образца, производится перерасчет, исходя из объема, внесенного в реакционную смесь раствора кДНК.
Полученные реакционные смеси в стрипах переносят в картридж для генерации микрокапель (в соответствующие лунки для образцов). Добавляют по 70 мкл «Droplet Generation Oil for Probes» (в соответствующие лунки для масла). Картридж загружают в прибор для генерации микрокапель «QX200 Droplet Generator)), где генерируется, обычно, 10000-15000 микрокапель, в каждой из которых будет проводиться ПЦР. Полученные взвеси микрокапель переносят в 96-луночный планшет и, после запаивания лунок с помощью прибора «РХ1 PCR Plate Sealer», загружают в амплификатор «С1000 Touch Thermal Cycler with 96-Deep Well Reaction Module».
Для рассчитанных праймеров была оптимизирована программа амплификации путем снижения температурного режима отжига праймеров с 60°С (согласно инструкции производителя) до 57°С и увеличения количества циклов с 40 (согласно инструкции производителя) до 45. Модифицированная программа амплификации включает: 95°С - 10 минут; 45 циклов: 94°С - 30 секунд, 57°С - 1 минута; 98°С - 10 минут. После окончания программы амплификации, 96-луночный планшет переносят в прибор «QX200 Droplet Digital PCR System» для учета результатов. Результаты учитывают с помощью программы управления прибором QuantaSoft Version 1.7.4.0917 (Bio-Rad, Temse, Бельгия). Программа, по заявлению производителя, непосредственно подсчитывает абсолютное количество выявленных копий гена в исследуемой реакционной смеси путем детекции флуоресцентного сигнала в каждой микрокапле. Вычисление ГЭ (копий)/мл проводят методом определения среднего арифметического значения для проб с разведениями кДНК, количество копий гена в которых в 2-100 раз меньше, чем количество сгенерированных прибором микрокапель для данного образца.
Заявленный способ успешно апробирован на 45 образцах назофарингеальных мазков от людей с подозрением на заболевание COVID-19 и назофарингеальных мазков от больного COVID-19, взятых в разгаре заболевания и на его спаде.
Сущность изобретения подтверждается следующими примерами.
Пример 1. Выявление SARS-CoV-2 в назофарингеальных мазках у пациентов с подозрением на заболевание COVID-19 не подтвержденным ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени.
Работу проводят в лаборатории, выполняющей молекулярно-биологические исследования биологического материала на наличие возбудителей инфекционных болезней, с соблюдением санитарно-эпидемиологических правил работы с микроорганизмами I-II групп патогенности.
Выделение тотальной РНК из исследуемого материала (назофарингеальных мазков) осуществляют с помощью коммерческого набора «РИБО-преп» (ФБУН ЦНИИ эпидемиологии Роспотребнадзора). Получение кДНК осуществляют с помощью коммерческого набора «РЕВЕРТА-L» (ФБУН ЦНИИ эпидемиологии Роспотребнадзора).
Полученную в результате обратной транскрипции кДНК дополнительно не разводят ввиду низкой исходной концентрации. К полученной в реакции обратной транскрипции кДНК объемом 20 мкл добавляют 20 мкл ДНК-буфера для получения суммарного объема 40 мкл, в соответствии с инструкцией производителя коммерческого набора «РЕВЕРТА-L», что соответствует 4-х кратному разведению исходного образца.
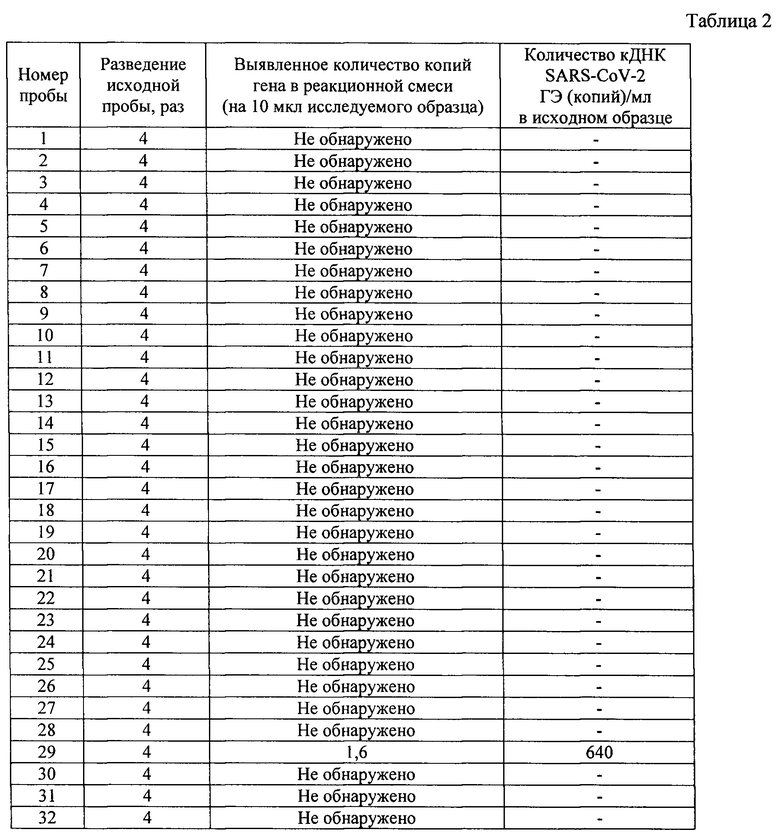
В качестве примера демонстрации возможностей цифровой капельной ПЦР было проведено исследование назофарингеальных мазков от 32 пациентов (таблица 2) с подозрением на заболевание COVID-19, не подтвержденным ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени (тест-система «Ампли-Сенс® Cov-Bat-FL» регистрационный №РЗН 2014/1987).
В одной из проб (№29) была выявлена кДНК SARS-CoV-2 в количестве 640 ГЭ (копий)/мл. В то время как в ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени «АмплиСенс® Cov-Bat-FL» в пробе №29 не выявлен вирус, т.к., согласно инструкции к набору реагентов, аналитическая чувствительность метода составляет 1×103 ГЭ/мл.
Данный пример показывает, что с помощью заявляемого способа возможно выявление SARS-CoV-2 в биологических жидкостях в концентрациях недоступных для обычных ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени.
Пример 2. Выявление различных геновариантов вида SARS-CoV-2 в назофарингеальных мазках у пациентов с заболеванием COVID-19.
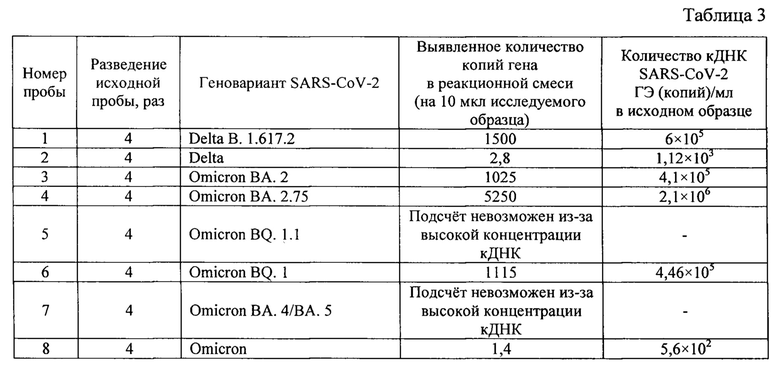
Выделение тотальной РНК, получение кДНК, постановку цкПЦР и учет результатов осуществляют как в Примере 1.
В заявляемом способе при помощи цкПЦР и разработанных праймеров и зонда возможно выявлять различные геноварианты коронавируса вида SARS-CoV-2 (таблица 3). Определение геноварианта предварительно проведено с помощью фрагментного секвенирования методом Сэнгера [11] в образцах кДНК определенных тест-системой «Ампли-Сенс® Cov-Bat-FL» (регистрационный №РЗН 2014/1987) как положительные.
Данный пример показывает, что заявленные праймеры, рассчитанные на стабильную область легко мутирующей последовательности гена S уханьского варианта вируса, успешно выявляют и другие новые геноварианты вируса.
Пример 3. Определение ГЭ в назофарингеальном мазке больного в динамике заболевания COVID-19.
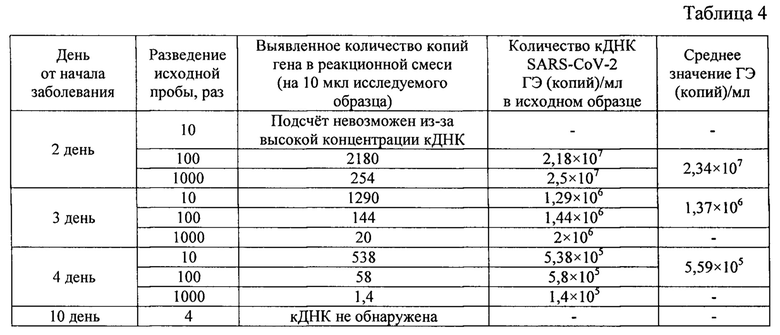
Выделение РНК, получение кДНК, постановку цкПЦР и учет результатов осуществляют как в Примере 1. Образцы полученного раствора кДНК дополнительно развели ТЕ-буфером (ЗАО «Евроген», РФ) в 10-1000 раз относительно концентрации исходного образца.
В качестве примера демонстрации возможностей цифровой капельной ПЦР для изучения динамики развития инфекционного процесса было проведено исследование назофарингеальных мазков пациента N, с заболеванием COVID-19. В различные дни от начала заболевания SARS-CoV-2 на фоне проводимого лечения отмечалось изменение вирусной нагрузки. Результаты представлены в таблице 4.
Разработанная методика позволяет наблюдать за изменением вирусной нагрузки в биологических жидкостях пациента в процессе развития заболевания и, в сочетании с изменением симптоматики, тяжести состояния пациента и физикальными исследованиями, может быть использована для оценки правильности примененных терапевтических схем.
Литература
1) ИНСТРУКЦИЯ ПО ПРИМЕНЕНИЮ набора реагентов для выявления и количественного определения РНК SARS-CoV-2 методом ОТ-ПЦР АмплиСенс® COVID-19-FL. -г.Москва, ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, 2021. - 53 с.
2) Пат. 2764021 Российская Федерация, МПК C12N 15/11 (2006.01), C12Q 1/68 (2006.01), G01N 33/00 (2006.01). Способ обнаружения РНК вируса SARS-CoV-2 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе / Тюменцев А.И., Тюменцева М.А., Преловская А.Н., Акимкин В.Г., заявитель и патентообладатель Федеральное бюджетное учреждение науки «Центральный научно-исследовательский институт эпидемиологии» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора) - №2021128585, заявл. 30.09.2021, опубл. 12.01.2022 Бюл. №2 - 30 с.
3) Пат. 2764022 Российская Федерация, МПК C12N 15/00 (2006.01), G01N 33/00 (2006.01). Способ обнаружения РНК вируса SARS-CoV-2 в ультранизких концентрациях и специфические олигонуклеотиды для использования в способе / Тюменцев А.И., Тюменцева М.А., Преловская А.Н., Акимкин В.Г., заявитель и патентообладатель Федеральное бюджетное учреждение науки «Центральный научно-исследовательский институт эпидемиологии» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора) - №2021128590, заявл. 30.09.2021, опубл. 12.01.2022 Бюл. №2 - 39 с.
4) Пат. 2764023 Российская Федерация, МПК C12N 15/11 (2006.01), C12Q 1/68 (2006.01), G01N 33/00 (2006.01). Система CRISPR-Cas12 для выявления РНК вируса SARS-CoV-2 в ультранизких концентрациях / Тюменцев А.И., Тюменцева М.А., Преловская А.Н., Акимкин В.Г., заявитель и патентообладатель Федеральное бюджетное учреждение науки «Центральный научно-исследовательский институт эпидемиологии» Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора) - №2021128583, заявл. 30.09.2021, опубл. 12.01.2022 Бюл. №2 - 30 с.
5) ddPCR: a more sensitive and accurate tool for SARS-CoV-2 detection in low viral load specimens / Tao Suo1, Xinjin Liu, Ming Guo et al. // Emerging Microbes & Infections. - 2020. -Vol. 9 - P. 1259-1268.
6) Instructions for Use Bio-Rad SARS-CoV-2 ddPCR Kit REF 12013743 - Pleasanton, Bio-Rad Laboratories, 2022. - 50 p.
7) Instructions for Use COVID-19 RT-Digital PCR Detection Kit. Catalog # CV0202 - San Diego, Gnomegen LLC, 2020. - 22 p.
8) Digital PCR Discriminates between SARS-CoV-2 Omicron Variants and Immune Escape Mutations / Steven С Holland et al. // Microbiology Spectrum. - 2023. - Vol. 11.
9) Optimization of duplex digital PCR for the measurement of SARS-CoV-2 RNA / Sang-Soo Lee at al. // Journal of Virological Methods. - 2024. - Vol. 326.
10) Rapid and accurate identification of SARS-CoV-2 Omicron variants using droplet digital PCR (RT-ddPCR) / Margaret G. Mills at al. // Journal of Clinical Virology. - 2022. - Vol. 154.
11) Sanger F., Niclein S., Coulson A.R. DNA sequencing with chain-terminating inhibitors // Proc Natl Acad Sci USA. - 1977. - T. 74. - C. 5463-5467.

Последовательности праймеров и зонда комплементарных р24144-24228 области S гена SARS-CoV-2
SEQ ID NO: 1 PR 55 F - 5'-TTGCCACCTTTGCTCACAGA-3'
SEQ ID NO: 2 PR 56 R - 5'-AAAGGTCCAACCAGAAGTGA-3'
SEQ ID NO: 3 Probe - 5'-FAM-ACACTTCTGCACTGTTAGCGGGTACA-BHQ1-3'
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ COVID-19
ddPCR.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-05-14">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2024-05-14</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>0</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2024-05-14</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное казенное учреждение
науки "Российский научно-исследовательский противочумный
институт "Микроб"" Федеральной службы по надзору в
сфере защиты прав потребителей и благополучия
человека</ApplicantName>
<ApplicantNameLatin>Russian Research Anti-Plague Institute
Microbe</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ выявления и количественного
определения геновариантов SARS-CoV-2 в динамике развития
инфекционного процесса с помощью цифровой капельной
ПЦР</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Severe acute respiratory syndrome-related
coronavirus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgccacctttgctcacaga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Severe acute respiratory syndrome-related
coronavirus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaggtccaaccagaagtga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Severe acute respiratory syndrome-related
coronavirus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acacttctgcactgttagcgggtaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Олигонуклеотиды для определения мутации S:delVYY143-145 SARS-CoV-2 | 2022 |
|
RU2795016C1 |
| Олигонуклеотиды для определения мутации S:delHV69-70 SARS-CoV-2 | 2022 |
|
RU2795014C1 |
| Олигонуклеотиды для определения мутации S:Ins214EPE SARS-CoV-2 | 2022 |
|
RU2795017C1 |
| Олигонуклеотиды для определения мутации S:N501Y SARS-CoV-2 | 2022 |
|
RU2791958C1 |
| Олигонуклеотиды для определения мутации S:L452R SARS-CoV-2 | 2022 |
|
RU2795018C1 |
| Олигонуклеотиды для определения мутации S:P681R SARS-CoV-2 | 2022 |
|
RU2795019C1 |
| Набор олигодезоксирибонуклетидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавирусов человека SARS и 2019-nCoV методом ОТ-ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2733665C1 |
| Набор олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда для идентификации РНК коронавируса человека 2019-nCoV методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2020 |
|
RU2734300C1 |
| Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2 и олигонуклеотидные праймеры для его реализации | 2021 |
|
RU2762759C1 |
| Набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для идентификации РНК коронавируса человека SARS-CoV-2 методом изотермической ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2021 |
|
RU2778855C1 |
Изобретение относится к медицинской микробиологии, молекулярной биологии. Описан способ выявления и количественного определения вируса SARS-CoV-2 с помощью цифровой капельной ПЦР, предусматривающий: выделение тотальной РНК, получение на ее основе комплементарной ДНК (кДНК), проведение цкПЦР с использованием комплементарных стабильному фрагменту области S гена SARS-CoV-2 праймеров и зонда SEQ ID NO: 1-3: SEQ ID NO: 1 PR 55 F – 5’-TTGCCACCTTTGCTCACAGA-3’, SEQ ID NO: 2 PR 56 R – 5’-AAAGGTCCAACCAGAAGTGA-3’, SEQ ID NO: 3 Probe – 5’-FAM-ACACTTCTGCACTGTTAGCGGGTACA-BHQ1-3’ с последующим учетом результатов при помощи программного обеспечения прибора для проведения цкПЦР: осуществляется подсчет абсолютного количества выявленных копий гена в исследуемой реакционной смеси при детекции флуоресцентного сигнала в каждой микрокапле. Изобретение позволяет выявлять вирус SARS-CoV-2 вне зависимости от геноварианта, а также количественное определение и динамику изменения его количества во время разных стадий инфекционного процесса и дает возможность сделать заключение на основе этих данных об эффективности терапии. 4 табл., 3 пр.
Способ выявления и количественного определения вируса SARS-CoV-2 с помощью цифровой капельной ПЦР, предусматривающий:
выделение тотальной РНК, получение на ее основе комплементарной ДНК (кДНК), проведение цкПЦР с использованием комплементарных стабильному фрагменту области S гена SARS-CoV-2 праймеров и зонда SEQ ID NO:1-3:
SEQ ID NO: 1 PR 55 F – 5’-TTGCCACCTTTGCTCACAGA-3’
SEQ ID NO: 2 PR 56 R – 5’-AAAGGTCCAACCAGAAGTGA-3’
SEQ ID NO: 3 Probe – 5’-FAM-ACACTTCTGCACTGTTAGCGGGTACA-BHQ1-3’
с последующим учетом результатов при помощи программного обеспечения прибора для проведения цкПЦР, осуществляется подсчет абсолютного количества выявленных копий гена в исследуемой реакционной смеси при детекции флуоресцентного сигнала в каждой микрокапле.
| Тест-система для обнаружения РНК вируса SARS-CoV-2 в биоматериале от животных, пищевых продуктах и объектах окружающей среды методом полимеразной цепной реакции в режиме реального времени | 2021 |
|
RU2756474C1 |
| Набор синтетических олигодезоксирибонуклеотидов и способ количественной оценки вирусной нагрузки SARS-CoV-2 в тканях различных органов методом количественной полимеразной цепной реакции в реальном времени | 2021 |
|
RU2761358C1 |
| US 20240191313 A1, 13.06.2024. | |||
Авторы
Даты
2025-05-12—Публикация
2024-05-30—Подача