Изобретение относится к биотехнологии, медицине, молекулярной биологии, генной инженерии и вирусологии, и может быть использовано для поиска важных мутаций вируса SARS-CoV-2 как при решении задач эпидемиологического надзора, так и в научно-исследовательских целях.
В конце 2019 г. в китайском городе Ухань впервые были зарегистрированы пациенты с вирусной пневмонией, вызванной неизвестным патогеном, впоследствии охарактеризованным как коронавирус SARS-CoV-2, способный вызывать инфекцию COVID-19. Исследования показали, что идентичность геномных последовательностей патогенов, собранных у ранних пациентов, составила более 99,98%. Примечательно, что вирус был похож на два других SARS-подобных коронавируса bat-SL-CoVZC45 и bat-SL-CoVZXC21, обнаруженных на территории восточного Китая в Чжоушане в 2018 году. В то же время, геном обнаруженного патогена оказался идентичен геномам SARS-CoV и MERS-CoV примерно на 79% и 50% соответственно. В силу сравнительно малого сходства с SARS-CoV, патоген был признан новым коронавирусом человека.
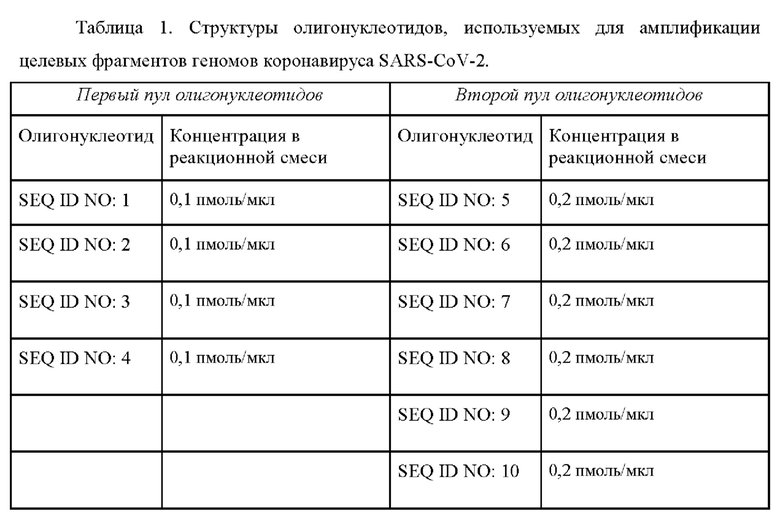
С момента начала пандемии COVID-19, вызываемой коронавирусом SARS-CoV-2, международное сообщество обеспокоено появлением мутаций, изменяющих биологические свойства патогена, например, повышающих его контагиозность или вирулентность. В частности, в конце 2020 года во всем мире было обнаружено несколько вызывающих озабоченность вариантов, включая «британский» (В. 1.1.7, ВОЗ предложила название «альфа»), «южноафриканский» (В. 1.351, «бета») и «бразильский» (Р.1, «гамма»). Эти варианты вируса были обозначены как вызывающие беспокойство в основном из-за того, что в некоторых географических регионах сообщалось об учащении случаев их передачи от человека к человеку, после чего они были обнаружены во многих странах мира. Например, «британский» вариант быстро распространился на Юго-Востоке Англии, где вызвал большое количество случаев заболевания COVID-19 и вскоре после этого был выявлен в США, став уже в апреле 2021 года доминирующим в стране. Было определено, что наличие мутации N501Y в гене, кодирующем S-гликопротеин (S-белок) вируса, повышает аффинность вирусного шипа к АСЕ2 рецептору человека, облегчая проникновение в клетку, и, таким образом, увеличивает трансмиссивность патогена. Точно так же варианты из Южной Африки и Бразилии, получившие названия по странам, где они были впервые выявлены, стали причиной крупных вспышек заболеваний в своих регионах. Эти варианты также вызывают озабоченность, поскольку они содержат мутацию Е484К в гене S-белка, которая снижает эффективность некоторых терапевтических моноклональных антител, ухудшает нейтрализацию вируса in vitro и может привести к потенциальному уходу от иммунного ответа, обусловленного ранее перенесенной инфекцией или вакцинацией. Несколько позже были обнаружены и другие эпидемиологически значимые штаммы, например, «индийский» (В. 1.617+, «дельта») и «калифорнийский» (В. 1.427/В. 1.429 «эпсилон»), также вызывающие беспокойство, в особенности первый. Кроме того, в России были выявлены и локальные штаммы «Сибирский» (В. 1.1.397+) и «Северо-западный» (В. 1.1.370.1), имеющие свои собственные ключевые отличия в гене S-белка, вероятно, требующие внимания.
При решении задач молекулярной диагностики нуклеиновых кислот, помимо методов полимеразной цепной реакции (ПЦР) и секвенирования первого поколения по методу Сэнгера, широко используется секвенирование следующего поколения (NGS, от англ. Next Generation Sequencing), потенциально позволяющее выявлять все изменения в геноме. Метод NGS играет незаменимую роль в обнаружении мутаций нового коронавируса SARS-CoV-2 и имеет более высокую точность по сравнению с широко применяемой технологией ОТ-ПЦР для обнаружения нуклеиновых кислот за счет одновременного прочтения протяженных участков генома и выявления сразу большого количества мутаций. Однако, существующий на данный момент механизм детекции важных мутаций и оперативного выявления значимых штаммов недостаточно эффективен, поскольку не все образцы патогена могут быть исследованы на наличие генетических изменений с помощью полногеномного секвенирования ввиду его высокой стоимости и большого объема необходимых работ в лаборатории. По мере развития технологий производительность секвенирующих платформ постоянно возрастает, а затраты на их работу становятся все меньше. Тем не менее, на текущем уровне технологического развития данного семейства методов, полногеномное секвенирование большого числа образцов сложно осуществить как в силу высоких материальных и трудовых затрат, так и в силу ограниченной возможности анализа очень больших массивов получаемых данных.
В качестве альтернативы полногеномному секвенированию, при решении ряда задач молекулярной диагностики может быть применено так называемое «таргетное» (или целевое) секвенирование представляющих наибольший интерес участков генома рассматриваемого патогена. Целевая область фрагментов ДНК целевого гена или области генома направленно амплифицируются, а затем подвергаются секвенированию NGS, так что стоимость значительно снижается, а необходимость последующего анализа данных также уменьшается. По сравнению с обычным методом NGS, таргетное секвенирование выполняется после целевой амплификации фрагментов генома, так что стоимость секвенирования и анализа данных значительно снижаются, а чувствительность обнаружения повышается. В настоящее время подобные подходы широко используются, например, для секвенирования экзома человека, отдельных генов, но редко применяется к патогенным микроорганизмам.
Как правило, методы секвенирования выполняют три функции во время эпидемических вспышек:
- идентификация неизвестных патогенов и определение идентичности новых патогенов в том случае, если будет получен весь или большая часть генома патогена;
- стадия развития эпидемической ситуации может использоваться для непосредственного обнаружения клинических образцов, динамического мониторинга состояния мутаций вирусного генома в режиме реального времени и помощи в диагностике ложно отрицательных образцов;
- стадия выявления эпидемической ситуации может использоваться для скрининга лекарственно-устойчивых участков и ежедневного выявления инфекций дыхательных путей, а также повышается точность диагностики заболеваний дыхательных путей.
Для того, чтобы идентифицировать исследуемый образец SARS-CoV-2 как представляющий интерес с эпидемиологической точки зрения, достаточно изучить несколько регионов гена, кодирующего S-белок, благодаря которому вирусная частица SARS-CoV-2 связывается с ангиотензинпревращающем ферментом 2 (рецептором АСЕ2), экспрессируемым клетками большинства тканей человека. При таргетном NGS секвенировании целевые регионы ДНК сначала амплифицируются с помощью специальных олигонуклеотидных праймеров, что позволяет изучать их затем избирательно. Далее, как правило, проводится этап лигирования служебных фрагментов олигонуклеотидов (адаптеров) с помощью особых дорогостоящих ферментов, и индексация с детекцией в режиме реального времени с помощью флуоресцентного интеркалирующего красителя.
Из уровня техники известны следующие протоколы для секвенирования геномов коронавируса SARS-CoV-2 и наборы реагентов, обеспечивающие выявление важных мутаций нового вируса:
Известно решение от Qiagen «QIAseq SARS-CoV-2 Primer Panel» для таргетной полногеномной пробоподготовки библиотек вируса SARS-CoV-2, что важно для геномного надзора и обнаружения генетических вариантов, в том числе новых. Панель праймеров QIAseq SARS-CoV-2 специально разработана для исследований генома коронавируса SARS-CoV-2. Этот набор в сочетании с набором QIAseq FX DNA Library UDI Kit представляет собой готовое решение для обогащения и секвенирования всего вирусного генома. Набор включает реагенты для обратной транскрипции РНК в кДНК и праймеры для специфического обогащения ее генома. Сама панель состоит из более чем 200 пар праймеров, охватывающих полный геном вируса размером ~29,900 п. н. [www.qiagen.com/ru/products/next-generation-sequencing/rna-sequencing/qiaseq-sars-cov-2-primer-panel/?clear=true#orderinginformation].
Существует решение «Swift Amplicon™ SARS-CoV-2 Panel», которое обеспечивает оптимальный охват и качество данных NGS на платформах секвенирования Illumina™. В этом наборе используется технология мультиплексной ПЦР, позволяющая конструировать библиотеку из цепи кДНК с использованием множества пар праймеров для покрытия полного вирусного генома длиной ~29,900 п. н. Праймеры были разработаны для эталонной последовательности NCBI NC 045512.2 (изолят коронавируса тяжелого острого респираторного синдрома 2 Ухань-Hu-l, полный геном) таким образом, чтобы отсутствовали нецелевые продукты, обусловленные амплификацией генома человека [swiftbiosci.com/swift-amplicon-sars-cov-2-panel/].
Известна панель «AmpliSeq for Illumina SARS-CoV-2 Research Panel» от компании Illumina, содержащая 247 ампликонов в 2 пулах, нацеленных на геном SARS-CoV-2. Панель обеспечивает более 99% покрытия генома коронавируса (длиной - 30 ООО п. н.) и охватывает все потенциальные серотипы [www.illumina.com/products/by-brand/ampliseq/community-panels/sars-cov-2.html].
Известна панель «Twist SARS-CoV-2 Research Panel» для обогащения мишеней для NGS секвенирования с целью обнаружения и характеризации вируса SARS-CoV-2. Панель нацелена на вирусный геном размером примерно 30,000 п. н. с использованием -1,000 зондов, разработанных для гибридизации с фрагментами генома SARS-CoV-2 (GenBank: MN908947.3) [www.twistbioscience.com/resources/product-sheet/twist-sars-cov-2-research-panel].
Панель «С1еапР1ех® SARS-CoV-2 FLEX» на основе ампликонов от Paragon Genomics предназначена для исследования и наблюдения за новым коронавирусом, обеспечивая полное покрытие генома вируса SARS-CoV-2. Панель FLEX построена на основе оригинальной панели SARS-CoV-2 и содержит дополнительные компоненты для более надежного поиска генетических вариантов, даже если вирус со временем мутирует. Это высоко мультиплексированная панель обогащения мишеней, охватывающая весь геном вируса SARS-CoV-2 (за исключением 92 нуклеотидных оснований на концах). Панель для исследований и наблюдения за SARS-CoV-2 позволяет провести полное секвенирование генома и эпидемиологические исследования нового вируса SARS-CoV-2, ответственного за пандемию COVID-19. С помощью технологии Paragon Genomics CleanPlex весь геном вируса может быть амплифицирован от РНК до готовых к последовательности библиотек за 5,5 часов [www.paragongenomics.com/product/cleanplex-sars-cov-2-flex-panel/].
Исследовательская панель «Ion AmpliSeq SARS-CoV-2» позволяет осуществлять высокоточное типирование вируса SARS-CoV-2, вызывающего COVID-19, менее, чем за сутки. Панель основана на технологии высокомультиплексной ПЦР AmpliSeq, что позволяет исследователям получить надежные данные секвенирования из небольшого количества стартового материала. «Ion AmpliSeq SARS-CoV-2» совместима с любыми секвенаторами, основанными на технологии Ion Torrent [www.thermofisher.com/ru/ra/home/global/forms/life-science/ampliseq-sars-cov-2-analysis-demo/thank-you.html].
Существует панель для NGS секвенирования гена S-белка «Swift Normalase Amplicon SARS-CoV-2», которая обеспечивает 100% покрытие S-гена, который кодирует спайковый белок в вирусе, вызывающем COVID-19. Панель позволяет определять появление новых вирусных штаммов, которые содержат мутации в гене S-белка, и определять их принадлежность к штаммам [swiftbiosci.com/swift-normalase-amplicon-sars-cov-2-s-gene-panel/].
Компания Thermo Fisher Scientific разработала и создала 26 тестов на основе ПЦР в реальном времени для выявления известных мутаций коронавируса «TaqMan SARS-CoV-2 Applied Biosystems», чтобы дать возможность исследователям комбинируя их создавать свою собственную панель для выявления мутаций. Это масштабируемое решение позволяет запускать несколько или сотни образцов для идентификации одной или нескольких мутаций - все на основе ПЦР в реальном времени [www.thermofisher.com/ru/ra/home/clinical/clinical-genomics/pathogen-detection-solutions/real-time-pcr-research-solutions-sars-cov-2/mutation-panel.html].
Существует способ получения кДНК из выделенных вирусных нуклеиновых кислот SARS-CoV-2 и последующего создания ампликонов с длиной 400 нуклеотидов, покрывающих вирусный геном, с использованием праймеров V3 nCov-2019 (ARTIC). За этим следует создание библиотеки, объединение эквивалентных объемов образцов и количественный анализ перед секвенированием на Illumina. Пул праймеров ARTIC V3 обеспечивает полногеномное секвенирование SARS-CoV-2. Пул праймеров ARTIC NGS был скорректирован для обеспечения специфической и равномерной амплификации геномных последовательностей SARS-CoV-2. Продукты ПЦР подходят для последующей подготовки библиотеки и анализа NGS. Этот продукт позволяет проводить селективную обогащающую амплификацию на основе ПЦР полного генома SARS-CoV-2 с использованием праймеров ARTIC, которые состоят из 218 праймеров, охватывающих весь вирусный геном SARS-CoV-2 (29,9 КБ). Каждая партия пула праймеров ARTIC NGS проходит валидацию, где одни и те же пулы праймеров используются для продукта «ARTIC SARS-CoV-2 WGS» [www.protocols.io/view/covid-19-artic-v3-illumina-library-construction-an-bgxjjxkn; eurofinsgenomics.eu/en/dna-ma-oligonucleotides/optimised-application-oligos/artic-ngs-primer-pool/].
Несмотря на то, что все вышеперечисленные решения позволяют выявлять мутации в геноме нового коронавируса, наиболее близким аналогом предлагаемого способа является протокол для NGS секвенирования гена S-белка «Swift Normalase Amplicon SARS-CoV-2». Процесс определения геномных последовательностей патогенов наподобие вируса SARS-CoV-2, согласно данному протоколу, включает в себя создание ампликонов с помощью мультиплексной ПЦР в одной пробирке в течение 2-х часов, поддерживает высокопроизводительные методы количественного анализа библиотеки (флуориметрический, электрофоретический), и оптимизирован для всех платформ секвенирования от компании Illumina.
Вместе с тем, все перечисленные решения, включая ближайший аналог, имеют общий недостаток, который выражается не только в высокой стоимости необходимых расходных материалов, а также в том, что секвенируются полные геном или S-ген вируса, и информация может быть избыточна, если целью стоит только выявление эпидемиологически значимых мутаций вируса, присущих к известным штаммам, вызывающим беспокойство.
Решения, основанные на ПЦР в реальном времени, также имеют свой недостаток -выявляются лишь конкретные мутации, без возможности исследования соседних участков в «горячих» областях, в которых могут произойти новые генетические изменения. Кроме того, выявление каждой мутации требует отдельной постановки ПЦР реакции, что увеличивает время исследования.
В настоящее время существует острая необходимость в получении эффективных и простых способах приготовления библиотек для целевого секвенирования важных фрагментов генома нового коронавируса SARS-CoV-2.
Технической задачей, на решение которой направлено заявляемое решение, является разработка эффективного способа пробоподготовки для фрагментарного NGS-секвенирования ряда регионов генома коронавируса S ARS-CoV-2, содержащих известные эпидемиологически значимые мутации.
Заявляемое изобретение позволяет преодолеть недостатки предшествующего уровня техники и в значительной мере уменьшает трудо- и финансовые затраты при пробоподготовке, объем получаемых данных секвенирования, что упрощает процесс анализа данных, и дополнительно повышает чувствительность и точность метода NGS.
Технический результат достигается за счет таргетного (целевого) секвенирования представляющих наибольший интерес участков генома вируса SARS-CoV-2, содержащих известные эпидемиологически значимые мутации, позволяющие также отнести изолят коронавируса к известным штаммам, в том числе штаммам, вызывающим обеспокоенность. Посредством разработанных праймеров, имеющих адаптерные подпоследовательности в своем составе, возможна амплификация целевых участков генома, и при этом не требуется проводить дорогостоящий этап лигирования адаптеров, используемый в ряде протоколов. При этом амплификацию проводят с использованием двух пулов олигонуклеотидных праймеров, где для первого пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1 4, с концентрацией в реакционной смеси каждого праймера 0,1 пмоль/мкл., а для второго пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 5 - 10, с концентрацией в реакционной смеси каждого праймера 0,2 пмоль/мкл. Амплифицированные фрагменты кДНК, после проведения секвенирования, позволяют выявить значимые мутации патогена в исследуемом образце.
Сложность выбора праймеров обусловлена требованием к их строгой видоспецифичности, необходимости обеспечить амплификацию в формате мультиплекса, отсутствия кросс-комплементарности между собой и с используемыми адаптерами, а также другими фрагментами генома коронавируса SARS-CoV-2 и генома человека.
Предложенные в изобретении синтетические олигонуклеотидные праймеры для обнаружения ряда эпидемиологически значимых мутаций в генетическом материале (РНК) коронавируса SARS-CoV-2 методом NGS секвенирования имеют следующую структуру: SEQ ID NO: 1-10. При этом олигонуклеотидные праймеры, имеющие структуры SEQ ID NO: 1, 3, 5, 7, 9 выполняют функции прямых праймеров, а олигонуклеотидные праймеры, имеющие структуры SEQ ID NO: 2, 4, 6, 8, 10, выполняют функции обратных праймеров.
Кроме того, олигонуклеотидные праймеры содержат в своем составе универсальные адаптерные последовательности, представленные в перечне последовательностей как SEQ ID NO: 11 и 12, позволяющие в дальнейшем облегчить индексацию продуктов амплификации. Адаптерные последовательности SEQ ID NO: 11, расположены на концах олигонуклеотидов SEQ ID NO: 1, 3, 5, 7, 9. Адаптерные последовательности SEQ ID NO: 12, расположены на концах олигонуклеотидов SEQ ID NO: 2, 4, 6, 8, 10.
При разработке применяемых в данном способе 5 пар праймеров (SEQ ID NO: 1-10) для таргетной амплификации фрагментов гена S-белка олигонуклеотидные последовательности были подобраны вручную с учетом имеющейся информации об известных эпидемиологически значимых мутациях. Этот процесс включал в себя загрузку множества последовательностей геномов коронавируса SARS-CoV-2 из баз данных NCBI и GISAID, выравнивание последовательностей с целью определения консервативных фрагментов, определение списка эпидемиологически значимых генетических изменений. Температуры плавления олигонуклеотидов и характер взаимодействий между ними определялись с помощью инструмента Multiple Primer Analyzer от Thermo Fisher Scientific (США). С помощью программы blastn оценивалась специфичность каждой полученной последовательности ко всем известным организмам, в частности, Homo sapiens, что позволяет исключить неспецифическое взаимодействие между праймерами и участками ДНК человека и других организмов. Кроме того, синтезированные олигонуклеотиды содержат дополнительные последовательности, облегчающие и удешевляющие процесс индексации продуктов амплификации. Расстояния между праймерами в парах были подобраны таким образом, чтобы длина исследуемого региона была в пределах 300 п. н., что позволяет избежать этапа отбора фрагментов с заданной длиной, и совместимо с длиной прочтений большинства наборов реагентов для секвенирования от компании Illumina.
Полученные олигонуклеотиды на одном конце имеют праймерную последовательность, комплементарную участкам генома, а на другом - адаптерные последовательности, следовательно, полученные после амплификации целевые фрагменты сразу фланкированы такими адаптерными последовательностями. Олигонуклеотиды, используемые для индексации, в свою очередь, комплементарны адаптерным последовательностям с одного конца и соответствуют индексам с другого.
В качестве индексных последовательностей используют олигонуклеотиды, несущие различные индексы, которые позволяют отличать данные секвенирования молекул из разных образцов. Примеры подобных индексных последовательностей доступны на сайтах производителей наборов реагентов для высокопроизводительного секвенирования.
Таким образом, в процессе индексации происходят этапы отжига олигонуклеотидов на концах целевых фрагментов и амплификации. После этого продукты амплификации подвергаются секвенированию методом NGS. Описанный подход приводит к тому, что стоимость исследования значительно снижается, уменьшаются объемы нуждающихся в анализе экспериментальных данных, а также возрастает чувствительность обнаружения. В настоящее время такие подходы широко распространены при клиническом секвенировании полного экзома человека, но редко применяются к патогенным микроорганизмам.
На основе имеющейся информации, определены регионы генома SARS-CoV-2, мутации в которых представляют наибольшую эпидемиологическую важность. Таргетное секвенирование данных участков позволяет кардинально снизить стоимость исследования и сократить объем генерируемых данных, включая соответствующий биоинформатический анализ.
По сравнению с известным протоколом пробоподготовки Illumina Nextera XT (https://www.illumina.com/products/by4ype/sequencing-kits/library-prep-kits/nextera-xt-dna.html), предлагаемый способ пробоподготовки позволяет избежать этапов тагментации и отбора фрагментов с необходимыми длинами, поскольку продукты амплификации изначально имеют длину от 216 до 377 пар нуклеотидов без учета адаптерных последовательностей (то есть совместимы с большинством версий наборов реагентов компании Illumina для ее секвенирующих платформ), а также содержат адаптерные последовательности для дальнейшей индексации, что существенно упрощает и удешевляет анализ полученных при секвенировании данных.
Заявляемое изобретение является результатом работы в рамках исследования генетического разнообразия коронавируса SARS-CoV-2, проделанной в ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (Москва, Россия). Представленный способ был отработан более чем на 500 образцах биологического материала, представляющего собой мазки в транспортной среде от пациентов из Москвы и Московской области с подозрениями на COVID-19, которые впоследствии были идентифицированы как положительные на наличие SARS-CoV-2 с помощью набора реагентов «AmpliSens® Cov-Bat-FL» (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), использованным в соответствии с инструкцией производителя.
Выделение РНК из клинического материала производилось с помощью комплекта реагентов в соответствии с инструкцией производителя. Для выделения РНК может быть использован комплекта реагентов РИБО-преп (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любой аналогичный коммерческий набор для выделения РНК.
Обратная транскрипция проводится с использованием набора реагентов РЕВЕРТА-L (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) или любого аналогичного коммерческого набора для обратной транскрипции в соответствии с инструкцией производителя. Далее, с полученной кДНК в качестве матрицы проводятся две реакции амплификации с использованием двух пулов специфических олигонуклеотидов (Таблица 1. Структуры олигонуклеотидов, используемых для амплификации целевых фрагментов геномов коронавируса SARS-CoV-2). Праймеры, помимо специфической области, комплементарной целевому фрагменту генома вируса, дополнительно содержат также особые последовательности, комплементарные последовательностям адаптеров, используемых при пробоподготовке к NGS секвенированию.
ПЦР-микс объемом 20 мкл на одну реакцию содержит 10 мкл ПЦР-смеси-2 blue (АмплиСенс, ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ, олигонуклеотиды в указанных в таблице концентрациях и воду milli-Q. ДНК-матрица добавляется в количестве 5 мкл, итоговый объем реакции составляет 25 мкл. Продуктами амплификации являются целевые участки гена, кодирующего S-белок вируса SARS-CoV-2. Длины получаемых фрагментов составляют 216-377 п. н. без учета длин адаптерных и индексных последовательностей, что обеспечивает их частичное или полное покрытие при использовании популярных наборов реагентов для секвенирования: MiSeq Reagent Kit v2 (300-cycles), MiSeq Reagent Kit v3 (600-cycle), HiSeq Rapid SBS Kit v2 (500 cycles) - все производства Illumina (США). Температурный профиль амплификации для получения ПЦР-продукта:
1. Денатурация проводится при 95°С в течение 2 минут;
2. 38 циклов амплификации:
a. 95°С - 30 сек;
b. 60°С - 20 сек;
c. 72°С - 60 сек.
3. Финальная элонгация: 72°С в течение 3 минут.
Длины полученных продуктов амплификации оцениваются с помощью электрофореза в агарозном геле. По интенсивности полос, соответствующих целевым фрагментам, оценивается приблизительное количество ПЦР-продуктов в каждом из двух пулов. Пропорционально полученным оценкам продукты амплификации первого и второго пулов объединяются в объемном отношении (n1*2):(n2*3), где n1 и n2 - оценки интенсивности для первого и второго пулов соответственно.
Затем проводится очистка ПЦР-продуктов от реакционной смеси с использованием магнитных частиц, например, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США) в объемном отношении 1:1.3.
После этого осуществляется индексация полученных фрагментов. Синтезированные олигонуклеотиды на одном конце содержат праймерную последовательность, комплементарную целевым участкам генома, а на другом -адаптерные последовательности двух типов: для прямых праймеров - SEQ ID NO: 11; для обратных праймеров - SEQ ID NO: 12, соответственно. Таким образом, полученные после амплификации целевые фрагменты будут сразу фланкированы адаптерными последовательностями, что существенно ускоряет и удешевляет пробоподготовку. Олигонуклеотиды, используемые для индексации, в свою очередь, на одном конце содержат последовательности, соответствующие индексам, а на другом последовательности, комплементарные упомянутым ранее адаптерным последовательностям.
Таким образом, в процессе индексации происходят этапы отжига олигонуклеотидов на концах целевых фрагментов и амплификации продукта. ПЦР-микс объемом 19 мкл на одну реакцию содержит 11.5 мкл ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ, 1 мкл интеркалирующего красителя EvaGreen® Dye (Biotium, США) для детекции амплификации в реальном времени, соответствующие олигонуклеотиды в концентрации 0.357 пмоль/мкл и воду milli-Q. ДНК-матрица добавляется в количестве 9 мкл, итоговый объем реакции составляет 28 мкл.
Температурный профиль индексации:
1. 98°С в течение 30 секунд;
2. 15 циклов:
a. 98°С в течение 10 секунд;
b. 65°С в течение 1 минуты 15 секунд (на этом этапе осуществляется детекция).
Далее повторно производится очистка от реакционной смеси с использованием магнитных частиц, например, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США) в объемном отношении 1:1.3.
В результате получаются готовые для секвенирования библиотеки, содержащие целевые фрагменты генома SARS-CoV-2, содержащие известные эпидемиологически значимые мутации, позволяющие также отнести изолят коронавируса к известным штаммам, в том числе, штаммам, вызывающим обеспокоенность.
Заявляемое изобретение поясняется рисунками, где:
Рисунок 1. Расположение участков гена S-белка вируса SARS-CoV-2, амплифицируемых с помощью разработанных наборов олигонуклеотидных праймеров. Также на рисунке отмечены мутации, представляющие наибольший интерес с эпидемиологической точки зрения ввиду их влияния на биологические свойства вируса.
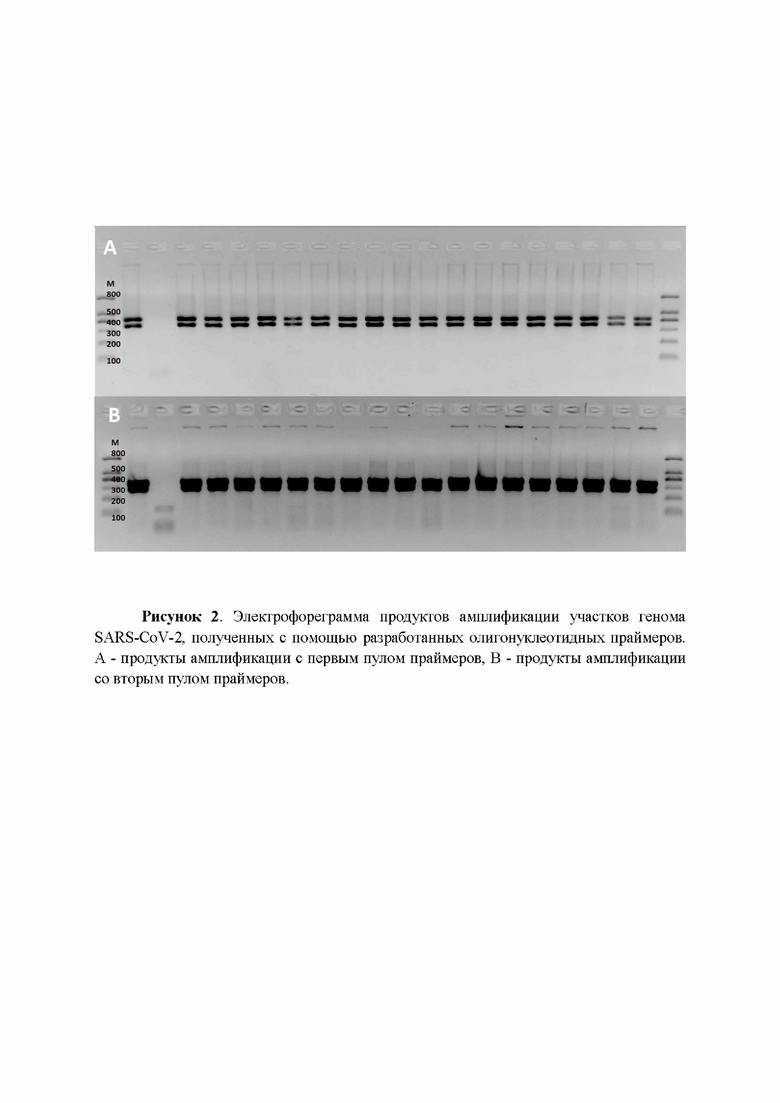
Рисунок 2. Электрофореграмма продуктов амплификации участков генома SARS-CoV-2, полученных с помощью разработанных олигонуклеотидных праймеров. А -продукты амплификации с первым пулом праймеров, В - продукты амплификации со вторым пулом праймеров.
Заявляемое изобретение также поясняется следующими примерами конкретного применения.
Пример 1. Обнаружение последовательности линии В. 1.1.7.
У пациента с симптомами ОРВИ был взят мазок из ротоглотки. С помощью набора реагентов РИБО-преп (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) из собранного материала была выделена РНК вируса, впоследствии подвергнутая обратной транскрипции с помощью набора PEBEPTA-L (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), оба этапа проводились согласно инструкциям производителя. Полученная кДНК была использована для амплификации целевых участков в двух пулах. ПЦР-микс объемом 20 мкл на одну реакцию был составлен из 10 мкл ПЦР-смеси-2 blue (АмплиСенс, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ и последовательностей, приведенных в перечне. В первом пуле в реакционную среду были добавлены олигонуклеотидные праймеры SEQ ID NO: 1-4 с концентрацией в реакционной смеси каждого праймера 0,1 пмоль/мкл. Во втором пуле в реакционную среду были добавлены олигонуклеотидные праймеры SEQ ID NO: 5-10 с концентрацией в реакционной смеси каждого праймера 0,2 пмоль/мкл.
Полученные смеси были доведены до объема 20 мкл с помощью воды Milli-Q. ДНК-матрица добавлялась в количестве 5 мкл, итоговый объем каждой реакции составлял 25 мкл.
Температурный профиль амплификации:
1. Денатурация проводится при 95°С в течение 2 минут;
2. 38 циклов амплификации:
a. 95°С - 30 сек;
b. 60°С - 20 сек;
c. 72°С - 60 сек.
3. Финальная элонгация: 72°С в течение 3 минут.
Полученные продукты амплификации, имеющие длину 216-377 п.н. без учета длин адаптерных и индексных последовательностей, были обнаружены методом электрофореза в агарозном геле, после чего пулы были объединены, а полученная смесь была очищена от реакционной среды с помощью магнитных частиц, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США). Затем, методом ПЦР в реальном времени была проведена индексация продуктов амплификации. Синтезированные олигонуклеотиды на одном конце содержат праймерную последовательность, комплементарную целевым участкам генома, а на другом - адаптерные последовательности двух типов: для прямых праймеров - SEQ ID NO: 11; для обратных праймеров - SEQ ID NO: 12, соответственно. ПЦР-микс объемом 19 мкл на одну реакцию был составлен из 11.5 мкл ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ, 1 мкл интеркалирующего красителя EvaGreen® Dye (Biotium, США) для детекции амплификации в реальном времени, соответствующих олигонуклеотидов в концентрации 0.357 пмоль/мкл и воды milli-Q. ДНК-матрица добавлялась в количестве 9 мкл, итоговый объем реакции составлял 28 мкл.
Температурный профиль индексации:
1. 98°С в течение 30 секунд;
2. 15 циклов:
a. 98°С в течение 10 секунд;
b. 65°С в течение 1 минуты 15 секунд (этап детекции).
Далее повторно была произведена очистка от реакционной смеси с использованием магнитных частиц, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США).
Индексированные последовательности были отсеквенированы на приборе Illumina MiSeq с использованием реагентов MiSeq Reagent Kit v3 (600-cycle). Результаты исследования были проанализированы с помощью утилит FastQC, bwa и samtools.
При изучении полученных данных был сделан вывод о наличии в исходной последовательности мутаций HV69-70, Y144, N501Y и A570D, что позволяет отнести ее к линии В. 1.1.7 («британский» штамм).
Пример 2. Обнаружение последовательности линии В. 1.351.
У пациента с симптомами ОРВИ был взят мазок из ротоглотки. С помощью набора реагентов РИБО-преп (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) из собранного материала была выделена РНК вируса, впоследствии подвергнутая обратной транскрипции с помощью набора PEBEPTA-L (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), оба этапа проводились согласно инструкциям производителя. Полученная кДНК была использована для амплификации целевых участков в двух пулах. ПЦР-микс объемом 20 мкл на одну реакцию был составлен из 10 мкл ПЦР-смеси-2 blue (АмплиСенс, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ и последовательностей, приведенных в перечне. В первом пуле в реакционную среду были добавлены олигонуклеотидные праймеры SEQ ID NO: 1-4 с концентрацией в реакционной смеси каждого праймера 0,1 пмоль/мкл. Во втором пуле в реакционную среду были добавлены олигонуклеотидные праймеры SEQ ID NO: 5-10 с концентрацией в реакционной смеси каждого праймера 0,2 пмоль/мкл. Полученные смеси были доведены до объема 20 мкл с помощью воды Milli-Q. ДНК-матрица добавлялась в количестве 5 мкл, итоговый объем каждой реакции составлял 25 мкл.
Температурный профиль амплификации:
1. Денатурация проводится при 95°С в течение 2 минут;
2. 38 циклов амплификации:
a. 95°С - 30 сек;
b. 60°С - 20 сек;
c. 72°С - 60 сек.
3. Финальная элонгация: 72°С в течение 3 минут.
Полученные продукты амплификации, имеющие длину 216-377 п. н. без учета длин адаптерных и индексных последовательностей, были обнаружены методом электрофореза в агарозном геле, после чего пулы были объединены, а полученная смесь была очищена от реакционной среды с помощью магнитных частиц, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США). Затем, методом ПЦР в реальном времени была проведена индексация продуктов амплификации. Синтезированные олигонуклеотиды на одном конце содержат праймерную последовательность, комплементарную целевым участкам генома, а на другом - адаптерные последовательности двух типов: для прямых праймеров - SEQ ID NO: 11; для обратных праймеров - SEQ ID NO: 12, соответственно. ПЦР-микс объемом 19 мкл на одну реакцию был составлен из 11.5 мкл ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ, 1 мкл интеркалирующего красителя EvaGreen® Dye (Biotium, США) для детекции амплификации в реальном времени, соответствующих олигонуклеотидов в концентрации 0.357 пмоль/мкл и воды milli-Q. ДНК-матрица добавлялась в количестве 9 мкл, итоговый объем реакции составлял 28 мкл.
Температурный профиль индексации:
1. 98°С в течение 30 секунд;
2. 15 циклов:
a. 98°С в течение 10 секунд;
b. 65°С в течение 1 минуты 15 секунд (этап детекции).
Далее повторно была произведена очистка от реакционной смеси с использованием магнитных частиц, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США).
Индексированные последовательности были отсеквенированы на приборе Illumina MiSeq с использованием реагентов MiSeq Reagent Kit v3 (600-cycle). Результаты исследования были проанализированы с помощью утилит FastQC, bwa и samtools. При изучении полученных данных был сделан вывод о наличии в исходной последовательности мутаций D80A, Е484К и N501Y, что позволяет отнести ее к линии В. 1.351 («южноафриканский» штамм).
Пример 3. Обнаружение последовательности линии В. 1.1.523.
У пациента с симптомами ОРВИ был взят мазок из ротоглотки. С помощью набора реагентов РИБО-преп (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия) из собранного материала была выделена РНК вируса, впоследствии подвергнутая обратной транскрипции с помощью набора PEBEPTA-L (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), оба этапа проводились согласно инструкциям производителя. Полученная кДНК была использована для амплификации целевых участков в двух пулах. ПЦР-микс объемом 20 мкл на одну реакцию был составлен из 10 мкл ПЦР-смеси-2 blue (АмплиСенс, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ и последовательностей, приведенных в перечне. В первом пуле в реакционную среду были добавлены олигонуклеотидные праймеры SEQ ID NO: 1-4 с концентрацией в реакционной смеси каждого праймера 0,1 пмоль/мкл. Во втором пуле в реакционную среду были добавлены олигонуклеотидные праймеры SEQ ID NO: 5-10 с концентрацией в реакционной смеси каждого праймера 0,2 пмоль/мкл. В первом и во втором пулах в реакционную среду были добавлены олигонуклеотиды SEQ ID NO: 1-4 и SEQ ID NO: 5-10 соответственно. Полученные смеси были доведены до объема 20 мкл с помощью воды Milli-Q. ДНК-матрица добавлялась в количестве 5 мкл, итоговый объем каждой реакции составлял 25 мкл.
Температурный профиль амплификации:
1. Денатурация проводится при 95°С в течение 2 минут;
2. 38 циклов амплификации:
a. 95°С - 30 сек;
b. 60°С - 20 сек;
c. 72°С - 60 сек.
3. Финальная элонгация: 72°С в течение 3 минут.
Полученные продукты амплификации, имеющие длину 216-377 п. н. без учета длин адаптерных и индексных последовательностей, были обнаружены методом электрофореза в агарозном геле, после чего пулы были объединены, а полученная смесь была очищена от реакционной среды с помощью магнитных частиц, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США). Затем, методом ПЦР в реальном времени была проведена индексация продуктов амплификации. Синтезированные олигонуклеотиды на одном конце содержат праймерную последовательность, комплементарную целевым участкам генома, а на другом - адаптерные последовательности двух типов: для прямых праймеров - SEQ ID NO: 11; для обратных праймеров - SEQ ID NO: 12, соответственно. ПЦР-микс объемом 19 мкл на одну реакцию был составлен из 11.5 мкл ПЦР-смеси-2 blue (ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, Россия), 2.5 мкл эквимолярной смеси четырех дезоксирибонуклеозидтрифосфатов со стоковой концентрацией 4.4 мМ, 1 мкл интеркалирующего красителя EvaGreen® Dye (Biotium, США) для детекции амплификации в реальном времени, соответствующих олигонуклеотидов в концентрации 0.357 пмоль/мкл и воды milli-Q. ДНК-матрица добавлялась в количестве 9 мкл, итоговый объем реакции составлял 28 мкл.
Температурный профиль индексации:
1. 98°С в течение 30 секунд;
2. 15 циклов:
a. 98°С в течение 10 секунд;
b. 65°С в течение 1 минуты 15 секунд (этап детекции).
Далее повторно была произведена очистка от реакционной смеси с использованием магнитных частиц, SpeedBeads™ magnetic carboxylate modified particles (Cytiva, США).
Индексированные последовательности были отсеквенированы на приборе Illumina MiSeq с использованием реагентов MiSeq Reagent Kit v3 (600-cycle). Результаты исследования были проанализированы с помощью утилит FastQC, bwa и samtools. При изучении полученных данных был сделан вывод о наличии в исходной последовательности мутаций Е484К, S494P и EFR156-158, что позволяет отнести ее к линии В.1.1.523.
Предложенное изобретение позволяет произвести пробоподготовку библиотек, содержащие целевые фрагменты генома SARS-CoV-2, содержащих известные эпидемиологически значимые мутации, с наименьшими трудовыми, временными и материальными затратами, не снижая качество и объемы произведенных исследований.
--->
Перечень последовательностей
ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
NUMBER OF SEQ ID NO: NOS: 12
SEQ ID NO: NO 1
63
DNA
artificial
SEQUENCE 1 ():
TCG TCG GCA GCG TCA GAT GTG TAT AAG AGA CAG TGT TTT TCT TGT TTT ATT GCC ACT AGT CTC 63
SEQ ID NO: NO 2
65
DNA
artificial
SEQUENCE 2 ():
GTC TCG TGG GCT CGG AGA TGT GTA TAA GAG ACA GTC TTA TTA TGT TAG ACT TCT CAG TGG AAG CA 65
SEQ ID NO: NO 3
62
DNA
artificial
SEQUENCE 3 ():
TCG TCG GCA GCG TCA GAT GTG TAT AAG AGA CAG GCT GGA TTT TTG GTA CTA CTT TAG ATT CG 62
SEQ ID NO: NO 4
64
DNA
artificial
SEQUENCE 4 ():
GTC TCG TGG GCT CGG AGA TGT GTA TAA GAG ACA GAA TCT ACC AAT GGT TCT AAA GCC GAA AAA C 64
SEQ ID NO: NO 5
53
DNA
artificial
SEQUENCE 5 ():
TCG TCG GCA GCG TCA GAT GTG TAT AAG AGA CAG GCT CCA GGG CAA ACT GGA AA 53
SEQ ID NO: NO 6
59
DNA
artificial
SEQUENCE 6 ():
GTC TCG TGG GCT CGG AGA TGT GTA TAA GAG ACA GCT GTA TGG TTG GTA ACC AAC ACC AT 59
SEQ ID NO: NO 7
56
DNA
artificial
SEQUENCE 7 ():
TCG TCG GCA GCG TCA GAT GTG TAT AAG AGA CAG CCA ACA ATT TGG CAG AGA CAT TG 56
SEQ ID NO: NO 8
59
DNA
artificial
SEQUENCE 8 ():
GTC TCG TGG GCT CGG AGA TGT GTA TAA GAG ACA GCG CCA AGT AGG AGT AAG TTG ATC TG 59
SEQ ID NO: NO 9
58
DNA
artificial
SEQUENCE 9 ():
TCG TCG GCA GCG TCA GAT GTG TAT AAG AGA CAG AAA CAC GTG CAG GCT GTT TAA TAG G 58
SEQ ID NO: NO 10
62
DNA
artificial
SEQUENCE 10 ():
GTC TCG TGG GCT CGG AGA TGT GTA TAA GAG ACA GCT ACT GAT GTC TTG GTC ATA GAC ACT GG 62
SEQ ID NO: NO 11
33
DNA
artificial
SEQUENCE 11 ():
TCG TCG GCA GCG TCA GAT GTG TAT AAG AGA CAG 33
SEQ ID NO: NO 12
34
DNA
artificial
SEQUENCE 12 ():
GTC TCG TGG GCT CGG AGA TGT GTA TAA GAG ACA G 34
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ секвенирования экзонов гена HLA-DPA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2837865C1 |
| Олигонуклеотиды для определения мутации S:N501Y SARS-CoV-2 | 2022 |
|
RU2791958C1 |
| Способ секвенирования экзонов гена HLA-DQA1 и набор синтетических олигонуклеотидов для его реализации | 2024 |
|
RU2833819C1 |
| Олигонуклеотиды для определения мутации S:L452R SARS-CoV-2 | 2022 |
|
RU2795018C1 |
| Олигонуклеотиды для определения мутации S:P681R SARS-CoV-2 | 2022 |
|
RU2795019C1 |
| Способ пробоподготовки образцов для типирования генов главного комплекса гистосовместимости HLA-A, HLA-B, HLA-C, HLA-DPB1, HLA-DQB1, HLA-DRB1 и олигонуклеотидные праймеры для его реализации | 2023 |
|
RU2829344C1 |
| Олигонуклеотиды для определения мутации S:delVYY143-145 SARS-CoV-2 | 2022 |
|
RU2795016C1 |
| Олигонуклеотиды для определения мутации S:Ins214EPE SARS-CoV-2 | 2022 |
|
RU2795017C1 |
| Олигонуклеотиды для определения мутации S:delHV69-70 SARS-CoV-2 | 2022 |
|
RU2795014C1 |
| Способ пробоподготовки образцов ДНК вируса гепатита В для полногеномного секвенирования и олигонуклеотидные праймеры для его реализации | 2023 |
|
RU2818585C1 |
Изобретение относится к биотехнологии, медицине, молекулярной биологии, генной инженерии и вирусологии. Описан способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2, включающий стадии: (a) обратной транскрипции РНК вируса; (b) ПЦР с применением двух пулов олигонуклеотидных праймеров, где для первого пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1-4, с концентрацией в реакционной смеси каждого праймера 0,1 пмоль/мкл, а для второго пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 5-10, с концентрацией в реакционной смеси каждого праймера 0,2 пмоль/мкл; (c) оценки получаемых в ходе амплификации ПЦР-продуктов с помощью электрофореза; (d) объединения ПЦР-продуктов двух пулов; (e) очистки ампликонов от реакционной смеси с использованием парамагнитных частиц с полимерным карбоксилированным покрытием; (f) индексации с использованием олигонуклеотидов, содержащих последовательности, комплементарные адаптерным последовательностям SEQ ID NO: 11-12, и индексные последовательности; (g) повторной очистки от реакционной смеси с использованием парамагнитных частиц с полимерным карбоксилированным покрытием. Представлен пул олигонуклеотидных праймеров для реализации способа по п. 1, имеющих структуру SEQ ID NO: 1-4, для выполнения функции праймеров, комплементарных участкам гена, кодирующего S-белок SARS-CoV-2, и пул олигонуклеотидных праймеров для реализации способа по п. 1, имеющих структуру SEQ ID NO: 5-10, для выполнения функции праймеров, комплементарных участкам гена, кодирующего S-белок SARS-CoV-2. Изобретение позволяет произвести пробоподготовку библиотек, содержащих целевые фрагменты генома SARS-CoV-2, содержащие известные эпидемиологически значимые мутации, с наименьшими трудовыми, временными и материальными затратами, не снижая качество и объемы произведенных исследований за счет применения. 3 н. и 6 з.п. ф-лы, 2 ил., 1 табл., 3 пр.
1. Способ пробоподготовки образцов изолятов коронавируса SARS-CoV-2, включающий стадии:
(a) обратной транскрипции РНК вируса;
(b) ПЦР с применением двух пулов олигонуклеотидных праймеров, где для первого пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1-4, с концентрацией в реакционной смеси каждого праймера 0,1 пмоль/мкл, а для второго пула применяют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 5-10, с концентрацией в реакционной смеси каждого праймера 0,2 пмоль/мкл;
(c) оценки получаемых в ходе амплификации ПЦР-продуктов с помощью электрофореза;
(d) объединения ПЦР-продуктов двух пулов;
(e) очистки ампликонов от реакционной смеси с использованием парамагнитных частиц с полимерным карбоксилированным покрытием;
(f) индексации с использованием олигонуклеотидов, содержащих последовательности, комплементарные адаптерным последовательностям SEQ ID NO: 11-12, и индексные последовательности;
(g) повторной очистки от реакционной смеси с использованием парамагнитных частиц с полимерным карбоксилированным покрытием.
2. Способ по п. 1, в котором амплификацию фрагментов кДНК в ПЦР осуществляют с использованием Таq-полимеразы.
3. Пул олигонуклеотидных праймеров для реализации способа по п. 1, имеющих структуру SEQ ID NO: 1-4, для выполнения функции праймеров, комплементарных участкам гена, кодирующего S-белок SARS-CoV-2.
4. Пул олигонуклеотидных праймеров для реализации способа по п. 1, имеющих структуру SEQ ID NO: 5-10, для выполнения функции праймеров, комплементарных участкам гена, кодирующего S-белок SARS-CoV-2.
5. Олигонуклеотидные праймеры по п. 3, где функции прямых праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 1, 3.
6. Олигонуклеотидные праймеры по п. 3, где функции обратных праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 2, 4.
7. Олигонуклеотидные праймеры по п. 4, где функции прямых праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 5, 7, 9.
8. Олигонуклеотидные праймеры по п. 4, где функции обратных праймеров выполняют олигонуклеотидные праймеры, имеющие структуру SEQ ID NO: 6, 8, 10.
9. Олигонуклеотидные праймеры по пп. 3 и 4, которые в своем составе имеют адаптерные последовательности, при этом адаптерные последовательности SF.Q ID NO: 11 расположены на концах олигонуклеотидных праймеров, имеющих структуру SEQ ID NO: 1, 3, 5, 7, 9, а адаптерные последовательности SEQ ID NO: 12 расположены на концах олигонуклеотидных праймеров, имеющих структуру SEQ ID NO: 2, 4, 6, 8, 10.
| Способ выявления кДНК коронавируса SARS-CoV-2 с помощью синтетических олигонуклеотидных праймеров в полимеразной цепной реакции | 2020 |
|
RU2727054C1 |
| CN 111455114 A, 28.07.2020 | |||
| CN 111139317 A, 12.05.2020 | |||
| Comparison of National RT-PCR Primers, Probes, and Protocols for SARS-CoV-2 Diagnostics, Johns Hopkins Center for Health Security, 13.04.2021, стр.1-5. | |||
Авторы
Даты
2021-12-22—Публикация
2021-06-30—Подача