1. ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННУЮ ЗАЯВКУ
В настоящей заявке испрашивается преимущество предварительной заявки на патент США №62/796 228, поданной 24 января 2019 г., озаглавленной «АВС-ТРАНСПОРТЕРЫ ДЛЯ ВЫСОКОЭФФЕКТИВНОГО ПРОИЗВОДСТВА РЕБАУДИОЗИДОВ», раскрытие которой полностью включено в настоящую заявку посредством ссылки.
2. ОБЛАСТЬ ТЕХНИКИ
Настоящее раскрытие относится к конкретным ABC-транспортерам, клеткам-хозяевам, содержащим их, и способам их использования для продукции стевиола и/или ребаудиозидов, включая ребаудиозид D и ребаудиозид М.
3. ОБЩИЕ СВЕДЕНИЯ
[0001] Желательно, чтобы подсластители с пониженной калорийностью, полученные из натуральных источников, ограничивали воздействие повышенного потребления сахара на здоровье. Растение стевия (Stevia rebaudiana Bertoni) производит множество сладких на вкус гликозилированных дитерпенов, называемых гликозидами стевиола. Из всех известных стевиоловых гликозидов Reb М обладает наивысшей эффективностью (примерно в 200-300 раз слаще сахарозы) и имеет наиболее привлекательный вкусовой профиль. Однако Reb М вырабатывается растением стевия только в незначительных количествах и составляет небольшую часть от общего содержания стевиоловых гликозидов (<1,0%), что делает нецелесообразным выделение Reb М из листьев стевии. Необходимы альтернативные методы получения Reb М. Одним из таких подходов является применение синтетической биологии для создания микроорганизмов (например, дрожжей), которые производят большие количества Reb М из устойчивых источников сырья.
[0002] Для экономичного производства продукта с использованием синтетической биологии каждый этап биоконверсии сырья в продукт должен иметь высокую эффективность преобразования (в идеале>90%). При разработке дрожжей для производства Reb М мы отметили, что цитозольное накопление Reb М подавляет метаболический путь стевиоловых гликозидов, сконструированный в дрожжах, тем самым ограничивая общий выход цикла ферментации. Это подавление, вероятно, связано с ингибированием продукта или ингибированием конечным продуктом одного или нескольких ферментов, участвующих в биосинтезе стевиоловых гликозидов. Соответственно, новые механизмы снятия ингибирования продукта необходимы для повышения эффективности преобразования биосинтетического производства Reb М.
4. РАСКРЫТИЕ ИЗОБРЕТЕНИЯ
[0003] В настоящем документе представлены генетически модифицированные клетки-хозяева, композиции и способы для улучшенного продуцирования Reb М. Эти композиции и способы частично основаны на экспрессии определенных гетерологических АВС-транспортеров в клетках-хозяевах, которые были генетически модифицированы для получения стевиоловых гликозидов, таких как Reb М. Эти ABC-транспортеры способны переносить определенные стевиоловые гликозиды, предпочтительно Reb М и/или родственный высокомолекулярный стевиолгликозид ребаудиозид D (Reb D), из цитозоля либо во внеклеточное пространство, либо в просвет субклеточных органелл, например, дрожжевую вакуоль. Секвестрация некоторых стевиоловых гликозидов, таких как Reb D и Reb М, увеличивает эффективность метаболического пути стевиоловых гликозидов за счет снятия ингибирования продукта, вызванного накоплением стевиоловых гликозидов.
[0004] В одном аспекте изобретения в настоящем документе представлены генетически модифицированные клетки-хозяева и способы их использования для получения промышленно полезных соединений. В одном аспекте в настоящем документе представлена генетически модифицированная клетка-хозяин, способная продуцировать один или более стевиоловых гликозидов, где клетка-хозяин содержит гетерологичную нуклеиновую кислоту, кодирующую ABC-транспортер, аминокислотная последовательность которого имеет по меньшей мере 80% идентичности последовательности аминокислотной последовательности, выбранной из последовательностей со следующими идентификаторами: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID: 28, SEQ ID NO: 29 и SEQ ID NO: 30.
[0005] В одном варианте изобретения ABC-транспортер имеет аминокислотную последовательность, имеющую последовательность, выбранную из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID. NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30. В другом варианте осуществления генетически модифицированные клетки-хозяева по изобретению содержат нуклеиновые кислоты, кодирующие геранилгеранилпирофосфатсинтазу (GGPPS), энт-копалилпирофосфатсинтазу (CPS), энт-кауренсинтазу (KS), энт-каурен-19-оксидазу (КО), 13-гидроксилазу энт-кауреновой кислоты (КАН), цитохром р450 редуктазу (CPR) и одну или несколько УДФ-глюкозилтрансфераз (UGT). В дополнительном варианте осуществления одна или несколько УДФ-глюкозилтрансфераз (UGT) выбраны из EUGT11. UGT85C2, UGT74G1, UGT91D like3, UGT76G1 и UGT40087. В дополнительном варианте осуществления изобретения геранилгеранилпирофосфатсинтаза (GGPPS) имеет аминокислотную последовательность, имеющую по меньшей мере 80% идентичность последовательности с SEQ ID NO: 9, энт-копалилпирофосфатсинтаза (CPS) имеет аминокислотную последовательность, имеющую по меньшей мере 80% идентичности последовательности с SEQ ID NO: 10, энт-кауренсинтаза (KS) имеет аминокислотную последовательность, имеющую по меньшей мере 80% идентичность последовательности с SEQ ID NO: 11, энт-каурен-19-оксидаза (КО) имеет аминокислотная последовательность, имеющая по меньшей мере 80% идентичности последовательности с SEQ ID NO: 12, 13-гидроксилаза энт-кауреновой кислоты (КАН) имеет аминокислотную последовательность, имеющую по меньшей мере 80% идентичность последовательности с SEQ ID NO: 13, цитохром р450 редуктаза (CPR) имеет аминокислотную последовательность, имеющую по меньшей мере 80% идентичность последовательности с SEQ ID NO: 14, и одна или несколько УДФ-глюкозилтрансфераз (UGT) имеют аминокислотную последовательность, имеющую по меньшей мере 80% идентичность последовательности с аминокислотой последовательностью, выбранная из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19, SEQ ID NO: 27.
[0006] В конкретном варианте осуществления изобретения геранилгеранилпирофосфатсинтаза (GGPPS) имеет аминокислотную последовательность SEQ ID NO: 9, энт-копалилпирофосфатсинтаза (CPS) имеет аминокислотную последовательность SEQ ID NO: 10, энт-кауренсинтаза (KS) имеет аминокислотную последовательность SEQ ID NO: 11, энт-каурен-19-оксидаза (КО) содержит аминокислотную последовательность SEQ ID NO: 12, 13-гидроксилаза энт-кауреновой кислоты (КАН) содержит аминокислотную последовательность SEQ ID NO: 13, редуктаза цитохрома р450 (CPR) содержит аминокислотную последовательность SEQ ID NO: 14, и одна или несколько УДФ-глюкозилтрансфераз (UGT) содержат аминокислотную последовательность, выбранную из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19 и SEQ ID NO: 27.
[0007] В одном из вариантов осуществления клетка-хозяин выбрана из клетки бактерии, гриба, водоросли, насекомого или растения. В другом варианте осуществления клетка-хозяин представляет собой клетку Saccharomyces cerevisiae.
[0008] В одном из вариантов осуществления ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 1.
[0009] В другом варианте осуществления ABC-транс портер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 2.
[0010] В другом варианте осуществления ABC-транс портер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 3.
[ООН] В еще одном воплощении ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 4.
[0012] В дополнительном воплощении ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 5.
[0013] В другом варианте осуществления ABC-транс портер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 6.
[0014] В другом варианте осуществления ABC-транс портер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 7.
[0015] В еще одном воплощении ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 8.
[0016] В еще одном воплощении ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 28.
[0017] В еще одном воплощении ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 29.
[0018] В еще одном воплощении ABC-транспортер имеет аминокислотную последовательность с идентификатором SEQ ID NO: 30.
[0019] В варианте осуществления изобретения один или несколько стевиоловых гликозидов выбирают из ребаудиозида A (Reb А), ребаудиозида В (Reb В), Reb D. ребаудиозида Е (Reb Е) или Reb М. В другом варианте осуществления один или несколько стевиоловых гликозидов включают Reb М.
[0020] В одном варианте осуществления большая часть одного или нескольких стевиоловых гликозидов накапливается в просвете органеллы. В другом варианте осуществления большая часть одного или нескольких стевиоловых гликозидов накапливается внеклеточно.
[0021] В другом аспекте изобретение обеспечивает последовательность нуклеиновой кислоты кассеты экспрессии гетерологичной нуклеиновой кислоты, которая экспрессирует ABC-транспортер. В одном из вариантов осуществления нуклеотидная последовательность кассеты экспрессии гетерологичной нуклеиновой кислоты имеет кодирующую последовательность с идентификатором SEQ ID NO: 20, SEQ ID NO: 21, SEQ ID NO: 22, SEQ ID NO: 23, SEQ ID NO: 24, SEQ ID NO: 25, SEQ ID NO: 26 или SEQ ID NO: 27, где кодирующая последовательность функционально связана с гетер о логичным промотором.
[0022] В другом аспекте изобретение обеспечивает способ получения стевиола или одного или нескольких стевиоловых гликозидов, включающий: культивирование популяции клеток-хозяев по изобретению в среде с источником углерода в условиях, подходящих для получения стевиола или одного или нескольких стевиоловых гликозидов, чтобы дать культуральный бульон; и выделение стевиола или одного или нескольких стевиоловых гликозидов из культурального бульона.
[0023] В другом аспекте изобретение обеспечивает способ получения Reb D, включающий: культивирование популяции клеток-хозяев по изобретению в среде с источником углерода в условиях, подходящих для получения Reb D с получением культурального бульона; и выделение указанного соединения Reb D из культуральной жидкости.
[0024] В другом аспекте изобретение обеспечивает способ получения Reb М, включающий: культивирование популяции клеток-хозяев по изобретению в среде с источником углерода в условиях, подходящих для получения Reb М с получением культурального бульона; и выделение указанного соединения Reb М из культурального бульона.
5. КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
[0025] На Фиг. 1 схематически показан ферментативный путь от нативного дрожжевого метаболита фарнезилпирофосфата (FPP) до стевиола.
[0026] На Фиг. 2 схематически показан ферментативный путь от стевиола до Ребаудиозида М.
[0027] Фиг. 3 представляет собой схему ДНК-конструкции посадочной площадки, используемой для вставки транспортеров в штаммы Reb М. Каждый конец конструкции содержит последовательность ДНК длиной 500 п. и. ниже дрожжевого гена SFM1 для облегчения гомологичной рекомбинации в этом локусе. Введение посадочной площадки в этот локус не приводит к удалению какого-либо гена. Посадочная площадка содержит полноразмерный промотор GAL1, за которым следует сайт узнавания эндонуклеазы F-CphI и терминатор из природного дрожжевого гена НЕМ 13.
[0028] Фиг. 4 представляет собой график процентного содержания Reb D+Reb М, обнаруженного в супернатанте. Штаммы дрожжей с разными сверхэкспрессированными переносчиками выращивали в микротитровальных планшетах. Здесь проиллюстрирован процент Reb D+Reb M (измеренный в мкмолях), который обнаруживается в супернатанте после удаления клеток. Родительский штамм не содержит сверхэкспрессированного транспортера. Количество Reb D+Reb М, измеренное в супернатанте, делится на количество Reb D+Reb М, измеренное во всем клеточном бульоне, чтобы получить процентное содержание Reb D+Reb М в супернатанте.
[0029] Фиг. 5 представляет собой график общего содержания стевиоловых гликозидов по отношению к исходному в цельноклеточном бульоне. Штаммы дрожжей с разными сверхэкспрессированными переносчиками выращивали в микротитровальных планшетах. На этой иллюстрации представлена общая сумма всех стевиоловых гликозидов (измеренная в мкмолях), обнаруженная в цельноклеточном бульоне (как в клетках, так и в супернатанте), относительно родительского штамма. Родительский штамм не содержит сверхэкспрессированного транспортера.
[0030] Фиг. 6 представляет собой график количества Reb D+Reb М по отношению к исходному в цельноклеточном бульоне. Штаммы дрожжей с разными сверхэкспрессированными переносчиками выращивали в микротитровальных планшетах. На этой иллюстрации показана сумма Reb D+Reb М (измеренная в мкмолях), обнаруженная в бульоне целых клеток (как в клетках, так и в супернатанте), относительно родительского штамма. Родительский штамм не содержит сверхэкспрессированного транспортера.
[0031] Фиг. 7 представляет собой график общего количества стевиоловых гликозидов относительно родительского в супернатанте. Штаммы дрожжей с разными сверхэкспрессированными переносчиками выращивали в микротитровальных планшетах. На этой иллюстрации представлена общая сумма всех стевиоловых гликозидов (измеренная в мкмолях), обнаруженная в супернатанте после удаления клеток, относительно родительского штамма. Родительский штамм не содержит сверхэкспрессированного транспортера.
[0032] На Фиг. 8 показан процент всех продуцируемых стевиоловых гликозидов, находящихся в супернатанте. Штаммы дрожжей с разными сверхэкспрессированными переносчиками выращивали в микротитровальных планшетах. Эта цифра показывает процент всех стевиоловых гликозидов, продуцируемых клетками (измеренный в мкмолях), который обнаруживается в супернатанте. Суммарное количество стевиоловых гликозидов, измеренное в супернатанте, делится на количество общих стевиоловых гликозидов, измеренных во всем клеточном бульоне, для получения процента общих стевиоловых гликозидов в супернатанте.
[0033] Фиг. 9 представляет собой график количества Reb D+Reb М по отношению к исходному в цельноклеточном бульоне. Штаммы дрожжей, экспрессирующие версии ВРТ1 с метками ЗФБ и без меток и транспортера Т4 Fungal 5, выращивали в микротитровальных планшетах. Сравнивались относительные активности версий транспортеров с метками ЗФБ и без меток. Данные показывают, что версии с метками ЗФБ вели себя аналогично непомеченным версиям транспортеров.
[0034] Фиг. 10 представляет собой набор микрофотографий светлопольных (А) и флуоресцентных (В) изображений дрожжей, экспрессирующих ВРТ1 с метками ЗФБ.
[0035] Фиг. 11 представляет собой набор микрофотографий светлопольных (А) и флуоресцентных (В) изображений дрожжей, экспрессирующих транспортер Т4 Fungal 5 с метками ЗФБ.
[0036] Фиг. 12 представляет собой график количества Reb М относительно родительского с Т4 Fungal 5 дикого типа в цельноклеточном бульоне. Штаммы дрожжей, экспрессирующие транспортеры T4_Fungal_5 и его варианты (Isolate_l-8), полученные с помощью подверженной ошибкам ПЦР и отбора, выращивали в микротитровальных планшетах. На этой иллюстрации показан титр Reb М (измеренный в мкмолях), который обнаруживается в цельноклеточном бульоне (как в клетках, так и в супернатанте) штаммов дрожжей, экспрессирующих мутагенизированные варианты транспортера T4_Fungal_5 (Isolate_l-8) по сравнению с немутагенизированным T4_Fungal_5. Данные демонстрируют, что экспрессия Isolates 1-8 приводила к улучшенной продукции Reb М штаммами дрожжей по сравнению с Т4 Fungal 5.
[0037] Фиг. 13 представляет собой график доли Reb М общих стевиоловых гликозидов относительно родительского с T4_Fungal_5 дикого типа в цельноклеточном бульоне. Штаммы дрожжей, экспрессирующие транспортеры Т4 Fungal 5 и его варианты (Isolate 1-8), полученные с помощью подверженной ошибкам ПЦР и отбора, выращивали в микротитровальных планшетах. На этой иллюстрации показано отношение Reb М к общей сумме всех стевиоловых гликозидов (измеренной в мкмолях), которое обнаруживается в цельноклеточном бульоне (как в клетках, так и в супернатанте) штаммов дрожжей, экспрессирующих мутагенизированные варианты транспортера T4_Fungal_5 (Isolate_l-8) по сравнению с немутагенизированным Т4 Fungal 5. Данные демонстрируют, что экспрессия Isolates 1-8 приводила к увеличению доли Reb М среди всех стевиоловых гликозидов по сравнению с транспортером T4_Fungal_5. Другими словами, Isolates_l-8 демонстрируют повышенное предпочтение субстрата для Reb М.
[0038] Фиг. 14 представляет собой график количества Reb М в цельноклеточном бульоне и фракции супернатанта, продуцируемой штаммами, экспрессирующими транспортеры T4_Fungal_5 или Fungal_5_muA. Штаммы дрожжей, экспрессирующие Т4 Fungal 5 или Fungal 5 muA под контролем PGAL3 (более низкая сила, чем PGAL1), выращивали в микротитровальных планшетах. На этой иллюстрации показан титр Reb М (измеренный в мкмолях), который обнаруживается в цельноклеточном бульоне (как в клетках, так и в супернатанте) и фракции супернатанта штаммов дрожжей. Данные подтверждают, что Fungal 5 muA действительно обеспечивает улучшенную производительность при экспрессии в дрожжевом штамме: на 30% больше Reb М в цельноклеточном бульоне и на 40% больше внеклеточного Reb М продуцируется штаммом с Fungal_5_muA, чем штаммом с T4_Fungal_5 дикого типа, когда оба транспортера были выражены при более низкой силе промотора.
[0039] Фиг. 15 представляет собой график количества Reb М относительно родительского с Fungal 5 muA в бульоне из цельных клеток. Штаммы дрожжей, экспрессирующие транспортер Fungal_5_muA и восемь его вариантов, в которых одна, две или три мутации были возвращены к последовательности T4_Fungal_5 дикого типа, выращивали в микротитровальных планшетах. На этой иллюстрации показан титр Reb М (измеренный в мкмолях), который обнаруживается в бульоне целых клеток (как в клетках, так и в супернатанте) штаммов дрожжей, экспрессирующих восемь вариантов Fungal_5_muA относительно Fungal_5_muA. Данные демонстрируют влияние различных мутаций на продукцию Reb М, особенно интересен положительный эффект реверсии E1320V. [0040] Фиг. 16 представляет собой график общего количества стевиоловых гликозидов относительно родительского с Fungal_5_muA в цельноклеточном бульоне. Штаммы дрожжей, экспрессирующие транспортер Fungal_5_muA и восемь его вариантов, в которых одна, две или три мутации были возвращены к последовательности Т4 Fungal 5 дикого типа, выращивали в микротитровальных планшетах. На этой иллюстрации представлена общая сумма всех стевиоловых гликозидов (измеренная в мкмолях), обнаруженная в цельноклеточном бульоне (как в клетках, так и в супернатанте) штаммов дрожжей, экспрессирующих восемь вариантов Fungal_5_muA относительно Fungal_5_muA. Данные демонстрируют влияние различных мутаций на производство TSG. Вместе с ФИГ. 15 он иллюстрирует не только различия в активности, но и предпочтение субстратов.
6. ПОДРОБНОЕ ОПИСАНИЕ ВАРИАНТОВ ОСУЩЕСТВЛЕНИЯ 6.1 Терминология
[0041] Используемый здесь термин «гетерологичный» относится к тому, что обычно не встречается в природе. Термин «гетерологичная нуклеотидная последовательность» относится к нуклеотидной последовательности, которая обычно не встречается в данной клетке в природе. Как таковая гетерологичная нуклеотидная последовательность может быть: (а) чужеродной для своей клетки-хозяина (т.е. «экзогенной» для клетки); (b) естественным образом возникающая в клетке-хозяине (т.е. «эндогенно»), но присутствующая в клетке в неестественном количестве (т.е. в большем или меньшем количестве, чем в клетке-хозяине в естественных условиях); или (с) естественным образом возникающая в клетке-хозяине, но располагающаяся вне ее естественного локуса. [0042] С другой стороны, термин «нативный» или «эндогенный», используемый здесь в отношении молекул и, в частности, ферментов и нуклеиновых кислот, означает молекулы, которые экспрессируются в организме, в котором они возникли, или встречаются в природе. Понятно, что экспрессия природных ферментов или полинуклеотидов может быть изменена в рекомбинантных микроорганизмах.
[0043] Используемый здесь термин «кассета экспрессии гетерологичной нуклеиновой кислоты» относится к последовательности нуклеиновой кислоты, которая содержит кодирующую последовательность, функционально связанную с одним или несколькими регуляторными элементами, достаточными для экспрессии кодирующей последовательности в клетке-хозяине. В одном варианте осуществления «кассета экспрессии ABC-транспортера» относится к кассете экспрессии гетерологичной нуклеиновой кислоты, в которой гетерологичная нуклеиновая кислота содержит кодирующую последовательность для полипептида АВС-транспортера. Неограничивающие примеры регуляторных элементов включают промоторы, энхансеры, сайленсеры, терминаторы и сигналы поли-А.
[0044] Используемые здесь термины «ABC-транспортер» и «АТФ-связывающий кассетный транспортер» относятся к суперсемейству мембранно-ассоциированных полипептидов, которые связывают гидролиз аденозинтрифосфата (АТФ) с перемещением различных субстратов через биологические мембраны.
[0045] Используемый здесь термин относится к АВС-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 1):

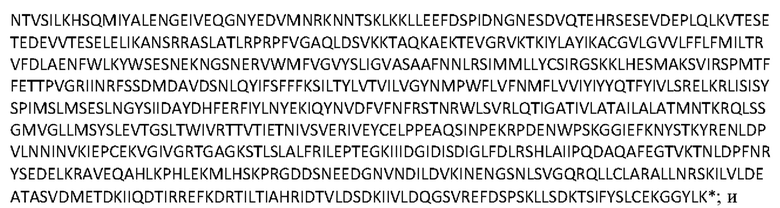
кодируется следующей последовательностью нуклеиновых кислот (идентификатор последовательности SEQ ID NO: 20):



[0046] Используемый здесь термин «T4 Fungal 1» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 2):
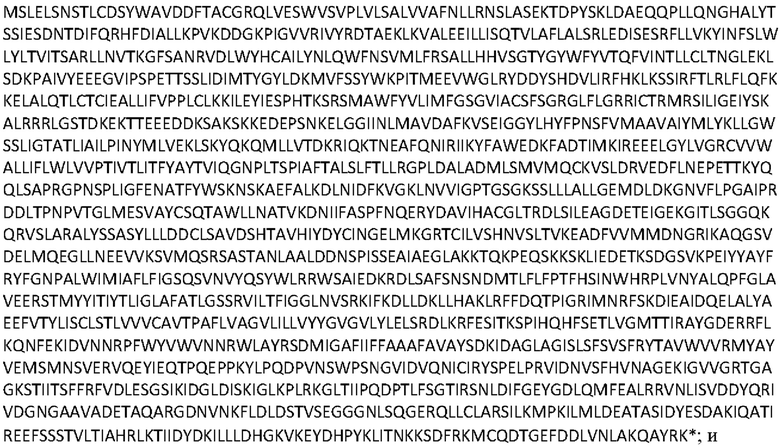
кодируется следующей последовательностью нуклеиновых кислот (идентификатор последовательности SEQ ID NO: 21):



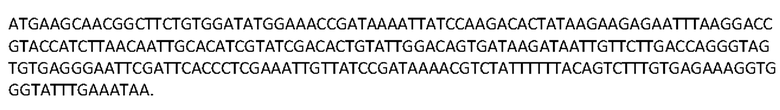
[0047] Используемый здесь термин «T4_Fungal_10» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 3):
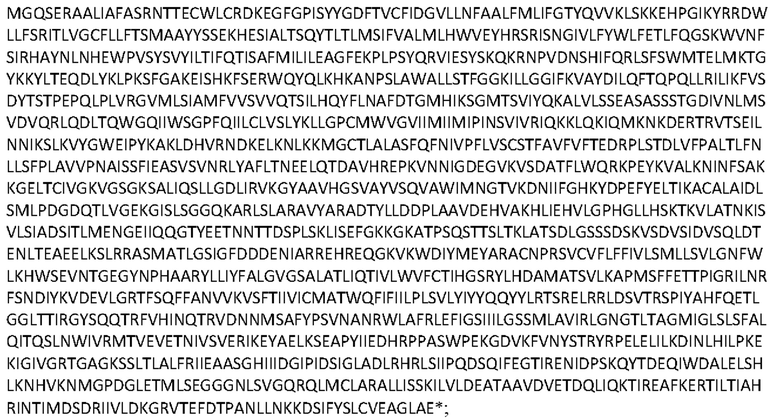
и кодируется следующей последовательностью нуклеиновых кислот (идентификатор последовательности SEQ ID NO: 22):


[0048] Используемый здесь термин «T4_Fungal_2» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 4):

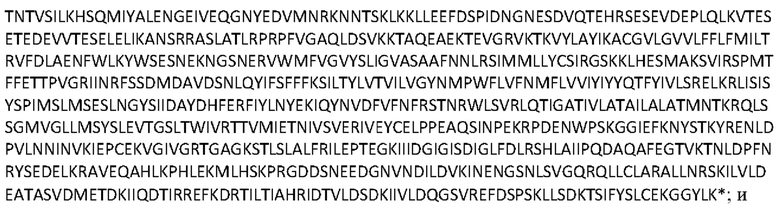
кодируется следующей последовательностью нуклеиновых кислот (идентификатор последовательности SEQ ID NO: 23):


[0049] Используемый здесь термин «T4 Fungal 3» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 5):





[0050] Используемый здесь термин «T4_Fungal_4» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 6):
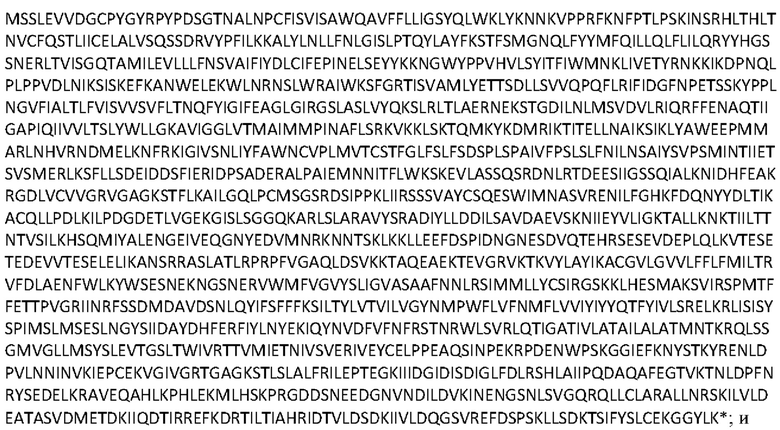
кодируется следующей последовательностью нуклеиновых кислот (идентификатор последовательности SEQ ID NO: 25):


[0051] Используемый здесь термин «T4_Fungal_5» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 7):


и кодируется следующей последовательностью нуклеиновых кислот (идентификатор последовательности SEQ ID NO: 26):


[0052] Используемый здесь термин «T4 Fungal 8» относится к ABC-транспортеру, имеющему следующую аминокислотную последовательность (SEQ ID NO: 8):





[0053] Используемый здесь термин «родительская клетка» относится к клетке, которая имеет идентичный генетический фон с генетически модифицированной клеткой-хозяином, описанной в данном документе, за исключением того, что она не содержит одну или несколько конкретных генетических модификаций, встроенных в модифицированную клетку-хозяин, например, одна или несколько модификаций, выбранных из группы, состоящей из: гетерологичной экспрессияифермента стевиолового пути, гетерологичной экспрессии фермента стевиолгликозидного пути, гетерологичной экспрессии геранилгеранилдифосфатсинтазы, гетерологичной экспрессии копалилдифосфатсинтазы, гетерологичной экспрессии каурен-синтазы, гетерологичной экспрессии кауреноксидазы (например, кауреноксидаза Pimm sativum), гетерологичной экспрессии стевиолсинтазы (гидроксилазы кауреновой кислоты), гетерологичной экспрессии редуктазы цитохрома Р450, гетерологичной экспрессии EUGT11, гетерологичной экспрессии UGT74G1, гетерологичной экспрессии UGT76G1, гетерологичной экспрессии UGT85C2, гетерологичной экспрессии UGT91D и гетерологичной экспрессии UGT40087 или его варианта.
[0054] Используемый здесь термин «встречающееся в природе» относится к тому, что встречается в природе. Например, ABC-транспортер, присутствующий в организме, который может быть выделен из источника в природе и который не был намеренно модифицирован человеком в лаборатории, является естественным ABC-транспортером. И наоборот, в контексте настоящего описания термин «не встречающийся в природе» относится к тому, что не встречается в природе, но создается в результате вмешательства человека.
[0055] Термин «среда» относится к культуральной среде и/или среде ферментации. [0056] Термин «ферментационная композиция» относится к композиции, которая включает генетически модифицированные клетки-хозяева и продукты или метаболиты, продуцируемые генетически модифицированными клетками-хозяевами. Примером ферментационной композиции является бульон для цельных клеток, который может представлять собой все содержимое сосуда (например, колбы, тарелки или ферментатора), включая клетки, водную фазу и соединения, полученные из генетически модифицированных клеток-хозяев.
[0057] Используемый здесь термин «продукция» обычно относится к количеству стевиола или стевиолгликозида, продуцируемого генетически модифицированной клеткой-хозяином, предоставленной в данном документе. В некоторых вариантах осуществления продукция выражается как выход стевиола или стевиолгликозида клеткой-хозяином. В других вариантах осуществления продукция выражается как продуктивность клетки-хозяина по продуцированию стевиола или стевиолгликозида.
[0058] Используемый здесь термин «продуктивность» относится к продукции стевиола или стевиолгликозида клеткой-хозяином, выраженной как количество продуцируемого стевиола или стевиолгликозида (по массе) на количество ферментационного бульона, в котором культивируется клетка-хозяин (по объему) с течением времени (в час). [0059] Используемый здесь термин «выход» относится к продукции стевиола или стевиолгликозида клеткой-хозяином, выраженной как количество продуцируемого стевиола или стевиолгликозида на количество источника углерода, потребляемого клеткой-хозяином, по массе.
[0060] Используемый здесь термин «неопределяемый уровень» соединения (например, Reb М, стевиоловых гликозидов или других соединений) означает уровень соединения, который слишком низок для измерения и/или анализа стандартным методом измерения соединение. Например, термин включает уровень соединения, который не определяется аналитическими методами, известными в данной области.
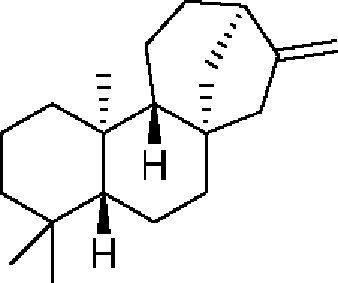
[0061] Термин «каурен» относится к соединению каурена, включая любой стереоизомер каурена. В конкретных вариантах осуществления термин относится к энантиомеру, известному в данной области как энт-каурен. В конкретных вариантах осуществления термин относится к соединению согласно следующей структуре:
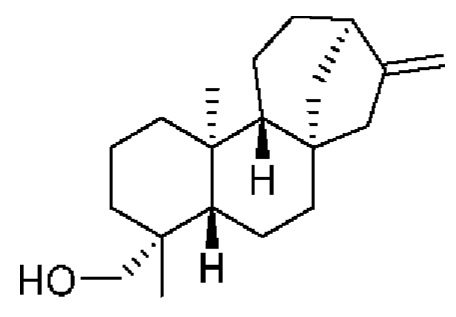
[0062] Термин «кауренол» относится к соединению кауренол, включая любой стереоизомер кауренола. В конкретных вариантах осуществления термин относится к энантиомеру, известному в данной области как энт-кауренол. В конкретных вариантах осуществления термин относится к соединению согласно следующей структуре.
[0063] Термин «кауренал» относится к составному кауреналу, включая любой стереоизомер кауренала. В конкретных вариантах осуществления термин относится к энантиомеру, известному в данной области как энт-кауренал. В конкретных вариантах осуществления термин относится к соединению согласно следующей структуре.
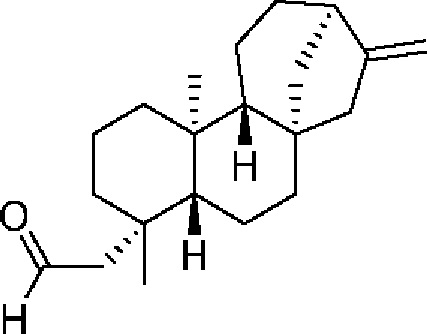
[0064] Термин «кауреновая кислота» относится к соединению кауреновой кислоты, включая любой стереоизомер кауреновой кислоты. В конкретных вариантах осуществления термин относится к энантиомеру, известному в данной области как энт-кауреновая кислота. В конкретных вариантах осуществления термин относится к соединению согласно следующей структуре.
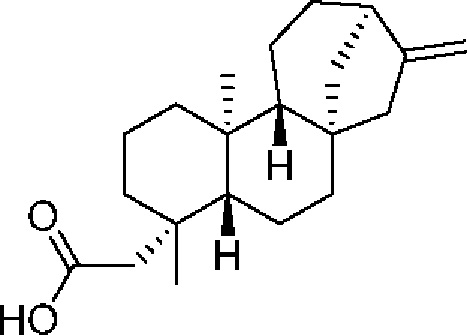
[0065] Термин «стевиол» относится к соединению стевиола, включая любой стереоизомер стевиола. В конкретных вариантах осуществления термин относится к соединению согласно следующей структуре.
ОН
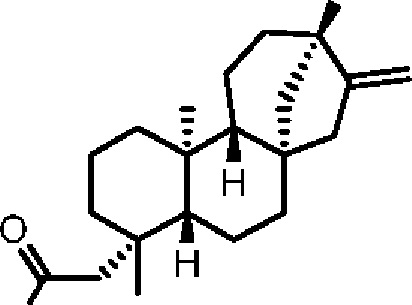
но
[0066] Используемый здесь термин «стевиоловый гликозид(ы)» относится к гликозиду стевиола, включая, но не ограничиваясь ими, природные стевиоловые гликозиды, например, стевиолмонозид, стевиолбиозид, рубузозид, дулкозид В, дулкозид А, ребаудиозид В, ребаудиозид G, стевиозид, ребаудиозид С, ребаудиозид F, ребаудиозид А, ребаудиозид I, ребаудиозид Е, ребаудиозид Н, ребаудиозид L, ребаудиозид К, ребаудиозид J, ребаудиозид М, ребаудиозид D, ребаудиозид N, ребаудиозид О, синтетические стевиоловые гликозиды, например, ферментативно стевиолгликозиды гликозиды и их комбинации.
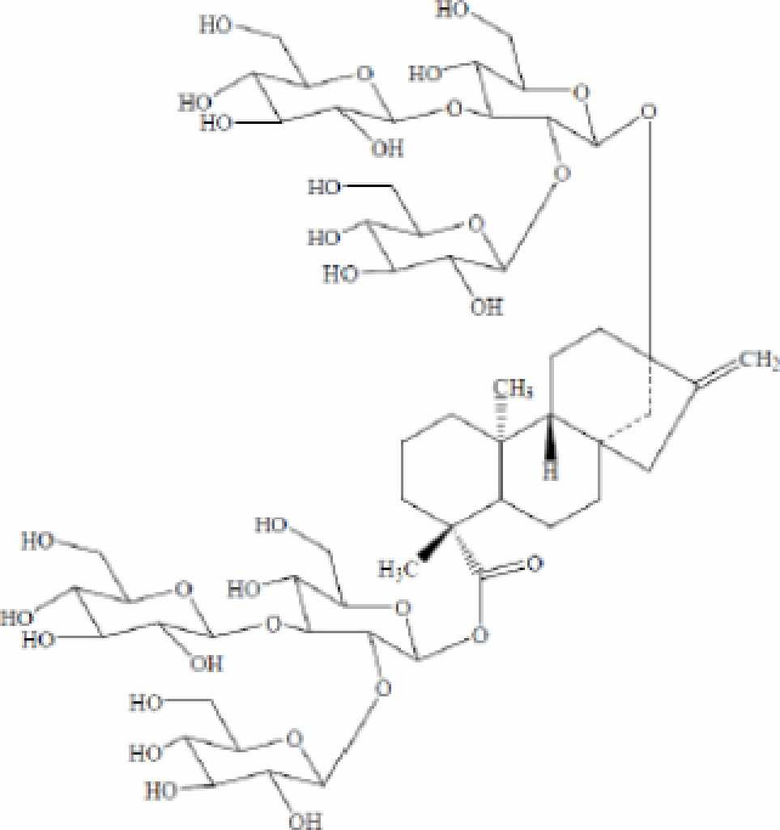
[0067] Используемый здесь термин «Ребаудиозид М» относится к соединению следующей структуры.
[0068] Используемый здесь термин «вариант» относится к полипептиду, отличающемуся от специально указанного «эталонного» полипептида (например, последовательности дикого типа) вставками, делениями, мутациями и/или заменами аминокислот, но сохраняет активность, которая по существу аналогичен эталонному полипептиду. В некоторых вариантах осуществления вариант создается методами рекомбинантной ДНК или мутагенезом. В некоторых вариантах осуществления вариантный полипептид отличается от своего эталонного полипептида заменой одного основного остатка на другой (например, Arg на Lys), заменой одного гидрофобного остатка на другой (например, Leu на Не) или заменой одного ароматического остатка для другого (например, Phe для Туг) и т.д. В некоторых вариантах осуществления варианты включают аналоги, в которых получены консервативные замены, приводящие к значительной структурной аналогии эталонной последовательности. Примеры таких консервативных замен, без ограничения, включают глутаминовую кислоту вместо аспарагиновой кислоты и наоборот, глугамин для аспарагина и наоборот, серии на треонин и наоборот, лизин на аргинин и наоборот, или любой из изолейцина, валина или лейцина друг для друга.
[0069] Используемый здесь термин«идентичность последовательностей» или «процент идентичности» в контексте двух или более последовательностей нуклеиновых кислот или белков относится к двум или более последовательностям или подпоследовательностям, которые являются одинаковыми или имеют определенный процент аминокислотных остатков или одинаковых нуклеотидов. Например, последовательность может иметь процентную идентичность по меньшей мере 60%, по меньшей мере 65%, по меньшей мере 70%, по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более высокую идентичность в указанной области для ссылки последовательность при сравнении и выравнивании для максимального соответствия в окне сравнения или обозначенной области при измерении с использованием алгоритма сравнения последовательностей или путем ручного выравнивания и визуального контроля. Например, процент идентичности определяется путем вычисления отношения количества идентичных нуклеотидов (или аминокислотных остатков) в последовательности, деленного на длину всех нуклеотидов (или аминокислотных остатков) за вычетом длин любых пробелов.
[0070] Для удобства степень идентичности между двумя последовательностями может быть установлена с использованием компьютерных программ и математических алгоритмов, известных в данной области техники. Такие алгоритмы, которые вычисляют процент идентичности последовательностей, обычно учитывают пробелы и несовпадения последовательностей в сравниваемой области. Для этой цели хорошо подходят программы, которые сравнивают и выравнивают последовательности, такие как Clustal W (Thompson et al, (1994) Nucleic Acids Res., 22: 4673-4680), ALIGN (Myers et al, (1988) CABIOS, 4: 11-17), FASTA (Pearson et al, (1988) PNAS, 85: 2444-2448; Pearson (1990), Methods Enzymol, 183: 63-98) и BLAST с пробелами (Altschul et al, (1997) Nucleic Acids Res., 25: 3389-3402). BLAST или BLAST 2.0 (Altschul et al, J. Mot. Biol 215: 403-10, 1990) доступны из нескольких источников, включая Национальный центр биологической информации (NCBI) и в Интернете, для использования в сочетании с программами анализа последовательностей BLASTP, BLASTN, BLASTX, TBLASTN и TBLASTX. Дополнительную информацию можно найти на веб-сайте NCBI.
[0071] В некоторых вариантах осуществления выравнивание последовательностей и расчеты процента идентичности могут быть определены с использованием программы BLAST с использованием ее стандартных параметров по умолчанию. Для выравнивания нуклеотидных последовательностей и расчетов идентичности последовательностей программа BLASTN используется со своими параметрами по умолчанию (штраф за открытие пробела=5, штраф за расширение пробела=2, нуклеиновое совпадение=2, нуклеиновое несоответствие=-3, ожидаемое значение=10.0, размер слова=11, максимальное количество совпадений в диапазоне запроса=0). Для выравнивания полипептидных последовательностей и расчетов идентичности последовательностей программа BLASTP используется с параметрами по умолчанию (Матрица выравнивания=BLOSUM62; Стоимость пробелов: Существование=11, Расширение=1; Композиционные корректировки=Условная оценка композиции, корректировка матрицы; Ожидаемое значение=10,0; Размер слова=6; Максимальное количество совпадений в диапазоне запроса=0). В качестве альтернативы можно использовать следующую программу и параметры: программное обеспечение Align Plus из Clone Manager Suite, версия 5 (Sci-Ed Software); Сравнение ДНК: глобальное сравнение, стандартная линейная матрица оценок, штраф за несоответствие=2, штраф за открытые пропуски=4, штраф за расширение пропусков=1. Сравнение аминокислот: Глобальное сравнение, Матрица оценок BLOSUM 62. В вариантах осуществления, описанных в данном документе, идентичность последовательности вычисляется с использованием программ BLASTN или BLASTP с использованием их параметров по умолчанию. В вариантах осуществления, описанных в данном документе, выравнивание последовательностей двух или более последовательностей выполняется с использованием Clustal W с использованием предложенных параметров по умолчанию (Отмена выравнивания входных последовательностей: нет; Mbed-подобное дерево направляющих кластеров: да; Mbed-подобная итерация кластеризации: да; число объединенных итераций: по умолчанию (0): Максимальное количество итераций направляющего дерева: по умолчанию; Максимальное количество итераций НММ: по умолчанию; Порядок: ввод).
6.2 ABC-транспортер, нуклеиновые кислоты, кассеты экспрессии и клетки-хозяева
[0072] В одном аспекте в настоящем документе представлены рекомбинантные нуклеиновые кислоты, которые экспрессируют ABC-транспортеры. АВС-транспортеры согласно изобретению могут быть идентифицированы поиском на основе последовательностей в отношении последовательностей известных АВС-транспортеров. Примерная база данных последовательностей известных АВС-транспортеров предоставлена (Ковальчук и Дриссен, Филогенетический анализ грибковых АВС-транспортеров (Kovalchuk and Driessen, Phylogenetic Analysis of Fungal ABC Transporters), BMC Genomics, 2010, 11:177). Базы данных АВС-транспортеров BLAST также могут быть созданы из дополнительных организмов. В предпочтительных вариантах осуществления базы данных грибковых последовательностей из (1) Hansenula polymorpha DL-1 (NRRL-Y-7560), (2) Yarrowia lipolytica ATCC 18945, (3) Arxula adeninivorans ATCC 76597, (4) S. cerevisiae CAT-1, (5) Lipomyces starkeyi ATCC 58690, (6) Kluyveromyces marxianus, (7) Kluyveromyces marxianus DMKU3-1042, (8) Komagataella phaffii NRRL Y-11430, (9) S. cerevisiae MBG3370, (10) S. cerevisiae MBG3373, (11) K. lactis ATCC 8585, (12) Candida utilis ATCC 22023, (13) Pichia pastoris ATCC 28485 и (14) Aspergillus oryzae NRRL5590 служат в качестве источников АВС-транспортеров изобретения.
[0073] Нуклеотидные последовательности ORF, полученные в результате геномного секвенирования de novo, сборки и аннотации различных организмов, анализируют с помощью алгоритма tblastn с использованием Biopython или любого другого подходящего программного обеспечения для анализа последовательностей. Алгоритм tblastn обеспечивает выравнивание белковых последовательностей известных АВС-транспортеров с транслированной ДНК нуклеотидных последовательностей ORF для каждого организма во всех 6 возможных рамках считывания с использованием BLAST. Примерные параметры BLAST являются стандартными с evalue=1е-25 (таблицы 4 и 5). Совпадения могут быть впоследствии отфильтрованы, чтобы обеспечить глобальное выравнивание не менее 2000 нуклеотидов.
[0074] В других вариантах осуществления изобретения весь протеом организма может быть извлечен из Uniprot с помощью Uniprot API, чтобы создать базу данных для поиска BLAST. Алгоритм blastp может применяться к базе данных, производной от Uniprot. В одном варианте осуществления параметры BLAST могут быть стандартными с evalue=0,001. В конкретных вариантах осуществления фильтрация может выполняться на основе процентного отсечения идентичности>40% и процентного отсечения выровненной длины>60%. В предпочтительных вариантах осуществления совпадения должны соответствовать по меньшей мере одной из 610 исходных последовательностей из ссылки.
[0075] После идентификации нуклеотидных последовательностей можно разработать праймеры для амплификации каждой полной ORF, амплифицированной с помощью ПЦР. Каждый праймер ПЦР в идеале должен иметь фланкирующую гомологию с последовательностями промотора и терминатора ДНК промотора и терминатора, используемых в кассете гетерологичной нуклеотидной экспрессии, добавленной к концам для облегчения гомологичной рекомбинации амплифицированного гена в сайт-мишень посадочной площадки для получения специфической кассеты экспрессии АВС-транспортера. Каждый ген ABC-транспортера может быть индивидуально трансформирован в виде единственной копии в родительский штамм дрожжей Reb М, описанный в данном документе, и подвергнут скринингу на способность увеличивать титры продукта при сверхэкспрессии in vivo.
[0076] В некоторых вариантах осуществления рекомбинантные нуклеиновые кислоты кодируют полипептид, аминокислотная последовательность которого представлена любой из последовательностей с идентификатором SEQ ID NO: 1-8. В некоторых вариантах осуществления рекомбинантная нуклеиновая кислота содержит нуклеотидную последовательность, представленную любой из последовательностей с идентификатором SEQ Ш NO: 20-27.
[0077] В настоящем документе также представлены клетки-хозяева, содержащие один или несколько полипептидов или нуклеиновых кислот АВС-транспортера, представленных в настоящем документе, которые способны продуцировать стевиоловые гликозиды. В некоторых вариантах осуществления клетки-хозяева могут продуцировать стевиоловые гликозиды из источника углерода в культуральной среде. В конкретных вариантах осуществления клетки-хозяева могут продуцировать стевиол из источника углерода в культуральной среде и могут дополнительно продуцировать Reb А или Reb D из стевиола. В конкретных вариантах осуществления клетки-хозяева могут дополнительно продуцировать Reb М из Reb D. В конкретных вариантах осуществления Reb D и/или Reb М транспортируются в просвет одной или нескольких органелл. В конкретных вариантах осуществления Reb D и/или Reb М транспортируются во внеклеточное пространство (т.е. супернатант).
[0078] В некоторых вариантах осуществления клетки-хозяева, экспрессирующие АВС-транспортеры в соответствии с вышеуказанными вариантами осуществления, продуцируют не менее 20%, не менее 30%, не менее 40%, не менее 50%, не менее 60%, не менее 70%, не менее 80%, по крайней мере, на 90% или, по крайней мере, на 100% больше общего стевиол гликозида (TSG) по сравнению с родительской клеткой-хозяином, в которой отсутствует кассета экспрессии АВС-транспортера.
[0079] В некоторых вариантах осуществления клетки-хозяева, экспрессирующие АВС-транспортеры в соответствии с вышеуказанными вариантами осуществления, продуцируют по меньшей мере на 20%, по меньшей мере на 30%, по меньшей мере на 40%, по меньшей мере на 50%, по меньшей мере на 60%, по меньшей мере на 70% или по меньшей мере на 75% больше TSG в супернатанте по сравнению с родительской клеткой-хозяином, в которой отсутствует кассета экспрессии АВС-транспортера. В конкретном варианте осуществления клетки-хозяева, экспрессирующие ABC-транспортеры в соответствии с вышеуказанными вариантами осуществления, продуцируют по меньшей мере в 2 раза, по меньшей мере в 3 раза, по меньшей мере в 4 или по меньшей мере в 5 раз больше TSG в супернатанте по сравнению с родительской клеткой-хозяином, в которой отсутствует кассета экспрессии АВС-транспортера.
[0080] В предпочтительных вариантах осуществления клетка-хозяин может включать один или несколько ферментативных путей, способных производить кауреновую кислоту, указанные пути взяты по отдельности или вместе. Как описано в данном документе, клетки-хозяева содержат гидроксилазу кауреновой кислоты Stevia rebaudiana, представленную в настоящем документе, способную превращать кауреновую кислоту в стевиол. В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать фарнезилдифосфат в геранилгеранилдифосфат.В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать геранилгеранилдифосфат в копалилдифосфат.В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать копалилдифосфат в каурен. В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать каурен в кауреновую кислоту. В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать стевиол в один или несколько гликозидов стевиола. В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один, два, три, четыре или более ферментов, вместе способных превращать стевиол в Reb А. В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать Reb А в Reb D. В некоторых вариантах осуществления клетка-хозяин дополнительно содержит один или несколько ферментов, способных превращать Reb D в Reb М. Полезные ферменты и нуклеиновые кислоты, кодирующие ферменты, известны специалистам. Особенно полезные ферменты и нуклеиновые кислоты описаны в разделах ниже и дополнительно описаны, например, в US 2014/0329281 Al, US 2014/0357588 Al, US 2015/0159188, WO 2016/038095 A2 и US 2016/0198748 Al.
[0081] В дополнительных вариантах осуществления клетки-хозяева дополнительно содержат один или несколько ферментов, способных производить геранилгеранилдифосфат из источника углерода. К ним относятся ферменты каскада реакций DXP и ферменты каскада реакций MEV. Полезные ферменты и нуклеиновые кислоты, кодирующие ферменты, известны специалистам в данной области. Примеры ферментов каждого пути описаны ниже и дополнительно описаны, например, в US 2016/0177341 Al, который полностью включен в настоящий документ посредством ссылки.
[0082] В некоторых вариантах осуществления изобретения клетки-хозяева включают один или несколько или все ферменты изопреноидного пути, выбранные из группы, состоящей из: (а) фермента, который конденсирует две молекулы ацетил-коэнзима А с образованием ацетоацетил-КоА (например, тиолаза ацетил-КоА); (b) фермента, который конденсирует ацетоацетил-КоА с другой молекулой ацетил-КоА с образованием 3-гидрокси-3-метилглутарил-КоА (ГМГ-КоА) (например, синтаза ГМГ-КоА); (с) фермент, превращающий HMG-CoA в мевалонат (например, HMG-CoA редуктаза); (d) фермент, превращающий мевалонат в мевалонат-5-фосфат (например, мевалонат-киназа); (е) фермент, превращающий мевалонат-5-фосфат в мевалонат-5-пирофосфат (например, фосфомевалонат киназа); (f) фермент, превращающий мевалонат 5-пирофосфат в изопентенил дифосфат (IPP) (например, мевалонат пирофосфат декарбоксилаза); (g) фермент, который превращает IPP в диметилаллил пирофосфат (DMAPP) (например, изомераза IPP); (h) полипренил синтаза, которая может конденсировать молекулы IPP и/или DMAPP с образованием полипрениловых соединений, содержащих более пяти углеродов; (i) фермент, который конденсирует IPP с DMAPP с образованием геранил пирофосфата (GPP) (например, синтаза GPP); (j) фермент, который конденсирует две молекулы IPP с одной молекулой DMAPP (например, синтаза FPP); (к) фермент, который конденсирует IPP с GPP с образованием фарнезилпирофосфата (FPP) (например, FPP-синтаза); (1) фермент, который конденсирует IPP и DMAPP с образованием геранилгеранилпирофосфата (GGPP); и (т) фермент, который конденсирует IPP и FPP с образованием GGPP. [0083] В некоторых вариантах осуществления дополнительные ферменты являются природными. В предпочтительных вариантах осуществления дополнительные ферменты гетерологичны. В некоторых вариантах осуществления два или более ферментов могут быть объединены в один полипептид.
6.3 Клеточные штаммы
[0084] Предлагаемые здесь подходящие композиции и способы для клеток-хозяев включают клетки архей, прокариот или эукариот.
[0085] Подходящие прокариотические хозяева включают, без ограничения, любые из множества грамположительных, грамотрицательных или грамположительных бактерий. Примеры включают, но не ограничиваются ими, клетки, принадлежащие к родам: Agrobacterium, Alicyclobacillus, Anabaena, Anacystis, Arthrobacter, Azobacter, Bacillus. В revi bacterium, Chromatium, Clostridium, Cory neb acterium, Enterobacter, Erwinia, Escheracillobacterium, Lactobactobacter, Erwinia, Escheracillobacterium, Lactobacterium, Microbacterium, Phormidium, Pseudomonas, Rhodobacter, Rhodopseudomonas, Rhodospirillum, Rhodococcus, Salmonella, Scenedesmun, Serratia, Shigella, Staphlococcus, Strepromyces, Synnecoccus и Zymomonas. Примеры прокариотических штаммов включают в себя, но не ограничиваются ими: Bacillus subtilis, Bacillus amyloliquefacines, Br evib acterium ammoniagenes, Brevibacterium immariophilum, Clostridium beigerinckii, Enterobacter sakazakii, Escherichia coli, Lactococcus lactis, Mesorhizobium loti, Pseudomonas aeruginosa, Pseudomonas mevalonii, Pseudomonas pudica, Rhodobacter capsulatus, Rhodobacter sphaeroides, Rhodospirillum rubrum, Salmonella enterica, Salmonella typhi, Salmonella typhimurium, Shigella dysenteriae, Shigella flexneri, Shigella sonnei и Staphylococcus aureus. В конкретном варианте осуществления клетка-хозяин представляет собой клетку Escherichia coli.
[0086] Подходящие хозяева архей включают, но не ограничиваются ими, клетки, принадлежащие к родам: Aeropyrum, Archaeglobus, Halobacterium, Methanococcus, Methanobacterium, Pyrococcus, Sulfolobus и Thermoplasma. Примеры штаммов архей включают, но не ограничиваются ими: Archaeoglobus fulgidus, Halobacterium sp., Methanococcus jannaschii, Methanobacterium thermoautotrophicum, Thermoplasma acidophilum, Thermoplasma volcanium, Pyrococcus horikoshii, Pyrococcus abyssi и Aeropyrum pernix.
[0087] Подходящие эукариотические хозяева включают, но не ограничиваются ими, клетки грибов, клетки водорослей, клетки насекомых и клетки растений. В некоторых вариантах осуществления дрожжи, используемые в настоящих способах, включают дрожжи, депонированные в хранилищах микроорганизмов (например, IFO, АТСС и т.д.) и принадлежащие к родам Aciculoconidium, Ambrosiozyma, Arthroascus, Arxiozyma, Ashbya, Babjevia, Bensingtonia, Botryoascus, Botryozyma, Brettanomyces, Bullera, Bulleromyces, Candida, Citeromyces, Clavispora, Cryptococcus, Cystofilobasidium, Debaryomyces, Dekkara, Dipodascopsis, Dipodascus, Eeniella, Endomycopsella, Eremascus, Eremothecium, Erythrobasidium, Fellomyces, Filobasidium, Galactomyces, Geotrichum, Guilliermondella, Hanseniaspora, Hansenula, Hasegawaea, Holtermannia, Hormoascus, Hyphopichia, Issatchenkia, Kloeckera, Kloeckeraspora, Kluyveromyces, Kondoa, Kuraishia, Kurtzmanomyces, Leucosporidium, Lipomyces, Lodderomyces, Malassezia, Metschnikowia, Mrakia, Myxozyma, Nadsonia, Nakazawaea, Nematospora, Ogataea, Oosporidium, Pachysolen, Phachytichospora, Phaffia, Pichia, Rhodosporidium, Rhodotorula, Saccharomyces, Saccharomycodes, Saccharomycopsis, Saitoella, Sakaguchia, Saturnospora, Schizoblastosporion, Schizosaccharomyces, Schwanniomyces, Sporidiobolus, Sporobolomyces, Sporopachydermia, Stephanoascus, Sterigmatomyces, Sterigmatosporidium, Symbiotaphrina, Sympodiomyces, Sympodiomycopsis, Torulaspora, Trichosporiella, Trichosporon, Trigonopsis, Tsuchiyaea, Udeniomyces, Waltomyces, Wickerhamia, Wickerhamiella, Williopsis, Yamadazyma, Yarrowia, Zygoascus, Zygosaccharomyces, Zygowilliopsis и Zygozyma, среди других.
[0088] В некоторых вариантах осуществления изобретения микроорганизм-хозяин представляет собой Saccharomyces cerevisiae, Pichia pastoris, Schizosaccharomyces pombe, Dekkera bruxellensis, Kluyveromyces lactis (ранее называвшийся Saccharomyces lactis), Kluveromyces marxianus, Arxula adeninustula polymorus (теперь известный как Pichiaa angienusa polymoris) или Pichia adeninustula polymorns. В некоторых вариантах осуществления изобретения микроорганизм-хозяин представляет собой штамм рода Candida, такой как Candida lipolytica, Candida guilliermondii, Candida krusei, Candida pseudotropicalis или Candida utilis.
[0089] В конкретном варианте осуществления микроорганизмом-хозяином является Saccharomyces cerevisiae. В некоторых вариантах осуществления изобретения хозяин представляет собой штамм Saccharomyces cerevisiae, выбранный из группы, состоящей из пекарских дрожжей, CBS 7959, CBS 7960, CBS 7961, CBS 7962, CBS 7963, CBS 7964, IZ-1904, ТА, BG-1, CR-1, SA-1, М-26, Y-904, РЕ-2, РЕ-5, VR-1, BR-1, BR-2, МЕ-2, VR-2, МА-3, МА-4, САТ-1, СВ-1, NR-1, ВТ-1 и AL-1. В некоторых вариантах осуществления микроорганизм-хозяин представляет собой штамм Saccharomyces cerevisiae, выбранный из группы, состоящей из РЕ-2, САТ-1, VR-1, BG-1, CR-1 и SA-1. В конкретном варианте осуществления штамм Saccharomyces cerevisiae представляет собой РЕ-2. В другом конкретном варианте осуществления штамм Saccharomyces cerevisiae представляет собой САТ-1. В другом конкретном варианте осуществления штамм Saccharomyces cerevisiae представляет собой BG-1.
[0090] В некоторых вариантах осуществления микроорганизм-хозяин представляет собой микроорганизм, который подходит для промышленной ферментации. В конкретных вариантах осуществления микроорганизм кондиционируется для существования в условиях высокой концентрации растворителя, высокой температуры, расширенного использования субстрата, ограничения питательных веществ, осмотического стресса из-за сахара и солей, кислотности, сульфита и бактериального загрязнения или их комбинаций, которые считаются стрессовыми условиями промышленная ферментационная среда.
6.4 Пути биосинтеза стевиола и стевиол гликозидов
[0091] В некоторых вариантах осуществления путь биосинтеза стевиола и/или путь биосинтеза стевиоловых гликозидов активируется в генетически модифицированных клетках-хозяевах, представленных в настоящем документе, путем инженерии клеток для экспрессии полинуклеотидов и/или полипептидов, кодирующих один или несколько ферментов этого пути. ФИГ. 1 иллюстрирует примерный путь биосинтеза стевиола.
[0092] Таким образом, в некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью геранилгеранилдифосфатсинтазы (GGPPS). В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью копалилдифосфатсинтазы или эит-копалилпирофосфатсинтазы (CDPS; также называемый энт-копалилпирофосфатсинтазой или CPS). В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью кауренсинтазы (KS; также называемой энт-кауренсинтазой). В конкретных вариантах осуществления генетически модифицированные клетки-хозяева, представленные в настоящем документе, содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий кауреноксидазной активностью (КО; также называемый энт-каурен-19-оксидазой), как описано в настоящем документе. В конкретных вариантах осуществления генетически модифицированные клетки-хозяева, представленные в настоящем документе, содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью полипептида гидроксилазы кауреновой кислоты (КАН; также называемый стевиолсинтазой), в соответствии с вариантами осуществления, представленными в настоящем документе. В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью цитохром Р450 редуктазы (CPR).
[0093] В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью UGT74G1. В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью UGT76G1. В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью UGT85C2. В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью UGT91D. В некоторых вариантах осуществления предложенные здесь генетически модифицированные клетки-хозяева содержат гетерологичный полинуклеотид, кодирующий полипептид, обладающий активностью UGTad. Как описано ниже, UGTad относится к уридиндифосфат-зависимой гликозилтрансферазе, способной переносить фрагмент глюкозы в положение С-2' 19-О-глюкозы Reb А с образованием Reb D.
[0094] В некоторых вариантах осуществления клетка-хозяин содержит вариантный фермент.В некоторых вариантах осуществления вариант может содержать до 15, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 аминокислотных замен относительно соответствующего полипептида. В некоторых вариантах осуществления вариант может содержать до 15, 10, 9, 8, 7, 6, 5, 4, 3, 2 или 1 консервативных аминокислотных замен относительно эталонного полипептида. В некоторых вариантах осуществления любая из описанных здесь нуклеиновых кислот может быть оптимизирована для клетки-хозяина, например, оптимизирована кодонами.
[0095] Примеры нуклеиновых кислот и ферментов пути биосинтеза стевиола и/или пути биосинтеза стевиоловых гликозидов описаны ниже.
6.4.1 Геранилгеранилдифосфатсинтаза (GGPPS)
[0096] Геранилгеранилдифосфатсинтазы (ЕС 2.5.1.29) катализируют превращение фарнезилпирофосфата в геранилгеранилдифосфат.Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (учетный номер ABD92926), Gibberella fujikuroi (учетный номер САА75568), Mus musculus (учетный номер ААН69913), Thalassiosira pseudonana (учетный номер ХР 002288339), Streptomyces clavuligerus (учетный номер ZP_05004570), Sulfiilobus acidocaldarius (учетный номер ВАА43200), Synechococcus sp.(учетный номер АВС98596), Arabidopsis thaliana (учетный номер NP 195399) и Blakeslea trispora (учетный номер AFC92798.1) и описанные в US 2014/0329281 Al. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот GGPPS. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов GGPPS.
6.4.2 Копалилдифосфатсинтаза (CDPS)
[0097] Копалилдифосфатсинтазы (ЕС 5.5.1.13) катализируют превращение геранилгеранилдифосфата в копалилдифосфат.Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (учетный номер ААВ87091), Streptomyces clavuligerus (учетный номер EDY51667), Bradyrhizobium japonicum (учетный номер ААС28895.1), Zea mays (учетный номер AY562490), Arabidopsis thaliana (учетный номер NM 116512) и Oryza sativa (учетный номер Q5MQ85.1) и описанные в US 2014/0329281 Al. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот CDPS. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов CDPS.
6.4.3 Каурен-синтаза (KS)
[0098] Каурен-синтазы (ЕС 4.2.3.19) катализируют превращение копалилдифосфата в каурен и дифосфат.Иллюстративные примеры ферментов включают ферменты Bradyrhizobium japonicum (учетный номер ААС28895.1), Phaeosphaeria sp. (учетный номер 013284), Arabidopsis thaliana (учетный номер Q9SAK2) и Picea glauca (учетный номер ADB55711.1) и описанные в US 2014/0329281 Al. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот KS. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов KS.
6.4.4 Бифункциональная копалилдифосфатсинтаза (CDPS) и кауренсинтаза (KS)
[0099] Также можно использовать бифункциональные ферменты CDPS-KS (ЕС 5.5.1.13 и ЕС 4.2.3.19). Иллюстративные примеры ферментов включают ферменты Phomopsis amygdali (учетный номер BAG30962), Physcomitrella patens (учетный номер BAF61135) и Gibberella fujikuroi (учетный номер Q9UVY5.1) и описанные в US 2014/0329281 Al, US 2014/0357588 Al, US 2015/0159188 и WO 2016/038095 A2. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот CDPS-KS. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов CDPS-KS.
6.4.5 Энт-кауреноксидаза (КО)
[00100] Энт-кауреноксидазы (ЕС 1.14.13.78; также называемые здесь кауреноксидазами) катализируют превращение каурена в кауреновую кислоту. Иллюстративные примеры ферментов включают ферменты Oryza sativa (учетный номер Q5Z5R4), Gibberella fujikuroi (учетный номер 094142), Arabidopsis thaliana (учетный номер Q93ZB2), Stevia rebaudiana (учетный номер AAQ63464.1) и Pisum sativum (номер Uniprot Q6XAF4) и описанные в US 2014/0329281 Al, US 2014/0357588 Al, US 2015/0159188 и WO 2016/038095 A2. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот КО. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов КО.
6.4.6 Стевиолсинтаза (КАН)
[00101] Стевиолсинтазы или гидроксилазы кауреновой кислоты (КАН) (ЕС 1.14.13) катализируют превращение кауреновой кислоты в стевиол. Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (учетный номер ACD93722), Stevia rebaudiana (SEQ ID NO: 10) Arabidopsis thaliana (учетный номер NP_197872), Vitis vinifera (учетный номер XP 002282091) и Medicago trunculata (учетный номер ABC59076) и описанные в US 2014/0329281 A1,US 2014/0357588 Al, US 2015/0159188 и WO 2016/038095 A2. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот КАН. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов КАН.
6.4.7 Редуктаза цитохрома Р450 (CPR)
[00102] Редуктазы цитохрома Р450 (ЕС 1.6.2.4) необходимы для активности КО и/или КАН, указанных выше. Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (учетный номер АВВ88839) Arabidopsis thaliana (учетный номер NP_194183), Gibberella fujikuroi (учетный номер САЕ09055) и Artemisia annua (учетный номер АВС47946.1) и описанные в US 2014/0329281 Al, US 2014/0357588 Al, US 2015/0159188 и WO 2016/038095 A2. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот CPR. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов CPR.
6.4.8 УДФ гликозилтрансфераза 74G1 (UGT74G1)
[00103] UGT74G1 способен функционировать как уридин-5'-дифосфоглюкозил: трансфераза стевиол 19-СООН и как уридин-5'-дифосфоглюкозил: стевиол-13-О-глюкозид 19-СООН трансфераза. Как показано на Фиг. 1, UGT74G1 способен превращать стевиол в 19-гликозид. UGT74G1 также способен превращать стевиолмонозид в рубузозид. UGT74G1 также может превращать стевиолбиозид в стевиозид. Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (например, Richman et al, 2005, Plant J.41: 56-67 и US 2014/0329281 и WO 2016/038095 A2 и учетный номер AAR06920.1). Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот UGT74G1. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов UGT74G1.
6.4.9 УДФ гликозилтрансфераза 76G1 (UGT76G1)
[00104] UGT76G1 способен переносить фрагмент глюкозы в С-3 'C-13-О-глюкозы акцепторной молекулы, стевиол-1,2 гликозида. Таким образом, UGT76G1 способен функционировать как уридин 5'-дифосфоглюкозил: стевиол 13-0-1,2 глюкозид С-3' глюкозилтрансфераза и уридин 5'-дифосфоглюкозил: стевиол-19-О-глюкоза, 13-0-1,2 биозид С-3' глюкозилтрансфераза. UGT76G1 способен превращать стевиолбиозид в Reb В. UGT76G1 также способен превращать стевиозид в Reb A. UGT76G1 также способен преобразовывать Reb D в Reb М. Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (например, ферменты Richman et al, 2005, Plant J'.41: 56-67 и US 2014/0329281 Al и WO 2016/038095 A2 и учетный номер AAR06912.1). Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот UGT76G1. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов UGT76G1.
6.4.10 УДФ гликозилтрансфераза 85С2 (UGT85C2)
[00105] UGT85C2 способен функционировать как уридин-5'-дифосфоглюкозил: стевиол 13-ОН трансфераза и уридин-5'-дифосфоглюкозил: стевиол-19-О-глюкозид 13-ОН трансфераза. UGT85C2 способен превращать стевиол в стевиолмонозид, а также способен превращать 19-гликозид в рубузозид. Иллюстративные примеры ферментов включают ферменты Stevia rebaudiana (например, Richman et al, 2005, Plant JA\: 56-67 и US 2014/0329281 Al и WO 2016/038095 A2 и учетный номер AAR06916.1). Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот UGT85C2. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов UGT85C2.
6.4.11 УДФ-гликозилтрансфераза 91D (UGT91D)
[00106] UGT91D способен функционировать как уридин-5'-дифосфоглюкозил:стевиол-13-О-глюкозид трансфераза, перенося молекулу глюкозы на С-2' 13-О-глюкозы акцепторной молекулы, стевиол-13-О-глюкозида (стевиолмонозида), для получения стевиолбиозида. UGT91D также способен функционировать как уридин-5'-дифосфоглюкозил: рубузозидтрансфераза, перенося фрагмент глюкозы на С-2 '13-О-глюкозы акцепторной молекулы, рубузозида, с получением стевиозида. UGT91D также обозначается как UGT91D2, UGT91D2e или UGT91D-like3. Иллюстративные примеры ферментов UGT91D включают ферменты Stevia rebaudiana (например, ферменты с последовательностью UGT с учетным номером АСЕ87855.1, US 2014/0329281 Al, WO 2016/038095 А2 и SEQ ID NO: 7). Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одной из этих нуклеиновых кислот UGT91D. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих ферментов UGT91D.
6.4.12 Уридиндифосфат-зависимая гликозилтрансфераза, способная превращать Reb А в Reb D (UGTad)
[00107] Уридиндифосфат-зависимая гликозилтрансфераза (UGTad) способна переносить фрагмент глюкозы в положение С-2' 19-О-глюкозы Reb А с образованием Reb D. UGTad также способен переносить фрагмент глюкозы в положение С-2' 19-О-глюкозы стевиозида для образования Reb Е. Полезные примеры UGT включают Os UGT 91С1 из Oryza sativa (также обозначаемый как EUGT11 в Houghton-Lars en et al, WO 2013/022989 A2; XP 015629141.1) и S1UGT 101249881 из Solanum lycopersicum также упоминается как UGTSL2 в Markosyan et al, WO2014/193888 Al; XP 004250485.1). Дополнительные полезные UGT включают в себя UGT40087 (XP 004982059.1; как описано в WO 2018/031955), sr.UGT_9252778, Bd_UGT10840 (ХР 003560669.1), HvUGTVl (BAJ94055.1), Bd_UGT10850 (ХР 010230871.1) и Ob_UGT91Bl_like (ХР 006650455.1).
Любой UGT или вариант UGT можно использовать в композициях и способах, описанных в данном документе. Нуклеиновые кислоты, кодирующие эти ферменты, применимы в клетках и способах, представленных здесь. В некоторых вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, имеющей по меньшей мере 80%, 85%, 90% или 95% идентичности последовательности по меньшей мере с одним из таких UGT. В определенных вариантах осуществления в настоящем документе представлены клетки и способы с использованием нуклеиновой кислоты, кодирующей полипептид, имеющий идентичность последовательности по меньшей мере на 80%, 85%, 90%, 95% по меньшей мере с одним из этих UGT. В некоторых вариантах осуществления в настоящем документе предусмотрена нуклеиновая кислота, кодирующая вариант UGT, описанный в настоящем документе.
6.5 Путь MEV Производство FPP и/или GGPP
[00108] В некоторых вариантах осуществления предложенная здесь генетически модифицированная клетка-хозяин содержит один или несколько гетерологичных ферментов пути MEV, полезных для образования FPP и/или GGPP. В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который конденсирует ацетил-КоА с малонил-КоА с образованием ацетоацетил-КоА. В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который конденсирует две молекулы ацетил-КоА с образованием ацетоацетил-КоА. В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который конденсирует ацетоацетил-КоА с ацетил-КоА с образованием ГМГ-КоА. В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который превращает ГМГ-КоА в мевалонат.В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который фосфорилирует мевалонат до мевалонат-5-фосфата. В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который превращает мевалонат-5-фосфат в мевалонат-5-пирофосфат.В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который превращает мевалонат-5-пирофосфат в изопентенилпирофосфат.В некоторых вариантах осуществления один или несколько ферментов пути MEV содержат фермент, который превращает изопентенилпирофосфат в диметилаллилдифосфат.
[00109] В некоторых вариантах осуществления один или несколько ферментов пути MEV выбраны из группы, состоящей из ацетил-КоА-тиолазы, ацетоацетил-КоА-синтетазы, ГМГ-КоА-синтазы, ГМГ-КоА-редуктазы, мевалонаткиназы, фосфомевалонаткиназы, мевалонатпирофосфатдекарбонатной кислоты и изопентилдифосфата:диметилаллилдифосфата-изомеразы (IDI или IPP-изомеразы). В некоторых вариантах осуществления, что касается фермента пути MEV, способного катализировать образование ацетоацетил-КоА, генетически модифицированная клетка-хозяин содержит либо фермент, который конденсирует две молекулы ацетил-КоА с образованием ацетоацетил-КоА, например ацетил-КоА тиолаза; или фермент, который конденсирует ацетил-КоА с малонил-КоА с образованием ацетоацетил-КоА, например ацетоацетил-КоА-синтазы. В некоторых вариантах осуществления генетически модифицированная клетка-хозяин содержит как фермент, который конденсирует две молекулы ацетил-КоА с образованием ацетоацетил-КоА, например, ацетил-КоА тиолазу; и фермент, который конденсирует ацетил-КоА с малонил-КоА с образованием ацетоацетил-КоА, например ацетоацетил-КоА-синтазы.
[00110] В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих более одного фермента пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих два фермента пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих фермент, который может превращать ГМГ-КоА в мевалонат, и фермент, который может превращать мевалонат в мевалонат-5-фосфат. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих три фермента пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих четыре фермента пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих пять ферментов пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих шесть ферментов пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит одну или несколько гетерологичных нуклеотидных последовательностей, кодирующих семь ферментов пути MEV. В некоторых вариантах осуществления клетка-хозяин содержит множество гетерологичных нуклеиновых кислот, кодирующих все ферменты пути MEV.
[00111] В некоторых вариантах осуществления генетически модифицированная клетка-хозяин дополнительно содержит гетерологичную нуклеиновую кислоту, кодирующую фермент, который может превращать изопентенилпирофосфат (IPP) в диметилаллилпирофосфат (DMAPP). В некоторых вариантах осуществления генетически модифицированная клетка-хозяин дополнительно содержит гетерологичную нуклеиновую кислоту, кодирующую фермент, который может конденсировать молекулы IPP и/или DMAPP с образованием полипренильного соединения. В некоторых вариантах осуществления генетически модифицированная клетка-хозяин дополнительно содержит гетерологичную нуклеиновую кислоту, кодирующую фермент, который может модифицировать IPP или полипренил с образованием изопреноидного соединения, такого как FPP.
6.5.1 Превращение ацетил-КоА в ацетоацетил-КоА
[00112] В некоторых вариантах осуществления генетически модифицированная клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может конденсировать две молекулы ацетил-кофермента А с образованием ацетоацетил-КоА, например, ацетил-КоА-тиолазы. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (РЕГИОН NC 000913: 2324131.2325315; Escherichia coli), (D49362; Paracoccus denitrificans) и (L20428; Saccharomyces cerevisiae).
[00113] Ацетил-КоА тиолаза катализирует обратимую конденсацию двух молекул ацетил-КоА с образованием ацетоацетил-КоА, но эта реакция термодинамически невыгодна; Тиолиз ацетоацетил-КоА предпочтительнее синтеза ацетоацетил-КоА. Ацетоацетил-КоА-синтаза (AACS) (также называемая ацетил-КоА: малонил-КоА-ацилтрансфераза; ЕС 2.3.1.194) конденсирует ацетил-КоА с малонил-КоА с образованием ацетоацетил-КоА. В отличие от тиолазы ацетил-КоА, синтез ацетоацетил-КоА, катализируемый AACS, по существу является энергетически выгодной реакцией из-за связанного декарбоксилирования малонил-КоА. Кроме того, AACS не проявляет активности тиолиза в отношении ацетоацетил-КоА, и, таким образом, реакция необратима.
[00114] В клетках-хозяевах, содержащих ацетил-СоА тиолазу и гетерологичную ADA и/или фосфотрансацетилазу (РТА), обратимая реакция, катализируемая ацетил-СоА тиолазой, которая способствует тиолизу ацетоацетил-СоА, может привести к большому пулу ацетил-СоА. Принимая во внимание обратимую активность ADA, этот пул ацетил-КоА может, в свою очередь, подталкивать ADA к обратной реакции превращения ацетил-КоА в ацетальдегид, тем самым уменьшая преимущества, обеспечиваемые ADA в отношении продукции ацетил-КоА. Точно так же активность РТА обратима, и, таким образом, большой пул ацетил-СоА может подтолкнуть РТА к обратной реакции превращения ацетил-СоА в ацетилфосфат.Следовательно, в некоторых вариантах осуществления, чтобы обеспечить сильное притяжение ацетил-КоА для запуска прямой реакции ADA и РТА, в пути MEV генетически модифицированной клетки-хозяина, представленной в настоящем документе, используется ацетоацетил-КоА-синтаза для образования ацетоацетил-КоА из ацетил-КоА и малонил-КоА.
[00115] В некоторых вариантах осуществления AACS происходит от Streptomyces sp., штамм CL190 (Okamura et al., Proc Natl Acad Sci USA 107 (25): 11265-70 (2010). Репрезентативные нуклеотидные последовательности AACS Streptomyces sp., штамм CL190 включает инвентарный номер АВ540131.1. Репрезентативные белковые последовательности AACS Streptomyces sp., штамм CL190 включает учетные номера D7URV0, ВАJ10048. Другие ацетоацетил-СоА-синтазы, применимые для композиций и способов, представленных в настоящем документе, включают, но не ограничиваются ими, Streptomyces sp.(АВ183750; КО-3988 BAD86806); S. anulatus, штамм 9663 (FN 178498; САХ48662); Streptomyces sp.КО-3988 (АВ212624; ВАЕ78983); Actinoplanes sp.A40644 (AB113568; BAD07381); Streptomyces sp.С (NZACEW010000640; ZP_05511702): Nocardiopsis dassonvillei DSM 43111 (NZ_ABUI01000023; ZP_04335288); Mycobacterium ulcerans Agy99 (NC 008611; YP 907152); Mycobacterium marinum M (NC 010612; YP 001851502); Streptomyces sp.Mgl (NZ DS570501; ZP 05002626); Streptomyces sp.AA4 (NZ_ACEV01000037; ZP_05478992); S. roseosporus NRRL 15998 (NZ ABYB01000295: ZP_04696763); Streptomyces sp.ACTE (NZADFD01000030; ZP_06275834); S viridochromogenes DSM 40736 (NZ ACEZ01000031; ZP 05529691); Frankia sp.CcI3 (NC 007777; YP 480101); Nocardia brasiliensis (NC 018681; YP 006812440.1); nAustwickia chelonae (NZBAGZ01000005; ZP 10950493.1). Дополнительные подходящие ацетоацетил-КоА-синтазы включают те, что описаны в публикациях патентных заявок США №№2010/0285549 и 2011/0281315, содержание которых полностью включено посредством ссылки.
[00116] Ацетоацетил-КоА-синтазы, также используемые в композициях и способах, представленных в настоящем документе, включают те молекулы, которые, как говорят, являются «производными» любой из ацетоацетил-КоА-синтаз, описанных в данном документе. Такое «производное» имеет следующие характеристики: (1) оно имеет существенную гомологию с любой из ацетоацетил-КоА-синтаз, описанных здесь; и (2) способен катализировать необратимую конденсацию ацетил-КоА с малонил-КоА с образованием ацетоацетил-КоА. Считается, что производное ацетоацетил-КоА синтазы имеет "существенную гомологию" с ацетоацетил-КоА синтазой, если аминокислотная последовательность производного по крайней мере на 80%, более предпочтительно по крайней мере на 90%, а наиболее предпочтительно по крайней мере на 95% совпадает с аминокислотной последовательностью ацетоацетил-КоА синтазы.
6.5.2 Превращение ацетоацетил-КоА в ГМГ-КоА
[00117] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может конденсировать ацетоацетил-КоА с другой молекулой ацетил-КоА с образованием 3-гидрокси-3-метилглутарил-КоА (ГМГ-КоА), например, ГМГ- КоА-синтаза. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (NC 001145; комплемент 19061.20536; Saccharomyces cerevisiae), (Х96617; Saccharomyces cerevisiae), (X83882; Arabidopsis thaliana), (AB037907; Kit007302; Homo sapiens) и (NC 002758, Locus tag SAV2546, GenelD 1122571; Staphylococcus aureus).
6.5.3 Превращение ГМГ-КоА в мевалонат
[00118] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может превращать ГМГ-КоА в мевалонат, например, ГМГ-КоА редуктазу. В некоторых вариантах осуществления ГМГ-КоА редуктаза представляет собой НАДН-использующую гидроксиметилглутарил-КоА редуктазу- Ко А редуктазу. ГМГ-Ко А-редуктазы (ЕС 1.1.1.34: ЕС 1.1.1.88) катализируют восстановительное деацилирование (8)-ГМГ-КоА до (R)-мевалоната, и их можно разделить на два класса: ГМГ класса I и класса П. Класс I включает ферменты эукариот и большинства архей, а класс II включает редуктазы ГМГ-КоА некоторых прокариот и архей. Помимо расхождения в последовательностях, ферменты двух классов также различаются по своей кофакторной специфичности. В отличие от ферментов класса I, которые используют исключительно НАДФН, редуктазы ГМГ-КоА класса II различаются по способности различать НАДФН и НАДН. См., например, Hedl и др., Journal of Bacteriology (Журнал бактериологии) 186 (7): 1927-1932 (2004). Ниже приведены особенности кофакторов для некоторых редуктаз ГМГ-КоА класса П.
Особенности кофакторов для некоторых редуктаз ГМГ-КоА класса II
[00119] Подходящие ГМГ-КоА-редуктазы для композиций и способов, представленных в настоящем документе, включают ГМГ-КоА-редуктазы, которые способны использовать НАДН в качестве кофактора, например, ГМГ-КоА-редуктазу из P. mevalonii, A. fulgidus или S. aureus. В конкретных вариантах осуществления ГМГ-КоА редуктаза способна использовать только НАДН в качестве кофактора, например, ГМГ-КоА редуктазу из Р. mevalonii, S. pomeroyi или D. acidovorans.
[00120] В некоторых вариантах осуществления ГМГ-КоА редуктаза, использующая НАДН, происходит из Pseudomonas mevalonii. Последовательность гена mvaA дикого типа Pseudomonas mevalonii, который кодирует ГМГ-КоА редуктазу (ЕС 1.1.1.88), была описана ранее. См. Beach and Rodwell, J. Bacteriol. 171:2994-3001 (1989). Репрезентативные нуклеотидные последовательности mvaA Pseudomonas mevalonii включают учетный номер М24015. Репрезентативные белковые последовательности ГМГ-КоА-редуктазы Pseudomonas mevalonii включают учетные номера ААА25837, Р13702, MVAA PSEMV. [00121] В некоторых вариантах осуществления ГМГ-КоА редуктаза, использующая НАДН, получена из Silicibacter pomeroyi. Репрезентативные нуклеотидные последовательности ГМГ-КоА редуктазы Silicibacter pomeroyi включают учетный номер NC 006569.1. Репрезентативные белковые последовательности ГМГ-КоА-редуктазы Silicibacter pomeroyi включают учетный номер YP 164994.
[00122] В некоторых вариантах осуществления ГМГ-КоА редуктаза, использующая НАДН, получена из Delftia acidovorans. Репрезентативная нуклеотидная
последовательность ГМГ-КоА редуктазы Delftia acidovorans включает NC 010002 REGION: комплемент (319980..321269). Репрезентативные последовательности белка ГМГ-КоА-редуктазы Delftia acidovorans включают учетный номер YP 001561318. [00123] В некоторых вариантах осуществления ГМГ-КоА-редуктазы, использующие НАДН, происходят из Solanum tuberosum (Crane et al., J. Plant Physiol. 159: 1301-1307 (2002)).
[00124] ГМГ-КоА-редуктазы, использующие НАДН, также полезные в композициях и способах, представленных в настоящем документе, включают те молекулы, которые, как говорят, являются «производными» любой из использующих НАДН ГМГ-КоА-редуктаз, описанных в данном документе, например, из P.mevalonii, S. pomeroyi и D. acidovorans. Такое «производное» имеет следующие характеристики: (1) оно имеет существенную гомологию с любой из описанных здесь редуктаз ГМГ-КоА, использующих НАДН; и (2) способен катализировать восстановительное деацилирование(8)-ГМГ-КоА до (R) -мевалоната, предпочтительно используя НАДН в качестве кофактора. Производное ГМГ-КоА-редуктазы, использующей НАДН, имеет «существенную гомологию» с ГМГ-КоА-редуктазой, использующей НАДН, если аминокислотные последовательности производного составляют не менее 80%, а более предпочтительно не менее 90%, и наиболее предпочтительно, по крайней мере, 95%, как и у ГМГ-КоА редуктазы, использующей НАДН.
[00125] В данном контексте фраза «с использованием НАДН» означает, что ГМГ-КоА редуктаза, использующая НАДН, является селективной в отношении НАДН по сравнению с НАДН в качестве кофактора, например, демонстрируя более высокую специфическую активность для НАДН, чем для НАДН. В некоторых вариантах осуществления селективность по НАДН в качестве кофактора выражается как отношение й:Са/НАДН^/ £сагГНАДФ1Я В некоторых вариантах осуществления ГМГ-КоА-редуктаза, использующая НАДН, имеет отношение ксщ(:НАДЯ)/ кст(^АДФЯ) по меньшей мере 5, 10, 15, 20, 25 или более 25. В некоторых вариантах осуществления ГМГ-КоА-редуктаза, использующая НАДН, использует исключительно НАДН. Например, ГМГ-КоА редуктаза, использующая НАДН, которая использует НАДН исключительно, проявляет некоторую активность с НАДН, поставляемым в качестве единственного кофактора in vitro, и не проявляет обнаруживаемой активности, когда НАДФН предоставляется в качестве единственного кофактора. Любой метод определения кофакторной специфичности, известный в данной области, может быть использован для идентификации ГМГ-КоА редуктаз, предпочитающих НАДН в качестве кофактора, включая те, которые описаны Kim et al, Protein Science 9: 1226-1234 (2000); и Wilding et al., J. Bacteriol. 182 (18): 5147-52 (2000), содержание которой полностью включено в настоящий документ.
[00126] В некоторых вариантах осуществления изобретения ГМГ-КоА редуктаза, использующая НАДН, сконструирована так, чтобы быть селективной в отношении НАДН по сравнению с НАПД, например, посредством сайт-направленного мутагенеза кармана связывания кофактора. Способы конструирования НАДН-селективности описаны в Watanabe et al, Microbiology 153:3044-3054 (2007), а методы определения кофакторной специфичности ГМГ-КоА редуктаз описаны в Kim et al, Protein Sci. 9: 1226-1234 (2000), содержание которых полностью включено в настоящее описание посредством ссылки.
[00127] В некоторых вариантах осуществления ГМГ-КоА редуктаза, использующая НАДН, происходит от вида-хозяина, который изначально включает путь разложения мевалоната, например, от вида-хозяина, который катаболизирует мевалонат в качестве своего единственного источника углерода. В этих вариантах осуществления ГМГ-КоА-редуктаза, использующая НАДН, которая обычно катализирует окислительное ацилирование интернализованного (R) -мевалоната до (8)-ГМГ-КоА внутри его нативной клетки-хозяина, используется для катализа обратной реакции, то есть, восстановительное деацилирование (8)-ГМГ-КоА до (К)-мевалоната в генетически модифицированной клетке-хозяине, включающей путь биосинтеза мевалоната. Прокариоты, способные расти на мевалонате в качестве единственного источника углерода, были описаны: Anderson et al., J. Bacteriol, 171 (12): 6468-6472 (1989); Beach et al., J. Bacteriol. 171: 2994-3001 (1989); Bensch et al., J. Biol. Chem. 245:3755-3762; Fimongnari et al., Biochemistry 4:2086-2090 (1965); Siddiqi et al., Biochem. Biophys. Res. Commun. 8:110-113 (1962); Siddiqi et al., J. Bacteriol. 93:207-214 (1967); and Takatsuji et al., Biochem. Biophys. Res. Commun. 110:187-193 (1983), содержание которой полностью включено в настоящий документ посредством ссылки. [00128] В некоторых вариантах осуществления представленных здесь композиций и способов клетка-хозяин содержит как HMGr, использующую НАДН, так и ГМГ-КоА-редуктазу, использующую НАДФН. Иллюстративные примеры нуклеотидных последовательностей, кодирующих ГМГ-КоА-редуктазу, использующую НАДФН, включают, помимо прочего: (NM 206548; Drosophila melanogaster), (NC 002758, метка локуса SAV2545, GenelD 1122570; Staphylococcus aureus), (ABO 15627; Streptomyces sp.КО 3988), (AX 128213, обеспечивающий последовательность, кодирующую усеченную ГМГ-КоА-редуктазу; Saccharomyces cerevisiae) и (NC_001145: комплемент (115734.118898: Saccharomyces cerevisiae).
6.5.4 Превращение мевалоната в мевалонат-5-фосфат
[00129] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может превращать мевалонат в мевалонат-5-фосфат, например мевалонаткиназу. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (L77688; Arabidopsis thaliana) и (Х55875; Saccharomyces cerevisiae).
6.5.5 Превращение мевалонат-5-фосфата в мевалонат-5-пирофосфат
[00130] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может превращать мевалонат-5-фосфат в мевалонат-5-пирофосфат, например, фосфомевалонаткиназу. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (AF429385; Hevea brasiliensis), (NM_006556; Homo sapiens) и (NC_001145. комплемент 712315.713670; Saccharomyces cerevisiae).
6.5.6 Превращение мевалонат-5-пирофосфата в IPP
[00131] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может превращать мевалонат-5-пирофосфат в изопентенилдифосфат (1РР)>например, мевалонатпирофосфатдекарбоксилазу. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (Х97557; Saccharomyces cerevisiae), (AF290095; Enterococcus faecium) и (U49260; Homo sapiens).
6.5.7 Преобразование IPP в DMAPP
[00132] В некоторых вариантах осуществления клетка-хозяин дополнительно содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может преобразовывать IPP, генерируемый посредством пути MEV, в диметилаллилпирофосфат (DMAPP), например, изомеразу IPP. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (NC_000913, 3031087.3031635; Escherichia coli) и (AF082326; Haematococcus pluvialis).
6.5.8 Полипренилсинтазы
[00133] В некоторых вариантах осуществления клетка-хозяин дополнительно содержит гетерологичную нуклеотидную последовательность, кодирующую полипренилсинтазу, которая может конденсировать молекулы IPP и/или DMAPP с образованием полипренильных соединений, содержащих более пяти атомов углерода. [00134] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может конденсировать одну молекулу IPP с одной молекулой DMAPP с образованием одной молекулы геранилпирофосфата («GPP»), например, GPP-синтазы. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (AF513111; Abies grandis), (AF513112; Abies grandis), (AF513113: Abies grandis), (AY534686; Antirrhinum majus), (AY534687; Antirrhinum majus), (Y17376: Arabidopsis thaliana), (AE016877, Locus AP11092; Bacillus cereus; ATCC 14579), (AJ243739; Citrus sinensis), (AY534745; Clarkia breweri), (AY953508; Ipspini), (DQ286930; Lycopersicon esculentum), (AF182828; Mentha x piperita), (AF182827; Mentha x piperita), (MPI249453; Mentha x piperita), (PZE431697, Locus CAD24425; Paracoccus zeaxanthinifaciens), (AY866498; Picrorhiza kurrooa), (AY351862; Vitis vinifera) и (AF203881, Locus AAF12843; Zymomonas mobilis).
[00135] В некоторых вариантах осуществления клетка-хозяин содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может конденсировать две молекулы IPP с одной молекулой DMAPP или добавлять молекулу IPP к молекуле GPP с образованием молекулы фарнезилпирофосфата («FPP»), например, FPP-синтаза. Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (ATU80605; Arabidopsis thaliana), (ATHFPS2R; Arabidopsis thaliana), (AAU36376; Artemisia annua), (AF461050; Bos taurus), (D00694; Escherichia coli K-12), (AE009951, Locus AAL95523; Fusobacterium nucleatum subsp.nucleatum ATCC 25586), (GFFPPSGEN; Gibberella fujikuroi), (CP000009, Locus AAW60034; Gluconobacter oxydans 621H), (AF019892; Helianthus annuus), (HUMFAPS; Homo sapiens), (KLPFPSQCR; Kluyveromyces lactis), (LAU15777; Lupinus albus), (LAU20771; Lupinus albus), (AF309508; Mus musculus), (NCFPPSGEN; Neurospora crassa), (PAFPS1; Parthenium argentatum), (PAFPS2; Parthenium argentatum), (RATFAPS; Rattus norvegicus), (YSCFPP; Saccharomyces cerevisiae), (D89104; Schizosaccharomyces pombe), (CP000003, Locus AAT87386; Streptococcus pyogenes), (CP000017, Locus AAZ51849; Streptococcus pyogenes), (NC 008022, Locus YP 598856; Streptococcus pyogenes MGAS10270), (NC 008023, Locus YP 600845; Streptococcus pyogenes MGAS2096), (NC 008024, Locus YP_602832; Streptococcus pyogenes MGAS 10750), (MZEFPS; Zea mays), (AE000657, Locus AAC06913; Aquifex aeolicus VF5), (NM_202836; Arabidopsis thaliana), (D84432, Locus BAA12575; Bacillus subtilis), (U12678, Locus AAC28894; Bradyrhizobium japonicum USDA 110), (BACFDPS; Geobacillus stear other mophilus), (NC_002940, Locus NP_873754: Haemophilus ducreyi 35000HP), (L42023, Locus AAC23087; Haemophilus influenzae Rd KW20), (J05262; Homo sapiens), (YP 395294; Lactobacillus sakei subsp.sakei 23K), (NC_005823, Locus YP_000273; Leptospira interrogans serovar Copenhageni str. Fiocruz Ll-130), (АВ003187; Micrococcus luteus), (NC_002946, Locus YV_20W6%; Neisseria gonorrhoeae FA 1090), (U00090, Locus AAB91752; Rhizobium sp.NGR234), (J05091; Saccharomyces cerevisae), (CP000031, локус AAV93568; Silicibacter pomeroyi DSS-3), (AE008481, локус AAK99890; Streptococcus pneumoniae R6) и (NC_004556, локус NP 779706; Xylella fastidiosa Temeculal).
[00136] В некоторых вариантах осуществления клетка-хозяин дополнительно содержит гетерологичную нуклеотидную последовательность, кодирующую фермент, который может объединять IPP и DMAPP или IPP и FPP с образованием геранилгеранилпирофосфата («GGPP»). Иллюстративные примеры нуклеотидных последовательностей, кодирующих такой фермент, включают, но не ограничиваются ими: (ATHGERPYRS; Arabidopsis thaliana), (ВТ005328; Arabidopsis thaliana), (NM_119845; Arabidopsis thaliana), (NZ_AAJM01000380, Locus ZP_00743052; Bacillus thuringiensis serovar israelensis, ATCC 35646 sql563), (CRGGPPS; Catharanthus roseus), (NZ AABF02000074, Locus ZP 00144509; Fusobacterium nucleatum subsp.vincentii, ATCC 49256), (GFGGPPSGN; Gibberella fujikuroi), (AY371321; Ginkgo biloba), (ABO55496; Hevea brasiliensis), (AB017971; Homo sapiens), (MCI276129; Mucor circinelloides f. lusitanicus), (ABO 16044; Mus musculus), (ААВХ01000298, Locus NCU01427; Neurospora crassa), (NCU20940; Neurospora crassa), (NZ AAKL01000008, Locus ZP 00943566; Ralstonia solanacearum UW551), (AB118238; Rattus norvegicus), (SCU31632; Saccharomyces cerevisiae), (ABO 16095; Synechococcus elongates), (SAGGPS; Sinapis alba), (SSOGDS; Sulfolobus acidocaldarius), (NC_007759, Locus YP_461832; Syntrophus aciditrophicus SB), (NC_006840, Locus YP_204095; Vibrio fischeri ESI 14), (NM_112315; Arabidopsis thaliana), (ERWCRTE; Pantoea agglomerans), (D90087, Locus BAA14124; Pantoea ananatis), (X52291, Locus CAA36538; Rhodobacter capsulatus), (AF195122, Locus AAF24294; Rhodobacter sphaeroides) и (NC_004350, Locus NP_721015; Streptococcus mutans UA159).
[00137] Хотя примеры ферментов мевалонатного пути описаны выше, в некоторых вариантах осуществления ферменты пути DXP можно использовать в качестве альтернативного или дополнительного пути для продуцирования DMAPP и IPP в клетках-хозяевах, композициях и способах, описанных в данном документе. Ферменты и нуклеиновые кислоты, кодирующие ферменты пути DXP, хорошо известны и охарактеризованы в данной области, например, WO 2012/135591 А2.
6.6 Способы получения стевиоловых гликозидов
[00138] В другом аспекте в настоящем документе предоставляется способ получения гликозида стевиола, который включает стадии: (а) культивирования популяции любой из генетически модифицированных клеток-хозяев, описанных в данном документе, которые способны продуцировать гликозид стевиола в среде с источником углерода в условиях, подходящих для получения гликозидного соединения стевиола; и (b) выделение указанного стевиолгликозидного соединения из среды.
[00139] В некоторых вариантах осуществления генетически модифицированная клетка-хозяин продуцирует повышенное количество стевиолового гликозида по сравнению с родительской клеткой, не содержащей одну или несколько модификаций, или родительской клеткой, содержащей только подмножество одной или нескольких модификаций генетически модифицированной клетки-хозяина, но в остальном генетически идентичной. В некоторых вариантах осуществления увеличенное количество составляет по меньшей мере 1%, 5%, 10%, 15%, 20%, 25%, 30%, 35%, 40%, 45%, 50%, 55%, 60%, 65. %, 70%, 75%, 80%, 85%, 90%, 95%, 100% или более 100%, как измерено, например, по выходу, объему производства и/или производительности, в граммах на литр клеточной культуры, миллиграммах на грамм веса сухих клеток, на единицу объема клеточной культуры, на единицу веса сухих клеток, на единицу объема клеточной культуры в единицу времени или на единицу веса сухих клеток в единицу времени.
[00140] В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который превышает примерно 1 грамм на литр ферментационной среды. В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который превышает примерно 5 граммов на литр ферментационной среды. В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который превышает примерно 10 грамм на литр ферментационной среды. В некоторых вариантах осуществления стевиоловый гликозид производится в количестве от около 10 до около 50 граммов, от около 10 до около 15 граммов, более чем около 15 граммов, более чем около 20 граммов, более чем около 25 граммов или более чем около 30 грамм на литр клеточной культуры.
[00141] В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который превышает примерно 50 миллиграммов на грамм сухой массы клетки. В некоторых таких воплощениях гликозид стевиола производится в количестве от около 50 до около 1500 миллиграмм, более чем около 100 миллиграмм, более чем около 150 миллиграмм, более чем около 200 миллиграмм, более чем около 250 миллиграмм, более чем около 500 миллиграмм, более чем около 750 миллиграмм или более чем около 1000 миллиграмм на грамм сухого веса клеток.
[00142] В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который составляет по меньшей мере примерно на 10%, по меньшей мере примерно на 15%, по меньшей мере примерно на 20%, по меньшей мере примерно на 25%, по меньшей мере примерно на 30%, по меньшей мере примерно на 35%, по меньшей мере примерно на 40%, по меньшей мере примерно на 45%, по меньшей мере примерно на 50%, по меньшей мере примерно на 60%, по меньшей мере примерно на 70%, по меньшей мере примерно на 80%, по меньшей мере примерно на 90%, по меньшей мере примерно в 2 раза, по меньшей мере примерно в 2,5 раза, по меньшей мере примерно в 5 раз, по меньшей мере примерно в 10 раз, по меньшей мере примерно в 20 раз, по меньшей мере примерно в 30 раз, по меньшей мере примерно в 40 раз, по меньшей мере примерно в 50 раз, по меньшей мере примерно в 75 раз, по меньшей мере примерно в 100 раз, по меньшей мере примерно в 200 раз, по меньшей мере примерно в 300 раз, по меньшей мере примерно в 400 раз, по меньшей мере примерно в 500 раз или по меньшей мере примерно в 1000 раз или более, выше, чем уровень стевиолгликозида, продуцируемого родительской клеткой, в расчете на единицу объема клеточной культуры.
[00143] В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который составляет по меньшей мере примерно на 10%, по меньшей мере примерно на 15%, по меньшей мере примерно на 20%, по меньшей мере примерно на 25%, по меньшей мере примерно на 30%, по меньшей мере примерно на 35%, по меньшей мере примерно на 40%, по меньшей мере примерно на 45%, по меньшей мере примерно на 50%, по меньшей мере примерно на 60%, по меньшей мере примерно на 70%, по меньшей мере примерно на 80%, по меньшей мере примерно на 90%, по меньшей мере примерно в 2 раза, по меньшей мере примерно в 2,5 раза, по меньшей мере примерно в 5 раз, по меньшей мере примерно в 10 раз, по меньшей мере примерно в 20 раз, по меньшей мере примерно в 30 раз, по меньшей мере примерно в 40 раз, по меньшей мере примерно в 50 раз, по меньшей мере примерно в 75 раз, по меньшей мере примерно в 100 раз, по меньшей мере примерно в 200 раз, по меньшей мере примерно в 300 раз, по меньшей мере примерно в 400 раз, по меньшей мере примерно в 500 раз или по меньшей мере примерно в 1000 раз или более, выше, чем уровень стевиолгликозида, продуцируемого родительской клеткой, в расчете на единицу массы сухой клетки.
[00144] В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который составляет по меньшей мере примерно на 10%, по меньшей мере примерно на 15%, по меньшей мере примерно на 20%, по меньшей мере примерно на 25%, по меньшей мере примерно на 30%, по меньшей мере примерно на 35%, по меньшей мере примерно на 40%, по меньшей мере примерно на 45%, по меньшей мере примерно на 50%, по меньшей мере примерно на 60%, по меньшей мере примерно на 70%, по меньшей мере примерно на 80%, по меньшей мере примерно на 90%, по меньшей мере примерно в 2 раза, по меньшей мере примерно в 2,5 раза, по меньшей мере примерно в 5 раз, по меньшей мере примерно в 10 раз, по меньшей мере примерно в 20 раз, по меньшей мере примерно в 30 раз, по меньшей мере примерно в 40 раз, по меньшей мере примерно в 50 раз, по меньшей мере примерно в 75 раз, по меньшей мере примерно в 100 раз, по меньшей мере примерно в 200 раз, по меньшей мере примерно в 300 раз, по меньшей мере примерно в 400 раз, по меньшей мере примерно в 500 раз или по меньшей мере примерно в 1000 раз или более, выше, чем уровень стевиолгликозида, продуцируемого родительской клеткой, в расчете на единицу объема клеточной культуры в единицу времени. [00145] В некоторых вариантах осуществления клетка-хозяин продуцирует повышенный уровень стевиолового гликозида, который составляет по меньшей мере примерно на 10%, по меньшей мере примерно на 15%, по меньшей мере примерно на 20%, по меньшей мере примерно на 25%, по меньшей мере примерно на 30%, по меньшей мере примерно на 35%, по меньшей мере примерно на 40%, по меньшей мере примерно на 45%, по меньшей мере примерно на 50%, по меньшей мере примерно на 60%, по меньшей мере примерно на 70%, по меньшей мере примерно на 80%, по меньшей мере примерно на 90%, по меньшей мере примерно в 2 раза, по меньшей мере примерно в 2,5 раза, по меньшей мере примерно в 5 раз, по меньшей мере примерно в 10 раз, по меньшей мере примерно в 20 раз, по меньшей мере примерно в 30 раз, по меньшей мере примерно в 40 раз, по меньшей мере примерно в 50 раз, по меньшей мере примерно в 75 раз, по меньшей мере примерно в 100 раз, по меньшей мере примерно в 200 раз, по меньшей мере примерно в 300 раз, по меньшей мере примерно в 400 раз, по меньшей мере примерно в 500 раз или по меньшей мере примерно в 1000 раз или более, выше, чем уровень стевиолгликозида, продуцируемого родительской клеткой, в расчете на единицу сухой массы клетки в единицу времени. [00146] В большинстве вариантов осуществления продукция повышенного уровня стевиолгликозида клеткой-хозяином индуцируется присутствием индуцирующего соединения. Такой клеткой-хозяином можно легко манипулировать в отсутствие индуцирующего соединения. Затем добавляют индуцирующее соединение для индукции продукции повышенного уровня стевиолгликозида клеткой-хозяином. В других вариантах осуществления продукция повышенного уровня стевиолгликозида клеткой-хозяином индуцируется изменением условий культивирования, таких как, например, температура роста, состав среды и т.п.
6.7 Кулътуральные среды и условия
[00147] Материалы и методы для поддержания и роста микробных культур хорошо известны специалистам в области микробиологии или науки о ферментации (см., например, Bailey et al, Biochemical Engineering Fundamentals (Основы биохимической инженерии), второе издание, McGraw Hill, New York, 1986 г.). Необходимо учитывать подходящую культуральную среду, рН, температуру и требования к аэробным, микроаэробным или анаэробным условиям, в зависимости от конкретных требований клетки-хозяина, ферментации и процесса.
[00148] Способы получения стевиоловых гликозидов, представленные в настоящем документе, могут быть выполнены в подходящей культуральной среде (например, с добавлением пантотената или без него) в подходящем контейнере, включая, помимо прочего, планшет для культивирования клеток, планшет для микротитрования, колбу или ферментатор. Кроме того, способы могут быть выполнены в любом масштабе ферментации, известном в данной области, для поддержки промышленного производства микробных продуктов. Может быть использован любой подходящий ферментатор, включая ферментатор с мешалкой, ферментатор с эрлифтом, пузырьковый ферментатор или любую их комбинацию. В конкретных вариантах осуществления, использующих Saccharomyces cerevisiae в качестве клетки-хозяина, штаммы можно выращивать в ферментаторе, как подробно описано Kosaric и др. в Ullmann's Encyclopedia of Industrial Chemistry (Энциклопедии промышленной химии Ульманна), шестое издание, том 12, страницы 398-473, Wiley-VCH Verlag GmbH & Co. KDaA, Вайнхайм, Германия.
[00149] В некоторых вариантах осуществления культуральная среда представляет собой любую культуральную среду, в которой может существовать генетически модифицированный микроорганизм, способный продуцировать стевиоловый гликозид, т.е. поддерживать рост и жизнеспособность. В некоторых вариантах осуществления культуральная среда представляет собой водную среду, содержащую источники ассимилируемого углерода, азота и фосфата. Такая среда может также включать соответствующие соли, минералы, металлы и другие питательные вещества. В некоторых вариантах осуществления источник углерода и каждое из основных питательных веществ для клеток добавляются в ферментационную среду постепенно или непрерывно, и каждое необходимое питательное вещество поддерживается по существу на минимальном уровне, необходимом для эффективной ассимиляции растущими клетками, например, в соответствии с заданной кривой роста клеток, основанной на метаболической или дыхательной функции клеток, которые преобразуют источник углерода в биомассу.
[00150] Подходящие условия и подходящие среды для культивирования микроорганизмов хорошо известны в данной области. В некоторых вариантах осуществления подходящая среда дополняется одним или несколькими дополнительными агентами, такими как, например, индуктор (например, когда одна или несколько нуклеотидных последовательностей, кодирующих продукт гена, находятся под контролем индуцибельного промотора), репрессор (например, когда одна или несколько нуклеотидных последовательностей, кодирующих продукт гена, находятся под контролем репрессируемого промотора) или агента отбора (например, антибиотика для отбора микроорганизмов, содержащих генетические модификации).
[00151] В некоторых вариантах осуществления источник углерода представляет собой моносахарид (простой сахар), дисахарид, полисахарид, неферментируемый источник углерода или одну или несколько их комбинаций. Неограничивающие примеры подходящих моносахаридов включают глюкозу, галактозу, маннозу, фруктозу, ксилозу, рибозу и их комбинации. Неограничивающие примеры подходящих дисахаридов включают сахарозу, лактозу, мальтозу, трегалозу, целлобиозу и их комбинации. Неограничивающие примеры подходящих полисахаридов включают крахмал, гликоген, целлюлозу, хитин и их комбинации. Неограничивающие примеры подходящих неферментируемых источников углерода включают ацетат и глицерин.
[00152] Концентрация источника углерода, такого как глюкоза, в культуральной среде достаточна для стимулирования роста клеток, но не настолько высока, чтобы подавлять рост используемых микроорганизмов. Как правило, культивирование проводится с источником углерода, таким как глюкоза, добавляемым в количествах, обеспечивающих желаемый уровень роста и биомассы. В других вариантах осуществления концентрация источника углерода, такого как глюкоза, в культуральной среде больше примерно 1 г/л, предпочтительно больше примерно 2 г/л и более предпочтительно больше примерно 5 г/л. Кроме того, концентрация источника углерода, такого как глюкоза, в культуральной среде обычно составляет менее примерно 100 г/л, предпочтительно менее примерно 50 г/л и более предпочтительно менее примерно 20 г/л. Следует отметить, что ссылки на концентрации компонентов культуры могут относиться как к начальным, так и/или текущим концентрациям компонентов. В некоторых случаях может быть желательно позволить питательной среде истощиться от источника углерода во время культивирования.
[00153] Источники ассимилируемого азота, которые можно использовать в подходящей культуральной среде, включают, помимо прочего, простые источники азота, источники органического азота и сложные источники азота. Такие источники азота включают безводный аммиак, соли аммония и вещества животного, растительного и/или микробного происхождения. Подходящие источники азота включают, но не ограничиваются ими, гидролизаты белка, гидролизаты микробной биомассы, пептон, дрожжевой экстракт, сульфат аммония, мочевину и аминокислоты. Обычно концентрация источников азота в культуральной среде больше примерно 0,1 г/л, предпочтительно больше примерно 0,25 г/л и более предпочтительно больше примерно 1,0 г/л. Однако при превышении определенных концентраций добавление источника азота к культуральной среде не способствует росту микроорганизмов. В результате концентрация источников азота в культуральной среде составляет менее примерно 20 г/л, предпочтительно менее примерно 10 г/л и более предпочтительно менее примерно 5 г/л. Кроме того, в некоторых случаях может быть желательно позволить питательной среде истощиться от источников азота во время культивирования.
[00154] Эффективная питательная среда может содержать другие соединения, такие как неорганические соли, витамины, следовые количества металлов или стимуляторы роста. Такие другие соединения также могут присутствовать в источниках углерода, азота или минералов в эффективной среде или могут быть добавлены специально в среду.
[00155] Питательная среда также может содержать подходящий источник фосфата. Такие источники фосфатов включают источники как неорганических, так и органических фосфатов. Предпочтительные источники фосфатов включают, но не ограничиваются ими, фосфатные соли, такие как одно- или двухосновные фосфаты натрия и калия, фосфат аммония и их смеси. Обычно концентрация фосфата в культуральной среде больше примерно 1,0 г/л, предпочтительно больше примерно 2,0 г/л и более предпочтительно больше примерно 5,0 г/л. Однако при превышении определенных концентраций добавление фосфата в культуральную среду не способствует росту микроорганизмов. Соответственно, концентрация фосфата в культуральной среде обычно составляет менее примерно 20 г/л, предпочтительно менее примерно 15 г/л и более предпочтительно менее примерно 10 г/л.
[00156] Подходящая культуральная среда может также включать источник магния, предпочтительно в форме физиологически приемлемой соли, такой как гептагидрат сульфата магния, хотя можно использовать другие источники магния в концентрациях, которые вносят аналогичные количества магния. Обычно концентрация магния в культуральной среде больше примерно 0,5 г/л, предпочтительно больше примерно 1,0 г/л и более предпочтительно больше примерно 2,0 г/л. Однако при превышении определенных концентраций добавление магния в культуральную среду не способствует росту микроорганизмов. Соответственно, концентрация магния в культуральной среде обычно составляет менее примерно 10 г/л, предпочтительно менее примерно 5 г/л и более предпочтительно менее примерно 3 г/л. Кроме того, в некоторых случаях может быть желательно позволить культуральной среде истощиться от источника магния во время культивирования.
[00157] В некоторых вариантах осуществления культуральная среда может также включать биологически приемлемый хелатирующий агент, такой как дигидрат тринатрийцитрата. В таком случае концентрация хелатирующего агента в культуральной среде больше примерно 0,2 г/л, предпочтительно больше примерно 0,5 г/л и более предпочтительно больше примерно 1 г/л. Однако при превышении определенных концентраций добавление хелатирующего агента к культуральной среде не способствует росту микроорганизмов. Соответственно, концентрация хелатирующего агента в культуральной среде обычно составляет менее примерно 10 г/л, предпочтительно менее примерно 5 г/л и более предпочтительно менее примерно 2 г/л.
[00158] Культуральная среда может также изначально включать биологически приемлемую кислоту или основание для поддержания желаемого рН культуральной среды. Биологически приемлемые кислоты включают, но не ограничиваются ими, соляную кислоту, серную кислоту, азотную кислоту, фосфорную кислоту и их смеси. Биологически приемлемые основания включают, но не ограничиваются ими, гидроксид аммония, гидроксид натрия, гидроксид калия и их смеси. В некоторых вариантах осуществления используемое основание представляет собой гидроксид аммония.
[00159] Культуральная среда может также включать биологически приемлемый источник кальция, включая, но не ограничиваясь этим, хлорид кальция. Обычно концентрация источника кальция, такого как хлорид, дигидрат кальция, в культуральной среде находится в диапазоне от примерно 5 мг/л до примерно 2000 мг/л, предпочтительно в диапазоне от примерно 20 мг/л до примерно 1000 мг/л и более предпочтительно в диапазоне от примерно 50 мг/л до примерно 500 мг/л.
[00160] Питательная среда также может включать хлорид натрия. Обычно концентрация хлорида натрия в культуральной среде находится в диапазоне от примерно 0,1 г/л до примерно 5 г/л, предпочтительно в диапазоне от примерно 1 г/л до примерно 4 г/л, а более предпочтительно в диапазоне от примерно 2 г/л до примерно 4 г/л.
[00161] В некоторых вариантах осуществления культуральная среда может также включать следовые количества металлов. Такие следовые количества металлов могут быть добавлены в культуральную среду в виде исходного раствора, который для удобства может быть приготовлен отдельно от остальной культуральной среды. Обычно количество такого раствора микроэлементов металлов, добавляемого в культуральную среду, составляет более примерно 1 мл/л, предпочтительно более примерно 5 мл/л и более предпочтительно более примерно 10 мл/л. Однако при превышении определенных концентраций добавление следов металлов в культуральную среду не способствует росту микроорганизмов. Соответственно, количество такого раствора следов металлов, добавляемого в культуральную среду, обычно составляет менее примерно 100 мл/л, предпочтительно менее примерно 50 мл/л и более предпочтительно менее примерно 30 мл/л. Следует отметить, что, в дополнение к добавлению следов металлов в основной раствор, отдельные компоненты могут быть добавлены отдельно, каждый в пределах диапазонов, независимо от количества компонентов, продиктованных указанными выше диапазонами раствора следов металлов.
[00162] Культуральная среда может включать другие витамины, такие как пантотенат, биотин, кальций, пантотенат, инозит, пиридоксин-HCl и тиамин-HCl. Такие витамины могут быть добавлены в культуральную среду в виде исходного раствора, который для удобства может быть приготовлен отдельно от остальной культуральной среды. Однако при превышении определенных концентраций добавление витаминов в культуральную среду не способствует росту микроорганизмов.
[00163] Описанные здесь способы ферментации могут выполняться в обычных режимах культивирования, которые включают, но не ограничиваются, периодический, периодический процесс с подпиткой, рециркуляцию клеток, непрерывный и полунепрерывный. В некоторых вариантах осуществления ферментацию проводят в периодическом режиме с подпиткой. В таком случае некоторые компоненты среды истощаются во время культивирования, включая пантотенат во время производственной стадии ферментации. В некоторых вариантах осуществления в культуру могут быть добавлены относительно высокие концентрации таких компонентов в начале, например, стадии продуцирования, так что рост и/или продуцирование стевиоловых гликозидов поддерживается в течение периода времени, прежде чем потребуются добавления. Предпочтительные диапазоны этих компонентов поддерживаются на протяжении всей культуры путем внесения добавок по мере того, как уровни истощаются в культуре. Уровни компонентов в культуральной среде можно контролировать, например, путем периодического отбора проб культуральной среды и анализа концентраций. В качестве альтернативы, как только стандартная процедура культивирования разработана, добавления могут быть сделаны через определенные интервалы времени, соответствующие известным уровням, в определенные периоды времени в культуре. Как будет понятно специалистам в данной области, скорость потребления питательного вещества увеличивается во время культивирования по мере увеличения плотности клеток в среде. Более того, чтобы избежать попадания чужеродных микроорганизмов в культуральную среду, добавление проводят с использованием известных в данной области методов асептического добавления. Кроме того, во время культивирования можно добавить небольшое количество пеногасителя.
[00164] Температура культуральной среды может быть любой температурой, подходящей для роста генетически модифицированных клеток и/или продукции стевиолового гликозида. Например, перед инокуляцией культуральной среды инокулятом, культуральная среда может быть доведена до температуры в диапазоне от примерно 20°С до примерно 45°С, предпочтительно до температуры в диапазоне от примерно 25°С до примерно 40°С, и более предпочтительно в диапазоне от примерно 28°С до примерно 32°С.
[00165] рН культуральной среды можно контролировать путем добавления кислоты или основания в культуральную среду. В таких случаях, когда аммиак используется для регулирования рН, он также удобно служит источником азота в культуральной среде. Предпочтительно рН поддерживается от примерно 3,0 до примерно 8,0, более предпочтительно от примерно 3,5 до примерно 7,0 и наиболее предпочтительно от примерно 4,0 до примерно 6,5.
[00166] В некоторых вариантах осуществления концентрация источника углерода, такая как концентрация глюкозы в культуральной среде, отслеживается во время культивирования. Концентрацию глюкозы в культуральной среде можно контролировать с помощью известных методов, таких как, например, использование теста фермента глюкозооксидазы или жидкостной хроматографии высокого давления, которые можно использовать для мониторинга концентрации глюкозы в супернатанте, например, в бесклеточном компоненте питательной среды. Концентрация источника углерода обычно поддерживается ниже уровня, при котором происходит ингибирование роста клеток. Хотя такая концентрация может варьироваться от организма к организму, для глюкозы как источника углерода ингибирование роста клеток происходит при концентрациях глюкозы, превышающих примерно 60 г/л, и может быть легко определено испытанием. Соответственно, когда глюкоза используется в качестве источника углерода, глюкоза предпочтительно подается в ферментатор и поддерживается ниже пределов обнаружения. В качестве альтернативы, концентрация глюкозы в культуральной среде поддерживается в диапазоне от примерно 1 г/л до примерно 100 г/л, более предпочтительно в диапазоне от примерно 2 г/л до примерно 50 г/л, и наболее предпочтительно в диапазоне от примерно 5 г/л до примерно 20 г/л. Хотя концентрацию источника углерода можно поддерживать на желаемых уровнях путем добавления, например, по существу чистого раствора глюкозы, приемлемо и может быть предпочтительным поддержание концентрации источника углерода в культуральной среде путем добавления аликвот исходной питательной среды. Использование аликвот исходной культуральной среды может быть желательным, поскольку концентрации других питательных веществ в среде (например, источников азота и фосфата) могут поддерживаться одновременно. Аналогичным образом, концентрации следов металлов можно поддерживать в культуральной среде путем добавления аликвот раствора следовых количеств металлов.
[00167] Другие подходящие ферментационные среды и методы описаны, например, в WO 2016/196321.
6.8 Составы для ферментации
[00168] В другом аспекте в настоящем документе представлены составы для ферментации, содержащие генетически модифицированную клетку-хозяина, описанную в данном документе, и стевиоловые гликозиды, полученные из генетически модифицированной клетки-хозяина. Составы для ферментации могут дополнительно содержать среду. В некоторых вариантах осуществления составы для ферментации содержат генетически модифицированную клетку-хозяин и дополнительно содержат Reb A, Reb D и Reb М. В определенных вариантах осуществления ферментационные композиции, представленные в настоящем документе, содержат Reb М в качестве основного компонента стевиоловых гликозидов, полученных из генетически модифицированной клетки-хозяина. В некоторых вариантах осуществления составы для ферментации содержат Reb A, Reb D и Reb М в соотношении по меньшей мере 1:7:50. В некоторых вариантах осуществления составы для ферментации содержат Reb A, Reb D и Reb М в соотношении по меньшей мере от 1:7:50 до 1:100:1000. В некоторых вариантах осуществления составы для ферментации содержат соотношение по меньшей мере от 1:7:50 до 1:200:2000. В некоторых вариантах осуществления соотношение Reb A, Reb D и Reb М основано на общем содержании стевиоловых гликозидов, связанных с генетически модифицированной клеткой-хозяином и средой. В некоторых вариантах осуществления соотношение Reb A, Reb D и Reb М основано на общем содержании стевиоловых гликозидов в среде. В некоторых вариантах осуществления соотношение Reb A, Reb D и Reb М основано на общем содержании стевиоловых гликозидов, связанных с генетически модифицированной клеткой-хозяином.
[00169] В некоторых вариантах осуществления ферментационные композиции, представленные в настоящем документе, содержат Reb М2 на неопределяемом уровне. В некоторых вариантах осуществления составы для ферментации, представленные в настоящем документе, содержат неприродные стевиоловые гликозиды в неопределяемом уровне.
6.9 Восстановление стевиоловых гликозидов
[00170] Как только стевиоловый гликозид продуцируется клеткой-хозяином, он может быть выделен или изолирован для последующего использования с использованием любых подходящих способов разделения и очистки, известных в данной области. В некоторых вариантах осуществления осветленную водную фазу, содержащую стевиоловый гликозид, отделяют от ферментации центрифугированием. В других вариантах осуществления осветленная водная фаза, содержащая стевиоловый гликозид, отделяется от ферментации путем добавления деэмульгатора в реакцию ферментации. Наглядные примеры деэмульгаторов включают флокулянты и коагулянты.
[00171] Стевиоловый гликозид, продуцируемый в этих клетках, может присутствовать в супернатанте культуры и/или быть связанным с клетками-хозяевами. В вариантах осуществления, где часть стевиоловых гликозидов связана с клеткой-хозяином, выделение стевиоловых гликозидов может включать способ улучшения высвобождения стевиоловых гликозидов из клеток. В некоторых вариантах осуществления это может принимать форму промывки клеток горячей водой или буферной обработки, с поверхностно-активным веществом или без него, а также с добавлением буферов или солей или без них. В некоторых вариантах осуществления температура представляет собой любую температуру, которая считается подходящей для высвобождения стевиоловых гликозидов. В некоторых вариантах осуществления температура находится в диапазоне от 40 до 95°С; или от 60 до 90°С; или от 75 до 85°С. В некоторых вариантах осуществления температура составляет 40, 45, 50, 55, 65, 70, 75, 80, 85, 90 или 95°С. В некоторых вариантах осуществления изобретения физическое или химическое разрушение клетки используется для увеличения высвобождения стевиоловых гликозидов из клетки-хозяина. Альтернативно и/или впоследствии стевиоловый гликозид в культуральной среде может быть извлечен с использованием операций блока выделения, включая, помимо прочего, экстракцию растворителем, очистку мембраны, концентрирование мембраны, адсорбцию, хроматографию, выпаривание, химическую дериватизацию, кристаллизацию и сушку.
6.10 Способы создания генетически модифицированных клеток
[00172] В настоящем документе также представлены способы получения клетки-хозяина, которая была создана с помощью генной инженерии и содержала одну или несколько модификаций, описанных выше, например, одну или несколько нуклеиновых гетерологичных нуклеиновых кислот, кодирующих гидроксилазу кауреновой кислоты Stevia rebaudiana, и/или ферменты биосинтетического пути, например, для соединения стевиоловых гликозидов. Экспрессия гетерологичного фермента в клетке-хозяине может быть достигнута путем введения в клетки-хозяева нуклеиновой кислоты, содержащей нуклеотидную последовательность, кодирующую фермент, под контролем регуляторных элементов, которые обеспечивают экспрессию в клетке-хозяине. В некоторых вариантах осуществления нуклеиновая кислота представляет собой внехромосомную плазмиду. В других вариантах осуществления нуклеиновая кислота представляет собой вектор хромосомной интеграции, который может интегрировать нуклеотидную последовательность в хромосому клетки-хозяина. В других вариантах осуществления нуклеиновая кислота представляет собой линейный фрагмент двухцепочечной ДНК, которая может интегрировать посредством гомологии нуклеотидную последовательность в хромосому клетки-хозяина.
[00173] Нуклеиновые кислоты, кодирующие эти белки, могут быть введены в клетку-хозяина любым способом, известным специалисту в данной области, без ограничения (см., например, Hinnen et al. (1978) Proc. Национальной академии наук USA 75:1292-3; Cregg et al. (1985) Mol. Cell. Biol. 5:3376-3385; Goeddel et al. eds, 1990, Methods in Enzymology (Методы в энзимологии), т.185, Academic Press, Inc., CA; Krieger, 1990, Gene Transfer and Expression ~ A Laboratory Manual (Перенос и экспрессия генов - лабораторное руководство), Stockton Press, NY; Sambrook et al., 1989, Molecular Cloning - A Laboratory Manual (Молекулярное клонирование - лабораторное руководство), Cold Spring Harbor Laboratory, NY; and Ausubel et at, eds., Current Edition, Current Protocols in Molecular Biology, Greene Publishing Associates and Wiley Interscience, NY). Примеры методик включают, но не ограничиваются ими, сферопластинг, электропорацию, трансформацию, опосредованную PEG 1000, и трансформацию, опосредованную ацетатом лития или хлоридом лития.
[00174] Количество фермента в клетке-хозяине может быть изменено путем изменения транскрипции гена, кодирующего фермент. Это может быть достигнуто, например, путем изменения числа копий нуклеотидной последовательности, кодирующей фермент (например, путем использования вектора экспрессии с большим или меньшим числом копий, содержащего нуклеотидную последовательность, или путем введения дополнительных копий нуклеотидной последовательности в геном клетки-хозяина, или путем удаления или нарушения нуклеотидной последовательности в геноме клетки-хозяина), путем изменения порядка кодирующих последовательностей в полицистронной мРНК оперона или разбиения оперона на отдельные гены, каждый из которых имеет свои собственные элементы управления, или путем увеличения силы промотора или оператора, с которым операбельно связана нуклеотидная последовательность. В качестве альтернативы или в дополнение, количество копий фермента в клетке-хозяине может быть изменено путем изменения уровня трансляции мРНК, кодирующей фермент.Этого можно достичь, например, изменяя стабильность мРНК, изменяя последовательность сайта связывания рибосомы, изменяя расстояние или последовательность между сайтом связывания рибосомы и стартовым кодоном кодирующей последовательности фермента, изменяя всю межцистронную область, расположенную "выше" или рядом с 5'-стороной стартового кодона кодирующей области фермента, стабилизируя З'-конец транскрипта мРНК с помощью шпилек и специализированных последовательностей, изменяя использование кодонов фермента, изменяя экспрессию используемых тРНК редких кодонов в биосинтезе фермента и/или повышении стабильности фермента, например, посредством мутации его кодирующей последовательности.
[00175] Активность фермента в клетке-хозяине может быть изменена различными способами, включая, в частности, экспрессию модифицированной формы фермента, обладающей повышенной или пониженной растворимостью в клетке-хозяине, экспрессию измененной формы фермента, в которой отсутствует домен, через который ингибируется активность фермента, экспрессия измененной формы фермента, которая имеет более высокий или более низкий Kcat или более низкий или более высокий Km для субстрата, или экспрессия измененной формы фермента, которая в большей или меньшей степени подвержена обратному или прямому регулированию со стороны другой молекулы в пути.
[00176] В некоторых вариантах осуществления нуклеиновая кислота, используемая для генетической модификации клетки-хозяина, содержит один или несколько селектируемых маркеров, полезных для отбора трансформированных клеток-хозяев и для оказания селективного давления на клетку-хозяина для сохранения чужеродной ДНК.
[00177] В некоторых вариантах осуществления селектируемый маркер представляет собой маркер устойчивости к антибиотикам. Иллюстративные примеры маркеров устойчивости к антибиотикам включают, но не ограничиваются ими, продукты генов BLA, NAT], PAT, AUR1-C, PDR4, SMR1, CAT, мышиного dhfr, HPH, DSDA, KANR и SH BLE. Продукт гена BLA из E.coli придает устойчивость к бета-лактамным антибиотикам (например, цефалоспоринам узкого спектра действия, цефамицинам и карбапенемам (эртапенем), цефамандолу и цефоперазону) и ко всем антиграмм-отрицательным бактериальным пенициллинам, кроме темоциллина; продукт гена NAT1 из S. noursei придает устойчивость к норсеотрицину; продукт гена PAT из S. viridochromogenes Ти94 придает устойчивость к биалофосу; продукт гена AURI-C из Saccharomyces cerevisiae придает устойчивость к Auerobasidin А (АЬА); продукт гена PDR4 придает устойчивость к церуленину; продукт гена SMR1 придает устойчивость к сульфометуронметилу; продукт гена CAT из транспозона Тп9 придает устойчивость к хлорамфениколу; продукт гена dhfr мыши придает устойчивость к метотрексату; продукт гена HPH Klebsiella pneumonia придает устойчивость к гигромицину В; продукт tqu&DSDA Е. coli позволяет клеткам расти на чашках с D-серином в качестве единственного источника азота; ген KANR транспозона Тп903 придает устойчивость к G418; а продукт гена SHBLE из Streptoalloteichus hindustanus придает устойчивость к зеоцину (блеомицину). В некоторых вариантах осуществления маркер устойчивости к антибиотикам удаляется после выделения генетически модифицированной клетки-хозяина, описанной в настоящем документе.
[00178] В некоторых вариантах осуществления селектируемый маркер устраняет ауксотрофию (например, пищевую ауксотрофию) у генетически модифицированного микроорганизма. В таких вариантах осуществления родительский микроорганизм включает функциональное нарушение в одном или нескольких генных продуктах, которые функционируют в пути биосинтеза аминокислот или нуклеотидов, и когда не функциональность делает родительскую клетку неспособной к росту в среде без добавления одного или нескольких питательных веществ. Такие генные продукты включают, но не ограничиваются ими, генные продукты HIS3, LEU2, LYS1, LYS2, МЕТ 15, TRP1, ADE2 и URA3 в дрожжах. Затем ауксотрофный фенотип может быть спасен путем трансформации родительской клетки экспрессирующим вектором или конструкцией хромосомной интеграции, кодирующей функциональную копию разрушенного генного продукта, и генерируемая генетически модифицированная клетка-хозяин может быть выбрана на основе потери ауксотрофного фенотипа родительская ячейка. Использование генов URA3, TRP1 и LYS2 в качестве селектируемых маркеров имеет заметное преимущество, поскольку возможны как положительный, так и отрицательный отбор. Положительный отбор осуществляется ауксотрофной комплементацией мутаций URA3, TRP1 и LYS2, тогда как отрицательный отбор основан на специфических ингибиторах, например, 5-фтороротовой кислоте (FOA), 5-фторантраниловой кислоте и аминоадипиновой кислоте (аАА), соответственно, которые предотвращают рост прототрофных штаммов, но допускают рост мутантов URA3, TRP1 и LYS2 соответственно. В других вариантах осуществления селектируемый маркер устраняет другие нелетальные недостатки или фенотипы, которые можно идентифицировать известным методом селекции.
[00179] В данном документе описаны конкретные гены и белки, которые можно использовать в способах, композициях и организмах согласно настоящему раскрытию; однако будет признано, что абсолютная идентичность таких генов не является необходимой. Например, могут быть выполнены изменения в конкретном гене или полинуклеотиде, включающем последовательность, кодирующую полипептид или фермент, и проведен скрининг на активность. Обычно такие изменения включают консервативные мутации и молчащие мутации. Такие модифицированные или мутированные полинуклеотиды и полипептиды могут быть проверены на экспрессию функционального фермента с использованием способов, известных в данной области. [00180] Из-за присущей вырожденности генетического кода другие полинуклеотиды, которые кодируют по существу такие же или функционально эквивалентные полипептиды, также могут быть использованы для клонирования и экспрессии полинуклеотидов. кодирующих такие ферменты.
[00181] Как будет понятно специалистам в данной области, может быть выгодно модифицировать кодирующую последовательность для усиления ее экспрессии в конкретном хозяине. Генетический код дублируется 64 возможными кодонами, но большинство организмов обычно используют подмножество этих кодонов. Кодоны, которые чаще всего используются у вида, называются оптимальными кодонами, а те, которые не используются очень часто, классифицируются как редкие или редко используемые кодоны. Кодоны могут быть заменены, чтобы отразить предпочтительное использование кодонов хозяином, в процессе, который иногда называют «оптимизацией кодонов» или «контролем смещения кодонов вида». Оптимизация кодонов для других клеток-хозяев может быть легко определена с использованием таблиц использования кодонов или может быть выполнена с использованием коммерчески доступного программного обеспечения, такого как CodonOp (www.idtdna.com/CodonOptfrom) от Integrated DNA Technologies.
[00182] Оптимизированные кодирующие последовательности, содержащие кодоны, предпочтительные для конкретного прокариотического или эукариотического хозяина (Murray et al., 1989, Nucl Acids Res. 17:477-508), могут быть получены, например, для увеличения скорости трансляции или для получения рекомбинантных РНК-транскриптов с желательными свойствами, такими как более длительный период полураспада, по сравнению с транскриптами, полученными из неоптимизированной последовательности. Стоп-кодоны трансляции также могут быть изменены для отражения предпочтений хозяина. Например, типичными стоп-кодонами для S. cerevisiae и млекопитающих являются UAA и UGA, соответственно. Типичным стоп-кодоном для однодольных растений является UGA, тогда как насекомые и Е. coli обычно используют UAA в качестве стоп-кодона (Dalphin et at, 1996, Nucl Acids Res. 24: 216-8).
[00183] Специалисты в данной области поймут, что из-за вырожденной природы генетического кода множество молекул ДНК, различающихся своими нуклеотидными последовательностями, можно использовать для кодирования данного фермента согласно описанию изобретения. Нативная последовательность ДНК, кодирующая биосинтетические ферменты, описанные выше, упоминается здесь просто для иллюстрации варианта осуществления настоящего описания, и это описание включает молекулы ДНК любой последовательности, которые кодируют аминокислотные последовательности полипептидов и белков ферментов, используемых в способах описания изобретения. Подобным образом полипептид обычно может переносить одну или несколько аминокислотных замен, делеций и вставок в своей аминокислотной последовательности без потери или значительной потери желаемой активности. Описание включает такие полипептиды с аминокислотными последовательностями, отличными от описанных здесь конкретных белков, при условии, что модифицированные или вариантные полипептиды обладают ферментативной анаболической или катаболической активностью эталонного полипептида. Кроме того, аминокислотные последовательности, кодируемые последовательностями ДНК, показанными в данном документе, просто иллюстрируют варианты осуществления настоящего описания.
[00184] Кроме того, описание охватывает гомологи ферментов, применимых для композиций и способов, представленных в настоящем документе. В некоторых вариантах осуществления два белка (или область белков) по существу гомологичны, если аминокислотные последовательности содержат по меньшей мере примерно 30%, 40%, 50%, 60%, 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичных областей. Чтобы определить процент идентичности двух аминокислотных последовательностей или двух последовательностей нуклеиновых кислот, последовательности выравнивают для целей оптимального сравнения (например, могут быть введены пробелы в одной или обеих из первой и второй аминокислотной или нуклеиновой последовательностей для оптимального выравнивания и негомологичные последовательности можно не принимать во внимание для целей сравнения). В одном варианте осуществления длина эталонной последовательности, выровненной для целей сравнения, составляет по меньшей мере 30%, обычно по меньшей мере 40%, более типично по меньшей мере 50%, еще более типично по меньшей мере 60% и даже более типично по меньшей мере 70%, 80%, 90%, 100% длины эталонной последовательности. Затем сравнивают аминокислотные остатки или нуклеотиды в соответствующих положениях аминокислот или положениях нуклеотидов. Если положение в первой последовательности занято тем же аминокислотным остатком или нуклеотидом, что и соответствующее положение во второй последовательности, то молекулы идентичны в этом положении (в данном случае "идентичность" аминокислот или нуклеиновых кислот эквивалентна "гомологии" аминокислот или нуклеиновых кислот). Процент идентичности между двумя последовательностями является функцией количества идентичных положений, общих для последовательностей, с учетом количества пробелов и длины каждого пробела, которые необходимо ввести для оптимального выравнивания двух последовательностей.
[00185] Когда термин «гомологичный» используется в отношении белков или пептидов, считается, что неидентичные положения остатков часто отличаются консервативными аминокислотными заменами. «Консервативная аминокислотная замена» представляет собой замену, при которой аминокислотный остаток заменен другим аминокислотным остатком, имеющим боковую цепь (R-группу) с аналогичными химическими свойствами (например, зарядом или гидрофобностью). Как правило, консервативная аминокислотная замена существенно не изменяет функциональные свойства белка. В случаях, когда две или более аминокислотных последовательностей отличаются друг от друга консервативными заменами, процент идентичности последовательностей или степень гомологии может быть увеличена для корректировки консервативного характера замены. Способы выполнения этой регулировки хорошо известны специалистам в данной области (см., например, Pearson W.R., 1994, Methods inMol Biol 25: 365-89).
[00186] Каждая из следующих шести групп содержит аминокислоты, которые являются консервативными заменами друг друга: 1) серии (S), треонин (Т); 2) аспарагиновая кислота (D), глутаминовая кислота (Е); 3) аспарагин (N), глутамин (Q); 4) аргинин (R), лизин (К); 5) изолейцин (I), лейцин (L), аланин (А), валин (V) и 6) фенилаланин (F), тирозин (Y), триптофан (W).
[00187] Гомология последовательностей полипептидов, которую также называют процентной идентичностью последовательностей, обычно измеряется с использованием программного обеспечения для анализа последовательностей. Типичным алгоритмом, используемым для сравнения последовательности молекулы с базой данных, содержащей большое количество последовательностей от разных организмов, является компьютерная программа BLAST. При поиске в базе данных, содержащей последовательности от большого числа различных организмов, обычно сравнивают аминокислотные последовательности.
[00188] Кроме того, любой из генов, кодирующих вышеупомянутые ферменты (или любые другие, упомянутые здесь (или любой из регуляторных элементов, которые контролируют или модулируют их экспрессию)), можно оптимизировать методами генетической/белковой инженерии, такими как направленная эволюция или рациональный мутагенез, которые известны специалистам в данной области техники. Такое действие позволяет специалистам в данной области оптимизировать экспрессию и активность ферментов в дрожжах.
[00189] Кроме того, гены, кодирующие эти ферменты, могут быть идентифицированы от других видов грибов и бактерий и могут быть экспрессированы для модуляции этого пути. Различные организмы могут служить источниками этих ферментов, включая, помимо прочего, Saccharomyces spp., включая S. cerevisiae и S. uvarum, Kluyveromyces spp., включая К. thermotolerans, К. lactis и К. marxianus, Pichia spp., Hansenula spp., включая H. polymorpha, Candida spp., Trichosporon spp., Yamadazyma spp., включая Y. spp.stipitis, Torulasporapretoriensis, Issatchenkia orientalis, Schizosaccharomyces spp., включая S. pombe, Cryptococcus spp., Aspergillus spp., Neurospora spp.или Ustilago spp.Источники генов анаэробных грибов включают, но не ограничиваются ими, Piromyces spp., Orpinomyces spp.или Neocallimastix spp.Источники прокариотических ферментов, которые можно использовать, включают, но не ограничиваются ими, Escherichia coli, Zymomonas mobilis, Staphylococcus aureus, Bacillus spp., Clostridium spp., Cory neb acterium spp., Pseudomonas spp., Lactococcus spp., Enterobacter spp.и Salmonella spp.
[00190] Методы, известные специалистам в данной области, могут быть подходящими для идентификации дополнительных гомологичных генов и гомологичных ферментов. Как правило, аналогичные гены и/или аналогичные ферменты можно идентифицировать с помощью функционального анализа, и они будут иметь функциональное сходство. Методы, известные специалистам в данной области, могут быть подходящими для идентификации аналогичных генов и аналогичных ферментов. Например, для идентификации гомологичных или аналогичных UDP-гликозилтрансфераз, КАН или любых генов, белков или ферментов биосинтетического пути методы могут включать, но не ограничиваются ими, клонирование гена с помощью ПЦР с использованием праймеров на основе опубликованной последовательности гена/представляющий интерес фермент, или с помощью вырожденной ПЦР с использованием вырожденных праймеров, предназначенных для амплификации консервативной области среди представляющего интерес гена. Кроме того, специалист в данной области может использовать методы для идентификации гомологичных или аналогичных генов, белков или ферментов с функциональной гомологией или сходством. Методы включают исследование клетки или клеточной культуры на каталитическую активность фермента с помощью ферментных анализов in vitro на указанную активность (например, как описано здесь или в Kiritani, К., Branched-Chain Amino Acids Methods Enzymology (Методы этимологии для аминокислот с разветвленной цепью), 1970), затем выделение фермента с указанной активностью посредством очистки, определения белковой последовательности фермента с помощью таких методов, как деградация по Эдману, конструирование праймеров ПЦР для вероятной последовательности нуклеиновой кислоты, амплификация указанной последовательности ДНК посредством ПЦР и клонирование указанной последовательности нуклеиновой кислоты. Для идентификации гомологичных или подобных генов и/или гомологичных или подобных ферментов, аналогичных генов и/или аналогичных ферментов или белков методы также включают сравнение данных, касающихся гена или фермента-кандидата, с такими базами данных, как BRENDA, KEGG или MetaCYC. Ген или фермент-кандидат могут быть идентифицированы в вышеупомянутых базах данных в соответствии с изложенными здесь идеями.
7. ПРИМЕРЫ
Пример 1. Методы трансформации дрожжей
[00191] Каждая конструкция ДНК была интегрирована в Saccharomyces cerevisiae (CEN.PK2) с использованием стандартных методов молекулярной биологии для оптимизированной трансформации ацетатом лития. Вкратце, клетки выращивали в течение ночи в среде дрожжевого экстракта с пептон-декстрозой (YPD) при 30°С при встряхивании (200 об/мин), разбавляли до OD600 0,1 в 100 мл YPD и выращивали до OD600 0,6-0,8. Для каждой трансформации 5 мл культуры собирали центрифугированием, промывали в 5 мл стерильной воды, снова центрифугировали, ресуспендировали в 1 мл 100 мМ ацетата лития и переносили в микроцентрифужную пробирку. Клетки центрифугировали (13000 g) в течение 30 секунд, супернатант удаляли и клетки ресуспендировали в смеси для трансформации, состоящей из 240 мкл 50% ПЭГ, 36 мкл 1 М ацетата лития, 10 мкл вареной ДНК спермы лосося и 74 мкл донорской ДНК. Донорская ДНК включала плазмиду, несущую ген эндонуклеазы F-CphI, экспрессируемую для экспрессии под дрожжевым промотором TDH3 (см. пример 4). После теплового шока при 42°С в течение 40 минут клетки выделяли в течение ночи в среде YPD, содержащей соответствующий антибиотик, для отбора клеток, которые поглотили плазмиду F-Cphl. После восстановления в течение ночи клетки ненадолго центрифугировали и высевали на среду YPD, содержащую соответствующий антибиотик, для отбора клеток, которые поглотили плазмиду F-Cphl. Интеграцию ДНК подтверждали с помощью ПЦР колоний с праймерами, специфичными для интеграции.
Пример 2: Получение основного штамма дрожжей, способного к высокой гидравлической проницаемости к фарнезилпирофосфату (FPP) и изопреноиду фарнезену
[00192] Штамм, продуцирующий фарнезен, был создан из штамма Saccharomyces cerevisiae дикого типа (CEN.PK2) путем экспрессии генов мевалонатного пути под контролем промоторов GAL1 или GAL10. Этот штамм включал следующие хромосомно интегрированные гены мевалонатного пути из S. cerevisiae: ацетил-КоА тиолаза ГМГ-КоА синтаза, ГМГ-КоА редуктаза мевалонаткиназа, фосфомевалонаткиназа, мевалонатпирофосфатдекарбоксилаза и IPP:DMAPP изомераза. Кроме того, штамм содержал множество копий фарнезен-синтазы из Artemisia annua, также под контролем промоторов GAL1 или GAL10. Все описанные здесь гетерологичные гены были оптимизированы по кодонам с использованием общедоступных или других подходящих алгоритмов. Штамм также содержал делецию гена GAL80, а ген ERG9, кодирующий сквален-синтазу, был понижен путем замены нативного промотора на промотор дрожжевого гена МЕТЗ (Westfall и др., Труды Национальной академии наук США 109(3), 2012, стр. Е111-Е118). Примеры того, как создать штаммы S. cerevisiae с высокой гидравлической проницаемостью к изопреноидам, описаны в патенте США №8,415,136 и патенте США №8,236,512, которые полностью включены в настоящий документ.
Пример 3. Получение базового штамма дрожжей, способного к высокой гидравлической проницаемости к Reb М
[00193] На ФИГ. 1 показан пример пути биосинтеза от FPP к стевиолу. На ФИГ.2 показан примерный биосинтетический путь от стевиола до гликозида Reb М. Чтобы преобразовать штамм на основе фарнезена, описанный выше, в тот, который иметь высокую гидравлическую проницаемость к изопреноид каурену С20, в геном были интегрированы четыре копии геранилгеранилпирофосфатсинтазы (GGPPS), за которыми следовали две копии копалилдифосфатсинтазы и одна копия кауренсинтазы. На этом этапе все копии фарнезенсинтазы были удалены из штамма. Как только было подтверждено, что новый штамм вырабатывает энт-каурен, оставшиеся гены для превращения энт-каурена в Reb М были вставлены в геном. В таблице 1 перечислены все гены и промоторы, используемые для преобразования FPP в Reb М. Каждый ген после кауренсинтазы был интегрирован в виде единой копии, за исключением фермента Sr.KAH, для которого были интегрированы две копии гена. Штамм, содержащий все гены, описанные в таблице 1, в первую очередь продуцировал Reb М.
Таблица 1. Гены, промоторы и аминокислотные последовательности ферментов, используемых для преобразования FPP в RebM
Первые 65 аминокислот удалены и заменены метионином
Пример 4. Создание штамма для скрининга переносчиков стевиоловых гликозидов.
[00194] Для быстрого скрининга переносчиков стевиоловых гликозидов in vivo в штамме, продуцирующем Reb М, в описанный выше штамм вставляли посадочную площадку. Посадочная площадка состояла из 500 п. н. последовательностей ДНК, нацеленных на локус, на обоих концах конструкции в геномной области ниже открытой рамки считывания SFM1 (см. Фиг. 3). Внутри посадочная площадка содержала промотор GAL1 и терминатор дрожжей, фланкирующий сайт узнавания эндонуклеазы (F-Cphl).
Пример 5: Условия культивирования дрожжей
[00195] Колонии дрожжей со сверхэкспрессированным белком-переносчиком собирали в 96-луночные микротитрационные планшеты, содержащие среду Bird Seed Media (BSM, первоначально описанную van Hoek et al., Biotechnology andBioengineering (Биотехнология и биоинженерия) 68 (5), 2000, pp.517-523) с 20 мкг/л сахарозы, 3,75 г/л сульфата аммония и 1 г / л лизина. Клетки культивировали при 28°С в инкубаторе для микротитровальных планшетов с высокой емкостью, встряхивая при 1000 об/мин и влажности 80% в течение 3 дней, пока культуры не достигли исчерпания углерода. Насыщенные для роста культуры пересевали в свежие чашки, содержащие BSM с 40 г/л сахарозы и 3,75 г/л сульфата аммония, взяв 14,4 мкл из насыщенных культур и разбавив 360 мкл свежей среды. Клетки в продукционной среде культивировали при 30°С в высокопроизводительном шейкере для микротитровальных планшетов при 1000 об/мин и влажности 80% в течение дополнительных 3 дней перед экстракцией и анализом.
Пример 6. Условия приготовления пробы цельноклеточного бульона для анализа стевиоловых гликозидов
[00196] Для анализа количества всех стевиоловых гликозидов, продуцируемых в культуре, по завершении культивирования бульон для цельных клеток разбавляли 628 мкл 100% этанола, герметично закрывали фольгой и встряхивали при 1250 об / мин в течение 30 секунд для экстракции стевиоловых гликозидов. 314 мкл воды добавляли непосредственно в каждую лунку для разбавления экстракта. Планшет быстро центрифугировали для получения осадка. Внутренний стандарт, 208 мкл смеси этанол: вода 50:50, содержащей 0,48 мг / л ребаудиозида N, переносили в новый аналитический планшет объемом 250 мкл и в аналитический планшет добавляли 2 мкл смеси культура / этанол. Перед анализом планшет закрывали фольгой.
Пример 7. Условия приготовления пробы супернатанта культуры для анализа стевиоловых гликозидов
[00197] Для анализа количества всех продуцируемых и экскретируемых в культуральную среду стевиоловых гликозидов после завершения культивирования бульон целых клеток центрифугировали в течение 5 минут при 2000х g для осаждения клеток. Аликвоту 240 мкл полученного супернатанта переносили в пустой 96-луночный планшет для микротитрования. Образцы супернатанта разбавляли 480 мкл 100% этанола, герметично закрывали фольгой и встряхивали при 1250 об/мин в течение 30 секунд для экстракции стевиоловых гликозидов. Для разбавления экстракта в каждую лунку добавляли 240 мкл воды. Планшет быстро центрифугировали для осаждения любых твердых частиц. Внутренний стандарт, 208 мкл смеси этанол: вода 50:50, содержащей 0,48 мг/л ребаудиозида N, переносили в новый аналитический планшет объемом 250 мкл и в аналитический планшет добавляли 2 мкл смеси культура / этанол. Перед анализом планшет закрывали фольгой.
Пример 8: Аналитические методы
[00198] Образцы для измерения стевиоловых гликозидов анализировали с помощью масс-спектрометра (Agilent 6470-QQQ) с автоматическим пробоотборником системы RapidFire 365 с картриджем С8, используя конфигурации, показанные в таблицах 2 и 3.
Таблица 2. Конфигурация системы RapidFire 365
Таблица 3. Конфигурации метода 6470-QQQ MS
[00199] Площади пиков из хроматограммы масс-спектрометра использовали для построения калибровочной кривой. Молярные соотношения соответствующих соединений определяли путем количественного определения количества в молях каждого соединения посредством внешней калибровки с использованием аутентичного стандарта, а затем выбирая соответствующие соотношения.
Пример 9. Скрининг транспортеров, способных увеличивать титры стевиоловых гликозидов in vivo
[00200] В штамме, продуцирующем Reb М, без экспрессии дополнительных переносчиков, приблизительно 80% стевиоловых гликозидов с более высокой молекулярной массой Reb D и Reb М были связаны с биомассой (см. Фиг. 4). Эта ассоциация биомассы, вероятно, связана с тем, что Reb D и Reb М не эффективно транспортируются из клетки и удерживаются в цитоплазме. Накопление Reb D и Reb М может привести к ингибированию продукта, что уменьшит поток углерода через метаболический путь стевиоловых гликозидов. Следовательно, ожидается, что экспрессия одного или нескольких транспортеров, которые будут транспортировать стевиоловые гликозиды (особенно Reb D и Reb М) из цитоплазмы в среду (супернатант), ослабит ингибирование продукта и, таким образом, увеличит поток углерода через этот путь, что приведет к увеличению титры стевиоловых гликозидов. Чтобы идентифицировать переносчики, способные экспортировать стевиоловые гликозиды с более высокой молекулярной массой из клетки и, таким образом, снимать ингибирование продукта, мы проверили ряд переносчиков, идентифицированных из различных грибов, на способность увеличивать общие титры стевиоловых гликозидов, особенно титр гликозидов с более высокой молекулярной массой (т.е. Reb D и Reb М).
[00201] Все белки, аннотированные как переносчики из генома S. cerevisiae. амплифицировали с помощью ПЦР с использованием CEN.PK2 в качестве источника геномной ДНК. Каждый праймер для ПЦР имел 40 п. н. фланкирующей гомологии к PGAL1 и последовательностям ДНК терминатора дрожжей в посадочной площадке (см. Фиг. 3), добавленным к концам для облегчения гомологичной рекомбинации амплифицированного гена в посадочную площадку. В дополнение к скринингу всех эндогенных транспортных белков S. cerevisiae, обнаруженных в CEN.PK2, был проведен расширенный биоинформатический поиск белков-переносчиков ABC из небольшого числа грибов и дополнительных штаммов S. cerevisiae.
[00202] Чтобы создать библиотеку грибковых АВС-транспортеров, мы сначала получили аминокислотные последовательности из публикации Ковальчука и Дриссена «Филогенетический анализ грибковых АВС-транспортеров» (Ковальчук и Дриссен, ВМС Genomics, 11, 2010, стр. 177-197) в Филогенетический анализ АВС-транспортеров проведен для 27 видов грибов. Из этого литературного источника было выбрано всего 610 аминокислотных последовательностей, которые включали все переносчики, обозначенные как принадлежащие подсемействам АВС-С, ABC-D и ABC-G. Затем мы разработали собственные базы данных BLAST для следующих грибов: (1) Hansenula polymorpha DL-1 (NRRL-Y-7560), (2) Yarrowia lipolytica ATCC 18945, (3)Arxula adeninivorans ATCC 76597, (4)5'. cerevisiae CAT-1, (5) Lipomyces starkeyi ATCC 58690, (6) Kluyveromyces marxianus, (7) Kluyveromyces marxianus DMKU3-1042, (8) Komagataella phaffii NRRL Y-11430, (9) S. cerevisiae MBG3370, (10) S. cerevisiae MBG3373, (11) Kluyveromyces lactis ATCC 8585, (12) Candida utilis ATCC 22023, (13) Pichia pastoris ATCC 28485 и (14) Aspergillus oryzae NRRL5590.
[00203] Для организмов, у которых у нас уже были собственные нуклеотидные последовательности ORF, полученные в результате усилий по геномному секвенированию, сборке и аннотации de novo, мы применили tBLASTn с помощью Biopython. Алгоритм tBLASTn позволял быстро выравнивать белковые последовательности (в данном случае 610 семенных последовательностей от Ковальчука и Дриссена (ВМС Genomics, 11, 2010, стр. 177-197)) с транслированной ДНК нуклеотидных последовательностей ORF для каждого организма во всех шести возможных рамках считывания с использованием BLAST. Параметры tBLASTn были стандартными с evalue=1 е"25 (см. таблицу 4). Все вычисления выполнялись через API-интерфейс biopython (версия 1.70, загруженная из PyPI) с использованием Python 2.7.12 и Ubuntu 16.04.5 LTS (GNU/Linux 4.4.0-138-generic х86_64). Совпадения были впоследствии отфильтрованы, чтобы обеспечить глобальное выравнивание не менее 2000 нуклеотидов. Все совпадения, соответствующие этим критериям, переводились на следующий этап рабочего процесса.
Таблица 4. Параметры tBLASTn по умолчанию
[00204] Для остальных организмов, для которых не было собственной геномной последовательности, весь протеом организма был получен от Uniprot с использованием Uniprot API, чтобы создать базу данных для поиска BLASTp.В большинстве случаев в Uniprot была точная запись для вида, для которого у нас была собственная геномная ДНК, но в других случаях было близкое, но не точное совпадение с собственными штаммами грибов. В последних случаях мы полагались на высокую вероятность того, что последовательности генов будут достаточно похожи, чтобы праймеры, разработанные для сравнения Uniprot, по-прежнему амплифицировали бы внутреннюю геномную ДНК. Затем мы применили BLASTp с помощью Biopython к базе данных, производной от Uniprot. Параметры BLAST были стандартными, с оценкой=0,001 (см. Таблицу 5). Последующая фильтрация была выполнена на основе процентного отсечения идентичности>40% и процентного отсечения выровненной длины>60%. Все вычисления выполнялись через API-интерфейс biopython (версия 1.70, загруженная из PyPI) с использованием Python 2.7.12 и Ubuntu 16.04.5 LTS (GNU/Linux 4.4.0-138-generic х86 64). Совпадения должны соответствовать по крайней мере одной из 610 исходных последовательностей из ссылки. Затем совпадения были преобразованы в нуклеотидную последовательность с помощью службы сопоставления идентификаторов Uniprot ID в идентификаторы EMBL. Европейская лаборатория молекулярной биологии позволяет извлекать нуклеотидные последовательности из записи Uniprot. Мы использовали любые обращения, соответствующие этим критериям, на следующем этапе рабочего процесса.
Таблица 5. Параметры BLASTp по умолчанию
[00205] После того, как все нуклеотидные последовательности были идентифицированы, были сконструированы праймеры для амплификации каждой полной ORF с помощью ПЦР. Каждый праймер для ПЦР имел 40 п. н. фланкирующей гомологии к PGAL1 и последовательностям ДНК терминатора дрожжей в посадочной площадке (Фиг. 3), добавленным к концам для облегчения гомологичной рекомбинации амплифицированного гена в посадочную площадку. Каждый ген транспортера был индивидуально трансформирован в виде единственной копии в штамм дрожжей, продуцирующих Reb М, описанный выше, и подвергнут скринингу на способность увеличивать титры продукта при сверхэкспрессии in vivo.
Пример 11: Сверхэкспрессия транспортеров, приводящая к увеличению продукции стевиоловых гликозидов in vivo
[00206] Скрининг транспортеров S. cerevisiae in vivo обнаружил восемь транспортеров, которые статистически увеличивали продукцию общего стевиолгликозида (TSG) при сверхэкспрессии, по сравнению с родительским штаммом Reb М, который не содержал сверхэкспрессируемого транспортера (см. Фиг. 5). TSG рассчитывали как сумму в микромолях всех стевиоловых гликозидов, продуцируемых клеткой (при измерении экстракцией из цельноклеточного бульона). Все идентифицированные транспортеры попадают в класс транспортеров, известных как ABC-транспортеры. Сверхэкспрессия этих транспортеров увеличивала TSG с 20% до двукратного по сравнению с родительским. Повышение TSG за счет сверхэкспрессии транспортера может быть связано с повышенным транспортом всех стевиоловых гликозидов или только подмножества стевиоловых гликозидов. Таким образом, данные также были проанализированы, чтобы определить влияние сверхэкспрессии переносчика только на более высокомолекулярные стевиоловые гликозиды Reb D и Reb М. Из восьми переносчиков, которые увеличивали TSG, семь из них также увеличивали общую продукцию Reb D и Reb М, как показано на ФИГ.6. Увеличение Reb D и Reb М со сверхэкспрессией транспортеров варьировало от 30% до двукратного увеличения.
Пример 12: Внеклеточный и внутриклеточный транспорт стевиоловых гликозидов
[00207] Семь из восьми штаммов Reb М, несущих сверхэкспрессированные транспортеры, приводящие к увеличению общего количества стевиоловых гликозидов во всем клеточном бульоне, также увеличивали общее содержание стевиоловых гликозидов в супернатанте (Фиг. 7). В то время как четыре транспортера увеличивали общее количество стевиоловых гликозидов во всем клеточном бульоне почти в два раза (ФИГ. 5), типичное увеличение TSG в супернатанте было меньше и составляло от 35 до 70% (ФИГ. 7). Однако транспортер Т4 Fungal 5 увеличивал TSG в супернатанте примерно в пять раз (Фиг. 7). Данные, представленные на ФИГ. 5 и 7, демонстрируют, что штаммы с некоторыми сверхэкспрессированными транспортерами производят больше в пересчете на TSG, но увеличение TSG не всегда отражается линейным увеличением TSG в супернатанте.
[00208] Подробный анализ доли общих продуцируемых стевиоловых гликозидов, находящихся в супернатанте (Фиг. 8), показывает, что большинство транспортеров (шесть из восьми) показали более низкую долю TSG в супернатанте по сравнению с родительским. Это говорит о том, что переносчики удаляли стевиоловые гликозиды из цитозоля, тем самым снимая ингибирование продукта и обеспечивая большее образование продукта, но они не переносили стевиоловые гликозиды в среду. Вместо этого эти переносчики, скорее всего, транспортируют стевиоловые гликозиды в вакуоль или какой-либо другой клеточный компартмент.Напротив, транспортер T4_Fungal_5 приводил к тому, что почти 100% продуцируемого TSG находилось в супернатанте (Фиг. 8). Это указывает на то, что Т4 Fungal 5, вероятно, является транспортером плазматической мембраны, который способен удалять стевиоловые гликозиды из цитоплазмы клетки и переносить их из клетки в среду. Кроме того, данные на ФИГ.4 показывают, что транспортер T4_Fungal_5 переносит стевиоловые гликозиды с более высокой молекулярной массой Reb D и Reb М из клетки в среду; действительно, почти 100% как Reb D, так и Reb М находились во фракции супернатанта.
[00209] Одним из результатов скрининга транспортеров оказался эндогенный АВС-транспортер S. cerevisiae ВРТ1. Этот белок аннотирован в базе данных генома Saccharomyces для локализации в вакуоли. Транспортеры Т4 Fungal 2 и Т4 Fungal 4 имеют белковые последовательности, которые на 99% идентичны CEN.PK2 ВРТ1 и происходят из штаммов S. cerevisiae САТ-1 и MBG3373, соответственно; они являются аллелями ВРТ1. Все другие транспортеры на 30-43% идентичны по последовательности белка ВРТ1 и представляют собой новые ABC-транспортеры, которые могут переносить стевиоловые гликозиды через мембраны (см. Таблицу 6). Из оставшихся не-ВРТ1 транспортеров, которые переносят стевиоловые гликозиды, ни одна из белковых последовательностей не идентична более чем на 53% любому другому белку, что показывает, что остальные пять белков являются уникальными последовательностями.
Таблица 6. Процентная идентичность всех транспортеров, увеличивающих титры стевиоловых гликозидов
Пример 13: Клеточная локализация BPT1 и T4_Fungal_5
[00210] Чтобы определить клеточную локализацию сверхэкспрессированных белков ВРТ1 и Т4 Fungal 5 в штаммах, продуцирующих Reb М, мы создали гибридные белки транспортер-ЗФБ. Каждый белок транспортера (ВРТ1 или T4_Fungal_5) имел белок ЗФБ, слитый с С-концом транспортера; слитые белки-переносчики ЗФБ экспрессировались через промотор GAL1 и содержали терминатор дрожжей. Штаммы конструировали, как описано в Примере 4, с той лишь разницей, что слитый белок транспортер-ЗФБ использовали вместо простого белка транспортера. Клетки с правильно интегрированными конструкциями транспортер-ЗФБ подтверждали с помощью ПЦР колоний, культивировали, как в примере 5, и подтверждали их активность, эквивалентную активности штаммов, содержащих транспортер без С-концевой метки ЗФБ (Фиг. 9).
[00211] Чтобы визуализировать локализацию белка с помощью ЗФБ, клетки размножали, как в Примере 5, но собирали через 2 дня в производственной среде для наблюдения. Клетки дважды промывали равными объемами ФСБ (фосфатно-солевого буфера) и затем ресуспендировали до ODeoo 1,0 в ФСБ. Клетки фиксировали с использованием 1% агарозных подушечек, установленных на предметном стекле, и визуализировали при 100-кратном увеличении с масляной иммерсией с использованием стандартного флуоресцентного микроскопа при возбуждении 488 нм или в ярком поле. Клетки, экспрессирующие ВРТ1, меченный на С-конце ЗФБ, демонстрировали паттерны флуоресценции, согласующиеся с локализацией гибридного белка в вакуоли (Фиг. 10). Это был ожидаемый результат, поскольку сообщалось, что ВРТ1 обычно локализуется в вакуоли у дрожжей (Sharma et al., Eukaryot. Cell 1 (3), 2002, стр. 391-400). Меченный на С-конце белок Т4 Fungal 5 показал другую локализацию ЗФБ, что согласуется с локализацией белка на плазматической мембране (Фиг. 11).
Пример 14: Направленная эволюция белка T4_Fungal_5 с использованием подверженной ошибкам ПЦР и селекции роста
[00212] Транспортер Т4 Fungal 5 активно удаляет как Reb D, так и Reb М из цитоплазмы (см. ФИГ.4). Reb D является непосредственным субстратом для Reb М (ФИГ. 2), таким образом, удаление Reb D из цитозоля снижает общее количество Reb М, продуцируемого дрожжами. Таким образом, Т4 Fungal 5 подвергался ферментной эволюции для увеличения как его общей активности, так и его специфичности в отношении Reb М. Кодирующая последовательность ДНК (CDS) T4_Fungal_5 была подвергнута мутагенезу с помощью подверженной ошибкам ПЦР с использованием набора для произвольного мутагенеза GeneMorph II (Agilent Technologies, Inc.) и полученную библиотеку ДНК трансформировали в дрожжевой штамм Reb М, аналогичный тому, который использовался в скрининге переносчиков, упомянутом в примере 11, но имеющий две дополнительные копии UGT76G1, обе экспрессируемые под промоторами GAL1. Дополнительную трансформацию с использованием транспортера T4_Fungal_5 дикого типа проводили в качестве контроля. Преобразования выполняли, как описано в Примере 1. После восстановления в течение ночи культуры переносили в производственную среду с добавлением селективного антибиотика для продолжения роста. Определяли OD6oo культур и проводили серийные разведения культур свежей продукционной средой, содержащей антибиотик, во избежание углеродного голодания. Образцы культуры отбирали ежедневно для обоих исходных архивов глицерина и высевали для образования отдельных колоний на чашки с агаром YPD, содержащие антибиотик. Титры TSG и Reb М 88 колоний из каждого суточного образца оценивали и сравнивали с использованием методов, описанных в примерах 6, 7 и 8. На основании этих данных был идентифицирован момент времени, когда был выявлен самый высокий процент колоний, продуцирующих TSG, равный или больший, чем у контрольного штамма (экспрессирующего Т4 Fungal 5 дикого типа). Дополнительные колонии с этого момента времени высевали из глицерина, и 900 колоний были отобраны и подвергнуты скринингу. Скрининг выявил восемь изолятов, которые увеличили титры Reb М на 26% до 47% и увеличили соотношение Reb М / TSG на 10% по сравнению с контролем (Фиг. 12 и 13). Данные на ФИГ. 12 и 13 показывают, что мутации, идентифицированные в транспортере Т4 Fungal 5, увеличивают как общую активность в отношении стевиоловых гликозидов, так и специфичность в отношении Reb М.
[00213] Секвенирование гена T4_Fungal_5 по Сэнгеру показало, что все восемь изолятов содержали одинаковые замены нуклеиновых кислот, в результате чего были заменены четыре аминокислоты: V666A, Y942N, L956P и E1320V. Этот мутантный аллель был назван «Fungal 5 muA». Чтобы проверить причинную связь Fungal 5 muA с улучшенным титром и специфичностью, мутантный аллель был амплифицирован из одного из изолятов и повторно введен в родительский штамм. Полученный штамм повторил фенотипы и продемонстрировал применение Fungal_5_muA для улучшения продукции и специфичности стевиоловых гликозидов. Когда Т4 Fungal 5 и Fungal 5 muA экспрессировались под более слабым промотором GAL3, штамм с Fungal 5 muA продуцировал на 30% больше Reb М в цельноклеточном бульоне и на 40% больше внеклеточного Reb М, чем штамм с T4_Fungal_5 дикого типа (Фиг. 14), что соответствует более ранним данным.
Пример 15: Дальнейшее улучшение Fungal_5_muA
[00214] Для дальнейшего улучшения Fungal_5_muA путем удаления потенциально вредных мутаций мы создали дополнительные варианты мутанта Т4 Fungal 5 с одной, двумя или тремя аминокислотными заменами, идентифицированными в Fungal S muA, и ввели их в штамм дрожжей, используемый для скрининга библиотеки мутагенеза Т4 Fungal 5 в Примере 14. Хотя однократная реверсия V666A в Fungal 5 muA оказала незначительное влияние на продукцию TSG или Reb М, реверсия E1320V была полезной, и тройной мутант V666A Y942N L956P продуцировал на 14% больше TSG и на 12% больше Reb М, чем штамм Fungal 5 muA (Фиг. 15 и 16). Однако дальнейшая реверсия L956P в тройном мутанте (V666A Y942N) привела к 10% снижению Reb М и 19% снижению продуцирования TSG по сравнению с тройным мутантом V666A Y942N L956P. По сравнению со штаммом Fungal_5_muA, единственный мутантный штамм Y942N продуцировал на 21% больше TSG, но на 10% меньше количества Reb М. Эти данные демонстрируют, что мутация Y942N улучшала общую активность Т4 Fungal 5 в экспорте стевиоловых гликозидов, но оказывала отрицательное влияние на его специфичность в отношении Reb М.
[00215] Все публикации, патенты и патентные заявки, упомянутые в этом описании, включены в данный документ посредством ссылки, как если бы каждая отдельная публикация, патент или патентная заявка была специально и отдельно указана как включенная посредством ссылки. Хотя вышеизложенные варианты осуществления были описаны подробно в целях иллюстрации и примера для ясности понимания, для специалиста в данной области техники будет ясно в свете раскрытия настоящего изобретения, что в изобретение могут быть внесены определенные изменения и модификации без отступления от сущности или объема прилагаемой формулы изобретения.
--->
SEQUENCE LISTING
<110> AMYRIS, INC.
<120> ABC TRANSPORTERS FOR THE HIGH EFFICIENCY PRODUCTION OF
REBAUDIOSIDES
<130> 107345.00779
<140> PCT/US2020/014859
<141> 2020-01-23
<150> US 62/796,228
<151> 2019-01-24
<160> 30
<170> PatentIn version 3.5
<210> 1
<211> 1559
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: CEN.PK.BPT1 ABC-transporter
<400> 1
Met Ser Ser Leu Glu Val Val Asp Gly Cys Pro Tyr Gly Tyr Arg Pro
1 5 10 15
Tyr Pro Asp Ser Gly Thr Asn Ala Leu Asn Pro Cys Phe Ile Ser Val
20 25 30
Ile Ser Ala Trp Gln Ala Val Phe Phe Leu Leu Ile Gly Ser Tyr Gln
35 40 45
Leu Trp Lys Leu Tyr Lys Asn Asn Lys Val Pro Pro Arg Phe Lys Asn
50 55 60
Phe Pro Thr Leu Pro Ser Lys Ile Asn Ser Arg His Leu Thr His Leu
65 70 75 80
Thr Asn Val Cys Phe Gln Ser Thr Leu Ile Ile Cys Glu Leu Ala Leu
85 90 95
Val Ser Gln Ser Ser Asp Arg Val Tyr Pro Phe Ile Leu Lys Lys Ala
100 105 110
Leu Tyr Leu Asn Leu Leu Phe Asn Leu Gly Ile Ser Leu Pro Thr Gln
115 120 125
Tyr Leu Ala Tyr Phe Lys Ser Thr Phe Ser Met Gly Asn Gln Leu Phe
130 135 140
Tyr Tyr Met Phe Gln Ile Leu Leu Gln Leu Phe Leu Ile Leu Gln Arg
145 150 155 160
Tyr Tyr His Gly Ser Ser Asn Glu Arg Leu Thr Val Ile Ser Gly Gln
165 170 175
Thr Ala Met Ile Leu Glu Val Leu Leu Leu Phe Asn Ser Val Ala Ile
180 185 190
Phe Ile Tyr Asp Leu Cys Ile Phe Glu Pro Ile Asn Glu Leu Ser Glu
195 200 205
Tyr Tyr Lys Lys Asn Gly Trp Tyr Pro Pro Val His Val Leu Ser Tyr
210 215 220
Ile Thr Phe Ile Trp Met Asn Lys Leu Ile Val Glu Thr Tyr Arg Asn
225 230 235 240
Lys Lys Ile Lys Asp Pro Asn Gln Leu Pro Leu Pro Pro Val Asp Leu
245 250 255
Asn Ile Lys Ser Ile Ser Lys Glu Phe Lys Ala Asn Trp Glu Leu Glu
260 265 270
Lys Trp Leu Asn Arg Asn Ser Leu Trp Arg Ala Ile Trp Lys Ser Phe
275 280 285
Gly Arg Thr Ile Ser Val Ala Met Leu Tyr Glu Thr Thr Ser Asp Leu
290 295 300
Leu Ser Val Val Gln Pro Gln Phe Leu Arg Ile Phe Ile Asp Gly Leu
305 310 315 320
Asn Pro Glu Thr Ser Ser Lys Tyr Pro Pro Leu Asn Gly Val Phe Ile
325 330 335
Ala Leu Thr Leu Phe Val Ile Ser Val Val Ser Val Phe Leu Thr Asn
340 345 350
Gln Phe Tyr Ile Gly Ile Phe Glu Ala Gly Leu Gly Ile Arg Gly Ser
355 360 365
Leu Ala Ser Leu Val Tyr Gln Lys Ser Leu Arg Leu Thr Leu Ala Glu
370 375 380
Arg Asn Glu Lys Ser Thr Gly Asp Ile Leu Asn Leu Met Ser Val Asp
385 390 395 400
Val Leu Arg Ile Gln Arg Phe Phe Glu Asn Ala Gln Thr Ile Ile Gly
405 410 415
Ala Pro Ile Gln Ile Ile Val Val Leu Thr Ser Leu Tyr Trp Leu Leu
420 425 430
Gly Lys Ala Val Ile Gly Gly Leu Val Thr Met Ala Ile Met Met Pro
435 440 445
Ile Asn Ala Phe Leu Ser Arg Lys Val Lys Lys Leu Ser Lys Thr Gln
450 455 460
Met Lys Tyr Lys Asp Met Arg Ile Lys Thr Ile Thr Glu Leu Leu Asn
465 470 475 480
Ala Ile Lys Ser Ile Lys Leu Tyr Ala Trp Glu Glu Pro Met Met Ala
485 490 495
Arg Leu Asn His Val Arg Asn Asp Met Glu Leu Lys Asn Phe Arg Lys
500 505 510
Ile Gly Ile Val Ser Asn Leu Ile Tyr Phe Ala Trp Asn Cys Val Pro
515 520 525
Leu Met Val Thr Cys Ser Thr Phe Gly Leu Phe Ser Leu Phe Ser Asp
530 535 540
Ser Pro Leu Ser Pro Ala Ile Val Phe Pro Ser Leu Ser Leu Phe Asn
545 550 555 560
Ile Leu Asn Ser Ala Ile Tyr Ser Val Pro Ser Met Ile Asn Thr Ile
565 570 575
Ile Glu Thr Ser Val Ser Met Glu Arg Leu Lys Ser Phe Leu Leu Ser
580 585 590
Asp Glu Ile Asp Asp Ser Phe Ile Glu Arg Ile Asp Pro Ser Ala Asp
595 600 605
Glu Arg Ala Leu Pro Ala Ile Glu Met Asn Asn Ile Thr Phe Leu Trp
610 615 620
Lys Ser Lys Glu Val Leu Thr Ser Ser Gln Ser Gly Asp Asn Leu Arg
625 630 635 640
Thr Asp Glu Glu Ser Ile Ile Gly Ser Ser Gln Ile Ala Leu Lys Asn
645 650 655
Ile Asp His Phe Glu Ala Lys Arg Gly Asp Leu Val Cys Val Val Gly
660 665 670
Arg Val Gly Ala Gly Lys Ser Thr Phe Leu Lys Ala Ile Leu Gly Gln
675 680 685
Leu Pro Cys Met Ser Gly Ser Arg Asp Ser Ile Pro Pro Lys Leu Ile
690 695 700
Ile Arg Ser Ser Ser Val Ala Tyr Cys Ser Gln Glu Ser Trp Ile Met
705 710 715 720
Asn Ala Ser Val Arg Glu Asn Ile Leu Phe Gly His Lys Phe Asp Gln
725 730 735
Asp Tyr Tyr Asp Leu Thr Ile Lys Ala Cys Gln Leu Leu Pro Asp Leu
740 745 750
Lys Ile Leu Pro Asp Gly Asp Glu Thr Leu Val Gly Glu Lys Gly Ile
755 760 765
Ser Leu Ser Gly Gly Gln Lys Ala Arg Leu Ser Leu Ala Arg Ala Val
770 775 780
Tyr Ser Arg Ala Asp Ile Tyr Leu Leu Asp Asp Ile Leu Ser Ala Val
785 790 795 800
Asp Ala Glu Val Ser Lys Asn Ile Ile Glu Tyr Val Leu Ile Gly Lys
805 810 815
Thr Ala Leu Leu Lys Asn Lys Thr Ile Ile Leu Thr Thr Asn Thr Val
820 825 830
Ser Ile Leu Lys His Ser Gln Met Ile Tyr Ala Leu Glu Asn Gly Glu
835 840 845
Ile Val Glu Gln Gly Asn Tyr Glu Asp Val Met Asn Arg Lys Asn Asn
850 855 860
Thr Ser Lys Leu Lys Lys Leu Leu Glu Glu Phe Asp Ser Pro Ile Asp
865 870 875 880
Asn Gly Asn Glu Ser Asp Val Gln Thr Glu His Arg Ser Glu Ser Glu
885 890 895
Val Asp Glu Pro Leu Gln Leu Lys Val Thr Glu Ser Glu Thr Glu Asp
900 905 910
Glu Val Val Thr Glu Ser Glu Leu Glu Leu Ile Lys Ala Asn Ser Arg
915 920 925
Arg Ala Ser Leu Ala Thr Leu Arg Pro Arg Pro Phe Val Gly Ala Gln
930 935 940
Leu Asp Ser Val Lys Lys Thr Ala Gln Lys Ala Glu Lys Thr Glu Val
945 950 955 960
Gly Arg Val Lys Thr Lys Ile Tyr Leu Ala Tyr Ile Lys Ala Cys Gly
965 970 975
Val Leu Gly Val Val Leu Phe Phe Leu Phe Met Ile Leu Thr Arg Val
980 985 990
Phe Asp Leu Ala Glu Asn Phe Trp Leu Lys Tyr Trp Ser Glu Ser Asn
995 1000 1005
Glu Lys Asn Gly Ser Asn Glu Arg Val Trp Met Phe Val Gly Val
1010 1015 1020
Tyr Ser Leu Ile Gly Val Ala Ser Ala Ala Phe Asn Asn Leu Arg
1025 1030 1035
Ser Ile Met Met Leu Leu Tyr Cys Ser Ile Arg Gly Ser Lys Lys
1040 1045 1050
Leu His Glu Ser Met Ala Lys Ser Val Ile Arg Ser Pro Met Thr
1055 1060 1065
Phe Phe Glu Thr Thr Pro Val Gly Arg Ile Ile Asn Arg Phe Ser
1070 1075 1080
Ser Asp Met Asp Ala Val Asp Ser Asn Leu Gln Tyr Ile Phe Ser
1085 1090 1095
Phe Phe Phe Lys Ser Ile Leu Thr Tyr Leu Val Thr Val Ile Leu
1100 1105 1110
Val Gly Tyr Asn Met Pro Trp Phe Leu Val Phe Asn Met Phe Leu
1115 1120 1125
Val Val Ile Tyr Ile Tyr Tyr Gln Thr Phe Tyr Ile Val Leu Ser
1130 1135 1140
Arg Glu Leu Lys Arg Leu Ile Ser Ile Ser Tyr Ser Pro Ile Met
1145 1150 1155
Ser Leu Met Ser Glu Ser Leu Asn Gly Tyr Ser Ile Ile Asp Ala
1160 1165 1170
Tyr Asp His Phe Glu Arg Phe Ile Tyr Leu Asn Tyr Glu Lys Ile
1175 1180 1185
Gln Tyr Asn Val Asp Phe Val Phe Asn Phe Arg Ser Thr Asn Arg
1190 1195 1200
Trp Leu Ser Val Arg Leu Gln Thr Ile Gly Ala Thr Ile Val Leu
1205 1210 1215
Ala Thr Ala Ile Leu Ala Leu Ala Thr Met Asn Thr Lys Arg Gln
1220 1225 1230
Leu Ser Ser Gly Met Val Gly Leu Leu Met Ser Tyr Ser Leu Glu
1235 1240 1245
Val Thr Gly Ser Leu Thr Trp Ile Val Arg Thr Thr Val Thr Ile
1250 1255 1260
Glu Thr Asn Ile Val Ser Val Glu Arg Ile Val Glu Tyr Cys Glu
1265 1270 1275
Leu Pro Pro Glu Ala Gln Ser Ile Asn Pro Glu Lys Arg Pro Asp
1280 1285 1290
Glu Asn Trp Pro Ser Lys Gly Gly Ile Glu Phe Lys Asn Tyr Ser
1295 1300 1305
Thr Lys Tyr Arg Glu Asn Leu Asp Pro Val Leu Asn Asn Ile Asn
1310 1315 1320
Val Lys Ile Glu Pro Cys Glu Lys Val Gly Ile Val Gly Arg Thr
1325 1330 1335
Gly Ala Gly Lys Ser Thr Leu Ser Leu Ala Leu Phe Arg Ile Leu
1340 1345 1350
Glu Pro Thr Glu Gly Lys Ile Ile Ile Asp Gly Ile Asp Ile Ser
1355 1360 1365
Asp Ile Gly Leu Phe Asp Leu Arg Ser His Leu Ala Ile Ile Pro
1370 1375 1380
Gln Asp Ala Gln Ala Phe Glu Gly Thr Val Lys Thr Asn Leu Asp
1385 1390 1395
Pro Phe Asn Arg Tyr Ser Glu Asp Glu Leu Lys Arg Ala Val Glu
1400 1405 1410
Gln Ala His Leu Lys Pro His Leu Glu Lys Met Leu His Ser Lys
1415 1420 1425
Pro Arg Gly Asp Asp Ser Asn Glu Glu Asp Gly Asn Val Asn Asp
1430 1435 1440
Ile Leu Asp Val Lys Ile Asn Glu Asn Gly Ser Asn Leu Ser Val
1445 1450 1455
Gly Gln Arg Gln Leu Leu Cys Leu Ala Arg Ala Leu Leu Asn Arg
1460 1465 1470
Ser Lys Ile Leu Val Leu Asp Glu Ala Thr Ala Ser Val Asp Met
1475 1480 1485
Glu Thr Asp Lys Ile Ile Gln Asp Thr Ile Arg Arg Glu Phe Lys
1490 1495 1500
Asp Arg Thr Ile Leu Thr Ile Ala His Arg Ile Asp Thr Val Leu
1505 1510 1515
Asp Ser Asp Lys Ile Ile Val Leu Asp Gln Gly Ser Val Arg Glu
1520 1525 1530
Phe Asp Ser Pro Ser Lys Leu Leu Ser Asp Lys Thr Ser Ile Phe
1535 1540 1545
Tyr Ser Leu Cys Glu Lys Gly Gly Tyr Leu Lys
1550 1555
<210> 2
<211> 1620
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_1 ABC-transporter
<400> 2
Met Ser Leu Glu Leu Ser Asn Ser Thr Leu Cys Asp Ser Tyr Trp Ala
1 5 10 15
Val Asp Asp Phe Thr Ala Cys Gly Arg Gln Leu Val Glu Ser Trp Val
20 25 30
Ser Val Pro Leu Val Leu Ser Ala Leu Val Val Ala Phe Asn Leu Leu
35 40 45
Arg Asn Ser Leu Ala Ser Glu Lys Thr Asp Pro Tyr Ser Lys Leu Asp
50 55 60
Ala Glu Gln Gln Pro Leu Leu Gln Asn Gly His Ala Leu Tyr Thr Ser
65 70 75 80
Ser Ile Glu Ser Asp Asn Thr Asp Ile Phe Gln Arg His Phe Asp Ile
85 90 95
Ala Leu Leu Lys Pro Val Lys Asp Asp Gly Lys Pro Ile Gly Val Val
100 105 110
Arg Ile Val Tyr Arg Asp Thr Ala Glu Lys Leu Lys Val Ala Leu Glu
115 120 125
Glu Ile Leu Leu Ile Ser Gln Thr Val Leu Ala Phe Leu Ala Leu Ser
130 135 140
Arg Leu Glu Asp Ile Ser Glu Ser Arg Phe Leu Leu Val Lys Tyr Ile
145 150 155 160
Asn Phe Ser Leu Trp Leu Tyr Leu Thr Val Ile Thr Ser Ala Arg Leu
165 170 175
Leu Asn Val Thr Lys Gly Phe Ser Ala Asn Arg Val Asp Leu Trp Tyr
180 185 190
His Cys Ala Ile Leu Tyr Asn Leu Gln Trp Phe Asn Ser Val Met Leu
195 200 205
Phe Arg Ser Ala Leu Leu His His Val Ser Gly Thr Tyr Gly Tyr Trp
210 215 220
Phe Tyr Val Thr Gln Phe Val Ile Asn Thr Leu Leu Cys Leu Thr Asn
225 230 235 240
Gly Leu Glu Lys Leu Ser Asp Lys Pro Ala Ile Val Tyr Glu Glu Glu
245 250 255
Gly Val Ile Pro Ser Pro Glu Thr Thr Ser Ser Leu Ile Asp Ile Met
260 265 270
Thr Tyr Gly Tyr Leu Asp Lys Met Val Phe Ser Ser Tyr Trp Lys Pro
275 280 285
Ile Thr Met Glu Glu Val Trp Gly Leu Arg Tyr Asp Asp Tyr Ser His
290 295 300
Asp Val Leu Ile Arg Phe His Lys Leu Lys Ser Ser Ile Arg Phe Thr
305 310 315 320
Leu Arg Leu Phe Leu Gln Phe Lys Lys Glu Leu Ala Leu Gln Thr Leu
325 330 335
Cys Thr Cys Ile Glu Ala Leu Leu Ile Phe Val Pro Pro Leu Cys Leu
340 345 350
Lys Lys Ile Leu Glu Tyr Ile Glu Ser Pro His Thr Lys Ser Arg Ser
355 360 365
Met Ala Trp Phe Tyr Val Leu Ile Met Phe Gly Ser Gly Val Ile Ala
370 375 380
Cys Ser Phe Ser Gly Arg Gly Leu Phe Leu Gly Arg Arg Ile Cys Thr
385 390 395 400
Arg Met Arg Ser Ile Leu Ile Gly Glu Ile Tyr Ser Lys Ala Leu Arg
405 410 415
Arg Arg Leu Gly Ser Thr Asp Lys Glu Lys Thr Thr Glu Glu Glu Asp
420 425 430
Asp Lys Ser Ala Lys Ser Lys Lys Glu Asp Glu Pro Ser Asn Lys Glu
435 440 445
Leu Gly Gly Ile Ile Asn Leu Met Ala Val Asp Ala Phe Lys Val Ser
450 455 460
Glu Ile Gly Gly Tyr Leu His Tyr Phe Pro Asn Ser Phe Val Met Ala
465 470 475 480
Ala Val Ala Ile Tyr Met Leu Tyr Lys Leu Leu Gly Trp Ser Ser Leu
485 490 495
Ile Gly Thr Ala Thr Leu Ile Ala Ile Leu Pro Ile Asn Tyr Met Leu
500 505 510
Val Glu Lys Leu Ser Lys Tyr Gln Lys Gln Met Leu Leu Val Thr Asp
515 520 525
Lys Arg Ile Gln Lys Thr Asn Glu Ala Phe Gln Asn Ile Arg Ile Ile
530 535 540
Lys Tyr Phe Ala Trp Glu Asp Lys Phe Ala Asp Thr Ile Met Lys Ile
545 550 555 560
Arg Glu Glu Glu Leu Gly Tyr Leu Val Gly Arg Cys Val Val Trp Ala
565 570 575
Leu Leu Ile Phe Leu Trp Leu Val Val Pro Thr Ile Val Thr Leu Ile
580 585 590
Thr Phe Tyr Ala Tyr Thr Val Ile Gln Gly Asn Pro Leu Thr Ser Pro
595 600 605
Ile Ala Phe Thr Ala Leu Ser Leu Phe Thr Leu Leu Arg Gly Pro Leu
610 615 620
Asp Ala Leu Ala Asp Met Leu Ser Met Val Met Gln Cys Lys Val Ser
625 630 635 640
Leu Asp Arg Val Glu Asp Phe Leu Asn Glu Pro Glu Thr Thr Lys Tyr
645 650 655
Gln Gln Leu Ser Ala Pro Arg Gly Pro Asn Ser Pro Leu Ile Gly Phe
660 665 670
Glu Asn Ala Thr Phe Tyr Trp Ser Lys Asn Ser Lys Ala Glu Phe Ala
675 680 685
Leu Lys Asp Leu Asn Ile Asp Phe Lys Val Gly Lys Leu Asn Val Val
690 695 700
Ile Gly Pro Thr Gly Ser Gly Lys Ser Ser Leu Leu Leu Ala Leu Leu
705 710 715 720
Gly Glu Met Asp Leu Asp Lys Gly Asn Val Phe Leu Pro Gly Ala Ile
725 730 735
Pro Arg Asp Asp Leu Thr Pro Asn Pro Val Thr Gly Leu Met Glu Ser
740 745 750
Val Ala Tyr Cys Ser Gln Thr Ala Trp Leu Leu Asn Ala Thr Val Lys
755 760 765
Asp Asn Ile Ile Phe Ala Ser Pro Phe Asn Gln Glu Arg Tyr Asp Ala
770 775 780
Val Ile His Ala Cys Gly Leu Thr Arg Asp Leu Ser Ile Leu Glu Ala
785 790 795 800
Gly Asp Glu Thr Glu Ile Gly Glu Lys Gly Ile Thr Leu Ser Gly Gly
805 810 815
Gln Lys Gln Arg Val Ser Leu Ala Arg Ala Leu Tyr Ser Ser Ala Ser
820 825 830
Tyr Leu Leu Leu Asp Asp Cys Leu Ser Ala Val Asp Ser His Thr Ala
835 840 845
Val His Ile Tyr Asp Tyr Cys Ile Asn Gly Glu Leu Met Lys Gly Arg
850 855 860
Thr Cys Ile Leu Val Ser His Asn Val Ser Leu Thr Val Lys Glu Ala
865 870 875 880
Asp Phe Val Val Met Met Asp Asn Gly Arg Ile Lys Ala Gln Gly Ser
885 890 895
Val Asp Glu Leu Met Gln Glu Gly Leu Leu Asn Glu Glu Val Val Lys
900 905 910
Ser Val Met Gln Ser Arg Ser Ala Ser Thr Ala Asn Leu Ala Ala Leu
915 920 925
Asp Asp Asn Ser Pro Ile Ser Ser Glu Ala Ile Ala Glu Gly Leu Ala
930 935 940
Lys Lys Thr Gln Lys Pro Glu Gln Ser Lys Lys Ser Lys Leu Ile Glu
945 950 955 960
Asp Glu Thr Lys Ser Asp Gly Ser Val Lys Pro Glu Ile Tyr Tyr Ala
965 970 975
Tyr Phe Arg Tyr Phe Gly Asn Pro Ala Leu Trp Ile Met Ile Ala Phe
980 985 990
Leu Phe Ile Gly Ser Gln Ser Val Asn Val Tyr Gln Ser Tyr Trp Leu
995 1000 1005
Arg Arg Trp Ser Ala Ile Glu Asp Lys Arg Asp Leu Ser Ala Phe
1010 1015 1020
Ser Asn Ser Asn Asp Met Thr Leu Phe Leu Phe Pro Thr Phe His
1025 1030 1035
Ser Ile Asn Trp His Arg Pro Leu Val Asn Tyr Ala Leu Gln Pro
1040 1045 1050
Phe Gly Leu Ala Val Glu Glu Arg Ser Thr Met Tyr Tyr Ile Thr
1055 1060 1065
Ile Tyr Thr Leu Ile Gly Leu Ala Phe Ala Thr Leu Gly Ser Ser
1070 1075 1080
Arg Val Ile Leu Thr Phe Ile Gly Gly Leu Asn Val Ser Arg Lys
1085 1090 1095
Ile Phe Lys Asp Leu Leu Asp Lys Leu Leu His Ala Lys Leu Arg
1100 1105 1110
Phe Phe Asp Gln Thr Pro Ile Gly Arg Ile Met Asn Arg Phe Ser
1115 1120 1125
Lys Asp Ile Glu Ala Ile Asp Gln Glu Leu Ala Leu Tyr Ala Glu
1130 1135 1140
Glu Phe Val Thr Tyr Leu Ile Ser Cys Leu Ser Thr Leu Val Val
1145 1150 1155
Val Cys Ala Val Thr Pro Ala Phe Leu Val Ala Gly Val Leu Ile
1160 1165 1170
Leu Leu Val Tyr Tyr Gly Val Gly Val Leu Tyr Leu Glu Leu Ser
1175 1180 1185
Arg Asp Leu Lys Arg Phe Glu Ser Ile Thr Lys Ser Pro Ile His
1190 1195 1200
Gln His Phe Ser Glu Thr Leu Val Gly Met Thr Thr Ile Arg Ala
1205 1210 1215
Tyr Gly Asp Glu Arg Arg Phe Leu Lys Gln Asn Phe Glu Lys Ile
1220 1225 1230
Asp Val Asn Asn Arg Pro Phe Trp Tyr Val Trp Val Asn Asn Arg
1235 1240 1245
Trp Leu Ala Tyr Arg Ser Asp Met Ile Gly Ala Phe Ile Ile Phe
1250 1255 1260
Phe Ala Ala Ala Phe Ala Val Ala Tyr Ser Asp Lys Ile Asp Ala
1265 1270 1275
Gly Leu Ala Gly Ile Ser Leu Ser Phe Ser Val Ser Phe Arg Tyr
1280 1285 1290
Thr Ala Val Trp Val Val Arg Met Tyr Ala Tyr Val Glu Met Ser
1295 1300 1305
Met Asn Ser Val Glu Arg Val Gln Glu Tyr Ile Glu Gln Thr Pro
1310 1315 1320
Gln Glu Pro Pro Lys Tyr Leu Pro Gln Asp Pro Val Asn Ser Trp
1325 1330 1335
Pro Ser Asn Gly Val Ile Asp Val Gln Asn Ile Cys Ile Arg Tyr
1340 1345 1350
Ser Pro Glu Leu Pro Arg Val Ile Asp Asn Val Ser Phe His Val
1355 1360 1365
Asn Ala Gly Glu Lys Ile Gly Val Val Gly Arg Thr Gly Ala Gly
1370 1375 1380
Lys Ser Thr Ile Ile Thr Ser Phe Phe Arg Phe Val Asp Leu Glu
1385 1390 1395
Ser Gly Ser Ile Lys Ile Asp Gly Leu Asp Ile Ser Lys Ile Gly
1400 1405 1410
Leu Lys Pro Leu Arg Lys Gly Leu Thr Ile Ile Pro Gln Asp Pro
1415 1420 1425
Thr Leu Phe Ser Gly Thr Ile Arg Ser Asn Leu Asp Ile Phe Gly
1430 1435 1440
Glu Tyr Gly Asp Leu Gln Met Phe Glu Ala Leu Arg Arg Val Asn
1445 1450 1455
Leu Ile Ser Val Asp Asp Tyr Gln Arg Ile Val Asp Gly Asn Gly
1460 1465 1470
Ala Ala Val Ala Asp Glu Thr Ala Gln Ala Arg Gly Asp Asn Val
1475 1480 1485
Asn Lys Phe Leu Asp Leu Asp Ser Thr Val Ser Glu Gly Gly Gly
1490 1495 1500
Asn Leu Ser Gln Gly Glu Arg Gln Leu Leu Cys Leu Ala Arg Ser
1505 1510 1515
Ile Leu Lys Met Pro Lys Ile Leu Met Leu Asp Glu Ala Thr Ala
1520 1525 1530
Ser Ile Asp Tyr Glu Ser Asp Ala Lys Ile Gln Ala Thr Ile Arg
1535 1540 1545
Glu Glu Phe Ser Ser Ser Thr Val Leu Thr Ile Ala His Arg Leu
1550 1555 1560
Lys Thr Ile Ile Asp Tyr Asp Lys Ile Leu Leu Leu Asp His Gly
1565 1570 1575
Lys Val Lys Glu Tyr Asp His Pro Tyr Lys Leu Ile Thr Asn Lys
1580 1585 1590
Lys Ser Asp Phe Arg Lys Met Cys Gln Asp Thr Gly Glu Phe Asp
1595 1600 1605
Asp Leu Val Asn Leu Ala Lys Gln Ala Tyr Arg Lys
1610 1615 1620
<210> 3
<211> 1515
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_10 ABC-transporter
<400> 3
Met Gly Gln Ser Glu Arg Ala Ala Leu Ile Ala Phe Ala Ser Arg Asn
1 5 10 15
Thr Thr Glu Cys Trp Leu Cys Arg Asp Lys Glu Gly Phe Gly Pro Ile
20 25 30
Ser Tyr Tyr Gly Asp Phe Thr Val Cys Phe Ile Asp Gly Val Leu Leu
35 40 45
Asn Phe Ala Ala Leu Phe Met Leu Ile Phe Gly Thr Tyr Gln Val Val
50 55 60
Lys Leu Ser Lys Lys Glu His Pro Gly Ile Lys Tyr Arg Arg Asp Trp
65 70 75 80
Leu Leu Phe Ser Arg Ile Thr Leu Val Gly Cys Phe Leu Leu Phe Thr
85 90 95
Ser Met Ala Ala Tyr Tyr Ser Ser Glu Lys His Glu Ser Ile Ala Leu
100 105 110
Thr Ser Gln Tyr Thr Leu Thr Leu Met Ser Ile Phe Val Ala Leu Met
115 120 125
Leu His Trp Val Glu Tyr His Arg Ser Arg Ile Ser Asn Gly Ile Val
130 135 140
Leu Phe Tyr Trp Leu Phe Glu Thr Leu Phe Gln Gly Ser Lys Trp Val
145 150 155 160
Asn Phe Ser Ile Arg His Ala Tyr Asn Leu Asn His Glu Trp Pro Val
165 170 175
Ser Tyr Ser Val Tyr Ile Leu Thr Ile Phe Gln Thr Ile Ser Ala Phe
180 185 190
Met Ile Leu Ile Leu Glu Ala Gly Phe Glu Lys Pro Leu Pro Ser Tyr
195 200 205
Gln Arg Val Ile Glu Ser Tyr Ser Lys Gln Lys Arg Asn Pro Val Asp
210 215 220
Asn Ser His Ile Phe Gln Arg Leu Ser Phe Ser Trp Met Thr Glu Leu
225 230 235 240
Met Lys Thr Gly Tyr Lys Lys Tyr Leu Thr Glu Gln Asp Leu Tyr Lys
245 250 255
Leu Pro Lys Ser Phe Gly Ala Lys Glu Ile Ser His Lys Phe Ser Glu
260 265 270
Arg Trp Gln Tyr Gln Leu Lys His Lys Ala Asn Pro Ser Leu Ala Trp
275 280 285
Ala Leu Leu Ser Thr Phe Gly Gly Lys Ile Leu Leu Gly Gly Ile Phe
290 295 300
Lys Val Ala Tyr Asp Ile Leu Gln Phe Thr Gln Pro Gln Leu Leu Arg
305 310 315 320
Ile Leu Ile Lys Phe Val Ser Asp Tyr Thr Ser Thr Pro Glu Pro Gln
325 330 335
Leu Pro Leu Val Arg Gly Val Met Leu Ser Ile Ala Met Phe Val Val
340 345 350
Ser Val Val Gln Thr Ser Ile Leu His Gln Tyr Phe Leu Asn Ala Phe
355 360 365
Asp Thr Gly Met His Ile Lys Ser Gly Met Thr Ser Val Ile Tyr Gln
370 375 380
Lys Ala Leu Val Leu Ser Ser Glu Ala Ser Ala Ser Ser Ser Thr Gly
385 390 395 400
Asp Ile Val Asn Leu Met Ser Val Asp Val Gln Arg Leu Gln Asp Leu
405 410 415
Thr Gln Trp Gly Gln Ile Ile Trp Ser Gly Pro Phe Gln Ile Ile Leu
420 425 430
Cys Leu Val Ser Leu Tyr Lys Leu Leu Gly Pro Cys Met Trp Val Gly
435 440 445
Val Ile Ile Met Ile Ile Met Ile Pro Ile Asn Ser Val Ile Val Arg
450 455 460
Ile Gln Lys Lys Leu Gln Lys Ile Gln Met Lys Asn Lys Asp Glu Arg
465 470 475 480
Thr Arg Val Thr Ser Glu Ile Leu Asn Asn Ile Lys Ser Leu Lys Val
485 490 495
Tyr Gly Trp Glu Ile Pro Tyr Lys Ala Lys Leu Asp His Val Arg Asn
500 505 510
Asp Lys Glu Leu Lys Asn Leu Lys Lys Met Gly Cys Thr Leu Ala Leu
515 520 525
Ala Ser Phe Gln Phe Asn Ile Val Pro Phe Leu Val Ser Cys Ser Thr
530 535 540
Phe Ala Val Phe Val Phe Thr Glu Asp Arg Pro Leu Ser Thr Asp Leu
545 550 555 560
Val Phe Pro Ala Leu Thr Leu Phe Asn Leu Leu Ser Phe Pro Leu Ala
565 570 575
Val Val Pro Asn Ala Ile Ser Ser Phe Ile Glu Ala Ser Val Ser Val
580 585 590
Asn Arg Leu Tyr Ala Phe Leu Thr Asn Glu Glu Leu Gln Thr Asp Ala
595 600 605
Val His Arg Glu Pro Lys Val Asn Asn Ile Gly Asp Glu Gly Val Lys
610 615 620
Val Ser Asp Ala Thr Phe Leu Trp Gln Arg Lys Pro Glu Tyr Lys Val
625 630 635 640
Ala Leu Lys Asn Ile Asn Phe Ser Ala Lys Lys Gly Glu Leu Thr Cys
645 650 655
Ile Val Gly Lys Val Gly Ser Gly Lys Ser Ala Leu Ile Gln Ser Leu
660 665 670
Leu Gly Asp Leu Ile Arg Val Lys Gly Tyr Ala Ala Val His Gly Ser
675 680 685
Val Ala Tyr Val Ser Gln Val Ala Trp Ile Met Asn Gly Thr Val Lys
690 695 700
Asp Asn Ile Ile Phe Gly His Lys Tyr Asp Pro Glu Phe Tyr Glu Leu
705 710 715 720
Thr Ile Lys Ala Cys Ala Leu Ala Ile Asp Leu Ser Met Leu Pro Asp
725 730 735
Gly Asp Gln Thr Leu Val Gly Glu Lys Gly Ile Ser Leu Ser Gly Gly
740 745 750
Gln Lys Ala Arg Leu Ser Leu Ala Arg Ala Val Tyr Ala Arg Ala Asp
755 760 765
Thr Tyr Leu Leu Asp Asp Pro Leu Ala Ala Val Asp Glu His Val Ala
770 775 780
Lys His Leu Ile Glu His Val Leu Gly Pro His Gly Leu Leu His Ser
785 790 795 800
Lys Thr Lys Val Leu Ala Thr Asn Lys Ile Ser Val Leu Ser Ile Ala
805 810 815
Asp Ser Ile Thr Leu Met Glu Asn Gly Glu Ile Ile Gln Gln Gly Thr
820 825 830
Tyr Glu Glu Thr Asn Asn Thr Thr Asp Ser Pro Leu Ser Lys Leu Ile
835 840 845
Ser Glu Phe Gly Lys Lys Gly Lys Ala Thr Pro Ser Gln Ser Thr Thr
850 855 860
Ser Leu Thr Lys Leu Ala Thr Ser Asp Leu Gly Ser Ser Ser Asp Ser
865 870 875 880
Lys Val Ser Asp Val Ser Ile Asp Val Ser Gln Leu Asp Thr Glu Asn
885 890 895
Leu Thr Glu Ala Glu Glu Leu Lys Ser Leu Arg Arg Ala Ser Met Ala
900 905 910
Thr Leu Gly Ser Ile Gly Phe Asp Asp Asp Glu Asn Ile Ala Arg Arg
915 920 925
Glu His Arg Glu Gln Gly Lys Val Lys Trp Asp Ile Tyr Met Glu Tyr
930 935 940
Ala Arg Ala Cys Asn Pro Arg Ser Val Cys Val Phe Leu Phe Phe Ile
945 950 955 960
Val Leu Ser Met Leu Leu Ser Val Leu Gly Asn Phe Trp Leu Lys His
965 970 975
Trp Ser Glu Val Asn Thr Gly Glu Gly Tyr Asn Pro His Ala Ala Arg
980 985 990
Tyr Leu Leu Ile Tyr Phe Ala Leu Gly Val Gly Ser Ala Leu Ala Thr
995 1000 1005
Leu Ile Gln Thr Ile Val Leu Trp Val Phe Cys Thr Ile His Gly
1010 1015 1020
Ser Arg Tyr Leu His Asp Ala Met Ala Thr Ser Val Leu Lys Ala
1025 1030 1035
Pro Met Ser Phe Phe Glu Thr Thr Pro Ile Gly Arg Ile Leu Asn
1040 1045 1050
Arg Phe Ser Asn Asp Ile Tyr Lys Val Asp Glu Val Leu Gly Arg
1055 1060 1065
Thr Phe Ser Gln Phe Phe Ala Asn Val Val Lys Val Ser Phe Thr
1070 1075 1080
Ile Ile Val Ile Cys Met Ala Thr Trp Gln Phe Ile Phe Ile Ile
1085 1090 1095
Leu Pro Leu Ser Val Leu Tyr Ile Tyr Tyr Gln Gln Tyr Tyr Leu
1100 1105 1110
Arg Thr Ser Arg Glu Leu Arg Arg Leu Asp Ser Val Thr Arg Ser
1115 1120 1125
Pro Ile Tyr Ala His Phe Gln Glu Thr Leu Gly Gly Leu Thr Thr
1130 1135 1140
Ile Arg Gly Tyr Ser Gln Gln Thr Arg Phe Val His Ile Asn Gln
1145 1150 1155
Thr Arg Val Asp Asn Asn Met Ser Ala Phe Tyr Pro Ser Val Asn
1160 1165 1170
Ala Asn Arg Trp Leu Ala Phe Arg Leu Glu Phe Ile Gly Ser Ile
1175 1180 1185
Ile Ile Leu Gly Ser Ser Met Leu Ala Val Ile Arg Leu Gly Asn
1190 1195 1200
Gly Thr Leu Thr Ala Gly Met Ile Gly Leu Ser Leu Ser Phe Ala
1205 1210 1215
Leu Gln Ile Thr Gln Ser Leu Asn Trp Ile Val Arg Met Thr Val
1220 1225 1230
Glu Val Glu Thr Asn Ile Val Ser Val Glu Arg Ile Lys Glu Tyr
1235 1240 1245
Ala Glu Leu Lys Ser Glu Ala Pro Tyr Ile Ile Glu Asp His Arg
1250 1255 1260
Pro Pro Ala Ser Trp Pro Glu Lys Gly Asp Val Lys Phe Val Asn
1265 1270 1275
Tyr Ser Thr Arg Tyr Arg Pro Glu Leu Glu Leu Ile Leu Lys Asp
1280 1285 1290
Ile Asn Leu His Ile Leu Pro Lys Glu Lys Ile Gly Ile Val Gly
1295 1300 1305
Arg Thr Gly Ala Gly Lys Ser Ser Leu Thr Leu Ala Leu Phe Arg
1310 1315 1320
Ile Ile Glu Ala Ala Ser Gly His Ile Ile Ile Asp Gly Ile Pro
1325 1330 1335
Ile Asp Ser Ile Gly Leu Ala Asp Leu Arg His Arg Leu Ser Ile
1340 1345 1350
Ile Pro Gln Asp Ser Gln Ile Phe Glu Gly Thr Ile Arg Glu Asn
1355 1360 1365
Ile Asp Pro Ser Lys Gln Tyr Thr Asp Glu Gln Ile Trp Asp Ala
1370 1375 1380
Leu Glu Leu Ser His Leu Lys Asn His Val Lys Asn Met Gly Pro
1385 1390 1395
Asp Gly Leu Glu Thr Met Leu Ser Glu Gly Gly Gly Asn Leu Ser
1400 1405 1410
Val Gly Gln Arg Gln Leu Met Cys Leu Ala Arg Ala Leu Leu Ile
1415 1420 1425
Ser Ser Lys Ile Leu Val Leu Asp Glu Ala Thr Ala Ala Val Asp
1430 1435 1440
Val Glu Thr Asp Gln Leu Ile Gln Lys Thr Ile Arg Glu Ala Phe
1445 1450 1455
Lys Glu Arg Thr Ile Leu Thr Ile Ala His Arg Ile Asn Thr Ile
1460 1465 1470
Met Asp Ser Asp Arg Ile Ile Val Leu Asp Lys Gly Arg Val Thr
1475 1480 1485
Glu Phe Asp Thr Pro Ala Asn Leu Leu Asn Lys Lys Asp Ser Ile
1490 1495 1500
Phe Tyr Ser Leu Cys Val Glu Ala Gly Leu Ala Glu
1505 1510 1515
<210> 4
<211> 1559
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_2 ABC-transporter
<400> 4
Met Ser Ser Leu Glu Val Val Asp Gly Cys Pro Tyr Gly Tyr Arg Pro
1 5 10 15
Tyr Pro Asp Ser Gly Thr Asn Ala Leu Asn Pro Cys Phe Ile Ser Val
20 25 30
Ile Ser Ala Trp Gln Ala Val Phe Phe Leu Leu Ile Gly Ser Tyr Gln
35 40 45
Leu Trp Lys Leu Tyr Lys Asn Asn Lys Val Pro Pro Arg Phe Lys Asn
50 55 60
Phe Pro Thr Leu Pro Ser Lys Ile Asn Ser Arg His Leu Thr His Leu
65 70 75 80
Thr Asn Val Cys Phe Gln Ser Thr Leu Ile Ile Cys Glu Leu Ala Leu
85 90 95
Val Ser Gln Ser Ser Asp Arg Val Tyr Pro Phe Ile Leu Lys Lys Ala
100 105 110
Leu Tyr Leu Asn Leu Leu Phe Asn Leu Gly Ile Ser Leu Pro Thr Gln
115 120 125
Tyr Leu Ala Tyr Phe Lys Ser Thr Phe Ser Met Gly Asn Gln Leu Phe
130 135 140
Tyr Tyr Met Phe Gln Ile Leu Leu Gln Leu Phe Leu Ile Leu Gln Arg
145 150 155 160
Tyr Tyr His Gly Ser Ser Asn Glu Arg Leu Thr Val Ile Ser Gly Gln
165 170 175
Thr Ala Met Ile Leu Glu Val Leu Leu Leu Phe Asn Ser Val Ala Ile
180 185 190
Phe Ile Tyr Asp Leu Cys Ile Phe Glu Pro Ile Asn Glu Leu Ser Glu
195 200 205
Tyr Tyr Lys Lys Asn Gly Trp Tyr Pro Pro Val His Val Leu Ser Tyr
210 215 220
Ile Thr Phe Ile Trp Met Asn Lys Leu Ile Val Glu Thr Tyr Arg Asn
225 230 235 240
Lys Lys Ile Lys Asp Pro Asn Gln Leu Pro Leu Pro Pro Val Asp Leu
245 250 255
Asn Ile Lys Ser Ile Ser Lys Glu Phe Lys Ala Asn Trp Glu Leu Glu
260 265 270
Lys Trp Leu Asn Arg Asn Ser Leu Trp Arg Ala Ile Trp Lys Ser Phe
275 280 285
Gly Arg Thr Ile Ser Val Ala Met Leu Tyr Glu Thr Thr Ser Asp Leu
290 295 300
Leu Ser Val Val Gln Pro Gln Phe Leu Arg Ile Phe Ile Asp Gly Phe
305 310 315 320
Asn Pro Glu Thr Ser Ser Lys Tyr Pro Pro Leu Asn Gly Val Phe Ile
325 330 335
Ala Leu Thr Leu Phe Val Ile Ser Val Val Ser Val Phe Leu Thr Asn
340 345 350
Gln Phe Tyr Ile Gly Ile Phe Glu Ala Gly Leu Gly Ile Arg Gly Ser
355 360 365
Leu Ala Ser Leu Val Tyr Gln Lys Ser Leu Arg Leu Thr Leu Ala Glu
370 375 380
Arg Asn Glu Lys Ser Thr Gly Asp Ile Leu Asn Leu Met Ser Val Asp
385 390 395 400
Val Leu Arg Ile Gln Arg Phe Phe Glu Asn Ala Gln Thr Ile Ile Gly
405 410 415
Ala Pro Ile Gln Ile Ile Val Val Leu Thr Ser Leu Tyr Trp Leu Leu
420 425 430
Gly Lys Ala Val Val Gly Gly Leu Val Thr Met Ala Ile Met Met Pro
435 440 445
Ile Asn Ala Phe Leu Ser Arg Lys Val Lys Lys Leu Ser Lys Thr Gln
450 455 460
Met Lys Tyr Lys Asp Met Arg Ile Lys Thr Ile Thr Glu Leu Leu Asn
465 470 475 480
Ala Ile Lys Ser Ile Lys Leu Tyr Ala Trp Glu Glu Pro Met Met Ala
485 490 495
Arg Leu Asn His Val Arg Asn Asp Met Glu Leu Lys Asn Phe Arg Lys
500 505 510
Ile Gly Ile Val Ser Asn Leu Ile Tyr Phe Ala Trp Asn Cys Val Pro
515 520 525
Leu Met Val Thr Cys Ser Thr Phe Gly Leu Phe Ser Leu Phe Ser Asp
530 535 540
Ser Pro Leu Ser Pro Ala Ile Val Phe Pro Ser Leu Ser Leu Phe Asn
545 550 555 560
Ile Leu Asn Ser Ala Ile Tyr Ser Val Pro Ser Met Ile Asn Thr Ile
565 570 575
Ile Glu Thr Ser Val Ser Met Glu Arg Leu Lys Ser Phe Leu Leu Ser
580 585 590
Asp Glu Ile Asp Asp Ser Phe Ile Glu Arg Ile Asp Pro Ser Ala Asp
595 600 605
Glu Arg Ala Leu Pro Ala Ile Glu Met Asn Asn Ile Thr Phe Leu Trp
610 615 620
Lys Ser Lys Glu Val Leu Ala Ser Ser Gln Ser Gly Asp Asn Leu Arg
625 630 635 640
Thr Asp Glu Glu Ser Ile Ile Gly Ser Ser Gln Ile Ala Leu Lys Asn
645 650 655
Ile Asp His Phe Glu Ala Lys Arg Gly Asp Leu Val Cys Val Val Gly
660 665 670
Arg Val Gly Ala Gly Lys Ser Thr Phe Leu Lys Ala Ile Leu Gly Gln
675 680 685
Leu Pro Cys Met Ser Gly Ser Arg Asp Ser Ile Pro Pro Lys Leu Ile
690 695 700
Ile Arg Ser Ser Ser Val Ala Tyr Cys Ser Gln Glu Ser Trp Ile Met
705 710 715 720
Asn Ala Ser Val Arg Glu Asn Ile Leu Phe Gly His Lys Phe Asp Gln
725 730 735
Asn Tyr Tyr Asp Leu Thr Ile Lys Ala Cys Gln Leu Leu Pro Asp Leu
740 745 750
Lys Ile Leu Pro Asp Gly Asp Glu Thr Leu Val Gly Glu Lys Gly Ile
755 760 765
Ser Leu Ser Gly Gly Gln Lys Ala Arg Leu Ser Leu Ala Arg Ala Val
770 775 780
Tyr Ser Arg Ala Asp Ile Tyr Leu Leu Asp Asp Ile Leu Ser Ala Val
785 790 795 800
Asp Ala Glu Val Ser Lys Asn Ile Ile Glu Tyr Val Leu Ile Gly Lys
805 810 815
Thr Ala Leu Leu Lys Asn Lys Thr Ile Ile Leu Thr Thr Asn Thr Val
820 825 830
Ser Ile Leu Lys His Ser Gln Met Ile Tyr Ala Leu Glu Asn Gly Glu
835 840 845
Ile Val Glu Gln Gly Asn Tyr Glu Asp Val Met Asn Arg Lys Asn Asn
850 855 860
Thr Ser Lys Leu Lys Lys Leu Leu Glu Glu Phe Asp Ser Pro Ile Asp
865 870 875 880
Asn Gly Asn Glu Ser Asp Val Gln Thr Glu His Arg Ser Glu Ser Glu
885 890 895
Val Asp Glu Pro Leu Gln Leu Lys Val Thr Glu Ser Glu Thr Glu Asp
900 905 910
Glu Val Val Thr Glu Ser Glu Leu Glu Leu Ile Lys Ala Asn Ser Arg
915 920 925
Arg Ala Ser Leu Ala Thr Leu Arg Pro Arg Pro Phe Val Gly Ala Gln
930 935 940
Leu Asp Ser Val Lys Lys Thr Ala Gln Glu Ala Glu Lys Thr Glu Val
945 950 955 960
Gly Arg Val Lys Thr Lys Val Tyr Leu Ala Tyr Ile Lys Ala Cys Gly
965 970 975
Val Leu Gly Val Val Leu Phe Phe Leu Phe Met Ile Leu Thr Arg Val
980 985 990
Phe Asp Leu Ala Glu Asn Phe Trp Leu Lys Tyr Trp Ser Glu Ser Asn
995 1000 1005
Glu Lys Asn Gly Ser Asn Glu Arg Val Trp Met Phe Val Gly Val
1010 1015 1020
Tyr Ser Leu Ile Gly Val Ala Ser Ala Ala Phe Asn Asn Leu Arg
1025 1030 1035
Ser Ile Met Met Leu Leu Tyr Cys Ser Ile Arg Gly Ser Lys Lys
1040 1045 1050
Leu His Glu Ser Met Ala Lys Ser Val Ile Arg Ser Pro Met Thr
1055 1060 1065
Phe Phe Glu Thr Thr Pro Val Gly Arg Ile Ile Asn Arg Phe Ser
1070 1075 1080
Ser Asp Met Asp Ala Val Asp Ser Asn Leu Gln Tyr Ile Phe Ser
1085 1090 1095
Phe Phe Phe Lys Ser Ile Leu Thr Tyr Leu Val Thr Val Ile Leu
1100 1105 1110
Val Gly Tyr Asn Met Pro Trp Phe Leu Val Phe Asn Met Phe Leu
1115 1120 1125
Val Val Ile Tyr Ile Tyr Tyr Gln Thr Phe Tyr Ile Val Leu Ser
1130 1135 1140
Arg Glu Leu Lys Arg Leu Ile Ser Ile Ser Tyr Ser Pro Ile Met
1145 1150 1155
Ser Leu Met Ser Glu Ser Leu Asn Gly Tyr Ser Ile Ile Asp Ala
1160 1165 1170
Tyr Asp His Phe Glu Arg Phe Ile Tyr Leu Asn Tyr Glu Lys Ile
1175 1180 1185
Gln Tyr Asn Val Asp Phe Val Phe Asn Phe Arg Ser Thr Asn Arg
1190 1195 1200
Trp Leu Ser Val Arg Leu Gln Thr Ile Gly Ala Thr Ile Val Leu
1205 1210 1215
Ala Thr Ala Ile Leu Ala Leu Ala Thr Met Asn Thr Lys Arg Gln
1220 1225 1230
Leu Ser Ser Gly Met Val Gly Leu Leu Met Ser Tyr Ser Leu Glu
1235 1240 1245
Val Thr Gly Ser Leu Thr Trp Ile Val Arg Thr Thr Val Met Ile
1250 1255 1260
Glu Thr Asn Ile Val Ser Val Glu Arg Ile Val Glu Tyr Cys Glu
1265 1270 1275
Leu Pro Pro Glu Ala Gln Ser Ile Asn Pro Glu Lys Arg Pro Asp
1280 1285 1290
Glu Asn Trp Pro Ser Lys Gly Gly Ile Glu Phe Lys Asn Tyr Ser
1295 1300 1305
Thr Lys Tyr Arg Glu Asn Leu Asp Pro Val Leu Asn Asn Ile Asn
1310 1315 1320
Val Lys Ile Glu Pro Cys Glu Lys Val Gly Ile Val Gly Arg Thr
1325 1330 1335
Gly Ala Gly Lys Ser Thr Leu Ser Leu Ala Leu Phe Arg Ile Leu
1340 1345 1350
Glu Pro Thr Glu Gly Lys Ile Ile Ile Asp Gly Ile Gly Ile Ser
1355 1360 1365
Asp Ile Gly Leu Phe Asp Leu Arg Ser His Leu Ala Ile Ile Pro
1370 1375 1380
Gln Asp Ala Gln Ala Phe Glu Gly Thr Val Lys Thr Asn Leu Asp
1385 1390 1395
Pro Phe Asn Arg Tyr Ser Glu Asp Glu Leu Lys Arg Ala Val Glu
1400 1405 1410
Gln Ala His Leu Lys Pro His Leu Glu Lys Met Leu His Ser Lys
1415 1420 1425
Pro Arg Gly Asp Asp Ser Asn Glu Glu Asp Gly Asn Val Asn Asp
1430 1435 1440
Ile Leu Asp Val Lys Ile Asn Glu Asn Gly Ser Asn Leu Ser Val
1445 1450 1455
Gly Gln Arg Gln Leu Leu Cys Leu Ala Arg Ala Leu Leu Asn Arg
1460 1465 1470
Ser Lys Ile Leu Val Leu Asp Glu Ala Thr Ala Ser Val Asp Met
1475 1480 1485
Glu Thr Asp Lys Ile Ile Gln Asp Thr Ile Arg Arg Glu Phe Lys
1490 1495 1500
Asp Arg Thr Ile Leu Thr Ile Ala His Arg Ile Asp Thr Val Leu
1505 1510 1515
Asp Ser Asp Lys Ile Ile Val Leu Asp Gln Gly Ser Val Arg Glu
1520 1525 1530
Phe Asp Ser Pro Ser Lys Leu Leu Ser Asp Lys Thr Ser Ile Phe
1535 1540 1545
Tyr Ser Leu Cys Glu Lys Gly Gly Tyr Leu Lys
1550 1555
<210> 5
<211> 1638
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_3 ABC-transporter
<400> 5
Met Asn Ser Tyr Asn Glu Ser Ala Pro Thr Gly Cys Ser Phe Trp Asp
1 5 10 15
Asn Asp Asp Ile Ser Pro Cys Ile Arg Lys Ser Leu Leu Asp Ser Tyr
20 25 30
Leu Pro Ala Ala Ile Val Val Gly Ser Leu Leu Tyr Leu Leu Leu Ile
35 40 45
Gly Ala Gln Gln Ile Lys Thr His Arg Lys Leu Tyr Ala Lys Asp Glu
50 55 60
Thr Gln Pro Leu Leu Glu Pro Ala Asn Gly Ser Pro Thr Asp Tyr Ser
65 70 75 80
Asn Thr Tyr Gly Thr Ile Asp Tyr Glu Glu Glu Gln Ser Thr Ala Glu
85 90 95
Leu Thr Thr Ser Gln Lys His Phe Asp Ile Ser Arg Leu Glu Pro Leu
100 105 110
Lys Asp Asp Gly Thr Pro Leu Gly Leu Val Lys Tyr Val Gln Arg Asp
115 120 125
Gly Trp Glu Lys Val Lys Leu Ile Leu Glu Phe Val Ile Leu Ile Phe
130 135 140
Gln Leu Val Ile Ala Val Val Ala Leu Phe Val Pro Ser Leu Asn Gln
145 150 155 160
Glu Trp Glu Gly Tyr Lys Leu Thr Pro Ile Val Arg Val Phe Val Trp
165 170 175
Ile Phe Leu Phe Ala Leu Gly Ser Ile Arg Ala Leu Asn Lys Ser Gly
180 185 190
Pro Phe Pro Leu Ala Asn Ile Ser Leu Leu Tyr Tyr Ile Val Asn Ile
195 200 205
Val Pro Ser Ala Leu Ser Phe Arg Ser Val Leu Ile His Pro Gln Asn
210 215 220
Ser Gln Leu Val Asn Tyr Tyr Tyr Ser Phe Gln Phe Ile Asn Asn Thr
225 230 235 240
Leu Leu Phe Leu Leu Leu Gly Ser Ala Arg Val Phe Asp His Pro Ser
245 250 255
Val Leu Phe Asp Thr Asp Asp Gly Val Lys Pro Ser Pro Glu Asn Asn
260 265 270
Ser Asn Phe Phe Glu Ile Val Thr Tyr Ser Trp Ile Asp Pro Leu Ile
275 280 285
Phe Lys Ala Tyr Lys Thr Pro Leu Gln Phe Asn Asp Ile Trp Gly Leu
290 295 300
Arg Ile Asp Asp Tyr Ala Tyr Phe Leu Leu Arg Arg Phe Lys Asp Leu
305 310 315 320
Gly Phe Thr Arg Thr Phe Thr Tyr Lys Ile Phe Tyr Phe Ser Lys Gly
325 330 335
Asp Leu Ala Ala Gln Ala Leu Trp Ala Ser Ile Asp Ser Met Leu Ile
340 345 350
Phe Gly Pro Ser Leu Leu Leu Lys Arg Ile Leu Glu Tyr Val Asp Asn
355 360 365
Pro Gly Met Thr Ser Arg Asn Met Ala Trp Leu Tyr Val Leu Thr Met
370 375 380
Phe Phe Ile Gln Ile Ser Asp Ser Leu Val Ser Gly Arg Ser Leu Tyr
385 390 395 400
Leu Gly Arg Arg Val Cys Ile Arg Met Lys Ala Leu Ile Ile Gly Glu
405 410 415
Val Tyr Ala Lys Ala Leu Arg Arg Arg Met Thr Ser Pro Glu Glu Leu
420 425 430
Ile Glu Glu Val Asp Pro Lys Asp Gly Lys Ala Pro Ile Ala Asp Gln
435 440 445
Thr Ser Lys Glu Glu Ser Lys Ser Thr Glu Leu Gly Gly Ile Ile Asn
450 455 460
Leu Met Ala Val Asp Ala Ser Lys Val Ser Glu Leu Cys Ser Tyr Leu
465 470 475 480
His Phe Phe Val Asn Ser Phe Phe Met Ile Ile Val Ala Val Thr Leu
485 490 495
Leu Tyr Arg Leu Leu Gly Trp Ser Ala Leu Ala Gly Ser Ser Ser Ile
500 505 510
Leu Ile Leu Leu Pro Leu Asn Tyr Lys Leu Ala Ser Lys Ile Gly Glu
515 520 525
Phe Gln Lys Glu Met Leu Gly Ile Thr Asp Asn Arg Ile Gln Lys Leu
530 535 540
Asn Glu Ala Phe Gln Ser Ile Arg Ile Ile Lys Phe Phe Ala Trp Glu
545 550 555 560
Glu Asn Phe Ala Lys Glu Ile Met Lys Val Arg Asn Glu Glu Ile Arg
565 570 575
Tyr Leu Arg Tyr Arg Val Ile Val Trp Thr Cys Ser Ala Phe Val Trp
580 585 590
Phe Ile Thr Pro Thr Leu Val Thr Leu Ile Ser Phe Tyr Phe Tyr Val
595 600 605
Val Phe Gln Gly Lys Ile Leu Thr Thr Pro Val Ala Phe Thr Ala Leu
610 615 620
Ser Leu Phe Asn Leu Leu Arg Ser Pro Leu Asp Gln Leu Ser Asp Met
625 630 635 640
Leu Ser Phe Met Val Gln Ser Lys Val Ser Leu Asp Arg Val Gln Lys
645 650 655
Phe Leu Glu Glu Gln Glu Ser Asp Lys Tyr Glu Gln Leu Thr His Thr
660 665 670
Arg Gly Ala Asn Ser Pro Glu Val Gly Phe Glu Asn Ala Thr Leu Ser
675 680 685
Trp Asn Lys Gly Ser Lys Asn Asp Phe Gln Leu Lys Asp Ile Asp Ile
690 695 700
Ala Phe Lys Val Gly Lys Leu Asn Val Ile Ile Gly Pro Thr Gly Ser
705 710 715 720
Gly Lys Thr Ser Leu Leu Leu Gly Leu Leu Gly Glu Met Gln Leu Thr
725 730 735
Asn Gly Lys Ile Phe Leu Pro Gly Ser Thr Pro Arg Asp Glu Leu Ile
740 745 750
Pro Asn Pro Glu Thr Gly Met Thr Glu Ala Val Ala Tyr Cys Ser Gln
755 760 765
Ile Ala Trp Leu Leu Asn Asp Thr Val Lys Asn Asn Ile Val Phe Ala
770 775 780
Ala Pro Phe Asn Gln Gln Arg Tyr Asp Ala Val Ile Asp Ala Cys Gly
785 790 795 800
Leu Thr Arg Asp Leu Lys Val Leu Asp Ala Gly Asp Ala Thr Glu Ile
805 810 815
Gly Glu Lys Gly Ile Thr Leu Ser Gly Gly Gln Lys Gln Arg Val Ser
820 825 830
Leu Ala Arg Ala Leu Tyr Ser Asn Ala Arg His Val Leu Leu Asp Asp
835 840 845
Cys Leu Ser Ala Val Asp Ser His Thr Ala Ala Trp Ile Tyr Glu Asn
850 855 860
Cys Ile Thr Gly Pro Leu Met Lys Asp Arg Thr Cys Ile Leu Val Ser
865 870 875 880
His Asn Val Ala Leu Thr Val Arg Asp Ala Ala Trp Ile Val Ala Met
885 890 895
Asp Asn Gly Arg Val Leu Glu Gln Gly Thr Cys Glu Asp Leu Leu Ser
900 905 910
Ser Gly Ser Leu Gly His Asp Asp Leu Val Ser Thr Val Ile Ser Ser
915 920 925
Arg Ser Gln Ser Ser Val Asn Leu Lys Gln Leu Asn Val Ser Asp Thr
930 935 940
Ser Glu Ile His Gln Lys Leu Lys Lys Ile Ala Glu Ser Asp Lys Ala
945 950 955 960
Asp Gln Leu Asp Glu Glu Arg Leu Ser Pro Arg Gly Lys Leu Ile Glu
965 970 975
Asp Glu Thr Lys Ser Ser Gly Ala Val Ser Trp Glu Val Tyr Lys Phe
980 985 990
Tyr Gly Arg Ala Phe Gly Gly Val Phe Ile Trp Phe Val Phe Val Ala
995 1000 1005
Ala Phe Ala Ala Ser Gln Gly Ser Asn Ile Met Gln Ser Val Trp
1010 1015 1020
Leu Lys Ile Trp Ala Ala Ala Asn Asp Lys Leu Val Ser Pro Ala
1025 1030 1035
Phe Thr Met Ser Ile Asp Arg Ser Leu Asn Ala Leu Lys Glu Gly
1040 1045 1050
Phe Arg Ala Ser Val Ala Ser Val Glu Trp Ser Arg Pro Leu Gly
1055 1060 1065
Gly Glu Met Phe Arg Val Tyr Gly Glu Glu Ser Ser His Ser Ser
1070 1075 1080
Gly Tyr Tyr Ile Thr Ile Tyr Ala Leu Ile Gly Leu Ser Tyr Ala
1085 1090 1095
Leu Ile Ser Ala Phe Arg Val Tyr Val Val Phe Met Gly Gly Ile
1100 1105 1110
Val Ala Ser Asn Lys Ile Phe Glu Asp Met Leu Thr Lys Ile Phe
1115 1120 1125
Asn Ala Lys Leu Arg Phe Phe Asp Ser Thr Pro Ile Gly Arg Ile
1130 1135 1140
Met Asn Arg Phe Ser Lys Asp Thr Glu Ser Ile Asp Gln Glu Leu
1145 1150 1155
Ala Pro Tyr Ala Glu Gly Phe Ile Val Ser Val Leu Gln Cys Gly
1160 1165 1170
Ala Thr Ile Leu Leu Ile Cys Ile Ile Thr Pro Gly Phe Ile Val
1175 1180 1185
Phe Ala Ala Phe Ile Val Ile Ile Tyr Tyr Tyr Ile Gly Ala Leu
1190 1195 1200
Tyr Leu Ala Ser Ser Arg Glu Leu Lys Arg Tyr Asp Ser Ile Thr
1205 1210 1215
Val Ser Pro Ile His Gln His Phe Ser Glu Thr Leu Val Gly Val
1220 1225 1230
Thr Thr Ile Arg Ala Tyr Gly Asp Glu Arg Arg Phe Met Arg Gln
1235 1240 1245
Asn Leu Glu Lys Ile Asp Asn Asn Asn Arg Ser Phe Phe Tyr Leu
1250 1255 1260
Trp Val Ala Asn Arg Trp Leu Ala Leu Arg Val Asp Phe Val Gly
1265 1270 1275
Ala Leu Val Ser Leu Leu Ser Ala Ala Phe Val Met Leu Ser Ile
1280 1285 1290
Gly His Ile Asp Ala Gly Met Ala Gly Leu Ser Leu Ser Tyr Ala
1295 1300 1305
Ile Ala Phe Thr Gln Ser Ala Leu Trp Val Val Arg Leu Tyr Ser
1310 1315 1320
Val Val Glu Met Asn Met Asn Ser Val Glu Arg Leu Glu Glu Tyr
1325 1330 1335
Leu Asn Ile Asp Gln Glu Pro Asp Arg Glu Ile Pro Asp Asn Lys
1340 1345 1350
Pro Pro Ser Ser Trp Pro Glu Thr Gly Glu Ile Glu Val Asp Asp
1355 1360 1365
Val Ser Leu Arg Tyr Ala Pro Ser Leu Pro Lys Val Ile Lys Asn
1370 1375 1380
Val Ser Phe Lys Val Glu Pro Arg Ser Lys Ile Gly Ile Val Gly
1385 1390 1395
Arg Thr Gly Ala Gly Lys Ser Thr Ile Ile Thr Ala Phe Phe Arg
1400 1405 1410
Phe Val Asp Pro Glu Ser Gly Ser Ile Lys Ile Asp Gly Ile Asp
1415 1420 1425
Ile Thr Ser Ile Gly Leu Lys Asp Leu Arg Asn Ala Val Thr Ile
1430 1435 1440
Ile Pro Gln Asp Pro Thr Leu Phe Thr Gly Thr Ile Arg Ser Asn
1445 1450 1455
Leu Asp Pro Phe Asn Gln Tyr Ser Asp Ala Glu Ile Phe Glu Ser
1460 1465 1470
Leu Lys Arg Val Asn Leu Val Ser Thr Asp Glu Pro Thr Ser Gly
1475 1480 1485
Ser Ser Ser Asp Asn Ile Glu Asp Ser Asn Glu Asn Val Asn Lys
1490 1495 1500
Phe Leu Asn Leu Asn Asn Thr Val Ser Glu Gly Gly Ser Asn Leu
1505 1510 1515
Ser Gln Gly Gln Arg Gln Leu Thr Cys Leu Ala Arg Ser Leu Leu
1520 1525 1530
Lys Ser Pro Lys Ile Ile Leu Leu Asp Glu Ala Thr Ala Ser Ile
1535 1540 1545
Asp Tyr Asn Thr Asp Ser Lys Ile Gln Thr Thr Ile Arg Glu Glu
1550 1555 1560
Phe Ser Asp Ser Thr Ile Leu Thr Ile Ala His Arg Leu Arg Ser
1565 1570 1575
Ile Ile Asp Tyr Asp Lys Ile Leu Val Met Asp Ala Gly Arg Val
1580 1585 1590
Val Glu Tyr Asp Asp Pro Tyr Lys Leu Ile Ser Asp Gln Asn Ser
1595 1600 1605
Leu Phe Tyr Ser Met Cys Ser Asn Ser Gly Glu Leu Asp Thr Leu
1610 1615 1620
Val Lys Leu Ala Lys Glu Ala Phe Ile Ala Lys Arg Asn Lys Lys
1625 1630 1635
<210> 6
<211> 1559
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_4 ABC-transporter
<400> 6
Met Ser Ser Leu Glu Val Val Asp Gly Cys Pro Tyr Gly Tyr Arg Pro
1 5 10 15
Tyr Pro Asp Ser Gly Thr Asn Ala Leu Asn Pro Cys Phe Ile Ser Val
20 25 30
Ile Ser Ala Trp Gln Ala Val Phe Phe Leu Leu Ile Gly Ser Tyr Gln
35 40 45
Leu Trp Lys Leu Tyr Lys Asn Asn Lys Val Pro Pro Arg Phe Lys Asn
50 55 60
Phe Pro Thr Leu Pro Ser Lys Ile Asn Ser Arg His Leu Thr His Leu
65 70 75 80
Thr Asn Val Cys Phe Gln Ser Thr Leu Ile Ile Cys Glu Leu Ala Leu
85 90 95
Val Ser Gln Ser Ser Asp Arg Val Tyr Pro Phe Ile Leu Lys Lys Ala
100 105 110
Leu Tyr Leu Asn Leu Leu Phe Asn Leu Gly Ile Ser Leu Pro Thr Gln
115 120 125
Tyr Leu Ala Tyr Phe Lys Ser Thr Phe Ser Met Gly Asn Gln Leu Phe
130 135 140
Tyr Tyr Met Phe Gln Ile Leu Leu Gln Leu Phe Leu Ile Leu Gln Arg
145 150 155 160
Tyr Tyr His Gly Ser Ser Asn Glu Arg Leu Thr Val Ile Ser Gly Gln
165 170 175
Thr Ala Met Ile Leu Glu Val Leu Leu Leu Phe Asn Ser Val Ala Ile
180 185 190
Phe Ile Tyr Asp Leu Cys Ile Phe Glu Pro Ile Asn Glu Leu Ser Glu
195 200 205
Tyr Tyr Lys Lys Asn Gly Trp Tyr Pro Pro Val His Val Leu Ser Tyr
210 215 220
Ile Thr Phe Ile Trp Met Asn Lys Leu Ile Val Glu Thr Tyr Arg Asn
225 230 235 240
Lys Lys Ile Lys Asp Pro Asn Gln Leu Pro Leu Pro Pro Val Asp Leu
245 250 255
Asn Ile Lys Ser Ile Ser Lys Glu Phe Lys Ala Asn Trp Glu Leu Glu
260 265 270
Lys Trp Leu Asn Arg Asn Ser Leu Trp Arg Ala Ile Trp Lys Ser Phe
275 280 285
Gly Arg Thr Ile Ser Val Ala Met Leu Tyr Glu Thr Thr Ser Asp Leu
290 295 300
Leu Ser Val Val Gln Pro Gln Phe Leu Arg Ile Phe Ile Asp Gly Phe
305 310 315 320
Asn Pro Glu Thr Ser Ser Lys Tyr Pro Pro Leu Asn Gly Val Phe Ile
325 330 335
Ala Leu Thr Leu Phe Val Ile Ser Val Val Ser Val Phe Leu Thr Asn
340 345 350
Gln Phe Tyr Ile Gly Ile Phe Glu Ala Gly Leu Gly Ile Arg Gly Ser
355 360 365
Leu Ala Ser Leu Val Tyr Gln Lys Ser Leu Arg Leu Thr Leu Ala Glu
370 375 380
Arg Asn Glu Lys Ser Thr Gly Asp Ile Leu Asn Leu Met Ser Val Asp
385 390 395 400
Val Leu Arg Ile Gln Arg Phe Phe Glu Asn Ala Gln Thr Ile Ile Gly
405 410 415
Ala Pro Ile Gln Ile Ile Val Val Leu Thr Ser Leu Tyr Trp Leu Leu
420 425 430
Gly Lys Ala Val Ile Gly Gly Leu Val Thr Met Ala Ile Met Met Pro
435 440 445
Ile Asn Ala Phe Leu Ser Arg Lys Val Lys Lys Leu Ser Lys Thr Gln
450 455 460
Met Lys Tyr Lys Asp Met Arg Ile Lys Thr Ile Thr Glu Leu Leu Asn
465 470 475 480
Ala Ile Lys Ser Ile Lys Leu Tyr Ala Trp Glu Glu Pro Met Met Ala
485 490 495
Arg Leu Asn His Val Arg Asn Asp Met Glu Leu Lys Asn Phe Arg Lys
500 505 510
Ile Gly Ile Val Ser Asn Leu Ile Tyr Phe Ala Trp Asn Cys Val Pro
515 520 525
Leu Met Val Thr Cys Ser Thr Phe Gly Leu Phe Ser Leu Phe Ser Asp
530 535 540
Ser Pro Leu Ser Pro Ala Ile Val Phe Pro Ser Leu Ser Leu Phe Asn
545 550 555 560
Ile Leu Asn Ser Ala Ile Tyr Ser Val Pro Ser Met Ile Asn Thr Ile
565 570 575
Ile Glu Thr Ser Val Ser Met Glu Arg Leu Lys Ser Phe Leu Leu Ser
580 585 590
Asp Glu Ile Asp Asp Ser Phe Ile Glu Arg Ile Asp Pro Ser Ala Asp
595 600 605
Glu Arg Ala Leu Pro Ala Ile Glu Met Asn Asn Ile Thr Phe Leu Trp
610 615 620
Lys Ser Lys Glu Val Leu Ala Ser Ser Gln Ser Arg Asp Asn Leu Arg
625 630 635 640
Thr Asp Glu Glu Ser Ile Ile Gly Ser Ser Gln Ile Ala Leu Lys Asn
645 650 655
Ile Asp His Phe Glu Ala Lys Arg Gly Asp Leu Val Cys Val Val Gly
660 665 670
Arg Val Gly Ala Gly Lys Ser Thr Phe Leu Lys Ala Ile Leu Gly Gln
675 680 685
Leu Pro Cys Met Ser Gly Ser Arg Asp Ser Ile Pro Pro Lys Leu Ile
690 695 700
Ile Arg Ser Ser Ser Val Ala Tyr Cys Ser Gln Glu Ser Trp Ile Met
705 710 715 720
Asn Ala Ser Val Arg Glu Asn Ile Leu Phe Gly His Lys Phe Asp Gln
725 730 735
Asn Tyr Tyr Asp Leu Thr Ile Lys Ala Cys Gln Leu Leu Pro Asp Leu
740 745 750
Lys Ile Leu Pro Asp Gly Asp Glu Thr Leu Val Gly Glu Lys Gly Ile
755 760 765
Ser Leu Ser Gly Gly Gln Lys Ala Arg Leu Ser Leu Ala Arg Ala Val
770 775 780
Tyr Ser Arg Ala Asp Ile Tyr Leu Leu Asp Asp Ile Leu Ser Ala Val
785 790 795 800
Asp Ala Glu Val Ser Lys Asn Ile Ile Glu Tyr Val Leu Ile Gly Lys
805 810 815
Thr Ala Leu Leu Lys Asn Lys Thr Ile Ile Leu Thr Thr Asn Thr Val
820 825 830
Ser Ile Leu Lys His Ser Gln Met Ile Tyr Ala Leu Glu Asn Gly Glu
835 840 845
Ile Val Glu Gln Gly Asn Tyr Glu Asp Val Met Asn Arg Lys Asn Asn
850 855 860
Thr Ser Lys Leu Lys Lys Leu Leu Glu Glu Phe Asp Ser Pro Ile Asp
865 870 875 880
Asn Gly Asn Glu Ser Asp Val Gln Thr Glu His Arg Ser Glu Ser Glu
885 890 895
Val Asp Glu Pro Leu Gln Leu Lys Val Thr Glu Ser Glu Thr Glu Asp
900 905 910
Glu Val Val Thr Glu Ser Glu Leu Glu Leu Ile Lys Ala Asn Ser Arg
915 920 925
Arg Ala Ser Leu Ala Thr Leu Arg Pro Arg Pro Phe Val Gly Ala Gln
930 935 940
Leu Asp Ser Val Lys Lys Thr Ala Gln Glu Ala Glu Lys Thr Glu Val
945 950 955 960
Gly Arg Val Lys Thr Lys Val Tyr Leu Ala Tyr Ile Lys Ala Cys Gly
965 970 975
Val Leu Gly Val Val Leu Phe Phe Leu Phe Met Ile Leu Thr Arg Val
980 985 990
Phe Asp Leu Ala Glu Asn Phe Trp Leu Lys Tyr Trp Ser Glu Ser Asn
995 1000 1005
Glu Lys Asn Gly Ser Asn Glu Arg Val Trp Met Phe Val Gly Val
1010 1015 1020
Tyr Ser Leu Ile Gly Val Ala Ser Ala Ala Phe Asn Asn Leu Arg
1025 1030 1035
Ser Ile Met Met Leu Leu Tyr Cys Ser Ile Arg Gly Ser Lys Lys
1040 1045 1050
Leu His Glu Ser Met Ala Lys Ser Val Ile Arg Ser Pro Met Thr
1055 1060 1065
Phe Phe Glu Thr Thr Pro Val Gly Arg Ile Ile Asn Arg Phe Ser
1070 1075 1080
Ser Asp Met Asp Ala Val Asp Ser Asn Leu Gln Tyr Ile Phe Ser
1085 1090 1095
Phe Phe Phe Lys Ser Ile Leu Thr Tyr Leu Val Thr Val Ile Leu
1100 1105 1110
Val Gly Tyr Asn Met Pro Trp Phe Leu Val Phe Asn Met Phe Leu
1115 1120 1125
Val Val Ile Tyr Ile Tyr Tyr Gln Thr Phe Tyr Ile Val Leu Ser
1130 1135 1140
Arg Glu Leu Lys Arg Leu Ile Ser Ile Ser Tyr Ser Pro Ile Met
1145 1150 1155
Ser Leu Met Ser Glu Ser Leu Asn Gly Tyr Ser Ile Ile Asp Ala
1160 1165 1170
Tyr Asp His Phe Glu Arg Phe Ile Tyr Leu Asn Tyr Glu Lys Ile
1175 1180 1185
Gln Tyr Asn Val Asp Phe Val Phe Asn Phe Arg Ser Thr Asn Arg
1190 1195 1200
Trp Leu Ser Val Arg Leu Gln Thr Ile Gly Ala Thr Ile Val Leu
1205 1210 1215
Ala Thr Ala Ile Leu Ala Leu Ala Thr Met Asn Thr Lys Arg Gln
1220 1225 1230
Leu Ser Ser Gly Met Val Gly Leu Leu Met Ser Tyr Ser Leu Glu
1235 1240 1245
Val Thr Gly Ser Leu Thr Trp Ile Val Arg Thr Thr Val Met Ile
1250 1255 1260
Glu Thr Asn Ile Val Ser Val Glu Arg Ile Val Glu Tyr Cys Glu
1265 1270 1275
Leu Pro Pro Glu Ala Gln Ser Ile Asn Pro Glu Lys Arg Pro Asp
1280 1285 1290
Glu Asn Trp Pro Ser Lys Gly Gly Ile Glu Phe Lys Asn Tyr Ser
1295 1300 1305
Thr Lys Tyr Arg Glu Asn Leu Asp Pro Val Leu Asn Asn Ile Asn
1310 1315 1320
Val Lys Ile Glu Pro Cys Glu Lys Val Gly Ile Val Gly Arg Thr
1325 1330 1335
Gly Ala Gly Lys Ser Thr Leu Ser Leu Ala Leu Phe Arg Ile Leu
1340 1345 1350
Glu Pro Thr Glu Gly Lys Ile Ile Ile Asp Gly Ile Asp Ile Ser
1355 1360 1365
Asp Ile Gly Leu Phe Asp Leu Arg Ser His Leu Ala Ile Ile Pro
1370 1375 1380
Gln Asp Ala Gln Ala Phe Glu Gly Thr Val Lys Thr Asn Leu Asp
1385 1390 1395
Pro Phe Asn Arg Tyr Ser Glu Asp Glu Leu Lys Arg Ala Val Glu
1400 1405 1410
Gln Ala His Leu Lys Pro His Leu Glu Lys Met Leu His Ser Lys
1415 1420 1425
Pro Arg Gly Asp Asp Ser Asn Glu Glu Asp Gly Asn Val Asn Asp
1430 1435 1440
Ile Leu Asp Val Lys Ile Asn Glu Asn Gly Ser Asn Leu Ser Val
1445 1450 1455
Gly Gln Arg Gln Leu Leu Cys Leu Ala Arg Ala Leu Leu Asn Arg
1460 1465 1470
Ser Lys Ile Leu Val Leu Asp Glu Ala Thr Ala Ser Val Asp Met
1475 1480 1485
Glu Thr Asp Lys Ile Ile Gln Asp Thr Ile Arg Arg Glu Phe Lys
1490 1495 1500
Asp Arg Thr Ile Leu Thr Ile Ala His Arg Ile Asp Thr Val Leu
1505 1510 1515
Asp Ser Asp Lys Ile Ile Val Leu Asp Gln Gly Ser Val Arg Glu
1520 1525 1530
Phe Asp Ser Pro Ser Lys Leu Leu Ser Asp Lys Thr Ser Ile Phe
1535 1540 1545
Tyr Ser Leu Cys Glu Lys Gly Gly Tyr Leu Lys
1550 1555
<210> 7
<211> 1381
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_5 ABC-transporter
<400> 7
Met Thr Ser Pro Gly Ser Glu Lys Cys Thr Pro Arg Ser Asp Glu Asp
1 5 10 15
Leu Glu Arg Ser Glu Pro Gln Leu Gln Arg Arg Leu Leu Thr Pro Phe
20 25 30
Leu Leu Ser Lys Lys Val Pro Pro Ile Pro Lys Glu Asp Glu Arg Lys
35 40 45
Pro Tyr Pro Tyr Leu Lys Thr Asn Pro Leu Ser Gln Ile Leu Phe Trp
50 55 60
Trp Leu Asn Pro Leu Leu Arg Val Gly Tyr Lys Arg Thr Leu Asp Pro
65 70 75 80
Asn Asp Phe Tyr Tyr Leu Glu His Ser Gln Asp Ile Glu Thr Thr Tyr
85 90 95
Ser Asn Tyr Glu Met His Leu Ala Arg Ile Leu Glu Lys Asp Arg Ala
100 105 110
Lys Ala Arg Ala Lys Asp Pro Thr Leu Thr Asp Glu Asp Leu Lys Asn
115 120 125
Arg Glu Tyr Pro Lys Asn Ala Val Ile Lys Ala Leu Phe Leu Thr Phe
130 135 140
Lys Trp Lys Tyr Leu Trp Ser Ile Phe Leu Lys Leu Leu Ser Asp Ile
145 150 155 160
Val Leu Val Leu Asn Pro Leu Leu Ser Lys Ala Leu Ile Asn Phe Val
165 170 175
Asp Glu Lys Met Tyr Asn Pro Asp Met Ser Val Gly Arg Gly Val Gly
180 185 190
Tyr Ala Ile Gly Val Thr Phe Met Leu Gly Thr Ser Gly Ile Leu Ile
195 200 205
Asn His Phe Leu Tyr Leu Ser Leu Thr Val Gly Ala His Cys Lys Ala
210 215 220
Val Leu Thr Thr Ala Ile Met Asn Lys Ser Phe Arg Ala Ser Ala Lys
225 230 235 240
Ser Lys His Glu Tyr Pro Ser Gly Arg Val Thr Ser Leu Met Ser Thr
245 250 255
Asp Leu Ala Arg Ile Asp Leu Ala Ile Gly Phe Gln Pro Phe Ala Ile
260 265 270
Thr Val Pro Val Pro Ile Gly Val Ala Ile Ala Leu Leu Ile Val Asn
275 280 285
Ile Gly Val Ser Ala Leu Ala Gly Ile Ala Val Phe Leu Val Cys Ile
290 295 300
Val Val Ile Ser Ala Ser Ser Lys Ser Leu Leu Lys Met Arg Lys Gly
305 310 315 320
Ala Asn Gln Tyr Thr Asp Ala Arg Ile Ser Tyr Met Arg Glu Ile Leu
325 330 335
Gln Asn Met Arg Ile Ile Lys Phe Tyr Ser Trp Glu Asp Ala Tyr Glu
340 345 350
Lys Ser Val Val Thr Glu Arg Asn Ser Glu Met Ser Ile Ile Leu Lys
355 360 365
Met Gln Ser Ile Arg Asn Phe Leu Leu Ala Leu Ser Leu Ser Leu Pro
370 375 380
Ala Ile Ile Ser Met Val Ala Phe Leu Val Leu Tyr Gly Val Ser Asn
385 390 395 400
Asp Lys Asn Pro Gly Asn Ile Phe Ser Ser Ile Ser Leu Phe Ser Val
405 410 415
Leu Ala Gln Gln Thr Met Met Leu Pro Met Ala Leu Ala Thr Gly Ala
420 425 430
Asp Ala Lys Ile Gly Leu Glu Arg Leu Arg Gln Tyr Leu Gln Ser Gly
435 440 445
Asp Ile Glu Lys Glu Tyr Glu Asp His Glu Lys Pro Gly Asp Arg Asp
450 455 460
Val Val Leu Pro Asp Asn Val Ala Val Glu Leu Asn Asn Ala Ser Phe
465 470 475 480
Ile Trp Glu Lys Phe Asp Asp Ala Asp Asp Asn Asp Gly Asn Ser Glu
485 490 495
Lys Thr Lys Glu Val Val Val Thr Ser Lys Ser Ser Leu Thr Asp Ser
500 505 510
Ser His Ile Asp Lys Ser Thr Asp Ser Ala Asp Gly Glu Tyr Ile Lys
515 520 525
Ser Val Phe Glu Gly Phe Asn Asn Ile Asn Leu Thr Ile Lys Lys Gly
530 535 540
Glu Phe Val Ile Ile Thr Gly Pro Ile Gly Ser Gly Lys Ser Ser Leu
545 550 555 560
Leu Val Ala Leu Ala Gly Phe Met Lys Lys Thr Ser Gly Thr Leu Gly
565 570 575
Val Asn Gly Thr Met Leu Leu Cys Gly Gln Pro Trp Val Gln Asn Cys
580 585 590
Thr Val Arg Asp Asn Ile Leu Phe Gly Leu Glu Tyr Asp Glu Ala Arg
595 600 605
Tyr Asp Arg Val Val Glu Val Cys Ala Leu Gly Asp Asp Leu Lys Met
610 615 620
Phe Thr Ala Gly Asp Gln Thr Glu Ile Gly Glu Arg Gly Ile Thr Leu
625 630 635 640
Ser Gly Gly Gln Lys Ala Arg Ile Asn Leu Ala Arg Ala Val Tyr Ala
645 650 655
Asn Lys Asp Ile Ile Leu Leu Asp Asp Val Leu Ser Ala Val Asp Ala
660 665 670
Arg Val Gly Lys Leu Ile Val Asp Asp Cys Leu Thr Ser Phe Leu Gly
675 680 685
Asp Lys Thr Arg Ile Leu Ala Thr His Gln Leu Ser Leu Ile Glu Ala
690 695 700
Ala Asp Arg Val Ile Tyr Leu Asn Gly Asp Gly Thr Ile His Ile Gly
705 710 715 720
Thr Val Gln Glu Leu Leu Glu Ser Asn Glu Gly Phe Leu Lys Leu Met
725 730 735
Glu Phe Ser Arg Lys Ser Glu Ser Glu Asp Glu Glu Asp Val Glu Ala
740 745 750
Ala Asn Glu Lys Asp Val Ser Leu Gln Lys Ala Val Ser Val Val Gln
755 760 765
Glu Gln Asp Ala His Ala Gly Val Leu Ile Gly Gln Glu Glu Arg Ala
770 775 780
Val Asn Gly Ile Glu Trp Asp Ile Tyr Lys Glu Tyr Leu His Glu Gly
785 790 795 800
Arg Gly Lys Leu Gly Ile Phe Ala Ile Pro Thr Ile Ile Met Leu Leu
805 810 815
Val Leu Asp Val Phe Thr Ser Ile Phe Val Asn Val Trp Leu Ser Phe
820 825 830
Trp Ile Ser His Lys Phe Lys Ala Arg Ser Asp Gly Phe Tyr Ile Gly
835 840 845
Leu Tyr Val Met Phe Val Ile Leu Ser Val Ile Trp Ile Thr Ala Glu
850 855 860
Phe Val Val Met Gly Tyr Phe Ser Ser Thr Ala Ala Arg Arg Leu Asn
865 870 875 880
Leu Lys Ala Met Lys Arg Val Leu His Thr Pro Met His Phe Leu Asp
885 890 895
Val Thr Pro Met Gly Arg Ile Leu Asn Arg Phe Thr Lys Asp Thr Asp
900 905 910
Val Leu Asp Asn Glu Ile Gly Glu Gln Ala Arg Met Phe Leu His Pro
915 920 925
Ala Ala Tyr Val Ile Gly Val Leu Ile Leu Cys Ile Ile Tyr Ile Pro
930 935 940
Trp Phe Ala Ile Ala Ile Pro Pro Leu Ala Ile Leu Phe Thr Phe Ile
945 950 955 960
Thr Asn Phe Tyr Ile Ala Ser Ser Arg Glu Val Lys Arg Ile Glu Ala
965 970 975
Ile Gln Arg Ser Leu Val Tyr Asn Asn Phe Asn Glu Val Leu Asn Gly
980 985 990
Leu Gln Thr Leu Lys Ala Tyr Asn Ala Thr Ser Arg Phe Met Glu Lys
995 1000 1005
Asn Lys Arg Leu Leu Asn Arg Met Asn Glu Ala Tyr Leu Leu Val
1010 1015 1020
Ile Ala Asn Gln Arg Trp Ile Ser Val Asn Leu Asp Leu Val Ser
1025 1030 1035
Cys Cys Phe Val Phe Leu Ile Ser Met Leu Ser Val Phe Arg Val
1040 1045 1050
Phe Asp Ile Asn Ala Ser Ser Val Gly Leu Val Val Thr Ser Val
1055 1060 1065
Leu Gln Ile Gly Gly Leu Met Ser Leu Ile Met Arg Ala Tyr Thr
1070 1075 1080
Thr Val Glu Asn Glu Met Asn Ser Val Glu Arg Leu Cys His Tyr
1085 1090 1095
Ala Asn Lys Leu Glu Gln Glu Ala Pro Tyr Ile Met Asn Glu Thr
1100 1105 1110
Lys Pro Arg Pro Thr Trp Pro Glu His Gly Ala Ile Glu Phe Lys
1115 1120 1125
His Ala Ser Met Arg Tyr Arg Glu Gly Leu Pro Leu Val Leu Lys
1130 1135 1140
Asp Leu Thr Ile Ser Val Lys Gly Gly Glu Lys Ile Gly Ile Cys
1145 1150 1155
Gly Arg Thr Gly Ala Gly Lys Ser Thr Ile Met Asn Ala Leu Tyr
1160 1165 1170
Arg Leu Thr Glu Leu Ala Glu Gly Ser Ile Thr Ile Asp Gly Val
1175 1180 1185
Glu Ile Ser Gln Leu Gly Leu Tyr Asp Leu Arg Ser Lys Leu Ala
1190 1195 1200
Ile Ile Pro Gln Asp Pro Val Leu Phe Arg Gly Thr Ile Arg Lys
1205 1210 1215
Asn Leu Asp Pro Phe Gly Gln Asn Asp Asp Glu Thr Leu Trp Asp
1220 1225 1230
Ala Leu Arg Arg Ser Gly Leu Val Glu Gly Ser Ile Leu Asn Thr
1235 1240 1245
Ile Lys Ser Gln Ser Lys Asp Asp Pro Asn Phe His Lys Phe His
1250 1255 1260
Leu Asp Gln Thr Val Glu Asp Glu Gly Ala Asn Phe Ser Leu Gly
1265 1270 1275
Glu Arg Gln Leu Ile Ala Leu Ala Arg Ala Leu Val Arg Asn Ser
1280 1285 1290
Lys Ile Leu Ile Leu Asp Glu Ala Thr Ser Ser Val Asp Tyr Glu
1295 1300 1305
Thr Asp Ser Lys Ile Gln Lys Thr Ile Ser Thr Glu Phe Ser His
1310 1315 1320
Cys Thr Ile Leu Cys Ile Ala His Arg Leu Lys Thr Ile Leu Thr
1325 1330 1335
Tyr Asp Arg Ile Leu Val Leu Glu Lys Gly Glu Val Glu Glu Phe
1340 1345 1350
Asp Thr Pro Arg Val Leu Tyr Ser Lys Asn Gly Val Phe Arg Gln
1355 1360 1365
Met Cys Glu Arg Ser Glu Ile Thr Ser Ala Asp Phe Val
1370 1375 1380
<210> 8
<211> 1650
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: T4_Fungal_8 ABC-transporter
<400> 8
Met Ser Gly Ser Asn Ser Asn Ser Asn Leu Asp Ala Ile Ser Asp Ser
1 5 10 15
Cys Pro Phe Trp Arg Tyr Asp Asp Ile Thr Glu Cys Gly Arg Val Gln
20 25 30
Tyr Ile Asn Tyr Tyr Leu Pro Ile Thr Leu Val Gly Val Ser Leu Leu
35 40 45
Tyr Leu Phe Lys Asn Ala Ile Gln His Tyr Tyr Arg Lys Pro Gln Glu
50 55 60
Ile Lys Pro Ser Val Ala Ser Glu Leu Leu Gly Ser Asn Leu Thr Asp
65 70 75 80
Leu Pro Asn Glu Asn Lys Pro Leu Leu Ser Glu Ser Thr Gln Ala Leu
85 90 95
Tyr Thr Asn Pro Asp Ser Asn Lys Thr Gly Phe Ser Leu Lys Glu Glu
100 105 110
His Phe Ser Ile Asn Lys Val Thr Leu Thr Glu Ile His Ser Asn Lys
115 120 125
His Asp Ala Val Lys Ile Val Arg Arg Asn Trp Leu Glu Lys Leu Arg
130 135 140
Val Phe Leu Glu Trp Val Leu Cys Ala Leu Gln Leu Cys Ile Tyr Ile
145 150 155 160
Ser Val Trp Ser Lys Tyr Thr Asn Thr Gln Glu Asp Phe Pro Met His
165 170 175
Ala Ser Ile Ser Gly Leu Met Leu Trp Ser Leu Leu Leu Leu Val Val
180 185 190
Ser Leu Arg Leu Ala Asn Ile Asn Gln Asn Ile Ser Trp Ile Asn Ser
195 200 205
Gly Pro Gly Asn Leu Trp Ala Leu Ser Phe Ala Cys Tyr Leu Ser Leu
210 215 220
Phe Cys Gly Ser Val Leu Pro Leu Arg Ser Ile Tyr Ile Gly His Ile
225 230 235 240
Thr Asp Glu Ile Ala Ser Thr Phe Tyr Lys Leu Gln Phe Tyr Leu Ser
245 250 255
Leu Thr Leu Phe Leu Leu Leu Phe Thr Ser Gln Ala Gly Asn Arg Phe
260 265 270
Ala Ile Ile Tyr Lys Ser Thr Pro Asp Ile Thr Pro Ser Pro Glu Pro
275 280 285
Ile Val Ser Ile Ala Ser Tyr Ile Thr Trp Ala Trp Val Asp Lys Phe
290 295 300
Leu Trp Lys Ala His Gln Asn Tyr Ile Glu Met Lys Asp Val Trp Gly
305 310 315 320
Leu Met Val Glu Asp Tyr Ser Ile Leu Val Ile Lys Arg Phe Asn His
325 330 335
Phe Val Gln Asn Lys Thr Lys Ser Arg Thr Phe Ser Phe Asn Leu Ile
340 345 350
His Phe Phe Met Lys Phe Ile Ala Ile Gln Gly Ala Trp Ala Thr Ile
355 360 365
Ser Ser Val Ile Ser Phe Val Pro Thr Met Leu Leu Arg Arg Ile Leu
370 375 380
Glu Tyr Val Glu Asp Gln Ser Thr Ala Pro Leu Asn Leu Ala Trp Met
385 390 395 400
Tyr Ile Phe Leu Met Phe Leu Ala Arg Ile Leu Thr Ala Ile Cys Ala
405 410 415
Ala Gln Ala Leu Phe Leu Gly Arg Arg Val Cys Ile Arg Met Lys Ala
420 425 430
Ile Ile Ile Ser Glu Ile Tyr Ser Lys Ala Leu Arg Arg Lys Ile Ser
435 440 445
Pro Asn Ser Thr Lys Glu Pro Thr Asp Val Val Asp Pro Gln Glu Leu
450 455 460
Asn Asp Lys Gln His Val Asp Gly Asp Glu Glu Ser Ala Thr Thr Ala
465 470 475 480
Asn Leu Gly Ala Ile Ile Asn Leu Met Ala Val Asp Ala Phe Lys Val
485 490 495
Ser Glu Ile Cys Ala Tyr Leu His Ser Phe Ile Glu Ala Ile Ile Met
500 505 510
Thr Ile Val Ala Leu Phe Leu Leu Tyr Arg Leu Ile Gly Trp Ser Ala
515 520 525
Leu Val Gly Ser Ala Met Ile Ile Cys Phe Leu Pro Leu Asn Phe Lys
530 535 540
Leu Ala Ser Leu Leu Gly Thr Leu Gln Lys Lys Ser Leu Ala Ile Thr
545 550 555 560
Asp Lys Arg Ile Gln Lys Leu Asn Glu Ala Phe Gln Ala Ile Arg Ile
565 570 575
Ile Lys Phe Phe Ser Trp Glu Glu Asn Phe Glu Lys Asp Ile Gln Asn
580 585 590
Thr Arg Asp Glu Glu Leu Asn Met Leu Leu Lys Arg Ser Ile Val Trp
595 600 605
Ala Leu Ser Ser Leu Val Trp Phe Ile Thr Pro Ser Ile Val Thr Ser
610 615 620
Ala Ser Phe Ala Val Tyr Ile Tyr Val Gln Gly Gln Thr Leu Thr Thr
625 630 635 640
Pro Val Ala Phe Thr Ala Leu Ser Leu Phe Ala Leu Leu Arg Asn Pro
645 650 655
Leu Asp Met Leu Ser Asp Met Leu Ser Phe Val Ile Gln Ser Lys Val
660 665 670
Ser Leu Asp Arg Val Gln Glu Phe Leu Asn Glu Glu Glu Thr Lys Lys
675 680 685
Tyr Glu Gln Leu Thr Val Ser Arg Asn Lys Leu Gly Leu Gln Asn Ala
690 695 700
Thr Phe Thr Trp Asp Lys Asn Asn Gln Asp Phe Lys Leu Lys Asn Leu
705 710 715 720
Thr Ile Asp Phe Lys Ile Gly Lys Leu Asn Val Ile Val Gly Pro Thr
725 730 735
Gly Ser Gly Lys Thr Ser Leu Leu Met Gly Leu Leu Gly Glu Met Glu
740 745 750
Leu Leu Asn Gly Lys Val Phe Val Pro Ser Leu Asn Pro Arg Glu Glu
755 760 765
Leu Val Val Glu Ala Asp Gly Met Thr Asn Ser Ile Ala Tyr Cys Ser
770 775 780
Gln Ala Ala Trp Leu Leu Asn Asp Thr Val Arg Asn Asn Ile Leu Phe
785 790 795 800
Asn Ala Pro Tyr Asn Glu Asn Arg Tyr Asn Ala Val Ile Ser Ala Cys
805 810 815
Gly Leu Lys Arg Asp Phe Glu Ile Leu Ser Ala Gly Asp Gln Thr Glu
820 825 830
Ile Gly Glu Lys Gly Ile Thr Leu Ser Gly Gly Gln Lys Gln Arg Val
835 840 845
Ser Leu Ala Arg Ser Leu Tyr Ser Ser Ser Arg His Leu Leu Leu Asp
850 855 860
Asp Cys Leu Ser Ala Val Asp Ser His Thr Ala Leu Trp Ile Tyr Glu
865 870 875 880
Asn Cys Ile Thr Gly Pro Leu Met Glu Gly Arg Thr Cys Val Leu Val
885 890 895
Ser His Asn Val Ala Leu Thr Leu Lys Asn Ala Asp Trp Val Ile Ile
900 905 910
Met Glu Asn Gly Arg Val Lys Glu Gln Gly Glu Pro Val Glu Leu Leu
915 920 925
Gln Lys Gly Ser Leu Gly Asp Asp Ser Met Val Lys Ser Ser Ile Leu
930 935 940
Ser Arg Thr Ala Ser Ser Val Asn Ile Ser Glu Thr Asn Ser Lys Ile
945 950 955 960
Ser Ser Gly Pro Lys Ala Pro Ala Glu Ser Asp Asn Ala Asn Glu Glu
965 970 975
Ser Thr Thr Cys Gly Asp Arg Ser Lys Ser Ser Gly Lys Leu Ile Ala
980 985 990
Glu Glu Thr Lys Ser Asn Gly Val Val Ser Leu Asp Val Tyr Lys Trp
995 1000 1005
Tyr Ala Val Phe Phe Gly Gly Trp Lys Met Ile Ser Phe Leu Cys
1010 1015 1020
Phe Ile Phe Leu Phe Ala Gln Met Ile Ser Ile Ser Gln Ala Trp
1025 1030 1035
Trp Leu Arg Ala Trp Ala Ser Asn Asn Thr Leu Lys Val Phe Ser
1040 1045 1050
Asn Leu Gly Leu Gln Thr Met Arg Pro Phe Ala Leu Ser Leu Gln
1055 1060 1065
Gly Lys Glu Ala Ser Pro Val Thr Leu Ser Ala Val Phe Pro Asn
1070 1075 1080
Gly Ser Leu Thr Thr Ala Thr Glu Pro Asn His Ser Asn Ala Tyr
1085 1090 1095
Tyr Leu Ser Ile Tyr Leu Gly Ile Gly Val Phe Gln Ala Leu Cys
1100 1105 1110
Ser Ser Ser Lys Ala Ile Ile Asn Phe Val Ala Gly Ile Arg Ala
1115 1120 1125
Ser Arg Lys Ile Phe Asn Leu Leu Leu Lys Asn Val Leu Tyr Ala
1130 1135 1140
Lys Leu Arg Phe Phe Asp Ser Thr Pro Ile Gly Arg Ile Met Asn
1145 1150 1155
Arg Phe Ser Lys Asp Ile Glu Ser Ile Asp Gln Glu Leu Thr Pro
1160 1165 1170
Tyr Met Glu Gly Ala Phe Gly Ser Leu Ile Gln Cys Val Ser Thr
1175 1180 1185
Ile Ile Val Ile Ala Tyr Ile Thr Pro Gln Phe Leu Ile Val Ala
1190 1195 1200
Ala Ile Val Met Leu Leu Phe Tyr Phe Val Ala Tyr Phe Tyr Met
1205 1210 1215
Ser Gly Ala Arg Glu Leu Lys Arg Leu Glu Ser Met Ser Arg Ser
1220 1225 1230
Pro Ile His Gln His Phe Ser Glu Thr Leu Val Gly Ile Thr Thr
1235 1240 1245
Ile Arg Ala Phe Ser Asp Glu Arg Arg Phe Leu Val Asp Asn Met
1250 1255 1260
Lys Lys Ile Asp Asp Asn Asn Arg Pro Phe Phe Tyr Leu Trp Val
1265 1270 1275
Cys Asn Arg Trp Leu Ser Tyr Arg Ile Glu Leu Ile Gly Ala Leu
1280 1285 1290
Ile Val Leu Ala Ala Gly Ser Phe Ile Leu Leu Asn Ile Lys Ser
1295 1300 1305
Ile Asp Ser Gly Leu Ala Gly Ile Ser Leu Gly Phe Ala Ile Gln
1310 1315 1320
Phe Thr Asp Gly Ala Leu Trp Val Val Arg Leu Tyr Ser Asn Val
1325 1330 1335
Glu Met Asn Met Asn Ser Val Glu Arg Leu Lys Glu Tyr Thr Thr
1340 1345 1350
Ile Glu Gln Glu Pro Ser Asn Val Gly Ala Leu Val Pro Pro Cys
1355 1360 1365
Glu Trp Pro Gln Asn Gly Lys Ile Glu Val Lys Asp Leu Ser Leu
1370 1375 1380
Arg Tyr Ala Ala Gly Leu Pro Lys Val Ile Lys Asn Val Thr Phe
1385 1390 1395
Thr Val Asp Ser Lys Cys Lys Val Gly Ile Val Gly Arg Thr Gly
1400 1405 1410
Ala Gly Lys Ser Thr Ile Ile Thr Ala Leu Phe Arg Phe Leu Asp
1415 1420 1425
Pro Glu Thr Gly Tyr Ile Lys Ile Asp Asp Val Asp Ile Thr Thr
1430 1435 1440
Ile Gly Leu Lys Arg Leu Arg Gln Ser Ile Thr Ile Ile Pro Gln
1445 1450 1455
Asp Pro Thr Leu Phe Thr Gly Thr Leu Lys Thr Asn Leu Asp Pro
1460 1465 1470
Tyr Asn Glu Tyr Ser Glu Ala Glu Ile Phe Glu Ala Leu Lys Arg
1475 1480 1485
Val Asn Leu Val Ser Ser Glu Glu Leu Gly Asn Pro Ser Thr Ser
1490 1495 1500
Asp Ser Thr Ser Val His Ser Ala Asn Met Asn Lys Phe Leu Asp
1505 1510 1515
Leu Glu Asn Glu Val Ser Glu Gly Gly Ser Asn Leu Ser Gln Gly
1520 1525 1530
Gln Arg Gln Leu Ile Cys Leu Ala Arg Ser Leu Leu Arg Cys Pro
1535 1540 1545
Lys Val Ile Leu Leu Asp Glu Ala Thr Ala Ser Ile Asp Tyr Asn
1550 1555 1560
Ser Asp Ser Lys Ile Gln Ala Thr Ile Arg Glu Glu Phe Ser Asn
1565 1570 1575
Ser Thr Ile Leu Thr Ile Ala His Arg Leu Arg Ser Ile Ile Asp
1580 1585 1590
Tyr Asp Lys Ile Leu Val Met Asp Ala Gly Glu Val Lys Glu Tyr
1595 1600 1605
Asp His Pro Tyr Ser Leu Leu Leu Asn Arg Asp Ser Ile Phe Tyr
1610 1615 1620
His Met Cys Glu Asp Ser Gly Glu Leu Glu Val Leu Ile Gln Leu
1625 1630 1635
Ala Lys Glu Ser Phe Val Lys Lys Leu Asn Ala Asn
1640 1645 1650
<210> 9
<211> 320
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Bt.GGPPS
<400> 9
Met Leu Thr Ser Ser Lys Ser Ile Glu Ser Phe Pro Lys Asn Val Gln
1 5 10 15
Pro Tyr Gly Lys His Tyr Gln Asn Gly Leu Glu Pro Val Gly Lys Ser
20 25 30
Gln Glu Asp Ile Leu Leu Glu Pro Phe His Tyr Leu Cys Ser Asn Pro
35 40 45
Gly Lys Asp Val Arg Thr Lys Met Ile Glu Ala Phe Asn Ala Trp Leu
50 55 60
Lys Val Pro Lys Asp Asp Leu Ile Val Ile Thr Arg Val Ile Glu Met
65 70 75 80
Leu His Ser Ala Ser Leu Leu Ile Asp Asp Val Glu Asp Asp Ser Val
85 90 95
Leu Arg Arg Gly Val Pro Ala Ala His His Ile Tyr Gly Thr Pro Gln
100 105 110
Thr Ile Asn Cys Ala Asn Tyr Val Tyr Phe Leu Ala Leu Lys Glu Ile
115 120 125
Ala Lys Leu Asn Lys Pro Asn Met Ile Thr Ile Tyr Thr Asp Glu Leu
130 135 140
Ile Asn Leu His Arg Gly Gln Gly Met Glu Leu Phe Trp Arg Asp Thr
145 150 155 160
Leu Thr Cys Pro Thr Glu Lys Glu Phe Leu Asp Met Val Asn Asp Lys
165 170 175
Thr Gly Gly Leu Leu Arg Leu Ala Val Lys Leu Met Gln Glu Ala Ser
180 185 190
Gln Ser Gly Thr Asp Tyr Thr Gly Leu Val Ser Lys Ile Gly Ile His
195 200 205
Phe Gln Val Arg Asp Asp Tyr Met Asn Leu Gln Ser Lys Asn Tyr Ala
210 215 220
Asp Asn Lys Gly Phe Cys Glu Asp Leu Thr Glu Gly Lys Phe Ser Phe
225 230 235 240
Pro Ile Ile His Ser Ile Arg Ser Asp Pro Ser Asn Arg Gln Leu Leu
245 250 255
Asn Ile Leu Lys Gln Arg Ser Ser Ser Ile Glu Leu Lys Gln Phe Ala
260 265 270
Leu Gln Leu Leu Glu Asn Thr Asn Thr Phe Gln Tyr Cys Arg Asp Phe
275 280 285
Leu Arg Val Leu Glu Lys Glu Ala Arg Glu Glu Ile Lys Leu Leu Gly
290 295 300
Gly Asn Ile Met Leu Glu Lys Ile Met Asp Val Leu Ser Val Asn Glu
305 310 315 320
<210> 10
<211> 736
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Ent-Os.CDPS
<400> 10
Met Glu His Ala Arg Pro Pro Gln Gly Gly Asp Asp Asp Val Ala Ala
1 5 10 15
Ser Thr Ser Glu Leu Pro Tyr Met Ile Glu Ser Ile Lys Ser Lys Leu
20 25 30
Arg Ala Ala Arg Asn Ser Leu Gly Glu Thr Thr Val Ser Ala Tyr Asp
35 40 45
Thr Ala Trp Ile Ala Leu Val Asn Arg Leu Asp Gly Gly Gly Glu Arg
50 55 60
Ser Pro Gln Phe Pro Glu Ala Ile Asp Trp Ile Ala Arg Asn Gln Leu
65 70 75 80
Pro Asp Gly Ser Trp Gly Asp Ala Gly Met Phe Ile Val Gln Asp Arg
85 90 95
Leu Ile Asn Thr Leu Gly Cys Val Val Ala Leu Ala Thr Trp Gly Val
100 105 110
His Glu Glu Gln Arg Ala Arg Gly Leu Ala Tyr Ile Gln Asp Asn Leu
115 120 125
Trp Arg Leu Gly Glu Asp Asp Glu Glu Trp Met Met Val Gly Phe Glu
130 135 140
Ile Thr Phe Pro Val Leu Leu Glu Lys Ala Lys Asn Leu Gly Leu Asp
145 150 155 160
Ile Asn Tyr Asp Asp Pro Ala Leu Gln Asp Ile Tyr Ala Lys Arg Gln
165 170 175
Leu Lys Leu Ala Lys Ile Pro Arg Glu Ala Leu His Ala Arg Pro Thr
180 185 190
Thr Leu Leu His Ser Leu Glu Gly Met Glu Asn Leu Asp Trp Glu Arg
195 200 205
Leu Leu Gln Phe Lys Cys Pro Ala Gly Ser Leu His Ser Ser Pro Ala
210 215 220
Ala Ser Ala Tyr Ala Leu Ser Glu Thr Gly Asp Lys Glu Leu Leu Glu
225 230 235 240
Tyr Leu Glu Thr Ala Ile Asn Asn Phe Asp Gly Gly Ala Pro Cys Thr
245 250 255
Tyr Pro Val Asp Asn Phe Asp Arg Leu Trp Ser Val Asp Arg Leu Arg
260 265 270
Arg Leu Gly Ile Ser Arg Tyr Phe Thr Ser Glu Ile Glu Glu Tyr Leu
275 280 285
Glu Tyr Ala Tyr Arg His Leu Ser Pro Asp Gly Met Ser Tyr Gly Gly
290 295 300
Leu Cys Pro Val Lys Asp Ile Asp Asp Thr Ala Met Ala Phe Arg Leu
305 310 315 320
Leu Arg Leu His Gly Tyr Asn Val Ser Ser Ser Val Phe Asn His Phe
325 330 335
Glu Lys Asp Gly Glu Tyr Phe Cys Phe Ala Gly Gln Ser Ser Gln Ser
340 345 350
Leu Thr Ala Met Tyr Asn Ser Tyr Arg Ala Ser Gln Ile Val Phe Pro
355 360 365
Gly Asp Asp Asp Gly Leu Glu Gln Leu Arg Ala Tyr Cys Arg Ala Phe
370 375 380
Leu Glu Glu Arg Arg Ala Thr Gly Asn Leu Arg Asp Lys Trp Val Ile
385 390 395 400
Ala Asn Gly Leu Pro Ser Glu Val Glu Tyr Ala Leu Asp Phe Pro Trp
405 410 415
Lys Ala Ser Leu Pro Arg Val Glu Thr Arg Val Tyr Leu Glu Gln Tyr
420 425 430
Gly Ala Ser Glu Asp Ala Trp Ile Gly Lys Gly Leu Tyr Arg Met Thr
435 440 445
Leu Val Asn Asn Asp Leu Tyr Leu Glu Ala Ala Lys Ala Asp Phe Thr
450 455 460
Asn Phe Gln Arg Leu Ser Arg Leu Glu Trp Leu Ser Leu Lys Arg Trp
465 470 475 480
Tyr Ile Arg Asn Asn Leu Gln Ala His Gly Val Thr Glu Gln Ser Val
485 490 495
Leu Arg Ala Tyr Phe Leu Ala Ala Ala Asn Ile Phe Glu Pro Asn Arg
500 505 510
Ala Ala Glu Arg Leu Gly Trp Ala Arg Thr Ala Ile Leu Ala Glu Ala
515 520 525
Ile Ala Ser His Leu Arg Gln Tyr Ser Ala Asn Gly Ala Ala Asp Gly
530 535 540
Met Thr Glu Arg Leu Ile Ser Gly Leu Ala Ser His Asp Trp Asp Trp
545 550 555 560
Arg Glu Ser Asn Asp Ser Ala Ala Arg Ser Leu Leu Tyr Ala Leu Asp
565 570 575
Glu Leu Ile Asp Leu His Ala Phe Gly Asn Ala Ser Asp Ser Leu Arg
580 585 590
Glu Ala Trp Lys Gln Trp Leu Met Ser Trp Thr Asn Glu Ser Gln Gly
595 600 605
Ser Thr Gly Gly Asp Thr Ala Leu Leu Leu Val Arg Thr Ile Glu Ile
610 615 620
Cys Ser Gly Arg His Gly Ser Ala Glu Gln Ser Leu Lys Asn Ser Glu
625 630 635 640
Asp Tyr Ala Arg Leu Glu Gln Ile Ala Ser Ser Met Cys Ser Lys Leu
645 650 655
Ala Thr Lys Ile Leu Ala Gln Asn Gly Gly Ser Met Asp Asn Val Glu
660 665 670
Gly Ile Asp Gln Glu Val Asp Val Glu Met Lys Glu Leu Ile Gln Arg
675 680 685
Val Tyr Gly Ser Ser Ser Asn Asp Val Ser Ser Val Thr Arg Gln Thr
690 695 700
Phe Leu Asp Val Val Lys Ser Phe Cys Tyr Val Ala His Cys Ser Pro
705 710 715 720
Glu Thr Ile Asp Gly His Ile Ser Lys Val Leu Phe Glu Asp Val Asn
725 730 735
<210> 11
<211> 757
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Ent-Pg.KS
<400> 11
Met Lys Arg Glu Gln Tyr Thr Ile Leu Asn Glu Lys Glu Ser Met Ala
1 5 10 15
Glu Glu Leu Ile Leu Arg Ile Lys Arg Met Phe Ser Glu Ile Glu Asn
20 25 30
Thr Gln Thr Ser Ala Ser Ala Tyr Asp Thr Ala Trp Val Ala Met Val
35 40 45
Pro Ser Leu Asp Ser Ser Gln Gln Pro Gln Phe Pro Gln Cys Leu Ser
50 55 60
Trp Ile Ile Asp Asn Gln Leu Leu Asp Gly Ser Trp Gly Ile Pro Tyr
65 70 75 80
Leu Ile Ile Lys Asp Arg Leu Cys His Thr Leu Ala Cys Val Ile Ala
85 90 95
Leu Arg Lys Trp Asn Ala Gly Asn Gln Asn Val Glu Thr Gly Leu Arg
100 105 110
Phe Leu Arg Glu Asn Ile Glu Gly Ile Val His Glu Asp Glu Tyr Thr
115 120 125
Pro Ile Gly Phe Gln Ile Ile Phe Pro Ala Met Leu Glu Glu Ala Arg
130 135 140
Gly Leu Gly Leu Glu Leu Pro Tyr Asp Leu Thr Pro Ile Lys Leu Met
145 150 155 160
Leu Thr His Arg Glu Lys Ile Met Lys Gly Lys Ala Ile Asp His Met
165 170 175
His Glu Tyr Asp Ser Ser Leu Ile Tyr Thr Val Glu Gly Ile His Lys
180 185 190
Ile Val Asp Trp Asn Lys Val Leu Lys His Gln Asn Lys Asp Gly Ser
195 200 205
Leu Phe Asn Ser Pro Ser Ala Thr Ala Cys Ala Leu Met His Thr Arg
210 215 220
Lys Ser Asn Cys Leu Glu Tyr Leu Ser Ser Met Leu Gln Lys Leu Gly
225 230 235 240
Asn Gly Val Pro Ser Val Tyr Pro Ile Asn Leu Tyr Ala Arg Ile Ser
245 250 255
Met Ile Asp Arg Leu Gln Arg Leu Gly Leu Ala Arg His Phe Arg Asn
260 265 270
Glu Ile Ile His Ala Leu Asp Asp Ile Tyr Arg Tyr Trp Met Gln Arg
275 280 285
Glu Thr Ser Arg Glu Gly Lys Ser Leu Thr Pro Asp Ile Val Ser Thr
290 295 300
Ser Ile Ala Phe Met Leu Leu Arg Leu His Gly Tyr Asp Val Pro Ala
305 310 315 320
Asp Val Phe Cys Cys Tyr Asp Leu His Ser Ile Glu Gln Ser Gly Glu
325 330 335
Ala Val Thr Ala Met Leu Ser Leu Tyr Arg Ala Ser Gln Ile Met Phe
340 345 350
Pro Gly Glu Thr Ile Leu Glu Glu Ile Lys Thr Val Ser Arg Lys Tyr
355 360 365
Leu Asp Lys Arg Lys Glu Asn Gly Gly Ile Tyr Asp His Asn Ile Val
370 375 380
Met Lys Asp Leu Arg Gly Glu Val Glu Tyr Ala Leu Ser Val Pro Trp
385 390 395 400
Tyr Ala Ser Leu Glu Arg Ile Glu Asn Arg Arg Tyr Ile Asp Gln Tyr
405 410 415
Gly Val Asn Asp Thr Trp Ile Ala Lys Thr Ser Tyr Lys Ile Pro Cys
420 425 430
Ile Ser Asn Asp Leu Phe Leu Ala Leu Ala Lys Gln Asp Tyr Asn Ile
435 440 445
Cys Gln Ala Ile Gln Gln Lys Glu Leu Arg Glu Leu Glu Arg Trp Phe
450 455 460
Ala Asp Asn Lys Phe Ser His Leu Asn Phe Ala Arg Gln Lys Leu Ile
465 470 475 480
Tyr Cys Tyr Phe Ser Ala Ala Ala Thr Leu Phe Ser Pro Glu Leu Ser
485 490 495
Ala Ala Arg Val Val Trp Ala Lys Asn Gly Val Ile Thr Thr Val Val
500 505 510
Asp Asp Phe Phe Asp Val Gly Gly Ser Ser Glu Glu Ile His Ser Phe
515 520 525
Val Glu Ala Val Arg Val Trp Asp Glu Ala Ala Thr Asp Gly Leu Ser
530 535 540
Glu Asn Val Gln Ile Leu Phe Ser Ala Leu Tyr Asn Thr Val Asp Glu
545 550 555 560
Ile Val Gln Gln Ala Phe Val Phe Gln Gly Arg Asp Ile Ser Ile His
565 570 575
Leu Arg Glu Ile Trp Tyr Arg Leu Val Asn Ser Met Met Thr Glu Ala
580 585 590
Gln Trp Ala Arg Thr His Cys Leu Pro Ser Met His Glu Tyr Met Glu
595 600 605
Asn Ala Glu Pro Ser Ile Ala Leu Glu Pro Ile Val Leu Ser Ser Leu
610 615 620
Tyr Phe Val Gly Pro Lys Leu Ser Glu Glu Ile Ile Cys His Pro Glu
625 630 635 640
Tyr Tyr Asn Leu Met His Leu Leu Asn Ile Cys Gly Arg Leu Leu Asn
645 650 655
Asp Ile Gln Gly Cys Lys Arg Glu Ala His Gln Gly Lys Leu Asn Ser
660 665 670
Val Thr Leu Tyr Met Glu Glu Asn Ser Gly Thr Thr Met Glu Asp Ala
675 680 685
Ile Val Tyr Leu Arg Lys Thr Ile Asp Glu Ser Arg Gln Leu Leu Leu
690 695 700
Lys Glu Val Leu Arg Pro Ser Ile Val Pro Arg Glu Cys Lys Gln Leu
705 710 715 720
His Trp Asn Met Met Arg Ile Leu Gln Leu Phe Tyr Leu Lys Asn Asp
725 730 735
Gly Phe Thr Ser Pro Thr Glu Met Leu Gly Tyr Val Asn Ala Val Ile
740 745 750
Val Asp Pro Ile Leu
755
<210> 12
<211> 499
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Ps.KO
<400> 12
Met Asp Thr Leu Thr Leu Ser Leu Gly Phe Leu Ser Leu Phe Leu Phe
1 5 10 15
Leu Phe Leu Leu Lys Arg Ser Thr His Lys His Ser Lys Leu Ser His
20 25 30
Val Pro Val Val Pro Gly Leu Pro Val Ile Gly Asn Leu Leu Gln Leu
35 40 45
Lys Glu Lys Lys Pro His Lys Thr Phe Thr Lys Met Ala Gln Lys Tyr
50 55 60
Gly Pro Ile Phe Ser Ile Lys Ala Gly Ser Ser Lys Ile Ile Val Leu
65 70 75 80
Asn Thr Ala His Leu Ala Lys Glu Ala Met Val Thr Arg Tyr Ser Ser
85 90 95
Ile Ser Lys Arg Lys Leu Ser Thr Ala Leu Thr Ile Leu Thr Ser Asp
100 105 110
Lys Cys Met Val Ala Met Ser Asp Tyr Asn Asp Phe His Lys Met Val
115 120 125
Lys Lys His Ile Leu Ala Ser Val Leu Gly Ala Asn Ala Gln Lys Arg
130 135 140
Leu Arg Phe His Arg Glu Val Met Met Glu Asn Met Ser Ser Lys Phe
145 150 155 160
Asn Glu His Val Lys Thr Leu Ser Asp Ser Ala Val Asp Phe Arg Lys
165 170 175
Ile Phe Val Ser Glu Leu Phe Gly Leu Ala Leu Lys Gln Ala Leu Gly
180 185 190
Ser Asp Ile Glu Ser Ile Tyr Val Glu Gly Leu Thr Ala Thr Leu Ser
195 200 205
Arg Glu Asp Leu Tyr Asn Thr Leu Val Val Asp Phe Met Glu Gly Ala
210 215 220
Ile Glu Val Asp Trp Arg Asp Phe Phe Pro Tyr Leu Lys Trp Ile Pro
225 230 235 240
Asn Lys Ser Phe Glu Lys Lys Ile Arg Arg Val Asp Arg Gln Arg Lys
245 250 255
Ile Ile Met Lys Ala Leu Ile Asn Glu Gln Lys Lys Arg Leu Thr Ser
260 265 270
Gly Lys Glu Leu Asp Cys Tyr Tyr Asp Tyr Leu Val Ser Glu Ala Lys
275 280 285
Glu Val Thr Glu Glu Gln Met Ile Met Leu Leu Trp Glu Pro Ile Ile
290 295 300
Glu Thr Ser Asp Thr Thr Leu Val Thr Thr Glu Trp Ala Met Tyr Glu
305 310 315 320
Leu Ala Lys Asp Lys Asn Arg Gln Asp Arg Leu Tyr Glu Glu Leu Leu
325 330 335
Asn Val Cys Gly His Glu Lys Val Thr Asp Glu Glu Leu Ser Lys Leu
340 345 350
Pro Tyr Leu Gly Ala Val Phe His Glu Thr Leu Arg Lys His Ser Pro
355 360 365
Val Pro Ile Val Pro Leu Arg Tyr Val Asp Glu Asp Thr Glu Leu Gly
370 375 380
Gly Tyr His Ile Pro Ala Gly Ser Glu Ile Ala Ile Asn Ile Tyr Gly
385 390 395 400
Cys Asn Met Asp Ser Asn Leu Trp Glu Asn Pro Asp Gln Trp Ile Pro
405 410 415
Glu Arg Phe Leu Asp Glu Lys Tyr Ala Gln Ala Asp Leu Tyr Lys Thr
420 425 430
Met Ala Phe Gly Gly Gly Lys Arg Val Cys Ala Gly Ser Leu Gln Ala
435 440 445
Met Leu Ile Ala Cys Thr Ala Ile Gly Arg Leu Val Gln Glu Phe Glu
450 455 460
Trp Glu Leu Gly His Gly Glu Glu Glu Asn Val Asp Thr Met Gly Leu
465 470 475 480
Thr Thr His Arg Leu His Pro Leu Gln Val Lys Leu Lys Pro Arg Asn
485 490 495
Arg Ile Tyr
<210> 13
<211> 500
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Sr.KAH
<400> 13
Met Glu Ala Ser Tyr Leu Tyr Ile Ser Ile Leu Leu Leu Leu Ala Ser
1 5 10 15
Tyr Leu Phe Thr Thr Gln Leu Arg Arg Lys Ser Ala Asn Leu Pro Pro
20 25 30
Thr Val Phe Pro Ser Ile Pro Ile Ile Gly His Leu Tyr Leu Leu Lys
35 40 45
Lys Pro Leu Tyr Arg Thr Leu Ala Lys Ile Ala Ala Lys Tyr Gly Pro
50 55 60
Ile Leu Gln Leu Gln Leu Gly Tyr Arg Arg Val Leu Val Ile Ser Ser
65 70 75 80
Pro Ser Ala Ala Glu Glu Cys Phe Thr Asn Asn Asp Val Ile Phe Ala
85 90 95
Asn Arg Pro Lys Thr Leu Phe Gly Lys Ile Val Gly Gly Thr Ser Leu
100 105 110
Gly Ser Leu Ser Tyr Gly Asp Gln Trp Arg Asn Leu Arg Arg Val Ala
115 120 125
Ser Ile Glu Ile Leu Ser Val His Arg Leu Asn Glu Phe His Asp Ile
130 135 140
Arg Val Asp Glu Asn Arg Leu Leu Ile Arg Lys Leu Arg Ser Ser Ser
145 150 155 160
Ser Pro Val Thr Leu Ile Thr Val Phe Tyr Ala Leu Thr Leu Asn Val
165 170 175
Ile Met Arg Met Ile Ser Gly Lys Arg Tyr Phe Asp Ser Gly Asp Arg
180 185 190
Glu Leu Glu Glu Glu Gly Lys Arg Phe Arg Glu Ile Leu Asp Glu Thr
195 200 205
Leu Leu Leu Ala Gly Ala Ser Asn Val Gly Asp Tyr Leu Pro Ile Leu
210 215 220
Asn Trp Leu Gly Val Lys Ser Leu Glu Lys Lys Leu Ile Ala Leu Gln
225 230 235 240
Lys Lys Arg Asp Asp Phe Phe Gln Gly Leu Ile Glu Gln Val Arg Lys
245 250 255
Ser Arg Gly Ala Lys Val Gly Lys Gly Arg Lys Thr Met Ile Glu Leu
260 265 270
Leu Leu Ser Leu Gln Glu Ser Glu Pro Glu Tyr Tyr Thr Asp Ala Met
275 280 285
Ile Arg Ser Phe Val Leu Gly Leu Leu Ala Ala Gly Ser Asp Thr Ser
290 295 300
Ala Gly Thr Met Glu Trp Ala Met Ser Leu Leu Val Asn His Pro His
305 310 315 320
Val Leu Lys Lys Ala Gln Ala Glu Ile Asp Arg Val Ile Gly Asn Asn
325 330 335
Arg Leu Ile Asp Glu Ser Asp Ile Gly Asn Ile Pro Tyr Ile Gly Cys
340 345 350
Ile Ile Asn Glu Thr Leu Arg Leu Tyr Pro Ala Gly Pro Leu Leu Phe
355 360 365
Pro His Glu Ser Ser Ala Asp Cys Val Ile Ser Gly Tyr Asn Ile Pro
370 375 380
Arg Gly Thr Met Leu Ile Val Asn Gln Trp Ala Ile His His Asp Pro
385 390 395 400
Lys Val Trp Asp Asp Pro Glu Thr Phe Lys Pro Glu Arg Phe Gln Gly
405 410 415
Leu Glu Gly Thr Arg Asp Gly Phe Lys Leu Met Pro Phe Gly Ser Gly
420 425 430
Arg Arg Gly Cys Pro Gly Glu Gly Leu Ala Ile Arg Leu Leu Gly Met
435 440 445
Thr Leu Gly Ser Val Ile Gln Cys Phe Asp Trp Glu Arg Val Gly Asp
450 455 460
Glu Met Val Asp Met Thr Glu Gly Leu Gly Val Thr Leu Pro Lys Ala
465 470 475 480
Val Pro Leu Val Ala Lys Cys Lys Pro Arg Ser Glu Met Thr Asn Leu
485 490 495
Leu Ser Glu Leu
500
<210> 14
<211> 711
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: At.CPR
<400> 14
Met Ser Ser Ser Ser Ser Ser Ser Thr Ser Met Ile Asp Leu Met Ala
1 5 10 15
Ala Ile Ile Lys Gly Glu Pro Val Ile Val Ser Asp Pro Ala Asn Ala
20 25 30
Ser Ala Tyr Glu Ser Val Ala Ala Glu Leu Ser Ser Met Leu Ile Glu
35 40 45
Asn Arg Gln Phe Ala Met Ile Val Thr Thr Ser Ile Ala Val Leu Ile
50 55 60
Gly Cys Ile Val Met Leu Val Trp Arg Arg Ser Gly Ser Gly Asn Ser
65 70 75 80
Lys Arg Val Glu Pro Leu Lys Pro Leu Val Ile Lys Pro Arg Glu Glu
85 90 95
Glu Ile Asp Asp Gly Arg Lys Lys Val Thr Ile Phe Phe Gly Thr Gln
100 105 110
Thr Gly Thr Ala Glu Gly Phe Ala Lys Ala Leu Gly Glu Glu Ala Lys
115 120 125
Ala Arg Tyr Glu Lys Thr Arg Phe Lys Ile Val Asp Leu Asp Asp Tyr
130 135 140
Ala Ala Asp Asp Asp Glu Tyr Glu Glu Lys Leu Lys Lys Glu Asp Val
145 150 155 160
Ala Phe Phe Phe Leu Ala Thr Tyr Gly Asp Gly Glu Pro Thr Asp Asn
165 170 175
Ala Ala Arg Phe Tyr Lys Trp Phe Thr Glu Gly Asn Asp Arg Gly Glu
180 185 190
Trp Leu Lys Asn Leu Lys Tyr Gly Val Phe Gly Leu Gly Asn Arg Gln
195 200 205
Tyr Glu His Phe Asn Lys Val Ala Lys Val Val Asp Asp Ile Leu Val
210 215 220
Glu Gln Gly Ala Gln Arg Leu Val Gln Val Gly Leu Gly Asp Asp Asp
225 230 235 240
Gln Cys Ile Glu Asp Asp Phe Thr Ala Trp Arg Glu Ala Leu Trp Pro
245 250 255
Glu Leu Asp Thr Ile Leu Arg Glu Glu Gly Asp Thr Ala Val Ala Thr
260 265 270
Pro Tyr Thr Ala Ala Val Leu Glu Tyr Arg Val Ser Ile His Asp Ser
275 280 285
Glu Asp Ala Lys Phe Asn Asp Ile Asn Met Ala Asn Gly Asn Gly Tyr
290 295 300
Thr Val Phe Asp Ala Gln His Pro Tyr Lys Ala Asn Val Ala Val Lys
305 310 315 320
Arg Glu Leu His Thr Pro Glu Ser Asp Arg Ser Cys Ile His Leu Glu
325 330 335
Phe Asp Ile Ala Gly Ser Gly Leu Thr Tyr Glu Thr Gly Asp His Val
340 345 350
Gly Val Leu Cys Asp Asn Leu Ser Glu Thr Val Asp Glu Ala Leu Arg
355 360 365
Leu Leu Asp Met Ser Pro Asp Thr Tyr Phe Ser Leu His Ala Glu Lys
370 375 380
Glu Asp Gly Thr Pro Ile Ser Ser Ser Leu Pro Pro Pro Phe Pro Pro
385 390 395 400
Cys Asn Leu Arg Thr Ala Leu Thr Arg Tyr Ala Cys Leu Leu Ser Ser
405 410 415
Pro Lys Lys Ser Ala Leu Val Ala Leu Ala Ala His Ala Ser Asp Pro
420 425 430
Thr Glu Ala Glu Arg Leu Lys His Leu Ala Ser Pro Ala Gly Lys Asp
435 440 445
Glu Tyr Ser Lys Trp Val Val Glu Ser Gln Arg Ser Leu Leu Glu Val
450 455 460
Met Ala Glu Phe Pro Ser Ala Lys Pro Pro Leu Gly Val Phe Phe Ala
465 470 475 480
Gly Val Ala Pro Arg Leu Gln Pro Arg Phe Tyr Ser Ile Ser Ser Ser
485 490 495
Pro Lys Ile Ala Glu Thr Arg Ile His Val Thr Cys Ala Leu Val Tyr
500 505 510
Glu Lys Met Pro Thr Gly Arg Ile His Lys Gly Val Cys Ser Thr Trp
515 520 525
Met Lys Asn Ala Val Pro Tyr Glu Lys Ser Glu Asn Cys Ser Ser Ala
530 535 540
Pro Ile Phe Val Arg Gln Ser Asn Phe Lys Leu Pro Ser Asp Ser Lys
545 550 555 560
Val Pro Ile Ile Met Ile Gly Pro Gly Thr Gly Leu Ala Pro Phe Arg
565 570 575
Gly Phe Leu Gln Glu Arg Leu Ala Leu Val Glu Ser Gly Val Glu Leu
580 585 590
Gly Pro Ser Val Leu Phe Phe Gly Cys Arg Asn Arg Arg Met Asp Phe
595 600 605
Ile Tyr Glu Glu Glu Leu Gln Arg Phe Val Glu Ser Gly Ala Leu Ala
610 615 620
Glu Leu Ser Val Ala Phe Ser Arg Glu Gly Pro Thr Lys Glu Tyr Val
625 630 635 640
Gln His Lys Met Met Asp Lys Ala Ser Asp Ile Trp Asn Met Ile Ser
645 650 655
Gln Gly Ala Tyr Leu Tyr Val Cys Gly Asp Ala Lys Gly Met Ala Arg
660 665 670
Asp Val His Arg Ser Leu His Thr Ile Ala Gln Glu Gln Gly Ser Met
675 680 685
Asp Ser Thr Lys Ala Glu Gly Phe Val Lys Asn Leu Gln Thr Ser Gly
690 695 700
Arg Tyr Leu Arg Asp Val Trp
705 710
<210> 15
<211> 481
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: UGT85C2
<400> 15
Met Asp Ala Met Ala Thr Thr Glu Lys Lys Pro His Val Ile Phe Ile
1 5 10 15
Pro Phe Pro Ala Gln Ser His Ile Lys Ala Met Leu Lys Leu Ala Gln
20 25 30
Leu Leu His His Lys Gly Leu Gln Ile Thr Phe Val Asn Thr Asp Phe
35 40 45
Ile His Asn Gln Phe Leu Glu Ser Ser Gly Pro His Cys Leu Asp Gly
50 55 60
Ala Pro Gly Phe Arg Phe Glu Thr Ile Pro Asp Gly Val Ser His Ser
65 70 75 80
Pro Glu Ala Ser Ile Pro Ile Arg Glu Ser Leu Leu Arg Ser Ile Glu
85 90 95
Thr Asn Phe Leu Asp Arg Phe Ile Asp Leu Val Thr Lys Leu Pro Asp
100 105 110
Pro Pro Thr Cys Ile Ile Ser Asp Gly Phe Leu Ser Val Phe Thr Ile
115 120 125
Asp Ala Ala Lys Lys Leu Gly Ile Pro Val Met Met Tyr Trp Thr Leu
130 135 140
Ala Ala Cys Gly Phe Met Gly Phe Tyr His Ile His Ser Leu Ile Glu
145 150 155 160
Lys Gly Phe Ala Pro Leu Lys Asp Ala Ser Tyr Leu Thr Asn Gly Tyr
165 170 175
Leu Asp Thr Val Ile Asp Trp Val Pro Gly Met Glu Gly Ile Arg Leu
180 185 190
Lys Asp Phe Pro Leu Asp Trp Ser Thr Asp Leu Asn Asp Lys Val Leu
195 200 205
Met Phe Thr Thr Glu Ala Pro Gln Arg Ser His Lys Val Ser His His
210 215 220
Ile Phe His Thr Phe Asp Glu Leu Glu Pro Ser Ile Ile Lys Thr Leu
225 230 235 240
Ser Leu Arg Tyr Asn His Ile Tyr Thr Ile Gly Pro Leu Gln Leu Leu
245 250 255
Leu Asp Gln Ile Pro Glu Glu Lys Lys Gln Thr Gly Ile Thr Ser Leu
260 265 270
His Gly Tyr Ser Leu Val Lys Glu Glu Pro Glu Cys Phe Gln Trp Leu
275 280 285
Gln Ser Lys Glu Pro Asn Ser Val Val Tyr Val Asn Phe Gly Ser Thr
290 295 300
Thr Val Met Ser Leu Glu Asp Met Thr Glu Phe Gly Trp Gly Leu Ala
305 310 315 320
Asn Ser Asn His Tyr Phe Leu Trp Ile Ile Arg Ser Asn Leu Val Ile
325 330 335
Gly Glu Asn Ala Val Leu Pro Pro Glu Leu Glu Glu His Ile Lys Lys
340 345 350
Arg Gly Phe Ile Ala Ser Trp Cys Ser Gln Glu Lys Val Leu Lys His
355 360 365
Pro Ser Val Gly Gly Phe Leu Thr His Cys Gly Trp Gly Ser Thr Ile
370 375 380
Glu Ser Leu Ser Ala Gly Val Pro Met Ile Cys Trp Pro Tyr Ser Trp
385 390 395 400
Asp Gln Leu Thr Asn Cys Arg Tyr Ile Cys Lys Glu Trp Glu Val Gly
405 410 415
Leu Glu Met Gly Thr Lys Val Lys Arg Asp Glu Val Lys Arg Leu Val
420 425 430
Gln Glu Leu Met Gly Glu Gly Gly His Lys Met Arg Asn Lys Ala Lys
435 440 445
Asp Trp Lys Glu Lys Ala Arg Ile Ala Ile Ala Pro Asn Gly Ser Ser
450 455 460
Ser Leu Asn Ile Asp Lys Met Val Lys Glu Ile Thr Val Leu Ala Arg
465 470 475 480
Asn
<210> 16
<211> 460
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: UGT74G1
<400> 16
Met Ala Glu Gln Gln Lys Ile Lys Lys Ser Pro His Val Leu Leu Ile
1 5 10 15
Pro Phe Pro Leu Gln Gly His Ile Asn Pro Phe Ile Gln Phe Gly Lys
20 25 30
Arg Leu Ile Ser Lys Gly Val Lys Thr Thr Leu Val Thr Thr Ile His
35 40 45
Thr Leu Asn Ser Thr Leu Asn His Ser Asn Thr Thr Thr Thr Ser Ile
50 55 60
Glu Ile Gln Ala Ile Ser Asp Gly Cys Asp Glu Gly Gly Phe Met Ser
65 70 75 80
Ala Gly Glu Ser Tyr Leu Glu Thr Phe Lys Gln Val Gly Ser Lys Ser
85 90 95
Leu Ala Asp Leu Ile Lys Lys Leu Gln Ser Glu Gly Thr Thr Ile Asp
100 105 110
Ala Ile Ile Tyr Asp Ser Met Thr Glu Trp Val Leu Asp Val Ala Ile
115 120 125
Glu Phe Gly Ile Asp Gly Gly Ser Phe Phe Thr Gln Ala Cys Val Val
130 135 140
Asn Ser Leu Tyr Tyr His Val His Lys Gly Leu Ile Ser Leu Pro Leu
145 150 155 160
Gly Glu Thr Val Ser Val Pro Gly Phe Pro Val Leu Gln Arg Trp Glu
165 170 175
Thr Pro Leu Ile Leu Gln Asn His Glu Gln Ile Gln Ser Pro Trp Ser
180 185 190
Gln Met Leu Phe Gly Gln Phe Ala Asn Ile Asp Gln Ala Arg Trp Val
195 200 205
Phe Thr Asn Ser Phe Tyr Lys Leu Glu Glu Glu Val Ile Glu Trp Thr
210 215 220
Arg Lys Ile Trp Asn Leu Lys Val Ile Gly Pro Thr Leu Pro Ser Met
225 230 235 240
Tyr Leu Asp Lys Arg Leu Asp Asp Asp Lys Asp Asn Gly Phe Asn Leu
245 250 255
Tyr Lys Ala Asn His His Glu Cys Met Asn Trp Leu Asp Asp Lys Pro
260 265 270
Lys Glu Ser Val Val Tyr Val Ala Phe Gly Ser Leu Val Lys His Gly
275 280 285
Pro Glu Gln Val Glu Glu Ile Thr Arg Ala Leu Ile Asp Ser Asp Val
290 295 300
Asn Phe Leu Trp Val Ile Lys His Lys Glu Glu Gly Lys Leu Pro Glu
305 310 315 320
Asn Leu Ser Glu Val Ile Lys Thr Gly Lys Gly Leu Ile Val Ala Trp
325 330 335
Cys Lys Gln Leu Asp Val Leu Ala His Glu Ser Val Gly Cys Phe Val
340 345 350
Thr His Cys Gly Phe Asn Ser Thr Leu Glu Ala Ile Ser Leu Gly Val
355 360 365
Pro Val Val Ala Met Pro Gln Phe Ser Asp Gln Thr Thr Asn Ala Lys
370 375 380
Leu Leu Asp Glu Ile Leu Gly Val Gly Val Arg Val Lys Ala Asp Glu
385 390 395 400
Asn Gly Ile Val Arg Arg Gly Asn Leu Ala Ser Cys Ile Lys Met Ile
405 410 415
Met Glu Glu Glu Arg Gly Val Ile Ile Arg Lys Asn Ala Val Lys Trp
420 425 430
Lys Asp Leu Ala Lys Val Ala Val His Glu Gly Gly Ser Ser Asp Asn
435 440 445
Asp Ile Val Glu Phe Val Ser Glu Leu Ile Lys Ala
450 455 460
<210> 17
<211> 485
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: UGT91D_like3
<400> 17
Met Tyr Asn Val Thr Tyr His Gln Asn Ser Lys Ala Met Ala Thr Ser
1 5 10 15
Asp Ser Ile Val Asp Asp Arg Lys Gln Leu His Val Ala Thr Phe Pro
20 25 30
Trp Leu Ala Phe Gly His Ile Leu Pro Tyr Leu Gln Leu Ser Lys Leu
35 40 45
Ile Ala Glu Lys Gly His Lys Val Ser Phe Leu Ser Thr Thr Arg Asn
50 55 60
Ile Gln Arg Leu Ser Ser His Ile Ser Pro Leu Ile Asn Val Val Gln
65 70 75 80
Leu Thr Leu Pro Arg Val Gln Glu Leu Pro Glu Asp Ala Glu Ala Thr
85 90 95
Thr Asp Val His Pro Glu Asp Ile Pro Tyr Leu Lys Lys Ala Ser Asp
100 105 110
Gly Leu Gln Pro Glu Val Thr Arg Phe Leu Glu Gln His Ser Pro Asp
115 120 125
Trp Ile Ile Tyr Asp Tyr Thr His Tyr Trp Leu Pro Ser Ile Ala Ala
130 135 140
Ser Leu Gly Ile Ser Arg Ala His Phe Ser Val Thr Thr Pro Trp Ala
145 150 155 160
Ile Ala Tyr Met Gly Pro Ser Ala Asp Ala Met Ile Asn Gly Ser Asp
165 170 175
Gly Arg Thr Thr Val Glu Asp Leu Thr Thr Pro Pro Lys Trp Phe Pro
180 185 190
Phe Pro Thr Lys Val Cys Trp Arg Lys His Asp Leu Ala Arg Leu Val
195 200 205
Pro Tyr Lys Ala Pro Gly Ile Ser Asp Gly Tyr Arg Met Gly Leu Val
210 215 220
Leu Lys Gly Ser Asp Cys Leu Leu Ser Lys Cys Tyr His Glu Phe Gly
225 230 235 240
Thr Gln Trp Leu Pro Leu Leu Glu Thr Leu His Gln Val Pro Val Val
245 250 255
Pro Val Gly Leu Leu Pro Pro Glu Ile Pro Gly Asp Glu Lys Asp Glu
260 265 270
Thr Trp Val Ser Ile Lys Lys Trp Leu Asp Gly Lys Gln Lys Gly Ser
275 280 285
Val Val Tyr Val Ala Leu Gly Ser Glu Val Leu Val Ser Gln Thr Glu
290 295 300
Val Val Glu Leu Ala Leu Gly Leu Glu Leu Ser Gly Leu Pro Phe Val
305 310 315 320
Trp Ala Tyr Arg Lys Pro Lys Gly Pro Ala Lys Ser Asp Ser Val Glu
325 330 335
Leu Pro Asp Gly Phe Val Glu Arg Thr Arg Asp Arg Gly Leu Val Trp
340 345 350
Thr Ser Trp Ala Pro Gln Leu Arg Ile Leu Ser His Glu Ser Val Cys
355 360 365
Gly Phe Leu Thr His Cys Gly Ser Gly Ser Ile Val Glu Gly Leu Met
370 375 380
Phe Gly His Pro Leu Ile Met Leu Pro Ile Phe Gly Asp Gln Pro Leu
385 390 395 400
Asn Ala Arg Leu Leu Glu Asp Lys Gln Val Gly Ile Glu Ile Pro Arg
405 410 415
Asn Glu Glu Asp Gly Cys Leu Thr Lys Glu Ser Val Ala Arg Ser Leu
420 425 430
Arg Ser Val Val Val Glu Lys Glu Gly Glu Ile Tyr Lys Ala Asn Ala
435 440 445
Arg Glu Leu Ser Lys Ile Tyr Asn Asp Thr Lys Val Glu Lys Glu Tyr
450 455 460
Val Ser Gln Phe Val Asp Tyr Leu Glu Lys Asn Ala Arg Ala Val Ala
465 470 475 480
Ile Asp His Glu Ser
485
<210> 18
<211> 458
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: UGT76G1
<400> 18
Met Glu Asn Lys Thr Glu Thr Thr Val Arg Arg Arg Arg Arg Ile Ile
1 5 10 15
Leu Phe Pro Val Pro Phe Gln Gly His Ile Asn Pro Ile Leu Gln Leu
20 25 30
Ala Asn Val Leu Tyr Ser Lys Gly Phe Ser Ile Thr Ile Phe His Thr
35 40 45
Asn Phe Asn Lys Pro Lys Thr Ser Asn Tyr Pro His Phe Thr Phe Arg
50 55 60
Phe Ile Leu Asp Asn Asp Pro Gln Asp Glu Arg Ile Ser Asn Leu Pro
65 70 75 80
Thr His Gly Pro Leu Ala Gly Met Arg Ile Pro Ile Ile Asn Glu His
85 90 95
Gly Ala Asp Glu Leu Arg Arg Glu Leu Glu Leu Leu Met Leu Ala Ser
100 105 110
Glu Glu Asp Glu Glu Val Ser Cys Leu Ile Thr Asp Ala Leu Trp Tyr
115 120 125
Phe Ala Gln Ser Val Ala Asp Ser Leu Asn Leu Arg Arg Leu Val Leu
130 135 140
Met Thr Ser Ser Leu Phe Asn Phe His Ala His Val Ser Leu Pro Gln
145 150 155 160
Phe Asp Glu Leu Gly Tyr Leu Asp Pro Asp Asp Lys Thr Arg Leu Glu
165 170 175
Glu Gln Ala Ser Gly Phe Pro Met Leu Lys Val Lys Asp Ile Lys Ser
180 185 190
Ala Tyr Ser Asn Trp Gln Ile Leu Lys Glu Ile Leu Gly Lys Met Ile
195 200 205
Lys Gln Thr Lys Ala Ser Ser Gly Val Ile Trp Asn Ser Phe Lys Glu
210 215 220
Leu Glu Glu Ser Glu Leu Glu Thr Val Ile Arg Glu Ile Pro Ala Pro
225 230 235 240
Ser Phe Leu Ile Pro Leu Pro Lys His Leu Thr Ala Ser Ser Ser Ser
245 250 255
Leu Leu Asp His Asp Arg Thr Val Phe Gln Trp Leu Asp Gln Gln Pro
260 265 270
Pro Ser Ser Val Leu Tyr Val Ser Phe Gly Ser Thr Ser Glu Val Asp
275 280 285
Glu Lys Asp Phe Leu Glu Ile Ala Arg Gly Leu Val Asp Ser Lys Gln
290 295 300
Ser Phe Leu Trp Val Val Arg Pro Gly Phe Val Lys Gly Ser Thr Trp
305 310 315 320
Val Glu Pro Leu Pro Asp Gly Phe Leu Gly Glu Arg Gly Arg Ile Val
325 330 335
Lys Trp Val Pro Gln Gln Glu Val Leu Ala His Gly Ala Ile Gly Ala
340 345 350
Phe Trp Thr His Ser Gly Trp Asn Ser Thr Leu Glu Ser Val Cys Glu
355 360 365
Gly Val Pro Met Ile Phe Ser Asp Phe Gly Leu Asp Gln Pro Leu Asn
370 375 380
Ala Arg Tyr Met Ser Asp Val Leu Lys Val Gly Val Tyr Leu Glu Asn
385 390 395 400
Gly Trp Glu Arg Gly Glu Ile Ala Asn Ala Ile Arg Arg Val Met Val
405 410 415
Asp Glu Glu Gly Glu Tyr Ile Arg Gln Asn Ala Arg Val Leu Lys Gln
420 425 430
Lys Ala Asp Val Ser Leu Met Lys Gly Gly Ser Ser Tyr Glu Ser Leu
435 440 445
Glu Ser Leu Val Ser Tyr Ile Ser Ser Leu
450 455
<210> 19
<211> 436
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: UGT40087
<400> 19
Met Asp Ala Ser Asp Ser Ser Pro Leu His Ile Val Ile Phe Pro Trp
1 5 10 15
Leu Ala Phe Gly His Met Leu Ala Ser Leu Glu Leu Ala Glu Arg Leu
20 25 30
Ala Ala Arg Gly His Arg Val Ser Phe Val Ser Thr Pro Arg Asn Ile
35 40 45
Ser Arg Leu Arg Pro Val Pro Pro Ala Leu Ala Pro Leu Ile Asp Phe
50 55 60
Val Ala Leu Pro Leu Pro Arg Val Asp Gly Leu Pro Asp Gly Ala Glu
65 70 75 80
Ala Thr Ser Asp Ile Pro Pro Gly Lys Thr Glu Leu His Leu Lys Ala
85 90 95
Leu Asp Gly Leu Ala Ala Pro Phe Ala Ala Phe Leu Asp Ala Ala Cys
100 105 110
Ala Asp Gly Ser Thr Asn Lys Val Asp Trp Leu Phe Leu Asp Asn Phe
115 120 125
Gln Tyr Trp Ala Ala Ala Ala Ala Ala Asp His Lys Ile Pro Cys Ala
130 135 140
Leu Asn Leu Thr Phe Ala Ala Ser Thr Ser Ala Glu Tyr Gly Val Pro
145 150 155 160
Arg Val Glu Pro Pro Val Asp Gly Ser Thr Ala Ser Ile Leu Gln Arg
165 170 175
Phe Val Leu Thr Leu Glu Lys Cys Gln Phe Val Ile Gln Arg Ala Cys
180 185 190
Phe Glu Leu Glu Pro Glu Pro Leu Pro Leu Leu Ser Asp Ile Phe Gly
195 200 205
Lys Pro Val Ile Pro Tyr Gly Leu Val Pro Pro Cys Pro Pro Ala Glu
210 215 220
Gly His Lys Arg Glu His Gly Asn Ala Ala Leu Ser Trp Leu Asp Lys
225 230 235 240
Gln Gln Pro Glu Ser Val Leu Phe Ile Ala Leu Gly Ser Glu Pro Pro
245 250 255
Val Thr Val Glu Gln Leu His Glu Ile Ala Leu Gly Leu Glu Leu Ala
260 265 270
Gly Thr Thr Phe Leu Trp Ala Leu Lys Lys Pro Asn Gly Leu Leu Leu
275 280 285
Glu Ala Asp Gly Asp Ile Leu Pro Pro Gly Phe Glu Glu Arg Thr Arg
290 295 300
Asp Arg Gly Leu Val Ala Met Gly Trp Val Pro Gln Pro Ile Ile Leu
305 310 315 320
Ala His Ser Ser Val Gly Ala Phe Leu Thr His Gly Gly Trp Ala Ser
325 330 335
Thr Ile Glu Gly Val Met Ser Gly His Pro Met Leu Phe Leu Thr Phe
340 345 350
Leu Asp Glu Gln Arg Ile Asn Ala Gln Leu Ile Glu Arg Lys Lys Ala
355 360 365
Gly Leu Arg Val Pro Arg Arg Glu Lys Asp Gly Ser Tyr Asp Arg Gln
370 375 380
Gly Ile Ala Gly Ala Ile Arg Ala Val Met Cys Glu Glu Glu Ser Lys
385 390 395 400
Ser Val Phe Ala Ala Asn Ala Lys Lys Met Gln Glu Ile Val Ser Asp
405 410 415
Arg Asn Cys Gln Glu Lys Tyr Ile Asp Glu Leu Ile Gln Arg Leu Gly
420 425 430
Ser Phe Glu Lys
435
<210> 20
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 20
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 21
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 21
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 22
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 22
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 23
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 23
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 24
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 24
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 25
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 25
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 26
<211> 4680
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 26
atgtcttcac tagaagtggt agatgggtgc ccctatggat accgaccata tccagatagt 60
ggcacaaatg cattaaatcc atgttttata tcagtaatat ccgcctggca agccgtcttt 120
ttcctattga ttggtagcta tcaattgtgg aaactttata agaacaataa agtaccaccc 180
agatttaaga actttcctac attaccaagt aaaatcaaca gtcgacatct aacgcatttg 240
accaatgttt gctttcagtc cacgcttata atttgtgaac tggccttggt atcccaatct 300
agcgataggg tttatccatt tatactaaag aaggctctgt acttgaatct ccttttcaat 360
ttgggtattt ctctccctac tcaatactta gcttatttta aaagtacatt ttcaatgggc 420
aaccagcttt tctattacat gtttcaaatt cttctacagc tcttcttgat attgcagagg 480
tactatcatg gttctagtaa cgaaaggctt actgttatta gcggacaaac tgctatgatt 540
ttagaagtgc tccttctttt caattctgtg gcaattttta tttatgatct atgcattttt 600
gagccaatta acgaattatc tgaatactac aagaaaaatg ggtggtatcc ccccgttcat 660
gtactatcct atattacatt tatctggatg aacaaactga ttgtggaaac ttaccgtaac 720
aagaaaatca aagatcctaa ccagttacca ttgccgccag tagatctgaa tattaagtcg 780
ataagtaagg aatttaaggc taactgggaa ttggaaaaat ggttgaatag aaattctctt 840
tggagggcca tttggaagtc atttggtagg actatttctg tggctatgct gtatgaaacg 900
acatctgatt tactttctgt agtacagccc cagtttctac ggatattcat agatggtttg 960
aacccggaaa catcttctaa atatcctcct ttaaatggtg tatttattgc tctaaccctt 1020
ttcgtaatca gcgtggtttc tgtgttcctc accaatcaat tttatattgg aatttttgag 1080
gctggtttgg ggataagagg ctctttagct tctttagtgt atcagaagtc cttaagattg 1140
acgctagcag agcgtaacga aaaatctact ggtgacatct taaatttgat gtctgtggat 1200
gtgttaagga tccagcggtt tttcgaaaat gcccaaacca ttattggcgc tcctattcag 1260
attattgttg tattaacttc cctgtactgg ttgctaggaa aggctgttat tggagggttg 1320
gttactatgg ctattatgat gcctatcaat gccttcttat ctagaaaggt aaaaaagcta 1380
tcaaaaactc aaatgaagta taaggacatg agaatcaaga ctattacaga gcttttgaat 1440
gctataaaat ctattaaatt atacgcctgg gaggaaccta tgatggcaag attgaatcat 1500
gttcgtaatg atatggagtt gaaaaatttt cggaaaattg gtatagtgag caatctgata 1560
tattttgcgt ggaattgtgt acctttaatg gtgacatgtt ccacatttgg cttattttct 1620
ttatttagtg attctccgtt atctcctgcc attgtcttcc cttcattatc tttatttaat 1680
attttgaaca gtgccatcta ttccgttcca tccatgataa ataccattat agagacaagc 1740
gtttctatgg aaagattaaa gtcattccta cttagtgacg aaattgatga ttcgttcatc 1800
gaacgtattg atccttcagc ggatgaaaga gcgttacctg ctatagagat gaataatatt 1860
acatttttat ggaaatcaaa agaagtatta acatctagcc aatctggaga taatttgagg 1920
acagatgaag agtctattat cggatcttct caaattgcgt tgaagaatat cgatcatttt 1980
gaagcaaaaa ggggtgattt agtttgtgtt gttggtcggg taggagctgg taaatcaaca 2040
tttttgaagg caattcttgg tcaacttcct tgcatgagtg gttctaggga ctcgatacca 2100
cctaaactga tcattagatc atcgtctgta gcctactgtt cacaagaatc ctggataatg 2160
aacgcatctg taagagaaaa cattctattt ggtcacaagt tcgaccaaga ttattatgac 2220
ctcactatta aagcatgtca attgctaccc gatttgaaaa tactaccaga tggtgatgaa 2280
actttggtag gtgaaaaggg catttcccta tcaggcggtc agaaggcccg tctttcatta 2340
gccagagcgg tgtactcgag agcagatatt tatttgttgg atgacatttt atctgctgtt 2400
gatgcagaag ttagtaaaaa tattattgaa tatgttttga tcggaaagac ggctttatta 2460
aaaaataaaa caattatttt aactaccaat actgtatcaa ttttaaaaca ttcgcagatg 2520
atatatgcgc tagaaaacgg tgaaattgtt gaacaaggga attatgagga tgtaatgaac 2580
cgtaagaaca atacttcaaa actgaaaaaa ttactagagg aatttgattc tccgattgat 2640
aatggaaatg aaagcgatgt ccaaactgaa caccgatccg aaagtgaagt ggatgaacct 2700
ctgcagctta aagtaactga atcagaaact gaggatgagg ttgttactga gagtgaatta 2760
gaactaatca aagccaattc tagaagagct tctctagcta cgctaagacc tagacccttt 2820
gtgggagcac aattggattc cgtgaagaaa acggcgcaaa aggccgagaa gacagaggtg 2880
ggaagagtca aaacaaagat ttatcttgcg tatattaagg cttgtggagt tttaggtgtt 2940
gttttatttt tcttgtttat gatattaaca agggttttcg acttagcaga gaatttttgg 3000
ttaaagtact ggtcagaatc taatgaaaaa aatggttcaa atgaaagggt ttggatgttt 3060
gttggtgtgt attccttaat cggagtagca tcggccgcat tcaataattt acggagtatt 3120
atgatgctac tgtattgttc tattaggggt tctaagaaac tgcatgaaag catggccaaa 3180
tctgtaatta gaagtcctat gactttcttt gagactacac cagttggaag gatcataaac 3240
aggttctcat ctgatatgga tgcagtggac agtaatctac agtacatttt ctcctttttt 3300
ttcaaatcaa tactaaccta tttggttact gttatattag tcgggtacaa tatgccatgg 3360
tttttagtgt tcaatatgtt tttggtggtt atctatattt actatcaaac attttacatt 3420
gtgctatcta gggagctaaa aagattgatc agtatatctt actctccgat tatgtcctta 3480
atgagtgaga gcttgaacgg ttattctatt attgatgcat acgatcattt tgagagattc 3540
atctatctaa attatgaaaa aatccaatac aacgttgatt ttgtcttcaa ctttagatca 3600
acgaatagat ggttatccgt gagattgcaa actattggtg ctacaattgt tttggctact 3660
gcaatcttag cactagcaac aatgaatact aaaaggcaac taagttcggg tatggttggt 3720
ctactaatga gctattcatt agaggttaca ggttcattga cttggattgt aaggacaact 3780
gtgacgattg aaaccaacat tgtatcagtg gagagaattg ttgagtactg cgaattacca 3840
cctgaagcac agtccattaa ccctgaaaag aggccagatg aaaattggcc atcaaagggt 3900
ggtattgaat tcaaaaacta ttccacaaaa tacagagaaa atttggatcc agtgctgaat 3960
aatattaacg tgaagattga gccatgtgaa aaggttggga ttgttggcag aacaggtgca 4020
gggaagtcta cactgagcct ggcattattt agaatactag aacctaccga aggtaaaatt 4080
attattgacg gcattgatat atccgacata ggtctgttcg atttaagaag ccatttggca 4140
attattcctc aggatgcaca agcttttgaa ggtacagtaa agaccaattt ggaccctttc 4200
aatcgttatt cagaagatga acttaaaagg gctgttgagc aggcacattt aaagcctcat 4260
ctggaaaaaa tgctgcacag taaaccaaga ggtgatgatt ctaatgaaga ggatggcaat 4320
gttaatgata ttctggatgt caagattaat gagaacggta gtaacttgtc agtggggcaa 4380
agacaactac tatgtttggc aagagcgctg ctaaaccgtt ccaaaatatt ggtccttgat 4440
gaagcaacgg cttctgtgga tatggaaacc gataaaatta tccaagacac tataagaaga 4500
gaatttaagg accgtaccat cttaacaatt gcacatcgta tcgacactgt attggacagt 4560
gataagataa ttgttcttga ccagggtagt gtgagggaat tcgattcacc ctcgaaattg 4620
ttatccgata aaacgtctat tttttacagt ctttgtgaga aaggtgggta tttgaaataa 4680
<210> 27
<211> 4953
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic: nucleic acid sequence
<400> 27
atgtcaggtt caaattcgaa ttcaaatcta gatgcaataa gtgattcatg cccattttgg 60
cgctatgatg atattacaga gtgtggaaga gtgcagtata tcaattacta ccttccaata 120
acattggtag gcgtttctct cttgtattta ttcaaaaacg cgatccaaca ttattacaga 180
aagcctcaag aaattaagcc tagtgttgct tccgaattat tgggctcaaa tctcacagac 240
cttccgaatg aaaacaagcc tttactatcg gagagtacac aagcattata cactaatccg 300
gattcgaata agacaggatt ctctctaaaa gaggagcatt tctctataaa taaagttaca 360
cttacggaaa ttcattccaa taagcatgac gctgtgaaga tcgtaaggag aaactggctt 420
gaaaaattaa gagtgttctt agaatgggtt ctatgcgcct tacaactttg catctacatt 480
tcagtctggt cgaaatacac taatacccaa gaggatttcc caatgcacgc atctatctca 540
ggtctaatgt tatggtctct actcttgtta gtagtgtcat tgaggttggc aaacatcaac 600
cagaatataa gctggatcaa ttcaggaccg ggaaacttat gggccctttc atttgcatgt 660
tatctatcac tattctgcgg atccgttttg ccattgagat ctatctatat cggtcatatc 720
acagatgaaa ttgcatcaac attttataag ttgcaatttt acctaagttt gacactattc 780
ttgttacttt tcacctctca agcgggaaat cggtttgcca ttatctataa aagtacacca 840
gatataacac cgtctcctga acctattgtg tcgattgcaa gttatatcac ttgggcatgg 900
gtagataaat ttctttggaa agcgcatcaa aattatatcg aaatgaaaga tgtttggggt 960
ctaatggtgg aagactattc cattctcgta ataaagagat tcaatcattt tgttcagaat 1020
aaaaccaagt ctaggacatt ttcatttaac ttaatccact ttttcatgaa atttatcgcc 1080
attcaaggtg cctgggcaac aatttcgtca gttattagtt ttgttccaac aatgttgctc 1140
agacgtattt tggagtatgt tgaagatcaa tcaactgctc cattaaattt ggcttggatg 1200
tatatttttc ttatgttcct tgccagaatt ttaactgcca tatgtgctgc tcaggcgcta 1260
tttttaggga gaagggtttg tatcagaatg aaggctatca taatttctga aatctactcc 1320
aaggctttga gaagaaaaat ttctccaaat tccactaagg agccaactga tgtcgttgat 1380
ccacaggaat taaatgacaa acaacacgtt gatggagatg aagaatcagc aaccactgca 1440
aatcttggtg ctatcattaa tttgatggcg gtggatgctt tcaaagtatc cgaaatatgt 1500
gcgtatttgc actcctttat agaggcgatc atcatgacca ttgttgcatt attcctttta 1560
tatcggttaa taggctggtc tgctttagtt ggtagtgcaa tgattatttg cttcttacca 1620
ttgaacttca aacttgccag cttgttaggg acactccaaa agaaatcctt ggcaatcaca 1680
gataaaagaa ttcagaaact aaacgaagct ttccaggcca ttcgtattat caaattcttc 1740
tcttgggaag agaattttga aaaggacata caaaacacaa gggatgaaga attaaatatg 1800
cttttaaaaa ggtctatcgt ttgggctctt tcttctcttg tttggttcat taccccctct 1860
attgtcacat ccgcttcttt tgcagtctat atttatgtgc aaggccaaac tttaactact 1920
ccggtagcat ttactgcact atctctattt gctctactaa gaaatccgtt agacatgctt 1980
tctgatatgt tgtcttttgt tattcaatcc aaggtctctt tggatagagt ccaagaattt 2040
ttaaatgaag aggagacgaa aaagtatgag caattaaccg tatcaagaaa taaacttggg 2100
ttgcaaaacg ctacttttac atgggataaa aataatcaag atttcaagtt aaaaaaccta 2160
actattgatt tcaaaattgg gaaattaaac gttattgtag gtccaactgg atctggtaaa 2220
acatcattgt taatgggatt attgggtgaa atggagctat tgaacggaaa agttttcgtc 2280
ccttcgctca atcctaggga agagttggtt gtagaggccg atggaatgac taattcaatc 2340
gcgtactgct cccaagctgc ctggttgcta aatgatactg tcaggaacaa tattctattc 2400
aatgcgcctt ataatgagaa tagatataat gccgtcatct ctgcgtgtgg tttgaaacgc 2460
gacttcgaga tcttaagcgc tggtgatcag acagagattg gcgaaaaggg tataacactt 2520
tctggtggtc aaaaacaaag agtctcgttg gccagatcat tgtattcttc atcaagacat 2580
ttgctgttag atgattgttt gagtgccgta gactcgcaca cggccttatg gatctacgaa 2640
aattgtataa caggcccatt aatggaagga agaacatgtg tattggtttc tcacaatgtt 2700
gcattaactt taaaaaatgc agattgggtt atcattatgg aaaatggtag agtaaaagaa 2760
caaggcgaac cagtagaatt gctacagaag gggtcccttg gggatgactc catggtgaaa 2820
tcatcaattt tgtcccgtac ggcgtcctca gttaatattt cagaaactaa cagtaagatt 2880
tctagtggtc cgaaggctcc agcggaatcg gataatgcca atgaggagtc caccacctgt 2940
ggagatcgtt caaagtcaag cggcaagcta atcgctgaag aaacaaaatc aaacggtgtt 3000
gtttccctgg acgtctataa gtggtatgcc gtgtttttcg gtggatggaa gatgatatca 3060
tttttgtgtt tcattttctt gtttgcccaa atgatcagta tttcacaggc ctggtggttg 3120
cgtgcttggg cctccaacaa cactctaaaa gttttctcca accttggatt gcaaacaatg 3180
aggccattcg ctttgtcctt acaaggaaaa gaagcttctc ctgtgactct tagtgctgtt 3240
ttcccaaatg gcagtctaac aacagccacg gaaccaaatc actcgaacgc gtattatcta 3300
tcaatatatt tgggtattgg tgtattccag gctttatgtt catcttcgaa agcaattata 3360
aactttgtgg ccggtattag agcttccagg aaaatattca atttattgtt gaaaaatgtg 3420
ttatacgcca agctgagatt ttttgattct actccaatag gaagaataat gaacagattt 3480
tctaaagaca tcgaatcaat agatcaagaa ttgactcctt atatggaagg tgcatttggt 3540
tccttaatac aatgtgtttc cacaattatc gtcattgcat acattactcc ccaatttttg 3600
attgtcgcgg cgattgtcat gttattgttt tattttgttg cctactttta catgtcagga 3660
gcaagagaat taaagcgtct tgaatcgatg tcacgctctc ctattcatca gcacttctct 3720
gagactcttg tgggtatcac gactattcga gcattttctg acgagcggcg ttttctggtt 3780
gataatatga agaaaattga tgataataat aggcctttct tttacttatg ggtctgtaat 3840
agatggctat cttacagaat cgagctgata ggcgccctta ttgttttggc tgcaggtagt 3900
ttcatcttat tgaacataaa atcgatcgat tctggtttgg ccggtatttc attgggtttc 3960
gctatacaat ttaccgatgg tgccctttgg gttgttaggt tatattccaa cgttgaaatg 4020
aatatgaatt ccgtcgaaag gttaaaagag tacaccacca tcgagcaaga accttctaac 4080
gttggtgcct tggtacctcc ttgcgaatgg ccacaaaatg gtaaaatcga agtcaaggat 4140
ttatctttac gctatgcagc tggtctacca aaggttataa aaaatgtcac attcaccgtc 4200
gattcaaagt gtaaagtagg tattgttggc aggactggtg ctggtaaatc tactattatc 4260
acagcccttt tcagattctt agaccctgaa actggttata tcaaaatcga tgacgttgat 4320
ataacaacca ttggtttaaa acgtttgcgc caatctatca ctattattcc acaggaccca 4380
acccttttca ccggtacttt gaaaaccaat ctcgatccat acaacgaata ttcggaagct 4440
gaaattttcg aagctctaaa acgtgtcaac cttgtttcct cagaagaact tggtaatcct 4500
tctacttcgg attcaacctc ggtacattca gcaaatatga ataagttttt ggatttggaa 4560
aatgaagtca gtgaaggtgg ttccaacctc tcacaaggac aacgtcaatt gatatgtttg 4620
gcccgttcat tattgcggtg tccaaaggta attctacttg atgaagccac agcttcaatc 4680
gattataact cagactctaa aatccaggct actataaggg aagaattcag taatagtacc 4740
attctcacga ttgctcatcg tttacgatca attattgatt atgataaaat acttgttatg 4800
gatgctgggg aggttaaaga atatgatcat ccttactcct tattgttgaa tcgtgatagt 4860
atattctatc atatgtgtga agatagtgga gaattagaag tcttgataca attagccaaa 4920
gaatcatttg tcaaaaagct caatgcaaat tga 4953
<210> 28
<211> 1381
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Fungal_5_muA
<400> 28
Met Thr Ser Pro Gly Ser Glu Lys Cys Thr Pro Arg Ser Asp Glu Asp
1 5 10 15
Leu Glu Arg Ser Glu Pro Gln Leu Gln Arg Arg Leu Leu Thr Pro Phe
20 25 30
Leu Leu Ser Lys Lys Val Pro Pro Ile Pro Lys Glu Asp Glu Arg Lys
35 40 45
Pro Tyr Pro Tyr Leu Lys Thr Asn Pro Leu Ser Gln Ile Leu Phe Trp
50 55 60
Trp Leu Asn Pro Leu Leu Arg Val Gly Tyr Lys Arg Thr Leu Asp Pro
65 70 75 80
Asn Asp Phe Tyr Tyr Leu Glu His Ser Gln Asp Ile Glu Thr Thr Tyr
85 90 95
Ser Asn Tyr Glu Met His Leu Ala Arg Ile Leu Glu Lys Asp Arg Ala
100 105 110
Lys Ala Arg Ala Lys Asp Pro Thr Leu Thr Asp Glu Asp Leu Lys Asn
115 120 125
Arg Glu Tyr Pro Lys Asn Ala Val Ile Lys Ala Leu Phe Leu Thr Phe
130 135 140
Lys Trp Lys Tyr Leu Trp Ser Ile Phe Leu Lys Leu Leu Ser Asp Ile
145 150 155 160
Val Leu Val Leu Asn Pro Leu Leu Ser Lys Ala Leu Ile Asn Phe Val
165 170 175
Asp Glu Lys Met Tyr Asn Pro Asp Met Ser Val Gly Arg Gly Val Gly
180 185 190
Tyr Ala Ile Gly Val Thr Phe Met Leu Gly Thr Ser Gly Ile Leu Ile
195 200 205
Asn His Phe Leu Tyr Leu Ser Leu Thr Val Gly Ala His Cys Lys Ala
210 215 220
Val Leu Thr Thr Ala Ile Met Asn Lys Ser Phe Arg Ala Ser Ala Lys
225 230 235 240
Ser Lys His Glu Tyr Pro Ser Gly Arg Val Thr Ser Leu Met Ser Thr
245 250 255
Asp Leu Ala Arg Ile Asp Leu Ala Ile Gly Phe Gln Pro Phe Ala Ile
260 265 270
Thr Val Pro Val Pro Ile Gly Val Ala Ile Ala Leu Leu Ile Val Asn
275 280 285
Ile Gly Val Ser Ala Leu Ala Gly Ile Ala Val Phe Leu Val Cys Ile
290 295 300
Val Val Ile Ser Ala Ser Ser Lys Ser Leu Leu Lys Met Arg Lys Gly
305 310 315 320
Ala Asn Gln Tyr Thr Asp Ala Arg Ile Ser Tyr Met Arg Glu Ile Leu
325 330 335
Gln Asn Met Arg Ile Ile Lys Phe Tyr Ser Trp Glu Asp Ala Tyr Glu
340 345 350
Lys Ser Val Val Thr Glu Arg Asn Ser Glu Met Ser Ile Ile Leu Lys
355 360 365
Met Gln Ser Ile Arg Asn Phe Leu Leu Ala Leu Ser Leu Ser Leu Pro
370 375 380
Ala Ile Ile Ser Met Val Ala Phe Leu Val Leu Tyr Gly Val Ser Asn
385 390 395 400
Asp Lys Asn Pro Gly Asn Ile Phe Ser Ser Ile Ser Leu Phe Ser Val
405 410 415
Leu Ala Gln Gln Thr Met Met Leu Pro Met Ala Leu Ala Thr Gly Ala
420 425 430
Asp Ala Lys Ile Gly Leu Glu Arg Leu Arg Gln Tyr Leu Gln Ser Gly
435 440 445
Asp Ile Glu Lys Glu Tyr Glu Asp His Glu Lys Pro Gly Asp Arg Asp
450 455 460
Val Val Leu Pro Asp Asn Val Ala Val Glu Leu Asn Asn Ala Ser Phe
465 470 475 480
Ile Trp Glu Lys Phe Asp Asp Ala Asp Asp Asn Asp Gly Asn Ser Glu
485 490 495
Lys Thr Lys Glu Val Val Val Thr Ser Lys Ser Ser Leu Thr Asp Ser
500 505 510
Ser His Ile Asp Lys Ser Thr Asp Ser Ala Asp Gly Glu Tyr Ile Lys
515 520 525
Ser Val Phe Glu Gly Phe Asn Asn Ile Asn Leu Thr Ile Lys Lys Gly
530 535 540
Glu Phe Val Ile Ile Thr Gly Pro Ile Gly Ser Gly Lys Ser Ser Leu
545 550 555 560
Leu Val Ala Leu Ala Gly Phe Met Lys Lys Thr Ser Gly Thr Leu Gly
565 570 575
Val Asn Gly Thr Met Leu Leu Cys Gly Gln Pro Trp Val Gln Asn Cys
580 585 590
Thr Val Arg Asp Asn Ile Leu Phe Gly Leu Glu Tyr Asp Glu Ala Arg
595 600 605
Tyr Asp Arg Val Val Glu Val Cys Ala Leu Gly Asp Asp Leu Lys Met
610 615 620
Phe Thr Ala Gly Asp Gln Thr Glu Ile Gly Glu Arg Gly Ile Thr Leu
625 630 635 640
Ser Gly Gly Gln Lys Ala Arg Ile Asn Leu Ala Arg Ala Val Tyr Ala
645 650 655
Asn Lys Asp Ile Ile Leu Leu Asp Asp Ala Leu Ser Ala Val Asp Ala
660 665 670
Arg Val Gly Lys Leu Ile Val Asp Asp Cys Leu Thr Ser Phe Leu Gly
675 680 685
Asp Lys Thr Arg Ile Leu Ala Thr His Gln Leu Ser Leu Ile Glu Ala
690 695 700
Ala Asp Arg Val Ile Tyr Leu Asn Gly Asp Gly Thr Ile His Ile Gly
705 710 715 720
Thr Val Gln Glu Leu Leu Glu Ser Asn Glu Gly Phe Leu Lys Leu Met
725 730 735
Glu Phe Ser Arg Lys Ser Glu Ser Glu Asp Glu Glu Asp Val Glu Ala
740 745 750
Ala Asn Glu Lys Asp Val Ser Leu Gln Lys Ala Val Ser Val Val Gln
755 760 765
Glu Gln Asp Ala His Ala Gly Val Leu Ile Gly Gln Glu Glu Arg Ala
770 775 780
Val Asn Gly Ile Glu Trp Asp Ile Tyr Lys Glu Tyr Leu His Glu Gly
785 790 795 800
Arg Gly Lys Leu Gly Ile Phe Ala Ile Pro Thr Ile Ile Met Leu Leu
805 810 815
Val Leu Asp Val Phe Thr Ser Ile Phe Val Asn Val Trp Leu Ser Phe
820 825 830
Trp Ile Ser His Lys Phe Lys Ala Arg Ser Asp Gly Phe Tyr Ile Gly
835 840 845
Leu Tyr Val Met Phe Val Ile Leu Ser Val Ile Trp Ile Thr Ala Glu
850 855 860
Phe Val Val Met Gly Asn Phe Ser Ser Thr Ala Ala Arg Arg Leu Asn
865 870 875 880
Leu Lys Ala Met Lys Arg Val Leu His Thr Pro Met His Phe Leu Asp
885 890 895
Val Thr Pro Met Gly Arg Ile Leu Asn Arg Phe Thr Lys Asp Thr Asp
900 905 910
Val Leu Asp Asn Glu Ile Gly Glu Gln Ala Arg Met Phe Leu His Pro
915 920 925
Ala Ala Tyr Val Ile Gly Val Leu Ile Leu Cys Ile Ile Asn Ile Pro
930 935 940
Trp Phe Ala Ile Ala Ile Pro Pro Leu Ala Ile Pro Phe Thr Phe Ile
945 950 955 960
Thr Asn Phe Tyr Ile Ala Ser Ser Arg Glu Val Lys Arg Ile Glu Ala
965 970 975
Ile Gln Arg Ser Leu Val Tyr Asn Asn Phe Asn Glu Val Leu Asn Gly
980 985 990
Leu Gln Thr Leu Lys Ala Tyr Asn Ala Thr Ser Arg Phe Met Glu Lys
995 1000 1005
Asn Lys Arg Leu Leu Asn Arg Met Asn Glu Ala Tyr Leu Leu Val
1010 1015 1020
Ile Ala Asn Gln Arg Trp Ile Ser Val Asn Leu Asp Leu Val Ser
1025 1030 1035
Cys Cys Phe Val Phe Leu Ile Ser Met Leu Ser Val Phe Arg Val
1040 1045 1050
Phe Asp Ile Asn Ala Ser Ser Val Gly Leu Val Val Thr Ser Val
1055 1060 1065
Leu Gln Ile Gly Gly Leu Met Ser Leu Ile Met Arg Ala Tyr Thr
1070 1075 1080
Thr Val Glu Asn Glu Met Asn Ser Val Glu Arg Leu Cys His Tyr
1085 1090 1095
Ala Asn Lys Leu Glu Gln Glu Ala Pro Tyr Ile Met Asn Glu Thr
1100 1105 1110
Lys Pro Arg Pro Thr Trp Pro Glu His Gly Ala Ile Glu Phe Lys
1115 1120 1125
His Ala Ser Met Arg Tyr Arg Glu Gly Leu Pro Leu Val Leu Lys
1130 1135 1140
Asp Leu Thr Ile Ser Val Lys Gly Gly Glu Lys Ile Gly Ile Cys
1145 1150 1155
Gly Arg Thr Gly Ala Gly Lys Ser Thr Ile Met Asn Ala Leu Tyr
1160 1165 1170
Arg Leu Thr Glu Leu Ala Glu Gly Ser Ile Thr Ile Asp Gly Val
1175 1180 1185
Glu Ile Ser Gln Leu Gly Leu Tyr Asp Leu Arg Ser Lys Leu Ala
1190 1195 1200
Ile Ile Pro Gln Asp Pro Val Leu Phe Arg Gly Thr Ile Arg Lys
1205 1210 1215
Asn Leu Asp Pro Phe Gly Gln Asn Asp Asp Glu Thr Leu Trp Asp
1220 1225 1230
Ala Leu Arg Arg Ser Gly Leu Val Glu Gly Ser Ile Leu Asn Thr
1235 1240 1245
Ile Lys Ser Gln Ser Lys Asp Asp Pro Asn Phe His Lys Phe His
1250 1255 1260
Leu Asp Gln Thr Val Glu Asp Glu Gly Ala Asn Phe Ser Leu Gly
1265 1270 1275
Glu Arg Gln Leu Ile Ala Leu Ala Arg Ala Leu Val Arg Asn Ser
1280 1285 1290
Lys Ile Leu Ile Leu Asp Glu Ala Thr Ser Ser Val Asp Tyr Glu
1295 1300 1305
Thr Asp Ser Lys Ile Gln Lys Thr Ile Ser Thr Val Phe Ser His
1310 1315 1320
Cys Thr Ile Leu Cys Ile Ala His Arg Leu Lys Thr Ile Leu Thr
1325 1330 1335
Tyr Asp Arg Ile Leu Val Leu Glu Lys Gly Glu Val Glu Glu Phe
1340 1345 1350
Asp Thr Pro Arg Val Leu Tyr Ser Lys Asn Gly Val Phe Arg Gln
1355 1360 1365
Met Cys Glu Arg Ser Glu Ile Thr Ser Ala Asp Phe Val
1370 1375 1380
<210> 29
<211> 1381
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Fungal_5_muA2
<400> 29
Met Thr Ser Pro Gly Ser Glu Lys Cys Thr Pro Arg Ser Asp Glu Asp
1 5 10 15
Leu Glu Arg Ser Glu Pro Gln Leu Gln Arg Arg Leu Leu Thr Pro Phe
20 25 30
Leu Leu Ser Lys Lys Val Pro Pro Ile Pro Lys Glu Asp Glu Arg Lys
35 40 45
Pro Tyr Pro Tyr Leu Lys Thr Asn Pro Leu Ser Gln Ile Leu Phe Trp
50 55 60
Trp Leu Asn Pro Leu Leu Arg Val Gly Tyr Lys Arg Thr Leu Asp Pro
65 70 75 80
Asn Asp Phe Tyr Tyr Leu Glu His Ser Gln Asp Ile Glu Thr Thr Tyr
85 90 95
Ser Asn Tyr Glu Met His Leu Ala Arg Ile Leu Glu Lys Asp Arg Ala
100 105 110
Lys Ala Arg Ala Lys Asp Pro Thr Leu Thr Asp Glu Asp Leu Lys Asn
115 120 125
Arg Glu Tyr Pro Lys Asn Ala Val Ile Lys Ala Leu Phe Leu Thr Phe
130 135 140
Lys Trp Lys Tyr Leu Trp Ser Ile Phe Leu Lys Leu Leu Ser Asp Ile
145 150 155 160
Val Leu Val Leu Asn Pro Leu Leu Ser Lys Ala Leu Ile Asn Phe Val
165 170 175
Asp Glu Lys Met Tyr Asn Pro Asp Met Ser Val Gly Arg Gly Val Gly
180 185 190
Tyr Ala Ile Gly Val Thr Phe Met Leu Gly Thr Ser Gly Ile Leu Ile
195 200 205
Asn His Phe Leu Tyr Leu Ser Leu Thr Val Gly Ala His Cys Lys Ala
210 215 220
Val Leu Thr Thr Ala Ile Met Asn Lys Ser Phe Arg Ala Ser Ala Lys
225 230 235 240
Ser Lys His Glu Tyr Pro Ser Gly Arg Val Thr Ser Leu Met Ser Thr
245 250 255
Asp Leu Ala Arg Ile Asp Leu Ala Ile Gly Phe Gln Pro Phe Ala Ile
260 265 270
Thr Val Pro Val Pro Ile Gly Val Ala Ile Ala Leu Leu Ile Val Asn
275 280 285
Ile Gly Val Ser Ala Leu Ala Gly Ile Ala Val Phe Leu Val Cys Ile
290 295 300
Val Val Ile Ser Ala Ser Ser Lys Ser Leu Leu Lys Met Arg Lys Gly
305 310 315 320
Ala Asn Gln Tyr Thr Asp Ala Arg Ile Ser Tyr Met Arg Glu Ile Leu
325 330 335
Gln Asn Met Arg Ile Ile Lys Phe Tyr Ser Trp Glu Asp Ala Tyr Glu
340 345 350
Lys Ser Val Val Thr Glu Arg Asn Ser Glu Met Ser Ile Ile Leu Lys
355 360 365
Met Gln Ser Ile Arg Asn Phe Leu Leu Ala Leu Ser Leu Ser Leu Pro
370 375 380
Ala Ile Ile Ser Met Val Ala Phe Leu Val Leu Tyr Gly Val Ser Asn
385 390 395 400
Asp Lys Asn Pro Gly Asn Ile Phe Ser Ser Ile Ser Leu Phe Ser Val
405 410 415
Leu Ala Gln Gln Thr Met Met Leu Pro Met Ala Leu Ala Thr Gly Ala
420 425 430
Asp Ala Lys Ile Gly Leu Glu Arg Leu Arg Gln Tyr Leu Gln Ser Gly
435 440 445
Asp Ile Glu Lys Glu Tyr Glu Asp His Glu Lys Pro Gly Asp Arg Asp
450 455 460
Val Val Leu Pro Asp Asn Val Ala Val Glu Leu Asn Asn Ala Ser Phe
465 470 475 480
Ile Trp Glu Lys Phe Asp Asp Ala Asp Asp Asn Asp Gly Asn Ser Glu
485 490 495
Lys Thr Lys Glu Val Val Val Thr Ser Lys Ser Ser Leu Thr Asp Ser
500 505 510
Ser His Ile Asp Lys Ser Thr Asp Ser Ala Asp Gly Glu Tyr Ile Lys
515 520 525
Ser Val Phe Glu Gly Phe Asn Asn Ile Asn Leu Thr Ile Lys Lys Gly
530 535 540
Glu Phe Val Ile Ile Thr Gly Pro Ile Gly Ser Gly Lys Ser Ser Leu
545 550 555 560
Leu Val Ala Leu Ala Gly Phe Met Lys Lys Thr Ser Gly Thr Leu Gly
565 570 575
Val Asn Gly Thr Met Leu Leu Cys Gly Gln Pro Trp Val Gln Asn Cys
580 585 590
Thr Val Arg Asp Asn Ile Leu Phe Gly Leu Glu Tyr Asp Glu Ala Arg
595 600 605
Tyr Asp Arg Val Val Glu Val Cys Ala Leu Gly Asp Asp Leu Lys Met
610 615 620
Phe Thr Ala Gly Asp Gln Thr Glu Ile Gly Glu Arg Gly Ile Thr Leu
625 630 635 640
Ser Gly Gly Gln Lys Ala Arg Ile Asn Leu Ala Arg Ala Val Tyr Ala
645 650 655
Asn Lys Asp Ile Ile Leu Leu Asp Asp Ala Leu Ser Ala Val Asp Ala
660 665 670
Arg Val Gly Lys Leu Ile Val Asp Asp Cys Leu Thr Ser Phe Leu Gly
675 680 685
Asp Lys Thr Arg Ile Leu Ala Thr His Gln Leu Ser Leu Ile Glu Ala
690 695 700
Ala Asp Arg Val Ile Tyr Leu Asn Gly Asp Gly Thr Ile His Ile Gly
705 710 715 720
Thr Val Gln Glu Leu Leu Glu Ser Asn Glu Gly Phe Leu Lys Leu Met
725 730 735
Glu Phe Ser Arg Lys Ser Glu Ser Glu Asp Glu Glu Asp Val Glu Ala
740 745 750
Ala Asn Glu Lys Asp Val Ser Leu Gln Lys Ala Val Ser Val Val Gln
755 760 765
Glu Gln Asp Ala His Ala Gly Val Leu Ile Gly Gln Glu Glu Arg Ala
770 775 780
Val Asn Gly Ile Glu Trp Asp Ile Tyr Lys Glu Tyr Leu His Glu Gly
785 790 795 800
Arg Gly Lys Leu Gly Ile Phe Ala Ile Pro Thr Ile Ile Met Leu Leu
805 810 815
Val Leu Asp Val Phe Thr Ser Ile Phe Val Asn Val Trp Leu Ser Phe
820 825 830
Trp Ile Ser His Lys Phe Lys Ala Arg Ser Asp Gly Phe Tyr Ile Gly
835 840 845
Leu Tyr Val Met Phe Val Ile Leu Ser Val Ile Trp Ile Thr Ala Glu
850 855 860
Phe Val Val Met Gly Asn Phe Ser Ser Thr Ala Ala Arg Arg Leu Asn
865 870 875 880
Leu Lys Ala Met Lys Arg Val Leu His Thr Pro Met His Phe Leu Asp
885 890 895
Val Thr Pro Met Gly Arg Ile Leu Asn Arg Phe Thr Lys Asp Thr Asp
900 905 910
Val Leu Asp Asn Glu Ile Gly Glu Gln Ala Arg Met Phe Leu His Pro
915 920 925
Ala Ala Tyr Val Ile Gly Val Leu Ile Leu Cys Ile Ile Asn Ile Pro
930 935 940
Trp Phe Ala Ile Ala Ile Pro Pro Leu Ala Ile Pro Phe Thr Phe Ile
945 950 955 960
Thr Asn Phe Tyr Ile Ala Ser Ser Arg Glu Val Lys Arg Ile Glu Ala
965 970 975
Ile Gln Arg Ser Leu Val Tyr Asn Asn Phe Asn Glu Val Leu Asn Gly
980 985 990
Leu Gln Thr Leu Lys Ala Tyr Asn Ala Thr Ser Arg Phe Met Glu Lys
995 1000 1005
Asn Lys Arg Leu Leu Asn Arg Met Asn Glu Ala Tyr Leu Leu Val
1010 1015 1020
Ile Ala Asn Gln Arg Trp Ile Ser Val Asn Leu Asp Leu Val Ser
1025 1030 1035
Cys Cys Phe Val Phe Leu Ile Ser Met Leu Ser Val Phe Arg Val
1040 1045 1050
Phe Asp Ile Asn Ala Ser Ser Val Gly Leu Val Val Thr Ser Val
1055 1060 1065
Leu Gln Ile Gly Gly Leu Met Ser Leu Ile Met Arg Ala Tyr Thr
1070 1075 1080
Thr Val Glu Asn Glu Met Asn Ser Val Glu Arg Leu Cys His Tyr
1085 1090 1095
Ala Asn Lys Leu Glu Gln Glu Ala Pro Tyr Ile Met Asn Glu Thr
1100 1105 1110
Lys Pro Arg Pro Thr Trp Pro Glu His Gly Ala Ile Glu Phe Lys
1115 1120 1125
His Ala Ser Met Arg Tyr Arg Glu Gly Leu Pro Leu Val Leu Lys
1130 1135 1140
Asp Leu Thr Ile Ser Val Lys Gly Gly Glu Lys Ile Gly Ile Cys
1145 1150 1155
Gly Arg Thr Gly Ala Gly Lys Ser Thr Ile Met Asn Ala Leu Tyr
1160 1165 1170
Arg Leu Thr Glu Leu Ala Glu Gly Ser Ile Thr Ile Asp Gly Val
1175 1180 1185
Glu Ile Ser Gln Leu Gly Leu Tyr Asp Leu Arg Ser Lys Leu Ala
1190 1195 1200
Ile Ile Pro Gln Asp Pro Val Leu Phe Arg Gly Thr Ile Arg Lys
1205 1210 1215
Asn Leu Asp Pro Phe Gly Gln Asn Asp Asp Glu Thr Leu Trp Asp
1220 1225 1230
Ala Leu Arg Arg Ser Gly Leu Val Glu Gly Ser Ile Leu Asn Thr
1235 1240 1245
Ile Lys Ser Gln Ser Lys Asp Asp Pro Asn Phe His Lys Phe His
1250 1255 1260
Leu Asp Gln Thr Val Glu Asp Glu Gly Ala Asn Phe Ser Leu Gly
1265 1270 1275
Glu Arg Gln Leu Ile Ala Leu Ala Arg Ala Leu Val Arg Asn Ser
1280 1285 1290
Lys Ile Leu Ile Leu Asp Glu Ala Thr Ser Ser Val Asp Tyr Glu
1295 1300 1305
Thr Asp Ser Lys Ile Gln Lys Thr Ile Ser Thr Glu Phe Ser His
1310 1315 1320
Cys Thr Ile Leu Cys Ile Ala His Arg Leu Lys Thr Ile Leu Thr
1325 1330 1335
Tyr Asp Arg Ile Leu Val Leu Glu Lys Gly Glu Val Glu Glu Phe
1340 1345 1350
Asp Thr Pro Arg Val Leu Tyr Ser Lys Asn Gly Val Phe Arg Gln
1355 1360 1365
Met Cys Glu Arg Ser Glu Ile Thr Ser Ala Asp Phe Val
1370 1375 1380
<210> 30
<211> 1381
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic: Fungal_5_muA3
<400> 30
Met Thr Ser Pro Gly Ser Glu Lys Cys Thr Pro Arg Ser Asp Glu Asp
1 5 10 15
Leu Glu Arg Ser Glu Pro Gln Leu Gln Arg Arg Leu Leu Thr Pro Phe
20 25 30
Leu Leu Ser Lys Lys Val Pro Pro Ile Pro Lys Glu Asp Glu Arg Lys
35 40 45
Pro Tyr Pro Tyr Leu Lys Thr Asn Pro Leu Ser Gln Ile Leu Phe Trp
50 55 60
Trp Leu Asn Pro Leu Leu Arg Val Gly Tyr Lys Arg Thr Leu Asp Pro
65 70 75 80
Asn Asp Phe Tyr Tyr Leu Glu His Ser Gln Asp Ile Glu Thr Thr Tyr
85 90 95
Ser Asn Tyr Glu Met His Leu Ala Arg Ile Leu Glu Lys Asp Arg Ala
100 105 110
Lys Ala Arg Ala Lys Asp Pro Thr Leu Thr Asp Glu Asp Leu Lys Asn
115 120 125
Arg Glu Tyr Pro Lys Asn Ala Val Ile Lys Ala Leu Phe Leu Thr Phe
130 135 140
Lys Trp Lys Tyr Leu Trp Ser Ile Phe Leu Lys Leu Leu Ser Asp Ile
145 150 155 160
Val Leu Val Leu Asn Pro Leu Leu Ser Lys Ala Leu Ile Asn Phe Val
165 170 175
Asp Glu Lys Met Tyr Asn Pro Asp Met Ser Val Gly Arg Gly Val Gly
180 185 190
Tyr Ala Ile Gly Val Thr Phe Met Leu Gly Thr Ser Gly Ile Leu Ile
195 200 205
Asn His Phe Leu Tyr Leu Ser Leu Thr Val Gly Ala His Cys Lys Ala
210 215 220
Val Leu Thr Thr Ala Ile Met Asn Lys Ser Phe Arg Ala Ser Ala Lys
225 230 235 240
Ser Lys His Glu Tyr Pro Ser Gly Arg Val Thr Ser Leu Met Ser Thr
245 250 255
Asp Leu Ala Arg Ile Asp Leu Ala Ile Gly Phe Gln Pro Phe Ala Ile
260 265 270
Thr Val Pro Val Pro Ile Gly Val Ala Ile Ala Leu Leu Ile Val Asn
275 280 285
Ile Gly Val Ser Ala Leu Ala Gly Ile Ala Val Phe Leu Val Cys Ile
290 295 300
Val Val Ile Ser Ala Ser Ser Lys Ser Leu Leu Lys Met Arg Lys Gly
305 310 315 320
Ala Asn Gln Tyr Thr Asp Ala Arg Ile Ser Tyr Met Arg Glu Ile Leu
325 330 335
Gln Asn Met Arg Ile Ile Lys Phe Tyr Ser Trp Glu Asp Ala Tyr Glu
340 345 350
Lys Ser Val Val Thr Glu Arg Asn Ser Glu Met Ser Ile Ile Leu Lys
355 360 365
Met Gln Ser Ile Arg Asn Phe Leu Leu Ala Leu Ser Leu Ser Leu Pro
370 375 380
Ala Ile Ile Ser Met Val Ala Phe Leu Val Leu Tyr Gly Val Ser Asn
385 390 395 400
Asp Lys Asn Pro Gly Asn Ile Phe Ser Ser Ile Ser Leu Phe Ser Val
405 410 415
Leu Ala Gln Gln Thr Met Met Leu Pro Met Ala Leu Ala Thr Gly Ala
420 425 430
Asp Ala Lys Ile Gly Leu Glu Arg Leu Arg Gln Tyr Leu Gln Ser Gly
435 440 445
Asp Ile Glu Lys Glu Tyr Glu Asp His Glu Lys Pro Gly Asp Arg Asp
450 455 460
Val Val Leu Pro Asp Asn Val Ala Val Glu Leu Asn Asn Ala Ser Phe
465 470 475 480
Ile Trp Glu Lys Phe Asp Asp Ala Asp Asp Asn Asp Gly Asn Ser Glu
485 490 495
Lys Thr Lys Glu Val Val Val Thr Ser Lys Ser Ser Leu Thr Asp Ser
500 505 510
Ser His Ile Asp Lys Ser Thr Asp Ser Ala Asp Gly Glu Tyr Ile Lys
515 520 525
Ser Val Phe Glu Gly Phe Asn Asn Ile Asn Leu Thr Ile Lys Lys Gly
530 535 540
Glu Phe Val Ile Ile Thr Gly Pro Ile Gly Ser Gly Lys Ser Ser Leu
545 550 555 560
Leu Val Ala Leu Ala Gly Phe Met Lys Lys Thr Ser Gly Thr Leu Gly
565 570 575
Val Asn Gly Thr Met Leu Leu Cys Gly Gln Pro Trp Val Gln Asn Cys
580 585 590
Thr Val Arg Asp Asn Ile Leu Phe Gly Leu Glu Tyr Asp Glu Ala Arg
595 600 605
Tyr Asp Arg Val Val Glu Val Cys Ala Leu Gly Asp Asp Leu Lys Met
610 615 620
Phe Thr Ala Gly Asp Gln Thr Glu Ile Gly Glu Arg Gly Ile Thr Leu
625 630 635 640
Ser Gly Gly Gln Lys Ala Arg Ile Asn Leu Ala Arg Ala Val Tyr Ala
645 650 655
Asn Lys Asp Ile Ile Leu Leu Asp Asp Ala Leu Ser Ala Val Asp Ala
660 665 670
Arg Val Gly Lys Leu Ile Val Asp Asp Cys Leu Thr Ser Phe Leu Gly
675 680 685
Asp Lys Thr Arg Ile Leu Ala Thr His Gln Leu Ser Leu Ile Glu Ala
690 695 700
Ala Asp Arg Val Ile Tyr Leu Asn Gly Asp Gly Thr Ile His Ile Gly
705 710 715 720
Thr Val Gln Glu Leu Leu Glu Ser Asn Glu Gly Phe Leu Lys Leu Met
725 730 735
Glu Phe Ser Arg Lys Ser Glu Ser Glu Asp Glu Glu Asp Val Glu Ala
740 745 750
Ala Asn Glu Lys Asp Val Ser Leu Gln Lys Ala Val Ser Val Val Gln
755 760 765
Glu Gln Asp Ala His Ala Gly Val Leu Ile Gly Gln Glu Glu Arg Ala
770 775 780
Val Asn Gly Ile Glu Trp Asp Ile Tyr Lys Glu Tyr Leu His Glu Gly
785 790 795 800
Arg Gly Lys Leu Gly Ile Phe Ala Ile Pro Thr Ile Ile Met Leu Leu
805 810 815
Val Leu Asp Val Phe Thr Ser Ile Phe Val Asn Val Trp Leu Ser Phe
820 825 830
Trp Ile Ser His Lys Phe Lys Ala Arg Ser Asp Gly Phe Tyr Ile Gly
835 840 845
Leu Tyr Val Met Phe Val Ile Leu Ser Val Ile Trp Ile Thr Ala Glu
850 855 860
Phe Val Val Met Gly Asn Phe Ser Ser Thr Ala Ala Arg Arg Leu Asn
865 870 875 880
Leu Lys Ala Met Lys Arg Val Leu His Thr Pro Met His Phe Leu Asp
885 890 895
Val Thr Pro Met Gly Arg Ile Leu Asn Arg Phe Thr Lys Asp Thr Asp
900 905 910
Val Leu Asp Asn Glu Ile Gly Glu Gln Ala Arg Met Phe Leu His Pro
915 920 925
Ala Ala Tyr Val Ile Gly Val Leu Ile Leu Cys Ile Ile Asn Ile Pro
930 935 940
Trp Phe Ala Ile Ala Ile Pro Pro Leu Ala Ile Leu Phe Thr Phe Ile
945 950 955 960
Thr Asn Phe Tyr Ile Ala Ser Ser Arg Glu Val Lys Arg Ile Glu Ala
965 970 975
Ile Gln Arg Ser Leu Val Tyr Asn Asn Phe Asn Glu Val Leu Asn Gly
980 985 990
Leu Gln Thr Leu Lys Ala Tyr Asn Ala Thr Ser Arg Phe Met Glu Lys
995 1000 1005
Asn Lys Arg Leu Leu Asn Arg Met Asn Glu Ala Tyr Leu Leu Val
1010 1015 1020
Ile Ala Asn Gln Arg Trp Ile Ser Val Asn Leu Asp Leu Val Ser
1025 1030 1035
Cys Cys Phe Val Phe Leu Ile Ser Met Leu Ser Val Phe Arg Val
1040 1045 1050
Phe Asp Ile Asn Ala Ser Ser Val Gly Leu Val Val Thr Ser Val
1055 1060 1065
Leu Gln Ile Gly Gly Leu Met Ser Leu Ile Met Arg Ala Tyr Thr
1070 1075 1080
Thr Val Glu Asn Glu Met Asn Ser Val Glu Arg Leu Cys His Tyr
1085 1090 1095
Ala Asn Lys Leu Glu Gln Glu Ala Pro Tyr Ile Met Asn Glu Thr
1100 1105 1110
Lys Pro Arg Pro Thr Trp Pro Glu His Gly Ala Ile Glu Phe Lys
1115 1120 1125
His Ala Ser Met Arg Tyr Arg Glu Gly Leu Pro Leu Val Leu Lys
1130 1135 1140
Asp Leu Thr Ile Ser Val Lys Gly Gly Glu Lys Ile Gly Ile Cys
1145 1150 1155
Gly Arg Thr Gly Ala Gly Lys Ser Thr Ile Met Asn Ala Leu Tyr
1160 1165 1170
Arg Leu Thr Glu Leu Ala Glu Gly Ser Ile Thr Ile Asp Gly Val
1175 1180 1185
Glu Ile Ser Gln Leu Gly Leu Tyr Asp Leu Arg Ser Lys Leu Ala
1190 1195 1200
Ile Ile Pro Gln Asp Pro Val Leu Phe Arg Gly Thr Ile Arg Lys
1205 1210 1215
Asn Leu Asp Pro Phe Gly Gln Asn Asp Asp Glu Thr Leu Trp Asp
1220 1225 1230
Ala Leu Arg Arg Ser Gly Leu Val Glu Gly Ser Ile Leu Asn Thr
1235 1240 1245
Ile Lys Ser Gln Ser Lys Asp Asp Pro Asn Phe His Lys Phe His
1250 1255 1260
Leu Asp Gln Thr Val Glu Asp Glu Gly Ala Asn Phe Ser Leu Gly
1265 1270 1275
Glu Arg Gln Leu Ile Ala Leu Ala Arg Ala Leu Val Arg Asn Ser
1280 1285 1290
Lys Ile Leu Ile Leu Asp Glu Ala Thr Ser Ser Val Asp Tyr Glu
1295 1300 1305
Thr Asp Ser Lys Ile Gln Lys Thr Ile Ser Thr Glu Phe Ser His
1310 1315 1320
Cys Thr Ile Leu Cys Ile Ala His Arg Leu Lys Thr Ile Leu Thr
1325 1330 1335
Tyr Asp Arg Ile Leu Val Leu Glu Lys Gly Glu Val Glu Glu Phe
1340 1345 1350
Asp Thr Pro Arg Val Leu Tyr Ser Lys Asn Gly Val Phe Arg Gln
1355 1360 1365
Met Cys Glu Arg Ser Glu Ile Thr Ser Ala Asp Phe Val
1370 1375 1380
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| БИОСИНТЕТИЧЕСКОЕ ПОЛУЧЕНИЕ СТЕВИОЛОВОГО ГЛИКОЗИДА РЕБАУДИОЗИДА D4 ИЗ РЕБАУДИОЗИДА E | 2017 |
|
RU2764803C2 |
| ГИДРОЛИЗ СТЕВИОЛОВЫХ ГЛИКОЗИДОВ С ПОМОЩЬЮ БЕТА-ГЛЮКОЗИДАЗЫ | 2018 |
|
RU2775697C2 |
| РЕКОМБИНАНТНОЕ ПОЛУЧЕНИЕ СТЕВИОЛ-ГЛИКОЗИДОВ | 2014 |
|
RU2741103C2 |
| Ферменты лактазы с улучшенными свойствами при кислом pH | 2019 |
|
RU2814542C2 |
| Ферменты лактазы с улучшенными свойствами | 2018 |
|
RU2806709C2 |
| ФЕРМЕНТЫ ЛАКТАЗЫ С УЛУЧШЕННЫМИ СВОЙСТВАМИ | 2018 |
|
RU2788608C2 |
| Ферменты лактазы с улучшенной активностью при низких температурах | 2018 |
|
RU2800427C2 |
| СПОСОБЫ ПРИГОТОВЛЕНИЯ ИНТЕНСИВНЫХ ПОДСЛАСТИТЕЛЕЙ | 2018 |
|
RU2767792C2 |
| СПОСОБ ПОЛУЧЕНИЯ (-)-АМБРОКСИДА, ПРОДУКТ РЕАКЦИИ, СОДЕРЖАЩИЙ (-)-АМБРОКСИД, И ЕГО ПРИМЕНЕНИЕ | 2016 |
|
RU2727641C2 |
| ФЕРМЕНТЫ И ИХ ПРИМЕНЕНИЕ | 2016 |
|
RU2836734C1 |
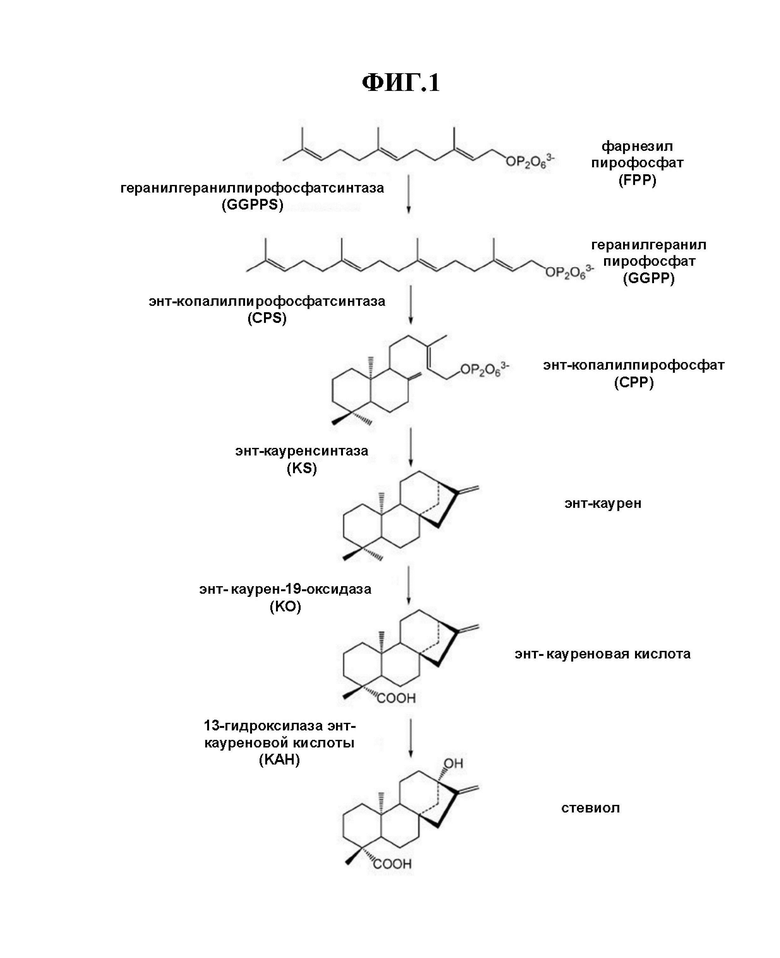
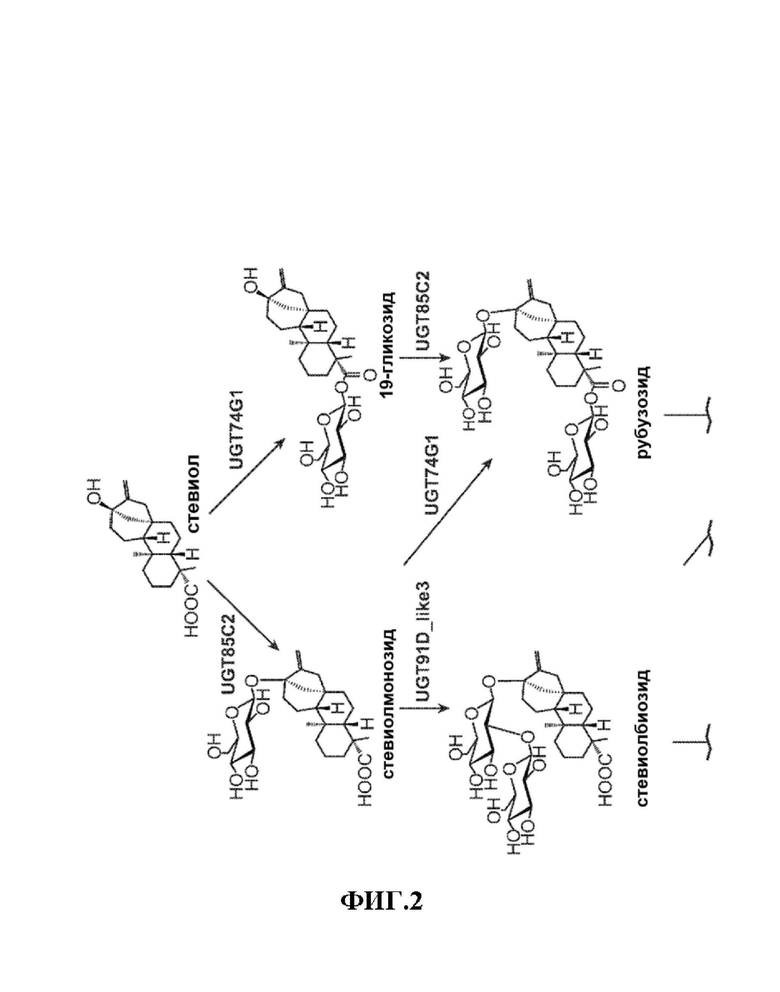
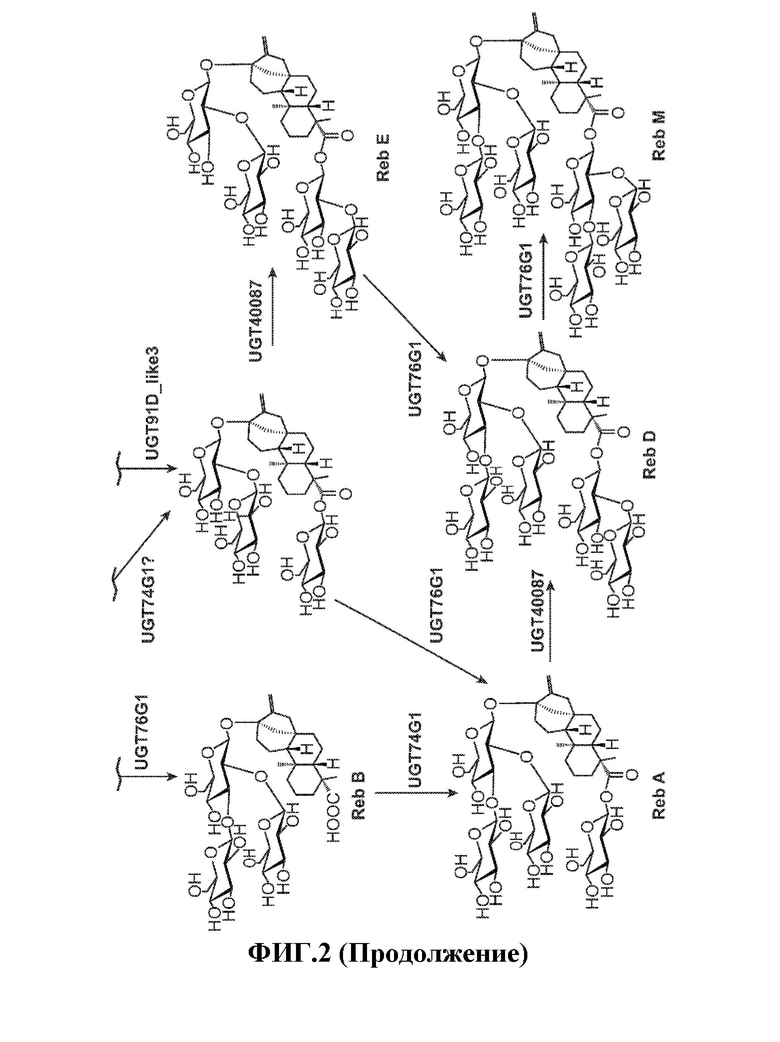

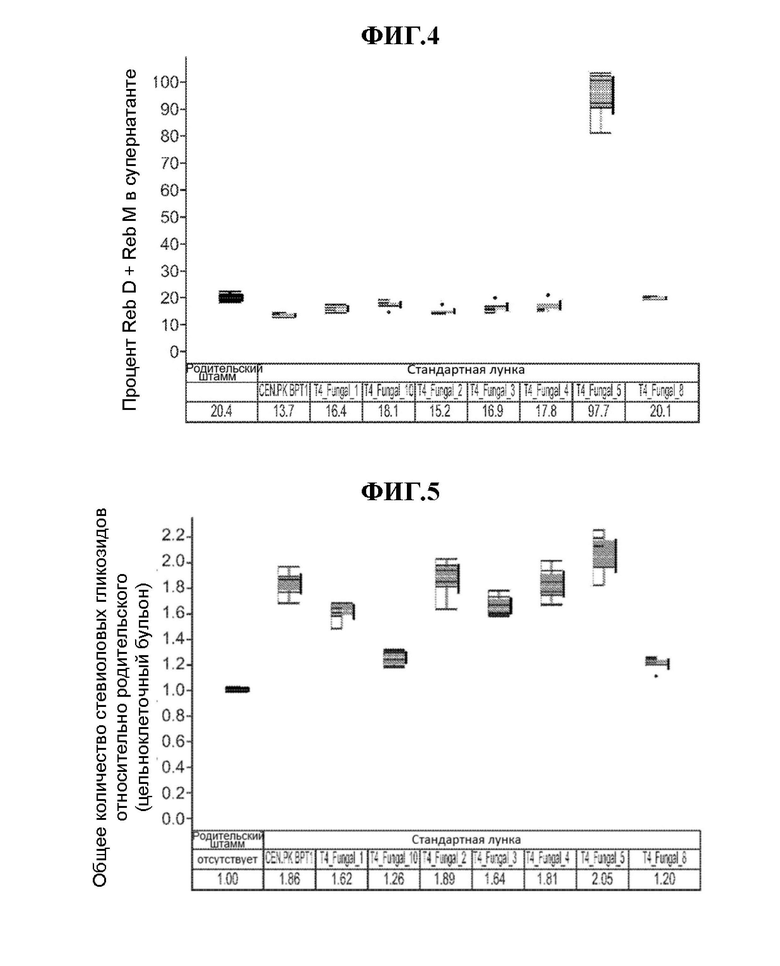
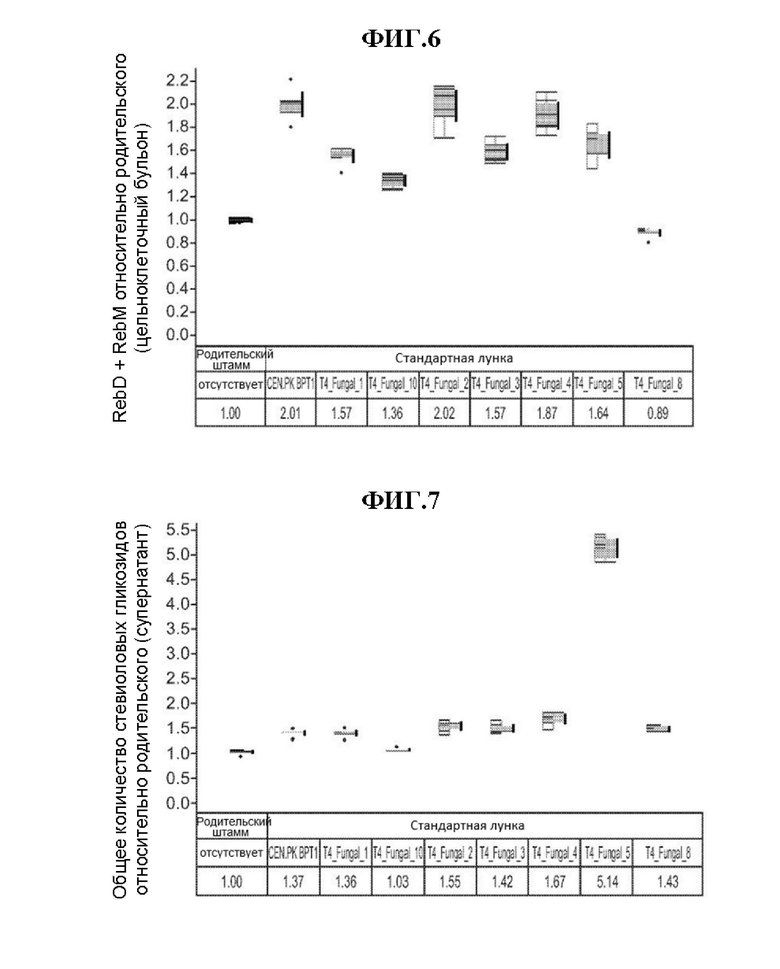
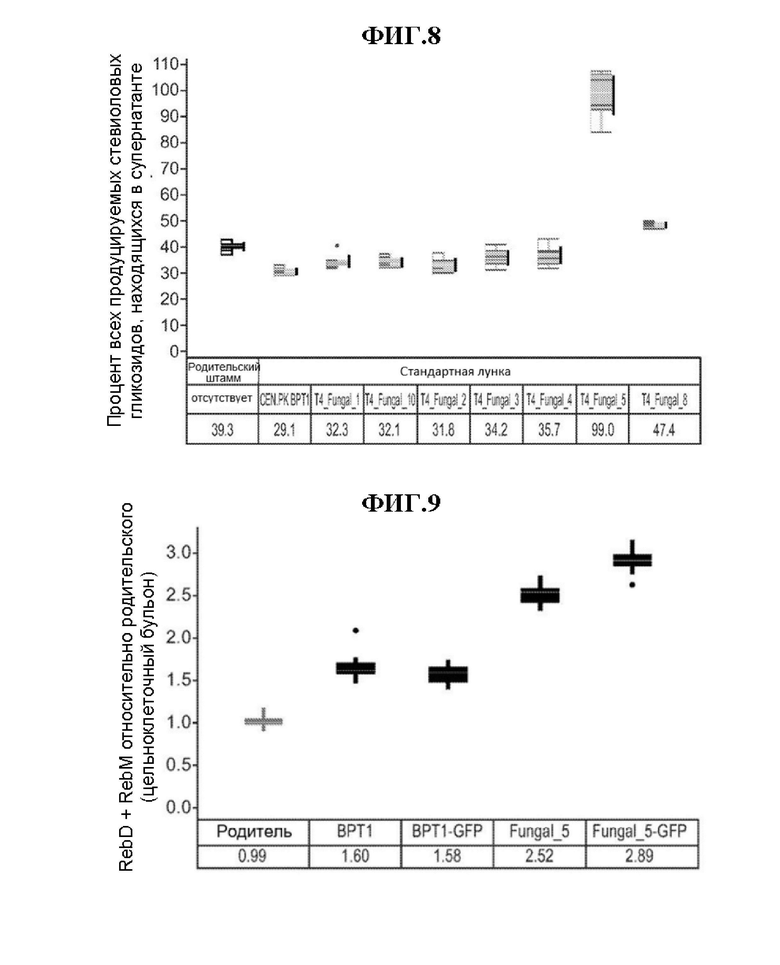

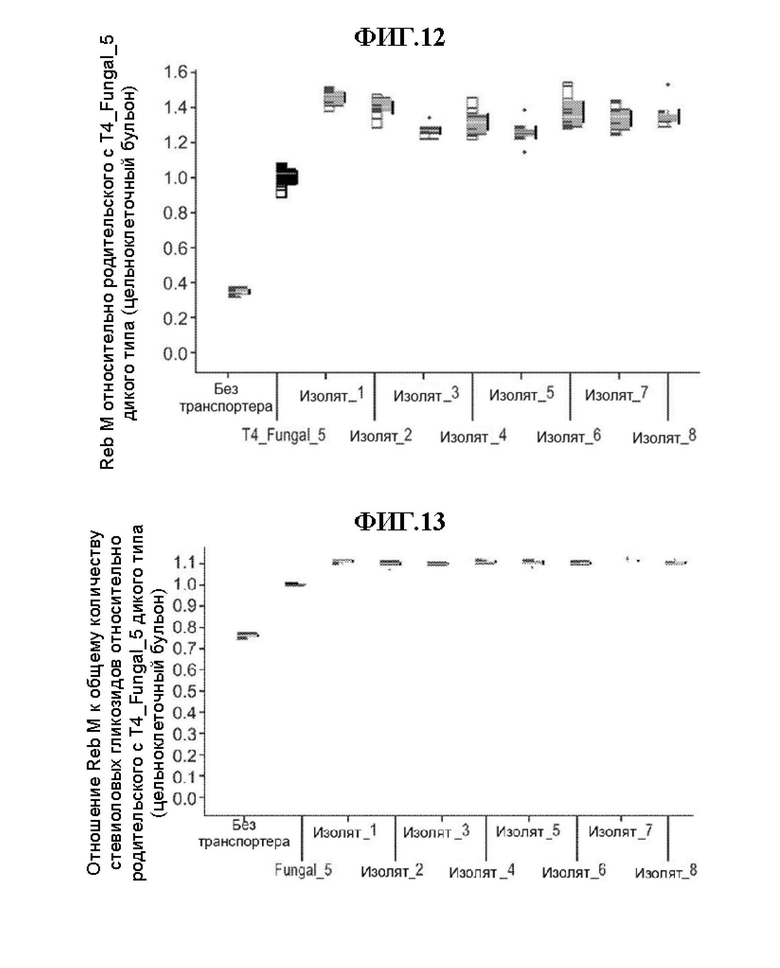
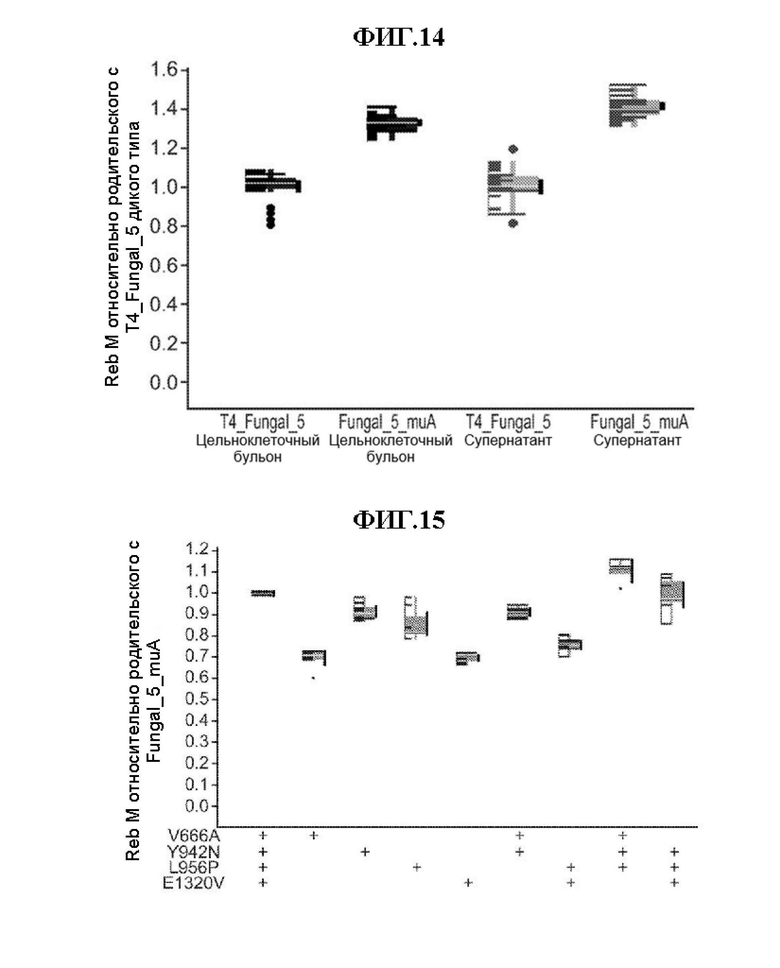
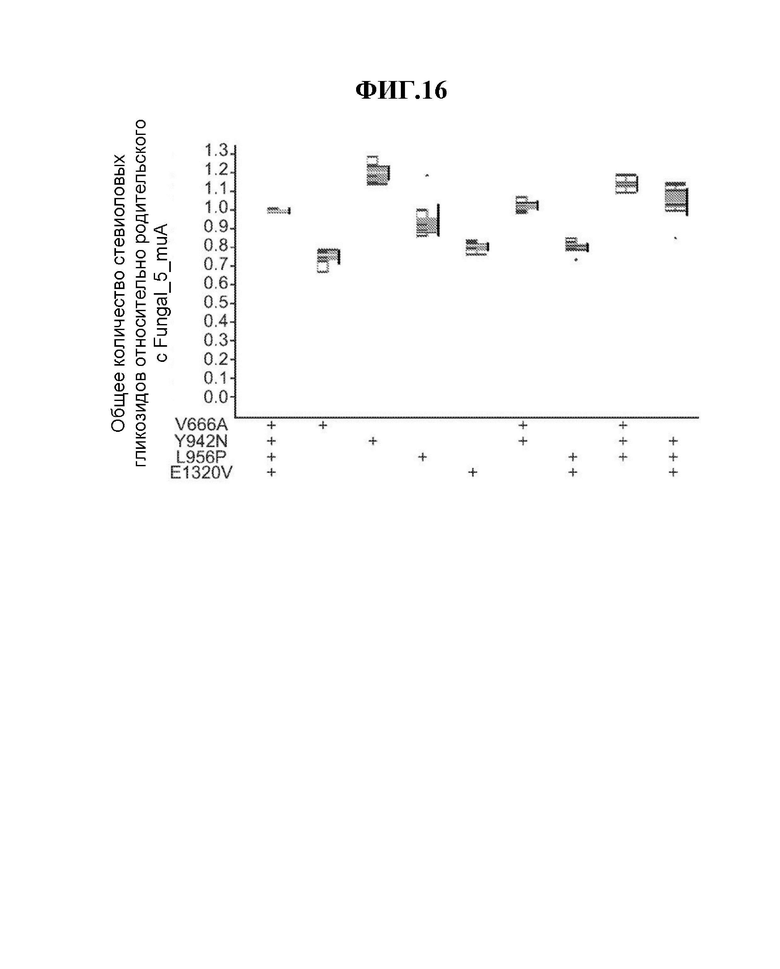
Изобретение относится к биотехнологии. Предложена генетически модифицированная клетка-хозяин, продуцирующая один или несколько стевиоловых гликозидов и содержащая гетерологичную нуклеиновую кислоту, кодирующую ABC-транспортер. Также предложены способ получения стевиола или одного или нескольких стевиоловых гликозидов, способ получения Reb D и получения Reb М, в которых используют указанную клетку-хозяина. Изобретение обеспечивает увеличение продукции общего стевиолгликозида (TSG), Reb D и Reb М. 4 н. и 26 з.п. ф-лы, 16 ил., 6 табл., 15 пр.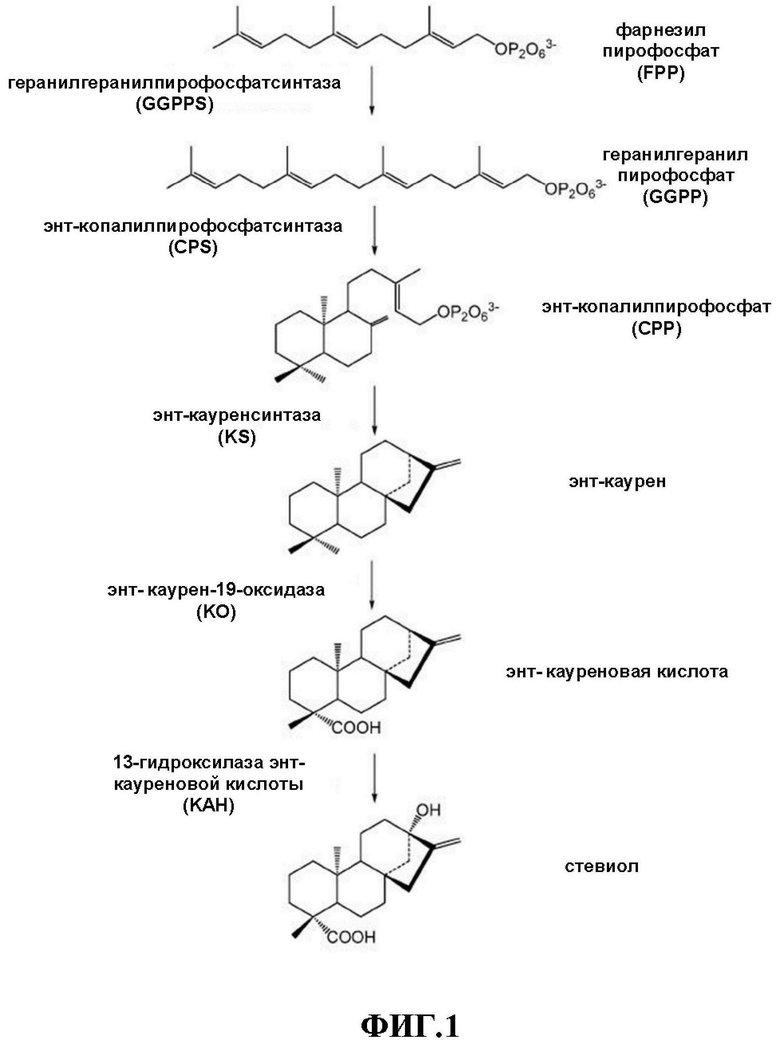
1. Генетически модифицированная клетка-хозяин, продуцирующая один или несколько стевиоловых гликозидов, где генетически модифицированная клетка-хозяин содержит гетерологичную нуклеиновую кислоту, кодирующую ABC-транспортер, и где генетически модифицированная клетка-хозяин с ABC-транспортером продуцирует больше общего гликозида стевиола (TSG) по сравнению с клеткой-хозяином, лишенной ABC-транспортера, при этом ABC-транспортер содержит аминокислотную последовательность, имеющую по меньшей мере 90% идентичности последовательности с аминокислотной последовательностью, выбранной из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7, SEQ ID NO: 8, SEQ ID NO: 28, SEQ ID NO: 29 и SEQ ID NO: 30.
2. Генетически модифицированная клетка-хозяин по п.1, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность, выбранную из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4, SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7 и SEQ ID NO: 8.
3. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, дополнительно содержащая нуклеиновую кислоту, кодирующую геранилгеранилпирофосфатсинтазу (GGPPS), энт-копалилпирофосфатсинтазу (CPS), энт-кауренсинтазу (KS), энт-каурен-19-оксидазу (KO), 13-гидроксилазу энт-кауреновой кислоты (KAH), цитохром p450 редуктазу (CPR) и одну или несколько УДФ-глюкозилтрансфераз (UGT).
4. Генетически модифицированная клетка-хозяин по п.3, отличающаяся тем, что одна или несколько УДФ-глюкозилтрансфераз (UGT) выбраны из группы, состоящей из UGT85C2, UGT74G1, UGT91D_like3, UGT76G1, EUGT11 и UGT40087.
5. Генетически модифицированная клетка-хозяин по п.4, отличающаяся тем, что геранилгеранилпирофосфатсинтаза (GGPPS) содержит аминокислотную последовательность, имеющую по меньшей мере 80% идентичность последовательности с идентификатором SEQ ID NO: 9, энт-копалилпирофосфатсинтаза (CPS) содержит аминокислотную последовательность, имеющую по меньшей мере 80% идентичности последовательности с идентификатором SEQ ID NO:10, энт-кауренсинтаза (KS) включает аминокислотную последовательность, имеющую по меньшей мере 80%-ную идентичность последовательности с идентификатором SEQ ID NO: 11, энт-каурен-19-оксидаза (KO) содержит аминокислотную последовательность, имеющую по меньшей мере 80%-ную идентичность последовательности с идентификатором SEQ ID NO: 12, 13-гидроксилаза энт-кауреновой кислоты (KAH) содержит аминокислотную последовательность, имеющую по меньшей мере 80%-ную идентичность последовательности с идентификатором SEQ ID NO: 13, редуктаза цитохрома p450 (CPR) содержит аминокислотную последовательность, имеющую по меньшей мере 80%-ную идентичность последовательности с идентификатором SEQ ID NO: 14, и одна или несколько УДФ-глюкозилтрансфераз (UGT) содержат аминокислотную последовательность, имеющую по меньшей мере 80% идентичности последовательности аминокислотной последовательности, выбранной из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19.
6. Генетически модифицированная клетка-хозяин по п.5, отличающаяся тем, что геранилгеранилпирофосфатсинтаза (GGPPS) содержит аминокислотную последовательность с идентификатором SEQ ID NO: 9, энт-копалилпирофосфатсинтаза (CPS) содержит аминокислотную последовательность с идентификатором SEQ ID NO: 10, энт-каурен-синтаза (KS) содержит аминокислотную последовательность с идентификатором SEQ ID NO: 11, энт-каурен-19-оксидаза (KO) включает аминокислотную последовательность с идентификатором SEQ ID NO: 12, 13-гидроксилаза энт-кауреновой кислоты (KAH) содержит аминокислотную последовательность с идентификатором SEQ ID NO: 13, редуктаза цитохрома p450 (CPR) содержит аминокислотную последовательность с идентификатором SEQ ID NO: 14, а одна или несколько УДФ-глюкозилтрансфераз (UGT) содержат аминокислотную последовательность, выбранную из группы, состоящей из последовательностей со следующими идентификаторами: SEQ ID NO: 15, SEQ ID NO: 16, SEQ ID NO: 17, SEQ ID NO: 18, SEQ ID NO: 19.
7. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что клетка-хозяин выбрана из бактериальной клетки, грибковой клетки, водорослевой клетки, клетки насекомого и клетки растения.
8. Генетически модифицированная клетка-хозяин по п.7, отличающаяся тем, что клетка-хозяин представляет собой клетку Saccharomyces cerevisiae.
9. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 1.
10. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 2.
11. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 3.
12. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 4.
13. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 5.
14. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 6.
15. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 7.
16. Генетически модифицированная клетка-хозяин по п.15, отличающаяся тем, что ABC-транспортер содержит одну или несколько аминокислотных замен относительно аминокислотной последовательности с идентификатором SEQ ID NO: 7.
17. Генетически модифицированная клетка-хозяин по п.16, отличающаяся тем, что одна или несколько аминокислотных замен выбраны из V666A, Y942N, L956P и E1320V.
18. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 8.
19. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 28.
20. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 29.
21. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что ABC-транспортер содержит аминокислотную последовательность, имеющую последовательность с идентификатором SEQ ID NO: 30.
22. Генетически модифицированная клетка-хозяин по любому из предшествующих пунктов, отличающаяся тем, что один или несколько стевиоловых гликозидов выбраны из группы, состоящей из Reb A, Reb B, Reb D, Reb E и Reb M.
23. Генетически модифицированная клетка-хозяин по п.22, отличающаяся тем, что один или несколько стевиоловых гликозидов содержат Reb M.
24. Способ получения стевиола или одного или нескольких стевиоловых гликозидов, включающий стадии:
(a) культивирование популяции клеток-хозяев по любому из пп.1-23 в среде с источником углерода в условиях, подходящих для получения стевиола или одного или нескольких стевиоловых гликозидов с получением культурального бульона; а также
(b) выделение указанного стевиола или одного или нескольких стевиоловых гликозидов из культуральной жидкости.
25. Способ получения Reb D, включающий стадии:
(a) культивирование популяции клеток-хозяев по любому из пп.1-23 в среде с источником углерода в условиях, подходящих для получения Reb D с получением культурального бульона; а также
(b) выделение указанного соединения Reb D из культурального бульона.
26. Способ получения Reb M, включающий стадии:
(a) культивирование популяции клеток-хозяев по любому из пп.1-23 в среде с источником углерода в условиях, подходящих для получения Reb M с получением культурального бульона; а также
(b) выделение указанного соединения Reb M из культурального бульона.
27. Генетически модифицированная клетка-хозяин по п.1 или 2, отличающаяся тем, что по меньшей мере 50% одного или нескольких стевиоловых гликозидов накапливаются в просвете органеллы.
28. Генетически модифицированная клетка-хозяин по п.1 или 2, отличающаяся тем, что по меньшей мере 50% одного или нескольких стевиоловых гликозидов накапливаются внеклеточно.
29. Генетически модифицированная клетка-хозяин по любому из пп.1-23, дополнительно содержащая УДФ-глюкозилтрансферазу (UGT), имеющую аминокислотную последовательность, имеющую по меньшей мере 80% идентичности последовательности с аминокислотной последовательностью с идентификатором SEQ ID NO: 18.
30. Генетически модифицированная клетка-хозяин по любому из пп.1-23, дополнительно содержащая УДФ-глюкозилтрансферазу (UGT), имеющую аминокислотную последовательность с идентификатором SEQ ID NO: 18.
| WO 2018211032 A1, 22.11.2018 | |||
| WO 2016055578 А1, 04.10.2016 | |||
| US 10017804 В2, 10.07.2018 | |||
| WO 2018031955 А2, 15.02.2018 | |||
| WO 2017025362 A1, 16.02.2017 | |||
| WO2008034648, 27.03.2008 | |||
| RU 2016120635 A, 06.12.2017. |
Авторы
Даты
2023-05-12—Публикация
2020-01-23—Подача