Область техники
[0001] Настоящее изобретение относится к новому активатору транскрипции, содержащему VP64 и участок активации транскрипции из R-трансактиватора (RTA). Кроме того, оно относится к комплексу модуля, узнающего последовательность нуклеиновой кислоты, специфически связывающегося с нуклеотидной последовательностью-мишенью в двухцепочечной ДНК, и вышеупомянутого активатора транскрипции.
Уровень техники
[0002] В последние годы, редактирование генома привлекает внимание в качестве способа модификации целевого гена и геномной области в различных видах. Например, опубликованы способ проведения рекомбинации в целевом локусе гена в ДНК в клетке растения или в клетке насекомого в качестве хозяина, с использованием нуклеазы с цинковыми пальцами (ZFN), в которой соединены связывающий ДНК домен с цинковыми пальцами и домен неспецифического расщепления ДНК (Патентный документ 1), и способ расщепления или модификации гена-мишени в конкретной нуклеотидной последовательности или в соседнем с ней участке с использованием TALEN, в которой соединены подобный активатору транскрипции (TAL) эффектор со связывающим ДНК модулем, который имеет патогенная для растений бактерия Xanthomonas, и ДНК-эндонуклеаза (Патентный документ 2). Кроме того, нуклеазу Cas9, полученную из Streptococcus pyogenes, широко используют в качестве мощного инструмента редактирования генома у эукариот, имеющих путь репарации двухцепочечных разрывов ДНК (DSB) (например, Патентный документ 3, Непатентные документы 1,2).
[0003] Способы сайт-специфической регуляции транскрипции также разработаны посредством использования способов редактирования генома. Например, опубликован способ активации или супрессии целевого гена, включающий связывание ZF или TALE, или белка или комплекса, в котором домен активации транскрипции или домен супрессии транскрипции (как правило, VP64 используют для активации, и KRAB используют для супрессии) слит с системой Cas9 (dCas9), лишенной способности расщеплять обе цепи двухцепочечной ДНК, с промоторной или энхансерной последовательностью целевого гена (например, Непатентный документ 3).
[0004] Однако, активация транскрипции с использованием VP64 имеет те проблемы, что достаточной способности к активации транскрипции не достигают посредством простого использованием одной молекулы VP64, и является необходимым связывание множества комплексов TALE-VP64 и dCas9-VP64/онРНК с одним геном (например, Непатентный документ 3). Для преодоления этой проблемы, например, опубликован способ с использованием активатора транскрипции, в котором другие факторы активации транскрипции (p65 и RTA) связаны с VP64 (например, Непатентный документ 4).
Список литературы
Патентные документы
[0005] PTL 1: WO 03/087341 A2
PTL 2: WO 2011/072246 A2
PTL 3: WO 2013/176772 Al
Непатентные документы
[0006] NPL 1: Mali P, et al, Science 339: 823-827 (2013)
NPL 2: Cong L, et al., Science 339: 819-823 (2013)
NPL 3: Hu J, et al, Nucleic Acids Res, 42: 4375-4390 (2014)
NPL 4: Chavez A, et al., Nat Methods, 12: 326-328 (2015)
Сущность изобретения
Техническая проблема
[0007] Однако, когда p65 и RTA связаны с VP64, их общая молекулярная масса становится большой. Таким образом, возникает та проблема, что нуклеиновая кислота, кодирующая комплекс системы CRISPR/Cas9 и активатор транскрипции, ограничена в отношении размера, и не может быть собрана в аденоассоциированном вирусном (AAV) векторе как объединенная нуклеиновая кислота. Соответственно, одной из проблем с опосредованной AAV доставкой является предоставление активатора транскрипции размером, который поддается сборке в векторе AAV и может в достаточной степени проявлять способность к активации транскрипции.
Решение проблемы
[0008] Авторы настоящего изобретения обратили внимание на множество белков, как известно, имеющих способность к активации транскрипции, и выдвинули изобретательскую идею, что активаторы, способные решать вышеупомянутую проблему, можно получать посредством подходящей комбинации таких белков. На основании этой идеи, они провели интенсивные исследования и обнаружили, что как уменьшения размера белка, так и все еще сохранения достаточной способности к активации транскрипции, можно достигать посредством объединения VP64 и RTA. На основании этого обнаружения, они провели дополнительные исследования и завершили настоящее изобретение.
[0009] Таким образом, настоящее изобретение относится к следующему.
[1] Активатор транскрипции, состоящий не более чем 200 аминокислот и содержащий VP64 и участок активации транскрипции из RTA.
[2] Активатор транскрипции по [1], где вышеупомянутый VP64 содержит
(1) аминокислотную последовательность, показанную в SEQ ID NO: 1,
(2) аминокислотную последовательность из (1), где 1 или несколько аминокислот делетированы, замещены и/или добавлены, или
(3) аминокислотную последовательность, на 90% или более идентичную аминокислотной последовательности из (1).
[3] Активатор транскрипции по [1] или [2], где вышеупомянутый участок активации транскрипции из RTA содержит
(4) последовательность, показанную в SEQ ID NO: 2,
(5) последовательность, показанную в SEQ ID NO: 3,
(6) аминокислотную последовательность по (4) или (5) где 1 или несколько аминокислоты делетированы, замещены и/или добавлены, или
(7) аминокислотную последовательность, на 90% или более идентичную аминокислотной последовательности из (4) или (5).
[4] Комплекс, содержащий модуль, узнающий последовательность нуклеиновой кислоты, специфически связывающийся с нуклеотидной последовательностью-мишенью в двухцепочечной ДНК, и активатор транскрипции по любому из [1] - [3], связанные друг с другом, и активирующий транскрипцию целевого гена в ДНК.
[5] Комплекс по [4], где вышеупомянутый модуль, узнающий последовательность нуклеиновой кислоты, содержит эффекторный белок CRISPR, лишенный способности расщеплять по меньшей мере одну цепь двухцепочечной ДНК.
[6] Комплекс по [5], где вышеупомянутый эффекторный белок CRISPR лишен способности расщеплять обе цепи двухцепочечной ДНК.
[7] Комплекс по [5] или [6], где эффекторный белок CRISPR происходит из Staphylococcus aureus или Campylobacter jejuni.
[8] Нуклеиновая кислота, кодирующая активатор транскрипции по любому из [1] - [3].
[9] Нуклеиновая кислота, кодирующая комплекс по любому из [4] - [7].
[10] Вектор, содержащий нуклеиновую кислоту по [8] или [9].
[11] Вектор по [10], где вышеупомянутый вектор представляет собой аденоассоциированный вирусный вектор.
[12] Способ активации транскрипции целевого гена в клетке, включающий
стадию введения комплекса по любому из [4] - [7], нуклеиновой кислоты по [8] или [9] или вектора по [10] или [11] в клетку.
[13] Способ по [12], где клетка представляет собой клетку млекопитающего.
[14] Способ по [13], где вышеупомянутое млекопитающее представляет собой человека.
Обеспечивающие преимущество эффекты изобретения
[0010] В соответствии с настоящим изобретением, представлен новый активатор транскрипции, имеющий размер, который поддается сборке в векторе AAV и может в достаточной степени проявлять способность к активации транскрипции. Кроме того, представлены комплекс модуля, узнающего последовательность нуклеиновой кислоты, специфически связывающегося с нуклеотидной последовательностью-мишенью в двухцепочечной ДНК, и вышеупомянутого активатора транскрипции, и способ активации транскрипции целевого гена в клетке с использованием этого комплекса.
Краткое описание чертежей
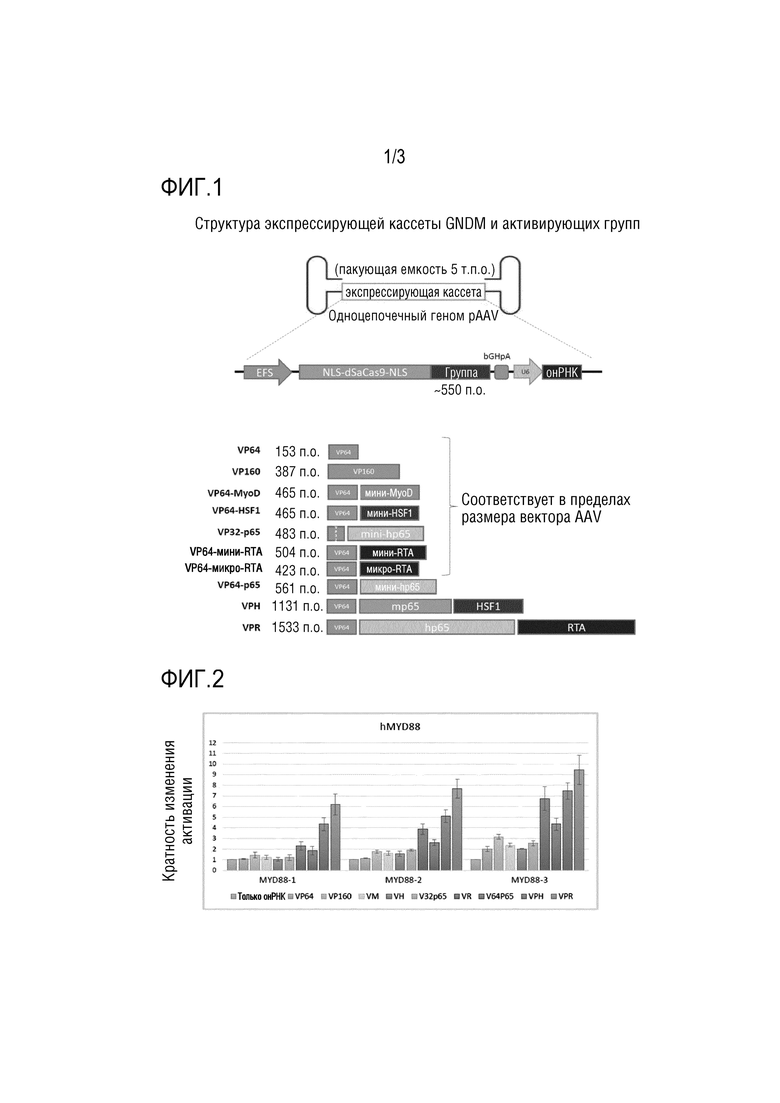
[0011] [фиг. l] На фигуре 1 показана структура вектора AAV и десяти активирующих групп, когда dSaCas9 используют в качестве эффекторного белка CRISPR. Количество оснований на фигуре указано по длине, включающей стоп-кодон.
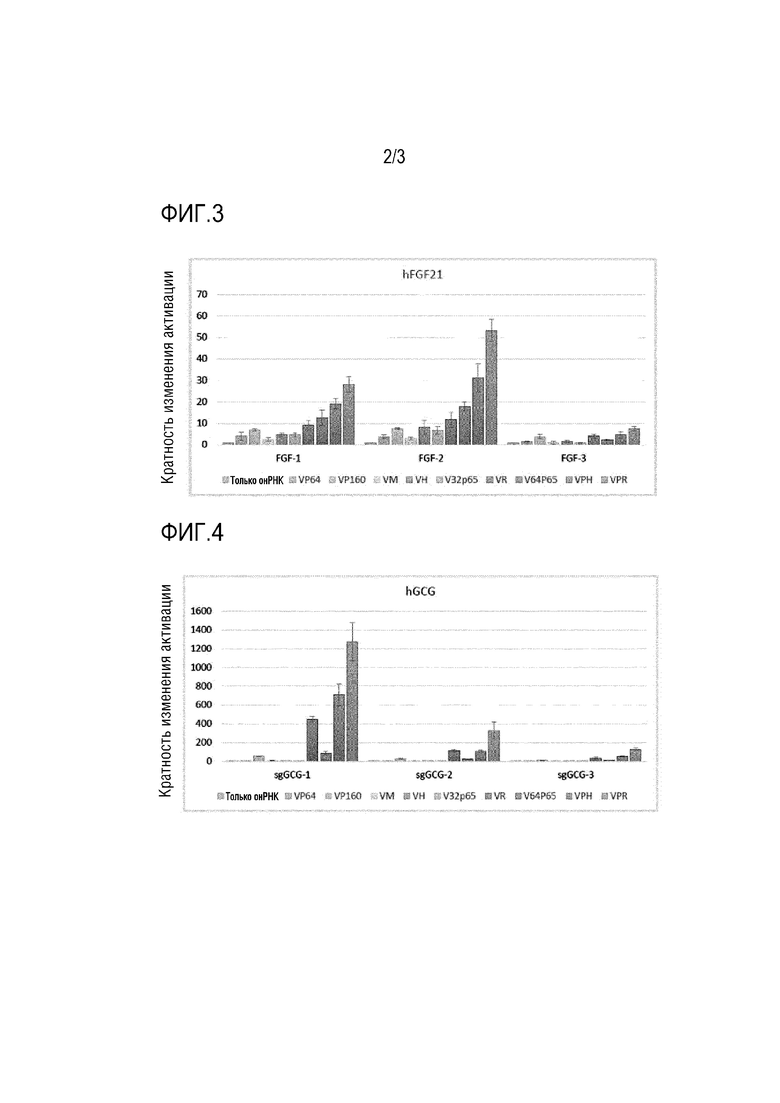
[фиг. 2] На фигуре 2 показана активация гена MYD88 посредством девяти активирующих групп. Для соответствующих нРНК, каждая столбчатая диаграмма показывает результаты для только онРНК, VP64, VP160, VM (VP64-MyoD), VH (VP64-HSF1), V32p65 (VP32-p65), VR (VP64-мини-RTA), V64P65 (VP64-p65), VPH и VPR, в этом порядке слева направо.
[Таблица 1]
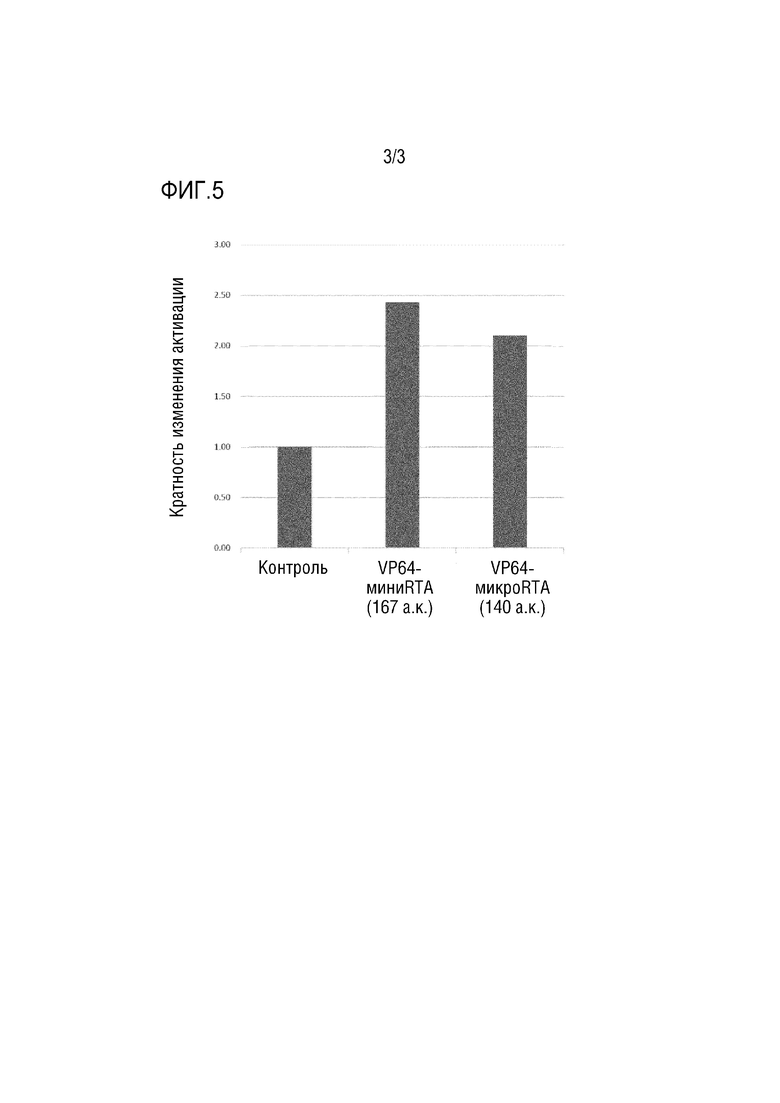
[фиг.3] На фигуре 3 показана активация гена FGF21 посредством девяти активирующих групп. Для соответствующих нРНК, каждая столбчатая диаграмма показывает результаты для только онРНК, VP64, VP160, VM (VP64-MyoD), VH (VP64-HSF1), V32p65 (VP32-p65), VR (VP64- мини-RTA), V64P65 (VP64-p65), VPH и VPR, в этом порядке слева направо.
[Таблица 2]
[фиг.4] На фигуре 4 показана активация гена GCG посредством девяти активирующих групп. Для соответствующих нРНК, каждая столбчатая диаграмма показывает результаты для только онРНК, VP64, VP160, VM (VP64-MyoD), VH (VP64-HSF1), V32p65 (VP32-p65), VR (VP64-мини-RTA), V64P65 (VP64-p65), VPH и VPR, в этом порядке слева направо.
[Таблица 3]
[фиг.5] На фигуре 5 показана активация гена MyD88 посредством VP64-мини-RTA и VP64-микро-RTA.
Описание вариантов осуществления
[0012] В рамках изобретения, неконкретизированные и конкретизированные формы единственного числа предназначены для включения форм как единственного числа, так и множественного числа, если в выражении явно не указано иное с использованием таких слов, как «только» «единственный» и/или «один». Кроме того, понятно, что термины «содержит», «содержащий», «включает» и/или «включающий», при использовании в настоящем описании, обозначают присутствие указанных признаков, стадий, операций, элементов, идей и/или компонентов, но сами по себе не препятствуют присутствию или добавлению одного или нескольких других их признаков, стадий, операций, элементов, компонентов, идей и/или групп.
[0013] Настоящее изобретение относится к новому активатору транскрипции, содержащему VP64 и участок активации транскрипции из R-трансактиватора (RTA) вируса Эпштейна-Барр (иногда в настоящем описании обозначенному как «активатор по настоящему изобретению»). Транскрипцию целевого гена можно активировать посредством активатора транскрипции по настоящему изобретению.
[0014] По настоящему изобретению, VP64 обозначает пептид, состоящий из 4 повторов в тандеме домена, состоящего из 437-го-447-го аминокислотных остатков из происходящего из вируса простого герпеса VP16 (DALDDFDLDML; SEQ ID NO: 21) с пептидным линкером, состоящим из глицина и серина (GS)
([DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]; SEQ ID NO: 1) (Beerli RR, et al, Proc Natl Acad Sci USA. 95(25): 14628-33 (1998)) или его вариант, имеющий способность к активности транскрипции. Примеры такого варианта включают аминокислотную последовательность, показанную в SEQ ID NO: 1, где 1 или несколько (например, 2, 3, 4, 5 или более) аминокислот делетированы, замещены и/или добавлены. Их конкретные примеры включают, но без ограничения, вариант, в котором линкерная часть является замещенным другим линкером (например, пептидным линкером, состоящим из G, S, GG, SG, GGG, GSG, GSGS (SEQ ID NO: 22), GSSG (SEQ ID NO: 23), GGGGS (SEQ ID NO: 24), GGGAR (SEQ ID NO: 25), GSGSGS (SEQ ID NO: 26) или SGQGGGGSG (SEQ ID NO: 27) и т.п.). Альтернативно, как вышеупомянутый вариант, можно упомянуть пептид, состоящий из аминокислотной последовательности, не менее, чем на 90% (например, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или выше) идентичной аминокислотной последовательности, показанной в SEQ ID NO: 1. Кроме того, пептид, состоящий из 10 повторов в тандеме вышеупомянутого домена (DALDDFDLDML; SEQ ID NO: 21)
([DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]-GS-[DALDDFDLDML]; SEQ ID NO:44), называют VP 160.
[0015] RTA представляет собой белок, состоящий из 605 аминокислотных остатков и имеющий способность к активации транскрипции (номер доступа в GenBank: CEQ33017) (SEQ ID NO: 4), и известно, что его C-концевой домен является важным для активации транскрипции (Hardwick JM, J Virol, 66(9):5500-8, 1992). В качестве вышеупомянутого домена, можно конкретно упомянуть область, состоящую из последовательности 493-й-605-й аминокислот RTA (SEQ ID NO: 2). Среди прочего, известно, что область, состоящая из последовательности 520-й-605-й аминокислот (SEQ ID NO: 3), является важной. Таким образом, RTA, содержащийся в активаторе по настоящему изобретению, предпочтительно, представляет собой участок активации транскрипции, содержащий аминокислотную последовательность, показанную в SEQ ID NO: 2 или SEQ ID NO: 3, или ее вариант, имеющий способность к активации транскрипции. Примеры такого варианта включают аминокислотную последовательность, показанную в SEQ ID NO: 2 или 3, где 1 или несколько (например, 2, 3, 4, 5 или более) аминокислот делетированы, замещены и/или добавлены. Конкретно, поскольку известно, что 564-й остаток лейцина, 566-й остаток лейцина, 570-й остаток лейцина, 578-й остаток лейцина, 581-й остаток фенилаланина и 582-й остаток лейцина в RTA являются важными для способности к активации транскрипции, вариант, в котором аминокислотные остатки, отличные от этих аминокислотных остатков делетированы, замещены и т.п., можно упомянуть, хотя и без ограничения этими модификациями. Альтернативно, в качестве вышеупомянутого варианта, можно упомянуть пептид, состоящий из аминокислотной последовательности, не менее, чем на 90% (например, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более), идентичной аминокислотной последовательности, показанной в SEQ ID NO: 2 или 3. В настоящем описании, пептид, состоящий из последовательности, показанной в SEQ ID NO: 2, иногда обозначен как «мини-RTA», а состоящий из последовательности, показанной в SEQ ID NO: 3, иногда обозначен как «микро-RTA».
[0016] Активатор по настоящему изобретению содержит VP64 и участок активации транскрипции из RTA. VP64 и RTA могут являться связанными посредством линкера (например, вышеупомянутого пептидного линкера) или непосредственно связанными без линкера. VP64 и участок активации транскрипции из RTA могут являться аранжированными в этом порядке от N-конца до C-конца или могут являться аранжированными в обратном порядке. Конкретные примеры активатора по настоящему изобретению включают аминокислотную последовательность, показанную в SEQ ID NO: 6 или 8, аминокислотную последовательность, показанную в SEQ ID NO: 6 или 8, где 1 или несколько (например, 2, 3, 4, 5 или более) аминокислот делетированы, замещены и/или добавлены, и активатор, содержащий аминокислотную последовательность, не менее, чем на 90% (например, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или более) идентичную аминокислотной последовательности, показанной в SEQ ID NO: 6 или 8.
[0017] Идентичность аминокислотной последовательности можно рассчитать с использованием алгоритма расчета NCBI BLAST (Национальный центр биотехнологической информации, Базовый инструментарий поиска локальных блоков) (https://blast.ncbi.nlm.nih.gov/Blast.cgi) и в следующих условиях (ожидание=10; пропуски разрешены; матрица=BLOSUM62; фильтрация=ВЫКЛ.). Понятно, что для определения идентичности последовательность по изобретению на протяжении всей ее длины сравнивают с другой последовательностью. Иными словами, идентичность по изобретению исключает сравнение коротких фрагментов (например, 1-3 аминокислот) последовательности по изобретению с другой последовательности, или наоборот.
[0018] Активатор по настоящему изобретению не является конкретно ограниченным, при условии, что он может активировать транскрипцию целевого гена. Для уменьшения размера, он, предпочтительно, состоит из не более, чем 200 (например, 200, 190, 180, 170, 169, 168, 167 или более) аминокислот и предпочтительно, не менее, чем 110 (например, 110, 120, 130, 135, 136, 137, 138, 139, 140 или менее) аминокислот. В предварительном варианте осуществления, используют активатор, состоящий из приблизительно 140 или приблизительно 167 аминокислот.
[0019] В другом варианте осуществления, настоящее изобретение относится к комплексу, в котором модуль, узнающий последовательность нуклеиновой кислоты, и активатор по настоящему изобретению связаны (далее в настоящем описании обозначенному как «комплекс по настоящему изобретению»).
[0020] По настоящему изобретению, «модуль, узнающий последовательность нуклеиновой кислоты», обозначает молекулу или комплекс молекул, имеющие способность специфически узнавать и связывать конкретную нуклеотидную последовательность (т.е., нуклеотидную последовательность-мишень) в цепи ДНК. Связывание модуля, узнающего последовательность нуклеиновой кислоты, с нуклеотидной последовательностью-мишенью позволяет активатору по настоящему изобретению, связанному с модулем, специфически действовать на участок-мишень двухцепочечной ДНК.
[0021] Комплекс по настоящему изобретению включает не только комплекс, состоящий из множества молекул, но также комплекс, имеющий модуль, узнающий последовательность нуклеиновой кислоты, и активатор по настоящему изобретению в одной молекуле, подобной слитому белку.
[0022] Нуклеотидная последовательность-мишень в двухцепочечной ДНК, подлежащей узнаванию модулем, узнающем последовательность нуклеиновой кислоты, в комплексе по настоящему изобретению, не является конкретно ограниченной, при условии, что модуль специфически связывается с ней, и может представлять собой любую последовательность в двухцепочечной ДНК. Длина нуклеотидной последовательности-мишени должна только являться достаточной для специфического связывания модуля, узнающего последовательность нуклеиновой кислоты. Например, при нацеливании на геномную ДНК млекопитающего, последовательность составляет, в соответствии с размером генома, предпочтительно, не менее, чем 12 нуклеотидов (например, 12 нуклеотидов, 15 нуклеотидов, 18 нуклеотидов, 19 нуклеотидов, 20 нуклеотидов или более) и не более чем 25 нуклеотидов (например, 25 нуклеотидов, 24 нуклеотидов, 23 нуклеотидов, 22 нуклеотидов или менее).
[0023] Примеры модуля, узнающего последовательность нуклеиновой кислоты, из комплекса по настоящему изобретению включают, но без ограничения, систему CRISPR-GNDM, в которой эффекторный белок CRISPR лишен способности расщеплять по меньшей мере одну цепь (предпочтительно, обе цепи) двухцепочечной ДНК, мотив с цинковыми пальцами, эффектор TAL, мотив PPR и т.п., так же как фрагмент, содержащий связывающий ДНК домен белка, способного к специфическому связыванию с ДНК, такого как фермент рестрикции, фактор транскрипции, РНК-полимераза и т.п. Предпочтительными являются система CRISPR-GNDM, мотив с цинковыми пальцами, эффектор TAL, мотив PPR и т.п., из которых система CRISPR-GNDM, в которой эффекторный белок CRISPR лишен способности расщеплять обе цепи двухцепочечной ДНК, является особенно предпочтительной.
[0024] Мотив с цинковыми пальцами составлен посредством связывания 3-6 различных звеньев цинковых пальцев типа Cys2His2 (1 палец узнает приблизительно 3 основания), и может узнавать нуклеотидную последовательность-мишень из 9-18 оснований. Мотив с цинковыми пальцами можно получать известным способом, таким как способ модульной сборки (Nat Biotechnol (2002) 20: 135-141), способ OPEN (Mol Cell (2008) 31: 294-301), способ CoDA (Nat Methods (2011) 8: 67-69), одногибридный способ в Escherichia coli (Nat Biotechnol (2008) 26:695-701) и т.п. На вышеупомянутый Патентный документ 1 можно ссылаться относительно подробностей получения мотива с цинковыми пальцами.
[0025] Эффектор TAL имеет модульную структуру повторов с приблизительно 34 аминокислотами в качестве звена, и с 12-м и 13-м аминокислотными остатками (называемыми RVD) из одного модуля, определяющими стабильность связывания и специфичность основания. Поскольку каждый модуль является в высокой степени независимым, эффектор TAL, специфический для нуклеотидной последовательности-мишени, можно получать посредством прямого присоединения модуля. Для эффектора TAL, разработаны способ получения с использованием открытых ресурсов (способ REAL (Curr Protoc Mol Biol (2012) Chapter 12: Unit 12.15), способ FLASH (Nat Biotechnol (2012) 30: 460-465) и способ Golden Gate (Nucleic Acids Res (2011) 39: e82) и т.д.), и эффектор TAL для нуклеотидной последовательности-мишени можно разрабатывать сравнительно удобно. На вышеупомянутый Патентный документ 2 можно ссылаться относительно подробностей получения эффектора TAL.
[0026] Мотив PPR составляют таким образом, что конкретную нуклеотидную последовательность узнают посредством добавления мотивов PPR, каждый из которых состоит из 35 аминокислот и узнает одно основание нуклеиновой кислоты, и узнает основание-мишень только посредством 1, 4 и ii(-2) аминокислот из каждого мотива. Состав мотива не имеет зависимости, и является свободным от влияния мотивов с обеих сторон. Таким образом, подобно эффектору TAL, белок PPR, специфический для нуклеотидной последовательности-мишени, можно получать посредством простого присоединения мотивов PPR. На WO 2011/111829 Al можно ссылаться относительно подробностей получения мотива PPR.
[0027] При использовании фрагмента из фермента рестрикции, фактора транскрипции, РНК-полимеразы и т.п., поскольку связывающие ДНК домены этих белков хорошо известны, фрагмент, содержащий этот домен и свободный от способности к расщеплению двухцепочечной ДНК, можно легко разрабатывать и конструировать.
[0028] Применительно к мотиву с цинковыми пальцами, получение множества фактически работоспособных мотивов с цинковыми пальцами не является простым, поскольку эффективность продукции цинкового пальца, который специфически связывается с нуклеотидной последовательностью-мишенью, не является высокой, и выбор цинкового пальца, имеющего высокую специфичность связывания, является сложным. В то время как эффектор TAL и мотив PPR имеют высокую степень свободы узнавания последовательности-мишени нуклеиновой кислоты, по сравнению с мотивом с цинковыми пальцами, остается проблема эффективности, поскольку большой белок необходимо разрабатывать и конструировать каждый раз, в соответствии с нуклеотидной последовательностью-мишенью. В отличие от этого, поскольку система CRISPR-GNDM узнает целевую последовательность двухцепочечной ДНК посредством направляющего нуклеотида, комплементарного нуклеотидной последовательности-мишени, на любую последовательность можно нацеливаться посредством простого синтеза олигонуклеотида, способного к специфическому формированию гибрида с нуклеотидной последовательностью-мишенью. Таким образом, в более предпочтительном варианте осуществления настоящего изобретения, систему CRISPR-GNDM используют в качестве модуля, узнающего последовательность нуклеиновой кислоты.
[0029] При использовании системы CRISPR-GNDM по настоящему изобретению, транскрипцию целевого гена можно достаточно активировать посредством привлечения мутантного эффекторного белка CRISPR, лишенного способности расщеплять по меньшей мере одну цепь (предпочтительно обе цепи) двухцепочечной ДНК (далее в настоящем описании обозначенного также как «эффекторный белок CRISPR»). Область регуляции транскрипции целевого гена может представлять собой любую область гена, при условии, что транскрипция гена активируется посредством привлечения эффекторного белка CRISPR и связанного с ним активатора по настоящему изобретению. Примеры такой области включают промоторную область и энхансерную область, интрон, экзон и т.п. целевого гена.
[0030] В настоящем описании, «система CRISPR-GNDM» обозначает систему, содержащую (a) эффекторный белок CRISPR класса 2 (например, dCas9 или dCpfl) или комплекс указанного эффекторного белка CRISPR и активатора по настоящему изобретению, и (b) направляющий нуклеотид (нН), который является комплементарным последовательности области регуляции транскрипции гена-мишени, что обеспечивает привлечение эффекторного белка CRISPR и связанного с ним регулятора транскрипции к области регуляции транскрипции гена-мишени. С использованием вышеупомянутой системы, активация транскрипции гена становится возможной посредством активатора по настоящему изобретению, связанного с эффекторным белком CRISPR.
[0031] «Эффекторный белок CRISPR» для использования по настоящему изобретению не является конкретно ограниченным, при условии, что он формирует комплекс с нН, узнает и связывает нуклеотидную последовательность-мишень в целевом гене и соседний с ней смежный с протоспейсером мотив (РАМ). Предпочтительной является Cas9 или Cpf1, или ее вариант. Примеры Cas9 включают, но без ограничения, происходящую из Streptococcus pyogene Cas9 (SpCas9; последовательность РАМ NGG (N представляет собой A, G, T или C, далее в настоящем описании так же), происходящую из Streptococcus thermophilus Cas9 (StCas9; последовательность РАМ NNAGAAW), происходящую из Neisseria meningitidis Cas9 (NmCas9; последовательность РАМ NNNNGATT), происходящую из Staphylococcus aureus Cas9 (SaCas9; последовательность РАМ: NNGRRT), происходящую из Campylobacter jejuni Cas9 (CjCas9; последовательность РАМ: NNNVRYM (V представляет собой A, G или C; R представляет собой A или G; Y представляет собой T или С; M представляет собой A или Q). С точки зрения размера, Cas9, предпочтительно, представляет собой SaCas9 или CjCas9, или ее вариант. Примеры Cpf1 включают, но без ограничения, происходящую из Francisella novicida Cpf1 (FnCpfl; последовательность РАМ NTT), происходящую из видов Acidaminococcus Cpfl (AsCpfl; последовательность РАМ NTTT), происходящую из бактерии Lachnospiraceae Cpfl (LbCpfl; последовательность РАМ NTTT) и т.п. В качестве эффекторного белка CRISPR для использования по настоящему изобретению, используют белок, в котором способность эффекторного белка CRISPR расщеплять по меньшей мере одну цепь (предпочтительно, обе цепи) двухцепочечной ДНК, инактивирована. Например, в случае SpCas9, можно использовать вариант, в котором 10-й остаток Asp переведен в остаток Ala и/или 840-й остаток His переведен в остаток Ala (вариант, лишенный способности расщеплять обе цепи двухцепочечной ДНК, иногда обозначенный как «dSpCas9»). Альтернативно, в случае SaCas9, можно использовать вариант, в котором 10-й остаток Asp переведен в остаток Ala и/или 556-й остаток Asp, 557-й остаток His, и/или 580-й остаток Asn переведен/переведены в остаток Ala (вариант, лишенный способности расщеплять обе цепи двухцепочечной ДНК, иногда обозначенный как «dSaCas9»). В случае CjCas9, можно использовать вариант, в котором 8-й остаток Asp переведен в остаток Ala и/или 559-й остаток His переведен в остаток Ala (вариант, лишенный способности расщеплять обе цепи двухцепочечной ДНК, иногда обозначенный как «dCjCas9»). В случае FnCpfl, можно использовать вариант, в котором 917-й остаток Asp переведен в остаток Ala и/или 1006-й остаток Glu переведен в остаток Ala. Кроме того, при условии, что способность связывания с нуклеотидной последовательностью-мишенью можно сохранять, можно использовать также вариант, в котором часть аминокислот из этих белков модифицирована. Примеры этого варианта включают укороченный вариант, в котором часть аминокислотной последовательности делетирована. Примеры такого варианта конкретно включают dSaCas9, в котором 721-ая - 745-ая аминокислоты делетированы (делетированная часть может являться замещенной вышеописанным пептидным линкером и т.п.) и т.п.
[0032] Вторым элементом системы CRISPR-GNDM по настоящему изобретению является направляющий нуклеотид (нН), содержащий нуклеотидную последовательность (далее в настоящем описании также обозначенную как «нацеливающая последовательность»), комплементарную нуклеотидной последовательности, соседней с РАМ целевой цепи в области регуляции транскрипции целевого гена. Когда эффекторный белок CRISPR представляет собой dCas9, нН представлен в форме химерного нуклеотида из укороченных crРНК и tracrРНК (т.е., одиночной направляющей РНК (онРНК)), или комбинации отдельных crРНК и tracrРНК. нН может быть представлен в форме РНК, ДНК или химеры ДНК/РНК. Таким образом, далее в настоящем описании, при условии, что это технически возможно, термины «онРНК», «crРНК» и «tracrРНК» используют для включения также соответствующей ДНК и химеры ДНК/РНК, в контексте настоящего изобретения.
[0033] «Целевая цепь» в настоящем описании обозначает цепь, формирующую гибрид с crРНК из нуклеотидной последовательности-мишени, и противоположная ей цепь, которая становится одноцепочечной при гибридизации с целевой цепью и crРНК, обозначена как «нецелевая цепь». Когда нуклеотидную последовательность-мишень необходимо выразить посредством одной из цепей (например, когда указана последовательность РАМ, когда показана связь расположения нуклеотидной последовательности-мишени и РАМ, и т.д.), ее представляют посредством последовательности нецелевой цепи.
[0034] Нацеливающая последовательность не является ограниченной, при условии, что она может специфически гибридизоваться с целевой цепью в области регуляции транскрипции целевого гена и привлекать эффекторный белок CRISPR и активатор по настоящему изобретению, связанные с ней, к области регуляции транскрипции. Например, когда dSaCas9 используют в качестве эффекторного белка CRISPR, нацеливающие последовательности, перечисленные в таблице 1, приведены в качестве примера. В таблице 1, в то время как описаны нацеливающие последовательности, состоящие из 21 нуклеотидов, длина нацеливающей последовательности, предпочтительно, составляет не менее, чем 12 нуклеотидов (например, 12 нуклеотидов, 15 нуклеотидов, 18 нуклеотидов, 19 нуклеотидов, 20 нуклеотидов или более), и не боле, чем 25 нуклеотидов (например, 25 нуклеотидов, 24 нуклеотидов, 23 нуклеотидов, 22 нуклеотидов или менее). В предпочтительном варианте осуществления, она составляет 21 нуклеотидов.
[0035] Когда Cas9 используют в качестве эффекторного белка CRISPR, нацеливающую последовательность можно разрабатывать, например, с использованием общедоступного веб-сайта для дизайна направляющего нуклеотида (CRISPR Design Tool, CRISPRdirect и т.д.), посредством перечисления 21-членных последовательностей, имеющих РАМ (например, NNGRRT для SaCas9), соседних с 3'-стороны с последовательностями CDS целевого гена. Последовательность-кандидат, имеющую небольшое количество участков неспецифического связывания в геноме хозяина, можно использовать в качестве нацеливающей последовательности. Когда программное обеспечение для дизайна направляющего нуклеотида, подлежащее использованию, не имеет функции поиска участков неспецифического связывания в геноме хозяина, поиск участков неспецифического связывания в геноме хозяина можно проводить посредством, например, проведения в геноме хозяине поиска Blast для 8-12 нуклеотидов (последовательности затравки с высокой способностью к различению нуклеотидной последовательности-мишени) с 3'-стороны от последовательности-кандидата. Даже когда используют эффекторный белок CRISPR, узнающий отличный РАМ, нацеливающую последовательность можно разрабатывать и получать сходным способом. Если не указано иное, в настоящем описании, нацеливающая последовательность показана как последовательность ДНК. Когда РНК используют в качестве нН, «T» следует читать как «U» в каждой последовательности.
[0036] [Таблица 4]
[0037] Любой из вышеупомянутых модулей, узнающих последовательность нуклеиновой кислоты, может быть представлен как белок, слитый с вышеупомянутым активатором по настоящему изобретению, или связывающий белок домен, такой как домен SH3, домен PDZ, домен GK, домен GB и т.п., и его партнер по связыванию могут являться слитыми с модулем, узнающим последовательность нуклеиновой кислоты, и активатором по настоящему изобретению, соответственно, и представленными в форме белкового комплекса посредством взаимодействия домена и его партнера по связыванию. Альтернативно, каждый из модуля, узнающего последовательность нуклеиновой кислоты, и активатора по настоящему изобретению могут являться слитыми с интеином, и они могут соединяться посредством связывания после синтеза белка.
[0038] Комплекс по настоящему изобретению, содержащий комплекс (включая слитый белок), в котором связаны модуль, узнающий последовательность нуклеиновой кислоты, и активатор по настоящему изобретению, можно приводить в контакт с двухцепочечной ДНК в форме ферментной реакции в бесклеточной системе. С учетом главной цели настоящего изобретения, нуклеиновую кислоту, кодирующую указанный комплекс, желательным образом вводят в клетку, имеющую целевую двухцепочечную ДНК (например, геномную ДНК). Таким образом, модуль, узнающий последовательность нуклеиновой кислоты, и активатор по настоящему изобретению, предпочтительно, представлены в форме нуклеиновой кислоты, кодирующей слитый из них белок, или в форме, способной к формированию комплекса в клетке-хозяине после трансляции в белок, с использованием связывающего домена, интеина и т.п., или в форме нуклеиновой кислоты, кодирующей каждый из них. Нуклеиновая кислота в настоящем описании может представлять собой ДНК или РНК. Когда она представляет собой ДНК, она, предпочтительно, представляет собой двухцепочечную ДНК, и представлена в форме экспрессирующего вектора, находящегося под контролем функционального промотора в клетке-хозяине. Когда она представляет собой РНК, она, предпочтительно, представляет собой одноцепочечную РНК.
[0039] Поскольку комплекс по настоящему изобретению, в котором связаны модуль, узнающий последовательность нуклеиновой кислоты, и активатор по настоящему изобретению, не сопровождается двухцепочечными разрывами ДНК (DSB), способ с использованием комплекса по настоящему изобретению можно использовать для широкого диапазона биологических материалов. Таким образом, клетки, подлежащие введению нуклеиновой кислоты, кодирующей модуль, узнающий последовательность нуклеиновой кислоты, и/или активатор по настоящему изобретению, могут включает клетки из любого вида, от бактерии из числа Escherichia coli и т.п., представляющих собой прокариоты, клеток микроорганизма, такого как дрожжи и т.п., представляющих собой низшие эукариоты, до клеток позвоночных, включая млекопитающих, таких как человек и т.п., и клетки высших эукариот, таких как насекомое, растение и т.п.
[0040] ДНК, кодирующую модуль, узнающий последовательность нуклеиновой кислоты, такой как мотив с цинковыми пальцами, эффектор TAL, мотив PPR, система CRISPR-GNDM и т.п., можно получать любым способом, упомянутым выше для каждого модуля. ДНК, кодирующую модуль, узнающий последовательность нуклеиновой кислоты, из фермента рестрикции, фактора транскрипции, РНК-полимеразы и т.п., можно клонировать, например, посредством синтеза олигоДНК праймера, перекрывающего область, кодирующую желательную часть белка (часть, содержащую связывающий ДНК домен), на основании информации о его последовательности кДНК, и амплификации способом RT-ПЦР с использованием, в качестве матрицы, тотальной РНК или фракции мРНК, полученной из продуцирующих белок клеток.
[0041] Мутантный эффекторный белок CRISPR можно получать, посредством введения в ДНК, кодирующую клонированный эффекторный белок CRISPR, мутации, переводящей аминокислотный остаток в участке, важном для активности расщепления ДНК (например, 10-й остаток Asp и 840-й остаток His для SpCas9, 10-й остаток Asp, 556-й остаток Asp, 557-й остаток His, 580-й остаток Asn для SaCas9, 8-й остаток Asp, 559-й остаток His для CjCas9, 917-й остаток Asp и 1006-й остаток Glu для FnCpfl и т.п., хотя и без ограничения ими) в другую аминокислоту.
[0042] Клонированную ДНК можно непосредственно, или после расщепления ферментом рестрикции, когда желательно, или после добавления пригодного линкера (например, вышеупомянутого пептидного линкера и т.д.), метки (например, метки HA, метки myc, метки MBP, метки FLAG и т.д.) и/или сигнала ядерной локализации (сигнала транспорта в каждую органеллу, когда целевая двухцепочечная ДНК представляет собой митохондриальную или хлоропластную ДНК), лигировать с ДНК, кодирующей модуль, узнающий последовательность нуклеиновой кислоты, для получения ДНК, кодирующей слитый белок. Альтернативно, каждую из ДНК, кодирующей модуль, узнающий последовательность нуклеиновой кислоты, и ДНК, кодирующей активатор по настоящему изобретению, можно сливать с ДНК, кодирующей связывающий домен или его партнер по связыванию, или обе ДНК можно сливать с ДНК, кодирующей разделенный интеин, при этом узнающий последовательность нуклеиновой кислоты конверсионный модуль и активатор по настоящему изобретению транслируются в клетке-хозяине с формированием комплекса. В этих случаях, линкер и/или сигнал ядерной локализации можно присоединять в подходящем положении одной или обеих ДНК, когда желательно. Когда комплекс по настоящему изобретению экспрессируют в форме слитого белка, активатор по настоящему изобретению может являться слитым с любым из N-конца и C-конца модуля, узнающего последовательность нуклеиновой кислоты, или составляющего его компонента (например, эффекторного белка CRISPR).
[0043] ДНК, кодирующую модуль, узнающий последовательность нуклеиновой кислоты, и/или активатор по настоящему изобретению, можно получать посредством химического синтеза цепи ДНК, или посредством соединения синтезированных частично перекрывающихся коротких цепей олигоДНК с использованием способа ПЦР и способа сборки Гибсона для конструирования ДНК, кодирующей его полную длину. Преимуществом конструирования полноразмерной ДНК посредством химического синтеза или комбинации способа ПЦР или способа сборки Гибсона является то, что можно осуществлять дизайн кодонов, подлежащих использованию в полноразмерной CDS, в соответствии с хозяином, в которого вводят ДНК. Ожидают, что при экспрессии гетерологичной ДНК, уровень экспрессии белка увеличивается посредством перевода последовательности его ДНК в кодоны, с высокой частотой используемые в организме-хозяине. В качестве данных частоты использования кодонов в подлежащем использованию хозяине, можно использовать, например, базу данных частоты использования генетического кода (http://www.kazusa.или.jp/codon/index.html), описанную на домашней странице Kazusa DNA Research Institute, или можно ссылаться на документы, показывающие частоту использования кодонов в каждом хозяине. Со ссылкой на полученные данные и последовательность ДНК, подлежащую введению, кодоны, для которых показана низкая частота использования в хозяине, среди кодонов, использованных для последовательности ДНК, можно переводить в кодон, который кодирует такую же аминокислоту, и для которого показана высокая частота использования.
[0044] РНК, кодирующую модуль, узнающий последовательность нуклеиновой кислоты, и/или активатор по настоящему изобретению, можно получать, например, посредством получения вектора, содержащего ДНК, кодирующую модуль и/или активатор, и ее транскрипции в мРНК посредством известной системы транскрипции in vitro с использованием вектора в качестве матрицы. Альтернативно, РНК можно также синтезировать химически.
[0045] Экспрессирующий вектор, содержащий ДНК, кодирующую активатор по настоящему изобретению или комплекс по настоящему изобретению, можно получать, например, посредством связывания ДНК ниже промотора в подходящем экспрессирующем векторе.
[0046] В качестве экспрессирующего вектора, используют происходящие из Escherichia coli плазмиды (например, pBR322, pBR325, pUC12, pUC13); происходящие из Bacillus subtilis плазмиды (например, pUB110, pTP5, pC194); происходящие из дрожжей плазмиды (например, pSH19, pSH15); плазмиды для экспрессии в клетках насекомых (например, pFast-Bac); плазмиды для экспрессии в клетках животных (например, pA1-11, pXT1, pRc/CMV, pRc/RSV, pcDNAI/Neo); бактериофаги, такие как λ-фаг и т.п.; вирусные векторы для насекомых, такие как бакуловирус и т.п. (например, BmNPV, AcNPV); вирусные векторы для животных, такие как ретровирус, вирус осповакцины, аденовирус, аденоассоциированный вирус (AAV) и т.п., и т.п. Принимая во внимание использование в генотерапии, AAV вектор используют предпочтительно, поскольку он может экспрессировать трансген в течение длительного времени и является безопасным, благодаря его происхождению из непатогенного вируса.
[0047] AAV вектор не является конкретно ограниченным, при условии, что его титр и эффективность инфекции являются достаточно надежными. Он составляет, предпочтительно, не более, чем приблизительно 5 т.п.о. (например, приблизительно 5 т.п.о., приблизительно 4,95 т.п.о., приблизительно 4,90 т.п.о., приблизительно 4,85 т.п.о., приблизительно 4,80 т.п.о., приблизительно 4,75 т.п.о., приблизительно 4,70 т.п.о. или ниже). Длина в аминокислотах активатора по настоящему изобретению, предпочтительно, составляет не более, чем 200 аминокислот. Таким образом, общую длину в основаниях нуклеиновой кислоты, кодирующей комплекс по настоящему изобретению, и нуклеиновой кислоты, кодирующей направляющий нуклеотид, можно легко разрабатывать ниже этого предела размера. Таким образом, активатор по настоящему изобретению имеет то преимущество, что сборка нуклеиновой кислоты, кодирующей комплекс по настоящему изобретению, и нуклеиновой кислоты, кодирующей направляющий нуклеотид, в отдельных AAV векторах не является необходимой.
[0048] Когда вирусный вектор используют в качестве экспрессирующего вектора, предпочтительно, используют вектор, происходящий из серотипа, подходящего для инфекции целевых ткани или органа. Принимая AAV вектор в качестве примера, является предпочтительным использование вектора на основе AAV 1, 2, 3, 4, 5, 7, 8, 9 или 10, когда центральная нервная система или сетчатка является мишенью, вектора на основе AAV 1, 3, 4, 6 или 9, когда сердце является мишенью, вектора на основе AAV 1, 5, 6, 9 или 10, когда легкое является мишенью, вектора на основе AAV 2, 3, 6, 7, 8 или 9, когда печень является мишенью, и вектора на основе AAV 1, 2, 6, 7, 8, 9, когда скелетная мышца является мишенью. Для лечения злокачественных опухолей, AAV 2 используют предпочтительно. Относительно серотипа AAV, можно ссылаться, например, на WO 2005/033321 A2 и т.п.
[0049] РНК, кодирующую модуль, узнающий последовательность нуклеиновой кислоты, и/или активатор по настоящему изобретению, можно вводить в клетку-хозяина способом микроинъекции, способом липофекции и т.п. Введение РНК можно проводить один раз или повторять множество раз (например, 2-5 раз) с подходящими интервалами.
[0050] Кроме того, множество областей ДНК в полностью различных участках могут служить мишенью. Таким образом, в одном варианте осуществления настоящего изобретения, можно использовать два или более видов модуля, узнающего последовательность нуклеиновой кислоты, которые специфически связываются с различными нуклеотидными последовательностями-мишенями (которые могут присутствовать в одном целевом гене, или в двух или более различных целевых генах, где целевые гены могут присутствовать на одной и той же хромосоме или различных хромосомах). В этом случае, каждый из этих модулей, узнающих последовательность нуклеиновой кислоты, и активатор по настоящему изобретению формируют комплекс. В этом случае, можно использовать общий активатор по настоящему изобретению. Например, когда систему CRISPR-GNDM используют в качестве модуля, узнающего последовательность нуклеиновой кислоты, используют общий комплекс из эффекторного белка CRISPR и активатора по настоящему изобретению (включая слитый белок), и две или более crРНК, или два или более вида химерных РНК из tracrРНК и каждой из двух или более crРНК, которая, соответственно, формирует комплементарную цепь с различной нуклеотидной последовательностью-мишенью, получают и используют в качестве нН. С другой стороны, когда мотив с цинковыми пальцами, эффектор TAL и т.п., используют в качестве модулей, узнающих последовательность нуклеиновой кислоты, например, активатор по настоящему изобретению может являться слитым с модулем, узнающим последовательность нуклеиновой кислоты, который специфически связывается с отличным нуклеотидом-мишенью.
[0051] ДНК, кодирующую нН, можно химически синтезировать с использованием синтезатора ДНК/РНК, на основании информации о ее последовательности. Например, ДНК, кодирующая нН для SaCas9, имеет дезоксирибонуклеотидную последовательность, кодирующую crРНК, содержащую нацеливающую последовательность, комплементарную области регуляции транскрипции целевого гена, и по меньшей мере часть области «повтора» (например, GUUUUAGUACUCUG; SEQ ID NO:31) нативной SacrРНК, и дезоксирибонуклеотидную последовательность, кодирующую tracrРНК, имеющую по меньшей мере часть области «антиповтора» (например, CAGAAUCUACUAAAAC; SEQ ID NO:32), комплементарной области повтора crРНК, и последующие области стебля-петли 1, линкера и стебля-петли 2 (AAGGCAAAAUGCCGUGUUUAUCACGUCAACUUGUUGGCGAGAUUUUUUU; SEQ ID NO:33) нативной SatracrРНК, необязательно, связанные посредством тетрапетли (например, GAAA). С другой стороны, ДНК, кодирующая нРНК для dCpf1, имеет дезоксирибонуклеотидную последовательность, кодирующую только crРНК, которая содержит нацеливающую последовательность, комплементарную области регуляции транскрипции целевого гена, и предшествующую 5'-ручку (например, AAUUUCUACUCUUGUAGAU; SEQ ID NO:34). Когда белок, отличный от SaCas9 и Cpf1, используют в качестве эффекторного белка CRISPR, tracrРНК для белка, подлежащего использованию, можно разрабатывать подходящим образом, на основании известной последовательности и т.п. ДНК, кодирующую эффекторный белок CRISPR, лигированный с ДНК, кодирующей активатор по настоящему изобретению, можно субклонировать в экспрессирующий вектор таким образом, чтобы указанные ДНК были локализованы под контролем промотора, который является функциональным в представляющей интерес клетке-хозяине.
[0052] ДНК, кодирующую нН (например, crРНК или химеру crРНК-tracrРНК), можно вводить в клетку-хозяина способом, сходным со способами, описанными выше, в зависимости от хозяина.
[0053] Альтернативно, РНК можно использовать вместо ДНК для доставки эффекторной молекулы CRISPR. В одном варианте осуществления, систему CRISPR-GNDM по настоящему изобретению, содержащую (a) комплекс по настоящему изобретению и (b) нН, содержащий нацеливающую последовательность, можно вводить в клетки или организмы - мишени в форме РНК, кодирующих (a) и (b) выше.
[0054] Например, вышеупомянутую РНК, кодирующую эффекторные молекулы выше, можно получать посредством транскрипции in vitro, и полученную мРНК можно очищать для доставки in vivo. Кратко, фрагмент ДНК, содержащий область CDS эффекторных молекул, можно клонировать ниже искусственного промотора из бактериофага, управляющего транскрипцией in vitro (например, промотора T7, T3 или SP6). РНК можно транскрибировать с промотора посредством добавления компонентов, необходимых для транскрипции in vitro, таких как полимераза T7, NTP и буферы IVT. При необходимости, РНК можно модифицировать для уменьшения иммунной стимуляции, увеличения трансляции и устойчивости к нуклеазе (например, кэпирование 5mCAP (m7G(5')ppp(5')G, ARCA; антиинвертированные аналоги кэп-структуры (3'O-Me-M7G(5')ppp(5')G), модификации 5-метилцитидина и псевдоуридина, 3'-поли A-хвост).
[0055] Альтернативно, комплекс эффекторного белка и нН, далее в настоящем описании названный нуклеопротеином (NP) (например, дезоксирибонуклеопротеином (DNP), рибонуклеопротеином (RNP)), можно использовать для доставки эффекторной молекулы CRISPR и нН. Кратко, полученный in vitro эффекторный белок CRISPR и транскрибированный in vitro или химически синтезированный нН смешивают в соответствующих соотношениях, и затем инкапсулируют в липидные наночастицы (LNP). Инкапсулированные LNP можно доставлять животному, страдающему от заболевания, или пациенту, и NP комплекс можно доставлять непосредственно в клетки или органы - мишени.
[0056] Эффекторный белок CRISPR можно экспрессировать в бактериях и можно очищать посредством аффинной колонки. Оптимизированную по кодонному составу для бактерий последовательность кДНК эффекторного белка CRISPR можно клонировать в плазмиды для экспрессии в бактериях, такие как вектор pE-SUMO от LifeSensors. Фрагмент кДНК можно метить с использованием небольшой пептидной последовательности, такой как пептиды HA, 6xHis, Мус или FLAG, либо на N-, либо на C-конце. Плазмиды можно вводить в экспрессирующие белок бактериальные штаммы, такие как E. coli B834 (DE3). После индукции, белок можно очищать с использованием связывания последовательностей небольшой пептидной метки с аффинной колонкой, такой как колонка Ni-NTA или аффинная колонка против FLAG. Присоединенную пептидную метку можно удалять посредством обработки протеазой TEV. Белок можно далее очищать посредством хроматографии на колонке HiLoad Superdex 200 16/60 (GE Health-care).
[0057] Альтернативно, эффекторный белок CRISPR можно экспрессировать в линиях клеток млекопитающих, таких как клетки CHO, COS, HEK293 и Hela. Например, оптимизированную по человеческому кодонному составу последовательность кДНК для белка CRISPR можно клонировать в экспрессирующие плазмиды для млекопитающих (например, pAl-11, pXTl, pRc/CMV, pRc/RSV, pcDNAI/Neo, pSRa); можно использовать векторы, происходящие из вируса животных, такого как ретровирус, вирус осповакцины, аденовирус, аденоассоциированный вирус, и т.д., и т.п. Фрагменты кДНК можно метить с использованием небольшой пептидной последовательности, такой как пептид HA, 6xHis, Мус или FLAG, либо на N-, либо на C-конце. Плазмиды можно вводить в экспрессирующие белок линии клеток млекопитающих. Через 2-3 суток после трансфекции, трансфицированные клетки можно собирать, и экспрессированный белок CRISPR можно очищать с использованием связывания последовательностей небольшой пептидной метки с аффинной колонкой, указанной выше.
[0058] Активатор по настоящему изобретению также можно получать способом, сходным с вышеупомянутым способом.
Примеры
[0059] Изобретение станет более полно понятным со ссылкой на следующие примеры, предоставляющие иллюстративные неограничивающие варианты осуществления изобретения.
[0060] Авторы настоящего изобретения разработали и сконструировали новые активирующие группы, которые являются достаточно маленькими для слияния с dSaCas9 и соответствия пределу размера вектора AAV 5 т.п.о., в то же время имея сравнимую или даже лучшую активность активации транскрипции, чем существующие активирующие группы (фигура 1). Существующие активирующие группы включают VP64 (50 а.к.), VP160 (130 а.к.), VPR (520 а.к.) и P300 (617 а.к.) (описаны в PMID:27214048/25730490). Из этих активирующих групп, только VP64 и VP160 удовлетворяют пределу размера вектора AAV при слиянии с dSaCas9.
[0061] Таким образом, авторы настоящего изобретения разработали, сконструировали и тестировали следующие семь новых активирующих групп, слитых с dSaCas9, и сравнили их активность трансактивации с тремя существующими группами (VP64, VP160 и VPR).
[0062] Аминокислотная и нуклеотидная последовательность полученных активирующих групп
1. VP64-мини-MYOD (154 а.к.) состоит из VP64 (курсив) и 1-100 а.к. из MYOD1 человека (жирный шрифт, PMID: 9710631), соединенных посредством линкера G-S-G-S (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDML GSGS MELLSPPLRDVDLTAPDGSLCSFATTDDFYDDPCFDSPDLRFFEDLDPRLMHVGALLKPEEHSHFPAAVHPAPGAREDEHVRAPSGHHQAGRCLLWACKA (SEQ ID NO:10)
gatgctttagacgattttgacttagatatgcttggttcagacgcgttagacgacttcgacctagacatgttaggctcagatgcattggacgacttcgatttagatatgttgggctccgatgccctagatgactttgatttggatatgctaggatctggtagcatggagctactgtcgccaccgctccgcgacgtagacctgacggcccccgacggctctctctgctcctttgccacaacggacgacttctatgacgacccgtgtttcgactccccggacctgcgcttcttcgaggacctggacccgcgcctgatgcacgtgggcgcgctcctgaaacccgaagagcactcgcacttccctgcggctgttcacccggcaccgggggcacgcgaggacgaacatgtcagggctcccagcggtcatcaccaggctggtcggtgtctgttgtgggcctgcaaggcg (SEQ ID NO:9)
[0063] 2. VP64-мини-HSF1 (154 а.к.) состоит из VP64 (курсив) и 430-529 а.к. из HSF1 человека (жирный шрифт, PMID:7760831), соединенных посредством линкера G-S-S-G (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDML GSSG PDLDSSLASIQELLSPQEPPRPPEAENSSPDSGKQLVHYTAQPLFLLDPGSVDTGSNDLPVLFELGEGSYFSEGDGFAEDPTISLLTGSEPPKAKDPTVS (SEQ ID NO:12)
gatgctttagacgattttgacttagatatgcttggttcagacgcgttagacgacttcgacctagacatgttaggctcagatgcattggacgacttcgatttagatatgttgggctccgatgccctagatgactttgatttggatatgctaggtagcagtgggcctgaccttgacagcagcctggccagtatccaagagctcctgtctccccaggagccccccaggcctcccgaggcagagaacagcagcccggattcagggaagcagctggtgcactacacagcgcagccgctgttcctgctggaccccggctccgtggacaccgggagcaacgacctgccggtgctgtttgagctgggagagggctcctacttctccgaaggggacggcttcgccgaggaccccaccatctccctgctgacaggctcggagcctcccaaagccaaggaccccactgtctcc (SEQ ID NO:11)
[0064] 3. VP32-мини-P65 (160 а.к.) состоит из VP32 (курсив) и 415-546 а.к. из P65 человека (жирный шрифт, PMID: 1732726), соединенных посредством линкера G-S-G-S (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDML GSGS PGPPQAVAPPAPKPTQAGEGTLSEALLQLQFDDEDLGALLGNSTDPAVFTDLASVDNSEFQQLLNQGIPVAPHTTEPMLMEYPEAITRLVTGAQRPPDPAPAPLGAPGLPNGLLSGDEDFSSIADMDFSALL (SEQ ID NO:14)
gatgcattggacgacttcgatttagatatgttgggctccgatgccctagatgactttgatttggatatgctaggatctggtagccctggacctccacaggctgtggctccaccagcccctaaacctacacaggccggcgagggcacactgtctgaagctctgctgcagctgcagttcgacgacgaggatctgggagccctgctgggaaacagcaccgatcctgccgtgttcaccgacctggccagcgtggacaacagcgagttccagcagctgctgaaccagggcatccctgtggcccctcacaccaccgagcccatgctgatggaataccccgaggccatcacccggctcgtgacaggcgctcagaggcctcctgatccagctcctgcccctctgggagcaccaggcctgcctaatggactgctgtctggcgacgaggacttcagctctatcgccgatatggatttctcagccttgctg (SEQ ID NO:13)
[0065] 4. VP64-мини-RTA (167 а.к.) состоит из VP64 (курсив) и 493-605 а.к. из активатора репликации и транскрипции вируса Эпштейна-Барр (жирный шрифт, RTA; PMID: 1323708), соединенных посредством линкера G-S-G-S (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDML GSGS PAPAVTPEASHLLEDPDEETSQAVKALREMADTVIPQKEEAAICGQMDLSHPPPRGHLDELTTTLESMTEDLNLDSPLTPELNEILDTFLNDECLLHAMHISTGLSIFDTSLF (SEQ ID NO:6)
gatgctttagacgattttgacttagatatgcttggttcagacgcgttagacgacttcgacctagacatgttaggctcagatgcattggacgacttcgatttagatatgttgggctccgatgccctagatgactttgatttggatatgctaggatctggtagcccagcgcccgcagtgactcccgaggccagtcacctgttggaagatcccgatgaagagaccagccaggctgtcaaagcccttcgggagatggccgatactgtgattccccagaaggaagaggctgcaatctgtggccaaatggacctttcccatccgcccccaaggggccatctggatgagctgacaaccacacttgagtccatgaccgaggatctgaacctggactcacccctgaccccggaattgaacgagattctggataccttcctgaacgacgagtgcctcttgcatgccatgcatatcagcacaggactgtccatcttcgacacatctctgttt (SEQ ID NO:5)
[0066] 5. VP64-мини-P65 (186 а.к.) состоит из VP64 (курсив) и 415-546 а.к. из P65 человека (жирный шрифт, PMID: 1732726), соединенных посредством линкера G-S-G-S (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDML GSGS PGPPQAVAPPAPKPTQAGEGTLSEALLQLQFDDEDLGALLGNSTDPAVFTDLASVDNSEFQQLLNQGIPVAPHTTEPMLMEYPEAITRLVTGAQRPPDPAPAPLGAPGLPNGLLSGDEDFSSIADMDFSALL (SEQ ID NO:16)
gatgctttagacgattttgacttagatatgcttggttcagacgcgttagacgacttcgacctagacatgttaggctcagatgcattggacgacttcgatttagatatgttgggctccgatgccctagatgactttgatttggatatgctaggatctggtagccctggacctccacaggctgtggctccaccagcccctaaacctacacaggccggcgagggcacactgtctgaagctctgctgcagctgcagttcgacgacgaggatctgggagccctgctgggaaacagcaccgatcctgccgtgttcaccgacctggccagcgtggacaacagcgagttccagcagctgctgaaccagggcatccctgtggcccctcacaccaccgagcccatgctgatggaataccccgaggccatcacccggctcgtgacaggcgctcagaggcctcctgatccagctcctgcccctctgggagcaccaggcctgcctaatggactgctgtctggcgacgaggacttcagctctatcgccgatatggatttctcagccttgctg (SEQ ID NO:15)
[0067] 6. VPH (376 а.к.) состоит из VP64 (курсив), 369-549 а.к. из P65 мыши (жирный шрифт) и 407-529 а.к. из HSF1 человека (подчеркнутый жирный шрифт), PMID: 25494202), соединенных посредством NLS (PKKKRKV) (SEQ ID NO:45) и/или линкера S-G-Q-G-G-G-G-S-G (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLSSGSPKKKRKVGSPSGQISNQALALAPSSAPVLAQTMVPSSAMVPLAQPPAPAPVLTPGPPQSLSAPVPKSTQAGEGTLSEALLHLQFDADEDLGALLGNSTDPGVFTDLASVDNSEFQQLLNQGVSMSHSTAEPMLMEYPEAITRLVTGSQRPPDPAPTPLGTSGLPNGLSGDEDFSSIADMDFSALLSQISSSGQGGGGSGFSVDTSALLDLFSPSVTVPDMSLPDLDSSLASIQELLSPQEPPRPPEAENSSPDSGKQLVHYTAQPLFLLDPGSVDTGSNDLPVLFELGEGSYFSEGDGFAEDPTISLLTGSEPPKAKDPTVS (SEQ ID NO:18)
gatgctttagacgattttgacttagatatgcttggttcagacgcgttagacgacttcgacctagacatgttaggctcagatgcattggacgacttcgatttagatatgttgggctccgatgccctagatgactttgatttggatatgctaagttccggatctccgaaaaagaaacgcaaagttggtagcccttcagggcagatcagcaaccaggccctggctctggcccctagctccgctccagtgctggcccagactatggtgccctctagtgctatggtgcctctggcccagccacctgctccagcccctgtgctgaccccaggaccaccccagtcactgagcgctccagtgcccaagtctacacaggccggcgaggggactctgagtgaagctctgctgcacctgcagttcgacgctgatgaggacctgggagctctgctggggaacagcaccgatcccggagtgttcacagacctggcctccgtggacaactctgagtttcagcagctgctgaatcagggcgtgtccatgtctcatagtacagccgaaccaatgctgatggagtaccccgaagccattacccggctggtgaccggcagccagcggccccccgaccccgctccaactcccctgggaaccagcggcctgcctaatgggctgtccggagatgaagacttctcaagcatcgctgatatggactttagtgccctgctgtcacagatttcctctagtgggcagggaggaggtggaagcggcttcagcgtggacaccagtgccctgctggacctgttcagcccctcggtgaccgtgcccgacatgagcctgcctgaccttgacagcagcctggccagtatccaagagctcctgtctccccaggagccccccaggcctcccgaggcagagaacagcagcccggattcagggaagcagctggtgcactacacagcgcagccgctgttcctgctggaccccggctccgtggacaccgggagcaacgacctgccggtgctgtttgagctgggagagggctcctacttctccgaaggggacggcttcgccgaggaccccaccatctccctgctgacaggctcggagcctcccaaagccaaggaccccactgtctcc (SEQ ID NO:17)
[0068] 7. VPR (510 а.к.) состоит из VP64 (курсив), 284-543 а.к. из P65 человека (жирный шрифт, PMID: 5970) и 416-605 а.к. из активатора репликации и транскрипции вируса Эпштейна-Барр (подчеркнутый жирный шрифт, RTA; PMID: 1323708), соединенных посредством NLS (PKKKRKV) и/или линкера G-S-G-S-G-S (подчеркнуто)
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLINSRSSGSPKKKRKVGSQYLPDTDDRHRIEEKRKRTYETFKSIMKKSPFSGPTDPRPPPRRIAVPSRSSASVPKPAPQPYPFTSSLSTINYDEFPTMVFPSGQISQASALAPAPPQVLPQAPAPAPAPAMVSALAQAPAPVPVLAPGPPQAVAPPAPKPTQAGEGTLSEALLQLQFDDEDLGALLGNSTDPAVFTDLASVDNSEFQQLLNQGIPVAPHTTEPMLMEYPEAITRLVTGAQRPPDPAPAPLGAPGLPNGLLSGDEDFSSIADMDFSALLGSGSGSRDSREGMFLPKPEAGSAISDVFEGREVCQPKRIRPFHPPGSPWANRPLPASLAPTPTGPVHEPVGSLTPAPVPQPLDPAPAVTPEASHLLEDPDEETSQAVKALREMADTVIPQKEEAAICGQMDLSHPPPRGHLDELTTTLESMTEDLNLDSPLTPELNEILDTFLNDECLLHAMHISTGLSIFDTSLF (SEQ ID NO:20)
gacgccctcgatgattttgaccttgacatgcttggttcggatgcccttgatgactttgacctcgacatgctcggcagtgacgcccttgatgatttcgacctggacatgctgattaactctAgaagttccggatctccgaaaaagaaacgcaaagttggtagccagtacctgcccgacaccgacgaccggcaccggatcgaggaaaagcggaagcggacctacgagacattcaagagCatcatgaagaagtcccccttcagcggccccaccgaccctagacctccacctagaagaatcgccgtgcccagcagatccagcgccagcgtgccaaaacctgccccccagccttaCcccttcaccagcagcctgagcaccatcaactacgacgagttccctaccatggtgttccccagcggccagatctctcaggcctctgctctggctccagcccctcctcaggtgctgcctcaggctcctgctcctgcaccagctccagccatggtgtctgcactggctcaggcaccagcacccgtgcctgtgctggctcctggacctccacaggctgtggctccaccagcccctaaacctacacaggccggcgagggcacactgtctgaagctctgctgcagctgcagttcgacgacgaggatctgggagccctgctgggaaacagcaccgatcctgccgtgttcaccgacctggccagcgtggacaacagcgagttccagcagctgctgaaccagggcatccctgtggcccctcacaccaccgagcccatgctgatggaataccccgaggccatcacccggctcgtgacaggcgctcagaggcctcctgatccagctcctgcccctctgggagcaccaggcctgcctaatggactgctgtctggcgacgaggacttcagctctatcgccgatatggatttctcagccttgctgggctctggcagcggcagccgggattccagggaagggatgtttttgccgaagcctgaggccggctccgctattagtgacgtgtttgagggccgcgaggtgtgccagccaaaacgaatccggccatttcatcctccaggaagtccatgggccaaccgcccactccccgccagcctcgcaccaacaccaaccggtccagtacatgagccagtcgggtcactgaccccggcaccagtccctcagccactggatccagcgcccgcagtgactcccgaggccagtcacctgttggaggatcccgatgaagagacgagccaggctgtcaaagcccttcgggagatggccgatactgtgattccccagaaggaagaggctgcaatctgtggccaaatggacctttcccatccgcccccaaggggccatctggatgagctgacaaccacacttgagtccatgaccgaggatctgaacctggactcacccctgaccccggaattgaacgagattctggataccttcctgaacgacgagtgcctcttgcatgccatgcatatcagcacaggactgtccatcttcgacacatctctgttt (SEQ ID NO:19)
[0069] 8. VP64-микро-RTA (140 а.к.) состоит из VP64 (курсив) и 520-605 а.к. из активатора репликации и транскрипции вируса Эпштейна-Барр (жирный шрифт, RTA; PMID: 1323708), соединенных посредством линкера G-S-G-S (подчеркнуто);
DALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDMLGSDALDDFDLDML GSGS REMADTVIPQKEEAAICGQMDLSHPPPRGHLDELTTTLESMTEDLNLDSPLTPELNEILDTFLNDECLLHAMHISTGLSIFDTSLF (SEQ ID NO:8)
gatgcactcgatgattttgacctcgatatgcttgggagtgatgcgctcgatgacttcgatttggatatgcttggatctgatgccctcgacgatttcgaccttgatatgctcgggtcagacgctttggatgactttgaccttgacatgctggggagcggctcccgggagatggctgacacagtaataccccaaaaagaggaggctgcgatttgtgggcagatggatttgtcccaccctccaccgagaggtcatcttgacgaattgacaacgacgctcgaatccatgaccgaggacctgaacctcgatagcccgctcacccccgagttgaatgagatcctggatacatttcttaatgatgagtgtttgcttcacgcaatgcatatttctacgggtcttagtattttcgacacgagcctgttt (SEQ ID NO:7)
[0070] Клонирование плазмид
Новые активирующие группы (AM) синтезировали посредством IDT и клонировали в вектор NUC9-dSaCas9. Слитые белки экспрессировали с промотора EFS.
использованная последовательность онРНК:
MYD88-1; GGTTCATACGGTCCTGCCCTC (SEQ ID NO:35)
MYD88-2; GGAGCCACAGTTCTTCCACGG (SEQ ID NO:36)
MYD88-3; CTCTACCCTTGAGGTCTCGAG (SEQ ID NO:37)
FGF21-1; TGCCAGATTCCAGTTGTCCAG (SEQ ID NO:38)
FGF21-2; ACATTCCTGAGTCTCAGAGAG (SEQ ID NO:39)
FGF21-3; GGCTAATTTCCTGGAGCCCCT (SEQ ID NO:40)
GCG-1; CTGTGAGGCTAAACAGAGCTG (SEQ ID NO:41)
GCG-2; GTCTCTCACCCAATATAAGCA (SEQ ID NO:42)
GCG-3; AAATCACTTAAGTTCTCTAAA (SEQ ID NO:43)
[0071] Трансфекция клеток
Клетки HEK293FT рассевали в 24-луночный планшет при 75000 клеток на лунку. 250 нг экспрессирующих слитый белок плазмид NUC9-dsaCas9-AM трансфицировали совместно с экспрессирующими онРНК плазмидами LvSG03 с использованием липофектамина 2000, в соответствии с инструкциями производителя. Через 24 часа, трансфицированные клетки подвергали отбору на пуромицине, и собирали на следующие сутки.
[0072] нуклеотидная последовательность dSaCas9;
atgaagcggaactacatcctgggcctggccatcggcatcaccagcgtgggctacggcatcatcgactacgagacacgggacgtgatcgatgccggcgtgcggctgttcaaagaggccaacgtggaaaacaacgagggcaggcggagcaagagaggcgccagaaggctgaagcggcggaggcggcatagaatccagagagtgaagaagctgctgttcgactacaacctgctgaccgaccacagcgagctgagcggcatcaacccctacgaggccagagtgaagggcctgagccagaagctgagcgaggaagagttctctgccgccctgctgcacctggccaagagaagaggcgtgcacaacgtgaacgaggtggaagaggacaccggcaacgagctgtccaccaaagagcagatcagccggaacagcaaggccctggaagagaaatacgtggccgaactgcagctggaacggctgaagaaagacggcgaagtgcggggcagcatcaacagattcaagaccagcgactacgtgaaagaagccaaacagctgctgaaggtgcagaaggcctaccaccagctggaccagagcttcatcgacacctacatcgacctgctggaaacccggcggacctactatgagggacctggcgagggcagccccttcggctggaaggacatcaaagaatggtacgagatgctgatgggccactgcacctacttccccgaggaactgcggagcgtgaagtacgcctacaacgccgacctgtacaacgccctgaacgacctgaacaatctcgtgatcaccagggacgagaacgagaagctggaatattacgagaagttccagatcatcgagaacgtgttcaagcagaagaagaagcccaccctgaagcagatcgccaaagaaatcctcgtgaacgaagaggatattaagggctacagagtgaccagcaccggcaagcccgagttcaccaacctgaaggtgtaccacgacatcaaggacattaccgcccggaaagagattattgagaacgccgagctgctggatcagattgccaagatcctgaccatctaccagagcagcgaggacatccaggaagaactgaccaatctgaactccgagctgacccaggaagagatcgagcagatctctaatctgaagggctataccggcacccacaacctgagcctgaaggccatcaacctgatcctggacgagctgtggcacaccaacgacaaccagatcgctatcttcaaccggctgaagctggtgcccaagaaggtggacctgtcccagcagaaagagatccccaccaccctggtggacgacttcatcctgagccccgtcgtgaagagaagcttcatccagagcatcaaagtgatcaacgccatcatcaagaagtacggcctgcccaacgacatcattatcgagctggcccgcgagaagaactccaaggacgcccagaaaatgatcaacgagatgcagaagcggaaccggcagaccaacgagcggatcgaggaaatcatccggaccaccggcaaagagaacgccaagtacctgatcgagaagatcaagctgcacgacatgcaggaaggcaagtgcctgtacagcctggaagccatccctctggaagatctgctgaacaaccccttcaactatgaggtggaccacatcatccccagaagcgtgtccttcgacaacagcttcaacaacaaggtgctcgtgaagcaggaagaagccagcaagaagggcaaccggaccccattccagtacctgagcagcagcgacagcaagatcagctacgaaaccttcaagaagcacatcctgaatctggccaagggcaagggcagaatcagcaagaccaagaaagagtatctgctggaagaacgggacatcaacaggttctccgtgcagaaagacttcatcaaccggaacctggtggataccagatacgccaccagaggcctgatgaacctgctgcggagctacttcagagtgaacaacctggacgtgaaagtgaagtccatcaatggcggcttcaccagctttctgcggcggaagtggaagtttaagaaagagcggaacaaggggtacaagcaccacgccgaggacgccctgatcattgccaacgccgatttcatcttcaaagagtggaagaaactggacaaggccaaaaaagtgatggaaaaccagatgttcgaggaaaagcaggccgagagcatgcccgagatcgaaaccgagcaggagtacaaagagatcttcatcaccccccaccagatcaagcacattaaggacttcaaggactacaagtacagccaccgggtggacaagaagcctaatagagagctgattaacgacaccctgtactccacccggaaggacgacaagggcaacaccctgatcgtgaacaatctgaacggcctgtacgacaaggacaatgacaagctgaaaaagctgatcaacaagagccccgaaaagctgctgatgtaccaccacgacccccagacctaccagaaactgaagctgattatggaacagtacggcgacgagaagaatcccctgtacaagtactacgaggaaaccgggaactacctgaccaagtactccaaaaaggacaacggccccgtgatcaagaagattaagtattacggcaacaaactgaacgcccatctggacatcaccgacgactaccccaacagcagaaacaaggtcgtgaagctgtccctgaagccctacagattcgacgtgtacctggacaatggcgtgtacaagttcgtgaccgtgaagaatctggatgtgatcaaaaaagaaaactactacgaagtgaatagcaagtgctatgaggaagctaagaagctgaagaagatcagcaaccaggccgagtttatcgcctccttctacaacaacgatctgatcaagatcaacggcgagctgtatagagtgatcggcgtgaacaacgacctgctgaaccggatcgaagtgaacatgatcgacatcacctaccgcgagtacctggaaaacatgaacgacaagaggccccccaggatcattaagacaatcgcctccaagacccagagcattaagaagtacagcacagacattctgggcaacctgtatgaagtgaaatctaagaagcaccctcagatcatcaaaaagggctaa (SEQ ID NO:28)
[0073] последовательность tracrРНК;
guuuuaguacucuggaaacagaaucuacuaaaacaaggcaaaaugccguguuuaucacgucaacuuguuggcgagauuuuuuu (SEQ ID NO:30)
[0074] Выделение РНК и анализ экспрессии генов
Для анализа экспрессии генов, трансфицированные клетки собирали через 48-72 час после трансфекции и лизировали в буфере RLT для выделения тотальной РНК с использованием набора RNeasy (Qiagen).
Для анализа Taqman, 1 мкг тотальной РНК использовали для получения кДНК с использованием высокопроизводительного набора TaqMan™ High-Capacity RNA-to-cDNA (Applied Biosystems) в объеме 10 мкл. Полученную кДНК разводили в 10 раз, и 3,33 мкл использовали на реакцию Taqman (общий объем 10 мкл на реакцию). Реакцию Taqman проводили с использованием готовой смеси Taqman для экспрессии генов (ThermoFisher) в Roche LightCycler 96 или LightCycler 480 и анализировали с использованием программного обеспечения для анализа LightCycler 96.
ID продуктов для зонда Taqman:
MYD88; Hs01573837_g1 (FAM)
FGF21: Hs00173927_m1
GCG: Hs01031536_m1
HPRT: Hs99999909_m1 (VIC PL)
Условия QПЦР Taqman:
Стадия 1; 95°C в течение 10 мин
Стадия 2; 95°C в течение 15 с
Стадия 3; 60°C в течение 30 с
Повтор стадии 2 и 3; 40 раз
[0075] Результат
Фигура 1. Структура вектора AAV и десяти активирующих групп
Вектор AAV авторов настоящего изобретения содержит dSaCas9, слитый с активирующими группами, показанными на диаграмме ниже. Слитые белки экспрессируются посредством промотора EFS, и онРНК экспрессируются с промотора U6. Получено семь новых активирующих групп; VP64-MyoD, VP64-HSF1, VP32-p65, VP64-мини-RTA, VP64-микро-RTA, VP64-p65 и VPH. Опубликованные активирующие группы (VP64, VP160 и VPR) также тестировали для сравнения. Предел размера вектора AAV составляет 5 т.п.о., и компоненты составляют в сумме 4,45 т.п.о., что оставляет место для слитых активирующих групп приблизительно 550 п.о. Таким образом, следующие семь активирующих групп соответствуют пределу размера вектора; VP64, Vp160, VP64-MyoD, VP64-HSF1, VP32-p65, VP64-мини-RTA и VP64-микро-RTA.
[0076] Фигура 2. Активация гена MYD88 посредством девяти активирующих групп
Функцию активации шести новых активирующих групп тестировали с использованием трех различных онРНК (MYD88-1, -2 и -3), нацеленных на промоторную область MYD88 человека. Три активирующие группы, VP64, VP160 и VPR, также тестировали для сравнения. Для всех трех тестированных онРНК, для VP64-RTA показана наилучшая активация гена из шести активирующих групп, соответствующих пределу размера вектора AAV.
[0077] Фигура 3. Активация гена FGF21 посредством девяти активирующих групп
Функцию активации шести новых активирующих групп тестировали с использованием трех различных онРНК (FGF-1, -2 и -3), нацеленных на промоторную область FGF21 человека. Три активирующие группы, VP64, VP160 и VPR, также тестировали для сравнения. Для всех трех тестированных онРНК, для VP64-RTA показана наилучшая активация гена из шести активирующих групп, соответствующих пределу размера вектора AAV.
[0078] Фигура 4. Активация гена GCG посредством девяти активирующих групп
Функцию активации шести новых активирующих групп тестировали с использованием трех различных онРНК (GCG-1, -2 и -3), нацеленных на промоторную область GCG человека. Три активирующие группы, VP64, VP160 и VPR, также тестировали для сравнения. Для всех трех тестированных онРНК, для VP64-RTA показана наилучшая активация гена из шести активирующих групп, соответствующих пределу размера вектора AAV.
[0079] Фигура 5. Активация гена MyD88 посредством VP64-мини-RTA и VP64-микро-RTA
Функцию активации VP64-мини-RTA (164 а.к.) и VP64-микро-RTA (140 а.к.) сравнивали для промотора MYD88 человека. Для VP64-микро-RTA показан уровень активации, сходный с VP64-мини-RTA. Использовали gMYD88_2.
[0080] Заключение
VP64-мини-RTA (мини-VR; 167 а.к., 501 п.о.) и VP64-микро-RTA (микро-VR; 140 а.к., 420 п.о.) авторов настоящего изобретения являются достаточно небольшими, чтобы соответствовать пределу размера вектора AAV (5 т.п.о.) в присутствии других элементов, таких как Cas9, онРНК и промоторы.
Таким образом, VP64-мини-RTA и VP64-микро-RTA являются мощными группами для использования с технологией CRISPR и системой доставки AAV.
[0081] Настоящая заявка основана на Предварительной патентной заявке США серийный No. 62/715432 (дата подачи: 7 августа 2018 г.), полное содержание которой приведено в настоящем описании в качестве ссылки.
| название | год | авторы | номер документа |
|---|---|---|---|
| НАПРАВЛЯЕМАЯ РНК РЕГУЛЯЦИЯ ТРАНСКРИПЦИИ | 2014 |
|
RU2690935C2 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМА CRISPR/CAS9 II ТИПА И ЕЕ ПРИМЕНЕНИЕ | 2022 |
|
RU2794774C1 |
| МОДУЛЯТОРЫ ТАУ И СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ИХ ДОСТАВКИ | 2017 |
|
RU2789459C2 |
| РЕДАКТИРОВАНИЕ ГЕНОВ С ИСПОЛЬЗОВАНИЕМ МОДИФИЦИРОВАННОЙ ДНК С ЗАМКНУТЫМИ КОНЦАМИ (зкДНК) | 2018 |
|
RU2811724C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ЛЕЧЕНИЯ БОЛЕЗНИ ХАНТИНГТОНА | 2015 |
|
RU2691102C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ЛЕЧЕНИЯ БОЛЕЗНИ ХАНТИНГТОНА | 2014 |
|
RU2693891C1 |
| НУКЛЕАЗА PaCas9 | 2018 |
|
RU2706298C1 |
| ОРТОГОНАЛЬНЫЕ БЕЛКИ CAS9 ДЛЯ РНК-НАПРАВЛЯЕМОЙ РЕГУЛЯЦИИ И РЕДАКТИРОВАНИЯ ГЕНОВ | 2021 |
|
RU2771583C1 |
| ИСКУССТВЕННАЯ МОДИФИКАЦИЯ ГЕНОМА ДЛЯ РЕГУЛЯЦИИ ЭКСПРЕССИИ ГЕНА | 2018 |
|
RU2767201C2 |
| ОРТОГОНАЛЬНЫЕ БЕЛКИ CAS9 ДЛЯ РНК-НАПРАВЛЯЕМОЙ РЕГУЛЯЦИИ И РЕДАКТИРОВАНИЯ ГЕНОВ | 2014 |
|
RU2704981C2 |
Настоящее изобретение относится к области биотехнологии, в частности к новым инструментам для технологии CRISPR/Cas. Изобретение раскрывает новый активатор транскрипции, содержащий комплекс VP64 и участка активации транскрипции из R-трансактиватора (RTA), длина последовательности которого была подобрана и оптимизирована таким образом, чтобы он поддавался сборке в аденоассоциированном вирусном (AAV) векторе, сохраняя при этом свою способность к активации процесса транскрипции генетической информации. 6 н. и 6 з.п. ф-лы, 5 ил., 4 табл.
1. Активатор транскрипции, состоящий из не более чем 200 аминокислот и содержащий
VP64 или его вариант, имеющий способность к активности транскрипции, и
участок активации транскрипции из RTA или его варианта, имеющего способность к активности транскрипции,
где указанный VP64 или его вариант, имеющий способность к активности транскрипции, содержит
(1) аминокислотную последовательность, показанную в SEQ ID NO: 1,
(2) аминокислотную последовательность из (1), где 1 или несколько аминокислот делетированы, замещены и/или добавлены, или
(3) аминокислотную последовательность, на 90% или более идентичную аминокислотной последовательности из (1), и
где указанный участок активации транскрипции из RTA или его варианта, имеющего способность к активности транскрипции, содержит
(4) последовательность, показанную в SEQ ID NO: 2,
(5) последовательность, показанную в SEQ ID NO: 3,
(6) аминокислотную последовательность из (4) или (5), где 1 или несколько аминокислот делетированы, замещены и/или добавлены, или
(7) аминокислотную последовательность, на 90% или более идентичную аминокислотной последовательности из (4) или (5).
2. Комплекс, активирующий транскрипцию целевого гена в ДНК, содержащий модуль, узнающий последовательность нуклеиновой кислоты, специфически связывающийся с нуклеотидной последовательностью-мишенью в двухцепочечной ДНК, и активатор транскрипции по п. 1, связанные друг с другом.
3. Комплекс по п. 2, где указанный модуль, узнающий последовательность нуклеиновой кислоты, содержит эффекторный белок CRISPR, лишенный способности расщеплять по меньшей мере одну цепь двухцепочечной ДНК.
4. Комплекс по п. 3, где указанный эффекторный белок CRISPR лишен способности расщеплять обе цепи двухцепочечной ДНК.
5. Комплекс по п. 3 или 4, где эффекторный белок CRISPR происходит из Staphylococcus aureus или Campylobacter jejuni.
6. Нуклеиновая кислота, кодирующая активатор транскрипции по п. 1.
7. Нуклеиновая кислота, кодирующая комплекс по любому из пп. 2-5.
8. Экспрессионный вектор, содержащий нуклеиновую кислоту по п. 6 или 7.
9. Вектор по п. 8, где указанный вектор представляет собой аденоассоциированный вирусный вектор.
10. Способ активации транскрипции целевого гена в клетке, включающий стадию введения комплекса по любому из пп. 2-5 или вектора по п. 8 или 9 в клетку.
11. Способ по п. 10, где клетка представляет собой клетку млекопитающего.
12. Способ по п. 11, где указанное млекопитающее представляет собой человека.
| CHAVEZ A | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Способ гальванического снятия позолоты с серебряных изделий без заметного изменения их формы | 1923 |
|
SU12A1 |
| HARDWICK J.M | |||
| et al.: "The Epstein-Barr virus R transactivator (Rta) contains a complex, potent activation domain with properties different from those of VP16", J.Virol., 1992, v | |||
| Приспособление для соединения пучка кисти с трубкою или втулкою, служащей для прикрепления ручки | 1915 |
|
SU66A1 |
| KIM E | |||
| et al.: "In vivo genome | |||
Авторы
Даты
2023-08-01—Публикация
2019-08-06—Подача