Изобретение относится к системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, и имеет отношение к области техники редактирования генома.
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
Технология редактирования генов позволяет модифицировать точки локализации последовательности ДНК. Например, первое поколение инструментов редактирования генома, нуклеазы цинковые пальцы (ZFN), второе поколение инструментов редактирования генома, такие как эффекторные нуклеазы, подобные активаторам транскрипции (TALEN), могут использоваться для трансформации геномов-мишеней. Однако эти способы сложны в конструировании, непросты в производстве, дороги и не универсальны.
Система CRISPR (короткие палиндромные повторы, регулярно расположенные группами)/Cas (CRISPR-ассоциированный белок) представляет собой врожденную иммунную систему, полученную из архей и бактерий, и служит инструментом редактирования генома третьего поколения. В отличие от предыдущих инструментов редактирования генома (распознавание белок-ДНК), этот метод использует принцип комплементарной пары оснований нуклеиновых кислот для определения последовательности ДНК-мишени и направления эффекторного белка Cas для выполнения сайт-специфического расщепления и отличается широкими возможностями применения, простой конструкцией, низкой стоимостью и высокой эффективностью. Белок Cas содержит множество различных эффекторных доменов, которые участвуют в различных процессах, таких как распознавание нуклеиновых кислот, стабилизация сложных структур и гидролиз фосфодиэфирных связей ДНК. Среди них система CRISPR/Cas9 типа II, полученная из Streptococcus pyogene Cas (SpCas9), стала наиболее широко используемой системой CRISPR/Cas вследствие ее высокой эффективности к расщеплению. Эта система идентифицирует и расщепляет последовательность мотива, смежного с протоспейсером (РАМ), то есть «NGG» на полинуклеотиде-мишени, оставляя выступ с тупым концом, влияя на редактирование генома.
В больших и разнообразных метагеномах, содержащих некультивируемые или даже неизвестные микроорганизмы, может существовать большое количество неисследованных систем CRISPR/Cas9, активность которых в прокариотах и эукариотах, а также в средах in vitro требует подтверждения.
В 2015 году команда доктора Бьюнг-Чана Кима выделила новый анаэробный штамм AL017 из фекалий лабораторных мышей линии C57BL/6J и проанализировала филогенетическую связь штамма с 16SrRNA генетической последовательностью прокариот и, при этом, обнаружила, что штамм тесно связан с Holdemanellabiformis DSM 3989Т, Faecalicoccuspleomorphus АТСС 29734Т, Faecalitaleacylindroides АТСС 27803Т и Allobaculumstercoricanis DSM 13633Т (гомология с последовательностей составляет 87,4%, 87,3%, 86,9% и 86,9% соответственно). На основании многочисленных таксономических данных этот вид считается новым родом семейства Erysipelothricaceae и назван Faecalibaculum rodentium Gen.nov., sp.nov. За последние пять лет ученые разных стран мира проводили исследования этого штамма в двух областях: микробная среда кишечника и диета с высоким содержанием жиров, и микробная среда кишечника и онкогенез. Однако не сообщалось о подобных исследованиях в области редактирования генома.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Цель изобретения состоит в том, чтобы преодолеть недостатки предшествующего уровня техники и представить систему редактирования генома CRISPR/Cas9 типа II, полученную из Faecalibaculum rodentium, где система редактирования генома имеет новые физические и химические свойства и может идентифицировать множество различных последовательностей РАМ (мотив, смежный с протоспейсером), включая NGTA и NNTA (N представляет собой А, С, G или Т).
Для достижения цели техническое решение, применяющееся в изобретении, заключается в следующем.
Система редактирования генома CRISPR/Cas9 типа II, отличающаяся тем, что система содержит белок Cas9, вспомогательные белки, crPHK (CRISPR РНК) и tracrPHK (трансактивированную CRISPR РНК) в функциональной форме комплекса РНП (комплекс рибонуклеопротеин) белка Cas9 и направляющей РНК, образованной путем гибридизации crPHK с tracrPHK;
при этом белок Cas9 представляет собой ДНК-эндонуклеазу, и белок Cas9 имеет аминокислотную последовательность, как показано в SEQ ID NO: 1, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98%, или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 1.
Белок Cas9 расщепляет двухцепочечную ДНК, комплементарную crPHK выше последовательности РАМ, с помощью нуклеазного домена, где нуклеазный домен выбран из HNH-подобного нуклеазного домена, RuvC-подобного нуклеазного домена или их комбинации.
Предпочтительно система редактирования генома CRISPR/Cas9 типа II получена из Faecalibaculum rodentim.
В некоторых вариантах осуществления термин «рибонуклеопротеиновый комплекс» предпочтительно относится к «белковому комплексу FrCas9» в соответствии с изобретением.
Мутации на нескольких ключевых аминокислотных сайтах специфического белка Cas9 подробно объясняются следующим образом. Мутация Е в А в положении 796 аминокислоты приведет к образованию нуклеазы никазы. Мутация N в А в положении 902 аминокислоты приведет к образованию нуклеазы никазы. Мутация Н в А в положении 1010 аминокислоты приведет к образованию нуклеазы никазы. Мутация D в А в положении 1013 аминокислоты приведет к образованию нуклеазы никазы.
Одновременная мутация Е в А в положении 796 аминокислоты и D в А в положении 1013 аминокислоты приведет к образованию нуклеазы Cas9, которая не обладает способностью к расщеплению, но сохраняет способность к связыванию (т.е. мертвая Cas9).
Вспомогательные белки включают вспомогательный белок Cas1, вспомогательный белок Cas2 и вспомогательный белок Csn2;
при этом вспомогательный белок Cas1 имеет аминокислотную последовательность, как показано в SEQ ID NO: 2, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 2;
при этом вспомогательный белок Cas2 имеет аминокислотную последовательность, как показано в SEQ ID NO: 3, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 3;
при этом вспомогательный белок Csn2 имеет аминокислотную последовательность,
как показано в SEQ ID NO: 4, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 4.
CRISPR РНК генерируется путем транскрипции массива CRISPR и имеет последовательность РНК, как показано в SEQ ID NO: 5, или последовательность РНК, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 5; при этом массив CRISPR содержит последовательность прямого повтора и спейсерную последовательность, при этом последовательность прямого повтора имеет последовательность нуклеиновой кислоты, как показано в SEQ ID NO: 6, или последовательность нуклеиновой кислоты, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 6; и при этом спейсерная последовательность имеет последовательность нуклеиновой кислоты, как показано в SEQ ID NO: 7, или последовательность нуклеиновой кислоты, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 7.
Трансактивированная CRISPR РНК содержит последовательность, комплементарную последовательности прямого повтора CRISPR РНК, и трансактивированная CRISPR РНК имеет последовательность нуклеиновой кислоты, как показано в SEQ ID NO: 8, или последовательность нуклеиновой кислоты, имеющую по меньшей мере 70%, 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 8.
Направляющая РНК, образованная путем гибридизации crPHK с tracrРНК, имеет каркас, состоящий из последовательности 7-24 нуклеотидов последовательности прямого повтора crPHK и последовательности tracrPHK; предпочтительно две части слиты линкером "GAAA", "TGAA" или "АААС" с образованием каркаса sgPHK; предпочтительно каркас sgPHK, образованный путем слияния последовательности прямого повтора crPHK из 20 нуклеотидов и полноразмерной последовательности tracrPHK с помощью «GAAA», показан в SEQ ID NO: 9, и на этой основе предпочтительно высокоэффективный вариант каркаса sgPHK представляет собой
каркас sgPHK, образованный путем слияния первых 18 14 нуклеотидов последовательности прямого повтора crPHK и последних 69-65 нуклеотидов tracrPHK с помощью линкерной последовательности («GAAA» и т.д.), предпочтительно выбран из следующих пяти каркасов:
(1) каркас sgPHK длиной 91 нуклеотид, который содержит 18 нуклеотидов последовательности прямого повтора и 69 нуклеотидов tracrPHK, как показано в SEQ ID NO: 10;
(2) каркас sgPHK длиной 89 нуклеотидов, который содержит 17 нуклеотидов последовательности прямого повтора и 68 нуклеотидов tracrPHK, как показано в SEQ ID NO: 11;
(3) каркас sgPHK длиной 87 нуклеотидов, который содержит 16 нуклеотидов последовательности прямого повтора и 67 нуклеотидов tracrPHK, как показано в SEQ ID NO: 12;
(4) каркас sgPHK длиной 85 нуклеотидов, который содержит 15 нуклеотидов последовательности прямого повтора и 66 нуклеотидов tracrPHK, как показано в SEQ ID NO: 13;
(5) каркас sgPHK длиной 83 нуклеотида, который содержит 14 нуклеотидов последовательности прямого повтора и 65 нуклеотидов tracrPHK, как показано в SEQ ID NO: 14;
необязательно каркас sgPHK имеет последовательность нуклеиновой кислоты, имеющую по меньшей мере 70%, 80%, 85%, 90%, 95%, 98% или 99% гомологии с любой из SEQ ID NO: 9-14.
Система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, связывает или расщепляет специфическую ДНК в биологическом процессе (например, в процессе редактирования генома) путем распознавания комплементарного спаривания направляющей РНК и мишени специфической ДНК, и при этом длина спаренной связывающей части направляющей РНК и мишени специфической ДНК составляет от 14 до 30 пар оснований (предпочтительно от 20 до 23 пар оснований); где специфическая ДНК представляет собой ДНК прокариот или эукариот.
Длина спаренной связывающей части направляющей РНК и мишени специфической ДНК составляет 21 пару оснований, 22 пары оснований или 23 пары оснований, при этом комплекс РНП высокочувствителен к несоответствию оснований на 14 пар оснований вблизи мотива, смежного с протоспейсером, и 14 пар оснований - это начальная область.
Мотив, смежный с протоспейсером, необходимый для функции связывания или расщепления ДНК, расположен на 5'-NNTA-3' ниже последовательности распознавания направляющей PHK/sgPHK.
В изобретении также предлагается применение вышеупомянутой системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, для редактирования ДНК, CRISPR активации или интерференции. Предпочтительно ДНК представляет собой прокариотическую или эукариотическую ДНК.
В качестве еще одного аспекта настоящего изобретения в нем дополнительно предлагается применение вышеуказанной системы редактирования генома CRISPR/Cas9 типа II для получения никазы, мертвой Cas9, базового редактора или основного редактора. Предпочтительно никазу, мертвый Cas9, базовый редактор или основной редактор получают в прокариотах или эукариотах.
По сравнению с предшествующим уровнем техники изобретение обеспечивает следующие полезные эффекты.
(1) Согласно изобретению, посредством биоинформатического анализа в Faecalibaculum rodentium обнаружена система редактирования генома CRSIPR/Cas9 типа II, указанная система редактирования генома применена для редактирования прокариотических или эукариотических генов и предоставляет новую возможность применения набора инструментов для редактирования генома.
(2) В изобретении представлена система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, которая обладает новыми физическими и химическими свойствами и может идентифицировать множество различных последовательностей РАМ, при этом специфическая последовательность РАМ, распознаваемая системой редактирования генома, расположена на 5'-NNTA-3' ниже последовательности распознавания направляющей PHK/sgPHK.
(3) В изобретении представлена система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, которая имеет высокую эффективность расщепления на одном и том же сайте ДНК по сравнению с наиболее распространенным SpCas9, в то время как отклонение от мишени ниже, чем у наиболее распространенного SpCas9, так что это более безопасный и эффективный инструмент редактирования генома.
(4) В изобретении представлена система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, в которой РАМ, распознаваемый на уровне системы редактирования генома, имеет характеристики палиндрома, и поэтому мишени распределяются «хвост к хвосту» в геноме, который имеет более высокую плотность, чем SpCas9.
(5) В изобретении представлена система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, которая обладает высокой гибкостью и может быть преобразована в базовый редактор и главный редактор, а также представляет собой инструмент редактирования генома, который можно широко применять при различных сценариях.
КРАТКОЕ ОПИСАНИЕ ИЗОБРАЖЕНИЙ
На Фигуре 1 представлена композиционная схема системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением.
На Фигуре 2 представлена структурная схема белка Cas9 системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением.
На Фигуре 3 (3A-3F) представлена схема прогнозирования вторичной структуры РНК и схема оптимального каркаса sgPHK для направляющей молекулы РНК, распознаваемой системой редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением. На Фигуре 3А показана полноразмерная gPHK длиной 95 нуклеотидов. На Фигуре 3В показан каркас sgPHK длиной 91 нуклеотид, который включает 18 нуклеотидов последовательности прямого повтора и 69 нуклеотидов tracrPHK. На Фигуре 3С показан каркас sgPHK длиной 89 нуклеотидов, который включает 17 нуклеотидов последовательности прямого повтора и 68 нуклеотидов tracrPHK. На Фигуре 3D показан каркас sgPHK длиной 87 нуклеотидов, который включает 16 нуклеотидов последовательности прямого повтора и 67 нуклеотидов tracrPHK. На Фигуре 3Е показан каркас sgPHK длиной 85 нуклеотидов, который включает 15 нуклеотидов последовательности прямого повтора и 66 нуклеотидов tracrPHK. На Фигуре 3F показан каркас sgPHK длиной 83 нуклеотида, который включает 14 нуклеотидов последовательности прямого повтора и 65 нуклеотидов tracrPHK.
На Фигуре 4 представлена диаграмма прокариотических последовательностей РАМ системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением.
На Фигуре 5 представлена схематическая диаграмма экспериментов по интерференции прокариот системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением.
На Фигуре 6 представлена схематическая диаграмма эукариотического расщепления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением. Фигура 6А представляет собой гелевую диаграмму ОДН-ПЦР. Фигура 6 В представляет собой пик последовательности Сэнгера.
На Фигуре 7 представлена диаграмма, показывающую оптимальную длину sgPHK в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением. Фигура 7А представляет собой график, показывающий эффективность расщепления sgPHK различной длины в сайте НЕК293 SITE2-T2. Фигура 7 В представляет собой график, показывающий эффективность расщепления sgPHK различной длины в сайте DNMT1-T3. Фигура 7С представляет собой график, показывающий эффективность расщепления sgPHK различной длины в сайте RNF2-T6.
На Фигуре 8 представлена схематическая диаграмма, показывающая оптимальную длину последовательности распознавания sgPHK системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением.
На Фигуре 9 представлена схематическая диаграмма, показывающая сравнение эффективности нацеливания и отклонения от мишени GUIDE-seq в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением по сравнению с SpCas9.
На Фигуре 10 представлена диаграмма, показывающая экспериментальные результаты базового редактора FrCas9-BE; при этом Фигура 10А представляет собой схематическую диаграмму и результаты подтверждения формы никазы FrCas9 с помощью ПЦР с точкой разрыва ОДН; Фигура 10В представляет собой окно редактирования для nFrCas9-BE4Gram; Фигура 10С представляет собой окно редактирования для nFrCas9-АВЕ7.10; Фигура 10D представляет собой диаграмму Венна патогенных мутаций в базе данных ClinVar, которая может быть однозначно скорректирована базовыми редакторами SpCas9 и FrCas9; на Фигуре 10Е показана эффективность редактирования С>Т для FrCas9-BE4Gam с одновременным использованием 2 sgPHK «хвост к хвосту».
На Фигуре 11 представлена диаграмма, показывающая распределение мишеней FrCas9 и SpCas9 в геноме человека, при этом на Фигуре 11А показано распределение 5'-GG-3' (представляющего SpCas9 РАМ) в геноме человека GRCh38; на Фигуре 11В показано распределение 5'-ТА-3' (представляющего FrCas9 РАМ) в геноме человека GRCh38.
На Фигуре 12 показано применение FrCa89-специфического, нацеленного на ТАТА-бокс, при CRISPR интерференции и CRISPR активации, где Фигура 12А представляет собой схематическую диаграмму ТАТА-бокса гена АВСА1, на который нацелен FrCas9; На Фигуре 12В показана CRISPR интерференция FrCas9 и SpCas9 путем нацеливания на ТАТА-бокс и CRISPR активация FrCas9 и SpCas9 путем нацеливания на ТАТА-бокс; На Фигуре 12С показана CRISPR активация FrCas9 и SpCas9 путем нацеливания на ТАТА-бокс.
На Фигуре 13 представлена схема системы редактирования генома по технологии Праймированного Редактирования (Prime Editing).
На Фигуре 14 показан эффект редактирования генома системы Праймированного Редактирования FrCas9-PE, верифицированный секвенированием по Сэнгеру, где измеренной последовательностью была CGAACACTCAAGGTAAT (SEQ ID NO: 33).
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Чтобы лучше объяснить цели, технические решения и преимущества изобретения, изобретение будет дополнительно описано ниже со ссылкой на конкретные варианты осуществления.
Система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, включает белок Cas9, вспомогательные белки, CRISPR РНК и трансактивированную CRISPR РНК, как показано на Фигуре 1. Белок Cas9 представляет собой ДНК-эндонуклеазу и имеет аминокислотную последовательность, как показано в SEQ ID NO: 1, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 1.
В качестве предпочтительного варианта осуществления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением белок Cas9 расщепляет двухцепочечную ДНК, комплементарную crPHK выше последовательности РАМ, с помощью нуклеазного домена, как показано на Фигуре 2. Нуклеазный домен выбран из HNH-подобного нуклеазного домена или RuvC-подобного нуклеазного домена.
Белок Cas9 (Faecalibaculum rodentium Cas9), сокращенно обозначаемый как белок FrCas9, содержит 1372 аминокислоты (SEQ ID NO: 1) и представляет собой многодоменную и многофункциональную ДНК-эндонуклеазу. Он эффективно расщепляет двухцепочечную ДНК, комплементарную sgPHK выше последовательности РАМ, с помощью нуклеазного домена, например, расщепляет цепь ДНК, комплементарную последовательности sgPHK, с помощью HNH-подобного нуклеазного домена или расщепляет некомплементарную цепь ДНК с помощью RuvC-подобного нуклеазного домена. При этом мутация Е в А в положении 796 аминокислоты приведет к образованию нуклеазы никазы; мутация N в А в положении 902 аминокислоты приведет к образованию нуклеазы никазы; мутация Н в А в положении 1010 аминокислоты приведет к образованию нуклеазы никазы; мутация D в А в положении 1013 аминокислоты приведет к образованию нуклеазы никазы; одновременная мутация Е в А в положении 796 аминокислоты и D в А в положении 1013 аминокислоты приведет к образованию нуклеазы Cas9, которая не обладает способностью к расщеплению, но сохраняет способность к связыванию.
В качестве предпочтительного варианта осуществления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением вспомогательные белки включают вспомогательный белок Cas1, вспомогательный белок Cas2 и вспомогательный белок Csn2. Вспомогательный белок Cas1 имеет аминокислотную последовательность, как показано в SEQ ID NO: 2, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 2. Вспомогательный белок Cas2 имеет аминокислотную последовательность, как показано в SEQ ID NO: 3, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 3. Вспомогательный белок Csn2 имеет аминокислотную последовательность, как показано в SEQ ID NO: 4, или аминокислотную последовательность, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 4.
Вспомогательные белки Casl, Cas2 и Csn2 в соответствии с изобретением участвуют в захвате экзогенного гена и созревании crPHK.
В качестве предпочтительного варианта осуществления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением CRISPR РНК создается путем транскрипции массива CRISPR. CRISPR РНК имеет последовательность РНК, как показано в SEQ ID NO: 5, или последовательность РНК, имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологии с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 5. Массив CRISPR содержит последовательность прямого повтора и спейсерную последовательность. Последовательность прямого повтора представлена в SEQ ID NO: 6. Спейсерная последовательность представлена в SEQ ID NO: 7.
CRISPR РНК (crPHK) в соответствии с изобретением направляет белок Cas на распознавание вторгшегося чужеродного генома в форме, комплементарной основаниям. Когда бактерии подвергаются вторжению бактериофага или вируса, короткий сегмент чужеродной ДНК интегрируется в качестве нового спейсера между последовательностями повтора спейсеров CRISPR в хромосоме хозяина, тем самым обеспечивая генетическую запись инфекции. Когда в организм снова вторгается чужеродный ген, массив CRISPR транскрибирует и продуцирует прекурсор crPHK (pre-crPHK) со спейсерной последовательностью на 5'-конце и длиной 30 пар оснований, комплементарной последовательности от чужеродного гена. 3'-конец представляет собой последовательность повтора длиной 36 пар оснований.
В качестве предпочтительного варианта осуществления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением трансактивированная CRISPR РНК содержит последовательность, комплементарную последовательности прямого повтора CRISPR РНК, как показано в SEQ ID NO: 8.
Трансактивированная crPHK (tracrPHK) согласно настоящему изобретению представляет собой РНК, которая не кодируется белком, и участвует в созревании crPHK и образовании sgPHK. Под действием tracrPHK и нуклеазы Cas9 pre-crPHK удаляет от 0 до 16 нуклеотидов выше спейсерной последовательности и от 12 до 29 нуклеотидов ниже последовательности повтора с образованием зрелой crPHK и связывает tracrPHK с образованием комплекса tracrPHK-crPHK, который содержит часть, распознающую чужеродную последовательность ДНК, и имеет длину от 14 до 30 пар оснований. Тетрапетля (например, последовательность «GAAA», «TGAA» или «АААС») из четырех оснований может быть добавлена между нижним концом crPHK и верхним концом tracrPHK для связывания tracrPHK и crPHK, чтобы образовать sgPHK, содержащую две выпуклые и три дуплексные структуры по направлению к верхнему концу а также структуру "шпилька" по направлению к нижнему концу, которую можно дополнительно разделить на часть, которая распознает чужеродную последовательность ДНК, и часть каркаса.
Расщепление эндонуклеазой можно дополнительно оптимизировать, регулируя длину части sgPHK, распознающей последовательность чужеродной ДНК, и длину tracrPHK. Ранние эксперименты по настоящему изобретению доказывают, что оптимальная длина части sgPHK, распознающей экзогенную последовательность ДНК, составляет 21 пару оснований, 22 пары оснований или 23 пары оснований (как показано на Фигуре 8), а оптимальная длина каркасной части - одна из следующих 5 типов, как показано на Фигурах 3 и 7: 91 нуклеотид (18 нуклеотидов прямого повтора crPHK+69 нуклеотидов tracrPHK), 89 нуклеотидов (17 нуклеотидов прямого повтора crPHK+68 нуклеотидов tracrPHK), 87 нуклеотидов (16 нуклеотидов прямого повтора crPHK+67 нуклеотидов). tracrPHK), 85 нуклеотидов (15 нуклеотидов прямого повтора crPHK+66 нуклеотидов tracrPHK) и 83 нуклеотида (14 нуклеотидов прямого повтора crPHK+65 нуклеотидов tracrPHK).
В качестве предпочтительного варианта осуществления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, связывает или расщепляет структуры ДНК, участвующие в процессе редактирования генома.
В качестве предпочтительного варианта осуществления системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением ДНК представляет собой ДНК прокариот или эукариот.
В некоторых вариантах осуществления изобретения FrCas9, согласно настоящему изобретению, может распознавать множество двухцепочечных разрывов ДНК (DSB), прилегающих к протоспейсеру (РАМ), непосредственно ниже последовательности нацеливания. Белку FrCas9 необходимы два важных фактора для распознавания последовательности нацеливания: один представляет собой нуклеотид, комплементарный спейсеру crPHK; а другой представляет собой последовательность мотива, смежного с протоспейсером (РАМ), примыкающую к комплементарной последовательности. Эксперимент по деплеции показывает, что FrCas9 оказывая эффект расщепления в прокариотической системе, и предварительно верифицирует третье и четвертое положения последовательности РАМ, распознаваемые этой недавно открытой системой CRISPR/Cas9 типа II, которые представляют собой ТА, как показано на Фигуре 4. Дополнительно подтверждено экспериментом по интерференции, что РАМ ниже последовательности нацеливания, распознаваемой FrCas9, представляет собой 5'-NNTA-3', как показано на Фигуре 5. Все вышеперечисленные РАМ верифицированы посредством эксперимента с эукариотами, как показано на Фигуре 6. Путем искусственного конструирования спейсерной последовательности в crPHK, эта система CRISPR-Cas9 может нацеливаться почти на все последовательности ДНК в геноме, представляющие интерес, производя сайт-специфический двухцепочечный разрыв с тупым концом (DSB). Восстановление DSB негомологичными концами приводит к небольшим случайным вставками/удалениями (инделам) в сайте расщепления для инактивации интересующего гена. В качестве альтернативы, с помощью высокоточной гомологичной репарации точные геномные модификации в сайте DSB могут быть выполнены с использованием шаблонов гомологичной репарации.
Большинство генетических заболеваний человека представляют собой мутации с одним основанием, которые не поддаются лечению традиционными способами. Базовый редактор - один из новейших и наиболее эффективных способов точного редактирования генома. Он характеризуется использованием белка CRISPR-Cas в форме разрыва для обнаружения специфических ДНК-мишеней и использованием ДНК-дезаминазы для модификации и мутации этой ДНК-мишени с целью исправления пораженных оснований без образования двухцепочечной фрагментации ДНК. В некоторых вариантах осуществления комбинация Е796А nFrCas9 с оптимизированным редактором цитидиновых оснований четвертого поколения BE4Gam может успешно сконструировать редактор цитозиновых оснований Е796А nFrCas9-BEGam (СВЕ), так, что пара оснований C:G в ДНК может быть мутирована в Т:А. Комбинация Е796А nFrCas9 с редактором адениновых оснований 7-го поколения АВЕ7.10 может успешно сконструировать редактор адениновых оснований Е796А nFrCas9-BABE7.10 (ABE), так, что пара оснований А:Т в ДНК может быть мутирована в G:C.
В некоторых вариантах осуществления специфический РАМ FrCas9 по настоящему изобретению представляет собой NNTA со специфическим нацеливанием на ТАТА-бокс (один из элементов, составляющих эукариотический промотор), так, что FrCas9 специфически нацеливается на ТАТА-бокс для создания уникального эффекта CRISPR интерференции (CRISPRi) / CRISPR активации (CRISPRa). Из них CRISPRi может быть достигнута путем прямого нацеливания на расщепление ТАТА-бокса с помощью активной формы FrCas9 и разрушение ТАТА-бокса или путем прямого связывания с ТАТА-боксом с помощью dFrCas9 (т.е., мертвого Cas9) без осуществления расщепления. CRISPRa может быть достигнута, помимо прочего, путем нацеливания dFrCas9-VP64 на сайт ТАТА-бокса.
Праймированное Редактирование (РЕ) - это совершенно новый инструмент для точного редактирования генома. С помощью этой технологии можно осуществить свободную замену единого основания и точную вставку и удаление нескольких оснований, значительно уменьшая вредные побочные продукты, создаваемые инделами в процессе редактирования генома, и значительно повышая точность редактирования. Принято считать, что это является значительным достижением в редактировании генома. В некоторых вариантах осуществления FrCas9 по настоящему изобретению слит с обратной транскриптазой, и соответствующая regPHK может быть сконструирована в соответствии с ее последовательностью РАМ для создания системы редактирования генома FrCas9-PE. В частности, две последовательности добавляются к последовательности regPHK на 3'-конце. Первая последовательность представляет собой сайт связывания праймера (PBS), который может быть комплементарен концу разорванной цепи-мишени ДНК для инициации процесса обратной транскрипции, а вторая последовательность представляет собой матрицу обратной транскрипции (RT матрица), которая осуществляет мутацию точки-мишени или мутацию вставки/удаления для достижения точного редактирования генома.
Еще одной целью изобретения является обеспечение применения системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, для редактирования прокариотических или эукариотических генов, CRISPR активации или CRISPR интерференции.
В качестве предпочтительного варианта применения в соответствии с изобретением система редактирования генома CRISPR/Cas9 типа II, полученная из Faecalibaculum rodentium, используется для связывания или расщепления структур ДНК, участвующих на уровне ДНК.
Пример 1
В примере была спрогнозирована вторичная структура РНК направляющей молекулы РНК, распознаваемая системой редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением, структура РНК после транскрипции tracrPHK и повтора была спрогнозирована с помощью моделирования процесса связывания двух РНК, полученная вторичная структура РНК показана на Фигуре 3.
(1) Материалы: спрогнозированные последовательности tracrPHK и повторов, а также спрогнозированные последовательности анти-повторов.
(2) Программное обеспечение: NUPACK (http://www.nupack.org/partition/new)
(3) Способ прогнозирования: процесс взаимодействия 1 мкл каждой из двух РНК при 37°С in vitro моделировался с помощью онлайн-приложения NUPACK, в результате была спрогнозирована вторичная структура полученной композиции РНК для получения вторичной структуры РНК, как показано на Фигуре 2.
Как показано на на Фигуре 3, pre-crPHK удаляла от 0 до 16 нуклеотидов по выше спейсерной последовательности и от 12 до 29 нуклеотидов ниже последовательности повтора под действием tracrPHK и нуклеазы Cas9 с образованием зрелой crPHK, которая сливалась с tracrPHK с образованием комплекса tracrPHK-crPHK, содержащего экзогенную последовательность ДНК, комплементарную спейсерной последовательности, при этом длина последовательности составляла от 14 до 30 пар оснований. Они были соединены с образованием sgPHK путем добавления тетрапетли из четырех оснований между нижним концом crPHK и верхним концом tracrPHK, содержащей две выпуклые и три дуплексные структуры по направлению к верхнему концу и три структуры "шпилька" по направлению к нижнему концу.
Пример 2
В примере мотив, смежный с протоспейсером (РАМ), распознаваемый системой редактирования генома CRTSPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в прокариотической системе представляет собой 5'-NNTA-3'.
(1) Материалы: гены, относящиеся к системе редактирования генома CRISPR/Cas9, спрогнозированные путем вышеописанной реализации.
(2) Способ проверки: в этом примере была сконструирована прокариотическая система проверки в отношении системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением для проверки эффекта ее расщепления и предварительного исследования распознанной последовательности РАМ с помощью второго поколения технологии секвенирования, при этом результат показан на Фигуре 4.
Подробные протоколы приведены ниже.
(a) Систему редактирования генома CRISPR/Cas9 типа II, полученного из Faecalibaculum rodentium (содержащую эндонуклеазу Cas9, вспомогательные белки Casl, Cas2, Csn2, массив CRISPR и некодирующую РНК tracrPHK), в соответствии с изобретением встраивали в вектор pACYC184, где белок Cas9 был подвергнут оптимизации кодонов Escherichia coli. Последовательности естественных спейсеров и последовательности спейсеров в библиотеке добавляли в массив CRISPR, а сильный гетерологичный промотор J23119 добавляли к белку Cas9 и массиву CRISPR для конструирования прокариотической экспрессионной плазмиды pACYC184-FrCas9.
(b) Семь случайных оснований (всего 16 384 вставки) были добавлены в спейсерную последовательность 3' библиотеки. Два сайта рестрикционных эндонуклеаз EcoRI и Ncol были выбраны из поликлонального MCS вектора pUC19. Библиотеку клонировали в вектор и конструировали плазмиду библиотеки мишени.
(c) Плазмиду, содержащую pACYC184-FrCas9 или пустую pACYC184 и библиотеку мишени, коэлектрически трансфицировали в Е. coli DH5a. После восстановления функций при 25°С в течение 2 ч их равномерно распределяли в среде SOB, обладающей двойной резистентностью к ампициллину натрия (100 мкг/мл) и хлорамфениколу (34 мкг/мл) для инкубации при 25°С в течение 30 часов, при этом плазмиду собирали щелочным лизисом.
(d) Была проведена ПЦР-амплификация на сайте, содержащем спейсерную последовательность и семь случайных оснований. Вторичное секвенирование проводили на двух концах продукта ПЦР путем добавления линкера. Рассчитывали значение деплеции РАМ (PPDV) относительно группы контроля отсутствия нагрузки. Последовательность РАМ системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, была создана с использованием Weblogo, где последовательность РАМ представляет собой 5'-NNTA-3'.
Фигура 3 представляет собой схематическую диаграмму консервативной последовательности РАМ, распознаваемой системой редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением в прокариотической системе. Для расчета значения деплеции РАМ (PPDV) относительно группы контроля отсутствия нагрузки был выполнен анализ секвенирования второго поколения ДНК библиотеки, полученной в ходе эксперимента по деплеции. Последовательность РАМ FrCas9, сгенерированная Weblogo, представляет собой 5'-NNTA-3'.
Пример 3
В примере посредством эксперимента по интерференции был верифицирован мотив, смежный с протоспейсером (РАМ), распознаваемый в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением, при этом были определены его способность к расщеплению на прокариотическом уровне и потенциальная способность к редактированию генома в эукариотах. Результаты экспериментов по интерференции показаны на Фигуре 5, при этом схематические диаграммы различных возможных последовательностей РАМ, распознаваемых системой редактирования генома CRISPR7Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением показаны на Фигуре 5.
(1) Материалы: pACYC184-FrCas9, плазмида библиотеки мишени, полученная в примере 2, и предварительно распознанный РАМ.
(2) Способ проверки: в этом примере мотив, смежный с протоспейсером (РАМ), распознаваемый системой редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением в прокариотической системе, дополнительно определяли с помощью интерференционного эксперимента.
Подробные протоколы приведены ниже.
(a) Всего было получено 16 комбинированных последовательностей путем добавления NNTA в положение 3' спейсерной последовательности, а плазмида-мишень была сконструирована путем клонирования в pUC19 через эндонуклеазные сайты рестрикции EcoRI и NcoI соответственно.
(b) 16 плазмид-мишеней соответственно трансфицировали в электрогенную компетентность Е. coli DH5a, содержащую FrCas9-родственные локусы, при этом плазмиды постепенно разбавляли после восстановления функций при 25°С в течение 2 часов. Плазм иды-мишени инкубировали в течение ночи при 25°С в среде SOB, обладающей двойной резистентностью к ампициллину натрия (100 мкг/мл) и хлорамфениколу (34 мкг/мл) с помощью дот-блоттинга, чтобы определить количество моноклональных бактерий.
Фигура 5 представляет собой схематическую диаграмму эксперимента по интерференции системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением. На основе NNTA, обнаруженного в эксперименте по деплеции, было сконструировано 16 плазмид-мишеней с различными комбинациями NN. Число моноклональных колоний наблюдали посредством интерференционного эксперимента. На Фигуре 5А крайний левый столбец был обозначен как одиночный FrCas9, а правая сторона была обозначена как плазмида-мишень, на которую нацелено расщепление FrCas9. Результаты показали, что количество колоний в правом столбце уменьшилось. Фигура 5В представляет собой статистическую диаграмму эффектов расщепления множества последовательностей РАМ, распознаваемых системой редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением и иллюстрирует, что количество колониеобразующих единиц (KOE/CFU) плазмид-мишеней 14 различных комбинаций NN снижено по сравнению с контрольной группой. Приведенные выше результаты показали, что мотив, смежный с протоспейсером (РАМ), распознанный недавно открытой системой CRISPR/Cas9 в прокариотической системе методом интерференционного эксперимента, представлял собой 5'-NNTA-3' (N представляет собой любое основание, выбранное из группы, состоящей из А, Т, С и G).
Пример 4
В примере посредством экспериментов по интерференции был верифицирован диапазон tracrPHK, необходимый для расщепления последовательностей-мишеней ДНК в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением, и результаты экспериментов по интерференции показаны на Фигуре 5С.
(1) Материалы: pACYC 184-FrCas9, плазмида-мишень, предварительно распознанный РАМ, полученный в примере 2.
(2) Способ проверки: в этом примере диапазон tracrPHK, необходимый для расщепления последовательности-мишени ДНК, был дополнительно определен с помощью системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в прокариотической системе.
Подробные протоколы приведены ниже.
(а) В общей сложности шесть генных последовательностей всех возможных некодирующих областей в Faecalibaculum rodentium дикого типа были клонированы в плазмиду-мишень методом клонирования Гибсона для конструирования плазмид-мишеней NC от 1 до 6, где NC6 образуется путем сплайсинга полной длины некодирующих областей, a NC от 1 до 5, соответственно, представляют возможные некодирующие области сегментов с первого по пятый. В каждую плазмиду NC был добавлен сильный гетерологичный промотор J23119 по направлению к верхней части некодирующей области, где "+" представлял собой прямую некодирующую область, а "-" представлял собой обратную некодирующую область, соответственно;
(b) Из pACYC184-FrCas9, полученного в Примере 2, были удалены все возможные некодирующие области с помощью метода гомологичной рекомбинации ПЦР, а белок, родственный CRISPR, и последовательность генов массива CRISPR были сохранены, таким образом сконструировав pACYC184-ΔFrCas9;
(c) 12 плазмид-мишеней соответственно трансфицировали в электрогенную компетентность E.coli DH5a, содержащую pACYC184-ΔFrCas9, при этом плазмиды постепенно разбавляли после восстановления функций при 25°С в течение 2 часов. Плазмиды-мишени инкубировали в течение ночи при 25°С в среде SOB, обладающий двойной резистентностью к ампициллину натрия (100 мкг/мл) и хлорамфениколу (34 мкг/ мл) с помощью дот-блоттинга, чтобы определить количество моноклональных бактерий.
Фигура 5С представляет собой схематическую диаграмму эксперимента по интерференции системы редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением при этом результаты показывают, что моноклональные колонии NC1 и NC6 были значительно меньше, чем колонии NC от 2 до 5, что указывает на то, что альтернативная некодирующая область в первом сегменте может оказать помощь FrCas9 в эффективном и нацеленном расщеплении последовательностей ДНК в Е. coli.
Пример 5
В примере посредством эксперимента с ОДН была верифицирована способность расщеплять последовательности-мишени ДНК в эукариотических клетках в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium. Результат ОДН-ПЦР показан на диаграмме на Фигуре 6А, а результат секвенирования по Сэнгеру показан на диаграмме на Фигуре 6В.
(1) Материалы: все аминокислотные последовательности, последовательности матрицы CRISPR, последовательности tracrPHK и распознанный РАМ редактируемого гена Faecalibaculum rodentium, полученные в примерах 1-4.
(2) Способ проверки: в этом примере способность расщеплять последовательности-мишени ДНК в эукариотических клетках была верифицирована посредством эксперимента с ОДН в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium.
Подробные протоколы приведены ниже.
(a) Оптимизированная последовательность белка FrCas9 человека была синтезирована и клонирована в эукариотический вектор РХ330 для создания плазмиды PX330-FrCas9.
(b) 30 пар оснований по направлению к верхнему концу NGTA, NATA, NCTA и NTTA гена RNF2 человека были выбраны в качестве последовательности-мишени. Массив CRISPR был сконструирован методом Гибсона. Сильный промотор эукариот человека U6 добавляли выше матрицы CRISPR, чтобы сконструировать плазмиду матрицы РХ330-FrCas9.
(c) Эукариотический сильный промотор U6, полученный от мыши, добавляли выше tracrPHK, показанной в примере 4, для конструирования плазмиды PX330-FrCas9-array-tracrRNA.
(d) Плазмида PX330-FrCas9-array-tracrRNA, нацеленная на различные сайты генов, сконструированная, как указано выше, с 2,5 мкг и 1,5 мкл ОДН, была электрически трансфицирована в клетки НЕК293Т в хорошем состоянии. Все клетки собирали через 72 часа для выделения ДНК.
(e) ОДН-ПЦР выполняли путем конструирования пары праймеров рядом с сайтом гена-мишени RNF2 и на последовательности ОДН, а также проводили электрофорез в агарозном геле для наблюдения за наличием полосы чтобы предварительно определить, произошло ли расщепление мишени или нет.
(f) Посредством секвенирования по Сэнгеру была верифицирована успешная вставка ОДН в сайт-мишень и было подтверждено, что FrCas9 обладает способностью редактировать ДНК-мишень в эукариотических клетках.
Результаты ОДН-ПЦР показаны на Фигуре 6А. NGTA, NATA, NCTA и NTTA имели полосы-мишени правильного размера. Как показано на Фигуре 8 с результатами секвенирования по Сэнгеру, положение вставки ОДН происходило в 3-4 пары оснований по направлению к верхнему концу РАМ, что указывает на то, что диапазон расщепления последовательности гена-мишени в FrCas9 соответствовал таковому в предыдущем SpCas9.
Пример 6
В примере посредством эксперимента с ОДН была верифицирована оптимальная длина части распознавания sgPHK в эукариотических клетках в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением, и результаты показаны на Фигуре 7.
(1) Материалы: все аминокислотные последовательности, последовательности sgPHK и распознанные РАМ редактируемого гена Faecalibaculum rodentium, полученные в примерах 1-4;
(2) Способ проверки: в этом примере оптимальная длина части распознавания sgPHK в эукариотических клетках в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, была верифицирована посредством эксперимента ОДН.
Подробные протоколы приведены ниже.
(a) Оптимизированная последовательность белка FrCas9, полученного от человека, была синтезирована и клонирована в эукариотический вектор РХ330 для конструирования плазмиды PX330-FrCas9.
(b) В качестве последовательности нацеливания была выбрана последовательность-мишень в 30 пар оснований выше GGTA рядом с сайтом-мишенью SpCas9, которая, как было обнаружено в предыдущем исследовании, обладала хорошим эффектом расщепления, при этом sgPHK с длиной распознавания от 19 до 23 пар оснований была сконструирована по методу Гибсона. Сильный промотор эукариот человека U6 добавляли по направлению к верхнему концу sgPHK для конструирования плазмиды PX330-FrCas9-sgRNA.
(c) Плазмида PX330-FrCas9-sgRNA, нацеленная на различные сайты генов, сконструированная, как указано выше, с использованием 2,5 мкг и 1,5 мкл ОДН, была электрически трансфицирована в клетки НЕК293Т в хорошем состоянии. Все клетки собирали через 72 часа для выделения ДНК.
(d) ОДН-ПЦР была выполнена путем конструирования пары праймеров рядом с сайтом гена-мишени и на последовательности ОДН, при этом был проведен электрофорез в агарозном геле для наблюдения за наличием полосы, чтобы предварительно определить, произошло ли расщепление мишени или нет, и сравнить интенсивность полос sgPHK с длиной распознавания от 19 до 23 пар оснований.
(e) Для количественного определения вставок в области мишени была сформирована высокопроизводительная библиотека Amplicon, при этом сравнивались эффекты расщепления sgPHK с длиной распознавания от 19 до 23 пар оснований.
Как показано на Фигуре 7, оптимальная длина распознавания sgPHK для FrCas9 составляет 21 пару оснований, 22 пары оснований или 23 пары оснований.
Пример 7
В примере посредством эксперимента с ОДН была верифицирована оптимальная длина каркаса sgPHK в эукариотических клетках в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением, и результаты показаны на Фигуре 8.
(1) Материалы: все аминокислотные последовательности, последовательности sgPHK и распознанные РАМ редактируемого гена Faecalibaculum rodentium, полученные в примерах 1-6.
(2) Способ проверки: в этом примере эксперимент с ОДН использовался для верификации оптимальной длины каркаса sgPHK эукариотических клеток в системе редактирования генома CRISPR7Cas9 типа II, полученной из Faecalibaculum rodentium.
Подробные протоколы приведены ниже.
(a) Оптимизированная последовательность белка FrCas9, полученного от человека, была синтезирована и клонирована в эукариотический вектор РХ330 для конструирования плазмиды PX330-FrCas9.
(b) В качестве последовательности нацеливания была выбрана последовательность-мишень в 30 пар оснований выше GGTA рядом с сайтом-мишенью SpCas9, которая, как было обнаружено в предыдущем исследовании, обладала хорошим эффектом расщепления, при этом sgPHK с длиной каркаса от 71 нуклеотида до 95 нуклеотидов была сконструирована по методу Гибсона. Сильный промотор эукариот человека U6 добавляли выше sgPHK для конструирования плазмиды PX330-FrCas9-sgPHK.
(c) Плазмида PX330-FrCas9-sgPHK, нацеленная на различные сайты генов, сконструированная, как указано выше, с использованием 2,5 мкг и 1,5 мкл ОДН, была электрически трансфицирована в клетки НЕК293Т в хорошем состоянии. Все клетки собирали через 72 часа для выделения ДНК.
(d) ОДН-ПЦР проводили путем конструирования пары праймеров рядом с сайтом гена-мишени и на последовательности ОДН, при этом был проведен электрофорез в агарозном геле для наблюдения за наличием полос, чтобы предварительно определить, произошло ли расщепление мишени или нет, и сравнить интенсивность полос sgPHK с длиной каркаса от 71 нуклеотида до 95 нуклеотидов.
(e) Для количественного определения вставок в области мишени была сформирована высокопроизводительная библиотека Amplicon, при этом сравнивались эффекты расщепления sgPHK с длиной каркаса от 71 нуклеотида до 95 нуклеотидов.
Как показано на Фигурах 8 и 3, оптимальный каркас sgPHK для FrCas9 составляет от 83 до 91 нуклеотида.
Пример 8
В примере посредством эксперимента с ОДН верифицировано, что эффект расщепления в эукариотических клетках в системе редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, выше, чем у SpCas9, а частота отклонения от мишени ниже, чем у SpCas9.
(1) Материалы: все аминокислотные последовательности, последовательность sgPHK длиной 22 пары оснований, последовательность 5'-GGTA-3' РАМ, аминокислотная последовательность SpCas9, последовательность sgPHK SpCas9 и последовательность SpCas9 5'-NGG-3' РАМ редактируемого гена Faecalibaculum rodentium, полученные в примерах 1-6;
(2) Способ проверки: в этом примере эксперимент GUIDE-seq использовали для проверки того, что эффект расщепления в ядерных клетках эукариотических клеток системой редактирования генома CRISPR/Cas9 типа II, полученной из Faecalibaculum rodentium, в соответствии с изобретением был выше, чем у SpCas9 и частота отклонения от мишени была ниже, чем у SpCas9.
Подробные протоколы приведены ниже.
(a) Оптимизированная последовательность белка FrCas9, полученного от человека, была синтезирована и клонирована в эукариотический вектор РХ330 для конструирования плазмиды PX330-FrCas9.
(b) В качестве последовательности нацеливания была выбрана последовательность-мишень в 30 пар оснований выше GGTA рядом с сайтом-мишенью SpCas9, которая, как было обнаружено в предыдущем исследовании, обладала хорошим эффектом расщепления, при этом sgPHK с длиной распознавания 22 пары оснований была сконструирована по методу Гибсона. Сильный промотор эукариот человека U6 добавляли выше sgPHK для конструирования плазмиды PX330-FrCas9-sgPHK и, одновременно, конструирования плазмиды PX330-SpCas9-sgPHK из 20 пар оснований sgPHK SpCas9.
(c) Плазмида PX330-FrCas9-sgPHK и плазмида PX330-SpCas9-sgPHK, нацеленные на различные сайты генов, сконструированные, как указано выше, с 2,5 мкг и 1,5 мкл ОДН, были электрически трансфицированы в клетки НЕК293Т в хорошем состоянии. Все клетки собирали через 72 часа для выделения ДНК.
(d) ОДН-ПЦР выполняли путем конструирования пары праймеров рядом с сайтом гена-мишени и на последовательности ОДН, при этом проводили электрофорез в агарозном геле для наблюдения за наличием полос, чтобы предварительно определить, произошло ли расщепление мишени или нет, и из полос ДНК создавали библиотеку GUIDE-seq.
(e) С помощью биоинформатического анализа сравнивались эффекты расщепления мишени и отклонения от мишени SpCas9 и FrCas9 в одном и том же сайте.
Как показано на Фигуре 9, FrCas9 находился на значении числа считываний мишени 3257 на сайте DYRK1A-T2, что выше, чем у SpCas9 при значении числа считываний мишени 2456. Между тем, отклонение от мишени в отношении FrCas9 не было обнаружено, в то время как SpCas9 показал отклонение от мишени на трех сайтах. FrCas9 находился на значении числа считываний мишени 34970 на сайте GRIB2B-T9, что было выше, чем у SpCas9 при значении числа считываний мишени 20434. Между тем в отношении FrCas9 отклонение от мишени не было обнаружено, в то время как SpCas9 показал отклонение от мишени на трех сайтах. Приведенные выше данные показали, что FrCas9 был белком Cas9, превосходящим SpCas9 по эффективности и специфичности расщепления.
Пример 9. FrCas9 можно использовать для Праймированного Редактирования (Prime Editing, РЕ)
Большинство генетических заболеваний человека связаны с мутациями, которые не поддаются лечению традиционными способами. Базовый редактор - один из новейших и наиболее эффективных способов точного редактирования генома. Он характеризуется использованием белка CRISPR-Cas в форме разрыва для обнаружения специфических ДНК-мишеней и использованием ДНК-дезаминазы для модификации и мутации этой ДНК-мишени с целью исправления пораженных оснований без образования двухцепочечной фрагментации ДНК. Редактор оснований цитозина (СВЕ) мутировал пару оснований C:G в ДНК в Т:А, а редактор оснований аденина (ABE) мутировал пару оснований А:Т в G:C.
Во-первых, три точечные мутации Е796А, Н1010А и D1013A соответственно были инкорпорированы для получения различных никаз FrCas9 (nFrCas9). Затем E796A nFrCas9 были объединены с оптимизированным редактором цитидиновых оснований четвертого поколения BE4Gam и редактором адениновых оснований седьмого поколения АВЕ7.10. Было замечено, что окно редактирования FrCas9-BE4Gam представляло собой 6-10-е основания (Фигура 10В), а окно FrCas9-ABE7.10 - 6-8-е основания (Фигура 10С). На основе вышеуказанных характеристик были рассчитаны области нацеливания FrCas9-BE4Gam и FrCas9-ABE7.10 в базах данных ClinVar. В отношении патогенных мутаций, которые можно было точно скорректировать с помощью FrCas9-BE4Gam, 90,38% (235/260) уникальных событий отличались от SpCas9-BE4Gam. В отношении патогенных мутаций, которые можно было точно скорректировать с помощью FrCas9-ABE7.10, 92,21% (1196/1297) уникальных событий отличались от SpCas9-ABE7.10 (Фигура 10D). Таким образом РАМ FrCas9, обогащенный ТА, значительно расширил мишени в геноме человека для редактора оснований для исправления мутаций, связанных с заболеванием человека.
Кроме того, РАМ FrCas9 (5'-NNTA-3') был палиндромным, что предполагало попарное существование sgPHK «хвост к хвосту» (Фигура 10Е). Этот признак может расширить возможности базовых редакторов FrCas9 путем одновременного изменения двух близких окон редактирования (Фигура 10Е) и увеличить распределение мишеней и плотность sgPHK FrCas9. Было рассчитано распределение 5'-GG-3' (представлено для SpCas9 РАМ) и 5'-ТА-3' (представлено для FrCas9 РАМ) в геномах человека (Фигуры 11А и 11 В). По сравнению с SpCas9 (медиана=5 пар оснований, среднее=8,66 пар оснований), FrCas9 показал более интенсивное распределение (медиана=1 пар оснований, среднее=6,16 пар оснований) в геномах человека, обеспечивая дополнительные применимые локусы.
Пример 10. FrCas9 может нацеливаться на ТАТА-бокс для модуляции экспрессии генов в качестве эффективного инструмента для активации и ингибирования CRISPR.
ТАТА-бокс (TATA-box/бокс Хогнесса) является одним из элементов, входящих в состав промотора эукариот. Его последовательный порядок: ТАТА(А/Т)А(А/Т) (нематричная последовательность цепочек). Он находится примерно на -30 пар оснований (от -25 до -32 пар оснований) выше точки инициации транскрипции у большинства эукариотических генов и в основном состоит из пар оснований А-Т, что является критерием отбора для определения инициации транскрипции генов. Транскрипция может начаться только после прочного связывания с ТАТА-боксом одного из сайтов связывания РНК-полимеразы. Поскольку последовательность РАМ FrCas9 представляет собой NNTA, она имеет естественные преимущества при нацеливании на ТАТА-бокс.
Интерференция FrCas9 CRISPR (CRISPRi) была протестирована в трех генах, промотированных ТАТА-боксом, АВСА1, UCP3 и RANKL (Фигура 12А). Расщепляя ТАТА-бокс, FrCas9 снижал экспрессию АВСА1, UCP3 и RANKL на 31,37%, 49,91% и 39,62% соответственно. В то же время, для прямого связывания с ТАТА-боксом также использовался dFrCas9 без осуществления расщепления, при этом экспрессия АВСА1, UCP3 и RANKL снизилась на 61,67%, 45,61% и 42,60% за счет прямого связывания dFrCas9 с ТАТА-боксом соответственно (Фигура 12 В). Таким образом, FrCas9 обладает уникальным потенциалом для эффективной инженерии генома генетических заболеваний, связанных с ТАТА-боксом.
Кроме того, была протестирована активация FrCas9 CRISPR (CRISPRa) с использованием dFrCas9-VP64, непосредственно нацеленного на ТАТА-бокс, при этом его эффективность сравнивалась с dSpCas9-VP64, нацеленным по направлению в вверх от ТАТА-бокса. Эксперименты CRISPRa проводились с генами АВСА1, SOD1, GH1 и BLM2. Результаты показали, что dFrCas9-VP64 обеспечивает эффективную активацию транскрипции. Более того, кратность активации dFrCas9-VP64 в АВСА1, GH1 и BLM2 была выше, чем таковая у dSpCas9-VP64, а кратность активации гена SOD1 была сравнима с таковой у dSpCas9-VP64 (Фигура 12С). Таким образом, FrCas9 является многообещающим инструментом для скрининга CRISPR благодаря своему уникальному 5'-NNTA-3' РАМ.
Пример 11. FrCas9 можно использовать для Праймированного Редактирования (Prime Editing, РЕ)
Праймированное Редактирование (РЕ) - это совершенно новый инструмент для точного редактирования генов. Эта технология может значительно уменьшить количество вредных побочных продуктов, образующихся вследствие инделов в процессе редактирования генов, значительно повысить точность редактирования и, в перспективе, преодолеть фундаментальные барьеры на пути лечения генетических заболеваний с помощью существующих методов редактирования генов. Система РЕ состоит из двух компонентов, таких как сконструированная направляющая РНК (regPHK) и белок первичного редактора. regPHK выполняют двойную функцию: имеют способность направлять отредактированный белок в сайт-мишень и содержат отредактированную последовательность матрицы. Белок главного редактора состоит из мутировавшей никазы Cas9 (которая разрывает только одну цепь ДНК) и слитой обратной транскриптазы. После того, как Cas9 расщепляет сайт-мишень, обратная транскриптаза использует regPHK в качестве матрицы для обратной транскрипции, тем самым производя требуемое редактирование в цепи ДНК, при этом скорректированная последовательность предпочтительно заменяет исходную геномную ДНК, тем самым постоянно редактируя сайт-мишень (Фигура 13).
Основываясь на биологических характеристиках FrCas9, был слит FrCas9 с обратной транскриптазой, была сконструирована соответствующая regPHK в соответствии с ее последовательностью РАМ, была создана система редактирования генов FrCas9-PE, при этом была оптимизирована ее эффективность редактирования генов. regPHK была сконструирована в соответствии с последовательностью РАМ FrCas9. По сравнению с sgPHK последовательность regPHK в области 3'-конца имеет две дополнительные последовательности. Первая последовательность представляет собой сайт связывания праймера (PBS), который может быть комплементарным концу разорванной цепи ДНК-мишени для инициации обратной транскрипции. При этом вторая последовательность представляет собой матрицу обратной транскрипции (RT матрица), которая осуществляет мутации точки-мишени или индел мутации для достижения точного редактирования генов. Предыдущие исследования показали, что длина матричных последовательностей PBS и RT значительно влияет на эффективность редактирования генов в системе РЕ, и зависит от локуса гена. Поэтому мы исследовали оптимизацию эффективности редактирования за счет комбинации матричных последовательностей PBS и RT разной длины.
На клеточном уровне был предварительно проверен эффект редактирования генов системой FrCas9-PE. В клетках НЕК293Т локус гена HEK293T-RNF2, который обычно используется для оценки эффективности редактирования генов CRISPR, был нацелен на верификацию функции редактирования генов системы FrCas9-PE. Наши текущие экспериментальные результаты показывают, что FrCas9-PE может оказывать эффекты, связанные с сайт-специфическим редактированием основания. После этого мы будем дополнительно трансформировать существующую FrCas9-PE, включая оптимизацию кодонов, оптимизацию положения и количества последовательностей ядерной локализации, модификацию обратной транскриптазы и т.д. В итоге была разработана новая система редактирования генома FrCas9-PE с высокой эффективностью редактирования базы, с меньшим количеством реакций, связанных с отклонением от мишени, и меньшим количеством побочных продуктов редактирования генов (инделов) (Фигура 14).
--->
Перечень последовательностей
SEQUENCE LISTING
<110> ZHUHAI SHU TONG MEDICAL TECHNOLOGY CO., LTD
<120> TYPE II CRISPR/CAS9 GENOME EDITING SYSTEM AND THE APPLICATION THEREOF
<130>
<160> 33
<170> PatentIn version 3.3
<210> 1
<211> 1372
<212> PRT
<213> Faecalibaculum rodentium
<400> 1
Met Cys Thr Lys Glu Ser Glu Lys Leu Asn Lys Asn Ala Asp Tyr Tyr
1 5 10 15
Ile Gly Leu Asp Met Gly Thr Ser Ser Ala Gly Trp Ala Val Ser Asp
20 25 30
Ser Glu Tyr Asn Leu Ile Arg Arg Lys Gly Lys Asp Leu Trp Gly Val
35 40 45
Arg Gln Phe Glu Glu Ala Lys Thr Ala Ala Glu Arg Arg Gly Phe Arg
50 55 60
Val Ala Arg Arg Arg Lys Gln Arg Gln Gln Val Arg Asn Arg Leu Leu
65 70 75 80
Ser Glu Glu Phe Gln Asn Glu Ile Thr Lys Ile Asp Ser Gly Phe Leu
85 90 95
Lys Arg Met Glu Asp Ser Arg Phe Val Ile Ser Asp Lys Arg Val Pro
100 105 110
Glu Lys Tyr Thr Leu Phe Asn Asp Ser Gly Tyr Thr Asp Val Glu Tyr
115 120 125
Tyr Asn Gln Tyr Pro Thr Ile Tyr His Leu Arg Lys Ala Leu Ile Glu
130 135 140
Ser Asn Glu Arg Phe Asp Ile Arg Leu Val Phe Leu Gly Ile His Ser
145 150 155 160
Leu Phe Gln His Pro Gly His Phe Leu Asp Lys Gly Asp Val Asp Thr
165 170 175
Asp Asn Thr Gly Pro Glu Glu Leu Ile Gln Phe Leu Glu Asp Cys Met
180 185 190
Asn Glu Ile Gln Ile Ser Ile Pro Leu Val Ser Asn Gln Lys Val Leu
195 200 205
Thr Asp Ile Leu Thr Asp Ser Arg Ile Thr Arg Arg Asp Lys Glu Gln
210 215 220
Gln Ile Leu Glu Ile Leu Gln Pro Asn Lys Glu Ser Lys Lys Ala Val
225 230 235 240
Ser Gln Phe Val Lys Val Leu Thr Gly Gln Lys Ala Lys Leu Gly Asp
245 250 255
Leu Ile Met Met Glu Asp Lys Asp Thr Glu Glu Tyr Lys Tyr Ser Phe
260 265 270
Ser Phe Arg Glu Lys Thr Leu Glu Glu Ile Leu Pro Asp Ile Glu Gly
275 280 285
Val Ile Asp Gly Leu Ala Leu Glu Tyr Ile Glu Ser Ile Tyr Ser Leu
290 295 300
Tyr Ser Trp Ser Leu Leu Asn Ser Tyr Met Lys Asp Thr Leu Thr Gly
305 310 315 320
His Tyr Tyr Ser Tyr Leu Ala Glu Ala Arg Val Ala Ala Tyr Asp Lys
325 330 335
His His Ser Asp Leu Val Lys Leu Lys Thr Leu Phe Arg Glu Tyr Ile
340 345 350
Pro Glu Glu Tyr Asp Asn Phe Phe Arg Lys Met Glu Lys Ala Asn Tyr
355 360 365
Ser His Tyr Ile Gly Ser Thr Glu Tyr Asp Gly Glu Lys Arg Cys Arg
370 375 380
Thr Ala Lys Ala Lys Gln Glu Asp Phe Tyr Lys Ser Ile Asn Lys Met
385 390 395 400
Leu Glu Lys Ile Pro Glu Cys Ser Glu Lys Thr Glu Ile Gln Lys Glu
405 410 415
Ile Ile Glu Gly Thr Phe Leu Leu Lys Gln Thr Gly Pro Gln Asn Gly
420 425 430
Phe Val Pro Asn Gln Leu Gln Leu Lys Glu Leu Arg Lys Ile Leu Gln
435 440 445
Asn Ala Ser Lys His Tyr Pro Phe Leu Thr Glu Lys Asp Glu Arg Asp
450 455 460
Met Thr Ala Ile Asp Arg Ile Glu Ala Leu Phe Ser Phe Arg Ile Pro
465 470 475 480
Tyr Tyr Ile Gly Pro Leu Lys Asn Thr Asp Asn Gln Gly His Gly Trp
485 490 495
Ala Val Arg Arg Asp Gly His Glu Gln Ile Pro Val Arg Pro Trp Asn
500 505 510
Phe Glu Glu Ile Ile Asp Glu Ser Ala Ser Ala Asp Leu Phe Ile Lys
515 520 525
Asn Leu Val Asn Ser Cys Thr Tyr Leu Arg Thr Glu Lys Val Leu Pro
530 535 540
Lys Ser Ser Leu Leu Tyr Gln Glu Phe Glu Val Leu Asn Glu Leu Asn
545 550 555 560
Asn Leu Arg Ile Asn Gly Met Tyr Pro Asp Glu Ile Gln Pro Gly Leu
565 570 575
Lys Arg Met Ile Phe Glu Gln Cys Phe Tyr Ser Gly Lys Lys Val Thr
580 585 590
Gly Lys Lys Leu Gln Leu Phe Leu Arg Ser Val Leu Thr Asn Ser Ser
595 600 605
Thr Glu Glu Phe Val Leu Thr Gly Ile Asp Lys Asp Phe Lys Ser Ser
610 615 620
Leu Ser Ser Tyr Lys Lys Phe Cys Glu Leu Phe Gly Val Lys Thr Leu
625 630 635 640
Asn Asp Thr Gln Lys Val Met Ala Glu Gln Ile Ile Glu Trp Ser Thr
645 650 655
Val Tyr Gly Asp Ser Arg Lys Phe Leu Lys Arg Lys Leu Glu Asp Asn
660 665 670
Tyr Pro Glu Leu Thr Asp Gln Gln Ile Arg Arg Ile Ala Gly Phe Lys
675 680 685
Phe Ser Glu Trp Gly Asn Leu Ser Arg Ala Phe Leu Glu Met Glu Gly
690 695 700
Tyr Lys Asp Glu Ala Gly Asn Pro Val Thr Ile Ile Arg Ala Leu Arg
705 710 715 720
Asp Thr Gln Lys Asn Leu Met Gln Leu Leu Ser Asn Asp Ser Ala Phe
725 730 735
Ala Lys Lys Leu Gln Glu Leu Asn Asp Tyr Val Thr Arg Asp Ile Trp
740 745 750
Ser Ile Glu Pro Asp Asp Leu Asp Gly Met Tyr Leu Ser Ala Pro Val
755 760 765
Arg Arg Met Ile Trp Gln Thr Phe Leu Ile Leu Arg Glu Val Val Asp
770 775 780
Thr Ile Gly Tyr Ser Pro Lys Lys Ile Phe Met Glu Met Ala Arg Gly
785 790 795 800
Glu Gln Glu Lys Lys Arg Thr Ala Ser Arg Lys Lys Gln Leu Ile Asp
805 810 815
Leu Tyr Lys Glu Ala Gly Met Lys Asn Asp Glu Leu Phe Gly Asp Leu
820 825 830
Glu Ser Leu Glu Glu Ala Gln Leu Arg Ser Lys Lys Leu Tyr Leu Tyr
835 840 845
Phe Arg Gln Met Gly Arg Asp Ile Tyr Ser Gly Lys Leu Ile Asp Phe
850 855 860
Met Asp Val Leu His Gly Asn Arg Tyr Asp Ile Asp His Ile His Pro
865 870 875 880
Gln Ser Lys Lys Lys Asp Asp Ser Leu Glu Asn Asn Leu Val Leu Thr
885 890 895
Ser Lys Asp Phe Asn Asn His Ile Lys Gln Asp Val Tyr Pro Ile Pro
900 905 910
Glu Gln Ile Gln Ser Arg Gln Lys Gly Phe Trp Ala Met Leu Leu Lys
915 920 925
Gln Gly Phe Met Ser Gln Glu Lys Tyr Asn Arg Leu Met Arg Thr Thr
930 935 940
Pro Phe Thr Asp Glu Glu Leu Ala Glu Phe Val Asn Arg Gln Leu Val
945 950 955 960
Glu Thr Arg Gln Gly Thr Lys Ala Ile Ile Ser Leu Ile Asn Gln Cys
965 970 975
Phe Pro Asp Ser Glu Val Val Tyr Val Lys Ala Gly Asn Thr Ser Asp
980 985 990
Phe Arg Gln Arg Phe Asp Ile Pro Lys Ser Arg Asp Leu Asn Asn Tyr
995 1000 1005
His His Ala Val Asp Ala Tyr Leu Asn Ile Val Val Gly Asn Val Tyr
1010 1015 1020
Asp Thr Lys Phe Thr Lys Asn Pro Ile Asn Phe Ile Lys Lys Met Arg
1025 1030 1035 1040
Lys Ser Gly Asn Leu His Ser Tyr Ser Leu Arg Arg Met Tyr Asp Phe
1045 1050 1055
Asn Val Gln Arg Gly Asp Gln Thr Ala Trp Val Ala Glu Asn Asp Thr
1060 1065 1070
Thr Leu Lys Thr Val Lys Lys Thr Ala Phe Lys Thr Ser Pro Met Val
1075 1080 1085
Thr Lys Arg Thr Tyr Glu Arg Lys Gly Gly Leu Ala Asp Ser Val Leu
1090 1095 1100
Ile Ala Ala Lys Lys Ala Lys Pro Gly Val His Leu Pro Val Lys Thr
1105 1110 1115 1120
Ser Asp Ser Arg Phe Ala Asn Gln Val Ser Thr Tyr Gly Gly Tyr Asp
1125 1130 1135
Asn Val Lys Gly Ser His Phe Phe Leu Val Glu His Gln Gln Lys Lys
1140 1145 1150
Lys Thr Ile Arg Ser Ile Glu Asn Val Pro Ile His Leu Lys Glu Lys
1155 1160 1165
Leu Lys Thr Lys Glu Glu Leu Glu His Tyr Cys Ala Gln Val Leu Gly
1170 1175 1180
Met Val Gln Pro Asp Val Arg Leu Thr Arg Ile Pro Met Tyr Ser Leu
1185 1190 1195 1200
Leu Leu Ile Asp Gly Tyr Tyr Tyr Tyr Leu Thr Gly Arg Thr Gly Gly
1205 1210 1215
Asn Leu Ser Leu Ser Asn Ala Val Glu Leu Cys Leu Pro Ala Lys Glu
1220 1225 1230
Gln Ala His Ile Arg Met Ile Ser Lys Ile Ala Gly Gly Arg Ser Thr
1235 1240 1245
Asp Ala Leu Ser Ala Glu Ala Lys Asp Asp Phe Arg Lys Lys Asn Leu
1250 1255 1260
Arg Leu Tyr Asp Glu Leu Ala Glu Lys His Arg Ser Thr Ile Phe Ser
1265 1270 1275 1280
Lys Arg Lys Asn Pro Ile Gly Pro Lys Leu Leu Lys Tyr Arg Glu Ala
1285 1290 1295
Phe Val Lys Gln Thr Ile Glu Asn Gln Cys Lys Val Ile Leu Gln Ile
1300 1305 1310
Leu Lys Leu Thr Ser Thr Asn Cys Lys Thr Ser Ala Asp Leu Lys Leu
1315 1320 1325
Ile Gly Gly Ser Gly Gln Glu Gly Val Met Ser Ile Ser Lys Leu Leu
1330 1335 1340
Arg Ala Glu Lys Tyr Ala Glu Phe Tyr Leu Ile Cys Gln Ser Pro Ser
1345 1350 1355 1360
Gly Ile Tyr Glu Thr Arg Lys Asn Leu Leu Thr Ile
1365 1370
<210> 2
<211> 294
<212> PRT
<213> Faecalibaculum rodentium
<400> 2
Met Thr Trp Arg Thr Ile Thr Ile Ser Ser His Ser Lys Leu Asp Tyr
1 5 10 15
Gln Met Gly Tyr Leu Val Val Arg Gly Glu Ser Ile Lys Arg Ile His
20 25 30
Leu Ser Glu Ile Ser Val Leu Ile Ile Glu Asn Thr Ala Val Ser Leu
35 40 45
Thr Ala Tyr Leu Val Ser Glu Leu Val Lys Asn Lys Ile Lys Leu Leu
50 55 60
Phe Cys Asp Glu Lys Arg Ser Pro Leu Ala Glu Val Ser Glu Leu Tyr
65 70 75 80
Gly Gly His Asp Ser Ser Asp Met Val Arg Lys Gln Ile Glu Ile Pro
85 90 95
Gln Glu Arg Lys Asp Ile Ala Trp Gln Ser Ile Ile Met Ser Lys Ile
100 105 110
Ser Asn Gln Phe Ala Val Leu His Asn Phe Asp Cys Pro Asn Gln Glu
115 120 125
Leu Leu Leu Gln Tyr Ile Asn Glu Val Leu Pro Gly Asp Val Thr Asn
130 135 140
Arg Glu Gly His Ala Ala Lys Val Tyr Phe Asn Ser Leu Phe Gly Lys
145 150 155 160
Ser Phe Tyr Arg Ala Ser Glu Cys Ala Leu Asn Ala Ala Leu Asn Tyr
165 170 175
Gly Tyr Ser Val Leu Leu Ser Ala Val Ser Arg Glu Ile Ala Gly Tyr
180 185 190
Gly Phe Leu Thr Gln Leu Gly Ile Phe His Asp Asn Cys Asp Asn Lys
195 200 205
Tyr Asn Leu Ser Cys Asp Leu Met Glu Pro Phe Arg Pro Val Val Asp
210 215 220
Tyr Leu Val Lys Ser Asn Ile Val Glu Val Phe Glu Lys Glu Gln Lys
225 230 235 240
Gln Lys Ile Leu Gln Leu Leu Gln Phe Lys Ile Gln Ile Asn Asp Arg
245 250 255
Gln Glu Thr Val Gln Asn Ala Ile Ser Ile Phe Val His Ser Val Leu
260 265 270
Asp Tyr Leu Leu Asp Pro Ser Val Tyr Ile Lys Val Pro Arg Ile Asp
275 280 285
Phe Thr Lys Asn Val Val
290
<210> 3
<211> 68
<212> PRT
<213> Faecalibaculum rodentium
<400> 3
Met Met Gln Glu Ser Val Tyr Cys Lys Leu Thr Thr Asn Gln Ser Ser
1 5 10 15
Ala Glu Thr Val Leu Lys Met Val Arg Ala Asn Lys Pro Pro Glu Gly
20 25 30
Leu Ile Gln Thr Leu Ile Ile Thr Glu Lys Gln Phe Ser Lys Met Asp
35 40 45
Phe Ile Leu Gly Gln Pro Asn Ser Asp Val Val Ala Thr Asp Glu Ser
50 55 60
Val Leu Asp Leu
65
<210> 4
<211> 223
<212> PRT
<213> Faecalibaculum rodentium
<400> 4
Met Arg Leu Leu Ile Asp Arg Leu Leu Leu Ser Ala Glu Leu Asn Ile
1 5 10 15
Asp Lys Ala Thr Thr Ile Ile Ile Glu Asn Pro Lys Ala Phe Arg Met
20 25 30
Val Ile Lys Asp Leu Ile Glu Gln Glu Asn Gly Gln Gly Gly Leu Leu
35 40 45
Arg Ile Val Glu Gly Asp Lys Glu Leu Cys Leu Ser Lys Ser Ala Ile
50 55 60
Leu Val Leu Asn Pro Tyr Leu Ala Asp Leu Asn Cys Arg Lys Phe Leu
65 70 75 80
Gln Leu Ala Tyr Ser Glu Leu Gln Ala Met Thr Gly Glu Phe Leu Glu
85 90 95
Asp Gln Ala Val Val Leu Ser Ala Met Thr Gly Tyr Leu Ser Lys Ile
100 105 110
Cys Asp Gln Ser Arg Phe Asp Phe Leu Glu Phe Ser Ala Ile Pro Asp
115 120 125
Trp Ala Ser Val Phe Lys Ala Trp Gly Leu Arg Phe Glu Gln Ala Ile
130 135 140
Pro Gly Leu Leu Pro Ser Leu Ile Gln Tyr Leu Gln Leu Ala Ala Thr
145 150 155 160
Phe Pro Gln Phe Lys Leu Ile Ile Phe Ile Asn Leu Lys Gln Tyr Leu
165 170 175
Leu Pro Glu Glu Gln Phe Glu Leu Phe Lys Met Ala Glu Tyr Leu Gln
180 185 190
Leu Lys Val Leu Leu Val Glu Ser Ala Gln Asn Tyr Lys Ser Asp Arg
195 200 205
Glu Asp Leu Ile Ile Ile Asp Lys Asp Leu Cys Glu Ile Gln Ser
210 215 220
<210> 5
<211> 20
<212> RNA
<213> Faecalibaculum rodentium
<400> 5
guuugagugu cuuguuaauu 20
<210> 6
<211> 36
<212> DNA
<213> Faecalibaculum rodentium
<400> 6
gtttgagtgt cttgttaatt cggaagtatt tcaaac 36
<210> 7
<211> 216
<212> DNA
<213> Faecalibaculum rodentium
<400> 7
gtttgagtgt cttgttaatt cgggagtagc tctctcgttt gagtgtcttg ttaattcgga 60
agtaagctca acttttgagt gtcttgttaa ttcggaagta tctcaaacgt ttgagtgtct 120
tgttaattca gaagtatttc aaacgtttga gtgtcttgtt aattcggaag tattccaaac 180
gtttgagtgt cttgttaatt cggaagtatt tcaaac 216
<210> 8
<211> 71
<212> RNA
<213> Faecalibaculum rodentium
<400> 8
aauuaacaag augaguucaa aucaggcucc uagagagauc cgaacuuacc uucauggcgg 60
gcauugugcc c 71
<210> 9
<211> 95
<212> RNA
<213> Faecalibaculum rodentium
<400> 9
guuugagugu cuuguuaauu gaaaaauuaa caagaugagu ucaaaucagg cuccuagaga 60
gauccgaacu uaccuucaug gcgggcauug ugccc 95
<210> 10
<211> 91
<212> RNA
<213> Faecalibaculum rodentium
<400> 10
guuugagugu cuuguuaaga aauuaacaag augaguucaa aucaggcucc uagagagauc 60
cgaacuuacc uucauggcgg gcauugugcc c 91
<210> 11
<211> 89
<212> RNA
<213> Faecalibaculum rodentium
<400> 11
guuugagugu cuuguuagaa auaacaagau gaguucaaau caggcuccua gagagauccg 60
aacuuaccuu cauggcgggc auugugccc 89
<210> 12
<211> 87
<212> RNA
<213> Faecalibaculum rodentium
<400> 12
guuugagugu cuuguugaaa aacaagauga guucaaauca ggcuccuaga gagauccgaa 60
cuuaccuuca uggcgggcau ugugccc 87
<210> 13
<211> 85
<212> RNA
<213> Faecalibaculum rodentium
<400> 13
guuugagugu cuugugaaaa caagaugagu ucaaaucagg cuccuagaga gauccgaacu 60
uaccuucaug gcgggcauug ugccc 85
<210> 14
<211> 83
<212> RNA
<213> Faecalibaculum rodentium
<400> 14
guuugagugu cuuggaaaca agaugaguuc aaaucaggcu ccuagagaga uccgaacuua 60
ccuucauggc gggcauugug ccc 83
<210> 15
<211> 79
<212> RNA
<213> Faecalibaculum rodentium
<400> 15
guuugagugu cugaaaagau gaguucaaau caggcuccua gagagauccg aacuuaccuu 60
cauggcgggc auugugccc 79
<210> 16
<211> 75
<212> RNA
<213> Faecalibaculum rodentium
<400> 16
guuugagugu gaaaaugagu ucaaaucagg cuccuagaga gauccgaacu uaccuucaug 60
gcgggcauug ugccc 75
<210> 17
<211> 71
<212> RNA
<213> Faecalibaculum rodentium
<400> 17
guuugaguga aagaguucaa aucaggcucc uagagagauc cgaacuuacc uucauggcgg 60
gcauugugcc c 71
<210> 18
<211> 69
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 18
ctacctgcat accgttatta acatatgaca actcaattaa acgccacatc catcggcgct 60
ttggtcggc 69
<210> 19
<211> 62
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 19
atgttaccac tggattgagg ataccgttat taacatatga caactcaatt aaactctggt 60
ac 62
<210> 20
<211> 69
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 20
tcagtttata tgagttacaa cgaacaccgt ttaattgagt tgtcatatgt taataacggt 60
attcaggta 69
<210> 21
<211> 69
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 21
ctacctacat accgttatta acatatgaca actcaattaa acgtcagcac ctgggacccc 60
gccaccgtg 69
<210> 22
<211> 34
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 22
gtttaattga gttgtcatat gttaataacg gtat 34
<210> 23
<211> 26
<212> DNA
<213> Artificial Sequence
<221> misc_feature
<222> (22)..(26)
<223> n is a, c, g, or t;r is a or g;w is a or t
<400> 23
tgttaccact ggattgaggt cnnrwr 26
<210> 24
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 24
tgttaccact ggattgaggt ctggta 26
<210> 25
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 25
aagaaccact ggattgaggt ctggct 26
<210> 26
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 26
gggaaccact ggattaaggt ccagat 26
<210> 27
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 27
agttaccaat ggattgagat ggggag 26
<210> 28
<211> 26
<212> DNA
<213> Artificial Sequence
<221> misc_feature
<222> (22)..(26)
<223> n is a, c, g, or t;r is a or g;w is a or t
<400> 28
aaaaaggaag gagttctttg tnnrwr 26
<210> 29
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 29
aaaaaggaag gagttctttg taggta 26
<210> 30
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 30
taaaaggaag gtgttctttg tggggt 26
<210> 31
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 31
gaaaaggaag gtgctctttg tgggtg 26
<210> 32
<211> 26
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 32
ggaaaaggtg gaattctttg ttggta 26
<210> 33
<211> 17
<212> DNA
<213> Artificial Sequence
<223> Synthetic
<400> 33
cgaacacctc aggtaat 17
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ЭКСПРЕССИРУЮЩИЕ CAS МЫШИНЫЕ ЭМБРИОНАЛЬНЫЕ СТВОЛОВЫЕ КЛЕТКИ И МЫШИ И ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2782358C2 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ DEMEQUINA SEDIMINICOLA | 2019 |
|
RU2722933C1 |
| Средство разрезания ДНК на основе Cas9 белка из бактерии Capnocytophaga ochracea | 2021 |
|
RU2778156C1 |
| СРЕДСТВО РАЗРЕЗАНИЯ ДНК НА ОСНОВЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA | 2019 |
|
RU2722934C1 |
| ПРИМЕНЕНИЕ CAS9 БЕЛКА ИЗ БАКТЕРИИ PASTEURELLA PNEUMOTROPICA ДЛЯ МОДИФИКАЦИИ ГЕНОМНОЙ ДНК В КЛЕТКАХ | 2019 |
|
RU2724470C1 |
| КОМПОЗИЦИИ И СПОСОБЫ, ПРЕДНАЗНАЧЕННЫЕ ДЛЯ ЛЕЧЕНИЯ ГЕМОГЛОБИНОПАТИЙ | 2018 |
|
RU2834231C2 |
| СИСТЕМА РЕДАКТИРОВАНИЯ ГЕНОМА ДРОЖЖЕЙ DEBARYOMYCES HANSENII НА ОСНОВЕ CRISPR/CAS9 | 2019 |
|
RU2710731C1 |
| Средство разрезания двунитевой ДНК с помощью Cas12d белка из Katanobacteria и гибридной РНК, полученной путем слияния направляющей CRISPR РНК и scout РНК | 2020 |
|
RU2771626C1 |
| СПОСОБ СОЗДАНИЯ НОВЫХ МУТАЦИЙ В ОРГАНИЗМАХ И ЕГО ПРИМЕНЕНИЕ | 2020 |
|
RU2833881C1 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
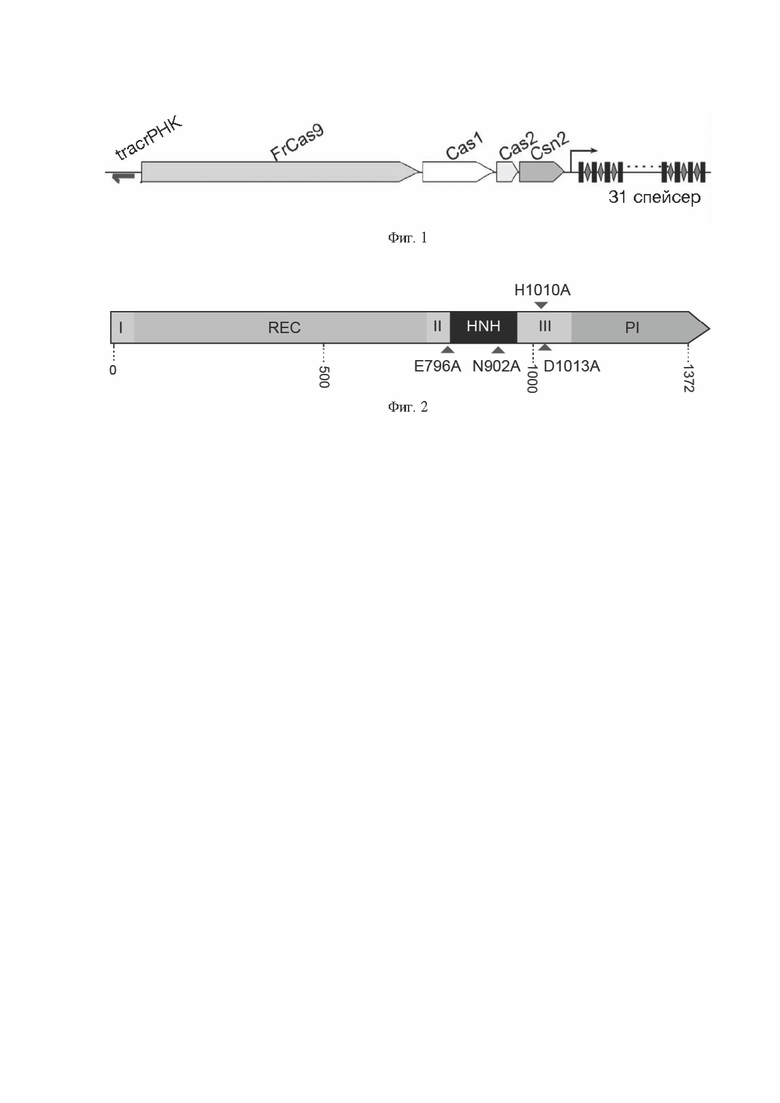

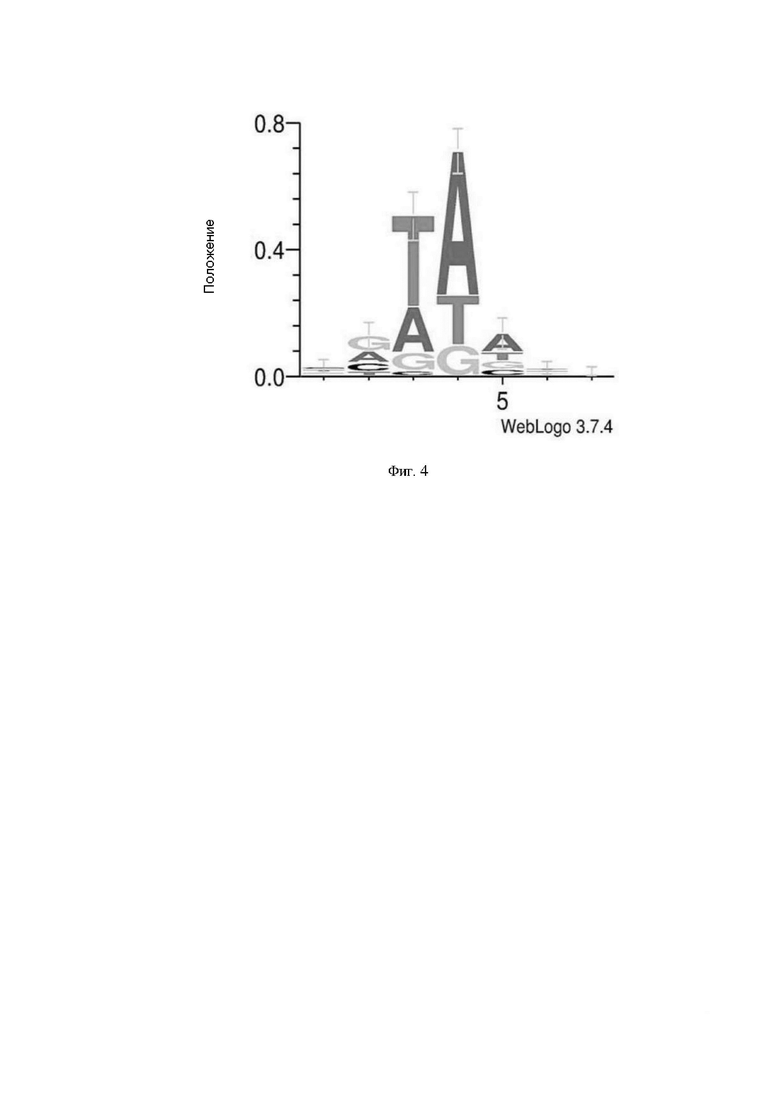
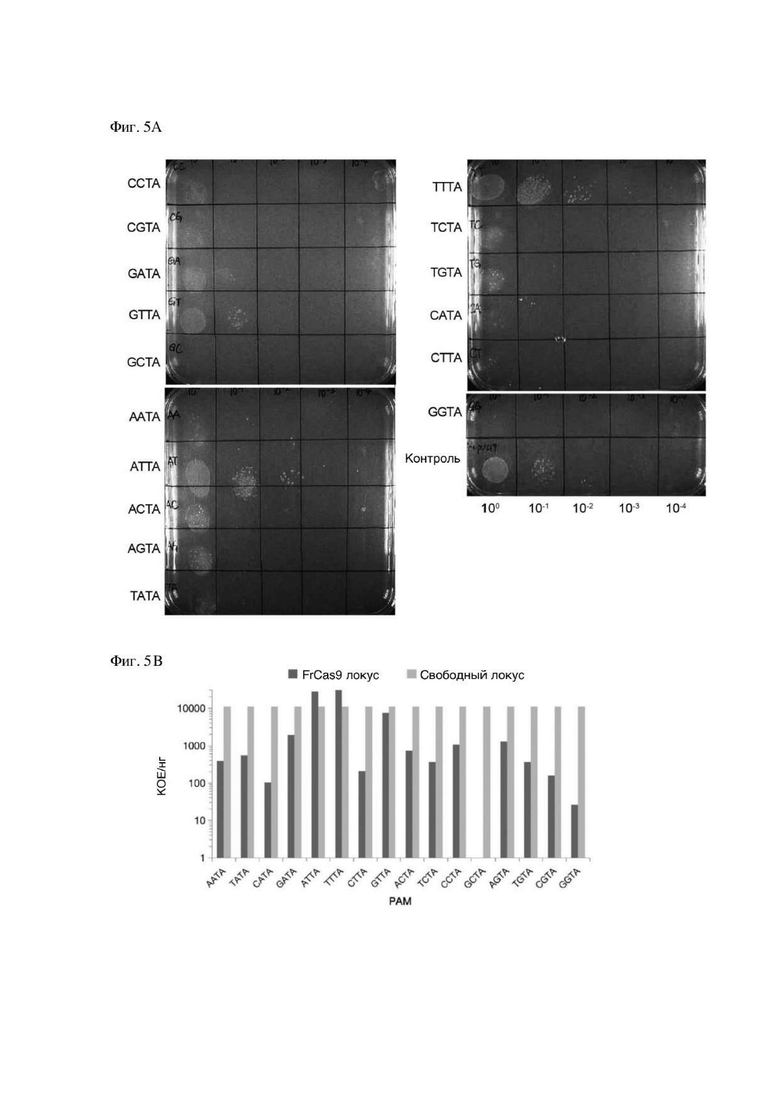
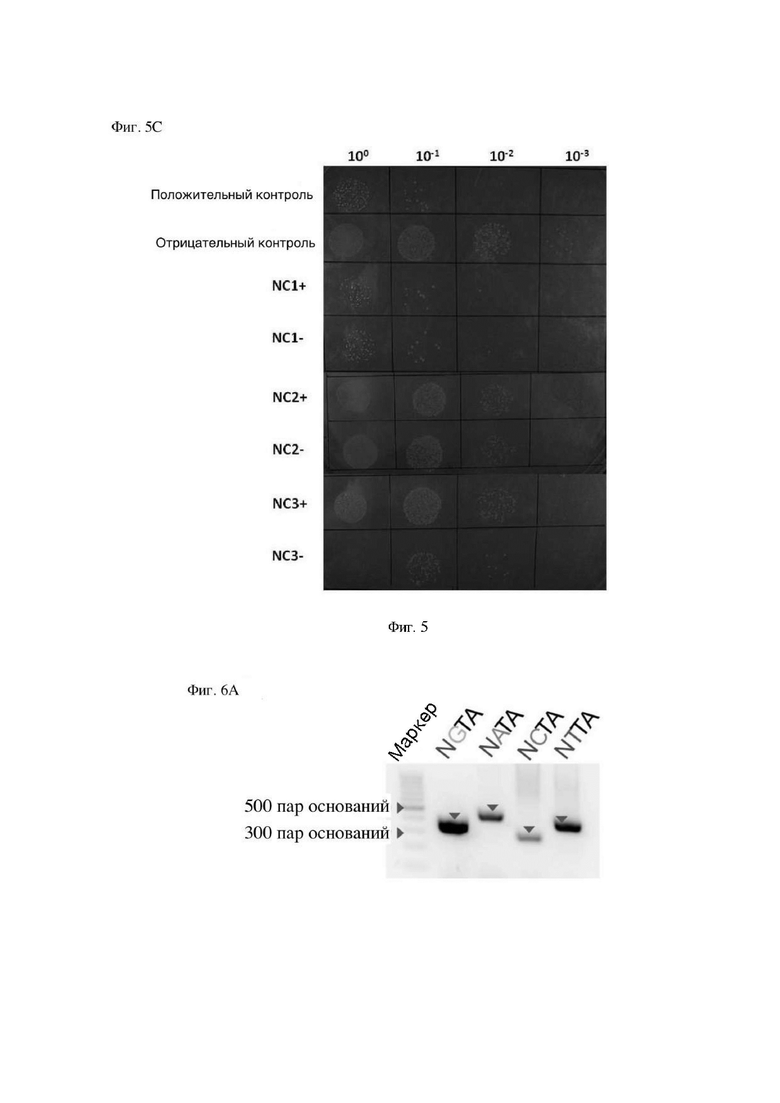

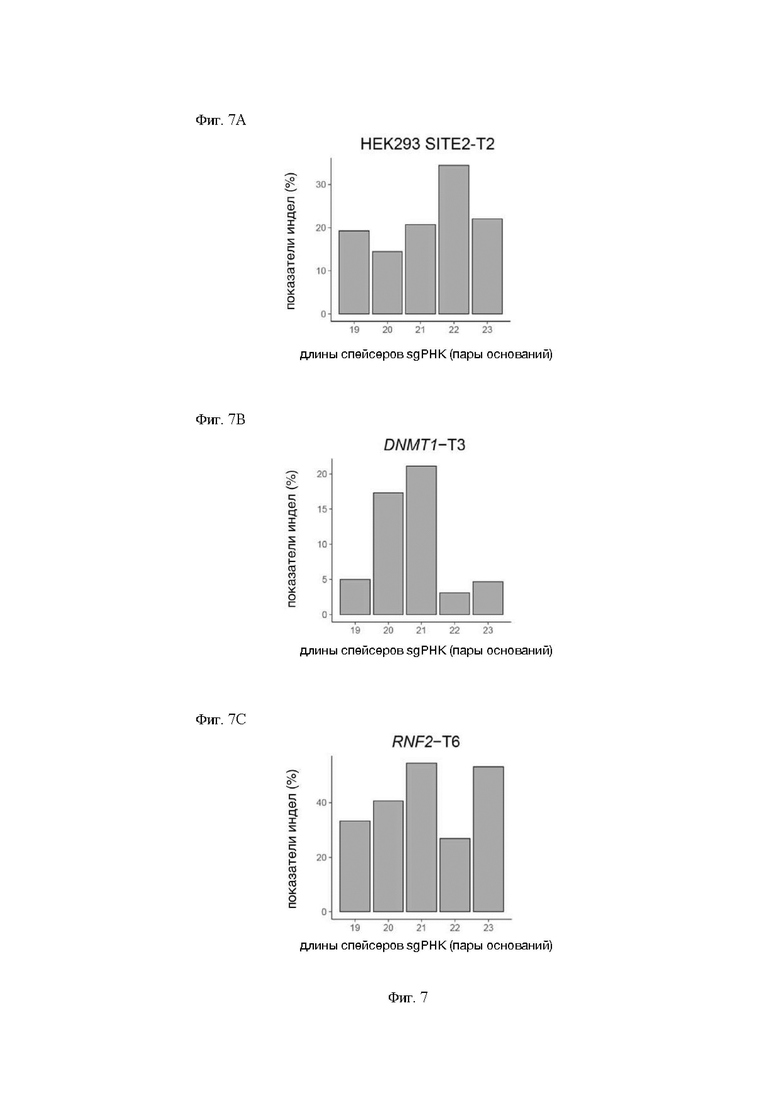


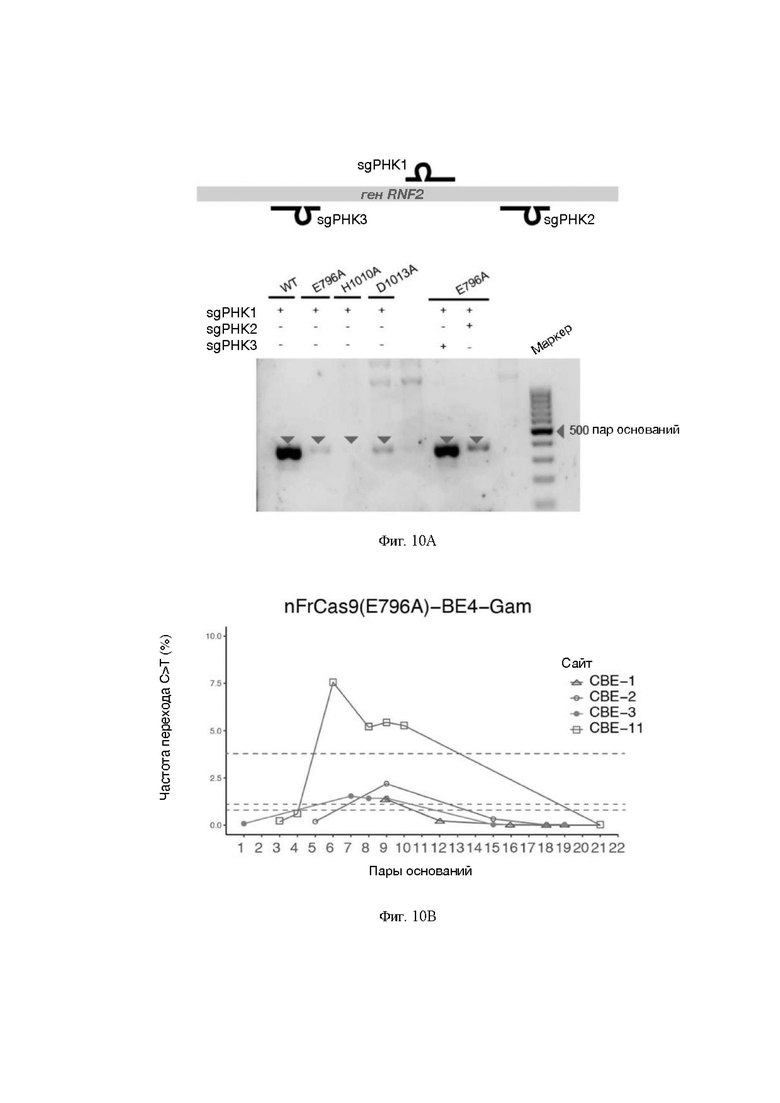
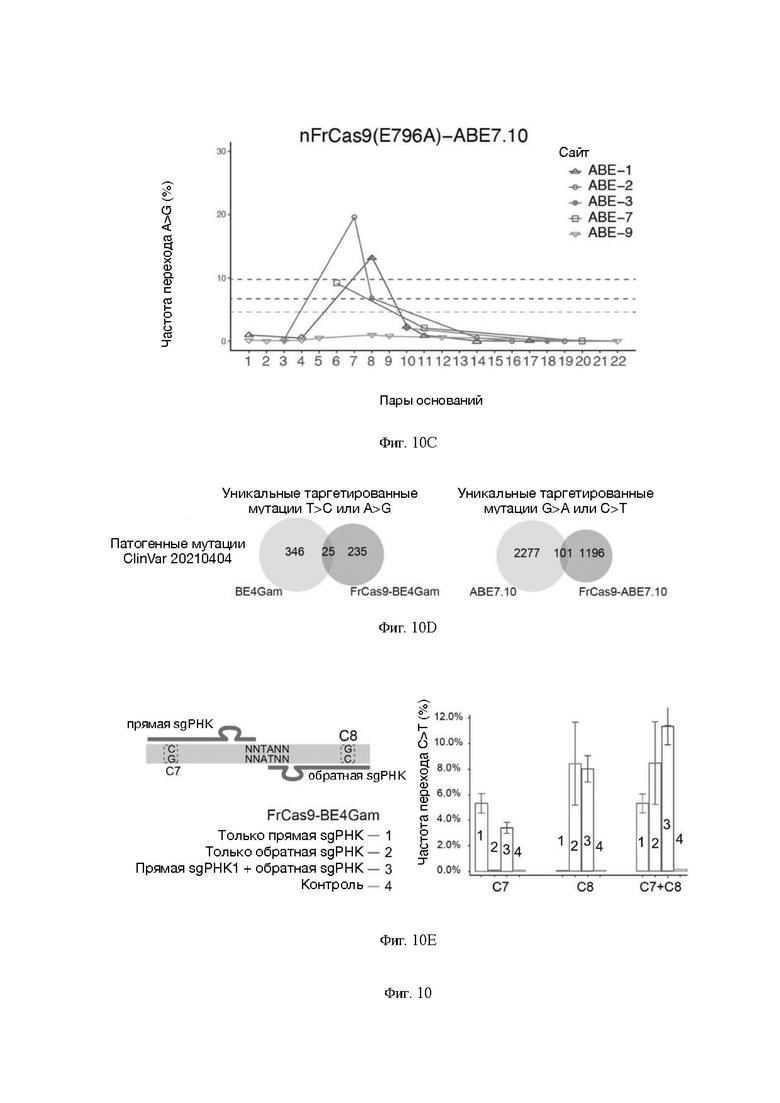
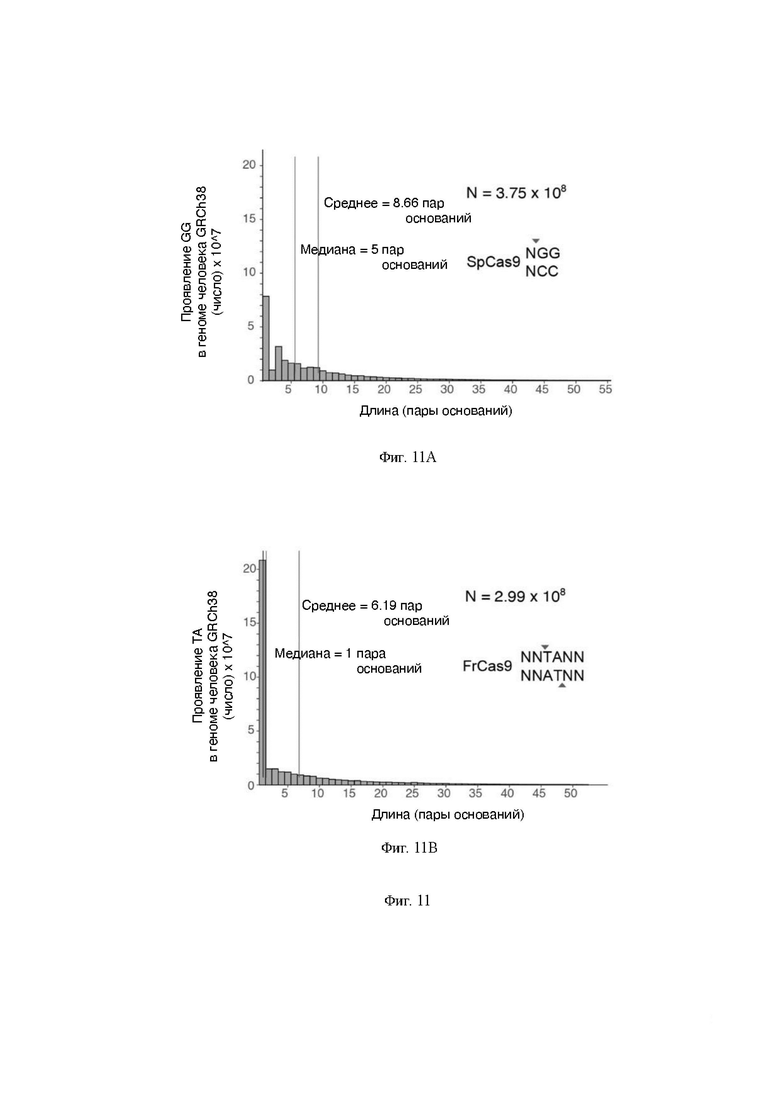
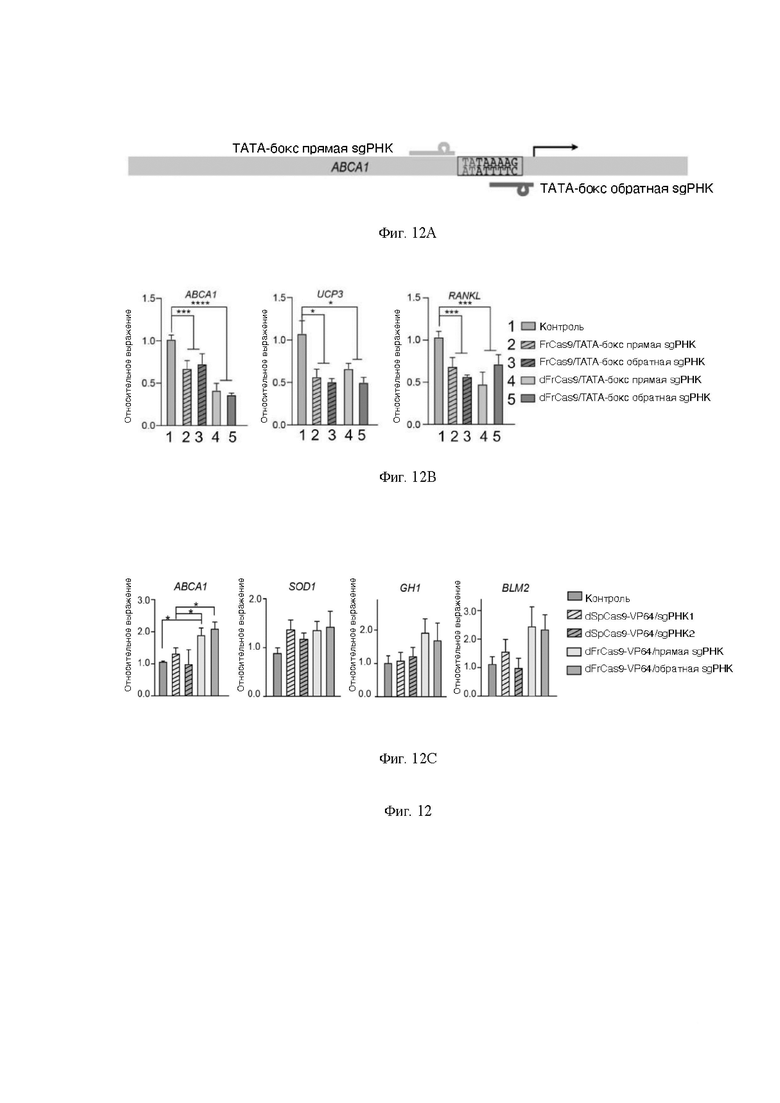
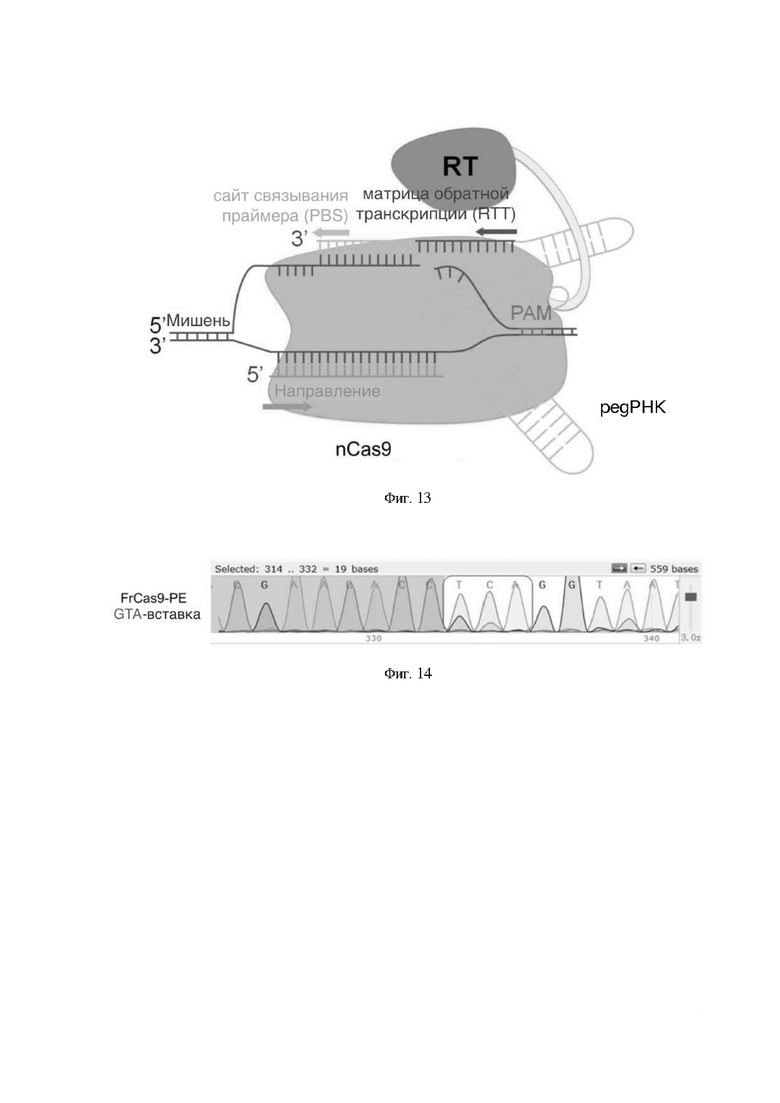
Изобретение относится к области биотехнологии, в частности к системе редактирования генома CRISPR/Cas9 типа II, отличающейся тем, что система содержит белок Cas9, вспомогательные белки, crРНК (CRISPR РНК) и tracrРНК (трансактивированную CRISPR РНК) в функциональной форме комплекса РНП (рибонуклеопротеиновый комплекс) белка Cas9 и направляющей РНК, образованной путем гибридизации crРНК с tracrРНК. Изобретение эффективно для редактирования ДНК. 9 н. и 13 з.п. ф-лы, 14 ил., 11 пр.
1. Система редактирования генома CRISPR/Cas9 типа II, отличающаяся тем, что система содержит белок Cas9, вспомогательные белки, crРНК (CRISPR РНК) и tracrРНК (трансактивированную CRISPR РНК) в функциональной форме комплекса РНП (рибонуклеопротеиновый комплекс) белка Cas9 и направляющей РНК, образованной путем гибридизации crРНК с tracrРНК;
при этом белок Cas9 представляет собой ДНК-эндонуклеазу и имеет аминокислотную последовательность, как показано в SEQ ID NO: 1, или аминокислотную последовательность, имеющую ту же функцию аминокислотной последовательности, как показано в SEQ ID NO: 1, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 1;
где CRISPR РНК создается путем транскрипции массива CRISPR и имеет последовательность РНК, как показано в SEQ ID NO: 5, или последовательность РНК, имеющую ту же функцию последовательности РНК, как показано в SEQ ID NO: 5, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 5; и
где трансактивированная CRISPR РНК содержит последовательность, комплементарную последовательности прямого повтора CRISPR РНК, а трансактивированная CRISPR РНК имеет последовательность нуклеиновой кислоты, как показано в SEQ ID NO: 8, или последовательность нуклеиновой кислоты, имеющую ту же функцию последовательности нуклеиновой кислоты, как показано в SEQ ID NO: 8, и имеющую по меньшей мере 70%, 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 8.
2. Система редактирования генома CRISPR/Cas9 типа II по п. 1, отличающаяся тем, что белок Cas9 расщепляет двухцепочечную ДНК, комплементарную crРНК выше последовательности PAM, с помощью нуклеазного домена, при этом нуклеазный домен выбран из HNH-подобного домена нуклеазы, RuvC-подобного домена нуклеазы или их комбинации.
3. Система редактирования генома CRISPR/Cas9 типа II по п. 1, отличающаяся тем, что вспомогательные белки включают вспомогательный белок Cas1, вспомогательный белок Cas2 и вспомогательный белок Csn2;
при этом вспомогательный белок Cas1 имеет аминокислотную последовательность, как показано в SEQ ID NO: 2, или аминокислотную последовательность, имеющую ту же функцию аминокислотной последовательности, как показано в SEQ ID NO: 2, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 2;
при этом вспомогательный белок Cas2 имеет аминокислотную последовательность, как показано в SEQ ID NO: 3, или аминокислотную последовательность, имеющую ту же функцию аминокислотной последовательности, как показано в SEQ ID NO: 3, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 3;
при этом вспомогательный белок Csn2 имеет аминокислотную последовательность, как показано в SEQ ID NO: 4, или аминокислотную последовательность, имеющую ту же функцию аминокислотной последовательности, как показано в SEQ ID NO: 4, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с аминокислотной последовательностью, как показано в SEQ ID NO: 4.
4. Система редактирования генома CRISPR/Cas9 типа II по п. 1, отличающаяся тем, что массив CRISPR содержит последовательность прямого повтора и спейсерную последовательность, где последовательность прямого повтора имеет последовательность нуклеиновой кислоты, как показано в SEQ ID NO: 6, или последовательность нуклеиновой кислоты, имеющую ту же функцию последовательности нуклеиновой кислоты, как показано в SEQ ID NO: 6, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 6; и где спейсерная последовательность имеет последовательность нуклеиновой кислоты, как показано в SEQ ID NO: 7, или последовательность нуклеиновой кислоты, имеющую ту же функцию последовательности нуклеиновой кислоты, как показано в SEQ ID NO: 7, и имеющую по меньшей мере 80%, 85%, 90%, 95%, 98% или 99% гомологию с последовательностью нуклеиновой кислоты, как показано в SEQ ID NO: 7.
5. Система редактирования генома CRISPR/Cas9 типа II по п. 1, отличающаяся тем, что направляющая РНК, образованная путем гибридизации crРНК с tracrРНК, имеет каркас, состоящий из последовательности 7–24 нуклеотидов последовательности прямого повтора crРНК и последовательности tracrРНК; предпочтительно две части слиты линкером "GAAA", "TGAA" или "AAAC" с образованием каркаса sgРНК;
предпочтительно каркас sgРНК, образованный путем слияния последовательности прямого повтора crРНК из 20 нуклеотидов и полноразмерной последовательности tracrРНК с помощью «GAAA», показан в SEQ ID NO: 9, и на этой основе предпочтительно высокоэффективным вариантом каркаса sgРНК является
каркас sgРНК, образованный путем слияния первых 18–14 нуклеотидов последовательности прямого повтора crРНК и последних 69–65 нуклеотидов tracrРНК с помощью линкерной последовательности, которая представляет собой GAAA, предпочтительно выбранной из следующих пяти каркасов:
(1) каркас sgРНК длиной 91 нуклеотид, который содержит 18 нуклеотидов последовательности прямого повтора и 69 нуклеотидов tracrРНК, как показано в SEQ ID NO: 10;
(2) каркас sgРНК длиной 89 нуклеотидов, который содержит 17 нуклеотидов последовательности прямого повтора и 68 нуклеотидов tracrРНК, как показано в SEQ ID NO: 11;
(3) каркас sgРНК длиной 87 нуклеотидов, который содержит 16 нуклеотидов последовательности прямого повтора и 67 нуклеотидов tracrРНК, как показано в SEQ ID NO: 12;
(4) каркас sgРНК длиной 85 нуклеотидов, который содержит 15 нуклеотидов последовательности прямого повтора и 66 нуклеотидов tracrРНК, как показано в SEQ ID NO: 13;
(5) каркас sgРНК длиной 83 нуклеотида, который содержит 14 нуклеотидов последовательности прямого повтора и 65 нуклеотидов tracrРНК, как показано в SEQ ID NO: 14;
необязательно каркас sgРНК имеет последовательность нуклеиновой кислоты, имеющую по меньшей мере 70%, 80%, 85%, 90%, 95%, 98% или 99% гомологию с любой из SEQ ID NO: 9 – 14.
6. Система редактирования генома CRISPR/Cas9 типа II по п. 1, отличающаяся тем, что система редактирования генома CRISPR/Cas9 типа II получена из Faecalibaculum rodentium, и в которой специфическая ДНК связывается или расщепляется в процессе редактирования генома, путем распознавания комплементарного спаривания направляющей РНК и мишени специфической ДНК, причем длина спаренной связывающей части направляющей РНК и мишени специфической ДНК составляет от 14 до 30 пар оснований;
ри этом специфическая ДНК представляет собой ДНК прокариот или эукариот.
7. Система редактирования генома CRISPR/Cas9 типа II по п. 6, отличающаяся тем, что длина спаренной связывающей части направляющей РНК и мишени специфической ДНК составляет 21 пару оснований, 22 пары оснований или 23 пары оснований, и при этом комплекс РНП очень чувствителен к несоответствию 14 пар оснований, близких к мотиву, смежному с протоспейсером, а 14 пар оснований являются затравочной областью.
8. Система редактирования генома CRISPR/Cas9 типа II по п. 7, в которой мотив, смежный с протоспейсером, необходимый для функции связывания или расщепления ДНК, представляет собой 5'-NNTA-3' по направлению к нижнему концу последовательности распознавания направляющей РНК/sgРНК, где N является A, T, C или G.
9. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп.1-8 для редактирования ДНК.
10. Применение по п. 9, где ДНК представляет собой прокариотическую или эукариотическую ДНК.
11. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для связывания ДНК.
12. Применение по п. 11, где ДНК представляет собой прокариотическую или эукариотическую ДНК.
13. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для CRISPR активации.
14. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для CRISPR интерференции.
15. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для получения никазы.
16. Применение по п. 15, где никазу получают в прокариоте или эукариоте.
17. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для получения мертвого Cas9.
18. Применение по п. 17, где мертвый Cas9 получают в прокариоте или эукариоте.
19. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для получения редактора оснований.
20. Применение по п. 19, где редактор оснований получают в прокариоте или эукариоте.
21. Применение системы редактирования генома CRISPR/Cas9 типа II по любому из пп. 1-8 для получения главного редактора.
22. Применение по п. 21, где главный редактор получают в прокариоте или эукариоте.
| База данных NCBI Reference Sequence: WP_217947137.1, 18.02.2022 | |||
| XUELIAN WANG et al., CRISPR-Cas9 System as a Versatile Tool for Genome Engineering in Human Cells, Mol Ther Nucleic Acids | |||
| Токарный резец | 1924 |
|
SU2016A1 |
| WO2018039145 A1, 01.03.2018 | |||
| US2015079681 A1, 19.03.2015 | |||
| ПУГАЧ К.С | |||
| и др | |||
| "CRISPR-системы адаптивного иммунитета прокариот", | |||
Авторы
Даты
2023-04-24—Публикация
2022-02-21—Подача