Область техники, к которой относится изобретение
Настоящее изобретение относится к области биохимии и может быть использовано в молекулярно-генетической диагностике. Предложенный способ обнаружения нуклеиновых кислот патогенов N. meningitidis, S. pneumoniae, Н. influenzae, Е. coli, L. monocytogenes, S. agalactiae, С.albicans, М. tuberculosis, S. aureus, HSV 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В предполагает проведение двух этапов исследования: 1) мультиплексная изотермическая амплификации с использованием петлевых праймеров и 2) последующая синглплексная детекция продуктов амплификации сенсорами на основе ДНКзимов. Данный способ обнаружения позволяет достичь высоких показателей чувствительности и специфичности и сократить время исследования до менее 2 часов, что может быть применено для диагностики инфекций, вызванных вышеперечисленными патогенами, а также для целей научных исследований.
Уровень техники
Обнаружение нуклеиновых кислот является одним из основных методов молекулярно-генетической диагностики инфекций: подход позволяет быстро идентифицировать возбудитель заболевания посредством амплификации специфического фрагмента нуклеиновой кислоты генома патогена-возбудителя, рибонуклеиновой (РНК) или дезоксирибонуклеиновой кислоты (ДНК). Скорость проведения исследования имеет особо важное значение для стремительно развивающихся воспалительных заболеваний с высоким уровнем инвалидизации, а мультиплексный подход - для инфекций, вызываемых широким спектром патогенов. Есть группа инфекций, для которых критичными являются оба параметра - и скорость реакции, и мультиплексный подход. Это, к примеру, инфекции центральной нервной системы.
Широкий спектр потенциальных возбудителей нейроинфекций, тяжесть последствий и размытость клинической картины становятся причиной использования большого набора тестов, используемых для выявления патогенов в спинномозговой жидкости (СМЖ). Подобные тестирования требуют наличия лабораторных условий и специально обученного персонала, а также функционирования полноценной клинико-диагностической лаборатории. Существует несколько основных подходов к исследованию СМЖ. Традиционные микробиологические методы, такие как посев, могут быть специфичны, однако они имеют низкую чувствительность, а постановка диагноза в случае их использования занимает несколько дней. Серологические тесты часто применяются в последнее время, однако они не позволяют различить острую форму инфекции от хронической. Молекулярно-генетические тесты, такие как полимеразная цепная реакция, намного удобнее и точнее, чем микробиологические посевы, к тому же могут определять патогены различной природы. Однако одним из недостатков серологических и молекулярно-биологических исследований является необходимость выбора целевых патогенов для проведения исследований, а также соответствующие материальные, временные и трудовые затраты. Все вышеобозначенные проблемы способны решить мультиплексные молекулярно-диагностические тест-системы.
На сегодняшний день к данному уровню техники можно отнести несколько существующих тест-систем.
В качестве ближайшего аналога может быть представлена система компании Biomerieux для мультиплексной детекции патогенов: панель ME (Meningitis/Encephalitis), позволяющая определять одновременно 14 патогенов центральной нервной системы в СМЖ (Е. coli, Н. influenzae, L. monocytogenes, N. meningitidis, S. agalactiae, S. pneumoniae, Cryptococcus neoformans/gattii, Cytomegalovirus, Coxsackievirus B, HSV 1/2, Human herpesvirus 6, Varicella Zoster virus и Parechovirus) по анализу кривых плавления после проведения полимеразной цепной реакции (ПЦР) в реальном времени, панель RP (Respiratory), позволяющая определять одновременно 21 патоген, панель для идентификации 43 возбудителей сепсиса BioFire BCID2 panel. Однако все эти панели предназначены для использования в закрытой тест-системе FilmArray, основанной на проведении полимеразной цепной реакции в режиме реального времени с накапливанием флуоресцентно меченого продукта амплификации. Данный способ детекции предполагает использование дорогостоящих реагентов, что препятствует массовизации метода для диагностики инфекций.
Известны диагностические системы компании «Т2 Biosystems», которые предназначены для прямой детекции ряда бактериальных патогенов (Enterococcus faecium, Е. coli, Klebsiella pneumoniae, Pseudomonas aeruginosa, Staphylococcus aureus) в различных биологических жидкостях, включая кровь, без предварительного выделения нуклеиновых кислот и их очистки. Реакции лизиса клеточных компонентов биологического материала и бактерий, этап амплификации и детекции ампликонов с помощью патентованной технологии Т2 магнитного резонанса проводятся автоматически в одном картридже в специальном аппарате (https://www.t2biosystems.com/t2direct-diagnostics-eu/t2bacteria-panel-eu/).
Еще одним изобретением для мультиплексной идентификации возбудителей инфекций являются тест-системы с наборами специфических праймеров для диагностики серии «Амплисенс»: «Амплисенс N.meningitidis/H. influenzae/S.pneumoniae-FL» для диагностики нейроинфекций и «Амплисенс
С.trachomatis/ M.genitalium/ Ureaplasma-FRT» для выявления ДНК возбудителей инфекций урогенитального тракта в клиническом материале методом ПЦР с гибридизационно-флуоресцентной детекцией (https://interlabservice.ru/catalog/reagents/?sid=1495&id=6505).
Главными недостатками всех известных систем являются их сложность, высокая стоимость, низкая мобильность по причине необходимости юстировки лазеров прибора при перемещении и длительность проведения анализа. Два последних изобретения, наборы реагентов «Амписенс» и диагностические системы компании «Т2 Biosystems», помимо того, что они также, как и панели Biomerieux, не могут быть использованы в местах предоставления медицинской помощи, нацелены на обнаружение ограниченного количества бактерий-возбудителей инфекционных заболеваний.
Таким образом, в результате поиска способа мультиплексного обнаружения 16 патоген-специфичных последовательностей нуклеиновых кислот не обнаружено.
Технической задачей является создание способа этиологической идентификации нуклеиновых кислот 16 возбудителей различных инфекций в относительно короткий промежуток времени, применение которого не требует использования дорогостоящих реагентов.
Описание иллюстраций
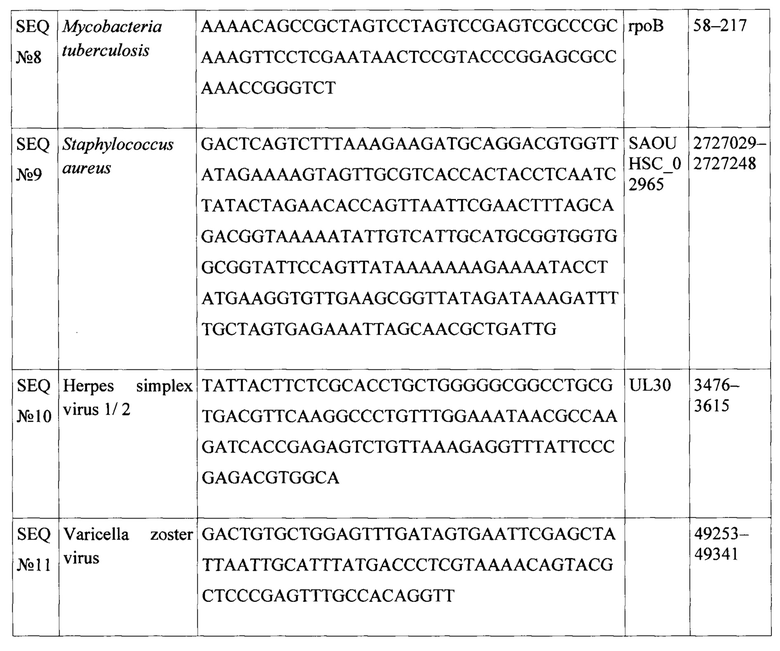
Фигура 1 - Схематическое изображение работы заявляемых петлевых праймеров для реакции изотермической амплификации [Luo G, et al. Stem-loop-primer assisted isothermal amplification enabling high-specific and ultrasensitive nucleic acid detection. Biosens Bioelectron. 2021 Jul 15;184:113239. doi: 10.1016/j.bios.2021.113239].
Фигура 2 - Схематическое изображение работы трехкомпонентной системы ДНКзимных сенсоров [Amanda J.C., et al. DNA antenna tile-associated deoxyribozymes sensor with improved sensitivity. Chembiochem. 2016 Nov 3; 17(21):2038-2041. doi: 10.1002/cbic.201600438].
Фигура 3 - Результаты детекции 16 патогенов при использовании заявляемых сенсоров электрофорез реакции амплификации.
Фигура 4 - Гель-электрофорез продуктов амплификации ДНК Е. coli. Слева направо: 3 технических повторности экспериментальной изотермической амплификации ДНК Е. coli, маркер веса Евроген 50+bp, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 5 - Гель-электрофорез продуктов амплификации ДНК S. pneumoniae. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК S. pneumoniae, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 6 - Гель-электрофорез продуктов амплификации ДНК N. meningitides. Слева направо: 3 технических повторности негативного контроля, 3 технических повторности из экспериментальной пробирки с добавленной ДНК N. meningitidis. Агароза 2%, 80 V.
Фигура 7 - Гель-электрофорез продуктов амплификации ДНК Н. influenzae. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК Н. influenzae, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 8 - Гель-электрофорез продуктов амплификации ДНК S. agalactiae. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК S. agalactiae, 3 технических повторности негативного контроля. Агароза 2%, 80 V
Фигура 9 - Гель-электрофорез продуктов амплификации ДНК L. monocytogenes. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК L. monocytogenes, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 10 - Гель-электрофорез продуктов амплификации ДНК S. aureus. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК S. aureus, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 11 - Гель-электрофорез продуктов амплификации М. tuberculosis. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК М. tuberculosis, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 12 - Гель-электрофорез продуктов амплификации ДНК С.albicans. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК С.albicans, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 13 - Гель-электрофорез продуктов амплификации ДНК HSV 1. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК HSV 1, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 14 - Гель-электрофорез продуктов амплификации ДНК HSV 2. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК HSV 2, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 15 - Гель-электрофорез продуктов амплификации ДНК Varicella Zoster virus. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК Varicella Zoster virus, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 16 - Гель-электрофорез продуктов амплификации ДНК Epstein-Barr virus. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК Epstein-Barr virus, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 17 - Гель-электрофорез продуктов амплификации Cytomegalovirus. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК Cytomegalovirus, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 18 - Гель-электрофорез продуктов амплификации Human herpesvirus 6. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК Human herpesvirus 6, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 19 - Гель-электрофорез продуктов амплификации Coxsackievirus В. Слева направо: 3 технических повторности из экспериментальной пробирки с добавленной ДНК Coxsackievirus В, 3 технических повторности негативного контроля. Агароза 2%, 80 V.
Фигура 20 - Результаты измерения флуоресцентного сигнала после гибридизации сенсоров к 16 патогенам, определяемым набором реагентов, с продуктом амплификации ДНК микроорганизма Н. influenzae. Идентификация нуклеиновых кислот лабораторного образца бактериального патогена при помощи заявляемого набора реагентов.
Фигура 21 - Результаты измерения флуоресцентного сигнала после гибридизации сенсоров к 16 определяемым набором реагентов патогенам с продуктом амплификации ДНК Human herpesvirus 6. Идентификация нуклеиновых кислот лабораторного образца вирусного патогена при помощи заявляемого набора реагентов.
Раскрытие изобретения
Настоящее техническое решение заключается в создании способа идентификации 16 возбудителей инфекций как бактериальной и вирусной, так и грибковой природы.
Техническим результатом создание тест-системы для определения этиологии инфекционных заболеваний, вызванных патогенами Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, Escherichia coli, Listeria monocytogenes, Streptococcus agalactiae, Candida albicans, Mycobacterium tuberculosis, Streptococcus aureus, Herpes simplex virus 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus B.
Технический результат достигается за счет того, что высокоспецифичные зонды, представленные ДНКзимами, при обнаружении целевой нуклеиновой кислоты возбудителя инфекционного заболевания (аналита) специфически связываются с ее участками и формируют гуаниновый квадруплекс (Г-4), обладающий ферментативной активностью пероксидазы, что позволяет выявлять специфичные фрагменты нуклеиновых кислот патогена посредством колориметрической реакции либо реакции с производством флуоресцентного продукта. В отсутствие аналита ДНКзимные сенсоры находятся преимущественно в диссоциированной форме. При добавлении в раствор аналита сенсоры собираются в комплексную структуру, которая и является ДНКзимом, способным связывать субстрат. Данный метод позволяет использовать различные субстраты и различные способы детектирования результата реакции - как колориметрические, так и световые.
С целью достижения высокой чувствительности и для облегчения потенциальной интеграции реагентов в мобильную тест-систему стадии детекции предшествует стадия изотермической амплификации с использованием петлевых праймеров, SPA (stem-loop-primer assisted amplification).
Суммарное время исследования проверки образца на 16 патогенов составляет около 90 минут.
Заявляемый набор реагентов нацелен на обнаружение последовательностей нуклеиновых кислот патогенов SEQ №1 - SEQ №15, что позволяет идентифицировать следующие патогены: N. meningitidis, S. pneumoniae, Н. influenzae, Е. coli, L. monocytogenes, S. agalactiae, C. albicans, M. tuberculosis, S. aureus, Herpes simplex virus 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В. В случае Coxsackievirus В, который является РНК-вирусом, последовательность представлена комплементарной последовательностью ДНК.
Олигонуклеотиды для изотермической амплификации специфической последовательности патогена представляют собой 15 наборов из четырех праймеров: SEQ №16-SEQ №75.
Г-4 зонды представлены олигонуклеотидами с последовательностями SEQ №76 - SEQ №120, содержащими участки, распознающие одноцепочечные нуклеиновые кислоты возбудителя инфекционного заболевания.
Новизна заявляемого технического решения заключается в том, что для получения информации о наличии нуклеиновой кислоты используется комбинация методов изотермической амплификации и гибридизации с ДНКзимными сенсорами в 15 отдельных пробирках с использованием недорогих реагентов, включая BST-полимеразу. Праймеры и сенсоры системы не различают Herpes simplex virus 1 и 2 типа, однако распознают любой из них. Для реализации каждой из стадий детекции были использованы праймеры для изотермической амплификации SPA, представленные в группе последовательностей SEQ №16 - SEQ №75, и последовательности ДНКзимных сенсоров, представленные в группе последовательностей SEQ №76 - SEQ №120. Данные реакции и реагенты могут быть как объединены в единой разработке для мультиплексной детекции, так и использованы для обнаружения нуклеиновых кислот и идентификации патогенов N. meningitidis, S. pneumoniae, Н. influenzae, Е. coli, L. monocytogenes, S. agalactiae, C. albicans, M. tuberculosis, S. aureus, Herpes simplex virus 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В в отдельных 15 пробирках.
Для обнаружения всех определяемых патогенов, кроме Coxsackievirus В, нужно в пробирке приготовить раствор следующего состава:
- буфер для BST-полимеразы 1х;
- сульфат магния 8 мМ;
- этиленгликоль 0,5%;
- дезоксирибонуклеотиды 1,4 мМ;
- короткие праймеры для соответствующего патогена (F и R из группы последовательностей SEQ№16 - SEQ №75 для поиска и амплификации фрагментов геномов патогенов SEQ №1 - SEQ №15) по 0,4 мкМ;
- петлевые праймеры для соответствующего патогена (LF и LR из группы последовательностей SEQ №16 - SEQ №75) по 1,6 мкМ;
- BST-полимераза 8U;
- нуклеиновые кислоты патогена в количестве, соответствующем 100 геномным эквивалентам (1000 геномным эквивалентам каждого из патогенов в случае мультиплексного образца нуклеиновых кислот);
- вода высокоочищенная до 50 мкл (при необходимости);
- термостабильная ревертаза (для обнаружения Coxsackievirus В).
Необходимо инкубировать полученную смесь при 65°С в течение 60 минут, далее 5 мкл смеси после амплификации добавить в раствор следующего состава:
- 50 мМ HEPES, рН 7,4;
- 70 мМ хлорида калия;
- 160 мМ хлорида натрия;
- 50 мМ хлорида магния;
- 1% ДМСО;
- 0,03% Тритон Х-100;
- соответствующий набор сенсоров в концентрации 1 мкМ каждой части (SEQ №76 - SEQ №120);
- субстрат для ДНКзимов: концентрация зависит от типа субстрата (например, флуорофор тиофлавин Т 0,5 мкМ).
Необходимо инкубировать полученную смесь в течение 5 минут, далее измерить результат окисления субстрата ДНКзимами в пробирке: в случае использования тиофлавина Т измерить флуоресценцию при длине волны возбуждения 450 нм и волны эмиссии 520 нм.
После завершения эксперимента результаты могут быть интерпретированы по наличию цветового или светового сигнала, количественно отражающего активность собранных ДНКзимных комплексов после обнаружения аналита. Такой порядок действий и такая репрезентация делают результат однозначным и позволяют использовать реагенты без ужесточения мер биологической безопасности. Использование одной пробы для обнаружения сразу 16 патогенов экономит время, затрачиваемое на получение результата.
Заявляемое техническое решение рекомендовано к использованию в медицинской клинической практике, эпидемиологии и научных исследованиях, а именно как устройство для мультиплексного обнаружения патоген-специфичных нуклеиновых кислот, что соответствует критерию «промышленная применимость».
Предлагаемое изобретение поясняется фигурами со схематическими изображениями стадий исследования и примерами использования (фигуры 1-21).
Создание тест-системы
Синтетические олигонуклеотиды произведены компаниями ДНК-Синтез, Россия и Евроген, Россия. Сконструированы последовательности гибридизационных ДНК-зондов на основе Г-4 (SEQ №76 - SEQ №120) для детекции одноцепочечной ДНК N meningitidis, S. pneumoniae, Н. influenzae, Е. coli, L. monocytogenes, S. agalactiae, C. albicans, M. tuberculosis, S. aureus, Herpes simplex virus 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В, а также петлевые праймеры для изотермической амплификации (SEQ №16 - SEQ №75).
Для фундаментальной разработки бинарных Г-4 зондов были проанализированы патоген-специфичные консервативные участки геномов N. meningitidis, S. pneumoniae, Н. influenzae, Е. coli, L. monocytogenes, S. agalactiae, C. albicans, M. tuberculosis, S. aureus, Herpes simplex virus 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В и выделены для посадки Г-4 зондов. В качестве спейсера использовали триэтиленгликоль или политимидин. Рабочая концентрация зондов составляет 1 мкМ для каждого компонента сенсорной системы.
Набор праймеров для каждого патогена из группы последовательностей SEQ№16 - SEQ №75 амплифицирует соответствующий фрагмент генома из набора последовательностей SEQ №1 - SEQ №15 в соответствии с протоколом реакции, указанным выше в разделе «Раскрытие изобретения». Чувствительность каждого набора праймеров для одного патогена составляет менее 10 геномных эквивалентов для образцов, содержащих нуклеиновые кислоты одного патогена, и менее 100 геномных эквивалентов для образцов, содержащих нуклеиновые кислоты двух и более патогенов. Каждый набор праймеров расфасован в 15 пробирок, уже содержащих все остальные составляющие реакционной смеси. Далее в такие наборы добавляют образцы выделенной из лабораторного образца нуклеиновой кислоты патогена. После инкубируют пробирки при 65°С в течение 60 минут.
Для визуализации в качестве субстрата нужно использовать флуорофор тиофлавин Т, который в результате связывания с Г-4 комплексом генерирует флуоресцентный сигнал. Для этого переносят по 5 мкл амплификационной смеси в пробирки для гибридизации с соответствующими ДНКзимными сенсорами, содержащими все необходимые для реакции ингредиенты, а через 5 минут замеряют уровень флуоресценции при помощи флуориметра Tecan при длине волны возбуждения 450 нм и длине волны эмиссии 520 нм. Результаты отражает фигура 3.
Полная длительность анализа со всеми манипуляциями исследователя составляет 90 минут.
Примеры использования тест-системы
Пример 1. Использование реагентов для определения лабораторных образцов патогенов
Изотермическая амплификация выделенных из патогенов нуклеиновых кислот -амплификация целевого патоген-специфического фрагмента (SEQ№1 - SEQ №15) нуклеиновых кислот лабораторных образцов культур
Для амплификации нуклеиновых кислот каждого патогена, кроме Coxsackievirus В, необходимо приготовить реакционную смесь следующего состава:
- буфер для BST-полимеразы 1х;
- сульфат магния 2 мМ;
- этиленгликоль 0,5%;
- дезоксирибонуклеотиды 1,4 мМ;
- короткие праймеры (F и R из группы последовательностей SEQ№16 - SEQ №75) по 0,4 мкМ;
- петлевые праймеры (LF и LR из группы последовательностей SEQ№16 - SEQ №75) по 0,8 мкМ;
- BST-полимераза 8U (полимераза, выделенная из Bacillus stearothermophilus);
- выделенная из лабораторной культуры патогена ДНК (в количестве, соответствующем 50 геномным эквивалентам (ГЭ)).
Для вируса Коксаки В необходимо приготовить реакционную смесь следующего состава:
- буфер для BST 1х;
- сульфат магния 2 мМ;
- этиленгликоль 0,5%;
- дезоксирибонуклеотиды 1,4 мМ;
- короткие праймеры (F и R) по 0,4 мкМ;
- петлевые праймеры (LF и LR) по 0,8 мкМ;
- термостабильная ревертаза 3U;
- BST-полимераза 8U;
- РНК 0,7 фг (соответствует около 100 ГЭ).
Реакция амплификации протекает при 65°С в течение 60 минут.
Пример 2. Идентификация неизвестных лабораторных образцов с помощью разработанных реагентов
Образец №1
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца бактериального патогена с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: Н. influenzae.
Результат идентификации методом ПЦР: Н. influenzae.
Образец №2
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца инактивированного вирусного патогена с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и длине волны эмиссии 520 нм (фигура 21).
Результат идентификации: Human herpesvirus 6.
Результат идентификации методом ПЦР: Human herpesvirus 6.
Образец №3
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца бактериального патогена с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: S. agalactiae
Результат идентификации методом ПЦР: S. agalactiae.
Образец №4
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца патогена грибов с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: С.albicans.
Результат идентификации методом ПЦР: С.albicans.
Образец №5
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца бактериального патогена с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: Е. coli.
Результат идентификации методом ПЦР: Е. coli.
Образец №6
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца бактериального патогена с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: L. monocytogenes.
Результат идентификации методом ПЦР: L. monocytogenes.
Образец №7
Проводили изотермическую амплификацию ДНК, выделенной из образца культуры клеток, зараженных неизвестным вирусным патогеном с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: Cytomegalovirus
Результат идентификации методом ПЦР: Cytomegalovirus.
Образец №8
Проводили изотермическую амплификацию ДНК, выделенной из образца культуры клеток, зараженных неизвестным вирусным патогеном с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: Epstein-Barr virus.
Результат идентификации методом ПЦР: Epstein-Barr virus.
Образец №9
Проводили изотермическую амплификацию ДНК, выделенной из образца культуры клеток, зараженных неизвестным вирусным патогеном с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: Varicella-zoster virus.
Результат идентификации методом ПЦР: Varicella-zoster virus.
Образец №10
Проводили изотермическую амплификацию ДНК, выделенной из неизвестного лабораторного образца бактериального патогена с использованием описанного выше протокола без добавления термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: М. tuberculosis.
Результат идентификации методом ПЦР: М. tuberculosis.
Образец №11
Проводили изотермическую амплификацию РНК, выделенной из образца культуры клеток, зараженных вирусом Коксаки с использованием протокола, описанного выше с добавлением термостабильной ревертазы. Полученную после амплификации смесь без предварительной очистки добавляли к ДНКзимным сенсорам по следующему протоколу:
- в СОХ-буфер добавляли 1 мкМ каждой из части сенсора, 0,5 мкМ тиофлавина, 5 мкл смеси после амплификации,
- все компоненты перемешивали,
- инкубировали в течение 5 минут при комнатной температуре,
- детектировали флуоресцентный сигнал при длине волны возбуждения 450 нм и волны эмиссии 520 нм (фигура 20).
Результат идентификации: Coxsackievirus В.
Результат идентификации методом ПЦР: Coxsackievirus В.
Пример 3: Пациент с респираторной инфекцией и с вестибулярными нарушениями - головокружением, рвотой, шумом и болью в ушах.
Для диагностики нейроинфекции исследовали спинномозговую жидкость при помощи тест-системы. Для этого выделяли нуклеиновые кислоты из 100 мкл спинномозговой жидкости, растворяли в 150 мкл воды и добавляли по 10 мкл полученного раствора в 15 пробирок с праймерами и растворами для амплификации (SEQ №16 - SEQ №75 для поиска и амплификации фрагментов геномов патогенов SEQ№1 - SEQ №15). После амплификации в течение 60 минут по 5 мкл амплификационных смесей добавляли в пробирки, содержащие соответствующие сенсоры (SEQ №76 - SEQ №120) и растворы для детекции. Через 5 минут после добавления замеряли уровень флуоресценции в каждой из 15 пробирок.
Длительность анализа составила 90 минут.
Результат исследования: в пробирке, где происходила гибридизация продукта амплификации с сенсорами для S. pneumoniae, показал флуоресцентный сигнал выше порогового.
Диагноз: пневмококковая инфекция, вызванная S. pneumoniae. Диагноз был подтвержден микробиологическими методами.
Пример 4: Пациент с подозрением на инфекцию центральной нервной системы с симптомами внезапной гипертермии, ознобом, вялостью, головной болью, спутанностью сознания и измененным поведением.
Для диагностики использовали образец спинномозговой жидкости поциента, полученной при помощи спинномозговой пункции в течение часа после госпитализации. Из 100 мкл спинномозговой жидкости выделяли нуклеиновые кислоты, затем растворяли в 150 мкл воды и добавляли по 10 мкл полученного раствора в 15 пробирок с праймерами и раствором для амплификации (SEQ№16 - SEQ №75 для поиска и амплификации фрагментов геномов патогенов SEQ№1 - SEQ№15). После амплификации по 5 мкл амплификационных смесей добавляли в пробирки, содержащие соответствующие сенсоры (SEQ№76 - SEQ №120) и растворы для детекции. Через 5 минут после добавления замеряли уровень флуоресценции в каждой из 15 пробирок.
Длительность анализа составила 90 минут.
Результат исследования: в пробирке, где происходила гибридизация продукта амплификации с сенсорами для идентификации Herpes simplex virus 1/2, показал флуоресцентный сигнал выше порогового.
Диагностическая специфичность зондов и чувствительность детекции аналитов составила 100%.
Диагноз: Herpes simplex virus  .
.
Диагноз был подтвержден методом ПЦР в реальном времени.
Пример 5. Пациент, демонстрирующий отказ от еды и питья, с бледностью кожи, рвотой, болями в мышцах, суставах, животе, судорожными подергиваниями отдельных мышц, головной болью, тахикардией, неустойчивостью артериального давления, ригидностью шеи, петехиями.
Брали спинномозговую жидкость, выделяли нуклеиновые кислоты и растворяли в 150 мкл воды, добавляли по 10 мкл полученного раствора в 15 пробирок с праймерами и растворами для амплификации из примера 1 (SEQ №16 - SEQ №75 для поиска и амплификации фрагментов геномов патогенов SEQ №1 - SEQ №15). После амплификации в течение 60 минут по 5 мкл амплификационных смесей добавляли в пробирки, содержащие соответствующие сенсоры (SEQ №76 - SEQ №120) и растворы для детекции. Через 5 минут после добавления замеряли уровень флуоресценции в каждой из 15 пробирок.
Длительность анализа составила 90 минут.
Результат исследования: в пробирке, где происходила гибридизация продукта амплификации с сенсорами для обнаружения N. meningitidis, показал флуоресцентный сигнал выше порогового.
Диагностическая специфичность зондов и чувствительность детекции аналитов составила 100%.
Диагноз: гнойный менингит, вызванный N. meningitidis.
Диагноз был подтвержден микробиологическими методами.
Пример 6. Пациент с гипертермией, плохим самочувствием, в анамнезе кашель.
При осмотре были выявлены симптомы инфекции нижних дыхательных путей. Был назначен посев мокроты. Далее выделяли нуклеиновые кислоты из чистой культуры и растворяли в 150 мкл воды, добавляли по 10 мкл полученного раствора в 15 пробирок с праймерами и растворами для амплификации из примера 1 (SEQ №16 - SEQ №75 для поиска и амплификации фрагментов геномов патогенов SEQ№1 - SEQ№15). После амплификации в течение 60 минут по 5 мкл амплификационных смесей добавляли в пробирки, содержащие соответствующие сенсоры (SEQ №76 - SEQ №120) и растворы для детекции. Через 5 минут после добавления замеряли уровень флуоресценции в каждой из 15 пробирок.
Длительность анализа составила 90 минут.
Результат исследования: в пробирке, где происходила гибридизация продукта амплификации с сенсорами для обнаружения S. aureus, показал флуоресцентный сигнал выше порогового.
Диагностическая специфичность зондов и чувствительность детекции аналитов составила 100%.
Диагноз: плеврит, вызванный S. aureus.
Диагноз был подтвержден микробиологическими методами.
Данные примеры демонстрируют, что определение этиологии инфекций при помощи ДНКзимных сенсоров показывает достоверные, подтверждаемые микробиологическими методами результаты.
Рабочие характеристики представленного набора реагентов:
1. Для визуализации реакции может быть использован любой субстрат, изменяющий свой цвет или испускающий флуоресцентный сигнал при окислении, в том числе диаминобензидин (ДАБ), 3,3',5,5'-тетраметилбензидин (ТМВ), 2,2-азино-бис (3-этилбензотиазолин-6-сульфокислота) (АБТС) и флуорофоры.
2. В качестве спейсера в ДНКзимных зондах может быть использован полиэтиленгликоль, триэтиленгликоль, гексаэтиленгликоль, политимидин.
3. Рабочая концентрация зондов может составлять 0,1-10 мкМ.
4. Для увеличения чувствительности тест-системы работа сенсоров совмещена с предварительной реакцией накопления аналита в виде изотермической амплификации. Реакция изотермической амплификации протекает при температуре 65°С в течение 60 минут в присутствии специфических петлевых праймеров.
5. Для использования в диагностике перед накоплением образец биологической жидкости с содержащимися в нем патогенами лизируется. Выделенные очищенные нуклеиновые кислоты используются в качестве матрицы для изотермической амплификации.
6. Заявляемый набор реагентов может быть помещен в любой формат тест-системы как для мультиплексной идентификации патогена, так и для синглплексных реакций.





--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Тест-система и
способ для обнаружения специфических фрагментов нуклеиновых кислот 16
патогенов с использованием изотермической реакции амплификации .xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-03-02">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2023125255</ApplicationNumberText>
<FilingDate>2023-03-02</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>123</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022123569</ApplicationNumberText>
<FilingDate>2022-03-02</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение "Центр стратегического планировния и
управления медико-биологическими рисками здоровью" Федерального
медико-биологического агентства</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Institution "Center
for Strategic Planning and Management of Biomedical Health
Risks" of the Federal Medical Biological
Agency</ApplicantNameLatin>
<InventionTitle languageCode="ru">Тест-система и способ для
обнаружения специфических фрагментов нуклеиновых кислот 16 патогенов
с использованием изотермической реакции амплификации
</InventionTitle>
<SequenceTotalQuantity>120</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>236</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..236</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacacaagaaatcggtttttcagccagaggcttatcgctttctgaagcc
attggccgtatgggcggtttgcaagatcgccgttctgatgcgcgtggtgtgtttgtgttccgctatacgc
cattggtggaattgccggcagaacgtcaggataaatggattgctcaaggttatggcagtgaggcagagat
tccaacggtatatcgtgtgaatatggctgatgcgcattcgctatttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>203</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..203</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctggaagaaaatcgctgacaagtggtactattttgatgtagaaggtgc
catgaagacaggctgggtcaagtacaaggacacttggtactacttagacgctaaagaaggcgccatggta
tcaaatgcctttatccagtcagcggacggaacaggctggtactacctcaaaccagacggaacactggcag
acaagccagagttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>196</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..196</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtgggttgtgagagactagaactctcgactgacggattaagagtccgc
tactctaccaactgagctaacaacccaactggacaactaaatcagagagtggtgggtcgtgaaggattcg
aaccttcgaccaacggattaaaagtccgctgctctaccgactgagctaacgaccccactgtattaaatgg
atttatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>204</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..204</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccatctcctgatgacgcatagtcagcccatcatgaatgttgctgtcga
tgacaggttgttacaaagggagaagggcatggcgagcgtacagctgcaaaatgtaacgaaagcctggggc
gaggtcgtggtatcgaaagatatcaatctcgatatccatgaaggtgaattcgtggtgtttgtcggaccgt
ctggctgcggtaaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>79</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..79</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Listeria
monocytogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaactgaagcaaaggatgcatctgcattccataaagaaaatttaattt
catccatggcaccaccagcatctccgcctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>215</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..215</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
agalactiae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtcgtgtagaagccttaacagatgtgattgaagcaatcactttttcaa
ctcaacatttaacaaataaggttagtcaagcaaatattgatatgggatttgggataactaagctagttat
tcgcattttagatccatttgcttcagttgattcaattaaagctcaagttaacgatgtaaaggcattagaa
caaaaagttttaacttatcctgattt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>106</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..106</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Candida albicans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctccagggtttgttaccttagactttaatgtcaaaagatcccttgttga
tccagatgatccaactgtcgaatctaaaagatcacctttatttttagatcttgatcc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>79</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..79</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaaacagccgctagtcctagtccgagtcgcccgcaaagttcctcgaata
actccgtacccggagcgccaaaccgggtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>232</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..232</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Staphylococcus
aureus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactcagtctttaaagaagatgcaggacgtggttatagaaaagtagttg
cgtcaccactacctcaatctatactagaacaccagttaattcgaactttagcagacggtaaaaatattgt
cattgcatgcggtggtggcggtattccagttataaaaaaagaaaatacctatgaaggtgttgaagcggtt
atagataaagattttgctagtgagaaattagcaacgctgattg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>113</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tattacttctcgcacctgctgggggcggcctgcgtgacgttcaaggccc
tgtttggaaataacgccaagatcaccgagagtctgttaaagaggtttattcccgagacgtggca</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>89</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..89</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactgtgctggagtttgatagtgaattcgagctattaattgcatttatg
accctcgtaaaacagtacgctcccgagtttgccacaggtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>242</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..242</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaattcgcgcatgatctcttcgaggtcaaaaacgttgctggaacgcag
ctctttctgcgagtaaagttccagtaccctgaagtcggtattttccagcgggtcgatatccagggcgatc
atgctgtcgacggtggagatactgctgaggtcaatcatgcgtttgaagaggtagtccacgtactcgtagg
ccgagttcccggcgatgaagatcttgaggctgggaagctgacattcctcagtg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>183</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..183</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggaataagcccccagacaggggagtgggcttgtttgtgacttcaccaa
aggtcagggcccaagggggttcgcgttgctaggccaccttctcagtccagcgcgtttacgtaagccagac
agcagccaattgtcagttctagggagggggaccactgcccctggtataaagtggtcctgcagct</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>93</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..93</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgagcgctaggttgaggatgatcgaaacgcctacacagaattttatgtt
tgtgacgagcgttattccttcgggtgtgacgtctggtgaaaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>80</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..80</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcgcctgttttataccccctctcccaactgtaacttagaagtaacac
acaccgatcaacagtcagcgtggcacaccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagaaatcggtttttcagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagcgaatgcgcatcagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>37</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..37</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acaccacgcgcatcagaacgtgaagccattggccgta</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgttccgctatacgccattggtcgttggaatctctgcctc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcccatcatgaatgttgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaattctggcctgtctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctaagtagtaccaagtgtccttgtggtactatttcaacgaagaaggt<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctaaagaaggcgccatggtcgtctggtttgaggtagt</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggttgtgagagactagaactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccatttaatacagtggggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agttgtccagttgggttgttagtcgactgacggattaagagt</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatcagagagtggtgggtcgtgcgttagctcagtcggtag</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccatctcctgatgacgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atttaccgcagccagacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggggcgaggtcgtggtattccgacaaacaccacgaatt</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattttgcagctgtacgctcgcagcccatcatgaatgttgct</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Listeria
monocytogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgcgcaacaaactgaagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Listeria
monocytogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcttttacgagagcacctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Listeria
monocytogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgtttcttttcgattggcgtctttttttcatccatggcaccacc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>51</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..51</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccacggagatgcagtgacaaatgttttggatttcttctttttctccaca
ac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
agalactiae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agaagccttaacagatgtga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
agalactiae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggataagttaaaaccttttgttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
agalactiae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagcttagttatcccaaatcccatagaagcaatcactttttcaactca<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attcgcattttagatccatttgcttgcctttacatcgttaacttgag</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Candida albicans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctccagttaaaagatttcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcatttctaccagtatcatcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>53</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..53</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Candida albicans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catctggatcaacaagggatctgaattcgggtttgttaccttagacttg
aatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>53</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..53</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Candida albicans</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaactgtcgaatctaaaagatcaccgaattcgggaatttgtgtgggat
taag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccgccagagcaaaacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cactcgaacgaatcggtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggaactttgcgggcgacgaattcccgctagtcctagtcc</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taacctgcgcgaaccacttgaggaattcacgtcaaggagtcacg</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Staphylococcus
aureus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtctttaaagaagatgcaggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgttgctaatttctcact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Staphylococcus
aureus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accgtctgctaaagttcgaattactagttgcgtcaccactac</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Staphylococcus
aureus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggtggcggtattccagttaataaccgcttcaacaccttc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttccgctcaacacggacta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccacgtctcgggaataaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaacgtcacgcaagcgaattcttacttctcgcacctgctg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>49</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..49</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccctgtttggaaataacgcgaattctctttaacagactctcggtgatc
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactgtgctggagtttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacctgtggcaaactcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctatataatatatagggactgtgctggagtttg</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctatataatatataggaacctgtggcaaactcg</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcgcgcatgatctcttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggaatgtcagcttcccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcgacccgctggaaaataccgttttgttgctggaacgcagctctt</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actgctgaggtcaatcatgcgtttttatcttcatcgccgggaactc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agaggaataagcccccagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accagaaatagctgcaggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q132">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagcaacgcgaacccccttggagtgggcttgtttgtgac</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcagtccagcgcgtttacgtctttataccaggggcagtgg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q136">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgctaggttgaggatgatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="69">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttttttcaccagacgtcacacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="70">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q140">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttatataatatataaacgctaggttgaggatgatc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="71">
<INSDSeq>
<INSDSeq_length>39</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttatataatatataaattttttcaccagacgtcacacc</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="72">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q144">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcgcctgttttataccccctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="73">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggtgtgccacgctgact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="74">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttatataatatataaagtgcgcctgttttataccccctc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="75">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttatataatatataaactggtgtgccacgctgact</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="76">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q152">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcccagggaggctagctcgtatagcggaacacaaacacacc</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="77">
<INSDSeq>
<INSDSeq_length>73</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..73</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agactagcagagaggtgacagtagtcagcttttttgccggcaattccac
caatggacaacgagaggaaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="78">
<INSDSeq>
<INSDSeq_length>62</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..62</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q156">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Neisseria
meningitidis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagcaatccatttatcctgacgttctttttttgctgactactgtcacc
tctctgctagtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="79">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcccagggaggctagctggtagtccgtcatgaactcttctttagttg<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="80">
<INSDSeq>
<INSDSeq_length>84</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..84</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q160">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagtccgttcgctgatgtccgtagcagtattttttctagattgcgtaa
gagttcgatataaaggcacaacgagaggaaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="81">
<INSDSeq>
<INSDSeq_length>61</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..61</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
pneumoniae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttttcggcaaacctgcttcatctgtttttttactgctacggacatcag
cgaacggactct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="82">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q164">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcccagggaggctagctctgatttagttgtccagttgggttgttag</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="83">
<INSDSeq>
<INSDSeq_length>76</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..76</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agactagcagagaggttcacagttgacgtaatttttttccttcacgacc
caccactctacaacgagaggaaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="84">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcagcggacttttaatccgttttttttttacgtcaactgtgaacctct
ctgctagtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="85">
<INSDSeq>
<INSDSeq_length>39</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggtagggttttttccaggctttcgttacattttgcagc</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="86">
<INSDSeq>
<INSDSeq_length>83</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..83</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q172">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgataccgttctcatctttacttctatgtcactacttatatttttttt
tcgataccacgacctcgccttttttggggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="87">
<INSDSeq>
<INSDSeq_length>98</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..98</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catggatatcgagattgatattttttttataactagtgacatagaagta
aagatgagaacgctatcatttttttgtacgctcgccatgcccttctccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="88">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q176">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Listeria
monocytogenes</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggtagggaaaaaatctttatggaatgcagatg</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="89">
<INSDSeq>
<INSDSeq_length>82</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..82</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgagcagagctagcgatccgttagctagtgacttgagtttctcaaaaaa
ccatggatgaaattaaattaaaaaagggaaggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="90">
<INSDSeq>
<INSDSeq_length>88</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..88</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q180">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagatgctggtggtgaaaaaagagaaactcaagtcactagctaacgga
tcgctagctctgctcgaaaaaacatcctttgcttcagtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="91">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
agalactiae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcccagggaggctagctcccaaatcccatatcaatatttgcttgac</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="92">
<INSDSeq>
<INSDSeq_length>85</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..85</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q184">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agactagcagagaggtgacagtagtcagcttttttggatctaaaatgcg
aataactagcttagttatacaacgagaggaaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="93">
<INSDSeq>
<INSDSeq_length>67</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Streptococcus
agalactiae</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttgagctttaattgaatcaactgaagcaaatttttttgctgactactg
tcacctctctgctagtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="94">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggttgggttttttgatcatctggatcaacaag</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="95">
<INSDSeq>
<INSDSeq_length>74</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..74</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgatattaatcatcaatctatctatcatccaccttttttatcttttaga
ttcgacagttgttttttgggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="96">
<INSDSeq>
<INSDSeq_length>82</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..82</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q192">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatctaaaaataaaggtgttttttggtggatgatagatagattgatgat
taatatcgttttttaagggatcttttgacatta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="97">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagggttgggttttttcgggcgactcggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="98">
<INSDSeq>
<INSDSeq_length>64</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..64</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q196">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caatttatataatcatattttacatattcttttttattcgaggaacttt
gttttttgggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="99">
<INSDSeq>
<INSDSeq_length>68</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..68</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q198">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgggtacggagttttttttgaatatgtaaaatatgattatataaattgt
ttttttaggactagcggct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="100">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q200">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcccagggaggctagctctgctaaagttcgaattaactggtgtt</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="101">
<INSDSeq>
<INSDSeq_length>80</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..80</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q202">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agactagcagagaggtgacagtagtcagcttttttgcatgcaatgacaa
tatttttaccgtcacaacgagaggaaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="102">
<INSDSeq>
<INSDSeq_length>60</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..60</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q204">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttataactggaataccgccaccaccttttttgctgactactgtcacctc
tctgctagtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="103">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q206">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggttgggttttttcgttatttccaaacagggaccc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="104">
<INSDSeq>
<INSDSeq_length>70</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..70</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q208">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaagagtctatatctacttcttctatatctagatttttttctcggtg
atcttggttttttgggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="105">
<INSDSeq>
<INSDSeq_length>72</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..72</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q210">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacctctttaacagactttttttctagatatagaagaagtagatataga
ctctttttttccttgaacgtcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="106">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q212">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagggttgggttttttaattaatagctcgaa</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="107">
<INSDSeq>
<INSDSeq_length>67</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q214">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagaagaattgagtactctataatagtagttttttagggtcataaatg
cttttttgggttgggacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="108">
<INSDSeq>
<INSDSeq_length>73</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..73</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q216">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcgtactgttttattttttctactattatagagtactcaattcttct
cattttttttcactatcaaactcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="109">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q218">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgacagcatgatcgccctggatatcgaccttttttgggtaggg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="110">
<INSDSeq>
<INSDSeq_length>48</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..48</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q220">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggttgggttttttcgctggaaaataccgacttcagggtactggaact<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="111">
<INSDSeq>
<INSDSeq_length>67</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q222">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaagagtctatatctacttcttctatatctttttttggtgtgtgtta
cttcttttttgggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="112">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q224">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcccagggaggctagcttttggtgaagtcacaaacaagccc</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="113">
<INSDSeq>
<INSDSeq_length>73</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..73</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q226">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtctagctgtctctctgctcagacgagcttttttttcccccttgggcc
ctgaccacaacgagaggaaacctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="114">
<INSDSeq>
<INSDSeq_length>56</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..56</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q228">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtggcctagcaacgcgaattttttaagctcgtctgagcagagagacag
ctagaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="115">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q230">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggttgggttttttcgctcgtcacaaacaccc</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="116">
<INSDSeq>
<INSDSeq_length>67</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q232">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccagatatagatgaagaagatatagagatatattttttacccgaagga
ataattttttgggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="117">
<INSDSeq>
<INSDSeq_length>69</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..69</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q234">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaccagacgtcattttttatatctctatatcttcttcatctatatctt
ttttataaaattctgtgtag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="118">
<INSDSeq>
<INSDSeq_length>29</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..29</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q236">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggttgggtttttttacttctaagtaccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="119">
<INSDSeq>
<INSDSeq_length>67</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q238">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaagagtctatatctacttcttctatatctttttttggtgtgtgtta
cttcttttttgggttggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="120">
<INSDSeq>
<INSDSeq_length>68</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..68</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q240">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgactgttgatcttttttagatatagaagaagtagatatagactcttt
ttttggagagggggtataa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Диагностическая тест-система для определения этиологии инфекционных заболеваний посредством обнаружения нуклеиновых кислот возбудителей инфекций | 2021 |
|
RU2798567C2 |
| Способ преимплантационного генетического тестирования болезни Гентингтона | 2024 |
|
RU2840728C1 |
| Способ преимплантационного генетического тестирования синдрома Мартина-Белл | 2022 |
|
RU2796834C1 |
| Способ преимплантационного генетического тестирования Синдрома Марфана | 2023 |
|
RU2837158C1 |
| СПОСОБ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799413C1 |
| Способ преимплантационного генетического тестирования болезни Штаргардта | 2023 |
|
RU2831940C1 |
| НАБОР ПРАЙМЕРОВ ДЛЯ ВЫЯВЛЕНИЯ STREPTOCOCCUS MUTANS МЕТОДОМ ИЗОТЕРМИЧЕСКОЙ ПЕТЛЕВОЙ АМПЛИФИКАЦИИ | 2022 |
|
RU2799414C1 |
| Способ преимплантационного генетического тестирования остеопетроза 4 типа | 2022 |
|
RU2795483C1 |
| Способ преимплантационного генетического тестирования ахондроплазии | 2022 |
|
RU2795482C1 |
| Способ преимплантационного генетического тестирования наследственной зонулярной катаракты | 2022 |
|
RU2799541C1 |
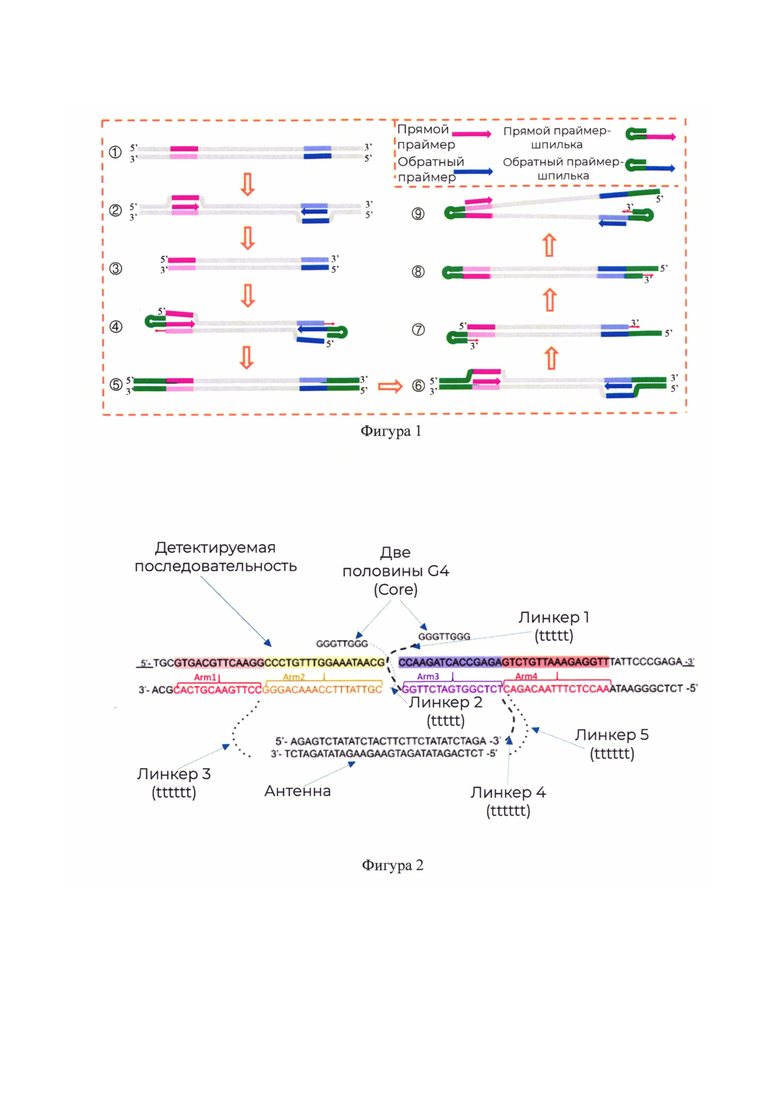


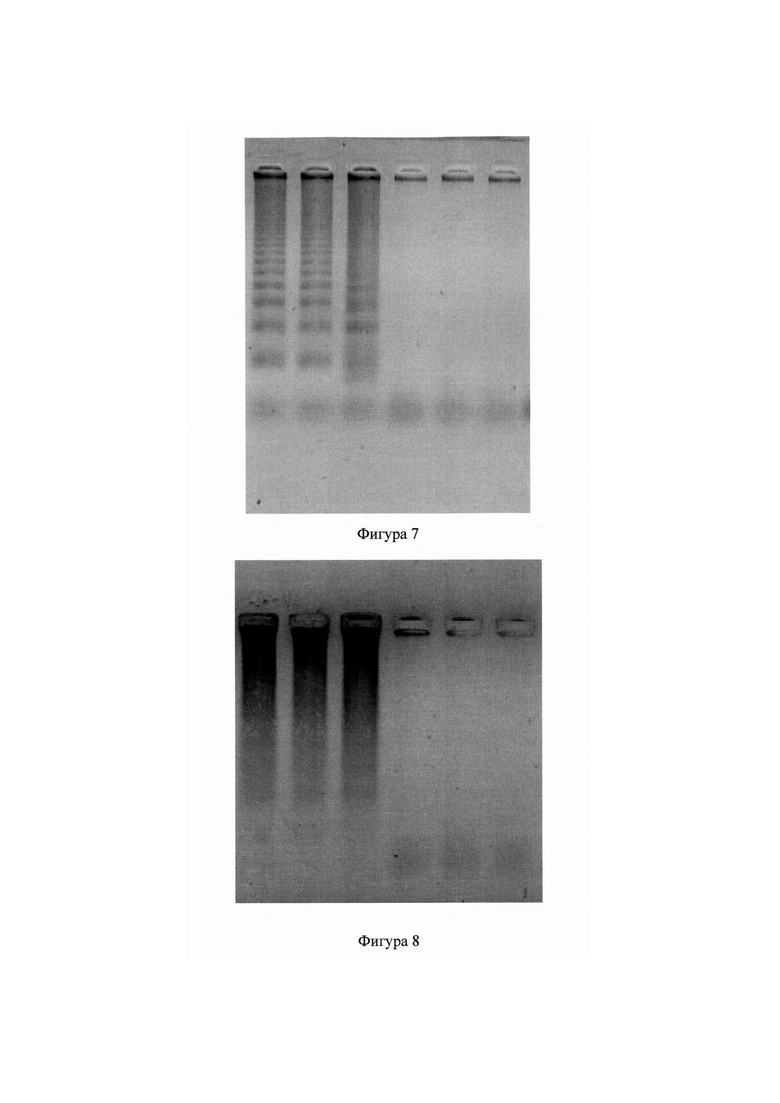
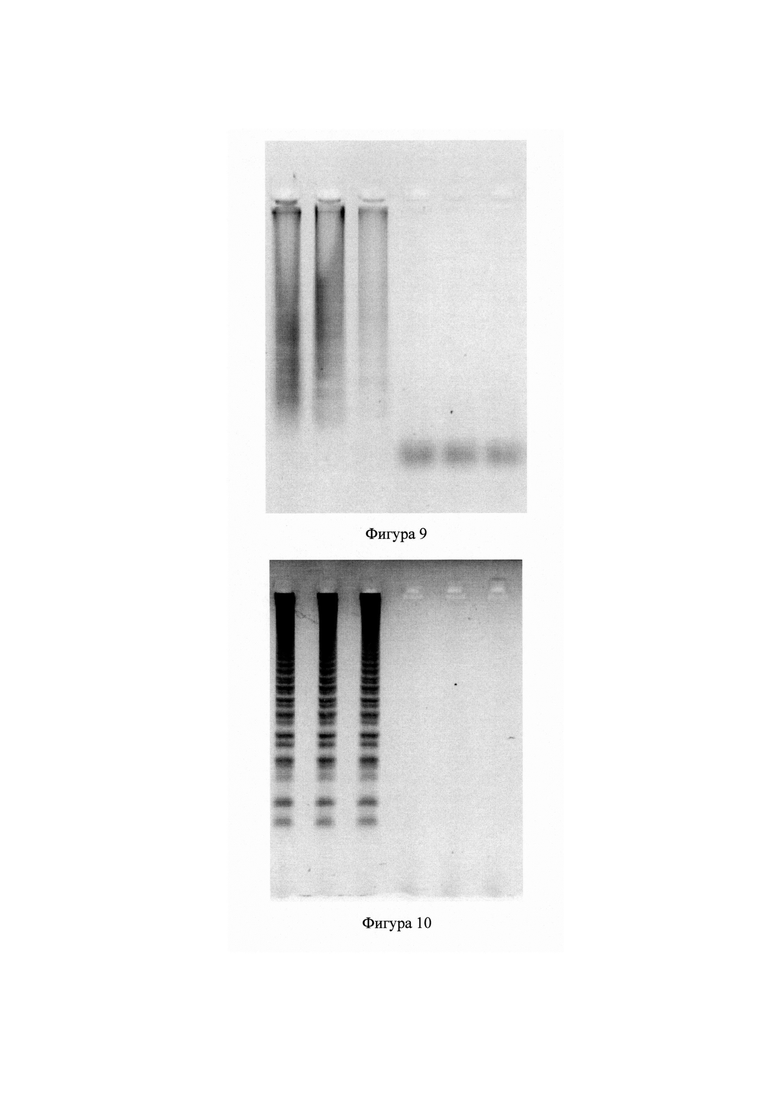
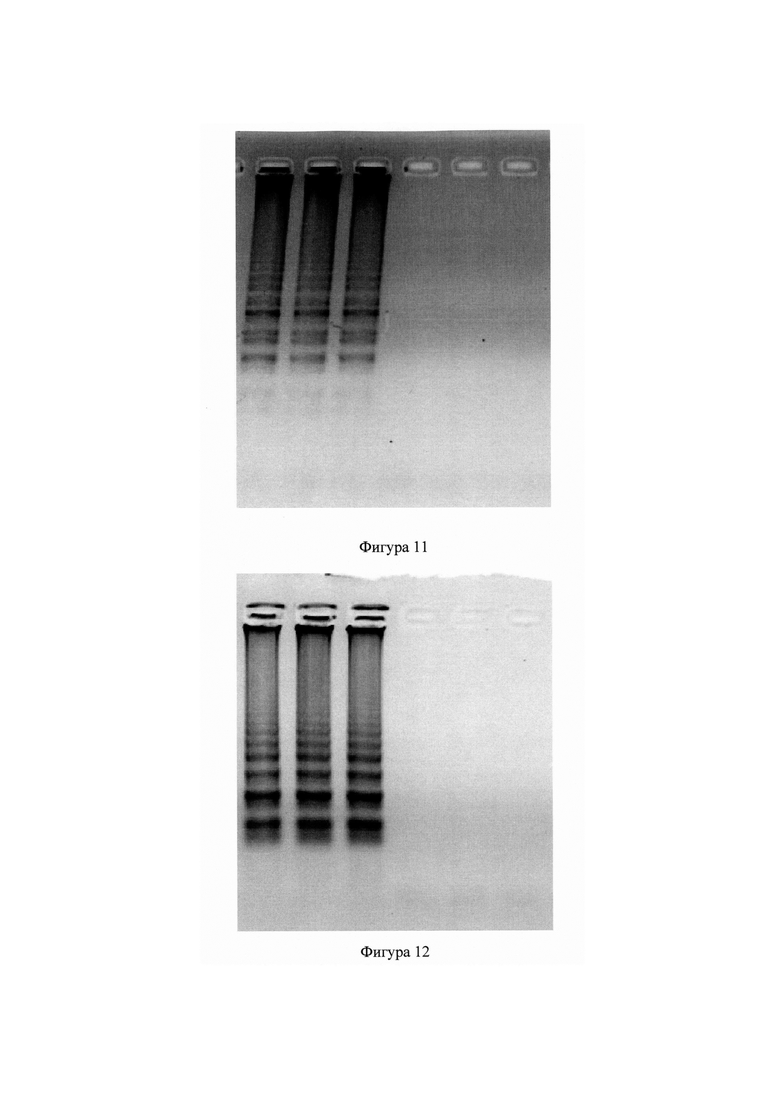
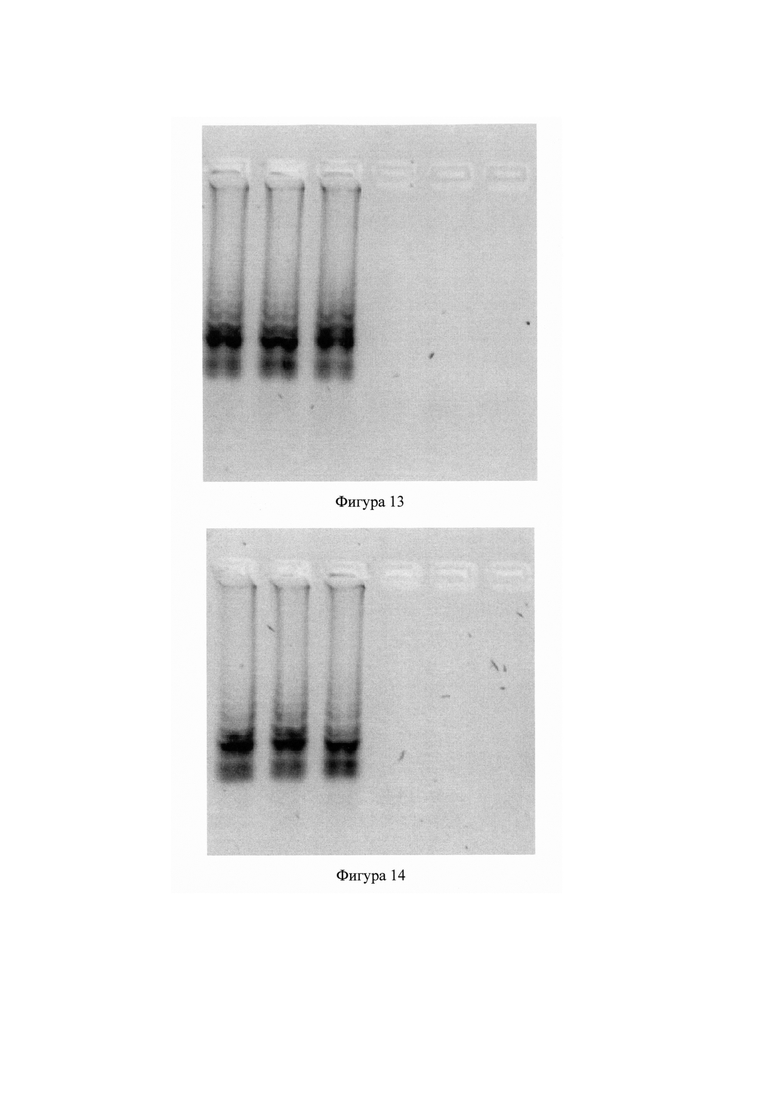
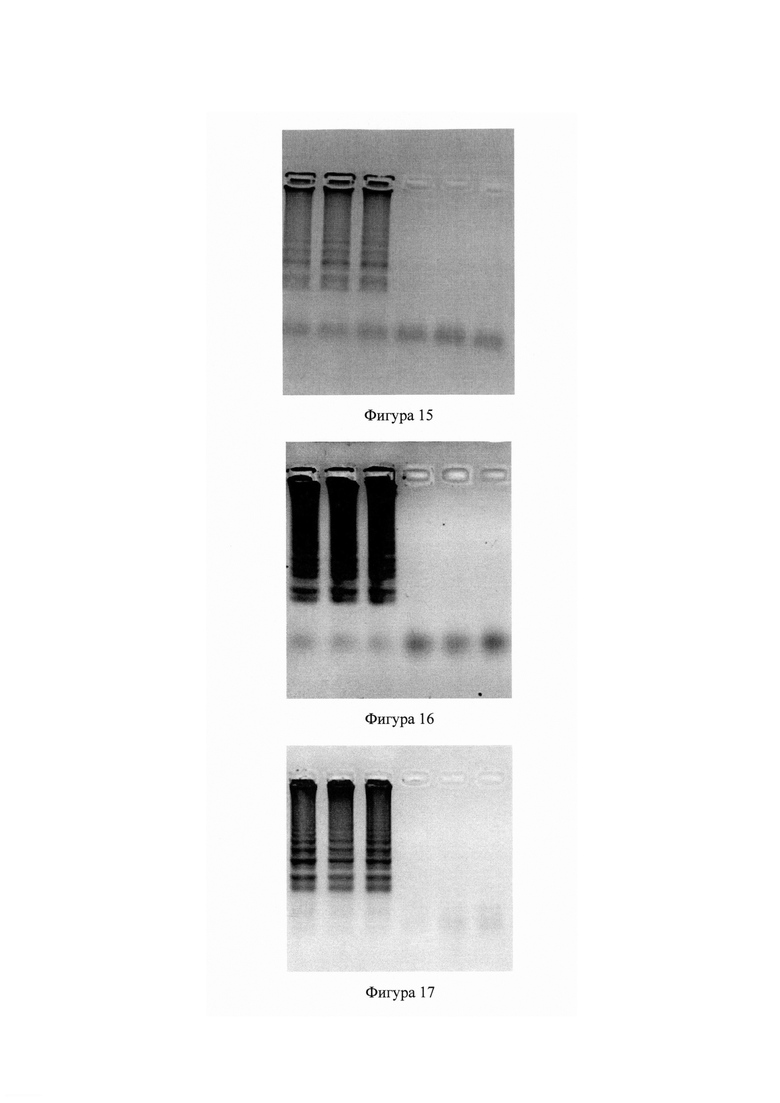
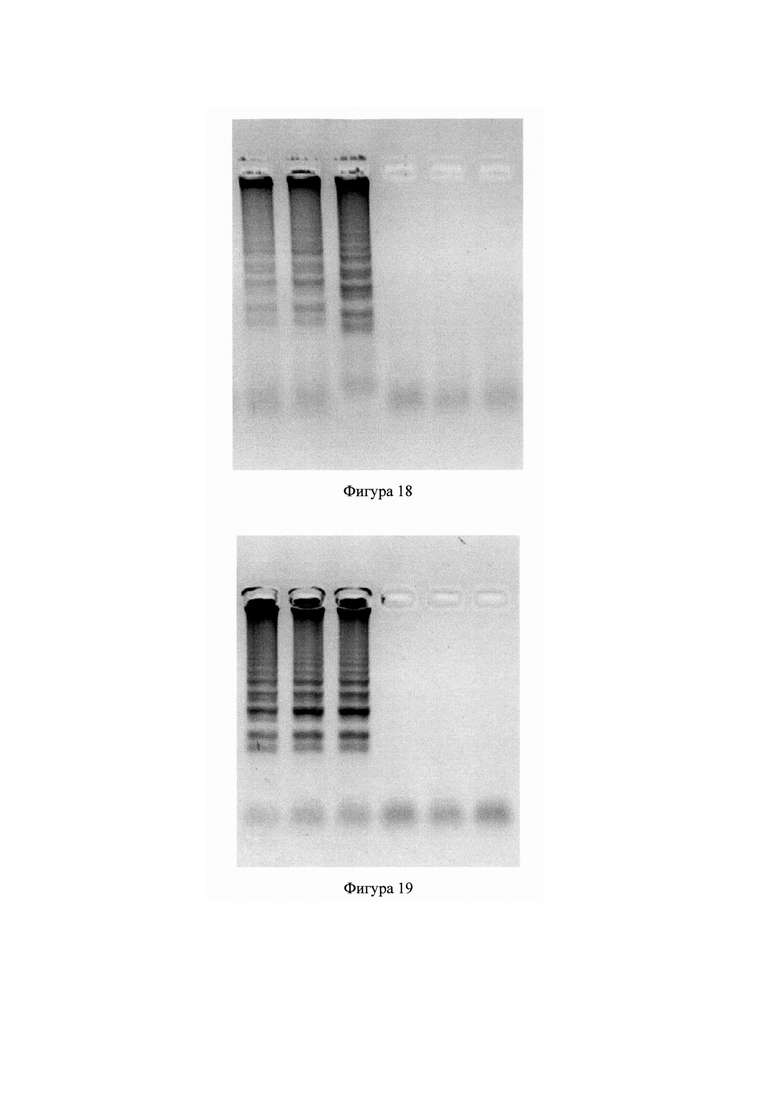
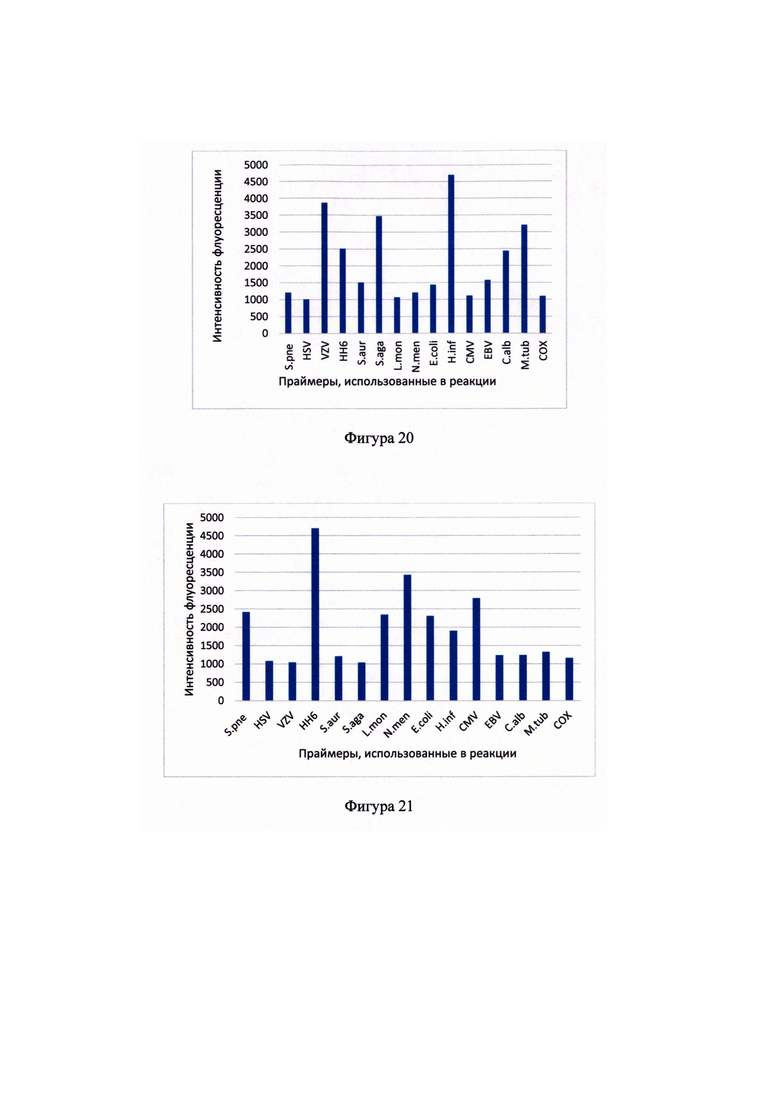
Изобретение относится к области биотехнологии. Предложена тест-система, содержащая набор реагентов для обнаружения в биологическом образце патогенов Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, Escherichia coli, Listeria monocytogenes, Streptococcus agalactiae, Candida albicans, Mycobacterium tuberculosis, Streptococcus aureus, Herpes simplex virus type 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В. Тест-система включает наборы олигонуклеотидов для изотермической амплификации специфических последовательностей геномов патогенов и ДНКзимные сенсоры, образующие гуаниновый комплекс Г-4. Тест-система используется в способе обнаружения указанных патогенов в биологическом образце. Изобретение направлено на детекцию продукта, накопленного методом циклической или изотермической ПЦР с использованием праймеров. Последующая визуализация реализуется посредством активации ферментативной активности гуанинового квадруплекса, который собирается при гибридизации ДНКзимных сенсоров с целевой последовательностью нуклеиновой кислоты (аналитом). Изобретение может быть использовано для диагностики инфекций, вызванных одним или несколькими указанными патогенами. 2 н.п. ф-лы, 21 ил., 1 табл., 6 пр.
1. Тест-система, содержащая набор реагентов для обнаружения в биологическом образце патогенов Neisseria meningitidis, Streptococcus pneumoniae, Haemophilus influenzae, Escherichia coli, Listeria monocytogenes, Streptococcus agalactiae, Candida albicans, Mycobacterium tuberculosis, Streptococcus aureus, Herpes simplex virus type 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus В, включающая:
А) наборы олигонуклеотидов для изотермической амплификации специфических последовательностей геномов патогенов, представленные в группе: SEQ ID NO: 16–19 Neisseria meningitides, SEQ ID NO: 20–23 Streptococcus pneumoniae, SEQ ID NO: 24–27 Haemophilus influenzae, SEQ ID NO: 28–31 Escherichia coli, SEQ ID NO: 32–35 Listeria monocytogenes, SEQ ID NO: 36–39 Streptococcus agalactiae, SEQ ID NO: 40–43 Candida albicans, SEQ ID NO: 44–47 Mycobacterium tuberculosis, SEQ ID NO: 48–51 Streptococcus aureus, SEQ ID NO: 52–55 Herpes simplex virus 1/2, SEQ ID NO: 56–59 Varicella Zoster virus, SEQ ID NO: 60–63 Cytomegalovirus, SEQ ID NO: 64–67 Epstein-Barr virus, SEQ ID NO: 68–71 Human herpesvirus 6, SEQ ID NO: 72–75 Coxsackievirus В и объединенные в 15 наборов по четыре олигонуклеотида в каждом, при этом упомянутые 15 наборов амплифицируют фрагменты геномов патогенов, представленные в группе последовательностей SEQ ID NO: 1–15;
Б) ДНКзимные сенсоры, образующие гуаниновый комплекс Г-4, содержащие участки, распознающие одноцепочечные нуклеиновые кислоты возбудителя инфекционного заболевания в биообразце, представленные в группе SEQ ID NO: 76–78 Neisseria meningitides, SEQ ID NO: 79–81 Streptococcus pneumoniae, SEQ ID NO: 82–84 Haemophilus influenzae, SEQ ID NO: 85–87 Escherichia coli, SEQ ID NO: 88–90 Listeria monocytogenes, SEQ ID NO: 91–93 Streptococcus agalactiae, SEQ ID NO: 94–96 Candida albicans, SEQ ID NO: 97–99 Mycobacterium tuberculosis, SEQ ID NO: 100–102 Streptococcus aureus, SEQ ID NO: 103–105 Herpes simplex virus 1/2, SEQ ID NO: 106–108 Varicella Zoster virus, SEQ ID NO: 109–111 Cytomegalovirus, SEQ ID NO: 112–114 Epstein-Barr virus, SEQ ID NO: 115–117 Human herpesvirus 6, SEQ ID NO: 118–120 Coxsackievirus В.
2. Способ обнаружения в биологическом образце возбудителей инфекций N. meningitidis, S. pneumoniae, Н. influenzae, Е. coli, L. monocytogenes, S. agalactiae, C. albicans, M. tuberculosis, S. aureus, HSV 1/2, Varicella Zoster virus, Cytomegalovirus, Epstein-Barr virus, Human herpesvirus 6, Coxsackievirus B, предусматривающий использование тест-системы по п. 1 путем выделения нуклеиновых кислот из биообразца, растворения нуклеиновых кислот в воде и добавления по 10 мкл полученного раствора в 15 пробирок с праймерами, представленными на участке SEQ ID NO: 16–75, для поиска и амплификации фрагментов геномов патогенов, которым соответствуют нуклеиновые кислоты, представленные на участке SEQ ID NO: 1–15, добавления после амплификации по 5 мкл амплификационных смесей в пробирки, содержащие соответствующие ДНКзимные сенсоры, представленные на участке SEQ ID NO: 76–120, оценки 5 мин спустя уровня флуоресценции в каждой из 15 пробирок.
| ГОРБЕНКО Д.А | |||
| и др | |||
| Пероксидазоподобные дезоксирибозимы для детекции бактериальных и вирусных патогенов, СОВРЕМЕННЫЕ ДОСТИЖЕНИЯ ХИМИКО-БИОЛОГИЧЕСКИХ НАУК В ПРОФИЛАКТИЧЕСКОЙ И КЛИНИЧЕСКОЙ МЕДИЦИНЕ / Сборник научных трудов Всероссийской научно-практической конференции с международным участием | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Под редакцией А.В | |||
| Силина, Л.Б | |||
| Гайковой | |||
Авторы
Даты
2023-12-28—Публикация
2023-03-03—Подача