Изобретение относится к области медицины, биотехнологии, микробиологии и молекулярной биологии, в частности к молекулярно-биологическим способам детекции микроорганизмов и предназначенным для их осуществления реагентам.
По оценкам исследования Глобального бремени болезней 2019 года (The Global Burden of Disease Study 2019), заболевания полости рта затрагивают около 3,5 миллиардов человек во всем мире, причем кариес постоянных зубов является наиболее распространенным заболеванием: 2,0 миллиарда человек страдают кариесом постоянных зубов и более 520 миллионов детей страдают кариесом молочных зубов. Вследствие недостаточной гигиены полости рта в сочетании с определенными привычками и стилем питания, задача сохранения здоровья зубов остается актуальной для большинства людей во всем мире.
Возникновение кариеса связано с локальным изменением рН на поверхности зуба под зубным налетом вследствие гликолиза углеводов, осуществляемого микроорганизмами. Накопление углеводов в ротовой полости является важным фактором, способствующим формированию биопленок (зубного налета) [Cagetti M.G., Mastroberardino S., Milia E., et al. The use of probiotic strains in caries prevention: a systematic review. Nutrients. 2013;5:2530-2550]. В полости рта обнаруживается множество бактерий, но в процессе формирования налета и последующей деминерализации эмали участвуют в основном кислотообразующие стрептококки.
Streptococcus mutans играет главную роль в разрушении зубов. Он является наиболее кислотообразующим представителем среди стрептококков полости рта и может существовать при низких значениях рН. Одним из важнейших биологических свойств S. mutans является его способность синтезировать экзополисахариды из сахарозы под действием множественных глюкозилтрансфераз и фруктозилтрансферазы. Эти ферменты катализируют синтез внеклеточных полимеров - глюкана и левана, которые действуют как сайты связывания для S. mutans, тем самым способствуя образованию биопленок и прикреплению стрептококков к гладкой поверхности зубов. Образующиеся липкие матриксы препятствуют диффузии большого количества молочной кислоты, образуемой стрептококками, что продлевает ее пребывание на поверхности зуба и ведет к деминерализации эмали, вызывая кариес. Кроме того, внеклеточные полисахариды, заполняя весь объем очага поражения, затрудняют процесс реминерализации, препятствуя поступлению в эмаль ионов кальция и фосфатов [Yoshida A., Kuramitsu Н.K. Multiple Streptococcus mutans genes are involved in biofilm formation. Appl Environ Microbiol. 2002;68:6283-6291].
Сообщается также о возникновении случаев геморрагических инсультов и подострого инфекционного эндокардита (опасного для жизни воспаления сердечных клапанов) в результате инфицирования кровотока S. mutans после стоматологических процедур [Bosshard P.P., Kronenberg А., Zbinden R., et al. Etiologic diagnosis of infective endocarditis by broad-range polymerase chain reaction: a 3-year experience. Clin Infect Dis. 2003;37:167-72; Mansour T.R., Alam Y., Dahbour L., et al. Streptococcus Mutans: A Potential Risk Factor in Recurrent Hemorrhagic Stroke. Cureus. 2017; 9(5): e1264]. При этом некоторые исследования также сообщают, что подгруппа штаммов Streptococcus mutans связана и с другими экстраоральными патологиями, такими как IgA-нефропатия и атеросклероз [Lemos J.A., Palmer S.R., Zeng L., et al. The Biology of Streptococcus mutans. Microbiology Spectrum. 2019; 7 (1): 26].
Своевременная диагностика кариеса, в частности выявление кариесогенных бактерий Streptococcus mutans, при проведении соответствующих профилактических и лечебных мероприятий, позволит избежать прогрессирования кариозного процесса. Следует отметить, что в связи с высокой заболеваемостью кариесом зубов, в частности, у детей различных возрастных групп, прогнозирование риска развития кариозного процесса является необходимой диагностической процедурой.
Согласно данным литературы, в настоящее время детекция Streptococcus mutans в биологических образцах проводится с помощью ряда микробиологических, иммунохимических и молекулярно-биологических методов анализа [Li Y., Saraithong P., Chen Z., et al. Comparison of Real-Time Quantitative PCR with a Chairside Test for Streptococcus Mutans Assessment. The Chinese journal of dental research. 2017; 20 (4): 199-210; Lima A.R., Herrera D.R., Francisco P.A., et al. Detection of Streptococcus mutans in symptomatic and asymptomatic infected root canals. Clinical Oral Investigations. 2021; 25 (6): 3535-3542].
Классическим подходом к выявлению бактерий является бактериологический посев. Для определения стрептококков вида Streptococcus mutans в наборах Dentocult SM Strip Mutans (Aidian Oy, Финляндия) используется бактериологический метод, который основан на выращивании бактерий на тестовых полосках. После сбора биологического образца (зубного налета или стимулированной слюны пациента) тестовая полоска с образцом помещается в пробирку с селективным бульоном и культивируется от 48 до 96 часов при температуре 35-37°С. Результат тестирования определяется визуально по интенсивности и плотности окраски тестовой полоски в синий цвет. Оценочная плотность S. mutans в образце определяется путем сопоставления плотности колонии на полоске с типовой схемой. При этом клетки эпителия из ротовой полости, попавшие в образец, также окрасятся в синий цвет в процессе инкубации, а некоторые загрязняющие бактерии могут окрасить культуральный бульон в другие цвета или сделать его мутным, что замедлит рост искомых S. mutans. Кроме того, на результаты тестирования могут повлиять такие факторы, как еда, курение, чистка зубов и прием антибиотиков. Таким образом, бактериологический метод анализа прост в интерпретации результатов, однако отличается низкой специфичностью и чувствительностью, и требует длительного времени для проведения тестирования подготовленным медицинским персоналом.
Наборы Saliva-Check Mutans (GC Corporation, Япония) основаны на применении иммунохроматографического метода для качественного анализа концентрации S. mutans в слюне. Биологический материал, смешанный с реагентами, помещается в специальное окно устройства. При высокой концентрации S. mutans в слюне, бактерии вступают в реакцию с маркированными золотом коллоидными антителами против S. mutans, что приводит к появлению красной линии в окне индикации. Оставшиеся антитела вступают в реакцию с иммуноглобулином в окне контроля и формируют контрольную красную линию. Если красная линия не появилась, результат отрицательный и концентрация S. mutans низкая. За сравнительно короткое время (15 минут) данный метод позволяет получить приблизительную оценку риска возникновения кариозных поражений. Однако визуальная оценка не позволяет получить точный результат тестирования, а стоимость одного анализа достаточно высока. Кроме того, в настоящее время набор снят с производства.
Из патента CN 106199001 В известен хемилюминесцентный набор реагентов для обнаружения Streptococcus mutans, включающий магнитные частицы с иммобилизованным на них иммуноглобулином G2a крысы, ванкомицин в качестве ферментной метки, люминесцентный субстрат, контрольные образцы суспензии бактерий Streptococcus mutans, буферный раствор и промывочный раствор. Выявление бактерий Streptococcus mutans с помощью этого набора требует специального оборудования и этапов длительного инкубирования, может проводиться только в лабораторных условиях, примерное время одного анализа составляет не менее 4 часов.
Обнаружение бактерий S. mutans также может осуществляться методом полимеразной цепной реакции, например, с использованием технологии Techne Prime Pro (Cole-Parmer, Великобритания), включающей тест-систему Techne DNA Streptococcus mutans и амплификатор для ПЦР в режиме реального времени Prime Pro 48.
Из заявки на патент US 20090305252 А1 известны наборы видоспецифичных праймеров для ПЦР для идентификации и количественного определения Streptococcus mutans и S. sanguinis в клинических образцах, а также олигонуклеотидные зонды и праймеры для использования в анализах, наборах и способах и сами анализы и способы для определения и оценки количества бактерий в полости рта человека и риска развития кариеса. В частности, из этого документа известны наборы праймеров для амплификации целевой последовательности S. mutans Sm479F/Sm479R или ее части с использованием ПЦР или другой технологии амплификации. Раскрыты следующие наборы праймеров: первый набор из двух праймеров TCGCGAAAAAGATAAACAAACA, GCCCCTTCACAGTTGGTTAG и второй набор из двух праймеров AAATAGATTAAGACGTAGAAAAGTTAATGGGTTCT, GTTGTGATGTTATGGAGGACGAGAT.
Метод ПЦР отличается высокой чувствительностью и позволяет проводить полуколичественный анализ проб в режиме реального времени. Путем многократного удвоения целевых участков ДНК в образце в присутствии флуоресцентных красителей происходит обнаружение бактерий. Время одного анализа составляет порядка 2 часов без учета времени, необходимого на подготовку пробы. Факторами, препятствующими использованию данного метода в стоматологической практике, являются высокая стоимость прибора и анализа, трудоемкость пробоподготовки и длительность анализа.
Изотермическая петлевая амплификация (loop-mediated isothermal amplification, LAMP) представляет собой быстрый, высокочувствительный и высокоспецифичный метод диагностики. Метод LAMP был разработан в 1998 г компанией Eiken Chemical Company в Японии, представляет собой альтернативу ПЦР и относится к группе методов, используемых в месте оказания медицинской помощи (point-of-care). Основными компонентами, использующимися для проведения LAMP, являются от четырех до шести специально подобранных праймеров для распознавания специфичных последовательностей гена, и ДНК-полимераза Bst, обладающая цепь-вытесняющей активностью. Особенностью LAMP является изотермический режим проведения реакции амплификации. Наиболее распространенными красителями, использующимися для LAMP, являются SYBR Green/Eva Green, гидроксинафтол голубой и кальцеин, рН-зависимые красители.
Из работы Нагашима с соавторами [Nagashima S., Yoshida A., Ansai Т., et al. Rapid detection of the cariogenic pathogens Streptococcus mutans and Streptococcus sobrinus using loop-mediated isothermal amplification. Oral Microbiol Immunol. 2007 Dec; 22(6): 361-8] известен набор праймеров для выявления Streptococcus mutans методом LAMP (показаны в направлении от 5' к 3'-концу):
F3: CGGGGCAAATTCCTGGTT
В3: AGCAATGTGCATGGAGCC
FIP: GCCCACGGTGGGTACTGGTA-CAGGTGAAGAAGCCGTAGC (F1c-F2)
BIP: GGTCATTGTGTGGCCAAGGGT-CTTGCCAAGACCTGTCTCC (В1-В2с)
LF: CCGTTAAATTGGCACAGACTCC
LB: GGTGACCTTAAGGGCATGATGG
Реакционную смесь, содержащую праймеры, реакционный буфер, Bst ДНК-полимеразу и ДНК-мишени, инкубировали в изотермических условиях при плюс 63°С с использованием обычного нагревательного блока и нагревали при температуре выше плюс 95°С в течение 2 мин для остановки реакции, результат получали менее чем за 1 ч. Продукты LAMP детектировали визуально при добавлении SYBR Green I. Предел обнаружения набора праймеров для S. mutans (с петлевыми праймерами) составлял одну клетку за 30-минутную реакцию в отличие от пределов обнаружения для систем идентификации на основе ПЦР, составляющих 25-100 клеток.
Из работы Китагавы с соавторами [Kitagawa М., Nagamine K., Oka Н., et al. Rapid detection of the Streptococcus mutans cnm gene by loop-mediated isothermal amplification. Anal Biochem. 2020; 605:113812] известен набор праймеров для выявления Streptococcus mutans методом LAMP (показаны в направлении от 5' к 3'-концу):
FIP: AACCATTAAGCTGGAGGTTCAGGAACTGCTTTGTCTTGCGT
BIP: CGTATAACCTGTTCCTCTGACTGTAATATTAAAGCAGGCGACAC
F3: CCAGTAATACTGTCATTGAAAGT
В3: CGCTTTGAGTTTGATGAGC
LF: GCAAGTATGTTGGTGATTTG
LB: CCTGAATTCTGCCAGTTAAC
Реакцию LAMP проводили с использованием реагента D для амплификации ДНК (Eiken Chemical, Япония), специфических праймеров, реагента для детекции флуоресценции (Eiken Chemical), бетаина и BHI бульона, содержащего колонии, выращенные из образцов слюны. Смесь инкубировали при плюс 64°С в течение 2 ч. Разработанный метод позволял обнаруживать cnm-положительные штаммы после короткого периода культивирования в присутствии порядка 1000 бактериальных клеток.
Той же исследовательской группе принадлежит патентный документ JP 2020068678 A, в котором приведены данные по разработке способа обнаружения cnm-положительных и РА-положительных штаммов S. mutans в образцах слюны и зубного налета. При этом метод идентификации включает в себя следующие этапы: биологический образец наносят на питательную среду для селективного культивирования штаммов S. mutans в течение 6-40 ч, затем выращенные бактериальные клетки переносят в жидкую среду, и далее проводят амплификацию целевой нуклеотидной последовательности S. mutans с помощью LAMP без предварительной экстракции нуклеиновых кислот с последующей визуализацией полученных результатов. Реакция протекает при плюс 64°С в течение 2 часов, результаты реакции визуализируют под ультрафиолетовой лампой. Этот способ длительный, в частности, требует длительной пробоподготовки.
В качестве праймеров использовали «SM-праймеры» для амплификации гена трансглюкозидазы и «cnm-праймеры» для амплификации гена белка cnm. Набор праймеров для амплификации гена трансглюкозидазы (называемый «SM-праймеры») включал (показаны в направлении от 5' к 3'-концу):
F3: GAAGTTTGTGCTTCTTCTGA
В3: GATAAGGCGGCATCTGAA
FIP: GCATAACTAAGGAAACTCCTTCACACGTATTTGTTACTGTTTTGTCAT
BIP: CTTGTATCAGCAACGTTTGCCTGCCAAACAGATGCACCTAA
LF: TATTACACACAAGCCAACT
LB: TGGGCCTGCGTTTGTTCTGT
Набор праймеров для амплификации гена белка cnm (называемый «cnm-праймеры») включал праймеры, указанные выше [Kitagawa М., Nagamine K., Oka Н., et al. Rapid detection of the Streptococcus mutans cnm gene by loop-mediated isothermal amplification. Anal Biochem. 2020; 605:113812].
В другом патентном документе JP 2005013055 A приведено описание способа идентификации кариесогенных бактерий S. mutans и S. sobrinus с использованием метода LAMP и набора специфических праймеров для детекции. Для обнаружения S. mutans проводят амплификацию части гена глюкозилтрансферазы gtfB. Набор праймеров включает (показаны в направлении от 5' к 3'-концу):
F3: AGCAGCCCATAAGAACCAAG
В3: AACATCTTGATCAGCCGCAGC
FIP: CAGCCCCTTCTTGATCGGAATGATCCGACCTTTACCTTTGAACCAC
BIP: CAGAGGGGAATTGATCTCCAGCCGCCCAGACACCTAAATAGCCCAG
LB: AGGCTATGCCAACCCTCAA
Реакционная смесь для проведения LAMP включает: образец ДНК, праймеры, dNTP, ДНК-полимеразу, реакционный буфер. Реакция проводится предпочтительно при плюс 65°С, от 10 до 40 мин. Амплифицированную ДНК можно обнаружить путем измерения степени мутности раствора, с помощью электрофореза или в присутствии в реакционной смеси флуоресценцентного интеркалирующего красителя с последующим измерением интенсивности флуоресценции. При амплификации ДНК S. mutans с использованием разработанного метода чувствительность обнаружения была примерно в 10 раз выше, чем в случае амплификации с помощью ПЦР.
Технической задачей, на решение которой направлено настоящее изобретение, является решение как минимум одной из вышеуказанных в уровне техники проблем.
Сущность изобретения
Техническим решением является использование описанного признаками в пунктах формулы изобретения.
Одной из задач, на решение которой направлено настоящее изобретение, являлось создание набора праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, позволяющего проводить детекцию упомянутого микроорганизма без пробоподготовки с высокой чувствительностью (детекция не менее 10-4 нг/мкл в течение получаса) и специфичностью (не менее 95%).
Задача решается тем, что разработаны и синтезированы наборы праймеров, позволяющие быстро, специфично и с высокой чувствительностью выявлять Streptococcus mutans методом изотермической петлевой амплификации. Одним из вариантов осуществления настоящего изобретения является набор, включающий праймеры с нуклеотидной последовательностью Seq Id No: 13-16, 54. Другим вариантом осуществления настоящего изобретения является набор, включающий праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55. Еще одним вариантом осуществления настоящего изобретения является набор, включающий праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58.
Одним из вариантов осуществления настоящего изобретения является набор, включающий праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55 и, дополнительно к этому, праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58.
Одним из вариантов осуществления изобретения является упомянутый набор праймеров, содержащий внутренние праймеры (FIP, BIP, Seq Id No: 13, 14 или 21, 22 или 23, 24), наружные праймеры (F3, В3, Seq Id No: 15, 16 или 19, 20 или 25, 26) и прямой петлевой праймер (LF, Seq Id No: 54 или 55 или 58) в молярном соотношении 8:8:1:1:4.
Изобретение иллюстрируется следующими графическими материалами:
На Фиг. 1 представлена структура основных праймеров для реакции изотермической петлевой амплификации.
На Фиг. 2 представлены результаты первичного тестирования наборов праймеров методом ПЦР в режиме реального времени. В скобках указано количество циклов амплификации Ct, которое понадобилось для детекции ДНК Streptococcus mutans с использованием определенного набора праймеров, в двух повторностях. А - График амплификации гена 16S рРНК. Кривые 3-4 - набор праймеров №1 (14,83; 14,85), кривые 5-6 - набор праймеров №2 (14,45; 14,64), кривые 1-2 - набор праймеров №3 (14,94; 14,84). Б - График амплификации гена UA159. Кривые 1-2 - набор праймеров из данных литературы (15,96; 15,85), кривые 4, 7 - набор праймеров №1 (17,40; 17,08), кривые 3, 8 - набор праймеров №2 (17,52; 17.41), кривые 5-6 - набор праймеров №3 (17,23; 17,76). В - График амплификации гена dexA. Кривые 1-2 - набор праймеров из данных литературы (16,17; 16,02), кривые 4-5 - набор праймеров №1 (18,31; 17,96), кривые 3, 6 - набор праймеров №2 (17,28; 16,66). Г - График амплификации гена spaP. Кривые 1-2 - набор праймеров из данных литературы (17,07; 17,05), кривые 4, 8 - набор праймеров №1 (19,20; 17,74), кривые 3, 5 - набор праймеров №2 (18,21; 18,39), кривые 6-7 - набор праймеров №3 (18,34; 18.42). Д - График амплификации гена cnm. Кривые 4, 6 - набор праймеров из данных литературы (39,25; 41,32), кривые 3, 5 - набор праймеров №1 (38,33; 40,65), кривые 1-2 - набор праймеров №2 (33,81; 33,98).
На Фиг. 3 представлены результаты тестирования гДНК Streptococcus mutans с использованием отечественного и зарубежного фермента ДНК-полимеразы Bst. Слева графики амплификации, справа - кривые плавления продукта амплификации. А - реакция, проведенная с использованием отечественного фермента UltraFast Bst ДНК-полимераза (ООО «Иннова плюс», Россия). Б - реакция, проведенная с использованием фермента Bst ДНК-полимераза Large fragment (New England Biolabs, Великобритания).
На Фиг. 4 представлены результаты тестирования праймеров на гДНК контрольных образцов модельных штаммов АТСС 25175 (А) и АТСС 35668 (Б) бактерии Streptococcus mutans.
На Фиг. 5 представлены результаты реакции изотермической петлевой амплификации в присутствии наборов праймеров к генам UA 159 (3.1) (амплификационные кривые 1-2), dexA (амплификационные кривые 5-6) и смеси двух наборов праймеров (амплификационные кривые 3-4) на гДНК штаммов Streptococcus mutans.
На Фиг. 6 представлены результаты тестирования набора реагентов до закладки на хранение (А) и спустя 2 недели (Б) хранения при 37°С. ОК - отрицательный контроль, «+» - наблюдается изменение окраски, «-» - отсутствует изменение окраски.
Детальное описание изобретения
С целью достижения высокого уровня чувствительности и специфичности набора реагентов, на основе биоинформатического анализа проводился поиск специфических праймеров. Праймеры для LAMP отличаются от классических праймеров для полимеразной цепной реакции. Они имеют оригинальную структуру: четыре праймера должны быть специфичны к шести участкам целевого фрагмента: F3 (прямой наружный праймер) комплементарен участку F3c, В3 (обратный наружный праймер) комплементарен участку В3с, FIP (прямой внутренний праймер) состоит из двух частей: Flc (5') и F2 (3'), комплементарных участкам F1 и F2c, BIP (обратный внутренний праймер) состоит из двух частей: В1 с (5') и В2 (3'), комплементарных участкам В1 и В2 с [Tomita N., et al. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nat Protoc. 2008; 3: 877-882]. Структура основных праймеров представлена на Фиг. 1.
В результате были подобраны четыре набора праймеров, позволяющие проводить детекцию Streptococcus mutans методом изотермической петлевой амплификации быстро, специфично и с высокой чувствительностью.
В одном из вариантов осуществления настоящего изобретения предлагается набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, включающий праймеры с нуклеотидной последовательностью Seq Id No: 13-16, 54. Использование этого набора приводит к выявлению геномной ДНК Streptococcus mutans, ложноположительные результаты отсутствуют, а выявление геномной ДНК соответствует 23-24 циклам или 11,5-12 минутам реакции.
В одном из вариантов осуществления настоящего изобретения предлагается набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, включающий праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55. Использование этого набора приводит к выявлению геномной ДНК Streptococcus mutans, ложноположительные результаты отсутствуют, а выявление геномной ДНК соответствует 19-22 циклам или 9,5-11 минутам реакции.
В одном из вариантов осуществления настоящего изобретения предлагается набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, включающий праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58. Использование этого набора приводит к выявлению геномной ДНК Streptococcus mutans, ложноположительные результаты отсутствуют, а выявление геномной ДНК соответствует 31-40 циклам или 15,5-20 минутам реакции.
В одном из вариантов осуществления настоящего изобретения предлагается набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, включающий праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55, дополнительно содержащий праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58. Использование этого набора приводит к выявлению геномной ДНК Streptococcus mutans, ложноположительные результаты отсутствуют, а выявление геномной ДНК соответствует 13-14 циклам или 6,5-7 минутам реакции.
В одном из вариантов осуществления настоящего изобретения предлагается набор праймеров, содержащий внутренние праймеры (FIP, В IP, Seq Id No: 13, 14, 21, 22), наружные праймеры (F3, В3, Seq Id No: 15, 16, 19, 20) и прямой петлевой праймер (LF, Seq Id No: 54, 55) в молярном соотношении 8:8:1:1:4. Молярное соотношение праймеров выбрано на основании литературных данных [Tomita N., Mori Y., Kanda H., Notomi Т. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nat Protoc. 2008;3:877-882; Huang W.E., Lim В., Hsu C.C., et al. RT-LAMP for rapid diagnosis of coronaviras SARS-CoV-2. Microb Biotechnol. 2020;13(4):950-961].
Набор праймеров по настоящему изобретению может входить в состав набора реагентов для выявления Streptococcus mutans методом изотермической петлевой амплификации. Например, набор реагентов может содержать также гидроксинафтол голубой (HNB), ДНК-полимеразу, 1 хреакционный буфер (20 мМ Tris-HCl, 10 мМ (NH4)2SO4,10 мМ KCl, 2 мМ MgSO4, 0.1% Тритон®-Х-100, рН 8,8), сульфат магния, дезоксинуклеотидтрифосфаты.
В стандартный набор реагентов для анализа методом петлевой изотермической амплификации входят: фермент, обладающий цепь-вытесняющей активностью (например, Bst ДНК-полимераза), дезоксинуклеотидтрифосфаты, специфические праймеры, реакционный буферный раствор, ионы магния, краситель.
Ключевым компонентом в реакционной смеси является фермент Bst ДНК-полимераза. В состав заявленного набора реагентов для выявления Streptococcus mutans методом LAMP входит фермент UltraFast Bst ДНК-полимераза производства ООО «Иннова плюс» (Россия). Проводилось сравнение UltraFast Bst ДНК-полимеразы с зарубежным аналогом - Bst ДНК-полимеразой Large fragment (New England Biolabs, Великобритания). В результате проведенного сравнительного эксперимента было показано, что активность отечественного фермента соответствует активности зарубежного аналога, который наиболее часто используется для проведения исследований методом изотермической петлевой амплификации. Данные, полученные в результате анализа методом LAMP с использованием фермента UltraFast Bst ДНК-полимеразы, характеризуются высокими показателями чувствительности (10-4 нг гДНК/реакцию) и специфичности (отсутствие ложноположительных результатов), кривые амплификации детектируются на 2-3 цикла раньше по сравнению с аналогом.
Набор праймеров по настоящему изобретению может применяться в способе выявления Streptococcus mutans методом изотермической петлевой амплификации. Например, способ может заключаться в том, что смешивают образец слюны, 0,12 мМ HNB, 8 или 12 ед. акт. UltraFast Bst ДНК-полимеразы, 1×реакционный буфер, 7 мМ сульфата магния, 1,4 мМ дезоксинуклеотидтрифосфатов, набор праймеров по любому из пп. 1-4, выдерживают при температуре 60-65°С в течение не менее 15 минут, при этом количество праймеров составляет 1,6 мМ для внутренних праймеров (FIP, BIP, Seq Id No: 13, 14 или 21, 22), 0,2 мМ для наружных праймеров (F3, В3, Seq Id No: 15, 16 или 19, 20) и 0,8 мМ для прямого петлевого праймера (LF, Seq Id No: 54 или 55), детектируют наличие в исследуемом образце ДНК Streptococcus mutans визуально по изменению окраски реакционной смеси с фиолетовой на голубую.
Аналитическая чувствительность набора реагентов, содержащего наборы праймеров по настоящему изобретению, при детекции методом LAMP в режиме реального времени составляет 4⋅10-6 нг/мкл, специфичность 100% (отсутствие ложноположительных результатов).
Способ с использованием наборов праймеров по настоящему изобретению позволяет выявлять ДНК Streptococcus mutans со специфичностью 100% и чувствительностью как минимум 4⋅10-4 нг/мкл без пробоподготовки (при добавлении образца слюны напрямую в пробирку с реакционной смесью). Время проведения анализа от 11 минут.
Сущность и практическая применимость настоящего изобретения поясняется следующими примерами.
Пример 1. Подбор праймеров для LAMP
Подбор праймеров для LAMP проводили с помощью специализированной программы Lamprim (ООО «Иннова плюс», Россия) [свидетельство о государственной регистрации программы для ЭВМ №2021668028]. Биоинформатический анализ состоял из следующих этапов: поиск, фильтрация, множественное выравнивание и кластеризация последовательностей выбранных генов бактерии Streptococcus mutans, находящихся в нуклеотидной базе данных NCBI, поиск консервативных участков, дизайн праймеров к консервативным последовательностям; проверка специфичности праймеров к целевым последовательностям ДНК бактерии Streptococcus mutans; проверка на отсутствие специфичности праймеров к нецелевым последовательностям.
На основе данных литературы для работы были выбраны пять генов: 16S рРНК [Momeni S.S., Ghazal Т., Grenett Н., et al. Streptococcus mutans serotypes and Collagen-Binding Proteins Cnm/Cbm in Children with Caries analyzed by PCR. Molecular Oral Microbiology. 2019;34(2):64-73; Wang Z.Y., Wang J.Q., Zhou Y., et al. Quantitative detection of Streptococcus mutans and bacteria of dental caries and no caries groups in permanent teeth from a north China population. Chin Med J (Engl). 2012;125(21):3880-4], UA159 [Chen Z., Saxena D., Caufield P.W., et al. Development of species-specific primers for detection of Streptococcus mutansin mixed bacterial samples. FEMS Microbiology Letters. 2007;272(2): 154-162], dexA [Igarashi Т., Yamamoto A., Goto N. Direct detection of Streptococcus mutans in human dental plaque by polymerase chain reaction. Oral Microbiology and Immunology. 1996;11(5):294-298], spaP [Ono Т., Hirota K., Nemoto K., et al. Detection of Streptococcus mutans by PCR amplification of spaP gene. Journal of Medical Microbiology. 1994;41(4):231-235], cnm [Kitagawa M., Nagamine K., Oka H., et al. Rapid detection of the Streptococcus mutans cnm gene by loop-mediated isothermal amplification. Anal Biochem. 2020; 605:113812].
В базе данных NCBI были отобраны все возможные последовательности, соответствующие целевым генам в ДНК Streptococcus mutans.
Программа Lamprim провела перебор всех возможных вариантов праймеров для выбранных последовательностей. С помощью программного обеспечения был выполнен расчет температуры плавления праймеров (Tm) наиболее современными термодинамическими методами с учетом концентрации компонентов, используемых в реакции; проведен термодинамический расчет наиболее стабильных вторичных структур (димеров и шпилек); исключены из анализа варианты наборов, не удовлетворяющие заданным критериям стабильности (изменение энергии Гиббса AG должно быть больше заданного порогового значения, Tm должна быть меньше заданного порогового значения). Наличие стабильных вторичных структур может приводить к замедлению реакции, амплификации неспецифичного продукта, и как следствие, к ложноположительным результатам. Качественный дизайн праймеров LAMP требует избегания как можно большего количества стабильных димеров и шпилек, образуемых в наборе.
В процессе дизайна праймеров проводили расчет основных характеристик праймеров, таких как температура плавления Tm, GC состав, значения изменений свободной энергии AG на 5' и 3' концах праймеров. Для более точного расчета температуры плавления Tm праймеров проводили ее корректировку с учетом концентрации компонентов реакции LAMP: средняя концентрация праймеров - 0,5 мкМ, K - 12 мМ, Tris - 20 мМ, Mg - 8 мМ, концентрация нуклеотидов дНТФ - 5,6 мМ.
Для учета вариабельности и консервативности позиций нуклеотидов в исследуемой последовательности была составлена дополнительная последовательность специальных символов, указывающая неконсервативные (встречаемость менее 90%) и консервативные нуклеотиды. Эти специальные последовательности также учитывались в процессе дизайна праймеров. С помощью автоматического подбора праймеров было получено более 8000 наборов для консенсусных последовательностей кластеров генов 16S рРНК, UA159, dexA, spaP, cnm бактерии Streptococcus mutans. Также анализировали способность последовательностей праймеров образовывать димеры и формировать шпилевидные трехмерные структуры. Для дальнейшей работы были отобраны наилучшие наборы праймеров к каждому таргетному гену.
Скорость реакции изотермической петлевой амплификации в основном зависит от внутренних праймеров (FIP, BIP). Изменение их структуры и концентрации позволяет уменьшить время детекции, что особенно важно при низкой концентрации ДНК [Hardinge P., Murray J.A.H. Full Dynamic Range Quantification using Loop-mediated Amplification (LAMP) by Combining Analysis of Amplification Timing and Variance between Replicates at Low Copy Number. Sci Rep.2020; 10(1): 916]. Включение линкера «tttt» между областями F1c и F2, а также областями B1c и В2 FIP и BIP повышает чувствительность реакции LAMP [Rabe В.А., Серко С. SARS-CoV-2 detection using isothermal amplification and a rapid, inexpensive protocol for sample inactivation and purification. Proc Natl Acad Sci USA. 2020;117: 24450-24458; Torres C., Vitalis E.A, Baker B.R., et al. LAVA: an open-source approach to designing LAMP (loop-mediated isothermal amplification) DNA signatures. BMC Bioinformatics. 2011; 12: 240].
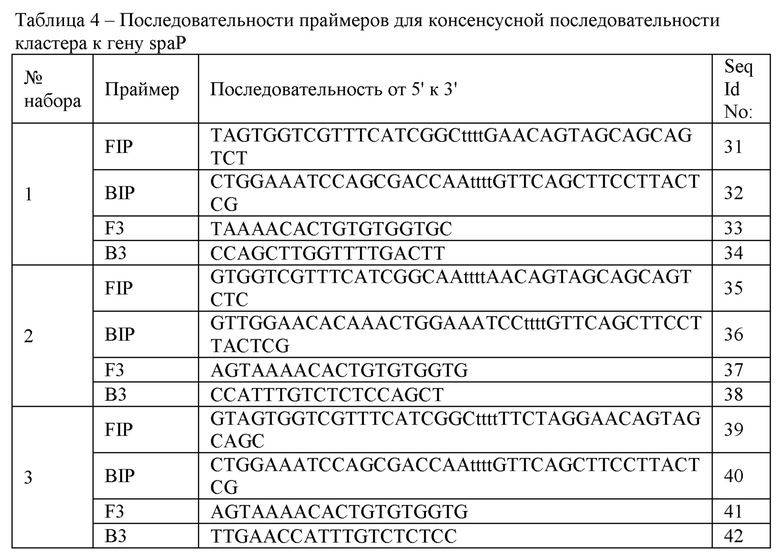
Последовательности праймеров представлены в таблицах: Таблица 1 - последовательности праймеров для консенсусной последовательности кластера к гену 16S рРНК, Таблица 2 - к гену UA159, Таблица 3 - к гену dexA, Таблица 4 - к гену spaP, Таблица 5 - к гену cnm.




В качестве праймеров сравнения были выбраны наборы к каждому гену на основе литературных данных (Таблица 6).

Проверку неспецифического взаимодействия праймеров с другими мишенями проводили в два этапа. На первом этапе в базе NCBI выполняли поиск последовательностей, которые являются комплементарными для F3/B3 праймеров из наборов, с помощью ресурса Primer-BLAST с активированной функцией проверки специфичности. На втором этапе весь набор праймеров, включая внутренние праймеры FIP и BIP, дополнительно проверяли на специфичность к обнаруженным на первом этапе последовательностям, комплементарным праймерам F3/B3, с помощью упомянутого ранее программного обеспечения Lamprim.
В результате проведенного анализа для подобранных наборов праймеров не было обнаружено специфичных участков в последовательностях нуклеиновых кислот других организмов.
Таким образом, были подобраны специфические наборы праймеров для детекции генов 16S рРНК, UA159, dexA, spaP, cnm бактерии Streptococcus mutans, которые не взаимодействуют с генами-мишенями других организмов.
После проведения биоинформатического поиска, специфичные наборы праймеров синтезировали и проводили первичное тестирование.
Пример 2. Тестирование наборов праймеров
Пример 2.1. Тестирование наборов праймеров при проведении ПЦР в реальном времени. Наборы праймеров, подобранные на основе литературных данных и с помощью программы Lamprim, тестировали при проведении полимеразной цепной реакции в режиме реального времени (рвПЦР). Условия проведения реакции ПЦР в режиме реального времени: начальная денатурация при температуре 95°С в течение 3 минут,
45 циклов:
денатурация при температуре 95°С в течение 15 секунд и
отжиг праймеров при температуре 58°С в течение 30 секунд,
элонгация при температуре 72°С в течение 30 секунд.
Результаты первичного тестирования наборов праймеров представлены в графическом виде на Фиг. 2, где разными цветами отмечены кривые амплификации наборов праймеров, подобранных биоинформатическим путем с применением программы Lamprim и из литературных источников.
Таким образом, отобранные для проведения исследования наборы праймеров специфично определяют участки генов 16S рРНК, UA159, dexA, spaP. Пороговое число циклов (Ct) в среднем соответствует 14-19 циклу амплификации. Детекция ДНК Streptococcus mutans с использованием праймеров к гену cnm была на низком уровне (Ct соответствует 33-41 циклам амплификации), что привело к исключению наборов праймеров к гену cnm из дальнейшего исследования.
В результате первичного тестирования праймеров к генам 16S рРНК, UA159, dexA, spaP, cnm бактерии Streptococcus mutans методом рвПЦР упомянутых генов с помощью соответствующих им наружных праймеров F3 и В3 было подтверждено специфичное взаимодействие подобранных наборов к таргетной ДНК.
Пример 2.2. Тестирование наборов праймеров при проведении изотермической петлевой амплификации. Следующим этапом тестирования праймеров был анализ работы наборов праймеров при проведении изотермической петлевой амплификации. Реакцию амплификации проводили при разной температуре от 60°С до 65°С. Количество геномной ДНК (гДНК) в реакционной смеси составляло 0,5 нг на реакцию (Таблица 7). В качестве отрицательного контроля для определения неспецифического сигнала реакции использовали деионизованную воду.
Наборы праймеров «отбраковывали» по следующим параметрам: отсутствие амплификации, ложноположительные результаты (ЛПР) во всех отрицательных контролях или же разница между выходом кривых амплификации в положительном и отрицательном контролях менее 30 циклов, что соответствует 12,5 минутам.


В результате скрининга методом изотермической петлевой реакции были отобраны наборы праймеров к гену UA159 №1 и №3 и к гену dexA №1 и №2.
При скрининге наборов праймеров к гену Cnm целевой продукт не детектировался ни при использовании подобранных нами праймеров, ни при использовании праймеров сравнения, взятых из литературного источника [Kitagawa М, Nagamine K, Oka Н, et al. Rapid detection of the Streptococcus mutans cnm gene by loop-mediated isothermal amplification. Anal Biochem. 2020; 605:113812].
Пример 2.3. Подбор дополнительных петлевых праймеров. Скорость амплификации и специфичность наборов праймеров могут быть улучшены добавлением в реакционную смесь дополнительных петлевых праймеров, сайты посадки которых расположены между участками праймеров F2, F1 и В1с, В2с.
Дизайн петлевых праймеров был выполнен программой Lamprim с использованием полученных ранее консенсусных последовательностей. В Таблице Таблица 8 представлены наилучшие последовательности петлевых праймеров к генам UA159, dexA по итогам биоинформатического подбора in silico с помощью программы Lamprim.

Постановку реакции изотермической петлевой амплификации с дополнительными петлевыми праймерами осуществляли при использовании реагентов, приведенных в Таблице Таблица 9, согласно следующему протоколу: 120 циклов длительностью 30 секунд при температуре плюс 63°С. В качестве матрицы использовали геномную ДНК бактерий Streptococcus mutans в количестве 0,1 нг на реакцию.

Результаты реакции изотермической амплификации представлены в Таблице Таблица 10.
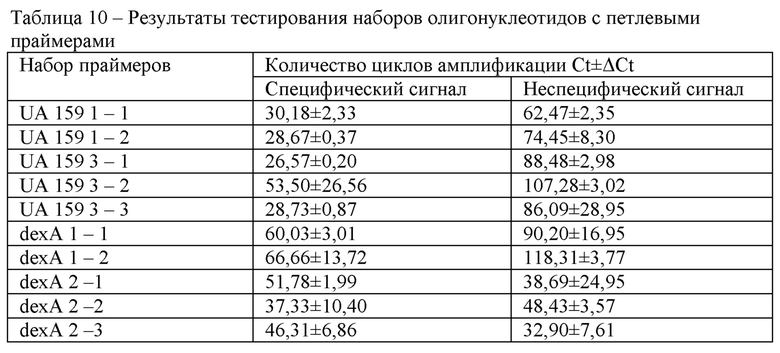
В результате проведения LAMP с помощью наборов праймеров к генам UA159 и dexA и с добавлением петлевых олигонуклеотидов, ранняя детекция (то есть наименьшее количество циклов амплификации) наблюдалась при использовании наборов UA 159 1 - 1, UA 159 1 - 2, UA 159 3 - 1, UA 159 3 - 3. Из-за повышенного уровня неспецифического сигнала набор UA 159 1 - 1 был исключен из дальнейшего анализа. Уровень детекции продукта на праймерах к гену dexA ниже по сравнению с геном UA159, неспецифический сигнал определяется на поздних циклах амплификации.
В результате сравнения данных тестирования наборов праймеров методом LAMP с добавлением (Таблица 10) и без добавления петлевых олигонуклеотидов (Таблица 7) был сделан вывод, что детекция целевого продукта при добавлении петлевых праймеров происходит быстрее и позволяет сократить время проведения анализа.
В результате экспериментального анализа подобранных биоинформатическим методом наборов праймеров наилучшие характеристики (Ct, уровень неспецифического сигнала), обеспечивающие специфичность реакции LAMP и высокую скорость детекции целевого продукта, были определены у наборов праймеров к гену UA 159 №1 с петлевым праймером LF - 2, №3 с петлевым праймером LF - 1. Кроме того, для дальнейших исследований наборов праймеров при их совместном использовании был выбран набор к гену dex №1 с петлевым праймером LF - 1.1. Финальные последовательности отобранных специфических праймеров приведены в Таблице Таблица 11.

Пример 3. Первичное тестирование наборов праймеров для выявления ДНК контрольного образца модельного штамма бактерии Streptococcus mutans
Ключевым компонентом в реакционной смеси является фермент Bst ДНК-полимераза. В состав заявленного набора реагентов для выявления Streptococcus mutans методом LAMP входит фермент UltraFast Bst ДНК-полимераза производства ООО «Иннова плюс» (Россия).
Проводилось сравнение UltraFast Bst ДНК-полимеразы с зарубежным аналогом - Bst ДНК-полимеразой Large fragment (New England Biolabs, Великобритания) при тестировании гДНК Streptococcus mutans методом LAMP в следующих условиях: состав реакционной смеси (Таблица 9), режим амплификации - 120 циклов по 30 сек при температуре плюс 63°С, инактивация при плюс 95°С в течение 2 мин. В результате проведенного сравнительного эксперимента было показано, что активность отечественного фермента соответствует активности зарубежного аналога, который наиболее часто используется для проведения исследований методом изотермической петлевой амплификации.
Данные, полученные в результате анализа методом LAMP с использованием фермента UltraFast Bst ДНК-полимеразы, характеризуются высокими показателями чувствительности (10-4 нг гДНК/реакцию) и специфичности (отсутствие ложноположительных результатов), кривые амплификации детектируются на 2-3 цикла раньше по сравнению с аналогом.
Результаты тестирования гДНК Streptococcus mutans с использованием отечественного и зарубежного фермента представлены в графическом виде на Фиг. 3.
Дальнейшую проверку подобранных специфических наборов праймеров в составе набора реагентов для выявления Streptococcus mutans, содержащего фермент UltraFast Bst ДНК-полимераза (ООО «Иннова плюс», Россия), методом LAMP проводили на матрице полногеномной ДНК целевой бактерии Streptococcus mutans.
Для проведения анализа была наработана кариесогенная бактерия Streptococcus mutans двух штаммов АТСС 25175 и АТСС 35668. Микроорганизмы с микробиологических петель высевали сначала на агаризованную, а затем в жидкую среду BHI (мозговая вытяжка из теленка - 7,5 г/л, вытяжка из мясного экстракта - 10 г/л, глюкоза - 2 г/л, пептон - 10 г/л, гидрофосфат натрия - 2,5 г/л, хлорид натрия - 5 г/л, агар микробиологический - 15 г/л, рН среды 7,4) и культивировали в течение 18 часов в орбитальном шейкере роторного типа при скорости 170 об/мин, далее 24 часа в аэробных условиях в термостатированном шейкере роторного типа при скорости 170 об/мин. После чего производили выделение гДНК с помощью коммерчески доступного набора реагентов Quick-DNA Fungal/Bacterial microprep kit (Zymo Research, США) в трех повторностях.
Первичное тестирование подобранных наборов праймеров (к гену UA 159 - №1 с петлевым праймером LF-2 или №3 с петлевым праймером LF-1, либо к гену dexA - с петлевым праймером LF - 1.1) проводили методом изотермической петлевой амплификации с детекцией в реальном времени с построением кривых плавления.
С целью увеличения эффективности реакции LAMP были подобраны оптимальные условия реакции, такие как температура проведения амплификации и концентрация ионов магния в реакционной смеси для достижения максимальной активности фермента, поскольку Bst ДНК-полимераза является магний-зависимым ферментом.
Температурный оптимум активности фермента Bst ДНК-полимераза составляет от плюс 60°С до плюс 65°С [Tangomo М., Hibbs J., Bonetti E.-J., et al. Robustness of a loop-mediated isothermal amplification reaction for diagnostic applications. FEMS Immunol. Med. Microbiol. 2011; 62(1):41-8]. Содержание ионов магния в реакционной смеси для проведения анализа может варьировать от 4 мМ до 10 мМ [https://international.neb.com/protocols/2014/11/21/typical-lamp-protocol-m0275]. Таким образом, оценивали эффективность каждого из отобранных наборов праймеров в реакции LAMP по скорости детекции целевого продукта (количеству циклов амплификации) при разных значениях температуры и концентрации солей магния (от 5 мМ до 8 мМ). Концентрации остальных компонентов реакции не менялись.
Реакцию амплификации проводили в двух повторностях для исследуемых образцов и отрицательного контроля, объем реакционной смеси - 25 мкл. В качестве исследуемых образцов использовали выделенную гДНК бактерий Streptococcus mutans штаммов АТСС 25175 и АТСС 35668 в концентрации 0,1 нг/мкл объемом 5 мкл на реакцию. В качестве отрицательного контроля добавляли деионизированную воду объемом 5 мкл на одну реакцию. Амплификацию проводили на приборе StepOne Real-Time PCR System (Applied Biosystems™, США), согласно протоколу: 120 циклов по 30 сек при температуре от 60°С до 65°С, инактивация при 95°С в течение 2 мин, с дальнейшим построением кривых плавления. Состав и концентрации компонентов реакционной смеси приведены в Таблице Таблица 12, концентрацию ионов магния в реакционной смеси варьировали от 5 мМ до 8 мМ.

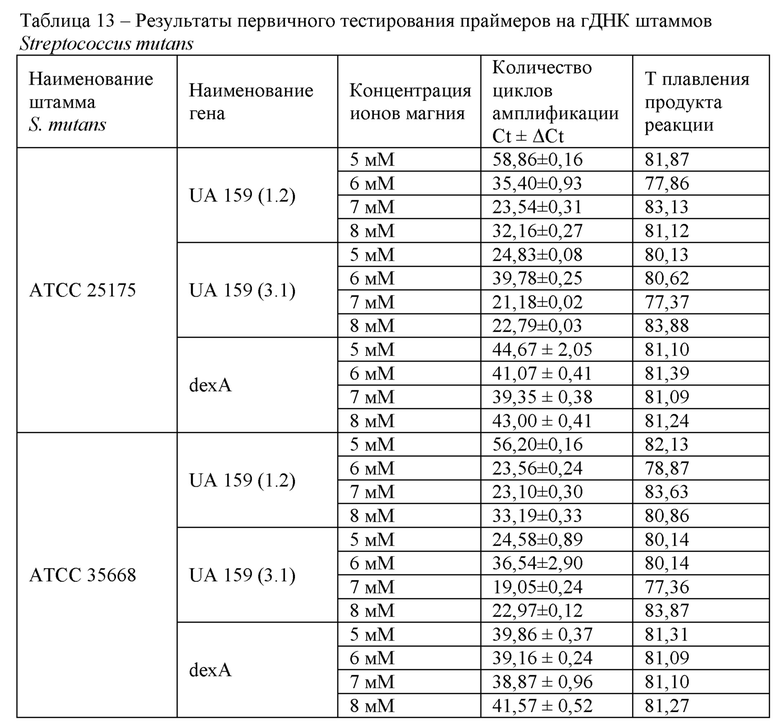
В таблице представлены результаты тестирования двух наборов праймеров к гену UA 159 (1.2) и UA 159 (3.1) и набора праймеров к гену dexA на матрице геномной ДНК Streptococcus mutans штаммов АТСС 25175 и АТСС 35668 (концентрация гДНК 0,1 нг/мкл) с различной концентрацией ионов магния в реакционной смеси (Таблица 13).
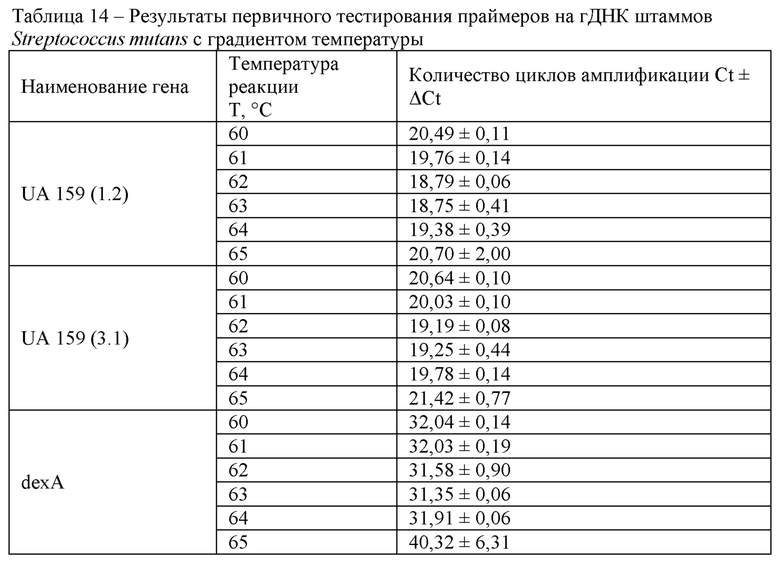
В таблице представлены результаты тестирования двух наборов праймеров к гену UA 159 (1.2) и UA 159 (3.1) и набора праймеров к гена dexA на матрице смеси геномной ДНК Streptococcus mutans штаммов АТСС 25175 и АТСС 35668 в концентрации 0,1 нг/мкл с различной температурой проведения реакции (Таблица 14).
Для большей наглядности результаты первичного тестирования праймеров на геномной ДНК Streptococcus mutans штаммов АТСС 25175 и АТСС 35668 представлены в графическом виде на Фиг. 4.
В результате проведенного тестирования было показано, что подобранные наборы праймеров определяли наличие целевой ДНК, ложноположительные результаты не детектировались. Время детекции образцов геномной ДНК при использовании набора праймеров к гену UA 159 (3.1) соответствовало 19-22 циклам или 9,5-11 минутам, при использовании набора праймеров к гену UA 159 (1.2) - 23-24 циклам или 11,5-12 минутам реакции, при использовании набора праймеров к гену dexA - 31-40 циклам или 15,5-20 минутам реакции. Оптимальные условия проведения реакции LAMP: изотермическая температура плюс 63°С, концентрация ионов магния в реакционной смеси 7 мМ.
С учетом подобранных оптимальных условий проведения реакции изотермической амплификации с наборами праймеров к генам UA 159 и dexA был проведен эксперимент с совместным присутствием обоих наборов праймеров в реакционной смеси (UA 159 (3.1) и dexA) при концентрации гДНК 0,1 нг/мкл, результат представлен в Таблице 15 и на Фиг. 5.

В результате проведенного эксперимента было показано, что наличие в реакционной смеси двух наборов праймеров к геному Streptococcus mutans позволяет детектировать гДНК на ранних циклах.
Пример 4. Аналитическая специфичность набора праймеров
Определение специфичности набора праймеров проводили на гомологичных и гетерологичных образцах бактерий.
В качестве гомологичных бактерий использовали Streptococcus salivarius, Streptococcus oralis, Streptococcus vestibularis, Streptococcus anginosus, Streptococcus sanguinus, выделенные из слюны человека и микробиологически охарактеризованные в Санкт-Петербургском НИИ эпидемиологии и микробиологии имени Пастера.
В качестве гетерологичных бактерий использовали: Escherichia coli; ДНК бактерий Borrelia burgdorferi sensu lato; полногеномная ДНК бактерии Borrelia afzelii размером 905394 п.н., номер в NCBI Reference Sequence СР000395 (Amplirun® Borrelia afzelii DNA control, производства Vircell S.L.); участок ДНК бактерии Borrelia burgdorferi размером 789 п.н., номер в NCBI Reference Sequence KF422803.1 (Amplirun® Borrelia burgdorferi DNA control, производства Vircell S.L.); четыре участка ДНК бактерии Borrelia garinii, размером 5313, 1367, 1351, 987 п.н., номер в NCBI Reference Sequence AY836549.1, D67018, U68005, D82846, соответственно (Amplirun® Borrelia garinii DNA control, производства Vircell S.L.).
Выделение ДНК из культур микроорганизмов проводили коммерческим набором Quick-DNA Fungal/Bacterial microprep kit (Zymo Research, США) в трех повторностях. Концентрацию гДНК определяли на спектрофотометре NanoDrop 2000 с определением параметров чистоты нуклеиновых кислот А260/280.
Реакцию LAMP проводили в следующих условиях: состав реакционной смеси (Таблица 9), режим амплификации - 120 циклов по 30 сек при температуре плюс 63°С, инактивация при плюс 95°С в течение 2 мин. В реакцию вносили по 5 мкл гДНК с концентрацией 1 нг/мкл, что является высокой концентрацией матрицы.
В результате тестирования не было определено ложноположительных результатов на ДНК гомологичных и гетерологичных организмов. Значение Ct для положительного контроля (ДНК Streptococcus mutans штаммов АТСС 25175 и АТСС 35668) с концентрацией 4×10-6 нг/мкл составило 17,64±0,13.
Пример 5. Аналитическая чувствительность набора реагентов
Пример 5.1. Аналитическая чувствительность набора реагентов при проведении LAMP с детекцией в режиме реального времени
Аналитическая чувствительность отражает наименьшее количество (концентрацию) обнаруживаемого вещества в исследуемом материале.
Определение аналитической чувствительности проводилось на модельных штаммах кариесогенной бактерии Streptococcus mutans АТСС 25175 и АТСС 35668.
Для проведения анализа Streptococcus mutans штаммов АТСС 25175 и АТСС 35668 культивировали для получения клеточной массы. Клетки микроорганизмов с микробиологических петель сначала высевали на агаризованную, а затем в жидкую среду для культивирования и культивировали, как описано выше (пример 3). Выделение геномной ДНК проводили с помощью коммерчески доступного набора реагентов Quick-DNA Fungal/Bacterial microprep kit (Zymo Research, США) в трех повторностях. Концентрацию гДНК определяли на спектрофотометре NanoDrop 2000 с определением параметров чистоты нуклеиновых кислот А260/280. Титрование матрицы проводили от 1 нг на реакцию (0,04 нг/мкл) до 10-6 нг на реакцию (4×10-8 нг/мкл) путем последовательных 10-кратных разведений.
Реакцию LAMP проводили в следующих условиях: состав реакционной смеси (Таблица 9), режим амплификации - 120 циклов по 30 сек при температуре плюс 63°С, инактивация при плюс 95°С в течение 2 мин.
В ходе исследования проводили сравнение коммерческого фермента Bst-ДНК полимераза большой фрагмент (NEB, Ipswich) и UltraFast Bst-ДНК полимераза (ООО «Иннова плюс», Россия) (Таблица 16).

В результате сравнительного анализа аналитической чувствительности с применением коммерческого и отечественного фермента Bst-ДНК полимеразы чувствительность наборов реагентов с коммерческим ферментом Bst-ДНК полимераза большой фрагмент (NEB, Великобритания) составила 4×10-5 нг/мкл, а с ферментом UltraFast Bst-ДНК полимераза (ООО «Иннова плюс», Россия) - 4×10-6 нг/мкл.
В следующем эксперименте были использованы две концентрации фермента UltraFast Bst-ДНК полимераза (ООО «Иннова плюс», Россия), соответствующие 8 ед. акт. и 12 ед. акт. Результаты представлены в таблице в виде числовых значений Ct (Таблица 17).

В результате проведенного исследования показано, что чувствительность набора реагентов составляет 4×10-6 нг/мкл. Время проведения реакции меньше при содержании в реакционной смеси 12 ед. акт. фермента.
Пример 5.2. Аналитическая чувствительность набора праймеров при проведении LAMP с визуальной детекцией
Параллельно с детекцией результата в режиме реального времени была проанализирована аналитическая чувствительность набора реагентов при проведении LAMP с визуальной детекцией с применением красителя гидроксинафтол голубого (HNB).
Реакцию LAMP проводили в следующих условиях: состав реакционной смеси (0,12 мМ HNB, 8 ед. акт. UltraFast Bst ДНК-полимеразы, 1 × реакционный буфер, 7 мМ сульфата магния, 1,4 мМ дезоксинуклеотидтрифосфатов, по 1,6 мМ внутренних праймеров (FIP, BIP), 0,2 мМ наружных (F3, В3), 0,8 мМ прямого петлевого праймера (LF)), режим амплификации - 120 циклов по 30 сек при температуре 63°С, инактивация при 95°С в течение 2 мин.
Для определения чувствительности набора реагентов образец геномной ДНК Streptococcus mutans анализировали в серии последовательных 10-кратных разведений в концентрации от 10 нг/реакцию (4⋅10-2 нг/мкл) до 10-5 нг/реакцию (4⋅10-7 нг/мкл). Результаты представлены на Фиг. 6А.
Кроме того, с целью моделирования эксперимента по оценке хранения составляющих набора реагентов в формате ускоренной температурной деградации набор реагентов помещали в термостат с температурой 37°С на 2 недели. 2 недели ускоренной температурной деградации при 37°С соответствуют сроку хранения 12 месяцев. Результаты представлены на Фиг. 6Б.
В результате тестирования установлено, что выявление геномной ДНК Streptococcus mutans при проведении LAMP с визуальной детекцией HNB с использованием заявленного набора реагентов наблюдается, начиная с разведения 4⋅10-4 нг/мкл, по изменению окрашивания реакционной смеси. Таким образом, предел аналитической чувствительности заявленного набора реагентов составляет 4⋅10-4 нг/мкл. Причем указанная аналитическая чувствительность сохраняется и после 2 недель ускоренного хранения набора реагентов при температуре 37°С, что свидетельствует о стабильности набора в течение срока хранения 12 месяцев при температуре минус 2±2°С.
Таким образом, у разработанного набора реагентов есть ряд преимуществ:
- в отличие от аналогов выявление Streptococcus mutans можно проводить без пробоподготовки, добавляя напрямую образец слюны в пробирку с реакционной смесью;
- детекцию результатов можно проводить без использования сложного оборудования по изменению цвета реакционной смеси за счет использования металлоиндикатора HNB;
- для автоматизации исследования детекцию также можно проводить с использованием амплификатора в реальном времени;
- аналитическая чувствительность набора реагентов при детекции методом LAMP в режиме реального времени составляет 4⋅10-6 нг/мкл (краситель EvaGreen);
- аналитическая чувствительность набора реагентов при визуальной детекции методом LAMP составляет 4⋅10-4 нг/мкл (краситель HNB);
- аналитическая специфичность составляет 100%;
- время реакции до момента определения предела чувствительности тест-системы составляет 9,5-11 минут, что значительно меньше времени проведения реакции ПЦР;
- специфические праймеры подобраны с использованием отечественного продукта, не имеющего аналогов в РФ, программы «Lamprim» ООО «Иннова плюс», Россия;
- в реакции LAMP используется фермент с цепь-вытесняющей активностью, UltraFast Bst ДНК-полимераза отечественного производства ООО «Иннова плюс», Россия.
Заявитель просит рассмотреть представленные материалы заявки «Набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации» на предмет выдачи патента на изобретение.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="2022134650_перечень.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2022-12-28">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022134650 </ApplicationNumberText>
<FilingDate>2022-12-27</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">ОБЩЕСТВО С ОГРАНИЧЕННОЙ
ОТВЕТСТВЕННОСТЬЮ "ИННОВА ПЛЮС"</ApplicantName>
<ApplicantNameLatin>Innova plus LLC</ApplicantNameLatin>
<InventionTitle languageCode="ru">Набор праймеров для выявления
Streptococcus mutans методом изотермической петлевой
амплификации</InventionTitle>
<SequenceTotalQuantity>62</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagagctttacgatccgaaaaccttttgcaatggacgaaagtctg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtgacggtagcttaccagaaagttttgggacctacgtattaccg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcagtagggaatcttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctttacgcccaataaatcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccactctcacacacgttcttgttttgtgaagaaggttttcggat</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtgacggtagcttaccagattttgggacctacgtattaccg</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaatggacgaaagtctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttacgcccaataaatccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agctttacgatccgaaaaccttcttttgcaatggacgaaagtctg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcacacagtgacggtagctttttgggacctacgtattaccg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcagtagggaatcttcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttacgcccaataaatccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgacaacgaactagtactgtggttttacattaaccagtgacagct</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctccataacatcacaactgcctattttttttgaacttgccgtactc</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttaatgggttctaagcgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctcaaagaatttctcggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacgaactagtactgtgggcttttacattaaccagtgacagc</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccataacatcacaactgcctacttttcctcaaagaatttctcggac</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttaatgggttctaagcgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtcaacgaagttgtcttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgacaacgaactagtactgtgggttttacattaaccagtgacagc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccataacatcacaactgcctacattttcctcaaagaatttctcggac</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gccttttgagttacatcgctcatttttggtatggcagtattggct</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q49">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgctgaggatatggaacagtcaattttccaccgtttcatttgacc</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctctgttgtaaccatactgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q53">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccttaaggtctgtctcttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q55">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacacgattgaccaaggctttttcagtcttggaattggtgg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q57">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgctgttgccatgctctatttttgtatctggtaaaacagccg</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q59">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgctacaaatcctttccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q61">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccataaccaccagtctcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q63">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tagtggtcgtttcatcggcttttgaacagtagcagcagtct</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q65">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggaaatccagcgaccaattttgttcagcttccttactcg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taaaacactgtgtggtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagcttggttttgactt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggtcgtttcatcggcaattttaacagtagcagcagtctc</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttggaacacaaactggaaatccttttgttcagcttccttactcg</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtaaaacactgtgtggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccatttgtctctccagct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtagtggtcgtttcatcggcttttttctaggaacagtagcagc</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctggaaatccagcgaccaattttgttcagcttccttactcg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtaaaacactgtgtggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttgaaccatttgtctctcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttaacagtaatggtgtcgccttttttcgctttgagtttgatgag</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcagaattcaggaacagtcttttaacctccagcttaatggtt</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actcagattaatgatggcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcgtaactaccaaatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taatggtgtcgcctgctttaatttttcagattaatgatggcggtaag</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>45</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..45</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagaattcaggaacagtcagaggttttcctgaacctccagcttaa</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttcatcgctgacggtat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgtaactaccaaatcacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaactacgtgccagcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctaactacgtgccagcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctaggcagaacatctatact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctaggcagaacatctatac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaaacaatacttggagtacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacaatacttggagtacgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atacttggagtacggcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgtcatgtctgttgaactatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctgtcatgtctgttgaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtttcaacctgcgaatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtttcaacctgcgaatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcatctgtttcaacctgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

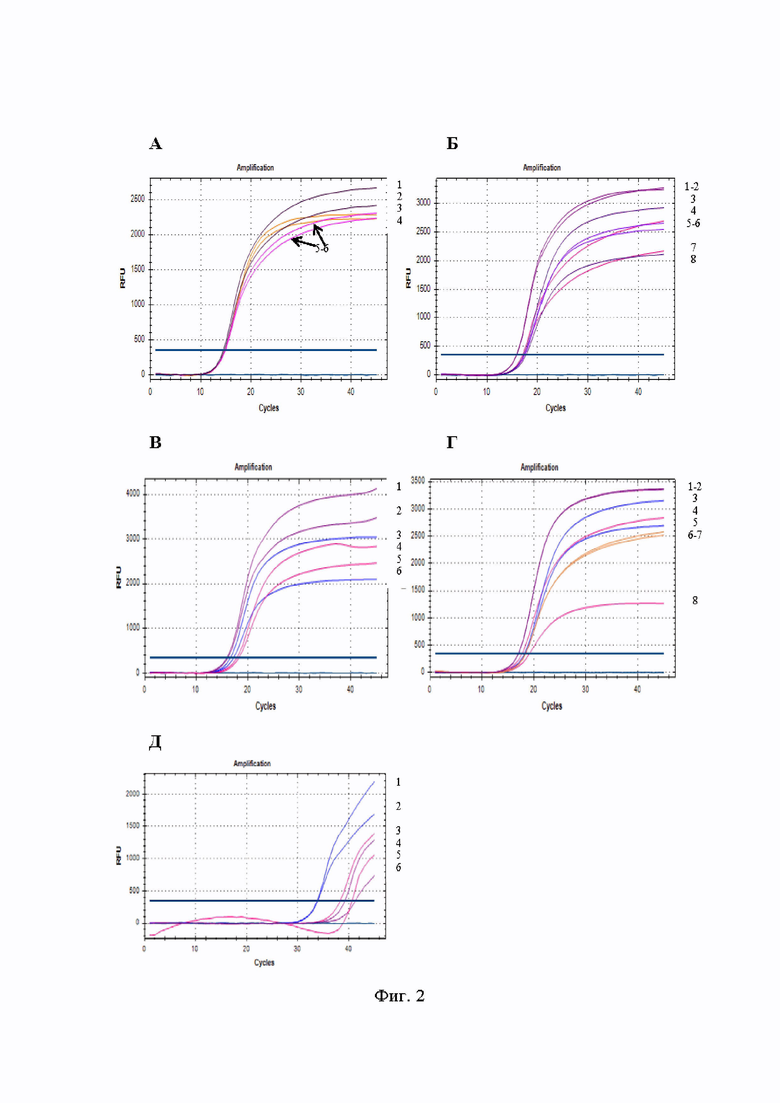
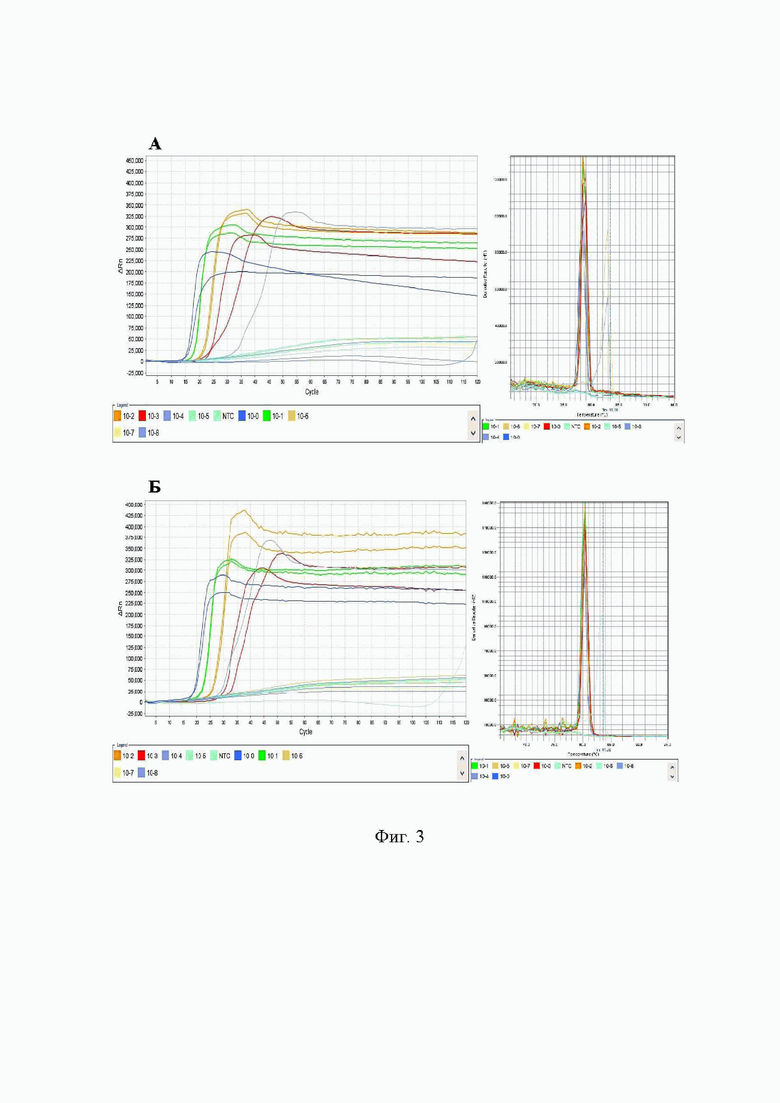



Изобретение относится к области медицины, биотехнологии, микробиологии и молекулярной биологии, в частности к молекулярно-биологическим способам детекции микроорганизмов. Предложен набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, выбранный из набора, включающего праймеры с нуклеотидной последовательностью Seq Id No: 13-16, 54, набора, включающего праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55, и набора, включающего праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58. Изобретение позволяет быстро выявлять Streptococcus mutans в образце слюны без пробоподготовки, с чувствительностью не ниже 10-4 нг/мкл и специфичностью 100%. 5 з.п. ф-лы, 6 ил., 17 табл., 5 пр.
1. Набор праймеров для выявления Streptococcus mutans методом изотермической петлевой амплификации, выбранный из набора, включающего праймеры с нуклеотидной последовательностью Seq Id No: 13-16, 54, набора, включающего праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55, и набора, включающего праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58.
2. Набор праймеров по п. 1, включающий праймеры с нуклеотидной последовательностью Seq Id No: 13-16, 54.
3. Набор праймеров по п. 1, включающий праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55.
4. Набор праймеров по п. 1, включающий праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58.
5. Набор праймеров по п. 1, включающий праймеры с нуклеотидной последовательностью Seq Id No: 19-22, 55 и, дополнительно, праймеры с нуклеотидной последовательностью Seq Id No: 23-26, 58.
6. Набор праймеров по любому из пп. 1-5, содержащий внутренние праймеры (FIP, BIP, Seq Id No: 13, 14 или 21, 22 или 23, 24), наружные праймеры (F3, B3, Seq Id No: 15, 16 или 19, 20 или 25, 26) и прямой петлевой праймер (LF, Seq Id No: 54 или 55 или 58) в молярном соотношении 8:8:1:1:4.
| JP 2005013055 A, 20.01.2005 | |||
| KITAGAWA М., et al | |||
| Rapid detection of the Streptococcus mutans cnm gene by loop-mediated isothermal amplification | |||
| Anal Biochem | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
| NAGASHIMA S., et al | |||
| Rapid detection of the cariogenic pathogens Streptococcus mutans and Streptococcus sobrinus using loop-mediated isothermal amplification | |||
| Oral | |||
Авторы
Даты
2023-07-05—Публикация
2022-12-27—Подача