Изобретение относится к области молекулярно-генетических методов идентификации микроорганизмов - наиболее значимых бактерий рода Bacillus: В. subtilis, В. megaterium и В. thuringiensis и может быть использовано при определении этих бактерий в составе коммерческих биопрепаратов.
Бактерии рода Bacillus, в особенности В. subtilis, часто используются в животноводстве в качестве пробиотиков. Также бактерии этого рода используются для обработки растений, поскольку некоторые виды бактерий, такие как В. subtilis и В. megaterium обладают свойствами антагониста патогенной микрофлоры почв, улучшают биодоступность питательных веществ, а бактерия В. thuringiensis обладает инсектицидным действием. Ввиду того, что эти виды бактерий обладают различными хозяйственными свойствами важно корректно их идентифицировать.
Идентифицировать эти бактерии морфологически крайне сложно, необходим более эффективный и быстрый метод дифференциации этих трех видов бактерий для верификации состава коммерчески доступных микробиологических биопрепаратов. Для этого был разработан метод дифференциации данных бактерий на основе ПЦР-ПДРФ (полимеразная цепная реакция-полиморфизм длины рестрикционных фрагментов). Для анализа использовался участок, который амплифицируют праймеры 337F (SEQ ID NO: 1) и 1100R (SEQ ID NO: 2). Первоначально был проведен биоинформатический анализ коммерчески доступных рестриктаз, способных формировать фрагменты, специфичные для каждого из трех видов бактерий: Bacillus subtilis, Bacillus megaterium, Bacillus thuringiensis.
Известен молекулярно-генетический способ идентификации прокариотических организмов на основе ПЦР (полимеразная цепная реакция) к участку hin-региона и последующего сравнения получившихся продуктов в гель-электрофорезе (патент 2486251, МПК C12Q 1/68 (2006.01)). На этапе ПЦР используются последовательности олигонуклеотидных праймеров: прямой праймер: GGAAGAGGGGTTCGACT; обратный праймер: AGAATGAAAATCTGGTG. Анализ результатов осуществляют методом горизонтального электрофореза. Данный метод используется для дифференциации Rizobium и других родов бактерий. Этот способ не позволяет проводить дифференциацию бактерий рода Bacillus.
Известен способ идентификации бактерий В. anthracis (патент 2204607, МПК C12Q 1/04 (2000.01), C12N 1/20 (2000.01)). Способ заключается в оценки культуральных свойств бактерии В. anthracis. Недостатком способа является невозможность идентификации трех видов бактерий рода Bacillus: В. subtilis, В. megaterium и В. thuringiensis
Известен способ идентификации бактерий В. thuringiensis (патент 2750913, МПК C12Q 1/04 (2006.01), G01N 33/50 (2006.01), C12N 1/20 (2006.01), C12R 1/07 (2006.01)). Способ заключается в проведении лизиса бактериальных клеток с последующим проведением электрофореза белков. Недостатком способа является невозможность идентификации двух других видов бактерий: В. subtilis и В. megaterium.
В качестве прототипа взят способ идентификации В. thuringiensis с помощью полимеразной цепной реакции с праймерами, специфичными к генам, кодирующими определенные токсины [Frederiksen, K., Rosenquist, H., Jorgensen, K., & Wilcks, Α. (2006). Occurrence of Natural Bacillus thuringiensis Contaminants and Residues of Bacillus thuringiensis-Based Insecticides on Fresh Fruits and Vegetables. Applied and Environmental Microbiology, 72(5), 3435-3440. doi:10.1128/aem.72.5.3435-3440.200]. Способ включал в себя выделение геномной ДНК, RAPD-анализ, проведение электрофореза в агарозном геле. Способ позволяет различать многие серотипы и некоторые штаммы В. thuringiensis, однако идентификация двух других видов бактерий: В. subtilis и В. megaterium при использовании данного способа не возможна.
Задачей настоящего изобретения является разработка молекулярно-генети-ческого метода идентификации бактерий рода Bacillus: В. subtilis, В. megaterium и В. thuringiensis, который ускорит и упростит идентификацию бактерий по сравнению с традиционными рутинными методами высевания на питательные среды, а также будет являться достоверным и точным, не требующим знаний морфологии бактерий. Данный молекулярно-генетический метод позволит проводить идентификацию бактерий в любой молекулярно-генетической лаборатории.
Существенным преимуществом заявляемого молекулярно-генетического метода идентификации бактерий является быстрота и точность определения. Предварительно был проведен анализ нуклеотидной последовательности гена 16S рРНК. Установлены отличия в нуклеотидной последовательности между В. subtilis, В. megaterium и В. thuringiensis, которые позволяют разработать метод экспресс-идентификации данных видов бактерий на основе рестриционного анализа гена 16S рРНК с использованием рестриктазы Alu I.
Идентификация бактерий в описываемом способе осуществляется на основе рестрикционного анализа предварительно амплифицированного гена 16S рРНК.
Технический результат - проведение идентификации бактерий видов В. subtilis, В. megaterium и В. thuringiensis без привлечения специалистов в области микробиологии стандартизированными методами молекулярной генетики, с возможностью проведения идентификационных процедур в течение 4,5-6 часов.
Технический результат достигается тем, что в способе идентификации бактерий рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis на основе рестрикционного анализа гена 16S рРНК, включающем выделение ДНК из предварительно собранного биологического материала, последующее проведение ПЦР, проведение электрофореза в агарозном геле, согласно изобретению, проводят ПЦР участка гена 16S рРНК с помощью праймеров: прямой 337F GACTCCTACGGGAGGCWGCAG, SEQ ID NO: 1 и обратный 1100R GGGTTGCGCTCGTTG, SEQ ID NO: 2 согласно Перечню последовательностей; затем осуществляют рестрикцию продуктов ПЦР, анализ полиморфизма длин рестрикционных фрагментов (ПДРФ) ампликона с помощью электрофореза, при рестрикционном анализе для идентификации используют рестриктазу Alu I, образующую уникальные фрагменты рестрикции для трех наиболее значимых бактерий рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis, где наличие фрагмента ДНК длиной 430 п.н. указывает на принадлежность к виду бактерий В. subtilis, длиной 526 п.н. - на принадлежность к виду бактерий В. megaterium, длиной 598 п.н - на принадлежность к виду бактерий - В. thuringiensis.
Способ идентификации бактерий рода В. subtilis, В. megaterium и В. thuringiensis на основе рестрикционного анализа предварительно амплифициро-ванного гена 16S рРНК реализуется следующим образом:
1. Отбирают нуждающиеся в идентификации микробиологические монопрепараты, содержащие предположительно бактерии рода Bacillus.
2. Производится выделение ДНК из полученных образцов, выделение ДНК можно проводить как с помощью коммерческих наборов отечественных и зарубежных производителей ("ДНК-технологии", "Синтол", "Zymo research", БиоСилика и др.), так и общепринятыми методами (фенол-хлороформная экстракция, ЦТАБ (цетилтриметиламмоний бромид)-метод).
3. Проводится ПЦР. Используют следующие температурные циклы: 95°С 2 мин, далее 30 циклов вида: 95°С 30 сек - денатурация, 54°С 30 сек - отжиг праймеров, 72°С 45 сек - элонгация. После чего проводят инкубирование смеси - 72°С в течение 5 мин. В качестве праймеров для амплификации используются: прямой 337F (SEQ ID NO: 1) GACTCCTACGGGAGGCWGCAG, обратный (SEQ ID NO: 2) 1100R GGGTTGCGCTCGTTG.
4. Продукты ПЦР подвергают рестрикции по следующей схеме: в пробирке на 0,2 мл смешивают 10 мкл ПЦР продукта, 1,5 мкл 10Х реакционного буфера, 10 ед. рестриктазы. Объем доводят до 15 мкл деионизированной водой. Смесь инкубируют в течение 2 часов при 37°С, после чего фермент инактивируют нагревом до 65°С в течение 20 мин. После чего проводят анализ полиморфизма длин рестрикционных фрагментов (ПДРФ) ампликона. Результаты рестрикционного анализа фиксируются с помощью метода электрофореза в 2% агарозном геле. В качестве рестриктазы используют Alu I. Фермент рестрикции образует уникальные фрагменты для трех представителей рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis (фиг. 2).
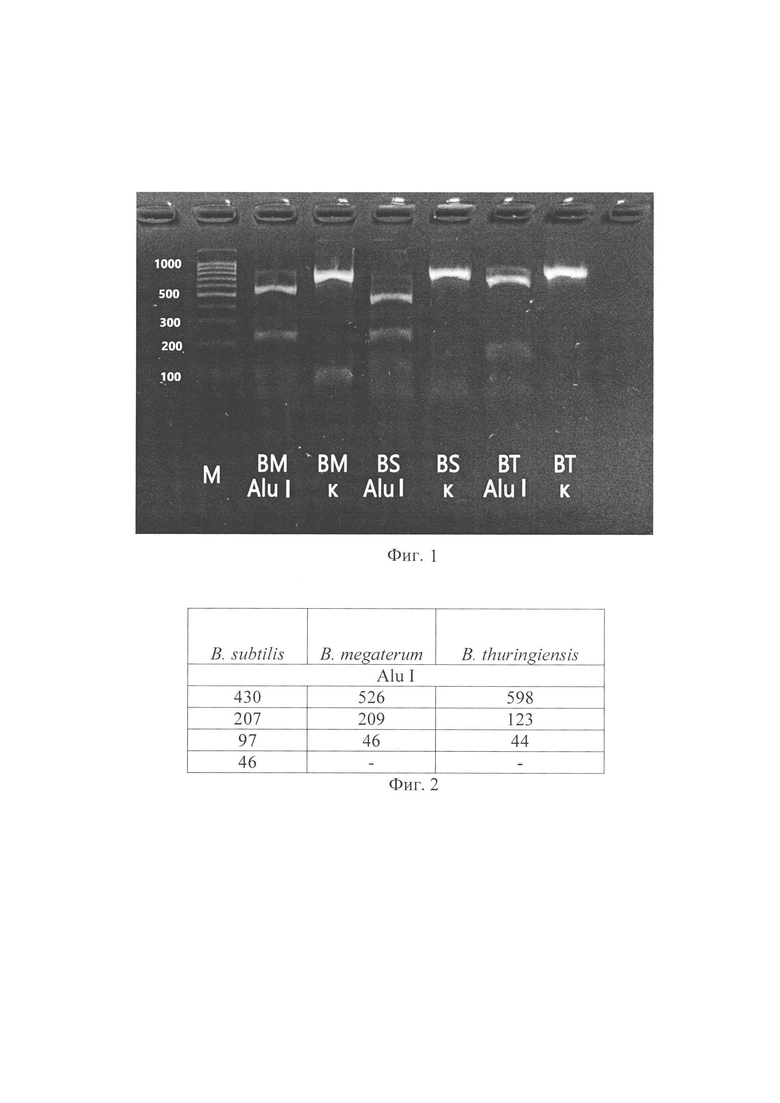
На фиг. 1 приведена электрофореграмма продуктов рестрикции ПЦР гена 16 рРНК; на фиг. 2 представлены длины ожидаемых фрагментов (п.н.) при расщеплении предварительно амплифицированной последовательности 16S рРНК рестриктазой Alu I.
Пример 1.
На электрофореграмме: M - маркер длин фрагментов ДНК, значения выражены в пар нуклеотидов (п.н.); ВМ - участок гена 16S рРНК бактерии В. megaterium из биопрепарата №1; BS - участок гена 16S рРНК бактерии В. subtilis из биопрепарата №2; ВТ - участок гена 16S рРНК бактерии В. thuringiensis из биопрепарата №3; К - продукт ПЦР до рестрикции; Alu I - продукт ПЦР после рестрикции с использованием фермента рестрикции Alu I.
При обработке рестриктазой Alu I продукта ПЦР образцов ДНК ВМ (В. megaterium), ВТ (В. thuringiensis) и BS (В. subtilis) образовались крупные фрагменты длиной 526 п. н., 430 п.н. и 598 п.н. для В. megaterium, В. subtilis и В. thuringiensis соответственно. Следовательно, в биопрепарате №1 содержатся бактерии В. megaterium, биопрепарате №2 - бактерии В. subtilis, биопрепарате №3 - бактерии В. thuringiensis.
Предлагаемый способ идентификации значимых для сельского хозяйства представителей рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis на основе рестрикционного анализа гена 16S рРНК является высокочувствительным, хорошо воспроизводимым, универсальным для хозяйственно значимых бактерий рода Bacillus и экономичным. В зависимости от способа выделения ДНК из биологических образцов анализ занимает от 4,5 до 6 часов. Анализ можно проводить в лабораториях, имеющих термоциклер, центрифугу, камеру для электрофореза и сопутствующие реактивы и расходные материалы.
--->
Перечень последовательностей
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ
идентификации значимых для сельского хозяйства представителей рода
Bacillus – B. subtilis, B. megaterium и B. thuringiensis на основе
рестрикционного анализа гена 16S рРНК.xml" softwareName="WIPO
Sequence" softwareVersion="2.3.0" productionDate="2023-07-03">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-07-03</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>16/23</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-07-03</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное образовательное учреждение высшего образования
"Воронежский государственный университет"</ApplicantName>
<ApplicantNameLatin>Federal'noe gosudarstvennoe byudjetnoe
obrazovatel'noe uchrejdenie vysshego obrazovaniya
"Voronejskii gosudarstvennyi
universitet"</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ идентификации значимых для
сельского хозяйства представителей рода Bacillus – B. subtilis, B.
megaterium и B. thuringiensis на основе рестрикционного анализа гена
16S рРНК</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus spp.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactcctacgggaggcwgcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Bacillus spp.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggttgcgctcgttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области микробиологии. Описан способ идентификации бактерий рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis на основе рестрикционного анализа включает выделение ДНК из биологического материала с последующим проведением ПЦР участка гена 16S рРНК с помощью праймеров 337F SEQ ID NO: 1 и 1100R SEQ ID NO: 2. Для идентификации при рестрикционном анализе используют рестриктазу Alu I, образующую уникальные фрагменты рестрикции для трех наиболее значимых для сельского хозяйства бактерий рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis. Различие в длинах больших фрагментов: 430, 526, 598 п.н у В. subtilis, В. megaterium и В. thuringiensis соответственно позволяет провести идентификацию данных бактерий. Изобретение позволяет идентифицировать данные виды бактерий быстро, точно и без привлечения специалиста в области микробиологии. Технический результат - проведение идентификации бактерий видов В. subtilis, В. megaterium и В. thuringiensis без привлечения специалистов в области микробиологии стандартизированными методами молекулярной генетики, с возможностью проведения идентификационных процедур в течение 4,5-6 часов. 2 ил., 1 пр.
Способ идентификации представителей рода Bacillus - В. subtilis, В. megaterium и В. thuringiensis на основе рестрикционного анализа гена 16S рРНК, включающий выделение ДНК из биологического материала, проведение ПЦР, проведение электрофореза в агарозном геле, отличающийся тем, что проводят ПЦР участка гена 16S рРНК с помощью праймеров: прямой 337F GACTCCTACGGGAGGCWGCAG, SEQ ID NO: 1 и обратный 1100R GGGTTGCGCTCGTTG, SEQ ID NO: 2; затем осуществляют рестрикцию продуктов ПЦР рестриктазой Alu I и анализ полиморфизма длин рестрикционных фрагментов ампликона с помощью электрофореза, где наличие фрагмента ДНК длиной 430 п.н указывает на принадлежность к виду бактерий В. subtilis, длиной 526 п.н. - на принадлежность к виду бактерий В. megaterium, длиной 598 п.н - на принадлежность к виду бактерий - B. thuringiensis.
| CN 101993953 A, 30.03.2011 | |||
| AU 2014202721 A1, 12.06.2014 | |||
| RU 2021101505 A, 26.07.2022. |
Авторы
Даты
2024-07-12—Публикация
2023-07-05—Подача