Изобретение относится к области молекулярной цитогенетики и может быть использовано для определения хромосомных транслокаций, делеций и амплификаций на основе флуоресцентной гибридизации ДНК-зонда с регионом генома протяженностью более 30 т.п.н., в различных биологических образцах клеток человека.
Флуоресцентная гибридизация in situ (FISH) - метод, который используется для пространственного обнаружения и количественного определения нуклеиновых кислот в их клеточном окружении. Он стал мощным цитогенетическим подходом к анализу клеток и тканей на уровне генома и транскриптома [1]. В диагностике FISH считается золотым стандартом для выявления клеток, содержащих хромосомные перестройки [2] или хромосомные аберрации [3; 4]. Метод основан на гибридизации специфичных к участку нуклеиновых кислот комплементарных зондов (обычно последовательностей ДНК) с их мишенью внутри клетки. Зонды могут быть нацелены на ДНК (метафазные и интерфазные хромосомы), а также на РНК, что делает возможным изучение геномных последовательностей и профилей экспрессии генов в отдельных клетках. Реакцию можно визуализировать непосредственно с помощью радиоактивно или флуоресцентно меченых зондов или косвенно с помощью гистохимических хромогенов, связывания с антигеном или взаимодействия биотин-стрептавидин.
Связывание комплементарных зондов позволяет обнаруживать и визуализировать интересующую мишень внутри клеток. Эта визуализация может быть использована в диагностических целях, таких как пренатальная и постнатальная диагностика хромосомных аберраций, при выявлении анеуплоидии, транслокации, инсерции или делеции [5]. Некоторые примеры заболеваний, диагностируемых с помощью FISH, включают муковисцидоз, мышечную дистрофию Дюшенна, синдром Прадера-Вилли, синдром Ангельмана, синдром Лежена, трисомии 13, 16, 18, 21, моносомию X, синдром Тернера и различные онкологические заболевания. Кроме того, ген-специфические мишени могут быть использованы для обнаружения амплификаций генов, часто обнаруживаемых в опухолевых клетках. Примером может служить обнаружение изменений числа копий гена рецептора 2 эпидермального фактора роста человека (HER2) в качестве прогностического биомаркера при раке молочной железы [6] и яичников [7; 8]. FISH дополнительно играет роль в видовой идентификации микроорганизмов [9]. Таким образом, метод FISH является распространенным инструментом в клеточных биологических исследованиях, а также в диагностических приложениях, поскольку он предоставляет ценную количественную информацию о распределении нуклеиновых кислот внутри отдельных клеток. Большим преимуществом метода является его способность предоставлять пространственную информацию о содержимом клетки, обеспечивая тем самым возможность исследования мозаицизма по хромосомным нарушениям в клеточной популяции [1].
В некоторых случаях в область молекулярной патологии и молекулярной онкологии помимо FISH внедряется также и метод хромогенной гибридизации in situ (CISH). Оба метода основаны на одной и той же идее гибридизации ДНК-зонда, специфичного к определенному региону генома на геномной ДНК без лизиса клеток [10]. Основное различие между методами заключается в характере детекции результатов: с помощью обнаружения хромогенной окраски в проходящем свете с использованием пероксидазы хрена (HRP) и хромогенного субстрата в случае CISH или с помощью эпифлуоресцентной детекции ДНК-зондов, меченных флуорофорами, при использовании FISH. В случае FISH мечение ДНК-зонда может быть прямым с использованием нуклеотидтрифосфатов, конъюгированных с флуорофорами, либо непрямым, когда в ДНК-зонд вводятся нуклеотидтрифосфаты, конъюгированные с биотином или дигоксигенином, к которым в последующем присоединяются стрептавидин, конъюгированный с флуорофором, или меченные флуорофором антитела к дигоксигенину, соответственно. В случае CISH используется непрямой вариант мечения с использованием зондов, меченных биотином, для обнаружения которых используют стрептавидин, конъюгированный с пероксидазой хрена (HRP-стрептавидин). Для зондов с конъюгированным дигоксигенином используют первичное антитело к дигоксигенину, а затем вторичное антитело, конъюгированное с HRP. Результаты CISH анализируются под микроскопом со светлым полем [11; 12], тогда как FISH-анализ требует использования флуоресцентной микроскопии.
Тестирование генетических маркеров часто играет ключевую роль в принятии решений, касающихся пациентов, от стратификации риска для пациентов до проведения соответствующего лечения. Оба метода успешно используются в медицине [13]. Уровень соответствия между результатами FISH и CISH, рассчитанный в группе из 4460 пациентов с диагнозом рак молочной железы, был на уровне 96%, что свидетельствует о том, что метод CISH сопоставим с FISH. Большинство источников согласны и сообщают о почти одинаковой эффективности FISH и CISH при диагностики амплификаций генов. Однако CISH может показать более низкую чувствительность для низкоуровневых амплификаций вследствие, например, полисомии хромосомы 17 [13]. Аналогичные наблюдения были представлены для перестроек гена ALK (тирозинкиназа рецептора ALK), протестированных с использованием FISH и CISH. Чувствительность CISH была на уровне 94,4%, а специфичность - 100%.
Существенным вопросом в методике FISH является выбор соответствующего зонда, который определяет ценность теста для пациента. Каждый тип FISH-зонда позволяет обнаруживать различные хромосомные аберрации. Наиболее часто используются ДНК-зонды следующих типов:
1. Полнохромосомные, окрашивающие всю хромосому. Применяются для обнаружения хромосомных транслокаций.
2. Локус-специфичные, гибридизующиеся с отдельным локусом на хромосоме, в том числе, центромеро-специфичные.
3. Мультилокусные ДНК-зонды, специфичные к определенным типам повторяющихся последовательностей (панцентромерные, пантеломерные).
Локус-специфичные ДНК-зонды можно использовать для оценки количества копий отдельного региона хромосомы или целой хромосомы. Последняя задача реализуется при оценке анеуплоидии с помощью центромеро-специфичных ДНК-зондов, или ДНК-зондов, специфичных для отдельных регионов хромосом.
Таким образом, флуоресцентная гибридизация in situ представляет собой метод, незаменимый для обнаружения различных хромосомных аномалий, таких как анеуплоидии, слияние генов, а также хромосомных амплификаций и делеций. Благодаря постепенно внедряемым усовершенствованиям метод дает важную информацию, которую можно использовать не только для пренатальной и постнатальной диагностики, но и для подбора лучших методов лечения. Вследствие универсальности FISH, данный метод можно использовать для цитогенетического анализа, а также для гематологических (например, лейкемии, лимфомы, множественной миеломы) и солидных опухолей (например, рака молочной железы, немелкоклеточного рака легких, колоректального рака).
Подбор праймеров и синтез ДНК-зондов.
Ключевыми характеристиками ДНК-зонда для использования в FISH диагностике являются следующие:
1. Специфическая гибридизация только с целевым регионом генома.
2. Флуоресцентный сигнал, уверенно видимый под флуоресцентным микроскопом при увеличении 630x как в интерфазных ядрах, так и на метафазных хромосомах.
Поэтому для специфической гибридизации ДНК-зонд должен разрабатываться на основе знания о последовательности ДНК целевого региона. Генерация достаточно интенсивного флуоресцентного сигнала на целевой ДНК возможна при гибридизации ДНК-зонда с регионом генома протяженностью более 30 т.п.н.
Известен способ специфического декорирования выбранных хромосом млекопитающих и их обнаружения, идентификации и/или количественного определения выбранных индивидуальных хромосом посредством супрессионной гибридизации in situ (CISS) [14]. CISS гибридизация основана на предварительной гибридизации меченой ДНК с избыточным количеством C0t-1 ДНК соответствующего вида. В результате меченная повторяющаяся ДНК формирует дуплексы с C0t-l ДНК и, таким образом, выводится из процесса гибридизации с ДНК метафазных хромосом. Метод используется для обнаружения хромосом, фрагментов хромосом или хромосомных аберраций.
Известен способ выявления внутрихромосомного дисбаланса в интерфазных ядрах с использованием in situ гибридизации региона хромосомы, на которую предположительно влияет указанный дисбаланс [15]. Для этого применяют два зонда, меченные различными флуорохромами - специфичные для длинного плеча и короткого плеча хромосомы, соответственно. Способ также используется для обнаружения раковых клеток в биологическом образце и для диагностики онкопатологий.
Известен способ обнаружения генных перестроек [16]. Разработана серия ДНК-зондов для использования в диагностике и мониторинге различных типов лейкозов с использованием методов нозерн-блот анализа и FISH. Эти зонды используются для обнаружения перестроек, таких как транслокации, с участием региона 11q23 с другими участками хромосом, включая 4q21, 6q27, 9p22, 19p13.3, как в делящихся лейкозных клетках, так и в интерфазных ядрах. Представлены результаты нозерн-блоттингового анализа, демонстрирующие, что ген MLL имеет несколько транскриптов, размер которых отличает лейкозные клетки от нормальных. Также раскрыты слитые белки с участием гена MLL, домены белка MLL и антитела против MLL.
Известен способ, основанный на обнаружении последовательности-мишени с использованием ДНК-зонда, который получен способом, включающим получение двухцепочечных полинуклеотидов, которые, содержат комплементарные последовательности-мишени и повторяющиеся последовательности, фрагментацию указанных полинуклеотидов на фрагменты, денатурирование в отдельные нити с последующей гибридизацией с флуорохромами с использованием ISH, FISH и CGH [17].
Известен способ для профилирования отдельных хромосом с использованием специфичных для мишени ДНК зондов в биологических образцах, таких как внеклеточная ДНК из периферической крови беременной женщины или больного раком [18]. Способ относится к генерации хромосомных профилей либо по отдельности, либо в комбинации (мультиплекс).
Недостатки аналогов. Для разработки ДНК-зондов ключевым этапом является получение фрагментов ДНК, комплементарных целевому региону, на который проводится разработка ДНК-зонда. Для этого часто используются либо микродиссектированные хромосомы, либо клоны бактериальных или дрожжевых искусственных хромосом (BAC или YAC). В первом случае полученные фрагменты изобилуют повторами и редко могут быть амплифицированы полностью с приемлемой равномерностью покрытия всего региона. Во втором случае, получение новых клонов искусственных хромосом затруднительно и чаще всего используются BAC из уже созданных библиотек.
Наиболее близким к заявленному способу является способ изготовления зондов с использованием ПЦР амплификации коротких участков ДНК, размером до 200 п.н., комплементарных исследуемым локусам генома человека, для гибридизации хромосом для диагностики хромосомных аберраций [19]. Недостатками прототипа являются использование большого количества специфических праймеров для достижения формирования зонда протяженностью более 30 т.п.н. для генерации достаточно интенсивного флуоресцентного сигнала на целевой ДНК при гибридизации ДНК-зонда с регионом генома. Поэтому, заявленный способ получения ДНК-зондов для флуоресцентной гибридизации in situ основан на использовании ПЦР длинных фрагментов, который позволяет благодаря комбинации taq и pfu полимераз амплифицировать фрагменты геномной ДНК до 15 т.п.н.
Технической задачей изобретения является разработка технологии получения ДНК-зондов для флуоресцентной гибридизации in situ на основе амплификации длинных фрагментов геномной ДНК с последующей флуоресценцией.
Поставленная задача решена путем разработки ДНК-зондов на основе амплификации длинных до 15 т.п.н. фрагментов геномной ДНК. Разработанная технология получения ДНК-зондов для флуоресцентной гибридизации in situ включает два модуля: 1) общую платформу в виде универсальных адаптеров, лигируемых к ДНК-библиотеке и обеспечивающих быструю амплификацию любой ДНК-библиотеки в стандартных условиях; 2) разрабатываемый под каждый ДНК-зонд набор фрагментов целевого региона генома, предварительно амплифицируемых перед их сборкой в геномную библиотеку и лигированием адаптеров.
Такой подход обеспечивает следующие преимущества метода:
1. Снижение затрат времени и трудозатрат на повторную амплификацию множества фрагментов при разработке и производстве ДНК-зонда.
2. Простота производства ДНК-зонда.
3. Унифицированные условия производства ДНК-зондов на различные регионы генома.
4. Защиту геномной библиотеки от контаминации после этапа лигирования адаптеров за счет амплификации только фрагментов ДНК, содержащих адаптеры.
Техническим результатом изобретения является разработка простого, дешевого и эффективного метода получения флуоресцентных ДНК-зондов для проведения флуоресцентной гибридизации in situ, по сравнению с существующими аналогами.
Описание сущности изобретения
Способ получения ДНК-зондов включает несколько этапов: 1) выбор целевых регионов генома; 2) подбор и синтез олигонуклеотидных праймеров на целевые регионы генома; 3) отработка условий ПЦР получения фрагментов целевых последовательностей; 4) получение библиотеки фрагментов ДНК и ее фрагментация; 5) лигирование адаптеров и амплификация библиотеки; 6) введение в геномную библиотеку флуоресцентной метки; 7) проверка специфичности и интенсивности ДНК-зонда в реакции FISH.
Подбор праймеров проводится на специфические регионы хромосом. Суммарная длина амплифицированных фрагментов ДНК для создания ДНК-зондов должна превышать 30 т.п.н., чтобы ДНК-зонд был хорошо визуализирован в нужном регионе хромосомы при проведении FISH-анализа. Поэтому получаемые фрагменты должны быть близко расположены на хромосоме, но не перекрываться, поэтому на каждые 10 т.п.н. подбирается одна пара праймеров. Для получения последовательности нуклеотидов используется геномный браузер UCSC (University of California, Santa Cruz), который содержит информацию о последовательностях геномов. В дальнейшем полученная последовательность нуклеотидов используется для подбора праймеров с помощью стандартных биоинформатических программ (Primer-BLAST), которые предоставляет Национальный центр биотехнологической информации США (англ. National Center for Biotechnological Information, NCBI). Для проверки термодинамических свойств праймеров используется программа Vector NTI Advance 11.5.
Праймеры подбираются с учетом следующих условий:
1) все праймеры должны иметь близкую температуру плавления и температуру отжига (±1°С);
2) длина праймера может варьировать от 18 до 24 н.;
3) внутренняя гомология не должна превышать 3 п.н.;
4) содержание GC-динуклеотидов может варьировать в диапазоне от 40% до 60%.
Исключаются праймеры с полипиримидиновыми или полипуриновыми протяженными участками, а также праймеры и сайты-мишени, температура плавления вторичной структуры которых выше, либо эквивалентна температуре плавления самого праймера. Кроме того, оценивается возможность гомо- и гетеродимеризации праймеров, так как это может привести к снижению выхода конечного продукта ПЦР.
Наработка ДНК-продуктов проводится с помощью ПЦР длинных фрагментов с использованием набора БиоМастер LR HS-ПЦР-Color (2×) (Биолабмикс, Россия). Наработка фрагментов проводится на амплификаторе SureCycler 8800 (Agilent Technologies, США). Для каждого набора праймеров подбирается индивидуальный режим, в зависимости от длины праймеров и чувствительности. Для обнаружения соответствующих бэндов проводится электрофорез в агарозном геле для всех амплифицированных фрагментов.
Далее проводится очистка ПЦР продукта после ПЦР длинных фрагментов c целью избавления от красителя с помощью переосаждения амплификатов этанолом.
Ферментативная фрагментация двухцепочечной ДНК необходима для образования фрагментов требуемой длины, для последующего построения качественных библиотек, поэтому используется от 10 до 1000 нг ДНК, растворенной в воде. Проведение фрагментации проводится с помощью набора реактивов FTP Display (ДНК Дисплей, Россия). Общий объем реакционной смеси должен составлять 50 мкл. Очистка полученных фрагментов проводится с помощью магнитных частиц Ampure XP (Beckman Coulter, США).
Лигирование адаптеров. В качестве адаптеров используются разработанные Y-образные адаптеры собственного дизайна, позволяющие эффективно амплифицировать большие количества целевых библиотек. Последовательности адаптеров: AdF - ATGGTGATTCTACTGTTTCTGTGACT, AdR - GTCACAGAATAATGCCGAGTCCA. Реакция лигирования проводится с помощью Т4 ДНК лигазы (Биолабмикс, Россия) в соответствии с рекомендациями производителя.
Получение ДНК-библиотек. Амплификация ДНК-библиотек проводится с помощью набора для проведения ПЦР БиоМастер HS-Taq ПЦР (2×) (Биолабмикс, Россия) в соответствии с рекомендациями производителя. Последовательности праймеров: PrF - ATGGTGATTCTACTGTTTCTGTGAC, PrR - TGGACTCGGCATTATTCTGTGAC. Концентрацию ДНК определяют с помощью флуориметра Qubit 4.0 (Thermo, США).
Мечение ДНК-библиотек. Мечение ДНК-библиотек проводят с помощью реакции ник-трансляции с использованием флуоресцентных красителей Flu-12-дУТФ и TAMRA-5-дУТФ (Биосан, Россия). Реакционная смесь содержит 5 мкл смеси дезоксирибонуклеозидтрифосфатов (0,5 mM дАТФ, дЦТФ, дГТФ, 0,1 mM дТТФ) (Биосан, Россия), 5 мкл 10× буфера для ДНК-полимеразы I (Thermo, США), 1 мкл Flu-12-дУТФ или TAMRA-5-дУТФ (1 нМ/мкл) (Биосан, Россия), 5 мкл бычьего сывороточного альбумина (0,5 мг/мл), 5 мкл ДНК-азы I (0,02 ед./мл) (Thermo, США), 1 мкл ДНК-полимеразы I (10 ед./мкл) (Thermo, США), 1 мкг ДНК-библиотеки. Объем смеси доводят до 50 мкл деионизированной водой. Реакцию проводят при 15°С в течение 2 часов, и останавливают добавлением 1 мкл 0,5М ЭДТА (pH 8,0).
Флуоресцентная in situ гибридизация. Препараты проводят через три смены раствора 2xSSC (0,3 M NaCl, 0,03 M цитрата натрия) по 5 минут при 37°С, после чего проводят инкубацию препарата в 0,01 % растворе пепсина (Sigma, США) в течение 10 минут при 37°С. Через 10 минут проводят постфиксацию препарата в 0,5% растворе параформальдегида (Sigma, США) при комнатной температуре в вытяжном шкафу также в течение 10 минут. Далее отмывают препарат от постфиксатора в фосфатно-солевом буфере (1xPBS). Следующим шагом является обезвоживание препарата по 5 минут в трёх растворах этанола (70 %, 80 %, 96 %) с увеличением концентрации. После инкубирования препараты подсушивают и на них наносится по 10 мкл ДНК-зондов в гибридизационной смеси, специфичных к определенным последовательностям. Препараты накрывают покровным стеклом и заклеивают по краям. Гибридизацию (75°С, 5 минут) и инкубацию (37°С, 24 часа) проводят во влажной камере с подогревом (Termobrite, Германия). После окончания инкубации проводится отмывка препаратов в растворе WT1 (0,4xSSC, 0,3% NP-40) 2 мин при 73°С и WT2 (2xSSC, 0,1% NP-40) 5 мин при 37°С. После отмывки препараты не высушивают и на них сразу наносят заключающий раствор Vectashield с красителем DAPI (Vectorlabs, США). После этого, препараты накрывают покровным стеклом, лишнюю жидкость убирают при помощи фильтровальной бумаги, стекло заклеивают по краям прозрачным лаком. Визуализацию связавшихся ДНК-зондов проводят при помощи флуоресцентного микроскопа.
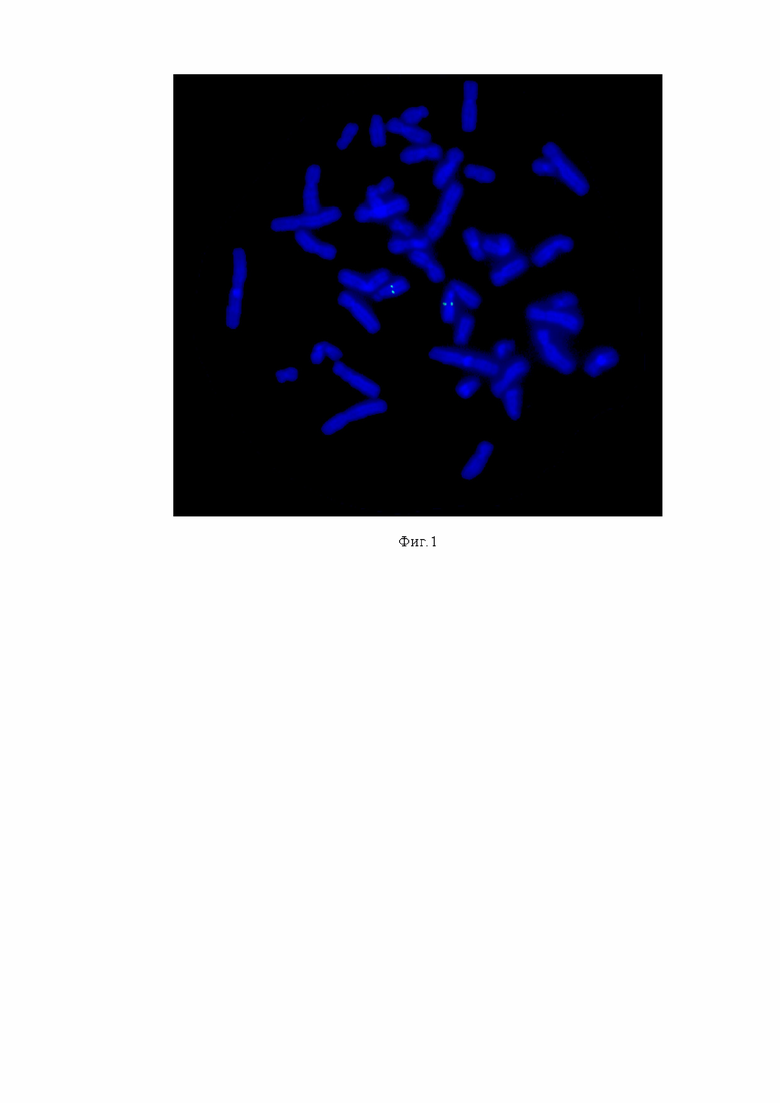
Эффективность разработанной технологии получения ДНК-зондов была подтверждена посредством разработки и получения с ее использованием ДНК-зонда на регион гена RB1 (13q14.2) (фиг.1).
Источники информации:
1. Huber D., Kaigala G.V. Rapid micro fluorescence in situ hybridization in tissue sections // Biomicrofluidics. 2018. Vol. 12. № 4. P. e042212.
2. Carter N.P., Ferguson-Smith M.A., Perryman M.T. et al. Reverse chromosome painting: a method for the rapid analysis of aberrant chromosomes in clinical cytogenetics // J. Med. Genet. 1992. Vol. 29. № 5. P. 299-307.
3. Weimer J., Kiechle M., Arnold N. FISH-microdissection (FISH-MD) analysis of complex chromosome rearrangements // Cytogenet. Cell Genet. 2000. Vol. 88. №№ 1-2. P. 114-118.
4. Cherif D., Bernard O., Berger R. Detection of single-copy genes by nonisotopic in situ hybridization on human chromosomes // Hum. Genet. 1989. Vol. 81. № 4. P. 358-362.
5. Meyer M.F., Gerresheim F., Pfeiffer A. et al. Association of polycystic ovary syndrome with an interstitial deletion of the long arm of chromosome 11 // Exp. Clin. Endocrinol. Diabetes. 2000. Vol. 108. № 8. P. 519-523.
6. Duffy M.J., Harbeck N., Nap M. et al. Clinical use of biomarkers in breast cancer: Updated guidelines from the European Group on Tumor Markers (EGTM) // Eur. J. Cancer. 2017. Vol. 75. P. 284-298.
7. Luo H., Xu X., Ye M. et al. The prognostic value of HER2 in ovarian cancer: A meta-analysis of observational studies // PLoS One. 2018. Vol. 13. № 1. P. e0191972.
8. Han C., Hsu J., Yao C. et al. HER2 gene amplification in primary mucinous ovarian cancer: a potential therapeutic target // Histopathology. 2010. Vol. 57. № 5. P. 763-774.
9. Frickmann H., Zautner A.E., Moter A. et al. Fluorescence in situ hybridization (FISH) in the microbiological diagnostic routine laboratory: a review // Crit. Rev. Microbiol. 2017. Vol. 43. №3. P. 263-293.
10. Summersgil B., Clarc J., Shipley J. Fluorescence and chromogenic in situ hybridization to detect genetic aberrations in formalin-fixed paraffin embedded material, including tissue microarrays // Nat Protoc. 2008. Vol. 3. P. 220-234.
11. Rosa F.E., Santos R.M., Rogatto S.R. et al. Chromogenic in situ hybridization compared with other approaches to evaluate HER2/Neu status in breast carcinomas // Braz. J. Med. Biol. Res. 2013. Vol. 46. P. 207-216.
12. Furrer D., Sanschagrin F., Jacob S. et al. Advantages and disadvantages of technologies for HER2 testing in breast cancer specimens: table 1 // Am. J Clin Pathol. 2015. Vol. 144. P. 686-703.
13. Saez A., Andreu F.J., Bare M.L. et al. HER-2 gene amplification by chromogenic in situ hybridisation (CISH) compared with fluorescence in situ hybridisation (FISH) in breast cancer - a study of two hundred cases // Breast. 2006. Vol. 15. P. 519-527.
14. Патент EP0444115 «In situ suppression hybridization and uses therefor», 10.07.2017г., авторы: Ward D.C., Lichter P., Cremer T. и Manuelidis L.
15. Патент US6605436 “Method and kit for detecting intrachromosome imbalance in interphase nuclei and their applications», 21.05.2003г., авторы: Truong K., Guilly Marie-No L., Malfoy B., Vielh Ph., Bourgeois C., Dutrillaux B.
16. Патент WO1993025713 “Compositions and methods for detecting gene rearrangements and translocations», 17.06.1993г., авторы Rowley J.D. и Diaz M.O.
17. Патент WO2007053245 «Methods and composition to generate unique sequence DNA probes, iabeling of DNA probes and the use of these probes», 20.09.2006г., авторы: Connelly M. C., Foulk B., Kagan M.T., Swennenhuis J.F., Terstappen L.W., Tibbe A.G. и Verrant J.A.
18. Патент EP3314024 «Method and system for multiplex profiling of chromosomes in biological samples using target-specific DNA probes», 23.06.2016г., автор Vallabhaneni Ramesh
19. Патент WO2018019610 «DNA probes for in situ hybridization on chromosomes», 14.07.2017, авторы: Weglöhner Wolfgang, Schindler Sabrina.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="2023-02МГ.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-11-17">
<ApplicantFileReference>2023-02</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное научное учреждение Томский национальный исследовательский
медицинский центр Российской академии наук</ApplicantName>
<ApplicantNameLatin>Federalnoe gosudarstvennoe byudjetnoe nauchnoe
uchrejdenie Tomskii nacionalnii issledovatelskii medicinskii centr
Rossiiskoi akademii nauk</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ получения панели ДНК-зондов
для определения хромосомных транслокаций, делеций и
амплификаций</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapien</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggtgattctactgtttctgtgact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapien</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcacagaataatgccgagtcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapien</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggtgattctactgtttctgtgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapien</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggactcggcattattctgtgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Настоящее изобретение относится к области биотехнологии, а именно к молекулярной цитогенетике. Описан способ получения панели ДНК-зондов для определения хромосомных транслокаций, делеций и амплификаций, включающий разработку панели ДНК-зондов с использованием длинных до 15 т.п.н. последовательностей ПЦР-амплификатов с последующим формированием ДНК-библиотек на диагностируемые участки генома человека и контрольные локусы, причем указанный способ включает стадии: выбор целевых регионов генома; подбор и синтез олигонуклеотидных праймеров на целевые регионы генома; наработка ДНК-продуктов с помощью ПЦР длинных фрагментов; получение библиотеки фрагментов ДНК с помощью метода ферментативной фрагментации; лигирование синтезированных адаптерных последовательностей и амплификация библиотеки с помощью синтезированных целевых праймерных последовательностей; введение в геномную библиотеку флуоресцентной метки; проверка специфичности и интенсивности ДНК-зонда в реакции FISH. Заявленный способ может быть использован для определения хромосомных транслокаций, делеций и амплификаций на основе флуоресцентной гибридизации ДНК-зонда с регионом генома протяженностью более 30 т.п.н., в различных биологических образцах клеток человека. 1 ил.
Способ получения панели ДНК-зондов для определения хромосомных транслокаций, делеций и амплификаций, включающий разработку панели ДНК-зондов с использованием длинных до 15 т.п.н. последовательностей ПЦР-амплификатов с последующим формированием ДНК-библиотек на диагностируемые участки генома человека и контрольные локусы, причем указанный способ включает стадии: выбор целевых регионов генома; подбор и синтез олигонуклеотидных праймеров на целевые регионы генома; наработка ДНК-продуктов с помощью ПЦР длинных фрагментов; получение библиотеки фрагментов ДНК с помощью метода ферментативной фрагментации; лигирование синтезированных адаптерных последовательностей SEQ ID № 1, 2 и амплификация библиотеки с помощью синтезированных целевых праймерных последовательностей SEQ ID № 3, 4; введение в геномную библиотеку флуоресцентной метки; проверка специфичности и интенсивности ДНК-зонда в реакции FISH.
| СПОСОБ ИДЕНТИФИКАЦИИ ХРОМОСОМНЫХ ТРАНСЛОКАЦИЙ, ПРИВОДЯЩИХ К РАЗВИТИЮ ЗЛОКАЧЕСТВЕННЫХ ЗАБОЛЕВАНИЙ КРОВИ (ЛЕЙКОЗОВ), С ИСПОЛЬЗОВАНИЕМ ОЛИГОНУКЛЕОТИДНОГО БИОЛОГИЧЕСКОГО МИКРОЧИПА (БИОЧИПА) | 2004 |
|
RU2286798C2 |
| CN 112391471 A, 23.02.2021 | |||
| CN 111647648 A, 11.09.2020 | |||
| CN 108753967 A, 06.11.2018 | |||
| СПОСОБ И УСТРОЙСТВО ДЛЯ ОПРЕДЕЛЕНИЯ СТРОЕНИЯ ХРОМОСОМ | 2005 |
|
RU2471871C2 |
| WO 2018019610 A1, 01.02.2018. | |||
Авторы
Даты
2024-10-07—Публикация
2023-11-17—Подача