Изобретение относится к ветеринарной микробиологии, в частности к идентификации видовой принадлежности тканей рыб с помощью полимеразной цепной реакции.
Известен способ видовой идентификации рыб семейства тресковых методом пцр в режиме реального времени, в котором выделяют ДНК из проб тканей рыб или из образца пищевого продукта (продовольственного сырья). Проводят анализ методом полимерной цепной реакции в режиме реального времени для выявления специфических последовательностей ДНК рыб с помощью синтетических олигонуклеотидных праймеров и зондов, установленных для каждого вида рыб. Готовят смесь для проведения Г'ПДР с гибридизационно-флуоресцентной детекцией продуктов ПЦР. Результаты интерпретируют на основании наличия/отсутствия пересечения кривой флуоресценции с установленной пороговой линией Threshold и значением порогового цикла, рассчитываемого программным обеспечением амилификатора. ДНК тресковых рыб (кроме пикши, Melanogrammus aeglefinus) считают обнаруженной, если для соответствующей пробирки/реактора значение порогового цикла не превышает 30. ДНК пикши (Melanogrammus aeglefinus) считают обнаруженной, если одновременно для пробирки/реактора с тест-системой для определения пикши (Melanogrammus aeglefinus) значение порогового цикла не превышает 30. Для определения путассу Micromesisteus poutassou) и сайды (Pollachius virens) флуоресцентный сигнал не регистрируется или значения порогового цикла превышают 30 в пробирках/реакторах с тест-системами. Предлагаемый способ позволяет провести идентификацию всех видов тресковых рыб в рамках одного анализа и получить результат анализа быстрее по сравнению с существующими методами (патент РФ №2748058, кл. C12Q 1/68, 2021 г.).
Наиболее близким по технической сущности является способ идентификации видовой принадлежности тканей животного в продовольственном сырье, кормах, включающий выделение ДНК из ткани животного сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, положительного контрольного образца в виде смеси, содержащую фрагменты геномов животного и бактериофага Т4 с нуклеотидной последовательностью:

и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный (патент РФ №2726433, кл. C12Q 1/68, 2020 г.).
Недостатком известного технического решения является отсутствие возможности идентифицировать ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в рыбных продуктах, в мясокостной рыбной муке и кормах.
Техническим результатом является расширение функциональных возможностей и повышение точности идентификации видовой принадлежности тканей рыб.
Технический результат достигается тем, что в способе идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в пробе, включающем выделение ДНК из пробы, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, при этом внутренний контрольный образец представлен в виде смеси, содержащей фрагменты генома ткани дальневосточной сардины, или иваси (Sardinops melanostictus), и бактериофага Т4 в соотношении 1:1, праймеры и зонды имеют следующие нуклеотидные последовательности:
T4F TACATATAAATCACGCAAAGC - прямой праймер
T4R TAGTATGGCTAATC iTATTGG - обратный праймер
Т4Р CY5 ACATTGGCACTGACCGAGTTC – зонд
Sm-F GGTAGAGCAATTCCCTGCGT – праймер
Sm-R CCGAGTCCCCTCATGGTTTT - праймер
Sm-Z R6G CATTGGACTATTAGGACCCGCA - зонд,
и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции – отрицательный.
В способе проба представляет собой пробу рыбны продуктов, мясо-косной рыбной муки, кормов.
Новизна заявляемого способа состоит в идентификации видовой принадлежности ткани рыб, с помощью полимеразной цепной реакции (ПЦР) с флуоресцентной детекцией в режиме реального времени, что в свою очередь позволяет с высокой точностью определить наличие их ингредиентов в сырых рыбных продуктах (частях туши, икре, полуфабрикатах и т.д.), в рыбных продуктах, подвергшихся кулинарной обработке, а также в мясокостной рыбной муке и кормах.
Признаки, отличающие заявляемое техническое решение от прототипа, направлены на достижение технического результата и не выявлены при изучении данной и смежной областей науки и техники и, следовательно, соответствуют критерию «изобретательский уровень».
Заявляемый способ рекомендовано использовать в специализированных ветеринарных, санитарно-эпидемиологических и сельскохозяйственных предприятиях, что соответствует критерию «промышленная применимость».
Способ идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в рыбных продуктах, в мясокостной рыбной муке в кормах, осуществляют следующим образом.
Для исследования наличия ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в сырых рыбных продуктах (частях туши, икре, полуфабрикатах и т.д.), в рыбных продуктах, подвергшихся кулинарной обработке, а также в мясокостной рыбной муке и кормах проводят полимеразную цепную реакцию с флуоресцентной детекцией с применением термоциклера типа Rotor-Gene Q при соответствующих температурно-временных режимах амплификации и измеряют накопление флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей: JOE/Yellow для специфического сигнала для ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), и Cy5/Red - для внутреннего контрольного образца. Интерпретацию результатов проводят на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции - отрицательный.
Для повышения точности идентификации ткани дальневосточной сардины, или иваси (Sardinops melanostictus), для внутреннего контрольного образца используют бактериофага Т4 с концентрацией 5×103 фаговых частиц па 1 мкл, если концентрация фаговых частиц отклоняется в большую или меньшую сторону, то наблюдаются повторное™ сомнительных образцов. Для положительного контрольного образца используют смесь содержащую фрагменты геномов ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), и бактериофага Т4 взятых в соотношении 1:1 со следующими нуклеотидными последовательностями:

Использование бактериофага Т4 со специфическими к нему праймерами и зондом обусловлено тем, что это позволяет контролировать корректное прохождение реакции в каждой пробирки, а также контролируется этап выделения ДНК из образцов. Кроме того, использование бактериофага Т4 повышает чувствительность и упрощает процесс идентификации ткани рыб в продуктах, а также улучшает качество процесса идентификации.
При конструировании праймеров и зонда основными требованиями были: степень гомологии (комплементарность) с выбранным участком гена; отсутствие самокоплементарных участков внутри олигонуклеотидов и комплементарности друг другу, чтобы не допускать возникновения устойчивых вторичных структур (димеров); близость значений температуры отжига праймеров.
Конструирование специфических праймеров и зонда осуществляли с помощью компьютерных программ на основании анализа нуклеотидных последовательностей референтных штаммов и изолятов, опубликованных на ресурсе GenBank и подбора условий для проведения ПЦР в реальном времени с применением разработанных праймеров и зонда, несущего флуорофор и тушитель, и комплементарного части амплифицируемого со специфическими праймерами фрагмента.
Праймеры, специфичные дальневосточной сардины, или иваси (Sardinops melanostictus), были отобраны на основе митохондриалыюй последовательности ДНК генома дальневосточной сардины, или иваси (Sardinops melanostictus), ген цитохром b (Sardinops melanostictus mitochondrial DNA, PRO-tRNA, D-Loop, 12S rRNA, partial and complete sequence, isolate: 2012-36, GenBank: AB361466.1 участок между 807 и 1042). Праймеры были спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность. Для детекции продуктов амплификации были подобраны олигонуклеотидные флуоресцентно-меченные зонды Sm-Z (комплементарный участку нуклеотид-ной последовательности, ограниченной позициями отжига праймеров Sm-F и Sm-R). Зонд был помечен красителем HEX. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР. Ни одна из выбранных последовательностей не обнаружена в геноме любых видов растений и животных, которые потенциально встречаются вблизи тех, которые определены в кормах и пищевых продуктах.
Ни одна из выбранных последовательностей:
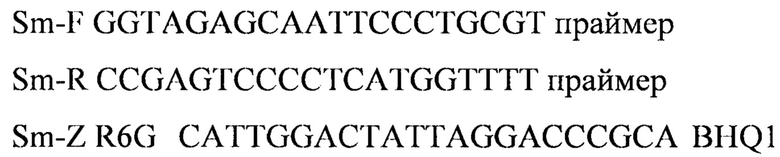 зонд, не обнаружена в геноме любых видов растений и животных, которые потенциально встречаются вблизи тех, которые определены в кормах и пищевых продуктах.
зонд, не обнаружена в геноме любых видов растений и животных, которые потенциально встречаются вблизи тех, которые определены в кормах и пищевых продуктах.
В качестве внутреннего контроля использовался бактериофаг Т4, имеющий геномную ДНК порядка 169-170 тысяч пар нуклеотидов (Enterobacteria phage Т4Т, complete genome GenBank: HM137666.1). В результате анализа был выбран участок между 400 и 500 нуклеотидами, содержащий уникальные иук-леотидные последовательности, рассчитаны первичные структуры олигонуклеотидных праймеров, фланкирующих выбранный участок генома. Праймеры были спроектированы с использованием Primer Express Software v3.0 (Applied Biosystems) и исследованы с использованием BLAST, чтобы подтвердить их специфичность.
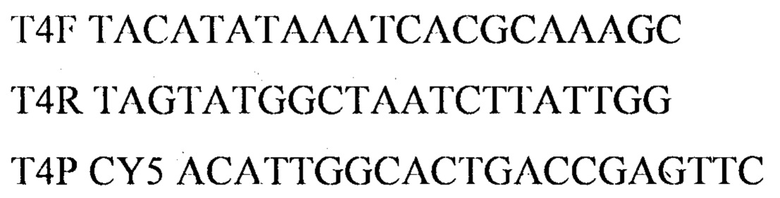
Для детекции продуктов амплификации подобран олигонуклеотидный флуоресцентно-меченный зонд Т4Р, комплементарный участку нуклеотидной последовательности, ограниченной позициями отжига праймеров T4F и T4R. Зонд был помечен красителем Су5. Используя программу "Oligo 6.0" описаны основные свойства рассчитанных олигонуклеотидов, определившие возможность их использования в ПЦР.
Пример конкретного осуществления способа идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в рыбных продуктах, в мясокостной рыбной муке и кормах
Для подтверждения эффективности способа были использованы сырые рыбные продукты (части туши, икра, полуфабрикаты и т.д.), рыбные продукты, подвергшихся кулинарной обработке, а также мясокостная рыбная мука и корма.
От пробы плотной консистенции отбирают на исследование общую пробу весом 10-50 г. Гранулированную или консервированную продукцию перед исследованием (10-20 г) растирают в ступке до гомогенного состояния.
Лабораторные пробы (20 - 40 мг) отбирают на исследование в одноразовые микропробирки вместимостью 1,5 мл в двух повторах. Отобранные лабораторные пробы направляют на выделения ДНК.
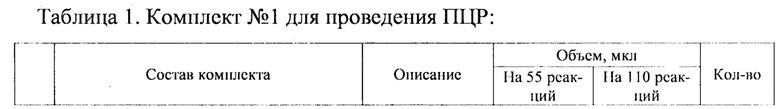
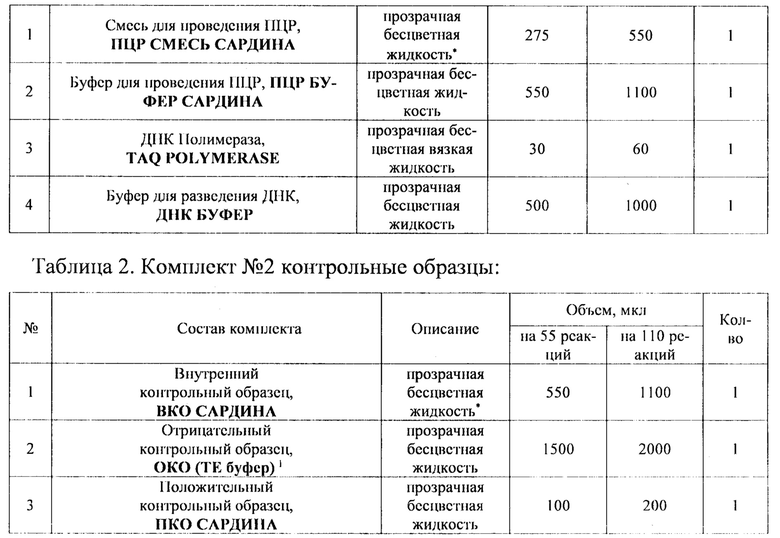
Исследование проводят с помощью набора реагентов «ПЦР-САРДИНА-ФАКТОР». Под словом «САРДИНА» авторы подразумевают набор реагентов используемых для выявления ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus). Набор состоит из комплекта реагентов для проведения мультиплексной ПЦР (комплект №1) и комплекта контрольных образцов (комплект №2). Набор выпускается в двух вариантах:
1) Для анализа 55 образцов (включая контрольные образцы)
2) Для анализа 110 образцов (включая контрольные образцы). Наборы используют в соответствии с инструкцией по применению
набора реагентов «ПЦР-САРДИНА-ФАКТОР» для определения ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), методом полимеразиой цепной реакции (ПЦР) с флуоресцентной детекцией в РВ ТУ 21.10.60-257-51062356-2021, для диагностики in vitro, http://www.vetfaktor.ru/. Состав набора приведен в Таблицах 1 и 2.

Исследования состоит из трех этапов:
• экстракция нуклеиновая кислота (НК);
• проведение реакции ПЦР РВ;
• учет результатов анализа.
Для экстракции (выделение) НК из исследуемых проб отбирают необходимое количество одноразовых пробирок объемом 1,5 мл, включая отрицательный контроль выделения. Во все пробирки с исследуемыми образцами, включая пробирку для отрицательного контрольного образца (ОКО), вносят по 10 мкл внутреннего контрольного образца (ВКО) для ткани дальневосточной сардины, или иваси, в качестве которого, используют бактериофаг Т4 с концентрацией 5×103 фаговых частиц на 1 мкл. Следующий этап, это подготовка образцов к проведению ПЦР. Общий объем реакционной смеси - 25 мкл, объем ДНК-пробы - 10 мкл.
Успешное прохождение реакции контролируют использованием положительного контрольного образца (ПКО) САРДИНА, ВКО САРДИНА и ДНК буфера. В качестве ПКО используют смесь содержащую фрагменты геномов ткани дальневосточной сардины, или иваси, и бактериофага Т4 взятых в соотношении 1:1.
В отдельной пробирке смешивают компоненты набора из расчета па каждую реакцию:

Перемешивают смесь на вортексе и сбрасывают капли кратковременным центрифугированием.
Отбирают необходимое количество пробирок для амплификации ДНК исследуемых и контрольных проб. Вносят по 15 мкл приготовленной реакционной смеси.
Помещают подготовленные для проведения ПЦР пробирки в ячейки ам-плификатора и используют программное обеспечение прибора. Далее проводят ПЦР РВ с флуоресцентной детекцией.
Параметры температурно-временного режима амплификации на приборе «Rotor-Gene Q» представлены в таблице 3.

Интерпретация результатов анализа. Полученные данные - кривые накопления флуоресцентного сигнала анализируются с помощью программного обеспечения используемого прибора для проведения Г1ЦР в соответствии с инструкцией производителя к прибору.
Учет результатов ПЦР РВ проводится по наличию или отсутствию пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией (что соответствует наличию или отсутствию значения порогового цикла «Ct» для исследуемого образца).
Результат считается достоверным в случае корректного прохождения положительных и отрицательных контролей амплификации и экстракции ДНК в соответствии с таблицей 4.
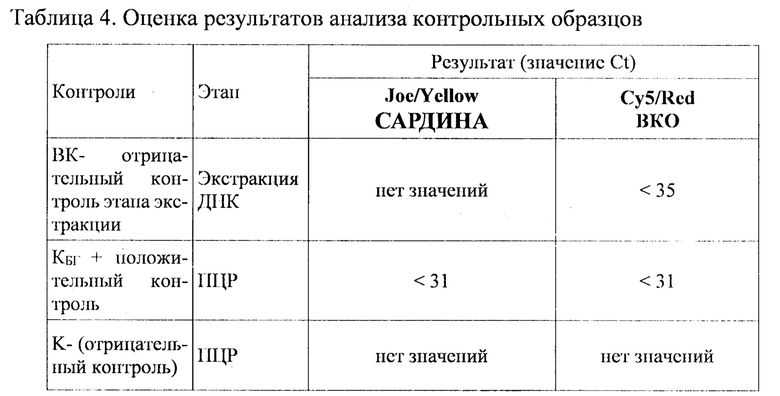
Появление любого значения Ct в таблице 4 результатов для отрицательного контроля этапа экстракции ВК- на канале JOE/Yellow и для отрицательного контроля этапа ПЦР К- на любом из каналов свидетельствует о наличии контаминации реактивов или образцов. В этом случае результаты анализа для всех проб считаются недействительными. Требуется повторить анализ всех проб, а также предпринять меры по выявлению и ликвидации источника контаминации.
Образцы, для которых значение Ct по каналу Cy5/Red отсутствует или превышает 35 цикл (и при этом не получен положительный результат на канале JOE/Yellow) требуют повторного проведения исследования с этапа экстракции ДНК. Задержка в значениях пороговых циклов для исследуемых образцов указывает на присутствие ингибиторов в пробе(ах) или на ошибки при экстракции ДНК или при постановке реакции ПЦР РВ.
В образце обнаружена ДНК ткани дальневосточной сардины или иваси, если наблюдается экспоненциальный рост сигнала на канале JOE/Yellow, при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4).
Если для исследуемого образца по каналам JOE/Yellow значение Ct определяется позднее 37 цикла при корректном прохождении положительных и отрицательных контролей, образец исследуется повторно с этапа экстракция ДНК. Если при повторной постановке Ct более 37 результат считается отрицательным.
Образец считается отрицательным (ДНК ткани дальневосточной сардины, или иваси, не обнаружена), если не определяется значение Ct (не наблюдается рост специфического сигнала) на канале JOE/Yellow, при этом значения Ct контрольных образцов находятся в пределах нормы (Табл. 4), а значение Ct по каналу Cy5/Red - менее 35.
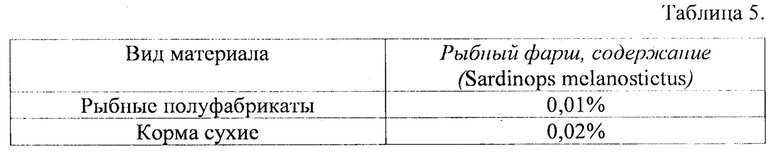
Для исследуемых образцов (сухой корм и рыбные полуфабрикаты)
предел точности содержания ткани дальневосточной сардины, или иваси, представлен в таблице 5.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName=" Способ
идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops
melanostictus) в пробе.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2023-06-23">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2023-06-23</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>1</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1</ApplicationNumberText>
<FilingDate>2024-03-19</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru"> Федеральное государственное
бюджетное образовательное учреждение высшего образования «Кубанский
государственный аграрный университет имени И.Т.
Трубилина»</ApplicantName><ApplicantNameLatin> Federalnoe
gosudarstvennoe byudzhetnoe obrazovatelnoe uchrezhdenie vysshego
obrazovaniya "Kubanskij gosudarstvennyj agrarnyj universitet imeni
I.T. Trubilina";</ApplicantNameLatin>
<InventionTitle languageCode="ru">=" Способ идентификации ДНК ткани
ткани дальневосточной сардины, или иваси (Sardinops melanostictus) в
пробе </InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>807…1042</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence> ggtagagcaattccctgcgt </INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>807…1042</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence> ccgagtcccctcatggtttt </INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>807…1042</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence> cattggactattaggacccgca </INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>400…500 </INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence> tacatataaatcacgcaaagc </INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>400…500</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence> tagtatggctaatcttattgg </INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>400…500</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence> аcattggcactgaccgagttc </INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Тест-система для идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в рыбных продуктах, в мясокостной рыбной муке и кормах с помощью полимеразной цепной реакции в режиме реального времени | 2023 |
|
RU2823069C1 |
| Способ идентификации ДНК ткани сайры тихоокеанской (Cololabis saira) в пробе | 2023 |
|
RU2838063C2 |
| Тест-система идентификации ДНК ткани сайры тихоокеанской (Cololabis saira) в рыбных продуктах, в мясокостной рыбной муке и кормах с помощью полимеразной цепной реакции в режиме реального времени | 2023 |
|
RU2822748C1 |
| Способ идентификации ДНК ткани японской скумбрии (Scomber japonicus) в рыбных продуктах, в мясокостной рыбной муке и кормах с помощью полимеразной цепной реакции в режиме реального времени | 2023 |
|
RU2814552C1 |
| Тест-система идентификации ДНК ткани японской скумбрии (Scomber japonicus) в рыбных продуктах, в мясокостной рыбной муке и кормах с помощью полимеразной цепной реакции в режиме реального времени | 2023 |
|
RU2816210C1 |
| Способ идентификации видовой принадлежности тканей крыс и мышей в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2742952C1 |
| Способ идентификации ДНК ткани ежа обыкновенного (Erinaceus europaeus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726433C1 |
| Способ идентификации ДНК ткани медведя (Ursus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726427C1 |
| Способ идентификации ДНК ткани кошки домашней (Felis silvestris catus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2728662C1 |
| Способ определения ДНК ткани дятла (Picidae) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2714287C1 |
Изобретение относится к ветеринарной микробиологии. Описан способ идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в пробе, включающий выделение ДНК из пробы сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, при этом внутренний контрольный образец представлен в виде смеси, содержащей фрагменты генома ткани дальневосточной сардины или иваси (Sardinops melanostictus) и бактериофага Т4 в соотношении 1:1, праймеры и зонды имеют следующие нуклеотидные последовательности: T4F TACATATAAATCACGCAAAGC – праймер; T4R TAGTATGGCTAATCTTATTGG – праймер; T4P CY5 ACATTGGCACTGACCGAGTTC – зонд; Sm-F GGTAGAGCAATTCCCTGCGT – праймер; Sm-R CCGAGTCCCCTCATGGTTTT – праймер; Sm-Z R6G CATTGGACTATTAGGACCCGCA – зонд, и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции – отрицательный. Проба представляет собой пробу рыбных продуктов, мясокостной рыбной муки, кормов. Изобретение служит для расширения функциональных возможностей и повышения точности идентификации видовой принадлежности тканей рыб. 1 з.п. ф-лы, 2 табл.
1. Способ идентификации ДНК ткани дальневосточной сардины, или иваси (Sardinops melanostictus), в пробе, включающий выделение ДНК из ткани животного сорбционным методом, постановку полимеразной цепной реакции с флуоресцентной детекцией с проведением 45 циклов амплификации в реальном времени с использованием специфичных для участка генома ДНК животного олигонуклеотидных праймеров, зондов, флуоресцентных красителей: для специфического сигнала для животного - JOE/Yellow и Cy5/Red - для внутреннего контрольного образца в виде суспензии бактериофага Т4 с концентрацией 5×103 фаговых частиц на 1 мкл, при этом внутренний контрольный образец представлен в виде смеси, содержащей фрагменты генома ткани дальневосточной сардины и бактериофага Т4 соотношении 1:1, праймеры и зонды имеют следующие нуклеотидные последовательности:
T4F TACATATAAATCACGCAAAGC – праймер
T4R TAGTATGGCTAATCTTATTGG – праймер
T4P CY5 ACATTGGCACTGACCGAGTTC – зонд
Sm-F GGTAGAGCAATTCCCTGCGT – праймер
Sm-R CCGAGTCCCCTCATGGTTTT – праймер
Sm-Z R6G CATTGGACTATTAGGACCCGCA – зонд,
и измерение накопления флуоресцентных сигналов по каналам соответствующих флуоресцентных красителей, проведение интерпретации результатов па основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, если кривые накопления флуоресцентного сигнала выходят до 35 цикла, то результат реакции считается положительным, а если кривые не пересекают пороговую линию или пересекают ее после 35 цикла, то результат реакции – отрицательный.
2. Способ по п.1, где проба представляет собой пробу рыбных продуктов, мясокостной рыбной муки, кормов.
| Способ идентификации ДНК ткани собаки домашней (Canis lupus familiaris) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2728612C1 |
| Способ идентификации ДНК ткани медведя (Ursus) в сухих кормах и мясных полуфабрикатах | 2019 |
|
RU2726427C1 |
| KR 1020110106172 A, 28.09.2011. | |||
Авторы
Даты
2025-02-21—Публикация
2023-06-23—Подача