Изобретение относится к области медицины, а именно к молекулярно-генетической диагностике и может быть использовано при определении риска развития привычного невынашивания беременности (ПНБ). Для прогнозирования риска невынашиваемости беременности в фиксированных формалином и помещённых в парафин тканей миометрия определяют уровень экспрессии генов микро- рибонуклеиновых кислот (микроРНК) miR-145 и miR-34a.
Одной из актуальных проблем современной клинической лабораторной генетики и биомедицинских исследований является поиск тканеспецифических и патогномоничных генетических маркеров, позволяющих проводить определение риска привычной невынашиваемости беременности.
Привычное невынашивание беременности является важной медико-социальной проблемой, затрагивающей до 1% женщин репродуктивного возраста. Несмотря на многофакторную природу данной проблемы, значительная часть случаев невынашивания остается без четко установленной причин, что делает актуальным интерес к изучению молекулярных механизмов, лежащих в основе нарушений имплантации эмбриона и ранних репродуктивных потерь.
В этом контексте особенно перспективным является исследование экспрессии микро-РНК — коротких некодирующих РНК, играющих одну из ключевых ролей в посттранскрипционной регуляции экспрессии генов.
МикроРНК играют важную роль во многих биологических событиях, таких как рост, деление, дифференциация, развитие, апоптоз и болезни. На сегодняшний день в клетках человека обнаружено более 1200 микроРНК (http://www.mirbase.org/). Эти микроРНК участвуют в регуляции более 60% генов человека. miRNA являются важными биомаркерами для классификации опухолей, диагностики, предсказания заболеваний и оценки прогноза. Обнаружение и количественное определение miRNA являются важными инструментами для обнаружения некоторых целевых генов и путей, изучения механизма заболевания, оценки безопасности и эффективности лекарств, диагностики и прогнозирования заболеваний (Баулина Н.М. и др., МикроРНК как биомаркеры прогрессирования рассеянного склероза, Неврология, нейропсихиатрия, психосоматика, 2023г., Т.15, с.4–7; Metcalf G. A., et al., MicroRNAs: circulating biomarkers for the early detection of imperceptible cancers via biosensor and machine-learning advances, Oncogene, 2024,43, р. 2135–2142).
Одним из наиболее часто применяемых методов оценки экспрессии микроРНК является способ секвенирования, включающий подготовку РНК-библиотек с присоединением специальных адаптеров, контроль длины фрагментов нуклеиновых кислот и определением нуклеотидной последовательности на высокопроизводительных секвенаторах Illumina (Loudig O., et al., Retrospective MicroRNA Sequencing: Complementary DNA Library Preparation Protocol Using Formalin-fixed Paraffin-embedded RNA, Journal of Visualized Experiments: JoVE, 2018, 135, р. 57471). Недостатками этого метода является необходимость использования дорогостоящего оборудования для секвенирования, дорогостоящих реагентов для подготовки библиотек для секвенирования и специального программного обеспечения и биоинформатических подходов для анализа полученных результатов, а также задействования высококвалифицированных специалистов для постановки методики секвенирования и анализа данных.
Другим методом оценки экспрессии генов микроРНК является использование коммерческих наборов для выделения РНК на специализированных силика-колонках с последующей ПЦР с использованием готовых наборов реагентов с использованием флуоресцентных зондов (Князева М.С. и др., Особенности профиля экспрессии микроРНК ткани анапластического рака щитовидной железы, Вопросы онкологии, 2021г., Т.67(1), с.70-76). Недостатками этого метода является использование дорогостоящих наборов реагентов для выделения микроРНК на силика-колонках, и использование дорогостоящих наборов с флуоресцентными метками.
Известно, что целевая РНК может служить шаблоном для ОТ-ПЦР (обратная транскрипция и последующая полимеразная цепная реакция). Основная проблема для прямой ОТ-ПЦР малых РНК связана с их размером - всего 20-27 нт, они почти такого же размера, как обычный праймер ПЦР, в то время как для экспоненциальной амплификации требуются два праймера. По этой причине первые анализы ОТ-ПЦР были разработаны для предшественников miRNA, которые более чем в два раза длиннее зрелых miRNA (Schmittgen et al. 2004; Jiang et al. 2005). Однако эта методология может не привести к точному представлению биологически релевантного профиля, поскольку уровни предшественников не всегда коррелируют с уровнями зрелых miRNA из-за быстрой обработки предшественников miRNA и долговечности активных miRNA, связанных с комплексом RISC (Bartel 2004; Jiang et al. 2005; Lao et al. 2007).
Один из подходов к амплификации miRNA основан на наличии только короткого перекрытия между праймером и последовательностями miRNA (Chen et al. 2005; Raymond et al. 2005; Raymond 2007; Sharbati-Tehrani et al. 2008; Sharbati-Tehrani & Einspanier 2008). К сожалению, обычные короткие праймеры ОТ-ПЦР не могут стабильно гибридизоваться при температурах, необходимых для этапа удлинения ПЦР. Более того, последовательности miRNA значительно различаются по содержанию GC, как среди разных видов miRNA и между 5' и 3' половинами отдельных miRNA (Hammond 2006). В результате праймеры, связывающиеся с различными miRNA, не будут одинаково эффективны в данных условиях, что ставит под угрозу как специфичность последовательности, так и эффективность амплификации ПЦР (Esquela-Kerscher & Slack 2004; Winkler et al. 2006). Дополнительной проблемой является сложность различения незначительных различий в последовательности и/или длине между различными изоформами miRNA (Sioud & Rosok 2004; Hammond 2006c). Для решения этих проблем используют удлиненные целевые праймеры, образующие короткие, но стабильные дуплексы с 3' концами miRNA. Одна группа исследователей использовала праймеры ОТ типа «стебель-и-петля», имеющие только 6 нт, комплементарных 3' концу целевых miRNAs, вместе с двумя другими праймерами ПЦР, где один праймер соответствовал 5′ концу целевой последовательности, а другой праймер соответствовал последовательности «стебель-и-петля», и зонды TaqMan (Chen et al. 2005). Вторая группа использовала комбинированные праймеры ОТ, которые включали 7-12 нт, комплементарных 3' концу целевых miRNAs, и дополнительную последовательность, кодирующую последовательность для второго праймера ПЦР, вместе с двумя другими праймерами ПЦР, где один праймер LNA-ДНК соответствовал 5' концу целевой последовательности, а другой праймер соответствовал дополнительной последовательности (Raymond et al. 2005; Raymond 2007). Третья группа использовала аналогичные комбинированные праймеры ОТ (но без модификаций LNA) вместе с тремя дополнительными праймерами ПЦР, где один праймер ПЦР, соответствующий 5'-концу целевой последовательности, был комбинированным праймером, а два других праймера соответствовали дополнительным последовательностям комбинированных праймеров (Sharbati-Tehrani et al. 2008; Sharbati-Tehrani & Einspanier 2008).
Быстро расширяющийся список различных методов обнаружения miRNA указывает на то, что ни одна из существующих технологий не является идеальной или не имеет явного преимущества перед конкурентами. Поскольку методы ОТ-ПЦР обладают очень хорошей чувствительностью, специфичностью последовательности и динамическим диапазоном, они часто используются в качестве метода выбора для профилирования экспрессии определенных miRNA, а также для проверки результатов, полученных другими распространенными методами, такими как микрочипы и нозерн-блот-анализы (Ahmed 2007).
В настоящее время известны способы диагностики, прогнозирования течения заболеваний, основанные на определении уровня экспрессии микроРНК в биологических материалах.
Известен способ анализа микроРНК в образцах биологического материала (RU2783509). Способ используют для количественного анализа специфических молекул микроРНК (миРНК) в образцах тотальной РНК, выделенной из материала биопсий. Для реакции обратной транскрипции (ОТ) используются оригинальный двухфланговый ОТ-праймер, при этом 5'-конец праймера связывает 8±2 оснований 5'-конца миРНК, а 3'-конец праймера связывает 6±2 оснований 3'-конца миРНК в направлении «навстречу» друг другу, полимеразная цепная реакция проводится с помощью двух оригинальных LNA модифицированных праймеров, причем структура обратного праймера исключает возможность его комплементарного взаимодействия с ОТ-праймером, детекция амплификации проводится в режиме реального времени по технологии TaqMan с помощью одного или двух ДНК-зондов, последовательность которых обеспечивает их отжиг на двух комплементарных цепях ампликона. Данный способ применим для анализа миРНК в биологическом материале, включая операционный материал, материал трепан- или тонкоигольной биопсии, соскобы эпителиев или материл жидкостной биопсии после выделения РНК любым доступным методом, обеспечивающим сохранность малых РНК молекул (<30 нуклеотидов).
В EP2699666B1 раскрыт способ обнаружения патологии системы органов у субъекта включает: a) измерение уровня по меньшей мере одной микроРНК, которой обогащена указанная система органов, в образце биологической жидкости, полученном у субъекта; b) измерение уровня нормирующей микроРНК в том же образце биологической жидкости, полученном у субъекта; c) расчет отношения уровней микроРНК, измеренных на этапах (а) и (b); d) сравнение отношения уровней микроРНК, рассчитанного на этапе (с), с соответствующим контрольным отношением и e) (i) идентификацию субъекта как имеющего патологию указанной системы органов в случае, когда отношение уровней микроРНК, рассчитанное на этапе (с), выше, чем соответствующее контрольное отношение, или (ii) идентификацию указанного субъекта как не имеющего патологии указанной системы органов в случае, когда отношение уровней микроРНК, рассчитанное на этапе (с), не выше, чем соответствующее контрольное отношение.
В настоящее время молекулярно-генетические методы диагностики используются для прогнозирования риска невынашивания беременности.
Известен способ (RU2566728) прогнозирования риска невынашивания первой половины беременности, состоящий в выделении ДНК из лейкоцитов периферической крови пациентки и проведении генотипирования, отличающийся тем, что при выявлении аллеля А полиморфизма rs-4680 гена катехол-О-метилтрансферазы СОМТ прогнозируют повышенный риск невынашивания беременности.
В RU 2454669 раскрыт способ прогнозирования риска невынашивания беременности у супружеских пар путем генотипирования на наличие полиморфизма гена SOD2, при котором носительство генотипа С/С или генотипа Т/С повышает риск невынашивания. Недостатком данного способа является необходимость обследования обоих супругов.
Способ прогнозирования привычного невынашивания беременности путем исследования иммуногенетического профиля лейкоцитов периферической крови в супружеской паре, отличающийся тем, что на основе показателей HLA-антигенов I, II классов определяют дискриминантную функцию F по формуле и при значении F > 0 прогнозируют привычное невынашивание беременности, а при F < 0 делают заключение об отсутствии угрозы невынашивания беременности. (RU2184968). Вопрос о роли HLA-антигенов является актуальным, однако требует дальнейшего углубленного изучения.
Способ (RU2532367) определения генетической предрасположенности к привычному невынашиванию беременности (ПНБ), основанный на анализе ДНК на полиморфизм генов: исследуют полиморфизмы 34V/L гена FXIII, М235Т гена AGT, 4a/b гена eNOS, PLA1/A2 гена GpIIIa и 353R/Q гена FVII; после установления генотипа по всем 5 генам определяют суммарное влияние выявленных аллелей путем подсчета суммарного балла в соответствии с формулой: Σ=0,19*(Amut FXIII)+0,16*(Amut AGT)+0,13*(Amut eNOS)-0,15*(Amut GpIIIa)-0,05*(Amut FVII), где Amut принимает значения от 0 до 2: 0 - отсутствие мутантных аллелей 34L гена FXIII, 235Т гена AGT, 4а гена eNOS, PLA2 гена GpIIIa и 353Q гена FVII соответственно (дикий генотип), 1 - наличие 1 мутантной аллели (гетерозиготный генотип), 2 - наличие 2 мутантных аллелей (мутантный генотип), значения 0,19; 0,16; 0,13; -0,15 и -0,05 являются коэффициентами, и при составлении заключения считают значение суммарного балла <0,16 баллов основанием для отнесения обследуемого лица к группе с низким риском, значение от 0,16 до 0,40 баллов - к группе со средним риском, а значение >0,40 баллов - к группе с высоким риском формирования ПНБ.
Ближайшим аналогом изобретения является способ оценки (RU2330071) предрасположенности к развитию привычного невынашивания беременности, включающий ДНК-диагностику на наличие полиморфизма С677Т гена метилентетрагидрофолатредуктазы (MTHFR) и составление заключения на основании данных о выявленном генотипе, отличающийся тем, что ДНК одновременно исследуют на наличие полиморфизма R353Q гена фактора VII свертывания крови, генотип определяют с учетом данных по двум исследуемым генам.
Однако, существенной проблемой в молекулярно-генетической диагностике является разрушение микроРНК в образцах биологических жидкостей и/или во время очистки малых РНК. Для этого используют дополнительные способы, подходящие для снижения или устранения разрушения микроРНК которые, например, включают, добавление ингибиторов РНКаз, применение гуанидинхлорида, гуанидинизотиоцианата, N-лауроилсаркозина, додецилсульфата натрия (ДСН) или их комбинации. Кроме того, при работе с образцами мочи риск разрушения микроРНК может быть понижен, если образец провел в мочевом пузыре мало времени (например, менее 4 часов). Ослабление разрушения микроРНК в образцах биологических жидкостей особенно важно, когда до количественной оценки микроРНК требуется хранение и транспортировка образца.
Известно, что наиболее используемым материалом для исследования человеческих тканей, как для диагностических, так и для научных целей, являются фиксированные в нейтральном формалине и заключённые в парафин образцы. Данный вид биоматериала имеет как преимущества в виде возможности длительного хранения при комнатной температуре, так и недостатки, включающие деградацию нуклеиновых кислот. Данные особенности учитываются при разработке методик исследования. Высокая стабильность микроРНК в биологических образцах, включая гистологически фиксированные ткани, делает их ценными биомаркерами для диагностики, прогноза и персонализированного подхода в лечении заболеваний (Князева М.С. и др., Особенности профиля экспрессии микроРНК ткани анапластического рака щитовидной железы, Вопросы онкологии, 2021г., Т.67(1), с.70-76).
Поэтому с быстрым развитием генетических технологий все больше таких молекулярно-генетических исследований используют в качестве рутинной практики для выявления геномных изменений, и определение микроРНК из материала парафиновых блоков у пациентов является актуальным вопросом для применения в клинической практике.
В настоящее время недостаточно известно о специфических микроРНК, задействованных в развитии привычного невынашивания беременности.
Таким образом, изучение экспрессии и разработка тест-систем для определения профиля специфических микроРНК является перспективным подходом в прогнозировании риска невынашивания беременности.
Выдвигаемая техническая проблема, на разрешение которой направлено заявляемое изобретение, является разработка способа для прогнозирования риска развития привычного невынашивания беременности путем определения специфических микроРНК (экспрессии генов miR-34a и miR-145) методом ПЦР в образцах тканей парафиновых блоков.
Выбор miR-34a и miR-145 для исследования обусловлен их ключевой ролью в регуляции пролиферации, апоптоза и миграции клеток. miR-145 играет важную роль в регуляции прогрессии опухолевых клеток, их миграции, инвазии и апоптоза за счет влияния на такие мишени, как c-Myc, Mucin-1, p70S6 киназа и инсулиновый рецептор. miR-34a участвует в процессах пролиферации и рассматривается как перспективный биомаркер для ранней диагностики, классификации и прогноза процессов, связанных с пролиферацией клеток.
Низкая экспрессия miR-145 и miR-34a в эндоцервиксе и эндометрии у женщин с привычным невынашиванием беременности (РПЛ) свидетельствует о нарушении клеточной пролиферации и иммунорегуляции, что может приводить к нарушению процесса инвазии трофобласта и ремоделированию спиральных артерий, что влияет на формирование плацентарного ложа.
Разработан способ прогнозирования риска развития привычного невынашивания беременности, включающий определение уровня относительной экспрессии генов miR-34a и miR-145 относительно референсного гена miR-16 в образцах тканей из парафиновых блоков методом ПЦР в режиме реального времени с применением олигонуклеотидных праймеров: для miR-145-5p использовали праймер для обратной транскрипции SEQ ID NO 1, прямой праймер SEQ ID NO 2 и обратный праймер SEQ ID NO 3, для miR-34a-5p - праймер для обратной транскрипции SEQ ID NO 4, прямой праймер SEQ ID NO 5 и обратный праймер SEQ ID NO 3 и для miR-16-5p - праймер для обратной транскрипции SEQ ID NO 6 прямой праймер SEQ ID NO 7 и обратный праймер SEQ ID NO 3, и при определении уровня относительной экспрессии гена miR-34a < 0,65 и/или относительной экспрессии гена miR-145 < 0,7 прогнозируют риск развития привычного невынашивания беременности, а при показателях относительной экспрессии гена miR-34a ≥ 0,65 и гена miR-145 ≥ 0,7 делают заключение об отсутствии угрозы развития привычного невынашивания беременности.
Техническим результатом предлагаемого изобретения является выявление специфичности генетических маркеров экспрессии микроРНК генов miR-34a и miR-145 в образцах тканей из парафиновых блоков с использованием набора праймеров SEQ ID NO 1- SEQ ID NO 7 для прогнозирования повышенного риска развития невынашивания беременности. Улучшение точности результаты за счёт достижения большей стабильности исследуемых микроРНК.
Для достижения указанного технического результата в способе оценки экспрессии генов микроРНК в биопсийном материале, включающем лабораторное исследование срезов с гистологических блоков, предлагается в качестве лабораторного исследования использовать двухстадийную количественную полимеразную цепную реакцию (ПЦР) с обратной транскрипцией, при этом выделяют из срезов общую РНК методом хлороформ-фенольной экстракции, ПЦР проводят для оценки экспрессии генов микроРНК с универсальным обратным праймером, используются универсализированные праймеры для обратной транскрипции с фрагментом последовательности, специфичным для выявляемой микроРНК, получают значения контрольных циклов (Cq), которые могут быть использованы для вычисления относительной экспрессии микроРНК генов miR-34a и miR-145 по методу дельта-дельта Cq относительно экспрессии референсной miR-16.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
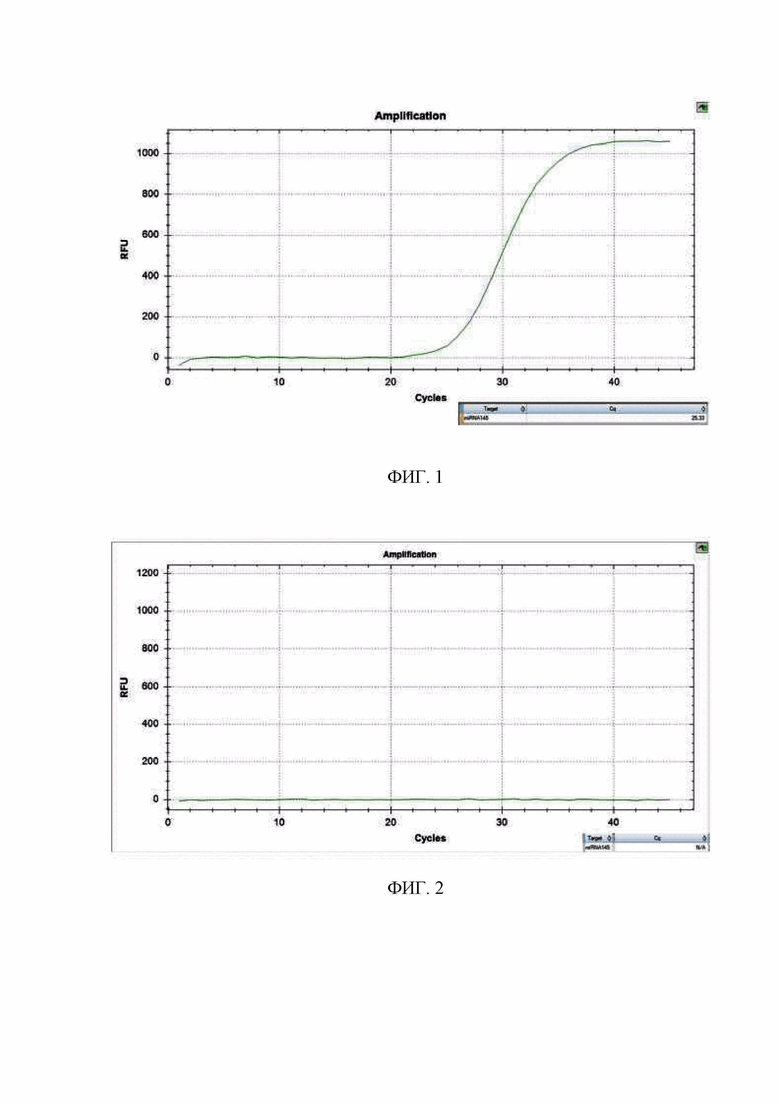
Фиг. 1. Пример выявленной экспрессии микроРНК.
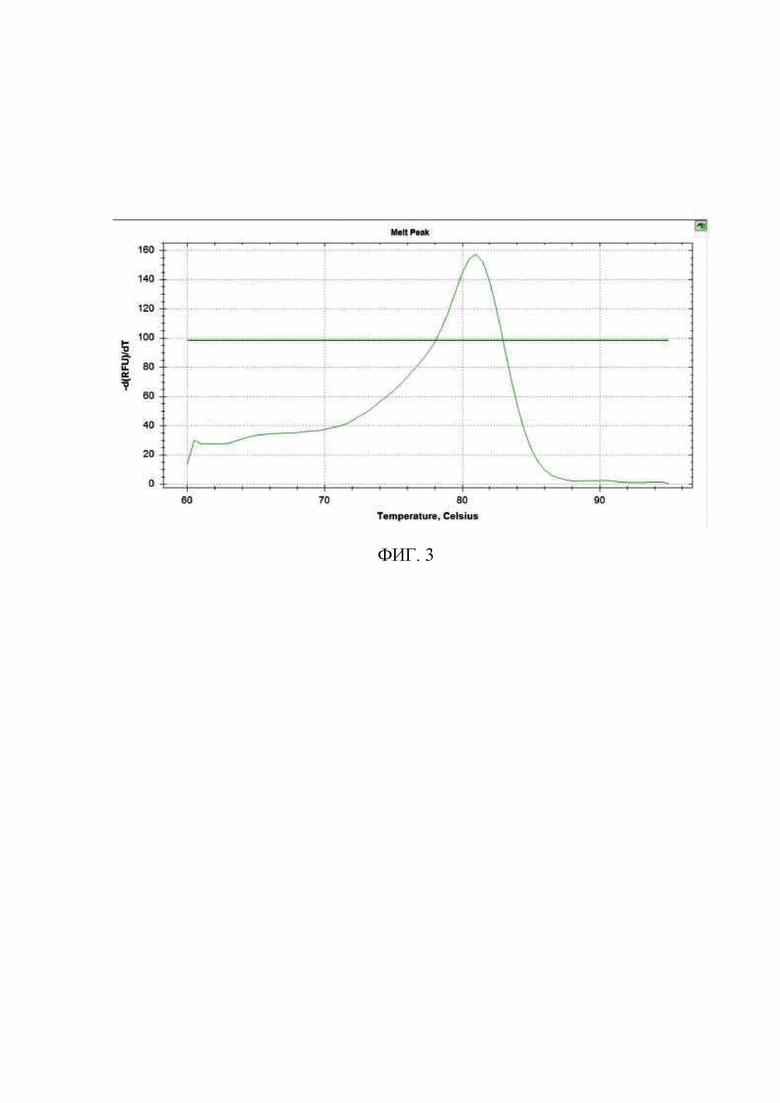
Фиг. 2. Пример отсутствия экспрессии микроРНК.
Фиг. 3. Кривая плавления специфичного ПЦР-продукта.
ОСУЩЕСТВЛЕНИЕ ИЗОБРЕТЕНИЯ
Способ осуществляют следующим образом.
Гистологические парафиновые блоки нарезают на микротоме с установленной толщиной срезов 5 микрометров, один срез помещают в микроцентрифужную пробирку объёмом 1.5 миллилитра.
Производят депарафинизацию методом двухкратной последовательной промывкой 1мл химически-чистого ксилола и 1 миллилитра 95% этанола, после чего высушивают при 60°С. Производится лизис депарафинизированной ткани в 200 мкл буфере следующего состава: 50 мM KCl, 0,5% додецилсульфат натрия, 10 мM Tris-HCl, 1 мM EDTA, 0,1 мг/мл протеиназы K) при 56° С в течении 12 часов с последующим нагреванием до 80° С в течении 15 минут. Полученный лизат центрифугируется 10 минут при 10000g в микроцентрифуге. Полученный супернатант отбирается для использовался для гуанидин-тиоционат-фенол-хлороформной экстракции суммарной РНК с помощью реагента ExtractRNA (Евроген, Россия) согласно инструкции. Наличие выделенной РНК контролируется по наличию осадка, видимого невооружённым глазом на дне микропробирки. Осадок растворяется в 100 мкл деионизированной стерильной воды и используется для постановки двухстадийной ПЦР с обратной транскрипцией.
Выделенная РНК используется в реакции обратной транскрипции с использованием праймеров для обратной транскрипции (ОТ) (Таблица 1) для определения уровня экспрессии генов miR-34a и miR-145 и miR-16.
Таблица 1
Праймеры для обратной транскрипции и ПЦР.
РНК
SEQ ID NO 1
SEQ ID NO 2
SEQ ID NO 3
SEQ ID NO 4
SEQ ID NO 5
SEQ ID NO 3
SEQ ID NO 6
SEQ ID NO 7
SEQ ID NO 3
Для исследуемого уровня специфической микроРНК проводят реакцию обратной транскрипции 3мкл выделенной РНК c использованием набора реактивов MMLV RT kit (Евроген, Россия) согласно инструкции. Реакцию проводят в объеме 20 мкл. В каждую постановку включают реакцию без ревертазы для контроля контаминации образца РНК примесью ДНК. В пробирке объёмом 0,2 мл смешивают следующие компоненты 3 мкл выделенной РНК, 2 мкл праймера для ОТ концентрацией 10 мкМ, объём реакционной смеси доводится до 11 мкл деионизированной стерильной водой. Смесь прогревают 2 минуты при 70 °C, перенесите затем переносят в лёд. Добавляют 9 мкл предварительно подготовленной смеси: 4 мкл 5Х First strand buffer; 2 мкл dNTP mix (10 мM each); 2 мкл DTT (20 mM); 1 мкл MMLV Reverse Transcriptase (содержит 100 единиц активности обратной транскриптазы). Смесь перемешивается пипетированием. Проводится инкубация при 37°C 60 минут, затем 70 °C 10 минут, после чего полученная смесь хранится во льду до измерения экспрессии методом ПЦР.
Изменение относительной экспрессии микроРНК оценивают методом ПЦР в режиме реального времени с интеркалирующем красителем SYBR Green I с использованием мастер-микса 5X qPCRmix-HS SYBR (Евроген, Росиия) согласно инструкции. Амплификацию выполняют в объёме 25 мкл при концентрации 200 нM прямого и универсально обратного праймера (Таблица 1). Постановка количественной ПЦР в режиме реального времени каждого образца РНК проводится в 3 повторах, для каждого образца и каждой определяемой микроРНК. Полученную ПЦР-смесь инкубируют в амплификаторе CFX 96 (Bio-Rad, США) по следующей программе: 5 мин при 95 °С, 40 циклов, включая денатурацию при 95 °С 10 с, отжиг при 59 °С 20 с, элонгация при 72 °С 20 с. Значения экспрессии генов микроРНК оценивают программой прибора CFX Manager (Bio-Rad, США) в режиме Регрессия (Regression), результаты получают в виде значения порогового цикла (Cq) флуоресцентной кривой.
Интерпретация: при наличии экспрессии микроРНК, определяется флуоресцентная кривая в течение 40 циклов ПЦР (определяется номер порогового цикла Cq), при отсутствии экспрессии специфической микроРНК пороговый цикл Cq не определяется, программа выдаёт значение N/A. Значение Cq используется для определения абсолютной, или относительной экспрессии микроРНК. Результаты, соответствующие значениям пороговых циклов N/A считаются отрицательными.
Для определения относительной экспрессии генов мкРНК использован метод 2-Δ ΔCq.
Относительную экспрессию (ОЭ) микроРНК рассчитывали по формуле:
ОЭ = Е–ΔΔ Cq (Е – эффективность амплификации, E = 2,0).
Нормализацию результатов проводили по уровню экспрессии miR-16 (рефересной) каждого образца по схеме, приведенной ниже:
(ΔCq34a=Cq34a −Cq miR-16),
(ΔCq145=Cq145 −Cq miR-16).
Относительная экспрессия (ОЭ), значение которой используется в алгоритме способа определения риска привычного невынашивания беременности определяется как ОЭ miR-145 =2−ΔCq145, ОЭ miR-34a =2−ΔCq34a .
Чувствительность и специфичность данного алгоритма составляет 84% и 91% соответственно.
Было проведено проспективное наблюдательное сравнительное исследование с участием женщин 118 женщин. Было выделено две группы: первая группа (n = 64) включала женщин с визуально неизмененной шейкой матки и привычным невынашиванием беременности (ПНБ) неясного генеза, а вторая группа (группа сравнения) (n = 54) состояла из здоровых беременных женщин без неблагоприятных репродуктивных исходов.
Критерии включения в группу ПНБ включали женщин в возрасте 18–45 лет с анамнезом двух и более выкидышей, неопределённой этиологией ПНБ, визуально неизмененной шейкой матки при осмотре в зеркалах и кольпоскопии, а также добровольное информированное согласие. Критерии исключения: морфологически измененная шейка матки (цервицит, рубцы, дисплазия, кисты), злокачественные новообразования репродуктивной системы, текущая беременность или лактация, тяжелые соматические заболевания, сахарный диабет, первичный и вторичный иммунодефицит, а также наличие в анамнезе кесарева сечения или миомэктомии. Группа сравнения отличалась отсутствием спонтанных выкидышей в анамнезе.
Образцы эндометрия были получены у женщин с ПНБ и контрольной группы через месяц после медикаментозного прерывания беременности на сроке 5–6 недель. Были собраны клинические и анамнестические данные, акушерско-гинекологический статус.
Пример 1: Пациентка Н., возраст 32 года, в анамнезе 3 самопроизвольных выкидыша, после обследования причины выкидышей не установлены. Проведено исследование уровня экспрессии генов miR-34a и miR-145 в образцах биоптата эндометрия. Уровень относительной экспрессии miR-34a составил 0.43, уровень относительной экспрессии miR-145 составил 0.58. В данном случае, уровень относительной экспрессии miR-34a < 0,65 и уровень относительной экспрессии гена miR-145 < 0,7, что позволяет прогнозировать высокий риск невынашивания беременности.
Пример 2: Пациентка М., возраст 29 лет, в анамнезе 2 самопроизвольных выкидыша, после обследования причины выкидышей не установлены. Проведено исследование уровня экспрессии генов miR-34a и miR-145 в образцах биоптата эндометрия. Уровень относительной экспрессии miR-34a составил 0.71, уровень относительной экспрессии miR-145 составил 0.4. В данном случае, уровень относительной экспрессии miR-34a > 0,65, но уровень относительной экспрессии гена miR-145 < 0,7, что позволяет прогнозировать высокий риск невынашивания беременности.
Пример 3: Пациентка А., возраст 35 лет, в анамнезе 3 самопроизвольных выкидыша, после обследования причины выкидышей не установлены. Проведено исследование уровня экспрессии генов miR-34a и miR-145 в образцах биоптата эндометрия. Уровень относительной экспрессии miR-34a составил 0.23, уровень относительной экспрессии miR-145 составил 0.92. В данном случае, уровень относительной экспрессии miR-145 > 0,7, но уровень относительной экспрессии гена miR-34а < 0,65, что позволяет прогнозировать высокий риск невынашивания беременности.
Пример 4: Пациентка С., возраст 24 года, акушерско-гинекологический анамнез не отягощён. Проведено исследование уровня экспрессии генов miR-34a и miR-145 в образцах биоптата эндометрия. Уровень относительной экспрессии miR-34a составил 0.65, уровень относительной экспрессии miR-145 составил 1.1. В данном случае, уровень относительной экспрессии miR-145 > 0,7, и уровень относительной экспрессии гена miR-34а = 0,65, что позволяет прогнозировать отсутствие риска невынашивания беременности.
Пример 5: Пациентка О., возраст 27 лет, акушерско-гинекологический анамнез не отягощён. Проведено исследование уровня экспрессии генов miR-34a и miR-145 в образцах биоптата эндометрия. Уровень относительной экспрессии miR-34a составил 0.84, уровень относительной экспрессии miR-145 составил 0.7. В данном случае, уровень относительной экспрессии miR-145 = 0,7, и уровень относительной экспрессии гена miR-34а > 0,65, что позволяет прогнозировать отсутствие риска невынашивания беременности.
Пример 6: Пациентка А., возраст 31 год, акушерско-гинекологический анамнез не отягощён. Проведено исследование уровня экспрессии генов miR-34a и miR-145 в образцах биоптата эндометрия. Уровень относительной экспрессии miR-34a составил 0.72, уровень относительной экспрессии miR-145 составил 0.88. В данном случае, уровень относительной экспрессии miR-145 > 0,7, и уровень относительной экспрессии гена miR-34а > 0,65, что позволяет прогнозировать отсутствие риска невынашивания беременности.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2025-02-26"
softwareVersion="2.3.0" softwareName="WIPO Sequence"
fileName="Pregnance_Loss_micRNAs.xml" dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0000</ApplicationNumberText>
<FilingDate>2025-02-26</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>0000</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0000</ApplicationNumberText>
<FilingDate>2025-02-26</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное научное учреждение «Российский научный центр хирургии имени
академика Б.В. Петровского»</ApplicantName>
<ApplicantNameLatin>Federal State Budgetary Research Institution
Russian research center of surgery named after academician B.V.
Petrovsky</ApplicantNameLatin>
<InventorName languageCode="ru">Николай Контантинович
Шахпазян</InventorName>
<InventorNameLatin>Nikolay K. Shakhpazyan</InventorNameLatin>
<InventionTitle languageCode="ru">Способ прогнозирования привычной
невынашиваемости беременности по экспрессии генов микроРНК в
биопсийном материале эндометрия</InventionTitle>
<SequenceTotalQuantity>7</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggtccgaggtattcgcactggatacgacagggat</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacaaggtccagttttcccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggtccgaggtattcgcactggatacgacacaacc</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacagtgtggcagtgtcttag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggtccgaggtattcgcactggatacgaccgccaa</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacagtgtagcagcacgtaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-143-5p методом количественной ПЦР и его применение | 2025 |
|
RU2838258C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837874C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-145-3p методом количественной ПЦР и его применение | 2025 |
|
RU2838260C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837872C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-3p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837873C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-mir-143-3p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837882C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-143-3p методом количественной ПЦР и его применение | 2025 |
|
RU2838257C1 |
| Набор реагентов для определения малых рибонуклеиновых кислот (микроРНК) при диагностике множественной миеломы | 2024 |
|
RU2836054C1 |
| Аналитическая система для диагностики рака лёгкого при помощи свободных и ассоциированных с клетками крови циркулирующих микроРНК | 2020 |
|
RU2755651C1 |
| Способ дифференциальной диагностики рака яичников, кистозных образований яичника и рака тела матки | 2024 |
|
RU2836527C1 |
Изобретение относится к области молекулярной биологии. Описан способ прогнозирования риска развития привычного невынашивания беременности, включающий определение уровня относительной экспрессии генов miR-34a и miR-145 относительно референсного гена miR-16 в образцах тканей из парафиновых блоков методом ПЦР в режиме реального времени с применением олигонуклеотидных праймеров. При определении уровня относительной экспрессии гена miR-34a < 0,65 и/или относительной экспрессии гена miR-145 < 0,7 прогнозируют риск развития привычного невынашивания беременности, а при показателях относительной экспрессии гена miR-34a ≥ 0,65 и гена miR-145 ≥ 0,7 делают заключение об отсутствии угрозы развития привычного невынашивания беременности. Техническим результатом предлагаемого изобретения является выявление специфичности генетических маркеров экспрессии микроРНК генов miR-34a и miR-145 в образцах тканей из парафиновых блоков. 3 ил., 1 табл., 6 пр.
Способ прогнозирования риска развития привычного невынашивания беременности, включающий определение уровня относительной экспрессии генов miR-34a и miR-145 относительно референсного гена miR-16 в образцах тканей из парафиновых блоков методом ПЦР в режиме реального времени с применением олигонуклеотидных праймеров: для miR-145-5p использовали праймер для обратной транскрипции SEQ ID NO 1, прямой праймер SEQ ID NO 2 и обратный праймер SEQ ID NO 3, для miR-34a-5p - праймер для обратной транскрипции SEQ ID NO 4, прямой праймер SEQ ID NO 5 и обратный праймер SEQ ID NO 3 и для miR-16-5p - праймер для обратной транскрипции SEQ ID NO 6 прямой праймер SEQ ID NO 7 и обратный праймер SEQ ID NO 3, и при определении уровня относительной экспрессии гена miR-34a < 0,65 и/или относительной экспрессии гена miR-145 < 0,7 прогнозируют риск развития привычного невынашивания беременности, а при показателях относительной экспрессии гена miR-34a ≥ 0,65 и гена miR-145 ≥ 0,7 делают заключение об отсутствии угрозы развития привычного невынашивания беременности.
| WO 2019154996 A1 15.08.2019 | |||
| Способ выявления наследственной предрасположенности к развитию задержки роста плода у курящих женщин | 2017 |
|
RU2646505C1 |
| Chen Y | |||
| et al | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Bioscience Reports, 2019, V.39(6), Найдено в сети интернет 12.03.2025 | |||
Авторы
Даты
2025-04-07—Публикация
2025-02-26—Подача