Область техники настоящего изобретения
Настоящее изобретение относится к области биотехнологии и молекулярной биологии и, в частности, относится к новой сконструированной рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, содержащей уникальную олигонуклеотидную вставку согласно SEQ ID NO:1 и имеющей последовательность согласно SEQ ID NO:4. Настоящее изобретение также относится к применению новой рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, в качестве положительного контроля при проведении полимеразной цепной реакции в реальном времени (ПЦР-РВ) для диагностики сердечно-сосудистых заболеваний и выявления риска их развития. Настоящее изобретение может быть использовано специалистами учреждений практического здравоохранения и в научно-исследовательских лабораториях.
Перечень последовательностей
К настоящему документу прилагается перечень последовательностей, поданный в формате XML.
Предшествующий уровень техники настоящего изобретения
Согласно данным Всемирной организации здравоохранения сердечно-сосудистые заболевания (ССЗ) являются основной причиной смерти во всем мире, от которой каждый год умирает 17,9 млн человек. Сердечно-сосудистые заболевания представляют собой группу болезней сердца и кровеносных сосудов, в которую входят ишемическая болезнь сердца, заболевания сосудов головного мозга, ревматическая болезнь сердца и другие патологии. Более четырех из пяти смертей от ССЗ происходит в результате инфаркта миокарда и инсульта, причем треть из этих случаев смерти носит преждевременный характер и происходит среди людей в возрасте до 70 лет.
Условием профилактики преждевременной смертности является выявление всех лиц, подверженных наиболее высокому риску ССЗ, и обеспечение предоставления им надлежащего лечения. Важнейшим условием предоставления необходимого лечения и консультирования всем, кому это необходимо, является обеспечение наличия в учреждениях первичного звена соответствующих лекарственных препаратов и базовых медицинских технологий, необходимых для диагностики и лечения неинфекционных заболеваний.
Таким образом, существует потребность в создании эффективных диагностических средств для диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
В статье Медведева Е.А. и др. Молекулярные биомаркеры в диагностике, стратификации риска и прогнозировании хронической сердечной недостаточности. Российский кардиологический журнал 2016, 8 (136): 86-91 http://dx.doi.org/10.15829/1560-4071-2016-8-86-91, указано, что последнее десятилетие широкое развитие получила концепция молекулярных биомаркеров, используемых в том числе и для диагностики сердечно-сосудистых заболеваний.
Поиск надежных молекулярных биомаркеров и разработка эффективных средств их обнаружения является текущей задачей последнего десятилетия.
В международной патентной публикации WO2011084460 A1 описан способ лечения или профилактики ишемии миокарда у нуждающегося в этом субъекта, включающий модуляцию экспрессии или активности одной или более микроРНК, выбранных из члена семейства miR-15, miR-21, miR-26a, let-7b, miR-199a, miR-214, miR-10a, miR-10b, miR-574, miR-320, miR-92a, miR-499, miR-101a, miR-101b, miR-125b, miR-126, члена семейства miR-30, miR-143, miR-145, miR-185, miR-34a, miR-1, miR-133, miR-210 и miR-29a, в клетках сердца субъекта. Таким образом, раскрыты семейства микроРНК, которые могут быть использованы в качестве молекулярных биомаркеров для сердечно-сосудистых заболеваний.
В международной патентной публикации WO2010135570 описан способ лечения или профилактики инфаркта миокарда, ремоделирования сердца и сердечной недостаточности у нуждающегося в этом субъекта, включающий модуляцию экспрессии или активности одной или более миРНК, выбранных из члена семейства let-7, miR-15b, miR-21, miR-199a, miR-199b, miR-214, miR-10a, miR-10b, miR-16, miR-146a, miR-146b, miR-221, miR-222, miR-497, miR-20a, miR-20b, miR-93, miR-101, miR-126, члена семейства miR-30, miR-143, miR-145, miR-150 и miR-29а, в клетках сердца субъекта.
Таким образом, определенные семейства микроРНК могут быть рассмотрены в качестве молекулярного биомаркера для диагностики сердечно-сосудистого заболевания и выявления риска его развития. Однако успех надежного и точного диагностирования состоит в выборе наиболее показательного молекулярного биомаркера, при этом проведение диагностики на основе конкретного выбранного наиболее показательного биомаркера существенно упрощает проведение диагностики.
Одним из современных методов диагностики заболеваний является полимеразная цепная реакция (ПЦР). Универсальность, высокая чувствительность и относительная простота исполнения сделали ПЦР незаменимой для решения различных задач клинической диагностики, таких как прямое обнаружение и идентификация возбудителей заболеваний, а также молекулярное типирование.
В патенте RU2662975 C1 описано определение микроРНК в плазме для обнаружения ранних стадий колоректального рака с использованием реакции ПЦР. В заявке RU2020108781 A раскрыт способ диагностики меланомы с помощью экзосомальных микроРНК с использованием реакции ПЦР. Однако согласно указанным публикациям проводят измерение относительного количества транскриптов микроРНК 143 путем анализа экспрессии гена искомой микроРНК.
В последние годы в мировой и отечественной практике стали широко использоваться методы молекулярно-биологического анализа, в частности, обратная транскрипция и полимеразная цепная реакция в режиме реального времени (ОТ-ПЦР-РВ).
В статье Xiangran Sun et al., miR-143-3p inhibits the proliferation, migration and invasion in osteosarcoma by targeting FOSL2, Scientific Reports | (2018) 8:606 | DOI:10.1038/s41598-017-18739-3, описано применение количественной ПЦР-РВ для сравнения уровня экспрессии miR-143-3p в нормальной и опухолевой ткани.
Однако для успешного проведения ПЦР-РВ и количественного определения молекулярного биомаркера необходима разработка надежного положительного контроля, как раскрыто в статье Доронин М. И. Конструирование плазмиды P JET 1.2 SF GFP-146S FMDV для получения положительного контроля при опосредованном определении концентрации 146S компонента вируса ящура в сырье для инактивированных вакцин. Актуальные вопросы ветеринарной биологии № 3 (55), 2022.
Таким образом, несмотря на высокий потенциал hsa-miR-143-3p как биомаркера для ее точного определения в клинических образцах (кровь, плазма, сыворотка) с использованием ПЦР-РВ необходима разработка эффективного и надежного положительного контроля.
Однако в области диагностики сердечно-сосудистых заболеваний и риска их развития и разработки надежных контрольных систем, таких как рекомбинантная плазмидная ДНК, содержащая последовательность выбранной микроРНК в качестве молекулярного биомаркера сердечно-сосудистого заболевания, на сегодняшний день не решена задача получения эффективной рекомбинантной плазмидной ДНК, кодирующей выбранную микроРНК, для применения в качестве положительного контроля при проведении ПЦР-РВ для диагностики сердечно-сосудистых заболеваний.
В статьях Shuhui Qi et. al., Knockdown of microRNA-143-5p by STTM technology affects eumelanin and pheomelanin production in melanocytes, Molecular Medicine RE POR TS 20: 2649-2656, 2019, и Hiroki Sanada et. al., Involvement of Dual Strands of miR-143 (miR-143-5p and miR-143-3p) and Their Target Oncogenes in the Molecular Pathogenesis of Lung Adenocarcinoma, Int. J. Mol. Sci. 2019, 20, 4482, описаны экспрессионные векторы для микроРНК, однако не предполагается их применение в качестве положительного контроля при проведении ПЦР-РВ для диагностики сердечно-сосудистых заболеваний.
Очевидно, что существует нерешенная задача предоставления эффективной рекомбинантной ДНК, кодирующей выбранную в качестве молекулярного биомаркера микроРНК, для применения в качестве положительного контроля при проведении ПЦР-РВ для диагностики сердечно-сосудистых заболеваний. Решение указанной задачи позволит создать средства диагностики для эффективной и надежной диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
Таким образом, разработка надежного положительного контроля для проведения ПЦР-РВ в целях диагностики сердечно-сосудистых заболеваний и выявления риска их развития, такого как рекомбинантная плазмидная ДНК, кодирующая последовательность выбранной микроРНК, в качестве молекулярного биомаркера, является критически важной задачей для стандартизации методов ПЦР-РВ с целью эффективной и надежной диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
Краткое раскрытие настоящего изобретения
Авторы настоящего изобретения обнаружили, что указанная задача может быть решена посредством конструирования рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, обладающей высокой стабильностью и обеспечивающей эффективное и надежное количественное определение микроРНК hsa-miR-143-3p.
Для создания рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, авторами настоящего изобретения был синтезирован уникальный олигонуклеотид, имеющий последовательность согласно SEQ ID NO:1, содержащий кДНК для микроРНК hsa-miR-143-3p.
При разработке данного уникального олигонуклеотида согласно SEQ ID NO:1 авторами настоящего изобретения была создана уникальная пара праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3.
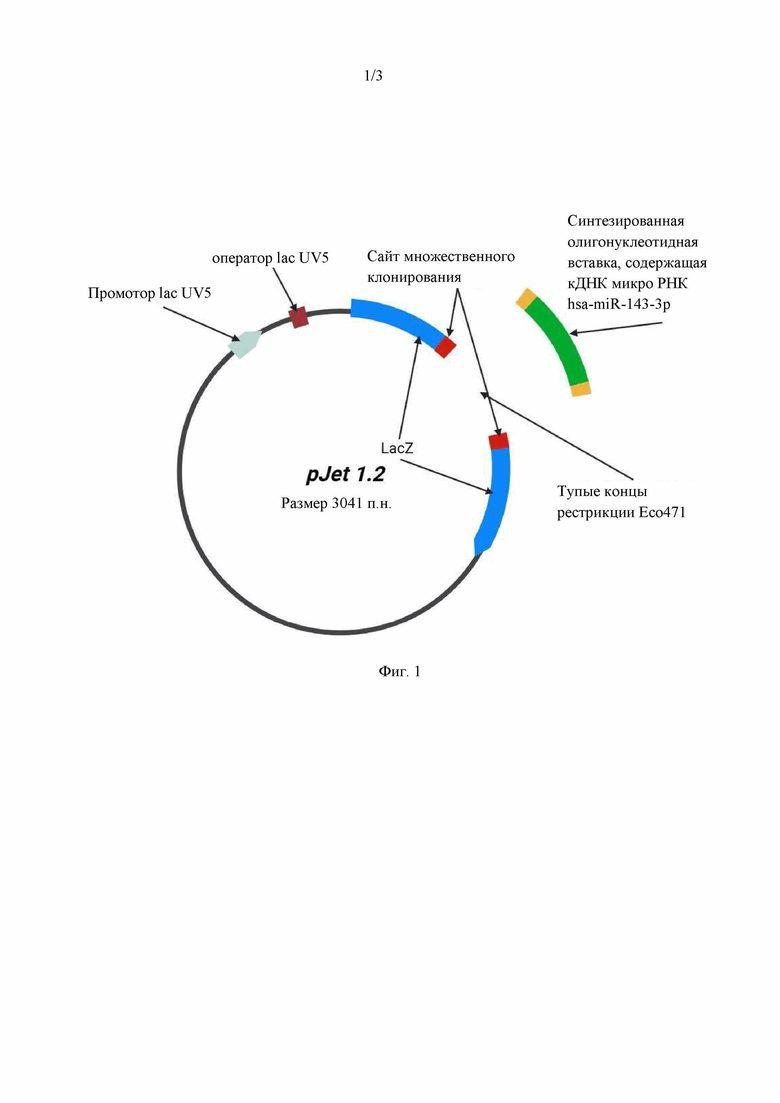
Таким образом, согласно первому аспекту настоящее изобретение относится к рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, где рекомбинантная плазмидная ДНК содержит олигонуклеотидную вставку с последовательностью согласно SEQ ID NO:1, кодирующую микроРНК hsa-miR-143-3p, фланкированную последовательностями сайта рестрикции Eco47I, и где рекомбинантная плазмидная ДНК имеет последовательность согласно SEQ ID NO:4 и расположение элементов, как представлено на фиг.1.
Полученная новая рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, имеет размер 3041 п.н.
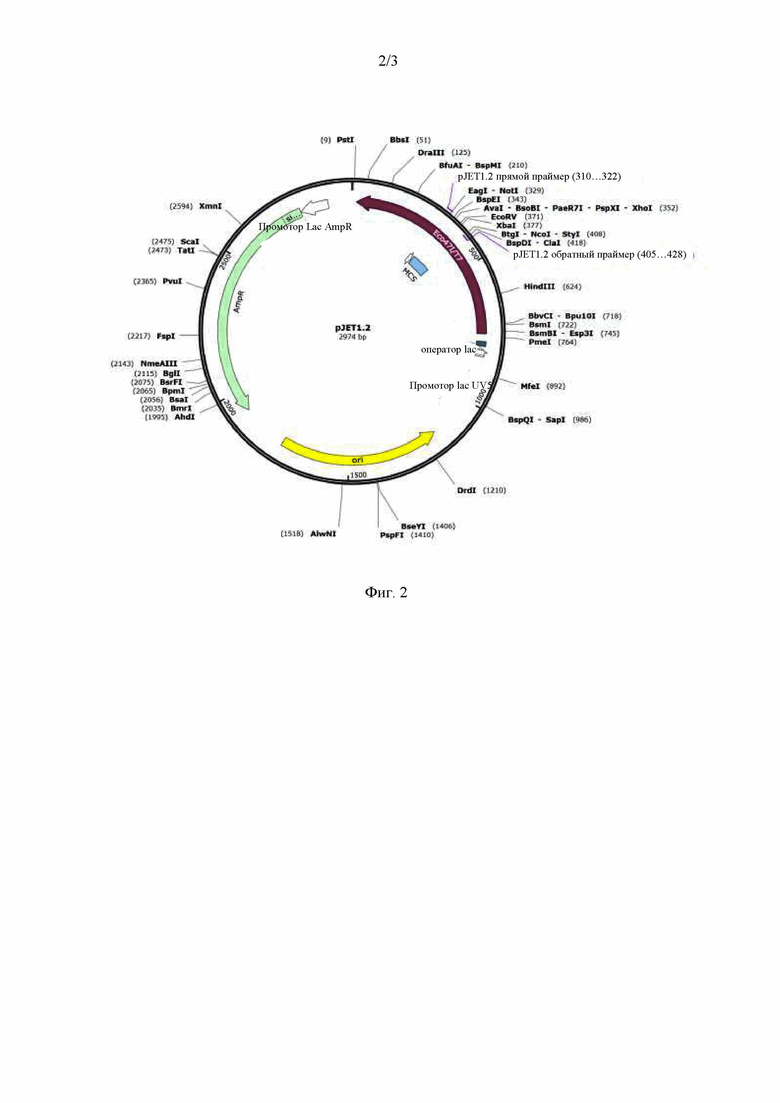
Согласно варианту осуществления первого аспекта настоящего изобретения рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, основана на плазмиде pJET1, расположение элементов которой представлено на фиг. 2.
Разработанная авторами новая рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, предназначена для диагностики сердечно-сосудистых заболеваний и определения риска их развития, в частности, в качестве положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития.
Согласно второму аспекту настоящее изобретение относится к применению рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, согласно первому аспекту настоящего изобретения в качестве положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития.
Настоящее изобретение также относится к уникальному набору праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3 для получения уникального олигонуклеотида согласно SEQ ID NO:1.
Технический результат настоящего изобретения состоит в следующем:
- получение новой эффективной и стабильной рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p,
- расширение арсенала средств для диагностики сердечно-сосудистых заболеваний и определения риска их развития,
- достижение эффективной амплификации микроРНК hsa-miR-143-3p in vitro,
- повышение амплификации микроРНК hsa-miR-143-3p in vitro,
- достижение стабильной и надежной амплификации микроРНК hsa-miR-143-3p in vitro,
- разработка эффективного и надежного положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития,
- достижение высокой диагностической чувствительности,
- достижение высокой диагностической специфичности,
- достижение высокой прогностичности диагностического результата,
- достижение высокой прогностичности отрицательного результата.
Краткое описание чертежей
На фиг. 1 представлена схема рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, согласно настоящему изобретению с указанием элементов плазмиды и их расположения.
На фиг. 2 представлена схема плазмиды pJET1.2 с указанием элементов плазмиды и их расположения.
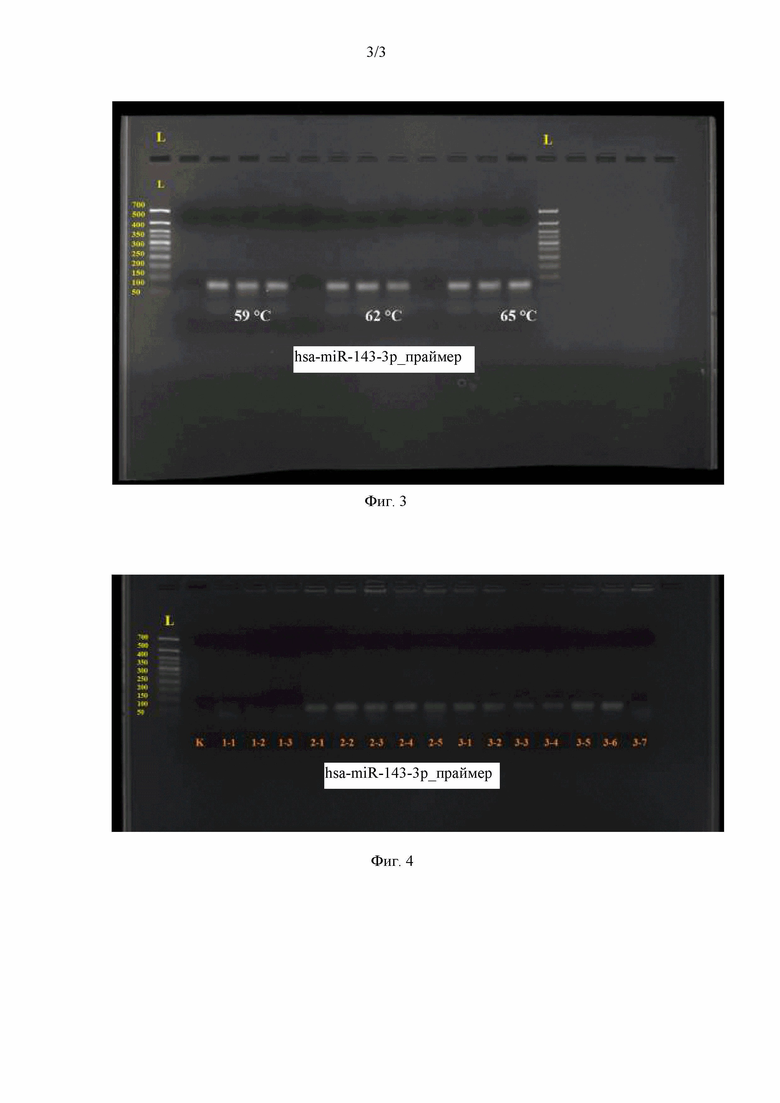
На фиг. 3 представлены результаты амплификации целевого фрагмента для лигирования.
На фиг. 4 представлены результаты ПЦР-теста.
Подробное раскрытие настоящего изобретения
Авторы настоящего изобретения обнаружили, что наиболее эффективным способом диагностики сердечно-сосудистых заболеваний и выявления риска их развития для указанного молекулярного маркера является применение ПЦР-РВ, что требует разработки эффективного и надежного положительного контроля.
Разработка надежных контрольных систем, таких как рекомбинантная плазмидная ДНК, содержащая последовательность выбранной микроРНК в качестве молекулярного биомаркера, является критически важной задачей для стандартизации методов ПЦР-РВ с целью эффективной и надежной диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
Решение указанной задачи позволит создать эффективные средства диагностики для эффективной и надежной диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
В ходе анализа опубликованных научно-исследовательских работ авторы настоящего изобретения пришли к выводу, что в качестве эффективного молекулярного биомаркера для диагностики сердечно-сосудистых заболеваний и выявления риска их развития можно применять микроРНК hsa-miR-143-3p.
Авторы настоящего изобретения обнаружили, что задача создания эффективного средства диагностики может быть решена посредством конструирования рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, обладающей высокой стабильностью и обеспечивающей эффективное и надежное количественное определение микроРНК hsa-miR-143-3p.
Для создания рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, авторами настоящего изобретения был синтезирован уникальный олигонуклеотид, имеющий последовательность согласно SEQ ID NO:1, содержащий кДНК для микроРНК hsa-miR-143-3p.
При разработке данного уникального олигонуклеотида согласно SEQ ID NO:1 авторами настоящего изобретения была создана уникальная пара праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3.
Разработка уникальной пары праймеров авторами настоящего изобретения позволила им создать уникальный олигонуклеотид согласно SEQ ID NO:1, который обеспечивает эффективную амплификацию с получением ампликонов с длиной, позволяющей проводить эффективное количественное определение.
В ходе многочисленных исследований авторами настоящего изобретения была выбрана плазмида pJET1.2, обладающая сайтами рестрикции Eco47I.
Авторами настоящего изобретения неожиданно обнаружено, что для получения стабильной и эффективной рекомбинантной ДНК-плазмиды, кодирующей микроРНК hsa-miR-143-3p, разработанная авторами настоящего изобретения уникальная олигонуклеотидная вставка должна быть фланкирована именно сайтами рестрикции Eco47I.
Разработанная авторами настоящего изобретения новая рекомбинантная плазмидная ДНК с последовательностью согласно SEQ ID NO:4 и размером 3041 п.н. обеспечивает in vitro амплификацию последовательности регуляторной микроРНК hsa-miR-143-3p и положительный контроль для проведения ПЦР-РВ на ее основе для диагностики сердечно-сосудистых заболеваний и определения риска их развития.
Выбор молекулярного биомаркера для диагностики сердечно-сосудистых заболеваний и выявления риска их развития
МикроРНК (miRNA) являются ключевыми регуляторами посттранскрипционной экспрессии генов, участвующими в широком спектре физиологических и патологических процессов, включая воспаление, метаболизм и регуляцию работы сердечно-сосудистой системы. Среди них hsa-miR-143-3p привлекает особое внимание благодаря своей роли в патогенезе сердечно-сосудистых заболеваний (ССЗ), являющихся одной из ведущих причин смертности в мире.
hsa-miR-143-3p представляет собой одну из двух зрелых форм микроРНК, происходящих из одного и того же гена miR-143. Она играет важную роль в регуляции экспрессии генов, участвующих в различных биологических процессах, таких как клеточный цикл, дифференцировка, апоптоз и метаболизм. Ниже приведено подробное описание hsa-miR-143-3p, включая его происхождение, механизмы действия, биологические функции и клиническое значение. hsa-miR-143-3p образуется в результате процессинга прекурсорной молекулы микроРНК (pre-miR-143), которая имеет шпилечную структуру. Прекурсорная молекула синтезируется в ядре из первичного транскрипта (pri-miR-143) с помощью фермента Drosha. Затем pre-miR-143 экспортируется в цитоплазму, где она разрезается ферментом Dicer, что приводит к образованию двух цепей: 3p (hsa-miR-143-3p) и 5p (hsa-miR-143-5p).
hsa-miR-143-3p функционирует как регулятор экспрессии генов на посттранскрипционном уровне. Он связывается с комплементарными участками в 3'-нетранслируемых областях (3'-UTR) мРНК-мишеней, что приводит к подавлению их экспрессии. Это подавление может происходить за счет деградации мРНК, а именно hsa-miR-143-3p способствует разрушению мРНК-мишеней. Согласно другому варианту связывание с мРНК hsa-miR-143-3p блокирует процесс трансляции, предотвращая синтез белка.
Исследования показывают, что hsa-miR-143-3p участвует в регуляции сосудистого тонуса, ремоделирования сосудов и воспалительных процессов, что делает ее перспективным биомаркером для ранней диагностики и прогнозирования риска ССЗ [Zhao W, Zhao SP, Zhao YH. MicroRNA-143/-145 in Cardiovascular Diseases. Biomed Res Int. 2015;2015:531740. doi:10.1155/2015/531740]. hsa-miR-143-3p играет важную роль в контроле дифференцировки сосудистых гладкомышечных клеток (ГМКС), а именно при фенотипическом переключении ГМКС, влияя на их пролиферацию, миграцию и способность к образованию атеросклеротических бляшек. Дисрегуляция этой микроРНК может приводить к атеросклеротическим изменениям и повышенному риску развития ишемической болезни сердца (ИБС) и инфаркта миокарда [Xin M, Small EM, Sutherland LB, Qi X, McAnally J, Plato CF, Richardson JA, Bassel-Duby R, Olson EN. MicroRNAs miR-143 and miR-145 modulate cytoskeletal dynamics and responsiveness of smooth muscle cells to injury. Genes Dev. 2009 Sep 15;23(18):2166-78. doi: 10.1101/gad.1842409. Epub 2009 Aug 31. PMID: 19720868; PMCID: PMC2751981.].
Также следует отметить противовоспалительное действие данной регуляторной молекулы. Экспериментальные данные свидетельствуют о том, что hsa-miR-143-3p модулирует сигнальные пути воспаления, такие как NF-κB и PI3K/AKT, подавляя экспрессию провоспалительных цитокинов (IL-6, TNF-α). Нарушение ее регуляции может способствовать развитию эндотелиальной дисфункции и хроническому воспалению сосудов, являющихся ключевыми механизмами прогрессии ССЗ [Wang Y, Zhang X, Wang J, et al. Inflammatory Periodontal Ligament Stem Cells Drive M1 Macrophage Polarization via Exosomal miR-143-3p-Mediated Regulation of PI3K/AKT/NF-κB Signaling. Stem Cells. 2023;41(2):184-199. doi:10.1093/stmcls/sxac087]. Отмечено влияние на метаболизм липидов. Было показано, что hsa-miR-143-3p регулирует гены, участвующие в метаболизме липопротеинов низкой плотности (ЛПНП) и триглицеридов, что делает ее важной мишенью для оценки риска дислипидемий и метаболического синдрома [Wang Y, Zhang X, Wang J, et al. Inflammatory Periodontal Ligament Stem Cells Drive M1 Macrophage Polarization via Exosomal miR-143-3p-Mediated Regulation of PI3K/AKT/NF-κB Signaling. Stem Cells. 2023;41(2):184-199. doi:10.1093/stmcls/sxac087].
Использование hsa-miR-143-3p в клинической практике имеет замечательные перспективы. Анализ уровней hsa-miR-143-3p в плазме крови может стать эффективным методом для раннего выявления лиц с высоким риском сердечно-сосудистых катастроф, таких как инфаркт миокарда, инсульт, разрыв аневризмы аорты.
hsa-miR-143-3p может быть использована как диагностический биомаркер. Повышенный или пониженный уровень hsa-miR-143-3p в биологических жидкостях может быть индикатором патологических изменений в сердечно-сосудистой системе задолго до появления клинических симптомов. hsa-miR-143-3p может быть использована также как прогностический маркер. Динамическое наблюдение за уровнем этой микроРНК позволяет оценивать эффективность терапии (например, статинов, антиагрегантов, противовоспалительных препаратов) и прогнозировать вероятность повторных сердечно-сосудистых событий.
В перспективе микроРНК могут быть использованы как терапевтическая мишень. Разработка молекулярных методов коррекции уровня hsa-miR-143-3p (например, антисмысловых олигонуклеотидов или малых молекул, регулирующих ее экспрессию) открывает перспективы персонализированной терапии атеросклероза и гипертонической болезни.
Учитывая всё сказанное выше, перед исследователями встаёт необходимость разработки точных методов анализа hsa-miR-143-3p. Однако несмотря на высокий потенциал hsa-miR-143-3p как биомаркера ее точное определение в клинических образцах (кровь, плазма, сыворотка) затруднено вариабельностью методов детекции и отсутствием надежного и эффективного положительного контроля. Поэтому разработка надежных контрольных систем, таких как рекомбинантная плазмидная ДНК, содержащая последовательность hsa-miR-143-3p, является критически важной задачей для стандартизации методов ПЦР-РВ.
Исследование hsa-miR-143-3p представляет собой важное направление в молекулярной кардиологии и персонализированной медицине. Разработка надежных методов детекции и контрольных систем для ее анализа позволит значительно повысить диагностическую точность, прогнозировать риски сердечно-сосудистых заболеваний и, возможно, открыть новые терапевтические стратегии для их профилактики и лечения.
Выбор методики проведения диагностики сердечно-сосудистых заболеваний
В последние годы в мировой и отечественной практике стали широко использоваться методы молекулярно-биологического анализа, в частности, обратная транскрипция и полимеразная цепная реакция в режиме реального времени (ОТ-ПЦР-РВ), для количественного анализа содержания молекулярного биомаркера.
В настоящее время существует потребность в разработке методики анализа ПЦР-РВ для количественного анализа содержания микроРНК в качестве молекулярного биомаркера с целью диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
Для повышения точности проводимого анализа необходимо применять дополнительный положительный контрольный образец микроРНК, для получения которого требуется использовать методы молекулярного клонирования, т.е. конструирование рекомбинантной плазмидной ДНК, кодирующей микроРНК, используемую в качестве молекулярного биомаркера, обеспечивающей эффективную, высокую, надежную и стабильную амплификацию микроРНК.
Готовую плазмиду трансфицируют в клетки. Для эффективной трансфекции нуклеиновая кислота должна достичь ядра клетки. Однако трансфицированный генетический материал может быть разрушен в процессе доставки в цитоплазме. Таким образом, полученная рекомбинантная плазмидная ДНК должна быть стабильной.
Конструирование рекомбинантной плазмидной ДНК согласно настоящему изобретению
Основная задача при конструировании рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, для достижения эффективной амплификации микроРНК hsa-miR-143-3p с целью применения полученной рекомбинантной плазмидной ДНК в качестве положительного контроля при проведении ПЦР-РВ состоит в создании уникальной олигонуклеотидной вставки, содержащей кДНК для микроРНК hsa-miR-143-3p.
Для создания уникальной олигонуклеотидной вставки, позволяющей создать рекомбинантную плазмидную ДНК, кодирующую микроРНК hsa-miR-143-3p, для достижения эффективной амплификации микроРНК hsa-miR-143-3p с целью применения полученной рекомбинантной плазмидной ДНК в качестве положительного контроля при проведении ПЦР-РВ авторами настоящего изобретения в ходе проведенных трудоемких исследований создана уникальная пара праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3 для амплификации кДНК для микроРНК hsa-miR-143-3p.
МикроРНК hsa-miR-143-3p имеет последовательность, как представлено в SEQ ID NO:5.
Таким образом, на основе последовательности микроРНК hsa-miR-143-3p для синтеза олигонуклеотидной вставки проводили реакцию амплификации с применением разработанной пары праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3.
В результате сконструирована уникальная олигонуклеотидная вставка с последовательностью согласно SEQ ID NO:1.
Основным компонентом для высокопроцессивного клонирования является векторная система, позволяющая осуществлять трансфекцию таргетного фрагмента в клетку. Плазмиды представляют собой двухцепочечные кольцевые небольшие по размерам молекулы ДНК, которые способны к автономному процессу репликации. Указанные нуклеотидные последовательности содержат генные регуляторные элементы для управления механизмами экспрессии больших фрагментов ДНК.
МикроРНК представляют собой короткие некодирующие цепочки РНК, состоящие из последовательности, содержащей приблизительно 21-23 нуклеотида.
Несмотря на то что плазмидные векторы получили широкое распространение для применения в диагностических и терапевтических целях и созданы банки данных для плазмидных векторов, на сегодняшний день имеется крайне мало сведений о плазмидных векторах, подходящих для получения рекомбинантной плазмидной ДНК, кодирующей микроРНК, обеспечивающих эффективную амплификацию микроРНК. Таким образом, выбор плазмидного вектора для микроРНК является сложной задачей, требующей трудоемких исследований.
В результате трудоемких исследований авторы настоящего изобретения обнаружили, что в качестве плазмидного вектора для конструирования рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, можно использовать вектор pJET1.2, схема которого представлена на фиг. 2, что является неожиданным, поскольку ранее данный вектор не описан для получения рекомбинантной плазмидной ДНК, кодирующей микроРНК.
Вектор pJET1.2 может функционировать в бактериальных клетках E.coli и обладает сайтами рестрикции Eco47I.
Эндонуклеазы рестрикции представляют собой ферменты, которые определяют специфические участки в ДНК и вносят разрывы в сайте узнавания, либо на определенном расстоянии от него. При этом рестриктазы осуществляют гидролиз фосфодиэфирной связи в середине молекулы нуклеиновой кислоты, что важно для проведения молекулярного клонирования.
Авторы настоящего изобретения несмотря на имеющиеся предпосылки по выбору сайтов рестрикции обнаружили в ходе трудоемких экспериментов, что для получения наиболее эффективной рекомбинантной плазмидной ДНК для микроРНК hsa-miR-143-3p синтезированный авторами настоящего изобретения уникальный олигонуклеотид должен быть фланкирован сайтами рестрикции Eco47I.
Специалисту в данной области техники очевидно, что сведения уровня техники и проведенные предварительные исследования не могут гарантировать получение стабильной эффективной рабочей рекомбинантной плазмидной ДНК, обеспечивающей эффективную, высокую и надежную амплификацию молекулярного биомаркера. Успешное конструирование рекомбинантной плазмидной ДНК может быть подтверждено исключительно в рамках экспериментов.
В ходе проведенных экспериментов авторы настоящего изобретения подтвердили, что им удалось сконструировать эффективную и стабильную рекомбинантную плазмидную ДНК, кодирующую микроРНК hsa-miR-143-3p, обеспечивающую эффективную, высокую и надежную амплификацию микроРНК hsa-miR-143-3p, что позволяет применять ее в качестве эффективного и надежного положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и выявления риска их развития.
Таким образом, согласно первому аспекту настоящее изобретение относится к рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, причем рекомбинантная плазмидная ДНК содержит олигонуклеотидную вставку с последовательностью согласно SEQ ID NO:1, кодирующую микроРНК hsa-miR-143-3p, фланкированную последовательностями сайта рестрикции Eco47I, и причем рекомбинантная плазмидная ДНК имеет последовательность согласно SEQ ID NO:4 и расположение элементов, как представлено на фиг.1.
Полученная новая рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, имеет размер 3041 п.н.
Согласно варианту осуществления первого аспекта настоящего изобретения рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, основана на плазмиде pJET1, расположение элементов которой представлено на фиг. 2.
Разработанная авторами новая рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, предназначена для диагностики сердечно-сосудистых заболеваний и определения риска их развития, в частности в качестве положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития.
Согласно второму аспекту настоящее изобретение относится к применению рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, согласно первому аспекту настоящего изобретения в качестве положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития.
Настоящее изобретение также относится к уникальному набору праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3 для получения уникального олигонуклеотида согласно SEQ ID NO:1.
Технический результат настоящего изобретения состоит в следующем:
- получение новой стабильной рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p,
- расширение арсенала средств для диагностики сердечно-сосудистых заболеваний и определения риска их развития,
- достижение эффективной амплификации микроРНК hsa-miR-143-3p in vitro,
- повышение амплификации микроРНК hsa-miR-143-3p in vitro,
- достижение стабильной и надежной амплификации микроРНК hsa-miR-143-3p in vitro,
- разработка эффективного и надежного положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития,
- достижение высокой диагностической чувствительности,
- достижение высокой диагностической специфичности,
- достижение высокой прогностичности диагностического результата,
- достижение высокой прогностичности отрицательного результата.
Перечень последовательностей, представленных в настоящем документе
TATCTCAGTTCGGTGTAGGTCGTTCGCTCCAAGCTGGGCTGTGTGCACGAACCCCCCGTTCAGCCCGACCGCTGCGCCTTATCCGGTAACTATCGTCTTGAGTCCAACCCGGTAAGACACGACTTATCGCCACTGGCAGCAGCCACTGGTAACAGGATTAGCAGAGCGAGGTATGTAGGCGGTGCTACAGAGTTCTTGAAGTGGTGGCCTAACTACGGCTACACTAGAAGGACAGTATTTGGTATCTGCGCTCTGCTGAAGCCAGTTACCTTCGGAAAAAGAGTTGGTAGCTCTTGATCCGGCAAACAAACCACCGCTGG
TAGCGGTGGTTTTTTTGTTTGCAAGCAGCAGATTACGCGCAGAAAAAAAGGATCTCAAGAAGATCCTTTGATCTTTTCTACGGGGTCTGACGCTCAGTGGAACGAAAACTCACGTTAAGGGATTTTGGTCATGAGATTATCAAAAAGGATCTTCACCTAGATCCTTTTAAATTAAAAATGAAGTTTTAAATCAATCTAAAGTATATATGAGTAAACTTGGTCTGACAGTTACCAATGCTTAATCAGTGAGGCACCTATCTCAGCGATCTGTCTATTTCGTTCATCCATAGTTGCCTGACTCCCCGTCGTGTAGATAACTA
CGATACGGGAGGGCTTACCATCTGGCCCCAGTGCTGCAATGATACCGCGAGACCCACGCTCACCGGCTCCAGATTTATCAGCAATAAACCAGCCAGCCGGAAGGGCCGAGCGCAGAAGTGGTCCTGCAACTTTATCCGCCTCCATCCAGTCTATTAATTGTTGCCGGGAAGCTAGAGTAAGTAGTTCGCCAGTTAATAGTTTGCGCAACGTTGTTGCCATTGCTACAGGCATCGTGGTGTCACGCTCGTCGTTTGGTATGGCTTCATTCAGCTCCGGTTCCCAACGATCAAGGCGAGTTACATGATCCCCCATGTTGTGCAAAAAAGCGGTTAGCTCCTTCGGTCCTCCGATCGTTGTCAGAAGTAAGTTGGCCGCAGTGTTATCACTCATGGTTATGGCAGCACTGCATAATTCTCTTACTGTCATGCCATCCGTAAGATGCTTTTCTGTGACTGGTGAGTACTCAACCAAGTCATTCTGAGAATAGTGTATGCGGCGACCGAGTTGCTCTTGCCCGGCGTCAATACGGGATAATACCGCGCCACATAGCAGAACTTTAAAAGTGCTCATCATTGGAAAACGTTCTTCGGGGCGAAAACTCTCAAGGATCTTACCGCTGTTGAGATCCAGTTCGATGTAACCCACTCGTGCACCCAACTGATCTTCAGCATCTTTTACTTTCACCAGCGTTTCTGGGTGAGCAAAAACAGGAAGGCAAAATGCCGCAAAAAAGGGAATAAGGGCGACACGGAAATGTTGAATACTCATACTCTTCCTTTTTCAATATTATTGAAGCATTTATCAGGGTTATTGTCTCATGAGCGGATACATATTTGAATGTATTTAGAAAAATAAACAAATAGGGGTTCCGCGCACATTTCCCCGAAAAGTGCCACCTGACGTCTAAGAAACCATTATTATCATGACATTAACCTATAAAAATAGGCGTATCACGAGGCC
Примеры
Пример 1. Получение рекомбинантной плазмидной ДНК согласно настоящему изобретению
Авторами настоящего изобретения создана уникальная пара праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3 для амплификации кДНК для микроРНК hsa-miR-143-3p.
Для синтеза олигонуклеотидной вставки на основе последовательности микроРНК hsa-miR-143-3p согласно SEQ ID NO:5 проводили реакцию амплификации с применением разработанной пары праймеров hsa-miR-143-3p_F с последовательностью согласно SEQ ID NO:2 и hsa-miR-143-3p_R с последовательностью согласно SEQ ID NO:3.
В результате амплификации получали ампликоны прогнозируемой длины 72 п.н. на каждой температуре отжига праймеров. Результаты представлены на фиг. 3.
Далее полоски, содержащие целевые фрагменты, вырезали скальпелем, и ДНК из указанных полосок выделяли с помощью набора QIAEX II Gel Extraction Kit (500).
В результате сконструировали уникальную олигонуклеотидную вставку с последовательностью согласно SEQ ID NO:1, содержащую кДНК для микроРНК hsa-miR-143-3p, для вставки в плазмидный вектор.
Ампликон кДНК обрабатывали фрагментом Кленова для затупления концов продукта и дальнейшего лигирования в место разрезания вектора pJET1.2.
Проводили лигирование полученной олигонуклеотидной вставки согласно SEQ ID NO:1 в вектор pJET1.2 (приобретен у Thermo Fisher Scientific) при помощи Т4 лигазы (приобретена у Thermo Fisher Scientific) по сайту эндонуклеазной рестрикции Eco47I, которая вносит разрыв с тупыми концами в месте разрезания.
Лигирование происходило при 14°С в течение 14-16 часов в термоциклере. Полученный продукт лигирования убирали на -20°С до трансформации E.coli.
Пример 2. Трансформация E.coli:
Три пробы использовали для лигирования: 2 пробы содержали компетентные клетки Dh5@, и одна проба содержала компетентные клетки NEB stable.
Три аликвоты по 10 мкл продуктов лигирования добавляли к компетентным клеткам и следовали следующему протоколу трасформации: 30 минут при 0°С, далее 1 минута при 42°С и затем образцы снова инкубировали 1 минуту при 0°С. Далее смесь продуктов лигирования и компетентных клеток добавляли в 1 мл подогретой до 37°С LB-среды без антибиотика. Клетки культивировали в LB-среде 1 час при 37°С.
После 30-секундного центрифугирования при 16000 g супернатант почти полностью удаляли, а осадок клеток с 100 мкл супернатанта переносили в чашки Петри на твёрдую агарозную среду содержащую агар-агар + 50 мкг ампициллина/мл среды + ИПТГ. Чашки Петри инкубировали 1-2 дня при 37°С.
Пример 3. Проведение полимеразной цепной реакции в реальном времени с применением рекомбинантной плазмидной ДНК согласно настоящему изобретению
После инкубирования в чашках Петри получившиеся колонии переносили в отдельные пробирки с 10 мкл mQ. Для скринингового ПЦР-теста отбирали 15 колоний и приготавливали необходимый набор праймеров hsa-miR-143-3p_F и hsa-miR-143-3p_R.
Результаты ПЦР-теста представлены на фиг. 4. По результатам ПЦР-теста полученные колонии были положительными и содержали ПЦР-продукт длиной 72 п.н. при использовании праймеров hsa-miR-143-3p_F и hsa-miR-143-3p_R по 10 мкМ.
Анализ ПЦР-РВ с применением рекомбинантной плазмидной ДНК согласно настоящему изобретению, полученной согласно примеру 1, в соответствии с методикой, описанной в статье Е.В. Воропаев и др., Принципы организации работы с нуклеиновыми кислотами в ПЦР-лаборатории, Журнал Гродненского государственного медицинского университета, № 4, 2012 г.
Полученные результаты подтвердили, что рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, для диагностики сердечно-сосудистых заболеваний и определения риска их развития, содержащая олигонуклеотидную вставку с последовательностью согласно SEQ ID NO:1, кодирующую микроРНК hsa-miR-143-3p, фланкированную последовательностями сайта рестрикции Eco47I, и имеющая последовательность согласно SEQ ID NO:4 и расположение элементов, как представлено на фиг.1, является эффективным и надежным положительным контролем, обеспечивающим высокую и надежную амплификацю, для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="плазмидная
ДНК1.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2025-02-25">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>hsa-miR-143-3p</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное научное учреждение «Российский научный центр хирургии имени
академика Б.В. Петровского»</ApplicantName>
<ApplicantNameLatin>Federalnoe gosudarstvennoe biudzhetnoe nauchnoe
uchrezhdenie Rossiiskii nauchnyi tsentr khirurgii imeni akademika
B.V. Petrovskogo</ApplicantNameLatin>
<InventionTitle languageCode="ru">РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК,
КОДИРУЮЩАЯ МИКРОРНК HSA-MIR-143-3P, И ЕЕ ПРИМЕНЕНИЕ В КАЧЕСТВЕ
ПОЛОЖИТЕЛЬНОГО КОНТРОЛЯ ПРИ ПЦР-РВ ДЛЯ ДИАГНОСТИКИ
СЕРДЕЧНО-СОСУДИСТЫХ ЗАБОЛЕВАНИЙ И ВЫЯВЛЕНИЯ РИСКА ИХ
РАЗВИТИЯ</InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>67</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..67</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggtccgaggtattcgcactggatacgacgagct
agatgaagcactgtagct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagagcgttgagatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>3041</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..3041</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcccctgcagccgaattatattatttttgccaaataatttttaacaaaa
gctctgaagtcttcttcatttaaattcttagatgatacttcatctggaaaattgtcccaattagtagcat
cacgctgtgagtaagttctaaaccatttttttattgttgtattatctctaatcttactactcgatgagtt
ttcggtattatctctatttttaacttggagcaggttccattcattgtttttttcatcatagtgaataaaa
tcaactgctttaacacttgtgcctgaacaccatatccatccggcgtaatacgactcactatagggagagc
ggccgccagatcttccggatggctcgagtttttcagcaagatgtcgtatccagtgcagggtccgaggtat
tcgcactggatacgacgagctagatgaagcactgtagctatctttctagaagatctcctacaatattctc
agctgccatggaaaatcgatgttcttcttttattctctcaagattttcaggctgtatattaaaacttata
ttaagaactatgctaaccacctcatcaggaaccgttgtaggtggcgtgggttttcttggcaatcgactct
catgaaaactacgagctaaatattcaatatgttcctcttgaccaactttattctgcattttttttgaacg
aggtttagagcaagcttcaggaaactgagacaggaattttattaaaaatttaaattttgaagaaagttca
gggttaatagcatccattttttgctttgcaagttcctcagcattcttaacaaaagacgtctcttttgaca
tgtttaaagtttaaacctcctgtgtgaaattgttatccgctcacaattccacacattatacgagccggaa
gcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacattaattgcgttgcgctcactgcc
aattgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcggccaacgcgcggggagaggc
ggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggc
gagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaa
catgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccatagg
ctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactat
aaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccgg
atacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagt
tcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgcct
tatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactgg
taacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggc
tacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggta
gctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcg
cagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaac
tcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaaat
gaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtga
ggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataact
acgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctc
cagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgc
ctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaac
gttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccggtt
cccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctcc
gatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctctt
actgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagt
gtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaacttt
aaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatcc
agttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgggt
gagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcat
actcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaa
tgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgacgtctaag
aaaccattattatcatgacattaacctataaaaataggcgtatcacgaggcc</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other RNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>uracil</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagatgaagcactgtagctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-3p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837873C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837872C1 |
| Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-145-5p, и ее применение в качестве положительного контроля при ПЦР-РВ для диагностики сердечно-сосудистых заболеваний и выявления риска их развития | 2025 |
|
RU2837874C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-145-3p методом количественной ПЦР и его применение | 2025 |
|
RU2838260C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-143-3p методом количественной ПЦР и его применение | 2025 |
|
RU2838257C1 |
| Набор олигодезоксирибонуклеотидных праймеров для диагностики сердечно-сосудистых заболеваний и выявления риска их развития на основе биомолекулярного маркера микроРНК hsa-miR-143-5p методом количественной ПЦР и его применение | 2025 |
|
RU2838258C1 |
| Тест-система "miR-M-SCREEN" для прогнозирования развития метастазов у больных колоректальным раком на основании уровня микро-РНК miR-26a и miR-143 в плазме крови | 2022 |
|
RU2786386C1 |
| Способ прогнозирования привычной невынашиваемости беременности по экспрессии генов микроРНК в биопсийном материале эндометрия | 2025 |
|
RU2837879C1 |
| ВАРИАНТЫ РЕКОМБИНАНТНЫХ AAV И ИХ ПРИМЕНЕНИЯ | 2015 |
|
RU2738421C2 |
| Набор реагентов для определения малых рибонуклеиновых кислот (микроРНК) при диагностике множественной миеломы | 2024 |
|
RU2836054C1 |
Настоящее изобретение относится к области биотехнологии и молекулярной биологии. Описана рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, причем рекомбинантная плазмидная ДНК содержит олигонуклеотидную вставку с последовательностью согласно SEQ ID NO:1, кодирующую микроРНК hsa-miR-143-3p, фланкированную последовательностями сайта рестрикции Eco47I, и причем рекомбинантная плазмидная ДНК имеет последовательность согласно SEQ ID NO:4 и расположение элементов, как представлено на фиг.1. Также описано применение рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, по п. 1 в качестве положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития. Технический результат заключается в расширении арсенала средств диагностики сердечно-сосудистых заболеваний и определения риска их развития. 2 н.п. ф-лы, 4 ил., 1 табл., 3 пр.
1. Рекомбинантная плазмидная ДНК, кодирующая микроРНК hsa-miR-143-3p, причем рекомбинантная плазмидная ДНК содержит олигонуклеотидную вставку с последовательностью согласно SEQ ID NO:1, кодирующую микроРНК hsa-miR-143-3p, фланкированную последовательностями сайта рестрикции Eco47I, и причем рекомбинантная плазмидная ДНК имеет последовательность согласно SEQ ID NO:4 и расположение элементов, как представлено на фиг. 1.
2. Применение рекомбинантной плазмидной ДНК, кодирующей микроРНК hsa-miR-143-3p, по п. 1 в качестве положительного контроля для проведения ПЦР-РВ с целью диагностики сердечно-сосудистых заболеваний и определения риска их развития.
| Gonzalez-Lopez P | |||
| et al | |||
| Канатное устройство для подъема и перемещения сыпучих и раздробленных тел | 1923 |
|
SU155A1 |
| International journal of molecular sciences, 2022, v | |||
| Прибор для равномерного смешения зерна и одновременного отбирания нескольких одинаковых по объему проб | 1921 |
|
SU23A1 |
| Способ прогнозирования развития рецидива у больных с вирусом папилломы человека и немышечно-инвазивным раком мочевого пузыря | 2022 |
|
RU2800265C1 |
| WO 2023105297 | |||
Авторы
Даты
2025-04-07—Публикация
2025-02-25—Подача