Изобретение относится к области медицины, в частности к клинической лабораторной диагностике, и предназначено для одновременного количественного определения микроРНК hsa-miR-16-5p и hsa-miR-21-5p в плазме венозной крови при диагностике множественной миеломы (ММ) с использованием молекулярно-генетических методов одновременного определения уровня экспрессии микроРНК в мультиплексной полимеразной цепной реакции в режиме «реального времени». Набор реагентов позволяет определить количество молекул микроРНК hsa-miR-16-5p и hsa-miR-21-5p в плазме крови. Соотношение их концентрации более 0,20 характерно для пациентов с множественной миеломой, в том числе с несекретирующим вариантом заболевания.
Согласно клиническим рекомендациям [Клинические рекомендации. Множественная миелома. Под ред. Поддубной И. В., Савченко В. Г., Каприна А. Д. – 2020.], основным лабораторным критерием множественной миеломы является обнаружение высокого уровня моноклонального иммуноглобулина, а также морфологическое и иммуногистохимическое исследование биоптата костного мозга и/или образцов вне костномозгового опухолевого субстрата. В случае отсутствия секреции опухолевыми клетками моноклональных иммуноглобулинов («несекретирующая» ММ), лабораторное подтверждение диагноза проводится путем исследования опухолевых клеток в биоптатах. Пациентам, с целью диагностики высокого риска тяжелого течения ММ, также рекомендуется проведение генетических тестов, таких как кариотипирование и флуоресцентная гибридизация in situ (FISH-метод), выявляющих нарушения количества и структуры хромосом, в том числе транслокации t(4;14), t(14;16), t(6;14), del17p13, t(11;14), del13. Однако, для выполнения данных анализов, необходимо получить достаточное количество опухолевых клеток с использованием инвазивных манипуляций и, кроме этого, результаты исследования не позволяют проводить количественную оценку.
Среди молекулярно-генетических маркеров, концентрация которых меняется в образцах костного мозга при ММ ранее описаны молекулы микроРНК hsa-miR-16-5p [Li F. et al. MicroRNA-15a/16-1 cluster located at chromosome 13q14 is down-regulated but displays different expression pattern and prognostic significance in multiple myeloma. Oncotarget. 2015. 6(35): 38270] и микроРНК hsa-miR-21-5p [Wang X. et al. Myeloma cell adhesion to bone marrow stromal cells confers drug resistance by microRNA-21 up-regulation. Leukemia & lymphoma. 2011. 52(10): 1991-1998].
Технологии секвенирования нуклеиновых кислот потенциально могут обнаружить несколько тысяч различных молекул РНК [Precazzini F, Detassis S, Imperatori AS, Denti MA, Campomenosi P. Measurements Methods for the Development of MicroRNA-Based Tests for Cancer Diagnosis. International Journal of Molecular Sciences. 2021. 22(3): 1176]. Альтернативная технология микрочипов также представляет собой высокопроизводительный метод, используемый для одновременного обнаружения широкого спектра РНК. Вместе с тем, классическое секвенирование по Сэнгеру, пиросеквенирование и микрочипы не обладают достаточной чувствительностью для количественного определения отдельных молекул РНК в тканях и биологических жидкостях человека. Для количественного высокочувствительного определения мРНК и микроРНК используются методы цифровой ПЦР, основанной на последовательном разведении образца во множестве отдельных капель, в которых проводятся реакции амплификации. Максимальное разведение образца, в котором еще детектируется, результат реакции отражает количество исходных молекул нуклеиновых кислот. Кроме того, для анализа РНК можно использовать высокочувствительное секвенирование «нового поколения» [Agnelli L. et al. Expanding the repertoire of miRNAs and miRNA-offset RNAs expressed in multiple myeloma by small RNA deep sequencing. Blood Cancer Journal. 2019. 9(3): 21].
К недостаткам всех вышеуказанных методов относятся отсутствие стандартизации, дорогостоящие оборудование и реактивы, технологическая сложность, трудоемкость и длительность выполнения тестирования, высокие требования к квалификации персонала, что повышает стоимость тестирования и ограничивает широкое использование данных методов в реальной практике клинико-диагностических лабораторий.
Альтернативным и широкодоступным вариантом количественного определения уровня отдельных молекул нуклеиновых кислот является полимеразная цепная реакция с детекцией результатов в режиме «реального времени» (ПЦР-РВ). Преимуществом ПЦР-РВ, наряду с высокой чувствительностью и специфичностью, является возможность использования широко распространенного стандартного оборудования для выполнения анализа в клинико-диагностических лабораториях медицинских организаций, что позволяет обеспечить большую доступность диагностики для населения.
Из практики лабораторных исследований также известно, что проведение ПЦР в мультиплексном варианте определения разных маркеров в одной аналитической постановке имеет ряд преимуществ перед ПЦР в отдельных аналитических сериях для каждого отдельного маркера, а именно: снижение риска контаминации образцов, уменьшение расхода реактивов, сокращение времени проведения анализа и постановки диагноза. Кроме того, стандартизованное одновременное определение отдельных маркеров позволяет оценивать дополнительные диагностические критерии, основанные на соотношении их концентраций более точно, чем при отдельном анализе маркеров в разных тест системах.
Учитывая способность клеток к высвобождению в плазму крови молекул микроРНК, а также их относительно высокую стабильность, описаны диагностические тесты, основанные на измерении уровня микроРНК в плазме венозной крови.
На сегодняшний день известны изобретения, основанные на анализе ряда микроРНК методом количественной ПЦР в реальном времени в плазме периферической крови человека, предназначенные для диагностики серозных пограничных цистаденом и цистаденокарценом яичника [Патент № 2688169 Российская федерация, МПК G 01 N 33/574, C 12Q 1/68. Количественная оценка hsa-miR-16-5p, hsa-miR-425-5p, hsa-miR-17-5p, hsa-miR-20a-5p, hsa-miR-101-3p, hsa-miR-30d-5p и hsa-miR-93-5p в плазме периферической крови женщин как способ неинвазивной диагностики серозных пограничных цистаденом и цистаденокарценом яичника : № 2018129534 : заявл. 14.08.2018 : опубл. 08.07.2019 / Тимофеева А.В., Чаговец В.В., Асатурова А.В. и др. – 25 с.] и выявления ранних стадий колоректального рака [Патент № 2611189 Российская Федерация, МПК C 12 Q 1/68. Определение микроРНК в плазме для обнаружения ранних стадий колоректального рака: № 2014120427 : заявл. 20.10.2012 : опубл. 21.02.2017 / Хиронелья И Кос Меритксель, Лосано Сальвателья Хуан Хосе, Кастельс И Гарангоу Антони и др. – 49 с.], однако данные изобретения не направлены на диагностику онкогематологических заболеваний, а также не включают возможность проведения анализа в мультиплексном режиме. Также известно о изобретении, предназначенном для выбора терапии для субъекта, имеющего B-клеточную неоплазию, при этом способ включает выявление с помощью количественной ПЦР или микроматричного анализа микроРНК сниженной экспрессии miR-629 [Патент № 2016138431 Российская Федерация, МПК C 12 Q 1/68. Композиции и способы для идентификации B-клеточных неоплазий, реагирующих на терапию посредством истощения популяции B-клеток : № 2016138431 : заявл. 04.03.2015 : опубл. 05.04.2018 / Кудзиора Майкл, Яо Ихун, Ранад Коустубх, Брохаун Филип З., Стрейчер Кати – 3 с.], однако данный способ предусматривает анализ лишь одной микроРНК для конкретной цели отбора пациентов, подходящих для терапии препаратами антител к CD19.
До сих пор не известно об использовании мультиплексных технологий ПЦР-РВ для одновременного количественного определения микроРНК hsa-miR-16-5p и hsa-miR-21-5p в плазме венозной крови при диагностике ММ.
Наиболее близким к заявляемому изобретению по совокупности признаков является набор реагентов для определения рибонуклеиновых кислот при диагностике лимфопролиферативных заболеваний для одновременного определения экспрессии мРНК генов ABL1 и мРНК ROR1, а также микроРНК miR-39, miR-155 и miR-15а для диагностики хронического лимфоцитарного лейкоза (ХЛЛ) [Патент № 2802064 Российская Федерация, МПК C 12 Q 1/68. Набор реагентов для определения рибонуклеиновых кислот при диагностике лимфопролиферативных заболеваний : № 2023100712 : заявл. 16.01.2023 : опубл. 22.08.2023 / Ольховский И.А., Горбенко А.С., Столяр М.А., Бахтина В.И. – 15 с. (прототип)].
Описанный вариант набора предусматривает выполнение полимеразной цепной реакции в реальном времени с использованием специфических праймеров для определения miR-15a и miR-155, при этом miR-155 детектируется одновременно с miR-39 в мультиплексной режиме посредством использования одной реакционной смеси. В качестве источника флуоресценции при регистрации уровня miR-155 и miR-39 используется TaqMan зонды на 5' конце применяют краситель флуоресцеин (FAM) и гексахлорфлуоресцеин (HEX), а для тушения флуоресценции используют Black Hole Quencher-1 (BHQ1) на 3' конце. Для регистрации уровня miR-15a используется оценка флуоресценции интеркалирующий красителя Eva Green.
Предметом настоящего изобретения является набор реактивов для мультиплексной ПЦР-РВ с целью одновременного обнаружения микроРНК hsa-miR-16-5p, hsa-miR-21-5p и cel-miR-39-3p в образцах плазмы венозной крови, что ранее не применялось в практике клинической лабораторной диагностики ММ.
Отличие заявляемого изобретения от прототипа заключается в том, что прототип не направлен на диагностику ММ, он не предусматривает возможность выявления микроРНК miR-16 и miR-21, а также в том, что в заявляемом изобретении в качестве флуорофоров для всех измеряемых микроРНК используются исключительно TaqMan зонды, имеющие большую специфичность за счет связывания с целевой последовательностью нуклеиновых кислот, чем описанные в прототипе интеркалирующие красители, которые связываются с любой двухцепочечной ДНК в ходе реакции ПЦР. Среди используемых реактивов в наборе прототипа нет стандартных образцов микроРНК с известным количественным уровнем (копий в мл), что исключает возможность построения калибровочных графиков для выполнения дальнейших количественных расчетов.
Технический результат заключается в возможности осуществлять все этапы выполнения анализа в одной аналитической серии.
Технический результат достигается тем, что в набор для диагностики множественной миеломы, включены все необходимые реактивы для выделения микроРНК, выполнения обратной транскрипции и мультиплексной амплификации. При этом для проведения реакции обратной транскрипции микроРНК (5'-3'-): используются следующие петлевые праймеры:
miR39-SL: GAAAGAAGGCGAGGAGCAGATCGAGGAAGAAGACGGAAGAATGTGCGTCTCGCCTTCTTTCCAAGCTGA (SEQ ID №10);
miR16-SL:
GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACCGCCAA (SEQ ID №11);
miR21-SL: CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTCAACAT (SEQ ID №12).
а для амплификации используются оригинальные комбинации специфических праймеров и зондов:
miR39-P: HEX-CGTCTCGCCTTCTTTCCA-BHQ1 (SEQ ID №1);
miR39-R: CGAGGAAGAAGACGGAAGAAT (SEQ ID №2);
miR39-F: GGTCACCGGGTGTAAATC (SEQ ID №3);
miR16-P: FAM-ACTGGATACGACCGCCAATA-BHQ1 (SEQ ID №4);
miR16-R: GTCGTATCCAGTGCAGGGT (SEQ ID №5);
miR16-F: AACAGTGTAGCAGCACGTAAA (SEQ ID №6);
miR21-P: FAM-GGCAATTCAGTTGAGTCAACA-BHQ1 (SEQ ID №7);
miR21-R: CTCAACTGGTGTCGTGGAGTC (SEQ ID №8);
miR21-F: TGTCCGCCTAGCTTATCAGAC (SEQ ID №9).
Заявленный вариант комплектации набора реагентов, рассчитанный на 50 реакций, включает флаконы и пробирки, содержание:
1. Реагенты для выделения РНК фенол-хлороформным методом (таблица 1):
а. Раствор 1 – 20 мл;
б. Раствор 2 – 40 мл;
в. Раствор 3 – 14 мл;
г. Раствор 4 – 45 мл;
д. Раствор 5 – 20 мл
е. раствор 6 – 1 мл;
ж. Раствор 7 – 50 мл;
з. Раствор 8 – 40 мл;
и. Раствор 9 – 8 мл;
2. две реакционные смеси для выполнения специфической обратной транскрипции микроРНК – «МикроРНК-39-21-ОТ» и «МикроРНК-16-ОТ» (таблица 2);
3. Ревертазу для проведения обратной транскрипции (5 ед/мкл) – 50 мкл;
4. реакционную смесь для определения экспрессии микроРНК cel-miR-39-3p и hsa-miR-21-5p (39-21-РВ) методом ПЦР-РВ - 1200 мкл (таблица 3);
5. реакционную смесь для определения экспрессии микроРНК hsa-miR-16-5p (16-РВ) методом ПЦР-РВ - 1200 мкл, (таблица 3);
6. ДНК-полимеразу Thermus aquaticus 5 ед/мкл (Taq-полимераза) - 100 мкл;
7. положительные контрольные образцы (ПКО) (стандартная кДНК hsa-miR-16-5p (4,8*108 копий/мкл), hsa-miR-21-5p и cel-miR-39-3p (4,8*109 копий/мкл)). Объем каждого ПКО – 500 мкл;
8. калибраторы (5 шт на каждую микроРНК: стандартные растворы кДНК hsa-miR-16-5p (4,8*109 копий/мкл, 4,8*108 копий/мкл, 4,8*107 копий/мкл, 4,8*106 копий/мкл, 4,8*105 копий/мкл), hsa-miR-21-5p и cel-miR-39-3p (1,75*109 копий/мкл, 1,75*108 копий/мкл, 1,75*107 копий/мкл, 1,75*106 копий/мкл, 1,75*105 копий/мкл)) для построения калибровочных графиков. Объем каждого калибратора – 25 мкл;
9. стандартный раствор микроРНК cel-miR-39-3p с концентрацией 109 копий/мкл;
10. отрицательный контрольный образец (ОКО, не содержит нуклеиновых кислот) – 500 мкл;
11. Колонки и собирательные пробирки для выделения РНК – 50 шт.
Положительные контрольные образцы (ПКО) представляют из себя кДНК микроРНК hsa-miR-16-5p и hsa-miR-21-5p. Отрицательный контрольный образец (ОКО) представляет собой деионизированную воду.
Таблица 1. Состав реактивов для выделения микроРНК и мРНК из образца крови
Таблица 2. Состав реакционных смесей на выполнение одной реакции обратной транскрипции микроРНК cel-miR-39, hsa-miR-16 и hsa-miR-21
Таблица 3. Состав реакционных смесей на выполнение одной реакции амплификации и детекции cel-miR-39, miR-21 и miR-16 в ПЦР-РВ
miR-21
Таблица 4. Нуклеотидные последовательности стандартных микроРНК
Набор позволяет определять микроРНК из одного образца крови объемом от 1,2 мл. При этом используются широко распространенные в клинико-диагностических лабораториях приборы и оборудование для ПЦР, что обеспечивает доступность тестирования для населения. Использование данного набора приводит к сокращению рабочего времени на выполнение лабораторных тестов и экономии расходных материалов в сравнении с раздельным использованием диагностических наборов для выявления отдельных молекул РНК.
Новым является то, что в набор для определения микроРНК дополнительно включены оригинальные реагенты для выделения микроРНК и реагенты для обратной транскрипции со специфическими петлевыми праймерами, а мультиплексную ПЦР-РВ проводят одновременно для двух маркеров, используя во всех реакционных смесях в качестве флуорофора TaqMan зонды.
Пример использования набора для выполнения анализа и оценки диагностического значения микроРНК miR-16 и miR-21 при множественной миеломе.
Образцы крови получали из локтевой вены и стабилизируют раствором ЭДТА К2 в объемном отношении 9:1. В процессе исследования кровь сохраняли в закрытых пластиковых пробирках при комнатной температуре не более 3 часов, с соблюдением общепринятых мер предосторожности при работе с венозной кровью и её компонентами. Для получения плазмы пробирку с венозной кровью центрифугировали при 3000 об/мин в течение 10 минут. Из надосадочной жидкости, которая представляет собой плазму крови, отбирали 200 мкл в чистую пробирку. Дальнейший анализ выполняли с полученной плазмой с использованием заявляемого набора реактивов в соответствии с технологическими этапами выполнения анализа, подробно представленных в таблице 5. В процессе выделения производится добавление внутреннего контроля (микроРНК cel-miR-39-3p) в исследуемую пробу для дополнительного контроля эффективности всех этапов анализа.
Таблица 5. Описание этапов определения уровня микроРНК с помощью заявляемого набора реагентов
Удаление фильтрата
Удаление фильтрата
Удаление фильтрата
Перенос колонки на пробирку
150 мкл
95°С - 5 минут; 95°С - 15 секунд, 58°С - 60 секунд (цикл повторить 50 раз).
Полученные данные – кривые накопления флуоресцентного сигнала и определение пороговых циклов (Ct) рассчитывают с помощью программного обеспечения используемого прибора для проведения ПЦР в режиме «реального времени» в соответствии с инструкцией к прибору.
Уровень экспрессии рибонуклеиновых кислот определяют в соответствии с калибровочными графиками для hsa-miR-16-5p, hsa-miR-21-5p и cel-miR-39-3p.
Коэффициент miR-21/miR-16 вычисляют по формуле:
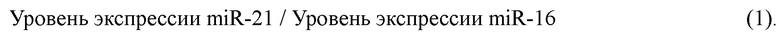
Интерпретация результатов возможна при выполнении следующих условий:
1) Ct микроРНК miR-39 для образца кДНК находится в диапазоне от 17 до 23 циклов.
2) Ct микроРНК miR-16 для образца ПКО находится в диапазоне от 16 до 19 циклов.
3) Ct микроРНК miR-21 для образца ПКО находится в диапазоне от 19 до 22 циклов.
4) Ct для всех микроРНК для ОКО не должен определяться.
Результаты тестирования клинических образцов крови представлены в таблице 6.
Таблица 6. Пример полученных результатов с использованием заявляемого набора реагентов при обследовании пациентов с ММ и группы контроля
miR-21/miR-16
miR-21/miR-16
Использование заявляемого набора позволило обнаружить увеличение уровня микроРНК miR-16 и снижение уровня микроРНК miR-21 в плазме образцов венозной крови пациентов с ММ. Коэффициент «К» отношения уровня экспрессии микроРНК miR-21 и микроРНК miR-16 у всех пациентов с диагнозом ММ значительно выше максимальной величины у доноров (0,20). Широкий диапазон вариации выявляемых РНК маркеров у пациентов с ММ не только позволяет выявить заболевание, но также представляет возможность оценить индивидуальные эпигенетические особенности опухолевых клеток и прогноз эффективности разных схем лечения.
Интерпретация результатов: при значениях коэффициента miR-21/miR-16 больше 0,20 с достоверностью 95% пациент относится к группе пациентов с ММ.
Пример использования набора при несекретирующей ММ
Пациентка ЛНА, 1952 г рождения. Клиническая манифестация заболевания с января 2023 г. - после осевой нагрузки на позвоночнике появились боли постоянные боли в спине. Лечилась самостоятельно без эффекта. Двигательная активность прогрессивно ухудшалось, вплоть до невозможности самостоятельно встать с кровати к июлю 2023 г. С 22.06 по 29.06.23 г. находилась на лечении в КМКБ №7 по поводу дорсопатии. 17.07.23 г обратилась в поликлинику по м/ж: лабораторно отрицательная динамика в виде нарастания уровня креатинина с 369 до 639, мочевины с 15.9 до 25.9 ммоль/л. Выписано направление на госпитализацию в нефрологическое отделение БСМП. С 20.07 по 03.08.23 г. пациентка находилась на госпитализации в отделении нефрологии КГБУЗ БСМП, где возникло подозрение на гематологическое заболевание. При поступлении в гематологическое отделение Красноярской краевой больницу клинический анализ крови - лейкоциты 23.8 тыс (с/я 6.2, лимфоциты 14.3, моноциты 1.7 тыс), эритроциты 2.72 млн, гемоглобин 89 г/л, тромбоциты 151 тыс. СОЭ 54 мм/час; - биохимический анализ крови - общий белок 61.6 г/л, мочевая к-та 630 мкмоль/л; - суточная потеря белка 0.56 г/сут. При электрофорезе патологические градиенты иммуноглобулинов сыворотки крови не обнаружены. 03.08.23 г. был выполнен анализ крови на определение микроРНК miR-16 и микроРНК miR-21 с использованием заявляемого набора реагентов. В результате получено значение коэффициента miR-21/miR-16 более 0,20, что соответствует уровню, характерному для множественной миеломы. Была выполнена стернальная пункция, где обнаружено 33% плазматических клеток. На основании этого результата был верифицирован диагноз симптоматической ММ. Инициирован 1 курс VCD. Проведена повторная стернальная пункция 14.09.2023, в результате молекулярно-цитологического исследования: в 5-7% клеток выявлена транслокация t(11;14) (q13 q23) с делецией 11q13 обнаружены делеция 13q14-q34, 16q23 (MAF), трипликации 1q21, делеция гена T353 и MYC.
Окончательный диагноз: Симптоматическая множественная миелома, не секретирующая моноклональный парапротеин, с распространенным остеодеструктивным процессом, IIIB стадия по Durie, S.Salmon) III ст. по ISS, III ст. по R-ISS, с транслокацией t(11;14) (q13 q23) с делецией 11q13 обнаружены делеция 13q14-q34, 16q23 (MAF), трипликации 1q21, делеция гена TP53 и MYC, впервые выявленная от 03.08.2023 г.
Таким образом, использование заявляемого набора реактивов способствовало своевременной постановке диагноза несекретирующая множественная миелома.
Сопоставительный анализ заявляемого набора реактивов с прототипом позволил выявить совокупность существенных по отношению к техническому результату отличительных признаков, изложенных в формуле. Следовательно, изобретение соответствует критерию «новизна».
Из литературы и практики молекулярно-генетических исследований в клинической лабораторной диагностике неизвестно о способе ПЦР-анализа для одновременного выявления экспрессии микроРНК hsa-miR-16-5p и hsa-miR-21-5p в плазме из одного образца крови при диагностике ММ, что позволяет сделать вывод о соответствии заявляемого решения критерию «изобретательский уровень». Преимуществом использования набора также является возможность выявления не секретирующих вариантов ММ, когда в периферической крови не удается выявить характерные моноклональные иммуноглобулины.
Отличие заявляемого набора реактивов от прототипа заключается в том, что:
1. Набор предусматривает возможность одновременной детекции микроРНК miR-39, miR-16 и miR-21 методом ПЦР в реальном времени.
2. Набор содержит во всех реакционных смесях в качестве флуорофора TaqMan зонды.
3. Набор содержит стандартные микроРНК в нескольких разведениях, предназначенные для построения калибровочных графиков для выполнения дальнейших расчетов.
4. Набор позволяет выполнять молекулярно-генетические исследования для диагностики ММ, в том числе и не секретирующем варианте заболевания.
При проведении тестирования использовалось стандарное оборудование клинико-диагностических лабораторий для ПЦР анализа, время проведения анализа составило 3 часа 40 мин для определения всех изучаемых молекул микроРНК.
Таким образом, в приведенном примере подтвержден заявленный технический результат, что свидетельствует о диагностических возможностях заявленного набора реактивов для диагностики множественной миеломы.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd" PUBLIC
"-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2024-05-10" softwareVersion="2.3.0"
softwareName="WIPO Sequence" fileName="sequence.xml" dtdVersion="V1_3"
nonEnglishFreeTextLanguageCode="ru" originalFreeTextLanguageCode="ru">
<ApplicationIdentification>
<IPOfficeCode/>
<ApplicationNumberText/>
<FilingDate>2024-05-10</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>sequence</ApplicantFileReference>
<ApplicantName languageCode="ru">Тимерман Анна Дмитриевна</ApplicantName>
<ApplicantNameLatin>Timerman Anna Dmitrievna</ApplicantNameLatin>
<InventionTitle languageCode="ru">Набор реагентов для определения
малых рибонуклеиновых кислот (микроРНК) при диагностике
множественной миеломы</InventionTitle>
<SequenceTotalQuantity>12</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>hex</INSDQualifier_value>
<NonEnglishQualifier_value>hex</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>bhq1</INSDQualifier_value>
<NonEnglishQualifier_value>bhq1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtctcgccttctttcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgaggaagaagacggaagaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtcaccgggtgtaaatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>fam</INSDQualifier_value>
<NonEnglishQualifier_value>fam</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>bhq1</INSDQualifier_value>
<NonEnglishQualifier_value>bhq1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>actggatacgaccgccaata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacagtgtagcagcacgtaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>fam</INSDQualifier_value>
<NonEnglishQualifier_value>fam</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>modified_base</INSDFeature_key>
<INSDFeature_location>21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mod_base</INSDQualifier_name>
<INSDQualifier_value>OTHER</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>bhq1</INSDQualifier_value>
<NonEnglishQualifier_value>bhq1</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggcaattcagttgagtcaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcaactggtgtcgtggagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtccgcctagcttatcagac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>69</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..69</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q49">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>stem_loop</INSDFeature_key>
<INSDFeature_location>1..61</INSDFeature_location>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaagaaggcgaggagcagatcgaggaagaagacggaagaatgtgcgtctcgccttctttccaagctga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>50</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..50</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>stem_loop</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgtatccagtgcagggtccgaggtattcgcactggatacgaccgccaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>43</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..43</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q51">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>stem_loop</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcaactggtgtcgtggagtcggcaattcagttgagtcaacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области молекулярной биологии. Описан способ для одновременного количественного определения не менее двух микроРНК в плазме венозной крови. Способ включает выделение РНК, проведение обратной транскрипции с праймерами и последующим проведением полимеразной цепной реакции в режиме «реального времени» с использованием подобранных праймеров и флуоресцентно-меченых TaqMan зондов, а также мультиплексного режима детекции одной из микроРНК вместе с miR-39, взятой в качестве маркера эффективности выделения РНК. Технический результат заключается в возможности осуществлять все этапы выполнения анализа в одной аналитической серии. 6 табл., 2 пр.
Способ для одновременного количественного определения не менее двух микроРНК в плазме венозной крови, включающий выделение РНК, проведение обратной транскрипции с праймерами SEQ ID NO: 10-12 и последующим проведением полимеразной цепной реакции в режиме «реального времени» с использованием праймеров и флуоресцентно- меченых TaqMan зондов SEQ ID NO: 1-9, а также мультиплексного режима детекции одной из микроРНК вместе с miR-39, взятой в качестве маркера эффективности выделения РНК, отличающийся тем, что в качестве диагностически значимых молекул в плазме венозной крови определяется концентрация микроРНК hsa-miR-16-5p и hsa-miR-21-5p, для выполнения обратной транскрипции используют петлевые праймеры SEQ ID NO: 10-12, при проведении полимеразной цепной реакции во всех реакционных смесях в качестве флуоресцентно-меченых зондов используют TaqMan зонды SEQ ID NO: 1,4,7, мультиплексный режим применяют для детекции микроРНК hsa-miR-21-5p и cel-miR-39-3p, используемой в качестве контроля процедуры выделения РНК, количество копий микроРНК, присутствующих в образцах плазмы венозной крови рассчитывают по калибровочным графикам, далее рассчитывают коэффициент соотношения уровня hsa-miR-21-5p к уровню hsa-miR-16-5p и при его значении более 0,20 приходят к заключению о лабораторном подтверждении диагноза множественной миеломы, в том числе с несекретирующим вариантом.
| СПОСОБ ДИФФЕРЕНЦИАЛЬНОЙ ДИАГНОСТИКИ РАКА ПРЕДСТАТЕЛЬНОЙ ЖЕЛЕЗЫ НА ОСНОВЕ АНАЛИЗА МИКРОРНК ПЛАЗМЫ КРОВИ | 2016 |
|
RU2618409C1 |
| Zhang L | |||
| et al | |||
| Способ получения продуктов уплотнения фенолов с альдегидами | 1920 |
|
SU361A1 |
| Oncology Letters, 2019, v | |||
| Способ использования делительного аппарата ровничных (чесальных) машин, предназначенных для мериносовой шерсти, с целью переработки на них грубых шерстей | 1921 |
|
SU18A1 |
| Жесткая автоматическая сцепка для железнодорожных вагонов, с головками для сквозных трубопроводов | 1926 |
|
SU4064A1 |
| Shao H | |||
| et al | |||
| Identification of characteristic genes and construction of regulatory network in gallbladder carcinoma.BMC Medical | |||
Авторы
Даты
2025-03-11—Публикация
2024-05-10—Подача