Изобретение относится к полинуклеотидным последовательностям, включающим генетические модификации гена PAR1 в положениях 3090 и/или 3329.
Активируемый протеазой рецептор 1 (PAR1) представляет собой рецептор тромбина, который относится к классу связанных с G-белком рецепторов (GCPR). Ген PAR1 находится в хромосоме 5q13, состоит из двух экзонов и занимает область длиной примерно 27 т.н. (тысяч нуклеотидов). PAR1 экспрессируется, в частности, в эндотелиальных клетках, гладкомышечных клетках, фибробластах, нейронах и человеческих тромбоцитах. В тромбоцитах PAR1 является важным рецептором трансдукции сигнала, который опосредует инициацию агрегации тромбоцитов.
Активация активируемых протеазой рецепторов (PAR) происходит через протеолитическое отщепление части N-конца PAR, благодаря чему высвобождается новая N-концевая последовательность, которая затем активирует рецептор.
При активации тромбоцитов центральную роль играют PAR1 и PAR4; активация этих рецепторов в тромбоцитах приводит к морфологическим изменениям, высвобождению аденозиндифосфата (АДФ) и агрегации тромбоцитов.
Связь коронарных болезней сердца с полиморфизмом одиночного нуклеотида (SNP) в промоторной области PAR1 не была подтверждена в случае группы корейских пациентов. Защитное действие в отношении возникновения венозных тромбоэмболий показано при другом исследовании для промоторных вариантов PAR1.
Последовательность человеческого гена PAR1 известна. Полинуклеотидная последовательность этого гена находится в базе данных по нуклеотидам NCBI под номером NM_001992. Точно также белковая последовательность доступна из базы данных по белкам NCBI под номером NP_001983. NCBI означает Международный информационный центр по биотехнологии (почтовый адрес: National Center for Biotechnology Information, National Library of Medicine, Building 38A, Bethesda, MD 20894, USA; Web-Adress: www.ncbi.nhm.nih.gov). Клонирование гена PAR1 описано, в частности, Schmidt и др., J. Biol. Chem., 271, 9307-9312 (1996).
Недавно обнаружены различные полиморфные формы гена PAR1, с помощью которых можно устанавливать более сильное предрасположение индивида к коронарным болезням сердца. Затронутые индивиды тем самым получили возможность за счет соответствующей адаптации к своему образу жизни своевременно противостоять этому фактору риска, который компенсируют, например, за счет усиления контроля за другими вредными влияниями, такими как курение, алкоголизм, обогащенная холестерином пища, высокое кровяное давление и пр.
Без знания более подробно описанных далее полиморфизмов PAR1 и их применения в соответствующих способах невозможны профилактические механизмы, касающиеся здоровья.
Варианты определенной нуклеотидной последовательности с заменами в отдельных положениях известны специалисту под понятием SNP (=полиморфизм одиночного нуклеотида).
Изобретение относится к выделенной полинуклеотидной последовательности гена PAR1, которая отличается тем, что в положении 3090 последовательности PAR1 согласно NM_001992, которая известна как уровень техники, имеется замена Т на С. Согласно предпочтительному варианту осуществления полинуклеотидная последовательность гена PAR1 с заменой Т на С в положении 3090 включает последовательность согласно SEQ ID №2, и согласно особенно предпочтительному варианту этой полинуклеотидной последовательности она состоит из последовательности, соответствующей SEQ ID №2.
Изобретение относится, далее, к выделенной полинуклеотидной последовательности гена PAR1, которая отличается тем, что в положении 3329 последовательности PAR1 согласно NM_001992, которая известна как уровень техники, имеется замена А на С. Согласно предпочтительному варианту осуществления полинуклеотидная последовательность гена PAR1 с заменой А на С в положении 3329 включает последовательность согласно SEQ ID №3, и согласно особенно предпочтительному варианту этой полинуклеотидной последовательности она состоит из последовательности, соответствующей SEQ ID №3.
Изобретение относится также к выделенной полинуклеотидной последовательности гена PAR1, которая отличается тем, что в положении 3090 последовательности PAR1 согласно NM_001992 имеется замена Т на С, и одновременно в положении 3329 этой последовательности PAR1 имеется замена А на C. Согласно предпочтительному варианту осуществления полинуклеотидная последовательность гена PAR1 с заменой Т на С в положении 3090 и одновременно имеющейся заменой А на С в положении 3329 включает последовательность согласно SEQ ID №4, и согласно особенно предпочтительному варианту этой полинуклеотидной последовательности, она состоит из последовательности соответствующей SEQ ID №4.
Изобретение относится также к выделенной части полинуклеотидной последовательности гена PAR1, состоящей из последовательности согласно SEQ ID №5.
Изобретение относится к выделенной части полинуклеотидной последовательности гена PAR1, которая отличается тем, что в положении 3090 последовательности PAR1 согласно NM_001992 имеется замена Т на С, состоящей из последовательности согласно SEQ ID №6.
Изобретение относится, кроме того, к выделенной части полинуклеотидной последовательности гена PAR1, которая отличается тем, что в положении 3329 последовательности PAR1 согласно NM_001992 имеется замена А на С, состоящей из последовательности согласно SEQ ID №7.
Изобретение относится также к выделенной части полинуклеотидной последовательности гена PAR1, которая отличается тем, что в положении 3090 последовательности PAR1 согласно NM_001992 имеется замена Т на С, и одновременно в положении 3329 указанной последовательности PAR1 имеется замена А на С, состоящей из последовательности согласно SEQ ID №8.
Изобретение включает, далее, получение полинуклеотидной последовательности гена кДНК PAR1 длиной 3592 пар оснований, которая может содержать или не содержать вышеуказанные полиморфизмы в положениях 3090 и 3329, индивидуально или в комбинации, отличающееся следующими стадиями способа:
а) получение человеческой ДНК, содержащей последовательность PAR1 согласно SEQ ID №2, и/или последовательность PAR1 согласно SEQ ID №3, и/или последовательность PAR1 согласно SEQ ID №4;
b) получение пары праймеров с последовательностью согласно SEQ ID №9 и SEQ ID №10;
с) амплификация полинуклеотидной последовательности PAR1 путем полимеразной цепной реакции (PCR);
d) выделение и/или очистка образовавшегося фрагмента длиной 3,56 т.н. со стадии с);
е) секвенирование фрагмента со стадии d).
Изобретение относится, кроме того, к получению полинуклеотидной последовательности согласно SEQ ID №5, SEQ ID №6, SEQ ID №7 или SEQ ID №8, отличающемуся следующими стадиями способа:
а) получение человеческой хромосомной ДНК, содержащей последовательность PAR1 согласно SEQ ID №1, и/или последовательность PAR1 согласно SEQ ID №2, и/или последовательность PAR1 согласно SEQ ID №3, и/или последовательность PAR1 согласно SEQ ID №4;
b) получение пары праймеров согласно SEQ ID №11 и SEQ ID №12;
с) амплификация фрагмента полинуклеотидной последовательности PAR1 путем полимеразной цепной реакции (PCR);
d) выделение и/или очистка образовавшегося фрагмента со стадии с);
е) секвенирование фрагмента со стадии d).
Изобретение относится, далее, к способу обнаружения наличия или отсутствия в гене PAR1 в положении 3090 последовательности согласно NM_001992 замены Т на С и/или в положении 3329 последовательности согласно NM_001992 замены А на С, включающему следующие стадии:
а) получение биологического материала, содержащего клетки человека;
b) получение хромосомной ДНК из материала со стадии а);
с) амплификация фрагмента полинуклеотида с помощью праймеров согласно SEQ ID №11 и SEQ ID №12 посредством полимеразной цепной реакции;
d) секвенирование фрагмента полинуклеотида со стадии с).
Далее, изобретение относится к способу обнаружения наличия или отсутствия в гене PAR в положении 3090 последовательности согласно NM_001992 замены Т на С и/или в положении 3329 последовательности согласно NM_001992 замены А на С, включающему следующие стадии:
а) получение биологического материала, содержащего клетки человека;
b) получение РНК из материала со стадии а);
с) транскрипция РНК на кДНК с помощью обратной транскриптазы;
d) при известных обстоятельствах, амплификация фрагмента полинуклеотида с помощью праймеров согласно SEQ ID №10 и SEQ ID №11 посредством полимеразной цепной реакции;
e) секвенирование кДНК со стадии с) и/или фрагмента полинуклеотида со стадии d).
Изобретение также относится к способу обнаружения наличия или отсутствия в гене PAR1 в положении 3090 последовательности согласно NM_001992 замены Т на С и/или в положении 3329 последовательности согласно NM_001992 замены А на С, включающему следующие стадии:
а) получение биологического материала, содержащего клетки человека;
b) получение хромосомной ДНК из материала со стадии а);
с) саузерн-блоттинг хромосомной ДНК со стадии b);
d) получение зонда согласно SEQ ID №5, и/или SEQ ID №6, и/или SEQ ID №7, и/или SEQ ID №8;
е) гибридизация саузерн-блота со стадии с) с зондом со стадии d) в строгих условиях гибридизации;
f) определение наличия или отсутствия генетической модификации в гене PAR1 в положении 3090 и/или 3329 согласно NM_001992 путем сравнения результатов гибридизации на стадии е).
Изобретение относится, далее, к способу обнаружения наличия или отсутствия в гене PAR1 в положении 3090 последовательности согласно NM_001992 замены Т на С и/или в положении 3329 последовательности согласно NM_001992 замены А на С, включающему следующие стадии:
а) получение биологического материала, содержащего клетки человека;
b) получение РНК из материала со стадии а);
с) назерн-блоттинг РНК со стадии b);
d) получение зонда согласно SEQ ID №5, и/или SEQ ID №6, и/или SEQ ID №7, и/или SEQ ID №8;
е) гибридизация назерн-блота со стадии с) с зондом со стадии d) в строгих условиях гибридизации;
f) определение наличия или отсутствия генетической модификации в гене PAR1 в положении 3090 и/или 3329 согласно NM_001992 путем сравнения результатов гибридизации.
Обнаружение генетических модификаций или полиморфизмов в гене PAR1 в положениях 3090 и/или 3329 можно использовать в качестве (а) генетического маркера для оценки риска возникновения мерцания предсердий, острого коронарного синдрома, кардиомиопатии и/или нестабильной стенокардии, в качестве (b) маркера для профилактической обработки в отношении мерцания предсердий, острого коронарного синдрома, кардиомиопатии и/или нестабильной стенокардии, носителя соответствующих генетических вариантов, в качестве (с) маркера для адаптации вводимой дозы фармацевтически активного вещества против мерцания предсердий, острого коронарного синдрома, кардиомиопатии и/или нестабильной стенокардии, в качестве (d) маркера для определения стратегии высокопроизводительного скрининга в целях идентификации фармацевтически активного вещества против мерцания предсердий, острого коронарного синдрома, кардиомиопатии и/или нестабильной стенокардии, в качестве (е) маркера для идентификации релевантных индивидов, соответственно пациентов, в целях клинических исследований для контроля совместимости, безопасности и эффективности фармацевтического вещества против мерцания предсердий, острого коронарного синдрома, кардиомиопатии и/или нестабильной стенокардии, и в качестве (f) основы для разработки тест-систем для анализа генетической модификации в гене PAR1 на уровне ДНК, РНК или белка.
Изобретение относится также к выделенной полинуклеотидной последовательности с количеством нуклеотидов от 21 до 50, отличающейся тем, что в ней содержится последовательность, соответствующая SEQ ID №11. Эта последовательность предпочтительно состоит из SEQ ID №11. Изобретение относится, далее, к выделенной полинуклеотидной последовательности с количеством нуклеотидов от 20 до 50, отличающейся тем, что она содержит последовательность, соответствующую SEQ ID №12. Эта последовательность предпочтительно состоит из SEQ ID №12.
Изобретение относится также к применению выделенной полинуклеотидной последовательности длиной от 21 до 50 нуклеотидов, которая включает последовательность согласно SEQ ID №11 или состоит из нее, в комбинации с выделенной полинуклеотидной последовательностью длиной от 20 до 50 нуклеотидов, которая включает последовательность согласно SEQ ID №12 или состоит из нее, для амплификации соответствующего фрагмента гена PAR1 посредством полимеразной цепной реакции (PCR). Это применение предпочтительно относится к амплификации фрагмента гена PAR1, который в положении 3090 последовательности согласно NM_001992 имеет замену Т на С и/или в положении 3329 последовательности согласно NM_001992 имеет замену А на С.
Кроме того, изобретение относится к набору компонентов, который содержит:
а) выделенную полинуклеотидную последовательность длиной от 21 до 50 нуклеотидов, которая включает последовательность согласно SEQ ID №11 или состоит из нее;
b) выделенную полинуклеотидную последовательность длиной от 20 до 50 нуклеотидов, которая включает последовательность согласно SEQ ID №12 или состоит из нее;
с) по меньшей мере один фермент для осуществления полимеразной цепной реакции (PCR);
d) смотря по обстоятельствам, вещества и/или растворы для проведения полимеразной цепной реакции;
е) смотря по обстоятельствам, полинуклеотидные последовательности, включающие ген PAR1 с заменой или нет в положении 3090 последовательности PAR1 согласно NM_001992 и/или в положении 3329 последовательности согласно NM_001992, соответственно, по всей длине и/или ее частях;
f) а также, смотря по обстоятельствам, реагенты для осуществления секвенирования.
Под набором компонентов, здесь и далее, понимают комбинацию указанных компонентов, которые пространственно объединены в одну функциональную единицу.
Изобретение относится, далее, к получению вышеописанного набора компонентов, причем:
а) получают выделенную полинуклеотидную последовательность длиной от 21 до 50 нуклеотидов, которая включает последовательность согласно SEQ ID №11 или состоит из нее;
b) получают выделенную полинуклеотидную последовательность длиной от 20 до 50 нуклеотидов, которая включает последовательность согласно SEQ ID №12 или состоит из нее;
с) получают фермент для осуществления полимеразной цепной реакции (PCR);
d) в случае необходимости, получают реагенты для осуществления секвенирования;
е) смотря по обстоятельствам, получают вещества и/или растворы для проведения полимеразной цепной реакции (PCR);
f) смотря по обстоятельствам, получают полинуклеотидные последовательности, включающие ген PAR1, с заменой или нет в положении 3090 последовательности PAR1 согласно NM_001992 Т на С и/или в положении 3329 последовательности согласно NM_001992 А на С соответственно, по всей длине и/или ее частях;
g) компоненты со стадий а)-f), соответственно, раздельно помещают в пригодные емкости;
h) емкости со стадии g), в случае необходимости, объединяют в одной или нескольких единицах упаковки.
Вышеописанный набор компонентов можно также использовать для амплификации фрагмента гена PAR1.
В нижеследующих вариантах осуществления подробнее обсуждаются технические аспекты изобретения.
Выделенные полинуклеотидные последовательности гена PAR1 можно получать, например, путем амплификации посредством полимеразной цепной реакции (PCR). Пригодные праймеры для этого представлены в виде SEQ ID №9 и SEQ ID №10.
PCR представляет собой осуществляемый in vitro способ, с помощью которого можно целенаправленно амплифицировать участки полинуклеотида, которые окружены двумя известными последовательностями. Для амплификации необходимы короткие однонитевые молекулы ДНК, которые комплементарны концам определенной последовательности матрицы ДНК или РНК (праймеры). ДНК-полимераза в соответствующих реакционных условиях и в присутствии дезоксинуклеотидтрифосфатов (дНТФ) удлиняет праймер вдоль однонитевой и денатурированной полинуклеотидной матрицы и, таким образом, синтезирует новые нити ДНК, последовательность которых комплементарна матрице. При этом температуру периодически изменяют, так что полинуклеотидные нити постоянно снова денатурируют, и праймеры могут присоединяться и удлиняться. Используют устойчивые к нагреву ДНК-полимеразы, например, Taq-полимеразу. Типичная смесь для PCR содержит, наряду с полинуклеотидной матрицей, два пригодных нуклеотида в качестве праймеров, например, в концентрации от 0,2 мкМ до 2 мкМ, далее, дНТФ, например, в концентрациях 200 мкМ на один дНТФ, далее, MgCl2 в концентрации 1-2 мМ и 1-10 Единиц устойчивой к нагреву ДНК-полимеразы, как, например, Taq-полимераза (полимераза из Thermus aquaticus). Устойчивая к нагреву ДНК-полимераза, а также компоненты для ее получения вместе с протоколами выпускаются многочисленными фирмами, как например: Roche Diagnostics, Clontech, Life Technologies, New England Biolabs, Promega, Stratagene и др.
Полинуклеотидная матрица для амплификации выделяемой полинуклеотидной последовательности может находиться в виде РНК или ДНК. Если полинуклеотидная матрица находится в виде РНК, перед непосредственной реакцией PCR ее транскрибируют посредством обратной транскриптазы на ДНК. Количество полинуклеотидной матрицы при осуществлении реакции PCR может составлять, например, 0,01-20 нг.
Получение полинуклеотидной матрицы осуществляют по известным специалисту способам для получения ДНК и/или РНК из биологического материала. Биологический материал, согласно данному контексту, должен включать, в частности, клетки ткани или органа (как, например, головной мозг, кровь, печень, селезенка, почки, сердце, кровеносные сосуды) позвоночного животного, включая человека, или клетки из эукариотической клеточной культуры (например, клетки Hela, клетки СНО, клетки 3Т3), или клетки, состоящие из бактерий или дрожжей, в которых изолируемая последовательность ДНК находится в клонированной форме.
Клетки тканевой системы или органа позвоночного животного, включая человека, можно получать путем взятия крови, пункции ткани или оперативных способов. Полинуклеотидную матрицу, исходя из этого, получают, например, путем переведения в растворимое состояние клеток, при известных обстоятельствах, концентрирования отдельных органелл, в особенности клеточного ядра, и получение ДНК или РНК осуществляют путем осаждения и центрифугирования.
Другой способ получения выделенных полинуклеотидных последовательностей гена PAR1 состоит в клонировании гена PAR1, последующей экспрессии в бактериях или дрожжах и очистки продуцированного полинуклеотида. Для получения фрагмента полинуклеотида, который можно клонировать, пригодна, например, вышеуказанная реакция PCR. В случае клонируемого фрагмента предпочтительно используют праймеры, которые в 5'-положении комплементарной последовательности содержат последовательность распознавания реакционного фермента. В случае обоих праймеров можно использовать, соответственно, одинаковые или разные последовательности распознавания для рестриктаз.
Обычными рестриктазами, например, являются BamHI (GGATCC), Clal (ATCGAT), EcoRI (GAATTC), EcoRV (GATATC), HindIII (AAGCTT), Ncol (CCATGG), SaII (GTCGAC), XbaI (TCTAG1).
Для клонирования вектор обрабатывают рестриктазами, которые соответствуют присоединенным к праймерам последовательностям распознавания. Фрагмент путем выделения и обработки с помощью этих рестриктаз посредством лигазы связывают с вектором. В качестве вектора нужно понимать молекулу ДНК, как, например, плазмида, бактериофаг или космида, с помощью которой можно клонировать гены или другие последовательности ДНК и для амплификации вводить в бактериальную или эукариотическую клетку. Векторами являются, например, молекулы ДНК, такие как pBR322, pUC18/19, pBluescript, pcDNA3.1. Векторы выпускаются специальными фирмами по биотехнологическому материалу, такими как Roche Diagnostics, New England Biolabs, Promega, Stratagene и др.
Указания, необходимые для осуществления реакции PCR, для получения полинуклеотидов или для проведения работ по клонированию, специалист может найти в форме рецептов и протоколов в стандартных руководствах, как например: в а) "Current Protocols in Molecular Biology" под редакцией Frederick M. Ausubel, Roger Brent, Robert E. Kingston, David D. Moore, J. G. Seidman, Kevin Struhl, стереотипное издание, постоянно модернизируемое, John Wiley and Sons, Inc., New York; в b) "Short Protocols in Molecular Biology", пятое издание, под редакцией Frederick M. Ausubel, Roger Brent, Robert E. Kingston, David D. Moore, J. G. Seidman, John A. Smith, Kevin Struhl, октябрь 2002, John Wiley and Sons, Inc., New York; или в с) "Molecular Cloning" J. Sambrock, E.F. Fritsch, T. Maniatis; Cold Spring Harbor Laboratory Press.
Получение пригодных праймерных последовательностей осуществляют, например, за счет их химического синтеза, который может проводиться по заказу на коммерческой основе такими компаниями, как MWG Biotech и др.
Человеческая кДНК из различных органов выпускается, например, компаниями Promega, Stratagene или др.
Секвенирование полинуклеотида осуществляют посредством известных специалисту рутинных способов с использованием лабораторных робототехнических устройств, выпускаемых фирмами, как, например, Life Technologies, Applied Biosystems, BioRad, или др.
Выделенные полинуклеотидные последовательности варианта PAR1, как и их фрагменты, также можно использовать для гибридизации в условиях различной жесткости. Жесткие условия представляют собой реакционные условия, которые влияют на специфичность гибридизации или присоединения двух однонитевых молекул нуклеиновой кислоты. Жесткость и вместе с тем также специфичность реакции можно повышать тем, что повышают температуру и снижают ионную силу. Условия слабой жесткости используют, например, когда гибридизацию осуществляют при комнатной температуре в растворе 2 х SSC. Условия высокой жесткости используют, когда гибридизацию проводят при температуре 68°С в растворе 0,1 х SSC/0,1% SDS (додецилсульфат натрия).
Гибридизация в жестких условиях в рамках настоящей заявки означает
1) гибридизацию маркированного зонда с исследуемым образцом при температуре 65°С (или, в случае олигонуклеотидов, при температуре на 5°С ниже температуры плавления) в течение ночи в 50 мМ Трис, рН=7,5, 1% NaCl, 1% додецилсульфата натрия, 10% декстрансульфата, 0,5 мг/мл денатурированной ДНК спермы лососевых;
2) промывку в течение 10 минут при комнатной температуре в растворе 2 х SSC;
3) промывку в течение 30 минут при температуре 65°С (или, в случае олигонуклеотидов, на 5°С ниже температуры плавления) в растворе 1 х SSC/1% SDS;
4) промывку в течение 30 минут при температуре 65°С (или, в случае олигонуклеотидов, на 5°С ниже температуры плавления) в растворе 0,2 х SSC/0,1% SDS;
5) промывку в течение 30 минут при температуре 65°С (или, в случае олигонуклеотидов, на 5°С ниже температуры плавления) в растворе 0,1 х SSC/0,1% SDS.
В качестве олигонуклеотидов для этого предусмотрены фрагменты ДНК с общей длиной 20 нуклеотидов. Температуру плавления рассчитывают по формуле:
T.пл.=2·(число А+Т)+4·(число G+C)°C.
Для приготовления раствора 2 х SSC, соответственно, 0,1 x SSC соответствующим образом разводят раствор 20 х SSC. Раствор 20 х SSC состоит из раствора 3 M NaCl/0,3 М цитрата натрия·2Н2О. SDS означает додецилсульфат натрия.
Для осуществления гибридизации, исследуемые полинуклеотиды после электрофоретического отделения и последующей денатурации переносят на нейлоновую или нитроцеллюлозную мембрану (саузерн-блот-ДНК; назерн-блот-РНК). Гибридизацию осуществляют с помощью зонда, который маркирован радиоактивно или другим образом, например с помощью флуоресцентных красителей. Зонд состоит из обычно однонитевой и/или денатурированной полинуклеотидной последовательности ДНК или РНК, которая связывается с комплементарной нуклеотидной последовательностью, в свою очередь, однонитевой и/или денатурированной исследуемой полинуклеотидной последовательностью ДНК или РНК.
Обнаружение полиморфизма одиночного нуклеотида гена PAR1 можно осуществлять с помощью предлагаемых согласно изобретению праймеров, а также путем SSCP-анализа. SSCP означает полиморфизм одиночной скрученной конформации. Речь идет при этом о способе электрофореза для идентификации одиночных замен пар оснований. Исследуемые полинуклеотиды амплифицируют с помощью маркированных праймеров путем полимеразной цепной реакции и после денатурации отделяют одиночные нити путем электрофореза в полиакриламидном геле (PAGE). Если исследуемые фрагменты ДНК имеют одиночные замены пар оснований, то они обладают различной конформацией, и поэтому с разной быстротой мигрируют в случае PAGE.
Веществами для осуществления реакции PCR являются, например, буферы, как Гепес или Трис, далее, dAPP, дГТФ, дТТФ, дЦТФ, а также Mg2+ и, при известных обстоятельствах, другие двухвалентные или одновалентные ионы. Растворы содержат эти вещества в растворенной форме.
ПРИМЕРЫ
Амплификация геномных областей гена PAR1
Для обнаружения нуклеотидной замены Т на С в положении 3090 и А на С в положении 3329 в последовательности PAR1 использовали следующие праймеры:
праймер 1: 5'-ACAGAGTGGAATAAGACAGAG-3' (SEQ ID №11)
праймер 2: 5'-CCAGTGCTAGCTTCTACTTAC-3' (SEQ ID №12).
Праймер 1 (SEQ ID №11) соответствует положениям 2767-2789 стандартной последовательности NM_001992. Праймер 2 происходит из экзона №1 гена PAR1.
Пропись PCR для амплификации
Используемые реагенты выпускаются фирмой Applied Biosystems (Foster City, США): 20 нг геномной ДНК; 1 Единица TaqGold ДНК-полимеразы; буфер для 1 х Taq полимеразы; 500 мкМ дНТФ; 2,5 мМ MgCl2; 200 нМ пары праймеров для каждой амплификации; Н2О до общего объема 5 мкл.
Программа амплификации путем PCR для генотипизации:
70°С в течение 30 секунд
65°С в течение 30 секунд
60°С в течение 30 секунд
56°С в течение 30 секунд
72°С в течение 30 секунд
4°С в течение 30 секунд
Идентификация SNP
Пропись для минисеквенирования и обнаружения SNP
Все реагенты выпускаются фирмой Applied Biosystems (Foster City, США): 2 мкл очищенного продукта PCR, 1,5 мкл набора BigDye-Terminator, 200 нМ праймера для секвенирования; Н2О до общего объема 10 мкл.
Программа амплификации для секвенирования:
55°С в течение 10 секунд
65°С в течение 4 минут
4°С в течение 30 секунд
Анализ продуктов секвенирования
Последовательности сначала анализировали с помощью программного обеспечения для анализа последовательностей (Applied Biosystems, Foster City, США) с получением "сырых" данных, затем осуществляли обработку данных с помощью Phred, Phrap, Polyphred и Consed. Phred, Phrap, Polyphred и Consed представляют собой программное обеспечение, описанное Phil Green из Вашингтонского Университета (http://www.genome.washington.edu).
Установление связи SNP PAR1 с коронарными заболеваниями
При клиническом исследовании изучали два полиморфизма PAR1 из 3'-некодирующей области гена в связи с тромботическими и сердечно-сосудистыми осложнениями в группе пациентов.
В дальнейшем используют следующие сокращения (все указанные положения относятся к положениям нуклеотида в стандартной последовательности NM_001992).
PAR1 T3090T: описывается группа людей, у которых на обоих аллелях гена PAR1 в положении 3090 имеется тимидин (Т). Эти люди являются гомозиготными относительно этого варианта PAR1.
PAR1 T3090С: описывается группа людей, у которых на одном аллеле гена PAR1 в положении 3090 имеется цитидин (С), на другом аллеле гена PAR1 в положении 3090 имеется тимидин (Т). Эти люди являются гетерозиготными относительно этого варианта PAR1.
PAR1 C3090C: описывается группа людей, у которых на обоих аллелях гена PAR1 в положении 3090 имеется цитидин (С). Эти люди являются гомозиготными относительно этого варианта PAR1.
PAR1 A3329A: описывается группа людей, у которых на обоих аллелях гена PAR1 в положении 3329 имеется аденозин (А). Эти люди являются гомозиготными относительно этого варианта PAR1.
PAR1 A3329C: описывается группа людей, у которых на одном аллеле гена PAR1 в положении 3329 имеется цитидин (С), на другом аллеле гена PAR1 в положении 3329 имеется аденозин (А). Эти люди являются гетерозиготными относительно этого варианта PAR1.
PAR1 C3329C: описывается группа людей, у которых на обоих аллелях гена PAR1 в положении 3329 имеется цитидин (С). Эти люди являются гомозиготными относительно этого варианта PAR1.
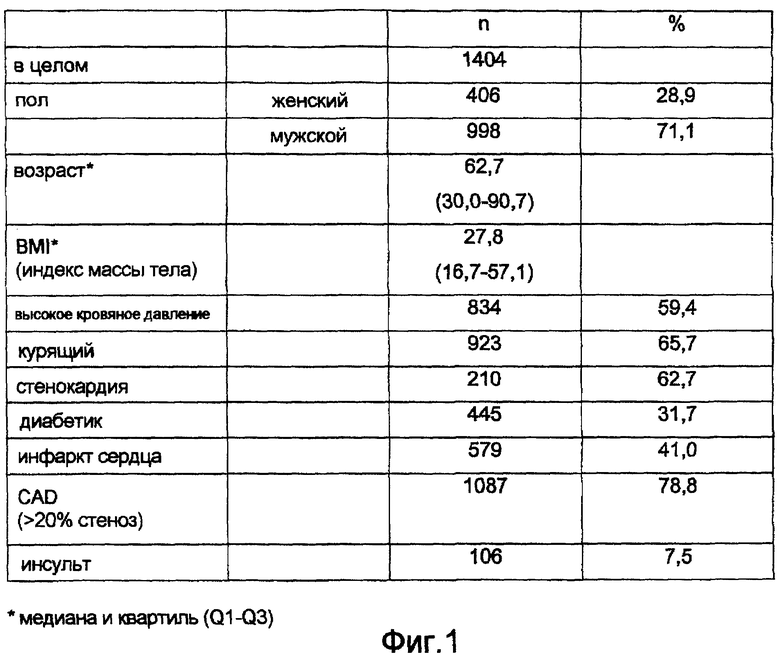
В проанализированной группе пациентов (фиг.1) обнаружены статистически значительные ассоциации гомозиготных носителей варианта PAR1 С3090С с мерцанием предсердий и кардиомиопатией. После осуществления логической регрессии смогли установить 1,97-кратно повышенный риск в отношении мерцания предсердий и 1,84-кратно повышенный риск для кардиомиопатии в случае гомозиготных носителей варианта PAR1 С3090С по сравнению с носителями вариантов PAR1 Т3090С/Т3090Т (фиг.3).
В случае носителей варианта PAR1 С3329С было обнаружено, что этот вариант ассоциируется с 2,35-кратно повышенным риском в отношении мерцания предсердий по сравнению с носителями вариантов PAR1 С3329А/А3329А. В случае носителей варианта PAR1 С3329С этот вариант, сверх того, является, по-видимому, защитным в отношении возникновения острого коронарного синдрома и нестабильной стенокардии. По сравнению с носителями вариантов PAR1 А3329С/А3329А носители варианта PAR1 С3329С имеют 2,78-кратно пониженный риск в отношении возникновения острого коронарного синдрома и/или нестабильной стенокардии (фиг.4).
Таким образом, с помощью предлагаемого согласно изобретению способа при использовании выделенной последовательности PAR1 соответствующего типа SNP или ее фрагмента в случае человека можно определять, существует ли соответствие группе риска в смысле представленных результатов.
Получение плазмидной ДНК
1 мл ночной культуры бактерий переносят в сосуд Эппендорфа и центрифугируют в биоцентрифуге Heraeus (5 минут со скоростью 5000 оборотов в минуту). Осадок клеток бактерий нужно ресуспендировать в 100 мкл охлажденного раствора I и затем выдержать в течение 5 минут на льду.
Раствор I: 25 мМ Трис-HCl, рН=8,0, 50 мМ глюкозы (стерильно отфильтровано), 10 мМ ЭДТУ (этилендиаминтетра-уксусная кислота), 100 мкг/мл рибонуклеазы А.
После добавления 200 мкл раствора II тщательно перемешивают. При этом приходят к щелочной денатурации ДНК.
Раствор II: 200 мМ NaOH, 1% SDS.
После последующей инкубации в течение 5 минут на льду к смеси добавляют 150 мкл раствора III. Снова перемешивают и инкубируют в течение следующих 15 минут на льду.
Раствор III: 3 М ацетата натрия (рН=4,8).
Путем 15-минутного центрифугирования в биоцентрифуге Heraeus со скоростью 12000 оборотов в минуту отделяют путем центрифугирования остатки клеток, геномную ДНК и денатурированные белки. Образующийся супернатант, в котором находится плазмидная ДНК, декантируют во второй сосуд Эппендорфа и смешивают с 1 мл 96%-ного этанола (соответственно, 300 мкл изопропанола). Смесь с осадком тщательно перемешивают и снова центрифугируют (15 минут со скоростью 12000 оборотов в минуту в центрифуге Heraeus). При этом происходит осаждение плазмидной ДНК. Выпавшую в осадок плазмидную ДНК промывают холодным, как лед, 70%-ным этанолом и затем высушивают на воздухе. После этого сухой осадок обрабатывают с помощью 50 мкл стерильной дистиллированной воды.
Осаждение спиртом ДНК
Смесь для осаждения: раствор ДНК, 1/10 объема 3 М раствора ацетата натрия (рН=5,4), 2-3 объема 96%-ного этанола (1 объем изопропанола).
Смесь тщательно перемешивают. Ее можно выдерживать при температуре -20°С, однако за счет этого выход осаждения не повышается. После центрифугирования в течение 20 минут со скоростью 12000 оборотов в минуту плазмидная ДНК осаждается.
Для устранения остатков используемого ацетата натрия плазмидную ДНК после осаждения промывают еще раз с помощью 1 мл 70%-ного этанола.
Экстракция ДНК фенолом
Раствор ДНК смешивают с равным объемом фенола (Rotiphenol®, уравновешен против буфера ТЕ, рН=7,6; Roth, Карлсруэ), встряхивают в течение 5 минут и центрифугируют со скоростью 5000 оборотов в минуту. Максимальная часть денатурированных теперь белков собирается на границе раздела фаз. Верхняя водная фаза содержит ДНК, и ее тщательно отделяют, затем смешивают со смесью хлороформа и изоамилового спирта (24:1) для удаления остатков фенола. После этого снова центрифугируют, водный супернатант удаляют и ДНК выделяют из раствора путем осаждения спиртом.
Очистка амплифицированных молекул ДНК
Очистку амплификатов ДНК осуществляют с помощью набора для очистки продуктов PCR (Qiagen). При этом удаляют молекулы-инициаторы, нуклеотиды (дНТФ), полимеразы и соли. Для этого реакционную смесь PCR смешивают с пятикратным объемом буфера РВ, тщательно перемешивают и вносят в колонку Qiaquick. Происходит селективное связывание амплифицированной ДНК с материалом колонки, дНТФ удаляют путем двукратной промывки с помощью 750 мкл буфера РЕ. Затем амплифицированную ДНК элюируют с помощью желательного объема воды, лучше всего с помощью такого же объема, как в случае исходного материала реакционной смеси PCR.
Расщепление ДНК рестриктазами
Смесь: 3 мкл ДНК, 2 мкл 10 х буфера для расщепления, 2,5-5 Ед. рестриктазы (например, EcoRI, BamHI, SaII, XbaI, Xhol и другие); доводят до объема 20 мкл с помощью дистиллированной воды.
Реакция расщепления, в зависимости от рестриктазы, протекает при температуре 25-55°С в течение 1-2 часов. Для анализа фрагменты разделяют путем электрофореза в агарозном или полиакриламидном геле параллельно стандарту длины. Если речь идет о двойном расщеплении, то к смеси сначала добавляют один фермент. Спустя один час аликвоту наносят на соответствующий гель; если произошло расщепление, то можно добавлять второй фермент.Если второй фермент не расщепляет в том же буфере для расщепления, то сначала требуется осаждение спиртом.
Электрофорез ДНК в агарозном геле
Агарозу (Roth) растворяют в желательной концентрации в 1 х буфере для агарозы и в микроволновом устройстве кипятят вплоть до полного растворения агарозы. Раствор затем выливают в герметизированную плоскую камеру для геля из плексигласа.
Образцы ДНК смешивают с 1/10 объема Ladeblau (50% об./об. глицерина; 50 мМ ЭДТУ; 0,005% мас./об. ВРВ [Merck, Дармштадт] и 0,005% ксиленцианола) и вносят пипеткой в "карманы" геля, которые получают с помощью гребенки.
Электрофорез осуществляют горизонтально при постоянном напряжении 80-140 В, в зависимости от величины геля, соответственно, расстояния между электродами, в 1 х буфере для агарозы в качестве рециркулирующего буфера.
1 х буфер для агарозы: 40 мМ Трис-HCl (рН=7,8), 5 мМ ацетата натрия, 1 мМ ЭДТУ.
Электрофорез ДНК в полиакриламидном геле
7,5%-ный раствор полиакриламидного геля: 0,94 мл 40%-ного исходного раствора акриламида-бисакриламида; 0,5 мл 10 х буфера ТВЕ (400 мМ Трис-HCl, рН=8,3; 200 мМ ацетата натрия, 20 мМ ЭДТУ), 0,25 мл 1% AMPS, 10 мкл TEMED, 3,33 мл дистиллированной воды.
Эту смесь для полимеризации (в течение примерно 10-20 минут) помещают между хорошо очищенными вертикальными стеклянными пластинами, которые закреплены в вертикальных приспособлениях. Гель функционирует в 1 х буфере ТВЕ при постоянном напряжении 140 В.
Секвенирование ДНК
1-2 мкг ДНК растворяют в 81 мкл дистиллированной воды и для денатурации добавляют 9 мкл 2 н. раствора NaOH. После инкубации в течение 10 минут при комнатной температуре смесь подвергается преципитации, причем для последующих реакций секвенирования важной является тщательная промывка полученного осадка ДНК с помощью холодного, как лед, 80%-ного этанола. К осадку добавляют 2 мкл 5 х буфера для секвеназы (200 мМ Трис-HCl, рН=7,5/100 мМ MgCl2/250 мМ NaCl), 1 мкл олигонуклеотида (1 пМ/мкл) и, наконец, дистиллированную воду до общего объема 10 мкл. Во время последующей инкубации в течение 30 минут на водяной бане при температуре 37°С приходят к гибридизации исходного олигонуклеотида с ДНК.
Добавляемые к смеси для гибридизации реагенты для реакции секвенирования: 1,0 мкл дитиотреитола (0,1 М), 2,0 мкл смеси для маркировки (в разведении 1:5), 0,5 мкл [α-35S]дАТФ, 2 мкл секвеназыТМ (13 Ед./мкл, United States Biochemical) (разводят в соотношении 1:8 с помощью буфера для разведения фермента).
Во время последующей инкубации в течение 5 минут при комнатной температуре происходит синтез комплементарной нити, причем за счет встраивания радиоактивно маркированного дАТФ происходит маркировка синтетической ДНК. Затем 3,5 мкл смеси для маркировки добавляют, соответственно, к 2,5 мкл четырех различных смесей для терминации. Следующая инкубация в течение 5 минут при температуре 37°С приводит к статистически распределенным реакциям прекращения синтеза комплементарной нити.
Реакции заканчиваются за счет добавки 4 мкл стоп-буфера, смеси денатурируют при температуре 80-90°С и затем наносят на 6%-ный денатурированный секвенирующий гель. После введения образцов, в течение 2-5 часов при мощности 30-50 Вт, соответственно, напряжении 1300-1600 В происходит основное протекание процесса. Затем гель фиксируют в 10%-ном растворе уксусной кислоты (15 минут), освобождают от остатков мочевины с помощью проточной воды и после этого высушивают (45 минут с помощью фена с теплым воздухом или в течение 2 часов в инкубаторе при температуре 70°С). Последующую авторадиографию осуществляют в течение 16-24 часов при температуре 4°С (пленка для рентгенографии Fujii Medical, 30 х 40; Kodak Scientific Imaging Film X-omat AR).
Исходный раствор смеси для маркировки: по 7,5 мкМ дАТФ, дТТФ, дГТФ, дЦТФ.
Смеси для терминации: по 80 мкМ дАТФ, дТТФ, дГТФ, дЦТФ и по 8 мкМ соответствующего ддНТФ.
Буфер для разведения секвеназы: 10 мМ Трис/HCl; рН=7,5; 5 мМ дитиотреитола, 0,5 мг/мл бычьего сывороточного альбумина.
Стоп-буфер: 95% формамида, 20 мМ ЭДТУ, 0,005% (мас./об.) ксиленцианола FF.
Автоматическое секвенирование ДНК
Смесь: 1 мкг плазмидной ДНК (в случае фрагментов PCR используют, например, 100 нг/500 нуклеотидов), 3-5 пмоль исходной молекулы (праймер PCR, по возможности с Т.пл. 55°С), 4 мкл готовой смеси терминатор-краситель (смесь FддНТФ-Ampli-TaqFS); дополнено до 20 мкл с помощью дистиллированной воды.
Реакционную смесь PCR [25 х (15 секунд при 94°С, 15 секунд при 50°С, 4 минуты при 60°С)] осаждают с помощью спирта и обрабатывают с помощью 4 мкл буфера для загрузки. После этого образцы денатурируют в течение 3 минут при температуре 95°С, подвергают центрифугированию и наносят на вертикальный полиакриламидный гель (длина 34 см, снабжен 24 параллельными блотами).
После возбуждения за счет лазерного излучения в атмосфере аргона при 488 нм красители эмиттируют свет различных длин волн между 525 нм и 605 нм, который, проходя через дифракционную решетку так называемого спектрографа, разлагается на его спектральные цвета. Затем осуществляют одновременное детектирование спектральных цветов с помощью поля элемента изображения высокого разрешения CCD-камеры. Регистрацию данных осуществляют с помощью компьютера (Macintosh Quadra/650 Macllcx Apple Share) и соответствующего оценочного программного обеспечения (PE Biosystems, Weiterstadt).
Гель для секвенирования: 30 г мочевины (Sigma), 21,5 мл дистиллированной воды, 6 мл 10 х ТВЕ. Смесь растворяют в колбе с широким горлом при температуре 50°С на нагревательном блоке и добавляют следующее: 9 мл 40%-ного бисакриламида (отфильтровано), 180 мкл 10% APS, 24 мкл Temed.
Полимеразная цепная реакция (PCR-реакция)
Можно использовать следующие ДНК-полимеразы:
Taq (Thermus aquaticus) ДНК-полимераза (рекомбинантная, Gibco/BRL) и 10 х буфер для PCR [200 мМ Трис/HCl (рН=8,4), 500 мМ KCl];
Tfl (Thermus flavus) ДНК-полимераза (Master AmpTM, Biozym, Oldendorf) и 20 х буфер для PCR [20 мМ (NH2)SO4, 1 М Трис/HCl (рН=9,0)]
Реакционная смесь PCR:
2,5 мкл в случае буфера для Tfl ДНК-полимеразы
1 Ед. для Tfl ДНК-полимеразы
При этом имеет значение: 1 Ед. катализирует перестройку 10 нМ дезоксирибонуклеозидтрифосфатов, при температуре 74°С в течение 30 минут, в не растворимый в кислоте ДНК-продукт.
Реакция PCR обычно начинается с так называемого "горячего старта": сначала смесь без полимеразы инкубируют при температуре 94°С для того, чтобы сделать возможной первую денатурацию ДНК. После достижения температуры 80°С к смеси добавляют ДНК-полимеразу, чтобы избежать неспецифической амплификации при еще низкой температуре. После этого протекает непосредственная реакция PCR в течение 25-35 циклов.
Для каждого цикла используют следующие реакционные условия:
Наконец, удлинение цепи осуществляют еще в течение 10 минут при температуре 72°С и, наконец, охлаждают.
Выделение общей РНК
Все стадии центрифугирования осуществляют со скоростью 13000 оборотов в минуту и при температуре 16°С. Клетки лизируют с помощью 600 мкл буфера для лизиса (100 RLT-буфер: 1 меркаптоэтанол). Клеточный лизат вносят в колонку QiaSchredder и центрифугируют в течение 2 минут. Элюат смешивают с 600 мкл 70%-ного этанола, тщательно перемешивают, в центрифужную миниколонку с RNAeasy вводят ДНК и центрифугируют в течение 15 секунд (происходит связывание РНК с матрицей из диоксида кремния). Колонку промывают три раза (один раз с помощью 700 мл буфера RW1 и два раза с помощью 500 мкл буфера RPE). Затем колонку переносят в автоклавированный сосуд Эппендорфа емкостью 1,5 мл и РНК элюируют с помощью 15 мкл дистиллированной воды. Средняя концентрация полученной таким образом общей РНК составляет 1 мкг/мкл.
Отделение РНК путем электрофореза в агарозном геле
Денатурирующий агарозный гель: 1 г агарозы, 37 мл дистиллированной воды, 10 мл 10 х МОРS (4-морфолинпропансульфо-кислота) (0,2 мM MOPS, 10 мМ ЭДТУ, 100 мМ ацетата натрия). Смесь кипятят и охлаждают до температуры 60°С. Добавляют 16 мл 37%-ного формальдегида.
После затвердевания ее используют вместе с рециркулирующим буфером для геля с РНК в аппаратуре для электрофореза. РНК наносят вместе со специальным буфером для образца.
Рециркулирующий буфер для геля с РНК: 40 мл 10 х MOPS, 65 мл 37%-ного формальдегида, 295 мл дистиллированной воды.
Буфер для образца РНК: 1-5 мкг РНК, 5 мкл NEW-буфера для РНК (7,5 мкл 37%-ного формальдегида, 4,5 мкл 10 х MOPS, 25,9 мкл формамида, 7,5 мкл дистиллированной воды), 2 мкл цветового маркера для формамида [50% (об./об.) глицерина, 1 мМ ЭДТУ (рН=8,0), 0,25% (об./об.) бромфенолового синего, 0,25% (об./об.) ксиленцианола].
Гель функционирует в течение примерно 3 часов при напряжении 80 В. Так как в настоящей работе речь идет об изолятах РНК эукариотов, на геле должны быть видимы 28S- и 18S-рРНК в виде доминирующих полос.
Обратная транскриптаза с MMLV-RT (вирус Moloney мышиной лейкемии - обратная транскриптаза)
Смесь для обратной транскриптазы: 5 мкг РНК, 100 мкм молекулы-инициатора.
Препарат РНК и молекулу-инициатор инкубируют в течение 10 минут при температуре 75°С, чтобы избежать возможных вторичных структурных образований в матрице РНК в качестве фактора помех для транскриптазы. Однако реакцию транскрипции обычно осуществляют также без этой стадии.
Реакционная смесь с обратной транскриптазой: 28 Ед. Rnasin (Promega), 25 мМ дНТФ, 5 мкл 10 х буфера для обратной транскриптазы [10 мМ Трис/HCl (рН=8,3), 75 мМ KCl, 3 мМ MgCl2], 50 Ед. обратной транскриптазы (StrataScriptTM, Stratagene); доведено до объема 50 мкл с помощью дистиллированной воды.
Для обратной транскрипции смесь инкубируют в течение 15 минут при температуре 42°С и в течение 45 минут при температуре 37°С. Более продолжительная инкубация при температуре 42°С в течение 2 часов с 30-секундным перерывом при температуре 55°С рекомендуется в случае более длинных матриц РНК. Последующая инкубация смеси в течение 5 минут при температуре 95°С приводит к инактивации обратной транскриптазы. Для реакции PCR тогда используют 5-20 мкл смеси с обратной транскриптазой.
Получение геномной ДНК из тканей
100 мг тканей разрушают в жидком азоте до получения порошка. К порошку из тканей в пробирке Falcon добавляют 6 мл буфера для реакции (к буферу незадолго до этого добавляют 30 мкл протеиназы К [20 мг/мл]) и инкубируют в течение ночи (12-18 часов) при температуре 56°С при осторожном встряхивании. После инкубации добавляют 100 мкл рибонуклеазы А (10 мкг/мкл) и инкубируют в течение часа при температуре 37°С при дальнейшем встряхивании. Сразу же после этого добавляют 4 мл фенола и пробирку в течение примерно 5 минут вручную переворачивают путем опрокидывания. Немедленно после этого добавляют 4 мл смеси хлороформа и изоамилового спирта и повторяют 5-минутное опрокидывание и затем центрифугируют в течение 15 минут (3000 оборотов в минуту). Супернатант осторожно отбирают и переносят в пробирку Falcon емкостью 10 мл. Если супернатант еще непрозрачный, нужно повторить экстракцию фенолом, в остальном снова добавляют 4 мл смеси хлороформа и изоамилового спирта, и пробирку в течение 5 минут вручную переворачивают путем опрокидывания, и после этого центрифугируют в течение 15 минут (3000 оборотов в минуту). Супернатант осторожно отбирают и повторяют экстракцию смесью хлороформа и изопропилового спирта. К полученному в конечном счете супернатанту добавляют 1/10 объема раствора ацетата натрия (3 М, рН=6) и 2,5-кратный объем этанола (99,8%-ный). Пробирку осторожно поворачивают до тех пор, пока ДНК не выпадет в осадок в виде клубка. Этот клубок ДНК с помощью стеклянного крючка переносят в примерно 25 мл этанола (70%-ный) и оставляют на 3 минуты в неподвижном состоянии. Промывку повторяют два раза. Затем ДНК высушивают на воздухе и при комнатной температуре растворяют в 0,5 мл дистиллированной воды.
Саузерн-блот
Отделение ДНК при помощи агарозного геля
Гель для разрушения нитей более крупных молекул ДНК (>6 т.н.) выдерживают в течение примерно 5 минут под воздействием коротковолнового ультрафиолетового излучения.
Гель для денатурации ДНК "прополаскивают" в течение 30 минут в растворе для денатурации.
Гель для нейтрализации "прополаскивают" в течение 30 минут в растворе для нейтрализации.
Структура блота (снизу вверх): гель, нейлоновая мембрана, сухая фильтровальная бумага, впитывающая бумага, пластина, масса (примерно 1 кг).
Блоттинг осуществляют в течение ночи с помощью 20 х SSC.
Мембрану промывают в течение 10 минут в 2 х SSC. Мембрану высушивают на фильтровальной бумаге. Нуклеиновую кислоту фиксируют путем обжига при температуре 80°С в течение 1 часа или путем УФ-сшивки (например, в "Stratalinker", автоматическая фиксация положения). Мембрану затем можно хранить вплоть до гибридизации.
За счет предварительной гибридизации мембраны в растворе для гибридизации в течение примерно 1-2 часов удаляют неспецифические места связывания на мембране.
Раствор для гибридизации: 5 х SSC, 5 х раствор Denhardt, 0,5% додецилсульфата натрия, 100 мкг/мл ДНК спермы сельдевых.
Раствор для денатурации: 0,5 М NaOH (20 г), 1 М NaCl.
Раствор для нейтрализации: 1,5 М NaCl/0,5 М Трис, рН=7,4.
20 х SSC: 3 М NaCl, 0,3 М цитрата натрия: 175,3 г NaCl, 88,2 г цитрата натрия х 2 Н2О, доливают до 1 л бидистиллированной водой; рН=7,0 устанавливают с помощью HCl.
50 х раствор Denhardt: 5 г фиколла 400, 5 г PVP (поливинилпирролидон), 5 г бычьего сывороточного альбумина, бидистиллированная вода до общего объема 500 мл.
Назерн-блот
Отделение РНК при помощи агарозного геля с формальдегидом
Структура блота (снизу вверх): гель, нейлоновая мембрана, сухая фильтровальная бумага, впитывающая бумага, пластина, масса (примерно 1 кг).
Блоттинг осуществляют в течение ночи с помощью 20 х SSC.
РНК фиксируют на фильтре путем обжига при температуре 80°С (1 час). Фильтр для деглиоксилирования РНК вносят в кипящий 20 мМ Трис, рН=8, и оставляют охлаждаться до комнатной температуры.
ОПИСАНИЕ ФИГУР
Фиг.1
Характеристика исследуемой группы.
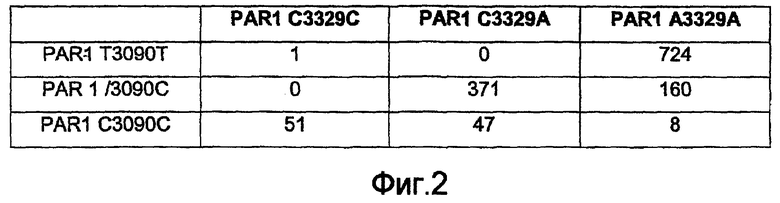
Фиг.2
Распределение вариантов PAR1 Т3090С и А3329С у 1362 анализируемых индивидов.
Фиг.3
Ассоциация варианта PAR1 С3090С с мерцанием предсердий и кардиомиопатией.
Фиг.4
Ассоциация варианта PAR1 С3229С с мерцанием предсердий, острым коронарным синдромом и нестабильной стенокардией.
Фиг.5
Полинуклеотидная последовательность кДНК человеческого гена PAR1 в 5'/3'-структуре. Последовательность соответствует последовательности, известной под номером NM_001992 в базе данных по нуклеотидам NCBI. Полученная последовательность идентична SEQ ID №1.
Фиг.6
Полинуклеотидная последовательность кДНК человеческого гена PAR1 в 5'/3'-структуре с полиморфизмом в положении 3090 последовательности согласно NM_001992, состоящим из замены Т на С. Представленная последовательность идентична SEQ ID №2.
Фиг.7
Полинуклеотидная последовательность кДНК человеческого гена PAR1 в 5'/3'-структуре с полиморфизмом в положении 3329 последовательности согласно NM_001992, состоящим из замены А на С. Представленная последовательность идентична SEQ ID №3.
Фиг.8
Полинуклеотидная последовательность кДНК человеческого гена PAR1 в 5'/3'-структуре с полиморфизмом в положении 3090 последовательности согласно NM_001992, состоящим из замены Т на С, а также с одновременно имеющимся вторым полиморфизмом в положении 3329 последовательности согласно NM_001992, состоящим из замены А на С. Представленная последовательность идентична SEQ ID №4.
Фиг.9
Полинуклеотидная последовательность фрагмента человеческого гена PAR1 в 5'/3'-структуре. Представленная последовательность идентична SEQ ID №5.
Фиг.10
Полинуклеотидная последовательность фрагмента человеческого гена PAR1 в 5'/3'-структуре с полиморфизмом в положении 3090 последовательности согласно NM_001992, состоящим из замены Т на С. Представленная последовательность идентична SEQ ID №6.
Фиг.11
Полинуклеотидная последовательность фрагмента человеческого гена PAR1 в 5'/3'-структуре с полиморфизмом в положении 3329 последовательности согласно NM_001992, состоящим из замены А на С. Представленная последовательность идентична SEQ ID №7.
Фиг.12
Полинуклеотидная последовательность фрагмента человеческого гена PAR1 в 5'/3'-структуре с полиморфизмом в положении 3090 последовательности согласно NM_001992, состоящим из замены Т на С, а также с одновременно имеющимся вторым полиморфизмом в положении 3329 последовательности согласно NM_001992, состоящим из замены А на С. Представленная последовательность идентична SEQ ID №8.
Фиг.13
Полинуклеотидная последовательность в 5'/3'-структуре 5'-конца кДНК человеческого гена PAR1. Представленная последовательность идентична SEQ ID №9.
Фиг.14
Полинуклеотидная последовательность в 5'/3'-структуре 3'-конца кДНК человеческого гена PAR1. Представленная последовательность идентична SEQ ID №10.
Фиг.15
Полинуклеотидная последовательность в 5'/3'-структуре кДНК человеческого гена PAR1, соответствующая положениям 2767-2789 согласно NM_001992. Представленная последовательность идентична SEQ ID №11.
Фиг.16
Полинуклеотидная последовательность в 5'/3'-структуре экзона №1 человеческого гена PAR1. Представленная последовательность идентична SEQ ID №12.
Фиг.17
Белковая последовательность человеческого рецептора PAR1. Последовательность соответствует последовательности, известной под номером NP_001983 базы данных по белкам NCBI. Представленная последовательность идентична SEQ ID №13.









| название | год | авторы | номер документа |
|---|---|---|---|
| ВЫСОКОПРОИЗВОДИТЕЛЬНЫЙ АНАЛИЗ ТРАНСГЕННЫХ ГРАНИЦ | 2012 |
|
RU2603265C2 |
| МОДИФИЦИРОВАННАЯ ТЕРМОСТАБИЛЬНАЯ ДНК-ПОЛИМЕРАЗА, СПОСОБ ЕЁ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЕ | 1997 |
|
RU2235773C2 |
| СПОСОБ ВЫЯВЛЕНИЯ ПАРВОВИРУСА B19 ЧЕЛОВЕКА В БИОЛОГИЧЕСКОМ ОБРАЗЦЕ (ВАРИАНТЫ) И НАБОР ДЛЯ ЕГО ОСУЩЕСТВЛЕНИЯ | 2002 |
|
RU2301263C2 |
| СПОСОБЫ, СИСТЕМЫ И КОМПОЗИЦИИ ДЛЯ ДЕТЕКЦИИ МИКРОБНОЙ ДНК ПРИ ПОМОЩИ ПЦР | 2012 |
|
RU2620953C2 |
| ГЕНЕТИЧЕСКАЯ МОДИФИКАЦИЯ MON89788 СОИ И СПОСОБЫ ЕЕ ОПРЕДЕЛЕНИЯ | 2006 |
|
RU2411720C2 |
| Способ анализа соматических мутаций в химерном гене BCR/ABL с использованием ОТ-ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом) | 2015 |
|
RU2609641C1 |
| Набор праймеров для идентификации РНК штаммов Puumala orthohantavirus, распространённых в Республике Татарстан | 2019 |
|
RU2720252C1 |
| СПОСОБ РАСШИРЕННОГО СКРИНИНГА ПРЕДРАСПОЛОЖЕННОСТИ К СЕРДЕЧНО-СОСУДИСТЫМ ЗАБОЛЕВАНИЯМ И БИОЧИП ДЛЯ ОСУЩЕСТВЛЕНИЯ ЭТОГО СПОСОБА | 2010 |
|
RU2453606C2 |
| СЕКВЕНИРОВАНИЕ ПОЛИНУКЛЕОТИДНЫХ БИБЛИОТЕК С ВЫСОКОЙ ПРОПУСКНОЙ СПОСОБНОСТЬЮ И АНАЛИЗ ТРАНСКРИПТОМОВ | 2018 |
|
RU2790291C2 |
| СПОСОБ ВЫЯВЛЕНИЯ МУТАЦИЙ В СЛОЖНЫХ СМЕСЯХ ДНК | 2014 |
|
RU2613489C2 |












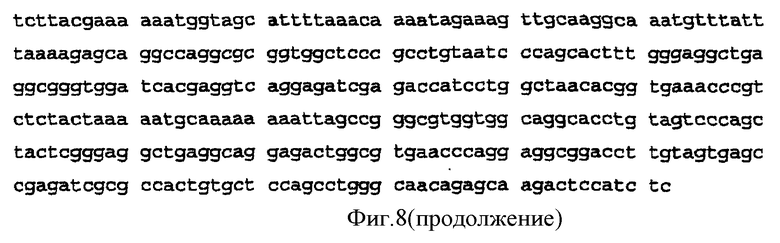





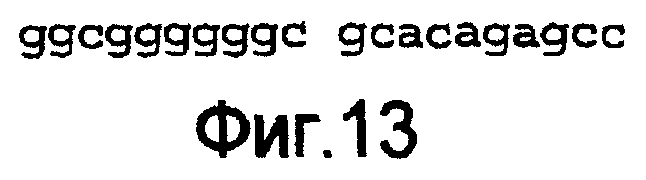
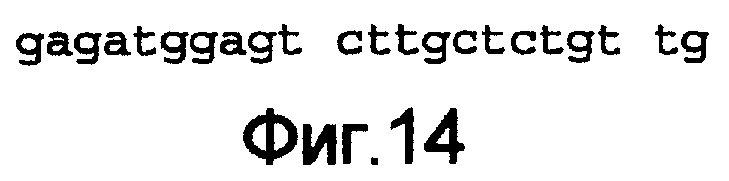
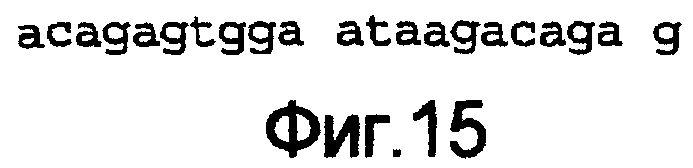
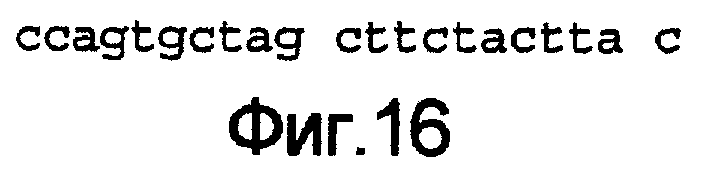

Изобретение относится к области молекулярной биологии и может быть использовано в медицинской генетике. Описан способ получения полиморфной области гена PAR1, которая может содержать замену Т на С в положении 3090 и/или замену А на С в положении 3329 полинуклеотидной последовательности гена дикого типа (NM_001992), с применением пары специфических праймеров, а также способ определения в названной области, полученной из ДНК-содержащего биологического образца, наличия или отсутствия указанных замен. Предложены полные наборы компонентов, применение которых обеспечивает как амплификацию полиморфной области гена PAR1 по изобретению, так и в случае необходимости, проведение дальнейшего анализа ее на наличие генетических модификаций в положениях 3090 и/или 3329. 6 с. и 1 з.п. ф-лы, 17 ил.
1. Способ получения изолированной полиморфной области гена PAR1 с последовательностью, выбранной из SEQ ID NO: 5, SEQ ID NO: 6, SEQ ID NO: 7 и SEQ ID NO: 8, включающий следующие стадии:
a) получение человеческой геномной ДНК, содержащей последовательность PAR1, выбранную из следующей группы: SEQ ID NO: 1, SEQ ID NO: 2, SEQ ID NO: 3, SEQ ID NO: 4;
b) получение пары праймеров согласно SEQ ID NO: 11 и SEQ ID NO: 12;
c) амплификация фрагмента полинуклеотидной последовательности PAR1 путем полимеразной цепной реакции (PCR) с праймерами согласно стадии b);
d) выделение и очистка образовавшегося фрагмента со стадии с);
e) секвенирование фрагмента со стадии d).
2. Способ обнаружения наличия или отсутствия в гене PAR1 замены Т на С в положении 3090 последовательности согласно NM_001992, включающий следующие стадии:
a) получение биологического материала, содержащего клетки человека;
b) получение геномной ДНК из материала со стадии а);
c) амплификация фрагмента полинуклеотида посредством полимеразной цепной реакции с праймерами согласно SEQ ID NO: 11 и SEQ ID NO: 12;
d) секвенирование фрагмента полинуклеотида со стадии с) и составление заключения.
3. Применение выделенной полинуклеотидной последовательности, состоящей из 21-50 последовательно расположенных в гене PAR1 нуклеотидов, отличающейся тем, что она содержит последовательность согласно SEQ ID NO: 11 или состоит из SEQ ID NO: 11, в комбинации с выделенной полинуклеотидной последовательностью, состоящей из 21-50 последовательно расположенных в гене PAR1 нуклеотидов, отличающейся тем, что она содержит последовательность согласно SEQ ID NO: 12 или состоит из SEQ ID NO: 12, для амплификации фрагмента гена PAR1 посредством полимеразной цепной реакции.
4. Применение по п.3, отличающееся тем, что оно предназначено для определения, имеет ли ген PAR1 замену Т на С в положении 3090 последовательности согласно NM_001992 и/или замену А на С в положении 3329 последовательности согласно NM_001992.
5. Набор компонентов для амплификации полиморфной области гена PAR1, содержащий:
a) полинуклеотидную последовательность, состоящую из 21-50 последовательно расположенных в гене PAR1 нуклеотидов, отличающуюся тем, что она содержит последовательность согласно SEQ ID NO: 11 или состоит из SEQ ID NO: 11;
b) полинуклеотидную последовательность, состоящую из 21-50 последовательно расположенных в гене PAR1 нуклеотидов, отличающуюся тем, что она содержит последовательность согласно SEQ ID NO: 12 или состоит из SEQ ID NO: 12;
c) по меньшей мере фермент с полимеразной активностью для осуществления полимеразной цепной реакции;
d) вещества и/или растворы для осуществления полимеразной цепной реакции (PCR); и
e) в случае необходимости, реагенты для осуществления секвенирования.
6. Способ получения набора компонентов для амплификации полиморфной области гена PAR1, согласно которому
a) получают полинуклеотидную последовательность, состоящую из 21-50 последовательно расположенных в гене PAR1 нуклеотидов, отличающуюся тем, что она содержит последовательность согласно SEQ ID NO: 11 или состоит из SEQ ID NO: 11;
b) получают полинуклеотидную последовательность, состоящую из 21-50 последовательно расположенных в гене PAR1 нуклеотидов, отличающуюся тем, что она содержит последовательность согласно SEQ ID NO: 12 или состоит из SEQ ID NO: 12;
c) получают фермент с полимеразной активностью для осуществления полимеразной цепной реакции;
d) готовят вещества и/или растворы для осуществления полимеразной цепной реакции (PCR);
e) готовят реагенты для осуществления секвенирования;
f) компоненты со стадий а)-е) соответственно раздельно помещают в пригодные емкости;
g) емкости со стадии f) в случае необходимости объединяют в одной или нескольких единицах упаковки.
7. Применение набора компонентов по п.5 или 6 для амплификации полиморфной области гена PAR1, а также в случае необходимости, дальнейшего анализа на наличие генетических модификаций в гене PAR1 в положениях 3090 и/или 3329 согласно NM_001992.
| PARK H-Y | |||
| ET AL., Clin | |||
| Exp | |||
| Parmacol | |||
| Physiol., 27(9), 690-693, 2000 | |||
| ARNAUD Е | |||
| ET AL., Arterioscler | |||
| Thromb | |||
| Vasc | |||
| Biol, 20 (2), 585-592, 2000. |
Авторы
Даты
2010-01-27—Публикация
2004-04-16—Подача