ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
[0001] Настоящая заявка испрашивает приоритет предварительной заявки США 61/487709, поданной 19 мая 2011 года, полное содержание которой включено в данный документ посредством ссылки.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
[0002] Данная заявка содержит перечень последовательностей.
ОБЛАСТЬ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
[0003] Настоящее изобретение относится к области молекулярных анализов. Более конкретно, настоящее изобретение относится к способу детекции незначительного количества микробной ДНК на основе ПЦР и композициям для осуществления детекции незначительного количества микробной ДНК.
ПРЕДПОСЫЛКИ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
[0004] Теоретически полимеразная цепная реакция (ПЦР) потенциально является наиболее чувствительной среди всех существующих способов для быстрой детекции микроорганизмов в анализируемом образце (например, в образце продуктов питания или клинической пробе). В тех случаях, когда требуется детекция широкого спектра патогенов и тех, которые трудно культивировать in vitro или которые требуют длительного периода культивирования, значение ПЦР особенно высоко. Например, путем нацеливания на консервативный ген, как, например, ген 16S рРНК для бактерий или 18S для эукариотических патогенных микроорганизмов, как, например, грибы, можно проводить рандомизированный скрининг или детекцию с использованием всего лишь одной реакции ПЦР. Однако использование на практике универсальной ПЦР для детекции незначительных количеств микроорганизмов все еще сопряжено со многими сложными задачами. Некоторые факторы, которые ограничивают практическое применение ПЦР при детекции микроорганизмов, включают присущую ПЦР чувствительность к воздействию ингибиторов, контаминации и экспериментальным условиям. Одной из наиболее сложных и давних проблем является эндогенная контаминация реактивов для ПЦР.
[0005] Реактивы для осуществления ПЦР, даже коммерчески доступные, получают из бактериальных источников. Реактивы, как таковые, в частности, Taq ДНК-полимеразы, неизбежно содержат некоторый уровень контаминирующей ДНК из источника бактерий. Контаминирующие ДНК обычно включают несколько штаммов или видов, которые нельзя идентифицировать, такие как Thermus aquaticus или Escherichia coli, хотя они имеют близкую гомологию с видами Pseudomonas fluorescens, Pseduomonas aeruginosa, Alcaligenes faecalis или Azotobacter vinelandii, которые все являются важными с клинической точки зрения. В результате этого в традиционной ПЦР часто происходит коамплификация данных контаминантов с целевой микробной ДНК с образованием чрезвычайно высокого уровня ложноположительных результатов, тем самым, представляя ПЦР-анализы настолько ненадежными, что их исключают из клинических применений.
[0006] За последние 20 лет предпринимались многочисленные попытки для решения данной проблемы. Некоторые примеры включают УФ-облучение, расщепление эндонуклеазой рестрикции, ультрафильтрацию и предварительную обработку реактивов ДНКазой I (ссыл. 20-23). К сожалению, все предыдущие попытки полностью устранить ложноположительные результаты либо провалились из-за присущих ограничений по условиям реакции, либо не смогли достигнуть требуемого уровня чувствительности из-за сопутствующего ингибирования ПЦР-реакции. Вследствие этого, как правило, считается, что универсальная ПЦР не релевантна с клинической точки зрения, при этом большинству исследователей остается продолжать следовать ПЦР или другим отличным от ПЦР способам, которые являются дорогостоящими, трудно оптимизируемыми и которые являются неудобными для осуществления.
[0007] Таким образом, все еще существует неудовлетворенная потребность в решении сложных задач, связанных с применением ПЦР для клинической детекции микроорганизмов.
КРАТКОЕ ОПИСАНИЕ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
[0008] В свете вышеизложенного, целью настоящего изобретения является обеспечение способа детекции микроорганизмов в образце на основе ПЦР, который является чувствительным, точным, недорогим и легким для осуществления.
[0009] Также целью настоящего изобретения является обеспечение реактивов и диагностических наборов для того, сделать возможным клинические применения молекулярных методов диагностики на основе ПЦР, в частности, в условиях лечения критических состояний.
[0010] Дополнительной целью настоящего изобретения является обеспечение системы для осуществления диагностического анализа на основе ПЦР для детекции микроорганизмов в клинических пробах и других биологических образцах.
[0011] Вышеизложенные цели настоящего изобретения удовлетворяет новый подход, называемый в данном документе PE-PCR, которая позволяет преодолеть давнюю проблему эндогенной контаминации реактивов для ПЦР.
[0012] Вкратце, подход PE-PCR подтверждает, что все предыдущие усилия решить проблему эндогенного контаминирования напрямую за счет попытки удалить или разрушить контаминанты привели к проблеме. В течение почти 20 лет такие прямые подходы приносили незначительный успех. В PE-PCR авторы обошли традиционное мышление и разработали непрямой подход, который оригинально обходит проблему эндогенной контаминации. Конкретно, подход PE-PCR использует гибридизационный ДНК-зонд для присоединения небактериальной последовательности к 5'-концу целевых матриц перед добавлением в реакционную смесь любого реактива для ПЦР. Таким образом, целевые матрицы отличаются от контаминирующих последовательностей по наличию 5'-концевых небактериальных последовательностей. С помощью праймера, комплементарного небактериальным меченым последовательностям, меченые матрицы можно селективно амплифицировать при помощи стандартной ПЦР. Поскольку небактериальный гибридизационный праймер (также известный как гибридизационный зонд) будет амплифицировать только 5'-концевые меченые матрицы, это превращает полученные из бактерий контаминирующие вещества в несуществующую проблему.
[0013] Соответственно, первый аспект настоящего изобретения ориентирован на способ селективной амплификации одной или нескольких целевых микробных ДНК в образце. В соответствии с данным аспектом настоящего изобретения в способах используют один или несколько гибридизационных ДНК-зондов, содержащих 5'-концевой участок, имеющий небактериальную последовательность, и 3'-концевой участок, имеющий последовательность, комплементарную участку целевой микробной ДНК. Способы, как правило, включают этапы гибиридизирования гибридизационного зонда (зондов) с целевой микробной ДНК в образце; удаления негибридизированного зонда (зонды) и несвязанного 3'-концевого участка целевой микробной ДНК; удлинения 3'-концов гибридизационных зондов и целевой микробной ДНК с образованием двухцепочечных, удлиненных с помощью праймера матриц; и осуществления способа ПЦР для селективной ампликации удлиненных с помощью праймера матриц при помощи набора праймеров, который включает по меньшей мере один праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационных зондов.
[0014] Второй аспект настоящего изобретения ориентирован на способ детекции микробной инфекции у субъекта. Способы в соответствии с данным аспектом настоящего изобретения также требуют множества ДНК-зондов, как описано выше. Они, как правило, включают этапы добавления гибридизационных зондов в образец, отобранный у субъекта; гибридизацию гибридизационных зондов с микробной ДНК в образце; удаления негибридизированных гибридизационных зондов и любой несвязанной 3'-концевой участок микробной ДНК; удлинения 3'-концов гибридизационных зондов и микробной ДНК с образованием двухцепочечных, удлиненных с помощью праймера матриц; амплифицирования удлиненных с помощью праймера матриц при помощи выполнения способа ПЦР с помощью набора праймеров, который включает по меньшей мере один прямой праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационного зонда; и анализа амплифицированных ПЦР-продуктов для определения присутствия или отсутствия микроорганизма.
[0015] Третий аспект настоящего изобретения ориентирован на гибридизационный зонд для создания удлиненной с помощью праймера ДНК-матрицы из целевой микробной ДНК в образце для селективной амплификации при помощи ПЦР. Гибридизационные зонды в соответствии с данным аспектом настоящего изобретения, как правило, содержат 5'-концевой участок, имеющий небактериальную последовательность, и 3'-концевой участок, имеющий последовательность, комплементарную участку целевого образца микроорганизма.
[0016] Четвертый аспект настоящего изобретения ориентирован на набор для создания удлиненной с помощью праймера ДНК-матрицы из целевой микробной ДНК для селективной ПЦР-амплификации и детекции. Наборы в соответствии с данным аспектом настоящего изобретения, как правило, включают множество гибридизационных зондов, как описано выше; и смесь 3'-5' экзонуклеазы и полимеразы Кленова, укомплектованные в удобном для использования виде.
[0017] Пятый аспект настоящего изобретения ориентирован на систему для детекции целевого микроорганизма в образце. Системы в соответствии с данным аспектом настоящего изобретения, как правило, содержат блок приема образца для приема образца; блок обработки образца для добавления реактивов в образец и поддерживания условий реакции; и блок анализа образца для анализа обработанного образца. Блок обработки образца предпочтительно автоматизирован и выполнен с возможностью осуществления этапов добавления множества гибридизационных зондов в образец и поддерживания условия, позволяющего гибридизационным зондам гибридизироваться с микробной ДНК в образце; добавления реактива для разрушения/удлинения для удаления негибридизированных гибридизационных зондов и несвязанного 3'-концевого участка гибридизированной целевой микробной ДНК, и для удлинения 3'-концов гибридизированных гибридизационных зондов и целевой микробной ДНК в направлении от 3' к 5' для создания двухцепочечных, удлиненных с помощью праймера матриц; и добавления реактивов для ПЦР и поддерживания условий реакции для осуществления амплификации удлиненных с помощью праймера матриц; и перемещения обработанного образца в блок анализа для анализа.
[0018] Другие аспекты и преимущества настоящего изобретения станут понятными из следующего описания и прилагаемой формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
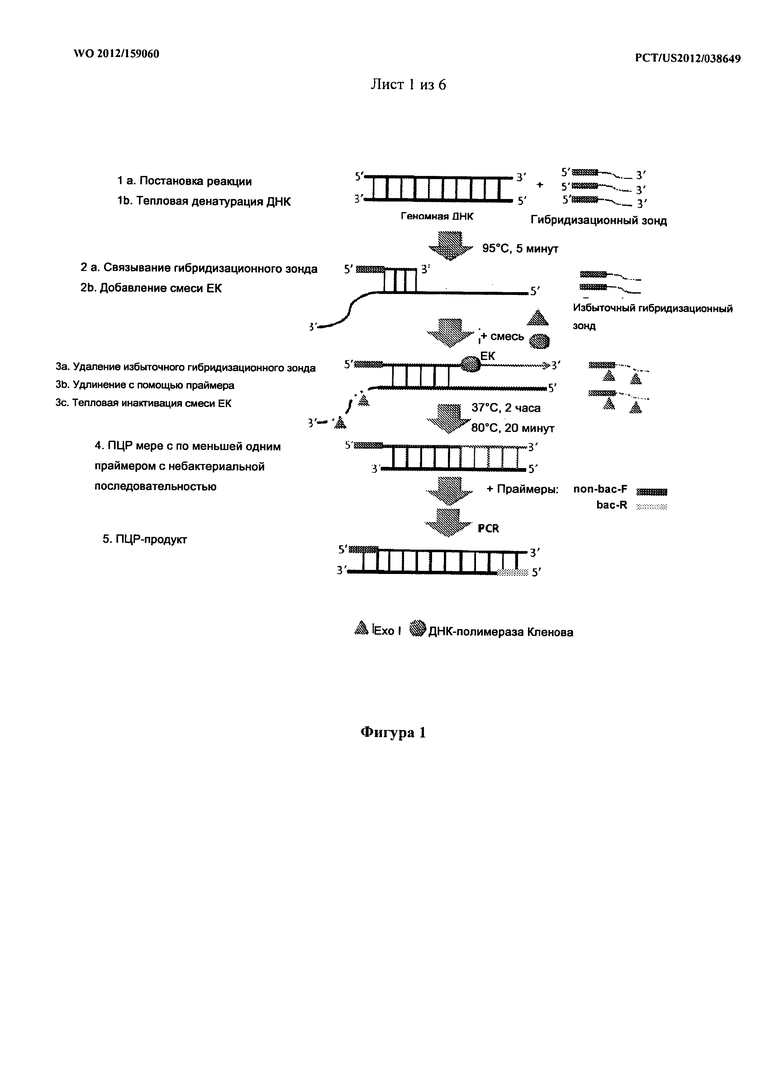
[0019] На фигуре 1 показано схематическое изображение способа PE-PCR в соответствии с вариантами осуществления настоящего изобретения.
[0020] На фигуре 2 показано изображение гель-электрофореза, иллюстрирующее, что коммерчески доступные полимеразы недостаточно чистые для чувствительной и специфичной универсальной амплификации бактериальной ДНК.
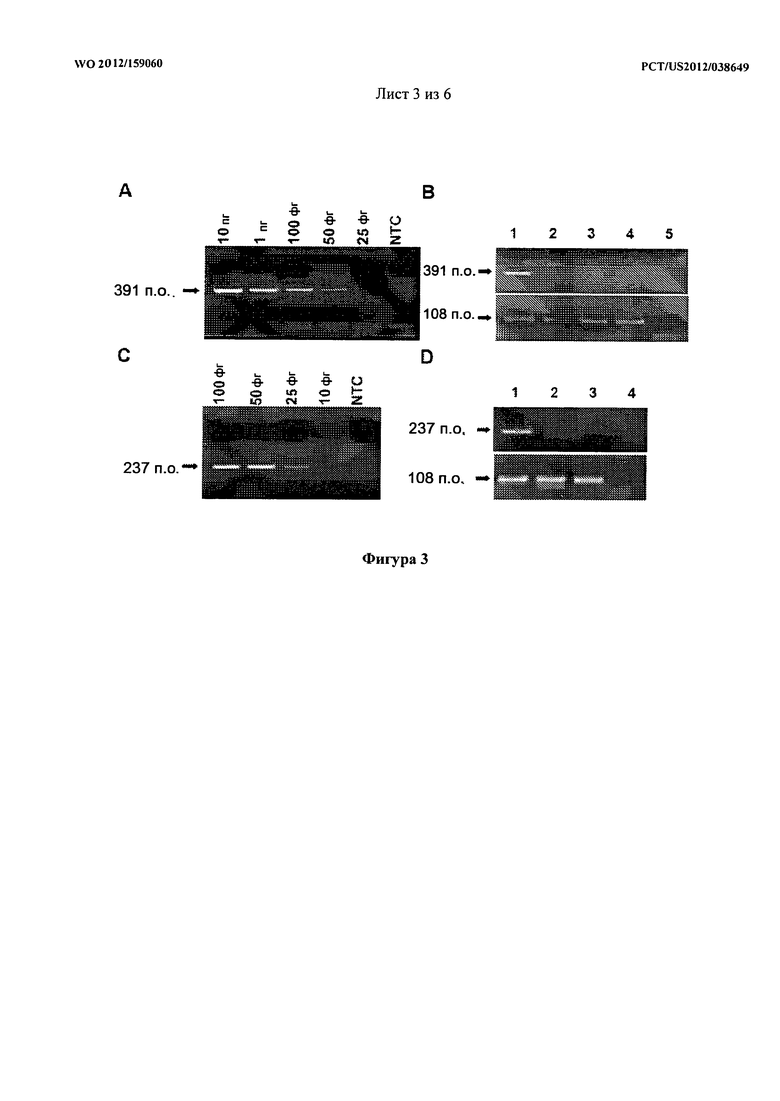
[0021] На фигуре 3 показаны изображения гель-электрофореза, демонстрирующие, что способы PE-PCR в соответствии с вариантами осуществления настоящего изобретения позволяют специфически амплифицировать целевую бактериальную ДНК без коамплификации контаминирующей бактериальной ДНК. (A) Указанное количество геномной ДНК S. aureus подвергали PE-PCR с использованием гибридизационного зонда M13-TstaG422 и набора праймеров M13 и TstaG765. (B) Геномную ДНК S. aureus (100 фг) подвергали PE-PCR (верхняя панель), как описано на панели А (полоса 1), в отсутствие праймера М13 (полоса 2) и при условии искусственной контаминации путем добавления 100 фг геномной ДНК S. aureus в смесь EK (полоса 3) или ПЦР-смеси (полоса 4). PE-PCR также осуществляли в отсутствие матричной ДНК (полоса 5). Присутствие геномной ДНК S. aureus подтверждали путем видоспецифичной ПЦР, в которой амплифицируется фрагмент хромосомной ДНК, специфичный для 5. aureus (нижняя панель). (C) Указанное количество геномной ДНК S. aureus использовали в качестве матрицы для универсальной PE-PCR с использованием гибридизационного зонда M13-16S-p201F и набора праймеров М13 и pi370. (D) Геномную ДНК S. aureus (100 фг) подвергали универсальной PE-PCR (верхняя панель), как описано на панели С (полоса 1), и при условии искусственной контаминации путем добавления 100 фг геномной ДНК S. aureus в смесь EK (полоса 2) или ПЦР-смеси (полоса 3). Универсальную PE-PCR также осуществляли в отсутствие матричной ДНК (полоса 4). Присутствие геномной ДНК S. aureus подтверждали при помощи видоспецифичной ПЦР для амплификации фрагмента хромосомной ДНК S. aureus (нижняя панель). NTC означает контроль без матрицы.
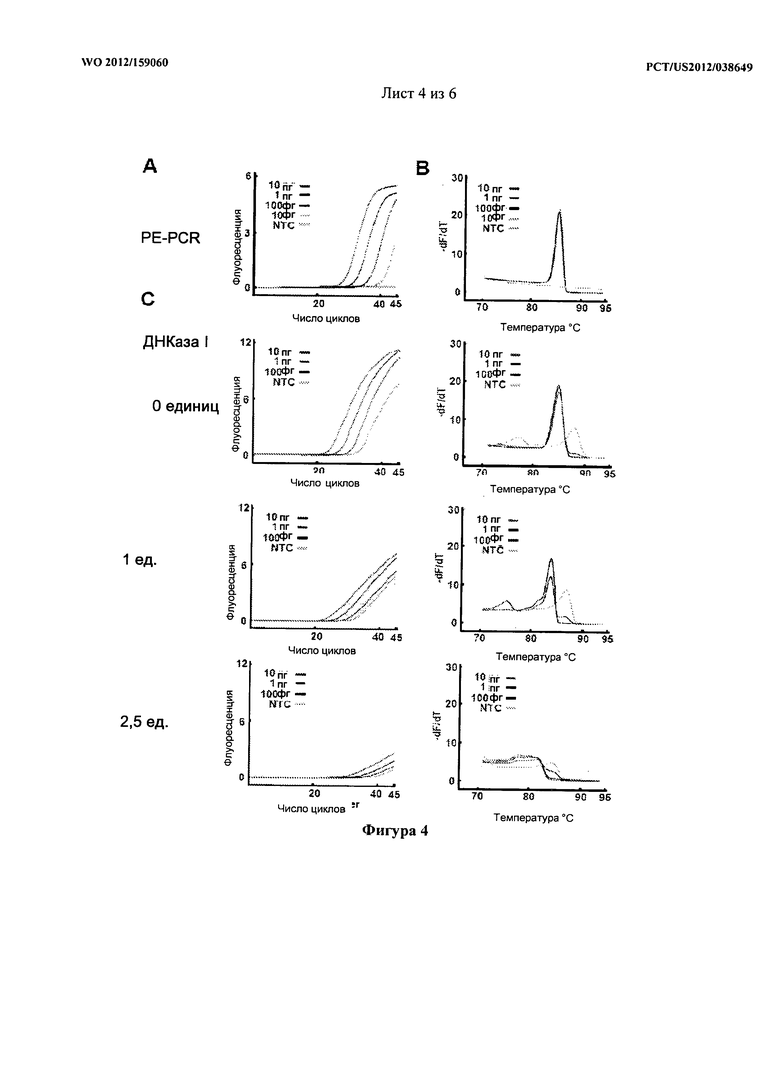
[0022] На фигуре 4 показано сравнение амплификации матричной бактериальной ДНК при универсальной PE-PCR в режиме реального времени и универсальной ПЦР в режиме реального времени после предварительной обработки реактивов для ПЦР ДНКазой I. (A) Указанные количества геномной ДНК S. aureus подвергали универсальной PE-PCR в режиме реального времени с использованием гибридизационного зонда М13-16S-p201F и набора праймеров М13 и р1370 в присутствии красителя LCGreen I plus для HRM. (C) В качестве альтернативы, реакционные ПЦР-смеси с или без предварительной обработки ДНКазой I (1 ед и 2,5 ед) использовали для ПЦР в режиме реального времени для амплификации указанных количеств геномной ДНК S. aureus. ПЦР-продукты подвергали HRMA с использованием прибора HR-1. Показаны амплификация (все фигуры в левом столбце) и графики производной (все фигуры в правом столбце). NTC означает контроль без матрицы.
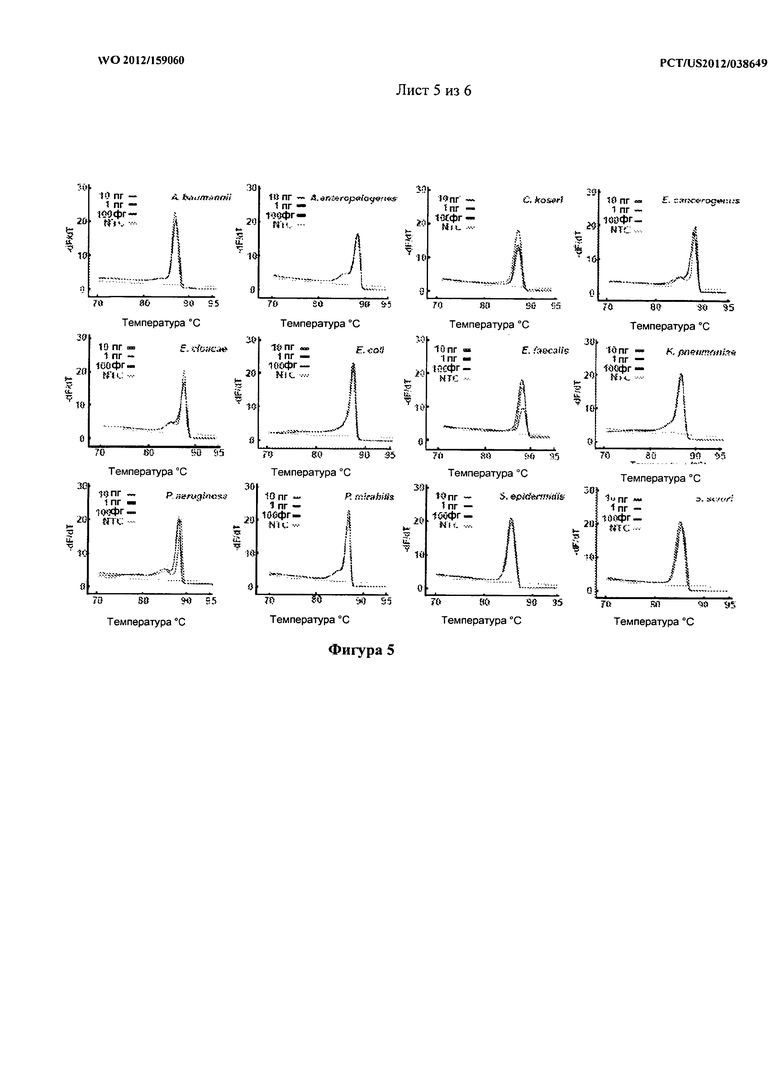
[0023] На фигуре 5 показаны графики кривых плавления для универсальной PE-PCR в режиме реального времени и HRM-анализа для 12 различных видов бактерий. Указанные количества геномной ДНК из 12 разных видов бактерий подвергали универсальной PE-PCR в режиме реального времени с использованием гибридизационного зонда M13-16S-p201F и набора праймеров М13 и р1370 в присутствии LCGreen I plus. ПЦР-продукт подвергали HRM-анализу, используя прибор HR-1, и показаны графики производной. NTC означает контроль без матрицы.
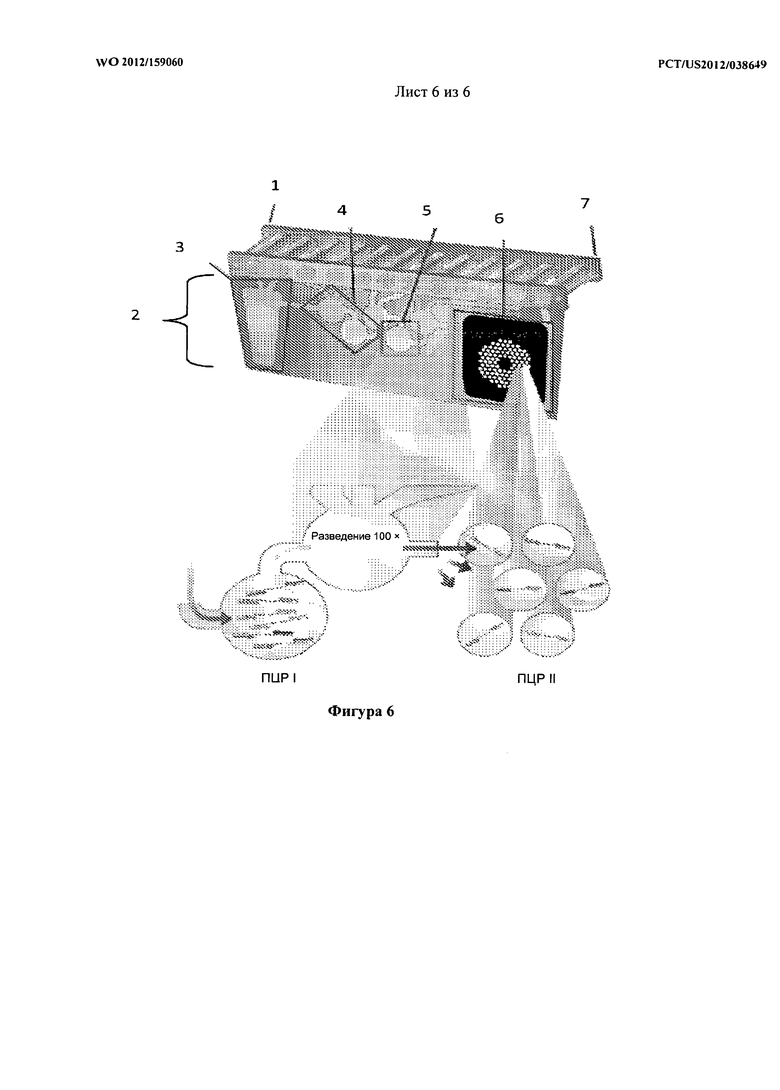
[0024] На фигуре 6 показано схематическое изображение системы в соответствии с вариантами осуществления настоящего изобретения.
ПОДРОБНОЕ ОПИСАНИЕ НАСТОЯЩЕГО ИЗОБРЕТЕНИЯ
[0025] Далее настоящее изобретение будет описано подробно посредством ссылки на конкретные варианты осуществления, как показано на прилагаемых фигурах. Несмотря на то, что настоящее изобретение будет описано с точки зрения конкретных вариантов осуществления и примеров, следует понимать, что варианты осуществления, раскрытые в данном документе, приведены только в целях иллюстрации, а специалисты в данной области техники могут вносить различные модификации и изменения, не отступая от сущности и объема настоящего изобретения, которые изложены в приложенной формуле изобретения. Определения
[0026] Если не указано иное, все выражения, используемые в данном документе, имеют нижеприведенные значения и, как правило, согласуются с тем же значением, которое имеют данные выражения для специалистов в данной области техники настоящего изобретения. В отношении определений и выражений данной области техники специалисты-практики, в частности, обращаются к Sambrook et al. (1989) Molecular Cloning: A Laboratory Manual (второе издание), Cold Spring Harbor Press, Plainview, N.Y. и Ausubel F M et al. (1993) Current Protocols in Molecular Biology, John Wiley & Sons, New York, N.Y. Следует понимать, что настоящее изобретение не ограничивается отдельной методикой, протоколами и описанными реактивами, поскольку они могут различаться.
[0027] Все публикации, цитированные в данном документе, прямо включены в данный документ посредством ссылки с целью описания и раскрытия композиций и методик, которые можно использовать в связи с настоящим изобретением.
[0028] Как используется в данном документе, выражение "ген" относится к последовательности нуклеиновой кислоты (например, ДНК), которая содержит кодирующие последовательности, необходимые для получения полипептида, предшественника или РНК (например, рРНК, тРНК). Полипептид может кодироваться кодирующей последовательностью полной длины или любым участком кодирующей последовательности, поскольку сохраняются требуемая активность или функциональные свойства (например, ферментативная активность, связывание лигандов, сигнальная трансдукция, иммуногенность и т.д.) полной длины или фрагмента. Выражение также охватывает кодирующую область структурного гена и последовательности, расположенные рядом с кодирующей областью как на 5', так и 3' концах, на расстоянии приблизительно 1 т.п.о. или более на любом конце, так что ген соответствует длине полной длины мРНК. Последовательности, расположенные 5' от кодирующей области и присутствующие в мРНК, называются 5'-нетранслируемыми последовательностями. Последовательности, расположенные 3' от или ниже кодирующей области и присутствующие в мРНК, называются 3'-нетранслируемыми последовательностями. Выражение "ген" охватывает как кДНК, так и геномные формы гена. Геномная форма или клон гена содержит кодирующую область, прерываемую некодирующими последовательностями, называемыми "интроны" или "вставочные области", или "вставочные последовательности". Интроны представляют собой сегменты гена, которые транскрибируются в ядерную РНК (гетерогенная ядерная РНК); интроны могут содержать регуляторные элементы, такие как энхансеры. Интроны удаляются или "вырезаются" из ядерного или первичного транскрипта; интроны, таким образом, отсутствуют в транскрипте матричной РНК (мРНК). МРНК функционирует на во время трансляции для определения последовательности или порядка аминокислот в растущем полипептиде.
[0029] "Зонды и/или праймеры", как используется в данном документе, могут представлять собой РНК или ДНК. ДНК может представлять собой либо кДНК, либо геномную ДНК. Полинуклеотидные зонды и праймеры представляют собой одно- или двухцепочечную ДНК или РНК, как правило, синтетические олигонуклеотиды, но могут быть созданы из клонированной кДНК или геномной последовательности, или ее комплементарных цепей. Аналитические зонды, как правило, будут составлять по меньшей мере 20 нуклеотидов в длину, хотя можно использовать несколько более короткие зонды (14-17 нуклеотидов). ПЦР-праймеры составляют по меньшей мере 5 нуклеотидов в длину, предпочтительно 15 или более нт, более предпочтительно 20-30 нт. Короткие полинуклеотиды можно использовать тогда, когда целью анализа является небольшая область гена. Для большого анализа генов, полинуклеотидный зонд может включать целый детектируемого сигнала, как, например, ферментом, биотином, радионуклидом, флуорофором, хемилюминесцентной меткой, парамагнитной частицей и им подобными, которые коммерчески доступны из многих источников, как, например, Molecular Probes, Inc., Юджин, Орегон и Amersham Corp., Арлингтон Хейтс, Иллинойс, с использованием методик, которые хорошо известны в данной области техники.
[0030] Считается, что молекулы ДНК имеют "5' концы" и "3' концы", потому что мононуклеотиды реагируют с образованием олигонуклеотидов или полинуклеотидов, таким образом, что 5' фосфат пентозного кольца одного мононуклеотида присоединяется к 3' кислороду соседнего в одном направлении через фосфодиэфирную связь. Таким образом, конец олигонуклеотида или полинуклеотида называют "5' концом", если его 5' фосфат не связан с 3' кислородом пентозного кольца мононуклеотида, и "3' концом", если его 3' кислород не связан с 5' фосфатом пентозного кольца последующего мононуклеотида. Как используется в данном документе, можно считать, что последовательность нуклеиновой кислоты также имеет 5' и 3' концы, даже если находятся внутри большего олигонуклеотида или полинуклеотида. Либо в линейной, либо в кольцевой молекуле ДНК, дискретные компоненты называются находящимися "выше" или 5' от находящихся "ниже" или 3' элементов. Данная терминология отражает тот факт, что транскрипция протекает в направлении от 5' к 3' вдоль цепи ДНК.
[0031] Как используется в данном документе, выражения "комплементарные" или "комплементарность" используют по отношению к полинуклеотидам (т.е., последовательностям нуклеотидов), связанных по правилам спаривания оснований. Например, последовательность "A-G-T" комплементарна последовательности "T-C-A". Комплементарность может быть "частичной", при которой только некоторые из оснований нуклеиновых кислот совпадают согласно правилам спаривания оснований. Или же между нуклеиновыми кислотами может быть "полная" или "абсолютная" комплементарность. Степень комплементарности между цепями нуклеиновых кислот оказывает значительные влияния на эффективность и силу гибридизации между цепями нуклеиновых кислот. Это представляет особенную важность для реакций амплификации, а также для способов детекции, которые зависят от образования связи между нуклеиновыми кислотами.
[0032] Как используется в данном документе, выражение "гибридизация" используют по отношению к спариванию комплементарных нуклеиновых кислот. На гибридизацию и силу гибридизации (т.е., силу ассоциации между нуклеиновыми кислотами) оказывают влияния такие факторы, как степень комплементарности между нуклеиновыми кислотами, жесткость применяемых условий, T.sub.m (Тпл) образовавшегося гибрида и соотношение G:C в пределах нуклеиновых кислот. Считается, что одиночная молекула, которая содержит спаренные комплементарные нуклеиновые кислоты в пределах ее структуры, "самогибридизируется."
[0033] "Амплификация" представляет собой частный случай репликации нуклеиновых кислот, включающий матричную специфичность. Ее следует отличать от неспецифичной матричной репликации (т.е., репликации, зависимой от матрицы, но независимой от специфичной матрицы). В данном случае матричная специфичность отличается от точности репликации (т.е., синтеза надлежащей полинуклеотидной последовательности) и (рибо- или дезоксирибо-) нуклеотидной специфичности. Матричную специфичность часто описывают с точки зрения "целевой" специфичности. Целевые последовательности являются "целями" в том смысле, что их необходимо отделить от другой нуклеиновой кислоты. Методы амплификации были разработаны в первую очередь для данного отделения.
[0034] Как используется в данном документе, выражение "полимеразная цепная реакция" ("ПЦР") относится к способу K.B. Mullis, патенты США №№4683195, 4683202 и 4965188, которые включены в данный документ посредством ссылки, где описывается способ увеличения концентрации сегмента целевой последовательности в смеси геномной ДНК без клонирования или очистки. Данный способ амплификации целевой последовательности состоит из введения значительного избытка двух олигонуклеотидных праймеров в смесь ДНК, содержащую требуемую целевую последовательность, за которым следует циклическое воздействие температуры в определенной последовательности в присутствии ДНК-полимеразы. Два праймера являются комплементарными соответствующим цепям двухцепочечной целевой последовательности. Для проведения амплификации смесь денатурируют и затем происходит отжиг праймеров с комплементарными последовательностями в пределах целевой молекулы. После отжига происходит удлинение праймеров с помощью полимеразы для того, чтобы образовалась новая пара комплементарных цепей. Этапы денатурации, отжиг праймеров и удлинение с помощью полимеразы могут повторяться много раз (т.е., денатурация, отжиг и удлинение составляют один "цикл"; может понадобиться множество "циклов") для получения высокой концентрации амплифицированного сегмента необходимой целевой последовательности. Длину амплифицированного сегмента необходимой целевой последовательности определяют по относительным положениям праймеров по отношению друг к другу, и таким образом, данная длина является контролируемым параметром. В силу аспекта повторения процесса, данный способ называют "полимеразной цепной реакцией" (далее в данном документе "ПЦР"). Так как необходимые амплифицированные сегменты целевой последовательности становятся преобладающими последовательностями (с точки зрения концентрации) в смеси, считается, что они "амплифицированы при помощи ПЦР".
[0035] Выражение "ПЦР в режиме реального времени", как используется в данном документ, означает, что сигнал, излучаемый при ПЦР-анализе, подвергается мониторингу на протяжении реакции в качестве индикатора получения ампликона на протяжении каждого цикла ПЦР-амплификации (т.е. в режиме "реального времени") в противоположность традиционным способам ПЦР, в которых сигнал анализа обнаруживается на конечном этапе ПЦР-реакции. ПЦР в режиме реального времени, как правило, основана на обнаружении и определении количества флуоресцентного репортера. Сигнал увеличивается прямо пропорционально количеству ПЦР-продукта в реакционной смеси. За счет регистрации количества испускания флуоресценции на каждом цикле, становится возможным осуществлять мониторинг ПЦР-реакции на протяжении экспоненциальной фазы, где первое значительное увеличение количества ПЦР-продукта коррелирует с первоначальным количеством целевой матрицы. В отношении общего описания "ПЦР в режиме реального времени" смотри Dehee et al. J. Virol. Meth. 102:37-51 (2002); и Aldea et al. J. Clin. Microbiol. 40:1060-1062 (2002) (ссылаясь на "LightCycler", где кинетическое определение количества в режиме реального времени дает возможность проводить измерения на протяжении log-линейной фазы ПЦР).
[0036] При помощи ПЦР становится возможным амплифицировать единственную копию специфичной целевой последовательности в геномной ДНК на уровне, поддающемся обнаружению посредством нескольких разных методик (например, гибридизации с меченым зондом; включения биотинилированных праймеров, за которой следует детекция конъюгата авидин-фермент; включения .sup.32P-меченных дезоксинуклеотидтрифосфатов, таких как дЦТФ или дАТФ, в амплифицированный сегмент). Помимо геномной ДНК можно амплифицировать последовательность любого олигонуклеотида или полинуклеотида с помощью соответствующего набора молекул праймеров. В частности, амплифицированные сегменты, созданные при помощи способа ПЦР, сами по себе являются эффективными матрицами для последующих ПЦР-амплификациий.
[0037] Как используется в данном документе, выражения "ПЦР-продукт", "ПЦР-фрагмент" и "продукт амплификации" относятся к полученной в результате смеси соединений после завершения двух или более циклов этапов ПЦР: денатурации, отжига и удлинения. Данные выражения охватывают случай, когда происходила амплификация одного или нескольких сегментов одной или нескольких целевых последовательностей
[0038] Как используется в данном документе, выражение "образец" используют в самом широком смысле. В некотором смысле, подразумевается, что он включает пробу или культуру, полученную из любого источника, а также биологические образцы и образцы окружающей среды. Биологические образцы могут быть получены из животных (включая людей) и охватывают жидкости, твердые вещества, ткани и газообразные вещества. Биологические образцы включают препараты крови, такие как плазма, сыворотка и им подобные. Образцы окружающей среды включают материалы окружающей среды, такие как проба с поверхности, почва, вода, кристаллы и промышленные образцы. Такие примеры не должны, однако, истолковываться как ограничивающие типы образцов, применимые к настоящему изобретению.
Общее описание подхода PE-PCR
[0039] Фигура 1 схематически иллюстрирует, как работает иллюстративный эксперимент PE-PCR. Начиная с этапа 1a, анализируемый образец, содержащий целевые ДНК-матрицы, такой как геномная ДНК микроорганизма, смешивают с избыточным количеством гибридизационных зондов. Каждый гибридизационный зонд состоит из 5'-концевого участка, имеющего небактериальную последовательность, за которым следует 3'-концевой участок, имеющий последовательность, комплементарную сайту в целевой ДНК-матрице.
[0040] На этапе 1b нагревают смесь образцов для денатурации ДНК-матриц, так, чтобы гибридизационные зонды могли гибридизироваться с целевыми ДНК-матрицами (этап 2a).
[0041] Как только гибридизационные зонды гибридизируются с предполагаемыми целями, в образец добавляют смесь 3'-концевой экзонуклеазы и ДНК-полимеразы Кленова (смесь EK) (этап 2b). Как показано на этапе 2a, если сайт гибридизации находится не в самом конце 3'-конца матрицы, образуется обтрепанный конец, так как небактериальный участок гибридизационного зонда не свяжется с матрицей.
[0042] На этапе 3a несвязанный участок 3'-конца целевой ДНК (обтрепанный конец) наряду с несвязанным избытком гибридизационных зондов расщепляются 3'-концевой экзонуклеазой. После расщепления экзонуклеазой создается выступ на 5'-конце цепи гибридизационного зонда.
[0043] На этапе 3b ДНК-полимераза Кленова будем затем удлинять как цепь гибридизационного зонда, так и матричную цепь в 5'>3' направлении с образованием двухцепочечных матриц с тупыми концами. Цепь, которая удлиняется со стороны гибридизационного зонда, будет иметь небактериальную меченую последовательность, расположенную на его 5'-конце, в то время как цепь, которая удлиняется со стороны целевой ДНК-матрицы будет иметь небактериальную меченую последовательность, расположенную на ее 3'-конце. Этот этап удлинения с помощью праймера можно завершить путем инактивации нагреванием смеси EK.
[0044] На данной стадии матрицы были мечены отличающимися последовательностями и готовы для амплификации при помощи ПЦР. Чтобы убедиться в том, что амплифицируются только меченые матрицы, набор праймеров, использованный для проведения ПЦР на этапе 4, должен включать по меньшей мере один прямой праймер (обозначенный на фигуре 1 как non-bac-F), который комплементарен небактериальной меченой последовательности, и обратный праймер (обозначенный на фигуре 1 как bac-R), который комплементарен последовательности на целевой ДНК-матрице ниже гибридизационного зонда.
[0045] Изложенный выше подход PE-PCR является простым, и его можно осуществить в одном реакционном сосуде без необходимости в каких-либо других предварительных обработках реактивов. Он обеспечивает готовое решение, которое разрешает давнюю проблему эндогенной контаминации бактериальной ДНК и является сопоставимым с существующими протоколами для ПЦР. На основе данного подхода изобретатели разработали различные способы, системы, композиции и наборы. Специалисты в данной области техники поймут, что подход PE-PCR можно преимущественно использовать в сочетании с любыми аналитическими методами, чтобы дополнительно определить количество или охарактеризовать ПЦР-продукты. Некоторые иллюстративные применения данного подхода могут включать методики молекулярной диагностики клинических проб для своевременной детекции бактериальной, вирусной или грибной инфекции. Другие применения могут включать применения в судебной экспертизе, обнаружении биологического оружия или определении количества археологического образца, но не ограничиваются ими. Гибридизационные зонды
[0046] В целом, гибридизационный зонд согласно вариантам осуществления настоящего изобретения представляет собой предпочтительно прямой праймер. Он имеет два компонента: один, кодирующий небактериальную последовательность, другой, кодирующий последовательность, комплементарную участку целевой ДНК. Небактериальная последовательность служит в качестве бактериальных контаминирующих веществ в реактивах для ПЦР. Комплементарная последовательность служит в качестве нацеливающей последовательности, которая связывает гибридизационный зонд с целевой ДНК в условиях гибридизации.
[0047] Правила конструирования эффективного гибридизационного зонда, как правило, известны в данной области техники. Гибридизационный зонд состоит из последовательности небактериальной ДНК на 5'-конце и последовательностей, комплементарних микробной целевой последовательности. Из-за возможного присутствия человеческой геномной ДНК на протяжении ПЦР-амплификации, 5'-концевая последовательность небактериальной ДНК преимущественно не комплементарна последовательности геномной ДНК человека. Похожий принцип подходит для применений, касающихся детекции бактериальной ДНК в образцах от других видов. 3'-концевые последовательности бактериальной ДНК являются комплементарными последовательностям целевого гена, которые повсеместно присутствуют в микробной ДНК. Tm 3'-бактериальной последовательности составляет по меньшей мере 37°C и самое большее 95°C. Для выбора последовательностей применяется общее правило конструирования праймеров, такое как отсутствие шпилек в последовательности.
[0048] Меченой последовательностью может быть последовательность, полученная из небактериального источника, или это может быть искусственно сконструированная последовательность. В некотором предпочтительном варианте осуществления меченая последовательность представляет собой вирусную последовательность, предпочтительно последовательность, полученную из фага M13.
[0049] Нацеливающая последовательность может быть уникальной последовательностью, которая связывает гибридизационный зонд с конкретной целевой матрицей или консервативной последовательностью, которая связывает гибридизационный зонд с широким спектром микробной ДНК. В некоторых иллюстративных вариантах осуществления нацеливающая последовательность представляет собой предпочтительно консервативную последовательность из гена 16S pPHK.
[0050] Гибридизационные зонды можно изготовить любыми способами, общеизвестными в данной области техники, включая, но без ограничений, химический синтез.
Способы детекции
[0051] Микробную ДНК, которая находится в образце в небольшом количестве, трудно обнаружить, ввиду ограничений способов детекции и приборов. Использование стандартной ПЦР для детекции небольшого количества ДНК в образце, как правило, включает целевые ДНК-матрицы, подвергающиеся селективной амплификации, в отличие от высокого уровня шумового фона. Их можно затем легко обнаружить за счет репликации целевой ДНК-матрицы до получения достаточно большого количества. Гибридизационные зонды, как описано выше, дают возможность для того, чтобы эффективно отличать целевую ДНК от фоновых помех и нежелательных контаминации, тем самым, обеспечивая селективную амплификацию целевой ДНК. После амплификации можно затем применять целый ряд аналитических инструментов для характеристики амплифицированных продуктов нуклеиновых кислот.
[0052] Соответственно, способы детекции микробной инфекции у субъекта в соответствии с вариантами осуществления настоящего изобретения, как правило, включают этапы, на которых добавляют гибридизационные зонды в образец, отобранный у субъекта; проводят гибридизацию гибридизационных зондов с микробной ДНК в образце; удаляют негибридизированные гибридизационные зонды и любой несвязанный 3'-концевой участок микробной ДНК; происходит удлинение 3'-концов гибридизационных зондов и микробной ДНК с образованием двухцепочечных, удлиненных с помощью праймера матриц; амплифицируют удлиненные с помощью праймера матрицы путем осуществления способа ПЦР с помощью набора праймеров, который включает по меньшей мере один прямой праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационного зонда; и анализируют амплифицированные ПЦР-продукты для определения присутствия или отсутствия микроорганизма.
[0053] Предпочтительно, образец берут из биологической жидкости субъекта, такой как кровь, моча, спинномозговая жидкость, слюна, мокрота или им подобные. Тип образца, который отбирают у субъекта, в частности, не ограничивается, но, как правило, будет соответствовать типу клинического состояния, которым, как предполагается, страдает субъект. Например, в предпочтительном варианте осуществления образцом является образец крови от субъекта, который, как полагают, страдает от бактериемия. В другом предпочтительном варианте осуществления образцом является спинномозговая жидкость от субъекта, который, как полагают, страдает от бактериальной инфекции, которую трудно культивировать in vitro (например, Chlamydia pneumoniae).
[0054] Для высвобождения ДНК из микроорганизмов типично необходима подготовка образца. Способы извлечения/выделения микробной ДНК, как правило, известны в данной области техники и могут быть преимущественно использованы для подготовки образцов. Однако такие подготовительные этапы не всегда необходимы, в зависимости от применения и конкретных целей анализа.
[0055] Гибридизация гибридизационных зондов с целевой ДНК, как правило, достигается при помощи нагревания образца сначала до верхней температуры плавления (Tm) для целевой ДНК так, чтобы они денатурировали до одноцепочечной ДНК, а затем постепенно охлаждают до нижней Tm, чтобы произошло связывание и отжиг. Подходящие условия гибридизации также известны в данной области техники, и их могут регулярно определять специалисты в данной области техники.
[0056] Удаление избытка гибридизационных зондов и 3'-концевого несвязанного участка целевой ДНК может достигаться при помощи 3'-экзонуклеазы. В предпочтительном варианте осуществления 3'-экзонуклеаза представляет собой Е. coli ехо I. Также можно использовать подходящим образом другую 3'-экзонуклеазу.
[0057] Удлинение матриц может достигаться за счет использования 5'-3'-полимеразы. В предпочтительном варианте осуществления используют 5'-3'-полимеразу Кленова.
[0058] 3'-экзонуклеаза и 3'-5'-полимераза для удобства могут обеспечиваться в одной смеси (смеси EK). Специалисты в данной области техники поймут, что оптимальное соотношение экзонуклеазы и полимеразы будет зависеть от конструкции гибридизационных зондов, а также от используемых конкретных экзонуклеазы и полимеразы. Такие соотношения можно определить путем регулярных экспериментов для оптимизации, и их предпочтительно провести до внедрения способа детекции в клинических условиях. Для подавления экзонуклеазной и полимеразной активностей, можно добавить стоп-реагент или образец можно просто нагреть для инактивации ферментов.
[0059] Способы ПЦР предпочтительно представляют собой стандартную ПЦР с использованием коммерчески доступной Taq ДНК-пояимеразы и набора праймеров, который включает по меньшей мере один прямой праймер, имеющий последовательность, комплементарную меченой последовательности гибридизационного зонда. Также можно использовать подходящим образом другие традиционные протоколы ПЦР, при условии, что протокол соответствует удлиненным с помощью праймера матрицам. Способы осуществления данных традиционных ПЦР, как правило, известны в данной области техники, и специалисты в данной области техники могут их адаптировать для работы с подходом PE-PCR настоящего изобретения. Например, можно также использовать ПЦР в режиме реального времени, чтобы одновременно амплифицировать удлиненные с помощью праймера матрицы и определить относительное содержание микробной ДНК.
[0060] Наконец, что не менее важно, амплифицированные ПЦР-продукты можно анализировать посредством многих традиционных аналитических способов, известных в данной области техники для характеристики и идентификации их происхождения. Например, для создания профиля кривых плавления к амплифицированным ПЦР-продуктам можно применить анализ кривых плавления. ДНК из разных микроорганизмов будут, как правило, обладать уникальным профилем кривых плавления, в силу отличающегося содержания нуклеотидов. Эти профили кривых плавления могут служить в качестве своего рода "фингерпринта" для идентификации происхождения образца.
[0061] Другие аналитические способы, которые можно использовать включают, но без ограничений, секвенирование, анализ с применением микрочипов, масс-спектроскопию, анализ кривых плавления, включая анализ кривых плавления с высокой разрешающей способностью, денатурирующую ВЭЖХ, капиллярный электрофорез, электрофорез в агарозном и/или полиакриламидном геле, анализ подвижности гетеродуплексов (НМА) и ЯМР-спектроскопию.
Системы и наборы
[0062] Для осуществления способа PE-PCR согласно настоящему изобретению, необходимы гибридизационные зонды и реактивы для удаления избытка зондов, а также удлинения гибридизированных зонда/матрицы, в дополнение к реактивам" для традиционной или ПЦР в режиме реального времени. Соответственно, наборы настоящего изобретения будут включать множество предварительно полученных гибридизационных зондов и реактивов для разрушения/удлинения (например, смесь EK) в удобной упаковке, как, например, во флаконе или картридже, но не ограничиваются ими. Набор может дополнительно включать инструкцию по применению для руководства по применению или дополнительные реактивы для облегчения ПЦР-амплификациий.
[0063] Для облегчения анализа с высокой производительностью, можно также сконструировать автоматизированные системы для осуществления способов детекции настоящего изобретения. На фигуре 6 показана блок-схема иллюстративной системы настоящего изобретения.
[0064] Ссылаясь на фигуру 6, система в соответствии с вариантами осуществления настоящего изобретения имеет блок приема образца 1 для приема образца, подлежащего анализу, блок обработки 2, выполненный с возможностью добавления различных реактивов в образец и поддерживания соответствующих условий реакции. Например, микробную ДНК можно выделить из образца в контейнере 3 в реакционные камеры 4 и 5, куда можно добавить гибридизационные зонды и смесь EK для образования удлиненных с помощью праймера матриц. Удлиненные с помощью праймера матрицы могут затем амплифицироваться при помощи ПЦР в камере 6 для ПЦР. Реакционная камера для ПЦР может затем направляться в блок анализа образца 7.
[0065] Блок обработки может быть выполнен посредством различных способов. Иллюстративными средствами реализации блока обработки может быть серия приводов, управляемых микрокомпьютером, или микроструйное устройство. Блок анализа можно также реализовать любыми способами в зависимости от конкретного вида анализа, подлежащего использованию. Например, это может быть масс-спектрометр, устройство для секвенирования ДНК, устройство для капиллярного электрофореза или аппарат для анализа температур плавления. Предпочтительно, системы настоящего изобретения адаптируют исходя из существующих автоматизированных систем для ПЦР, таких как система BD Мах™ от Becton Dickinson (Франклин Лейке, Нью-Джерси).
[0066] Неавтоматизированные системы можно также собрать с использованием имеющихся в наличии компонентов. Например, в иллюстративном варианте осуществления обработку образца можно осуществлять вручную или в отдельном блоке обработки/подготовки образца. Реакцию ПЦР можно осуществлять с использованием стандартного термоциклера, такого как система LightCycler® 480, изготовленная Roche Diagnostics Corporation (Индианаполис, Индиана). Анализ видовой идентификации можно осуществлять путем анализа кривых плавления с высокой разрешающей способностью с использованием системы LightScanner® 32 (Idaho Technologies, Солт-Лейк-Сити, Юта).
[0067] Для дополнительной помощи читающим в получении полного и глубокого понимания настоящего изобретения, ниже приводятся иллюстративные конкретные примеры.
ПРИМЕРЫ
Универсальная детекция бактерий, присутствующих в небольшом количестве, при помощи PE-PCR
[0068] Из коммерческих источников доступны несколько ДНК-полимераз "с низким фоновым уровнем ДНК" и HotStart Taq. В качестве отправного пункта сначала были исследованы 4 коммерчески доступных ДНК-полимераз "с низким фоновым уровнем ДНК" или HotStart Taq, чтобы продемонстрировать, что они не подходят для универсальной амплификации бактериальной ДНК с использованием универсального набора праймеров р201-р1370.
[0069] С помощью данных полимераз осуществляли классическую ПЦР для амплификации образца, содержащего 100 фг бактериальной геномной ДНК (соответствует 20 копиям бактериального генома) образцового Staphylococcus aureus и "контроля без матрицы (NTC)". Как показано на фигуре 2, значительное количество амплифицированного ДНК-продукта было обнаружено в реакции с NTC, отражая то, что она не отличается от реакции с образцом. Данный результат подтверждает, что коммерчески доступная Taq ДНК-полимераза и реактивы для ПЦР недостаточно чистые для универсального детекции бактериальной ДНК в клинических условиях, в которой обычно требуются пределы детекции на уровне фемтограмма.
[0070] В качестве проверки и подтверждения принципа действия подхода PE-PCR последовательно разведенную геномную ДНК S. aureus анализировали при помощи данного подхода. Конкретно, ген фактора элонгации трансляции Tu (Tuf) S. aureus выбирали в качестве мишени для PE-PCR-амплификации. Сконструировали гибридизационный зонд (M13-TstaG422) с последовательностью прямого праймера M13 на 5'-конце и последовательностями Tuf (номер доступа AF298796) на 3'-конце (таблица 1). После отжига M13-TstaG422 с матрицей ДНК для разрушения не связавшегося гибридизационного зонда и инициации удлинения с помощью праймера добавили смесь EK в реакционную смесь. Продукт удлинения праймера затем подвергали ПЦР-амплификации с использованием M13 и обратного праймера TstaG765, соответствующего геномным последовательностям Tuf. В результате этого был получен один ПЦР-продукт из 391 п.о. с 50 фг бактериальной ДНК, что соответствует 10 копиям генома S. aureus в образце. В отличие от этого, наличие ПЦР-продукта в NTC-контроле не наблюдали (Фигура 3А). Данный результат указывает на то, что данная PE-PCR является точной и чувствительной вплоть до диапазона фемтограммов.
[0071] Для имитации контаминирования реактива для ПЦР и фермента бактериальной ДНК, внесли соответственно 100 фг геномной ДНК S. aureus в смесь EK и реакционную смесь для ПЦР с Taq ДНК-полимеразой. Только тогда, когда матричная бактериальная ДНК присутствовала на протяжении удлинения праймера, произошло образование значительного количества PE-PCR-продукта (фигура 3B, полоса 1). Образования ПЦР-продукта не происходило (фигура 3B, полоса 5) в реакции с NTC или когда ПЦР осуществляли в отсутствие праймера M13 (фигура 3B, полоса 2). Примечательно, что PE-PCR облегчает амплификацию матричной бактериальной ДНК без коамплификации внесенной бактериальной ДНК.
[0072] Присутствие внесенной ДНК в смеси EK или реакционной смеси для ПЦР (фигура 3B, полосы 3 и 4) проверяли путем видоспецифичной к S. aureus ПЦР (фигура 3B, нижняя панель). Эти данные указывают на отсутствие влияния со стороны контаминирующей бактериальной ДНК в реактивах для ПЦР и ферментах на специфичную амплификацию матричной бактериальной ДНК и обеспечивают подтверждение принципа действия данного подхода PE-PCR.
[0073] С целью демонстрации пригодности PE-PCR для универсальной амплификации бактериальной ДНК, сконструировали универсальный гибридизационный зонд M13-16S-p201F для универсальной PE-PCR гена 16S рРНК, который является высококонсервативным среди различных видов бактерий. Последовательно разведенную геномную ДНК S. aureus подвергали универсальной PE-PCR с использованием гибридизационного зонда M13-16S-p201F, прямого праймера М13, обратного праймера р1370 (таблица 1) и обычно используемой HotStart Taq ДНК-полимеразы (Protech) без применения какой-либо предварительной обработки для деконтаминации. Был получен один ПЦР-продукт из 237 п.о., поэтому даже 10 фг матричной ДНК, соответствующей 2 копиям геномной ДНК S. aureus, можно обнаружить путем PE-PCR. Наличие ПЦР-продукта в NTC-контроле не наблюдали (Фигура 3C). Соответственно, протестировали геномные ДНК из целого ряда важных с клинической точки зрения видов бактерий, и было показано, что они могут быть амлифицированы при помощи универсальной PE-PCR.
[0074] Также имитировали контаминацию бактериальной ДНК путем внесения соответственно 100 фг геномной ДНК S. aureus в смесь EK и смесь для ПЦР с Taq ДНК полимеразой. Как и предполагалось, в присутствии 100 фг матричной бактериальной ДНК был получен положительный PE-PCR-сигнал (Фигура 3D, полоса 1), в то время как образования ПЦР-продукта не происходило при условии искусственного контаминирования и в реакции с NTC (Фигура 3D, полосы 2-4). Снова проверили присутствие внесенной ДНК в реакционной смеси при помощи видоспецифичной к S. aureus ПЦР (Фигура 3D, нижняя панель). Эти данные вместе доказывают, что данный подход PE-PCR делает возможным универсальную детекцию бактериальной ДНК даже в присутствии эндогенного контаминирования реактивами для ПЦР. Данное достижение до этого времени было недосягаемым.
[0075] Для выяснения предела детекции подхода, разные концентрации геномной ДНК S. aureus, варьирующей в диапазоне от 100 фг до 10 фг, подвергали универсальной PE-PCR. Вероятность получения положительного ПЦР-сигнала для 100, 50, 25 и 10 фг матричной бактериальной ДНК составляла соответственно 100%, 95%, 65% и 55% (n=20). Таким образом, чтобы специфично амплифицировать матричную бактериальную ДНК без снижения предела детекции и специфичности, с легкостью выполняют универсальную PE-PCR.
Специфичная детекция путем совмещения PE-PCR с анализом кривых плавления
[0076] Для дополнительного повышения эффективности PE-PCR, модифицировали протокол для внедрения ПЦР в режиме реального времени и HRM-анализа в универсальный способ детекции PE-PCR, описанный выше. С целью демонстрации, последовательно разведенную геномную ДНК S. aureus подвергали универсальной PE-PCR в режиме реального времени в присутствии красителя для HRM LCGreen I plus. Как было выявлено посредством графиков амплификации, 10 фг матричной ДНК привело к ампликон-специфичной амплификации, и к отсутствию ПЦР-продукта в реакции с NTC (Фигура 4A и 4B). Для уникальных содержаний нуклеотидов в ПЦР-ампликоне S. aureus был получен отличающийся график производной, в то время как никакого пика плавления для реакции с NTC не наблюдали. Вероятность получения положительного ПЦР-сигнала для 100, 50, 25 и 10 фг матричной ДНК составляла соответственно 100%, 90%, 50%, и 30% (n=10).
[0077] Для сравнения реактивы для ПЦР предварительно обработали ДНКазой I с последующей универсальной ПЦР-амплификацией матрицы геномной ДНК S. aureus с использованием пары праймеров р201 и p1370. Как было выявлено посредством амплификации и графиков производной (Фигура 4C), добавление ДНКазы I в значительной степени ингибировало ПЦР-амплификацию. При использовании 1 ед ДНКазы эндогенная контаминирующая ДНК удалилась не полностью. Увеличение концентрации ДНКазы I до 2,5 ед вызвало дополнительное ингибирование ПЦР и ограничило предел детекции для универсальной ПЦР-амплификации бактериальной ДНК.
[0078] Для дополнительного подтверждения данного подхода, применили универсальную PE-PCR в режиме реального времени для анализа дополнительных 12 распространенных видов бактерий (таблица 2). HRM-анализ ПЦР-продукта с использованием графиков производной и температуры плавления позволяет легко отличить большинство видов бактерий (фигура 5 и таблица 2). С использованием данного подхода секвенирование матриц можно по большей части исключить, чтобы обеспечить большую экономию средств и удобства.
МАТЕРИАЛ И СПОСОБ
Материалы
[0079] Exo I и ДНК-полимеразу Кленова приобрели у New England Biolab (Ипсуич, Массачусетс). Набор реактивов LCGreen I plus и прибор HR-1 приобрели у Idaho Technology (Солт-Лейк-Сити, Юта). HotStart Taq ДНК-полимеразу приобрели у Protech (Тайбэй, Тайвань). Fast Hot Start Taq ДНК-полимеразу приобрели у КАРА Biosystems (Вобурн, Массачусетс). ДНК-полимеразу Taq "с низким фоновым уровнем ДНК" приобрели у Takara (Сига, Япония). ULTRATOOLS Taq ДНК-полимеразу приобрели у Biotools Inc. (Мадрид, Испания). Капилляры для LightCycler приобрели у Roche Applied Science (Индианаполис, Индиана). ДНКазу I приобрели у Promega (Мэдисон, Висконсин). Бактериальные штаммы являлись клиническими изолятами, как описано ранее (ссылка 11). Полный перечень последовательностей праймеров и гибридизационных зондов показан в таблице 1.
Выделение бактериальной геномной ДНК
[0080] Бактериальную суспензию суточной культуры (4 мл) центрифугировали при 10000 об./мин. в минуту в течение 10 минут и осадок ресуспендировали в 4 мл раствора буфера I (25 мМ Трис-HCl, pH 7,5, 50 мМ глюкозы, 10 мМ ЭДТА и 40 мкг/мл лизостафина). Бактериальную суспензию инкубировали при 37°C в течение 2 часов и в бактериальную взвесь добавили реакционный буфер, содержащий 280 мкл 20% SDS и 40 мкл протеиназы К (10 мг/мл) и РНКазы А (10 мг/мл), и инкубировали при 55°C на протяжении ночи. После экстракции фенолом/хлороформом, супернатант переносили в чистую микроцентрифужную пробирку, и ДНК преципитировали при помощи этанола. После двух отмывок 75% этанолом, осадки ДНК ресуспендировали в воде для определения количества и последующих ПЦР-анализов. PE-PCR
[0081] Традиционную PE-PCR осуществляли в одной реакционной пробирке. Вкратце, этап отжига включал 20 мкл смеси для отжига, содержащей 8 мкл H2O, 5 мкл гибридизационного зонда (2 нг/мкл), 5 мкл бактериальной геномной ДНК в указанной концентрации и 2 мкл 10Х ПЦР-буфера. Реакционную смесь нагревали до 95°C в течение 5 минут и поддерживали при 37°C. Затем в смесь для отжига добавляли 11 мкл смеси EK, состоящей из 3 мкл H2O, 1 мкл 10Х ПЦР-буфера, 5 мкл дНТФ (2 мМ), 1 мкл ДНК-полимеразы Кленова (5 ед/мкл) и 1 мкл exo I (20 ед/мкл), и инкубировали при 37°C в течение 2 часов. После инактивации нагреванием при 80°C в течение 20 минут реакционную смесь доводили до 50 мкл путем добавления 14 мкл H2O, 2 мкл 10Х ПЦР-буфера, 1 мкл прямого праймера M13 (5 мкМ), 1 мкл обратного праймера (5 мкМ) и 1 мкл HotStart Taq ДНК-полимеразы (5 ед/мкл). Условия проведения ПЦР-циклов были следующие: 1 цикл при 95°C в течение 10 минут, 45 циклов при 95°C в течение 15 секунд и 60°C в течение 1 минуты. Для детекции внесенной геномной ДНК S. aureus, M13 и обратный праймер заменяли набором праймеров SA-F и SA-R (таблица 1), который специфично амплифицирует фрагмент геномной ДНК S. aureus (ссылка 43).
[0082] Для PE-PCR в режиме реального времени реакционную смесь пропорционально уменьшали до 8 мкл во время связывания гибридизационного зонда и удлинения праймера. Затем реакционную смесь доводили до 20 мкл путем добавления 1,5 мкл H2O, 2 мкл 10Х альбумина сыворотки крупного рогатого скота (BSA), 2 мкл 10Х LCGreen I plus, 0,8 мкл 10Х ПЦР-буфера, 0,4 мкл прямого праймера M13 (5 мкМ), 0,4 мкл обратного праймера (5 мкМ) и 0,5 мкл HotStart Taq ДНК-полимеразы (5 ед/мкл) и переносили в капиллярную трубку. ПЦР в режиме реального времени осуществляли с использованием прибора LightCycler 1.5, и условия циклов были следующими: 1 цикл при 95°C в течение 10 минут, 45 циклов при 95°C в течение 15 секунд и 60°C в течение 1 минуты при скорости перехода 20°C/с.
Получение и анализ кривых плавления с высокой разрешающей способностью
[0083] Стеклянные капилляры, содержащие продукты амплификации, переносили в прибор HR-1 (Idaho Technology), и ПЦР-фрагменты расплавляли при 64-96°C со скоростью 0,3°C/сек. Профили плавления оценивали при помощи программного обеспечения для HR-1 с нормализацией флуоресценции и наложенным отображением температур для совмещения кривых при 0%-20% флуоресценции.
Предварительная обработка ДНКазой I для деконтаминации и универсальной ПЦР-амплификации
[0084] Реактивы для ПЦР, содержащие 2 мкл 10Х BSA, 2 мкл дНТФ (2 мМ), 2 мкл 10Х LCGreen I plus, 1 мкл 10Х ПЦР-буфера, 0,5 мкл HotStart Taq ДНК-полимеразы (5 ед/мкл), инкубировали с ДНКазой I (1 ед. или 2,5 ед.) при 37°C в течение 30 минут. После тепловой инактивации ДНКазы I при 85°C в течение 15 минут, реакционную смесь доводили до 20 мкл путем добавления 1 мкл 10Х ПЦР-буфера, 2 мкл прямого праймера р201 (5 мкМ), 2 мкл обратного праймера p1370 (5 мкМ) и 5 мкл матричной ДНК. Условия проведения ПЦР-циклов были следующие: 1 цикл при 95°C в течение 10 минут, 45 циклов при 95°C в течение 15 секунд и 60°C в течение 1 минуты при скорости перехода 20°C/с. Иллюстративный диагностический набор
[0085] В предпочтительном применении PE-PCR можно использовать в качестве основы для клинического диагностического анализа для исследования на бактериемию или сепсис. В следующей таблице перечислены реактивы, которые можно включить в подобный диагностический набор:
праймера/зонда
ССЫЛКИ
Каждая из следующих ссылок включена в данный документ посредством ссылки.
1. Rice TW, Bernard GR (2005) Therapeutic intervention and targets for sepsis. Annu Rev Med 56: 225-248.
2 Weile J, Knabbe С (2009) Current applications and future trends of molecular diagnostics in clinical bacteriology. Anal Bioanal Chem 394: 731-742.
3. Klouche M, Schroder U (2008) Rapid methods for diagnosis of bloodstream infections. Clin Chem Lab Med 46: 888-908.
4. Wang Q, Wang S, Beutin L, Cao B, Feng L, et al. (2010) Development of a DNA microarray for detection and serotyping of enterotoxigenic Escherichia coli. J Clin Microbiol 48: 2066-2074.
5. Anthony RM, Brown TJ, French GL (2000) Rapid diagnosis of bacteremia by universal amplification of 23 S ribosomal DNA followed by hybridization to an oligonucleotide array. J Clin Microbiol 38: 781-788.
6. Tissari P, Zumla A, Tarkka E, Mero S, Savolainen L, et al. (2010) Accurate and rapid identification of bacterial species from positive blood cultures with a DNA-based microarray platform: an observational study. Lancet 375: 22430.
7. Tsalik EL, Jones D, Nicholson B, Waring L, Liesenfeld O, et al. (2010) Multiplex PCR to diagnose bloodstream infections in patients admitted from the emergency department with sepsis. J Clin Microbiol 48: 26-33.
8. Tseng CP, Cheng JC, Tseng CC, Wang C, Chen YL, et al. (2003) Broad-range ribosomal RNA real-time PCR after removal of DNA from reagents: melting profiles for clinically important bacteria. Clin Chem 49: 306-309.
9. Reno WL 3rd, McDaniel DO, Turner WW Jr., Williams MD (2001) Polymerase chain reaction for the detection of bactermia. Am Surg 67: 508-512.
10. Sleigh J, Cursons R, La Pine M (2001) Detection of bacteremia in critically ill patients using 16S rDNA polymerase chain reaction and DNA sequencing. Intensive Care Med 27: 1269-1273.
11. Cheng JC, Huang CL, Lin CC, Chen CC, Chang YC, et al. (2006) Rapid detection and identification of clinically important bacteria by high-resolution melting analysis after broad-range ribosomal RNA real-time PCR. Clin Chem 52: 1997-2004.
12 Won H, Rothman R, Ramachandran P, Hsieh YH, Kecojevic A, et al. (2010) Rapid identification of bacterial pathogens in positive blood culture bottles by use of a broad-based PCR assay coupled with high-resolution melt analysis. J Clin Microbiol 48: 3410-3413.
13. Yang S, Ramachandran P, Rothman R, Hsieh YH, Hardick A, et al. (2009) Rapid identification of biothreat and other clinically relevant bacterial species by use of universal PCR coupled with high-resolution melting analysis. J Clin Microbiol 47: 2252-2255.
14. Corless CE, Guiver M, Borrow R, Edwards-Jones V, Kaczmarski EB, et al. (2000) Contamination and sensitivity issues with a real-time universal 16S rRNA PCR. J Clin Microbiol 38: 1747-1752.
15. Miihl H, Kochern AJ, Disque C, Sakka SG (2010) Activity and DNA contamination of commercial polymerase chain reaction reagents for the universal 16S rDNA real-time polymerase chain reaction detection of bacterial pathogens in blood. Diagn Microbiol Infect Dis 66: 41-49.
16. Bottger EC (1990) Frequent contamination of Taq polymerase with DNA. Clin Chem 36:1258-1259.
17. Hughes MS, Beck LA, Skuce RA (1994) Identification and elimination of DNA sequences in Taq DNA polymerase. J Clin Microbiol 32: 2007-2008.
18. Maiwald M, Ditton H-J, Sonntag H-G, von Knebel Doeberitz M (1994) Characterization of contaminating DNA in Taq polymerase which occurs during amplification with a primer set for Legionella 5S ribosomal RNA. Mol Cell Probes 8: 11-14.
19. Sontakke S, Cadenas MB, Maggi RG, Diniz PP, Breitschwerdt EB (2009) Use of broad range 16S rDNA PCR in clinical microbiology. J Microbiol Methods 76: 217-225.
20. Ou CY, Moore JL, Schochetman G (1991) Use of UV irradiation to reduce false positivity in polymerase chain reaction. Biotechniques 10: 442-446.
21. Pandit L, Kumar S, Karunasagar I, Karunasagar I (2005) Diagnosis of partially treated culture-negative bacterial meningitis using 16S rRNA universal primers and restriction endonuclease digestion. J Med Microbiol 54: 539-542.
22. Glushkov SA, Bragin AG, Dymshits GM (2009) Decontamination of polymerase chain reaction reagents using DEAE-cellulose. Anal Biochem 393: 135-137.
23. Silkie SS, Tolcher MP, Nelson KL (2008) Reagent decontamination to eliminate false-positives in Escherichia coli qPCR. J Microbiol Methods 72: 275-282.
24. Philipp S, Huemer HP, Irschick EU, Gassner С (2010) Obstacles of multiplex real-time PCR for bacterial 16S rDNA: primer specifity and DNA decontamination of Taq polymerase. Transfus Med Hemother 37: 21-28.
25. Li D, Liu B, Chen M, Guo D, Guo X, et al. (2010) A multiplex PCR method to detect 14 Escherichia coli serogroups associated with urinary tract infections. J Microbiol Methods 82: 71-77.
26. Dierkes C, Ehrenstein B, Siebig S, Linde HJ, Reischl U, et al. (2009) Clinical impact of a commercially available multiplex PCR system for rapid detection of pathogens in patients with presumed sepsis. BMC Infect Dis 11: 126.
27. Wallet F, Nseir S, Baumann L, Herwegh S, Sendid B, et al. (2010) Preliminary clinical study using a multiplex real-time PCR test for the detection of bacterial and fungal DNA directly in blood. Clin Microbiol Infect 16: 774-779.
28. Lehmann LE, Hunfeld KP, Steinbrucker M, Brade V, Book M, et al. (2010) Improved detection of blood stream pathogens by real-time PCR in severe sepsis. Intensive Care Med 36: 49-56.
29. Steinman CR, Muralidhar B, Nuovo GJ, Rumore PM, Yu D, et al. (1997) Domain-directed polymerase chain reaction capable of distinguishing bacterial from host DNA at the single-cell level: characterization of a systematic method to investigate putative bacterial infection in idiopathic disease. Anal Biochem 244: 328-339.
30. McCabe KM, Zhang Y-H, Huang B-L, Eagar EA, McCabe ERB (1999) Bacterial species identification after DNA amplification with a universal primer pair. Mol Gene Metab66: 205-211.
31. Woo PC, Lau SK, Teng JL, Tse H, Yuen KY (2008) Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect 14: 908-934.
32. Cherkaoui A, Emonet S, Ceroni D, Candolfi B, Hibbs J, et al. (2009) Development and validation of a modified broad-range 16S rDNA PCR for diagnostic purposes in clinical microbiology. J Microbiol Methods 79: 227-231.
33. Champlot S, Berthelot C, Pruvost M, Bennett EA, Grange T, et al. (2010) An efficient multistrategy DNA decontamination procedure of PCR reagents for hypersensitive PCR applications. PLoS One 5: el3042.
34. Spangler R, Goddard NL, Thaler DS (2009) Optimizing Taq polymerase concentration for improved signal-to-noise in the broad range detection of low abundance bacteria. PLoSOne4:e7010.
35. Trevino S, Mahon CR (2000) Bacteremia. In: Mahon CR, Manuselis G, editors. Textbook of diagnostic microbiology. Philadelphia: W.B. Saunders Company, pp.998-1008.
36. Lin JH, Tseng CP, Chen YJ, Lin CY, Chang SS, et al. (2008) Rapid differentiation of influenza A virus subtypes and genetic screening for virus variants by high-resolution melting analysis. J Clin Microbiol 46: 1090-1097.
37. Tajiri-Utagawa E, Hara M, Takahashi K, Watanabe M, Wakita T (2009) Development of a rapid high-throughput method for high-resolution melting analysis for routine detection and genotyping of noroviruses. J Clin Microbiol 47: 435-440.
38. Robertson T, Bibby S, O'Rourke D, Belfiore T, Lambie H, et al. (2009) Characterization of Chlamydiaceae species using PCR and high resolution melt curve analysis of the 16S rRNA gene. J Appl Microbiol 107: 2017-2028.
39. Stephens AJ, Inman-Bamber J, Giffard PM, Huygens F (2008) High-resolution melting analysis of the spa repeat region of Staphylococcus aureus. Clin Chem 54: 432-436.
40. Weinstein MP, Reller LB, Murphy JR, Lichtenstein KA (1983) The clinical significance of positive blood cultures: A comprehensive analysis of 500 episodes of bacteremia and fungemia in adults. I. Laboratory and epidemiologic observations. Rev Infect Dis 5: 35-53.
41. Rello J, Quintana E, Mirelis B, Gurgui M, Net A, et al. (1993) Polymicrobial bacteremia in critically ill patients. Intensive Care Med 9: 22-25.
42. Montgomery JL, Sanford LN, Wittwer CT (2010) High-resolution DNA melting analysis in clinical research and diagnostics. Expert Rev Mol Diag 10: 219-240.
43. Martineau F, Picard FJ, Roy PH, Ouellette M, Bergeron MG (1998) Species-specific and ubiquitous-DNA-based assays for rapid identification of Staphylococcus aureus. J Clin Microbiol 36: 618-623.



Изобретение относится к биохимии. Изобретение обеспечивает способ селективной амплификации одной или нескольких целевых бактериальных ДНК в образце, включающий: гибридизацию множества гибридизационных зондов, выбранных из SEQ ID NO: 1 или SEQ ID NO: 6, с указанной целевой микробной ДНК, где каждый из указанных гибридизационных зондов содержит 5'-концевой участок, имеющий последовательность небактериальной ДНК, выбранную из вирусной последовательности или искусственно сконструированной последовательности, и 3'-концевой участок, имеющий последовательность, комплементарную участку последовательности гена бактериальной 16S rRNA; удаление негибридизированных гибридизационных зондов и любого несвязанного 3'-концевого участка целевой бактериальной ДНК; удлинение 3'-концов гибридизационных зондов и целевой бактериальной ДНК с образованием двухцепочечных удлиненных с помощью праймера матриц. Затем проводят ПЦР для селективного амплифицирования удлиненных с помощью праймера матриц при помощи набора праймеров, который включает по меньшей мере один праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационных зондов. Также обеспечивается способ детекции бактериальной инфекции у субъекта, включающий: добавление множества гибридизационных зондов, выбранных из SEQ ID NO: 1 или SEQ ID NO: 6, в образец субъекта, где каждый из указанных гибридизационных зондов содержит 5'-концевой участок, имеющий последовательность небактериальной ДНК, выбранную из вирусной последовательности или искусственно сконструированной последовательности, 3'-концевой участок, имеющий последовательность, комплементарную участку последовательности гена бактериальной 16S rRNA; гибридизацию гибридизационных зондов с бактериальной ДНК в образце; удаление негибридизированных гибридизационных зондов и любого несвязанного 3'-концевого участка бактериальной ДНК; удлинение 3'-концов гибридизационных зондов и бактериальной ДНК с образованием двухцепочечных удлиненных с помощью праймера матриц; амплифицирование удлиненных с помощью праймера матриц путем проведения способа ПЦР при помощи набора праймеров, который включает по меньшей мере один прямой праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационного зонда; и анализирование амплифицированных ПЦР-продуктов для определения присутствия или отсутствия бактерии. Также описан гибридизационный зонд для создания удлиненной с помощью праймера ДНК-матрицы из целевой бактериальной ДНК в образце для селективной амплификации с помощью способа ПЦР, при этом указанный гибридизационный зонд содержит: 5'-концевой участок, имеющий небактериальную последовательность, выбранную из вирусной последовательности и искусственно сконструированной последовательности; и 3'-концевой участок, имеющий последовательность, комплементарную участку последовательности гена бактериальной 16S rRNA, где указанный гибридизационный зонд выбран из SEQ ID NO: 1 или SEQ ID NO: 6. Описан набор, содержащий множество таких зондов. Способы настоящего изобретения основаны на новом подходе, согласно которому метят 5'-конец целевых ДНК-матриц небактериальной меченой последовательностью для того, чтобы отличить матрицы от эндогенных контаминирующих веществ, присутствующих в реактивах для ПЦР. 4 н. и 9 з.п. ф-лы, 6 ил., 2 табл., 1 пр.
1. Способ селективной амплификации одной или нескольких целевых бактериальных ДНК в образце, включающий:
гибридизацию множества гибридизационных зондов, выбранных из SEQ ID NO: 1 или SEQ ID NO: 6, с указанной целевой микробной ДНК, где каждый из указанных гибридизационных зондов содержит 5'-концевой участок, имеющий последовательность небактериальной ДНК, выбранную из вирусной последовательности или искусственно сконструированной последовательности, и 3'-концевой участок, имеющий последовательность, комплементарную участку последовательности гена бактериальной 16S rRNA;
удаление негибридизированных гибридизационных зондов и любого несвязанного 3'-концевого участка целевой бактериальной ДНК;
удлинение 3'-концов гибридизационных зондов и целевой бактериальной ДНК с образованием двухцепочечных удлиненных с помощью праймера матриц и
проведение способа ПЦР для селективного амплифицирования удлиненных с помощью праймера матриц при помощи набора праймеров, который включает по меньшей мере один праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационных зондов.
2. Способ по п. 1, отличающийся тем, что этап удаления и этап удлинения включают добавление смеси экзонуклеазы I и ДНК-полимеразы Кленова.
3. Способ по п. 1, отличающийся тем, что последовательность указанного 3'-концевого участка гибридизационных зондов комплементарна консервативному участку последовательности гена 16S рРНК.
4. Способ по п. 1, отличающийся тем, что указанный способ ПЦР выбирают из стандартной ПЦР или ПЦР в режиме реального времени.
5. Способ по п. 1, отличающийся тем, что указанный образец не обрабатывают от контаминирования бактериальной ДНК.
6. Способ детекции бактериальной инфекции у субъекта, включающий:
добавление множества гибридизационных зондов, выбранных из SEQ ID NO: 1 или SEQ ID NO: 6, в образец субъекта, где каждый из указанных гибридизационных зондов содержит 5'-концевой участок, имеющий последовательность небактериальной ДНК, выбранную из вирусной последовательности или искусственно сконструированной последовательности, 3'-концевой участок, имеющий последовательность, комплементарную участку последовательности гена бактериальной 16S rRNA;
гибридизацию гибридизационных зондов с бактериальной ДНК в образце;
удаление негибридизированных гибридизационных зондов и любого несвязанного 3'-концевого участка бактериальной ДНК;
удлинение 3'-концов гибридизационных зондов и бактериальной ДНК с образованием двухцепочечных удлиненных с помощью праймера матриц;
амплифицирование удлиненных с помощью праймера матриц путем проведения способа ПЦР при помощи набора праймеров, который включает по меньшей мере один прямой праймер, имеющий небактериальную последовательность, комплементарную небактериальной последовательности гибридизационного зонда; и
анализирование амплифицированных ПЦР-продуктов для определения присутствия или отсутствия бактерии.
7. Способ по п. 6, отличающийся тем, что указанный образец не обрабатывают от контаминации бактериальной ДНК.
8. Способ по п. 6, отличающийся тем, что последовательность указанного 3'-концевого участка гибридизационных зондов комплементарна консервативному участку последовательности гена 16S рРНК.
9. Гибридизационный зонд для создания удлиненной с помощью праймера ДНК-матрицы из целевой бактериальной ДНК в образце для селективной амплификации с помощью способа ПЦР, при этом указанный гибридизационный зонд содержит:
5'-концевой участок, имеющий небактериальную последовательность, выбранную из вирусной последовательности и искусственно сконструированной последовательности; и
3'-концевой участок, имеющий последовательность, комплементарную участку последовательности гена бактериальной 16S rRNA,
где указанный гибридизационный зонд выбран из SEQ ID NO: 1 или SEQ ID NO: 6.
10. Гибридизационный зонд по п. 9, отличающийся тем, что указанная небактериальная последовательность представляет собой последовательность прямого праймера М13.
11. Гибридизационный зонд по п. 9, отличающийся тем, что последовательность указанного 3'-концевого участка гибридизационного зонда комплементарна консервативной последовательности гена 16S рРНК.
12. Набор для создания удлиненной с помощью праймера ДНК-матрицы из целевой микробной ДНК для селективной ПЦР-амплификации и детекции, включающий:
множество гибридизационных зондов по п. 9.
13. Набор по п. 12, дополнительно включающий смесь 3'-5'-экзонуклеазы и ДНК-полимеразы Кленова.
| RU 2182176 C2, 10.05.2002 | |||
| WO 2006076650 A2, 20.07.2006. |
Авторы
Даты
2017-05-30—Публикация
2012-05-18—Подача